Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Targets for Cell Cycle/Checkpoint
- ATM/ATR(23)
- Aurora Kinase(17)
- Cdc42(4)
- Cdc7(3)
- Chk(14)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(77)
- E1 enzyme(1)
- G-quadruplex(11)
- Haspin(6)
- HMTase(1)
- Kinesin(23)
- Ksp(4)
- Microtubule/Tubulin(219)
- Mps1(15)
- Mitotic(7)
- RAD51(16)
- ROCK(60)
- Rho(13)
- PERK(10)
- PLK(33)
- PTEN(6)
- Wee1(7)
- PAK(20)
- Arp2/3 Complex(8)
- Dynamin(10)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Products for Cell Cycle/Checkpoint
- Cat.No. Nom du produit Informations
-
GC44730
proTAME
Pro-N-4-tosyl-L-arginine methyl ester
proTAME est une forme de promédicament perméable aux cellules de N-4-tosyl-L-arginine méthylester, un inhibiteur du complexe/cyclosome promoteur d'anaphase (APC/C), qui est converti en TAME par des estérases intracellulaires.
-
GC44732
Protopine (hydrochloride)
Protopine is an alkaloid found in Berberidaceae, Ranunculaceae, Rutaceae, Fumariaceae, and Papaveraceae with diverse biological activities.
-
GC49914
Pt(II)-NHC Complex 2C
Platinum(II)-N-Heterocyclic Carbene Complex 2C
An inducer of immunogenic cancer cell death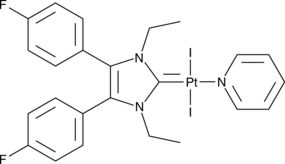
-
GC69776
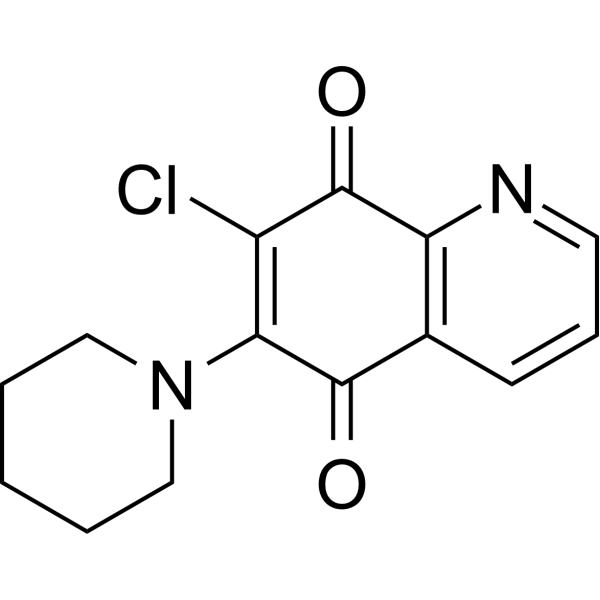
PT-262
PT-262 est un inhibiteur efficace de ROCK avec une valeur IC50 d'environ 5 μM. PT-262 induit la perte du potentiel membranaire mitochondrial et augmente l'activation de caspase-3 et l'apoptose cellulaire. PT-262 inhibe la phosphorylation d'ERK et CDC2 par une voie indépendante de p53. PT-262 bloque la fonction du cytosquelette cellulaire et la migration des cellules. PT-262 a une activité anticancéreuse.
-
GC44740
PtdIns-(1,2-dioctanoyl) (sodium salt)
DOPI, Phosphatidylinositol C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.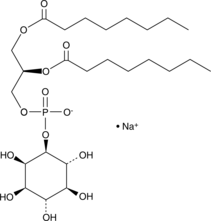
-
GC44743
PtdIns-(3)-P1 (1,2-dioctanoyl) (sodium salt)
DOPI3P1, Phosphatidylinositol3monophosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.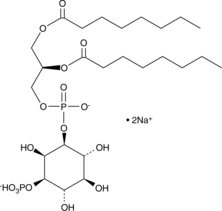
-
GC44748
PtdIns-(3,4,5)-P3 (1,2-dihexanoyl) (ammonium salt)
DHPI-3,4,5-P3, Phosphatidylinositol-3,4,5-triphosphate C-6, PIP3C-16
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.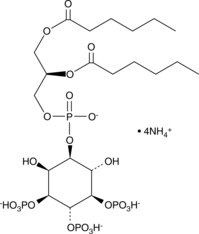
-
GC44750
PtdIns-(3,4,5)-P3 (1,2-dipalmitoyl) (sodium salt)
DPPI3,4,5P3, Phosphatidylinositol3,4,5triphosphate C16
The phosphatidylinositol phosphates represent a small percentage of total membrane phospholipids.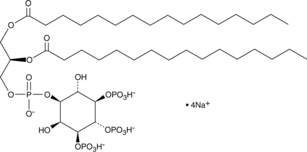
-
GC44753
PtdIns-(3,4,5)-P3-biotin (sodium salt)
Phosphatidylinositol3,4,5triphosphate C8biotin, PIP3biotin
The PtdIn phosphates play an important role in the generation and transduction of intracellular signals.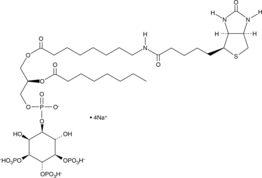
-
GC44759
PtdIns-(4)-P1 (1,2-dioctanoyl) (ammonium salt)
DOPI4P1, Phosphatidylinositol4phosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.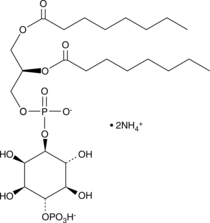
-
GC52484
Purified Ganglioside Mixture (bovine) (ammonium salt)
A mixture of purified bovine gangliosides

-
GC14707
Purvalanol A
NG 60
Le purvalanol A est un puissant inhibiteur de CDK, qui inhibe cdc2-cycline B, cdk2-cycline A, cdk2-cycline E, cdk4-cycline D1 et cdk5-p35 avec des IC50 de 4, 70, 35, 850, 75 nM, respectivement.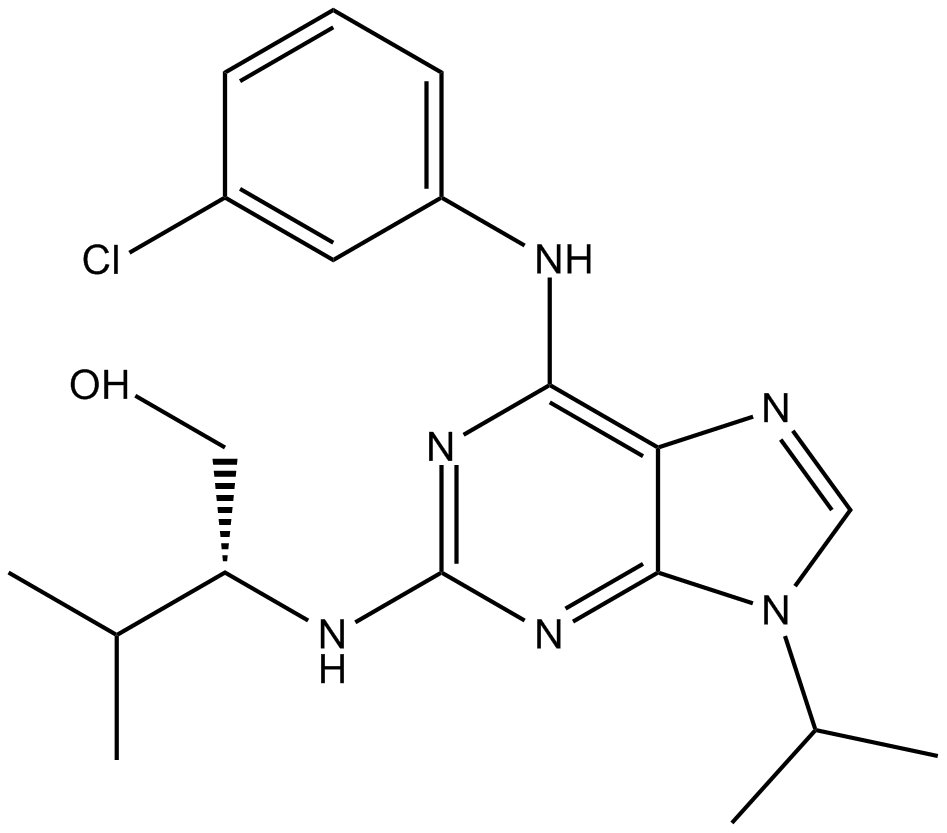
-
GC16268
Purvalanol B
NG 95; NG95; NG-95
Le purvalanol B (NG 95) est un CDK inhibiteur puissant, sélectif, réversible et compétitif pour l'ATP, avec des IC50 de 6 nM, 6 nM, 9 nM, 6 nM pour cdc2-cycline B, CDK2-cycline A, CDK2-cycline E et CDK5-p35, respectivement.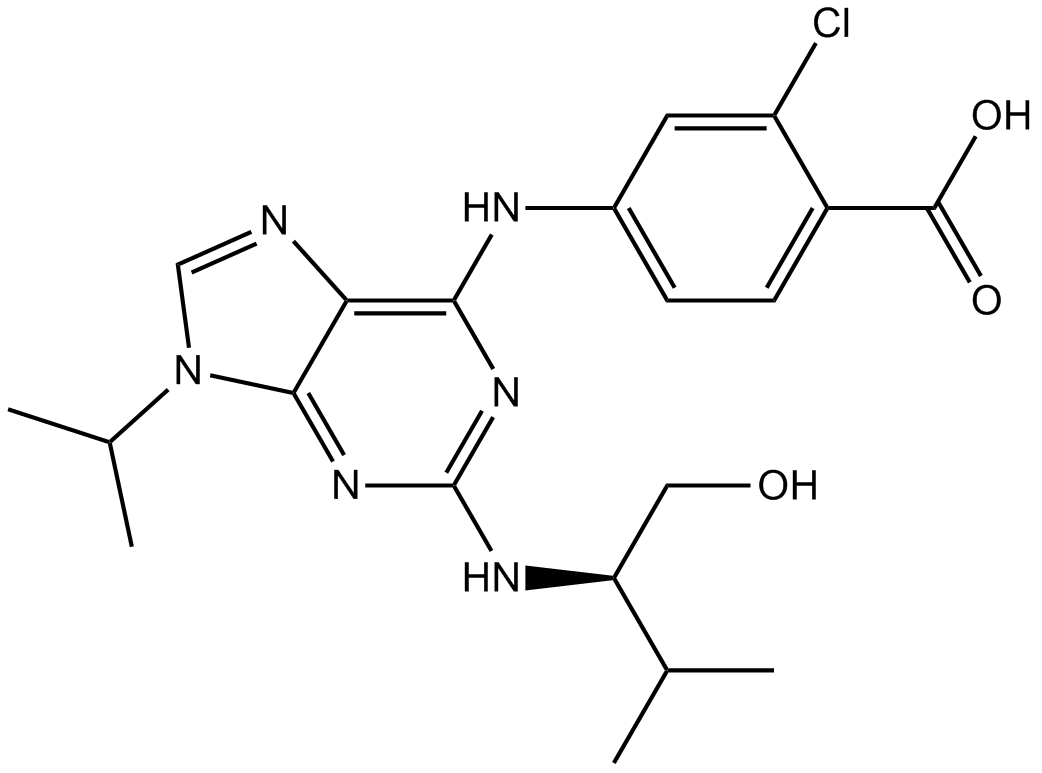
-
GC45552
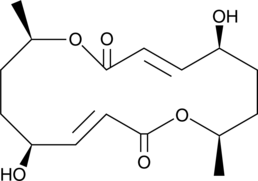
Pyrenophorol
Le pyrénophorol est un composé C9H12O3 isolé avec un faible rendement À partir de filtrats de culture du champignon phytopathogène Stemphylium radicinum, C.
-
GC15162
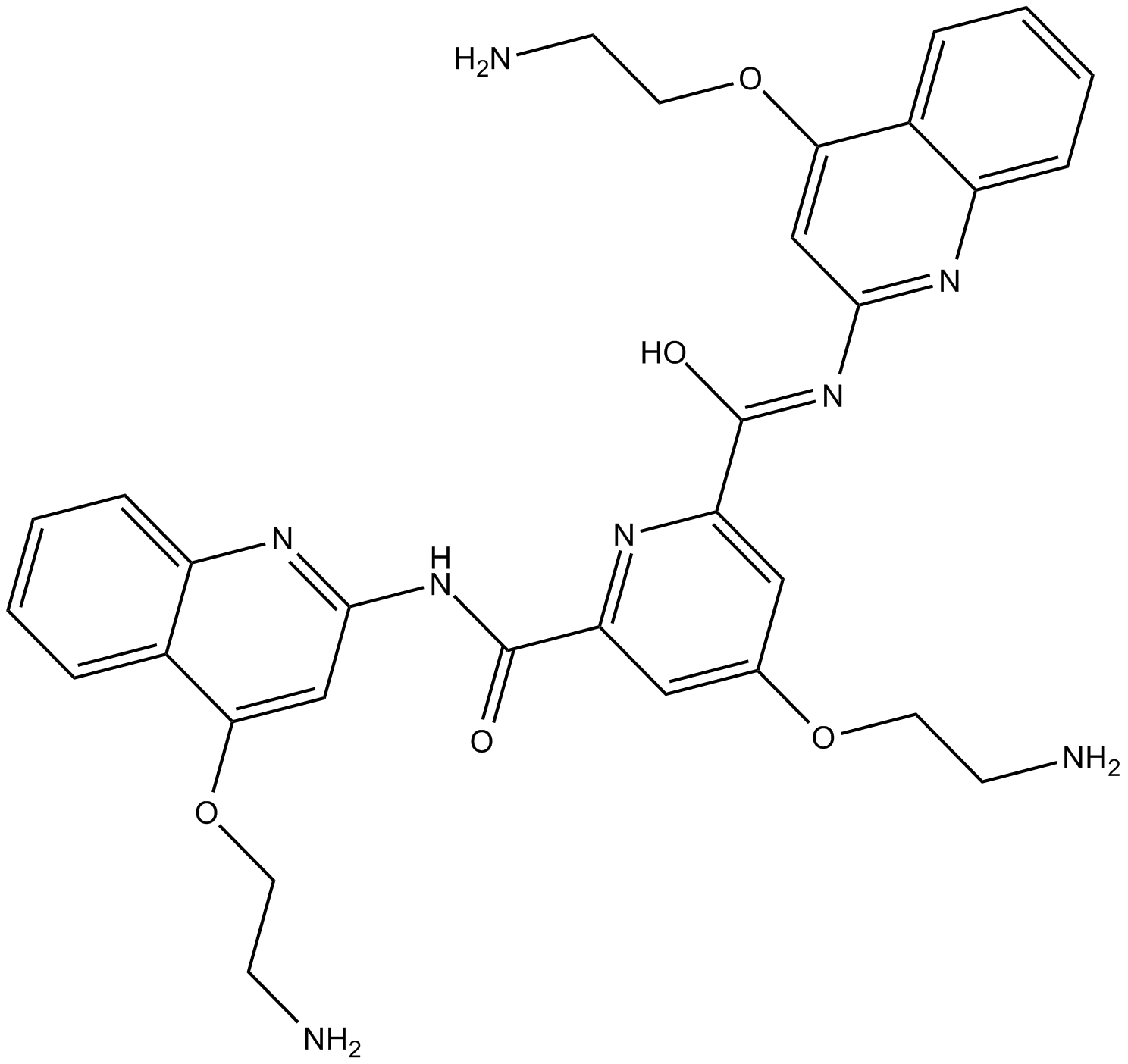
Pyridostatin
Pyridostatin est une petite molécule hautement sélective se liant aux G-quadruplex, conçue pour cibler les structures polymorphes de G-quadruplex indépendamment de la variabilité de séquence.
-
GC48016
Pyridostatin (trifluoroacetate salt)
RR82 TFA
La pyridostatine (RR82) TFA est un agent stabilisant de l'ADN G-quadruplex (Kd = 490 nM). La pyridostatine (sel de trifluoroacétate) favorise l'arrêt de la croissance des cellules cancéreuses humaines en induisant des dommages À l'ADN dépendant de la réplication et de la transcription. La pyridostatine (sel de trifluoroacétate) cible le proto-oncogène Src. La pyridostatine (sel de trifluoroacétate) a réduit les niveaux de protéine SRC et la motilité cellulaire dépendante du SRC dans les cellules cancéreuses du sein humain.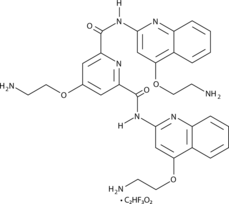
-
GC37043
Pyridostatin hydrochloride
RR82 hydrochloride
Le chlorhydrate de pyridostatine (RR82) est un agent stabilisant de l'ADN G-quadruplex (Kd = 490 nM). Le chlorhydrate de pyridostatine favorise l'arrêt de la croissance des cellules cancéreuses humaines en induisant des dommages À l'ADN dépendant de la réplication et de la transcription. Le chlorhydrate de pyridostatine cible le proto-oncogène Src. Le chlorhydrate de pyridostatine a réduit les niveaux de protéine SRC et la motilité cellulaire dépendante du SRC dans les cellules cancéreuses du sein humain.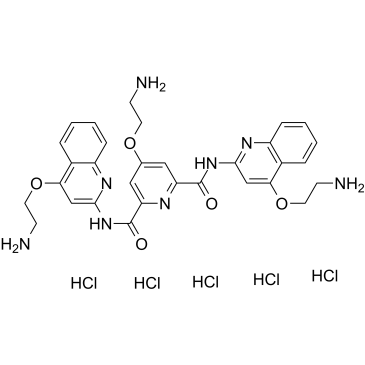
-
GC17801
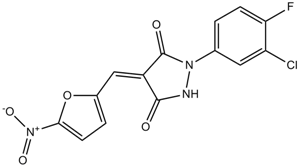
PYZD-4409
Le PYZD-4409 est un inhibiteur spécifique de l'enzyme activatrice de l'ubiquitine UBA1 avec une IC50 de 20 μM (cell-free enzymatic assay). Le PYZD-4409 induit la mort cellulaire dans les cellules malignes et inhibe préférentiellement la croissance clonogénique des cellules de la leucémie myéloÏde aiguë primaire.
-
GC52057
QN523
An anticancer agent

-
GC45903
Quazinone
Ro 13-6438
A PDE3 inhibitor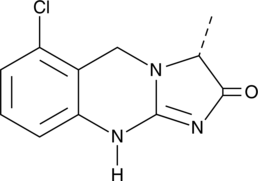
-
GC48019
Quercetin (hydrate)
A flavonoid with diverse biological activities
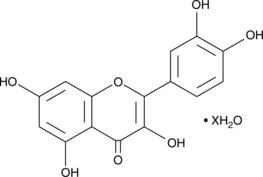
-
GC46210
Quinacrine mustard (hydrochloride)
NSC 3424
La moutarde de quinacrine (chlorhydrate) est un fluorochrome.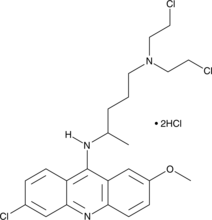
-
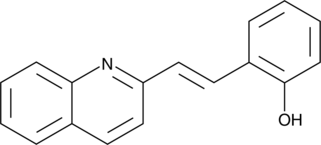
GC44800
Quininib
A CysLT1 and CysLT2 receptor antagonist
-
GC17400
R547
Ro 4584820
R547 est un inhibiteur de CDK puissant, sélectif et actif par voie orale, avec un Kis de 2 nM, 3 nM et 1 nM pour CDK1/cycline B, CDK2/cycline E et CDK4/cycline D1, respectivement.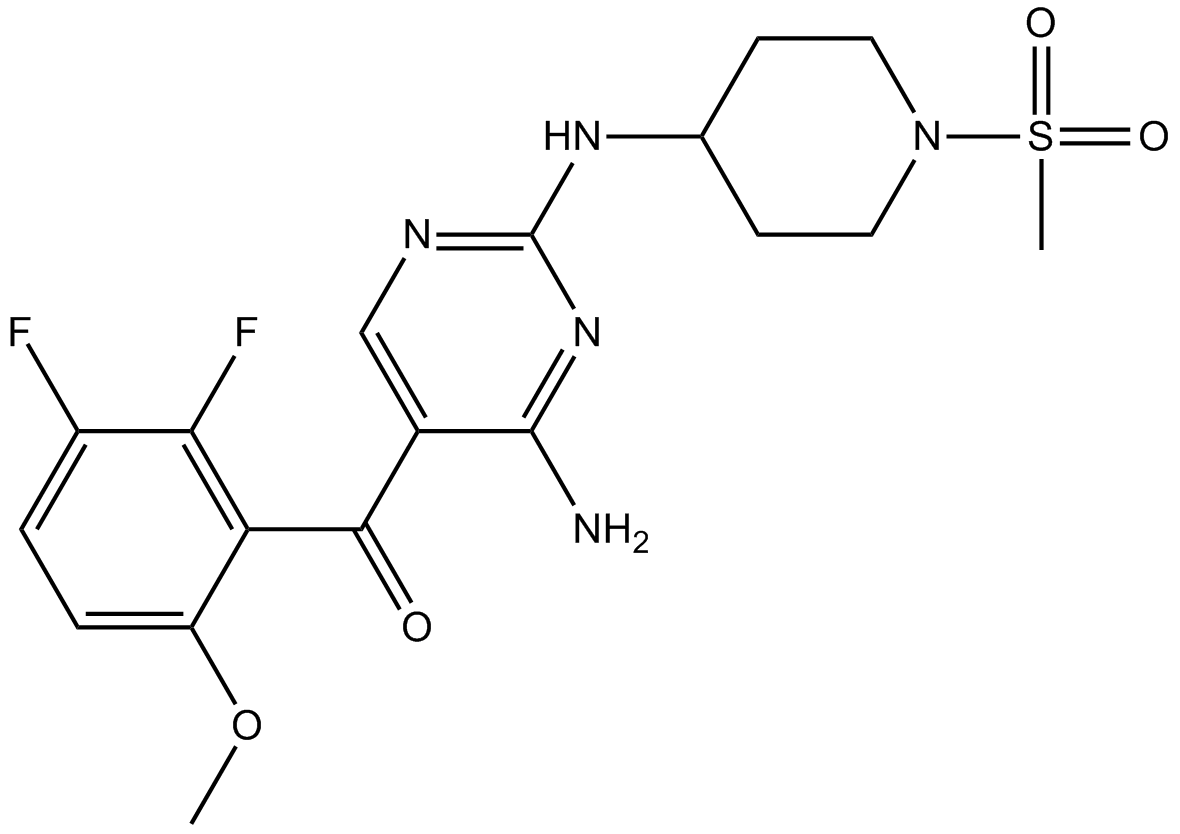
-
GC49771
rac-Hesperetin-d3
(±)-3',5,7-Trihydroxy-4'-methoxyflavanone-d3
An internal standard for the quantification of hesperetin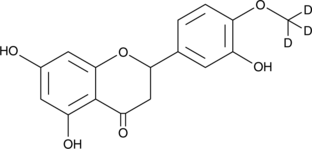
-
GC49108
Racecadotril-d5
Acetorphan-d5
An internal standard for the quantification of racecadotril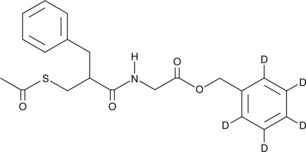
-
GC19306
RAD51 Inhibitor B02
B02
L'inhibiteur de RAD51 B02 (B02) est un inhibiteur de RAD51 humain avec une IC50 de 27,4 μM.
-
GC63165
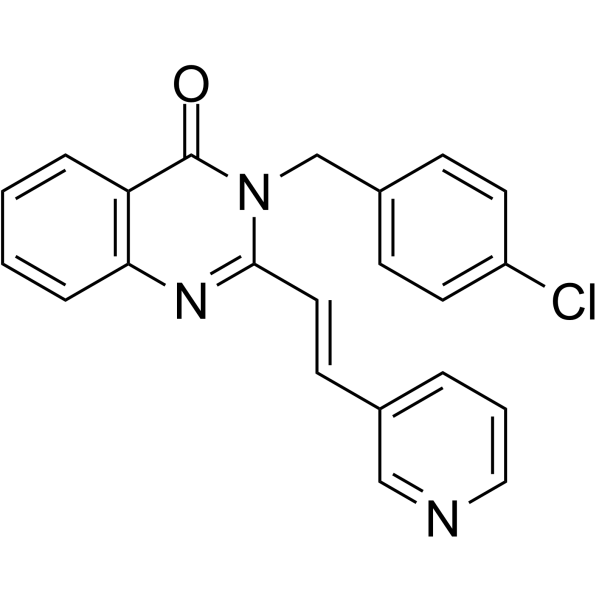
RAD51-IN-1
RAD51-IN-1, un dérivé de B02, est un puissant inhibiteur de RAD51. RAD51-IN-1 peut être utilisé pour la recherche sur le cancer.
-
GC65383
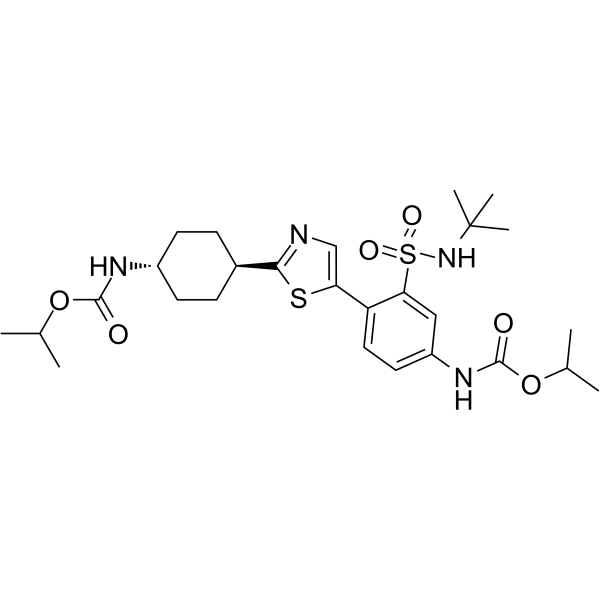
RAD51-IN-2
RAD51-IN-2 (composé exemple 67A) est un inhibiteur de RAD51 extrait du brevet WO2019/051465A1.
-
GC68416
RAD51-IN-3
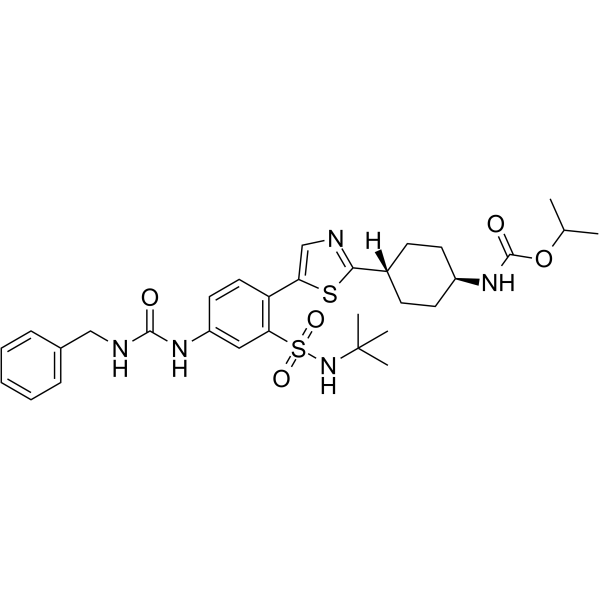
-
GC67888
RAD51-IN-5

-
GC52054
Ranitidine S-oxide
Le S-oxyde de ranitidine est le métabolite de la ranitidine.

-
GC52196
RGD Peptide
GRGDNP, HGlyArgGlyAspAsnProOH
Le peptide RGD agit comme un inhibiteur des interactions intégrine-ligand et joue un rÔle important dans l'adhésion, la migration, la croissance et la différenciation cellulaires.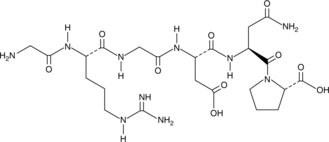
-
GC45798
Rhein-13C4
Rheic Acid-13C4
An internal standard for the quantification of rhein
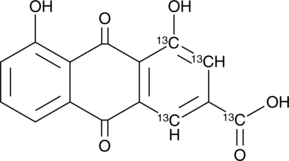
-
GC33310
Rho-Kinase-IN-1
Rho-Kinase-IN-1 est un inhibiteur de Rho kinase (ROCK) (valeurs Ki de 30,5 et 3,9 nM pour ROCK1 et ROCK2, respectivement) extrait de US20090325960A1, composé 1.008.
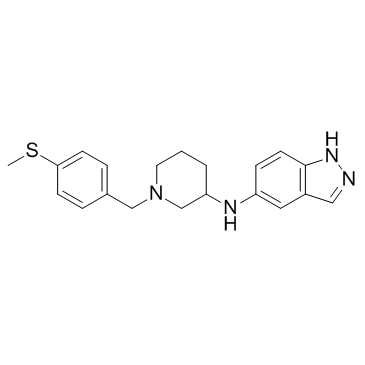
-
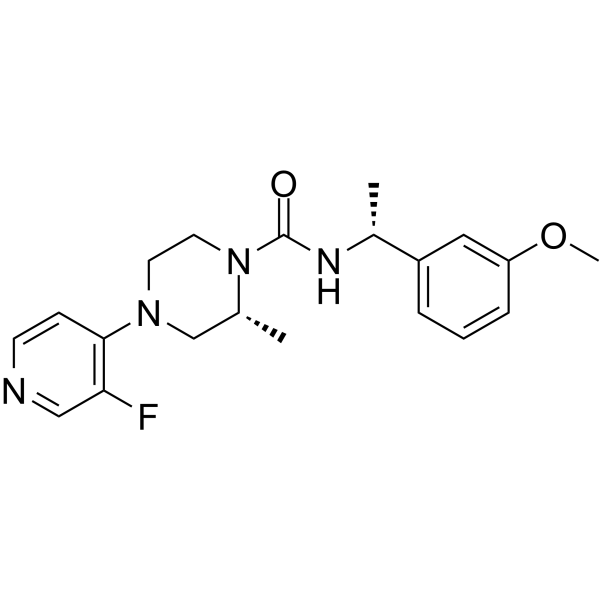
GC66050
Rho-Kinase-IN-2
La Rho-Kinase-IN-2 (Composé 23) est un inhibiteur de la Rho Kinase (ROCK) actif par voie orale, sélectif et pénétrant dans le système nerveux central (ROCK2 IC50 = 3 nM). Rho-Kinase-IN-2 peut être utilisé dans la recherche de Huntington'
-
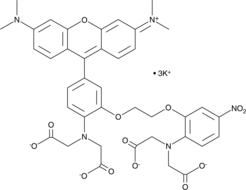
GC44829
Rhod-5N (potassium salt)
Rhod-5N is a low affinity fluorescent calcium probe (Kd = 320 μM).
-
GC18140
RI-1
A RAD51 inhibitor

-
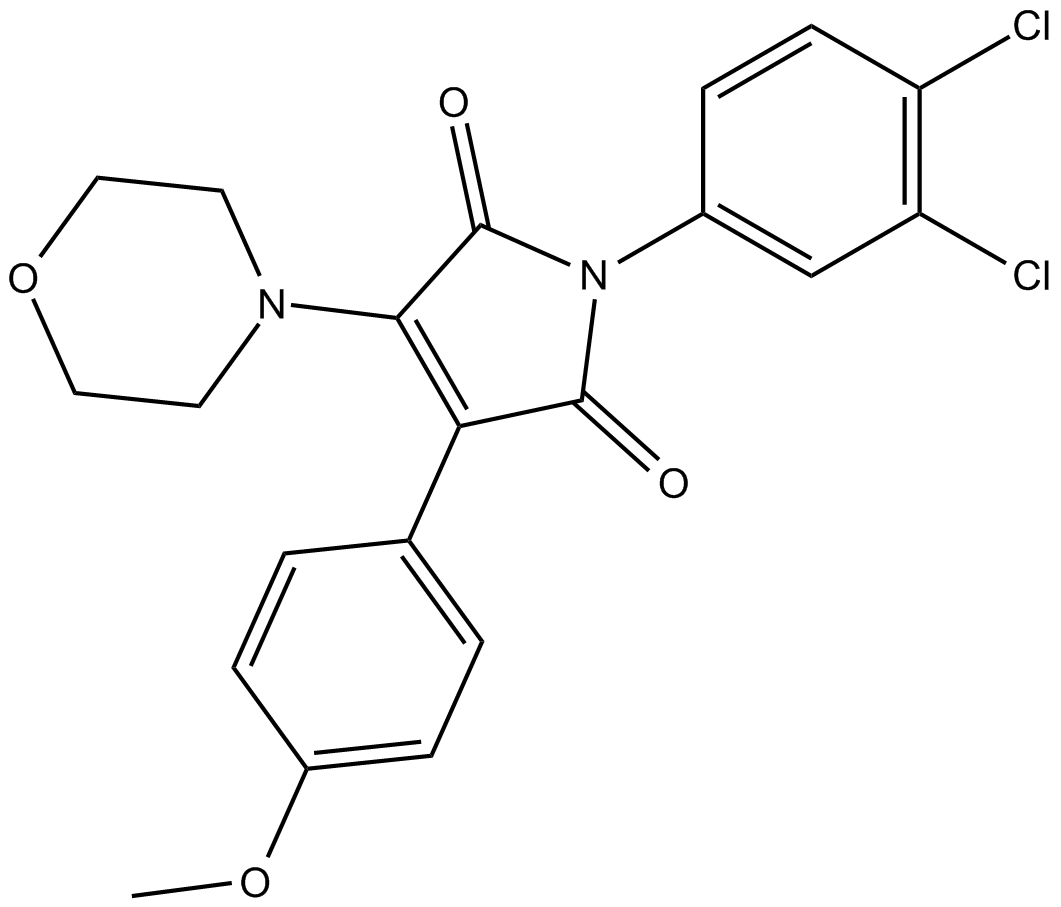
GC12752
RI-2
RI-2 est un inhibiteur réversible de RAD51, avec une IC50 de 44,17 μM, et inhibe spécifiquement la réparation par recombinaison homologue dans les cellules humaines.
-
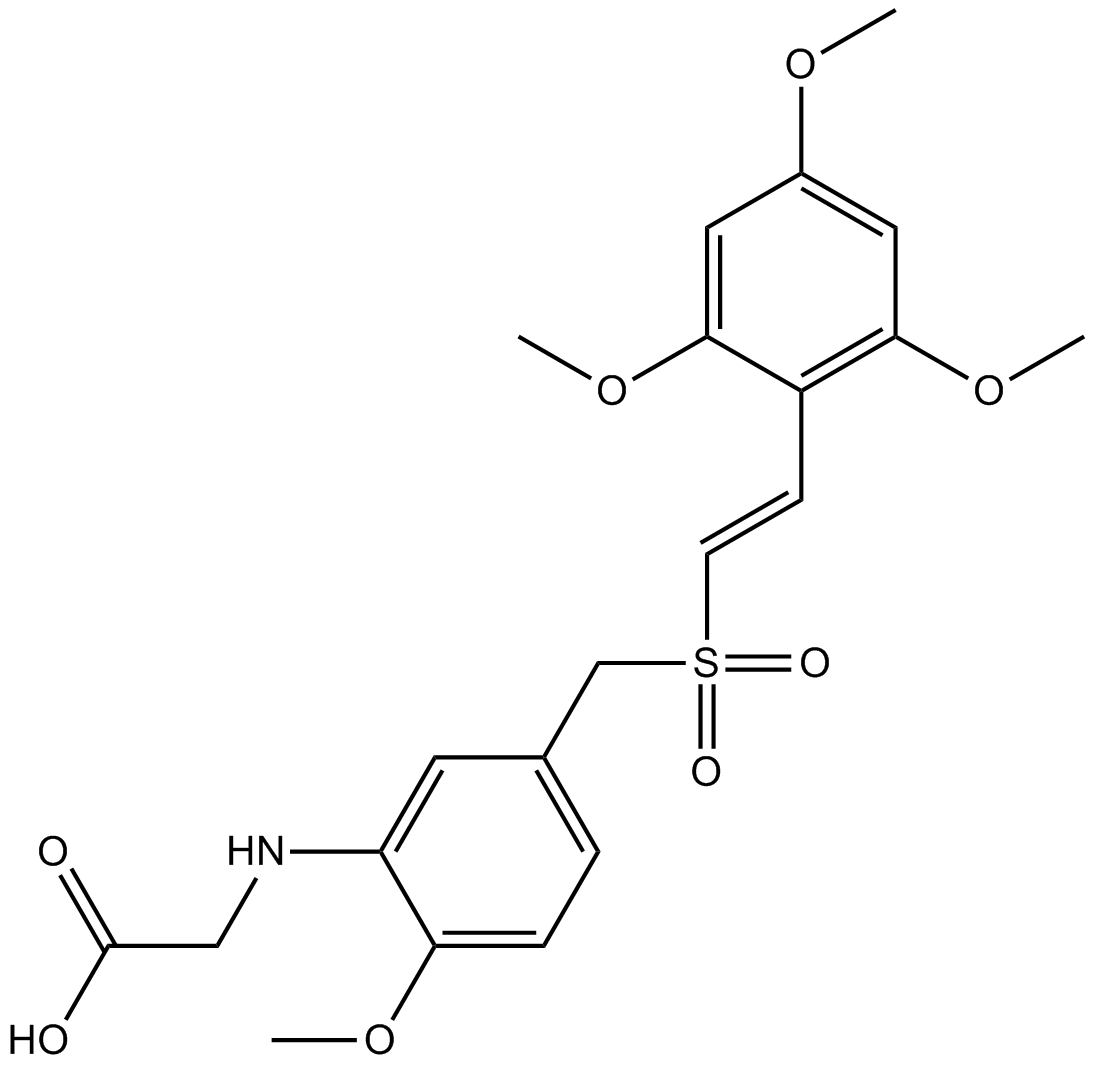
GC12415
Rigosertib
Rigosertib (ON-01910) est un inhibiteur multi-kinase et un agent anticancéreux sélectif, qui induit l'apoptose en inhibant la voie PI3 kinase/Akt, favorise la phosphorylation de l'histone H2AX et induit l'arrêt G2/M dans le cycle cellulaire. Le rigosertib est un inhibiteur sélectif et non compétitif de l'ATP de PLK1 avec une IC50 de 9 nM.
-
GC14358
Rigosertib (ON-01910,Estybon)
Rigosertib
Plk1 inhibitor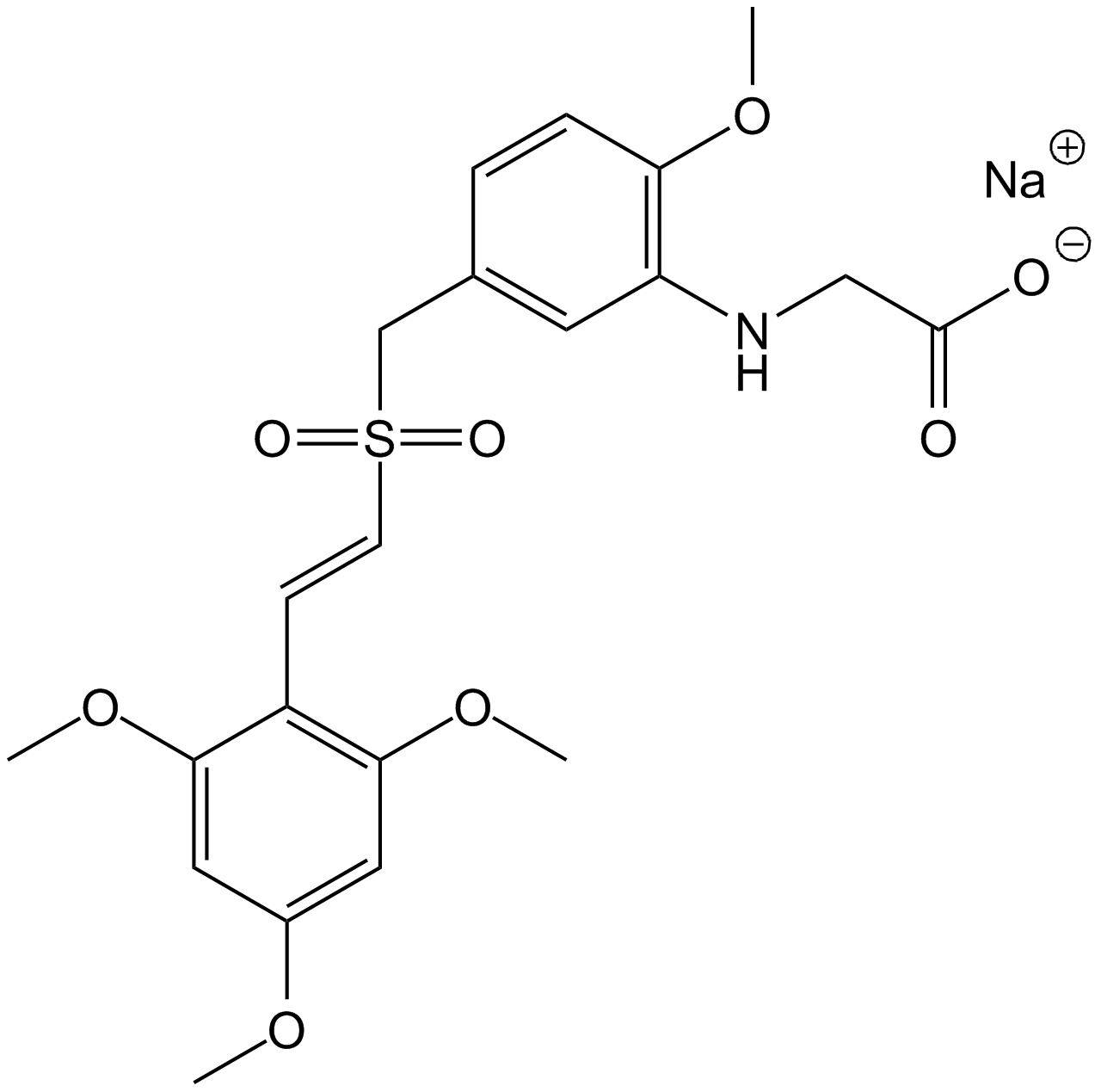
-
GC13580
Rigosertib sodium
Le rigosertib sodium (ON-01910 sodium) est un inhibiteur multi-kinase et un agent anticancéreux sélectif, qui induit l'apoptose par inhibition de la voie PI3K/Akt, favorise la phosphorylation de l'histone H2AX et induit l'arrêt G2/M dans le cycle cellulaire. Le rigosertib sodique est un inhibiteur sélectif et non compétitif de l'ATP de PLK1 avec une IC50 de 9 nM.
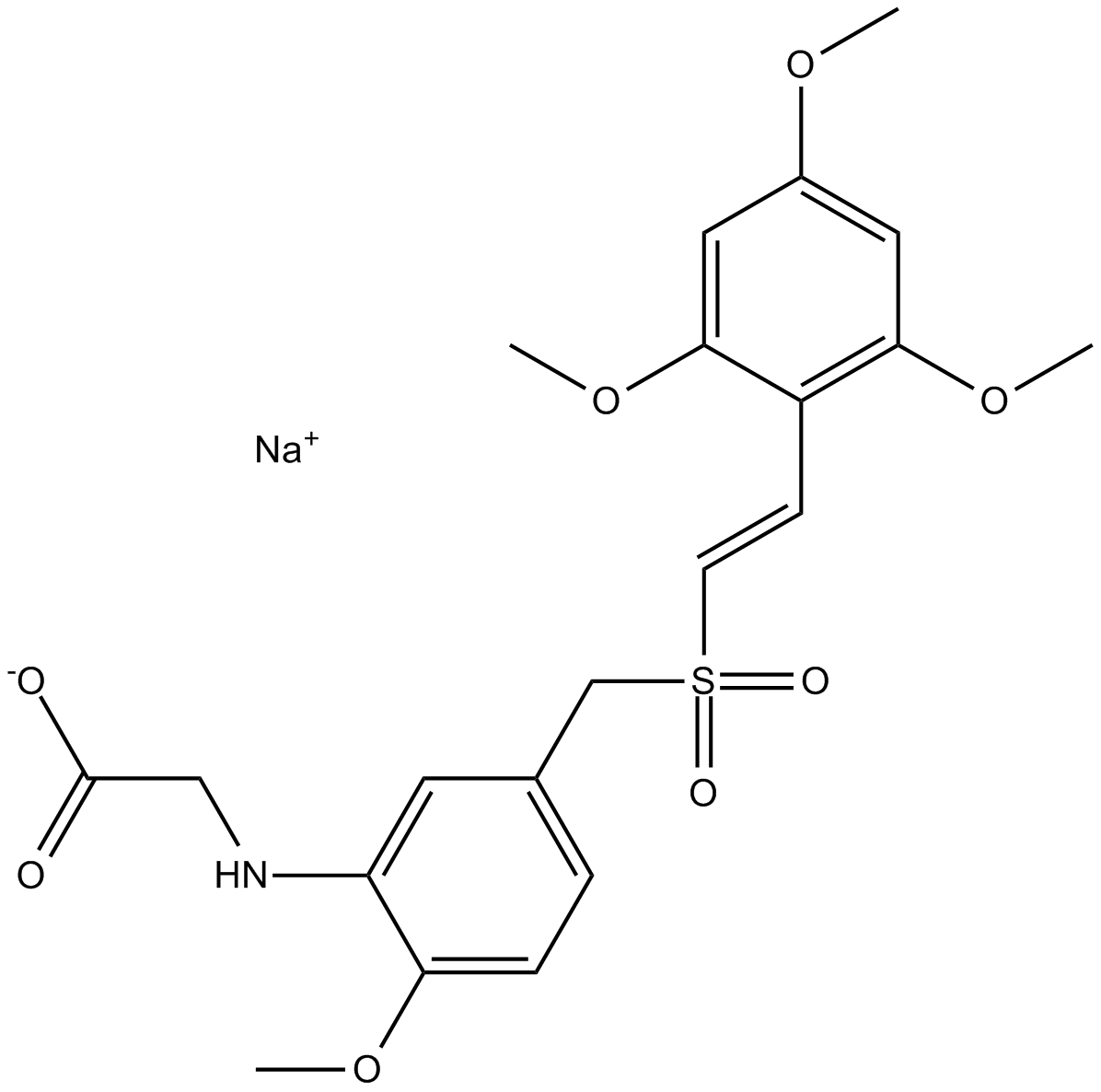
-
GC37534
Ripasudil free base
K-115 (free base)
La base libre de Ripasudil (base libre K-115) est un inhibiteur spécifique de ROCK, avec des IC50 de 19 et 51 nM pour ROCK2 et ROCK1, respectivement.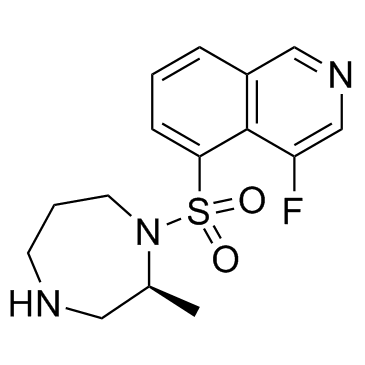
-
GC48401
Risuteganib (trifluoroacetate salt)
ALG-1001
An anti-integrin peptide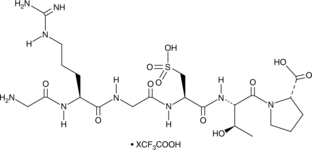
-
GC44846
RK-682 (calcium salt)
CI-010, TAN 1364B
Protein tyrosine phosphatases (PTPs) remove phosphate from tyrosine residues of cellular proteins.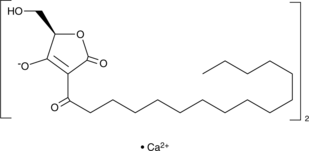
-
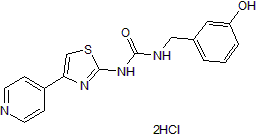
GC50092
RKI 1447 dihydrochloride
Le dichlorhydrate de RKI 1447 est un inhibiteur ROCK puissant et sélectif avec des IC50 de 14,5 et 6,2 nM pour ROCK1 et ROCK2, respectivement. Le dichlorhydrate de RKI 1447 supprime la croissance des cellules du carcinome colorectal et favorise l'apoptose.
-
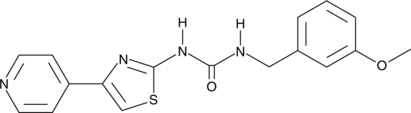
GC44847
RKI-1313
RKI-1313 est un inhibiteur de ROCK avec des IC50 de 34, 8 μM pour ROCK 1 et ROCK 2, respectivement. RKI-1313 montre peu d'effet sur les niveaux de phosphorylation des substrats ROCK, la migration, l'invasion ou la croissance indépendante de l'ancrage.
-
GC15437
RKI-1447
RKI 1447;RKI1447
A ROCK1 and ROCK2 inhibitor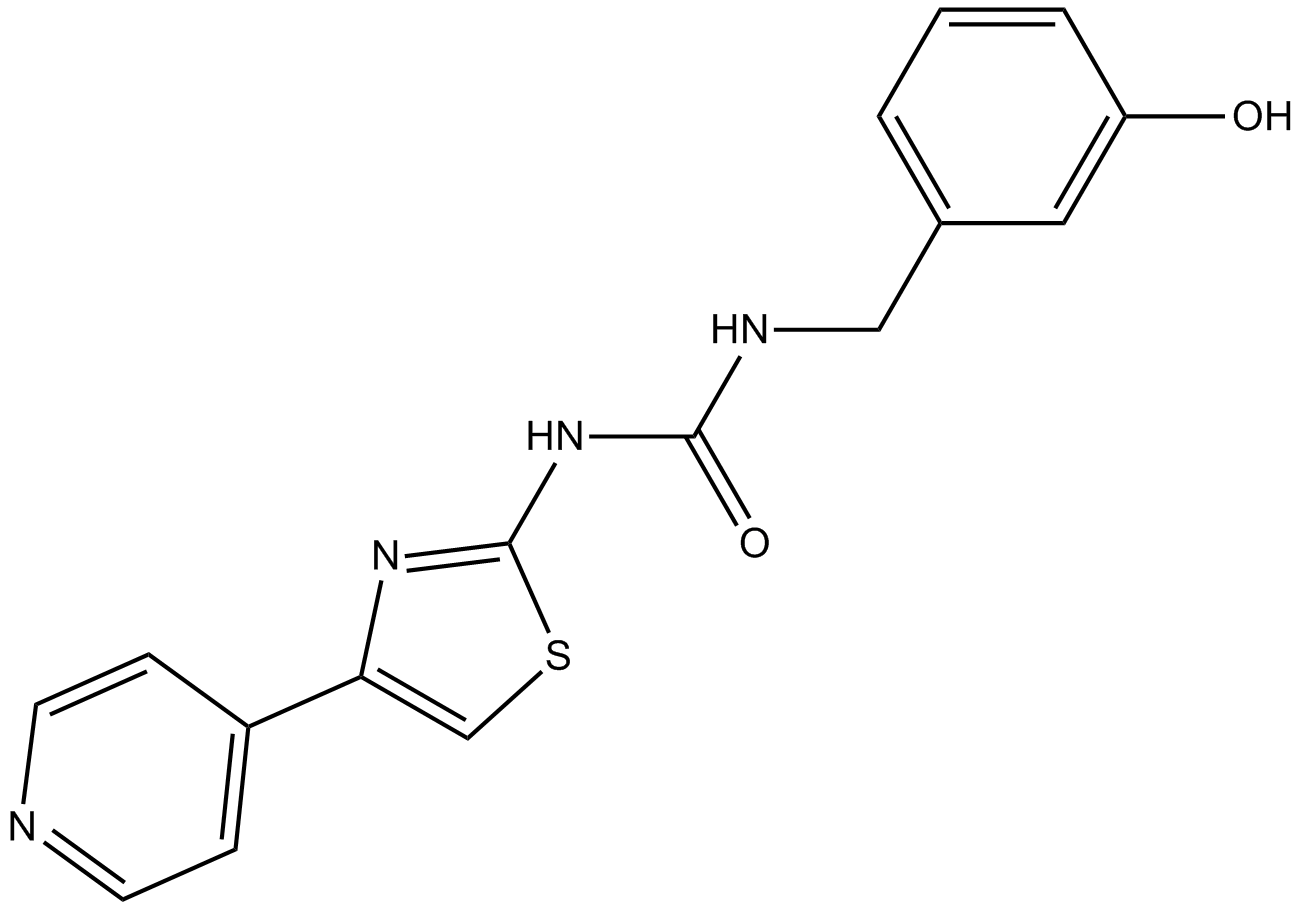
-
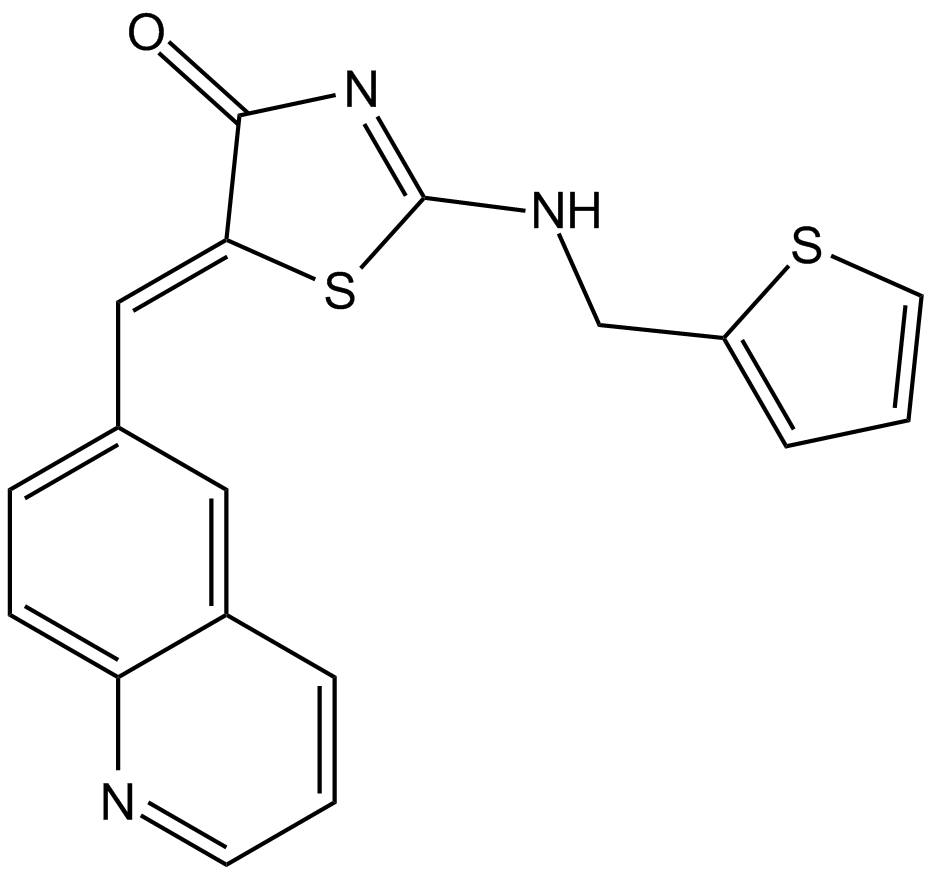
GC12348
Ro 3306
Le Ro 3306 est un inhibiteur puissant et sélectif de CDK1, avec un Kis de 20 nM, 35 nM et 340 nM pour CDK1, CDK1/cycline B1 et CDK2/cycline E, respectivement.
-
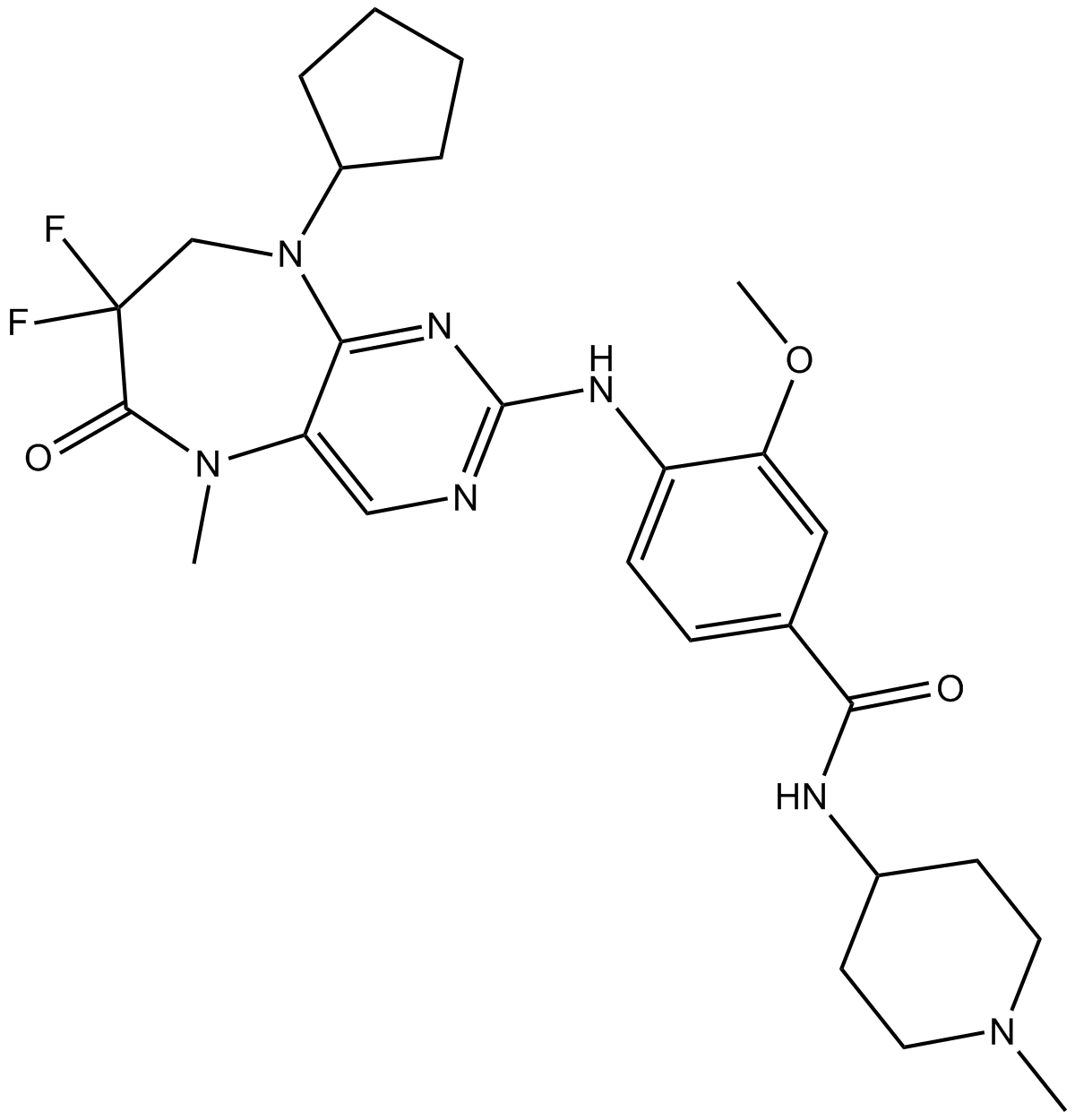
GC13903
Ro3280
Ro3280 est un inhibiteur puissant et hautement sélectif de PLK1 avec une IC50 et un Kd de 3 nM et 0,09 nM, respectivement, et n'a presque aucun effet sur PLK2 et PLK3.
-
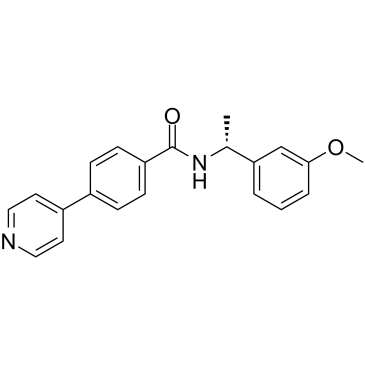
GC37551
ROCK inhibitor-2
L'inhibiteur de ROCK-2 est un double inhibiteur sélectif de ROCK1 et ROCK2 avec des IC50 de 17 nM et 2 nM, respectivement .
-
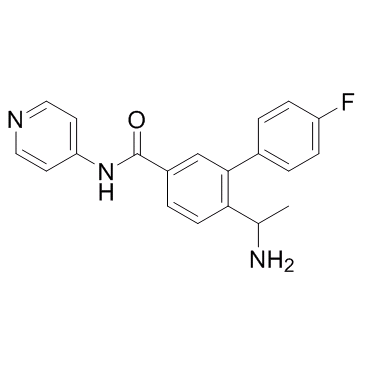
GC31952
ROCK-IN-1
ROCK-IN-1 est un puissant inhibiteur de ROCK, avec une IC50 de 1,2 nM pour ROCK2.
-
GC66338
ROCK1-IN-1
ROCK1-IN-1 est un inhibiteur de ROCK1 avec une valeur Ki de 540 nM. ROCK1-IN-1 peut être utilisé pour la recherche sur l'hypertension, le glaucome et la dysfonction érectile.
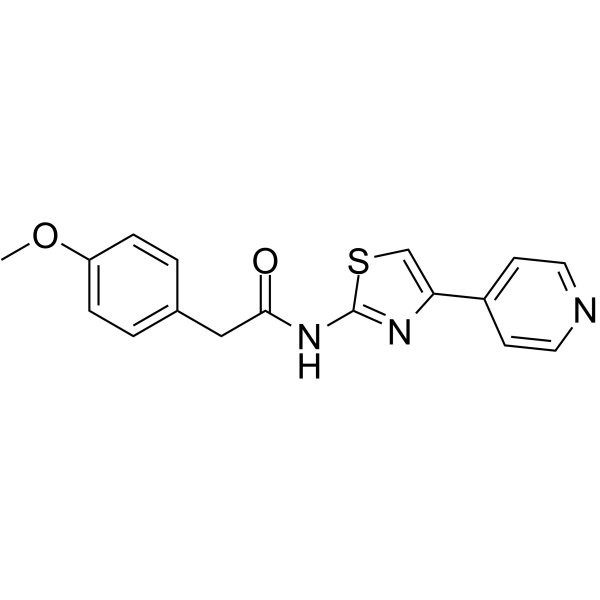
-
GC31883
ROCK2-IN-2
ROCK2-IN-2 est un inhibiteur sélectif de ROCK2 extrait du brevet US20180093978A1, Composé A-30, a une IC50 <1 μM.
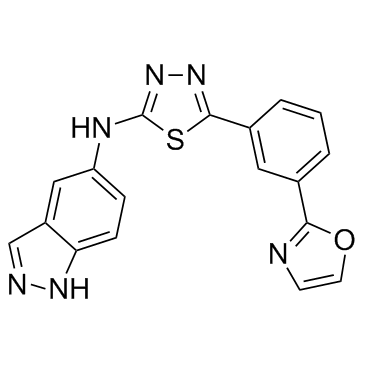
-
GC33169
Rosabulin (STA 5312)
La rosabuline (STA 5312) (STA 5312) est un inhibiteur de microtubules puissant et actif par voie orale qui inhibe l'assemblage des microtubules. La rosabuline (STA 5312) a une activité antitumorale À large spectre.
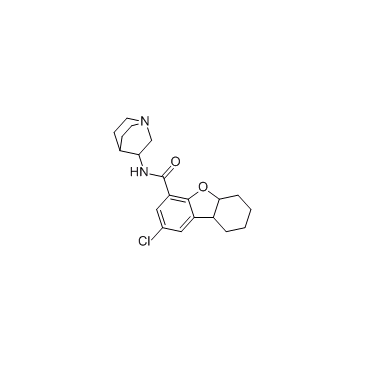
-
GC11401
Roscovitine (Seliciclib,CYC202)
Seliciclib
La roscovitine (Seliciclib, CYC202) (Roscovitine) est un inhibiteur sélectif et biodisponible des CDK par voie orale avec des CI50 de 0,2 μ ; M, 0,65 μ ; M et 0,7 μ ; M pour CDK5, Cdc2 et CDK2, respectivement.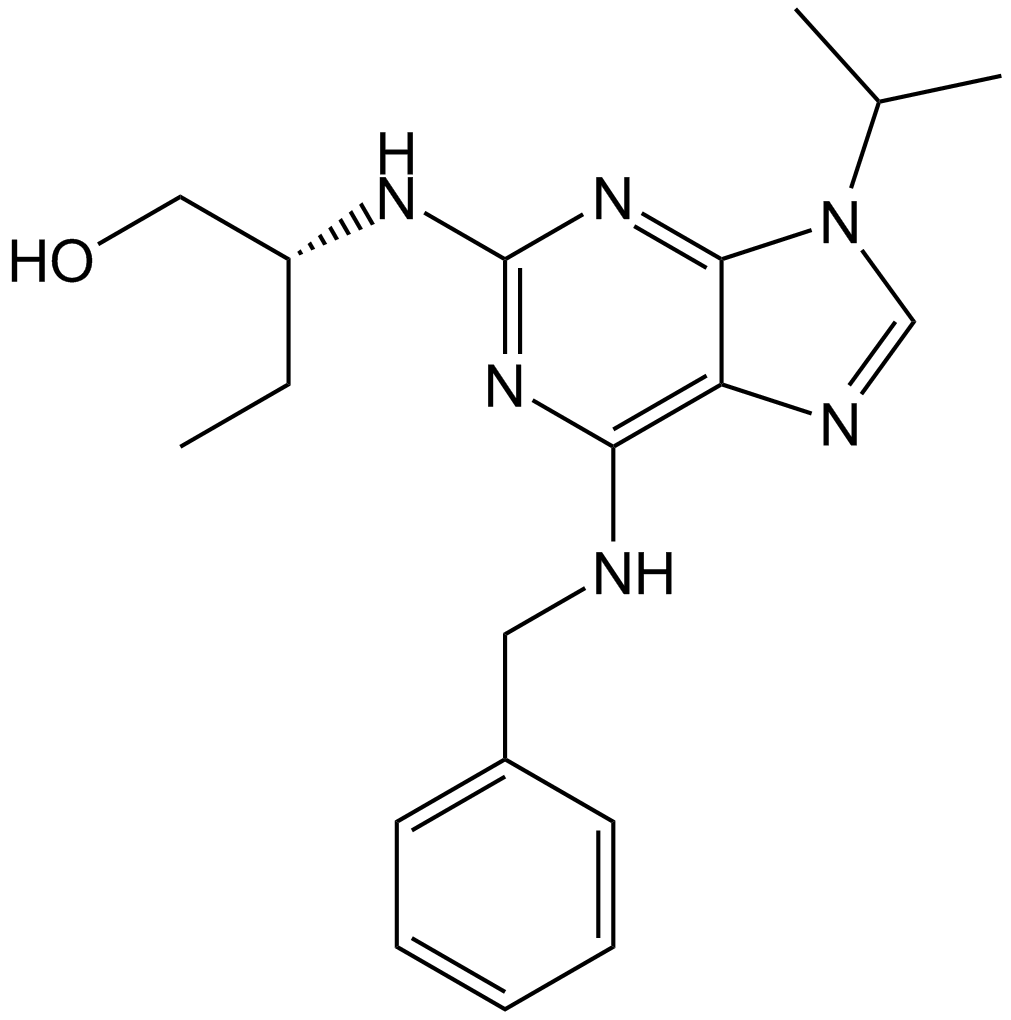
-
GC64278
RP-3500
RP-3500; ATR inhibitor 4
Le RP-3500 (inhibiteur d'ATR 4) est un inhibiteur sélectif de l'ATR kinase (ATRi) actif par voie orale avec une IC50 de 1,00 nM dans les tests biochimiques. Le RP-3500 présente une sélectivité de 30 fois pour l'ATR sur mTOR (IC50 = 120 nM) et une sélectivité > 2 000 fois sur les kinases ATM, DNA-PK et PI3Kα. Le RP-3500 a une puissante activité antitumorale.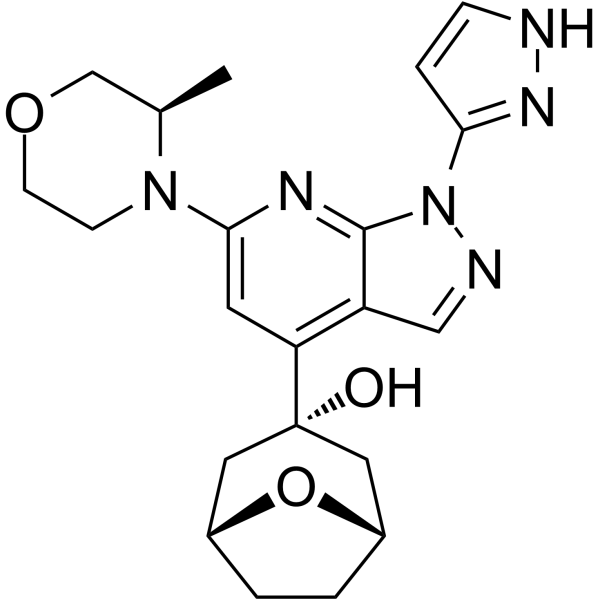
-
GC16265
RS-1
RS-1 est un activateur RAD51 et augmente également les efficacités d'activation médiées par CRISPR/Cas9.
-
GC18847
RSM-932A
TCD-717
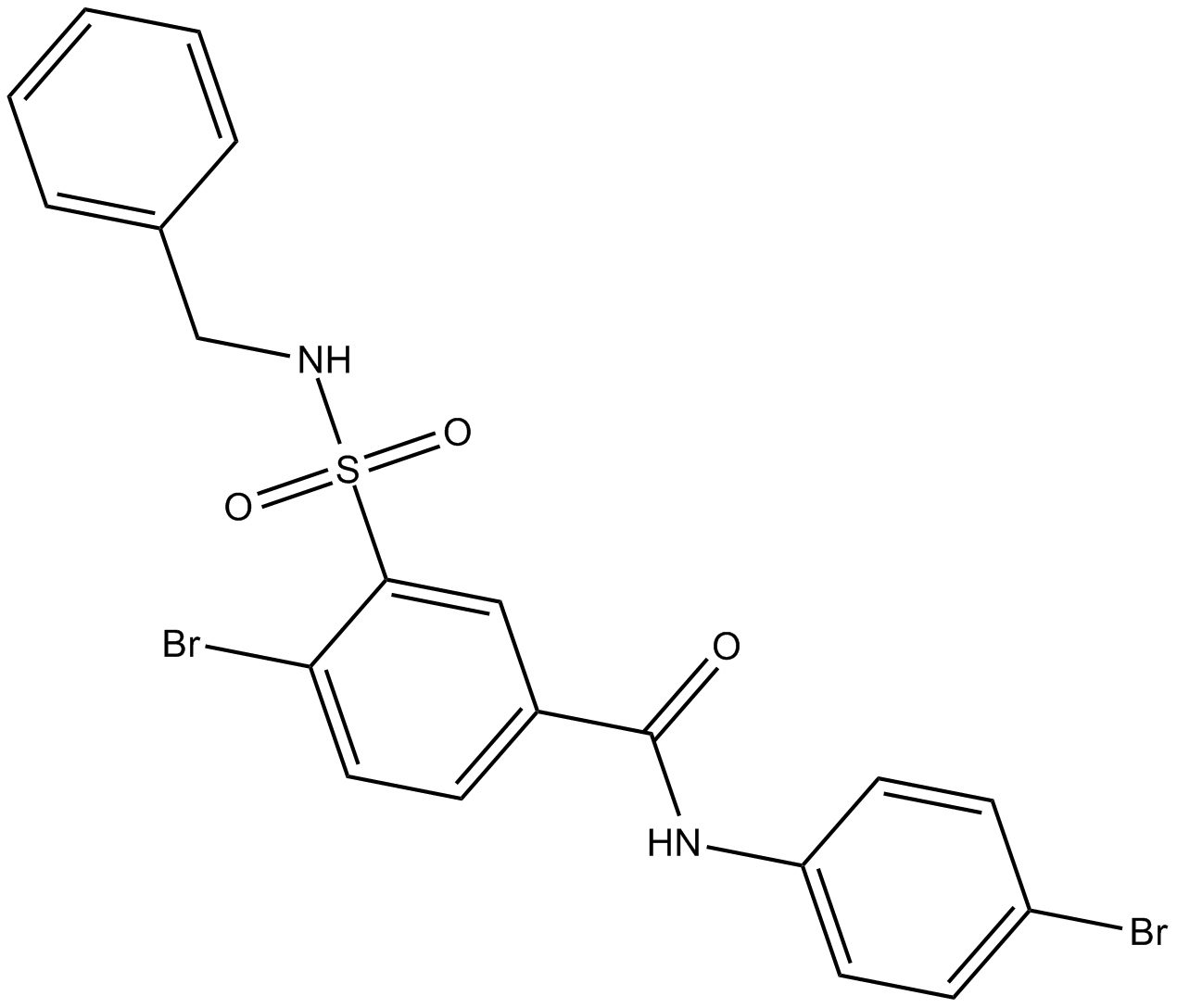
Le RSM-932A (TCD-717) est un inhibiteur spécifique de ChoKα avec des IC50 de 1 et 33 μM pour les enzymes humaines recombinantes ChoKα et ChoKβ, respectivement. RSM-932A agit comme le « premier composé chez l'homme » ciblant ChoKα. Le RSM-932A a une puissante activité antiproliférative in vitro et antitumorale in vivo contre les xénogreffes humaines chez la souris, montrant une efficacité élevée avec des profils de faible toxicité.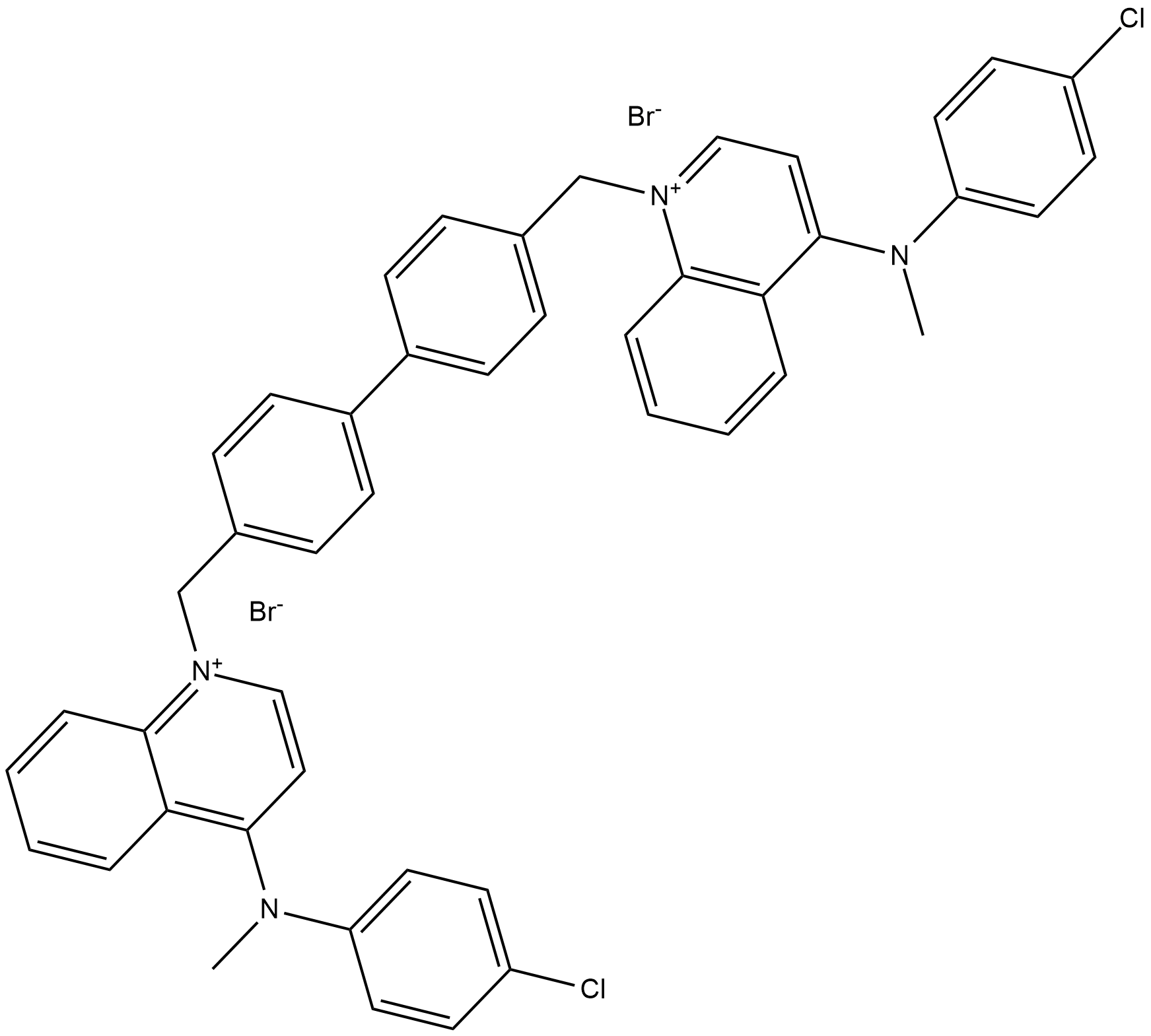
-
GC17150
Ryuvidine
Cdk4 Inhibitor III, Cyclic-dependent Kinase 4 Inhibitor III, SPS812
La ryuvidine est un puissant inhibiteur de la protéine 8 contenant le domaine SET (SETD8) avec une IC50 de 0,5 μM et supprime la monométhylation de H4K20 in vitro. La ryuvidine inhibe également CDK4 avec une IC50 de 6,0 μM et est cytotoxique contre une gamme de cellules cancéreuses humaines.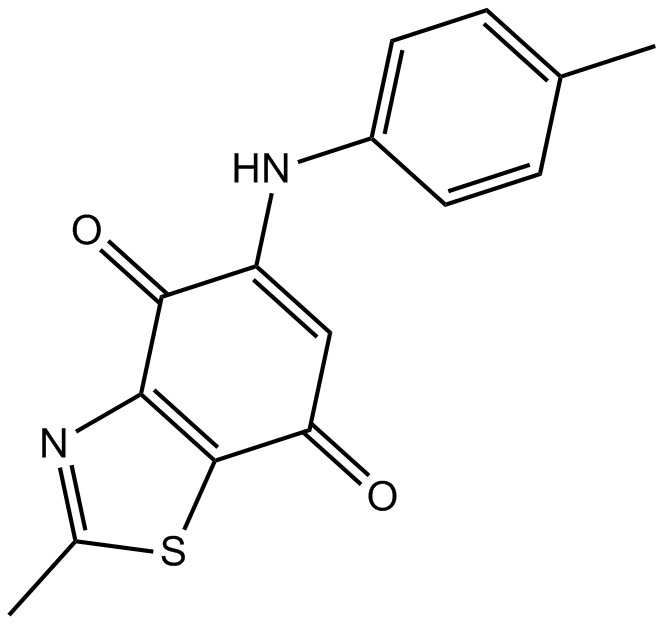
-
GC49532
S-(1,2-Dichlorovinyl)-Cysteine (hydrochloride)
S-(1,2-dichlorovinyl)-L-Cysteine, S-(trans-1,2-dichlorovinyl)-L-Cysteine, DCVC
A nephrotoxin and metabolite of trichloroethylene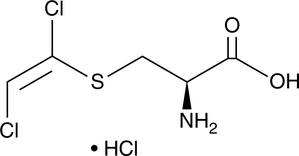
-
GC46224
S-Phenylcysteine
3-(phenylthio)-L-Alanine, (2R)-2-Amino-3-phenylsulfanylpropanoic acid
An adduct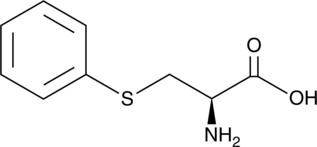
-
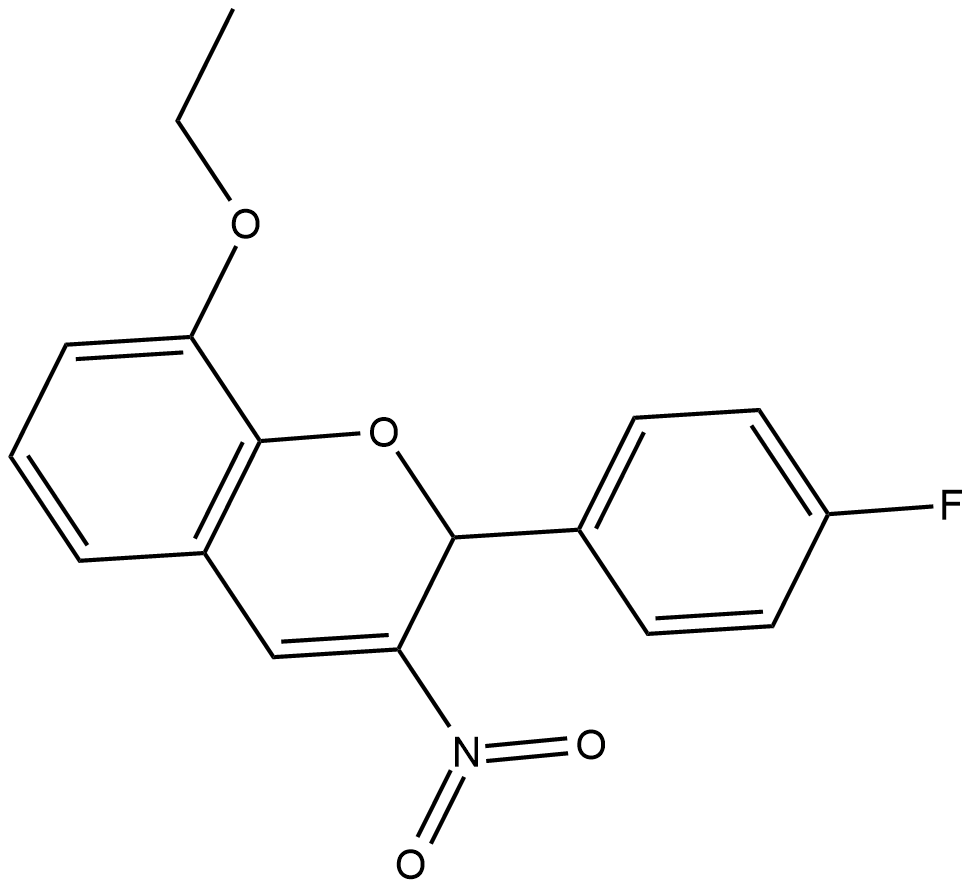
GC18852
S14161
D-Cyclins regulate the cell cycle by acting in a complex with cyclin dependent kinases (CDKs) to promote phosphorylation of the retinoblastoma protein and initiate cellular progression from the G1 to the S phase.
-
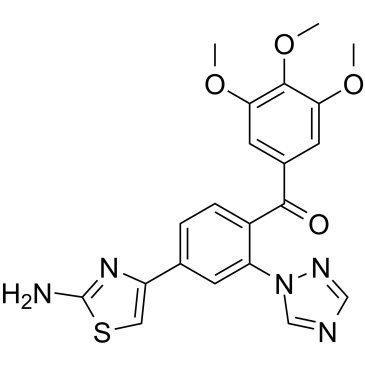
GC38945
S516
Le S516 (Composé 22) est un métabolite actif du CKD-516 et un puissant inhibiteur de la polymérisation de la tubuline avec une IC50 de 4,29 μM. S516 a une activité antitumorale marquée.
-
GC49093
Safflower Red
Carthamin Yellow
A red pigment with diverse biological activities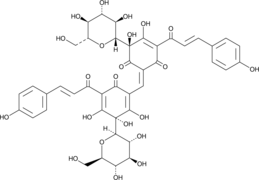
-
GC38845
sAJM589
sAJM589 est un inhibiteur de Myc qui perturbe puissamment l'hétérodimère Myc-Max avec une IC50 de 1,8 μM.
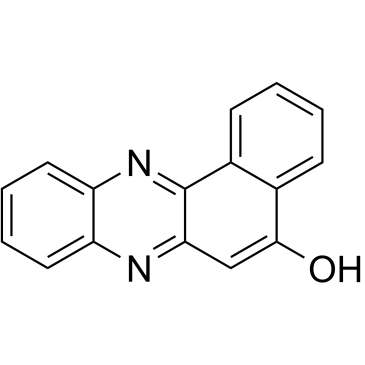
-
GC13759
SAR407899
SAR407899 est un inhibiteur ROCK sélectif, puissant et compétitif pour l'ATP, avec une IC50 de 135 nM pour ROCK-2 et une Kis de 36 nM et 41 nM pour ROCK-2 humain et de rat, respectivement.
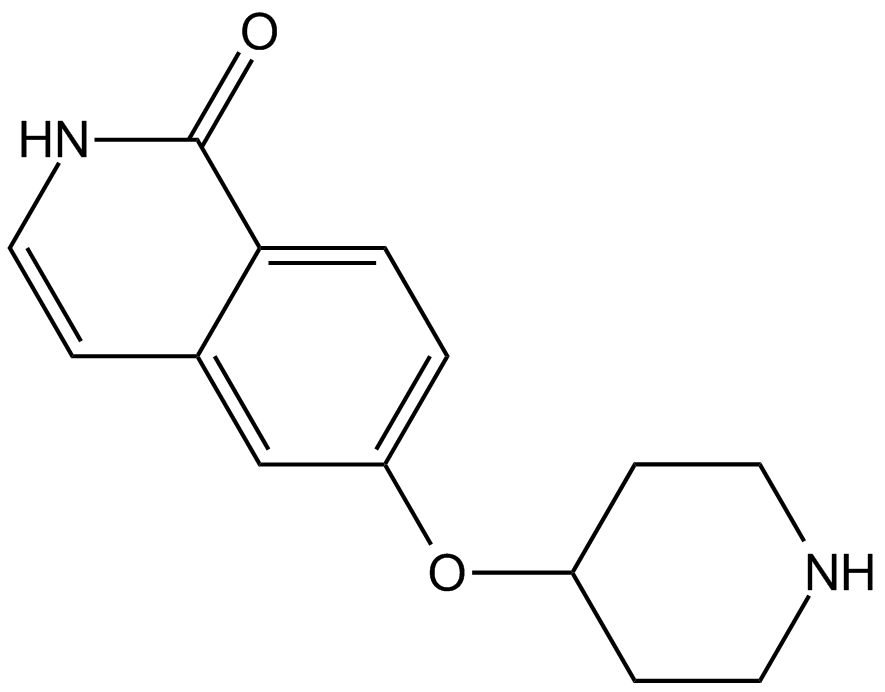
-
GC16289
SAR407899 hydrochloride
Le chlorhydrate de SAR407899 est un inhibiteur ROCK sélectif, puissant et compétitif pour l'ATP, avec une IC50 de 135 nM pour ROCK-2 et une Kis de 36 nM et 41 nM pour ROCK-2 humain et de rat, respectivement.
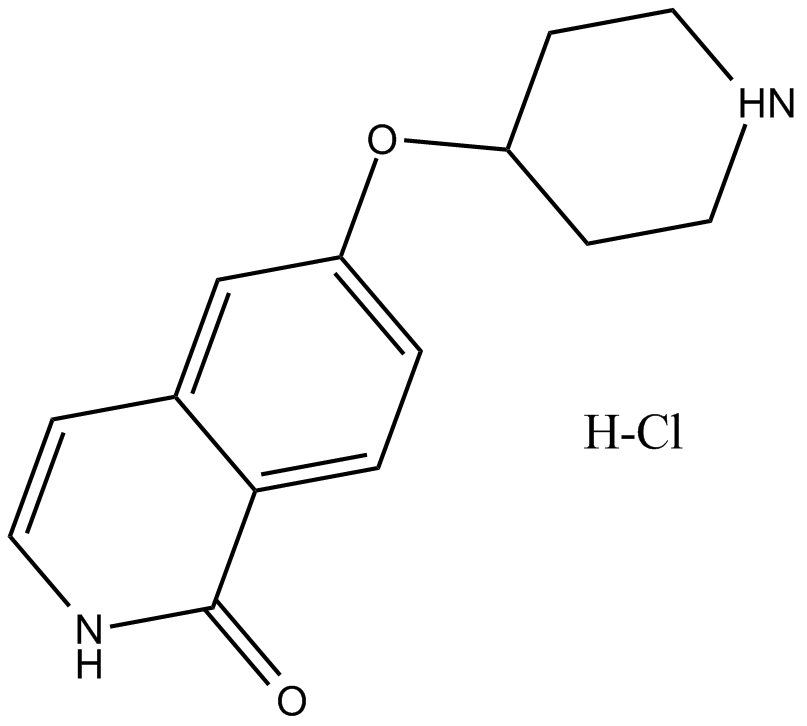
-
GC46218
Satratoxin G
A macrocyclic trichothecene mycotoxin
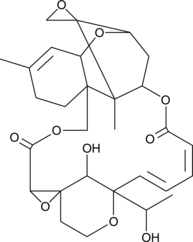
-
GC46219
Satratoxin H
A trichothecene mycotoxin
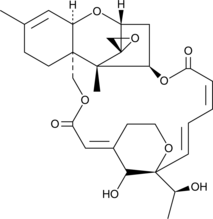
-
GC11022
SB 218078
Le SB 218078 est un inhibiteur puissant, sélectif, compétitif pour l'ATP et perméable aux cellules de la kinase 1 (Chk1) du point de contrôle qui inhibe la phosphorylation de cdc25C par Chk1 avec une IC50 de 15 nM. SB 218078 inhibe moins puissamment Cdc2 (IC50 de 250 nM) et PKC (IC50 de 1000 nM). SB 218078 provoque l'apoptose par des dommages à l'ADN et l'arrêt du cycle cellulaire.
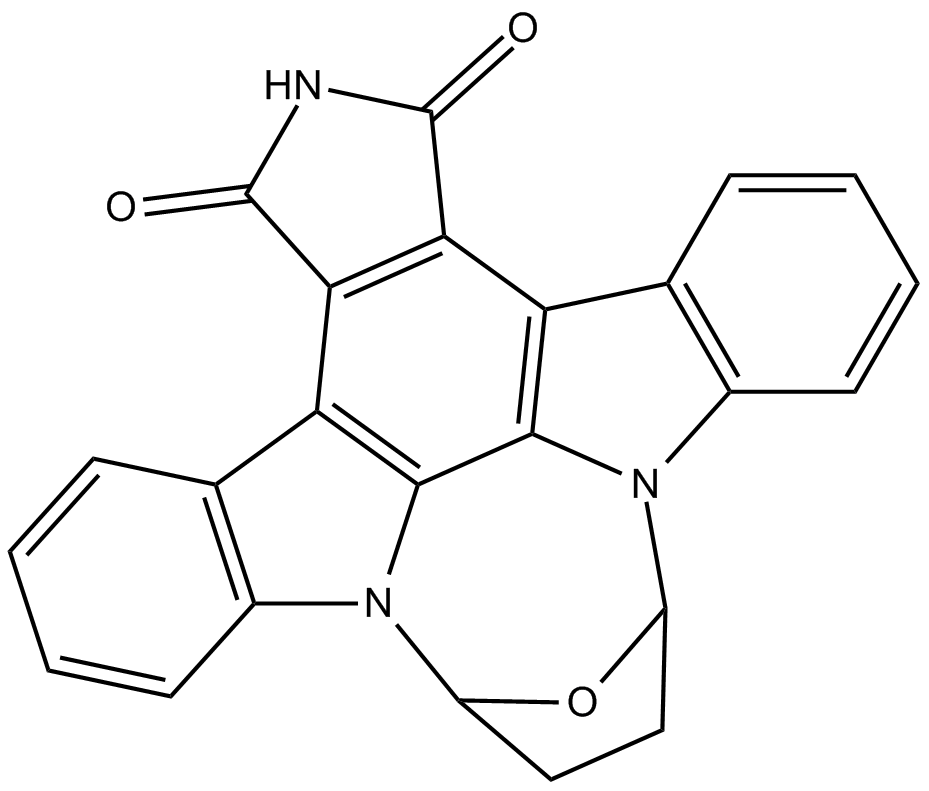
-
GC15170
SB 772077B dihydrochloride
Le dichlorhydrate de SB 772077B est un inhibiteur de Rho kinase à base d'aminofurazan (ROCK) avec des IC50 de 5,6 nM et 6 nM vers ROCK1 et ROCK2, respectivement.
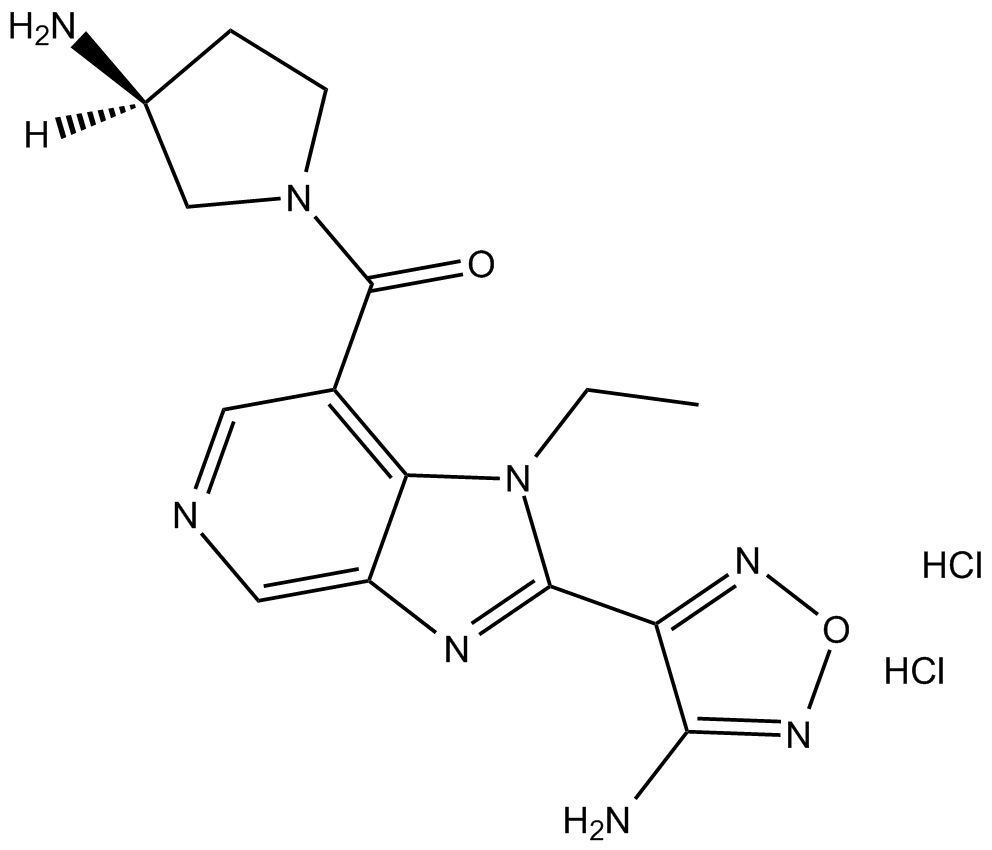
-
GC66437
SB-216
Le SB-216 est un puissant inhibiteur de polymérisation de la tubuline. Le SB-216 présente une forte puissance antiproliférative dans un panel de lignées cellulaires cancéreuses humaines, notamment le mélanome, le cancer du poumon et le cancer du sein. Le SB-216 peut être utilisé pour la recherche sur le cancer.
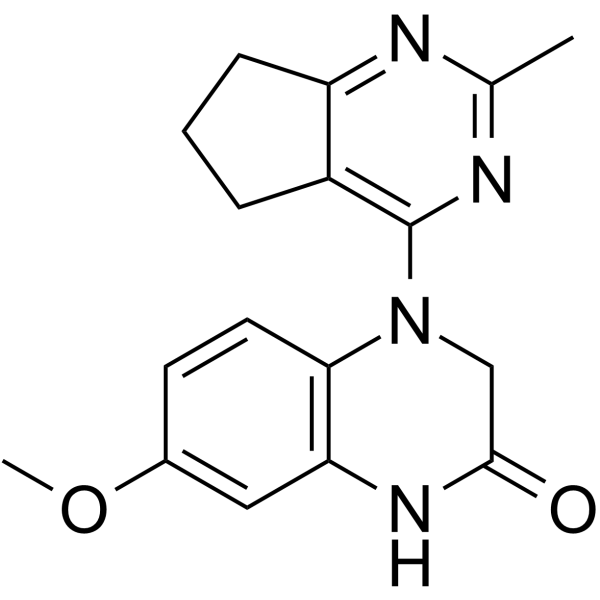
-
GC12064
SB1317
TG02, Zotiraciclib
A multi-kinase inhibitor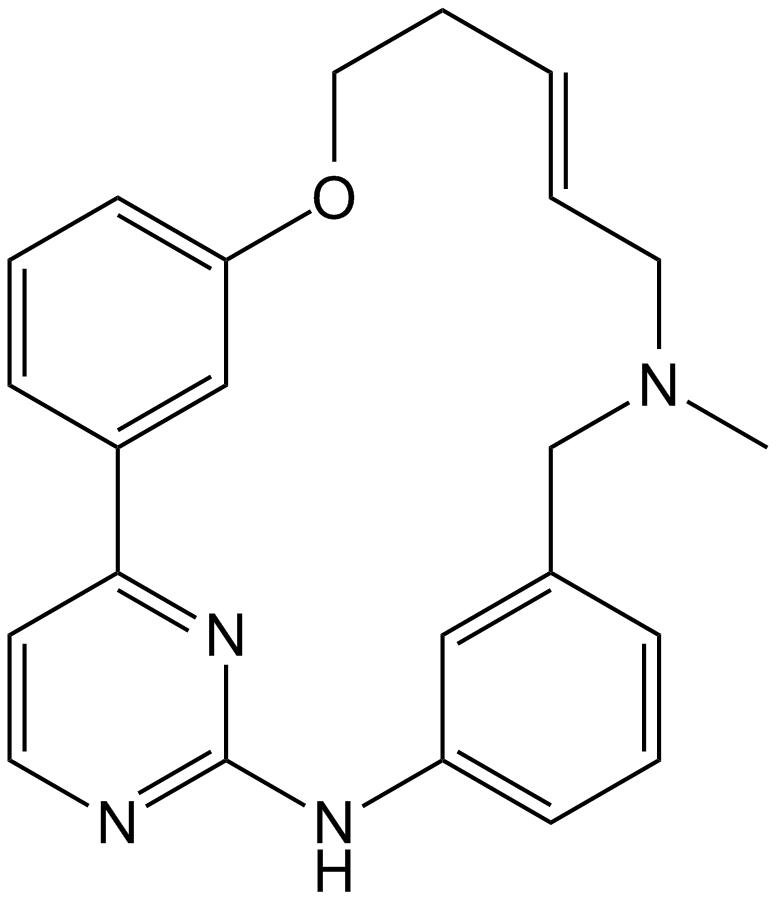
-
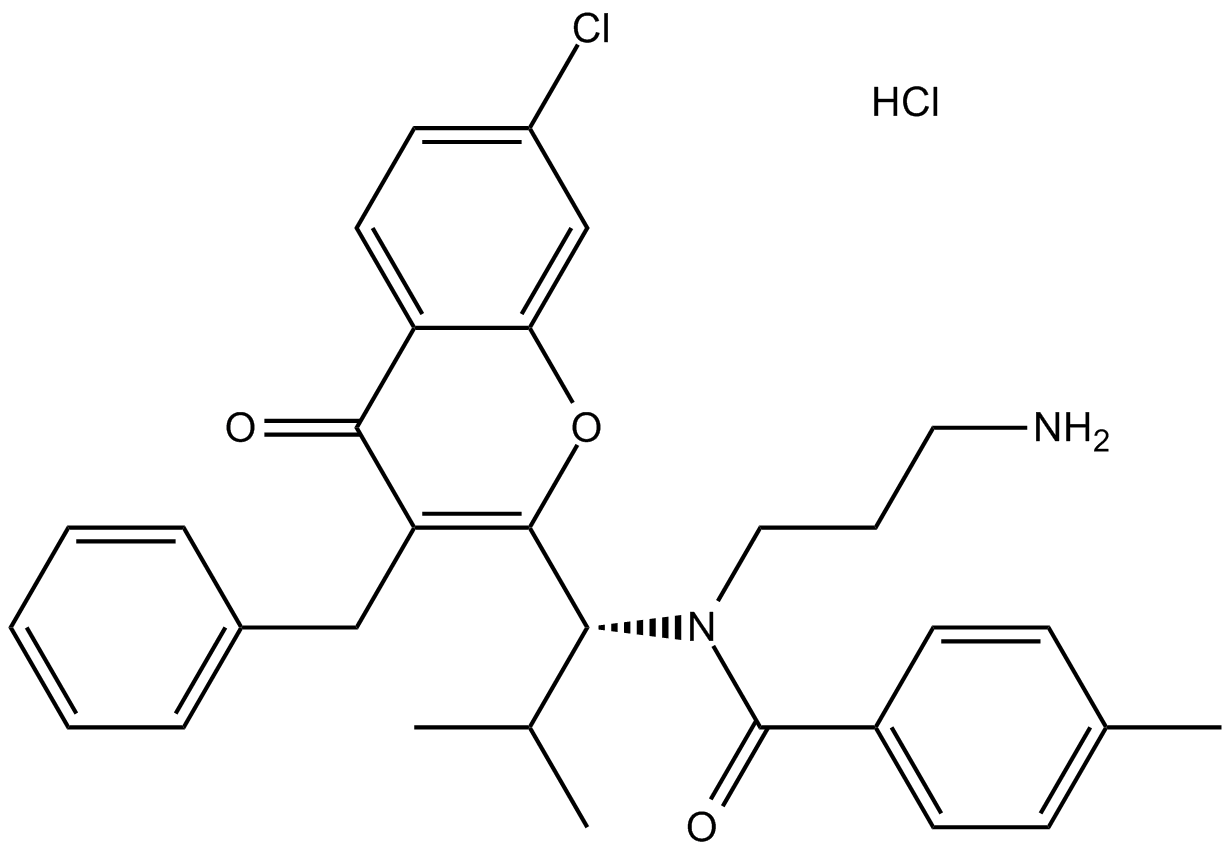
GC14600
SB743921
SB743921 est un puissant inhibiteur de la kinésine mitotique KSP (Eg5), avec un Ki de 0,1 nM.
-
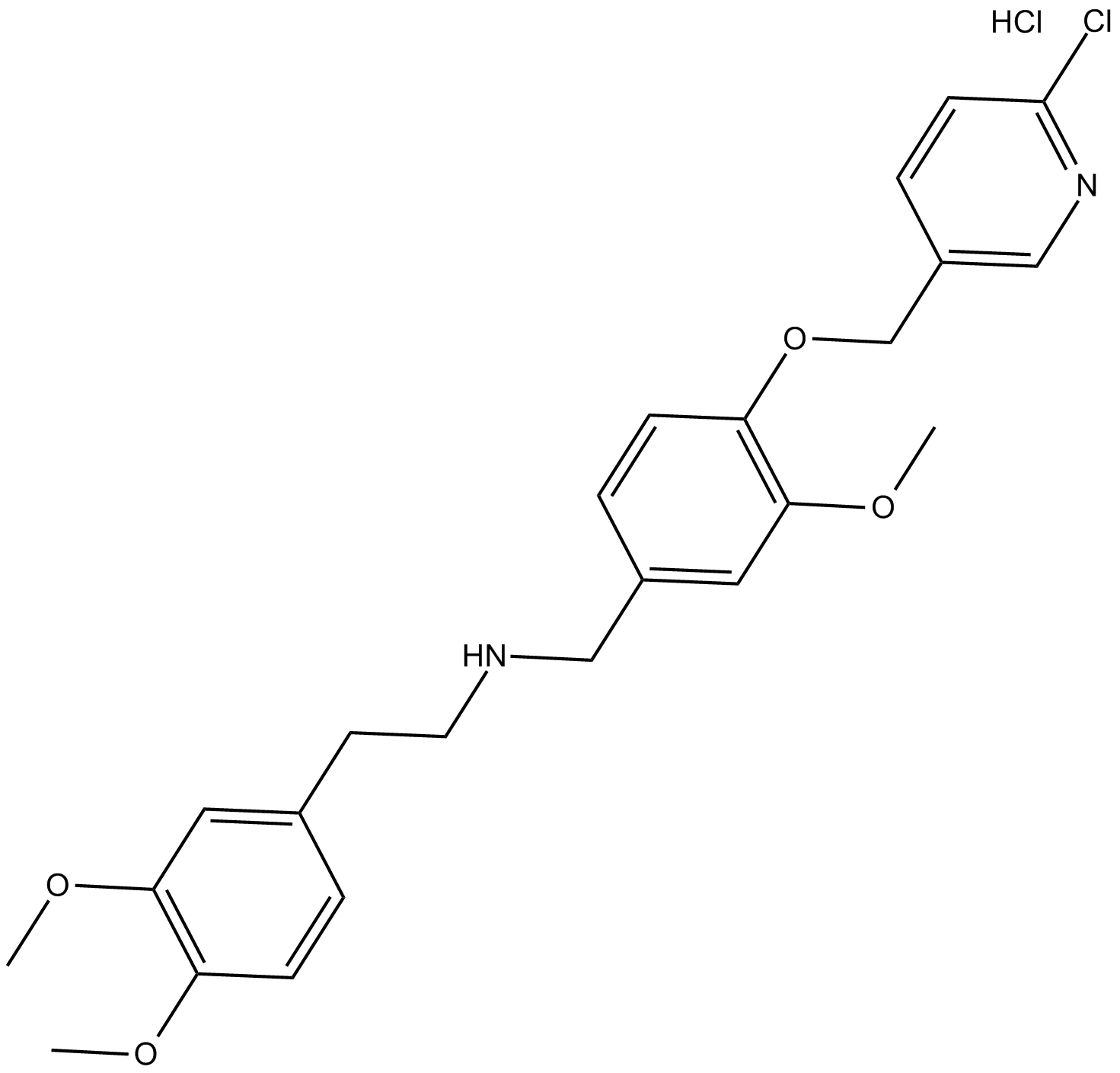
GC14644
SBE 13 HCl
SBE 13 HCl est un inhibiteur puissant et sélectif de Plk1, avec une IC50 de 200 pM ; Le SBE 13 HCl inhibe mal Plk2 (IC50>66μM) ou Plk3 (IC50=875nM).
-
GC37601
SBE13
SBE13 est un inhibiteur puissant et sélectif de Plk1, avec une IC50 de 200 pM ; SBE13 inhibe mal Plk2 (IC50> 66μM) ou Plk3 (IC50 = 875nM).
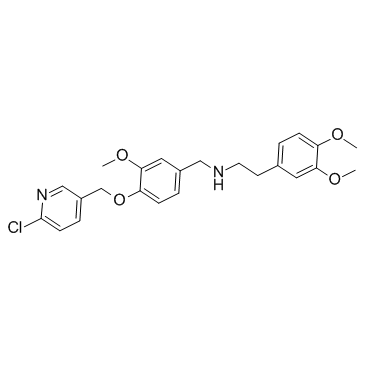
-
GC37606
SCH-1473759 hydrochloride
Le chlorhydrate de SCH-1473759 est un inhibiteur d'aurores avec des CI50 de 4 et 13 nM pour les aurores A et B, respectivement.
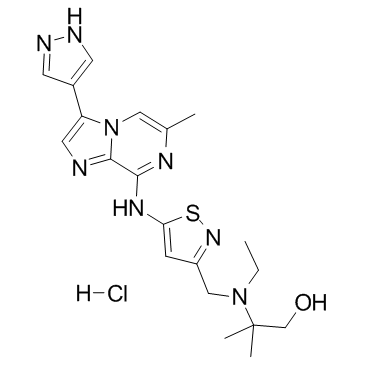
-
GC17792
SCH900776 S-isomer
SCH 900776 S-isomer;SCH-900776 S-isomer
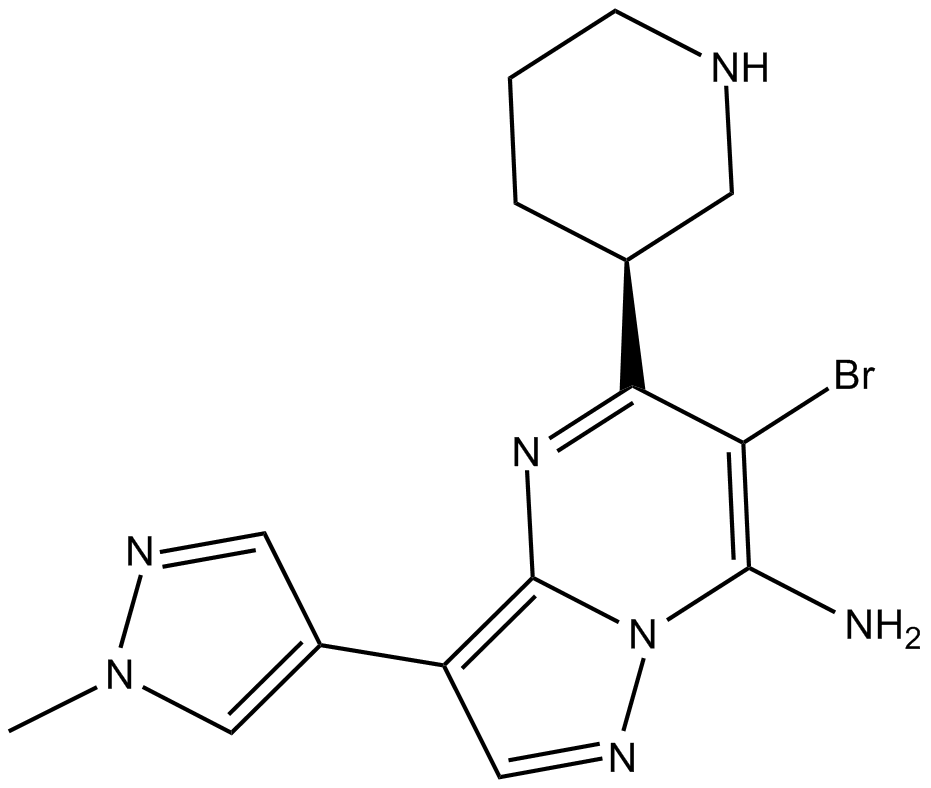
-
GN10091
Schaftoside
Apigenin 6-C-glucoside-8-C-arabinoside
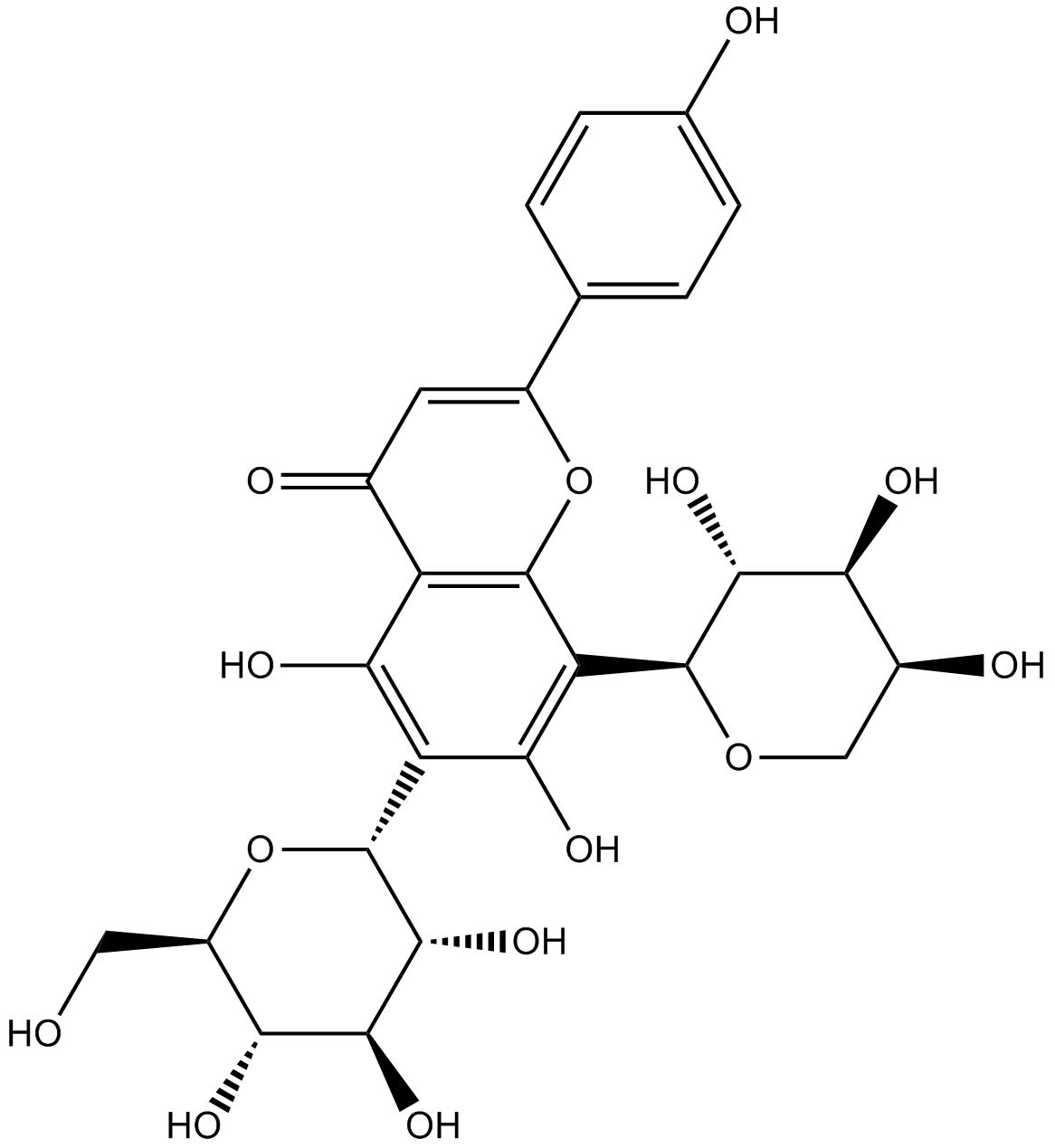
-
GC49674
Schizandrin
(+)-Schizandrin, Schizandrol A
La schizandrine (Schizandrin), un lignane dibenzocyclooctadiène, est isolée du fruit de Schisandra chinensis Baill.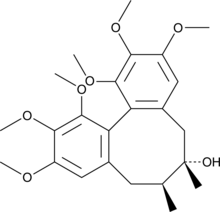
-
GC39798
Scoulerine
(–)-Scoulerine, l-Scoulerine, (S)-Scoulerine
La Scoulerine ((-)-Scoulerine), un alcaloÏde de l'isoquinoline, est un puissant composé antimitotique. La scoulerine est également un inhibiteur de BACE1 (enzyme de clivage 1 de la protéine précurseur de l'amyloÏde du site ß). La scoulerine inhibe la prolifération, arrête le cycle cellulaire et induit l'apoptose des cellules cancéreuses.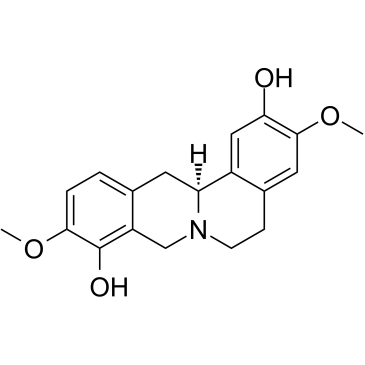
-
GC49723
SenTraGor™ Cell Senescence Reagent
GL13, SBB-A-B, SBB-Analogue (GL13) Biotin
A cellular senescence detection reagent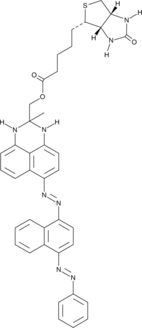
-
GC48076
Sesquicillin A
Sesquicillin
A fungal metabolite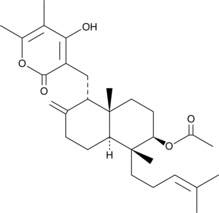
-
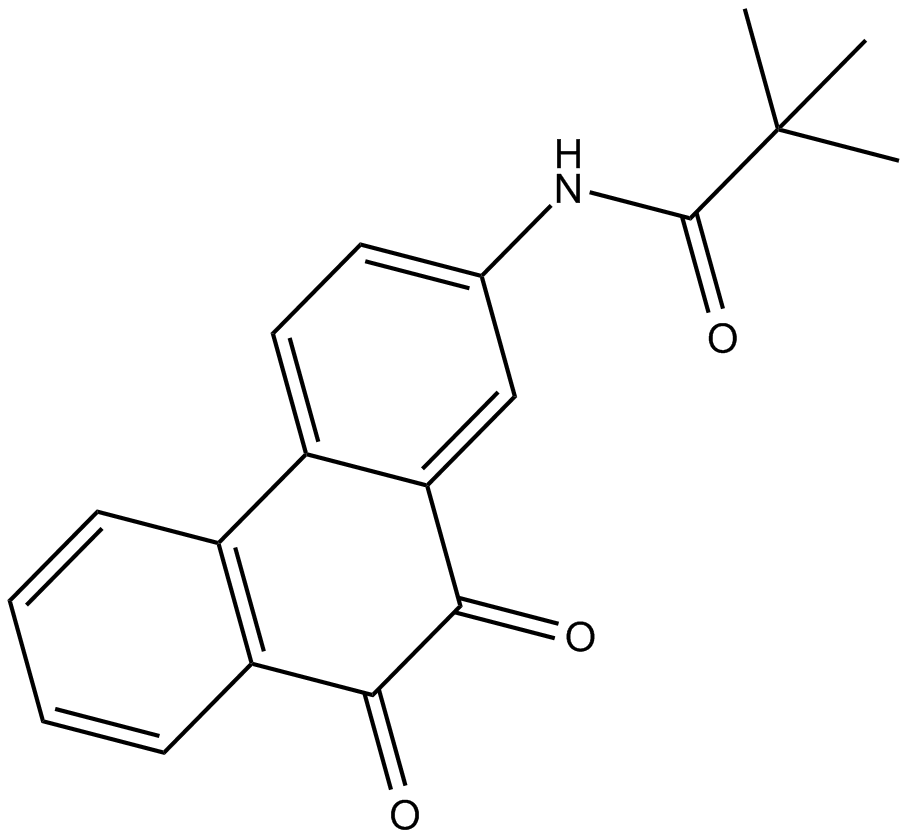
GC10019
SF1670
SF1670 est un inhibiteur puissant et spécifique de la phosphatase et de la tensine délétée sur le chromosome 10 (PTEN).
-
GC49713
SIKVAV (acetate)
Hexapeptide-10, Ser-Ile-Lys-Val-Ala-Val
A laminin α1-derived peptide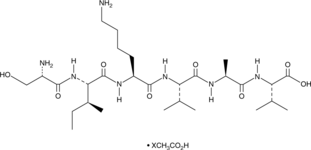
-
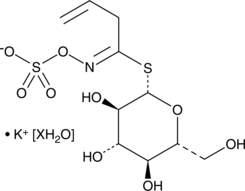
GC49002
Sinigrin (hydrate)
La sinigrine (hydrate) est un glucosinolate aliphatique naturel présent dans les plantes de la famille des Brassicacées.
-
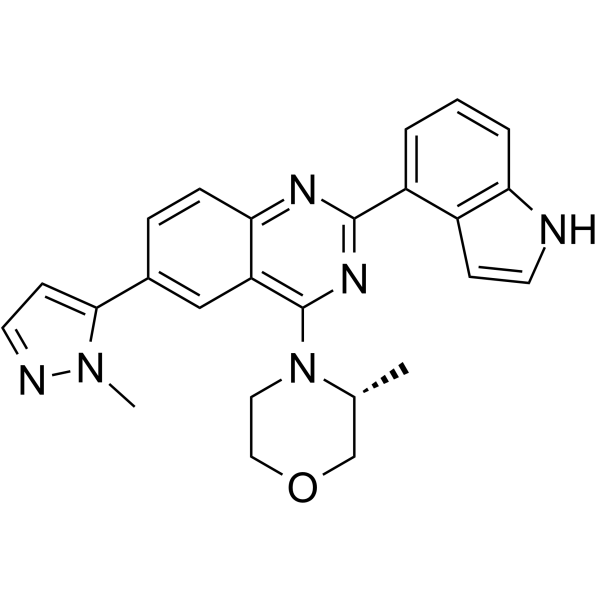
GC64539
SKLB-197
SKLB-197 a montré une valeur IC50 de 0,013 μM contre l'ATR mais une activité très faible ou nulle contre les autres 402 protéines kinases. Il a montré une puissante activité antitumorale contre les tumeurs déficientes en ATM À la fois in vitro et in vivo.
-
GC12827
SLx-2119
Belumosudil, SLx-2119
A ROCK2 inhibitor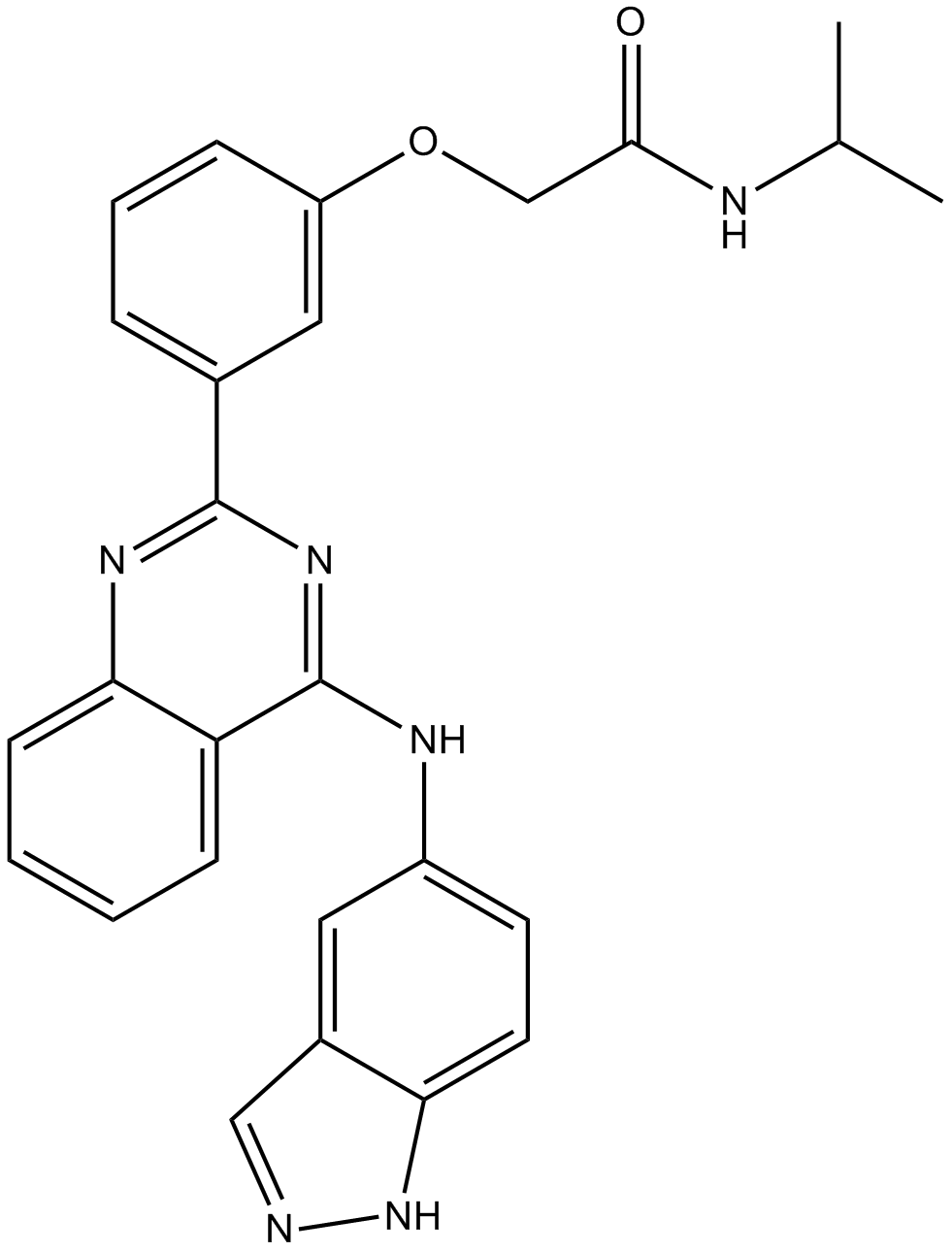
-
GC11396
SNS-032 (BMS-387032)
BMS-387032
SNS-032 (BMS-387032) (BMS-387032) est un inhibiteur puissant et sélectif de CDK2, CDK7 et CDK9 avec des IC50 de 38 nM, 62 nM et 4 nM, respectivement. SNS-032 (BMS-387032) a un effet antitumoral.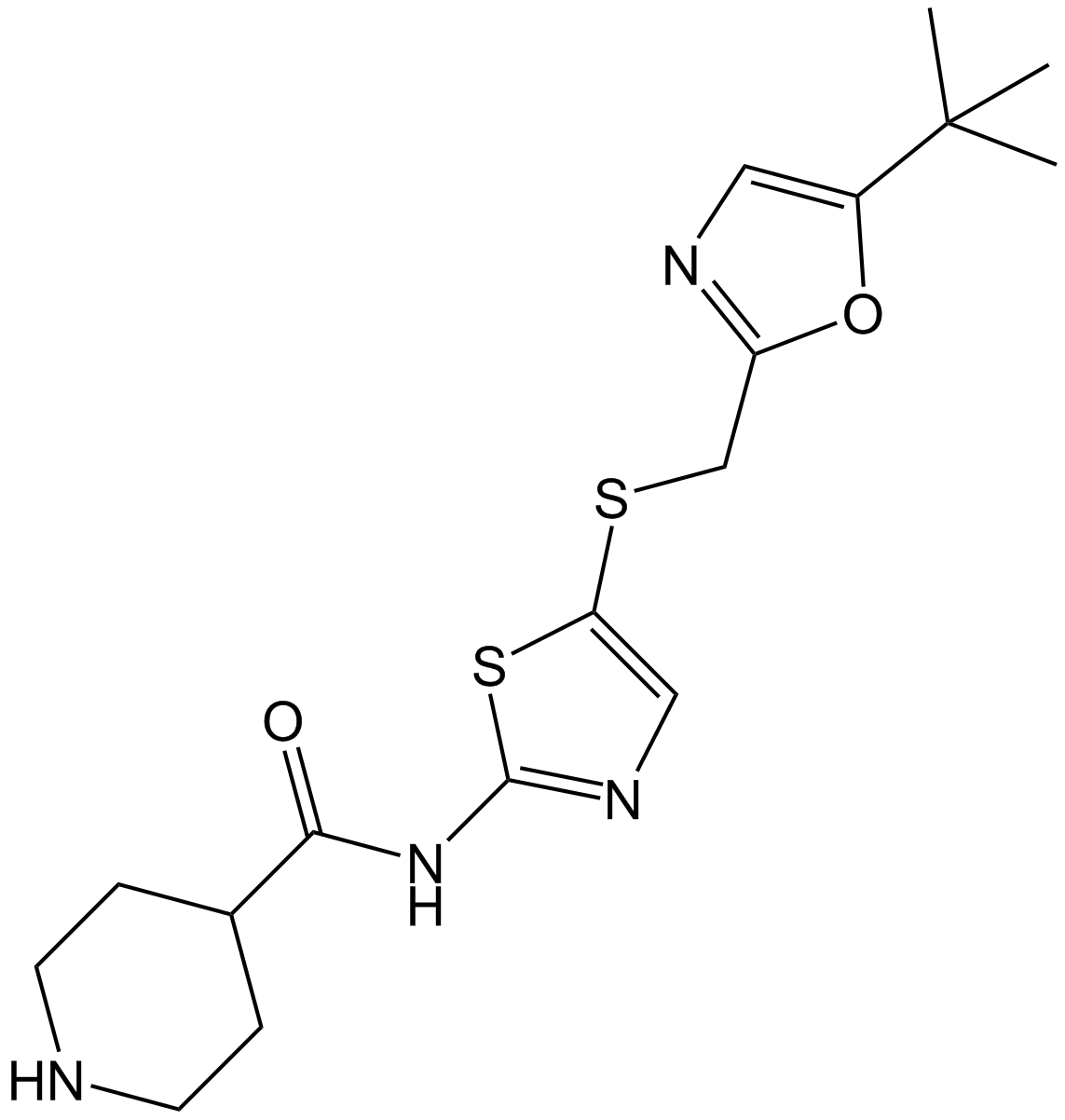
-
GC25940
SNS-314
SNS-314 is a potent and selective inhibitor of Aurora A, Aurora B and Aurora C with IC50 of 9 nM, 31 nM, and 3 nM, respectively. It is less potent to Trk A/B, Flt4, Fms, Axl, c-Raf and DDR2. Phase 1.
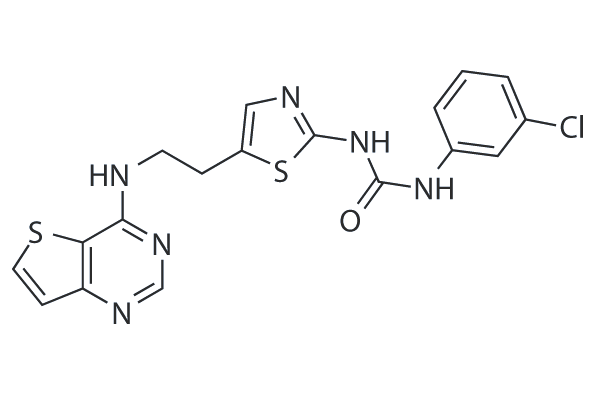
-
GC32935
Soblidotin (Auristatin PE)
Auristatin PE; TZT-1027
Soblidotin (Auristatine PE) (Auristatine PE) est un nouveau dérivé synthétique de la dolastatine 10 et un inhibiteur de la polymérisation de la tubuline.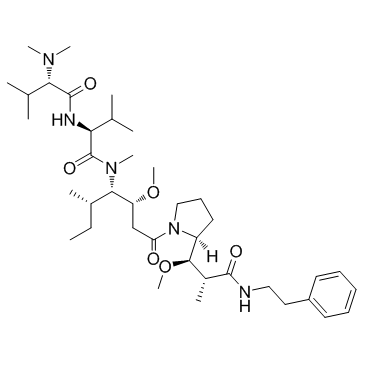
-
GC48084
Sodium 4-Phenylbutyrate-d11
Benzenebutanoic acid-d11, TriButyrate-d11
Le phénylbutyrate-d11 (sodium) est du 4-phénylbutyrate de sodium marqué au deutérium. Le 4-phénylbutyrate de sodium (4-PBA sodique) est un inhibiteur du stress des HDAC et du réticulum endoplasmique (RE), utilisé dans la recherche sur le cancer et les infections.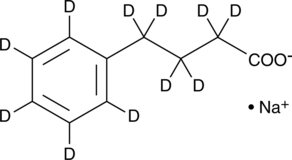
-
GC65887
SOP1812
Le SOP1812 est un dérivé de naphtalène diimide (ND) À activité anti-tumorale. SOP1812 se lie aux arrangements quadruplex (G4) et régule À la baisse plusieurs voies génétiques du cancer. SOP1812 montre une grande affinité pour hTERT G4 et HuTel21 G4 avec des valeurs KD de 4,9 et 28,4 nM, respectivement. SOP1812 peut être utilisé pour la recherche sur le cancer.
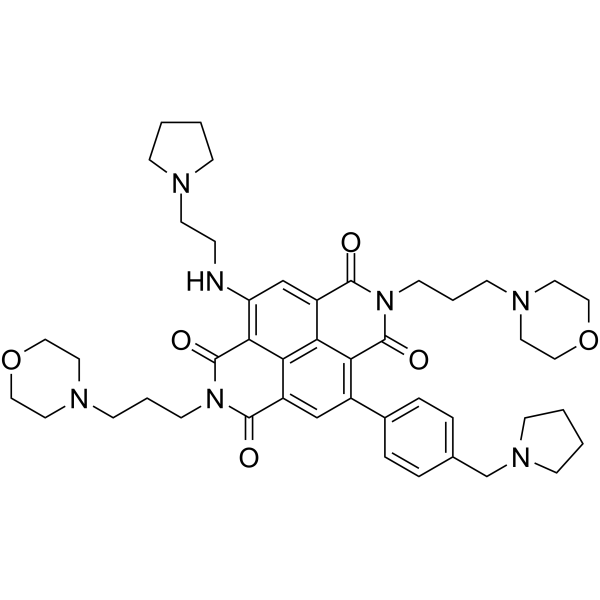
-
GC63351
Sovesudil
PHP-201; AMA0076
Le sovésudil (PHP-201) est un puissant inhibiteur de la Rho kinase (ROCK) À action locale, compétitif pour l'ATP, avec des CI50 de 3,7 et 2,3 nM pour ROCK-I et ROCK-II, respectivement.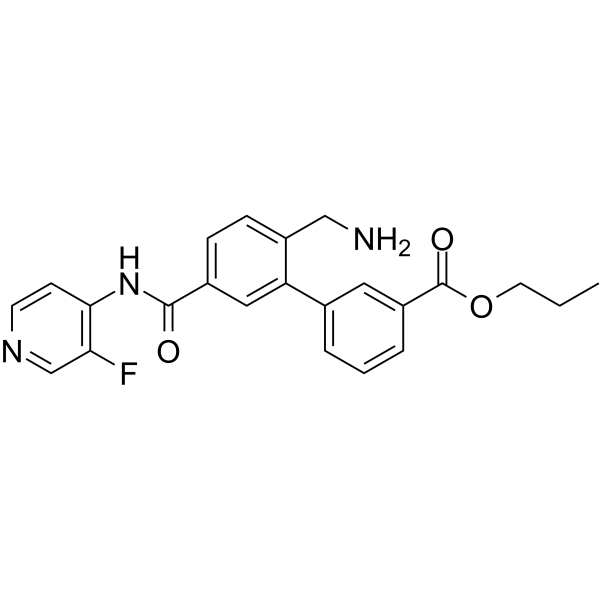
-
GC63716
Sovesudil hydrochloride
PHP-201 hydrochloride; AMA0076 hydrochloride
Le chlorhydrate de sovésudil (PHP-201) est un puissant inhibiteur de la Rho kinase (ROCK) À action locale, compétitif pour l'ATP, avec des CI50 de 3,7 et 2,3 nM pour ROCK-I et ROCK-II, respectivement.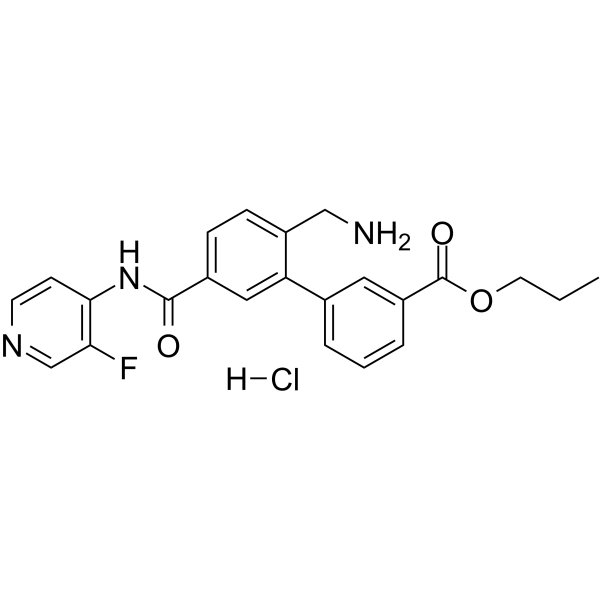
-
GC64019
Sovilnesib
AMG 650
Le sovilnésib (AMG 650) est un inhibiteur de la protéine de type kinésine KIF18A (WO2020132648). Le sovilnesib peut être utilisé pour la recherche sur le cancer.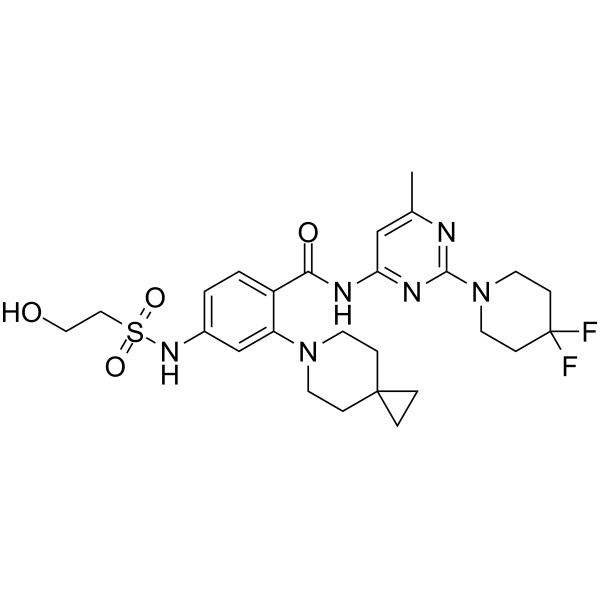
-
GC48092
Spinosad
LY232105, XDE-105
Le spinosad, mélange de spinosynes A et D connus comme produits de fermentation d'un actinomycète du sol (Saccharopolyspora spinosa), est un insecticide biologique neurotoxique À spectre d'action plus large.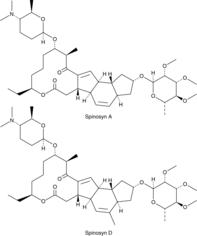
-
GC41258
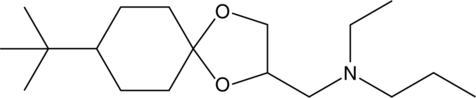
Spiroxamine
Spiroxamine is a tertiary amine fungicide and an inhibitor of δ14 reductase/δ8→δ7 isomerase.
-
GC44946
Sporidesmolide III
Sporidesmolide III is a cyclodepsipeptide originally isolated from P.