Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Ziele für Cell Cycle/Checkpoint
- ATM/ATR(23)
- Aurora Kinase(17)
- Cdc42(4)
- Cdc7(3)
- Chk(14)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(77)
- E1 enzyme(1)
- G-quadruplex(11)
- Haspin(6)
- HMTase(1)
- Kinesin(23)
- Ksp(4)
- Microtubule/Tubulin(219)
- Mps1(15)
- Mitotic(7)
- RAD51(16)
- ROCK(60)
- Rho(13)
- PERK(10)
- PLK(33)
- PTEN(6)
- Wee1(7)
- PAK(20)
- Arp2/3 Complex(8)
- Dynamin(10)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Produkte für Cell Cycle/Checkpoint
- Bestell-Nr. Artikelname Informationen
-
GC44730
proTAME
Pro-N-4-tosyl-L-arginine methyl ester
proTAME ist eine zellpermeable Prodrug-Form von N-4-Tosyl-L-Arginin-Methylester, einem Inhibitor des Anaphase-Promoting-Komplexes/Cyclosoms (APC/C), der durch intrazelluläre Esterasen in TAME umgewandelt wird.
-
GC44732
Protopine (hydrochloride)
Protopine is an alkaloid found in Berberidaceae, Ranunculaceae, Rutaceae, Fumariaceae, and Papaveraceae with diverse biological activities.
-
GC49914
Pt(II)-NHC Complex 2C
Platinum(II)-N-Heterocyclic Carbene Complex 2C
An inducer of immunogenic cancer cell death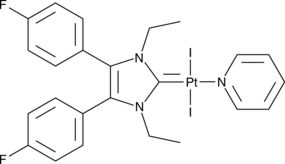
-
GC69776
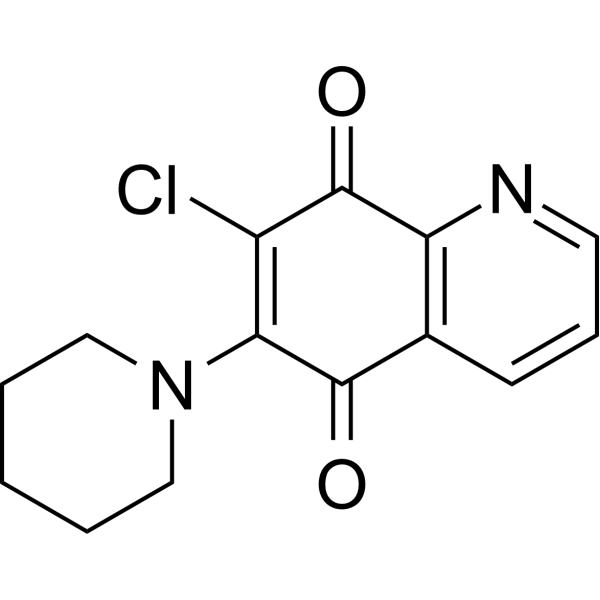
PT-262
PT-262 ist ein wirksamer ROCK-Inhibitor mit einem IC50-Wert von etwa 5 μM. PT-262 induziert den Verlust des mitochondrialen Membranpotenzials und erhöht die Aktivierung von Caspase-3 und Zellapoptose. PT-262 hemmt ERK und CDC2 Phosphorylierung auf einem p53-unabhängigen Weg. PT-262 blockiert die Zellskelett-Funktion und Zellmigration. PT-262 hat antikarzinogene Aktivität.
-
GC44740
PtdIns-(1,2-dioctanoyl) (sodium salt)
DOPI, Phosphatidylinositol C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.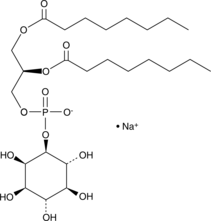
-
GC44743
PtdIns-(3)-P1 (1,2-dioctanoyl) (sodium salt)
DOPI3P1, Phosphatidylinositol3monophosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.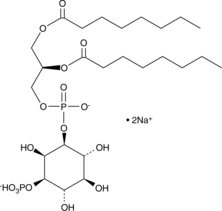
-
GC44748
PtdIns-(3,4,5)-P3 (1,2-dihexanoyl) (ammonium salt)
DHPI-3,4,5-P3, Phosphatidylinositol-3,4,5-triphosphate C-6, PIP3C-16
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.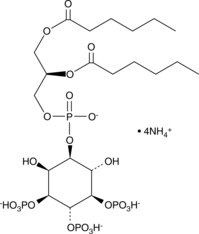
-
GC44750
PtdIns-(3,4,5)-P3 (1,2-dipalmitoyl) (sodium salt)
DPPI3,4,5P3, Phosphatidylinositol3,4,5triphosphate C16
The phosphatidylinositol phosphates represent a small percentage of total membrane phospholipids.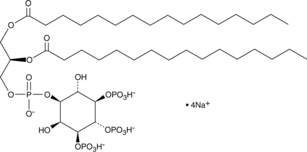
-
GC44753
PtdIns-(3,4,5)-P3-biotin (sodium salt)
Phosphatidylinositol3,4,5triphosphate C8biotin, PIP3biotin
The PtdIn phosphates play an important role in the generation and transduction of intracellular signals.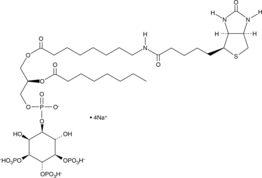
-
GC44759
PtdIns-(4)-P1 (1,2-dioctanoyl) (ammonium salt)
DOPI4P1, Phosphatidylinositol4phosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.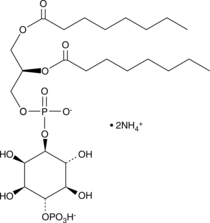
-
GC52484
Purified Ganglioside Mixture (bovine) (ammonium salt)
A mixture of purified bovine gangliosides

-
GC14707
Purvalanol A
NG 60
Purvalanol A ist ein potenter CDK-Inhibitor, der cdc2-Cyclin B, cdk2-Cyclin A, cdk2-Cyclin E, cdk4-Cyclin D1 und cdk5-p35 mit IC50-Werten von 4, 70, 35, 850 bzw. 75 nM hemmt.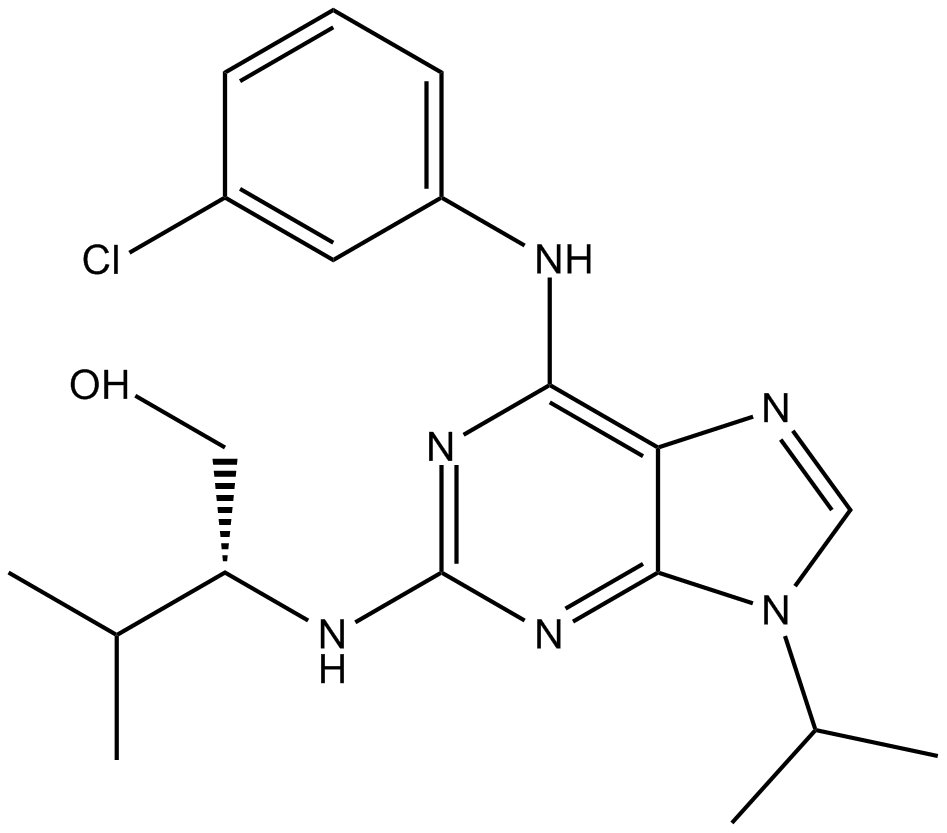
-
GC16268
Purvalanol B
NG 95; NG95; NG-95
Purvalanol B (NG 95) ist ein potenter, selektiver, reversibler und ATP-kompetitiver CDK-Inhibitor mit IC50-Werten von 6 nM, 6 nM, 9 nM, 6 nM fÜr cdc2-Cyclin B, CDK2-Cyclin A, CDK2-Cyclin E und CDK5-p35.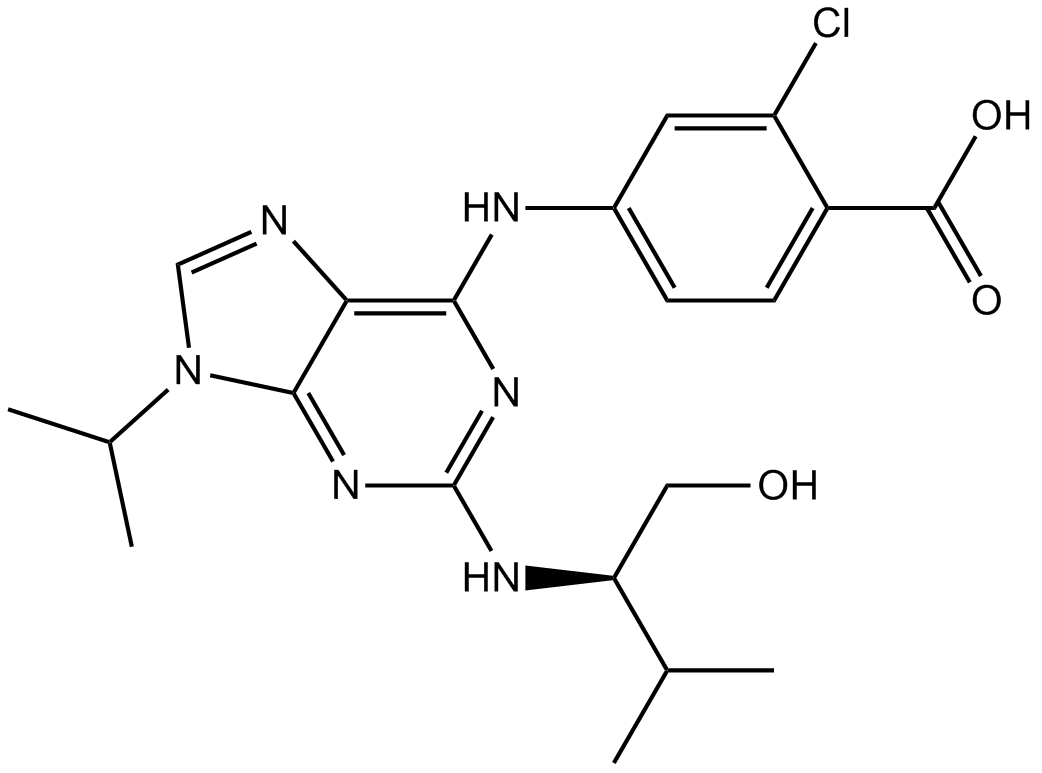
-
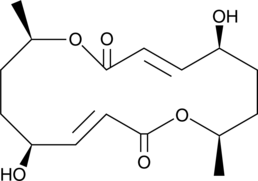
GC45552
Pyrenophorol
Pyrenophorol ist eine C9H12O3-Verbindung, die in geringer Ausbeute aus Kulturfiltraten des phytopathogenen Pilzes Stemphylium radicinum, C.
-
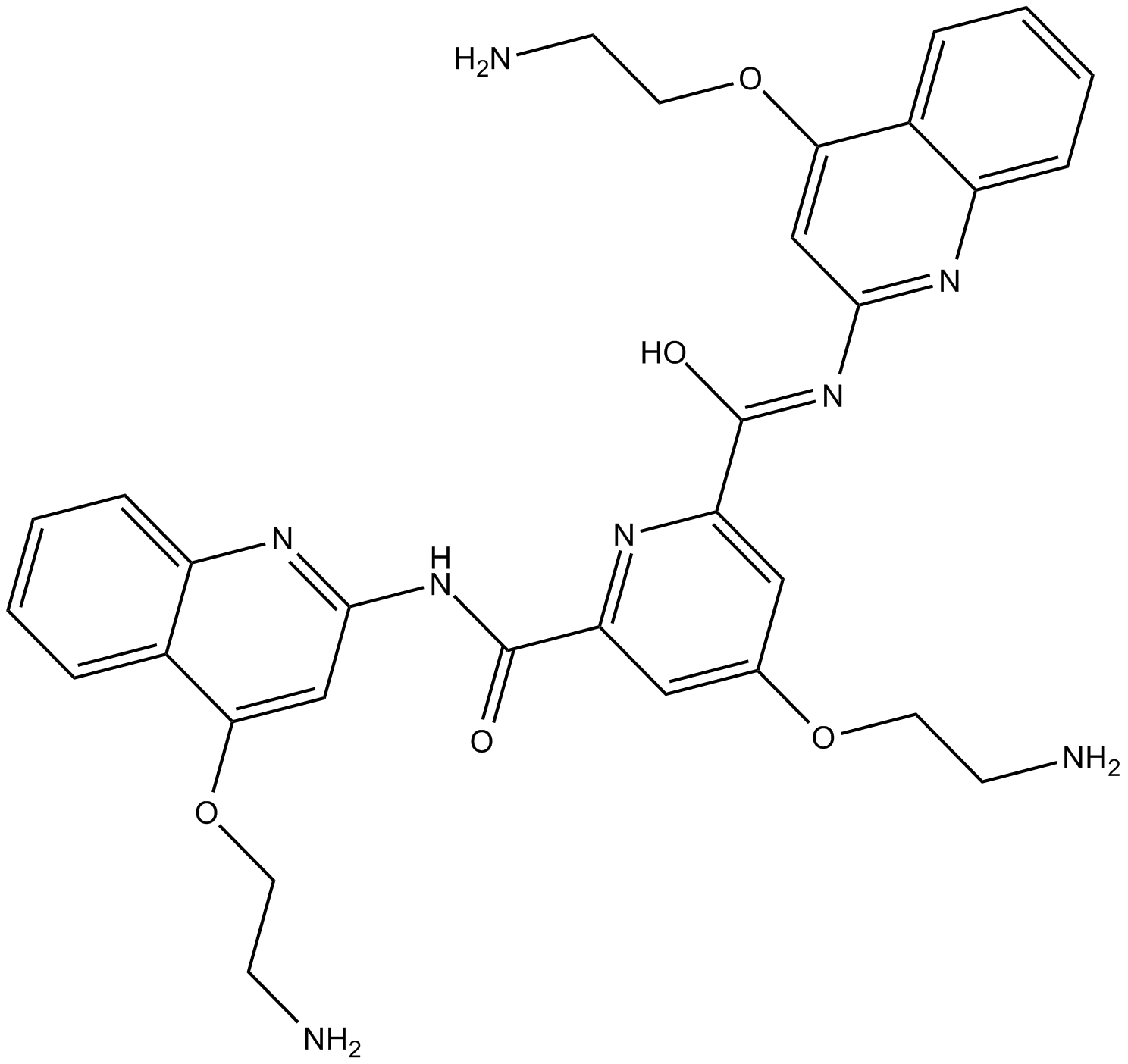
GC15162
Pyridostatin
Pyridostatin ist eine hochselektive kleine Molekülverbindung, die speziell zur Bindung an G-Quadruplex-Strukturen entwickelt wurde und unabhängig von der Sequenzvariabilität polymorphe G-Quadruplex-Strukturen anspricht.
-
GC48016
Pyridostatin (trifluoroacetate salt)
RR82 TFA
Pyridostatin (RR82) TFA ist ein G-Quadruplex-DNA-Stabilisierungsmittel (Kd = 490 nM). Pyridostatin (Trifluoracetatsalz) fÖrdert den Wachstumsstillstand in menschlichen Krebszellen, indem es replikations- und transkriptionsabhÄngige DNA-SchÄden induziert. Pyridostatin (Trifluoracetatsalz) zielt auf das Proto-Onkogen Src ab. Pyridostatin (Trifluoracetatsalz) reduzierte die SRC-Proteinspiegel und die SRC-abhÄngige ZellmotilitÄt in menschlichen Brustkrebszellen.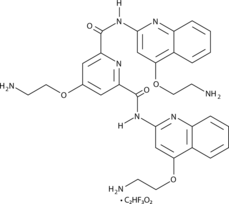
-
GC37043
Pyridostatin hydrochloride
RR82 hydrochloride
Pyridostatin (RR82)-Hydrochlorid ist ein G-Quadruplex-DNA-Stabilisierungsmittel (Kd = 490 nM). Pyridostatinhydrochlorid fÖrdert den Wachstumsstillstand in menschlichen Krebszellen, indem es replikations- und transkriptionsabhÄngige DNA-SchÄden induziert. Pyridostatin-Hydrochlorid zielt auf das Proto-Onkogen Src ab. Pyridostatinhydrochlorid reduzierte die SRC-Proteinspiegel und die SRC-abhÄngige ZellmotilitÄt in menschlichen Brustkrebszellen.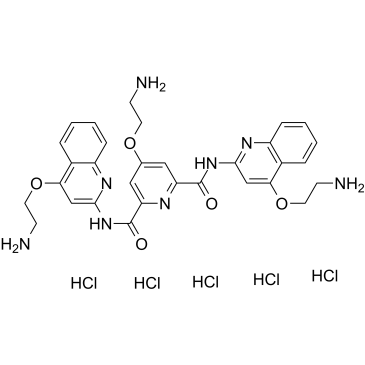
-
GC17801
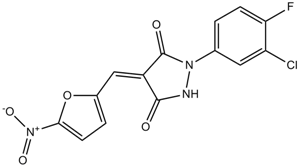
PYZD-4409
PYZD-4409 ist ein spezifischer Inhibitor des Ubiquitin-aktivierenden Enzyms UBA1 mit einer IC50 von 20 μM (zellfreier enzymatischer Assay). PYZD-4409 induziert den Zelltod in bÖsartigen Zellen und hemmt vorzugsweise das klonogene Wachstum von primÄren akuten myeloischen LeukÄmiezellen.
-
GC52057
QN523
An anticancer agent

-
GC45903
Quazinone
Ro 13-6438
A PDE3 inhibitor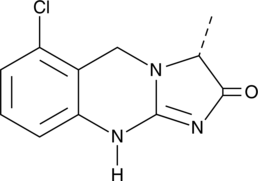
-
GC48019
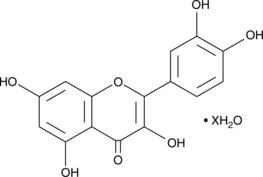
Quercetin (hydrate)
A flavonoid with diverse biological activities
-
GC46210
Quinacrine mustard (hydrochloride)
NSC 3424
Chinacrin-Senf (Hydrochlorid) ist ein Fluorochrom.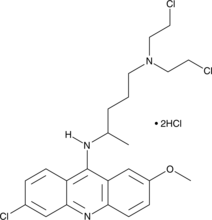
-
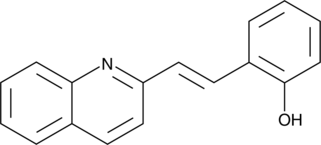
GC44800
Quininib
A CysLT1 and CysLT2 receptor antagonist
-
GC17400
R547
Ro 4584820
R547 ist ein potenter, selektiver und oral aktiver ATP-kompetitiver CDK-Inhibitor mit Kis von 2 nM, 3 nM und 1 nM fÜr CDK1/Cyclin B, CDK2/Cyclin E bzw. CDK4/Cyclin D1.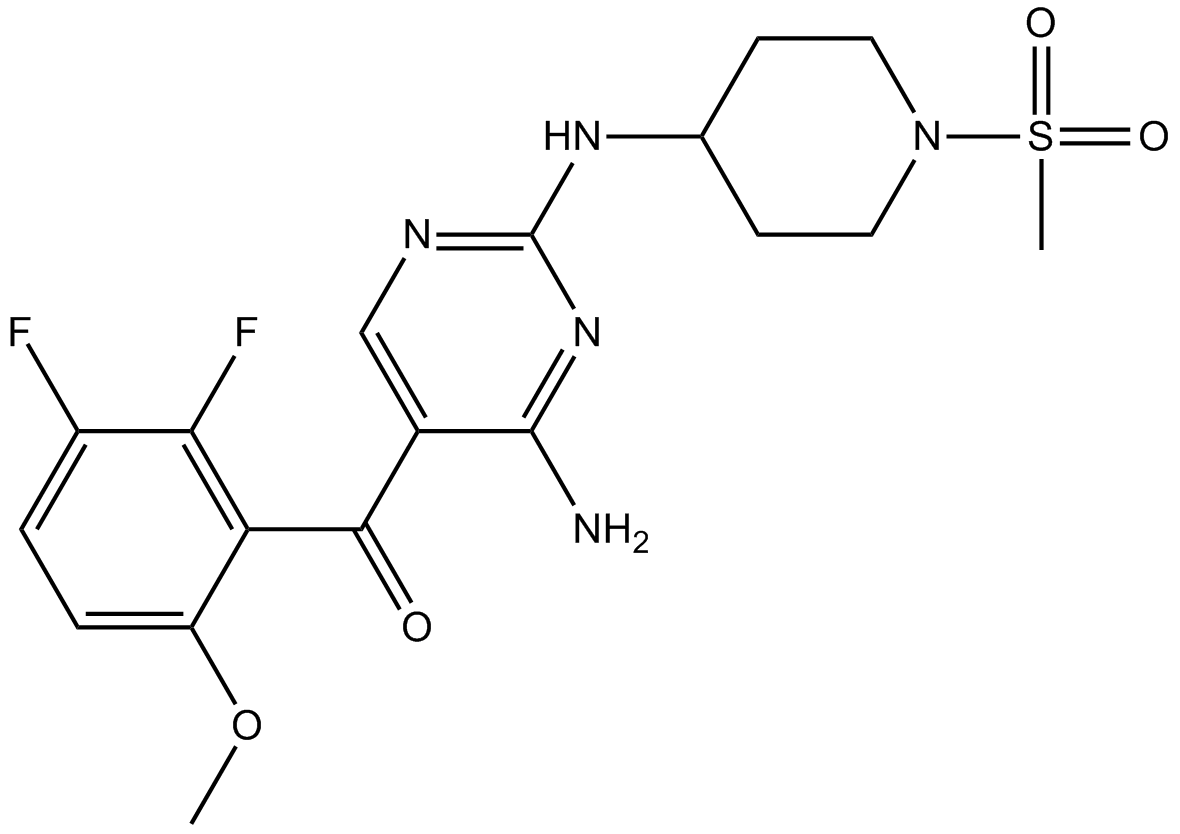
-
GC49771
rac-Hesperetin-d3
(±)-3',5,7-Trihydroxy-4'-methoxyflavanone-d3
An internal standard for the quantification of hesperetin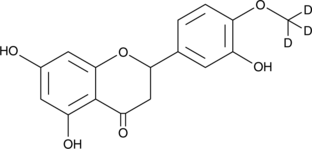
-
GC49108
Racecadotril-d5
Acetorphan-d5
An internal standard for the quantification of racecadotril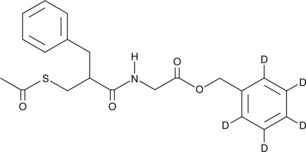
-
GC19306
RAD51 Inhibitor B02
B02
RAD51 Inhibitor B02 (B02) ist ein Inhibitor von humanem RAD51 mit einem IC50 von 27,4 μM.
-
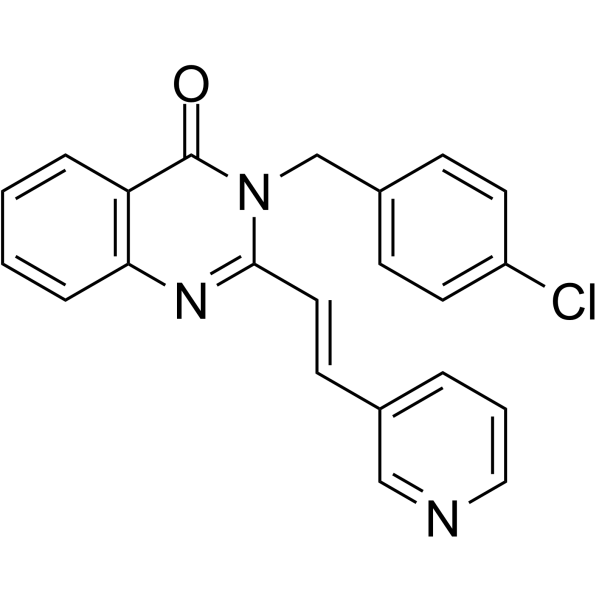
GC63165
RAD51-IN-1
RAD51-IN-1, ein Derivat von B02, ist ein starker Inhibitor von RAD51. RAD51-IN-1 kann fÜr die Krebsforschung verwendet werden.
-
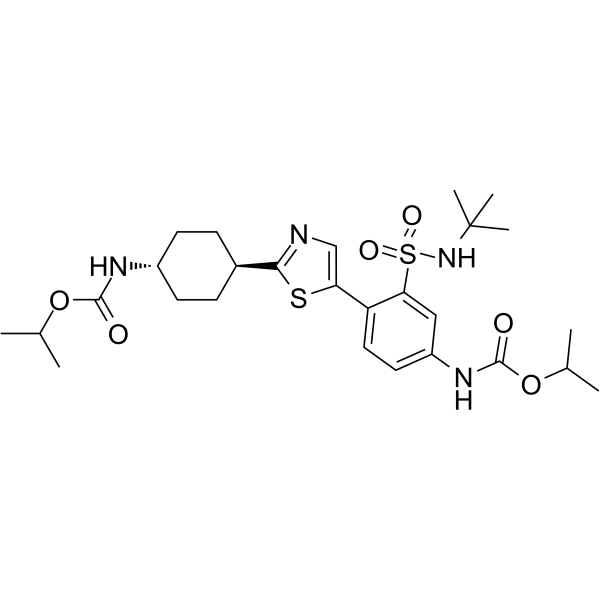
GC65383
RAD51-IN-2
RAD51-IN-2 (Verbindungsbeispiel 67A) ist ein RAD51-Inhibitor, der aus dem Patent WO2019/051465A1 extrahiert wurde.
-
GC68416
RAD51-IN-3
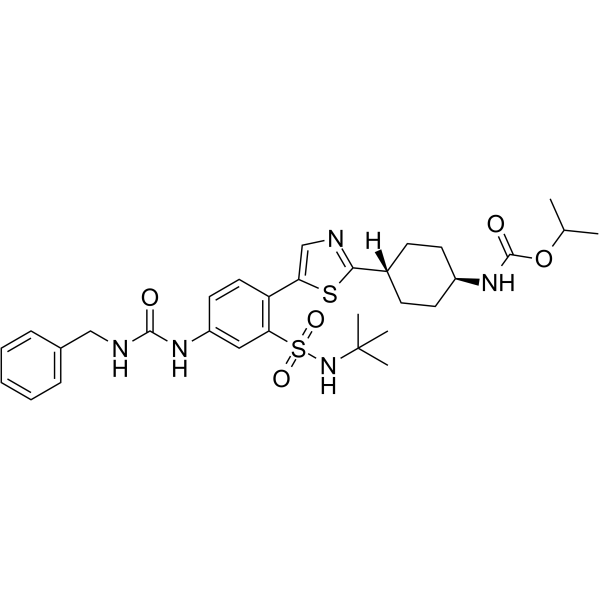
-
GC67888
RAD51-IN-5

-
GC52054
Ranitidine S-oxide
Ranitidin-S-Oxid ist der Metabolit von Ranitidin.

-
GC52196
RGD Peptide
GRGDNP, HGlyArgGlyAspAsnProOH
RGD-Peptid wirkt als Inhibitor von Integrin-Liganden-Wechselwirkungen und spielt eine wichtige Rolle bei ZelladhÄsion, Migration, Wachstum und Differenzierung.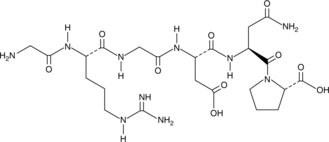
-
GC45798
Rhein-13C4
Rheic Acid-13C4
An internal standard for the quantification of rhein
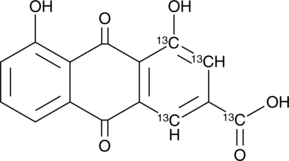
-
GC33310
Rho-Kinase-IN-1
Rho-Kinase-IN-1 ist ein Rho-Kinase (ROCK)-Inhibitor (Ki-Werte von 30,5 und 3,9 nM fÜr ROCK1 bzw. ROCK2), extrahiert aus US20090325960A1, Verbindung 1.008.
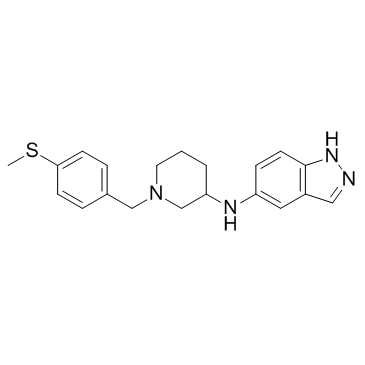
-
GC66050
Rho-Kinase-IN-2
Rho-Kinase-IN-2 (Verbindung 23) ist ein oral aktiver, selektiver und das Zentralnervensystem (ZNS) durchdringender Rho-Kinase (ROCK)-Inhibitor (ROCK2 IC50 = 3 nM). Rho-Kinase-IN-2 kann in Huntingtons Forschung verwendet werden.
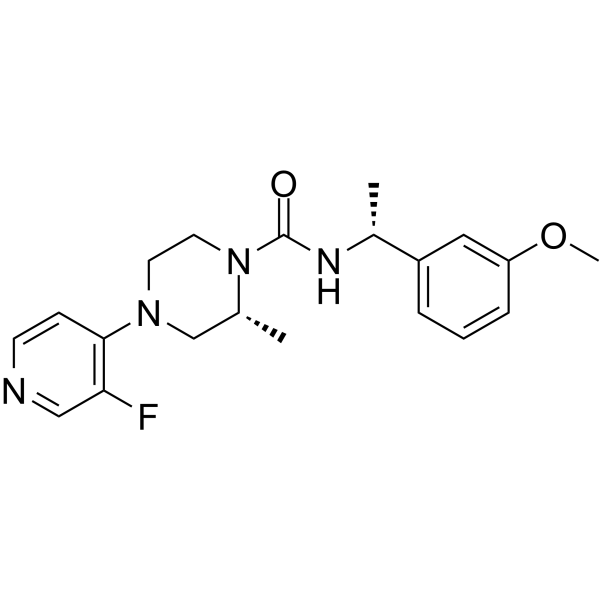
-
GC44829
Rhod-5N (potassium salt)
Rhod-5N is a low affinity fluorescent calcium probe (Kd = 320 μM).
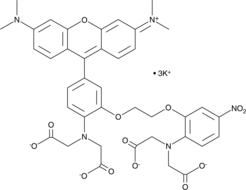
-
GC18140
RI-1
A RAD51 inhibitor

-
GC12752
RI-2
RI-2 ist ein reversibler RAD51-Inhibitor mit einem IC50-Wert von 44,17 μM und hemmt spezifisch die homologe Rekombinationsreparatur in menschlichen Zellen.
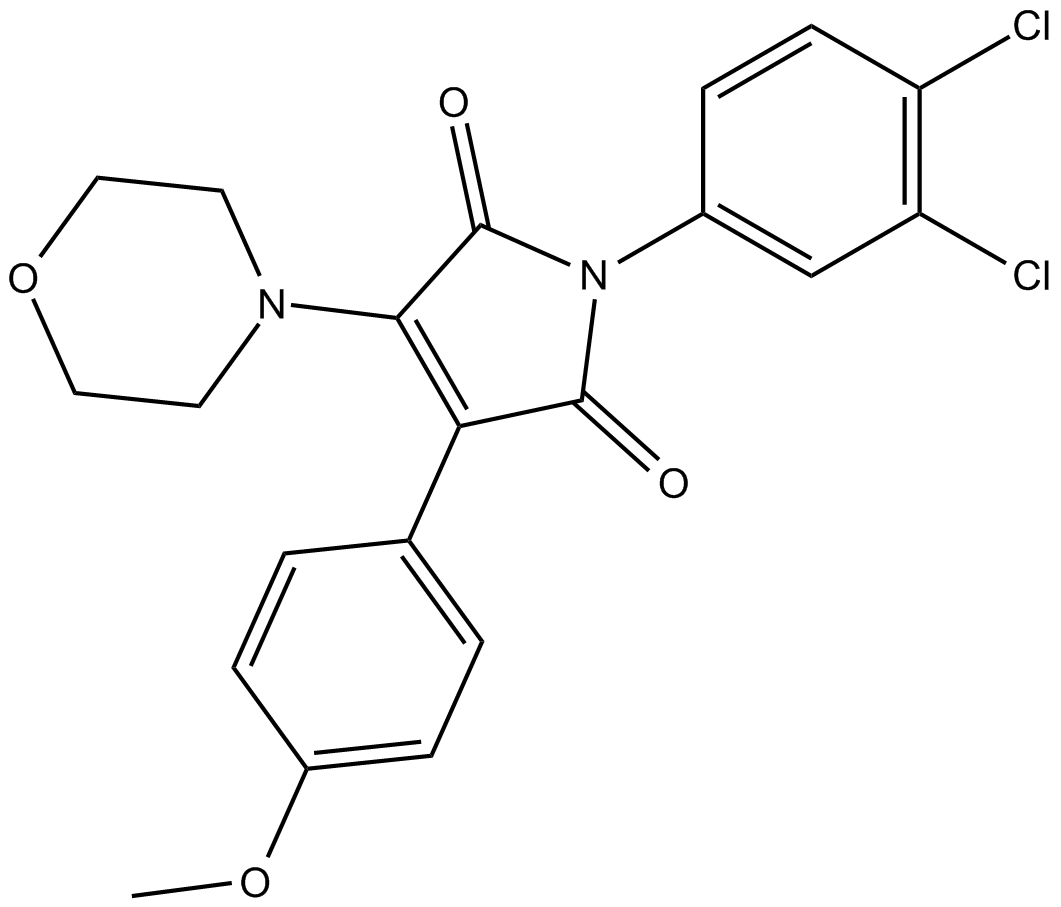
-
GC12415
Rigosertib
Rigosertib (ON-01910) ist ein Multi-Kinase-Inhibitor und ein selektiver Krebswirkstoff, der Apoptose durch Hemmung des PI3-Kinase/Akt-Signalwegs induziert, die Phosphorylierung von Histon H2AX fÖrdert und G2/M-Arrest im Zellzyklus induziert. Rigosertib ist ein selektiver und nicht ATP-kompetitiver Inhibitor von PLK1 mit einer IC50 von 9 nM.
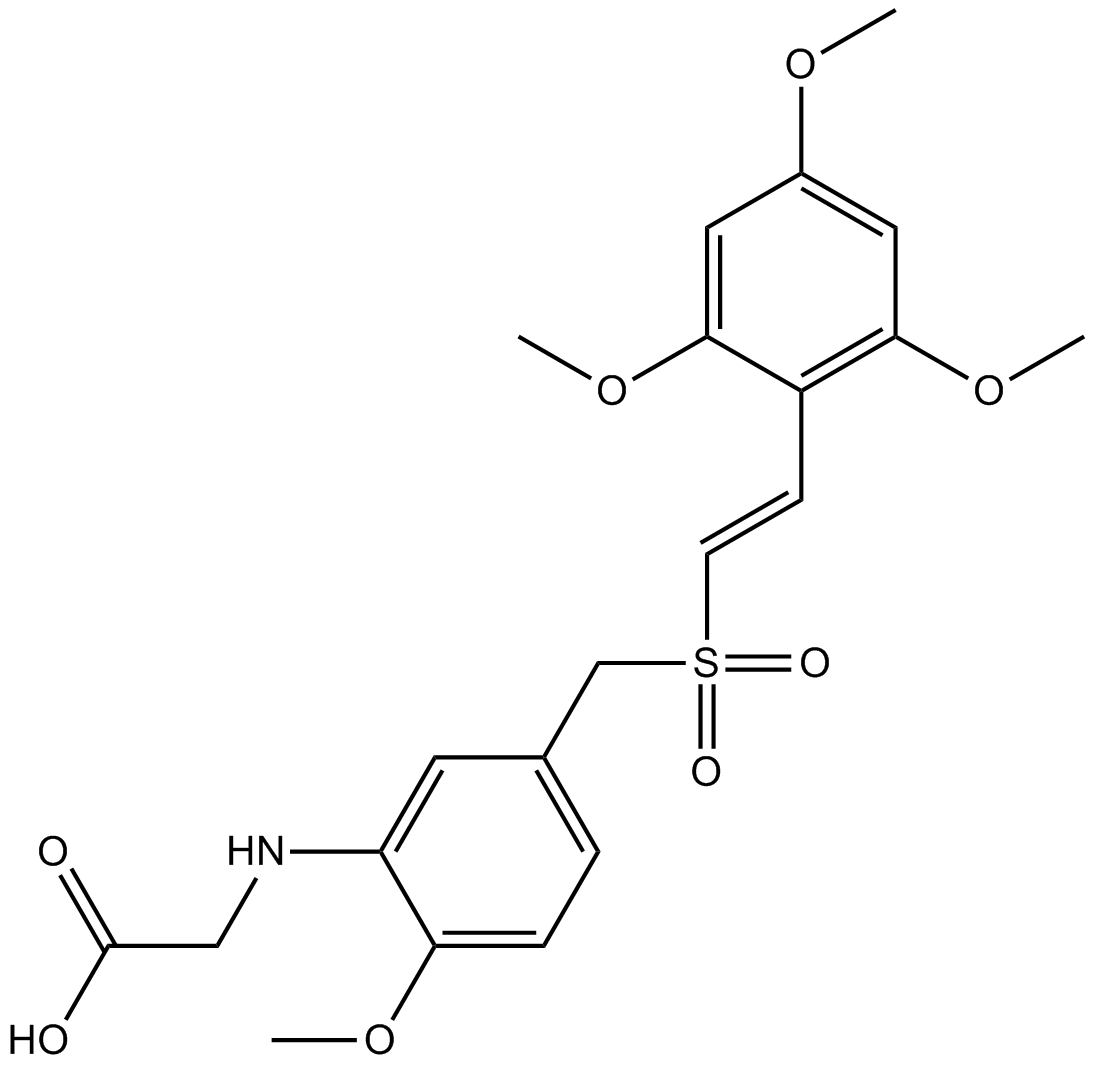
-
GC14358
Rigosertib (ON-01910,Estybon)
Rigosertib
Plk1 inhibitor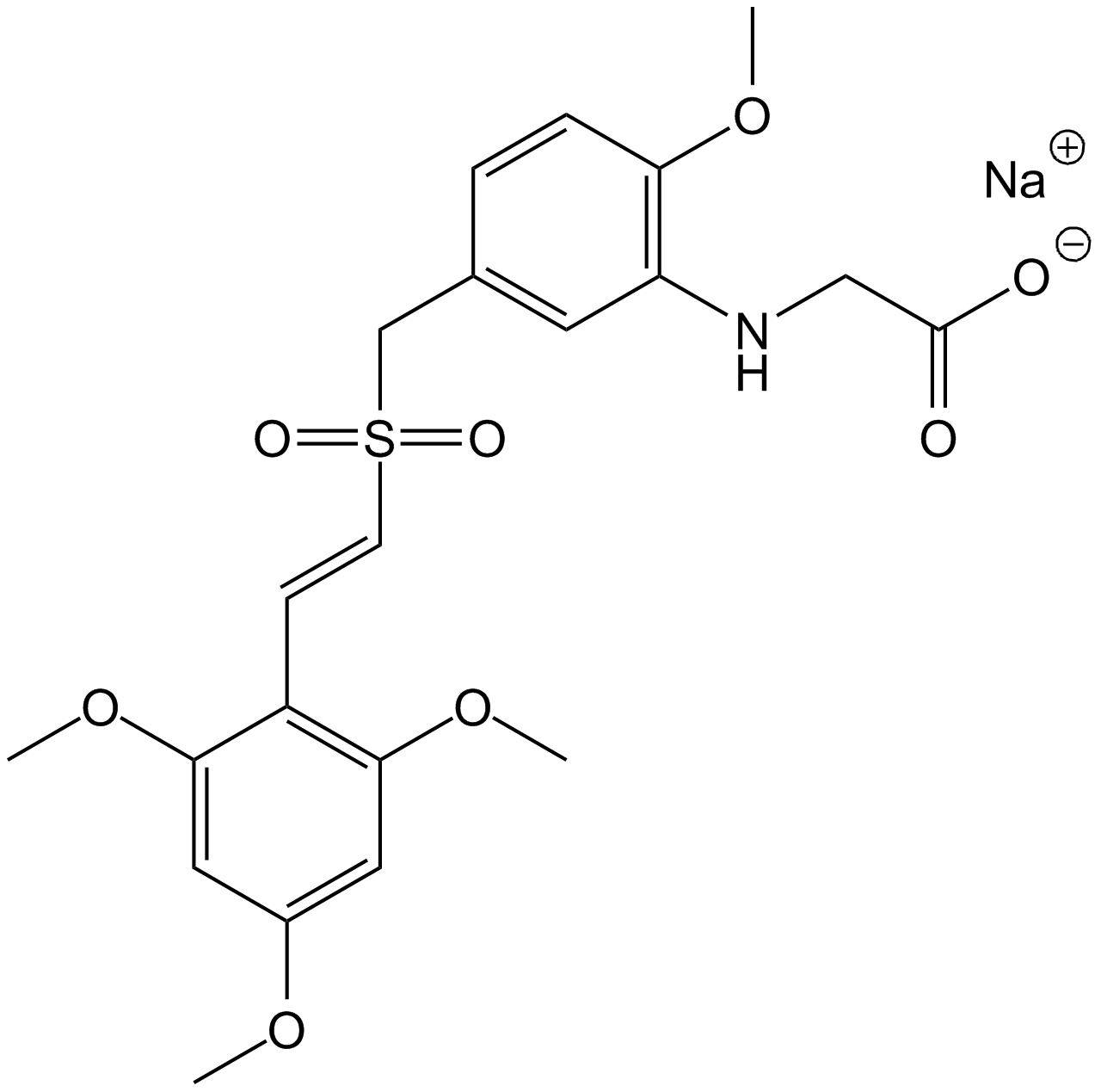
-
GC13580
Rigosertib sodium
Rigosertib-Natrium (ON-01910-Natrium) ist ein Multi-Kinase-Inhibitor und ein selektives Antikrebsmittel, das durch Hemmung des PI3K/Akt-Signalwegs Apoptose induziert, die Phosphorylierung von Histon H2AX fÖrdert und einen G2/M-Arrest im Zellzyklus induziert. Rigosertib-Natrium ist ein selektiver und nicht ATP-kompetitiver Inhibitor von PLK1 mit einer IC50 von 9 nM.
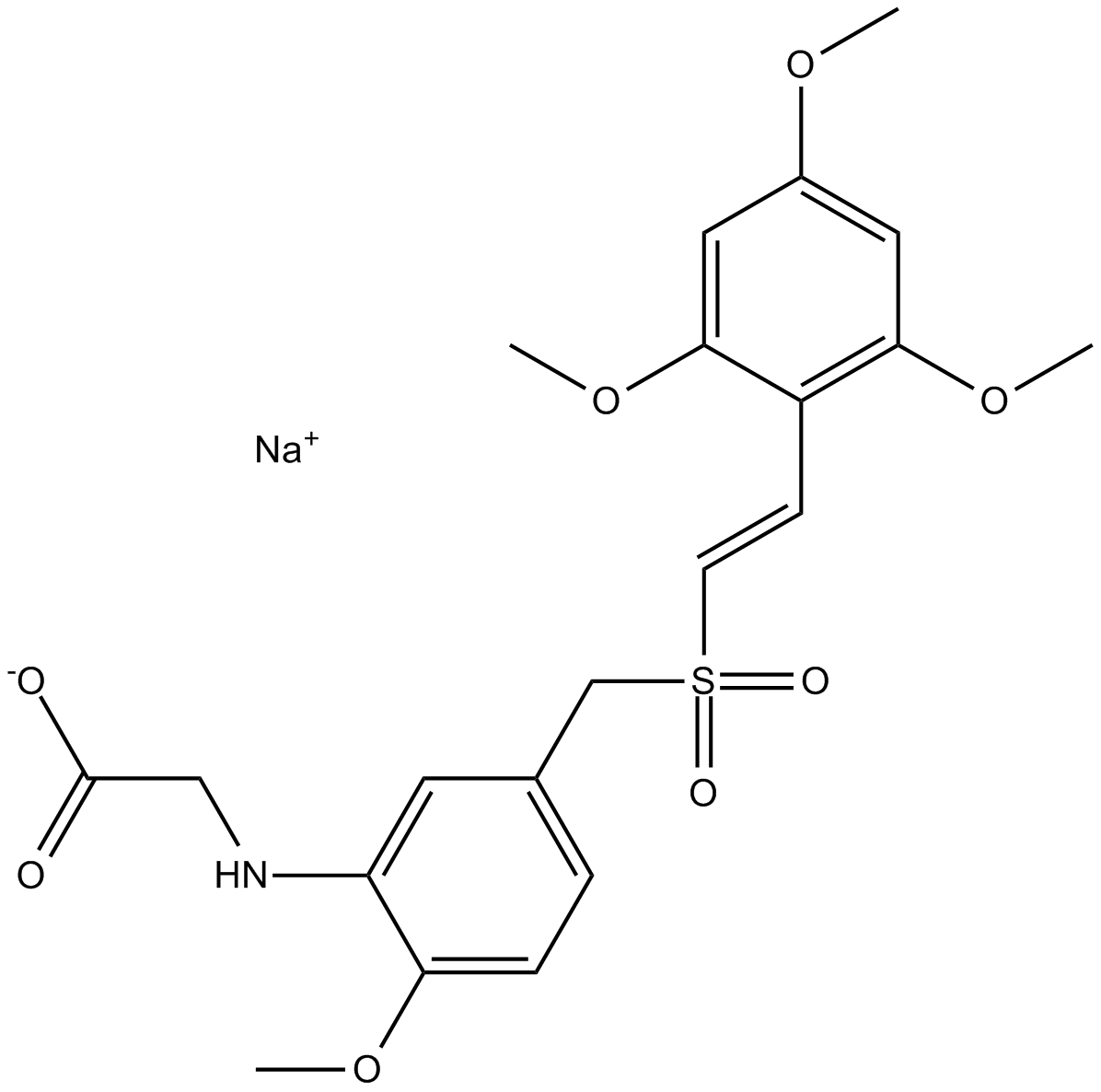
-
GC37534
Ripasudil free base
K-115 (free base)
Die freie Base Ripasudil (freie Base K-115) ist ein spezifischer Inhibitor von ROCK mit IC50-Werten von 19 und 51 nM fÜr ROCK2 bzw. ROCK1.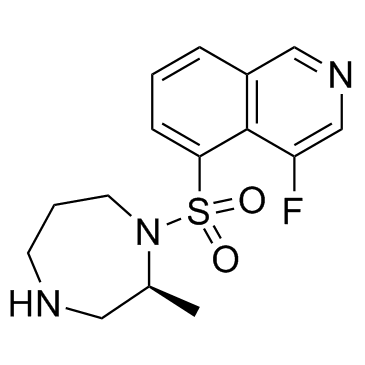
-
GC48401
Risuteganib (trifluoroacetate salt)
ALG-1001
An anti-integrin peptide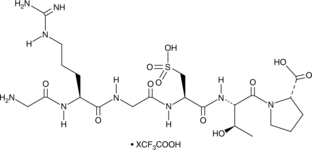
-
GC44846
RK-682 (calcium salt)
CI-010, TAN 1364B
Protein tyrosine phosphatases (PTPs) remove phosphate from tyrosine residues of cellular proteins.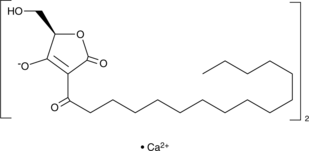
-
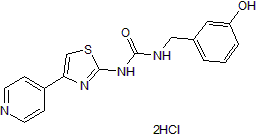
GC50092
RKI 1447 dihydrochloride
RKI 1447 Dihydrochlorid ist ein potenter und selektiver ROCK-Inhibitor mit IC50-Werten von 14,5 und 6,2 nM für ROCK1 bzw. ROCK2. RKI 1447 Dihydrochlorid unterdrückt das Wachstum kolorektaler Karzinomzellen und fördert die Apoptose.
-
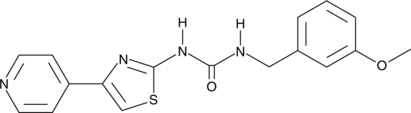
GC44847
RKI-1313
RKI-1313 ist ein ROCK-Inhibitor mit IC50-Werten von 34, 8 μM fÜr ROCK 1 bzw. ROCK 2. RKI-1313 zeigt wenig Wirkung auf die Phosphorylierungsgrade von ROCK-Substraten, Migration, Invasion oder verankerungsunabhÄngiges Wachstum.
-
GC15437
RKI-1447
RKI 1447;RKI1447
A ROCK1 and ROCK2 inhibitor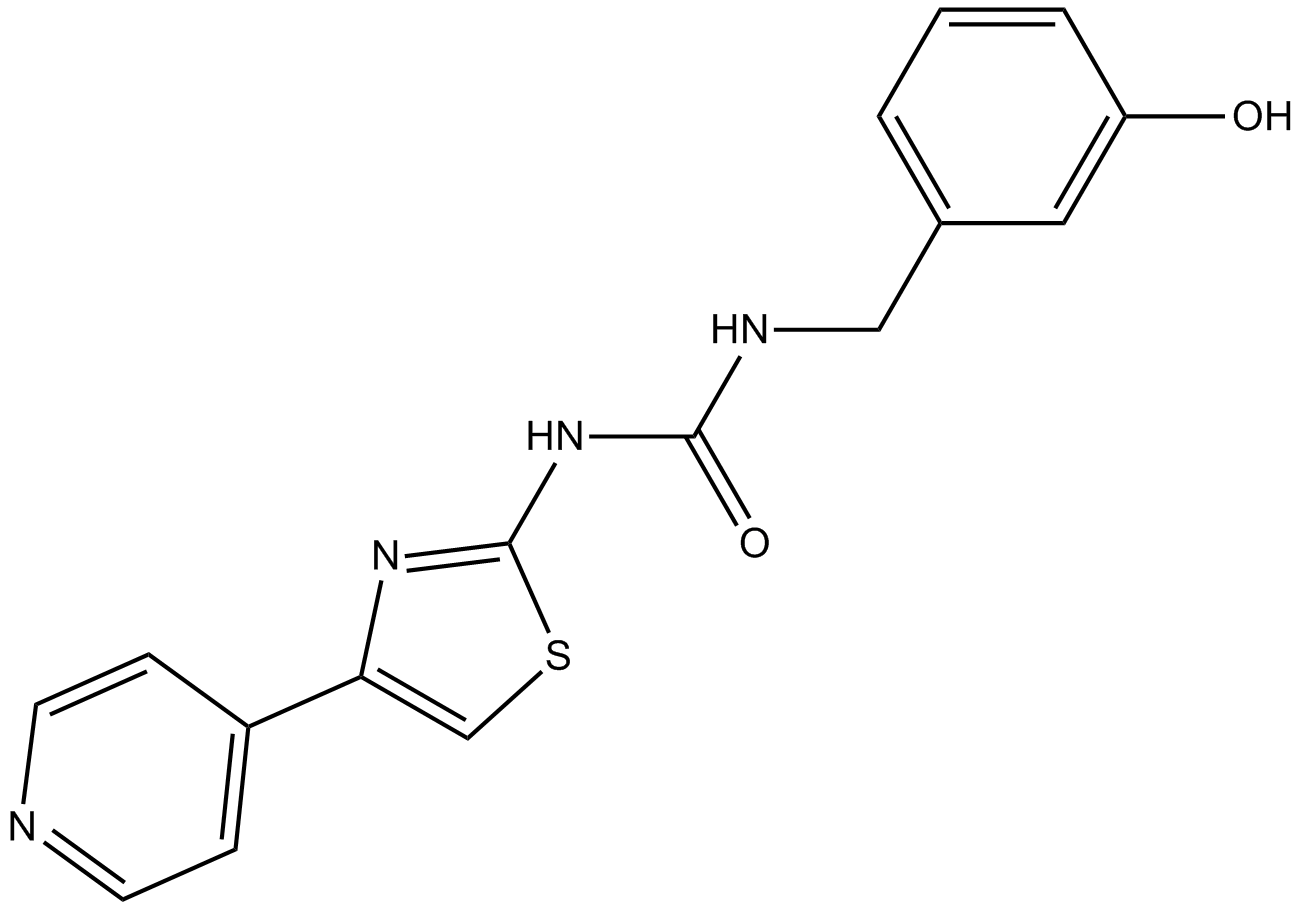
-
GC12348
Ro 3306
Ro 3306 ist ein potenter und selektiver Inhibitor von CDK1 mit Kis von 20 nM, 35 nM und 340 nM für CDK1, CDK1/Cyclin B1 bzw. CDK2/Cyclin E.
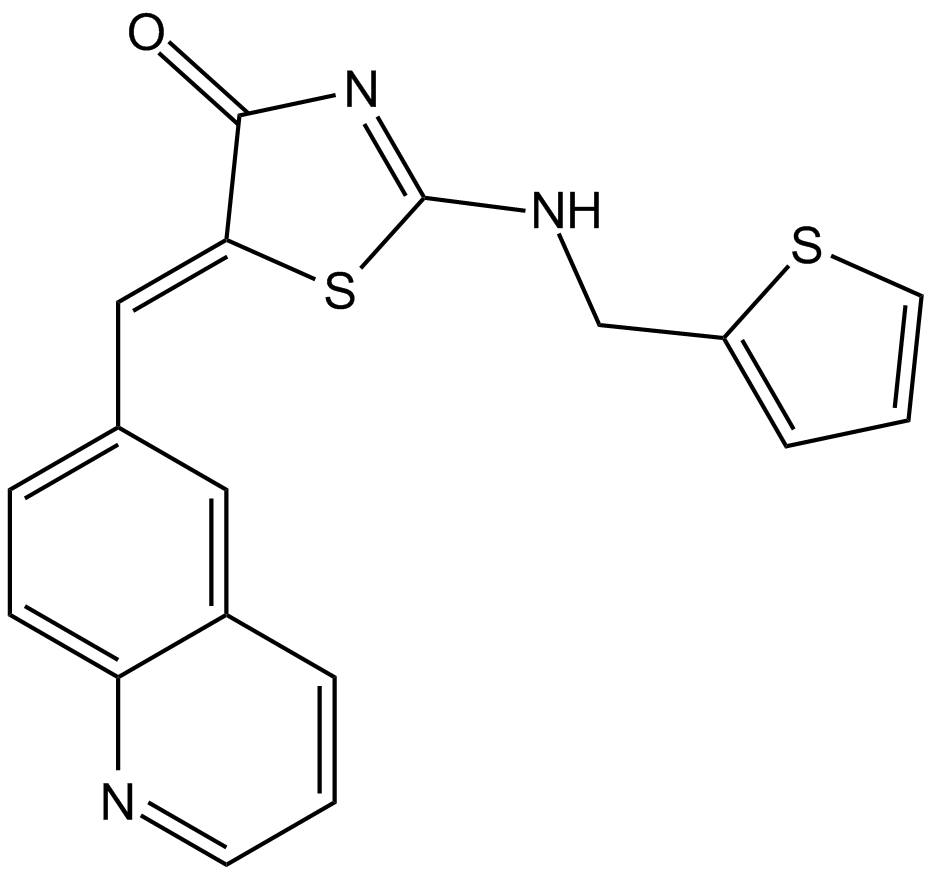
-
GC13903
Ro3280
Ro3280 ist ein potenter, hochselektiver Inhibitor von PLK1 mit einem IC50 und einem Kd von 3 nM bzw. 0,09 nM und hat nahezu keine Wirkung auf PLK2 und PLK3.
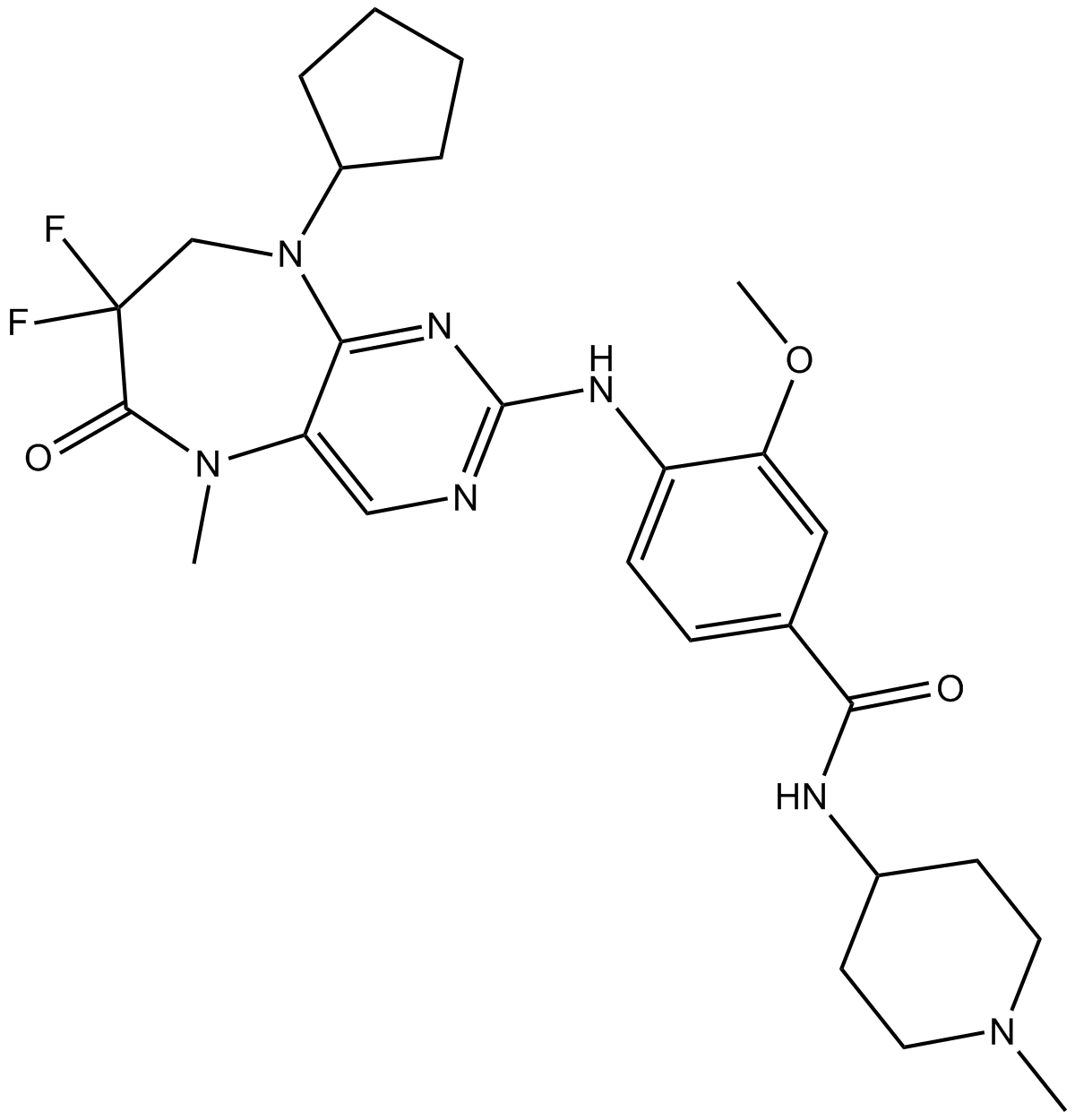
-
GC37551
ROCK inhibitor-2
ROCK-Inhibitor-2 ist ein selektiver dualer ROCK1- und ROCK2-Inhibitor mit IC50-Werten von 17 nM bzw. 2 nM.
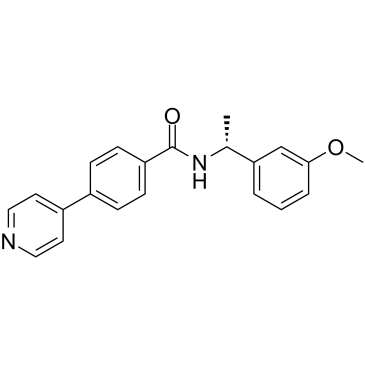
-
GC31952
ROCK-IN-1
ROCK-IN-1 ist ein potenter Inhibitor von ROCK mit einem IC50 von 1,2 nM fÜr ROCK2.
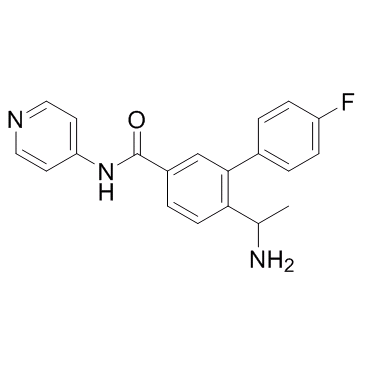
-
GC66338
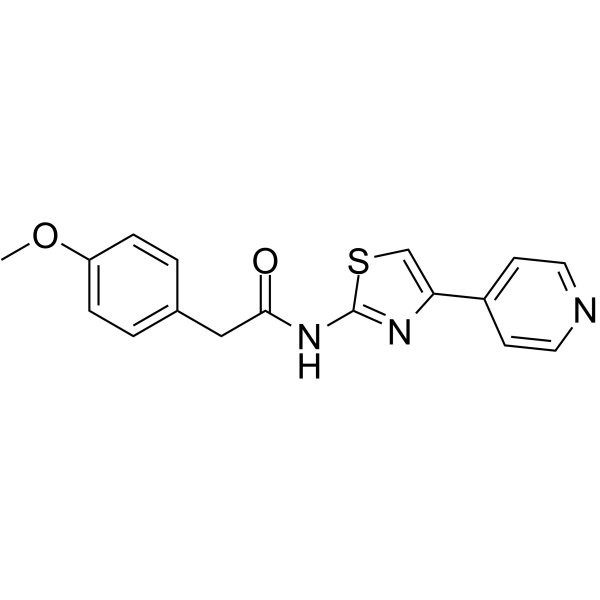
ROCK1-IN-1
ROCK1-IN-1 ist ein ROCK1-Inhibitor mit einem Ki-Wert von 540 nM. ROCK1-IN-1 kann zur Erforschung von Bluthochdruck, Glaukom und erektiler Dysfunktion eingesetzt werden.
-
GC31883
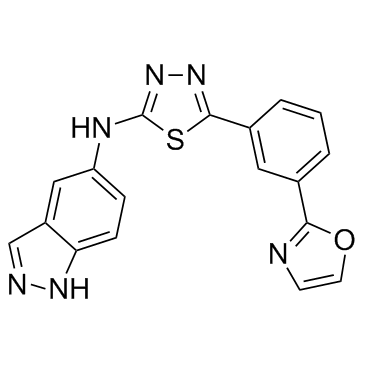
ROCK2-IN-2
ROCK2-IN-2 ist ein selektiver ROCK2-Inhibitor, extrahiert aus Patent US20180093978A1, Verbindung A-30, hat einen IC50 von <1 μM.
-
GC33169
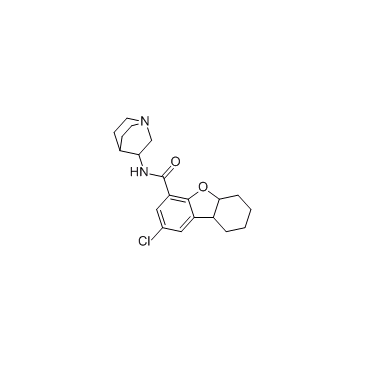
Rosabulin (STA 5312)
Rosabulin (STA 5312) (STA 5312) ist ein potenter und oral aktiver Mikrotubuli-Inhibitor, der die Mikrotubuli-Montage hemmt. Rosabulin (STA 5312) hat ein breites Spektrum an AntitumoraktivitÄt.
-
GC11401
Roscovitine (Seliciclib,CYC202)
Seliciclib
Roscovitin (Seliciclib, CYC202) (Roscovitin) ist ein oral bioverfÜgbarer und selektiver CDKs-Inhibitor mit IC50-Werten von 0,2 μM, 0,65 μM und 0,7 μM fÜr CDK5, Cdc2 bzw. CDK2.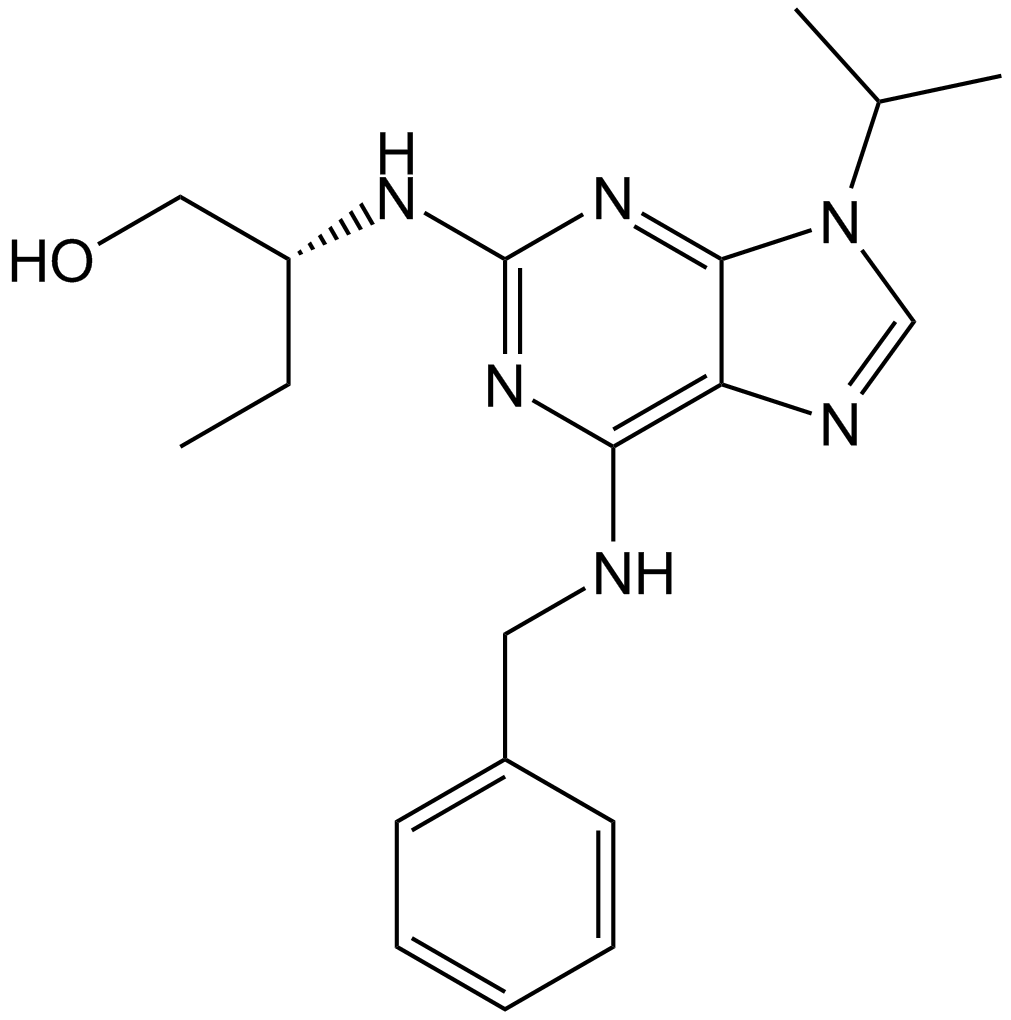
-
GC64278
RP-3500
RP-3500; ATR inhibitor 4
RP-3500 (ATR-Inhibitor 4) ist ein oral aktiver, selektiver ATR-Kinase-Inhibitor (ATRi) mit einem IC50-Wert von 1,00 nM in biochemischen Assays. RP-3500 zeigt eine 30-fache SelektivitÄt fÜr ATR gegenÜber mTOR (IC50=120 nM) und eine >2.000-fache SelektivitÄt gegenÜber ATM-, DNA-PK- und PI3Kα-Kinasen. RP-3500 hat eine starke AntitumoraktivitÄt.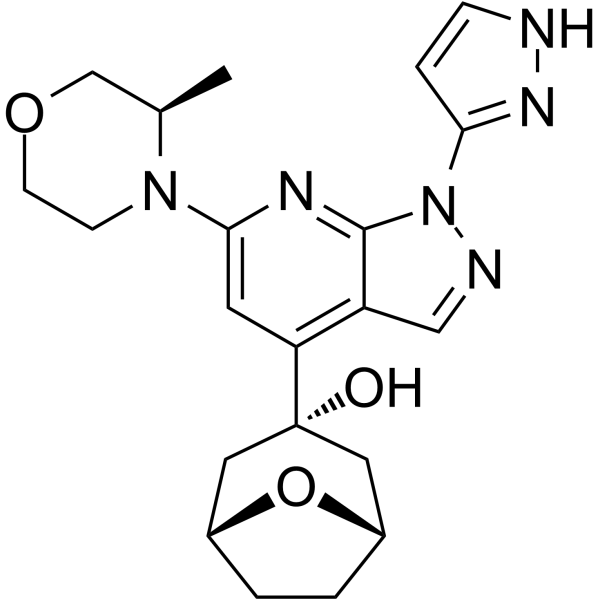
-
GC16265
RS-1
RS-1 ist ein RAD51-Aktivator und erhÖht auch die CRISPR/Cas9-vermittelte Knock-in-Effizienz.
-
GC18847
RSM-932A
TCD-717
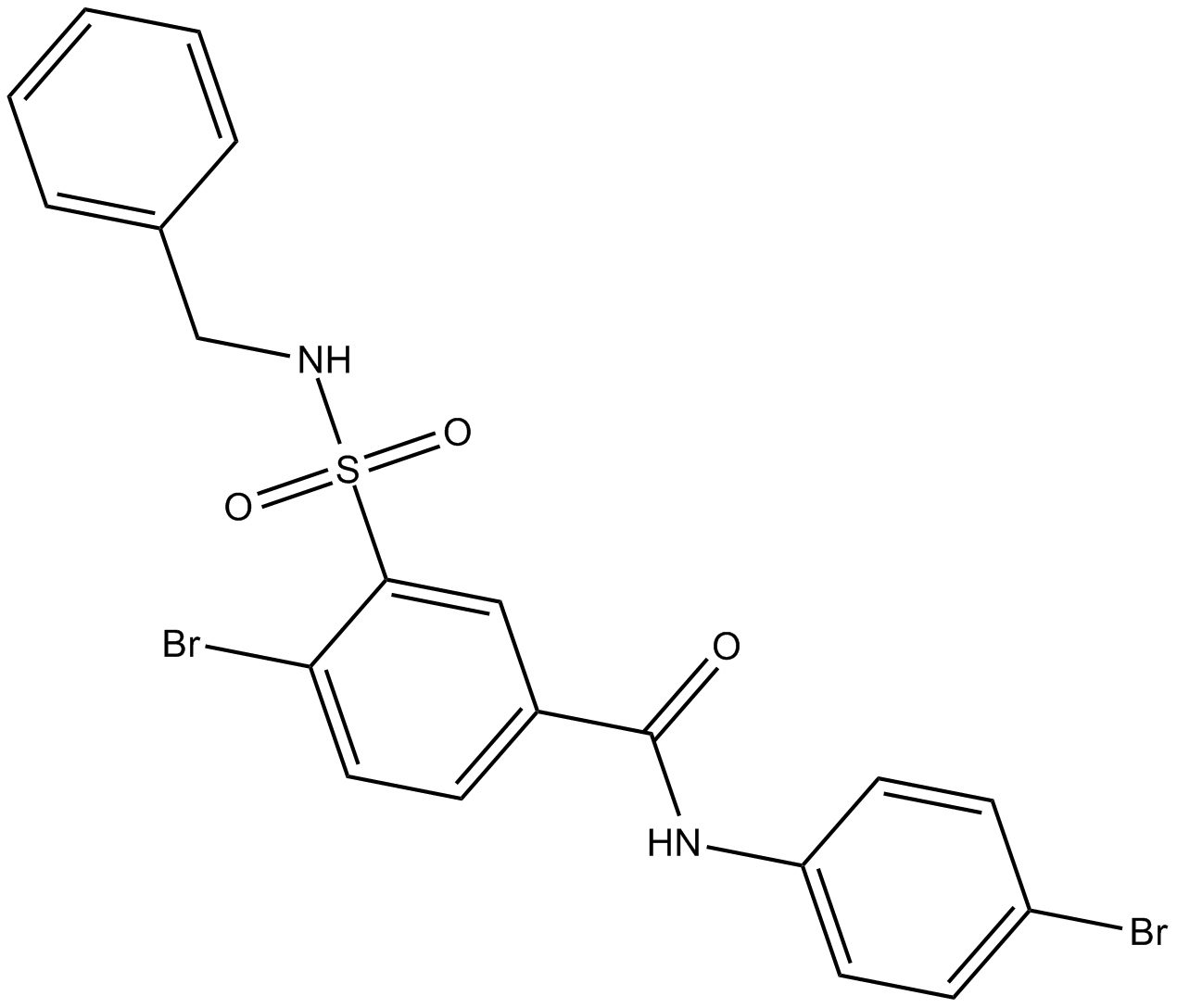
RSM-932A (TCD-717) ist ein spezifischer ChoKα-Inhibitor mit IC50-Werten von 1 bzw. 33 μM fÜr humane rekombinante ChoKα- bzw. ChoKβ-Enzyme. RSM-932A wirkt als die „erste Verbindung beim Menschen", die auf ChoKα abzielt. RSM-932A hat eine starke antiproliferative und in vivo antitumorale AktivitÄt gegen menschliche Xenotransplantate in MÄusen und zeigt eine hohe Wirksamkeit bei niedrigen ToxizitÄtsprofilen.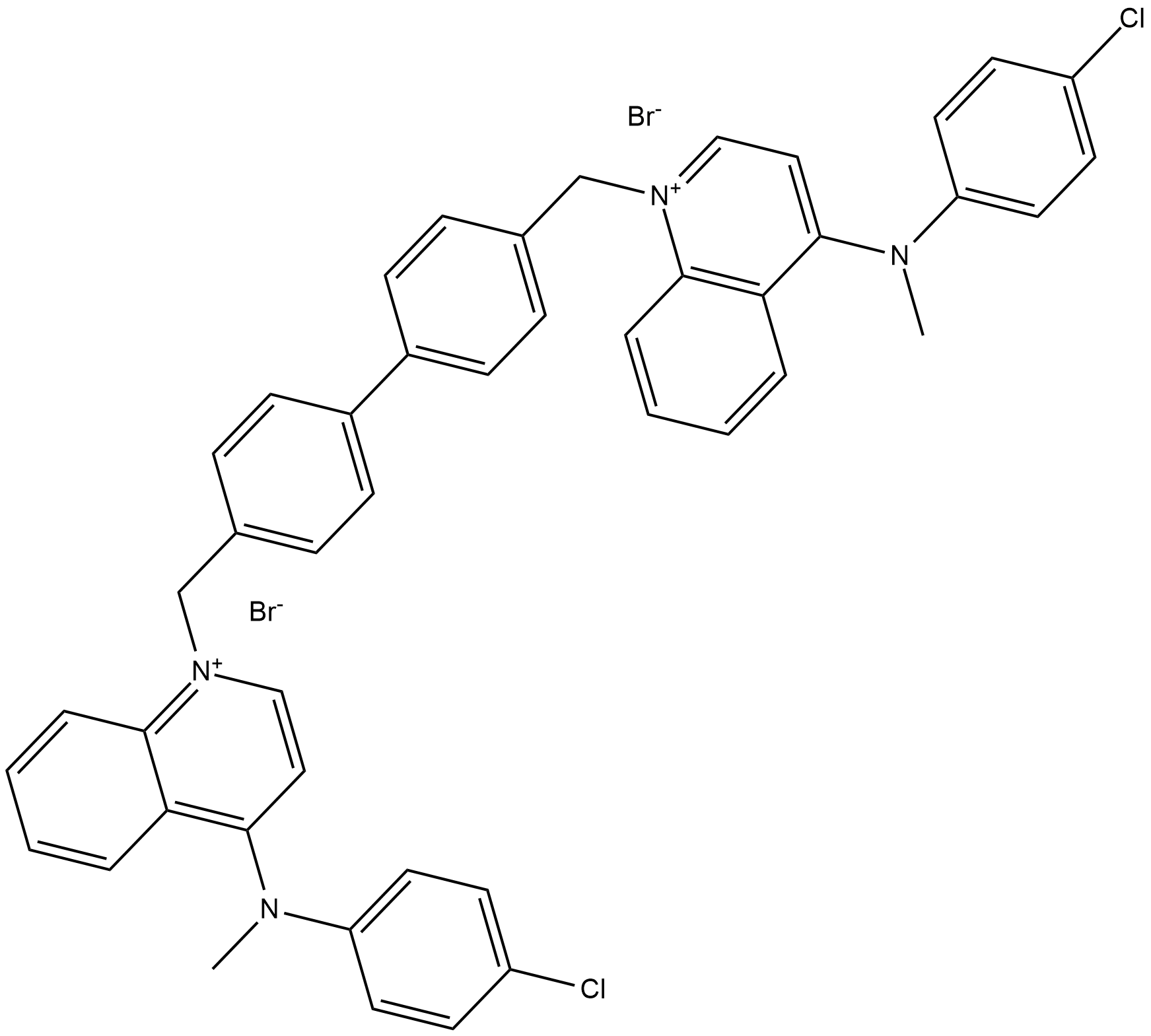
-
GC17150
Ryuvidine
Cdk4 Inhibitor III, Cyclic-dependent Kinase 4 Inhibitor III, SPS812
Ryuvidin ist ein potenter Inhibitor des SET-DomÄnen-enthaltenden Proteins 8 (SETD8) mit einem IC50 von 0,5 μM und unterdrÜckt die Monomethylierung von H4K20 in vitro. Ryuvidin hemmt auch CDK4 mit einem IC50 von 6,0 μM und ist zytotoxisch gegenÜber einer Reihe menschlicher Krebszellen.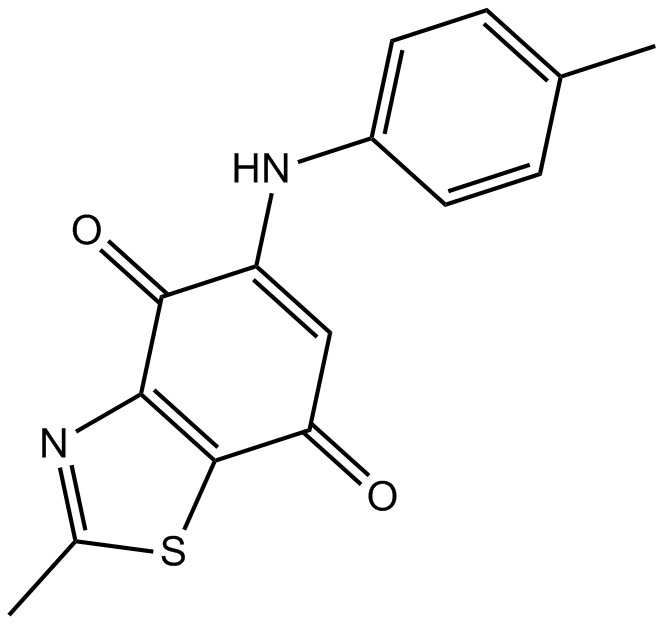
-
GC49532
S-(1,2-Dichlorovinyl)-Cysteine (hydrochloride)
S-(1,2-dichlorovinyl)-L-Cysteine, S-(trans-1,2-dichlorovinyl)-L-Cysteine, DCVC
A nephrotoxin and metabolite of trichloroethylene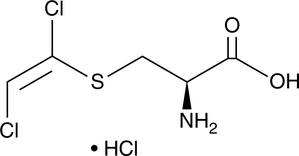
-
GC46224
S-Phenylcysteine
3-(phenylthio)-L-Alanine, (2R)-2-Amino-3-phenylsulfanylpropanoic acid
An adduct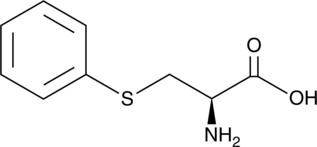
-
GC18852
S14161
D-Cyclins regulate the cell cycle by acting in a complex with cyclin dependent kinases (CDKs) to promote phosphorylation of the retinoblastoma protein and initiate cellular progression from the G1 to the S phase.
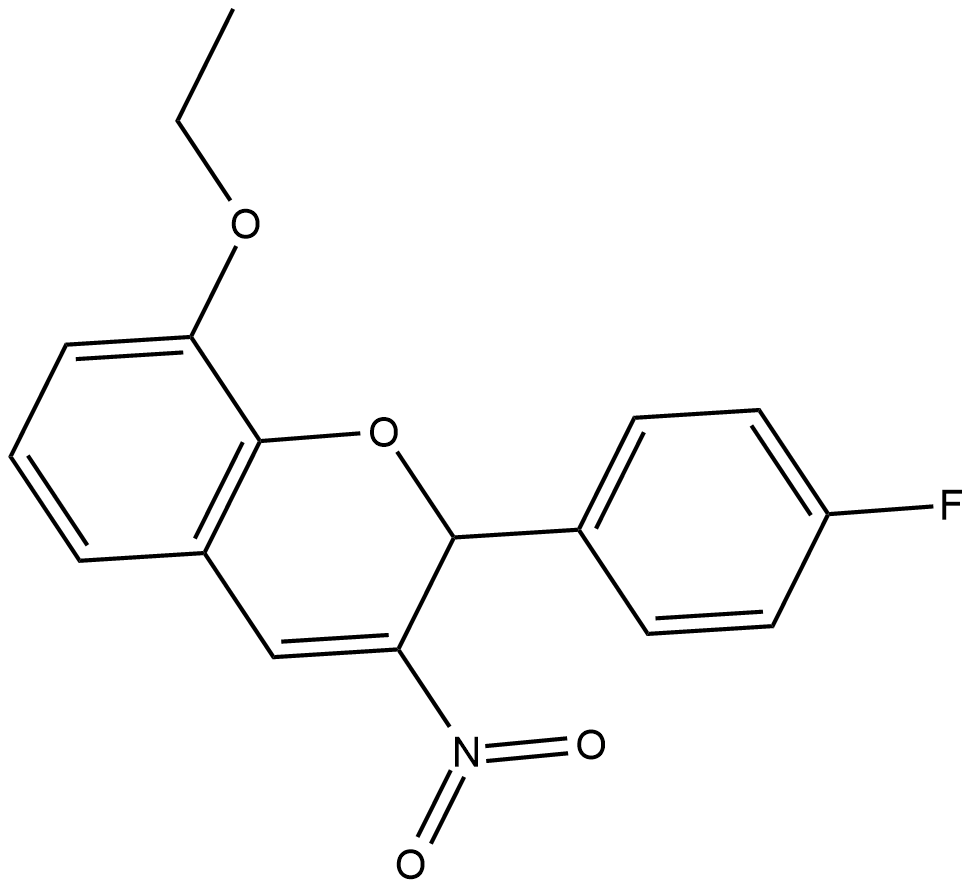
-
GC38945
S516
S516 (Verbindung 22) ist ein aktiver Metabolit von CKD-516 und ein potenter Tubulin-Polymerisationsinhibitor mit einem IC50 von 4,29 μM. S516 hat eine ausgeprÄgte AntitumoraktivitÄt.
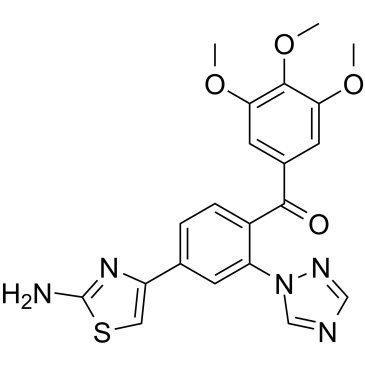
-
GC49093
Safflower Red
Carthamin Yellow
A red pigment with diverse biological activities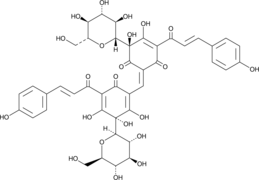
-
GC38845
sAJM589
sAJM589 ist ein Myc-Inhibitor, der das Myc-Max-Heterodimer mit einem IC50 von 1,8 μM stark stÖrt.
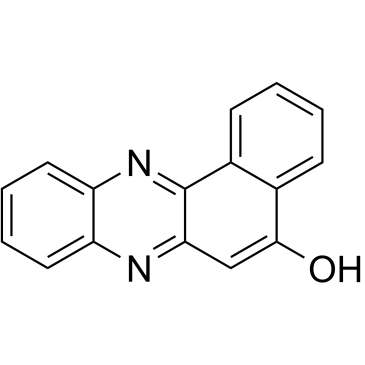
-
GC13759
SAR407899
SAR407899 ist ein selektiver, potenter und ATP-kompetitiver ROCK-Inhibitor mit einem IC50-Wert von 135 nM fÜr ROCK-2 und einem Kis von 36 nM bzw. 41 nM fÜr ROCK-2 aus Mensch und Ratte.
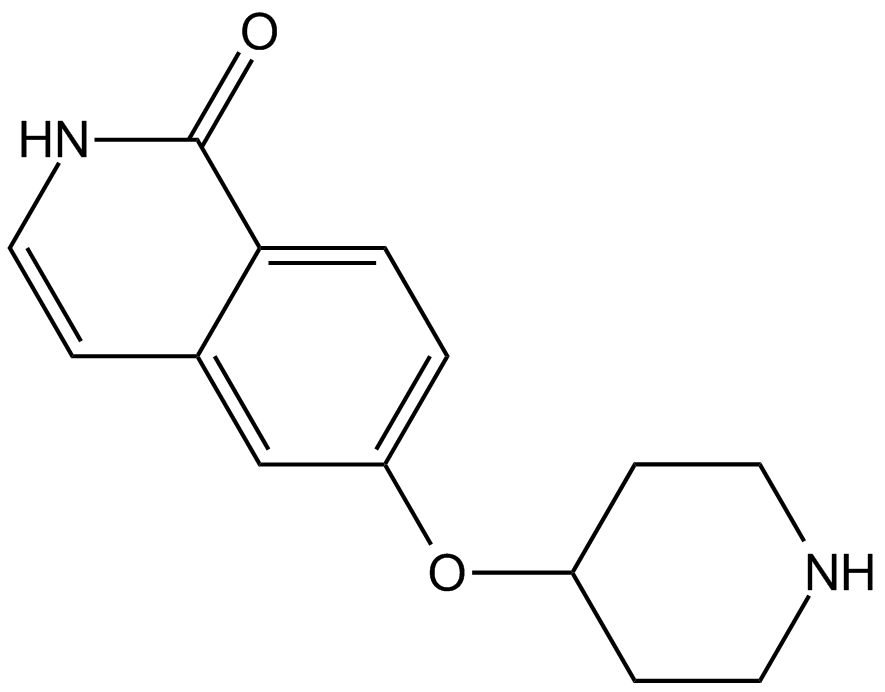
-
GC16289
SAR407899 hydrochloride
SAR407899-Hydrochlorid ist ein selektiver, potenter und ATP-kompetitiver ROCK-Inhibitor mit einem IC50-Wert von 135 nM fÜr ROCK-2 und einem Kis von 36 nM bzw. 41 nM fÜr ROCK-2 von Mensch und Ratte.
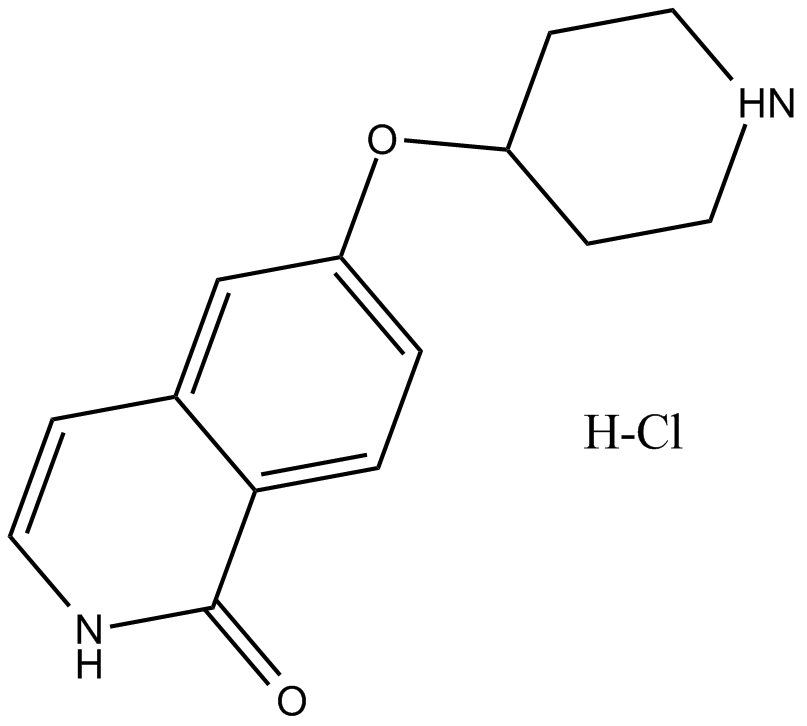
-
GC46218
Satratoxin G
A macrocyclic trichothecene mycotoxin
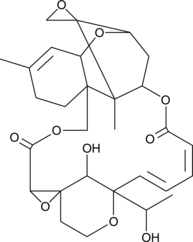
-
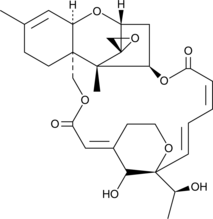
GC46219
Satratoxin H
A trichothecene mycotoxin
-
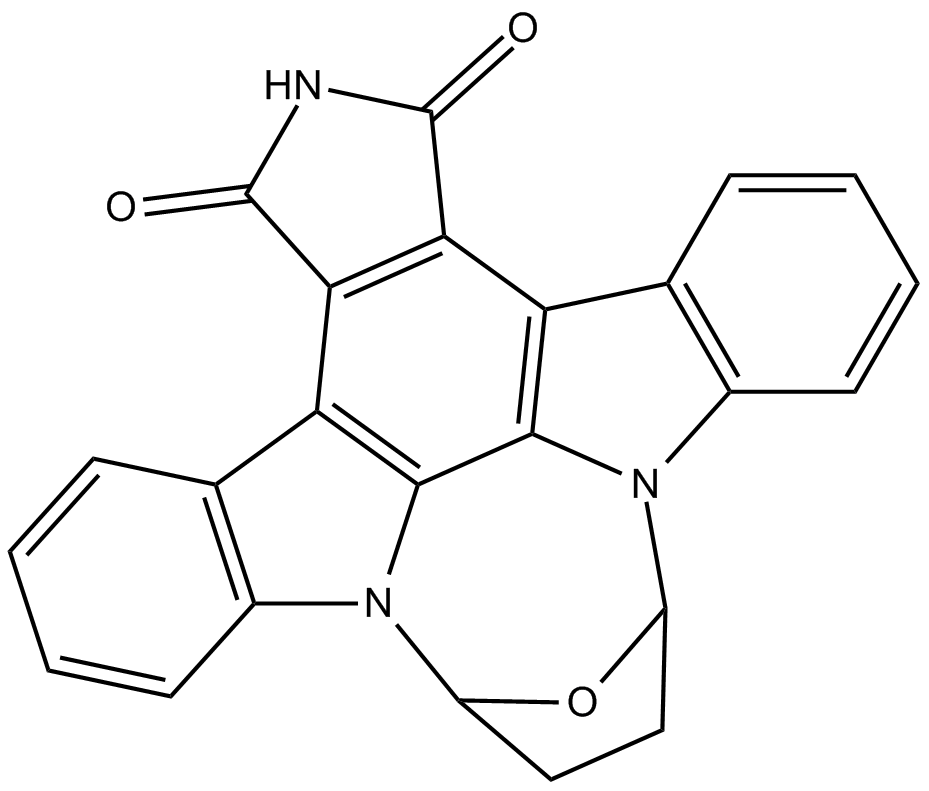
GC11022
SB 218078
SB 218078 ist ein potenter, selektiver, ATP-kompetitiver und zellgängiger Checkpoint-Kinase-1 (Chk1)-Inhibitor, der die Chk1-Phosphorylierung von cdc25C mit einem IC50 von 15 nM hemmt. SB 218078 hemmt weniger stark Cdc2 (IC50 von 250 nM) und PKC (IC50 von 1000 nM). SB 218078 verursacht Apoptose durch DNA-Schädigung und Zellzyklusarrest.
-
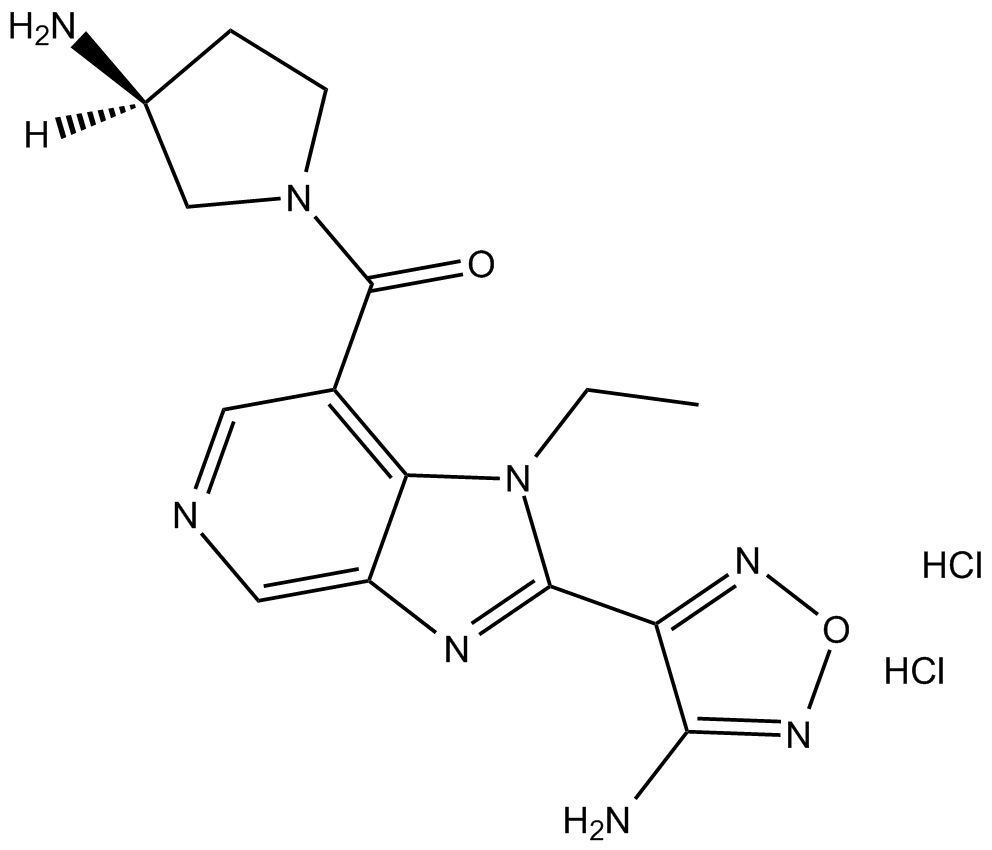
GC15170
SB 772077B dihydrochloride
SB 772077B Dihydrochlorid ist ein Aminofurazan-basierter Rho-Kinase (ROCK)-Inhibitor mit IC50-Werten von 5,6 nM und 6 nM für ROCK1 bzw. ROCK2.
-
GC66437
SB-216
SB-216 ist ein potenter Tubulin-Polymerisationsinhibitor. SB-216 zeigt eine starke antiproliferative Potenz in einer Reihe menschlicher Krebszelllinien, darunter Melanom, Lungenkrebs und Brustkrebs. SB-216 kann fÜr die Krebsforschung verwendet werden.
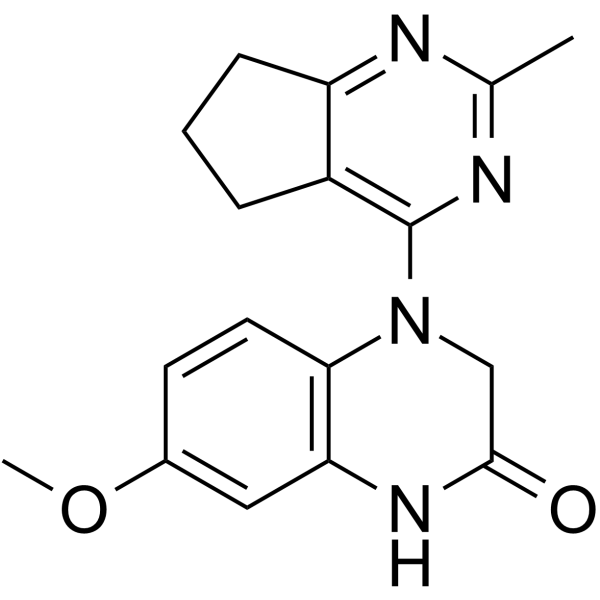
-
GC12064
SB1317
TG02, Zotiraciclib
A multi-kinase inhibitor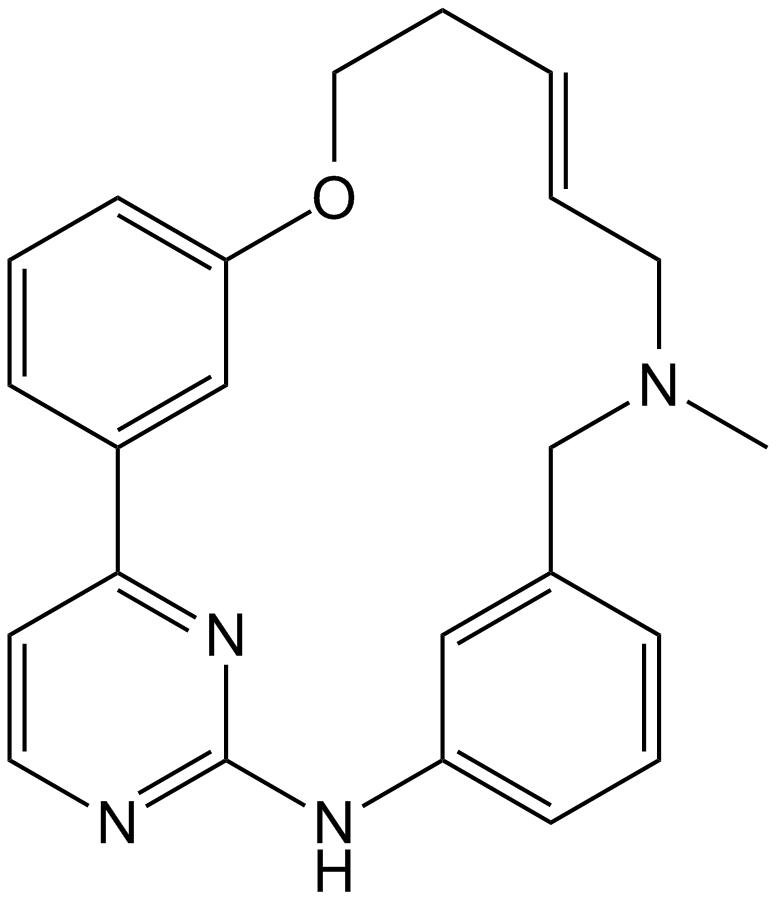
-
GC14600
SB743921
SB743921 ist ein potenter Inhibitor des mitotischen Kinesins KSP (Eg5) mit einem Ki von 0,1 nM.
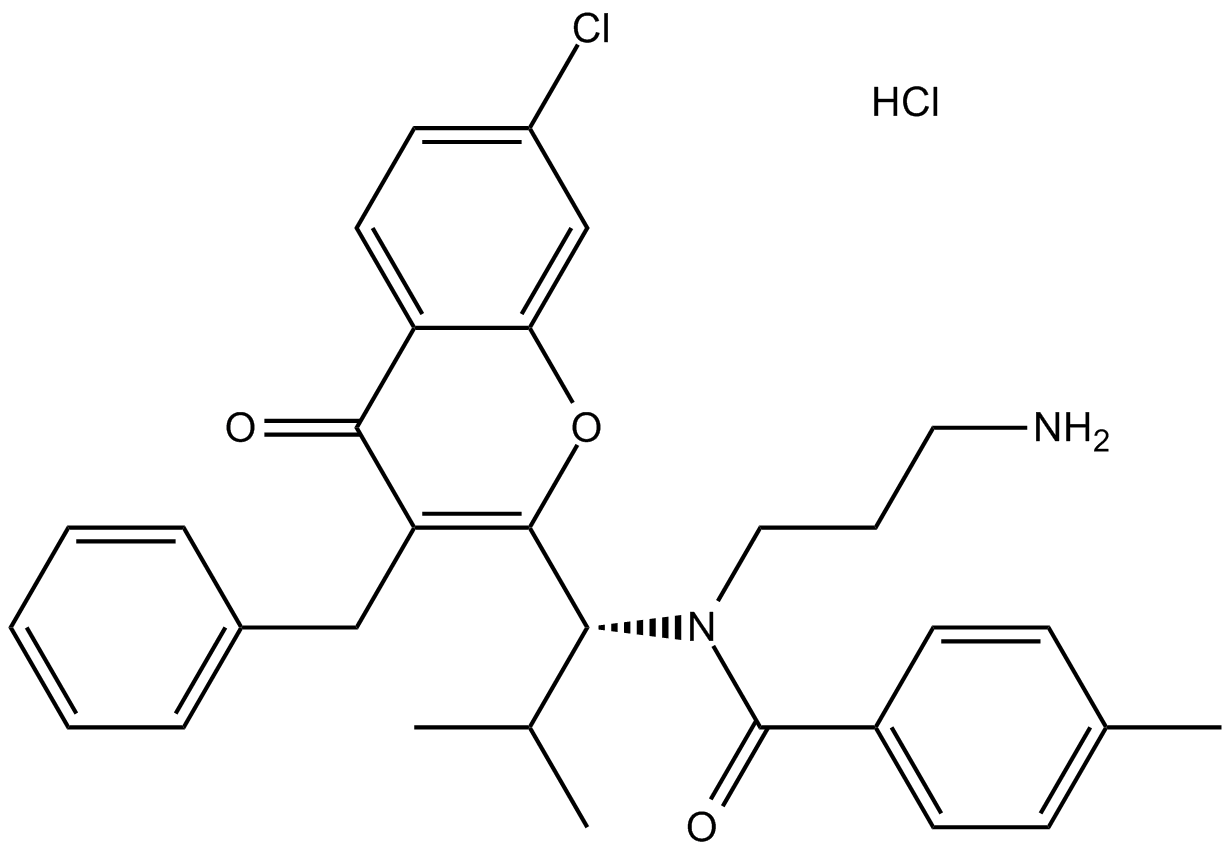
-
GC14644
SBE 13 HCl
SBE 13 HCl ist ein potenter und selektiver Plk1-Inhibitor mit einem IC50 von 200 pM; SBE 13 HCl hemmt Plk2 (IC50 > 66μ M) oder Plk3 (IC50 = 875 nM) schwach.
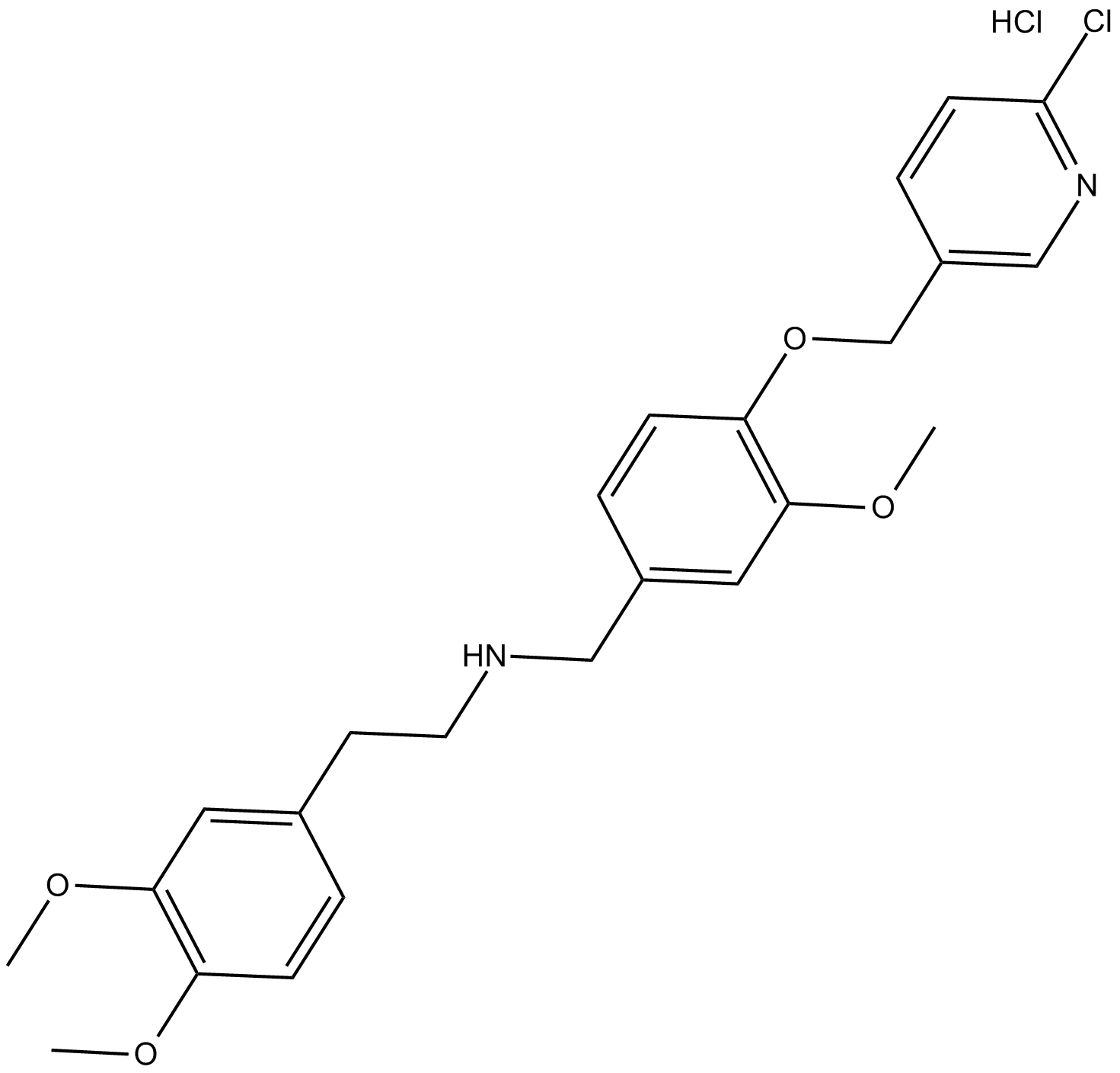
-
GC37601
SBE13
SBE13 ist ein potenter und selektiver Plk1-Inhibitor mit einem IC50 von 200 pM; SBE13 hemmt Plk2 (IC50>66μM) oder Plk3 (IC50=875nM) nur schwach.
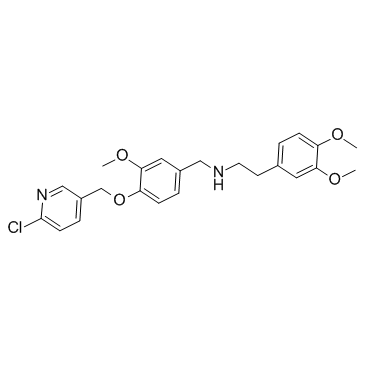
-
GC37606
SCH-1473759 hydrochloride
SCH-1473759-Hydrochlorid ist ein Aurora-Inhibitor mit IC50-Werten von 4 und 13 nM fÜr Aurora A bzw. B.
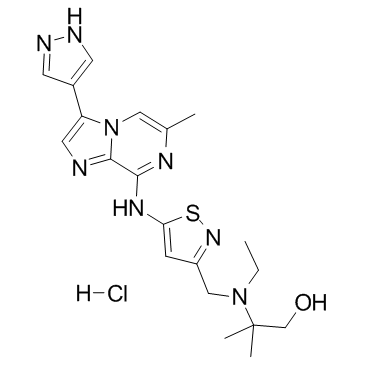
-
GC17792
SCH900776 S-isomer
SCH 900776 S-isomer;SCH-900776 S-isomer
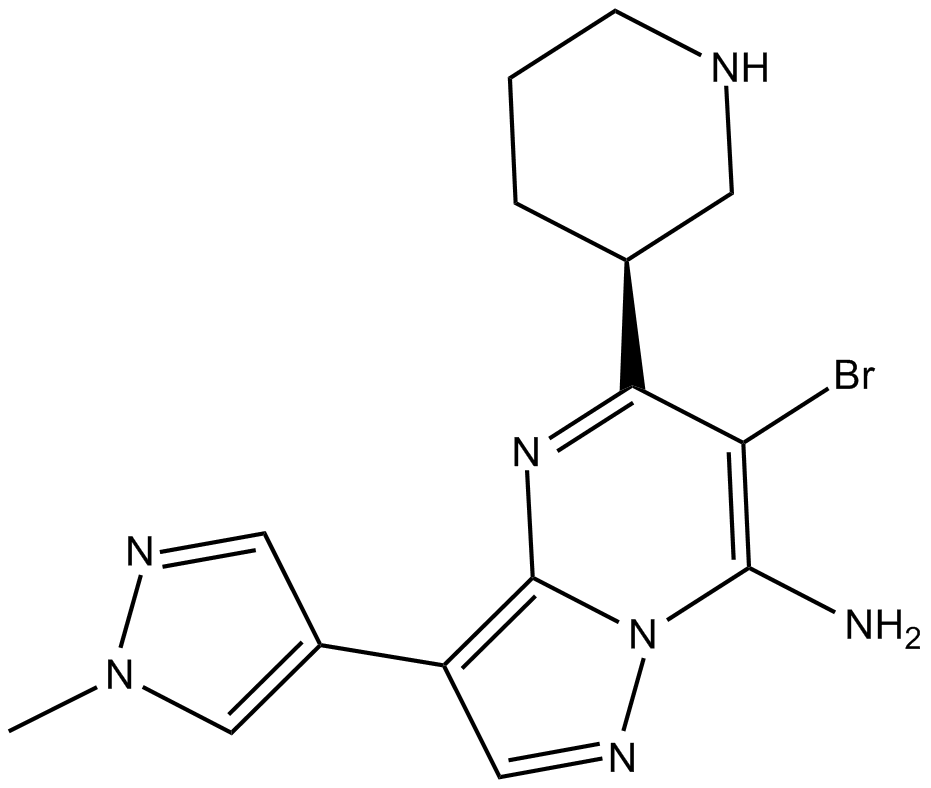
-
GN10091
Schaftoside
Apigenin 6-C-glucoside-8-C-arabinoside
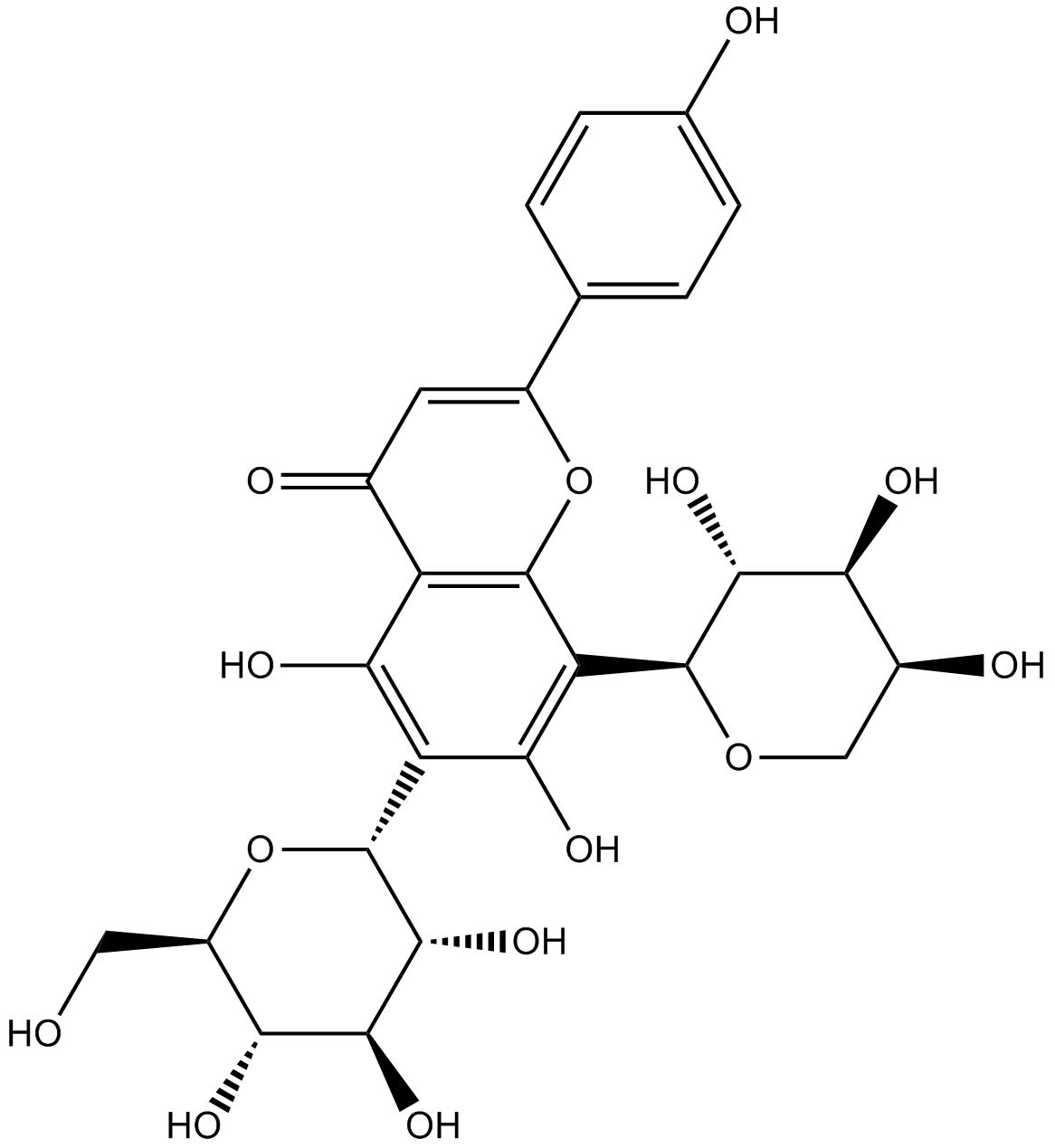
-
GC49674
Schizandrin
(+)-Schizandrin, Schizandrol A
Schizandrin (Schizandrin), ein Dibenzocyclooctadien-Lignan, wird aus der Frucht von Schisandra chinensis Baill isoliert.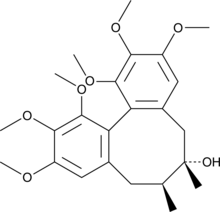
-
GC39798
Scoulerine
(–)-Scoulerine, l-Scoulerine, (S)-Scoulerine
Scoulerine ((-)-Scoulerine), ein Isochinolin-Alkaloid, ist eine starke antimitotische Verbindung. Scoulerine ist auch ein Inhibitor von BACE1 (ß-site amyloid precursor protein cleavingenzym 1). Scoulerine hemmt die Proliferation, hÄlt den Zellzyklus an und induziert Apoptose in Krebszellen.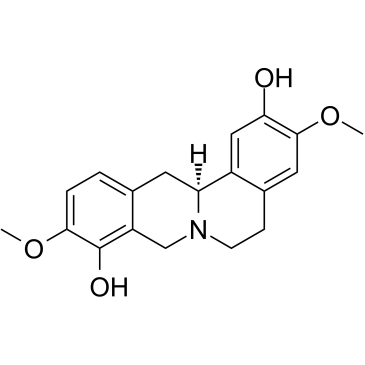
-
GC49723
SenTraGor™ Cell Senescence Reagent
GL13, SBB-A-B, SBB-Analogue (GL13) Biotin
A cellular senescence detection reagent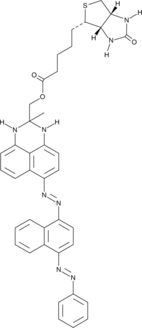
-
GC48076
Sesquicillin A
Sesquicillin
A fungal metabolite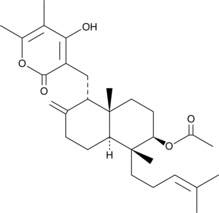
-
GC10019
SF1670
SF1670 ist ein potenter und spezifischer Phosphatase- und Tensin-Homolog, der auf dem Chromosom 10 (PTEN)-Inhibitor deletiert ist.
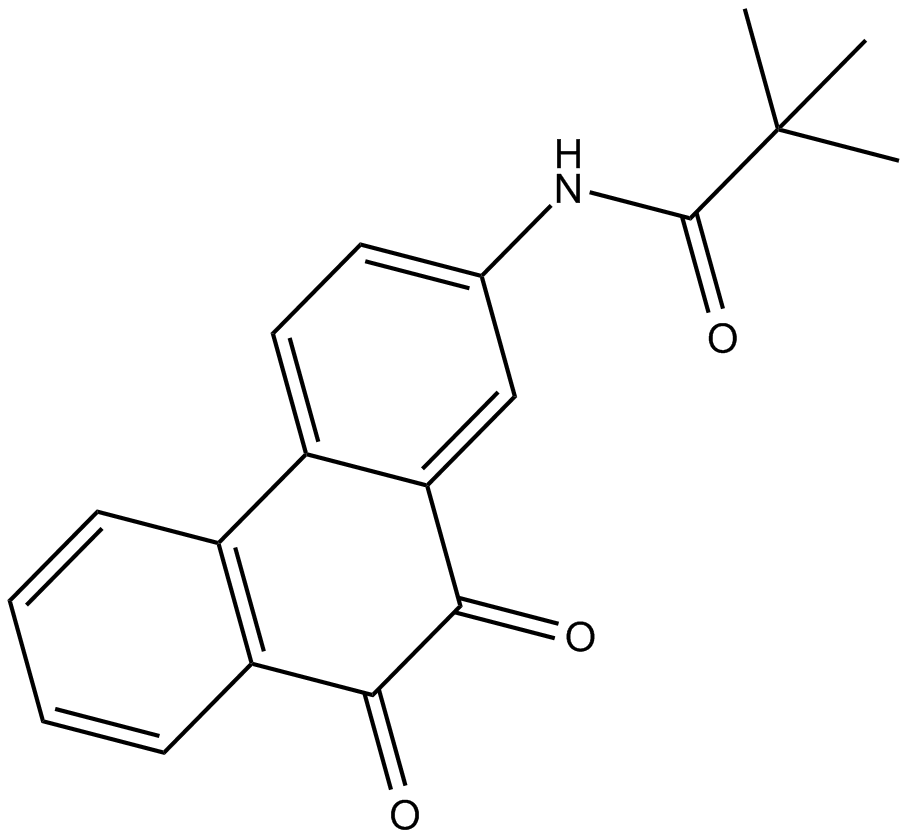
-
GC49713
SIKVAV (acetate)
Hexapeptide-10, Ser-Ile-Lys-Val-Ala-Val
A laminin α1-derived peptide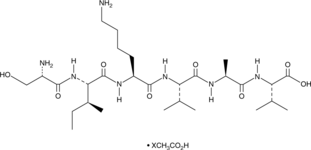
-
GC49002
Sinigrin (hydrate)
Sinigrin (Hydrat) ist ein natürliches aliphatisches Glucosinolat, das in Pflanzen der Familie Brassicaceae vorkommt.
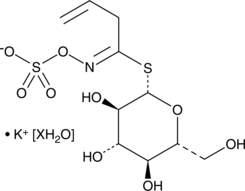
-
GC64539
SKLB-197
SKLB-197 zeigte einen IC50-Wert von 0,013 μM gegen ATR, aber sehr schwache oder keine AktivitÄt gegen andere 402-Proteinkinasen. Es zeigte sowohl in vitro als auch in vivo eine starke AntitumoraktivitÄt gegen ATM-defiziente Tumore.
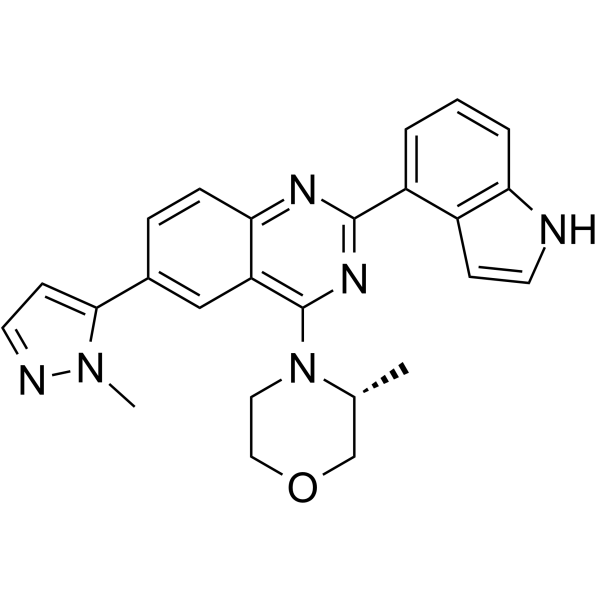
-
GC12827
SLx-2119
Belumosudil, SLx-2119
A ROCK2 inhibitor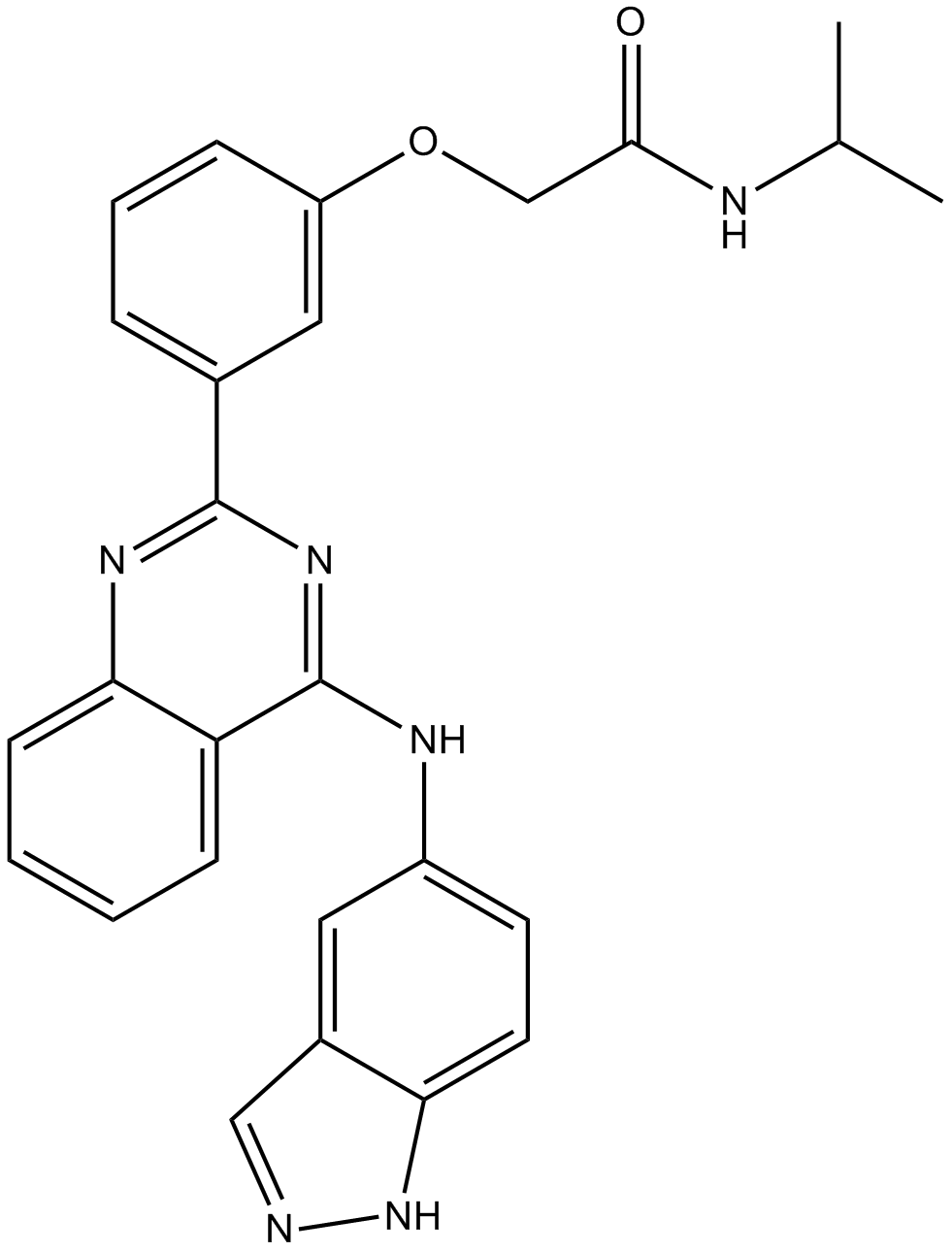
-
GC11396
SNS-032 (BMS-387032)
BMS-387032
SNS-032 (BMS-387032) (BMS-387032) ist ein potenter und selektiver Inhibitor von CDK2, CDK7 und CDK9 mit IC50s von 38 nM, 62 nM bzw. 4 nM. SNS-032 (BMS-387032) hat eine Antitumorwirkung.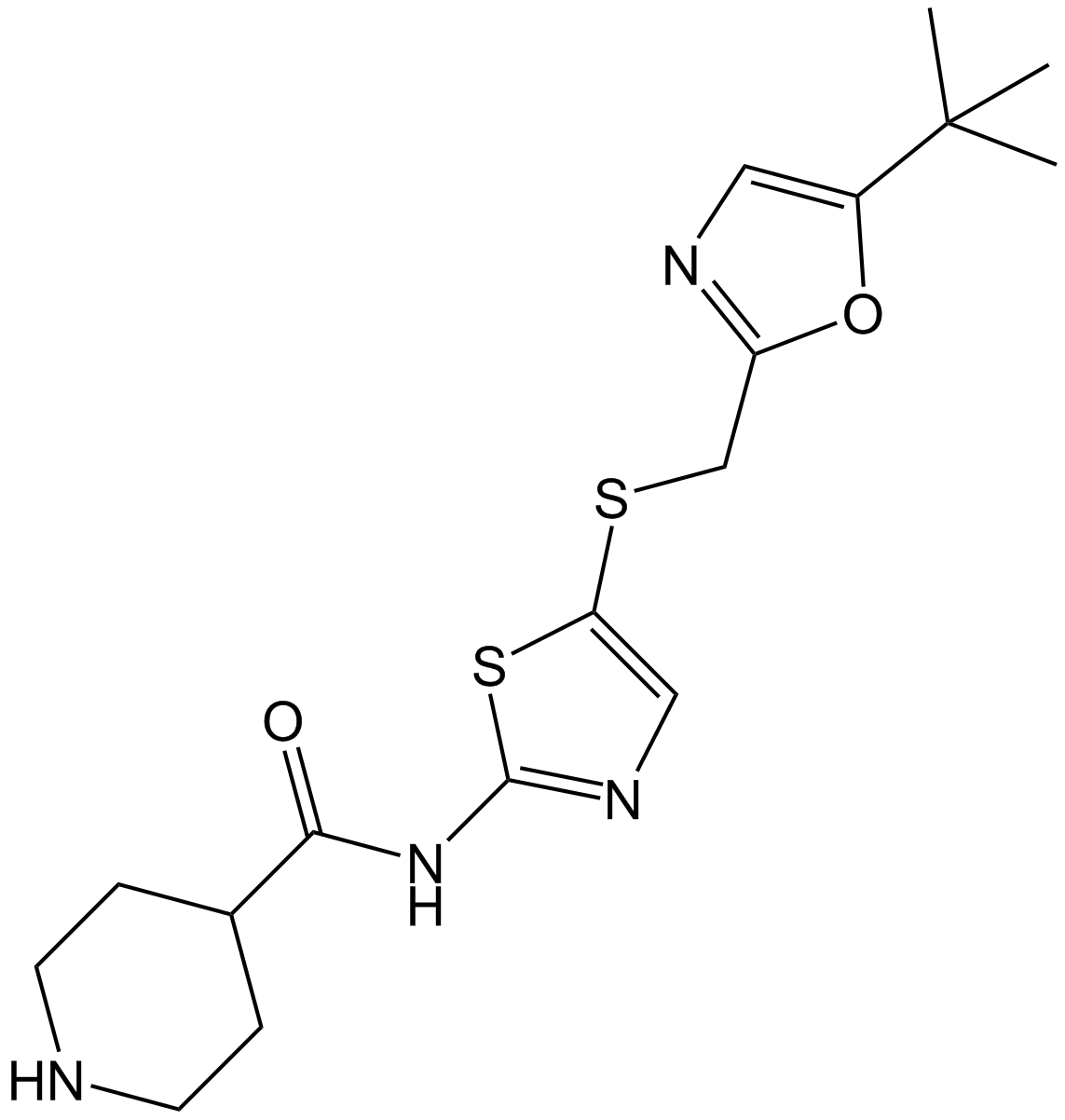
-
GC25940
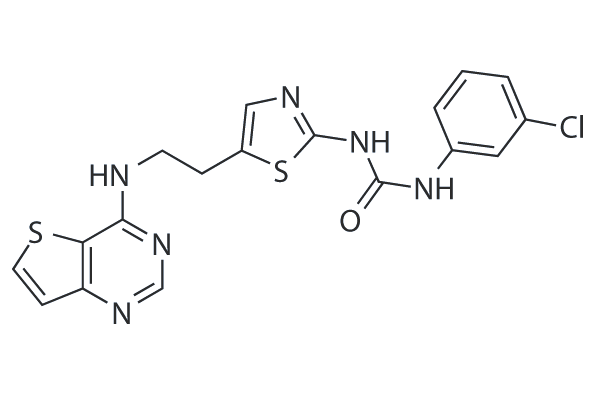
SNS-314
SNS-314 is a potent and selective inhibitor of Aurora A, Aurora B and Aurora C with IC50 of 9 nM, 31 nM, and 3 nM, respectively. It is less potent to Trk A/B, Flt4, Fms, Axl, c-Raf and DDR2. Phase 1.
-
GC32935
Soblidotin (Auristatin PE)
Auristatin PE; TZT-1027
Soblidotin (Auristatin PE) (Auristatin PE) ist ein neuartiges synthetisches Dolastatin-10-Derivat und ein Inhibitor der Tubulin-Polymerisation.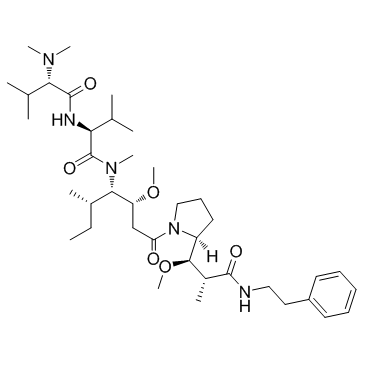
-
GC48084
Sodium 4-Phenylbutyrate-d11
Benzenebutanoic acid-d11, TriButyrate-d11
Phenylbutyrat-d11 (Natrium) ist mit Deuterium markiertes Natrium-4-phenylbutyrat. Natrium-4-phenylbutyrat (4-PBA-Natrium) ist ein Inhibitor von HDAC und Stress des endoplasmatischen Retikulums (ER), der in der Krebs- und Infektionsforschung eingesetzt wird.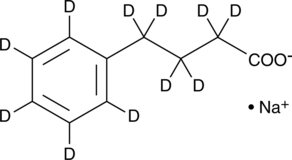
-
GC65887
SOP1812
SOP1812 ist ein Naphthalindiimid (ND)-Derivat mit Anti-Tumor-AktivitÄt. SOP1812 bindet an Quadruplex-Anordnungen (G4s) und reguliert mehrere Krebsgenwege herunter. SOP1812 zeigt eine große AffinitÄt zu hTERT G4 und HuTel21 G4 mit KD-Werten von 4,9 bzw. 28,4 nM. SOP1812 kann fÜr die Krebsforschung eingesetzt werden.
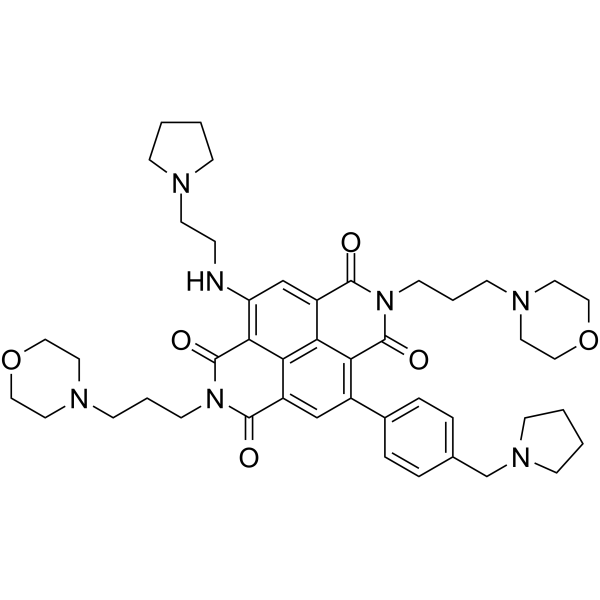
-
GC63351
Sovesudil
PHP-201; AMA0076
Sovesudil (PHP-201) ist ein potenter, ATP-kompetitiver, lokal wirkender Rho-Kinase (ROCK)-Hemmer mit IC50-Werten von 3,7 und 2,3 nM fÜr ROCK-I bzw. ROCK-II.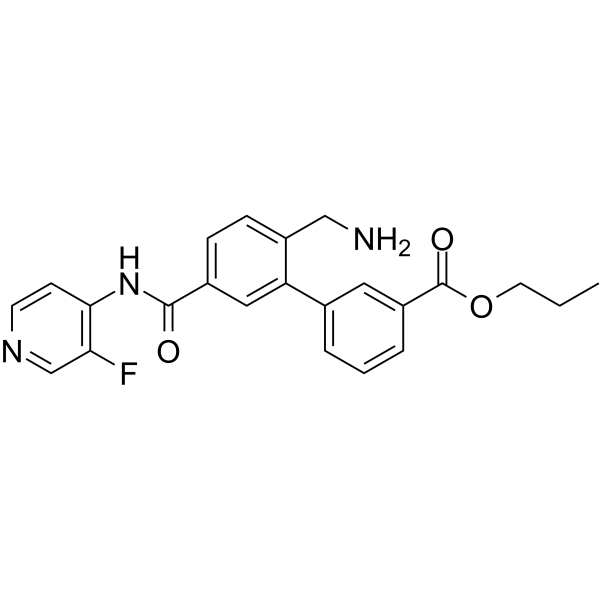
-
GC63716
Sovesudil hydrochloride
PHP-201 hydrochloride; AMA0076 hydrochloride
Sovesudil (PHP-201) Hydrochlorid ist ein potenter, ATP-kompetitiver, lokal wirkender Rho-Kinase (ROCK)-Hemmer mit IC50-Werten von 3,7 und 2,3 nM fÜr ROCK-I bzw. ROCK-II.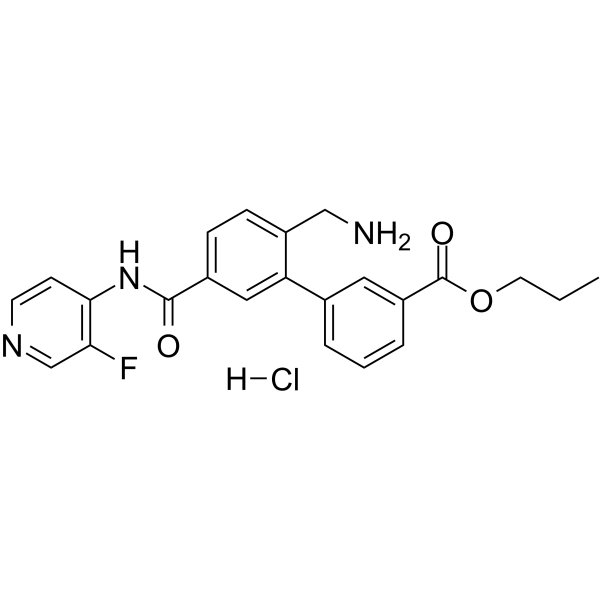
-
GC64019
Sovilnesib
AMG 650
Sovilnesib (AMG 650) ist ein Kinesin-Ähnlicher Protein-KIF18A-Inhibitor (WO2020132648). Sovilnesib kann zur Krebsforschung eingesetzt werden.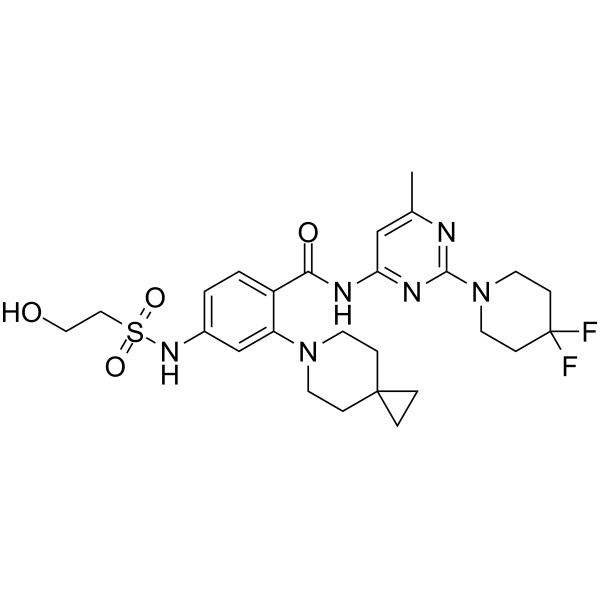
-
GC48092
Spinosad
LY232105, XDE-105
Spinosad, ein als Fermentationsprodukt eines Bodenaktinomyceten (Saccharopolyspora spinosa) bekanntes Gemisch der Spinosyn A und D, ist ein biologisches neurotoxisches Insektizid mit einem breiteren Wirkungsspektrum.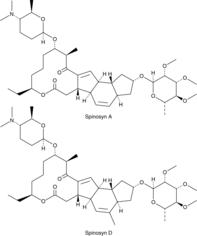
-
GC41258
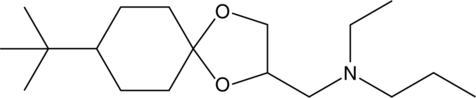
Spiroxamine
Spiroxamine is a tertiary amine fungicide and an inhibitor of δ14 reductase/δ8→δ7 isomerase.
-
GC44946
Sporidesmolide III
Sporidesmolide III is a cyclodepsipeptide originally isolated from P.