Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Targets for Cell Cycle/Checkpoint
- ATM/ATR(23)
- Aurora Kinase(17)
- Cdc42(4)
- Cdc7(3)
- Chk(14)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(77)
- E1 enzyme(1)
- G-quadruplex(11)
- Haspin(6)
- HMTase(1)
- Kinesin(23)
- Ksp(4)
- Microtubule/Tubulin(219)
- Mps1(15)
- Mitotic(7)
- RAD51(16)
- ROCK(60)
- Rho(13)
- PERK(10)
- PLK(33)
- PTEN(6)
- Wee1(7)
- PAK(20)
- Arp2/3 Complex(8)
- Dynamin(10)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Products for Cell Cycle/Checkpoint
- Cat.No. Nombre del producto Información
-
GC44730
proTAME
Pro-N-4-tosyl-L-arginine methyl ester
proTAME es una forma de prodroga permeable a la célula del éster metílico N-4-tosil-L-arginina, un inhibidor del complejo/ciclosoma promotor de anafase (APC/C), que se convierte en TAME por esterasas intracelulares.
-
GC44732
Protopine (hydrochloride)
Protopine is an alkaloid found in Berberidaceae, Ranunculaceae, Rutaceae, Fumariaceae, and Papaveraceae with diverse biological activities.
-
GC49914
Pt(II)-NHC Complex 2C
Platinum(II)-N-Heterocyclic Carbene Complex 2C
An inducer of immunogenic cancer cell death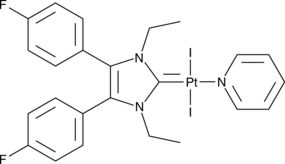
-
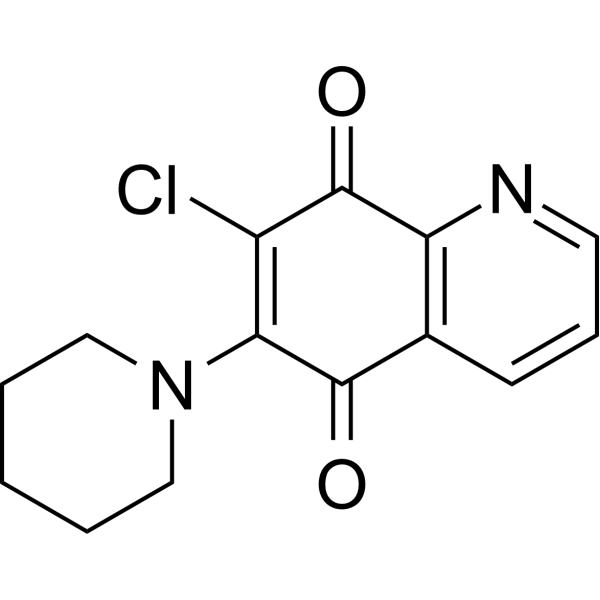
GC69776
PT-262
PT-262 es un inhibidor efectivo de ROCK con un valor IC50 de aproximadamente 5 μM. PT-262 induce la pérdida del potencial de membrana mitocondrial y aumenta la activación de caspasa-3 y la apoptosis celular. PT-262 inhibe la fosforilación de ERK y CDC2 a través de una vía independiente de p53. PT-262 bloquea la función del citoesqueleto celular y la migración celular. PT-262 tiene actividad anticancerígena.
-
GC44740
PtdIns-(1,2-dioctanoyl) (sodium salt)
DOPI, Phosphatidylinositol C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.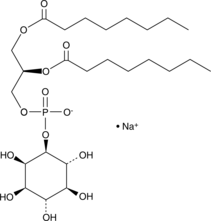
-
GC44743
PtdIns-(3)-P1 (1,2-dioctanoyl) (sodium salt)
DOPI3P1, Phosphatidylinositol3monophosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.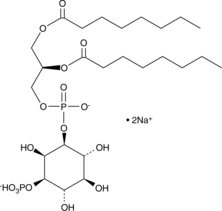
-
GC44748
PtdIns-(3,4,5)-P3 (1,2-dihexanoyl) (ammonium salt)
DHPI-3,4,5-P3, Phosphatidylinositol-3,4,5-triphosphate C-6, PIP3C-16
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.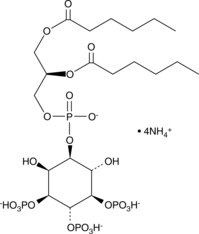
-
GC44750
PtdIns-(3,4,5)-P3 (1,2-dipalmitoyl) (sodium salt)
DPPI3,4,5P3, Phosphatidylinositol3,4,5triphosphate C16
The phosphatidylinositol phosphates represent a small percentage of total membrane phospholipids.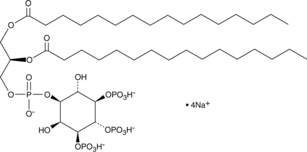
-
GC44753
PtdIns-(3,4,5)-P3-biotin (sodium salt)
Phosphatidylinositol3,4,5triphosphate C8biotin, PIP3biotin
The PtdIn phosphates play an important role in the generation and transduction of intracellular signals.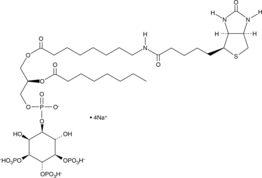
-
GC44759
PtdIns-(4)-P1 (1,2-dioctanoyl) (ammonium salt)
DOPI4P1, Phosphatidylinositol4phosphate C8
The phosphatidylinositol (PtdIns) phosphates represent a small percentage of total membrane phospholipids.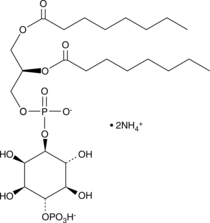
-
GC52484
Purified Ganglioside Mixture (bovine) (ammonium salt)
A mixture of purified bovine gangliosides

-
GC14707
Purvalanol A
NG 60
Purvalanol A es un potente inhibidor de CDK, que inhibe cdc2-ciclina B, cdk2-ciclina A, cdk2-ciclina E, cdk4-ciclina D1 y cdk5-p35 con IC50 de 4, 70, 35, 850, 75 nM, respectivamente.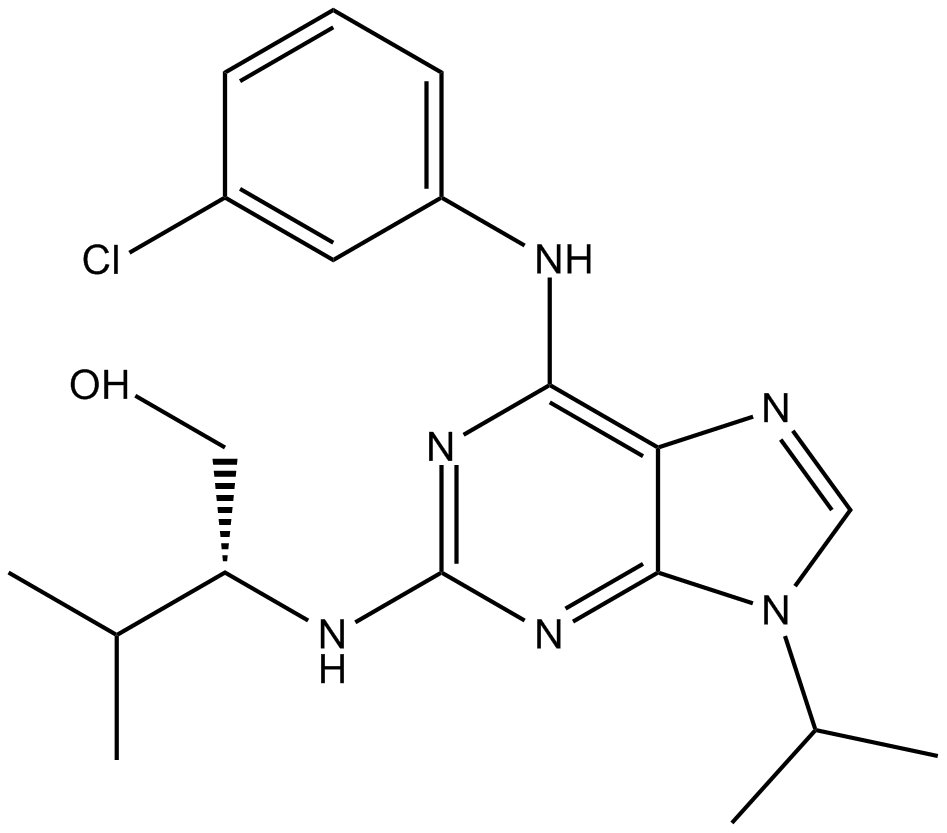
-
GC16268
Purvalanol B
NG 95; NG95; NG-95
Purvalanol B (NG 95) es un inhibidor de CDK potente, selectivo, reversible y competitivo con ATP, con IC50 de 6 nM, 6 nM, 9 nM, 6 nM para cdc2-ciclina B, CDK2-ciclina A, CDK2-ciclina E y CDK5-p35, respectivamente.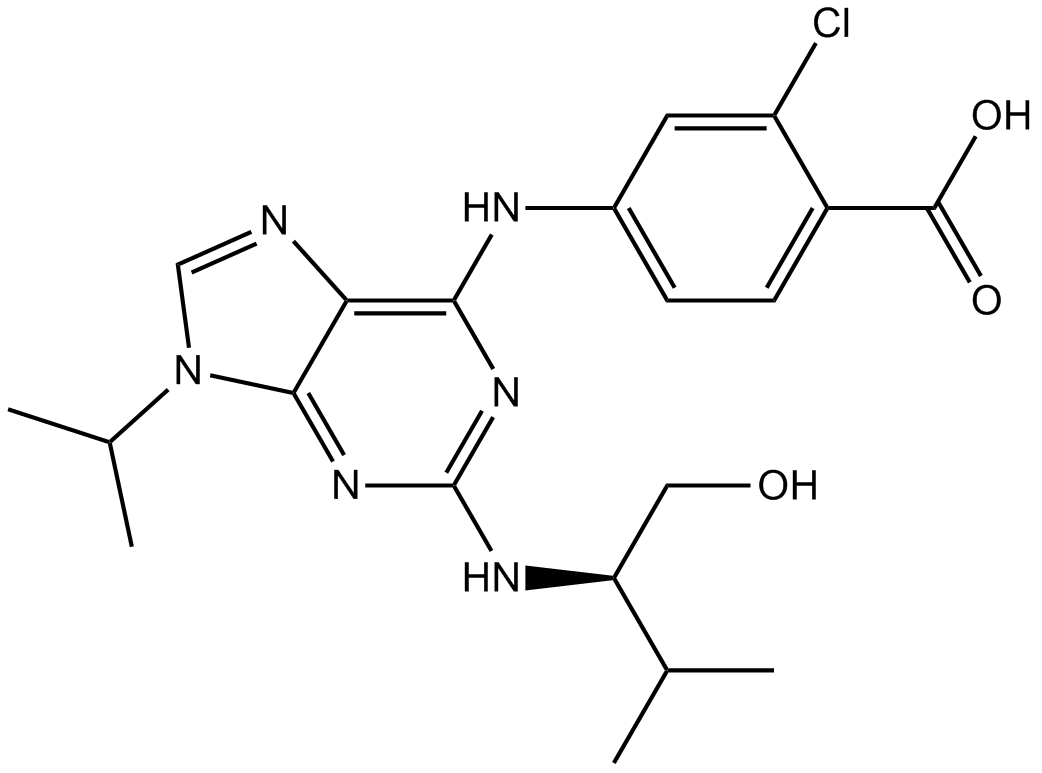
-
GC45552
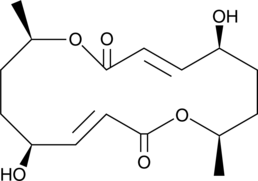
Pyrenophorol
El pirenoforol es un compuesto C9H12O3 aislado con bajo rendimiento de filtrados de cultivo del hongo fitopatÓgeno Stemphylium radicinum, C.
-
GC15162
Pyridostatin
Pyridostatin es una pequeña molécula altamente selectiva que se une a los G-cuádruplex y fue diseñada para dirigirse a estructuras polimórficas de G-cuádruplex independientemente de la variabilidad de secuencia.
-
GC48016
Pyridostatin (trifluoroacetate salt)
RR82 TFA
La piridostatina (RR82) TFA es un agente estabilizador del ADN cuÁdruplex G (Kd = 490 nM). La piridostatina (sal de trifluoroacetato) promueve la detenciÓn del crecimiento en células cancerosas humanas al inducir daÑos en el ADN dependientes de la replicaciÓn y la transcripciÓn. La piridostatina (sal de trifluoroacetato) se dirige al protooncogén Src. La piridostatina (sal de trifluoroacetato) redujo los niveles de proteÍna SRC y la motilidad celular dependiente de SRC en células de cÁncer de mama humano.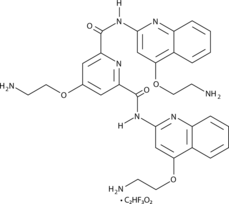
-
GC37043
Pyridostatin hydrochloride
RR82 hydrochloride
El clorhidrato de piridostatina (RR82) es un agente estabilizador del ADN cuÁdruplex G (Kd = 490 nM). El clorhidrato de piridostatina promueve la detenciÓn del crecimiento en células cancerosas humanas al inducir daÑos en el ADN dependientes de la replicaciÓn y la transcripciÓn. El clorhidrato de piridostatina se dirige al protooncogén Src. El clorhidrato de piridostatina redujo los niveles de proteÍna SRC y la motilidad celular dependiente de SRC en células de cÁncer de mama humano.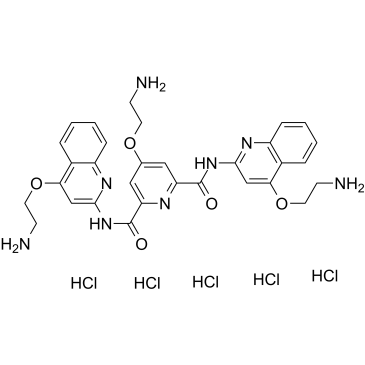
-
GC17801
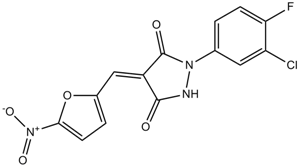
PYZD-4409
PYZD-4409 es un inhibidor especÍfico de la enzima activadora de ubiquitina UBA1 con una IC50 de 20 μM (ensayo enzimÁtico libre de células). PYZD-4409 induce la muerte celular en células malignas e inhibe preferentemente el crecimiento clonogénico de células de leucemia mieloide aguda primaria.
-
GC52057
QN523
An anticancer agent

-
GC45903
Quazinone
Ro 13-6438
A PDE3 inhibitor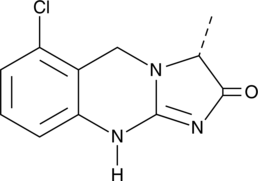
-
GC48019
Quercetin (hydrate)
A flavonoid with diverse biological activities
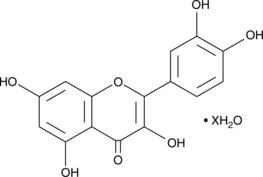
-
GC46210
Quinacrine mustard (hydrochloride)
NSC 3424
La mostaza de quinacrina (clorhidrato) es un fluorocromo.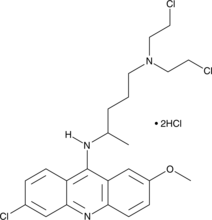
-
GC44800
Quininib
A CysLT1 and CysLT2 receptor antagonist
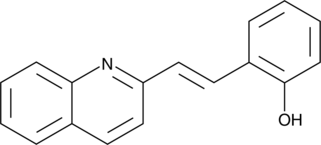
-
GC17400
R547
Ro 4584820
R547 es un inhibidor de CDK potente, selectivo y oralmente activo que compite con ATP, con Kis de 2 nM, 3 nM y 1 nM para CDK1/ciclina B, CDK2/ciclina E y CDK4/ciclina D1, respectivamente.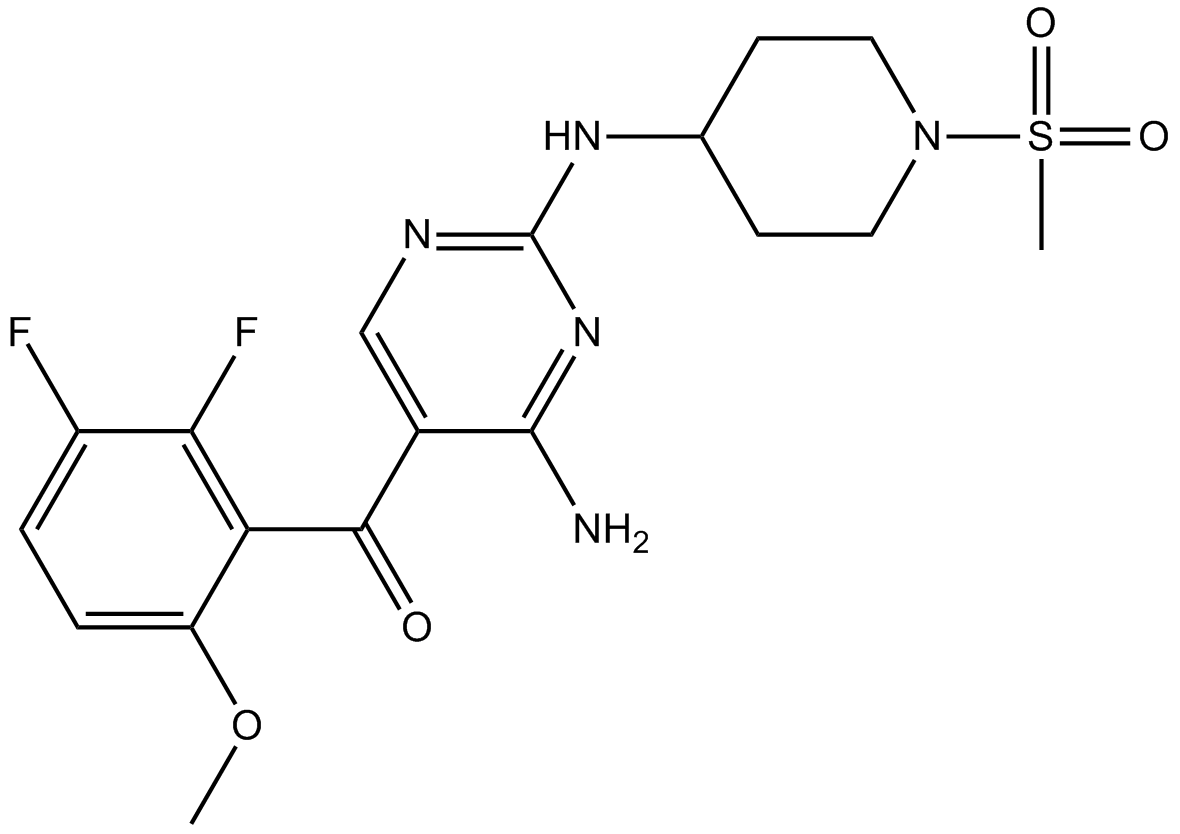
-
GC49771
rac-Hesperetin-d3
(±)-3',5,7-Trihydroxy-4'-methoxyflavanone-d3
An internal standard for the quantification of hesperetin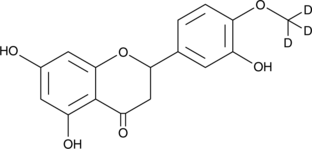
-
GC49108
Racecadotril-d5
Acetorphan-d5
An internal standard for the quantification of racecadotril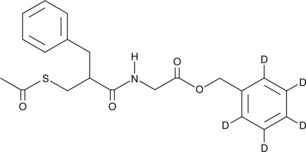
-
GC19306
RAD51 Inhibitor B02
B02
RAD51 Inhibitor B02 (B02) es un inhibidor de RAD51 humano con una IC50 de 27,4 μM.
-
GC63165
RAD51-IN-1
RAD51-IN-1, un derivado de B02, es un potente inhibidor de RAD51. RAD51-IN-1 se puede utilizar para la investigaciÓn del cÁncer.
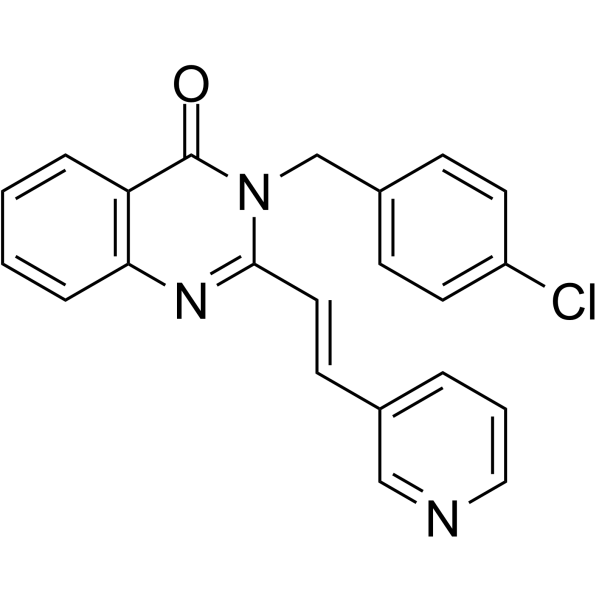
-
GC65383
RAD51-IN-2
RAD51-IN-2 (ejemplo compuesto 67A) es un inhibidor de RAD51 extraÍdo de la patente WO2019/051465A1.
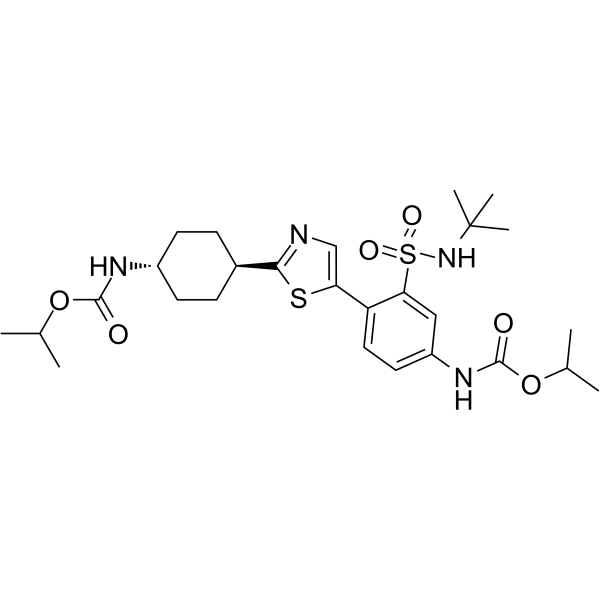
-
GC68416
RAD51-IN-3
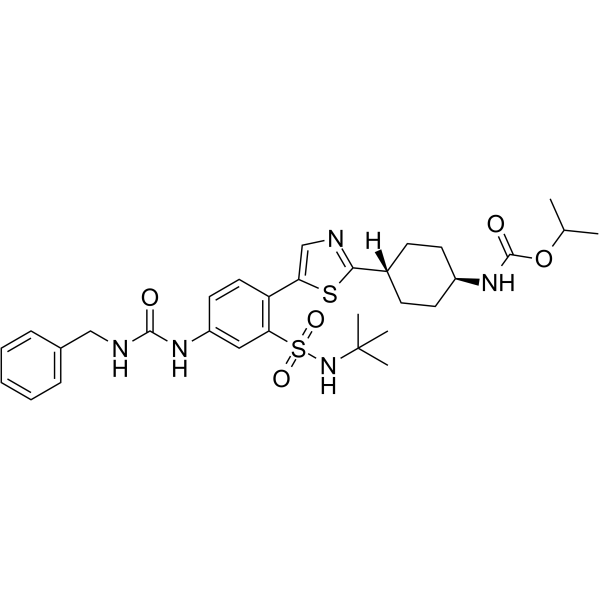
-
GC67888
RAD51-IN-5

-
GC52054
Ranitidine S-oxide
El S-Óxido de ranitidina es el metabolito de la ranitidina.

-
GC52196
RGD Peptide
GRGDNP, HGlyArgGlyAspAsnProOH
El péptido RGD actÚa como un inhibidor de las interacciones integrina-ligando y juega un papel importante en la adhesiÓn, migraciÓn, crecimiento y diferenciaciÓn celular.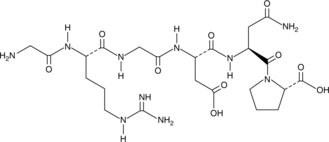
-
GC45798
Rhein-13C4
Rheic Acid-13C4
An internal standard for the quantification of rhein
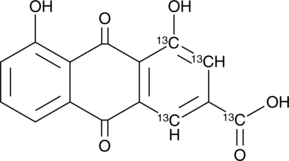
-
GC33310
Rho-Kinase-IN-1
Rho-Kinase-IN-1 es un inhibidor de la Rho quinasa (ROCK) (valores de Ki de 30,5 y 3,9 nM para ROCK1 y ROCK2, respectivamente) extraÍdo del documento US20090325960A1, compuesto 1.008.
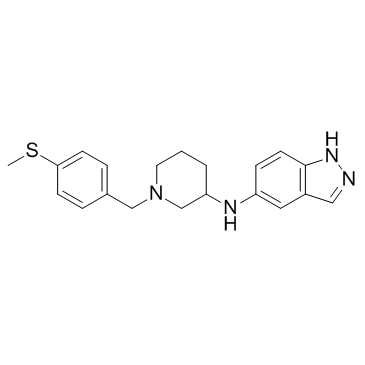
-
GC66050
Rho-Kinase-IN-2
Rho-Kinase-IN-2 (Compuesto 23) es un inhibidor de Rho Kinase (ROCK) activo por vÍa oral, selectivo y penetrante en el sistema nervioso central (SNC) (ROCK2 IC50 = 3 nM). Rho-Kinase-IN-2 se puede utilizar en la investigaciÓn de Huntington'.
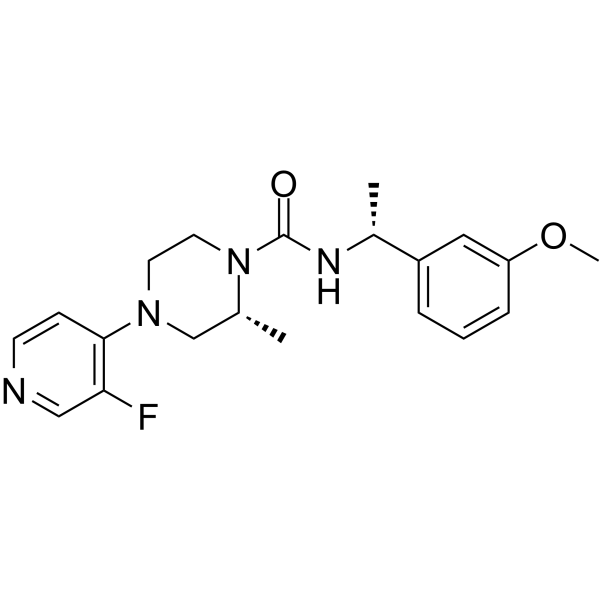
-
GC44829
Rhod-5N (potassium salt)
Rhod-5N is a low affinity fluorescent calcium probe (Kd = 320 μM).
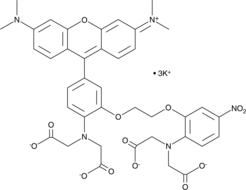
-
GC18140
RI-1
A RAD51 inhibitor

-
GC12752
RI-2
RI-2 es un inhibidor reversible de RAD51, con una IC50 de 44,17 μM, e inhibe especÍficamente la reparaciÓn de recombinaciÓn homÓloga en células humanas.
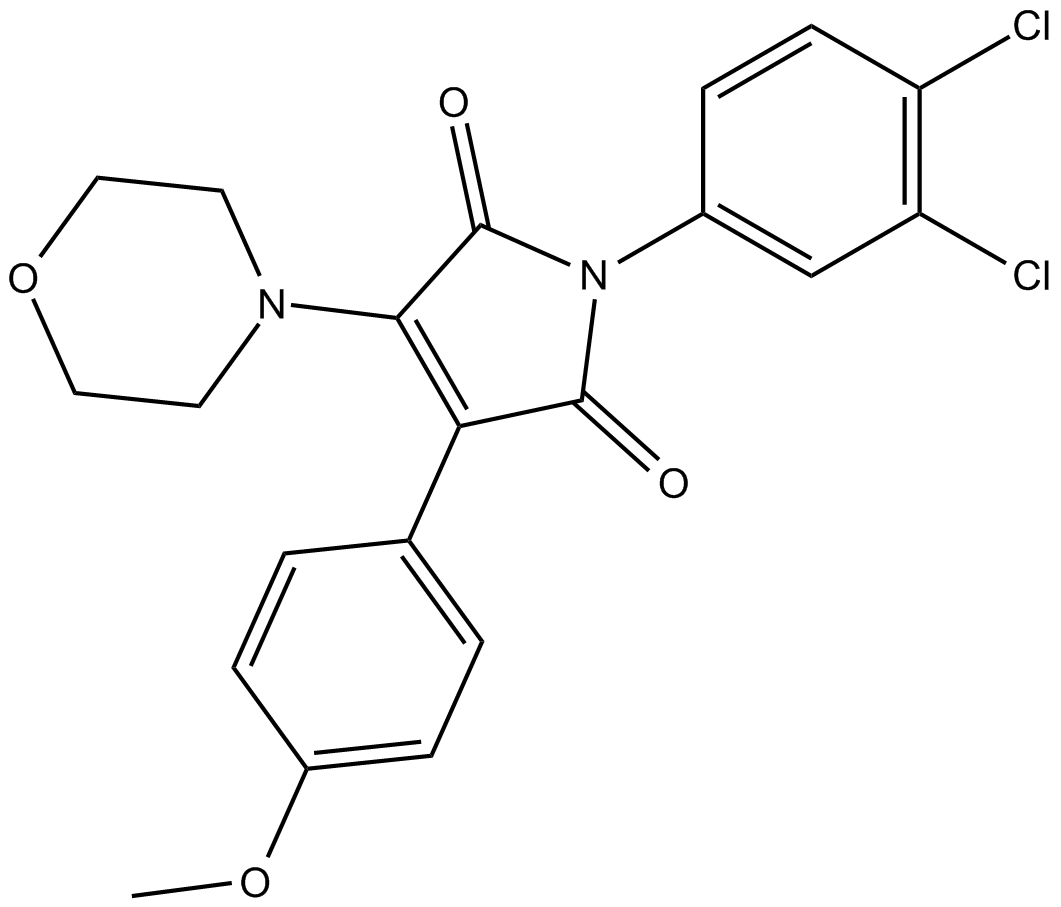
-
GC12415
Rigosertib
Rigosertib (ON-01910) es un inhibidor multicinasa y un agente anticancerÍgeno selectivo que induce la apoptosis mediante la inhibiciÓn de la vÍa PI3 cinasa/Akt, promueve la fosforilaciÓn de la histona H2AX e induce la detenciÓn de G2/M en el ciclo celular. Rigosertib es un inhibidor selectivo y no competitivo de ATP de PLK1 con una IC50 de 9 nM.
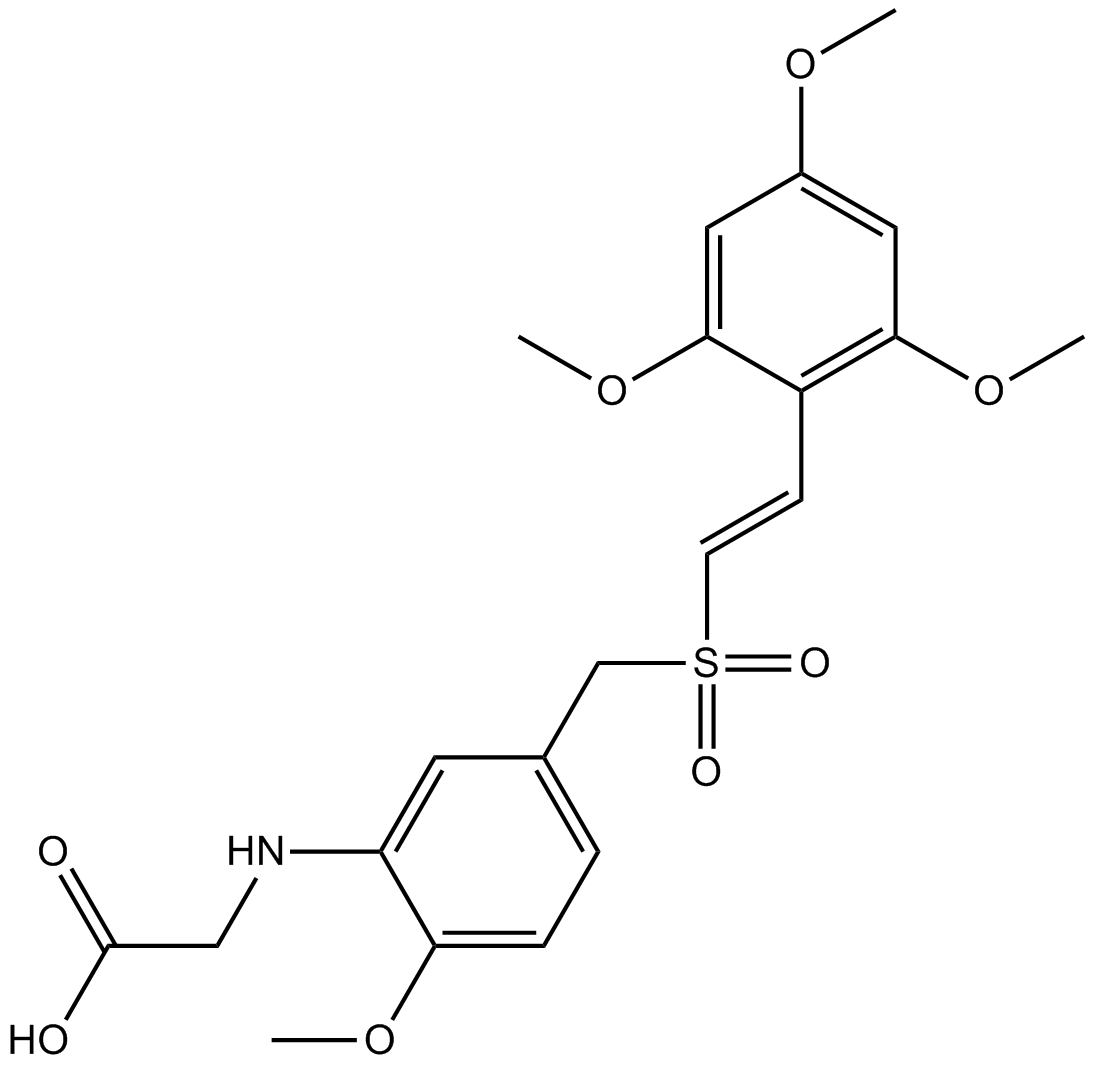
-
GC14358
Rigosertib (ON-01910,Estybon)
Rigosertib
Plk1 inhibitor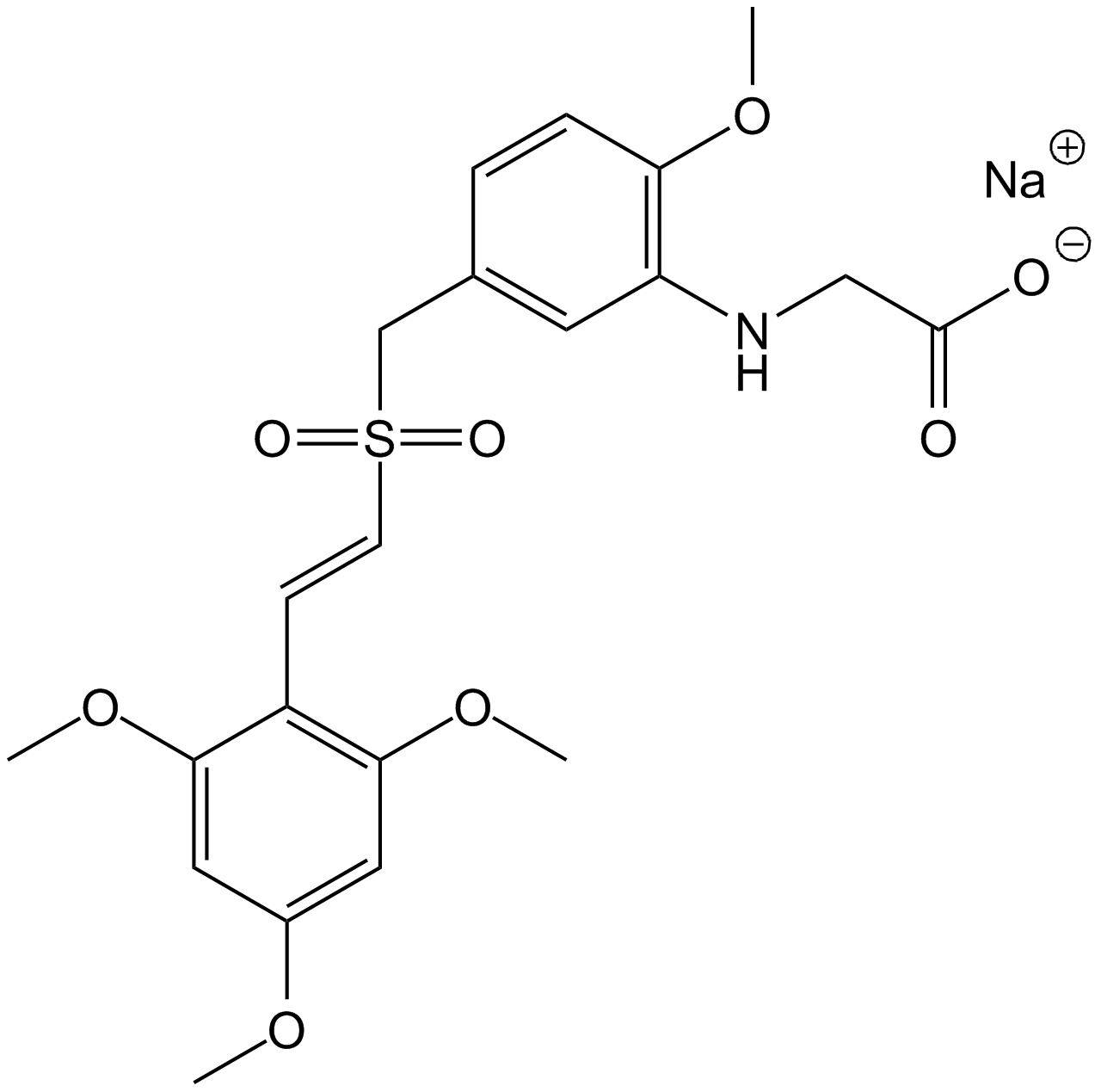
-
GC13580
Rigosertib sodium
El rigosertib sÓdico (ON-01910 sÓdico) es un inhibidor multicinasa y un agente anticancerÍgeno selectivo que induce la apoptosis mediante la inhibiciÓn de la vÍa PI3K/Akt, promueve la fosforilaciÓn de la histona H2AX e induce la detenciÓn de G2/M en el ciclo celular. Rigosertib sÓdico es un inhibidor selectivo y no competitivo de ATP de PLK1 con una IC50 de 9 nM.
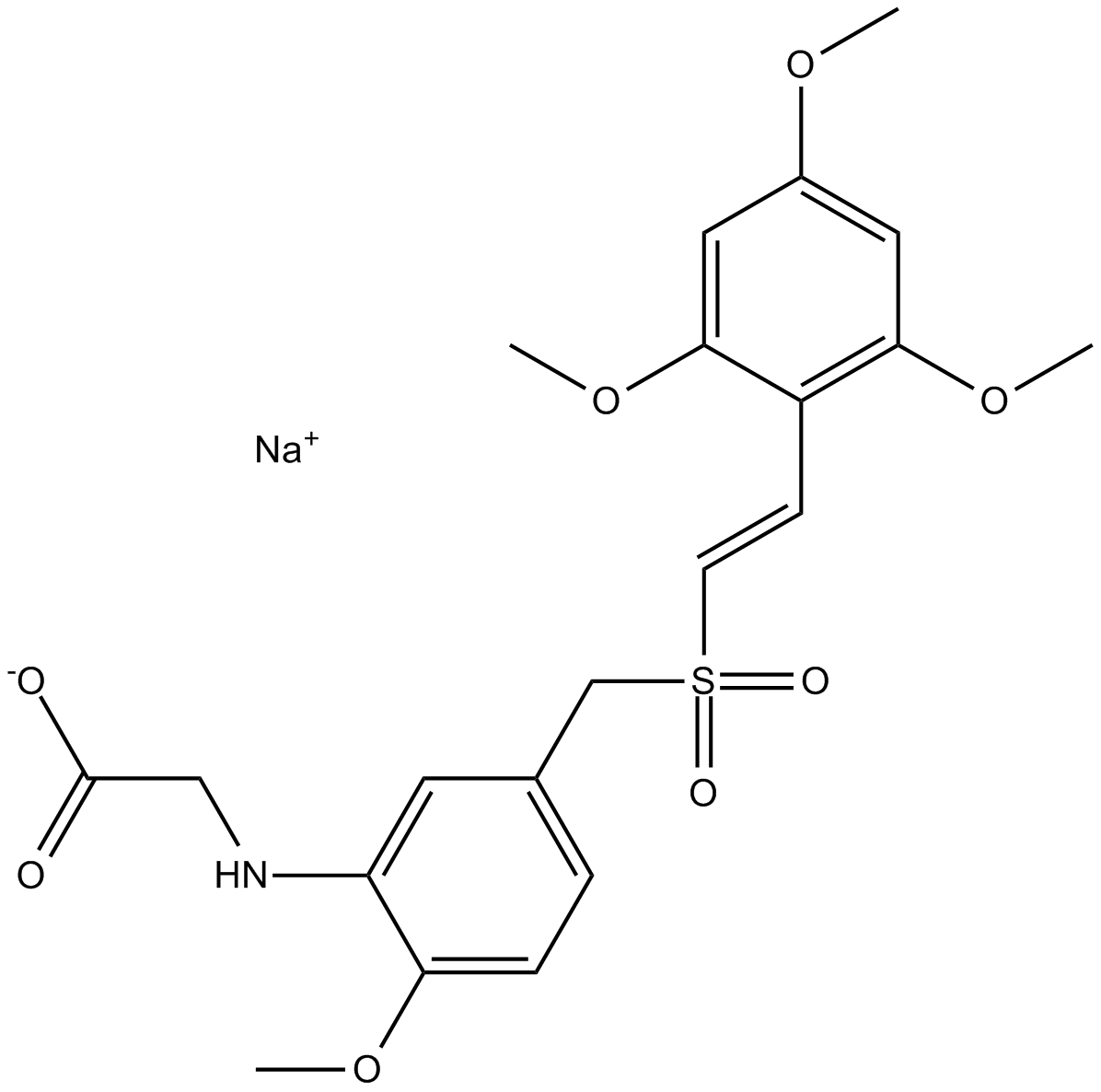
-
GC37534
Ripasudil free base
K-115 (free base)
La base libre de ripasudil (base libre K-115) es un inhibidor especÍfico de ROCK, con IC50 de 19 y 51 nM para ROCK2 y ROCK1, respectivamente.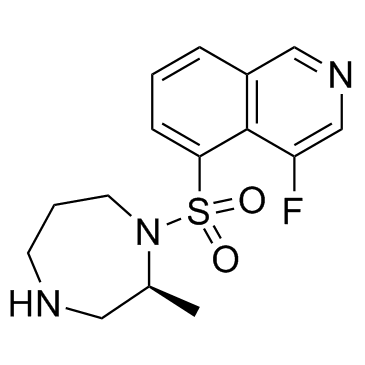
-
GC48401
Risuteganib (trifluoroacetate salt)
ALG-1001
An anti-integrin peptide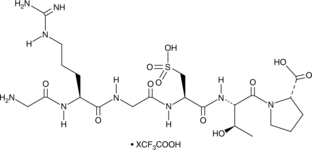
-
GC44846
RK-682 (calcium salt)
CI-010, TAN 1364B
Protein tyrosine phosphatases (PTPs) remove phosphate from tyrosine residues of cellular proteins.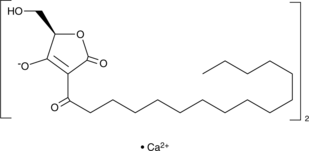
-
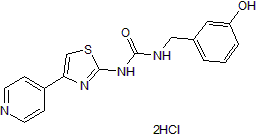
GC50092
RKI 1447 dihydrochloride
El diclorhidrato de RKI 1447 es un inhibidor de ROCK potente y selectivo con IC50 de 14,5 y 6,2 nM para ROCK1 y ROCK2, respectivamente. El diclorhidrato de RKI 1447 suprime el crecimiento de células de carcinoma colorrectal y promueve la apoptosis.
-
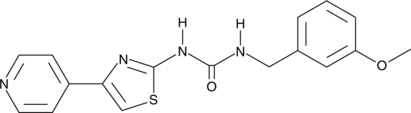
GC44847
RKI-1313
RKI-1313 es un inhibidor de ROCK con IC50 de 34, 8 μM para ROCK 1 y ROCK 2, respectivamente. RKI-1313 muestra poco efecto sobre los niveles de fosforilaciÓn de sustratos ROCK, migraciÓn, invasiÓn o crecimiento independiente del anclaje.
-
GC15437
RKI-1447
RKI 1447;RKI1447
A ROCK1 and ROCK2 inhibitor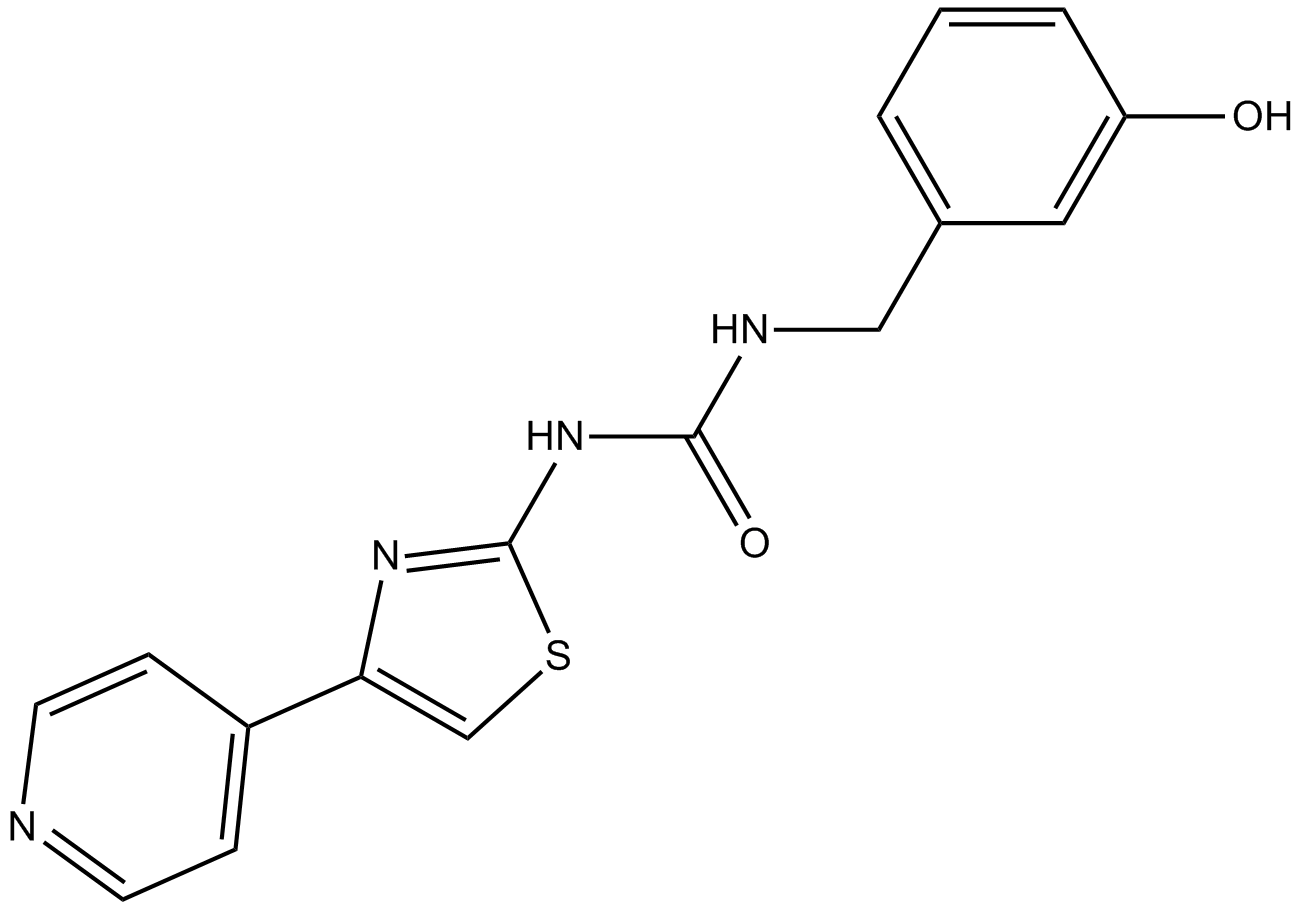
-
GC12348
Ro 3306
Ro 3306 es un inhibidor potente y selectivo de CDK1, con Kis de 20 nM, 35 nM y 340 nM para CDK1, CDK1/ciclina B1 y CDK2/ciclina E, respectivamente.
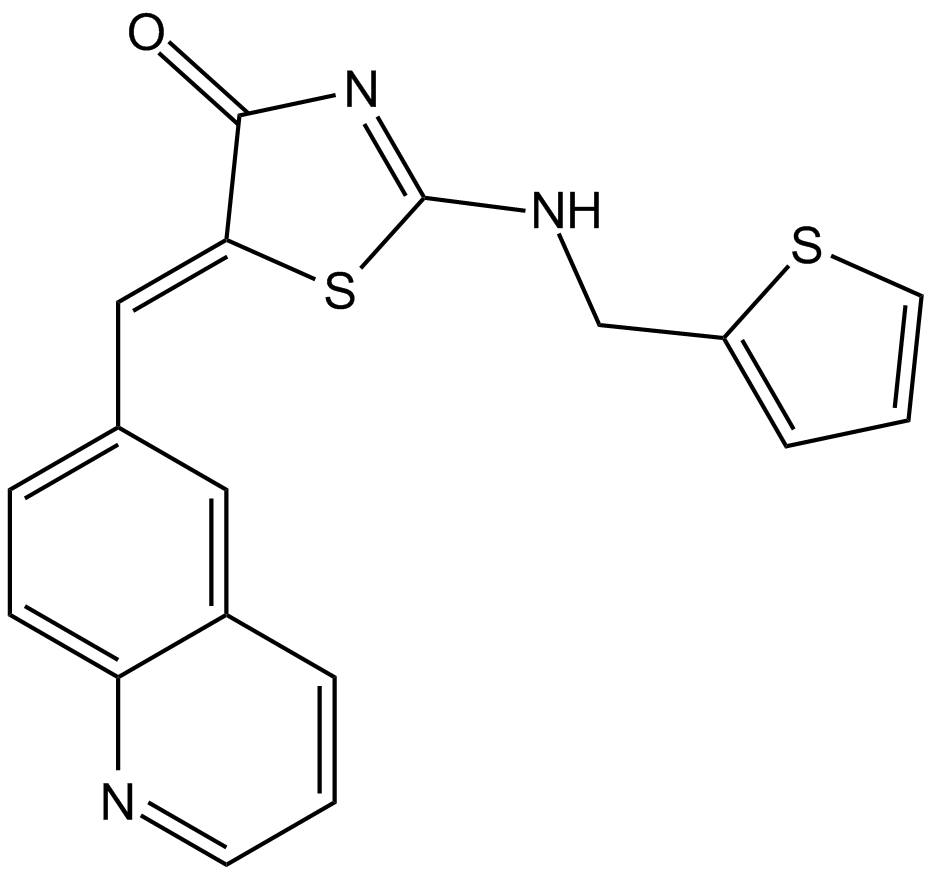
-
GC13903
Ro3280
Ro3280 es un inhibidor potente y altamente selectivo de PLK1 con una IC50 y una Kd de 3 nM y 0,09 nM, respectivamente, y casi no tiene efecto sobre PLK2 y PLK3.
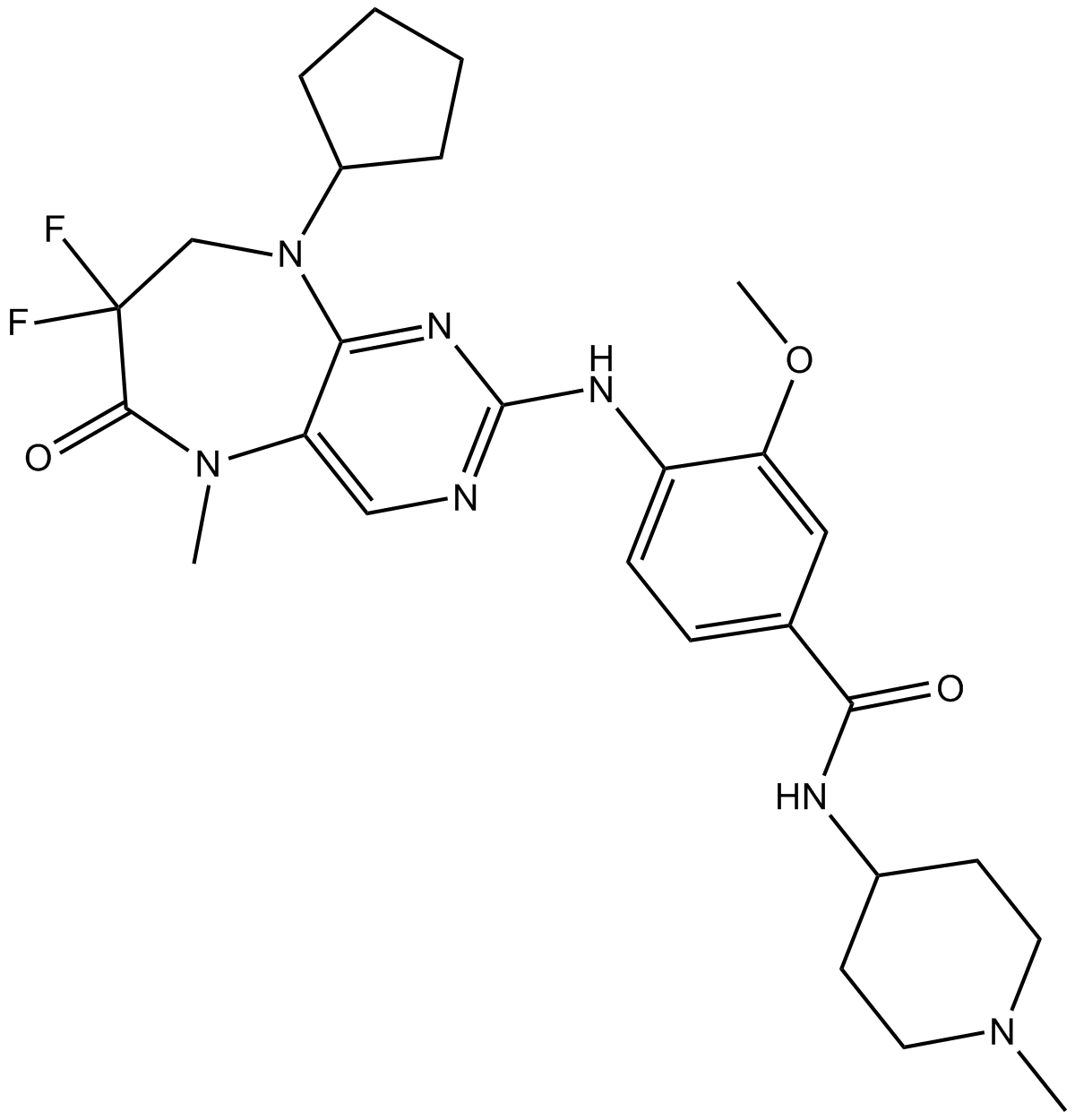
-
GC37551
ROCK inhibitor-2
El inhibidor de ROCK-2 es un inhibidor dual selectivo de ROCK1 y ROCK2 con IC50 de 17 nM y 2 nM, respectivamente.
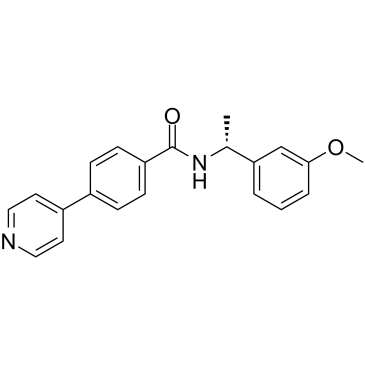
-
GC31952
ROCK-IN-1
ROCK-IN-1 es un potente inhibidor de ROCK, con una IC50 de 1,2 nM para ROCK2.
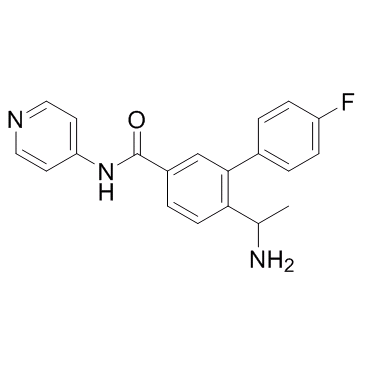
-
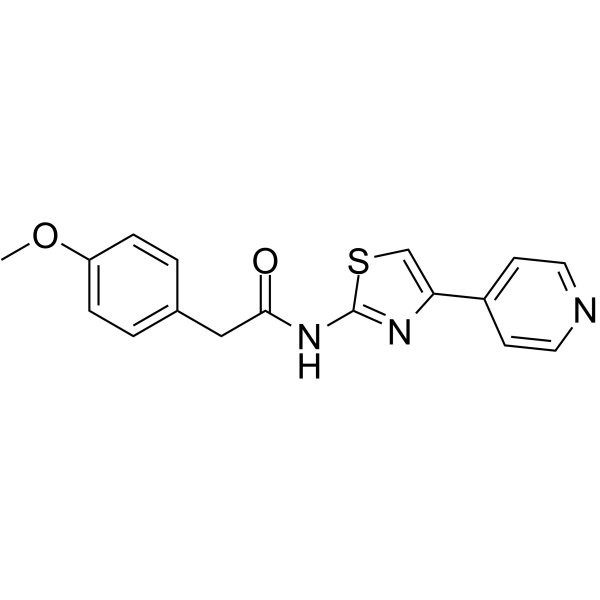
GC66338
ROCK1-IN-1
ROCK1-IN-1 es un inhibidor de ROCK1 con un valor Ki de 540 nM. ROCK1-IN-1 se puede utilizar para la investigaciÓn de la hipertensiÓn, el glaucoma y la disfunciÓn eréctil.
-
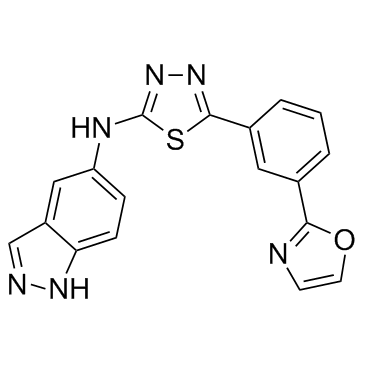
GC31883
ROCK2-IN-2
ROCK2-IN-2 es un inhibidor selectivo de ROCK2 extraÍdo de la patente US20180093978A1, Compound A-30, tiene un IC50 de <1 μM.
-
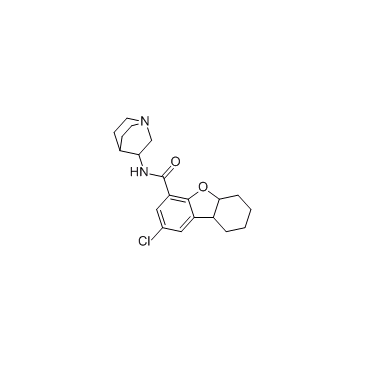
GC33169
Rosabulin (STA 5312)
Rosabulin (STA 5312) (STA 5312) es un inhibidor de microtÚbulos potente y activo por vÍa oral que inhibe el ensamblaje de microtÚbulos. Rosabulin (STA 5312) tiene actividad antitumoral de amplio espectro.
-
GC11401
Roscovitine (Seliciclib,CYC202)
Seliciclib
Roscovitine (Seliciclib,CYC202) (Roscovitine) es un inhibidor de CDK biodisponible por vÍa oral y selectivo con IC50 de 0,2 μM, 0,65 μM y 0,7 μM para CDK5, Cdc2 y CDK2, respectivamente.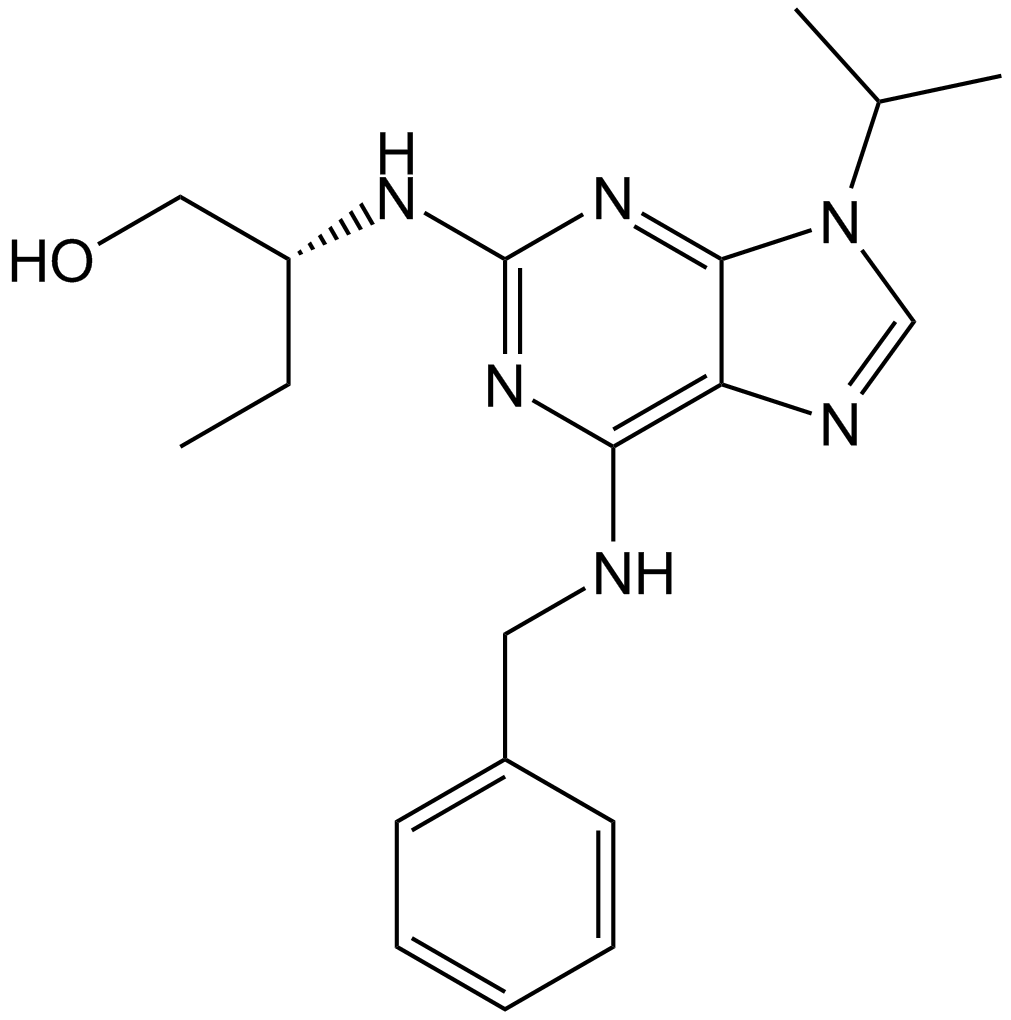
-
GC64278
RP-3500
RP-3500; ATR inhibitor 4
RP-3500 (inhibidor ATR 4) es un inhibidor selectivo de la cinasa ATR (ATRi) activo por vÍa oral con una IC50 de 1,00 nM en ensayos bioquÍmicos. RP-3500 muestra una selectividad de 30 veces para ATR sobre mTOR (IC50 = 120 nM) y una selectividad de >2000 veces para ATM, DNA-PK y PI3Kα quinasas. RP-3500 tiene una potente actividad antitumoral.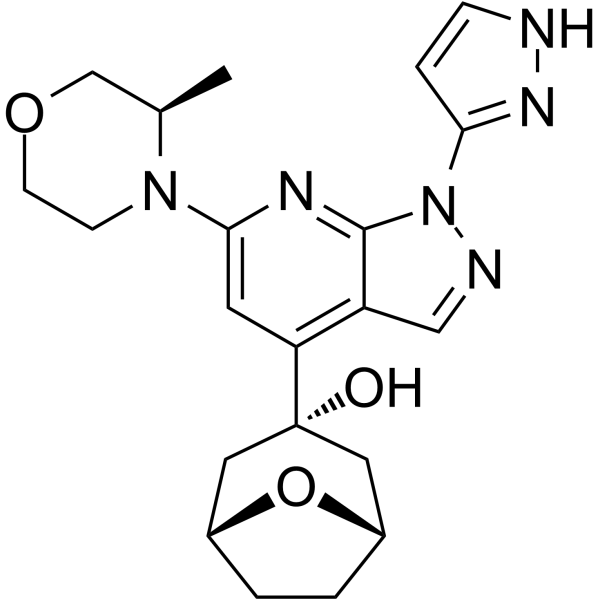
-
GC16265
RS-1
RS-1 es un activador RAD51 y también aumenta las eficiencias de activaciÓn mediadas por CRISPR/Cas9.
-
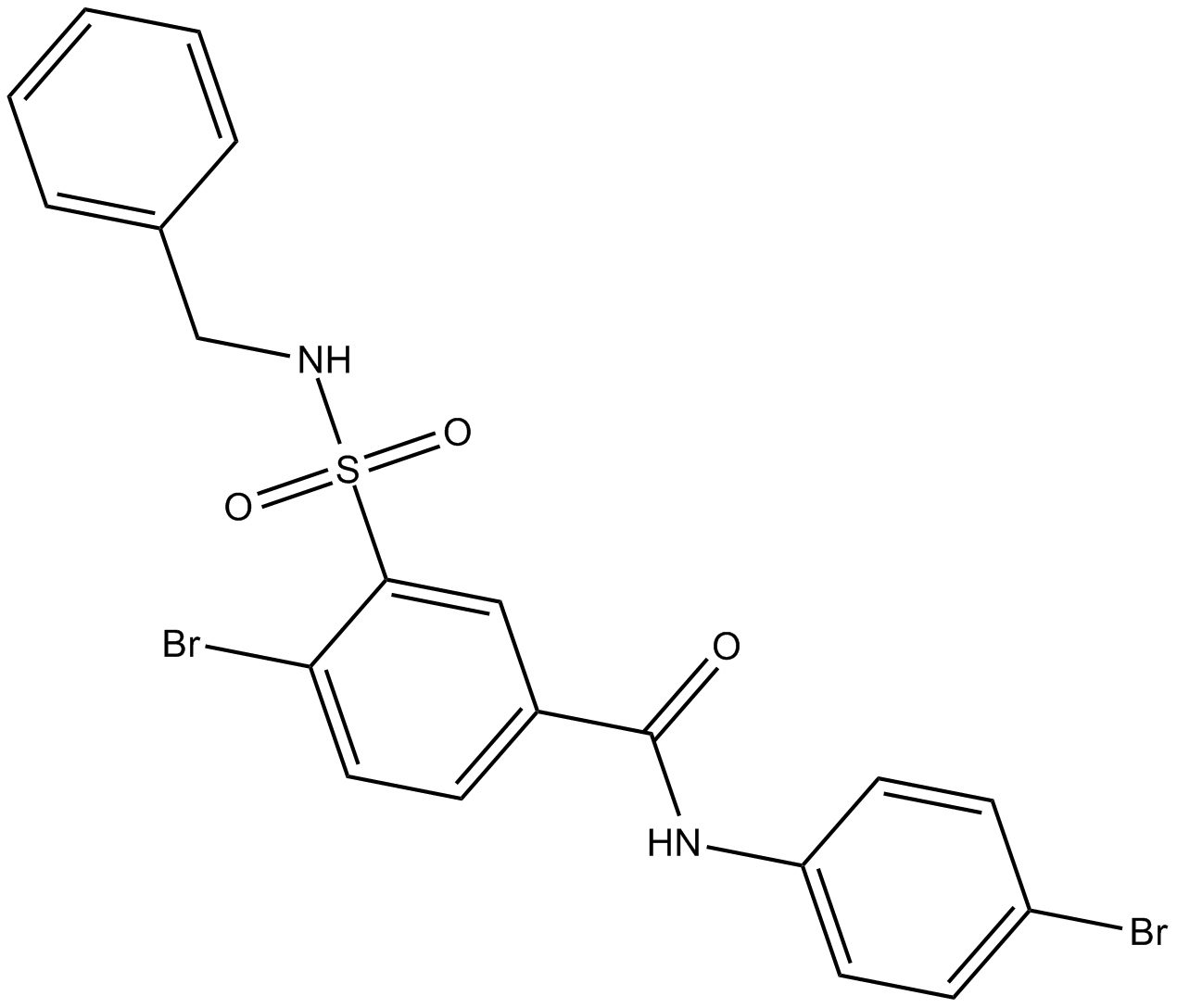
GC18847
RSM-932A
TCD-717
RSM-932ª es un inhibidor ChoKα.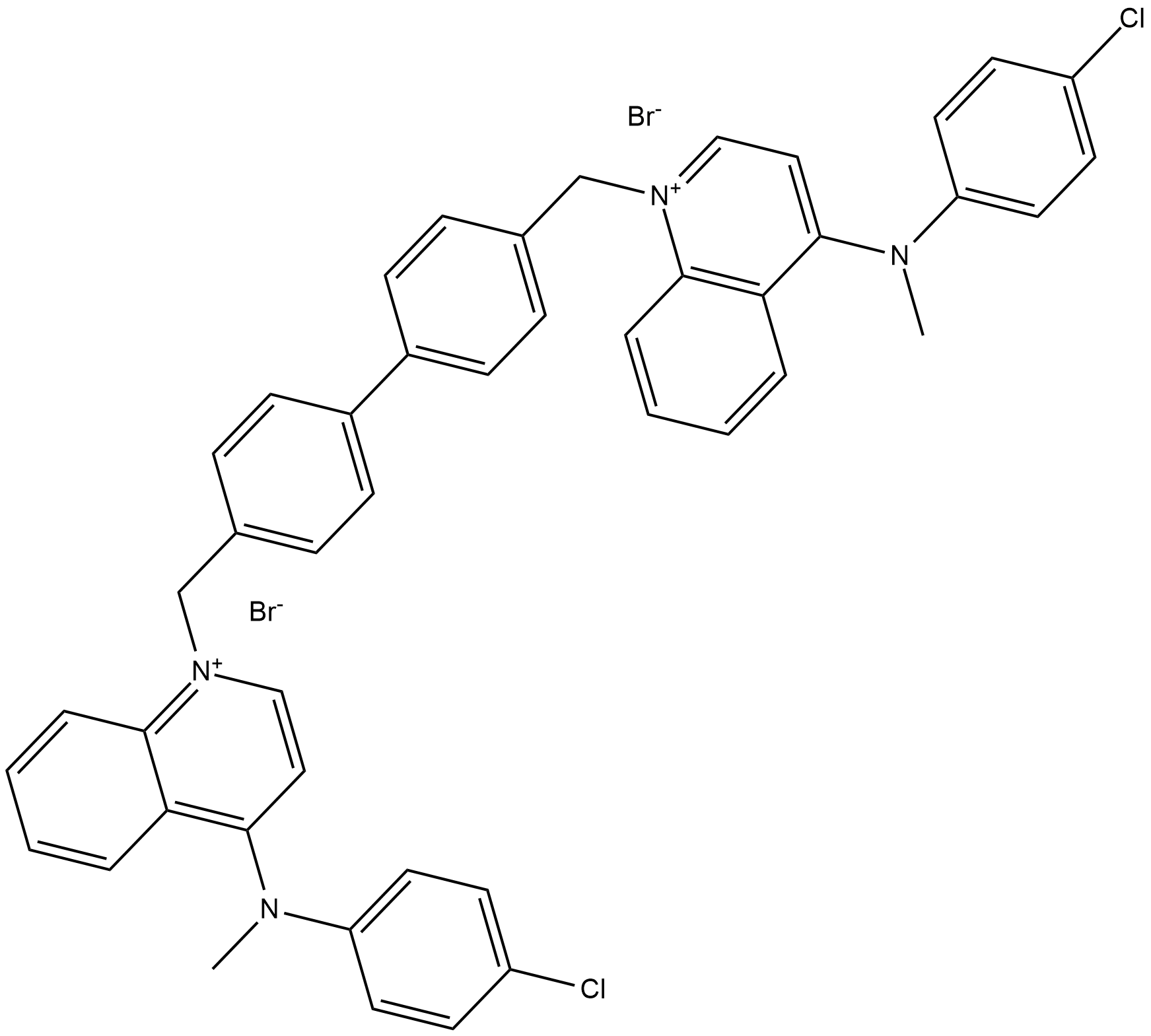
-
GC17150
Ryuvidine
Cdk4 Inhibitor III, Cyclic-dependent Kinase 4 Inhibitor III, SPS812
La ryuvidine es un potente inhibidor de la proteÍna 8 que contiene el dominio SET (SETD8) con una IC50 de 0,5 μM y suprime la monometilaciÓn de H4K20 in vitro. La ryuvidine también inhibe CDK4 con un IC50 de 6,0 μM y es citotÓxica contra una variedad de células cancerosas humanas.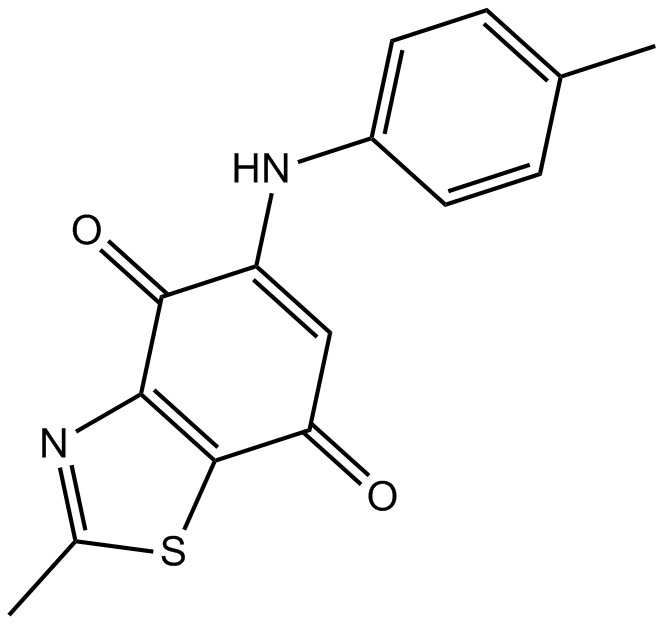
-
GC49532
S-(1,2-Dichlorovinyl)-Cysteine (hydrochloride)
S-(1,2-dichlorovinyl)-L-Cysteine, S-(trans-1,2-dichlorovinyl)-L-Cysteine, DCVC
A nephrotoxin and metabolite of trichloroethylene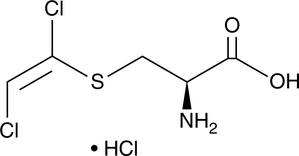
-
GC46224
S-Phenylcysteine
3-(phenylthio)-L-Alanine, (2R)-2-Amino-3-phenylsulfanylpropanoic acid
An adduct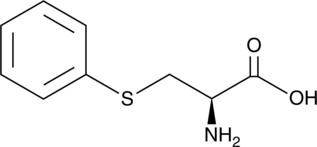
-
GC18852
S14161
D-Cyclins regulate the cell cycle by acting in a complex with cyclin dependent kinases (CDKs) to promote phosphorylation of the retinoblastoma protein and initiate cellular progression from the G1 to the S phase.
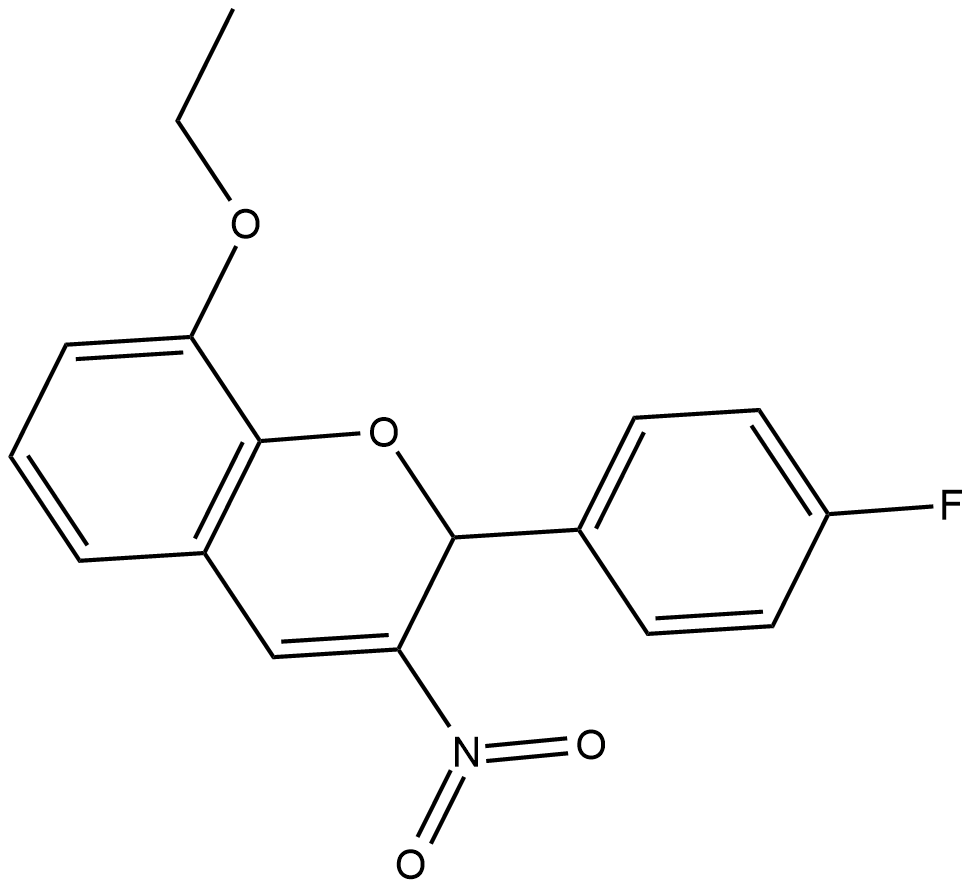
-
GC38945
S516
S516 (Compuesto 22) es un metabolito activo de CKD-516 y un potente inhibidor de la polimerizaciÓn de tubulina con una IC50 de 4,29 μM. S516 tiene marcada actividad antitumoral.
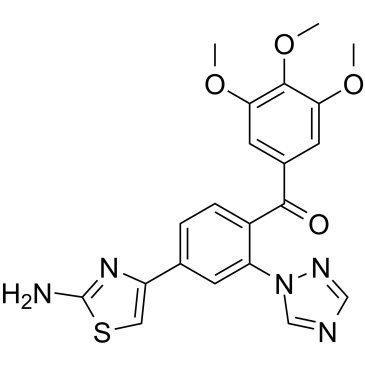
-
GC49093
Safflower Red
Carthamin Yellow
A red pigment with diverse biological activities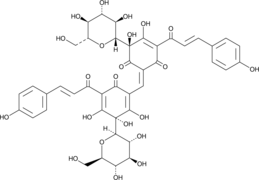
-
GC38845
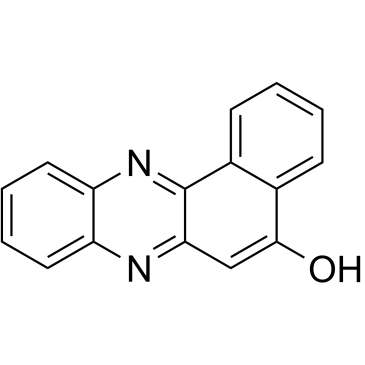
sAJM589
sAJM589 es un inhibidor de Myc que interrumpe potentemente el heterodÍmero Myc-Max con una IC50 de 1,8 μM.
-
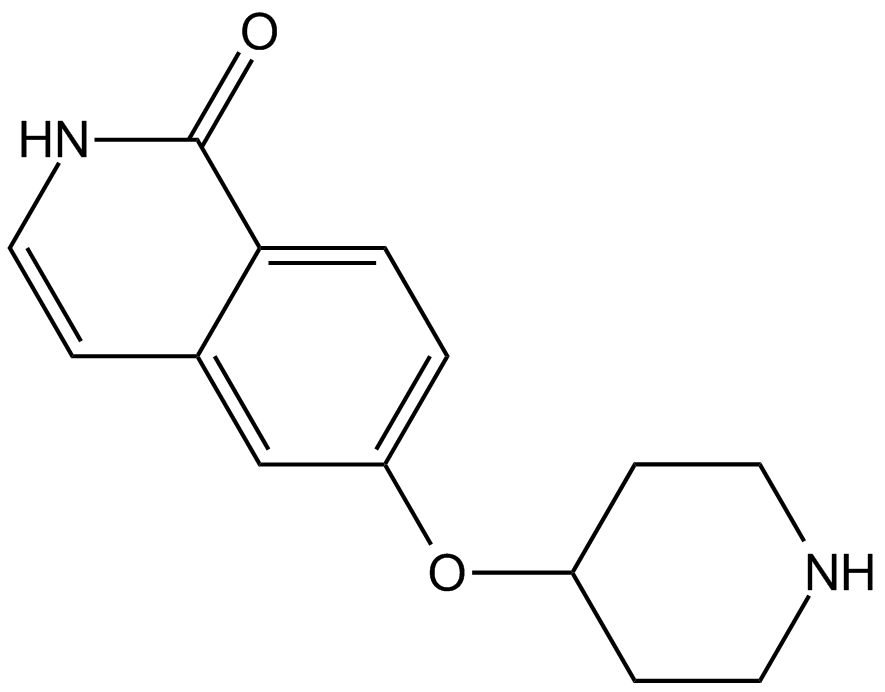
GC13759
SAR407899
SAR407899 es un inhibidor de ROCK selectivo, potente y competitivo con ATP, con una IC50 de 135 nM para ROCK-2 y Kis de 36 nM y 41 nM para ROCK-2 humano y de rata, respectivamente.
-
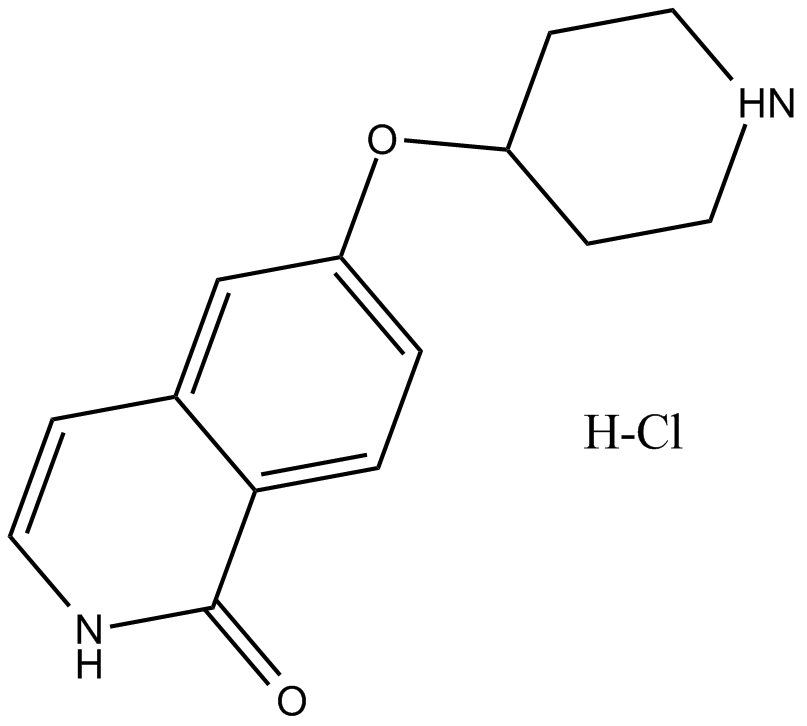
GC16289
SAR407899 hydrochloride
El clorhidrato de SAR407899 es un inhibidor de ROCK selectivo, potente y competitivo con ATP, con una IC50 de 135 nM para ROCK-2 y Kis de 36 nM y 41 nM para ROCK-2 humano y de rata, respectivamente.
-
GC46218
Satratoxin G
A macrocyclic trichothecene mycotoxin
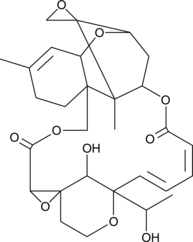
-
GC46219
Satratoxin H
A trichothecene mycotoxin
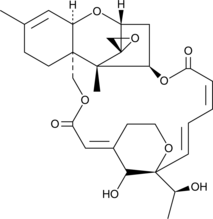
-
GC11022
SB 218078
SB 218078 es un inhibidor potente, selectivo, ATP-competitivo y permeable a las células del punto de control quinasa 1 (Chk1) que inhibe la fosforilación de Chk1 de cdc25C con una IC50 de 15 nM. SB 218078 inhibe menos potentemente Cdc2 (IC50 de 250 nM) y PKC (IC50 de 1000 nM). SB 218078 causa apoptosis por daño en el ADN y detención del ciclo celular.
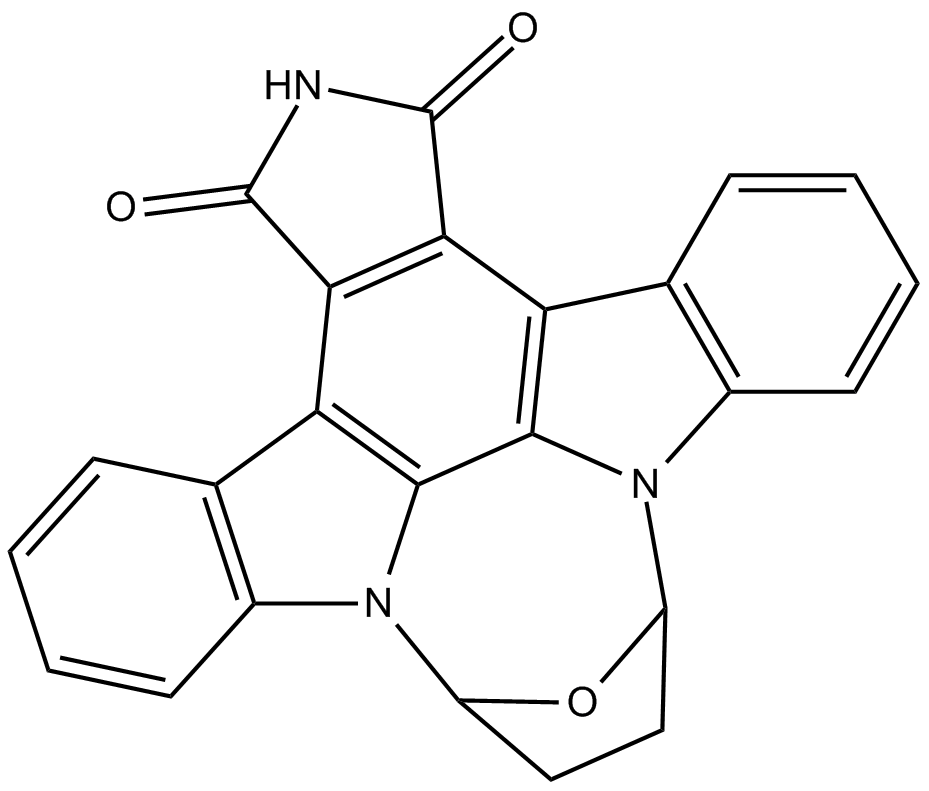
-
GC15170
SB 772077B dihydrochloride
El diclorhidrato de SB 772077B es un inhibidor de la quinasa Rho (ROCK) basado en aminofurazan con IC50 de 5,6 nM y 6 nM hacia ROCK1 y ROCK2, respectivamente.
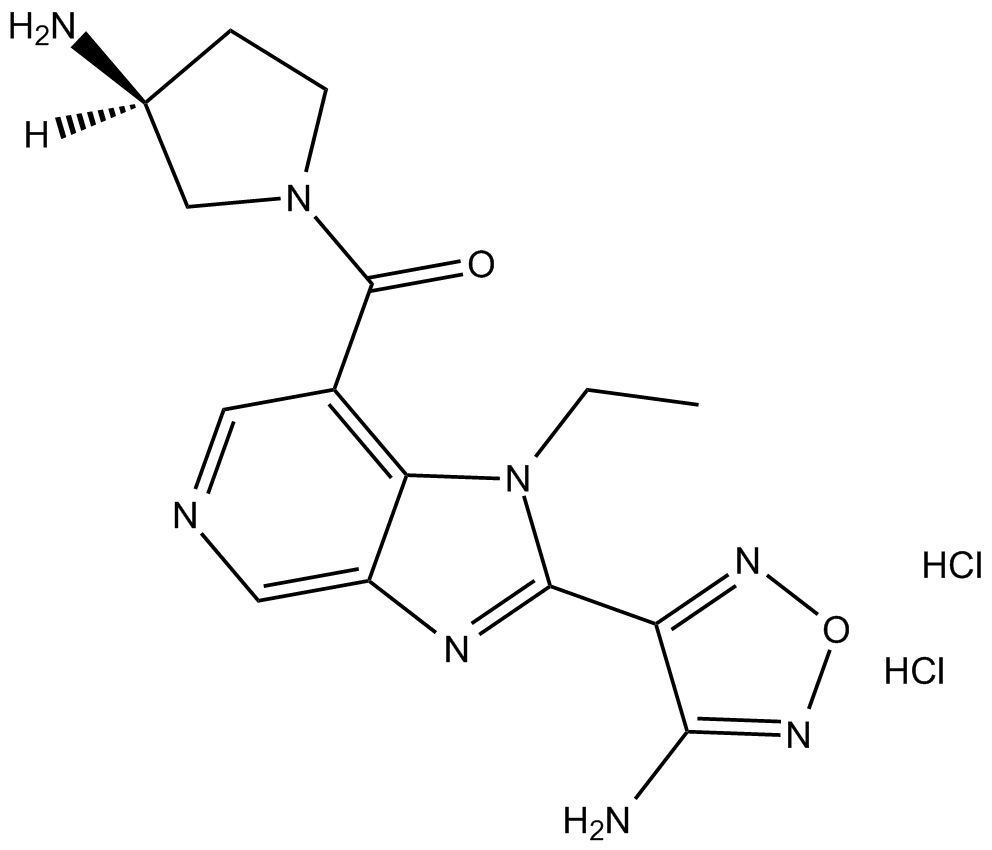
-
GC66437
SB-216
SB-216 es un potente inhibidor de la polimerizaciÓn de tubulina. SB-216 muestra una fuerte potencia antiproliferativa en un panel de lÍneas celulares de cÁncer humano, incluido el melanoma, el cÁncer de pulmÓn y el cÁncer de mama. SB-216 se puede utilizar para la investigaciÓn del cÁncer.
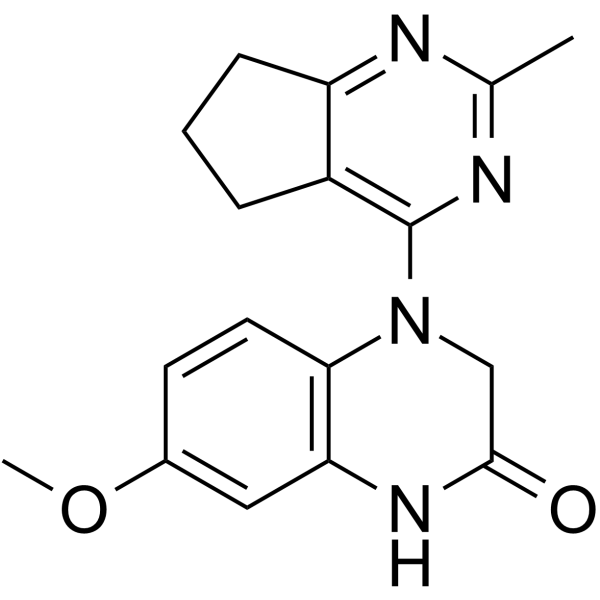
-
GC12064
SB1317
TG02, Zotiraciclib
A multi-kinase inhibitor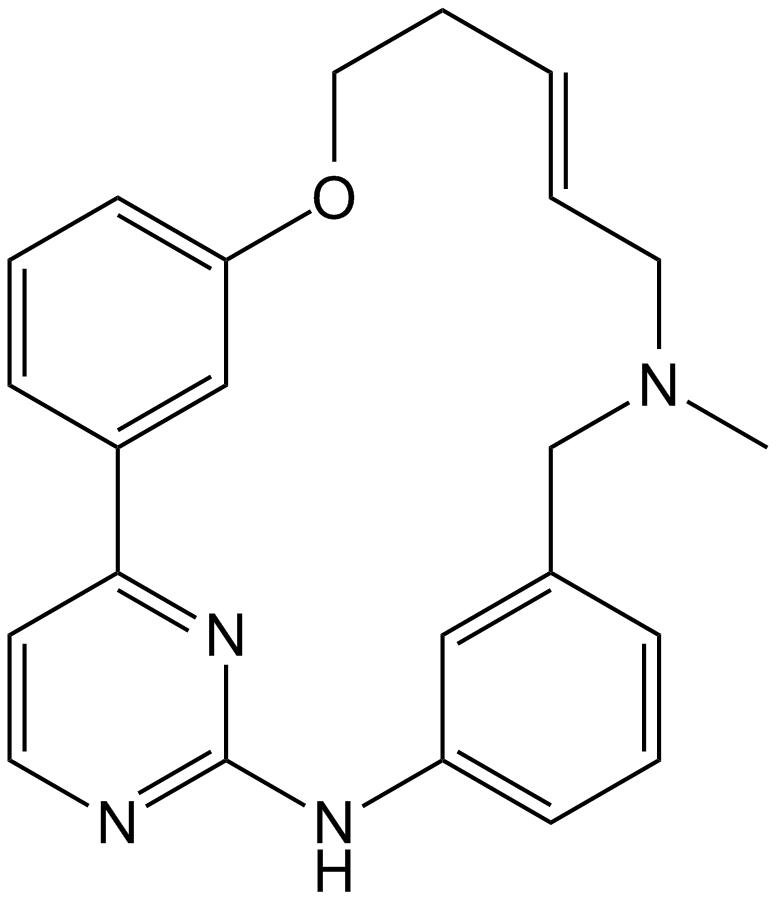
-
GC14600
SB743921
SB743921 es un potente inhibidor de la kinesina mitÓtica KSP (Eg5), con una Ki de 0,1 nM.
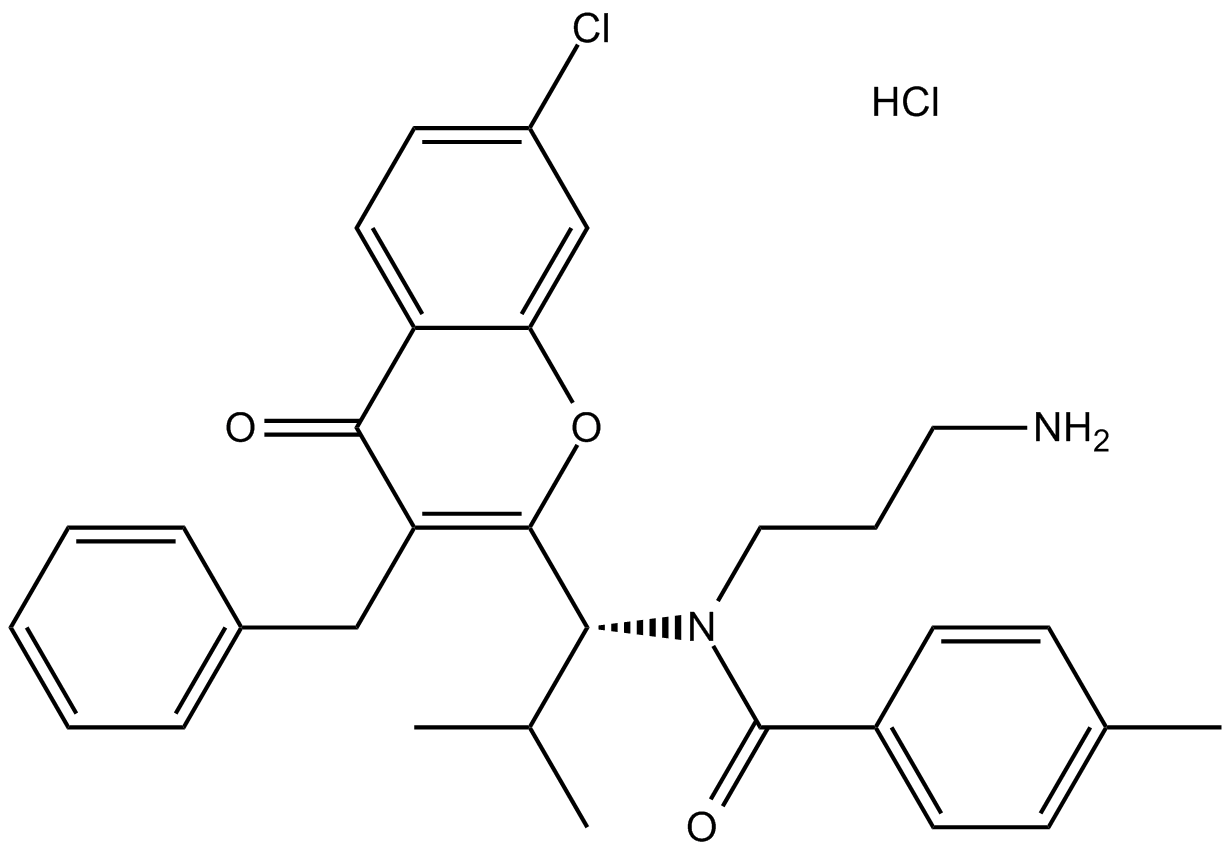
-
GC14644
SBE 13 HCl
SBE 13 HCl es un inhibidor de Plk1 potente y selectivo, con una IC50 de 200 pM; SBE 13 HCl inhibe pobremente Plk2 (IC50>66μ M) o Plk3 (IC50 = 875 nM).
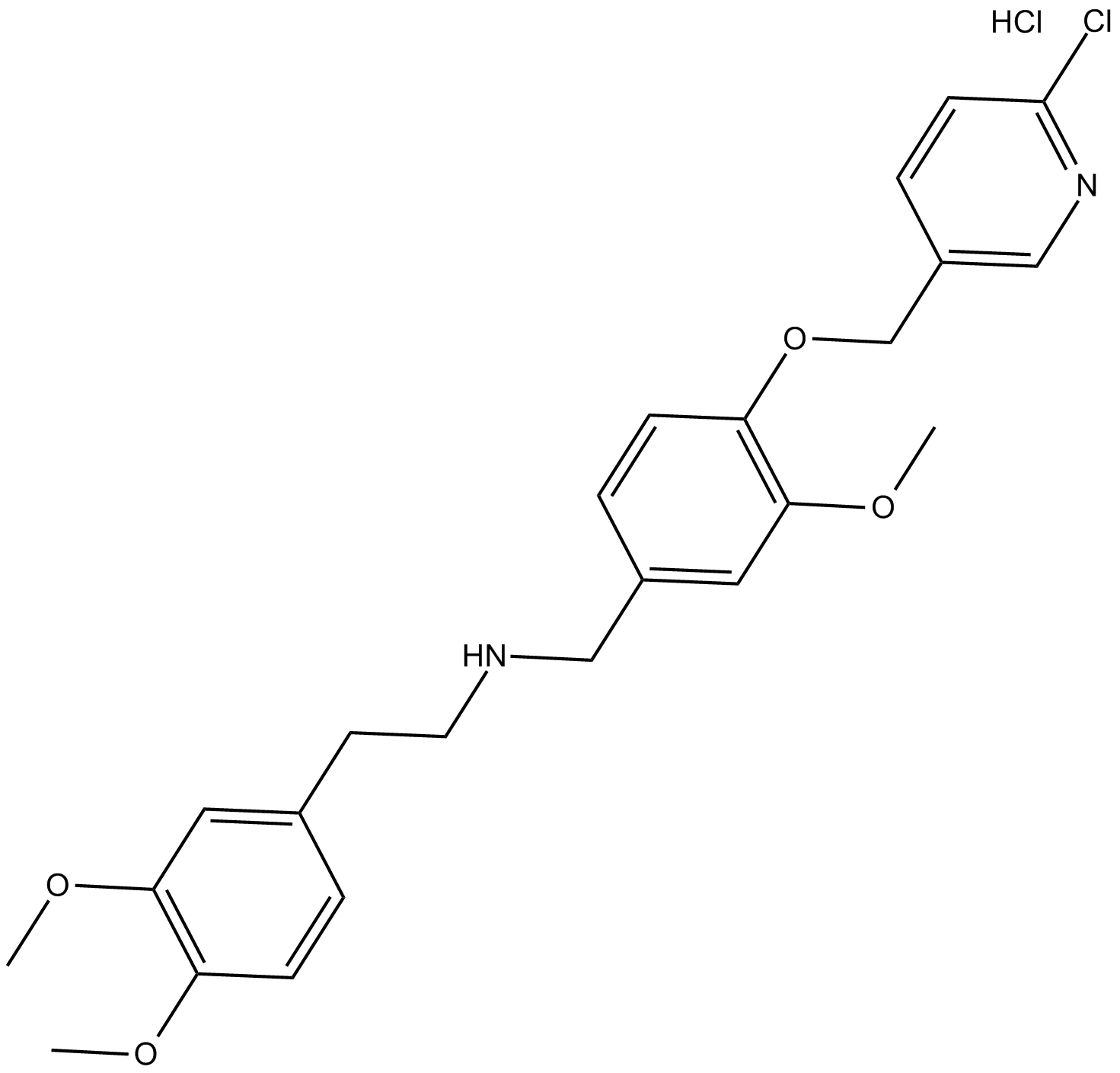
-
GC37601
SBE13
SBE13 es un inhibidor de Plk1 potente y selectivo, con una IC50 de 200 pM; SBE13 inhibe pobremente Plk2 (IC50>66μM) o Plk3 (IC50=875nM).
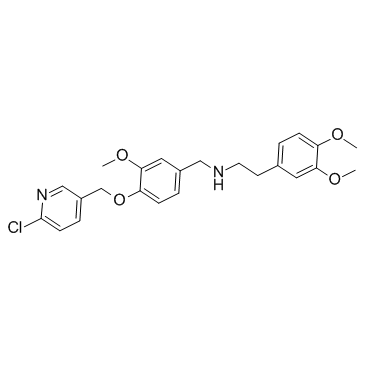
-
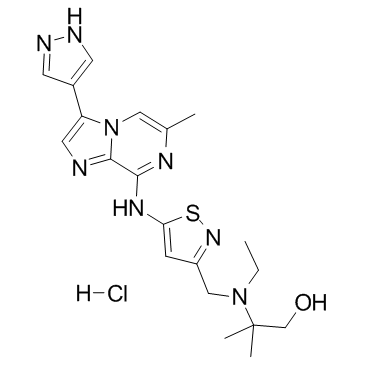
GC37606
SCH-1473759 hydrochloride
El clorhidrato de SCH-1473759 es un inhibidor de aurora con IC50 de 4 y 13 nM para aurora A y B, respectivamente.
-
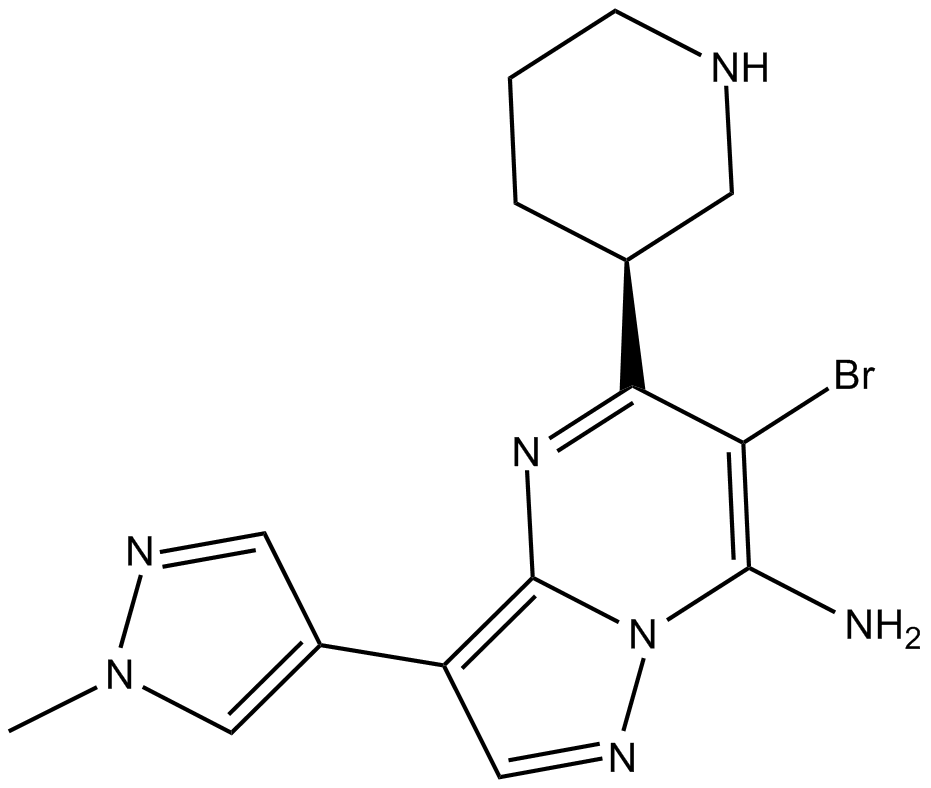
GC17792
SCH900776 S-isomer
SCH 900776 S-isomer;SCH-900776 S-isomer
-
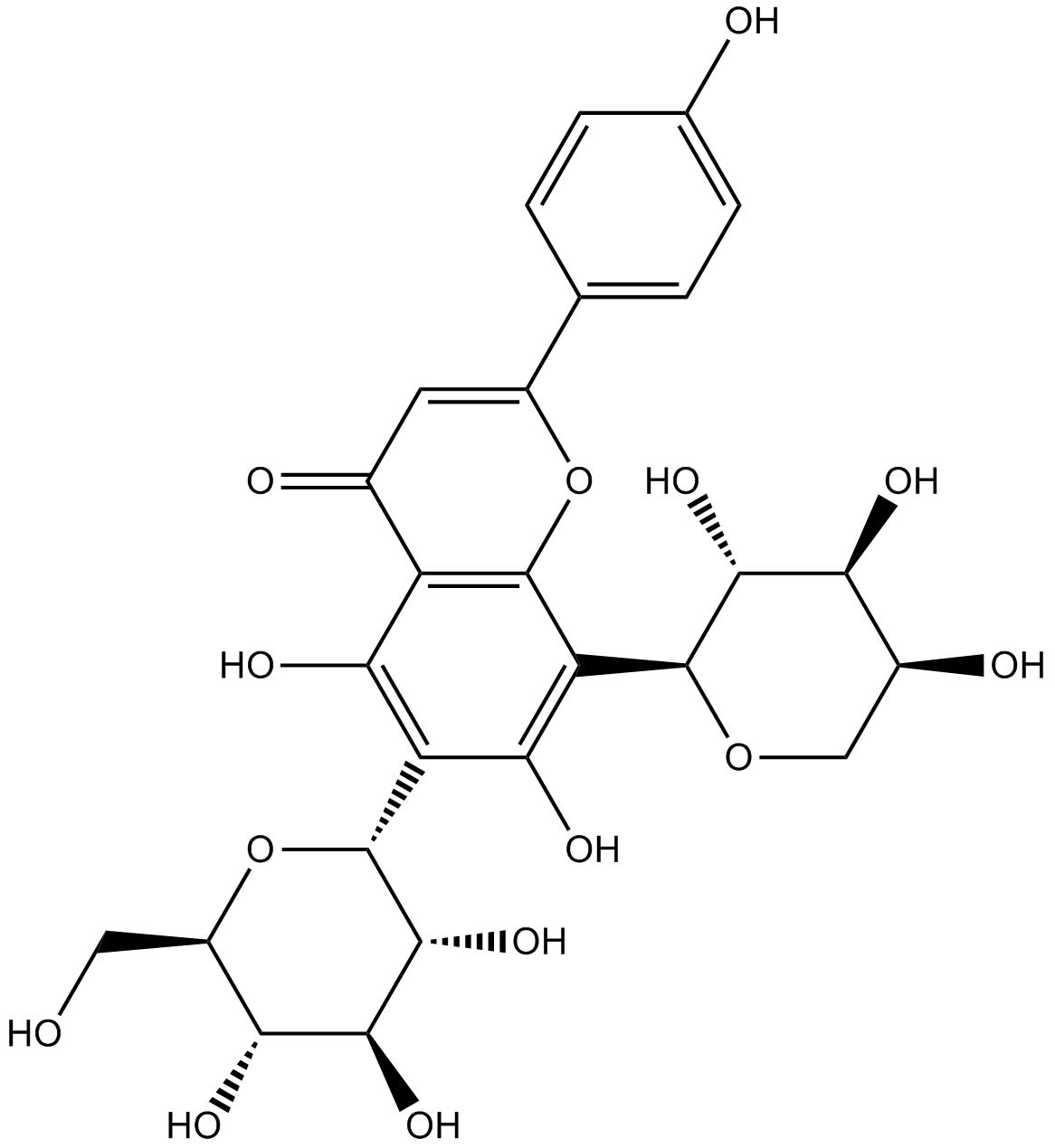
GN10091
Schaftoside
Apigenin 6-C-glucoside-8-C-arabinoside
-
GC49674
Schizandrin
(+)-Schizandrin, Schizandrol A
La esquizandrina (Schizandrin), un lignano dibenzociclooctadieno, se aÍsla del fruto de Schisandra chinensis Baill.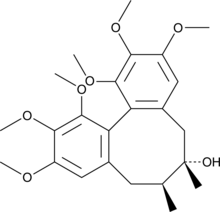
-
GC39798
Scoulerine
(–)-Scoulerine, l-Scoulerine, (S)-Scoulerine
Scoulerine ((-)-Scoulerine), un alcaloide de isoquinolina, es un potente compuesto antimitÓtico. Scoulerine también es un inhibidor de BACE1 (enzima 1 que escinde la proteÍna precursora de amiloide del sitio ß). Scoulerine inhibe la proliferaciÓn, detiene el ciclo celular e induce la apoptosis en las células cancerosas.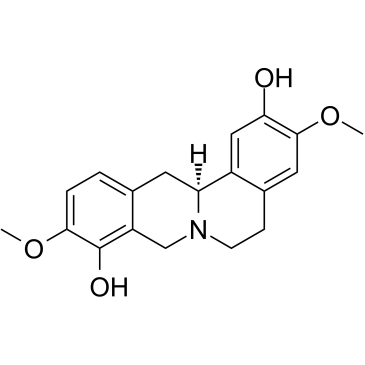
-
GC49723
SenTraGor™ Cell Senescence Reagent
GL13, SBB-A-B, SBB-Analogue (GL13) Biotin
A cellular senescence detection reagent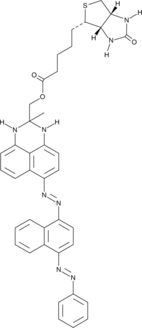
-
GC48076
Sesquicillin A
Sesquicillin
A fungal metabolite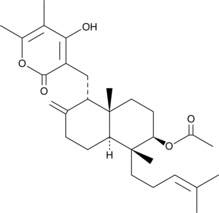
-
GC10019
SF1670
SF1670 es un potente y especÍfico inhibidor del homÓlogo de la fosfatasa y la tensina eliminado en el cromosoma 10 (PTEN).
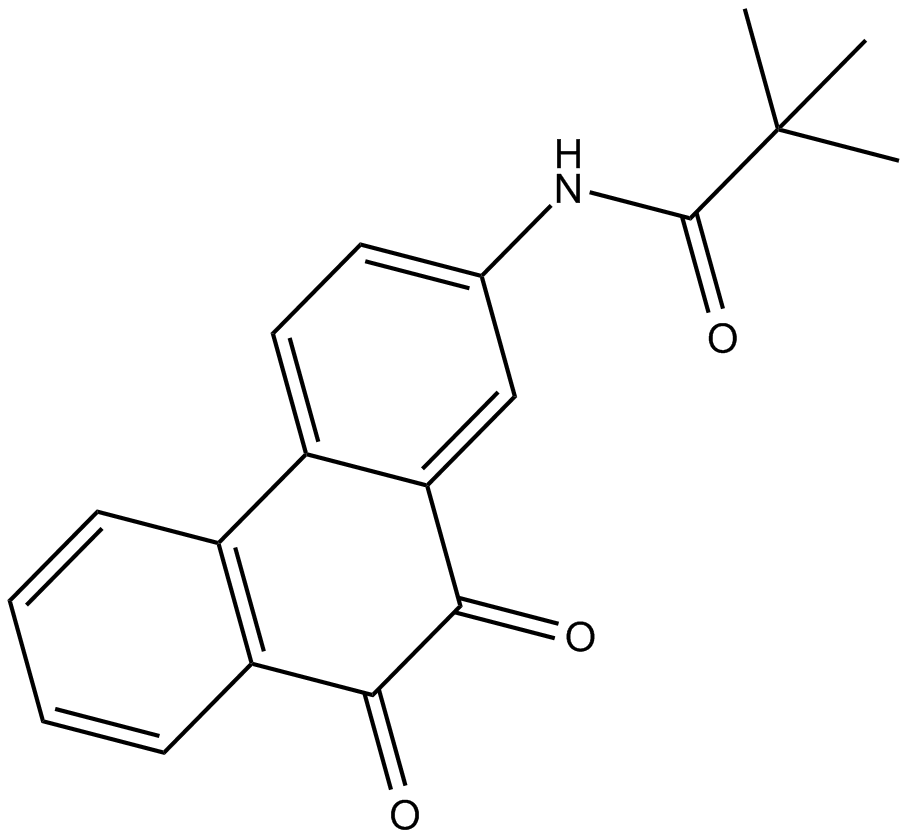
-
GC49713
SIKVAV (acetate)
Hexapeptide-10, Ser-Ile-Lys-Val-Ala-Val
A laminin α1-derived peptide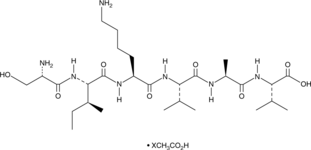
-
GC49002
Sinigrin (hydrate)
Sinigrin (hidrato) es un glucosinolato alifático natural presente en plantas de la familia Brassicaceae.
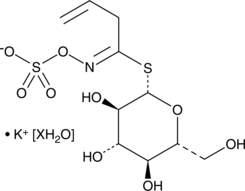
-
GC64539
SKLB-197
SKLB-197 mostrÓ un valor IC50 de 0,013 μM frente a ATR pero actividad muy débil o nula frente a otras proteÍnas quinasas 402. MostrÓ una potente actividad antitumoral contra tumores deficientes en ATM tanto in vitro como in vivo.
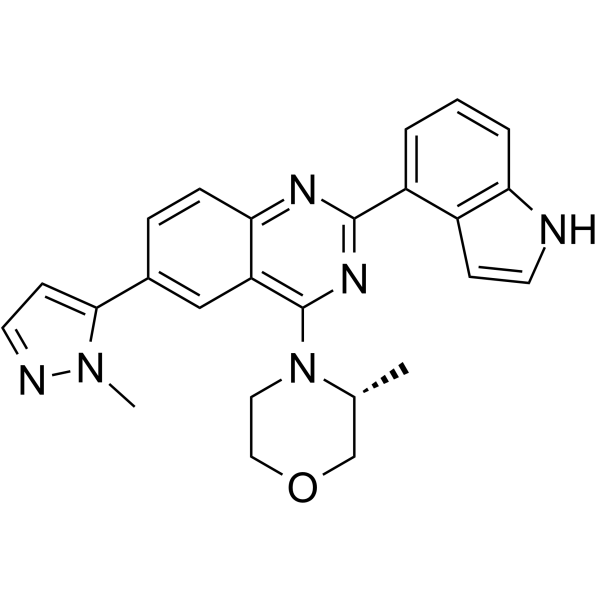
-
GC12827
SLx-2119
Belumosudil, SLx-2119
A ROCK2 inhibitor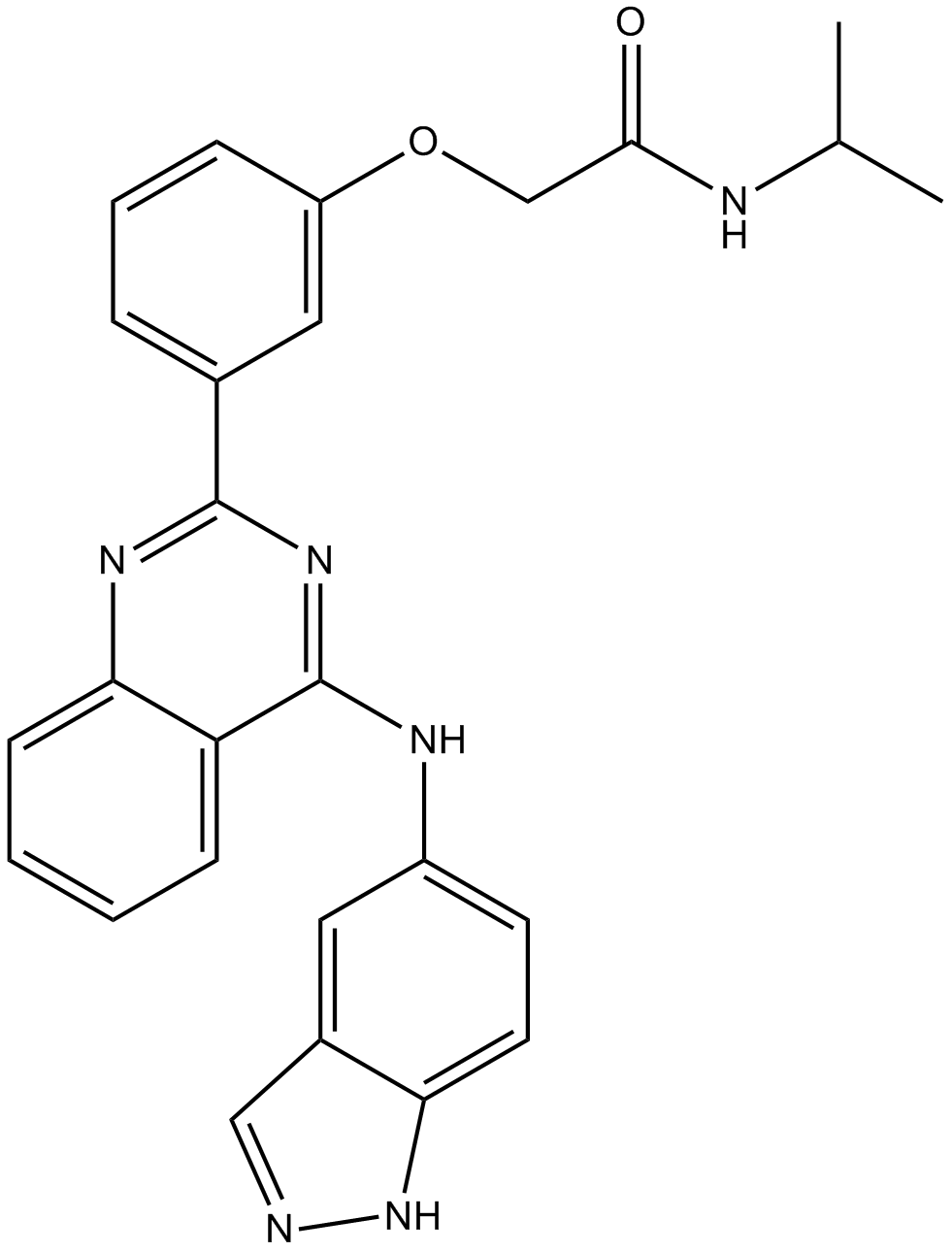
-
GC11396
SNS-032 (BMS-387032)
BMS-387032
SNS-032 (BMS-387032) (BMS-387032) es un inhibidor potente y selectivo de CDK2, CDK7 y CDK9 con IC50s de 38 nM, 62 nM y 4 nM, respectivamente. SNS-032 (BMS-387032) tiene efecto antitumoral.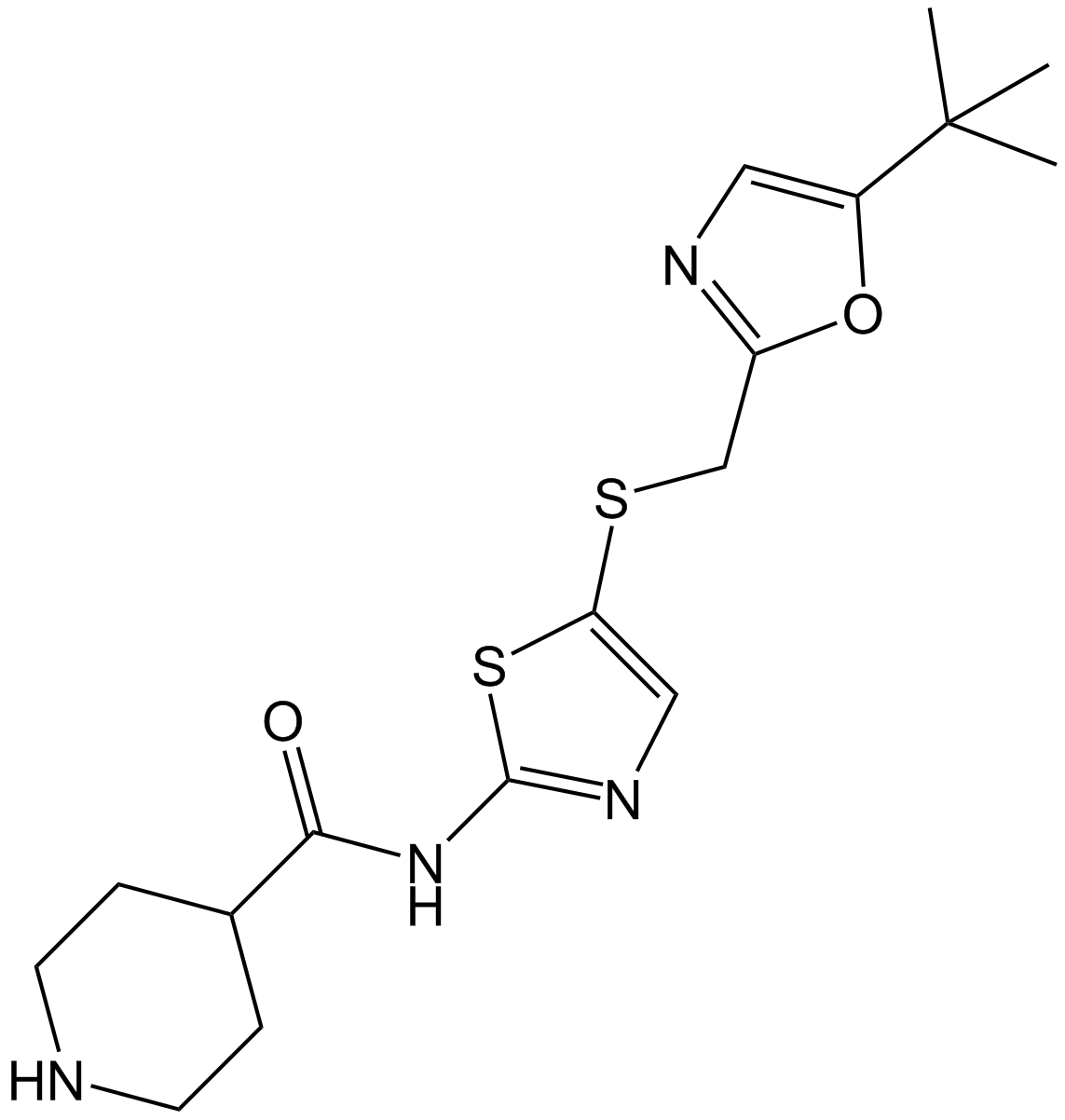
-
GC25940
SNS-314
SNS-314 is a potent and selective inhibitor of Aurora A, Aurora B and Aurora C with IC50 of 9 nM, 31 nM, and 3 nM, respectively. It is less potent to Trk A/B, Flt4, Fms, Axl, c-Raf and DDR2. Phase 1.
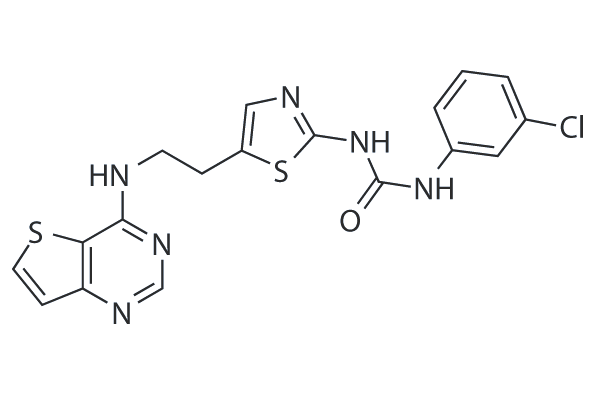
-
GC32935
Soblidotin (Auristatin PE)
Auristatin PE; TZT-1027
Soblidotin (Auristatin PE) (Auristatin PE) es un nuevo derivado sintético de Dolastatin 10 e inhibidor de la polimerizaciÓn de tubulina.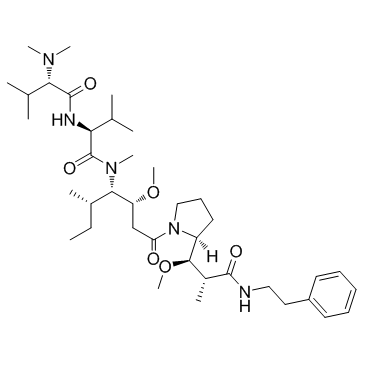
-
GC48084
Sodium 4-Phenylbutyrate-d11
Benzenebutanoic acid-d11, TriButyrate-d11
El fenilbutirato-d11 (sodio) es 4-fenilbutirato de sodio marcado con deuterio. El 4-fenilbutirato de sodio (4-PBA de sodio) es un inhibidor de la HDAC y el estrés del retÍculo endoplÁsmico (ER), que se utiliza en la investigaciÓn del cÁncer y las infecciones.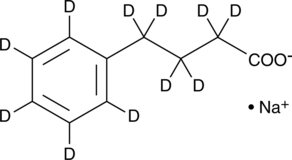
-
GC65887
SOP1812
SOP1812 es un derivado de diimida de naftaleno (ND) con actividad antitumoral. SOP1812 se une a arreglos cuÁdruplex (G4) y regula a la baja varias vÍas de genes del cÁncer. SOP1812 muestra una gran afinidad por hTERT G4 y HuTel21 G4 con valores de KD de 4,9 y 28,4 nM, respectivamente. SOP1812 se puede utilizar para la investigaciÓn del cÁncer.
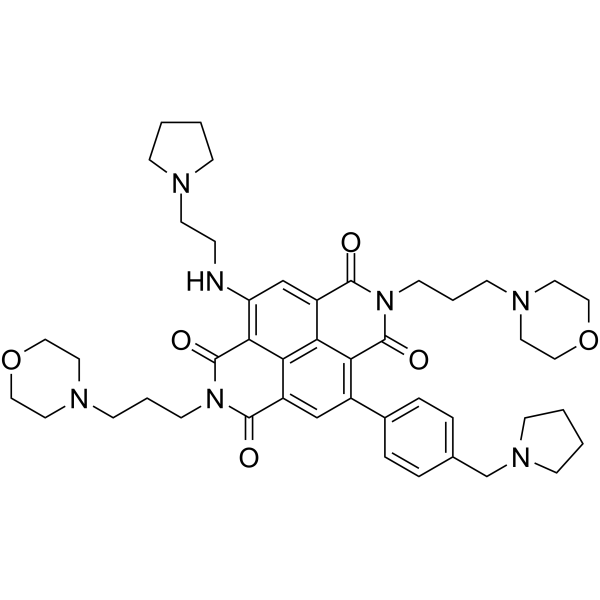
-
GC63351
Sovesudil
PHP-201; AMA0076
Sovesudil (PHP-201) es un potente inhibidor de la Rho quinasa (ROCK) de acciÓn local, competitivo con ATP, con IC50 de 3,7 y 2,3 nM para ROCK-I y ROCK-II, respectivamente.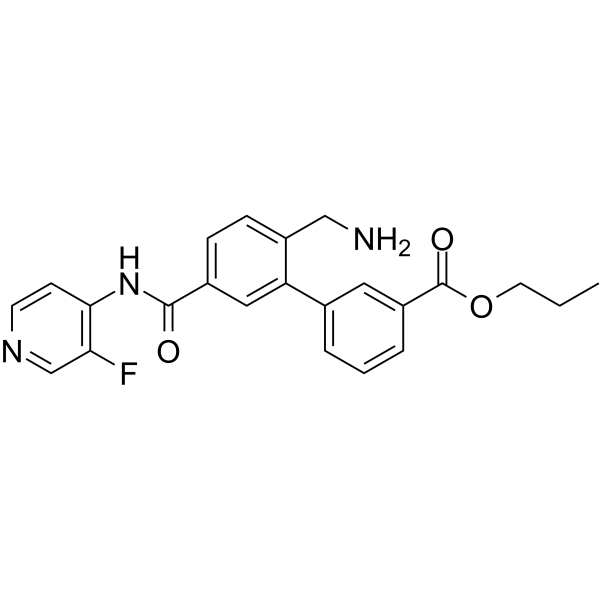
-
GC63716
Sovesudil hydrochloride
PHP-201 hydrochloride; AMA0076 hydrochloride
El clorhidrato de sovesudil (PHP-201) es un potente inhibidor de la Rho quinasa (ROCK) de acciÓn local, competitivo con ATP, con IC50 de 3,7 y 2,3 nM para ROCK-I y ROCK-II, respectivamente.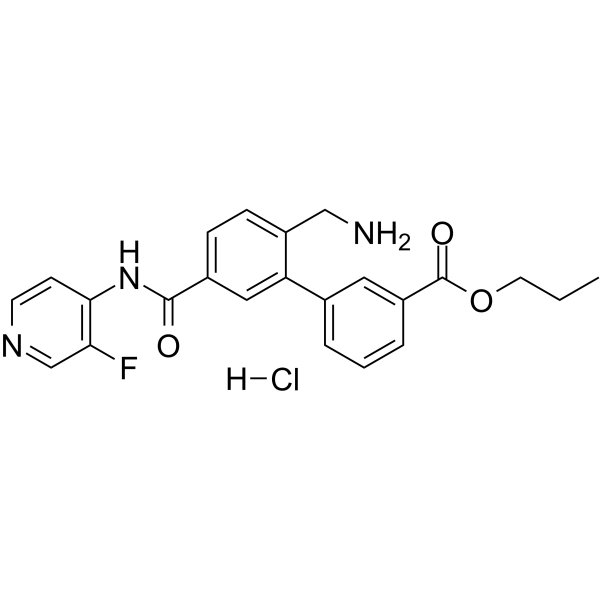
-
GC64019
Sovilnesib
AMG 650
Sovilnesib (AMG 650) es un inhibidor de la proteÍna KIF18A similar a la cinesina (WO2020132648). Sovilnesib se puede utilizar para la investigaciÓn del cÁncer.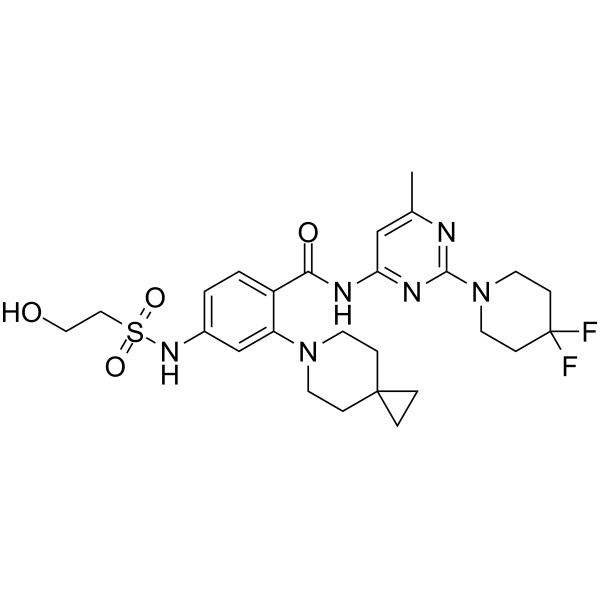
-
GC48092
Spinosad
LY232105, XDE-105
Spinosad, una mezcla de espinosinas A y D conocidas como productos de fermentaciÓn de un actinomiceto del suelo (Saccharopolyspora spinosa), es un insecticida neurotÓxico biolÓgico con un espectro de acciÓn mÁs amplio.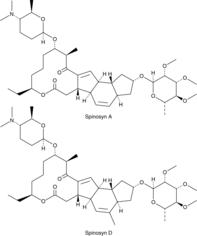
-
GC41258
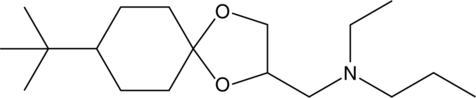
Spiroxamine
Spiroxamine is a tertiary amine fungicide and an inhibitor of δ14 reductase/δ8→δ7 isomerase.
-
GC44946
Sporidesmolide III
Sporidesmolide III is a cyclodepsipeptide originally isolated from P.