Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
GC43274
Citromycetin
La citromycétine est un composé polycétide aromatique de Penicillium spp d'origine marine et terrestre australienne.

-
GC63393
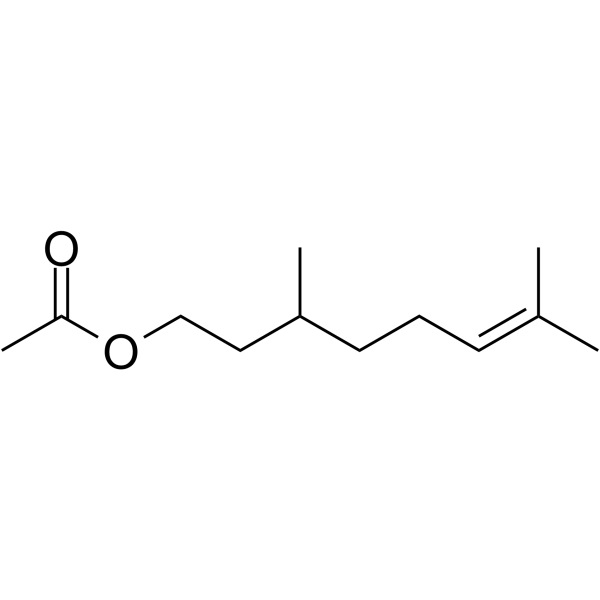
Citronellyl acetate
L'acétate de citronellyle est un produit monoterpénique du métabolisme secondaire des plantes, avec une activité antinociceptive.
-
GC52367
Citrullinated Vimentin (G146R) (R144 + R146) (139-159)-biotin Peptide
A biotinylated and citrullinated mutant vimentin peptide

-
GC52370
Citrullinated Vimentin (R144) (139-159)-biotin Peptide
A biotinylated and citrullinated vimentin peptide

-
GN10219
Ciwujianoside-B
-
GC64649
Cjoc42
Cjoc42 est un composé capable de se lier À la gankyrine. Cjoc42 inhibe l'activité de la gankyrine de manière dose-dépendante. Cjoc42 empêche la diminution des niveaux de protéine p53 normalement associée À des quantités élevées de gankyrine. Cjoc42 restaure la transcription dépendante de p53 et la sensibilité aux dommages À l'ADN.
-
GC39485
CK2/ERK8-IN-1
CK2/ERK8-IN-1 est un double inhibiteur de caséine kinase 2 (CK2) (Ki de 0,25 μM) et ERK8 (MAPK15, ERK7) avec des IC50 de 0,50 μM. CK2/ERK8-IN-1 se lie également À PIM1, HIPK2 (protéine kinase 2 interagissant avec l'homéodomaine) et DYRK1A avec Kis de 8,65 μM, 15,25 μM et 11,9 μM, respectivement. CK2/ERK8-IN-1 a une efficacité pro-apoptotique.
-
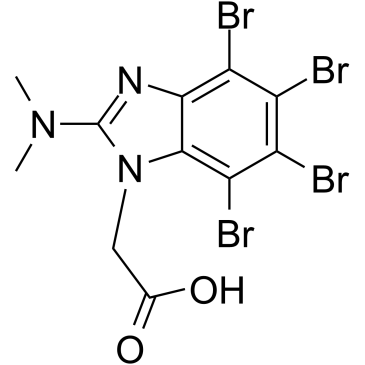
GC49556
Cl-Necrostatin-1
A RIPK1 inhibitor
-
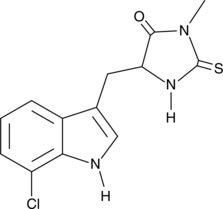
GC47098
CL2-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
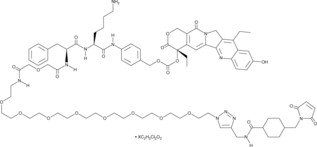
GC60714
CL2A-SN-38
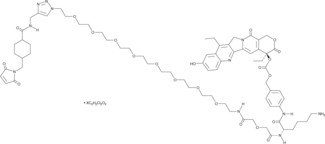
CL2A-SN-38 est un conjugué médicament-lieur composé d'un puissant inhibiteur de l'ADN topoisomérase I SN-38 et d'un lieur CL2A pour fabriquer un conjugué anticorps-médicament (ADC).
-
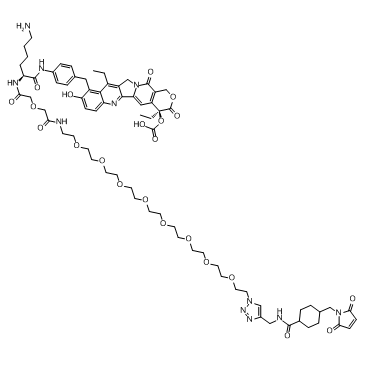
GC52469
CL2A-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
GC10509
Cladribine
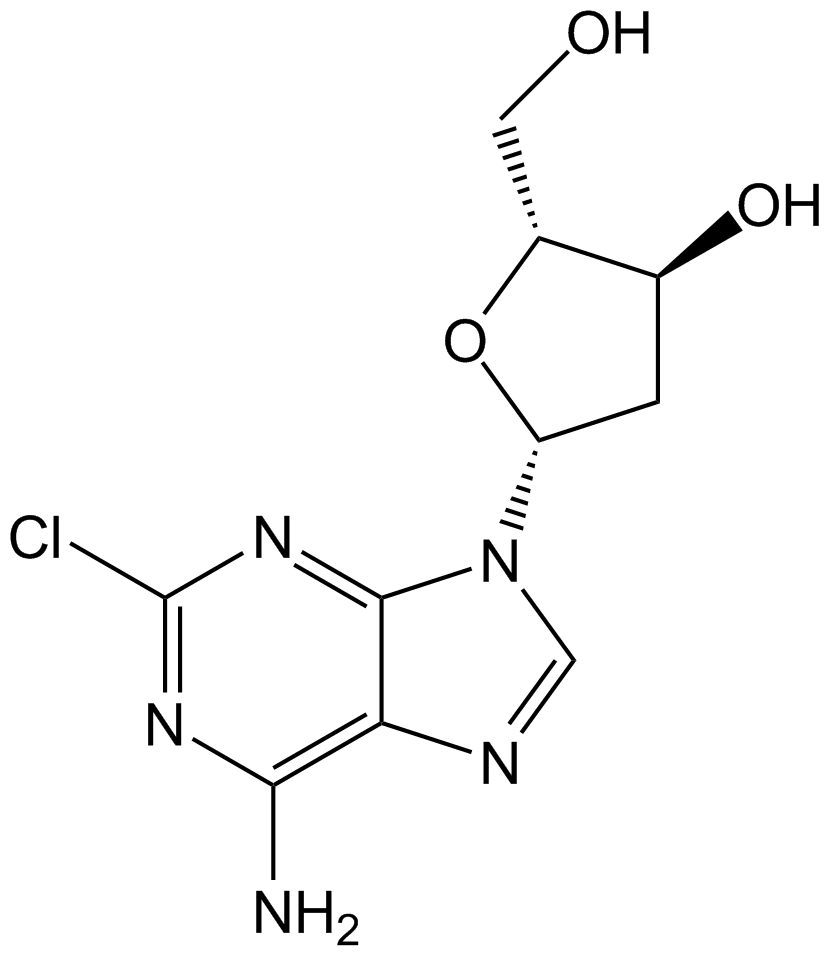
La cladribine (2-chloro-2′-désoxyadénosine), un analogue nucléosidique de la purine, est un inhibiteur de l'adénosine désaminase actif par voie orale.
-
GC60111
Clitocine
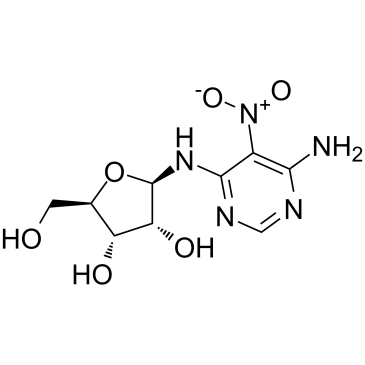
La clitocine, un analogue nucléosidique de l'adénosine isolé du champignon, est un agent de lecture puissant et efficace. La clitocine agit comme un suppresseur de mutations non-sens et peut induire la production de protéine p53 dans des cellules hébergeant des allèles mutés p53 non-sens. La clitocine peut induire l'apoptose dans les cellules cancéreuses humaines multirésistantes en ciblant Mcl-1. Activité anticancéreuse.
-
GC15219
Clofarabine
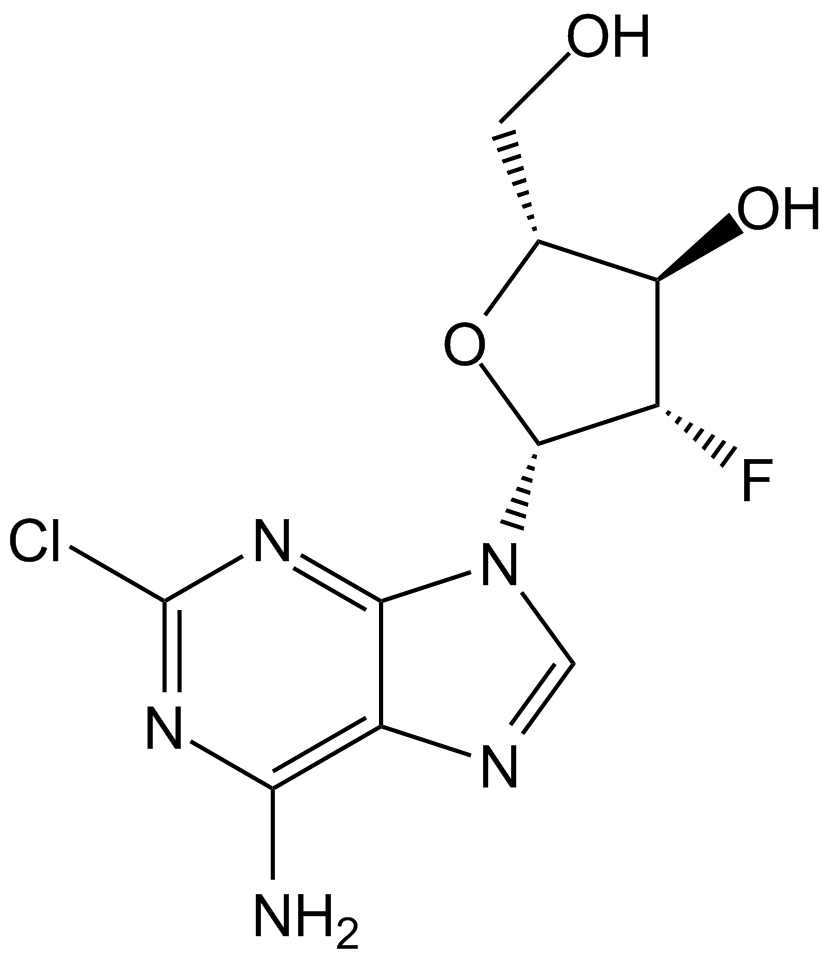
La clofarabine, un analogue nucléosidique pour la recherche sur le cancer, est un puissant inhibiteur de la ribonucléotide réductase (IC50 = 65 nM) en se liant au site allostérique de la sous-unité régulatrice.
-
GC10813
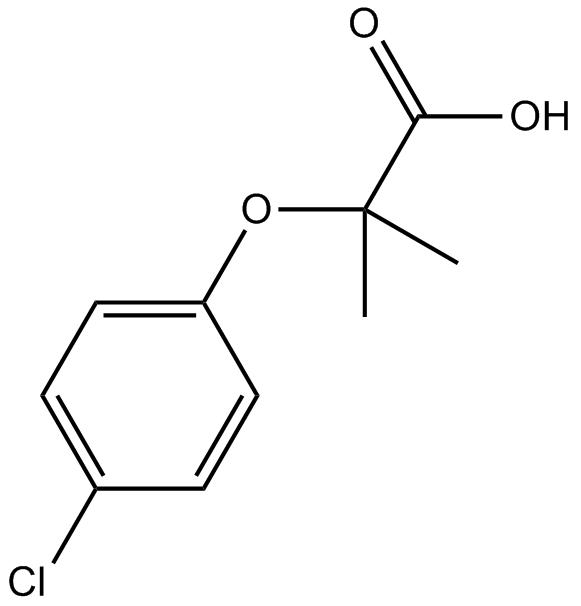
Clofibric Acid
L'acide clofibrique (acide chlorofibrinique), le métabolite pharmaceutiquement actif des régulateurs lipidiques Clofibrate, Etofibrate et Etofyllinclofibrate, est un agoniste PPARα qui présente des effets hypolipidémiants.
-
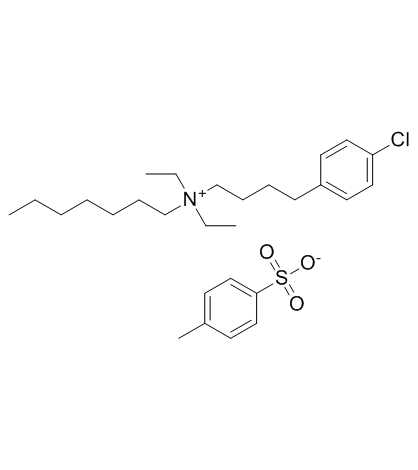
GC32587
Clofilium tosylate
Le tosylate de clofilium, un inhibiteur des canaux potassiques, induit l'apoptose des cellules de la leucémie promyélocytaire humaine (HL-60) via l'activation insensible À Bcl-2 de la caspase-3.
-
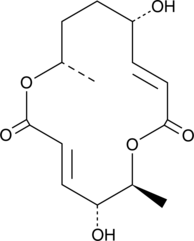
GC47105
Clonostachydiol
Le clonostachydiol est un macrodiolide anthelminthique du champignon Clonostachys cylindrospora (souche FH-A 6607).
-
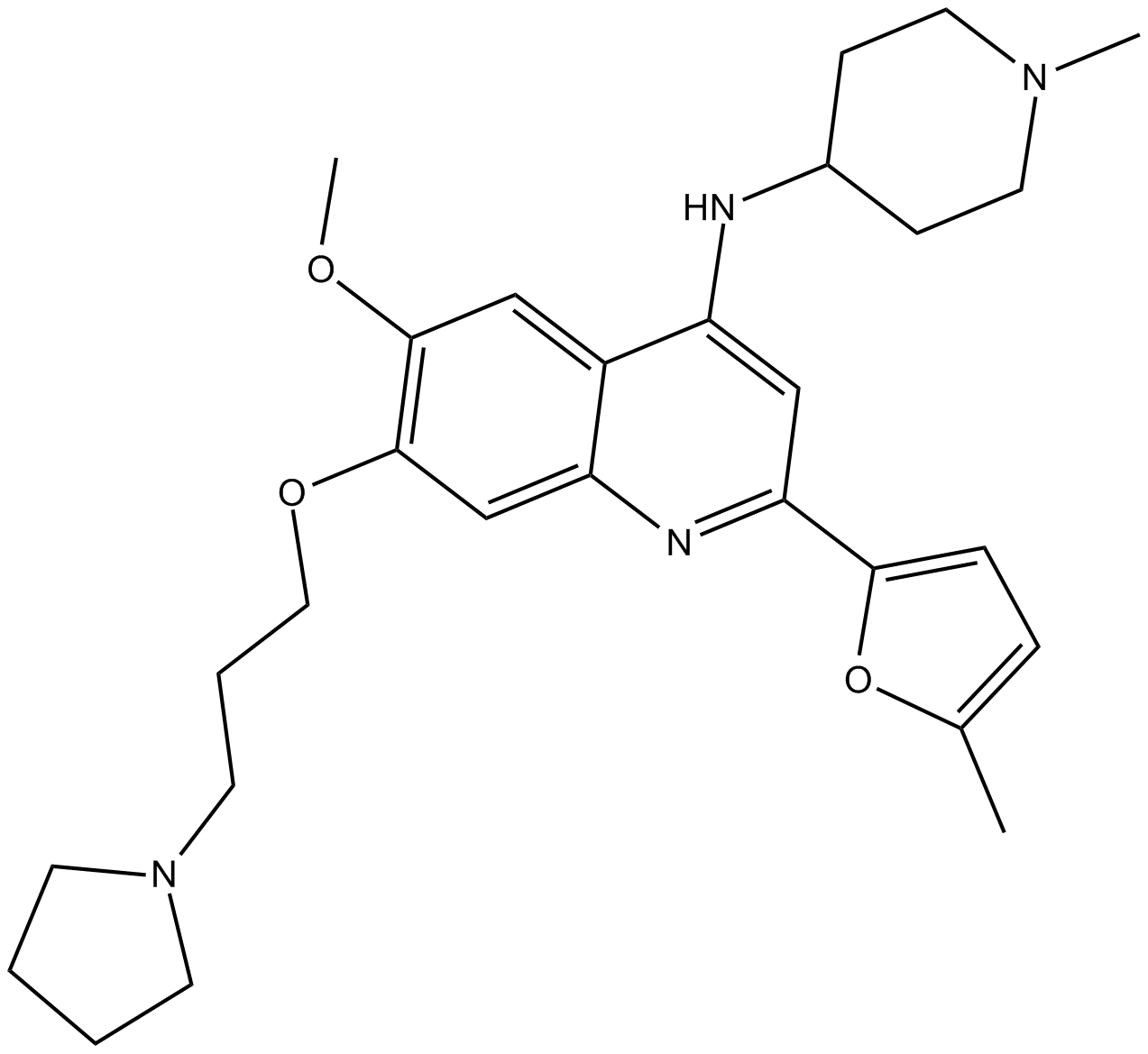
GC12367
CM-272
Le CM-272 est un inhibiteur double G9a/ADN méthyltransférases (DNMT) de premier ordre, puissant, sélectif, compétitif avec le substrat et réversible, doté d'activités antitumorales. CM-272 inhibe G9a, DNMT1, DNMT3A, DNMT3B et GLP avec des IC50 de 8 nM, 382 nM, 85 nM, 1200 nM et 2 nM, respectivement. Le CM-272 inhibe la prolifération cellulaire et favorise l'apoptose, induisant des gènes stimulés par l'IFN et la mort cellulaire immunogène.
-
GC62347
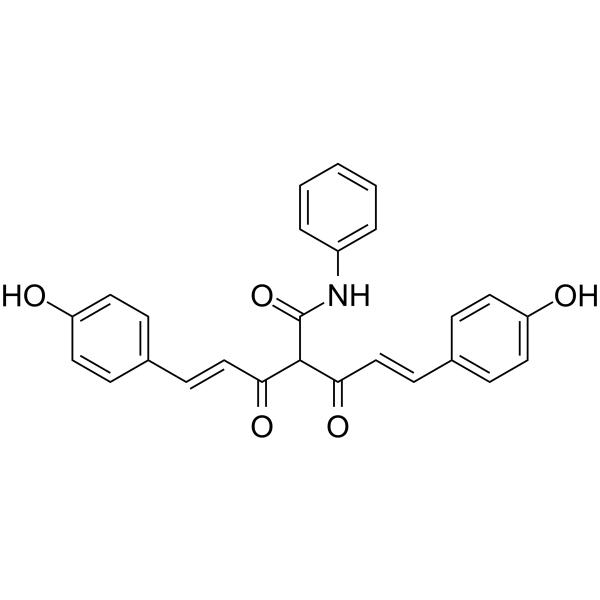
CMC2.24
CMC2.24 (TRB-N0224), un agent tricarbonylméthane actif par voie orale, est efficace contre la tumeur pancréatique chez la souris en inhibant l'activation de Ras et sa voie effectrice en aval ERK1/2.
-
GC61567
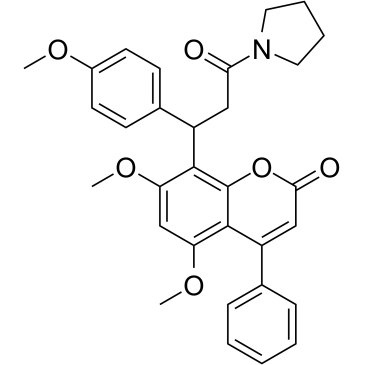
CMLD-2
CMLD-2, un inhibiteur de l'interaction HuR-ARE, se lie de manière compétitive À la protéine HuR, perturbant son interaction avec les ARNm contenant des éléments riches en adénine-uridine (ARE) (Ki = 350 nM). CMLD-2 induit l'apoptose et présente une activité antitumorale dans différentes cellules cancéreuses telles que les lignées cellulaires du cancer du cÔlon, du pancréas, de la thyroÏde et du poumon. L'antigène Hu R (HuR) est une protéine de liaison À l'ARN, peut réguler la stabilité et la traduction des ARNm cibles.
-
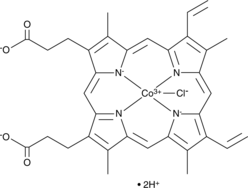
GC49096
Cobaltic Protoporphyrin IX (chloride)
An inducer of HO-1 activity
-
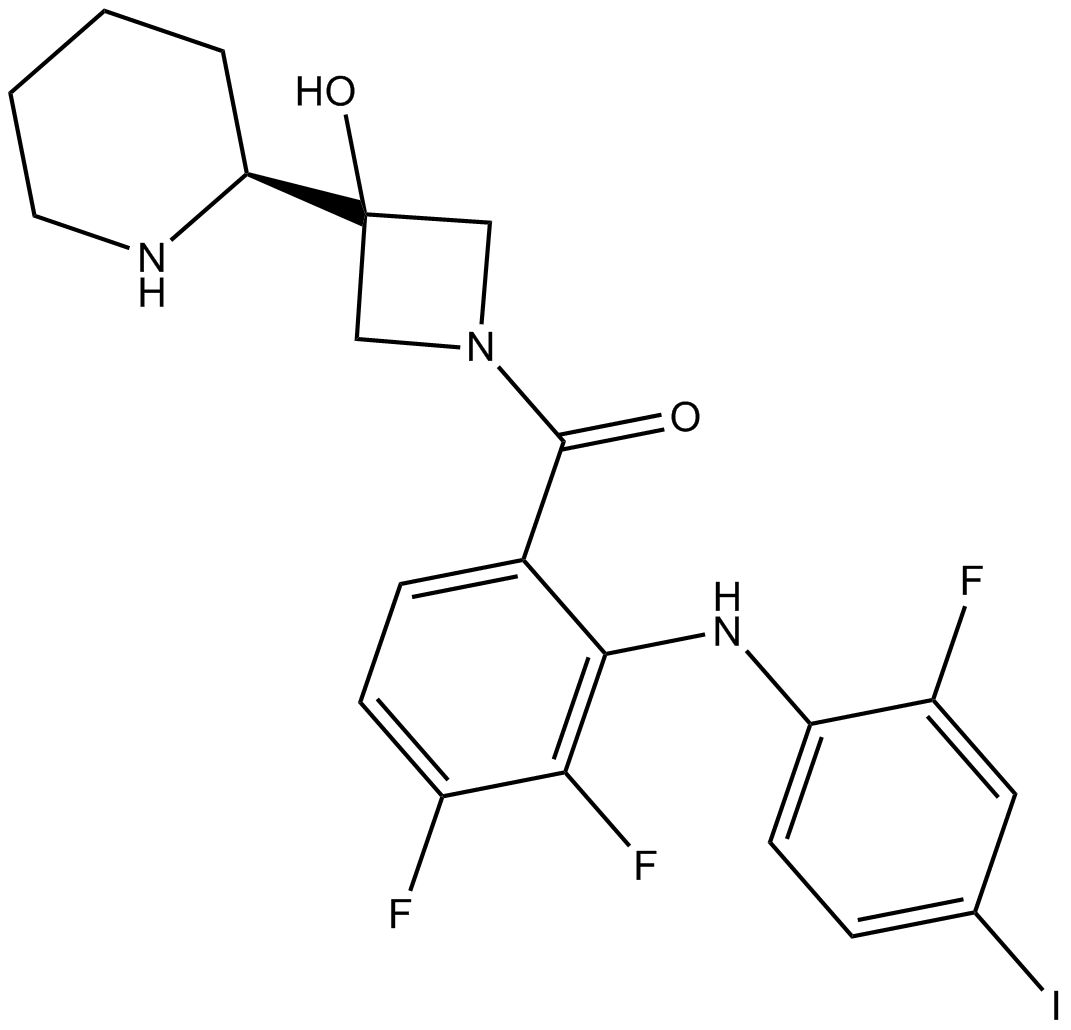
GC10033
Cobimetinib
A potent, orally available MEK1 inhibitor
-
GC43297
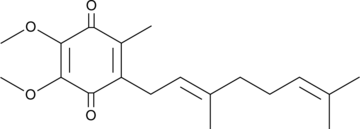
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
-
GC62192
COG1410
COG1410 est un peptide dérivé de l'apolipoprotéine E.
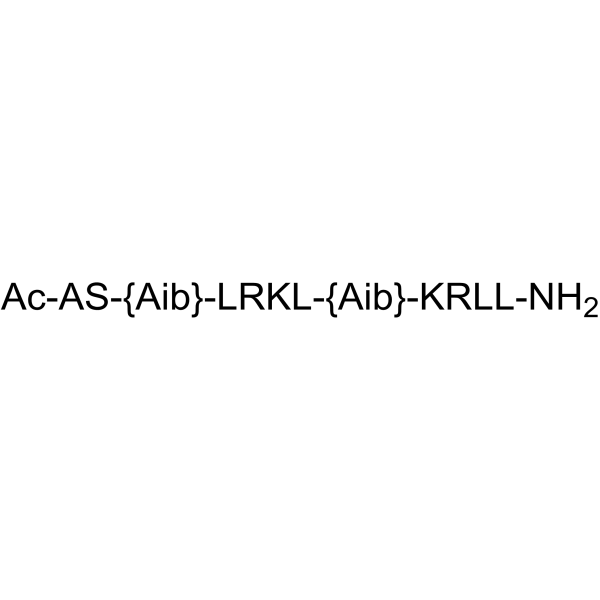
-
GC40664
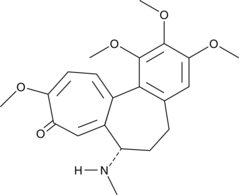
Colcemid
Colcemid est un inhibiteur cytosquelettique qui induit l'arrêt mitotique en phase G2/M ou l'arrêt méiotique lors de la rupture de la vésicule (GVBD) dans les cellules mammifères ou les ovocytes, respectivement.
-
GN10123
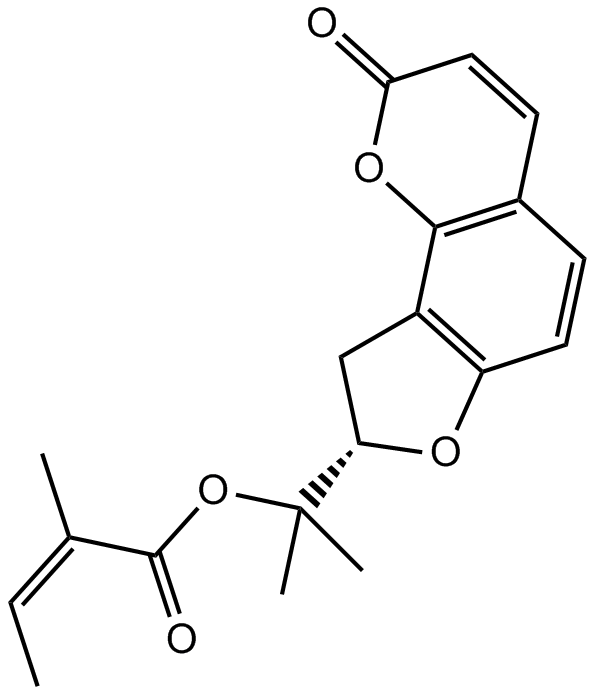
Columbianadin
-
GC49454
Complex 3
A fluorescent copper complex with anticancer activity
-
GC18572
Concanavalin A
La concanamycine A appartient aux concanamycines, une famille d'antibiotiques macrolides isolés de Streptomyces diastatochromogenes qui sont des inhibiteurs hautement actifs et sélectifs de la vacuolaire proton-ATPase (v-[H+]ATPase).

-
GC18832
Conglobatin
Conglobatin is a dimeric macrolide dilactone originally isolated from S.
-
GC48483
Conglobatin B
A bacterial metabolite
-
GC48497
Conglobatin C1
A bacterial metabolite
-
GC38376
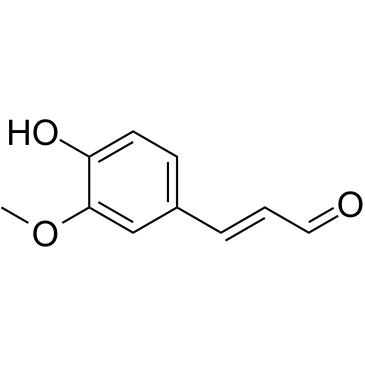
Coniferaldehyde
Le coniféraldéhyde (férulaldéhyde) est un inducteur efficace de l'hème oxygénase-1 (HO-1).
-
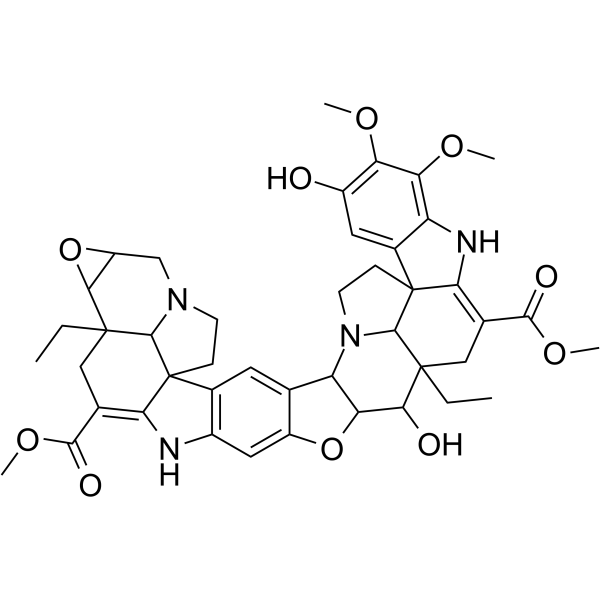
GC63379
Conophylline
La conophylline est un vinca alcaloÏde extrait des feuilles d'une plante tropicale Ervatamia microphylla.
-
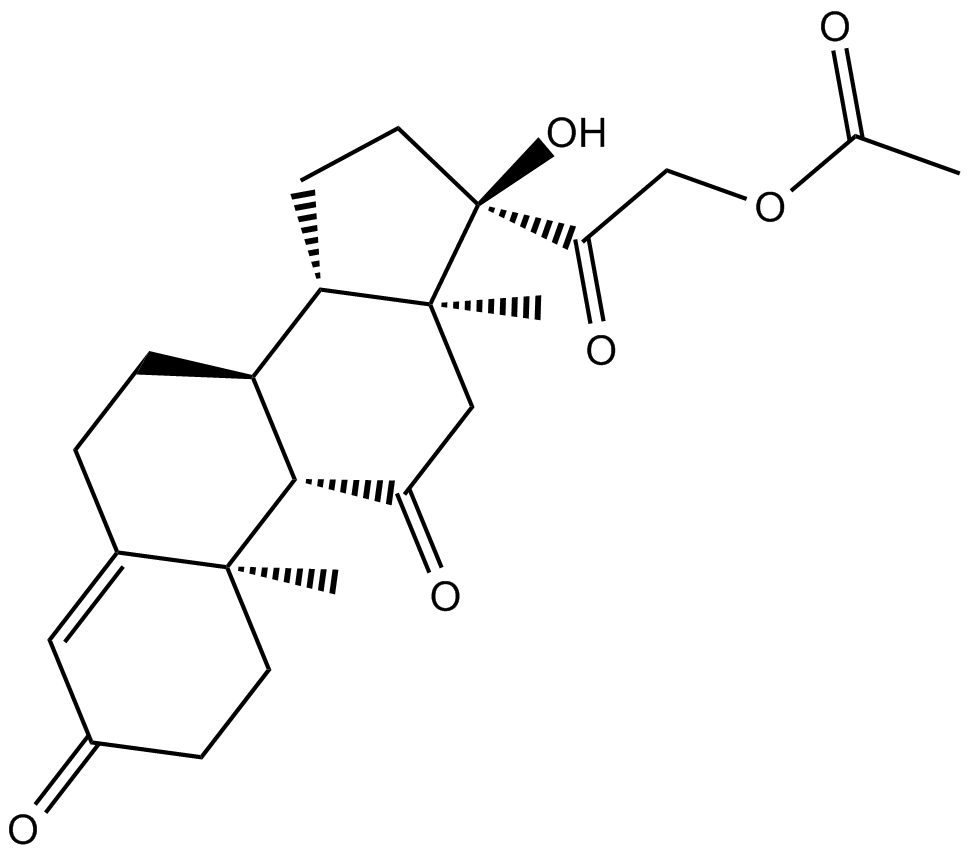
GC16772
Cortisone acetate
Acétate de cortisone (acétate de cortisone 21), un métabolite oxydé du cortisol (un glucocorticoÏde).
-
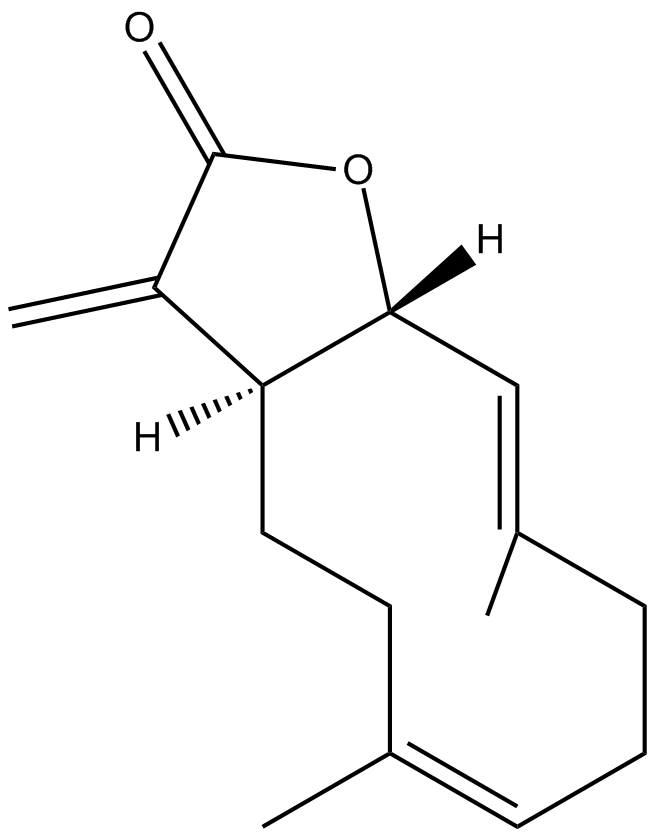
GC16116
Costunolide
A natural sesquiterpene lactone
-
GC15225
COTI-2
COTI-2, un médicament anticancéreux À faible toxicité, est un activateur de troisième génération disponible par voie orale des formes mutantes p53. COTI-2 agit À la fois en réactivant p53 mutant et en inhibant la voie PI3K/AKT/mTOR. COTI-2 induit l'apoptose dans plusieurs lignées cellulaires tumorales humaines. COTI-2 présente une activité antitumorale dans le HNSCC par le biais de mécanismes dépendants et indépendants de p53. COTI-2 convertit p53 mutant en conformation de type sauvage.
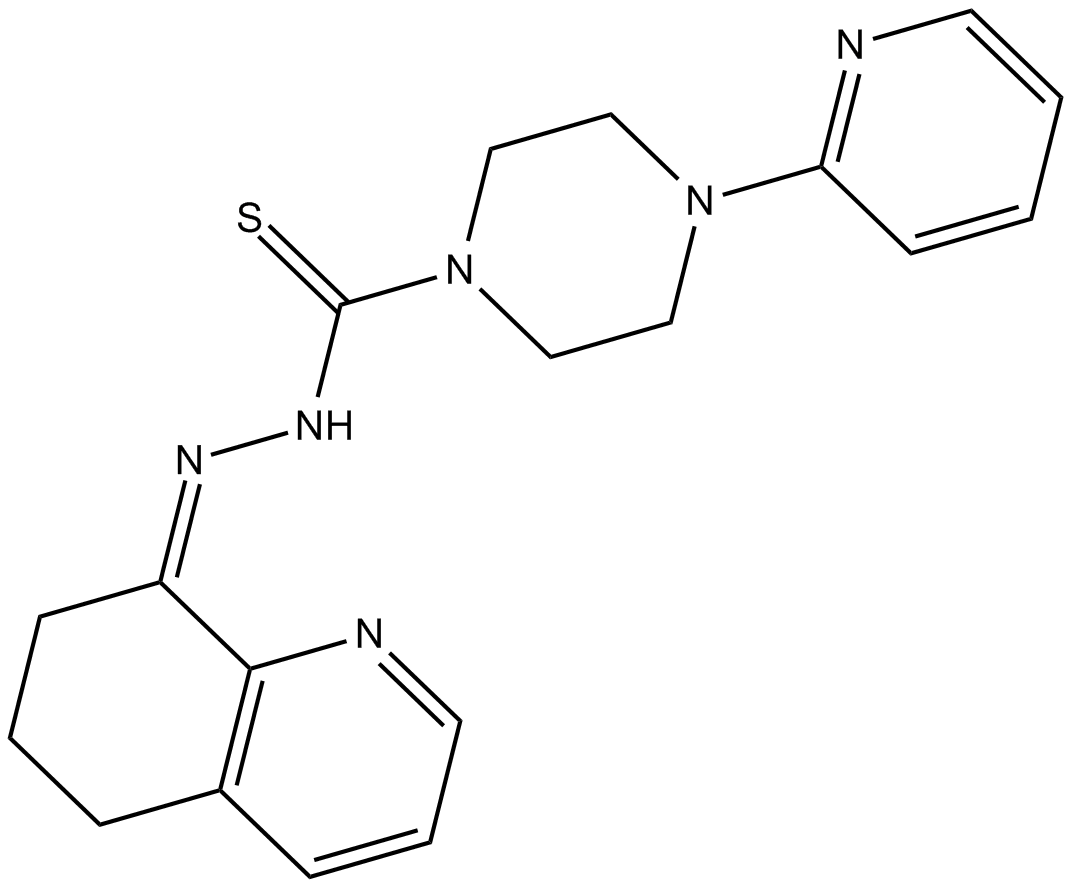
-
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
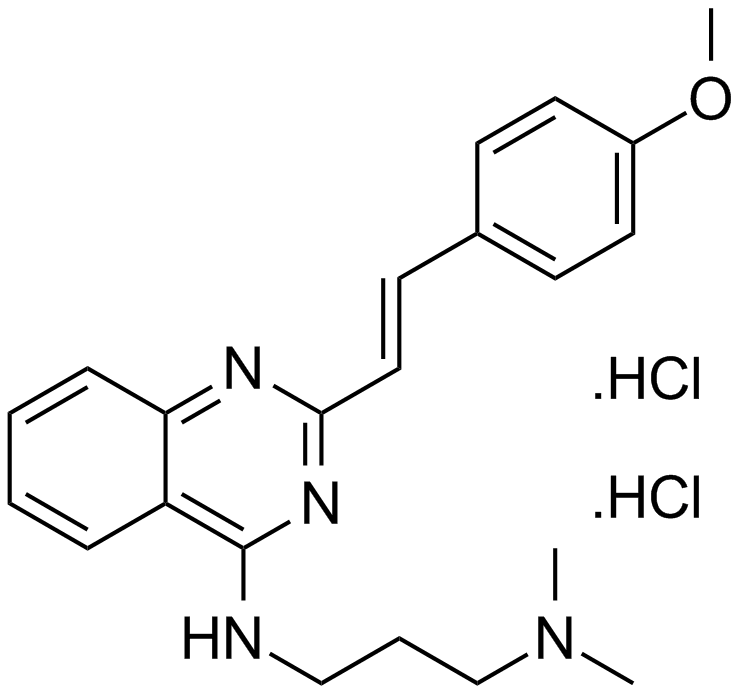
-
GC13091
CP-724714
Le CP-724714 est un inhibiteur de tyrosine kinase ErbB2 (HER2) puissant, sélectif et actif par voie orale, avec une IC50 de 10 nM. CP-724714 affiche une sélectivité marquée contre l'EGFR kinase (IC50 = 6400 nM). Le CP-724714 inhibe puissamment l'autophosphorylation du récepteur ErbB2 dans les cellules intactes. Activités antitumorales.
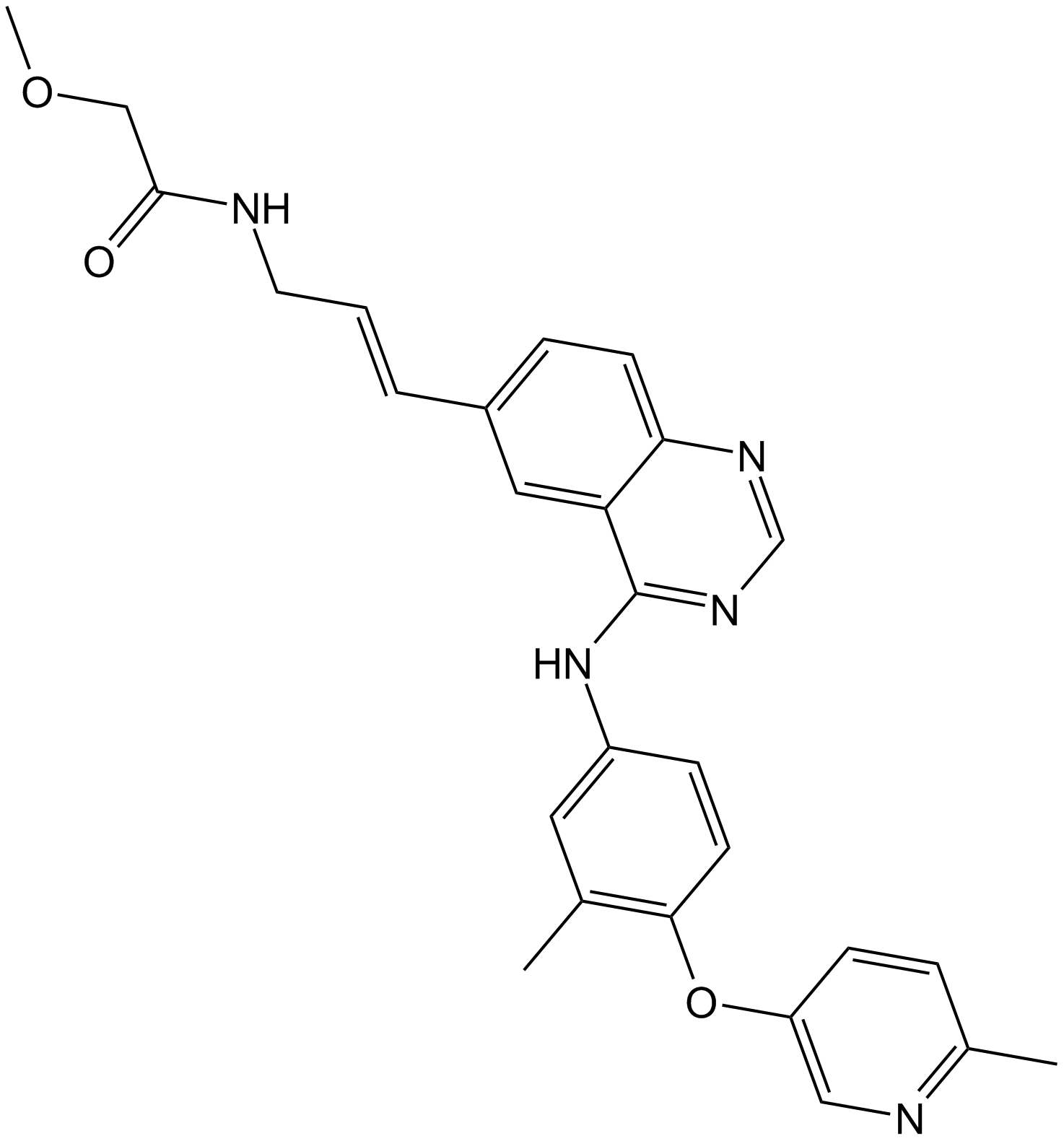
-
GC14500
CPI-1189
Le CPI-1189 est un inhibiteur de libération de TNF-α aux propriétés antioxydantes et neuroprotectrices.
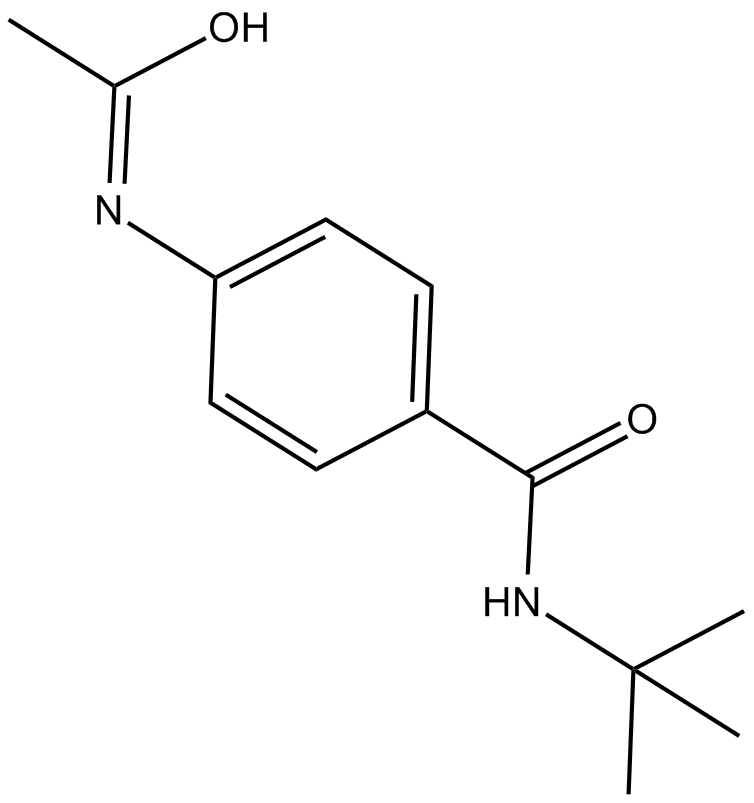
-
GC14699
CPI-203
Le CPI-203 est un nouvel inhibiteur puissant, sélectif et perméable aux cellules du bromodomaine BET, avec une valeur IC50 d'environ 37 nM (test BRD4 α-screen).
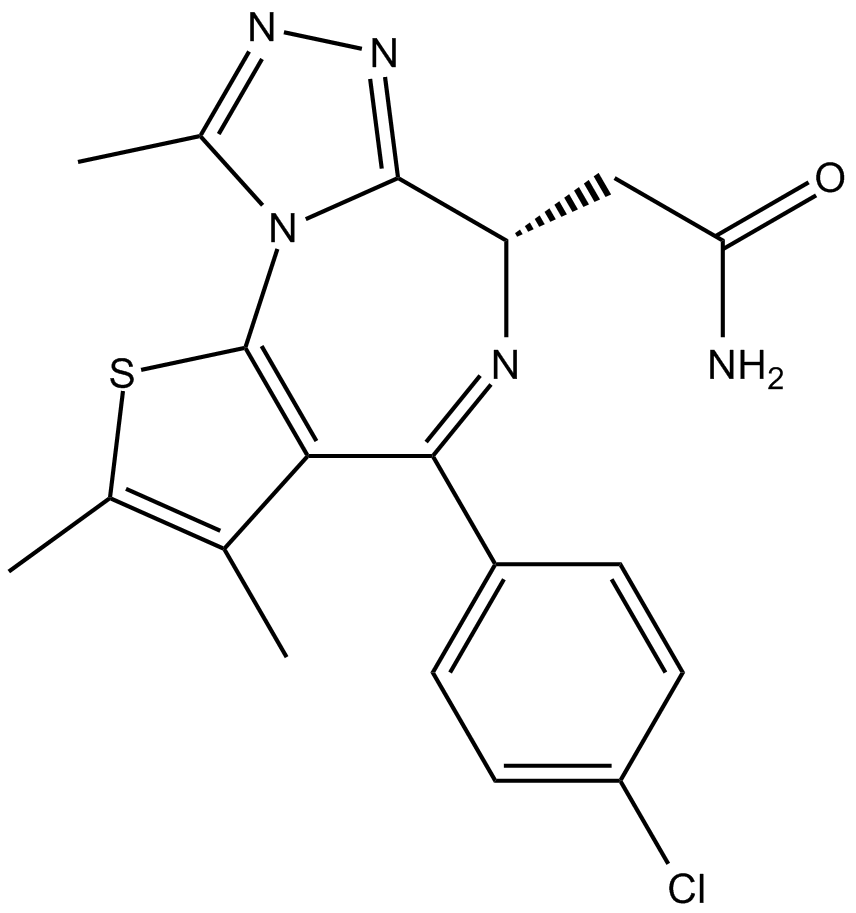
-
GC10021
CPI-360
EZH2 inhibitor
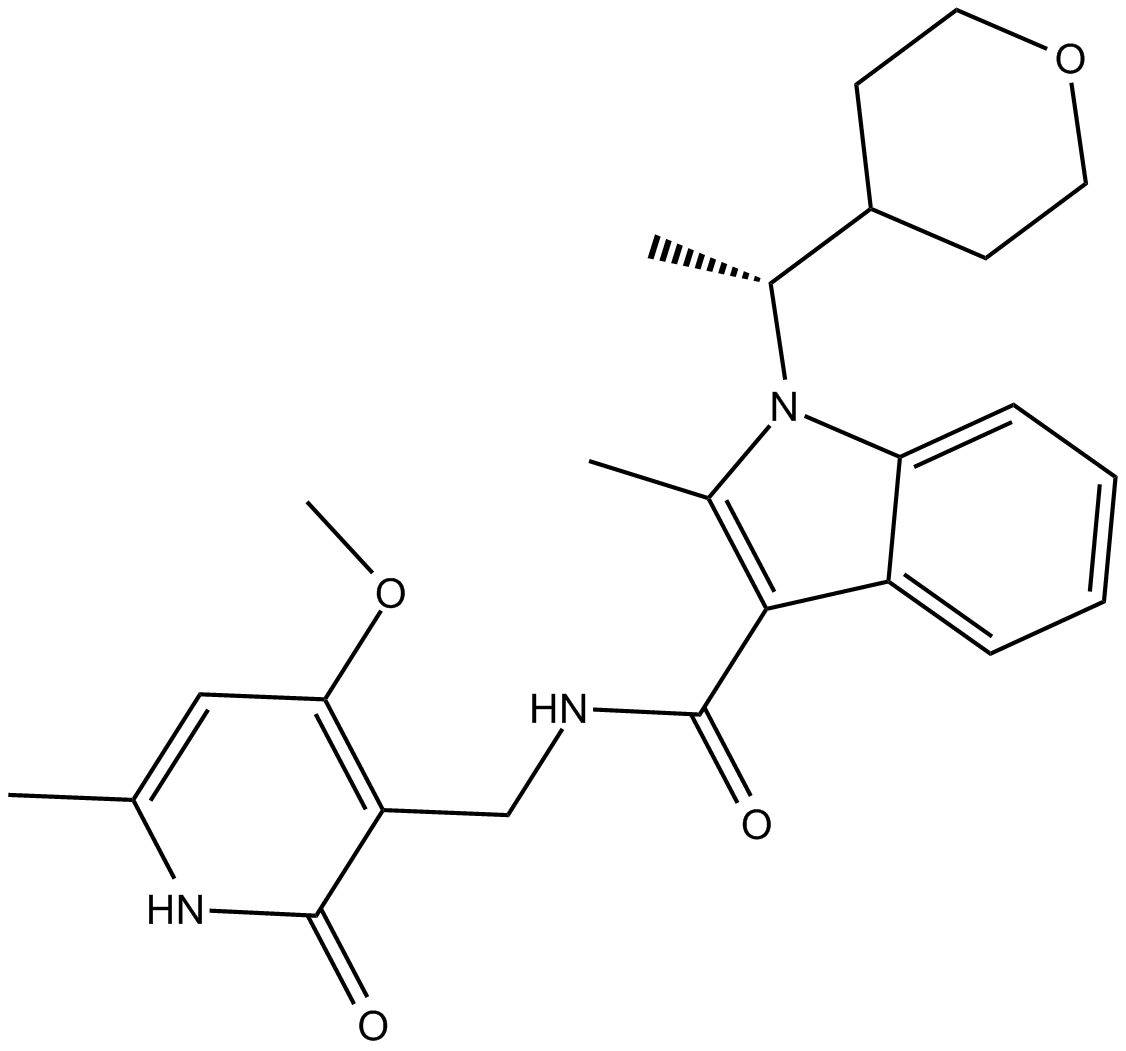
-
GC14921
CPI-613
An inhibitor of α-ketoglutarate dehydrogenase
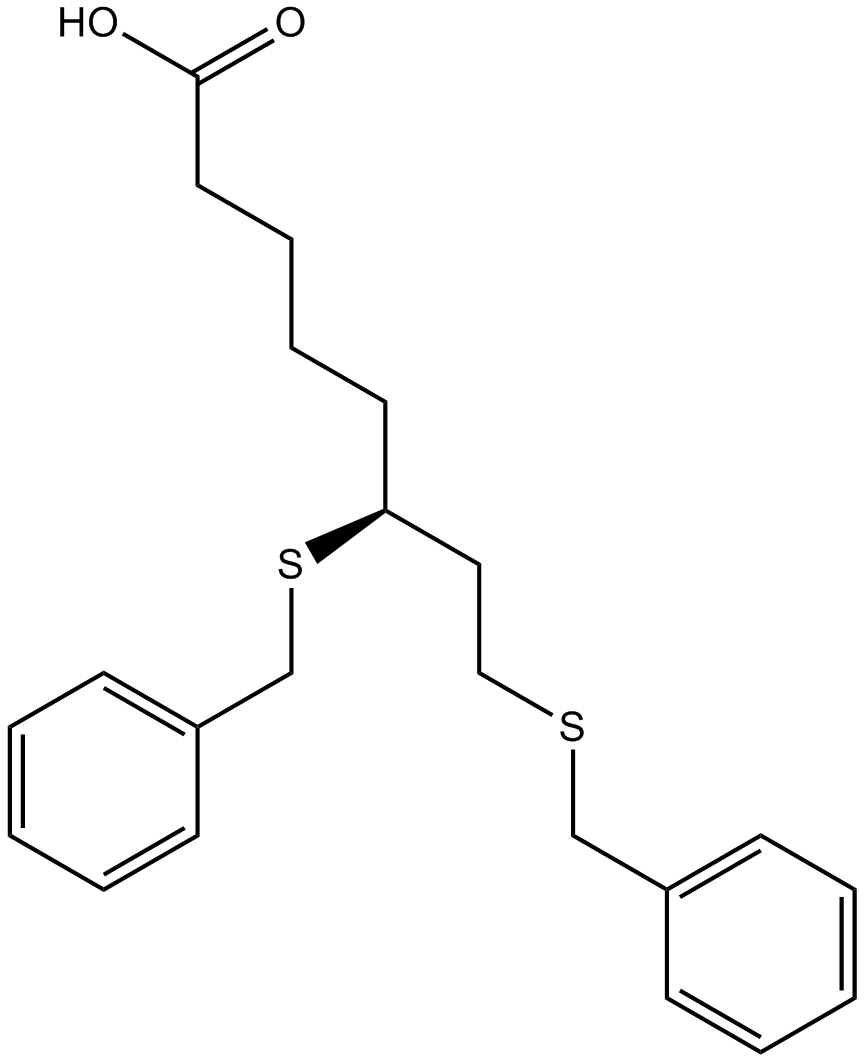
-
GC39365
CPTH2
CPTH2 est un puissant inhibiteur de l'histone acétyltransférase (HAT). CPTH2 inhibe sélectivement l'acétylation de l'histone H3 par Gcn5. CPTH2 induit l'apoptose et diminue le caractère invasif d'une lignée cellulaire de carcinome rénal À cellules claires (ccRCC) par l'inhibition de l'acétyltransférase p300 (KAT3B).
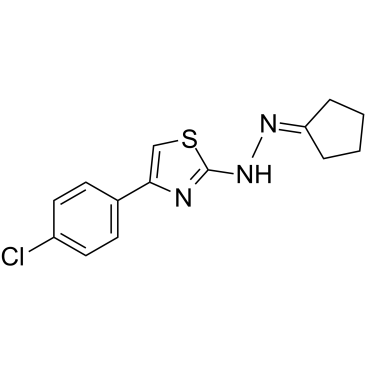
-
GC35747
Crebanine
La crébanine, un alcaloÏde de Stephania venosa, induit l'arrêt de G1 et l'apoptose dans les cellules cancéreuses humaines.
-
GC34543
cRIPGBM
cRIPGBM, un dérivé pro-apoptotique de RIPGBM, un inducteur sélectif de type cellulaire de l'apoptose dans les cellules souches cancéreuses GBM (CSC) en se liant À la protéine kinase 2 interagissant avec le récepteur (RIPK2), avec une CE50 de 68 nM dans les cellules GBM-1.
-
GC13838
CRT 0066101
PKD inhibitor
-
GC35750
CRT0066101 dihydrochloride
Le dichlorhydrate de CRT0066101 est un inhibiteur puissant et spécifique de la PKD avec des valeurs IC50 de 1, 2,5 et 2 nM pour PKD1, 2 et 3 respectivement.
-
GC45414
CRT0066854
CRT0066854 est un inhibiteur atypique puissant et sélectif des isoenzymes PKC.
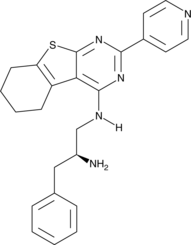
-
GC14355
CRT5
Le CRT5, un pyrazine benzamide, est un inhibiteur puissant et sélectif des trois isoformes de PKD dans les cellules endothéliales traitées avec le VEGF (IC50 = 1, 2 et 1,5 nM pour PKD1, PKD2 et PKD3, respectivement).
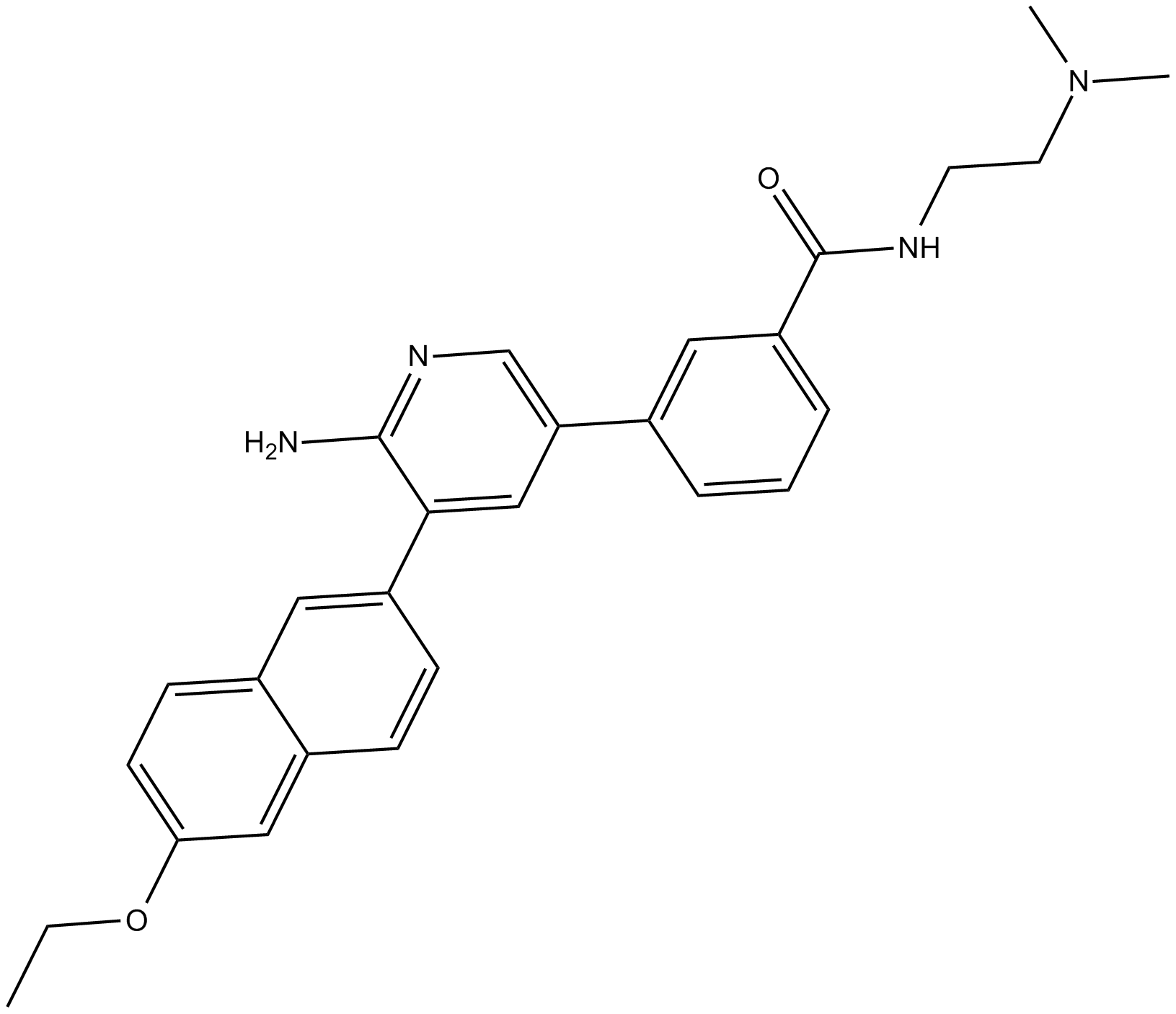
-
GC32911
CTX1
CTX1 est un activateur de p53 qui surmonte la répression de p53 médiée par HdmX. CTX1 présente une puissante activité anticancéreuse dans un système modèle de leucémie myéloÏde aiguë (LMA) chez la souris.
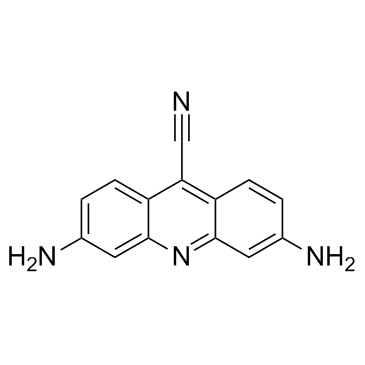
-
GN10535
Cucurbitacin B
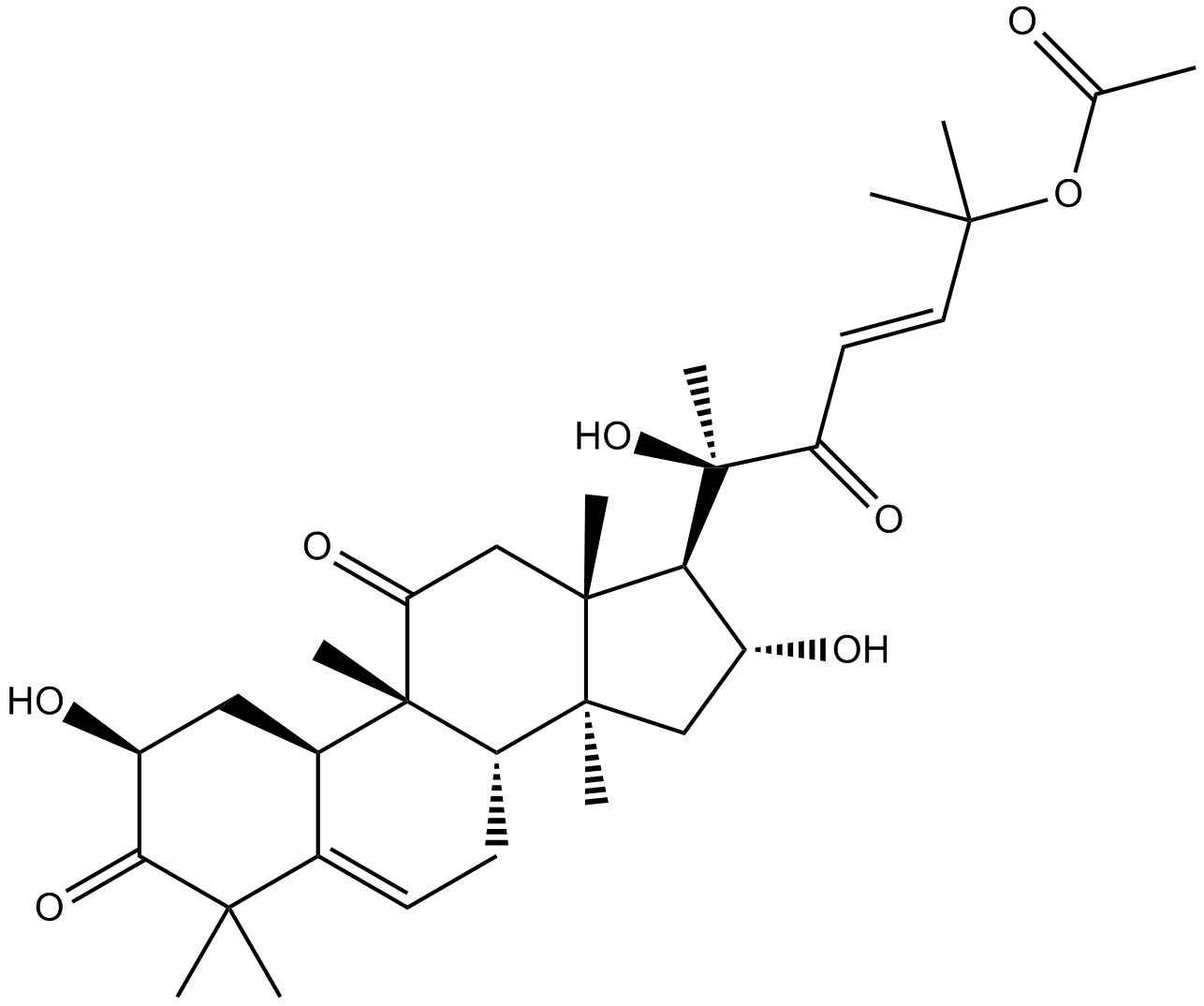
-
GC35758
Cucurbitacin IIa
La cucurbitacine IIa est un triterpène isolé de Hemsleya amalils Diels, induit l'apoptose des cellules cancéreuses, réduit l'expression de la survivine, réduit la phospho-histone H3 et augmente la PARP clivée dans les cellules cancéreuses.
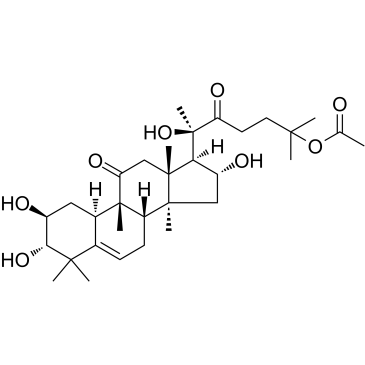
-
GN10788
Cucurbitacin IIb
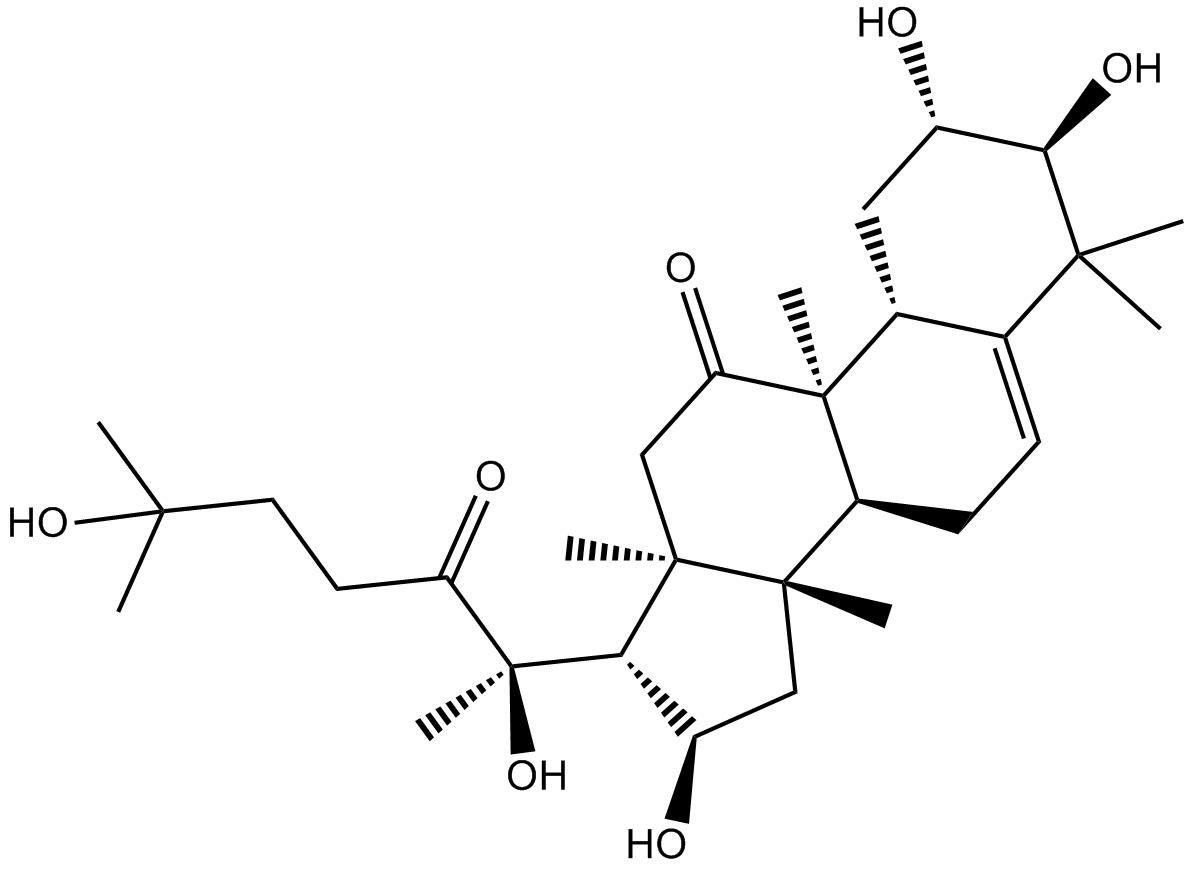
-
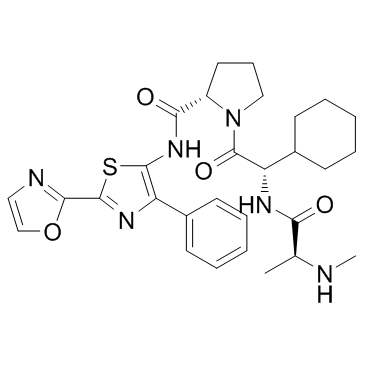
GC32781
CUDC-427 (GDC-0917)
CUDC-427 (GDC-0917) est un puissant antagoniste IAP pan-sélectif de deuxième génération, utilisé pour le traitement de divers cancers.
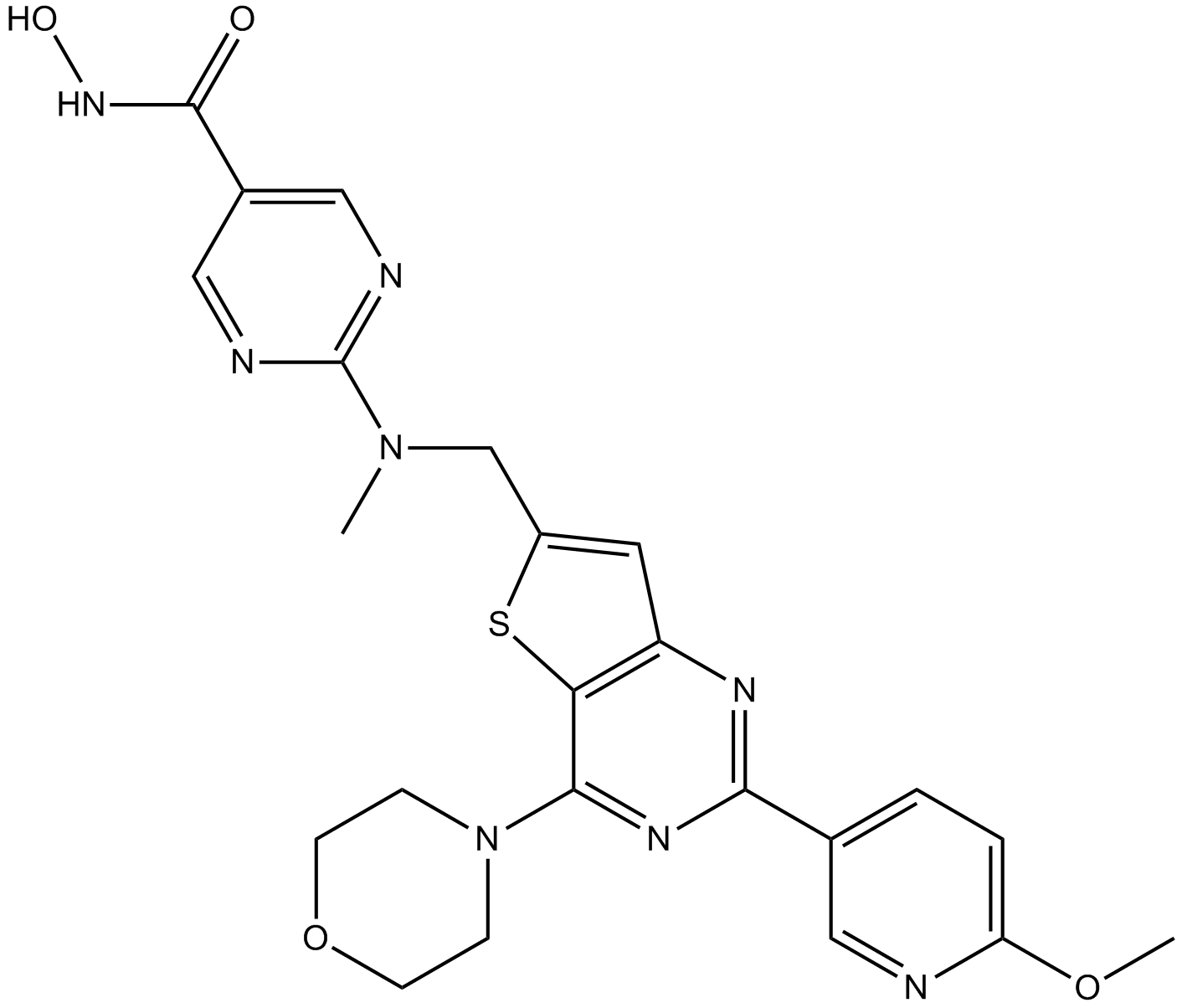
-
GC12115
CUDC-907
A dual inhibitor of HDACs and PI3Ks
-
GC11217
CUR 61414
CUR 61414 est un nouvel inhibiteur de la voie de signalisation Hedgehog puissant et perméable aux cellules (IC50 \u003d 100-200 nM).
-
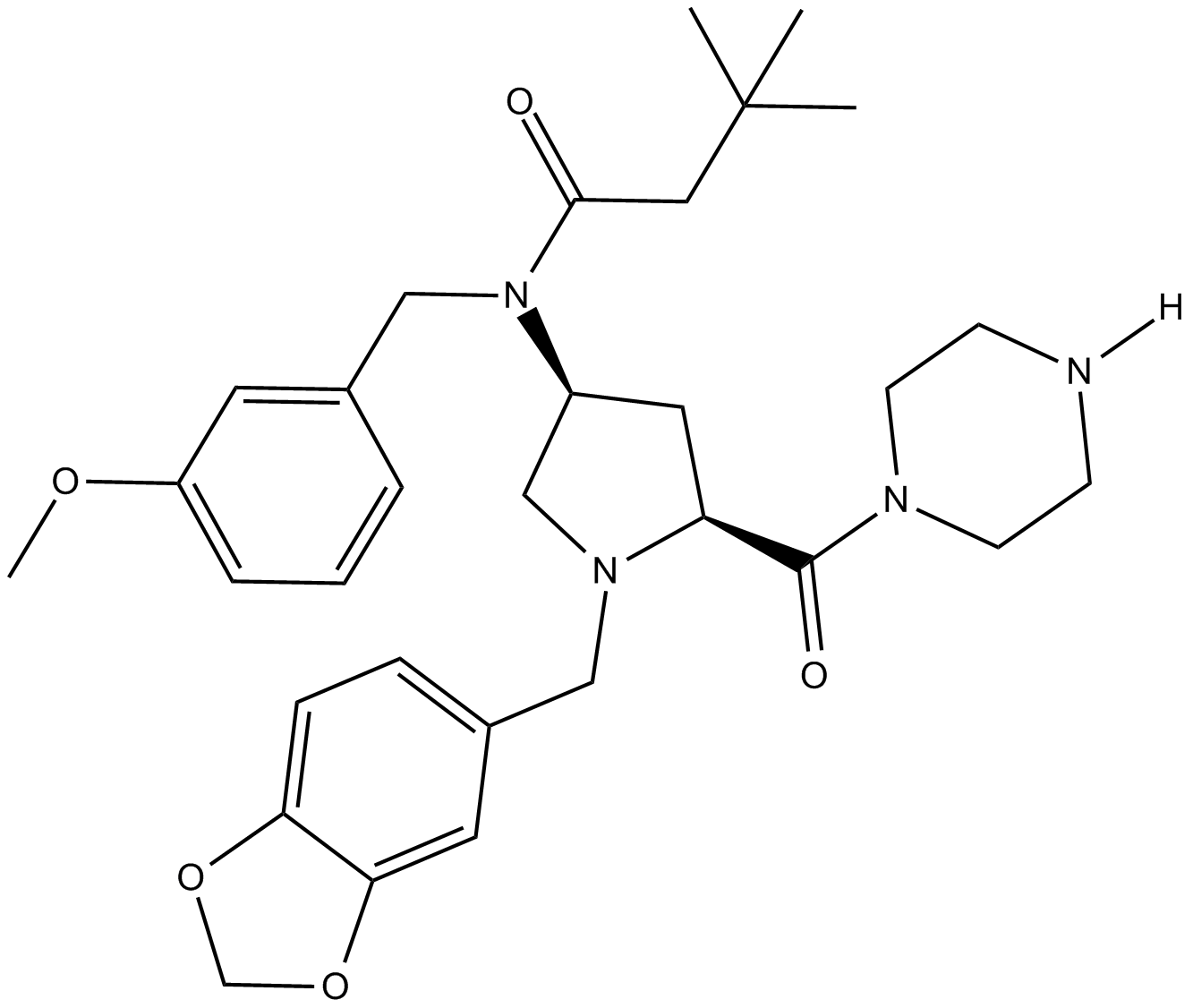
GC14787
Curcumin
Un pigment jaune avec diverses activités biologiques
-
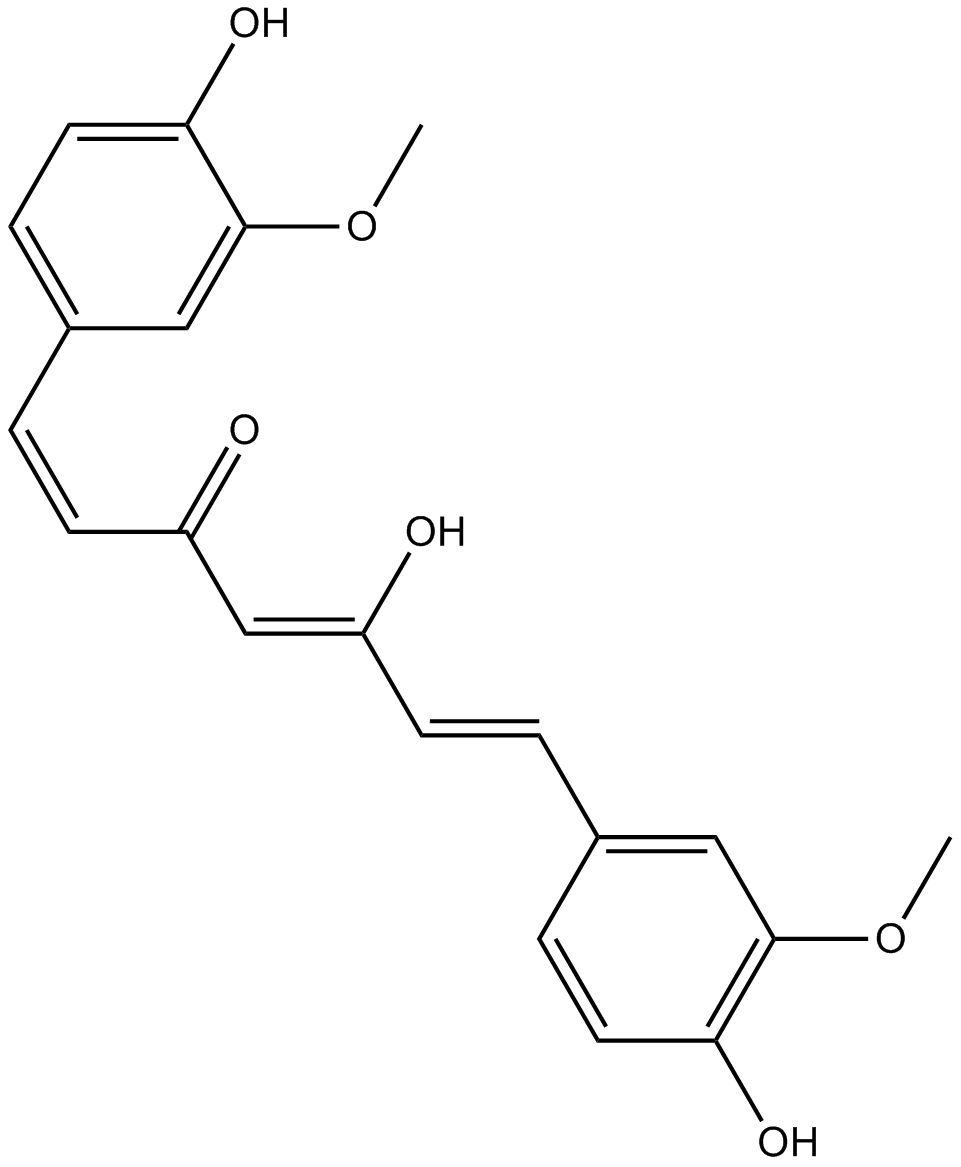
GC40226
Curcumin-d6
La curcumine D6 (Diferuloylmethane D6) est une curcumine marquée au deutérium (jaune curcuma).
-
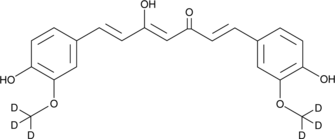
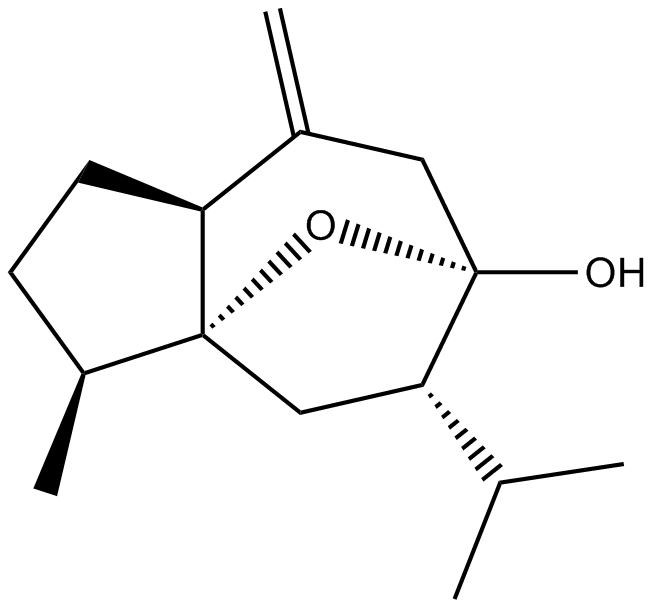
GN10521
Curcumol
-
GC66356
Cusatuzumab
Le cusatuzumab est un anticorps monoclonal humain αCD70. Le cusatuzumab montre une activité de cytotoxicité avec une amélioration cellulaire dépendante des anticorps. Le cusatuzumab réduit les cellules souches leucémiques (LSC) et déclenche les signatures génétiques liées À la différenciation myéloÏde et À l'apoptose. Le cusatuzumab a le potentiel pour la recherche sur la leucémie myéloÏde aiguë (LMA).

-
GC63967
Cycleanine
La cycléanine est un puissant antagoniste sélectif du calcium vasculaire.
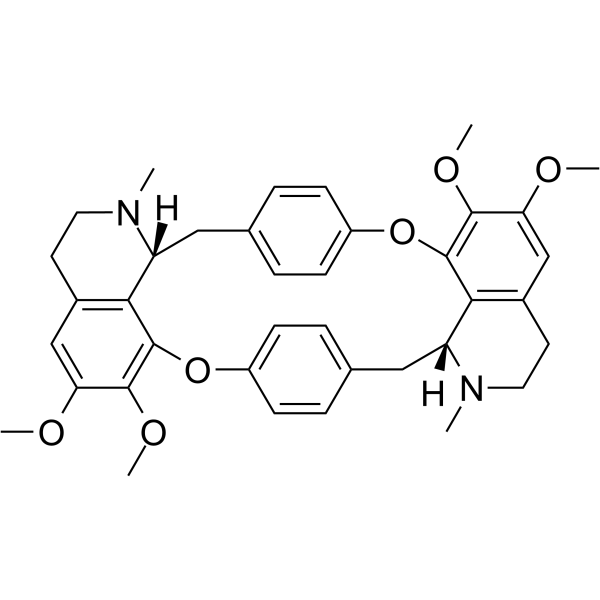
-
GC49716
Cyclo(RGDyK) (trifluoroacetate salt)
A cyclic peptide ligand of αVβ3 integrin
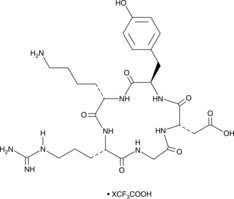
-
GC17198
Cycloheximide
Cycloheximide is an antibiotic that inhibits protein synthesis at the translation level, acting exclusively on cytoplasmic (80s) ribosomes of eukaryotes.
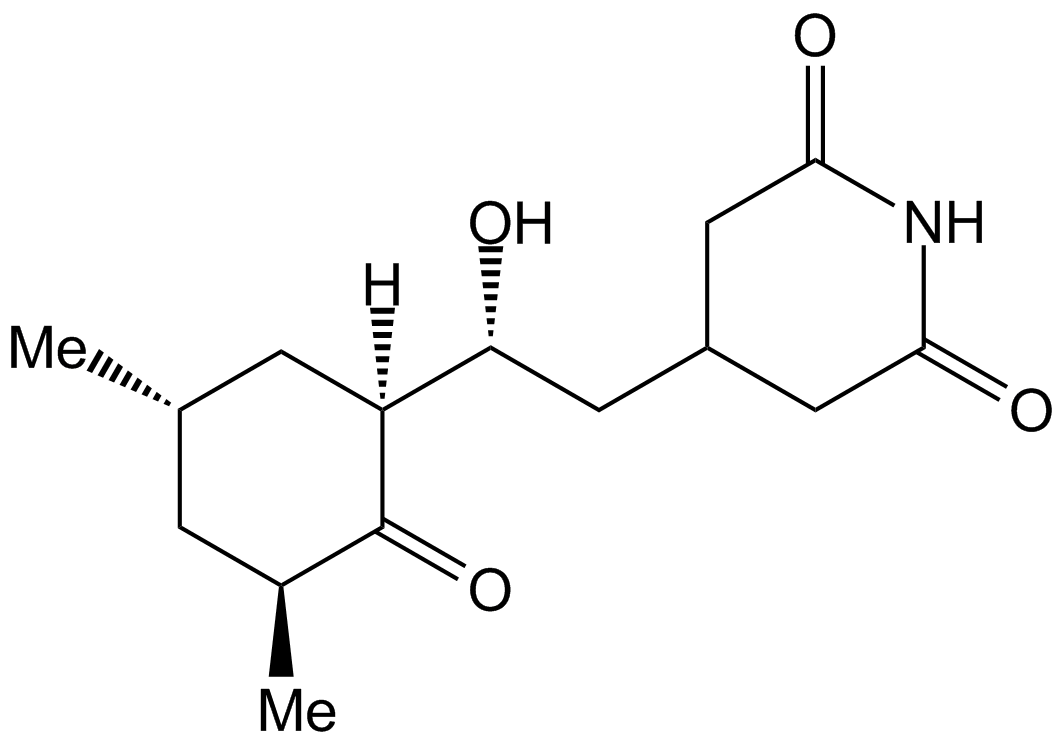
-
GC43346
Cyclopamine-KAAD
La cyclopamine-KAAD, un inhibiteur de la signalisation hedgehog, est un antagoniste lissé.

-
GC47148
Cyclophosphamide-d4
Le cyclophosphamide-d4 est le cyclophosphamide marqué au deutérium. Le cyclophosphamide est un agent alkylant synthétique apparenté chimiquement aux moutardes azotées À activité antinéoplasique, un immunosuppresseur.
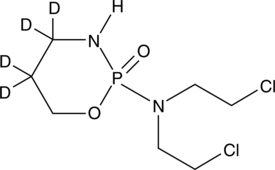
-
GC38419
Cyclovirobuxine D
La cyclovirobuxine D (CVB-D) est le principal composant actif de la médecine traditionnelle chinoise Buxus microphylla.
-
GC33330
Cynaropicrin
La cynaropicrine est une lactone sesquiterpénique qui peut inhiber la libération du facteur de nécrose tumorale (TNF-α) avec des CI50 de 8,24 et 3,18 μM pour les cellules macrophages murines et humaines, respectivement. La cynaropicrine inhibe également l'augmentation du facteur de dégradation du cartilage (MMP13) et supprime la signalisation NF-κB.
-
GC65565
Cyproheptadine
La cyproheptadine est un antagoniste des récepteurs 5-HT2A puissant et actif par voie orale, avec des effets antidépresseurs et antisérotoninergiques.
-
GC33779
Cysteamine (β-Mercaptoethylamine)
-
GC13502
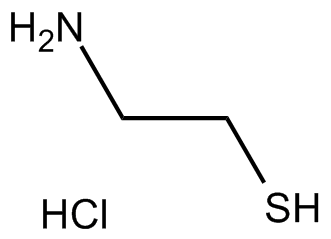
Cysteamine HCl
La cystéamine HCl (chlorhydrate de 2-aminoéthanethiol) est un agent actif par voie orale pour le traitement de la cystinose néphropathique et un antioxydant.
-
GC17050
CYT387
A potent inhibitor of JAK1 and JAK2
-
GC11383
CYT997 (Lexibulin)
CYT997 (Lexibuline) (CYT-997) est un inhibiteur de polymérisation de la tubuline puissant et actif par voie orale avec des CI50 de 10 À 100 nM dans les lignées cellulaires cancéreuses ; avec une puissante activité cytotoxique et perturbatrice vasculaire in vitro et in vivo. CYT997 (Lexibulin) induit l'apoptose cellulaire et induit la génération de ROS mitochondriales dans les cellules GC.
-
GC13070
Cytarabine
Agent cytotoxique, bloque la synthèse de l'ADN.
-
GC43356
CytoCalcein™ Violet 450
CytoCalcein? Violet 450 is a fluorogenic dye used to assess cell viability.
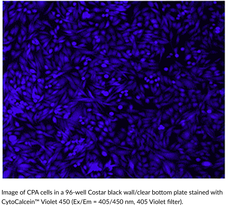
-
GC43357
CytoCalcein™ Violet 500
CytoCalcein? Violet 500 is a fluorogenic dye used to assess cell viability.
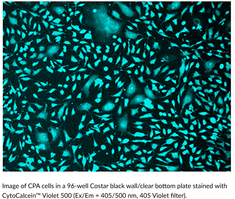
-
GC43361
Cytostatin (sodium salt)
Cytostatin is a natural antitumor inhibitor of cell adhesion to extracellular matrix, blocking adhesion of B16 melanoma cells to laminin and collagen type IV in vitro (IC50s = 1.3 and 1.4 μg/ml, respectively) and B16 cells metastatic activity in mice.
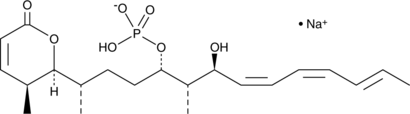
-
GC43368
D,L-1′-Acetoxychavicol Acetate
D,L-1′-Acetoxychavicol acetate is a natural compound first isolated from the rhizomes of ginger-like plants.
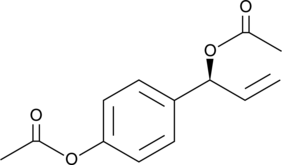
-
GC66824
D-α-Tocopherol Succinate
Le D-α-le succinate de tocophérol (succinate de vitamine E) est un tocophérol antioxydant et une forme saline de la vitamine E. Le D-α-le succinate de tocophérol inhibe la cytotoxicité induite par le cisplatine. D-α-Le succinate de tocophérol peut être utilisé pour la recherche sur le cancer.
-
GC12256
D-Mannitol
Le D-Mannitol est un diurétique osmotique et un faible vasodilatateur rénal.
-
GC60796
D-Trimannuronic acid
L'acide D-trimannuronique, un oligomère d'alginate est extrait des algues.
-
GC13202
D4476
Inhibitor of CK1 and ALK5
-
GC17851
D609
A competitive inhibitor of PC-specific PLC
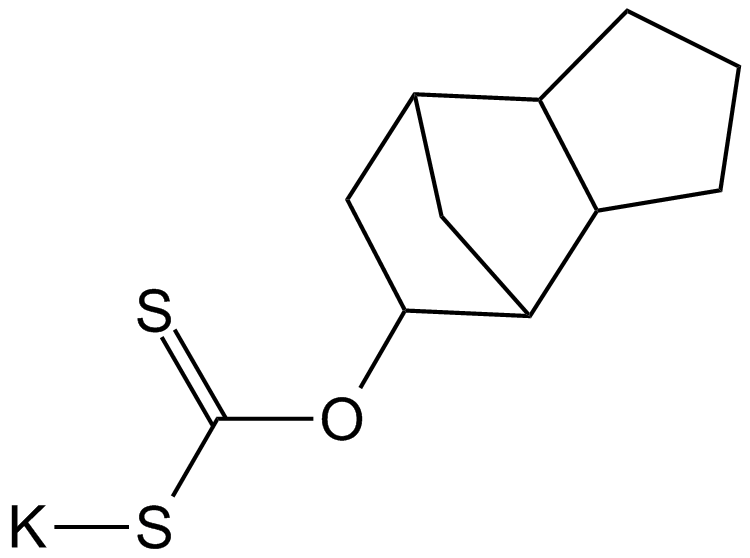
-
GC50296
D9
D9 est un inhibiteur puissant et sélectif de la thiorédoxine réductase (TrxR), avec une EC50 de 2,8 nM. D9 a la capacité d'inhiber la prolifération tumorale À la fois in vitro et in vivo.
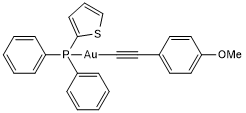
-
GC18421
Dabcyl-YVADAPV-EDANS
Dabcyl-YVADAPV-EDANS is a fluorogenic substrate for caspase-1.
-
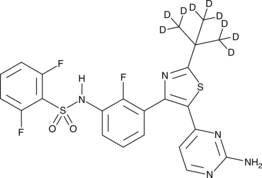
GC47166
Dabrafenib-d9
Dabrafenib-d9 (GSK2118436A-d9) est le Dabrafenib marqué au deutérium. Dabrafenib (GSK2118436A) est un inhibiteur compétitif de l'ATP de Raf avec des IC50 de 5 nM et 0,6 nM pour C-Raf et B-RafV600E, respectivement.
-
GC14485
Dacarbazine
La dacarbazine est un agent alkylant antinéoplasique non spécifique du cycle cellulaire. La dacarbazine inhibe la réponse lymphoblastique T et B, avec des valeurs d'IC50 de 50 et 10 μg/ml, respectivement. La dacarbazine peut être utilisée pour la recherche du mélanome malin métastatique.
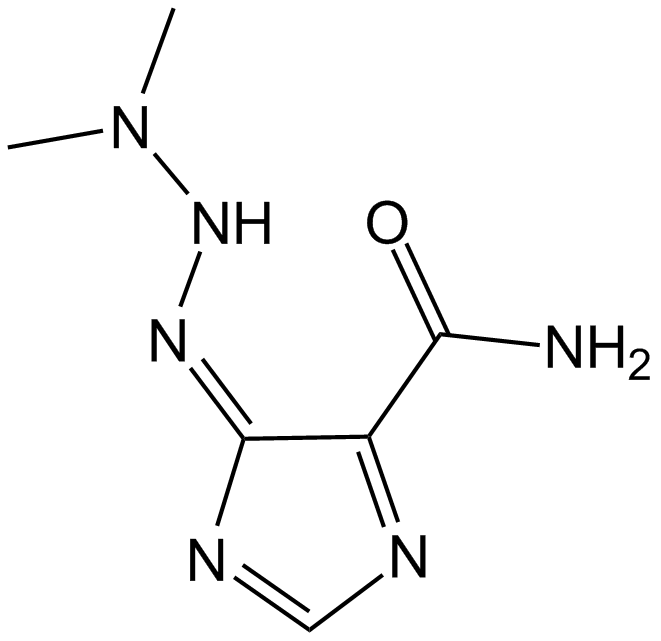
-
GC47167
Dacarbazine-d6
La dacarbazine-d6 (imidazole carboxamide-d6) est la dacarbazine marquée au deutérium. La dacarbazine (DTIC-Dome ; DTIC) est un agent antinéoplasique. Il a une activité importante contre les mélanomes.
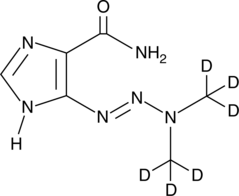
-
GC68305
Dacetuzumab

-
GC10225
Dacomitinib (PF299804, PF299)
Dacomitinib (PF299804, PF299) (PF-00299804) est un inhibiteur spécifique et irréversible de la famille des kinases ERBB avec des IC50 de 6 nM, 45,7 nM et 73,7 nM pour EGFR, ERBB2 et ERBB4, respectivement.
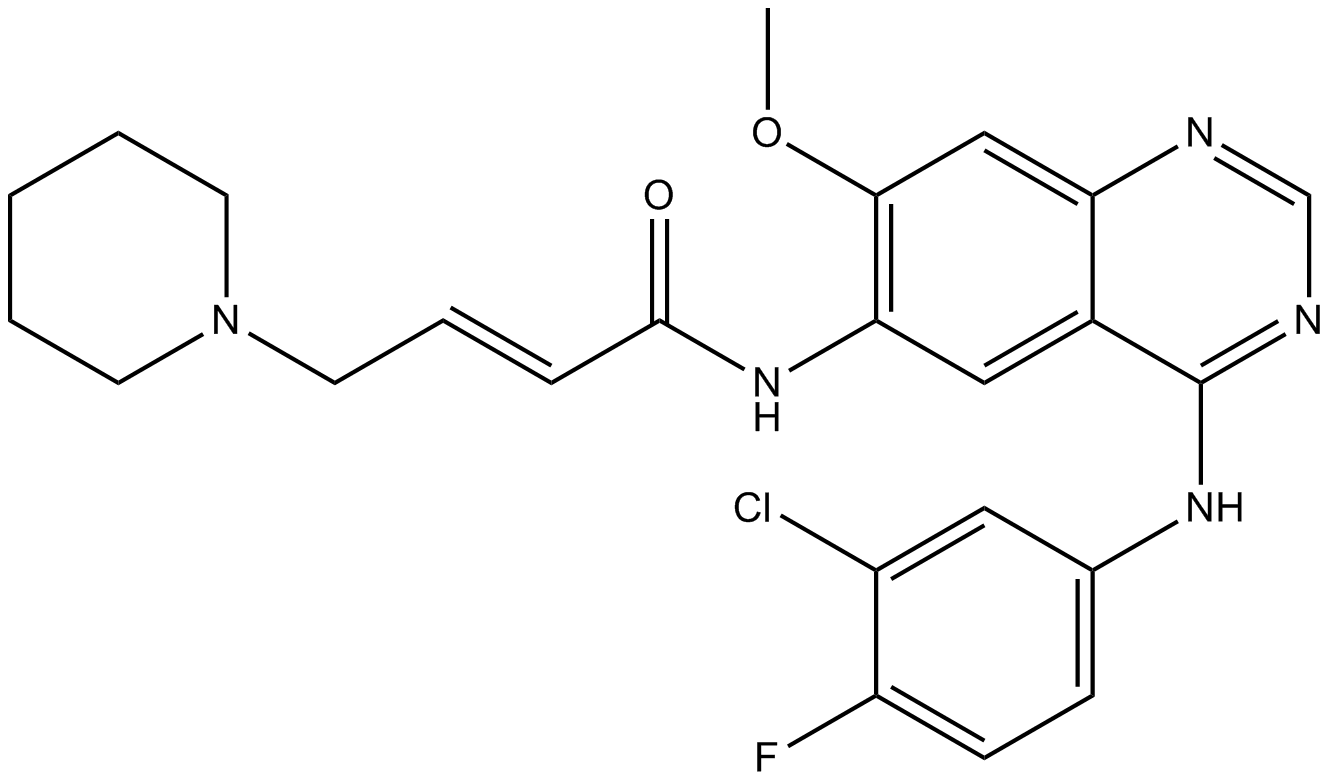
-
GC15211
Damnacanthal
Le damnacanthal est une anthraquinone isolée de la racine de Morinda citrifolia.
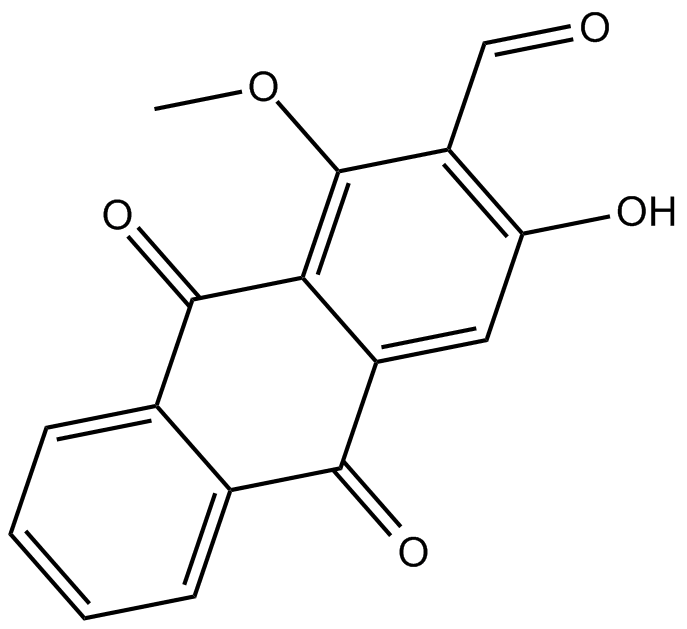
-
GN10318
Danshensu
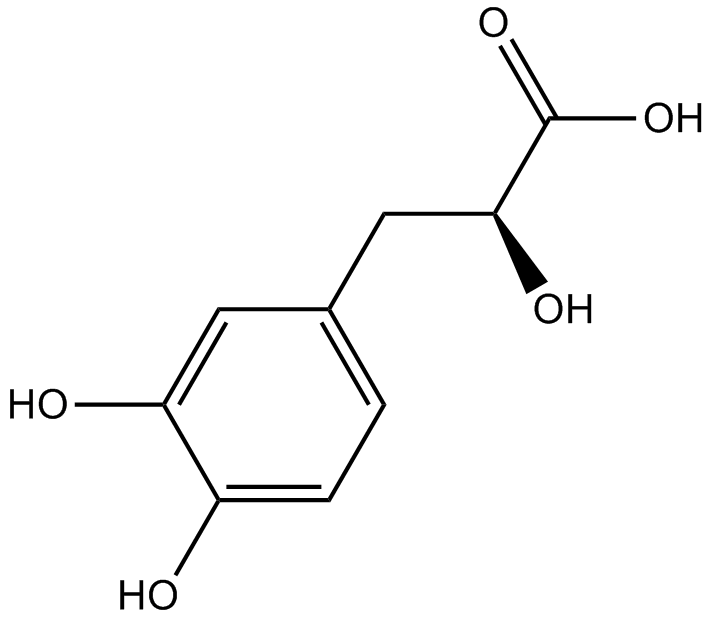
-
GC34010
Danshensu (Dan shen suan A)
Danshensu, an active ingredient of Salvia miltiorrhiza, shows wide cardiovascular benefit by activating Nrf2 signaling pathway.
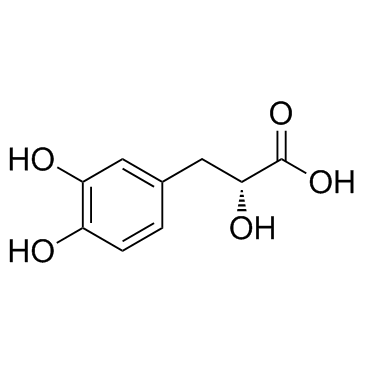
-
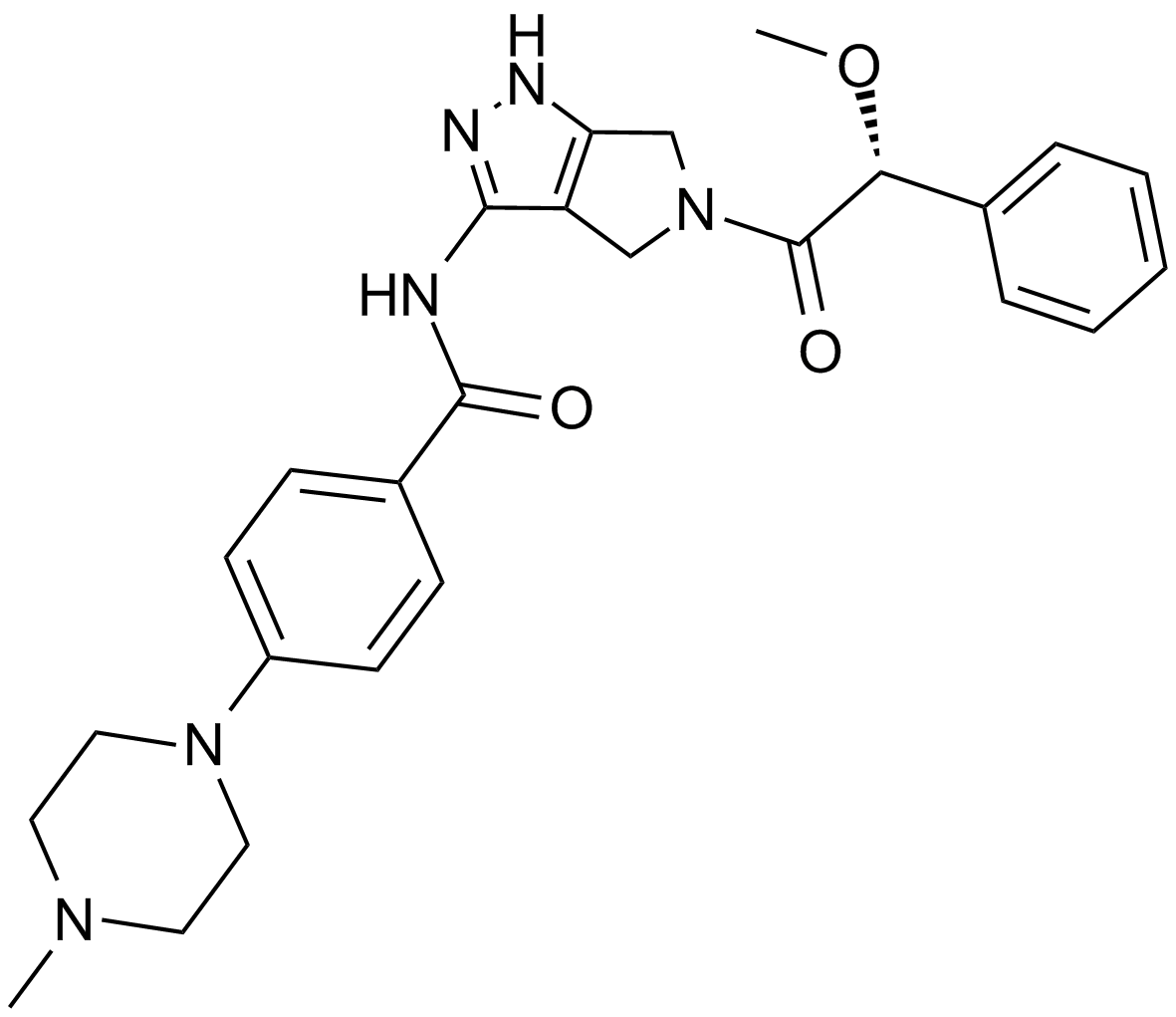
GC15217
Danusertib (PHA-739358)
A pan-Aurora kinase and Abl inhibitor
-
GC17650
DAPK Substrate Peptide
A DAPK1 peptide substrate
-
GC49883
DAPK Substrate Peptide (trifluoroacetate salt)
A DAPK1 peptide substrate

-
GC12942
DAPT (GSI-IX)
Inhibiteur de la γ-sécrétase
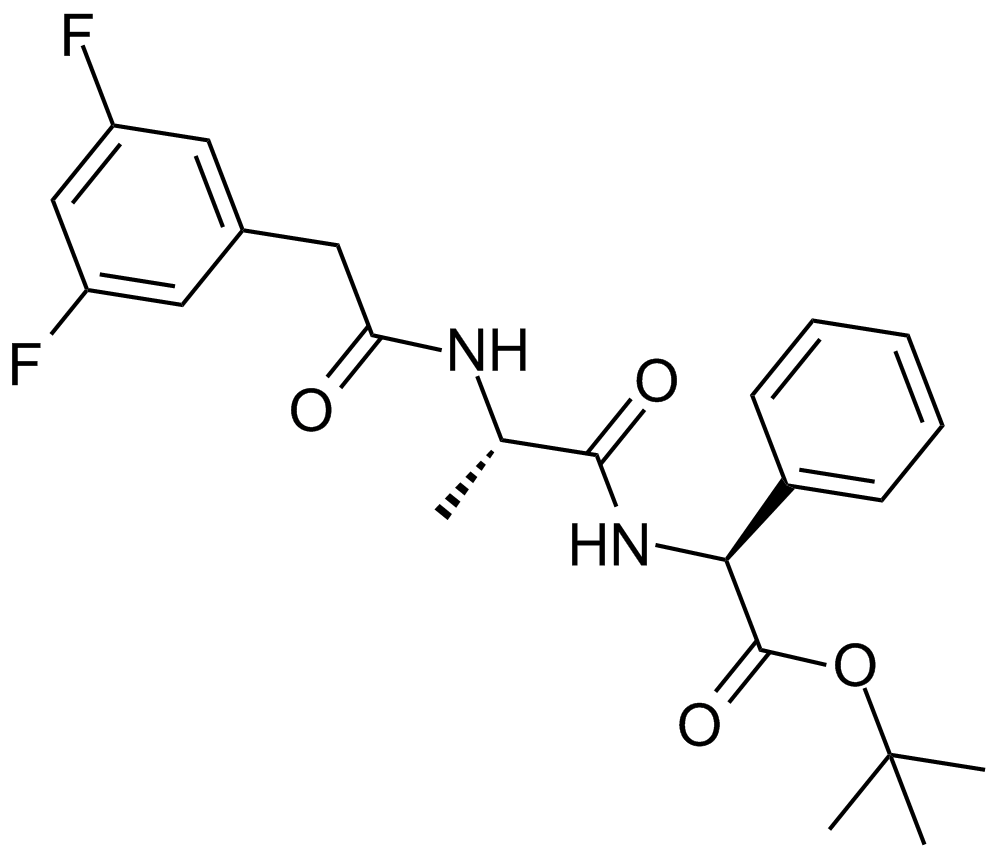
-
GC43379
Darinaparsin
La darinaparsine (ZIO-101), un arsenical organique, est un agent ciblant les mitochondries. La dariparsine induit l'apoptose dans les cellules ancéreuses et a des effets anticancéreux.
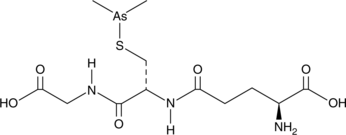
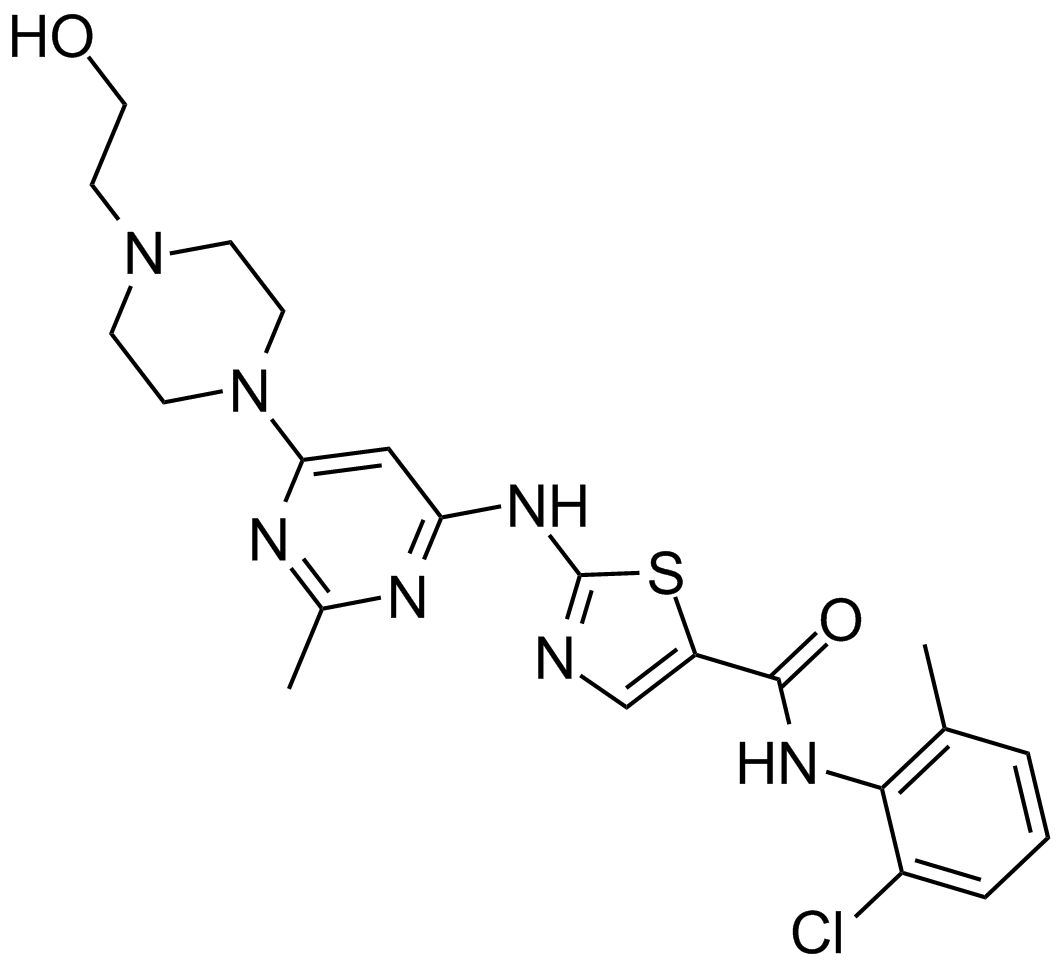
-
GC15568
Dasatinib (BMS-354825)
Un inhibiteur d'Abl et de Src.
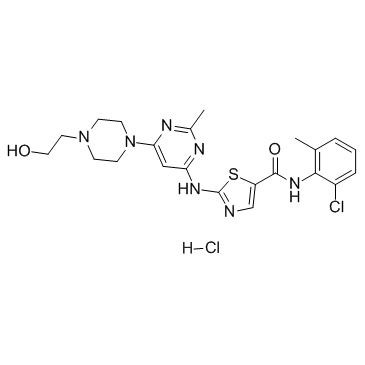
-
GC35812
Dasatinib hydrochloride
Le chlorhydrate de dasatinib (BMS-354825) est un inhibiteur double Src/Bcr-Abl très puissant, compétitif pour l'ATP, actif par voie orale et doté d'une puissante activité antitumorale. Les Kis sont respectivement de 16 h et 30 h pour Src et Bcr-Abl. Le chlorhydrate de dasatinib inhibe Bcr-Abl et Src avec des IC50 <1,0 nM et 0,5 nM, respectivement. Le chlorhydrate de dasatinib induit également l'apoptose et l'autophagie.
-
GC10354
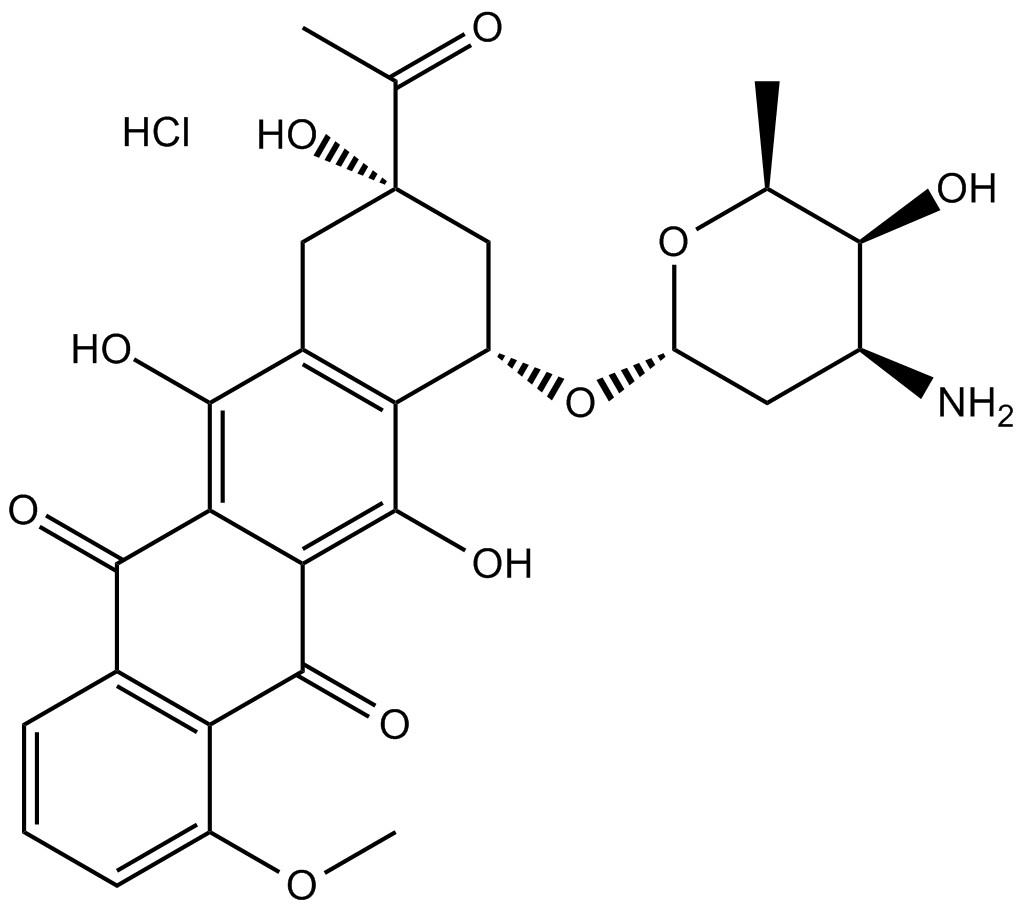
Daunorubicin HCl
Le chlorhydrate de daunorubicine (daunomycine) est un inhibiteur de la topoisomérase II doté d'une puissante activité antitumorale.