Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Ziele für Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Produkte für Apoptosis
- Bestell-Nr. Artikelname Informationen
-
GC43274
Citromycetin
Citromycetin ist eine aromatische Polyketidverbindung aus dem australischen Meerwasser stammenden und terrestrischen Penicillium spp.

-
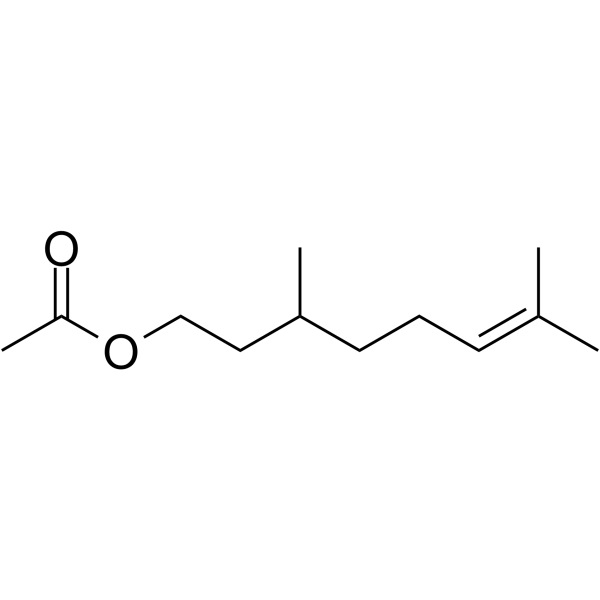
GC63393
Citronellyl acetate
Citronellylacetat ist ein Monoterpenprodukt des SekundÄrstoffwechsels von Pflanzen mit antinozizeptiver Wirkung.
-
GC52367
Citrullinated Vimentin (G146R) (R144 + R146) (139-159)-biotin Peptide
A biotinylated and citrullinated mutant vimentin peptide

-
GC52370
Citrullinated Vimentin (R144) (139-159)-biotin Peptide
A biotinylated and citrullinated vimentin peptide

-
GN10219
Ciwujianoside-B
-
GC64649
Cjoc42
Cjoc42 ist eine Verbindung, die an Gankyrin binden kann. Cjoc42 hemmt die Gankyrin-AktivitÄt dosisabhÄngig. Cjoc42 verhindert die Abnahme des p53-Proteinspiegels, der normalerweise mit hohen Gankyrin-Mengen verbunden ist. Cjoc42 stellt die p53-abhÄngige Transkription und Empfindlichkeit gegenÜber DNA-SchÄden wieder her.
-
GC39485
CK2/ERK8-IN-1
CK2/ERK8-IN-1 ist ein dualer Caseinkinase 2 (CK2) (Ki von 0,25 μM) und ERK8 (MAPK15, ERK7) Inhibitor mit IC50s von 0,50 μM. CK2/ERK8-IN-1 bindet auch an PIM1, HIPK2 (homÖodomÄnen-interagierende Proteinkinase 2) und DYRK1A mit Kis von 8,65 μM, 15,25 μM bzw. 11,9 μM. CK2/ERK8-IN-1 hat pro-apoptotische Wirksamkeit.
-
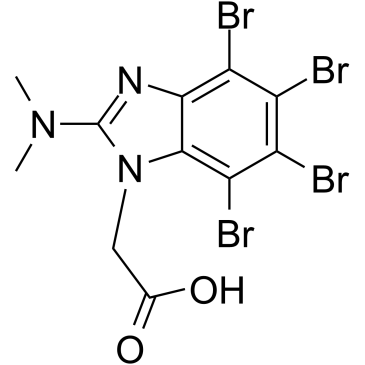
GC49556
Cl-Necrostatin-1
A RIPK1 inhibitor
-
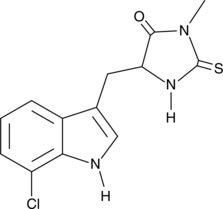
GC47098
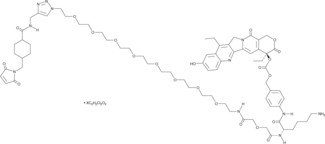
CL2-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
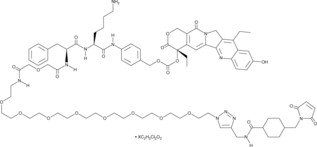
GC60714
CL2A-SN-38
CL2A-SN-38 ist ein Wirkstoff-Linker-Konjugat, das aus einem potenten DNA-Topoisomerase-I-Inhibitor SN-38 und einem Linker CL2A zur Herstellung eines AntikÖrper-Wirkstoff-Konjugats (ADC) besteht.
-
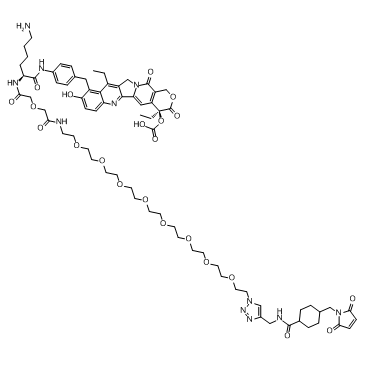
GC52469
CL2A-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
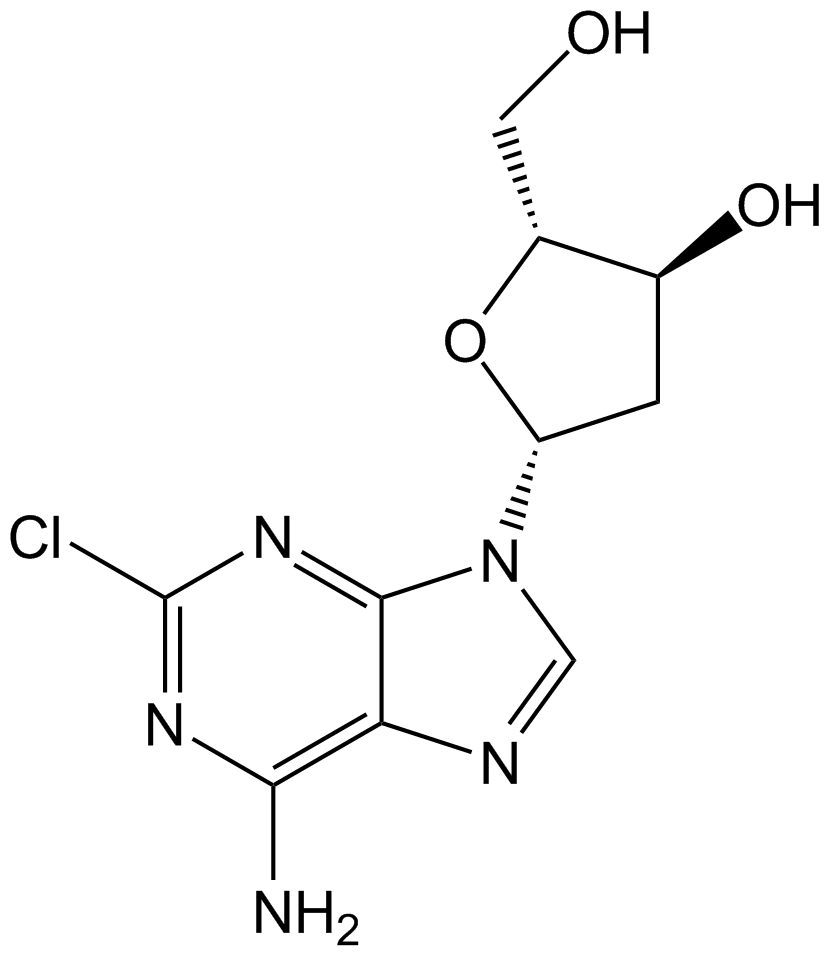
GC10509
Cladribine
Cladribin (2-Chlor-2′-desoxyadenosin), ein Purin-Nukleosid-Analogon, ist ein oral wirksamer Adenosin-Desaminase-Hemmer.
-
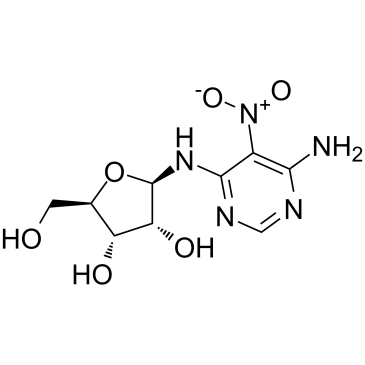
GC60111
Clitocine
Clitocin, ein aus Pilzen isoliertes Adenosin-Nucleosid-Analogon, ist ein starkes und wirksames Durchlesemittel. Clitocin wirkt als UnterdrÜcker von Nonsense-Mutationen und kann die Produktion von p53-Protein in Zellen induzieren, die Nonsense-mutierte p53-Allele beherbergen. Clitocin kann Apoptose in multiresistenten menschlichen Krebszellen induzieren, indem es auf Mcl-1 abzielt. AktivitÄt gegen Krebs.
-
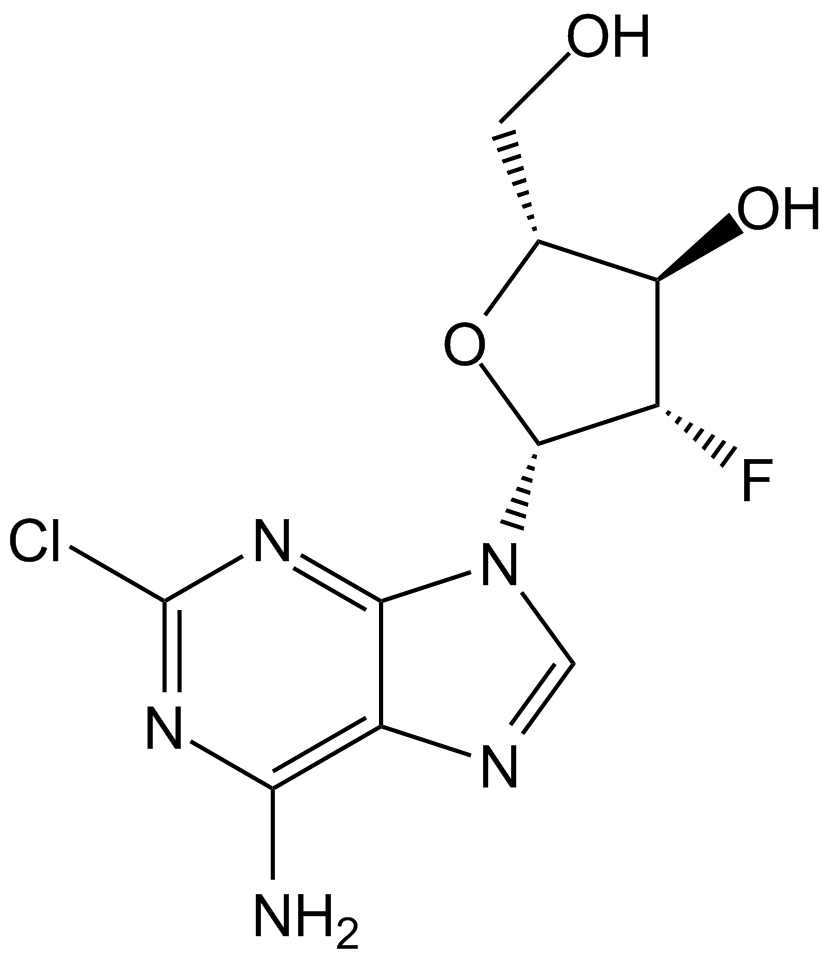
GC15219
Clofarabine
Clofarabin, ein Nukleosid-Analogon fÜr die Krebsforschung, ist ein potenter Inhibitor der Ribonukleotid-Reduktase (IC50=65 nM), indem es an die allosterische Stelle der regulatorischen Untereinheit bindet.
-
GC10813
Clofibric Acid
ClofibrinsÄure (ChlorofibrinsÄure), der pharmazeutisch aktive Metabolit der Lipidregulatoren Clofibrat, Etofibrat und Etofyllinclofibrat, ist ein PPARα-Agonist, der hypolipidÄmische Wirkungen zeigt.
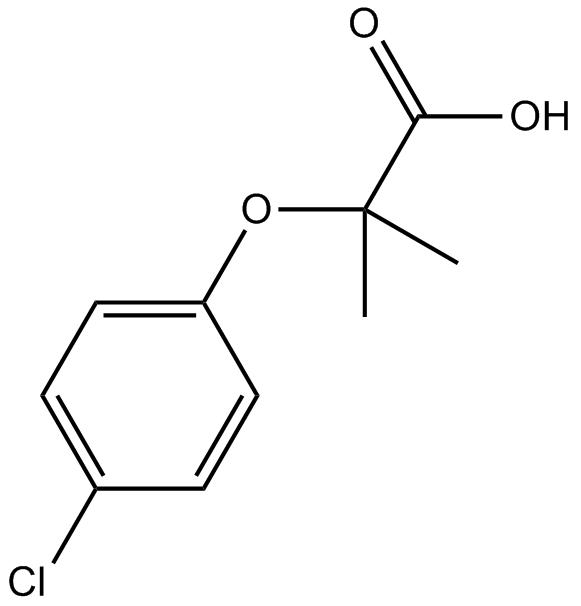
-
GC32587
Clofilium tosylate
Clofiliumtosylat, ein Kaliumkanalblocker, induziert die Apoptose menschlicher PromyelozytenleukÄmie (HL-60)-Zellen Über Bcl-2-unempfindliche Aktivierung von Caspase-3.
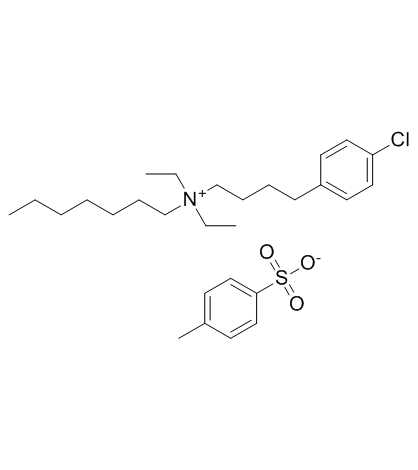
-
GC47105
Clonostachydiol
Clonostachydiol ist ein anthelmintisches Makrodiolid aus dem Pilz Clonostachys cylindrospora (Stamm FH-A 6607).
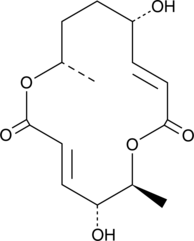
-
GC12367
CM-272
CM-272 ist ein erstklassiger, potenter, selektiver, substratkompetitiver und reversibler dualer G9a/DNA-Methyltransferase (DNMTs)-Inhibitor mit AntitumoraktivitÄten. CM-272 hemmt G9a, DNMT1, DNMT3A, DNMT3B und GLP mit IC50-Werten von 8 nM, 382 nM, 85 nM, 1200 nM bzw. 2 nM. CM-272 hemmt die Zellproliferation und fÖrdert die Apoptose, wodurch IFN-stimulierte Gene und immunogener Zelltod induziert werden.
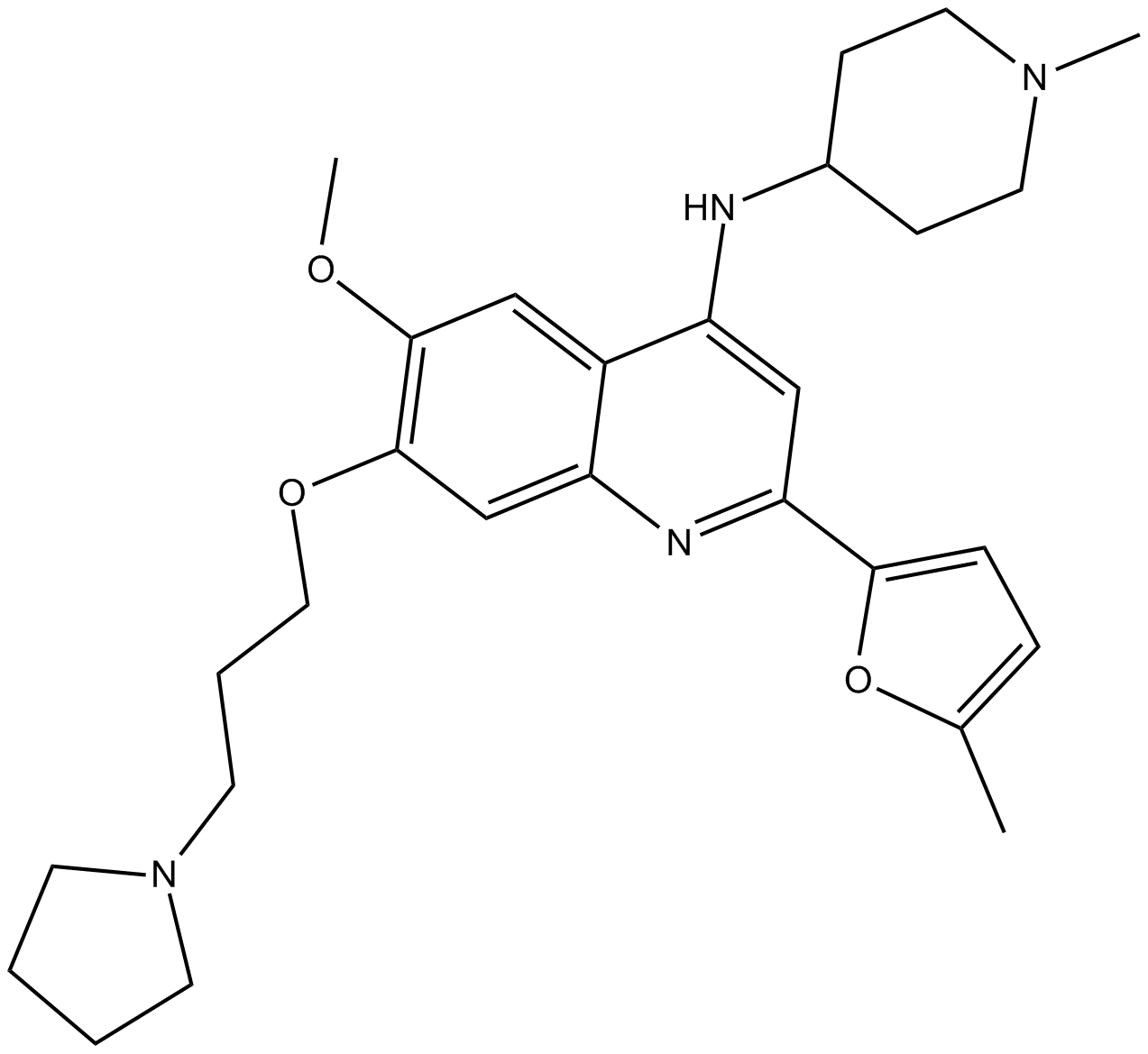
-
GC62347
CMC2.24
CMC2.24 (TRB-N0224), ein oral aktiver Tricarbonylmethan-Wirkstoff, ist bei MÄusen wirksam gegen BauchspeicheldrÜsentumoren, indem er die Ras-Aktivierung und den nachgeschalteten Effektor-ERK1/2-Weg hemmt.
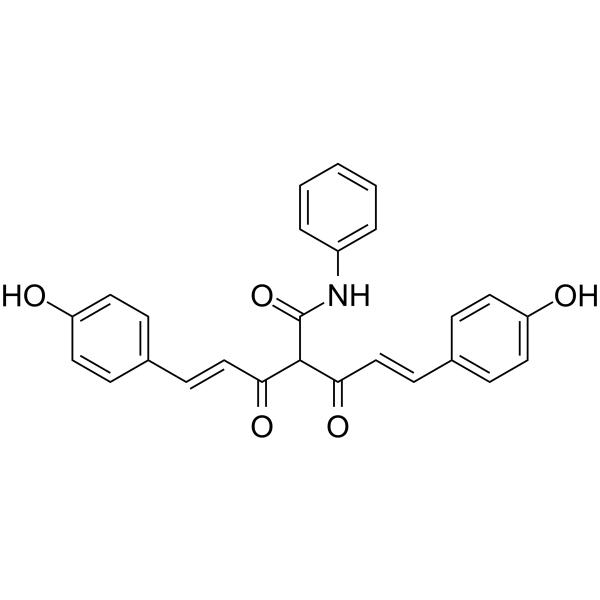
-
GC61567
CMLD-2
CMLD-2, ein Inhibitor der HuR-ARE-Wechselwirkung, bindet kompetitiv an das HuR-Protein, wodurch seine Wechselwirkung mit Adenin-Uridin-reichen Elementen (ARE)-enthaltenden mRNAs (Ki = 350 nM) unterbrochen wird. CMLD-2 induziert Apoptose und zeigt AntitumoraktivitÄt in verschiedenen Krebszellen wie Dickdarm-, BauchspeicheldrÜsen-, SchilddrÜsen- und Lungenkrebs-Zelllinien. Hu-Antigen R (HuR) ist ein RNA-bindendes Protein, das die StabilitÄt und Translation von Ziel-mRNAs regulieren kann.
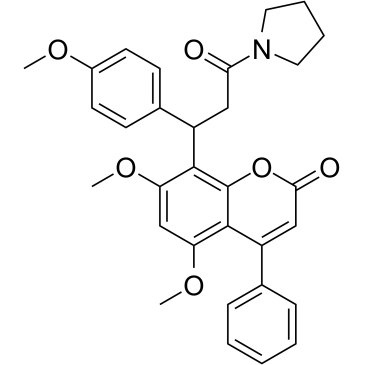
-
GC49096
Cobaltic Protoporphyrin IX (chloride)
An inducer of HO-1 activity
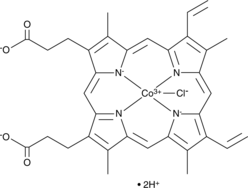
-
GC10033
Cobimetinib
A potent, orally available MEK1 inhibitor
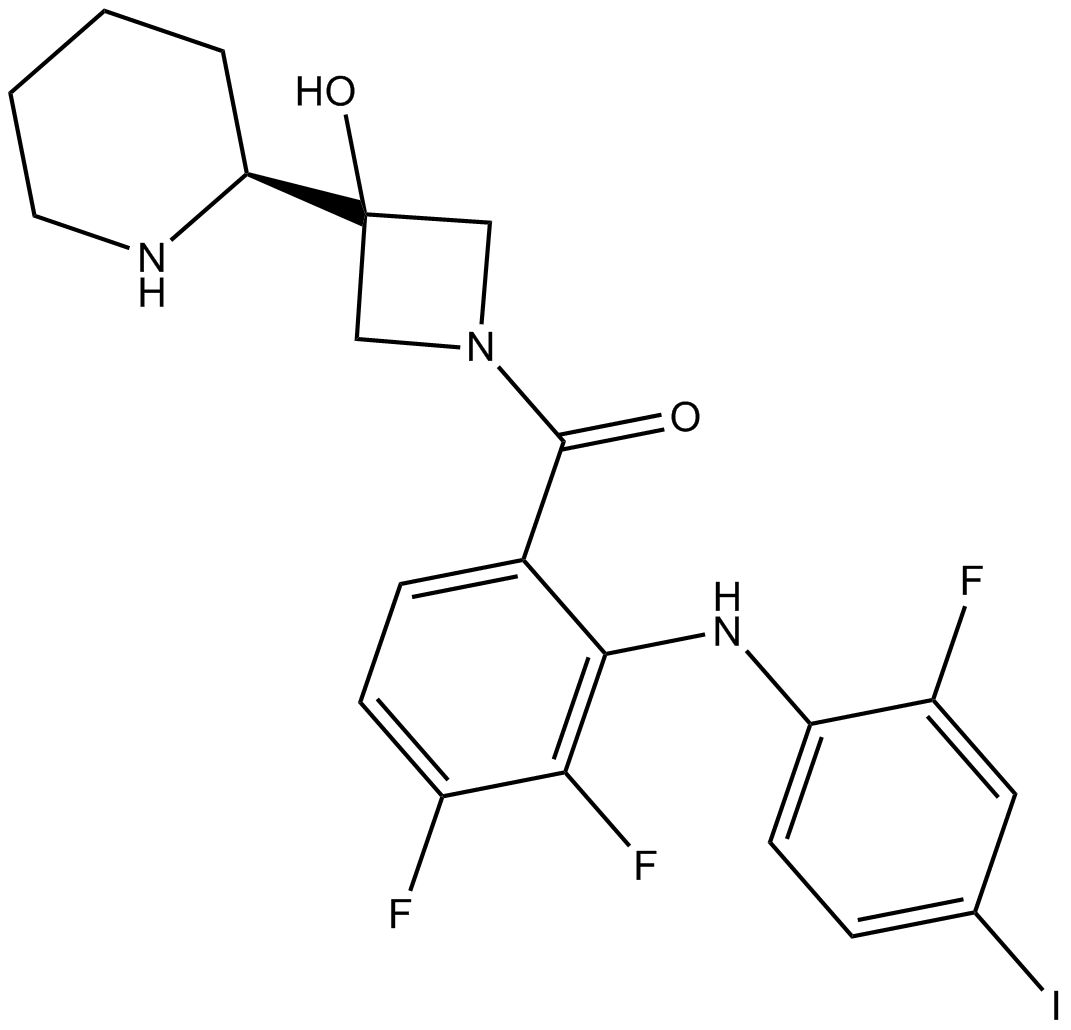
-
GC43297
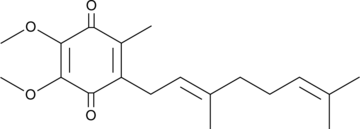
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
-
GC62192
COG1410
COG1410 ist ein von Apolipoprotein E abgeleitetes Peptid.
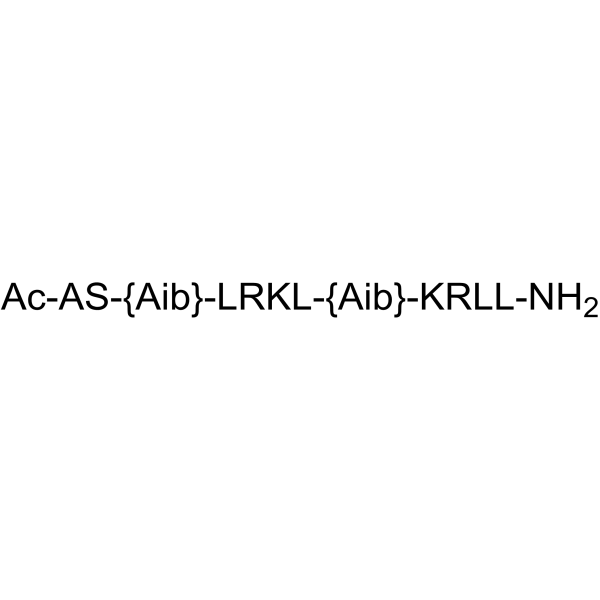
-
GC40664
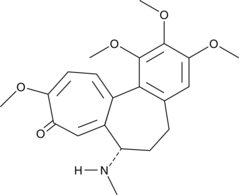
Colcemid
Colcemid ist ein Zytoskelett-Inhibitor, der in Säugetierzellen oder Oozyten eine mitotische Arrestierung in der G2/M-Phase oder eine meiotische Arrestierung in der Vesikelruptur (GVBD)-Phase induziert.
-
GN10123
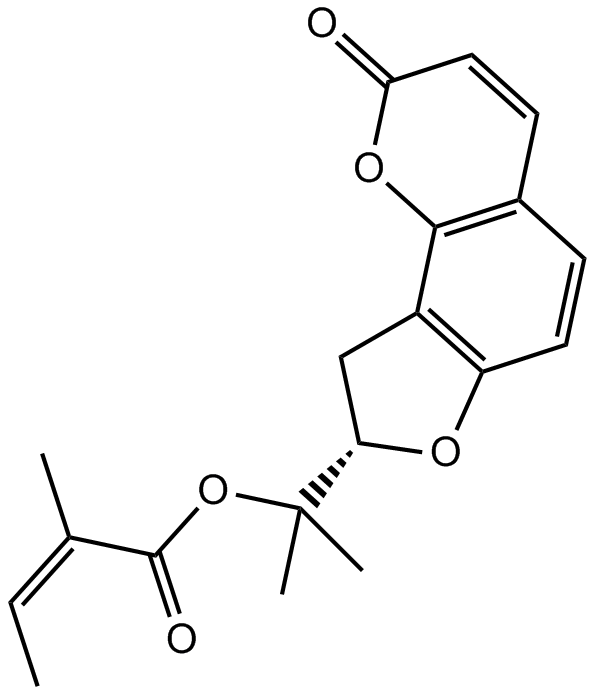
Columbianadin
-
GC49454
Complex 3
A fluorescent copper complex with anticancer activity
-
GC18572
Concanavalin A
Concanamycin A gehört zu den Concanamycinen, einer Familie von Makrolid-Antibiotika, die aus Streptomyces diastatochromogenes isoliert wurden und hochaktive und selektive Hemmstoffe der Vakuolären Protonen-ATPase (v-[H+]ATPase) sind.

-
GC18832
Conglobatin
Conglobatin is a dimeric macrolide dilactone originally isolated from S.
-
GC48483
Conglobatin B
A bacterial metabolite
-
GC48497
Conglobatin C1
A bacterial metabolite
-
GC38376
Coniferaldehyde
Coniferaldehyd (Ferulaldehyd) ist ein wirksamer Induktor der HÄmoxygenase-1 (HO-1).
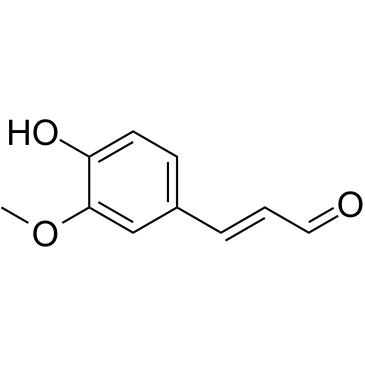
-
GC63379
Conophylline
Conophyllin ist ein Vinca-Alkaloid, das aus BlÄttern der tropischen Pflanze Ervatamia microphylla gewonnen wird.
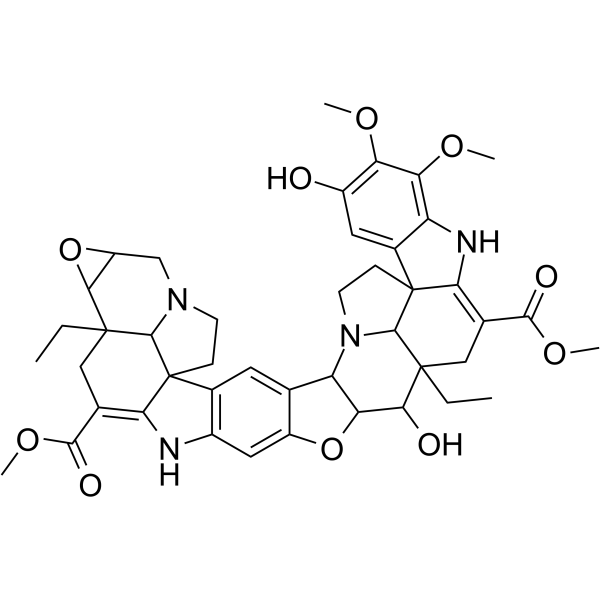
-
GC16772
Cortisone acetate
Cortisonacetat (Cortison 21-Acetat), ein oxidierter Metabolit von Cortisol (ein Glucocorticoid).
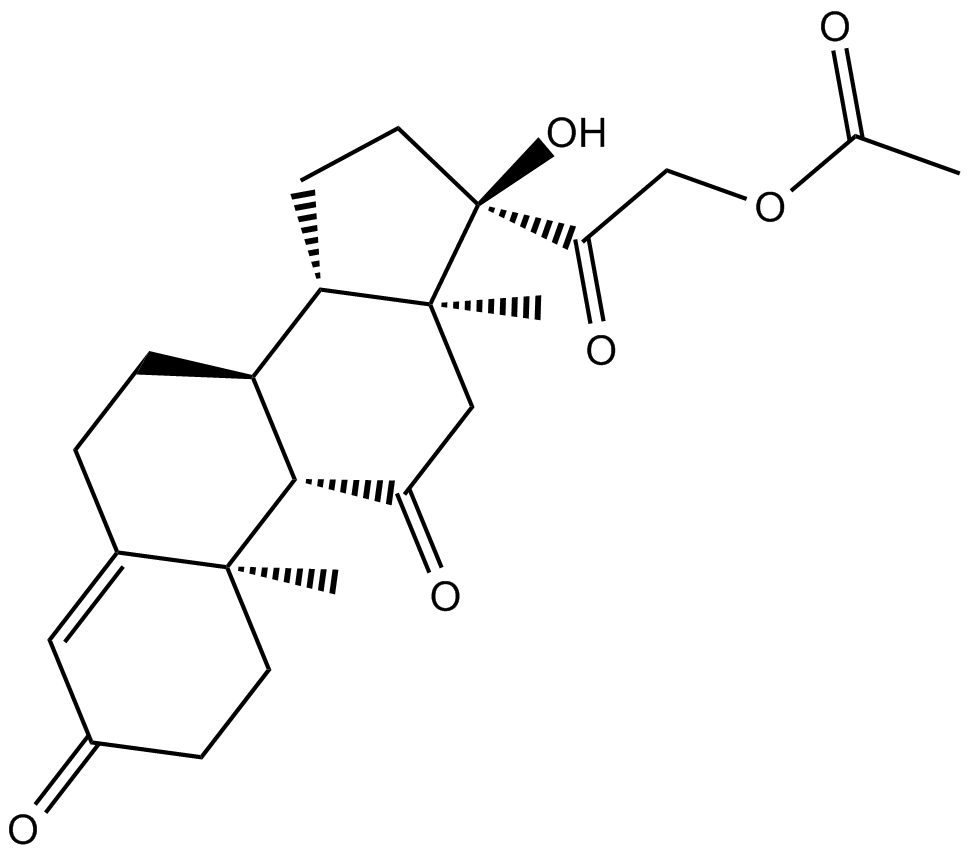
-
GC16116
Costunolide
A natural sesquiterpene lactone
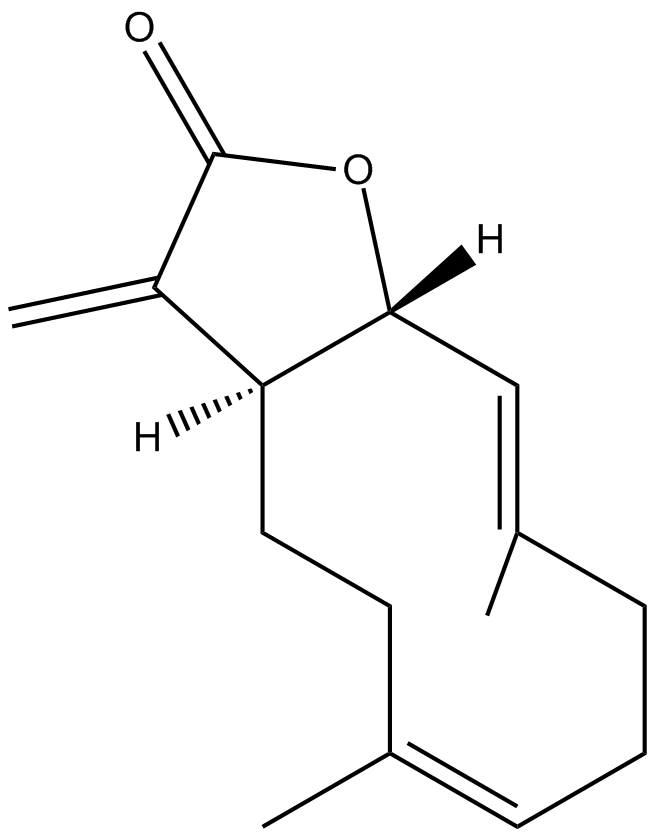
-
GC15225
COTI-2
COTI-2, ein Anti-Krebs-Medikament mit geringer ToxizitÄt, ist ein oral verfÜgbarer Aktivator der dritten Generation von p53-Mutantenformen. COTI-2 wirkt sowohl durch die Reaktivierung des mutierten p53 als auch durch die Hemmung des PI3K/AKT/mTOR-Signalwegs. COTI-2 induziert Apoptose in mehreren menschlichen Tumorzelllinien. COTI-2 zeigt AntitumoraktivitÄt bei HNSCC durch p53-abhÄngige und -unabhÄngige Mechanismen. COTI-2 wandelt mutiertes p53 in die Wildtyp-Konformation um.
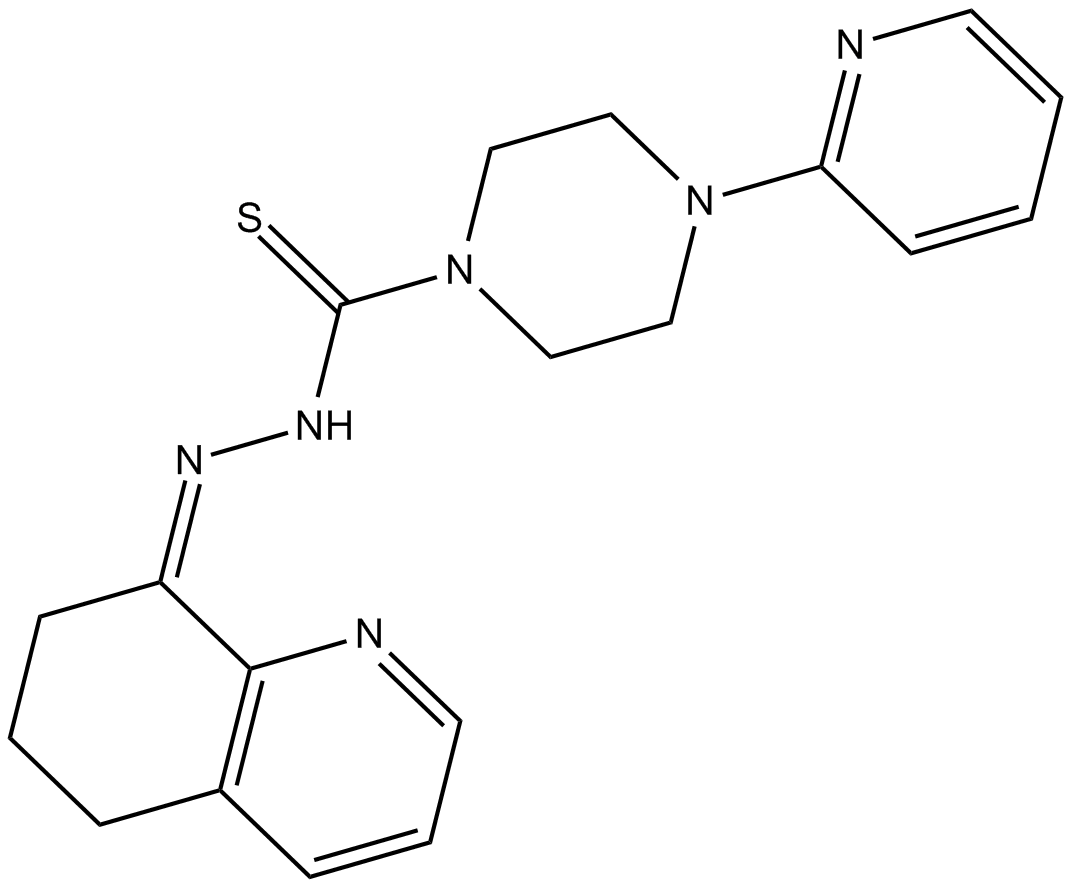
-
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
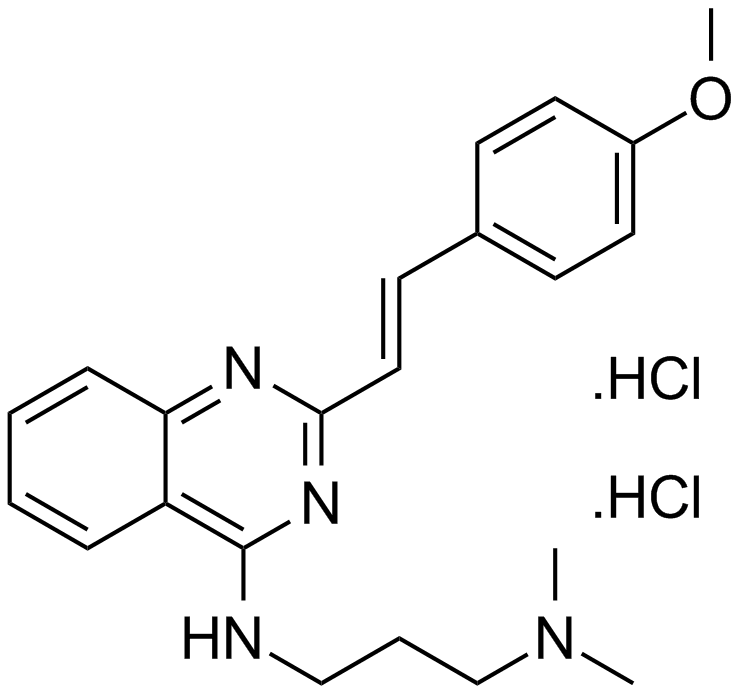
-
GC13091
CP-724714
CP-724714 ist ein potenter, selektiver und oral aktiver ErbB2 (HER2)-Tyrosinkinase-Inhibitor mit einem IC50 von 10 nM. CP-724714 zeigt eine ausgeprÄgte SelektivitÄt gegenÜber EGFR-Kinase (IC50 = 6400 nM). CP-724714 hemmt wirksam die Autophosphorylierung des ErbB2-Rezeptors in intakten Zellen. Antitumor-AktivitÄten.
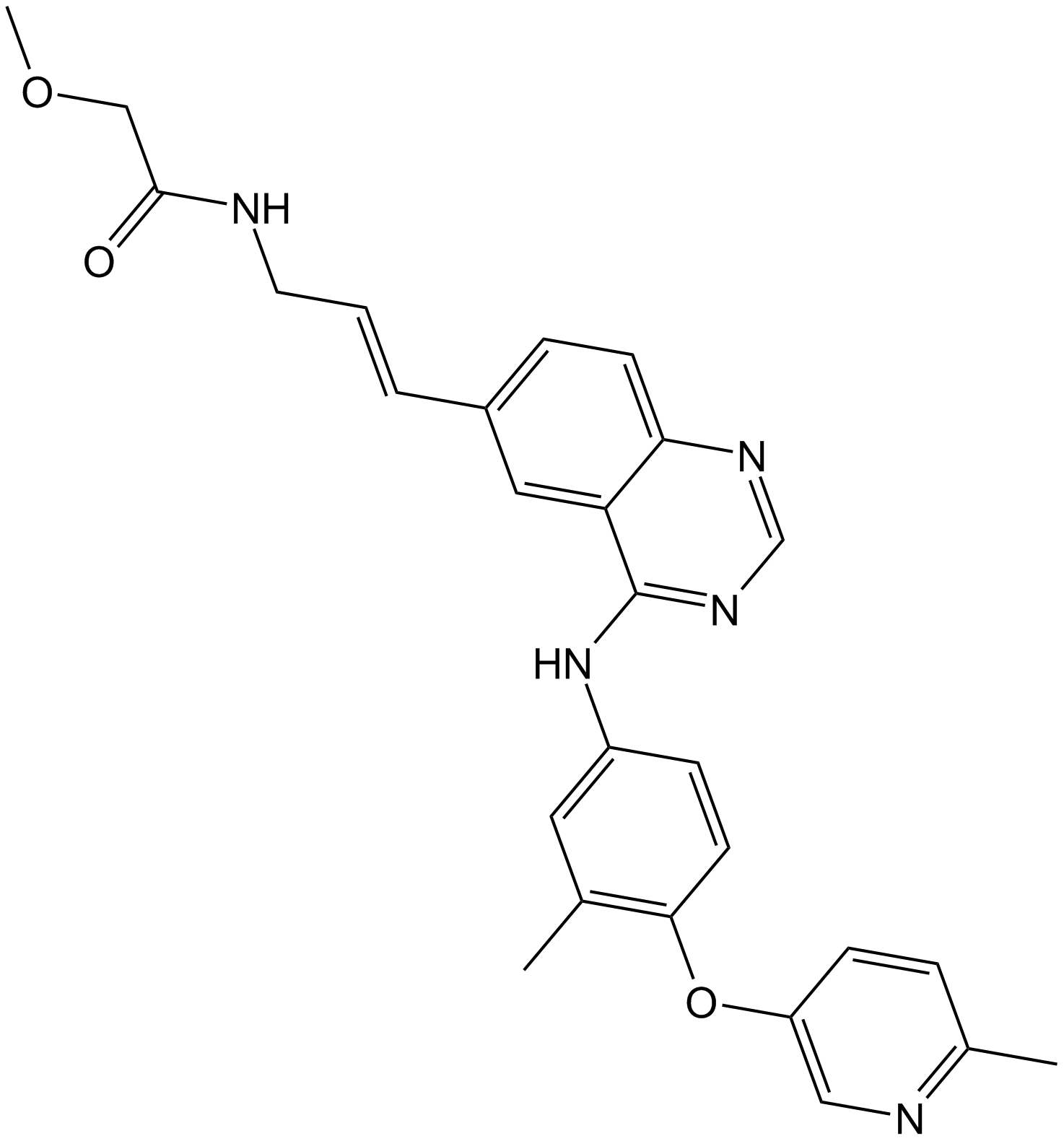
-
GC14500
CPI-1189
CPI-1189 ist ein Inhibitor der Freisetzung von TNF-α mit antioxidativen und neuroprotektiven Eigenschaften.
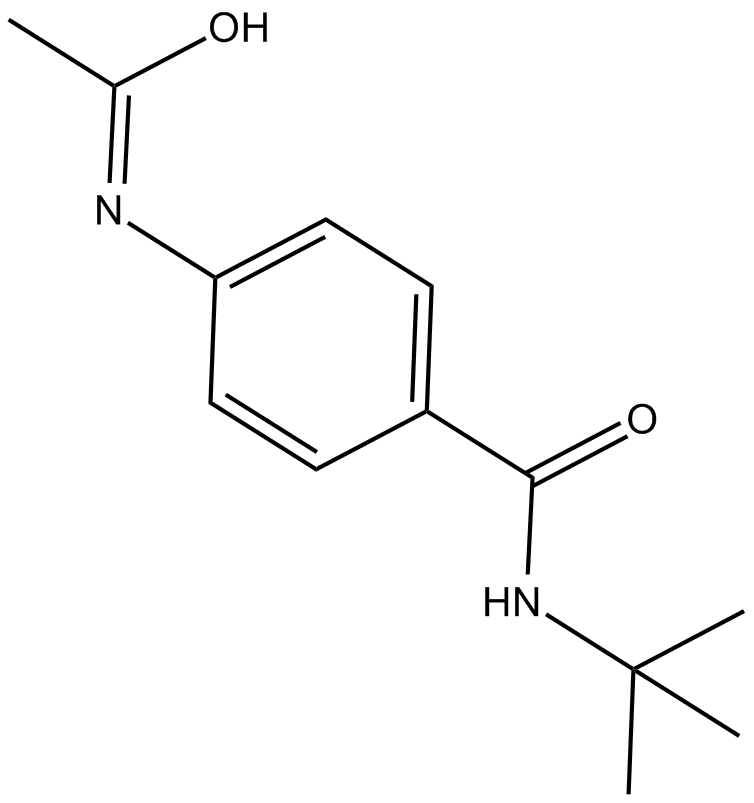
-
GC14699
CPI-203
CPI-203 ist ein neuartiger potenter, selektiver und zellgÄngiger Inhibitor der BET-BromodomÄne mit einem IC50-Wert von ca. 37 nM (BRD4 α-Screen-Assay).
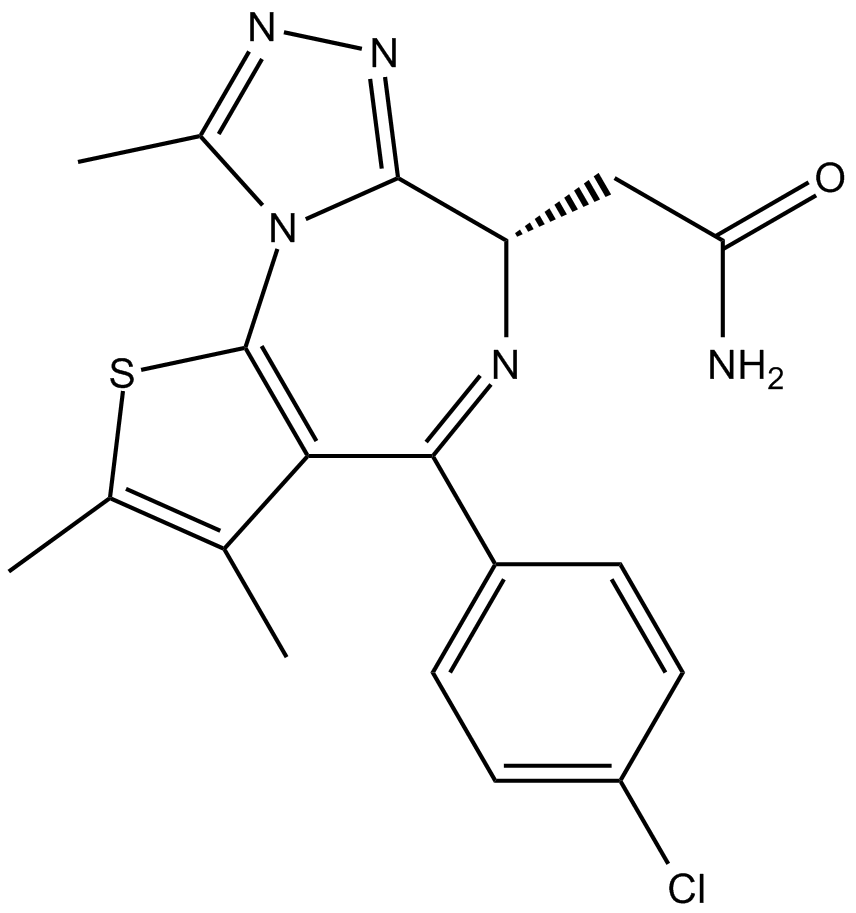
-
GC10021
CPI-360
EZH2 inhibitor
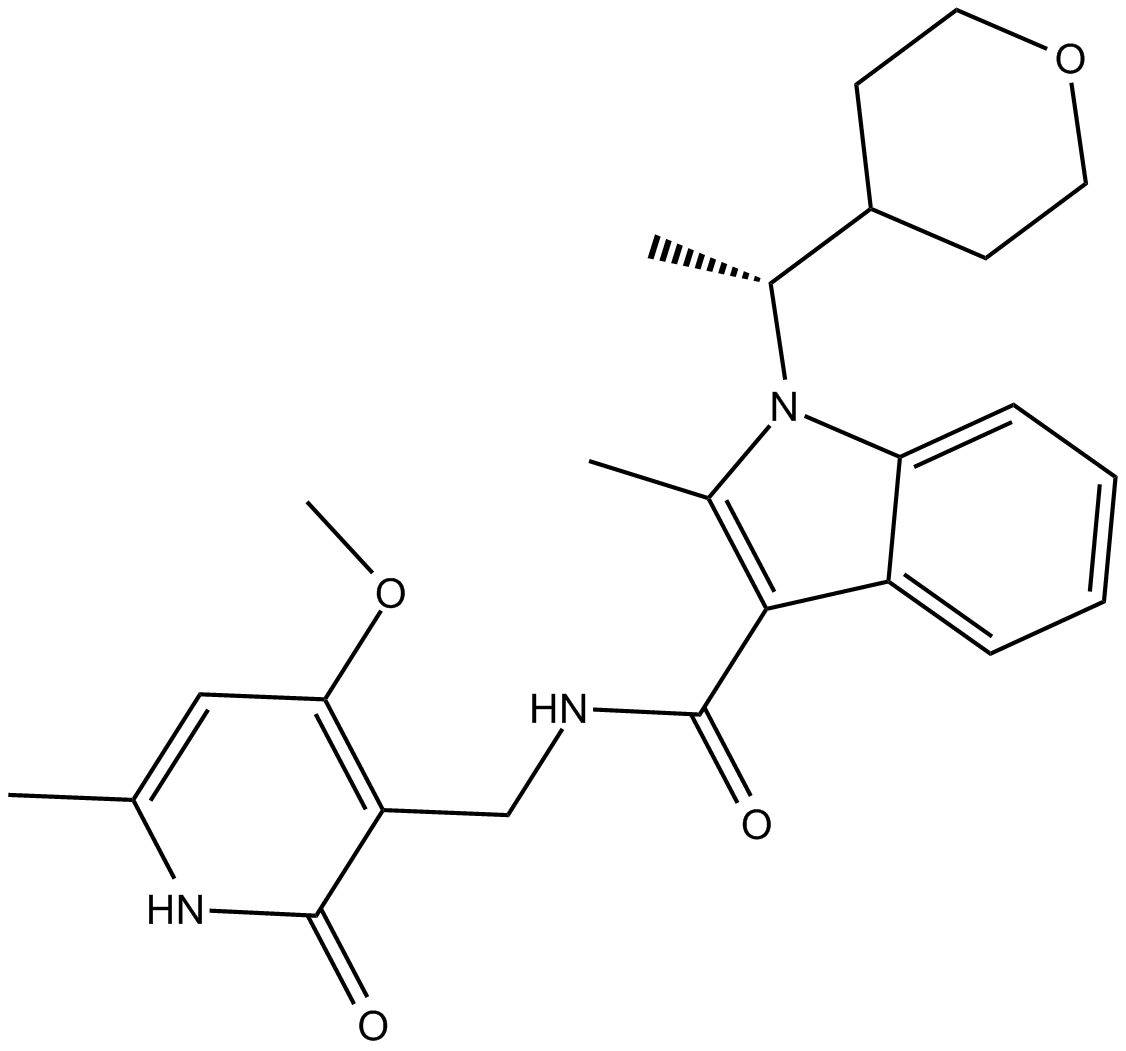
-
GC14921
CPI-613
An inhibitor of α-ketoglutarate dehydrogenase
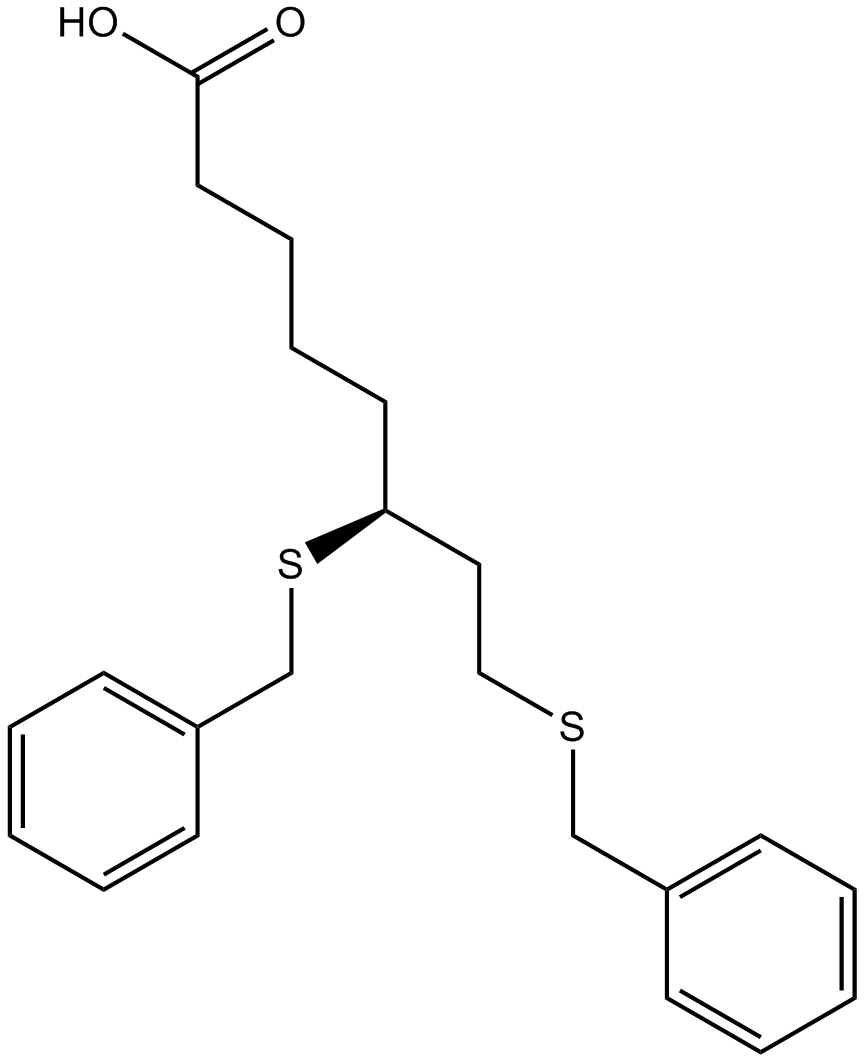
-
GC39365
CPTH2
CPTH2 ist ein potenter Hemmer der Histonacetyltransferase (HAT). CPTH2 hemmt selektiv die Acetylierung von Histon H3 durch Gcn5. CPTH2 induziert Apoptose und verringert die InvasivitÄt einer Zelllinie des klarzelligen Nierenkarzinoms (ccRCC) durch die Hemmung der Acetyltransferase p300 (KAT3B).
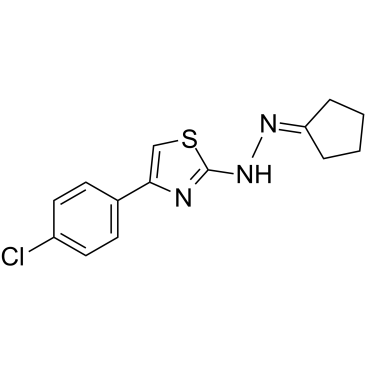
-
GC35747
Crebanine
Crebanin, ein Alkaloid aus Stephania venosa, induziert G1-Arrest und Apoptose in menschlichen Krebszellen.
-
GC34543
cRIPGBM
cRIPGBM, ein proapoptotisches Derivat von RIPGBM, einem zelltypselektiven Induktor der Apoptose in GBM-Krebsstammzellen (CSCs) durch Bindung an Rezeptor-interagierende Proteinkinase 2 (RIPK2), mit einem EC50 von 68 nM in GBM-1-Zellen.
-
GC13838
CRT 0066101
PKD inhibitor
-
GC35750
CRT0066101 dihydrochloride
CRT0066101 Dihydrochlorid ist ein potenter und spezifischer PKD-Inhibitor mit IC50-Werten von 1, 2,5 und 2 nM fÜr PKD1, 2 bzw. 3.
-
GC45414
CRT0066854
CRT0066854 ist ein potenter und selektiver atypischer PKC-Isoenzym-Inhibitor.
-
GC14355
CRT5
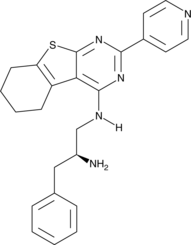
CRT5, ein Pyrazinbenzamid, ist ein potenter und selektiver Inhibitor fÜr alle drei Isoformen von PKD in mit VEGF behandelten Endothelzellen (IC50s = 1, 2 und 1,5 nM fÜr PKD1, PKD2 bzw. PKD3).
-
GC32911
CTX1
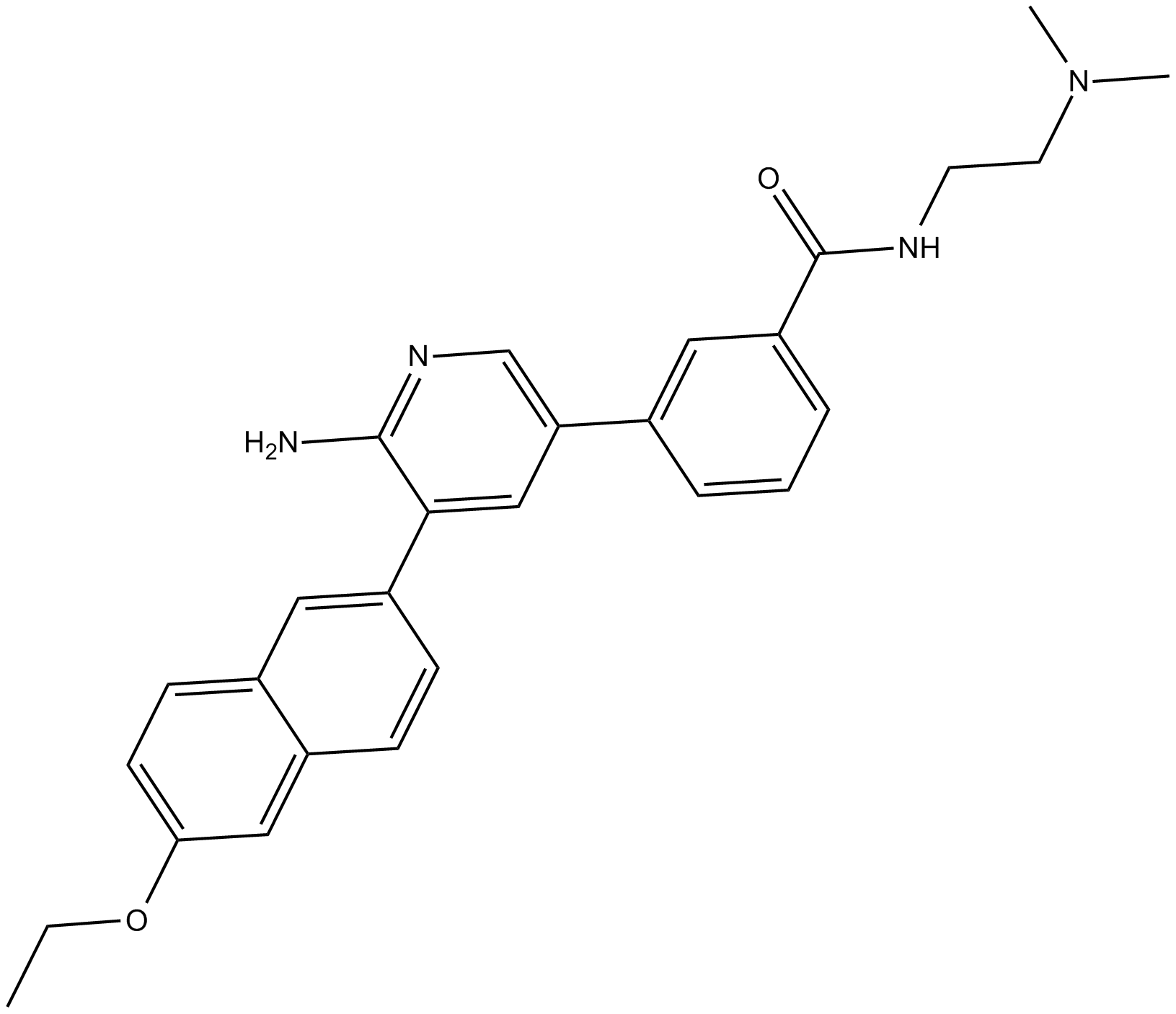
CTX1 ist ein p53-Aktivator, der die HdmX-vermittelte p53-Repression Überwindet. CTX1 zeigt in einem Maus-Modellsystem fÜr akute myeloische LeukÄmie (AML) eine starke Anti-Krebs-AktivitÄt.
-
GN10535
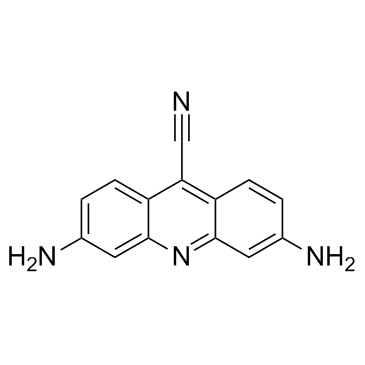
Cucurbitacin B
-
GC35758
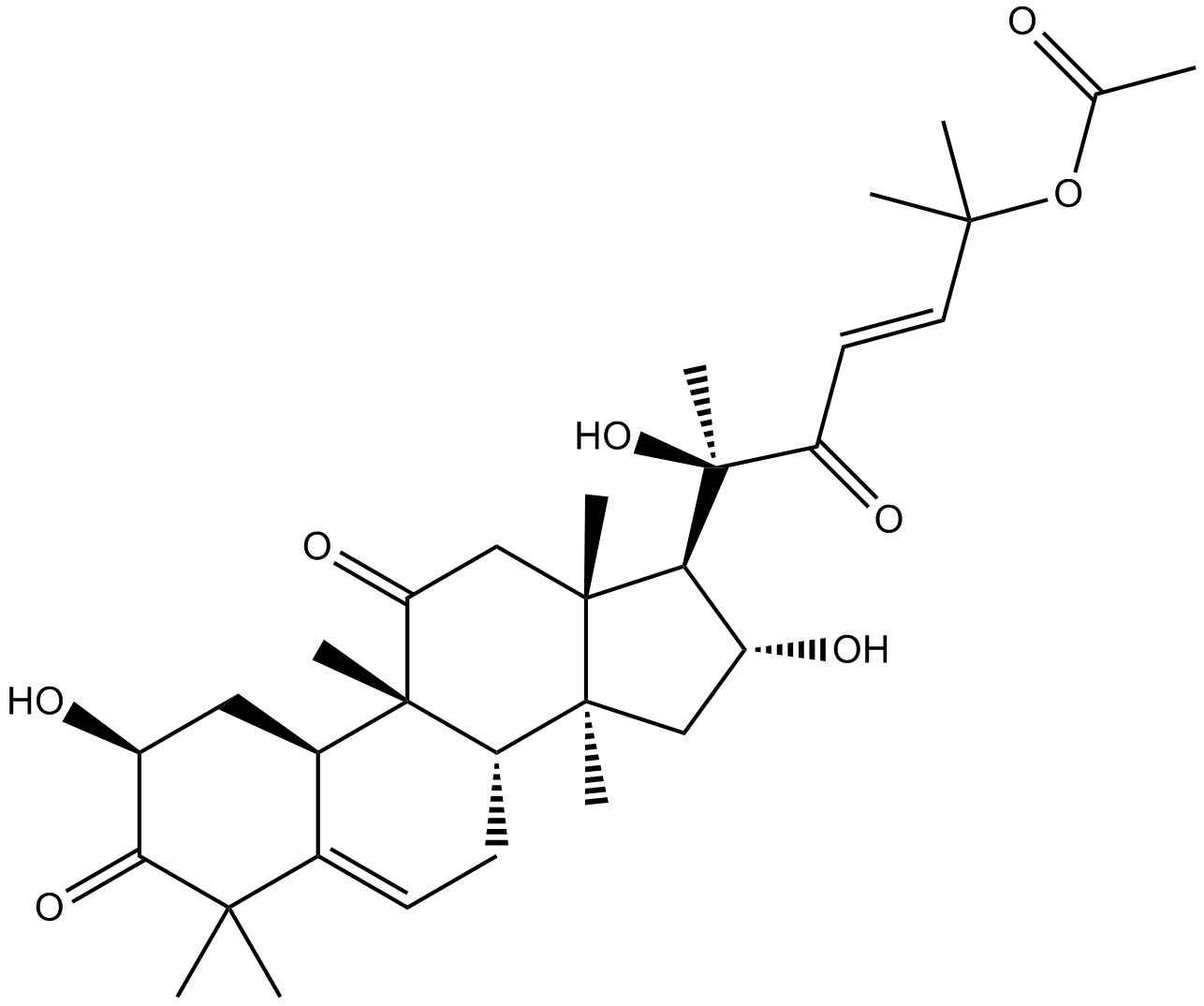
Cucurbitacin IIa
Cucurbitacin IIa ist ein aus Hemsleya Amalils Diels isoliertes Triterpen, das die Apoptose von Krebszellen induziert, die Expression von Survivin reduziert, das Phospho-Histon H3 reduziert und das gespaltene PARP in Krebszellen erhÖht.
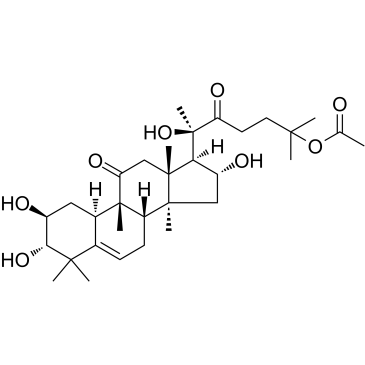
-
GN10788
Cucurbitacin IIb
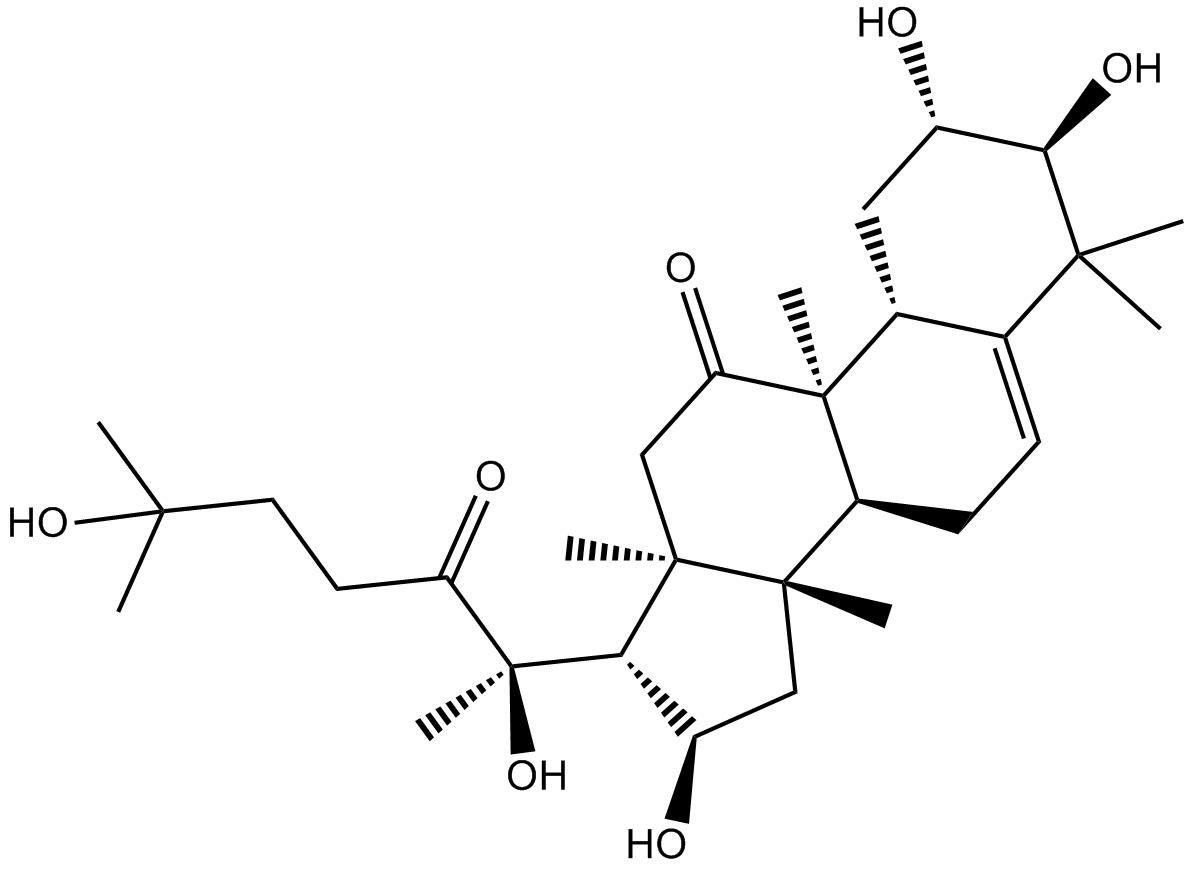
-
GC32781
CUDC-427 (GDC-0917)
CUDC-427 (GDC-0917) ist ein potenter panselektiver IAP-Antagonist der zweiten Generation, der zur Behandlung verschiedener Krebsarten eingesetzt wird.
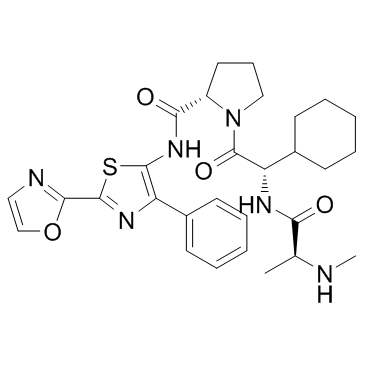
-
GC12115
CUDC-907
A dual inhibitor of HDACs and PI3Ks
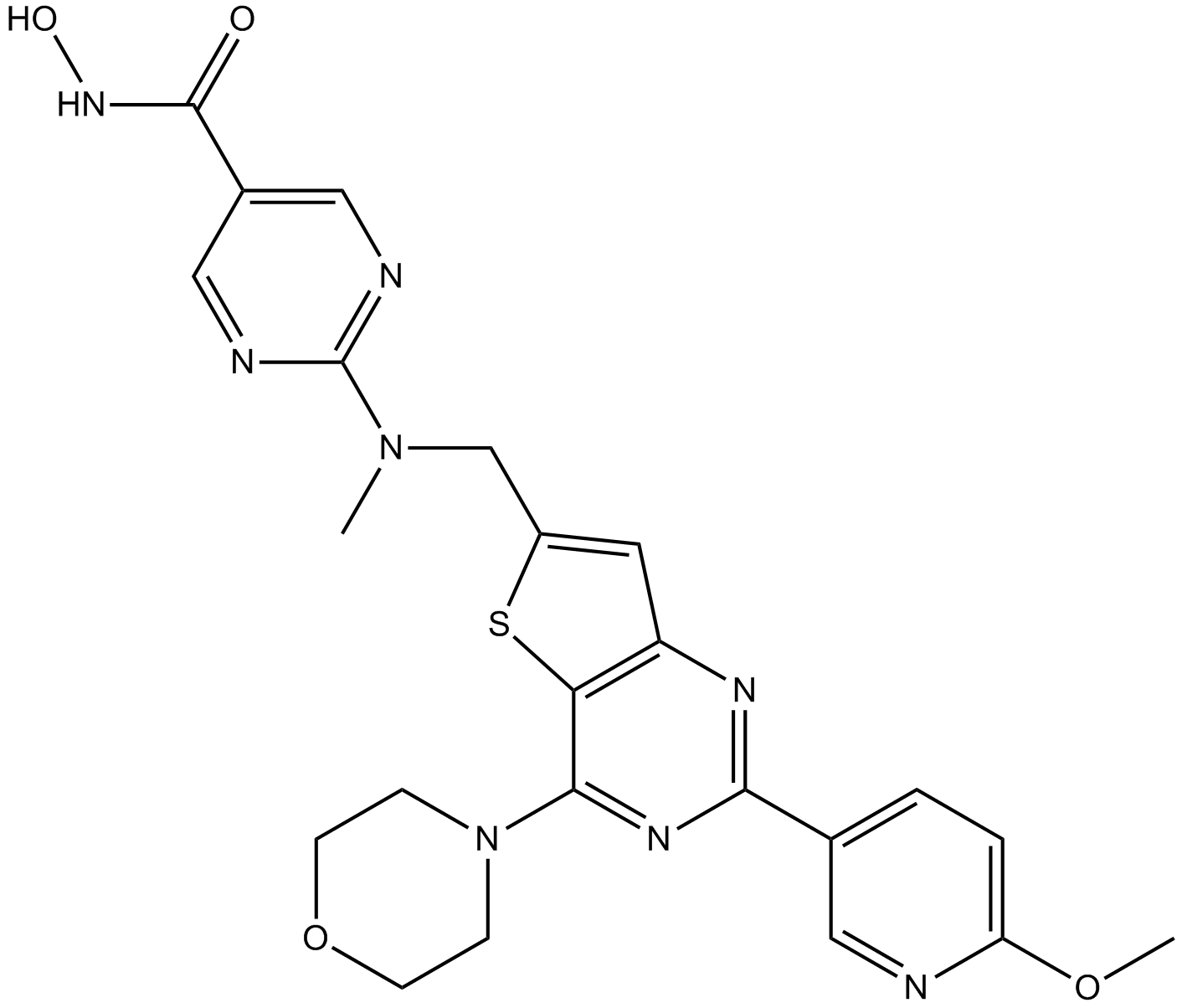
-
GC11217
CUR 61414
CUR 61414 ist ein neuartiger, potenter und zellgängiger Inhibitor des Hedgehog-Signalwegs (IC50 \u003d 100-200 nM).
-
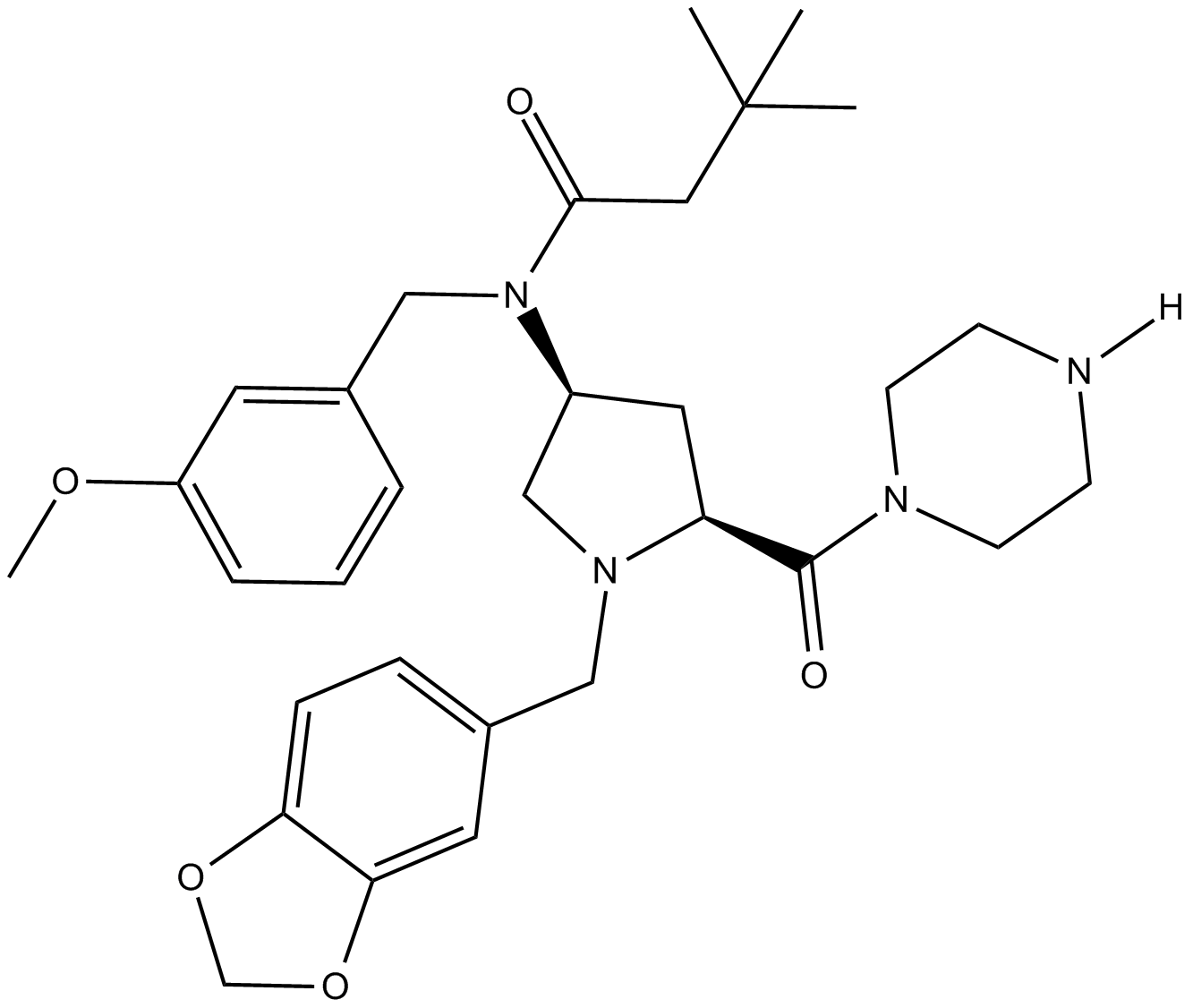
GC14787
Curcumin
Ein gelbes Pigment mit vielfältigen biologischen Aktivitäten.
-
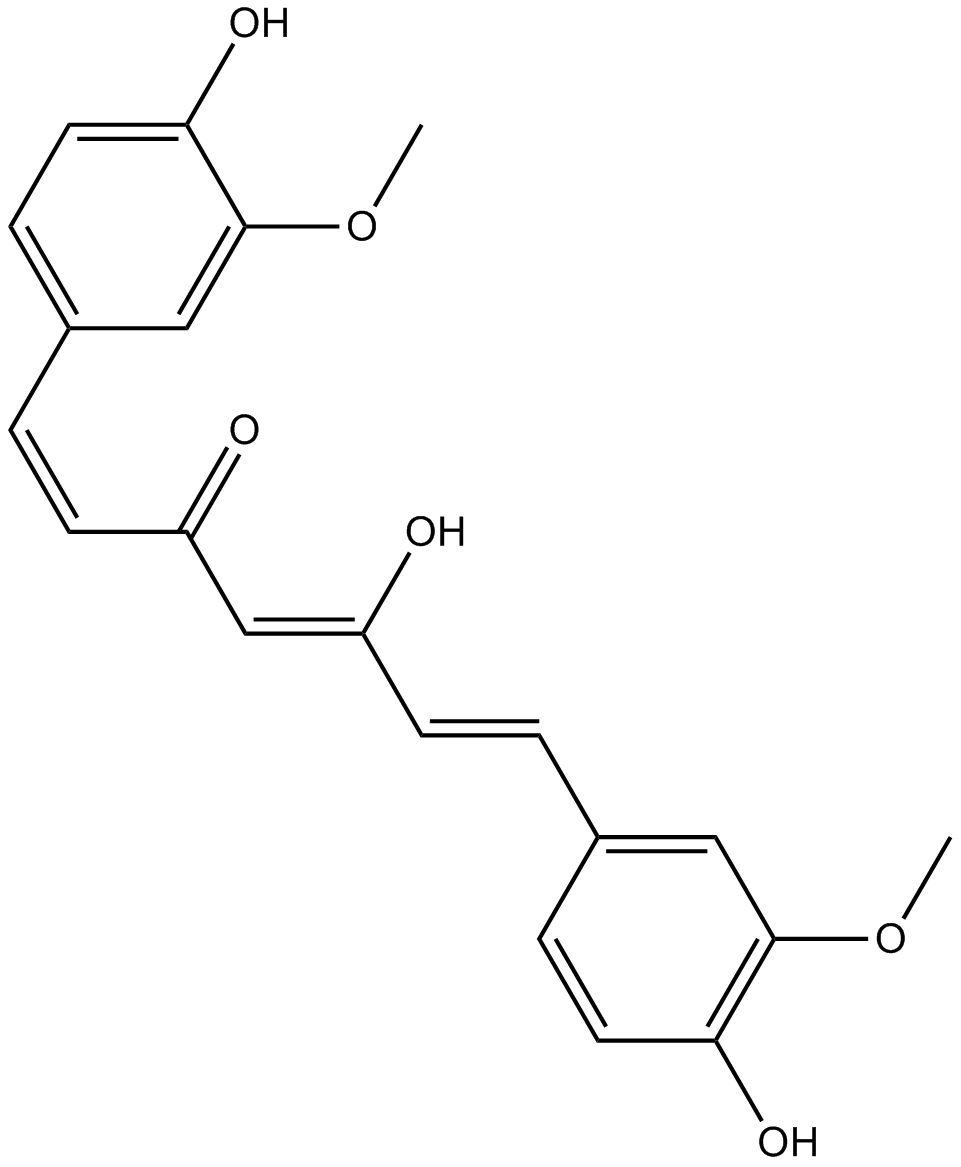
GC40226
Curcumin-d6
Curcumin D6 (Diferuloylmethan D6) ist ein Deuterium mit der Bezeichnung Curcumin (gelbe Kurkuma).
-
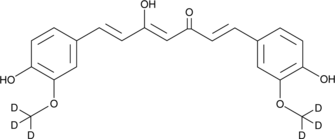
GN10521
Curcumol
-
GC66356
Cusatuzumab
Cusatuzumab ist ein humaner αCD70 monoklonaler AntikÖrper. Cusatuzumab zeigt zytotoxische AktivitÄt mit verstÄrkter antikÖrperabhÄngiger zellulÄrer AktivitÄt. Cusatuzumab reduziert LeukÄmie-Stammzellen (LSCs) und lÖst Gensignaturen im Zusammenhang mit myeloischer Differenzierung und Apoptose aus. Cusatuzumab hat das Potenzial fÜr die Erforschung der akuten myeloischen LeukÄmie (AML).

-
GC63967
Cycleanine
Cycleanin ist ein potenter gefÄßselektiver Calciumantagonist.
-
GC49716
Cyclo(RGDyK) (trifluoroacetate salt)
A cyclic peptide ligand of αVβ3 integrin
-
GC17198
Cycloheximide
Cycloheximide is an antibiotic that inhibits protein synthesis at the translation level, acting exclusively on cytoplasmic (80s) ribosomes of eukaryotes.
-
GC43346
Cyclopamine-KAAD
Cyclopamin-KAAD, ein Hedgehog-Signalweg-Inhibitor, ist ein geglÄtteter Antagonist.

-
GC47148
Cyclophosphamide-d4
Cyclophosphamid-d4 ist das mit Deuterium markierte Cyclophosphamid. Cyclophosphamid ist ein synthetisches Alkylierungsmittel, das chemisch mit Stickstofflost mit antineoplastischer AktivitÄt, einem Immunsuppressivum, verwandt ist.
-
GC38419
Cyclovirobuxine D
Cyclovirobuxin D (CVB-D) ist der Hauptwirkstoff der traditionellen chinesischen Medizin Buxus microphylla.
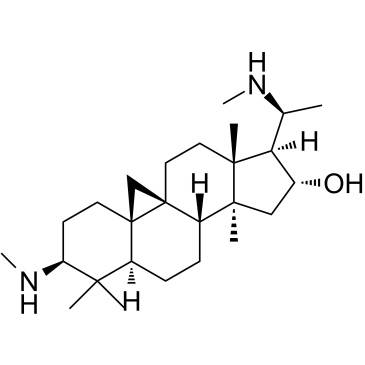
-
GC33330
Cynaropicrin
Cynaropicrin ist ein Sesquiterpenlacton, das die Freisetzung des Tumornekrosefaktors (TNF-α) mit IC50-Werten von 8,24 bzw. 3,18 μM fÜr murine bzw. menschliche Makrophagenzellen hemmen kann. Cynaropicrin hemmt auch den Anstieg des Knorpelabbaufaktors (MMP13) und unterdrÜckt die NF-κB-SignalÜbertragung.
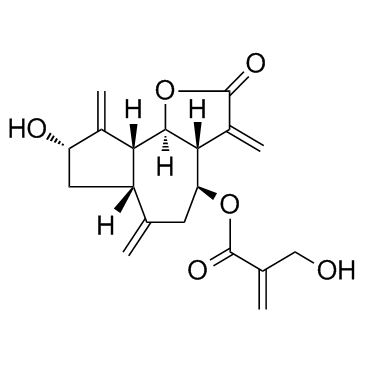
-
GC65565
Cyproheptadine
Cyproheptadin ist ein potenter und oral aktiver 5-HT2A-Rezeptorantagonist mit antidepressiver und antiserotonerger Wirkung.
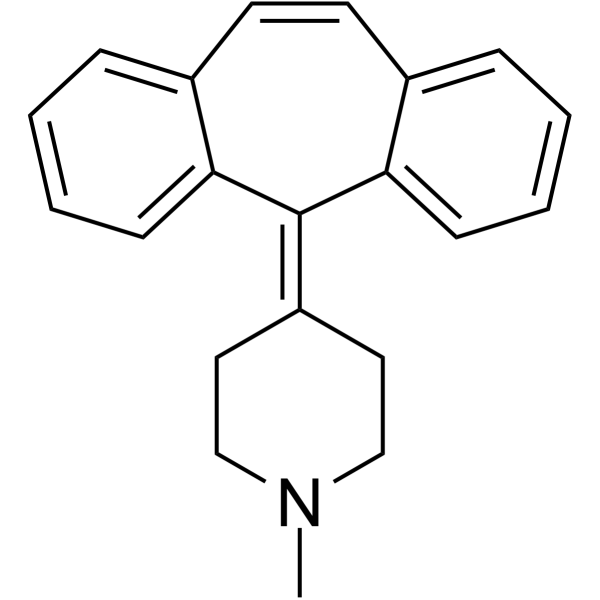
-
GC33779
Cysteamine (β-Mercaptoethylamine)
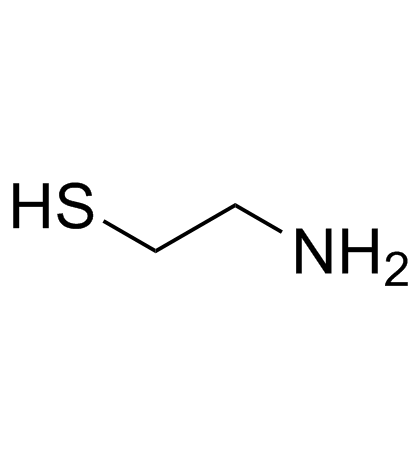
-
GC13502
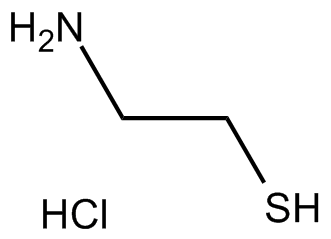
Cysteamine HCl
Cysteamin-HCl (2-Aminoethanthiol-Hydrochlorid) ist ein oral aktiver Wirkstoff zur Behandlung von nephropathischer Cystinose und ein Antioxidans.
-
GC17050
CYT387
A potent inhibitor of JAK1 and JAK2
-
GC11383
CYT997 (Lexibulin)
CYT997 (Lexibulin) (CYT-997) ist ein potenter und oral aktiver Tubulin-Polymerisationshemmer mit IC50-Werten von 10-100 nM in Krebszelllinien; mit starker zytotoxischer und gefÄßzerstÖrender AktivitÄt in vitro und in vivo. CYT997 (Lexibulin) induziert Zellapoptose und induziert die mitochondriale ROS-Erzeugung in GC-Zellen.
-
GC13070
Cytarabine
Zytotoxisches Mittel, blockiert die DNA-Synthese.
-
GC43356
CytoCalcein™ Violet 450
CytoCalcein? Violet 450 is a fluorogenic dye used to assess cell viability.
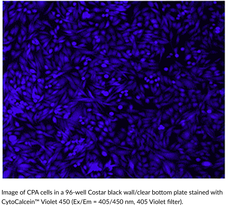
-
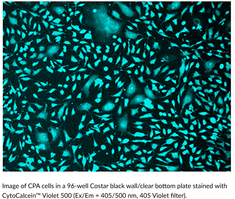
GC43357
CytoCalcein™ Violet 500
CytoCalcein? Violet 500 is a fluorogenic dye used to assess cell viability.
-
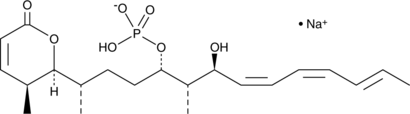
GC43361
Cytostatin (sodium salt)
Cytostatin is a natural antitumor inhibitor of cell adhesion to extracellular matrix, blocking adhesion of B16 melanoma cells to laminin and collagen type IV in vitro (IC50s = 1.3 and 1.4 μg/ml, respectively) and B16 cells metastatic activity in mice.
-
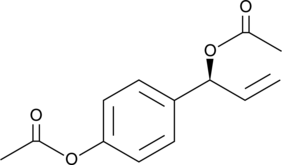
GC43368
D,L-1′-Acetoxychavicol Acetate
D,L-1′-Acetoxychavicol acetate is a natural compound first isolated from the rhizomes of ginger-like plants.
-
GC66824
D-α-Tocopherol Succinate
D-&7#945;-Tocopherolsuccinat (Vitamin-E-Succinat) ist ein antioxidatives Tocopherol und eine Salzform von Vitamin E. D-α-Tocopherolsuccinat hemmt die Cisplatin-induzierte Zytotoxizität. D-α-Tocopherol Succinate kann für die Krebsforschung verwendet werden.
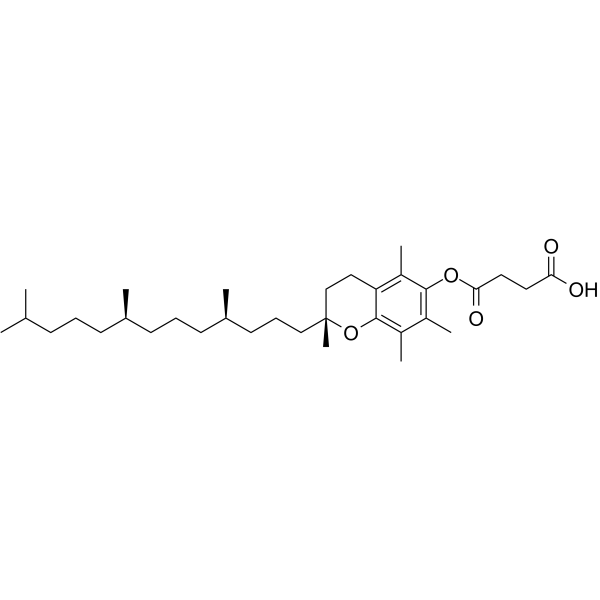
-
GC12256
D-Mannitol
D-Mannit ist ein osmotisches Diuretikum und ein schwacher renaler Vasodilatator.
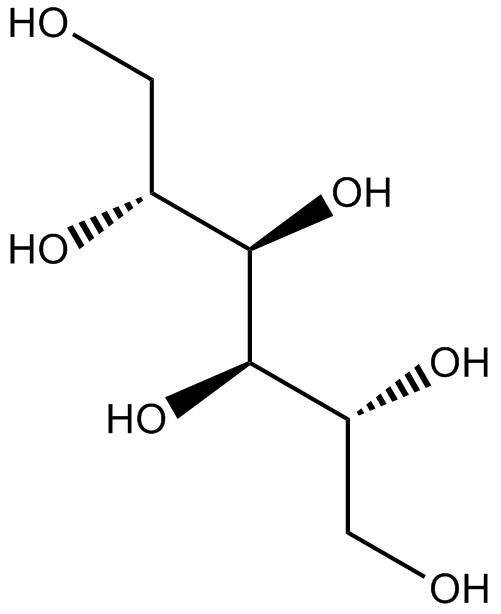
-
GC60796
D-Trimannuronic acid
D-TrimannuronsÄure, ein Alginatoligomer, wird aus Meeresalgen extrahiert.
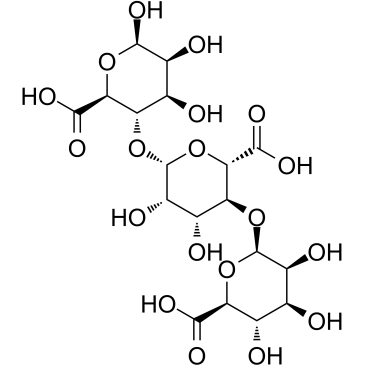
-
GC13202
D4476
Inhibitor of CK1 and ALK5
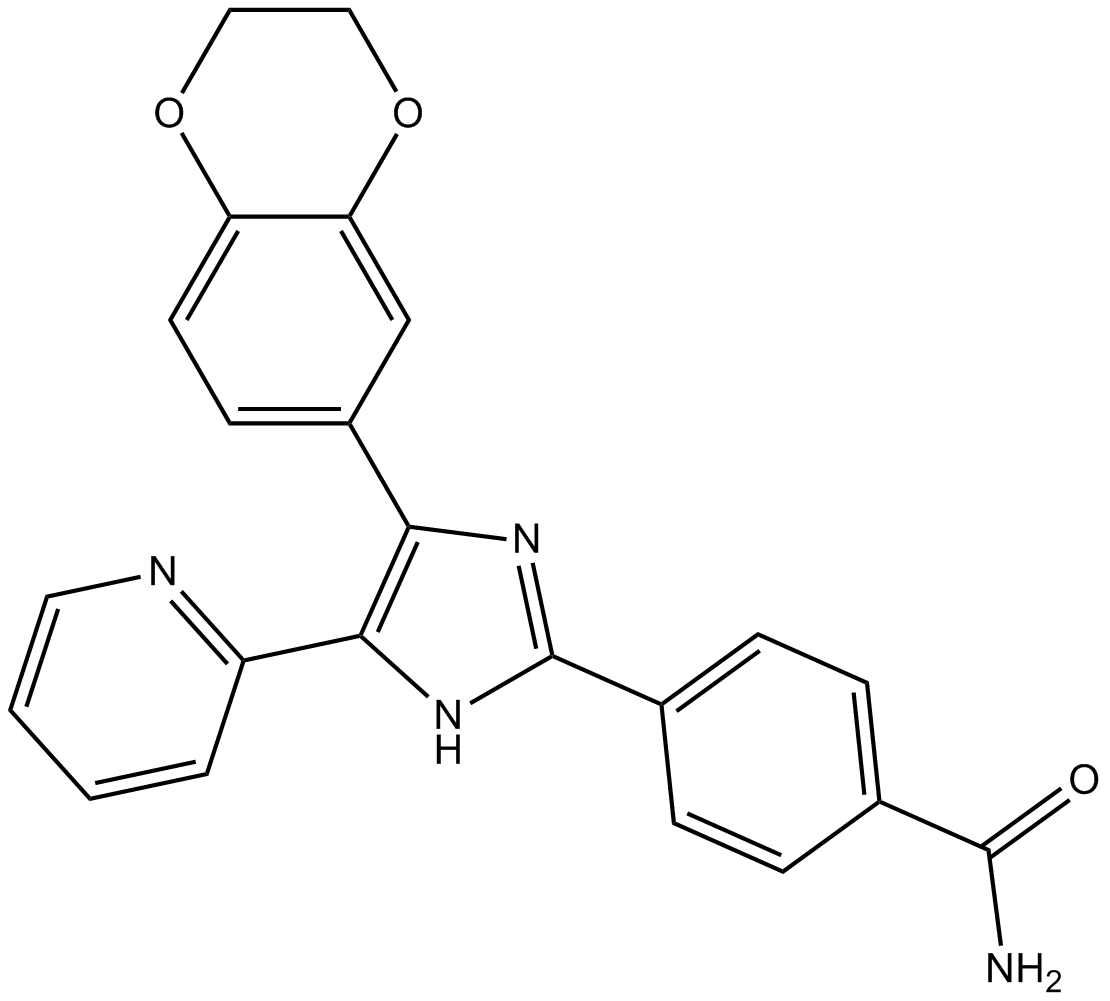
-
GC17851
D609
A competitive inhibitor of PC-specific PLC
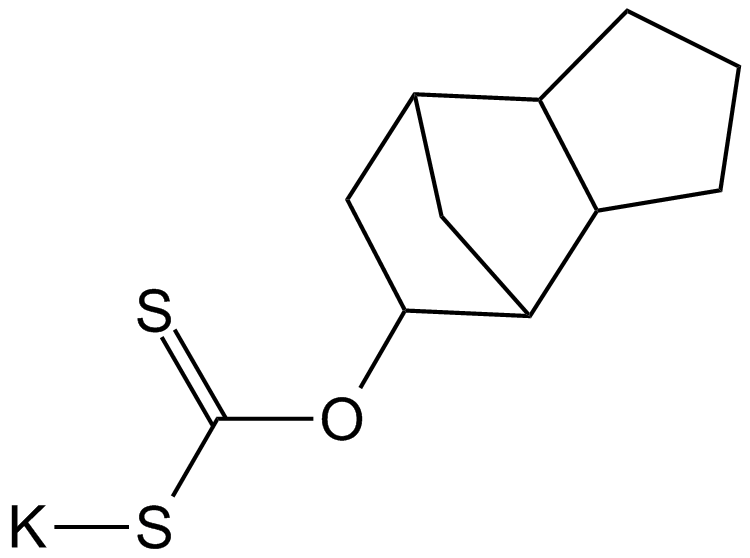
-
GC50296
D9
D9 ist ein potenter und selektiver Inhibitor der Thioredoxinreduktase (TrxR) mit einem EC50-Wert von 2,8 nM. D9 hat die FÄhigkeit, die Tumorproliferation sowohl in vitro als auch in vivo zu hemmen.
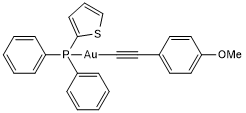
-
GC18421
Dabcyl-YVADAPV-EDANS
Dabcyl-YVADAPV-EDANS is a fluorogenic substrate for caspase-1.
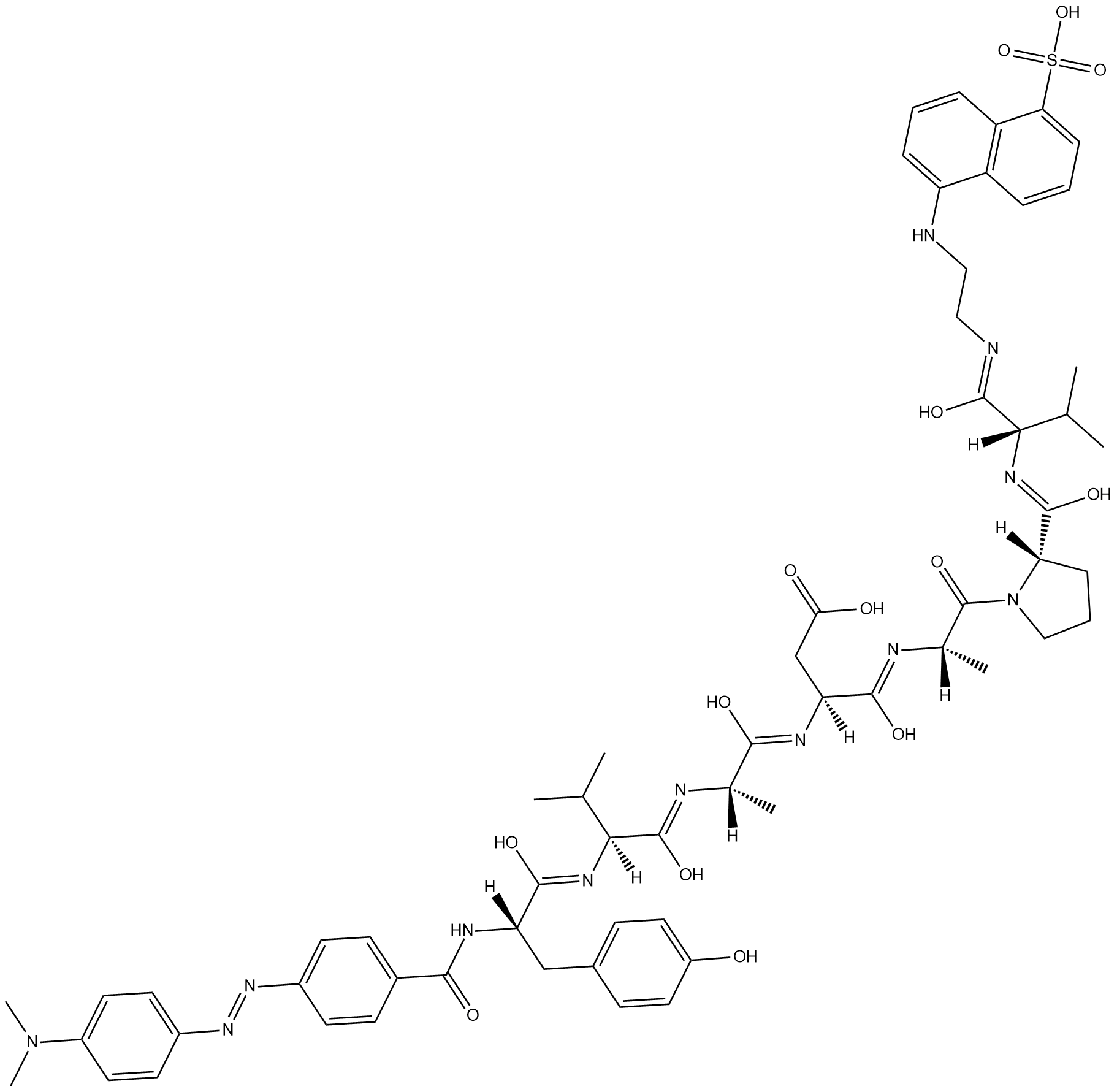
-
GC47166
Dabrafenib-d9
Dabrafenib-d9 (GSK2118436A-d9) ist das Deuterium-markierte Dabrafenib. Dabrafenib (GSK2118436A) ist ein ATP-kompetitiver Inhibitor von Raf mit IC50-Werten von 5 nM und 0,6 nM fÜr C-Raf bzw. B-RafV600E.
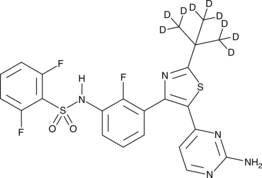
-
GC14485
Dacarbazine
Dacarbazin ist ein fÜr den Zellzyklus unspezifisches antineoplastisches Alkylierungsmittel. Dacarbazin hemmt die T- und B-lymphoblastische Reaktion mit IC50-Werten von 50 bzw. 10 μg/ml. Dacarbazin kann fÜr die Erforschung des metastasierten malignen Melanoms verwendet werden.
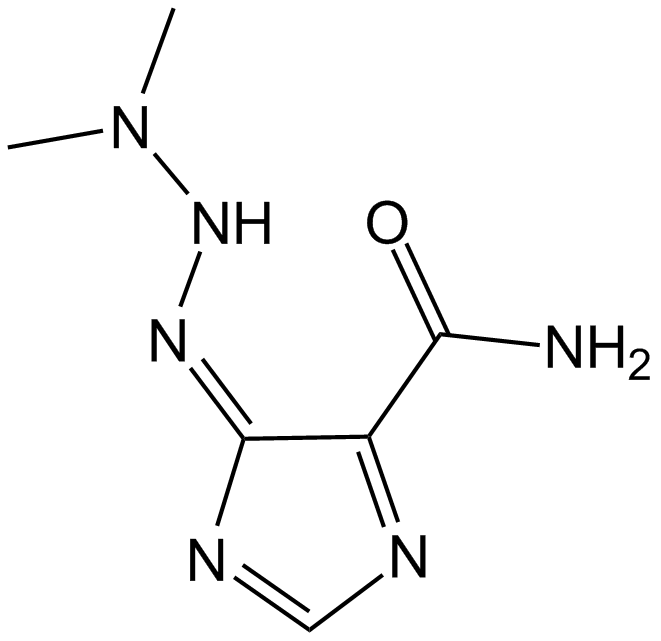
-
GC47167
Dacarbazine-d6
Dacarbazin-d6 (Imidazolcarboxamid-d6) ist das mit Deuterium bezeichnete Dacarbazin. Dacarbazin (DTIC-Dome; DTIC) ist ein antineoplastisches Mittel. Es hat eine signifikante AktivitÄt gegen Melanome.
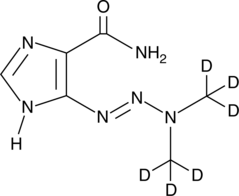
-
GC68305
Dacetuzumab

-
GC10225
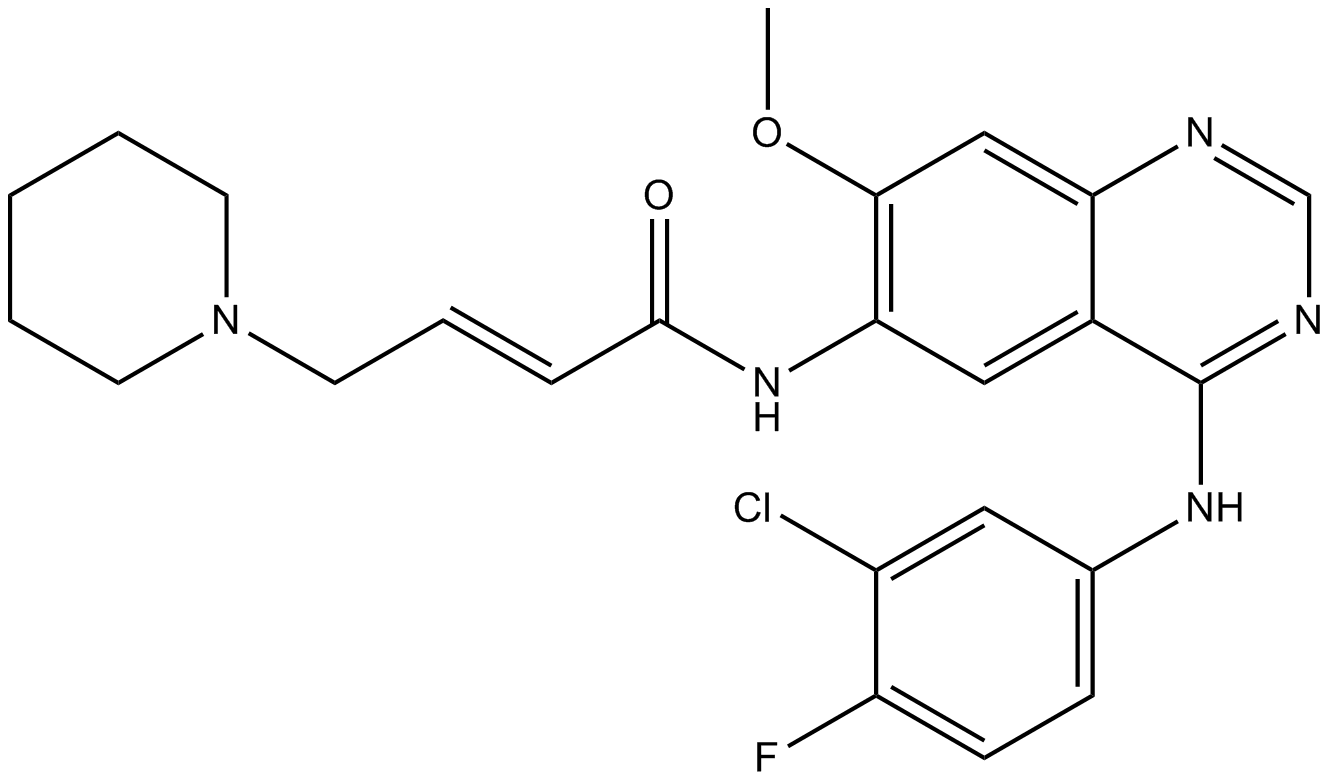
Dacomitinib (PF299804, PF299)
Dacomitinib (PF299804, PF299) (PF-00299804) ist ein spezifischer und irreversibler Inhibitor der ERBB-Familie von Kinasen mit IC50-Werten von 6 nM, 45,7 nM und 73,7 nM fÜr EGFR, ERBB2 bzw. ERBB4.
-
GC15211
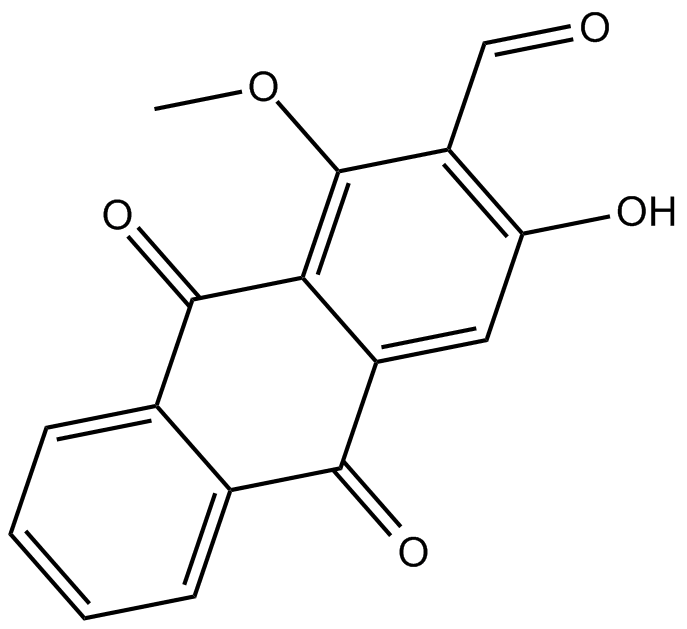
Damnacanthal
Damnacanthal ist ein Anthrachinon, das aus der Wurzel von Morinda citrifolia isoliert wird.
-
GN10318
Danshensu
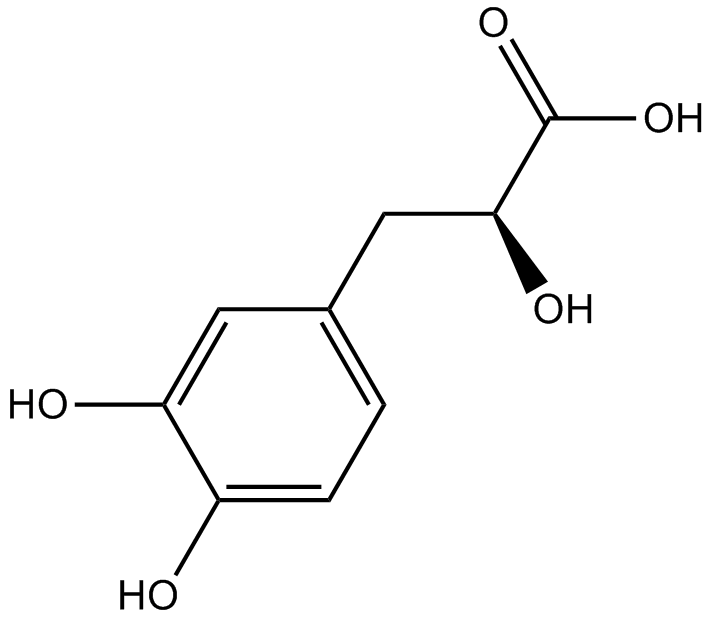
-
GC34010
Danshensu (Dan shen suan A)
Danshensu, an active ingredient of Salvia miltiorrhiza, shows wide cardiovascular benefit by activating Nrf2 signaling pathway.
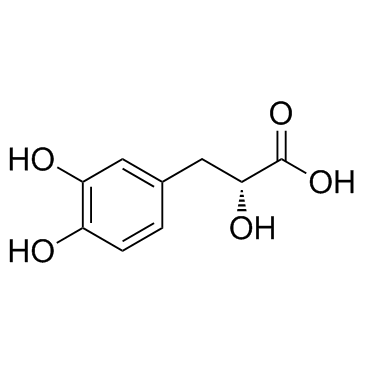
-
GC15217
Danusertib (PHA-739358)
A pan-Aurora kinase and Abl inhibitor
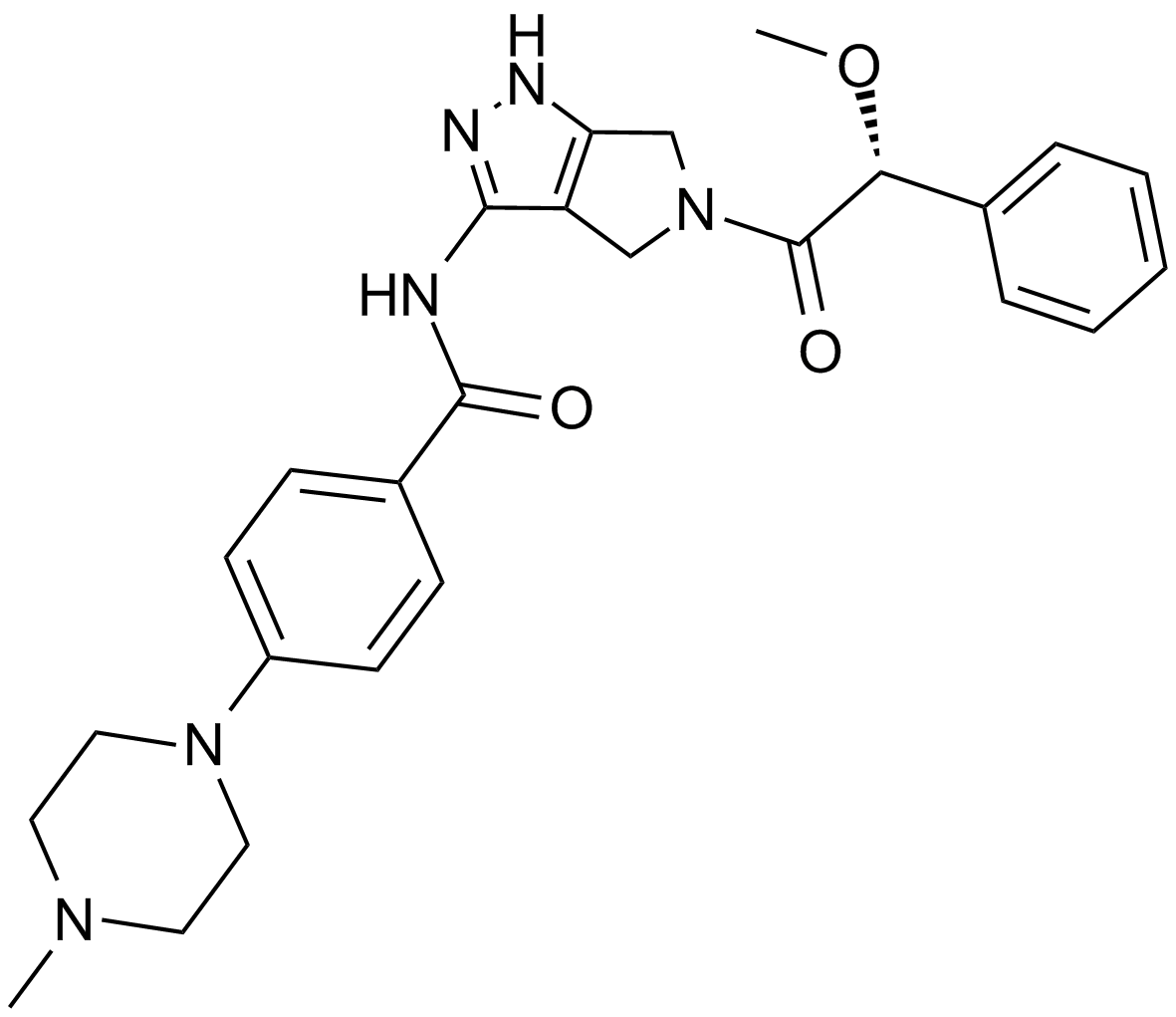
-
GC17650
DAPK Substrate Peptide
A DAPK1 peptide substrate
-
GC49883
DAPK Substrate Peptide (trifluoroacetate salt)
A DAPK1 peptide substrate

-
GC12942
DAPT (GSI-IX)
Inhibitor von γ-Sekretase
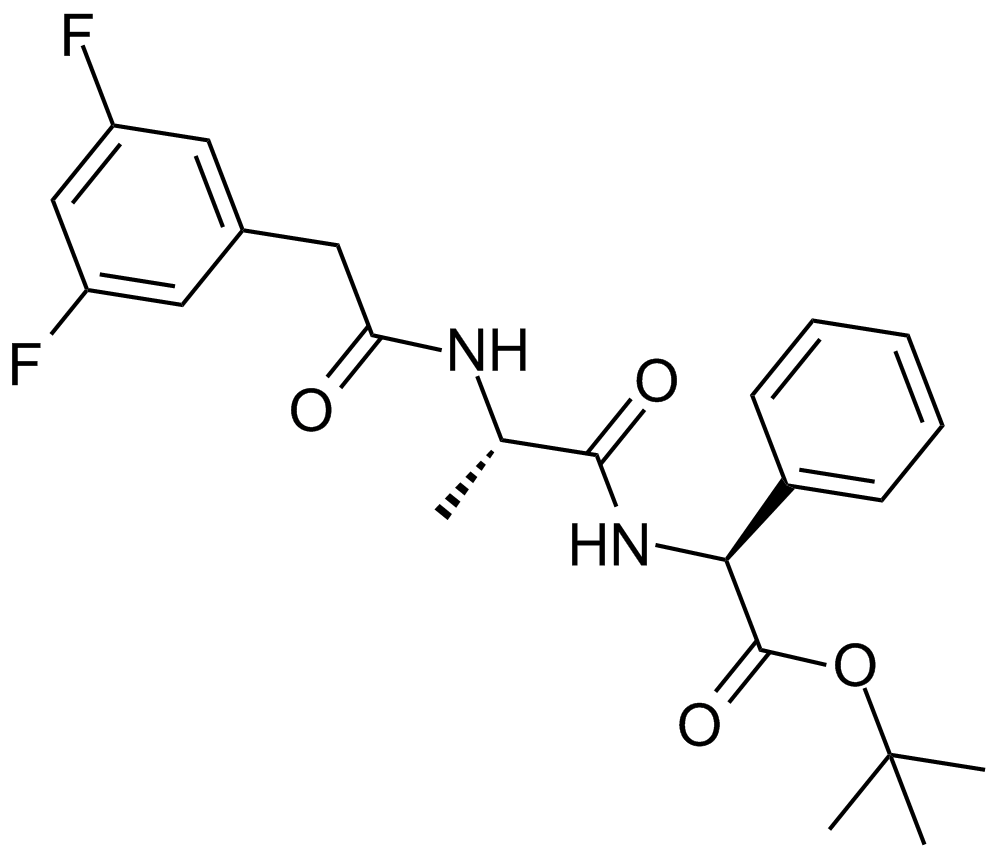
-
GC43379
Darinaparsin
Darinaparsin (ZIO-101), ein organisches Arsen, ist ein auf die Mitochondrien gerichteter Wirkstoff. Darinaparsin induziert Apoptose in Krebszellen und hat Antikrebswirkungen.
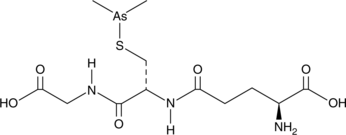
-
GC15568
Dasatinib (BMS-354825)
Ein Hemmstoff von Abl und Src.
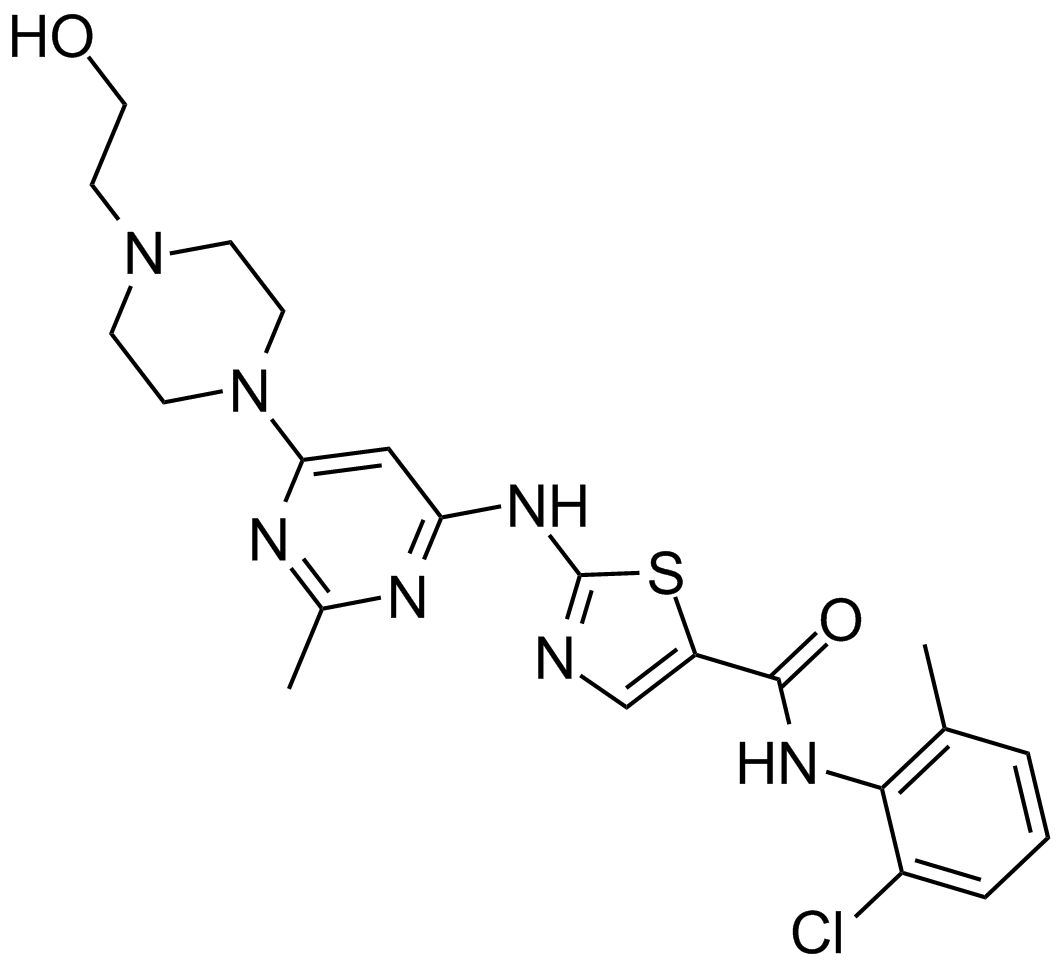
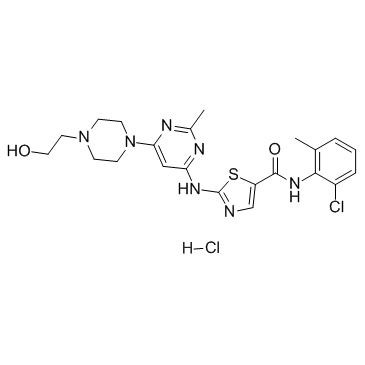
-
GC35812
Dasatinib hydrochloride
Dasatinib (BMS-354825) Hydrochlorid ist ein hochpotenter, ATP-kompetitiver, oral aktiver dualer Src/Bcr-Abl-Inhibitor mit starker AntitumoraktivitÄt. Die Kis sind 16 pM und 30 pM fÜr Src bzw. Bcr-Abl. Dasatinibhydrochlorid hemmt Bcr-Abl und Src mit IC50-Werten von < 1,0 nM bzw. 0,5 nM. Dasatinibhydrochlorid induziert auch Apoptose und Autophagie.
-
GC10354
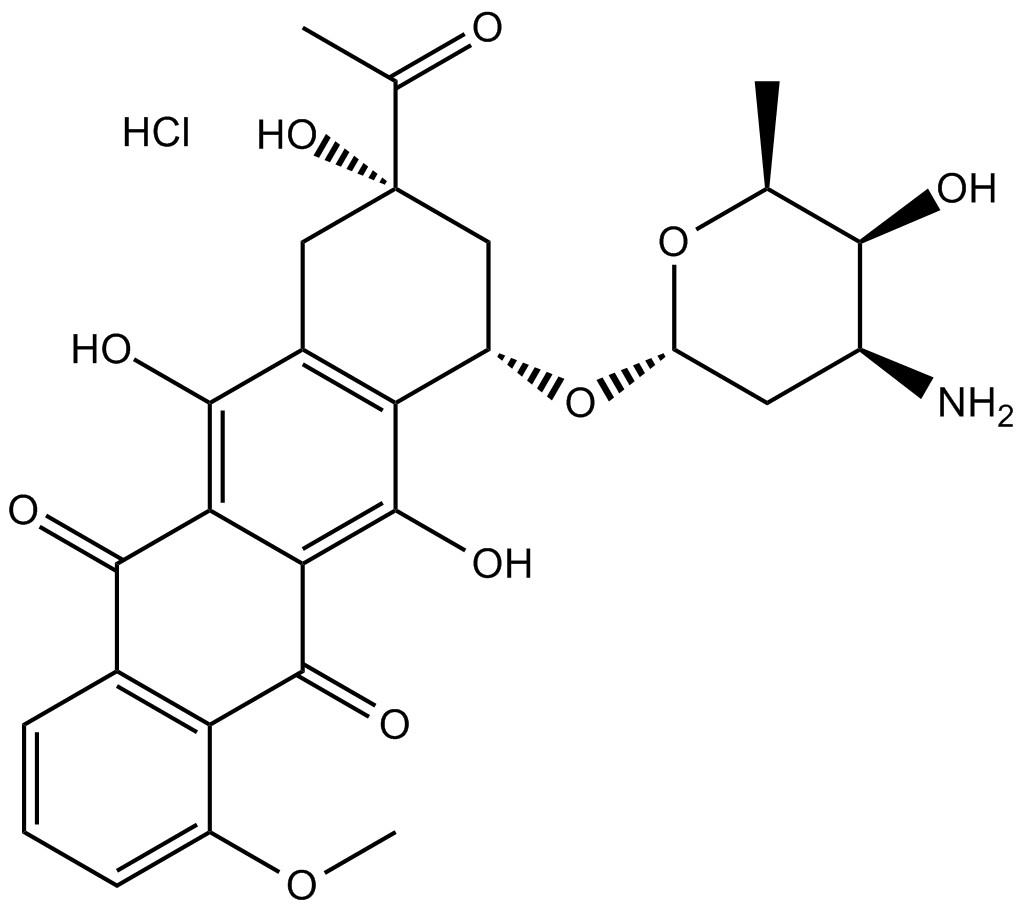
Daunorubicin HCl
Daunorubicin (Daunomycin)-Hydrochlorid ist ein Topoisomerase-II-Inhibitor mit starker AntitumoraktivitÄt.