DNA Damage/DNA Repair
- MTH1(4)
- PARP(58)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(94)
- Nucleoside Antimetabolite/Analogue(136)
- Telomerase(14)
- Topoisomerase(133)
- tankyrase(5)
- Antifolate(32)
- CDK(233)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(62)
- DNA Alkylator/Crosslinker(64)
- DNA/RNA Synthesis(388)
- Eukaryotic Initiation Factor (eIF)(22)
- IRE1(22)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(53)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nom du produit Informations
-
GC31950
Halofuginone hydrobromide (RU-19110 (hydrobromide))
Le bromhydrate d'halofuginone (RU-19110), un dérivé de la fébrifugine, est un inhibiteur compétitif de la prolyl-ARNt synthétase avec un Ki de 18,3 nM.
-
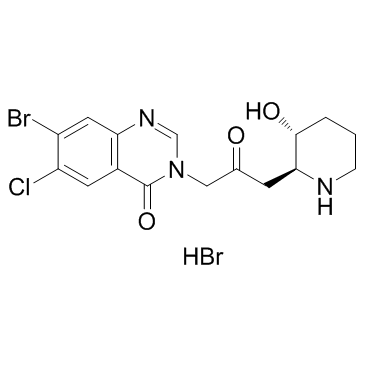
GC62328
HBV-IN-4
HBV-IN-4, un dérivé de la phtalazinone, est un inhibiteur de la réplication de l'ADN du VHB puissant et actif par voie orale avec une IC50 de 14 nM.
-
GC36211
HBX 19818
HBX 19818 est un inhibiteur spécifique de la protéase 7 spécifique À l'ubiquitine (USP7), avec une IC50 de 28,1 μM.
-
GC19190
HDAC-IN-4
AZD 9468
HDAC-IN-4 est un inhibiteur de HDAC de classe I puissant, sélectif et actif par voie orale avec des IC50 de 63 nM, 570 nM et 550 nM pour HDAC1, HDAC2 et HDAC3, respectivement. L'HDAC-IN-4 n'a aucune activité contre l'HDAC de classe II. HDAC-IN-4 a une activité antitumorale.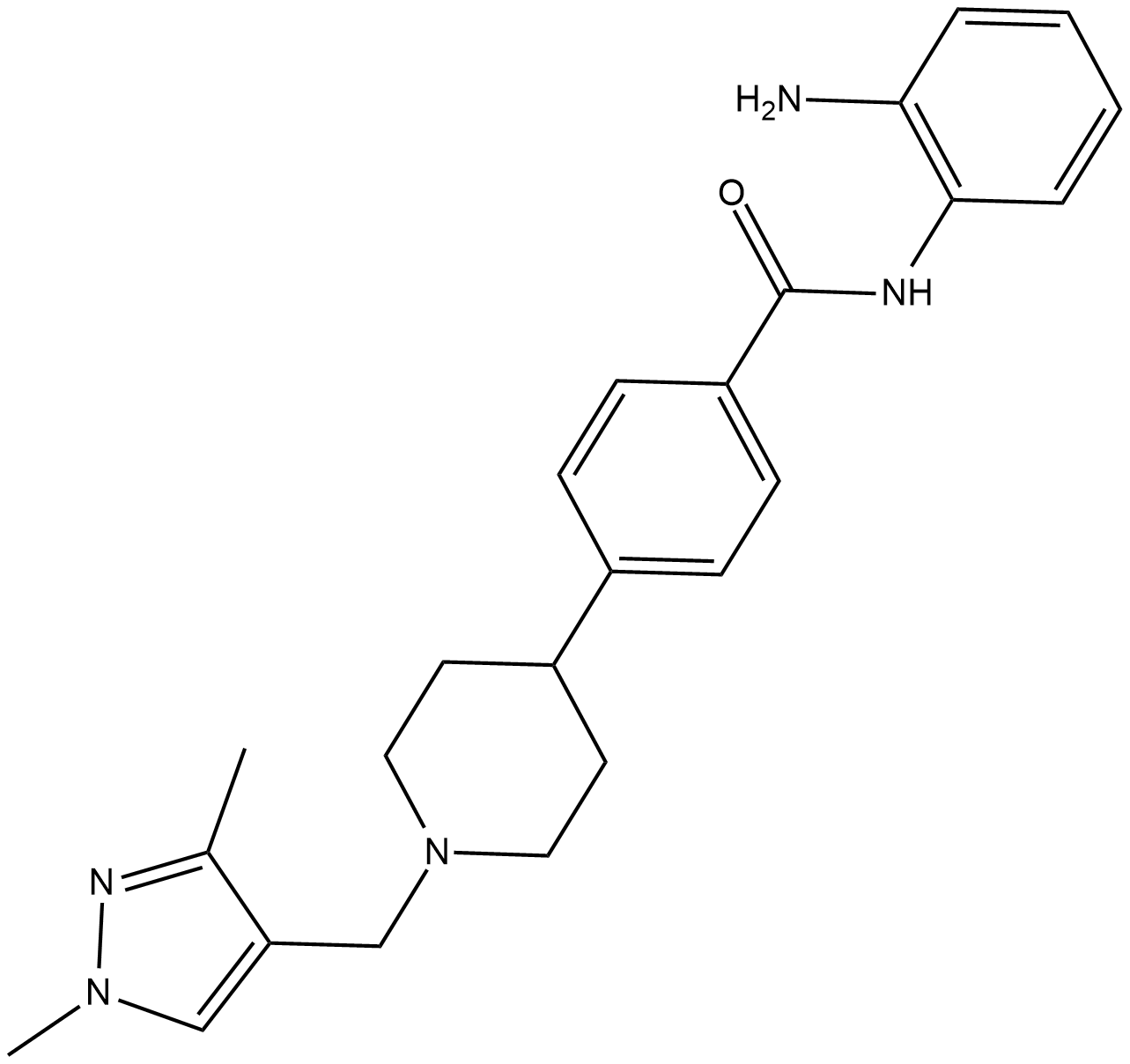
-
GC66052
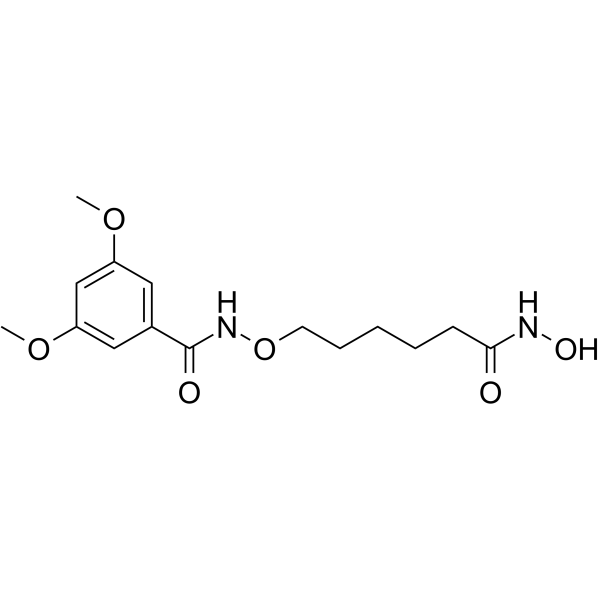
HDAC-IN-40
HDAC-IN-40 est un puissant inhibiteur de HDAC À base d'alcoxyamide avec des valeurs Ki de 60 nM et 30 nM pour HDAC2 et HDAC6, respectivement. HDAC-IN-40 avait des effets antitumoraux.
-
GC33395
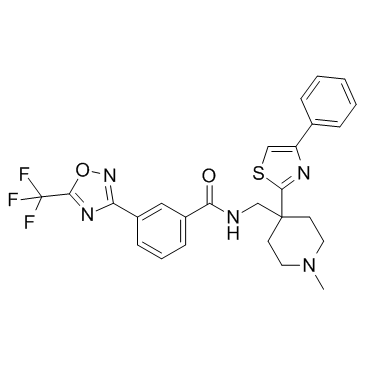
HDAC-IN-5
HDAC-IN-5 est un inhibiteur de l'histone désacétylase (HDAC).
-
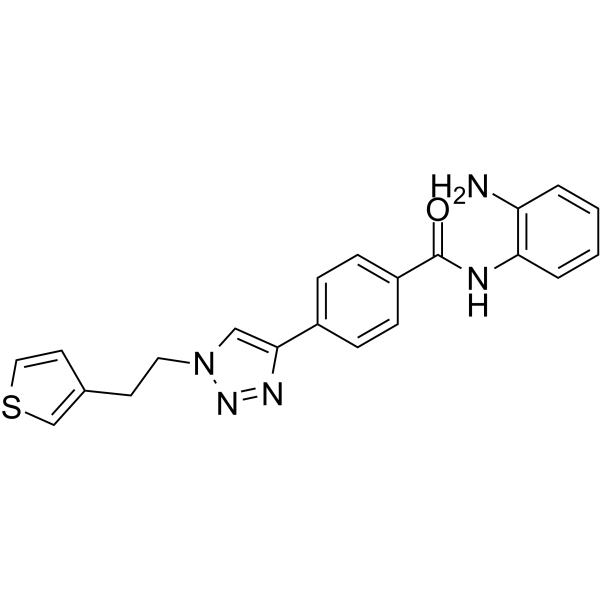
GC68010
HDAC3-IN-T247
-
GC41085
HDAC6 Inhibitor
Histone Deacetylase 6
L'inhibiteur HDAC6 est un inhibiteur HDAC6 puissant et sélectif (IC50 = 36 nM). L'inhibiteur d'HDAC6 inhibe faiblement les autres isoformes d'HDAC. L'inhibiteur HDAC6 inhibe l'accumulation d'acyl-tubuline dans les cellules avec une valeur IC50 de 210 nM.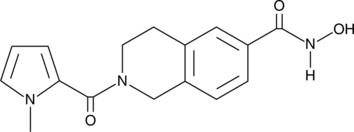
-
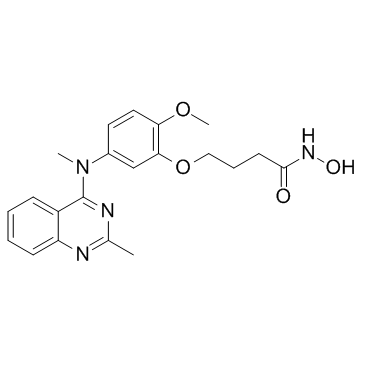
GC33317
HDAC6-IN-1
HDAC6-IN-1 est un inhibiteur puissant et sélectif de HDAC6 avec une IC50 de 17 nM et présente une sélectivité de 25 fois et 200 fois par rapport À HDAC1 (IC50 = 422 nM) et HDAC8 (IC50 = 3398 nM), respectivement.
-
GC19189
HDAC8-IN-1
MDK-7933
HDAC8-IN-1 est un inhibiteur de HDAC8 avec une IC50 de 27,2 nM.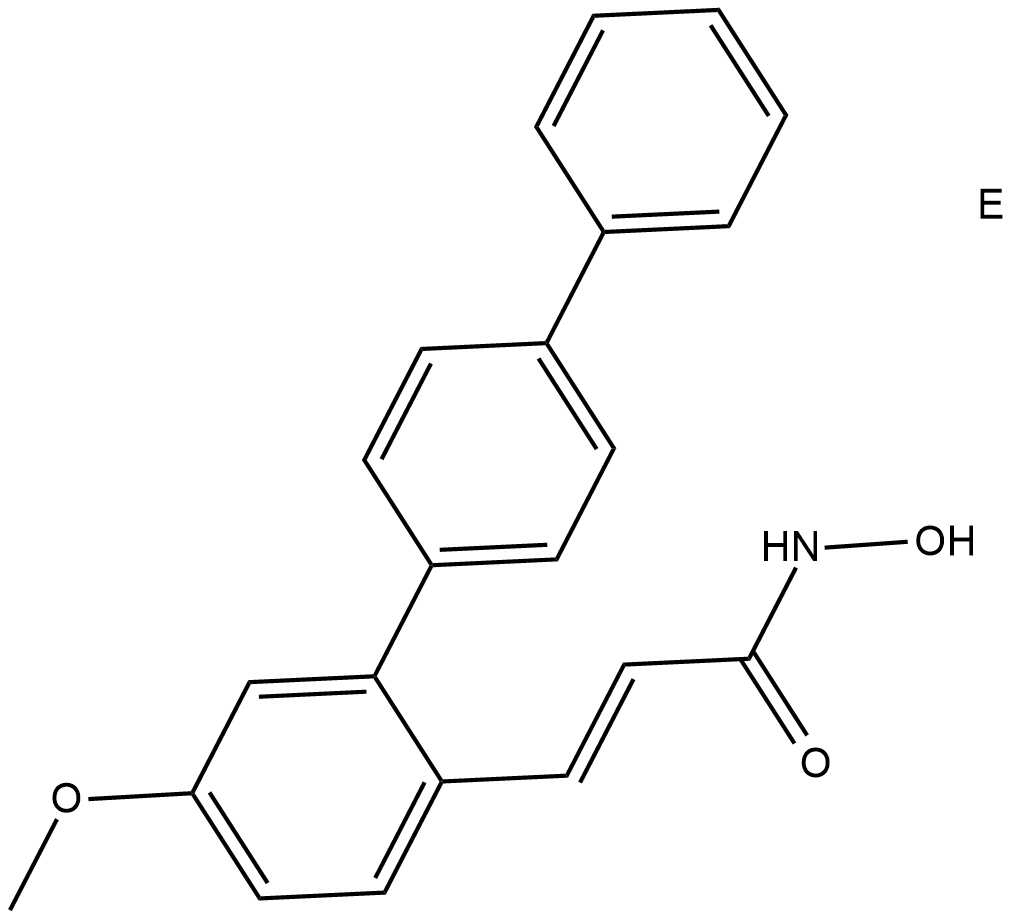
-
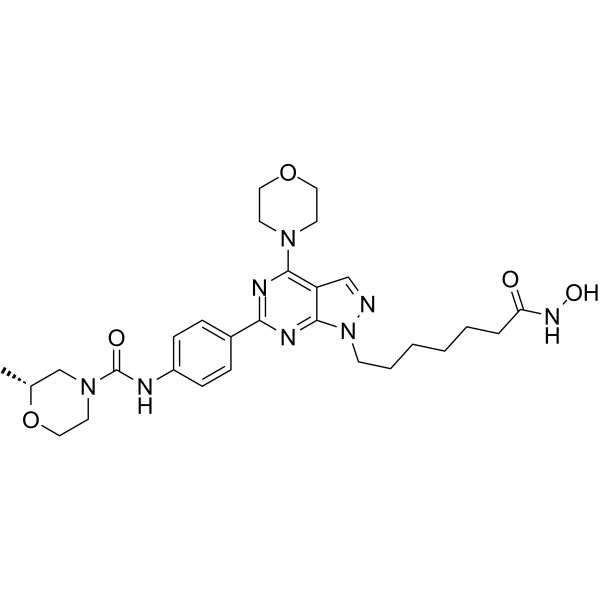
GC65460
HDACs/mTOR Inhibitor 1
HDACs/mTOR Inhibitor 1 est un double inhibiteur cible des histones désacétylases (HDAC) et de la cible mammifère de la rapamycine (mTOR) pour le traitement des hémopathies malignes, avec des IC50 de 0,19 nM, 1,8 nM, 1,2 nM et > 500 nM pour HDAC1, HDAC6, mTOR et PI3Kα, respectivement. L'inhibiteur HDACs/mTOR 1 stimule l'arrêt du cycle cellulaire en phase G0/G1 et induit l'apoptose des cellules tumorales avec une faible toxicité in vivo.
-
GC39279
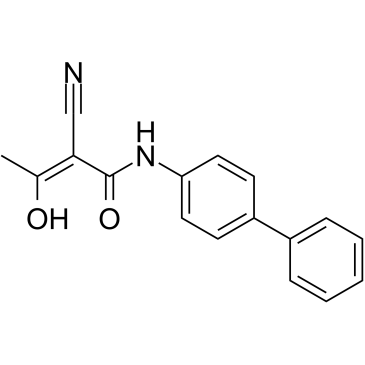
hDHODH-IN-1
hDHODH-IN-1 est un inhibiteur de la dihydroorotate déshydrogénase humaine (hDHODH).
-
GC48388
Heliquinomycin
NSC 702208, Rubymycin
A bacterial metabolite with diverse biological activities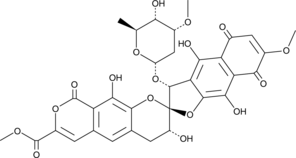
-
GC39266
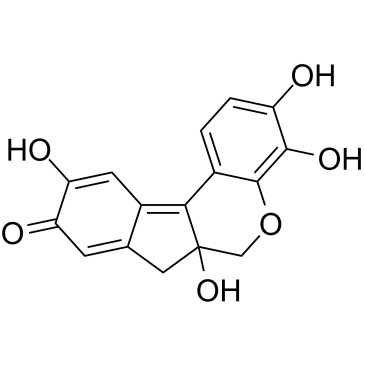
Hematein
L'hématéine est un produit d'oxydation de l'hématoxyline qui agit comme un colorant. L'hématéine est un inhibiteur allostérique de la caséine kinase II avec une IC50 de 0,74 μM. L'hématéine inhibe la phosphorylation d'Akt/PKB Ser129, la voie Wnt/TCF et augmente l'apoptose dans les cellules cancéreuses du poumon.
-
GC40103
Herboxidiene
GEX1A, Tan 1609
Herboxidiene, en tant qu'agent antitumoral puissant, cible l'unité SF3B du spliceosome. Herboxidiene induit également l'arrêt du cycle cellulaire en G1 et G2/M dans une ligne cellulaire de fibroblastes humains normaux WI-38.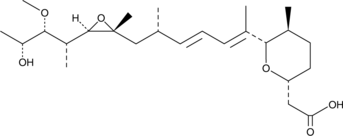
-
GC12201
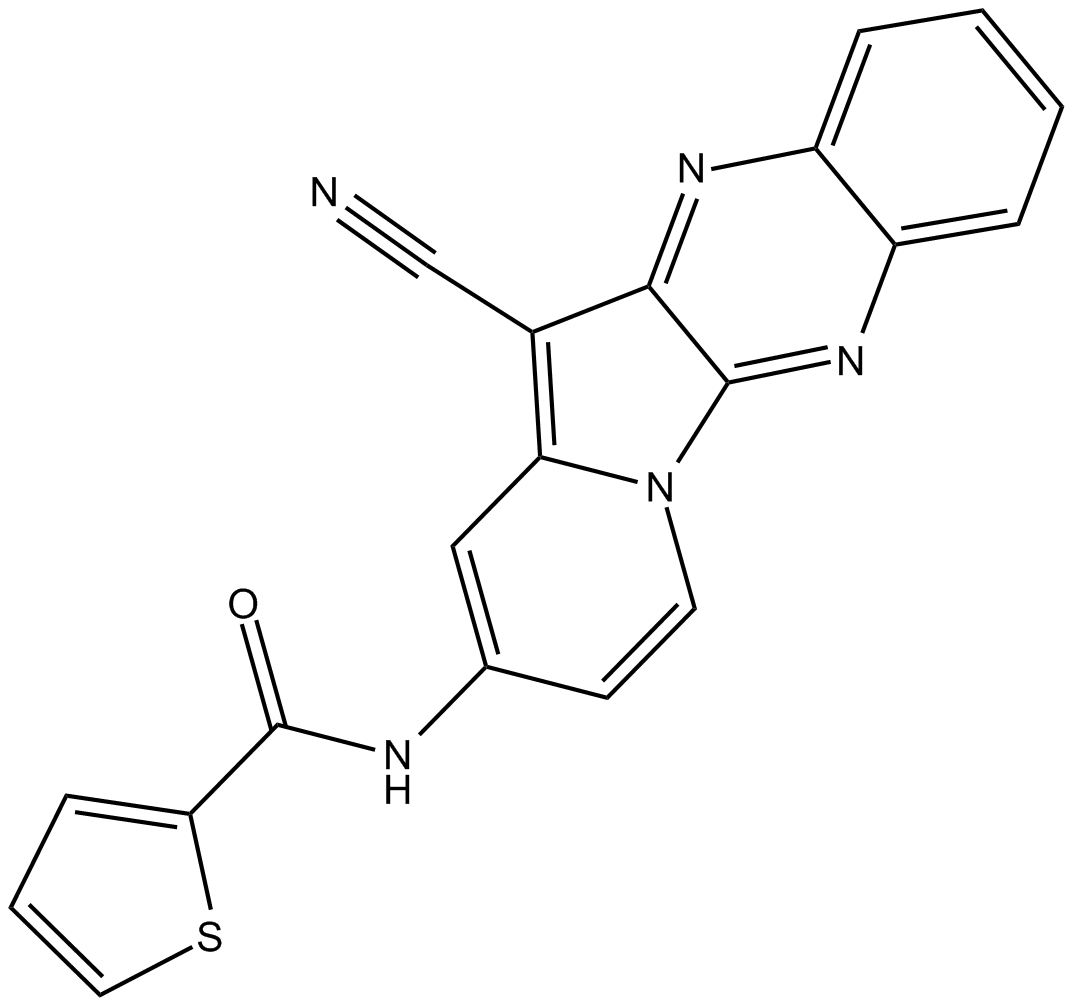
HI TOPK 032
HI TOPK 032 est un inhibiteur de TOPK puissant et spécifique.
-
GC11302
Hinokitiol
NSC 18804, β-Thujaplicin, 4-isopropyl Tropolone
L'hinokitiol est un composant des huiles essentielles isolées de Chymacyparis obtusa, réduit l'expression de Nrf2 et diminue l'expression de l'ARNm et des protéines de DNMT1 et UHRF1, avec des activités anti-infectieuses, anti-oxydantes et anti-tumorales.
-
GC12334
HNHA
Histone Deacetylase Inhibitor VI
HNHA est un puissant inhibiteur de l'histone désacétylase (HDAC). HNHA arrête le cycle cellulaire en phase G1/S via l'induction de p21. HNHA inhibe la croissance tumorale et la néovascularisation tumorale. HNHA peut être un puissant agent anticancéreux contre le cancer du sein.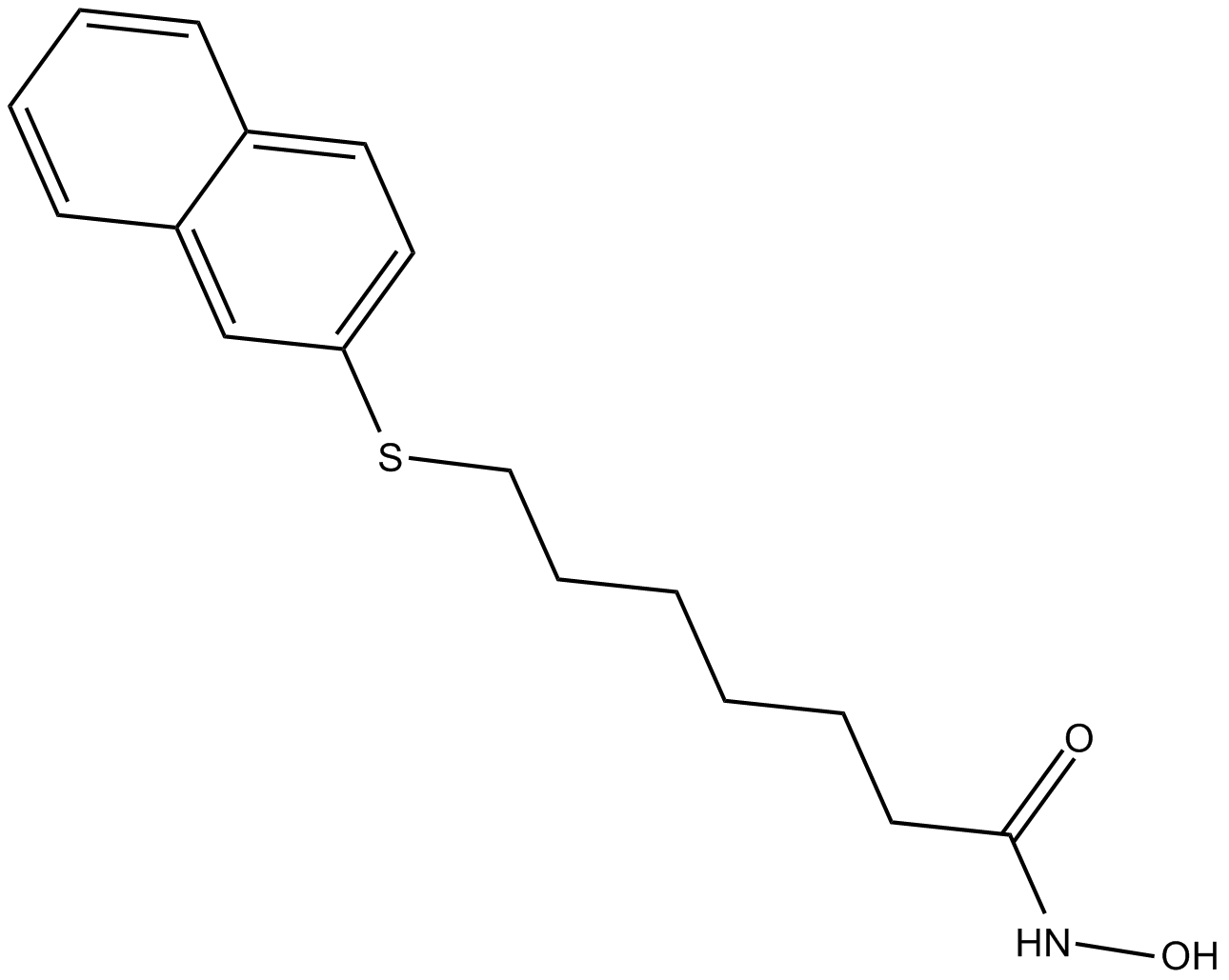
-
GC11574
HPOB
HPOB est un inhibiteur très puissant et sélectif de HDAC6 avec une IC50 de 56 nM. HPOB affiche > 30 fois moins puissant contre les autres HDAC. HPOB améliore l'efficacité des agents anticancéreux endommageant l'ADN dans les cellules transformées, mais pas dans les cellules normales. HPOB ne bloque pas l'activité de liaison À l'ubiquitine de HDAC6.
-
GC63854
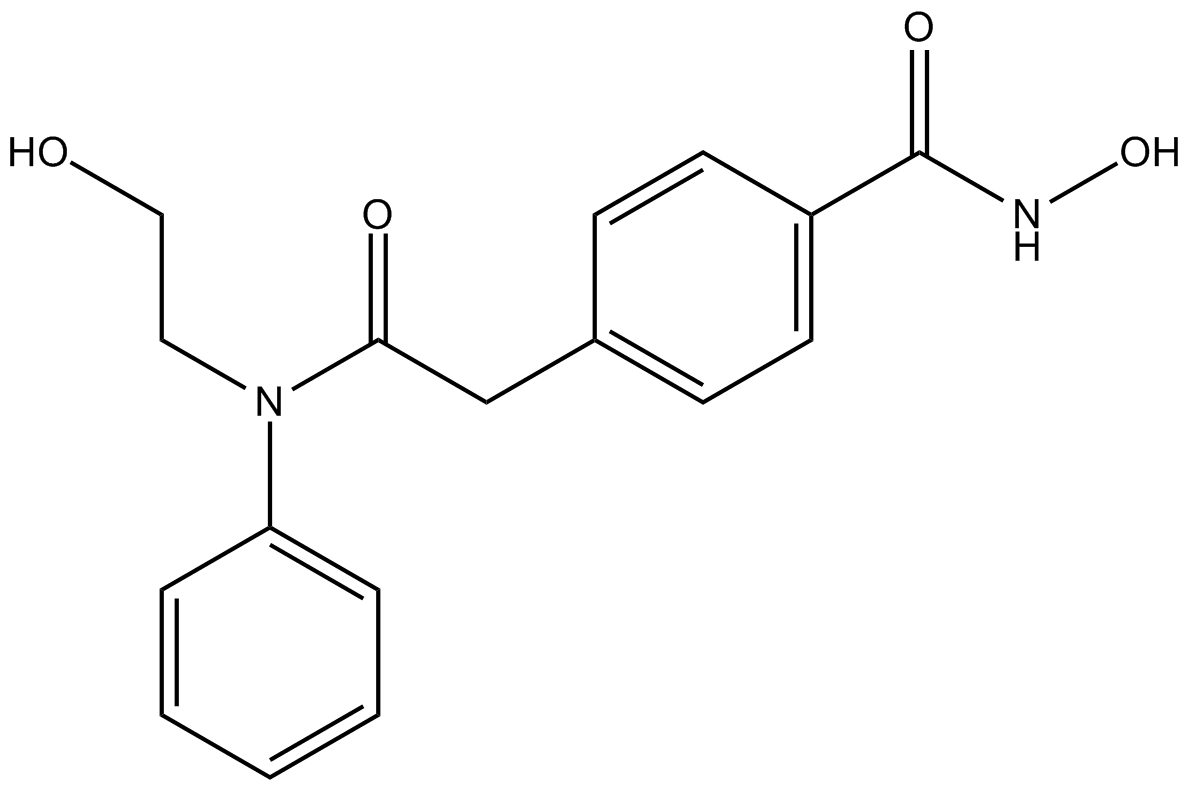
HQ461
HQ461 est une colle moléculaire qui favorise l'interaction CDK12-DDB1 pour déclencher la dégradation de la cycline K. La dégradation de la cycline K médiée par HQ461 altère la fonction de CDK12, entraÎnant une diminution de la phosphorylation du substrat de CDK12, une régulation négative des gènes de réponse aux dommages À l'ADN et la mort cellulaire.
-
GC62572
hSMG-1 inhibitor 11e
L'inhibiteur de hSMG-1 11e est un inhibiteur puissant et sélectif de la hSMG-1 kinase avec une IC50 de 900 fois la sélectivité par rapport À mTOR (IC50 de 45 nM), PI3Kα/γ (IC50 de 61 nM et 92 nM) et CDK1/CDK2 (IC50 de 32 μM et 7,1 μM).
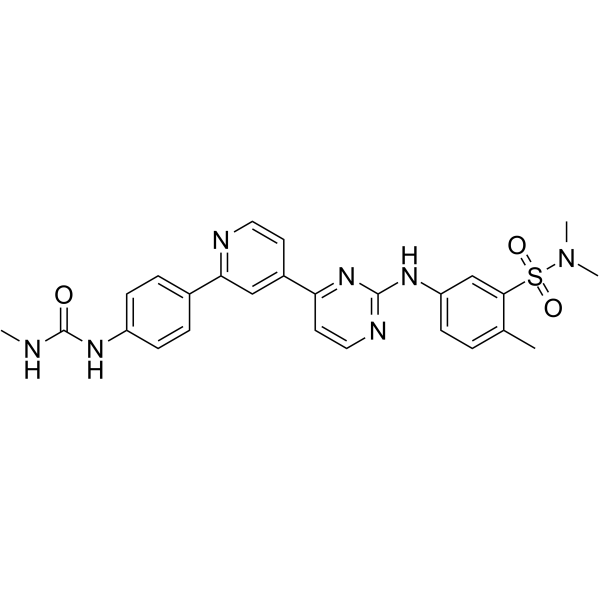
-
GC61925
hSMG-1 inhibitor 11j
L'inhibiteur de hSMG-1 11j, un dérivé de la pyrimidine, est un inhibiteur puissant et sélectif de hSMG-1, avec une IC50 de 0,11 nM. L'inhibiteur de hSMG-1 11j présente une sélectivité > 455 fois pour hSMG-1 sur mTOR (IC50 = 50 nM), PI3Kα/γ (IC50 = 92/60 nM) et CDK1/CDK2 (IC50 = 32/7,1 μM). L'inhibiteur de hSMG-1 11j peut être utilisé pour la recherche sur le cancer.
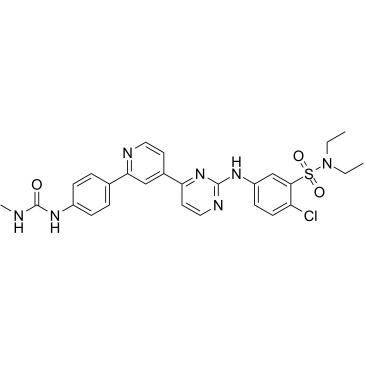
-
GC16843
Hydroxyurea
NCI C04831, NSC 32065
L'hydroxyurée est un inducteur de l'apoptose cellulaire qui inhibe la synthèse de l'ADN par inhibition de la ribonucléotide réductase. L'hydroxyurée présente une activité anti-orthopoxvirus.
-
GC49694
Hygromycin A
(-)-Hygromycin A, Totomycin
An antibiotic
-
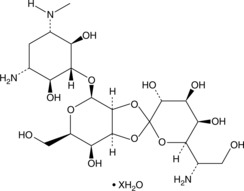
GC48445
Hygromycin B (hydrate)
An aminoglycoside antibiotic
-
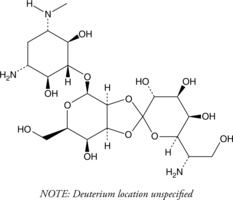
GC49267
Hygromycin B-d4
An internal standard for the quantification of hygromycin
-
GC14503
IC261
SU5607
IC261 est un inhibiteur sélectif de CK1 compétitif pour l'ATP, avec des IC50 de 1 μM, 1 μM, 16 μM pour Ckiδ, Ckiε et Ckiα1, respectivement.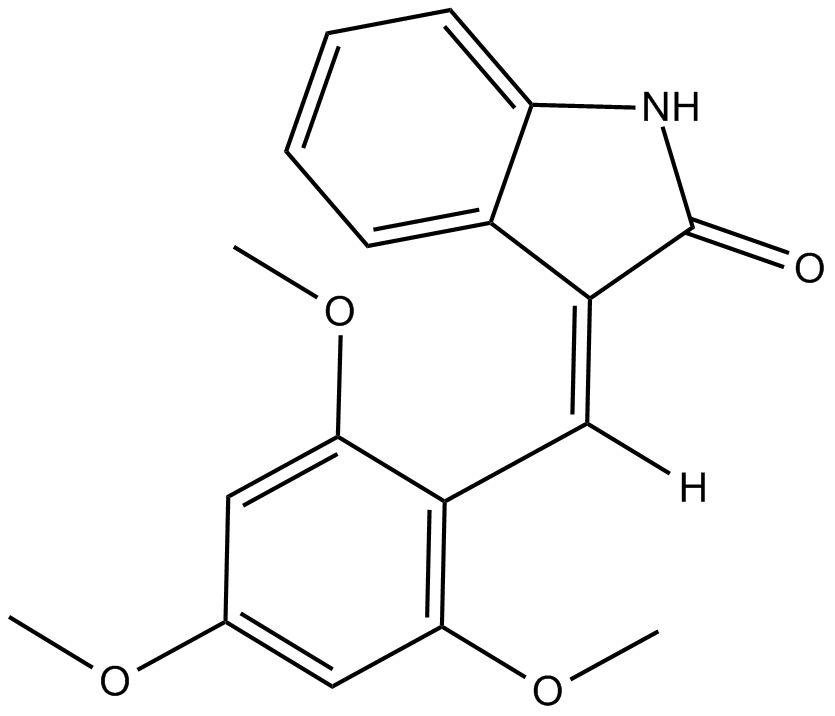
-
GC14969
Idarubicin HCl
4Demethoxydaunorubicin, 4DMD, NSC 256439
Idarubicin HCl est un médicament antileucémique anthracycline.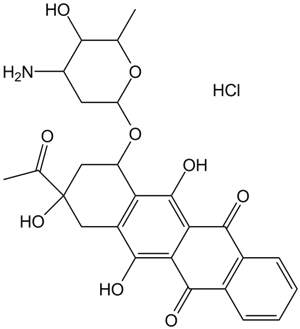
-
GC16003
Ifosfamide
Isophosphamide, NSC 109724
L'ifosfamide est un agent chimiothérapeutique alkylant ayant une activité contre un large éventail de tumeurs.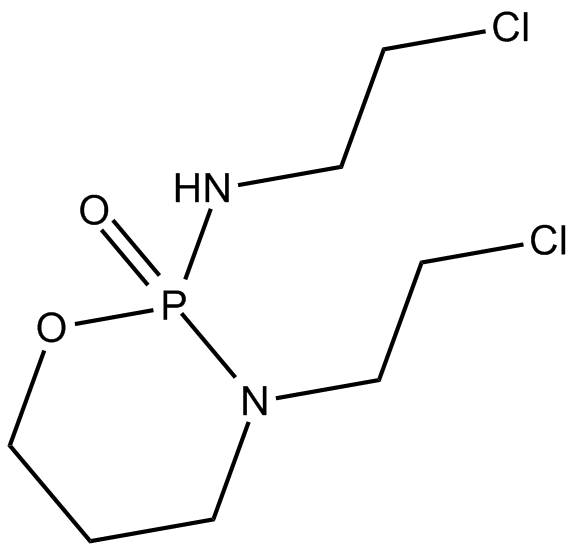
-
GC60929
Ilaprazole sodium
L'ilaprazole (IY-81149) sodique est un inhibiteur de la pompe À protons actif par voie orale.
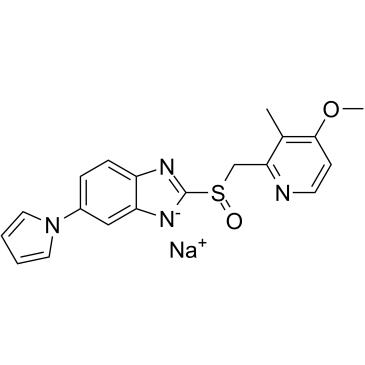
-
GC41340
Illudin M
DR-15977, (-)-Illudin M, NSC 400978, NSC 626370
Illudin M est un sesquiterpène fongique cytotoxique qui peut être isolé du milieu de culture des champignons Omphalotus olearius. Illudin M peut alkyler l'ADN. Illudin M a des activités anti-tumorales.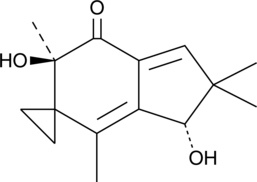
-
GC18100
Illudin S
Lampterol,NSC 400979,NSC 626369
Illudin S, un Illudin cytotoxique, est un sesquiterpène naturel avec de fortes activités antitumorales et antivirales.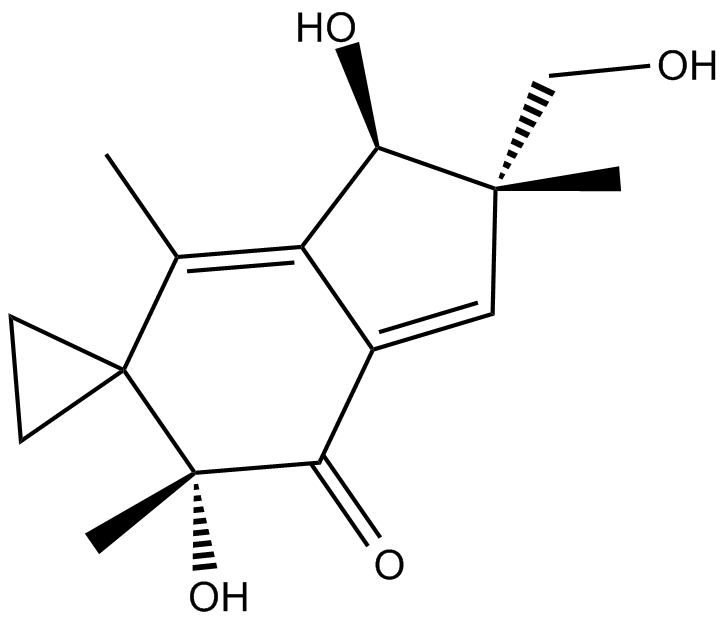
-
GC62137
IMT1
IMT1 est un inhibiteur spécifique et non compétitif de l'ARN polymérase mitochondriale humaine (POLRMT) de première classe.
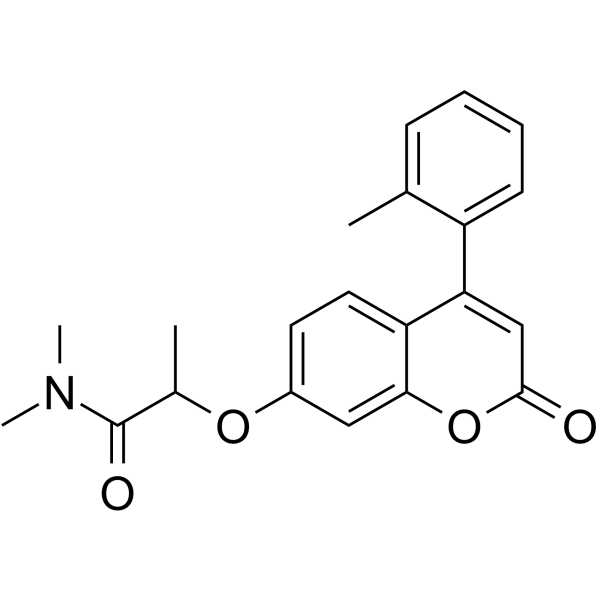
-
GC65939
Indimitecan
LMP776
L'indimitecan (LMP776) est un inhibiteur de la topoisomérase I (Top1) ayant des activités anticancéreuses.
-
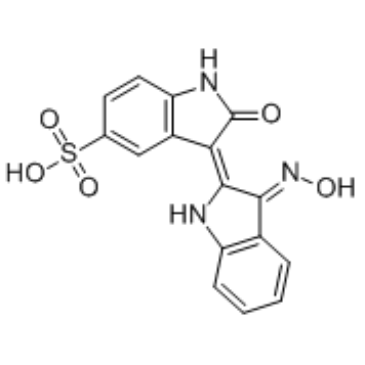
GC36312
Indirubin-3'-monoxime-5-sulphonic acid
L'acide indirubine-3'-monoxime-5-sulfonique est un inhibiteur puissant et sélectif de CDK1, CDK5 et GSK-3β avec des IC50 de 5 nM, 7 nM et 80 nM, respectivement.
-
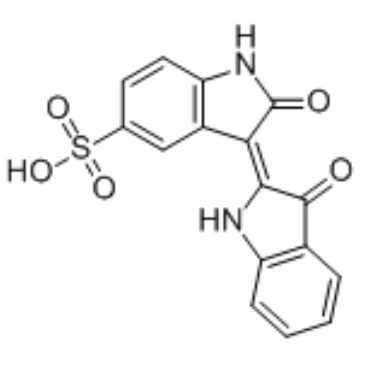
GC36313
Indirubin-5-sulfonate
L'indirubine-5-sulfonate est un inhibiteur de la kinase cycline-dépendante (CDK), avec des valeurs IC50 de 55 nM, 35 nM, 150 nM, 300 nM et 65 nM pour CDK1/cycline B, CDK2/cycline A, CDK2/cycline E, CDK4/cycline D1 et CDK5/p35, respectivement. L'indirubine-5-sulfonate présente également une activité inhibitrice contre GSK-3β.
-
GC52513
Indolimine-214
Isoamylindolimine
A genotoxic gut microbiota metabolite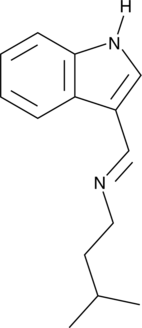
-
GC33205
Indotecan (LMP-400)
LMP-400; NSC-724998
L'indotecan (LMP-400) (LMP-400) est un puissant inhibiteur de la topoisomérase 1 (Top1) avec des valeurs IC50 de 300, 1200, 560 nM pour les lignées cellulaires P388, HCT116, MCF-7, respectivement.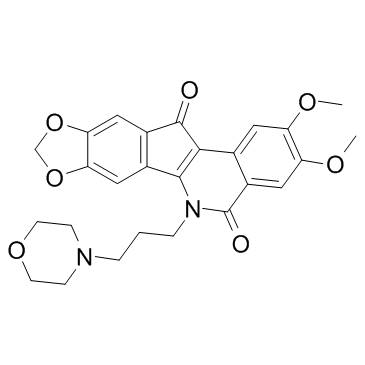
-
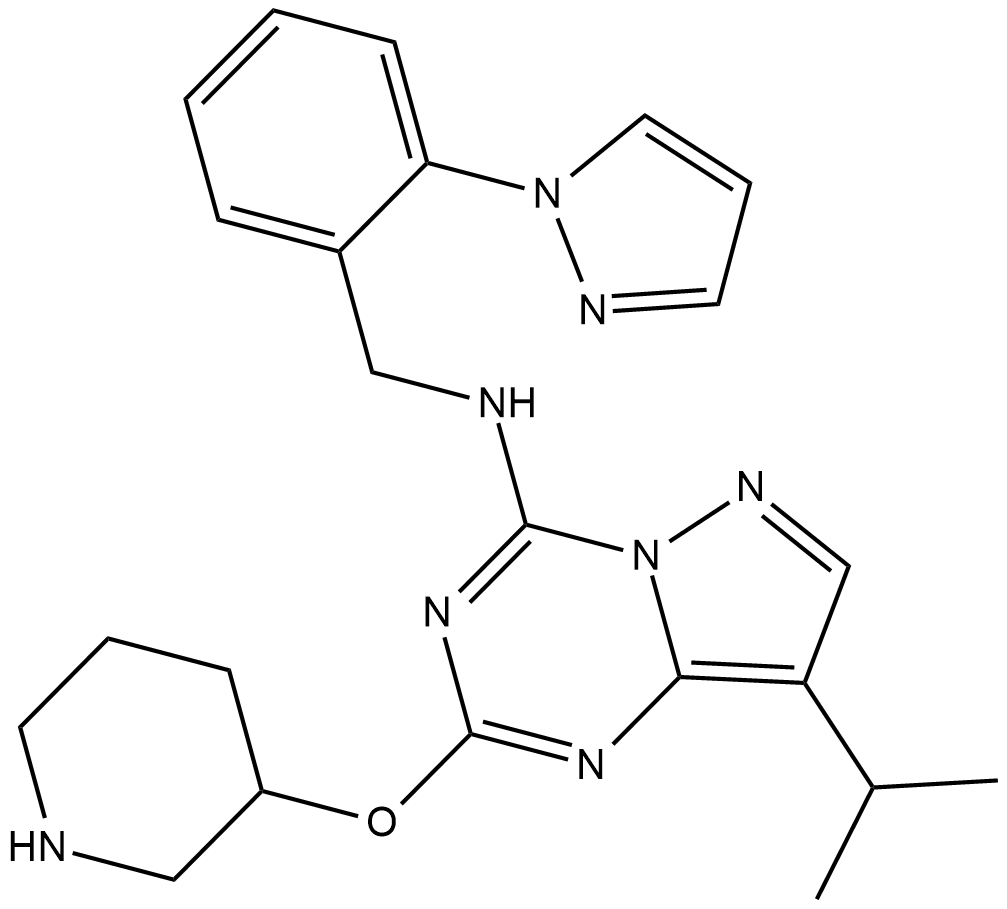
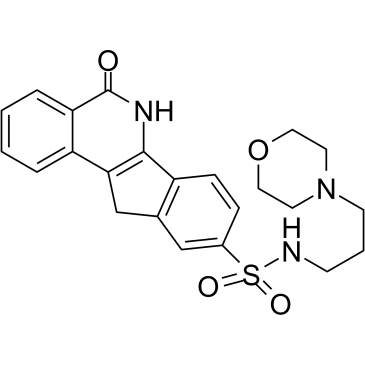
GC38385
INO-1001
-
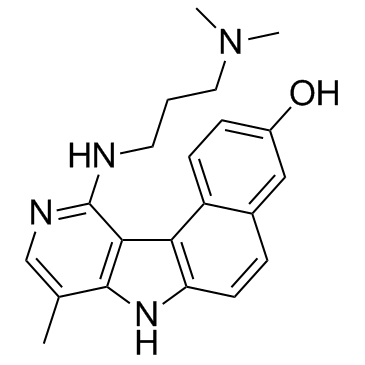
GC33165
Intoplicine
L'intoplicine (RP 60475), un dérivé antitumoral de la série 7H-benzo[e]pyrido[4,3-b]indole, est un inhibiteur de l'ADN topoisomérase I et II. L'intoplicine se lie fortement À l'ADN (KA = 2 x 105/M) et augmente ainsi la longueur de l'ADN linéaire.
-
GC63354
Intoplicine dimesylate
RP 60475 dimesylate
Le dimésylate d'intoplicine (RP 60475), un dérivé antitumoral de la série 7H-benzo[e]pyrido[4,3-b]indole, est un inhibiteur de l'ADN topoisomérase I et II. Le dimésylate d'intoplicine se lie fortement À l'ADN (KA = 2 x 105/M) et augmente ainsi la longueur de l'ADN linéaire.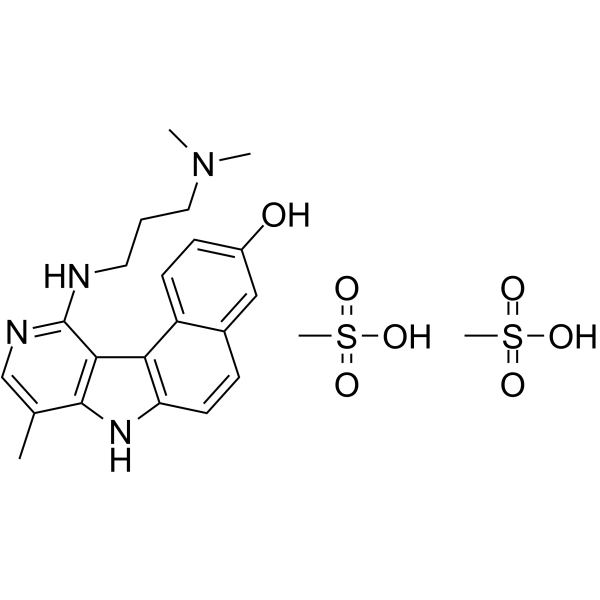
-
GC63023
Ipivivint
Ipivivint (composé 38) est un puissant inhibiteur de la kinase de type CDC (CLK) avec des CE50 de 1 nM, 7 nM pour CLK2 et CLK3, respectivement. Ipivivint inhibe la voie Wnt (EC50 = 13 nM).
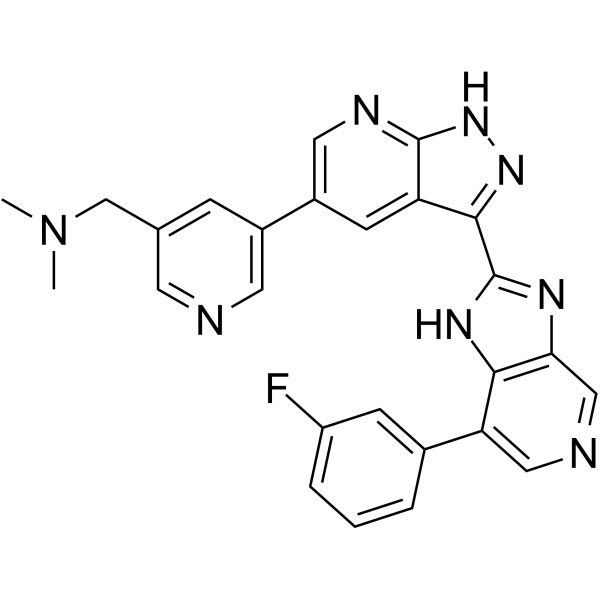
-
GC52175
IQA
CGP 029482
-
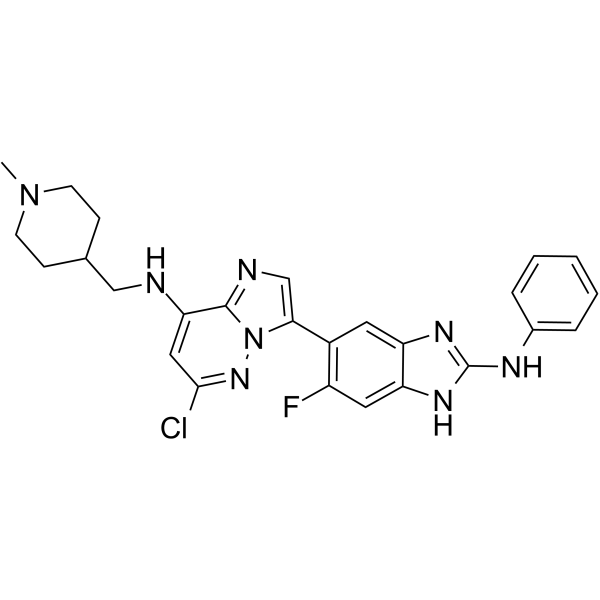
GC63513
IRE1α kinase-IN-1
IRE1α ; la kinase-IN-1 est une IRE1α hautement sélective; (ERN1), avec une IC50 de 77 nM.
-
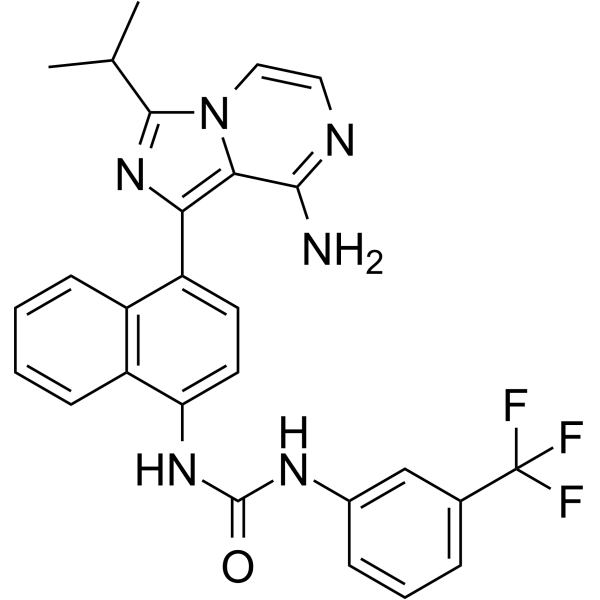
GC64232
IRE1α kinase-IN-2
IRE1α ; la kinase-IN-2 est une puissante IRE1α ; inhibiteur de kinase, avec une CE50 de 0,82 μM. IRE1α ; la kinase-IN-2 inhibe IRE1α ; autophosphorylation de la kinase (IC50=3,12 μM). IRE1α ; La kinase-IN-2 inhibe l'épissage de l'ARNm de XBP1 dans les lignées cellulaires WT.
-
GC11473
Irinotecan
Inhibiteur de la topoisomérase I
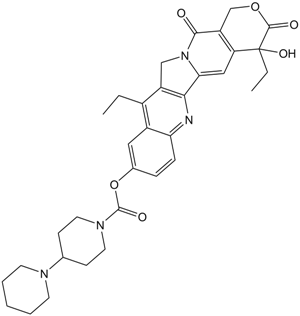
-
GC11048
Irinotecan HCl Trihydrate
Irinotecan HCl Trihydrate ((+)-Irinotecan HCl Trihydrate) est un inhibiteur de la topoisomérase I avec une activité antitumorale.
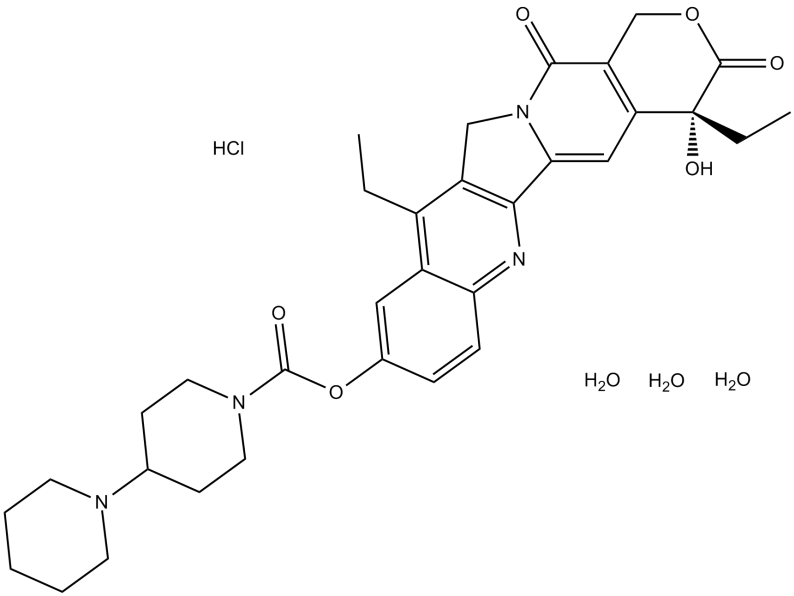
-
GC36329
Irinotecan hydrochloride
Le chlorhydrate d'irinotécan ((+)-chlorhydrate d'irinotécan) est un inhibiteur de la topoisomérase I principalement utilisé pour traiter le cancer du cÔlon et le cancer du rectum.
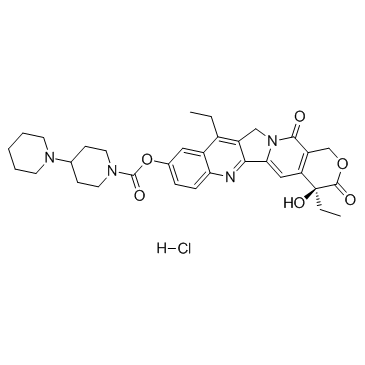
-
GC30554
Isocytosine
L'isocytosine est une nucléobase non naturelle et un isomère de la cytosine.
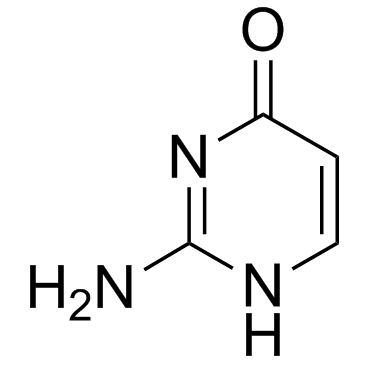
-
GC33662
Isoguanine
Crotonoside, 2-hydroxy-6-Aminopurine, 2-Hydroxyadenine, NSC 241501, 2-Oxoadenine
L'isoguanine est une base purique qui est un isomère de la guanine.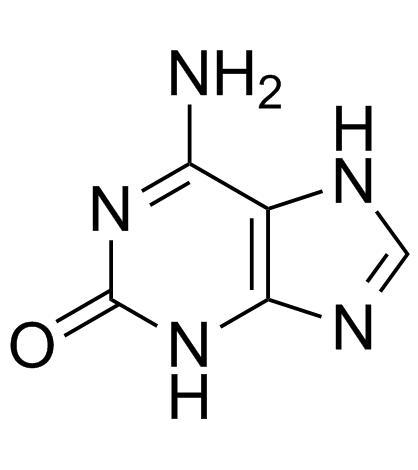
-
GC39140
Isopimpinellin
NSC 217988, NSC 401288
Isopimpinellin, un composé oralement actif isolé des racines de Pimpinella saxifrage.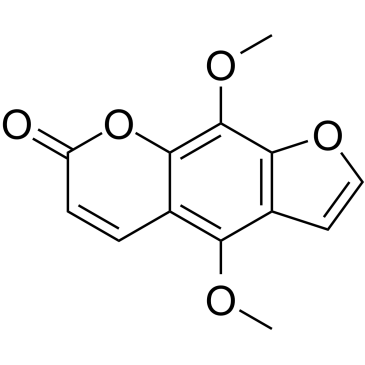
-
GC10834
Isoxanthopterin
NSC 118090,NSC 614991
interferes with RNA and DNA synthesis
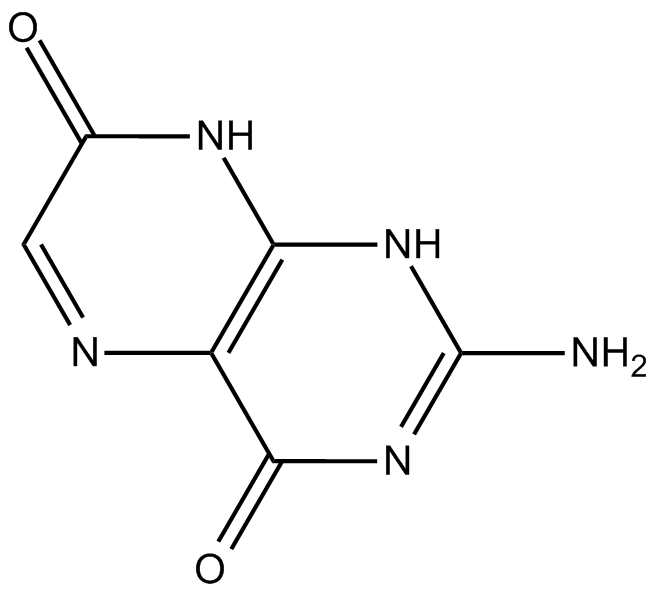
-
GC14597
ITSA-1 (ITSA1)
ITSA-1 (ITSA1) est un activateur de l'histone désacétylase (HDAC) et contrecarre l'arrêt du cycle cellulaire induit par la trichostatine A (TSA), l'acétylation des histones et l'activation de la transcription.
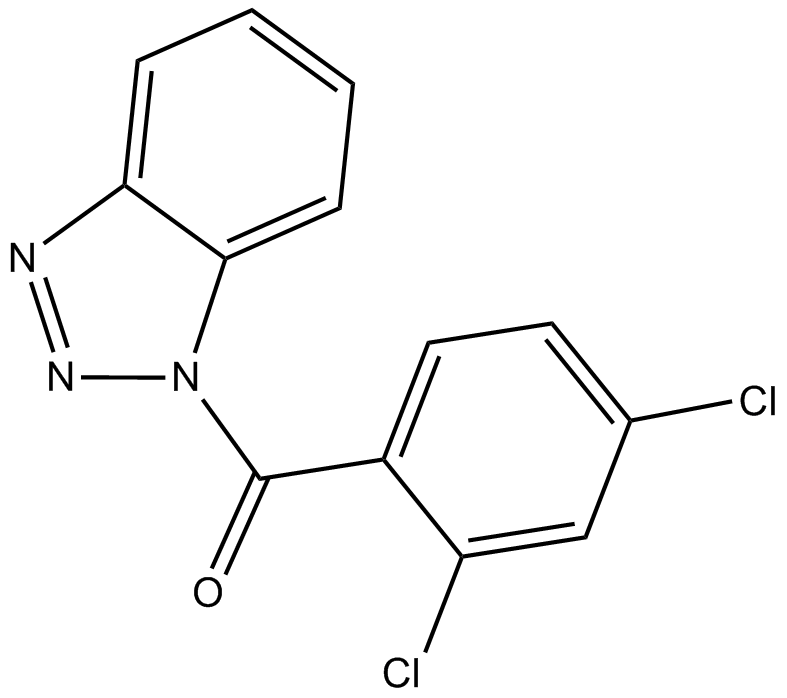
-
GC14797
IU1
Usp14 inhibitor
A deubiquitinating enzyme inhibitor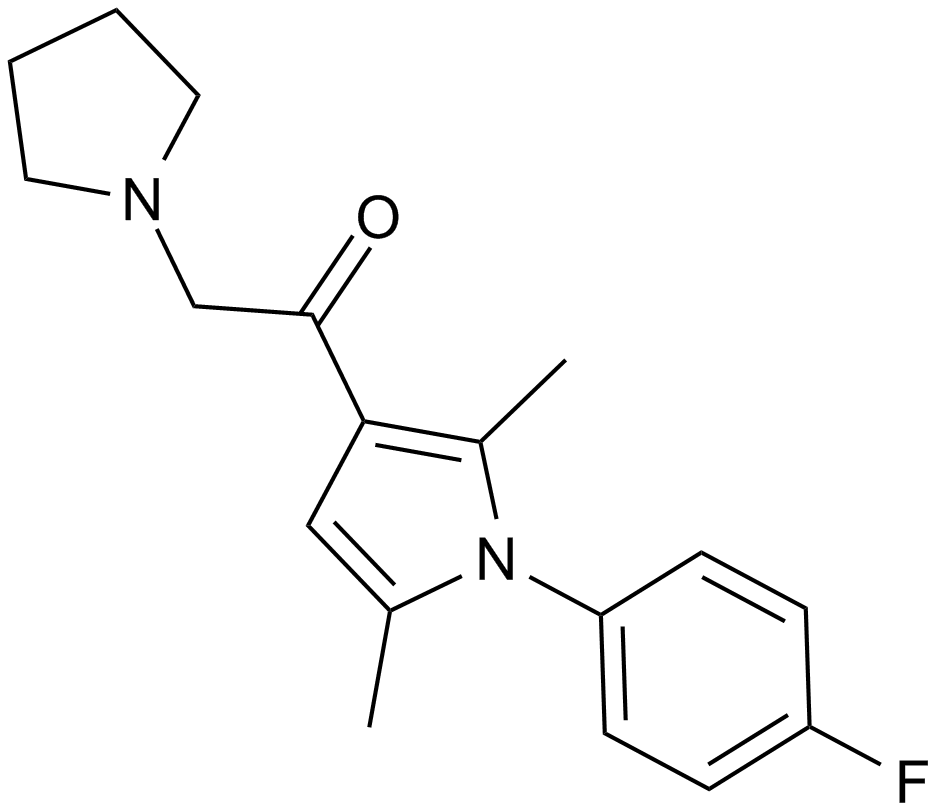
-
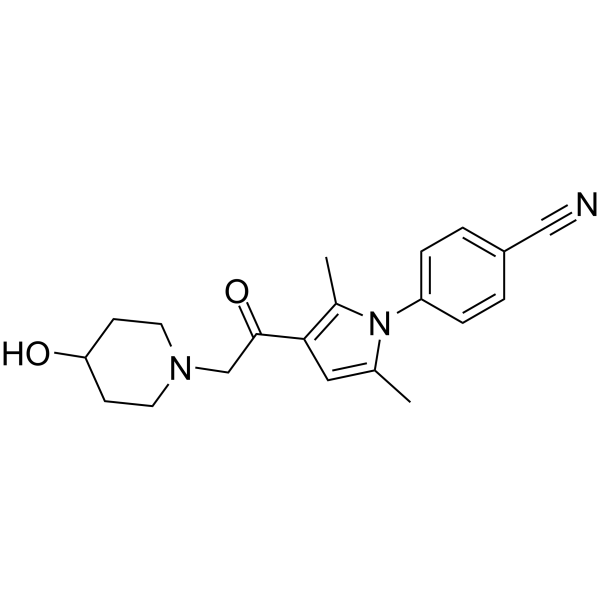
GC63027
IU1-248
IU1-248, un dérivé de IU1, est un inhibiteur puissant et sélectif de l'USP14 avec une IC50 de 0,83μM.
-
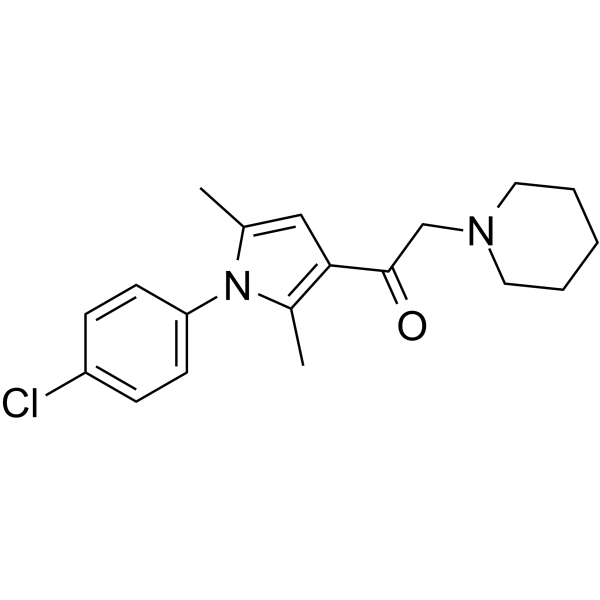
GC64956
IU1-47
IU1-47 est un inhibiteur puissant et spécifique de l'USP14 avec une IC50 de 0,6 μM.
-
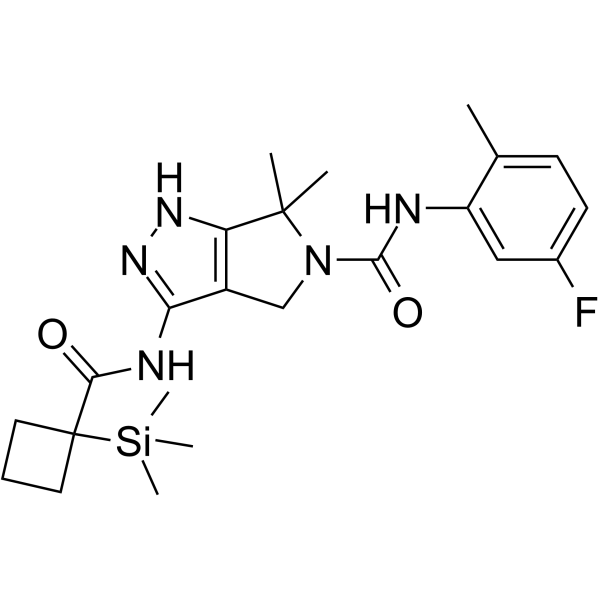
GC63460
IV-361
IV-361 est un inhibiteur de CDK7 actif et sélectif par voie orale (Ki≤50 nM). IV-361 a une activité anticancéreuse (US20190256531A1).
-
GC16454
IWP-2
IWP 2
IWP-2 is a inhibitor of the Wnt signaling pathway, with an IC50 value of 27 nM. IWP-2 is also an ATP-competitive inhibitor of CK1δ, with an IC50 value of 40 nM.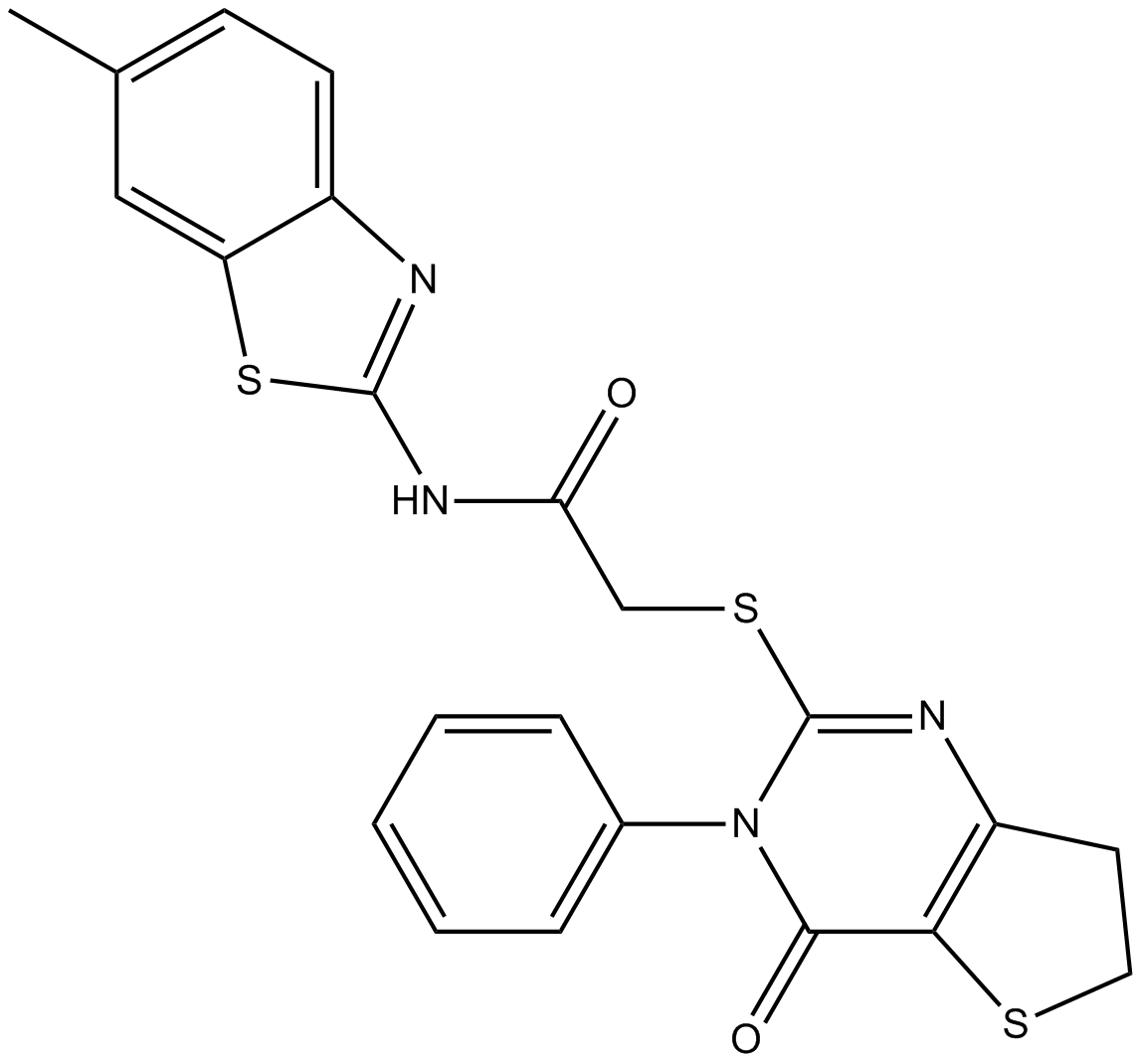
-
GC63785
IXA4
IXA4 est un activateur IRE1/XBP1s hautement sélectif et non toxique.
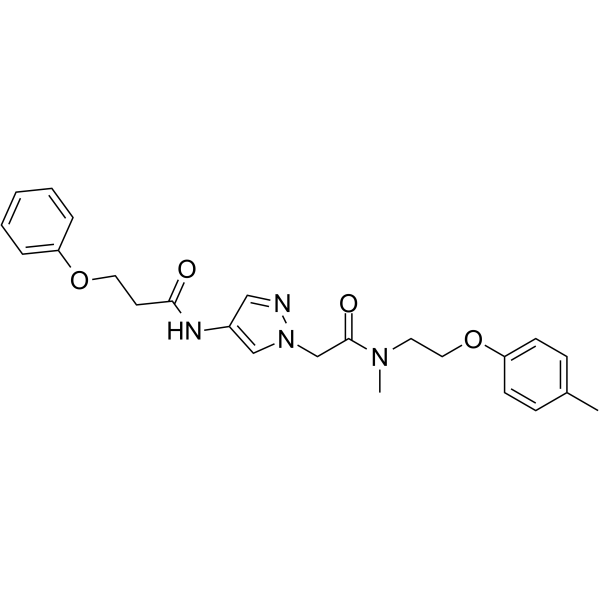
-
GC34629
J22352
J22352 est un inhibiteur HDAC6 de type PROTAC (protéolyse-ciblage des chimères) et hautement sélectif avec une valeur IC50 de 4,7 nM. J22352 favorise la dégradation de HDAC6 et induit des effets anticancéreux en inhibant l'autophagie et en déclenchant la réponse immunitaire antitumorale dans les cancers du glioblastome, et en conduisant À la restauration de l'activité antitumorale de l'hÔte en réduisant l'activité immunosuppressive de PD-L1.
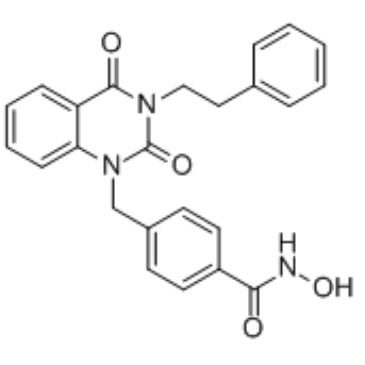
-
GC64959
JAK/HDAC-IN-1
JAK/HDAC-IN-1 est un puissant double inhibiteur de JAK2/HDAC, présente des activités antiprolifératives et proapoptotiques dans plusieurs lignées cellulaires hématologiques. JAK/HDAC-IN-1 montre des IC50 de 4 et 2 nM pour JAK2 et HDAC, respectivement.
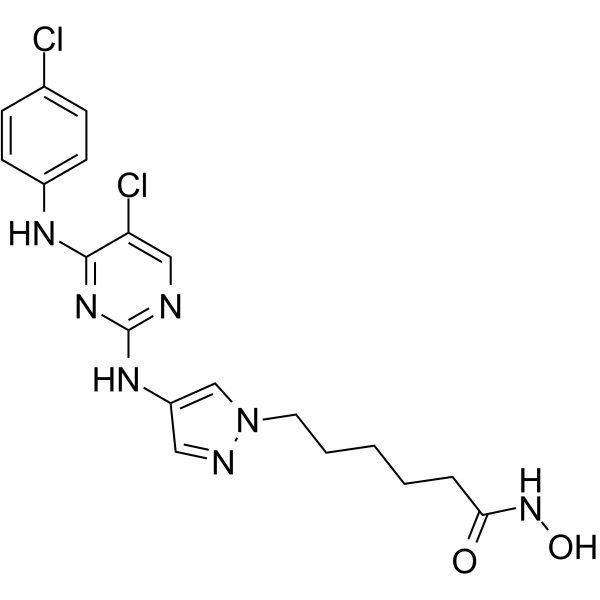
-
GC36367
JH-RE-06
JH-RE-06, un puissant inhibiteur de l'interface REV1-REV7 (IC50=0,78 μM ; Kd=0,42 μM), cible REV1 qui interagit avec la sous-unité REV7 de POLζ. JH-RE-06 perturbe la synthèse de la translésion mutagène (TLS) en empêchant le recrutement de POLζ mutagène. JH-RE-06 améliore la chimiothérapie.
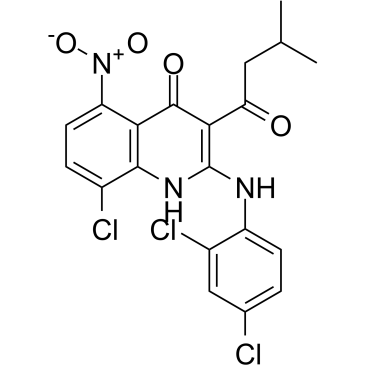
-
GC65471
JH-XI-10-02
JH-XI-10-02 est un PROTAC connecté par des ligands pour Cereblon et CDK. JH-XI-10-02 est un dégradeur de CDK8 PROTAC hautement puissant et sélectif, avec une IC50 de 159 nM. JH-XI-10-02 provoque une dégradation protéasomale, sans affecter les niveaux d'ARNm de CDK8. JH-XI-10-02 ne montre aucun effet sur CDK19.
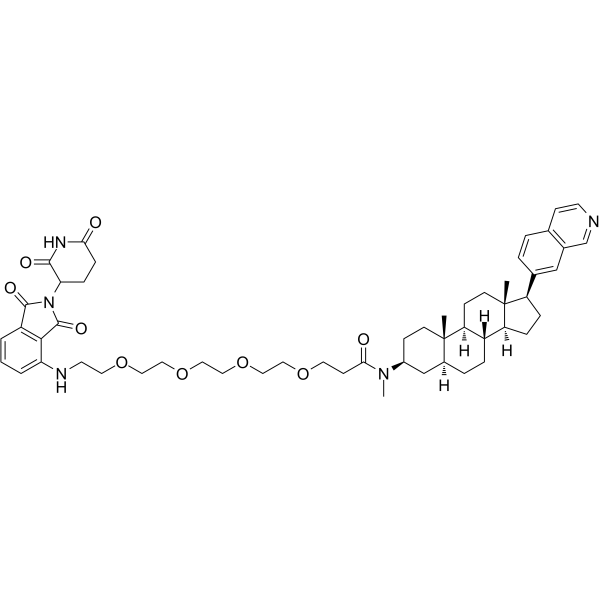
-
GC65577
JH-XVI-178
JH-XVI-178 est un inhibiteur très puissant et sélectif de CDK8/19 qui affiche une faible clairance et des propriétés pharmacocinétiques orales modérées.

-
GC12612
JNJ-7706621
JNJ7706621, JNJ 7706621
A dual inhibitor of CDKs and Aurora kinases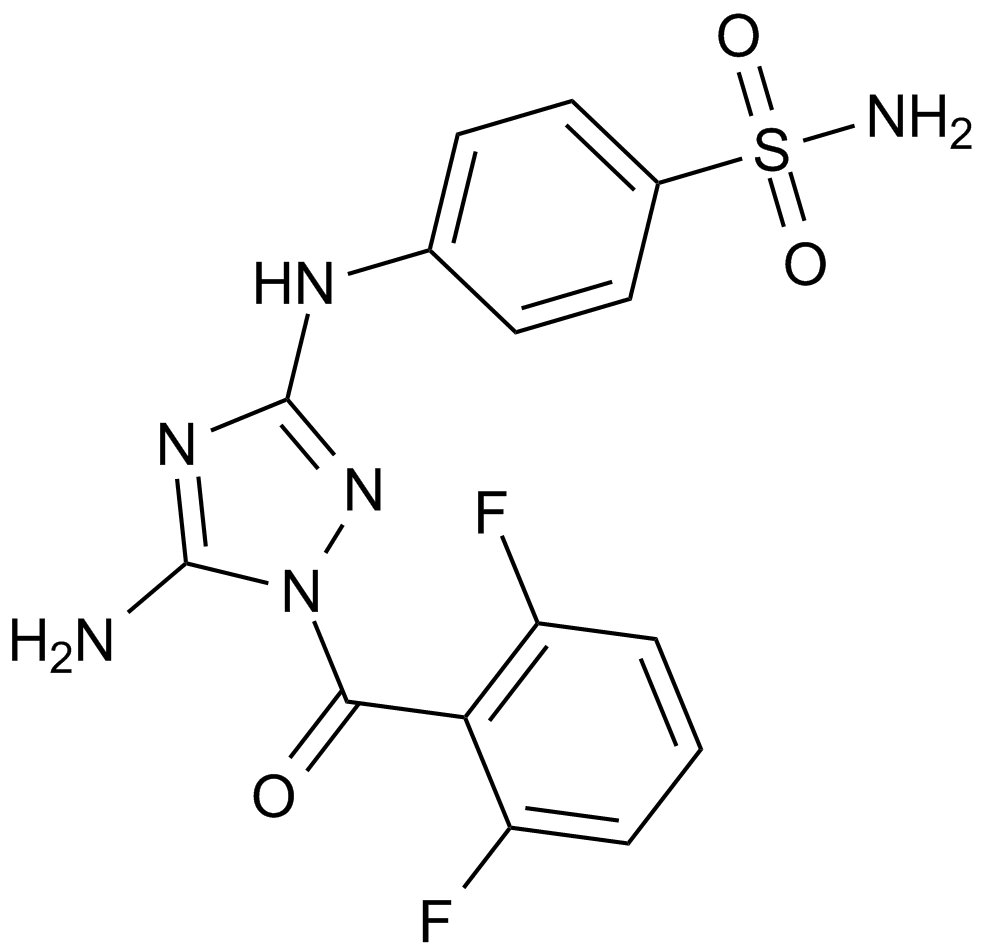
-
GC18734
JS-K
JS-K est un donneur de NO qui réagit avec le glutathion pour générer du NO À pH physiologique. JS-K inhibe la prolifération, induit l'apoptose et perturbe le cycle cellulaire des cellules de la leucémie lymphoblastique aiguë Jurkat T.
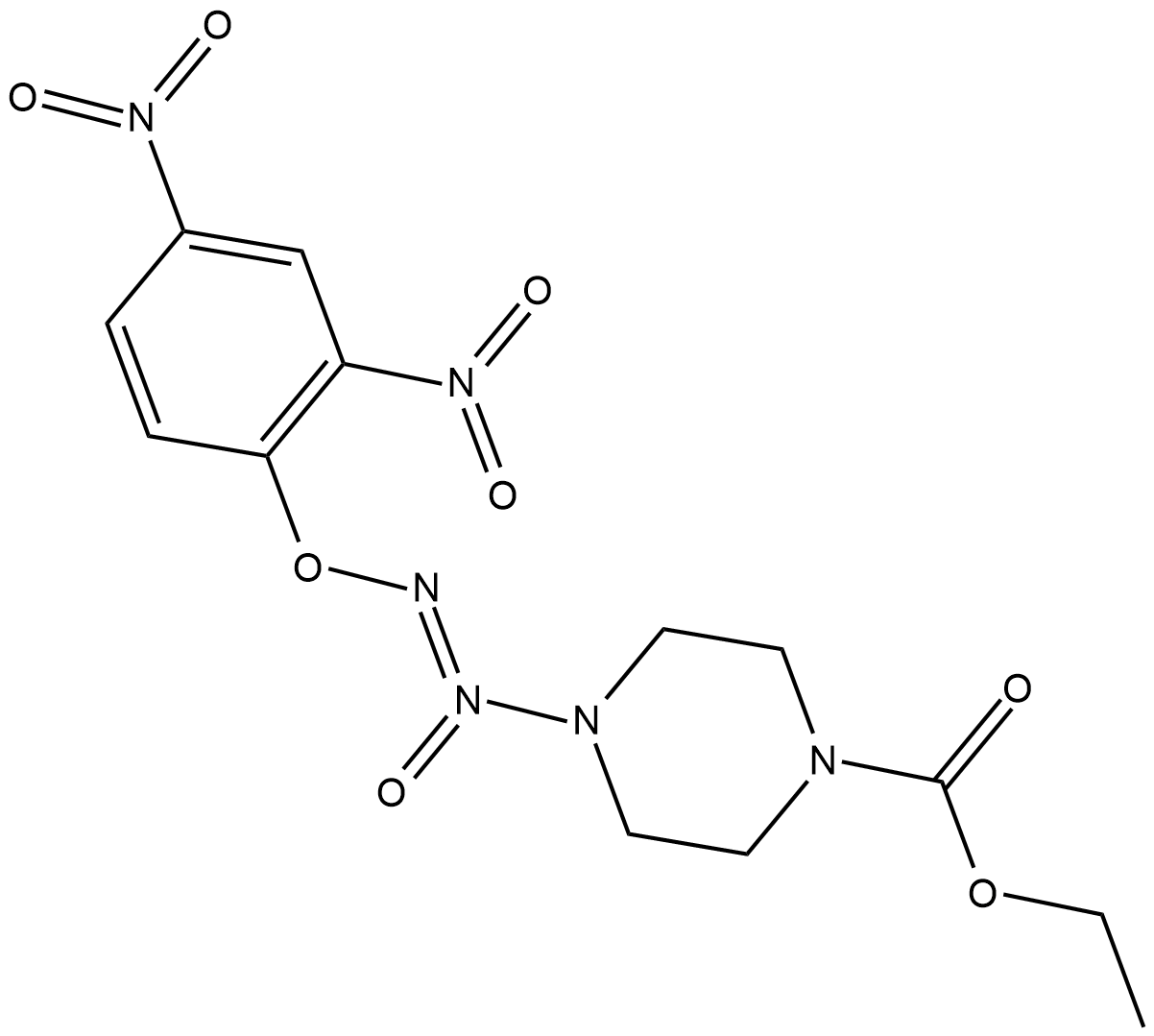
-
GC34204
JSH-150
JSH-150 est un inhibiteur de CDK9 hautement sélectif et puissant avec une IC50 de 1 nM.
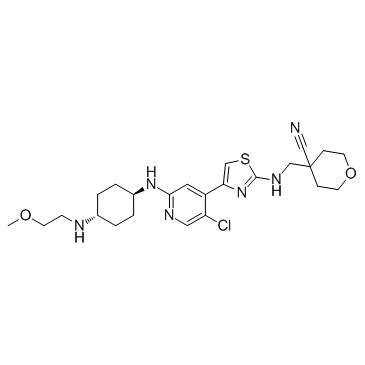
-
GC50117
JTE 607 dihydrochloride
Le dichlorhydrate de JTE 607, un inhibiteur hautement sélectif de la synthèse des cytokines inflammatoires, protège du choc endotoxinique chez la souris.
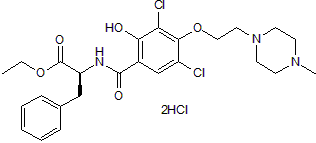
-
GC13978
JW 74
JW 74 antagonise l'activation induite par LiCl de la signalisation canonique Wnt avec une IC50 de 420 nM.
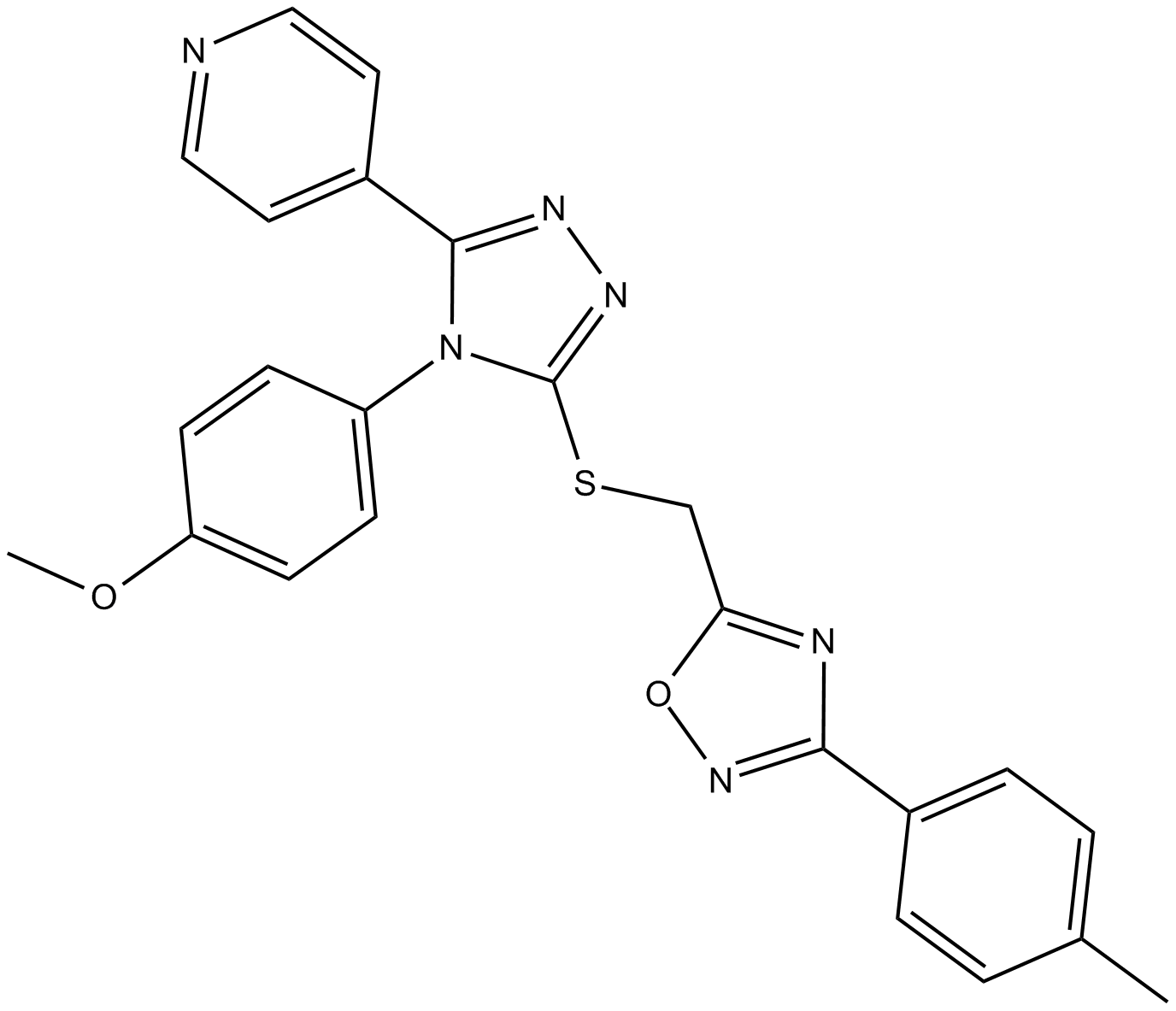
-
GC34195
K-756
Le K-756 est un inhibiteur direct et sélectif de la tankyrase (TNKS), qui inhibe l'activité d'ADP-ribosylation de TNKS1 et TNKS2 avec des IC50 de 31 et 36 nM, respectivement.
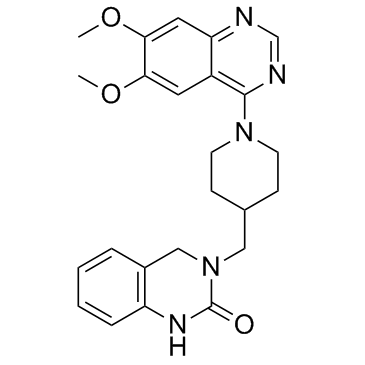
-
GC41384
K-TMZ
K-TMZ is a DNA alkylating agent.
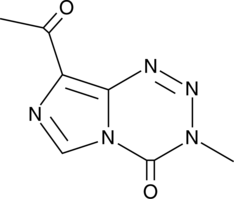
-
GC14230
K03861
K03861 (K03861) est un inhibiteur de CDK2 de type II avec un Kd de 8,2 nM. K03861 (K03861) inhibe l'activité de CDK2 en entrant en compétition avec la liaison des cyclines activatrices.
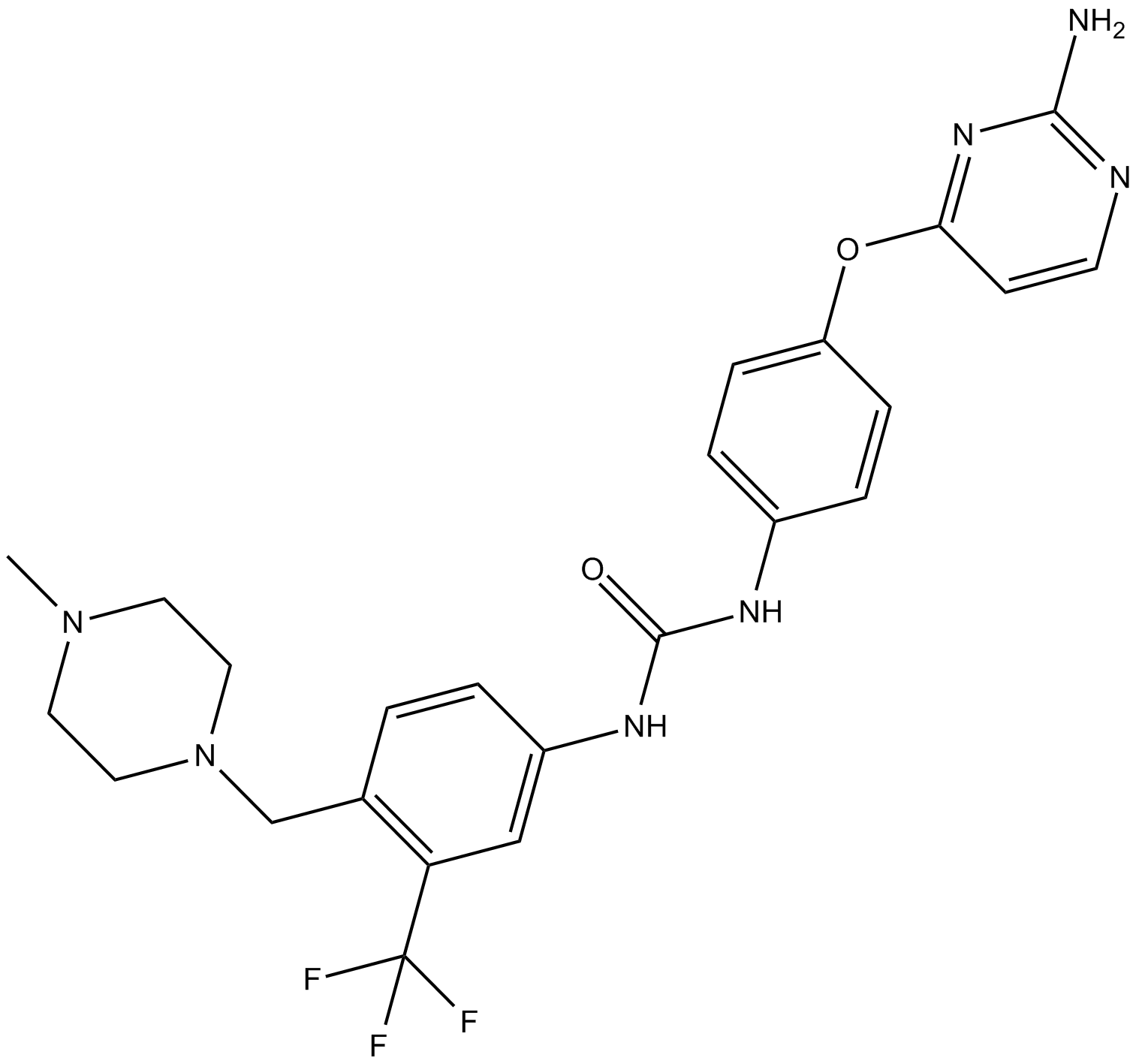
-
GC47521
K22
An antiviral agent
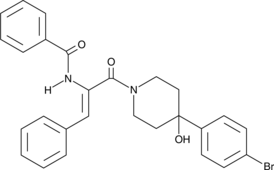
-
GC34144
Karenitecin (Cositecan)
Karenitecin (Cositecan) (Cositecan) est un inhibiteur de la topoisomérase I, avec une puissante activité anticancéreuse.
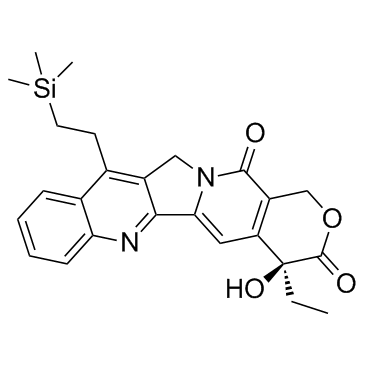
-
GC62392
KB-0742 dihydrochloride
Le dichlorhydrate de KB-0742 est un inhibiteur de CDK9 puissant, sélectif et actif par voie orale avec une IC50 de 6 nM pour CDK9/cycline T1. Le dichlorhydrate de KB-0742 est sélectif pour CDK9/cycline T1 avec une sélectivité > 50 fois supérieure aux autres CDK kinases. Le dichlorhydrate de KB-0742 a une puissante activité anti-tumorale.
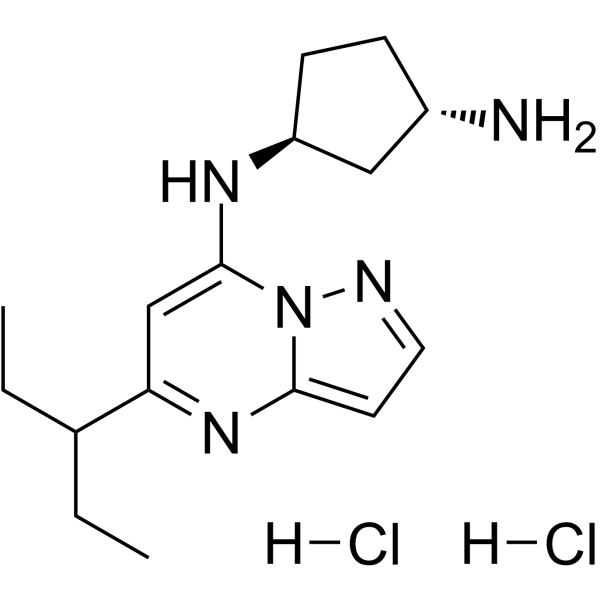
-
GC14182
Kenpaullone
9Bromopaullone, NSC 664704
La kenpaullone est un puissant inhibiteur de CDK1/cycline B et GSK-3β, avec des IC50 de 0,4 μM et 23 nM, et inhibe également CDK2/cycline A, CDK2/cycline E et CDK5/p25 avec des IC50 de 0,68 μM, 7,5 μM, 0,85 μM, respectivement. La kenpaullone, une petite molécule inhibitrice de KLF4, réduit l'auto-renouvellement des cellules souches du cancer du sein et la motilité cellulaire in vitro.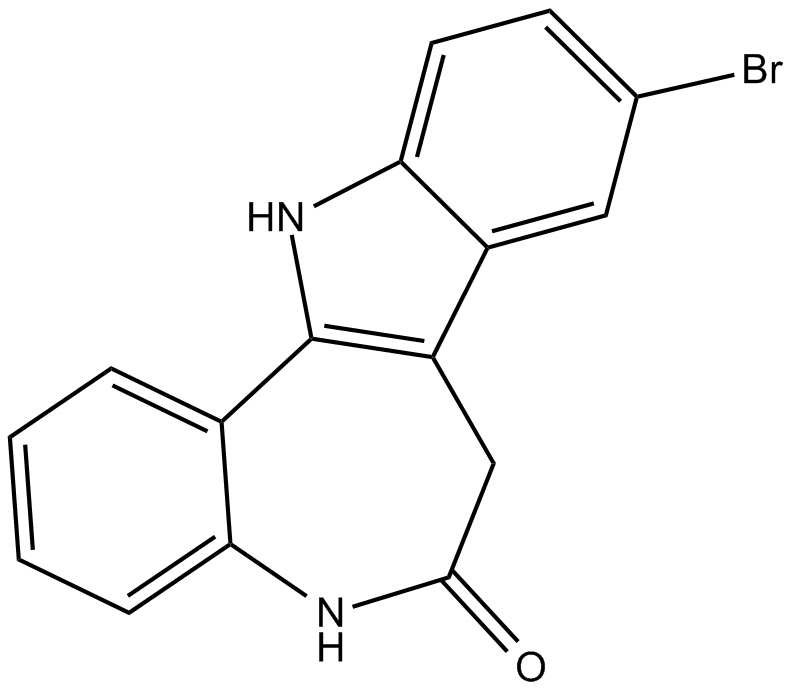
-
GC11774
KH CB19
KH CB19 est un puissant inhibiteur de la CLK (cdc2-like kinase) (CLK1 IC50 \u003d 19,7 nM ; CLK3 IC50 \u003d 530 nM).
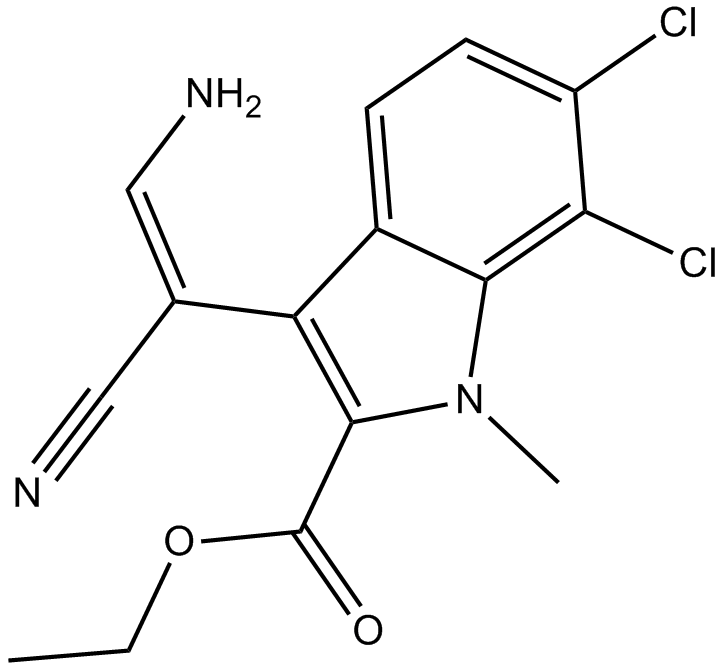
-
GC63943
KH-CB20
KH-CB20, un mélange E/Z, est un inhibiteur puissant et sélectif de CLK1 et de l'isoforme étroitement apparentée CLK4, avec une IC50 de 16,5 nM pour CLK1.
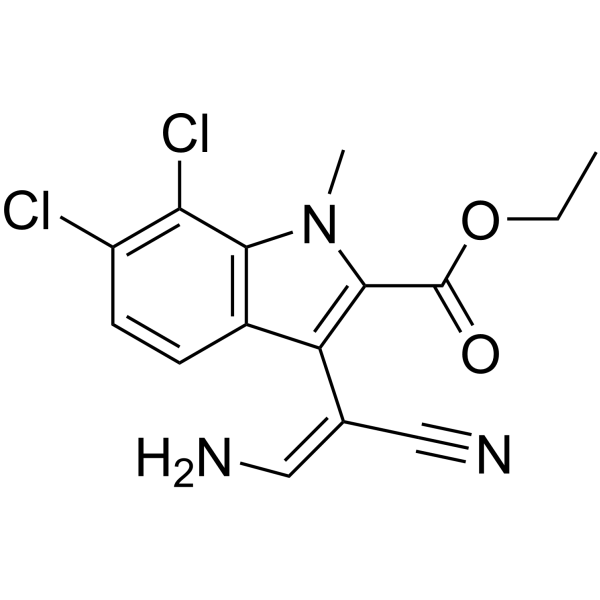
-
GC64238
KIRA-7
KIRA-7, un composé d'imidazopyrazine, se lie À la kinase IRE1α (IC50 de 110 nM) pour inhiber de manière allostérique son activité RNase.
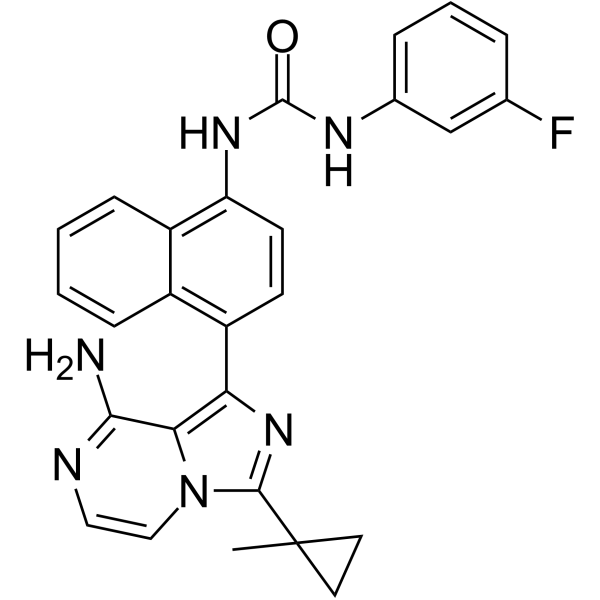
-
GC19212
KIRA6
IRE1 Inhibitor IV, IRE1α Kinase Inhibiting RNase Attenuator 6
KIRA6 est un inhibiteur avancé de la RNase kinase IRE1α À petite molécule avec une IC50 de 0,6 μM.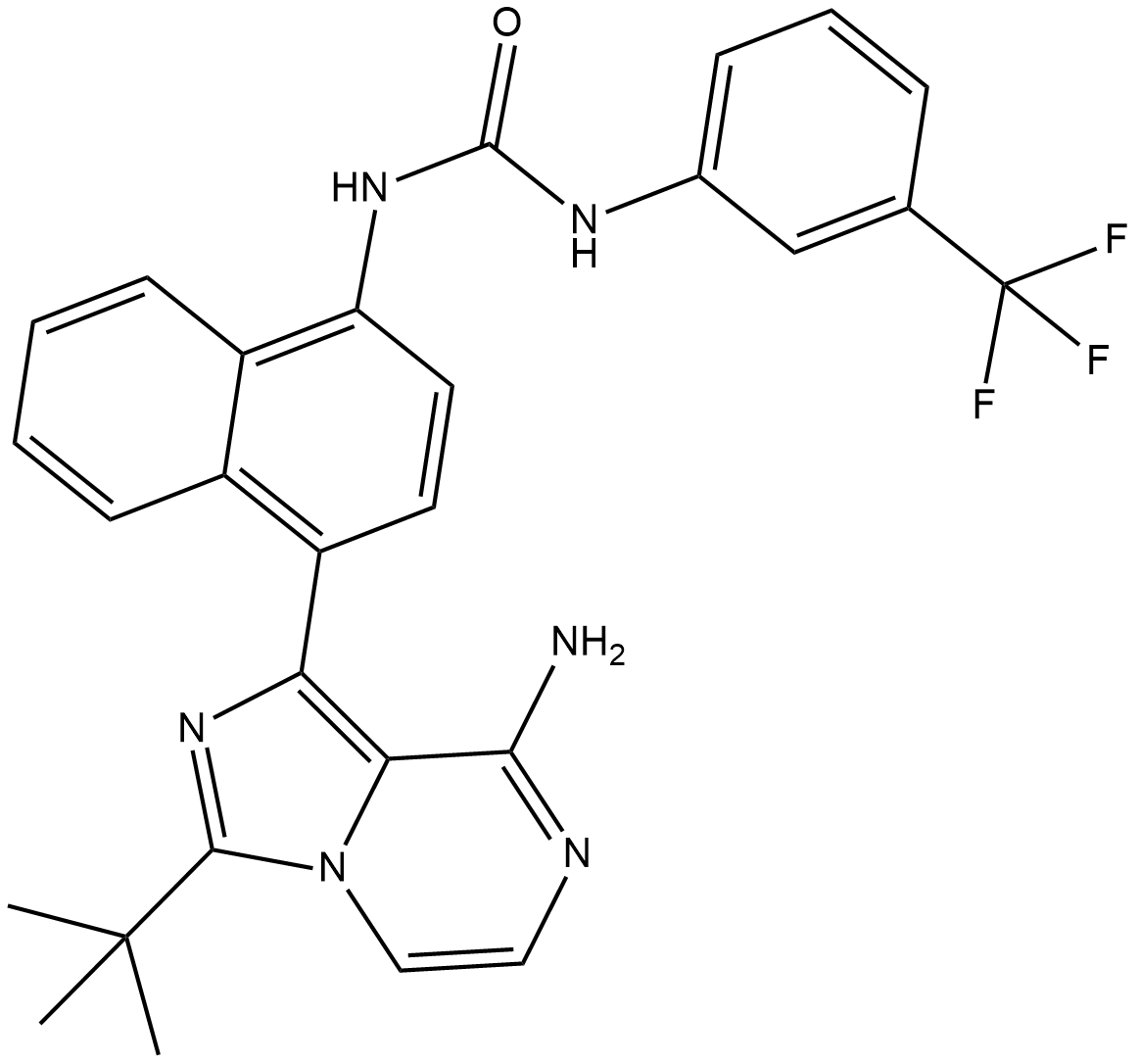
-
GC31879
Kira8
AMG-18
Kira8 (AMG-18) est un inhibiteur monosélectif de IRE1α qui atténue de manière allostérique l'activité IRE1α RNase avec une IC50 de 5,9 nM.
-
GC65907
KSQ-4279
USP1-IN-1
KSQ-4279 (USP1-IN-1, Formule I) est un inhibiteur de USP1 et PARP (extrait du brevet WO2021163530).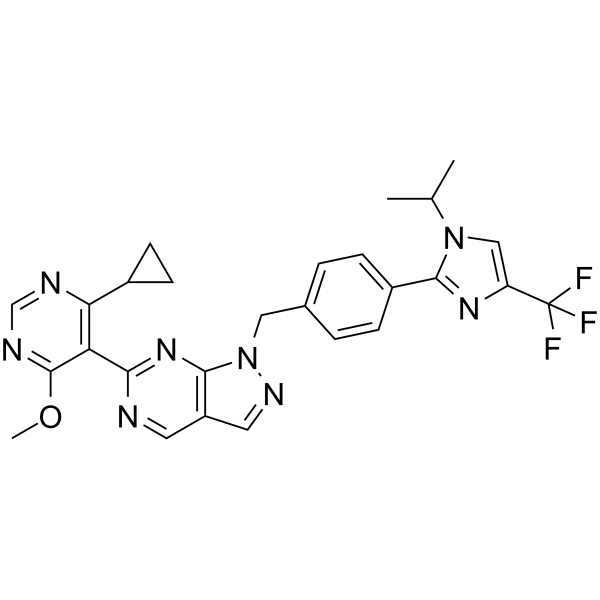
-
GC25552
KT-531
KT-531 (KT531) is a potent, selective HDAC6 inhibitor with IC50 of 8.5 nM, displays 39-fold selectivity over other HDAC isoforms.

-
GC33404
KU 59403
KU 59403 est un puissant inhibiteur d'ATM, avec des valeurs IC50 de 3 nM, 9,1 μM et 10 μM pour ATM, DNA-PK et PI3K, respectivement.
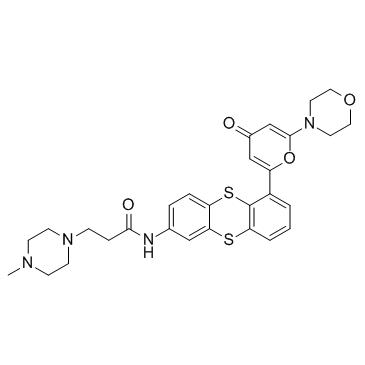
-
GC14346
KU-0060648
KU-0060648 est un double inhibiteur de PI3K et DNA-PK avec des IC50 de 4 nM, 0,5 nM, 0,1 nM, 0,594 nM et 8,6 nM pour PI3Kα, PI3Kβ, PI3Kγ, PI3Kδ et DNA-PK, respectivement.
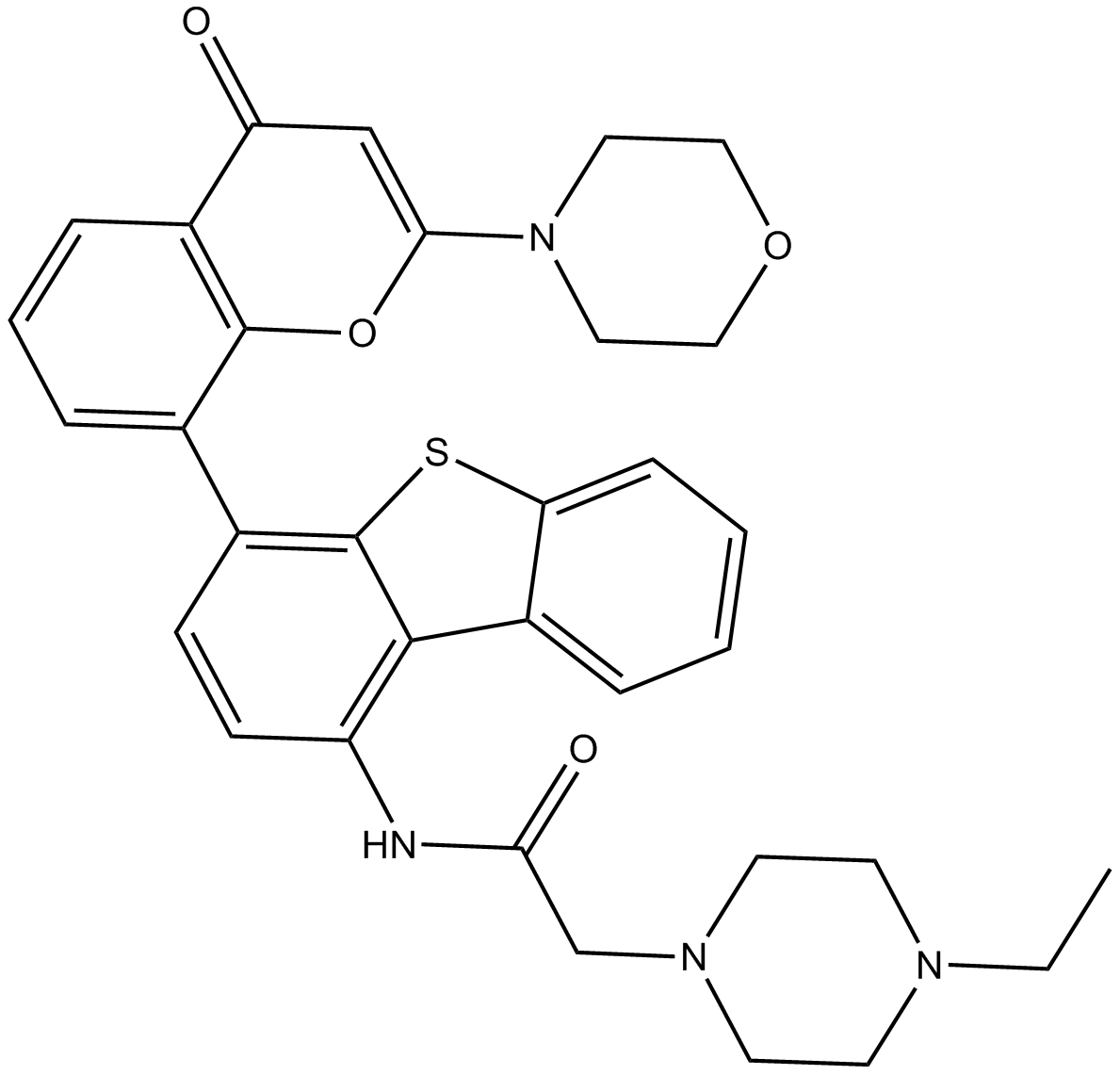
-
GC12054
KU-60019
KU60019;KU 60019
Le KU-60019 est un inhibiteur spécifique de la kinase ATM amélioré avec une IC50 de 6,3 nM.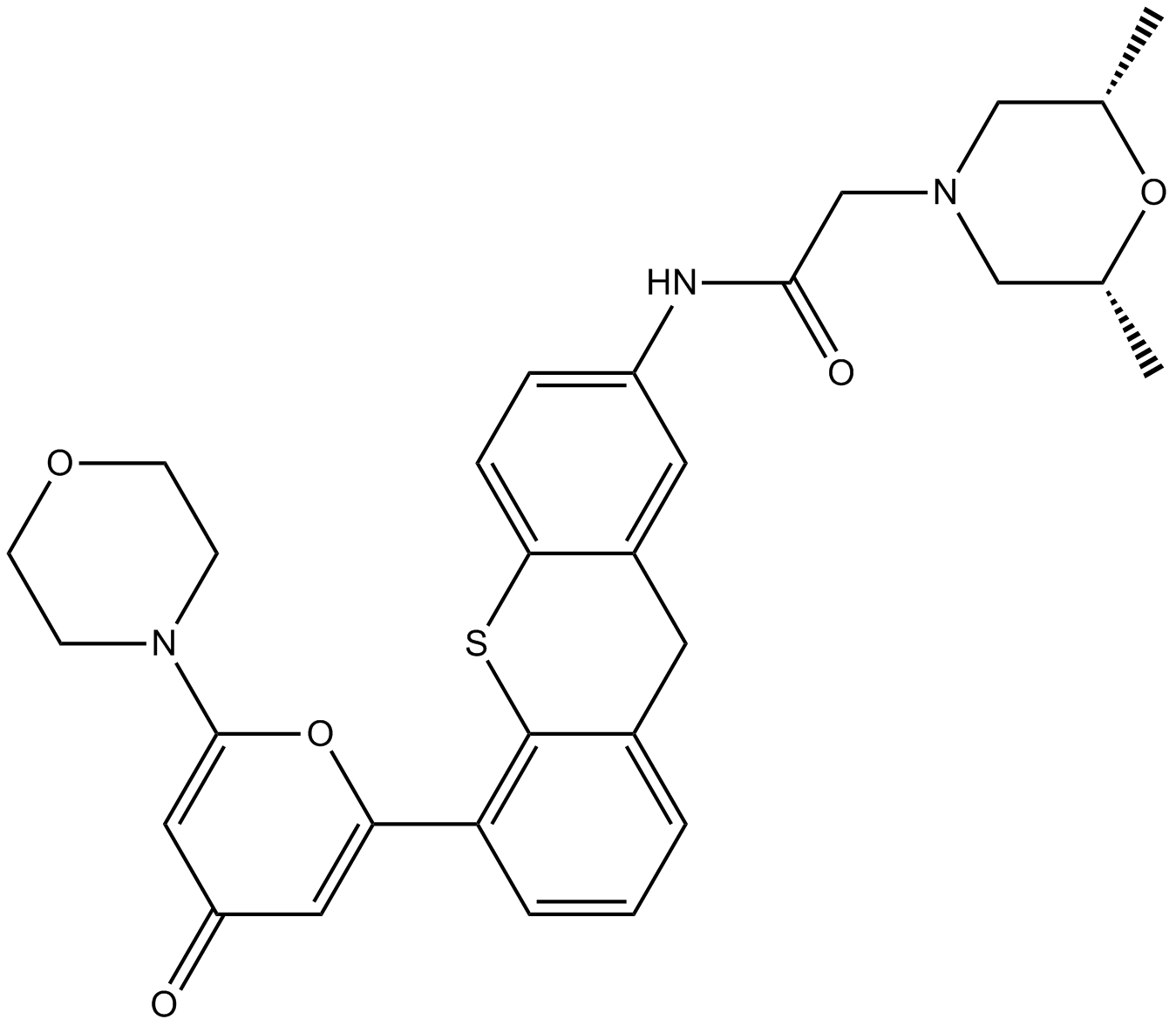
-
GC50532
KuWal151
Potent and selective CLK inhibitor
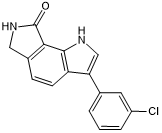
-
GC15743
L-755,507
Le L-755,507 est un puissant agoniste sélectif du β3-AR avec une IC50 de 35 nM.
-
GC66622
L-Guanosine
La L-guanosine est la configuration L de la guanosine. La guanosine est un nucléoside purique ayant une activité anti-herpèsvirus.
-
GC49381
L-Leucine-d10
Leu-d10, Pentanoic Acid-d10
An internal standard for the quantification of L-leucine
-
GC49368
L-Methionine-d3
(S)-Methionine-d3
La L-méthionine-d3 est la L-méthionine marquée au deutérium.
-
GC49384
L-Proline-d3
L-(–)-Proline-d3, (S)-(–)-Proline-d3
An internal standard for the quantification of L-proline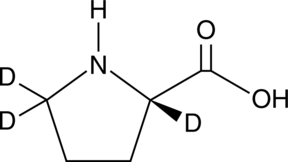
-
GC12131
L189
L189 est un inhibiteur de l'ADN ligase. L189 a un effet inhibiteur pour l'ADN Ligase I, III et IV avec des valeurs IC50 de 5 μM, 9 μM et 5 μM, respectivement. L189 n'a pas de cytotoxicité et augmente individuellement la mort cellulaire. L189 peut être utilisé pour la recherche sur le cancer.
-
GC18186
L67
DNA Ligase Inhibitor
L67 (DNA Ligase Inhibitor) est un inhibiteur compétitif de l'ADN ligase qui inhibe efficacement les ADN ligases I/III (les deux IC50 sont de 10 μM). L67 (inhibiteur d'ADN ligase) peut causer des dommages À l'ADN nucléaire en réduisant les niveaux d'ADN mitochondrial et en augmentant les niveaux de ROS générés par les mitochondries. L67 (DNA Ligase Inhibitor) active également la voie de l'apoptose dépendante de la caspase 1 dans les cellules cancéreuses, peut être utilisé dans la recherche sur le cancer.
-
GC67785
L82

-
GC68436
L82-G17

-
GC39632
Laflunimus
Le laflunimus (HR325) est un agent immunosuppresseur et un analogue du métabolite actif du léflunomide A77 1726.
-
GC62460
LCAHA
LCA hydroxyamide
LCAHA (LCA hydroxyamide) est un inhibiteur de la déubiquitinase USP2a avec des IC50 de 9,7 μM et 3,7 μM dans les tests Ub-AMC et Di-Ub, respectivement. LCAHA déstabilise la cycline D1 et induit l'arrêt G0/G1 en inhibant la déubiquitinase USP2a.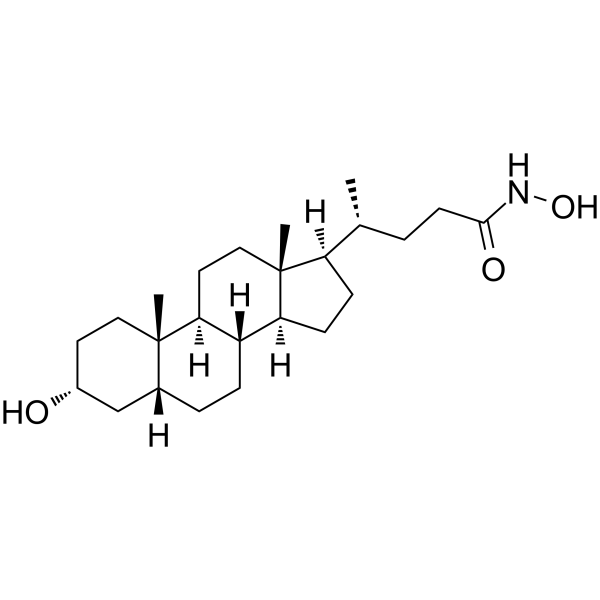
-
GC17067
LDC000067
LDC067
LDC000067 est un inhibiteur hautement spécifique de CDK9 avec une valeur IC50 de 44 ± 10 nM in vitro.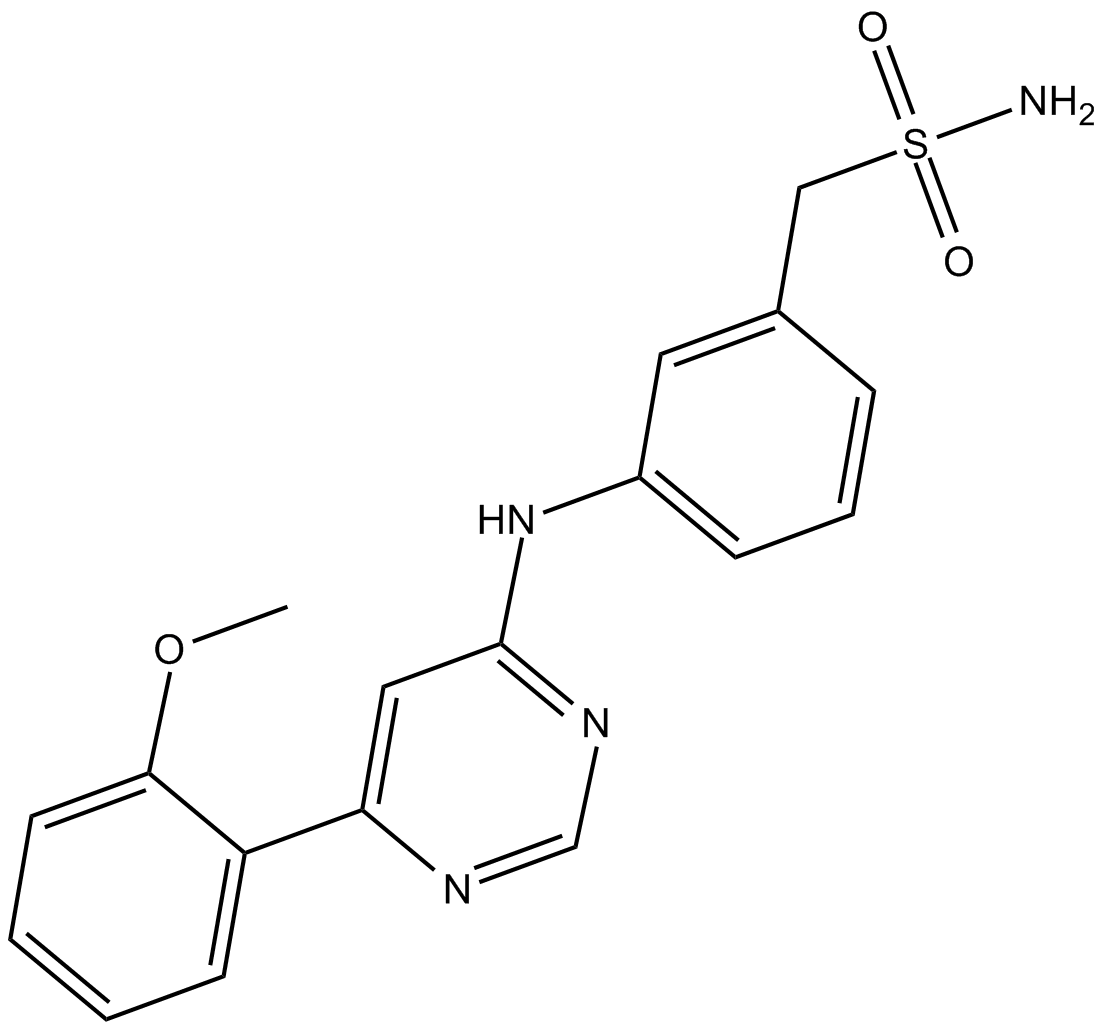
-
GC19219
LDC4297
LDC4297 est un inhibiteur puissant et sélectif de CDK7 avec une IC50 de 0,13 nM.