Immunology/Inflammation
The immune and inflammation-related pathway including the Toll-like receptors pathway, the B cell receptor signaling pathway, the T cell receptor signaling pathway, etc.
Toll-like receptors (TLRs) play a central role in host cell recognition and responses to microbial pathogens. TLR4 initially recruits TIRAP and MyD88. MyD88 then recruits IRAKs, TRAF6, and the TAK1 complex, leading to early-stage activation of NF-κB and MAP kinases [1]. TLR4 is endocytosed and delivered to intracellular vesicles and forms a complex with TRAM and TRIF, which then recruits TRAF3 and the protein kinases TBK1 and IKKi. TBK1 and IKKi catalyze the phosphorylation of IRF3, leading to the expression of type I IFN [2].
BCR signaling is initiated through ligation of mIg under conditions that induce phosphorylation of the ITAMs in CD79, leading to the activation of Syk. Once Syk is activated, the BCR signal is transmitted via a series of proteins associated with the adaptor protein B-cell linker (Blnk, SLP-65). Blnk binds CD79a via non-ITAM tyrosines and is phosphorylated by Syk. Phospho-Blnk acts as a scaffold for the assembly of the other components, including Bruton’s tyrosine kinase (Btk), Vav 1, and phospholipase C-gamma 2 (PLCγ2) [3]. Following the assembly of the BCR-signalosome, GRB2 binds and activates the Ras-guanine exchange factor SOS, which in turn activates the small GTPase RAS. The original RAS signal is transmitted and amplified through the mitogen-activated protein kinase (MAPK) pathway, which including the serine/threonine-specific protein kinase RAF followed by MEK and extracellular signal related kinases ERK 1 and 2 [4]. After stimulation of BCR, CD19 is phosphorylated by Lyn. Phosphorylated CD19 activates PI3K by binding to the p85 subunit of PI3K and produce phosphatidylinositol-3,4,5-trisphosphate (PIP3) from PIP2, and PIP3 transmits signals downstream [5].
Central process of T cells responding to specific antigens is the binding of the T-cell receptor (TCR) to specific peptides bound to the major histocompatibility complex which expressed on antigen-presenting cells (APCs). Once TCR connected with its ligand, the ζ-chain–associated protein kinase 70 molecules (Zap-70) are recruited to the TCR-CD3 site and activated, resulting in an initiation of several signaling cascades. Once stimulation, Zap-70 forms complexes with several molecules including SLP-76; and a sequential protein kinase cascade is initiated, consisting of MAP kinase kinase kinase (MAP3K), MAP kinase kinase (MAPKK), and MAP kinase (MAPK) [6]. Two MAPK kinases, MKK4 and MKK7, have been reported to be the primary activators of JNK. MKK3, MKK4, and MKK6 are activators of P38 MAP kinase [7]. MAP kinase pathways are major pathways induced by TCR stimulation, and they play a key role in T-cell responses.
Phosphoinositide 3-kinase (PI3K) binds to the cytosolic domain of CD28, leading to conversion of PIP2 to PIP3, activation of PKB (Akt) and phosphoinositide-dependent kinase 1 (PDK1), and subsequent signaling transduction [8].
References
[1] Kawai T, Akira S. The role of pattern-recognition receptors in innate immunity: update on Toll-like receptors[J]. Nature immunology, 2010, 11(5): 373-384.
[2] Kawai T, Akira S. Toll-like receptors and their crosstalk with other innate receptors in infection and immunity[J]. Immunity, 2011, 34(5): 637-650.
[3] Packard T A, Cambier J C. B lymphocyte antigen receptor signaling: initiation, amplification, and regulation[J]. F1000Prime Rep, 2013, 5(40.10): 12703.
[4] Zhong Y, Byrd J C, Dubovsky J A. The B-cell receptor pathway: a critical component of healthy and malignant immune biology[C]//Seminars in hematology. WB Saunders, 2014, 51(3): 206-218.
[5] Baba Y, Matsumoto M, Kurosaki T. Calcium signaling in B cells: regulation of cytosolic Ca 2+ increase and its sensor molecules, STIM1 and STIM2[J]. Molecular immunology, 2014, 62(2): 339-343.
[6] Adachi K, Davis M M. T-cell receptor ligation induces distinct signaling pathways in naive vs. antigen-experienced T cells[J]. Proceedings of the National Academy of Sciences, 2011, 108(4): 1549-1554.
[7] Rincón M, Flavell R A, Davis R A. The Jnk and P38 MAP kinase signaling pathways in T cell–mediated immune responses[J]. Free Radical Biology and Medicine, 2000, 28(9): 1328-1337.
[8] Bashour K T, Gondarenko A, Chen H, et al. CD28 and CD3 have complementary roles in T-cell traction forces[J]. Proceedings of the National Academy of Sciences, 2014, 111(6): 2241-2246.
Targets for Immunology/Inflammation
- Cyclic GMP-AMP Synthase(1)
- Apoptosis(137)
- 5-Lipoxygenase(18)
- TLR(106)
- Papain(2)
- PGDS(1)
- PGE synthase(26)
- SIKs(10)
- IκB/IKK(83)
- AP-1(2)
- KEAP1-Nrf2(47)
- NOD1(1)
- NF-κB(265)
- Interleukin Related(129)
- 15-lipoxygenase(2)
- Others(10)
- Aryl Hydrocarbon Receptor(35)
- CD73(16)
- Complement System(46)
- Galectin(30)
- IFNAR(19)
- NO Synthase(78)
- NOD-like Receptor (NLR)(37)
- STING(84)
- Reactive Oxygen Species(434)
- FKBP(14)
- eNOS(4)
- iNOS(24)
- nNOS(21)
- Glutathione(37)
- Adaptive Immunity(144)
- Allergy(129)
- Arthritis(25)
- Autoimmunity(134)
- Gastric Disease(64)
- Immunosuppressants(27)
- Immunotherapeutics(3)
- Innate Immunity(411)
- Pulmonary Diseases(76)
- Reactive Nitrogen Species(43)
- Specialized Pro-Resolving Mediators(42)
- Reactive Sulfur Species(24)
Products for Immunology/Inflammation
- Cat.No. Nom du produit Informations
-
GC49887
7-(β-Hydroxyethyl)theophylline-d6
An internal standard for the quantification of 7-(β-hydroxyethyl)theophylline
-
GC45707
7-Azido-4-methylcoumarin
A fluorescent H2S probe
-
GC40978
7-epi Maresin 1
7-epi Maresin 1 is the inactive 7(S) epimer of Maresin 1, which contains a 7(R) hydroxyl group.
-
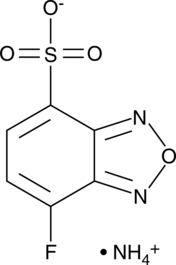
GC49312
7-Fluoro-2,1,3-benzoxadiazole-4-sulfonate (ammonium salt)
Le 7-fluoro-2,1,3-benzoxadiazole-4-sulfonate (sel d'ammonium) est un marqueur fluorescent.
-
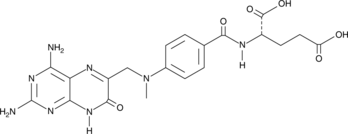
GC49051
7-hydroxy Methotrexate
Le 7-hydroxy méthotrexate est un métabolite majeur du méthotrexate
-
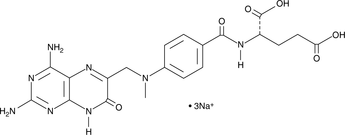
GC42608
7-hydroxy Methotrexate (sodium salt)
7-hydroxy Methotrexate (7-hydroxy MTX) is a phase I metabolite of MTX, which is converted by hepatic aldehyde oxidases.
-
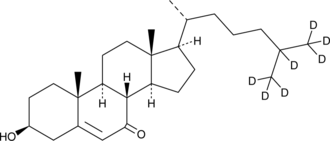
GC46241
7-keto Cholesterol-d7
7-keto Cholesterol is a bioactive sterol and a major oxysterol component of oxidized LDL
-
GC48880
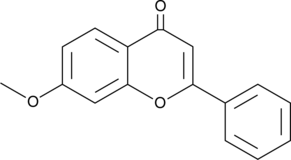
7-Methoxyflavone
La 7-méthoxyflavone est un composé isolé de Zornia brasiliensis.
-
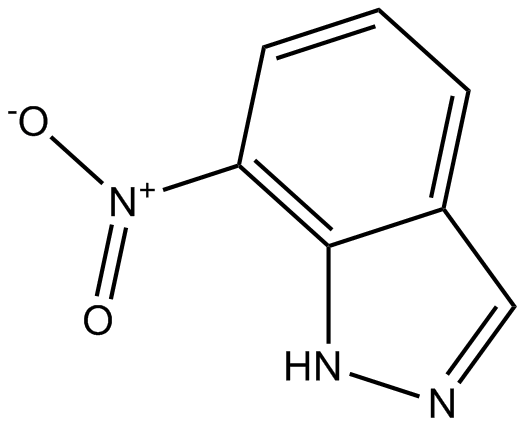
GC13982
7-NINA
non-selective NOS inhibitor
-
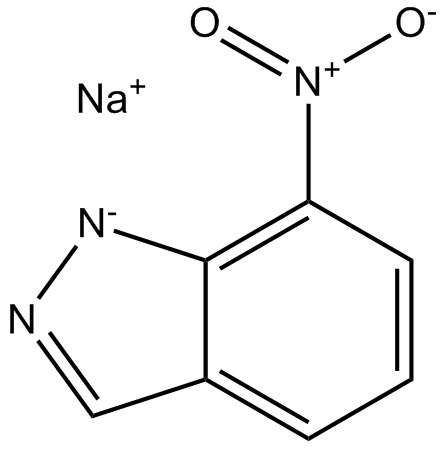
GC12309
7-Nitroindazole
Le 7-nitroindazole est un inhibiteur sélectif de la nNOS avec des effets antinociceptifs et cardiovasculaires.
-
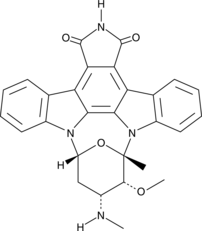
GC42616
7-oxo Staurosporine
7-oxo Staurosporine is an antibiotic originally isolated from S.
-
GC52211
7-[1-(1H)-Tetrazolylacetamido]desacetoxycephalosporanic Acid
A potential impurity in commercial preparations of cefazolin
![7-[1-(1H)-Tetrazolylacetamido]desacetoxycephalosporanic Acid Chemical Structure 7-[1-(1H)-Tetrazolylacetamido]desacetoxycephalosporanic Acid Chemical Structure](/media/struct/GC5/GC52211.png)
-
GC41136
8(S),15(S)-DiHETE
8(S),15(S)-DiHETE is formed when 15(S)-HETE is subjected to further oxidation by 15-LO.
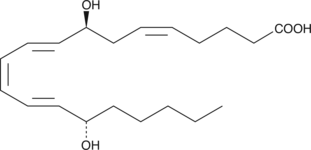
-
GC42623
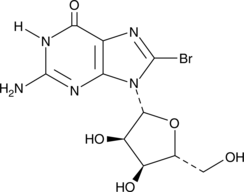
8-Bromoguanosine
8-Bromoguanosine is a brominated derivative of guanosine.
-
GC35199
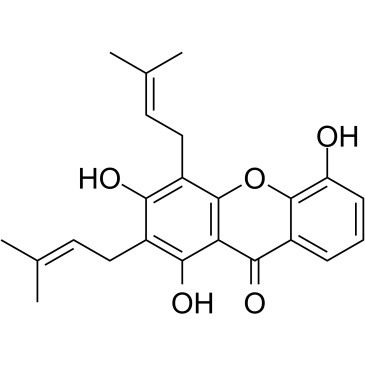
8-Deoxygartanin
La 8-désoxygartanine, une xanthone prénylée de G.
-
GC42627
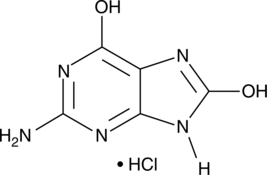
8-Hydroxyguanine (hydrochloride)
8-Hydroxyguanine is produced by oxidative degradation of DNA by hydroxyl radical.
-
GC18426
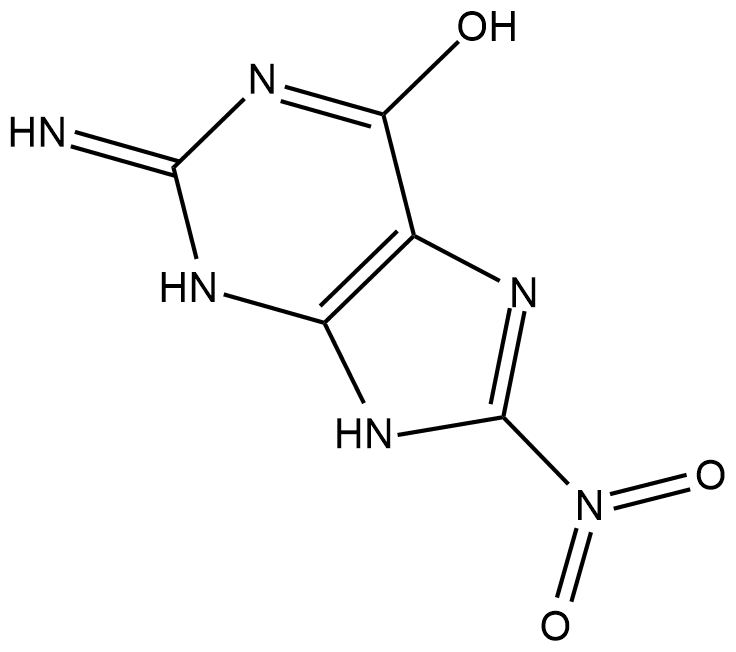
8-Nitroguanine
8-Nitroguanine is a nitrative guanine derivative formed by oxidative damage to the guanine base in DNA by reactive nitrogen species (RNS) during inflammation and in vitro by reaction of DNA with peroxynitrite and other RNS reagents.
-
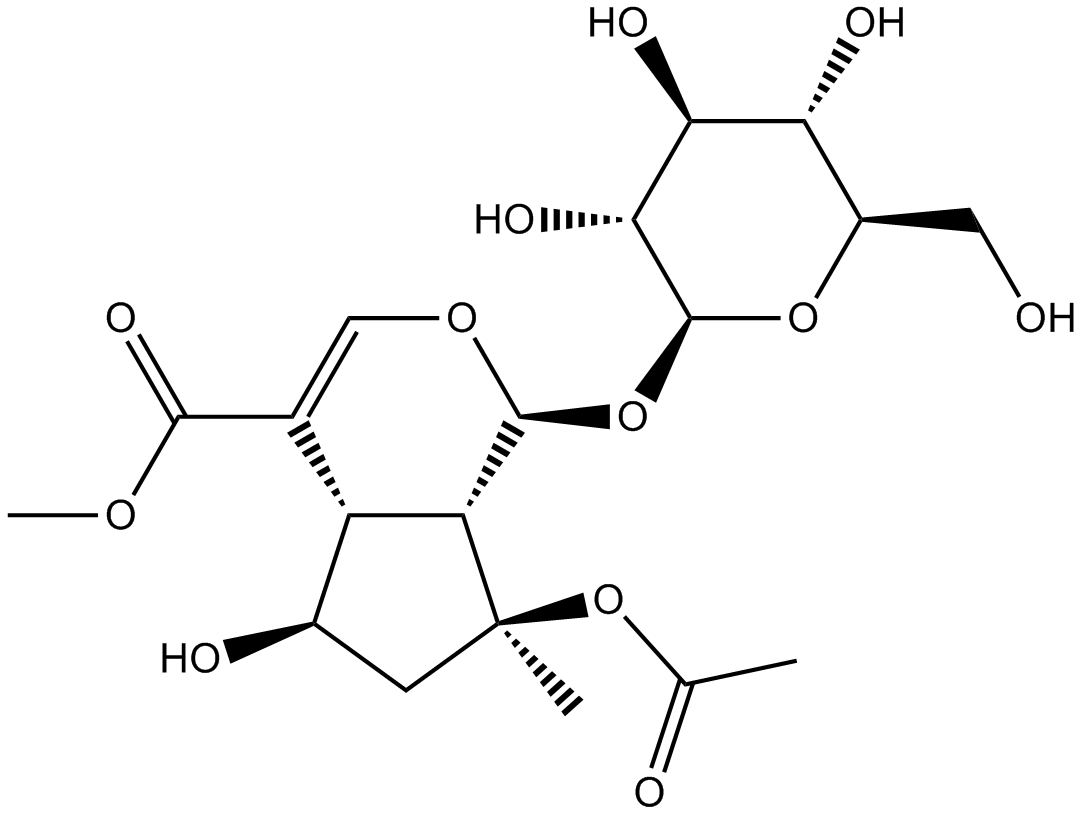
GN10212
8-O-Acetyl shanzhiside methyl ester
-
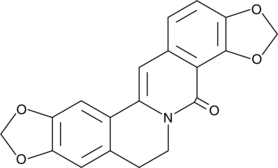
GC49275
8-Oxycoptisine
La 8-oxycoptisine est un alcaloÏde protoberbérine naturel À activité anticancéreuse.
-
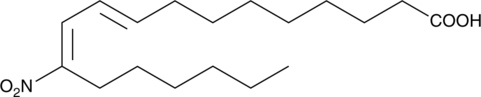
GC46748
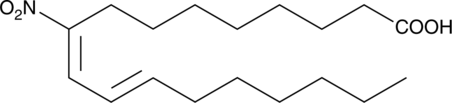
9(E),11(E)-12-nitro Conjugated Linoleic Acid
A nitrated fatty acid
-
GC46749
9(E),11(E)-9-nitro Conjugated Linoleic Acid
A nitrated fatty acid
-
GC42633
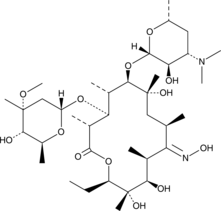
9(E)-Erythromycin A oxime
(9E)-Erythromycin A oxime is a metabolite of the semisynthetic antibiotic roxithromycin .
-
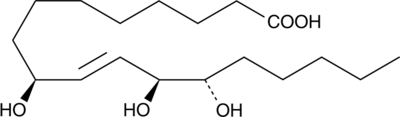
GC46753
9(S),12(S),13(S)-TriHOME
An oxylipin
-
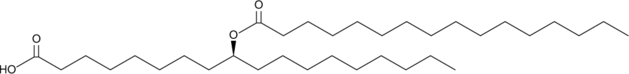
GC40324
9(S)-PAHSA
9-PAHSA is a newly identified endogenous lipid that belongs to a collection of branched fatty acid esters of hydroxy fatty acids (FAHFAs).
-
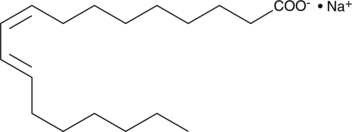
GC46757
9(Z),11(E)-Conjugated Linoleic Acid (sodium salt)
An isomer of linoleic acid
-
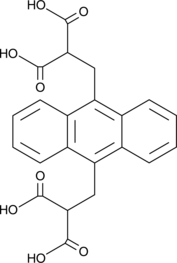
GC42642
9,10-Anthracenediyl-bis(methylene)dimalonic Acid
L'acide 9,10-anthracènediyl-bis(méthylène)dimalonique (ABMDMA) est un colorant biologique et un indicateur utilisé pour détecter la génération d'oxygène singulet (SOG). L'acide 9,10-anthracènediyl-bis(méthylène)dimalonique est un dérivé hydrosoluble de l'anthracène. L'acide 9,10-anthracènediyl-bis(méthylène)dimalonique peut être photoblanchi par de l'oxygène singulet en son endoperoxyde correspondant. Cette réaction peut être contrÔlée par spectrophotométrie en enregistrant la diminution de l'absorbance À 400 nm.
-
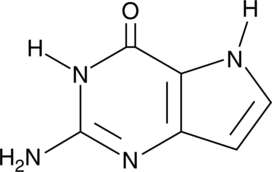
GC42644
9-Deazaguanine
9-Deazaguanine is an analog of guanine that acts as an inhibitor of purine nucleoside phosphorylase (PNP; Kd = 160 nM).
-
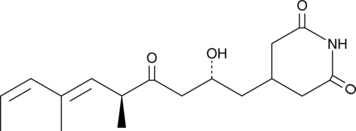
GC42648
9-Methylstreptimidone
9-Methylstreptimidone is a microbial metabolite originally isolated from Streptomyces sp.
-
GC42649
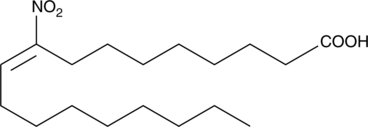
9-Nitrooleate
Nitrated unsaturated fatty acids, such as 10- and 12-nitrolinoleate, cholesteryl nitrolinoleate, and nitrohydroxylinoleate, represent a new class of endogenous lipid-derived signalling molecules.
-
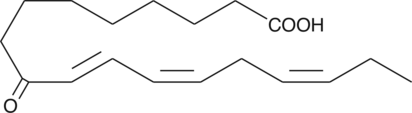
GC42653
9-OxoOTrE
9-OxoOTrE is produced by the oxidation of 9-HpOTrE.
-
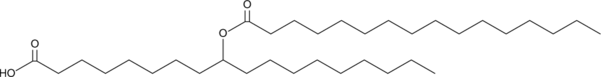
GC40325
9-PAHSA
Branched fatty acid esters of hydroxy fatty acids (FAHFAs) are newly identified endogenous lipids regulated by fasting and high-fat feeding and associated with insulin sensitivity.
-
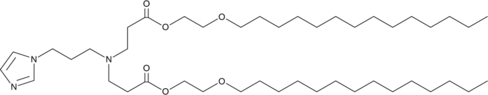
GC49237
93-O17O
A cationic lipidoid
-
GC49238
93-O17S
A cationic lipidoid
-
GC45960
9c(i472)
9c(i472) est un puissant inhibiteur de 15-LOX-1 (15-lipoxygénase-1) avec une valeur IC50 de 0,19 μM.
-
GC45930
A 26771B
A macrolide antibiotic
-
GC42660
A-39183A
A-39183A is an active component of the A-39183 antibiotic complex produced by aerobic fermentation of Streptomyces NRRL 12049.
-
GC42661
A-54556B
A-54556B is a natural acyldepsipeptide (ADEP) antibiotic isolated from the fermentation broth of S.
-
GC18936
A-83016F
A-83016F is an aurodox antibiotic isolated from the actinomycete species A83016.
-
GC49309
A-943931 (hydrochloride hydrate)
A histamine H4 receptor antagonist
-
GC35214
A-9758
A-9758 est un ligand RORγ et un agoniste inverse RORγt puissant et sélectif (IC50 = 5 nM), et présente une puissance robuste contre la libération d'IL-17A.
-
GP10060
a-MSH, amide
L'a-MSH (α-Mélanocyte-Stimulant Hormone), amide, active le récepteur de la mélanocortine-1 et active la signalisation du AMP cyclique (cAMP) via un transporteur de protéines G, peut synthétiser la mélanine.
-
GC19496
AAPH
AAPH is a water-soluble azo compound

-
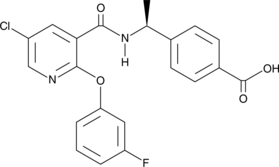
GC42666
AAT-008
AAT-008 est un antagoniste des récepteurs de la prostaglandine EP4 puissant, sélectif et actif par voie orale avec Kis de 0,97 et 6,1 nM pour l'EP4 humain recombinant et l'EP4 de rat recombinant, respectivement .
-
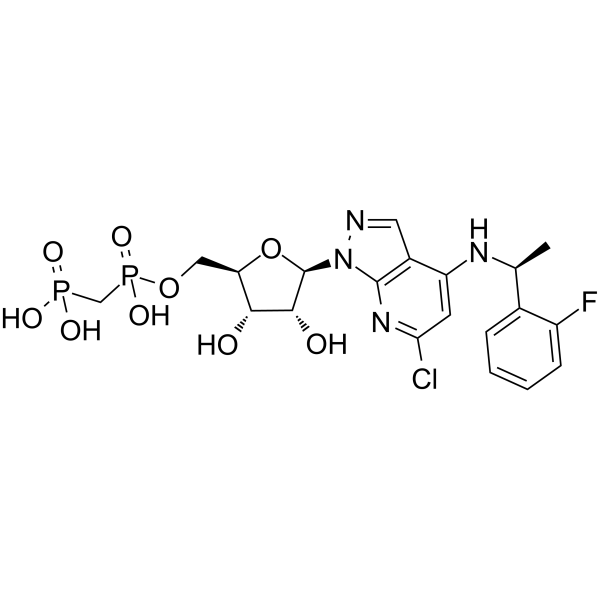
GC65293
AB-680
AB-680 est un inhibiteur très puissant, réversible et sélectif de CD73 (une ecto-nucléotidase), avec un Ki de 4,9 pM pour hCD73, affiche une sélectivité > 10 000 fois supérieure À celle des ecto-nucléotidases apparentées CD39. Activité anti-tumorale.
-
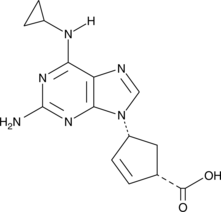
GC42667
Abacavir Carboxylate
Abacavir carboxylate is an inactive metabolite of the HIV-1 reverse transcriptase inhibitor abacavir.
-
GC46767
Abacavir-d4
An internal standard for the quantification of abacavir
-
GC49766
Abafungin
L'abafungine, un agent antifongique, inhibe la transméthylation en position C-24 de la chaÎne latérale du stérol, catalysée par l'enzyme stérol-C-24-méthyltransférase.
-
GC42668
ABC34
ABC34 is an inactive control probe for JJH260, the inhibitor of androgen-induced gene 1 (AIG1), an enzyme that hydrolyzes fatty acid esters of hydroxy fatty acids (FAHFAs).
-
GC49745
ABT-263-d8
ABT-263-d8 est le deutérium marqué Navitoclax. Navitoclax (ABT-263) est un inhibiteur de la protéine de la famille Bcl-2 puissant et actif par voie orale qui se lie À plusieurs protéines anti-apoptotique de la famille Bcl-2, telles que Bcl-xL, Bcl-2 et Bcl-w, avec un Ki de moins supérieure À 1 nM.
-
GC42683
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp is a fluorescence-quenched peptide substrate for human neutrophil elastase (kcat/Km = 531 mM-1s-1).
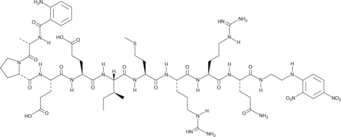
-
GC52499
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp (trifluoroacetate salt)
A sensitive substrate for neutrophil elastase
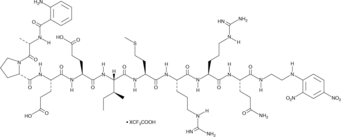
-
GC42684
Abz-Val-Ala-Asp-Nva-Arg-Asp-Arg-Gln-EDDnp (trifluoroacetate salt)
Abz-Val-Ala-Asp-Nva-Arg-Asp-Arg-Gln-EDDnp is a fluorescence-quenched peptide substrate for human proteinase 3 (kcat/Km = 1,570 mM-1s-1).
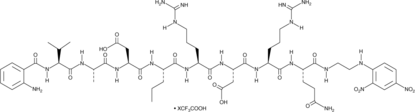
-
GC42685
Ac-ANW-AMC
Ac-ANW-AMC est un substrat fluorogène pour l'immunoprotéasome.
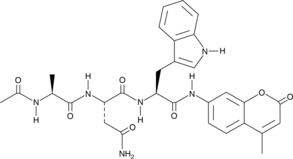
-
GC49704
Ac-FLTD-CMK (trifluoroacetate salt)
An inhibitor of caspase-1, -4, -5, and -11

-
GA20621
Ac-muramic acid
Un composant des parois cellulaires bactériennes. Pour un dérivé protégé de MurNAc, voir Q-1005.

-
GC52372
Ac-VDVAD-AFC (trifluoroacetate salt)
A fluorogenic substrate for caspase-2
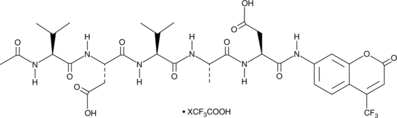
-
GC15598
Ac2-26
Ac2-26, un peptide N-terminal actif de l'annexine A1 (AnxA1), atténue les lésions pulmonaires aiguës induites par l'ischémie-reperfusion.
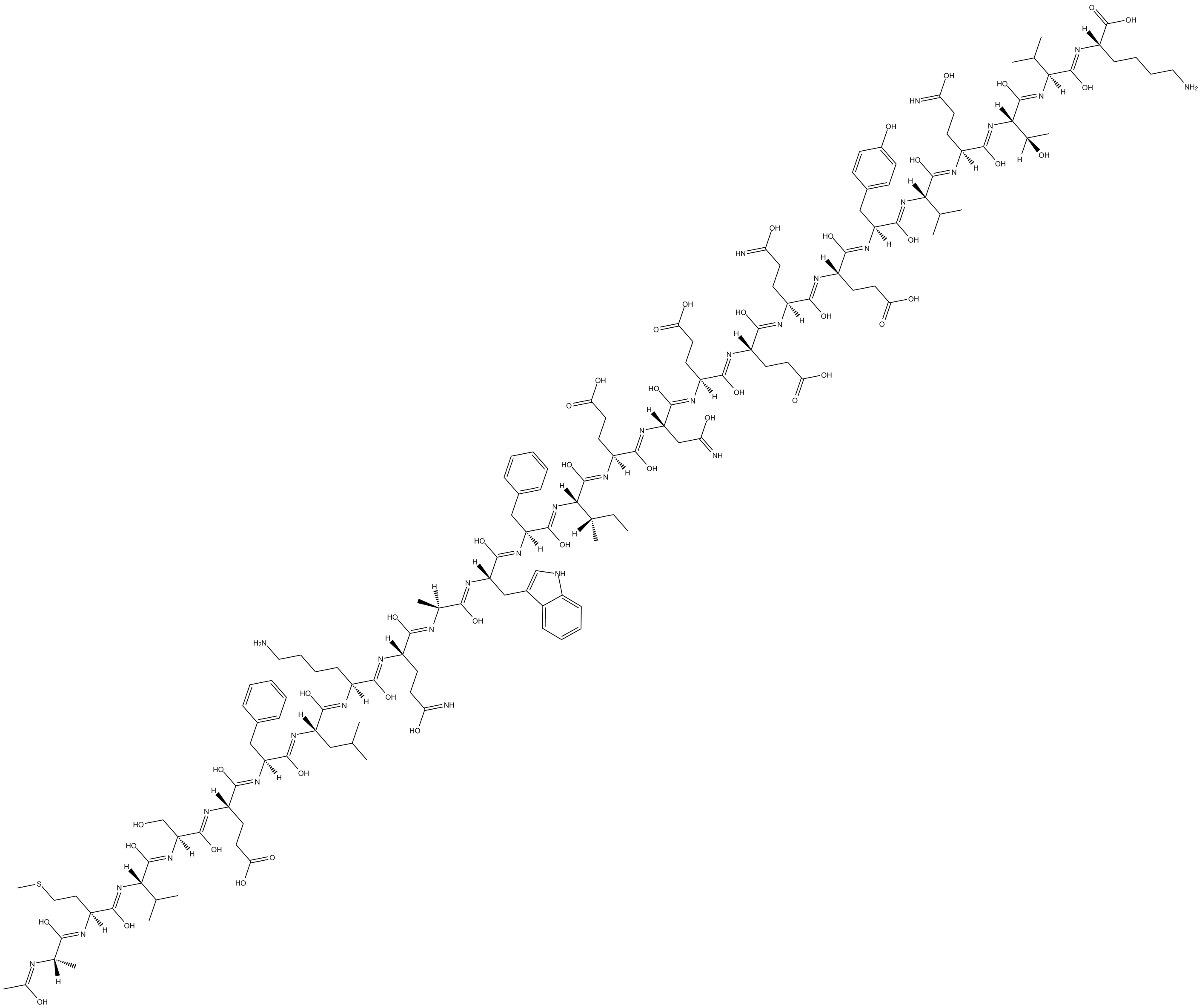
-
GC49263
Ac2-26 (human) (ammonium salt)
An annexin A1-mimetic peptide

-
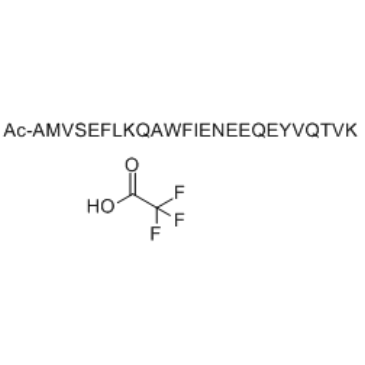
GC35224
Ac2-26 TFA
Ac2-26 TFA, un peptide N-terminal actif de l'annexine A1 (AnxA1), atténue les lésions pulmonaires aiguës induites par l'ischémie-reperfusion.
-
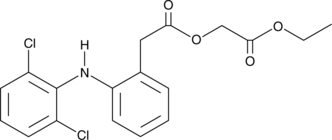
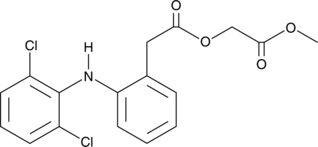
GC42691
Aceclofenac ethyl ester
A potential impurity found in commercial preparations of aceclofenac
-
GC42692
Aceclofenac methyl ester
A potential impurity in commercial preparations of aceclofenac
-
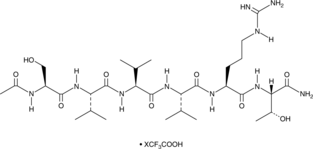
GC49911
Acetyl Hexapeptide-38 (trifluoroacetate salt)
A hexapeptide
-
GC42700
Acetyl Pentapeptide-1
Acetyl pentapeptide-1 is a pentapeptide that decreases IL-8 secretion in human keratinocytes when used in combination with acetyl hexapeptide-36 and acetyl hexapeptide-38.

-
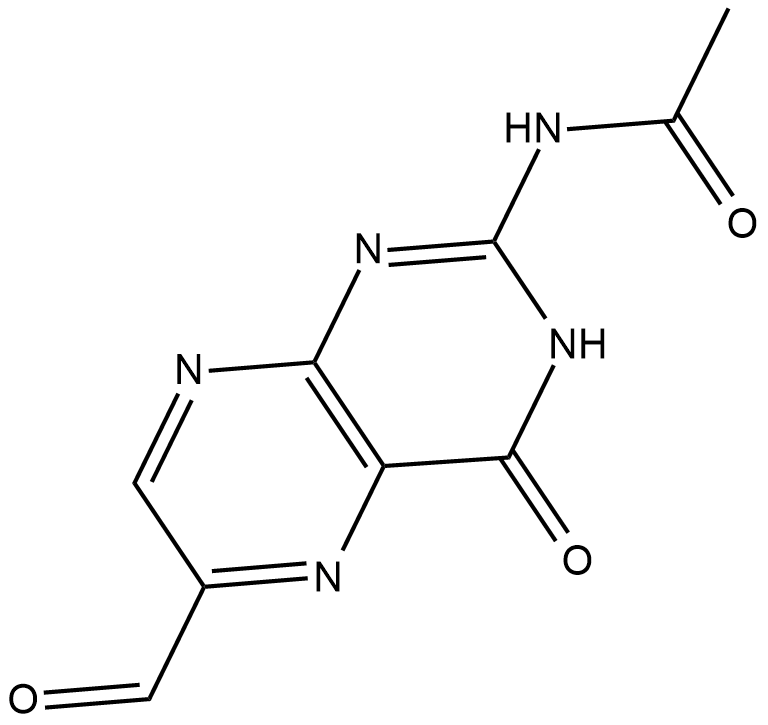
GC18443
Acetyl-6-formylpterin
Acetyl-6-formylpterin is an inhibitor of mucosal-associated invariant T (MAIT) cell activation.
-
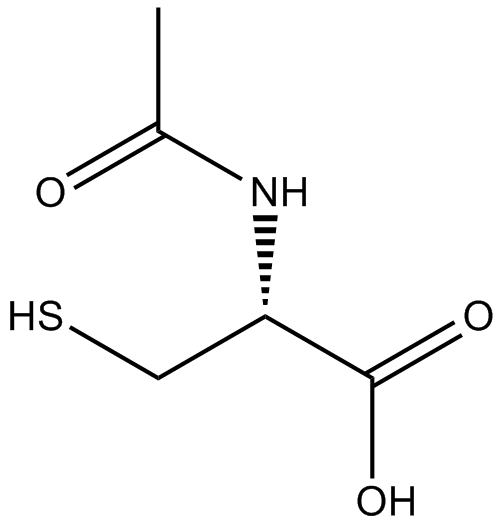
GC11786
Acetylcysteine
L'acétylcystéine est le dérivé N-acétyl de la CYSTEINE.
-
GC17416
ACHP
IκB kinase inhibitor
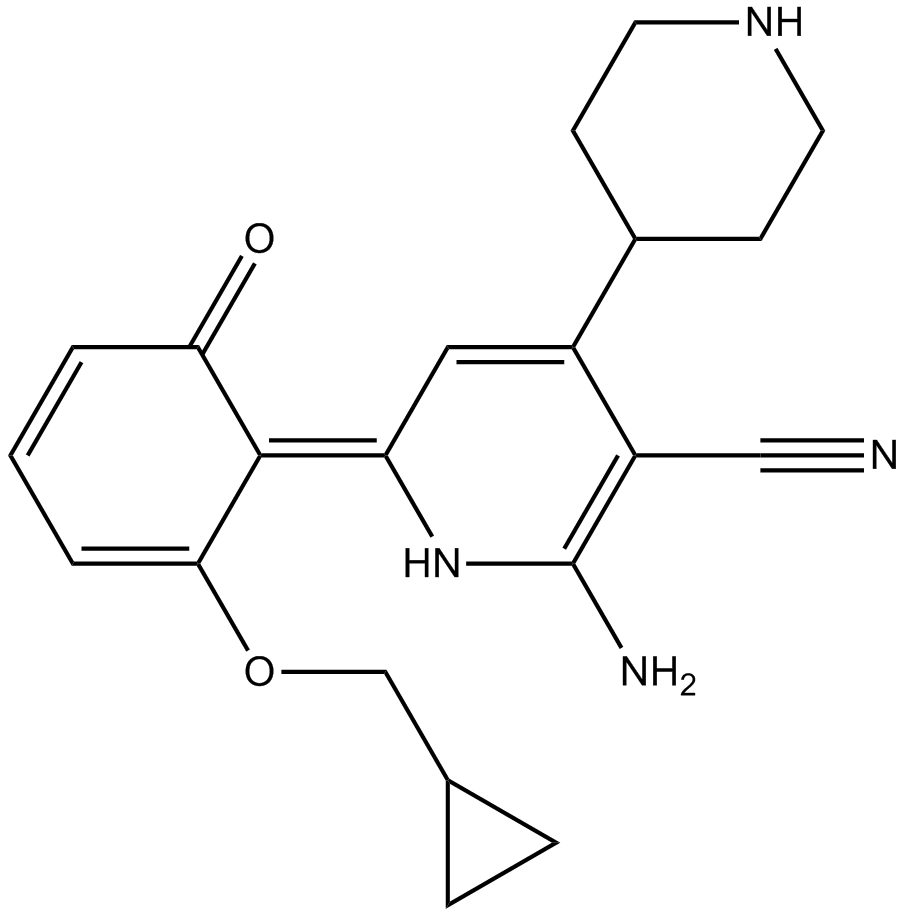
-
GN10536
Aconine
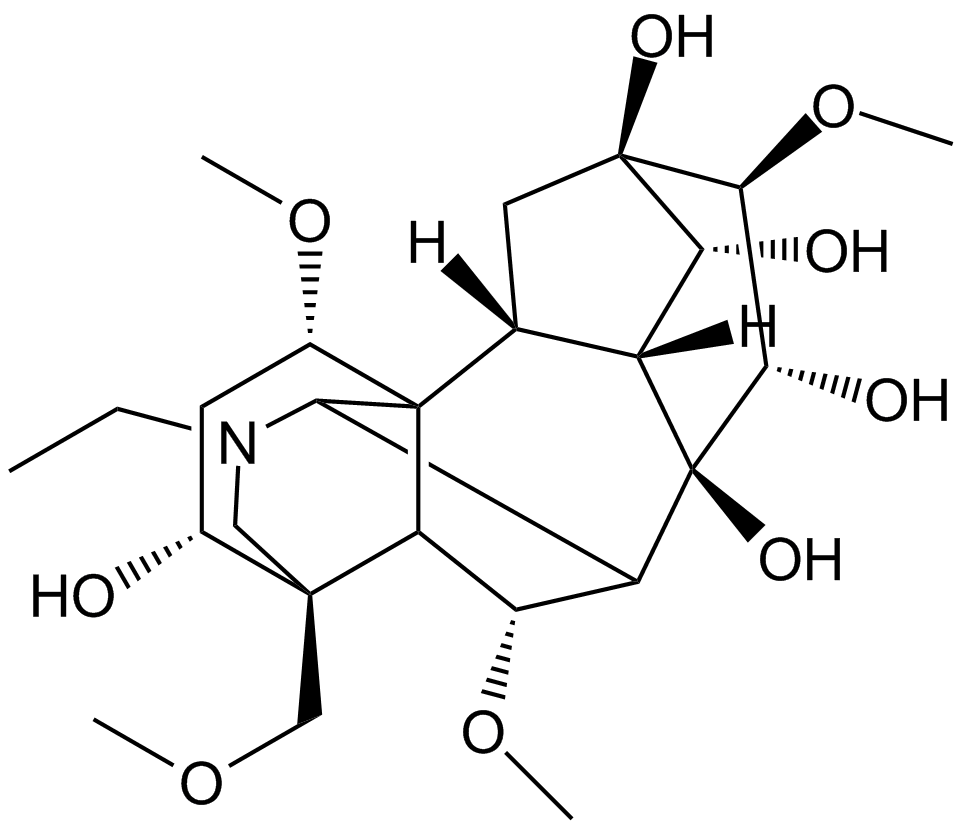
-
GC49804
Acridine
An azaarene
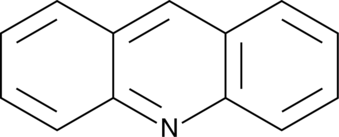
-
GC40645
Actarit
Actarit, un composé antirhumatismal actif par voie orale, a le potentiel de traiter l'arthrite induite par le collagène de type II .
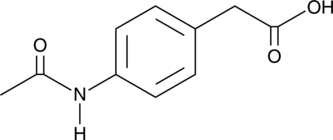
-
GC41316
Actinopyrone A
Actinopyrone A is a pyrone isolated from S.
-
GC48842
Actiphenol
A bacterial metabolite with antiviral activity
-
GC46797
Acyclovir-d4
An internal standard for the quantification of acyclovir
-
GC46798
Adapalene-d3
An internal standard for the quantification of adapalene
-
GC46805
Adefovir-d4
An internal standard for the quantification of adefovir
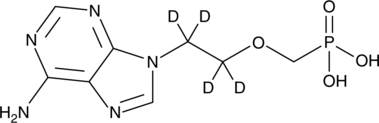
-
GC31693
Adelmidrol
L'adelmidrol exerce d'importants effets anti-inflammatoires qui dépendent en partie de PPARγ.
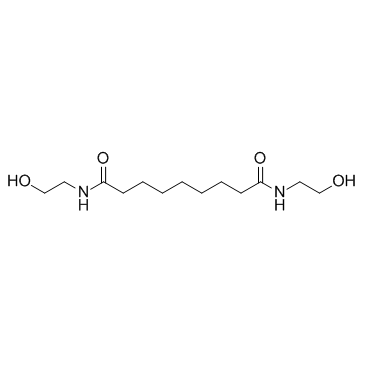
-
GC49285
Adenosine 5’-methylenediphosphate (hydrate)
An inhibitor of ecto-5’-nucleotidase
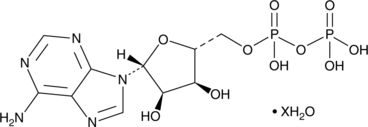
-
GC60040
ADH-503
ADH-503 ((Z)-Leukadherin-1 choline) est un agoniste CD11b actif par voie orale et allostérique. L'ADH-503 entraÎne la repolarisation des macrophages associés À la tumeur, la réduction du nombre de cellules myéloÏdes immunosuppressives infiltrant la tumeur et améliore les réponses des cellules dendritiques.
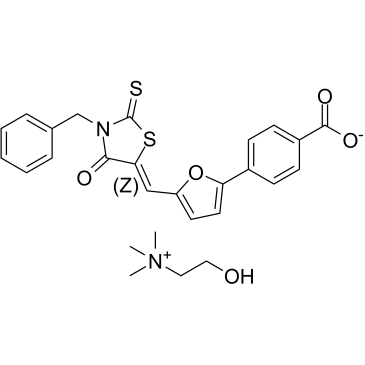
-
GC42742
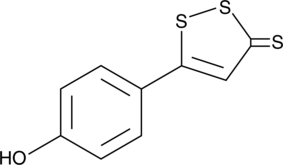
ADT-OH
L'ADT-OH est un donneur libérant du sulfure d'hydrogène. L'ADT-OH induit l'apoptose et inhibe le développement du mélanome in vivo en régulant À la hausse le FADD. ADT-OH a le potentiel pour la recherche sur les maladies cancéreuses.
-
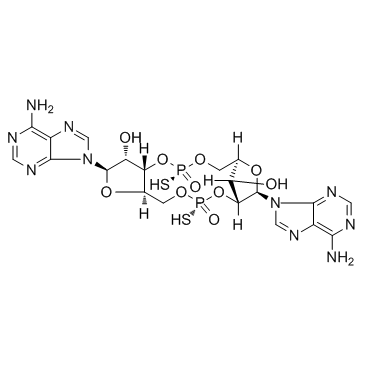
GC31649
ADU-S100 (ML RR-S2 CDA)
ADU-S100 (ML RR-S2 CDA) (MIW815), un activateur du stimulateur des gènes de l'interféron (STING), conduit À une régression tumorale et À une immunité puissantes et systémiques.
-
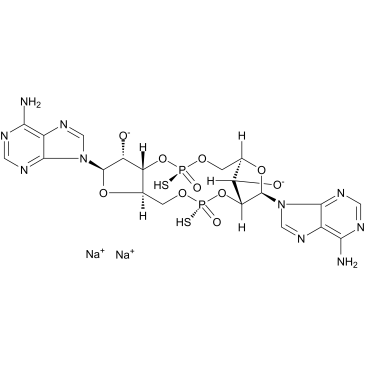
GC39161
ADU-S100 disodium salt
Le sel disodique ADU-S100 (sel disodique MIW815) est un activateur du stimulateur des gènes de l'interféron (STING).
-
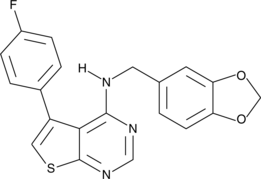
GC42743
AEM1
AEM1 est un inhibiteur de Nrf2. AEM1 réduit les expressions des gènes dépendants de Nrf2 dans les cellules A549 et inhibe la croissance des cellules A549 in vitro et in vivo.
-
GC63485
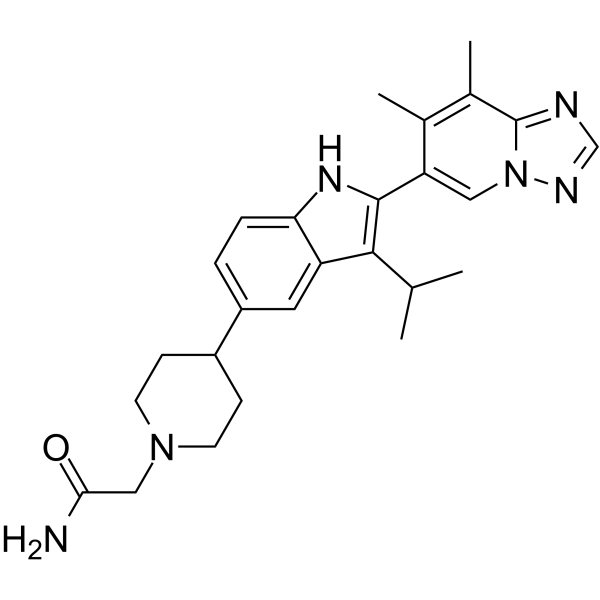
Afimetoran
L'afimetoran est un antagoniste des récepteurs de type péage, qui peut être utilisé dans la recherche de maladies inflammatoires et auto-immunes.
-
GC66396
Agatolimod
L'agatolimod (ODN 2006), un ODN de classe B (oligodésoxynucléotide), est un agoniste du TLR9. L'agatolimod est également une séquence CpG optimale pour l'homme. L'agatolimod stimule une très forte production de NO2 et d'IL-6 dans les cellules HD11. L'agatolimod peut être utilisé pour la recherche sur le cancer du sein. Séquence : 5' ; -tcgtcgttttgtcgttttgtcgtt-3' ;.

-
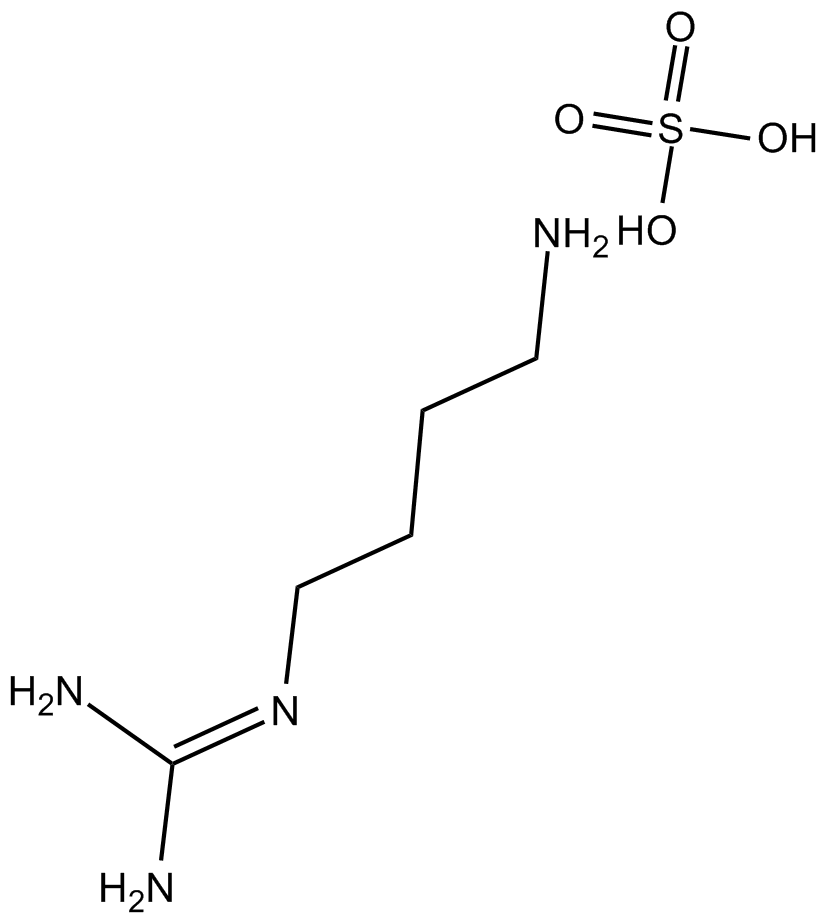
GC16831
Agmatine sulfate
Le sulfate d'agmatine exerce une action modulatrice sur de multiples cibles moléculaires, telles que les systèmes de neurotransmetteurs, les canaux ioniques et la synthèse de l'oxyde nitrique.
-
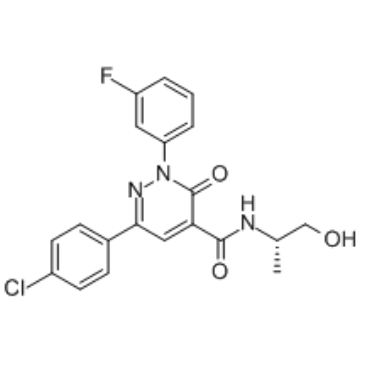
GC35271
AHR antagonist 1
L'antagoniste AHR 1 (antagoniste AHR 1) est un antagoniste des récepteurs d'aryle hydrocarbure (AHR).
-
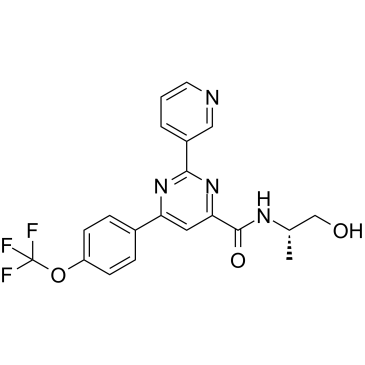
GC39669
AHR antagonist 2
L'antagoniste 2 de l'AHR est un puissant antagoniste du récepteur des arylhydrocarbures (AHR), extrait du brevet WO2019101641A1, exemple de composé 1, avec des IC50 de 0,885 et 2,03 nM pour l'AhR humain et de souris.
-
GC64275
AHR antagonist 5
L'antagoniste AHR 5, un antagoniste puissant et actif par voie orale des récepteurs d'aryle hydrocarbure (AHR) extrait du brevet WO2018195397, exemple 39, a une IC50 < 0,5 μμ. L'antagoniste AHR 5 inhibe significativement la croissance tumorale en combinaison avec l'inhibiteur de point de contrÔle anti-PD-1.
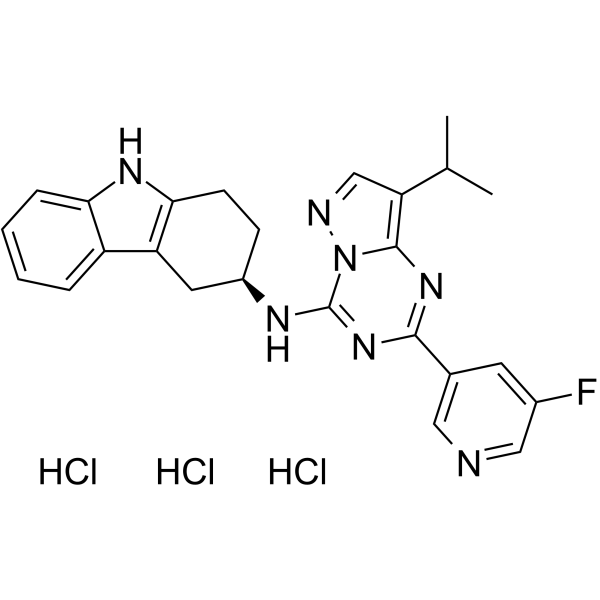
-
GC64208
AHR antagonist 5 free base
La base libre de l'antagoniste 5 de l'AHR est un inhibiteur sélectif et actif par voie orale du récepteur des aryl-hydrocarbures (AHR). La base libre de l'antagoniste 5 de l'AHR empêche efficacement la translocation de l'AHR du cytoplasme vers le noyau. La base libre de l'antagoniste 5 de l'AHR est hautement sélective pour l'AHR par rapport aux autres récepteurs, transporteurs et kinases.
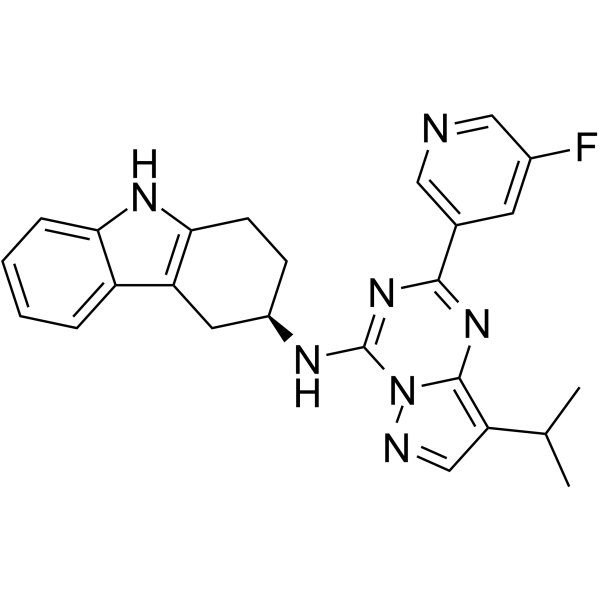
-
GC16120
AI-3
Nrf2/Keap1 and Keap1/Cul3 interaction inhibitor
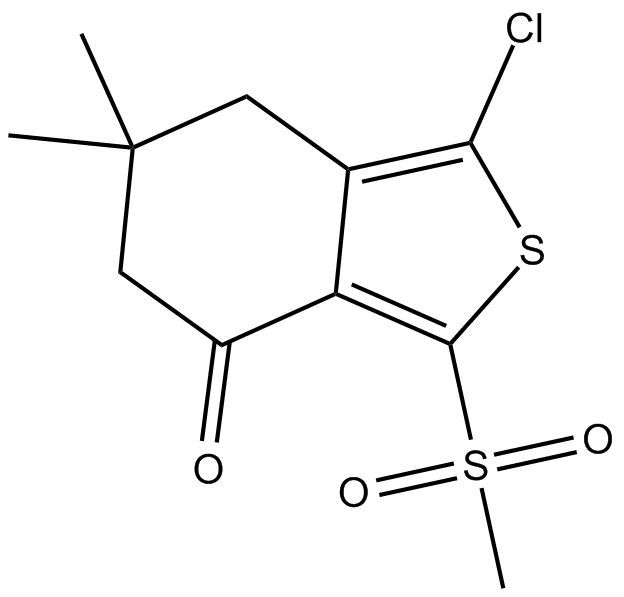
-
GC46821
Ajoene
L'ajoène, un composé dérivé de l'ail, est un agent antithrombotique et antifongique.
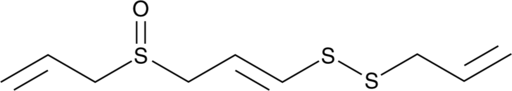
-
GC45681
AKP-11
An S1P1 receptor agonist
-
GC42763
Alamethicin F50
Alamethicin F50 is a peptaibol isolated from Trichoderma.
-
GC49773
Albendazole sulfone-d3
An internal standard for the quantification of albendazole sulfone
-
GC48848
Albendazole-d7
L'albendazole-d7 (SKF-62979-d7) est l'albendazole marqué au deutérium.
-
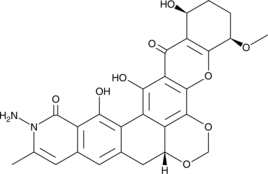
GC41080
Albofungin
Albofungin is a xanthone isolated from A.
-
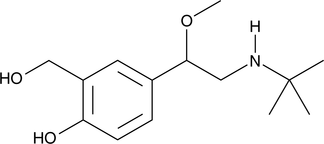
GC42764
Albuterol methyl ether
Salbutamol (albuterol) is a selective β2-adrenergic partial agonist that is used as a bronchodilator.
-
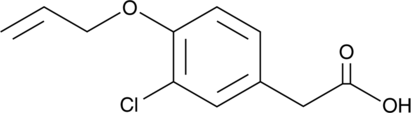
GC46826
Alclofenac
An NSAID
-
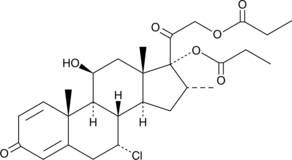
GC40638
Alclometasone Dipropionate
Le dipropionate d'alclométasone (Sch 22219) est un composé stéroÏdien.
-
GN10298
Alibiflorin
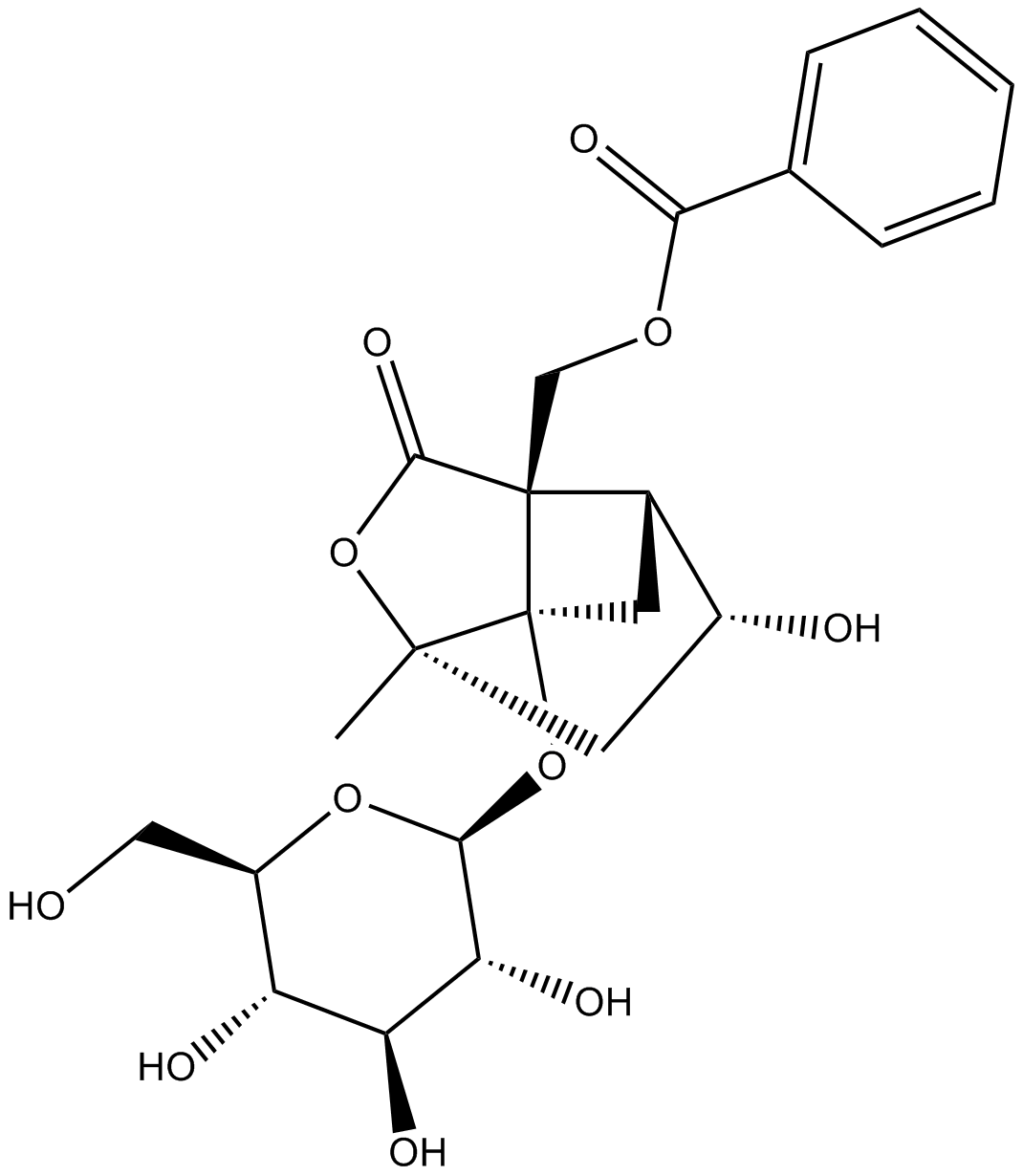
-
GC42769
all-trans Retinoyl β-D-Glucuronide
all-trans Retinoyl β-D-glucuronide is a metabolite of all-trans retinoic acid formed by the UDP-glucuronosyltransferase (UGT) system.