Histone Methyltransferase
Histone methyltransferases are a group of enzymes that catalyze the methylation of histone lysine and arginine by adding methyl groups to specific histone arginine or lysine residues. Histone methyltransferaes can be classified into 3 classes, including SET domain lysine methyltransferases, non-SET domain lysine methyltransferases and arginine methyltransferases (PRMTs), all of which use S-adenosylmethionine as a cosubstrate for the transfer of the methyl group. Aberrant histone methylation has been associated with a wide range of human cancers (such as hematological malignancies), which leads to the development of novel cancer chemotherapies targeting cancer-associated histone methyltransferases (more than 20 lysine methyltransferases and 9 arginine methyltransferases in humans).
Products for Histone Methyltransferase
- Cat.No. Nombre del producto Información
-
GC45908
(R)-BAY-598
An inhibitor of SMYD2
-
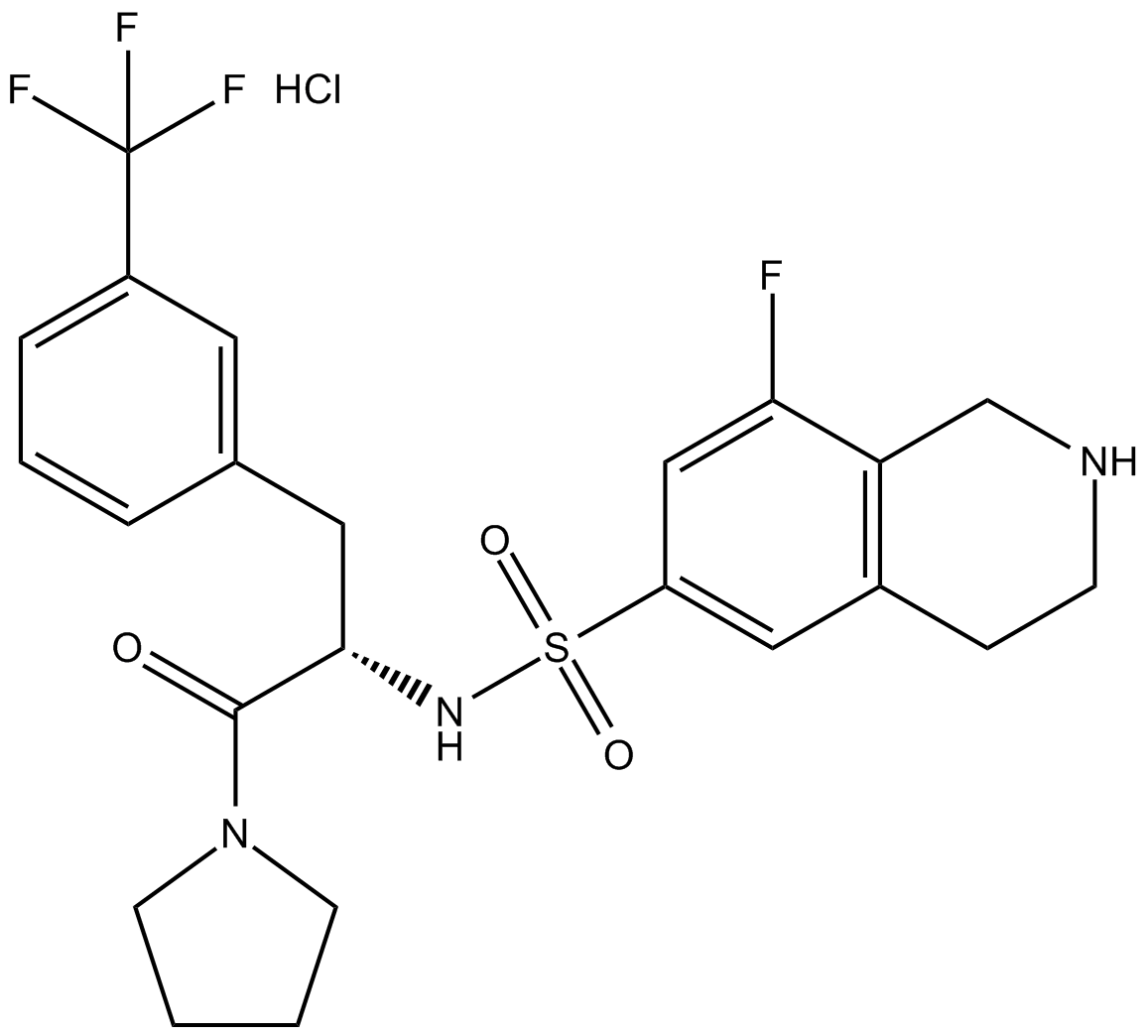
GC11340
(R)-PFI 2 hydrochloride
El clorhidrato de PFI-2 ((R)-(R)-PFI 2 clorhidrato) es un dominio SET potente y selectivo que contiene un inhibidor de la lisina metiltransferasa 7 (SETD7).
-
GC65045
(S)-MRTX-1719
(S)-MRTX-1719 (ejemplo 16-7) es el enantiÓmero S de MRTX-1719. (S)-MRTX-1719 es un inhibidor del complejo PRMT5/MTA, con una IC50 de 7070 nM.
-
GC13634
(S)-PFI-2 (hydrochloride)
Negative control of (R)-PFI 2 hydrochloride
-
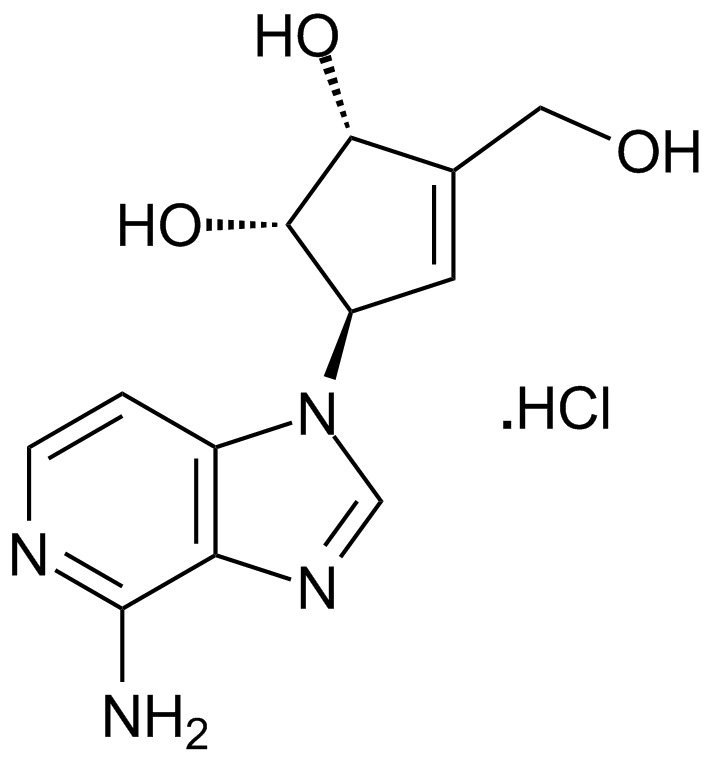
GC17907
3-Deazaneplanocin A (DZNep) hydrochloride
Un inhibidor de la lisina metiltransferasa EZH2.
-
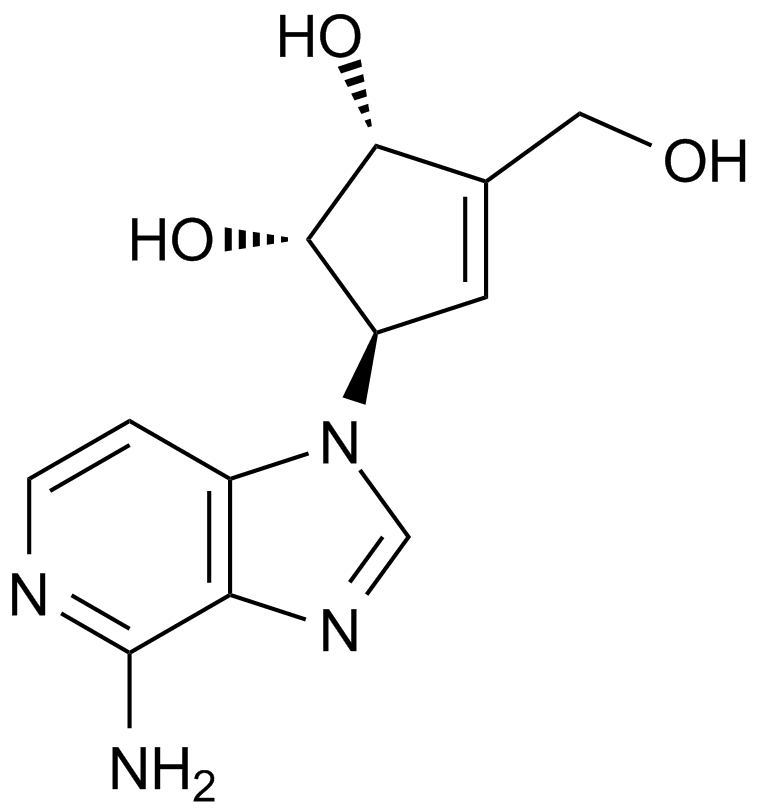
GC13145
3-Deazaneplanocin,DZNep
An inhibitor of lysine methyltransferase EZH2
-
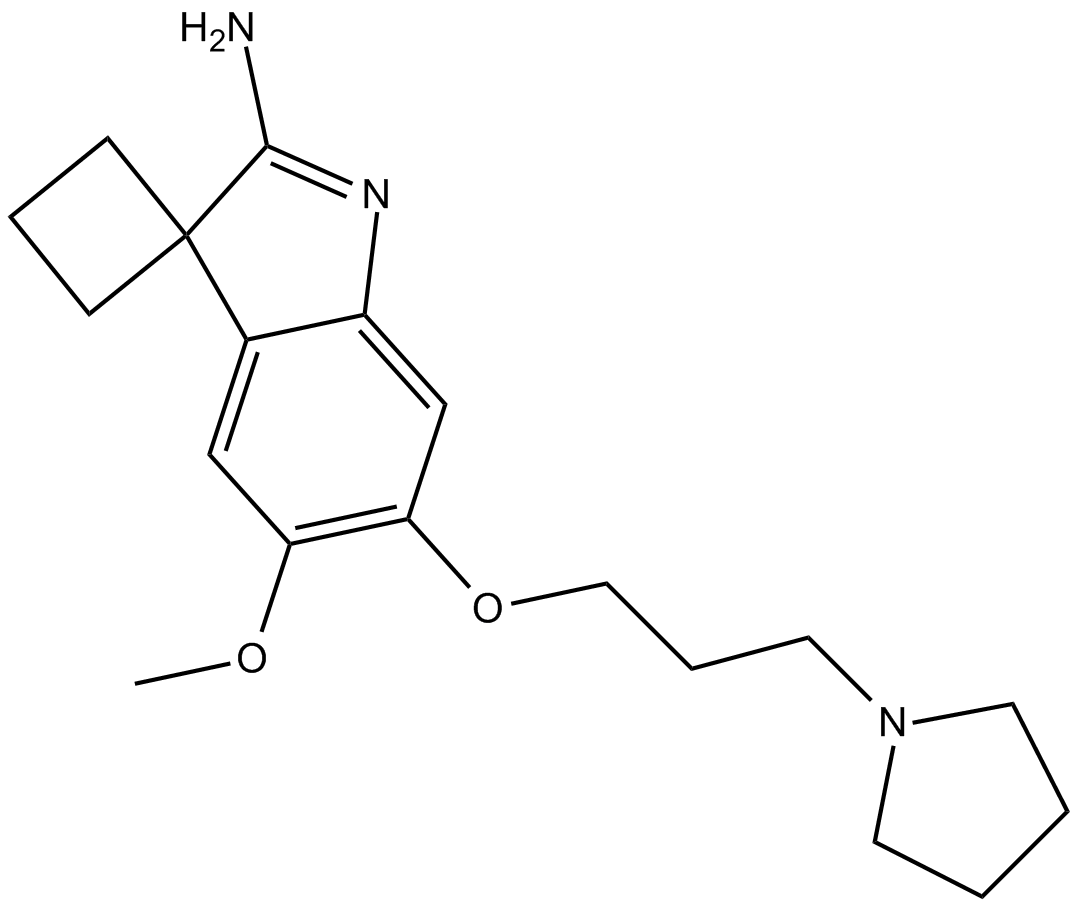
GC16015
A 366
A 366 es un inhibidor de histona metiltransferasa G9a potente, altamente selectivo y competitivo con péptidos con IC50 de 3,3 y 38 nM para G9a y GLP (EHMT1), respectivamente. A 366 muestra una selectividad de \u003e1000 veces sobre otras 21 metiltransferasas. A 366 también es un potente inhibidor nanomolar de la interacción Spindlin1-H3K4me3 (IC50 \u003d 182,6 nM). A 366 muestra alta afinidad por el receptor H3 de histamina humano (Ki \u003d 17 nM) y muestra selectividad de subtipo entre los subconjuntos de las familias de receptores histaminérgicos y dopaminérgicos.
-
GC32861
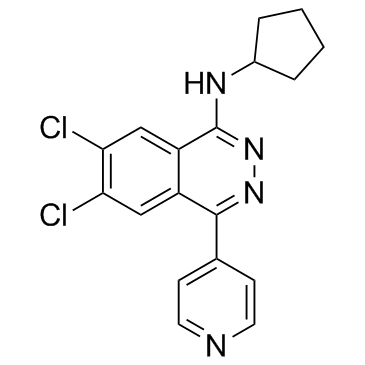
A-196
A-196 es un inhibidor potente y selectivo de SUV420H1 y SUV420H2 con valores IC50 de 25 nM y 144 nM, respectivamente. A-196 inhibe SUV4-20 bioquÍmicamente de manera competitiva con el sustrato. A-196 representa una sonda quÍmica de primera clase de SUV4-20 para investigar el papel de las histonas metiltransferasas en la integridad genÓmica.
-
GC33187
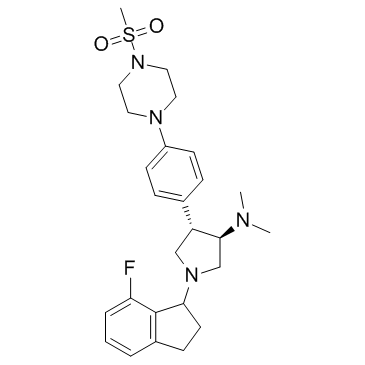
A-395 (A395)
A-395 (A395) es un antagonista de las interacciones proteÍna-proteÍna del complejo represivo 2 de Polycomb (PRC2) que inhibe potentemente el complejo trimérico PRC2 (EZH2-EED-SUZ12) con una IC50 de 18 nM.
-
GC30503
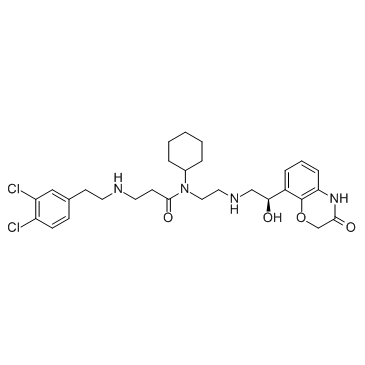
A-893
A-893 es un inhibidor celular activo de la metiltransferasa SMYD2, con una IC50 de 2,8 nM.
-
GC16509
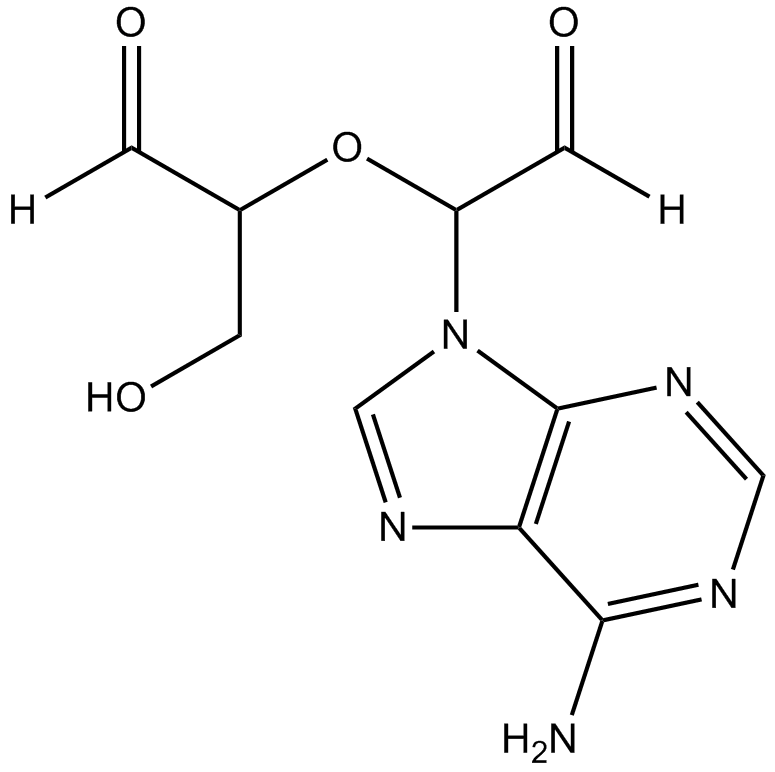
Adox
Adox, un anÁlogo de nucleÓsido de purina, es un potente inhibidor de la S-adenosilhomocisteÍna hidrolasa (SAHH) (Ki = 3,3 nM). El dialdehÍdo de adenosina exhibe una potente actividad antitumoral in vivo y puede usarse para la investigaciÓn del cÁncer.
-
GC17275
AMI-1
A cell permeable inhibitor of PRMTs
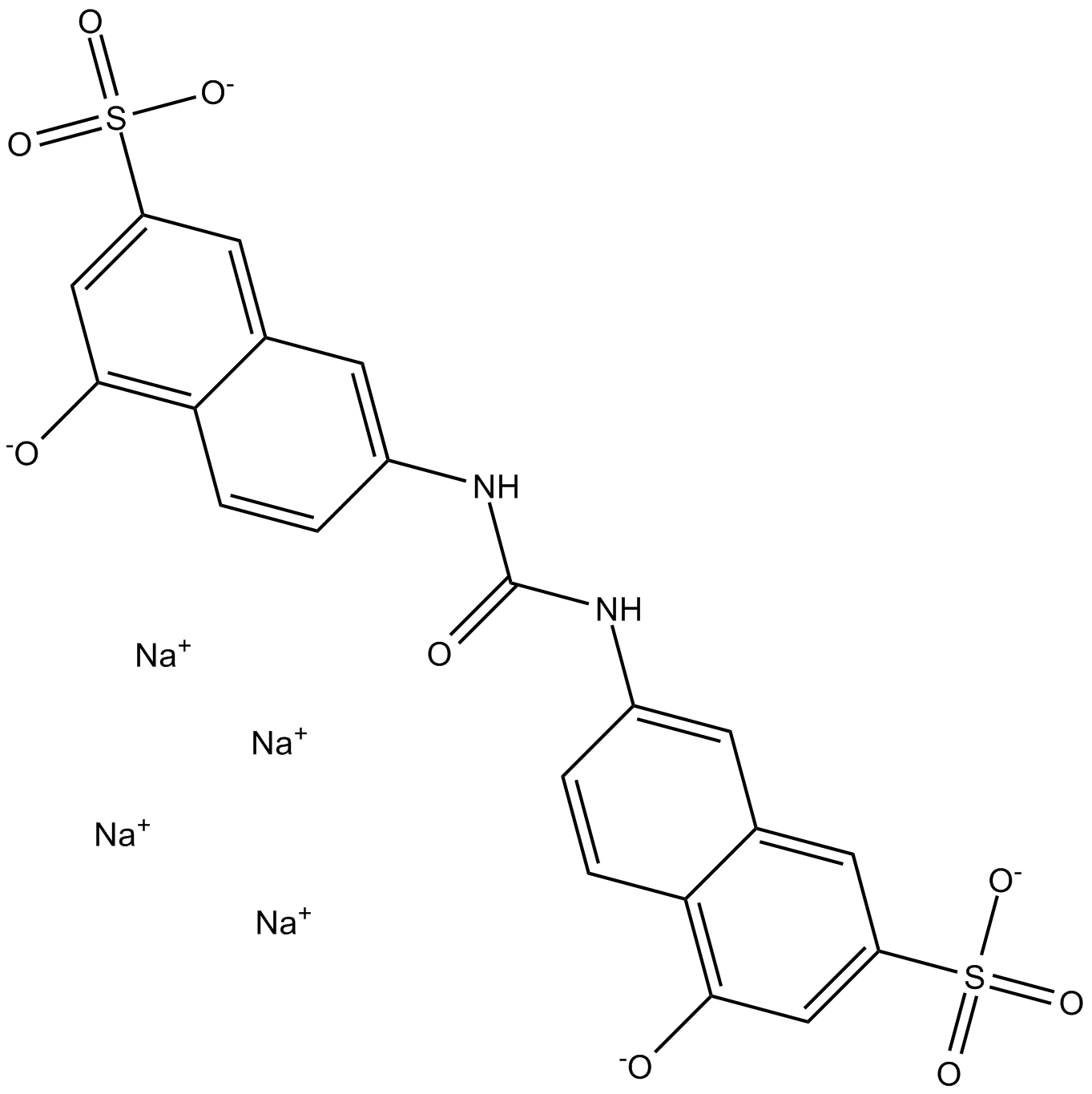
-
GC39840
AMI-1 free acid
El Ácido libre de AMI-1 es un inhibidor potente, permeable a las células y reversible de la proteÍna arginina N-metiltransferasas (PRMT), con IC50 de 8,8 μM y 3,0 μM para PRMT1 humano y levadura-Hmt1p, respectivamente. El Ácido libre de AMI-1 ejerce efectos inhibidores de PRMT al bloquear la uniÓn de péptido-sustrato.
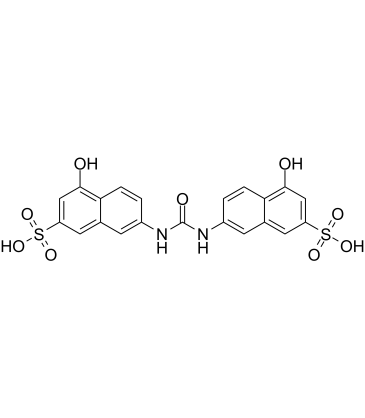
-
GC17546
AMI5
La eosina Y (disÓdica) es una molécula de colorante rojo Ácido soluble.
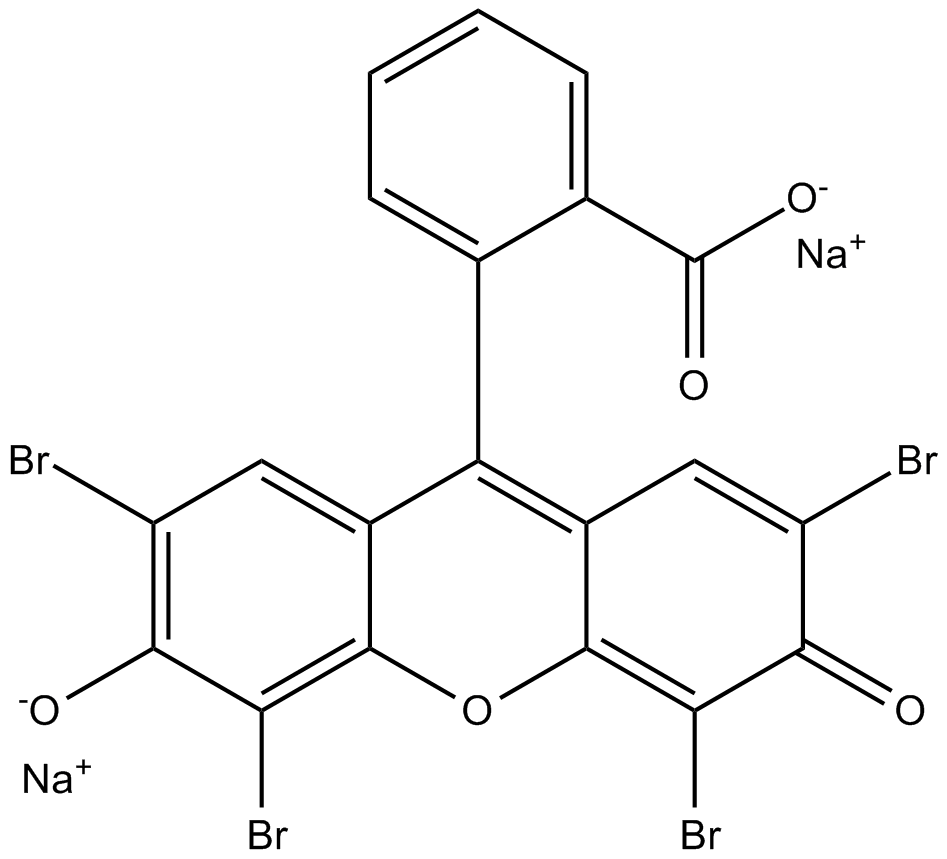
-
GC42790
Amodiaquine
Amodiaquina es un compuesto antipalúdico aminoquinolínico.
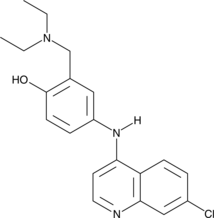
-
GC60579
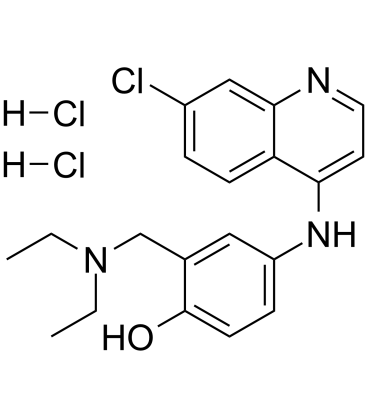
Amodiaquine dihydrochloride
El diclorhidrato de amodiaquina (diclorhidrato de amodiaquina), una clase de agente antipalÚdico de 4-aminoquinolina, es un inhibidor potente y activo por vÍa oral de la histamina N-metiltransferasa con una Ki de 18,6 nM.
-
GC10905
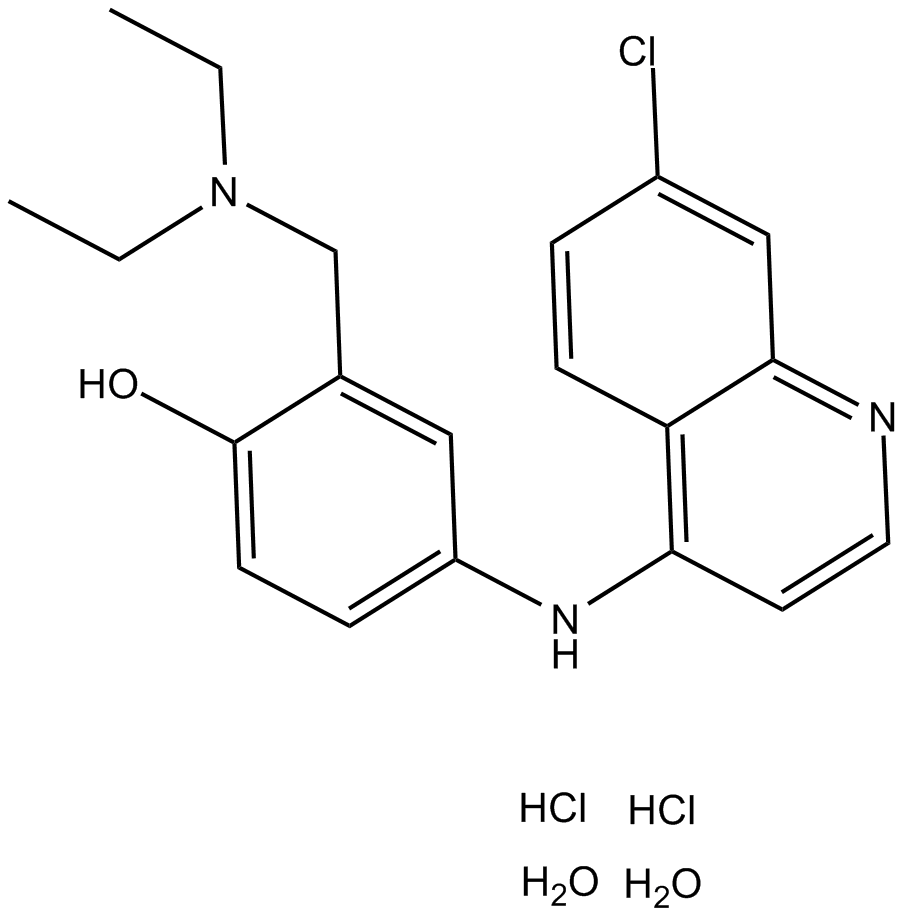
Amodiaquine dihydrochloride dihydrate
El dihidrocloruro de amodiaquina dihidrato (Dihidrocloruro de amodiaquina dihidrato), una clase de 4-aminoquinolina de agente antipalÚdico, es un inhibidor potente y activo por vÍa oral de la histamina N-metiltransferasa.
-
GC65918
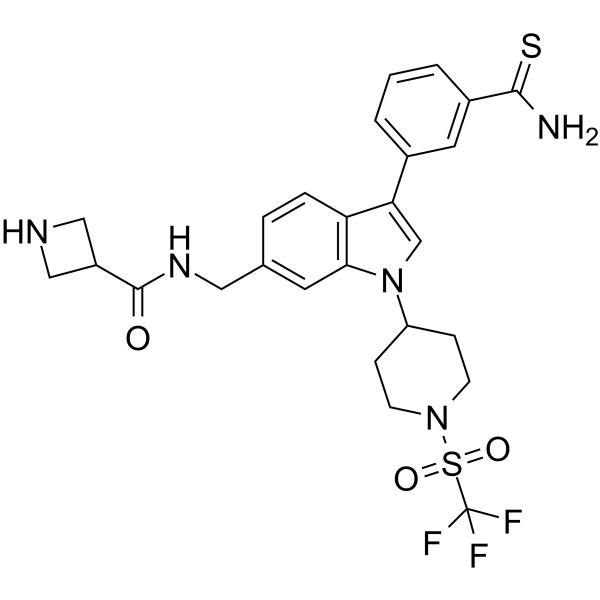
AS-85
AS-85 es un potente inhibidor de la histona metiltransferasa ASH1L (IC50=0,6 μM) con actividad antileucémica. AS-85 se une fuertemente al dominio SET ASH1L, con el valor Kd de 0.78μM.
-
GC62615
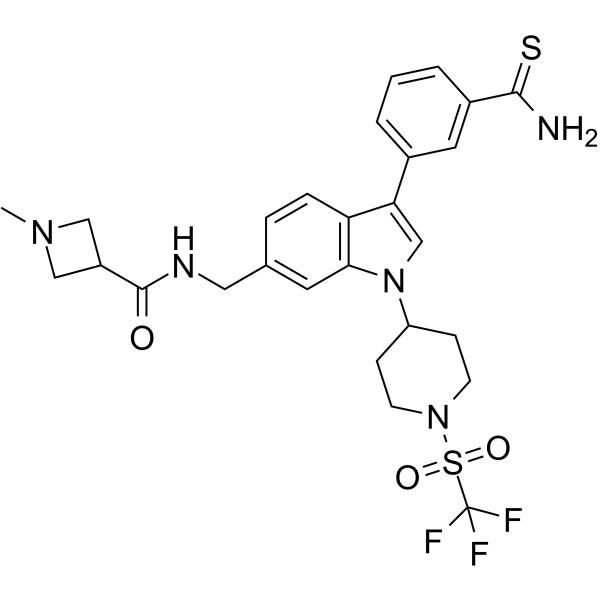
AS-99
AS-99 es un inhibidor de histona metiltransferasa ASH1L selectivo, potente y de primera clase (IC50 = 0,79μ M, Kd = 0,89μ M) con actividad antileucémica. AS-99 bloquea la proliferaciÓn celular, induce la apoptosis y la diferenciaciÓn, regula a la baja los genes diana de la fusiÓn MLL y reduce la carga de leucemia in vivo.
-
GC62849
AS-99 TFA
AS-99 TFA es un inhibidor de histona metiltransferasa ASH1L selectivo, potente y de primera clase (IC50=0,79μM, Kd=0,89μM) con actividad antileucémica. AS-99 TFA bloquea la proliferaciÓn celular, induce la apoptosis y la diferenciaciÓn, regula a la baja los genes diana de la fusiÓn MLL y reduce la carga de leucemia in vivo.
-
GC13744
AZ505
AZ505 es un inhibidor potente y selectivo de SMYD2 con una IC50 de 0,12 μM.
-
GC13103
AZ505 ditrifluoroacetate
El ditrifluoroacetato AZ505 es un inhibidor potente y selectivo de SMYD2 con IC50 de 0,12 μM.
-
GC64124
AZ506
AZ506 es un potente inhibidor de SMYD2 con una IC50 de 17 nM. AZ506 inhibe la actividad de la metiltransferasa de SMYD2 en las células, lo que provoca una disminuciÓn de la seÑal de metilaciÓn mediada por SMYD2.
-
GC18159
BAY-598
BAY-598 es un inhibidor selectivo de molécula pequeÑa de SMYD2 con una IC50 de 27 nM.
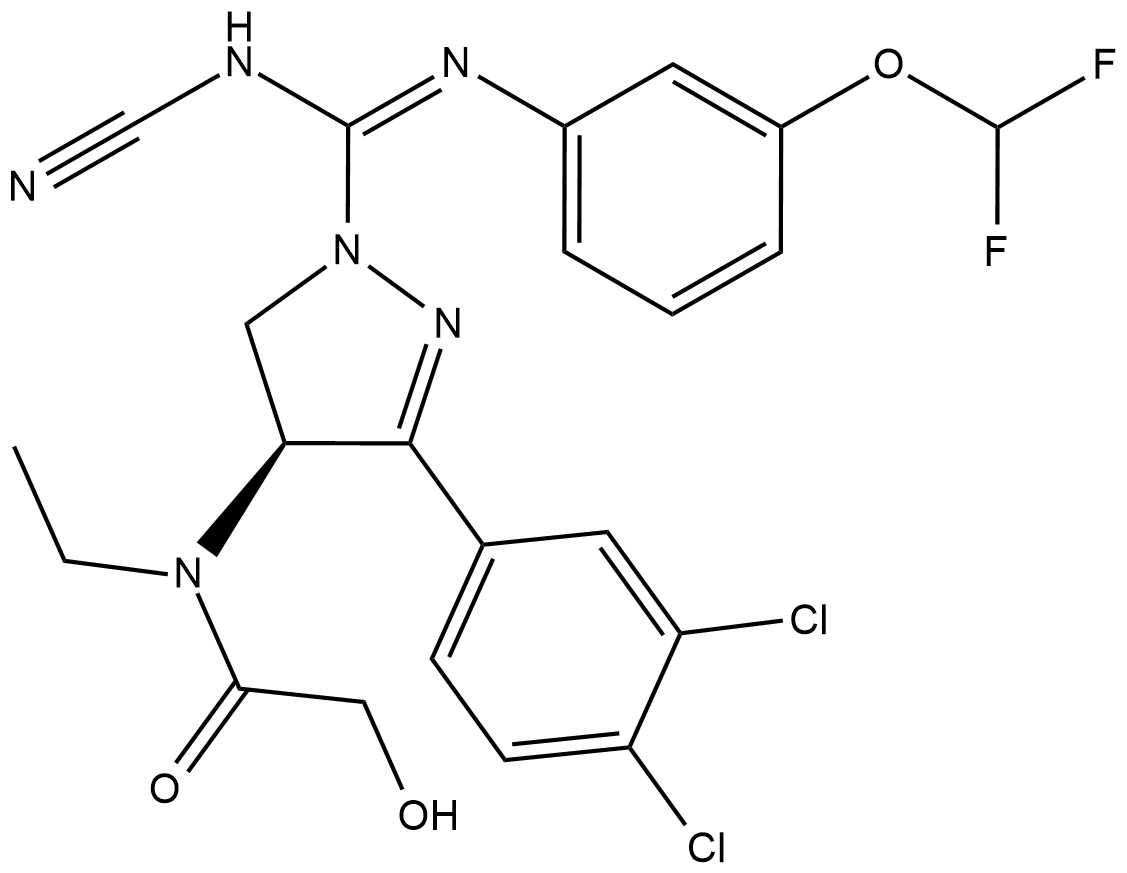
-
GC45389
BAY-6035
BAY-6035 es un inhibidor potente, selectivo y competitivo de sustrato de SMYD3. BAY-6035 inhibe la metilaciÓn del péptido MEKK2 con una IC50 de 88 nM.
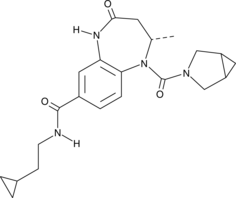
-
GC18161
BCI-121
BCI-121 es un inhibidor de SMYD3 que impide la proliferaciÓn de células cancerosas.
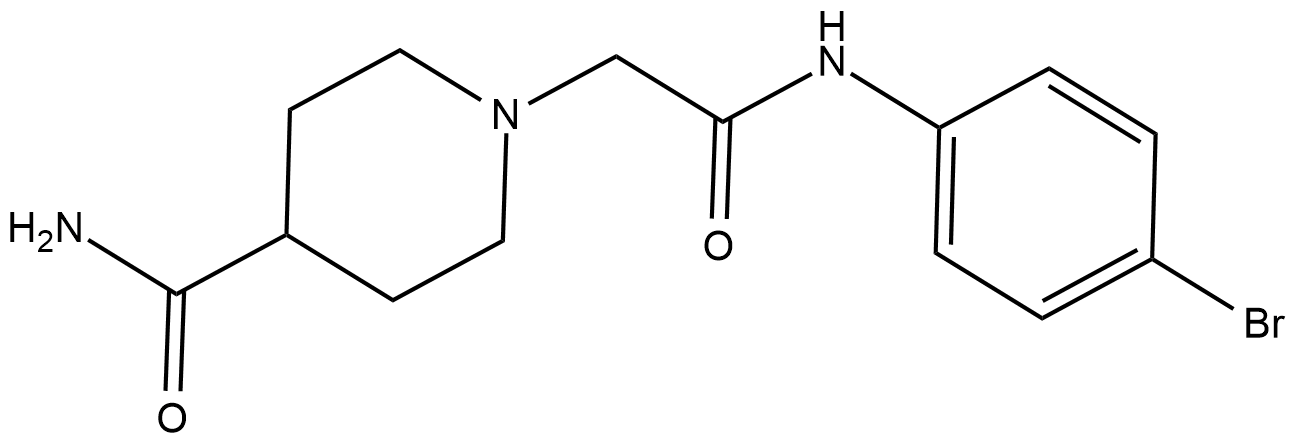
-
GC50540
BI 9321
Nuclear receptor-binding SET domain (NSD) 3 antagonist; selectively binds PWWP1 domain
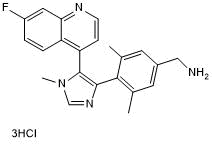
-
GC39663
BI-9321 trihydrochloride
El trihidrocloruro de BI-9321 es un antagonista del dominio SET 3 (NSD3)-PWWP1 potente, selectivo y celularmente activo con un valor de Kd de 166 nM. El trihidrocloruro de BI-9321 es inactivo frente a NSD2-PWWP1 y NSD3-PWWP2. El triclorhidrato de BI-9321 interrumpe especÍficamente las interacciones de histonas del dominio NSD3-PWWP1 con un IC50 de 1,2 μM en células U2OS.
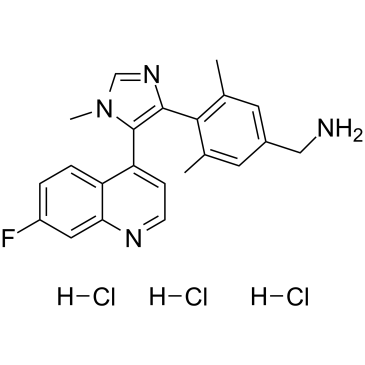
-
GC48463
Bisubstrate Inhibitor 78
An inhibitor of NNMT
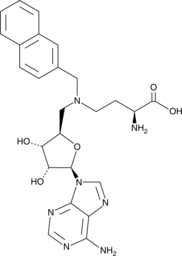
-
GC12171
BIX 01294
An inhibitor of G9a histone methyltransferase
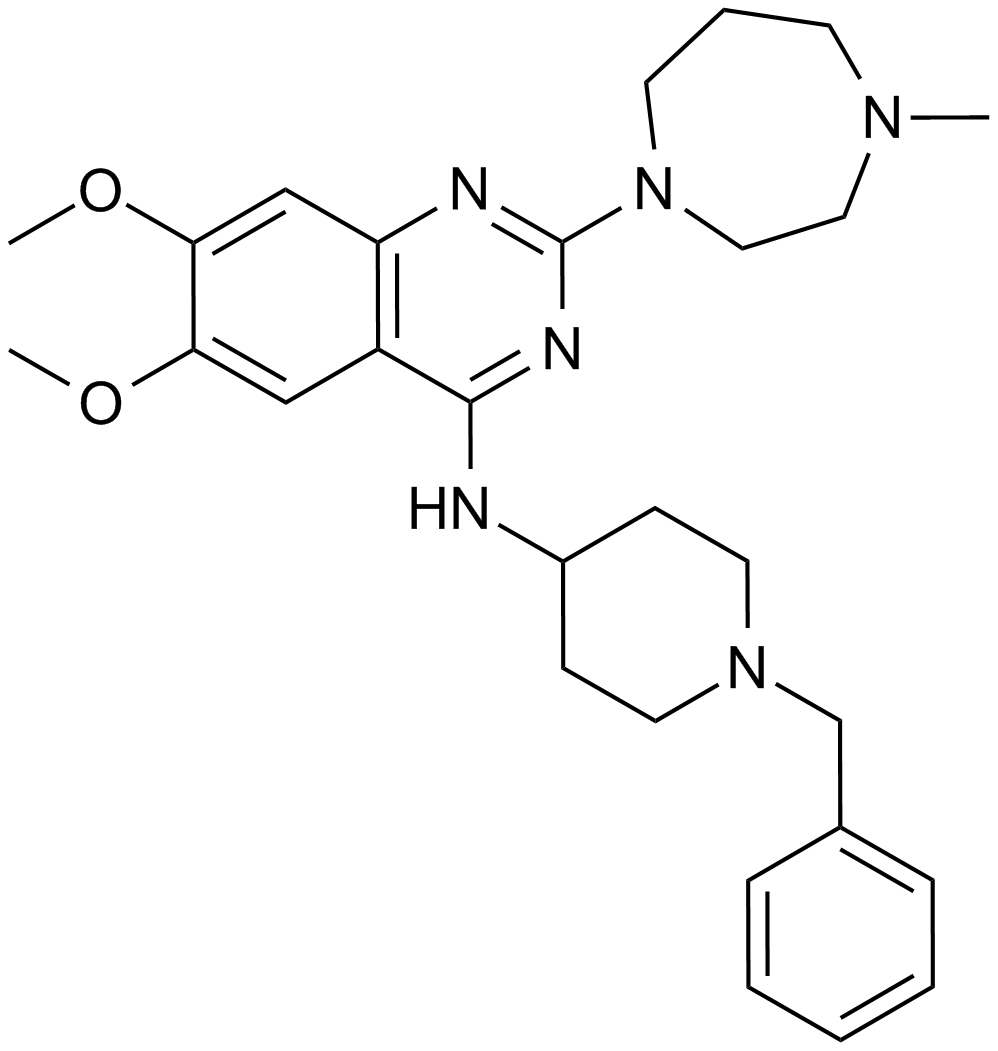
-
GC33301
BIX-01338 hydrate (BIX01338 hydrate)
BIX-01338 hidrato (BIX01338 hidrato) es un inhibidor de la histona lisina metiltransferasa.
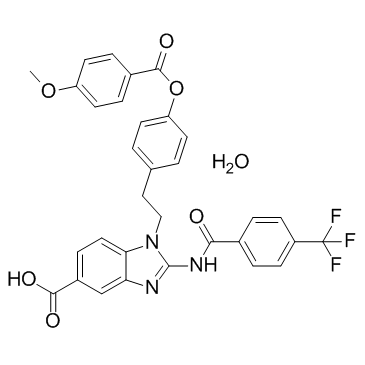
-
GC42944
BIX01294 (hydrochloride hydrate)
The methylation of lysine residues on histones plays a central role in determining euchromatin structure and gene expression.
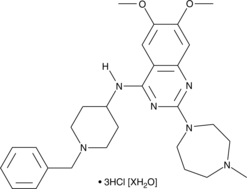
-
GC63731
BRD0639
BRD0639 es el primer inhibidor de su clase de la interacciÓn PRMT5-adaptador de sustrato. BRD0639 es un agente competitivo con motivo de uniÓn a PRMT5 (PBM) que puede respaldar los estudios de las actividades de PRMT5 dependientes de PBM.
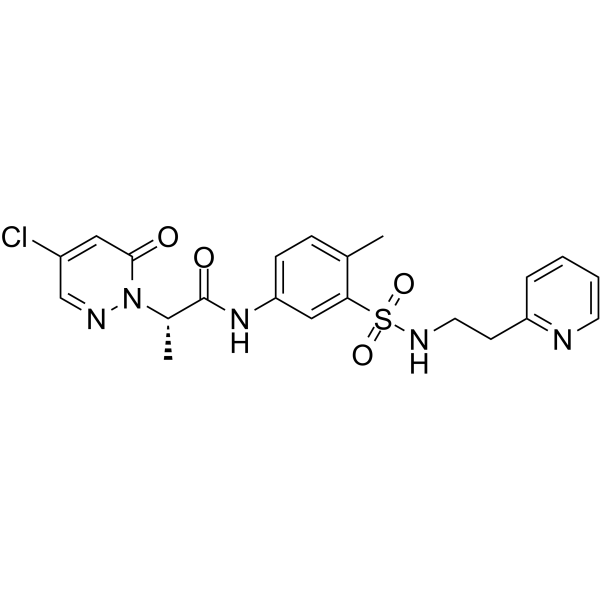
-
GC10259
BRD4770
BRD4770 es un inhibidor de la histona metiltransferasa G9a. BRD4770 reduce la dimetilaciÓn y la trimetilaciÓn de la lisina 9 en la histona H3 (H3K9) con una CE50 de 5 μM y tiene un efecto menor o menor hacia H3K27me3, H3K36me3, H3K4me3 y H3K79me3. BRD4770 puede activar la vÍa mutada de ataxia telangiectasia (ATM) e inducir la senescencia celular.
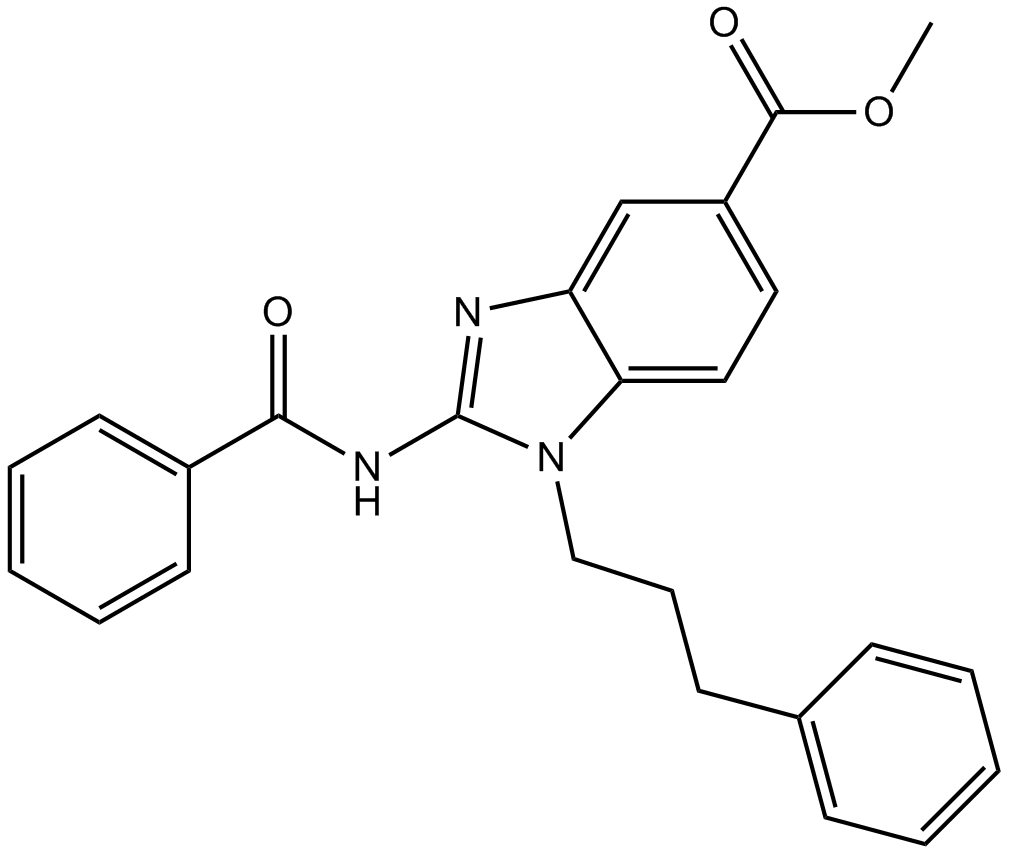
-
GC32988
BRD9539
BRD9539 es un inhibidor de la histona metiltransferasa G9a con una IC50 de 6,3 μM. BRD9539 también inhibe la actividad de PRC2 y es inactivo frente a SUV39H1, NSD2 y DNMT1.
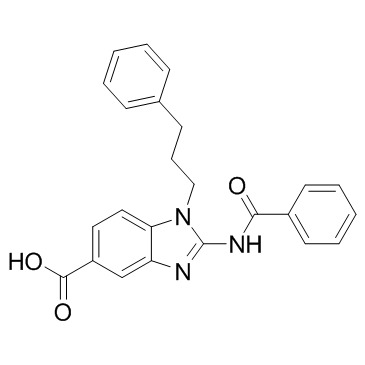
-
GC16130
C 21
Protein arginine methyltransferase 1 (PRMT1) inhibitor

-
GC12005
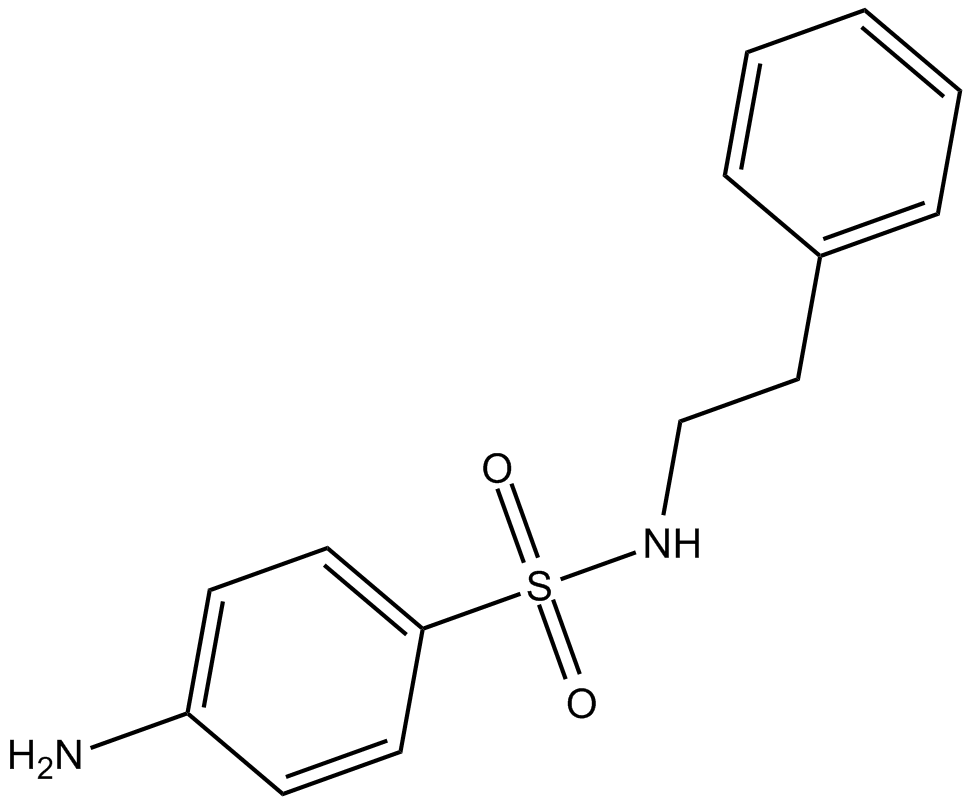
C7280948
C7280948 es un inhibidor selectivo y potente de la proteína metiltransferasa 1 (PRMT1) con un valor IC50 de 12,75 μM.
-
GC34108
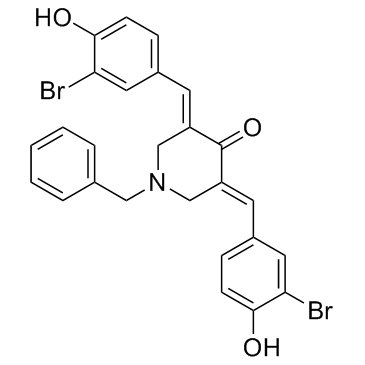
CARM1-IN-1
CARM1-IN-1 es un potente y especÍfico inhibidor de CARM1 (arginina metiltransferasa 1 asociada a coactivador) con IC50 de 8,6 uM; muestra una actividad muy baja frente a PRMT1 y SET7 (IC50 > 600 uM).
-
GC34424
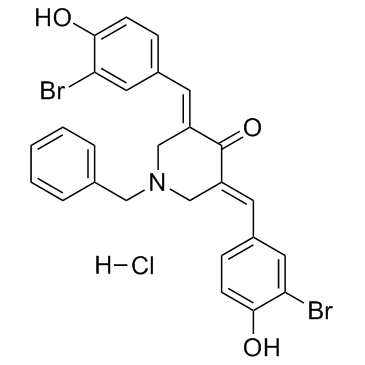
CARM1-IN-1 hydrochloride
El clorhidrato de CARM1-IN-1 es un inhibidor potente y especÍfico de CARM1 (arginina metiltransferasa 1 asociada a coactivador) con IC50 de 8,6 uM; muestra una actividad muy baja frente a PRMT1 y SET7 (IC50 > 600 uM).
-
GC18949
CAY10677
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.
-
GC14936
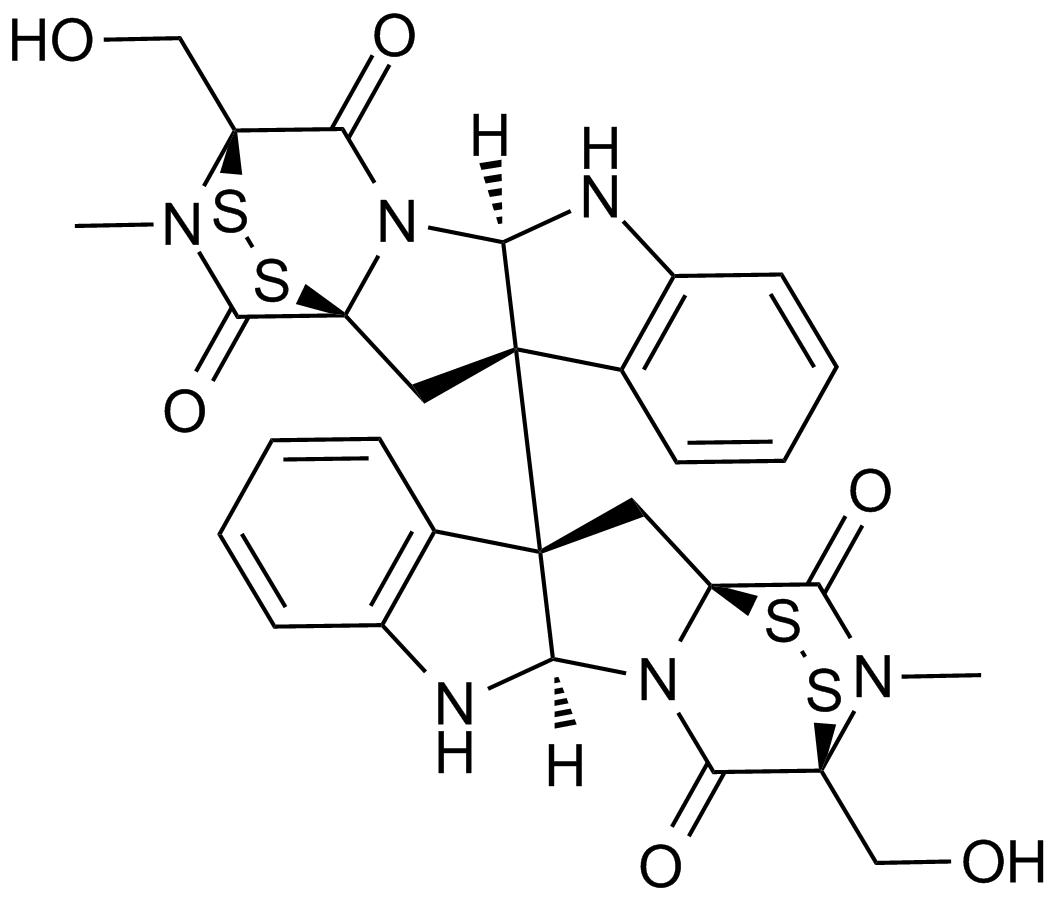
Chaetocin
Inhibitor of lys9-specific HMTs
-
GC18577
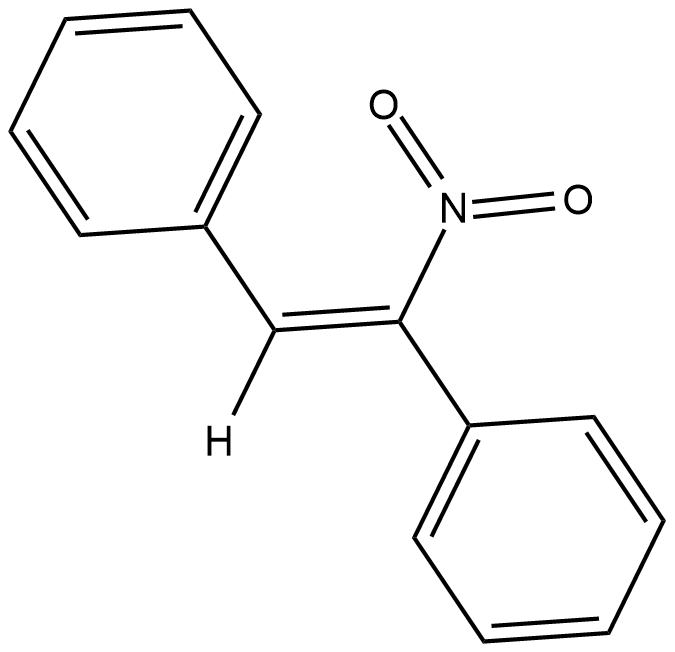
CID-2818500
An inhibitor of PRMT1
-
GC12367
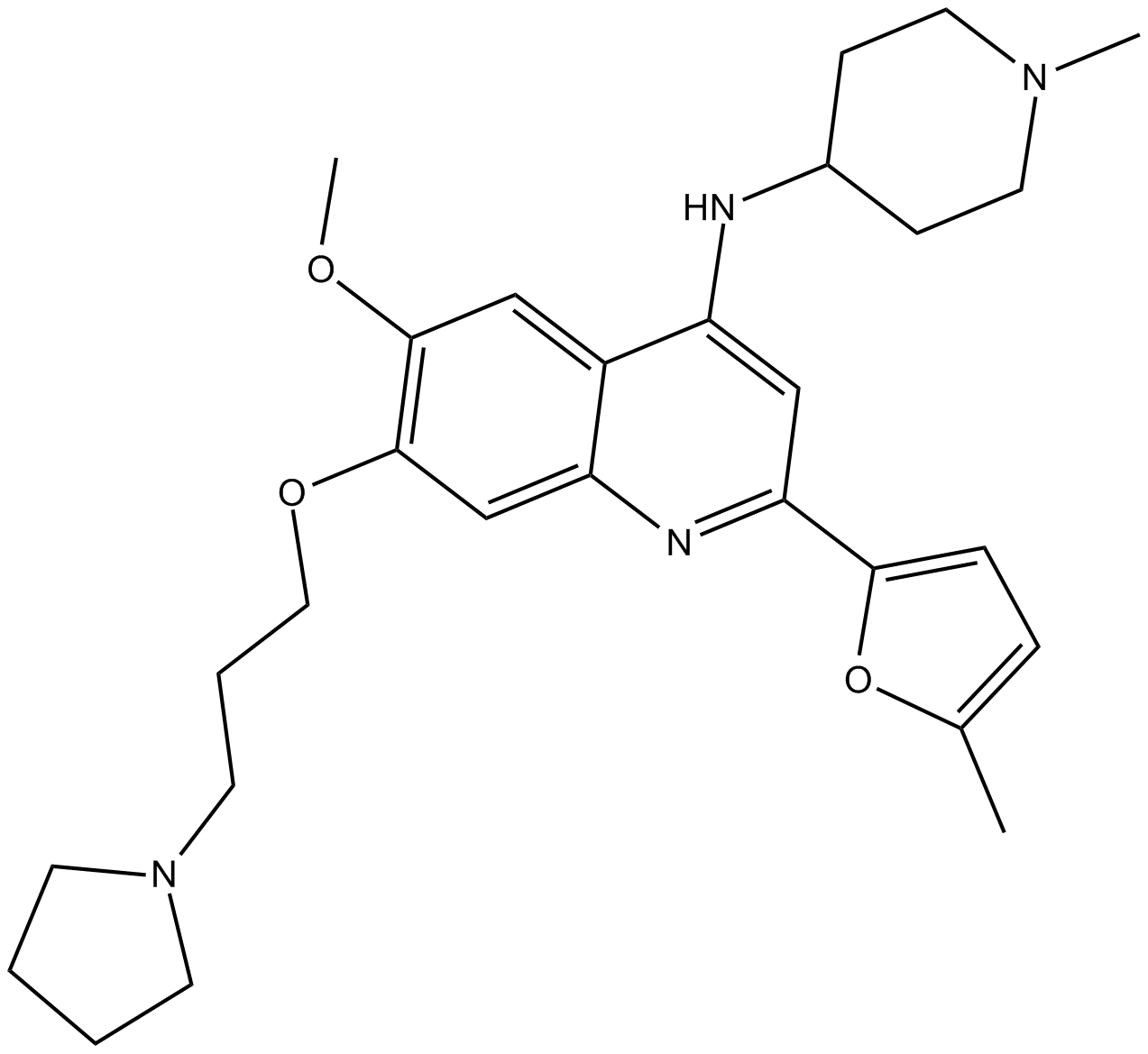
CM-272
CM-272 es un inhibidor dual G9a/DNA metiltransferasas (DNMT) de primer nivel, potente, selectivo, competitivo con sustrato y reversible con actividades antitumorales. CM-272 inhibe G9a, DNMT1, DNMT3A, DNMT3B y GLP con IC50 de 8 nM, 382 nM, 85 nM, 1200 nM y 2 nM, respectivamente. CM-272 inhibe la proliferaciÓn celular y promueve la apoptosis, induciendo genes estimulados por IFN y muerte celular inmunogénica.
-
GC33320
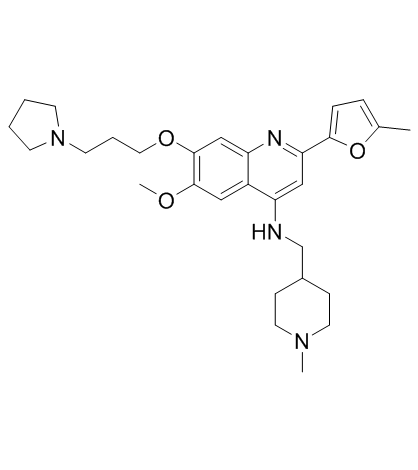
CM-579
CM-579 es el primer inhibidor dual reversible de su clase de G9a y DNMT, con valores IC50 de 16 nM, 32 nM para G9a y DNMT, respectivamente. Tiene una potente actividad celular in vitro en una amplia gama de células cancerosas.
-
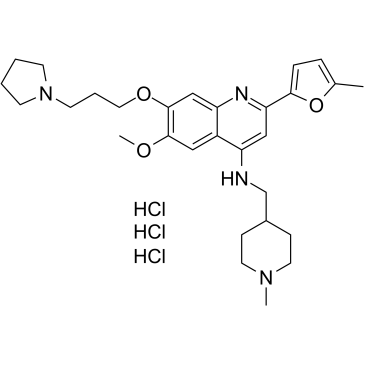
GC35714
CM-579 trihydrochloride
El trihidrocloruro de CM-579 es el primer inhibidor dual reversible de su clase de G9a y DNMT, con valores IC50 de 16 nM, 32 nM para G9a y DNMT, respectivamente. Tiene una potente actividad celular in vitro en una amplia gama de células cancerosas.
-
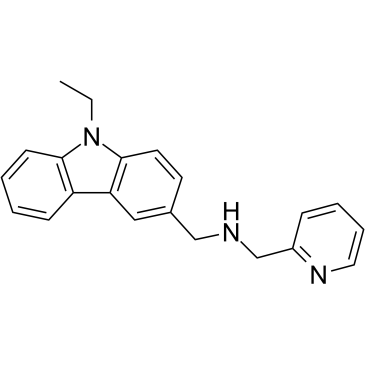
GC39665
CMP-5
CMP-5 es un inhibidor potente, especÍfico y selectivo de PRMT5, mientras que no muestra actividad contra las enzimas PRMT1, PRMT4 y PRMT7. CMP-5 bloquea selectivamente S2Me-H4R3 al inhibir la actividad de la metiltransferasa PRMT5 en las preparaciones de histonas. CMP-5 previene la transformaciÓn de linfocitos B impulsada por el virus de Epstein-Barr (EBV), pero no afecta a las células B normales.
-
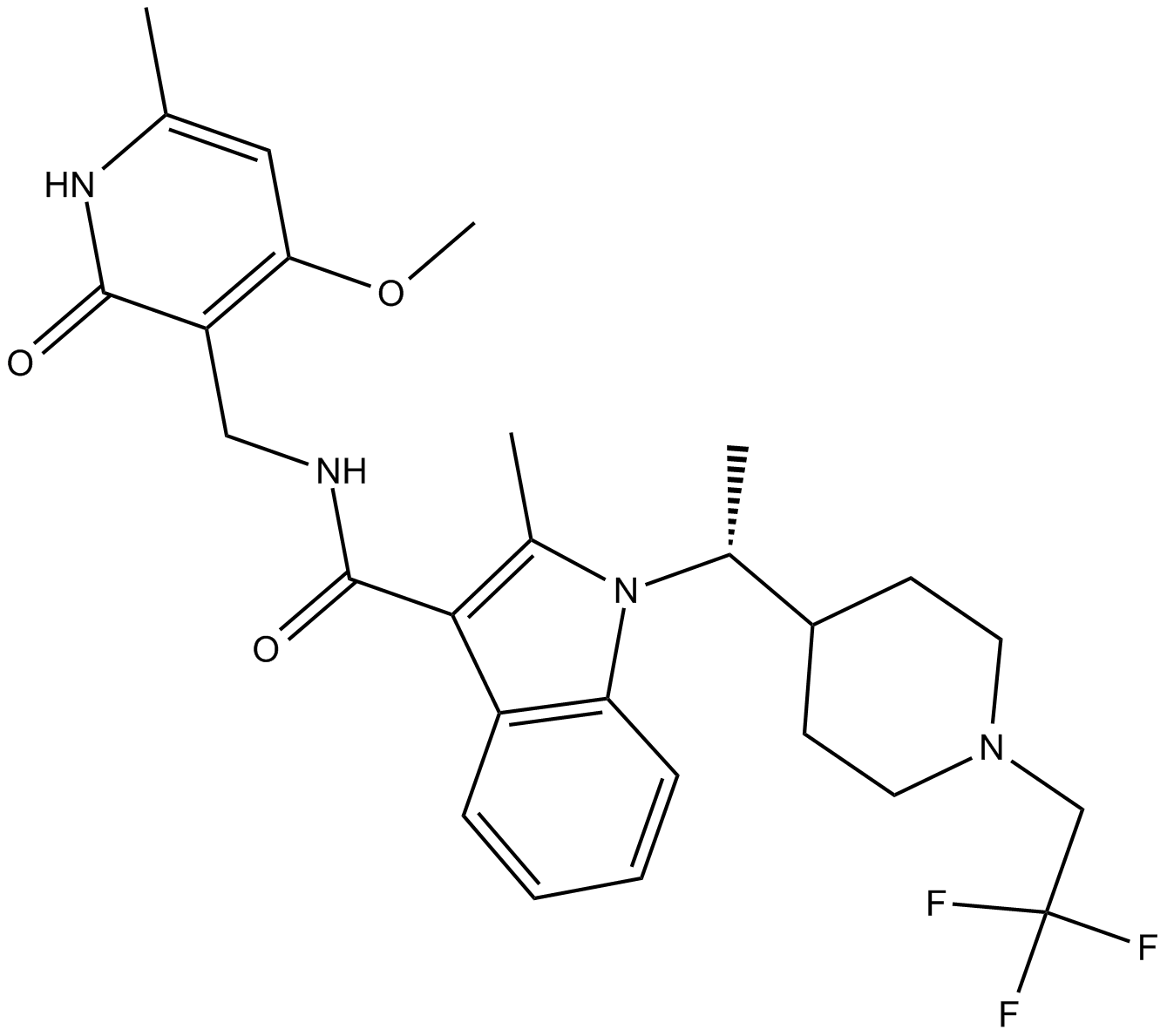
GC16298
CPI-1205
EZH2 inhibitor
-
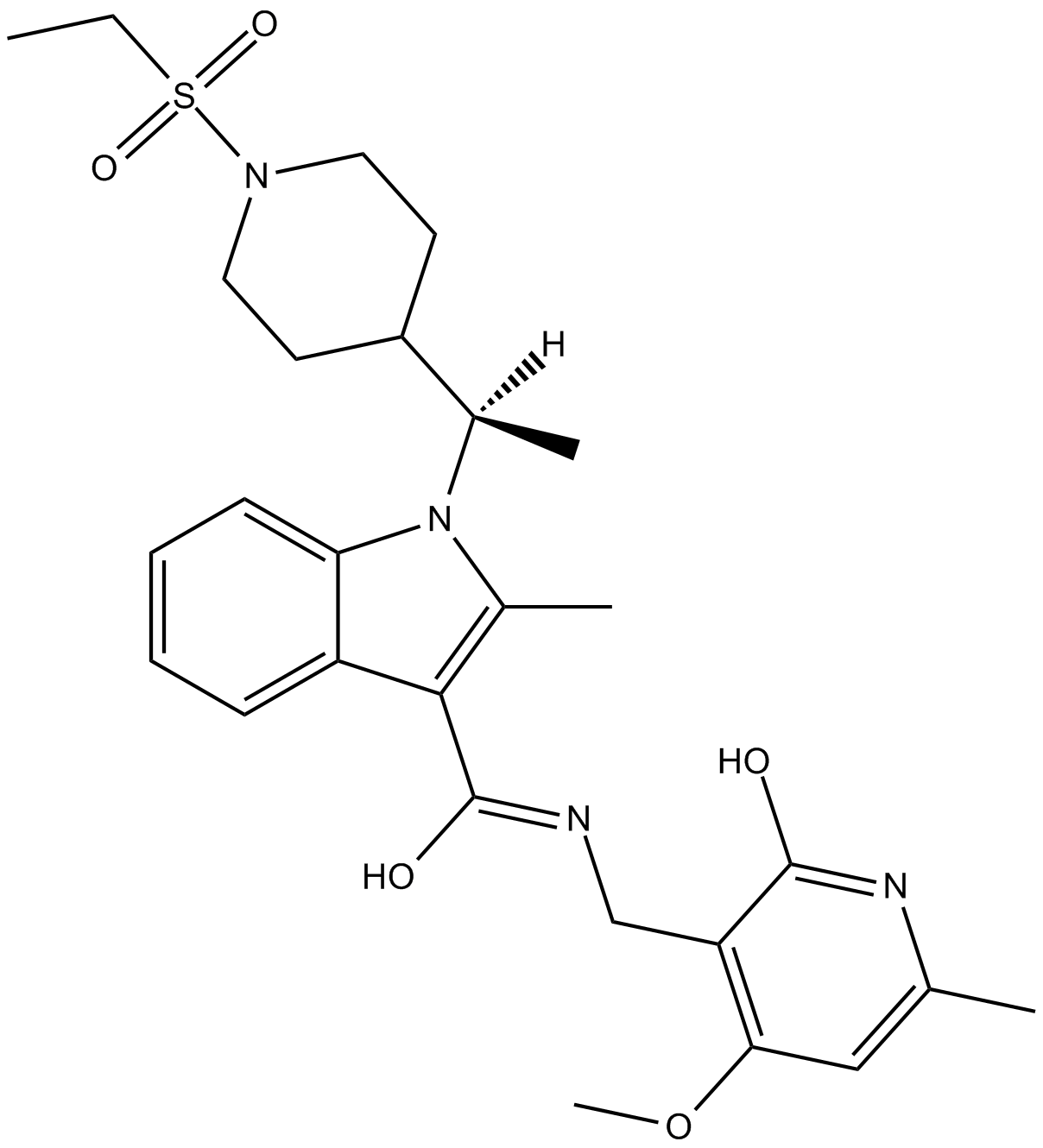
GC16599
CPI-169
EZH2 inhibitor
-
GC10021
CPI-360
EZH2 inhibitor
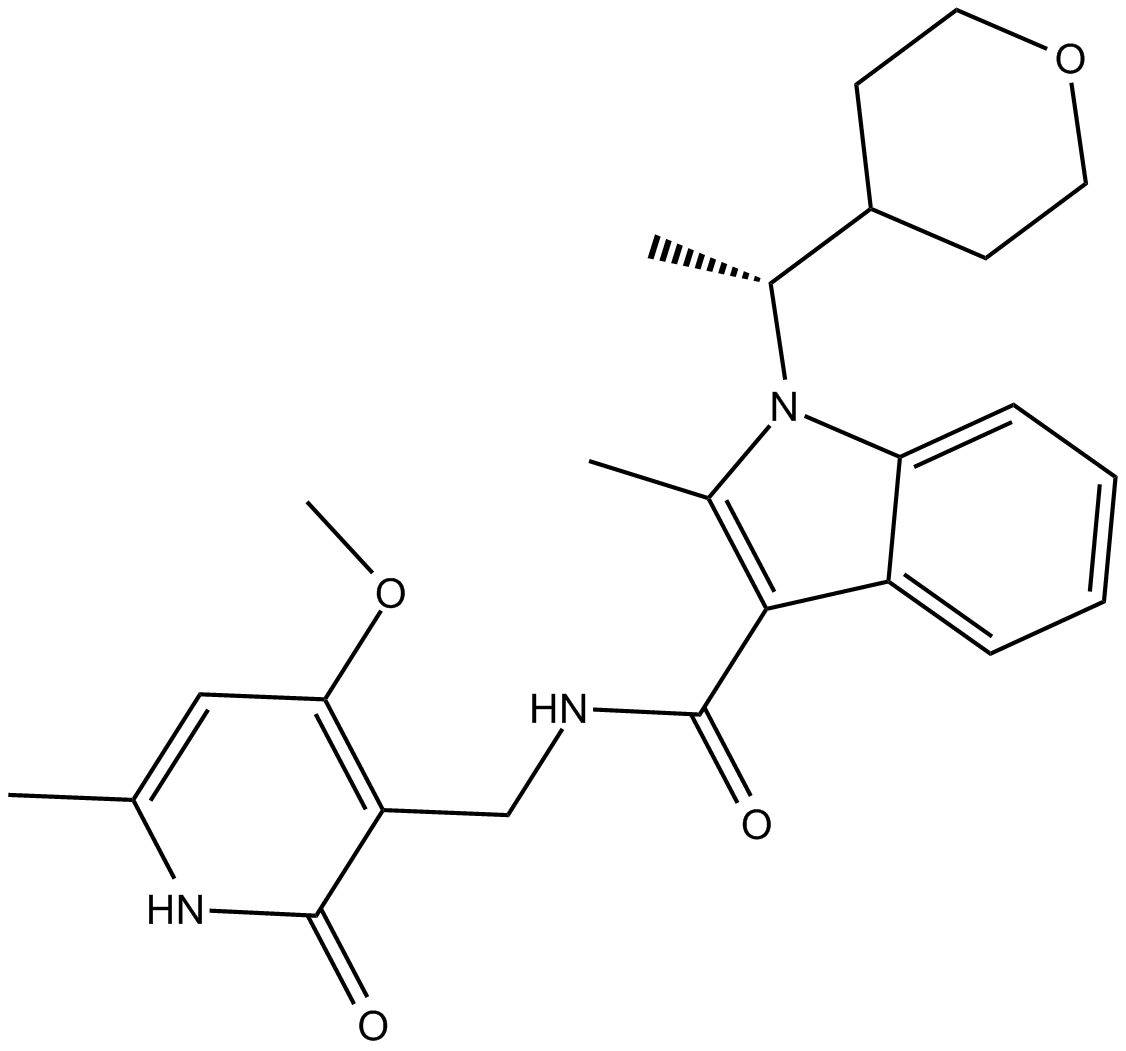
-
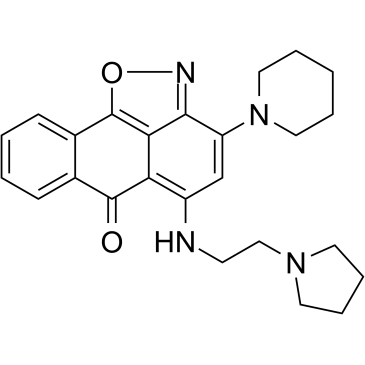
GC35742
CPUY074020
CPUY074020 es un inhibidor potente y biodisponible por vÍa oral de la histona metiltransferasa G9a, con una IC50 de 2,18 μM. CPUY074020 posee actividad antiproliferativa.
-
GC43354
Cysmethynil
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.

-
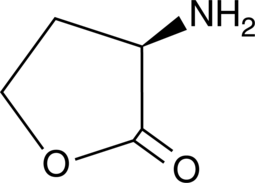
GC48967
D-Homoserine lactone
An enantiomer of L-homoserine lactone
-
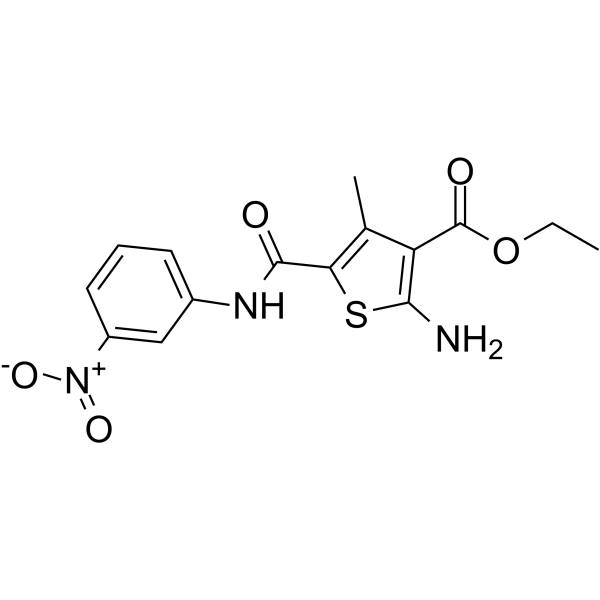
GC65186
DC-S239
DC-S239 es un inhibidor selectivo de histona metiltransferasa SET7 con un valor IC50 de 4,59 μM. DC-S239 también muestra selectividad para DNMT1, DOT1L, EZH2, NSD1, SETD8 y G9a. DC-S239 tiene actividad anticancerÍgena.
-
GC63662
DCLX069
DCLX069 es un inhibidor selectivo de la proteÍna arginina metiltransferasa 1 (PRMT1) con un valor IC50 de 17,9 μM. DCLX069 se muestra menos activo frente a PRMT4 y PRMT6. DCLX069 tiene efectos anticancerÍgenos.
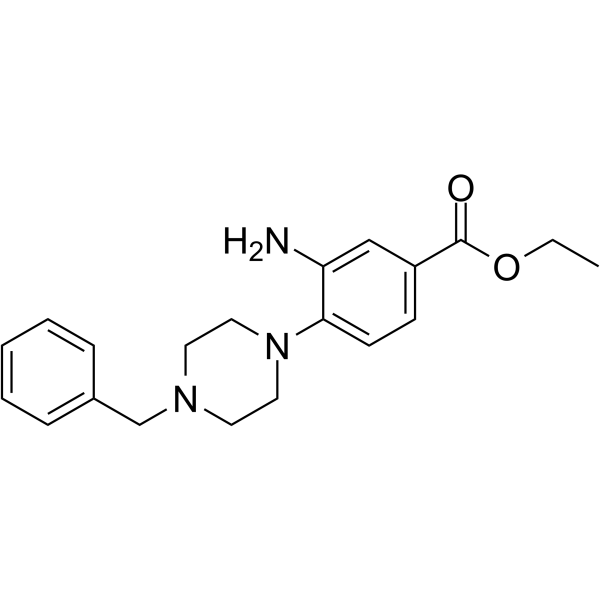
-
GC35816
DC_C66
DC_C66 es un inhibidor selectivo de la arginina metiltransferasa 1 (CARM1) asociado a un coactivador permeable a las células con una IC50 de 1,8 μM. DC_C66 tiene una buena selectividad para CARM1 contra PRMT1 (IC50 = 21 μM), PRMT6 (IC50 = 47 μM) y PRMT5.
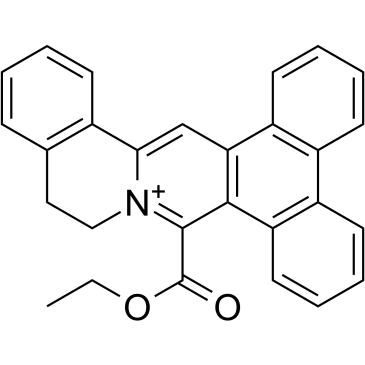
-
GC67863
DDO-2093 dihydrochloride

-
GC33208
Dot1L-IN-1
Dot1L-IN-1 es un inhibidor de Dot1L muy potente, selectivo y estructuralmente novedoso con una Ki de 2 pM.
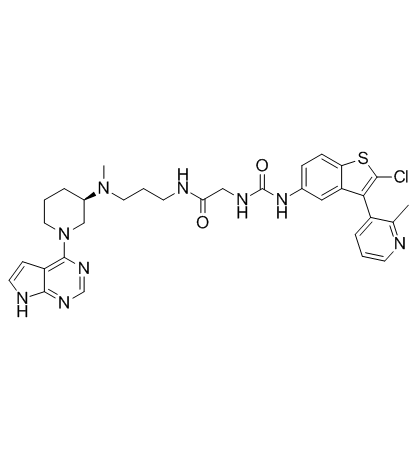
-
GC30530
Dot1L-IN-2
Dot1L-IN-2 es un inhibidor potente, selectivo y biodisponible por vÍa oral de Dot1L (una histona metiltransferasa), con una IC50 y una Ki de 0,4 nM y 0,08 nM, respectivamente.
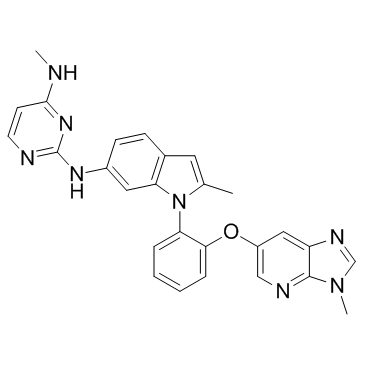
-
GC62613
Dot1L-IN-4
Dot1L-IN-4 es un potente disruptor del inhibidor de la proteÍna similar a 1 silenciador telomérico (DOT1L) con un IC50 SPA DOT1L de 0,11 nM.
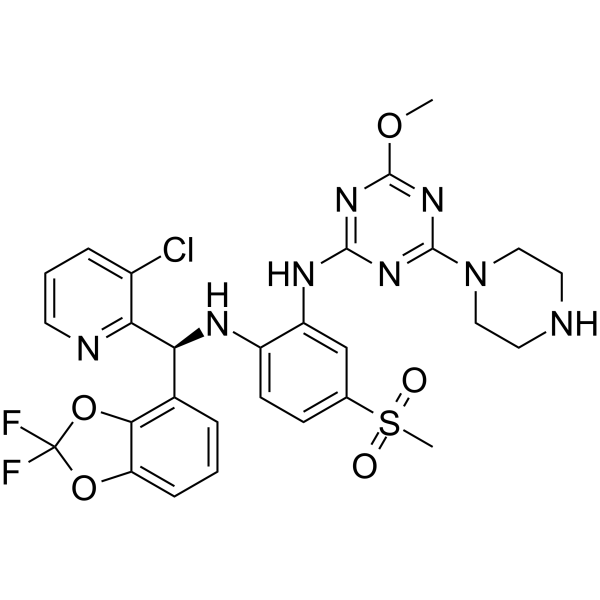
-
GC65962
Dot1L-IN-5
Dot1L-IN-5 es un potente disruptor del inhibidor de la proteÍna similar al silenciador telomérico 1 (DOT1L) con un IC50 SPA DOT1L de 0,17 nM.
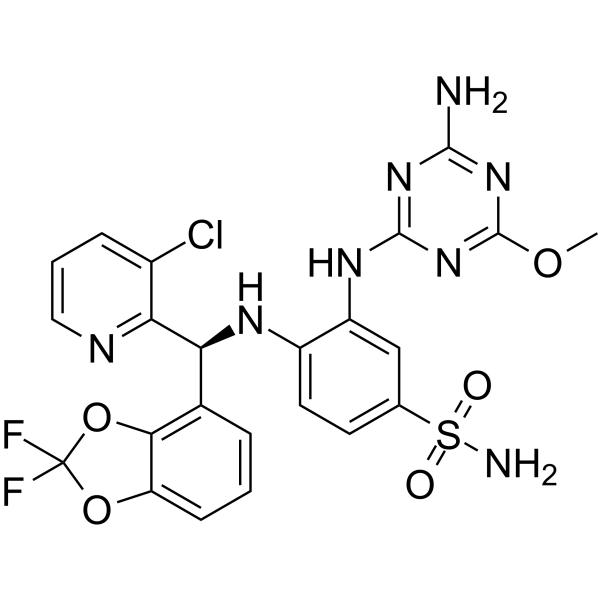
-
GC45927
DS-437
DS-437 es un inhibidor dual de PRMT5/7 (CI50 de PRMT5/7 = 6 μM).
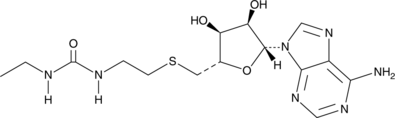
-
GC35914
DW14800
DW14800 es un inhibidor de la proteÍna arginina metiltransferasa 5 (PRMT5), con una IC50 de 17 nM. DW14800 reduce los niveles de H4R3me2s y mejora la transcripciÓn de HNF4α, pero no altera la expresiÓn de PRMT5. Actividad anticancerÍgena.
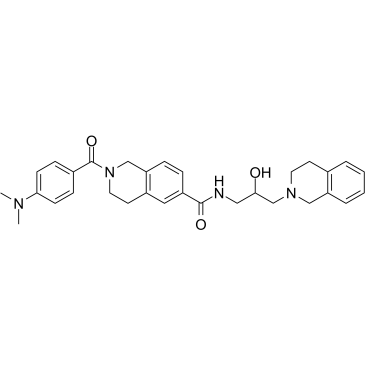
-
GC33220
EBI-2511
EBI-2511 es un inhibidor de EZH2 muy potente y activo por vÍa oral, con una IC50 de 6 nM en lÍneas celulares Pfeffiera, respectivamente.
-
GC19130
EED226
EED226 es un inhibidor del complejo represivo Polycomb 2 (PRC2), que se une al bolsillo K27me3 en el desarrollo del ectodermo embrionario (EED) y muestra una fuerte actividad antitumoral en el modelo de ratones con xenoinjerto. EED226 es un inhibidor de EED potente, selectivo y biodisponible por vÍa oral. EED226 inhibe PRC2 con una IC50 de 23,4 nM cuando se utiliza el péptido H3K27me0 como sustrato en los ensayos enzimÁticos in vitro.
-
GC67934
EEDi-5285
-
GC34567
EHMT2-IN-1
EHMT2-IN-1 es un potente inhibidor de EHMT, con IC50 de todos <100 nM para el péptido EHMT1, el péptido EHMT2 y el EHMT2 celular. Se utiliza en la investigaciÓn de trastornos sanguÍneos o cÁncer.
-
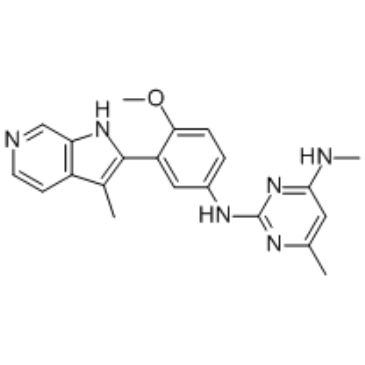
GC34568
EHMT2-IN-2
EHMT2-IN-2 es un potente inhibidor de EHMT, con IC50 de todos <100 nM para el péptido EHMT1, el péptido EHMT2 y el EHMT2 celular. Se utiliza en la investigaciÓn de enfermedades de la sangre o cÁncer.
-
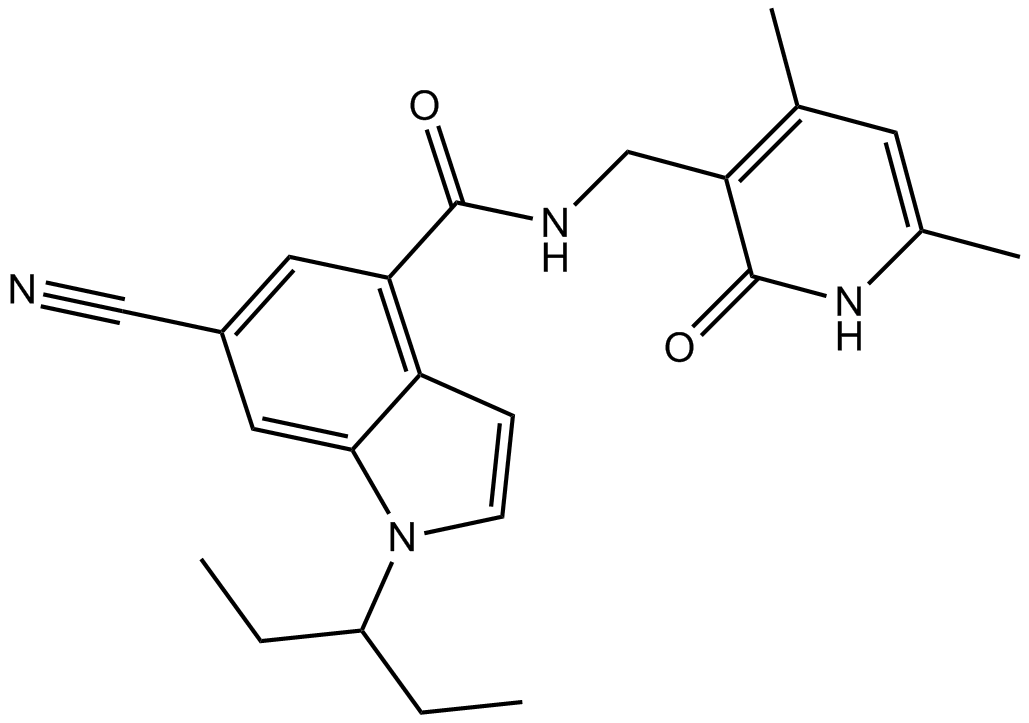
GC14756
EI1
EI1 (KB-145943) es un inhibidor de EZH2 potente y selectivo con una IC50 de 15 nM y 13 nM para EZH2 (WT) y EZH2 (Y641F), respectivamente.
-
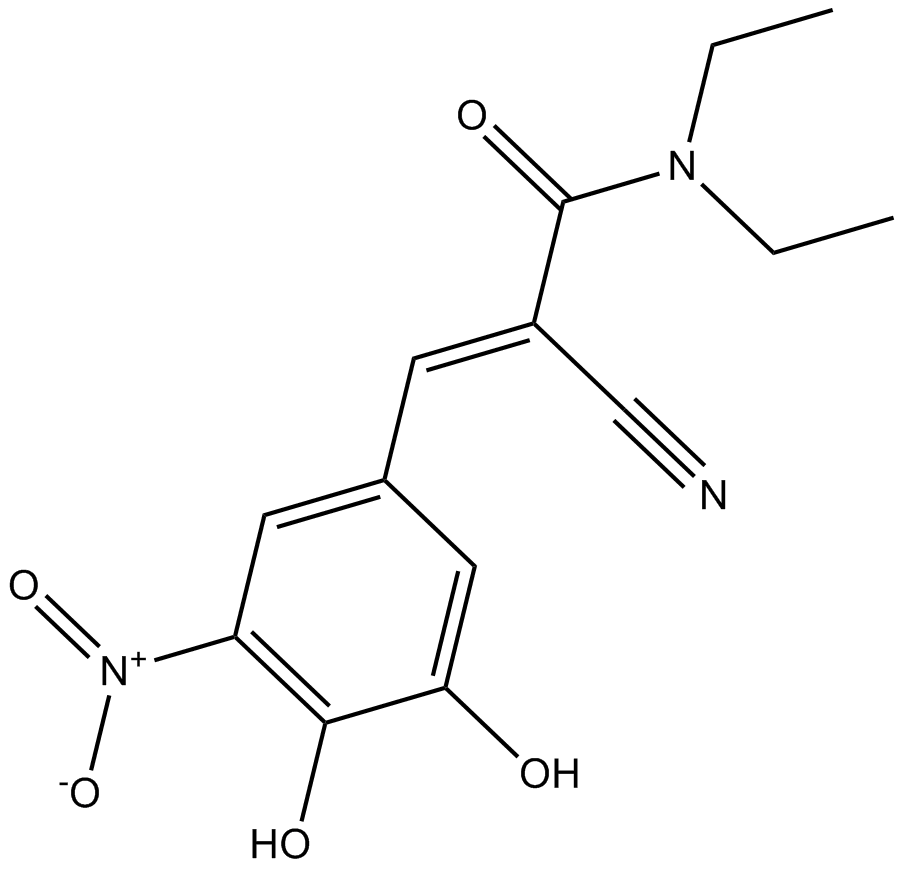
GC17334
Entacapone
A reversible COMT inhibitor
-
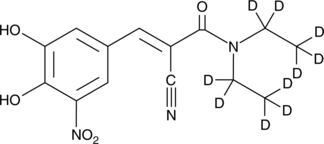
GC47294
Entacapone-d10
Entacapona-d10 es el deuterio etiquetado como entacapona.
-
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
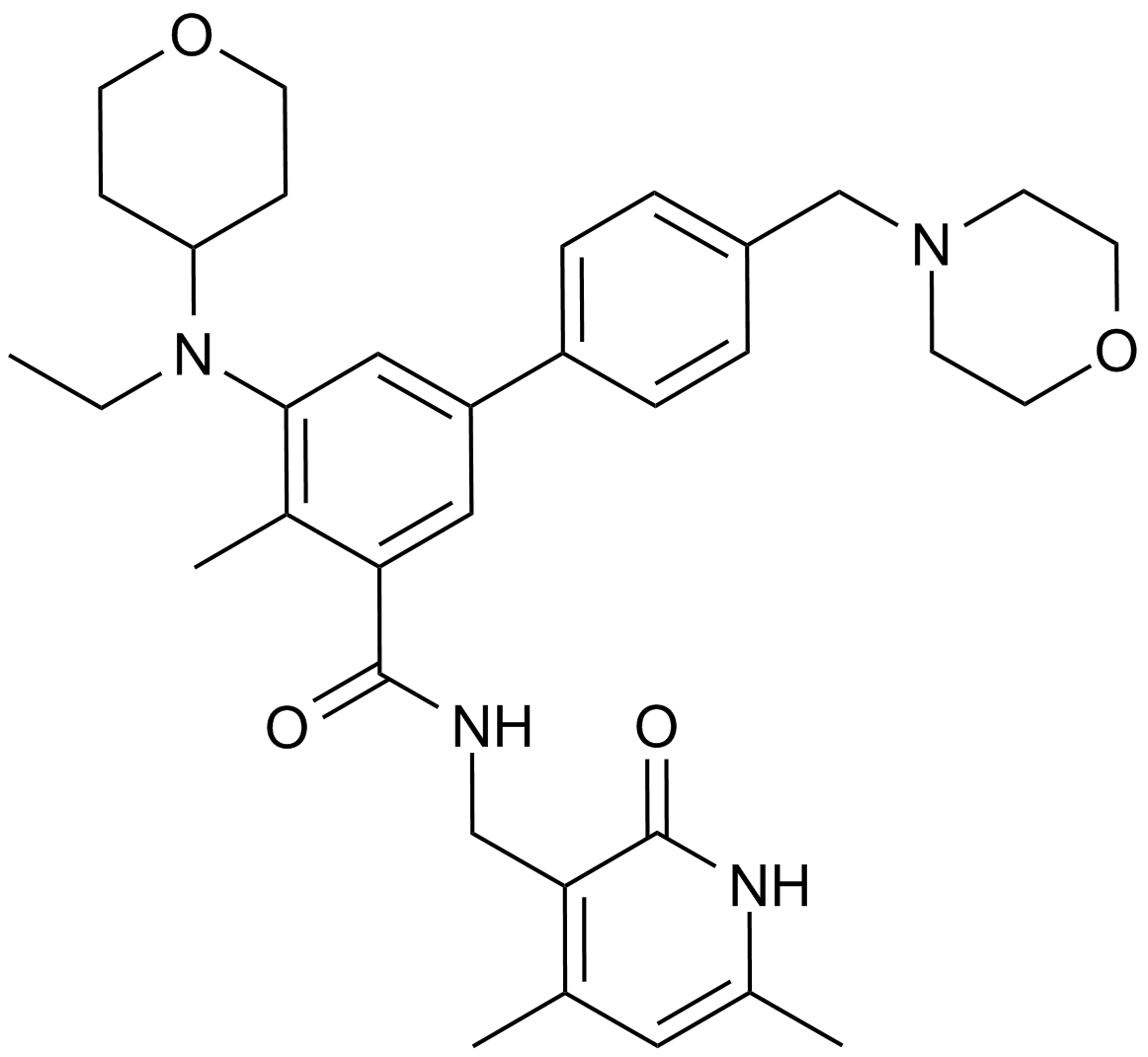
-
GC64343
EPZ-719
EPZ-719 es un nuevo y potente inhibidor de SETD2 (CI50 = 0,005 μM) con una alta selectividad sobre otras histonas metiltransferasas.
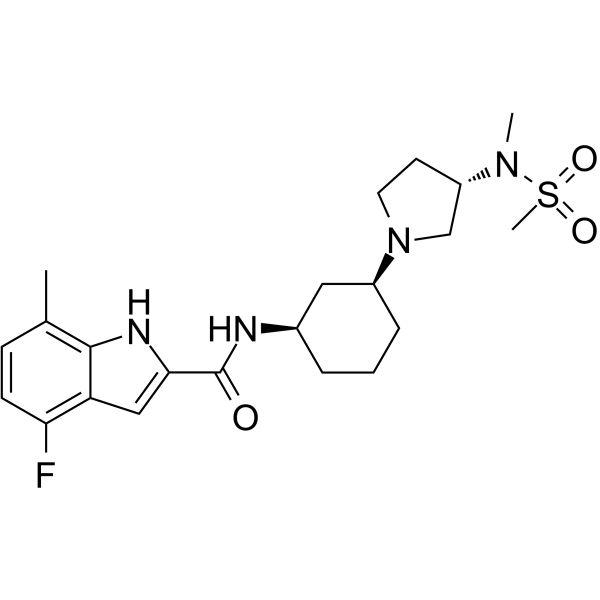
-
GC13383
EPZ004777
A potent inhibitor of DOT1L
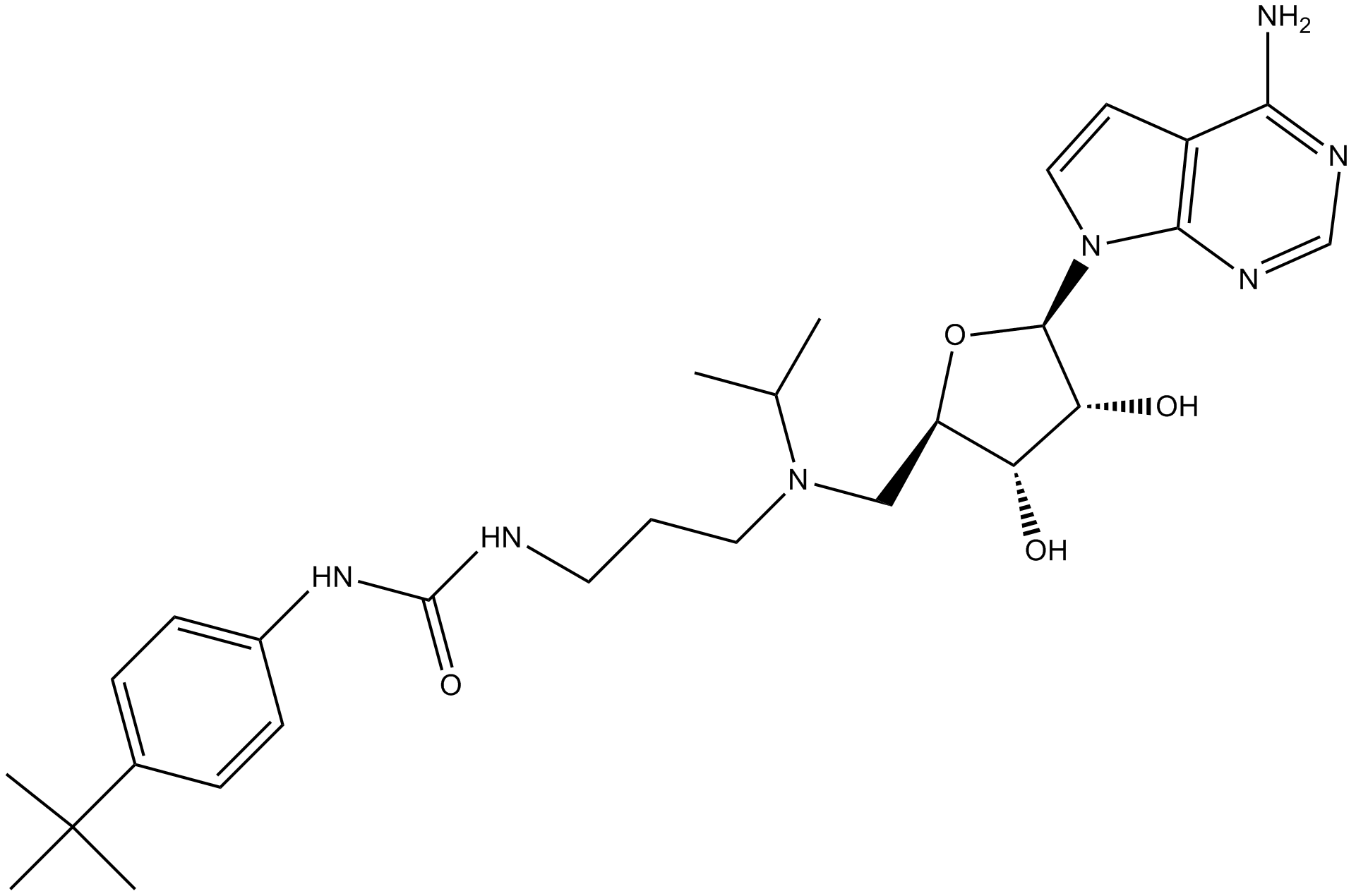
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
GC15259
EPZ004777 HCl
EPZ004777 HCl es un potente inhibidor DOT1L selectivo con una IC50 de 0,4 nM.
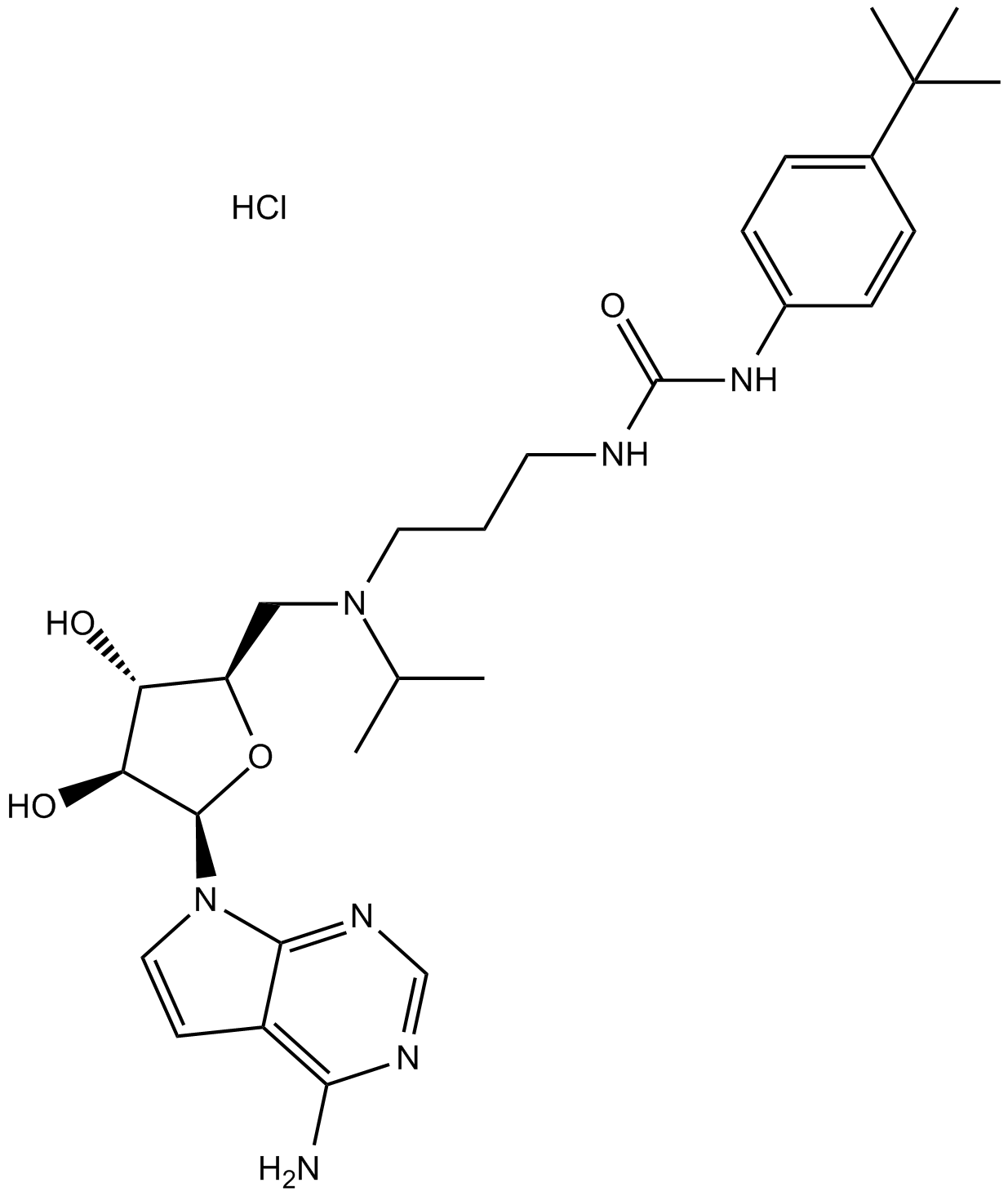
-
GC13878
EPZ005687
A potent, selective inhibitor of EZH2
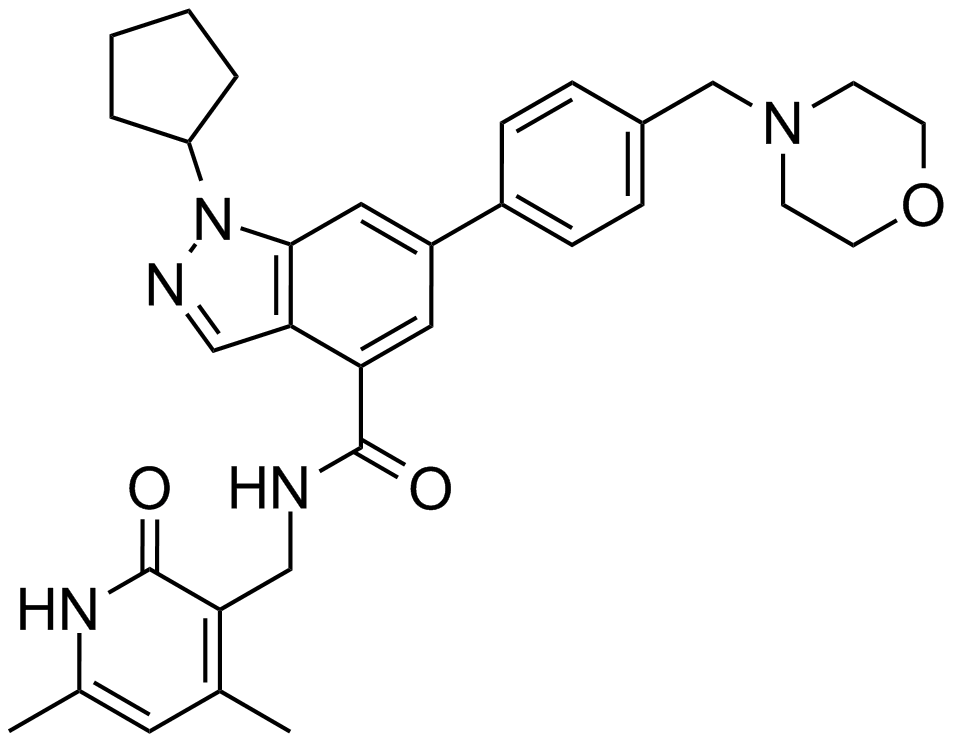
-
GC19141
EPZ011989
EPZ011989 es un inhibidor potente y oralmente activo de Zeste Homolog 2 (EZH2) con estabilidad metabÓlica. EPZ011989 tiene una inhibiciÓn inhibitoria para EZH2 con un valor Ki de <3 nM. EPZ011989 muestra una fuerte inhibiciÓn de la marca de metilo y actividad antitumoral. EPZ011989 se puede utilizar para la investigaciÓn de varios tipos de cÁncer.
-
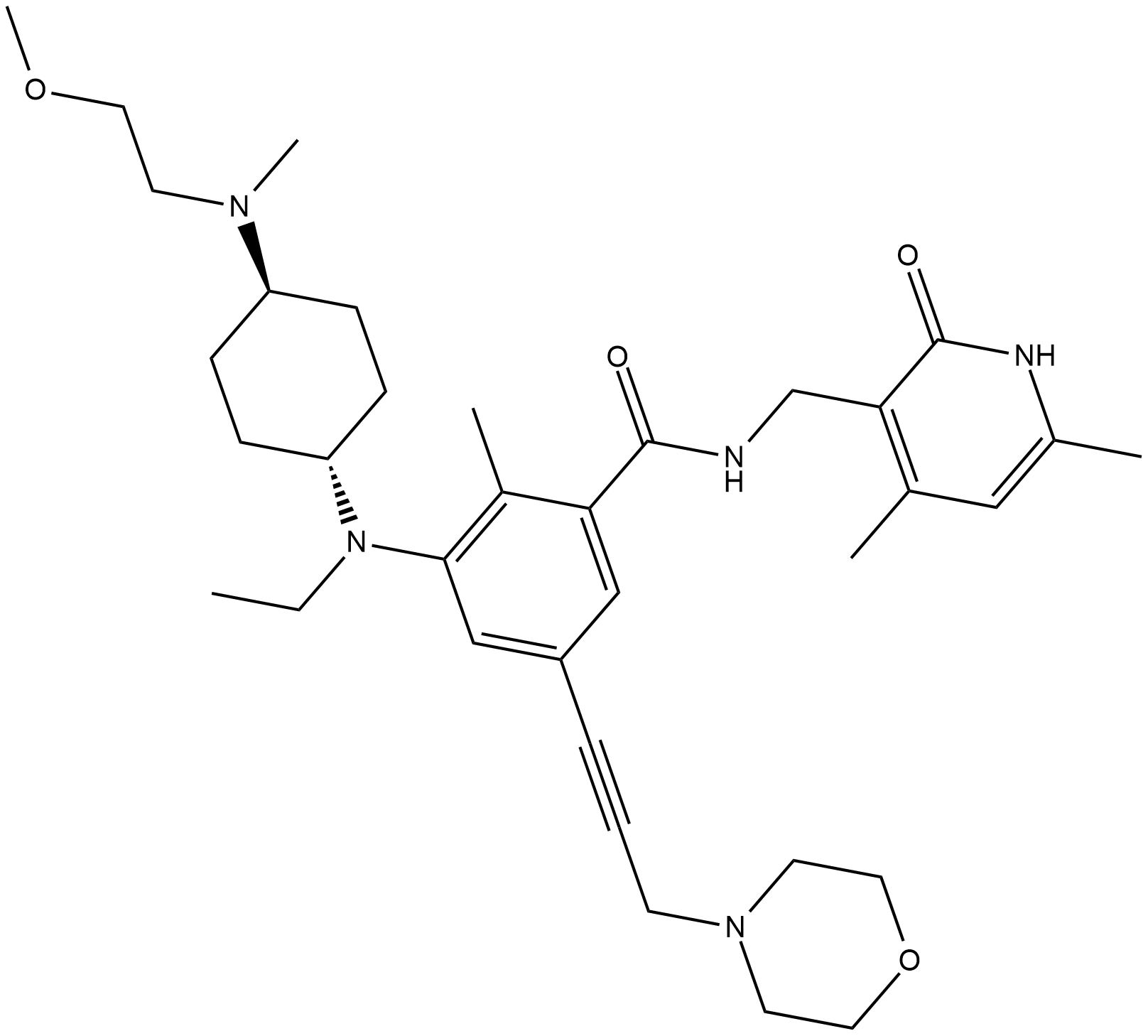
GC34136
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
El trifluoroacetato EPZ-011989 es un inhibidor potente y oralmente activo de Zeste Homolog 2 (EZH2) con estabilidad metabÓlica. El trifluoroacetato EPZ-011989 tiene una inhibiciÓn inhibitoria para EZH2 con un valor Ki de <3 nM. El trifluoroacetato EPZ-011989 muestra una fuerte inhibiciÓn de la marca de metilo y actividad antitumoral. El trifluoroacetato EPZ-011989 se puede utilizar para la investigaciÓn de varios tipos de cÁncer.
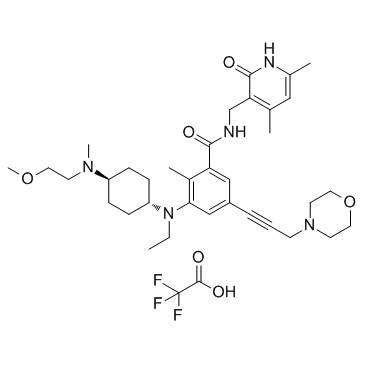
-
GC15302
EPZ015666
EPZ015666 (GSK3235025) es un inhibidor de PRMT5 disponible por vÍa oral con una IC50 de 22 nM.
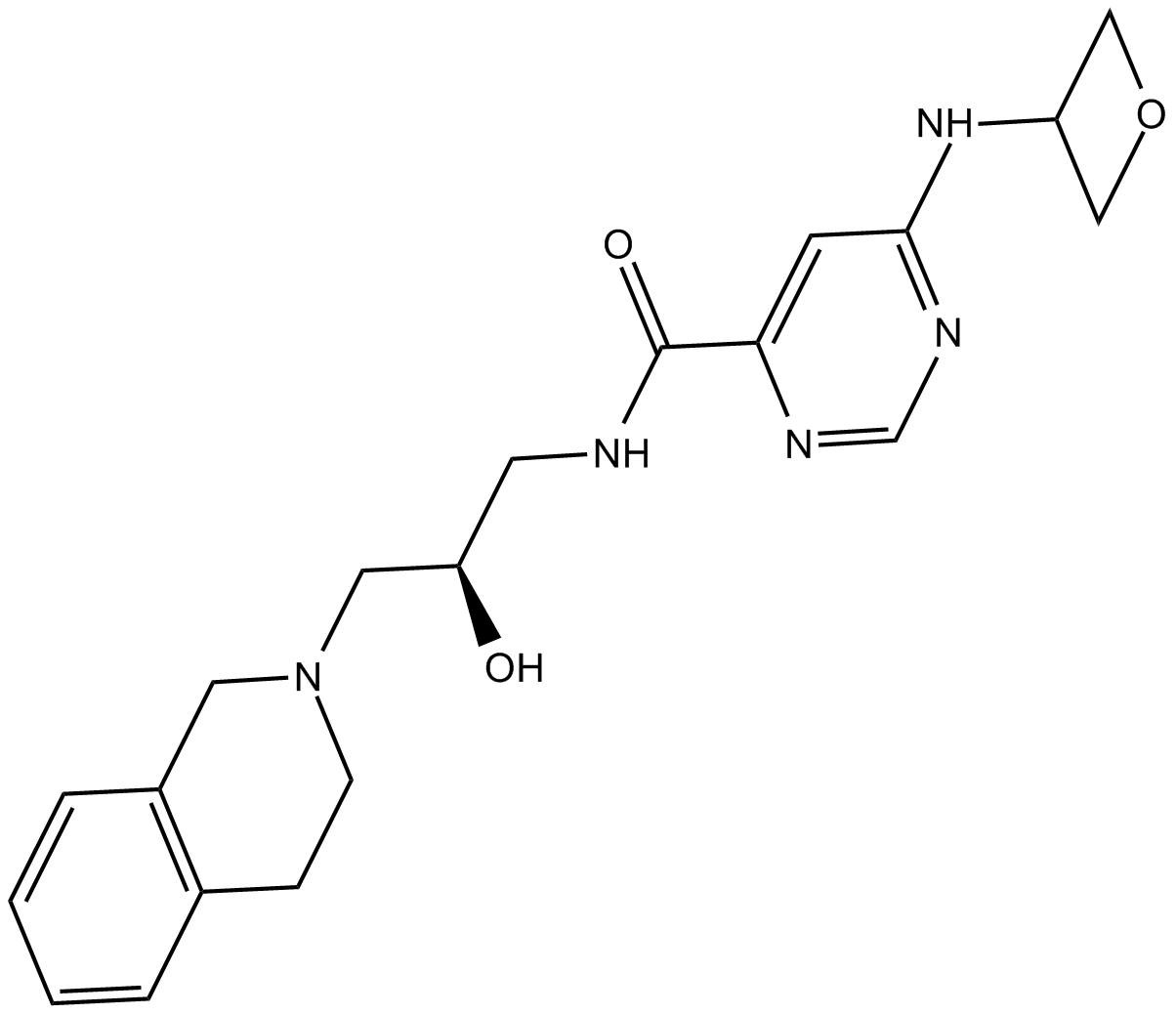
-
GC15102
EPZ020411
EPZ020411 es un inhibidor selectivo de PRMT6 con una IC50 de 10 nM, tiene una selectividad de >10 veces para PRMT6 sobre PRMT1 y PRMT8. EPZ020411 se puede utilizar para la investigaciÓn del cÁncer.
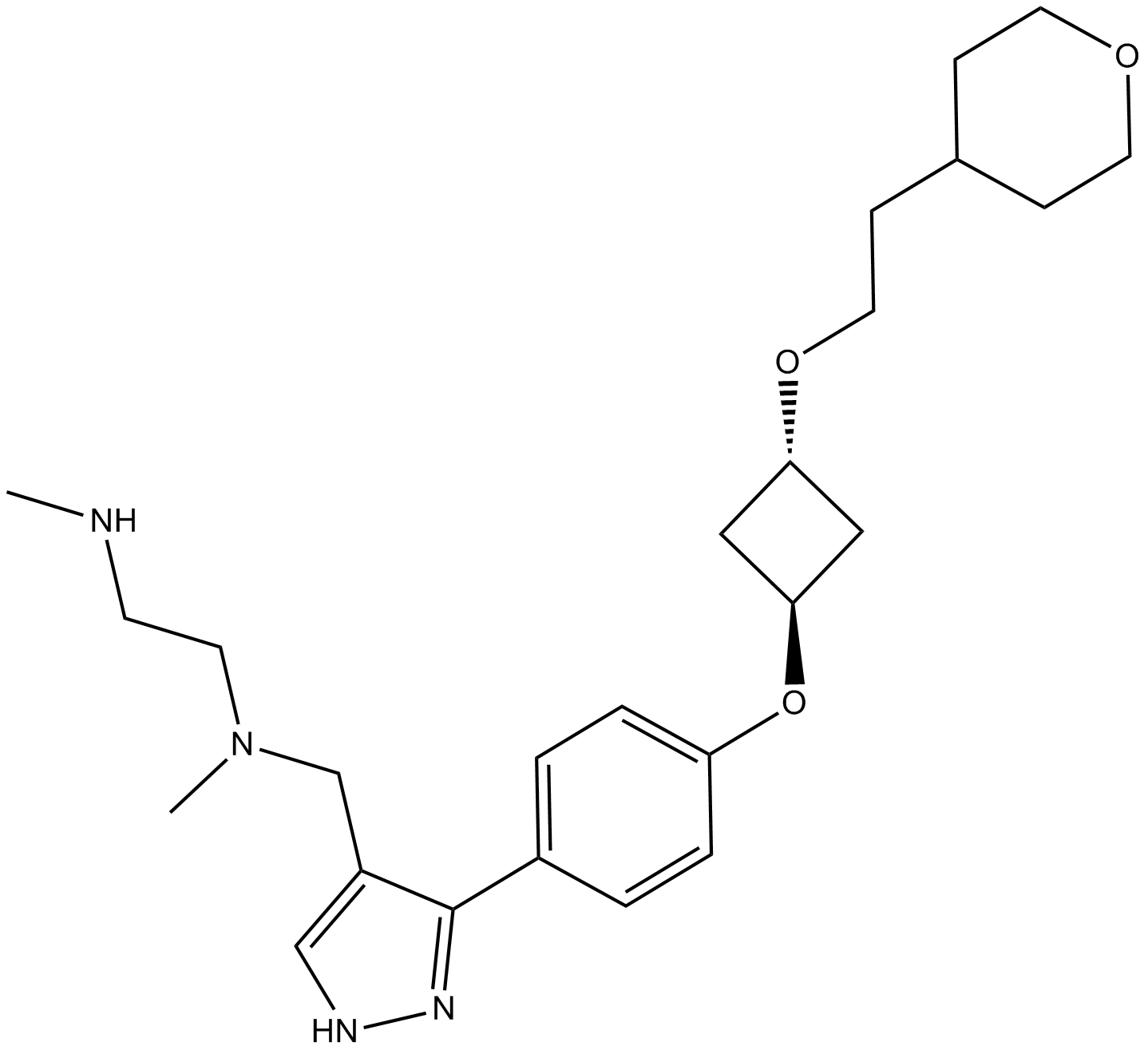
-
GC36000
EPZ020411 hydrochloride
El clorhidrato de EPZ020411 es un inhibidor selectivo de PRMT6 con una IC50 de 10 nM, tiene una selectividad de >10 veces para PRMT6 sobre PRMT1 y PRMT8. El clorhidrato de EPZ020411 se puede utilizar para la investigaciÓn del cÁncer.
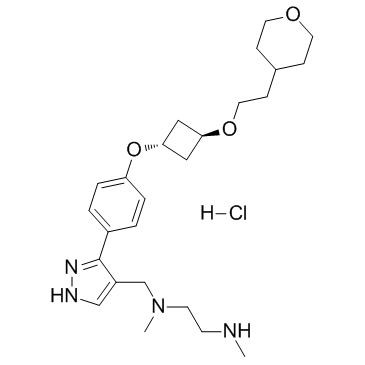
-
GC16224
EPZ031686
EPZ031686 es un inhibidor de SMYD3 potente y activo por vÍa oral y con un valor IC50 de 3 nM. EPZ031686 se puede utilizar para la investigaciÓn del cÁncer.
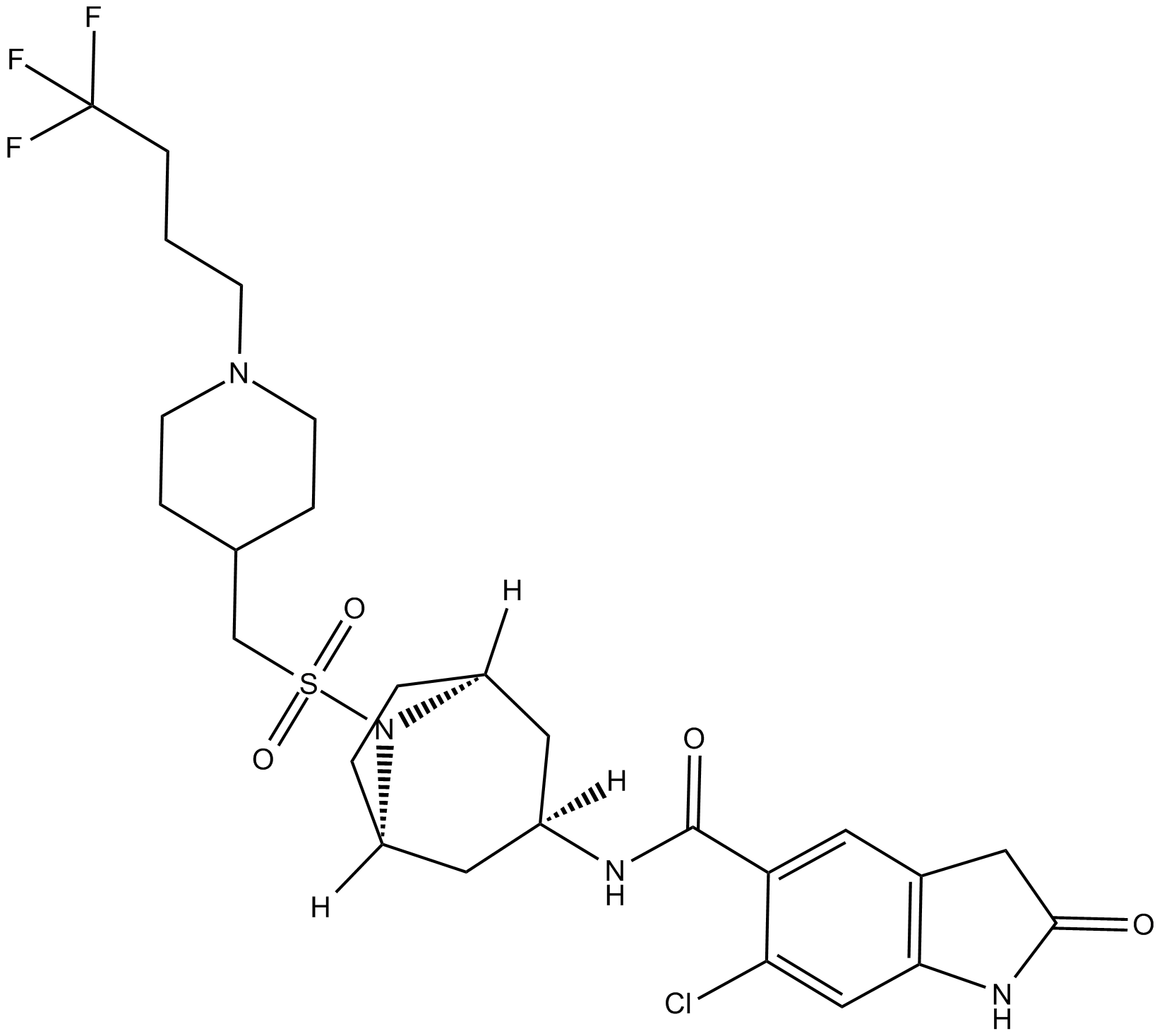
-
GC12932
EPZ5676
A highly potent DOT1L inhibitor
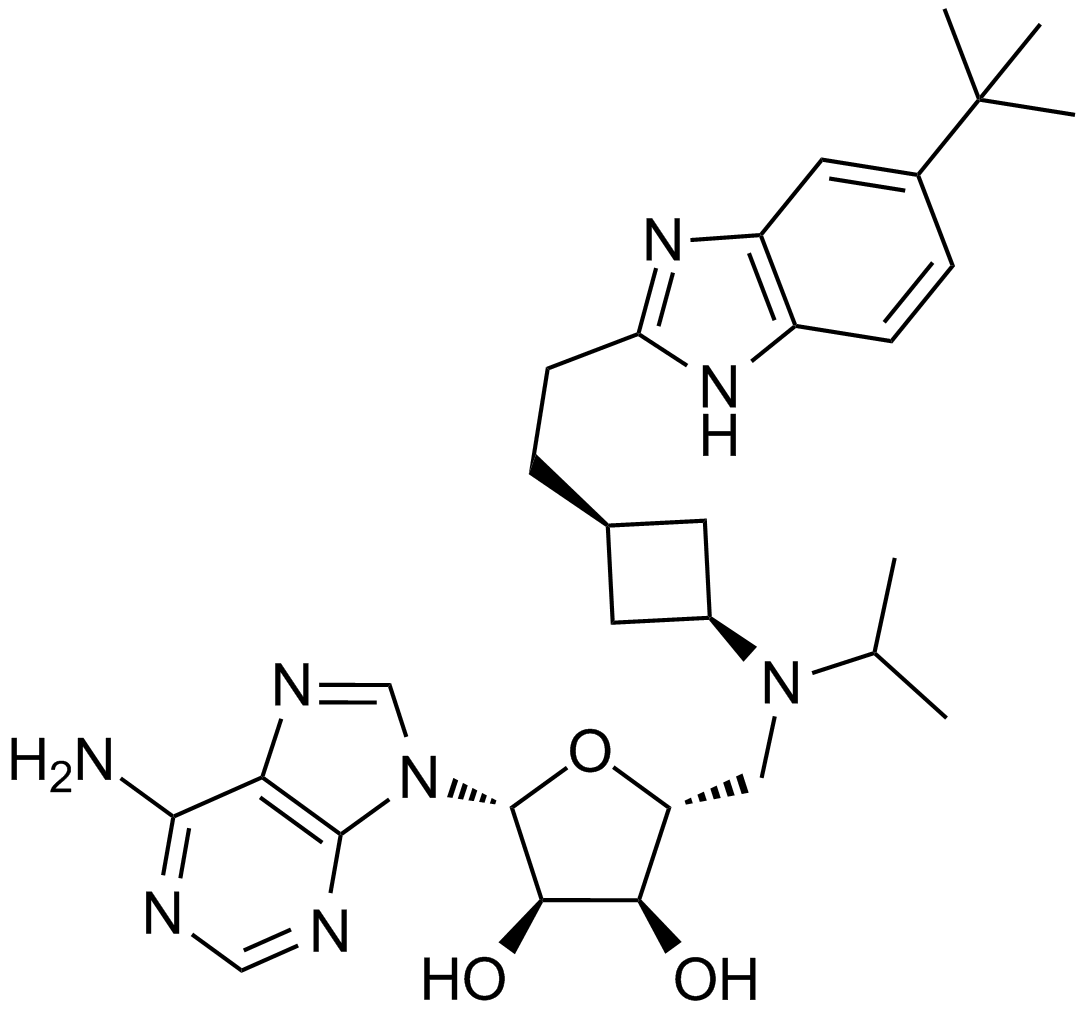
-
GC65980
EZH2-IN-13
EZH2-IN-13 es un potente inhibidor de EZH2; para obtener mÁs informaciÓn, consulte el compuesto 73 en la patente WO2017139404. EZH2-IN-13 se puede utilizar para estudiar cÁnceres o lesiones precancerosas asociadas con la actividad de EZH2.
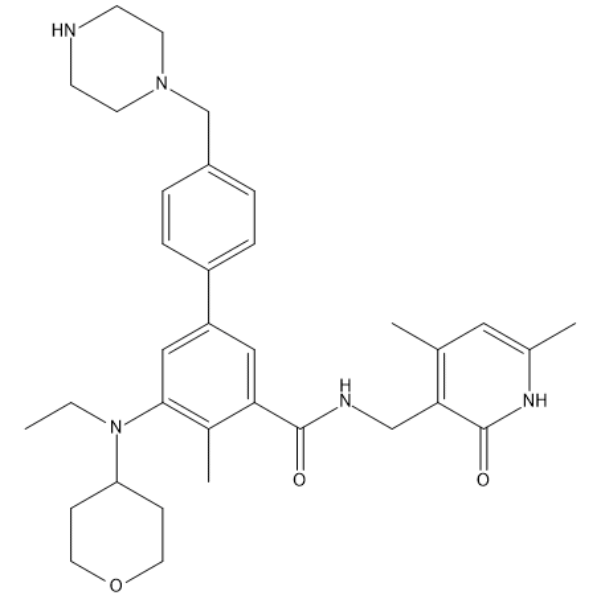
-
GC19149
EZM 2302
EZM 2302 es un inhibidor de la arginina metiltransferasa 1 asociada al coactivador (CARM1) con una IC50 de 6nM.
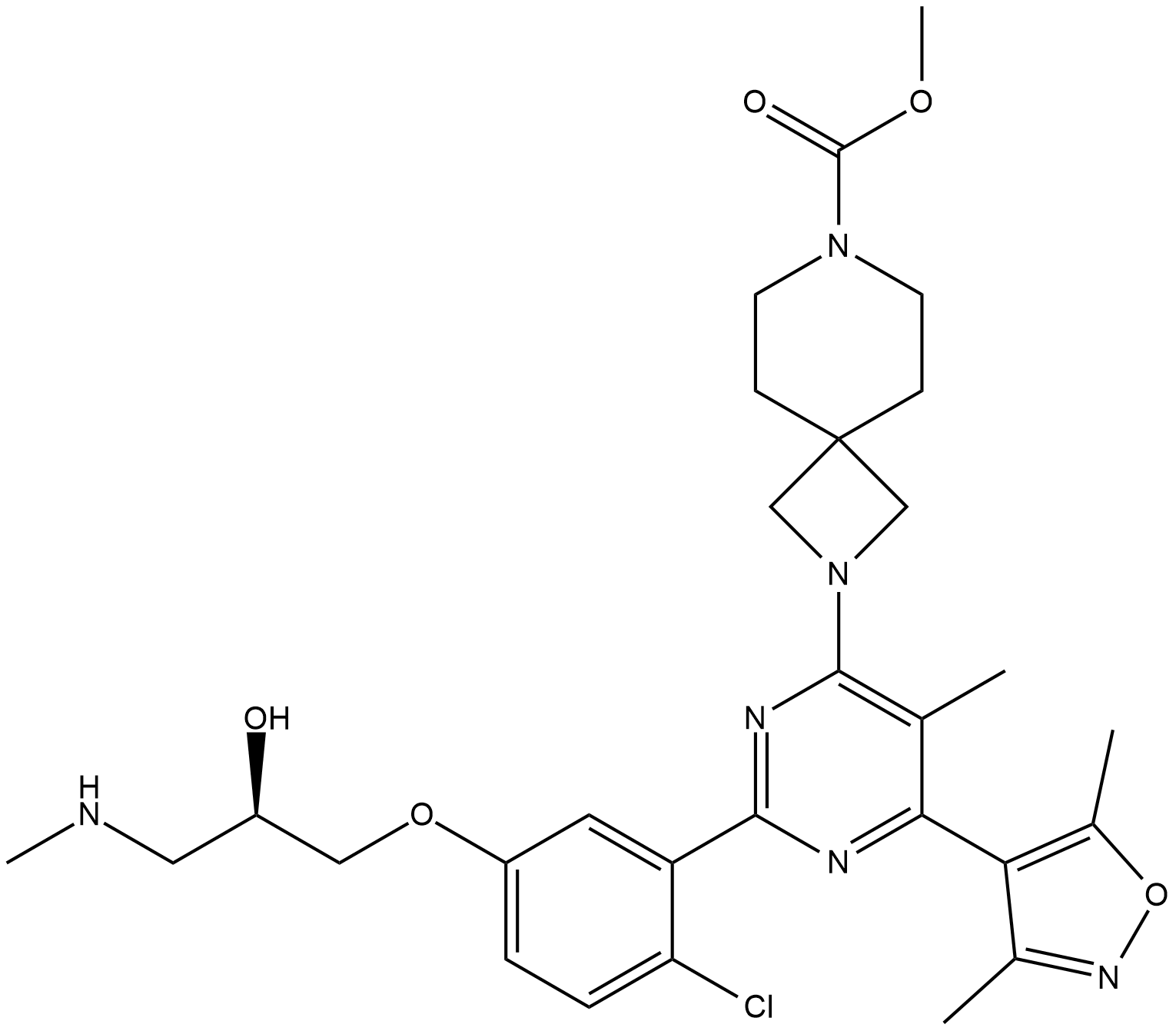
-
GC64900
EZM0414
EZM0414 es un inhibidor potente, selectivo y biodisponible por vÍa oral de SETD2 (IC50 = 18 nM en el ensayo bioquÍmico de SETD2; IC50 = 34 nM en el ensayo celular). EZM0414 se puede utilizar para la investigaciÓn del mieloma mÚltiple en recaÍda o refractario y el linfoma difuso de células B grandes.
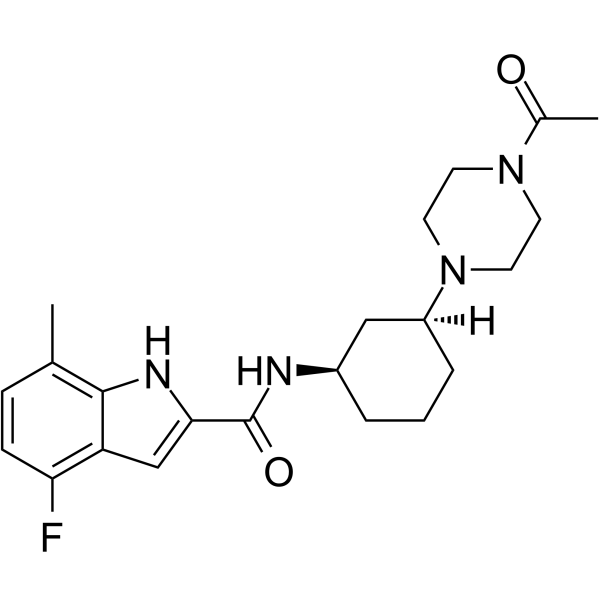
-
GC63545
FTX-6058
FTX-6058 ((S)-FTX-6058) es un inhibidor potente y activo por vÍa oral del desarrollo del ectodermo embrionario (EED).
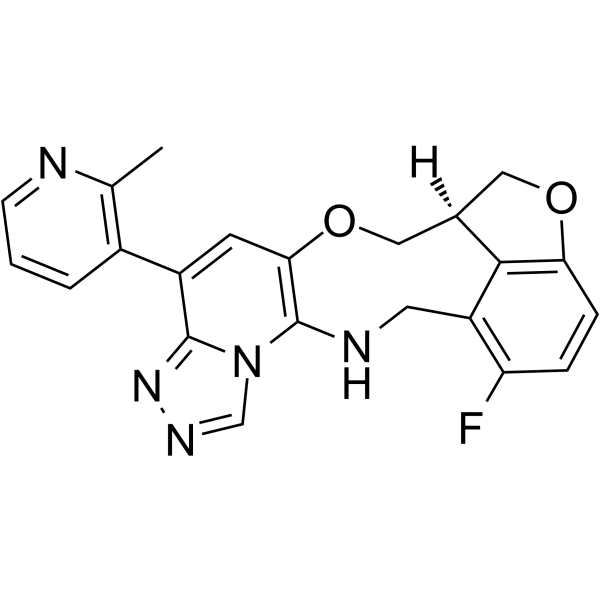
-
GC63546
FTX-6058 hydrochloride
El clorhidrato de (S)-pociredir ((S)-FTX-6058) es un inhibidor potente y oralmente activo del desarrollo del ectodermo embrionario (EED).
-
GC43715
Furamidine (hydrochloride)
La furamidina (clorhidrato) (DB75 diclorhidrato) es un inhibidor selectivo de la proteÍna arginina metiltransferasa 1 (PRMT1) con una IC50 de 9,4 μM.
-
GC38062
Gambogenic acid
El Ácido gambigénico es un ingrediente activo del gamboge, con actividad anticancerÍgena. El Ácido gambigénico actÚa como un inhibidor eficaz de EZH2, se une de manera especÍfica y covalente a Cys668 dentro del dominio EZH2-SET e induce la ubiquitinaciÓn de EZH2.
-
GC36168
GNA002
GNA002 es un inhibidor de EZH2 (potenciador del homÓlogo zeste 2) muy potente, especÍfico y covalente con una IC50 de 1,1 μM. GNA002 puede unirse de manera especÍfica y covalente a Cys668 dentro del dominio EZH2-SET, lo que desencadena la degradaciÓn de EZH2 a través del término COOH de la ubiquitinaciÓn mediada por la proteÍna que interactÚa con Hsp70 (CHIP). GNA002 reduce de manera eficiente la trimetilaciÓn de H3K27 mediada por EZH2, reactiva los genes supresores de tumores silenciados por el complejo represor polycomb 2 (PRC2).
-
GC50697
GSK 591 dihydrochloride
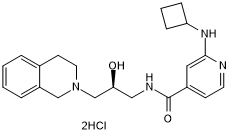
-
GC15783
GSK126
Un inhibidor selectivo de EZH2
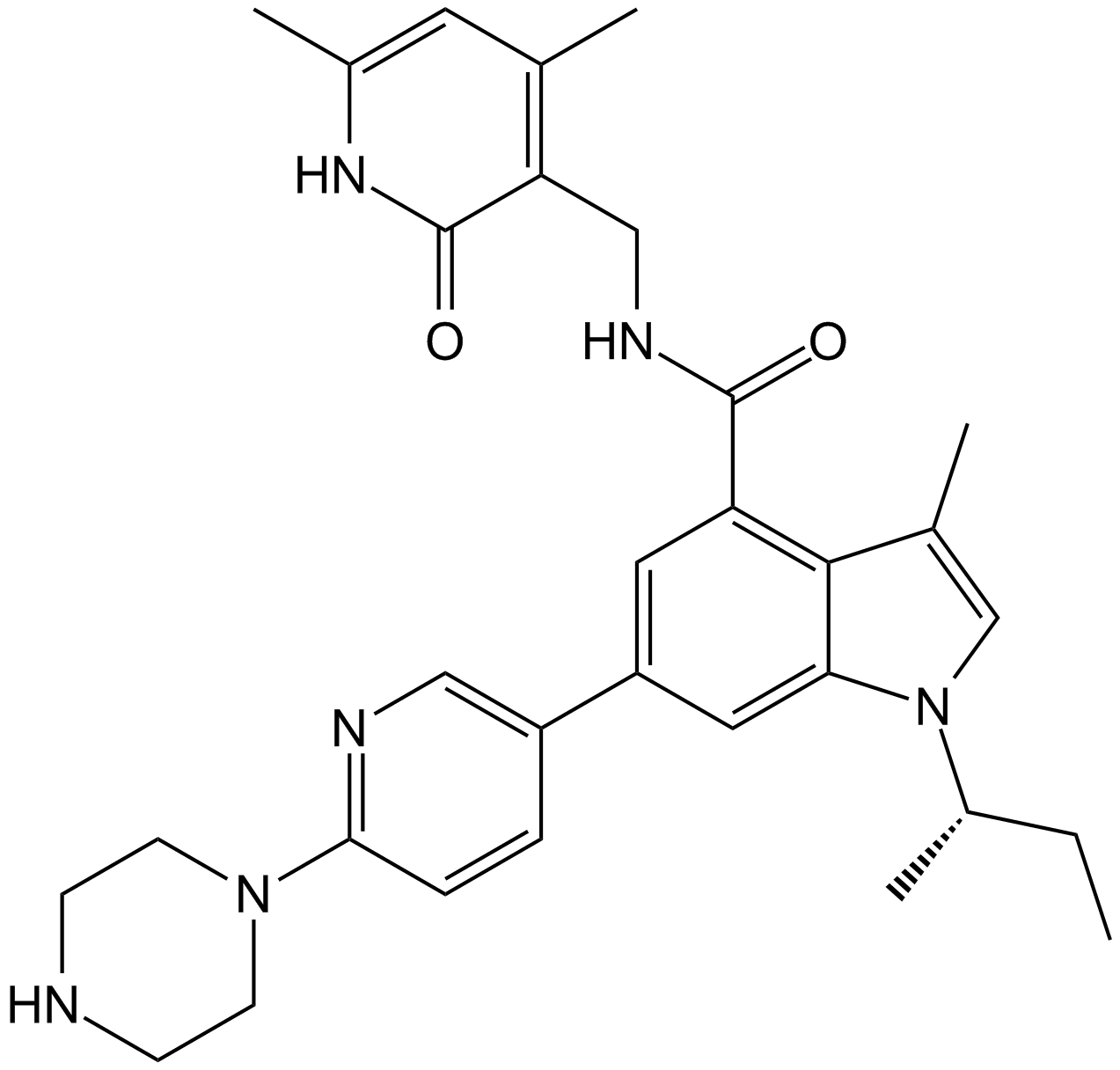
-
GC19181
GSK2807 Trifluoroacetate
El trifluoroacetato GSK2807 es un inhibidor potente, selectivo y competitivo de SAM de SMYD3, con una Ki de 14 nM y una IC50 de 130 nM.
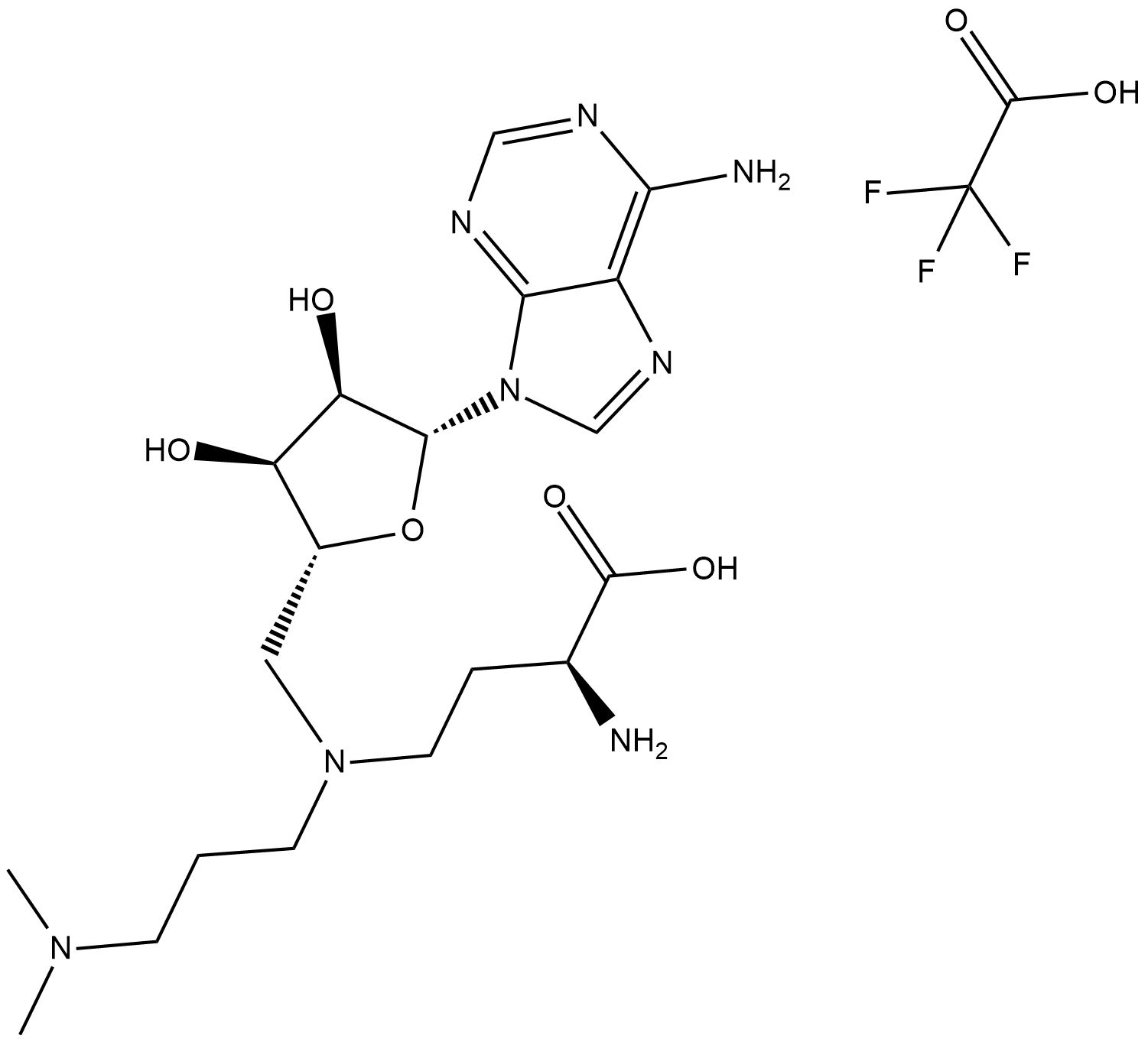
-
GC32693
GSK3326595 (EPZ015938)
GSK3326595 (EPZ015938) (EPZ015938) es un inhibidor reversible, selectivo y potente de la proteÍna arginina metiltransferasa 5 (PRMT5) con una IC50 de 6,2 nM.
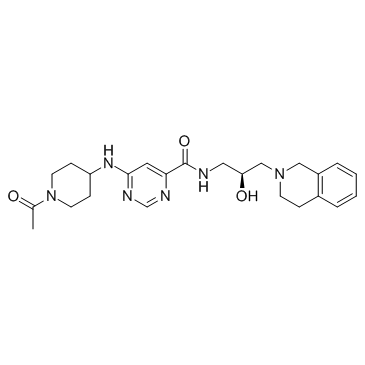
-
GC36191
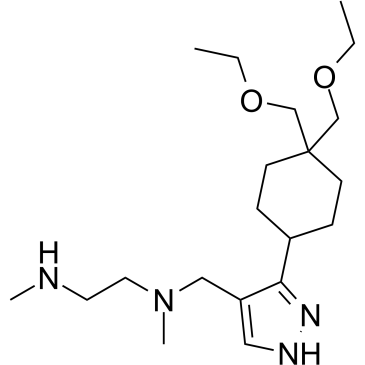
GSK3368715
GSK3368715 (EPZ019997) es un inhibidor de la proteÍna arginina metiltransferasas (PRMT) tipo I activo por vÍa oral, reversible y S-adenosil-L-metionina (SAM) no competitivo (IC50 = 3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM ( PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715 (EPZ019997) produce un cambio en los estados de metilaciÓn de la arginina, altera el uso de exones y tiene una fuerte actividad anticancerÍgena.
-
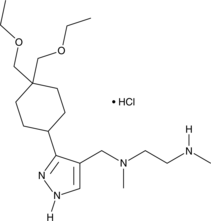
GC49398
GSK3368715 (hydrochloride)
An inhibitor of type I PRMTs
-
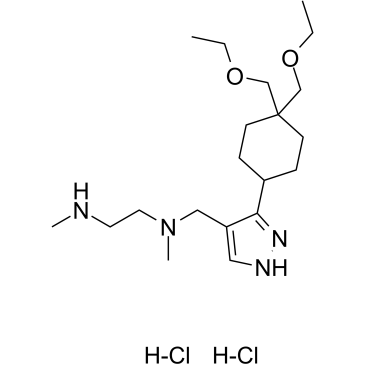
GC36192
GSK3368715 dihydrochloride
El diclorhidrato de GSK3368715 (diclorhidrato de EPZ019997) es un inhibidor de la proteÍna arginina metiltransferasas (PRMT) tipo I activo por vÍa oral, reversible y S-adenosil-L-metionina (SAM) no competitivo (IC50 = 3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM (PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). El diclorhidrato de GSK3368715 (diclorhidrato de EPZ019997) produce un cambio en los estados de metilaciÓn de la arginina, altera el uso del exÓn y tiene una fuerte actividad anticancerÍgena.
-
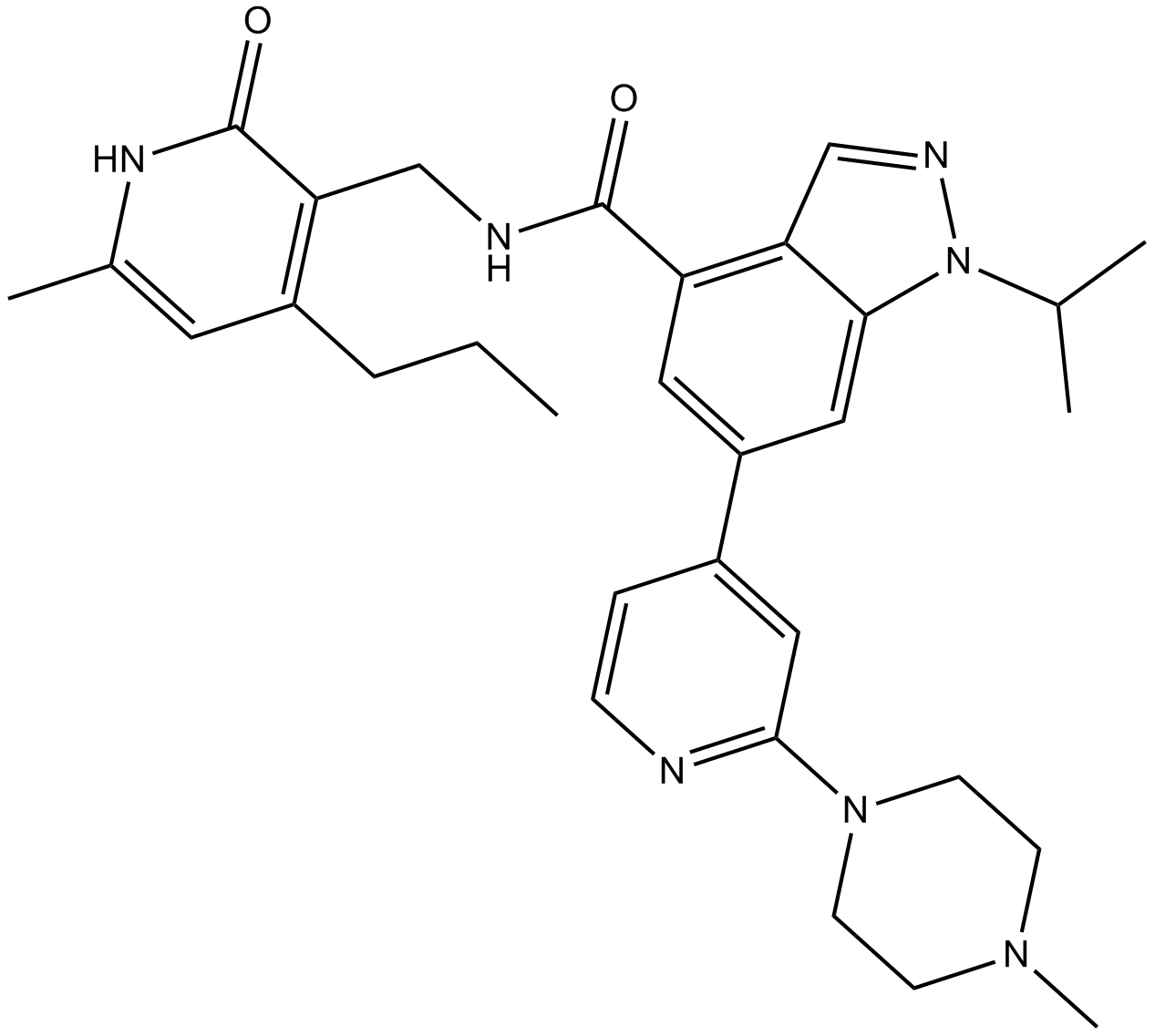
GC17534
GSK343
A selective, cell-permeable EZH2 inhibitor
-
GC11414
GSK503
GSK503 es un inhibidor potente y especÍfico de la metiltransferasa EZH2 con valores de Kiapp de 3 a 27 nM.