TGF-β / Smad Signaling
Transforming growth factor-beta (TGF-beta) is a multifunctional cytokine that regulates proliferation, migration, differentiation, and survival of many different cell types. Deletion or mutation of different members of the TGF-β family have been shown to cause vascular remodeling defect and absence of mural cell formation, leading to embryonic lethality or severe vascular disorders. TGF-β induces smooth muscle differentiation via Notch or SMAD2 and SMAD3 signaling in ES cells or in a neural crest stem cell line. TGF-β binds to TGF-βRI and to induce phosphorylation of SMAD2/3, thereby inhibiting proliferation, tube formation, and migration of endothelial cells (ECs).
TGF-β is a pluripotent cytokine with dual tumour-suppressive and tumour-promoting effects. TGF-β induces the epithelial-to-mesenchymal transition (EMT) leading to increased cell plasticity at the onset of cancer cell invasion and metastasis.
Targets for TGF-β / Smad Signaling
Products for TGF-β / Smad Signaling
- Cat.No. Nombre del producto Información
-
GC44729
Prostratin
An HIV reactivator
-
GC37014
Protein Kinase C 19-31
La proteína quinasa C 19-31, un inhibidor peptídico de la proteína quinasa C (PKC), derivado del dominio regulador de pseudosustrato de PKCa (residuos 19-31) con una serina en la posición 25 que reemplaza a la alanina de tipo salvaje, se usa como péptido sustrato de la proteína quinasa C para probar la actividad de la proteína quinasa C.

-
GC37015
Protein Kinase C 19-36
La proteína quinasa C 19-36 es un inhibidor peptídico de pseudosustrato de la proteína quinasa C (PKC), con una IC50 de 0,18 μM.

-
GC66006
Protein kinase inhibitor H-7
El inhibidor de la proteÍna quinasa H-7 es un potente inhibidor de la proteÍna quinasa C (PKC) y de la proteÍna quinasa dependiente de nucleÓtidos cÍclicos, con una Ki de 6 μM para PKC.
-
GC11803
Pseudo RACK1
Activator of protein kinase C
-
GC69776
PT-262
PT-262 es un inhibidor efectivo de ROCK con un valor IC50 de aproximadamente 5 μM. PT-262 induce la pérdida del potencial de membrana mitocondrial y aumenta la activación de caspasa-3 y la apoptosis celular. PT-262 inhibe la fosforilación de ERK y CDC2 a través de una vía independiente de p53. PT-262 bloquea la función del citoesqueleto celular y la migración celular. PT-262 tiene actividad anticancerígena.
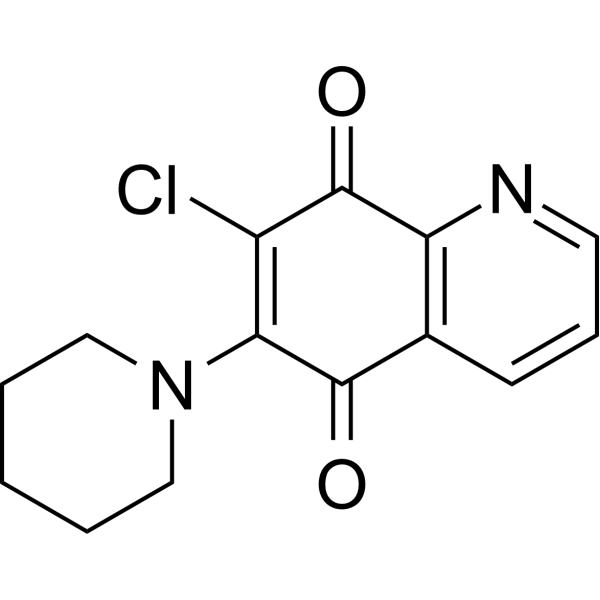
-
GC14583
R 59-022
R 59-022 (DKGI-I) es un inhibidor de diacilglicerol quinasa (IC50 = 2,8 μM).
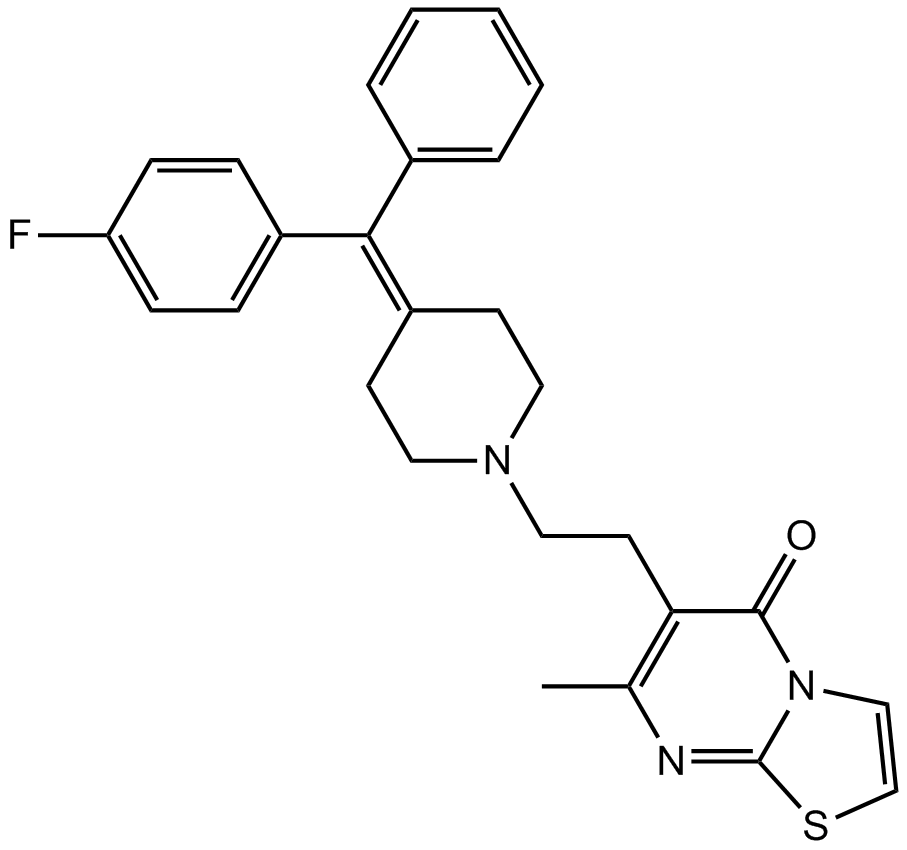
-
GC69792
R 59-022 hydrochloride
R 59-022 (DKGI-I) hidrocloruro es un inhibidor de DGK (IC50: 2.8 µM). R 59-022 hidrocloruro inhibe la fosforilación de OAG a OAPA. R 59-022 hidrocloruro es un antagonista del receptor 5-HT y puede activar la proteína quinasa C (PKC). R 59-022 aumenta la producción de diacilglicerol inducida por trombina en plaquetas e inhibe la producción de ácido fosfatídico en neutrófilos.
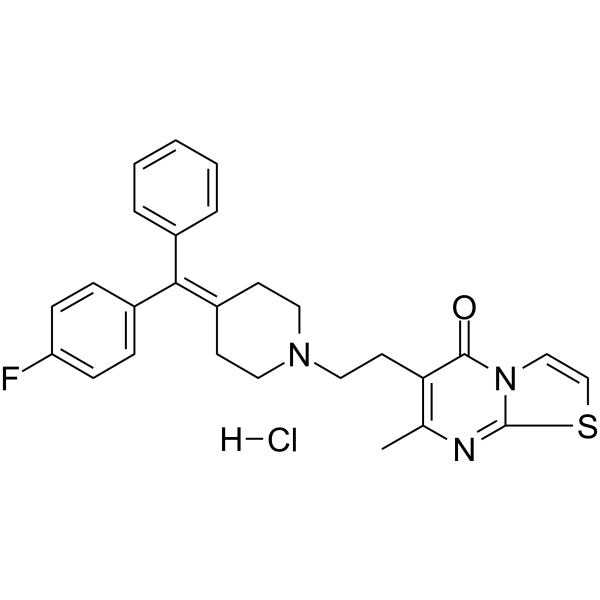
-
GC32840
R-268712
R-268712 es un inhibidor de ALK-5 selectivo y activo por vÍa oral, con una IC50 de 2,5 nM.
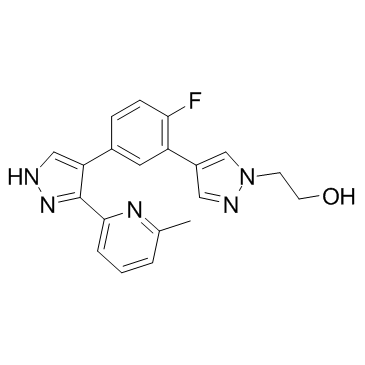
-
GC18246
R-59-949
R-59-949 es un inhibidor de pan diacilglicerol quinasa (DGK) con una IC50 de 300 nM. El R-59-949 inhibe fuertemente la actividad de las DGK α y γ de tipo I y atenúa moderadamente la actividad de las DGK θ y κ de tipo II. El R-59-949 activa la proteína quinasa C (PKC) al aumentar los niveles del ligando endógeno diacilglicerol.
-
GC11140
Radotinib(IY-5511)
Bcr-Abl tyrosine kinase inhibitor
-
GC16793
RepSox
Un inhibidor selectivo de ALK5.
-
GC33310
Rho-Kinase-IN-1
Rho-Kinase-IN-1 es un inhibidor de la Rho quinasa (ROCK) (valores de Ki de 30,5 y 3,9 nM para ROCK1 y ROCK2, respectivamente) extraÍdo del documento US20090325960A1, compuesto 1.008.
-
GC66050
Rho-Kinase-IN-2
Rho-Kinase-IN-2 (Compuesto 23) es un inhibidor de Rho Kinase (ROCK) activo por vÍa oral, selectivo y penetrante en el sistema nervioso central (SNC) (ROCK2 IC50 = 3 nM). Rho-Kinase-IN-2 se puede utilizar en la investigaciÓn de Huntington'.
-
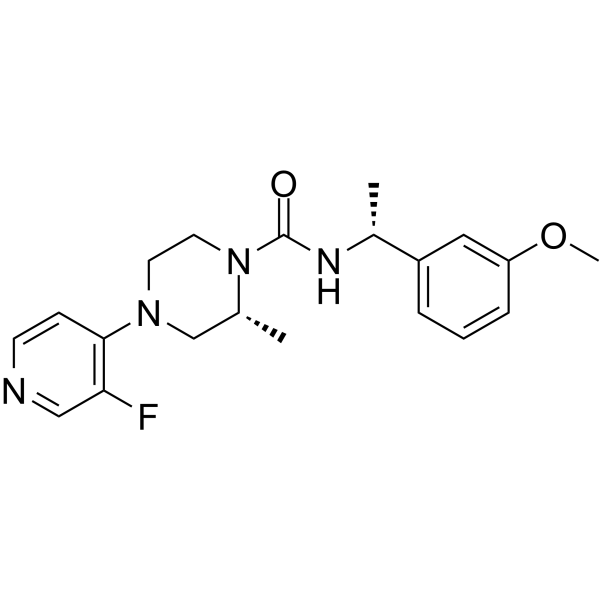
GC37534
Ripasudil free base
La base libre de ripasudil (base libre K-115) es un inhibidor especÍfico de ROCK, con IC50 de 19 y 51 nM para ROCK2 y ROCK1, respectivamente.
-
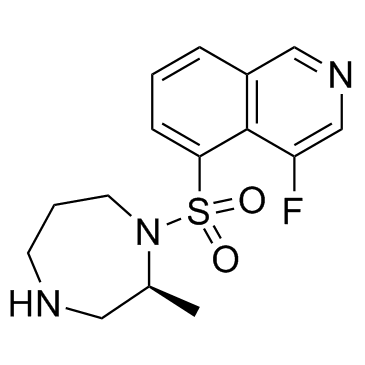
GC17322
Ro 31-8220
Ro 31-8220 es un potente inhibidor de PKC, con IC50 de 5, 24, 14, 27, 24 y 23 nM para PKCα, PKCβI, PKCβII, PKCγ, PKCε y PKC de cerebro de rata, respectivamente.
-
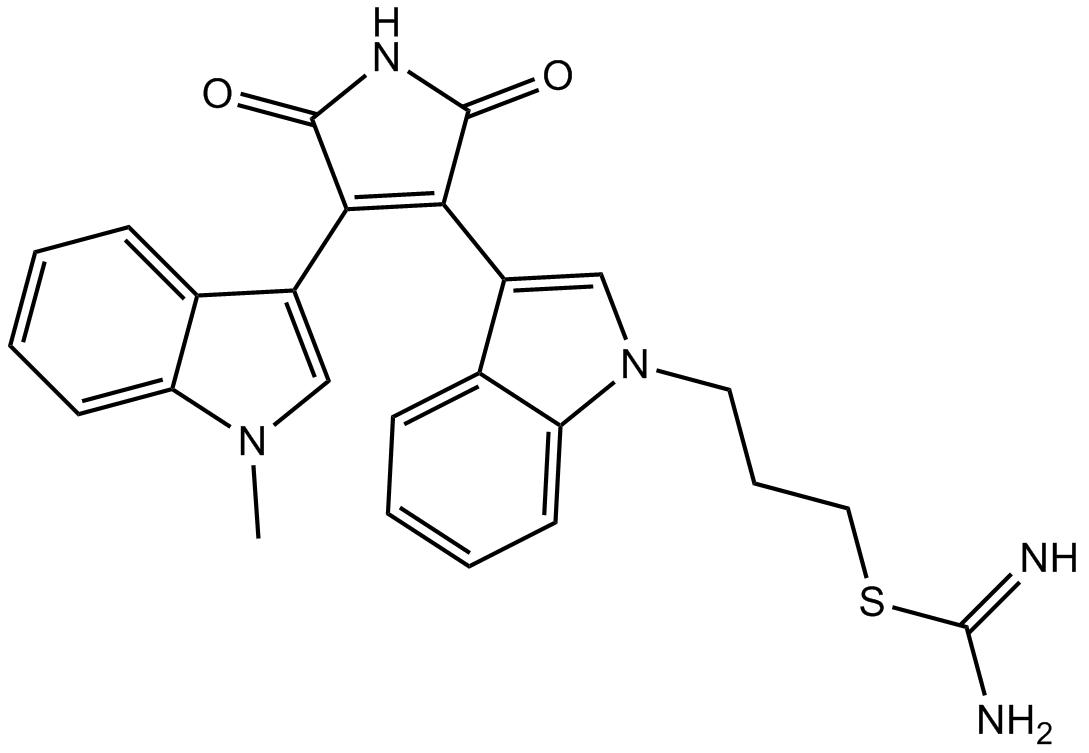
GC17014
Ro 31-8220 Mesylate
Pan-PKC inhibitor
-
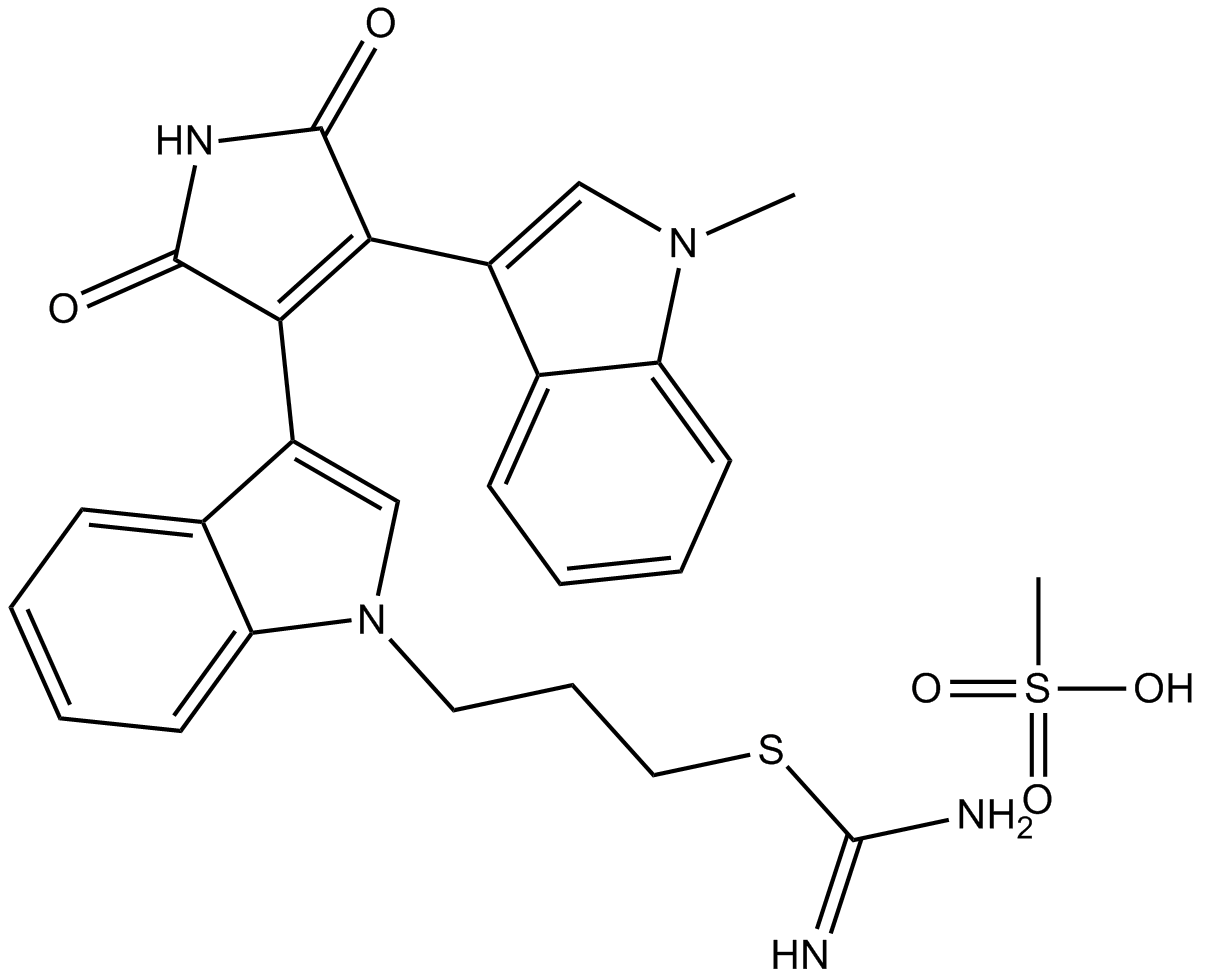
GC10029
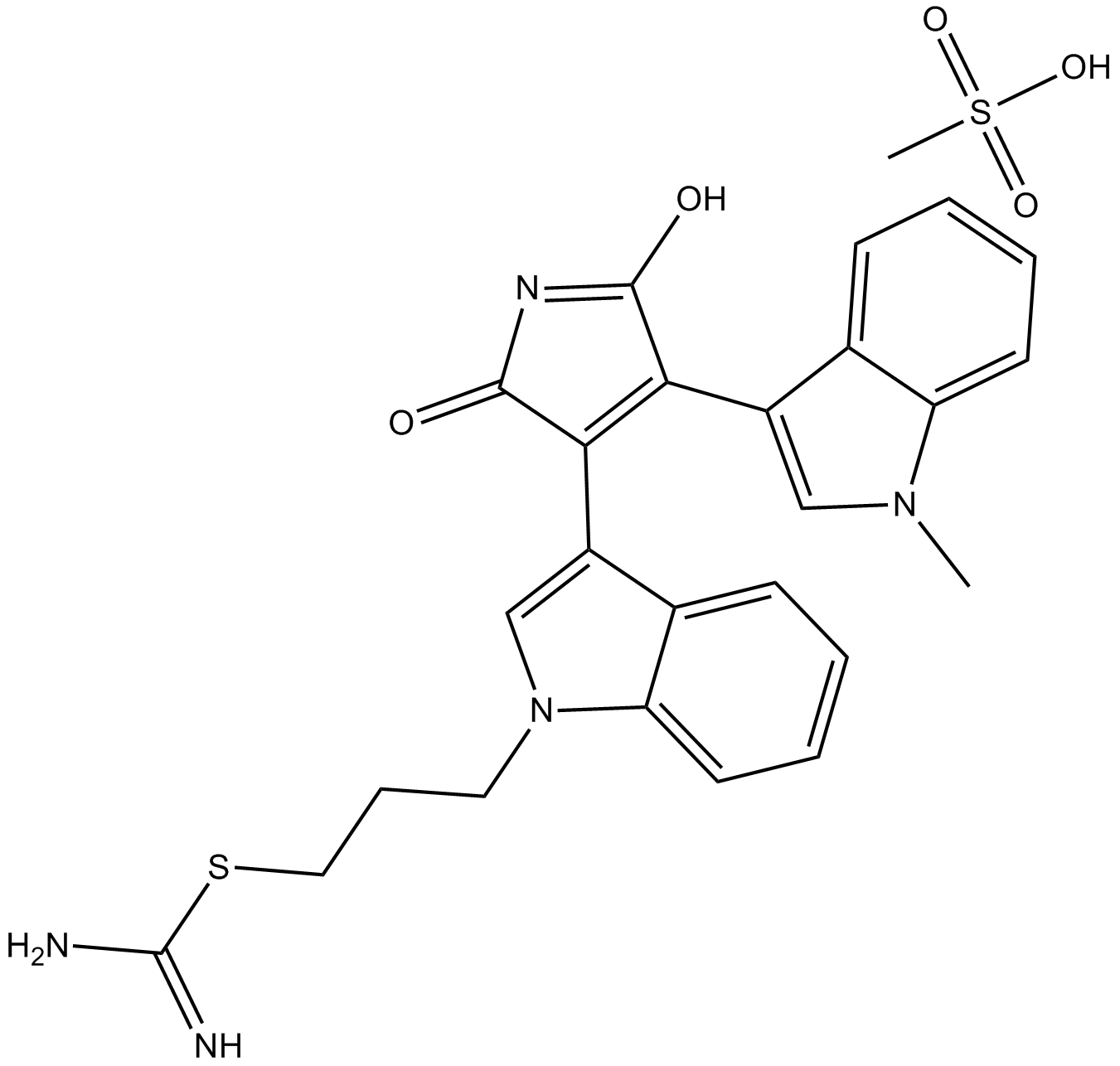
Ro 31-8220 methanesulfonate
RO 31-8220 El metanesulfonato es un potente inhibidor de PKC, con IC50 de 5, 24, 14, 27, 24 y 23 nm para PKC&#####. offlineefficient_models_2022q2.mdand rat brain PKC, respectively.en_es_2021q4.md
-
GC13677
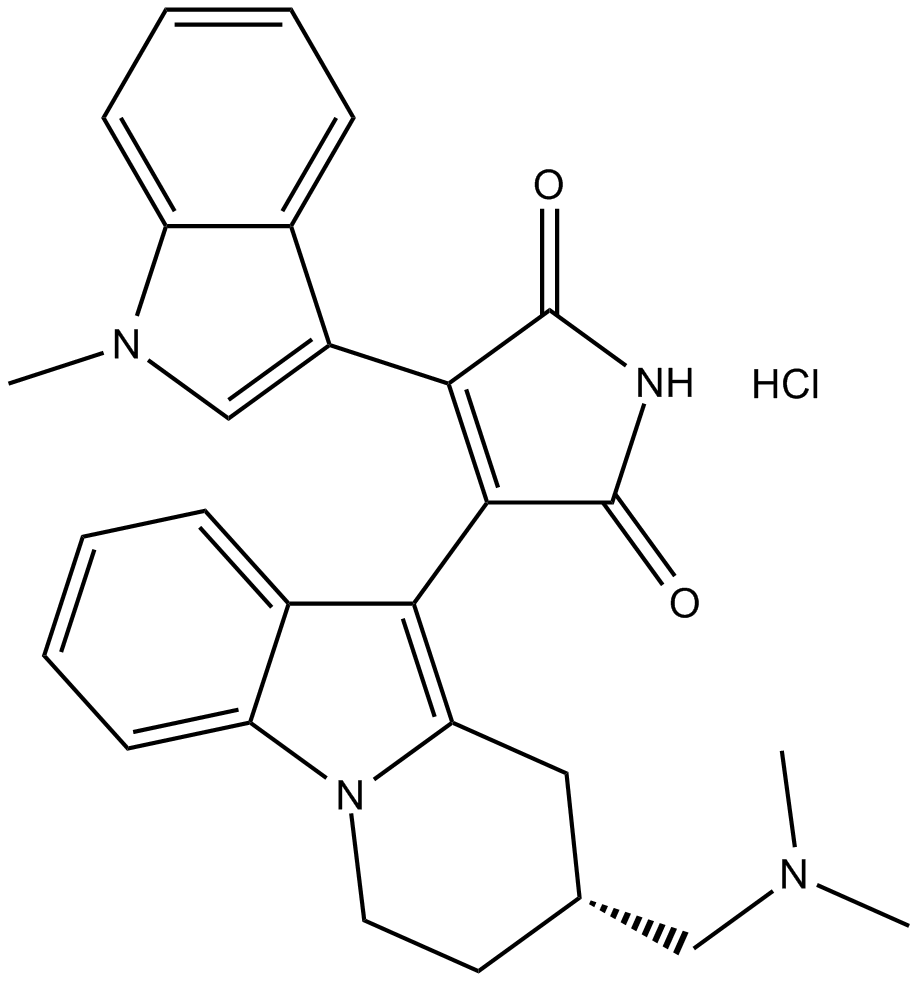
Ro 32-0432 hydrochloride
El clorhidrato de Ro 32-0432 es un inhibidor de PKC potente, selectivo, competitivo con ATP y activo por vÍa oral.
-
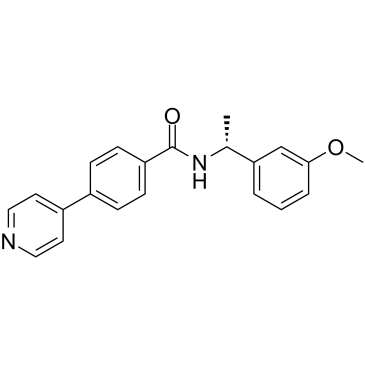
GC37551
ROCK inhibitor-2
El inhibidor de ROCK-2 es un inhibidor dual selectivo de ROCK1 y ROCK2 con IC50 de 17 nM y 2 nM, respectivamente.
-
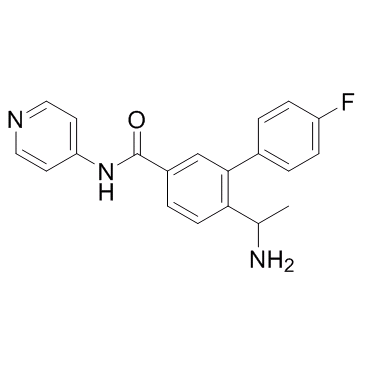
GC31952
ROCK-IN-1
ROCK-IN-1 es un potente inhibidor de ROCK, con una IC50 de 1,2 nM para ROCK2.
-
GC66338
ROCK1-IN-1
ROCK1-IN-1 es un inhibidor de ROCK1 con un valor Ki de 540 nM. ROCK1-IN-1 se puede utilizar para la investigaciÓn de la hipertensiÓn, el glaucoma y la disfunciÓn eréctil.
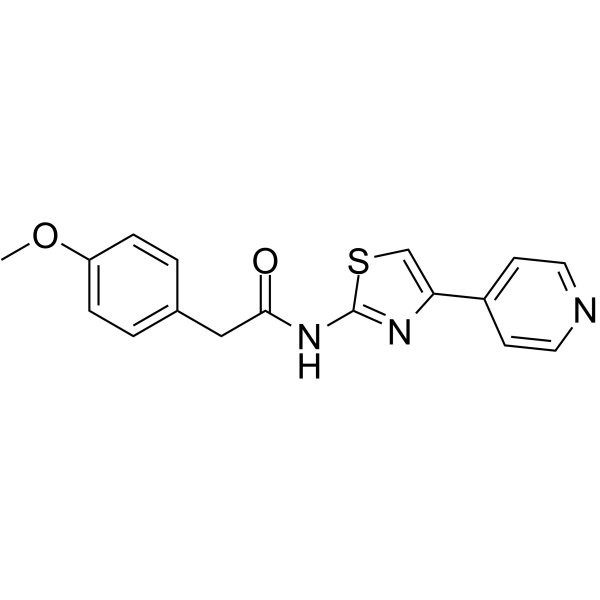
-
GC31883
ROCK2-IN-2
ROCK2-IN-2 es un inhibidor selectivo de ROCK2 extraÍdo de la patente US20180093978A1, Compound A-30, tiene un IC50 de <1 μM.
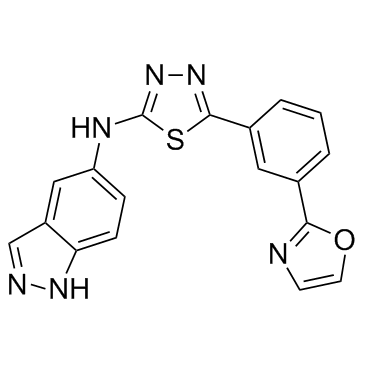
-
GC10820
Rottlerin
Rottlerin, un producto natural purificado de Mallotus Philippinensis, es un inhibidor especÍfico de PKC, con valores de IC50 para PKCδ de 3-6 μM, PKCα,β,γ de 30-42 μM, PKCε,η,ζ de 80-100 μM.
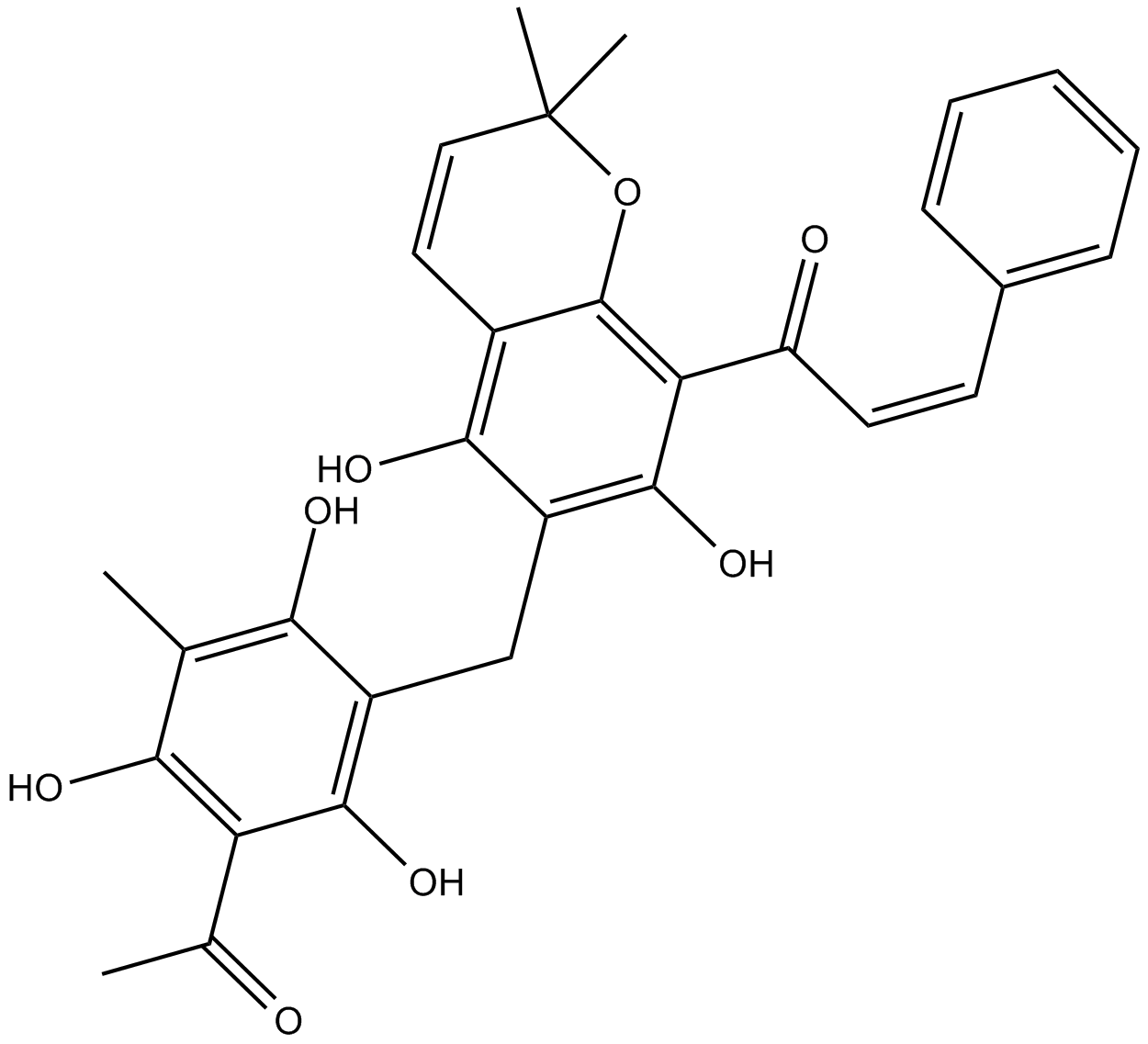
-
GC63177
Ruboxistaurin
Ruboxistaurin (LY333531) es un inhibidor beta selectivo de PKC activo por vÍa oral (Ki = 2 nM).

-
GC16405
Safingol
Safingol es un inhibidor de la PKC (proteÍna quinasa C) lisoesfingolÍpido.
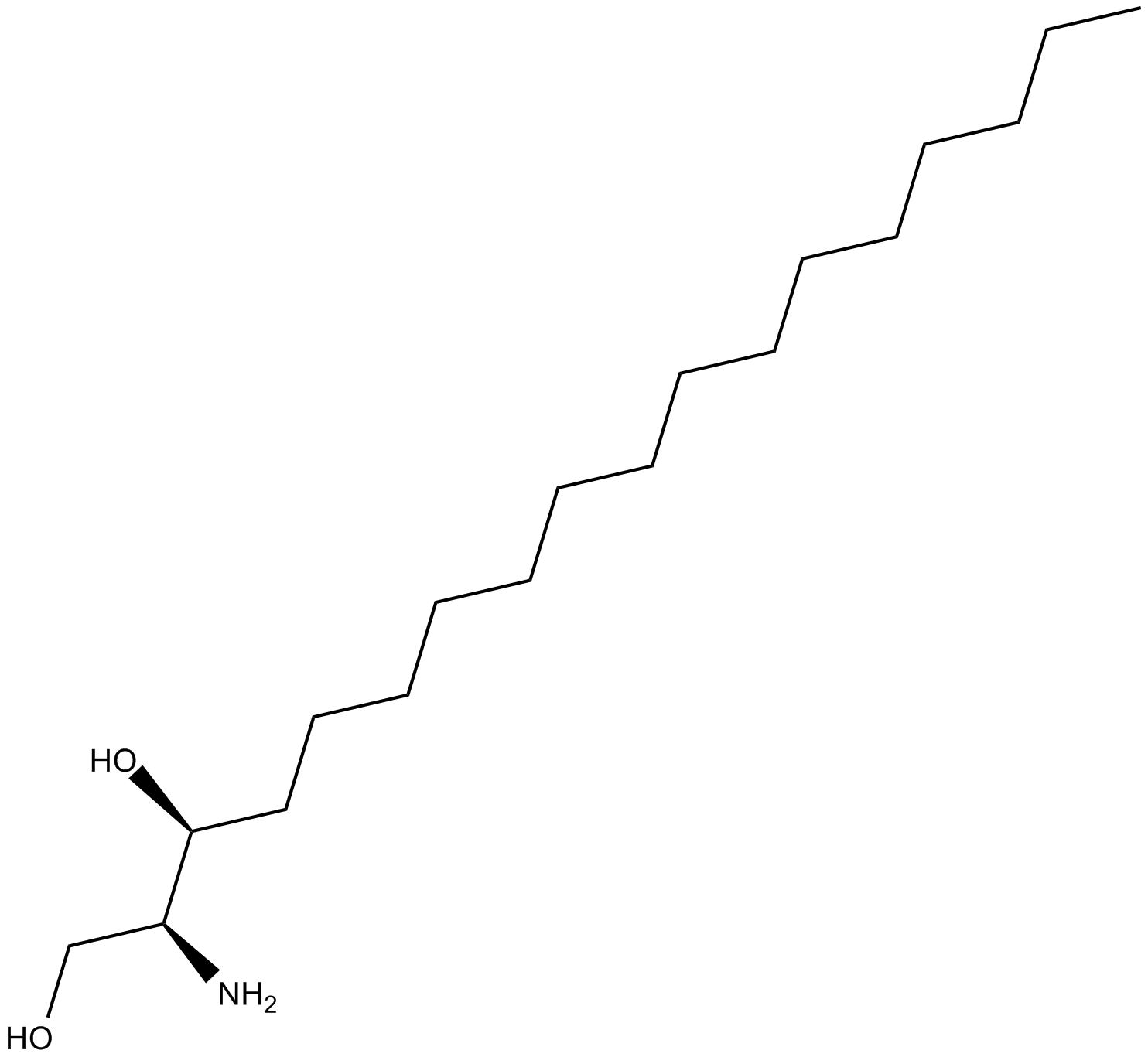
-
GC63439
Sangivamycin
La sangivamicina (NSC 65346), un anÁlogo de nucleÓsido, es un potente inhibidor de la proteÍna quinasa C (PKC) con una Ki de 10 μM. La sangivamicina tiene una potente actividad antiproliferativa contra una variedad de cÁnceres humanos.
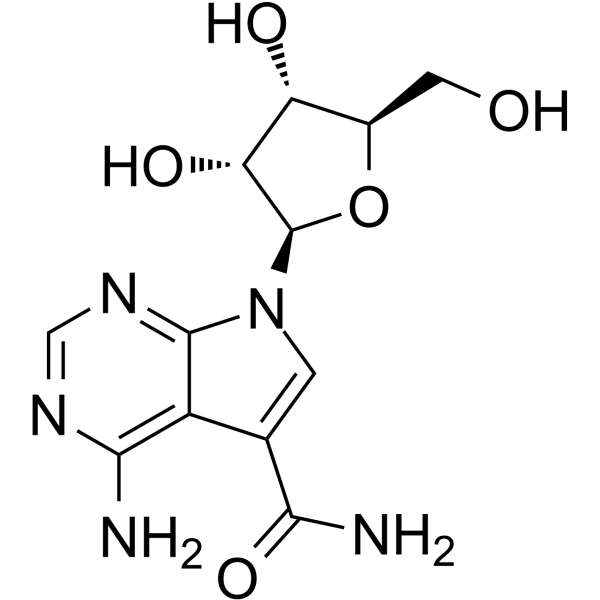
-
GC13759
SAR407899
SAR407899 es un inhibidor de ROCK selectivo, potente y competitivo con ATP, con una IC50 de 135 nM para ROCK-2 y Kis de 36 nM y 41 nM para ROCK-2 humano y de rata, respectivamente.
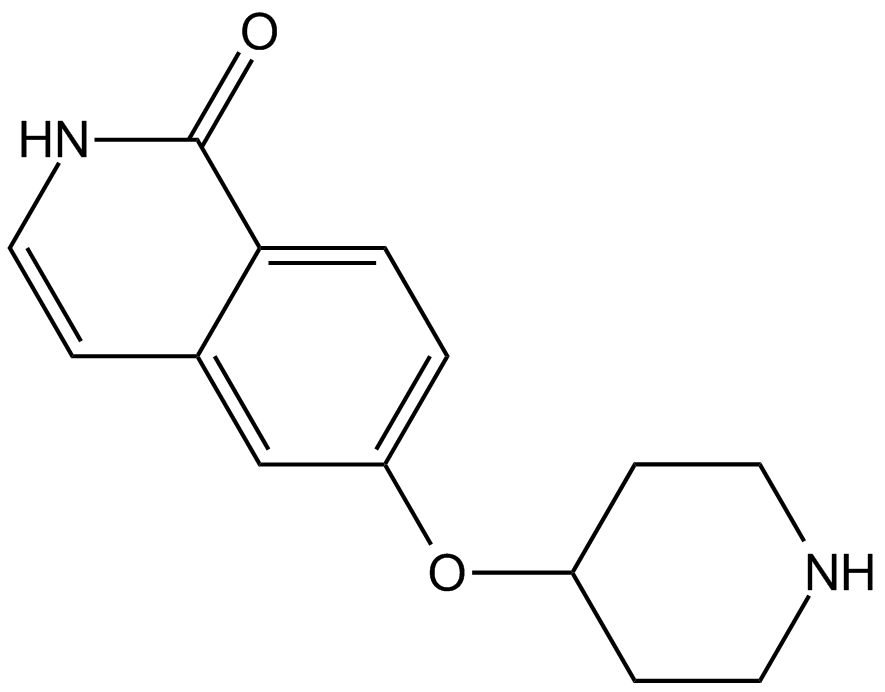
-
GC16289
SAR407899 hydrochloride
El clorhidrato de SAR407899 es un inhibidor de ROCK selectivo, potente y competitivo con ATP, con una IC50 de 135 nM para ROCK-2 y Kis de 36 nM y 41 nM para ROCK-2 humano y de rata, respectivamente.
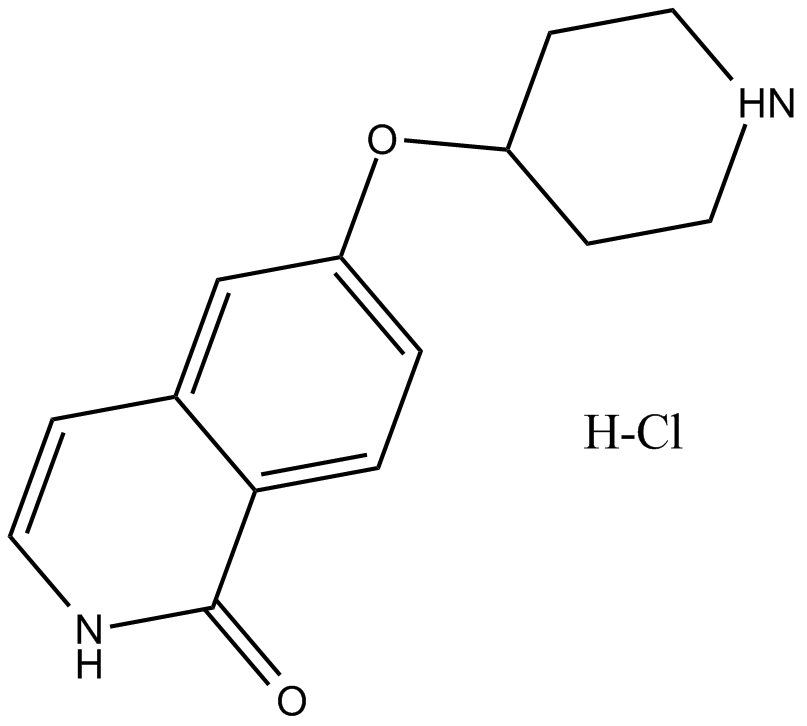
-
GC15197
Saracatinib (AZD0530)
Saracatinib (AZD0530) (AZD0530) es un potente inhibidor de la familia Src con IC50 de 2,7 a 11 nM para c-Src, Lck, c-YES, Lyn, Fyn, Fgr y Blk. Saracatinib (AZD0530) muestra una alta selectividad sobre otras tirosina quinasas.
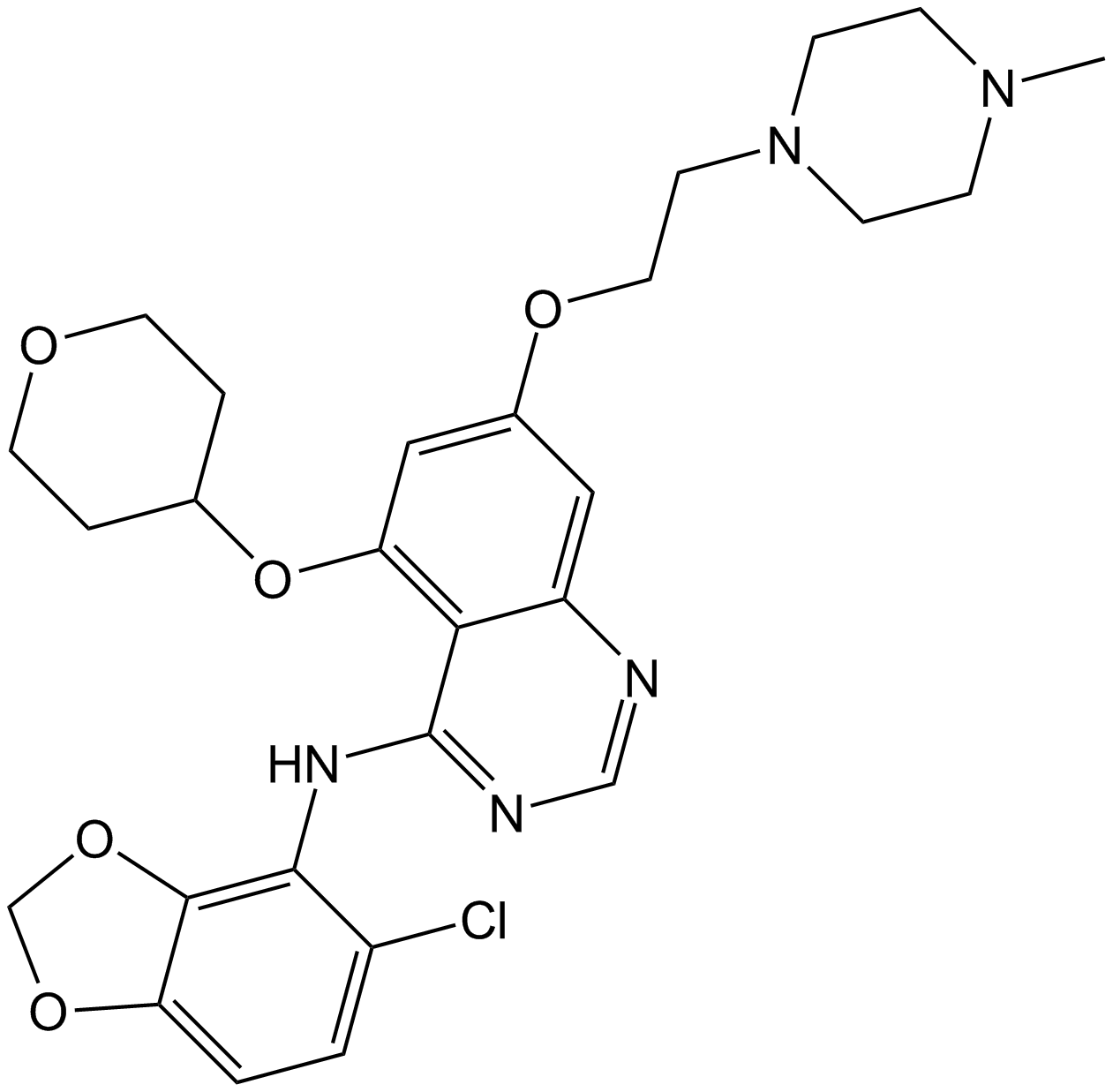
-
GC11022
SB 218078
SB 218078 es un inhibidor potente, selectivo, ATP-competitivo y permeable a las células del punto de control quinasa 1 (Chk1) que inhibe la fosforilación de Chk1 de cdc25C con una IC50 de 15 nM. SB 218078 inhibe menos potentemente Cdc2 (IC50 de 250 nM) y PKC (IC50 de 1000 nM). SB 218078 causa apoptosis por daño en el ADN y detención del ciclo celular.
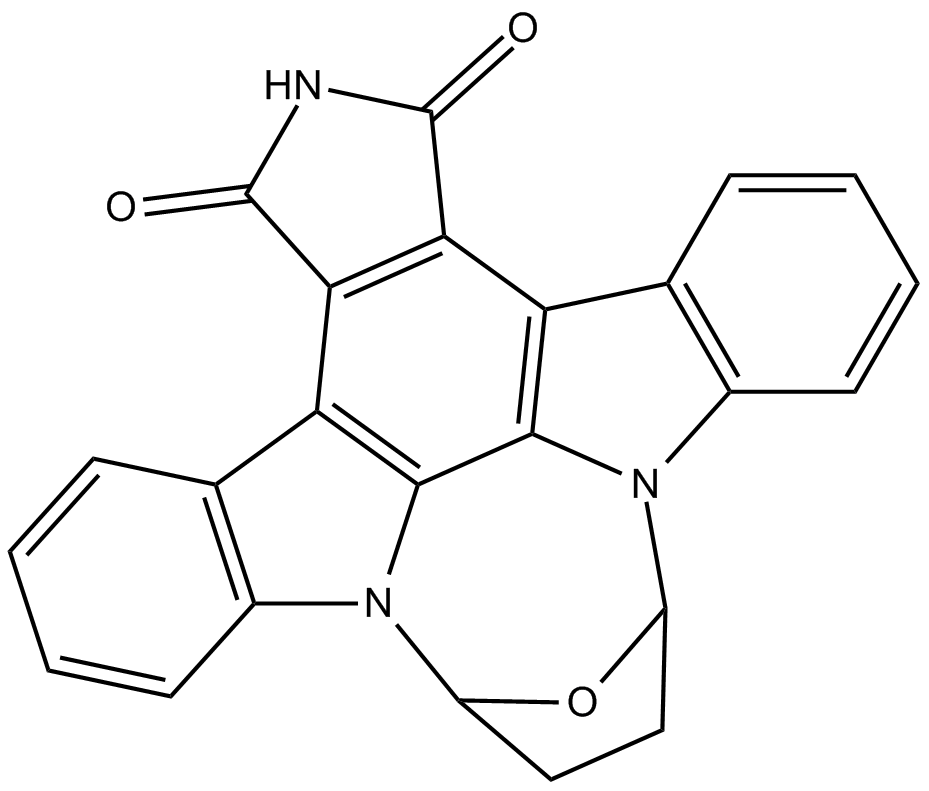
-
GC11545
SB 431542
SB-431542, a small molecule inhibitor
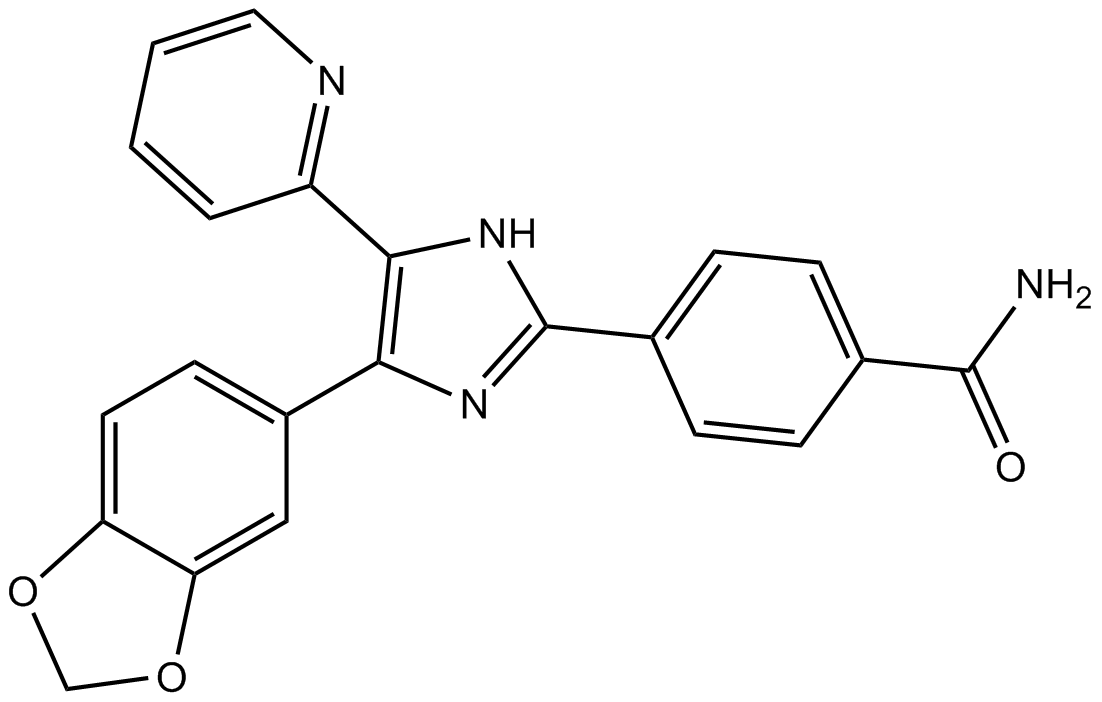
-
GC15170
SB 772077B dihydrochloride
El diclorhidrato de SB 772077B es un inhibidor de la quinasa Rho (ROCK) basado en aminofurazan con IC50 de 5,6 nM y 6 nM hacia ROCK1 y ROCK2, respectivamente.
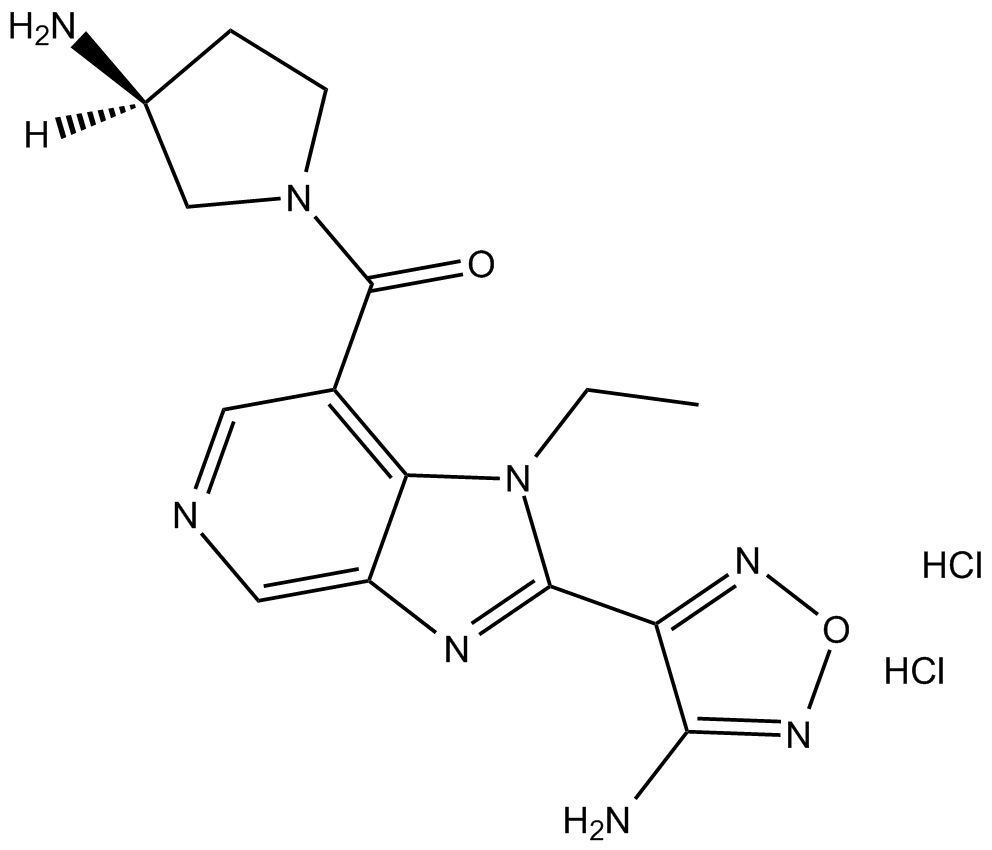
-
GC10421
SB-505124 hydrochloride
Inhibidor de los receptores ALK4, ALK5 y ALK7.
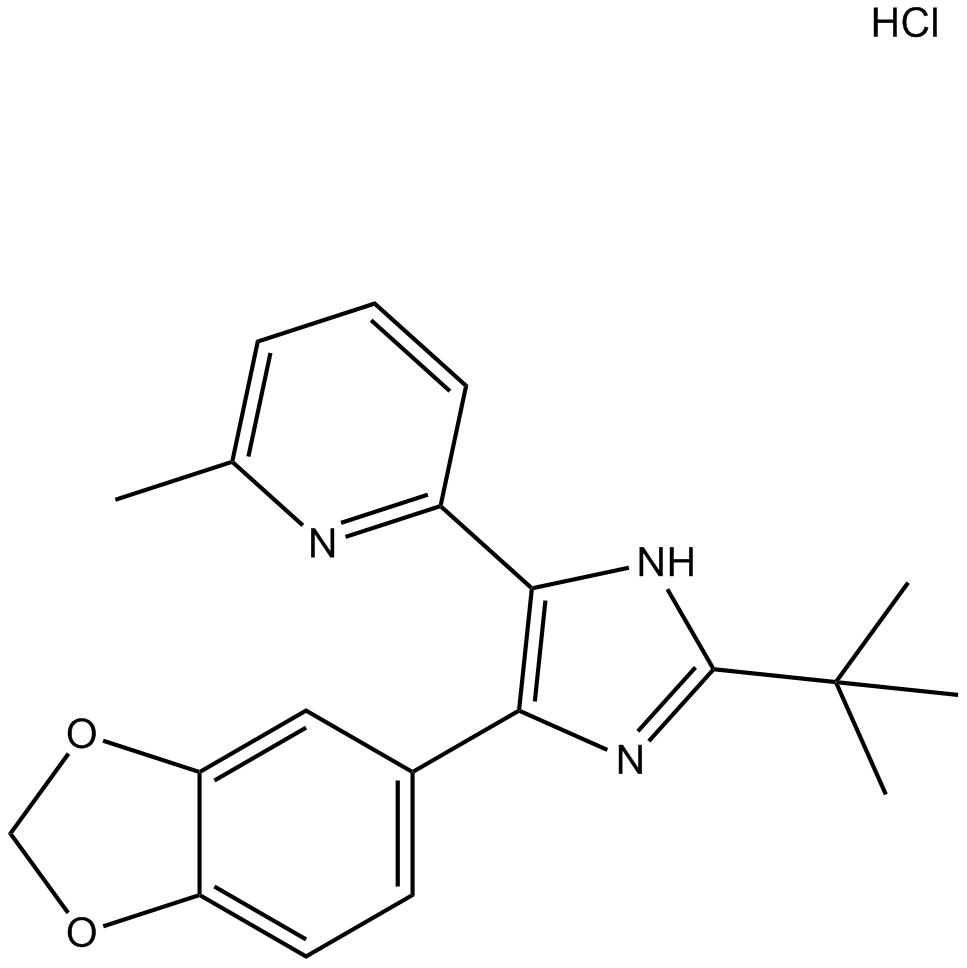
-
GC13769
SB505124
SB505124 es un inhibidor selectivo de los receptores tipo I del receptor TGF-β (ALK4, ALK5, ALK7), con IC50 de 129 nM y 47 nM para ALK4, ALK5, respectivamente, pero no inhibe ALK1, 2, 3 o 6.
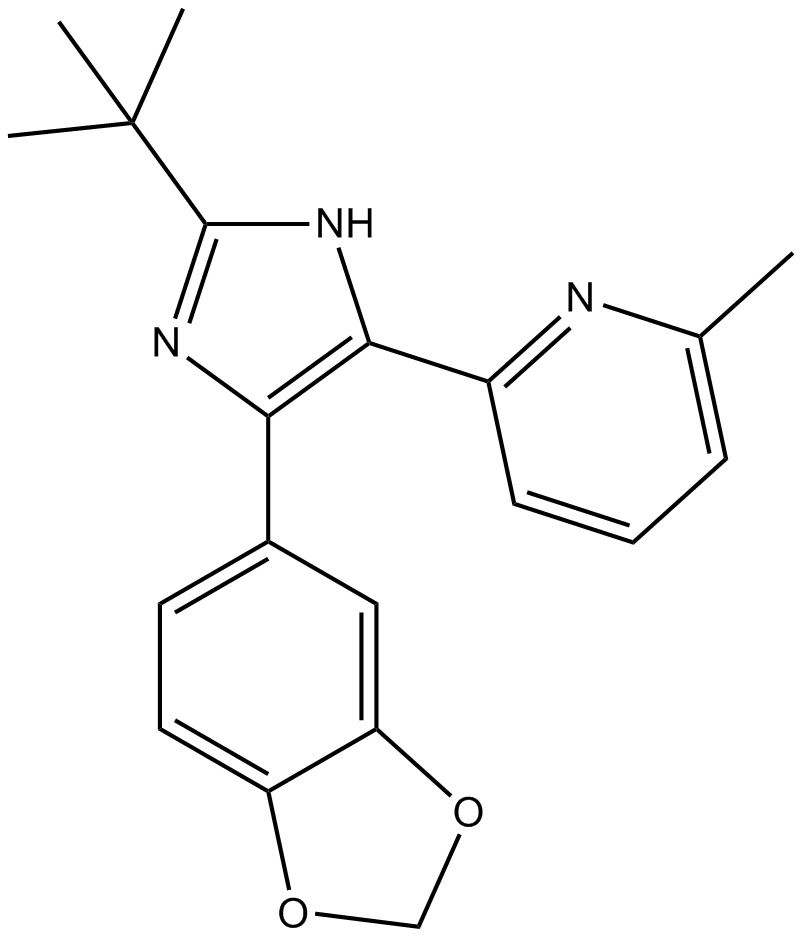
-
GC14349
SB525334
SB525334 es un inhibidor potente y selectivo del receptor del factor de crecimiento transformante β1 (ALK5) con una IC50 de 14,3 nM.
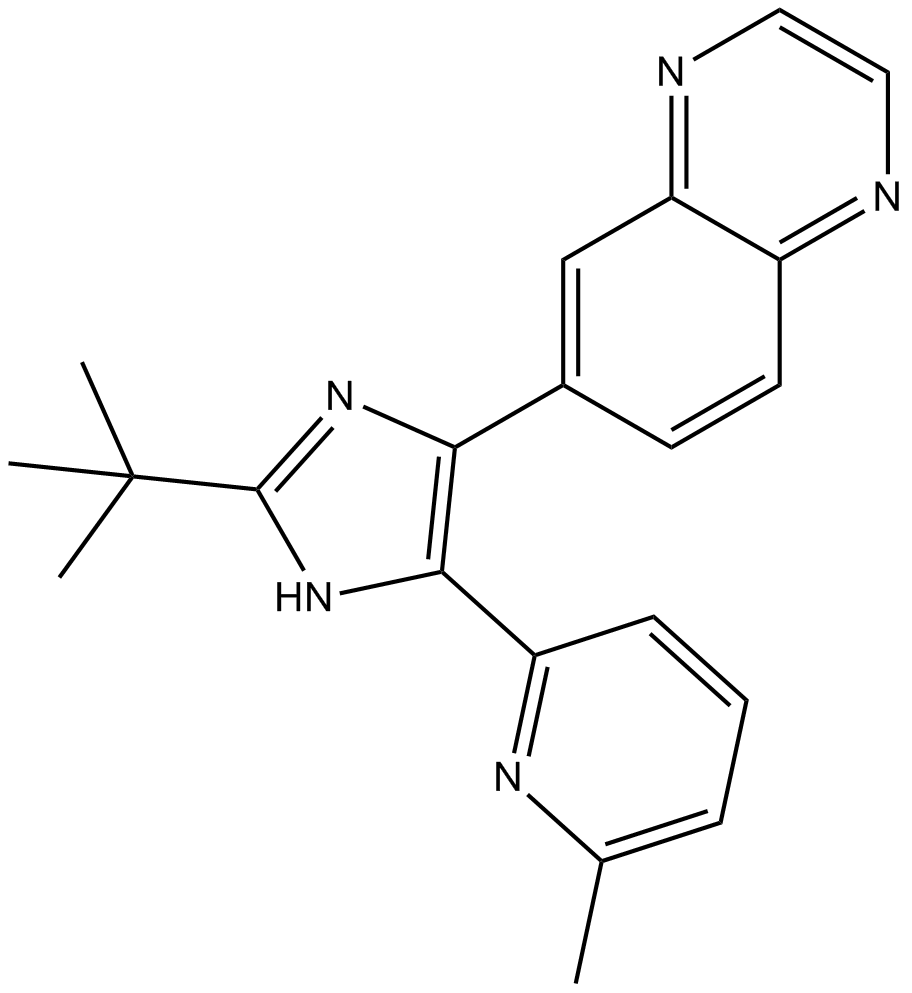
-
GC13320
SC-10
Protein kinase C activator
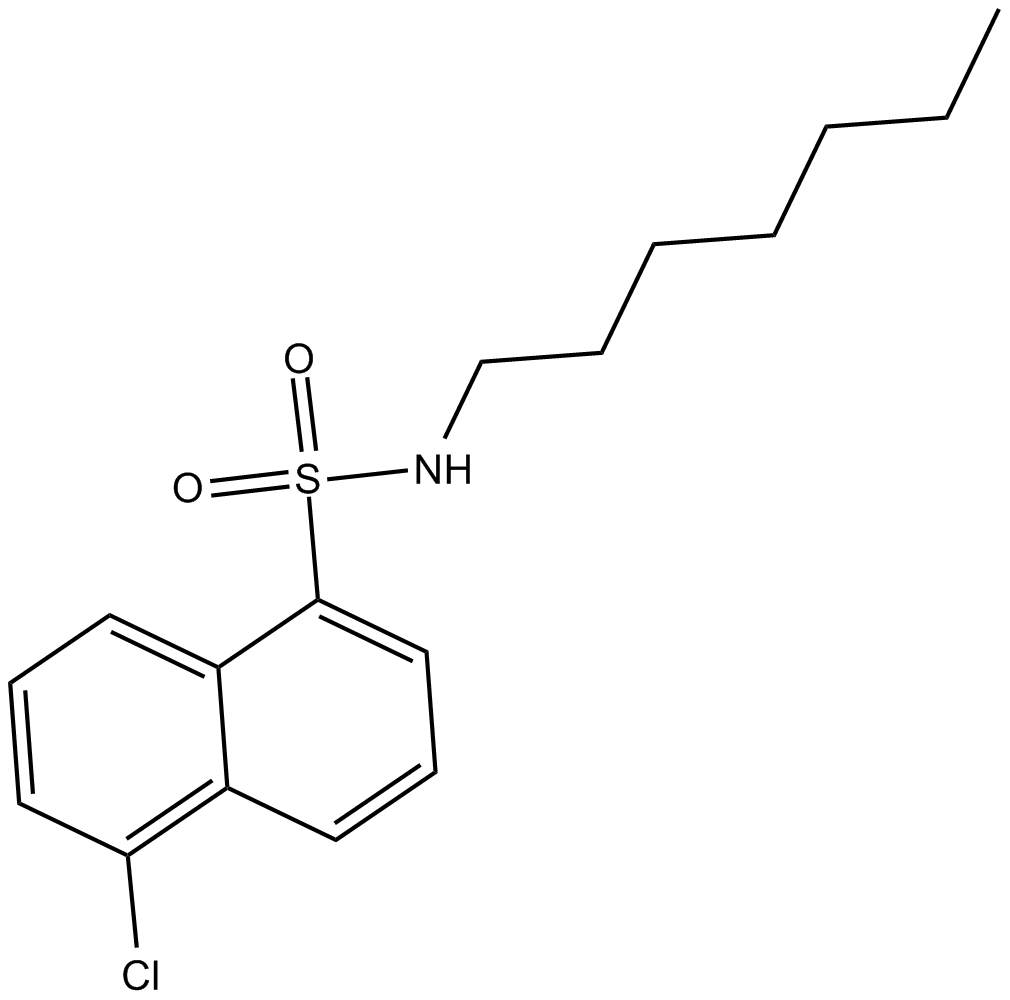
-
GC18055
SC-9
SC-9 es un activador de PKC en presencia de Ca2+.
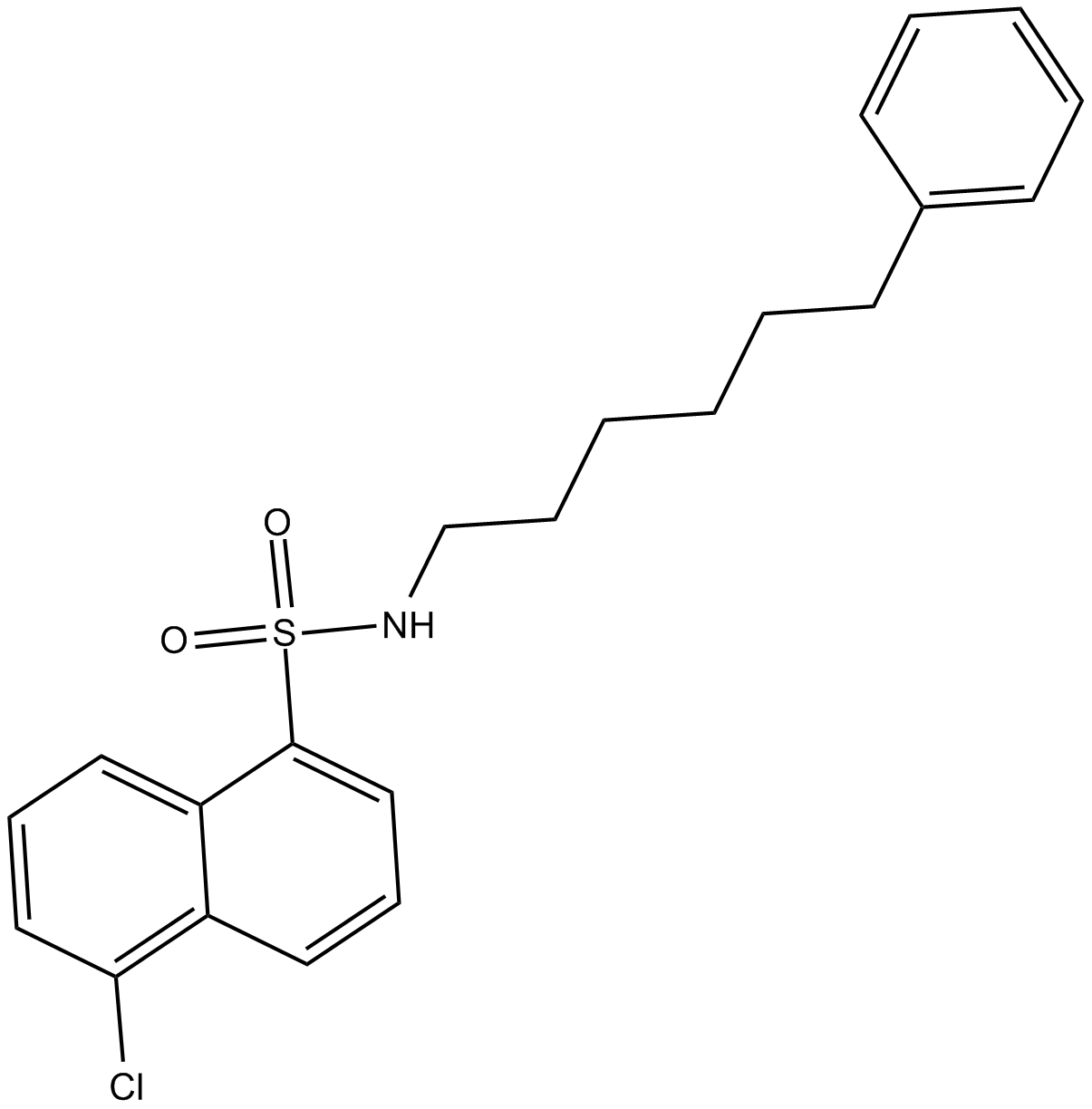
-
GC13904
SD-208
A potent, selective inhibitor of TGF-βRI
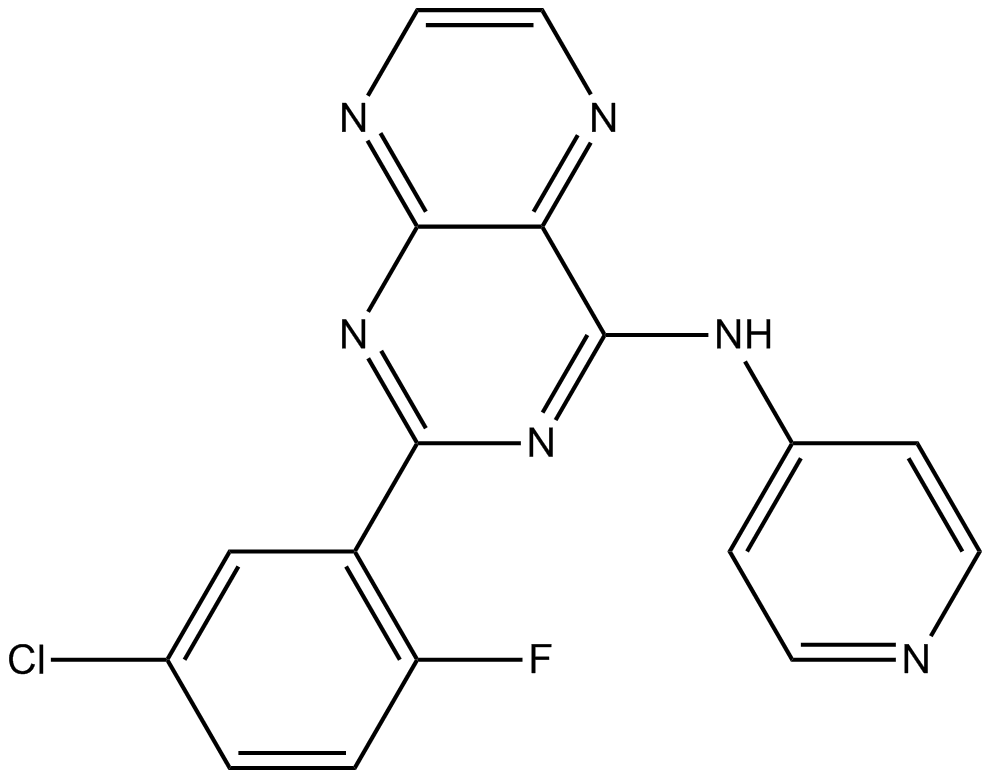
-
GC17191
SIS3 HCl
SIS3HCl es un inhibidor potente y selectivo de Smad3 con una IC50 de 3 μM para la fosforilación de Smad3.
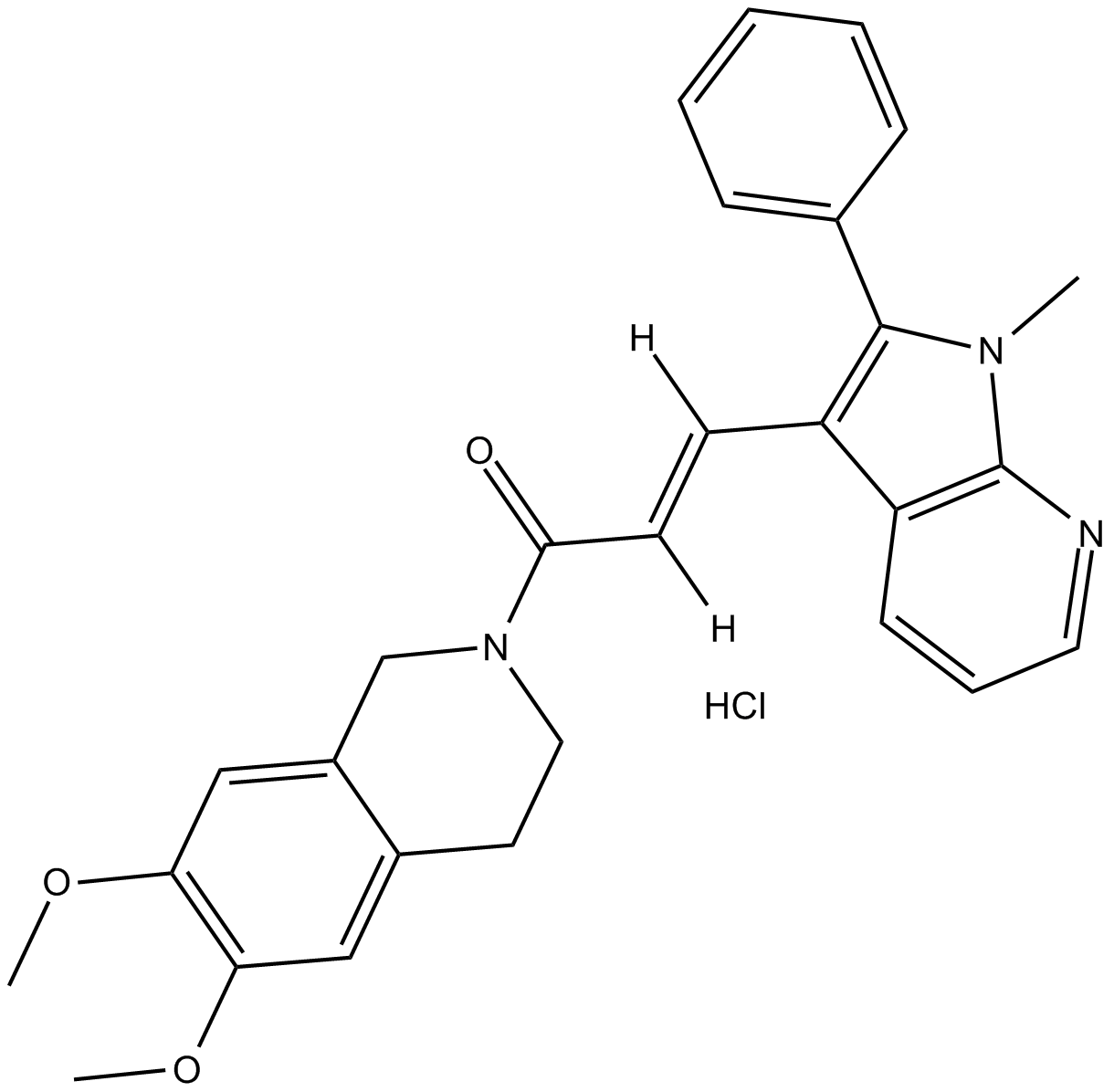
-
GC69903
SJ000063181
SJ000063181 es un activador efectivo de la señal BMP, con una EC50 ≤1 µM. SJ000063181 puede ser utilizado como sonda química para explorar la señalización BMP, ya que puede penetrar en embriones de pez cebra.
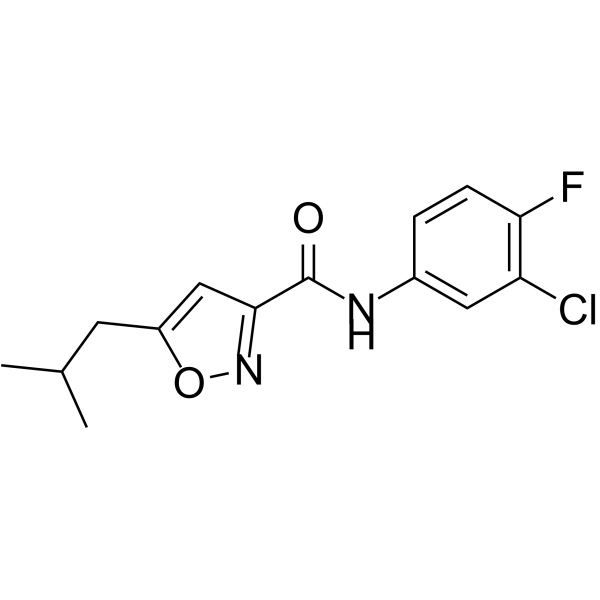
-
GC32788
SJ000291942
SJ000291942 es un activador de la vÍa de seÑalizaciÓn de proteÍnas morfogenéticas Óseas canÓnicas (BMP). Las BMP son miembros de la familia de moléculas de seÑalizaciÓn secretadas del factor de crecimiento transformante beta (TGFβ).
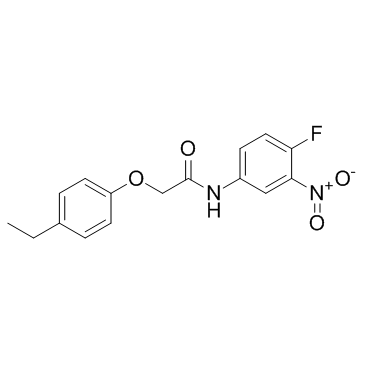
-
GC19447
SM 16
SM 16 es un inhibidor de la cinasa ALK5/ALK4 con Kis de 10 y 1,5 nM, respectivamente.

-
GC44903
SMAD3 Inhibitor, SIS3
Inhibidor de SMAD3, SIS3 es un inhibidor potente y selectivo de la fosforilaciÓn de Smad3.
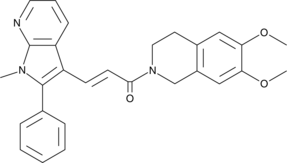
-
GC65592
SNIPER(ABL)-020
SNIPER(ABL)-020, conjugando Dasatinib (inhibidor ABL) con Bestatina (ligando IAP) con un enlazador, induce la reducciÓn de la proteÍna BCR-ABL.
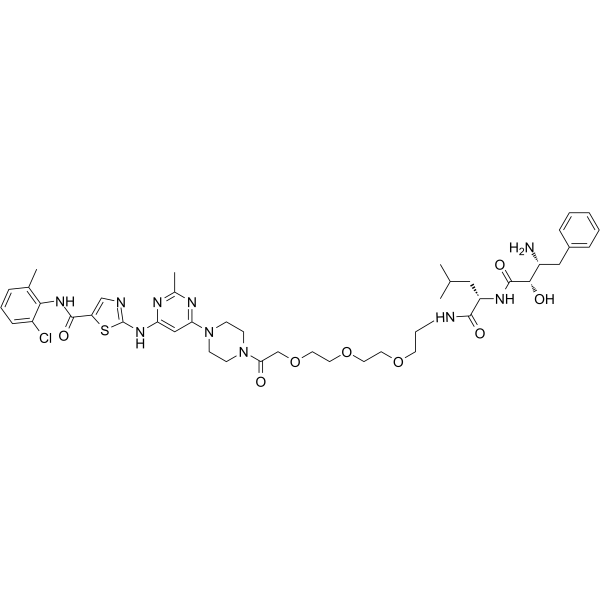
-
GC15520
Sotrastaurin (AEB071)
Sotrastaurin (AEB071) (AEB071) es un inhibidor PAN-PKC potente y oralmente activo, con KIS de 0.22 nm, 0.64 nm, 0.95 nm, 1.8 nm, 2.1 nm y 3.2 nm para PKC&&&&&&&&&&&&&&&&77TRES. offlineefficient_models_2022q2.md;, PKCδen_es_2021q4.mdand PKCε, respectively.en_es_2021q4.md
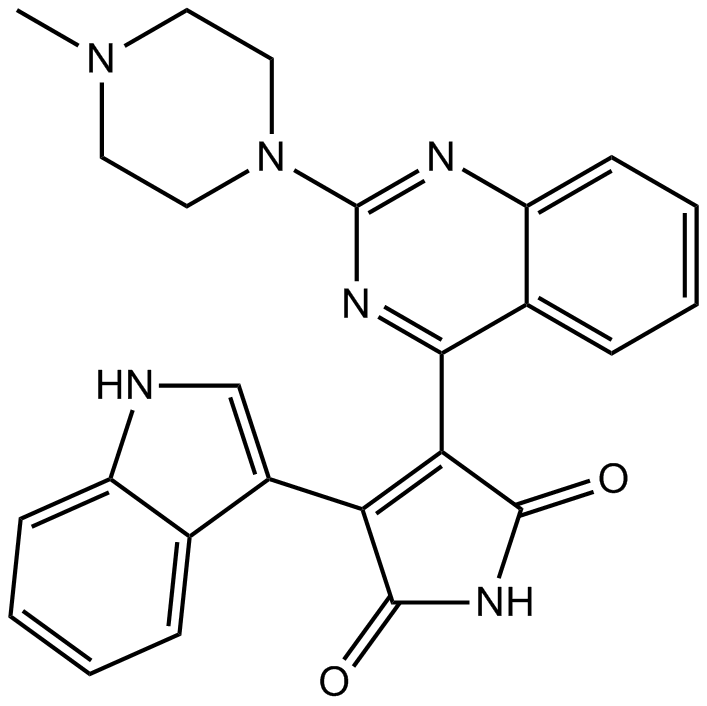
-
GC10722
SR-3677
SR-3677 es un inhibidor de ROCK-II potente y selectivo con una IC50 de ~3 nM.
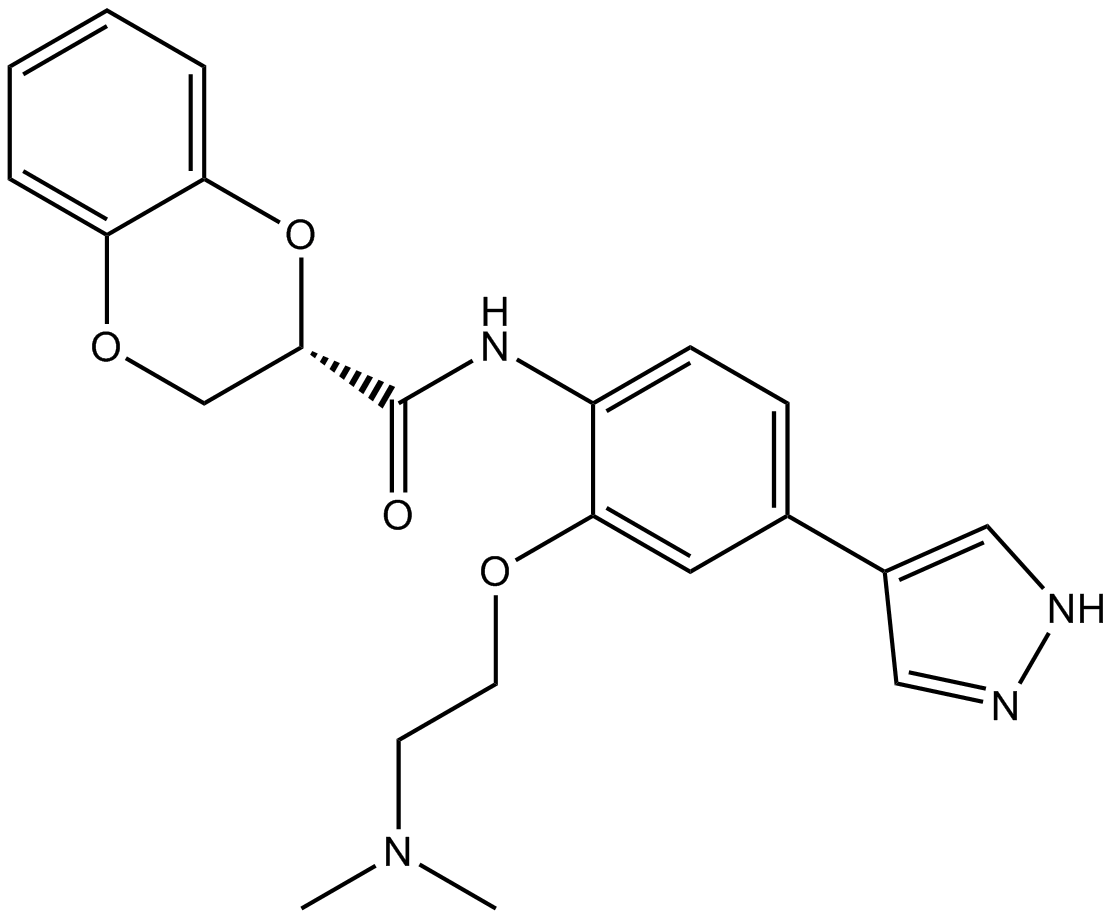
-
GC19338
SRI-011381 hydrochloride
El clorhidrato de SRI-011381 es un agonista de seÑalizaciÓn de TGF-β activo por vÍa oral, exhibe efectos neuroprotectores.
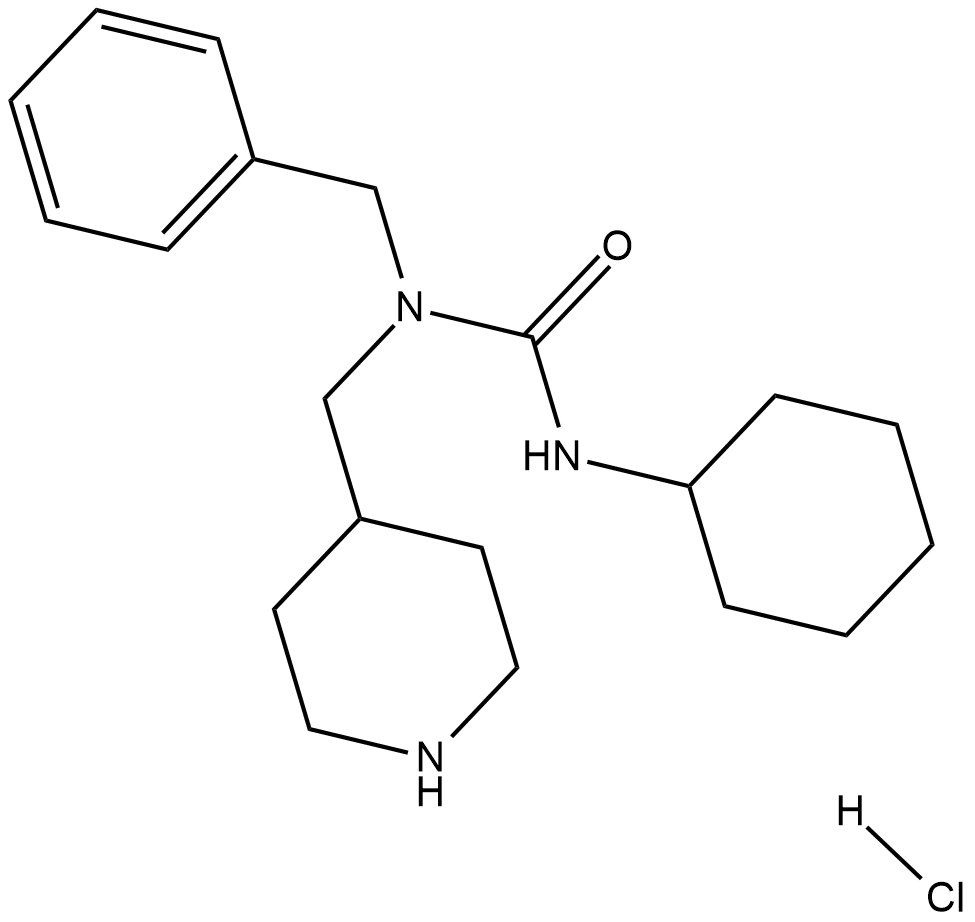
-
GC15299
Staurosporine(CGP 41251)
A potent inhibitor of protein kinase C
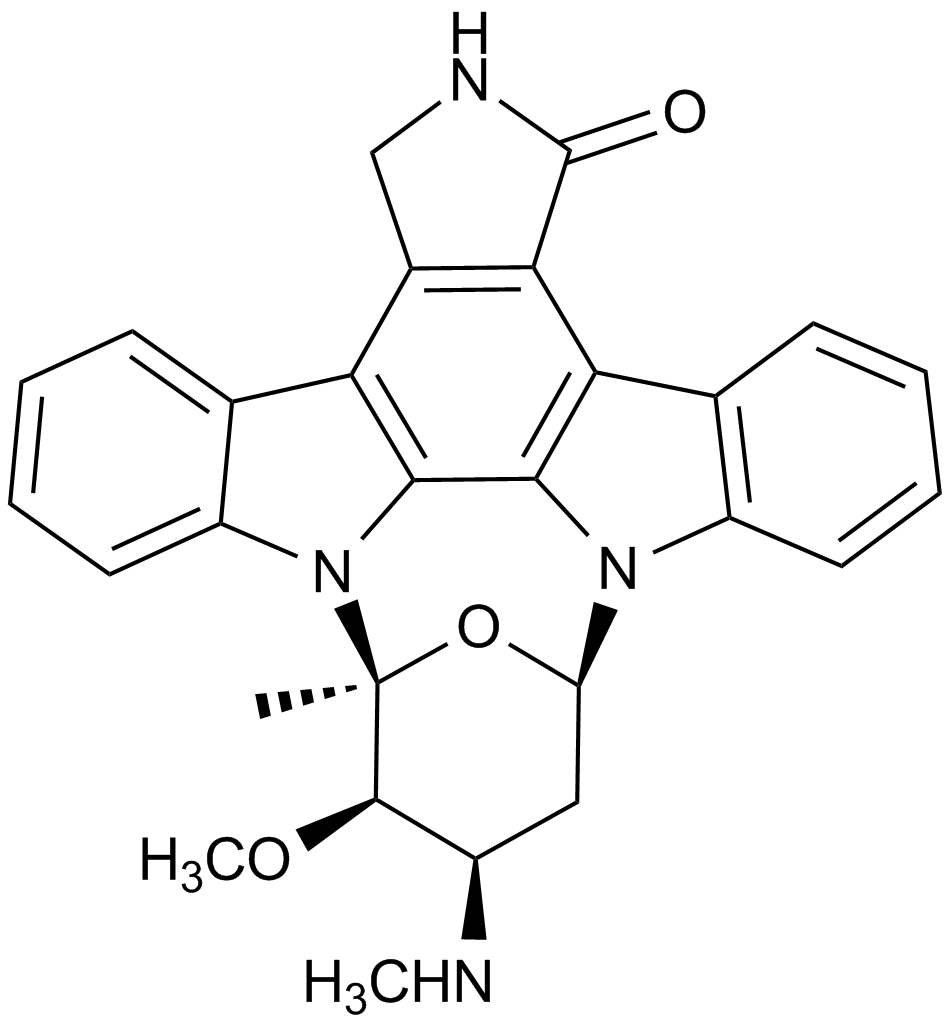
-
GC69981
SY-LB-35
SY-LB-35 es un agonista efectivo del receptor de proteína morfogenética ósea (BMP). SY-LB-35 puede estimular significativamente el aumento en la cantidad y actividad celular de las células musculares C2C12, haciendo que el ciclo celular se transforme hacia las fases S y G2/M. SY-LB-35 estimula tanto la vía de señalización intracelular típica Smad como las no típicas PI3K/Akt, ERK, p38 y JNK.
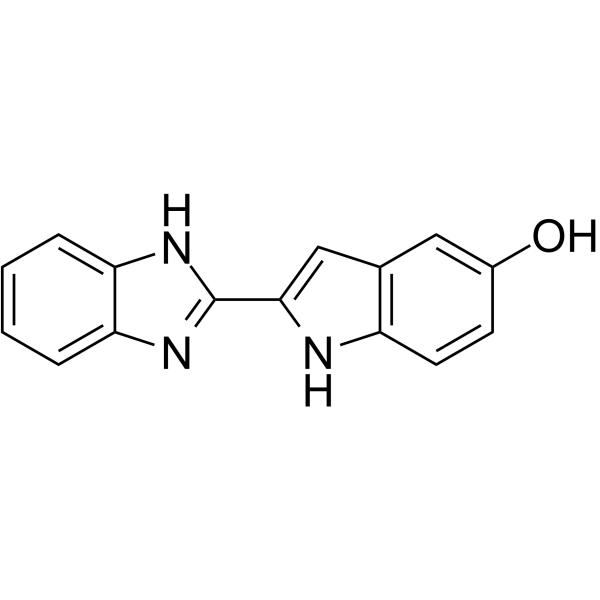
-
GC16821
TAE226(NVP-TAE226)
TAE226 (NVP-TAE226) (TAE226) es un potente inhibidor dual de FAK e IGF-1R competitivo con ATP con IC50 de 5,5 nM y 140 nM, respectivamente.
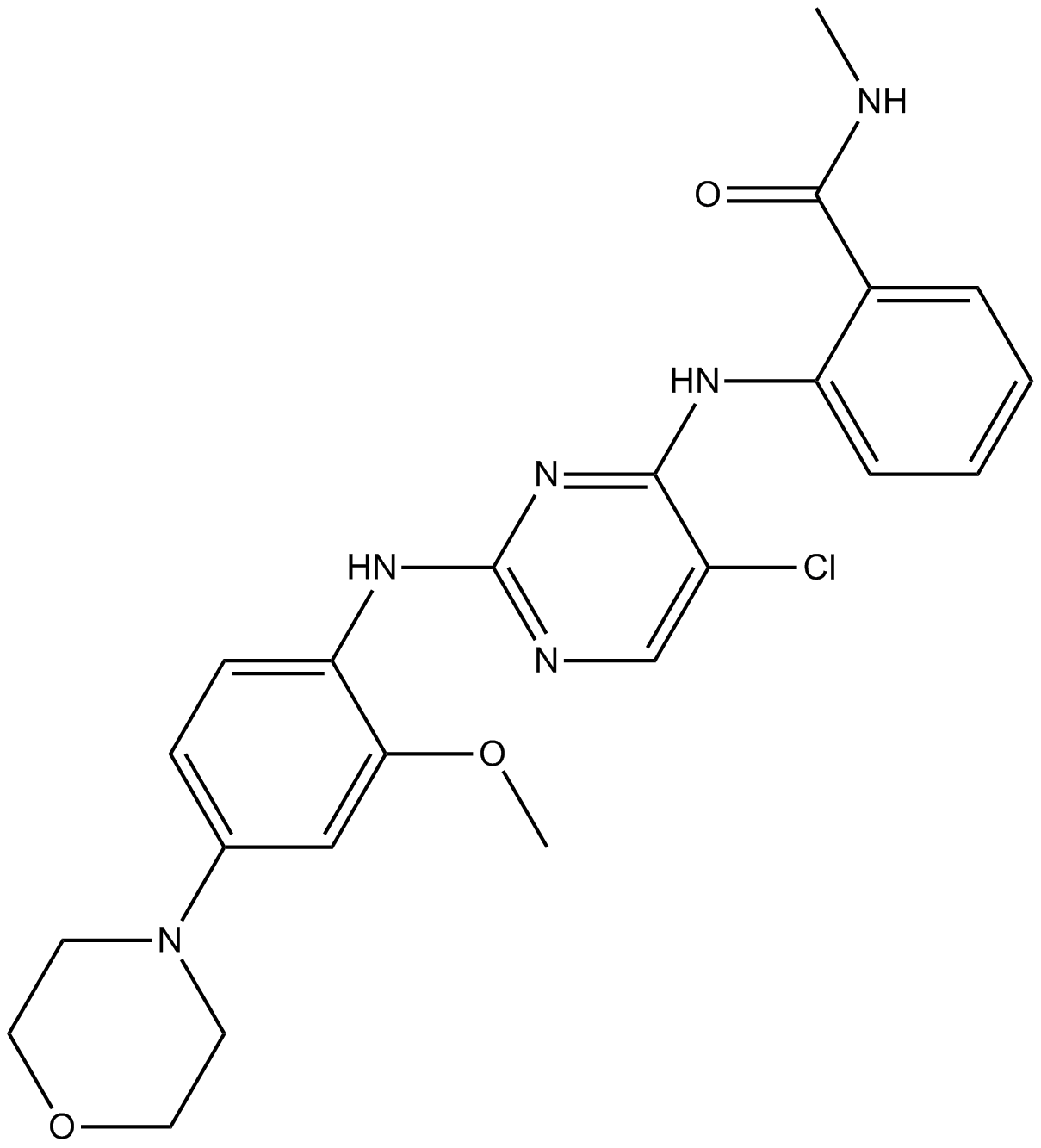
-
GC17901
Tamoxifen
Tamoxifeno (TAM) actúa como un regulador selectivo del receptor de estrógeno (SERM), inhibiendo los efectos del estrógeno en las células mamarias mientras potencialmente estimula la actividad estrogénica en células encontradas en diferentes tejidos.
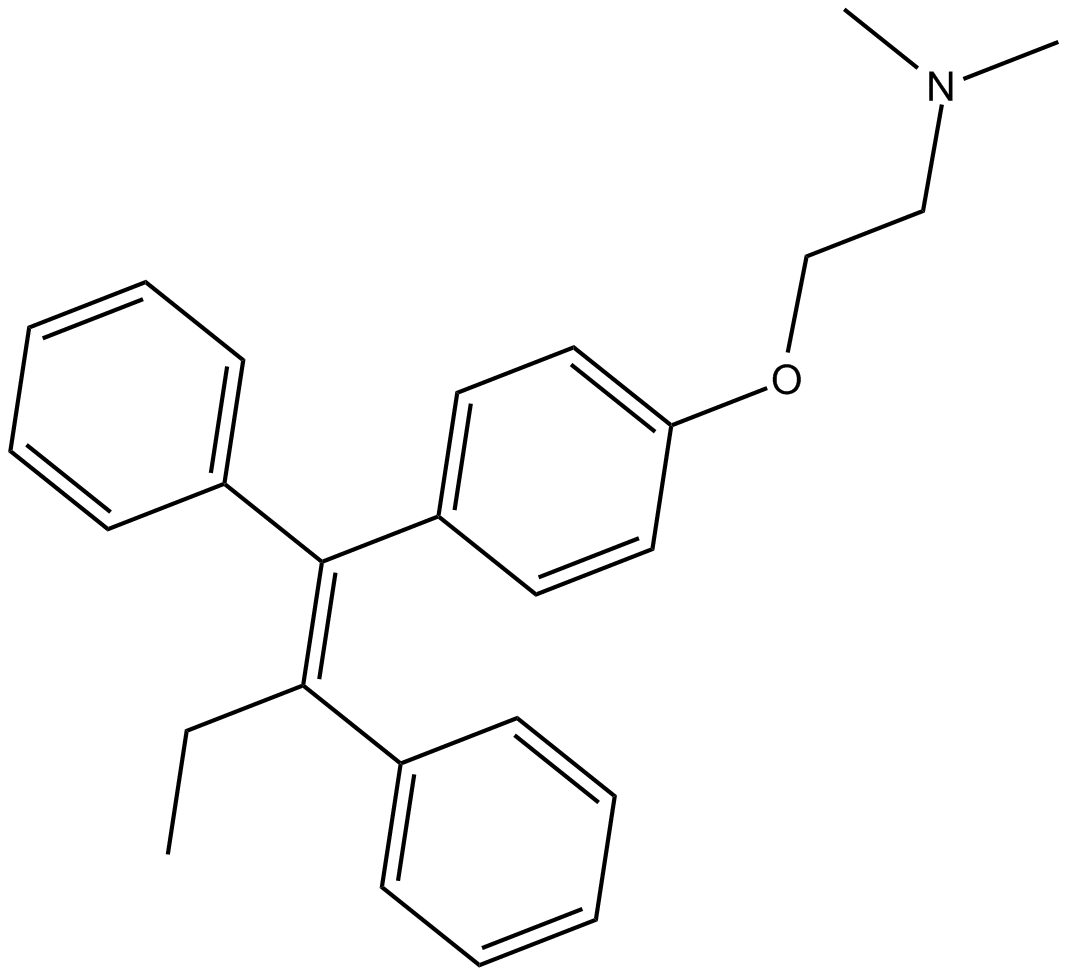
-
GC10682
TAS 301
TAS 301 es un inhibidor de la migración y proliferación de células de músculo liso e inhibe la activación de PKC inducida por PDGF.
-
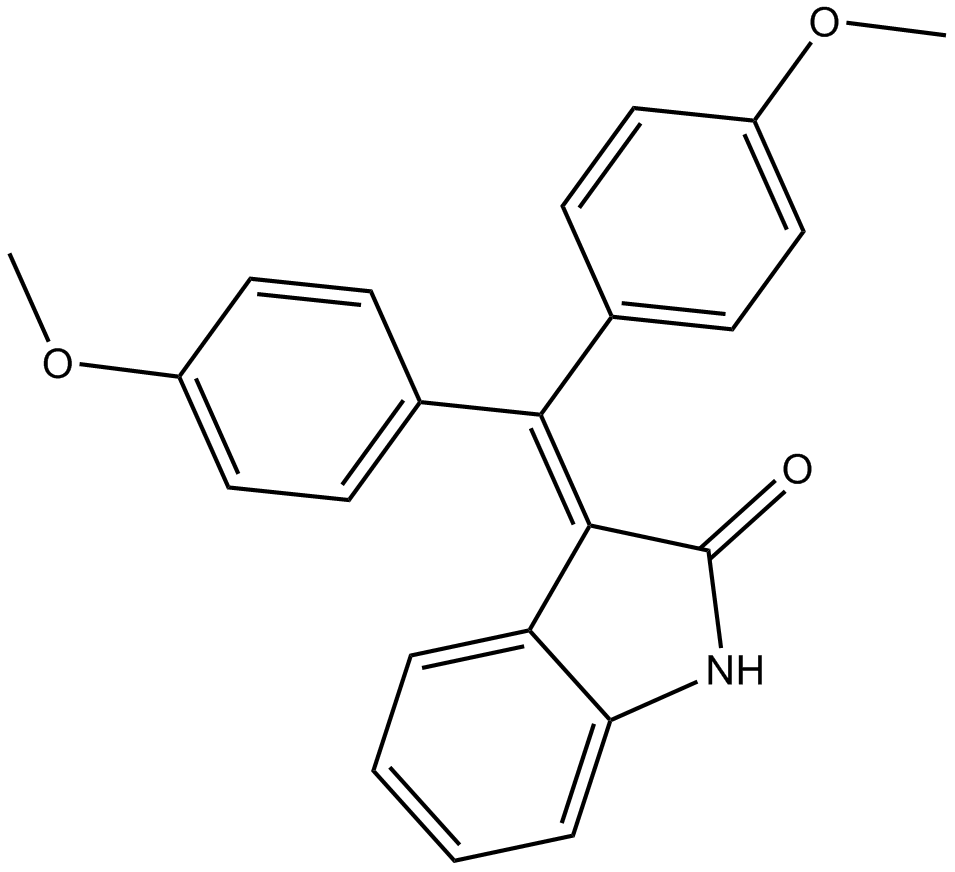
GC11181
Thiazovivin
La tiazovivina es un potente inhibidor de ROCK, que puede proteger las células madre embrionarias humanas. La tiazovivina mejora la eficiencia de la generaciÓn de iPSC.
-
GC26000
TP0427736 HCl
TP0427736 is a potent inhibitor of ALK5 kinase activity with an IC50 of 2.72 nM and this effect is 300-fold higher than the inhibitory effect on ALK3 (IC50 = 836 nM for ALK3). It also inhibits Smad2/3 phosphorylation in A549 cells induced by TGF-β1 with an IC50 value of 8.68 nM.
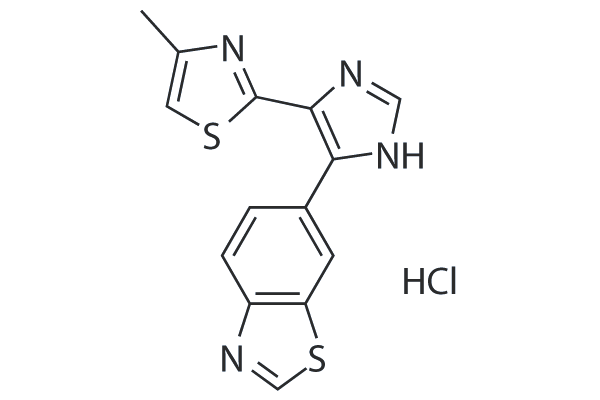
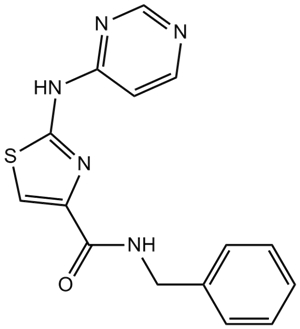
-
GC70050
Trabedersen
Trabedersen (AP 12009) es un oligonucleótido antisentido que inhibe específicamente TGF-β2 (TGF-beta/Smad). Trabedersen se puede utilizar en la investigación de tumores cerebrales malignos y otros tumores sólidos que sobreexpresan TGF-β2, como los tumores de piel, páncreas y colon.

-
GC40928
Trimethylamine N-oxide
El óxido de trimetilamina (TMAO) es un producto de la oxidación de TMA por la mono-oxigenasa 3 que contiene flavina en el hígado.
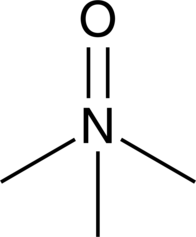
-
GC16678
UCN-02
UCN-02 (7-epi-Hydroxystaurosporine) es un inhibidor selectivo de la proteÍna quinasa C (PKC) producido por la cepa N-12 de Streptomyces, con IC50 de 62 nM y 250 nM para PKC y proteÍna quinasa A (PKA), respectivamente. UCN-02 (7-epi-Hydroxystaurosporine) muestra un efecto citotÓxico en el crecimiento de las células HeLa S3.
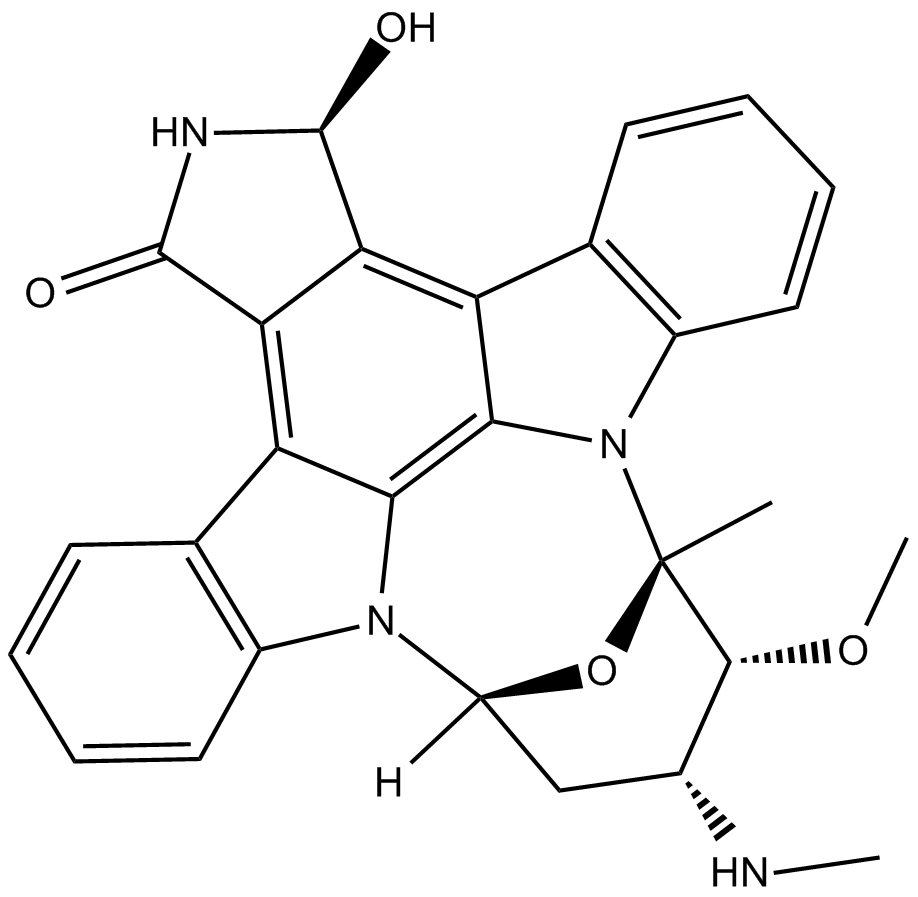
-
GC37880
Vactosertib Hydrochloride
El clorhidrato de vactosertib (clorhidrato EW-7197) es un potente inhibidor de la quinasa 5 similar al receptor de activina (ALK5) activo por vÍa oral y competitivo con ATP con una IC50 de 12,9 nM. El clorhidrato de vactosertib también inhibe ALK2 y ALK4 (IC50 de 17,3 nM) en concentraciones nanomolares. El clorhidrato de vactosertib tiene una potente actividad antimetastÁsica y un efecto anticancerÍgeno.
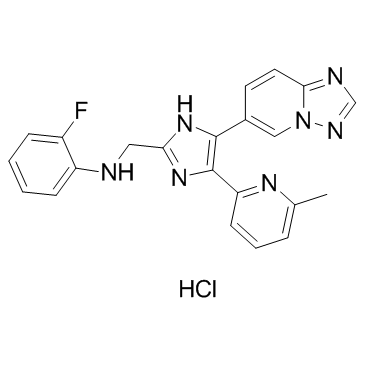
-
GC16934
Valrubicin
La valrubicina es un agente de quimioterapia, inhibe la activaciÓn de PKC inducida por TPA y PDBu con IC50 de 0,85 y 1,25 μM, respectivamente, y tiene actividad antitumoral y antiinflamatoria.
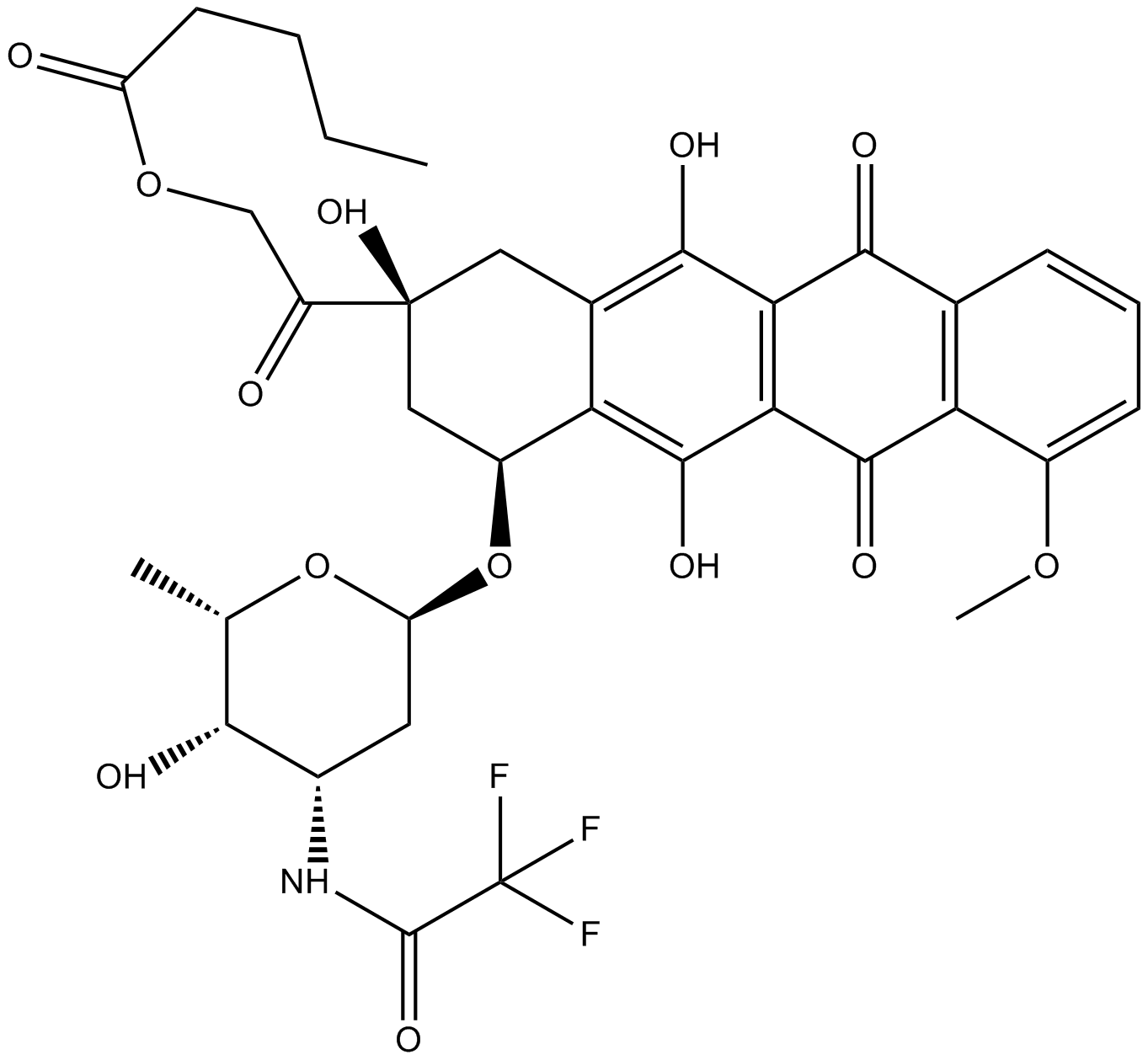
-
GC70110
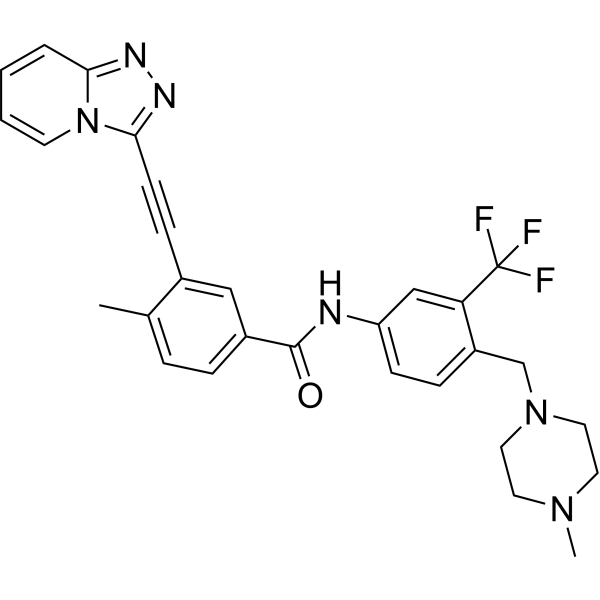
Vamotinib
Vamotinib (PF-114) es un inhibidor de la tirosina quinasa efectivo, selectivo y con actividad oral. Vamotinib inhibe la autofosforilación de BCR/ABL y BCR/ABL-T315I. Vamotinib induce apoptosis celular. Vamotinib muestra actividad antiproliferativa y antitumoral. Vamotinib tiene potencial en la investigación de leucemia mieloide crónica resistente al cromosoma Filadelfia positivo (Ph+).
-
GC13232
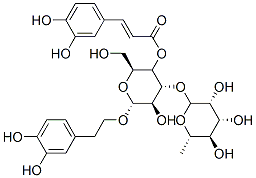
Verbascoside
El verbascoside se aÍsla de Acanthus mollis, actÚa como un inhibidor de PKC competitivo con ATP, con un IC50 de 25 μM, y tiene actividad antitumoral, antiinflamatoria y antineuropÁtica.
-
GC31912
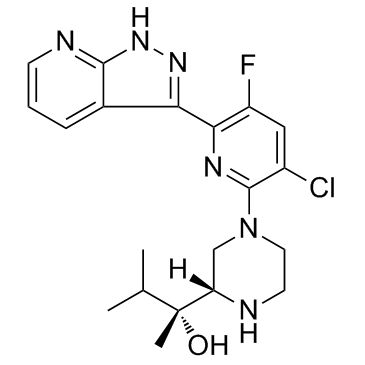
VTX-27
VTX-27 es un inhibidor selectivo de la proteÍna quinasa C θ (PKC θ), con Kis de 0,08 nM y 16 nM para PKC θ y PKC δ.
-
GC26055
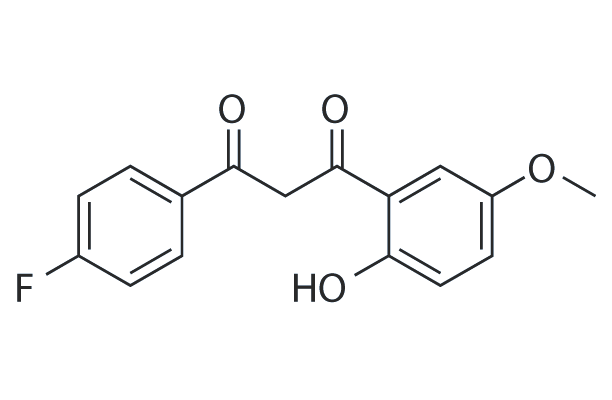
WAY-301398
WAY-301398 (6-methoxynaphthalene) can bind to protein kinase C (PKC).
-
GC26065
WAY-354831
WAY-354831 exhibits antibacterial activity.
-
GC10970
WP1130
WP1130 (WP1130) es un inhibidor de la deubiquitinasa permeable a las células (DUB), que inhibe directamente la actividad DUB de USP9x, USP5, USP14 y UCH37. Se ha demostrado que WP1130 regula a la baja las proteÍnas antiapoptÓticas Bcr-Abl y JAK2.
-
GC62146
XST-14
XST-14 es un inhibidor de ULK1 potente, competitivo y altamente selectivo con una IC50 de 26,6 nM. XST-14 induce la inhibiciÓn de la autofagia al reducir la fosforilaciÓn del sustrato aguas abajo de ULK1. XST-14 induce la apoptosis en células de carcinoma hepatocelular (CHC) y tiene efectos antitumorales.
-
GC15712
Y-27632
Un inhibidor de ROCK, potente y selectivo.
-
GC37947
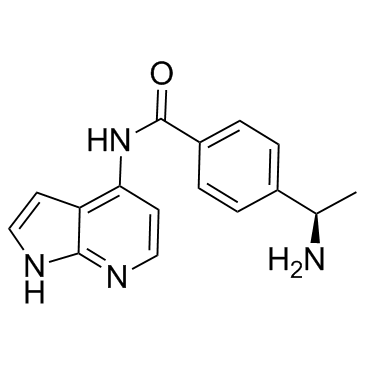
Y-33075
Y-33075 es un inhibidor selectivo de ROCK derivado de Y-27632 y es mÁs potente que Y-27632, con una IC50 de 3,6 nM.
-
GC13423
Y-39983 dihydrochloride
ROCK family inhibitor
-
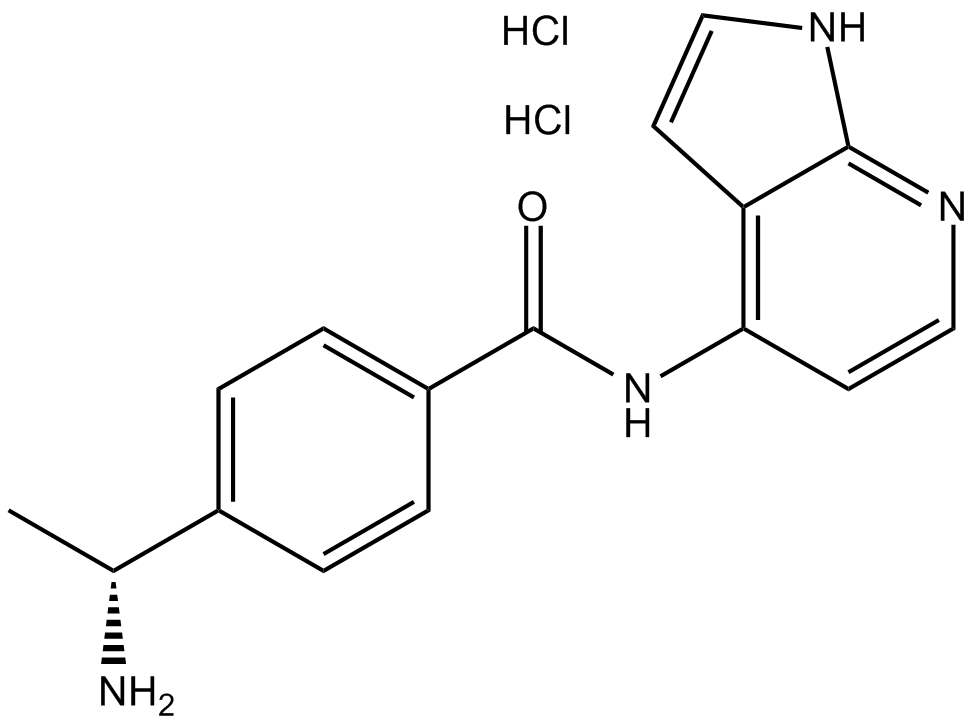
GC70166
Z-Arg-SBzl
Z-Arg-SBzl es el sustrato para la activación de la proteína C en vacas y humanos.
-
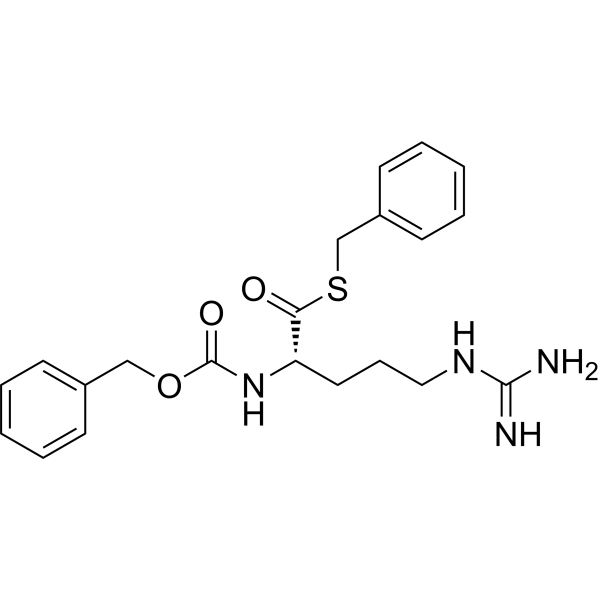
GC33120
ZINC00881524
ZINC00881524 es un inhibidor de ROCK.
-
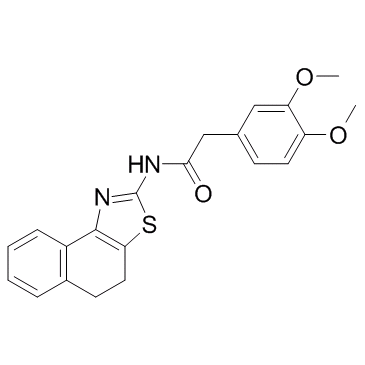
GC13273
ZIP
ZIP es un inhibidor peptÍdico selectivo de PKMζ.
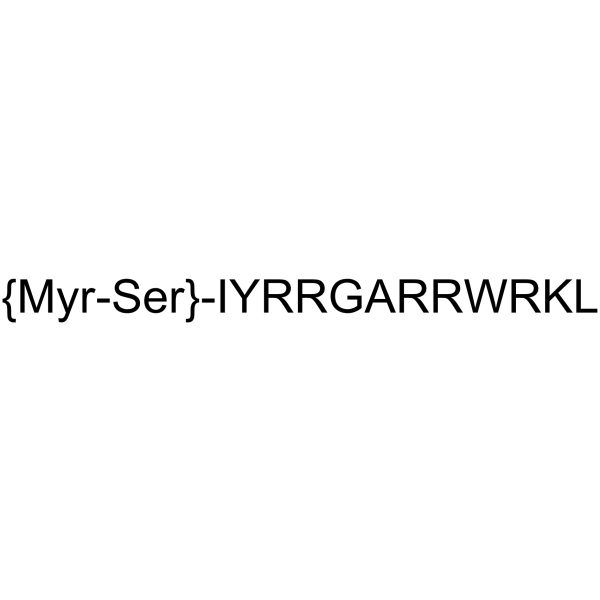
-
GC13461
ZIP (SCRAMBLED)
ZIP (SCRAMBLED) es un péptido de control codificado para el péptido inhibidor zeta (ZIP).
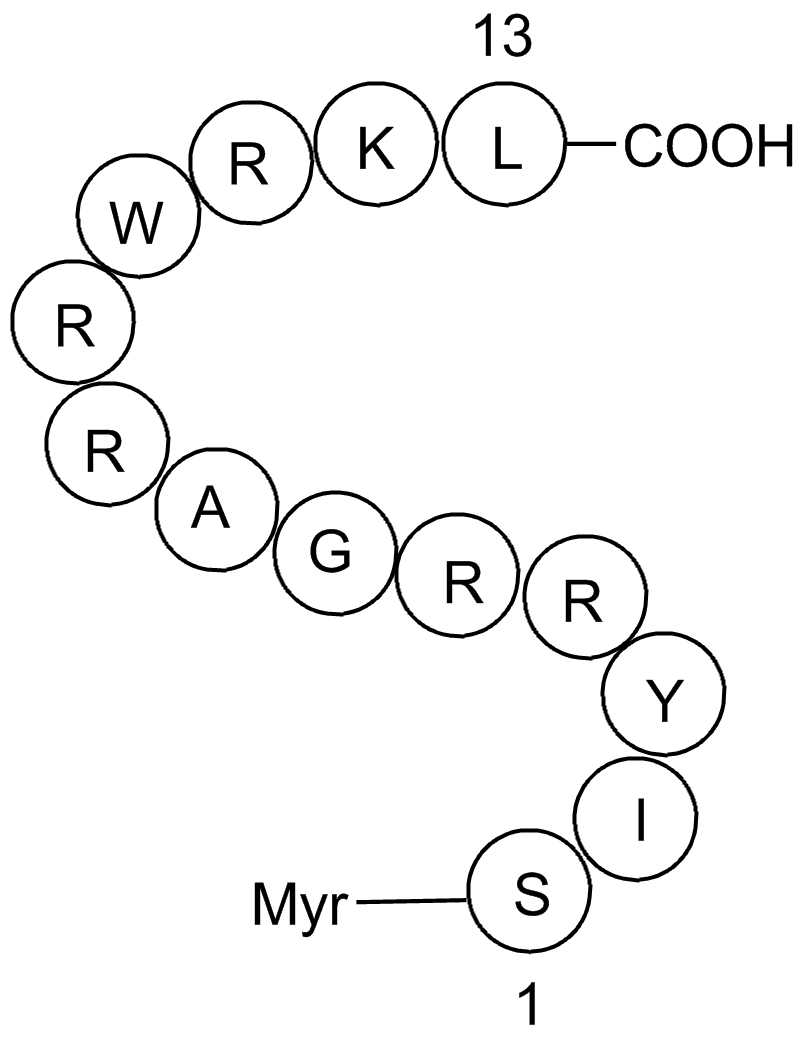
-
GC16510
ZIP, Biotinylated
ZIP with a biotin moiety covalently attached
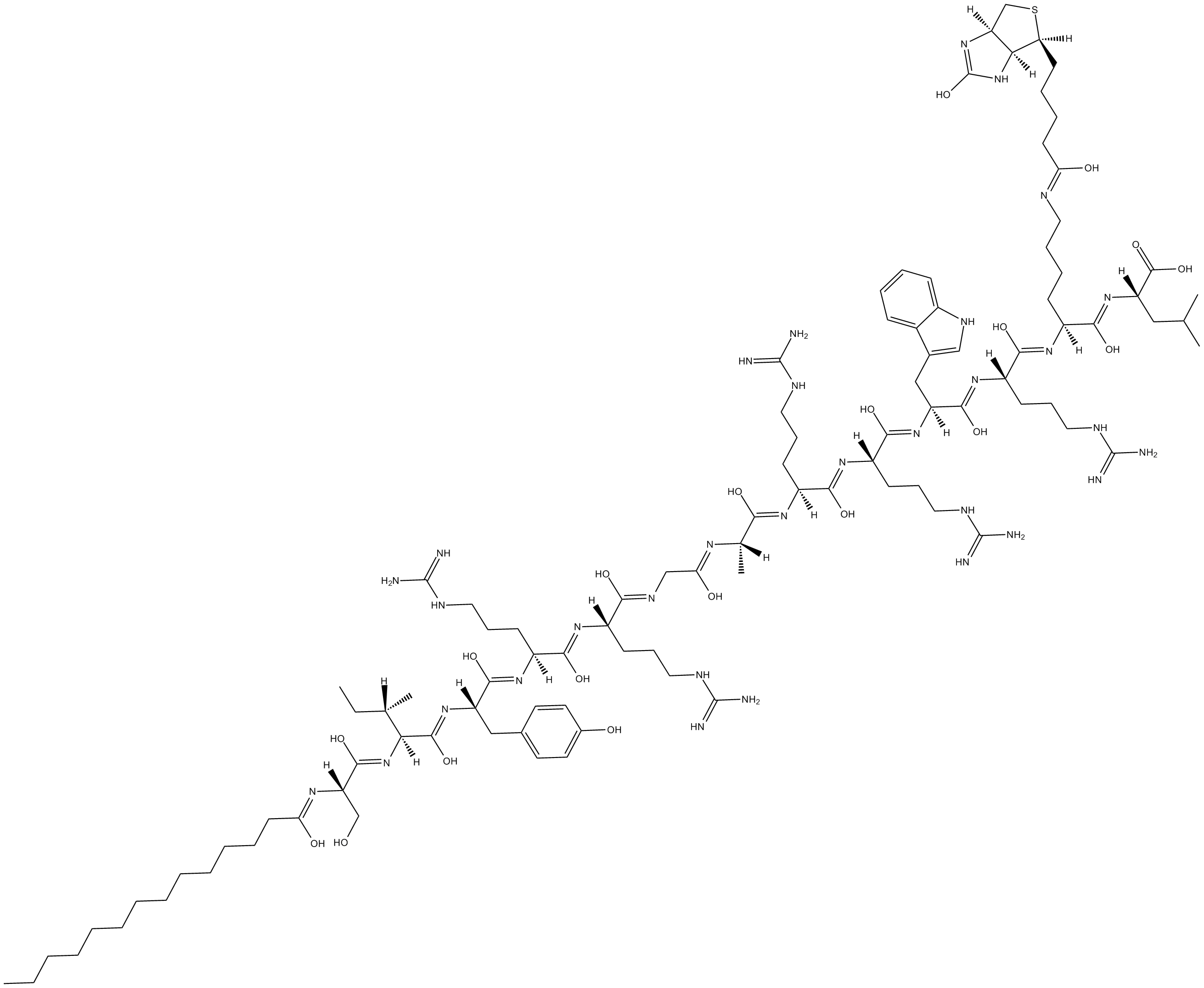
-
GC14701
Zoledronic Acid
El Ácido zoledrÓnico (zoledronato) es un bisfosfonato (BP) de tercera generaciÓn, con una potente actividad antirresortiva.
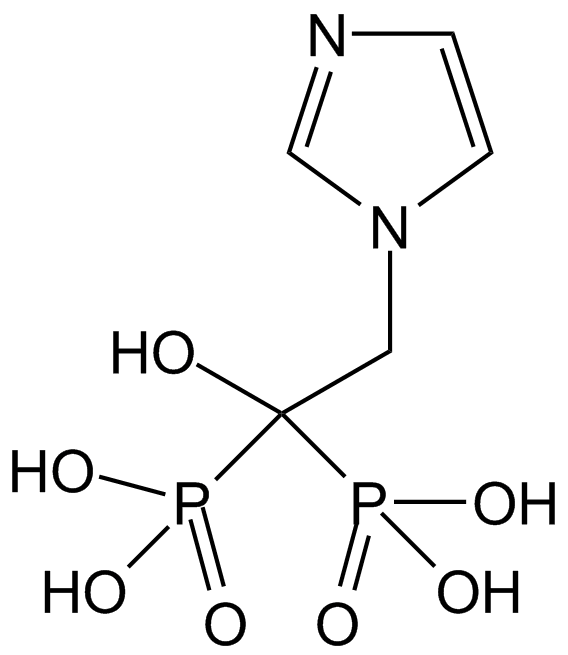
-
GC17727
[Ala107]-MBP (104-118)
[Ala107]-MBP (104-118) es un inhibidor peptídico no competitivo de la proteína quinasa C (PKC), con una IC50 que oscila entre 46 y 145 μM.
![[Ala107]-MBP (104-118) Chemical Structure [Ala107]-MBP (104-118) Chemical Structure](/media/struct/GC1/GC17727.png)
-
GC15345
[Ala113]-MBP (104-118)
[Ala113]-MBP (104-118) es un inhibidor peptídico no competitivo de la proteína quinasa C (PKC), con una IC50 que oscila entre 28 y 62 μM.
![[Ala113]-MBP (104-118) Chemical Structure [Ala113]-MBP (104-118) Chemical Structure](/media/struct/GC1/GC15345.png)
-
GC10343
α-Amyloid Precursor Protein Modulator
α-El modulador de proteÍna precursora de amiloide es un activador de proteÍna quinasa C (PKC) derivado de benzolactama permeable a las células con una Ki de 11,9 nM.
-
GC62478
ζ-Stat
ζ-Stat (NSC37044) es un inhibidor de PKC-ζ especÍfico y atÍpico, con una IC50 de 5 μM. ζ-Stat puede reducir la proliferaciÓn de lÍneas celulares de melanoma e inducir la apoptosis, y tiene actividad antitumoral in vitro.