Ubiquitination/ Proteasome
- Autophagy(1071)
- DUB(20)
- E1 Activating(1)
- E2 conjugating(1)
- E2 Ligases(0)
- E3 Ligase(8)
- Proteasome(96)
- p97(11)
- Mitophagy(77)
- ULK(9)
Produkte für Ubiquitination/ Proteasome
- Bestell-Nr. Artikelname Informationen
-
GN10779
Reserpine
NSC 59272, NSC 237659, Rausedil
-
GC14553
Resveratrol
(E)Resveratrol
Ein Polyphenol mit vielfältigen biologischen Aktivitäten.
-
GC14651
Reversine
A purine derivative
-
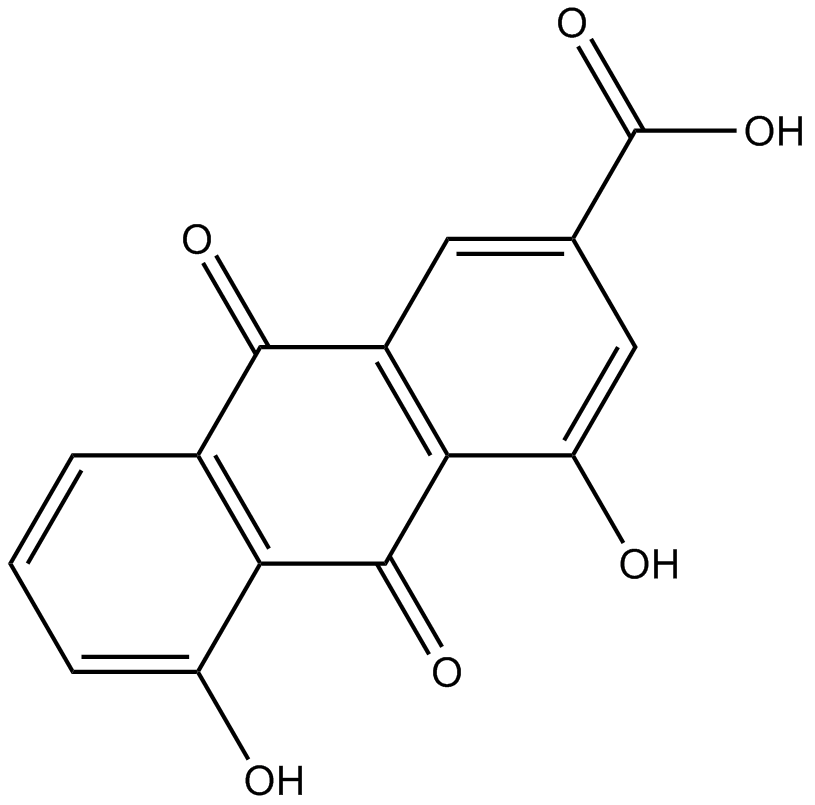
GN10475
Rhein
NSC 38629, Rheic Acid
-
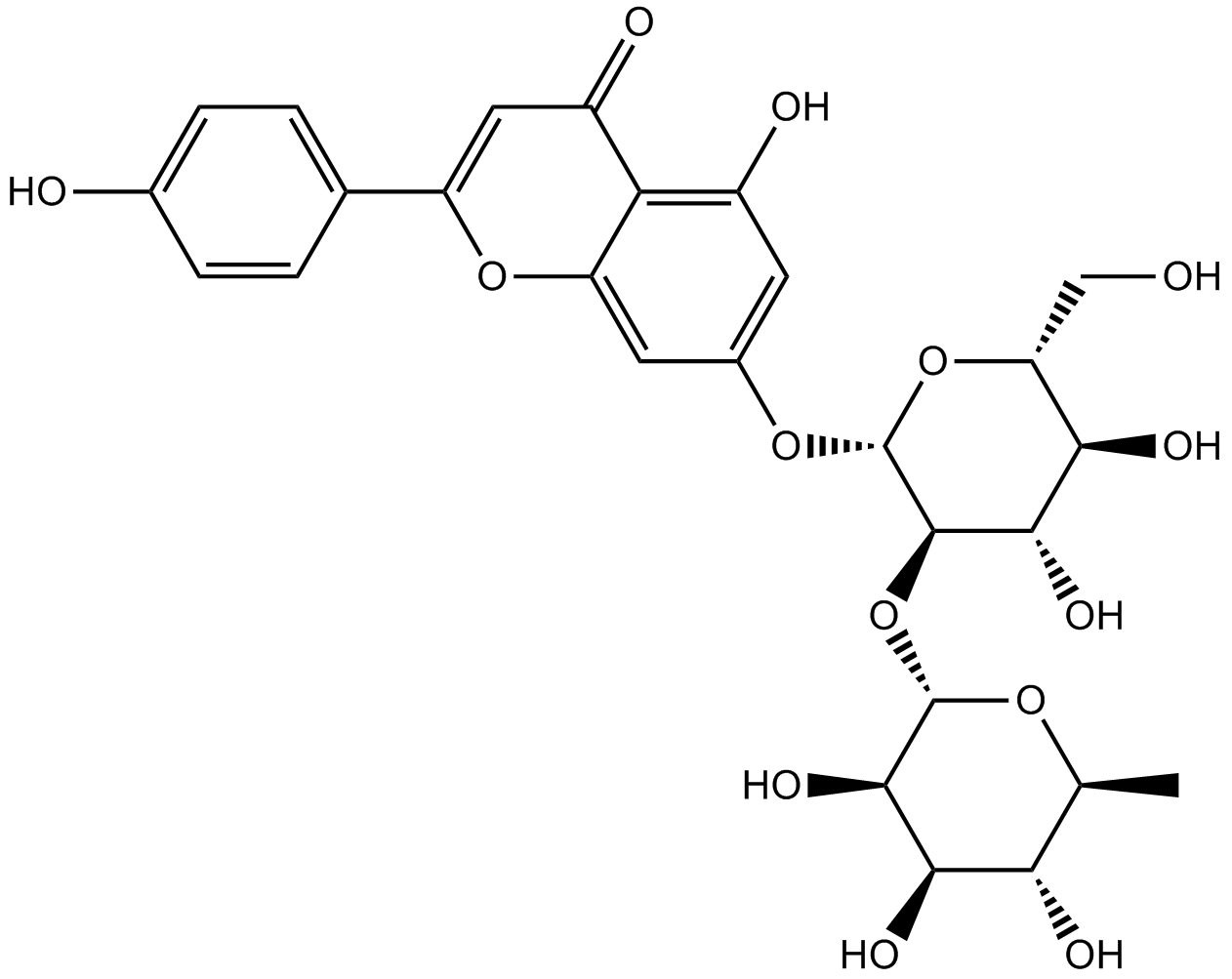
GN10784
Rhoifolin
Apigenin 7-O-Neohesperidoside
-
GC13071
Ridaforolimus (Deforolimus, MK-8669)
AP23573, Deforolimus, Ridaforolimus
Ridaforolimus (Deforolimus, MK-8669) (MK-8669) ist ein potenter und selektiver mTOR-Inhibitor; hemmt die Phosphorylierung des ribosomalen Proteins S6 mit einem IC50 von 0,2 nM in HT-1080-Zellen.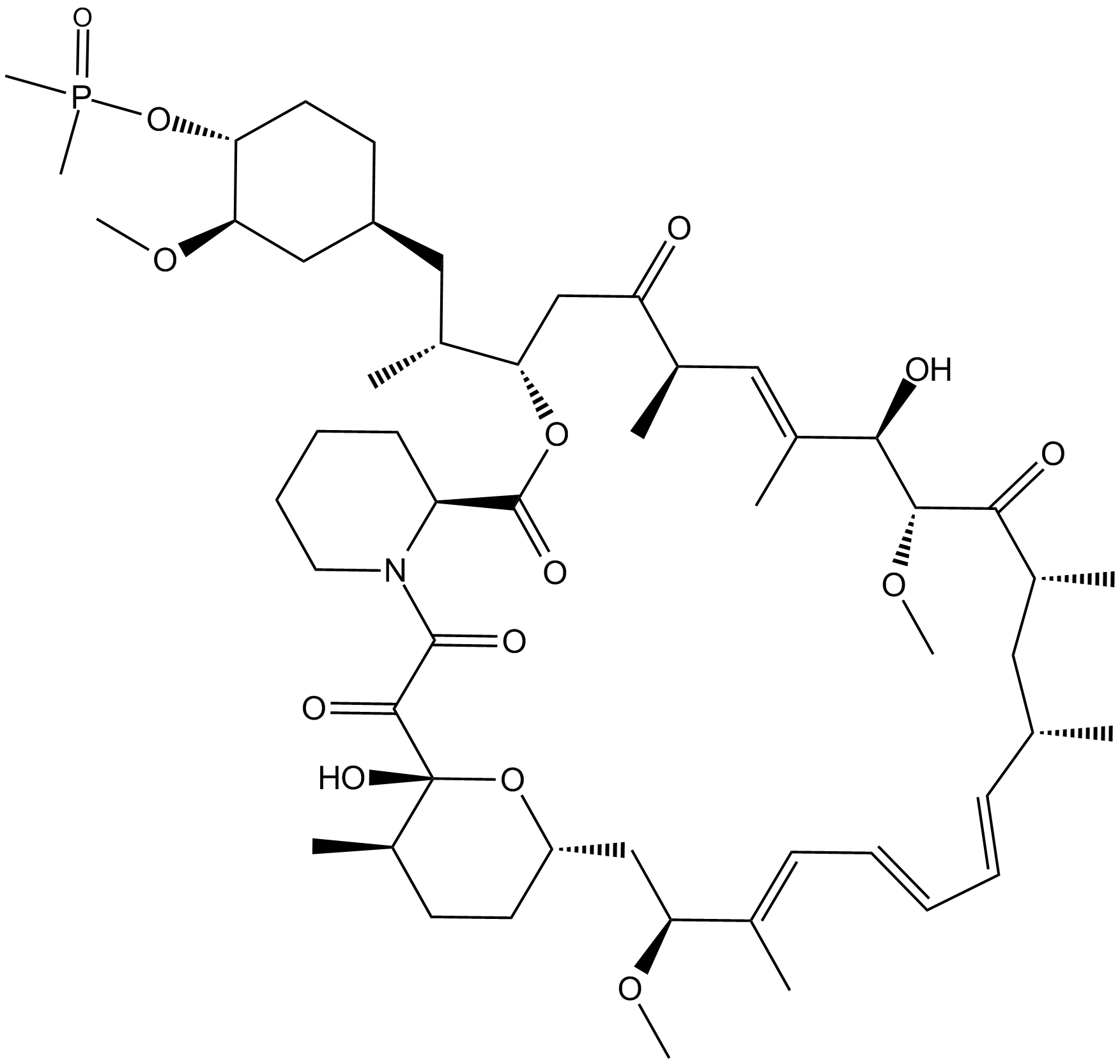
-
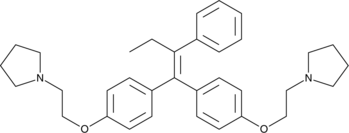
GC44839
Ridaifen-B
An analog of tamoxifen
-
GC60325
Rilmenidine
Oxaminozoline, NSC 664312
Rilmenidin, ein innovatives Antihypertonikum, ist ein oral aktiver, selektiver I1-Imidazolin-Rezeptor-Agonist.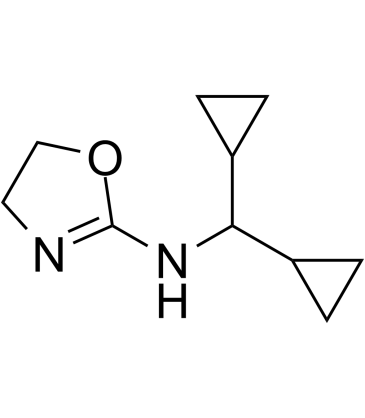
-
GC48052
Rilmenidine-d4
Oxaminozoline-d4
An internal standard for the quantification of rilmenidine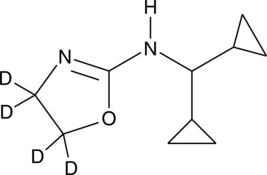
-
GC33775
Rimacalib (SMP 114)
SMP 114
Rimacalib (SMP 114) (SMP 114) ist ein Ca2+/Calmodulin-abhÄngiger Proteinkinase II (CaMKII)-Inhibitor mit IC50-Werten von ~1 μ M fÜr CaMKIIα bis ~30 μM fÜr CaMKIIγ.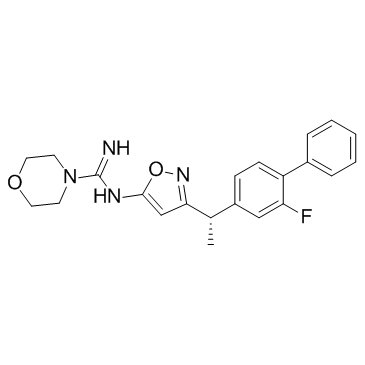
-
GC12793
RITA (NSC 652287)
2,5bis(5hydroxymethyl2thienyl) Furan, NSC 652287, Reactivation of p53 and Induction of Tumor Cell Apoptosis
An inhibitor of the p53-HDM-2 interaction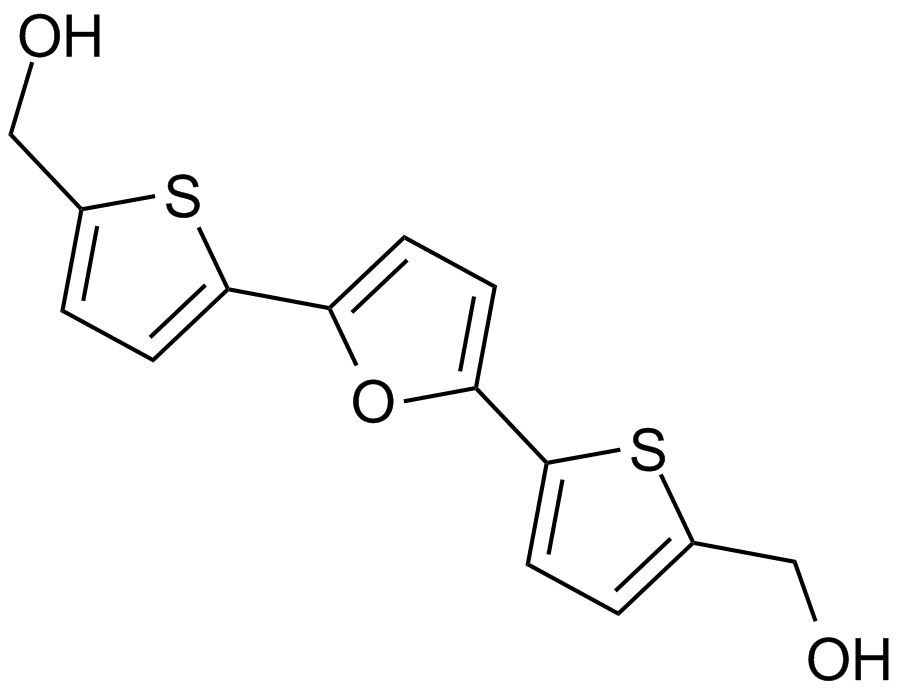
-
GC32901
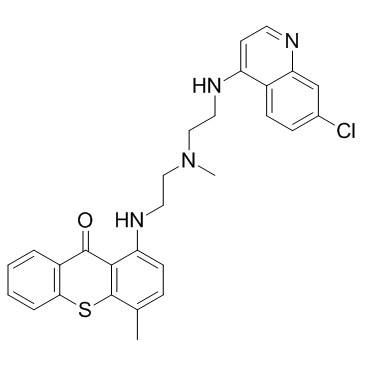
ROC-325
ROC-325 ist ein potenter und oral aktiver Autophagie-Hemmer mit einer starken Anti-Krebs-AktivitÄt. ROC-325 induziert die EntsÄuerung von Lysosomen, die Akkumulation von Autophagosomen und einen gestÖrten autophagischen Fluss. ROC-325 induziert auch die Apoptose von Nierenzellkarzinomen.
-
GC14998
Rocilinostat (ACY-1215)
Ricolinostat
A selective inhibitor of HDAC6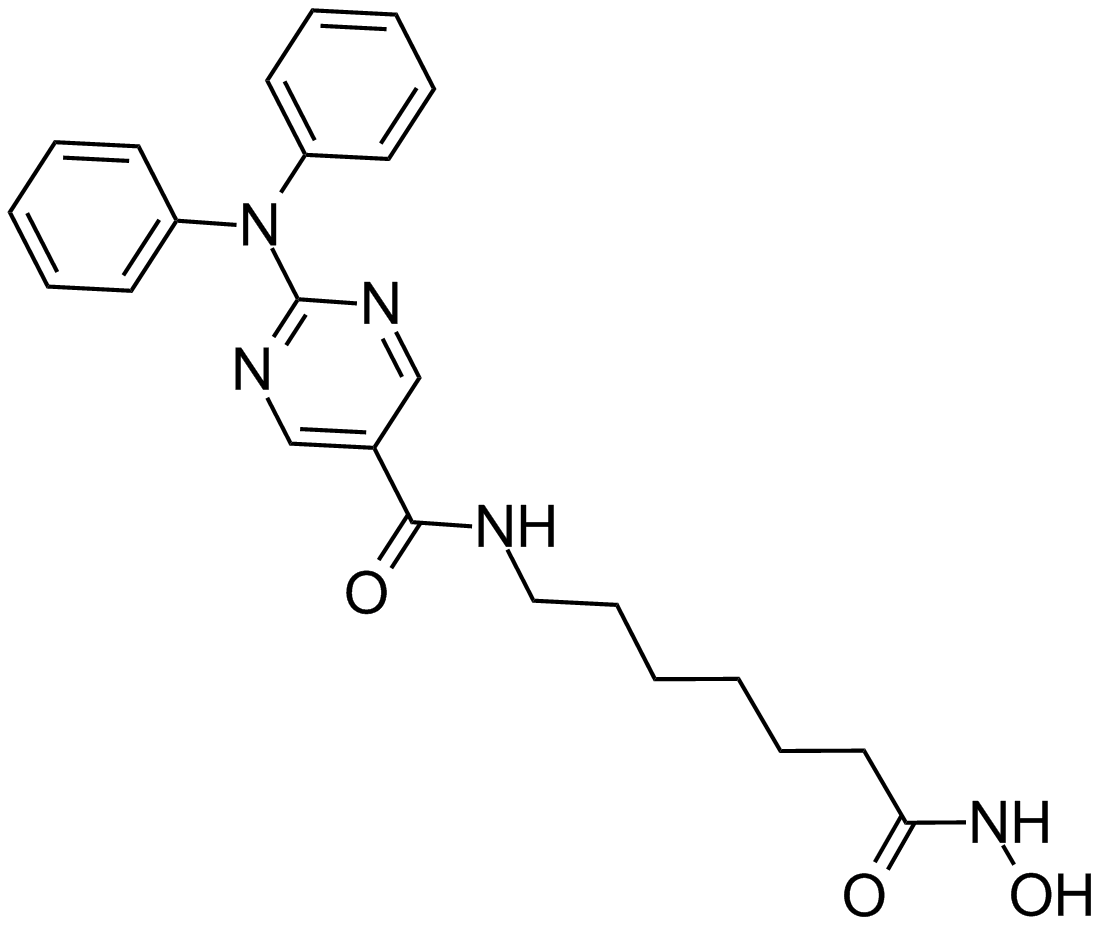
-
GC16444
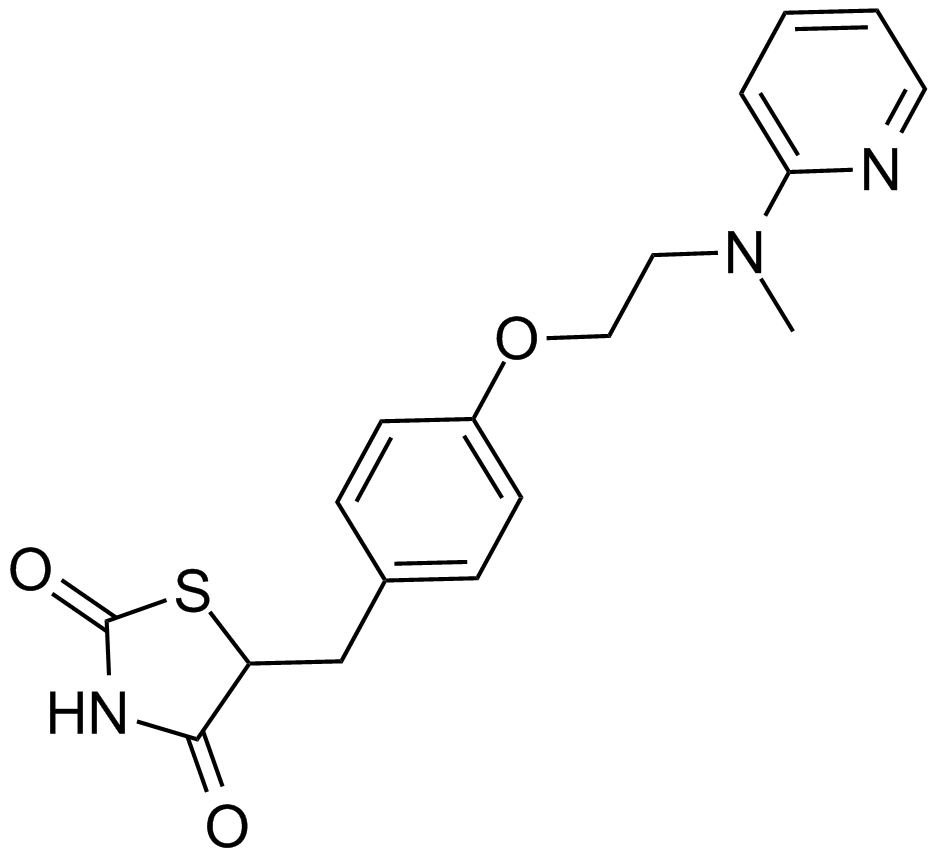
Rosiglitazone
BRL 49653
Ein PPARγ-Agonist
-
GC11318
Rosiglitazone maleate
BRL 49653C
A PPARγ agonist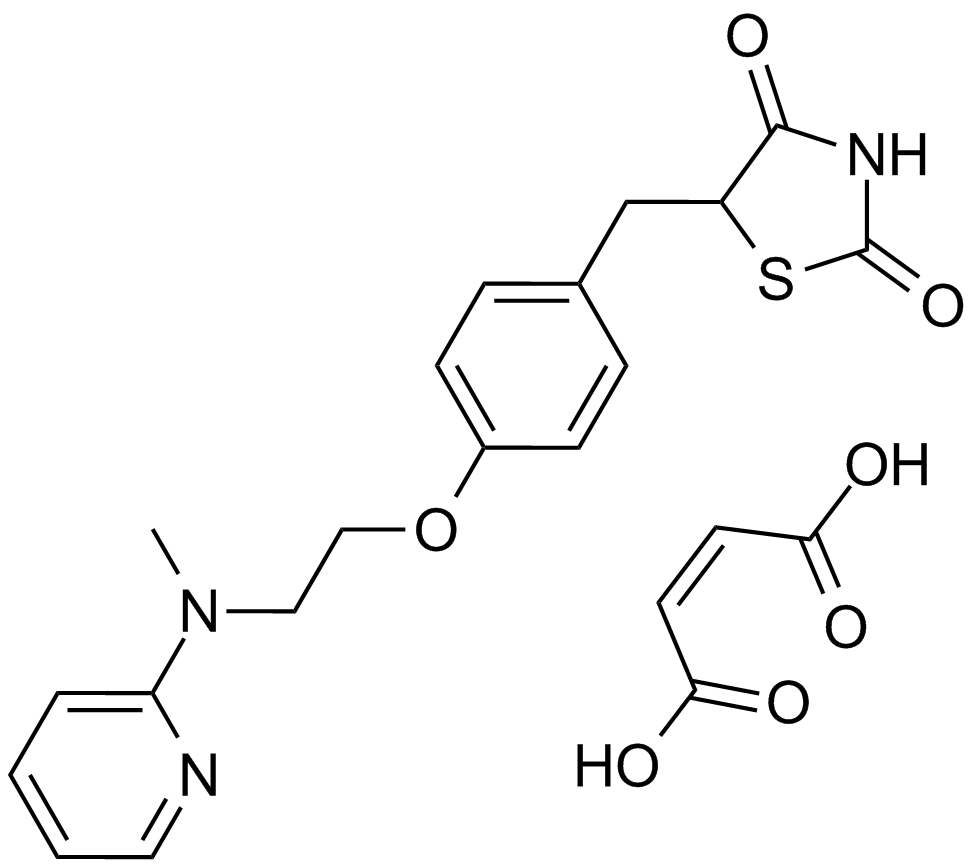
-
GC16359
Rosuvastatin
ZD 4522
Rosuvastatin (ZD 4522) ist ein kompetitiver HMG-CoA-Reduktase-Inhibitor mit einem IC50 von 11 nM.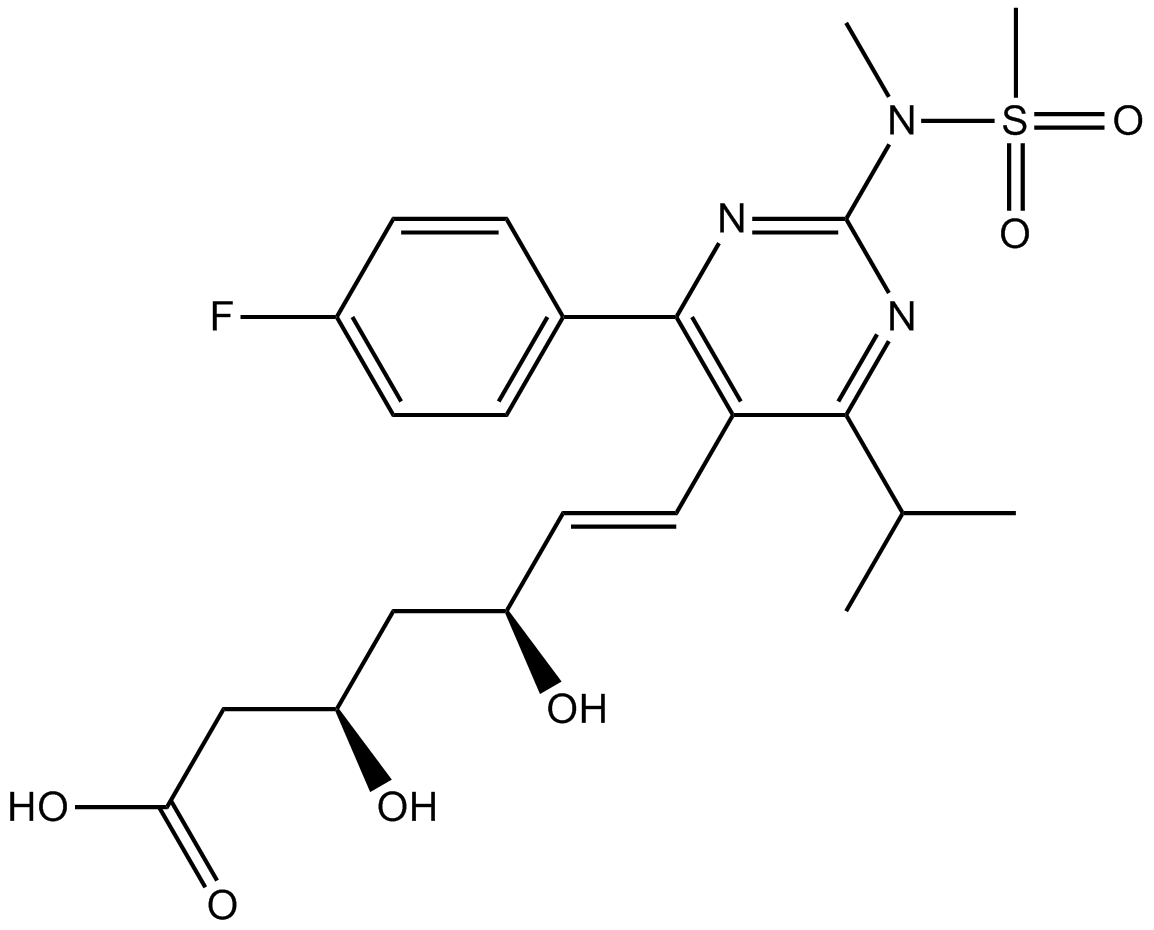
-
GC15736
Rosuvastatin Calcium
ZD 4522
Rosuvastatin Calcium ist ein kompetitiver HMG-CoA-Reduktase (HMGCR)-Hemmer mit einem IC50-Wert von 11 nM.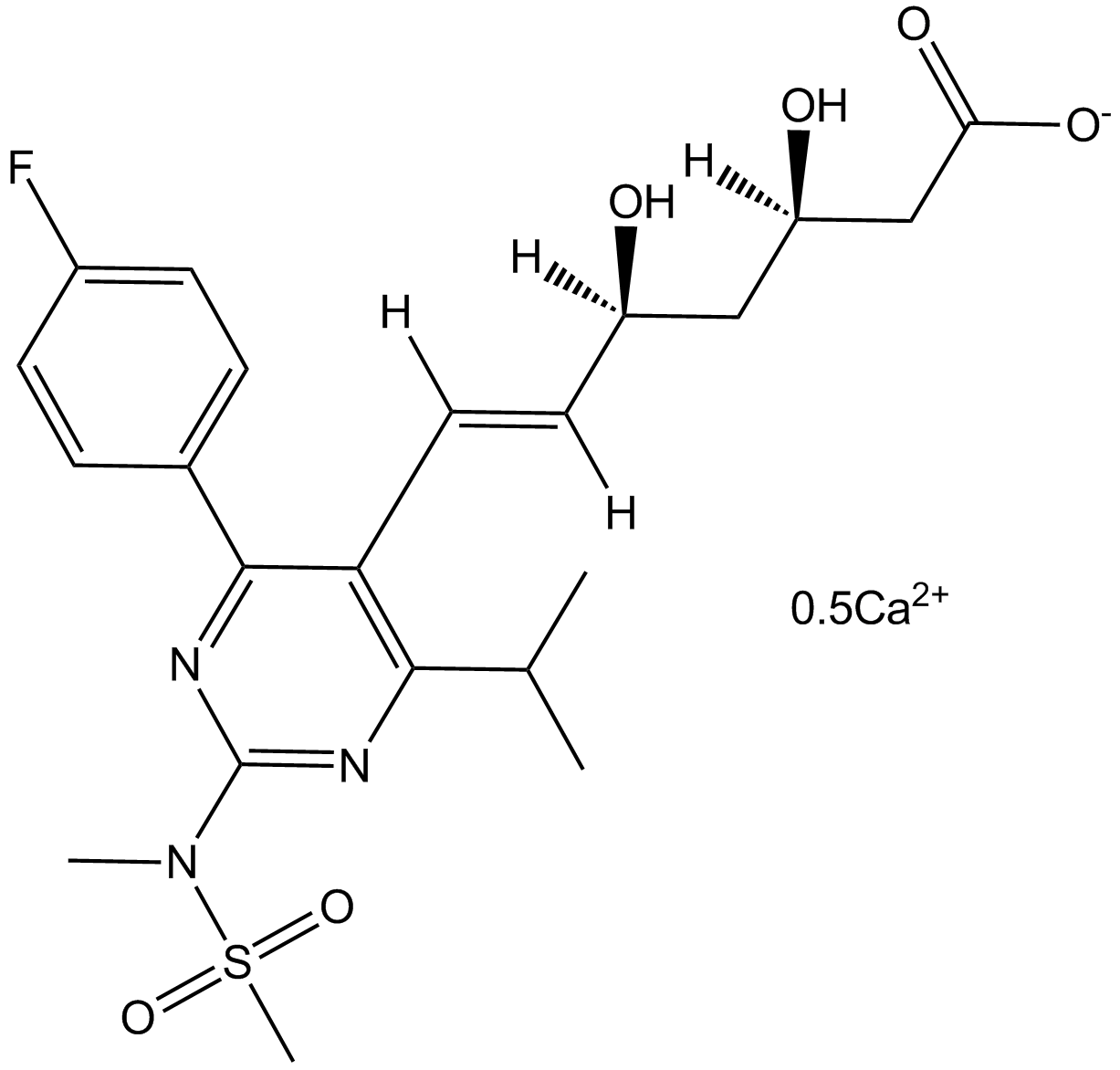
-
GC61251
Rosuvastatin D3
ZD 4522 D3
Rosuvastatin D3 (ZD 4522 D3) ist ein mit Deuterium gekennzeichnetes Rosuvastatin.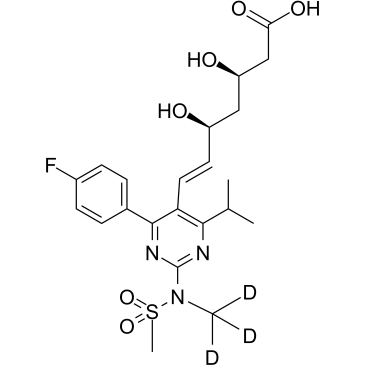
-
GC37559
Rosuvastatin D3 Sodium
Rosuvastatin D3 Natrium ist mit Deuterium markiertes Rosuvastatin, das ein kompetitiver Inhibitor der HMG-CoA-Reduktase mit einem IC50-Wert von 11 nM ist.
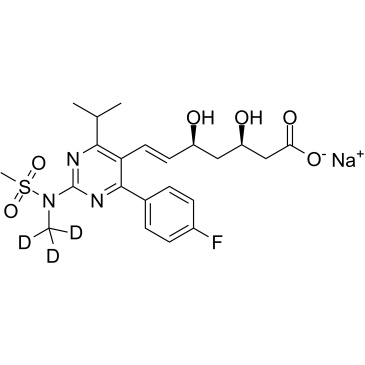
-
GC37560
Rosuvastatin D6 Calcium
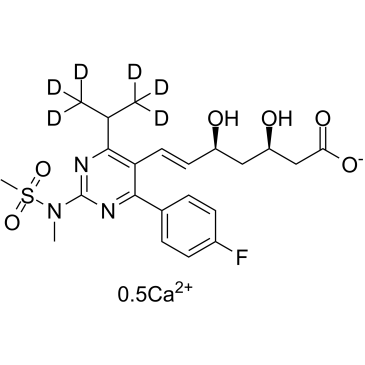
-
GC37561
Rosuvastatin D6 Sodium
Rosuvastatin D6 Natrium ist mit Deuterium markiertes Rosuvastatin, das ein kompetitiver Inhibitor der HMG-CoA-Reduktase mit einem IC50-Wert von 11 nM ist.
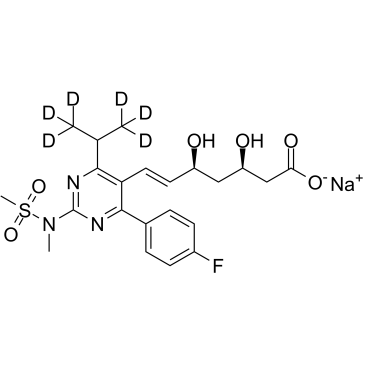
-
GC16775
Rotenone
Nicouline, NSC 8505, NSC 26258, Tubatoxin
Inhibitor der Elektronentransportkette des mitochondrialen Komplexes I
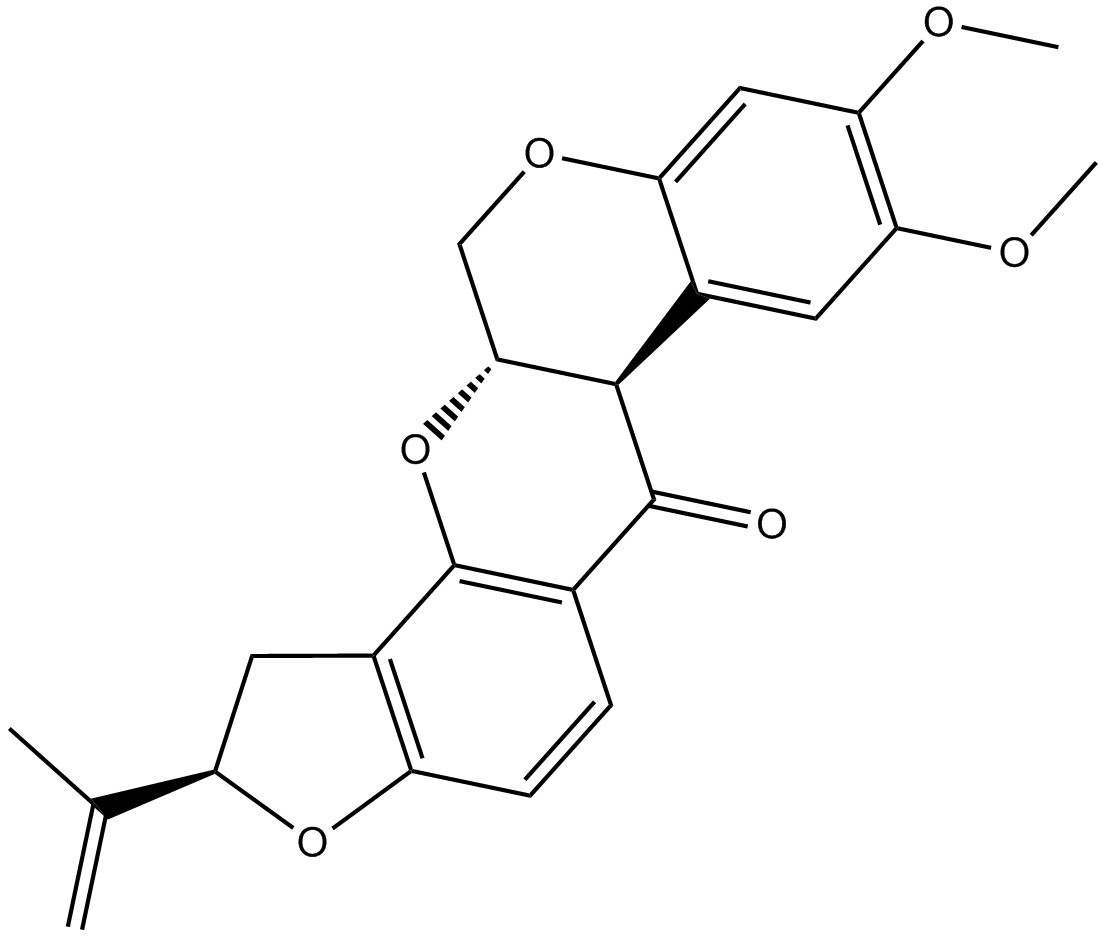
-
GC10820
Rottlerin
Mallotoxin, NSC 94525, NSC 56346
Rottlerin, ein aus Mallotus Philippinensis gereinigtes Naturprodukt, ist ein spezifischer PKC-Inhibitor mit IC50-Werten fÜr PKCδ von 3-6 μM, PKCα,β,γ von 30-42 μM, PKCε,η,ζ von 80-100 μM.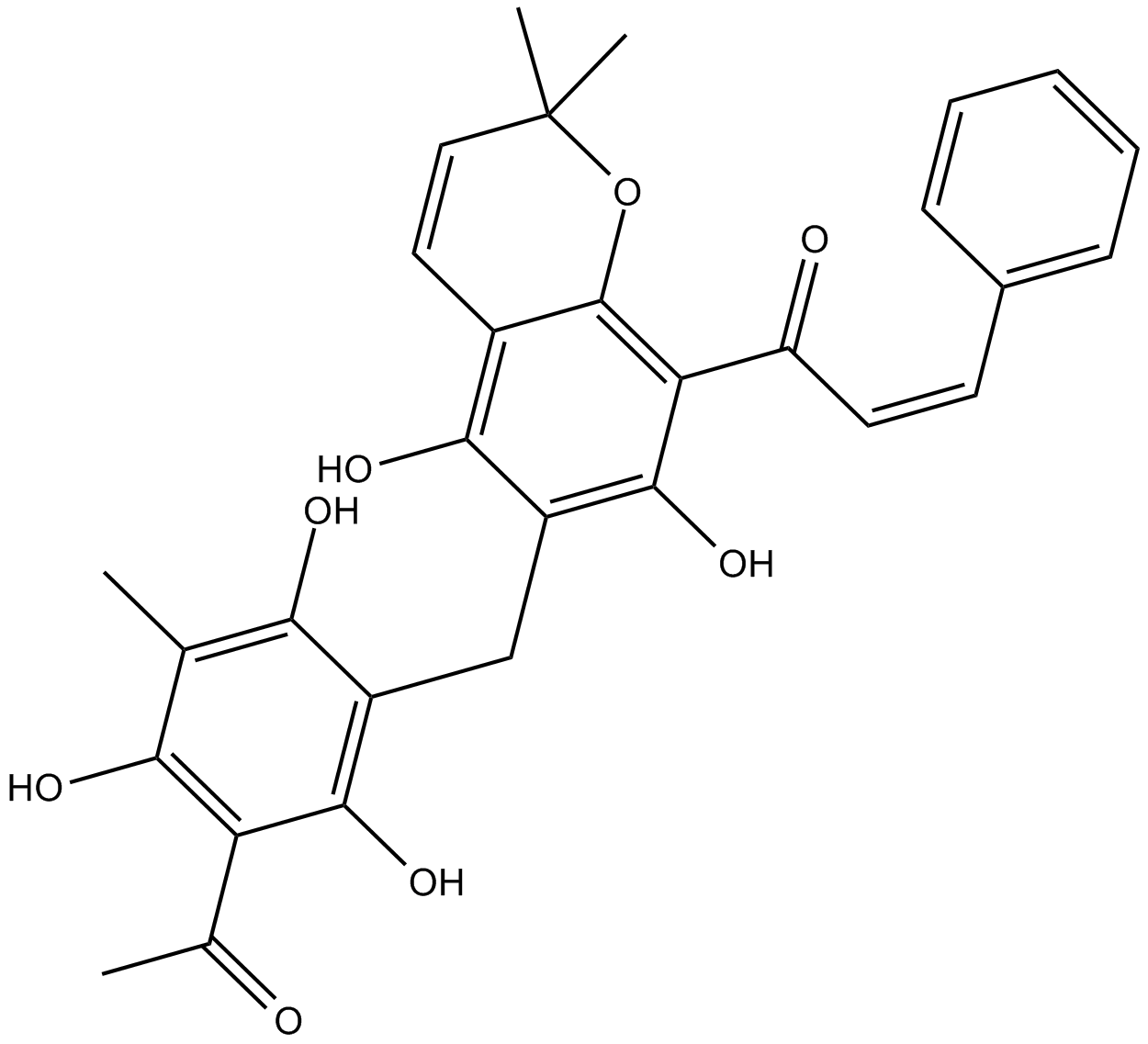
-
GC32407
Rpn11-IN-1
Capzimin intermediate
Rpn11-IN-1 (Capzimin-Zwischenprodukt) ist ein potenter und selektiver Inhibitor der Proteasom-Untereinheit Rpn11 mit einem IC50 von 390 nM.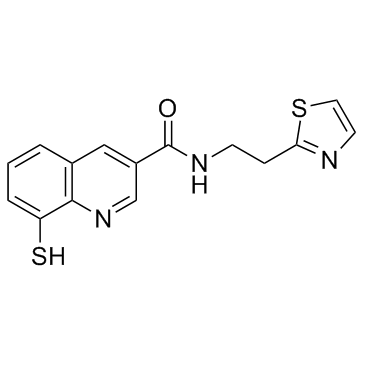
-
GC61256
Rupatadine D4 fumarate
UR-12592 D4 fumarate
Rupatadin-D4-Fumarat (UR-12592 D4-Fumarat) ist ein Deuterium-markiertes Rupatadin-Fumarat.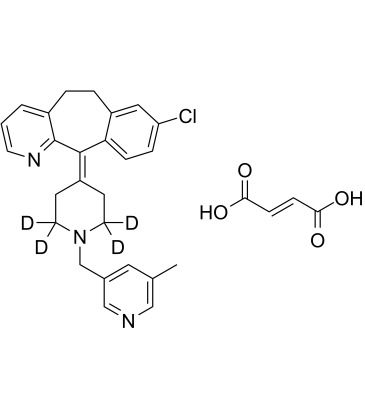
-
GC11033
Rupatadine Fumarate
Rupatadin (UR-12592) Fumarat ist ein potenter, oral aktiver und lang anhaltender dualer PAF/H1-Antagonist mit einem Kis von 0,55 μM bzw. 0,1 μM.
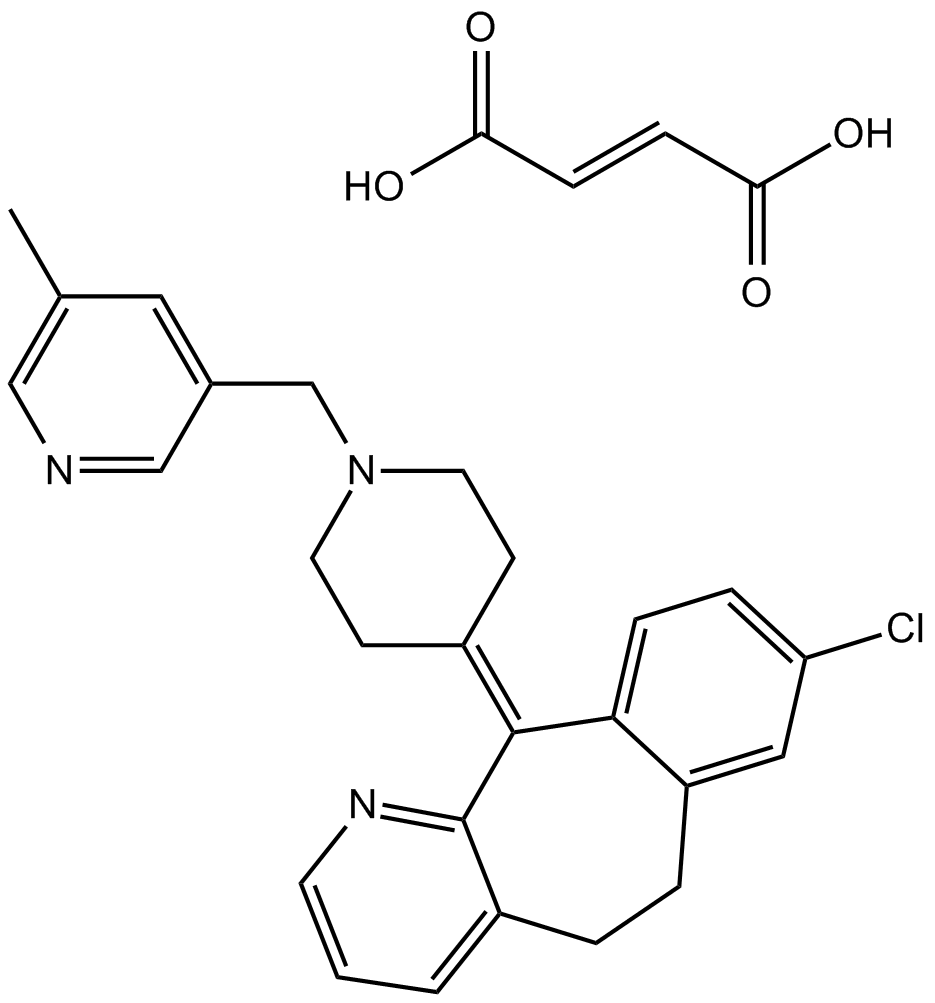
-
GN10693
Rutin
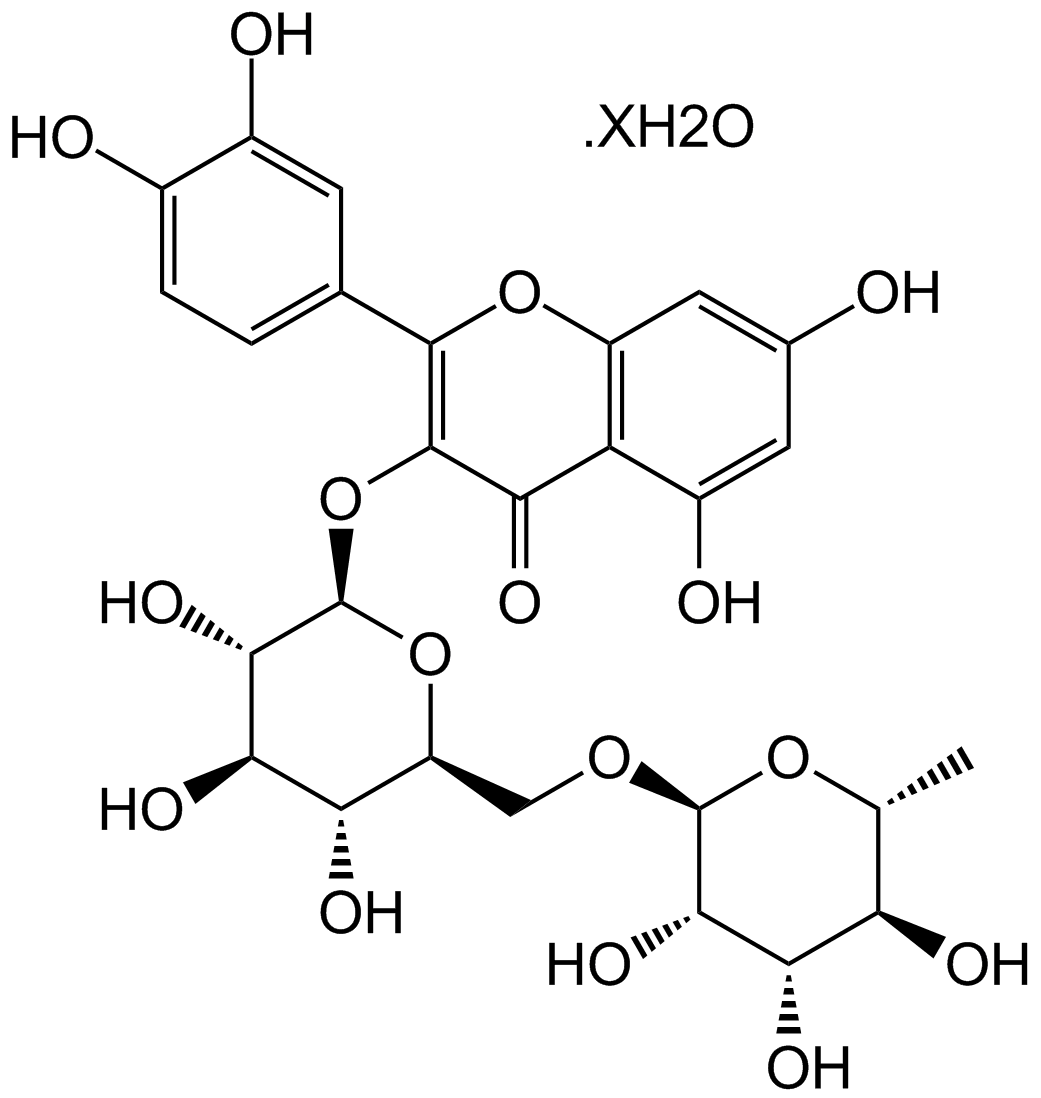
-
GC44856
Rutin (hydrate)
Quercetin-3-O-rutinoside, Rutoside, Sophorin
Rutin (Rutosid) Trihydrat ist ein multifunktionales natÜrliches Flavonoid-Glykosid.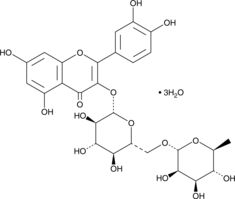
-
GC14191
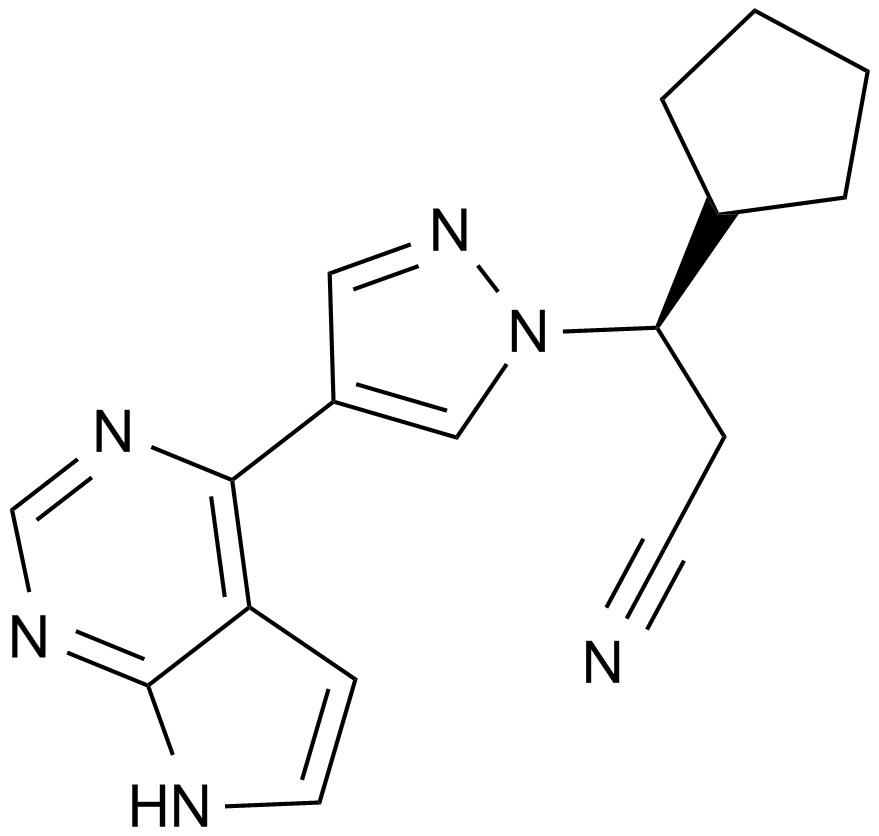
Ruxolitinib (INCB018424)
INCB 018424
Ein potenter, selektiver JAK1/JAK2-Inhibitor
-
GC16124
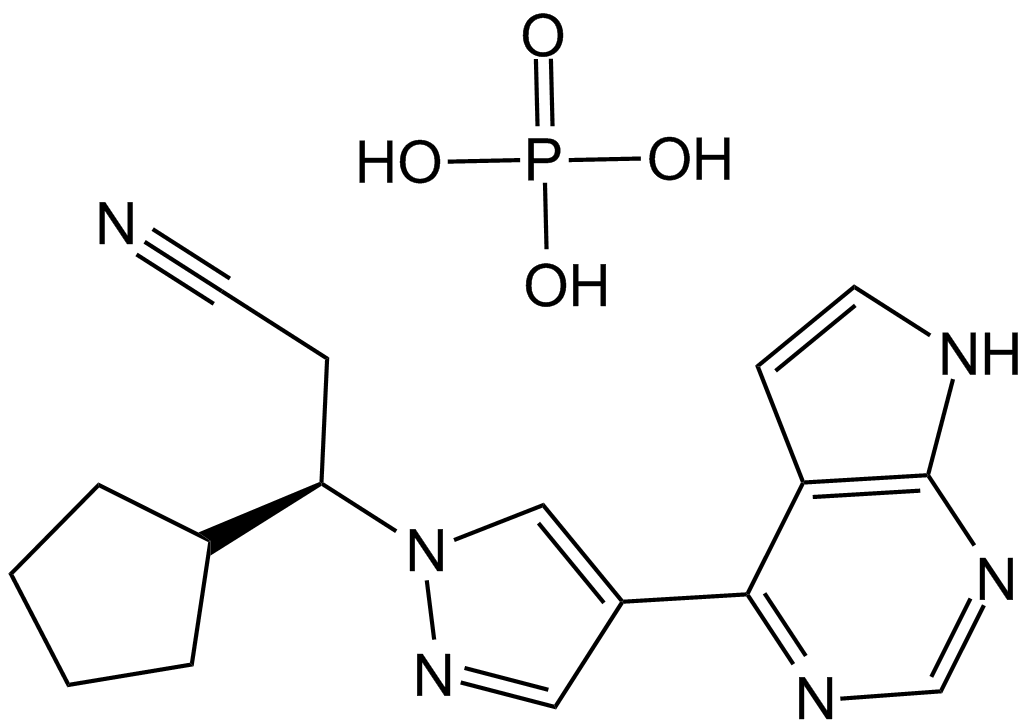
Ruxolitinib phosphate
INC 424 (phosphate), INCB 018424 (phosphate)
Ein potenter und selektiver JAK1/JAK2-Inhibitor
-
GC37575
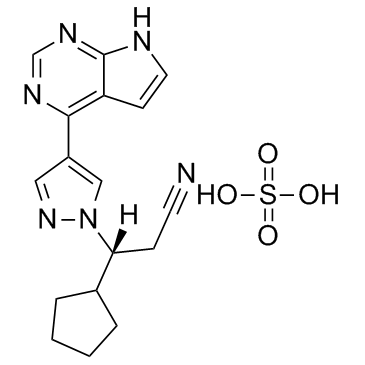
Ruxolitinib sulfate
Ruxolitinib-Sulfat (INCB018424-Sulfat) ist der erste potente, selektive JAK1/2-Inhibitor, der mit IC50-Werten von 3,3 nM/2,8 nM auf den Markt kommt, und hat eine > 130-fache SelektivitÄt fÜr JAK1/2 gegenÜber JAK3.
-
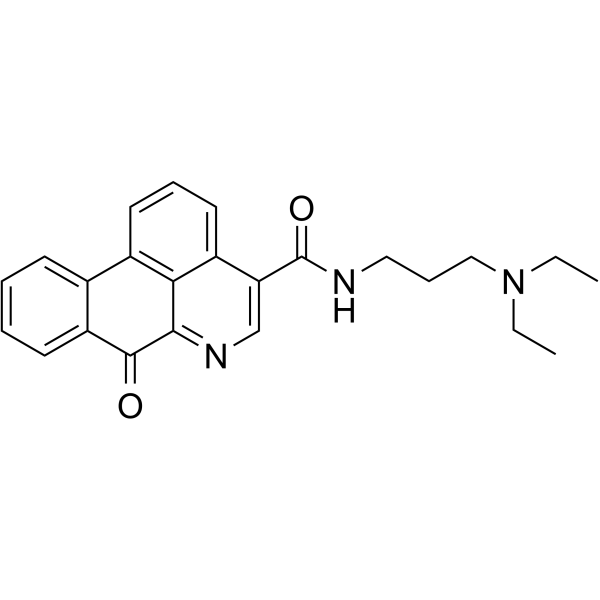
GC65307
S130
S130 ist ein hochaffiner, selektiver Inhibitor von ATG4B (einer wichtigen Cysteinprotease) mit einem IC50 von 3,24 μM. S130 unterdrÜckt den Autophagiefluss.
-
GC38451
S29434
NMDPEF
S29434 (NMDPEF) ist ein potenter, kompetitiver, selektiver und zellgÄngiger Inhibitor der Chinonreduktase 2 (QR2) mit IC50-Werten im Bereich von 5 bis 16 nM fÜr humanes QR2 auf verschiedenen Organisationsebenen und hat eine gute SelektivitÄt fÜr QR2 gegenÜber QR1.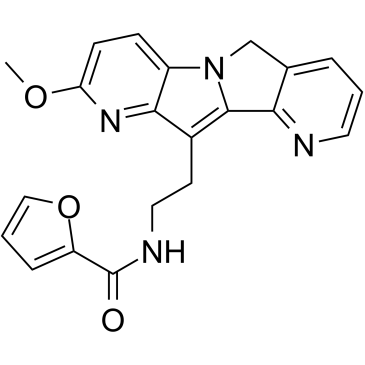
-
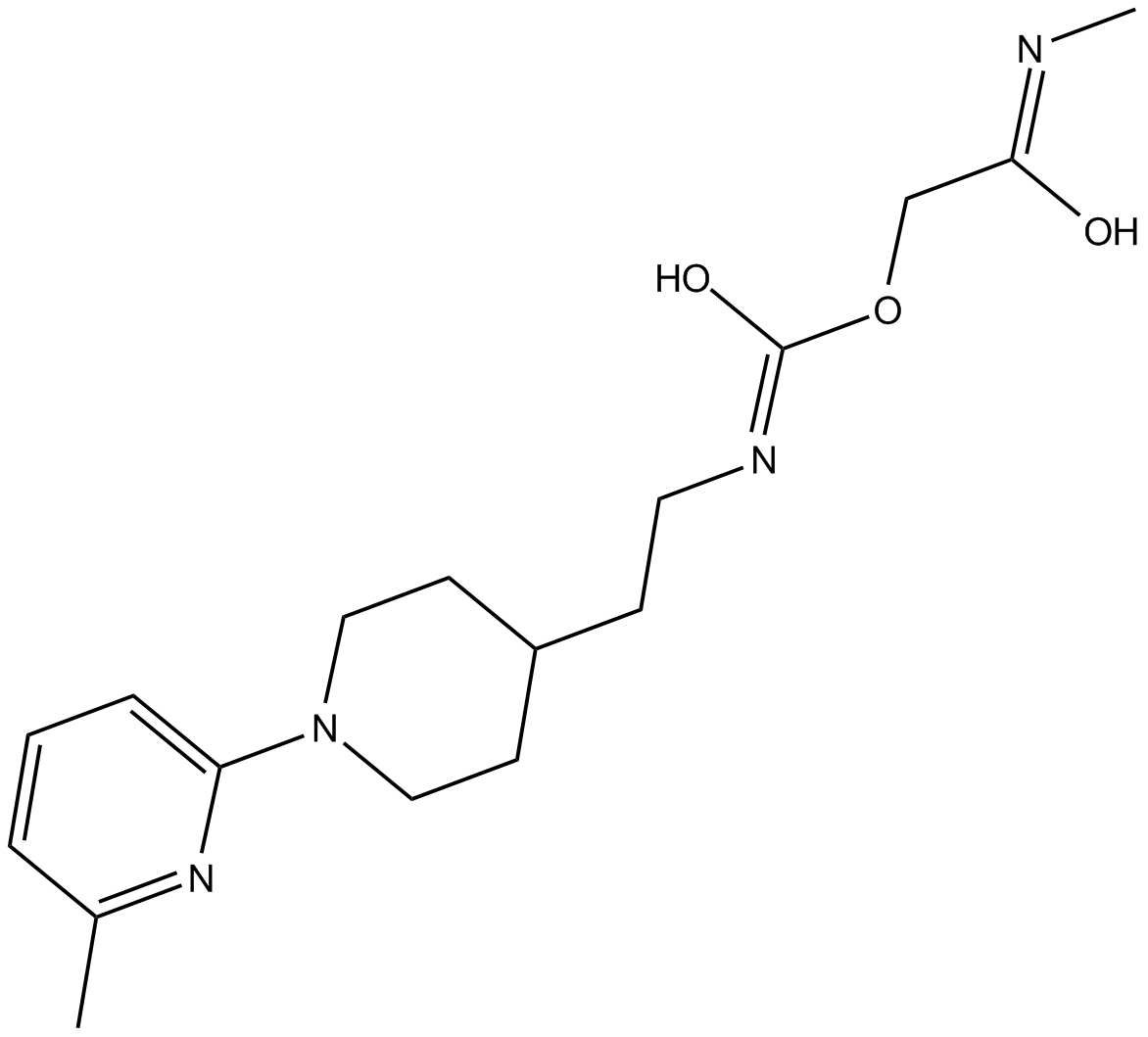
GC15077
SA 47
SA 47 ist ein selektiver und wirksamer Inhibitor von FettsÄureamidhydrolase (FAAH) und Carbamat.
-
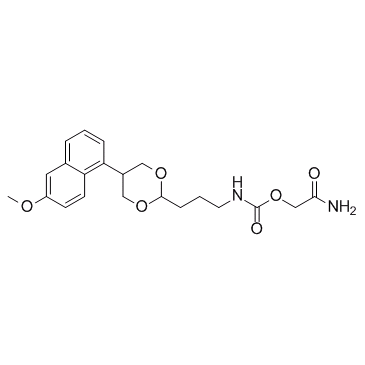
GC31239
SA72
SA72 ist ein hochselektiver Hemmer der FettsÄureamidhydrolase (FAAH).
-
GC13974
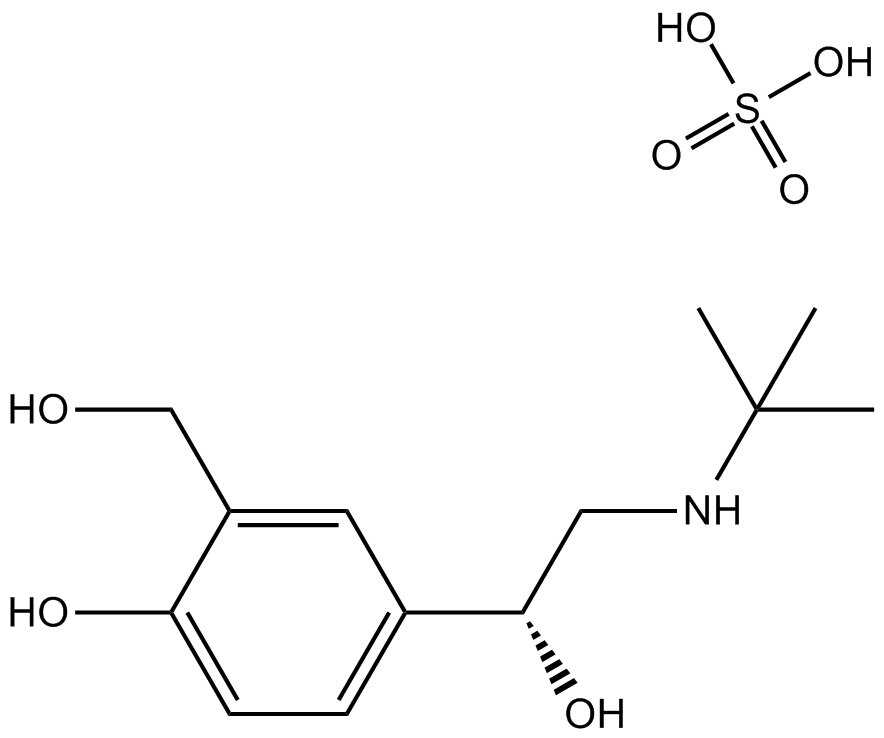
Salbutamol Sulfate
Salbutamol-Hemisulfat (Albuterol-Hemisulfat) ist ein kurz wirkender β2-adrenerger Rezeptor-AgonistTarget: &7#946;2-adrenerger RezeptorSalbutamol-Hemisulfat (Albuterol-Hemisulfat) ist ein kurz wirkender, selektiver beta2-adrenergischer Rezeptor-Agonist und wird zur Behandlung von COPD eingesetzt.
-
GC16508
Salicylic acid
2-Hydroxybenzoic Acid, NSC 180
SalicylsÄure (2-HydroxybenzoesÄure) hemmt die AktivitÄt der Cyclooxygenase-2 (COX-2) unabhÄngig von der Aktivierung des Transkriptionsfaktors (NF-κB).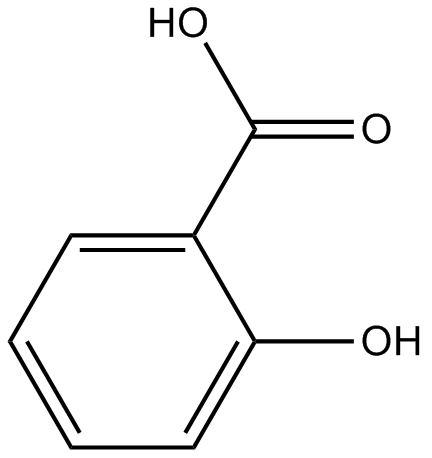
-
GC64032
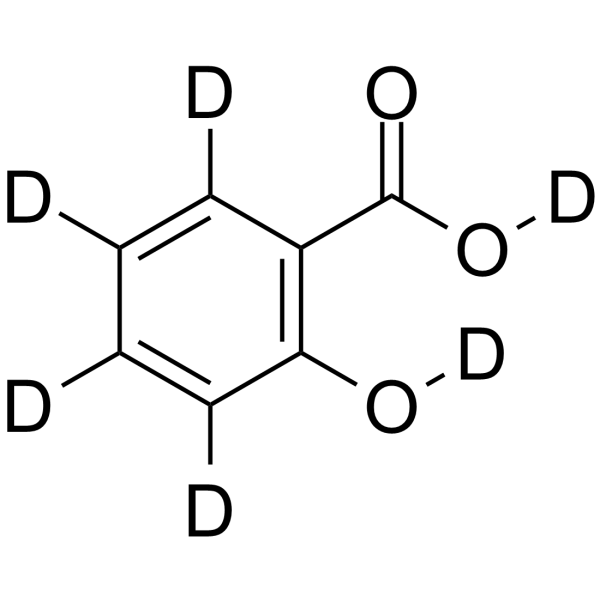
Salicylic acid-d6
SalicylsÄure-D6 (2-HydroxybenzoesÄure-D6) ist eine mit Deuterium markierte SalicylsÄure.
-
GC14882
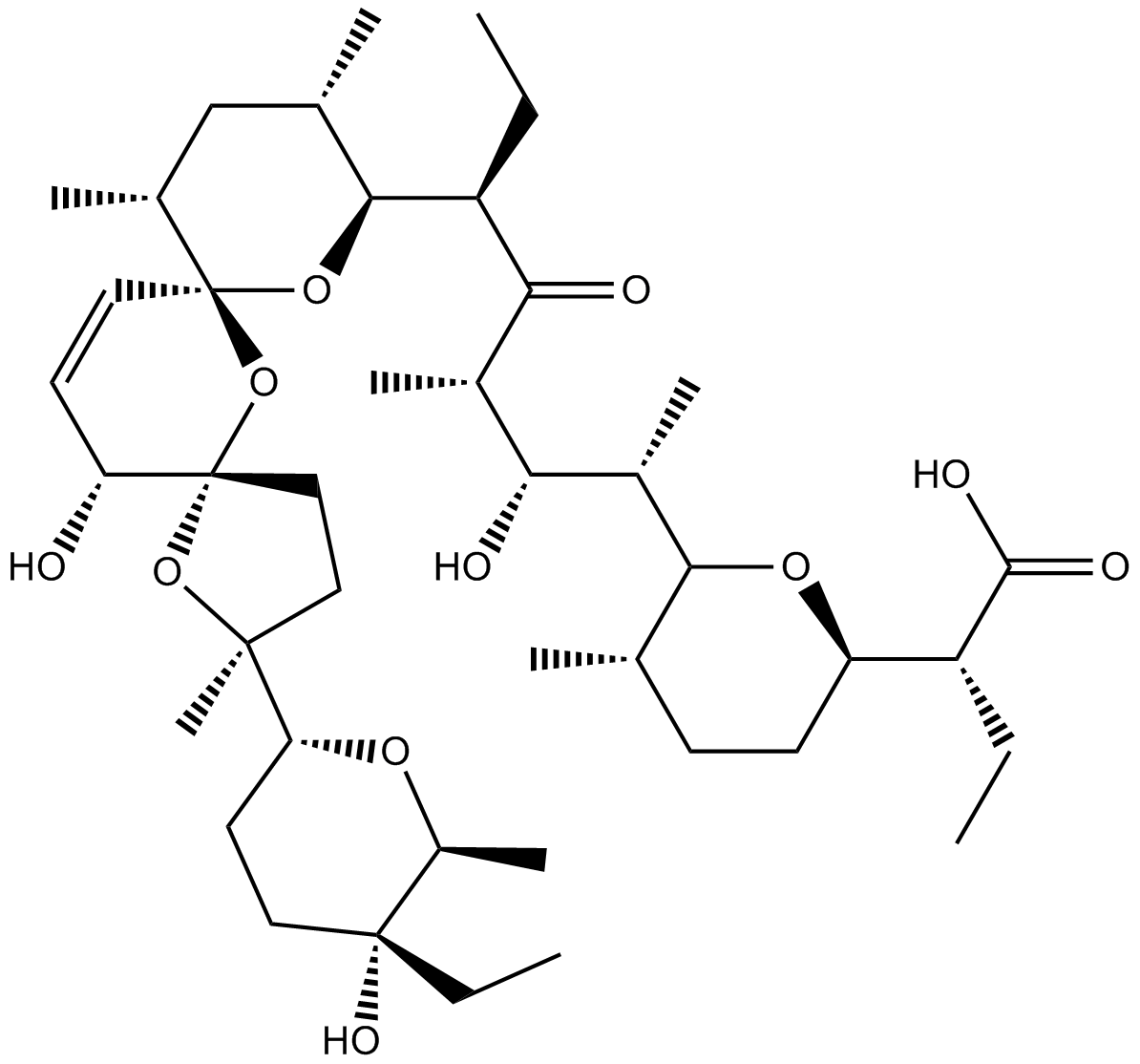
Salinomycin
A selective cancer stem cell inhibitor
-
GC18107
Salinomycin sodium salt
A selective cancer stem cell inhibitor
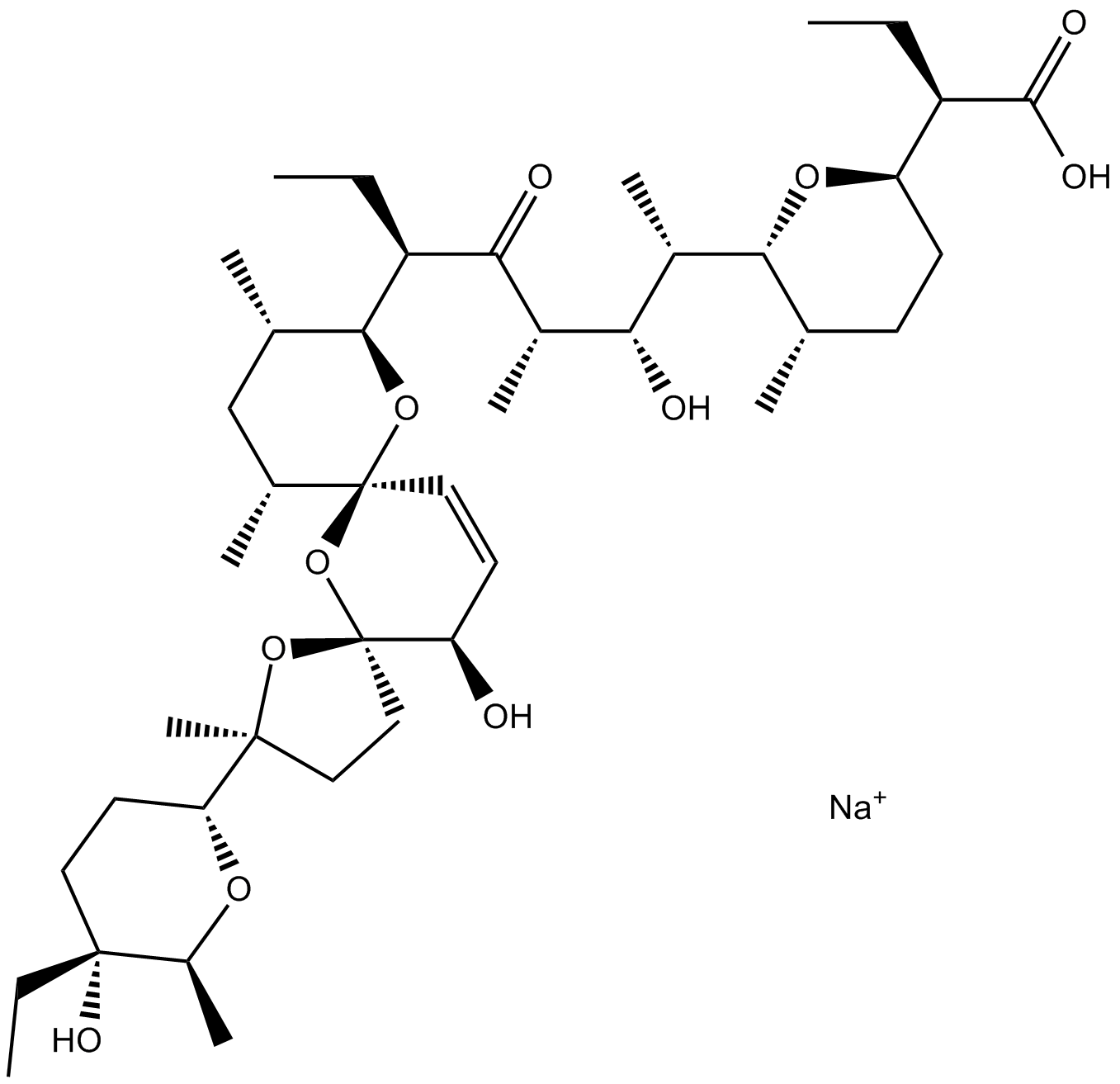
-
GC10486
Salinosporamide A (NPI-0052, Marizomib)
Marizomib, ML-858, NPI-0052
An orally bioactive proteasome inhibitor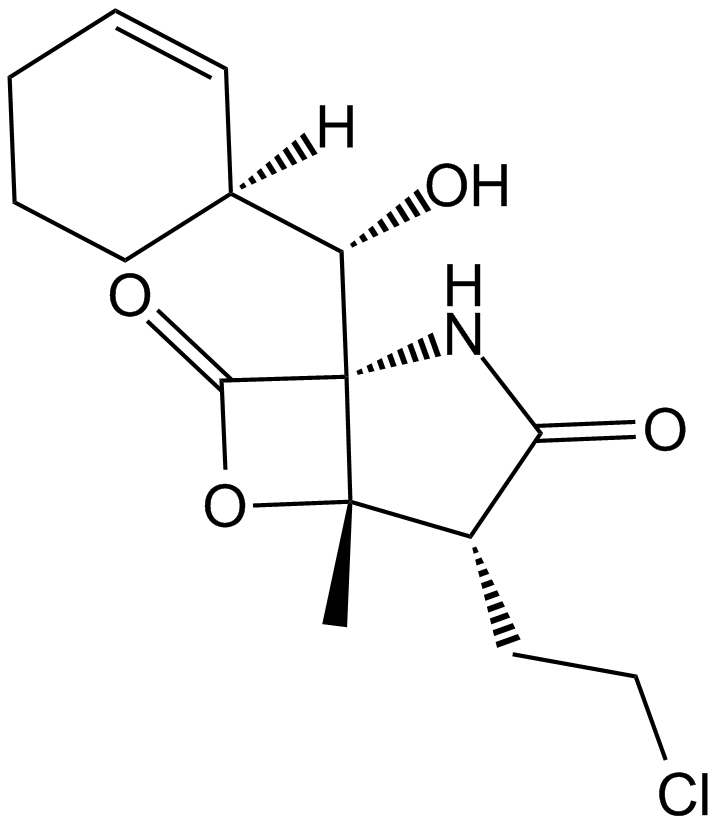
-
GC10528
Salirasib
FTS, Salirasib
A Ras inhibitor with anti-cancer and anti-atherogenic activity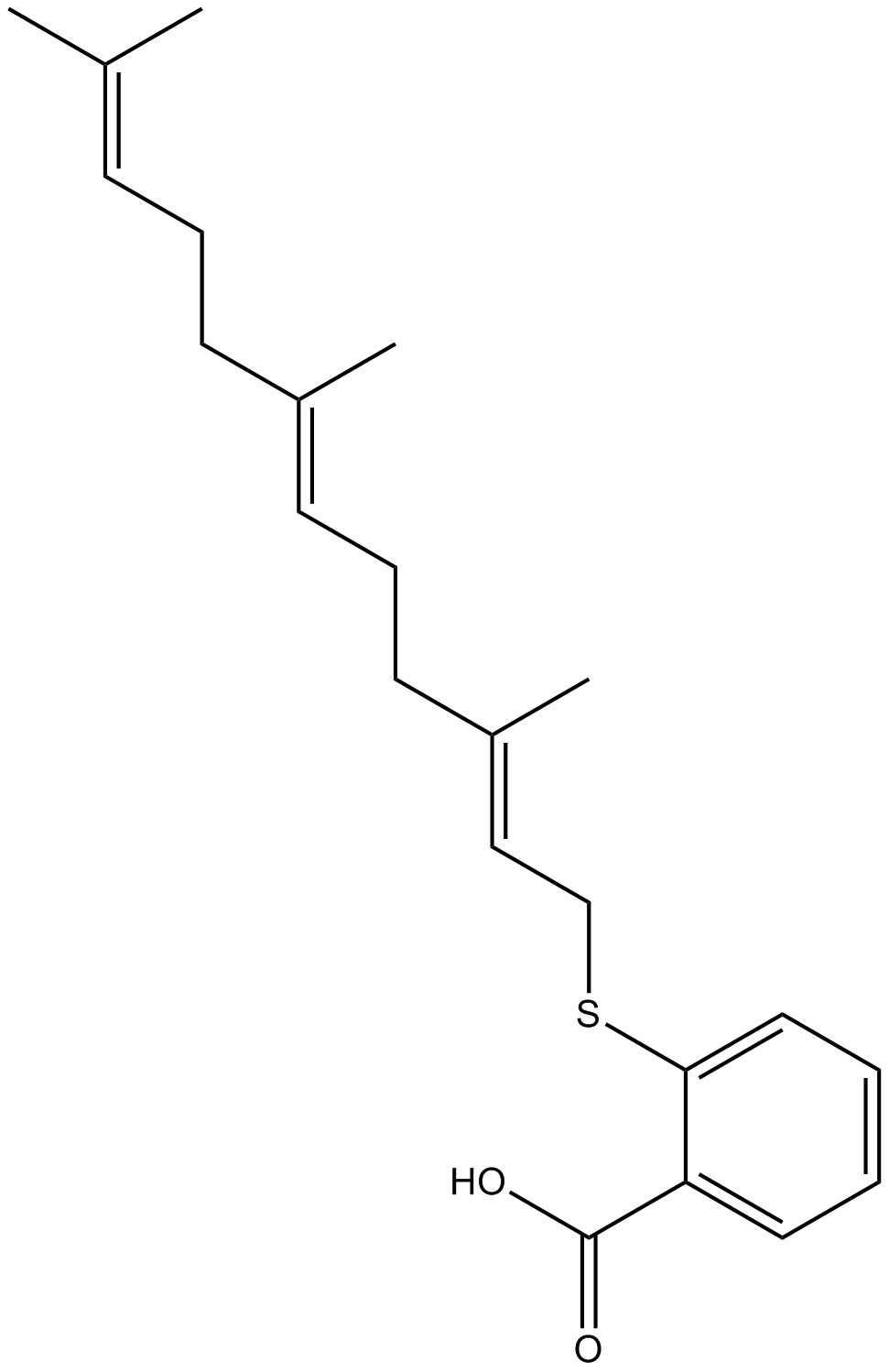
-
GC17331
Salubrinal
Salubrinal ist ein zellgÄngiger und selektiver Inhibitor der eIF2α-Dephosphorylierung.
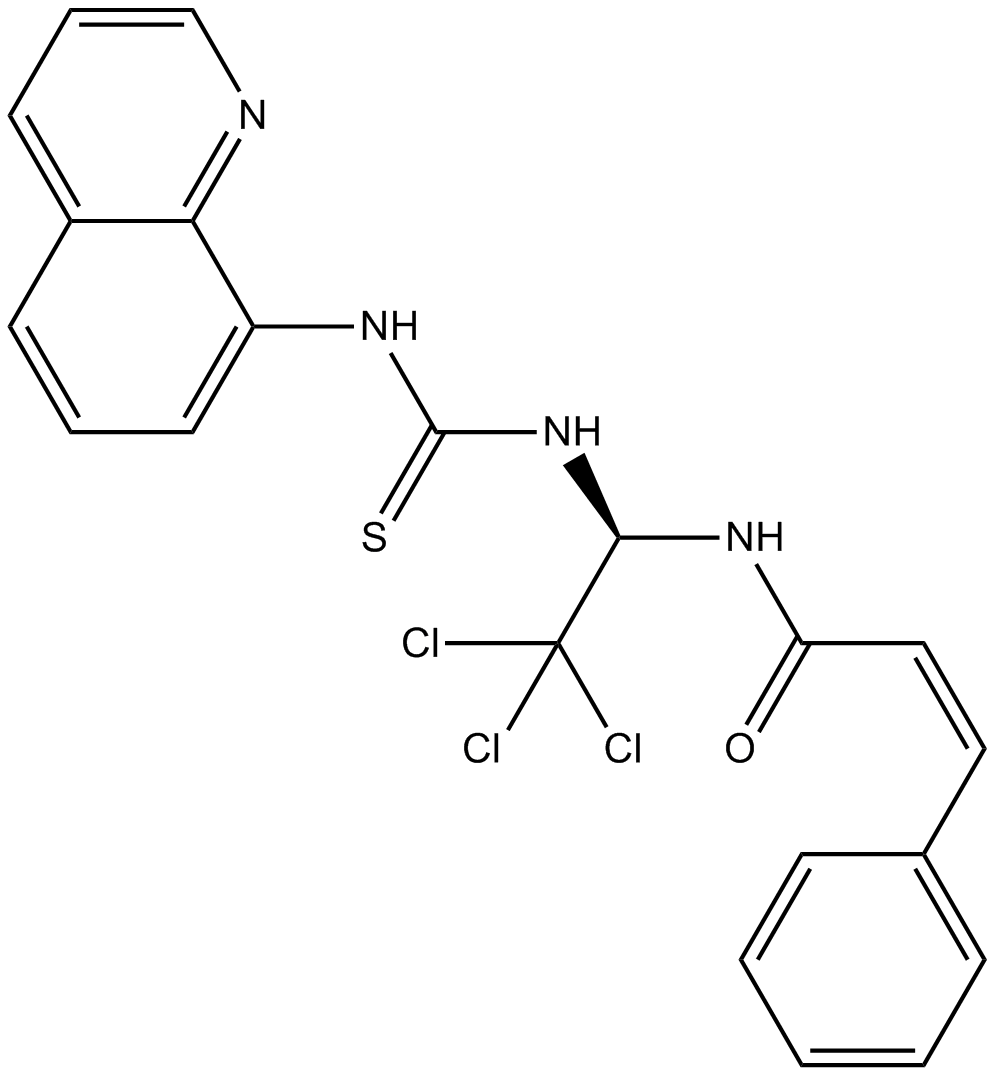
-
GN10344
Salvianolic acid B
Lithospermate B
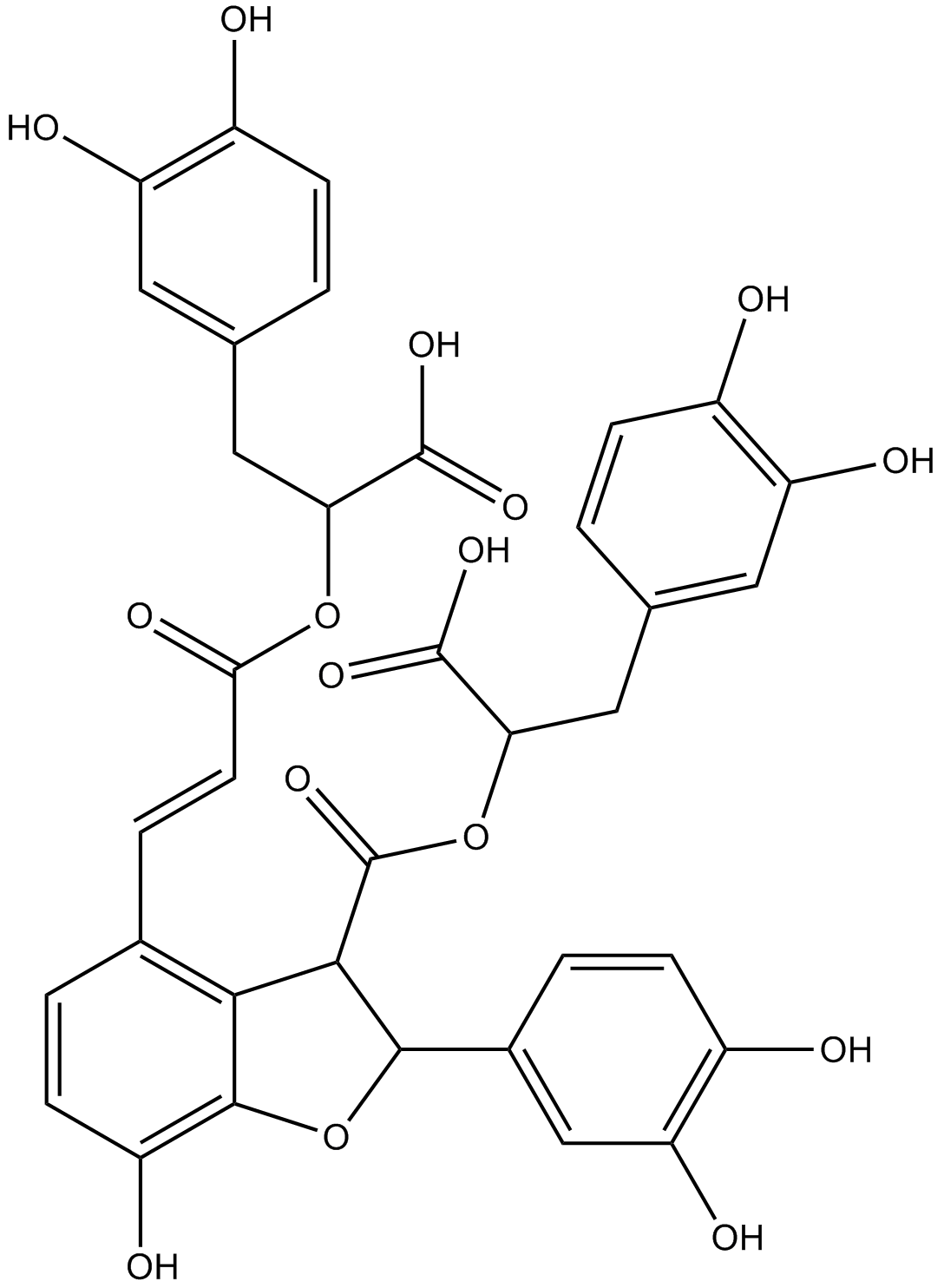
-
GC61263
Salvigenin
5-hydroxy-4',6,7-Trimethoxyflavone, 7-O-Methylpectolinarigenin, Psathyrotin
Salvigenin ist eine natÜrliche polyphenolische Verbindung mit neuroprotektiver Wirkung.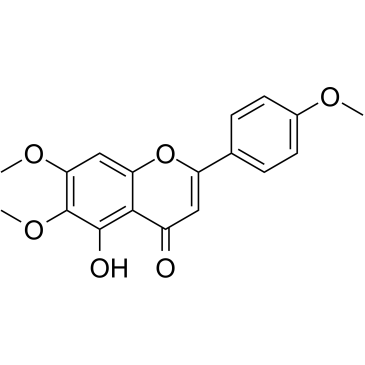
-
GN10717
Sanguinarine
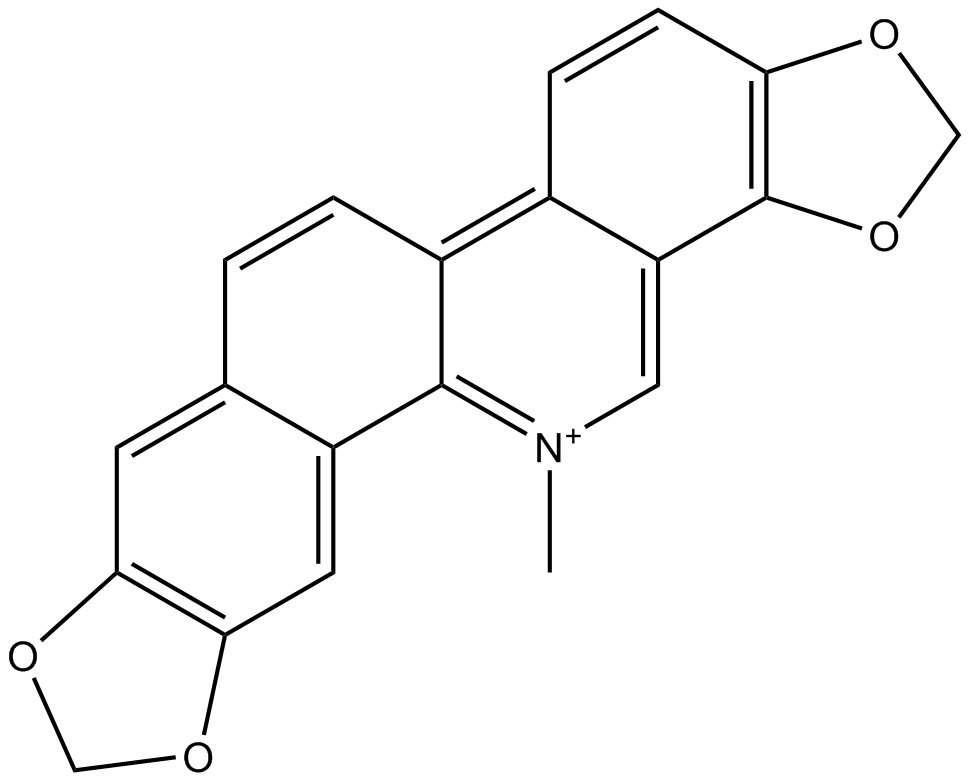
-
GC13442
Sanguinarine chloride
Sanguinarin (Sanguinarin)-Chlorid, ein Benzophenanthridin-Alkaloid, das aus der Wurzel von Sanguinaria Canadensis gewonnen wird, kann die Apoptose stimulieren, indem es die Produktion von reaktiven Sauerstoffspezies (ROS) aktiviert. Sanguinarin-induzierte Apoptose ist mit der Aktivierung von JNK und NF-κB assoziiert.
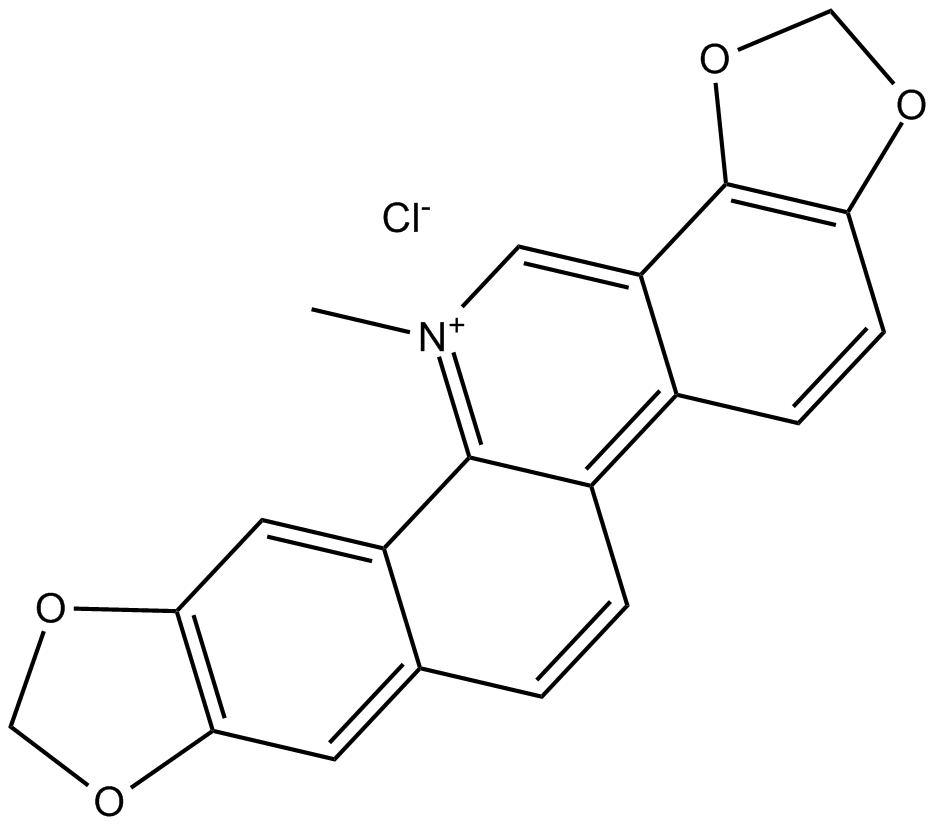
-
GC15785
Saquinavir mesylate
Ro 318959/003
An HIV protease inhibitor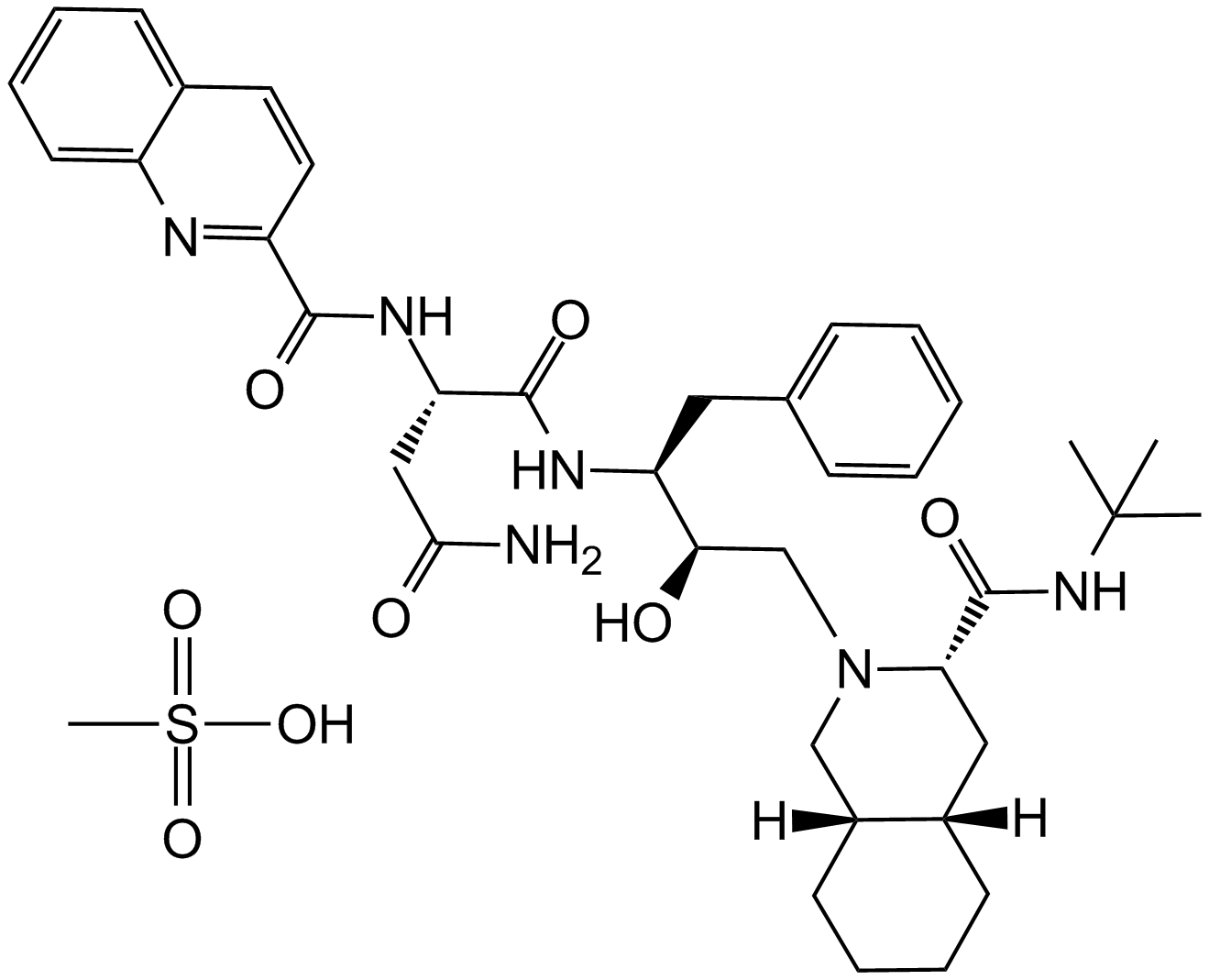
-
GC11471
SAR405
SAR405 ist ein erstklassiger, selektiver und ATP-kompetitiver Vps34-Inhibitor der Isoform PI3K Klasse III (PIK3C3) (IC50=1,2 nM; Kd=1,5 nM). SAR405 hemmt die Autophagie, die entweder durch Hunger oder durch mTOR-Hemmung induziert wird. AktivitÄt gegen Krebs.
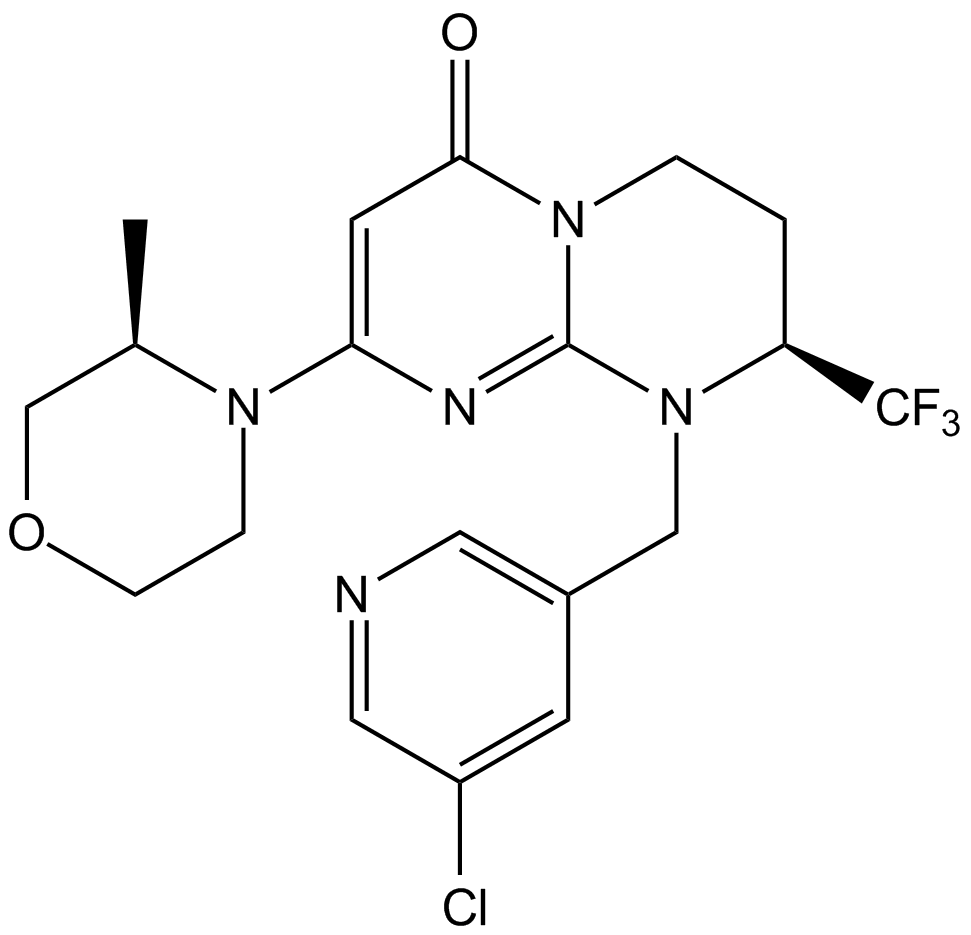
-
GC15197
Saracatinib (AZD0530)
AZD 0530
Saracatinib (AZD0530) (AZD0530) ist ein potenter Inhibitor der Src-Familie mit IC50-Werten von 2,7 bis 11 nM fÜr c-Src, Lck, c-YES, Lyn, Fyn, Fgr und Blk. Saracatinib (AZD0530) zeigt eine hohe SelektivitÄt gegenÜber anderen Tyrosinkinasen.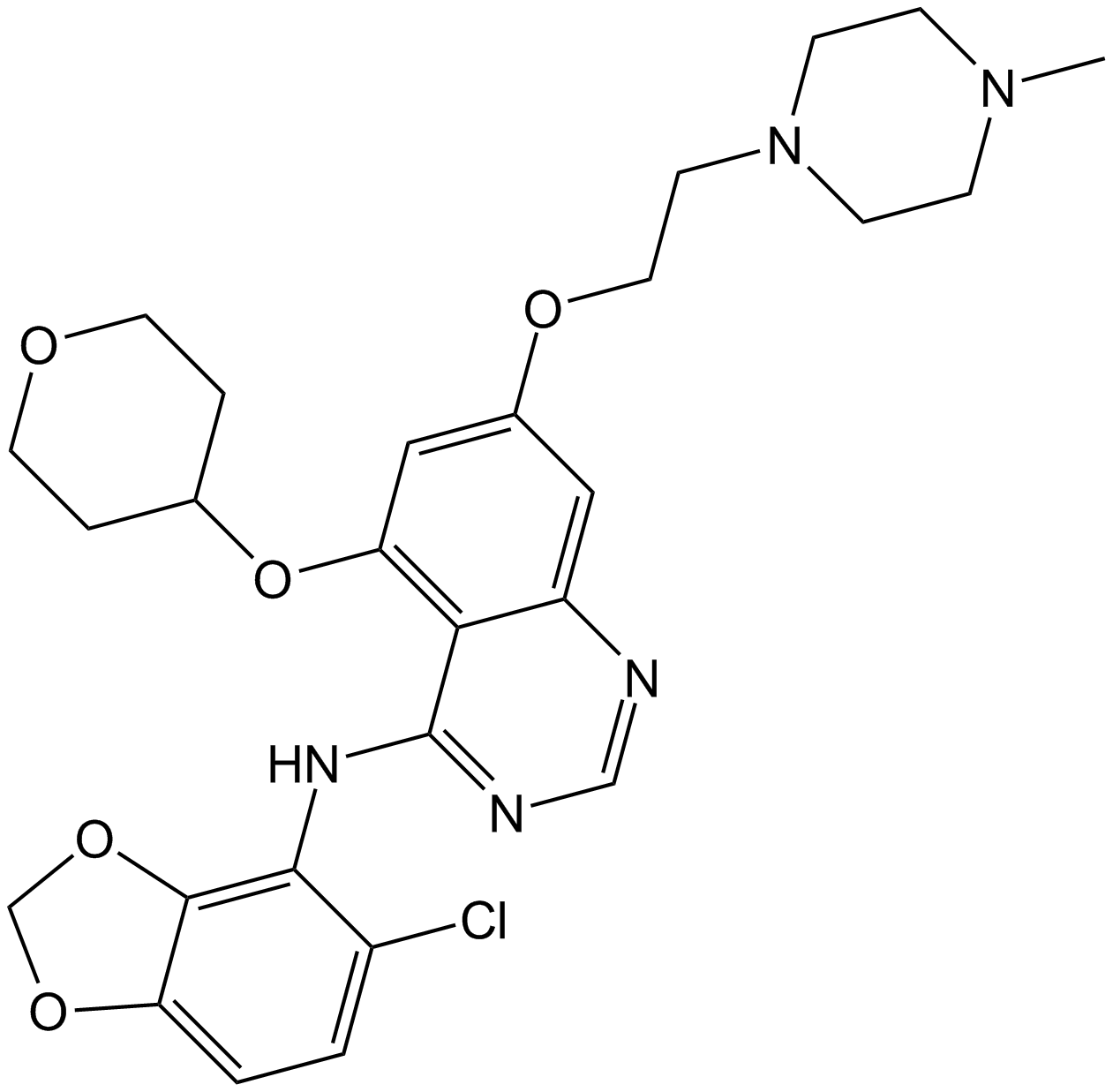
-
GC13595
SB 203580
SB 203580; RWJ 64809
SB 203580 is a specific inhibitor of p38-MAPK (Mitogen-activated Protein Kinase) pathway .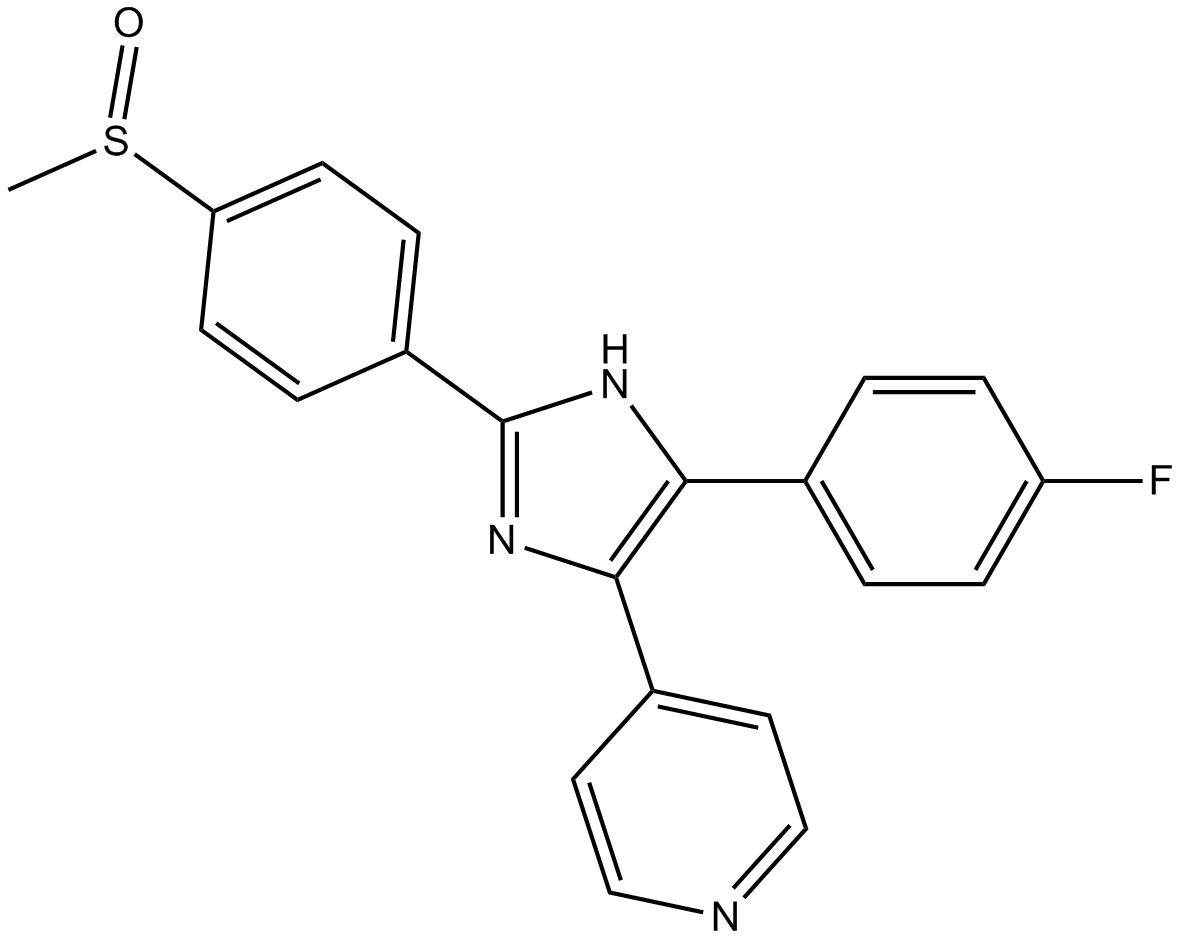
-
GC13001
SB 203580 hydrochloride
PB 203580, RWJ 64809
Adezmapimod (SB 203580) Hydrochlorid ist ein selektiver und ATP-kompetitiver p38-MAPK-Inhibitor mit IC50-Werten von 50 nM und 500 nM fÜr SAPK2a/p38 bzw. SAPK2b/p38β2.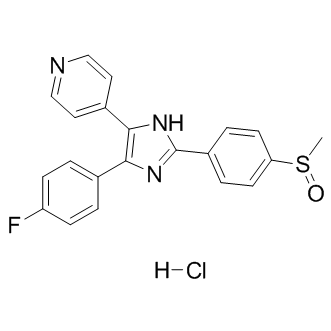
-
GC10054
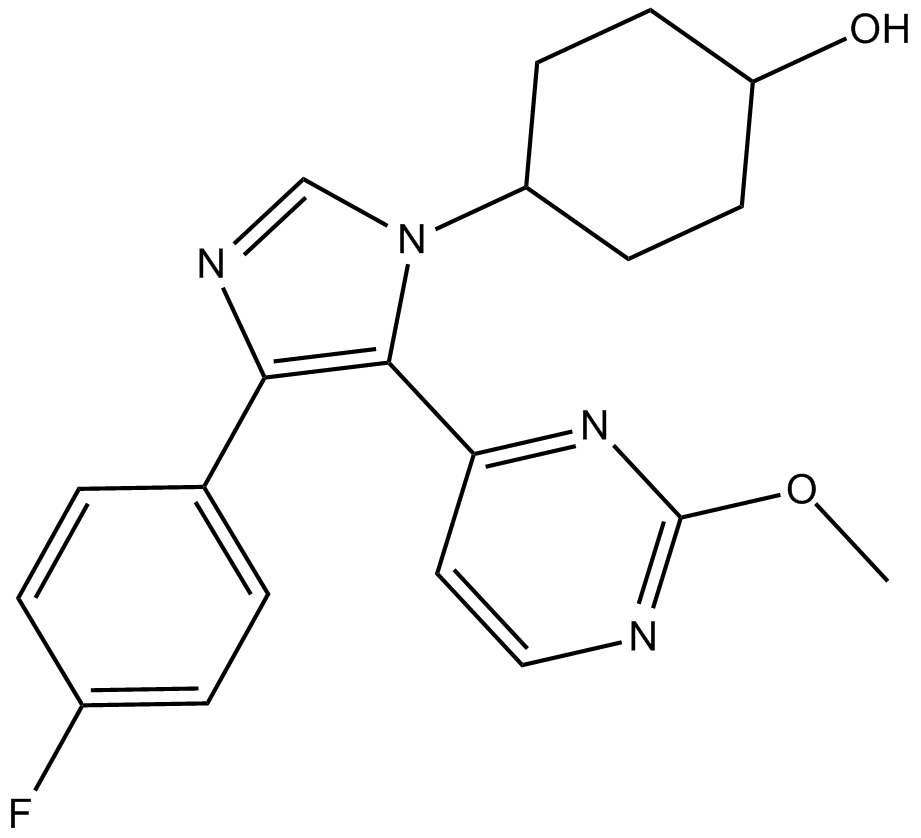
SB 239063
A selective p38 MAPK inhibitor
-
GC37595
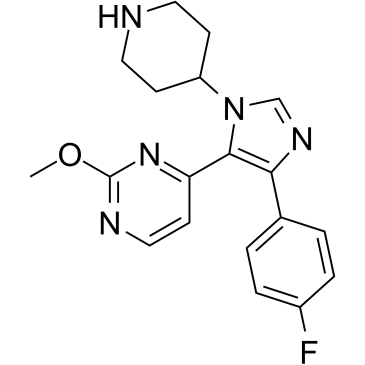
SB 242235
SB-242235 ist ein potenter und selektiver p38-MAP-Kinase-Inhibitor mit einem IC50-Wert von 1,0 μM in primÄren menschlichen Chondrozyten.
-
GC13968
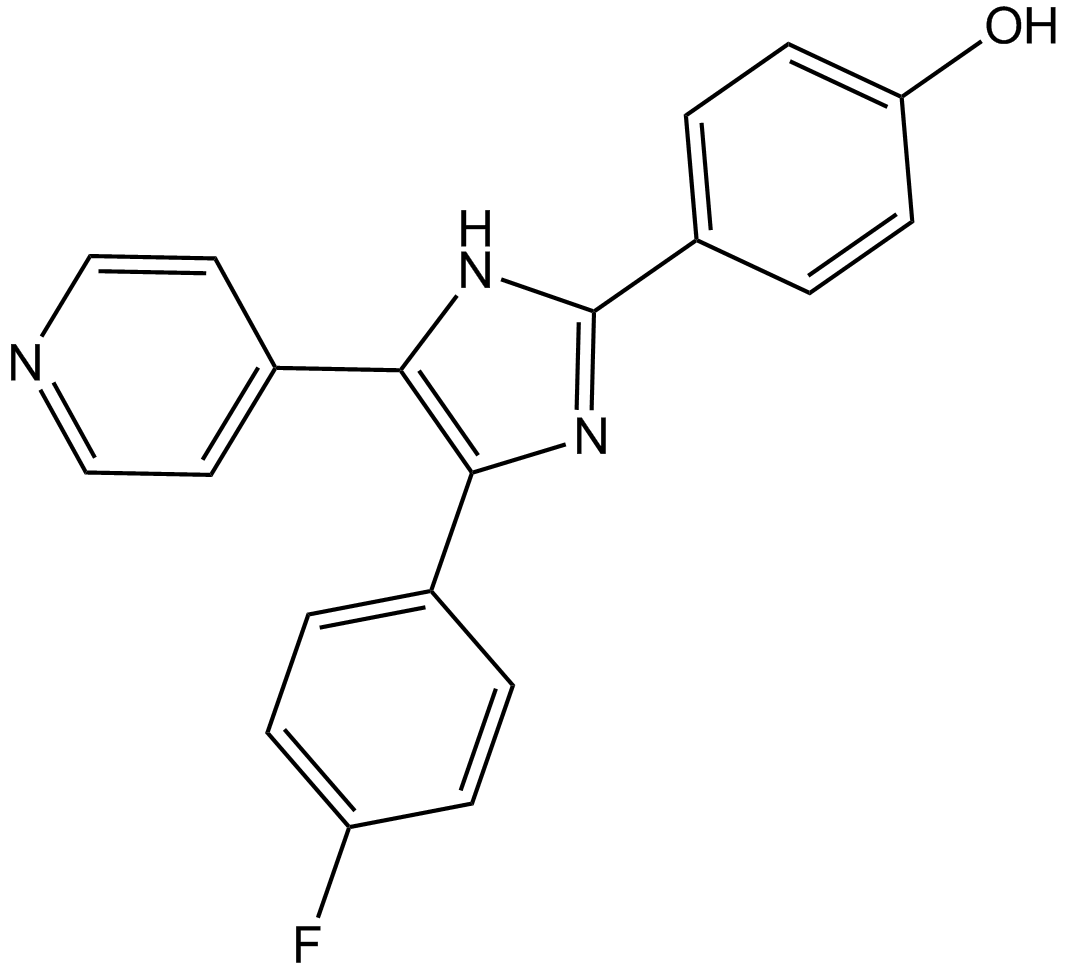
SB202190 (FHPI)
SB202190 (FHPI) ist ein selektiver p38 MAP-Kinase-Inhibitor mit IC50-Werten von 50 nM und 100 nM für p38α bzw. p38β2.
-
GC14644
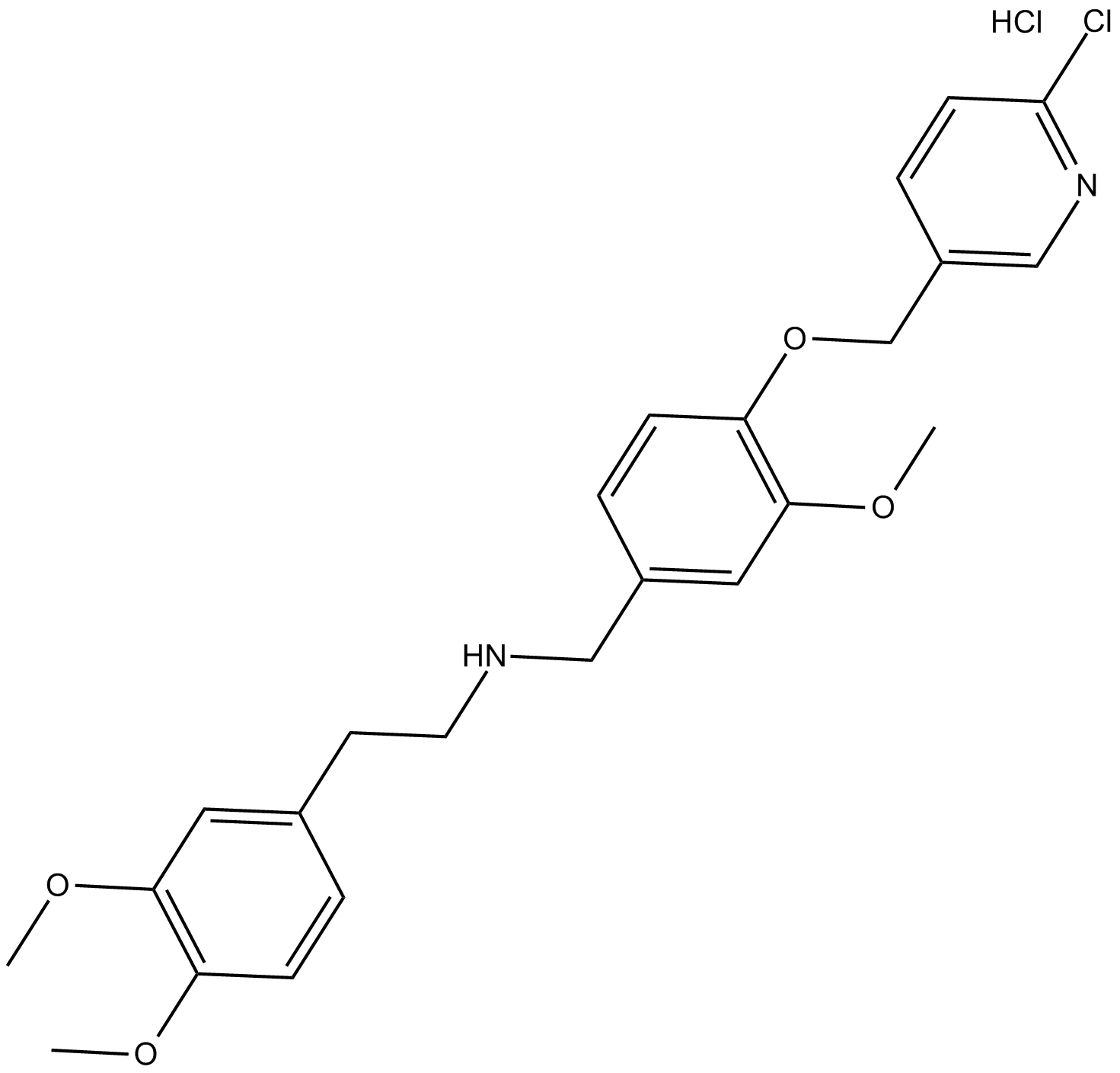
SBE 13 HCl
SBE 13 HCl ist ein potenter und selektiver Plk1-Inhibitor mit einem IC50 von 200 pM; SBE 13 HCl hemmt Plk2 (IC50 > 66μ M) oder Plk3 (IC50 = 875 nM) schwach.
-
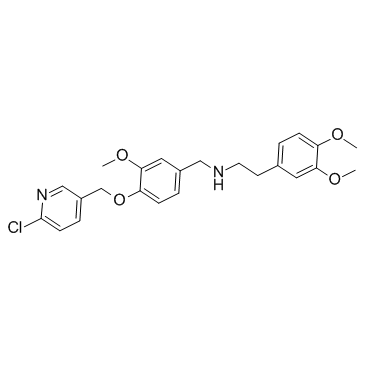
GC37601
SBE13
SBE13 ist ein potenter und selektiver Plk1-Inhibitor mit einem IC50 von 200 pM; SBE13 hemmt Plk2 (IC50>66μM) oder Plk3 (IC50=875nM) nur schwach.
-
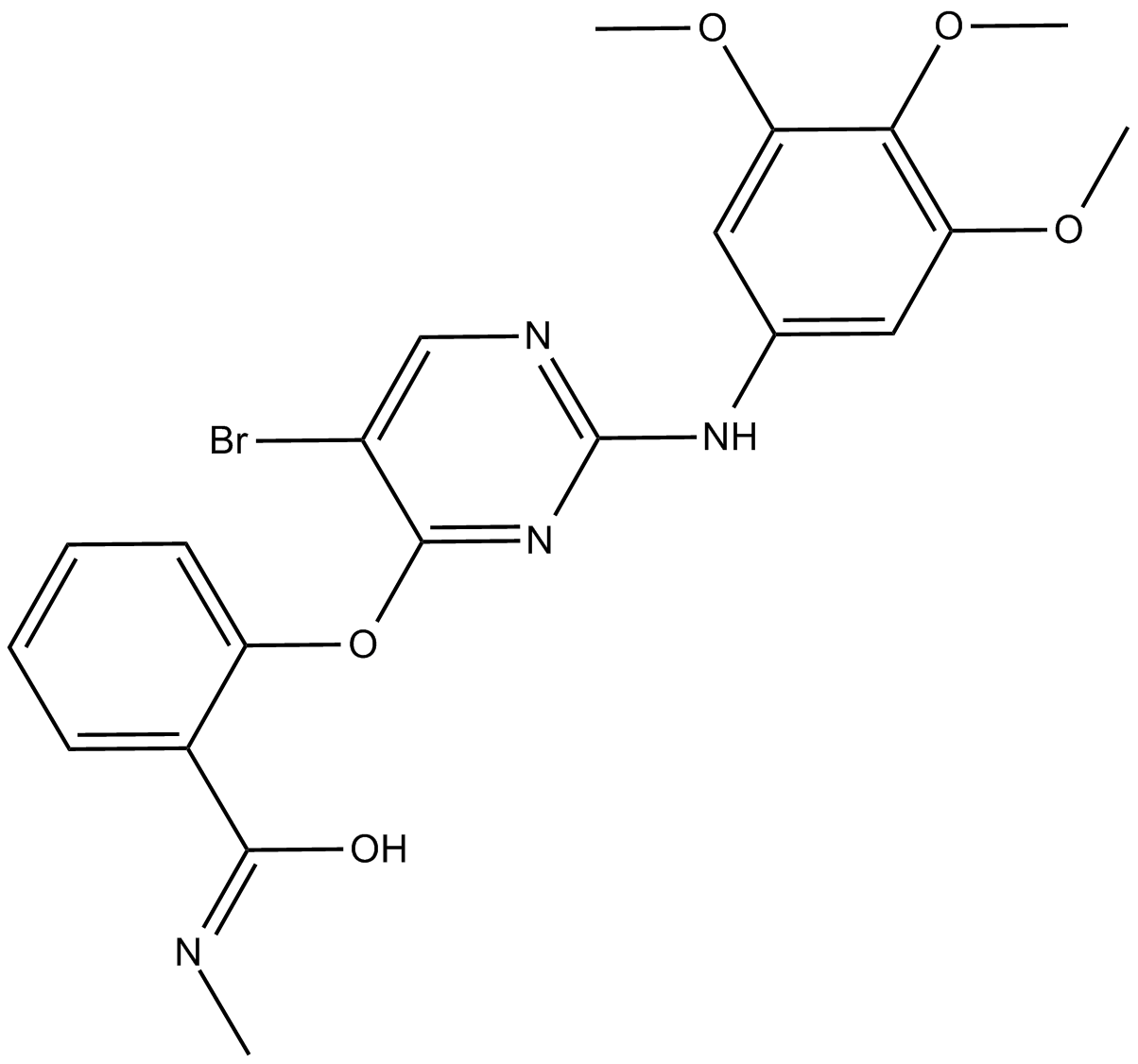
GC15654
SBI-0206965
SBI-0206965 ist ein potenter, selektiver und zellgÄngiger Autophagie-Kinase-ULK1-Inhibitor mit IC50-Werten von 108 nM fÜr ULK1-Kinase und 711 nM fÜr die hoch verwandte Kinase ULK2.
-
GC19451
SBI-0640756
SBI-756
SBI-0640756 (SBI-756) ist ein Inhibitor von eIF4G1 und unterbricht den eIF4F-Komplex.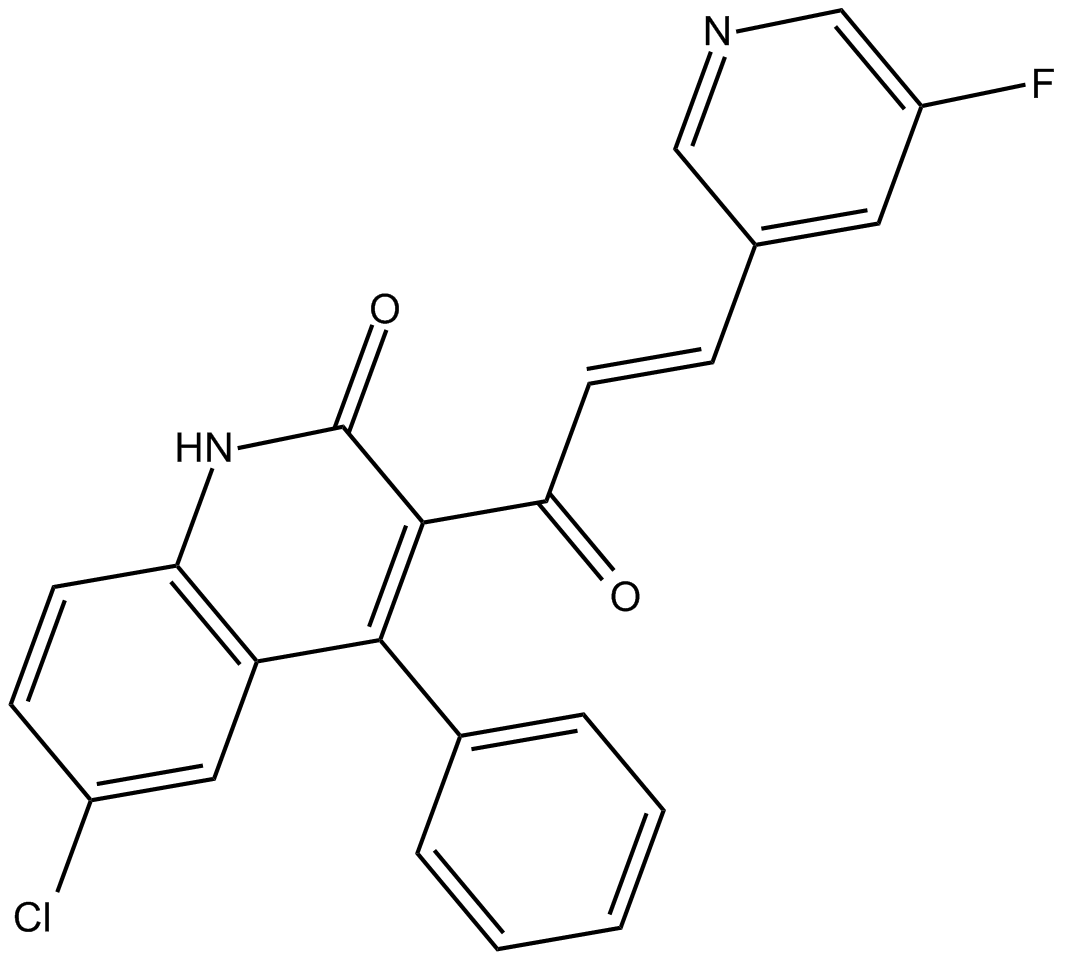
-
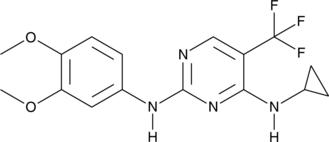
GC48689
SBP-7455
Ein Dualhemmer von ULK1 und ULK2.
-
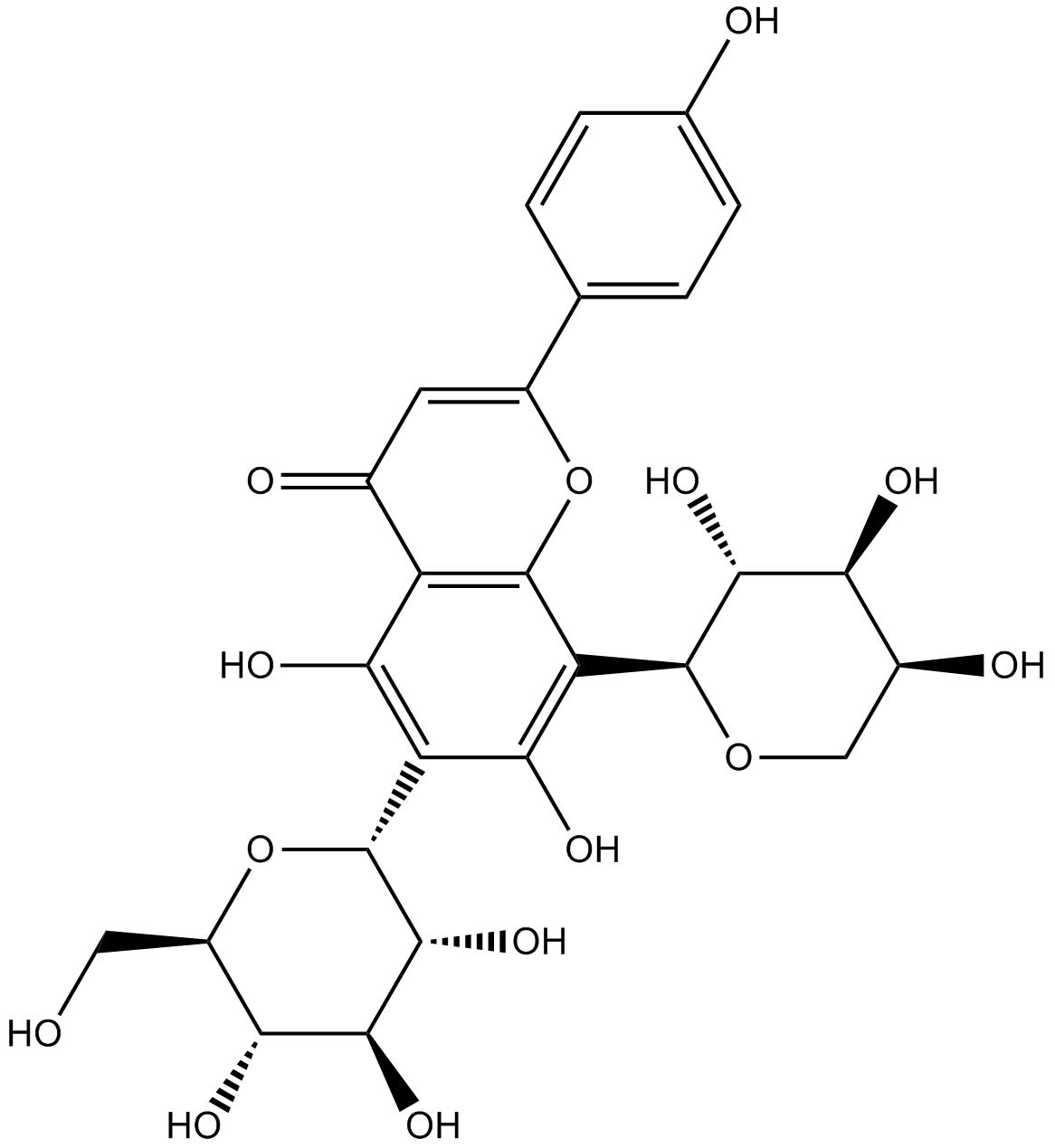
GN10091
Schaftoside
Apigenin 6-C-glucoside-8-C-arabinoside
-
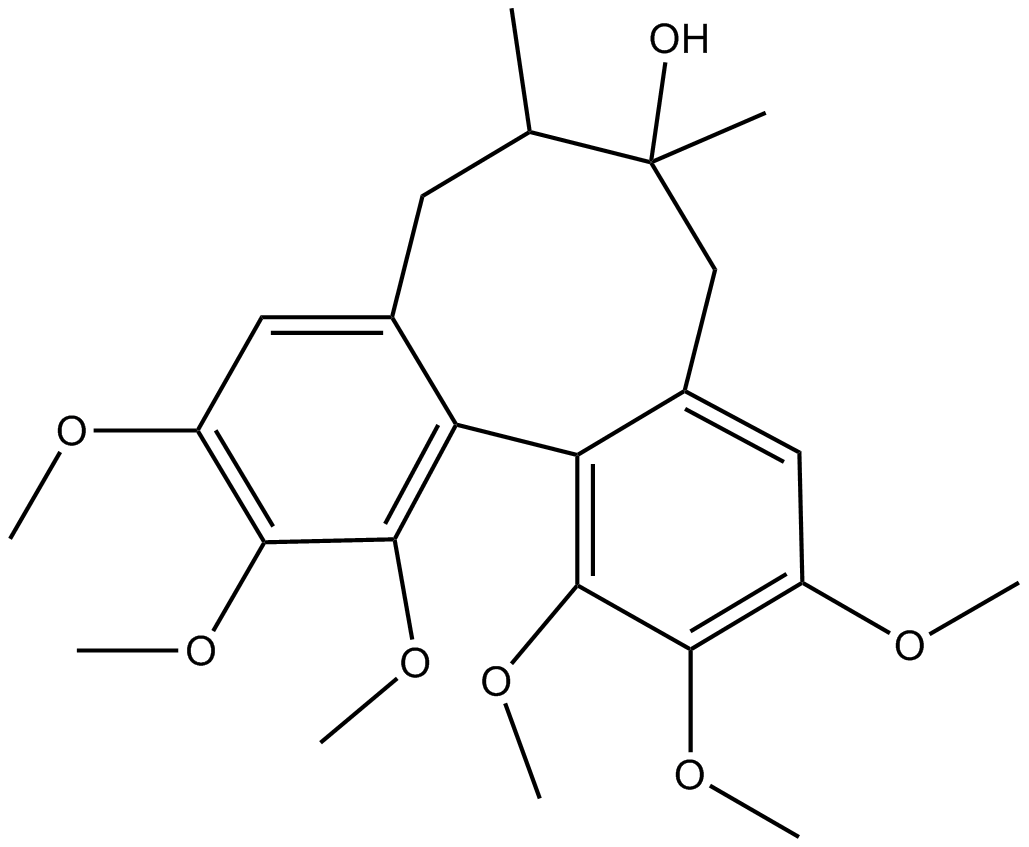
GN10425
Schisandrin A
Deoxyschizandrin, Wuweizisu A
-
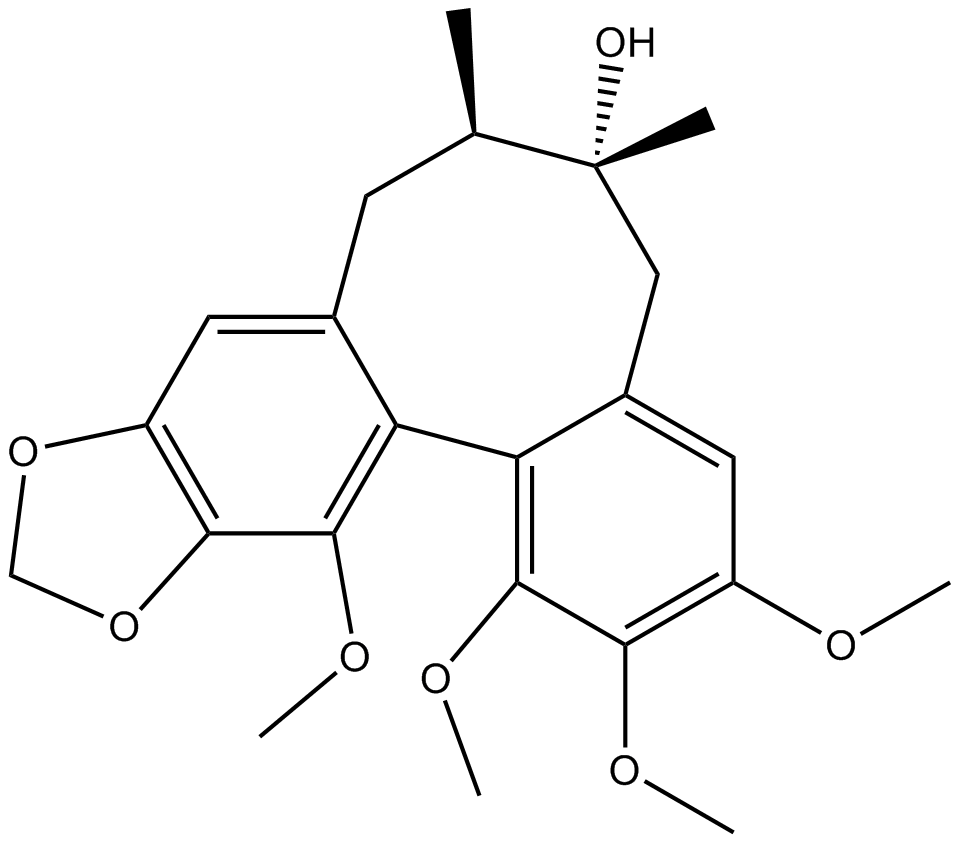
GN10474
Schisandrol B
Besigomsin, Gomisin A, Wuweizichun B
-
GN10132
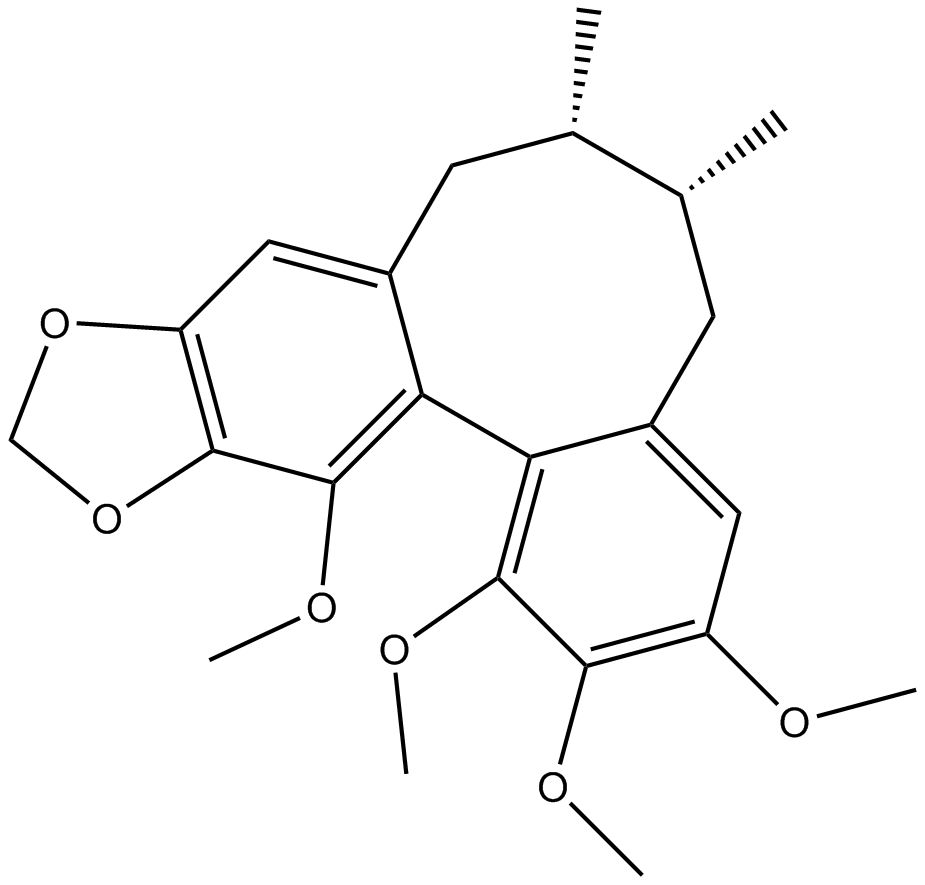
Schizandrin B
Wuweizisu B
-
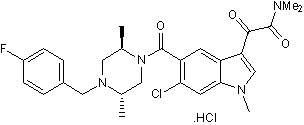
GC50040
SCIO 469 hydrochloride
Selective p38 MAPK inhibitor
-
GC15315
Scriptaid
Scriptide; GCK1026
HDAC inhibitor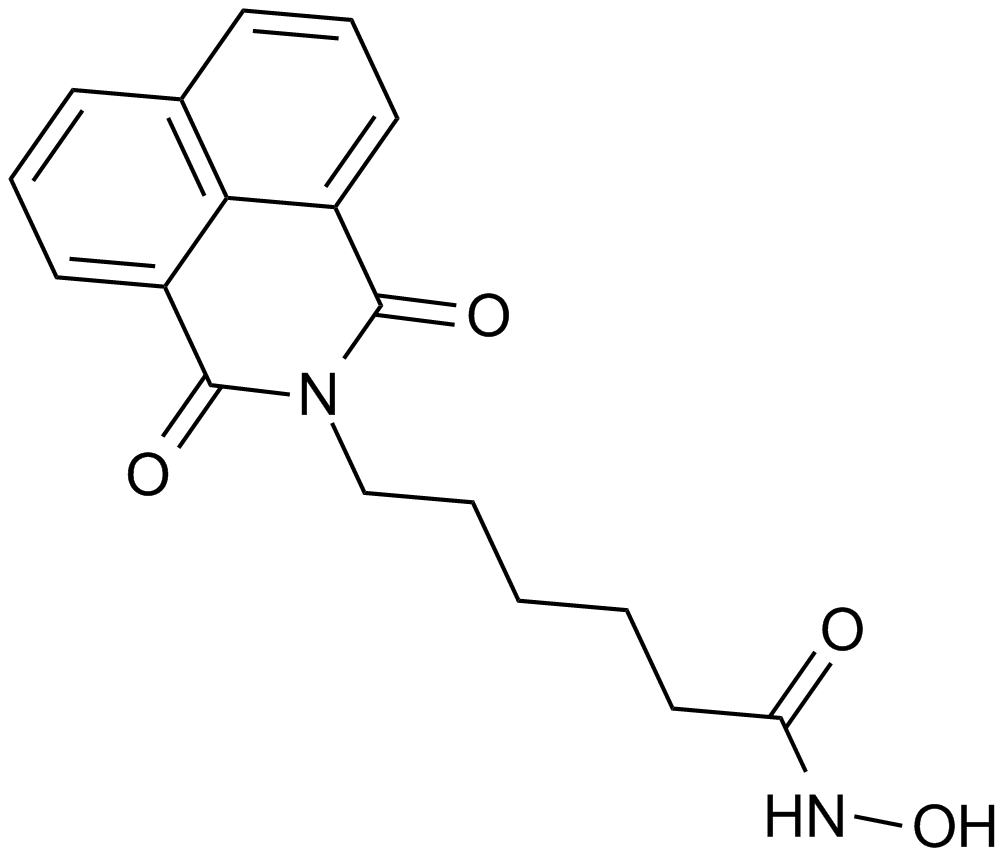
-
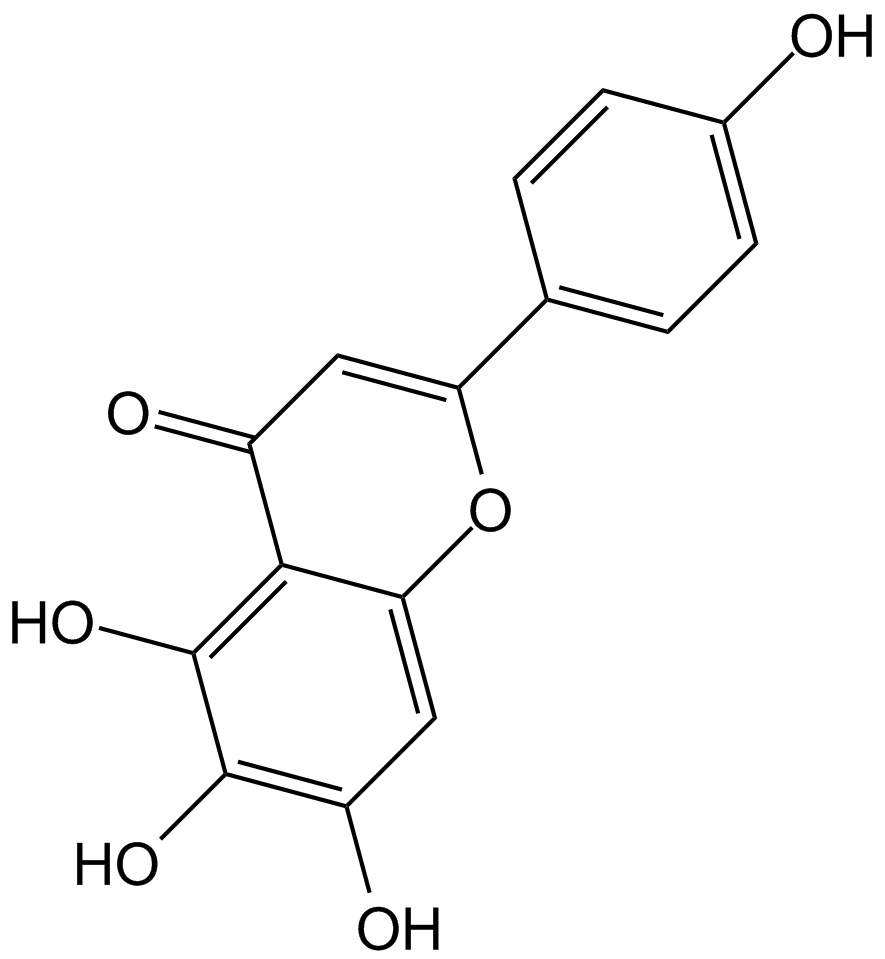
GN10633
Scutellarein
6-Hydroxyapigenin
-
GC19325
SD 0006
SD-006
SD 0006 (SD-06) ist ein oral aktiver, selektiver, ATP-kompetitiver und potenter Diarylpyrazol-Inhibitor der p38α-MAP-Kinase mit einem IC50-Wert von 110 nM fÜr p38α.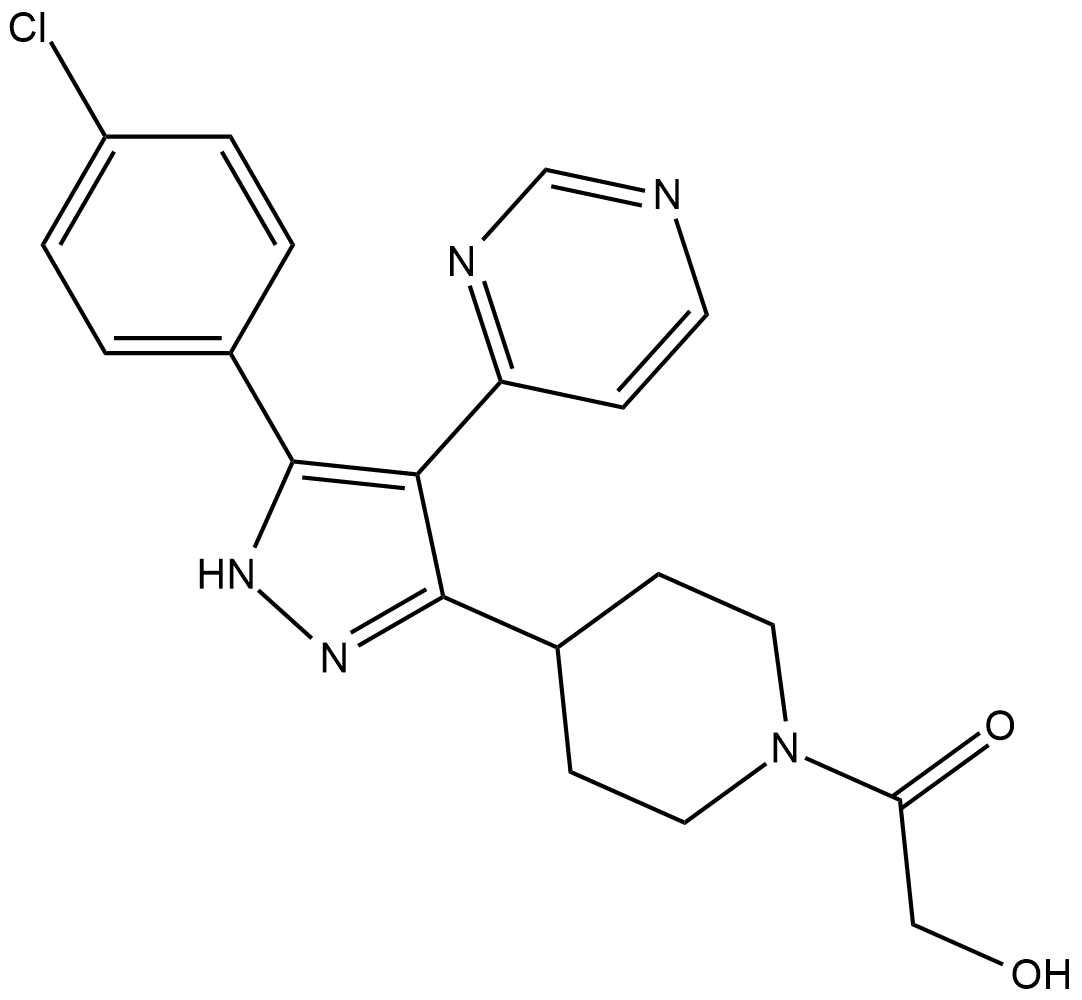
-
GC30343
Selenomethionine (Seleno-DL-methionine)
Selenomethionin (Seleno-DL-Methionin) ist eine natÜrlich vorkommende AminosÄure, die Selen enthÄlt und eine Übliche natÜrliche Nahrungsquelle fÜr Selen ist.
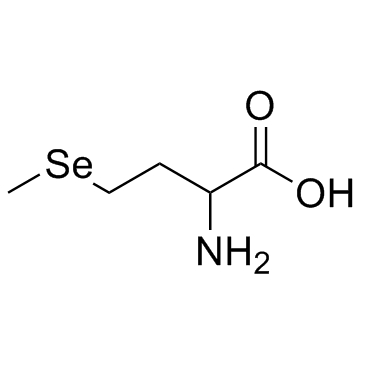
-
GC30787
Sertindole (Lu 23-174)
Sertindol (Lu 23-174) (Lu 23-174) ist ein oral aktiver Antagonist von 5-HT2A, 5-HT2C, Dopamin D2 und αl-adrenergen Rezeptoren.
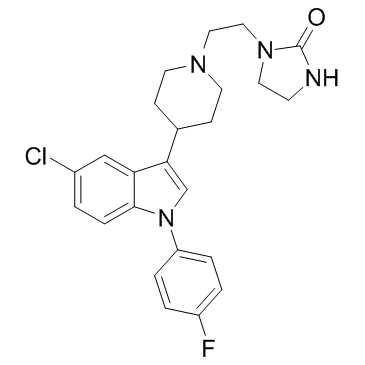
-
GC37630
Sevelamer
Sevelamer ist ein phosphatbindendes Medikament zur Behandlung von HyperphosphatÄmie bei Patienten mit chronischer Nierenerkrankung; besteht aus Polyallylamin, das mit Epichlorhydrin vernetzt ist.
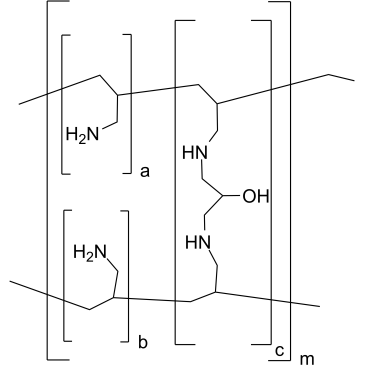
-
GC11885
Sevelamer HCl
Sevelamer HCl ist ein phosphatbindendes Medikament zur Behandlung von HyperphosphatÄmie bei Patienten mit chronischer Nierenerkrankung; besteht aus Polyallylamin, das mit Epichlorhydrin vernetzt ist.
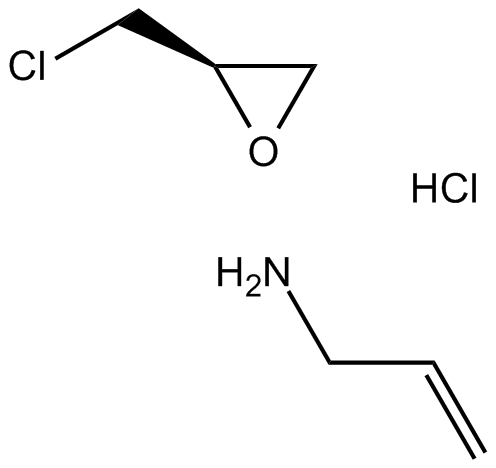
-
GC10019
SF1670
SF1670 ist ein potenter und spezifischer Phosphatase- und Tensin-Homolog, der auf dem Chromosom 10 (PTEN)-Inhibitor deletiert ist.
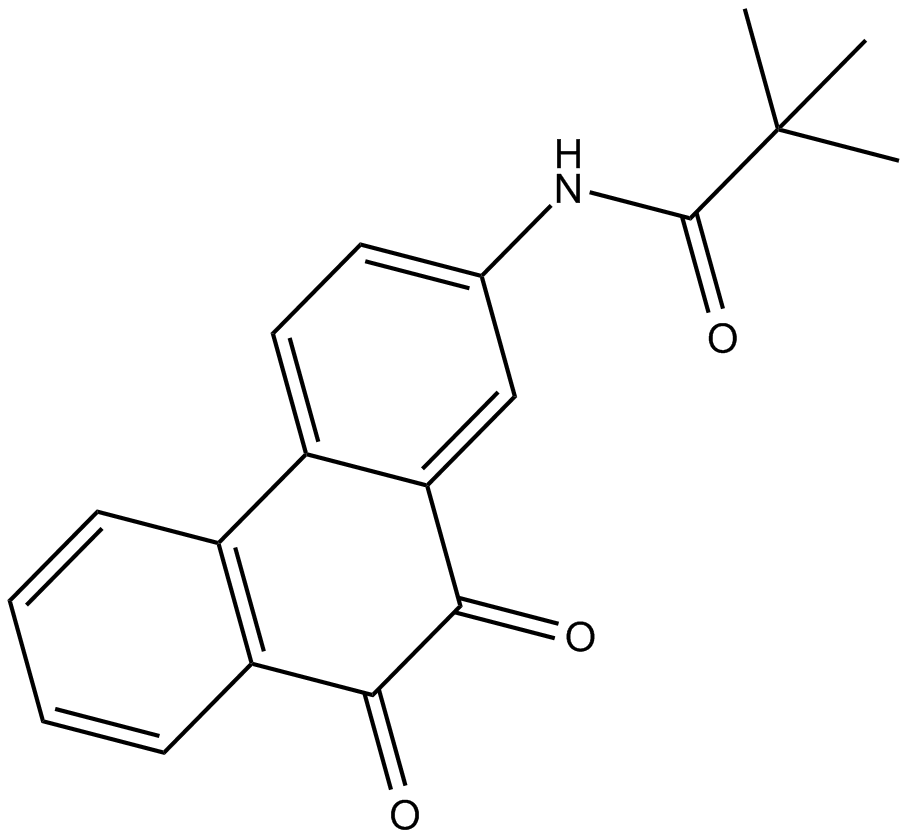
-
GC16497
SGI-1776 free base
SGI1776,SGI 1776
A potent inhibitor of Pim kinases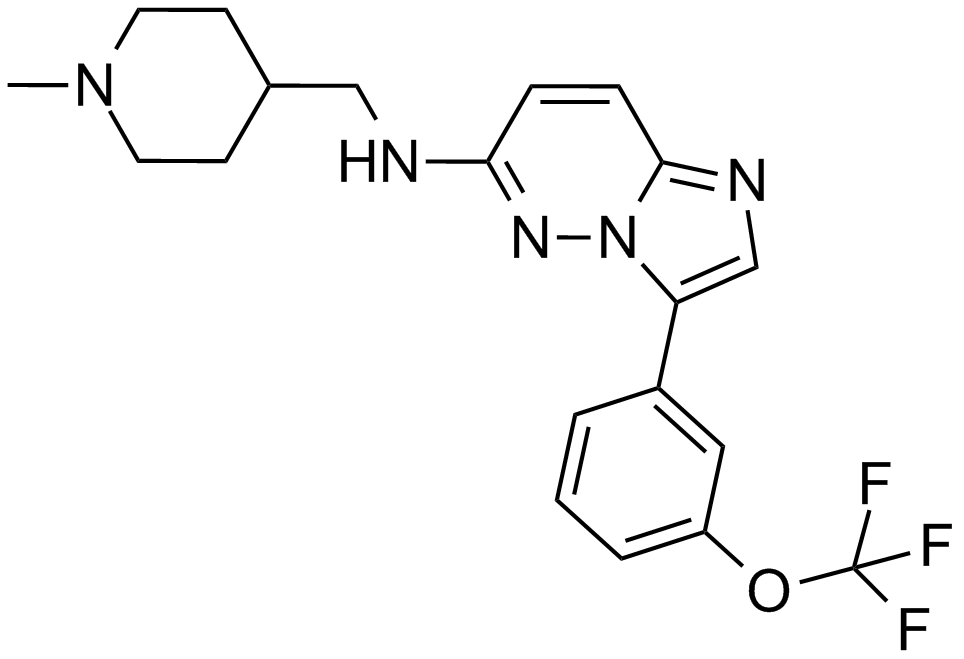
-
GC11369
Sildenafil
A PDE5 inhibitor
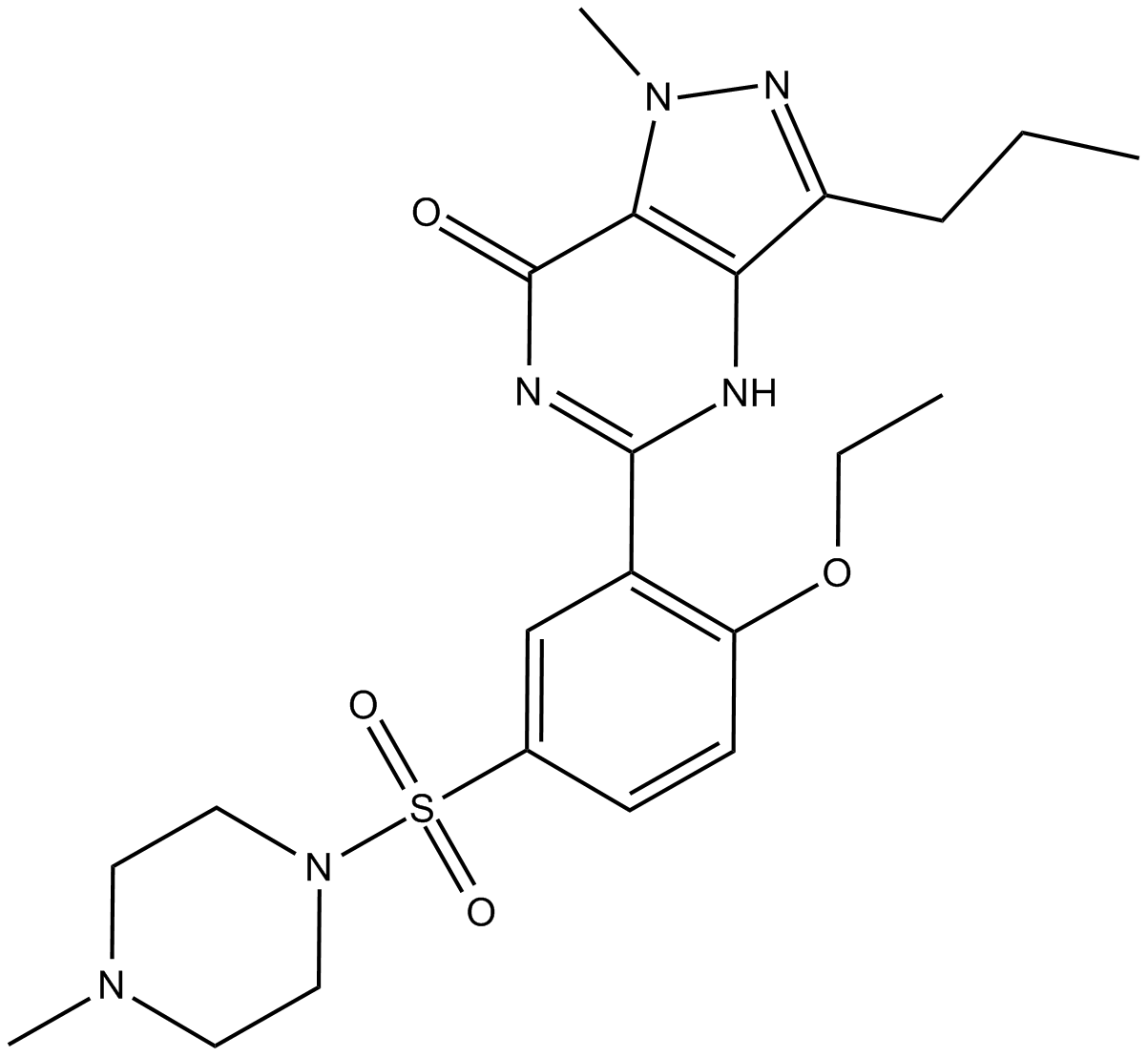
-
GC17685
Sildenafil Citrate
Apodefil, Tonafil, UK 92480
A PDE5 inhibitor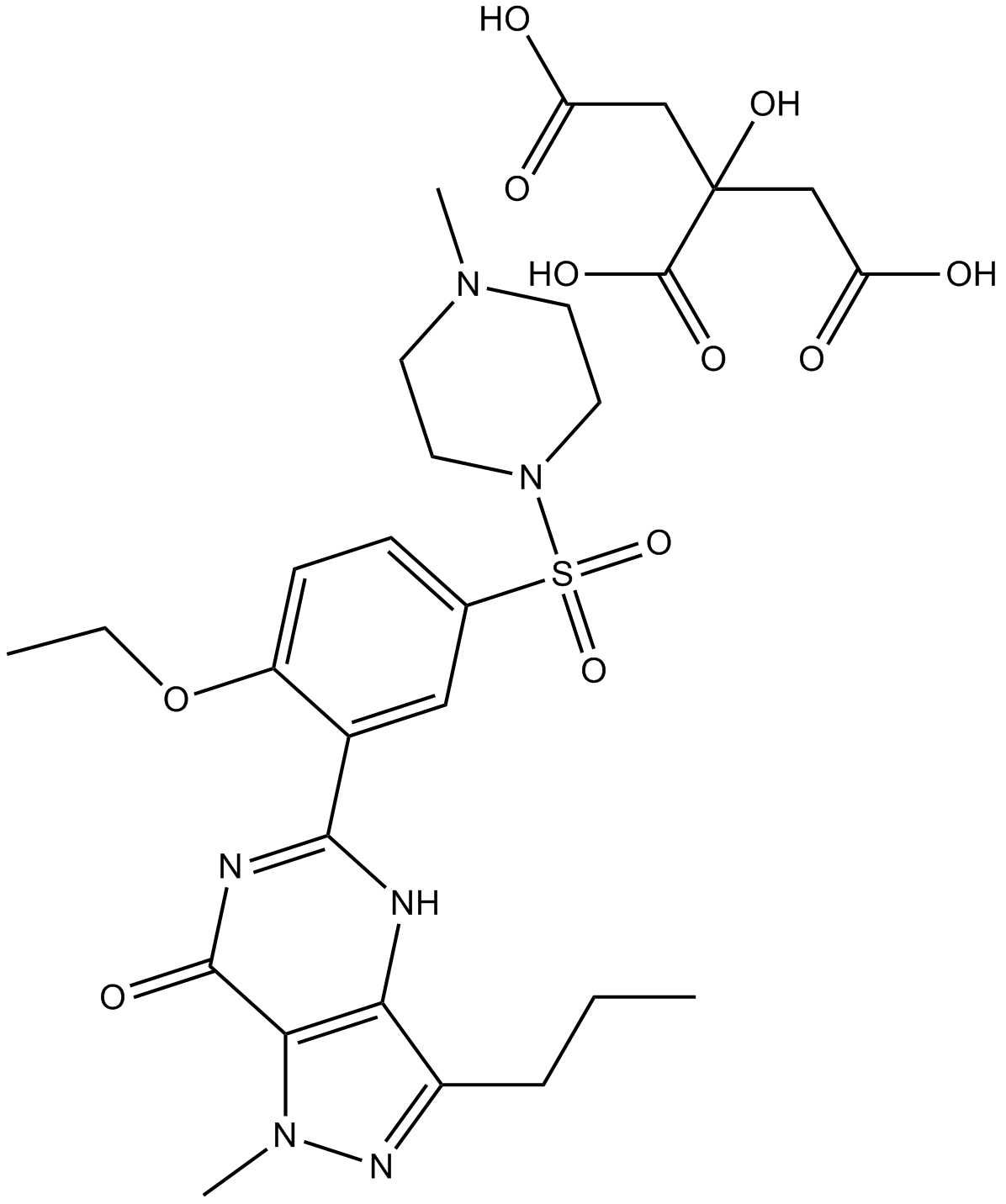
-
GN10292
Silibinin
Silibinin, Silibinin A, Silybin A
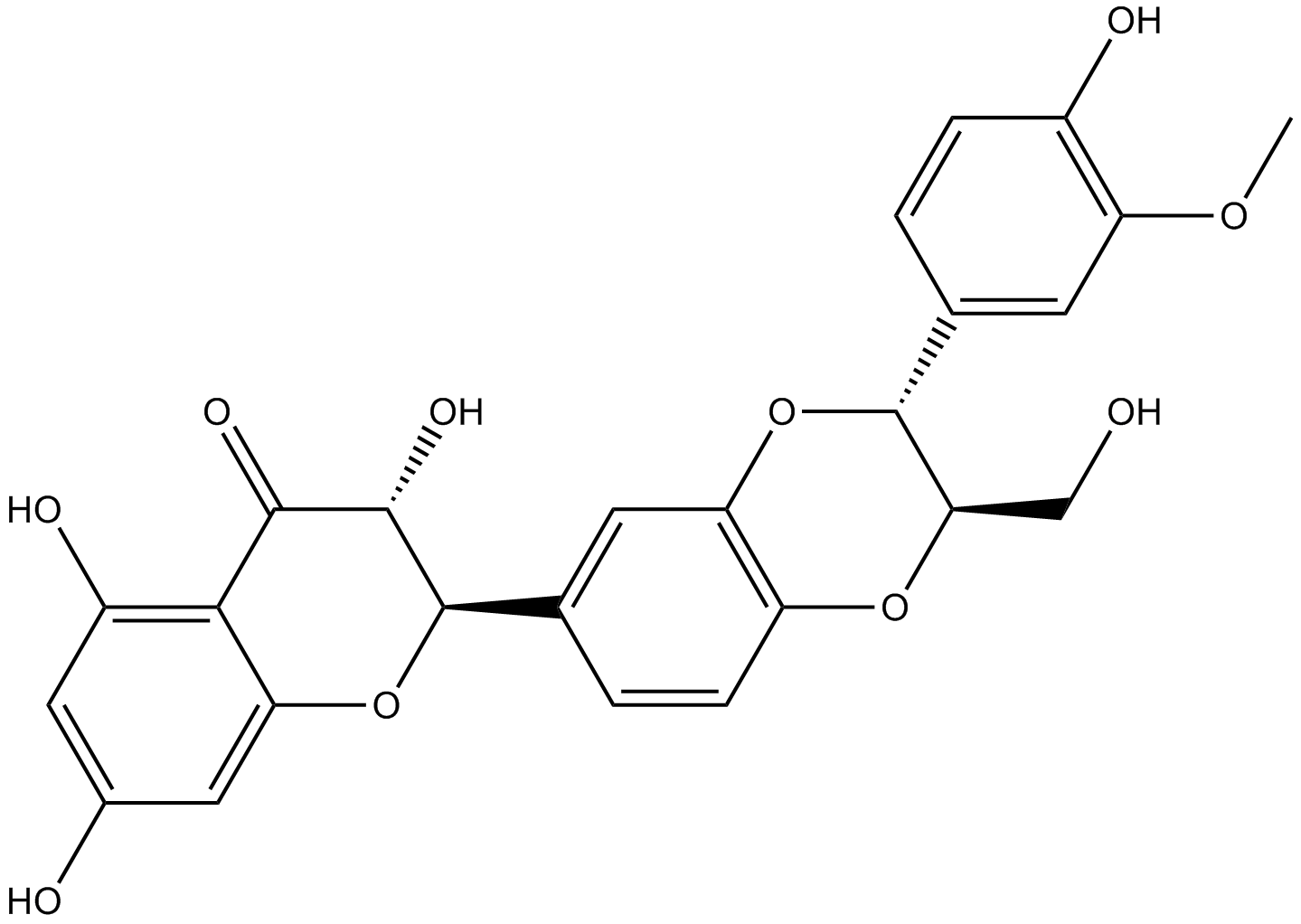
-
GC11455
Silvestrol
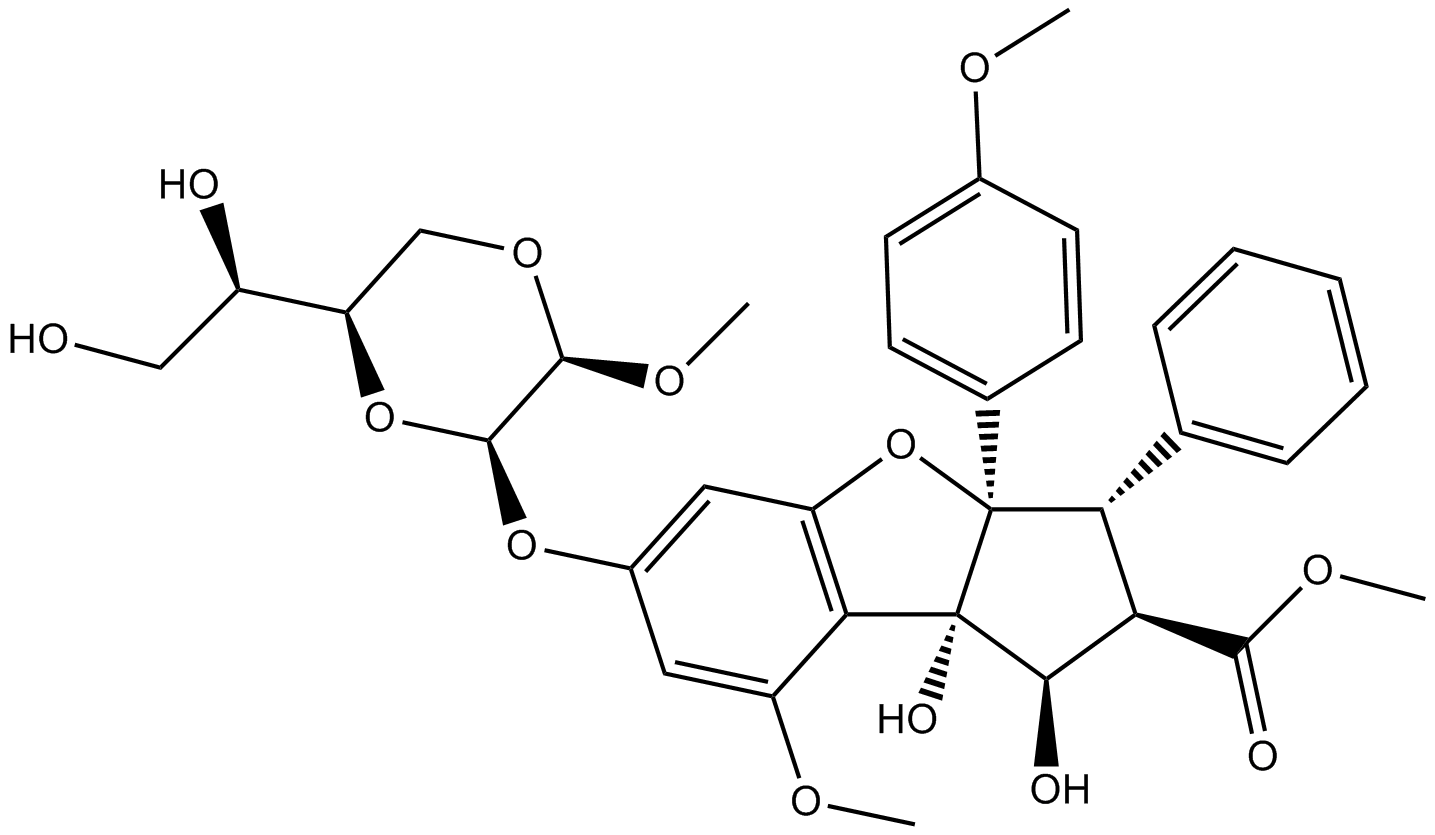
-
GC10585
Simvastatin (Zocor)
MK-733, SVA
Simvastatin (Zocor) (MK 733) ist ein kompetitiver Inhibitor von HMG-CoA-Reduktase mit einem Ki-Wert von 0,2 nM.
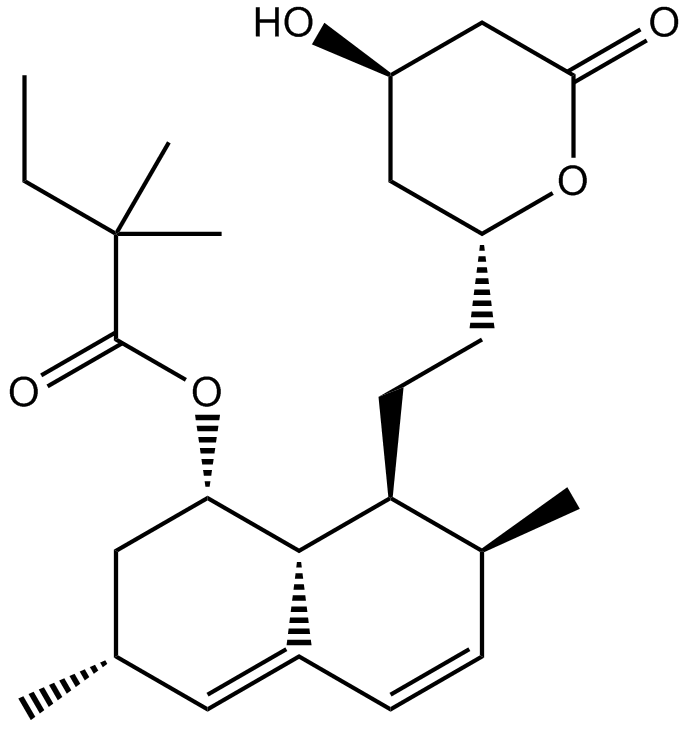
-
GN10213
Sinomenine Hydrochloride
Cucoline, NSC 76021
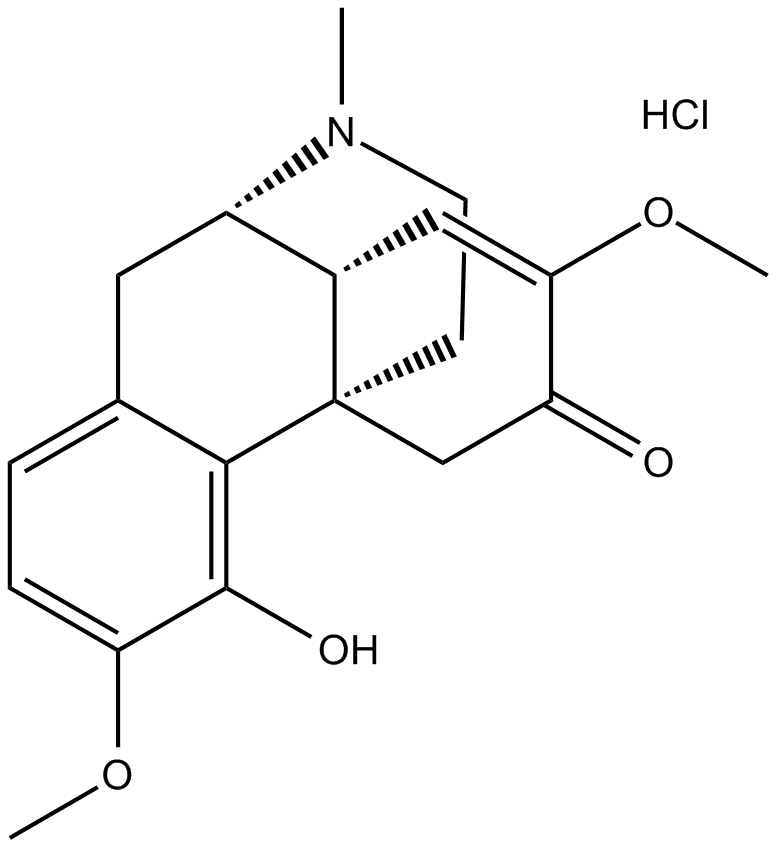
-
GC14945
Sirtinol
Sir Two Inhibitor Naphthol
Inhibitor of sirtuin deacetylases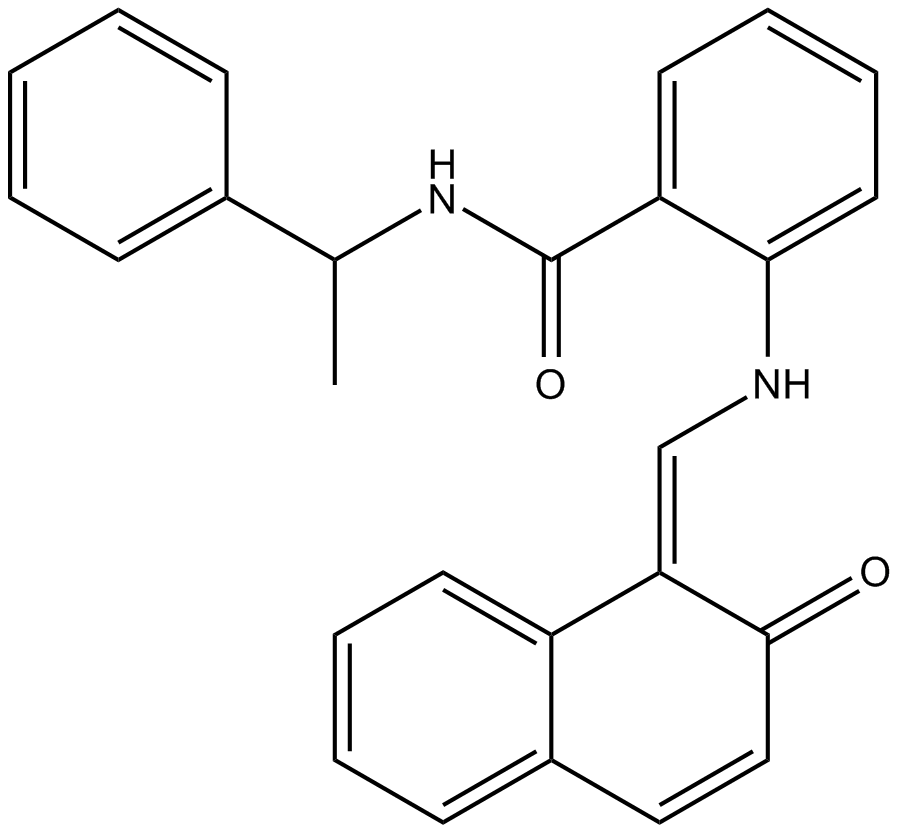
-
GC31317
Sitagliptin (MK0431)
Sitagliptin (MK0431) (MK-0431) ist ein potenter Inhibitor von DPP4 mit einem IC50 von 19 nM in Caco-2-Zellextrakten.
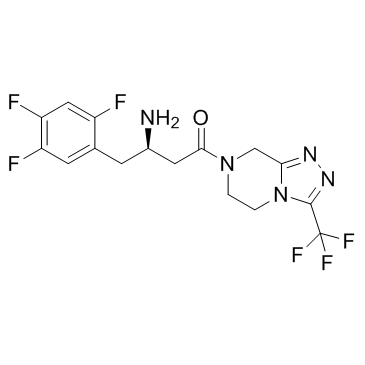
-
GC12235
Sitagliptin (phosphate)
INN,MK-431,ONO-5435
Sitagliptin (Phosphat) (MK-0431-Phosphat) ist ein potenter Inhibitor von DPP4 mit einem IC50 von 19 nM in Caco-2-Zellextrakten.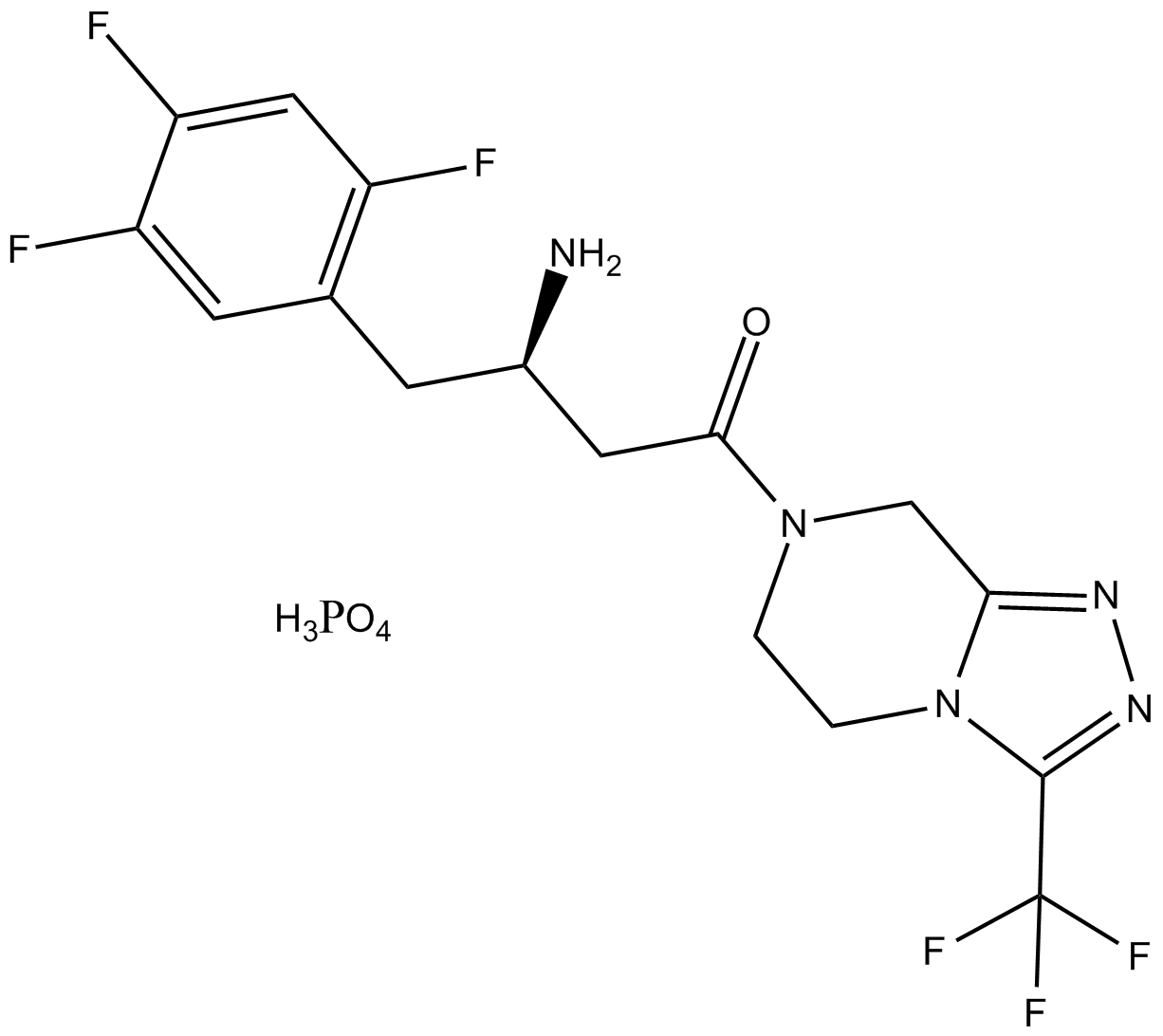
-
GC10298
Sitagliptin phosphate monohydrate
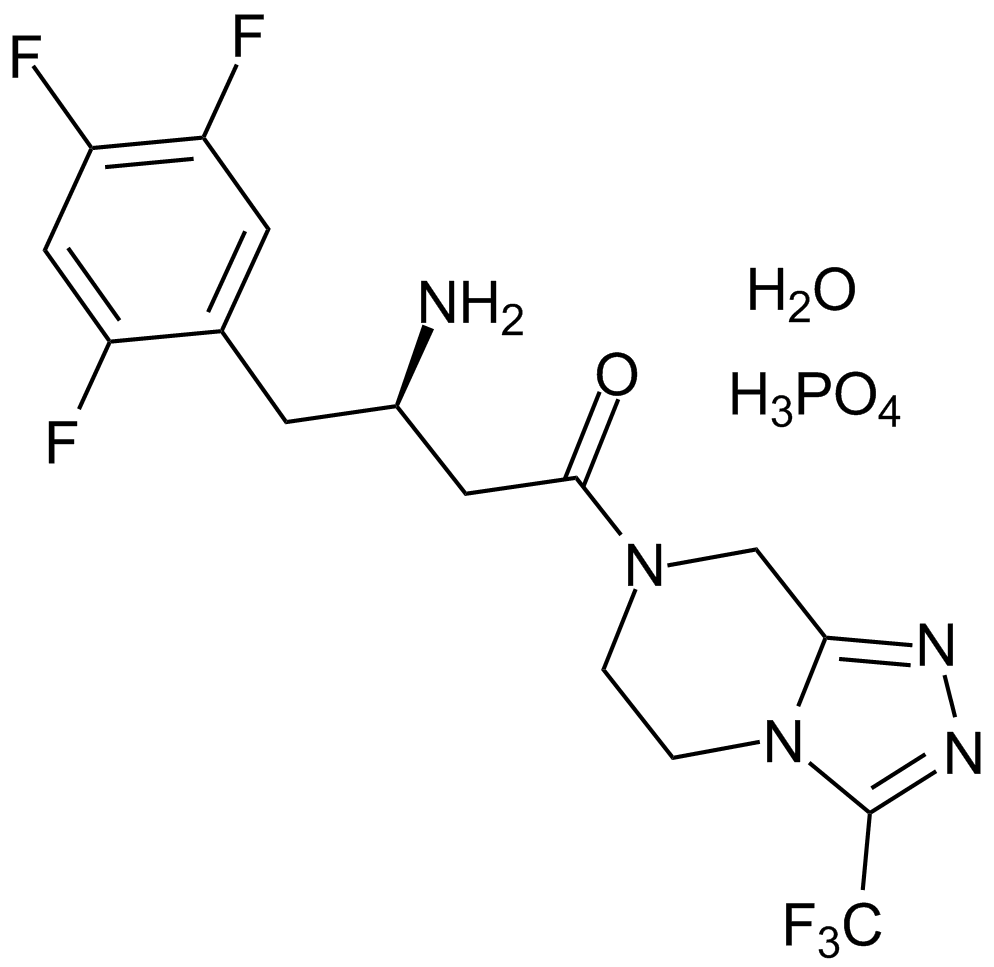
-
GC17698
SJB2-043
An inhibitor of the USP1-UAF1 complex and SARS-CoV-2 PLpro
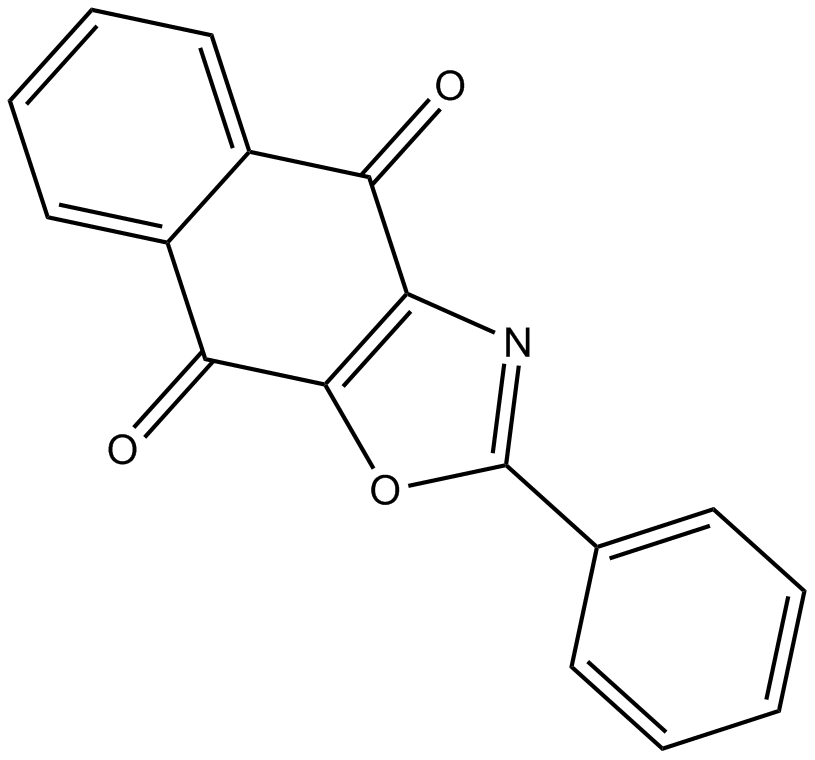
-
GC13401
SJB3-019A
SJB3-019A ist ein wirksamer und neuartiger USP1-Inhibitor, der fÜnfmal wirksamer als SJB2-043 bei der FÖrderung des ID1-Abbaus und der ZytotoxizitÄt in K562-Zellen mit einem IC50 von 0,0781 μM ist.
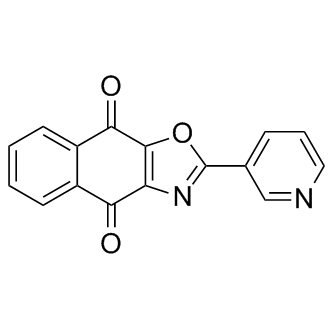
-
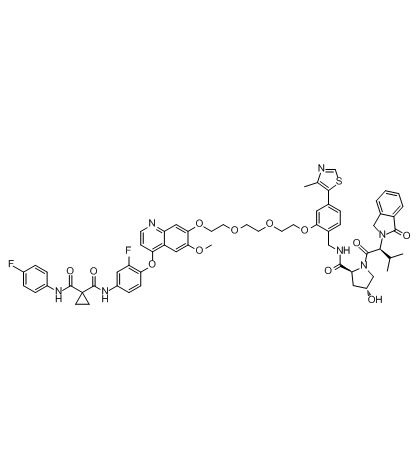
GC33229
SJFα
SJFα ist ein 13-atomiger Linker PROTAC basierend auf einem von Hippel-Lindau-Liganden. SJFα baut p38α mit einem DC50 von 7,16 nM ab, ist aber beim Abbau von p38δ weit weniger wirksam (DC50\u003d299 nM) und baut die anderen p38-Isoformen (β und γ) bei Konzentrationen bis zu 2,5 µM nicht ab.
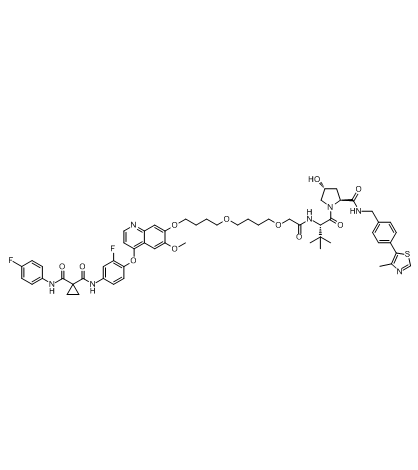
-
GC33238
SJFδ
SJFδ ist ein 10-atomiger Linker PROTAC basierend auf einem von Hippel-Lindau-Liganden. SJFδ baut p38δ mit einem DC50 von 46,17 nM ab, baut aber p38α, p38β oder p38γ nicht ab.
-
GC30646
Skatole(3-Methylindole)
3-Methyl-1H-indole
Skatol (3-Methylindol) wird von Darmbakterien produziert, reguliert die Zellfunktionen des Darmepithels durch Aktivierung von Arylkohlenwasserstoffrezeptoren und p38.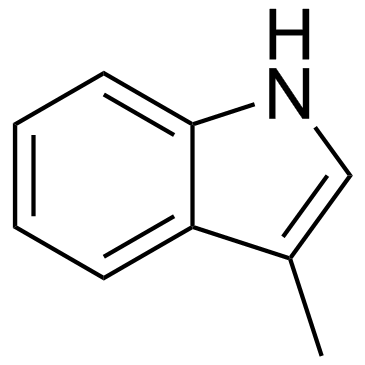
-
GC13578
Skepinone-L
Skepinone L
An inhibitor of p38 MAPK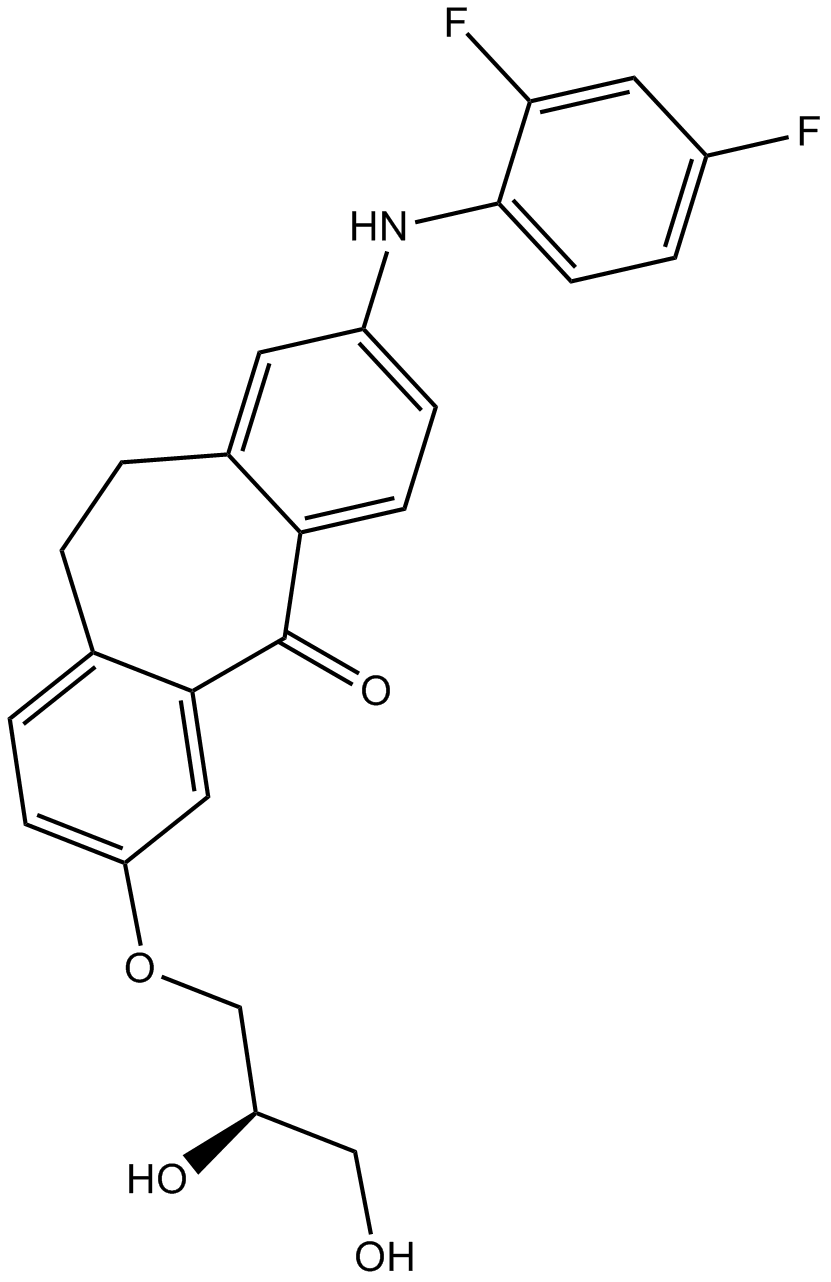
-
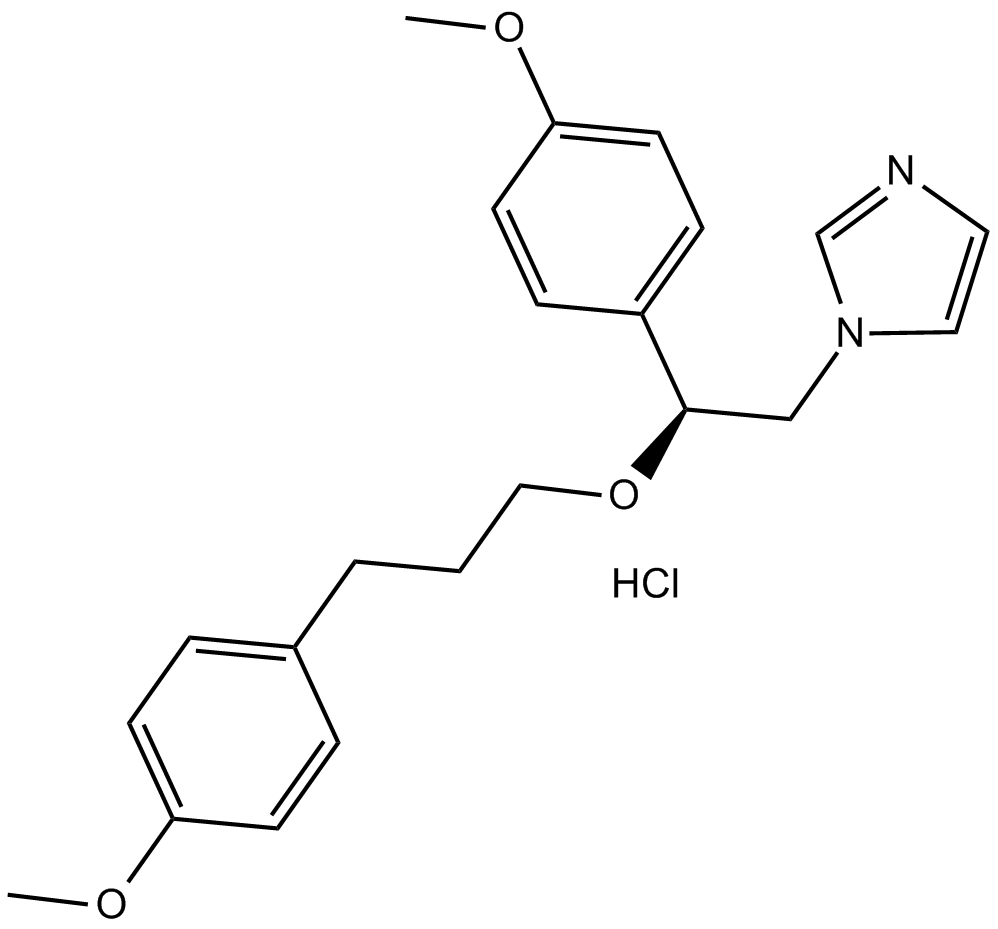
GC14641
SKF 96365 hydrochloride
SKF 96365 Hydrochlorid ist ein potenter TRP-Kanalblocker und ein im Handel erhältlicher Ca2+-Entry (SOCE)-Inhibitor.
-
GC37646
SKF-86002
SKF-86002 ist ein oral aktiver p38-MAPK-Inhibitor mit entzÜndungshemmenden, antiarthritischen und analgetischen AktivitÄten.
-
GC14272
SMER 28
SMER 28 ist ein positiver Regulator der Autophagie, der über einen mTOR-unabhängigen Mechanismus wirkt. SMER 28 verhindert die Ansammlung von Amyloid-Beta-Peptid.
-
GC15492
SMER 3
SCFMET30 Inhibitor, MET30 Antagonist
SMER 3, ein Rapamycin-Enhancer, ist ein selektiver Skp1-Cullin-F-Box (SCF)Met30-Ubiquitin-Ligase-Inhibitor. SMER 3 verstärkt die wachstumshemmende Wirkung von Rapamycin durch Hemmung von SCFMet30.
-
GC34117
SMER18
SMER18 ist ein kleiner MolekÜlverstÄrker von Rapamycin, der als mTOR-unabhÄngiger Autophagie-Induktor wirkt.
-
GN10449
SN-38 (NK012)
7Ethyl10Hydroxycamptothecin, 7ethyl10hydroxy20(S)Camptothecin, NK 012
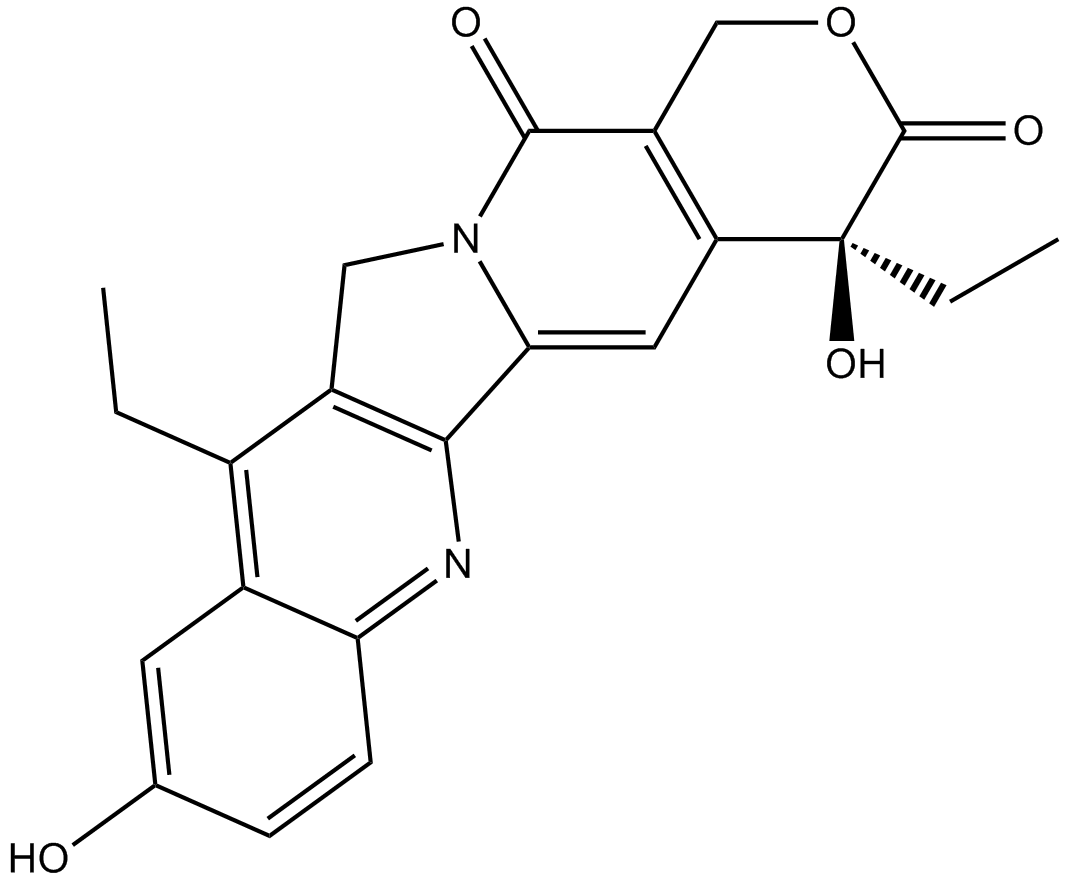
-
GC13946
SNX-2112
PF-04928473
A potent Hsp90 inhibitor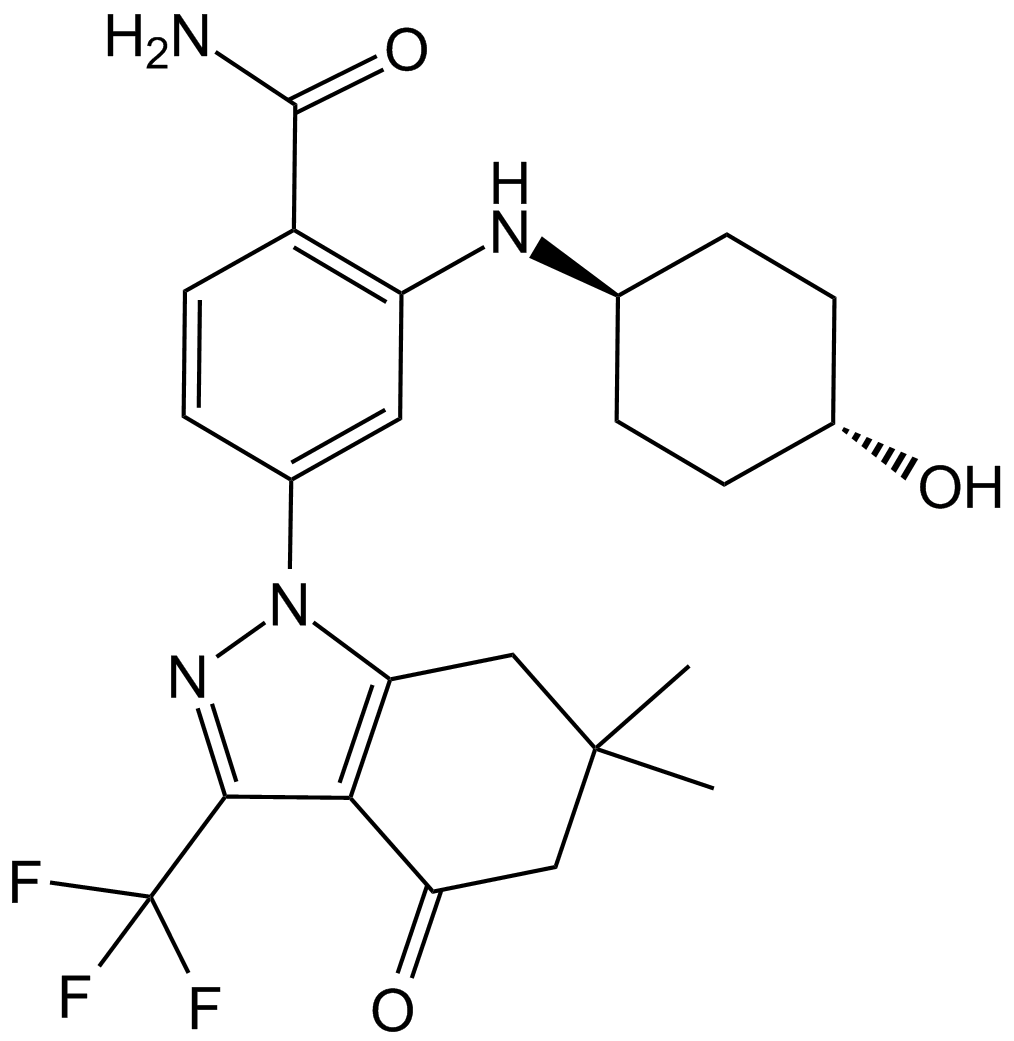
-
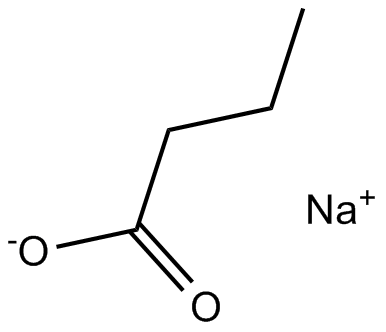
GC15857
Sodium butyrate
Butyric Acid
Histone deacetylase inhibitor
-
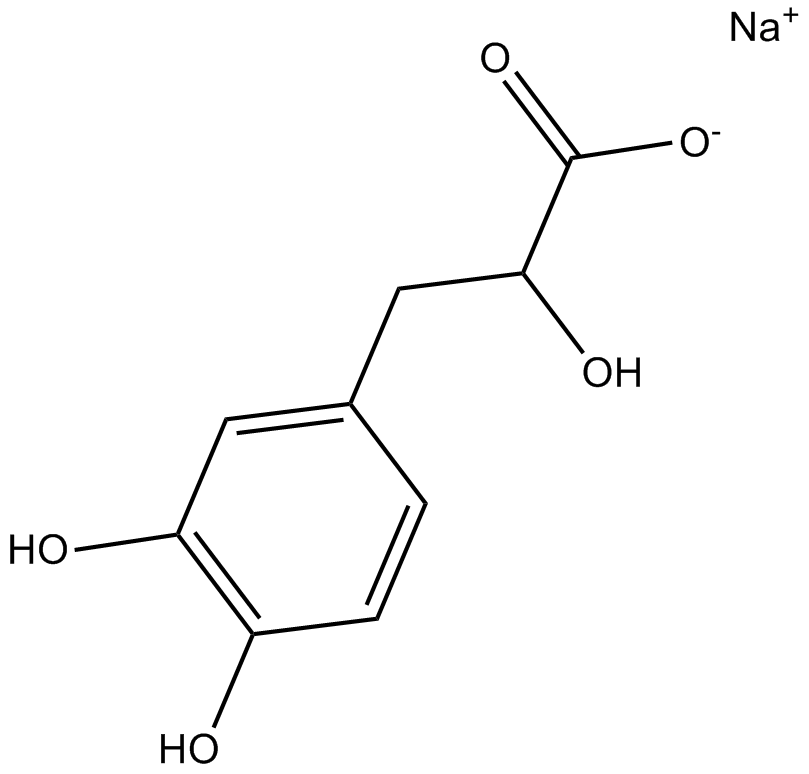
GN10100
Sodium Danshensu
-
GC15002
Sodium Phenylbutyrate
Benzenebutanoic Acid, NSC 657802, TriButyrate
A chemical chaperone