Epigenetic Reader Domain
Epigenetic regulators of gene expression and chromatin state include so-called writers, erasers, and readers of chromatin modifications.Well-characterized examples of reader domains include bromodomains typically binding acetyllysine and chromatin organization modifier (chromo), malignant brain tumor (MBT), plant homeodomain (PHD), and Tudor domains generally associating with methyllysine. Research on epigenetic readers has been tremendously influenced by the discovery of selective inhibitors targeting the bromodomain and extraterminal motif (BET) family of acetyl-lysine readers. The human genome encodes 46 proteins containing 61 bromodomains clustered into eight families. Distinct experimental approaches are used to identify the first BET inhibitors, GSK 525762A and (+)-JQ-1.
The Polycomb group (PcG) protein, enhancer of zeste homologue 2 (EZH2), has an essential role in promoting histone H3 lysine 27 trimethylation (H3K27me3) and epigenetic gene silencing. This function of EZH2 is important for cell proliferation and inhibition of cell differentiation, and is implicated in cancer progression. Cyclin-dependent kinases regulate epigenetic gene silencing through phosphorylation of EZH2. In many types of cancers including lymphomas and leukemia, EZH2 is postulated to exert its oncogenic effects via aberrant histone and DNA methylation, causing silencing of tumor suppressor genes.
p300/CBP is not only a transcriptional adaptor but also a histone acetyltransferase.
Targets for Epigenetic Reader Domain
Products for Epigenetic Reader Domain
- Cat.No. Nom du produit Informations
-
GC61595
(+)-JQ-1-aldehyde
(+)-JQ-1-aldéhyde est la forme aldéhyde de (+)-JQ1. (+)-JQ-1-aldéhyde peut être utilisé comme précurseur pour synthétiser les PROTAC, qui ciblent les bromodomaines BET.
-
GC34958
(+)-JQ1 PA
(+)-JQ1 PA est un dérivé du bromodomaine et de l'inhibiteur extra-terminal (BET) JQ1, avec une IC50 de 10,4 nM.
-
GC13822
(-)-JQ1
A selective inhibitor of BET bromodomains
-
GC63791
(R)-(-)-JQ1 Enantiomer
L'énantiomère (R)-(-)-JQ1 est le stéréoisomère de (+)-JQ1. (+)-JQ1 diminue puissamment l'expression des deux gènes cibles de BRD4, alors que l'énantiomère (R)-(-)-JQ1 n'a aucun effet.
-
GC34443
(R)-BAY1238097
(R)-BAY1238097 est l'isomère R avec une activité plus faible de BAY1238097. BAY1238097 est un inhibiteur puissant et sélectif de la liaison BET aux histones et possède une forte activité anti-proliférative dans différents modèles AML (leucémie myéloÏde aiguë) et MM (myélome multiple) grÂce À la régulation À la baisse des niveaux de c-Myc et de son transcriptome en aval.
-
GC34446
(rac)-BAY1238097
(Rac)-BAY1238097 est un inhibiteur de BET, avec une IC50 de 1,02 μM pour BRD4. Utilisé dans la recherche sur le cancer.
-
GC33198
(S)-JQ-35 (TEN-010)
(S)-JQ-35 (TEN-010) (TEN-010) est un inhibiteur des protéines contenant des bromodomaines de la famille Bromodomain et Extra-Terminal (BET) avec une activité antinéoplasique potentielle.
-
GC35175
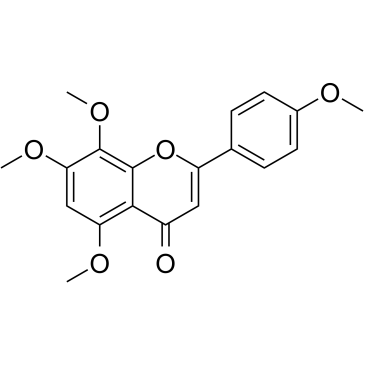
6-Demethoxytangeretin
La 6-déméthoxytangérétine est un flavonoÏde d'agrumes isolé de Citrus depressa.
-
GC62279
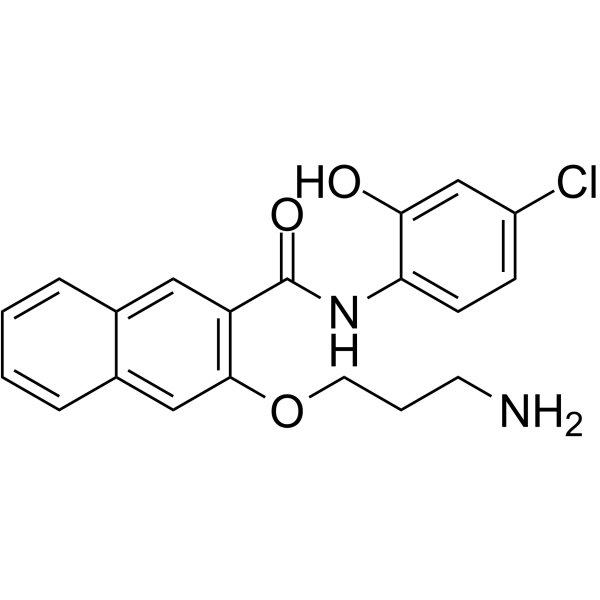
653-47
653-47, un potentialisateur, potentialise significativement l'activité inhibitrice de la protéine de liaison À l'élément de réponse À l'AMPc (CREB) de 666-15. Le 653-47 est également un inhibiteur CREB très faible avec une IC50 de 26,3 μM.
-
GC62144
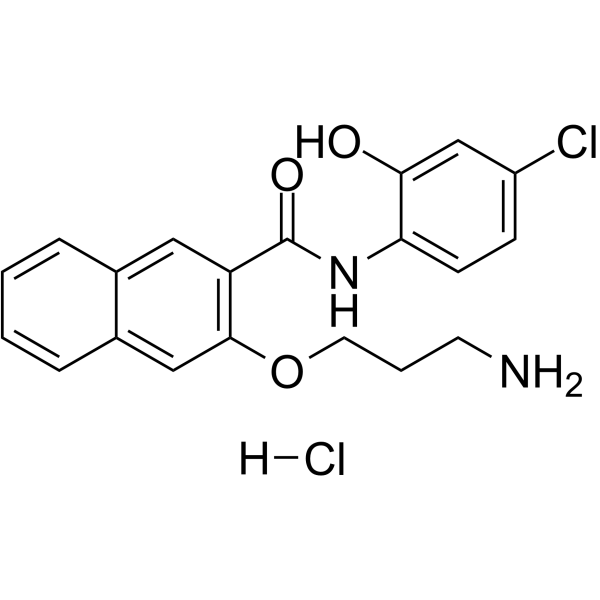
653-47 hydrochloride
Le chlorhydrate de 653-47, un potentialisateur, potentialise de manière significative l'activité inhibitrice de la protéine de liaison À l'élément de réponse À l'AMPc (CREB) de 666-15. Le chlorhydrate de 653-47 est également un très faible inhibiteur de CREB avec une IC50 de 26,3 μM.
-
GC32689
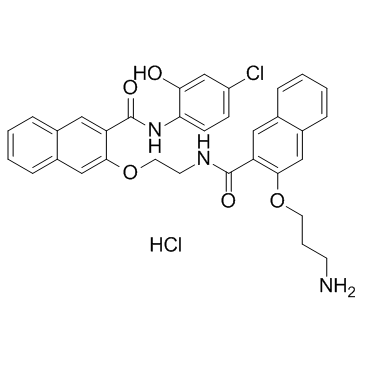
666-15
666-15 est un inhibiteur CREB puissant et sélectif avec une IC50 de 81 nM. 666-15 supprime la croissance tumorale dans un modèle de xénogreffe de cancer du sein.
-
GC32677
A-485
A-485 est un inhibiteur catalytique puissant et sélectif de p300/CBP avec des IC50 de 9,8nM et 2,6nM pour p300 et CBP histone acétyltransférase (HAT), respectivement.
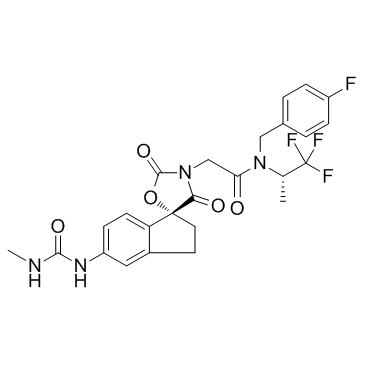
-
GC33280
A1874
A1874 est un PROTAC À base de nutline (ligand MDM2) et dégradant BRD4 avec un DC50 de 32 nM (induit la dégradation de BRD4 dans les cellules). Efficace pour inhiber la prolifération de nombreuses lignées cellulaires cancéreuses.
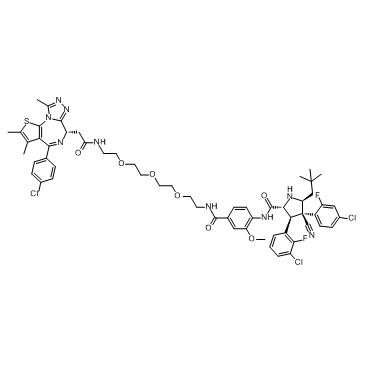
-
GC19409
ABBV-744
ABBV-744 est un inhibiteur sélectif et actif par voie orale, premier de sa catégorie, du domaine BDII des protéines de la famille BET avec des valeurs IC50 allant de 4 À 18 nM pour BRD2, BRD3, BRD4 et BRDT.
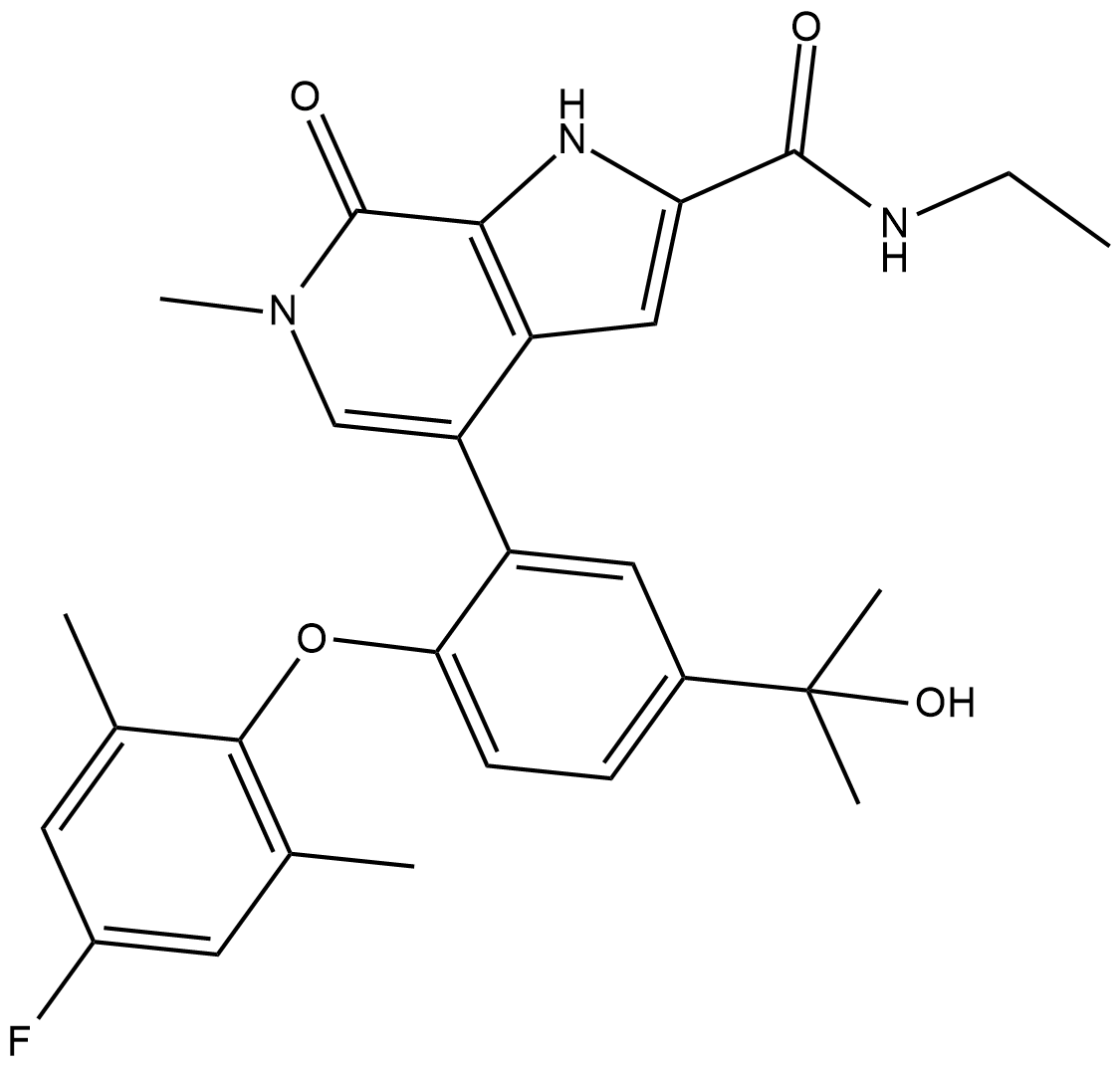
-
GC35227
ACBI1
ACBI1 est un dégradeur puissant et coopératif de SMARCA2, SMARCA4 et PBRM1 avec des DC50 de 6, 11 et 32 nM, respectivement. ACBI1 est un dégradeur de PROTAC. ACBI1 montre une activité anti-proliférative. ACBI1 induit l'apoptose.
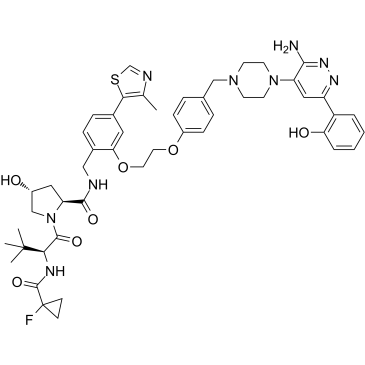
-
GC35297
Alobresib
L'alobrésib (GS-5829) est un inhibiteur du bromodomaine BET, qui représente un agent thérapeutique hautement efficace contre le carcinome séreux utérin (USC) récurrent/résistant À la chimiothérapie surexprimant c-Myc.
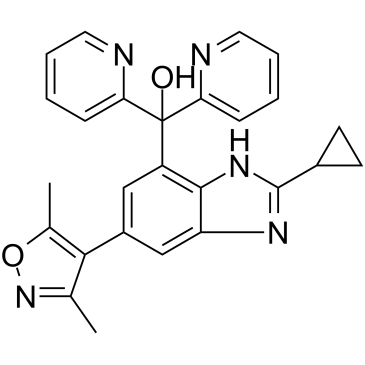
-
GC67912
Amredobresib

-
GC17284
Anacardic acid
A histone acetyltransferase inhibitor
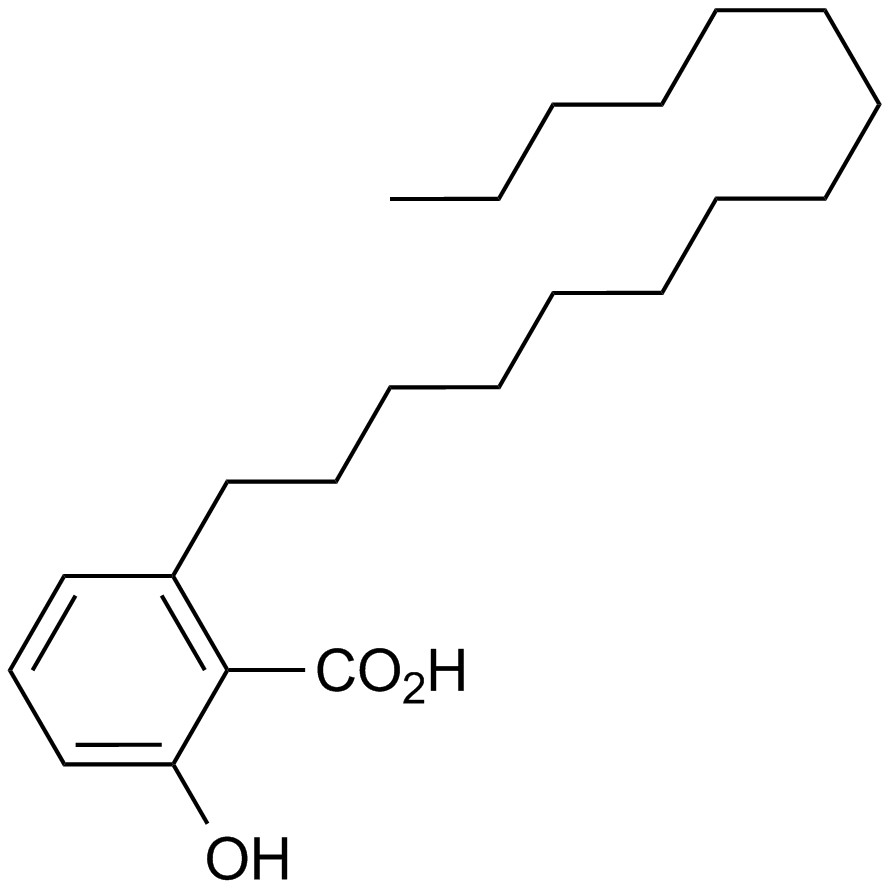
-
GC32685
ARV-771
ARV-771 est un puissant BET PROTAC basé sur la ligase E3 de Hippel-Lindau avec des Kd de 34 nM, 4,7 nM, 8,3 nM, 7,6 nM, 9,6 nM et 7,6 nM pour BRD2(1), BRD2(2), BRD3( 1), BRD3(2), BRD4(1) et BRD4(2), respectivement.
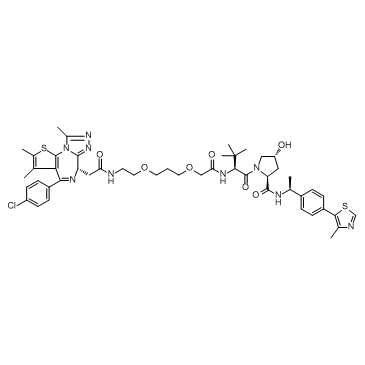
-
GC19038
ARV-825
ARV-825 est un PROTAC connecté par des ligands pour Cereblon et BRD4. ARV-825 se lie À BD1 et BD2 de BRD4 avec des Kd de 90 et 28 nM, respectivement.
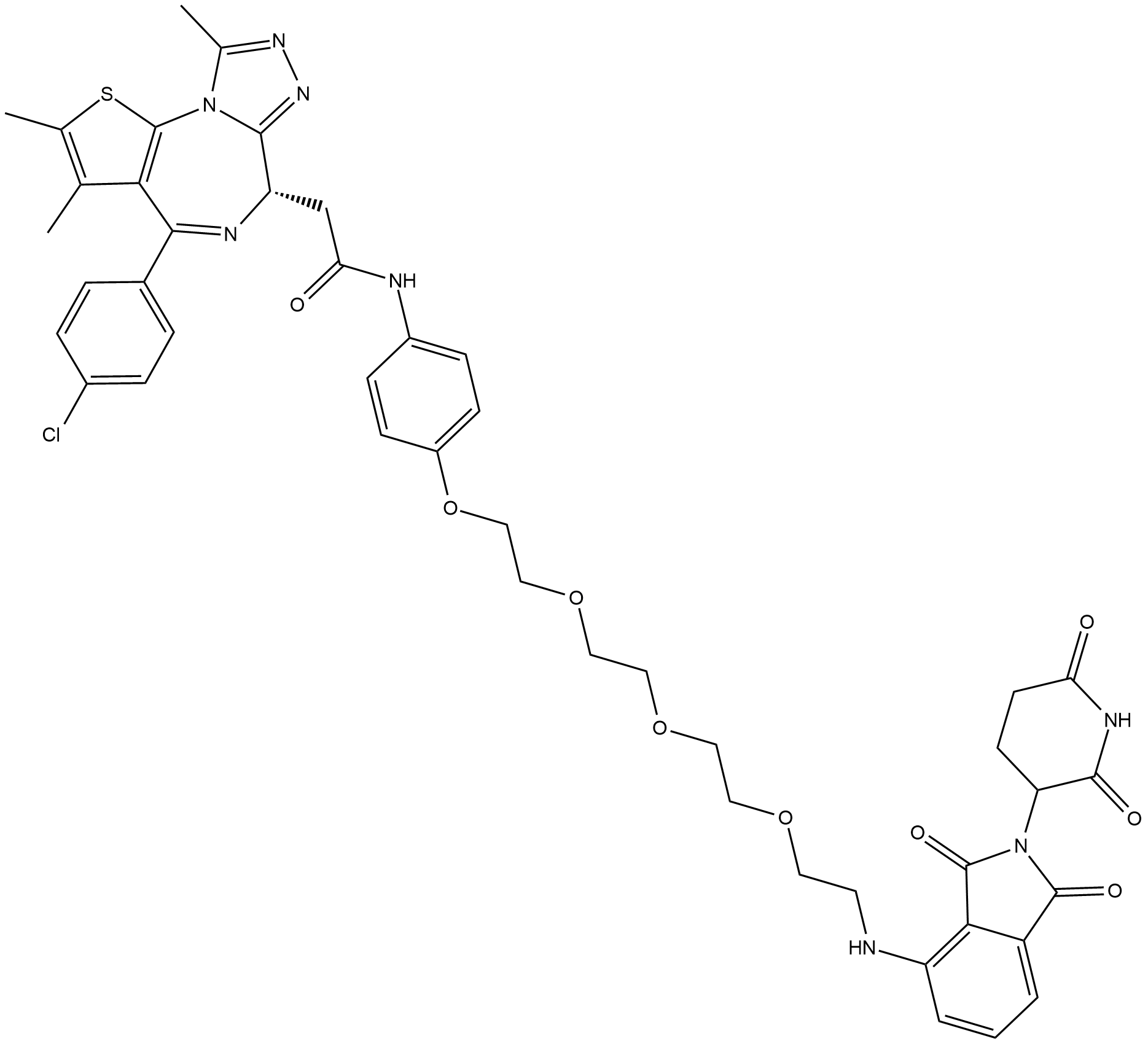
-
GC64271
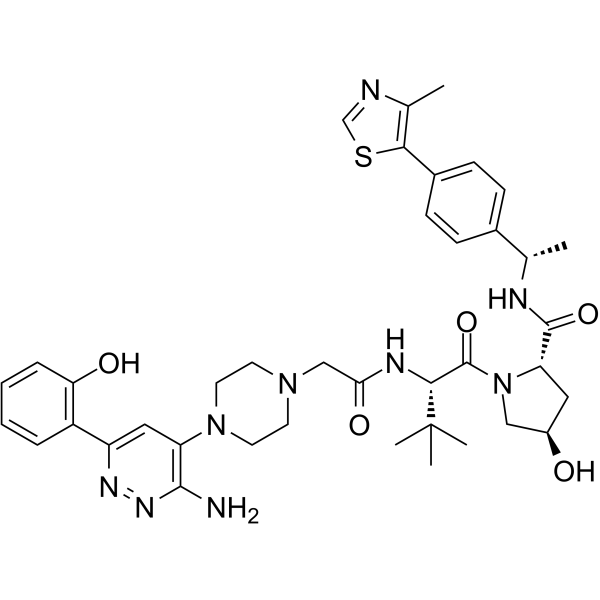
AU-15330
AU-15330 est un dégradeur chimère ciblant la protéolyse (PROTAC) des sous-unités SWI/SNF ATPase, SMARCA2 et SMARCA4. L'AU-15330 induit une puissante inhibition de la croissance tumorale dans les modèles de xénogreffe du cancer de la prostate et agit en synergie avec l'enzalutamide, un antagoniste de l'AR. L'AU-15330 induit une rémission de la maladie dans des modèles de cancer de la prostate résistant À la castration (CRPC) sans toxicité.
-
GC35448
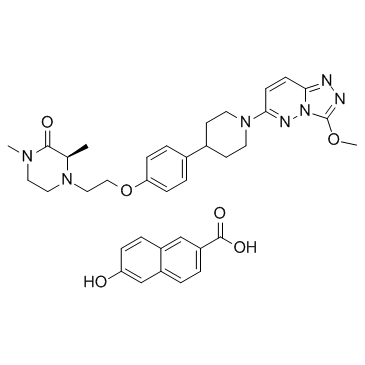
AZD5153 6-Hydroxy-2-naphthoic acid
AZD5153 L'acide 6-hydroxy-2-naphtoÏque est l'acide 6-hydroxy-2-naphtoÏque de l'AZD5153. AZD5153 est un inhibiteur de bromodomaine BET/BRD4 puissant, sélectif et disponible par voie orale; perturbe BRD4 avec une IC50 de 1,7 nM.
-
GC18508
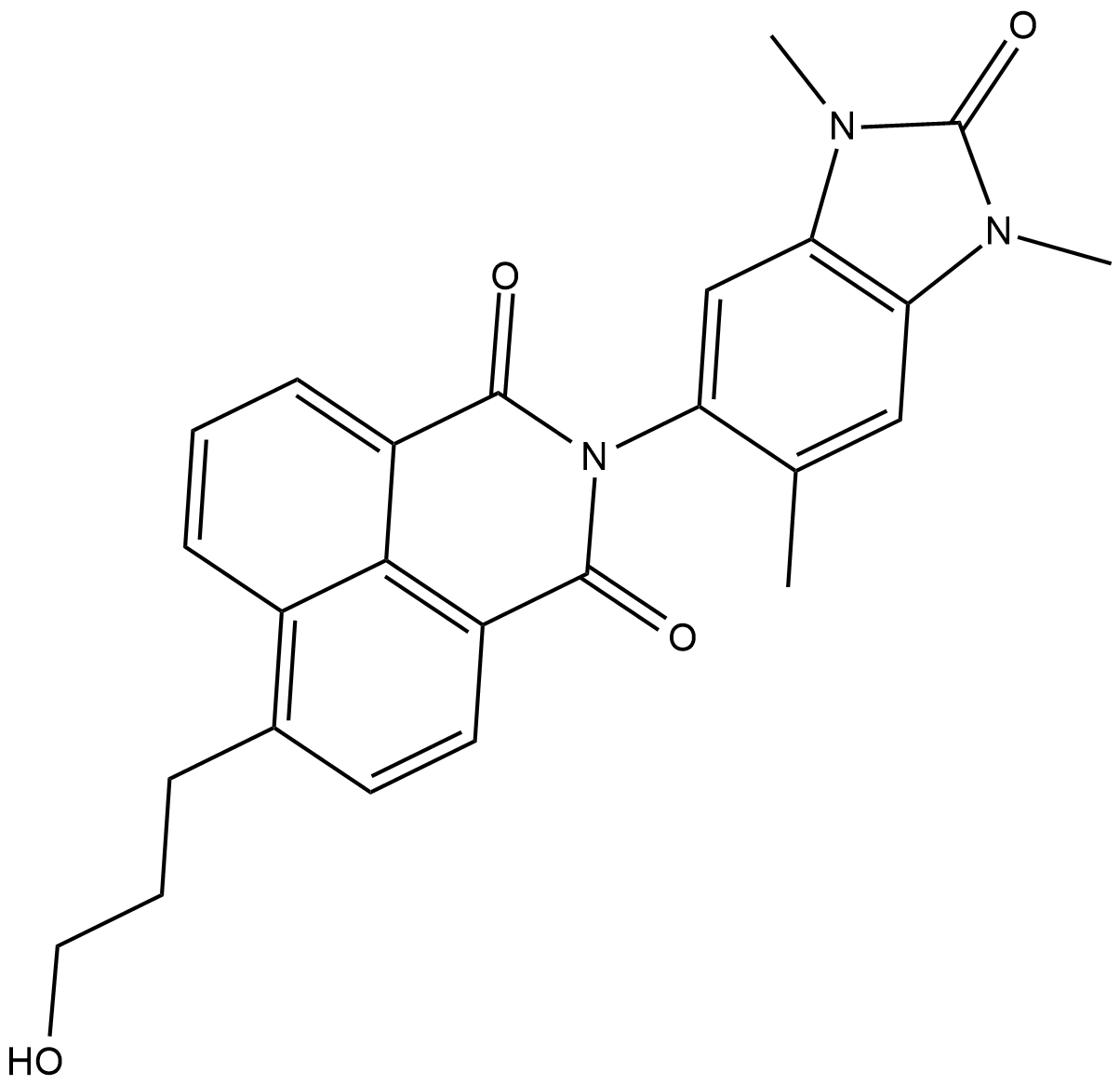
BAY-299
BAY-299 est un double inhibiteur très puissant avec des IC50 de 67 nM pour les bromodomaines BRPF2 (BD), 8 nM pour TAF1 BD2 et 106 nM pour TAF1L BD2.
-
GC33104
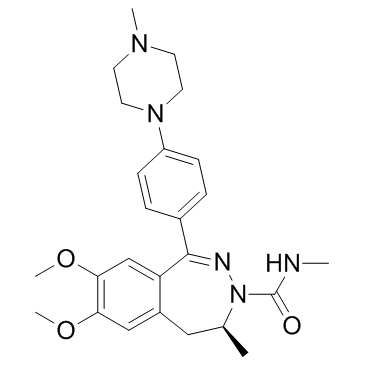
BAY1238097
BAY1238097 est un inhibiteur puissant et sélectif de la liaison de BET aux histones et possède une forte activité antiproliférative dans différents modèles de LMA (leucémie myéloÏde aiguë) et de MM (myélome multiple) grÂce À la régulation À la baisse des niveaux de c-Myc et de son transcriptome en aval (IC50 < 100nM).
-
GC63694
BAZ1A-IN-1
BAZ1A-IN-1 est un puissant inhibiteur de BAZ1A (protéine contenant du bromodomaine). BAZ1A-IN-1 montre une valeur KD de 0,52 μM contre le bromodomaine BAZ1A. BAZ1A-IN-1 montre une bonne activité anti-viabilité contre les lignées cellulaires cancéreuses exprimant un niveau élevé de BAZ1A, mais une activité faible ou nulle contre les cellules cancéreuses avec un faible niveau d'expression de BAZ1A.
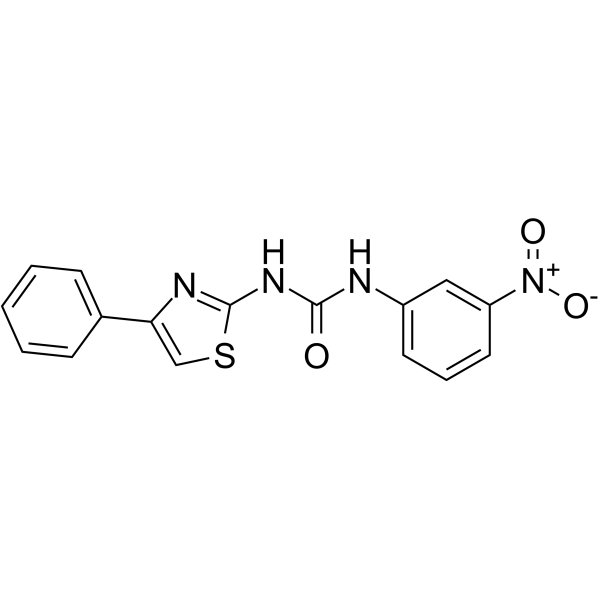
-
GC15175
BAZ2-ICR
BAZ2-ICR est un inhibiteur des bromodomaines BAZ2A/B puissant, sélectif, actif sur les cellules et actif par voie orale avec des IC50 de 130 nM et 180 nM, et des Kd de 109 nM et 170 nM, respectivement. BAZ2-ICR montre une sélectivité de 10 À 15 fois pour la liaison de BAZ2A/B sur CECR2 et une sélectivité > 100 fois sur tous les autres bromodomaines. BAZ2-ICR est une sonde chimique épigénétique.
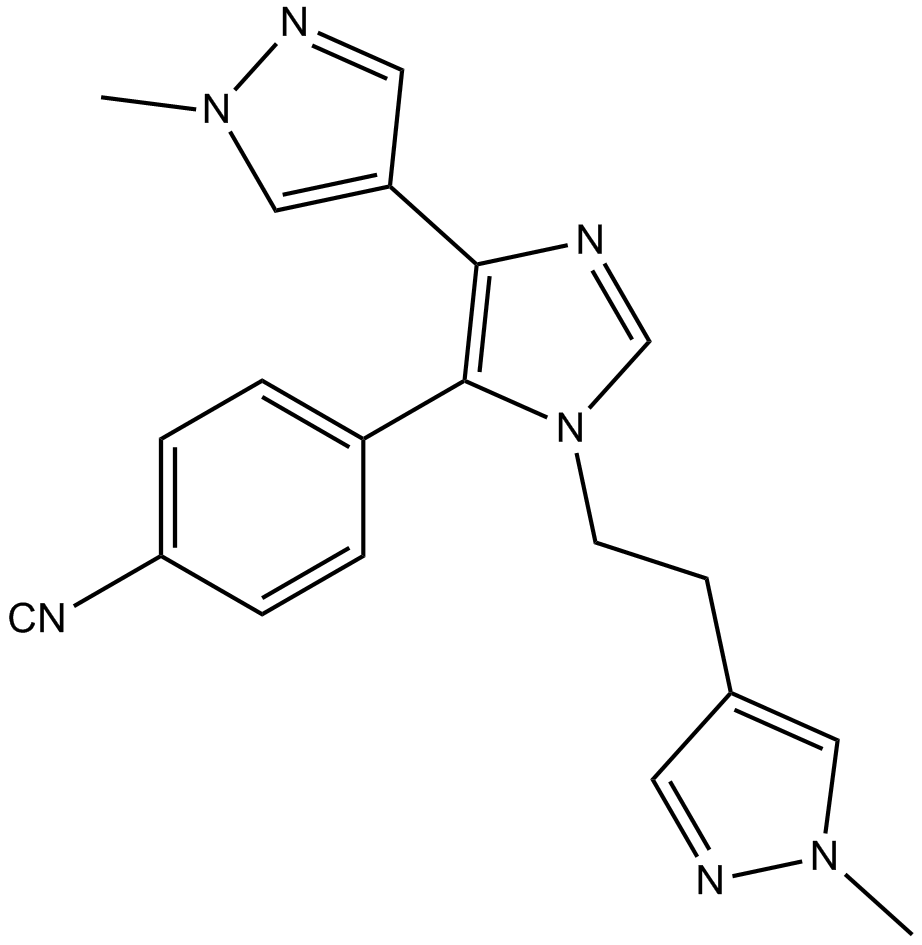
-
GC16299
BET bromodomain inhibitor
L'inhibiteur de bromodomaine BET est un puissant inhibiteur de BET extrait du brevet WO/2015/153871A2, exemple de composé 11.
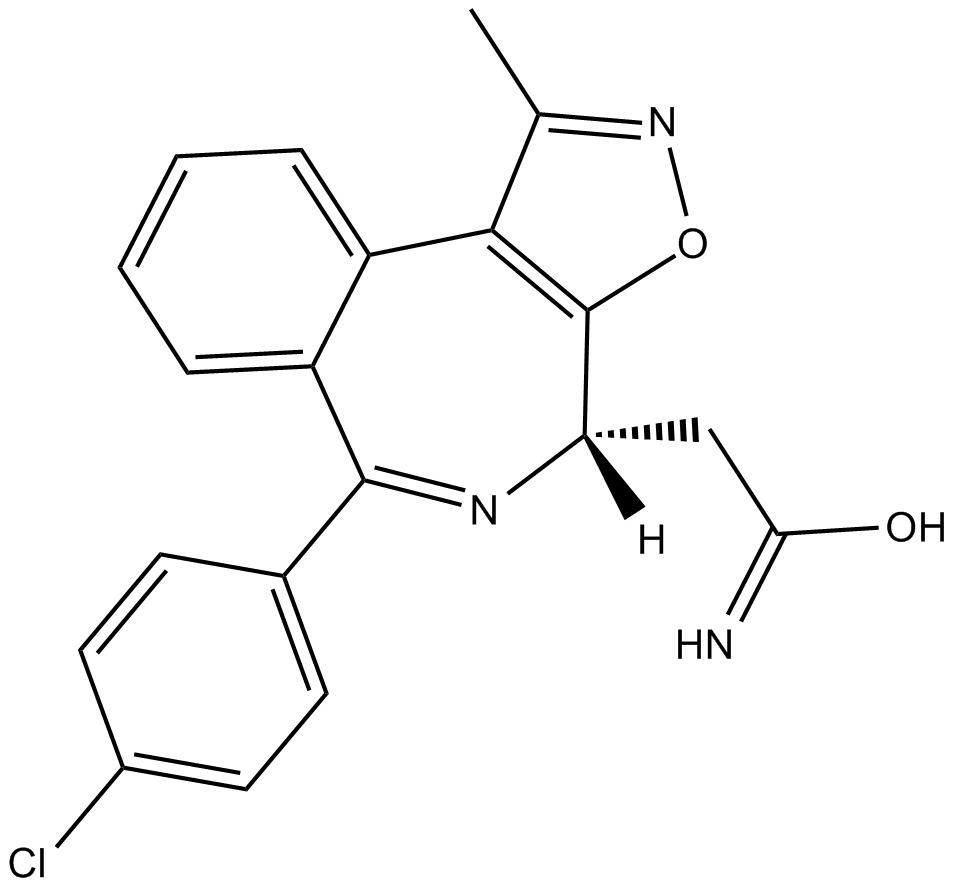
-
GC63528
BET bromodomain inhibitor 1
L'inhibiteur 1 du bromodomaine BET est un inhibiteur sélectif du bromodomaine et du bromodomaine extra-terminal (BET) actif par voie orale avec une IC50 de 2,6 nM pour BRD4. L'inhibiteur de bromodomaine BET 1 se lie À BRD2(2), BRD3(2), BRD4(1), BRD4(2) et BRDT(2) avec des affinités élevées (valeurs Kd de 1,3 nM, 1,0 nM, 3,0 nM, 1,6 nM, 2,1 nM, respectivement). l'inhibiteur de bromodomaine 1 a une activité anticancéreuse.
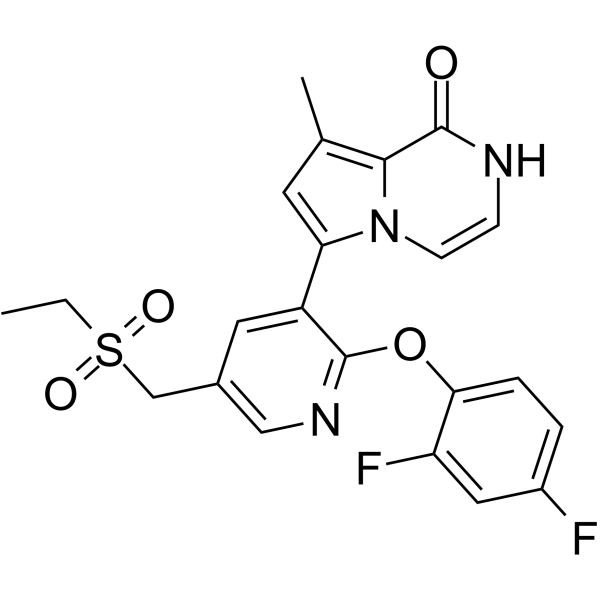
-
GC35505
BET-BAY 002
BET-BAY 002 est un puissant inhibiteur de BET; montre son efficacité dans un modèle de myélome multiple.
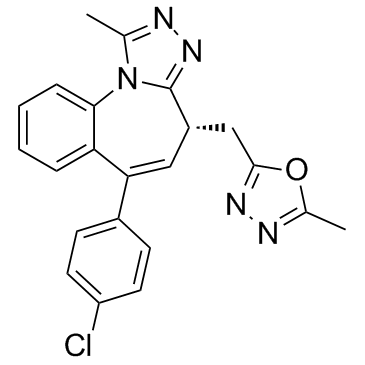
-
GC35506
BET-BAY 002 S enantiomer
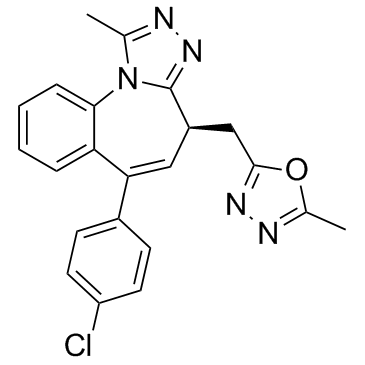
-
GC31831
BET-IN-1
BET-IN-1 est un pan-inhibiteur des huit bromodomaines BET et une sélectivité par rapport À d'autres protéines représentatives contenant des bromodomaines.
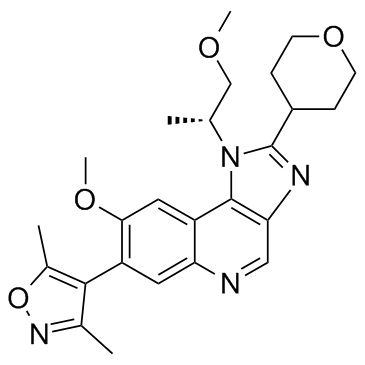
-
GC65506
BETd-246
BETd-246 est un inhibiteur du bromodomaine BET (BRD) de deuxième génération et basé sur PROTAC, connecté par des ligands pour Cereblon et BET, présentant une sélectivité, une puissance et une activité antitumorale supérieures.
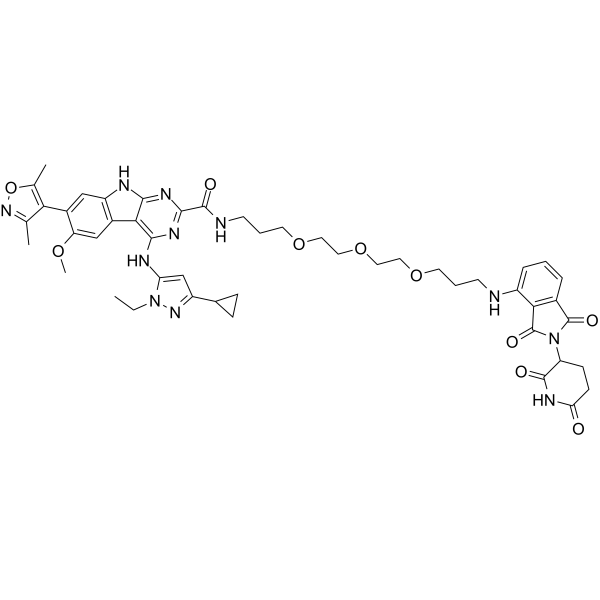
-
GC32791
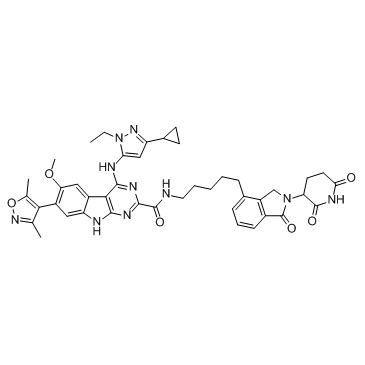
BETd-260 (ZBC 260)
BETd-260 (ZBC 260) (ZBC 260) est un PROTAC connecté par des ligands pour Cereblon et BET, avec aussi peu que 30 pM contre la protéine BRD4 dans la lignée cellulaire de leucémie RS4;11. BETd-260 (ZBC 260) supprime puissamment la viabilité cellulaire et induit de manière robuste l'apoptose dans les cellules de carcinome hépatocellulaire (HCC).
-
GC12450
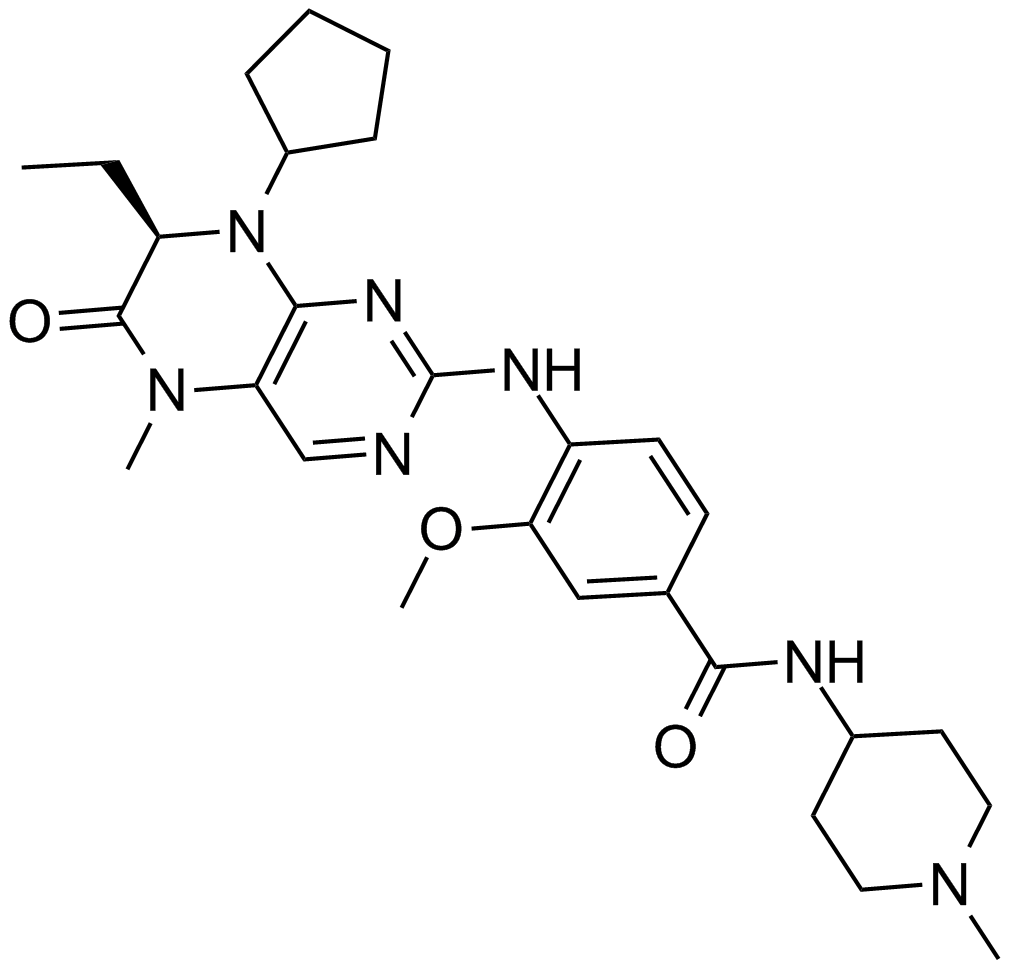
BI 2536
Un inhibiteur puissant de Plk1
-
GC16726
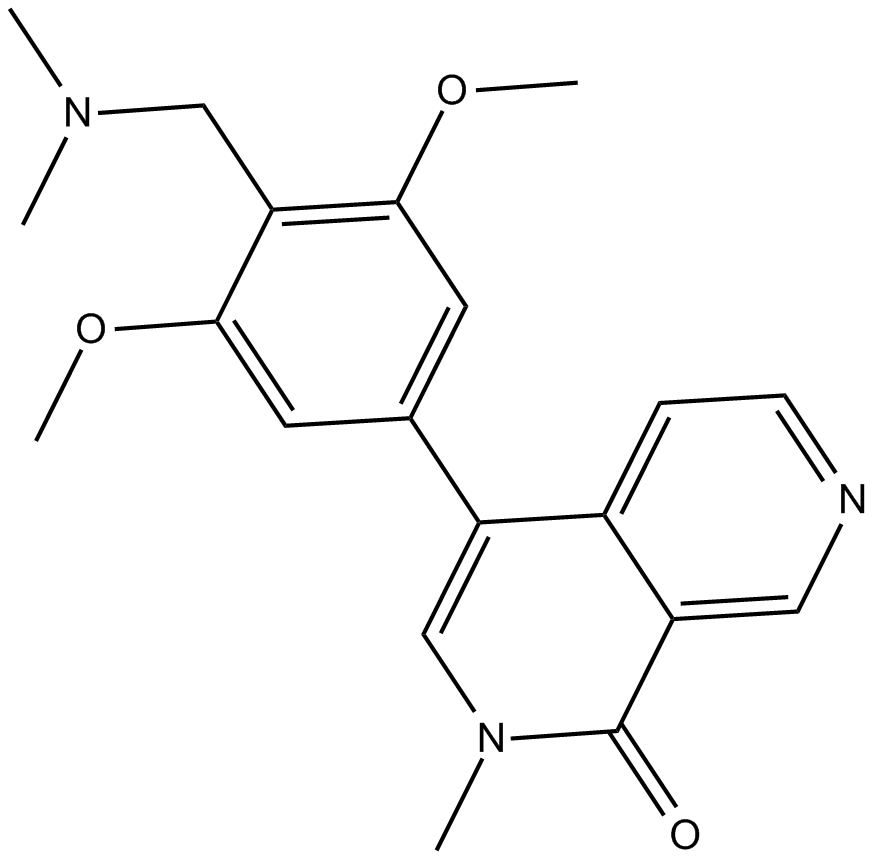
BI-7273
BI-7273 est un inhibiteur de BRD9 sélectif et perméable aux cellules, avec une IC50 et un Kd de 19 et 0,75 nM; montre également un effet élevé sur BRD7, avec une IC50 et un Kd de 117 nM et 0,3 nM.
-
GC16817
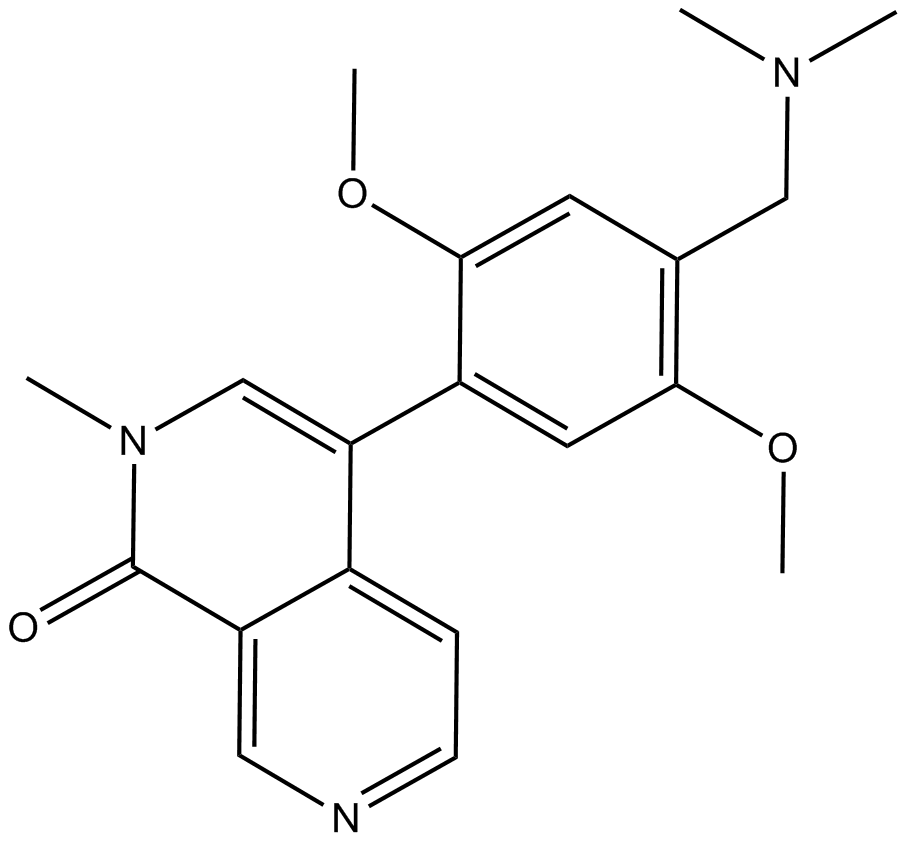
BI-9564
Le BI-9564 est un inhibiteur des bromodomaines BRD9/BRD7 puissant, sélectif et perméable aux cellules, avec des IC50 de 75 nM et 3,4 μM et des Kd de 14 nM et 239 nM, respectivement. Le BI-9564 a une IC50 > 100 μM pour la famille BET.
-
GC64789
Biotinylated-JQ1
Biotinylé-JQ1 (biotine-JQ1) est un dérivé biotinylé de JQ1 avec une haute affinité pour le bromodomaine de BRD4. Le JQ1 biotinylé inhibe la prolifération des cellules de myélome multiple MM1.S avec une EC50 de 0,4μM.
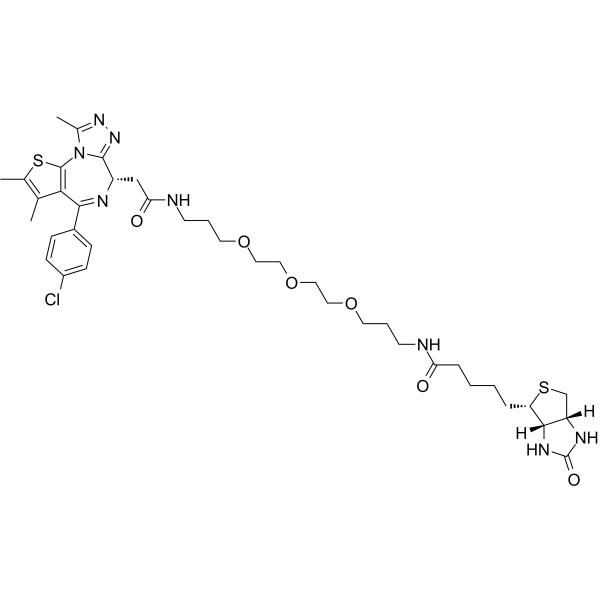
-
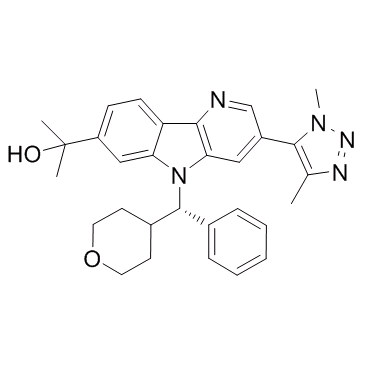
GC32812
BMS-986158
Le BMS-986158 est un puissant inhibiteur de BET avec des IC50 de 6,6 et 5nM dans les cellules NCI-H211 du cancer du poumon À petites cellules (SCLC) et les cellules MDA-MB231 du cancer du sein triple négatif (TNBC), respectivement.
-
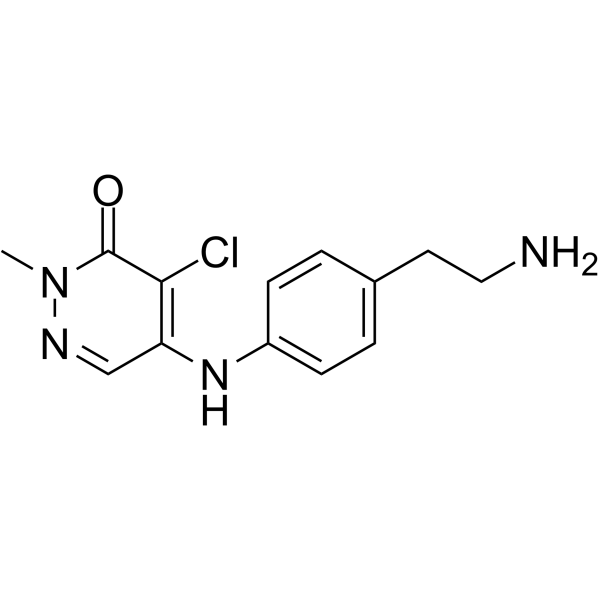
GC64538
BPTF-IN-BZ1
BPTF-IN-BZ1, un inhibiteur de BPTF, possède une puissance élevée (Kd = 6,3 nM).
-
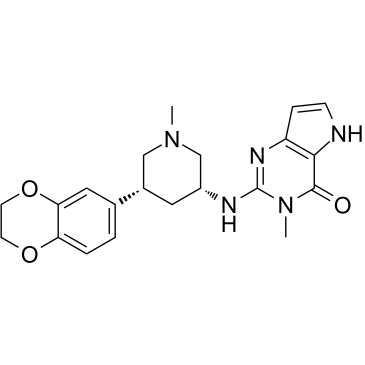
GC35552
BRD-IN-3
BRD-IN-3 ((R,R)-36n) est un inhibiteur très puissant du bromodomaine PCAF (BRD), avec une IC50 de 7 nM. BRD-IN-3 présente également une activité contre GCN5 et FALZ.
-
GC33017
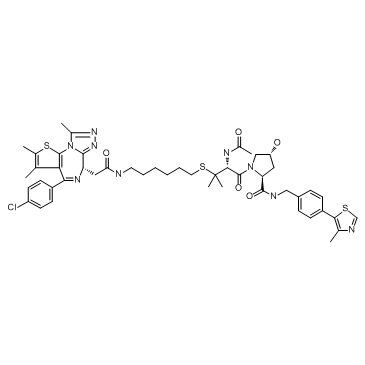
BRD4 degrader AT1
Le dégradeur de BRD4 AT1 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BRD4 en tant que dégradeur de Brd4 hautement sélectif, avec un Kd de 44 nM pour Brd4BD2 dans les cellules.
-
GC64960
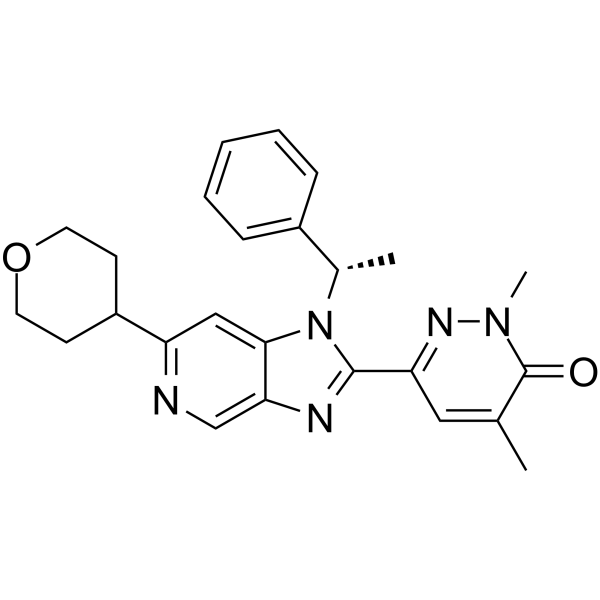
BRD4 Inhibitor-10
BRD4 Inhibitor-10 est un puissant inhibiteur de BRD4-BD1 extrait du brevet WO2015022332A1, Composé II-25, a une IC50 de 8 nM.
-
GC65569
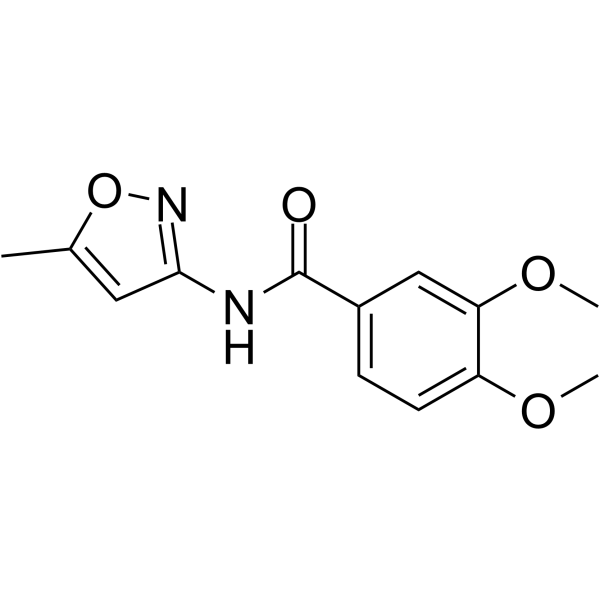
BRD4 Inhibitor-24
BRD4 Inhibitor-24 (composé 3U) est un puissant inhibiteur de BRD4, BRD4 Inhibitor-24 présente une activité antitumorale contre les cellules MCF7 et K652, avec des valeurs IC50 de 33,7 et 45,9 μM, respectivement (extrait du brevet CN107721975A).
-
GC66441
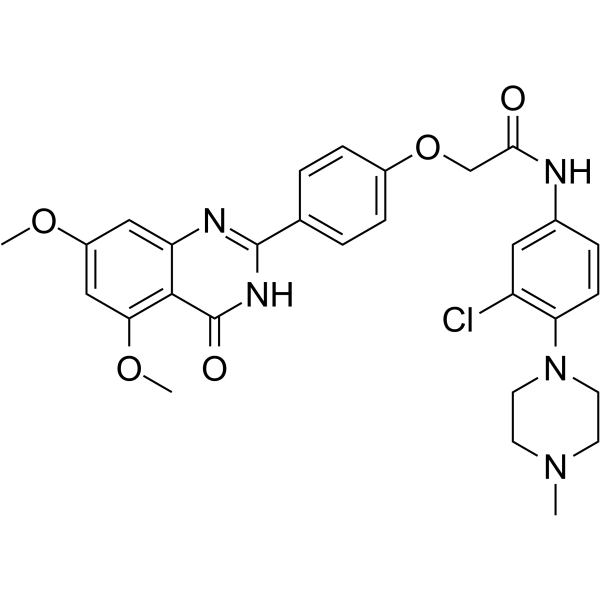
BRD4/CK2-IN-1
BRD4/CK2-IN-1 est le premier inhibiteur actif À double cible hautement efficace et oral de BRD4/CK2 (protéine 4/caséine kinase 2 contenant du bromodomaine), avec des IC50 de 180 nM et 230 nM pour BRD4 et CK2, respectivement. BRD4/CK2-IN-1 a une forte activité anticancéreuse sans toxicités évidentes. BRD4/CK2-IN-1 induit l'apoptose et la mort cellulaire associée À l'autophagie dans le cancer du sein triple négatif (TNBC)
-
GC34505
BRD7-IN-1
BRD7-IN-1, un dérivé modifié de BI7273 (inhibiteur de BRD7/9), se lie À un ligand VHL via un lieur pour former un PROTAC VZ185 (VZ185 contre BRD7/9 avec DC50 de 4,5 et 1,8 nM, respectivement).
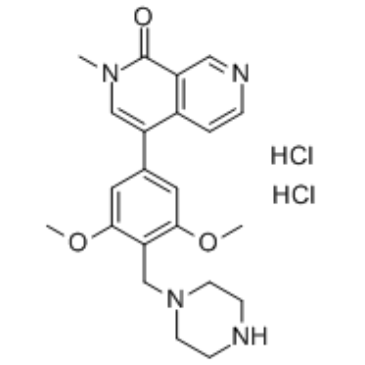
-
GC64541
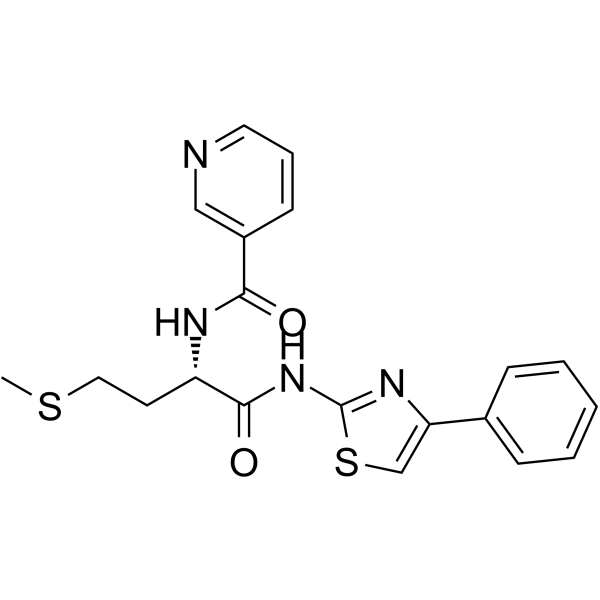
BRM/BRG1 ATP Inhibitor-2
BRM/BRG1 ATP Inhibitor-2 est un inhibiteur de BRG1/BRM ATPase pour le traitement des troubles liés au BAF.
-
GC35557
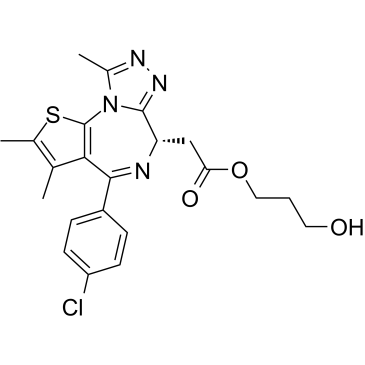
Bromodomain IN-1
Bromodomain IN-1 est un inhibiteur de Bromodomain extrait du brevet WO2016069578A1, composé 4 .
-
GC16531
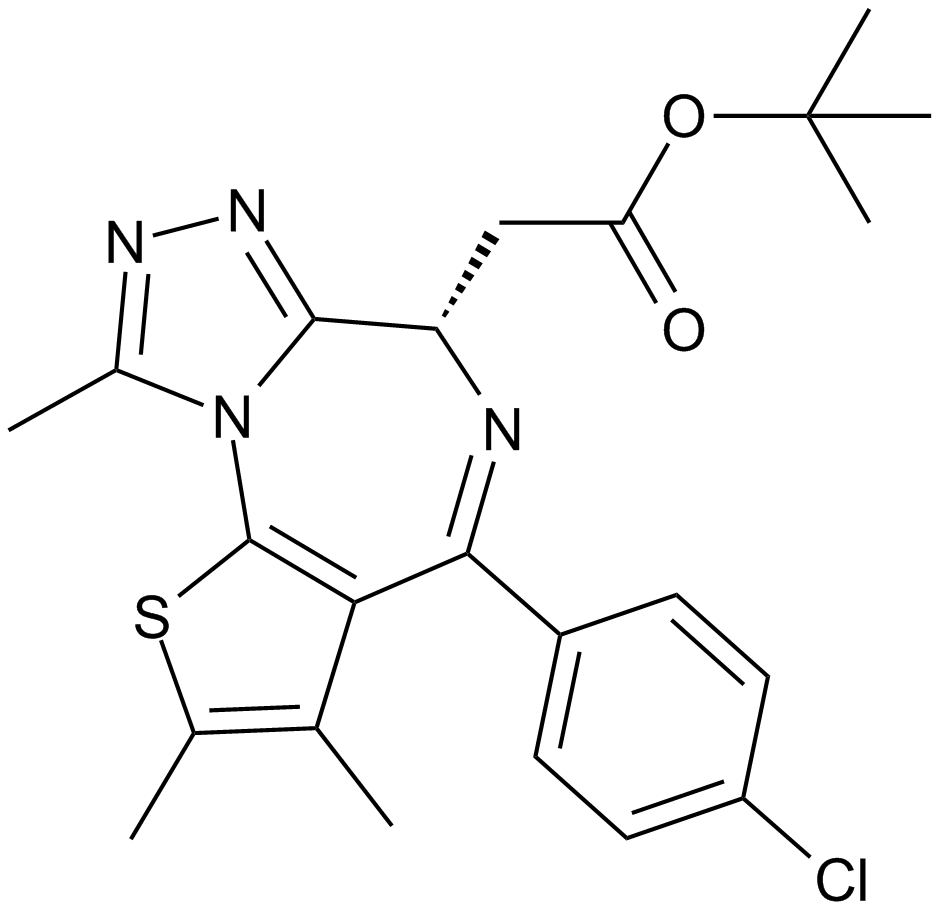
Bromodomain Inhibitor, (+)-JQ1
A selective inhibitor of BET bromodomains
-
GC35558
Bromodomain inhibitor-8
L'inhibiteur de bromodomaine-8 (intermédiaire 21) est un inhibiteur de bromodomaine BET pour le traitement des maladies auto-immunes et inflammatoires.
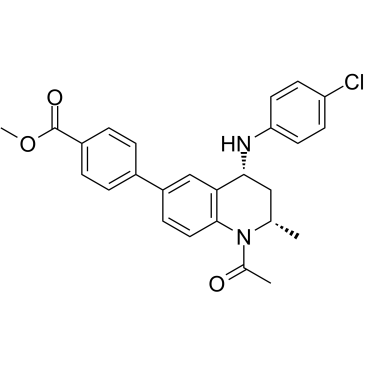
-
GC10402
Bromosporine
A non-specific bromodomain inhibitor
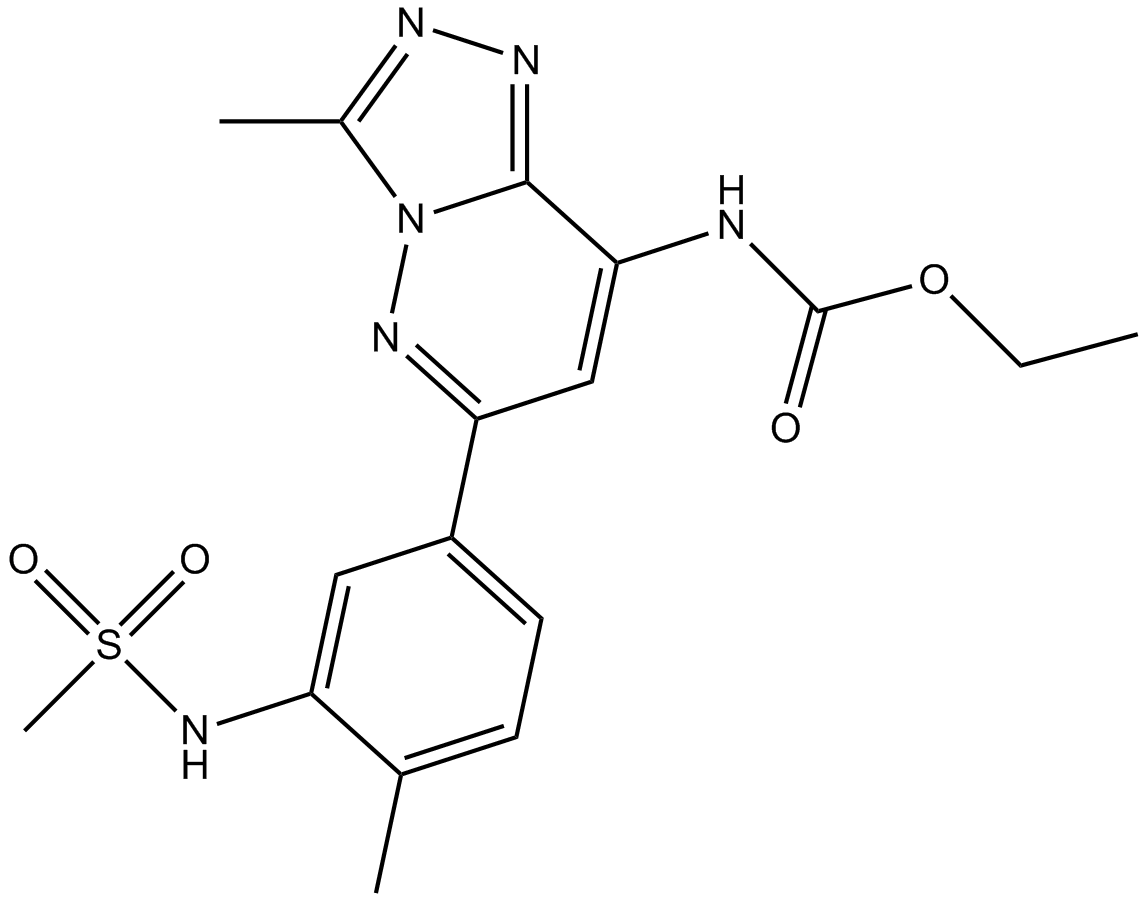
-
GC12733
C646
C646 est un inhibiteur sélectif et compétitif de l'histone acétyltransférase p300 avec Ki de 400 nM, et est moins puissant pour les autres acétyltransférases.
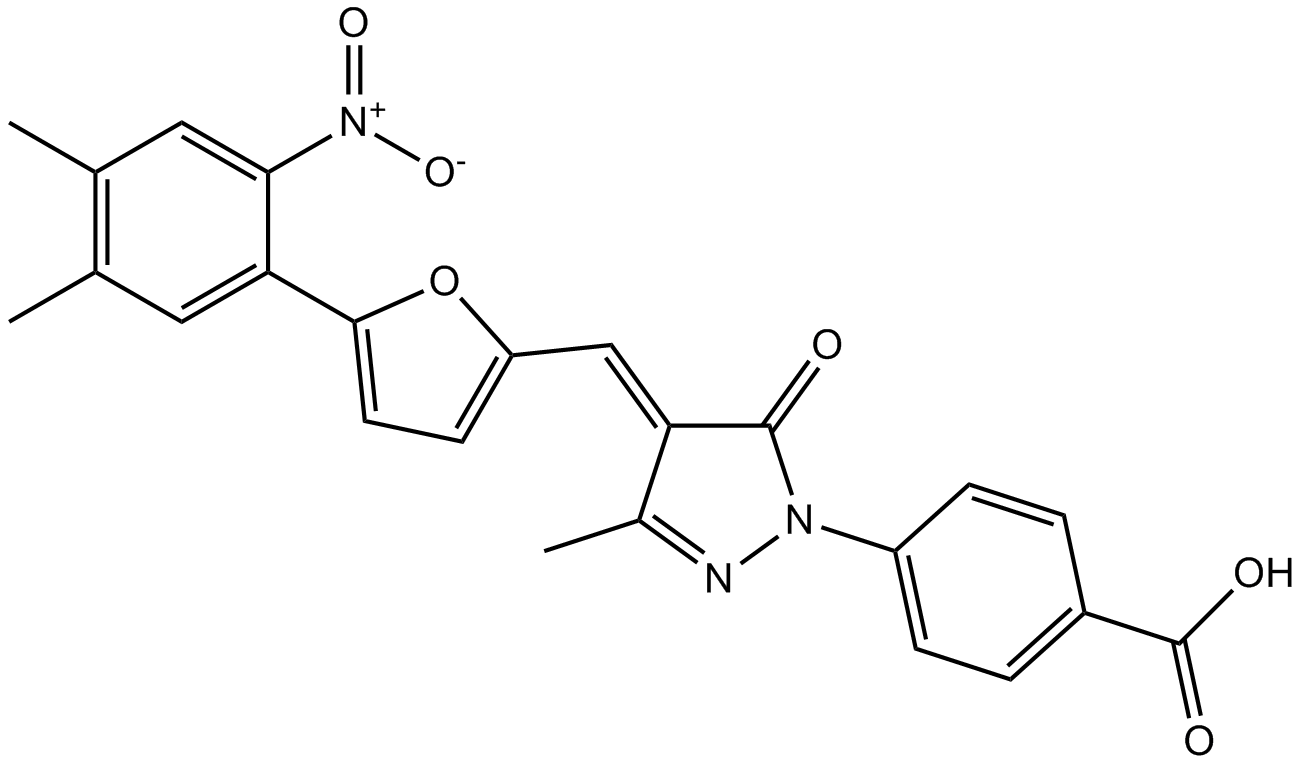
-
GC34517
CBP-IN-1
CBP-IN-1 (CCS1477) est un inhibiteur oral, puissant et sélectif du bromodomaine p300/CBP.
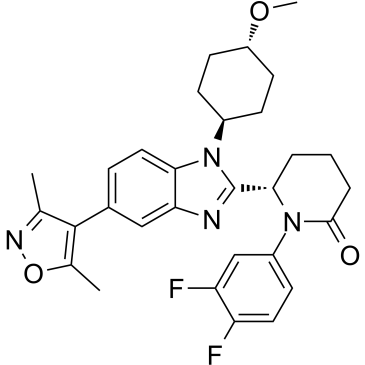
-
GC61577
CBP/p300-IN-1
CBP/p300-IN-1 est un inhibiteur de bromodomaine CBP/EP300.
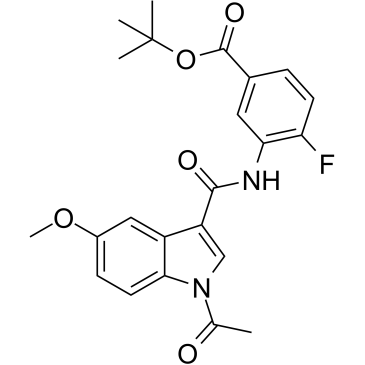
-
GC64696
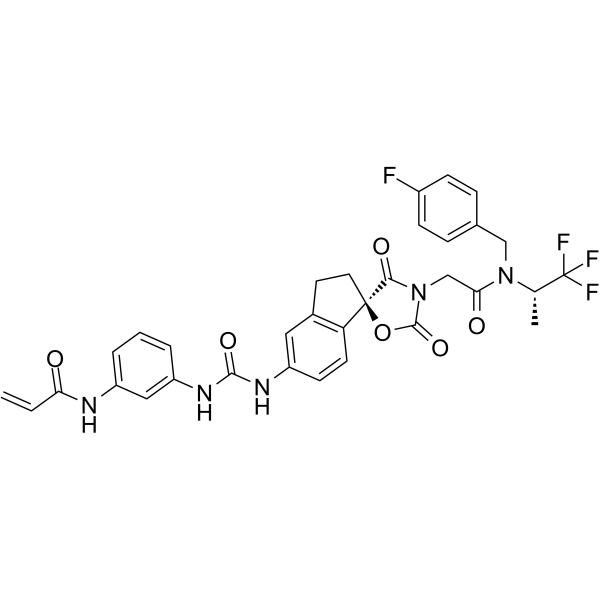
CBP/p300-IN-12
CBP/p300-IN-12 est un inhibiteur covalent puissant et sélectif des histones acétyltransférases p300 (IC50 de 166 nM) et CBP. CBP/p300-IN-12 diminue les niveaux de H3K27Ac des cellules PC-3 (EC50 de 37 nM). CBP/p300-IN-12 forme un adduit covalent avec C1450.
-
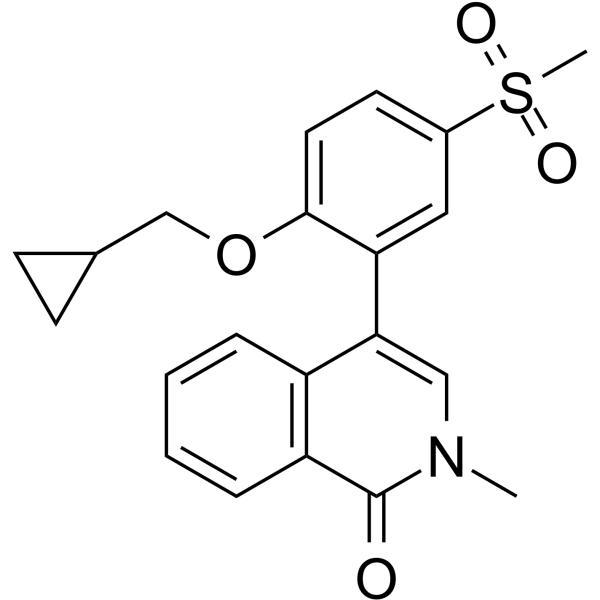
GC62330
CC-90010
Le CC-90010 (composé 1) est un inhibiteur de BET réversible et actif par voie orale. CC-90010 est appliqué dans l'étude des tumeurs solides avancées.
-
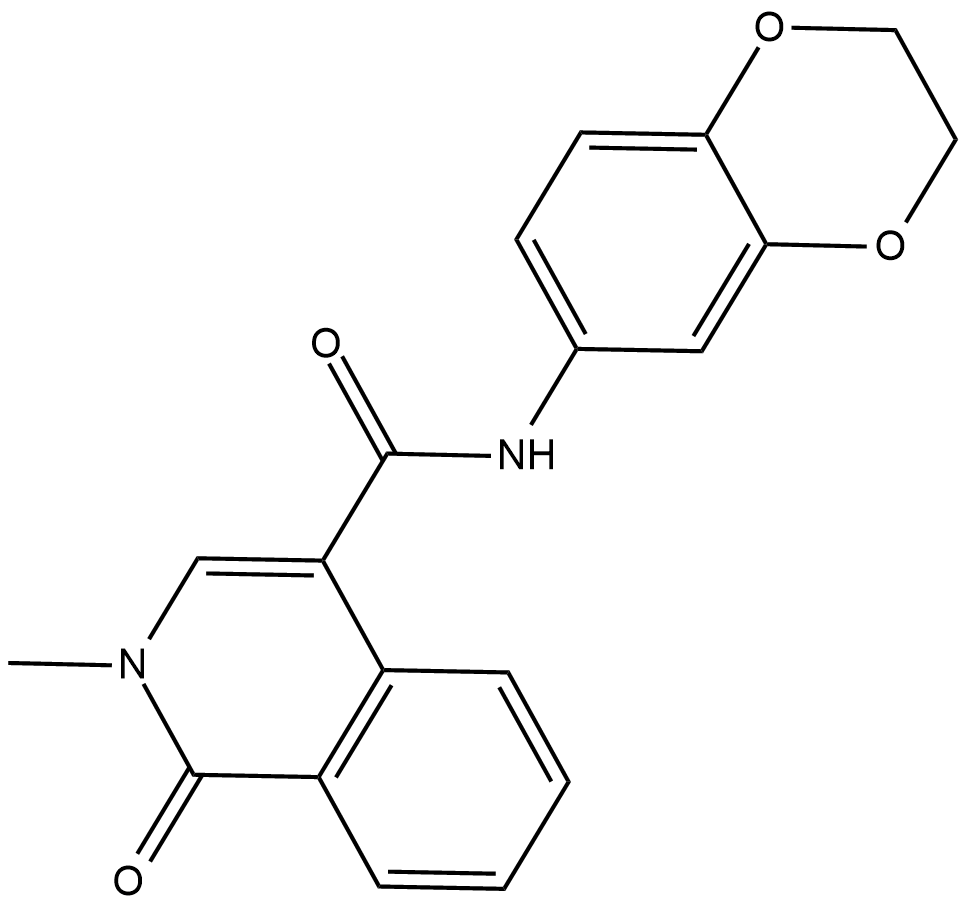
GC19098
CeMMEC1
CeMMEC1 est un inhibiteur de BRD4 et a également une forte affinité pour TAF1, avec une IC50 de 0,9 μM pour TAF1 et un Kd de 1,8 μM pour TAF1 (2).
-
GC33028
CF53
CF53 est un inhibiteur très puissant, sélectif et actif par voie orale de la protéine BET, avec un Ki <1 nM, un Kd de 2,2 nM et une IC50 de 2 nM pour BRD4 BD1. CF53 se lie À la fois aux domaines BD1 et BD2 des protéines BRD2, BRD3, BRD4 et BRDT BET avec des affinités élevées, très sélectives par rapport aux protéines ne contenant pas de bromodomaine BET. CF53 présente une puissante activité anti-tumorale À la fois in vitro et in vivo.
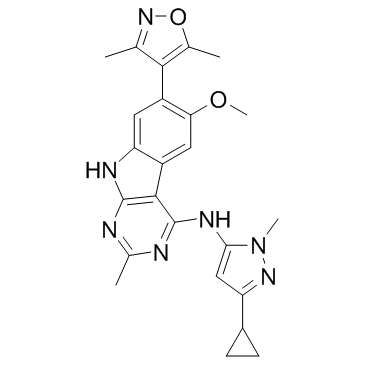
-
GC65602
CFT8634
Le CFT8634 est un dégradeur ciblant BRD9 extrait du brevet WO2021178920A1 composé 174. Le CFT8634 peut être utilisé pour la recherche du sarcome synovial et des tumeurs solides délétées par SMARCB1.
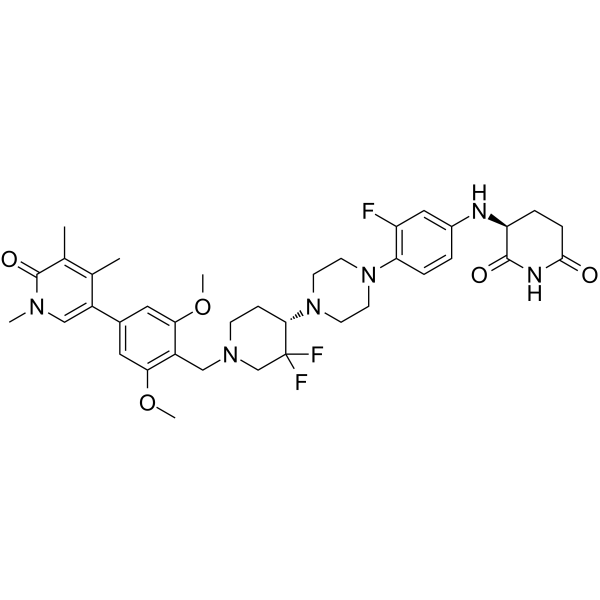
-
GC16599
CPI-169
EZH2 inhibitor
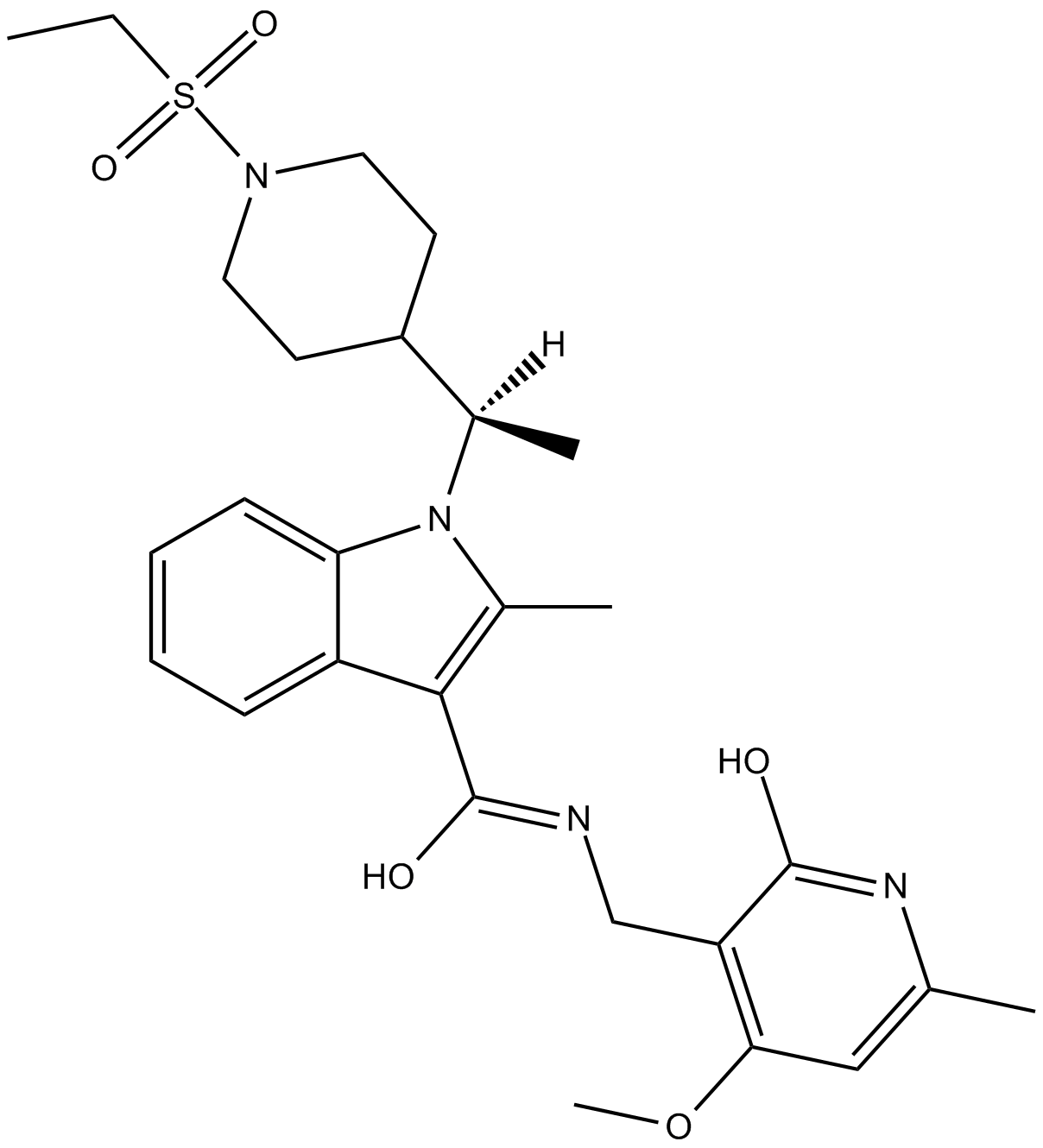
-
GC14699
CPI-203
Le CPI-203 est un nouvel inhibiteur puissant, sélectif et perméable aux cellules du bromodomaine BET, avec une valeur IC50 d'environ 37 nM (test BRD4 α-screen).
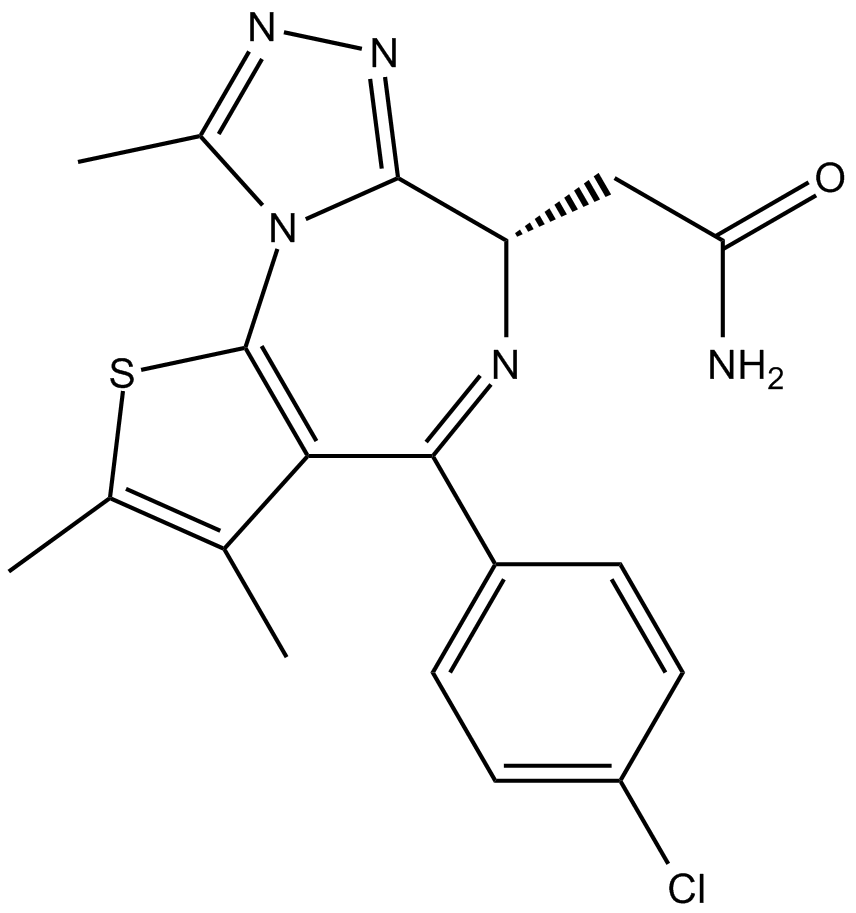
-
GC10382
CPI-637
CPI-637 est un inhibiteur sélectif et puissant du bromodomaine CBP/EP300 avec des valeurs IC50 de 0,03 μM, 0,051 μM et 11,0 μM pour CBP, EP300 et BRD4 BD-1, respectivement, et une CE50 de 0,3 μM pour CBP.
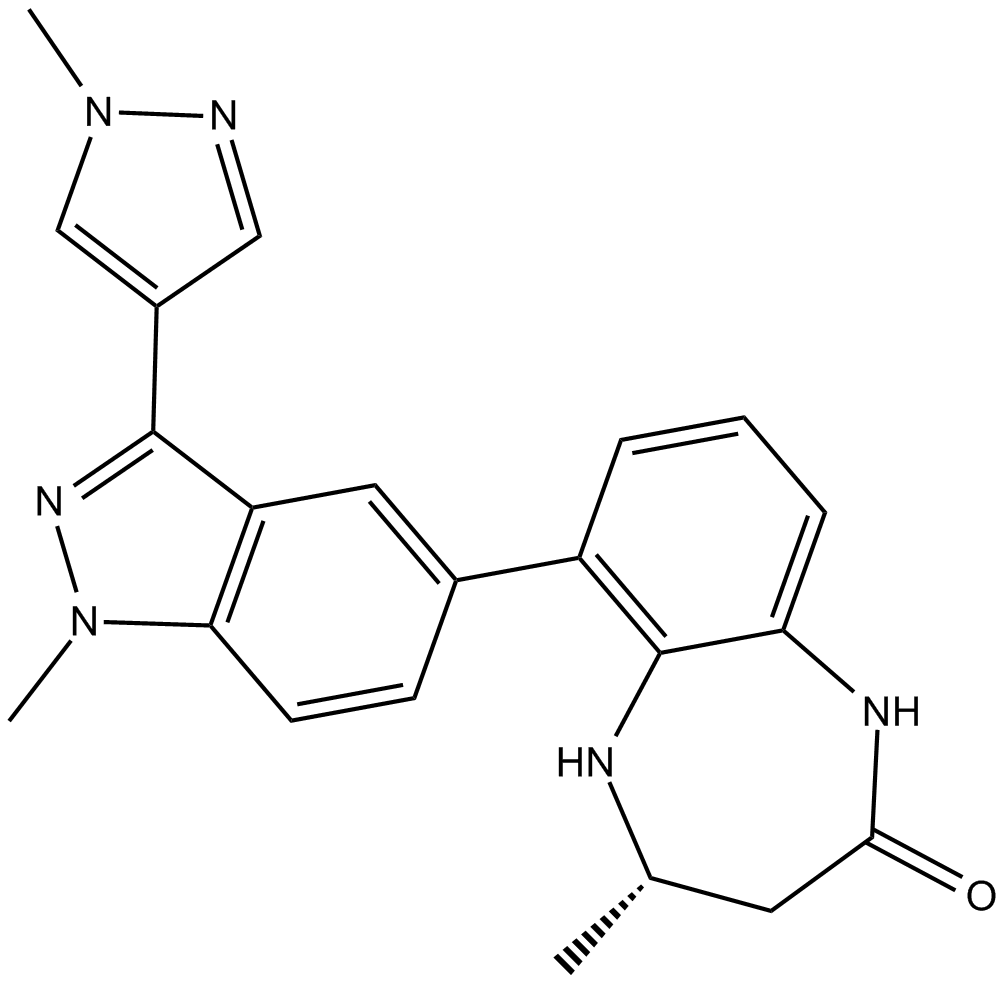
-
GC14787
Curcumin
Un pigment jaune avec diverses activités biologiques
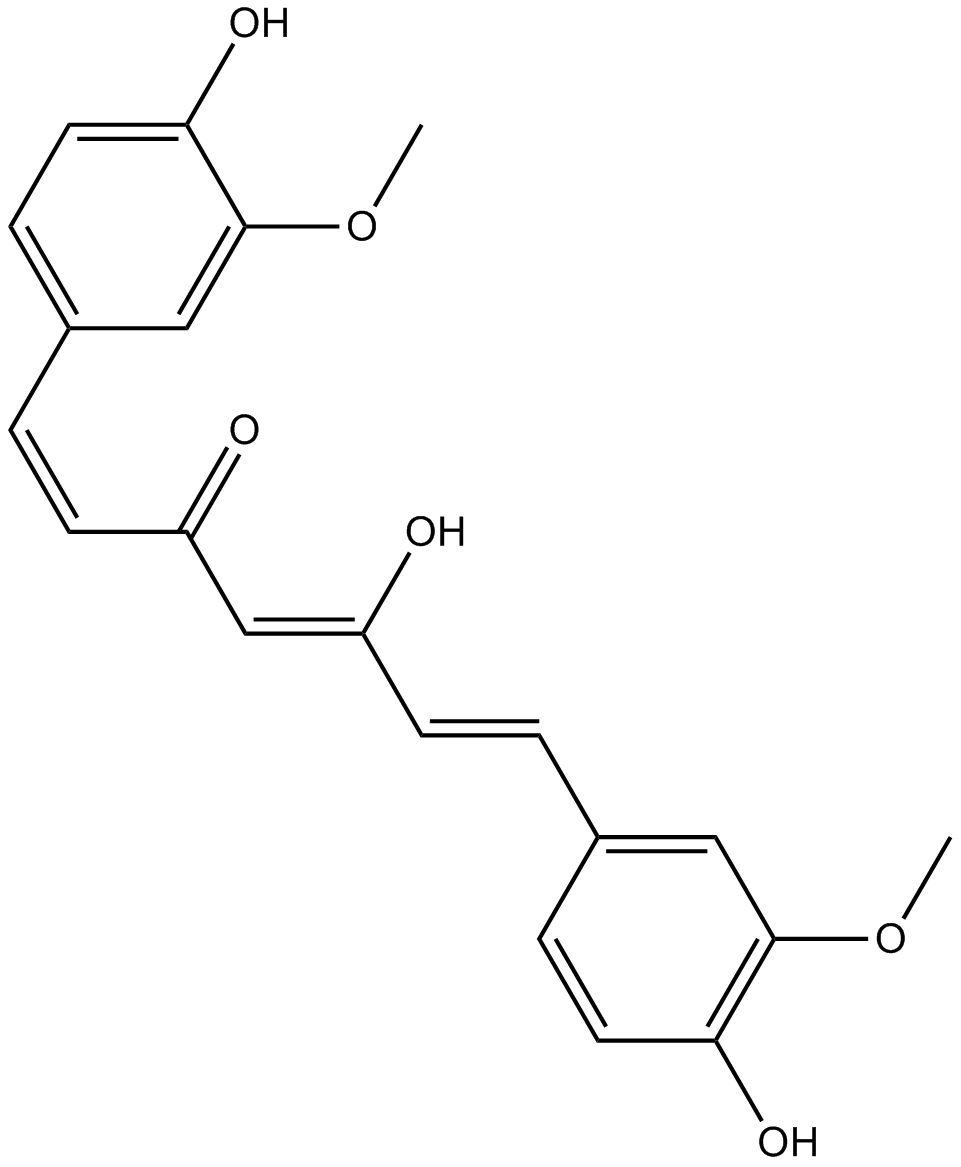
-
GC40226
Curcumin-d6
La curcumine D6 (Diferuloylmethane D6) est une curcumine marquée au deutérium (jaune curcuma).
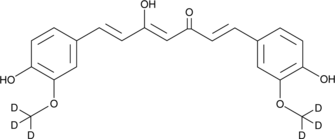
-
GC19119
dBET1
dBET1 est un PROTAC relié par des ligands pour Cereblon et BRD4 avec une CE50 de 430 nM. dBET1 est un PROTAC composé de (+)-JQ1 lié au NSC 527179 avec un lieur.
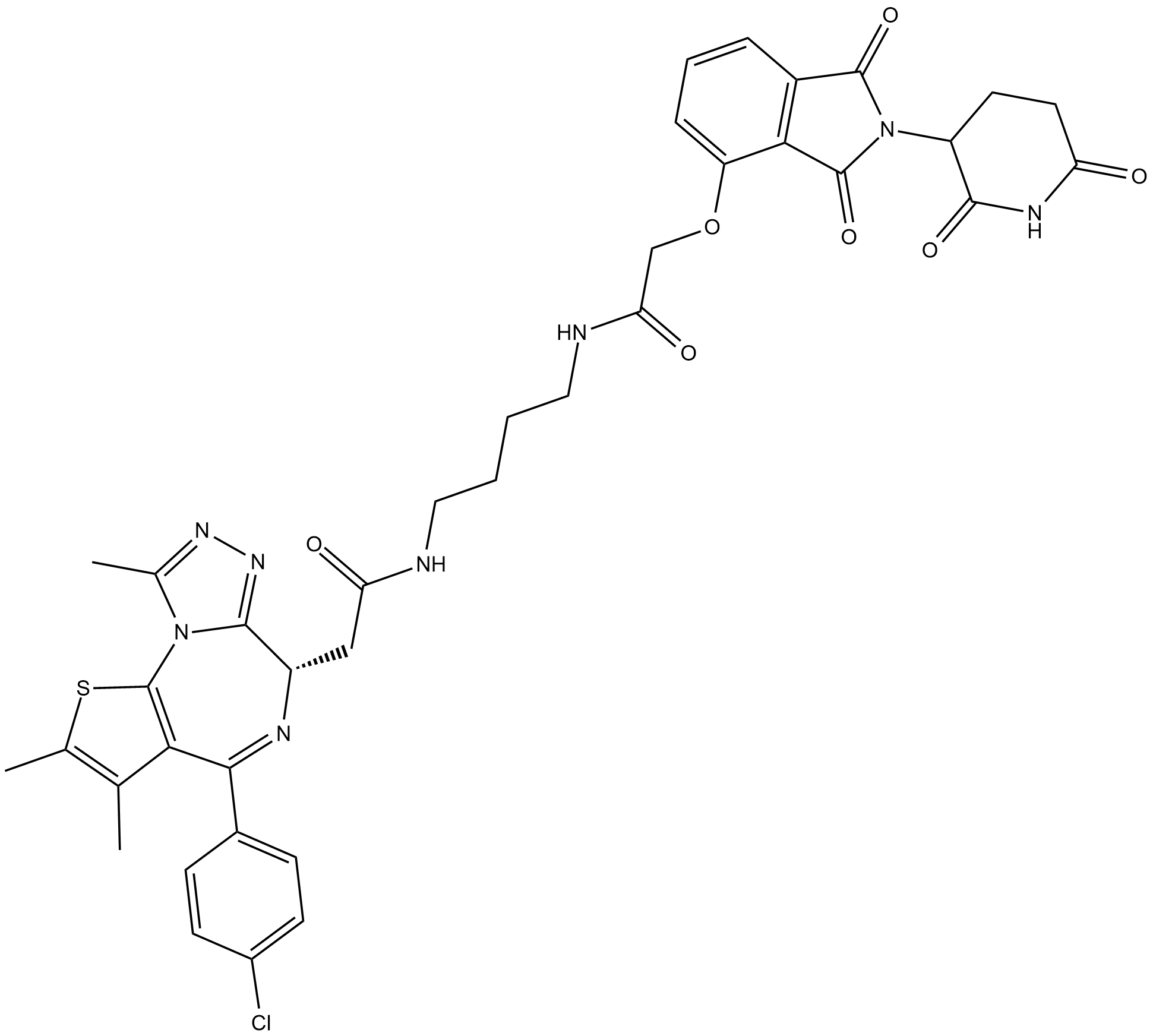
-
GC35815
dBET57
dBET57 est un dégradeur puissant et sélectif de BRD4BD1 basé sur la technologie PROTAC. dBET57 intervient dans le recrutement de l'ubiquitine ligase CRL4Cereblon E3, avec un DC50/5h de 500 nM pour BRD4BD1, et est inactif sur BRD4BD2.
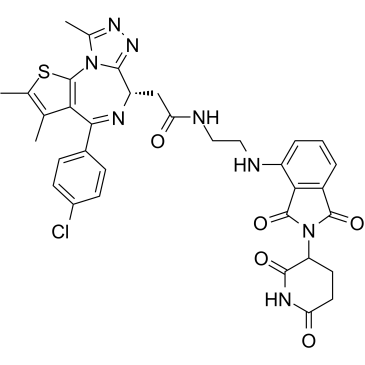
-
GC32719
dBET6
dBET6 est un PROTAC très puissant, sélectif et perméable aux cellules connecté par des ligands pour Cereblon et BET, avec une IC50 de 14 nM, et a une activité antitumorale.
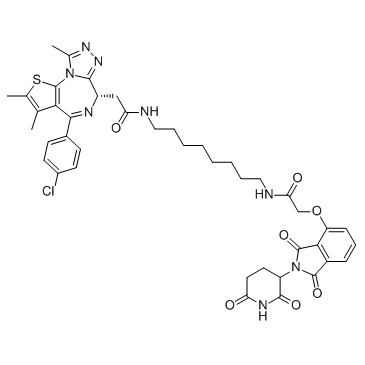
-
GC62211
dCBP-1
Le dCBP-1 est un dégradeur hétérobifonctionnel puissant et sélectif de p300/CBP basé sur le ligand Cereblon. Le dCBP-1 est exceptionnellement puissant pour tuer les cellules de myélome multiple et supprime l'activité de l'amplificateur oncogène À l'origine de l'expression de MYC.
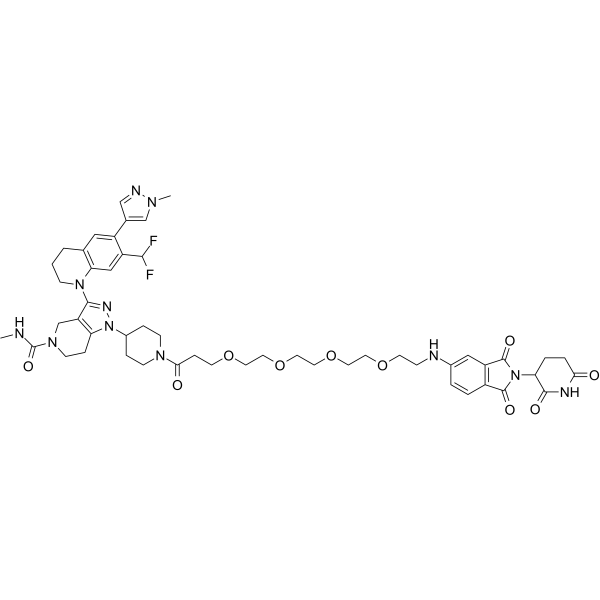
-
GC35904
dTRIM24
dTRIM24 est un dégradeur bifonctionnel sélectif de TRIM24 basé sur PROTAC, composé de ligands pour von Hippel-Lindau et TRIM24.
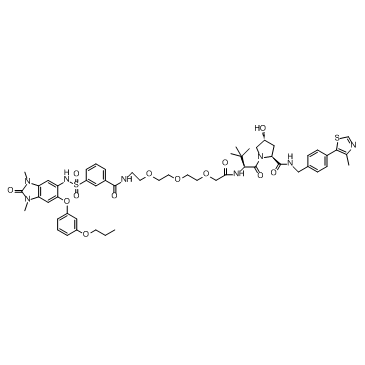
-
GC33403
E-7386
E-7386 est un modulateur CBP/bêta-caténine actif par voie orale.
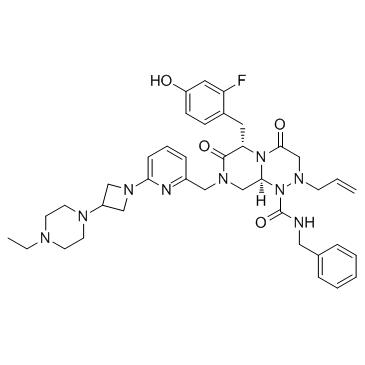
-
GC31245
EML 425
EML425 est un inhibiteur puissant et sélectif de la protéine de liaison CREB (CBP)/p300 avec des IC50 de 2,9 et 1,1 μM, respectivement.
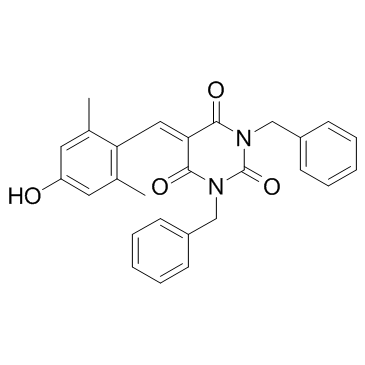
-
GC50182
ent-LP 99
ent-LP 99 est un inhibiteur puissant et sélectif de BRD7 et BRD9 avec un KD de 99 nM pour BRD9.
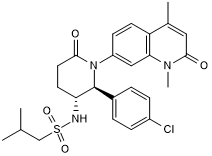
-
GC64821
FHD-286
Le FHD-286 est un inhibiteur de BRG1/BRM ATPase pour le traitement des troubles liés au BAF tels que la leucémie myéloÏde aiguë.
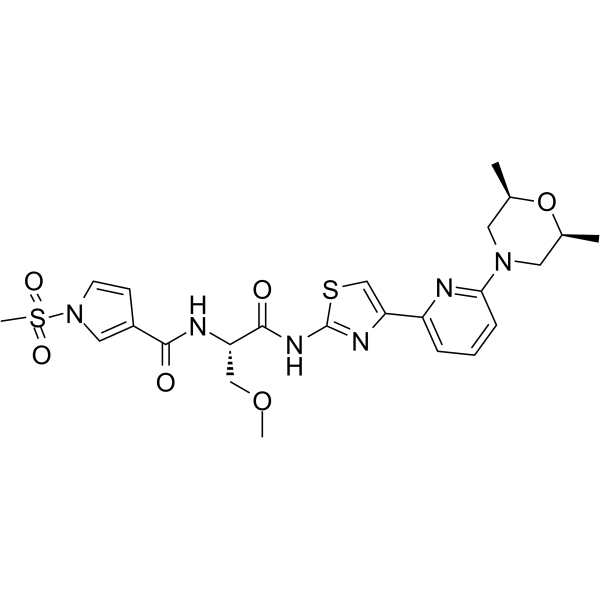
-
GC65160
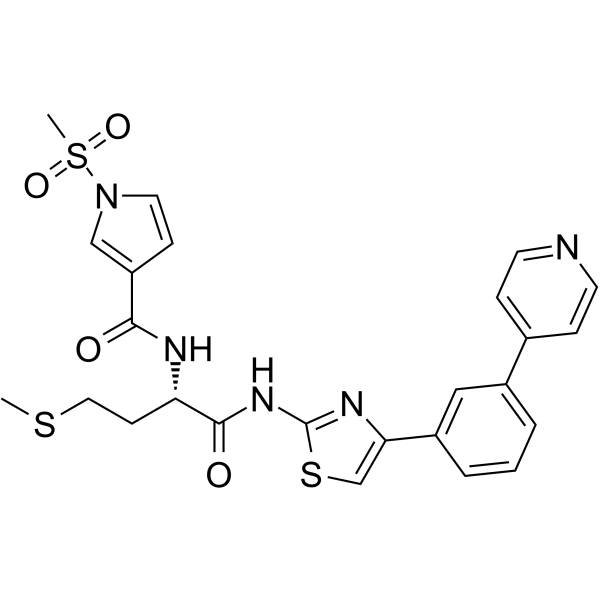
FHT-1015
FHT-1205 est un puissant inhibiteur de SMARCA4/SMARCA2 ATPase (BRG1 et BRM) avec des CI50 ≤ 10 nM (WO2020160180A1 ; composé 67).
-
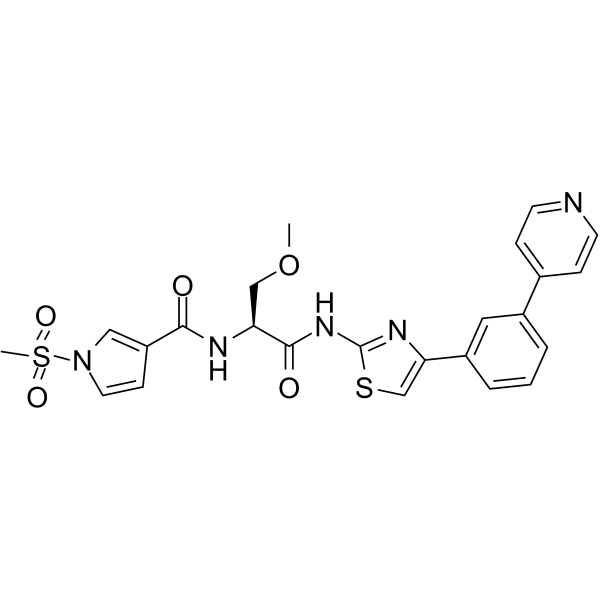
GC64777
FHT-1204
Le FHT-1204 est un puissant inhibiteur de SMARCA4/SMARCA2 ATPase (BRG1 et BRM) avec des CI50 ≤ 10 nM (WO2020160180A1 ; composé 70).
-
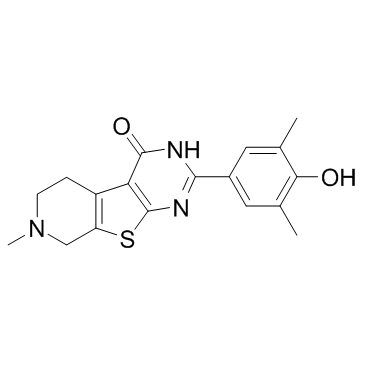
GC33015
FL-411 (BRD4-IN-1)
FL-411 (BRD4-IN-1) est un inhibiteur puissant et sélectif de BRD4 avec une IC50 de 0,43± ; 0,09 μ ; M pour BRD4(1).
-
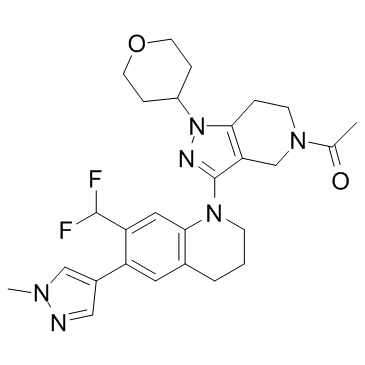
GC32960
GNE-049
GNE-049 est un inhibiteur de CBP très puissant et sélectif avec une IC50 de 1,1 nM dans le test TR-FRET. GNE-049 inhibe également BRET et BRD4(1) avec des IC50 de 12 nM et 4200 nM, respectivement.
-
GC68439
GNE-064

-
GC33212
GNE-207
GNE-207 est un inhibiteur puissant, sélectif et biodisponible par voie orale du bromodomaine de CBP, avec une IC50 de 1 nM, présente un indice sélectif de> 2500 fois contre BRD4 (1). GNE-207 montre une excellente puissance CBP, avec une CE50 de 18 nM pour l'expression de MYC dans les cellules MV-4-11.
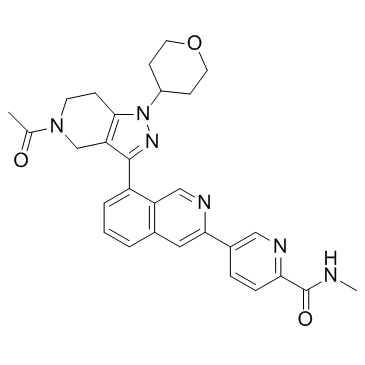
-
GC32747
GNE-272
GNE-272 est un inhibiteur puissant et sélectif de CBP/EP300 avec des valeurs IC50 de 0,02, 0,03 et 13 μM pour CBP, EP300 et BRD4, respectivement. GNE-272 est également une sonde sélective in vivo pour CBP/EP300.
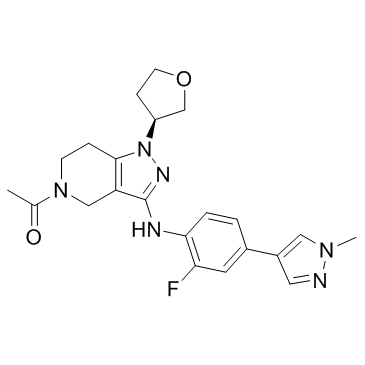
-
GC65326
GNE-375
GNE-375 est un inhibiteur de BRD9 puissant et hautement sélectif avec une IC50 de 5 nM. GNE-375 montre > 100 fois sélectif pour BRD9 sur BRD4, TAF1 et CECR2. GNE-375 diminue la liaison de BRD9 À la chromatine.
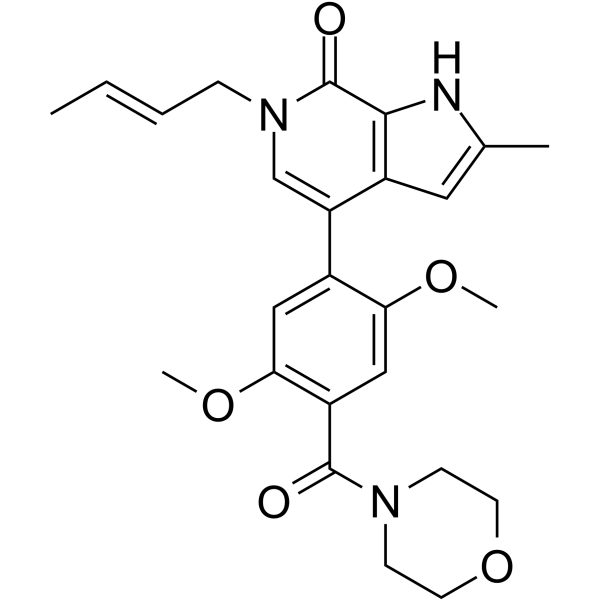
-
GC32081
GNE-781
Le GNE-781 est un inhibiteur de la CBP actif par voie orale, très puissant et sélectif avec une IC50 de 0,94 nM dans le test TR-FRET. GNE-781 inhibe également BRET et BRD4(1) avec des IC50 de 6,2 nM et 5100 nM, respectivement. GNE-781 affiche une activité antitumorale dans un modèle de xénogreffe MOLM-16 AML.
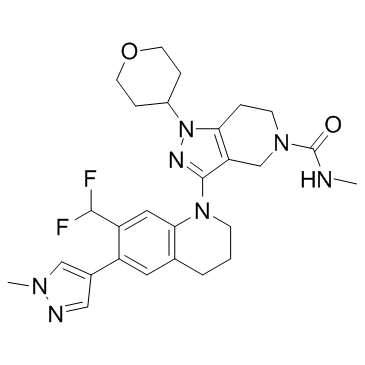
-
GC33204
GS-626510
Le GS-626510 est un inhibiteur des bromodomaines de la famille BET puissant et actif par voie orale, avec des valeurs Kd de 0,59 À 3,2 nM pour BRD2/3/4, avec des valeurs IC50 de 83 nM et 78 nM pour BD1 et BD2, respectivement.
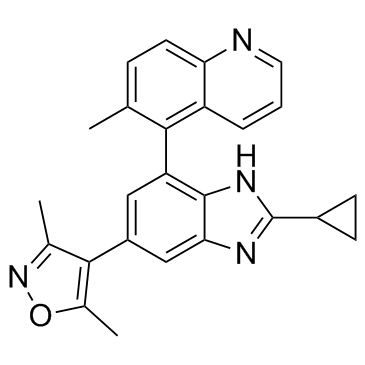
-
GC31731
GSK 4027
GSK 4027 est une sonde chimique pour le bromodomaine PCAF/GCN5 avec un pIC50 de 7,4 ± 0,11 pour PCAF dans un essai de transfert d'énergie par résonance de fluorescence (TR-FRET) résolu en temps.
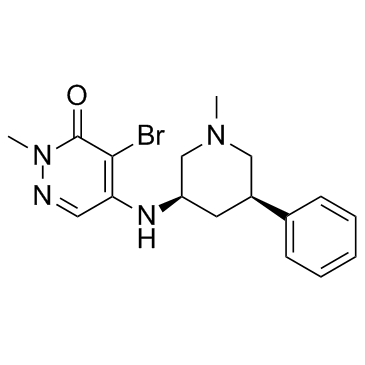
-
GC12440
GSK 5959
GSK 5959 est un inhibiteur de bromodomaine BRPF1 puissant, sélectif et perméable aux cellules avec une IC50 d'environ 80 nM.
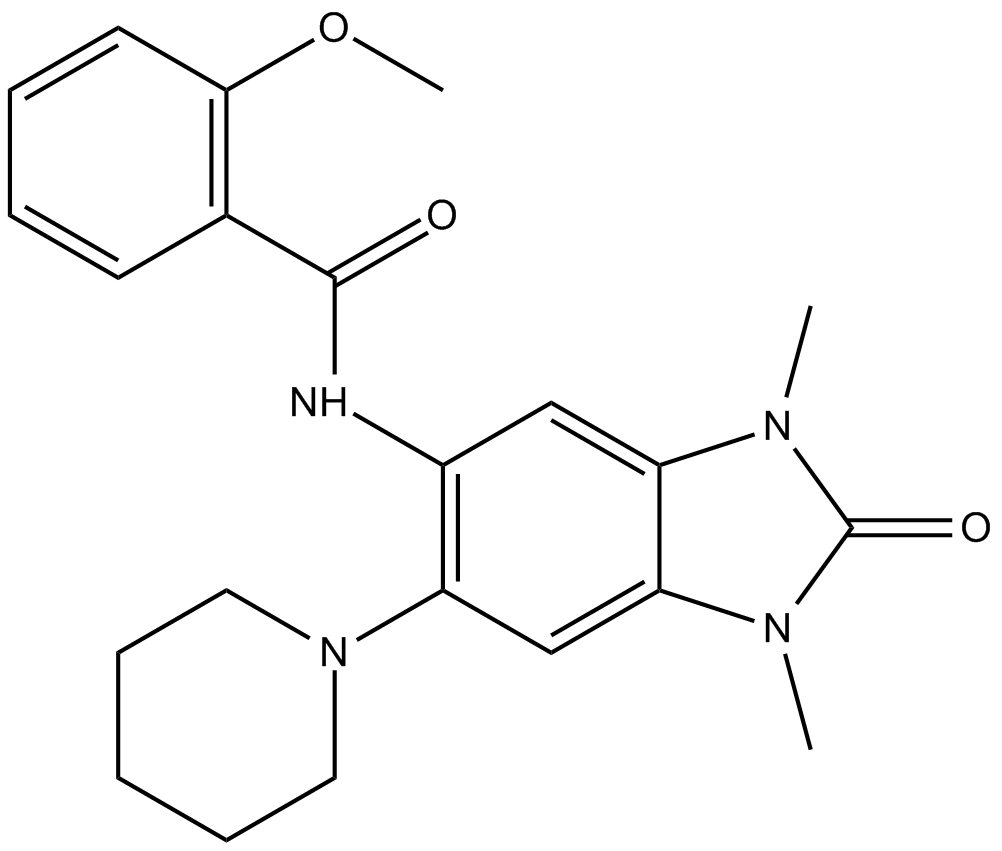
-
GC50378
GSK 9311 hydrochloride
Le chlorhydrate de GSK 9311, un analogue moins actif du GSK6853, peut être utilisé comme témoin négatif. Le chlorhydrate de GSK 9311 inhibe le bromodomaine BRPF avec des valeurs de pIC50 de 6,0 et 4,3 pour BRPF1 et BRPF2, respectivement.
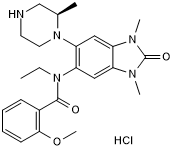
-
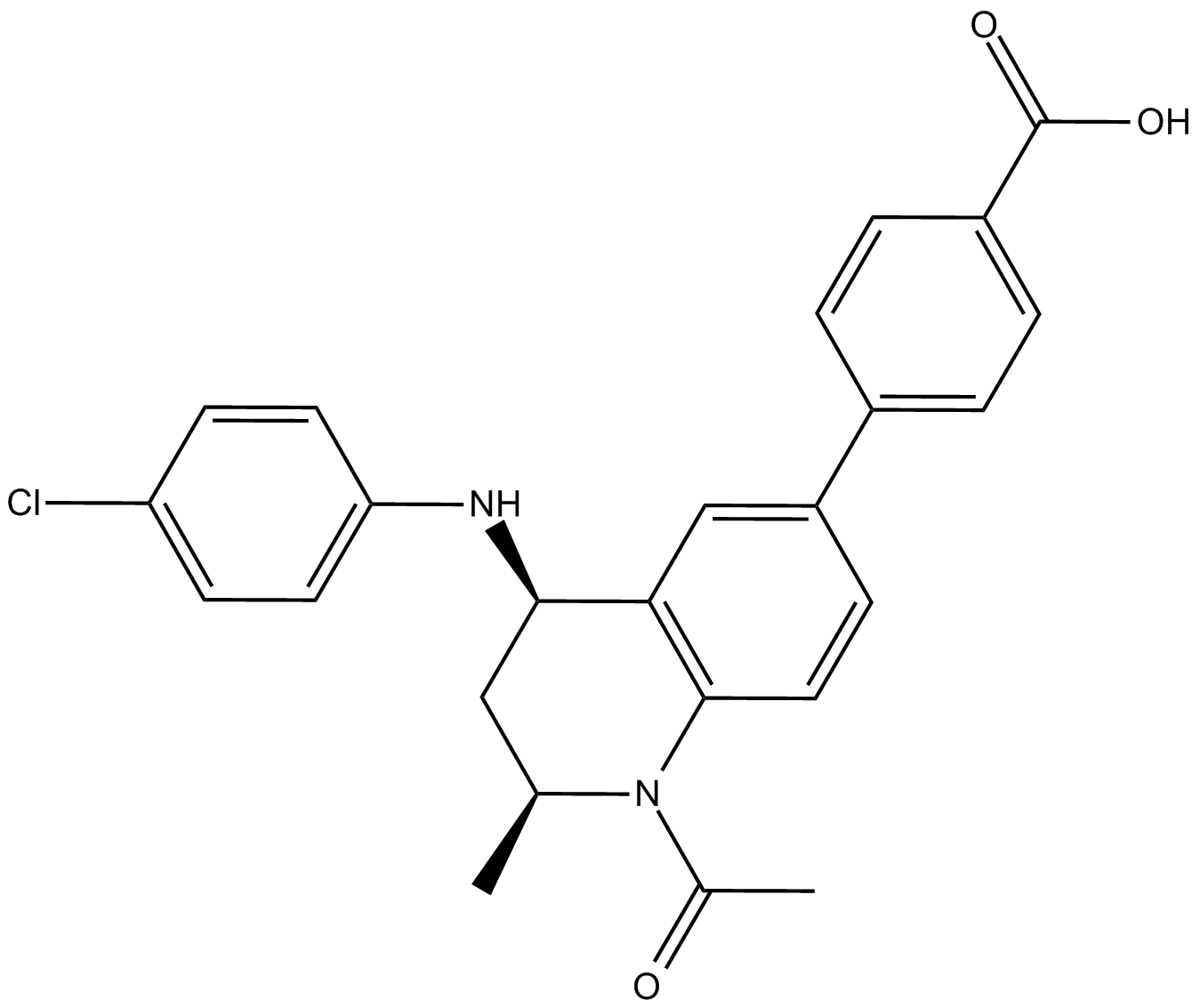
GC14063
GSK1324726A
GSK1324726A est un nouvel inhibiteur puissant et sélectif des protéines BET avec une affinité élevée pour BRD2 (IC50 = 41 nM), BRD3 (IC50 = 31 nM) et BRD4 (IC50 = 22 nM).
-
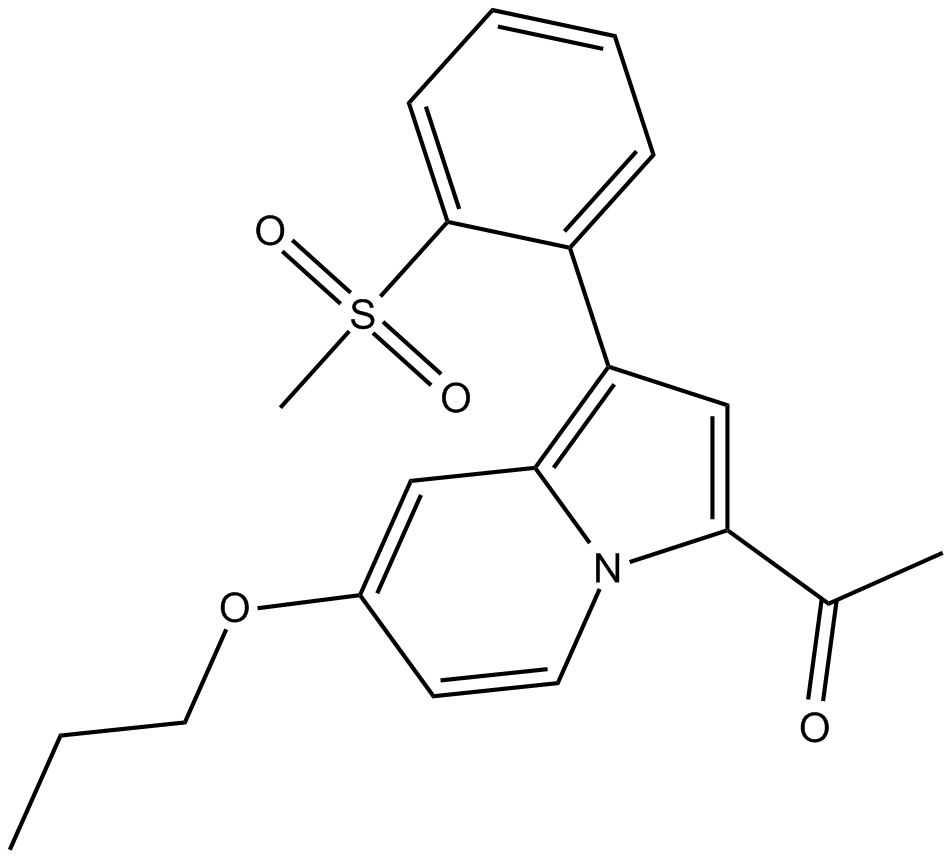
GC15789
GSK2801
GSK2801 est un inhibiteur des bromodomaines BAZ2A et BAZ2B puissant, sélectif, actif par voie orale et actif sur les cellules, avec des valeurs de Kd de 136 nM et 257 nM, respectivement. GSK2801 montre une sélectivité >50 fois pour BAZ2A/B par rapport À BRD4.
-
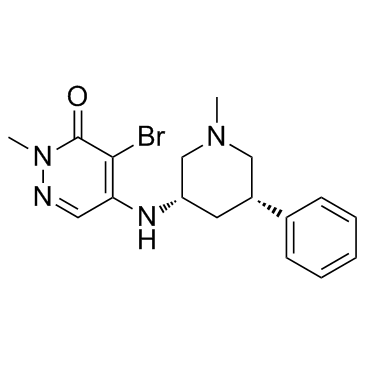
GC30714
GSK4028
GSK4028 est le contrÔle négatif énantiomère de GSK4027, qui est une sonde chimique bromodomaine PCAF/GCN5, le pIC50 de GSK4028 est de 4,9 dans un test de transfert d'énergie par résonance de fluorescence (TR-FRET) résolu en temps.
-
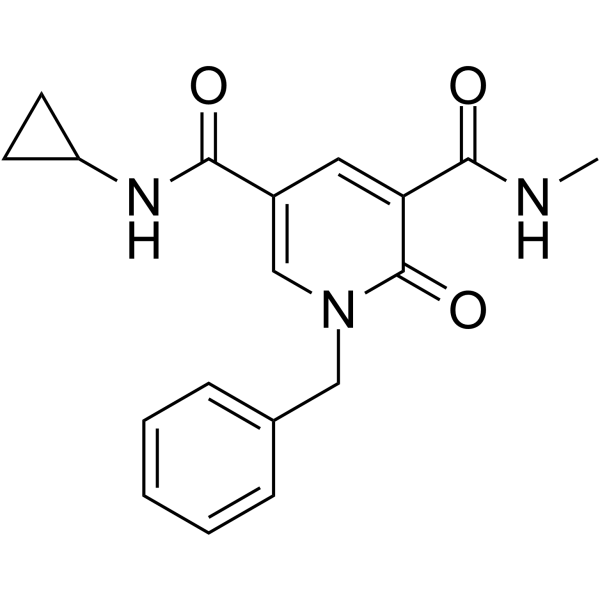
GC62312
GSK620
GSK620 est un inhibiteur pan-BD2 puissant et actif par voie orale avec une excellente sélectivité large, une bonne capacité de développement et des pharmacocinétiques orales in vivo.
-
GC13025
GSK6853
GSK6853 est un inhibiteur puissant et sélectif du bromodomaine BRPF1.
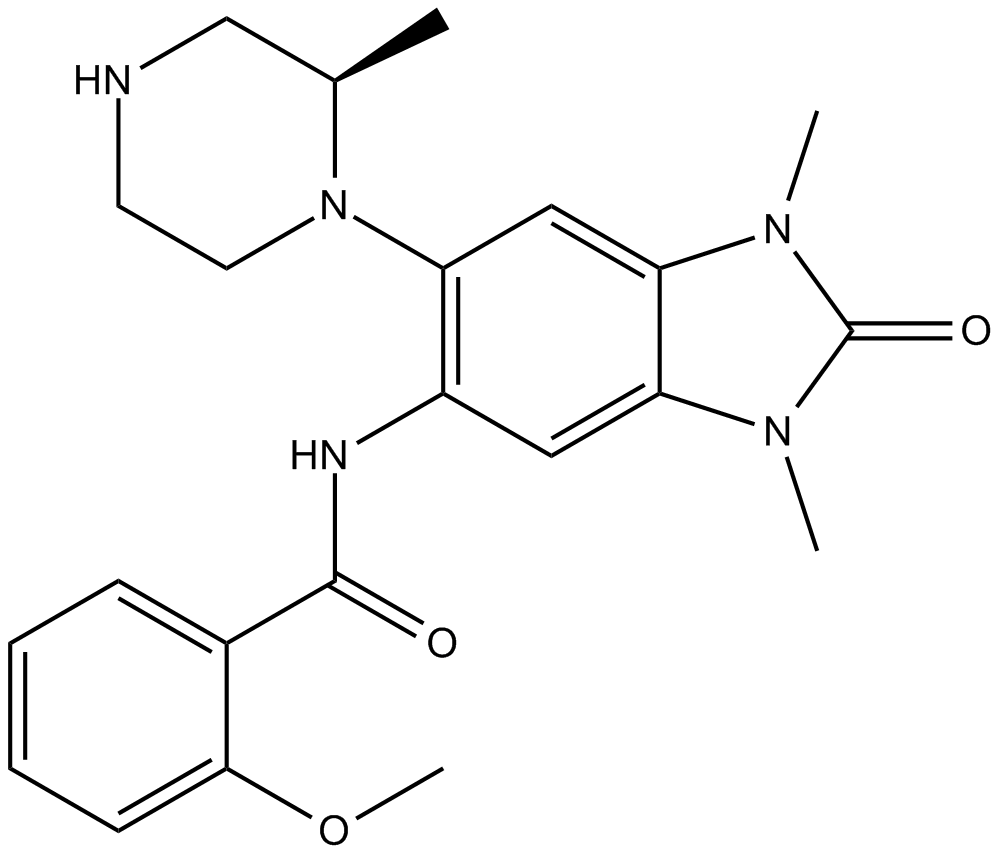
-
GC62654
GSK778
GSK778 (iBET-BD1) est un inhibiteur de bromodomaine BD1 puissant et sélectif des protéines BET, avec des IC50 de 75 nM (BRD2 BD1), 41 nM (BRD3 BD1), 41 nM (BRD4 BD1) et 143 nM (BRDT BD1) , respectivement.
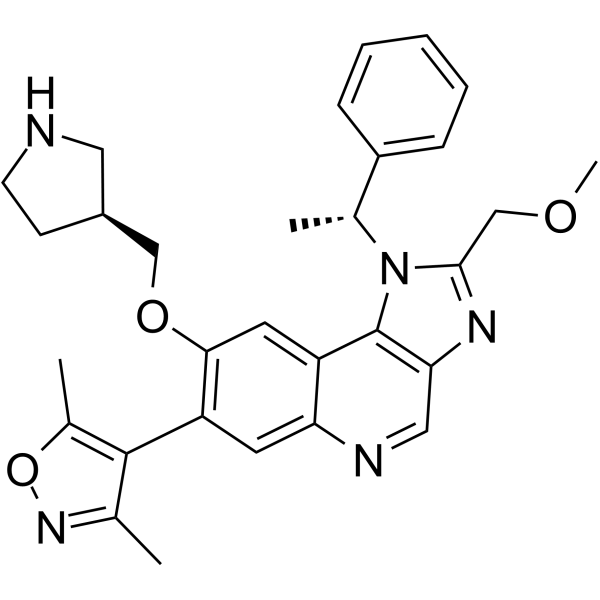
-
GC38049
GSK8573
GSK8573 est un composé témoin inactif pour GSK2801 (inhibiteur compétitif de l'acétyl-lysine des bromodomaines BAZ2A et BAZ2B). GSK8573 a une activité de liaison À BRD9 avec une valeur Kd de 1,04 μM et est inactif contre BAZ2A/B et d'autres familles de bromodomaines. GSK8573 peut être utilisé comme composé de contrÔle négatif structurellement apparenté dans des expériences biologiques.
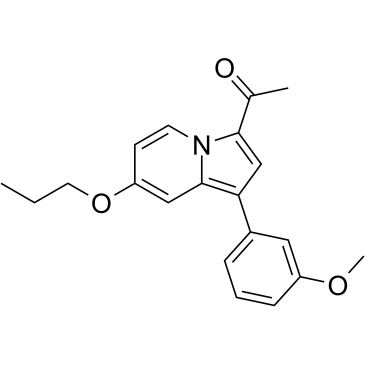
-
GC60184
GSK8814
GSK8814 est une sonde chimique et un inhibiteur de bromodomaine puissant, sélectif et ATAD2/2B, avec une constante de liaison pKd = 8,1 et un pKi = 8,9 dans BROMOscan. GSK8814 se lie À ATAD2 et BRD4 BD1 avec des pIC50 de 7,3 et 4,6, respectivement. GSK8814 montre une sélectivité de 500 fois pour ATAD2 sur BRD4 BD1.
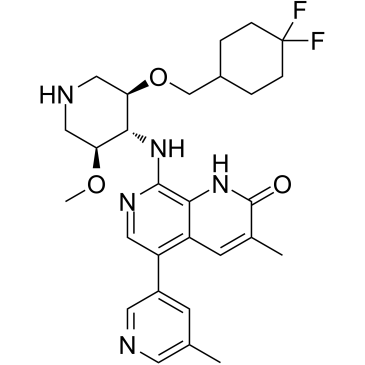
-
GC33324
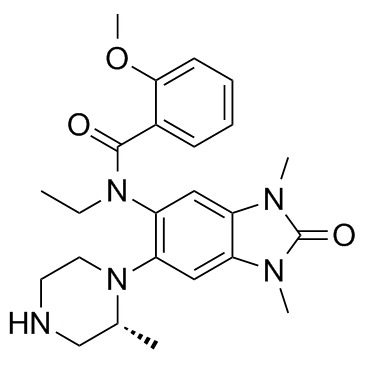
GSK9311
Le GSK9311, un analogue moins actif du GSK6853, peut être utilisé comme témoin négatif. GSK9311 inhibe le bromodomaine BRPF avec des valeurs de pIC50 de 6,0 et 4,3 pour BRPF1 et BRPF2, respectivement.
-
GC33211
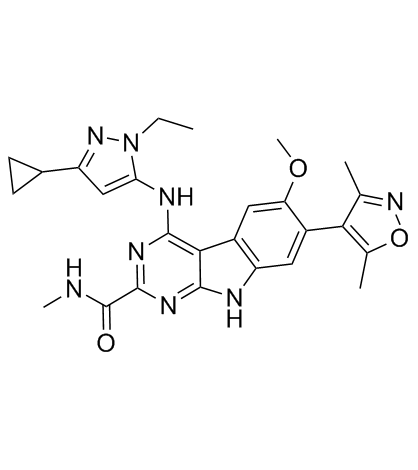
HJB97
HJB97 est un inhibiteur BET de haute affinité avec Kis de 0,9 nM (BRD2 BD1), 0,27 nM (BRD2 BD2), 0,18 nM (BRD3 BD1), 0,21 nM (BRD3 BD2), 0,5 nM (BRD4 BD1), 1,0 nM (BRD4 BD2), respectivement. HJB97 est utilisé pour la conception d'un dégradeur potentiel de PROTAC BET et a une activité antitumorale.
-
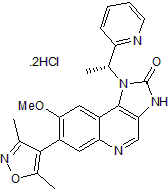
GC50070
I-BET 151 dihydrochloride
Le dichlorhydrate I-BET 151 (dichlorhydrate GSK1210151A) est un inhibiteur du bromodomaine BET qui inhibe BRD4, BRD2 et BRD3 avec des pIC50 de 6,1, 6,3 et 6,6, respectivement.
-
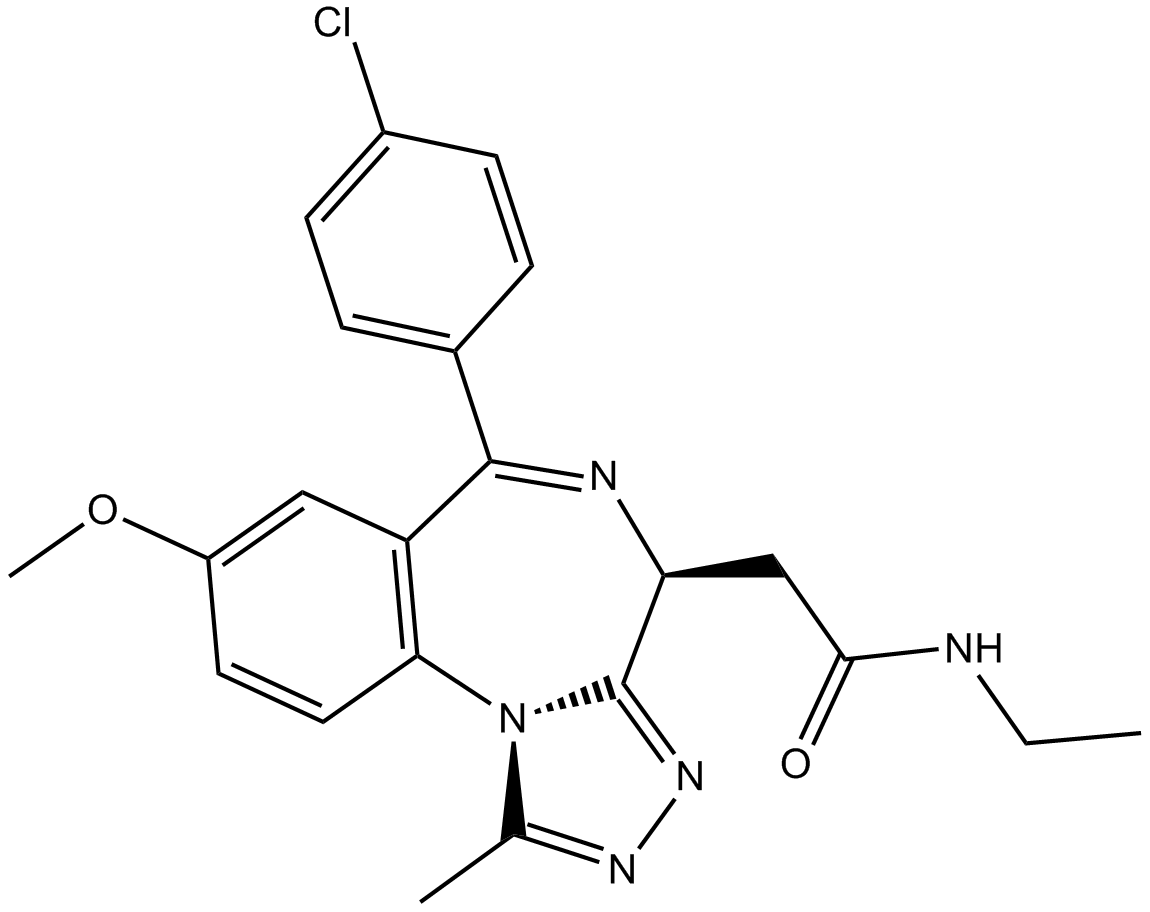
GC17073
I-BET-762
I-BET-762 (I-BET762; GSK525762) est un inhibiteur de bromodomaine BET avec IC50 de 32,5-42,5 nM.
-
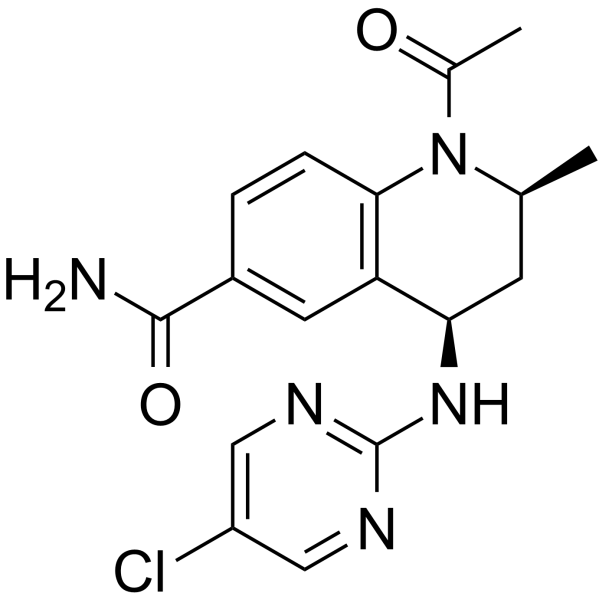
GC15747
I-BET151 (GSK1210151A)
I-BET151 (GSK1210151A) (GSK1210151A) est un inhibiteur du bromodomaine BET qui inhibe BRD4, BRD2 et BRD3 avec pIC50 de 6,1, 6,3 et 6,6, respectivement.
-
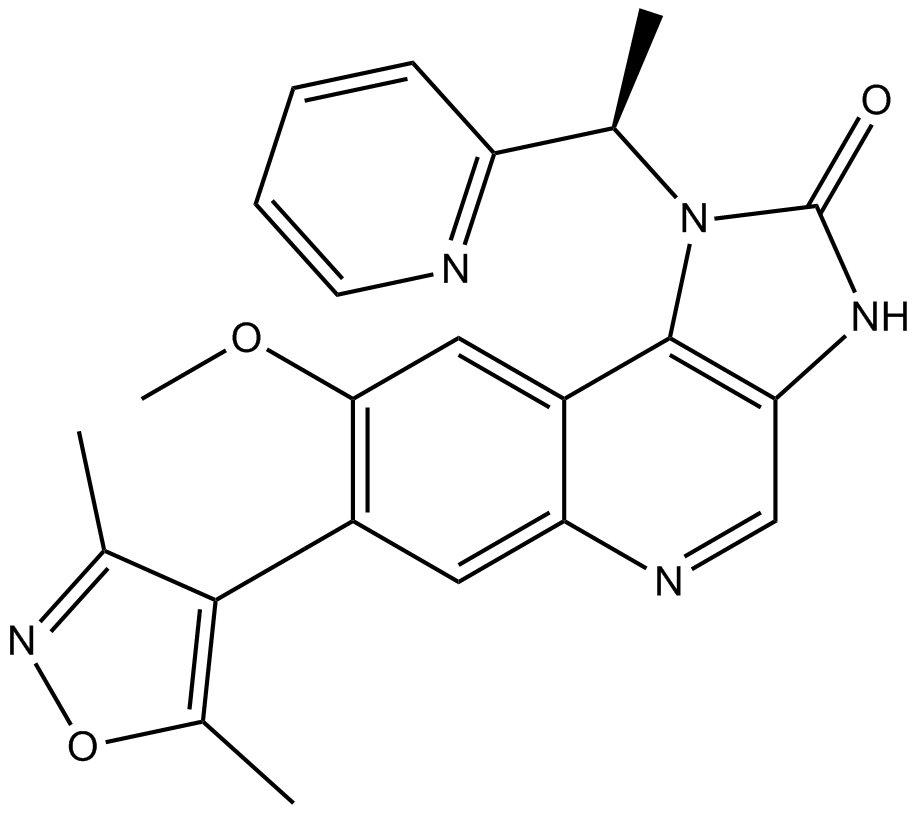
GC64297
I-BET567
I-BET567 est un inhibiteur puissant et oralement actif du candidat pan-BET avec des pIC50 de 6,9 et 7,2 pour BRD4 BD1 et BD2, respectivement.
-
GC12588
I-BRD9
I-BRD9 est une sonde chimique cellulaire sélective de la protéine 9 contenant un bromodomaine (BRD9) avec une valeur de pIC50 de 7,3 μM.