MAPK Signaling

Targets for MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Products for MAPK Signaling
- Cat.No. Nombre del producto Información
-
GC32900
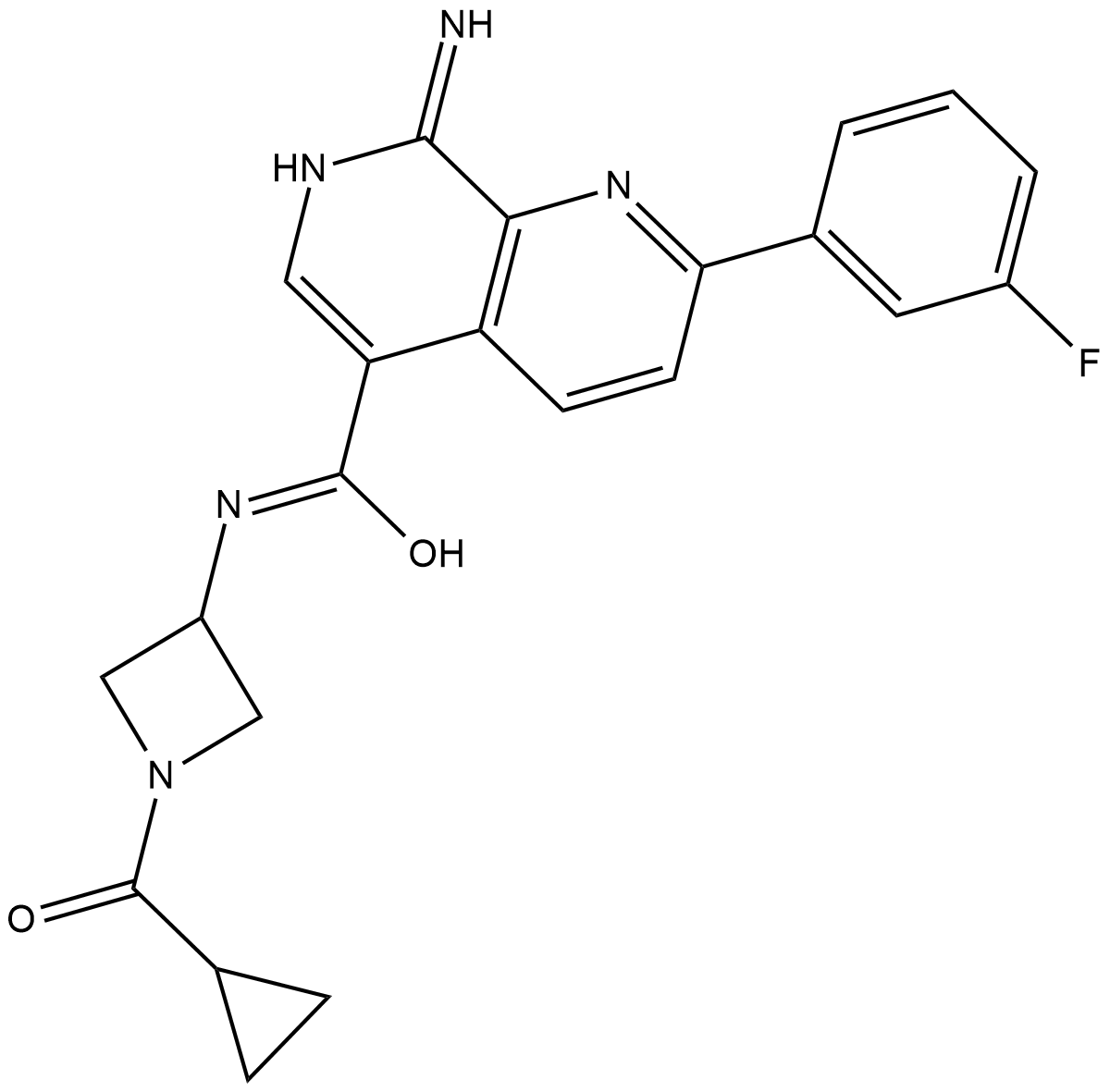
CC-90003
CC-90003 es un inhibidor irreversible y selectivo de ERK 1/2 con actividad antitumoral.
-
GC10446
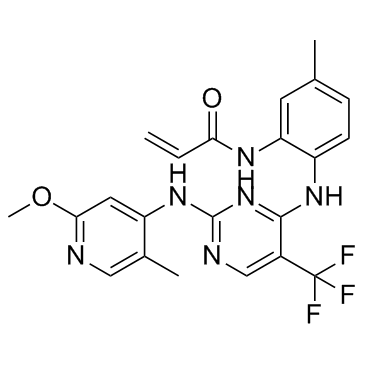
CC-930
A potent JNK inhibitor
-
GC63441
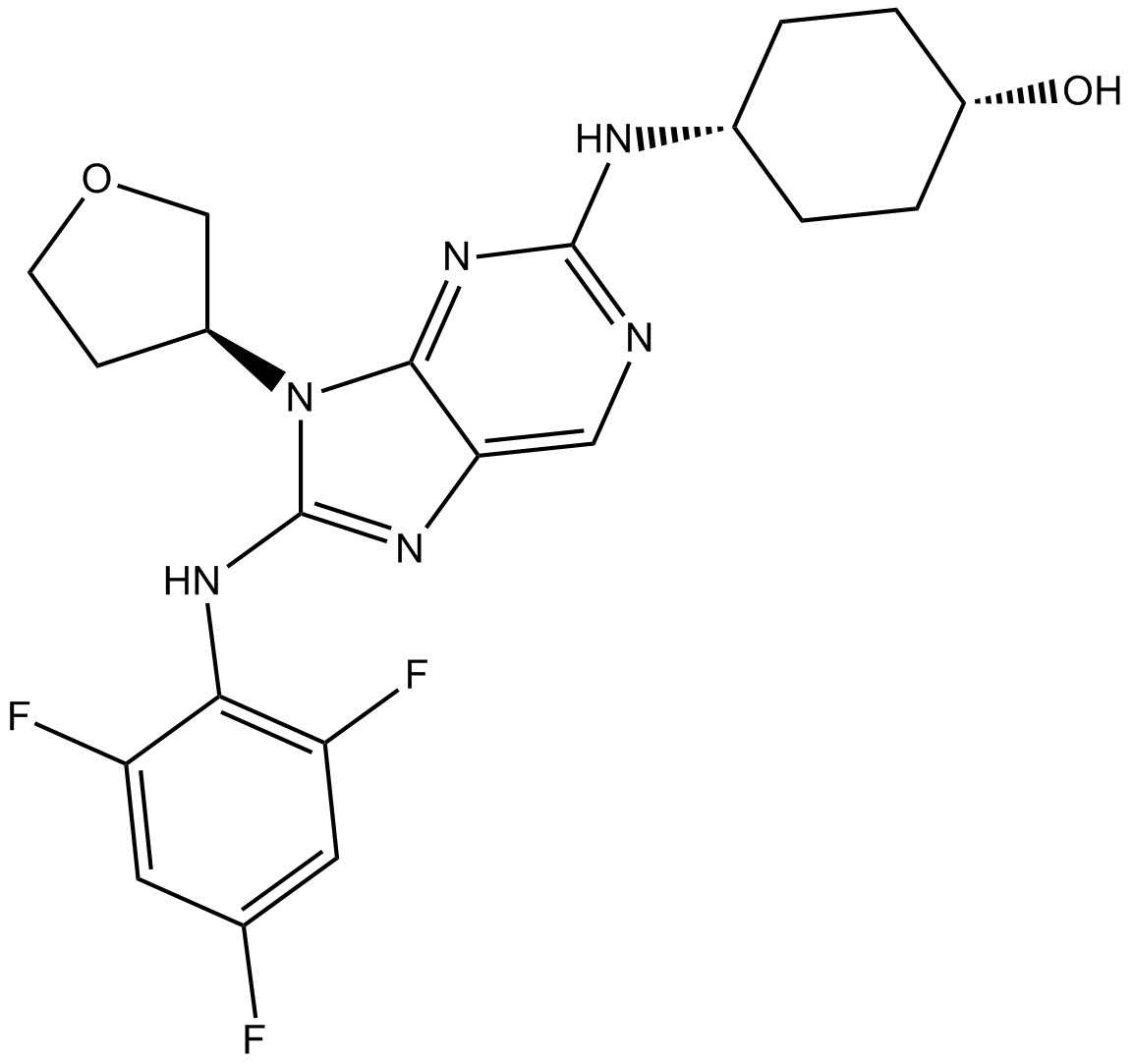
CC-99677
CC-99677 es un inhibidor potente, covalente e irreversible de la vÍa de la proteÍna cinasa-2 (MK2) de la proteÍna activada por mitÓgeno (MAP) cinasa activada en ensayos bioquÍmicos (IC50 = 156,3 nM) y basados en células (EC50 = 89 nM ).
-
GC19090
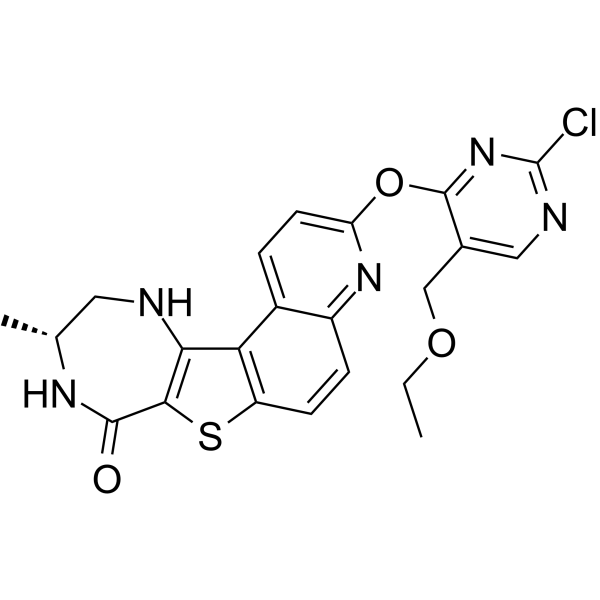
CCG215022
CCG215022 es un inhibidor de las quinasas del receptor acoplado a proteÍna G (GRK) con IC50 de 0,15 ± 0,07 μM, 0,38 ± 0,06 μM y 3,9 ± 1 μM para GRK2, GRK5 y GRK1, respectivamente.
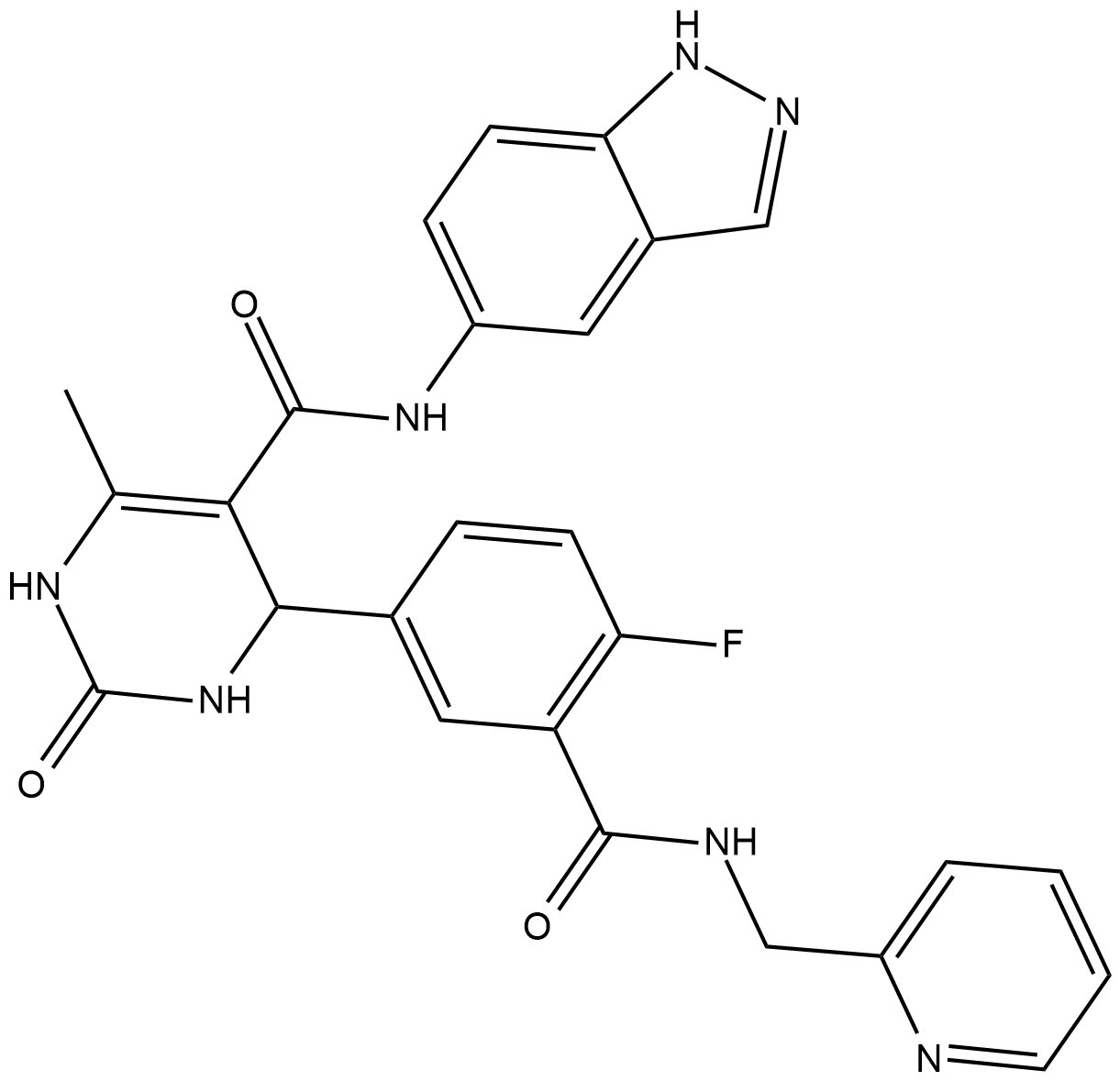
-
GC19091
CCT196969
CCT196969 es un inhibidor de pan-Raf, que inhibe B-Raf, BRafV600E y CRAF con IC50 de 0,1, 0,04 y 0,01 μM, respectivamente.
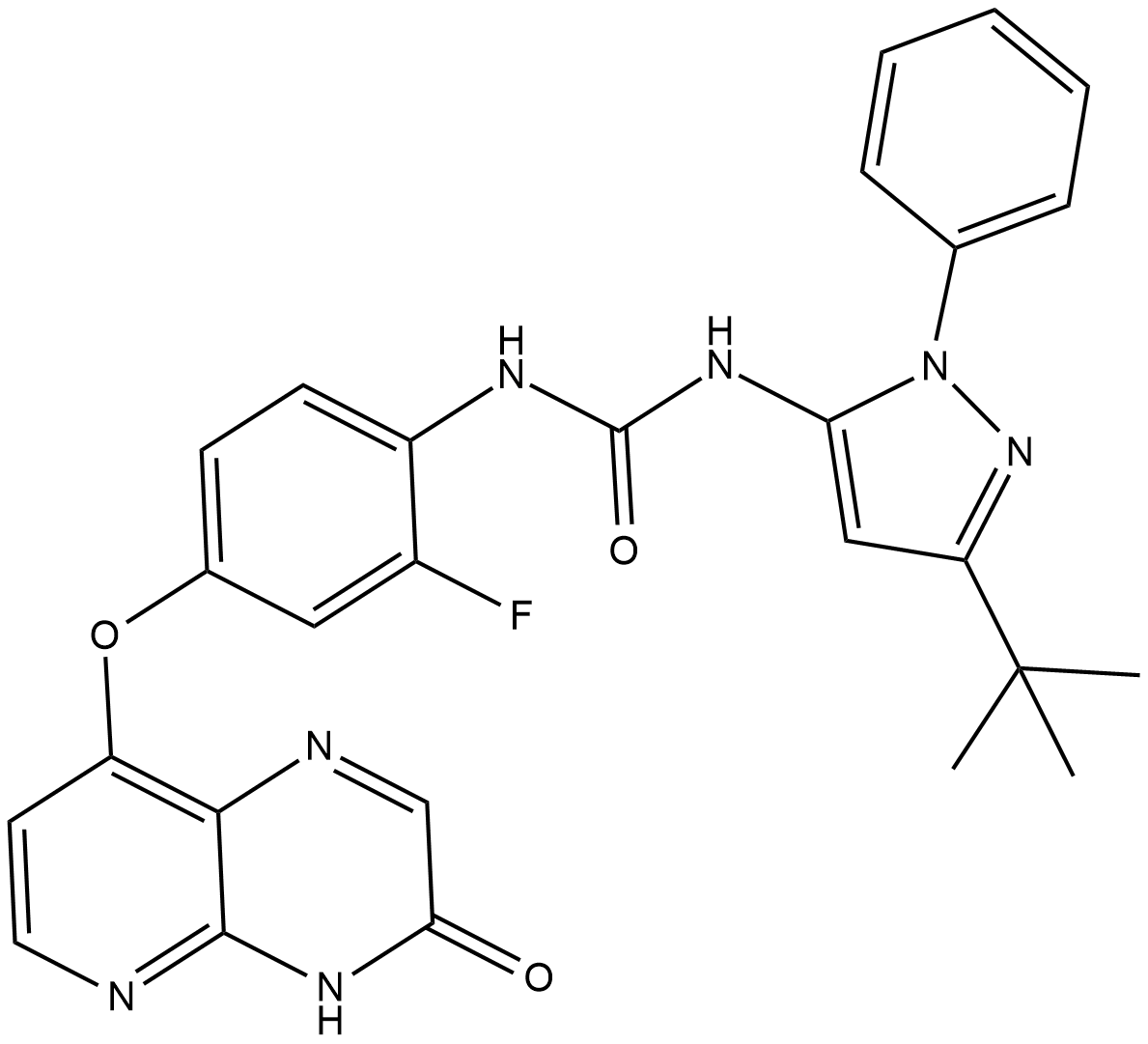
-
GC40054
CCT241161
CCT241161 es un inhibidor pan-RAF activo por vÍa oral con IC50 de 3, 6, 10, 15 y 30 nM para LCK, CRAF, SRC, V600E-BRAF y BRAF, respectivamente. CCT241161 muestra una buena actividad en los melanomas mutantes BRAF y NRAS. CCT241161 también exhibe actividad proliferativa de células anticancerÍgenas.
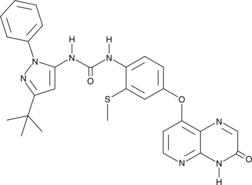
-
GC61865
Cearoin
La cearoÍna aumenta la autofagia y la apoptosis a través de la producciÓn de ROS y la activaciÓn de ERK.
-
GC12841
CEP 1347
CEP 1347 es un inhibidor de la vía JNK/SAPK con efectos neuroprotectores.
-
GC16845
CEP-32496
CEP-32496 (CEP-32496; RXDX-105) es un inhibidor muy potente y eficaz por vÍa oral de BRAFV600E con una Kd de 14 nM.
-
GC17860
CEP-32496 hydrochloride
A potent inhibitor of B-Raf
-
GC15525
cGMP Dependent Kinase Inhibitor Peptide
El péptido inhibidor de la quinasa dependiente de cGMP es un inhibidor competitivo de ATP de la proteÍna quinasa dependiente de cGMP (PKG), con una Ki de 86 μM.
-
GC10666
CGP 57380
CGP 57380 es un compuesto de pirazolo-pirimidina permeable a las células que actÚa como un inhibidor selectivo de Mnk1 con IC50 de 2,2 μM, pero no tiene actividad inhibidora contra p38, JNK1, ERK1/2, PKC o quinasas similares a Src.
-
GC35674
Chicanine
La chicanina es un compuesto de lignano de Schisandra chinesis, inhibe la fosforilaciÓn inducida por LPS de p38 MAPK, ERK 1/2 e IκB-α, con actividad antiinflamatoria.
-
GC35684
CHMFL-EGFR-202
CHMFL-EGFR-202 es un inhibidor potente e irreversible de la quinasa mutante del receptor del factor de crecimiento epidérmico (EGFR), con IC50 de 5,3 nM y 8,3 nM para las quinasas EGFR T790M y WT EGFR mutantes resistentes a los medicamentos, respectivamente. CHMFL-EGFR-202 muestra una selectividad de ~10 veces por EGFR L858R/T790M frente al EGFR de tipo salvaje en las células. CHMFL-EGFR-202 adopta una conformaciÓn de uniÓn inactiva covalente \CHMFL-EGFR-202 adopts a covalent "DFG-in-C-helix-out" inactive binding conformation with EGFR, with strong antiproliferative effects against EGFR mutant-driven nonsmall-cell lung cancer (NSCLC) cell lines.en_es_2021q4.md
-
GC12532
CHPG
CHPG es un agonista selectivo de mGluR5 y atenÚa el estrés oxidativo y la inflamaciÓn inducidos por SO2 a través de la vÍa TSG-6/NF-κB en células microgliales BV2.
-
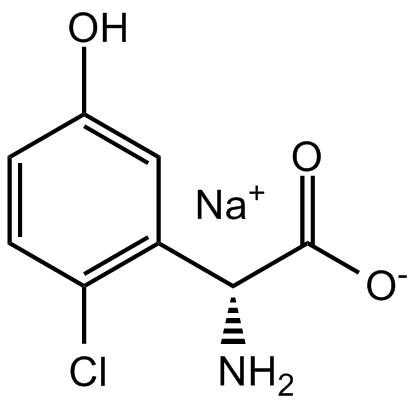
GC17963
CHPG Sodium salt
La sal sÓdica de CHPG es un agonista selectivo de mGluR5 y atenÚa el estrés oxidativo y la inflamaciÓn inducidos por SO2 a través de la vÍa TSG-6/NF-κB en células microgliales BV2.
-
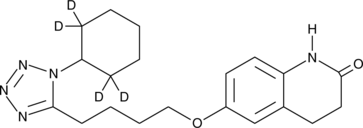
GC47088
Cilostazol-d4
An internal standard for the quantification of cilostazol
-
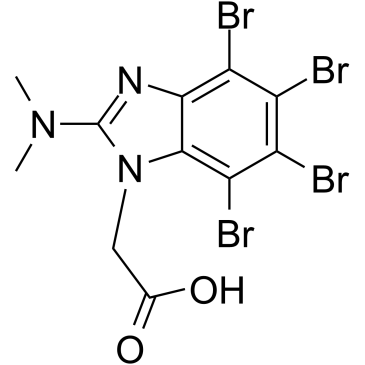
GC39485
CK2/ERK8-IN-1
CK2/ERK8-IN-1 es un inhibidor dual de caseÍna quinasa 2 (CK2) (Ki de 0,25 μM) y ERK8 (MAPK15, ERK7) con IC50 de 0,50 μM. CK2/ERK8-IN-1 también se une a PIM1, HIPK2 (proteÍna quinasa 2 que interactÚa con el homeodominio) y DYRK1A con Kis de 8,65 μM, 15,25 μM y 11,9 μM, respectivamente. CK2/ERK8-IN-1 tiene eficacia proapoptÓtica.
-
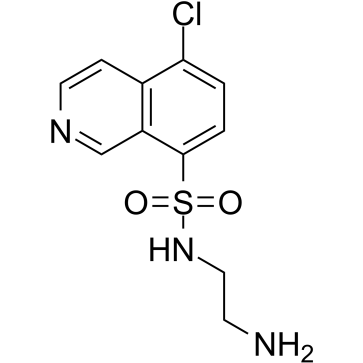
GC38755
CKI-7
CKI-7 es un inhibidor de la caseÍna cinasa 1 (CK1) potente y competitivo con ATP con una IC50 de 6 μM y una Ki de 8,5 μM. CKI-7 es un inhibidor selectivo de la quinasa Cdc7. CKI-7 también inhibe SGK, S6 quinasa-1 ribosomal (S6K1) y proteÍna quinasa-1 activada por mitÓgeno y estrés (MSK1). CKI-7 tiene un efecto mucho mÁs débil sobre la caseÍna quinasa II y otras proteÍnas quinasas.
-
GC14693
CMK
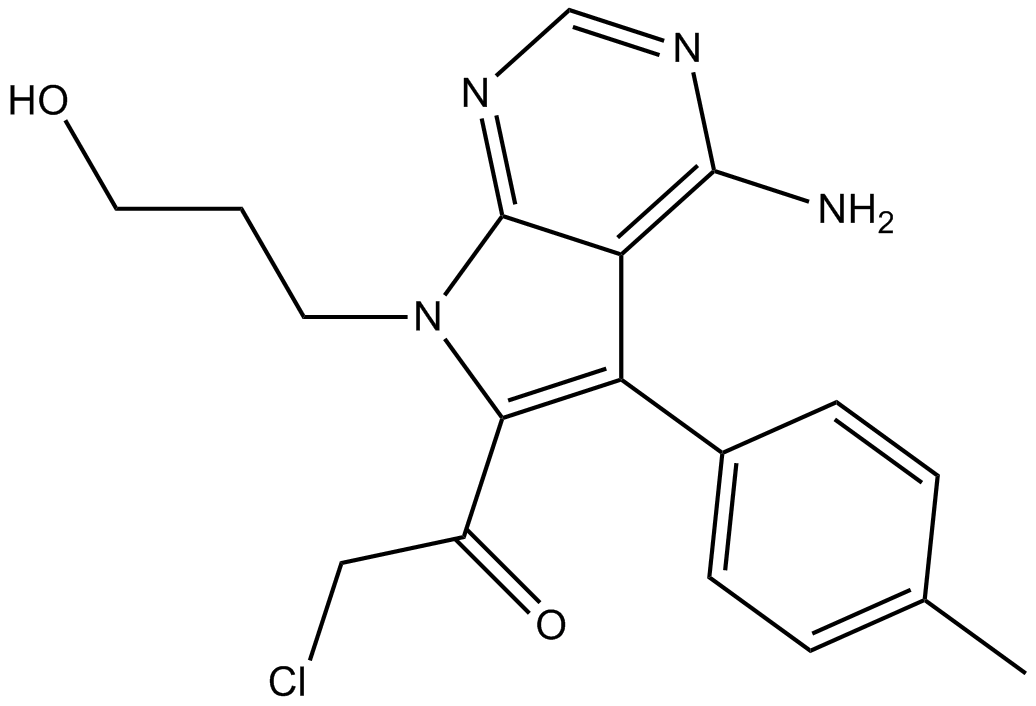
-
GC11180
CMPD-1
CMPD-1 es un inhibidor de la fosforilación de MK2 mediado por p38 MAPK selectivo y no competitivo con ATP con Ki aparente (Kiapp) de 330 nM.
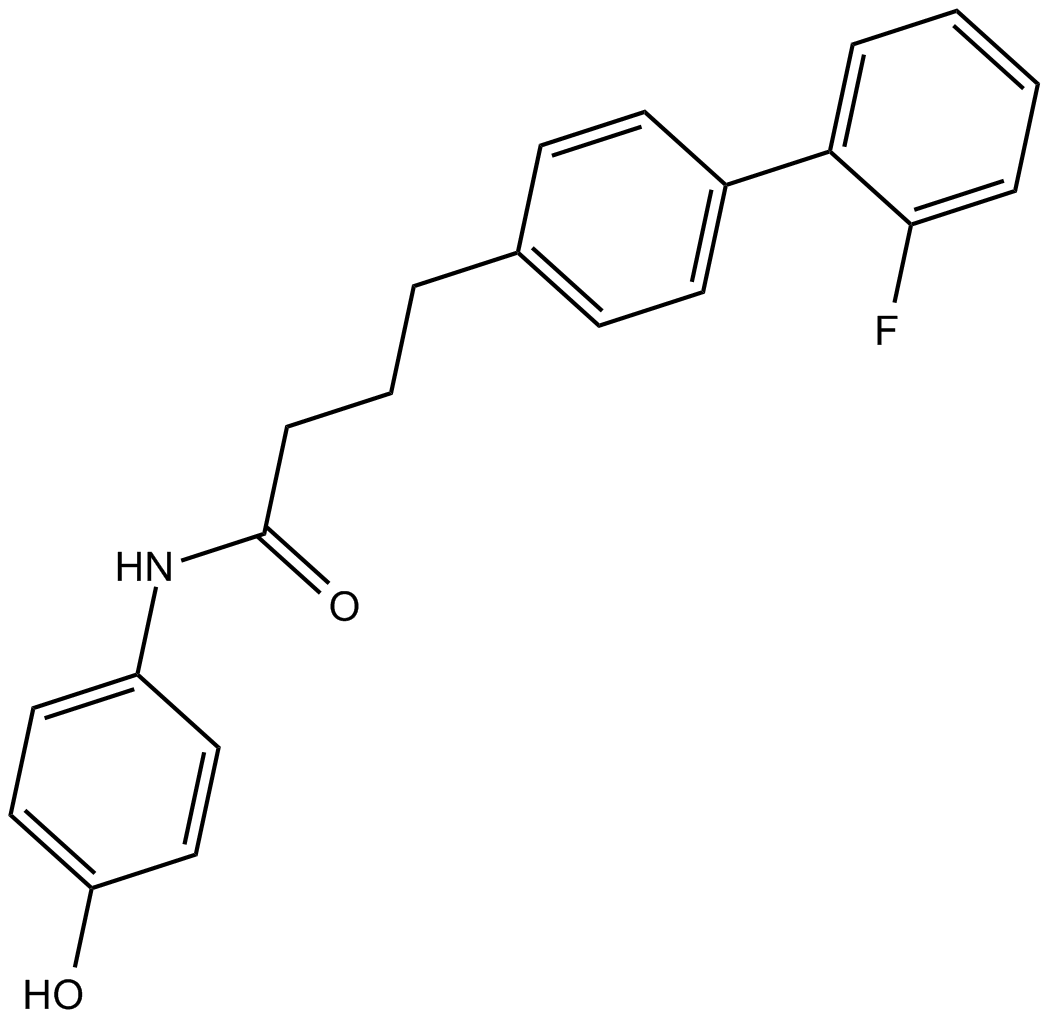
-
GC10033
Cobimetinib
A potent, orally available MEK1 inhibitor
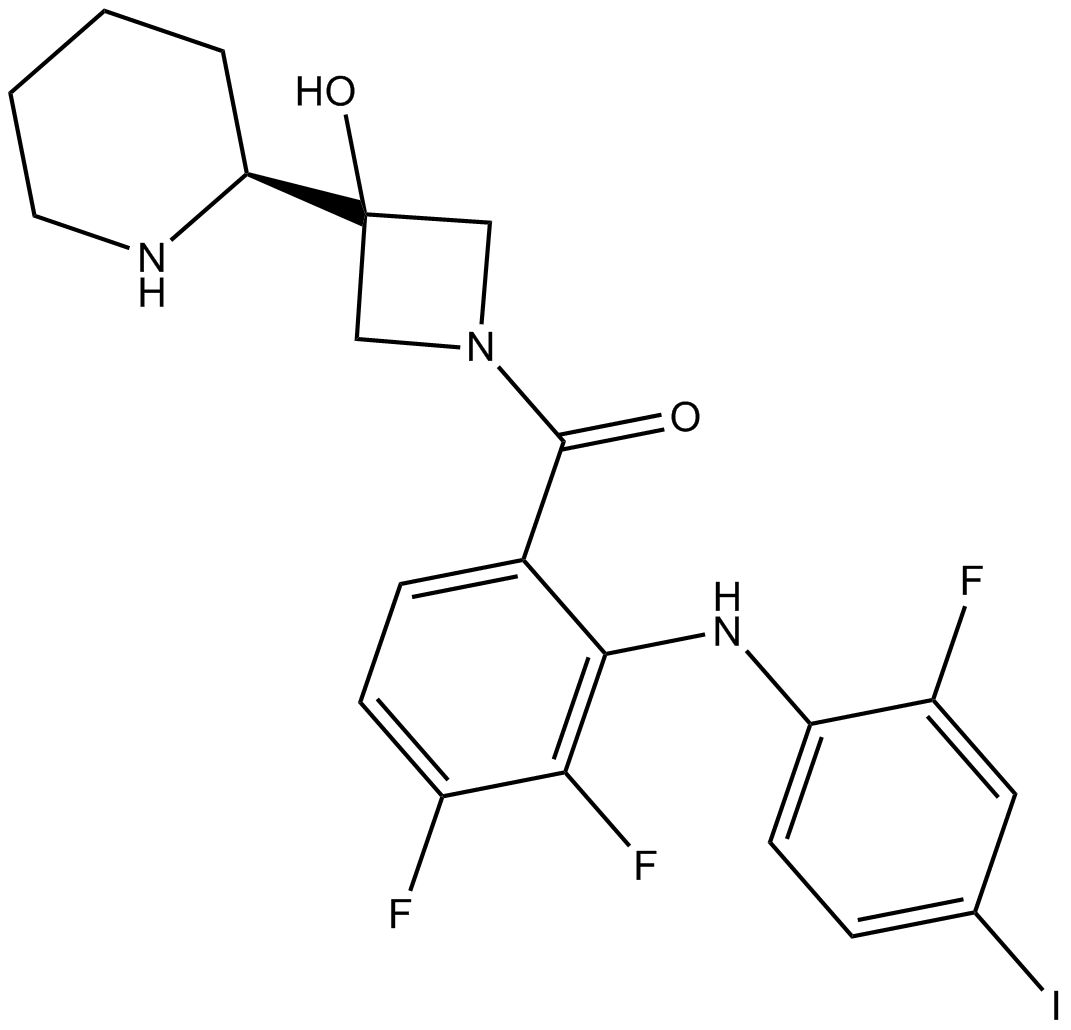
-
GC14337
Cobimetinib (R-enantiomer)

-
GC17426
Cobimetinib (racemate)

-
GC35719
Cobimetinib hemifumarate
El hemifumarato de cobimetinib es un nuevo inhibidor selectivo de MEK1 y el valor IC50 frente a MEK1 es de 4,2 nM.
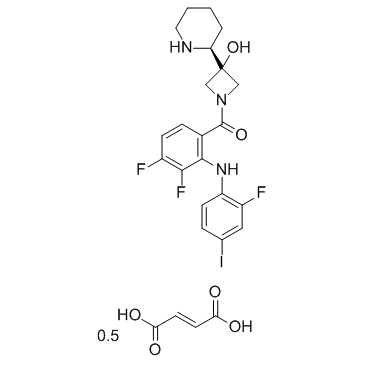
-
GC25289
CompK
CompK (compound K), a potent and selective hematopoietic progenitor kinase 1 (HPK1 or MAP4K1) small molecule inhibitor, significantly improves human T-cell functions, with enhanced T-cell receptor avidity to recognize tumor-associated antigens and tumor cytolytic activity by CD8+ T cells.
-
GC46123
Comprehensive Kinase Screening Library
For screening of a variety of kinase inhibitors

-
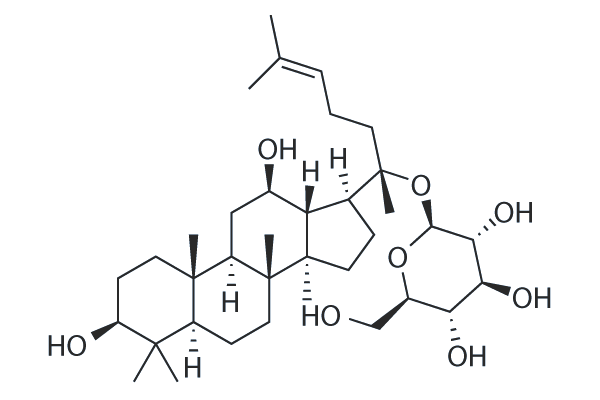
GN10409
cor-nuside
-
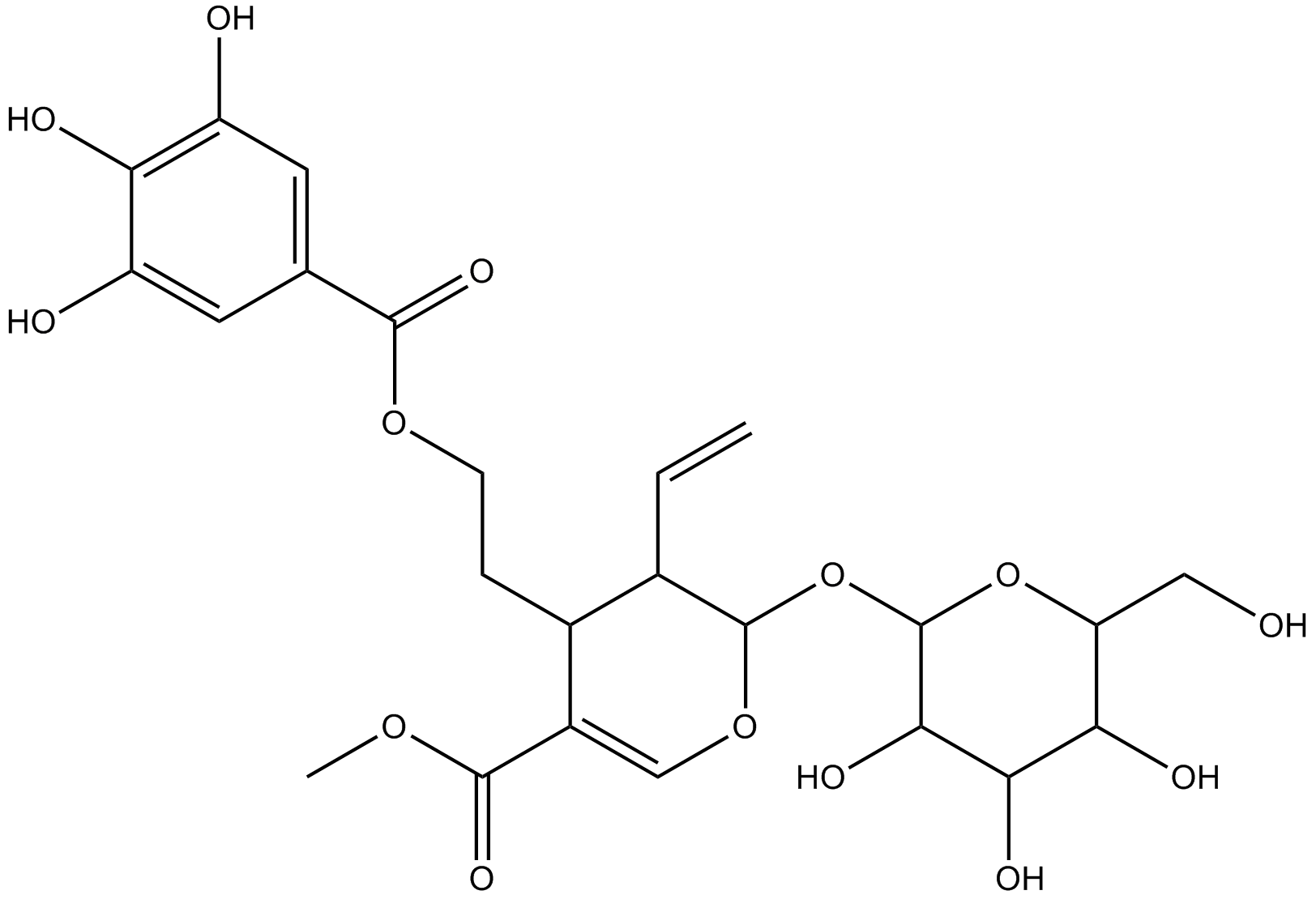
GN10481
Corynoxeine
-
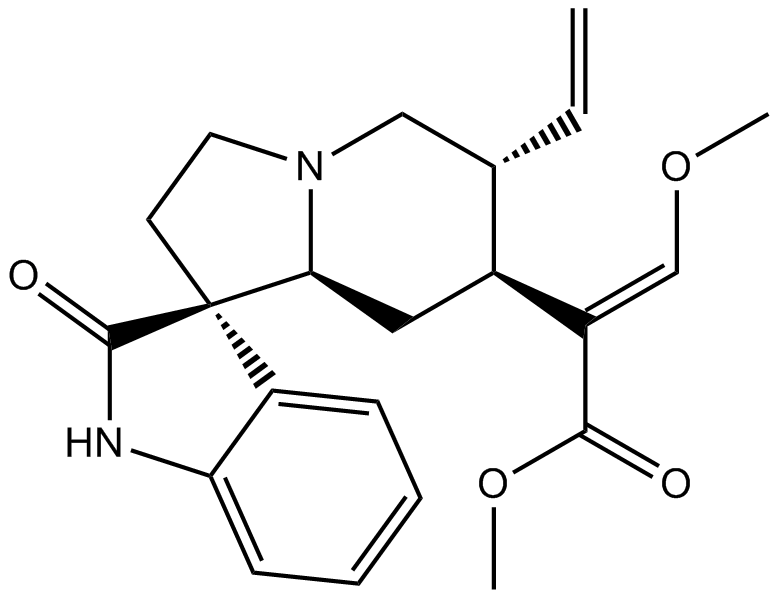
GC35734
Cot inhibitor-1
El inhibidor de Cot-1 (compuesto 28) es un inhibidor selectivo de la cinasa loci-2 (Tpl2) de progresiÓn tumoral con una IC50 de 28 nM.
-
GC35735
Cot inhibitor-2
El inhibidor de Cot-2 es un inhibidor de Cot (Tpl2/MAP3K8) potente, selectivo y activo por vÍa oral con una IC50 de 1,6 nM. El inhibidor de Cot-2 inhibe la producciÓn de TNF-α en sangre entera humana estimulada con LPS con una IC50 de 0,3 μM.
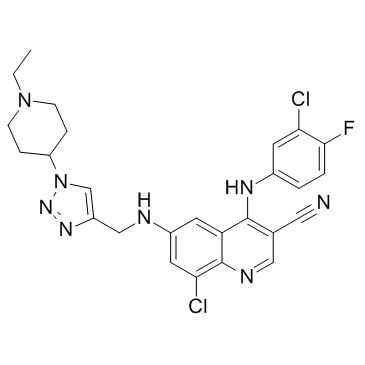
-
GC31525
CREBtide
CREBtide, un péptido sintético de 13 aminoÁcidos, se ha informado como sustrato de PKA.
-
GC43332
Cuspin-1
The Survival of Motor Neurons (SMN) protein participates in RNA splicing.
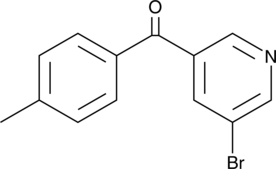
-
GC50181
CW 008
PKA signaling activator; promotes osteogenesis from hMSCs

-
GC43339
Cyclic di-AMP (sodium salt)
Cyclic di-AMP (c-di-AMP) is a second messenger produced by bacteria but not by mammals.
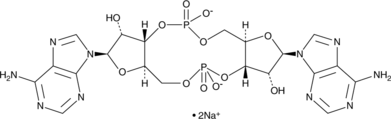
-
GC43341
Cyclic GMP
Guanosine 3',5'-cyclic monophosphate (cyclic GMP or cGMP) is a second messenger that is biosynthesized from GTP by guanylate cyclases.
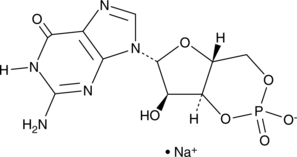
-
GC30057
D-JNKI-1 (AM-111)
D-JNKI-1 (AM-111) (AM-111) es un inhibidor peptÍdico de JNK muy potente y permeable a las células.

-
GC15187
Dabrafenib (GSK2118436)
Dabrafenib (GSK2118436) (GSK2118436A) es un inhibidor de Raf competitivo con ATP con IC50 de 5 nM y 0,6 nM para C-Raf y B-RafV600E, respectivamente.
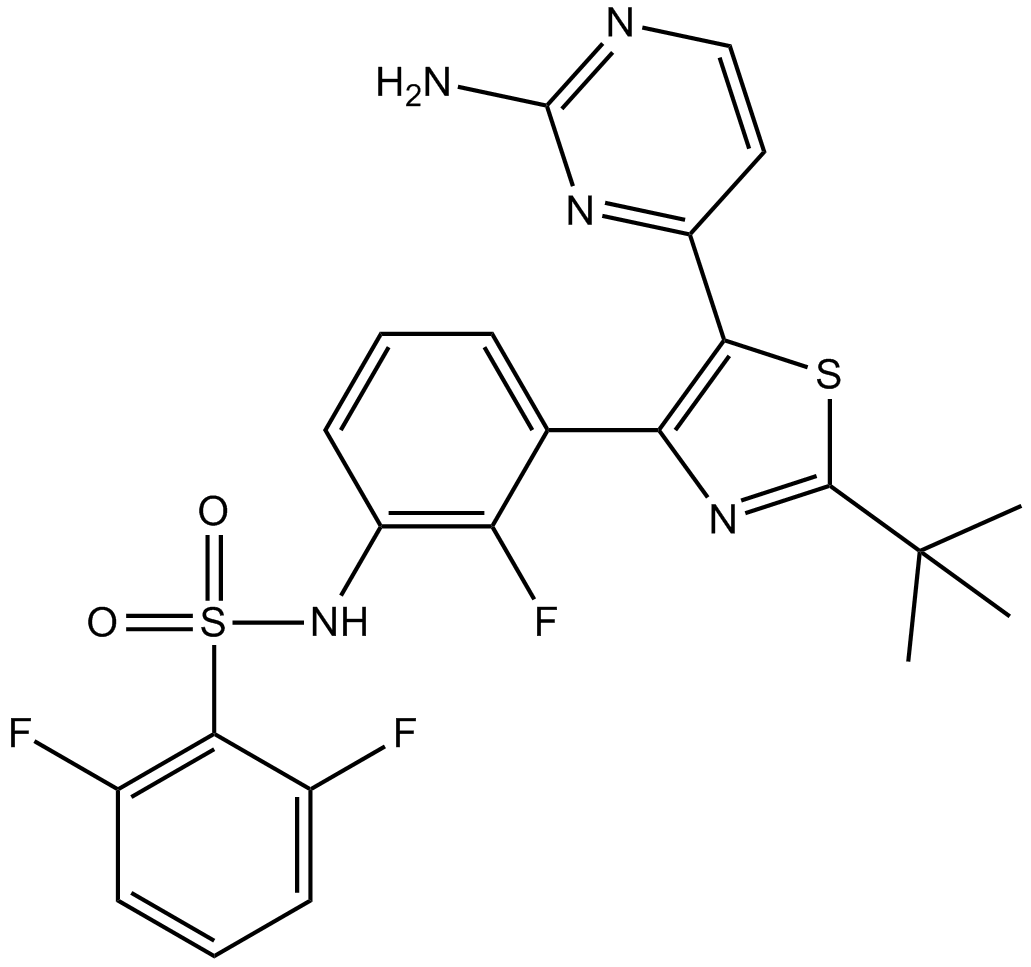
-
GC12486
Dabrafenib Mesylate (GSK-2118436)
A Raf kinase inhibitor
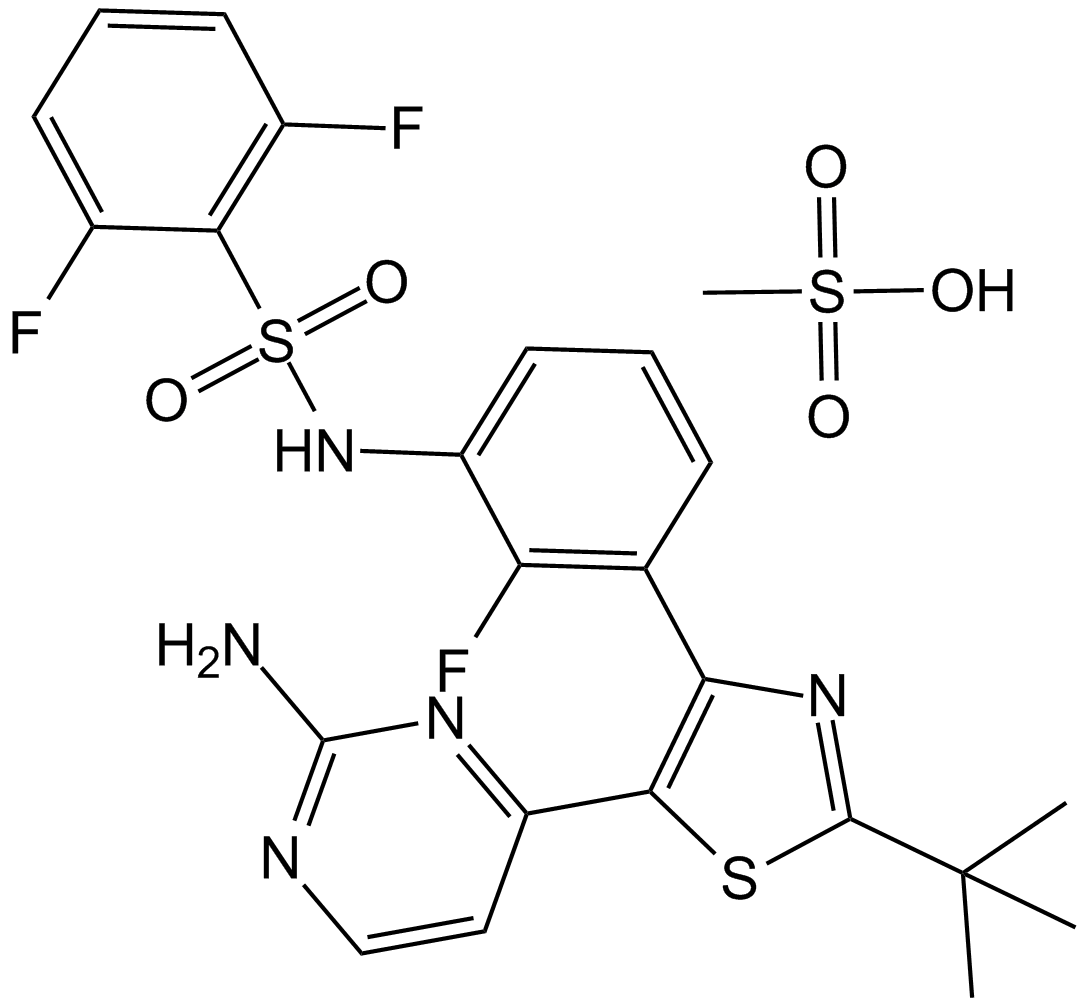
-
GC47166
Dabrafenib-d9
Dabrafenib-d9 (GSK2118436A-d9) es el deuterio etiquetado como Dabrafenib. Dabrafenib (GSK2118436A) es un inhibidor de Raf competitivo con ATP con IC50 de 5 nM y 0,6 nM para C-Raf y B-RafV600E, respectivamente.
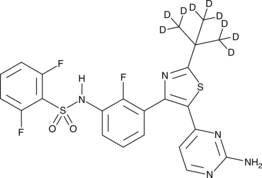
-
GN10336
Daphnetin
-
GC14114
DB07268
A potent inhibitor of JNK1
-
GC15477
DBM 1285 dihydrochloride
p38 MAPK inhibitor
-
GC16795
DCA
DCA es un regulador metabólico en las mitocondrias de las células cancerosas con actividad anticancerígena. DCA inhibe PDHK, lo que resulta en una disminución del ácido láctico en el microambiente tumoral. DCA aumenta la generación de especies reactivas de oxígeno (ROS) y promueve la apoptosis de las células cancerosas. DCA también funciona como inhibidor de NKCC.

-
GC18666
Debromohymenialdisine
Damaged DNA in humans is detected by sensor proteins that transmit a signal through checkpoint kinases (Chks) Chk1 and Chk2.
-
GC35832
Dehydrocorydaline chloride
El cloruro de dehidrocoridalina (cloruro de 13-metilpalmatina) es un alcaloide que regula la expresiÓn proteica de Bax, Bcl-2; activa caspasa-7, caspasa-8 e inactiva PARP.
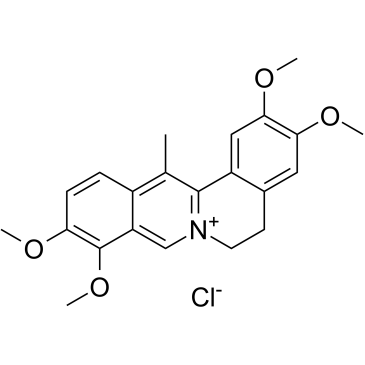
-
GC16214
DEL-22379
DEL-22379 es un inhibidor de dimerizaciÓn de ERK. DEL-22379 se une fÁcilmente a ERK2 con una Kd estimada en el rango micromolar bajo, aunque la uniÓn es detectable incluso a concentraciones nanomolares bajas. La dimerizaciÓn de ERK2 se inhibe progresivamente con una IC50 de ~0,5 μM.
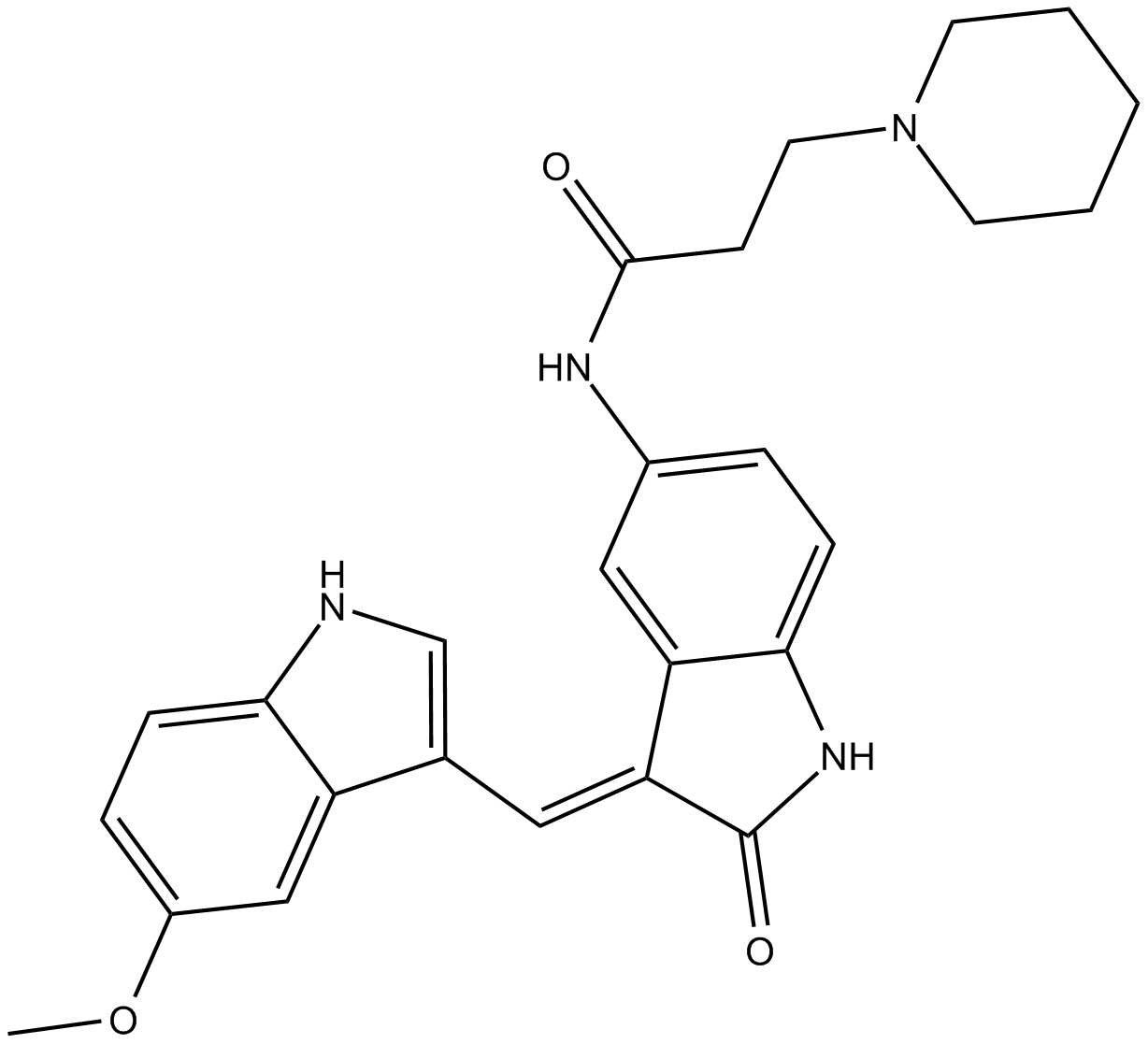
-
GC32254
Deltonin
Deltonin, una saponina esteroidal, aislada de Dioscorea zingiberensis Wright, con actividad antitumoral; Deltonin inhibe la activaciÓn de ERK1/2 y AKT.
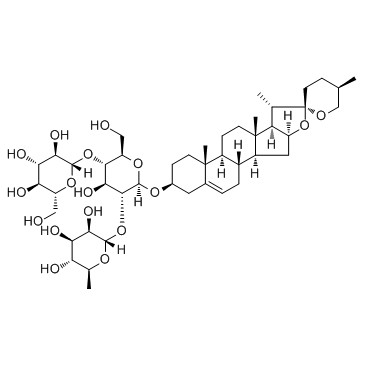
-
GC12824
Dibutyryl-cAMP, sodium salt
La sal sÓdica de dibutiril-cAMP (sal sÓdica de dibutiril-cAMP) es un anÁlogo del AMP cÍclico estabilizado (cAMP) y un activador selectivo de la PKA.
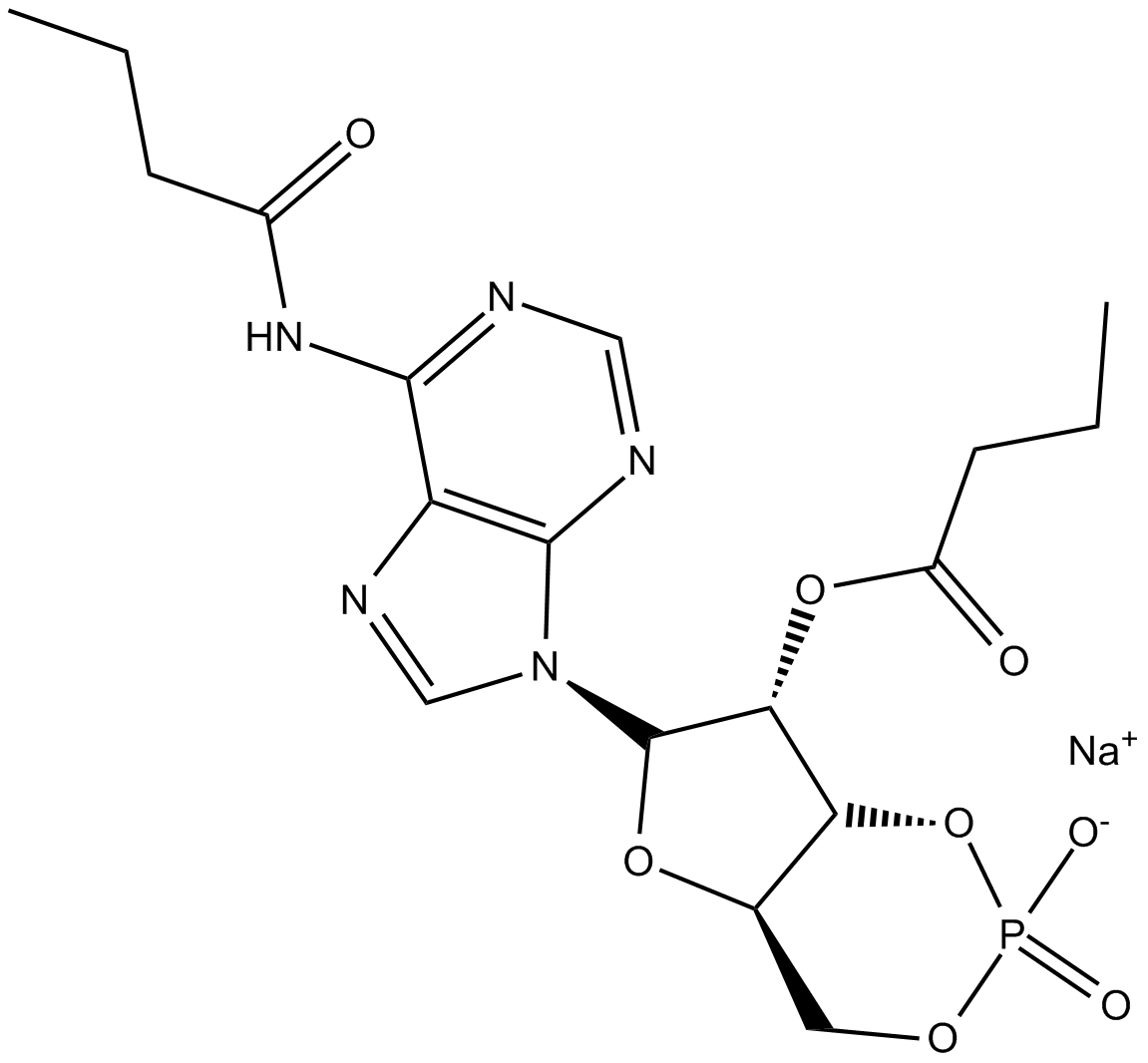
-
GC38319
Dihydrocaffeic acid
El Ácido dihidrocaféico es un Ácido fenÓlico que se encuentra en Gynura bicolor, reduce la fosforilaciÓn de MAPK p38 y previene el daÑo de la piel inducido por los rayos UVB.
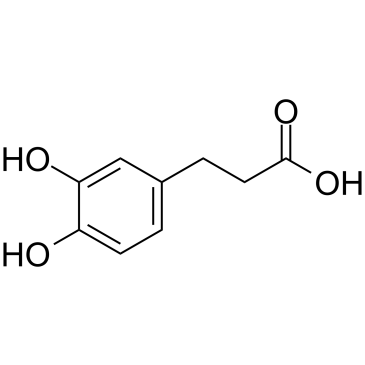
-
GC31697
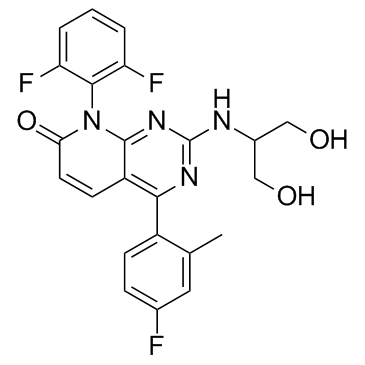
Dilmapimod (SB-681323)
Dilmapimod (SB-681323) (SB-681323) es un potente inhibidor de p38 MAPK que potencialmente suprime la inflamaciÓn en la enfermedad pulmonar obstructiva crÓnica.
-
GC30913
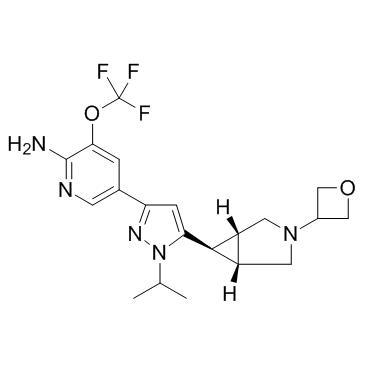
DLK-IN-1
DLK-IN-1 es un inhibidor selectivo activo por vÍa oral de la cremallera quinasa de leucina dual (DLK, MAP3K12), con una Ki de 3 nM.
-
GC61466
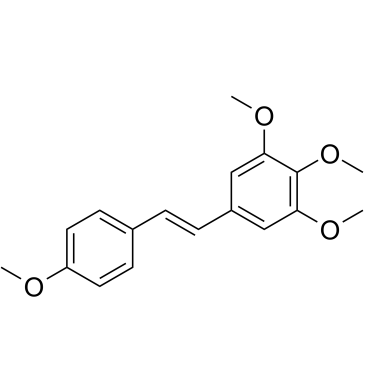
DMU-212
DMU-212 es un derivado metilado del Resveratrol, con actividades antimitÓticas, antiproliferativas, antioxidantes y promotoras de la apoptosis. DMU-212 induce la detenciÓn mitÓtica mediante la inducciÓn de la apoptosis y la activaciÓn de la proteÍna ERK1/2. DMU-212 tiene actividad oral.
-
GC65298
DMX-5804
DMX-5804 es un inhibidor de MAP4K4 potente, activo por vÍa oral y selectivo, con una IC50 de 3 nM, una pIC50 de 8,55 para MAP4K4 humana, menos potente en MINK1/MAP4K6 (pIC50, 8,18) y TNIK/MAP4K7 (pIC50, 7,96) .
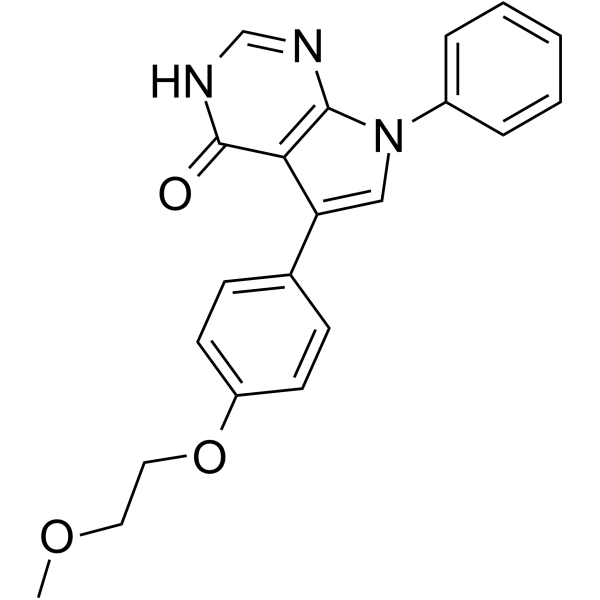
-
GC13244
DTP3
DTP3 TFA es un inhibidor potente y selectivo de GADD45β/MKK7. DTP3 TFA se dirige a un mÓdulo esencial de supervivencia celular selectivo del cÁncer aguas abajo de la vÍa NF-κB.
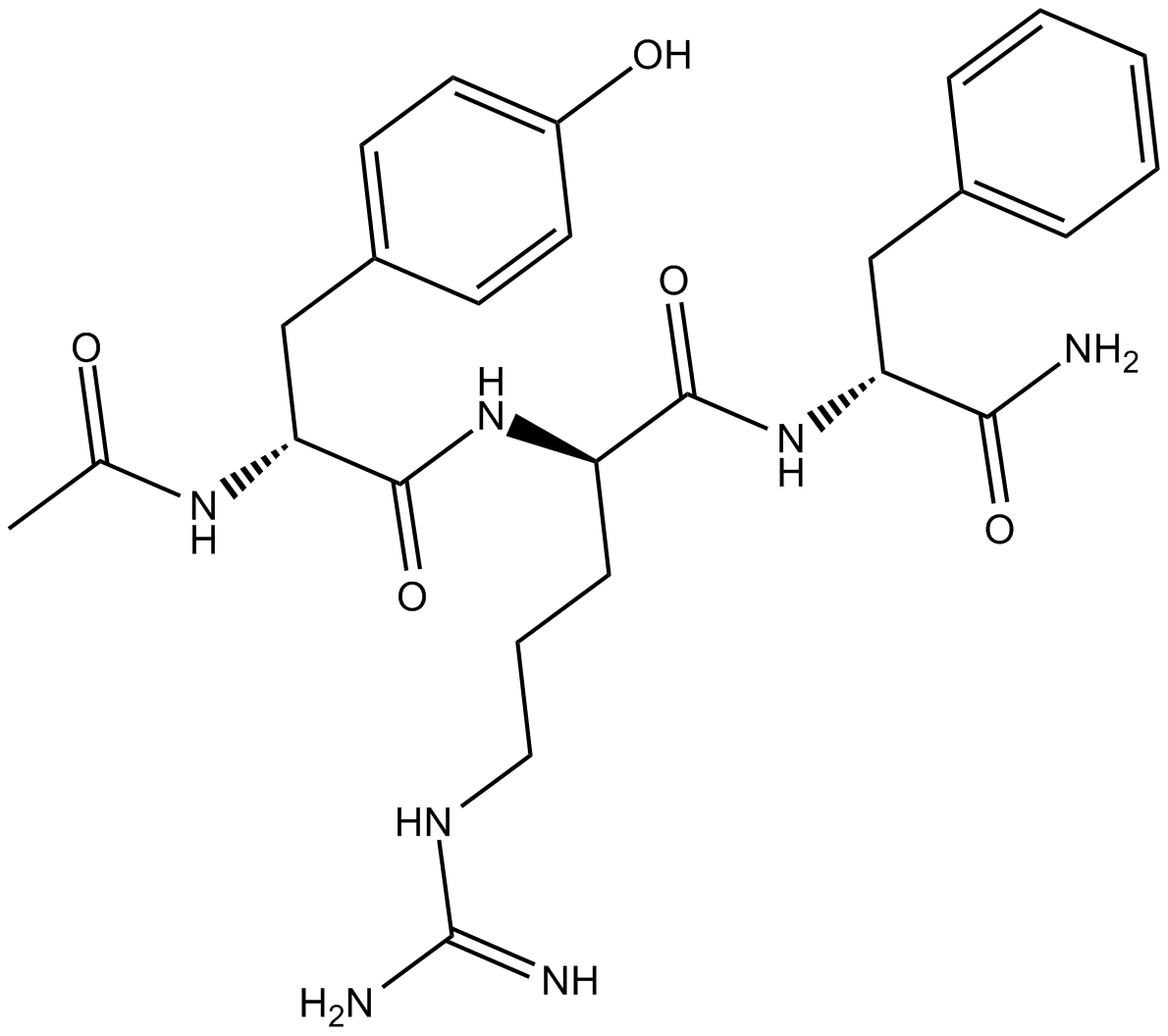
-
GC38202
DTP3 TFA
DTP3 TFA es un inhibidor potente y selectivo de GADD45β/MKK7 (proteÍna quinasa 7 activada por mitÓgeno/β inducible por daÑo en el crecimiento y detenciÓn del crecimiento). DTP3 TFA se dirige a un mÓdulo esencial de supervivencia celular selectivo del cÁncer aguas abajo de la vÍa NF-κB.
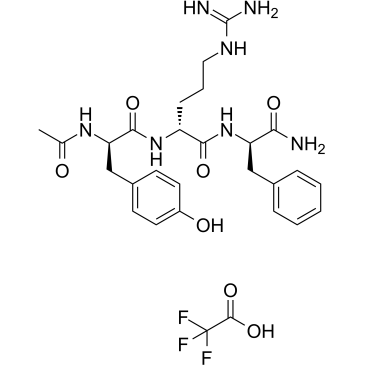
-
GC64496
EF24
EF24 es un anÁlogo de curcumina con mayor eficacia antitumoral y biodisponibilidad oral a través de la desactivaciÓn de la vÍa de seÑalizaciÓn MAPK/ERK en el carcinoma de células escamosas oral (OSCC). El tratamiento con EF24 aumenta los niveles de caspasa 3 y 9 activadas y disminuye las formas fosforiladas de MEK1 y ERK.
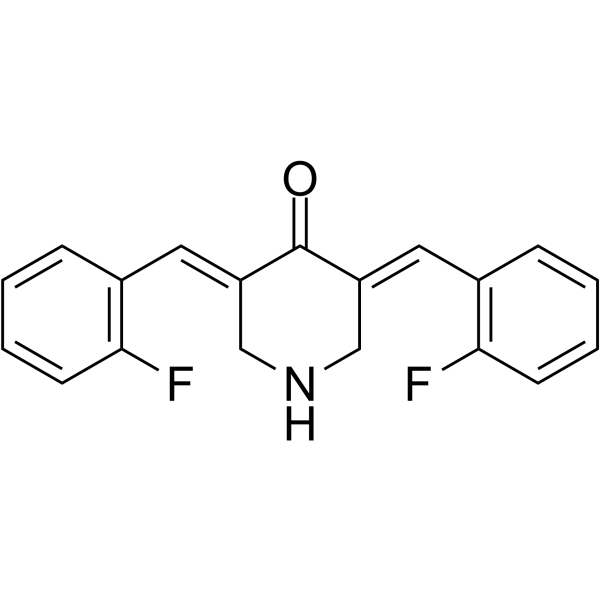
-
GC10030
EHop-016
Inhibidor de la GTPasa Rac1/Rac3, potente y específico.
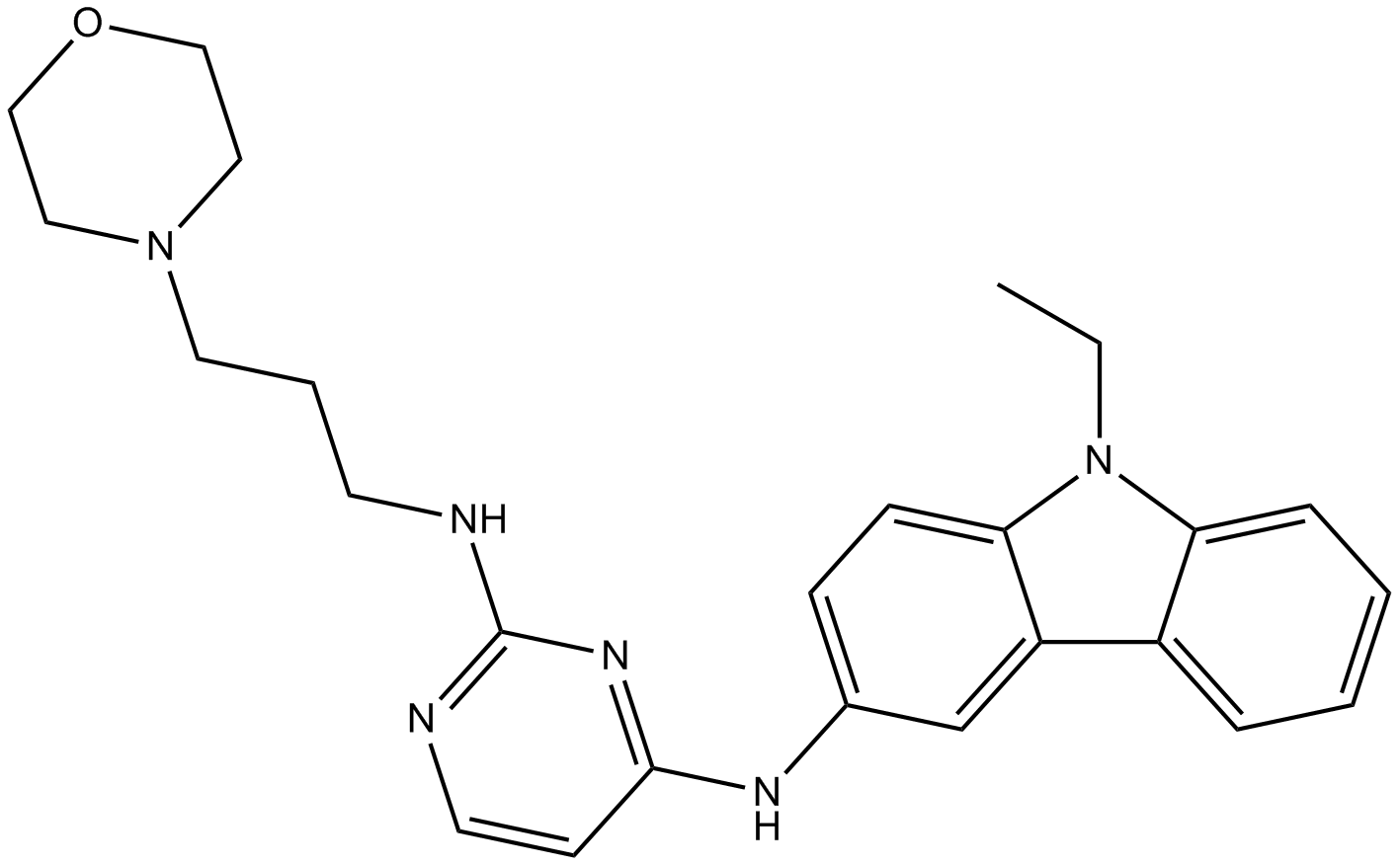
-
GC43610
Enniatin A1
La enniatina A1 aislada de las micotoxinas de Fusarium es un hexadepsipéptido cÍclico que consta de Ácidos D-α-hidroxiisovalérico alternados y N-metil-L-aminoÁcidos. Enniatin A1 posee propiedades anticancerÍgenas mediante la inducciÓn de apoptosis y la interrupciÓn de la vÍa de seÑalizaciÓn de ERK. Enniatin A1 inhibe ACAT con un IC50 de 49 μM en microsomas de hÍgado de rata.
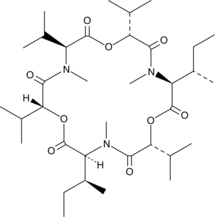
-
GC17693
Enniatin B
La enniatina B es una micotoxina de Fusarium.
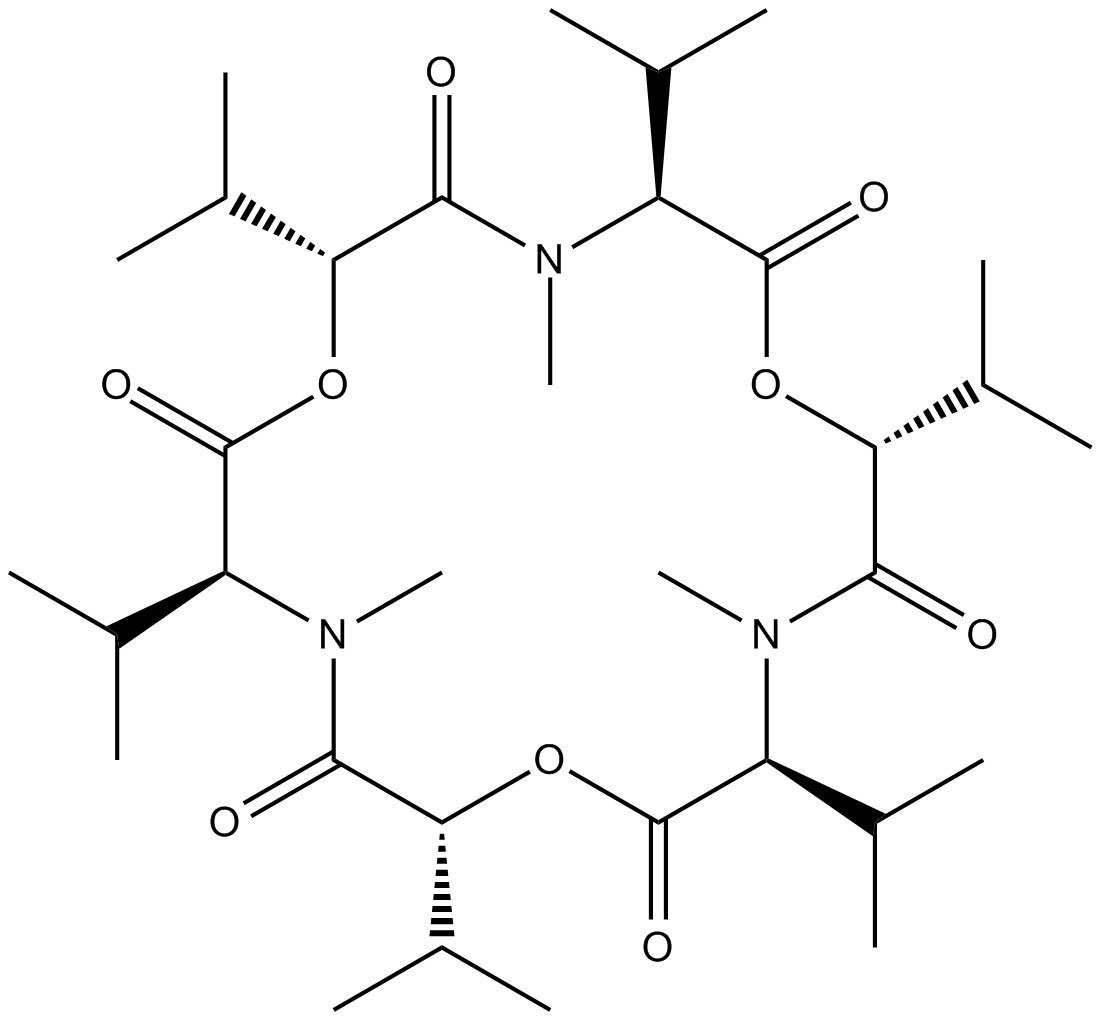
-
GC43611
Enniatin B1
La enniatina B1 es una micotoxina de Fusarium.
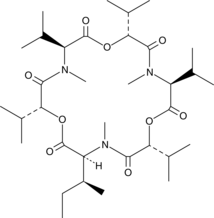
-
GC10403
EO 1428
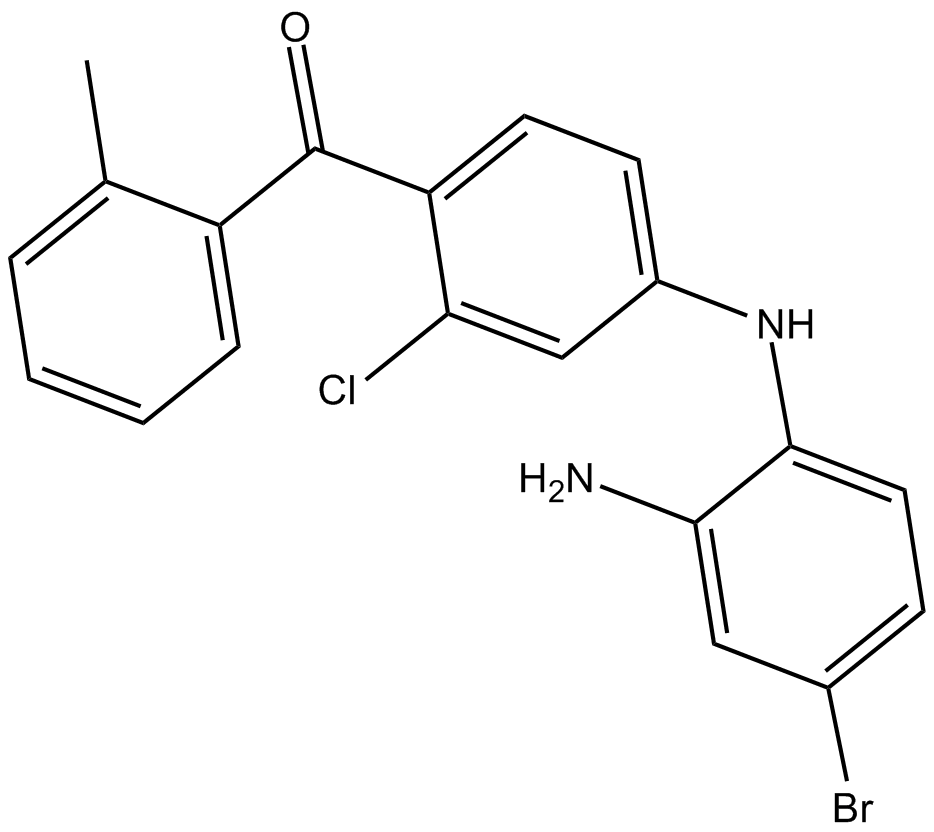
EO 1428 es un inhibidor altamente especÍfico de p38 de la clase aminobenzofenona.
-
GC43624
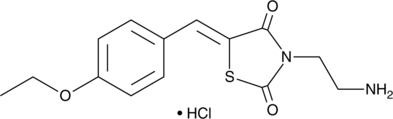
ERK Inhibitor
ERK inhibitor is a cell-permeable inhibitor that binds ERK2 near its docking domain (KD = 5 μM).
-
GC33206
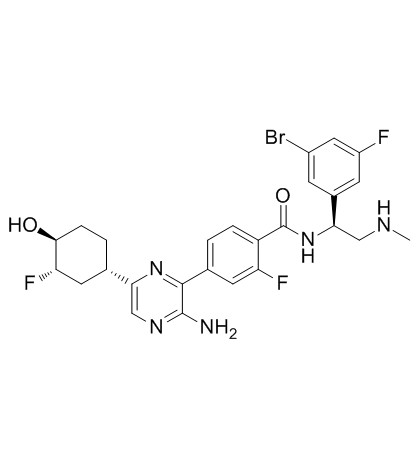
ERK-IN-1
ERK-IN-1 (compuesto B) es un inhibidor de ERK1 y ERK2 disponible por vÍa oral en el tratamiento de una enfermedad proliferativa caracterizada por mutaciones activadoras en la vÍa MAPK. La actividad estÁ particularmente relacionada con el tratamiento del CPNM con mutaciÓn en KRAS, el CPNM con mutaciÓn en BRAF, el cÁncer de pÁncreas con mutaciÓn en KRAS, el cÁncer colorrectal (CCR) con mutaciÓn en KRAS y el cÁncer de ovario con mutaciÓn en KRAS. El clorhidrato de ERK-IN-1 también puede inhibir la RAF.
-
GC62229
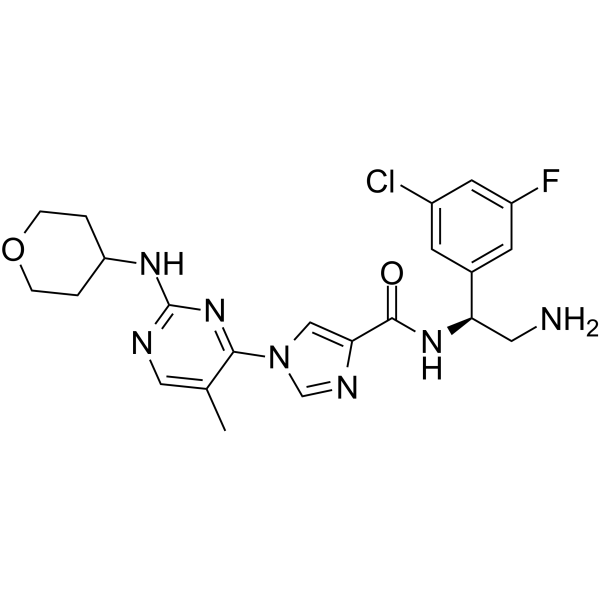
ERK-IN-3
ERK-IN-3 es un inhibidor potente y oralmente activo de ERK. ERK-IN-3 inhibe ERK1/2 con valores IC50 nM bajos de un solo dÍgito. ERK-IN-3 se puede utilizar para la investigaciÓn de cÁnceres provocados por mutaciones RAS.
-
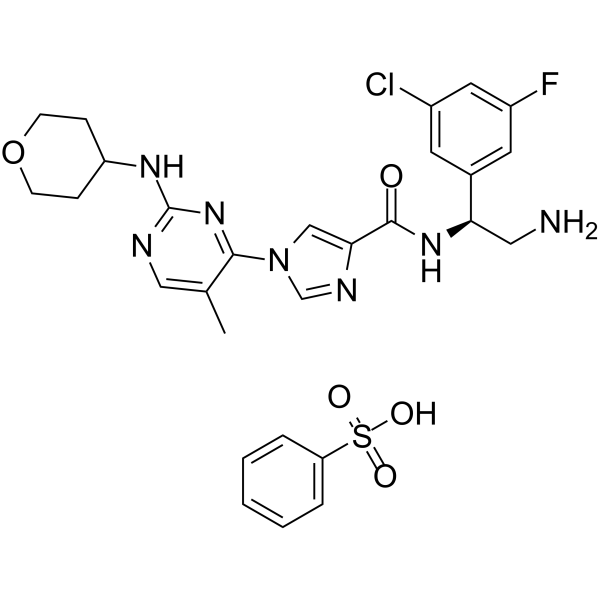
GC63459
ERK-IN-3 benzenesulfonate
El bencenosulfonato de ERK-IN-3 es un inhibidor potente y oralmente activo de ERK. El bencenosulfonato de ERK-IN-3 inhibe ERK1/2 con valores IC50 nM bajos de un solo dÍgito. El bencenosulfonato ERK-IN-3 se puede utilizar para la investigaciÓn de cÁnceres provocados por mutaciones RAS.
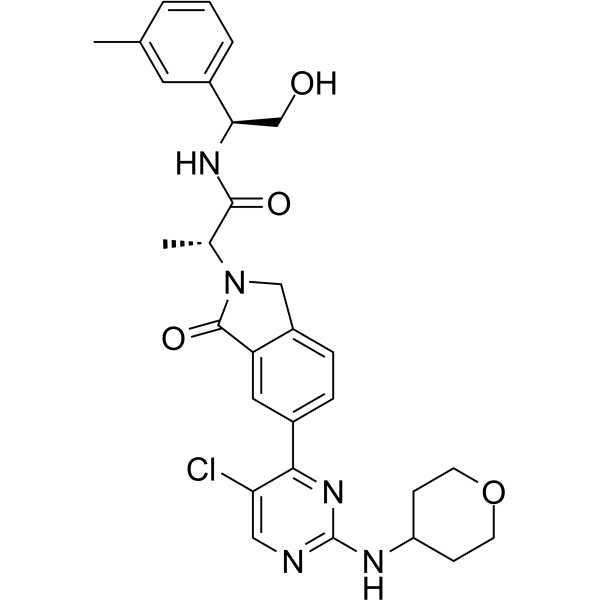
-
GC65510
ERK1/2 inhibitor 1
El inhibidor 1 de ERK1/2 es un potente inhibidor de ERK1/2 biodisponible por vÍa oral, que muestra una inhibiciÓn del 60 % a 1 nM y una IC50 de 3,0 nM contra ERK1 y ERK2, respectivamente.
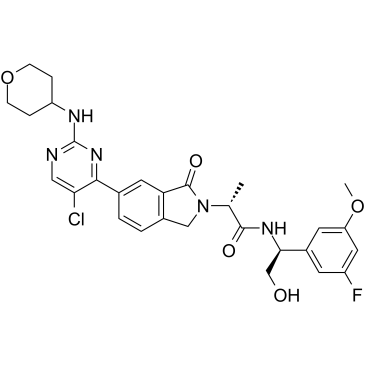
-
GC60812
ERK1/2 inhibitor 2
El inhibidor 2 de ERK1/2 (Ejemplo 1) es un potente inhibidor dual de ERK1/2. El inhibidor 2 de ERK1/2 tiene actividad anticancerÍgena.
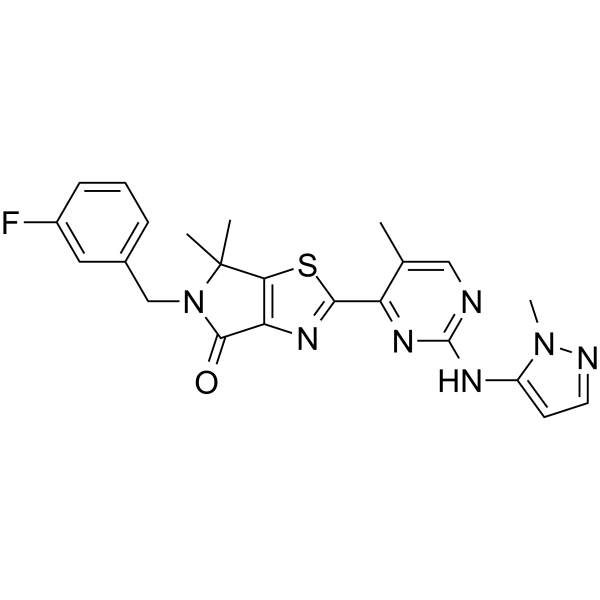
-
GC67905
ERK1/2 inhibitor 7
-
GC32796
ERK5-IN-1
ERK5-IN-1 es un potente inhibidor de ERK5 con una IC50 de 87±7 nM. ERK5-IN-1 también inhibe LRRK2[G2019S] con un IC50 de 26 nM.
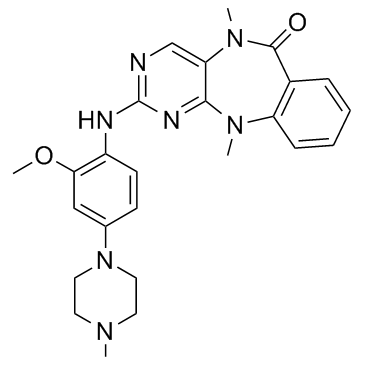
-
GC36002
ERK5-IN-2
ERK5-IN-2 es un inhibidor selectivo de ERK5 submicromolar activo por vÍa oral con IC50 de 0,82 μM, 3 μM para ERK5 y ERK5 MEF2D, respectivamente.
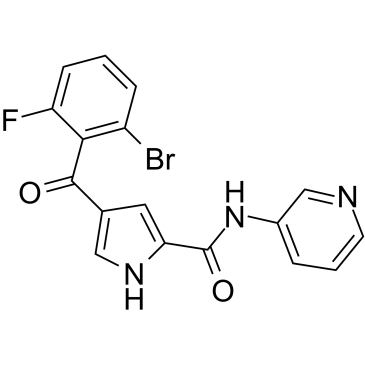
-
GP23419
ESAT6
Early Secretory Target Mycobacterium Tuberculosis Recombinant

-
GC36006
Esculentoside H
La talidomida-O-PEG4-amina es un conjugado enlazador-ligando ligasa E3 sintetizado que incorpora el ligando cereblon basado en talidomida y un enlazador utilizado en la tecnologÍa PROTAC.
-
GC38708
Esculin
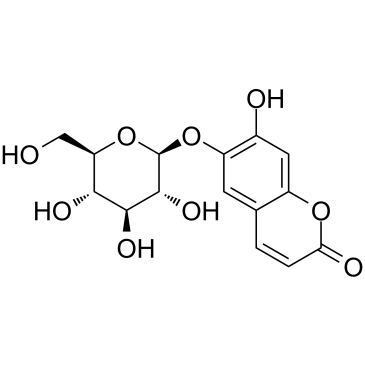
-
GC33033
ETC-206
ETC-206 es un inhibidor selectivo de MNK1 y MNK2 con IC50 de 64 nM y 86 nM, respectivamente.
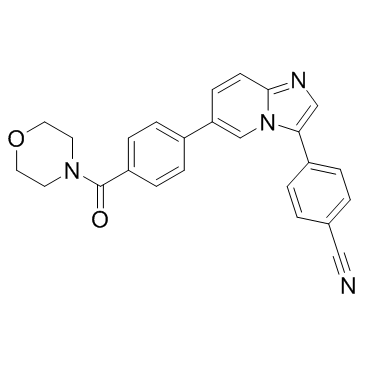
-
GC61941
Eudesmin
Eudesmin ((-)-Eudesmin) altera la diferenciaciÓn adipogénica a través de la inhibiciÓn de la vÍa de seÑalizaciÓn S6K1.
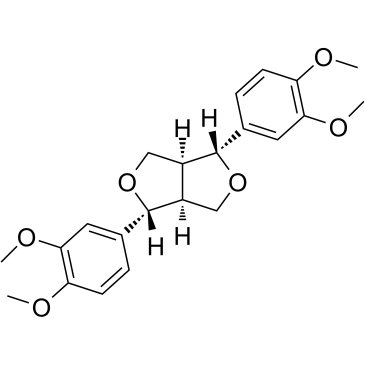
-
GC65329
EW-7195
EW-7195 es un inhibidor potente y selectivo de ALK5 (TGFβR1) con una IC50 de 4,83 nM. EW-7195 tiene una selectividad de >300 veces para ALK5 sobre p38α. EW-7195 inhibe eficazmente la seÑalizaciÓn de Smad inducida por TGF-β1, la transiciÓn epitelial a mesenquimatosa (EMT) y la metÁstasis del tumor de mama en el pulmÓn.
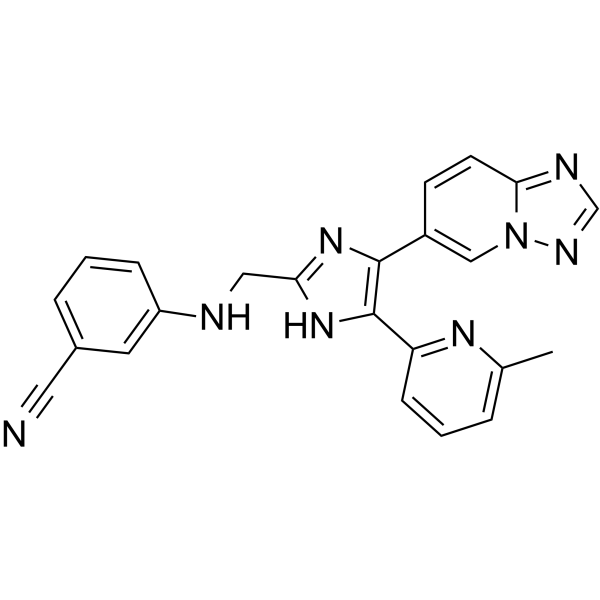
-
GC67964
Exarafenib
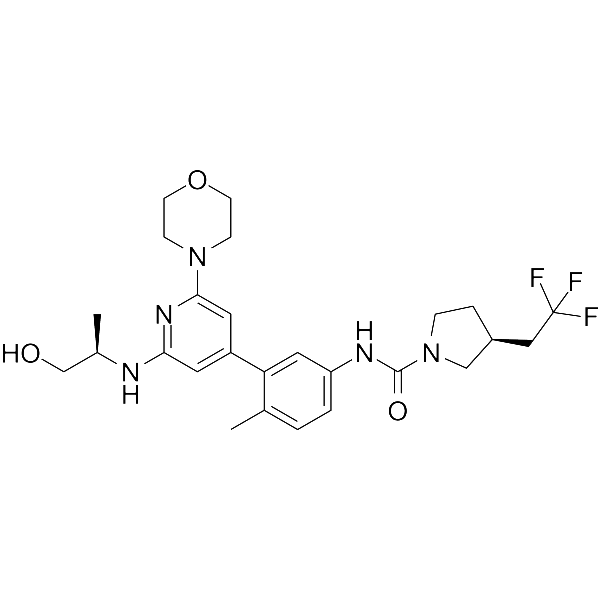
-
GC13869
Fasudil
Fasudil (HA-1077; AT877) es un inhibidor inespecÍfico de RhoA/ROCK y también tiene un efecto inhibitorio sobre las proteÍnas quinasas, con una Ki de 0,33 μM para ROCK1, IC50 de 0,158 μM y 4,58 μM, 12,30 μM, 1,650 μM para ROCK2 y PKA , PKC, PKG, respectivamente. Fasudil es también un potente antagonista y vasodilatador de los canales de Ca2+.

-
GC14289
Fasudil (HA-1077) HCl
Fasudil (HA-1077; AT877) El clorhidrato es un inhibidor rhoa/roca inespecÍfico y también tiene un efecto inhibitorio en las proteÍnas quinasas, con un KI de 0.33 μ m para rock115&&&&7777 que offlineefficient_models_2022q2.md;M, 1.650 μM for ROCK2 and PKA, PKC, PKG, respectively.en_es_2021q4.mdFasudil (HA-1077) HCl is also a potent Ca2+ channel antagonist and vasodilator.en_es_2021q4.md
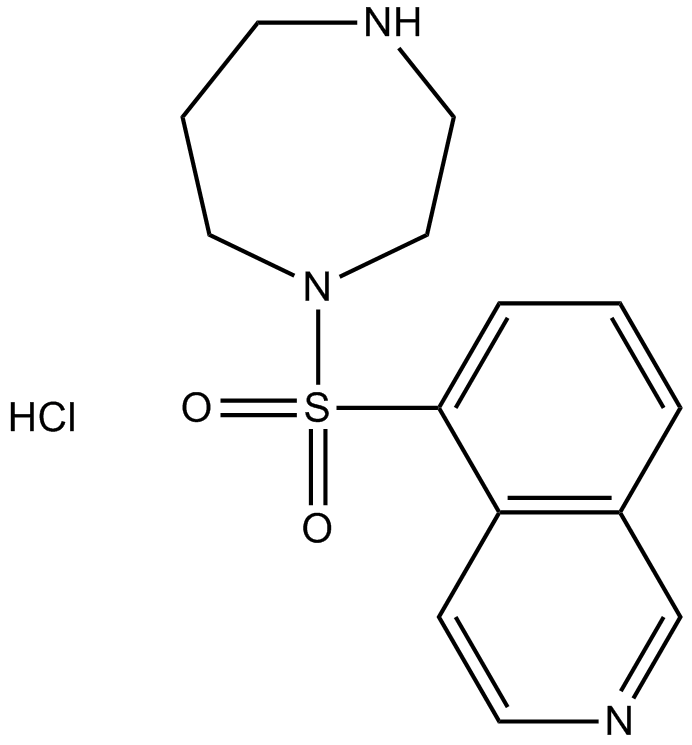
-
GC40484
Ferulic Acid methyl ester
El éster metÍlico del Ácido ferÚlico (Ferulato de metilo) es un derivado del Ácido ferÚlico, aislado de Stemona tuberosa, con propiedades antiinflamatorias y antioxidantes.
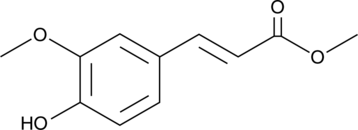
-
GC25420
Fipexide hydrochloride
Fipexide (hydrochloride) is a psychoactive drug of the piperazine class.
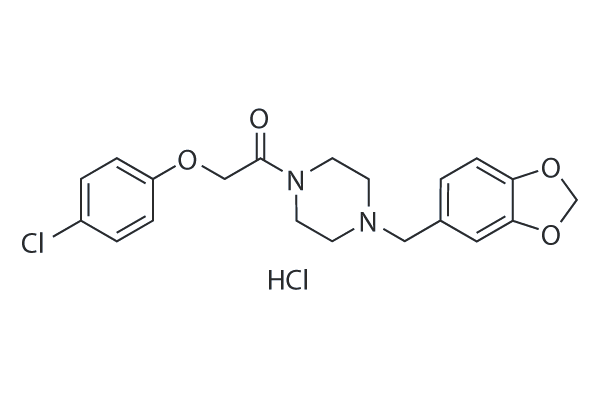
-
GP23504
FLRT3 Human
Fibronectin Leucine Rich Transmembrane Protein 3 Human Recombinant

-
GC14125
FMK
An inhibitor of RSK2
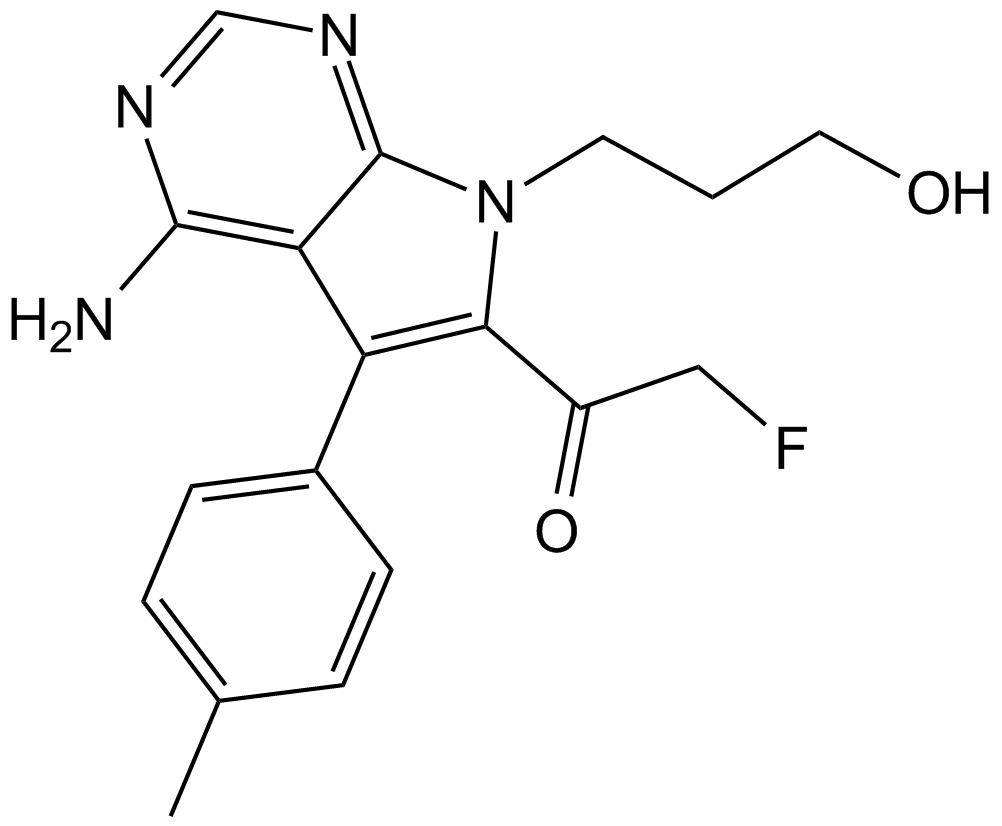
-
GC34197
FMK-MEA
FMK-MEA es un potente y selectivo inhibidor de la quinasa ribosomal S6 p90 (RSK).
-
GC32411
FR 167653 (FR 167653 sulfate)
FR 167653 (FR 167653 sulfato) (FR 167653 (FR 167653 sulfato) sulfato), un inhibidor p38 MAPK selectivo y activo por vÍa oral, es un potente supresor de TNF-α y IL-1β producciÓn a través de la inhibiciÓn especÍfica de la actividad de p38 MAPK.
-
GC31058
FR 167653 free base
La base libre FR 167653, un inhibidor de p38 MAPK selectivo y activo por vÍa oral, es un potente supresor de la producciÓn de TNF-α e IL-1β a través de la inhibiciÓn especÍfica de la actividad de p38 MAPK.
-
GC10647
FR 180204
FR 180204 es un inhibidor de ERK selectivo y competitivo con ATP. FR 180204 inhibe ERK1 y ERK2 con IC50 de 0,51 μM (Ki = 0,31 μM) y 0,33 μM (Ki = 0,14 μM), respectivamente.
-
GC12970
Furosemide
La furosemida es un inhibidor potente y activo por vÍa oral del cotransportador Na+/K+/2Cl-(NKCC), NKCC1 y NKCC2.
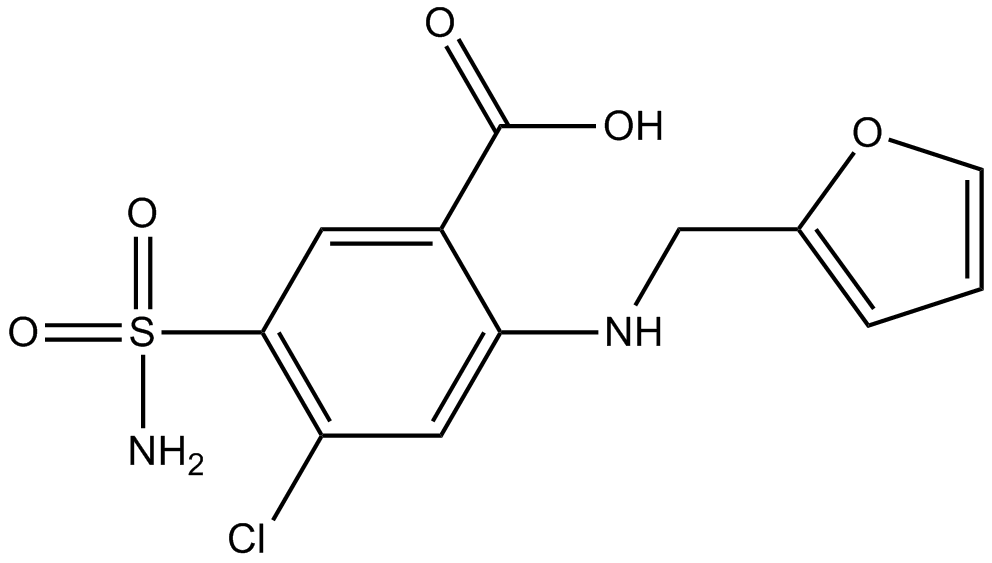
-
GC36091
Furosemide sodium
La furosemida sÓdica es un inhibidor potente y activo por vÍa oral del cotransportador Na+/K+/2Cl-(NKCC), NKCC1 y NKCC2.
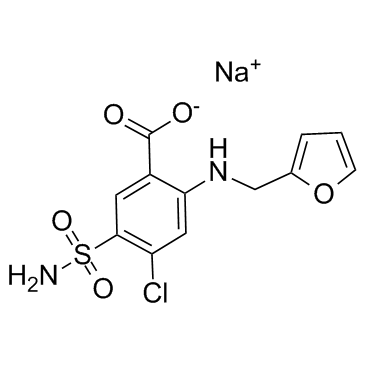
-
GC43734
Ganglioside GQ1b Mixture (sodium salt)
Ganglioside GQ1b is a tetrasialoganglioside that contains two sialic acid residues linked to an inner galactose unit.
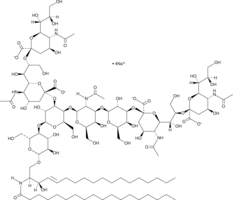
-
GC14247
GDC-0623
GDC-0623 (RG 7421) es un potente inhibidor no competitivo de ATP de MEK1 (Ki =0,13 nM, +ATP) y muestra una potencia 6 veces mÁs débil frente a HCT116 (KRAS (G13D), EC50 =42 nM) frente a A375 (BRAFV600E, EC50 =7 nM).
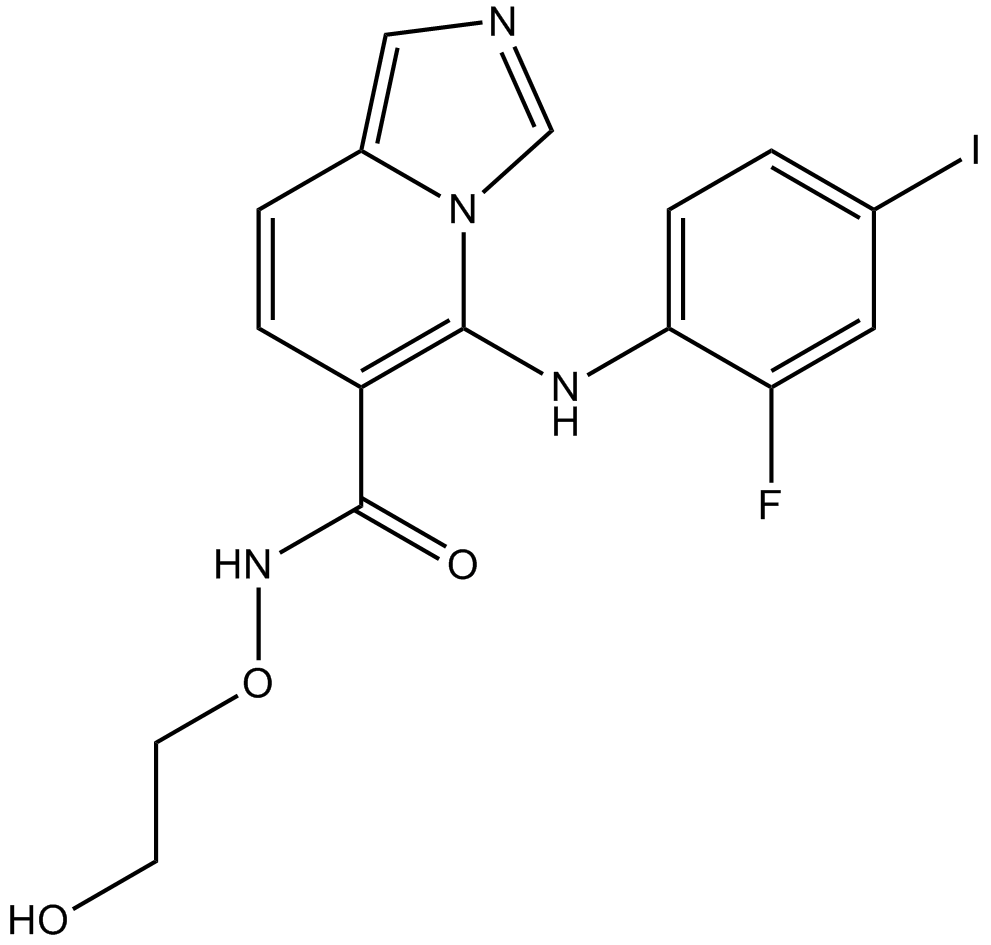
-
GC10505
GDC-0879
GDC-0879 es un inhibidor de B-Raf potente y selectivo con una IC50 de 0,13 nM.
-
GC12006
GDC-0994
GDC-0994 (GDC-0994) es un inhibidor de la cinasa ERK activo por vÍa oral con una IC50 de 6,1 nM y 3,1 nM para ERK1 y ERK2, respectivamente.
-
GN10307
Ginsenoside Re

-
GC17255
Gliotoxin
An immunosuppressive mycotoxin with diverse biological effects
-
GC33123
GNE 220
GNE-220 es un inhibidor potente y selectivo de MAP4K4 con un IC50 de 7 nM.
-
GC34208
GNE 220 Hydrochloride
GNE 220 (clorhidrato) es un inhibidor potente y selectivo de MAP4K4, con un IC50 de 7 nM.
-
GC19173
GNE-3511
GNE-3511 es un inhibidor de la cremallera quinasa de leucina dual (DLK) biodisponible y penetrante en el cerebro activo por vÍa oral con una Ki de 0,5 nM.
-
GC18257
GNE-495
GNE-495 es un inhibidor de MAP4K4 potente y selectivo con una IC50 de 3,7 nM.