Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
أهداف لـ نبسب؛ Cell Cycle/Checkpoint
- ATM/ATR(26)
- Aurora Kinase(47)
- Cdc42(4)
- Cdc7(4)
- Chk(16)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(91)
- E1 enzyme(1)
- G-quadruplex(14)
- Haspin(7)
- HMTase(1)
- Kinesin(26)
- Ksp(6)
- Microtubule/Tubulin(243)
- Mps1(15)
- Mitotic(11)
- RAD51(18)
- ROCK(71)
- Rho(13)
- PERK(11)
- PLK(37)
- PTEN(8)
- Wee1(7)
- PAK(21)
- Arp2/3 Complex(8)
- Dynamin(12)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
منتجات لـ نبسب؛ Cell Cycle/Checkpoint
- القط. رقم اسم المنتج بيانات
-
GC43546
Dnp-PLALWAR (trifluoroacetate salt)
Dnp-PLALWAR is a fluorogenic substrate for matrix metalloproteinase-1 (MMP-1) and MMP-8.
-
GC43547
Dnp-PLAYWAR (trifluoroacetate salt)
Dnp-PLAYWAR is a fluorogenic substrate for matrix metalloproteinase-8 (MMP-8) and MMP-26.
-
GC43550
Dnp-PLGMWSR (trifluoroacetate salt)
Dnp-PLGMWSR is a fluorogenic substrate for matrix metalloproteinase-2 (MMP-2) and MMP-9.
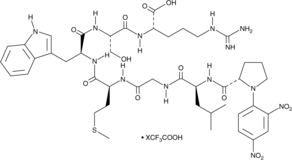
-
GC43552
Dnp-RPLALWRS (trifluoroacetate salt)
Dnp-RPLALWRS is a fluorogenic substrate for matrix metalloproteinase-7 (MMP-7).
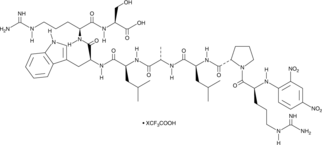
-
GC16684
Docetaxel
دوسيتاكسيل هو دواء مضاد للأورام يعمل عن طريق تثبيط تحلل الأنابيب الدقيقة والتخفيف من آثار تعبير جينات bcl-2 و bcl-xL.
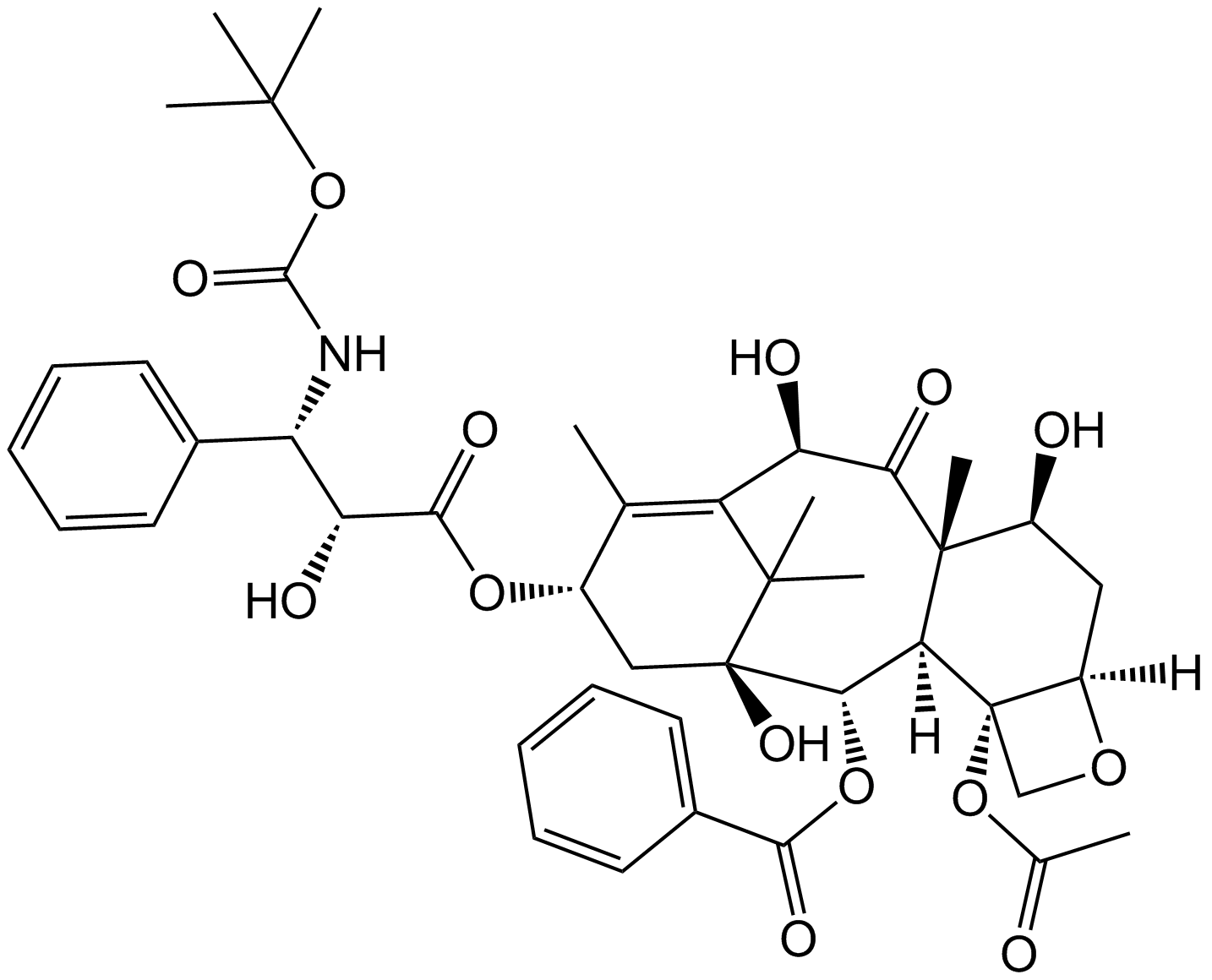
-
GC48968
Docetaxel (hydrate)
An analog of taxol with antitumor properties
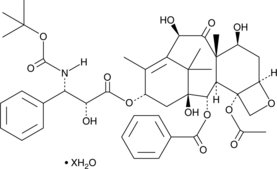
-
GC12550
Docetaxel Trihydrate
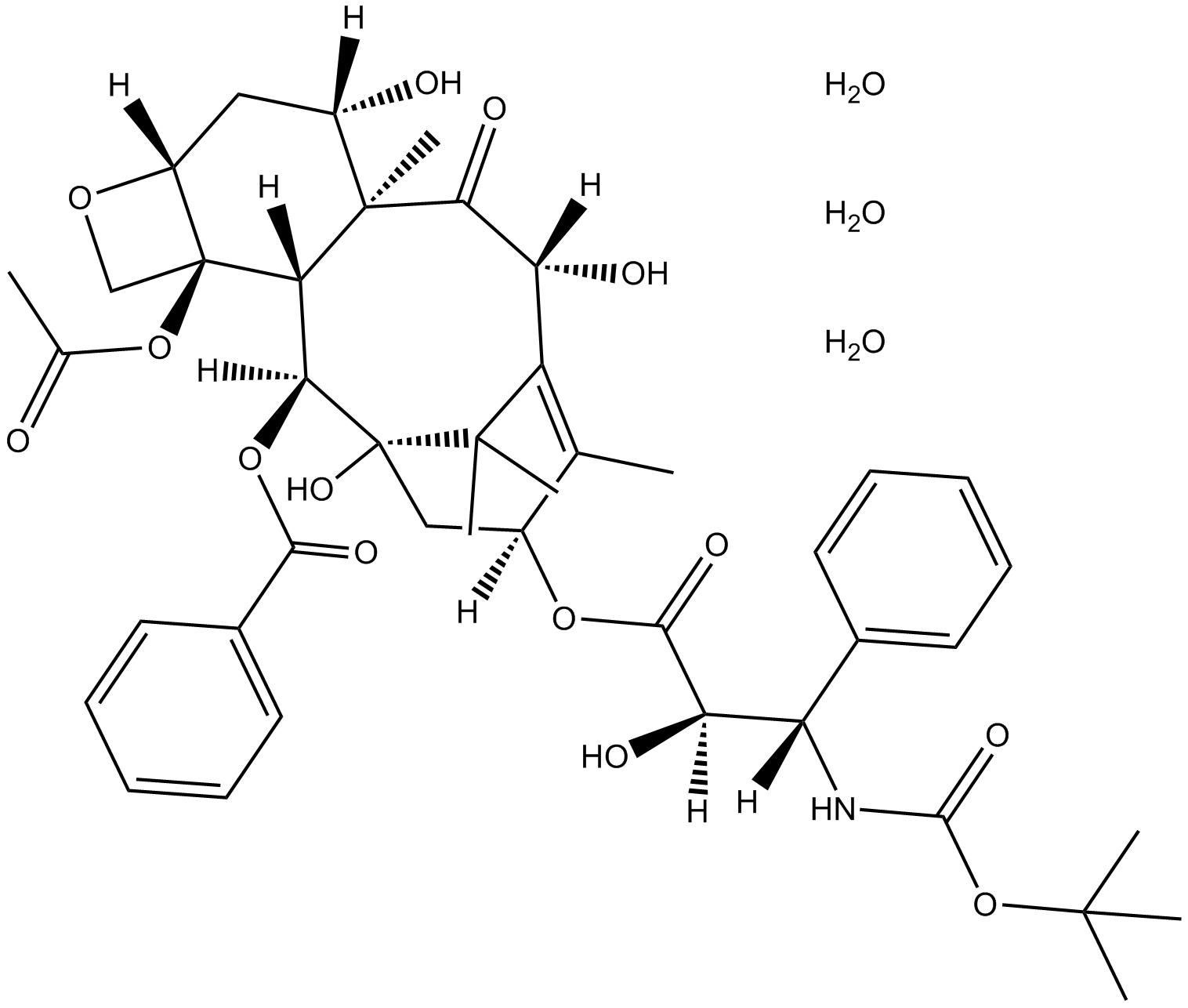
-
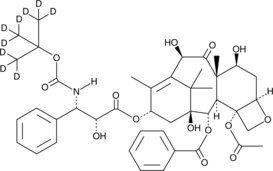
GC47250
Docetaxel-d9
Docetaxel-d9 (RP-56769-d9) هو الديوتيريوم المسمى Docetaxel. Docetaxel (RP-56769) هو مثبط لإزالة البلمرة في الأنابيب الدقيقة ، مع IC 50 من 0.2 ميكرومتر. يخفف Docetaxel من تأثيرات التعبير الجيني bcl-2 و bcl-xL. يوقف Docetaxel دورة الخلية عند G2 / M ويؤدي إلى موت الخلايا المبرمج. Docetaxel له نشاط مضاد للسرطان.
-
GC52095
Dodecyltrimethylammonium (bromide)
Dodecyltrimethylammonium (بروميد) (DTAB) هو خافض للتوتر السطحي.

-
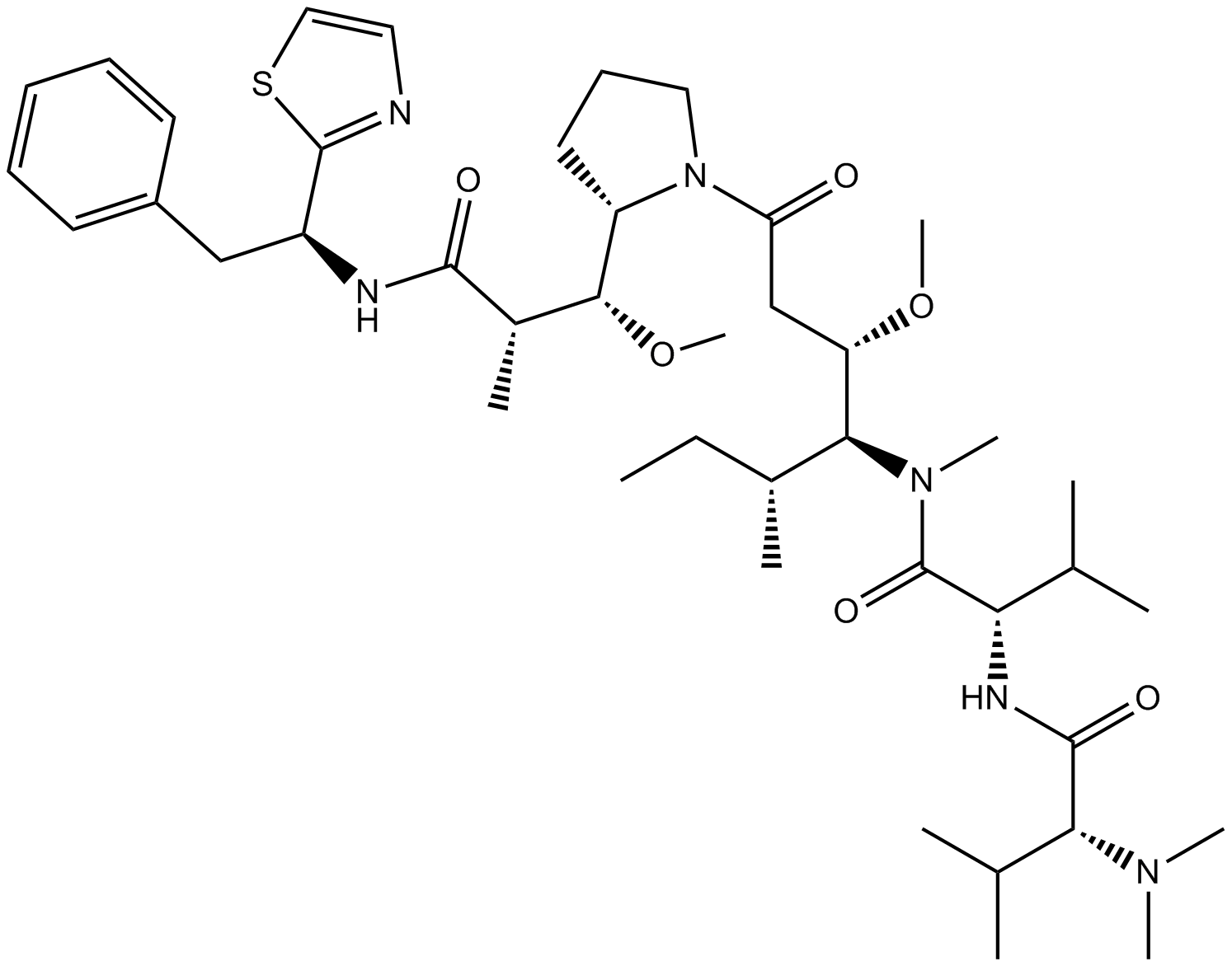
GC16273
Dolastatin 10
Dolastatin 10 (DLS 10) هو ببتيد قوي مضاد للمضادات يثبط بلمرة التوبولين
-
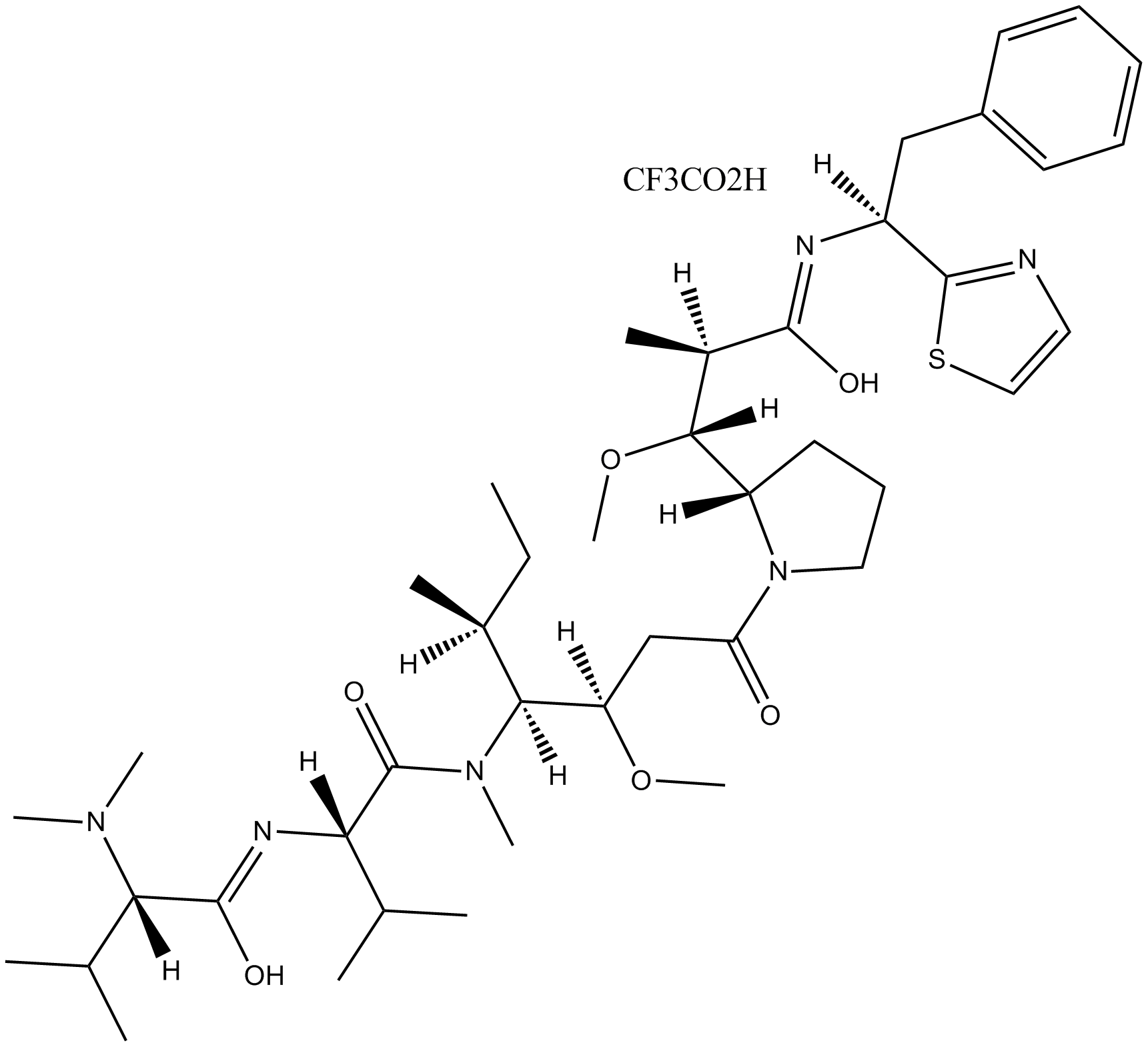
GC10557
Dolastatin 10 trifluoroacetate
Antitumor agent
-
GA21397
Dolastatin 15
Dolastatin 15 (DLS 15) ، ديسبيبتيد مشتق من Dolabella auricularia ، هو عامل مضاد قوي يرتبط بنيوياً بعامل مضاد التوبولين Dolastatin 10. Dolastatin 15 يحفز توقف دورة الخلية وموت الخلايا المبرمج في خلايا المايلوما المتعددةيمكن استخدام Dolastatin 15 باعتباره سمًا خلويًا لـ ADC

-
GC43567
DPC-AJ1951 (trifluoroacetate salt)
DPC-AJ1951 is a peptide agonist of the parathyroid hormone (PTH)/PTH-related peptide receptor (PPR; EC50 = 0.15 nM in HEK293 cells expressing human PPR).
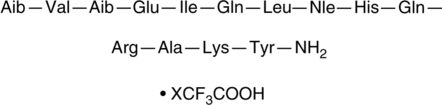
-
GC64927
Drp1-IN-1
Drp1-IN-1 (comp A-7) هو مثبط بروتين شبيه بالدينامين 1 (Drp1) ، مع IC50 من 0.91 ميكرومتر
-
GC50020
Dynamin inhibitory peptide, myristoylated
الببتيد المثبط لـ DynaMin ، myristoylated هو مثبط DynaMin للتدخل في ارتباط amphiphysin بالدينامين
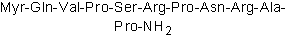
-
GC10395
Dynasore
ديناسور، كمثبط للـ GTPase، يمكنه بسرعة وبشكل قابل للاسترداد تثبيط نشاط الدينامين، مما يحول دون عملية الانتهاء من التغذية.
-
GC17208
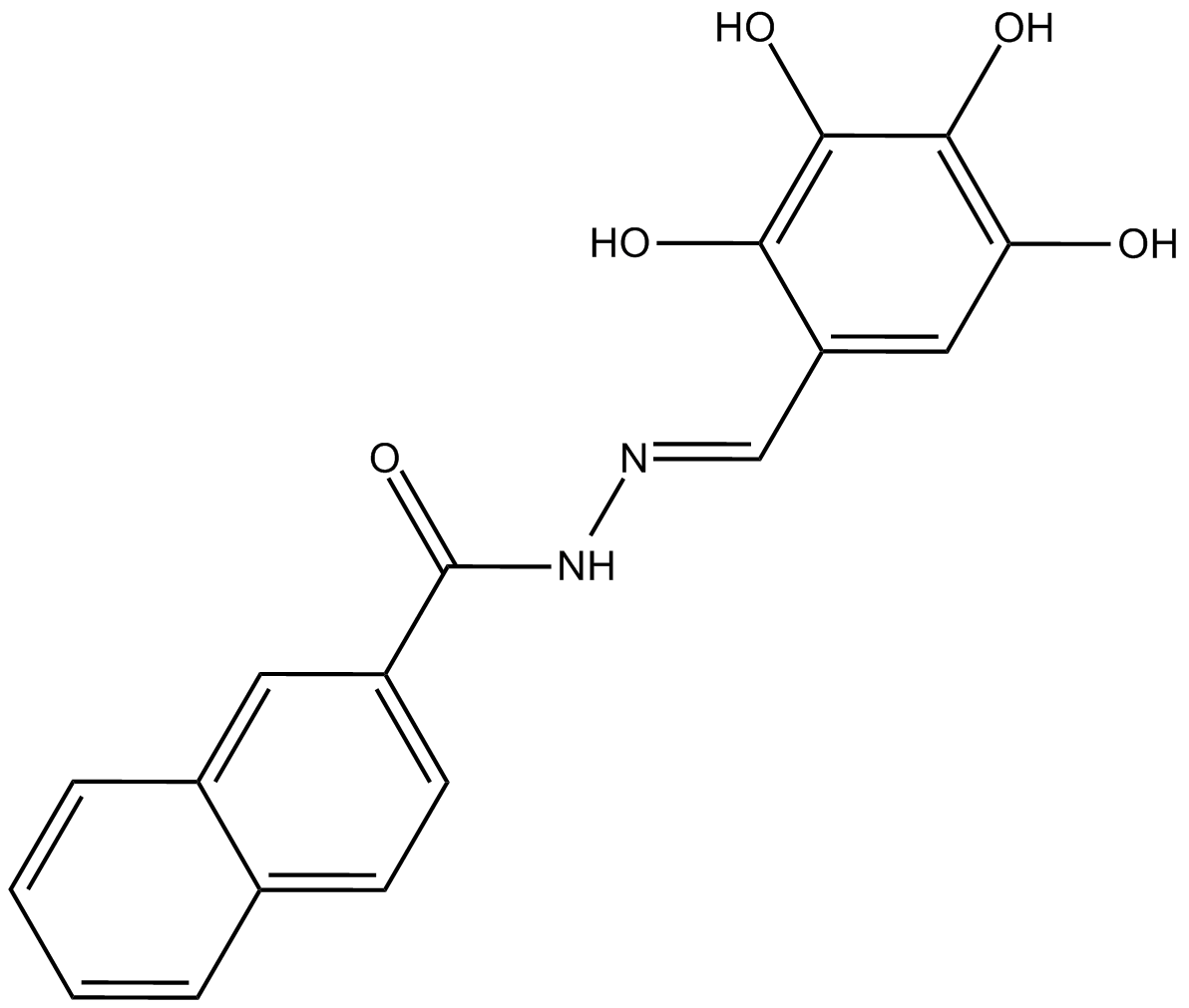
Dyngo-4a
Hydroxy Dynasore (Dyngo-4a) ، التناظرية الهيكلية لـ Dynasore ، هو محسّن للفاعلية ، وانخفاض السمية الخلوية ومثبط دينامين ملزم غير محدد مع قيم IC50 من 0.38 μ ؛ M و 2.3μ ؛ M لدينامين الدماغ I ودينامين الفئران المؤتلف الثاني ، على التوالي .
-
GC66056
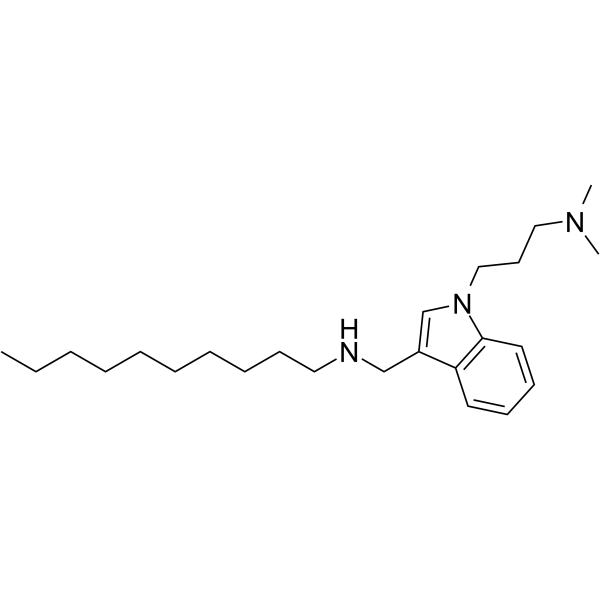
Dynole 2-24
Dynole 2-24 هو مثبط GTPase قائم على الإندول (IC50 = 0.56 μ ؛ M للدينامين I). Dynole 2-24 غير سام ويظهر فاعلية متزايدة ضد الدينامين الأول والثاني في المختبر وفي الخلايا (IC₅₀ (CME) = 1.9 μ ؛ M). يُظهر Dynole 2-24 أيضًا انتقائية بمقدار 4.4 ضعفًا للدينامين I. يعتبر Dynole 2-24 مثبطًا نشطًا داخل الخلية للالتقام الخلوي بوساطة الكلاذرين. CME: الالتقام الخلوي Clathrin بوساطة
-
GC47279
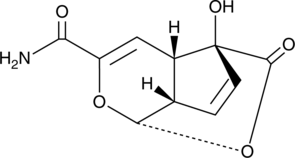
Echinosporin
A bacterial metabolite with antibacterial and anticancer activities
-
GC35964
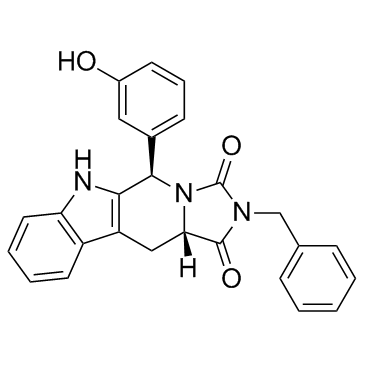
Eg5 Inhibitor V, trans-24
مثبط Eg5 V ، trans-24 هو مثبط قوي ومحدد لـ kinesin Eg5 مع IC50 من 0.65 ميكرومتر ، ويمكن استخدامه في أبحاث السرطان
-
GC10681
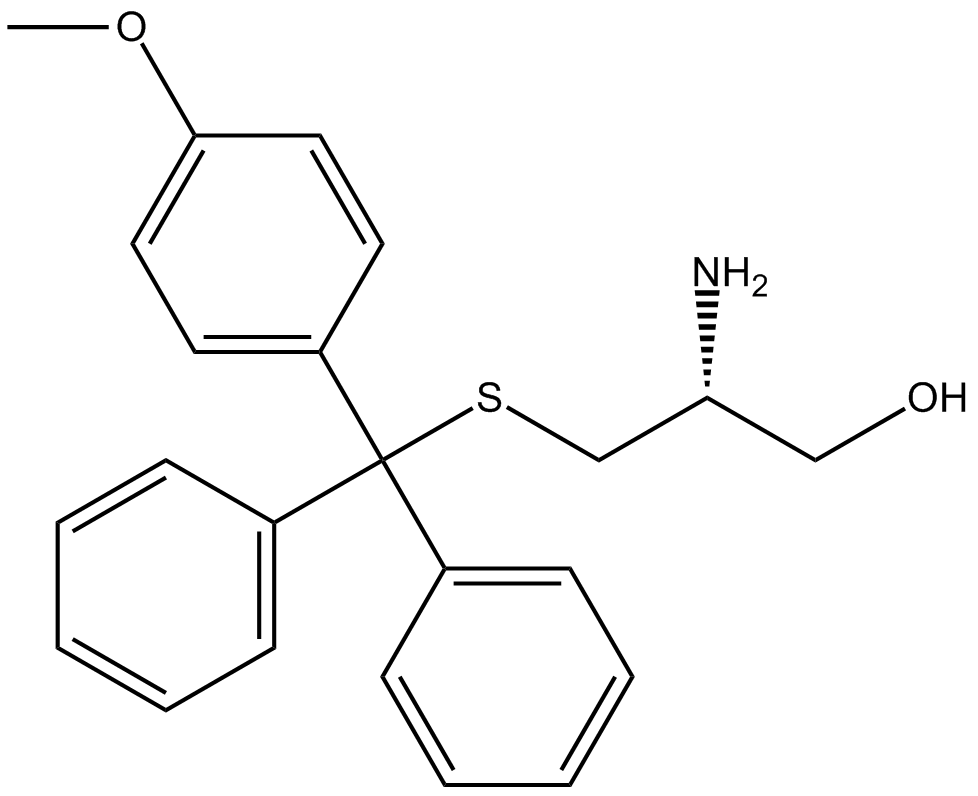
Eg5-I
potent inhibitor of Eg5
-
GC43588
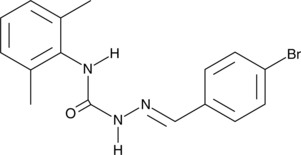
EGA
EGA هو مثبط انتقائي لمسارات تهريب الإندوسومول التي يتم استغلالها بواسطة العديد من السموم والفيروسات
-
GC10030
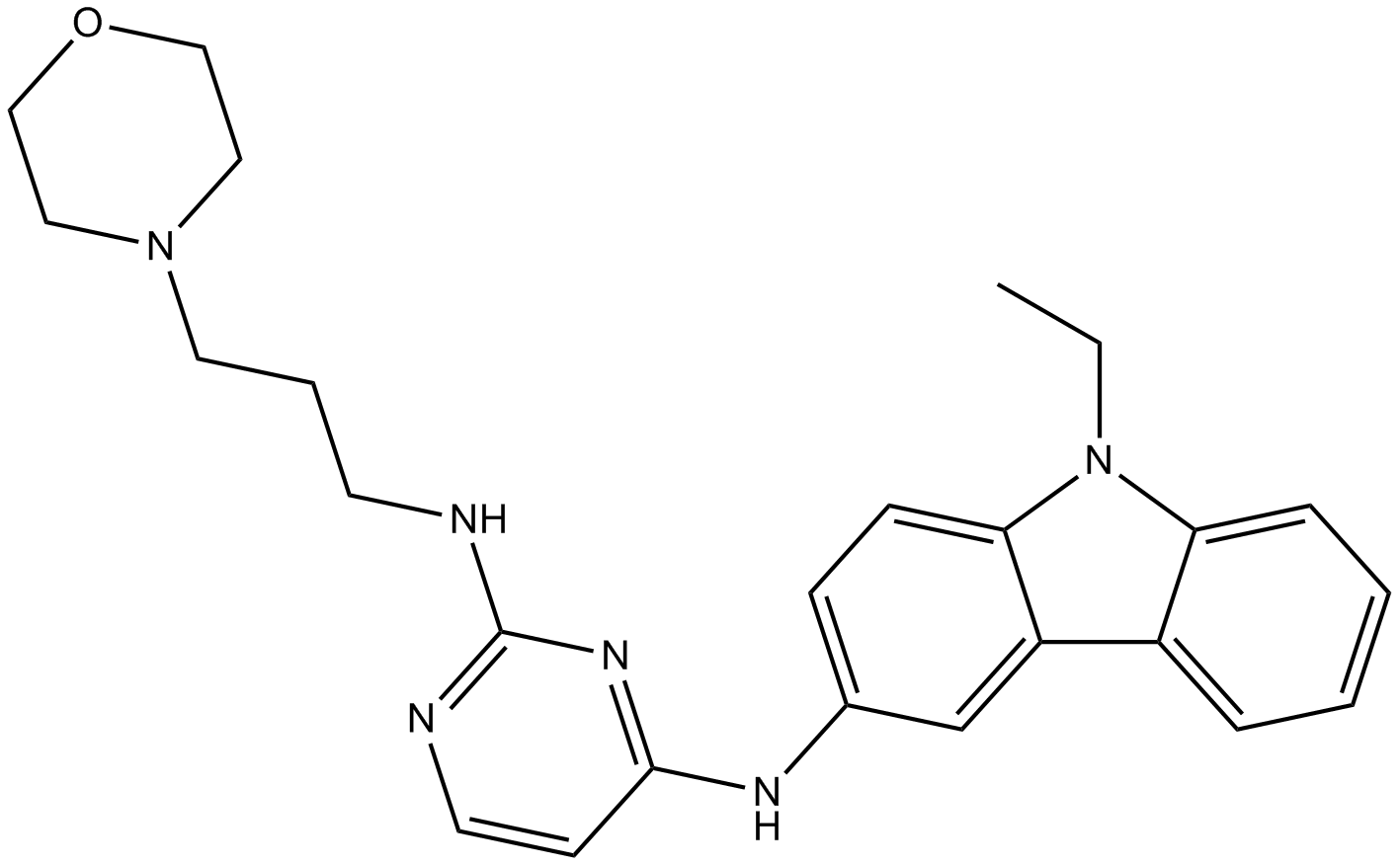
EHop-016
مثبط Rac1 / Rac3 GTPase ، فعال ومحدد بشكل كبير
-
GC25372
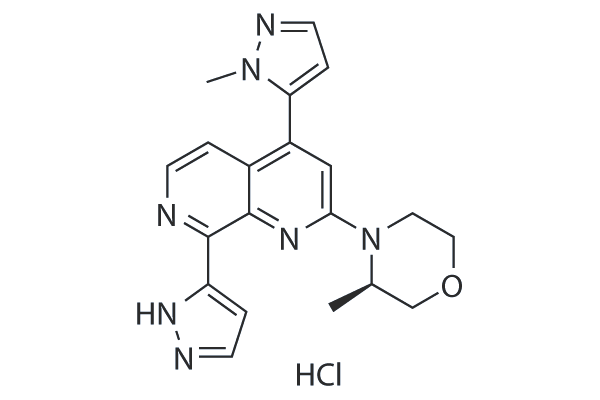
Elimusertib (BAY-1895344) hydrochloride
Elimusertib (BAY-1895344) hydrochloride is a potent, highly selective and orally available ATR inhibitor with an IC50 of 7 nM.
-
GC16827
ELR510444
ELR510444 هو معطل جديد للأنابيب الدقيقة ؛ يمنع تكاثر الخلايا MDA-MB-231 مع IC50 من 30.9 نانومتر ؛ ليس ركيزة لناقل عقاقير P-glycoprotein ويحتفظ بالنشاط في خطوط الخلايا IIII-tubulin-overexpressing
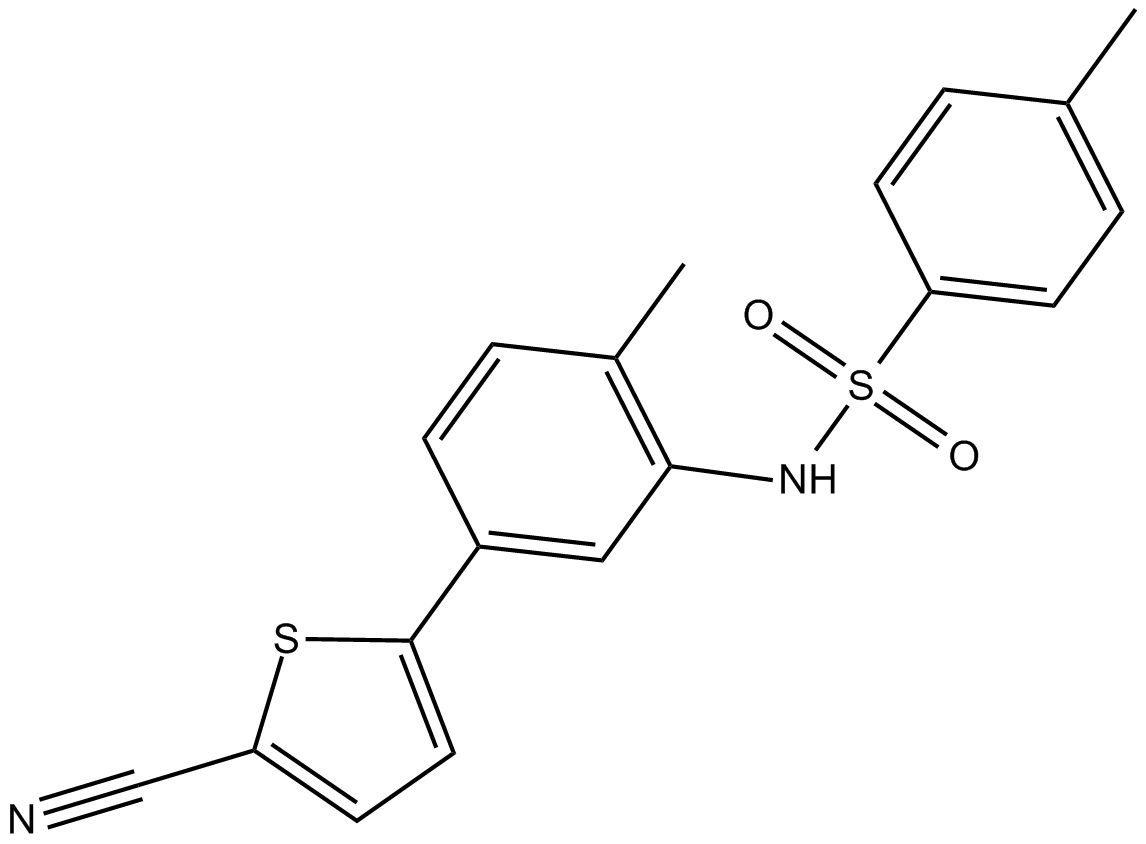
-
GC19466
Eltanexor (KPT-8602)
A second-generation exportin-1 inhibitor
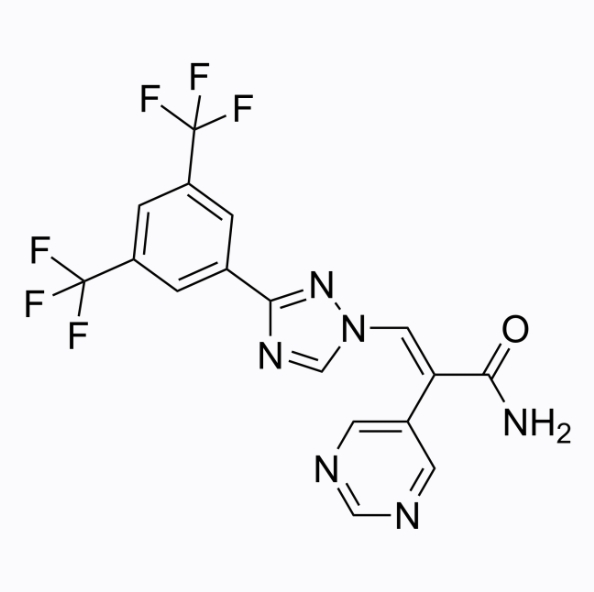
-
GC19213
Eltanexor Z-isomer
Eltanexor Z-isomer (KPT-8602 Z-isomer) هو الأيزومر الأقل نشاطًا لـ KPT-8602
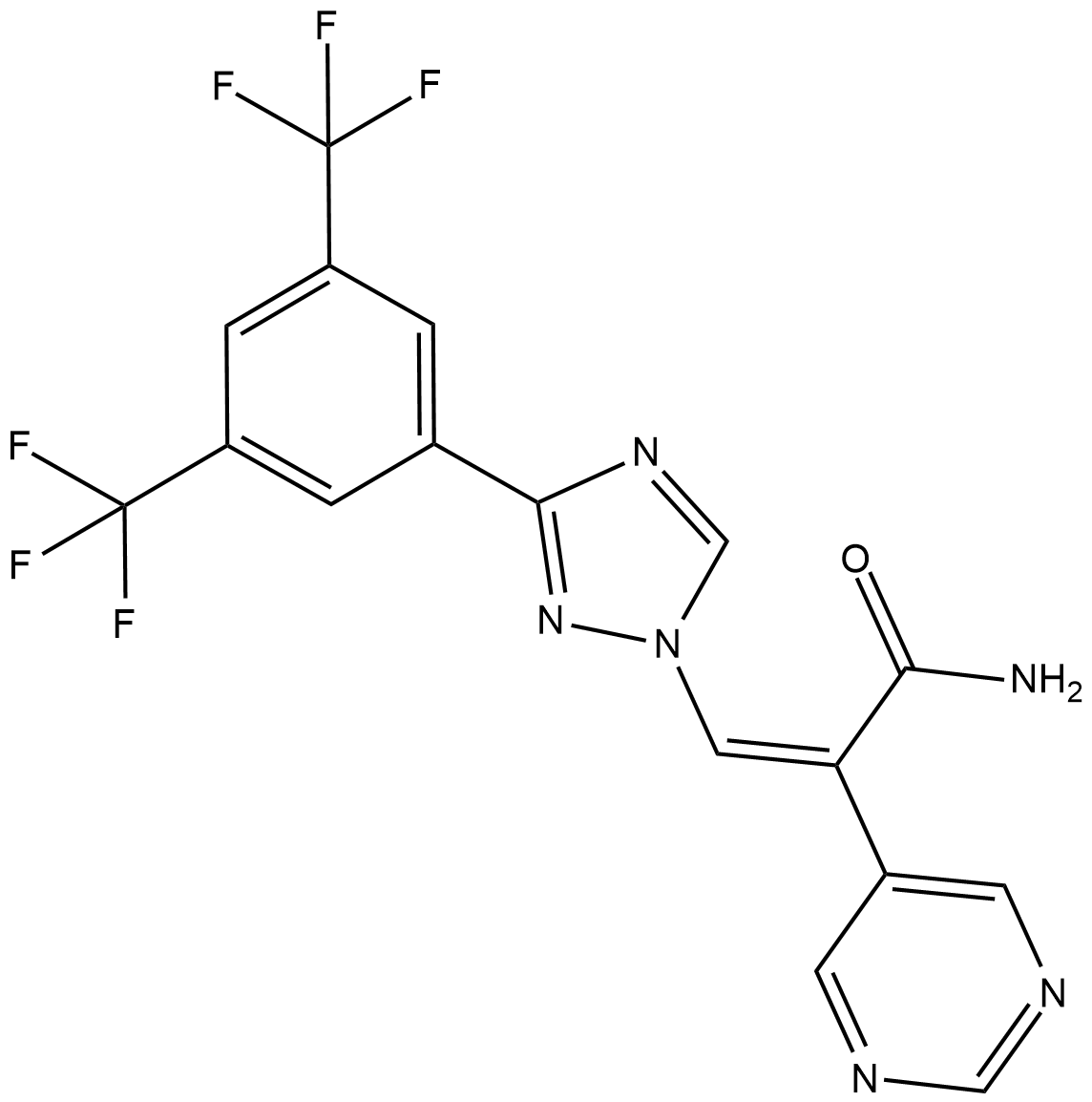
-
GC18165
EMD534085
EMD534085 هو مثبط قوي وانتقائي لـ kinesin-5 الانقسامي مع IC50 من 8 نانومتر
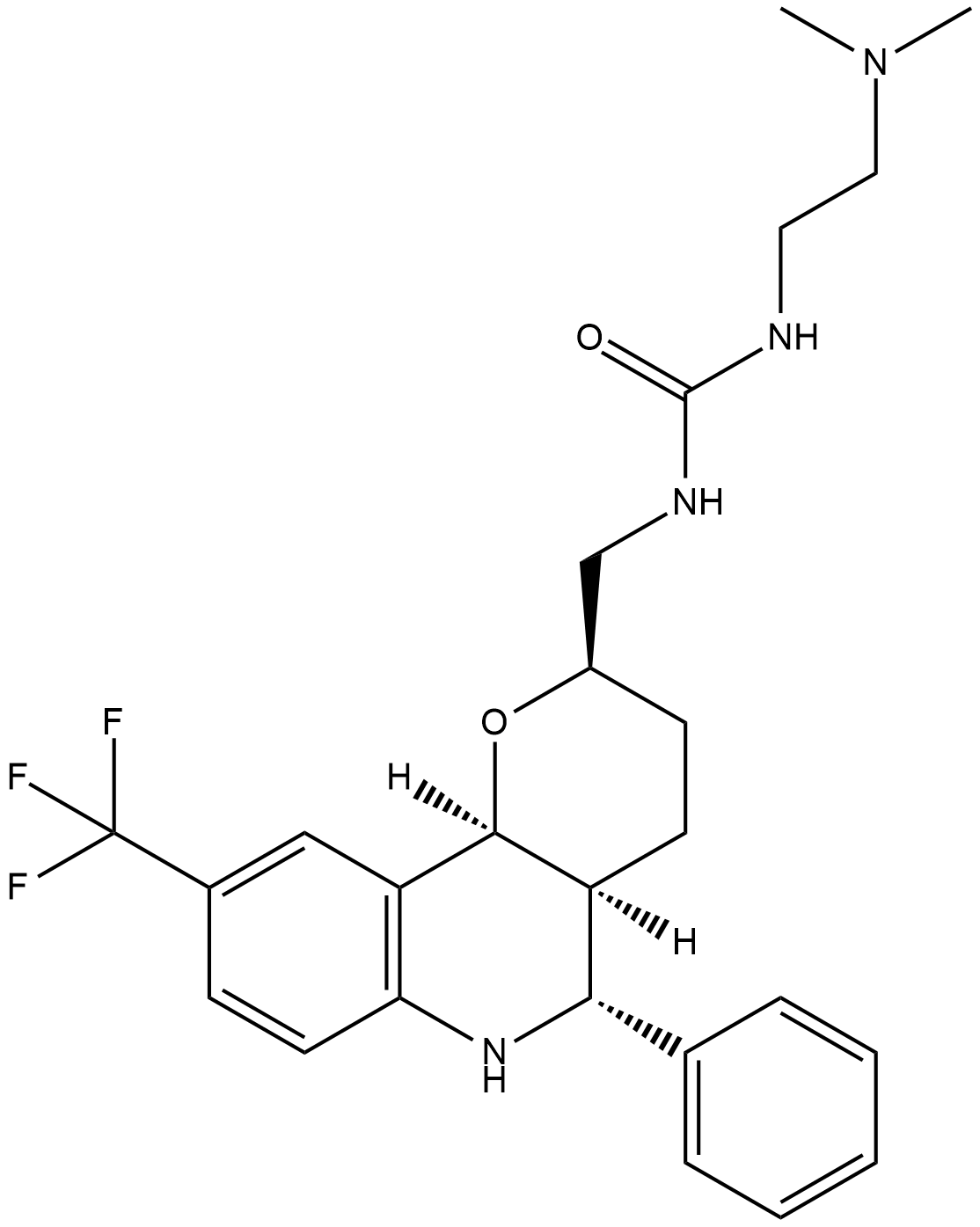
-
GC34073
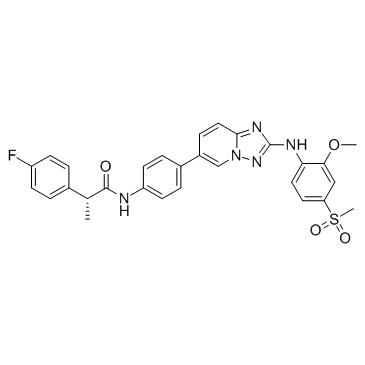
Empesertib (BAY 1161909)
Empesertib (BAY 1161909) (BAY 1161909) هو مثبط قوي لـ Mps1 ، مع IC50 أقل من 1 نانومتر.
-
GC49124
EN219
EN219 عبارة عن رابطة تساهمية تركيبية انتقائية بشكل معتدل ضد سيستين N-terminal (C8) لـ RNF114 مع IC50 من 470 نانومتر. EN219 يمنع التواجد الذاتي بوساطة RNF114 وانتشار p21.
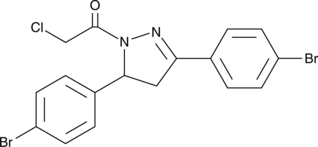
-
GC62287
EN4
EN4 عبارة عن رابطة تساهمية تستهدف السيستين 171 (C171) من MYCEN4 انتقائي لـ c-MYC على N-MYC و L-MYCيثبط EN4 نشاط النسخ MYC ، ويقلل من أهداف MYC ، ويضعف تكوين الأورام
-
GC19010
Endosidin 2
مثبط قابل للنفاذية الخلوية لعملية الإفراز وإعادة التدوير في الغشاء الخلوي.
-
GC43605
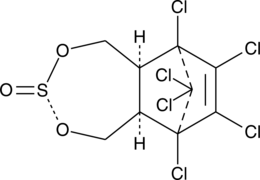
Endosulfan II
Endosulfan II is an organochlorine insecticide and a stereoisomer of endosulfan I.
-
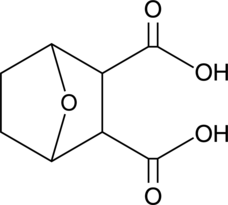
GC47291
Endothall
An herbicide
-
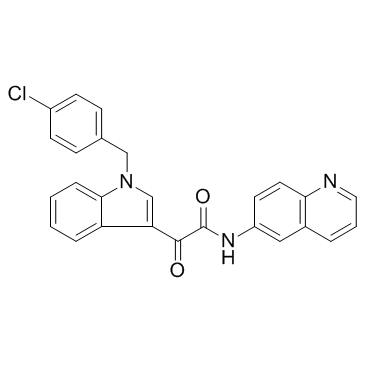
GC33125
Entasobulin
Entasobulin هو مثبط β-tubulin بلمرة مع نشاط محتمل مضاد للسرطان
-
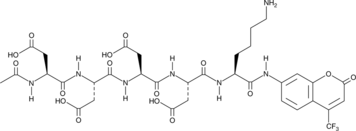
GC43614
Enteropeptidase Fluorogenic Substrate
Enteropeptidase fluorogenic substrate is a substrate for enteropeptidase that contains a 7-amino-4-trifluoromethylcoumarin (AFC) moiety.
-
GC48995
Enteropeptidase Fluorogenic Substrate (trifluoroacetate salt)
A fluorogenic substrate of enteropeptidase
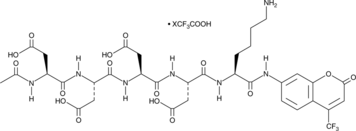
-
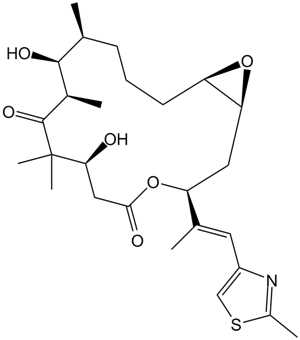
GC11202
Epothilone A
Epothilone A هو مثبط تنافسي لربط [3H] باكليتاكسيل ببوليمرات التوبولين ، مع كي من 0.6-1.4 ميكرومتر
-
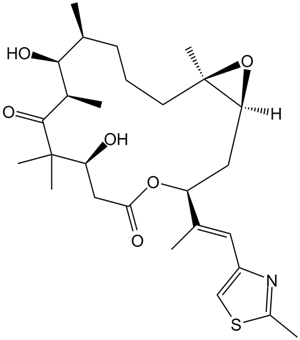
GC17240
Epothilone B (EPO906, Patupilone)
Epothilone B (EPO906 ، Patupilone) هو مثبت للأنابيب الدقيقة مع Ki 0.71μ ؛ M.
-
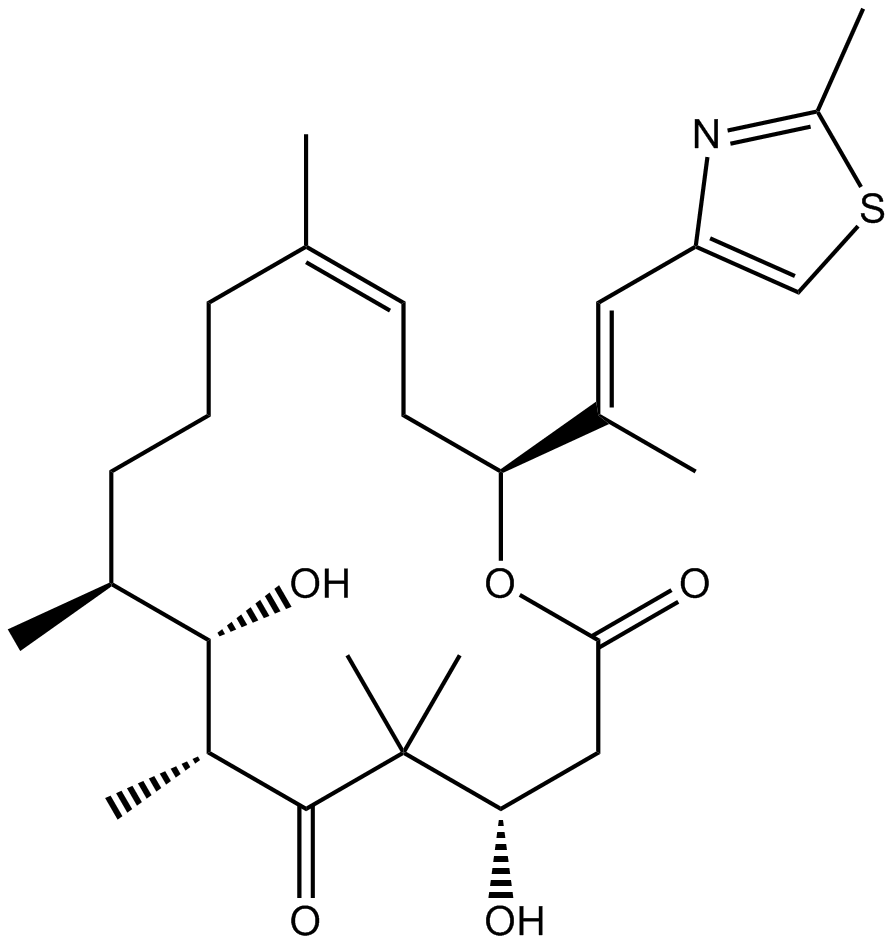
GC17440
Epothilone D
A microtubule-stabilizing agent
-
GC14354
Eribulin
Eribulin (E7389) هو عامل استهداف للأنابيب الدقيقة يستخدم في البحث عن سرطان الثدي النقيلييمنع Eribulin تكاثر الخلايا السرطانية عن طريق ربط بروتينات الأنابيب الدقيقة والأنابيب الدقيقة
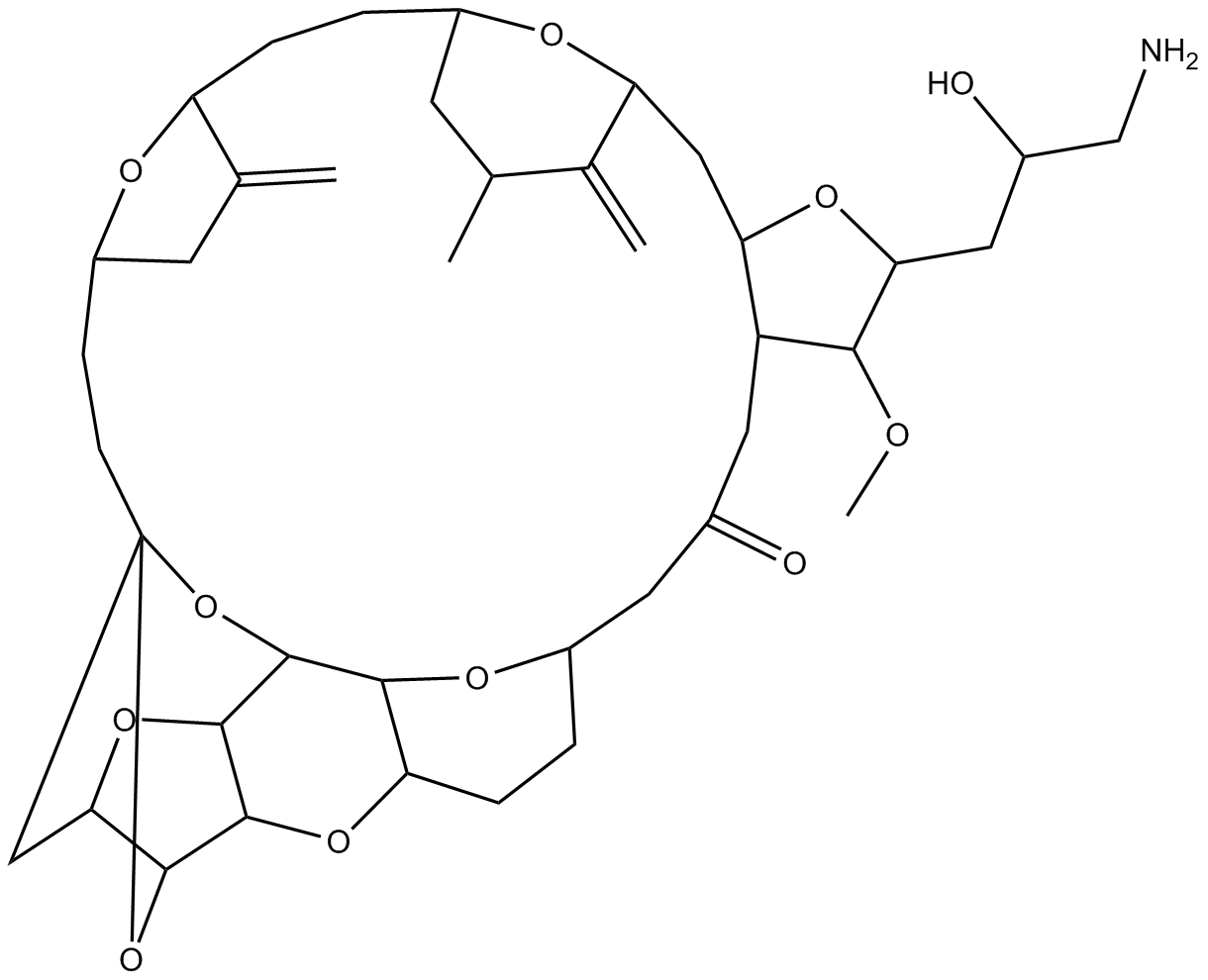
-
GC12483
Eribulin mesylate
مشتق اصطناعي للهاليكوندرين بي
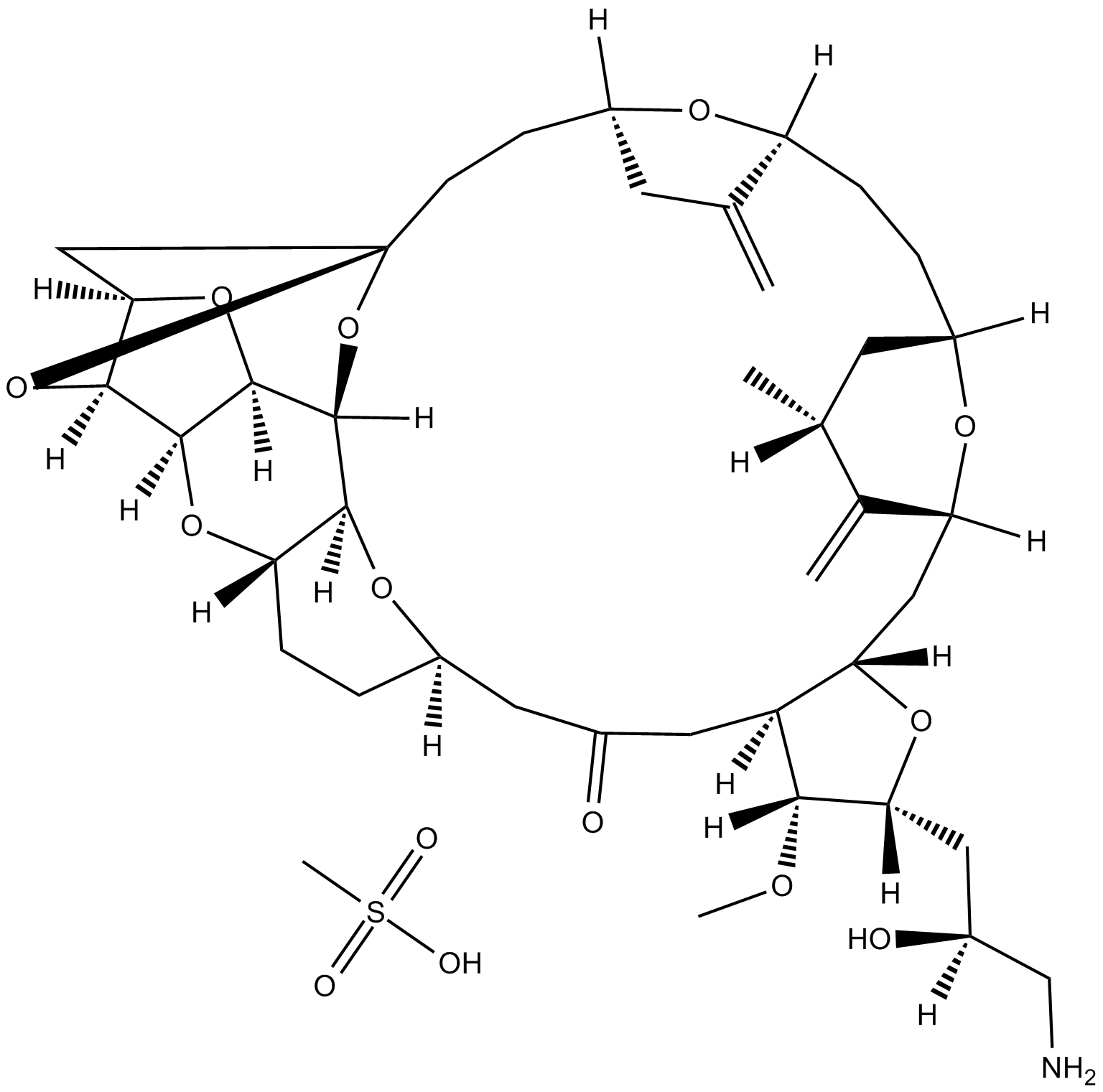
-
GC63845
Eribulin-d3 mesylate
Eribulin-d3 mesylate هو الديوتيريوم المسمى Eribulin mesylateEribulin mesylate هو عامل استهداف للأنابيب الدقيقة يستخدم في أبحاث السرطان
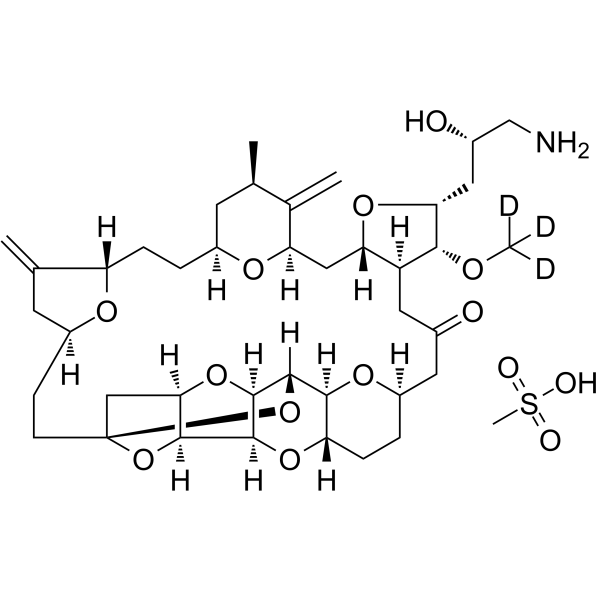
-
GC43625
Erucin
Erucin (ERU) هو isothiocyanate وفير بشكل خاص في الجرجير
-
GC67333
Estramustine
Estramustine هو عامل مضاد للأورام. يزيل Estramustine بلمرة microtnbules عن طريق الارتباط بـ tubulin 1 ، ويعرض نشاطًا مضادًا للمضادات بقيمة IC50 تبلغ حوالي 16 μ ؛ M للانقسام من خلايا DU 145. يمنع Estramustine الخلايا عند الانقسام في طعوم أورام البروستاتا.
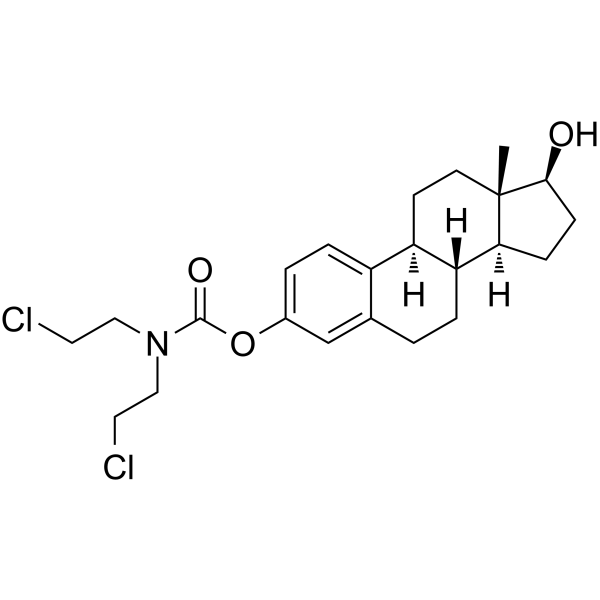
-
GC32816
Estramustine phosphate sodium
إستراموستين فوسفات الصوديوم ، نظير استراديول ، هو عامل علاج كيميائي فعال عن طريق الفم
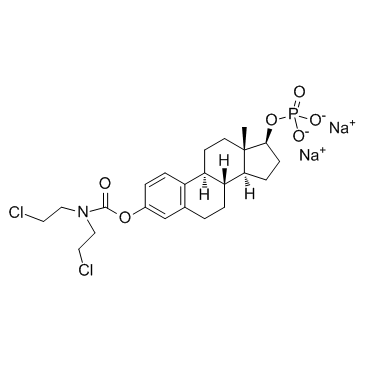
-
GC52061
Ethylene Glycol monododecyl ether
A nonionic surfactant

-
GC10196
ETP-46464
ETP-46464 هو مثبط فعال لـ mTOR و ATR مع IC50s 0.6 و 14 نانومتر ، على التوالي
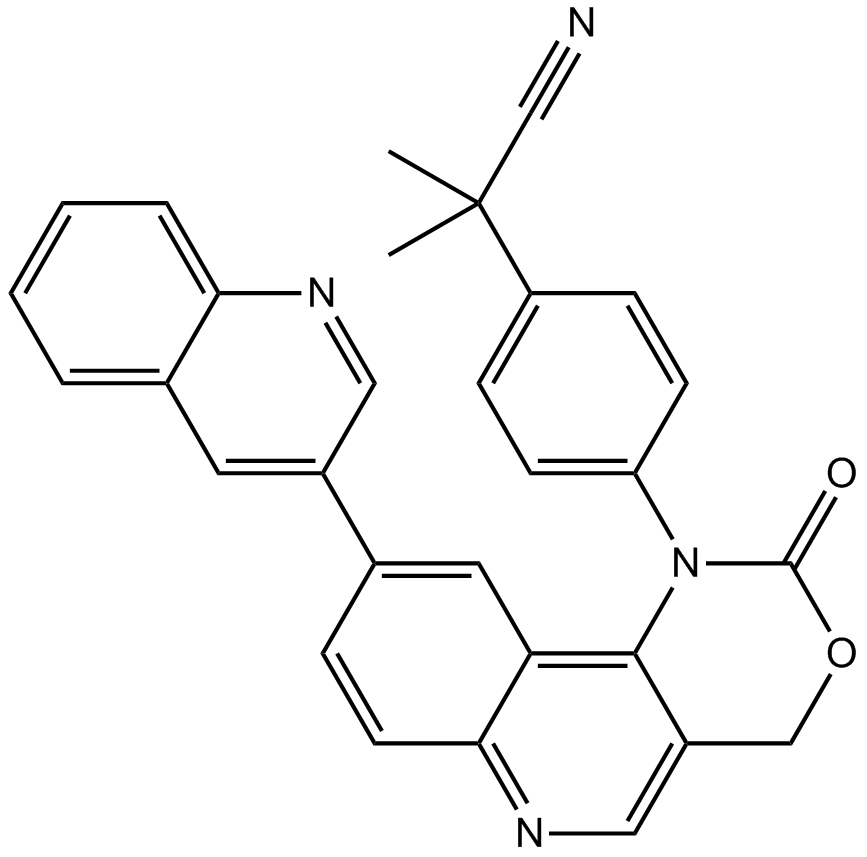
-
GC18693
Fascaplysin (chloride)
Fascaplysin (كلوريد) هو صبغة حمراء مضادة للميكروبات وسامة للخلايا ، يمكن أن تأتي من الإسفنج البحري (Fascaplysin (chloride) opsis sp.

-
GC13869
Fasudil
Fasudil (HA-1077 ؛ AT877) هو مثبط RhoA / ROCK غير محدد وله أيضًا تأثير مثبط على كينازات البروتين ، مع Ki 0.33 ميكرومتر لـ ROCK1 و IC50s من 0.158 ميكرومتر و 4.58 ميكرومتر و 12.30 ميكرومتر و 1.650 ميكرومتر لـ ROCK2 و PKA ، PKC ، PKG ، على التواليFasudil هو أيضًا مضاد قوي للقنوات Ca2 + وموسع للأوعية

-
GC14289
Fasudil (HA-1077) HCl
Fasudil (HA-1077؛ AT877) Hydrochloride هو مثبط RhoA / ROCK غير محدد وله أيضًا تأثير مثبط على كينازات البروتين ، مع Ki 0.33 μ ؛ M لـ ROCK1 ، IC50s من 0.158 μ ؛ M و 4.58 &88 ، 128888956 ؛ ؛ M ، 1.650 μ ؛ M لـ ROCK2 و PKA ، PKC ، PKG ، على التوالي. Fasudil (HA-1077) حمض الهيدروكلوريك هو أيضًا مضاد فعال لقناة Ca2+ وموسع للأوعية.
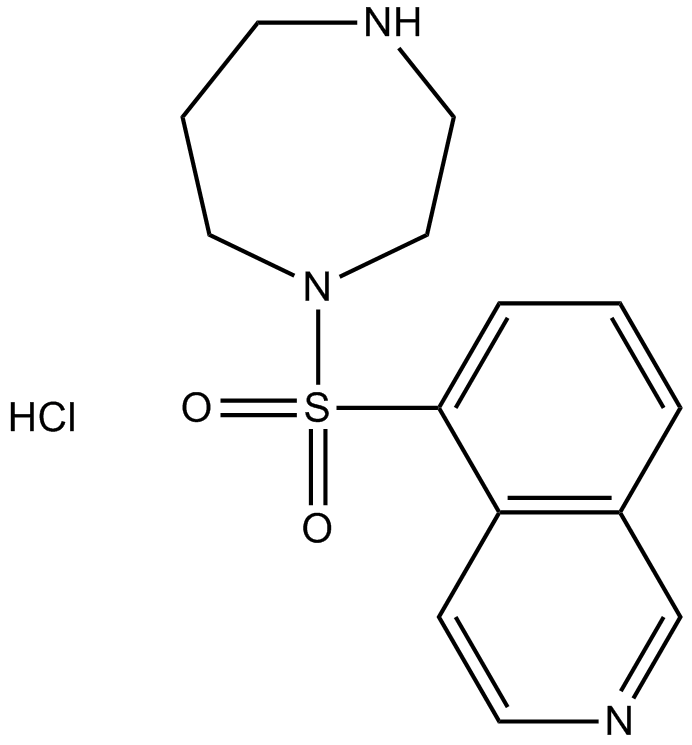
-
GC12319
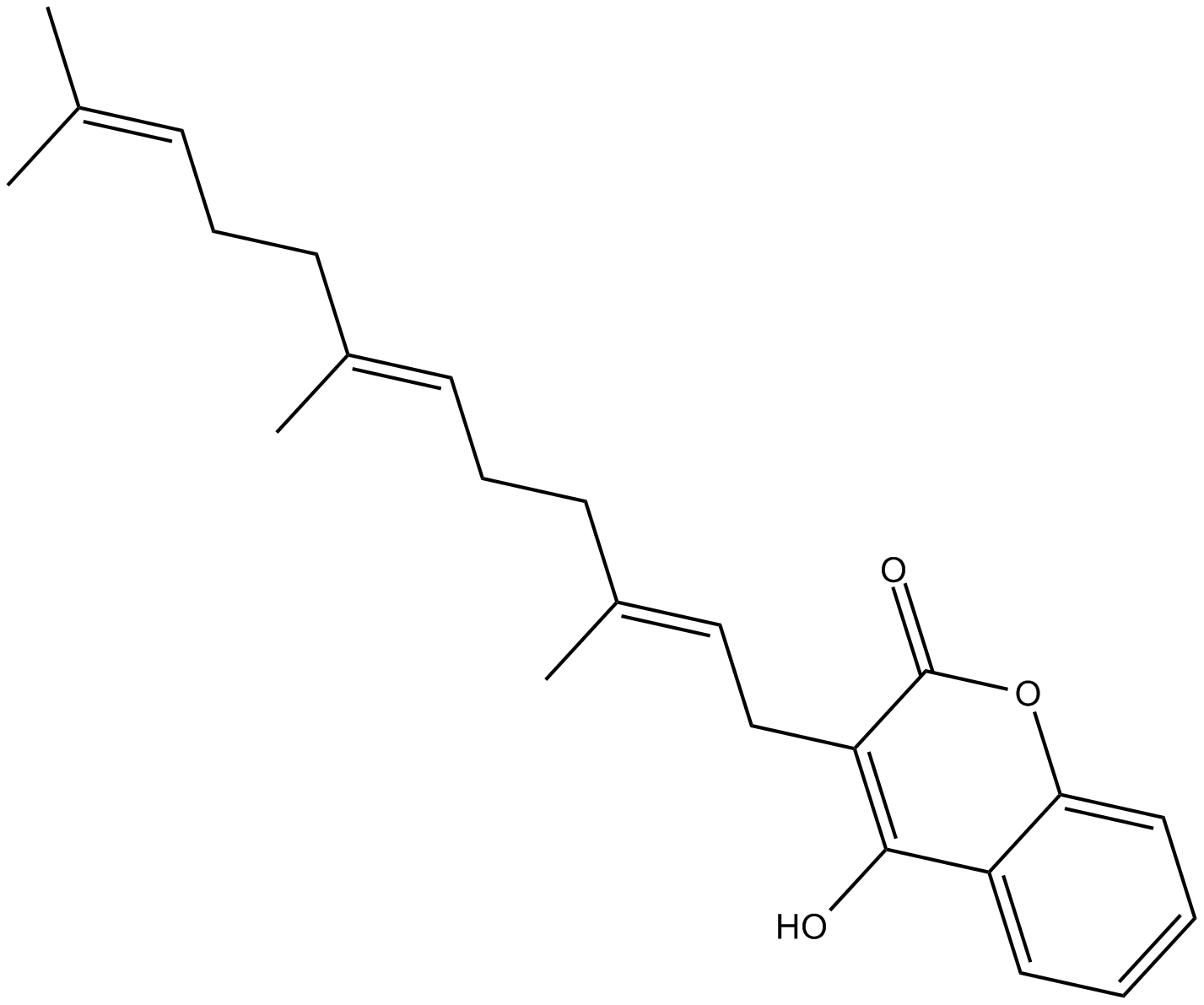
Ferulenol
Ferulenol ، مشتق sesquiterpene prenylated coumarin ، يثبط على وجه التحديد اختزال السكسينات يوبيكوينون عند مستوى دورة يوبيكوينون
-
GC43666
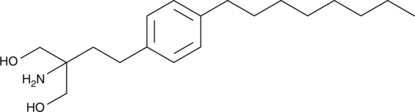
Fingolimod
Fingolimod (FTY720 free base) هو مضاد سفينجوزين 1-فوسفات (S1P) مع IC 50 من 0.033 نانومتر في خلايا K562 و NK
-
GC14807
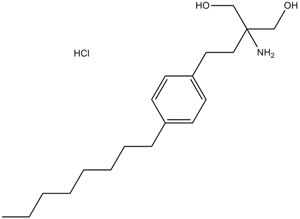
Fingolimod(FTY720)
فينغوليمود (FTY720) HCl (FTY720)، وهو مشتق من السفينجوزين، هو عامل تعديل لمستقبلات سفينجوزين 1- فسفات (S1P) قوية.
-
GC49344
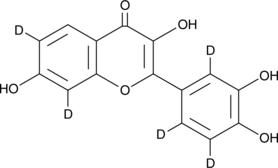
Fisetin-d5
An internal standard for the quantification of fisetin
-
GC49695
FKBP52 Monoclonal Antibody (Clone Hi52C)
For immunochemical analysis of FKBP52

-
GC43674
FlAsH-EDT2
FlAsH-EDT2 عبارة عن كاشف لوضع العلامات على البروتين
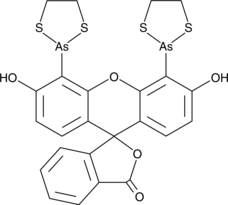
-
GC16063
Flavopiridol
An inhibitor of cyclin-dependent kinases
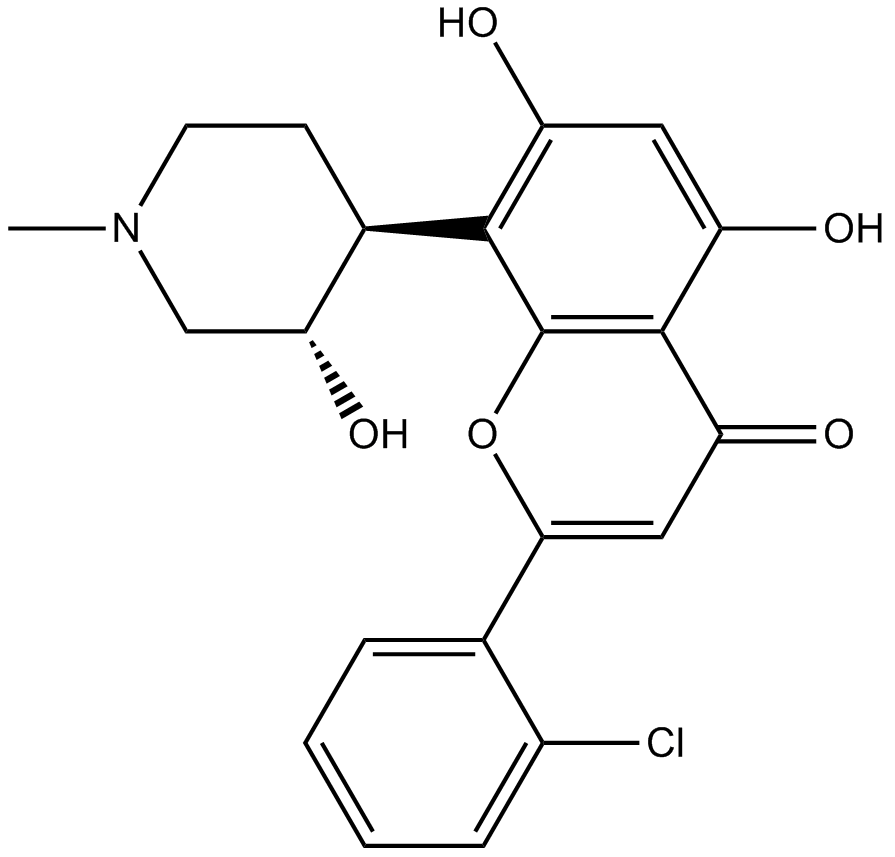
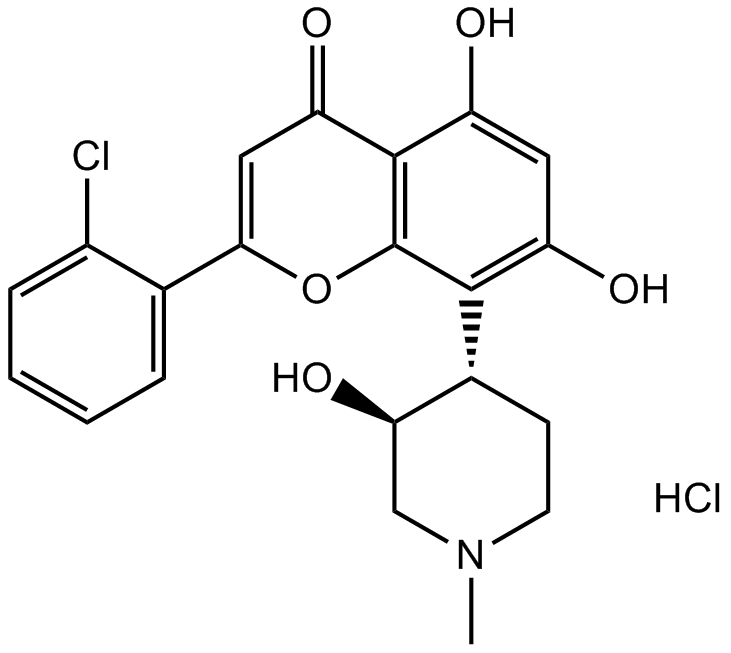
-
GC16425
Flavopiridol hydrochloride
Flavopiridol Hydrochloride (Alvocidib Hydrochloride) هو مثبط واسع لـ CDK ، يتنافس مع ATP لتثبيط CDK بما في ذلك CDK1 ، CDK2 ، CDK4 مع IC50s من 30 ، 170 ، 100 نانومتر ، على التوالي
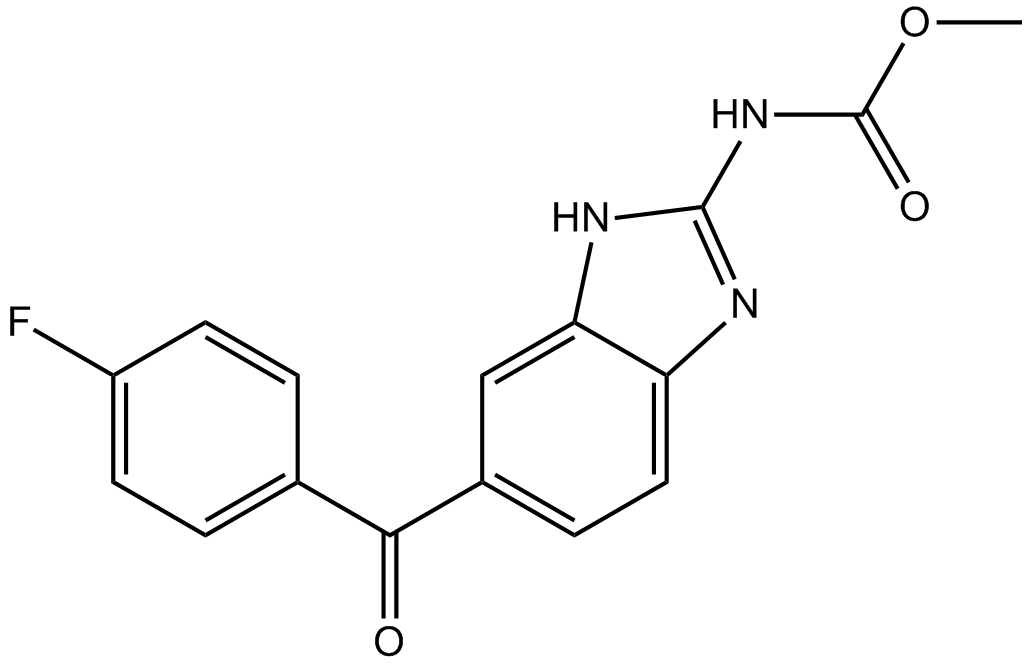
-
GC13210
Flubendazole
Flubendazole هو دواء مضاد للديدان آمن وفعال ، يستخدم على نطاق واسع للديدان للإنسان والقوارض والحيوانات المجترة
-
GC16727
Flutax 1
A fluorescent taxol derivative used for direct imaging of the microtubule cytoskeleton
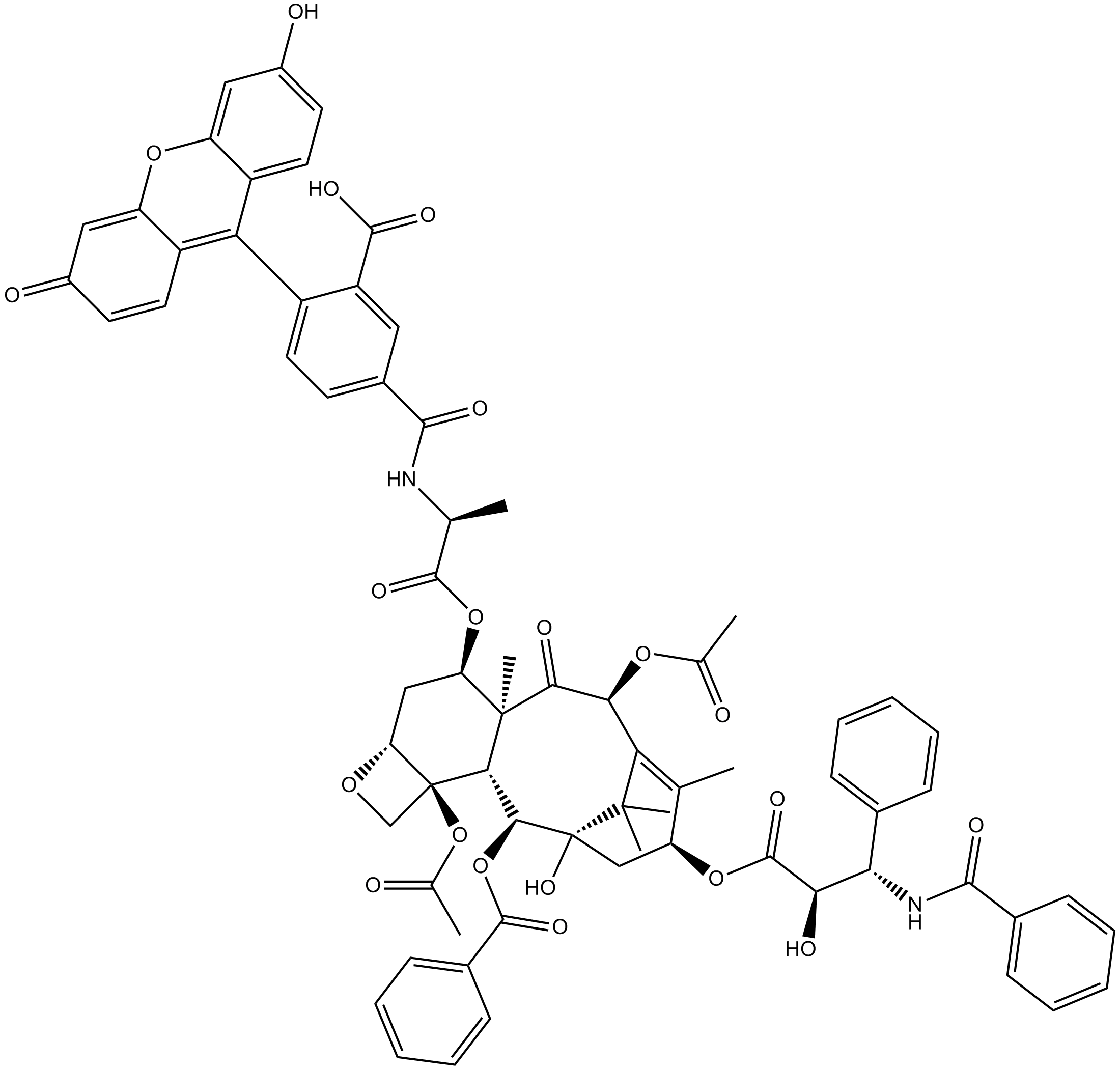
-
GC50397
Flutax 2
Green fluorescent taxol derivative; binds microtubules
-
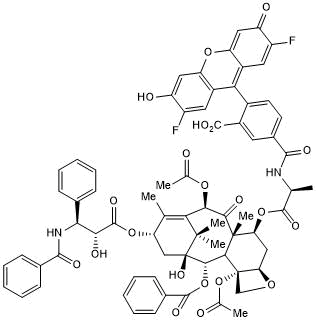
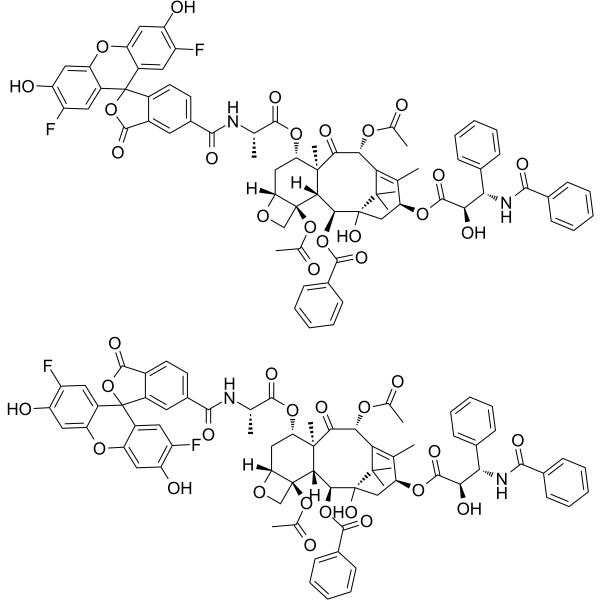
GC66242
Flutax-2 (5/6-mixture)
Flutax-2 (خليط 5/6) هو مشتق فلوري نشط من تاكسول. يرتبط Flutax-2 (خليط 5/6) بالبلمرة α ؛ β ؛ - ثنائيات التوبولين. Flutax-2 (خليط 5/6) قادر على تثبيت الأنابيب الدقيقة من T. gallinae و T. fetus trophozoites.
-
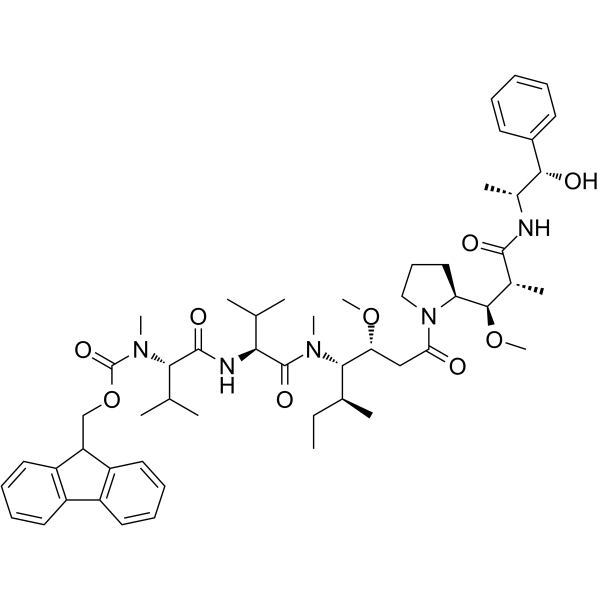
GC63596
Fmoc-MMAE
Fmoc-MMAE عبارة عن مركب وقائي أحادي ميثيل أوريستاتين E (MMAE) ، وهو مثبط قوي لتوبيولينيمكن استخدام Fmoc-MMAE في تركيب ADC
-
GC12308
Fosbretabulin (Combretastatin A4 Phosphate (CA4P)) Disodium
Fosbretabulin (Combretastatin A4 Phosphate (CA4P)) ثنائي الصوديوم (CA 4DP) هو عامل زعزعة استقرار توبيولين. Fosbretabulin (Combretastatin A4 Phosphate (CA4P)) ثنائي الصوديوم هو الدواء الأولي Combretastatin A4 الذي يستهدف بشكل انتقائي الخلايا البطانية ، ويحث على تراجع الأورام الحديثة للورم ، ويقلل من تدفق الدم إلى الورم ويسبب نخر الورم المركزي.
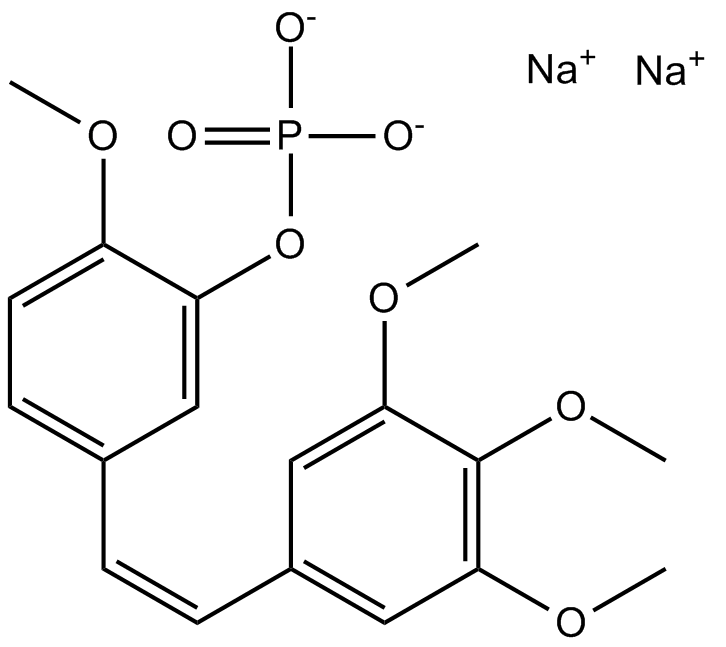
-
GC49089
FR900359
A cyclic depsipeptide and an inhibitor of Gαq, Gα11, and Gα14
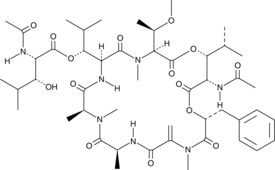
-
GC10251
FRAX1036
FRAX1036 هو مثبط PAK مع Kis 23.3 نانومتر و 72.4 نانومتر و 2.4 ميكرومتر لـ PAK1 و PAK2 و PAK4 ، على التوالي
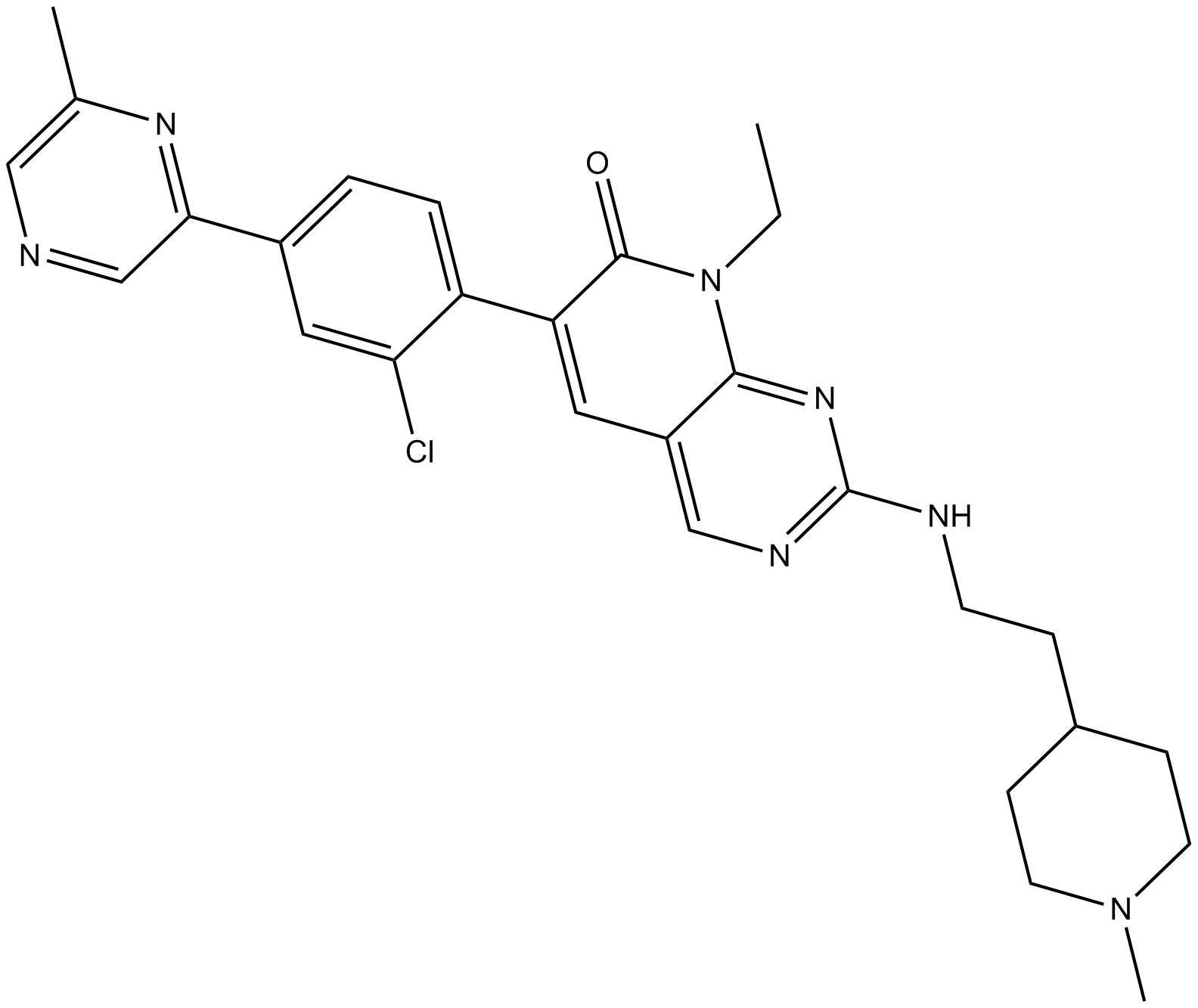
-
GC16075
FRAX486
FRAX486 هو مثبط كيناز (PAK) منشط p21 مع IC50s 14 و 33 و 39 نانومتر لـ PAK1 و PAK2 و PAK3 ، على التوالي
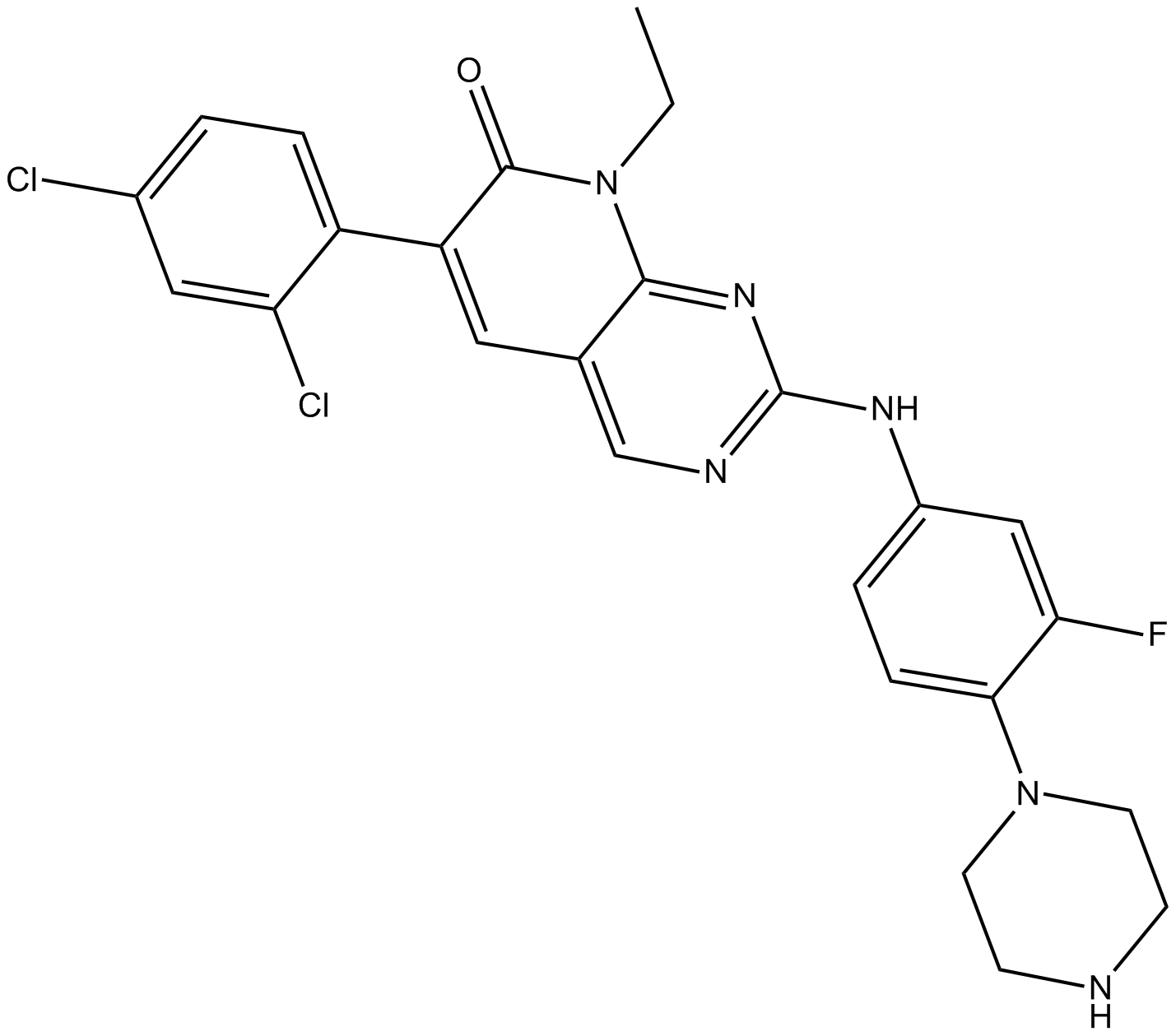
-
GC12261
FRAX597
FRAX597 عبارة عن مثبط قوي للكينازات المنشطة من المجموعة الأولى p21 (PAKs) مع IC50 من 8 و 13 و 19 نانومتر لـ PAK1 و 2 و 3
-
GC52288
Fumonisin B1-13C34
An internal standard for the quantification of fumonisin B1
-
GC47378
Fumonisin B2-13C34
An internal standard for the quantification of fumonisin B2
-
GC18720
G-5555
G-5555 هو مثبط قوي للكيناز 1 (PAK1) منشط p21 مع Kis 3.7 نانومتر و 11 نانومتر لـ PAK1 و PAK2 ، على التوالي
-
GC36095
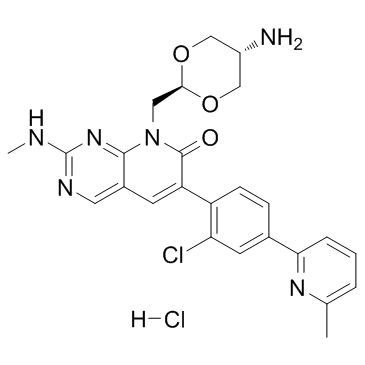
G-5555 hydrochloride
هيدروكلوريد G-5555 هو مثبط قوي وانتقائي للكيناز 1 (PAK1) منشط p21 مع Ki 3.7 نانومتر
-
GC40016
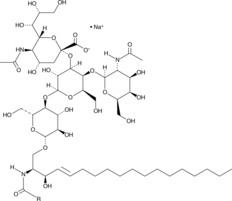
Ganglioside GM2 Mixture (sodium salt)
Ganglioside GM2 is a glycosphingolipid component of cellular membranes, primarily the plasma membrane.
-
GC43732
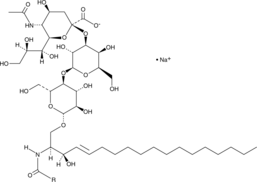
Ganglioside GM3 Mixture (sodium salt)
Ganglioside GM3 is a monosialoganglioside that demonstrates antiproliferative and proapoptotic effects in tumor cells by modulating cell adhesion, proliferation, and differentiation.
-
GC49322
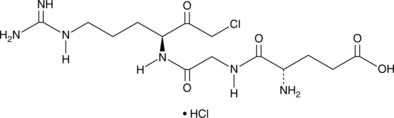
GGACK (hydrochloride)
A uPA and factor Xa inhibitor
-
GC38783
Ginkgolic acid C17:1
حمض الجنكوليك C17: 1 ، المستخرج من أوراق الجنكة بيلوبا ، يمنع تنشيط STAT3 التأسيسي والمحفز من خلال تحريض PTEN و SHP-1 التيروزين الفوسفاتيزحمض الجنكوليك C17: 1 له تأثيرات مضادة للسرطان
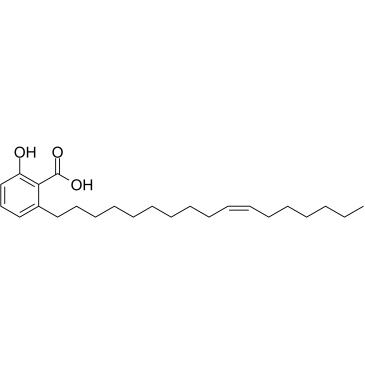
-
GC49437
Gliotoxin-13C13
An internal standard for the quantification of gliotoxin
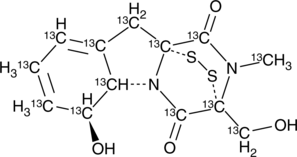
-
GC41563
Globosuxanthone A
Globosuxanthone A هو ثنائي هيدروكسانثينون ذو نشاط مضاد للفطريات واضح تجاه Fusarium graminearum و Fusarium solani و Botrytis cinerea بقيم MIC تبلغ 4 و 8 و 16 ميكروغرام / مل على التوالي
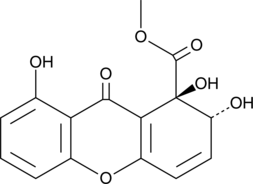
-
GC45668
Gly-Arg-AMC (hydrochloride)
A fluorogenic substrate for cathepsin C
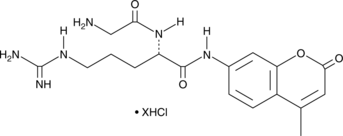
-
GC49687
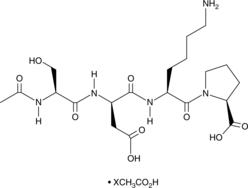
Goralatide (acetate)
A tetrapeptide regulator of hematopoiesis
-
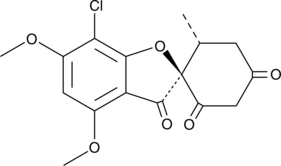
GC48720
Griseofulvic Acid
A metabolite of griseofulvin
-
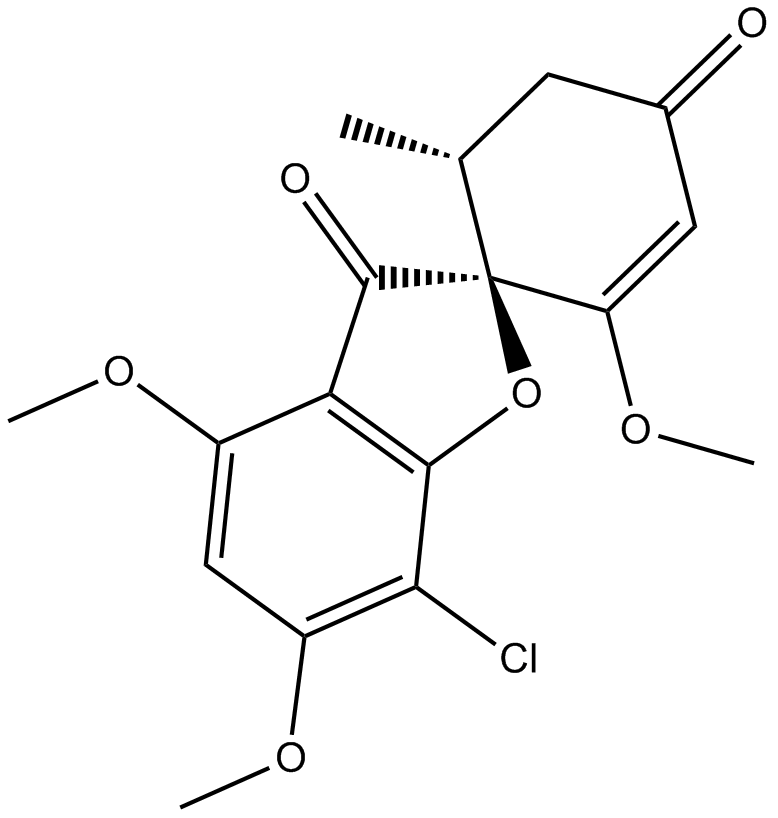
GC13484
Griseofulvin
Griseofulvin (Gris-PEG ؛ Grifulvin) هو منتج فطري حلزوني طبيعي يستخدم في علاج الفطريات الجلدية الفطرية ؛ دواء مضاد للفطريات
-
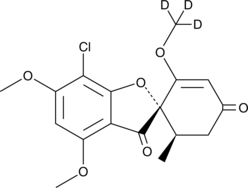
GC48688
Griseofulvin-d3
An internal standard for the quantification of griseofulvin
-
GC49744
GRP94 Monoclonal Antibody (Clone 9G10)
For immunochemical detection of GRP94

-
GC38791
GSK-25
GSK-25 هو مثبط ROCK1 قوي وانتقائي ومتوفر بيولوجيًا عن طريق الفم (IC50 = 7 نانومتر)
-
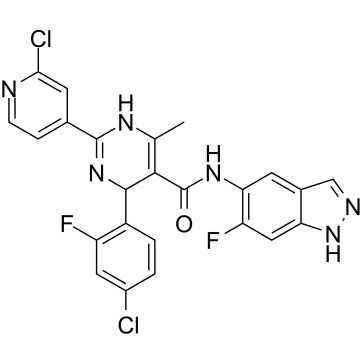
GC15659
GSK-923295
An inhibitor of CENP-E
-
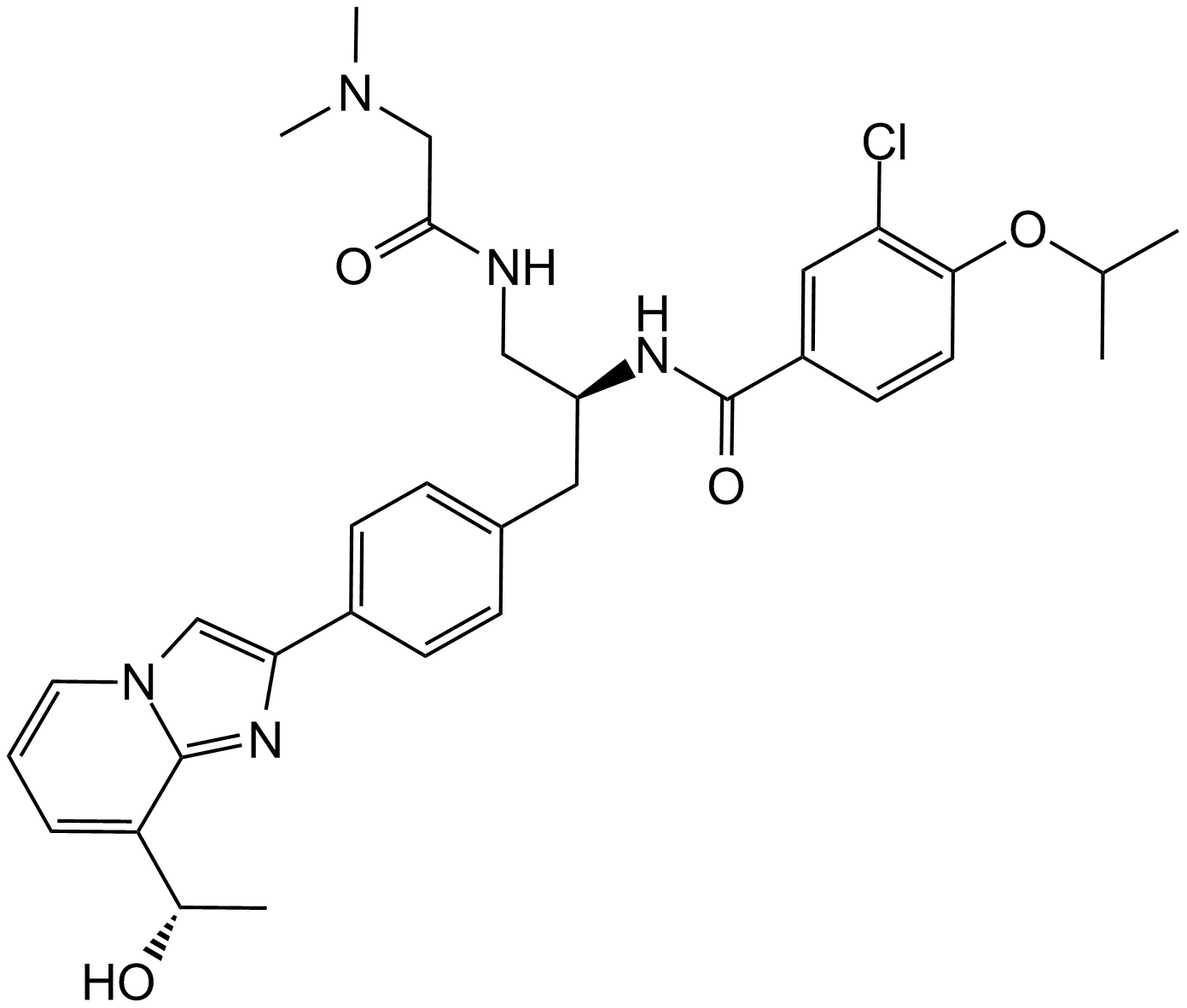
GC19177
GSK180736A
GSK180736A هو مثبط قوي للكيناز الملفوف المرتبط بـ Rho (ROCK1) مع IC50 من 100 نانومتر
-
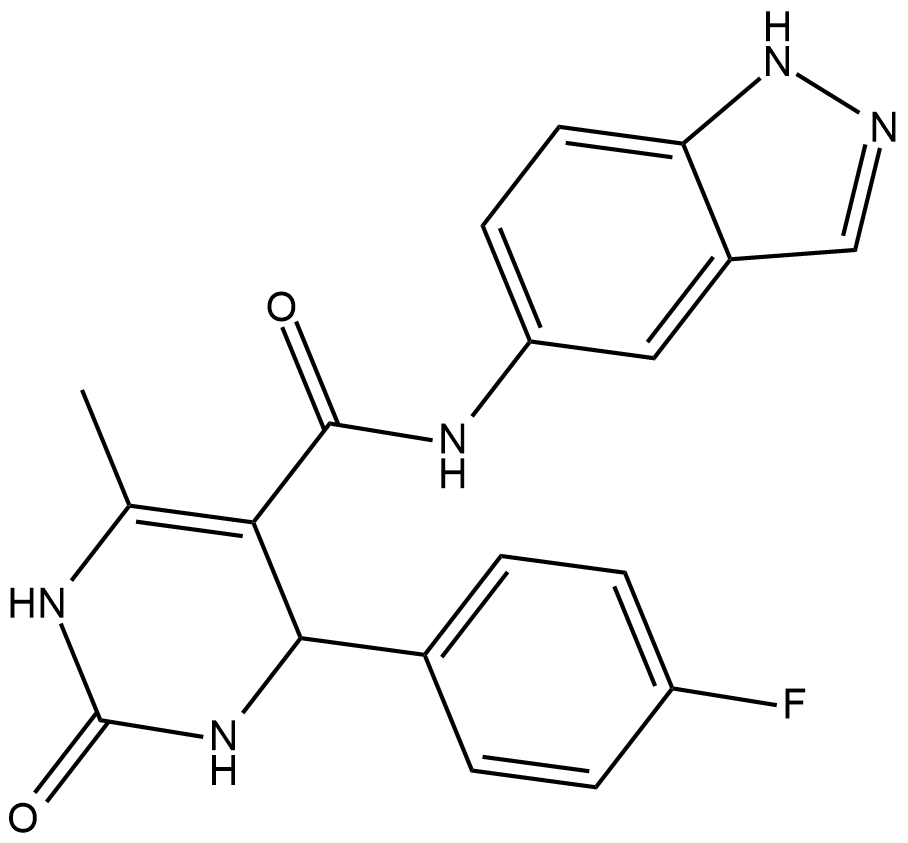
GC17450
GSK2606414
مثبط انتقائي لبروتين PERK
-
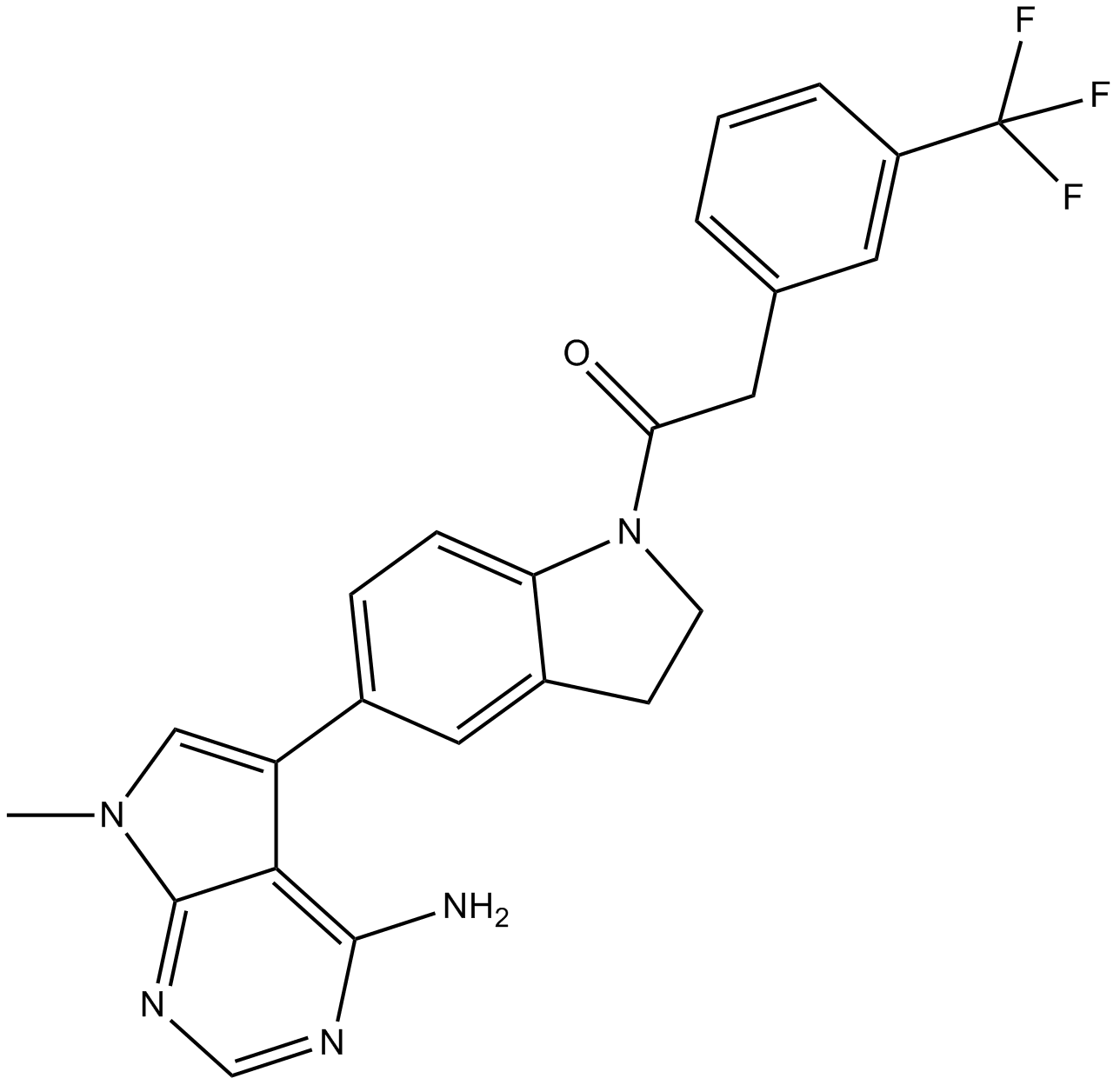
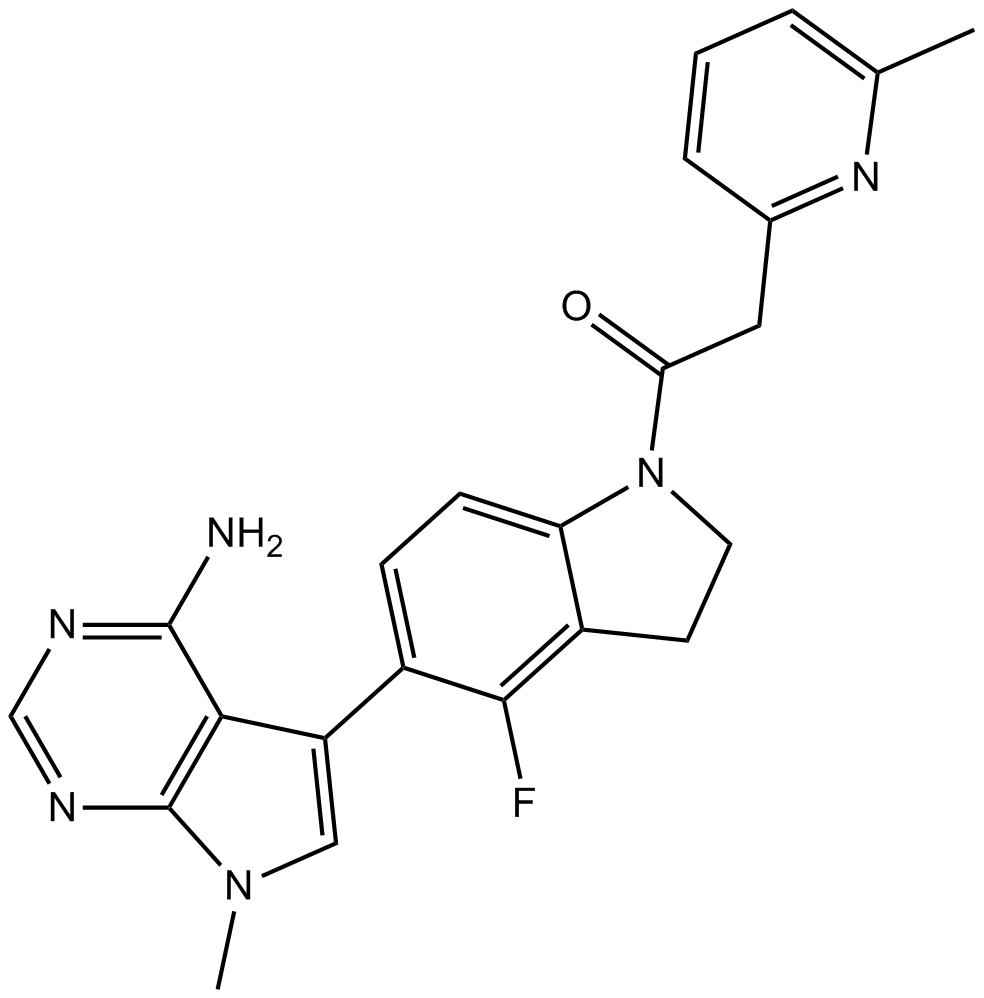
GC11068
GSK2656157
GSK2656157 هو مثبط انتقائي وتنافسي لـ ATP لبروتين كيناز R (PKR) على غرار شبكة الإندوبلازمية كيناز (PERK) مع IC 50 من 0.9 نانومتر
-
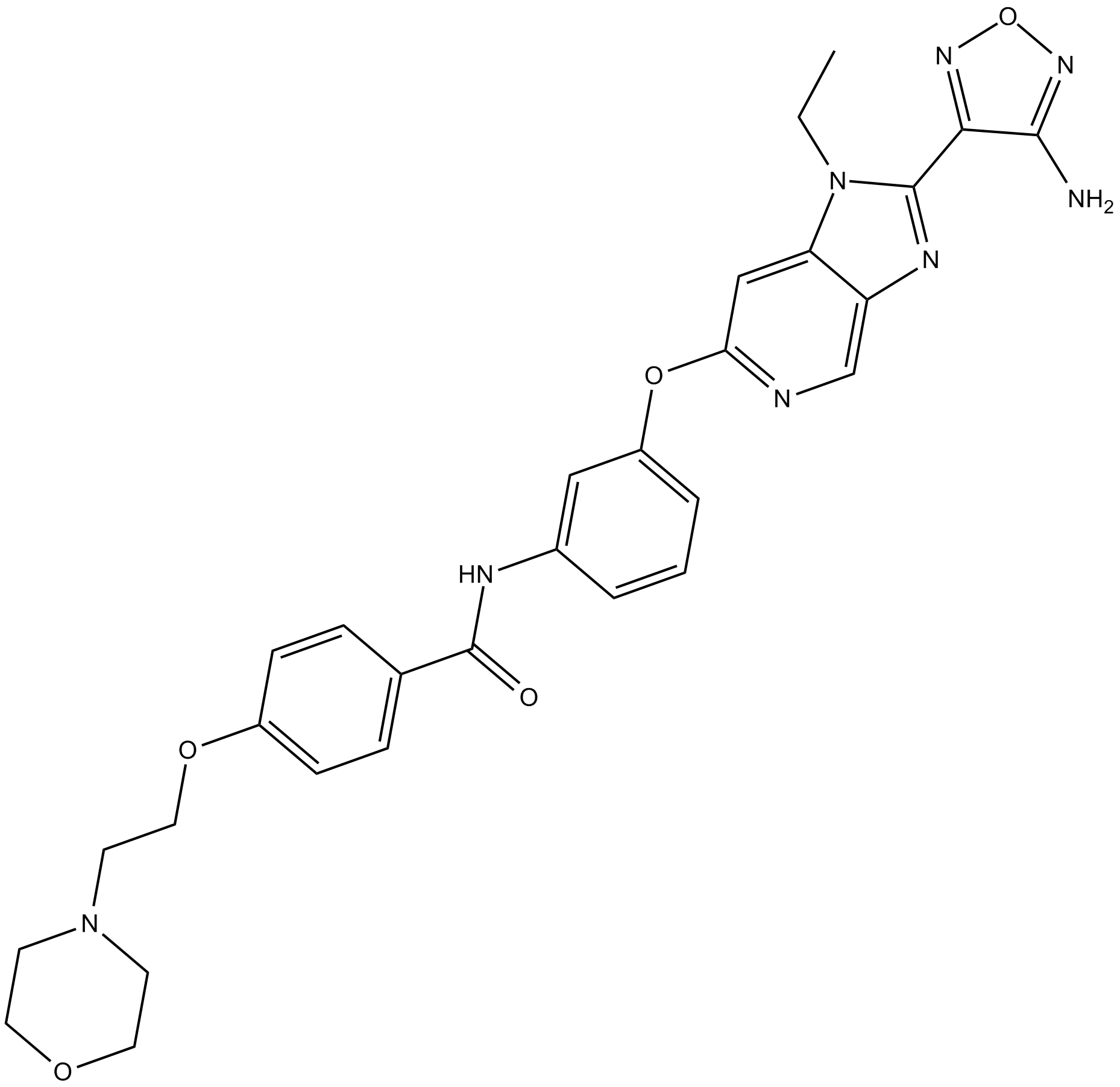
GC17936
GSK269962A
GSK269962A (GSK 269962) هو مثبط قوي للصخور مع IC50s 1.6 و 4 نانومتر للإنسان المؤتلف ROCK1 و ROCK2 على التوالي
-
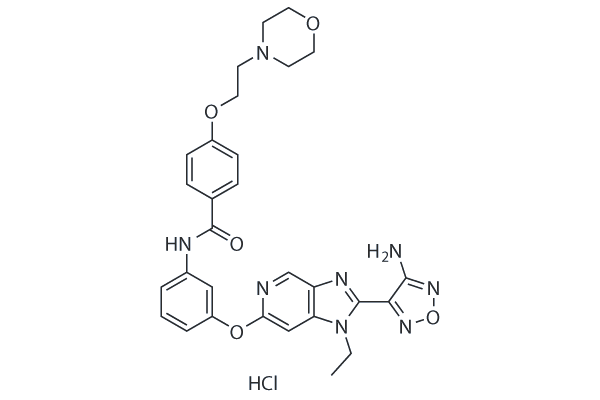
GC25482
GSK269962A HCl
GSK269962A HCl (GSK269962B, GSK269962) is a selective ROCK(Rho-associated protein kinase) inhibitor with IC50 values of 1.6 and 4 nM for ROCK1 and ROCK2, respectively.
-
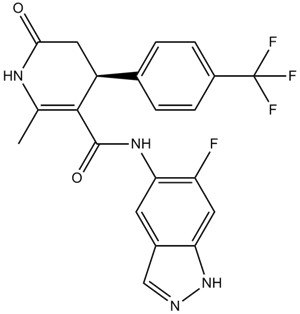
GC18119
GSK429286A
GSK429286A هو مثبط انتقائي لـ ROCK1 بقيمة IC50 تبلغ 14 نانومتر
-
GC12335
GSK461364
GSK461364 هو مثبط للكيناز 1 (PLK1) شبيه بالبولو انتقائي وقابل للانعكاس وتنافسي ATP بقيمة Ki تبلغ 2.2 نانومتر
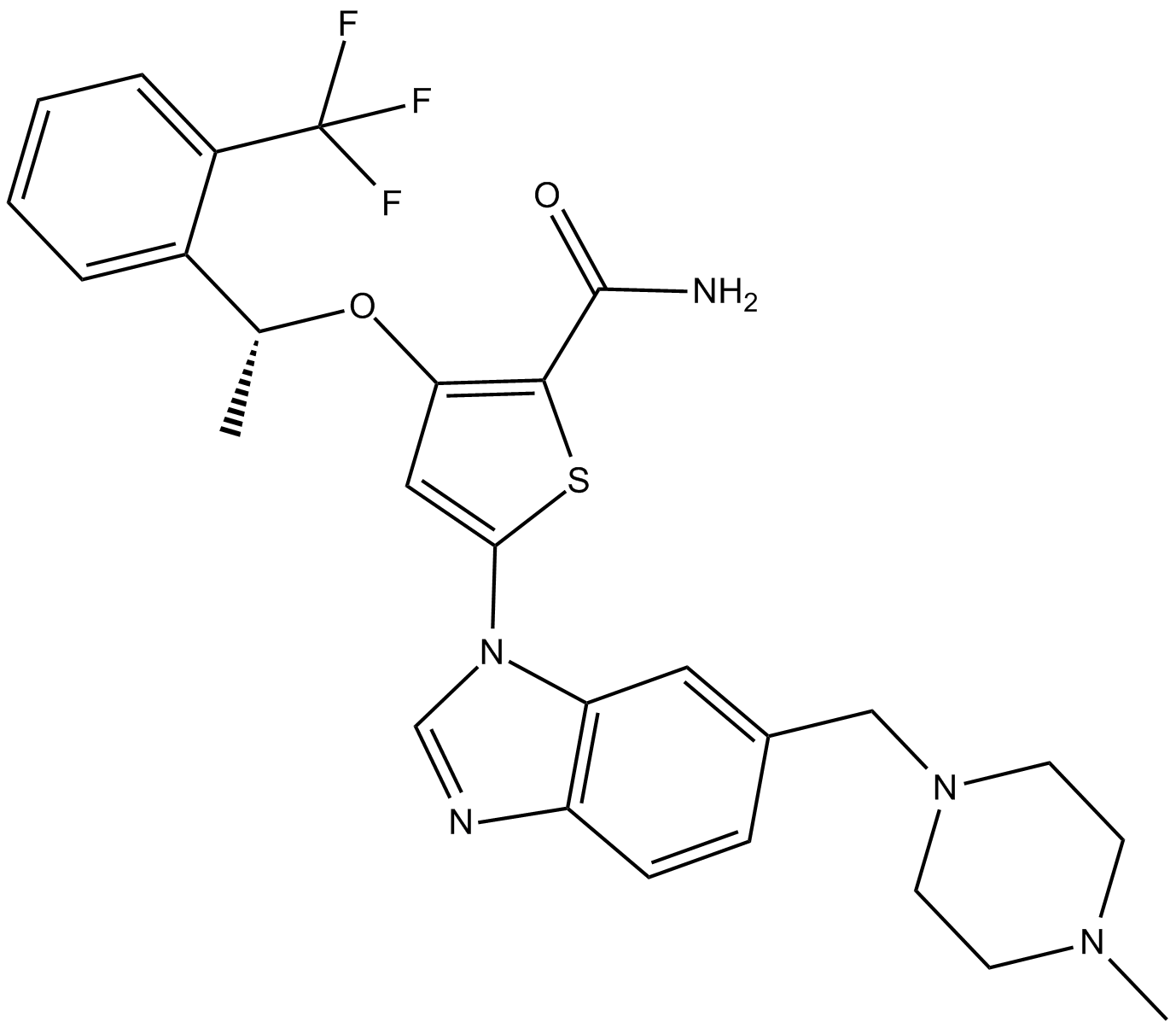
-
GC49280
GSK579289A
A Plk1 inhibitor
-
GC60891
GW406108X
GW406108X هو مثبط Kif15 (Kinesin-12) محدد مع IC50 يبلغ 0.82 ميكرومتر في فحوصات ATPaseيُظهر GW406108X ، مثبط الالتهام الذاتي القوي ، تثبيط ATP التنافسي ضد ULK1 مع pIC50 من 6.37 (427 نانومتر)GW406108X يثبط نشاط كيناز ULK1 ويمنع التدفق الذاتي ، دون التأثير على كينازات الإشارات المنبع mTORC1 و AMPK
-
GC11621
GW843682X
A reversible polo-like kinase inhibitor
-
GC16545
GX-674
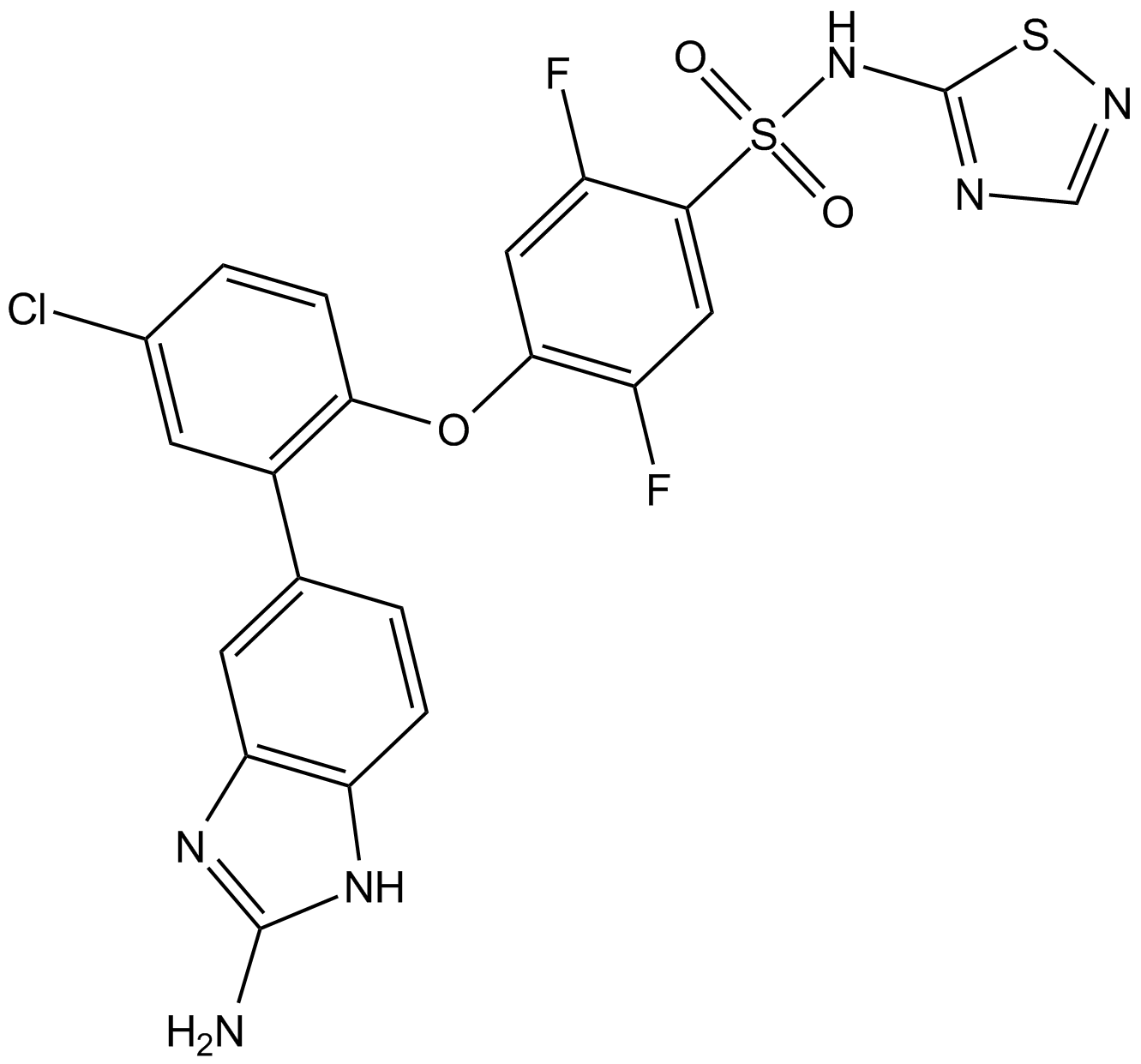
GX-674 عبارة عن قناة صوديوم قوية ، معتمدة على الحالة ، انتقائية ذات جهد إسوي الشكل 1.7 (Nav1.7) مع IC50 من 0.1 نانومتر عند -40 مللي فولت
-
GC36207
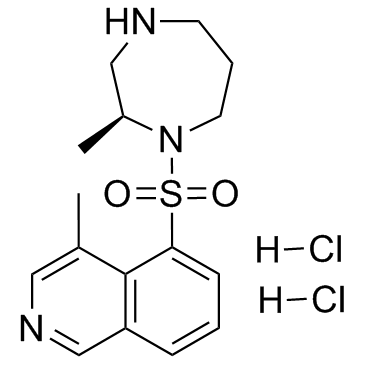
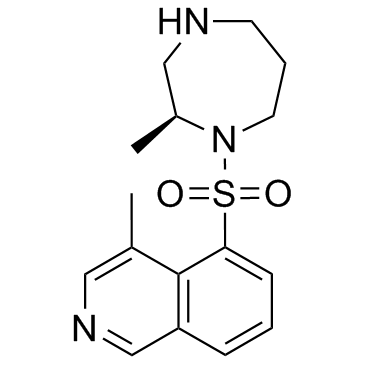
H-1152
H-1152 هو مثبط ROCK انتقائي منفذ للغشاء ، بقيمة Ki تبلغ 1.6 نانومتر ، وقيمة IC50 تبلغ 12 نانومتر لـ ROCK2
-
GC36208
H-1152 dihydrochloride
ثنائي هيدروكلوريد H-1152 هو مثبط ROCK انتقائي قابل للنفاذ بالغشاء ، بقيمة Ki تبلغ 1.6 نانومتر ، وقيمة IC50 تبلغ 12 نانومتر لـ ROCK2