KEAP1-Nrf2
The KEAP1-Nrf2 pathway is the major regulator of cytoprotective responses to chemical and/or oxidative stresses caused by reactive oxygen species (ROS) and electrophiles. Nrf2 (nuclear factor erythroid 2-related factor 2) is a transcription factor that binds together with small Maf proteins to the antioxidant response element (ARE) in the regulatory regions of cellular defense enzyme genes leading to the activation of a wide rang of cell defense processes; while KEAP1 (Kelch ECH associating protein 1), a negative repressor of Nrf2, is an adaptor protein for a Cul3-based ubiqutitin E3 ligase that binds to Nrf2 and promotes its degradation by the ubiquitin proteasome pathway.
Produkte für KEAP1-Nrf2
- Bestell-Nr. Artikelname Informationen
-
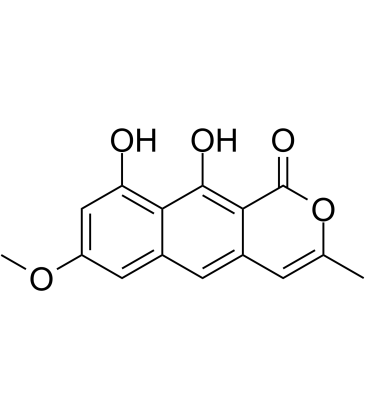
GN10654
(+)-Corynoline
-
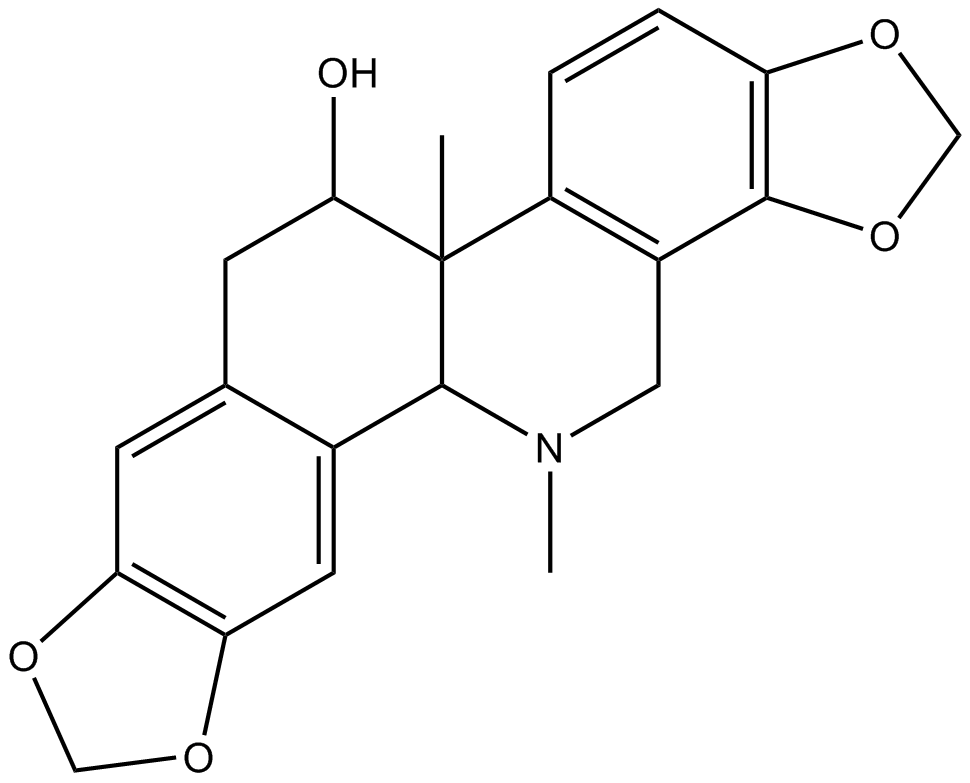
GC31691
(+)-DHMEQ
(+)-DHMEQ ist ein Aktivator des antioxidativen Transkriptionsfaktors Nrf2.
-
GC65610
(R)-5-Hydroxy-1,7-diphenyl-3-heptanone
(R)-5-Hydroxy-1,7-diphenyl-3-heptanon ist ein Diarylheptanoid, das in Alpinia officinarum vorkommt.

-
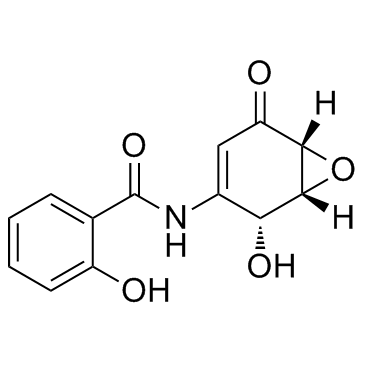
GC12545
2-HBA
2-HBA ist ein starker Induktor der NAD(P)H:Chinon-Akzeptor-Oxidoreduktase 1 (NQO1), die auch Caspase-3 und Caspase-10 aktivieren kann.
-
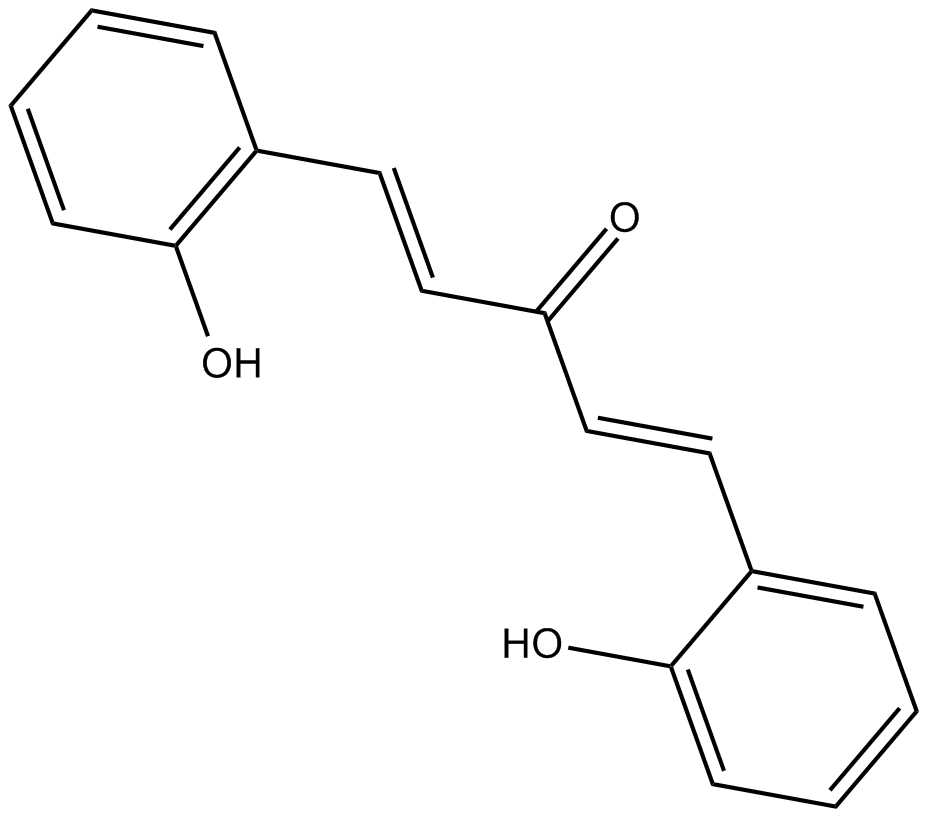
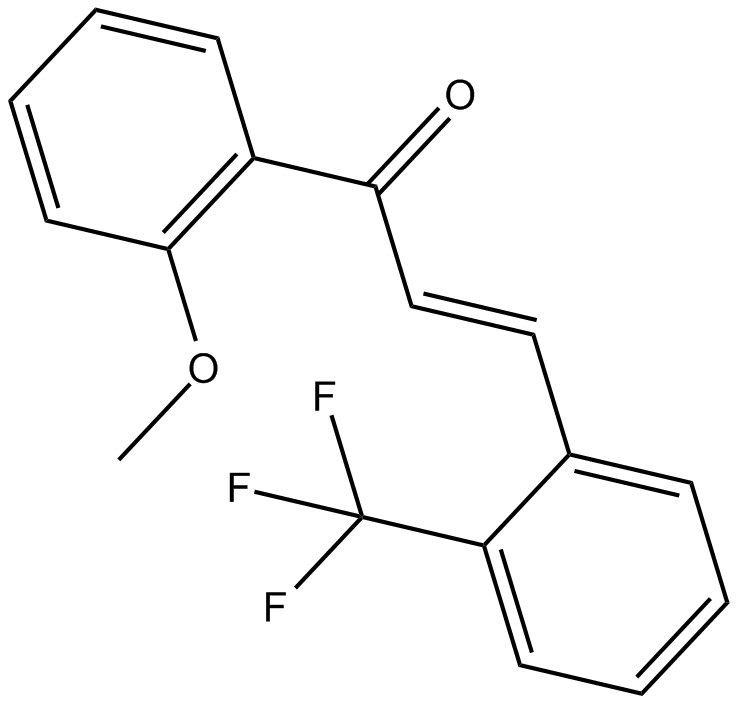
GC15355
2-Trifluoromethyl-2'-methoxychalcone
Nrf2 activator
-
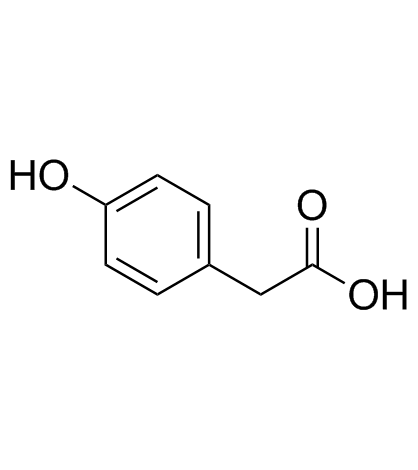
GC33815
4-Hydroxyphenylacetic acid
4-HydroxyphenylessigsÄure, ein wichtiger, von Mikrobiota stammender Metabolit von Polyphenolen, ist an der antioxidativen Wirkung beteiligt.
-
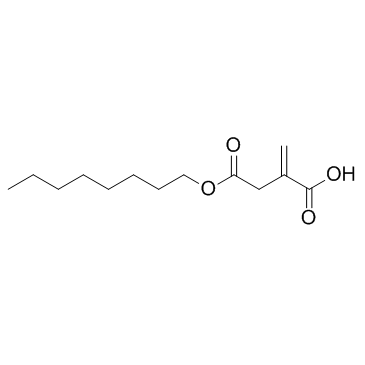
GC31648
4-Octyl Itaconate
4-Octyl-Itakonat (4-OI) ist ein zellpermeables Itakonat-Derivat. Itakonat und 4-Octyl-Itakonat hatten eine ähnliche Thiol-Reaktivität, wodurch sich 4-Octyl-Itakonat als geeigneter Ersatz für Itakonat zur Untersuchung seiner biologischen Funktion eignet.
-
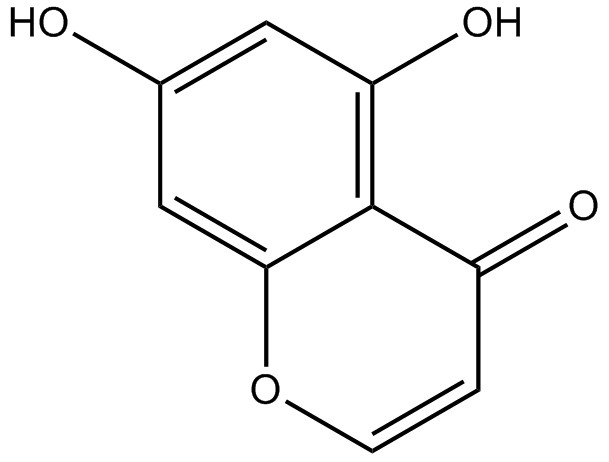
GN10629
5,7-dihydroxychromone
-
GC16120
AI-3
Nrf2/Keap1 and Keap1/Cul3 interaction inhibitor
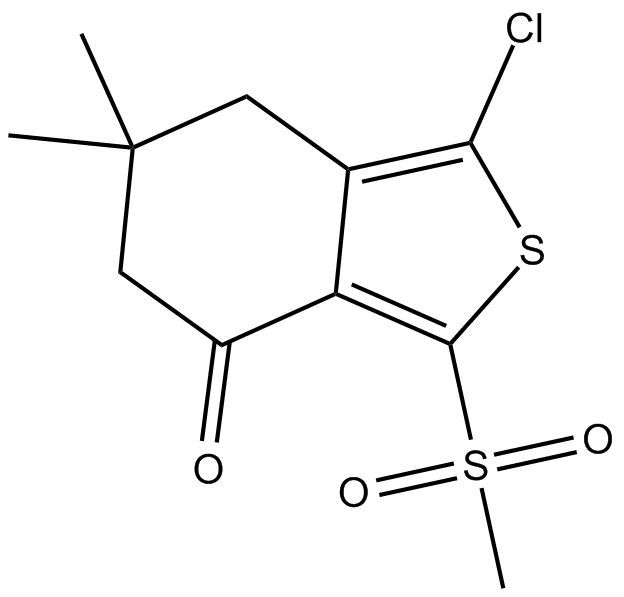
-
GN10415
Astilbin
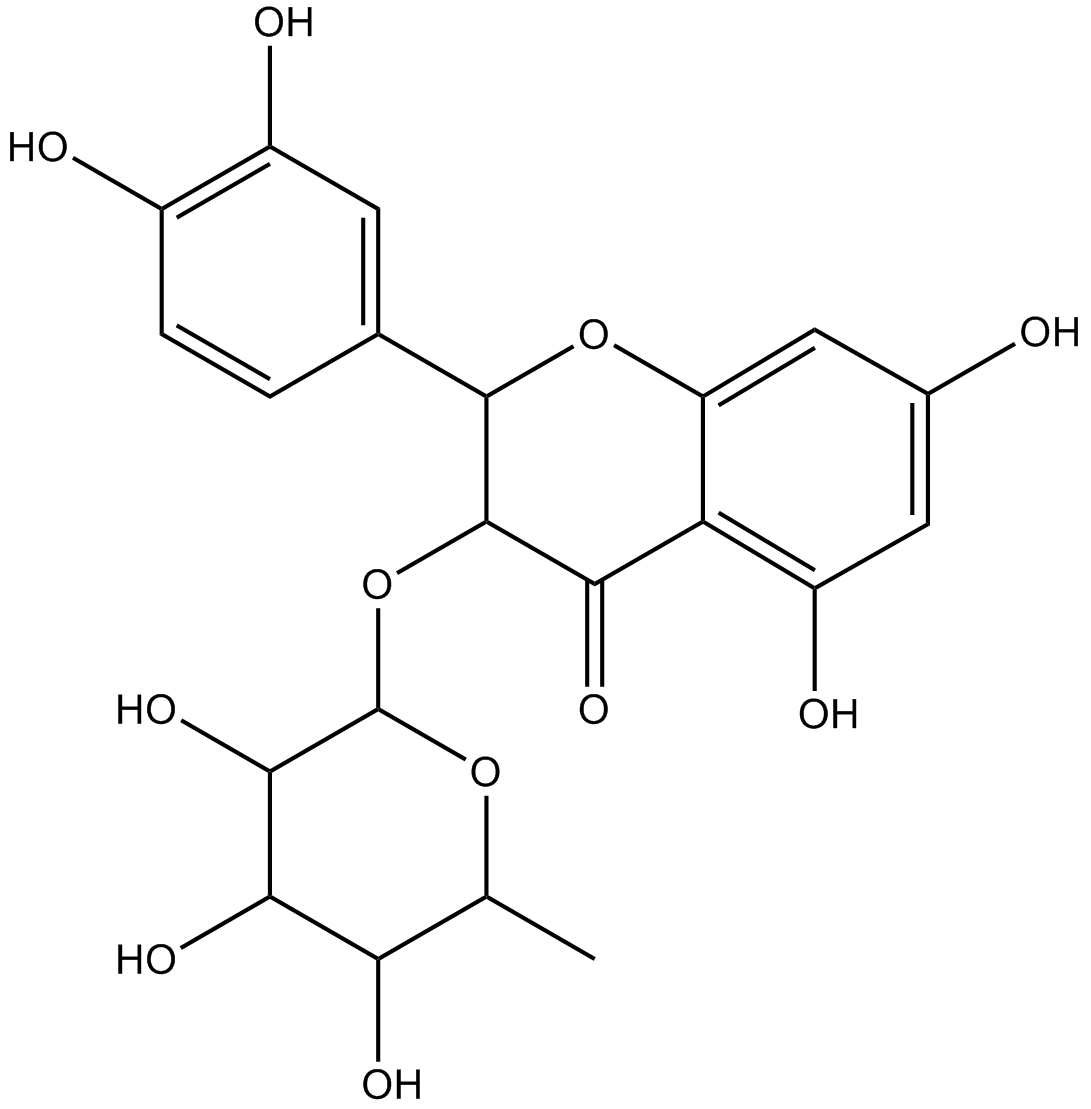
-
GC15371
Bardoxolone
An anti-inflammatory compound that activates Nrf2/ARE signaling
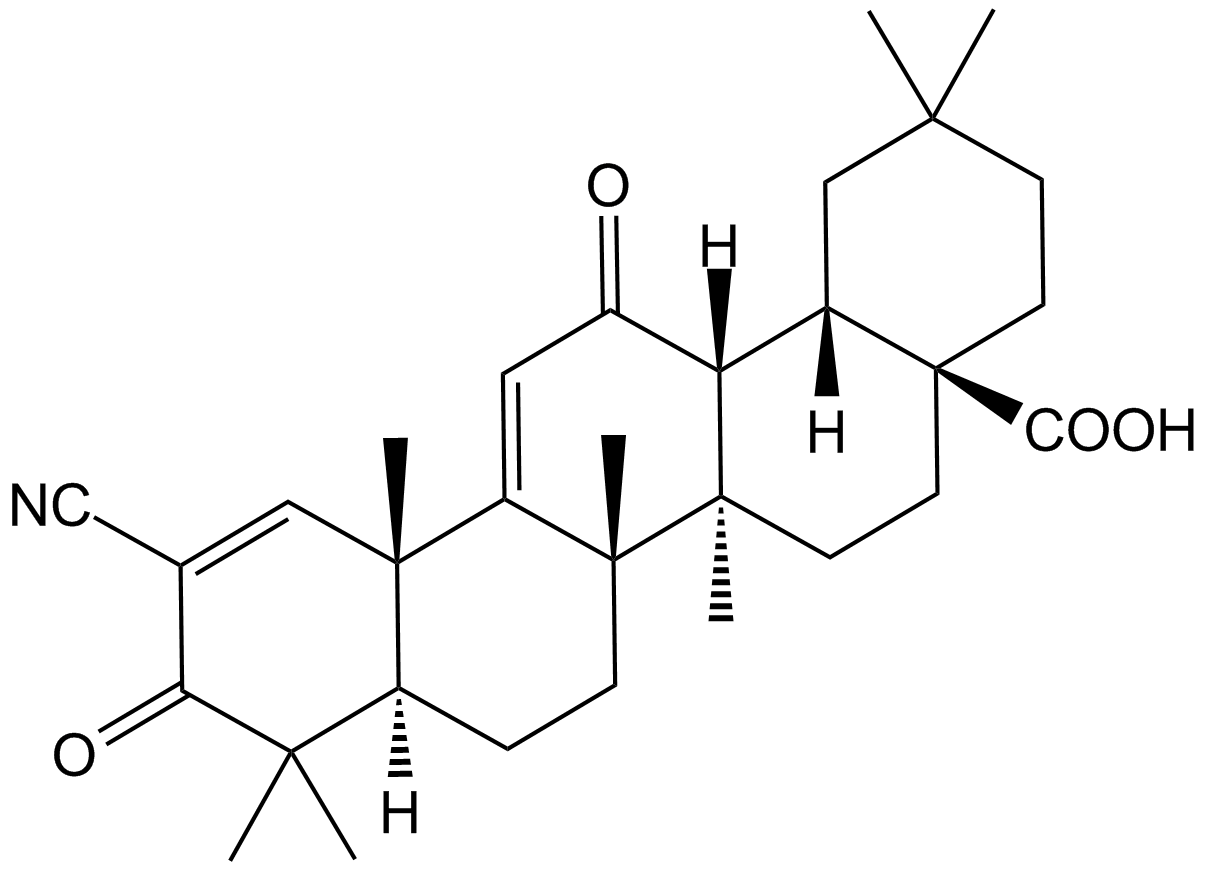
-
GC11572
Bardoxolone methyl
A synthetic triterpenoid with potent anticancer and antidiabetic activity
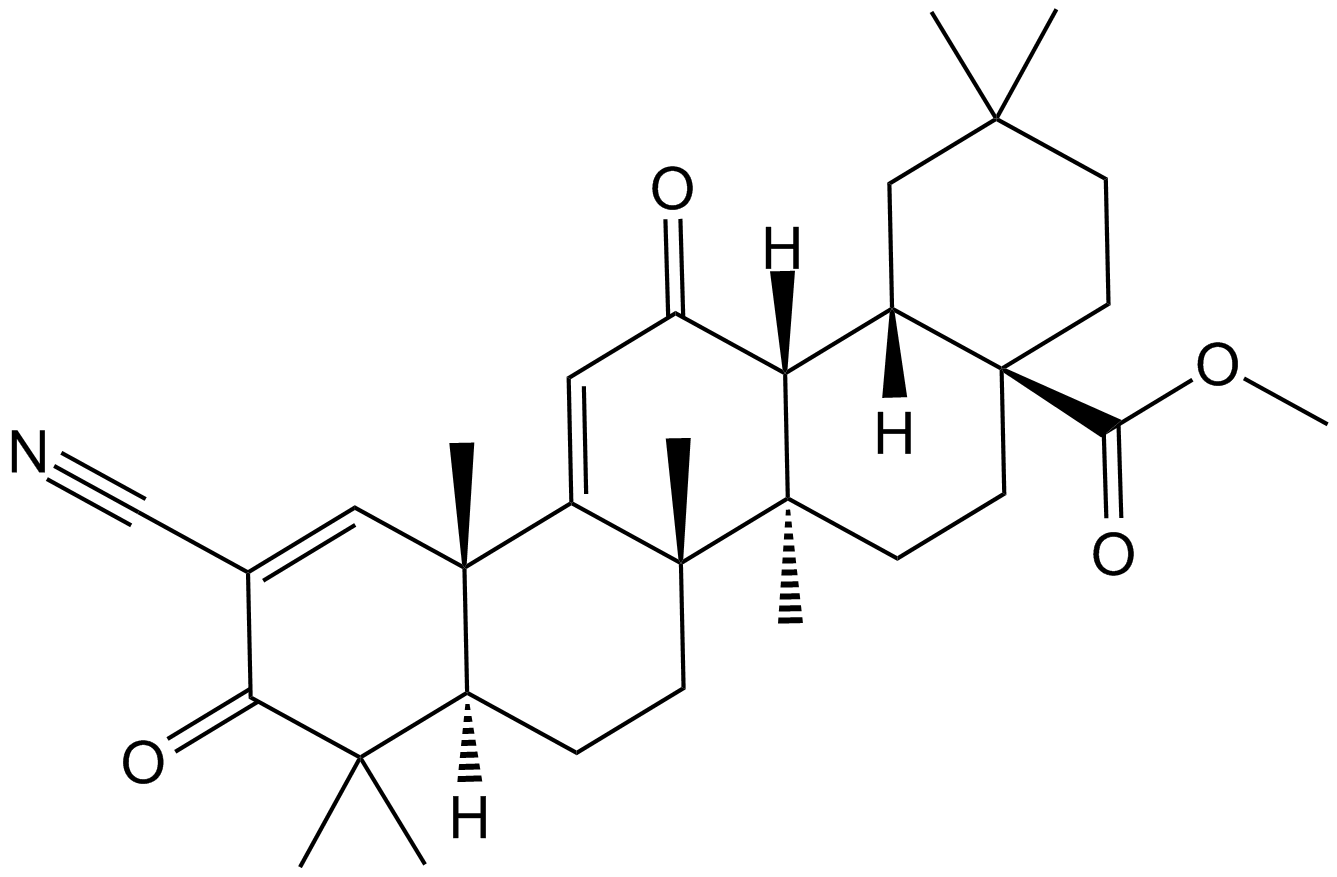
-
GC34070
Brusatol (NSC 172924)
Brusatol (NSC 172924) (NSC172924) ist ein einzigartiger Inhibitor des Nrf2-Signalwegs, der ein breites Spektrum von Krebszellen fÜr Cisplatin und andere Chemotherapeutika sensibilisiert. Brusatol (NSC 172924) verstÄrkt die Wirksamkeit der Chemotherapie, indem es den Nrf2-vermittelten Abwehrmechanismus hemmt. Brusatol (NSC 172924) kann zu einem adjuvanten Chemotherapeutikum entwickelt werden. Brusatol (NSC 172924) erhÖht die zellulÄre Apoptose.
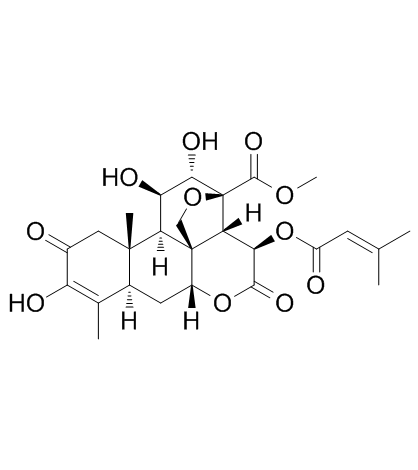
-
GC61636
CBR-470-2
CBR-470-2, ein Glycin-substituiertes Analogon, kann die NRF2-Signalgebung aktivieren.
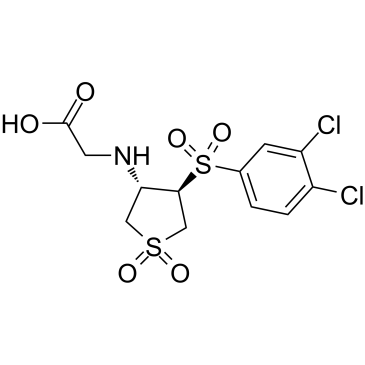
-
GC35629
CDDO-dhTFEA
CDDO-dhTFEA (RTA dh404) ist eine synthetische Oleanan-Triterpenoid-Verbindung, die Nrf2 wirksam aktiviert und den entzÜndungsfÖrdernden Transkriptionsfaktor NF-κB hemmt.
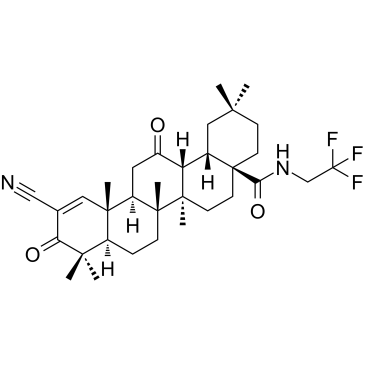
-
GC35630
CDDO-EA
CDDO-EA ist ein mit NF-E2 verwandter Faktor 2/antioxidatives Response-Element (Nrf2/ARE)-Aktivator.

-
GC32723
CDDO-Im (RTA-403)
CDDO-Im (RTA-403) (RTA-403) ist ein Aktivator von Nrf2 und PPAR, mit Kis von 232 und 344 nM fÜr PPARα und PPARγ.
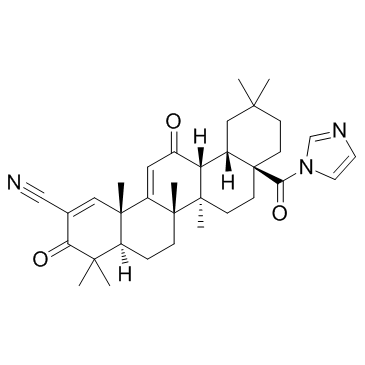
-
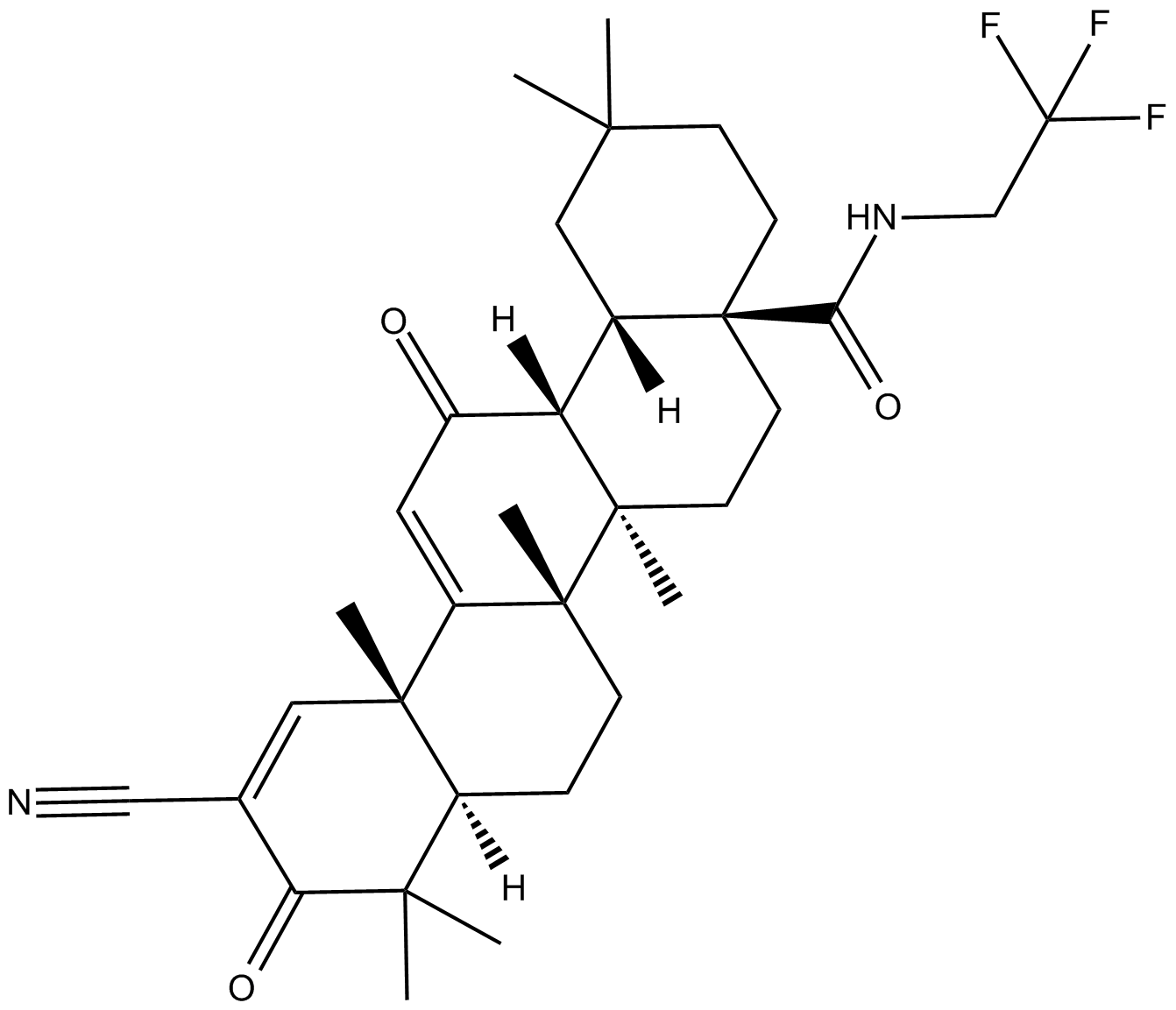
GC16625
CDDO-TFEA
Nrf2 activator
-
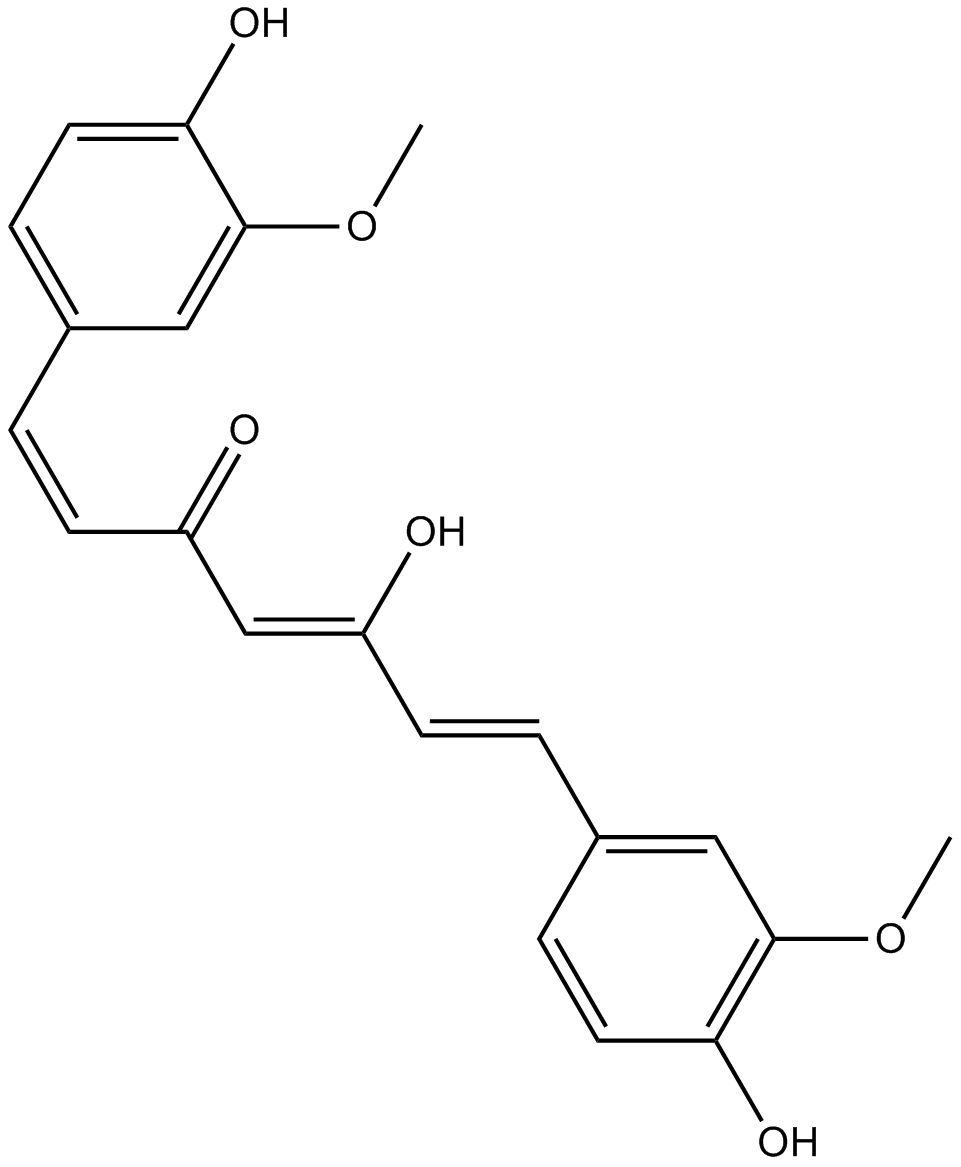
GC14787
Curcumin
Ein gelbes Pigment mit vielfältigen biologischen Aktivitäten.
-
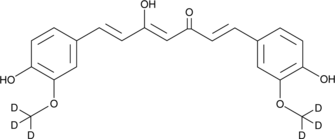
GC40226
Curcumin-d6
Curcumin D6 (Diferuloylmethan D6) ist ein Deuterium mit der Bezeichnung Curcumin (gelbe Kurkuma).
-
GN10318
Danshensu
-
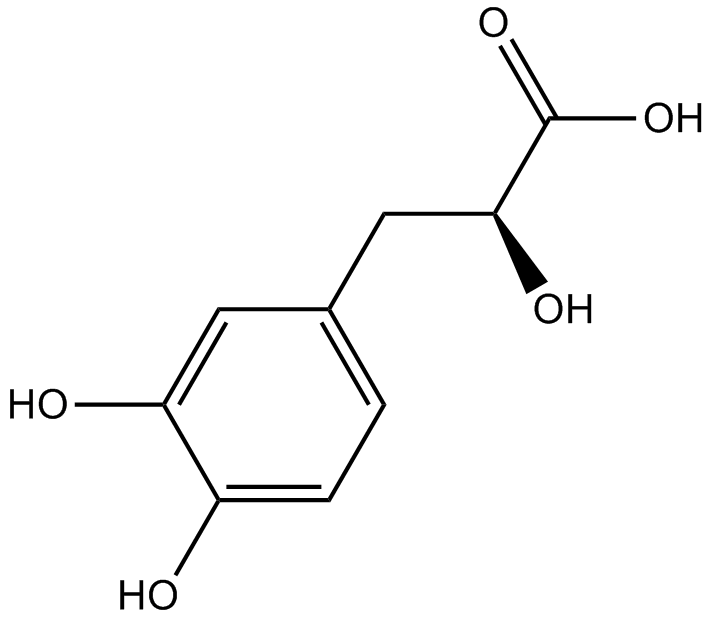
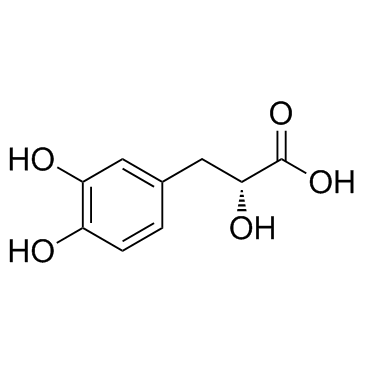
GC34010
Danshensu (Dan shen suan A)
Danshensu, an active ingredient of Salvia miltiorrhiza, shows wide cardiovascular benefit by activating Nrf2 signaling pathway.
-
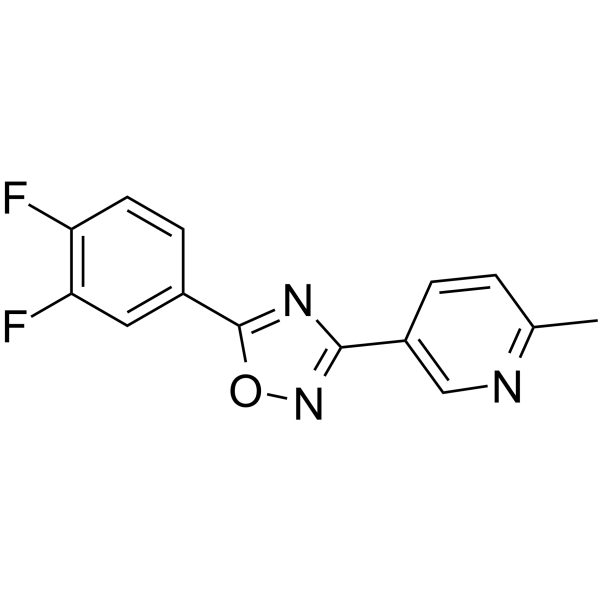
GC64971
DDO-7263
DDO-7263, ein 1,2,4-Oxadiazol-Derivat, ist ein potenter Nrf2-ARE-Aktivator.
-
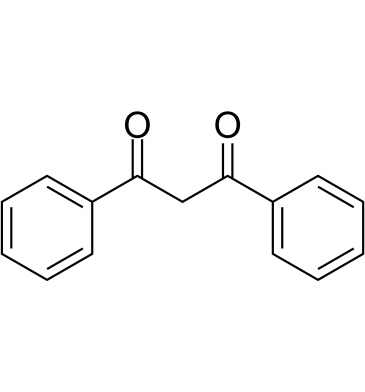
GC61419
Dibenzoylmethane
Dibenzoylmethan, ein Nebenbestandteil von SÜßholz, aktiviert Nrf2 und beugt verschiedenen Krebsarten und oxidativen SchÄden vor.
-
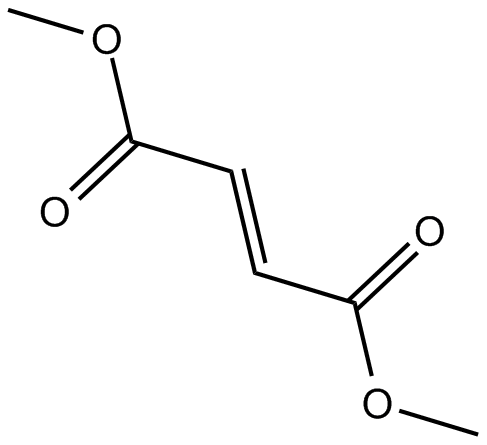
GC16590
Dimethyl Fumarate
Dimethylfumarat (DMF) ist ein oral aktiver und in das Gehirn eindringender Nrf2-Aktivator und induziert eine Hochregulierung der antioxidativen Genexpression.
-
GC62936
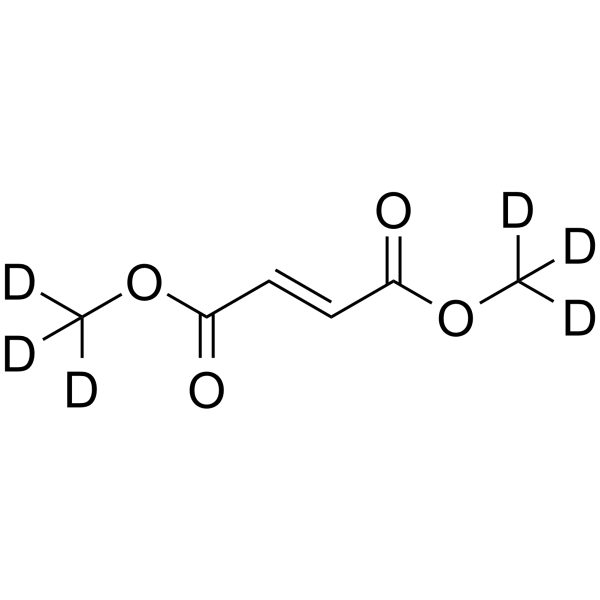
Dimethyl fumarate D6
Dimethylfumarat D6 ist ein mit Deuterium gekennzeichnetes Dimethylfumarat.
-
GC25351
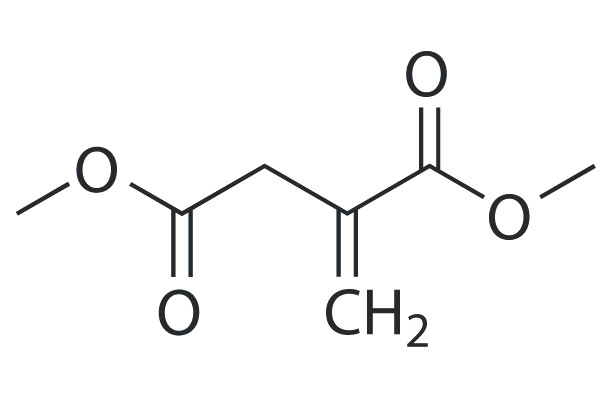
Dimethyl itaconate
Dimethyl itaconate can reprogram neurotoxic to neuroprotective primary astrocytes through the regulation of LPS-induced Nod-like receptor protein 3 (NLRP3) inflammasome and nuclear factor 2/heme oxygenase-1 (NRF2/HO-1) pathways.
-
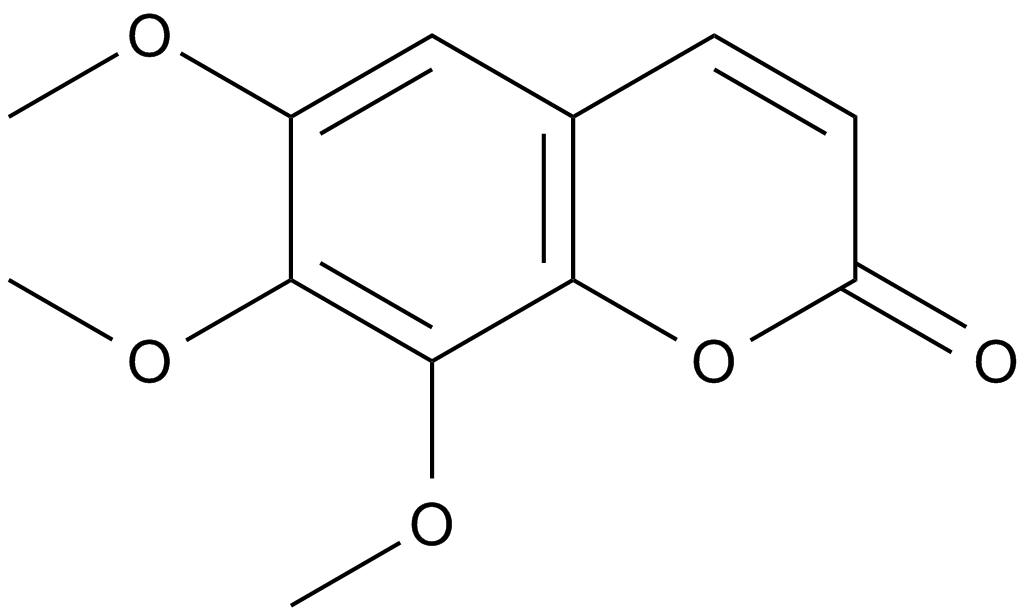
GN10531
Dimethylfraxetin
-
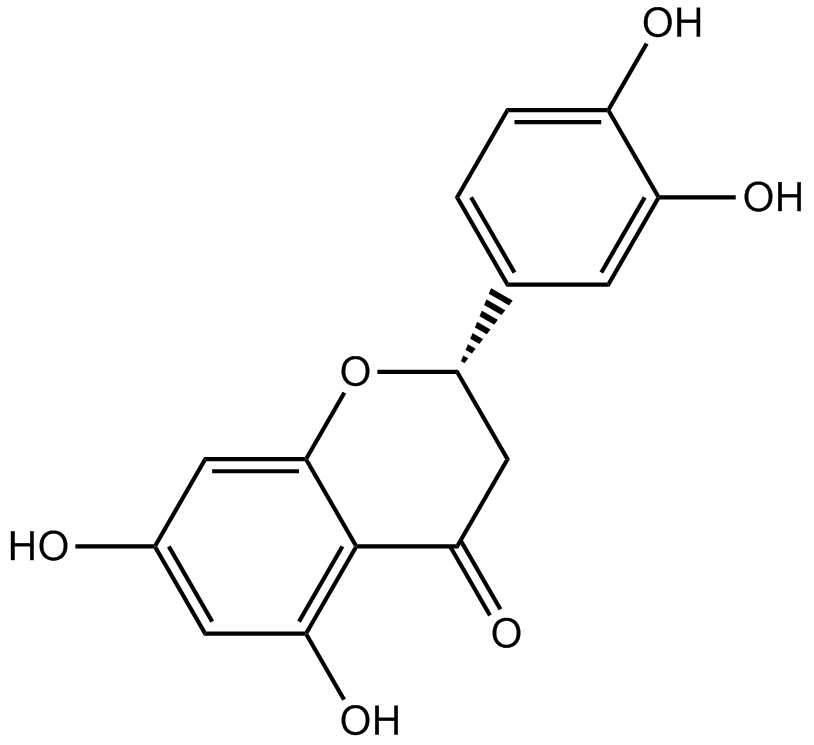
GN10470
Eriodictyol
-
GC62957
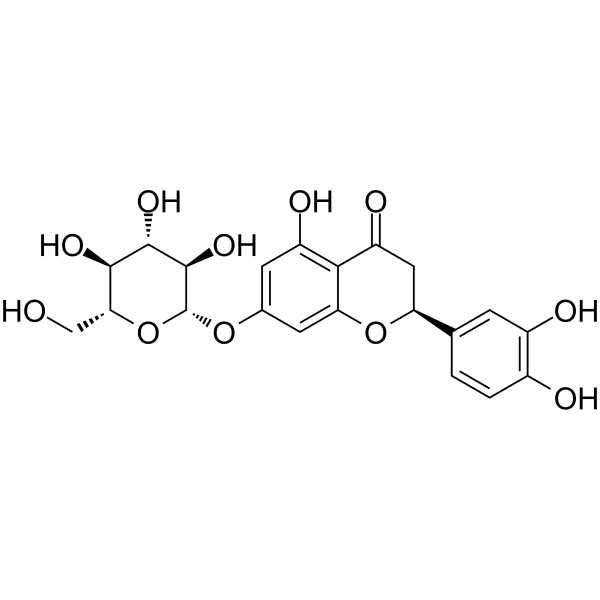
Eriodictyol-7-O-glucoside
Eriodictyol-7-O-Glucosid (Eriodictyol 7-O-β-D-Glucosid), ein Flavonoid, ist ein starker FÄnger freier Radikale.
-
GC15605
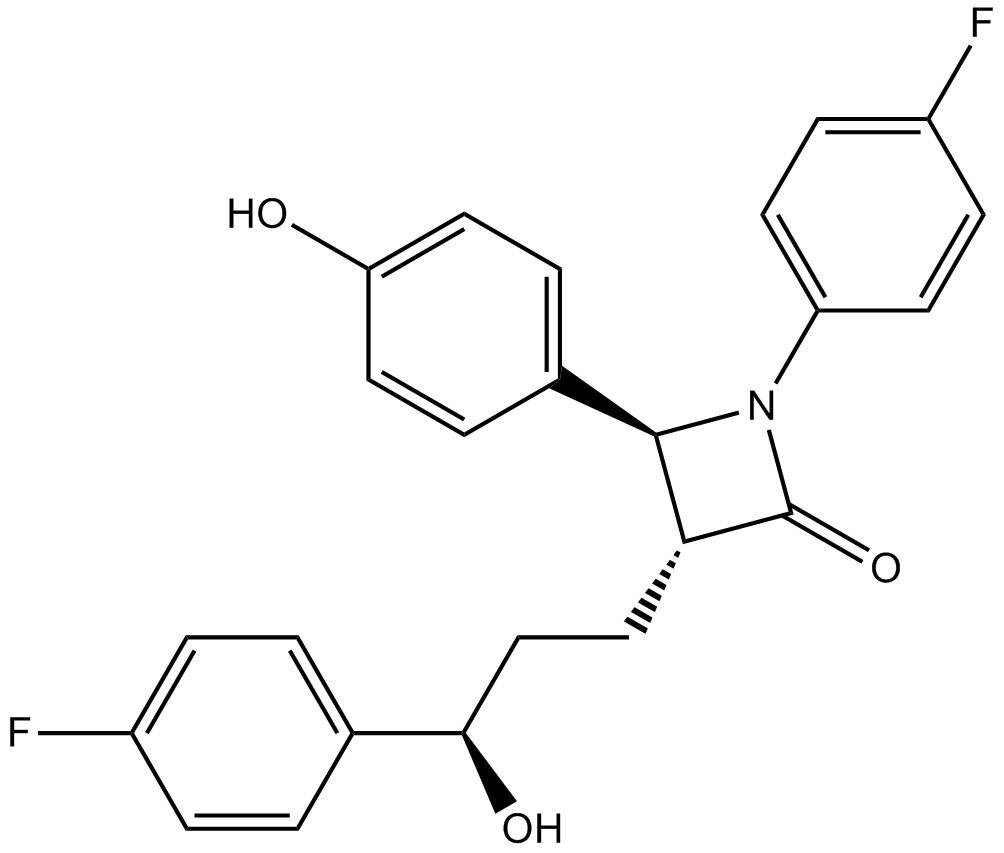
Ezetimibe
Ezetimib (SCH 58235) ist ein starker Hemmer der Cholesterinabsorption.
-
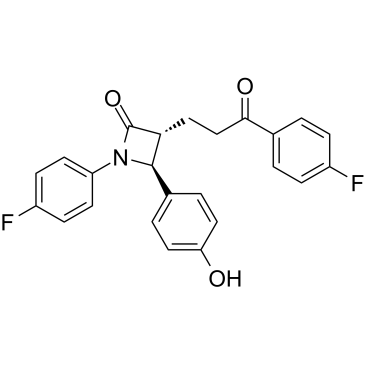
GC60159
Ezetimibe ketone
Ezetimib-Keton (EZM-K) ist ein Phase-I-Metabolit von Ezetimib.
-
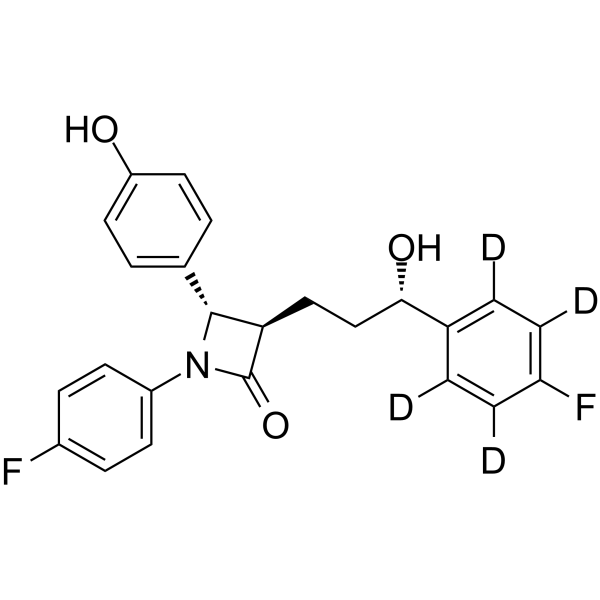
GC66354
Ezetimibe-d4-1
Ezetimib-d4 ist Deuterium mit der Bezeichnung Ezetimib. Ezetimib (SCH 58235) ist ein starker Hemmer der Cholesterinabsorption. Ezetimib ist ein C1-like1 (NPC1L1)-Hemmer von Niemann-Pick und ein starker Nrf2-Aktivator.
-
GN10741
Garcinone D
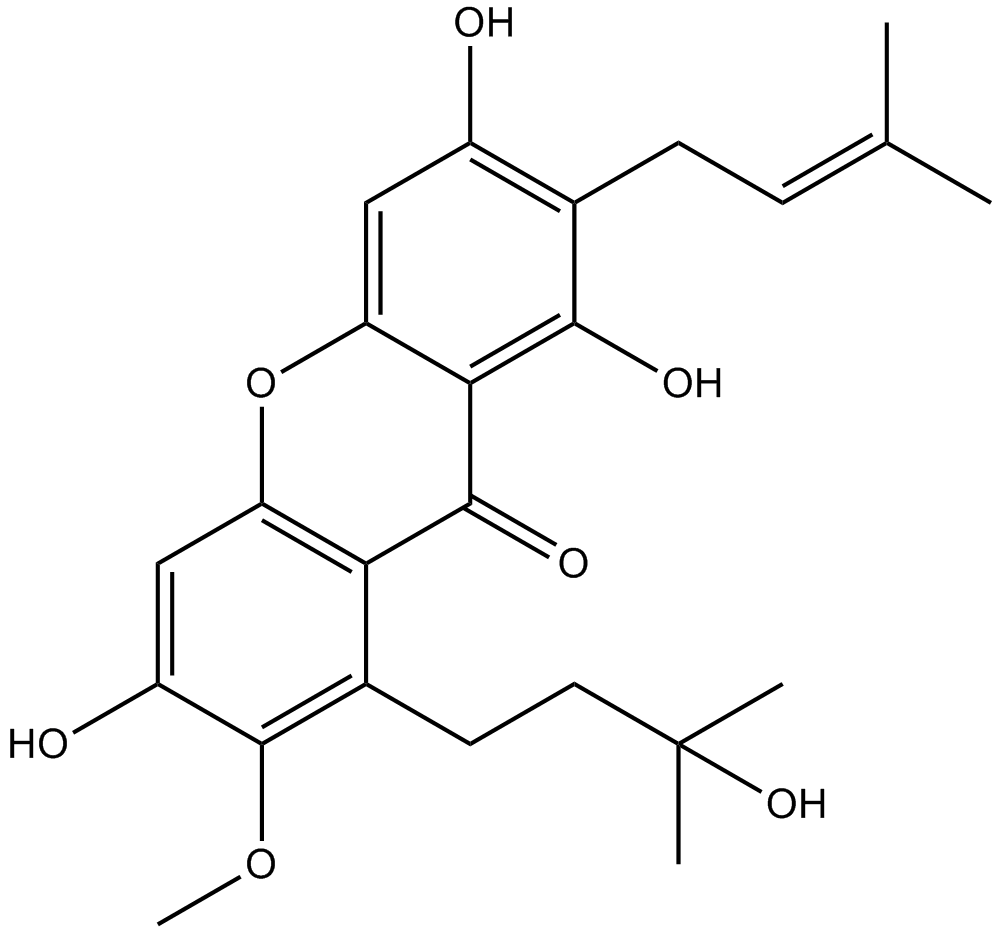
-
GN10038
Ginsenoside Rh3
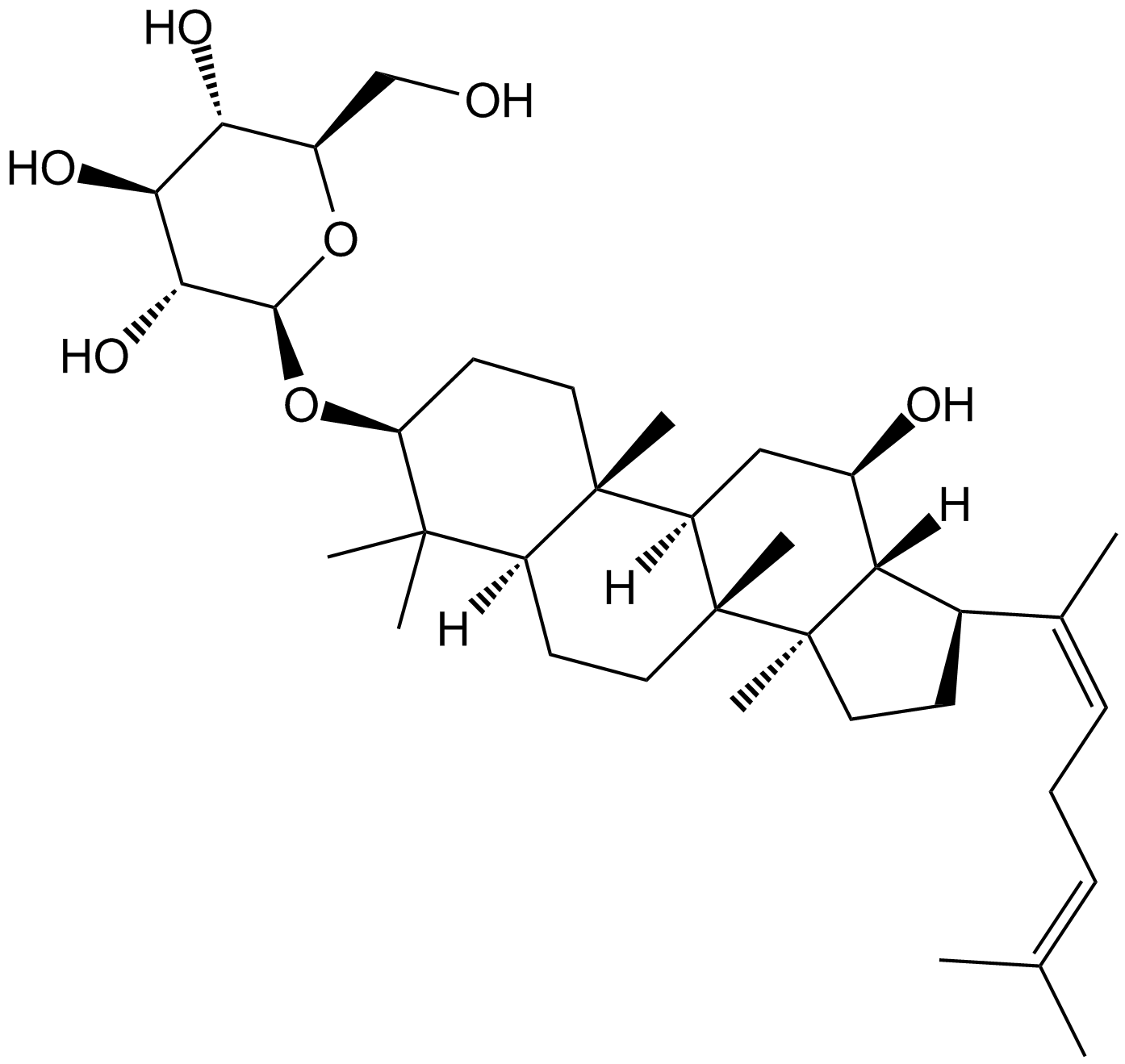
-
GC33092
Hesperin
Hesperin ist ein bioaktiver Inhaltsstoff, der in japanischem Meerrettich (Wasabi) vorkommt und sich als Nrf2-Aktivator erwiesen hat.
-
GC11302
Hinokitiol
Hinokitiol ist ein Bestandteil Ätherischer Öle, die aus Chymacyparis obtusa isoliert wurden, reduziert die Nrf2-Expression und verringert die mRNA- und Proteinexpression von DNMT1 und UHRF1 mit antiinfektiÖsen, antioxidativen und AntitumoraktivitÄten.

-
GC66004
K67
K67 hemmt spezifisch die Interaktion zwischen Keap1 und S349-phosphoryliertem p62. K67 verhindert, dass p-p62 die Bindung von Keap1 und Nrf2 blockiert. K67 hemmt wirksam die Proliferation von HCC-Kulturen mit hochzellulÄrem S351-phosphoryliertem p62, indem es die von Keap1 angetriebene Ubiquitinierung und den Abbau von Nrf2 wiederherstellt.
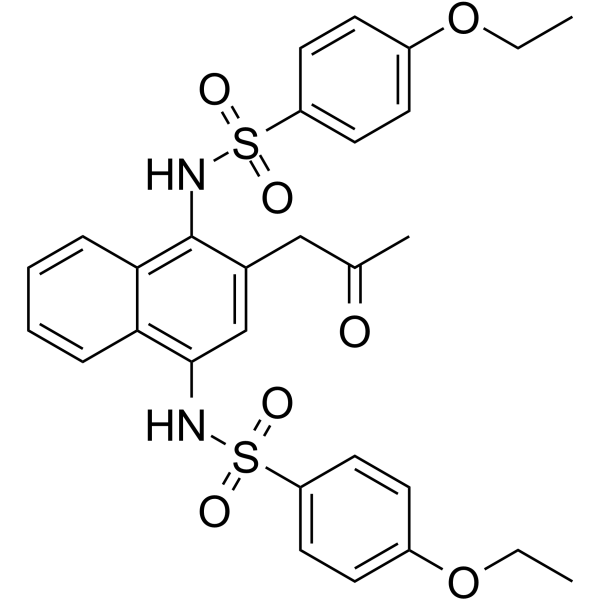
-
GC36390
Keap1-Nrf2-IN-1
Keap1-Nrf2-IN-1 ist ein Protein-Protein-Interaktionsinhibitor von Keap1 (Kelch-like ECH-associated protein 1)-Nrf2 (nuclear factor erythroid 2-related factor 2) und mit einem IC50-Wert von 43 nM fÜr das Keap1-Protein.
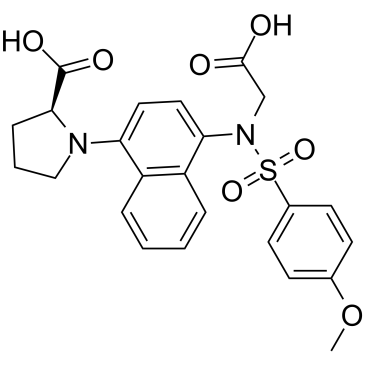
-
GC31321
KI696
KI696 ist eine hochaffine Sonde, die die Keap1/NRF2-Interaktion unterbricht.
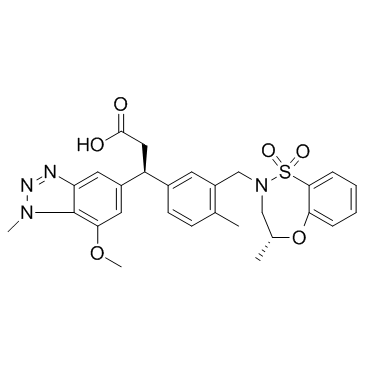
-
GC34642
KI696 isomer
Das KI696-Isomer ist das weniger aktive Isomer von KI696.
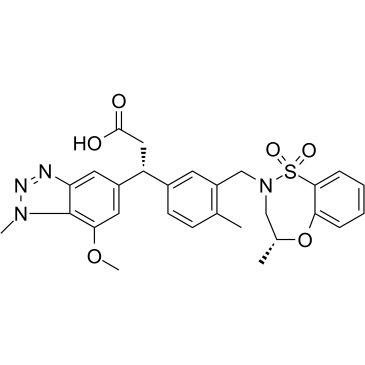
-
GC44006
Kinsenoside
Kinsenosid ist ein aus Pflanzen der Gattung Anoectochilus isolierter Hauptwirkstoff und weist viele biologische AktivitÄten und pharmakologische Wirkungen auf.
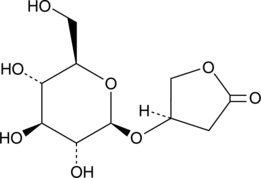
-
GN10491
Mangiferin
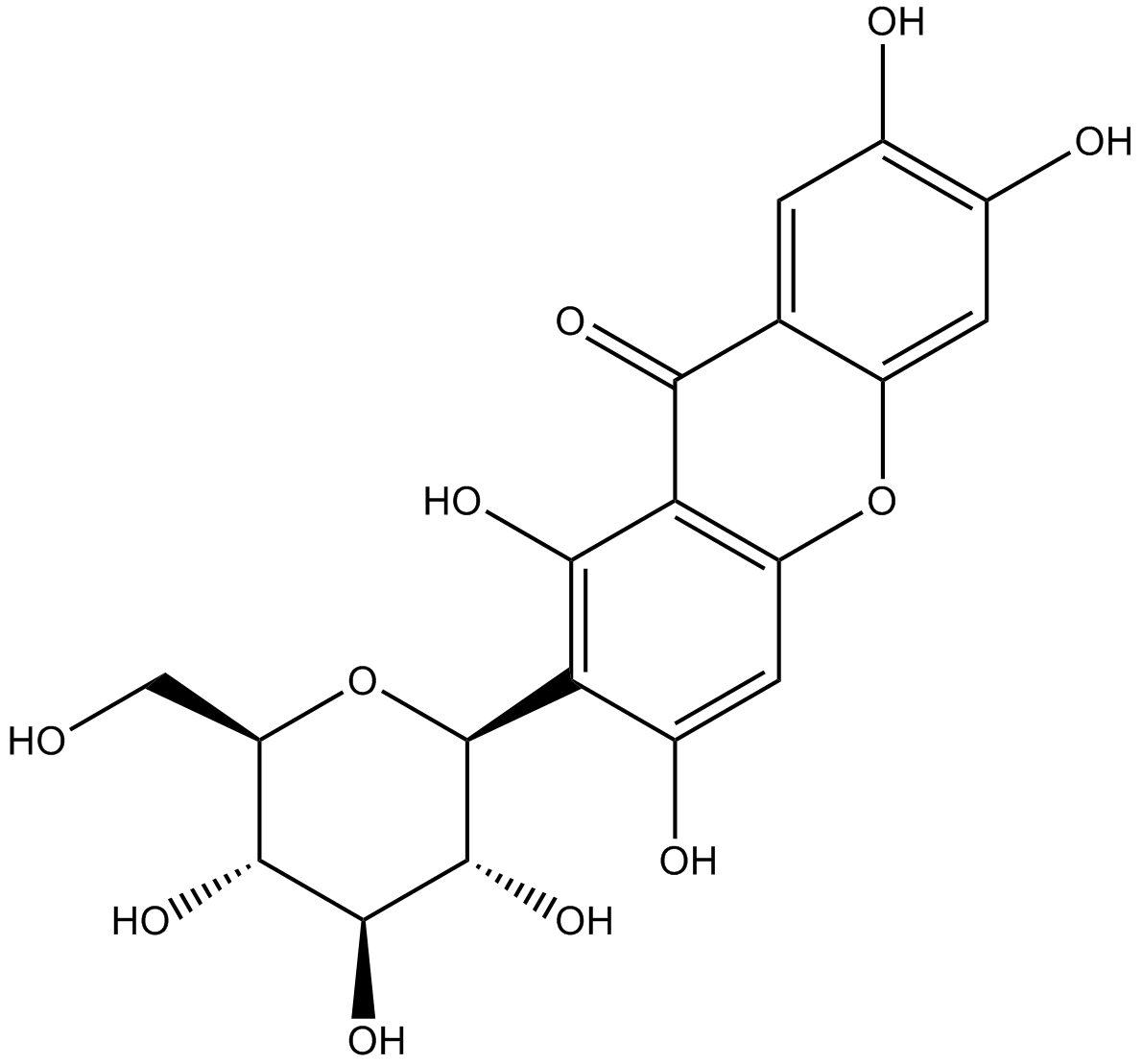
-
GC31783
Methyl 3,4-dihydroxybenzoate (Protocatechuic acid methyl ester)
Methyl-3,4-dihydroxybenzoat (Protocatechinsäuremethylester) (Protocatechinsäuremethylester; Methylprotokatechuat) ist ein wichtiger Metabolit von antioxidativen Polyphenolen, die in grünem Tee gefunden werden.

-
GC38819
ML334
ML334 ist ein potenter, zellgÄngiger Aktivator von NRF2 durch Hemmung der Keap1-NRF2-Protein-Protein-Wechselwirkung. ML334 bindet an die Kelch-DomÄne von Keap1 mit einem Kd von 1 μM. ML334 stimuliert die NRF2-Expression und Kerntranslokation und induziert die AktivitÄt von Antioxidans-Response-Elementen (ARE).
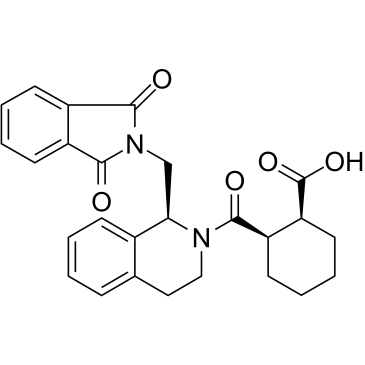
-
GC19254
ML385
ML385 ist ein spezifischer nuklearer Erythroid-2-bezogener Faktor 2 (NRF2)-Inhibitor mit einem IC50-Wert von 1,9 μM.
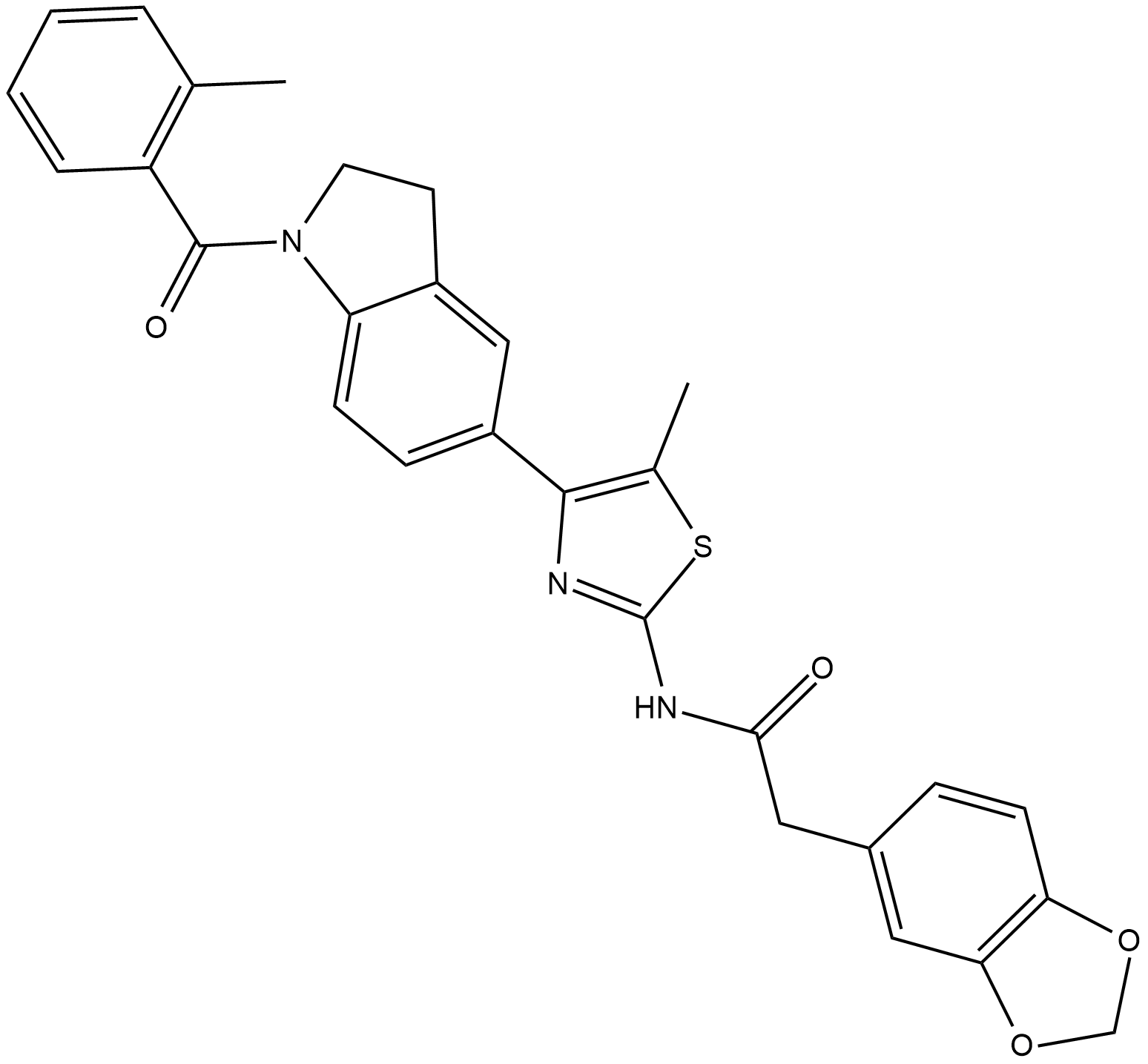
-
GC13058
NK 252
NK 252 ist ein potenzieller Nrf2-Aktivator, der ein großes Nrf2-aktivierendes Potenzial aufweist.
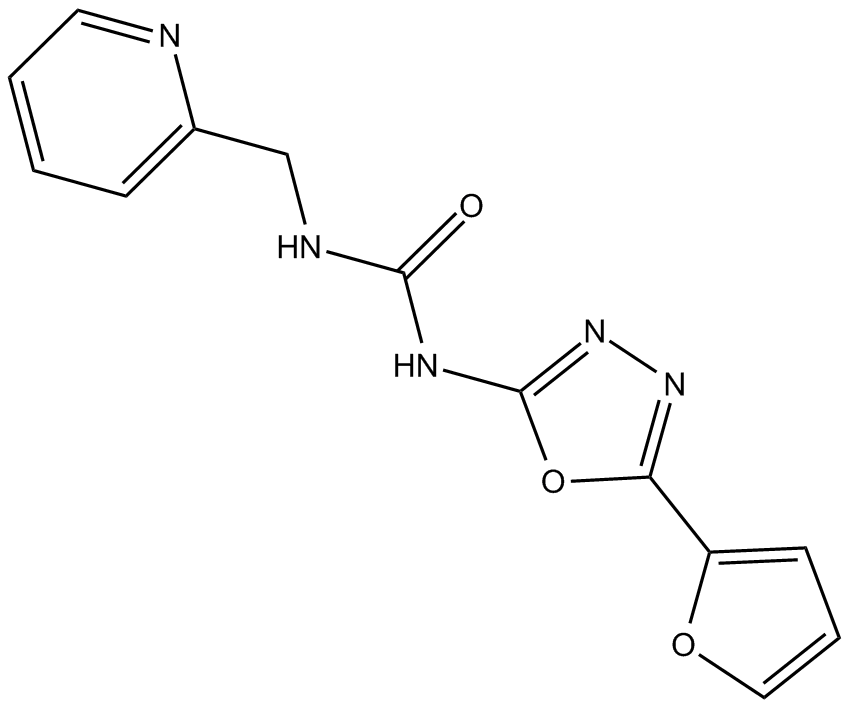
-
GC69590
Nrf2 activator-2
Nrf2-Aktivator-2 (Verbindung O15) is a derivative of sulforaphane, an effective Nrf2 activator with an EC50 of 2.9 μM in 293 T cells. Nrf2 activator-2 effectively inhibits the interaction between Keap1 and Nrf2, thereby showing an activating effect on Nrf2. Nrf2 activator-2 significantly reduces the level of ubiquitinated Nrf2 in cells.
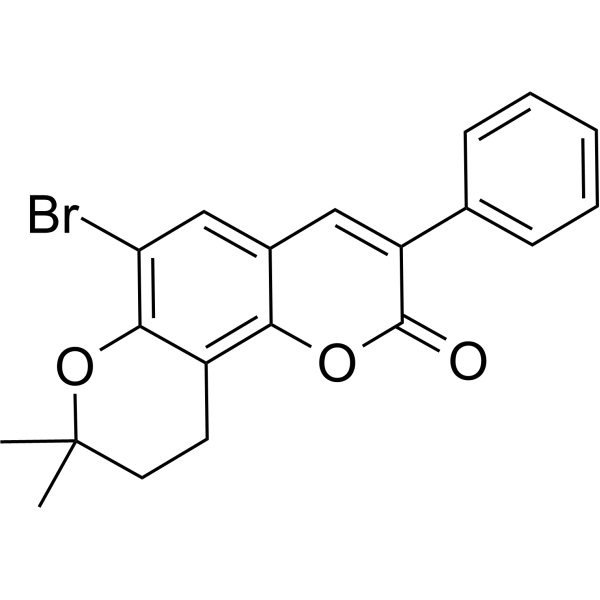
-
GC66054
Nrf2 activator-4
Nrf2-Aktivator-4 (Verbindung 20a) ist ein hochpotenter, oral aktiver Nrf2-Aktivator mit einem EC50-Wert von 0,63 μM. Nrf2-Aktivator-4 unterdrÜckt reaktive Sauerstoffspezies gegen oxidativen Stress in Mikroglia. Nrf2-Aktivator-4 stellt die Lern- und GedÄchtnisbeeintrÄchtigung in einem Scopolamin-induzierten Mausmodell effektiv wieder her.
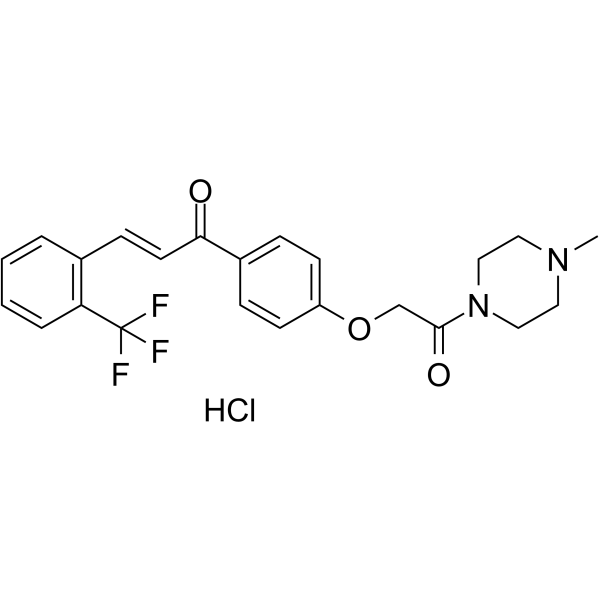
-
GC36773
Nrf2-IN-1
Nrf2-IN-1 ist ein Inhibitor des Kernfaktors Erythroid 2-verwandter Faktor 2 (Nrf2). Nrf2-IN-1 wurde fÜr die Erforschung der akuten myeloischen LeukÄmie (AML) entwickelt.
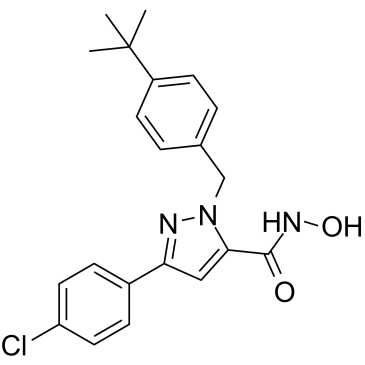
-
GC12821
Oltipraz
Oltipraz hat eine zeitabhÄngige hemmende Wirkung auf die HIF-1α-Aktivierung, wobei die HIF-1α-Induktion bei Konzentrationen von ≥ 10 μM vollstÄndig aufgehoben wird, die IC50 von Oltipraz fÜr die HIF-1α-Hemmung betrÄgt 10 μM. Oltipraz ist ein starker Nrf2-Aktivator.
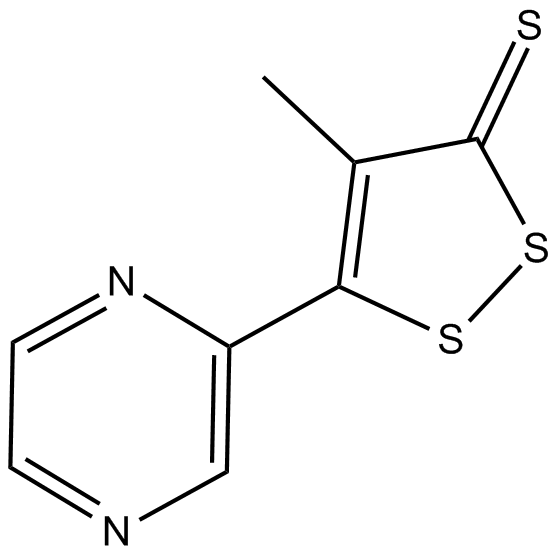
-
GC13693
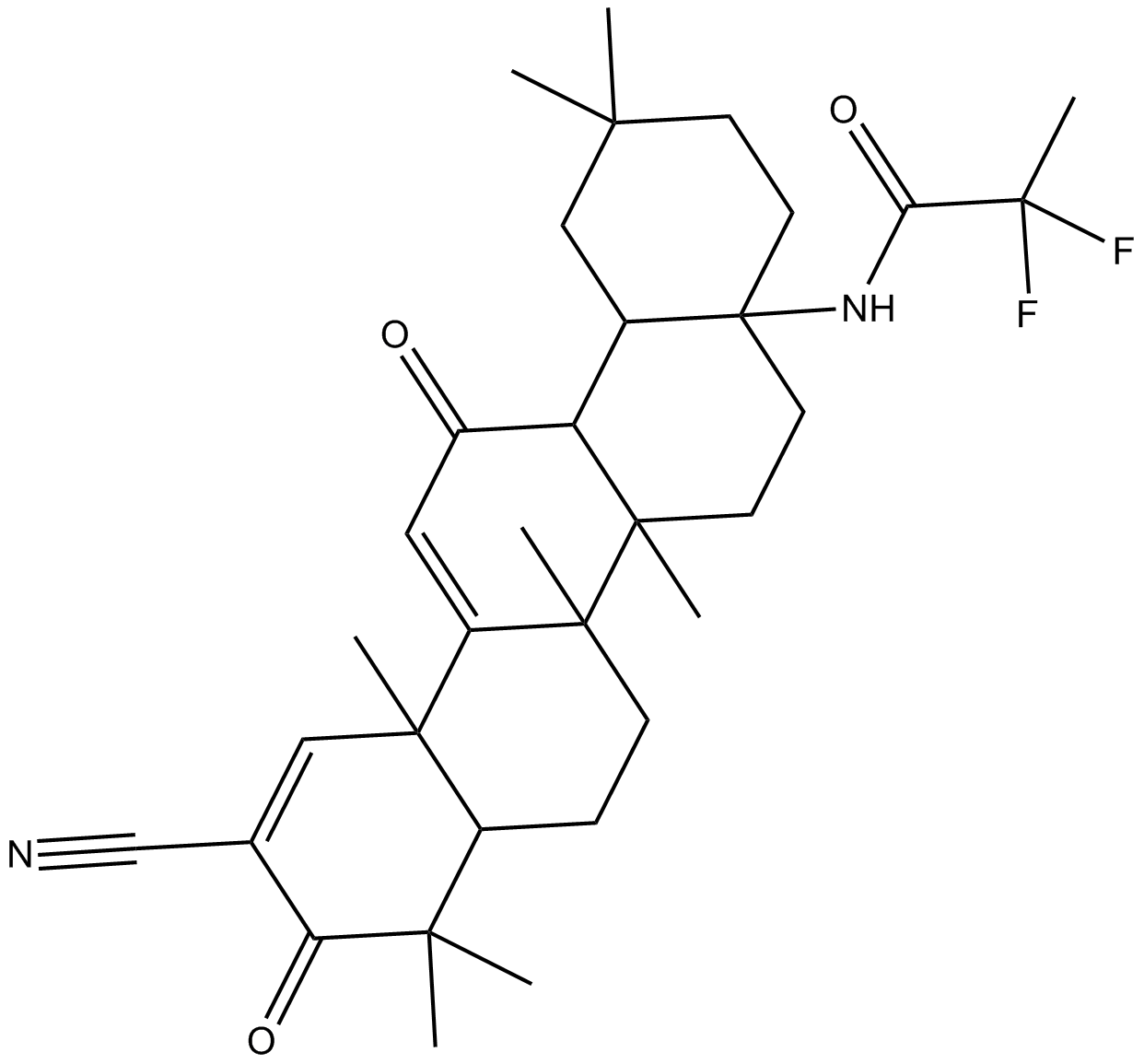
Omaveloxolone (RTA-408)
Omaveloxolon (RTA-408) (RTA 408) ist ein antioxidativer EntzÜndungsmodulator (AIM), der Nrf2 aktiviert und Stickstoffmonoxid (NO) unterdrÜckt.
-
GC15457
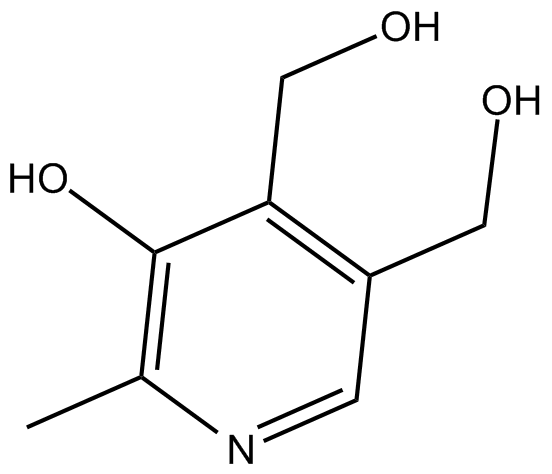
Pyridoxine
Pyridoxin (Pyridoxol) ist ein Pyridinderivat.
-
GC17393
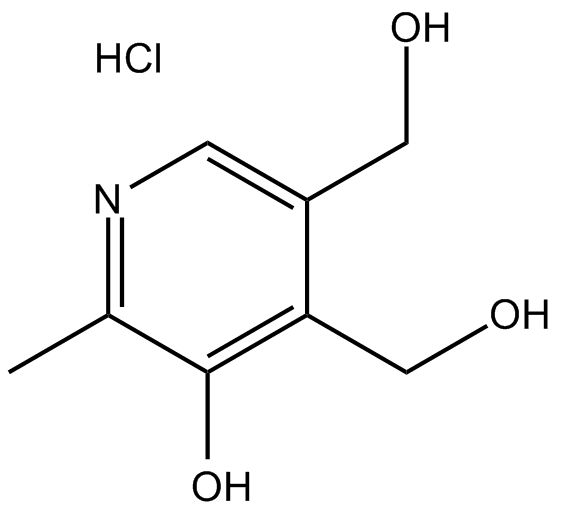
Pyridoxine HCl
Pyridoxin-HCl (Pyridoxol; Vitamin B6) ist ein Pyridin-Derivat.
-
GC50242
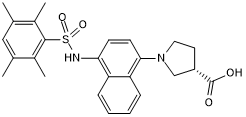
RA 839
Nrf2-Aktivator; hemmt die Nrf2/Keap1-Interaktion.
-
GC63933
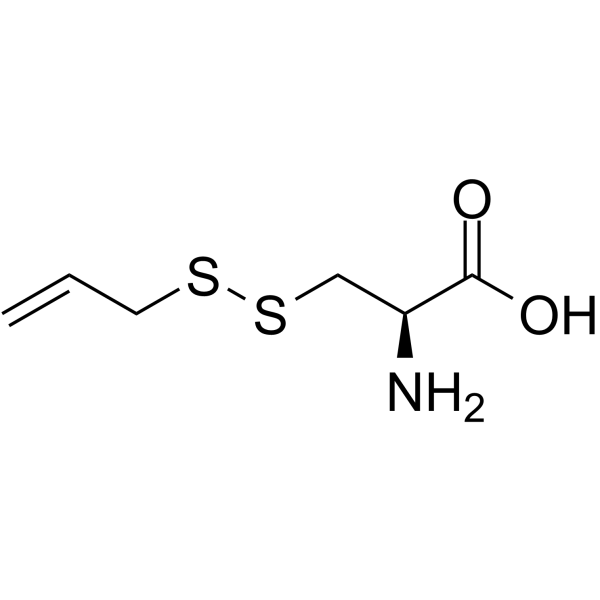
S-Allylmercaptocysteine
S-Allylmercaptocystein, eine aus Knoblauch gewonnene organische Schwefelverbindung, wirkt bei verschiedenen Lungenerkrankungen entzÜndungshemmend und antioxidativ.
-
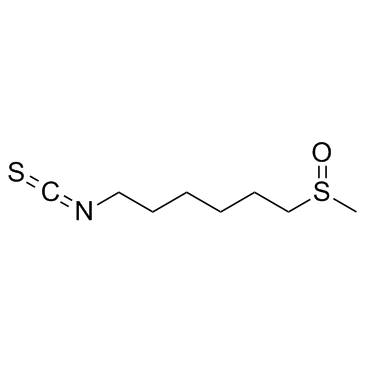
GC14016
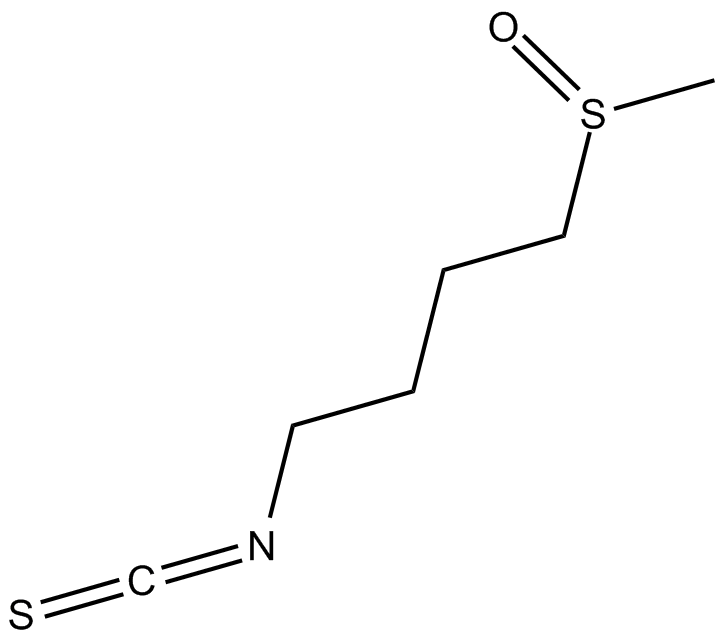
Sulforaphane
Sulforaphan (SFN), auch bekannt als [1-Isocyanato-4-(methylsulfinyl)butan].
-
GC14352
TAT 14
TAT 14 ist ein 14-meres Peptid, das als Nrf2-Aktivator mit entzündungshemmender Wirkung wirkt.

-
GC34057
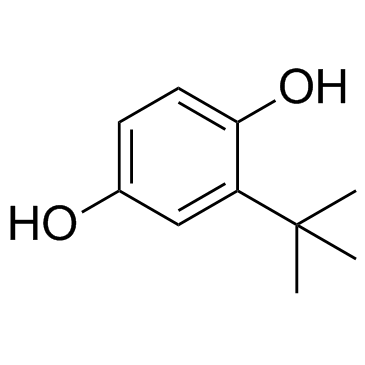
TBHQ (tert-Butylhydroquinone)
TBHQ (tert-Butylhydrochinon) (tert-Butylhydrochinon) ist ein weit verbreiteter Nrf2-Aktivator, der durch Aktivierung von Nrf2 vor Doxorubicin (DOX)-induzierter KardiotoxizitÄt schÜtzt. TBHQ (tert-Butylhydrochinon) (tert-Butylhydrochinon) ist ebenfalls ein ERK-Aktivator; rettet Dehydrocorydalin (DHC)-induzierte Hemmung der Zellproliferation bei Melanomen.
-
GC61338
Toralactone
Toralacton, isoliert aus Cassia obtusifolia, vermittelt den Leberschutz Über einen Nrf2-abhÄngigen antioxidativen Mechanismus.