Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
GC15355
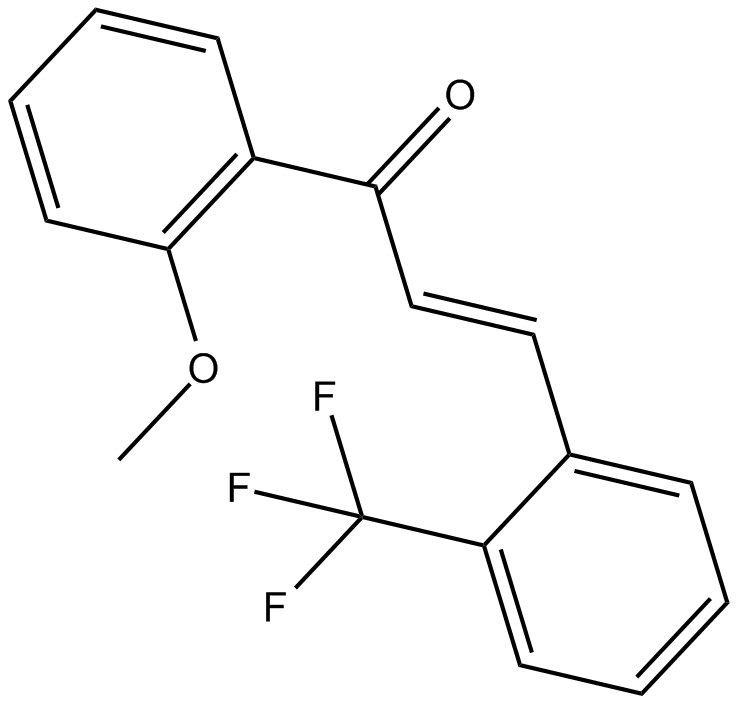
2-Trifluoromethyl-2'-methoxychalcone
Nrf2 activator
-
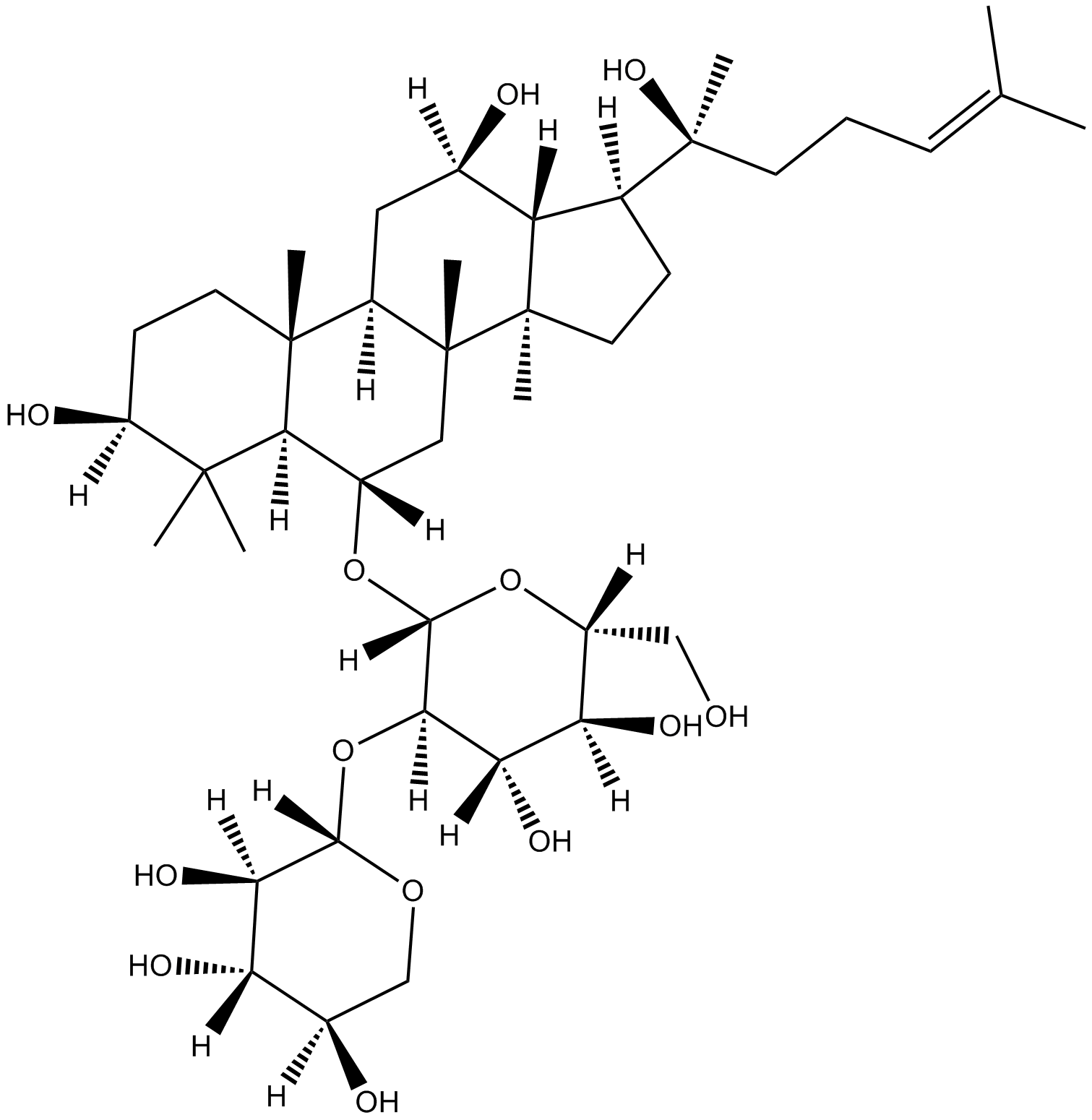
GN10800
20(S)-NotoginsenosideR2
-
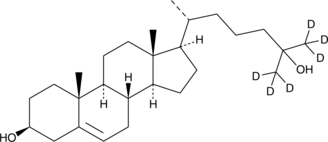
GC46528
25-hydroxy Cholesterol-d6
An internal standard for the quantification of 25hydroxy cholesterol
-
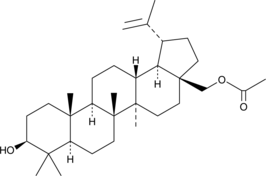
GC48482
28-Acetylbetulin
A lupane triterpenoid with anti-inflammatory and anticancer activities
-
GC35112
3'-Hydroxypterostilbene
Le 3'-hydroxyptérostilbène est un analogue du ptérostilbène. Le 3'-hydroxyptérostilbène inhibe la croissance des cellules COLO 205, HCT-116 et HT-29 avec des CI50 de 9,0, 40,2 et 70,9 μM, respectivement. Le 3'-hydroxyptérostilbène régule À la baisse de manière significative les voies de signalisation PI3K/Akt et MAPK et inhibe efficacement la croissance des cellules cancéreuses du cÔlon humain en induisant l'apoptose et l'autophagie. Le 3'-hydroxyptérostilbène peut être utilisé pour la recherche sur le cancer.
-
GC12791
3,3'-Diindolylmethane
A phytochemical with antiradiation and chemopreventative effects

-
GC42237
3,5-dimethyl PIT-1
PtdIns-(3,4,5)-P3 (PIP3) serves as an anchor for the binding of signal transduction proteins bearing pleckstrin homology (PH) domains such as phosphatidylinositol 3-kinase (PI3K) or PTEN.
-
GC64762
3,6-Dihydroxyflavone
La 3,6-dihydroxyflavone est un agent anticancéreux. La 3,6-dihydroxyflavone diminue en fonction de la dose et du temps la viabilité cellulaire et induit l'apoptose en activant la cascade de caspases, en clivant la poly (ADP-ribose) polymérase (PARP). La 3,6-dihydroxyflavone augmente le stress oxydatif intracellulaire et la peroxydation lipidique.
-
GC46583
3-Amino-2,6-Piperidinedione
An active metabolite of (±)-thalidomide
-
GC49849
3-Aminosalicylic Acid
A salicylic acid derivative
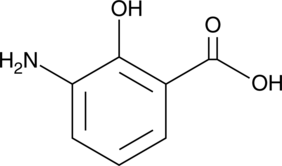
-
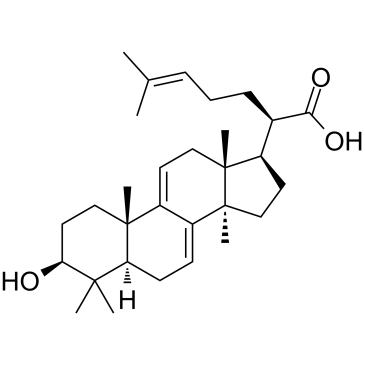
GC35106
3-Dehydrotrametenolic acid
L'acide 3-déhydrotraméténolique, isolé du sclérote de Poria cocos, est un inhibiteur de la lactate déshydrogénase (LDH). L'acide 3-déhydrotraméténolique favorise la différenciation adipocytaire in vitro et agit comme un sensibilisateur À l'insuline in vivo. L'acide 3-déhydrotraméténolique induit l'apoptose et possède une activité anticancéreuse.
-
GC68537
3-IN-PP1
3-IN-PP1 est un inhibiteur de la protéine kinase D (PKD). Il présente une activité d'inhibition efficace et étendue sur PKD1, PKD2 et PKD3, avec des valeurs IC50 respectives de 108, 94 et 108 nM. 3-IN-PP1 est également un agent anticancéreux à large spectre qui a un effet inhibiteur sur la croissance de diverses cellules cancéreuses. Il peut être utilisé dans la recherche sur le cancer.
-
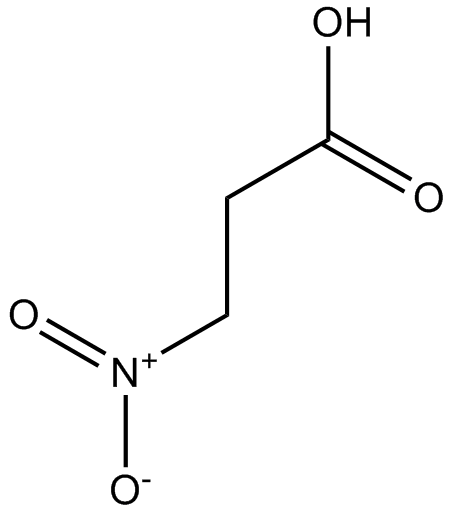
GC17394
3-Nitropropionic acid
L'acide 3-nitropropionique (β-acide nitropropionique) est un inhibiteur irréversible de la succinate déshydrogénase.
-
GC35099
3-O-Acetyloleanolic acid
L'acide 3-O-acétyloléanolique (3AOA), un dérivé de l'acide oléanolique isolé des graines de Vigna sinensis K., induit le cancer et présente également une activité anti-angiogenèse.
-
GC60507
3-O-Methylgallic acid
L'acide 3-O-méthylgallique (acide 3,4-dihydroxy-5-méthoxybenzoÏque) est un métabolite anthocyanique et possède une puissante capacité antioxydante. L'acide 3-O-méthylgallique inhibe la prolifération des cellules Caco-2 avec une valeur IC50 de 24,1 μM. L'acide 3-O-méthylgallique induit également l'apoptose cellulaire et a des effets anticancéreux.
-
GC32767
3BDO
3BDO est un nouvel activateur mTOR qui peut également inhiber l'autophagie.
-
GC45354
4β-Hydroxywithanolide E
4β-Hydroxywithanolide E, isolé de Physalis peruviana L.
-
GC48437
4'-Acetyl Chrysomycin A
A bacterial metabolite with antibacterial and anticancer activities
-
GC42346
4-bromo A23187
Le 4-Bromo A23187 est un analogue halogéné de l'ionophore calcique hautement sélectif A-23187.
-
GC42401
4-hydroperoxy Cyclophosphamide
Un analogue activé de la cyclophosphamide.
-
GC30896
4-Hydroxybenzyl alcohol
L'alcool 4-hydroxybenzylique est un composé phénolique largement répandu dans divers types de plantes.
-
GC33815
4-Hydroxyphenylacetic acid
L'acide 4-hydroxyphénylacétique, un métabolite majeur des polyphénols issu du microbiote, est impliqué dans l'action antioxydante.
-
GC35138
4-Methyldaphnetin
La 4-méthyldaphnétine est un précurseur dans la synthèse des dérivés de la 4-méthyl coumarine. La 4-méthyldaphnétine a des effets anti-prolifératifs et inducteurs d'apoptose puissants et sélectifs sur plusieurs lignées cellulaires cancéreuses. La 4-méthyldaphnétine possède une propriété de piégeage des radicaux et inhibe fortement la peroxydation des lipides membranaires.
-
GC68231
4-Methylsalicylic acid

-
GC31648
4-Octyl Itaconate
Le 4-octyl itaconate (4-OI) est un dérivé d'itaconate perméable aux cellules. L'itaconate et le 4-octyl itaconate ont une réactivité similaire avec les thiols, ce qui fait du 4-octyl itaconate un substitut approprié pour étudier sa fonction biologique.
-
GC49127
4-oxo Cyclophosphamide
An inactive metabolite of cyclophosphamide
-
GC45352
4-oxo Withaferin A
La withaferine A 4-oxo est l'analogue de la withaferine A. La withaferine A est un withanolide isolé de Withania somnifera. 4-oxo Withaferin A a le potentiel pour la recherche sur le myélome multiple.
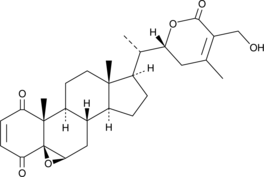
-
GC45353
4-oxo-27-TBDMS Withaferin A
La 4-oxo-27-TBDMS Withaferin A, un dérivé de la withaferine A, présente de puissants effets antiprolifératifs sur les cellules tumorales. La 4-oxo-27-TBDMS Withaferin A induit l'apoptose des cellules tumorales. 4-oxo-27-TBDMS Withaferin A est un agent anticancéreux.
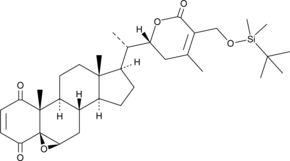
-
GC60525
4-Vinylphenol (10%w/w in propylene glycol)
Le 4-vinylphénol se trouve dans l'herbe médicinale Hedyotis diffusa Willd, le riz sauvage et est également le métabolite de l'acide p-coumarique et férulique par les bactéries lactiques du vin. Le 4-vinylphénol induit l'apoptose et inhibe la formation de vaisseaux sanguins et supprime la croissance invasive des tumeurs mammaires in vivo.
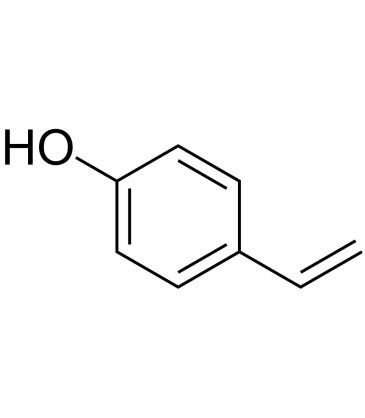
-
GC10468
4EGI-1
4EGI-1 est un inhibiteur de l'interaction eIF4E/eIF4G, avec un Kd de 25 μM contre la liaison eIF4E.
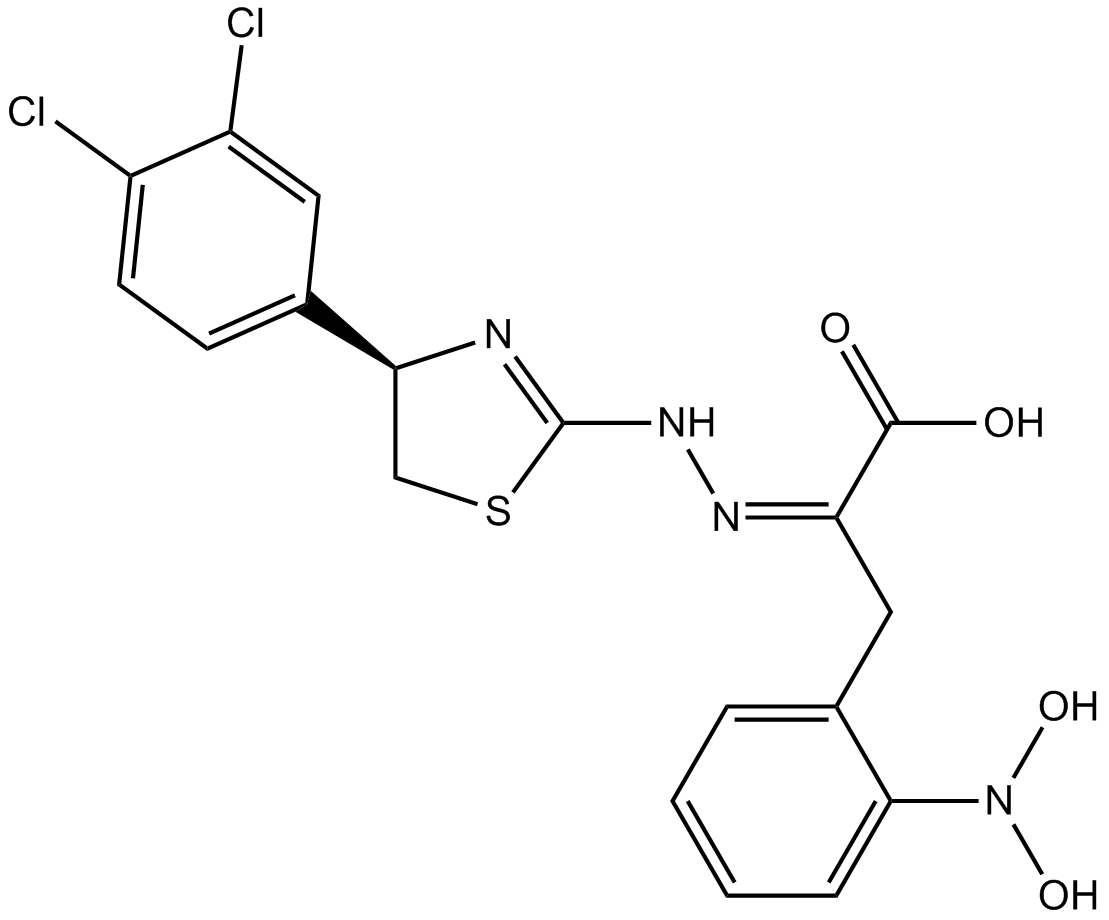
-
GC35150
5,7,4'-Trimethoxyflavone
La 5,7,4'-triméthoxyflavone est isolée de Kaempferia parviflora (KP) qui est une célèbre plante médicinale de Thaïlande. La 5,7,4'-triméthoxyflavone induit l'apoptose, comme en témoignent les augmentations de la phase sous-G1, la fragmentation de l'ADN, la coloration annexine-V/PI, le rapport Bax/Bcl-xL, l'activation protéolytique de la caspase-3 et la dégradation de la poly (ADP-ribose) polymérase (PARP). La 5,7,4'-triméthoxyflavone est significativement efficace pour inhiber la prolifération des cellules cancéreuses gastriques humaines SNU-16 d'une manière dépendante de la concentration.
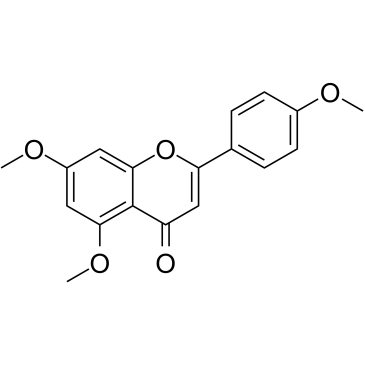
-
GN10629
5,7-dihydroxychromone
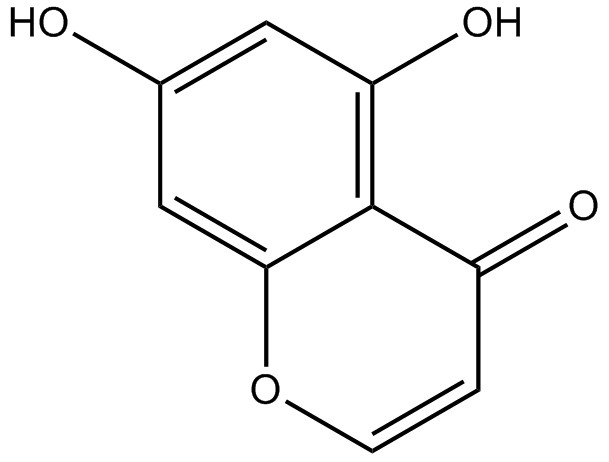
-
GC63972
5,7-Dimethoxyflavanone
La 5,7-diméthoxyflavanone montre une puissante activité antimutagène contre la mutagenèse MeIQ dans le test d'Ames utilisant le S.
-
GC52227
5-(3',4'-Dihydroxyphenyl)-γ-Valerolactone
An active metabolite of various polyphenols
-
GC35147
5-(N,N-Hexamethylene)-amiloride
Le 5-(N,N-Hexaméthylène)-amiloride (Hexaméthylène amiloride) dérive d'un amiloride et est un puissant inhibiteur de l'échangeur Na+/H+, qui diminue le pH intracellulaire (pHi) et induit l'apoptose dans les leucémies. cellules.
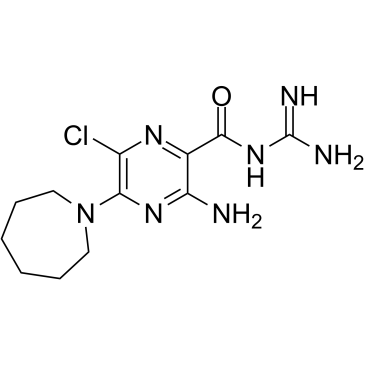
-
GC45356
5-Aminolevulinic Acid (hydrochloride)
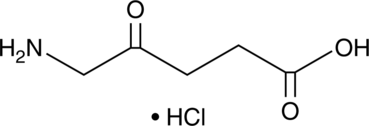
-
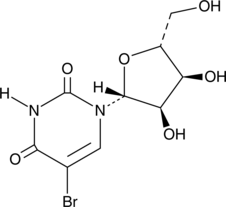
GC46681
5-Bromouridine
A brominated uridine analog
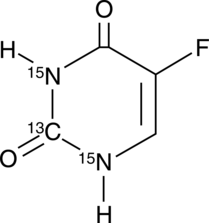
-
GC42545
5-Fluorouracil-13C,15N2
5-Fluorouracil-13C,15N2 is intended for use as an internal standard for the quantification of 5-flurouracil by GC- or LC-MS.
-
GC46705
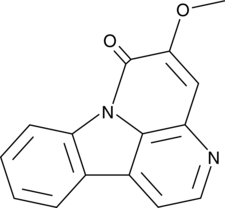
5-Methoxycanthinone
La 5-méthoxycanthinone est un inhibiteur actif par voie orale des souches de Leishmania.
-
GC42586
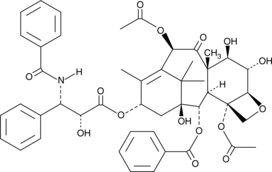
6α-hydroxy Paclitaxel
6α-hydroxy Paclitaxel est un métabolite primaire du Paclitaxel. 6α-hydroxy Le paclitaxel conserve un effet dépendant du temps sur les polypeptides transporteurs d'anions organiques 1B1/SLCO1B1 (OATP1B1) avec une puissance d'inhibition similaire À celle du paclitaxel, alors qu'il n'a plus montré d'inhibition dépendante du temps de l'OATP1B3. 6α-hydroxy Paclitaxel peut être utilisé pour la recherche sur le cancer.
-
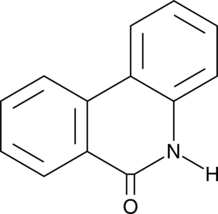
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
-
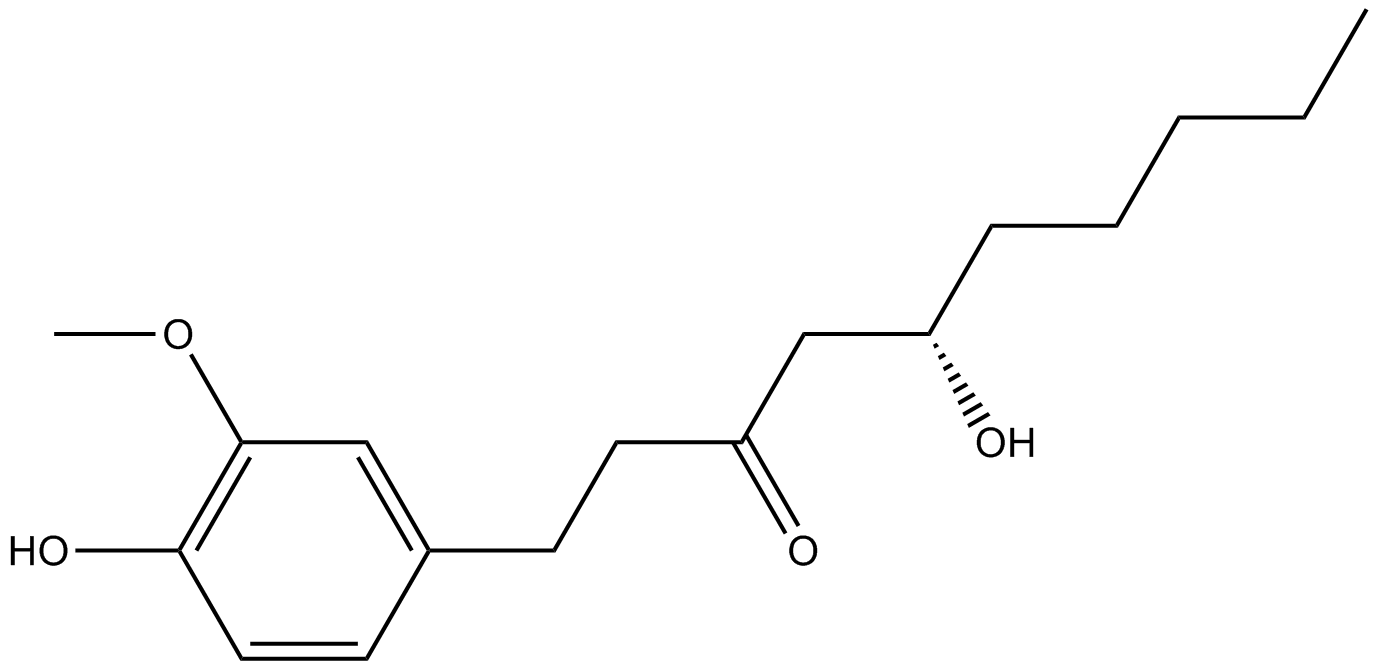
GN10093
6-gingerol
-
GC49429
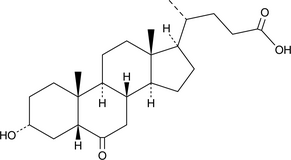
6-keto Lithocholic Acid
A metabolite of lithocholic acid
-
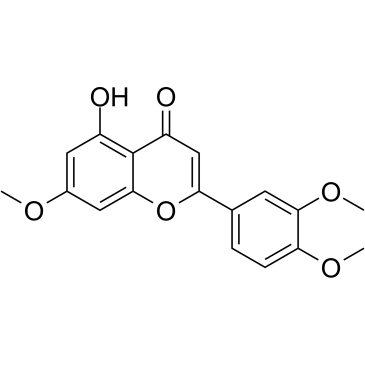
GC35184
7,3',4'-Tri-O-methylluteolin
La 7,3',4'-tri-O-méthyllutéoline (5-hydroxy-3',4',7-triméthoxyflavone), un composé flavonoÏde, possède de puissants effets anti-inflammatoires dans la lignée cellulaire de macrophages induite par le LPS médiée par l'inhibition de libération de médiateurs inflammatoires, NO, PGE2 et cytokines pro-inflammatoires.
-
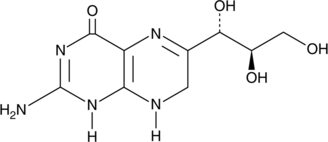
GC45673
7,8-Dihydroneopterin
La 7,8-dihydronéoptérine, un marqueur de l'inflammation, induit l'apoptose cellulaire dans les astrocytes et les neurones via l'amélioration de l'expression de l'oxyde nitrique synthase (iNOS).
-
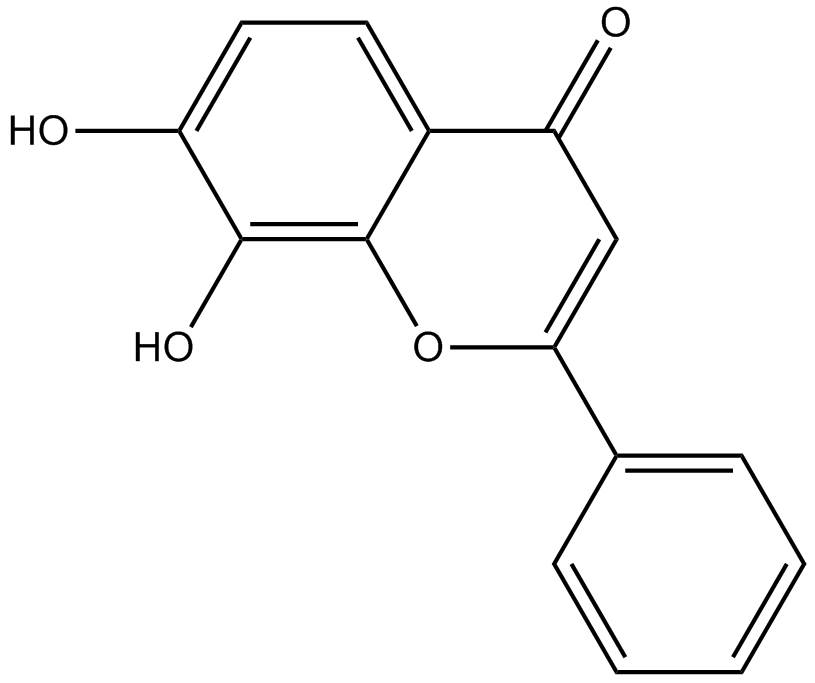
GC16853
7,8-Dihydroxyflavone
La 7,8-dihydroxyflavone est un agoniste puissant et sélectif de TrkB qui imite les actions physiologiques du facteur neurotrophique dérivé du cerveau (BDNF).
-
GC42616
7-oxo Staurosporine
7-oxo Staurosporine is an antibiotic originally isolated from S.
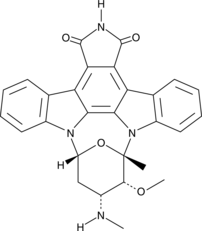
-
GC16037
7BIO
7BIO (7-Bromoindirubin-3-Oxime) est le dérivé de l'indirubine.
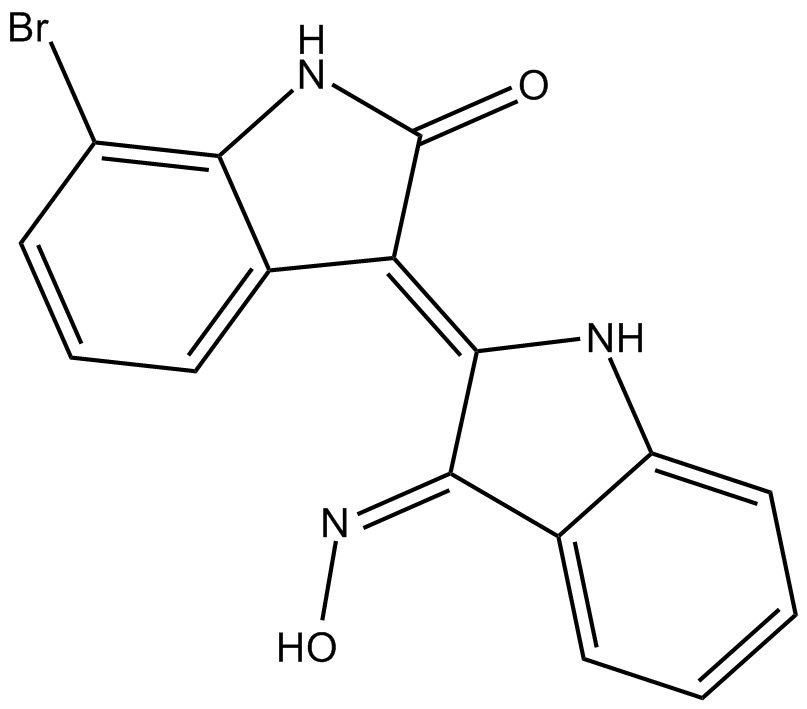
-
GC46741
8(E),10(E),12(Z)-Octadecatrienoic Acid
A conjugated PUFA

-
GC42622
8-bromo-Cyclic AMP
8-bromo-Cyclic AMP is a brominated derivative of cAMP that remains long-acting due to its resistance to degradation by cAMP phosphodiesterase.
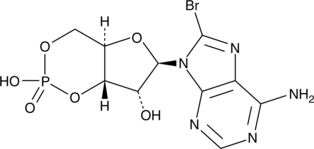
-
GC49275
8-Oxycoptisine
La 8-oxycoptisine est un alcaloÏde protoberbérine naturel À activité anticancéreuse.
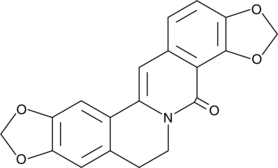
-
GC17119
8-Prenylnaringenin
La 8-prénylnaringénine est un prénylflavonoïde isolé des cônes de houblon (Humulus lupulus), avec une cytotoxicité. La 8-prénylnaringénine a une activité anti-proliférative contre les cellules cancéreuses du côlon HCT-116 via l'induction de l'apoptose médiée par les voies intrinsèques et extrinsèques. La 8-prénylnaringénine favorise également la récupération de l'atrophie musculaire induite par l'immobilisation par l'activation de la voie de phosphorylation Akt chez la souris .
-
GC41642
9(E),11(E),13(E)-Octadecatrienoic Acid
9(E),11(E),13(E)-Octadecatrienoic acid (β-ESA) is a conjugated polyunsaturated fatty acid that is found in plant seed oils and in mixtures of conjugated linolenic acids synthesized by the alkaline isomerization of linolenic acid.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
GC40785
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC40710
9(Z),11(E),13(E)-Octadecatrienoic Acid methyl ester
9Z,11E,13E-octadecatrienoic acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC39152
9-ING-41
Le 9-ING-41 est un inhibiteur sélectif et compétitif de la glycogène synthase kinase-3β (GSK-3β) À base de maléimide avec une IC50 de 0,71 μM. 9-ING-41 conduit de manière significative À l'arrêt du cycle cellulaire, À l'autophagie et À l'apoptose dans les cellules cancéreuses. 9-ING-41 a une activité anticancéreuse et a le potentiel d'améliorer les effets antitumoraux des médicaments chimiothérapeutiques.
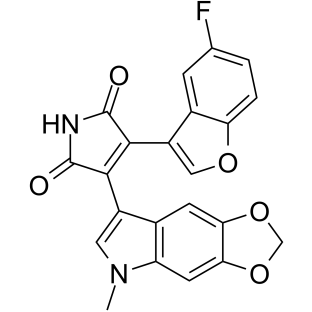
-
GN10035
9-Methoxycamptothecin
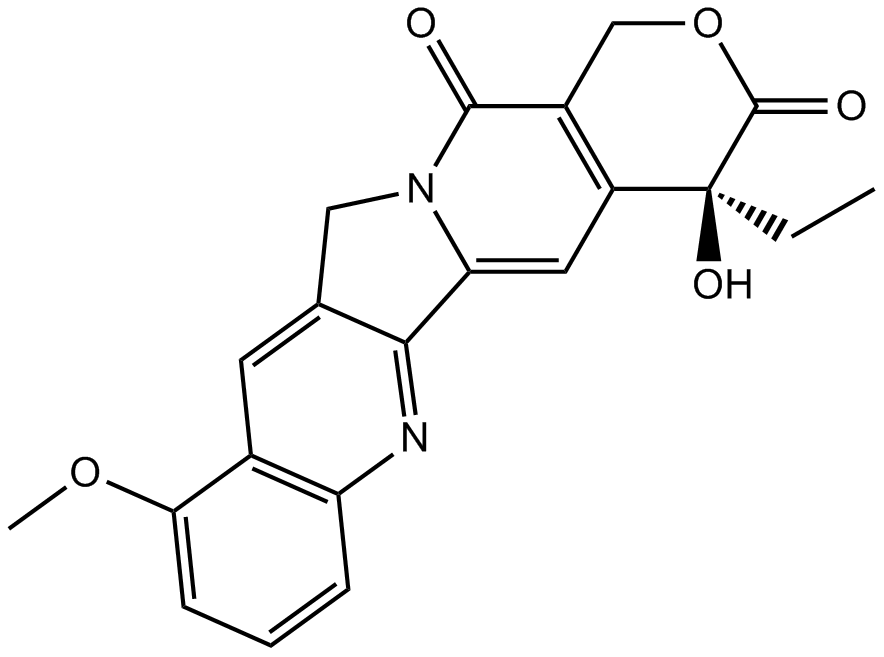
-
GC45960
9c(i472)
9c(i472) est un puissant inhibiteur de 15-LOX-1 (15-lipoxygénase-1) avec une valeur IC50 de 0,19 μM.
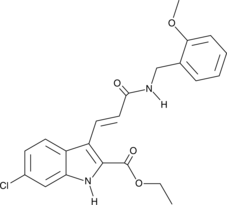
-
GC50465
A 410099.1
High affinity XIAP antagonist; active in vivo
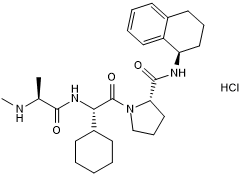
-
GC17512
A-1155463
A-1155463 est un inhibiteur BCL-XL très puissant et sélectif avec une CE50 de 70 nM dans la cellule Molt-4.
-
GC16278
A-1210477
A-1210477 est un inhibiteur puissant et sélectif de MCL-1 avec un Ki de 0,45 nM. A-1210477 se lie spécifiquement À MCL-1 et favorise l'apoptose des cellules cancéreuses de manière dépendante de MCL-1.
-
GC17513
A-1331852
A-1331852 est un inhibiteur sélectif de BCL-XL disponible par voie orale avec un Ki inférieur À 10 pM.
-
GC60544
A-192621
A-192621 est un antagoniste des récepteurs de l'endothéline B (ETB) puissant, non peptidique, actif par voie orale et sélectif avec une IC50 de 4,5 nM et un Ki de 8,8 nM.
-
GC32981
A-385358
A-385358 est un inhibiteur sélectif de Bcl-XL avec Kis de 0,80 et 67 nM pour Bcl-XL et Bcl-2, respectivement.
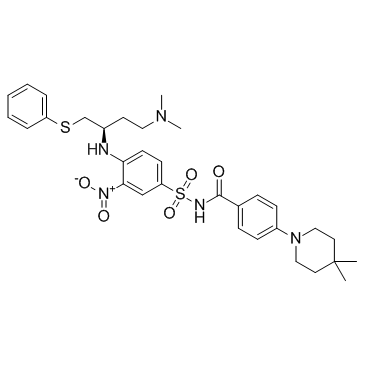
-
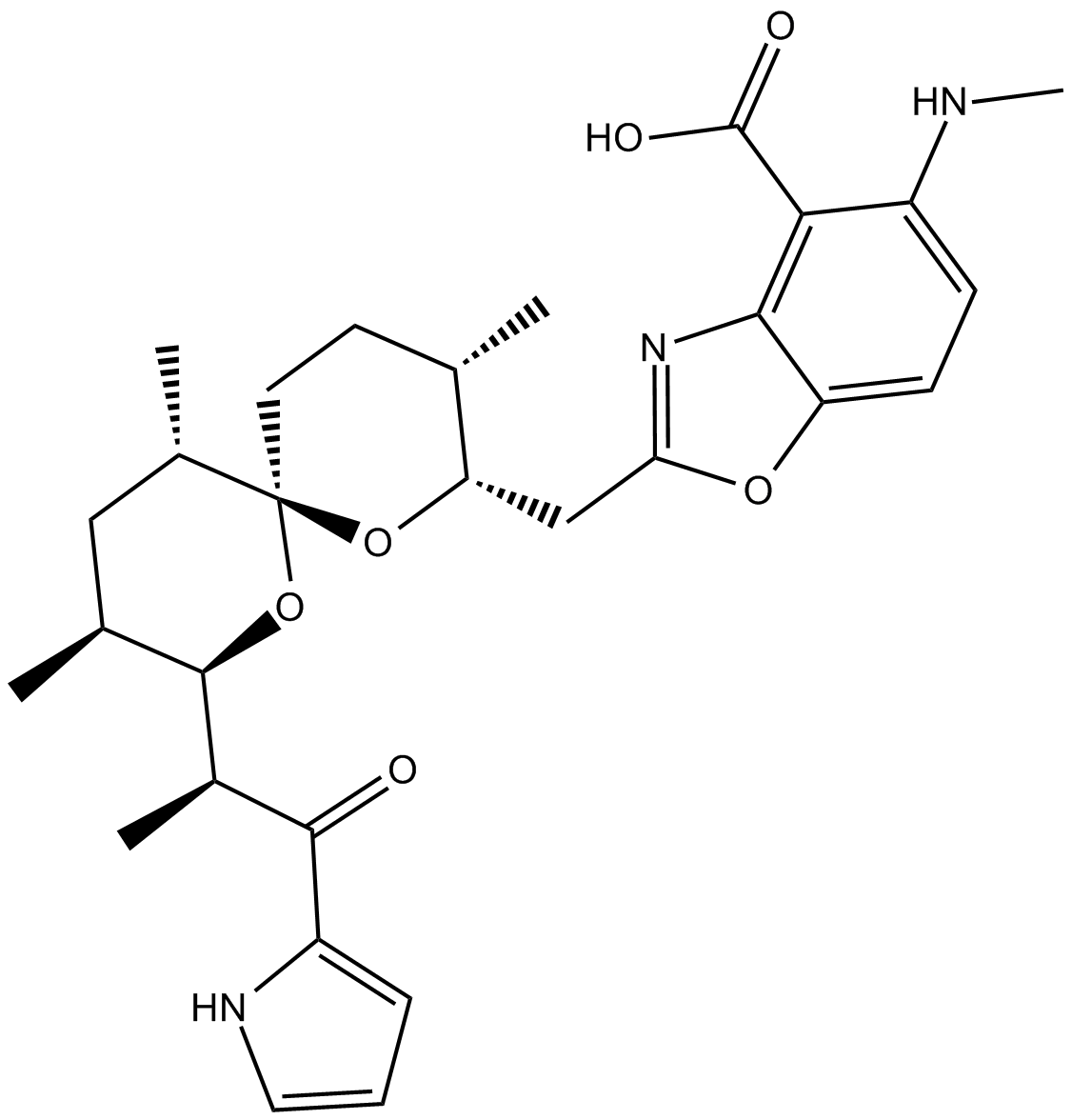
GC11200
A23187
A23187 (A-23187) est un antibiotique et un ionophore de cations divalents unique (comme le calcium et le magnésium).
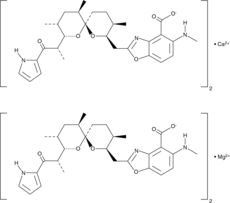
-
GC42659
A23187 (calcium magnesium salt)
A23187 is a divalent cation ionophore.
-
GC35216
AAPK-25
AAPK-25 est un double inhibiteur Aurora/PLK puissant et sélectif avec une activité anti-tumorale, qui peut provoquer un retard mitotique et arrêter les cellules dans une prométaphase, reflétée par la phosphorylation de l'histone H3Ser10 du biomarqueur et suivie d'une augmentation de l'apoptose. AAPK-25 cible Aurora-A, -B et -C avec des valeurs de Kd allant de 23 À 289 nM, ainsi que PLK-1, -2 et -3 avec des valeurs de Kd allant de 55 À 456 nM.
-
GC13805
Abacavir
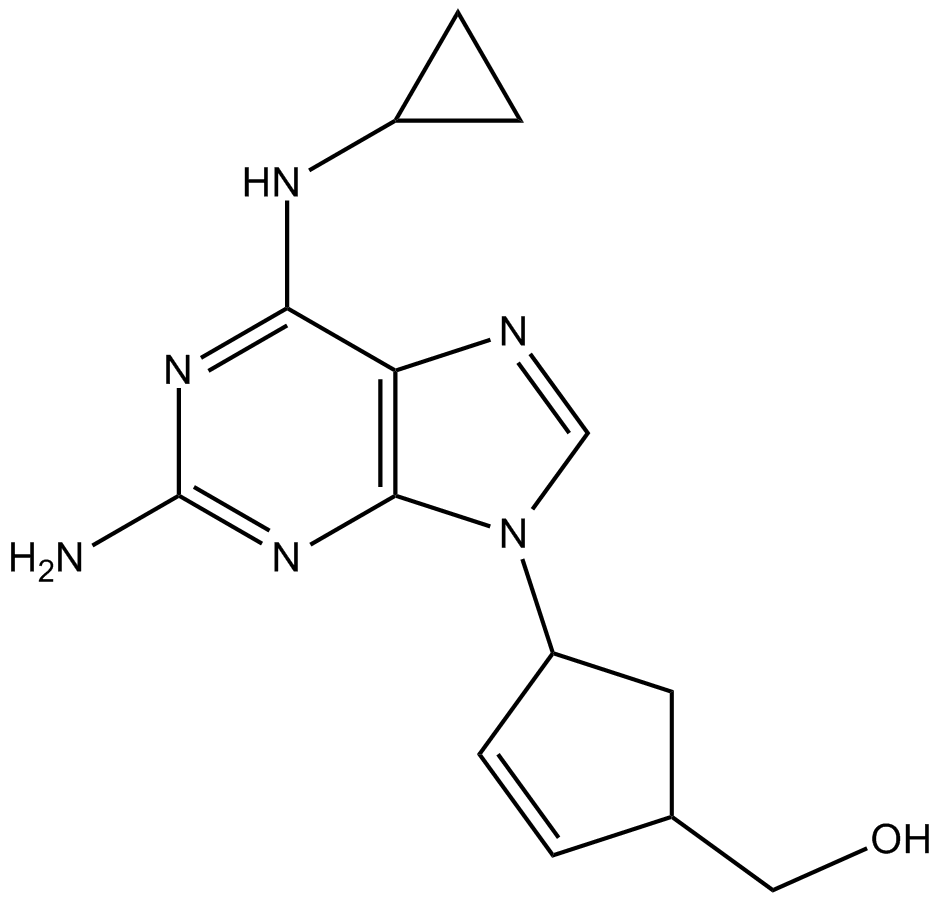
-
GC64674
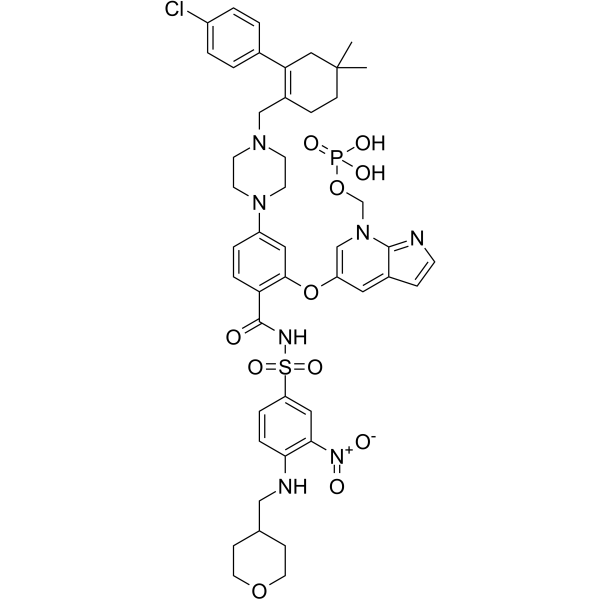
ABBV-167
ABBV-167 est un promédicament phosphate du vénétoclax inhibiteur de BCL-2.
-
GC60548
ABT-100
ABT-100 est un inhibiteur de farnésyltransférase puissant, hautement sélectif et actif par voie orale. ABT-100 inhibe la prolifération cellulaire (IC50 de 2,2 nM, 3,8 nM, 5,9 nM, 6,9 nM, 9,2 nM, 70 nM et 818 nM pour EJ-1, DLD-1, MDA-MB-231, HCT-116, MiaPaCa- 2, PC-3 et cellules DU-145, respectivement), augmente l'apoptose et diminue l'angiogenèse. ABT-100 possède une activité antitumorale À large spectre.
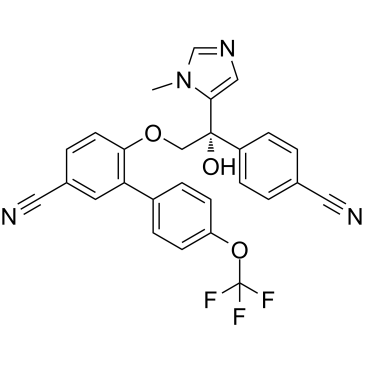
-
GC14069
ABT-199
Un inhibiteur de Bcl-2
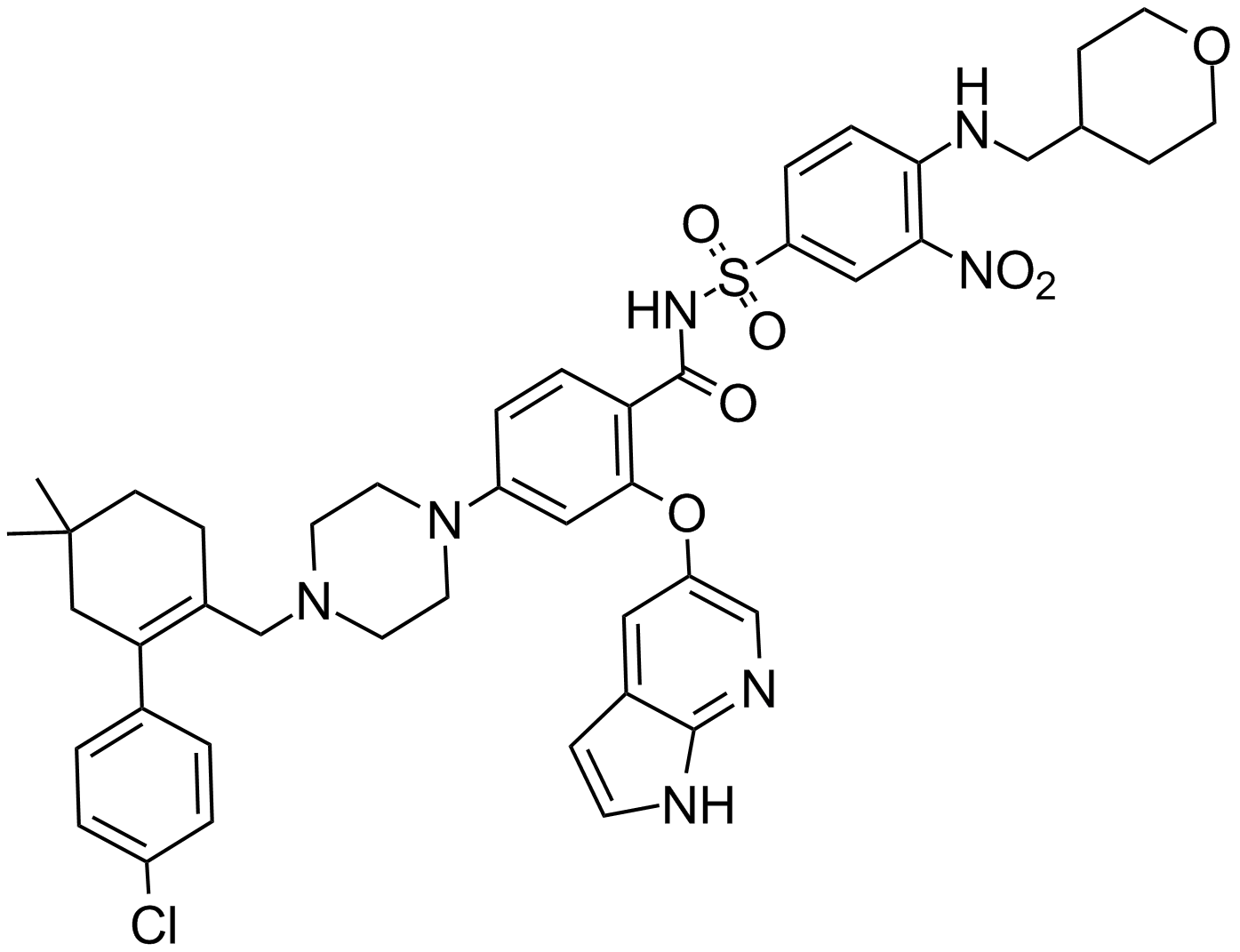
-
GC12405
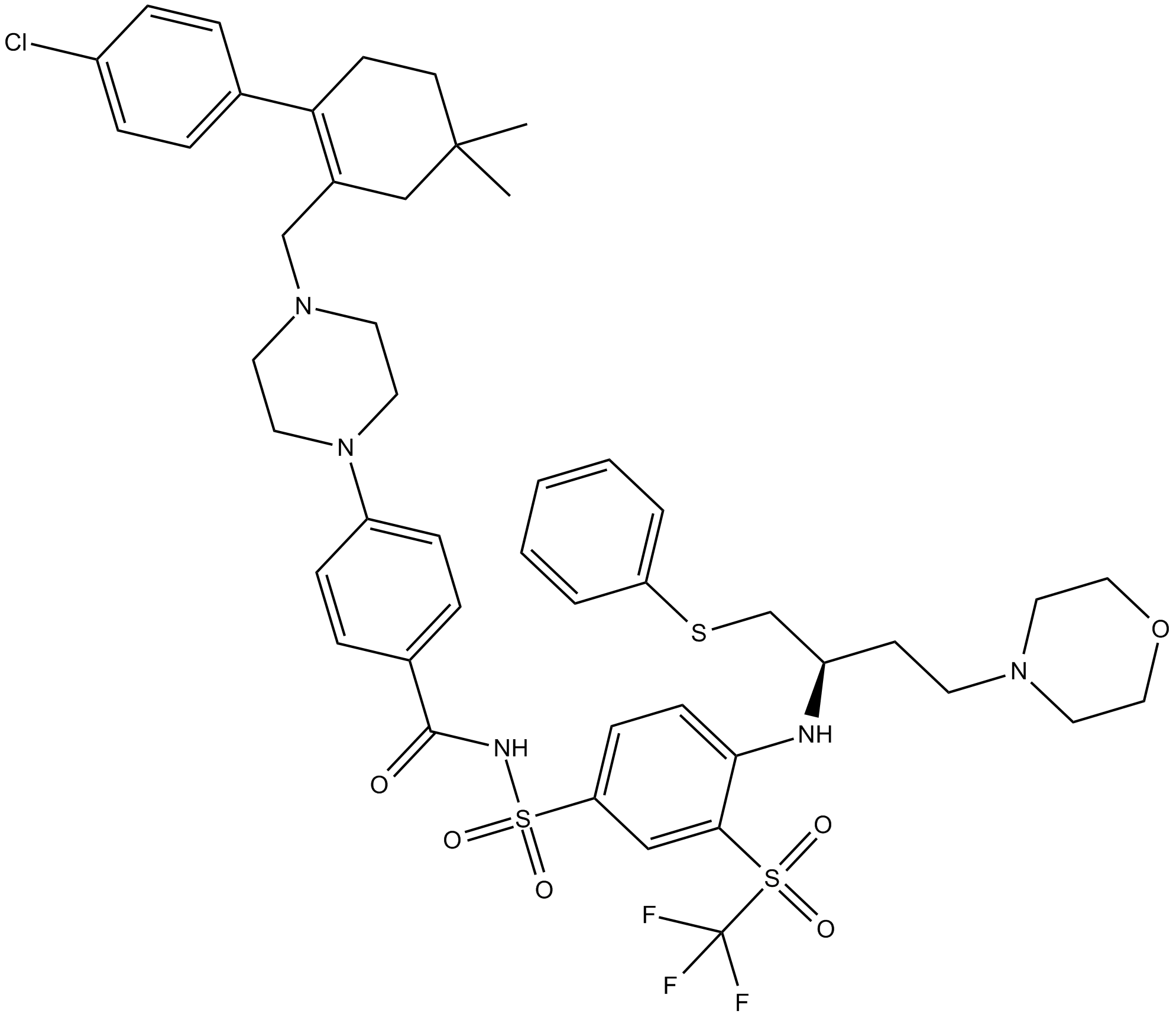
ABT-263 (Navitoclax)
Un inhibiteur des protéines de la famille Bcl-2.
-
GC49745
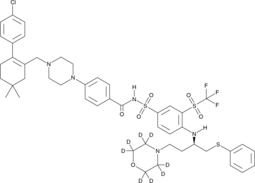
ABT-263-d8
ABT-263-d8 est le deutérium marqué Navitoclax. Navitoclax (ABT-263) est un inhibiteur de la protéine de la famille Bcl-2 puissant et actif par voie orale qui se lie À plusieurs protéines anti-apoptotique de la famille Bcl-2, telles que Bcl-xL, Bcl-2 et Bcl-w, avec un Ki de moins supérieure À 1 nM.
-
GC17234
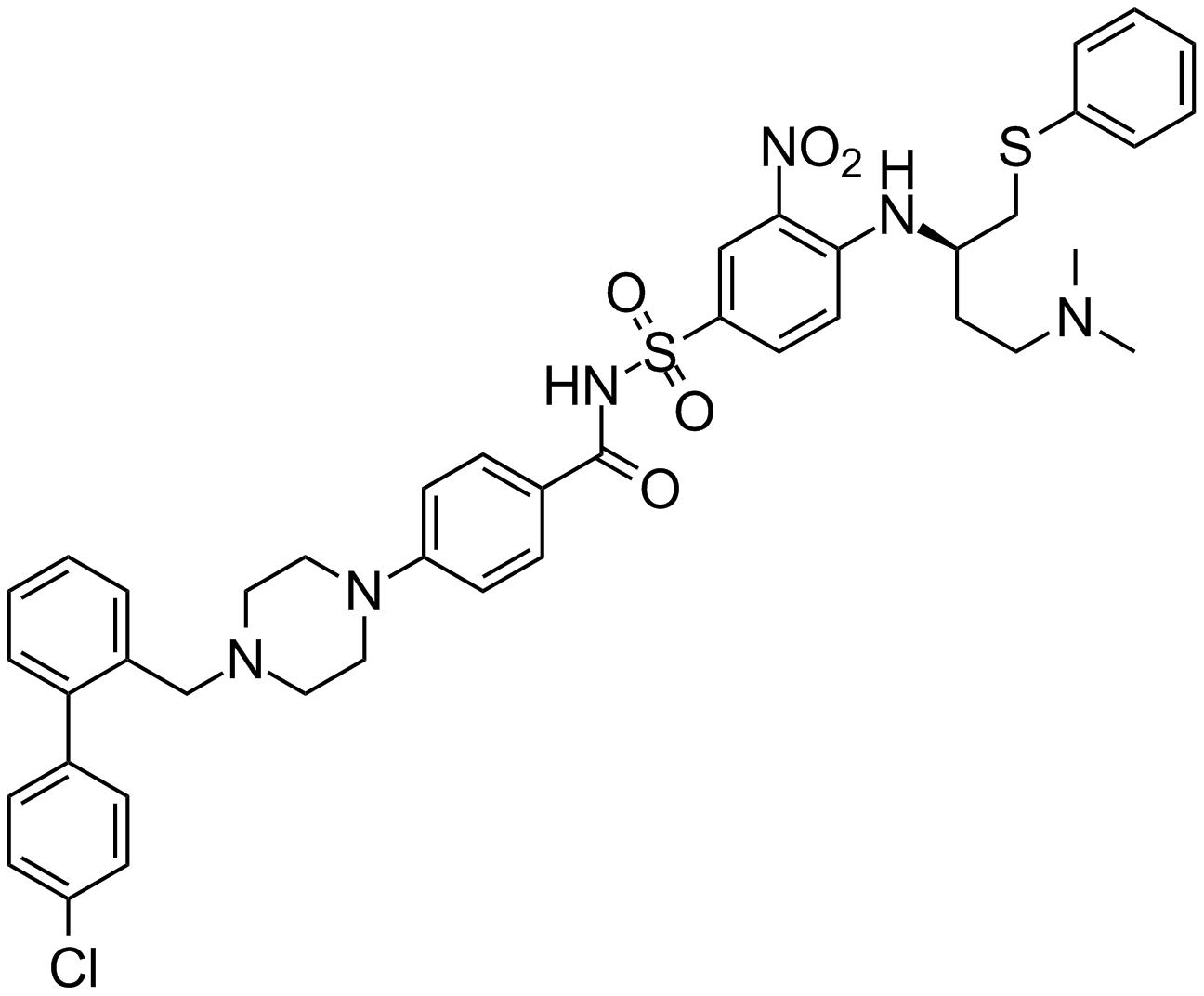
ABT-737
An inhibitor of anti-apoptotic Bcl-2 proteins
-
GA20494
Ac-Asp-Glu-Val-Asp-pNA
The cleavage of the chromogenic caspase-3 substrate Ac-DEVD-pNA can be monitored at 405 nm.

-
GC17602
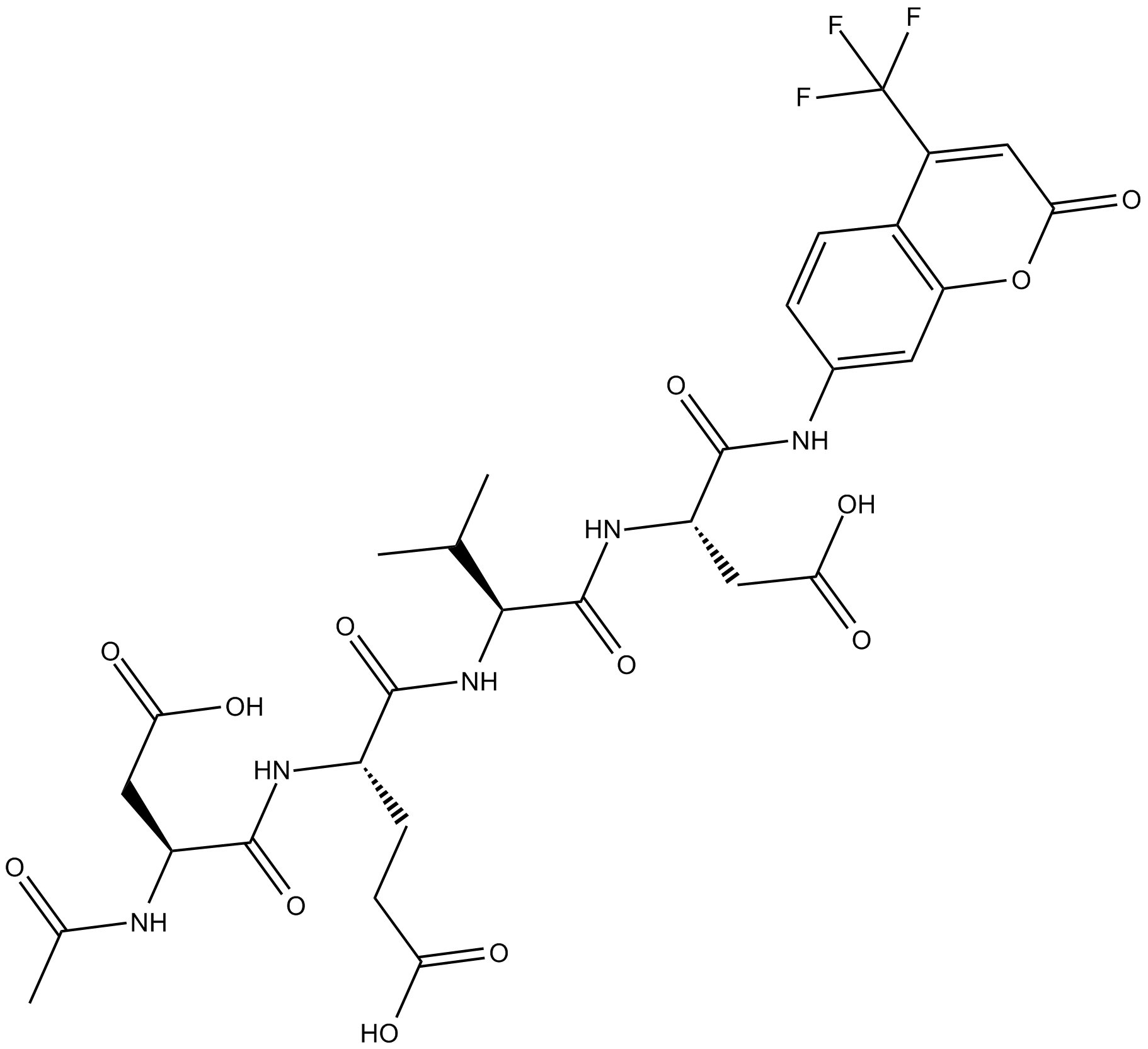
Ac-DEVD-AFC
Ac-DEVD-AFC est un substrat fluorogène (Λex = 400 nm, Λem = 530 nm).
-
GC32695
Ac-DEVD-CHO
Ac-DEVD-CHO est un inhibiteur spécifique de la caspase-3 avec une valeur Ki de 230 pM.
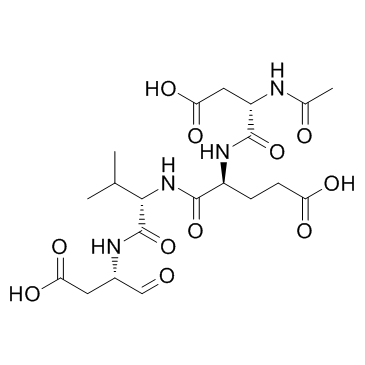
-
GC48470
Ac-DEVD-CHO (trifluoroacetate salt)
A dual caspase3/caspase7 inhibitor
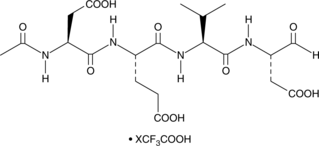
-
GC10951
Ac-DEVD-CMK
cell-permeable, and irreversible inhibitor of caspase
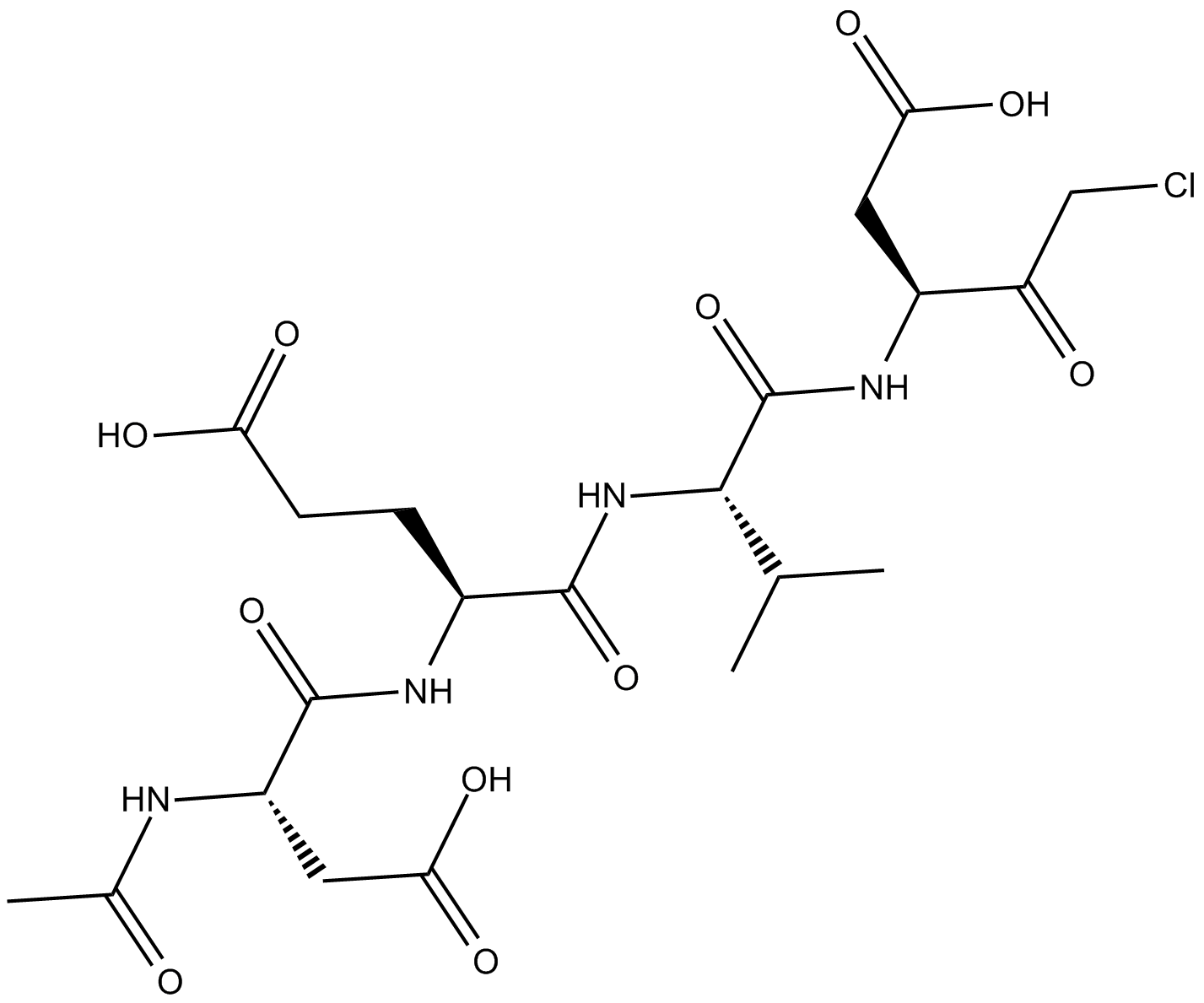
-
GC42689
Ac-DNLD-AMC
Ac-WLA-AMC est un substrat fluorogène de la caspase-3.
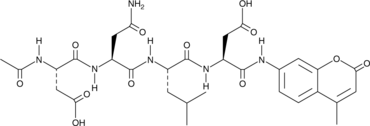
-
GC65107
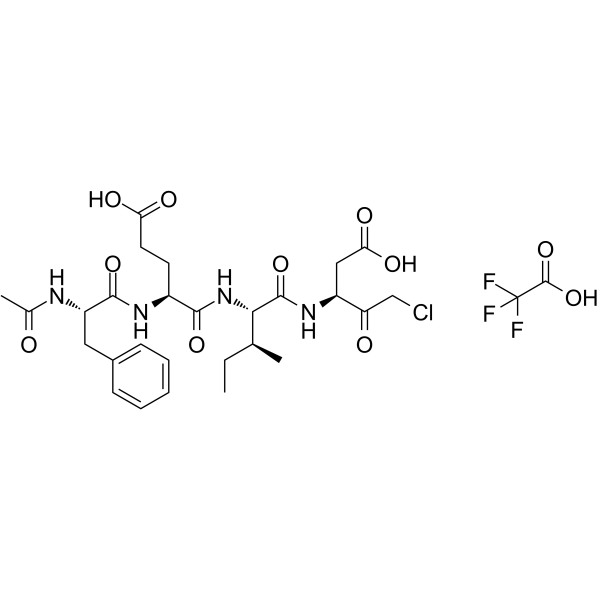
Ac-FEID-CMK TFA
Ac-FEID-CMK TFA est un puissant inhibiteur peptidique dérivé de GSDMEb spécifique au poisson zèbre.
-
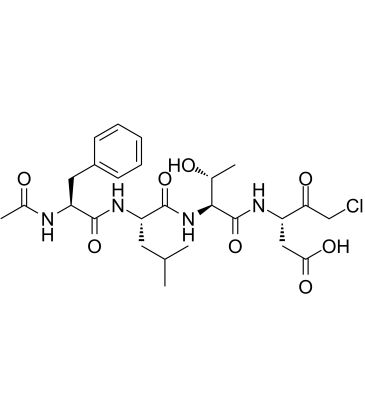
GC60558
Ac-FLTD-CMK
Ac-FLTD-CMK, un inhibiteur dérivé de la gasdermine D (GSDMD), est un inhibiteur spécifique des caspases inflammatoires.
-
GC49704
Ac-FLTD-CMK (trifluoroacetate salt)
An inhibitor of caspase-1, -4, -5, and -11

-
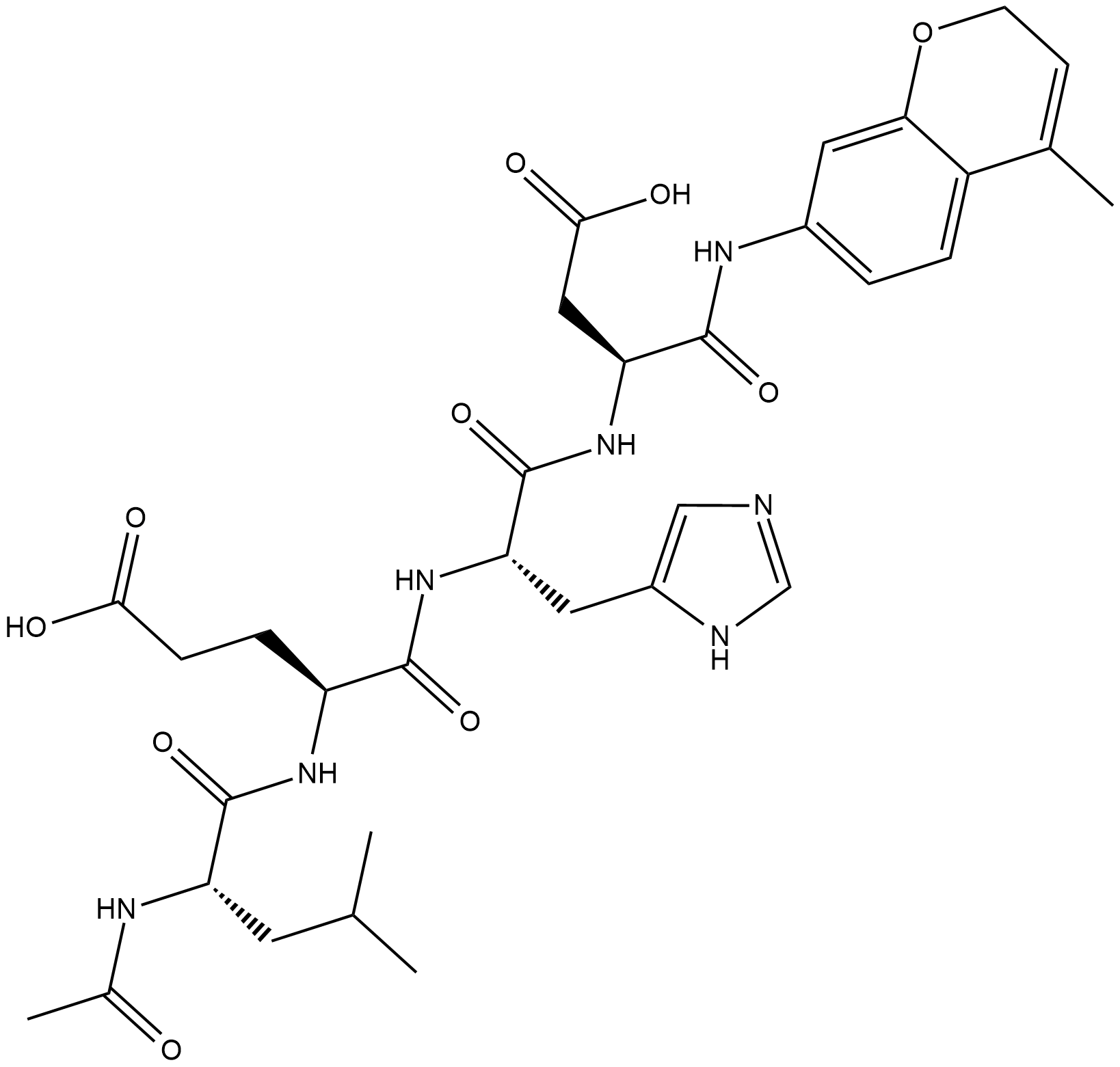
GC18226
Ac-LEHD-AMC (trifluoroacetate salt)
Ac-LEHD-AMC (sel de trifluoroacétate) est un substrat fluorogène pour la caspase-9 (Excitation : 341 nm ; Emission : 441 nm).
-
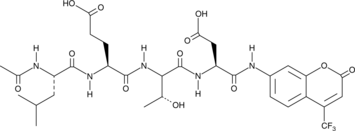
GC40556
Ac-LETD-AFC
Ac-LETD-AFC est un substrat fluorogène de la caspase-8.
-
GC13400
Ac-VDVAD-AFC
Ac-VDVAD-AFC est un substrat fluorescent spécifique À la caspase. Ac-VDVAD-AFC peut mesurer l'activité de type caspase-3 et l'activité caspase-2 et peut être utilisé pour la recherche sur les tumeurs et le cancer.
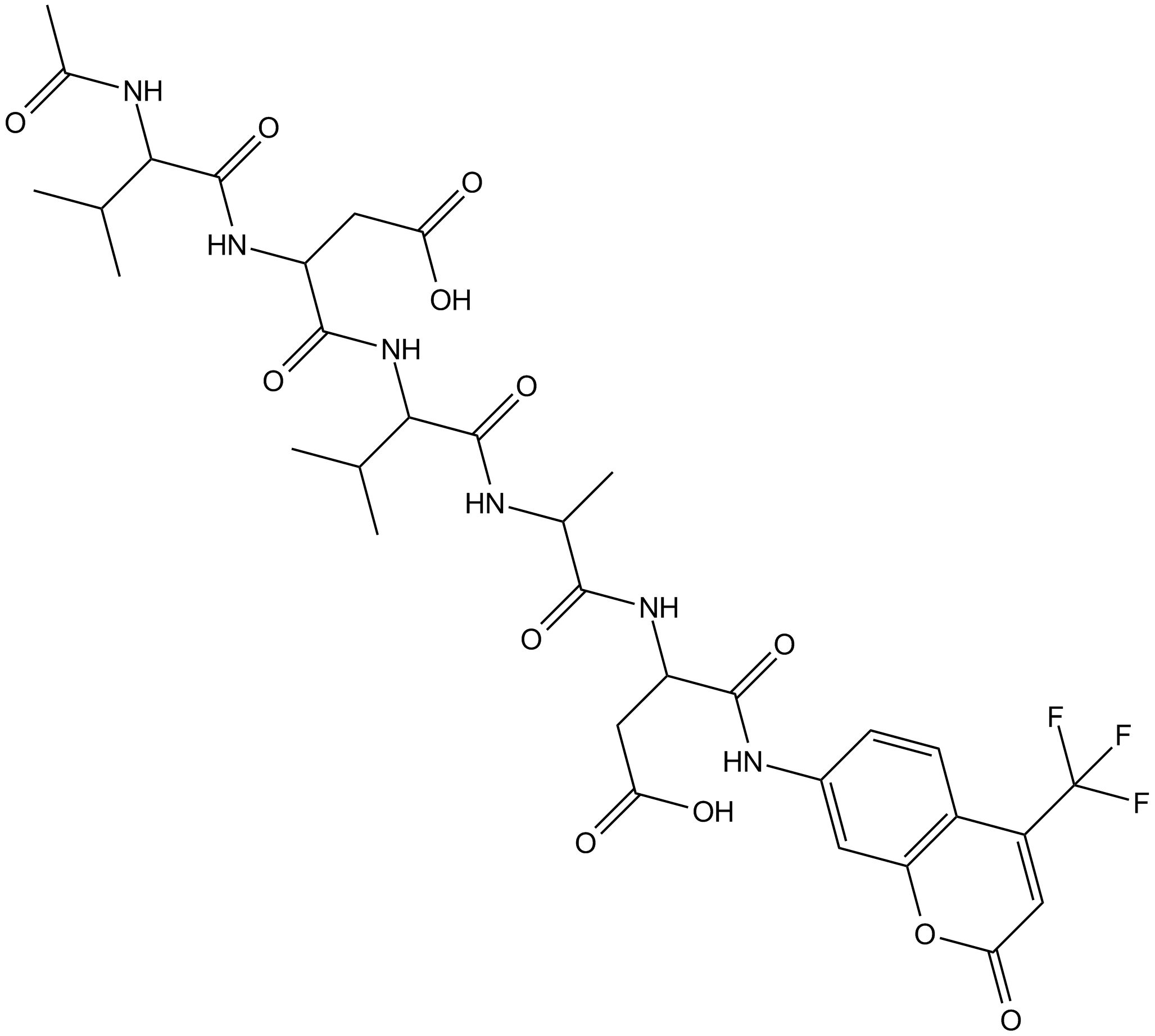
-
GC52372
Ac-VDVAD-AFC (trifluoroacetate salt)
A fluorogenic substrate for caspase-2
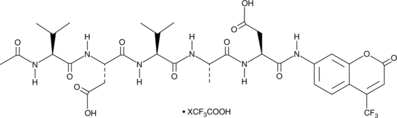
-
GC48974
Ac-VEID-AMC (ammonium acetate salt)
A caspase-6 fluorogenic substrate
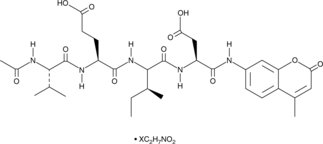
-
GC18021
Ac-YVAD-CHO
Ac-YVAD-CHO (L-709049) est un inhibiteur puissant, réversible et spécifique de l'enzyme de conversion de l'interleukine-lβ tétrapeptidique (ICE) avec des valeurs de Ki chez la souris et l'homme de 3,0 et 0,76 nM.
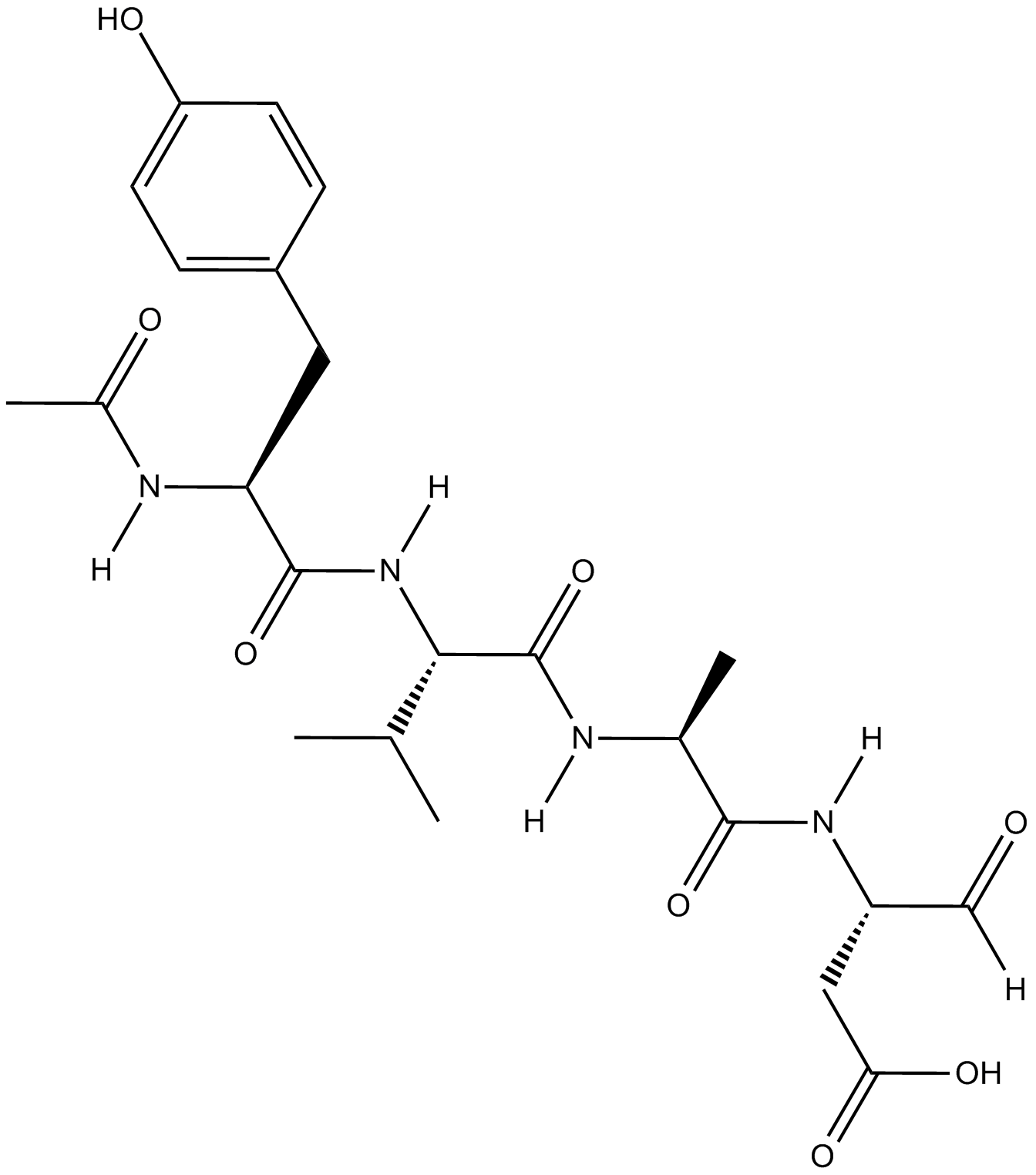
-
GC42721
Ac-YVAD-CMK
Ac-YVAD-CMK est un inhibiteur irréversible sélectif de la caspase-1 (Ki=0,8nM), qui peut empêcher l'activation de la cytokine pro-inflammatoire IL-1β. Ac-YVAD-CMK peut réduire la réponse inflammatoire et induire un effet neuroprotecteur durable.
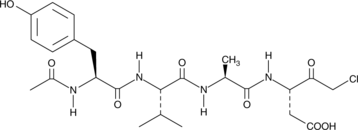
-
GC35227
ACBI1
ACBI1 est un dégradeur puissant et coopératif de SMARCA2, SMARCA4 et PBRM1 avec des DC50 de 6, 11 et 32 nM, respectivement. ACBI1 est un dégradeur de PROTAC. ACBI1 montre une activité anti-proliférative. ACBI1 induit l'apoptose.
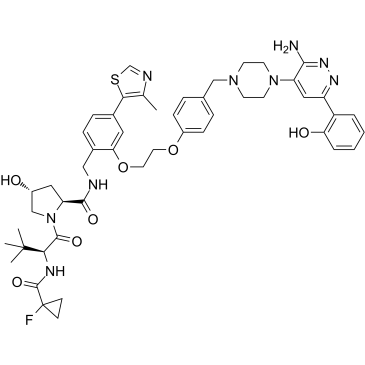
-
GN10341
Acetate gossypol
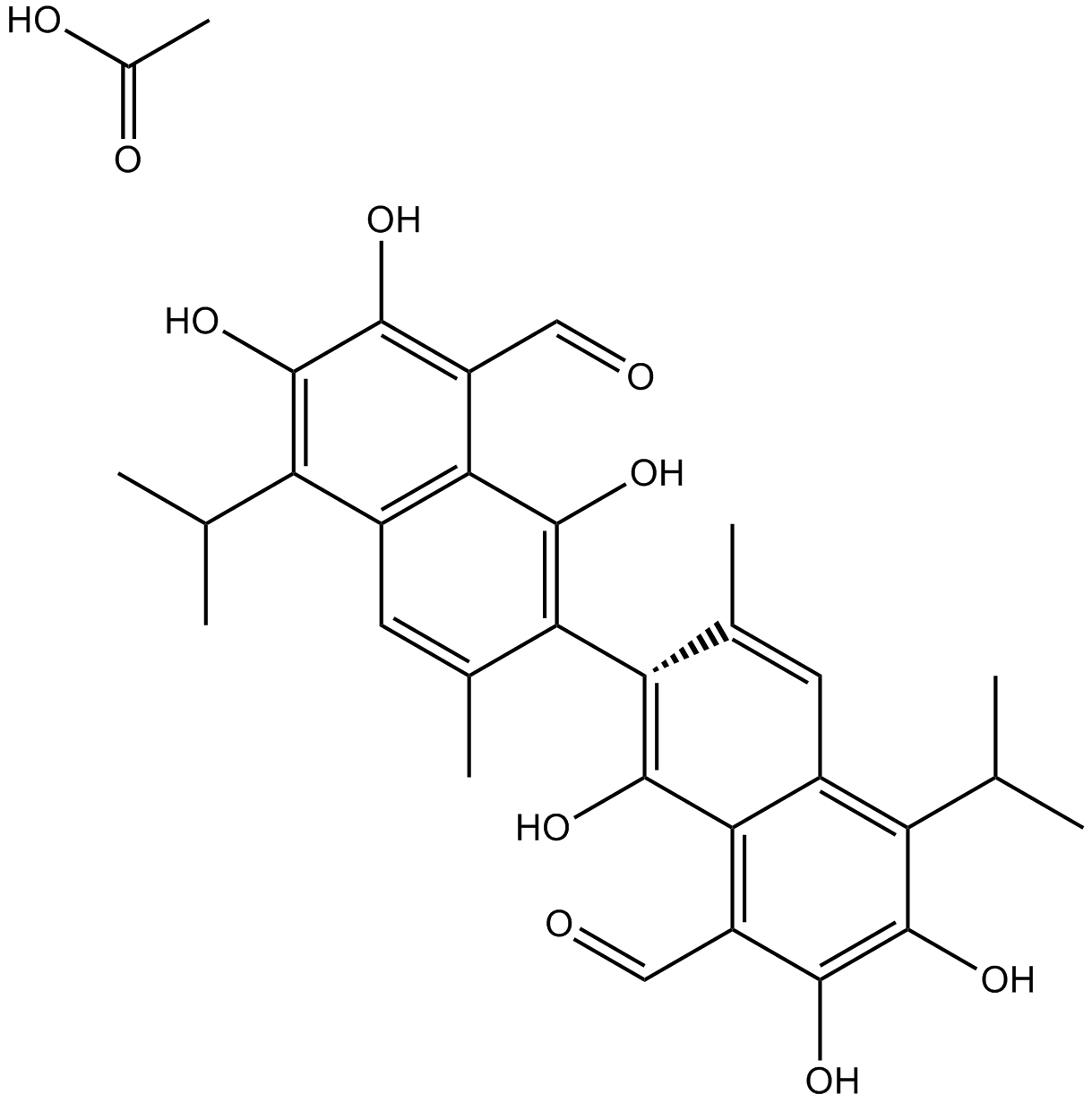
-
GC11786
Acetylcysteine
L'acétylcystéine est le dérivé N-acétyl de la CYSTEINE.
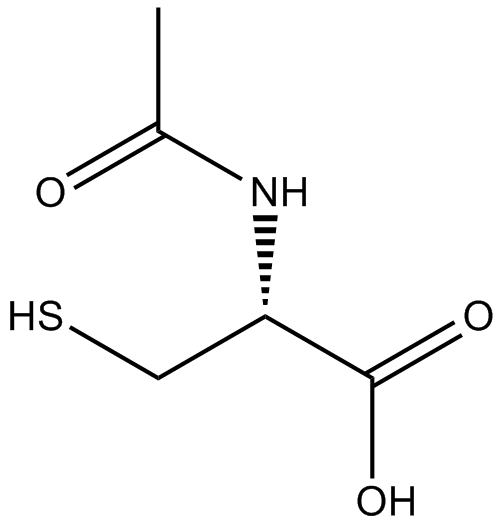
-
GC17094
Acitretin
L'acitrétine (Ro 10-1670) est un rétinoÏde systémique de deuxième génération qui a été utilisé dans le traitement du psoriasis.
-
GC35242
Actein
L'actéine est un glycoside triterpénique isolé des rhizomes de Cimicifuga foetida. L'actéine supprime la prolifération cellulaire, induit l'autophagie et l'apoptose en favorisant l'activation des ROS/JNK et en atténuant la voie AKT dans le cancer de la vessie humaine. L'actéine a peu de toxicité in vivo.
-
GC16866
Actinomycin D
Un bloqueur de transcription interagissant avec l'ADN ayant une activité anti-cancer.

-
GC16350
Actinonin
L'actinonine ((-)-Actinonine) est un agent antibactérien naturel produit par Actinomyces.
-
GC16362
AD57 (hydrochloride)
polypharmacological cancer therapeutic that inhibits RET.
-
GC34214
Adalimumab (Anti-Human TNF-alpha, Human Antibody)
Adalimumab (Anti-TNF-alpha humain, anticorps humain) est un anticorps monoclonal IgG1 humain ciblant le facteur de nécrose tumorale α (TNF-α).



