Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Targets for Cell Cycle/Checkpoint
- ATM/ATR(26)
- Aurora Kinase(47)
- Cdc42(4)
- Cdc7(4)
- Chk(16)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(91)
- E1 enzyme(1)
- G-quadruplex(14)
- Haspin(7)
- HMTase(1)
- Kinesin(26)
- Ksp(6)
- Microtubule/Tubulin(243)
- Mps1(15)
- Mitotic(11)
- RAD51(18)
- ROCK(71)
- Rho(13)
- PERK(11)
- PLK(37)
- PTEN(8)
- Wee1(7)
- PAK(21)
- Arp2/3 Complex(8)
- Dynamin(12)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Products for Cell Cycle/Checkpoint
- Cat.No. Nombre del producto Información
-
GC43546
Dnp-PLALWAR (trifluoroacetate salt)
Dnp-PLALWAR is a fluorogenic substrate for matrix metalloproteinase-1 (MMP-1) and MMP-8.
-
GC43547
Dnp-PLAYWAR (trifluoroacetate salt)
Dnp-PLAYWAR is a fluorogenic substrate for matrix metalloproteinase-8 (MMP-8) and MMP-26.
-
GC43550
Dnp-PLGMWSR (trifluoroacetate salt)
Dnp-PLGMWSR is a fluorogenic substrate for matrix metalloproteinase-2 (MMP-2) and MMP-9.
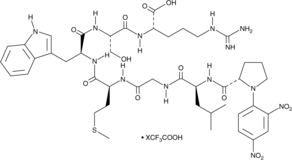
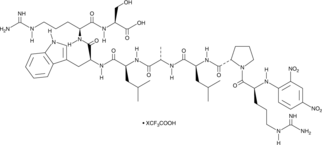
-
GC43552
Dnp-RPLALWRS (trifluoroacetate salt)
Dnp-RPLALWRS is a fluorogenic substrate for matrix metalloproteinase-7 (MMP-7).
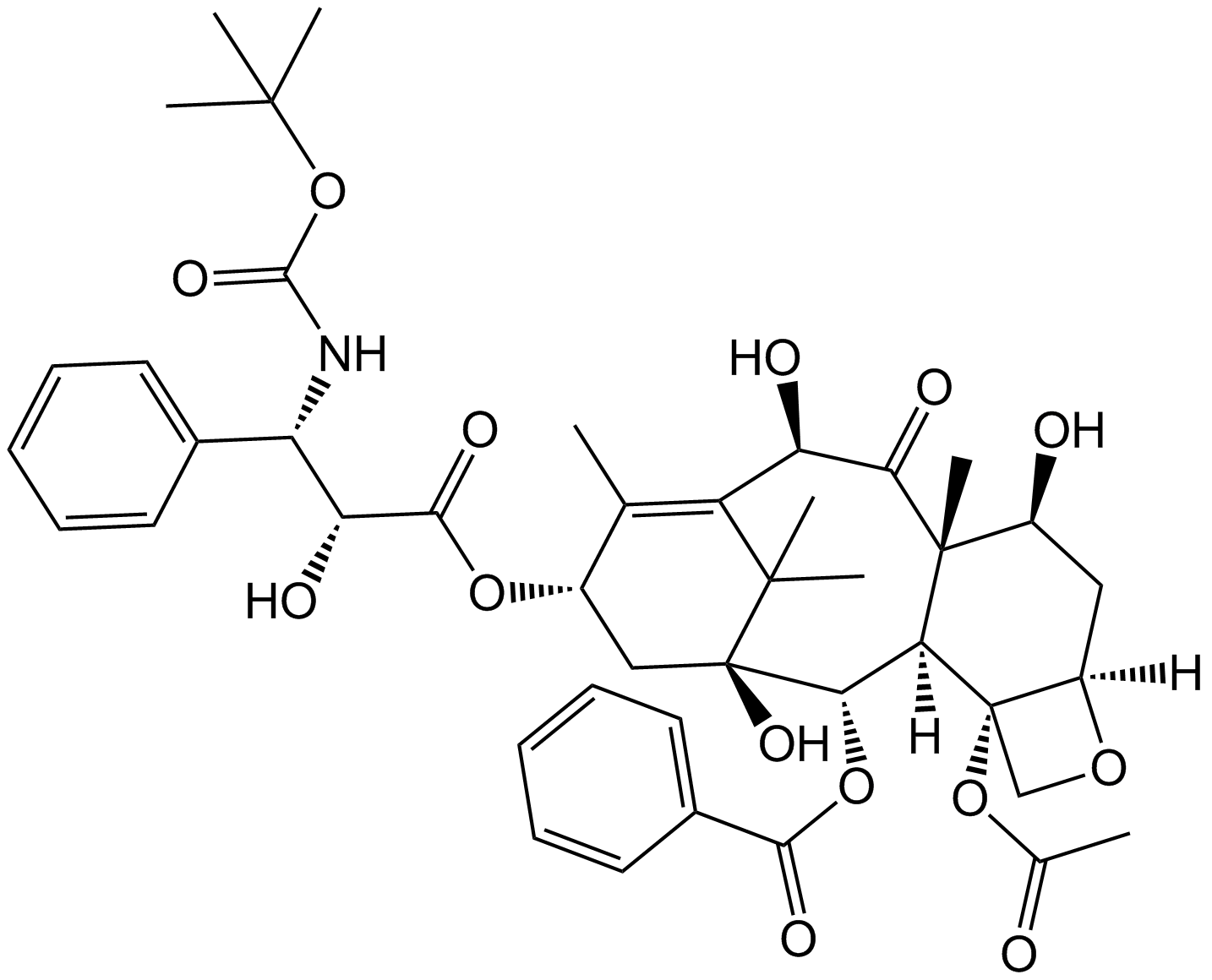
-
GC16684
Docetaxel
Docetaxel es un fármaco antineoplásico que inhibe la despolimerización de los microtúbulos y atenúa los efectos de la expresión génica bcl-2 y bcl-xL.
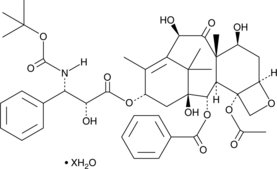
-
GC48968
Docetaxel (hydrate)
An analog of taxol with antitumor properties
-
GC12550
Docetaxel Trihydrate
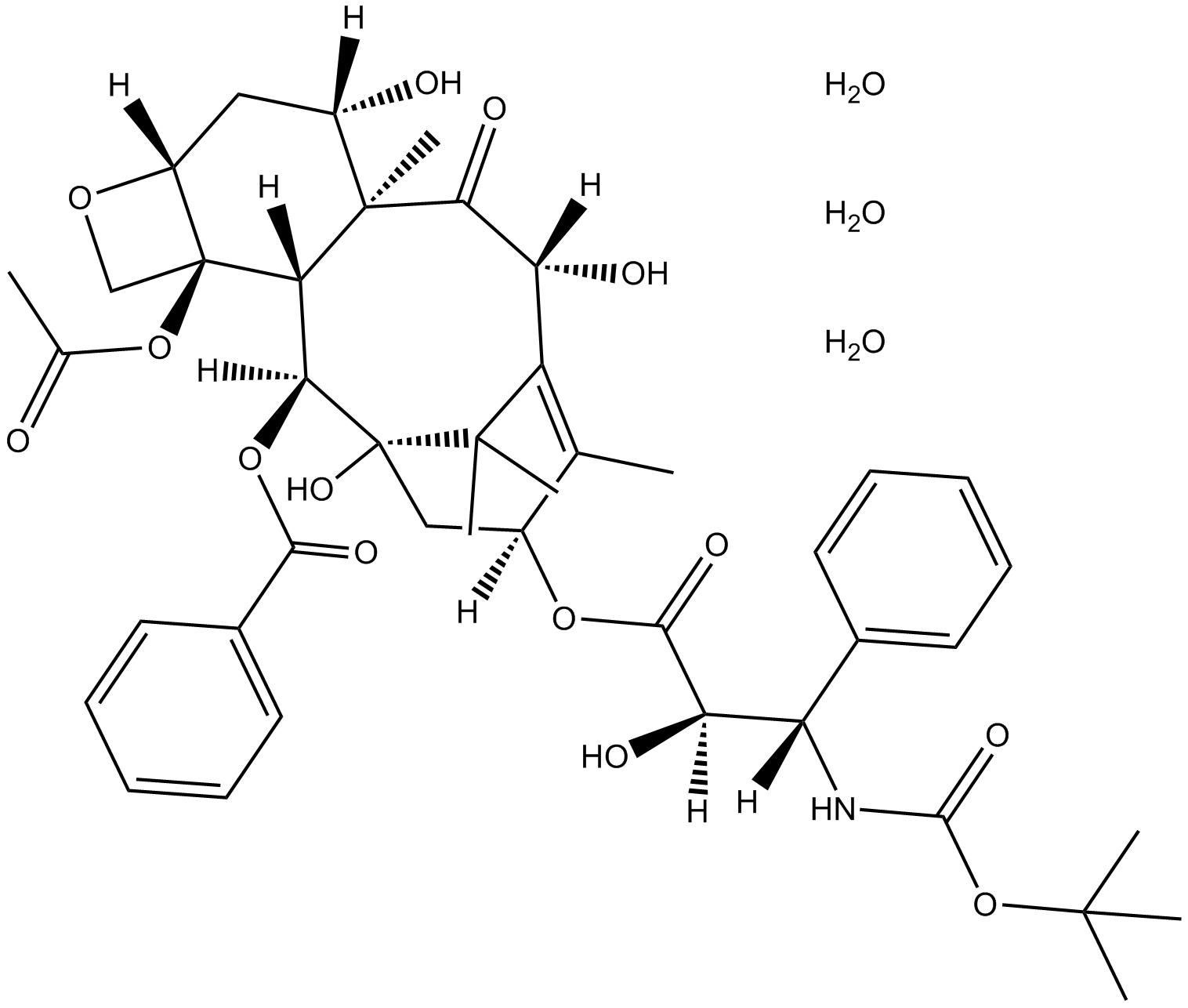
-
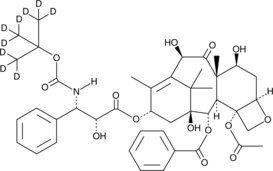
GC47250
Docetaxel-d9
Docetaxel-d9 (RP-56976-d9) es el docetaxel etiquetado con deuterio. Docetaxel (RP-56976) es un inhibidor de la despolimerizaciÓn de microtÚbulos, con una IC50 de 0,2 μM. Docetaxel atenÚa los efectos de la expresiÓn de los genes bcl-2 y bcl-xL. Docetaxel detiene el ciclo celular en G2/M y conduce a la apoptosis celular. El docetaxel tiene actividad anticancerÍgena.
-
GC52095
Dodecyltrimethylammonium (bromide)
El dodeciltrimetilamonio (bromuro) (DTAB) es un tensioactivo.

-
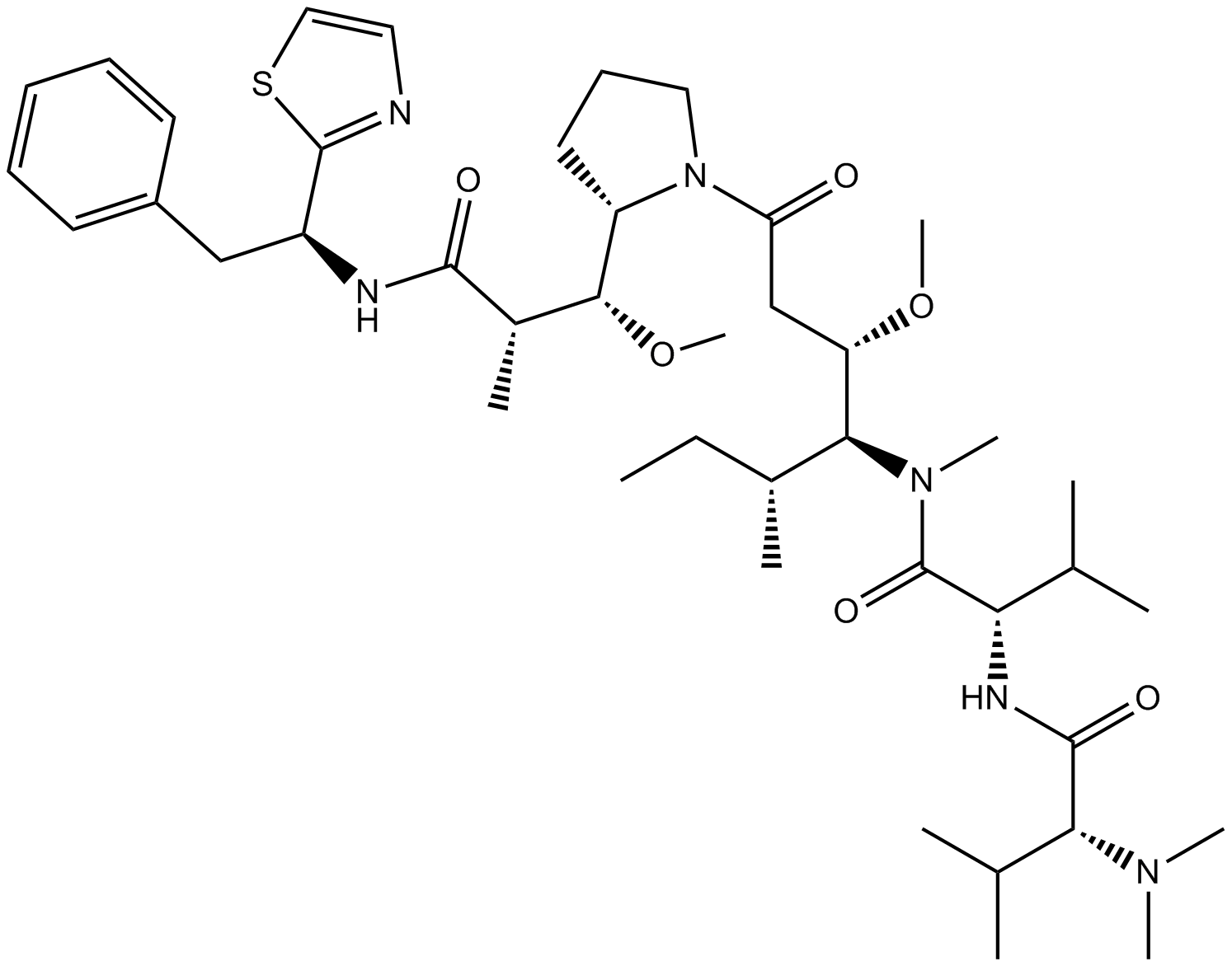
GC16273
Dolastatin 10
La dolastatina 10 (DLS 10) es un potente péptido antimitÓtico que inhibe la polimerizaciÓn de la tubulina.
-
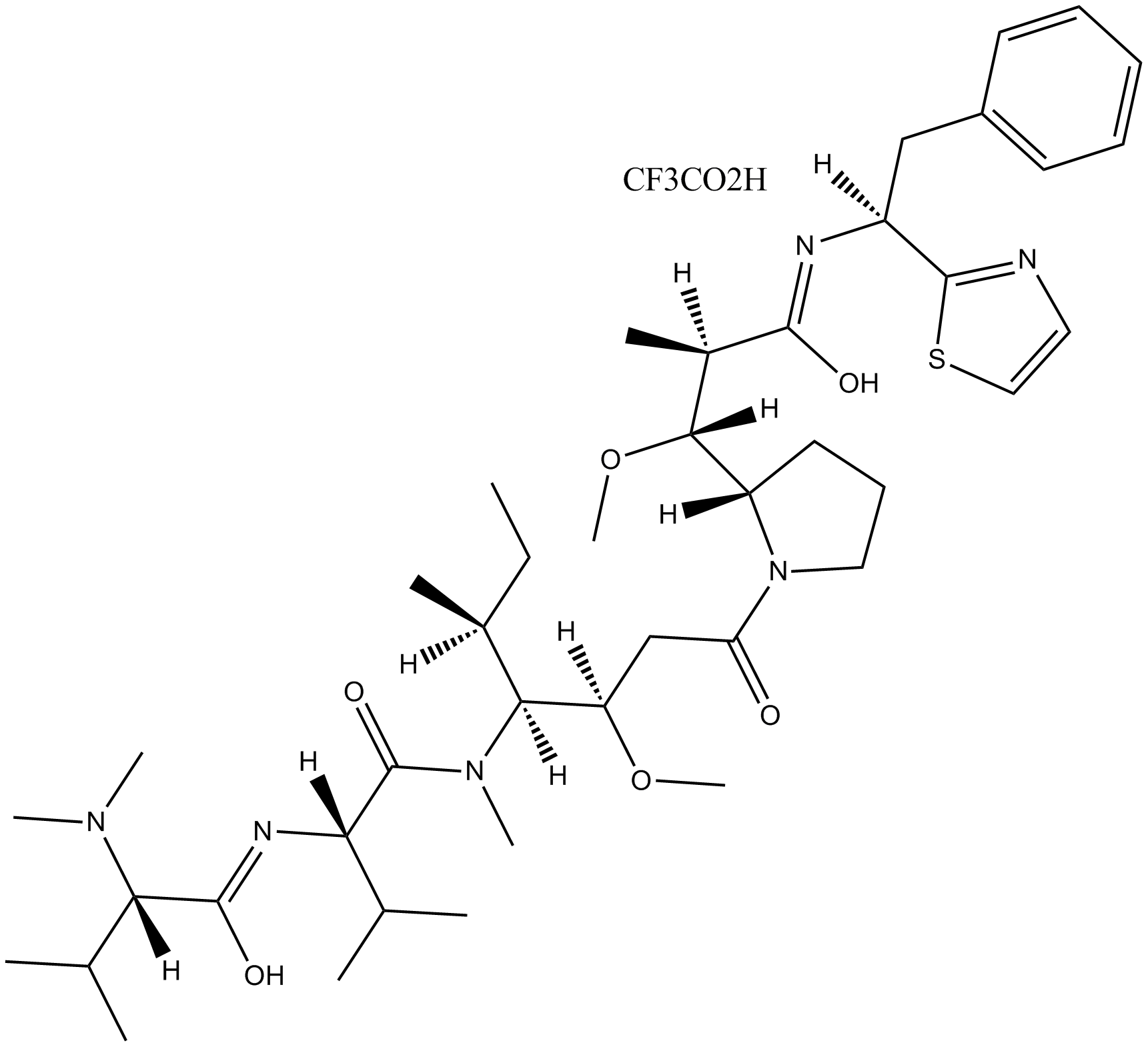
GC10557
Dolastatin 10 trifluoroacetate
Antitumor agent
-
GA21397
Dolastatin 15
La dolastatina 15 (DLS 15), un depsipéptido derivado de Dolabella auricularia, es un potente agente antimitÓtico estructuralmente relacionado con el agente antitubulina Dolastatina 10. La dolastatina 15 induce la detenciÓn del ciclo celular y la apoptosis en células de mieloma mÚltiple. La dolastatina 15 se puede utilizar como citotoxina ADC.

-
GC43567
DPC-AJ1951 (trifluoroacetate salt)
DPC-AJ1951 is a peptide agonist of the parathyroid hormone (PTH)/PTH-related peptide receptor (PPR; EC50 = 0.15 nM in HEK293 cells expressing human PPR).
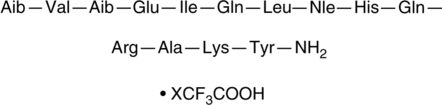
-
GC64927
Drp1-IN-1
Drp1-IN-1 (comp A-7) es un inhibidor de la proteÍna similar a la dinamina-1 (Drp1), con una IC50 de 0,91 μM.
-
GC50020
Dynamin inhibitory peptide, myristoylated
El péptido inhibidor de DynaMin, miristoilado, es un inhibidor de DynaMin que interfiere con la uniÓn de la anfifisina con la dinamina.
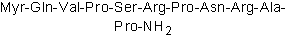
-
GC10395
Dynasore
Dynasore, como inhibidor de la GTPasa, puede inhibir rápida y reversiblemente la actividad de la dinamina, lo que previene la endocitosis.
-
GC17208
Dyngo-4a
Hydroxy Dynasore (Dyngo-4a), un anÁlogo estructural de Dynasore, es un inhibidor de dinamina de uniÓn no especÍfica, baja citotoxicidad y potencia mejorada con valores IC50 de 0,38 μM y 2,3μM para dinamina cerebral I y dinamina de rata recombinante II, respectivamente .
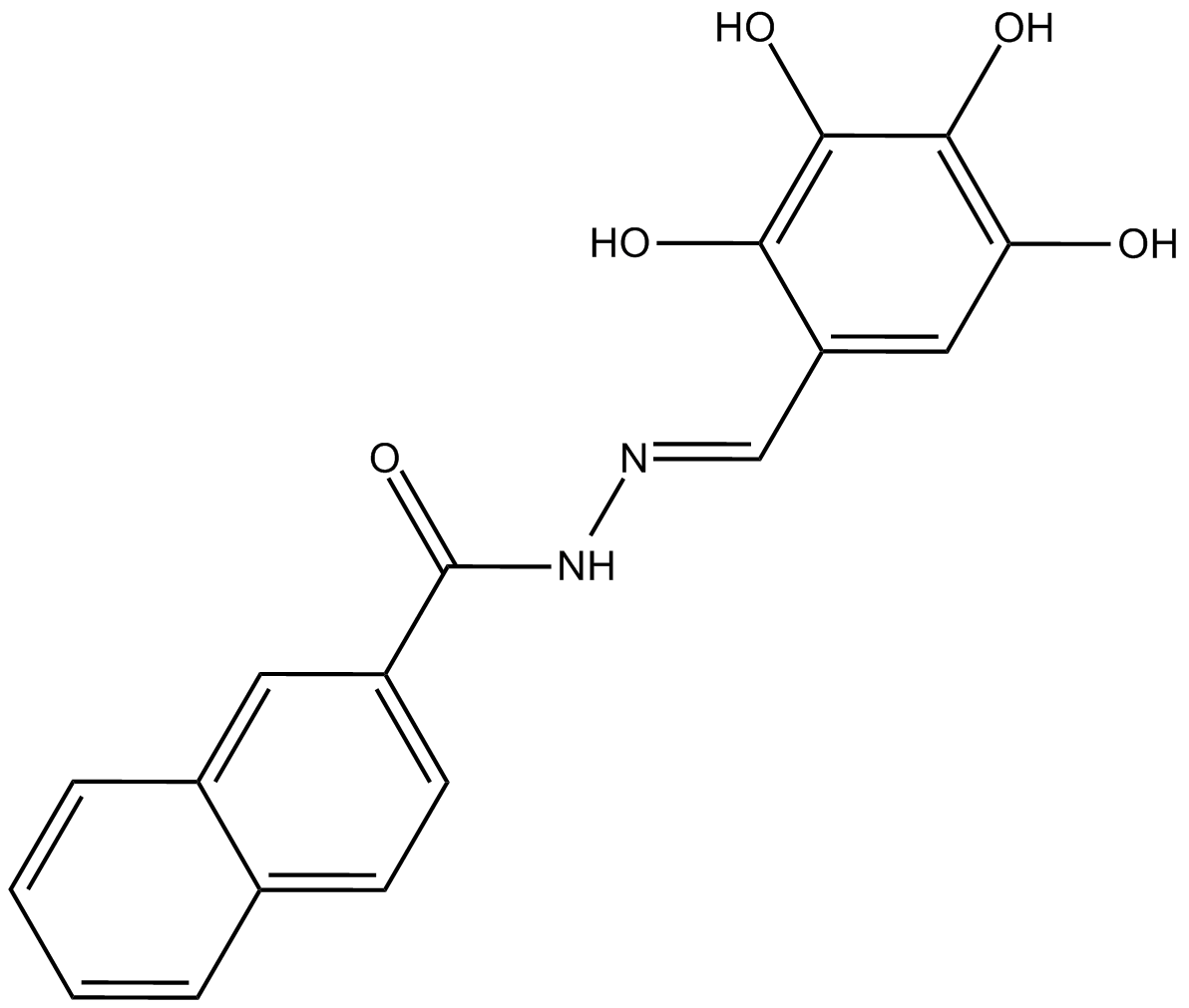
-
GC66056
Dynole 2-24
Dynole 2-24 es un inhibidor de dinamina GTPasa basado en indol (IC50 = 0,56 μ M para dinamina I). Dynole 2-24 no es tÓxico y muestra una mayor potencia contra la dinamina I y II in vitro y en células (IC₅₀(CME)=1,9 μM). Dynole 2-24 también muestra una selectividad de 4,4 veces para la dinamina I. Dynole 2-24 es un inhibidor activo en la célula de la endocitosis mediada por clatrina. CME: endocitosis mediada por clatrina
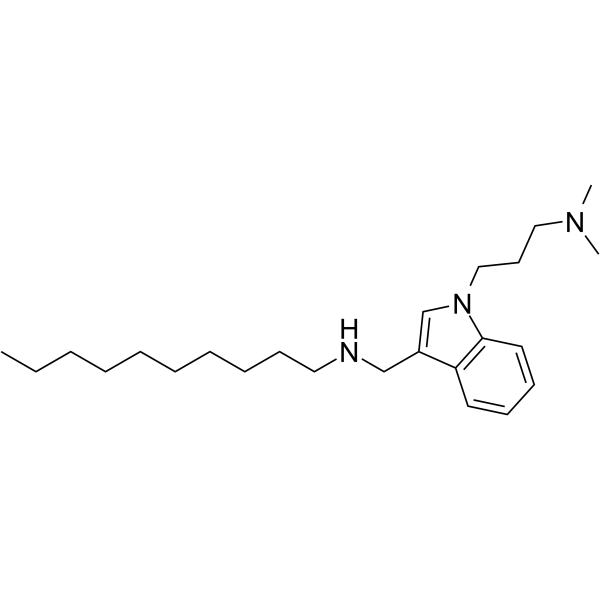
-
GC47279
Echinosporin
A bacterial metabolite with antibacterial and anticancer activities
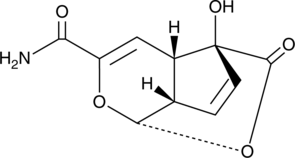
-
GC35964
Eg5 Inhibitor V, trans-24
Eg5 Inhibitor V, trans-24 es un potente y especÍfico inhibidor de cinesina Eg5 con un IC50 de 0,65 μM, y se puede utilizar en la investigaciÓn del cÁncer.
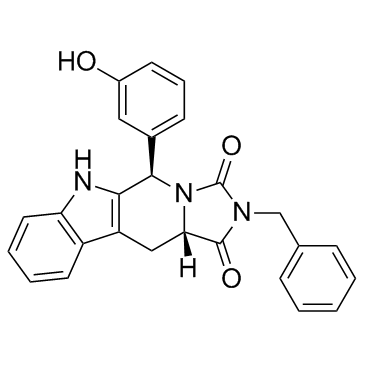
-
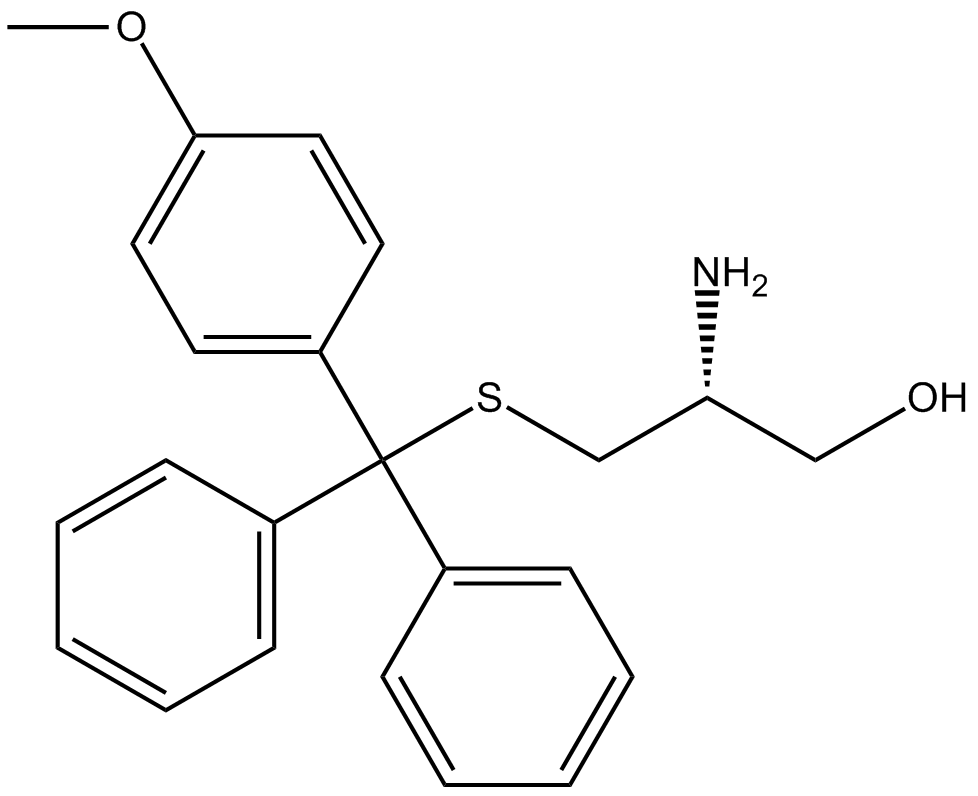
GC10681
Eg5-I
potent inhibitor of Eg5
-
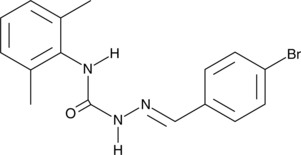
GC43588
EGA
EGA es un inhibidor selectivo de las vÍas de trÁfico de endosomol explotadas por mÚltiples toxinas y virus.
-
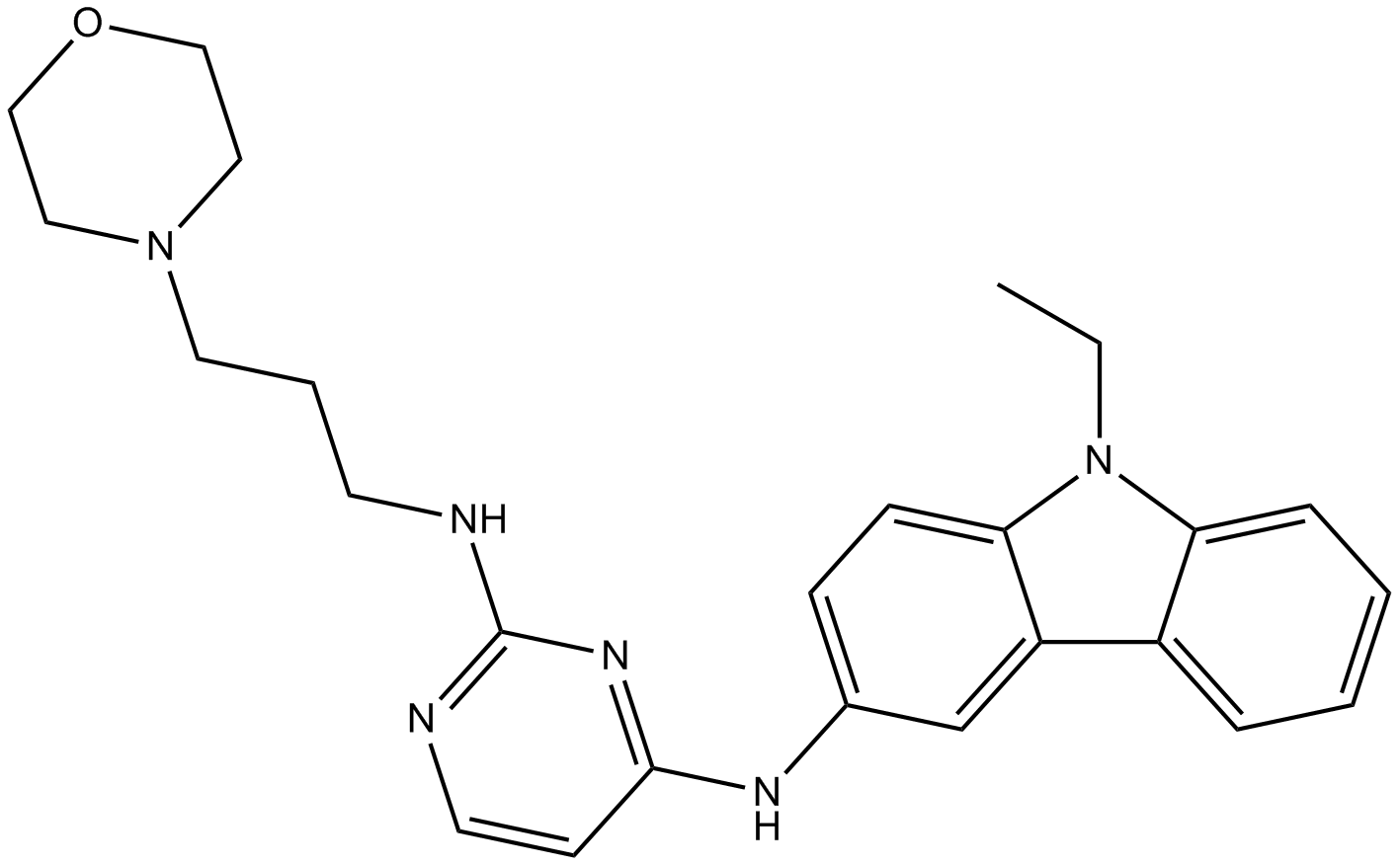
GC10030
EHop-016
Inhibidor de la GTPasa Rac1/Rac3, potente y específico.
-
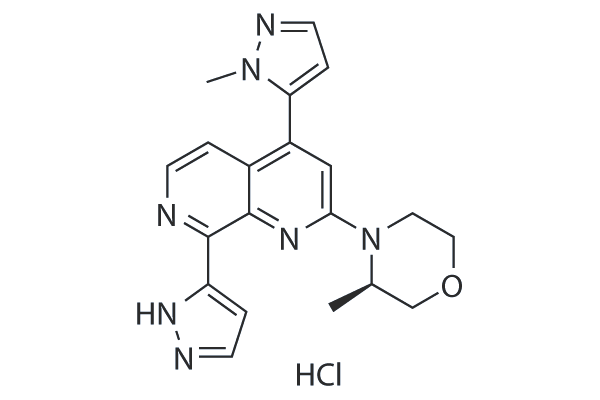
GC25372
Elimusertib (BAY-1895344) hydrochloride
Elimusertib (BAY-1895344) hydrochloride is a potent, highly selective and orally available ATR inhibitor with an IC50 of 7 nM.
-
GC16827
ELR510444
ELR510444 es un nuevo disruptor de microtÚbulos; inhibe la proliferaciÓn de células MDA-MB-231 con IC50 de 30,9 nM; no es un sustrato para el transportador de fÁrmacos de la glicoproteÍna P y retiene la actividad en lÍneas celulares que sobreexpresan βIII-tubulina.
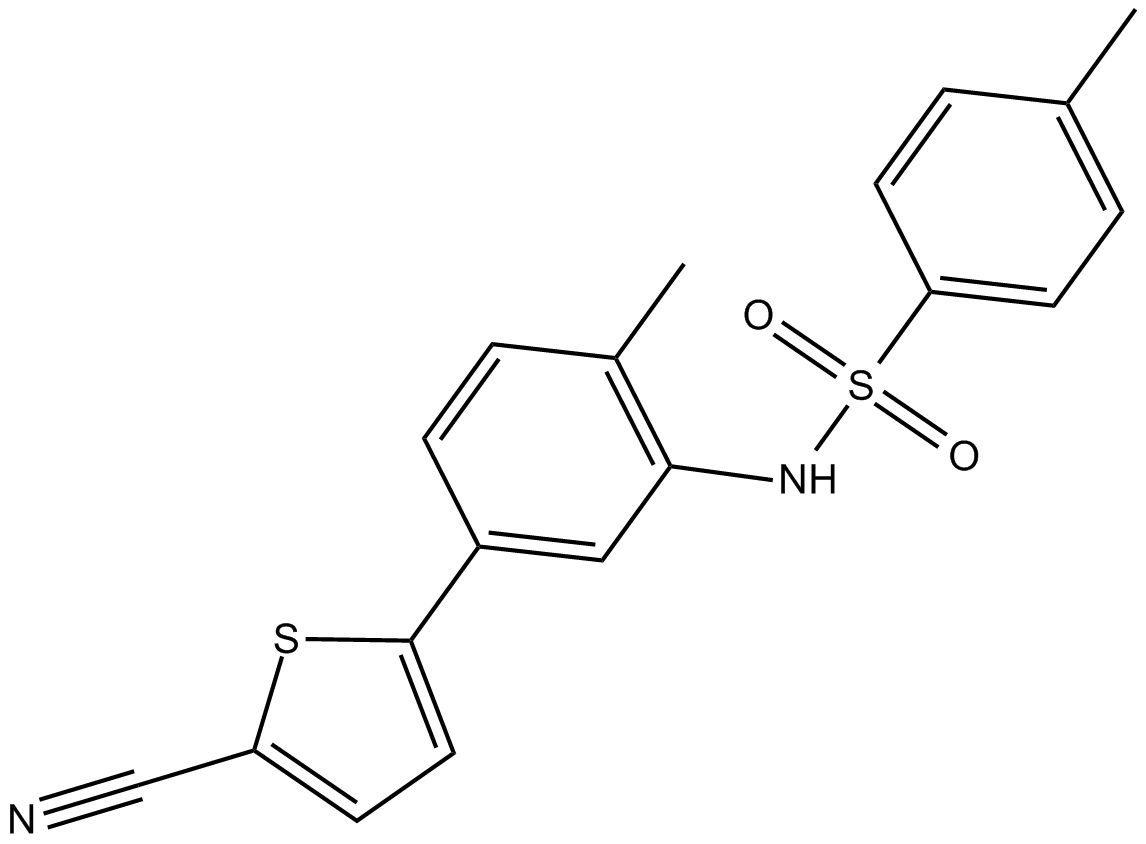
-
GC19466
Eltanexor (KPT-8602)
A second-generation exportin-1 inhibitor
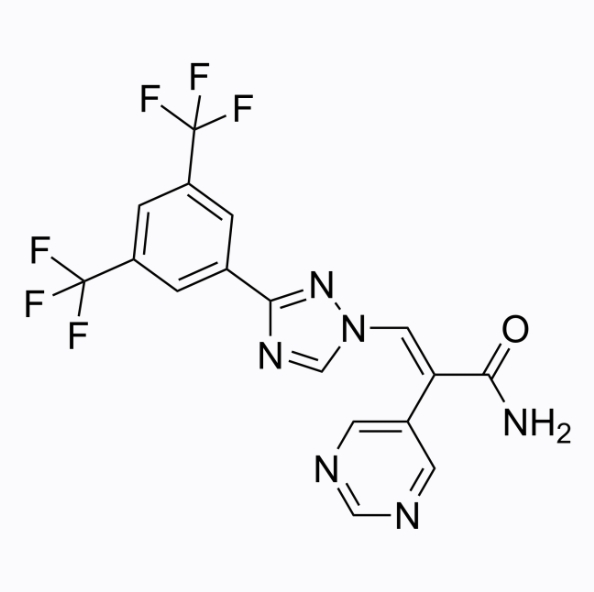
-
GC19213
Eltanexor Z-isomer
Eltanexor Z-isÓmero (KPT-8602 Z-isÓmero) es el isÓmero menos activo de KPT-8602.
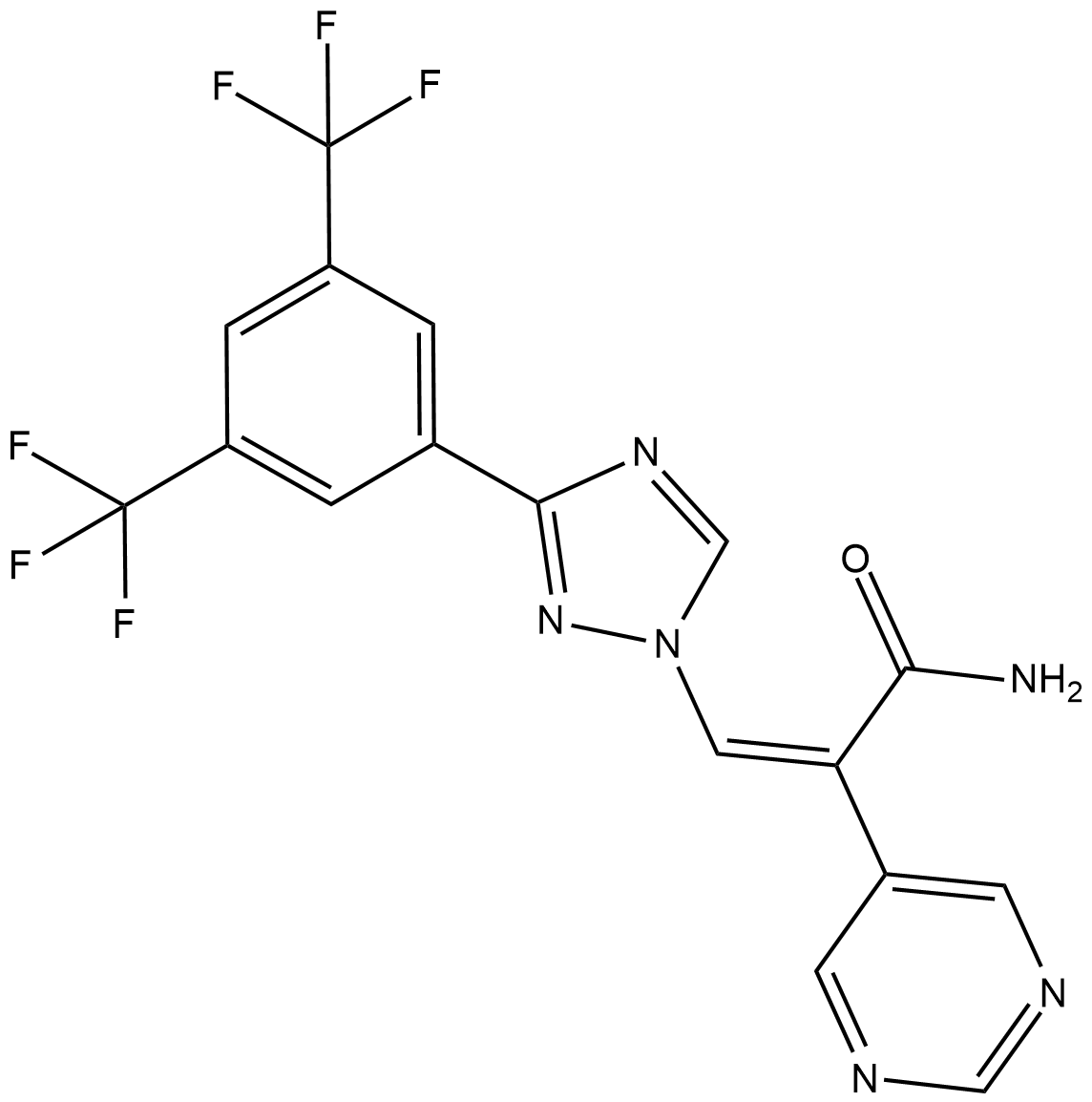
-
GC18165
EMD534085
EMD534085 es un inhibidor potente y selectivo de la kinesina-5 mitÓtica con una IC50 de 8 nM.
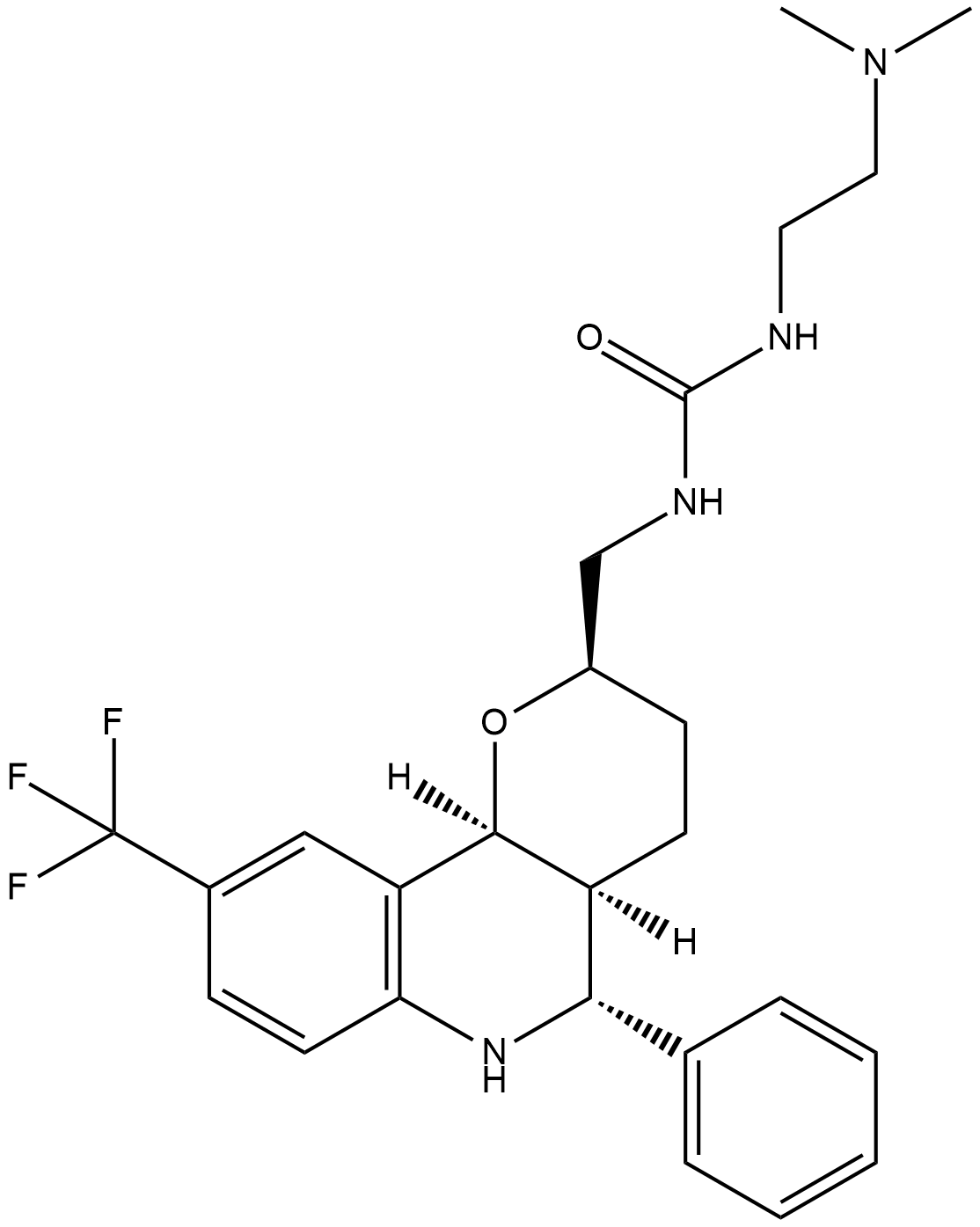
-
GC34073
Empesertib (BAY 1161909)
Empesertib (BAY 1161909) (BAY 1161909) es un potente inhibidor de Mps1, con una IC50 de < 1 nM.
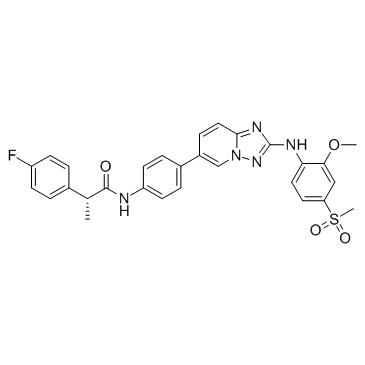
-
GC49124
EN219
EN219 es un ligando covalente sintético moderadamente selectivo frente a una cisteÍna N-terminal (C8) de RNF114 con una IC50 de 470 nM. EN219 inhibe la autoubiquitinaciÓn mediada por RNF114 y la ubiquitinaciÓn de p21.
-
GC62287
EN4
EN4 es un ligando covalente que se dirige a la cisteÍna 171 (C171) de MYC. EN4 es selectivo para c-MYC sobre N-MYC y L-MYC. EN4 inhibe la actividad transcripcional de MYC, regula a la baja los objetivos de MYC y altera la tumorigénesis.
-
GC19010
Endosidin 2
Un inhibidor permeable a la célula de la exocitosis y el reciclaje endosomal.
-
GC43605
Endosulfan II
Endosulfan II is an organochlorine insecticide and a stereoisomer of endosulfan I.
-
GC47291
Endothall
An herbicide
-
GC33125
Entasobulin
La entasobulina es un inhibidor de la polimerizaciÓn de la β-tubulina con potencial actividad anticancerÍgena.
-
GC43614
Enteropeptidase Fluorogenic Substrate
Enteropeptidase fluorogenic substrate is a substrate for enteropeptidase that contains a 7-amino-4-trifluoromethylcoumarin (AFC) moiety.
-
GC48995
Enteropeptidase Fluorogenic Substrate (trifluoroacetate salt)
A fluorogenic substrate of enteropeptidase
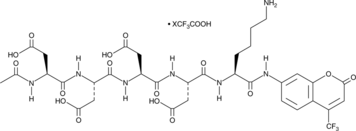
-
GC11202
Epothilone A
La epotilona A es un inhibidor competitivo de la uniÓn de [3H] paclitaxel a los polÍmeros de tubulina, con una Ki de 0,6-1,4 μM.
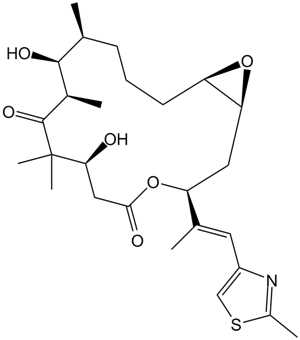
-
GC17240
Epothilone B (EPO906, Patupilone)
La epotilona B (EPO906, patupilona) es un estabilizador de microtÚbulos con una Ki de 0,71μM.
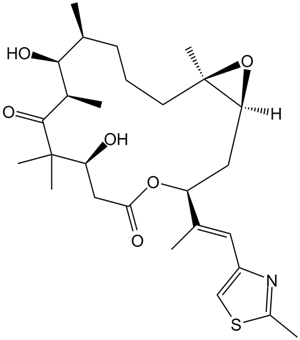
-
GC17440
Epothilone D
A microtubule-stabilizing agent
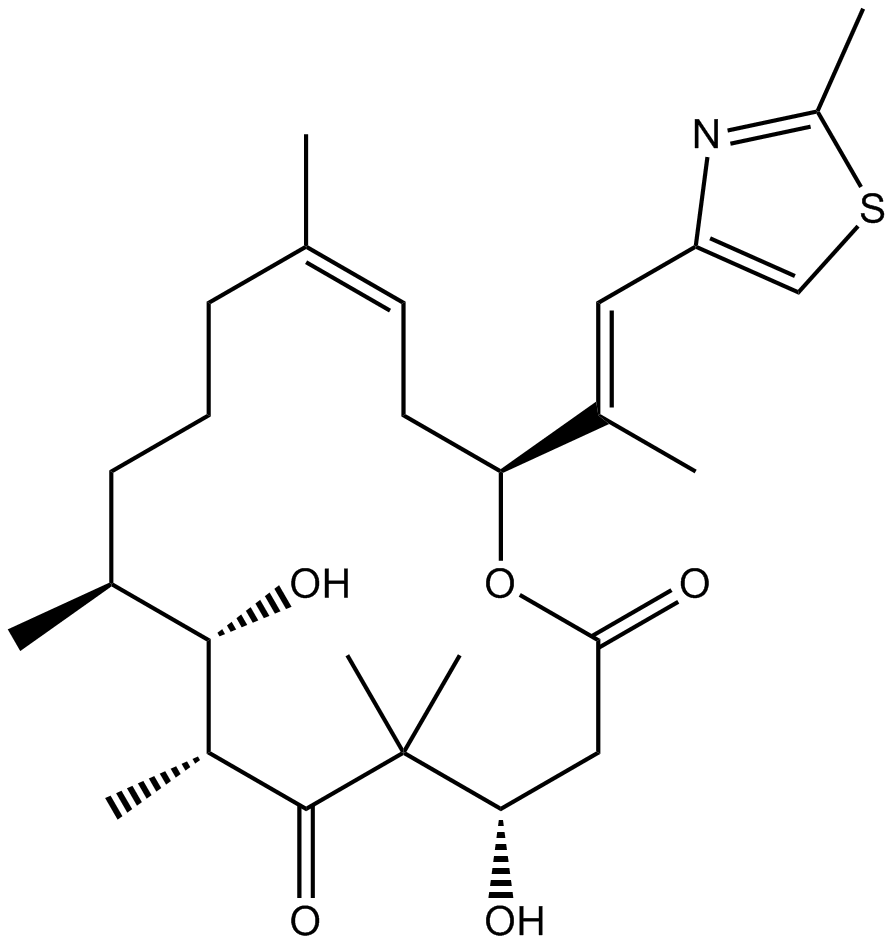
-
GC14354
Eribulin
Eribulina (E7389) es un agente dirigido a los microtÚbulos que se utiliza para la investigaciÓn del cÁncer de mama metastÁsico. La eribulina inhibe la proliferaciÓn de células cancerosas al unirse a las proteÍnas de los microtÚbulos y los microtÚbulos.
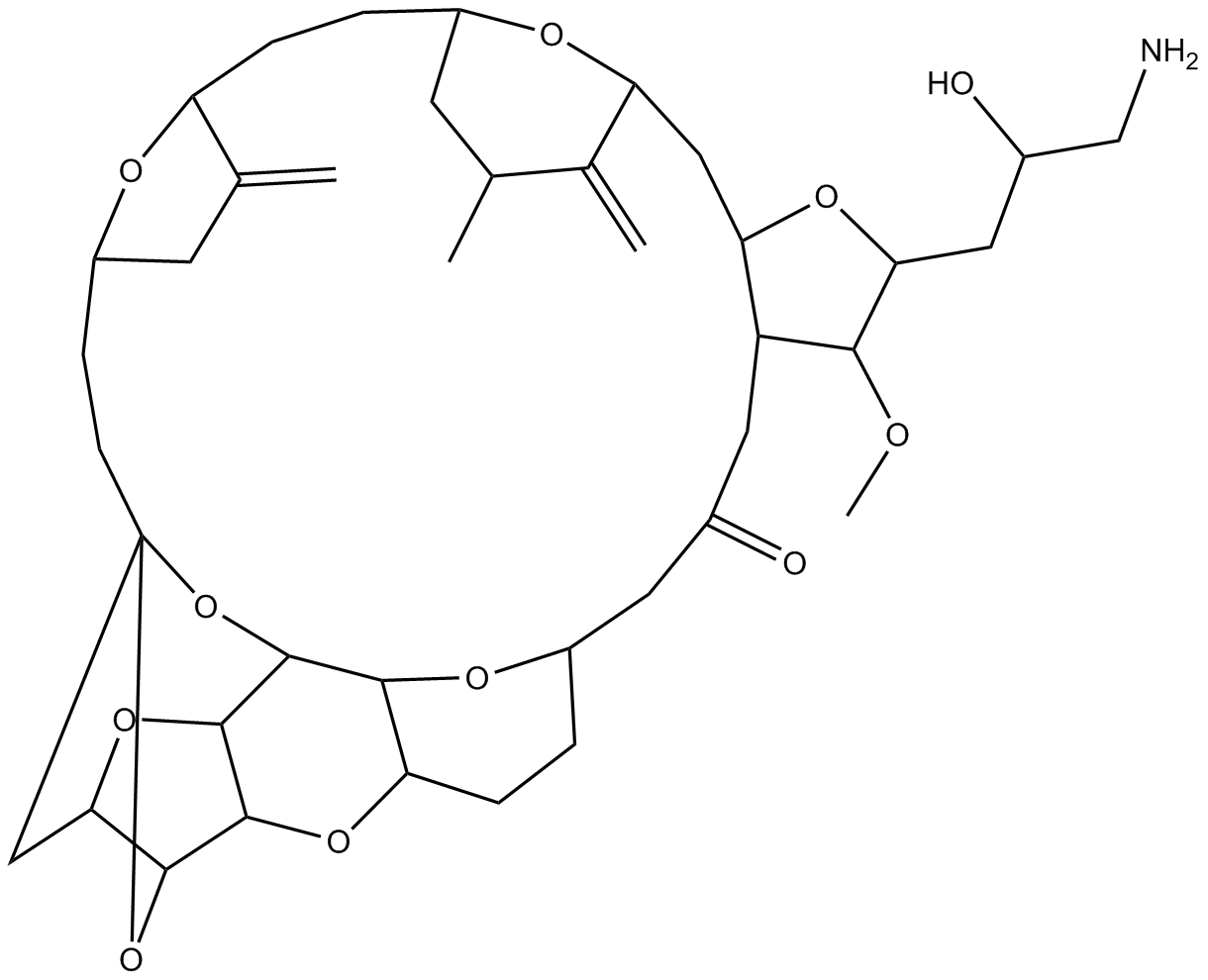
-
GC12483
Eribulin mesylate
análogo sintético de la halichondrina B
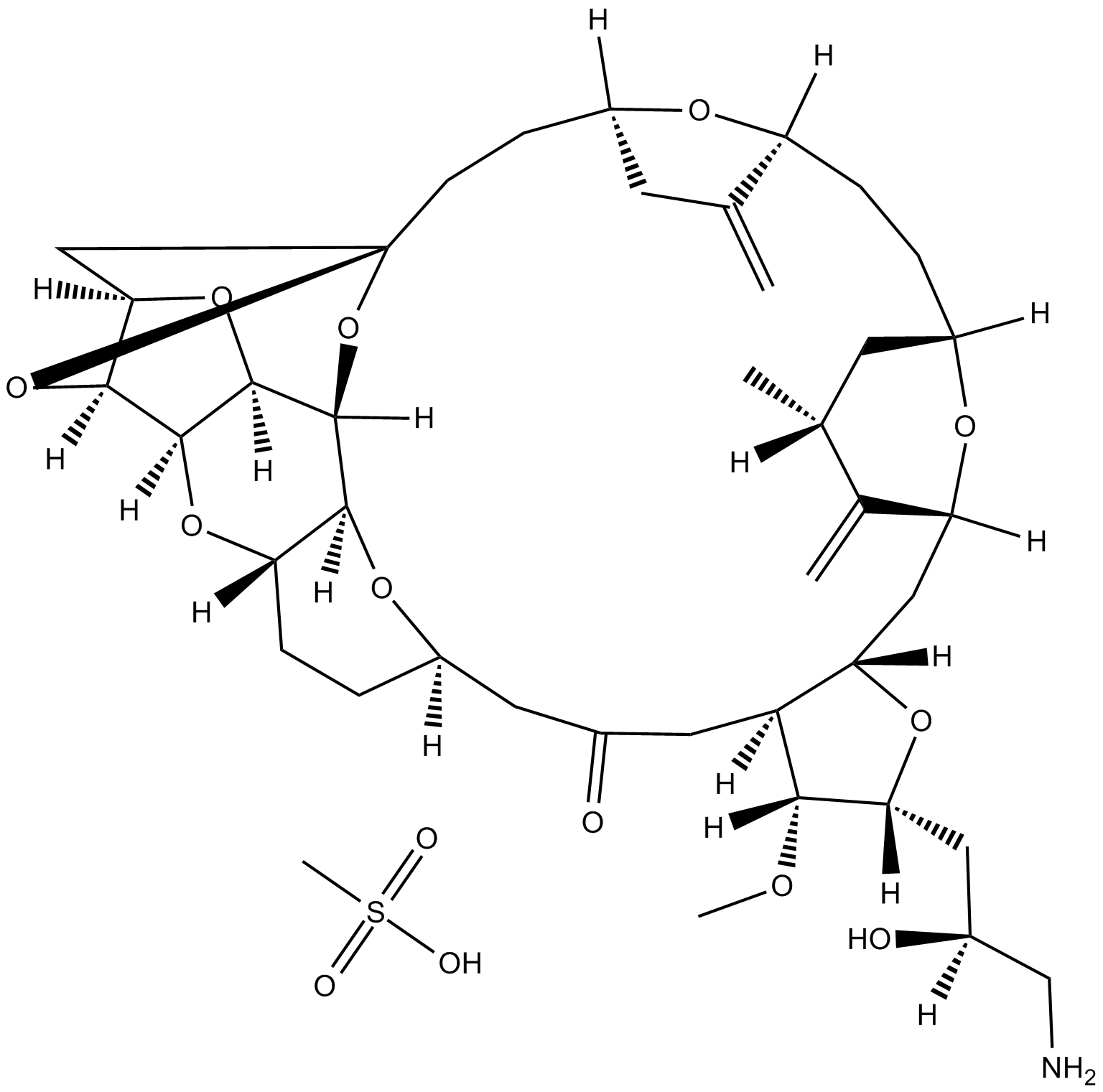
-
GC63845
Eribulin-d3 mesylate
El mesilato de eribulina-d3 es un mesilato de eribulina marcado con deuterio. El mesilato de eribulina es un agente dirigido a los microtÚbulos que se utiliza para la investigaciÓn del cÁncer.
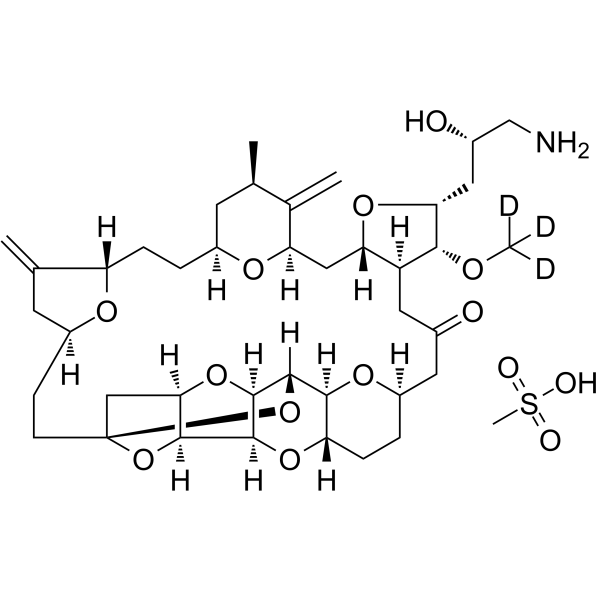
-
GC43625
Erucin
Erucin (ERU) es un isotiocianato particularmente abundante en la rÚcula.
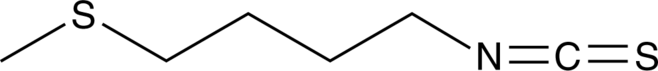
-
GC67333
Estramustine
La estramustina es un agente antineoplásico. La estramustina despolimeriza los microtúbulos al unirse a la tubulina 1, exhibe actividad antimitótica con un valor IC50 de ~16 μM para la mitosis de las células DU 145. La estramustina bloquea las células en la mitosis en xenoinjertos de tumores de próstata.
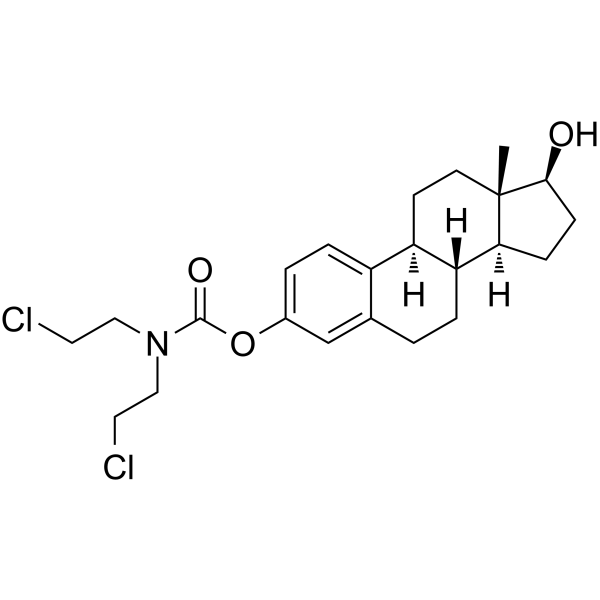
-
GC32816
Estramustine phosphate sodium
El fosfato sÓdico de estramustina, un anÁlogo del estradiol, es un agente quimioterÁpico antimicrotubular activo por vÍa oral.
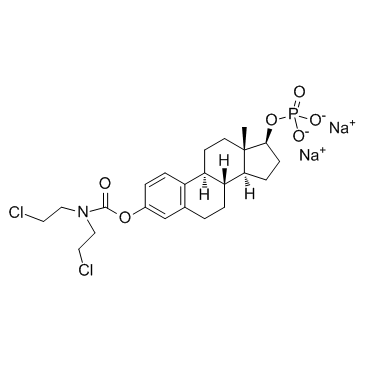
-
GC52061
Ethylene Glycol monododecyl ether
A nonionic surfactant

-
GC10196
ETP-46464
ETP-46464 es un inhibidor eficaz de mTOR y ATR con IC50 de 0,6 y 14 nM, respectivamente.
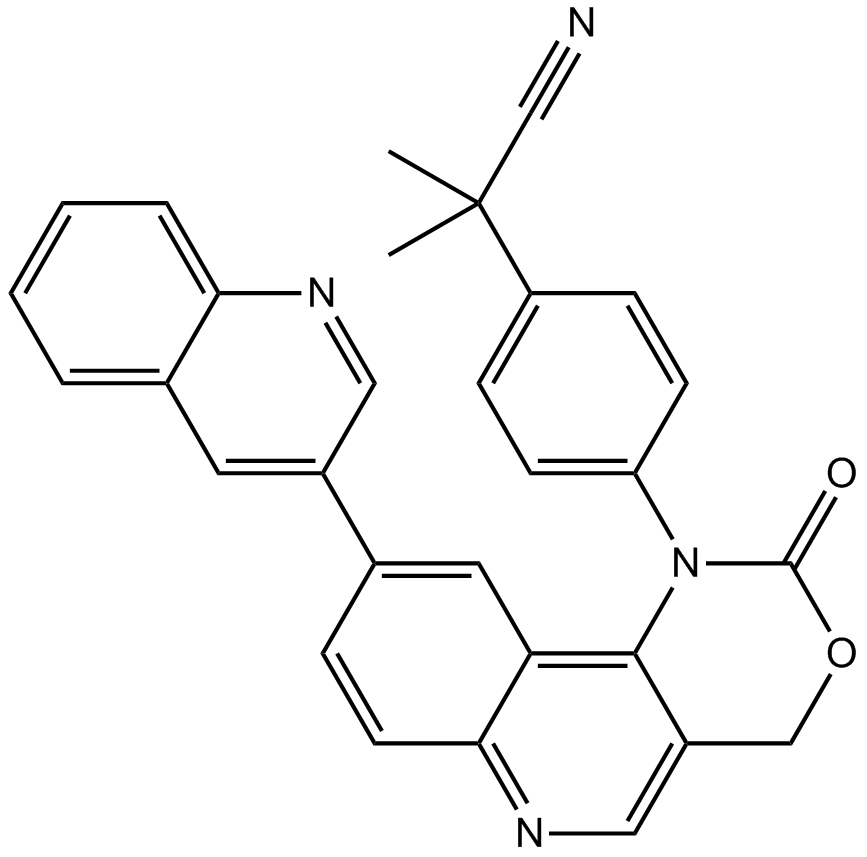
-
GC18693
Fascaplysin (chloride)
La fascaplysin (cloruro) es un pigmento rojo antimicrobiano y citotÓxico, que puede provenir de la esponja marina (Fascaplysin (cloruro)opsis sp.

-
GC13869
Fasudil
Fasudil (HA-1077; AT877) es un inhibidor inespecÍfico de RhoA/ROCK y también tiene un efecto inhibitorio sobre las proteÍnas quinasas, con una Ki de 0,33 μM para ROCK1, IC50 de 0,158 μM y 4,58 μM, 12,30 μM, 1,650 μM para ROCK2 y PKA , PKC, PKG, respectivamente. Fasudil es también un potente antagonista y vasodilatador de los canales de Ca2+.

-
GC14289
Fasudil (HA-1077) HCl
Fasudil (HA-1077; AT877) El clorhidrato es un inhibidor rhoa/roca inespecÍfico y también tiene un efecto inhibitorio en las proteÍnas quinasas, con un KI de 0.33 μ m para rock115&&&&7777 que offlineefficient_models_2022q2.md;M, 1.650 μM for ROCK2 and PKA, PKC, PKG, respectively.en_es_2021q4.mdFasudil (HA-1077) HCl is also a potent Ca2+ channel antagonist and vasodilator.en_es_2021q4.md
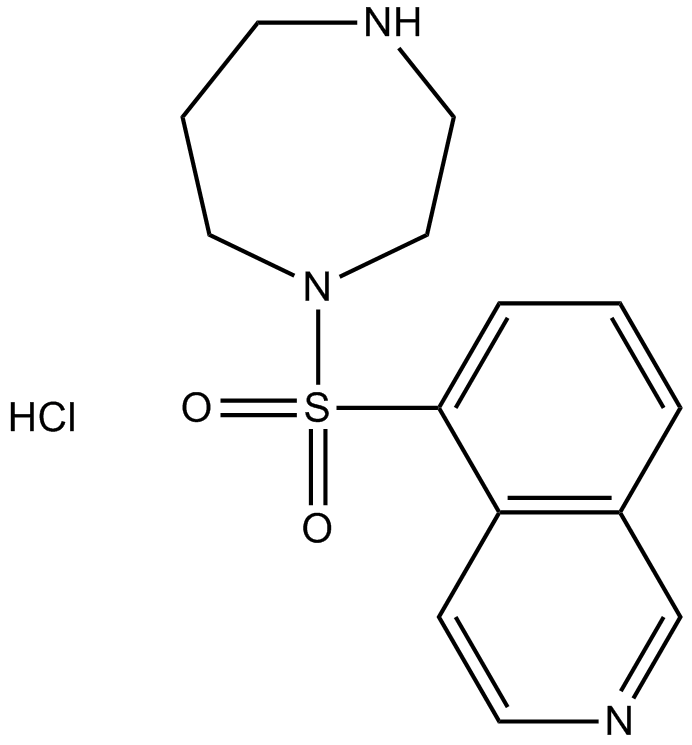
-
GC12319
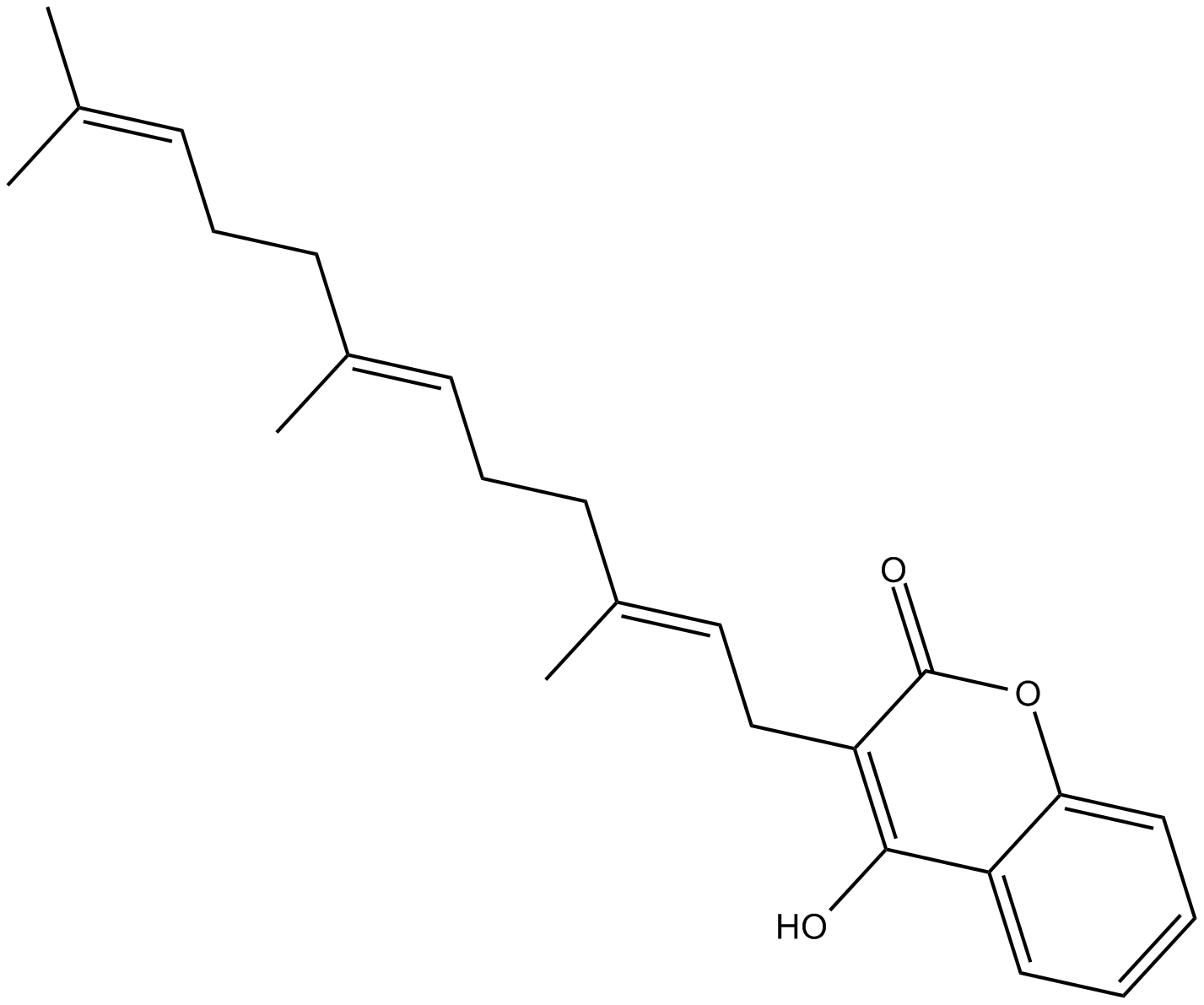
Ferulenol
El ferulenol, un derivado cumarÍnico prenilado sesquiterpénico, inhibe especÍficamente la succinato ubiquinona reductasa al nivel del ciclo de la ubiquinona.
-
GC43666
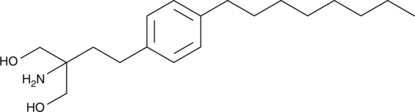
Fingolimod
El fingolimod (base libre FTY720) es un antagonista de la esfingosina 1-fosfato (S1P) con una IC50 de 0,033 nM en células K562 y NK.
-
GC14807
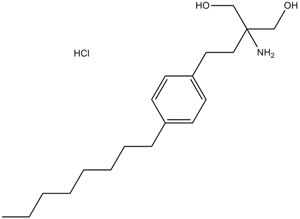
Fingolimod(FTY720)
Fingolimod (FTY720) HCl (FTY720), un análogo de la esfingosina, es un potente modulador de los receptores de fosfato de esfingosina 1 (S1P).
-
GC49344
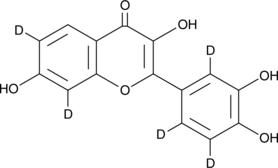
Fisetin-d5
An internal standard for the quantification of fisetin
-
GC49695
FKBP52 Monoclonal Antibody (Clone Hi52C)
For immunochemical analysis of FKBP52

-
GC43674
FlAsH-EDT2
FlAsH-EDT2 es un reactivo de marcaje de proteÍnas.
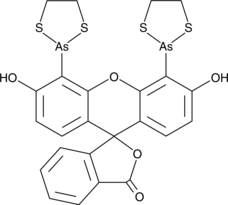
-
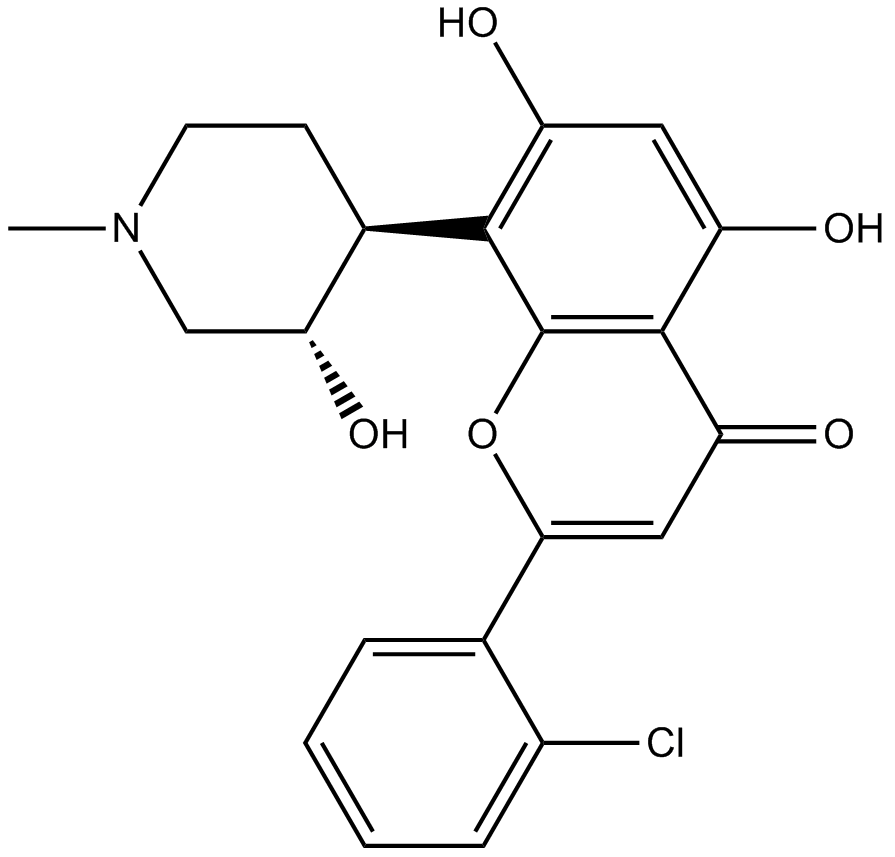
GC16063
Flavopiridol
An inhibitor of cyclin-dependent kinases
-
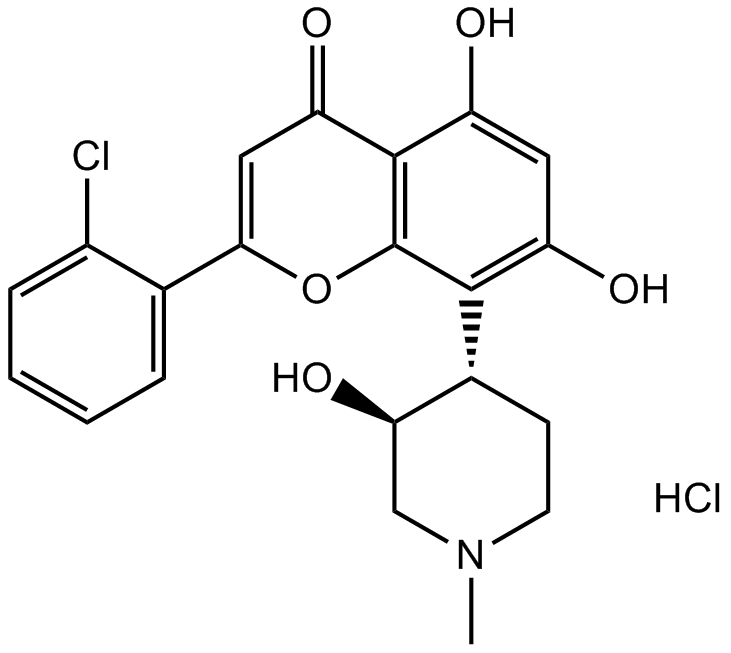
GC16425
Flavopiridol hydrochloride
El clorhidrato de flavopiridol (clorhidrato de alvocidib) es un amplio inhibidor de CDK, que compite con el ATP para inhibir las CDK, incluidas CDK1, CDK2, CDK4 con IC50 de 30, 170 y 100 nM, respectivamente.
-
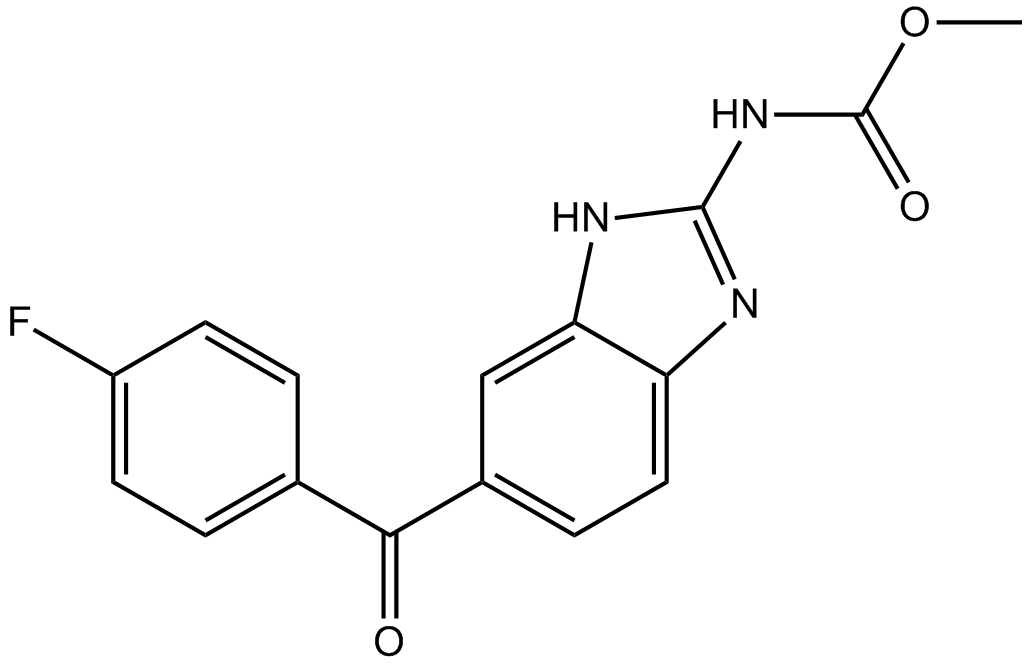
GC13210
Flubendazole
El flubendazol es un fÁrmaco antihelmÍntico seguro y eficaz, que se usa ampliamente como antihelmÍntico para humanos, roedores y rumiantes.
-
GC16727
Flutax 1
A fluorescent taxol derivative used for direct imaging of the microtubule cytoskeleton
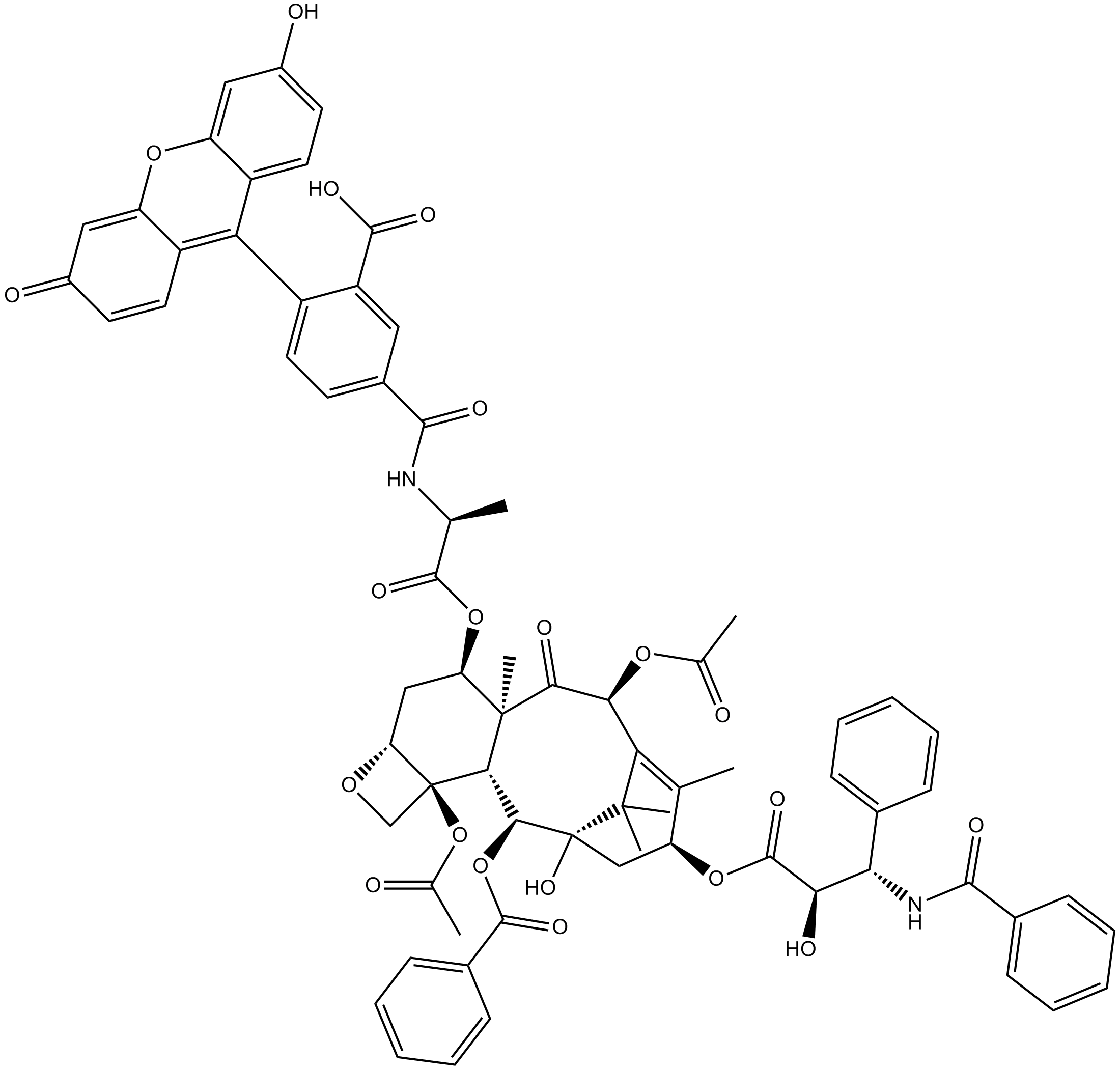
-
GC50397
Flutax 2
Green fluorescent taxol derivative; binds microtubules
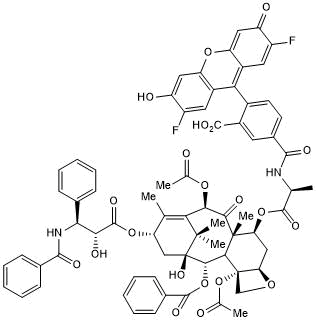
-
GC66242
Flutax-2 (5/6-mixture)
Flutax-2 (mezcla 5/6) es un derivado fluorescente activo de Taxol. Flutax-2 (mezcla 5/6) se une a los dÍmeros de tubulina polimerizados. Flutax-2 (mezcla 5/6) es capaz de estabilizar microtÚbulos de trofozoÍtos intactos de T. gallinae y T. fetus.
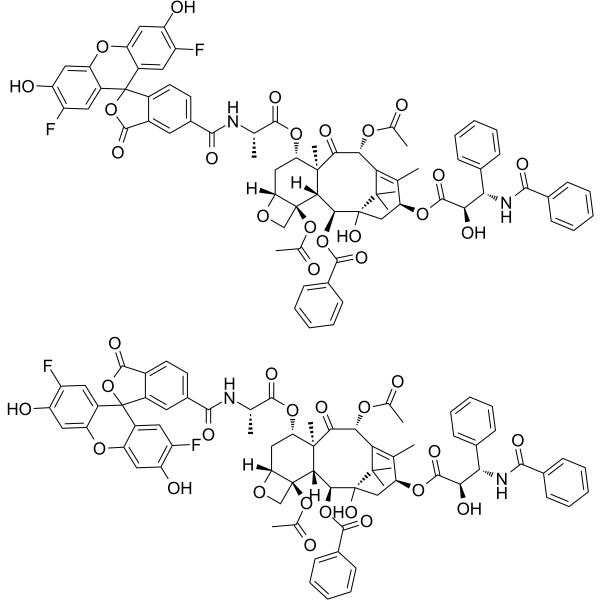
-
GC63596
Fmoc-MMAE
Fmoc-MMAE es una monometil auristatina E conjugada con un grupo protector (MMAE), que es un potente inhibidor de la tubulina. Fmoc-MMAE se puede utilizar en la sÍntesis de ADC.
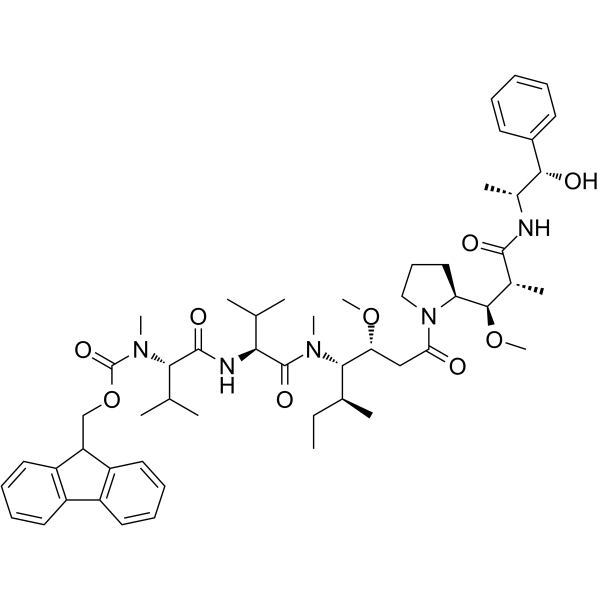
-
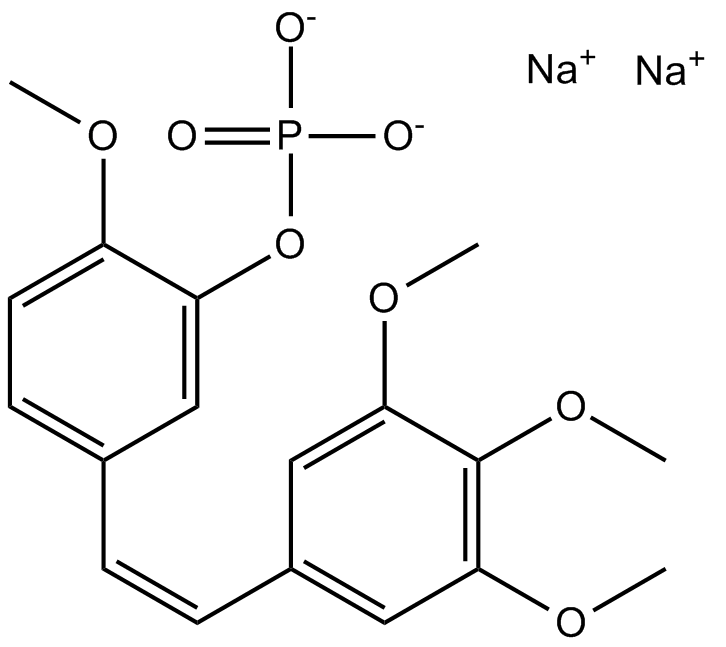
GC12308
Fosbretabulin (Combretastatin A4 Phosphate (CA4P)) Disodium
Fosbretabulin (Combretastatina A4 Fosfato (CA4P)) DisÓdico (CA 4DP) es un agente desestabilizador de tubulina. La fosbretabulina (fosfato de combretastatina A4 (CA4P)) disÓdica es el profÁrmaco de combretastatina A4 que se dirige selectivamente a las células endoteliales, induce la regresiÓn de los neovasos tumorales nacientes, reduce el flujo sanguÍneo tumoral y causa necrosis tumoral central.
-
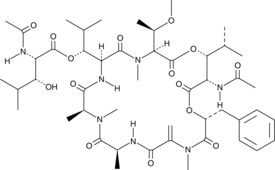
GC49089
FR900359
A cyclic depsipeptide and an inhibitor of Gαq, Gα11, and Gα14
-
GC10251
FRAX1036
FRAX1036 es un inhibidor de PAK con Kis de 23,3 nM, 72,4 nM y 2,4 μM para PAK1, PAK2 y PAK4, respectivamente.
-
GC16075
FRAX486
FRAX486 es un inhibidor de la quinasa activada por p21 (PAK) con IC50 de 14, 33 y 39 nM para PAK1, PAK2 y PAK3, respectivamente.
-
GC12261
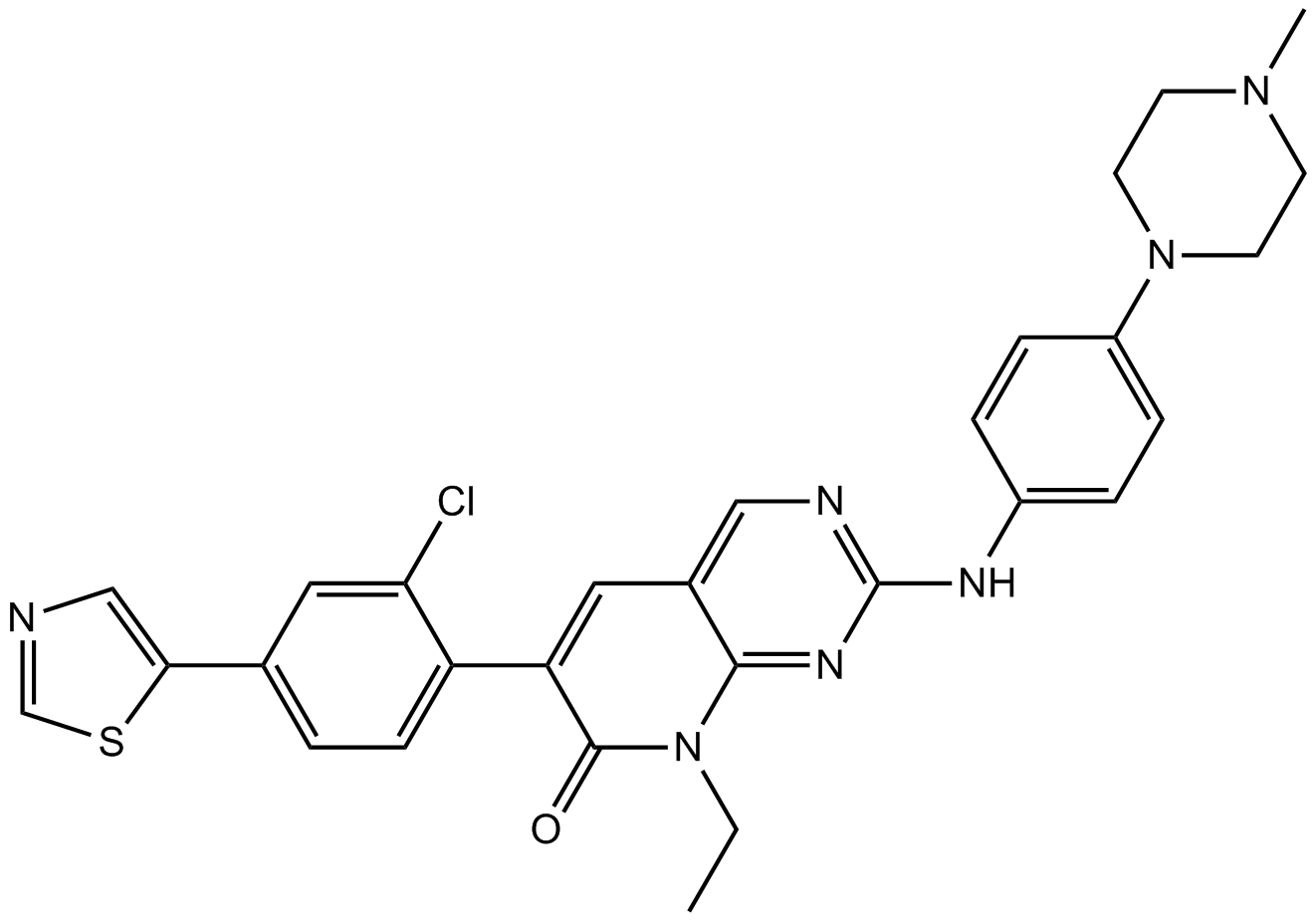
FRAX597
FRAX597 es un potente inhibidor de quinasas activadas por p21 (PAK) del grupo I con IC50 de 8, 13 y 19 nM para PAK1, 2 y 3.
-
GC52288
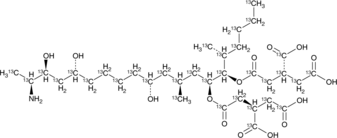
Fumonisin B1-13C34
An internal standard for the quantification of fumonisin B1
-
GC47378
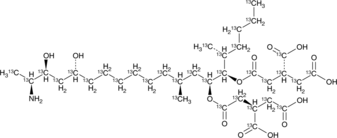
Fumonisin B2-13C34
An internal standard for the quantification of fumonisin B2
-
GC18720
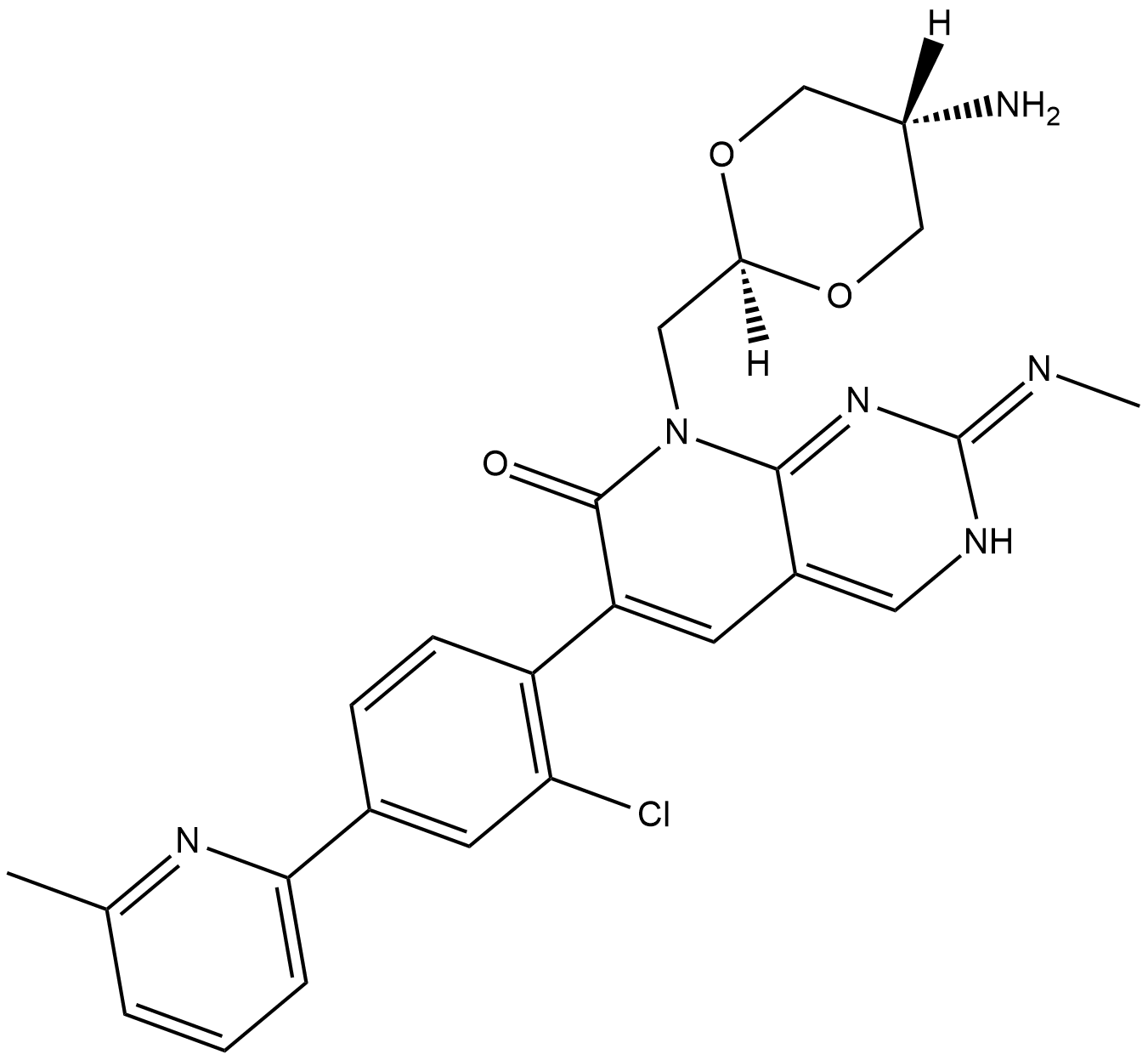
G-5555
G-5555 es un potente inhibidor de la quinasa 1 activada por p21 (PAK1) con Kis de 3,7 nM y 11 nM para PAK1 y PAK2, respectivamente.
-
GC36095
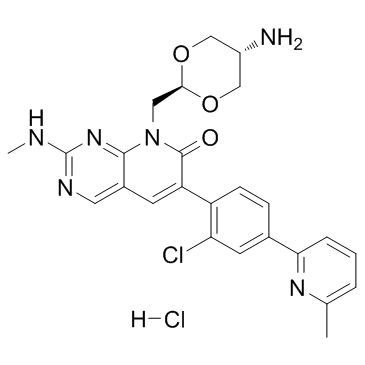
G-5555 hydrochloride
El clorhidrato de G-5555 es un inhibidor potente y selectivo de la quinasa 1 activada por p21 (PAK1) con una Ki de 3,7 nM.
-
GC40016
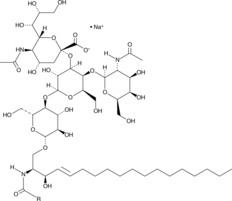
Ganglioside GM2 Mixture (sodium salt)
Ganglioside GM2 is a glycosphingolipid component of cellular membranes, primarily the plasma membrane.
-
GC43732
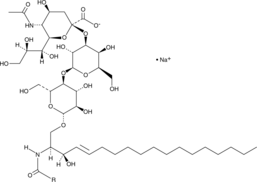
Ganglioside GM3 Mixture (sodium salt)
Ganglioside GM3 is a monosialoganglioside that demonstrates antiproliferative and proapoptotic effects in tumor cells by modulating cell adhesion, proliferation, and differentiation.
-
GC49322
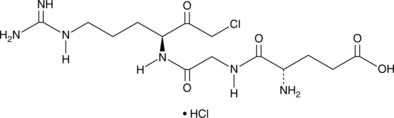
GGACK (hydrochloride)
A uPA and factor Xa inhibitor
-
GC38783
Ginkgolic acid C17:1
El Ácido ginkgolic C17:1, extraÍdo de las hojas de Ginkgo biloba, suprime la activaciÓn constitutiva e inducible de STAT3 a través de la inducciÓn de PTEN y SHP-1 tirosina fosfatasa. El Ácido ginkgolic C17:1 tiene efectos anticancerÍgenos.
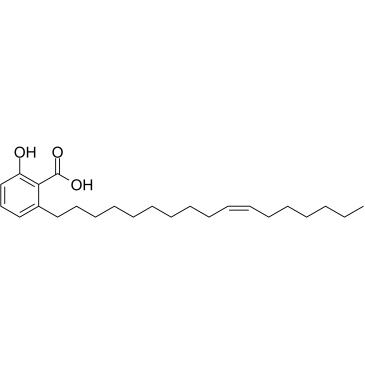
-
GC49437
Gliotoxin-13C13
An internal standard for the quantification of gliotoxin
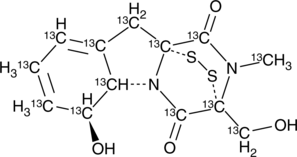
-
GC41563
Globosuxanthone A
Globosuxanthone A es una dihidroxantenona con actividad antifÚngica evidente frente a Fusarium graminearum, Fusarium solani y Botrytis cinerea con valores de MIC de 4, 8 y 16 μg/mL, respectivamente.
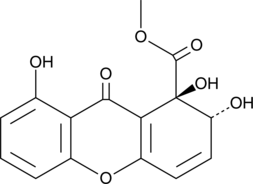
-
GC45668
Gly-Arg-AMC (hydrochloride)
A fluorogenic substrate for cathepsin C
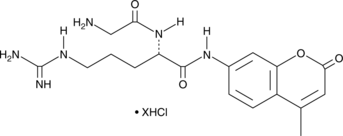
-
GC49687
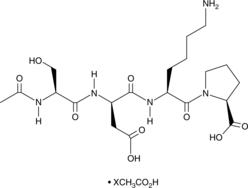
Goralatide (acetate)
A tetrapeptide regulator of hematopoiesis
-
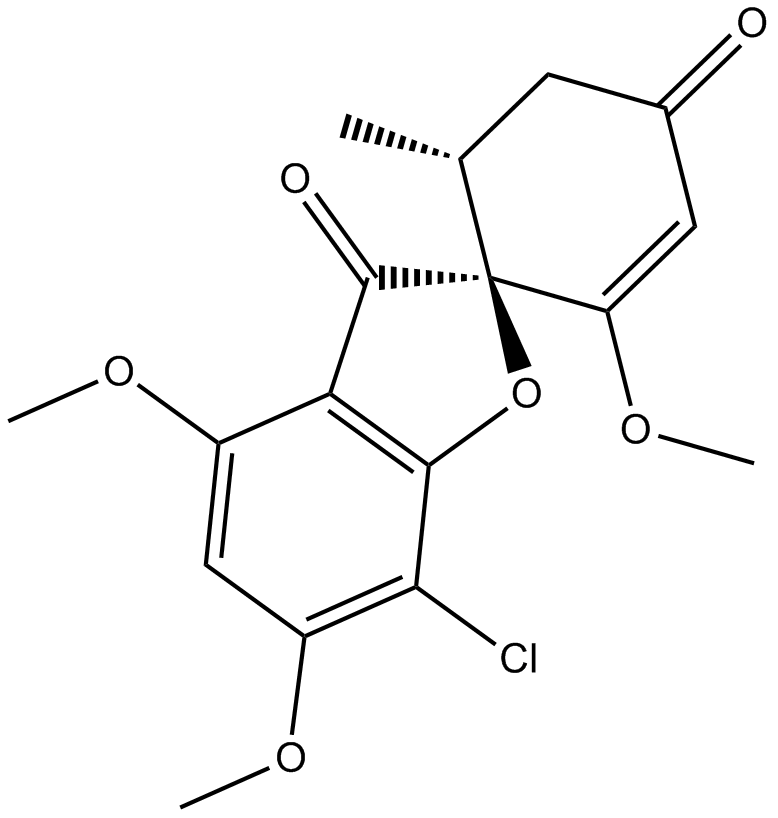
GC48720
Griseofulvic Acid
A metabolite of griseofulvin
-
GC13484
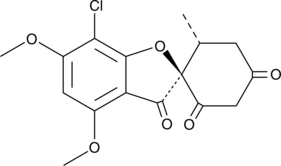
Griseofulvin
La griseofulvina (Gris-PEG; Grifulvin) es un producto natural fÚngico espirocÍclico utilizado en el tratamiento de dermatofitos fÚngicos; Medicamento antifÚngico.
-
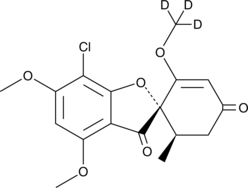
GC48688
Griseofulvin-d3
An internal standard for the quantification of griseofulvin
-
GC49744
GRP94 Monoclonal Antibody (Clone 9G10)
For immunochemical detection of GRP94

-
GC38791
GSK-25
GSK-25 es un inhibidor de ROCK1 potente, selectivo y biodisponible por vÍa oral (IC50 = 7 nM).
-
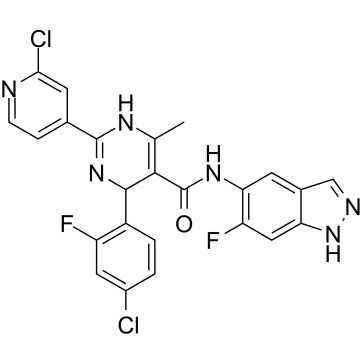
GC15659
GSK-923295
An inhibitor of CENP-E
-
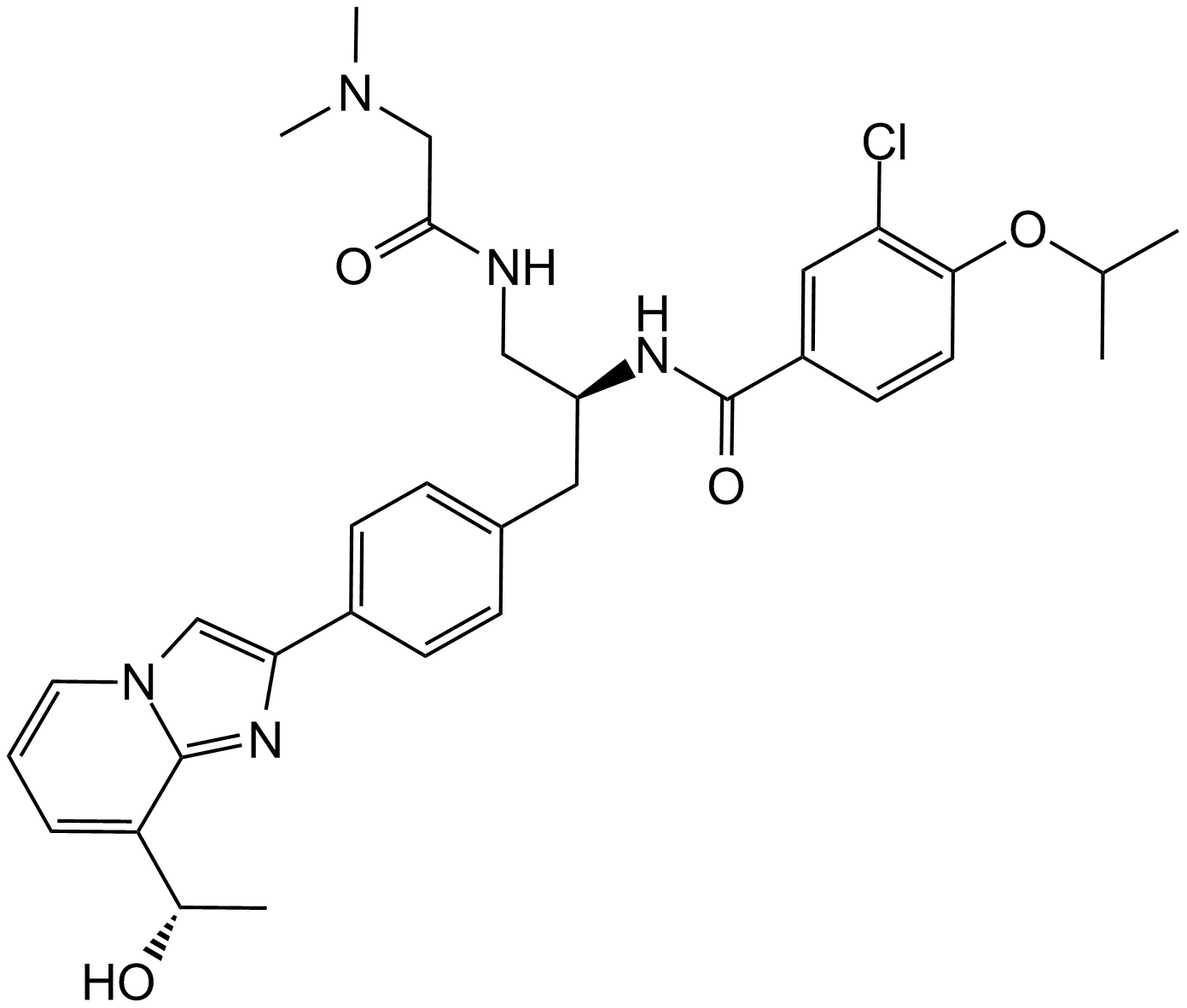
GC19177
GSK180736A
GSK180736A es un potente inhibidor de la quinasa 1 en espiral asociada a Rho (ROCK1) con una IC50 de 100 nM.
-
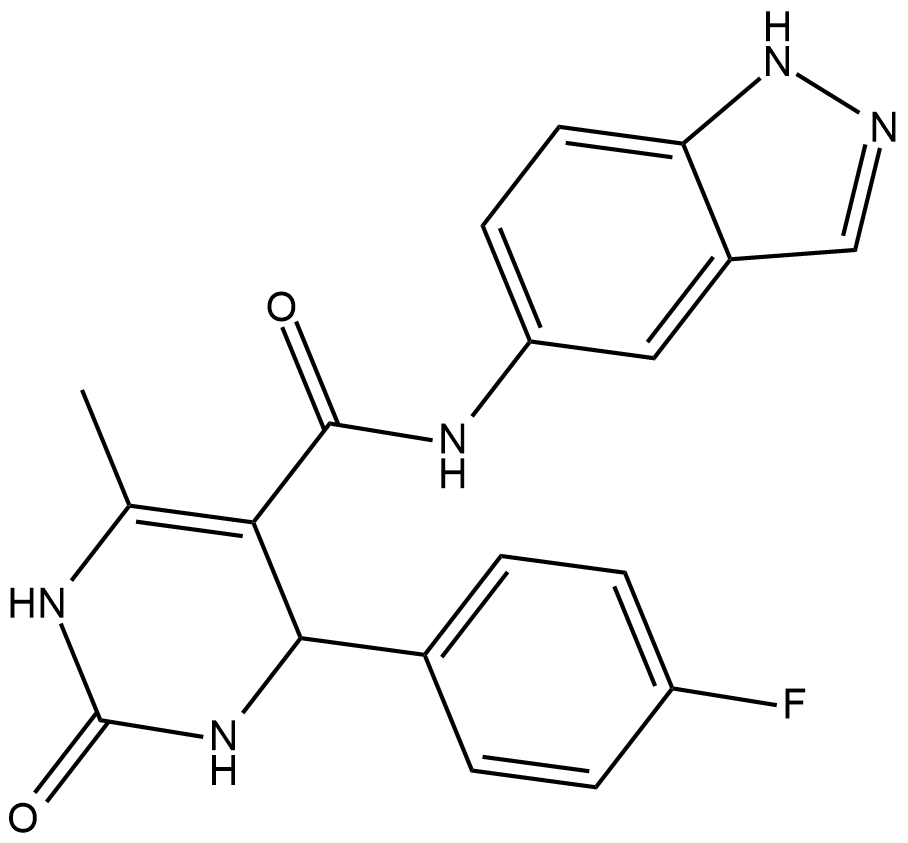
GC17450
GSK2606414
Un inhibidor selectivo de PERK
-
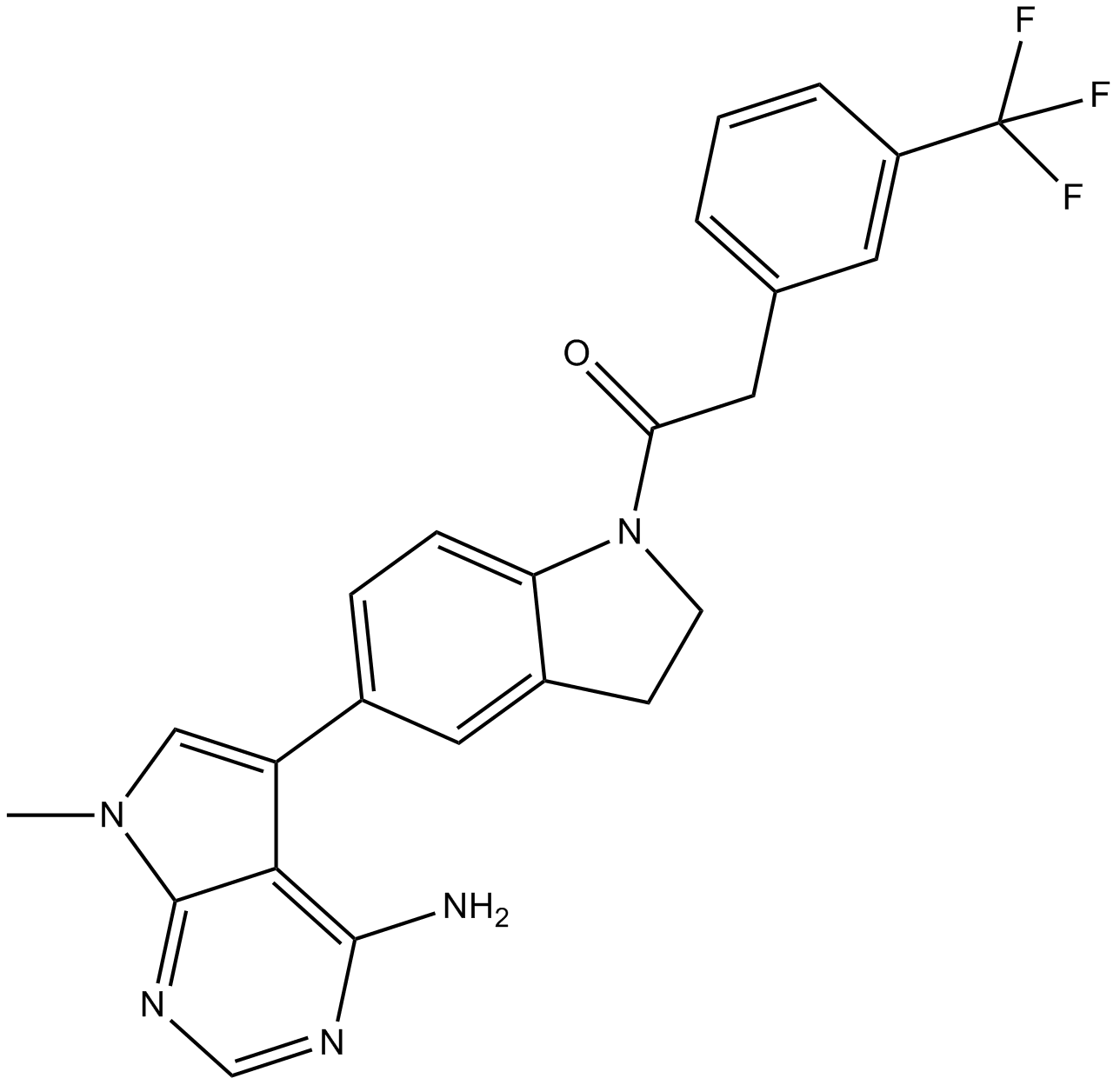
GC11068
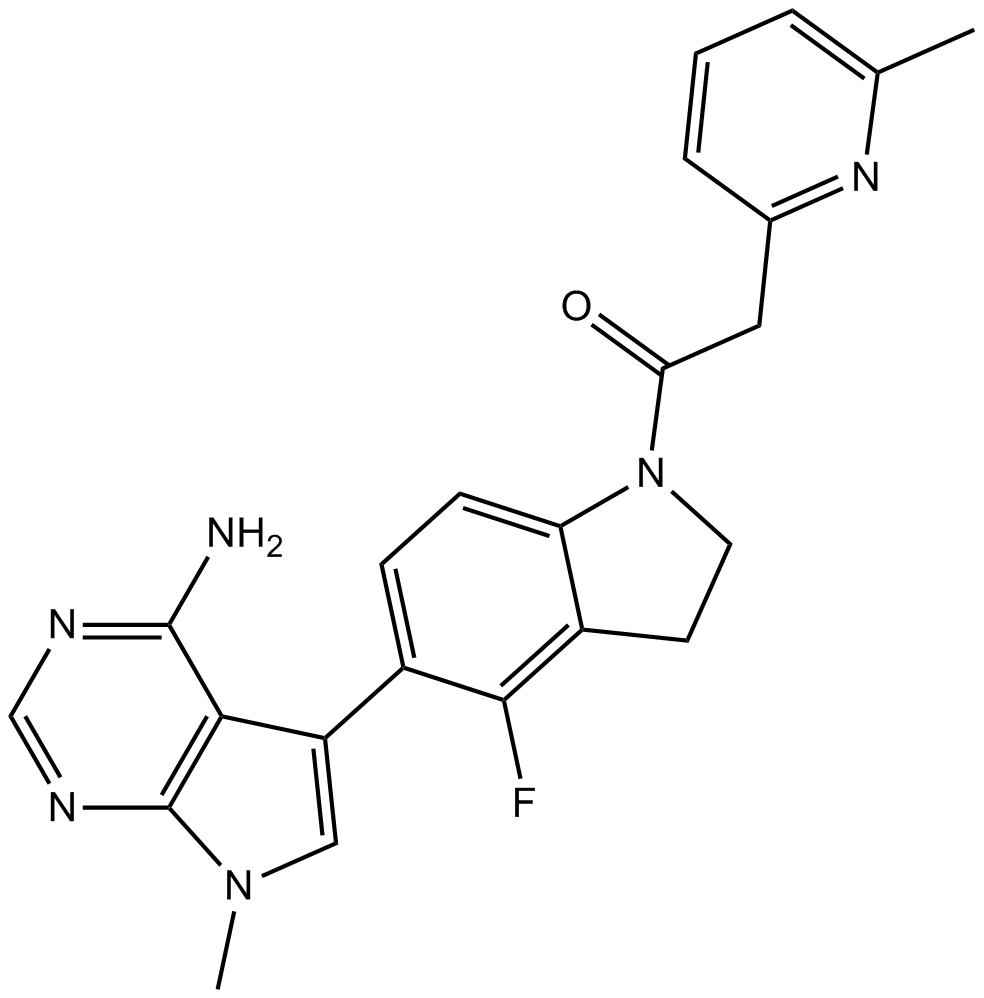
GSK2656157
GSK2656157 es un inhibidor selectivo y competitivo con ATP de la quinasa del retÍculo endoplÁsmico (PERK) similar a la proteÍna quinasa R (PKR) con una IC50 de 0,9 nM.
-
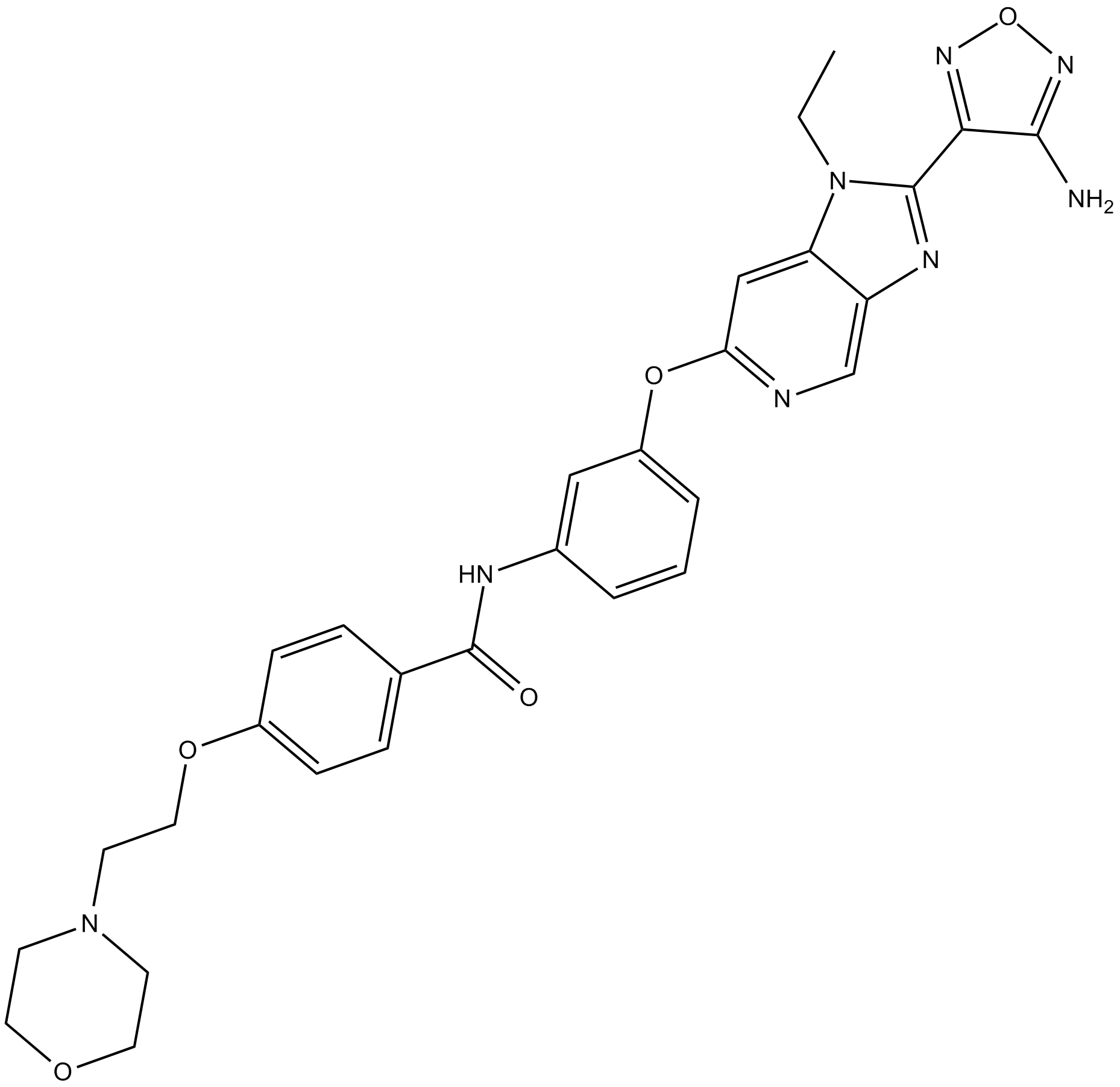
GC17936
GSK269962A
GSK269962A (GSK 269962) es un potente inhibidor de ROCK con IC50 de 1,6 y 4 nM para ROCK1 y ROCK2 humanos recombinantes, respectivamente.
-
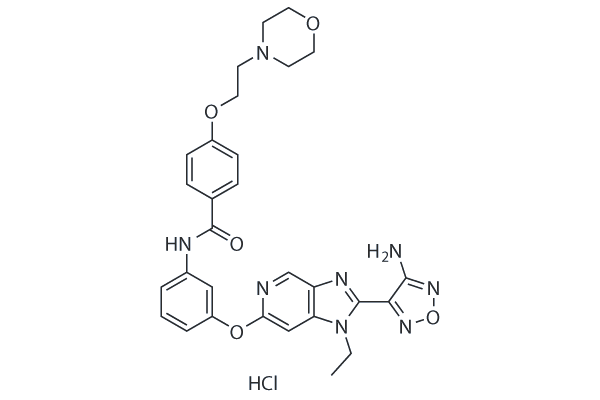
GC25482
GSK269962A HCl
GSK269962A HCl (GSK269962B, GSK269962) is a selective ROCK(Rho-associated protein kinase) inhibitor with IC50 values of 1.6 and 4 nM for ROCK1 and ROCK2, respectively.
-
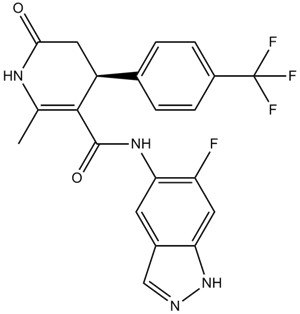
GC18119
GSK429286A
GSK429286A es un inhibidor selectivo de ROCK1 con un valor IC50 de 14 nM.
-
GC12335
GSK461364
GSK461364 es un inhibidor de la quinasa 1 (PLK1) tipo Polo, selectivo, reversible y competitivo con ATP, con un valor de Ki de 2,2 nM.
-
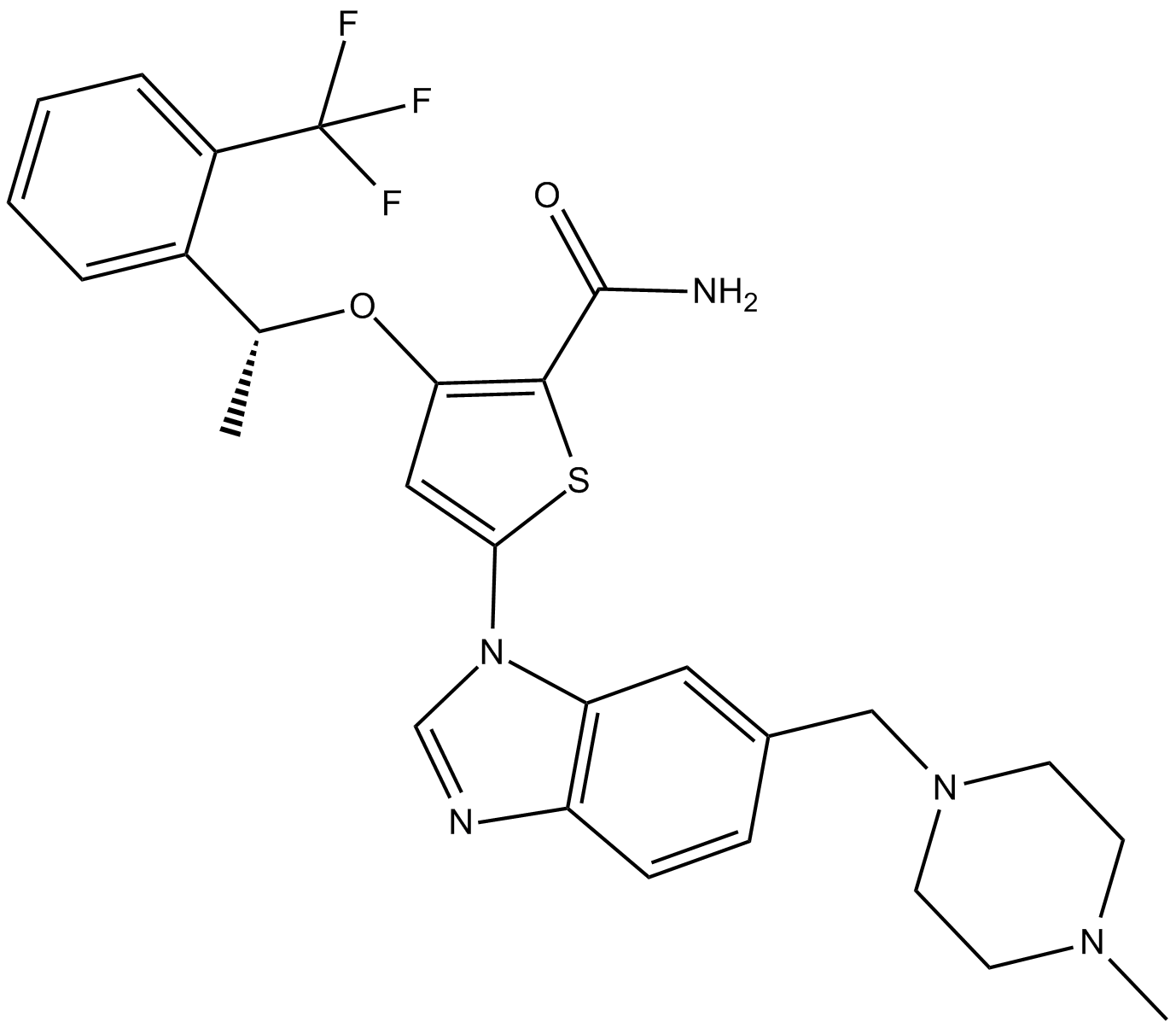
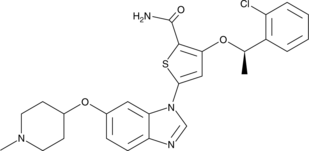
GC49280
GSK579289A
A Plk1 inhibitor
-
GC60891
GW406108X
GW406108X es un inhibidor especÍfico de Kif15 (Kinesin-12) con una IC50 de 0,82 uM en ensayos de ATPasa. GW406108X, un potente inhibidor de la autofagia, muestra una inhibiciÓn competitiva de ATP frente a ULK1 con un pIC50 de 6,37 (427 nM). GW406108X inhibe la actividad de la quinasa ULK1 y bloquea el flujo autofÁgico, sin afectar las quinasas de seÑalizaciÓn aguas arriba mTORC1 y AMPK.
-
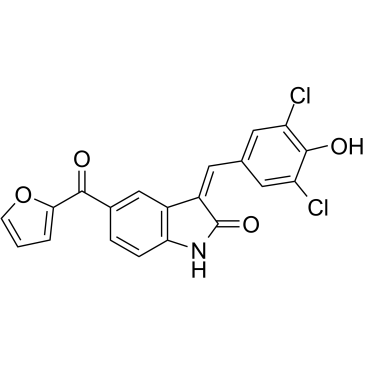
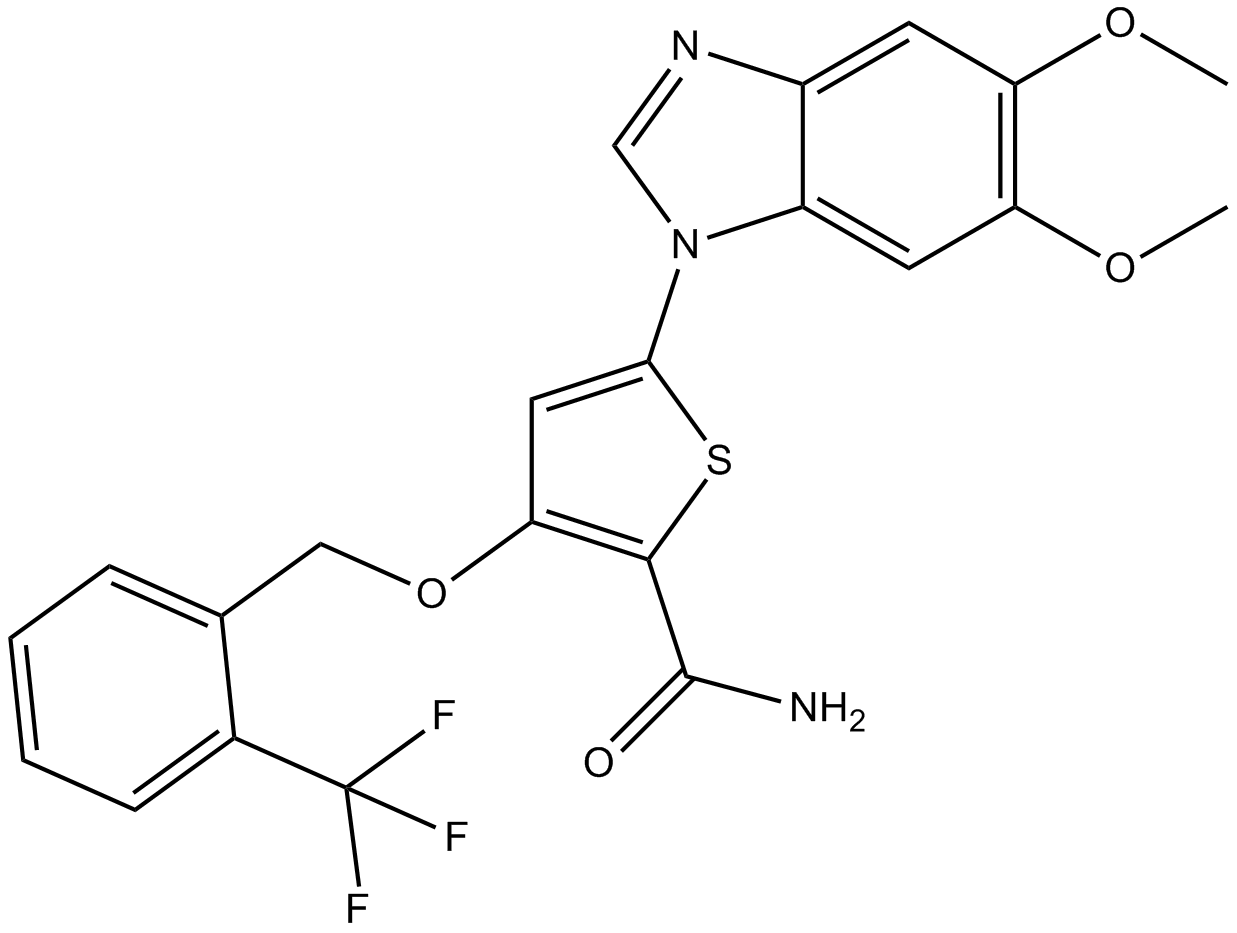
GC11621
GW843682X
A reversible polo-like kinase inhibitor
-
GC16545
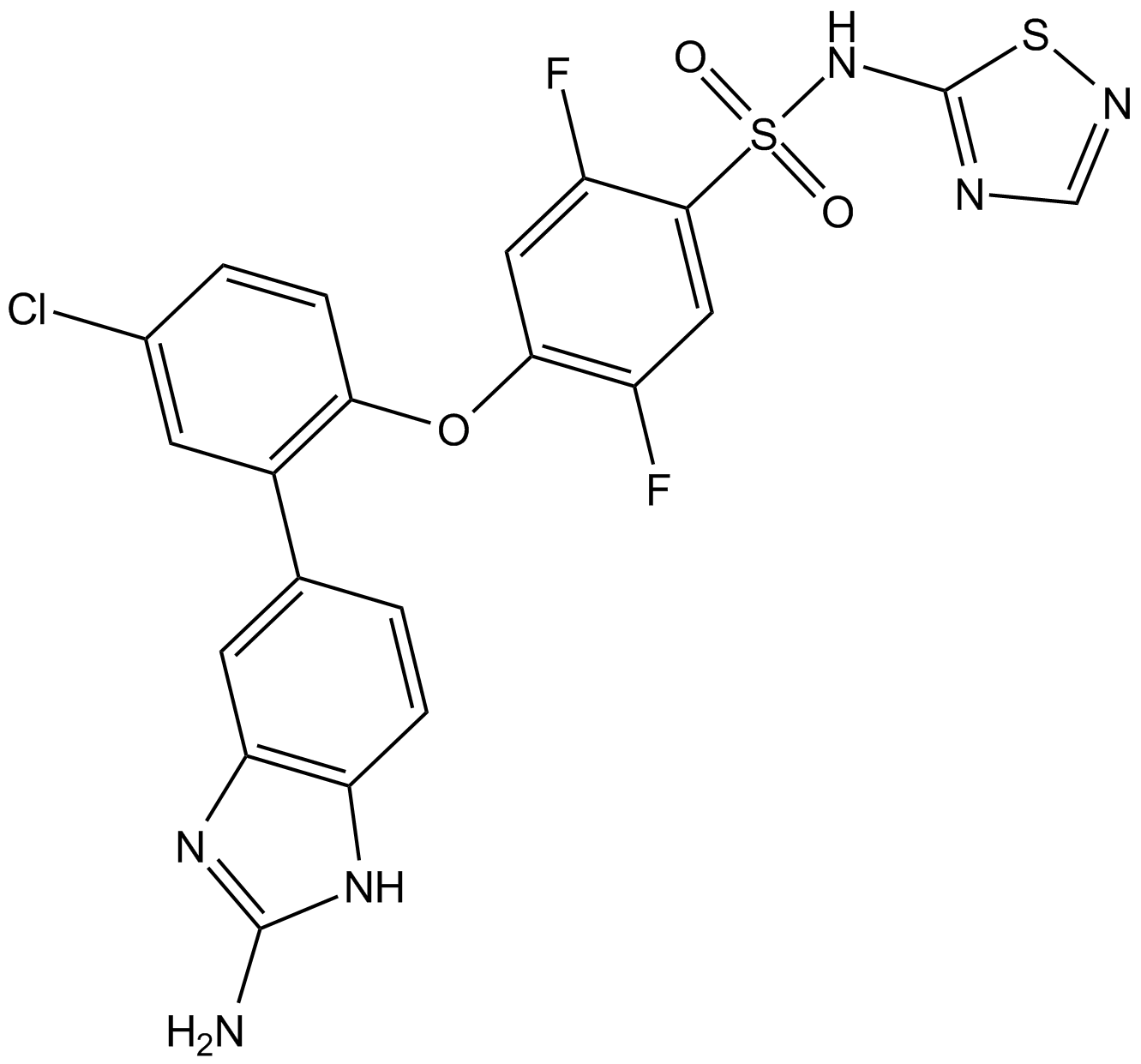
GX-674
GX-674 es un potente antagonista del canal de sodio 1.7 (Nav1.7) controlado por voltaje, dependiente del estado y selectivo de las isoformas con una IC50 de 0,1 nM a -40 mV.
-
GC36207
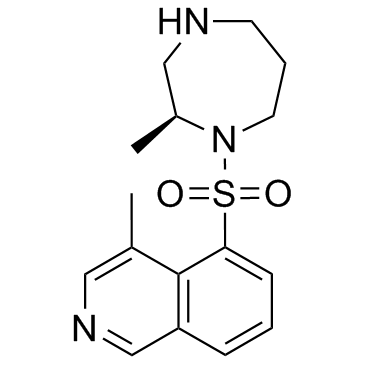
H-1152
H-1152 es un inhibidor de ROCK selectivo y permeable a la membrana, con un valor Ki de 1,6 nM y un valor IC50 de 12 nM para ROCK2.
-
GC36208
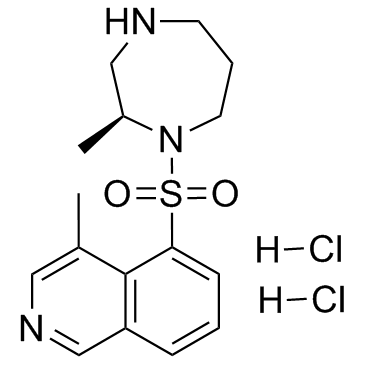
H-1152 dihydrochloride
El diclorhidrato de H-1152 es un inhibidor de ROCK selectivo y permeable a la membrana, con un valor Ki de 1,6 nM y un valor IC50 de 12 nM para ROCK2.