MAPK Signaling

Targets for MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Products for MAPK Signaling
- Cat.No. Nombre del producto Información
-
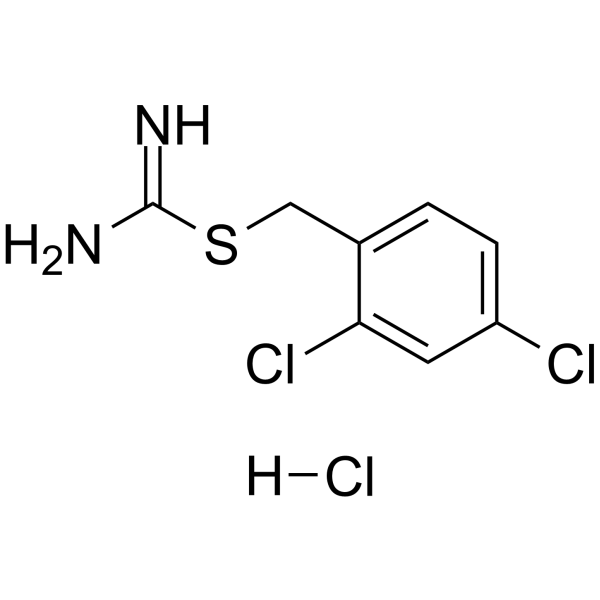
GC36652
MPT0B392
MPT0B392, un derivado de quinolina activo por vÍa oral, induce la activaciÓn de la quinasa N-terminal (JNK) de c-Jun, lo que lleva a la apoptosis. MPT0B392 inhibe la polimerizaciÓn de tubulina y desencadena la inducciÓn de la detenciÓn mitÓtica, seguida de la pérdida del potencial de la membrana mitocondrial y la escisiÓn de las caspasas mediante la activaciÓn de JNK y, en Última instancia, conduce a la apoptosis. Se ha demostrado que MPT0B392 es un nuevo agente despolimerizante de microtÚbulos y mejora la citotoxicidad de sirolimus en células leucémicas agudas resistentes a sirolimus y en la lÍnea celular multirresistente.
-
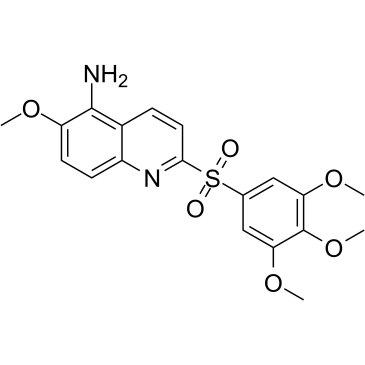
GC64292
MS432
MS432 es un degradador PROTAC de reclutamiento de von Hippel-Lindau basado en PD0325901, el primero en su clase y altamente selectivo para MEK1 y MEK2. MS432 muestra una buena exposiciÓn al plasma en ratones, exhibiendo valores de DC50 de 31 nM y 17 nM para MEK1, MEK2 en células HT29 respectivamente.
-
GC65297
MW-150
MW150 (MW01-18-150SRM) es un inhibidor selectivo, penetrante del SNC y activo por vÍa oral de p38α MAPK con una Ki de 101 nM.
-
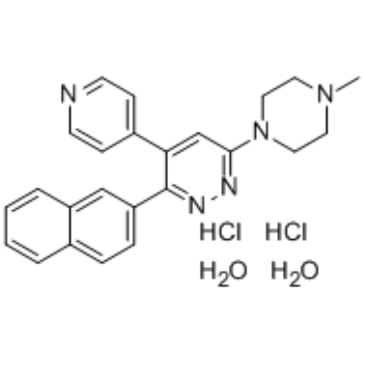
GC34918
MW-150 dihydrochloride dihydrate
El dihidrocloruro de MW-150 dihidrato (MW01-18-150SRM dihidrocloruro de dihidrato) es un inhibidor selectivo, penetrante del SNC y activo por vÍa oral de p38α MAPK con una Ki de 101 nM.
-
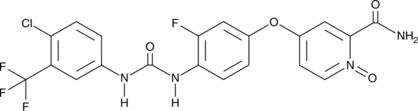
GC49686
N-desmethyl Regorafenib N-oxide
An active metabolite of regorafenib
-
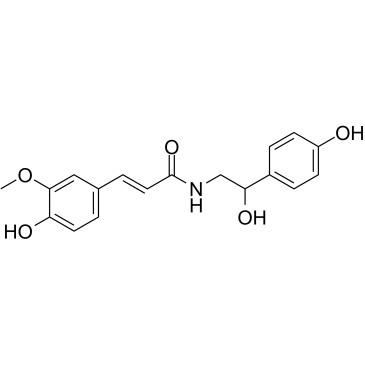
GC60262
N-Feruloyloctopamine
La N-feruloiloctopamina es un constituyente antioxidante. La N-feruloiloctopamina disminuye significativamente los niveles de fosforilación de Akt y p38 MAPK.
-
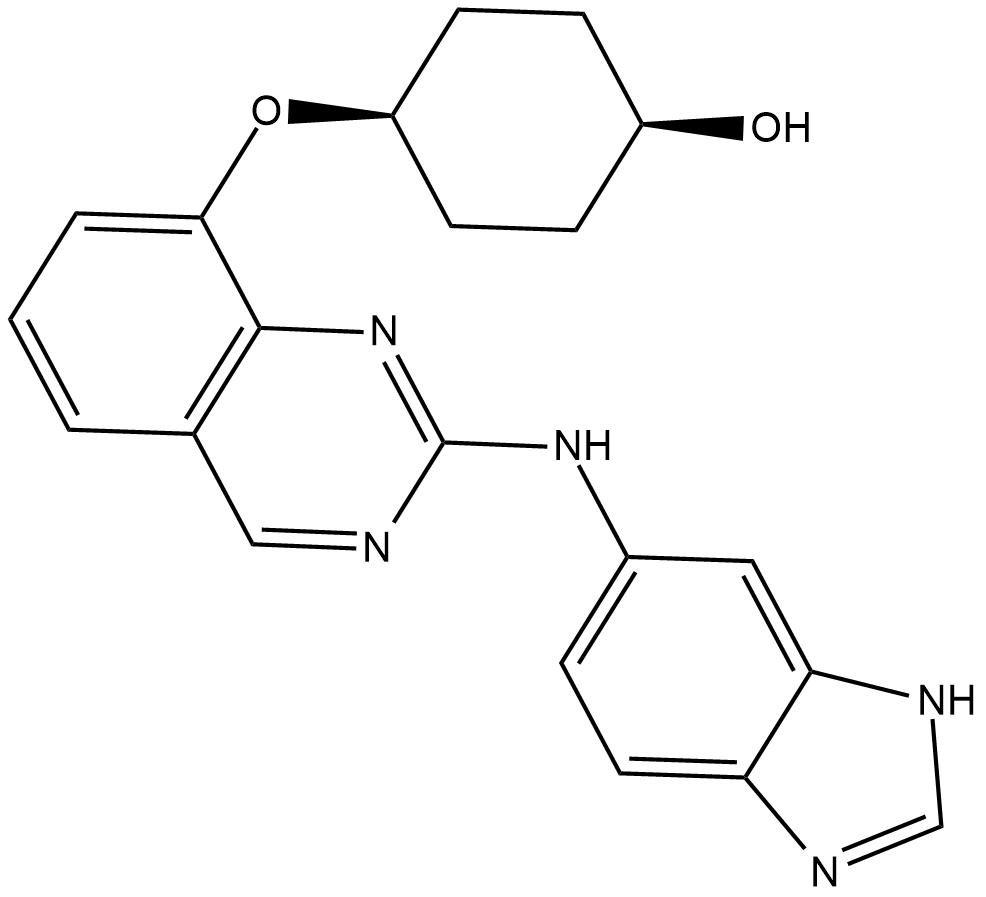
GC19260
NCB-0846
NCB-0846 es un inhibidor de TNIK disponible por vÍa oral con un IC50 de 21nM.
-
GC69543
NecroIr1
NecroIr1 es un compuesto de iridio (III) que induce la necrosis en células resistentes al cisplatino (A549R) en cáncer de pulmón. NecroIr1 se acumula selectivamente en las mitocondrias, lo que causa estrés oxidativo y pérdida del potencial de membrana mitocondrial (MMP). NecroIr1 puede activar la quinasa serina-treonina receptora interactiva proteína 3 (RIPK3) y la pseudocinasa con dominio similar a mezcla de linaje celular (MLKL), regulando así la expresión de CDK4.
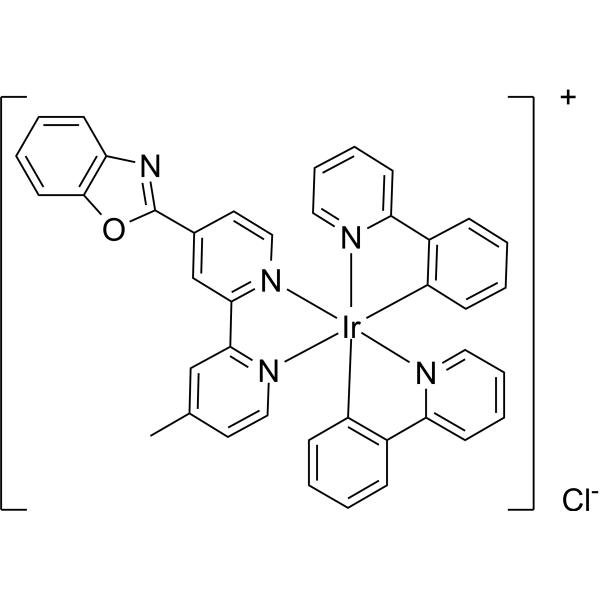
-
GC69544
NecroIr2
NecroIr2 es un compuesto de iridio (III) que induce la necrosis en células resistentes al cisplatino (A549R) en cáncer de pulmón. NecroIr2 se acumula selectivamente en las mitocondrias, lo que causa estrés oxidativo y pérdida del potencial de membrana mitocondrial (MMP). NecroIr2 puede activar la quinasa serina-treonina receptora interactiva con RIPK3 y el dominio similar a quinasa pseudocinasa mixta MLKL, regulando la expresión de CDK4.
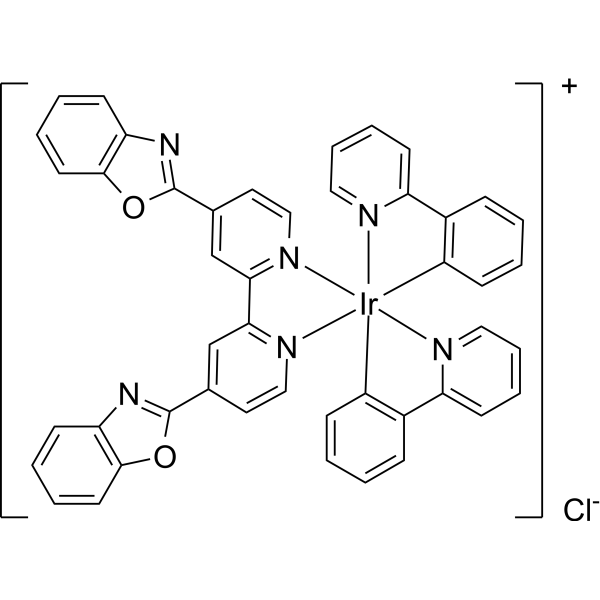
-
GC47771
NG 25 (hydrochloride hydrate)
An inhibitor of MAP4K2 and TAK1
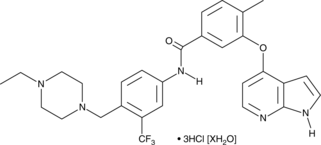
-
GC13514
NG25
An inhibitor of MAP4K2 and TAK1

-
GC17577
NQDI 1
A selective inhibitor of ASK1
-
GC10069
NSC 23766 trihydrochloride
El trihidrocloruro de NSC 23766 es un inhibidor de la activaciÓn de Rac1.
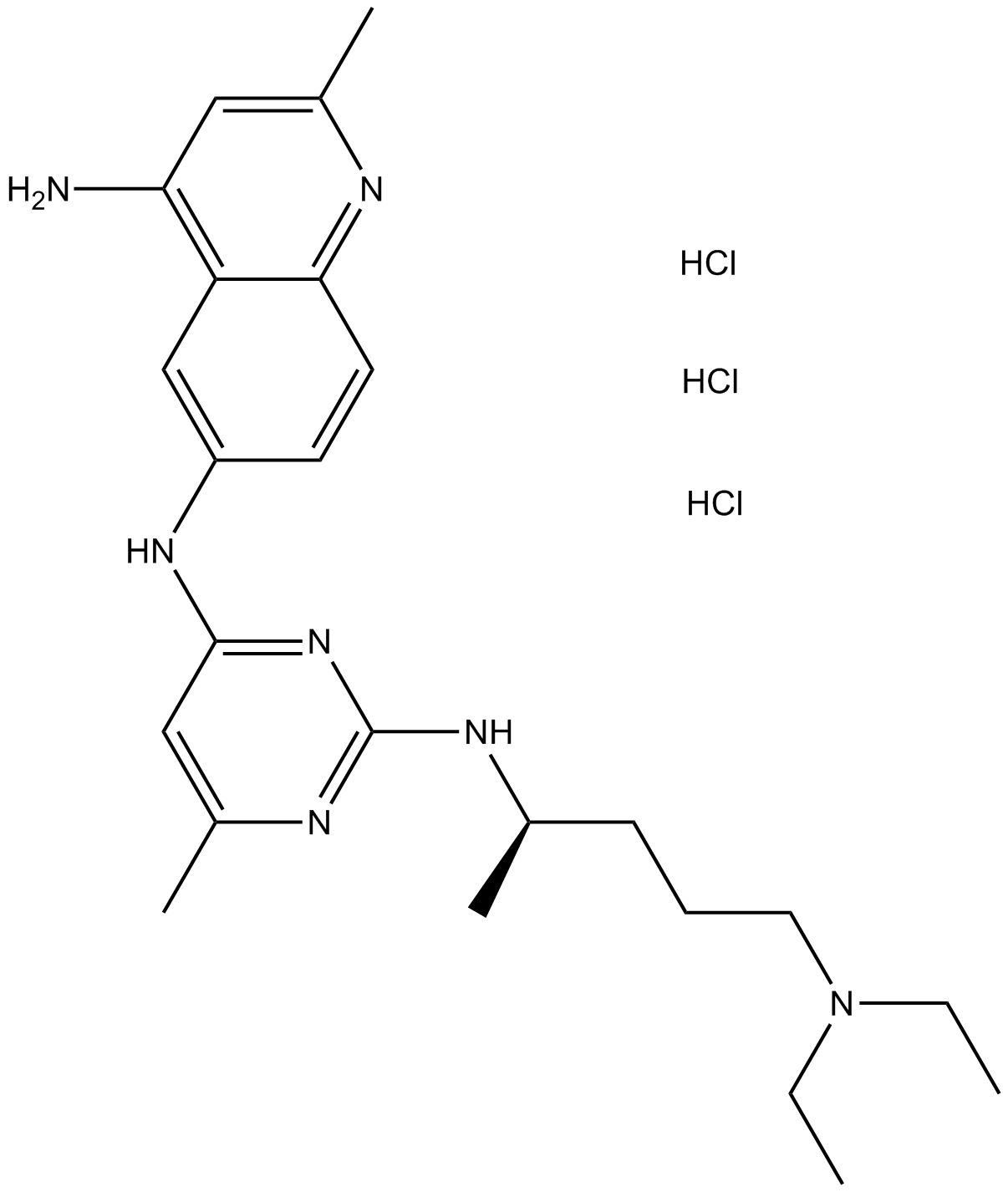
-
GC49186
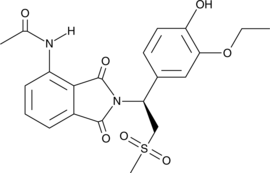
O-Demethyl Apremilast
An active metabolite of apremilast
-
GC18951
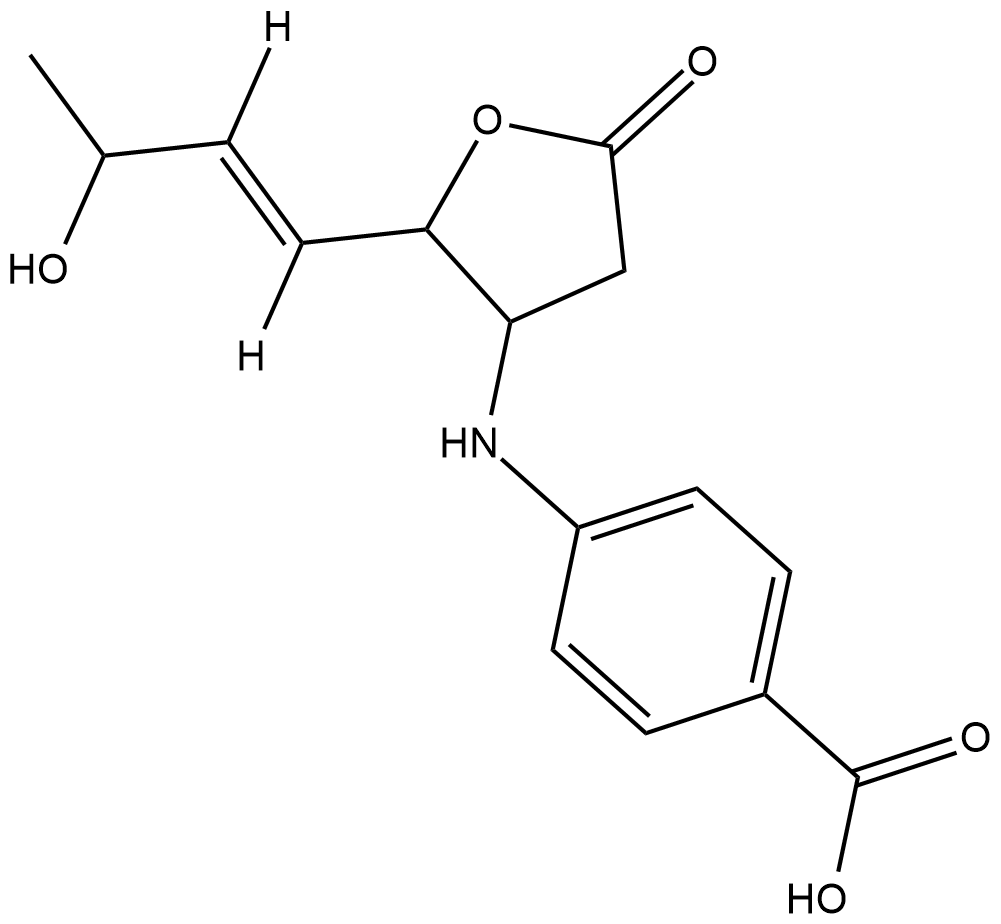
Obscurolide A1
Obscurolide A1 is a natural butyrolactone isolated from S.
-
GC48652
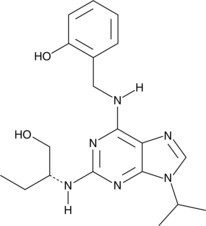
Olomoucine II
A CDK inhibitor
-
GC33857
Omtriptolide
Omtriptolide (PG490-88) es un profÁrmaco derivado de triptolide purificado de la hierba china.
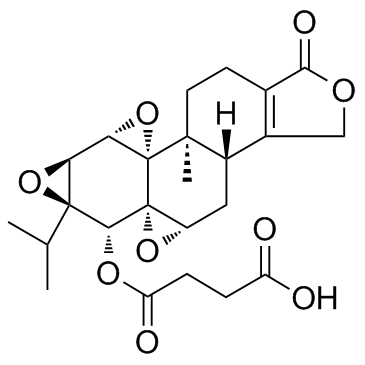
-
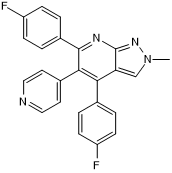
GC50316
Org 48762-0
Selective p38α/β inhibitor; orally bioavailable
-
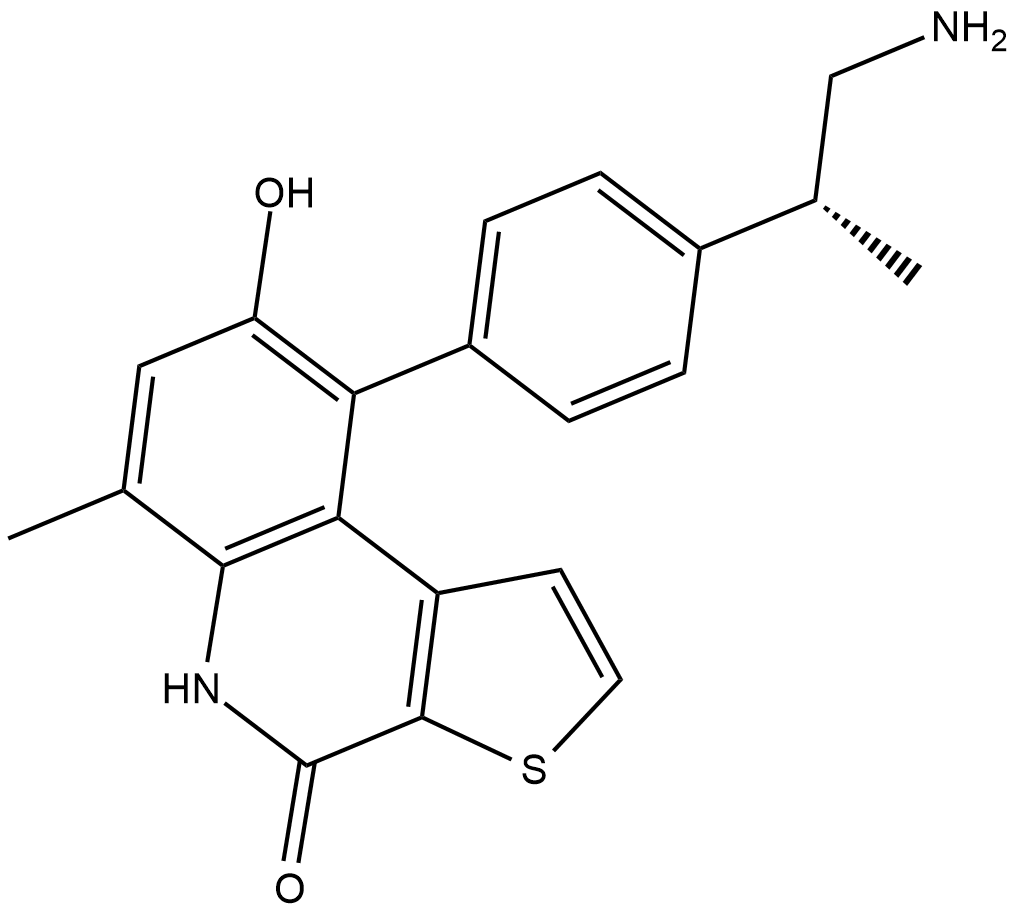
GC12304
OTS514
OTS514 es un inhibidor de TOPK muy potente con una IC50 de 2,6 nM. OTS514 suprime fuertemente el crecimiento de células cancerosas positivas para TOPK. OTS514 induce la detenciÓn del ciclo celular y la apoptosis.
-
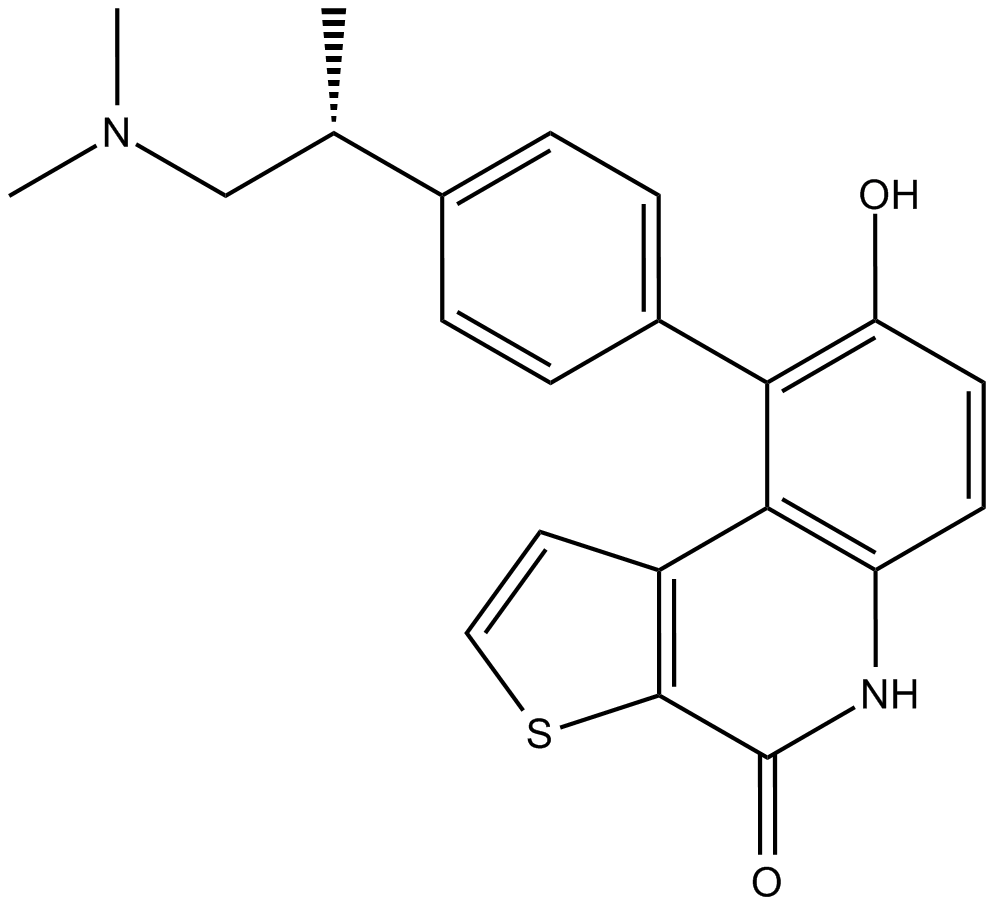
GC16511
OTS964
OTS964 es un inhibidor de TOPK (proteÍna quinasa originada en células asesinas activada por linfocina T) activo por vÍa oral, de alta afinidad y selectivo con una IC50 de 28 nM. OTS964 también es un potente inhibidor de la cinasa dependiente de ciclina CDK11, que se une a CDK11B con una Kd de 40 nM.
-
GC69639
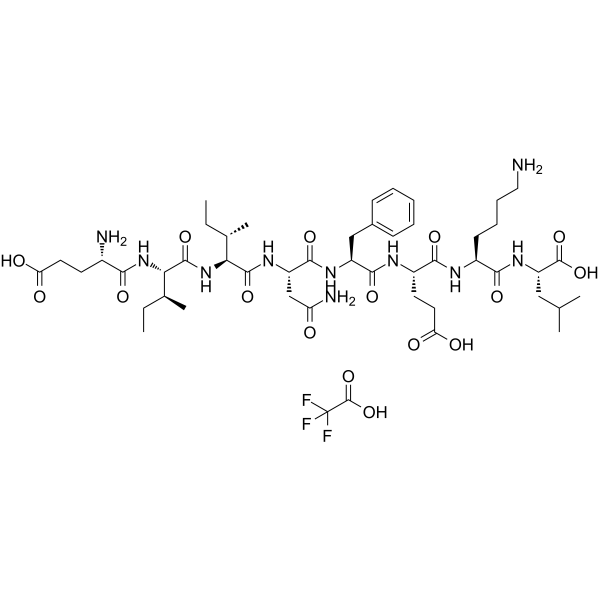
OVA-E1 peptide TFA
El péptido OVA-E1 TFA es una variante antagonista de SIINFEKL [OVA (257-264)]. El péptido OVA-E1 activa las cascadas p38 y JNK en células del timo tanto mutantes como salvajes.
-
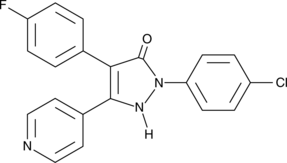
GC44530
p38 MAPK Inhibitor
p38 MAPK inhibitor is a potent inhibitor of p38 MAP kinase (IC50 = 35 nM).
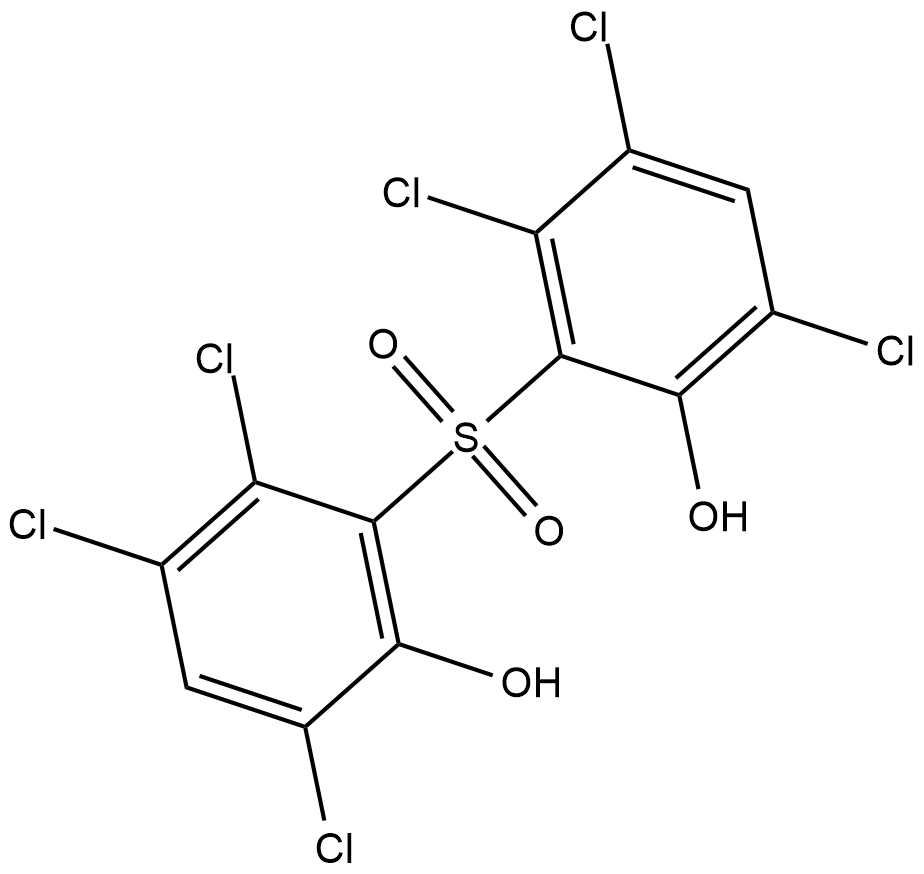
-
GC18602
p38 MAPK Inhibitor IV
p38 MAPK Inhibitor IV es un p38α altamente especÍfico competitivo con ATP; inhibidor de MAPK con IC50 de 0,13 y 0,55 μ M para p38α y p38β MAPK, respectivamente.
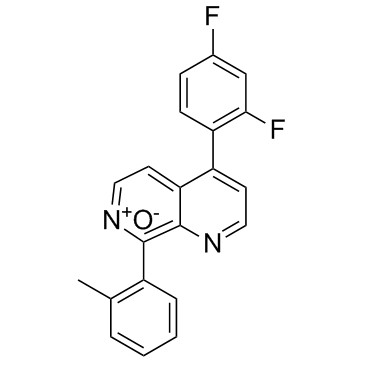
-
GC33008
p38 MAPK-IN-1
p38 MAPK-IN-1 (Compuesto 4) es un nuevo inhibidor potente y selectivo de p38 MAPK con IC50 de 68 nM.
-
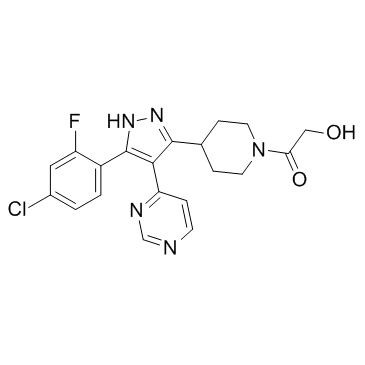
GC30609
p38 MAPK-IN-2
p38 MAPK-IN-2 es un inhibidor de la quinasa p38.
-
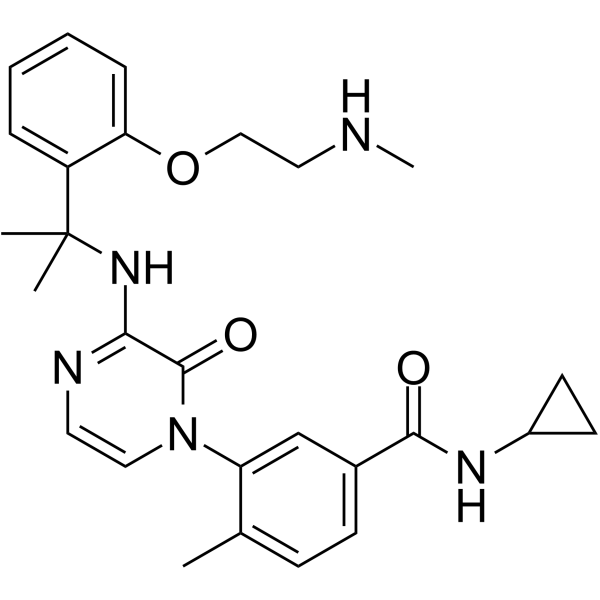
GC62404
p38α inhibitor 2
p3&8#945; el inhibidor 2 es un p3&8#945 altamente potente y selectivo; Inhibidor de MAPK, con un pIC50 de 9,6.
-
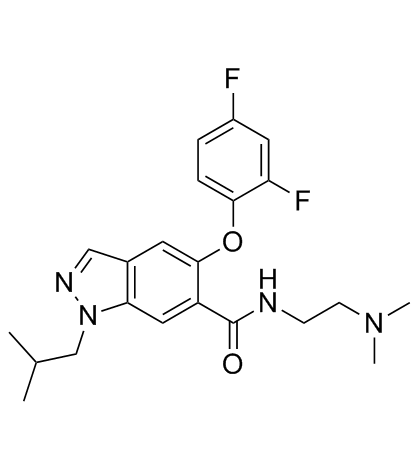
GC31988
p38α inhibitor 1
p38&alfa; el inhibidor 1 es un inhibidor de p38α extraído de la patente WO 2008076265 A1.
-
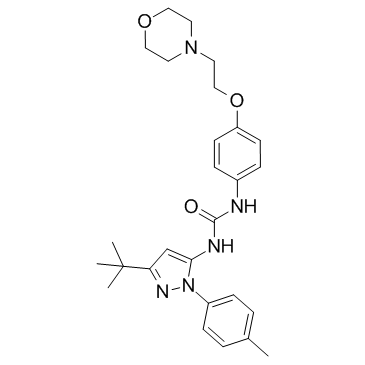
GC30158
p38-α MAPK-IN-1
p38-&alfa; MAPK-IN-1 es un inhibidor de MAPK14 (p38-α), con IC50 de 2300 nM en el ensayo de desplazamiento EFC y 5500 nM en el ensayo HTRF.
-
GN10752
Pachymic acid
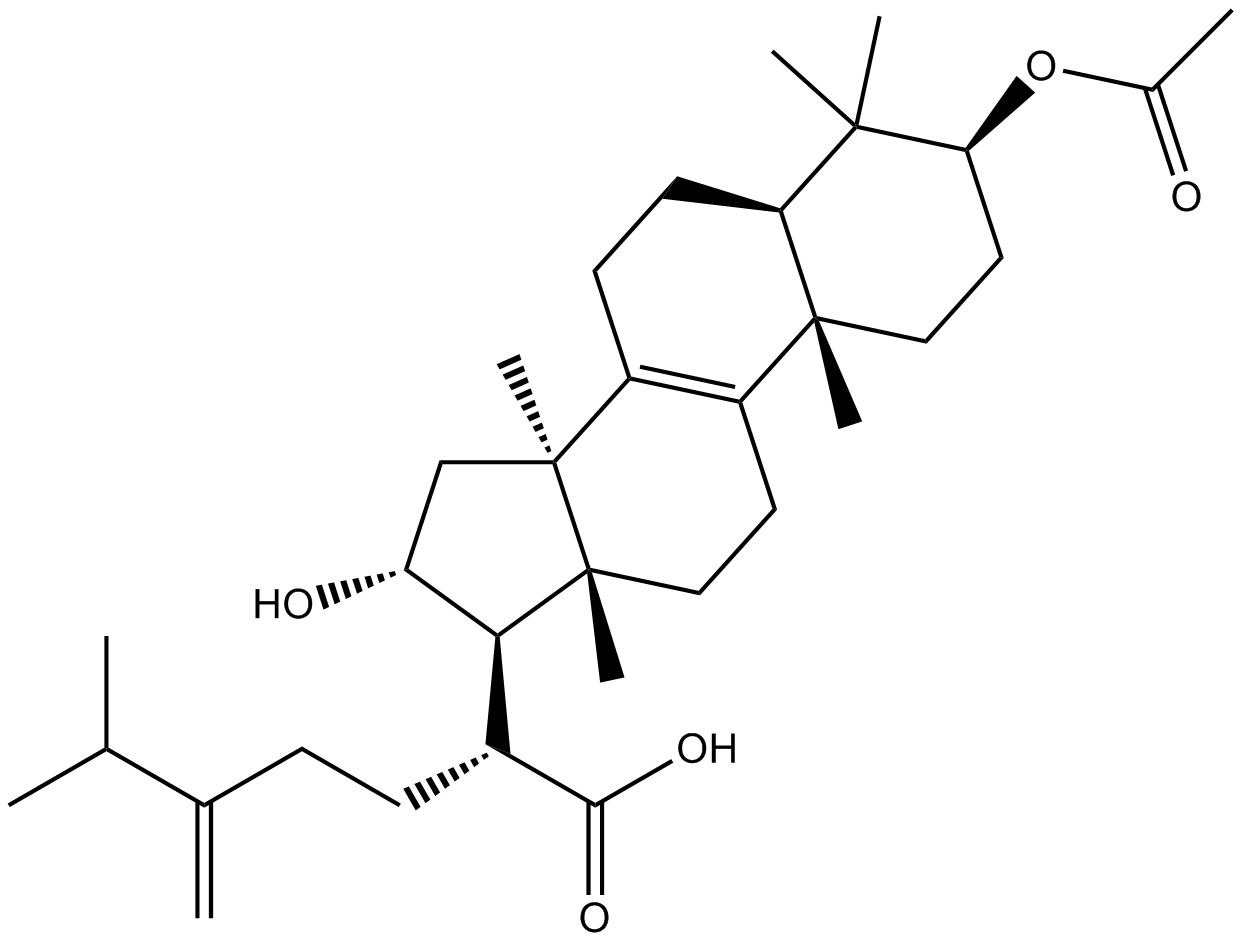
-
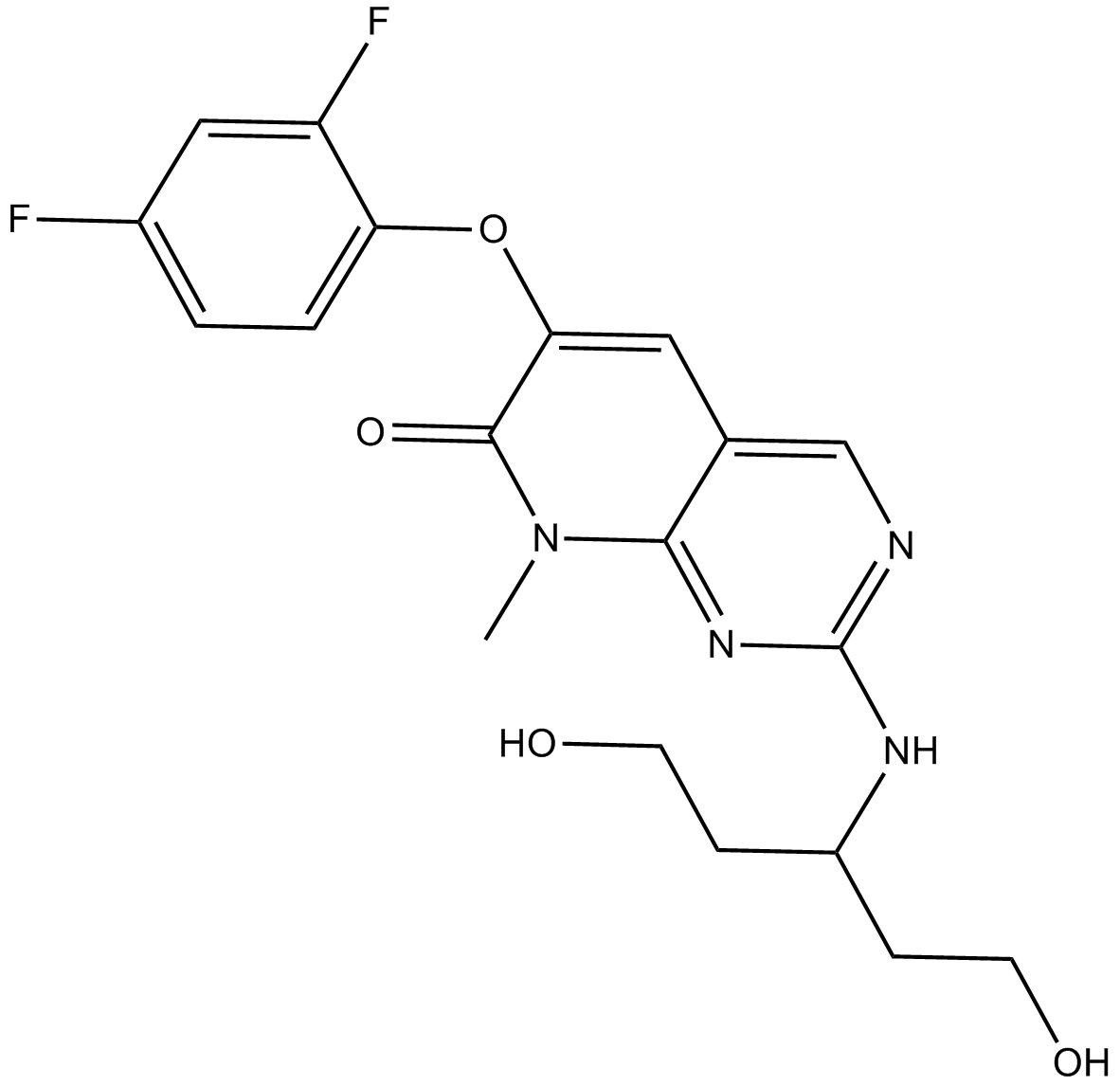
GC15814
Pamapimod (R-1503, Ro4402257)
Pamapimod (R-1503, Ro4402257) (Ro4402257) es un inhibidor potente, selectivo y oralmente activo de p38 MAPK con IC50 de 14 nM y 480 nM y Kis de 1,3 nM y 120 nM para p38α y p38β, respectivamente.
-
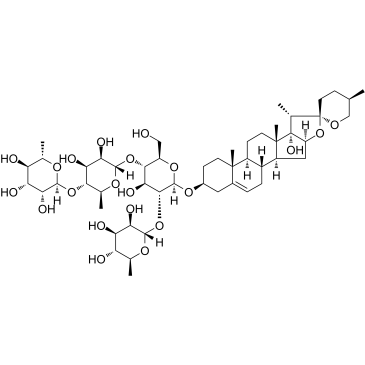
GC36855
Paris saponin VII
Paris saponin VII (Chonglou Saponin VII) es una saponina esteroide aislada de las raÍces y rizomas de Trillium tschonoskii Maxim. La apoptosis inducida por saponina VII de Paris en células K562/ADR estÁ asociada con Akt/MAPK y la inhibiciÓn de P-gp. La saponina VII de Paris atenÚa el potencial de la membrana mitocondrial, aumenta la expresiÓn de proteÍnas relacionadas con la apoptosis, como Bax y el citocromo c, y disminuye los niveles de expresiÓn de proteÍnas de Bcl-2, caspasa-9, caspasa-3, PARP-1 y p- Act. Paris saponin VII induce una autofagia robusta en células K562/ADR y proporciona una base bioquÍmica en el tratamiento de la leucemia.
-
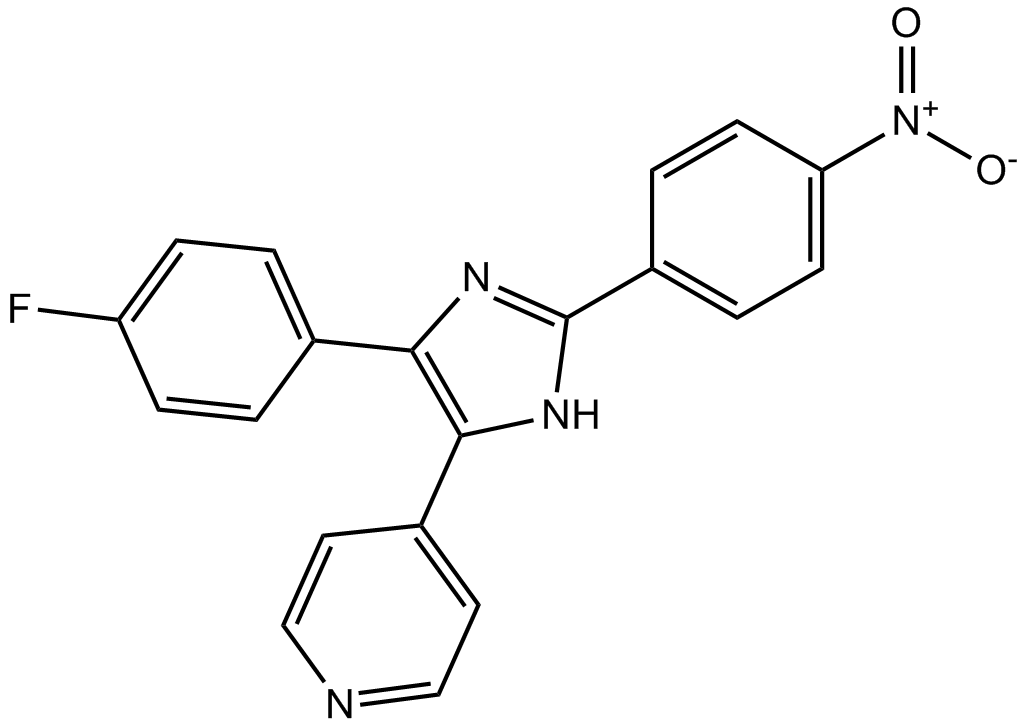
GC17737
PD 169316
PD 169316 es un inhibidor potente, permeable a las células y selectivo de la MAP quinasa p38, con una IC50 de 89 nM. PD169316 inhibe selectivamente la actividad quinasa de la p38 fosforilada sin impedir que las quinasas corriente arriba fosforilen la p38. PD169316 muestra actividad antiviral contra Enterovirus71. PD169316 muestra actividad antiviral contra Enterovirus71.
-
GC15646
PD 184161
PD 184161 es un inhibidor de MEK activo por vía oral.
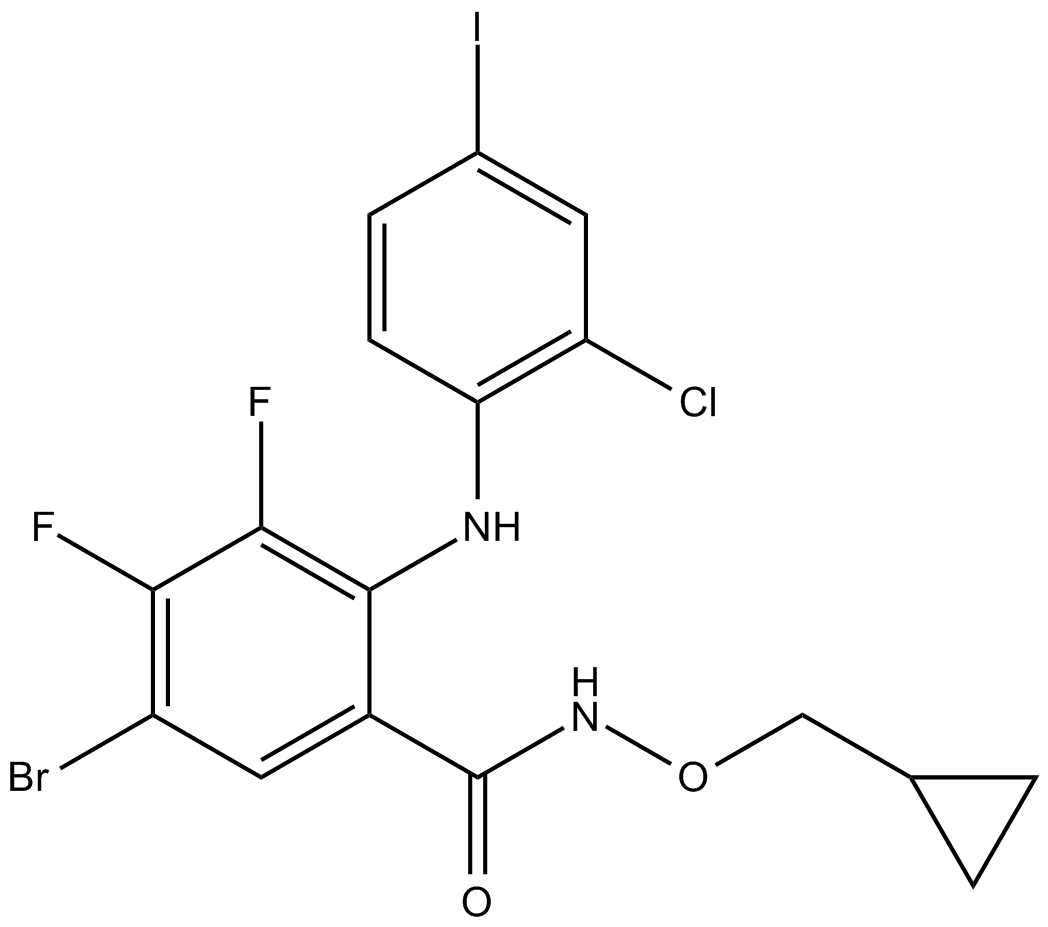
-
GC17964
PD 198306
PD 198306 es un inhibidor selectivo de MAPK/ERK-quinasa (MEK).
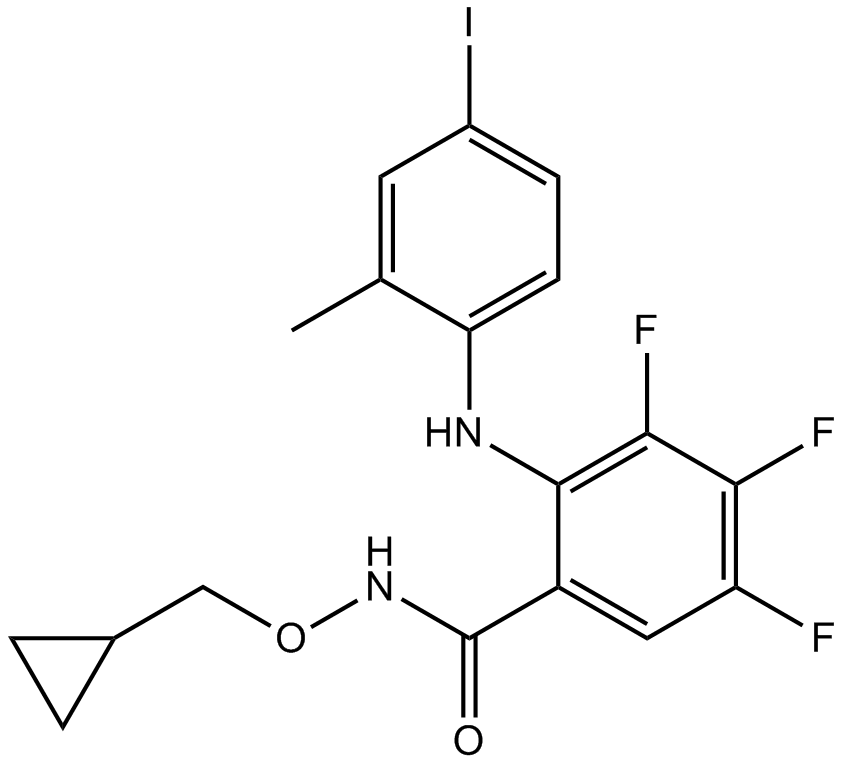
-
GC17485
PD 334581
PD 334581 es un inhibidor de MEK1.
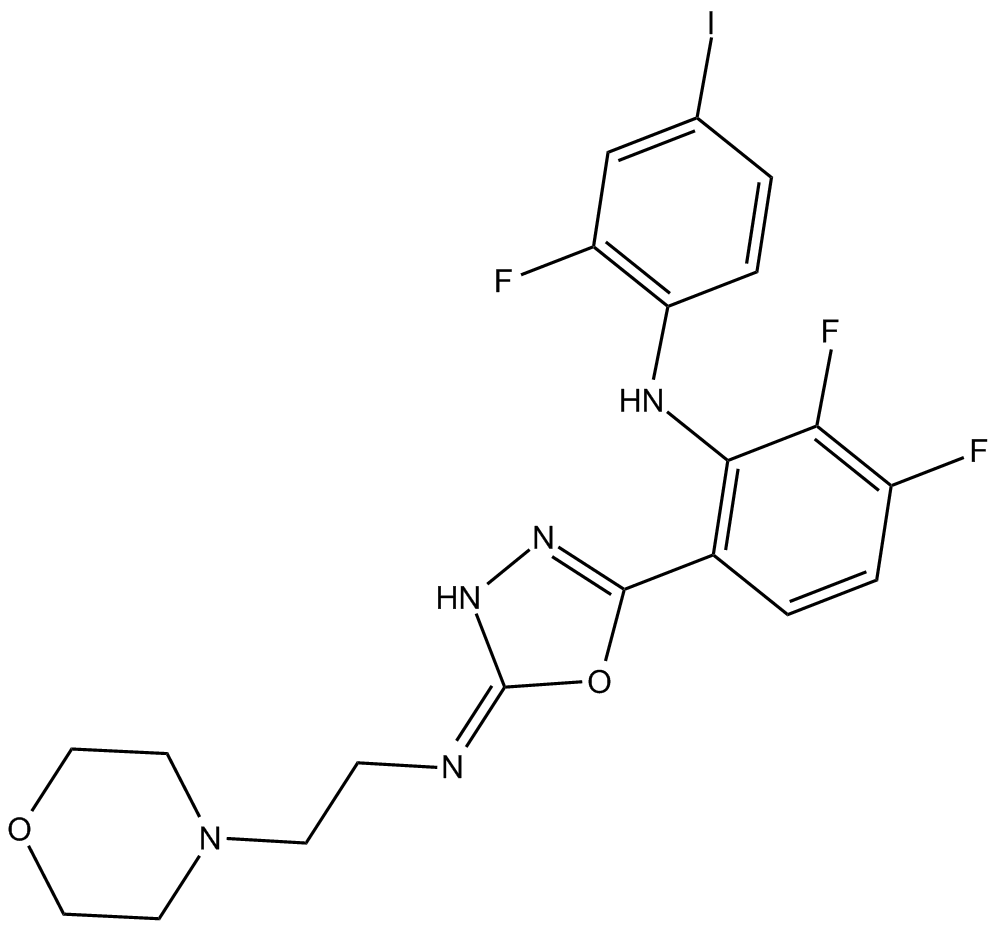
-
GC10397
PD0325901
Un inhibidor de MEK que sostiene la renovación de células madre.
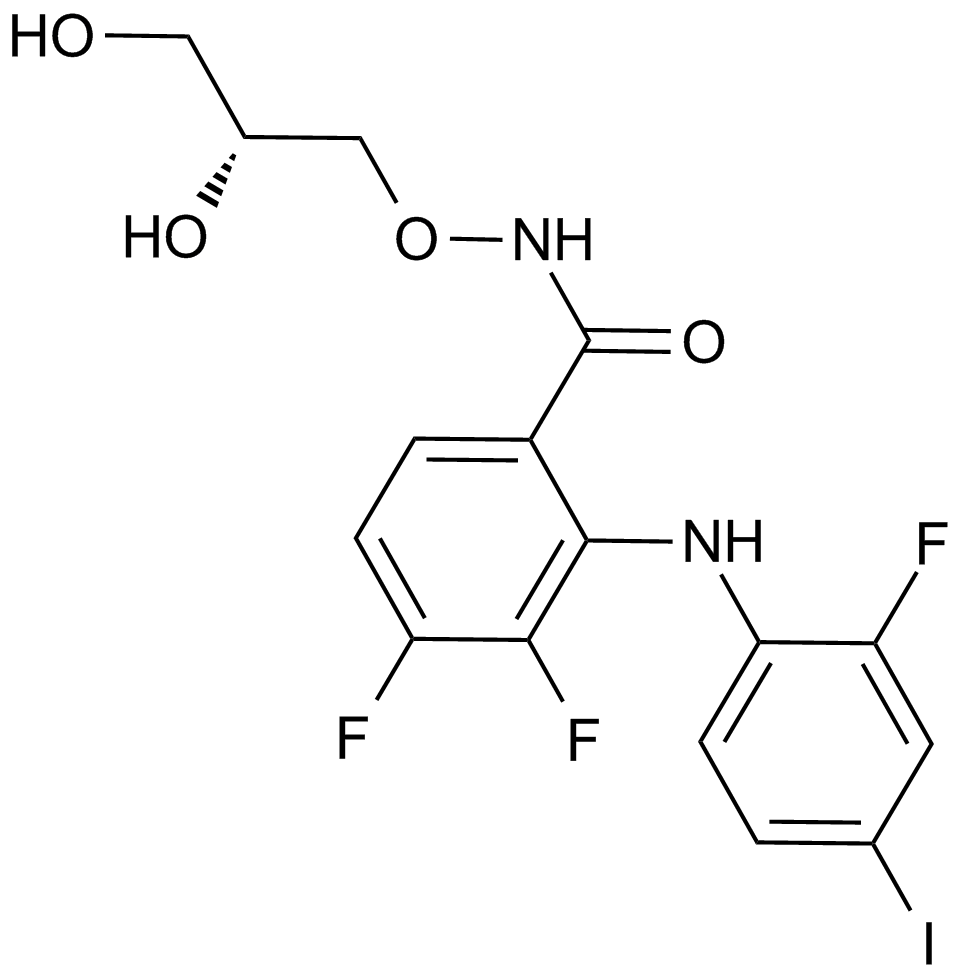
-
GC63554
PD0325901-O-C2-dioxolane
PD0325901-O-C2-dioxolano tiene la porciÓn principal del inhibidor de MEK PD0325901. PD0325901-O-C2-dioxolano y un ligando de VHL o CRBN E3 ligasa pueden usarse en la sÍntesis del degradador MEK1/2.
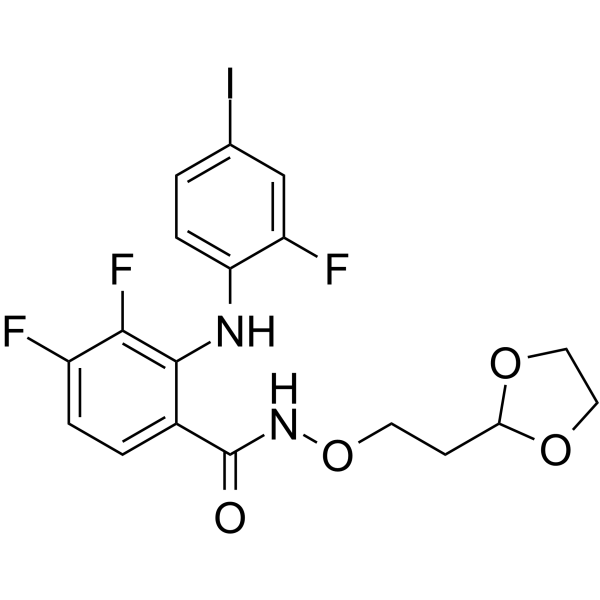
-
GC12989
PD184352 (CI-1040)
PD184352 (CI-1040) (PD 184352) es un inhibidor de molécula pequeÑa altamente especÍfico activo por vÍa oral de MEK con una IC50 de 17 nM para MEK1.
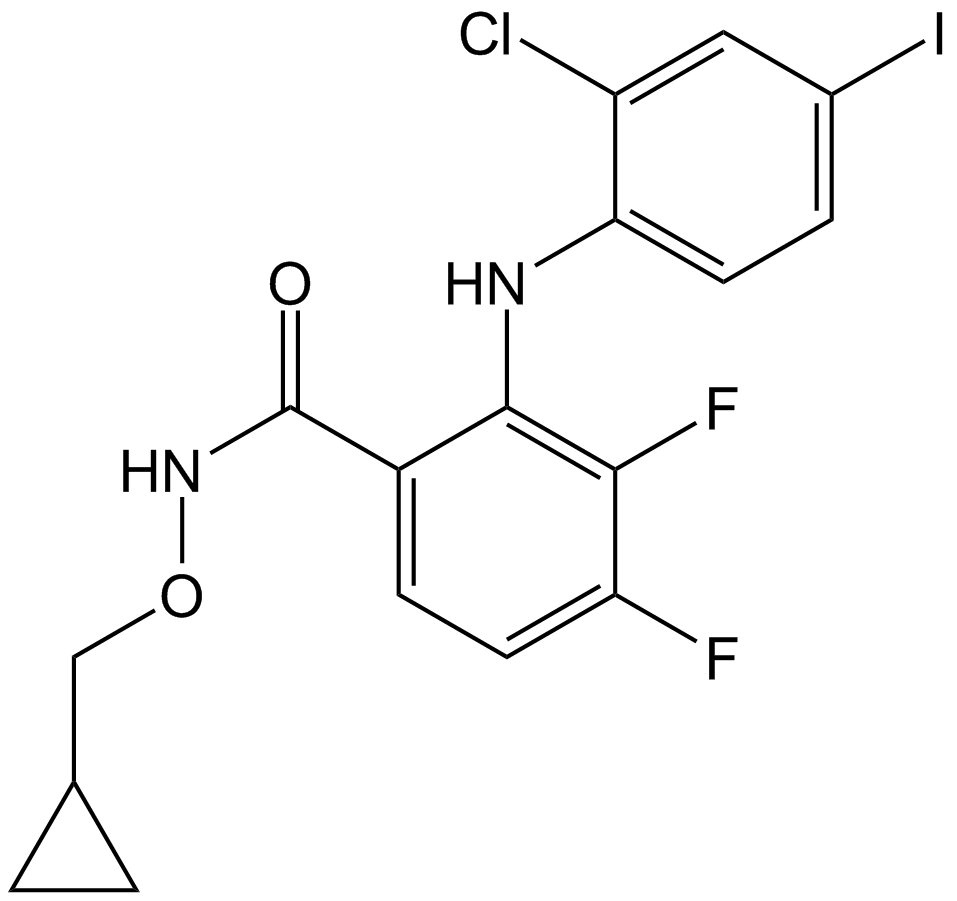
-
GC10110
PD318088
PD318088 es un inhibidor de MEK1/2 potente, alostérico y no competitivo de ATP, un anÁlogo de PD184352. PD318088 se une simultÁneamente con ATP en una regiÓn del sitio activo de MEK1 que estÁ adyacente al sitio de uniÓn de ATP. PD318088 se puede utilizar para la investigaciÓn del cÁncer.
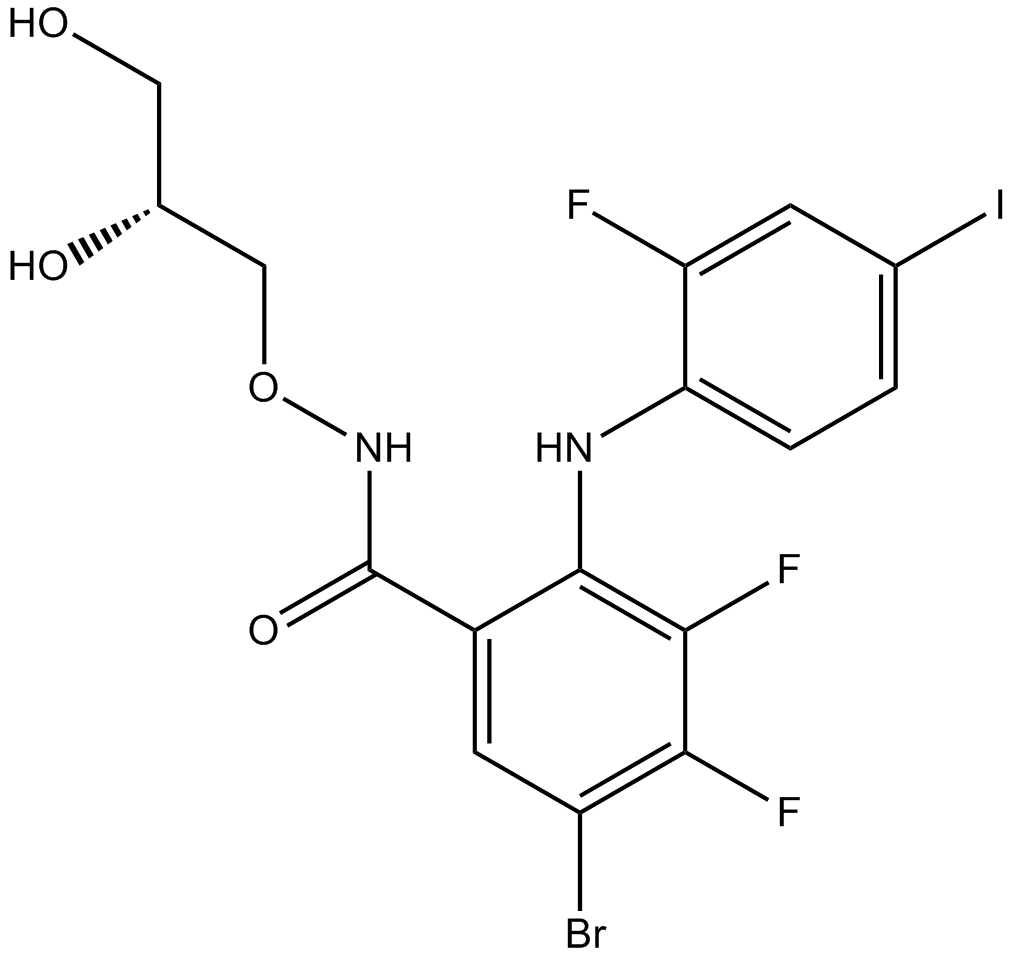
-
GC12819
PD98059
PD98059 es un inhibidor de MEK potente y selectivo con una IC50 de 5 μM. PD98059 se une a la forma inactiva de MEK, evitando asÍ la activaciÓn de MEK1 (IC50 de 2-7 μM) y MEK2 (IC50 de 50 μM) por quinasas corriente arriba. PD98059 es un inhibidor de la seÑalizaciÓn de ERK1/2. PD98059 es un ligando para el receptor de hidrocarburo de arilo (AHR) y suprime la uniÓn de TCDD (IC50 de 4 μM) y la transformaciÓn de AHR (IC50 de 1 μM). PD98059 también inhibe la autofagia.
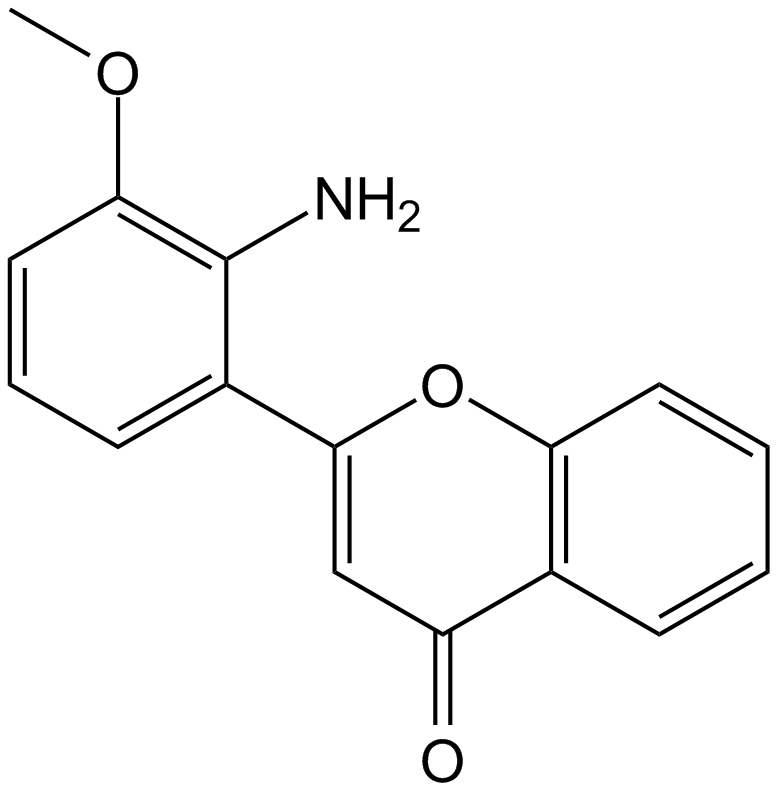
-
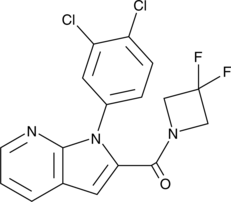
GC47931
PDE4B Inhibitor
A PDE4B inhibitor
-
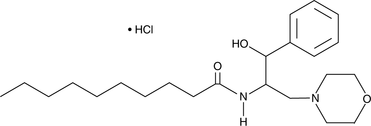
GC44587
PDMP (hydrochloride)
PDMP is a ceramide analog first prepared in a search for inhibitors of glucosylceramide synthase.
-
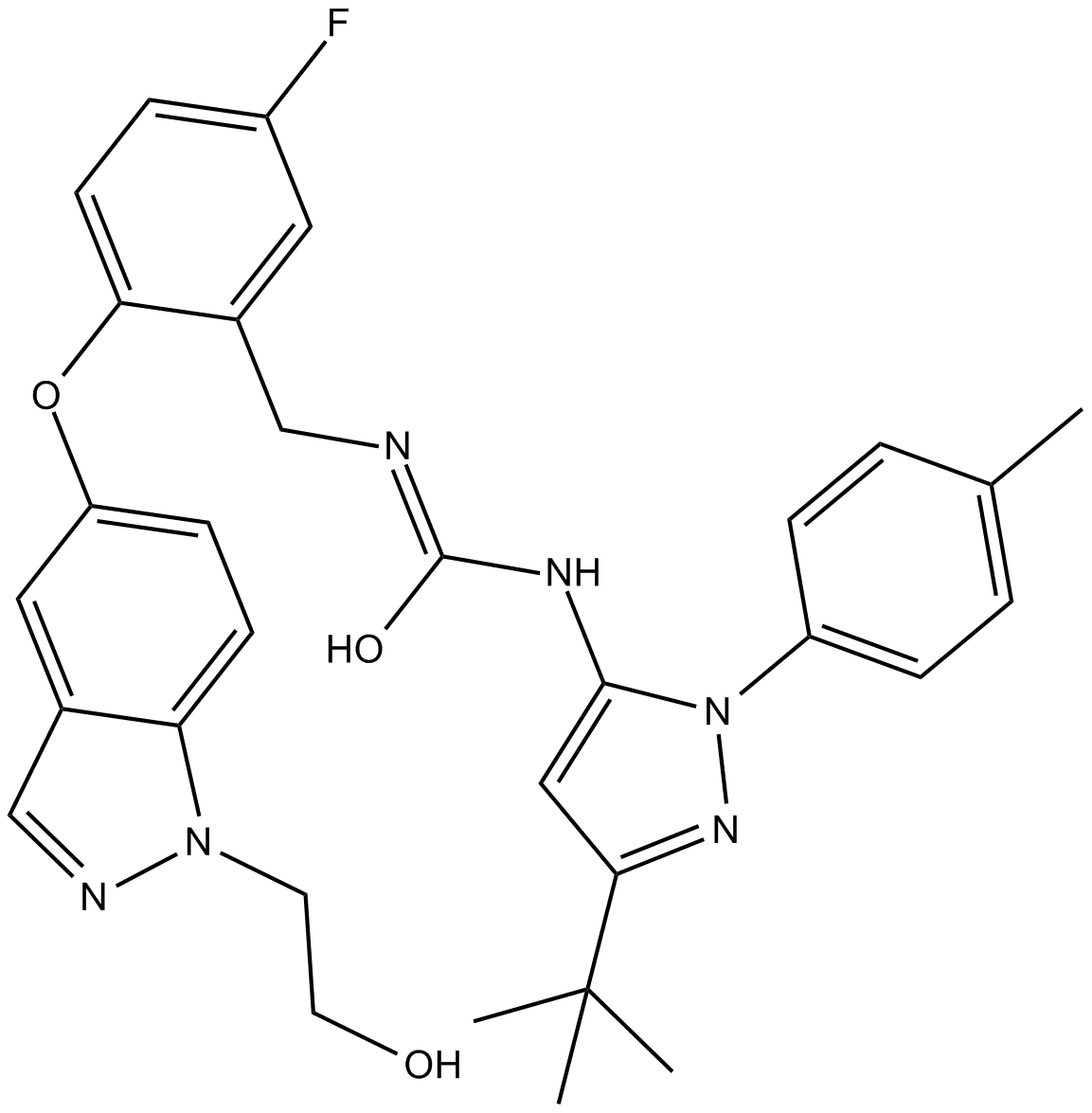
GC11857
Pexmetinib (ARRY-614)
Pexmetinib (ARRY-614) es un potente inhibidor dual Tie-2 y p38 MAPK, con IC50 de 1 nM, 35 nM y 26 nM para Tie-2, p38α y p38β, respectivamente, y se pueden utilizar en la investigaciÓn de la leucemia mieloide aguda.
-
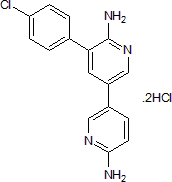
GC50252
PF 06260933 dihydrochloride
MAP4K4 (HGK) inhibitor; also inhibits MINK and TNIK
-
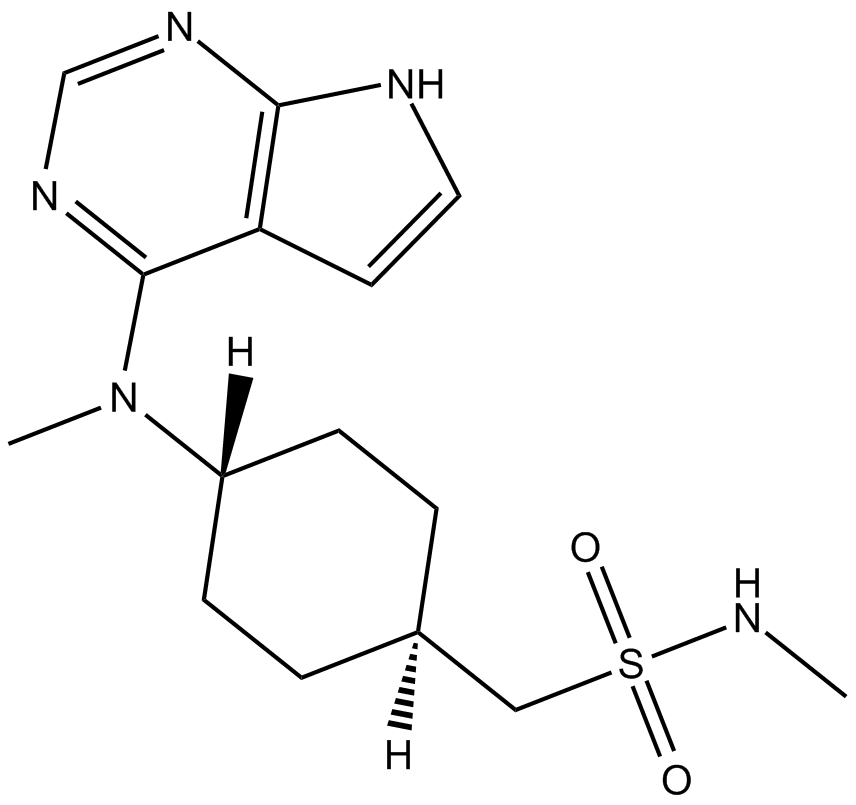
GC14938
PF-03394197(Oclacitinib)
Novel Janus kinase inhibitor
-
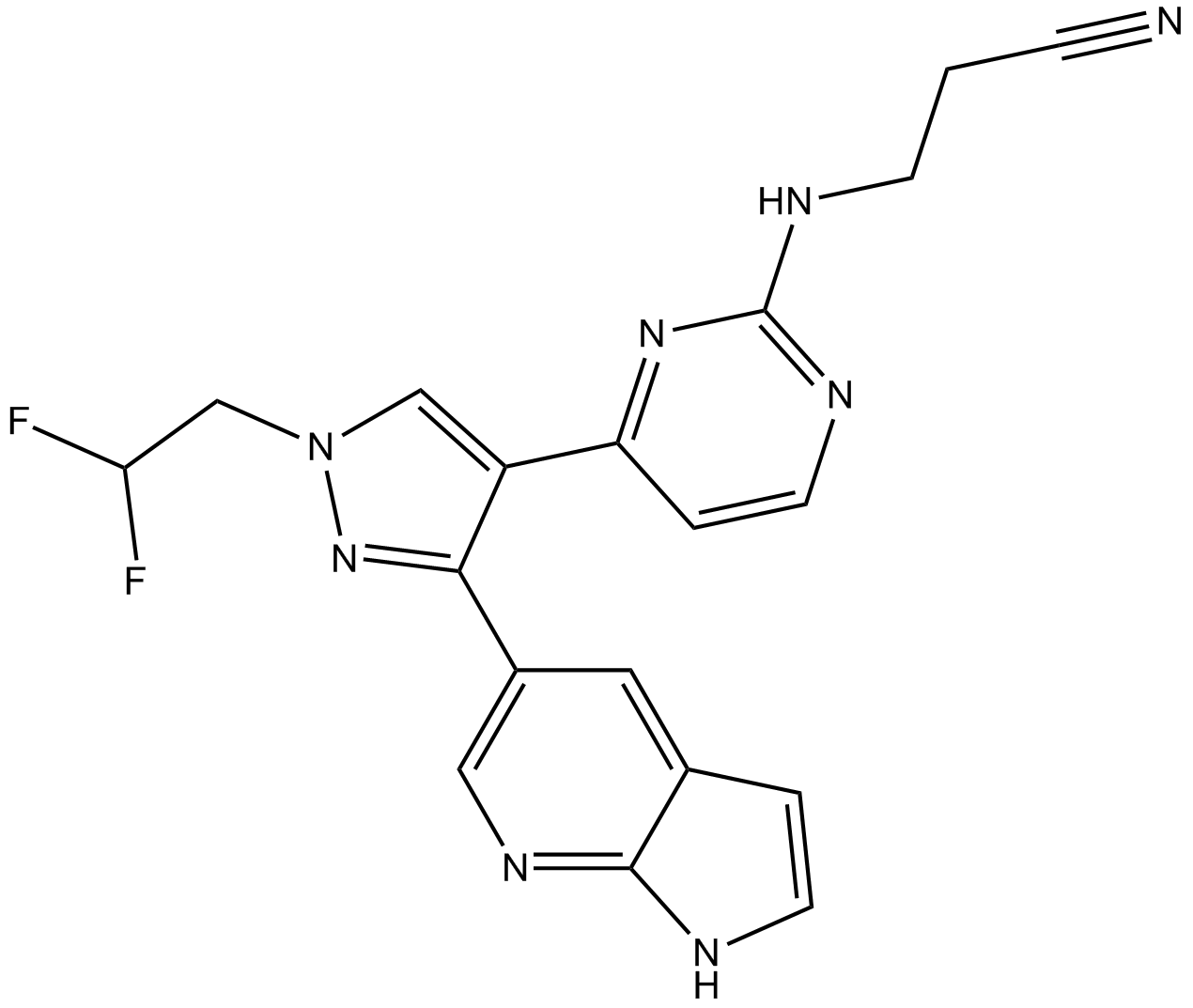
GC15821
PF-04880594
PF-04880594 es un inhibidor RAF potente y selectivo. PF-04880594 inhibe BRAF y CRAF de tipo salvaje y mutante. PF-04880594 muestra actividad antitumoral.
-
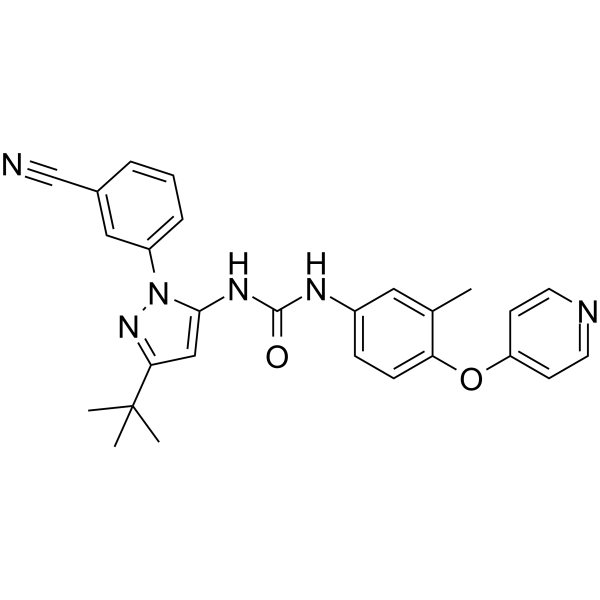
GC63144
PF-05381941
PF-05381941 es un potente inhibidor dual de TAK1/p38α, con IC50 de 156 y 186 nM, respectivamente.
-
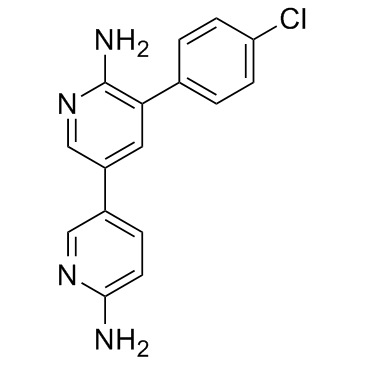
GC31675
PF-06260933
PF-06260933 es un inhibidor altamente selectivo y activo por vÍa oral de MAP4K4 con IC50 de 3,7 y 160 nM para quinasa y célula, respectivamente.
-
GC14645
PF-3644022
PF-3644022 es un inhibidor de MAPKAPK2 (MK2) potente, selectivo, activo por vÍa oral y competitivo con ATP con una IC50 de 5,2 nM y una Ki de 3 nM.
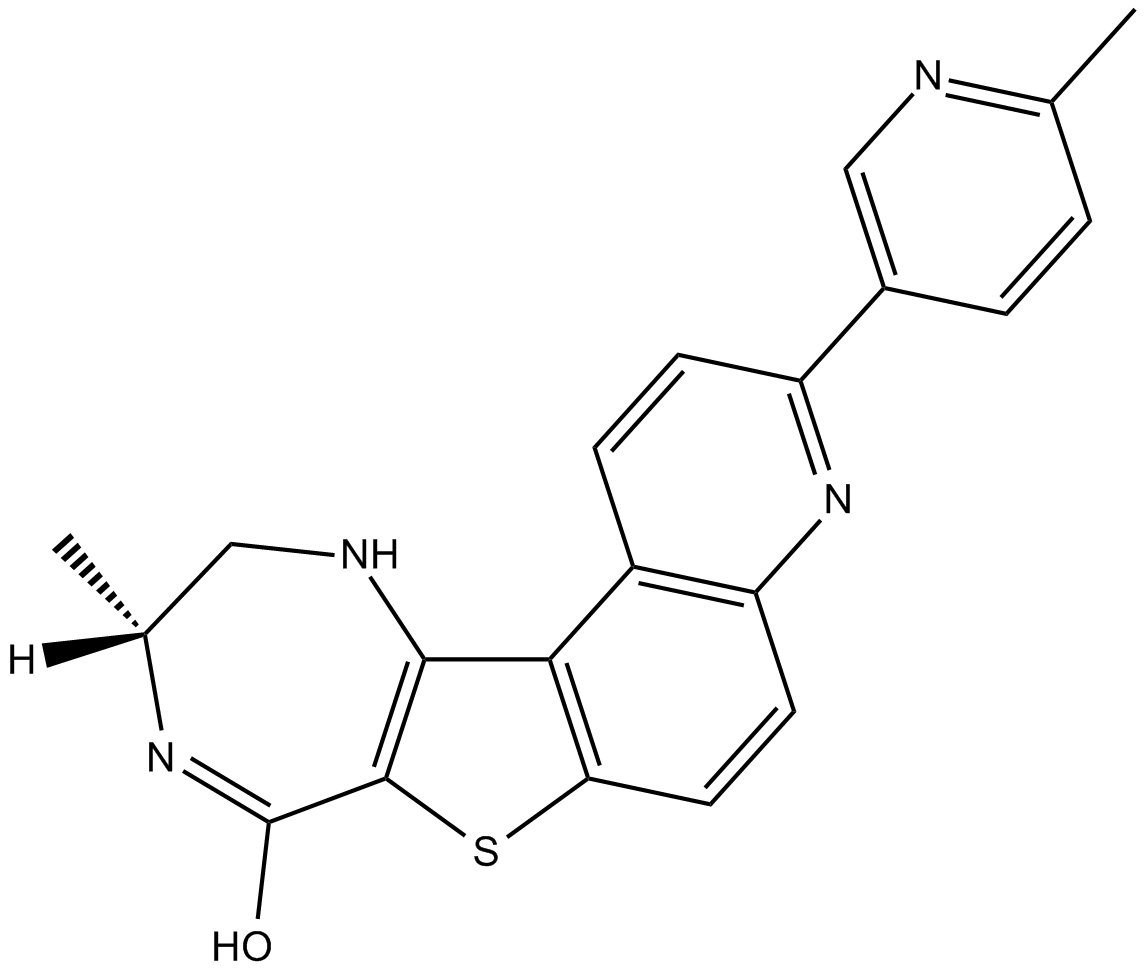
-
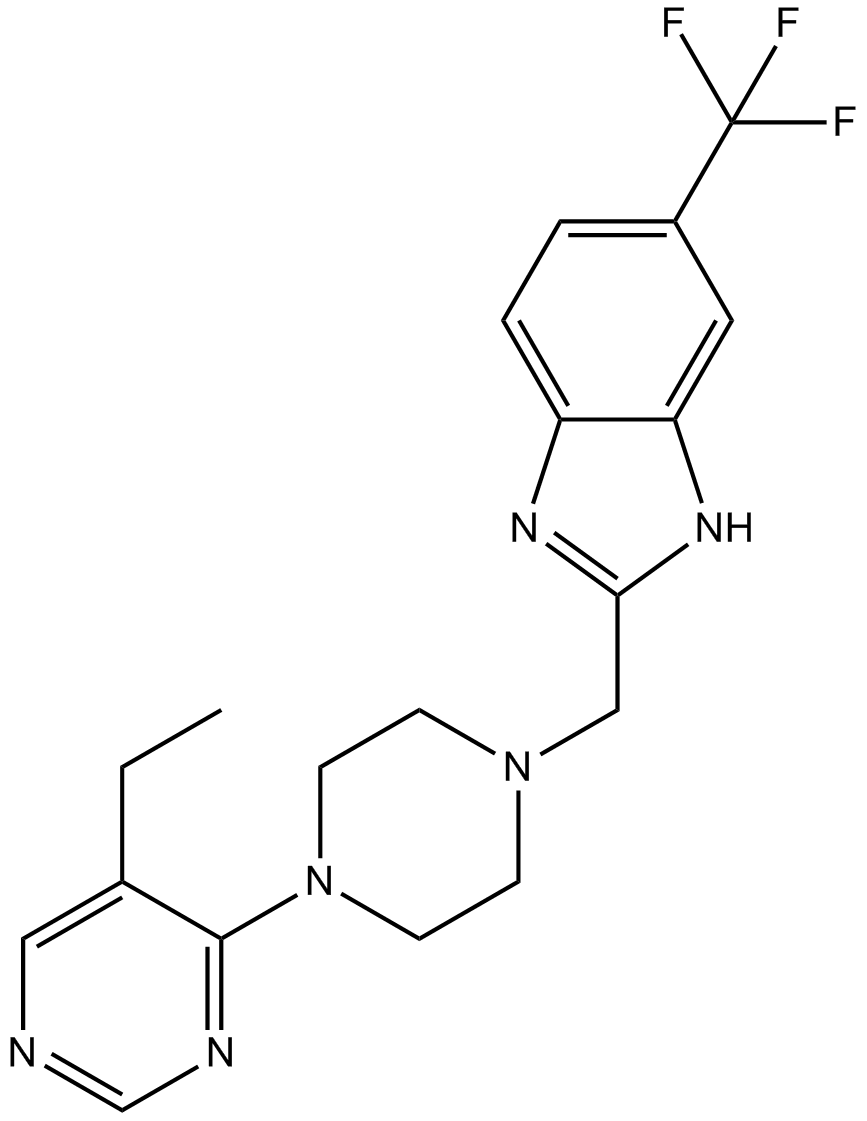
GC10689
PF-4708671
PF-4708671, es un nuevo inhibidor de S6K1 permeable a las células, que inhibe específicamente la isoforma S6K1 con una Ki de 20 nM y una IC50 de 160 nM.
-
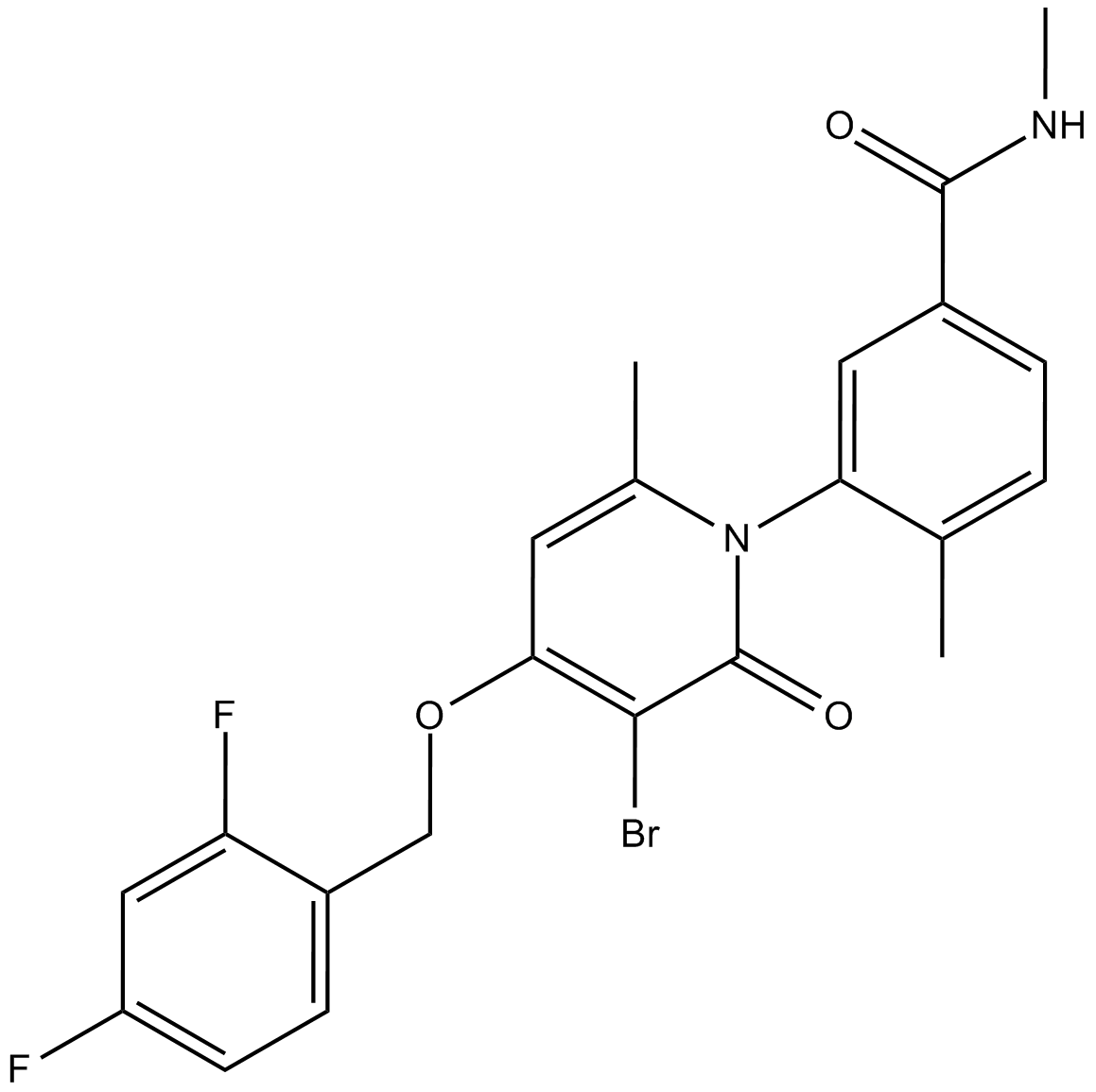
GC10261
PH-797804
PH-797804 es un inhibidor de p38α/p38β selectivo, competitivo con ATP (CI50 = 26 nM y Ki = 5,8 nM para p38α; Ki = 40 nM para p38β) y no inhibe JNK2.
-
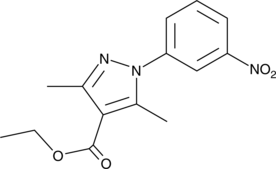
GC41635
Phosphodiesterase 4 Inhibitor
Phosphodiesterase 4 (PDE4) inhibitor is an inhibitor of PDE4 with an IC50 value of 0.10 μM for the human recombinant enzyme.
-
GC18050
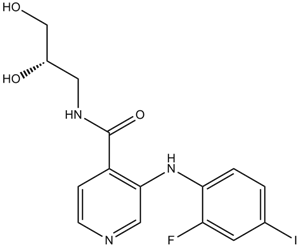
Pimasertib (AS-703026)
Pimasertib (AS-703026) (AS703026) es un inhibidor de MEK1/2 alostérico disponible por vÍa oral no competitivo ATP altamente selectivo.
-
GC10282
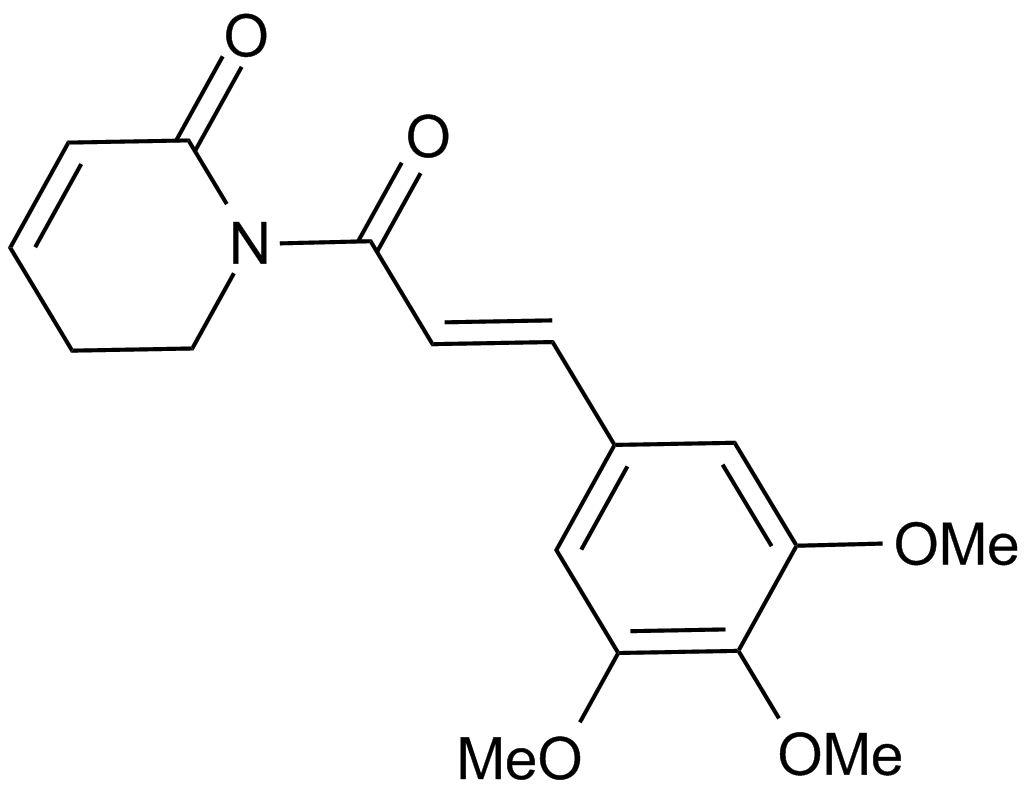
Piperlongumine
An alkaloid with anticancer and antioxidant activities
-
GC44653
PKA Inhibitor (5-24)
El inhibidor de PKA (5-24) es un inhibidor peptÍdico sintético, competitivo y potente de la PKA (proteÍna quinasa dependiente de AMPc), con una Ki de 2,3 nM.

-
GC48329
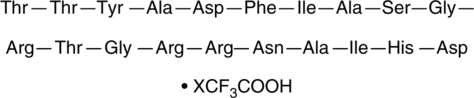
PKA Inhibitor (5-24) (trifluoroacetate salt)
A synthetic peptide inhibitor of PKA
-
GP10075
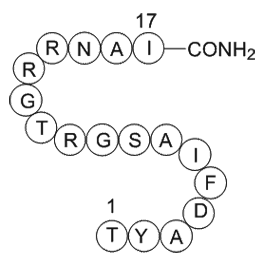
PKA inhibitor fragment (6-22) amide
A synthetic peptide inhibitor of PKA
-
GC61512
PKA Inhibitor Fragment (6-22) amide TFA
Fragmento inhibidor de PKA (6-22) amida TFA es un inhibidor de la proteÍna quinasa A (PKA) dependiente de cAMP, con una Ki de 2,8 nM.

-
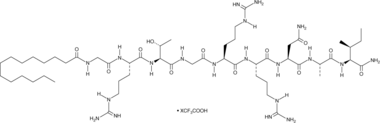
GC49265
PKI (14-22) amide (myristoylated) (trifluoroacetate salt)
A PKA inhibitor
-
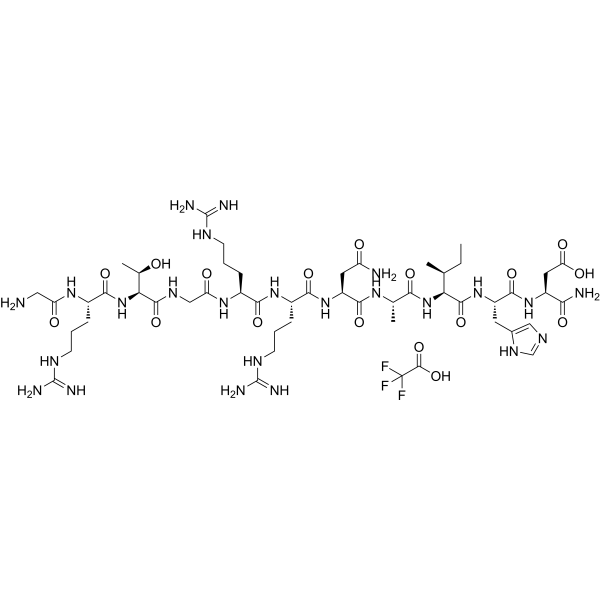
GC69715
PKI (14-24)amide TFA
PKI (14-24)amide TFA es un inhibidor efectivo de PKA. En el citosol celular, PKI (14-24)amide inhibe fuertemente la actividad de la proteína quinasa dependiente del AMP cíclico.
-
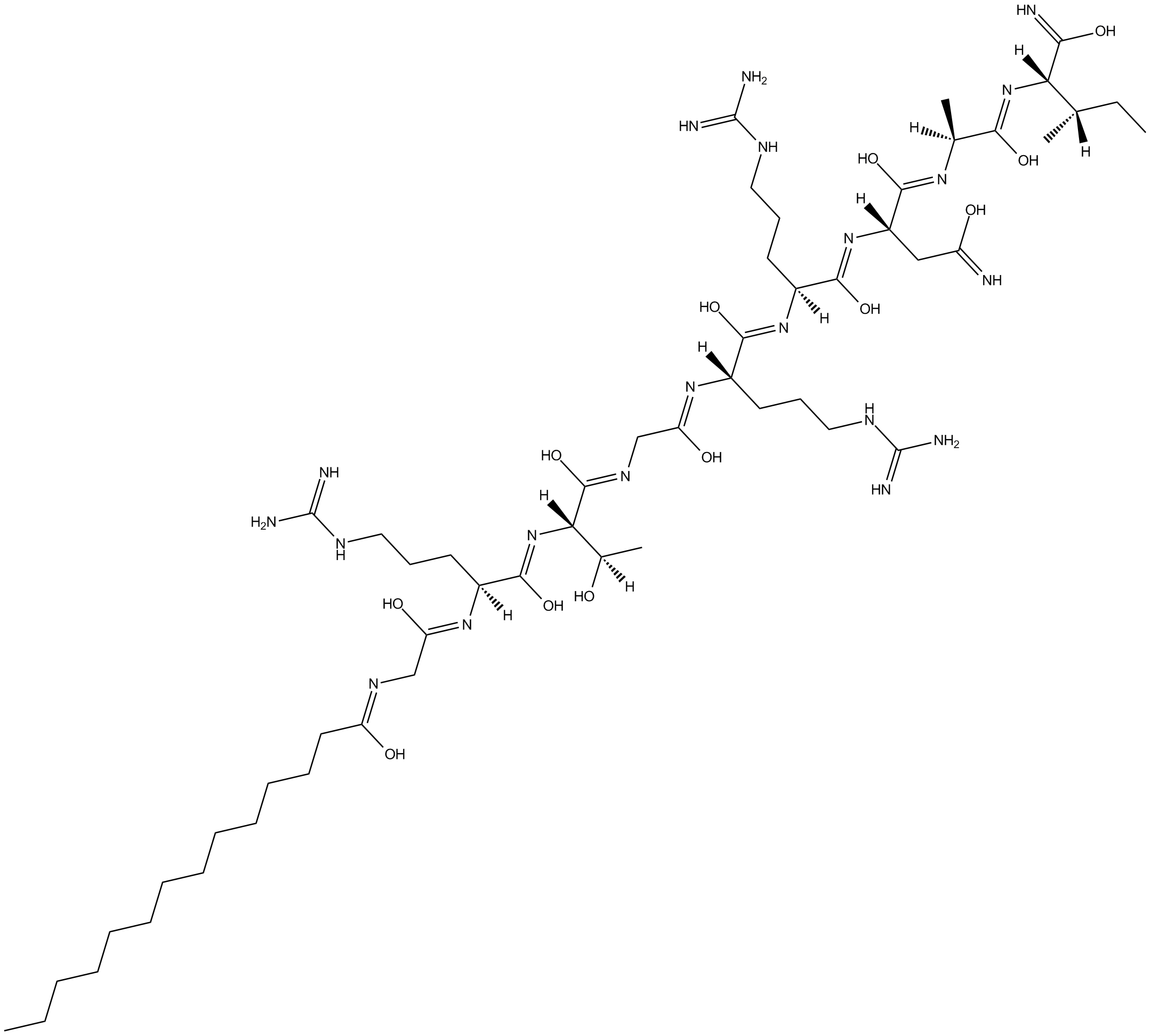
GC12321
PKI 14-22 amide, myristoylated
PKI 14-22 amida, miristoilada es un potente inhibidor de PKA dependiente de cAMP. PKI 14-22 amida, miristoilada reduce la respuesta fagocítica mediada por IgG y también inhibe la adhesión de neutrófilos.
-
GC61849
PKI 14-22 amide,myristoylated TFA
PKI 14-22 amida, miristoilado TFA es un potente inhibidor de PKA dependiente de cAMP. PKI 14-22 amida, miristoilado TFA reduce la respuesta fagocÍtica mediada por IgG y también inhibe la adhesiÓn de neutrÓfilos.

-
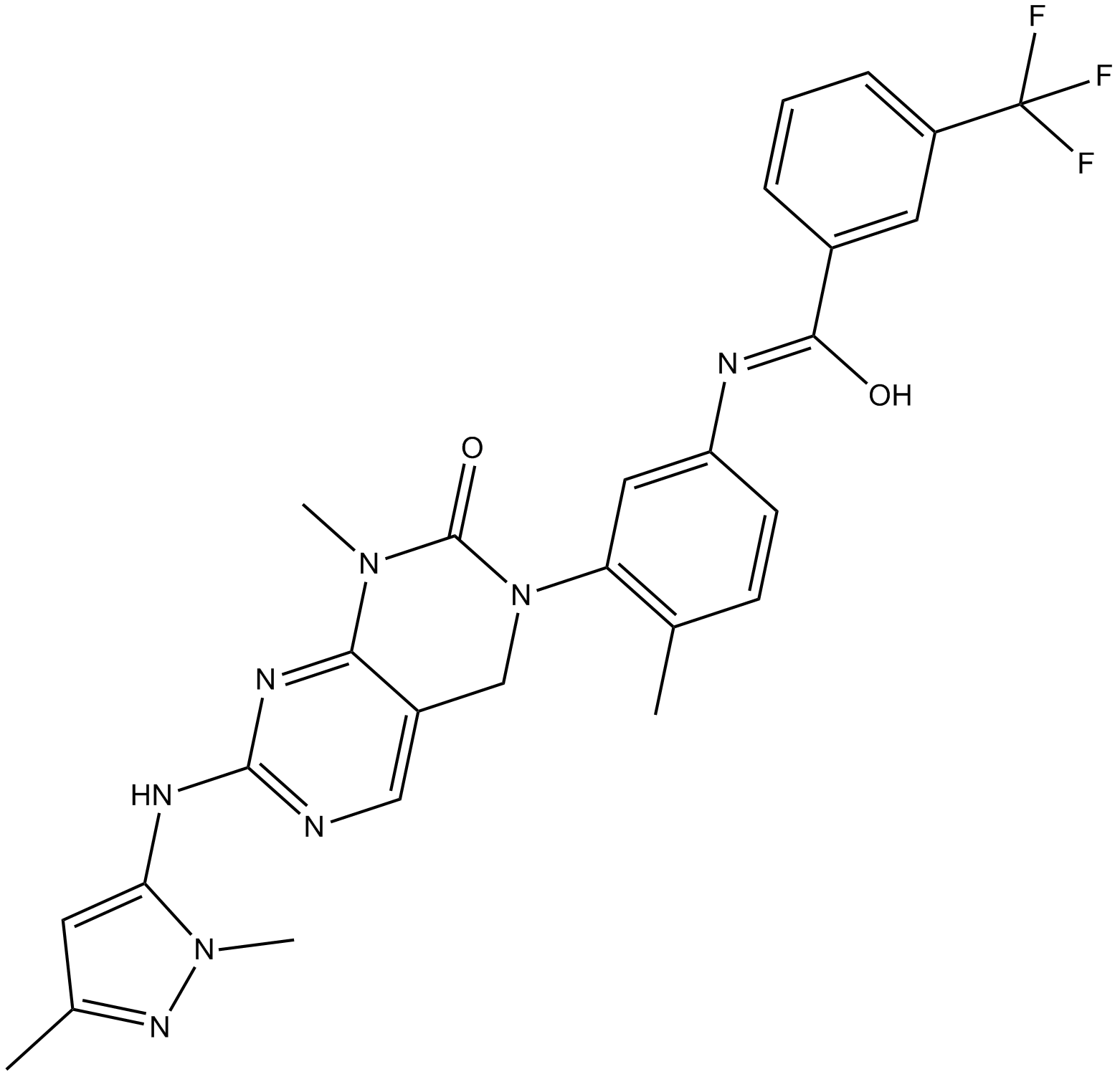
GC17407
Pluripotin
Pluripotin es un inhibidor dual de ERK1 y RasGAP con KD de 98 nM y 212 nM, respectivamente. Pluripotin también inhibe RSK1, RSK2, RSK3 y RSK4 con IC50 de 0,5, 2,5, 3,3 y 10,0 μM, respectivamente.
-
GC14732
PLX-4720
An orally-available inhibitor of the B-raf mutant B-RafV600E
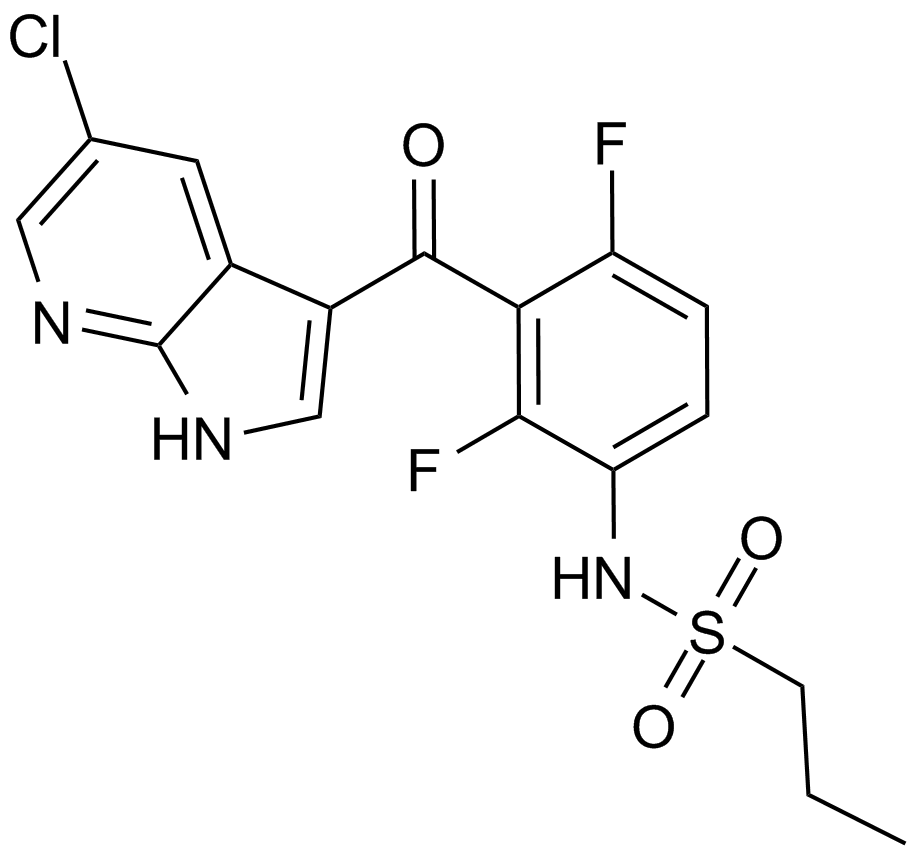
-
GC62206
PLX-4720-d7
PLX-4720-d7 es el deuterio etiquetado como PLX-4720. PLX-4720 es un inhibidor potente y selectivo deB-RafV600Econuna IC50de 13 nM en un ensayo libre de células, igualmente potente que c-Raf-1 (mutaciones Y340D y Y341D) y selectividad 10 veces mayor para B-RafV600E que el B-Raf de tipo salvaje.
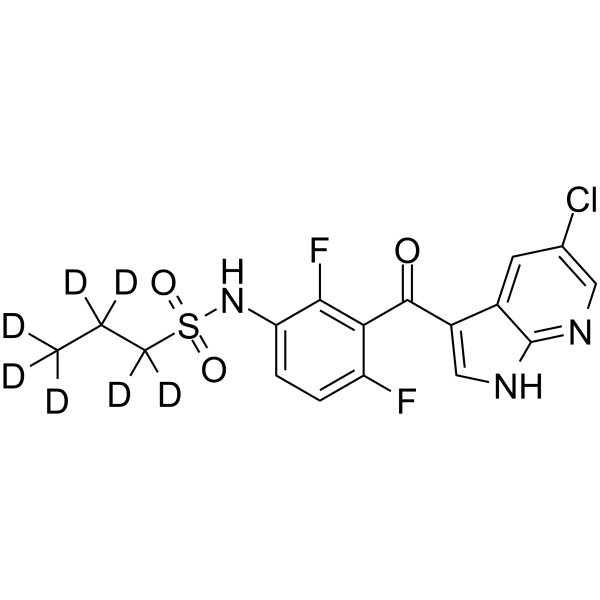
-
GC10324
PLX7904
PLX7904 es un inhibidor de BRAF potente y selectivo, con IC50 de aproximadamente 5 nM contra BRAFV600E en células mutantes que expresan RAS.
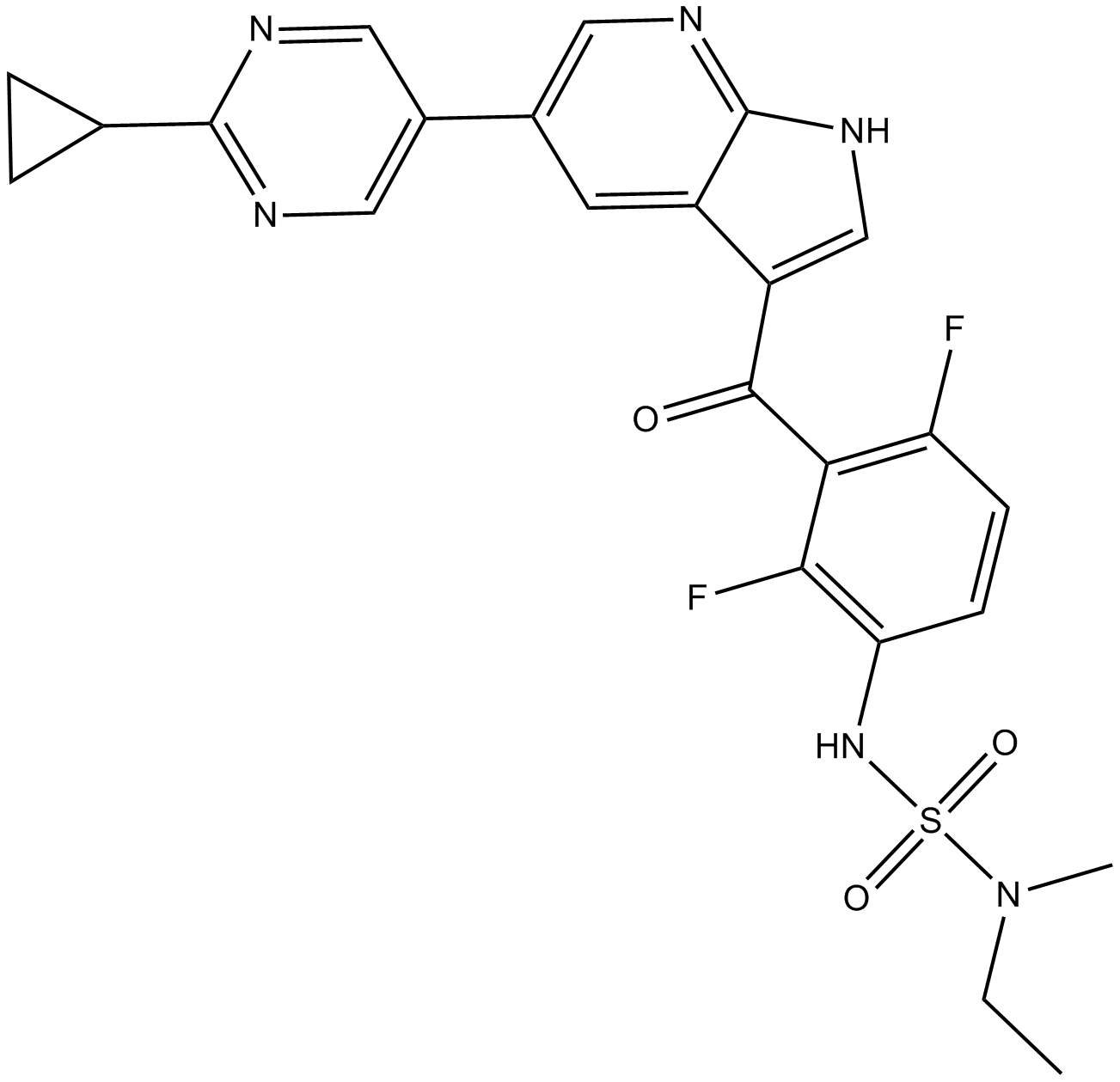
-
GC64828
PLX7922
PLX7922, un inhibidor de RAF, puede unirse a BRAFV600E. PLX7922 inhibe pERK en lÍneas celulares BRAFV600E y activa pERK en lÍneas celulares mutantes NRAS.
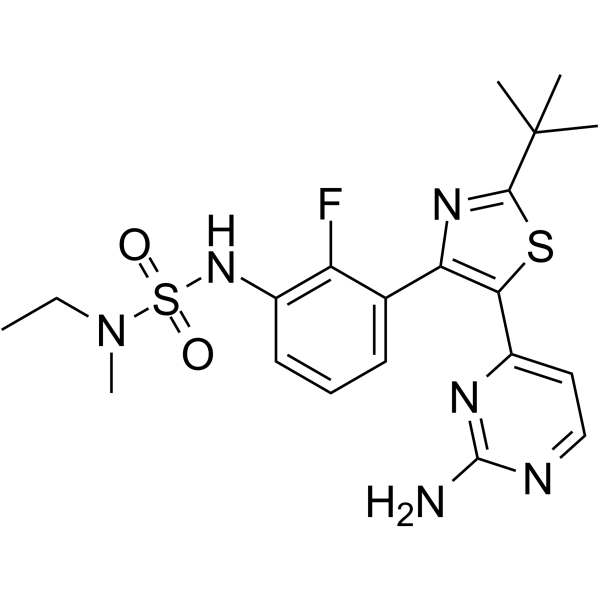
-
GC32686
PLX8394
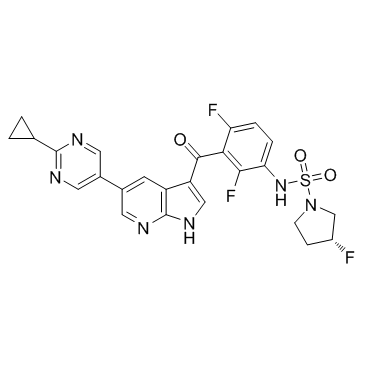
-
GN10709
Polyphyllin A
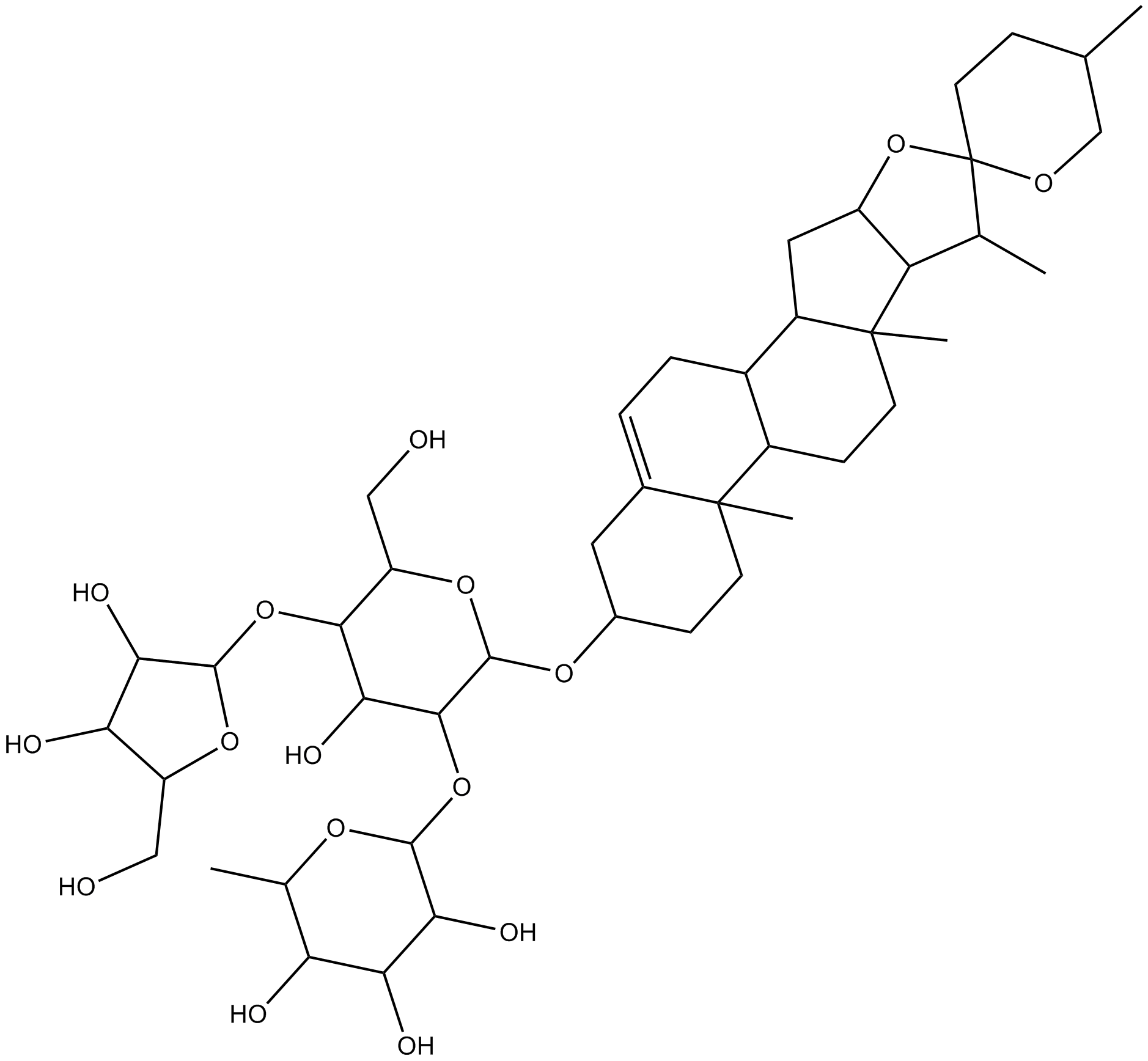
-
GC44673
PQ-10
PQ-10 es un potente inhibidor de la fosfodiesterasa 10A (PDE10A) con IC50 y ED50 de 4,6 nM y 13 mg/kg, respectivamente.
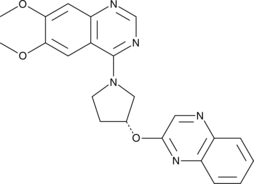
-
GC65479
PROTAC B-Raf degrader 1
El degradador 1 de PROTAC B-Raf (compuesto 2) es una quimera dirigida a la proteÓlisis (PROTAC) para la degradaciÓn de B-Raf basada en el ligando Cereblon con actividad anticancerÍgena.
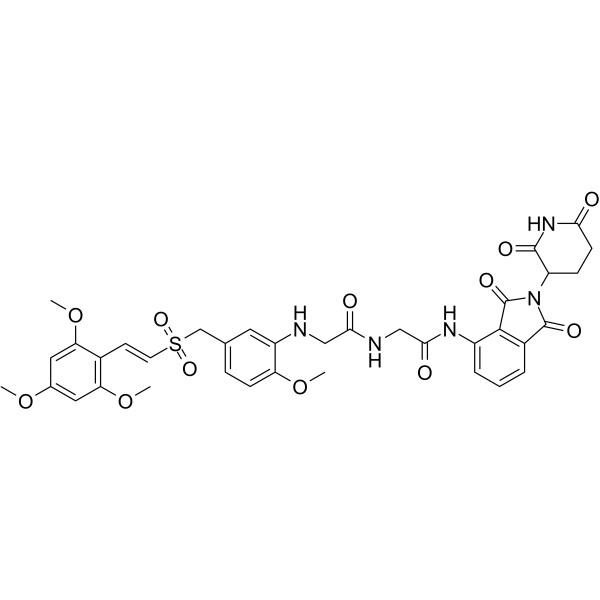
-
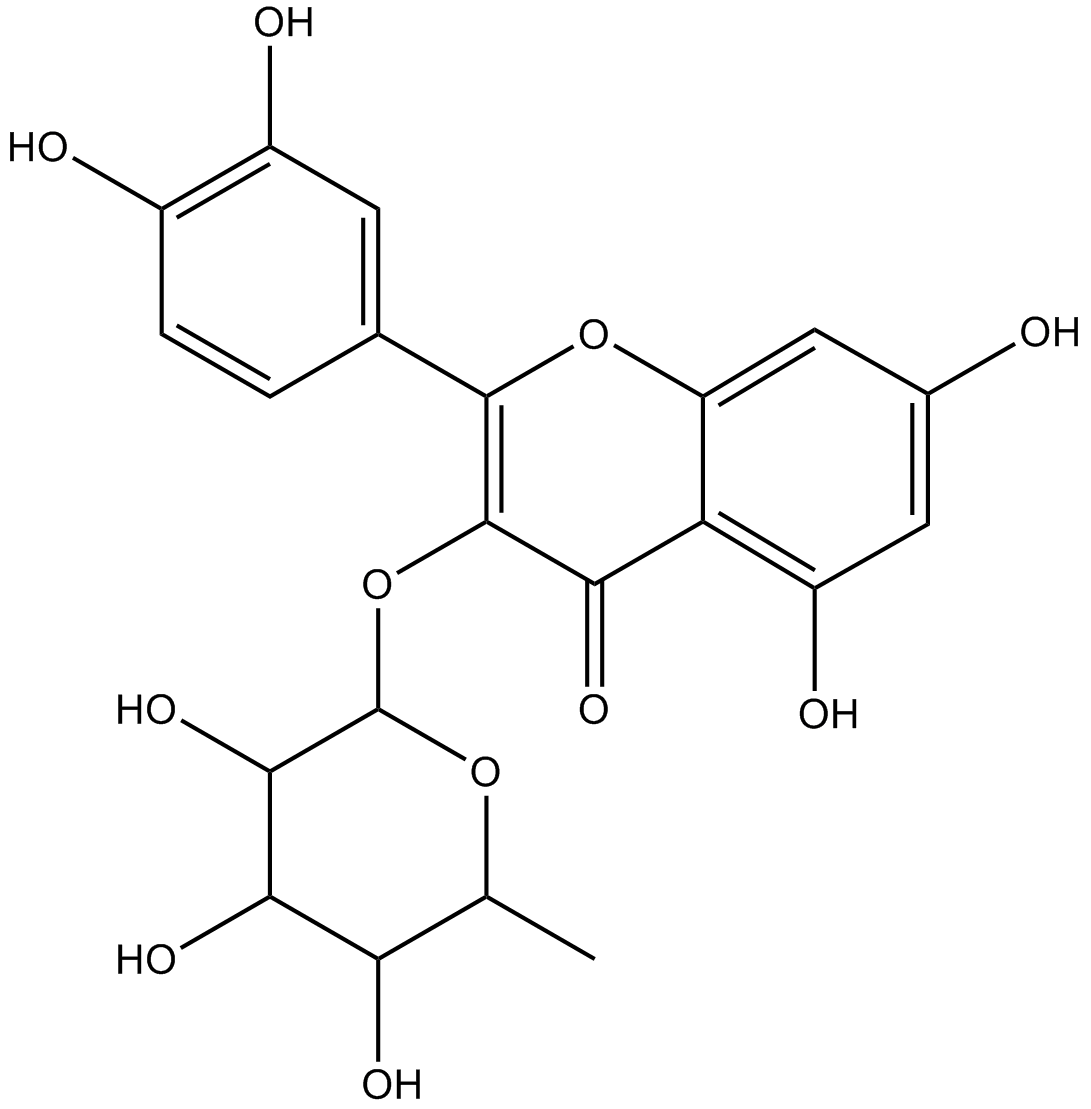
GN10814
Quercitrin
-
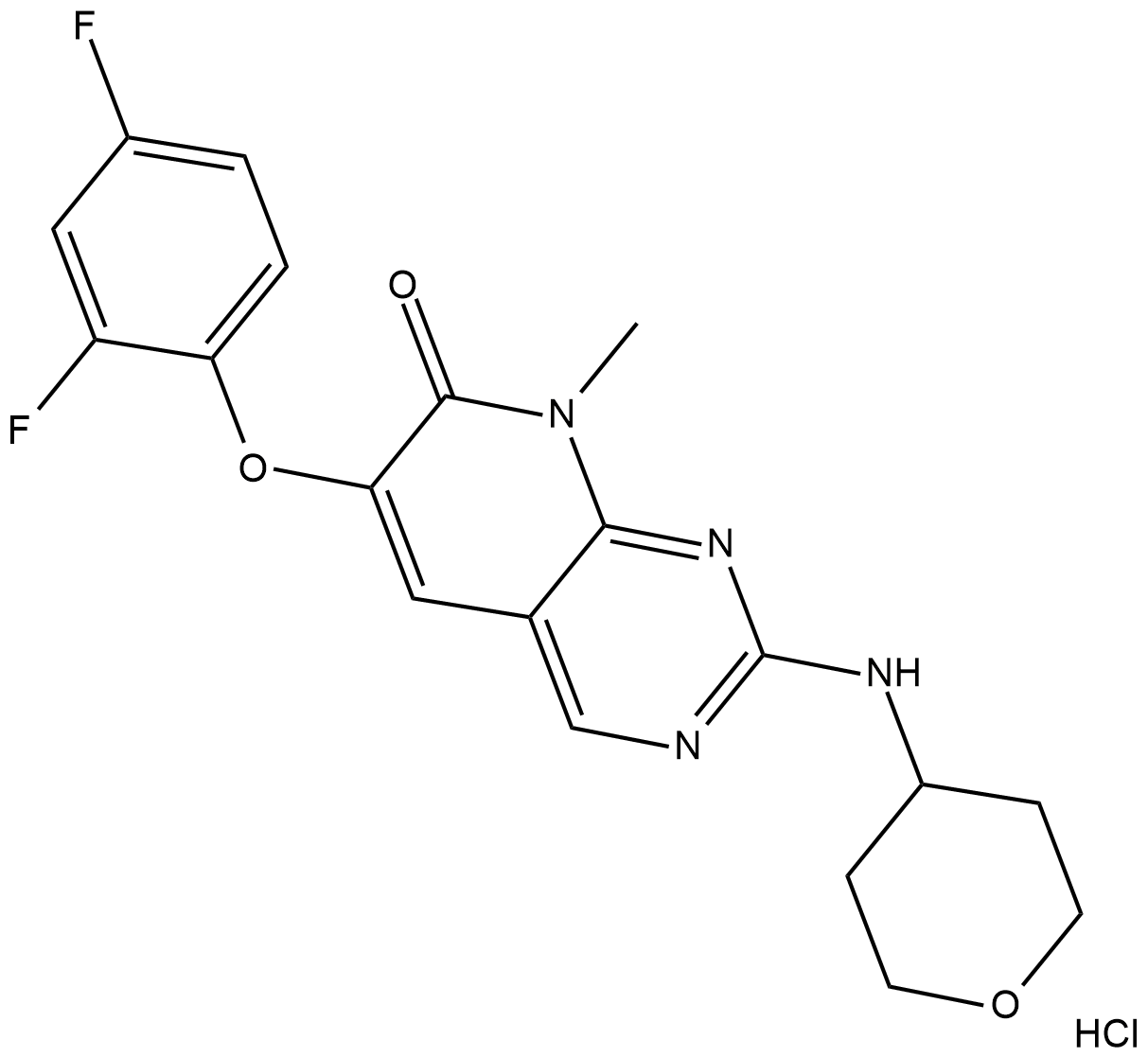
GC19304
R1487 Hydrochloride
El clorhidrato de R1487 es un inhibidor de p38α muy potente y selectivo, con valores de Kd de 0,2 nM y 29 nM para p38α y p38β, respectivamente.
-
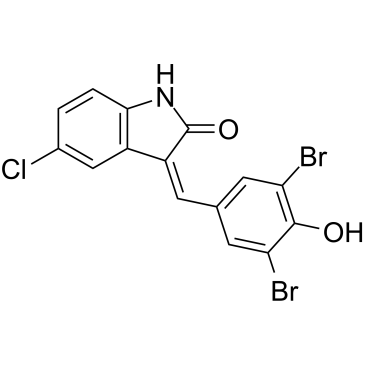
GC61485
Raf inhibitor 2
El inhibidor de raf 2 es un potente inhibidor de quinasa de raf (IC50<1,0 μM), compuesto 32, extraÍdo de la patente EP1003721B1. El inhibidor de Raf 2 se puede utilizar para la investigaciÓn del cÁncer.
-
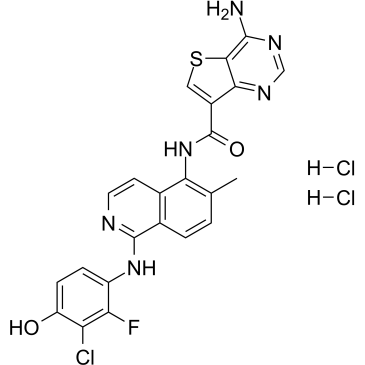
GC37068
RAF mutant-IN-1
RAF mutant-IN-1 es un inhibidor de la quinasa RAF, extraÍdo de la patente WO2019107987A1, con valores IC50 de 21 nM, 30 nM y 392 nM para C-RAF 340D/Y341D, B-RAFV600E y B-RAFWT, respectivamente.
-
GC15818
RAF265
RAF265 es un potente inhibidor de RAF/VEGFR2.
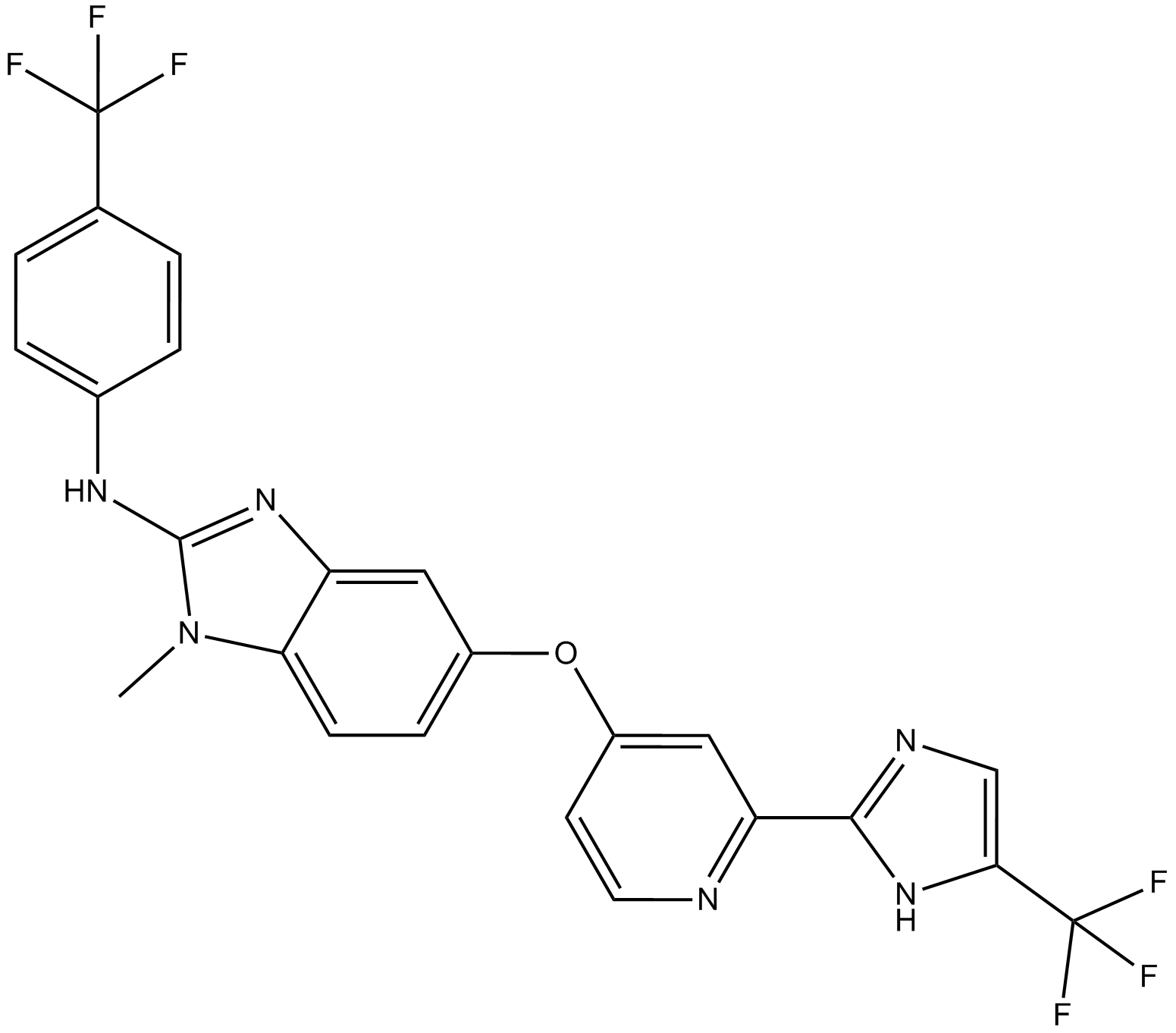
-
GC19307
RAF709
RAF709 es un inhibidor de RAF potente, selectivo y eficaz con IC50 de 0,4 nM y 0,5 nM para BRAF y CRAF, respectivamente. Eficacia antitumoral.
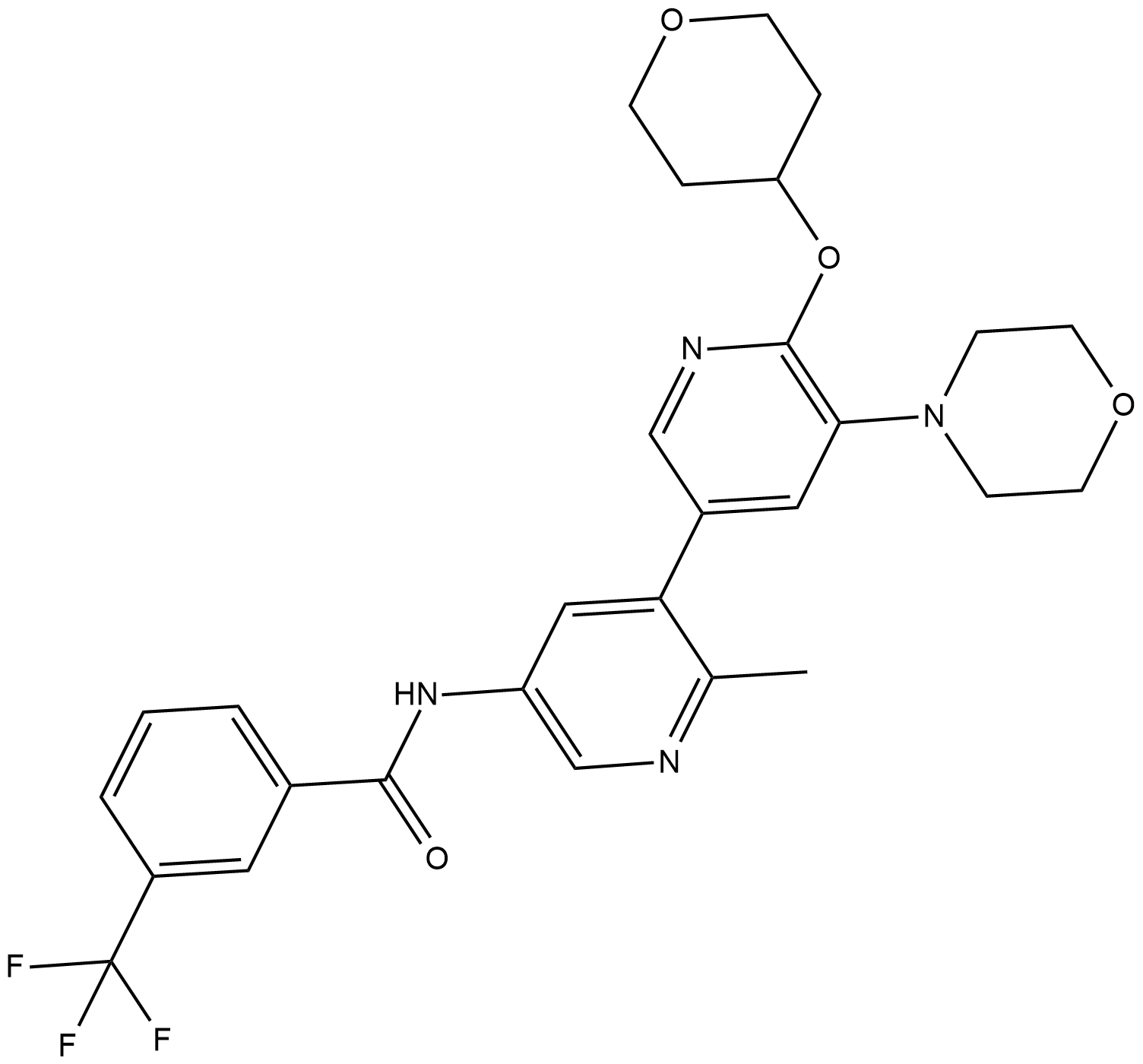
-
GC62504
RAS/RAS-RAF-IN-1
RAS/RAS-RAF-IN-1 es un potente inhibidor de RAS y RAS-RAF. RAS/RAS-RAF-IN-1 tiene una KD de 5,0 μμ-15 μμ para la afinidad de uniÓn de la ciclofilina A (CYPA). RAS/RAS-RAF-IN-1 tiene actividad antitumoral.
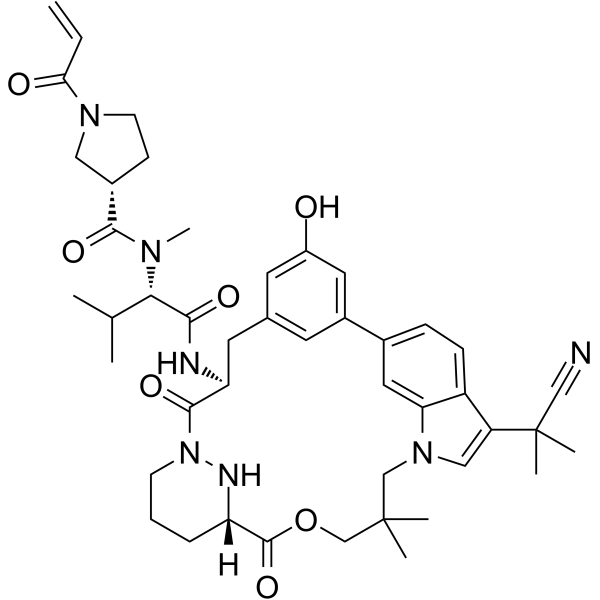
-
GC37071
Ravoxertinib hydrochloride
El clorhidrato de ravoxertinib (clorhidrato de GDC-0994) es un inhibidor selectivo biodisponible por vÍa oral para la actividad de la quinasa ERK con IC50 de 6,1 nM y 3,1 nM para ERK1 y ERK2, respectivamente.
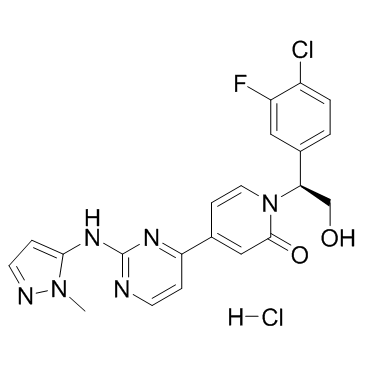
-
GC16872
Refametinib
Refametinib (BAY 869766; RDEA119) es un inhibidor de MEK1/MEK2 alostérico selectivo, potente, no competitivo con ATP, disponible por vÍa oral con IC50 de 19 nM y 47 nM, respectivamente.
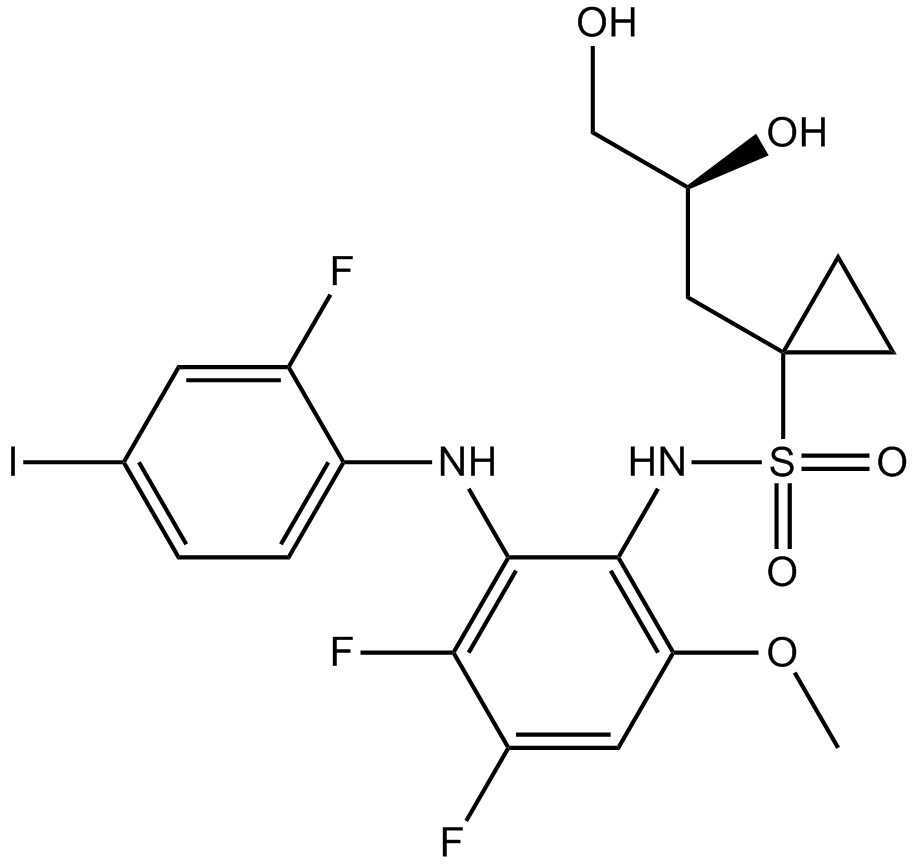
-
GC37516
Refametinib R enantiomer
El enantiómero de refametinib R es un inhibidor de MEK extraído de la patente WO2007014011A2, compuesto 1022, tiene una CE50 de 2,0-15 nM.
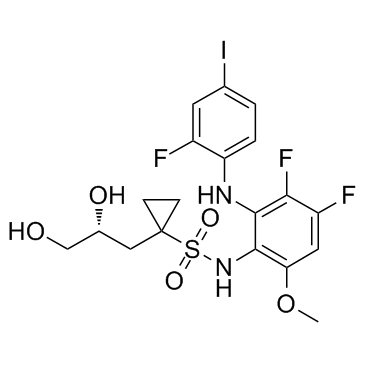
-
GC14606
Regorafenib hydrochloride
A multi-kinase inhibitor
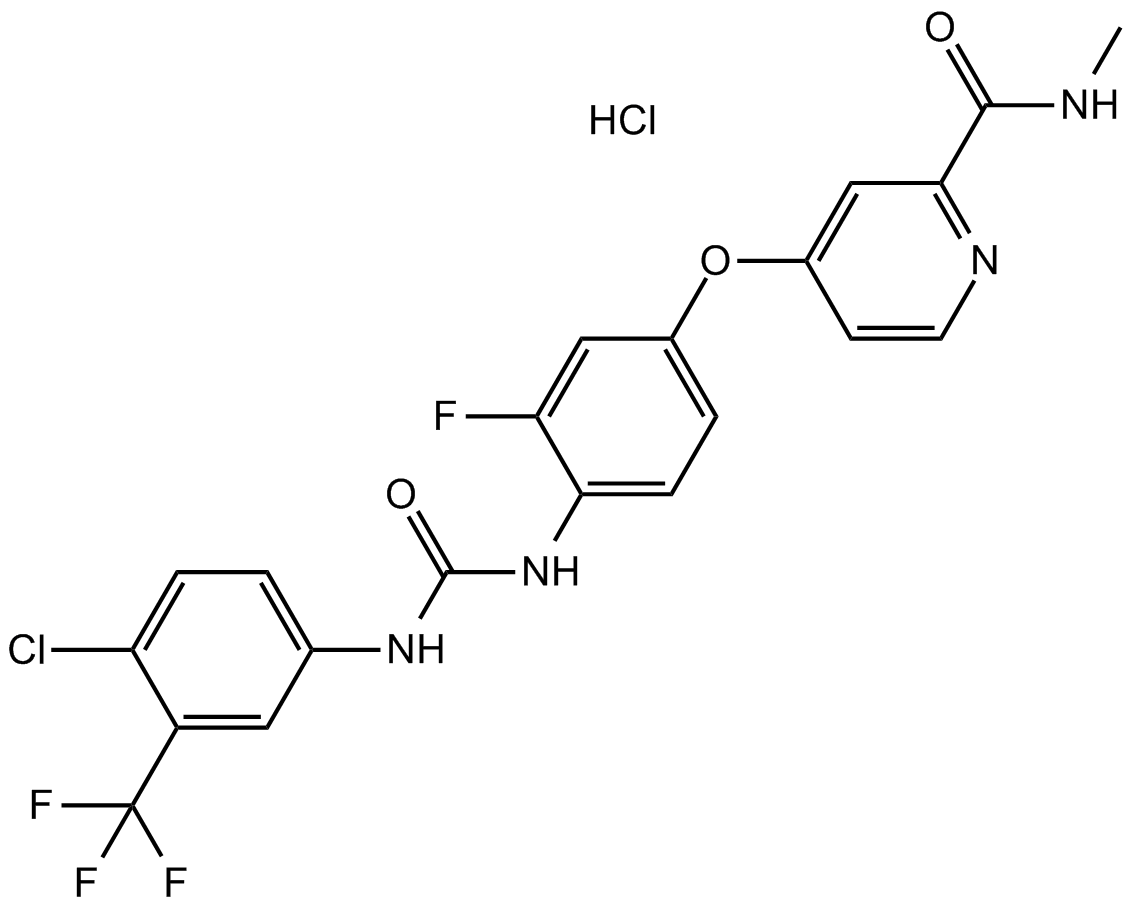
-
GC14534
Regorafenib monohydrate
A multi-kinase inhibitor
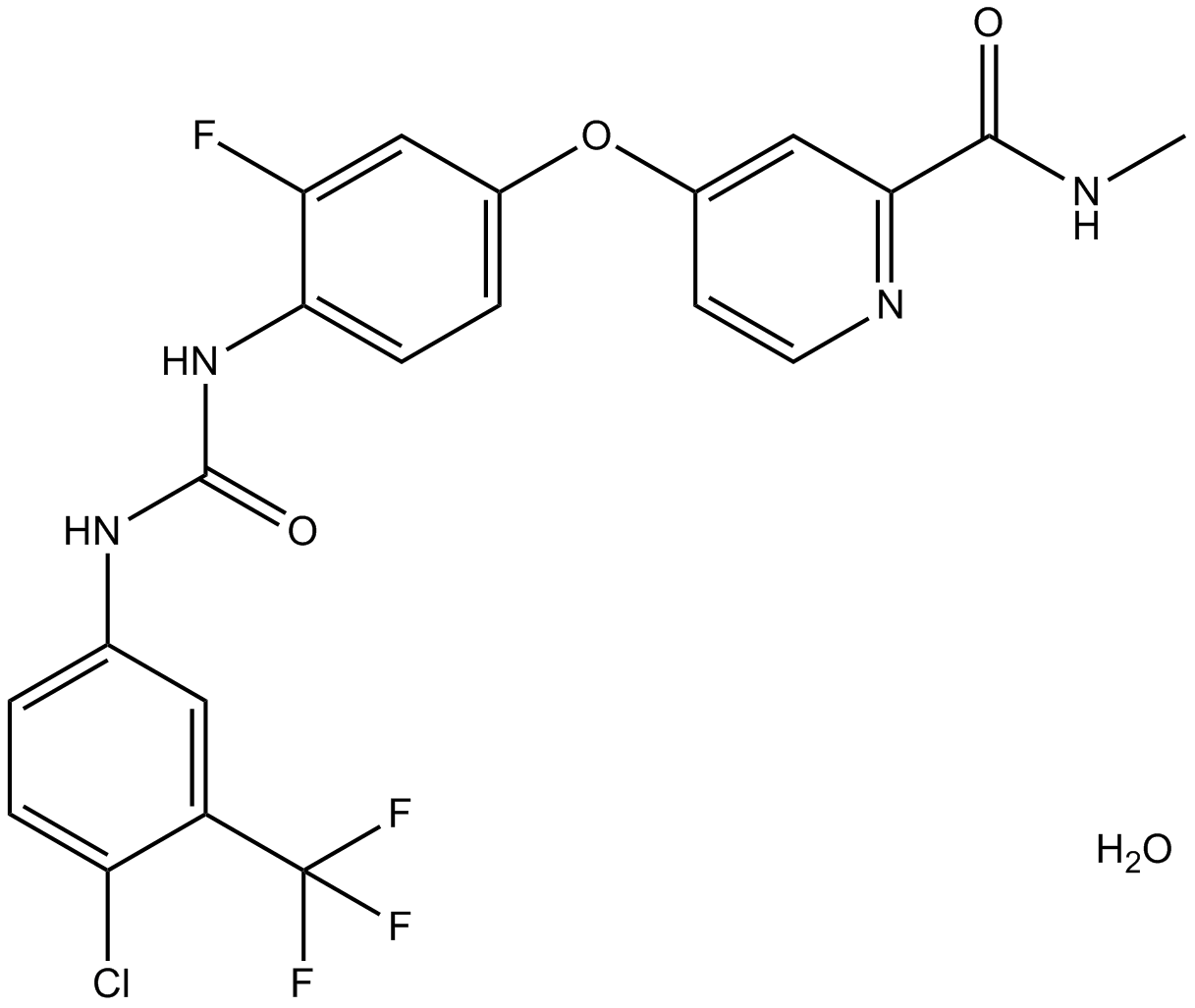
-
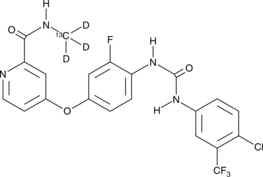
GC40213
Regorafenib-13C-d3
Regorafenib-13C-d3 is intended for use as an internal standard for the quantification of regorafenib by GC- or LC-MS.
-
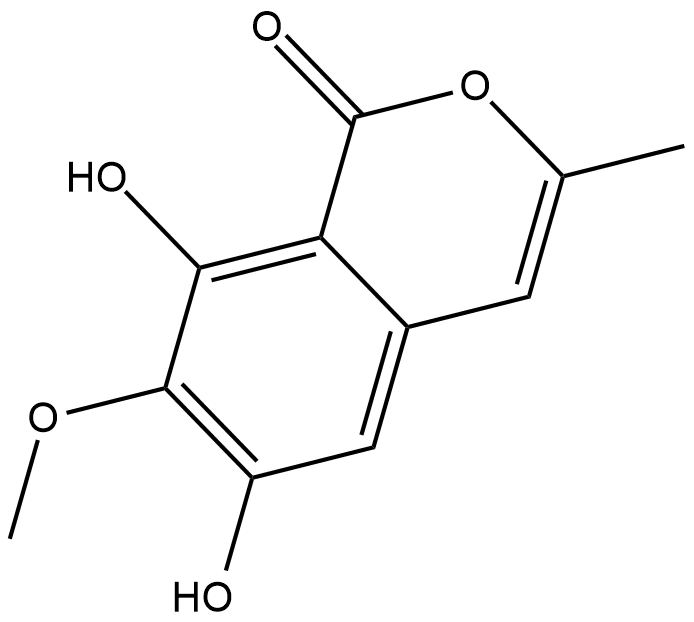
GC18751
Reticulol
Reticulol is an isocoumarin derivative produced by certain species of Streptomyces that inhibits cAMP phosphodiesterase (IC50 = 41 uM).
-
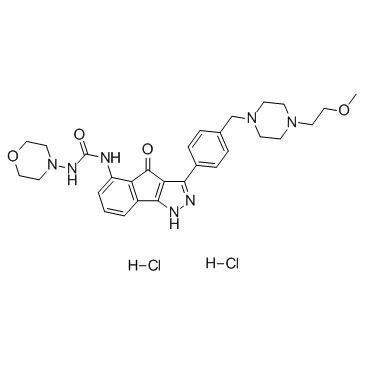
GC37522
RGB-286638
RGB-286638 es un inhibidor de CDK que inhibe la actividad quinasa de ciclina T1-CDK9, ciclina B1-CDK1, ciclina E-CDK2, ciclina D1-CDK4, ciclina E-CDK3 y p35-CDK5 con IC50 de 1, 2, 3 , 4, 5 y 5 nM, respectivamente; también inhibe GSK-3β, TAK1, Jak2 y MEK1, con IC50 de 3, 5, 50 y 54 nM.
-
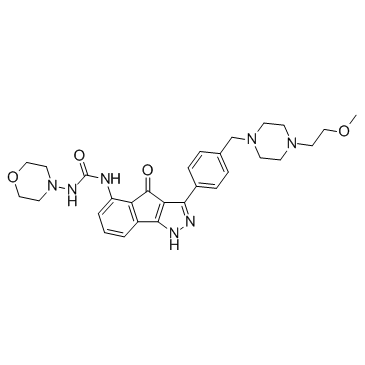
GC37523
RGB-286638 free base
RGB-286638 es un inhibidor de CDK que inhibe la actividad quinasa de ciclina T1-CDK9, ciclina B1-CDK1, ciclina E-CDK2, ciclina D1-CDK4, ciclina E-CDK3 y p35-CDK5 con IC50 de 1, 2, 3 , 4, 5 y 5 nM, respectivamente; también inhibe GSK-3β, TAK1, Jak2 y MEK1, con IC50 de 3, 5, 50 y 54 nM.
-
GN10784
Rhoifolin
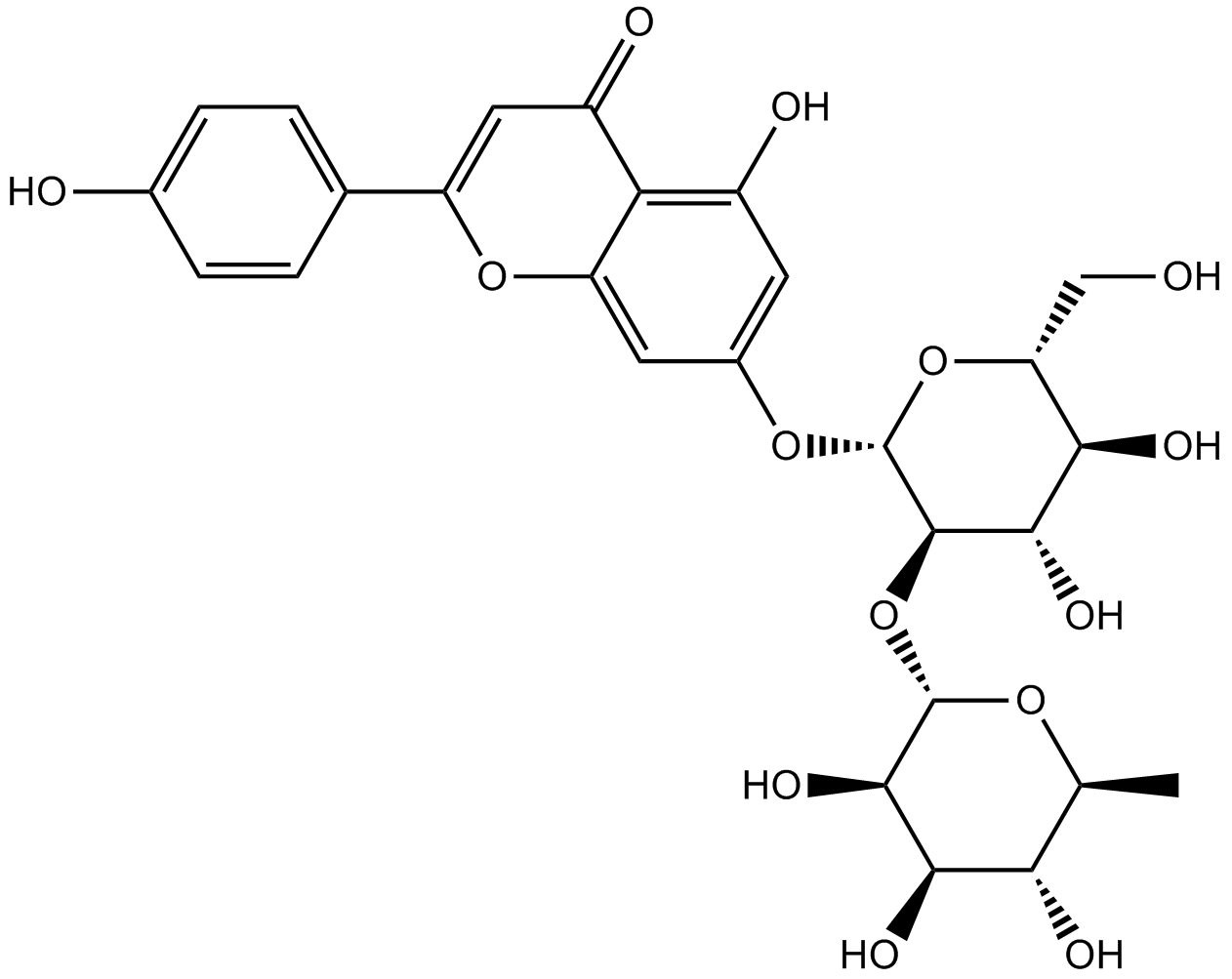
-
GC50502
RI-STAD 2
AKAP disruptor; selectively binds PKA-RI with high affinity and blocks its interaction with AKAP; cell permeable
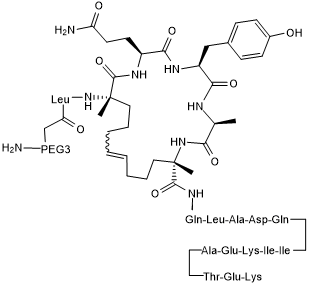
-
GC62448
Rineterkib hydrochloride
El clorhidrato de rineterkib (compuesto B) es un inhibidor de ERK1 y ERK2 disponible por vÍa oral en el tratamiento de una enfermedad proliferativa caracterizada por mutaciones activadoras en la vÍa MAPK. La actividad estÁ particularmente relacionada con el tratamiento del CPNM con mutaciÓn en KRAS, el CPNM con mutaciÓn en BRAF, el cÁncer de pÁncreas con mutaciÓn en KRAS, el cÁncer colorrectal (CCR) con mutaciÓn en KRAS y el cÁncer de ovario con mutaciÓn en KRAS. El clorhidrato de rineterkib también puede inhibir la RAF.
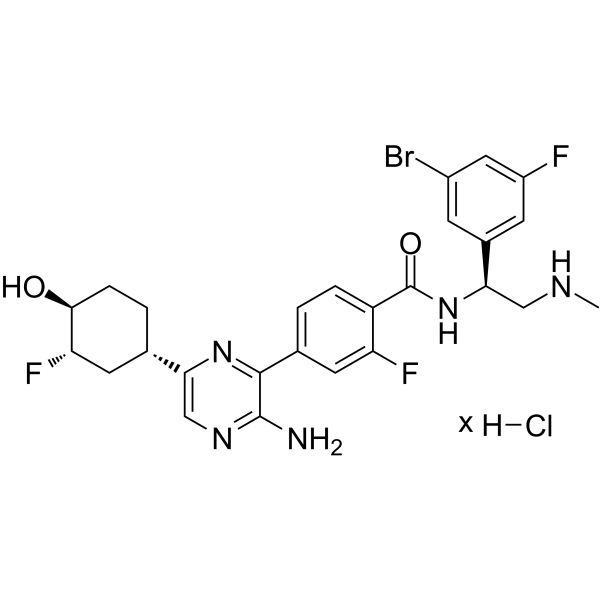
-
GC69821
RLX-33
RLX-33 es un antagonista efectivo, selectivo y con permeabilidad a la barrera hematoencefálica de la familia de péptidos relaxina 3 (RXFP3), que también bloquea la fosforilación de ERK1/2 inducida por relaxina 3, con IC50 para RXFP3, ERK1 y ERK2 de 2.36 μM, 7.82 μM y 13.86 μM respectivamente. RLX-33 puede bloquear el aumento en la ingesta alimentaria en ratas inducido por el agonista selectivo R3/I5 de RXFP3. RLX-33 se puede utilizar en investigaciones sobre síndrome metabólico.
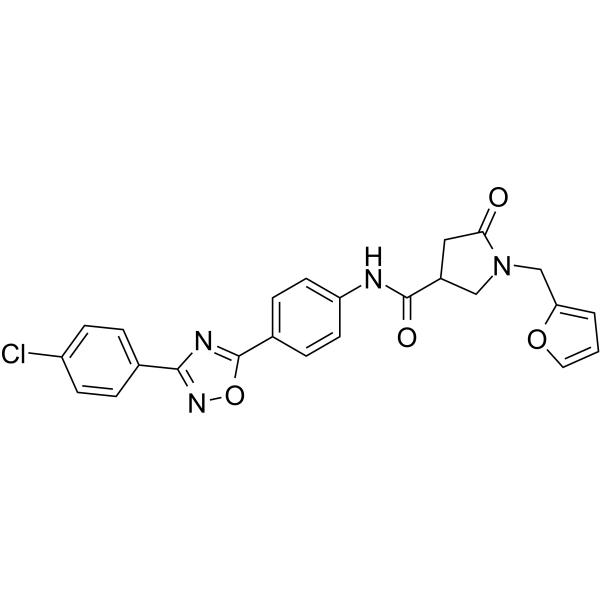
-
GC50294
RMM 46
RMM 46 es un inhibidor covalente selectivo y reversible de las quinasas de la familia MSK/RSK.
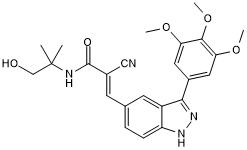
-
GC12141
RO4987655
RO4987655 es un inhibidor de MEK altamente selectivo y activo por vÍa oral con una IC50 de 5,2 nM para la inhibiciÓn de MEK1/MEK2.
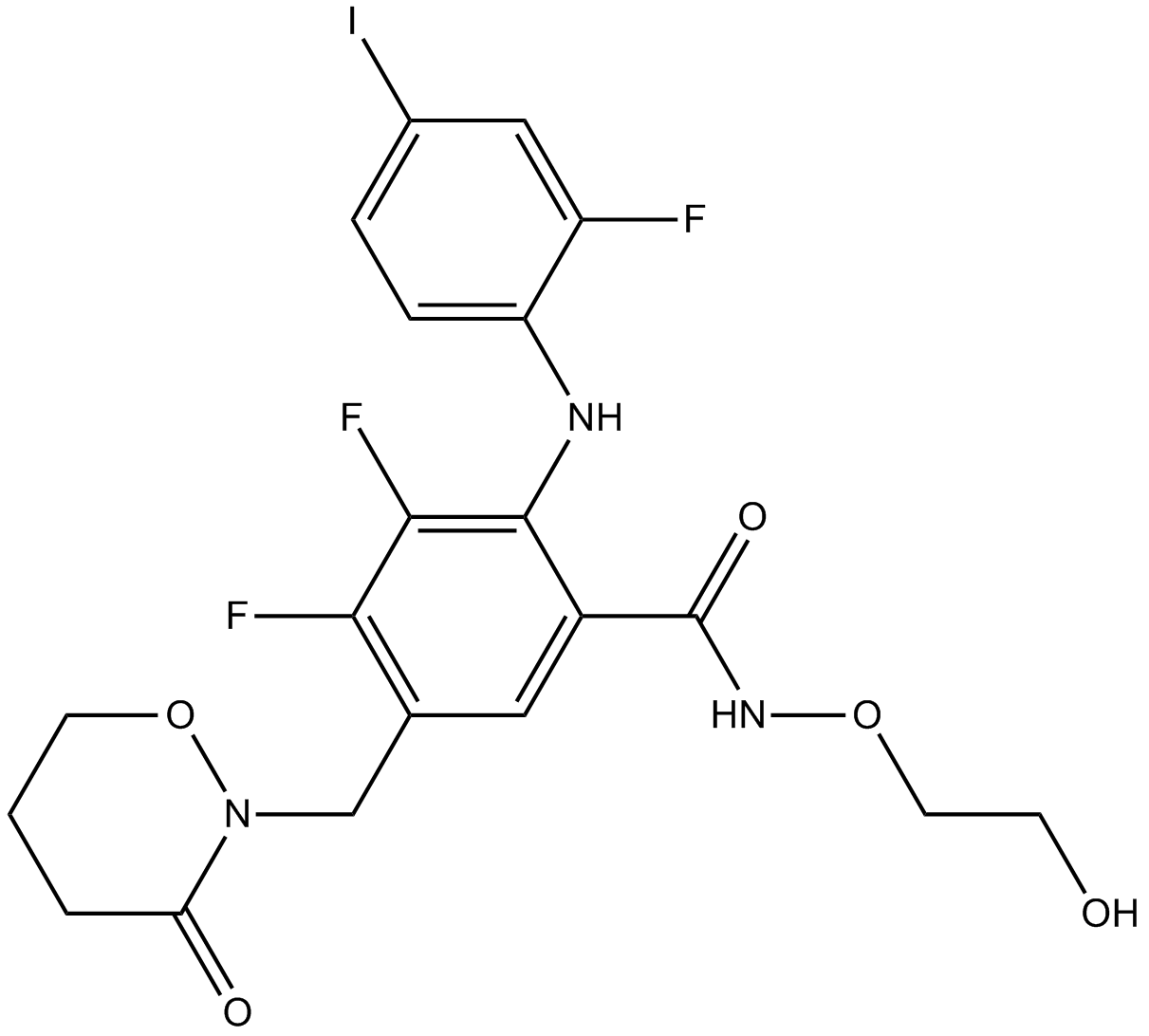
-
GC12406
RO5126766(CH5126766)
RO5126766(CH5126766) (CH5126766) es un inhibidor dual MEK/RAF primero en su clase que inhibe alostéricamente BRAFV600E, CRAF, MEK y BRAF (IC50: 8.2, 56, 160 nM y 190 nM, respectivamente).
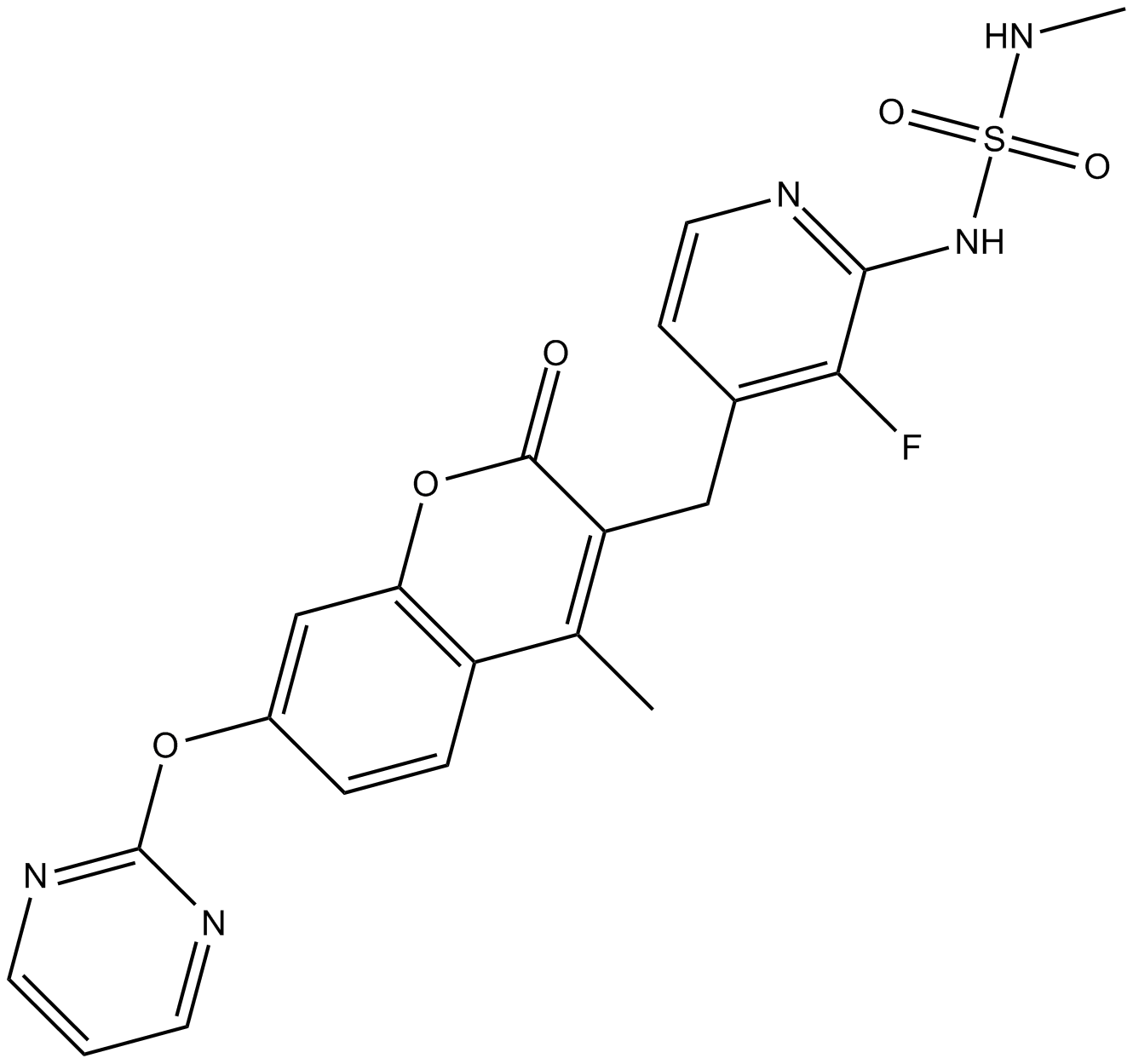
-
GC38609
Rotundic acid
Ácido rotÚndico, un triterpenoide obtenido de I.
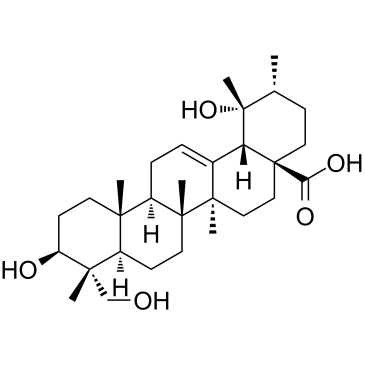
-
GC15611
Rp-8-Br-PET-cGMPS
cGMP-dependent protein kinase (PKG) inhibitor
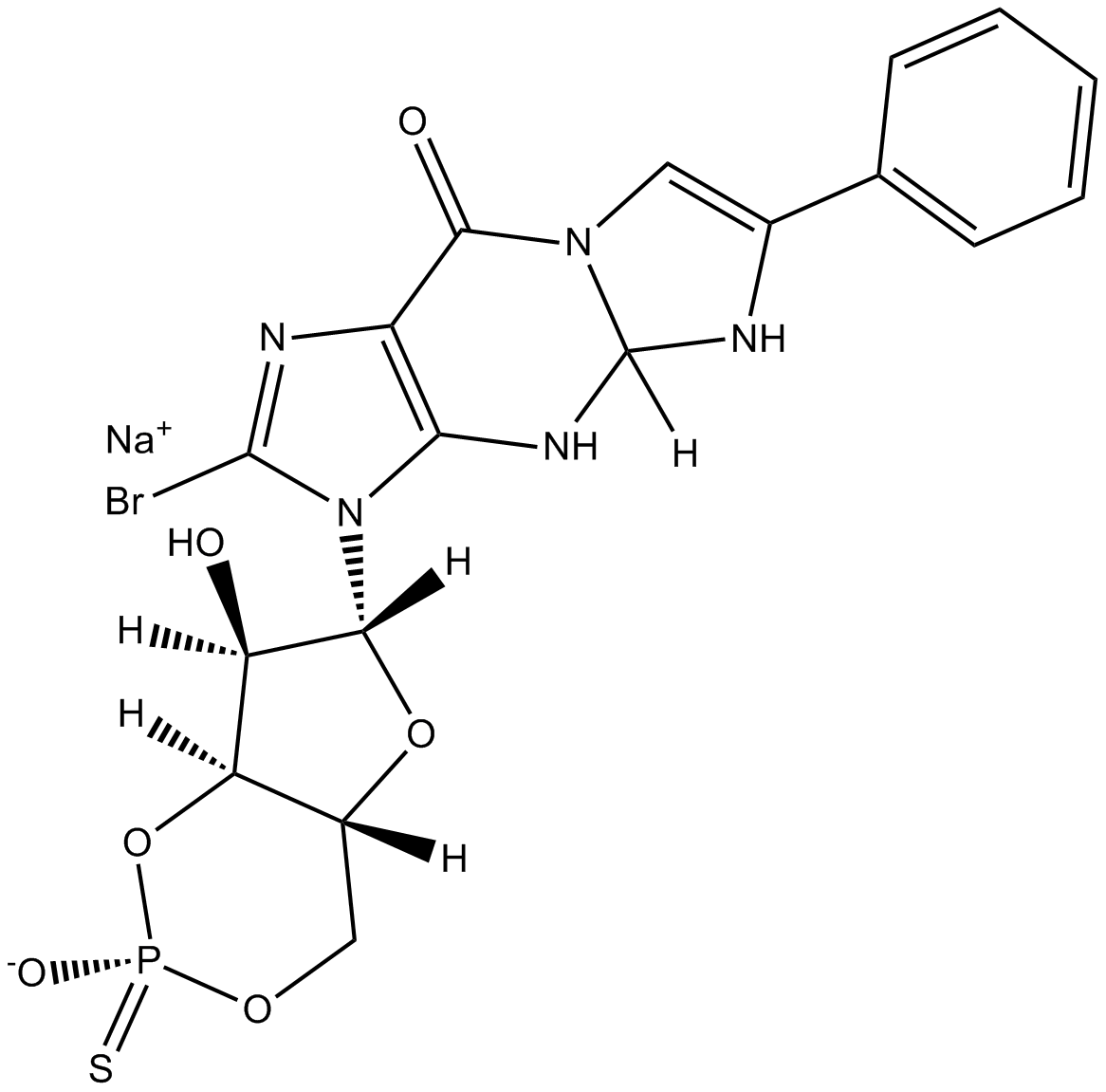
-
GC44852
Rp-8-bromo-Cyclic AMPS (sodium salt)
Rp-8-bromo-Cyclic AMPS (Rp-8-bromo-cAMPS) is a cell-permeable cAMP analog that combines an exocyclic sulfur substitution in the equatorial position of the cyclophosphate ring with a bromine substitution in the adenine base of cAMP.
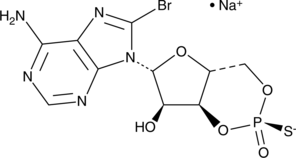
-
GC44853
Rp-8-CPT-Cyclic AMP (sodium salt)
Rp-8-CPT-Cyclic AMP (sal de sodio), un anÁlogo de cAMP, es un antagonista potente y competitivo de la activaciÓn inducida por cAMP de PKA I y II dependiente de cAMP.
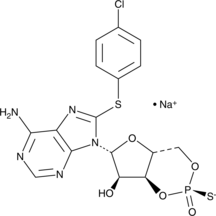
-
GC63176
Rp-cAMPS sodium salt
La sal sÓdica de Rp-cAMPS, un anÁlogo de cAMP, es una activaciÓn potente y competitiva inducida por cAMP de antagonistas de PKA I y II dependientes de cAMP (Kis de 12,5 μM y 4,5 μM, respectivamente).
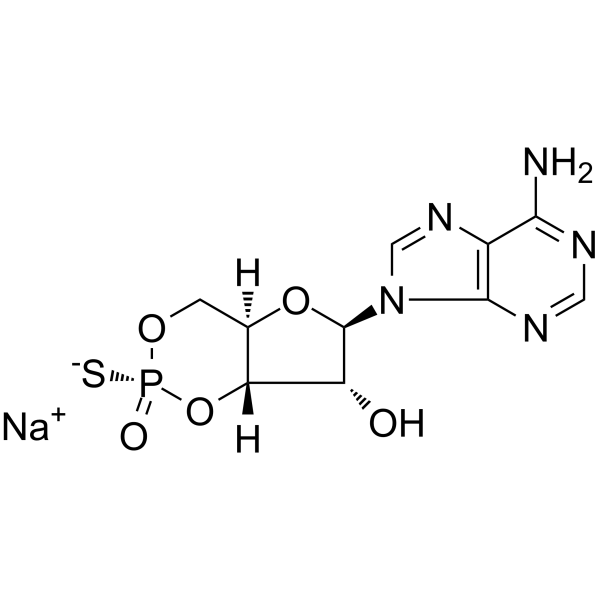
-
GC62269
RRD-251
RRD-251 es un inhibidor de la interacciÓn de la proteÍna supresora de tumores de retinoblastoma (Rb)-Raf-1, con potentes actividades antiproliferativas, antiangiogénicas y antitumorales.