MAPK Signaling

Ziele für MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Produkte für MAPK Signaling
- Bestell-Nr. Artikelname Informationen
-
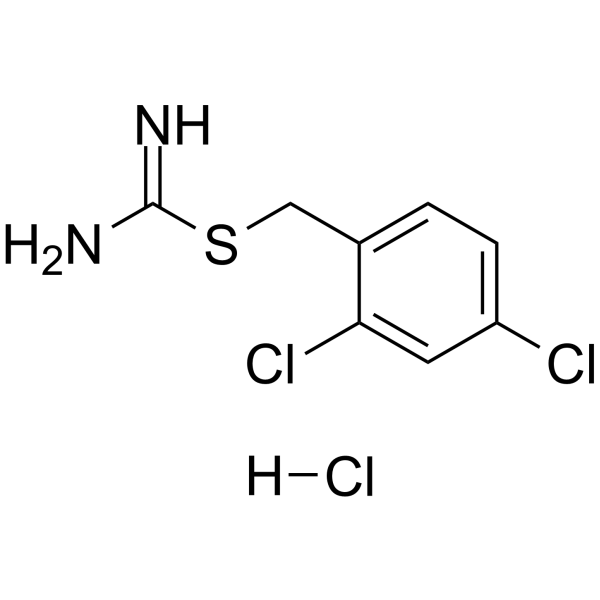
GC36652
MPT0B392
MPT0B392, ein oral aktives Chinolinderivat, induziert die Aktivierung der c-Jun-N-terminalen Kinase (JNK), was zu Apoptose fÜhrt. MPT0B392 hemmt die Tubulinpolymerisation und lÖst die Induktion des mitotischen Arrests aus, gefolgt von einem Verlust des mitochondrialen Membranpotentials und einer Caspasenspaltung durch Aktivierung von JNK und fÜhrt schließlich zur Apoptose. MPT0B392 erweist sich als neuartiges Mikrotubuli-depolymerisierendes Mittel und verstÄrkt die ZytotoxizitÄt von Sirolimus in Sirolimus-resistenten akuten LeukÄmiezellen und der mehrfach resistenten Zelllinie.
-
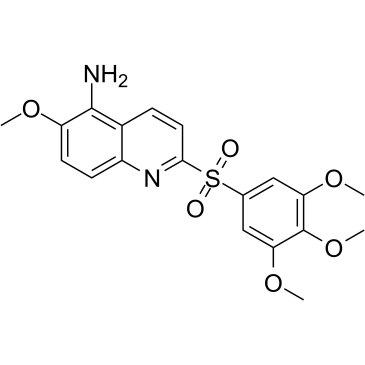
GC64292
MS432
MS432 ist ein erstklassiger und hochselektiver PD0325901-basierter von Hippel-Lindau rekrutierender PROTAC-Abbauer fÜr MEK1 und MEK2. MS432 zeigt eine gute Plasmaexposition bei MÄusen und zeigt DC50-Werte von 31 nM und 17 nM fÜr MEK1 bzw. MEK2 in HT29-Zellen.
-
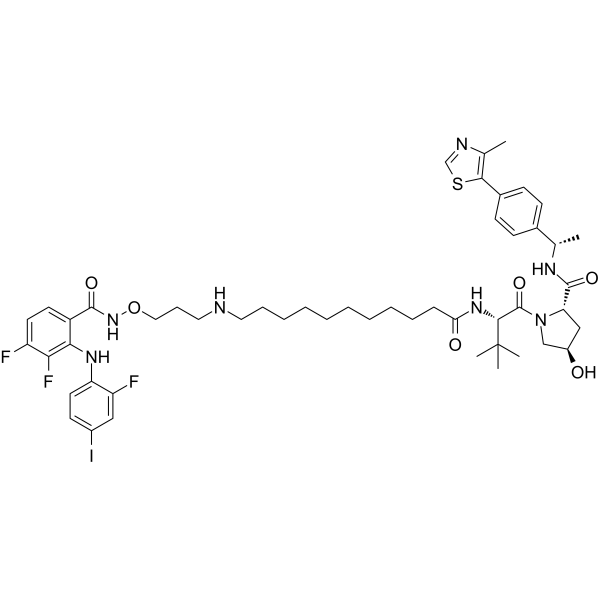
GC65297
MW-150
MW150 (MW01-18-150SRM) ist ein selektiver, das ZNS durchdringender und oral wirksamer Inhibitor von p38α-MAPK mit einem Ki von 101 nM.
-
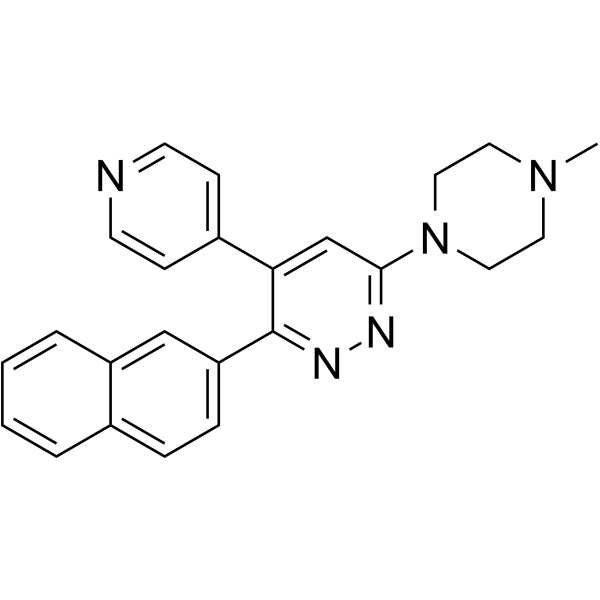
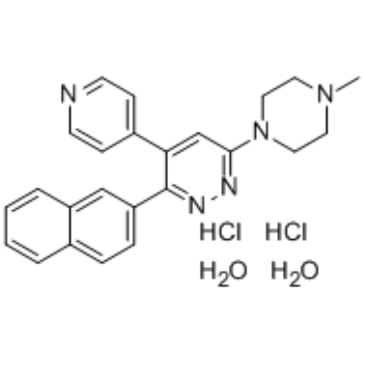
GC34918
MW-150 dihydrochloride dihydrate
MW-150-Dihydrochlorid-Dihydrat (MW01-18-150SRM-Dihydrochlorid-Dihydrat) ist ein selektiver, das ZNS durchdringender und oral aktiver Inhibitor von p38α-MAPK mit einem Ki von 101 nM.
-
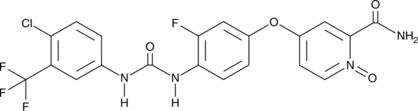
GC49686
N-desmethyl Regorafenib N-oxide
An active metabolite of regorafenib
-
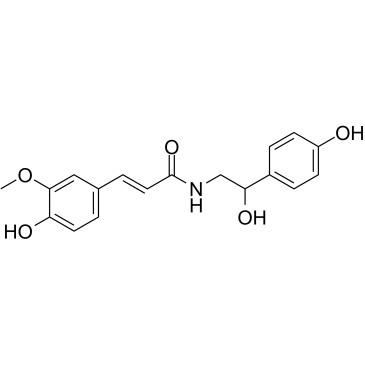
GC60262
N-Feruloyloctopamine
N-Feruloyloctopamin ist ein Antioxidansbestandteil. N-Feruloyloctopamin verringert signifikant die Phosphorylierungsgrade von Akt und p38 MAPK.
-
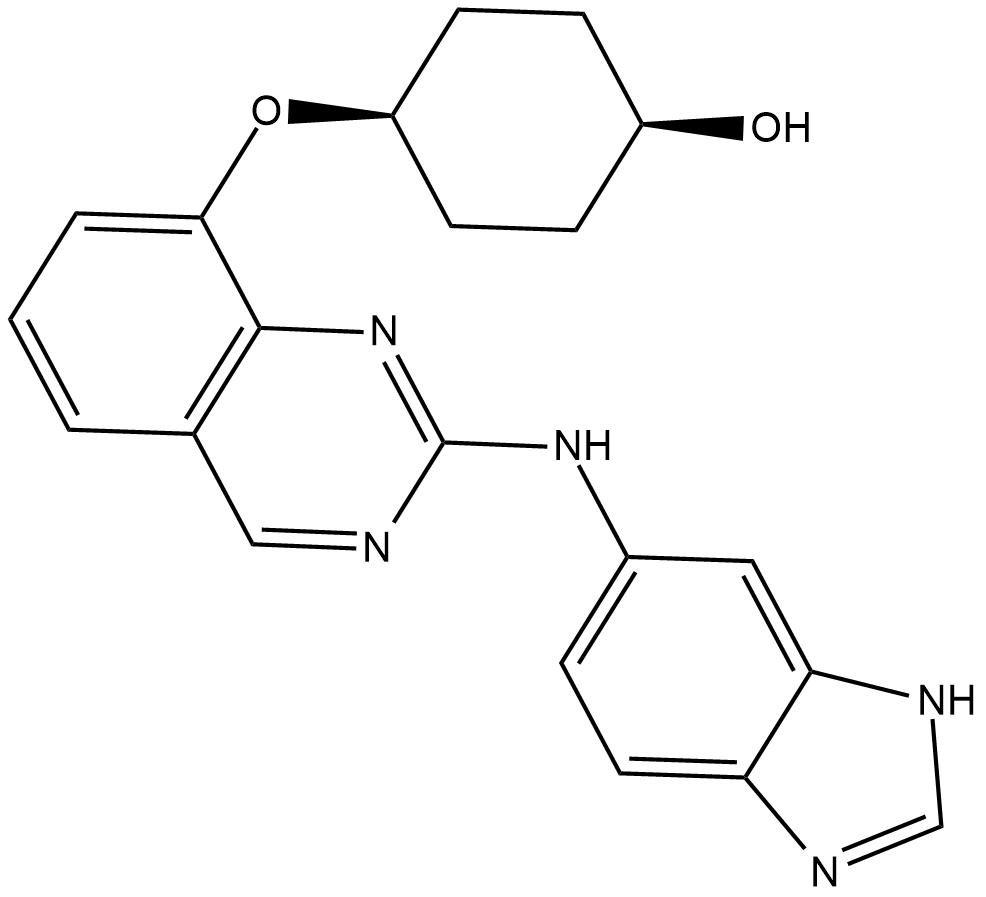
GC19260
NCB-0846
NCB-0846 ist ein oral verfÜgbarer TNIK-Inhibitor mit einem IC50 von 21nM.
-
GC69543
NecroIr1
NecroIr1 ist eine Iridium (III)-Verbindung und ein Induktor von Nekrose in Cisplatin-resistenten Lungenkrebszellen (A549R). NecroIr1 reichert sich selektiv in den Mitochondrien an, was zu oxidativem Stress und Verlust des mitochondrialen Membranpotenzials (MMP) führt. NecroIr1 aktiviert die Serin/Threonin-Kinasen Rezeptor-interagierendes Protein Kinase 3 (RIPK3) und Pseudo-Kinase mit Mischlinien-Struktur-Domäne ähnlich wie Kinase (MLKL), reguliert die Expression von CDK4.
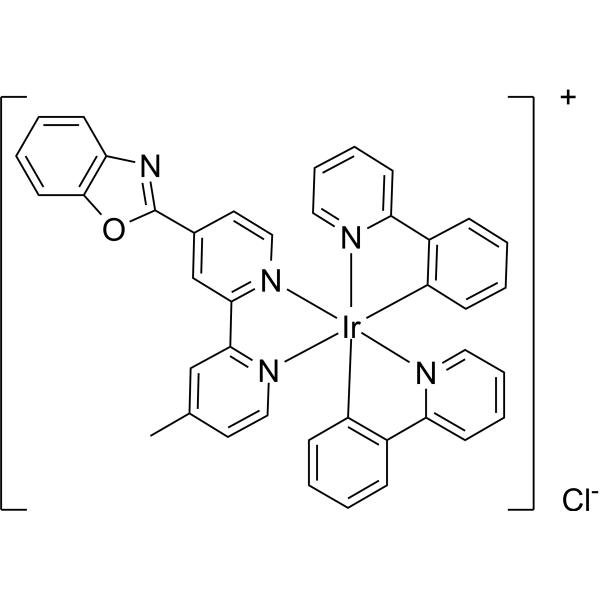
-
GC69544
NecroIr2
NecroIr2 ist eine Iridium (III) Verbindung und ein Induktor von Nekrose in Cisplatin-resistenten Lungenkrebszellen (A549R). NecroIr2 reichert sich selektiv in den Mitochondrien an, was zu oxidativem Stress und dem Verlust des mitochondrialen Membranpotenzials (MMP) führt. NecroIr2 aktiviert die Serin/Threonin-Kinasen Rezeptor-interagierendes Protein Kinase 3 (RIPK3) und Pseudo-Kinase mit Mischlinien-Struktur-Domäne ähnlich wie Kinase (MLKL), reguliert die Expression von CDK4.
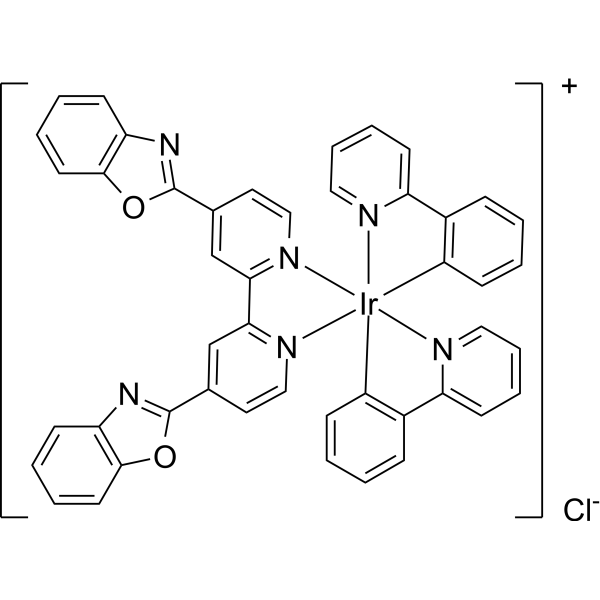
-
GC47771
NG 25 (hydrochloride hydrate)
An inhibitor of MAP4K2 and TAK1
-
GC13514
NG25
An inhibitor of MAP4K2 and TAK1

-
GC17577
NQDI 1
A selective inhibitor of ASK1
-
GC10069
NSC 23766 trihydrochloride
NSC 23766 Trihydrochlorid ist ein Inhibitor der Rac1-Aktivierung.
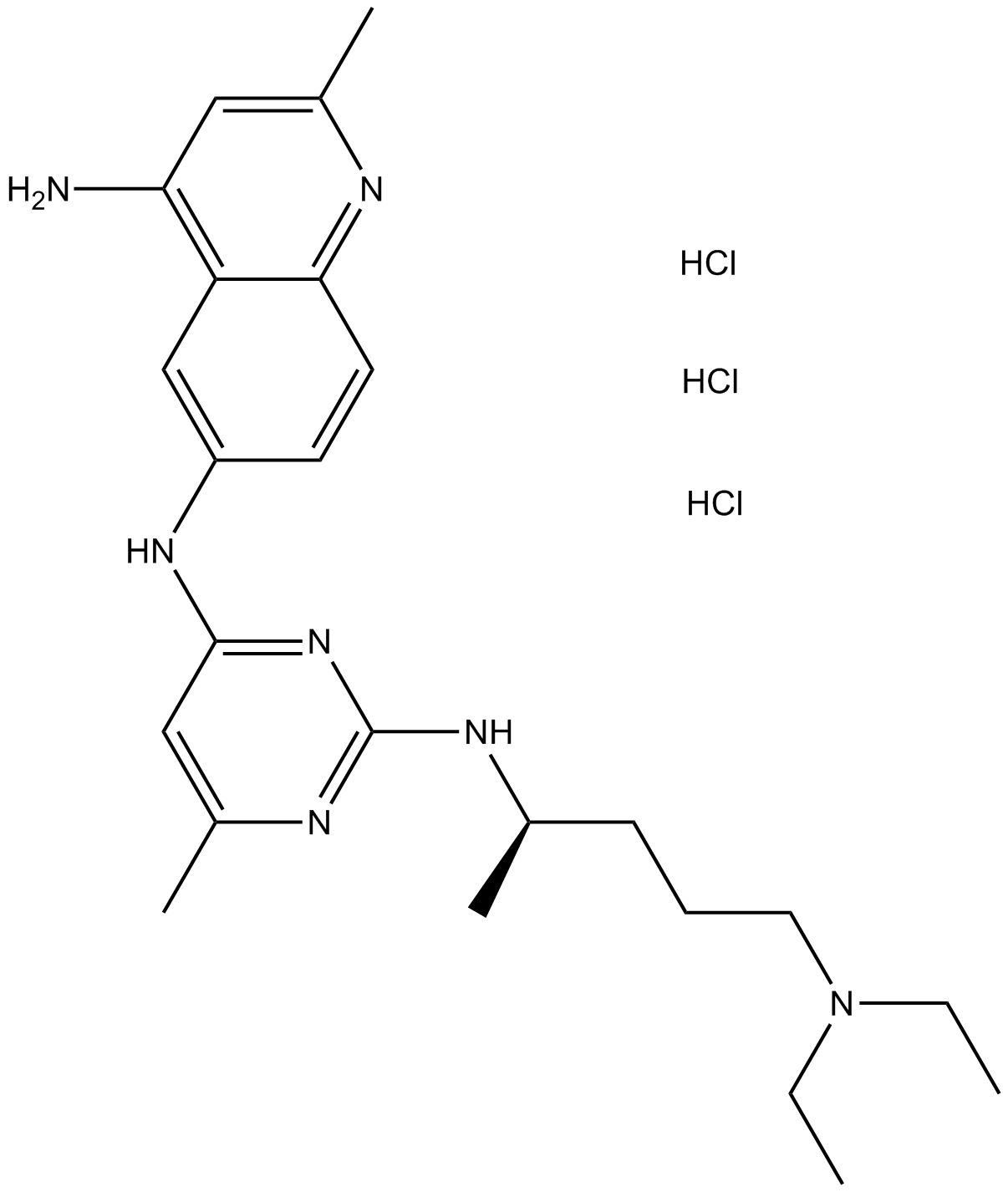
-
GC49186
O-Demethyl Apremilast
An active metabolite of apremilast
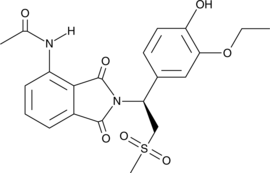
-
GC18951
Obscurolide A1
Obscurolide A1 is a natural butyrolactone isolated from S.
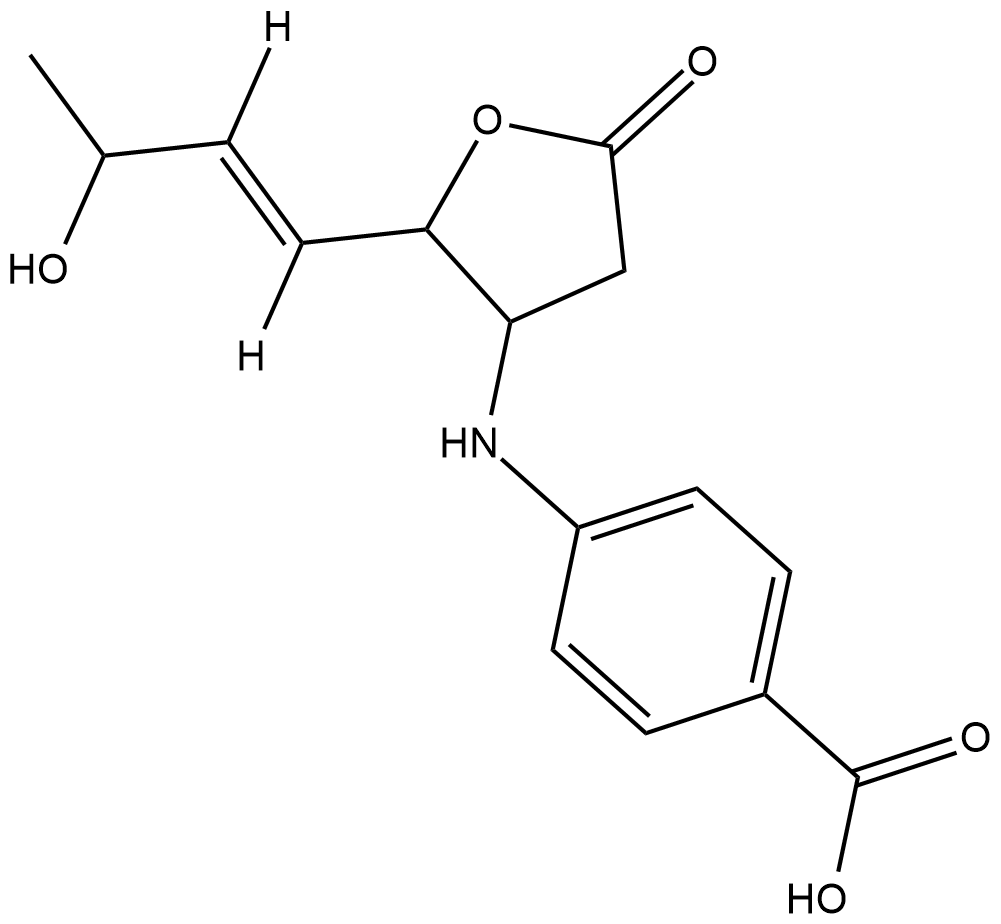
-
GC48652
Olomoucine II
A CDK inhibitor
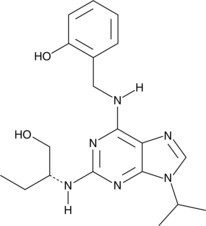
-
GC33857
Omtriptolide
Omtriptolid (PG490-88) ist ein abgeleitetes Prodrug von Triptolid, gereinigt aus dem chinesischen Kraut.
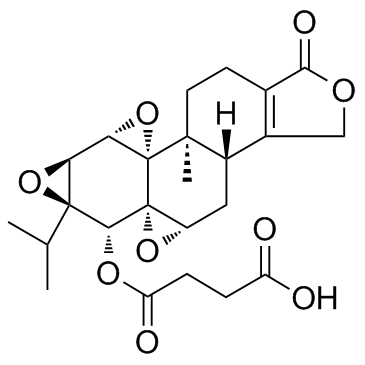
-
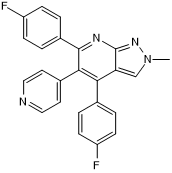
GC50316
Org 48762-0
Selective p38α/β inhibitor; orally bioavailable
-
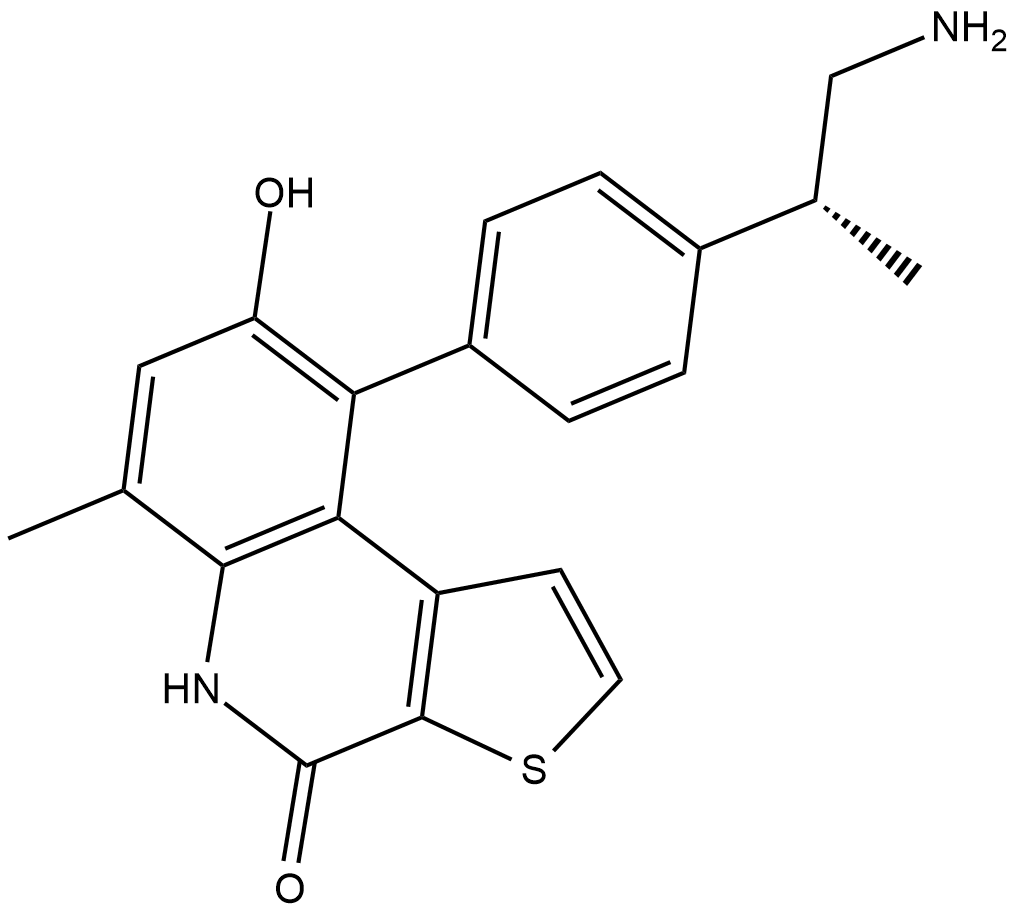
GC12304
OTS514
OTS514 ist ein hochpotenter TOPK-Inhibitor mit einem IC50 von 2,6 nM. OTS514 unterdrÜckt stark das Wachstum von TOPK-positiven Krebszellen. OTS514 induziert Zellzyklusarrest und Apoptose.
-
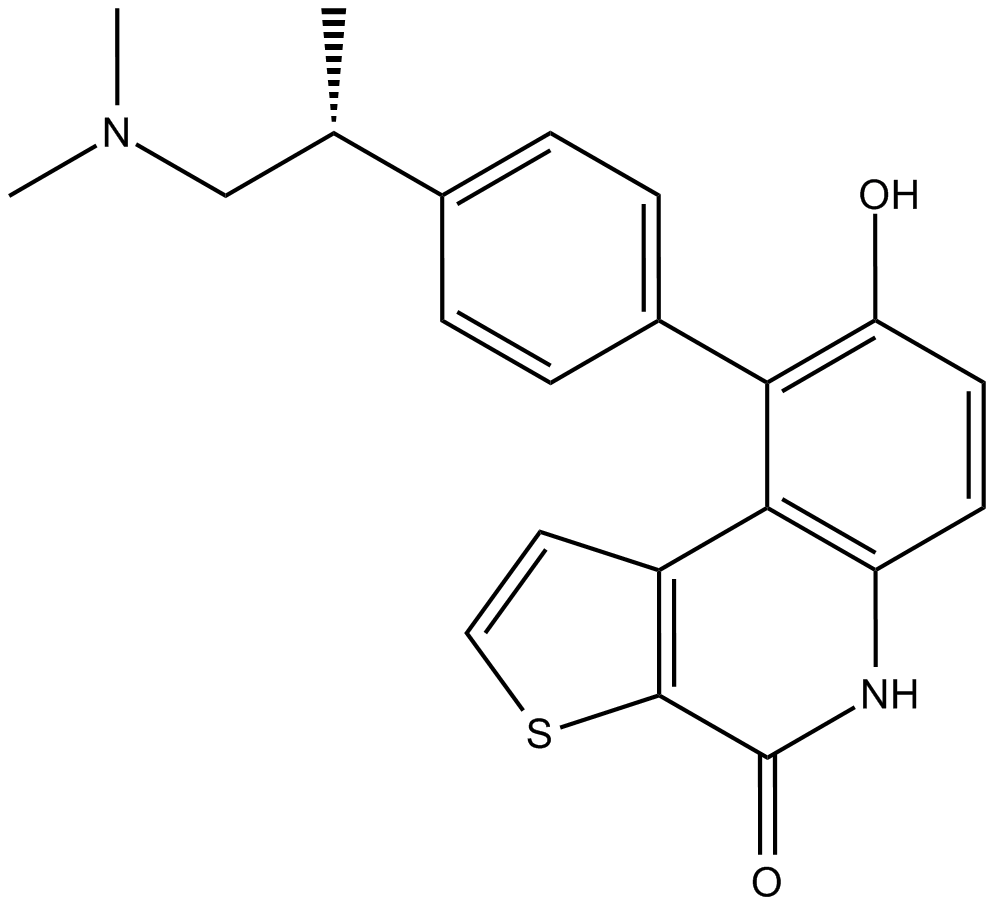
GC16511
OTS964
OTS964 ist ein oral aktiver, hochaffiner und selektiver TOPK-Inhibitor (T-lymphokin-activated killer cell-originated protein kinase) mit einem IC50 von 28 nM. OTS964 ist auch ein potenter Inhibitor der Cyclin-abhÄngigen Kinase CDK11, die mit einer Kd von 40 nM an CDK11B bindet.
-
GC69639
OVA-E1 peptide TFA
OVA-E1 Peptid TFA ist ein Antagonist-Mutant von SIINFEKL [OVA (257-264)]. OVA-E1 Peptid aktiviert die p38 und JNK Kaskade in mutierten und wildtypischen Thymozyten.
-
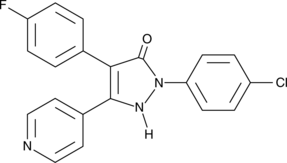
GC44530
p38 MAPK Inhibitor
p38 MAPK inhibitor is a potent inhibitor of p38 MAP kinase (IC50 = 35 nM).
-
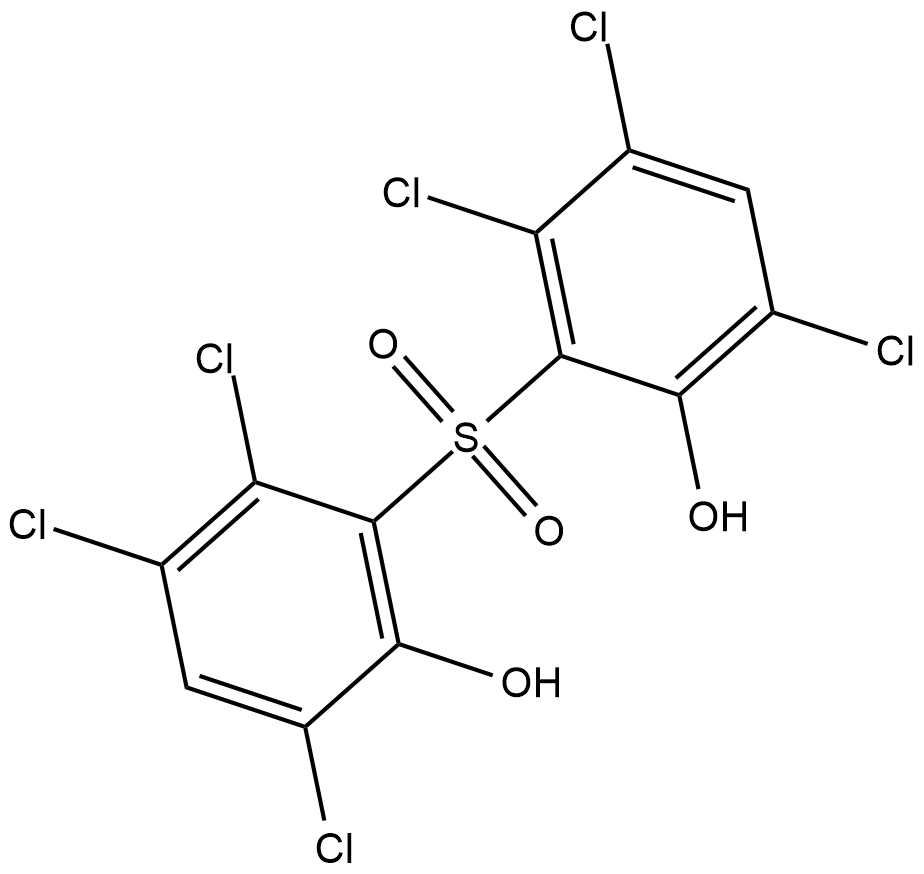
GC18602
p38 MAPK Inhibitor IV
p38 MAPK Inhibitor IV ist ein hochspezifisches ATP-kompetitives p38α MAPK-Inhibitor mit IC50s von 0,13 und 0,55 μ M fÜr p38α und p38β MAPK bzw.
-
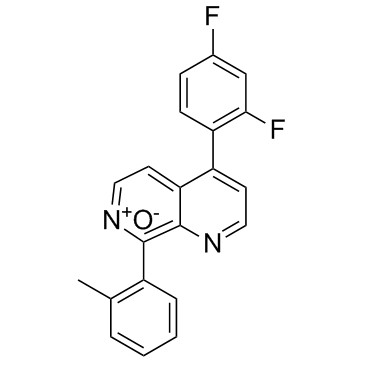
GC33008
p38 MAPK-IN-1
p38 MAPK-IN-1 (Verbindung 4) ist ein neuer potenter und selektiver Inhibitor von p38 MAPK mit einem IC50 von 68 nM.
-
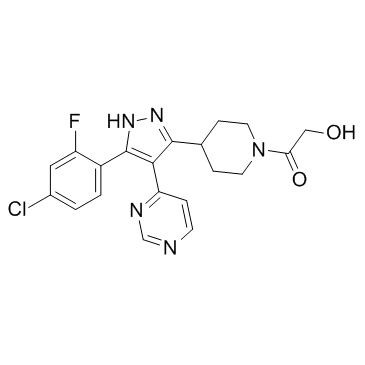
GC30609
p38 MAPK-IN-2
p38 MAPK-IN-2 ist ein Inhibitor der p38-Kinase.
-
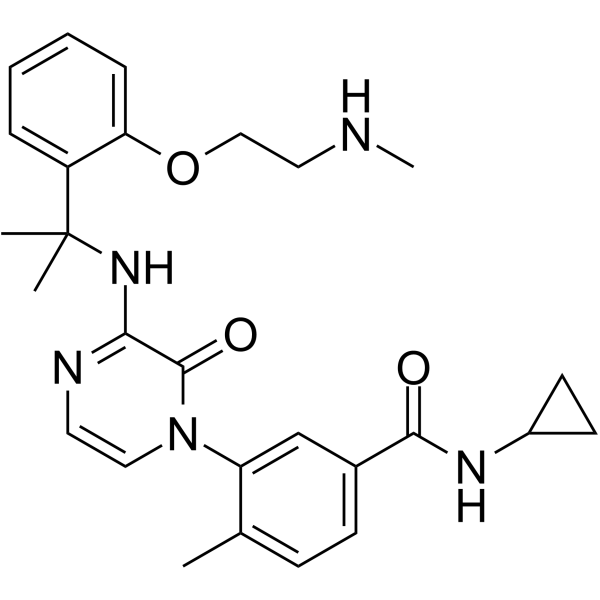
GC62404
p38α inhibitor 2
p3&8#945; Inhibitor 2 ist ein hochwirksames und selektives p3&8#945; MAPK-Inhibitor mit einem pIC50 von 9,6.
-
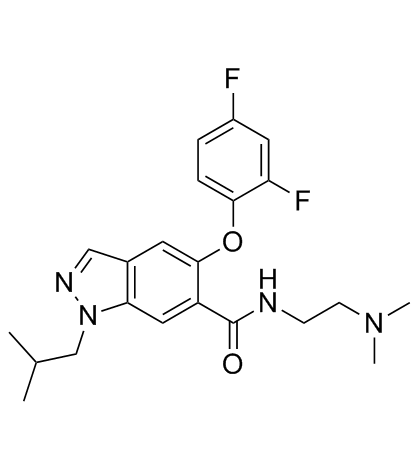
GC31988
p38α inhibitor 1
p38α Inhibitor 1 ist ein p38α-Inhibitor, extrahiert aus dem Patent WO 2008076265 A1.
-
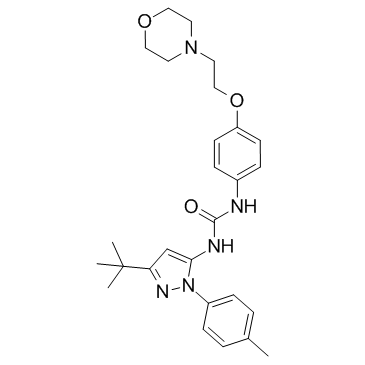
GC30158
p38-α MAPK-IN-1
p38-α MAPK-IN-1 ist ein Inhibitor von MAPK14 (p38-α) mit einem IC50-Wert von 2300 nM im EFC-Verdrängungsassay und 5500 nM im HTRF-Assay.
-
GN10752
Pachymic acid
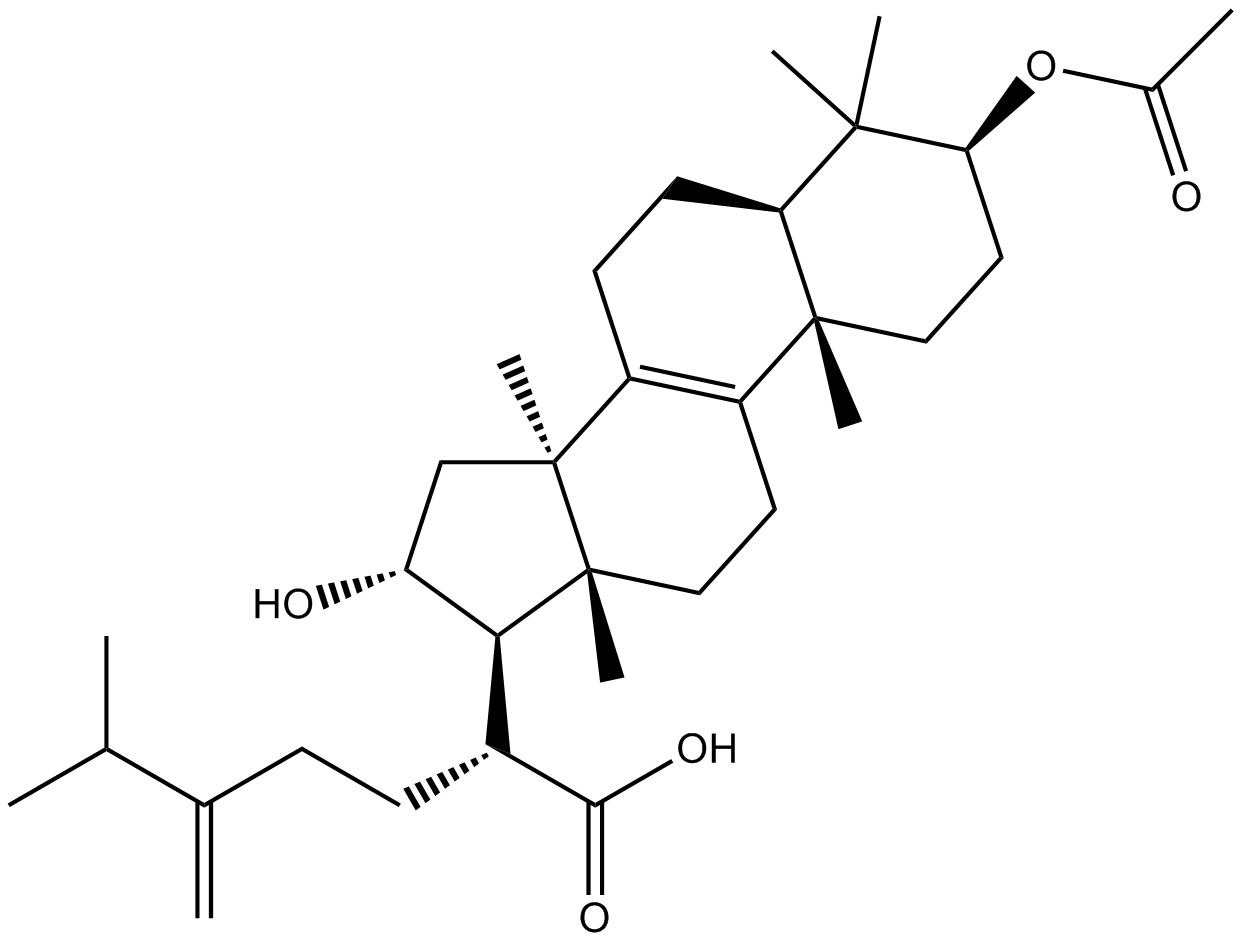
-
GC15814
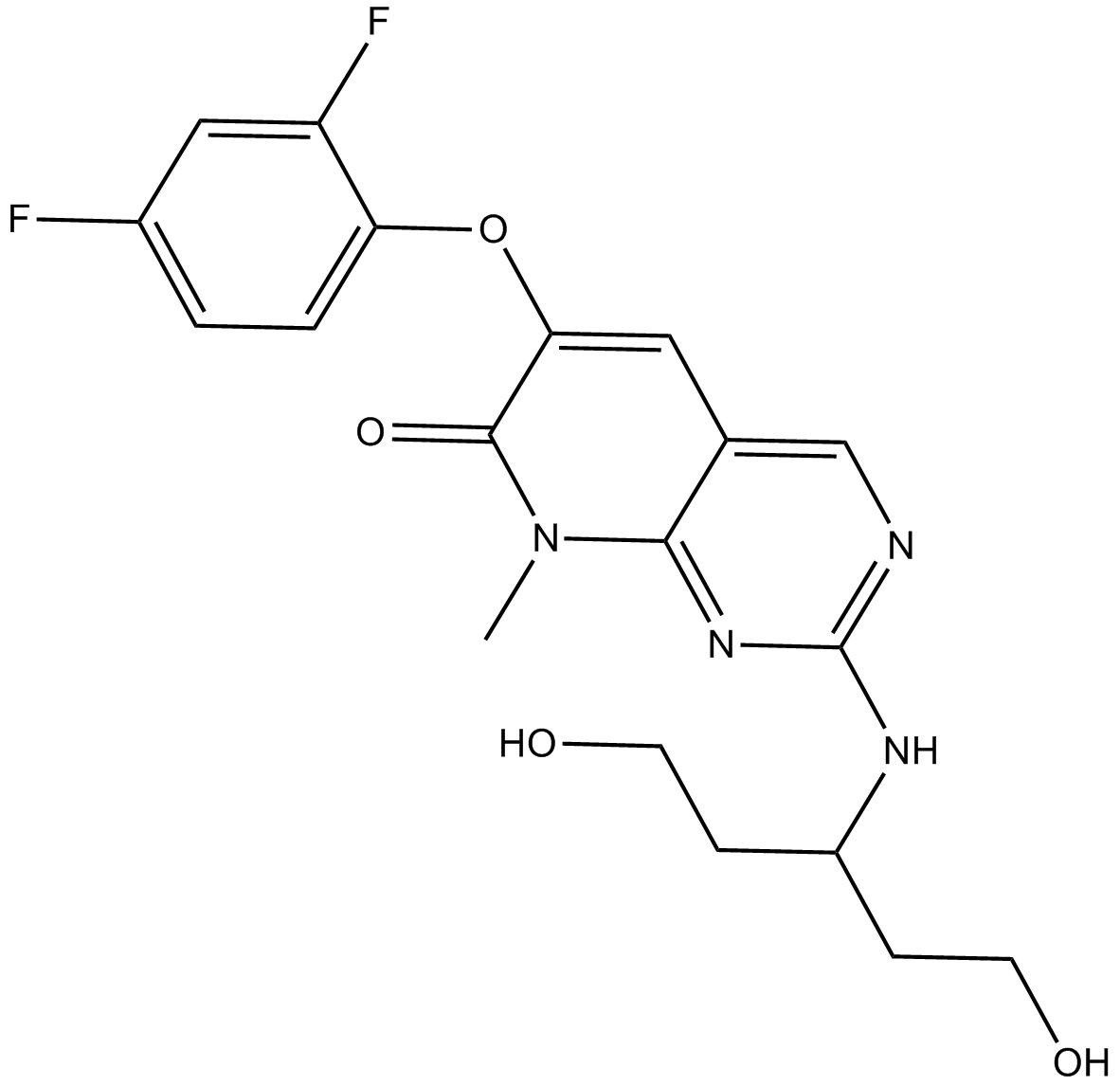
Pamapimod (R-1503, Ro4402257)
Pamapimod (R-1503, Ro4402257) (Ro4402257) ist ein potenter, selektiver und oral aktiver p38-MAPK-Inhibitor mit IC50s von 14 nM und 480 nM und Kis von 1,3 nM und 120 nM fÜr p38α bzw. p38&7#946;.
-
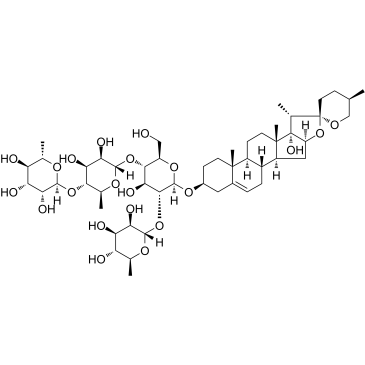
GC36855
Paris saponin VII
Paris Saponin VII (Chonglou Saponin VII) ist ein steroidales Saponin, das aus den Wurzeln und Rhizomen von Trillium tschonoskii Maxim isoliert wird. Paris-Saponin-VII-induzierte Apoptose in K562/ADR-Zellen ist mit Akt/MAPK und der Hemmung von P-gp assoziiert. Paris-Saponin VII schwÄcht das mitochondriale Membranpotential ab, erhÖht die Expression von Apoptose-verwandten Proteinen wie Bax und Cytochrom c und verringert die Proteinexpressionsniveaus von Bcl-2, Caspase-9, Caspase-3, PARP-1 und p- Akt. Paris-Saponin VII induziert eine robuste Autophagie in K562/ADR-Zellen und liefert eine biochemische Grundlage fÜr die Behandlung von LeukÄmie.
-
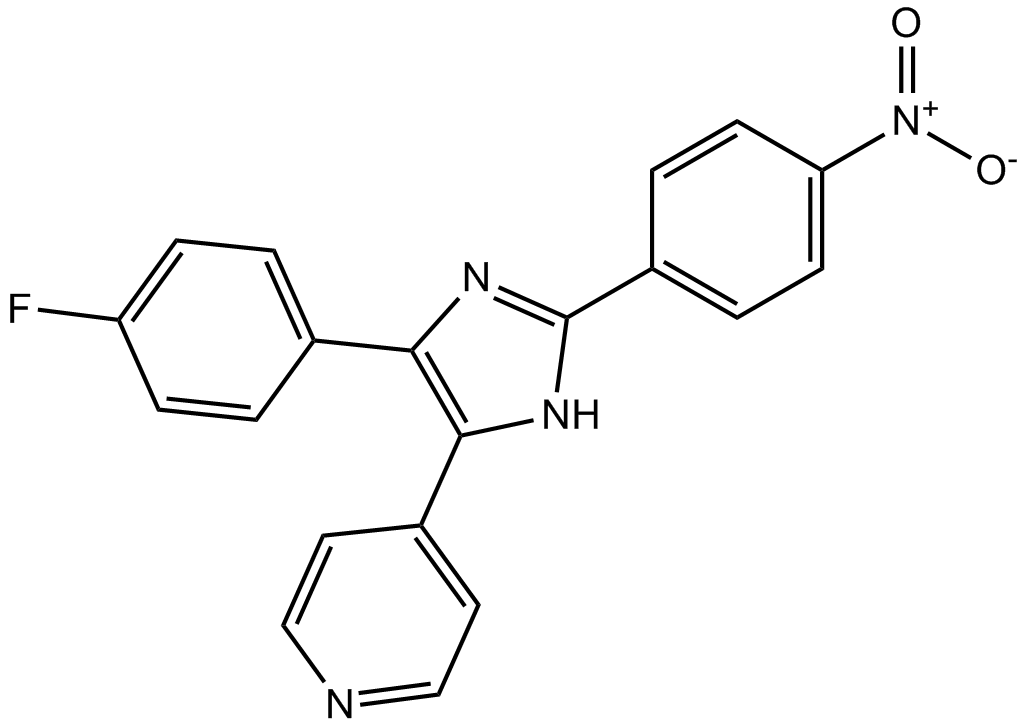
GC17737
PD 169316
PD 169316 ist ein potenter, zellgÄngiger und selektiver p38-MAP-Kinase-Inhibitor mit einem IC50-Wert von 89 nM. PD169316 hemmt selektiv die KinaseaktivitÄt des phosphorylierten p38, ohne stromaufwÄrts gelegene Kinasen daran zu hindern, p38 zu phosphorylieren. PD169316 zeigt antivirale AktivitÄt gegen Enterovirus71. PD169316 zeigt antivirale AktivitÄt gegen Enterovirus71.
-
GC15646
PD 184161
PD 184161 ist ein oral aktiver MEK-Inhibitor.
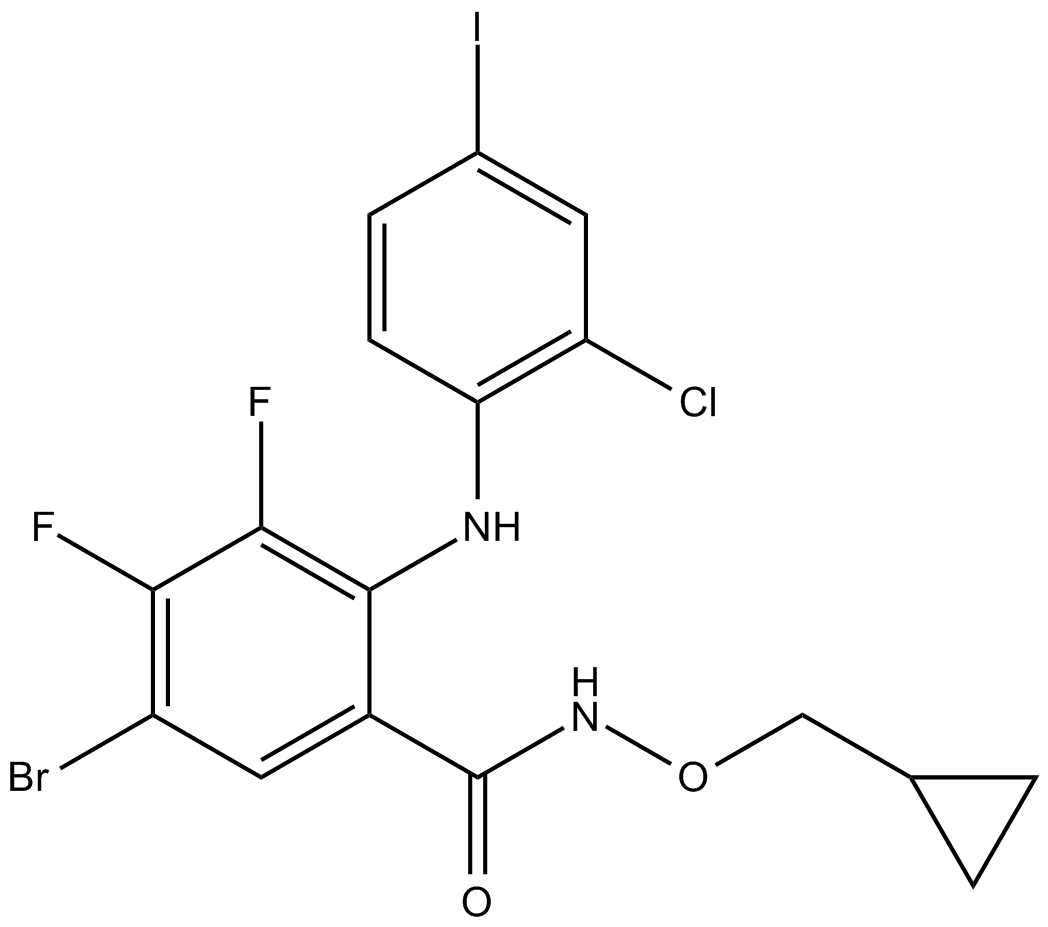
-
GC17964
PD 198306
PD 198306 ist ein selektiver MAPK/ERK-Kinase (MEK)-Inhibitor.
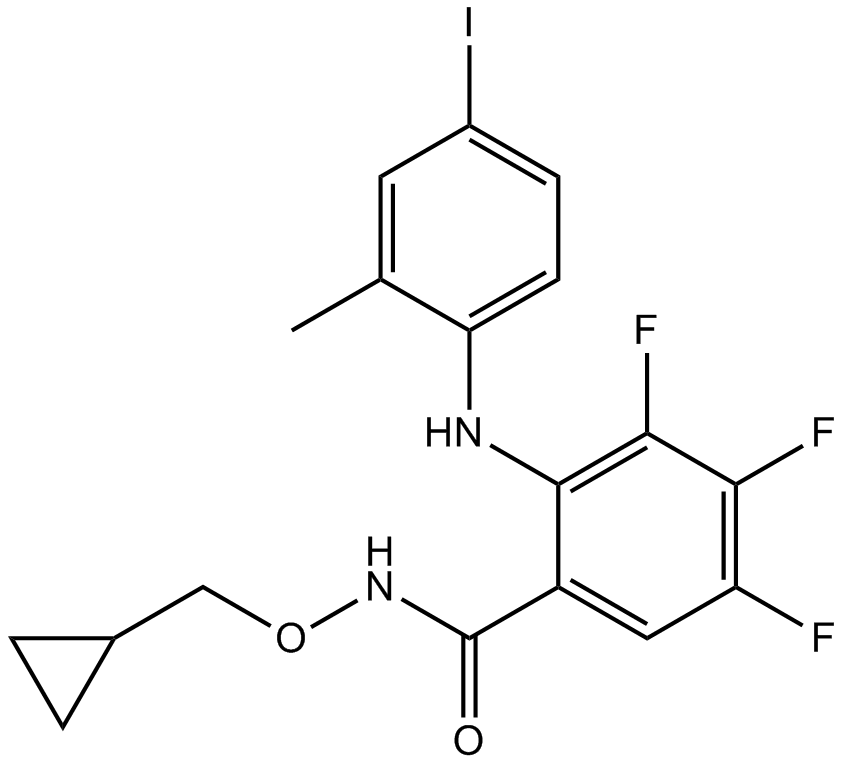
-
GC17485
PD 334581
PD 334581 ist ein MEK1-Inhibitor.
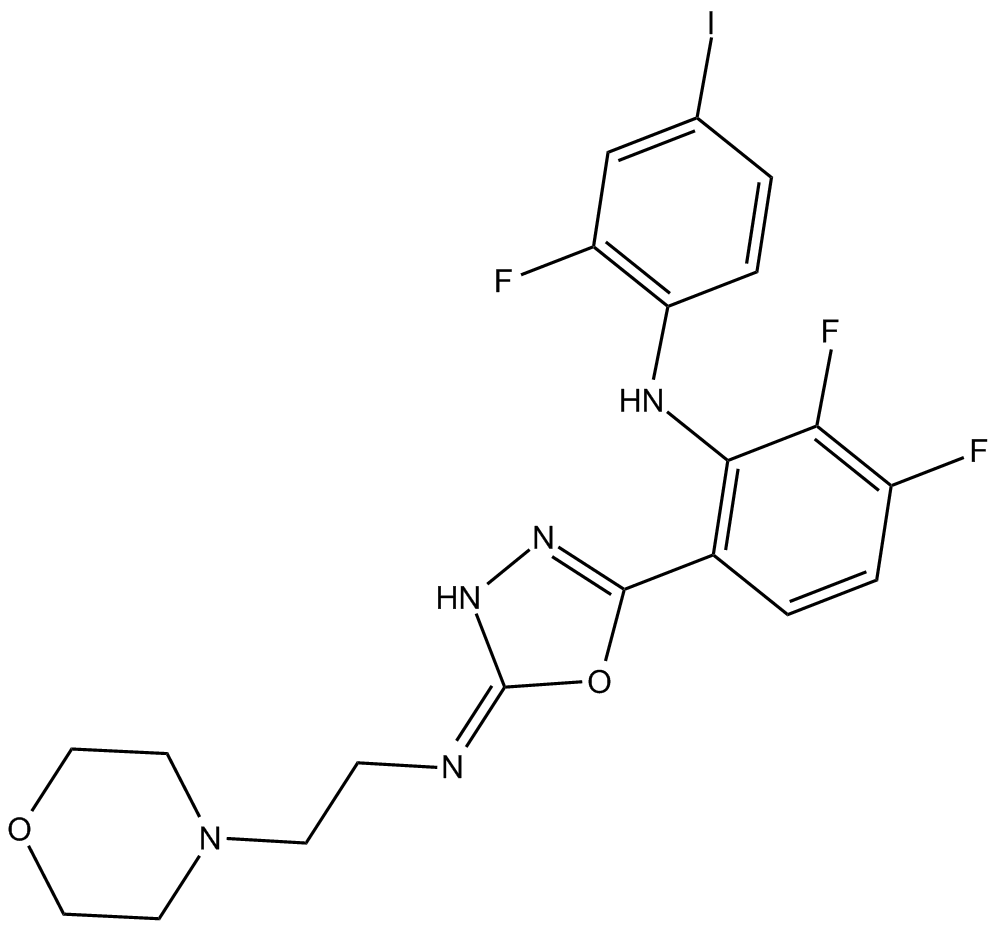
-
GC10397
PD0325901
Ein MEK-Inhibitor, der die Stammzellenerneuerung aufrechterhält.
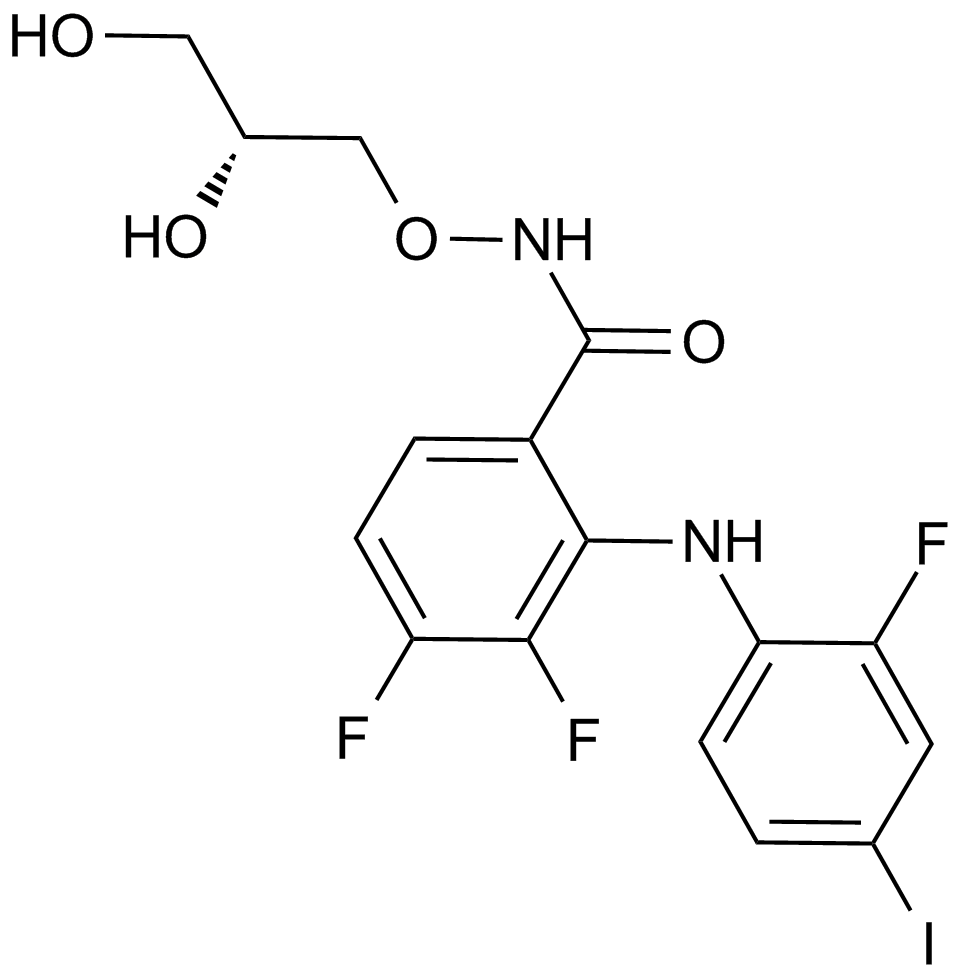
-
GC63554
PD0325901-O-C2-dioxolane
PD0325901-O-C2-Dioxolan hat den Hauptanteil des MEK-Inhibitors PD0325901. PD0325901-O-C2-Dioxolan und ein Ligand der VHL- oder CRBN-E3-Ligase kÖnnen bei der Synthese des MEK1/2-Abbaumittels verwendet werden.
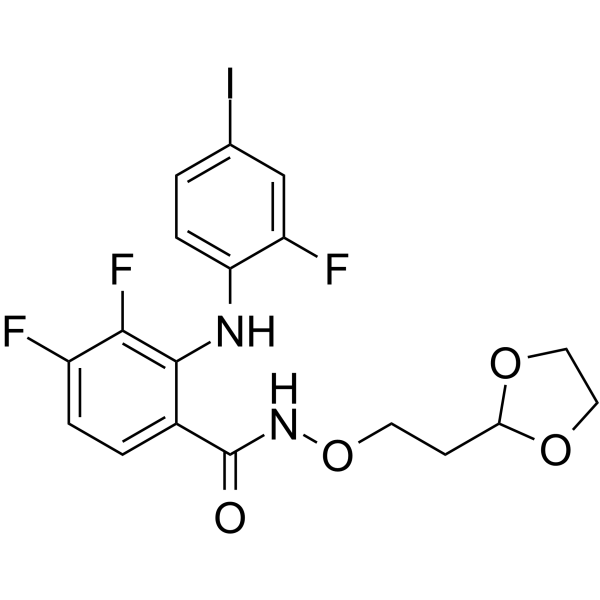
-
GC12989
PD184352 (CI-1040)
PD184352 (CI-1040) (PD 184352) ist ein oral aktiver, hochspezifischer, niedermolekularer Inhibitor von MEK mit einem IC50 von 17 nM fÜr MEK1.
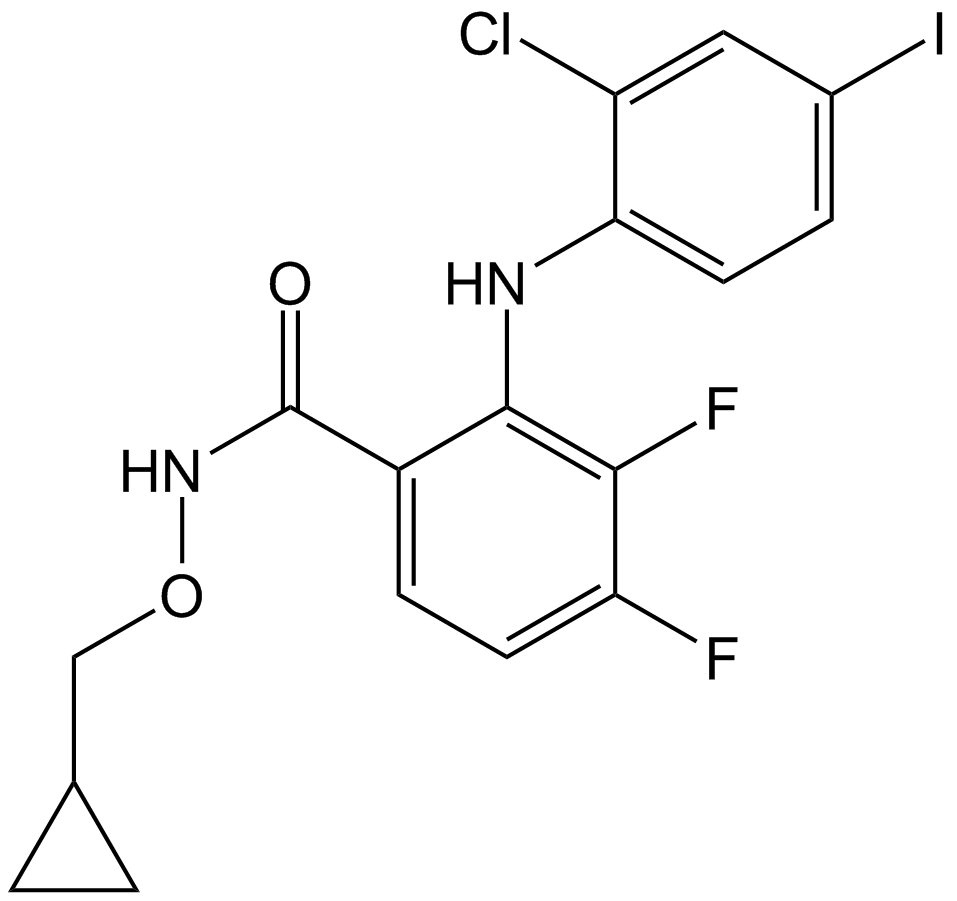
-
GC10110
PD318088
PD318088 ist ein potenter, allosterischer und nicht-ATP-kompetitiver MEK1/2-Inhibitor, ein Analogon von PD184352. PD318088 bindet gleichzeitig mit ATP in einer Region der aktiven Stelle von MEK1, die an die ATP-Bindungsstelle angrenzt. PD318088 kann fÜr die Krebsforschung verwendet werden.
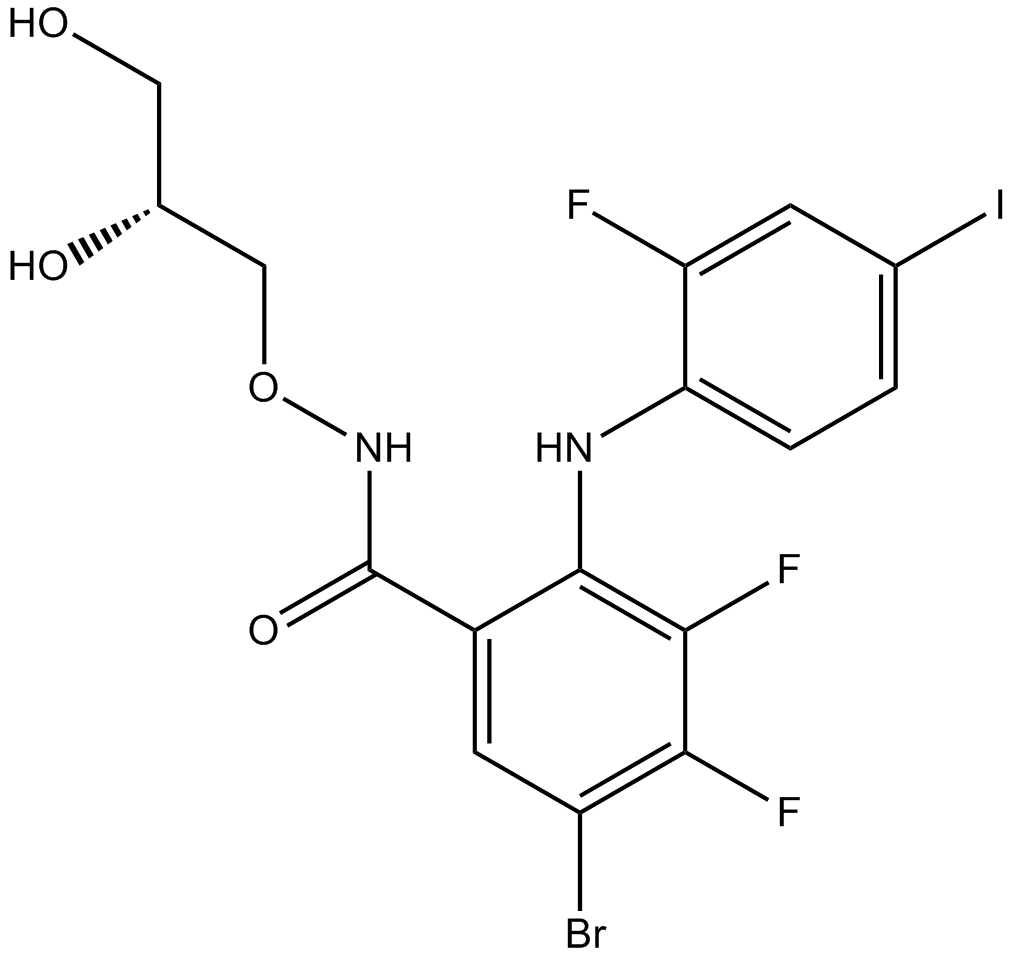
-
GC12819
PD98059
PD98059 ist ein potenter und selektiver MEK-Inhibitor mit einem IC50 von 5 μM. PD98059 bindet an die inaktive Form von MEK und verhindert dadurch die Aktivierung von MEK1 (IC50 von 2-7 μM) und MEK2 (IC50 von 50 μM) durch vorgeschaltete Kinasen. PD98059 ist ein ERK1/2-Signalweg-Inhibitor. PD98059 ist ein Ligand fÜr den Arylhydrocarbonrezeptor (AHR) und unterdrÜckt die TCDD-Bindung (IC50 von 4 μM) und die AHR-Transformation (IC50 von 1 μM). PD98059 hemmt auch die Autophagie.
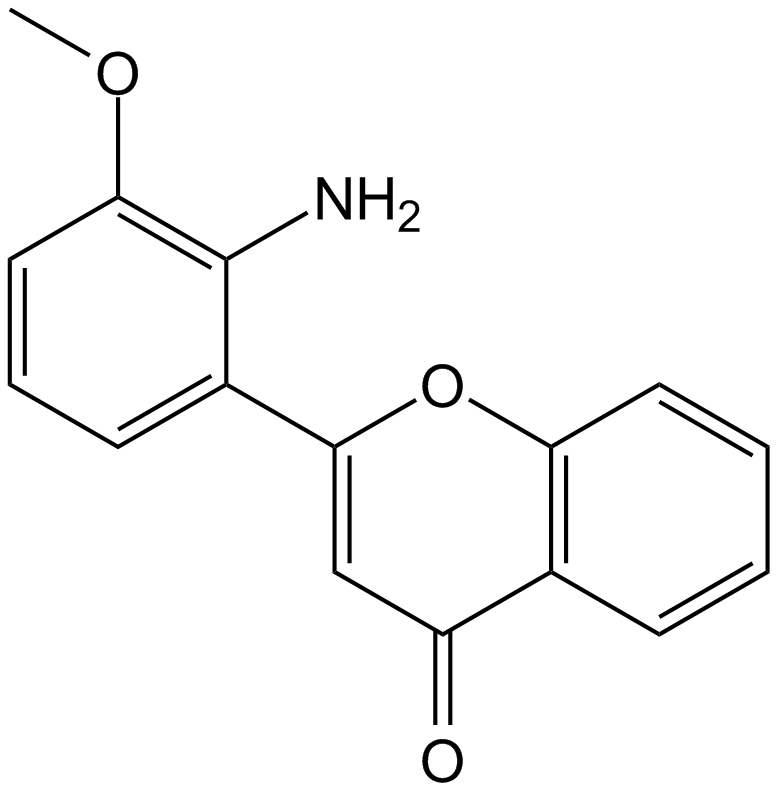
-
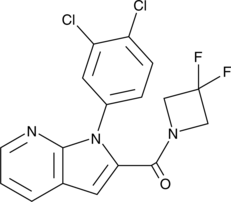
GC47931
PDE4B Inhibitor
A PDE4B inhibitor
-
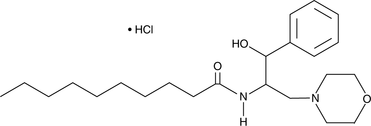
GC44587
PDMP (hydrochloride)
PDMP is a ceramide analog first prepared in a search for inhibitors of glucosylceramide synthase.
-
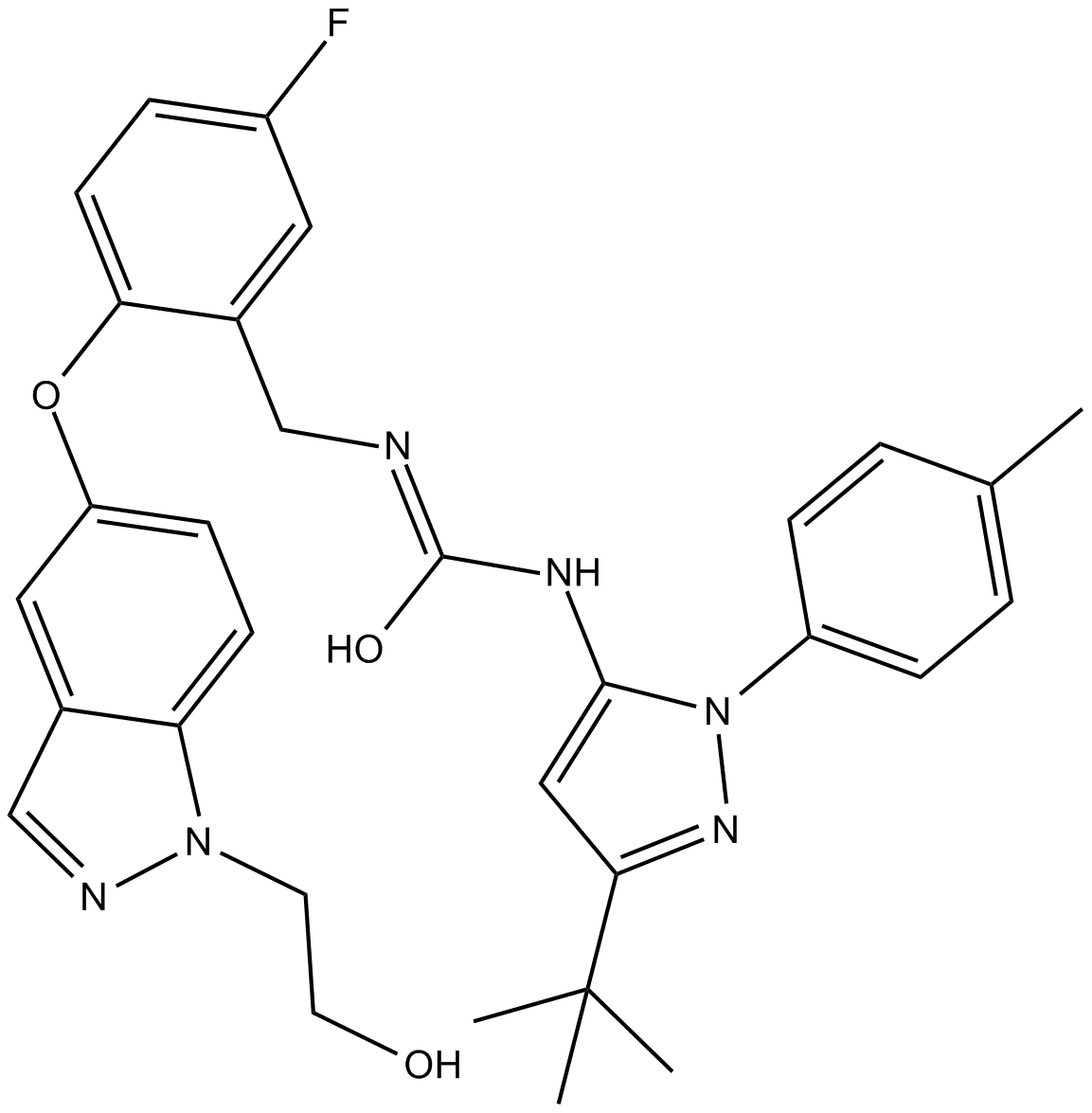
GC11857
Pexmetinib (ARRY-614)
Pexmetinib (ARRY-614) ist ein potenter Tie-2- und p38-MAPK-Dual-Inhibitor mit IC50-Werten von 1 nM, 35 nM und 26 nM fÜr Tie-2, p38α bzw. p38β und kÖnnen bei der Erforschung akuter myeloischer LeukÄmie verwendet werden.
-
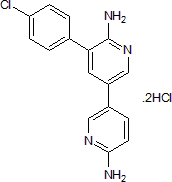
GC50252
PF 06260933 dihydrochloride
MAP4K4 (HGK) inhibitor; also inhibits MINK and TNIK
-
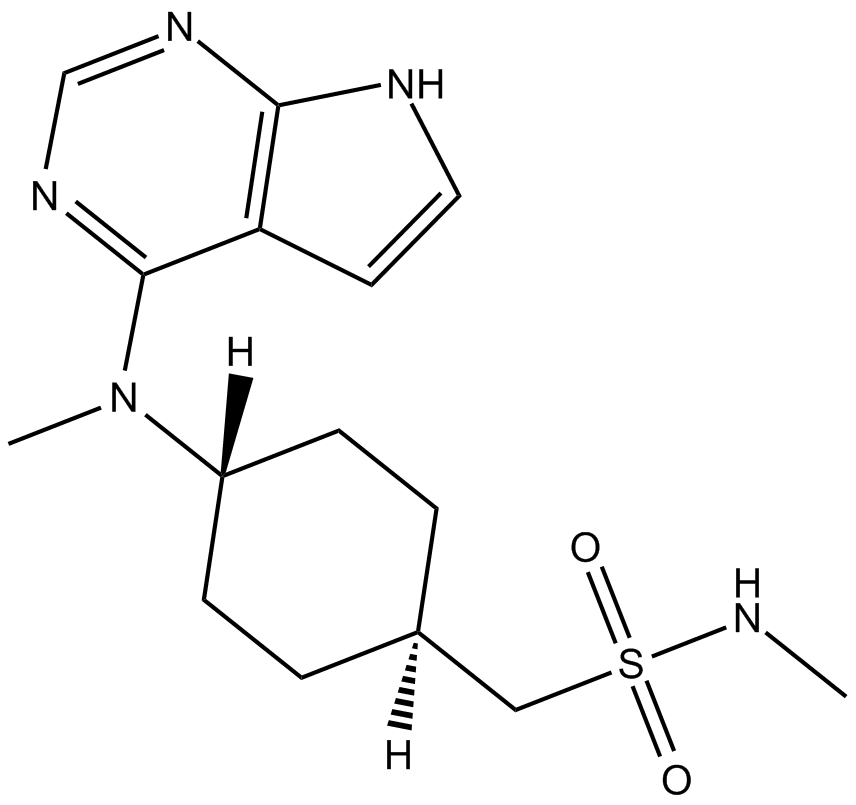
GC14938
PF-03394197(Oclacitinib)
Novel Janus kinase inhibitor
-
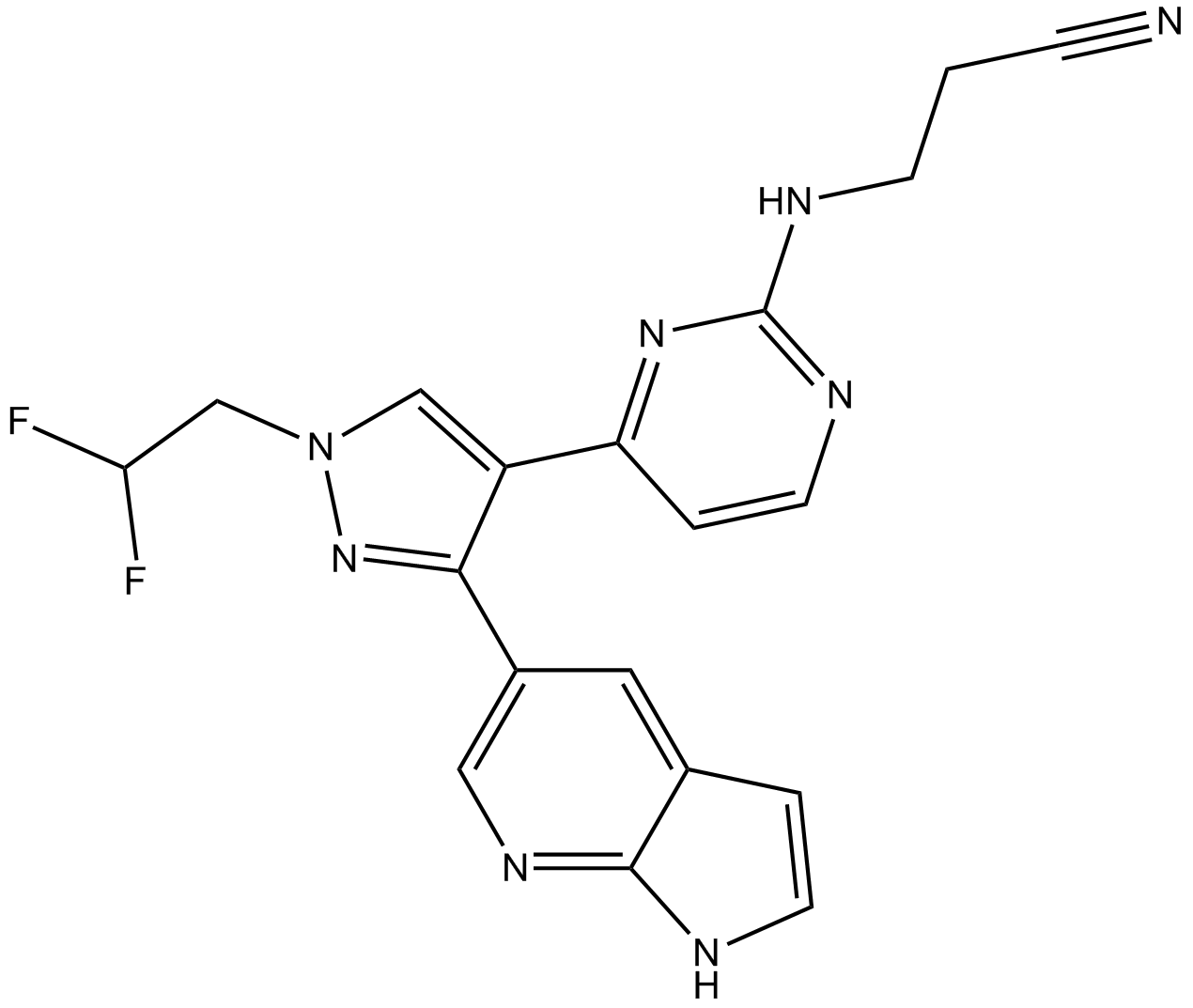
GC15821
PF-04880594
PF-04880594 ist ein potenter und selektiver RAF-Inhibitor. PF-04880594 hemmt sowohl Wildtyp- als auch Mutanten-BRAF und CRAF. PF-04880594 zeigt AntitumoraktivitÄt.
-
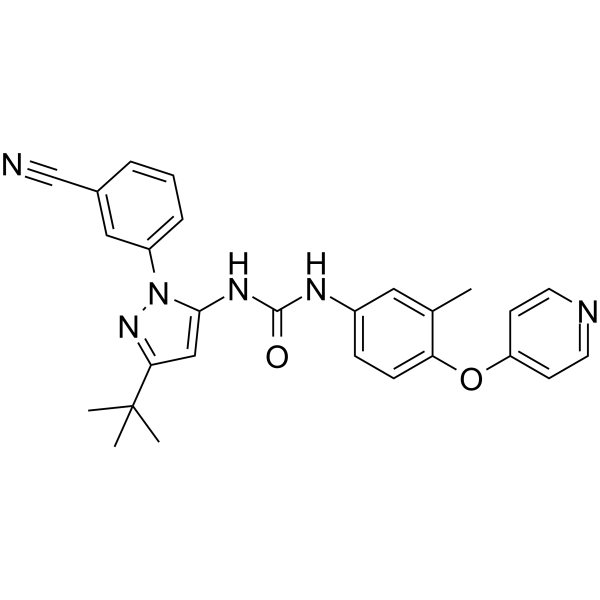
GC63144
PF-05381941
PF-05381941 ist ein potenter dualer Inhibitor von TAK1/p38α mit IC50-Werten von 156 bzw. 186 nM.
-
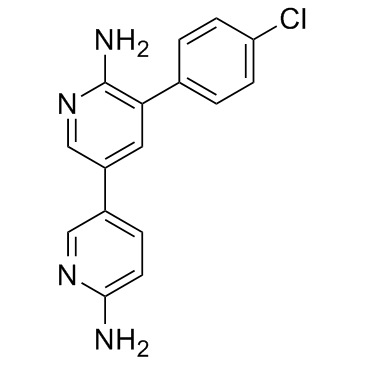
GC31675
PF-06260933
PF-06260933 ist ein oral aktiver und hochselektiver Inhibitor von MAP4K4 mit IC50-Werten von 3,7 und 160 nM fÜr Kinase bzw. Zelle.
-
GC14645
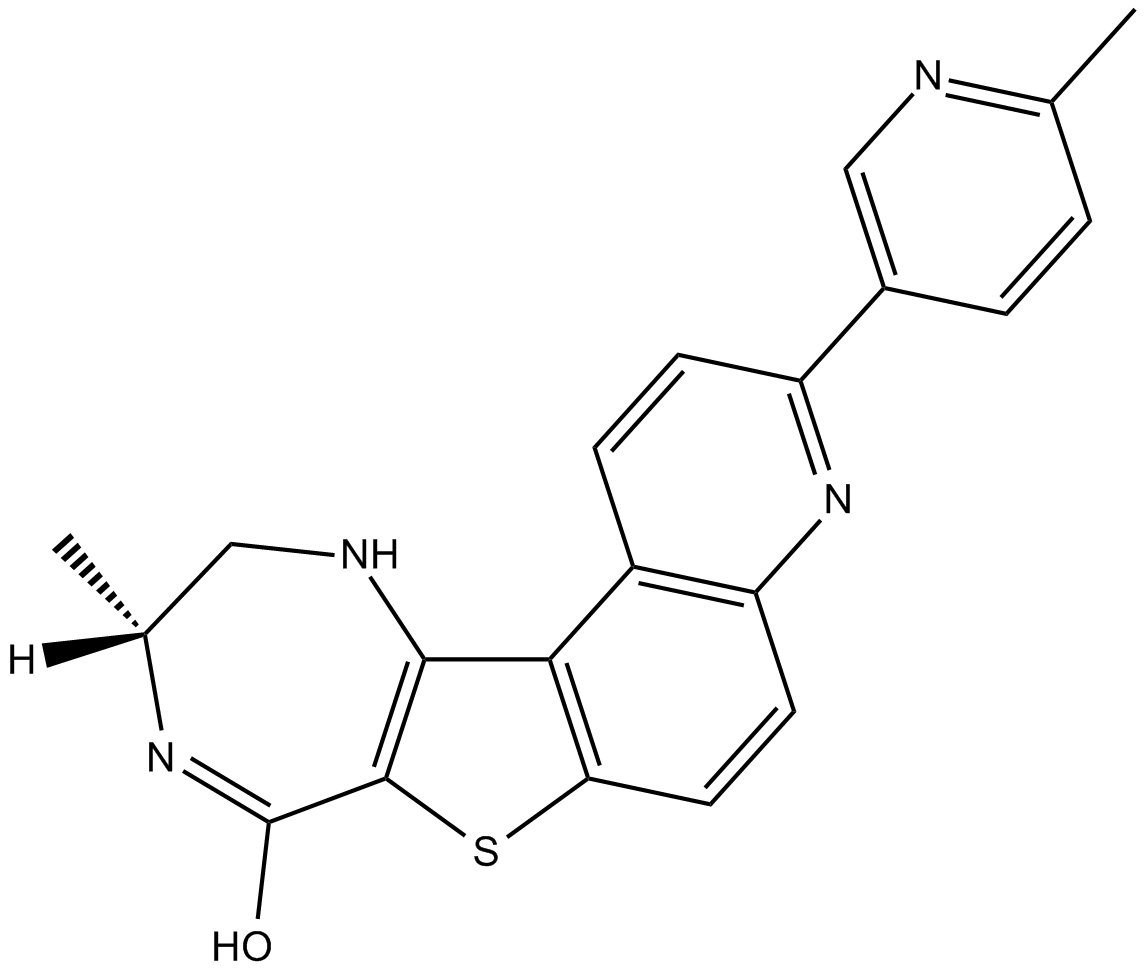
PF-3644022
PF-3644022 ist ein potenter, selektiver, oral aktiver und ATP-kompetitiver MAPKAPK2 (MK2)-Inhibitor mit einem IC50 von 5,2 nM und einem Ki von 3 nM.
-
GC10689
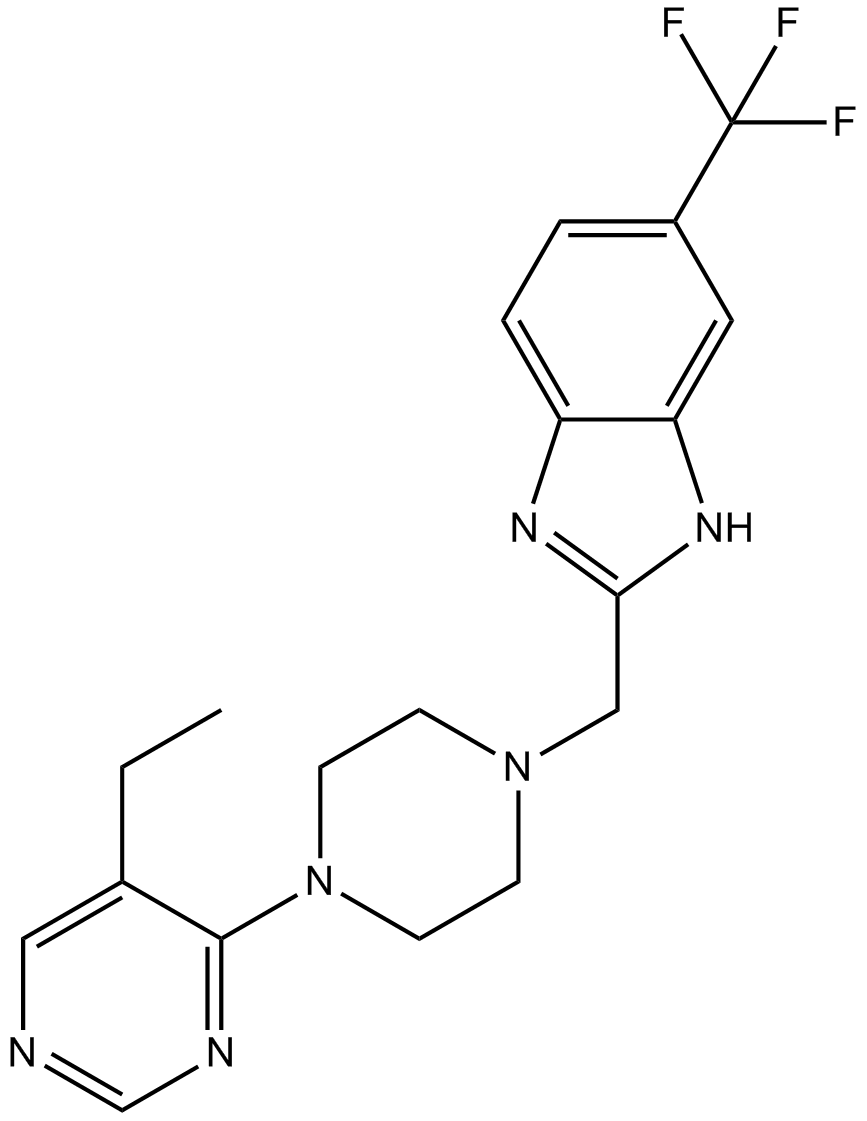
PF-4708671
PF-4708671 ist ein potenter zellgÄngiger S6K1-Inhibitor mit einem Ki von 20 nM und einem IC50 von 160 nM.
-
GC10261
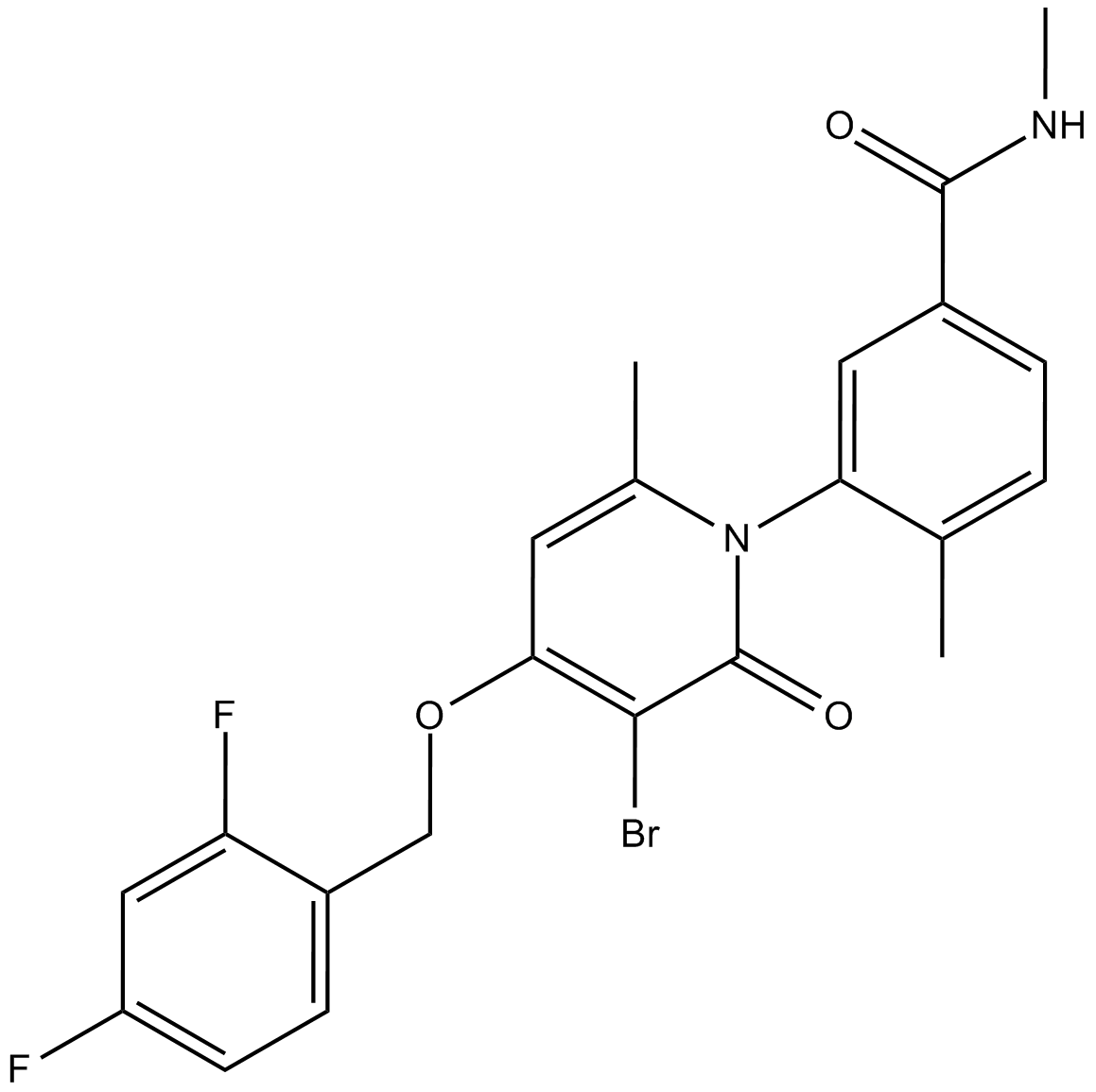
PH-797804
PH-797804 ist ein ATP-kompetitiver, selektiver p38α/p38β-Inhibitor (IC50=26 nM und Ki=5,8 nM fÜr p38α; Ki=40 nM fÜr p38β) und hemmt JNK2 nicht.
-
GC41635
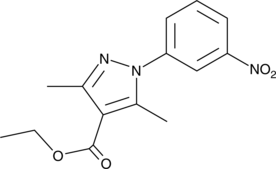
Phosphodiesterase 4 Inhibitor
Phosphodiesterase 4 (PDE4) inhibitor is an inhibitor of PDE4 with an IC50 value of 0.10 μM for the human recombinant enzyme.
-
GC18050
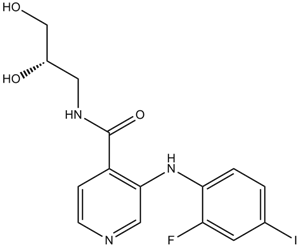
Pimasertib (AS-703026)
Pimasertib (AS-703026) (AS703026) ist ein hochselektiver, ATP-nicht-kompetitiver, allosterischer, oral verfÜgbarer MEK1/2-Inhibitor.
-
GC10282
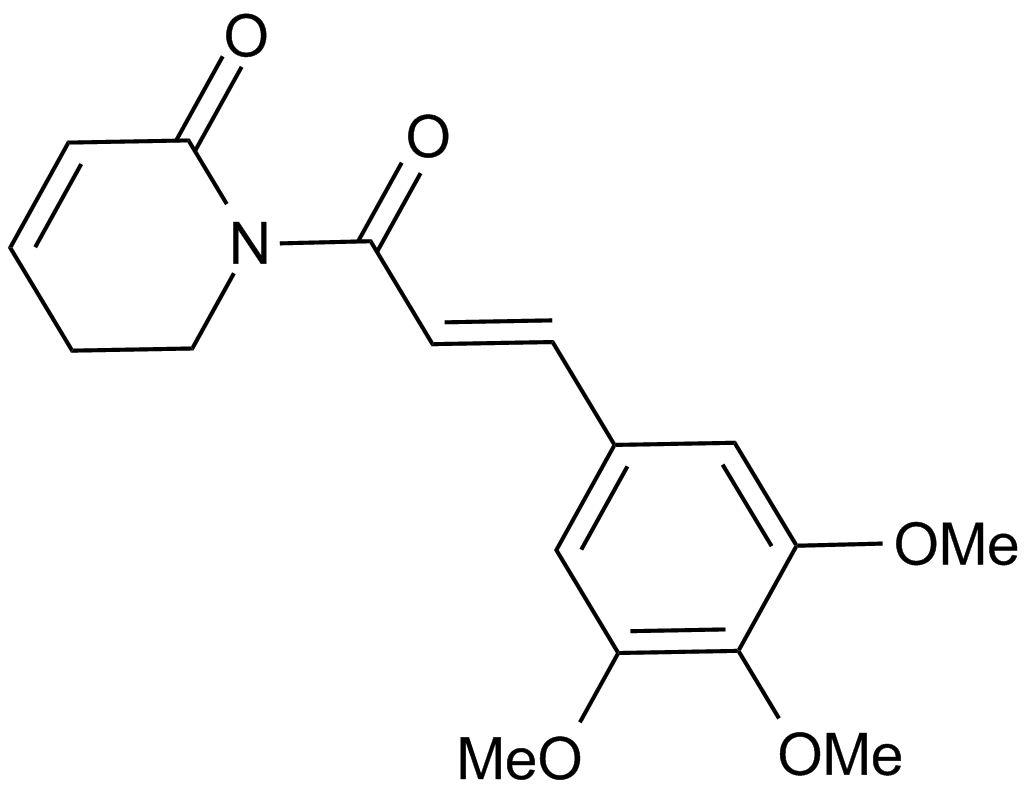
Piperlongumine
An alkaloid with anticancer and antioxidant activities
-
GC44653
PKA Inhibitor (5-24)
PKA-Inhibitor (5-24) ist ein potenter, kompetitiver und synthetischer Peptid-Inhibitor von PKA (cAMP-abhÄngige Proteinkinase) mit einem Ki von 2,3 nM.

-
GC48329
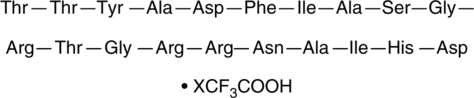
PKA Inhibitor (5-24) (trifluoroacetate salt)
A synthetic peptide inhibitor of PKA
-
GP10075
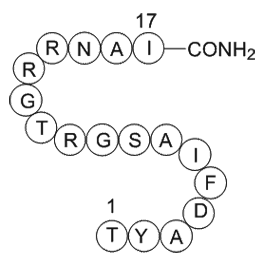
PKA inhibitor fragment (6-22) amide
A synthetic peptide inhibitor of PKA
-
GC61512
PKA Inhibitor Fragment (6-22) amide TFA
PKA-Inhibitorfragment (6-22) Amid TFA ist ein Inhibitor der cAMP-abhÄngigen Proteinkinase A (PKA) mit einem Ki von 2,8 nM.

-
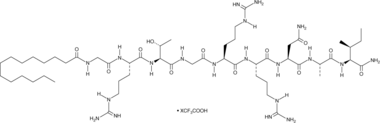
GC49265
PKI (14-22) amide (myristoylated) (trifluoroacetate salt)
A PKA inhibitor
-
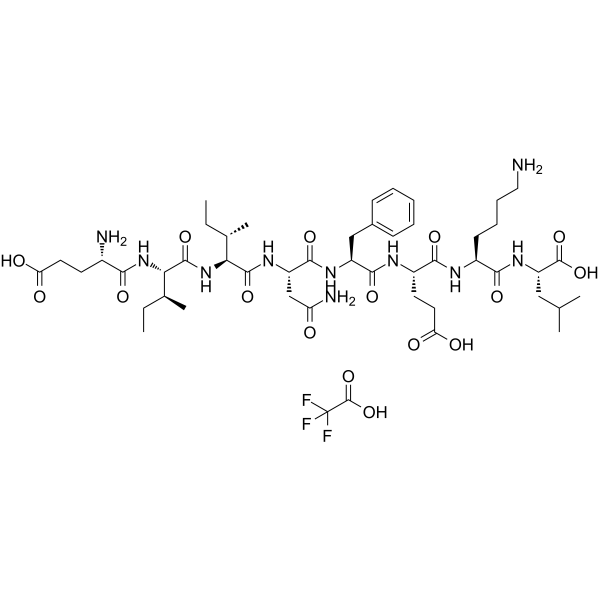
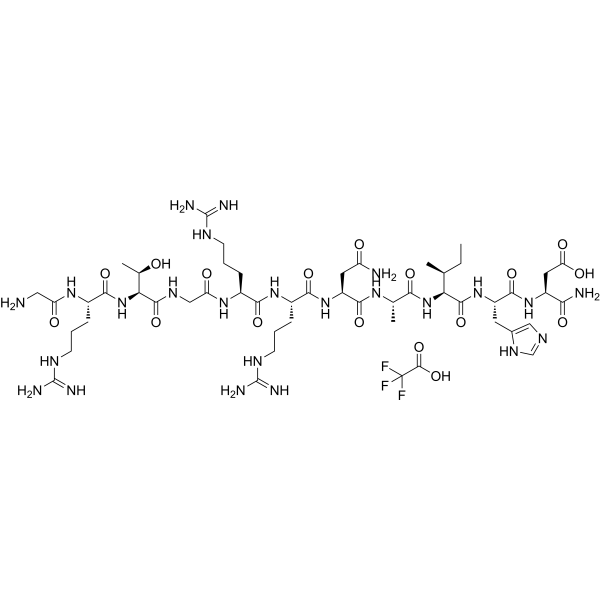
GC69715
PKI (14-24)amide TFA
PKI (14-24)amid TFA ist ein wirksamer PKA-Inhibitor. PKI (14-24)amid hemmt die Aktivität der zyklischen AMP-abhängigen Proteinkinase in Zellhomogenaten stark.
-
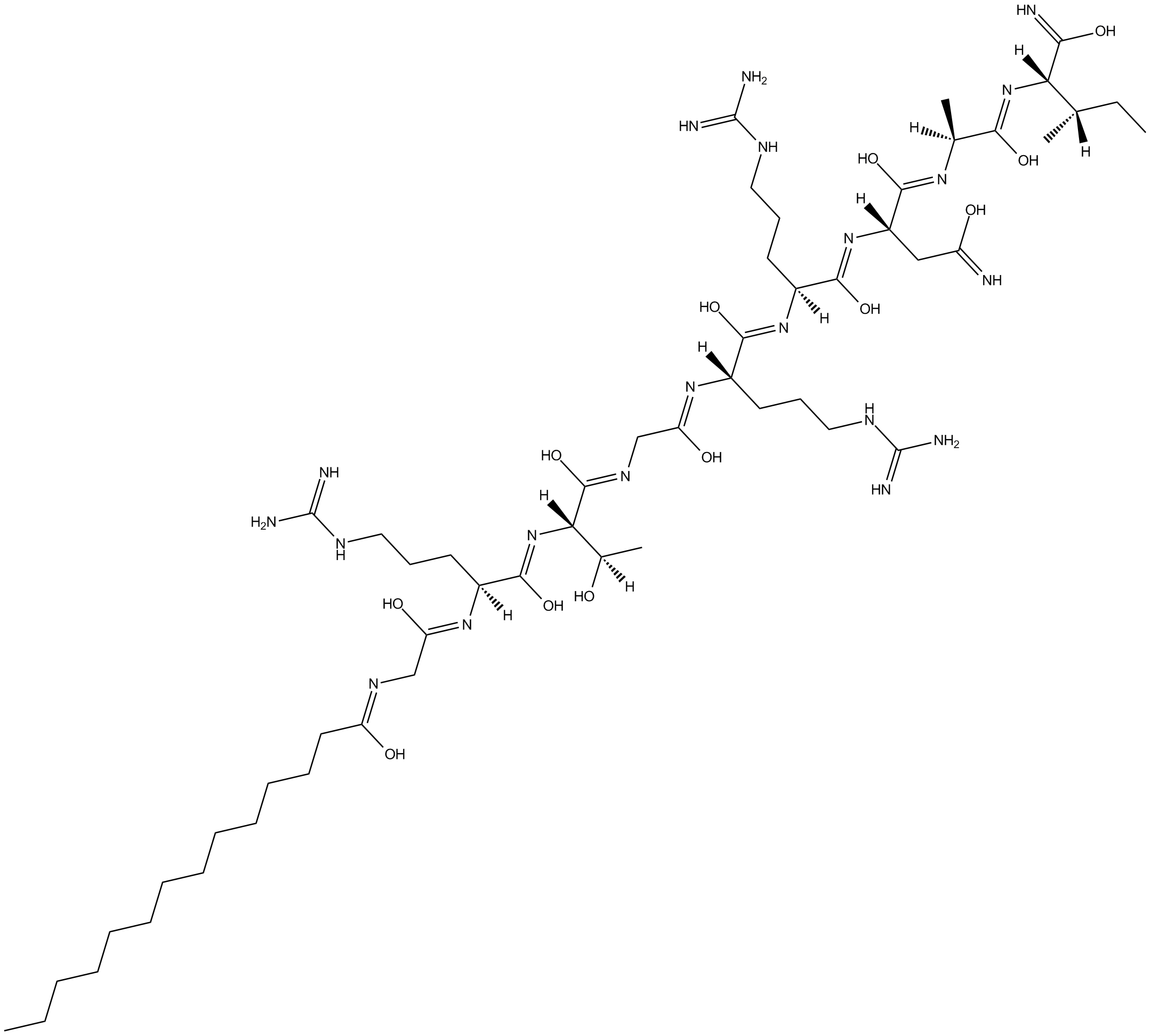
GC12321
PKI 14-22 amide, myristoylated
PKI 14-22 Amid, myristoyliert, ist ein potenter cAMP-abhängiger PKA-Inhibitor. PKI 14-22-Amid, myristoyliert reduziert die IgG-vermittelte phagozytische Reaktion und hemmt auch die Adhäsion von Neutrophilen.
-
GC61849
PKI 14-22 amide,myristoylated TFA
PKI 14-22 Amid, myristoyliertes TFA ist ein potenter cAMP-abhÄngiger PKA-Inhibitor. PKI 14-22 Amid, myristoyliertes TFA reduziert die IgG-vermittelte phagozytische Antwort und hemmt auch die AdhÄsion von Neutrophilen.

-
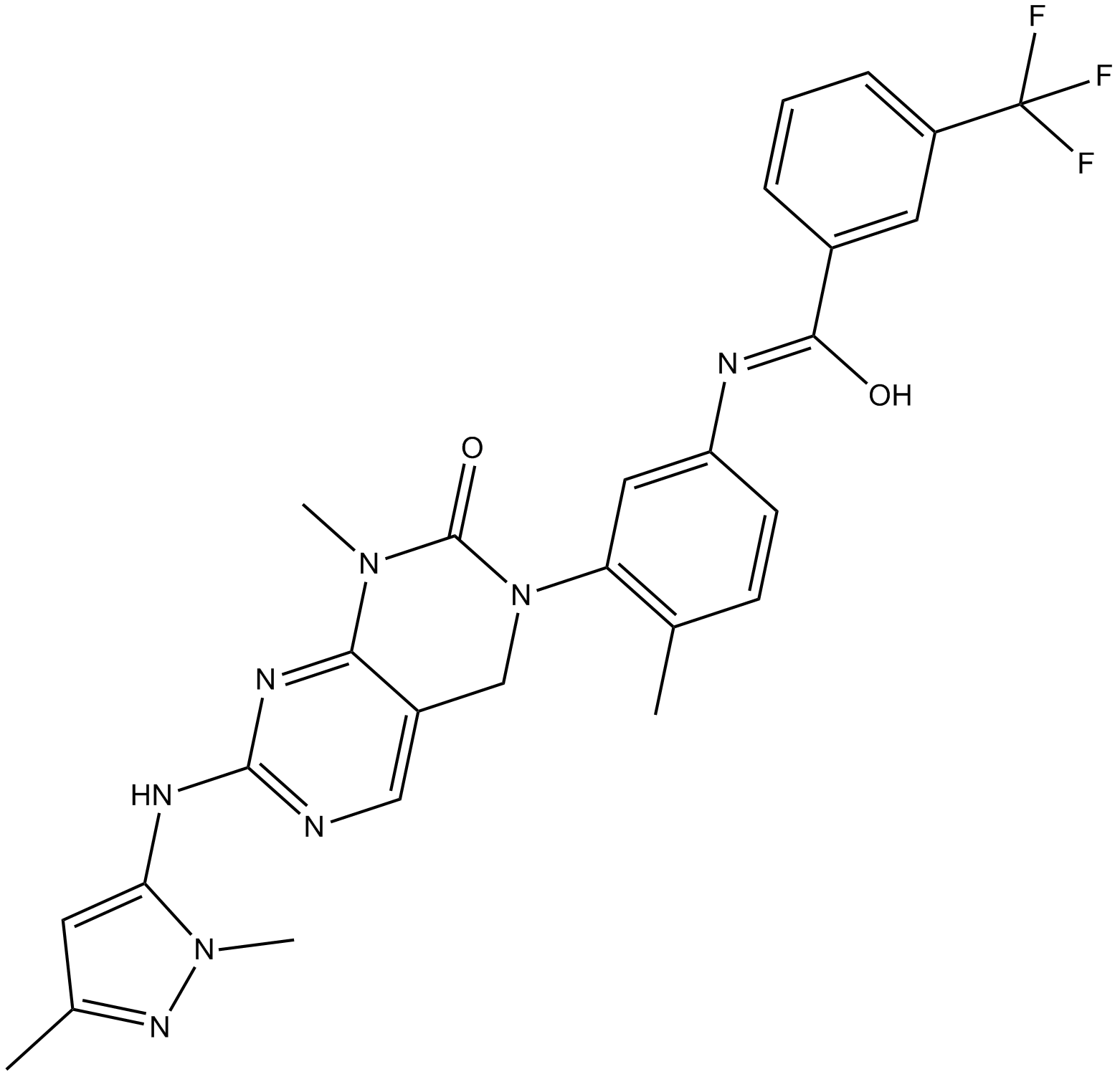
GC17407
Pluripotin
Pluripotin ist ein dualer Inhibitor von ERK1 und RasGAP mit KDs von 98 nM bzw. 212 nM. Pluripotin hemmt auch RSK1, RSK2, RSK3 und RSK4 mit IC50-Werten von 0,5, 2,5, 3,3 bzw. 10,0 μM.
-
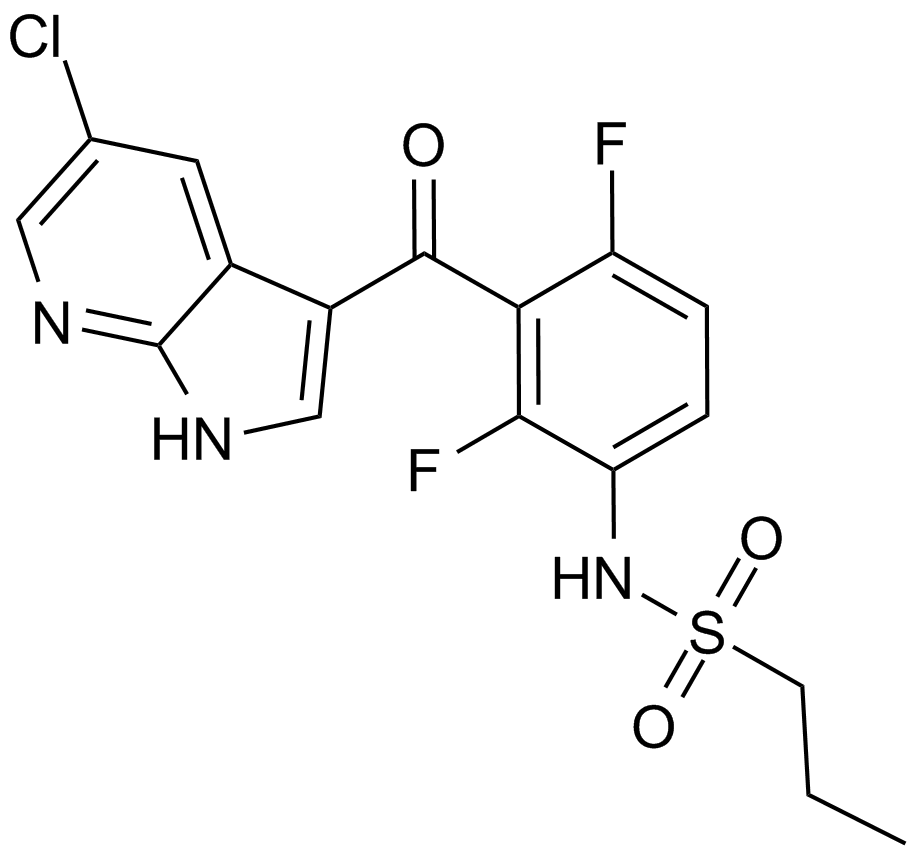
GC14732
PLX-4720
An orally-available inhibitor of the B-raf mutant B-RafV600E
-
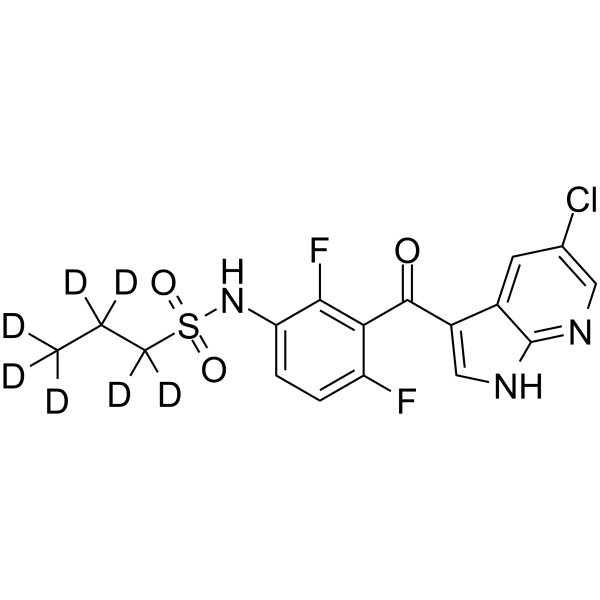
GC62206
PLX-4720-d7
PLX-4720-d7 ist das Deuterium mit der Bezeichnung PLX-4720. PLX-4720 ist ein potenter und selektiver Inhibitor von B-RafV600E mit einem IC50-Wert von 13 nM in einem zellfreien Assay, ebenso potent wie c-Raf-1 (Y340D- und Y341D-Mutationen) und einer 10-fachen SelektivitÄt fÜr B-RafV600E als Wildtyp B-Raf.
-
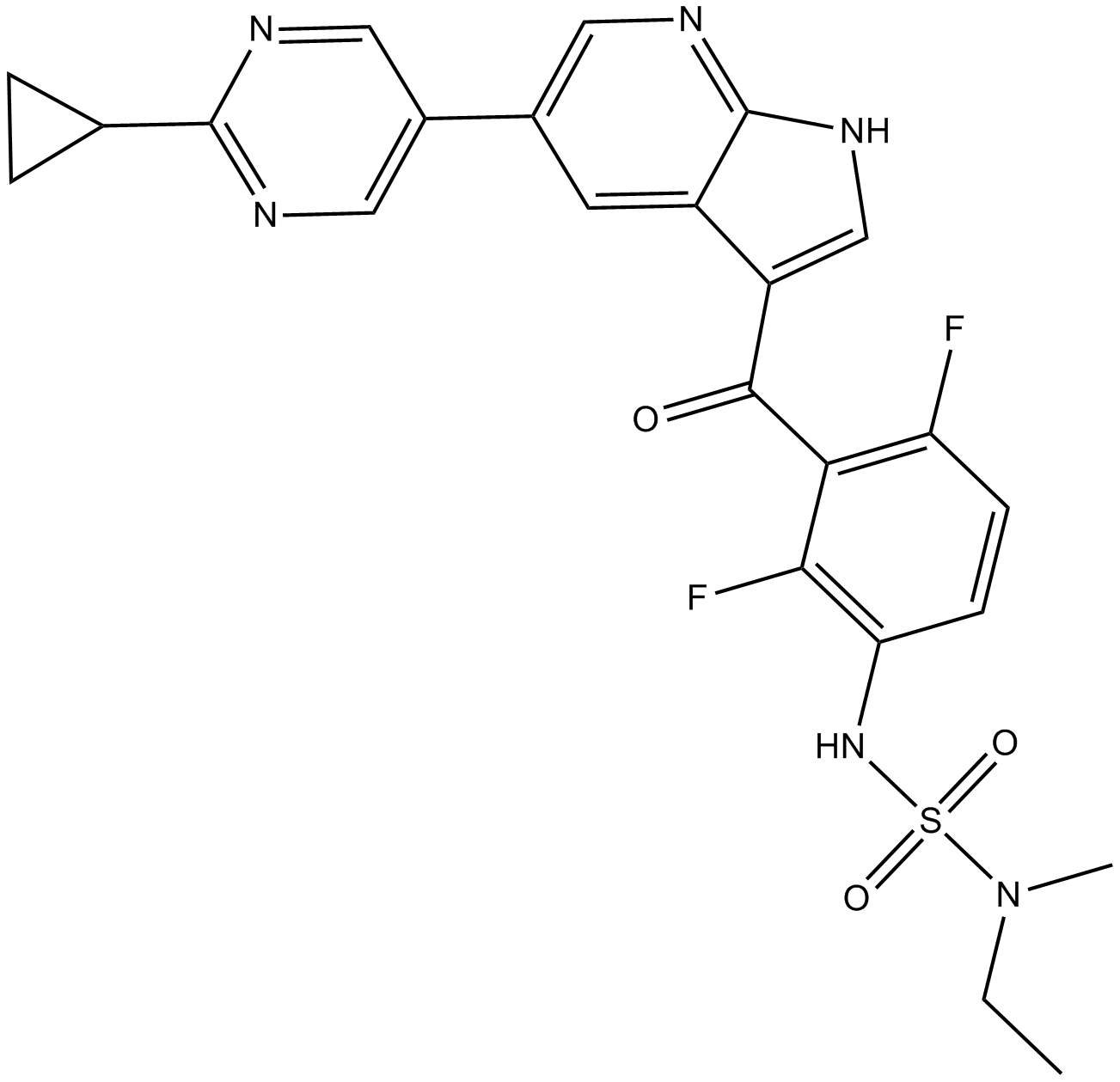
GC10324
PLX7904
PLX7904 ist ein potenter und selektiver BRAF-Inhibitor mit einem IC50-Wert von ca. 5 nM gegen BRAFV600E in mutierten RAS-exprimierenden Zellen.
-
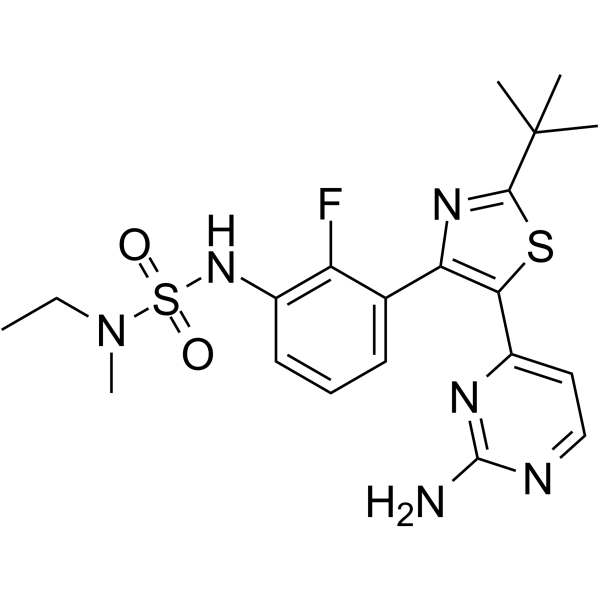
GC64828
PLX7922
PLX7922, ein RAF-Inhibitor, kann an BRAFV600E binden. PLX7922 hemmt pERK in BRAFV600E-Zelllinien und aktiviert pERK in mutierten NRAS-Zelllinien.
-
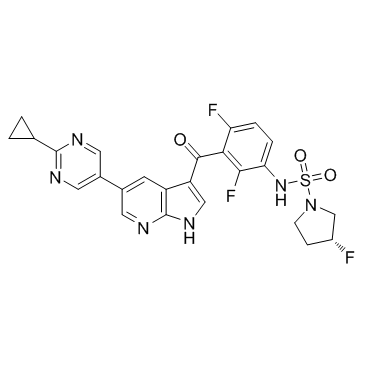
GC32686
PLX8394
-
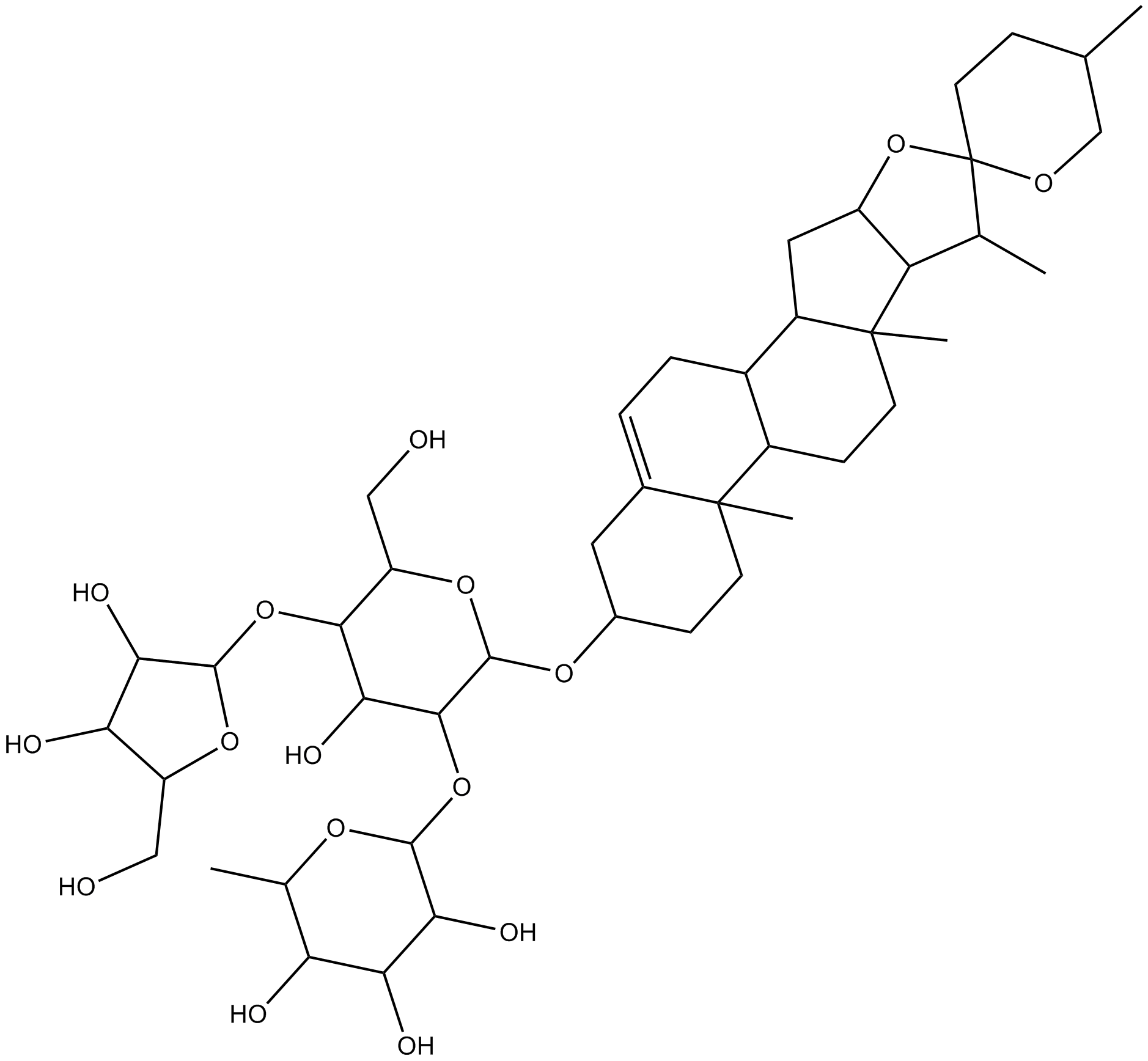
GN10709
Polyphyllin A
-
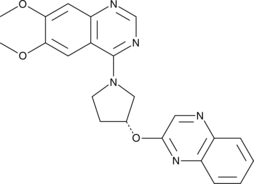
GC44673
PQ-10
PQ-10 ist ein potenter Inhibitor der Phosphodiesterase 10A (PDE10A) mit IC50 und ED50 von 4,6 nM bzw. 13 mg/kg.
-
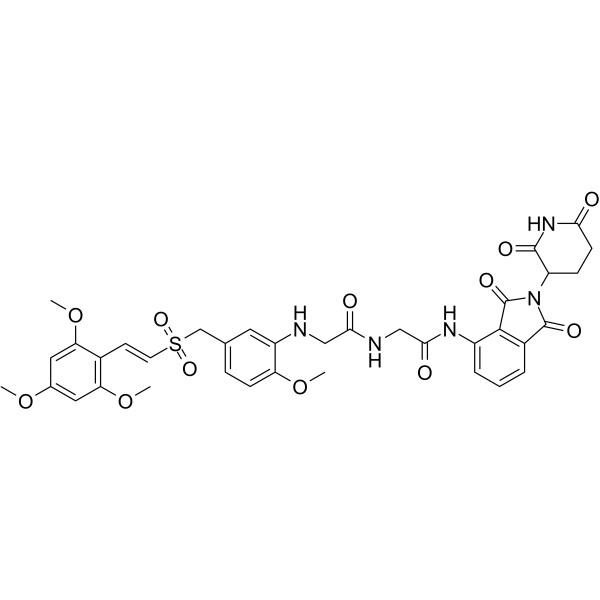
GC65479
PROTAC B-Raf degrader 1
PROTAC B-Raf-Degrader 1 (Verbindung 2) ist eine Proteolyse-Targeting-ChimÄre (PROTAC) fÜr den Abbau von B-Raf auf der Basis von Cereblon-Liganden mit Anti-Krebs-AktivitÄt.
-
GN10814
Quercitrin
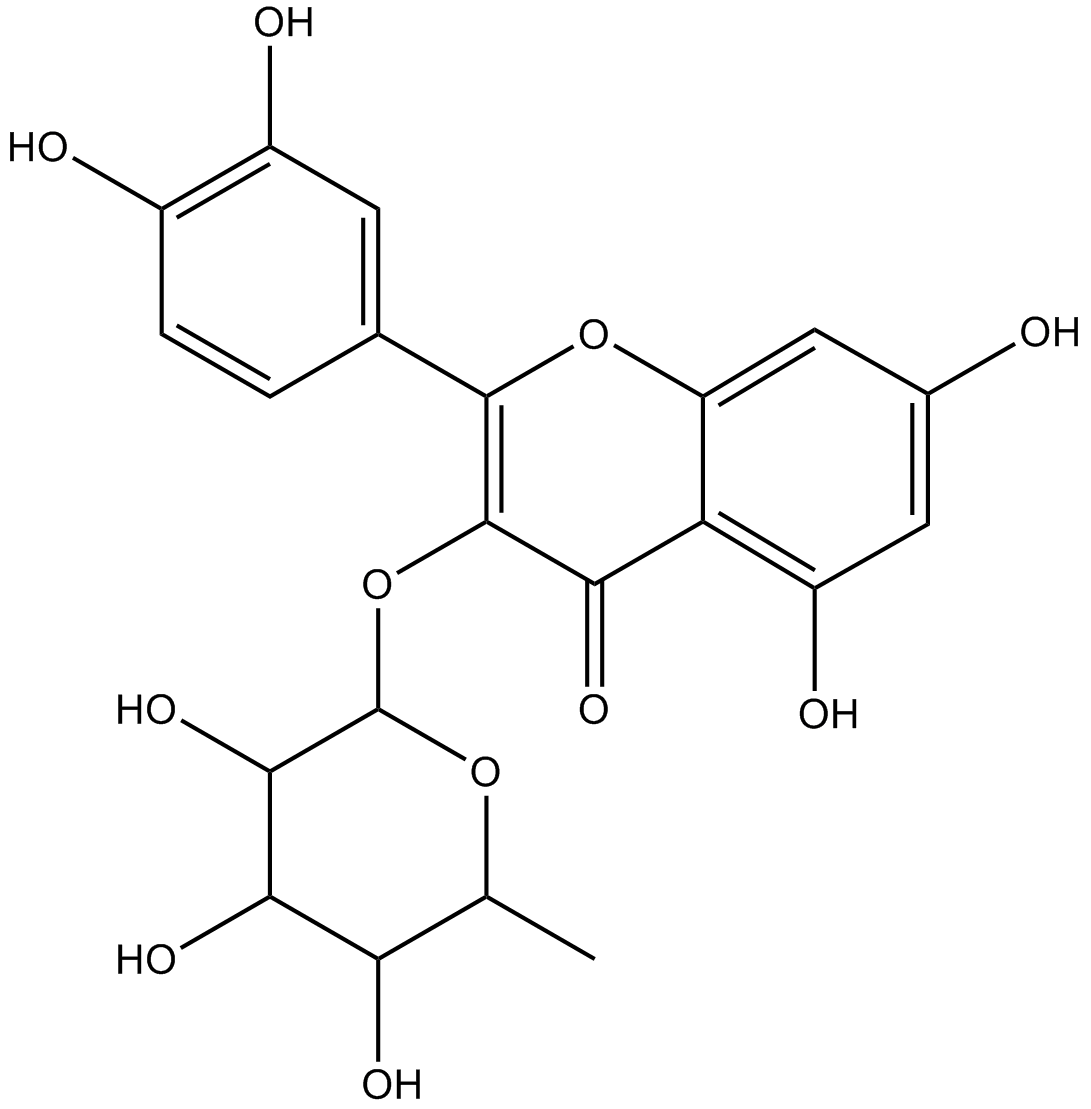
-
GC19304
R1487 Hydrochloride
R1487-Hydrochlorid ist ein hochwirksamer und selektiver p38α-Inhibitor mit Kd-Werten von 0,2 nM und 29 nM fÜr p38α bzw. p38β.
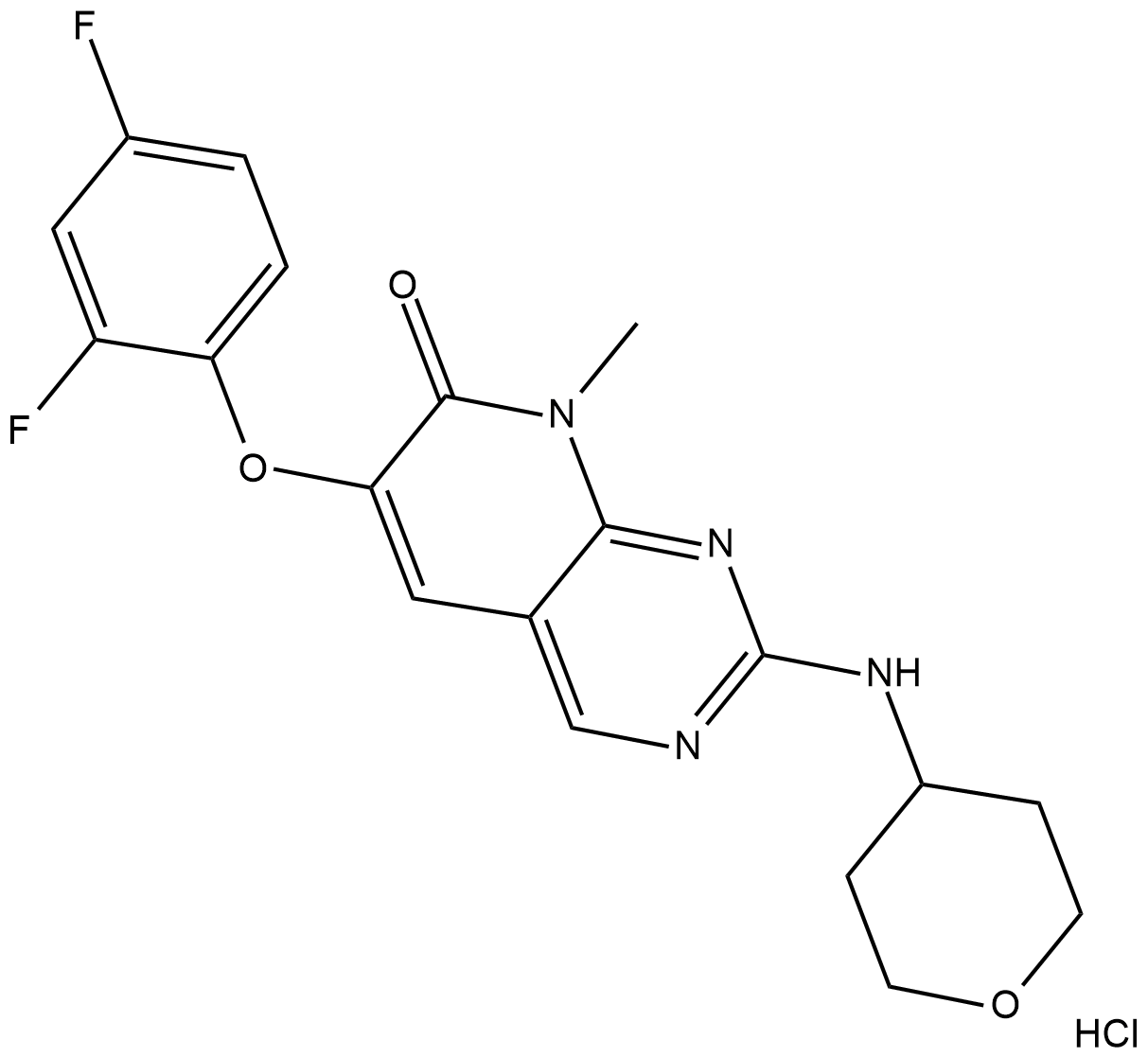
-
GC61485
Raf inhibitor 2
Raf-Inhibitor 2 ist ein potenter Raf-Kinase (IC50 < 1,0 μM)-Inhibitor, Verbindung 32, extrahiert aus dem Patent EP1003721B1. Der Raf-Inhibitor 2 kann fÜr die Krebsforschung eingesetzt werden.
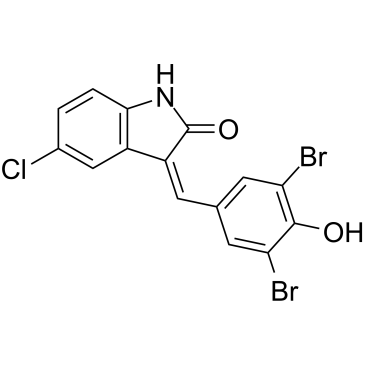
-
GC37068
RAF mutant-IN-1
Die RAF-Mutante-IN-1 ist ein RAF-Kinase-Inhibitor, extrahiert aus dem Patent WO2019107987A1, mit IC50-Werten von 21 nM, 30 nM und 392 nM fÜr C-RAF 340D/Y341D, B-RAFV600E bzw. B-RAFWT.
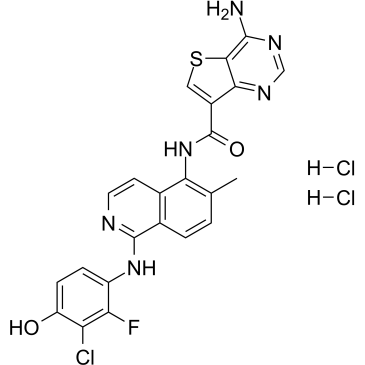
-
GC15818
RAF265
RAF265 ist ein potenter RAF/VEGFR2-Inhibitor.
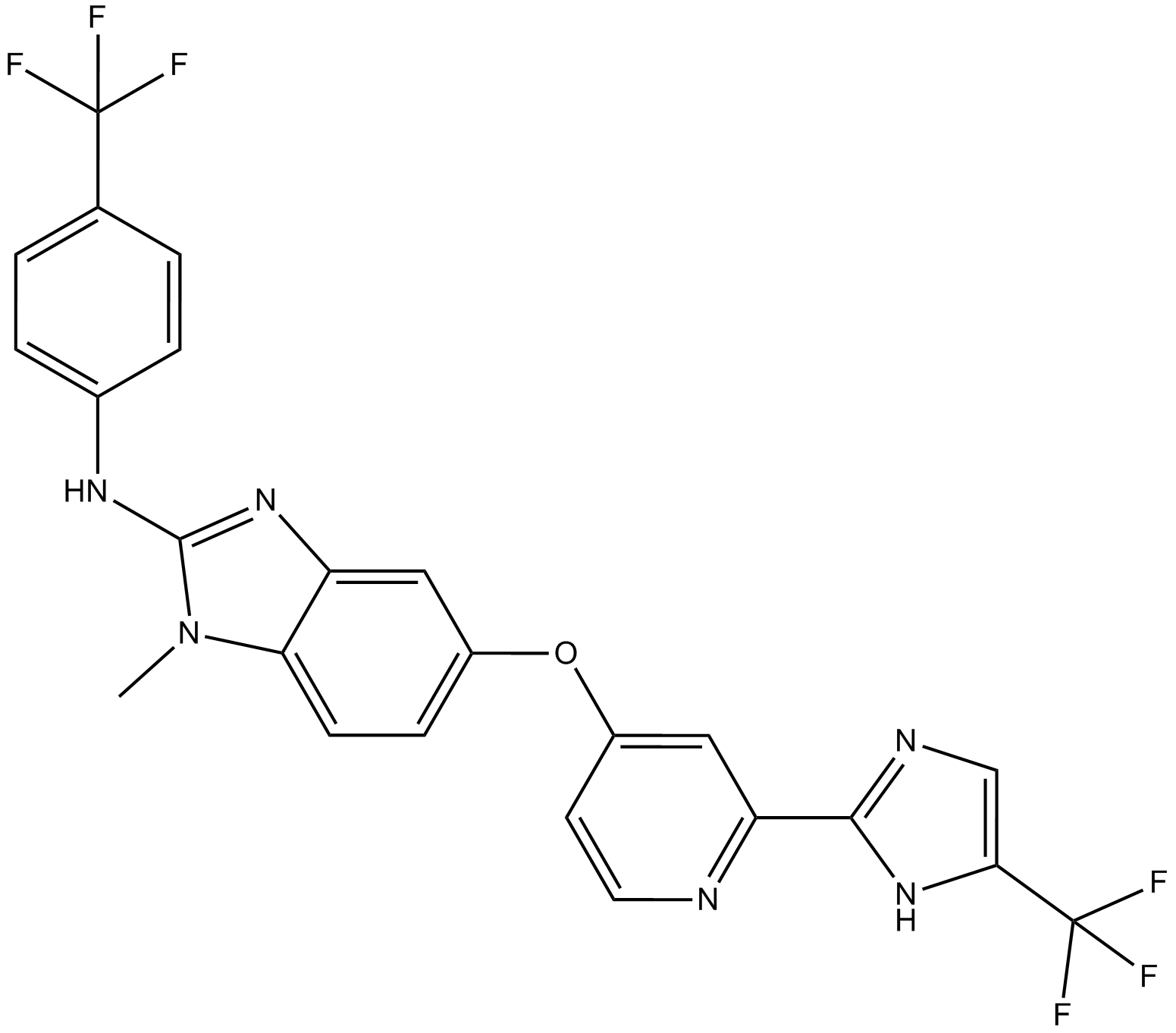
-
GC19307
RAF709
RAF709 ist ein potenter, selektiver und wirksamer RAF-Inhibitor mit IC50-Werten von 0,4 nM bzw. 0,5 nM fÜr BRAF und CRAF. Antitumorwirksamkeit.
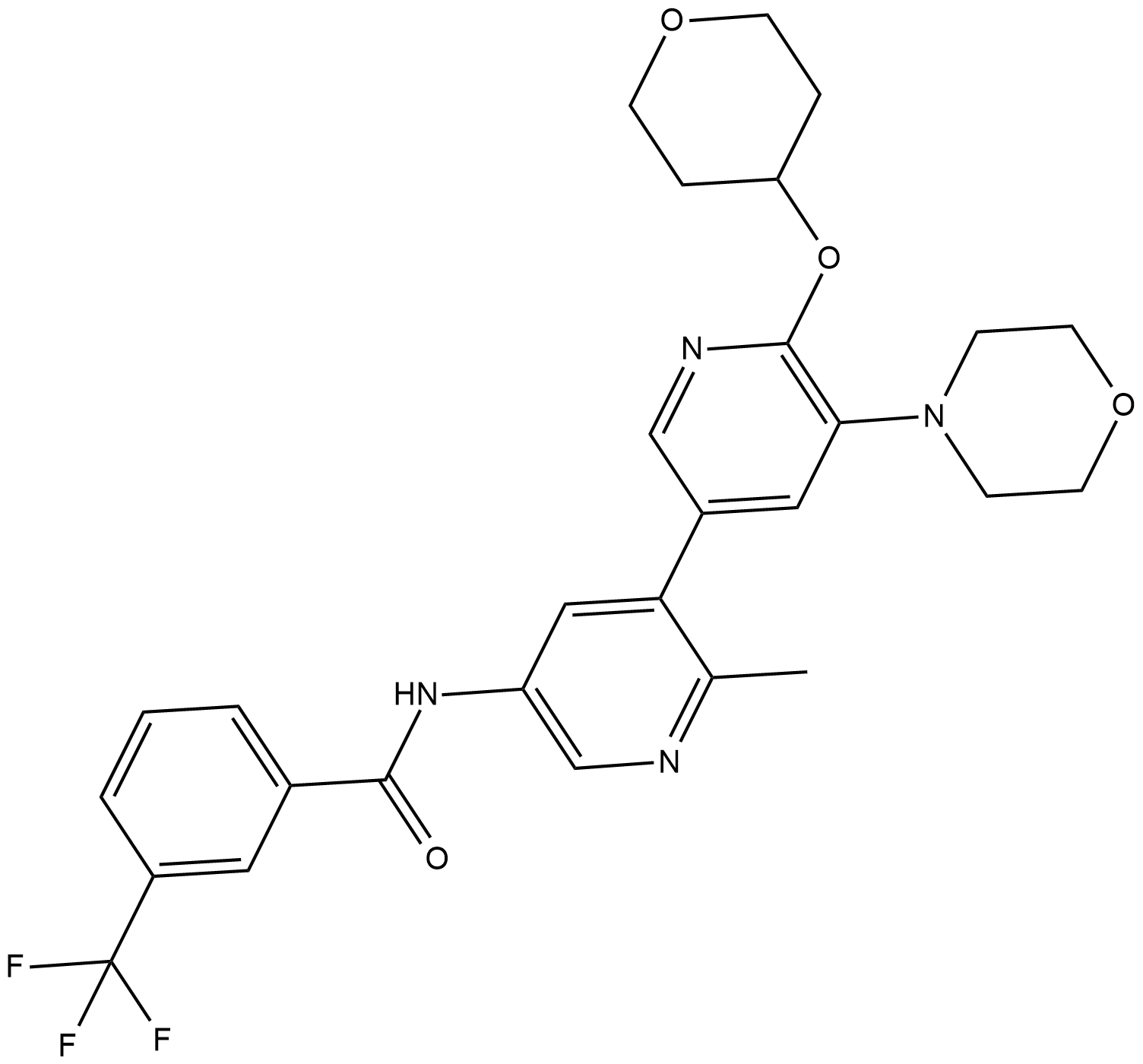
-
GC62504
RAS/RAS-RAF-IN-1
RAS/RAS-RAF-IN-1 ist ein potenter RAS- und RAS-RAF-Inhibitor. RAS/RAS-RAF-IN-1 hat eine KD von 5,0 μM-15 μM fÜr die BindungsaffinitÄt von Cyclophilin A (CYPA). RAS/RAS-RAF-IN-1 hat AntitumoraktivitÄt.
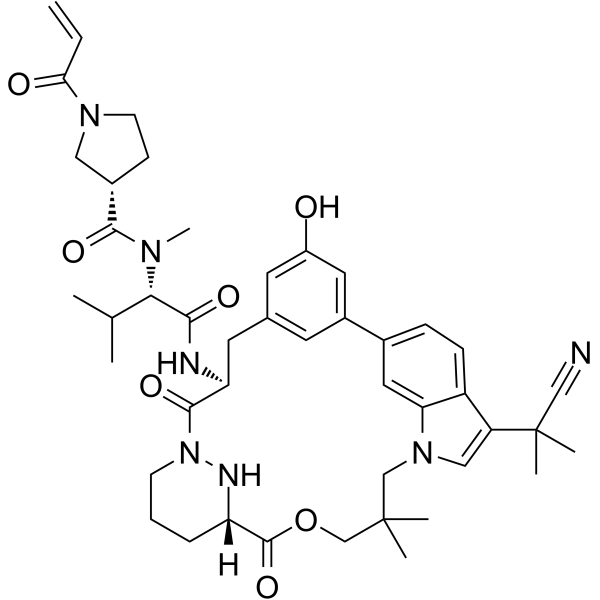
-
GC37071
Ravoxertinib hydrochloride
Ravoxertinib-Hydrochlorid (GDC-0994-Hydrochlorid) ist ein oral bioverfÜgbarer Inhibitor, der selektiv fÜr die ERK-Kinase-AktivitÄt mit IC50 von 6,1 nM und 3,1 nM fÜr ERK1 bzw. ERK2 ist.
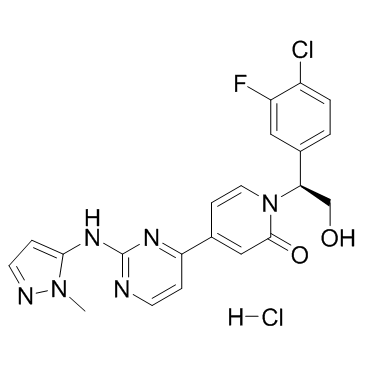
-
GC16872
Refametinib
Refametinib (BAY 869766; RDEA119) ist ein oral verfÜgbarer, potenter, nicht ATP-kompetitiver, selektiver, allosterischer MEK1/MEK2-Inhibitor mit IC50-Werten von 19 nM bzw. 47 nM.
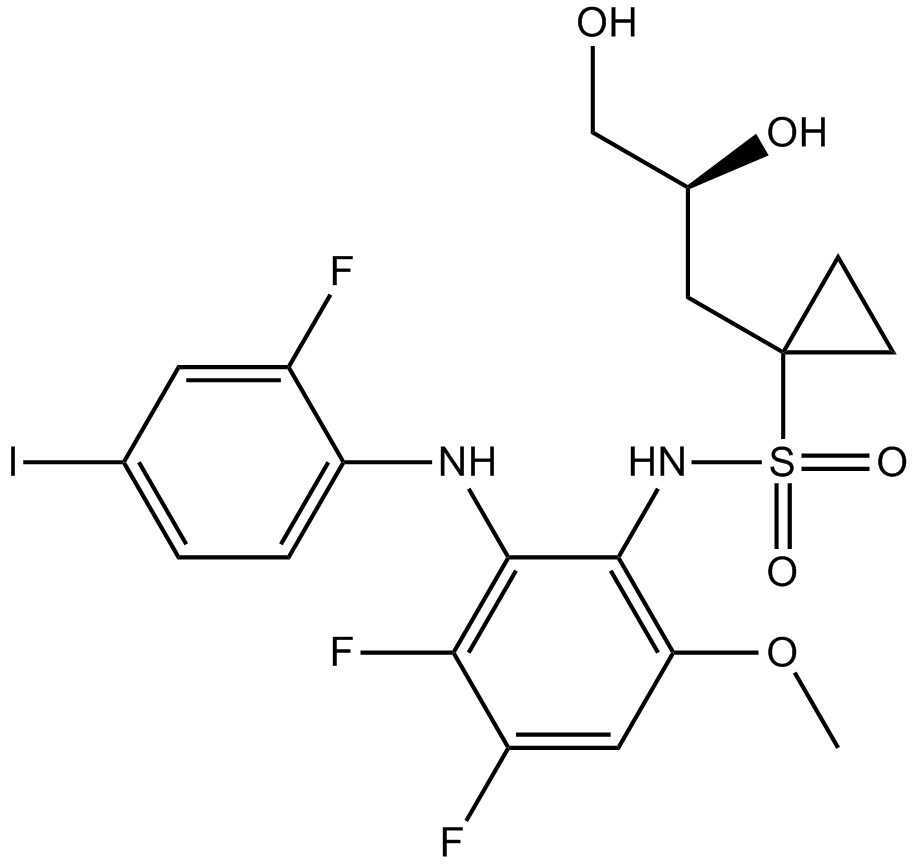
-
GC37516
Refametinib R enantiomer
Refametinib-R-Enantiomer ist ein MEK-Inhibitor, extrahiert aus Patent WO2007014011A2, Verbindung 1022, hat einen EC50-Wert von 2,0–15 nM.
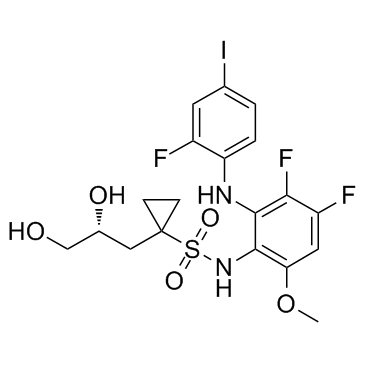
-
GC14606
Regorafenib hydrochloride
A multi-kinase inhibitor
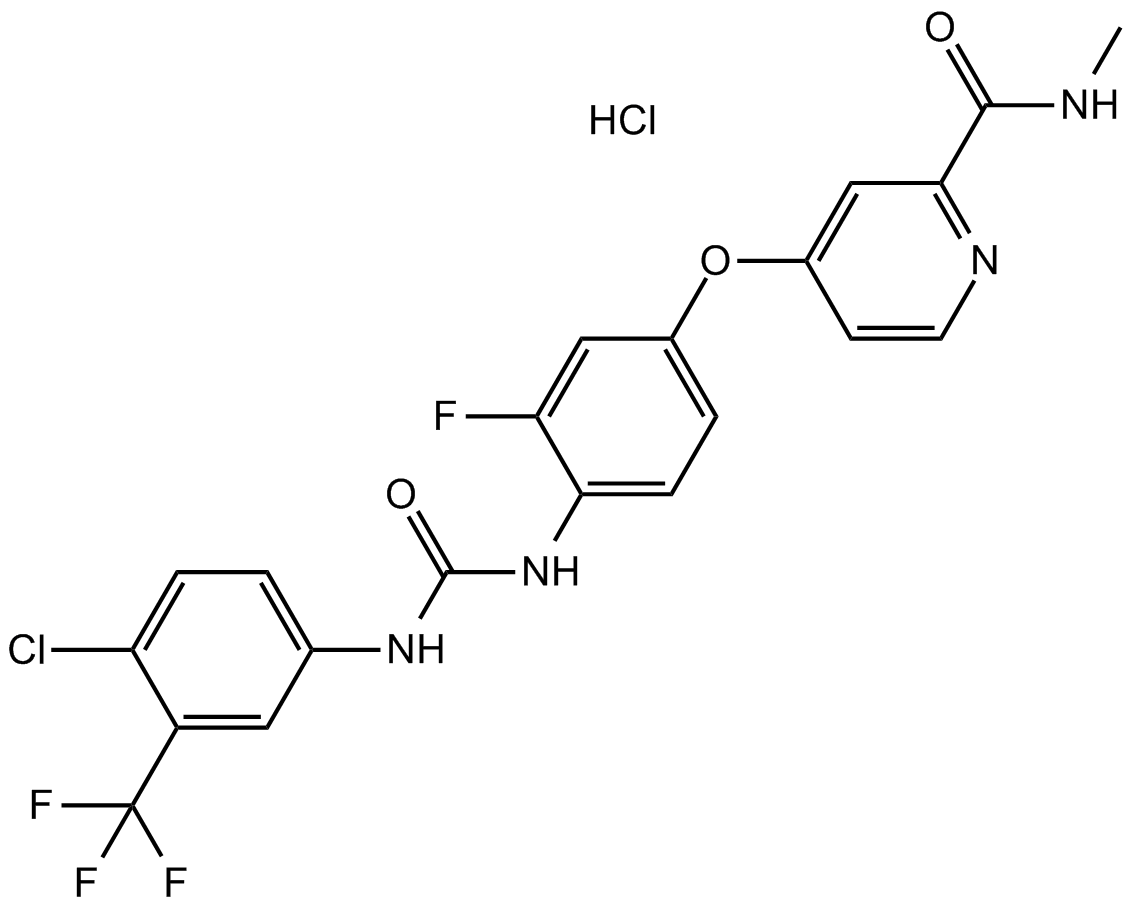
-
GC14534
Regorafenib monohydrate
A multi-kinase inhibitor
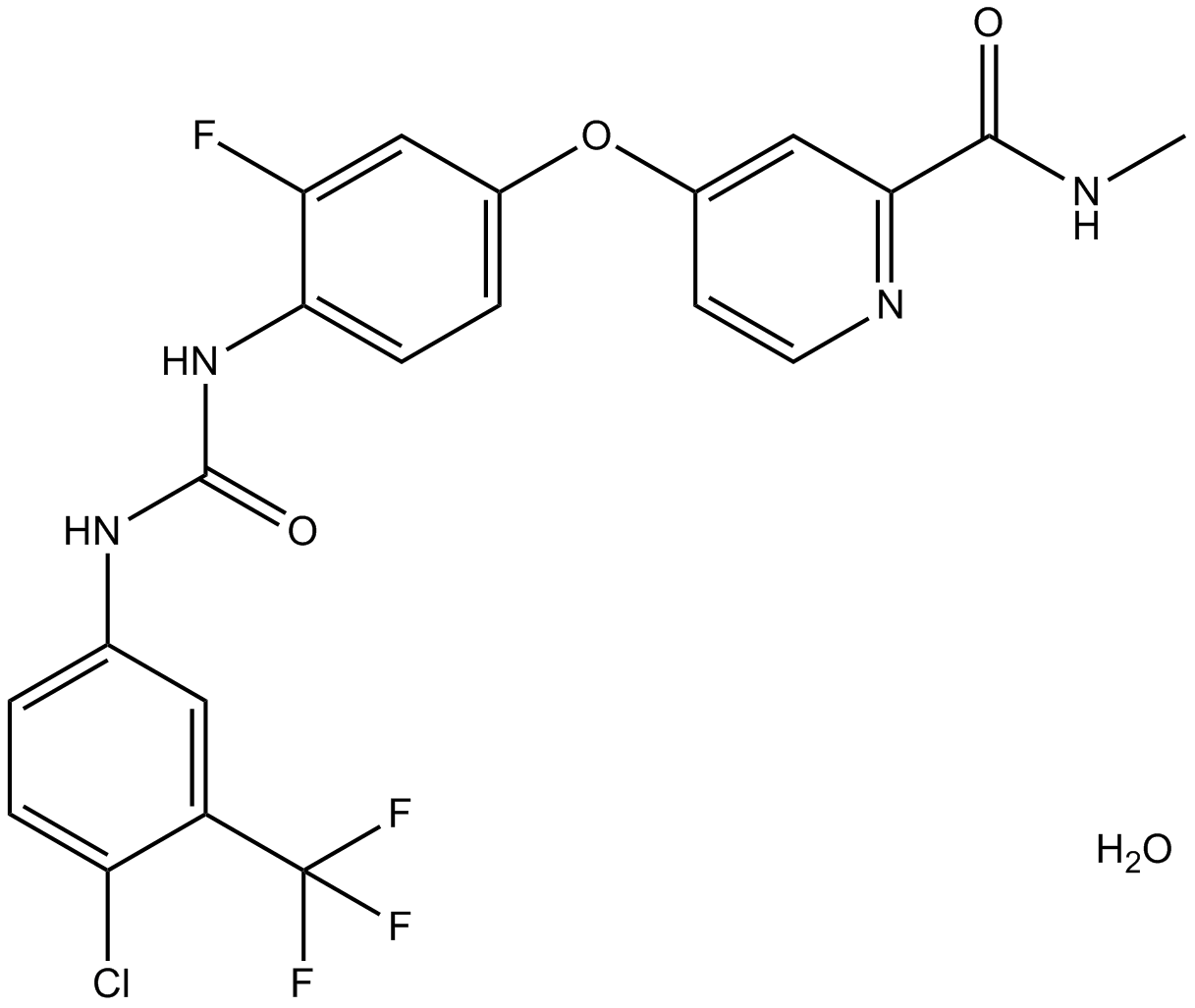
-
GC40213
Regorafenib-13C-d3
Regorafenib-13C-d3 is intended for use as an internal standard for the quantification of regorafenib by GC- or LC-MS.
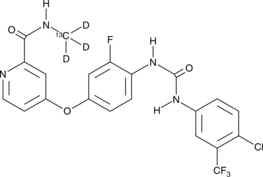
-
GC18751
Reticulol
Reticulol is an isocoumarin derivative produced by certain species of Streptomyces that inhibits cAMP phosphodiesterase (IC50 = 41 uM).
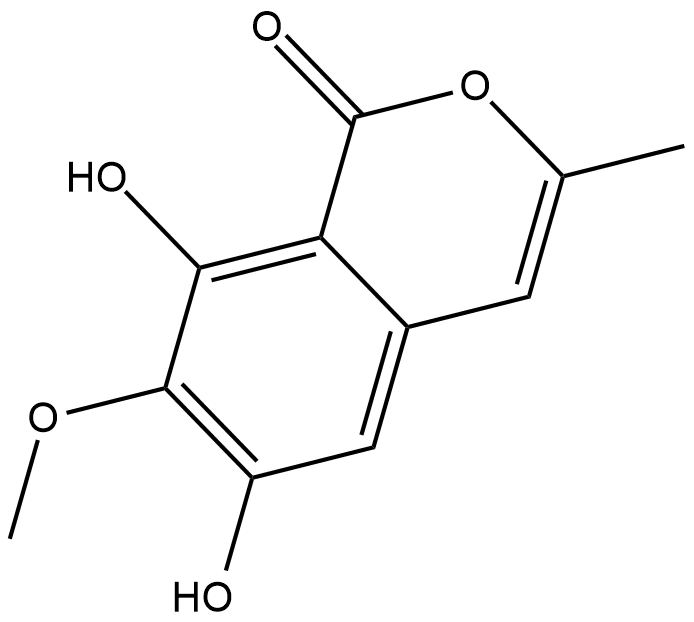
-
GC37522
RGB-286638
RGB-286638 ist ein CDK-Inhibitor, der die KinaseaktivitÄt von Cyclin T1-CDK9, Cyclin B1-CDK1, Cyclin E-CDK2, Cyclin D1-CDK4, Cyclin E-CDK3 und p35-CDK5 mit IC50-Werten von 1, 2, 3 hemmt , 4, 5 bzw. 5 nM; hemmt auch GSK-3β, TAK1, Jak2 und MEK1 mit IC50-Werten von 3, 5, 50 und 54 nM.
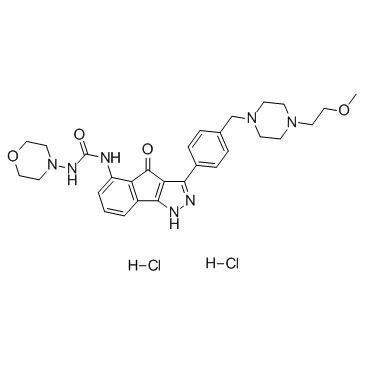
-
GC37523
RGB-286638 free base
RGB-286638 ist ein CDK-Inhibitor, der die KinaseaktivitÄt von Cyclin T1-CDK9, Cyclin B1-CDK1, Cyclin E-CDK2, Cyclin D1-CDK4, Cyclin E-CDK3 und p35-CDK5 mit IC50-Werten von 1, 2, 3 hemmt , 4, 5 bzw. 5 nM; hemmt auch GSK-3β, TAK1, Jak2 und MEK1 mit IC50-Werten von 3, 5, 50 und 54 nM.
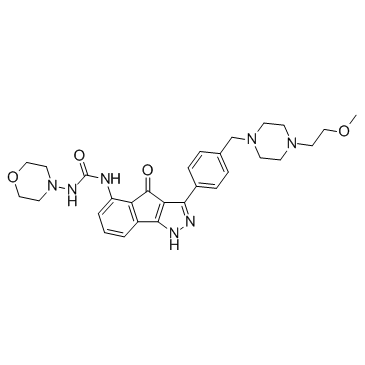
-
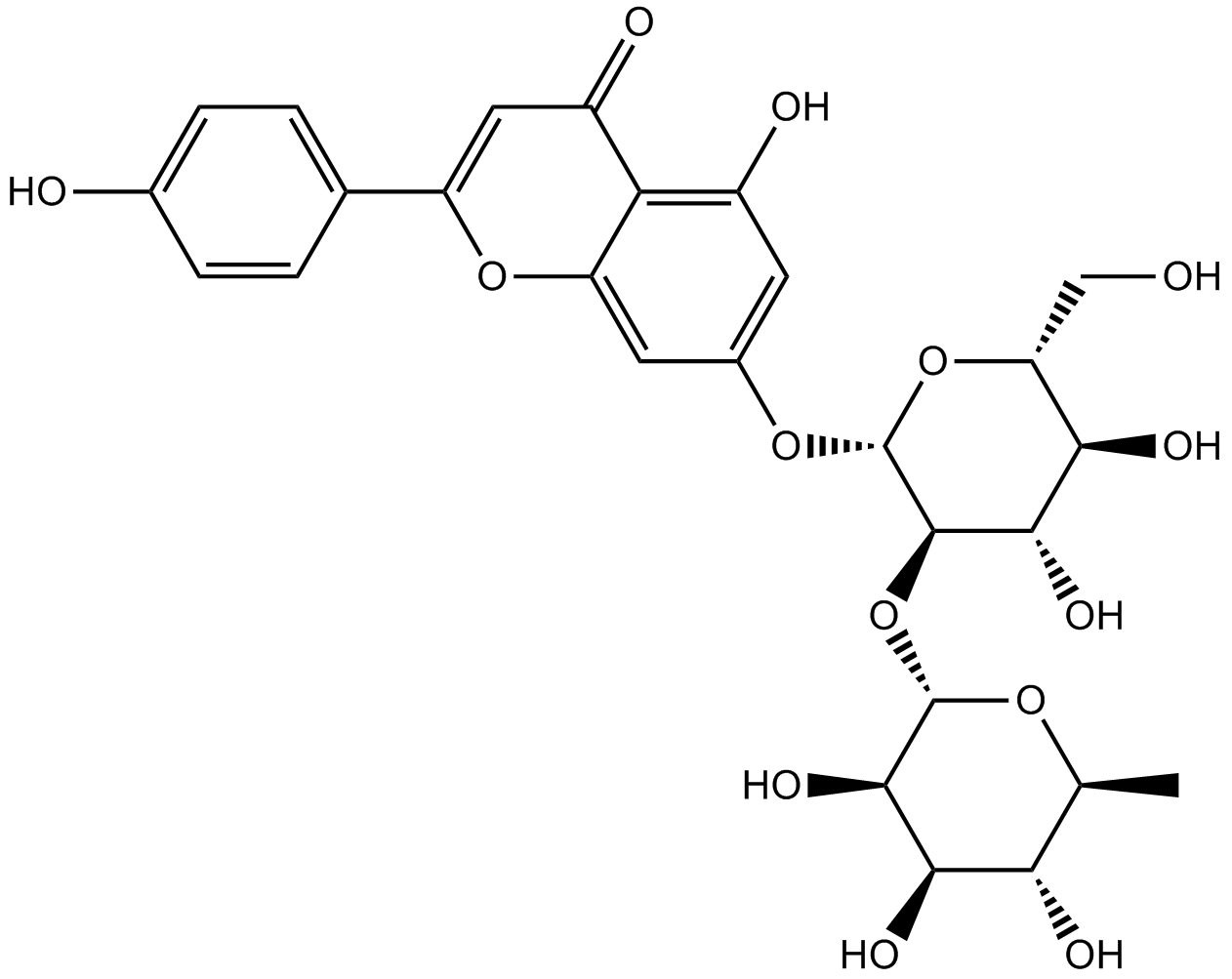
GN10784
Rhoifolin
-
GC50502
RI-STAD 2
AKAP disruptor; selectively binds PKA-RI with high affinity and blocks its interaction with AKAP; cell permeable
-
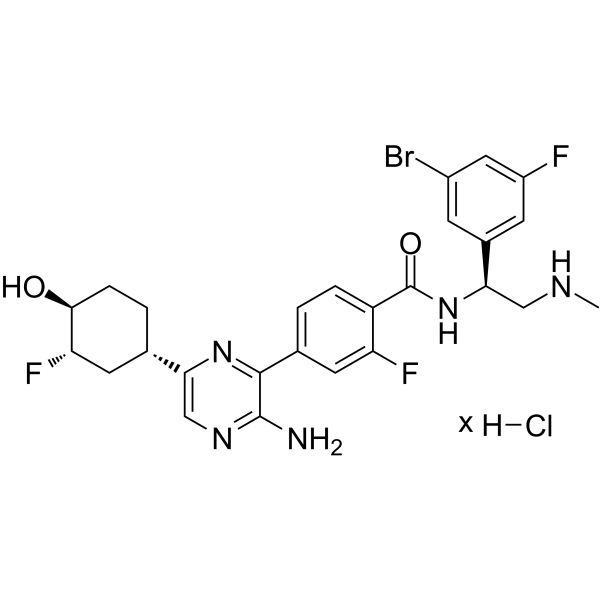
GC62448
Rineterkib hydrochloride
Rineterkib-Hydrochlorid (Verbindung B) ist ein oral verfÜgbarer ERK1- und ERK2-Inhibitor zur Behandlung einer proliferativen Erkrankung, die durch aktivierende Mutationen im MAPK-Signalweg gekennzeichnet ist. Die AktivitÄt steht insbesondere im Zusammenhang mit der Behandlung von KRAS-mutiertem NSCLC, BRAF-mutiertem NSCLC, KRAS-mutiertem BauchspeicheldrÜsenkrebs, KRAS-mutiertem Darmkrebs (CRC) und KRAS-mutiertem Eierstockkrebs. Rineterkibhydrochlorid kann auch RAF hemmen.
-
GC69821
RLX-33
RLX-33 ist ein wirksamer und selektiver Antagonist des Relaxin-Familienpeptids 3 (RXFP3), der auch die durch Relaxin 3 induzierte ERK1/2-Phosphorylierung blockiert. Die IC50-Werte für RXFP3, ERK1 und ERK2 Phosphorylierung betragen jeweils 2,36 μM, 7,82 μM und 13,86 μM. RLX-33 kann die erhöhte Nahrungsaufnahme von Ratten blockieren, die durch den RXFP3-selektiven Agonisten R3/I5 induziert wird. RLX-33 kann zur Erforschung des metabolischen Syndroms eingesetzt werden.
-
GC50294
RMM 46
RMM 46 ist ein selektiver und reversibler kovalenter Inhibitor für Kinasen der MSK/RSK-Familie.
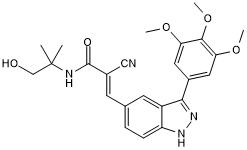
-
GC12141
RO4987655
RO4987655 ist ein oral aktiver und hochselektiver MEK-Inhibitor mit einem IC50 von 5,2 nM zur Hemmung von MEK1/MEK2.
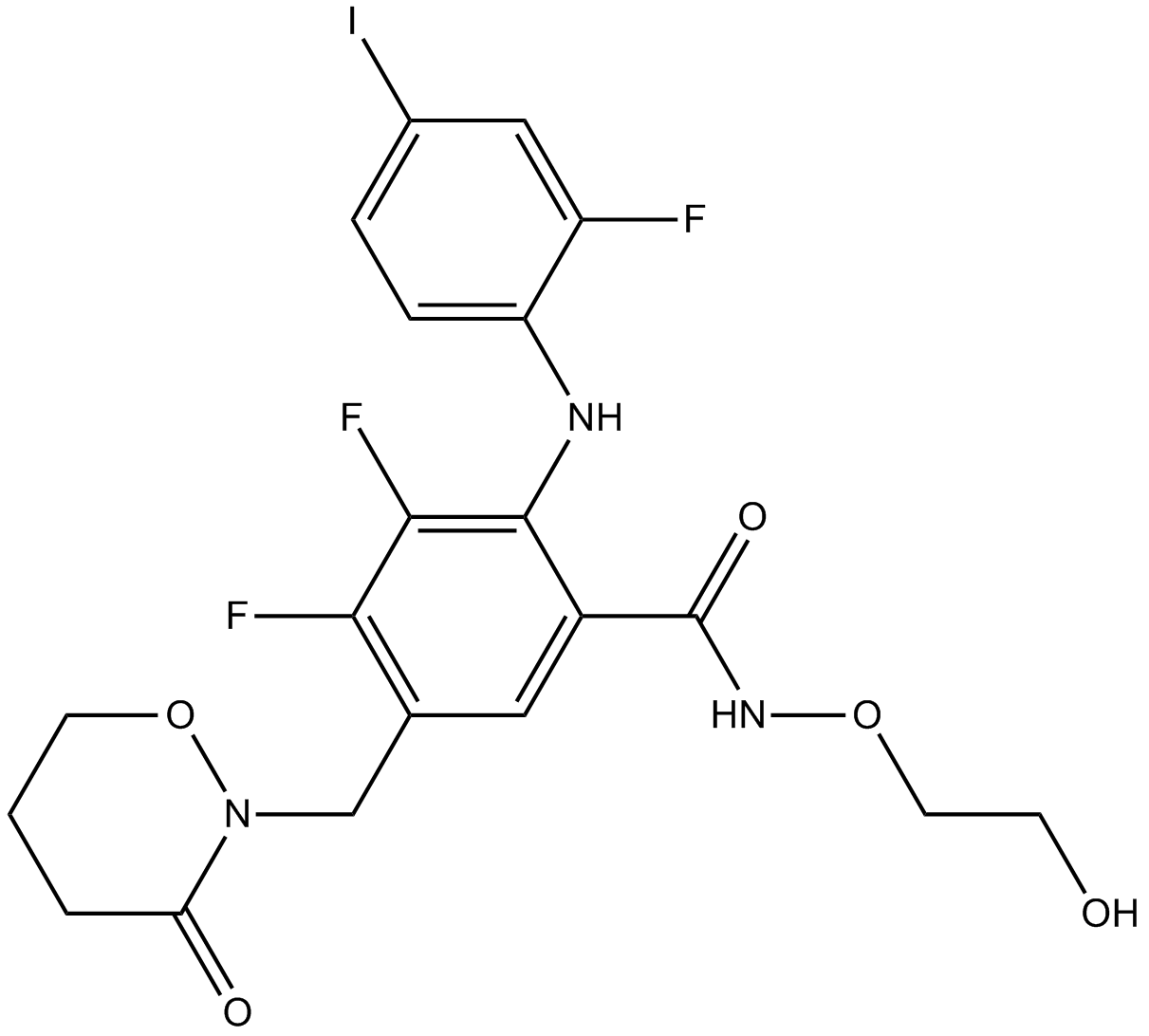
-
GC12406
RO5126766(CH5126766)
RO5126766(CH5126766) (CH5126766) ist ein erstklassiger dualer MEK/RAF-Inhibitor, der BRAFV600E, CRAF, MEK und BRAF allosterisch hemmt (IC50: 8,2, 56, 160 nM bzw. 190 nM).
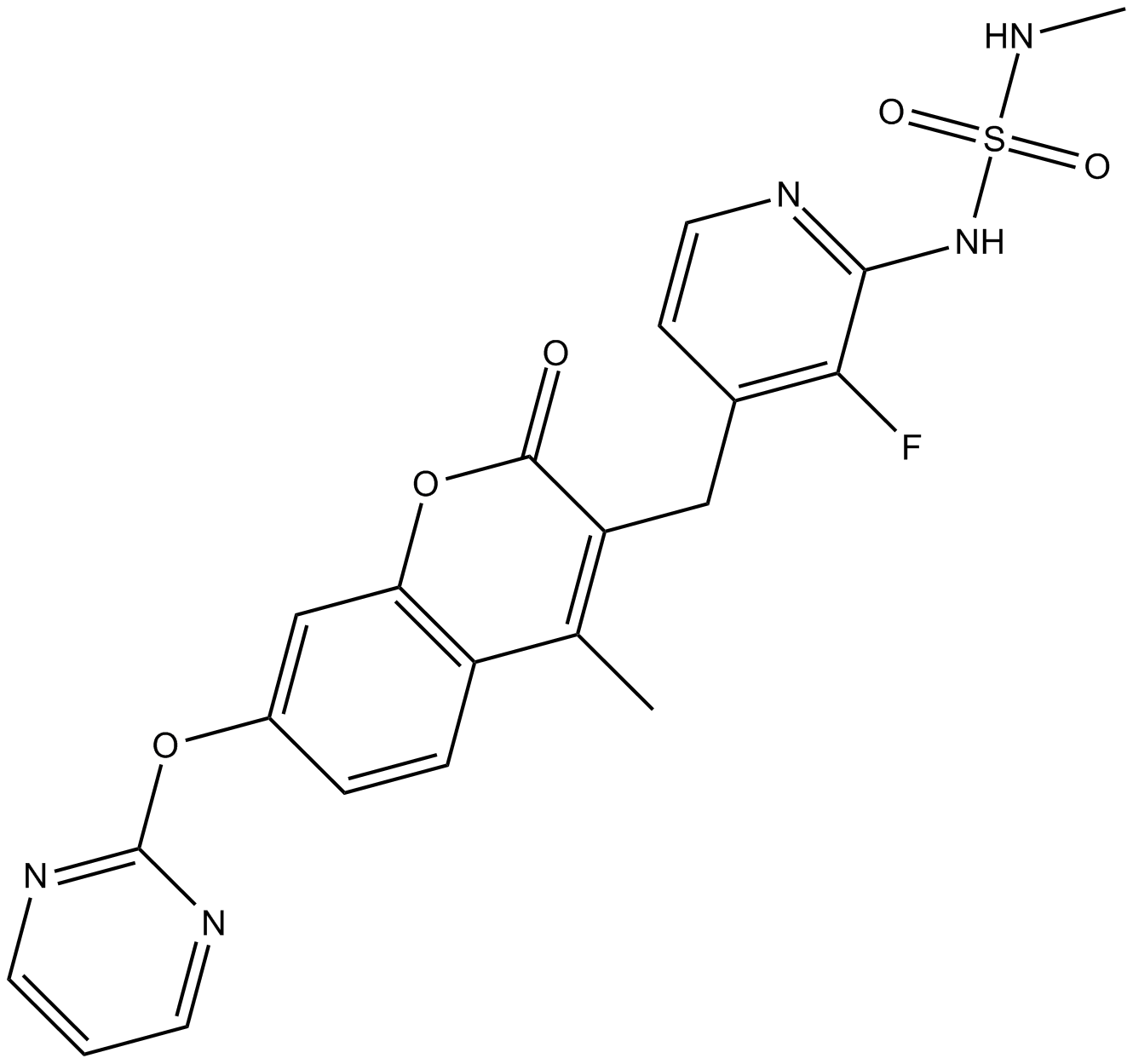
-
GC38609
Rotundic acid
RotundinsÄure, ein Triterpenoid aus I.
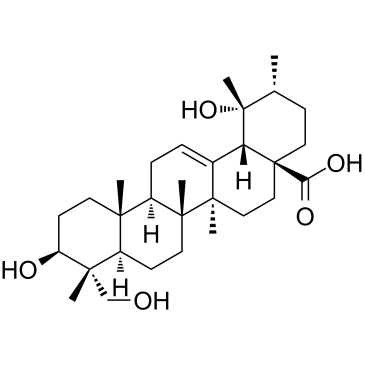
-
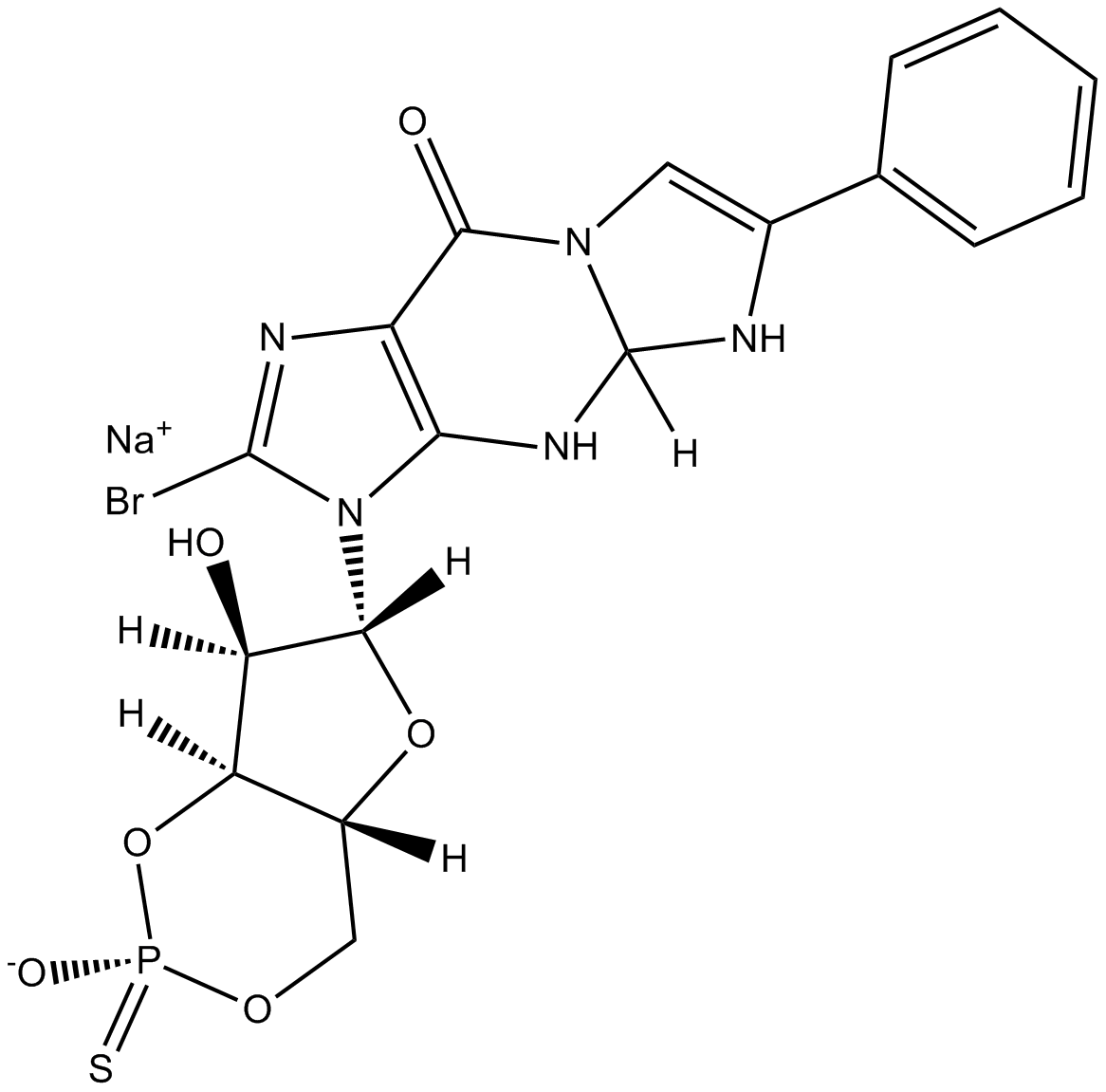
GC15611
Rp-8-Br-PET-cGMPS
cGMP-dependent protein kinase (PKG) inhibitor
-
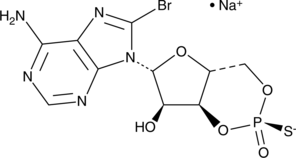
GC44852
Rp-8-bromo-Cyclic AMPS (sodium salt)
Rp-8-bromo-Cyclic AMPS (Rp-8-bromo-cAMPS) is a cell-permeable cAMP analog that combines an exocyclic sulfur substitution in the equatorial position of the cyclophosphate ring with a bromine substitution in the adenine base of cAMP.
-
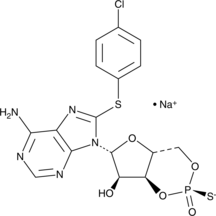
GC44853
Rp-8-CPT-Cyclic AMP (sodium salt)
Rp-8-CPT-cyclisches AMP (Natriumsalz), ein cAMP-Analogon, ist ein potenter und kompetitiver Antagonist der cAMP-induzierten Aktivierung von cAMP-abhÄngigen PKA I und II.
-
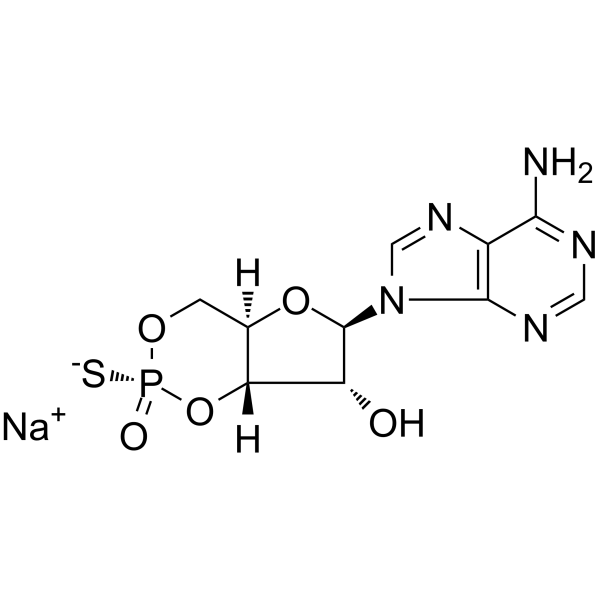
GC63176
Rp-cAMPS sodium salt
Rp-cAMPS-Natriumsalz, ein cAMP-Analog, ist ein potenter, kompetitiver cAMP-induzierter Aktivierung von cAMP-abhÄngigen PKA-I- und -II-Antagonisten (Kis von 12,5 μM bzw. 4,5 μM).
-
GC62269
RRD-251
RRD-251 ist ein Inhibitor der Retinoblastom-Tumorsuppressorprotein (Rb)-Raf-1-Interaktion mit starken antiproliferativen, antiangiogenen und AntitumoraktivitÄten.