Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Targets for Cell Cycle/Checkpoint
- ATM/ATR(26)
- Aurora Kinase(47)
- Cdc42(4)
- Cdc7(4)
- Chk(16)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(91)
- E1 enzyme(1)
- G-quadruplex(14)
- Haspin(7)
- HMTase(1)
- Kinesin(26)
- Ksp(6)
- Microtubule/Tubulin(243)
- Mps1(15)
- Mitotic(11)
- RAD51(18)
- ROCK(71)
- Rho(13)
- PERK(11)
- PLK(37)
- PTEN(8)
- Wee1(7)
- PAK(21)
- Arp2/3 Complex(8)
- Dynamin(12)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Products for Cell Cycle/Checkpoint
- Cat.No. Nombre del producto Información
-
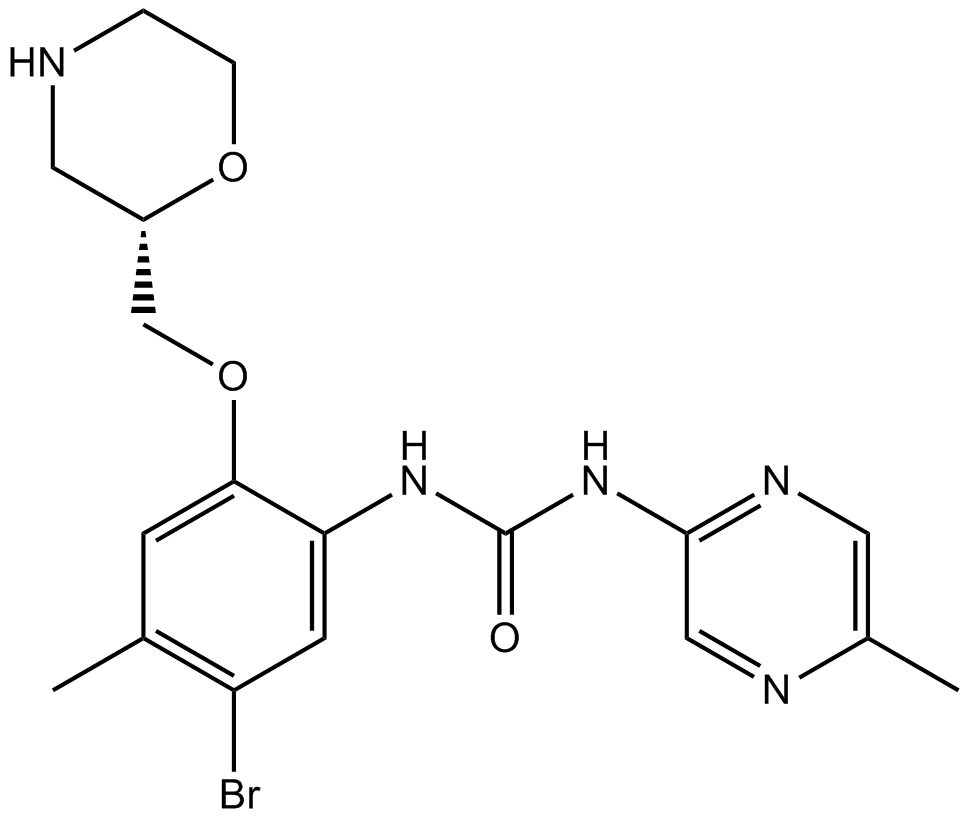
GA21951
H-4-Nitro-Phe-OEt . HCl
A building block

-
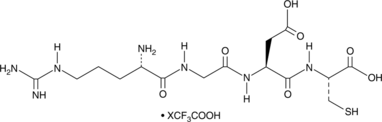
GC49305
H-Arg-Gly-Asp-Cys-OH (trifluoroacetate salt)
An RGD-containing tetrapeptide
-
GA22303
H-D-Pro-Phe-Arg-pNA . 2 HCl
pFR-pNA, chromogenic substrate for the determination of plasma kallikrein-like activity.
CAS Number (net): 64816-19-9.

-
GC45471
H-Gly-Arg-pNA (hydrochloride)
-
GC49605
H-Gly-Pro-Hyp-OH (acetate)
A peptide DPP-4 inhibitor
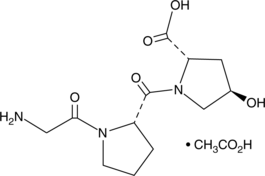
-
GC43803
HA-1077 (hydrochloride)
Fasudil (HA-1077; AT877) dihidrocloruro es un inhibidor rhoa/roca inespecÍfico y también tiene un efecto inhibitorio en las proteÍnas quinasas, con un KI de 0.33 ⋼ m para el rock111&&&&&&77TRESTO. offlineefficient_models_2022q2.md;M, 1.650 μM for ROCK2 and PKA, PKC, PKG, respectively.en_es_2021q4.mdHA-1077 (hydrochloride) is also a potent Ca2+ channel antagonist and vasodilator.en_es_2021q4.md
-
GC43805
HAPC-Chol
HAPC-Chol is a cationic cholesterol.
-
GC49855
Harmalol (hydrochloride hydrate)
A β-carboline alkaloid and an active metabolite of harmaline
-
GC49915
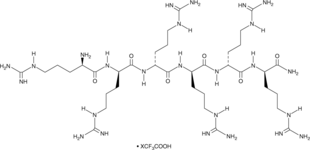
Hexa-D-Arginine (trifluoroacetate salt)
A furin inhibitor
-
GC45639
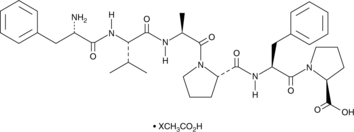
Hexapeptide-11 (acetate)
A synthetic hexapeptide
-
GC43832
Histone H3 (21-44)-GK-biotin amide (trifluoroacetate salt)
Histone H3 (21-44)-GK-biotin is a peptide fragment of histone H3 that corresponds to amino acid residues 22-45 of the human histone H3.3 sequence and is biotinylated via a C-terminal GK linker.
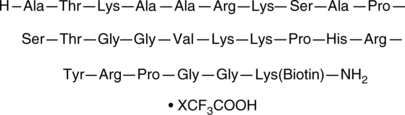
-
GC13928
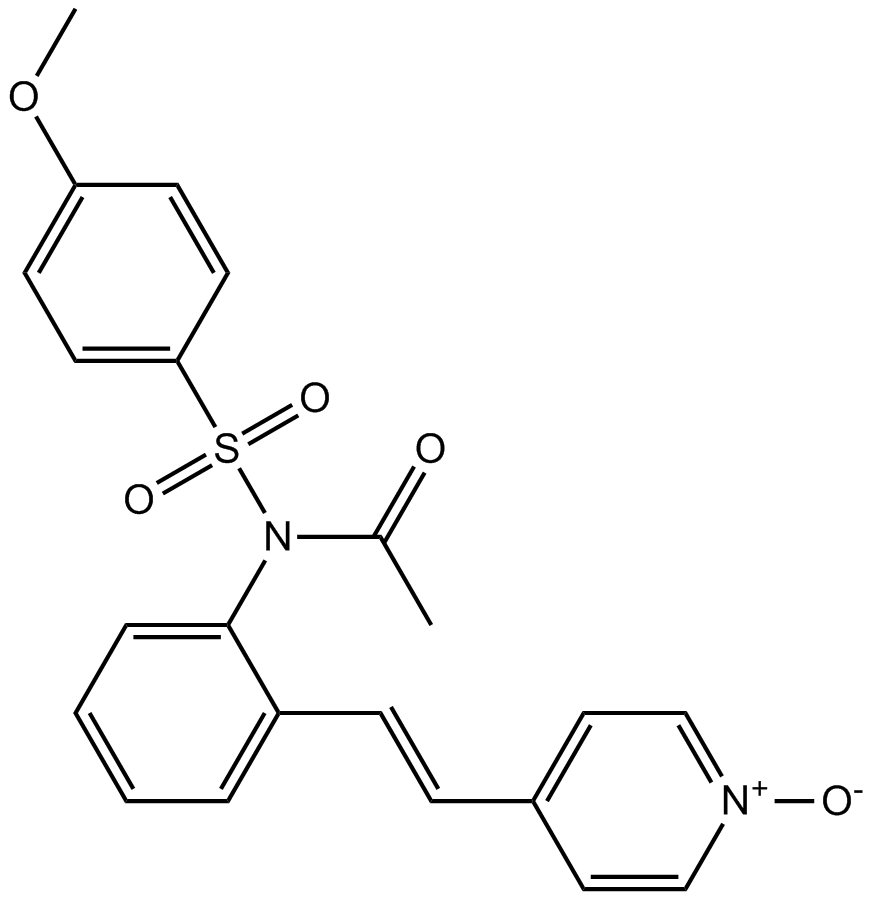
HMN-214
HMN-214, un profÁrmaco biodisponible por vÍa oral de HMN-176, es un inhibidor de la quinasa-1 tipo polo (plk1), con actividad antitumoral.
-
GC62594
hnRNPK-IN-1
hnRNPK-IN-1 es un ligando de uniÓn a la ribonucleoproteÍna K nuclear heterogénea (hnRNPK) con valores de Kd de 4,6 μM y 2,6 μM medidos con SPR y MST, respectivamente. hnRNPK-IN-1 inhibe la transcripciÓn de c-myc al interrumpir la uniÓn de hnRNPK y el promotor de c-myc. hnRNPK-IN-1 induce la apoptosis de las células Hela y tiene fuertes actividades antitumorales.
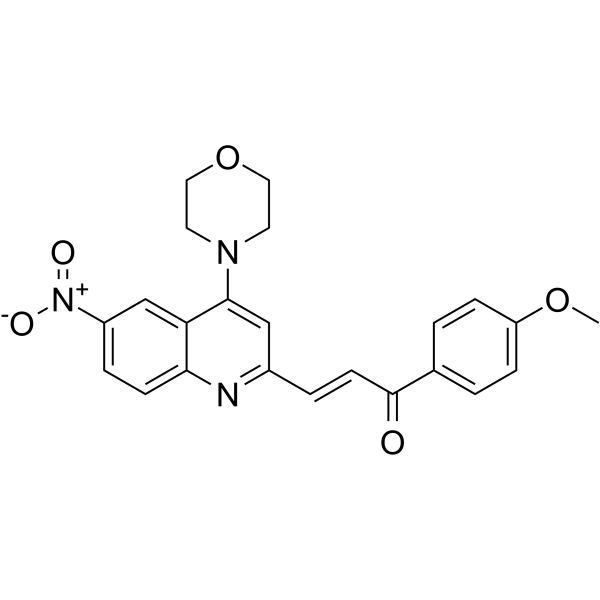
-
GC49742
Hsf1 Monoclonal Antibody (Clone 10H8)
For immunochemical analysis of Hsf1

-
GC47436
HT-2 Toxin-13C22
An internal standard for the quantification of HT-2 toxin
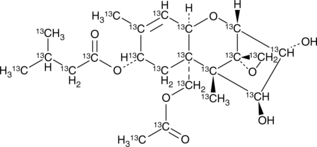
-
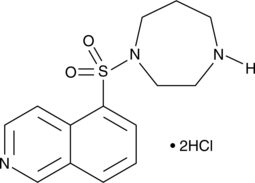
GC10901
Hydroxyfasudil
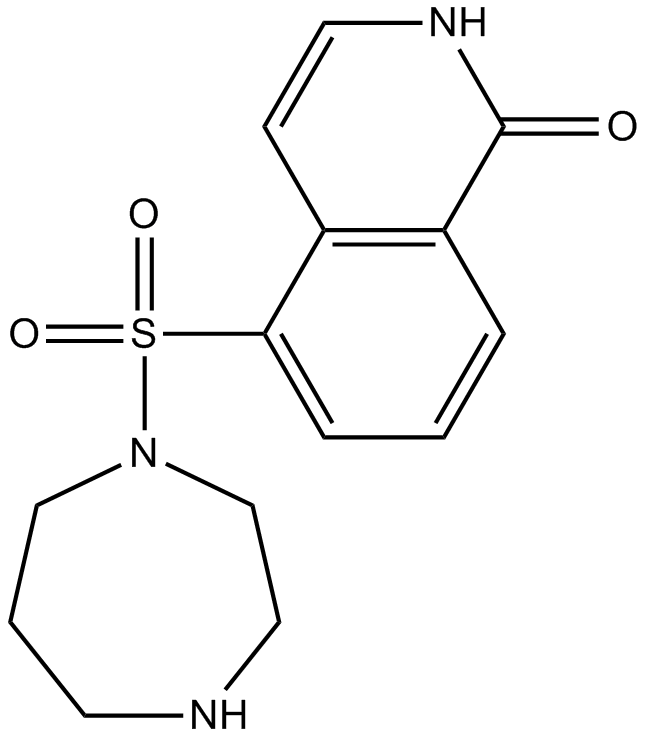
-
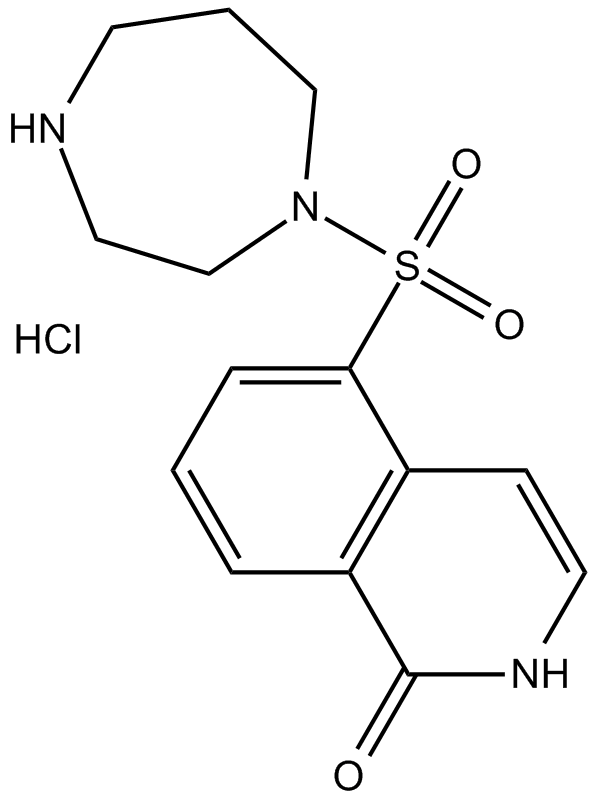
GC10338
Hydroxyfasudil hydrochloride
A ROCK inhibitor
-
GC32912
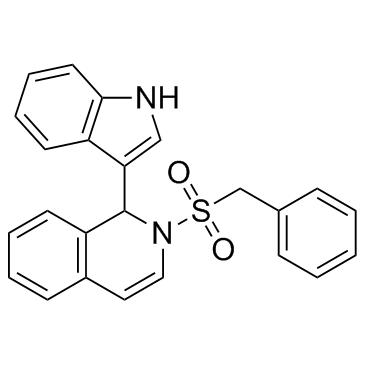
IBR2
IBR2 es un inhibidor potente y especÍfico de RAD51 e inhibe la reparaciÓn de roturas de doble cadena de ADN mediada por RAD51. IBR2 interrumpe la multimerizaciÓn de RAD51, acelera la degradaciÓn de la proteÍna RAD51 mediada por proteasoma, inhibe el crecimiento de células cancerosas e induce la apoptosis.
-
GC45743
ICAAc
A solvatochromic fluorophore and pH probe
-
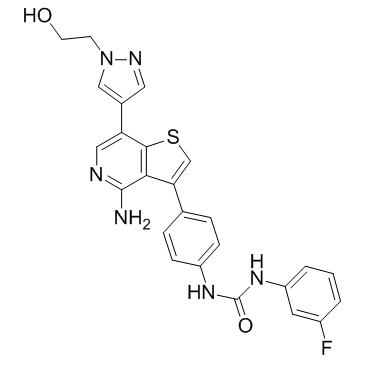
GC34159
Ilorasertib (ABT-348)
Ilorasertib (ABT-348) (ABT-348) es un inhibidor de aurora potente, activo por vÍa oral y competitivo con ATP con IC50 de 116, 5, 1 nM para aurora A, aurora B, aurora C, respectivamente. Ilorasertib (ABT-348) también es un potente inhibidor de VEGF, PDGF. Ilorasertib (ABT-348) tiene potencial para la investigaciÓn de la leucemia mieloide aguda (LMA) y el sÍndrome mielodisplÁsico (SMD).
-
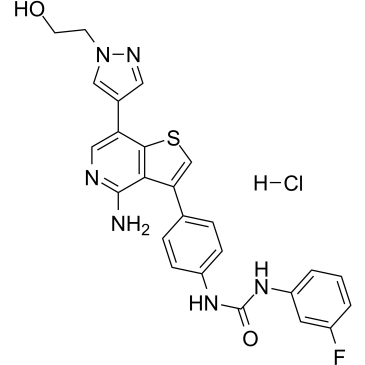
GC38519
Ilorasertib hydrochloride
El clorhidrato de ilorasertib (ABT-348) es un inhibidor de la aurora potente, oralmente activo y competitivo con ATP con IC50 de 116, 5, 1 nM para aurora A, aurora B, aurora C, respectivamente. El clorhidrato de ilorasertib también es un potente inhibidor de VEGF, PDGF. El clorhidrato de ilorasertib tiene potencial para la investigaciÓn de la leucemia mieloide aguda (LMA) y el sÍndrome mielodisplÁsico (SMD).
-
GC36308
Indibulin
Indibulin (ZIO 301), un inhibidor del ensamblaje de tubulina aplicable por vÍa oral, muestra una potente actividad anticancerÍgena con una neurotoxicidad mÍnima.
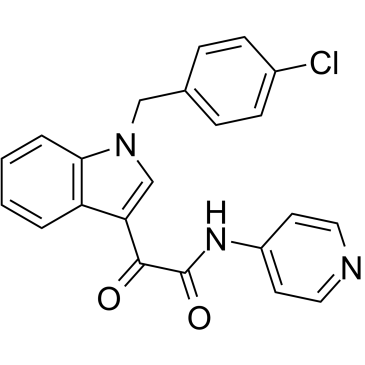
-
GC52513
Indolimine-214
A genotoxic gut microbiota metabolite
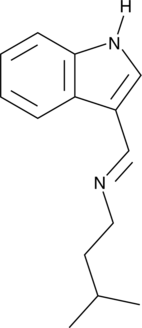
-
GC49460
ING-2 AM
A cell-permeable fluorescent sodium indicator

-
GC10334
INH6
INH6 es un potente inhibidor de Nek2/Hec1; inhibe el crecimiento de células HeLa con una IC50 de 2,4 μM.
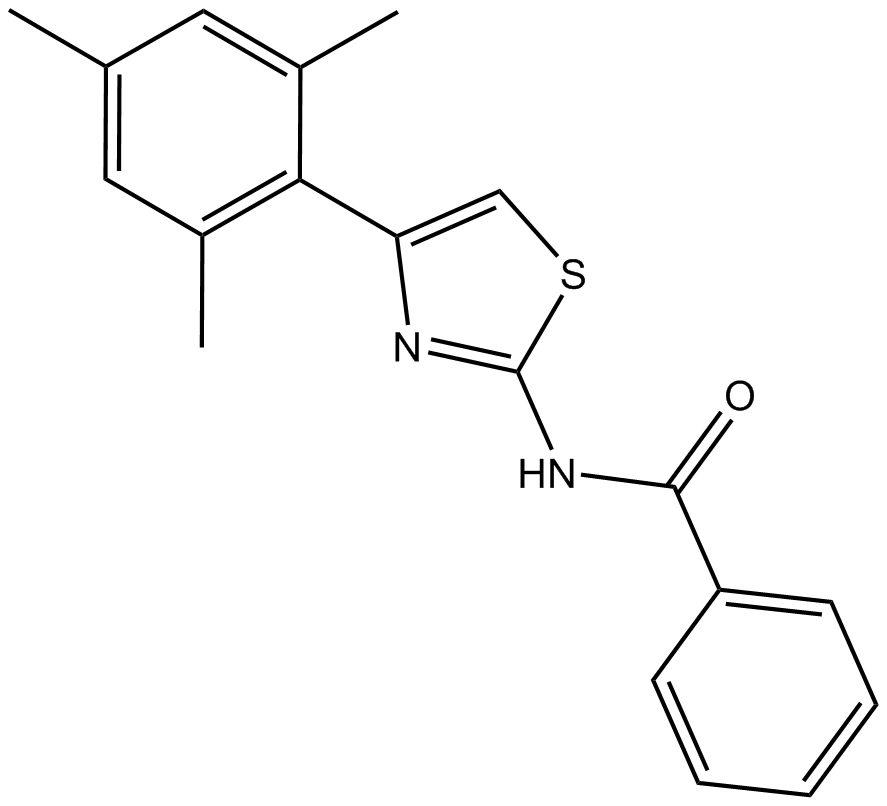
-
GC48387
Inostamycin A
A bacterial metabolite with anticancer activity
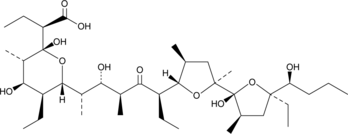
-
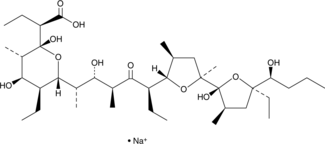
GC52472
Inostamycin A (sodium salt)
A bacterial metabolite with anticancer activity
-
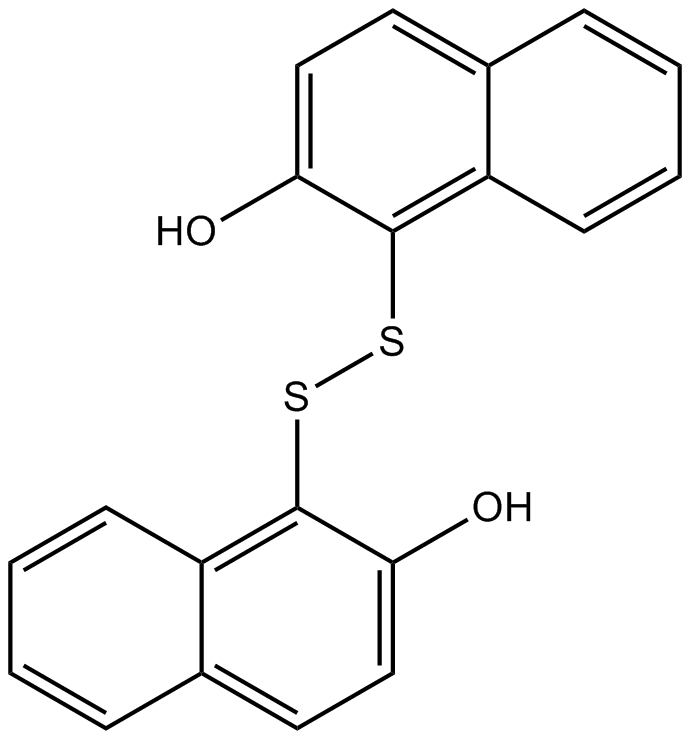
GC16225
IPA-3
IPA-3 es un inhibidor selectivo de PAK1 competitivo no ATP con IC50 de 2,5 μM, y no muestra inhibiciÓn de las PAK del grupo II (PAK 4-6).
-
GC49468
IPG-1 AM
A cell-permeable fluorescent potassium indicator
-
GC49469
IPG-1 TMA
A cell-impermeable fluorescent potassium indicator

-
GC49463
IPG-2 AM
A cell-permeable fluorescent potassium indicator
-
GC49466
IPG-2 TMA
A cell-impermeable fluorescent potassium indicator
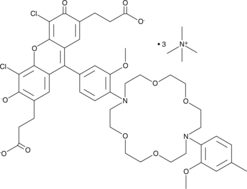
-
GC65188
IRES-C11
IRES-C11 es un inhibidor de la traducciÓn del sitio de entrada al ribosoma interno (IRES) c-MYC espectfico. IRES-C11 bloquea la interacciÓn de un factor de acciÓn trans c-MYC IRES necesario, la ribonucleoproteÍna nuclear heterogénea A1, con su IRES. IRES-C11 no inhibe BAG-1, XIAP y p53 IRESes.
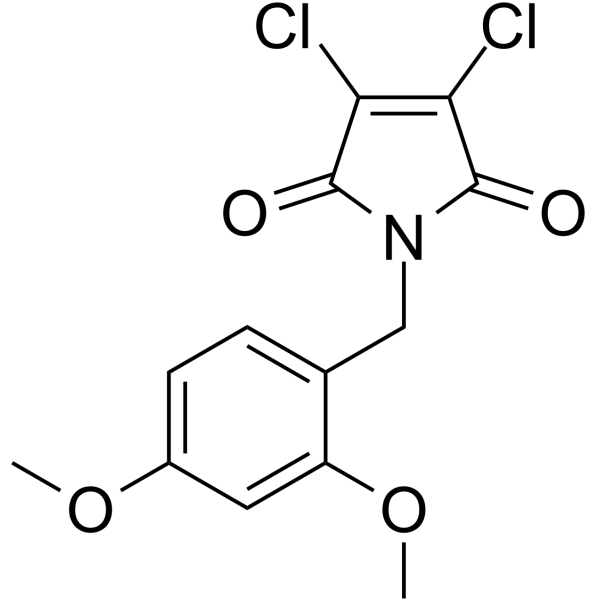
-
GC15215
Iso-Olomoucine
inactive stereoisomer of the Cdk5 inhibitor olomoucine
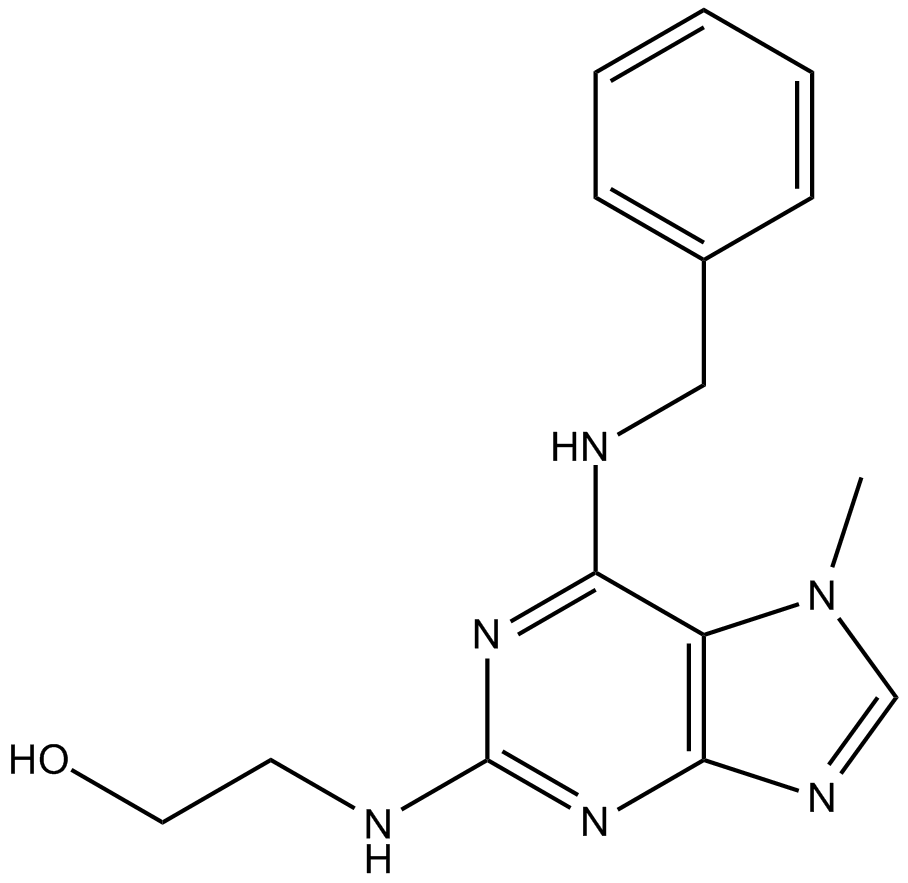
-
GC40901
Isogarcinol
Isogarcinol is a natural polyisoprenylated benzophenone first isolated from plant species in the genus Garcinia.
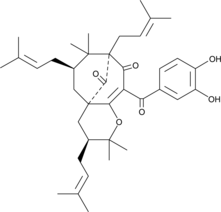
-
GC43917
Isopropyl Benzenesulfonate
Isopropyl benzenesulfonate is a sulfonate ester genotoxic impurity found in active pharmaceutical ingredients.
-
GC49142
Isorhoifolin
La isorhoifolina es un glucÓsido flavonoide de las hojas de periploca nigrescens.
-
GC43922
Isovaleryl-L-carnitine (chloride)
La isovaleril-L-carnitina (cloruro), un producto del catabolismo de la L-leucina, es un potente activador de la proteinasa dependiente de Ca2+- (calpaÍna) de los neutrÓfilos humanos.
-
GC13394
Ispinesib (SB-715992)
Ispinesib (SB-715992) es un inhibidor especÍfico de la proteÍna del huso de kinesina (KSP), con una Ki app de 1,7 nM.
-
GC10581
ISRIB
PERK signaling inhibitor
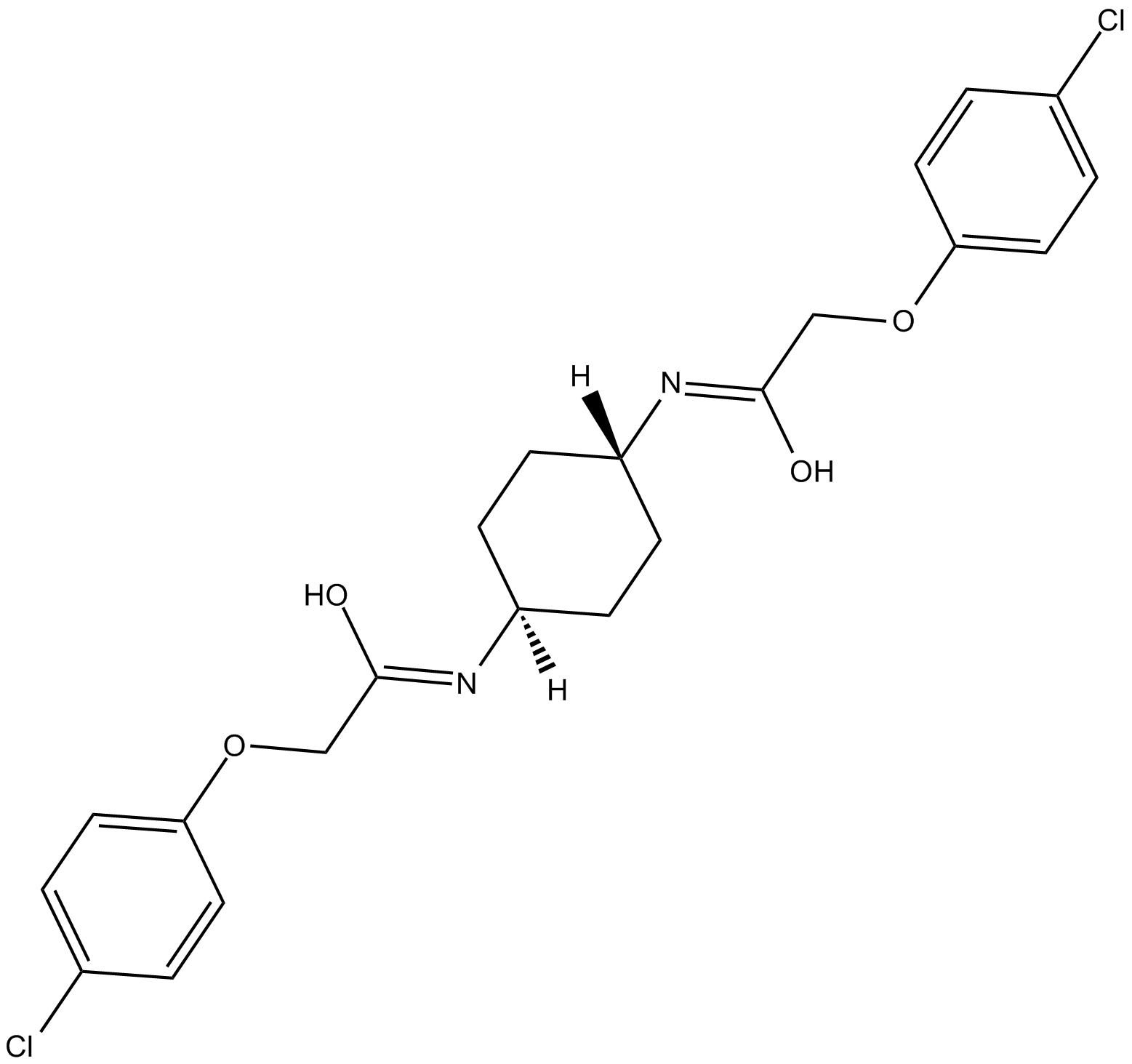
-
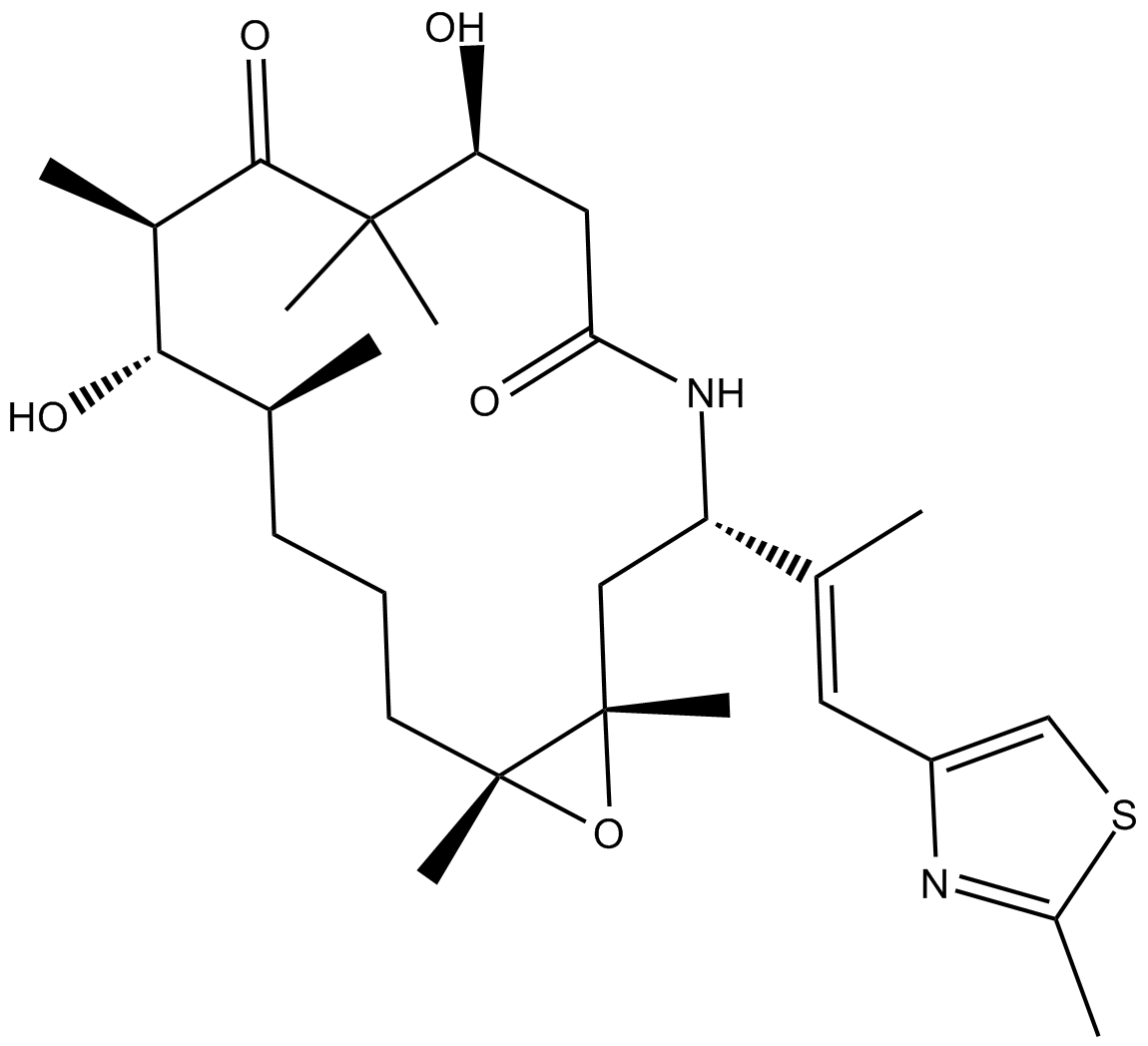
GC16869
Ixabepilone
A broad-spectrum anticancer agent
-
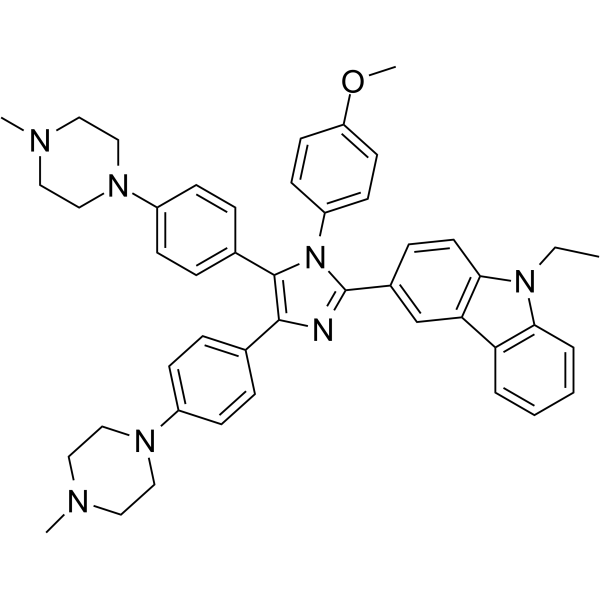
GC65331
IZCZ-3
IZCZ-3 es un potente inhibidor de la transcripciÓn de c-MYC con actividad antitumoral.
-
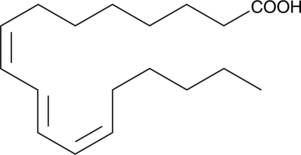
GC41645
Jacaric Acid
Jacaric acid is a conjugated polyunsaturated fatty acid first isolated from seeds of Jacaranda plants.
-
GC18699
JCP174
JCP174 es un inhibidor de la palmitoil proteÍna tioesterasa-1 (TgPPT1), una depalmitoilasa del parÁsito T.
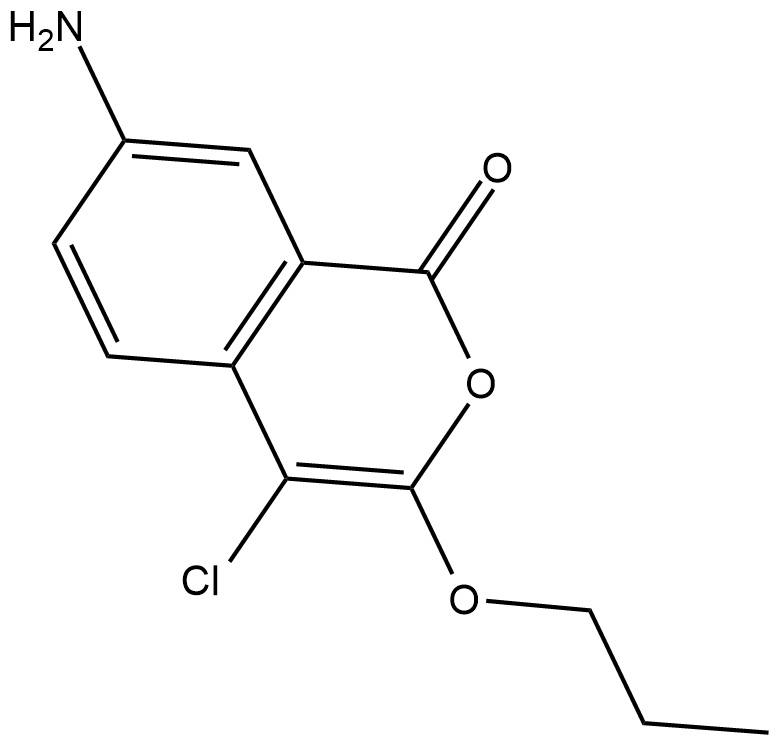
-
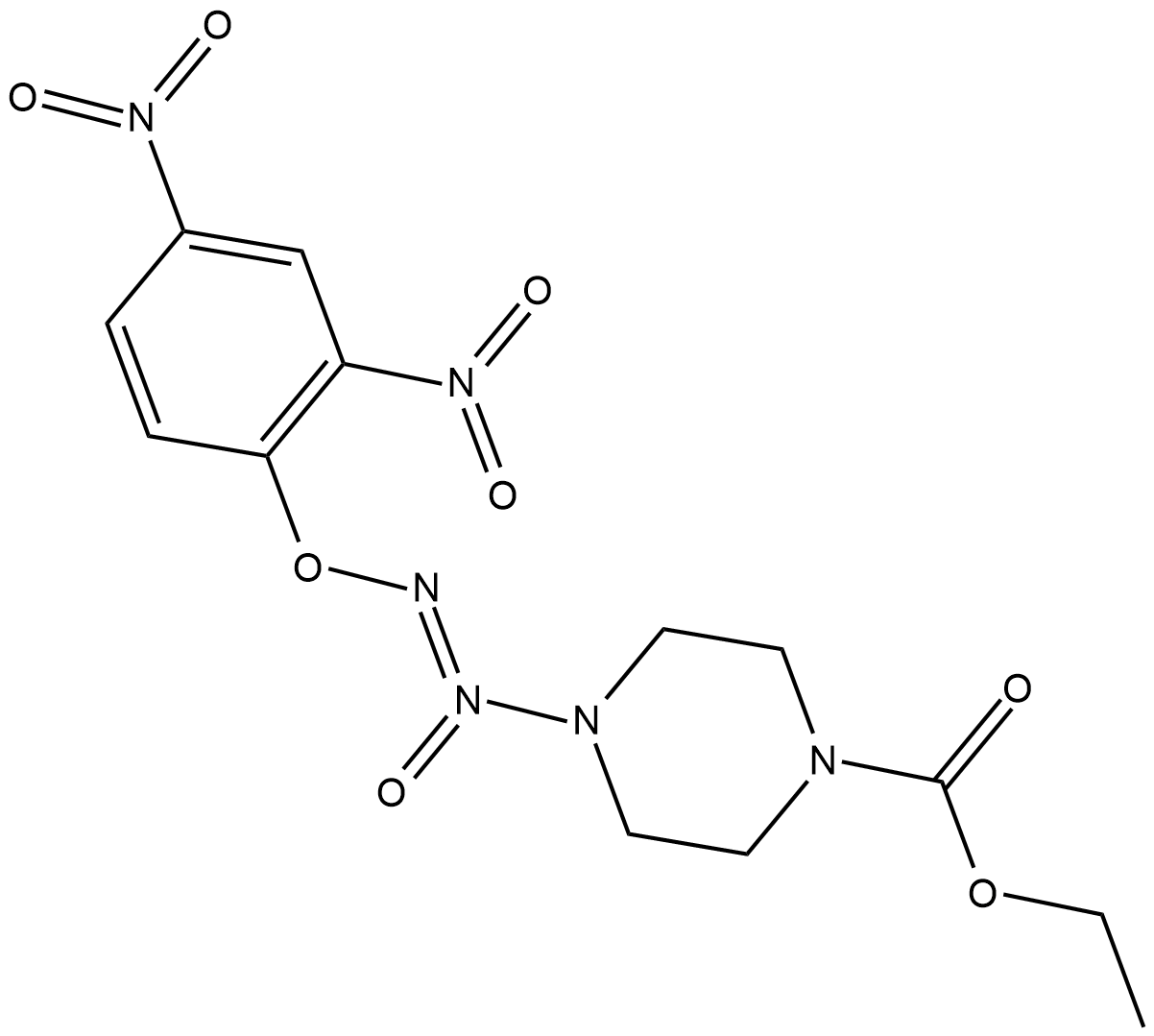
GC18734
JS-K
JS-K es un donante de NO que reacciona con el glutatiÓn para generar NO a pH fisiolÓgico. JS-K inhibe la proliferaciÓn, induce la apoptosis e interrumpe el ciclo celular de las células de leucemia linfoblÁstica aguda Jurkat T.
-
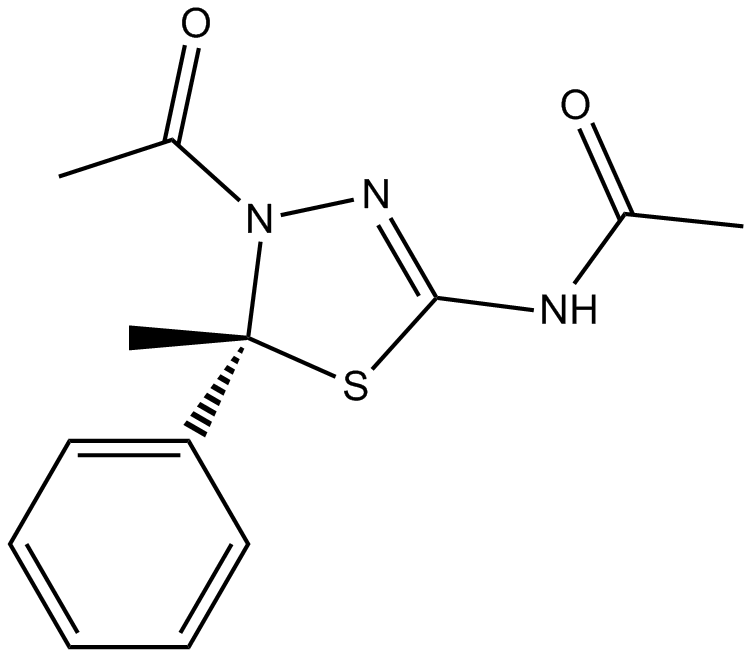
GC16167
K 858
K858 Racemic es un inhibidor no competitivo de ATP de la kinesina Eg5 con un IC50 de 1.3 μM.
-
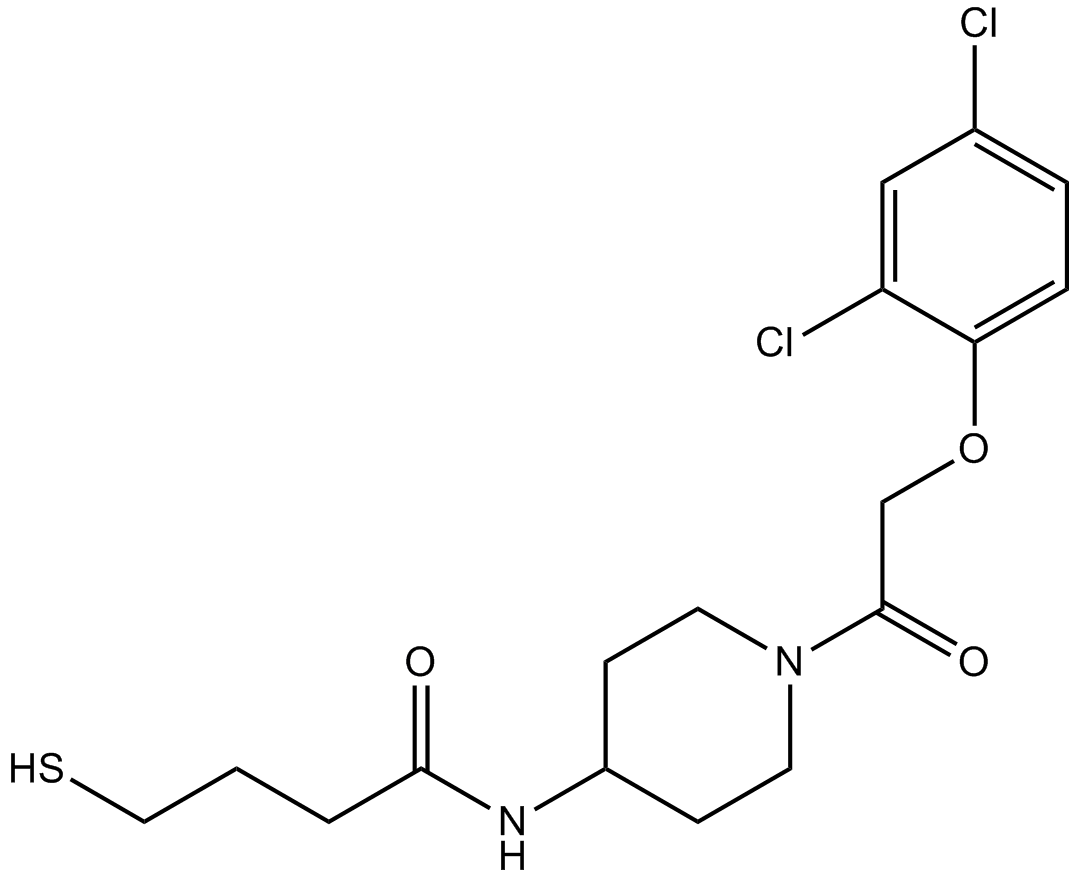
GC17388
K-Ras(G12C) inhibitor 6
El inhibidor 6 de K-Ras(G12C) es un inhibidor alostérico irreversible de K-Ras(G12C).
-
GC14230
K03861
K03861 (K03861) es un inhibidor de CDK2 de tipo II con Kd de 8,2 nM. K03861 (K03861) inhibe la actividad de CDK2 al competir con la uniÓn de las ciclinas activadoras.
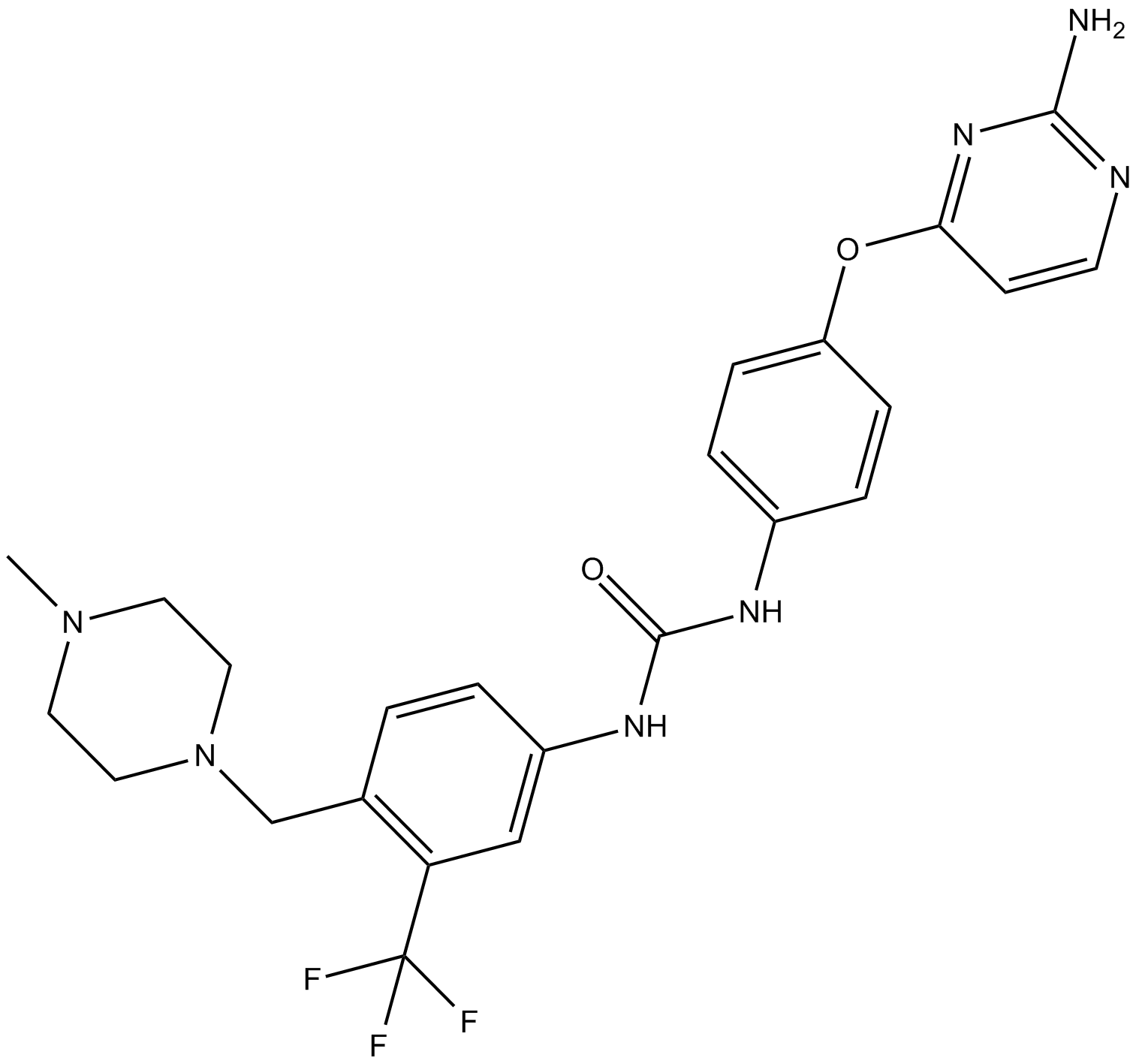
-
GC43994
KAPA (hydrochloride)
Biotin is a growth factor that plays an important role in numerous reactions that are critical to microorganisms, plants, and animals.
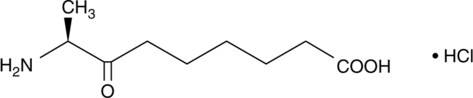
-
GC43995
Kazusamycin B
Kazusamycin B is a bacterial metabolite originally isolated from Streptomyces.
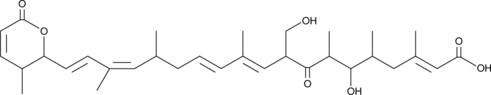
-
GC14182
Kenpaullone
La kenpaulona es un potente inhibidor de CDK1/ciclina B y GSK-3β, con IC50 de 0,4 μM y 23 nM, y también inhibe CDK2/ciclina A, CDK2/ciclina E y CDK5/p25 con IC50 de 0,68 μM, 7,5 μM, 0,85 μM, respectivamente. La kenpaulona, una molécula pequeÑa inhibidora de KLF4, reduce la autorrenovaciÓn de las células madre del cÁncer de mama y la motilidad celular in vitro.
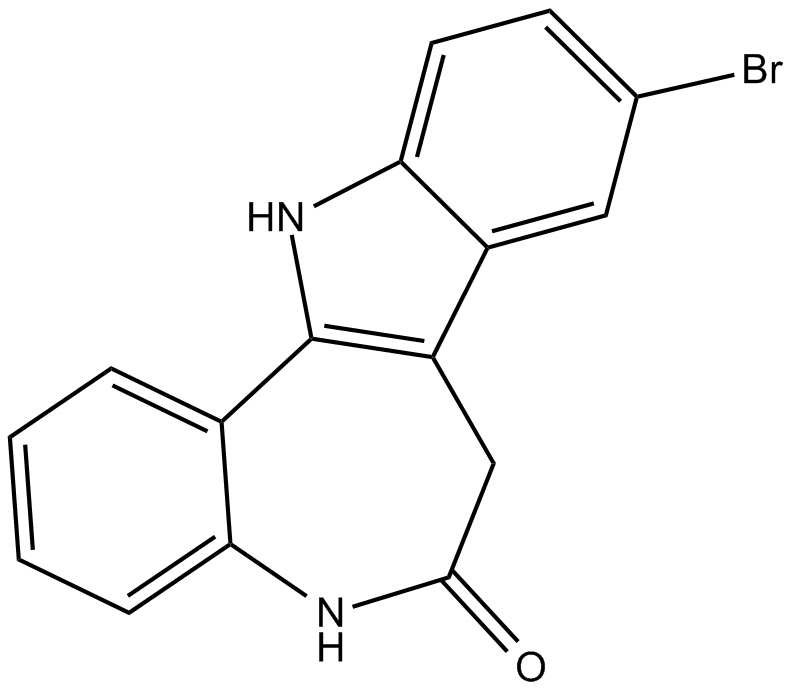
-
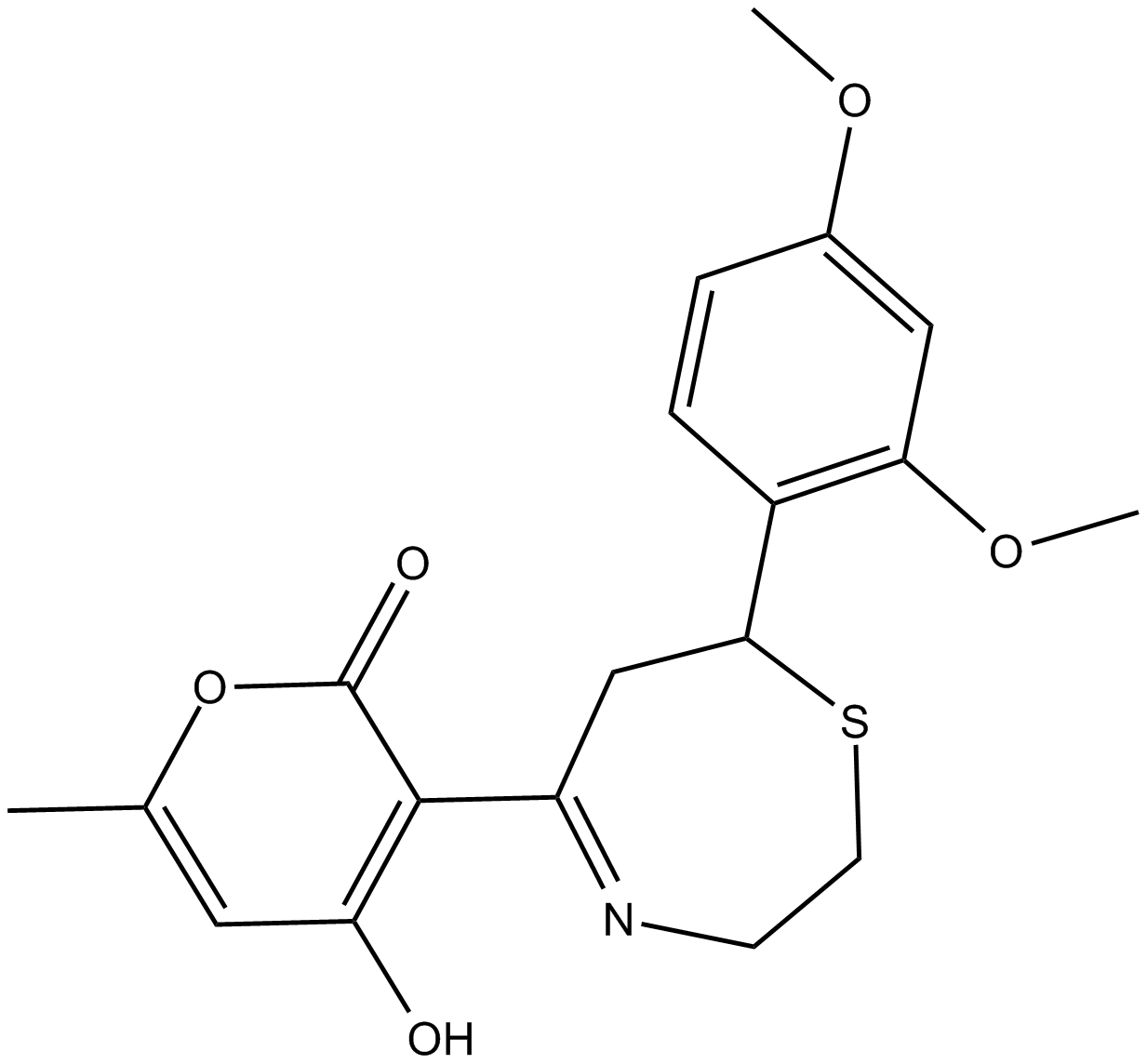
GC14589
KF 38789
KF 38789 es un inhibidor selectivo de la unión de P-selectina-PSGL-1.
-
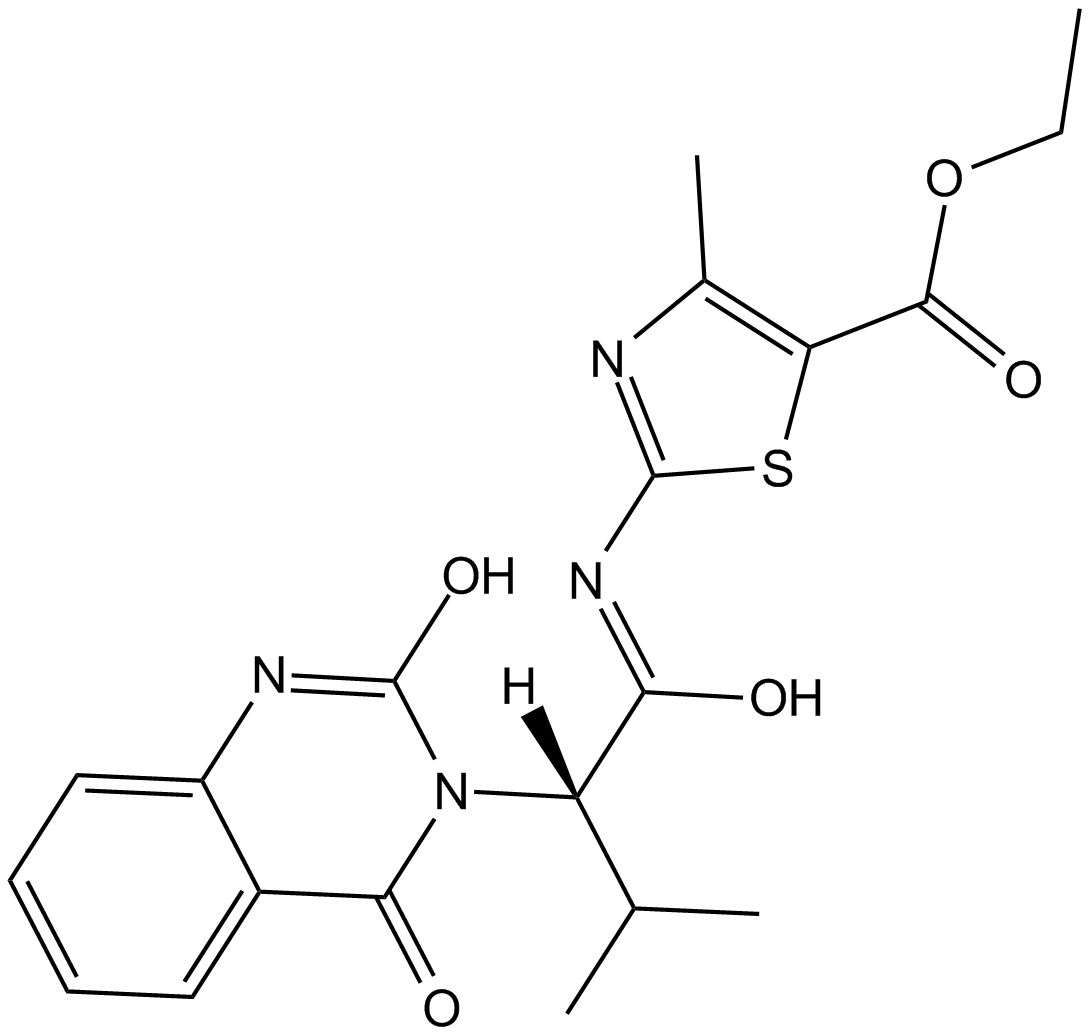
GC16603
Kif15-IN-1
Kif15-IN-1 es un inhibidor del miembro 15 de la familia Kinesina mitÓtica (Kif15) y se utiliza para la investigaciÓn de enfermedades de proliferaciÓn celular.
-
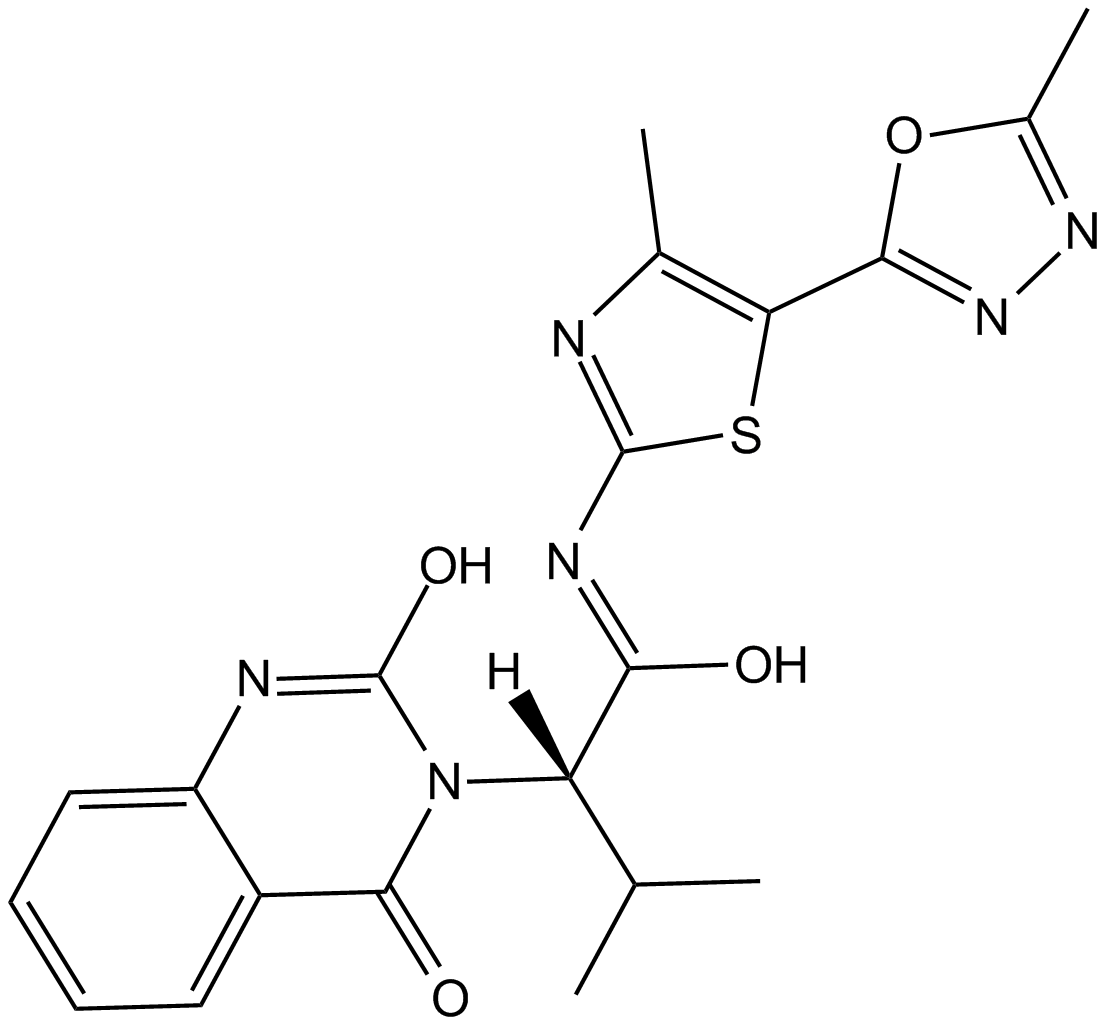
GC17994
Kif15-IN-2
Kif15-IN-2 es un inhibidor de la cinesina mitÓtica Kif15 y se utiliza para la investigaciÓn de enfermedades de proliferaciÓn celular.
-
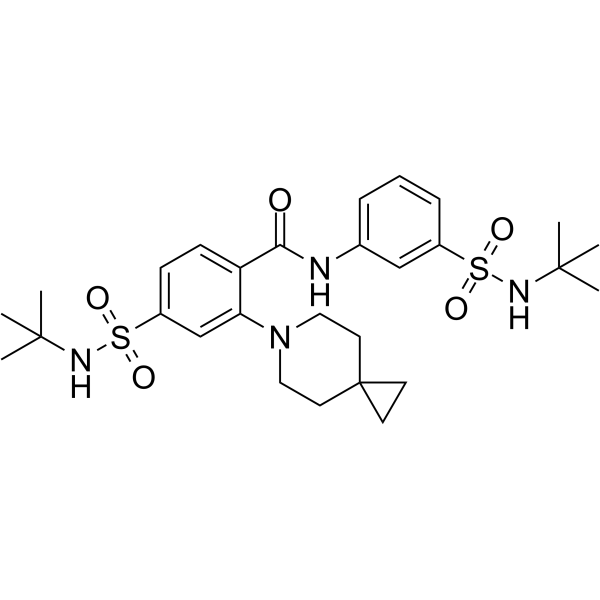
GC64085
KIF18A-IN-1
KIF18A-IN-1 es un inhibidor de kinesina mitÓtica KIF18A extraÍdo de la patente WO2021026098A1 ejemplo 100-13.
-
GC64808
KIF18A-IN-2
KIF18A-IN-2 es un potente inhibidor de KIF18A (IC50=28 nM). KIF18A-IN-2 causa un arresto mitÓtico significativo y aumenta el nÚmero de células mitÓticas en los tejidos tumorales. KIF18A-IN-2 se puede utilizar para investigar el cÁncer.
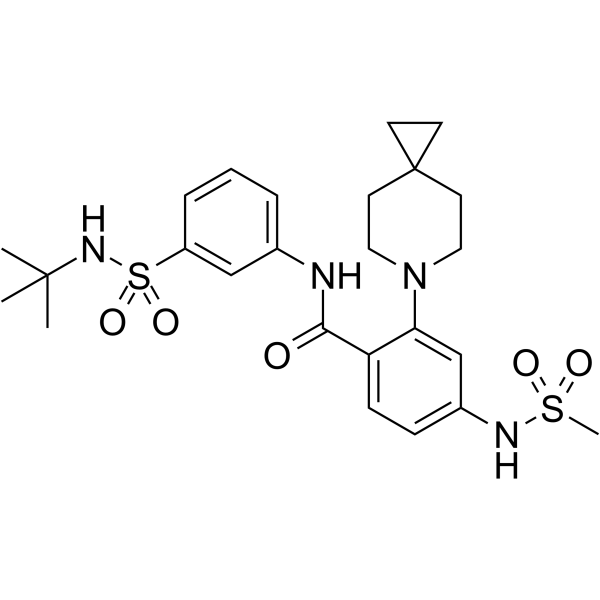
-
GC64857
KIF18A-IN-3
KIF18A-IN-3 es un potente inhibidor de KIF18A (IC50=61 nM). KIF18A-IN-3 causa un arresto mitÓtico significativo y aumenta el nÚmero de células mitÓticas en los tejidos tumorales. KIF18A-IN-3 se puede utilizar para investigar el cÁncer.
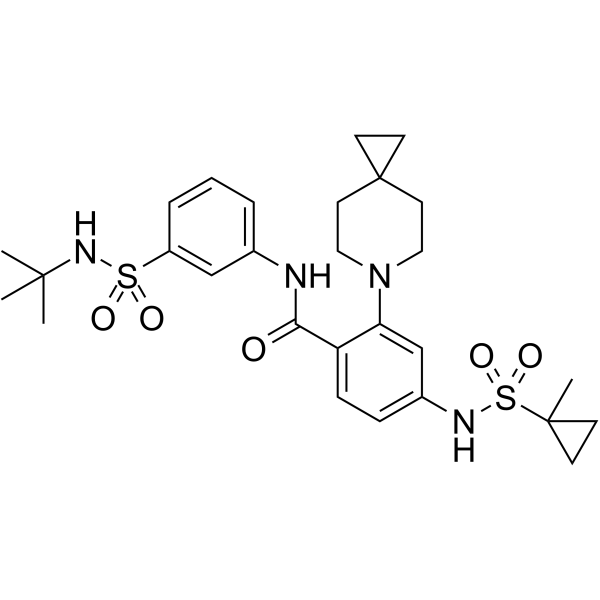
-
GC69333
KIF18A-IN-7
KIF18A-IN-7 (Compuesto 22) es un inhibidor de KIF18A con actividad oral, que inhibe la actividad ATPasa dependiente de microtúbulos de KIF18A, con una IC50 de 9.4 nM.
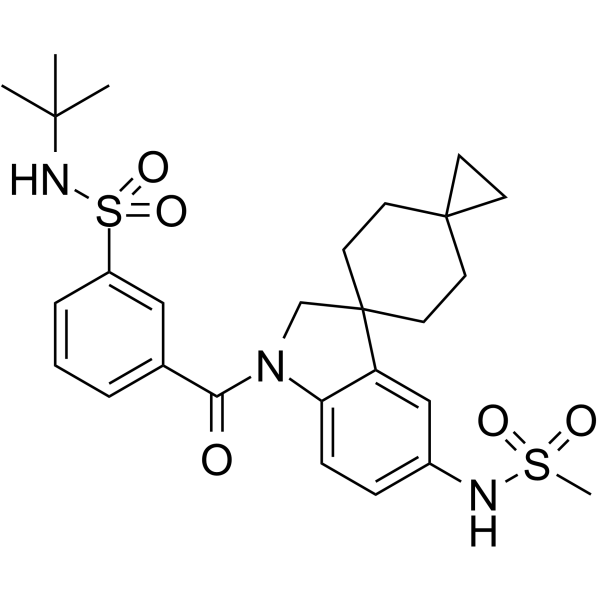
-
GC14413
KJ Pyr 9
KJ Pyr 9 es un inhibidor de MYC con una Kd de 6,5 nM en un ensayo in vitro.
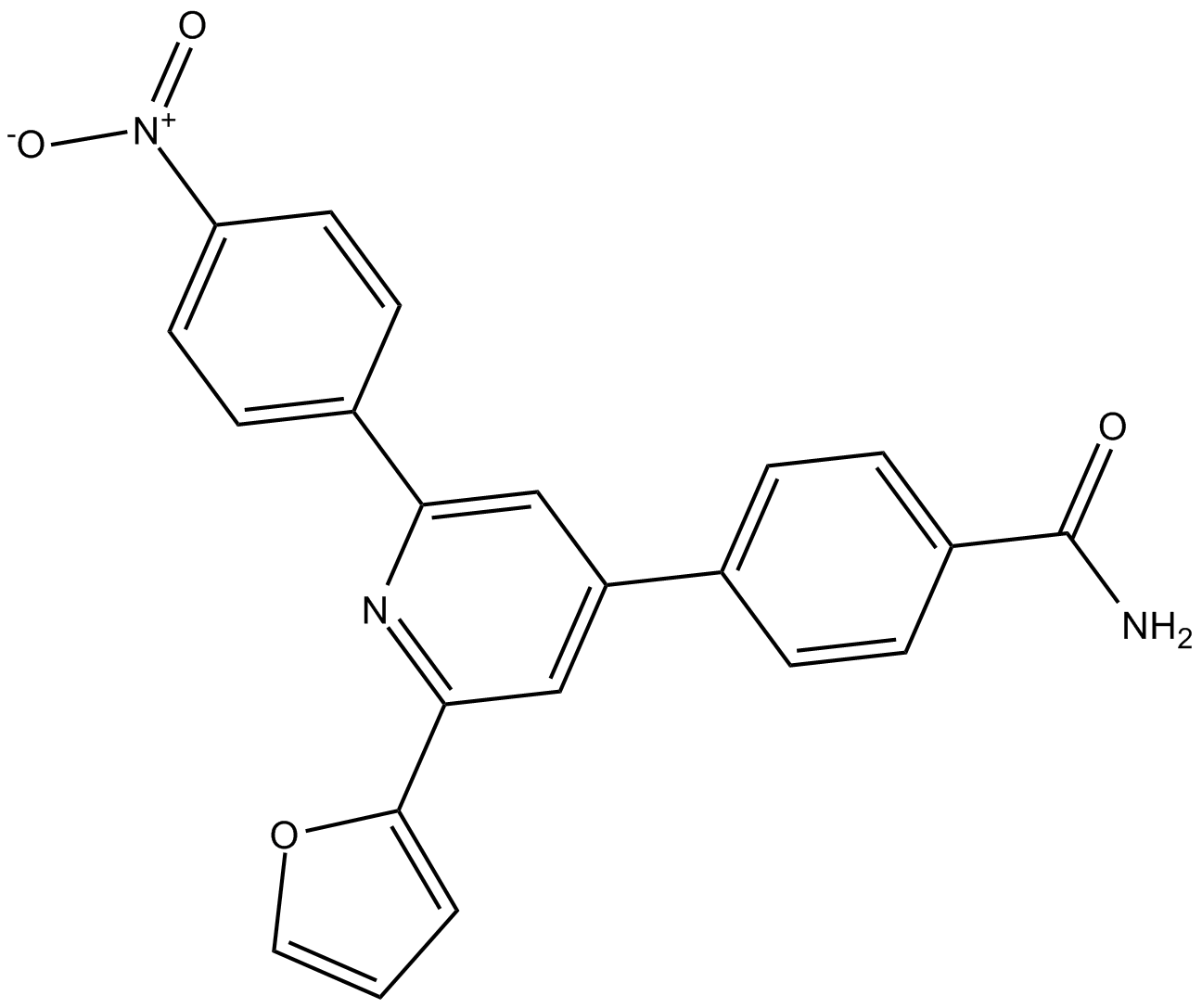
-
GC14517
KPT-185
CRM1 inhibitor,selective and irrversible
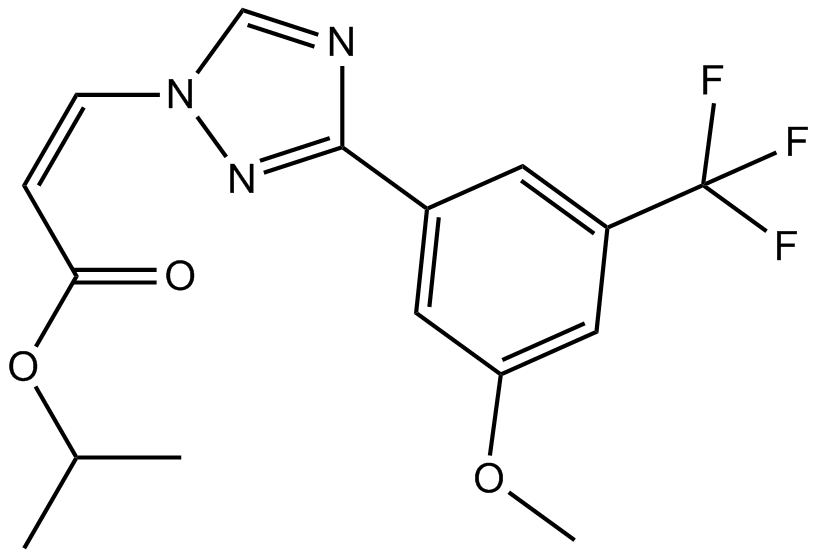
-
GC12142
KPT-276
inhibitor of nuclear export (SINE) and CRM1, orally bioavailable
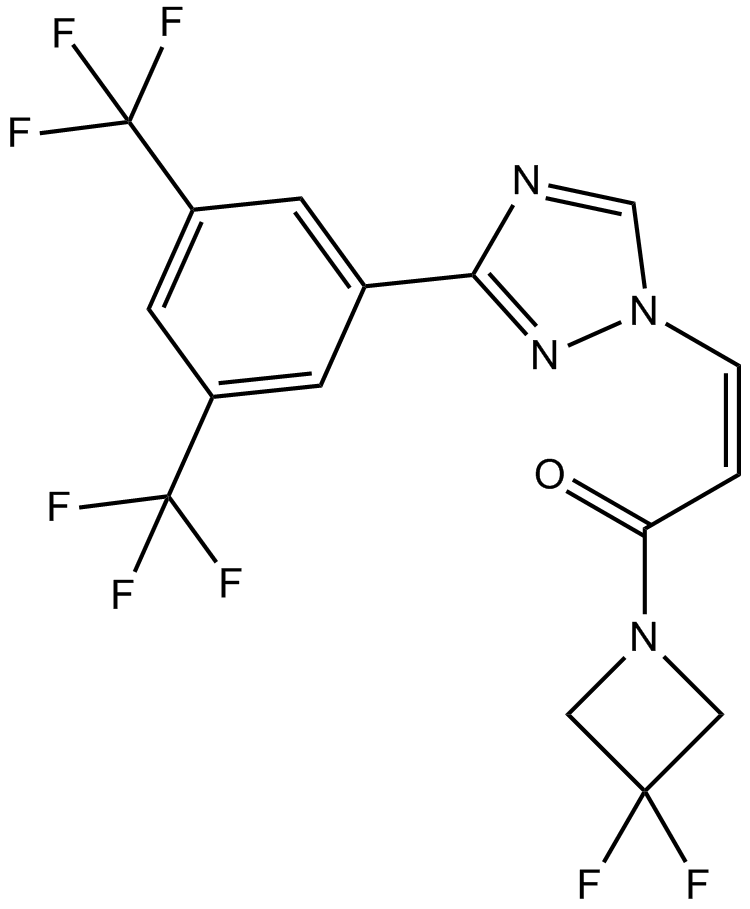
-
GC12467
KPT-330
Inhibidor de CRM1, oralmente biodisponible y selectivo.
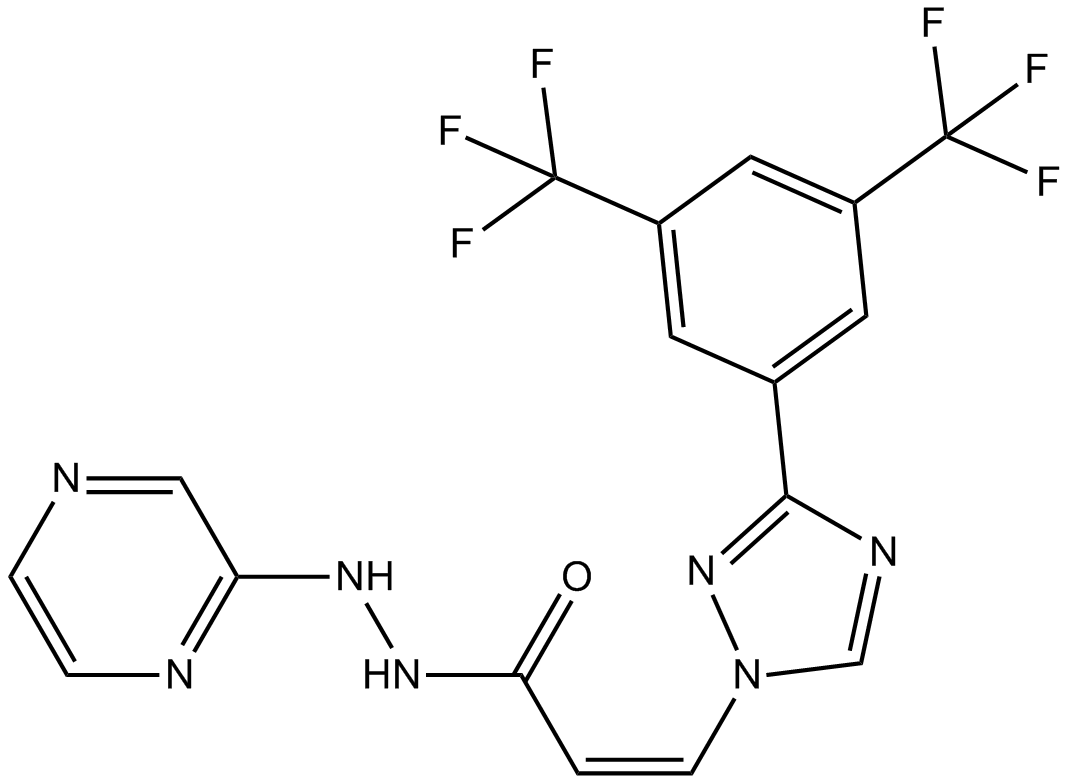
-
GC14615
KPT-9274
Orally acitve allosteric inhibitor of PAK4
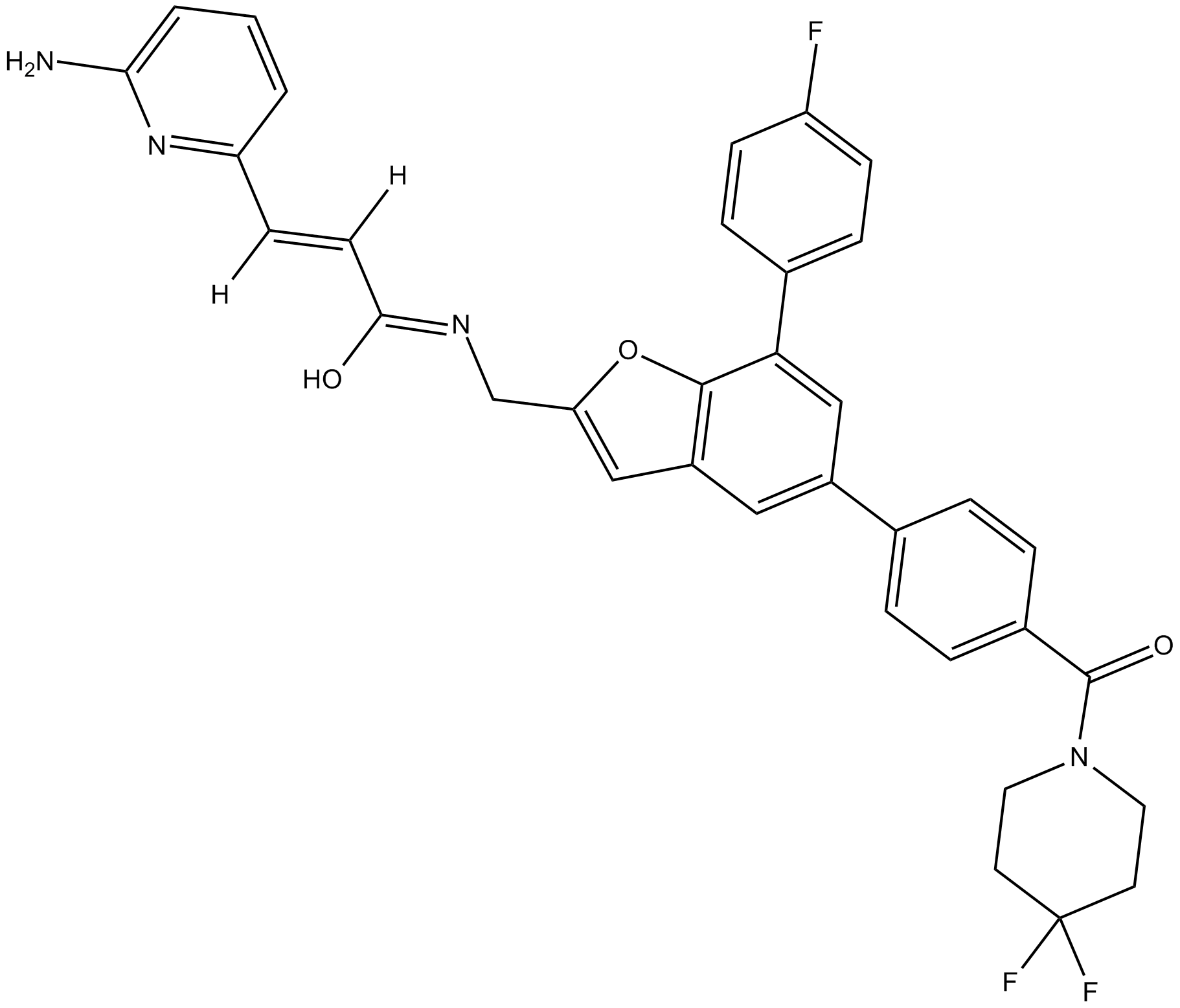
-
GC18909
KRIBB3
KRIBB3 is an Hsp27 and microtubule inhibitor that inhibits migration and invasion of MDA-MB-231 cells in vitro in an Hsp27-dependent manner.
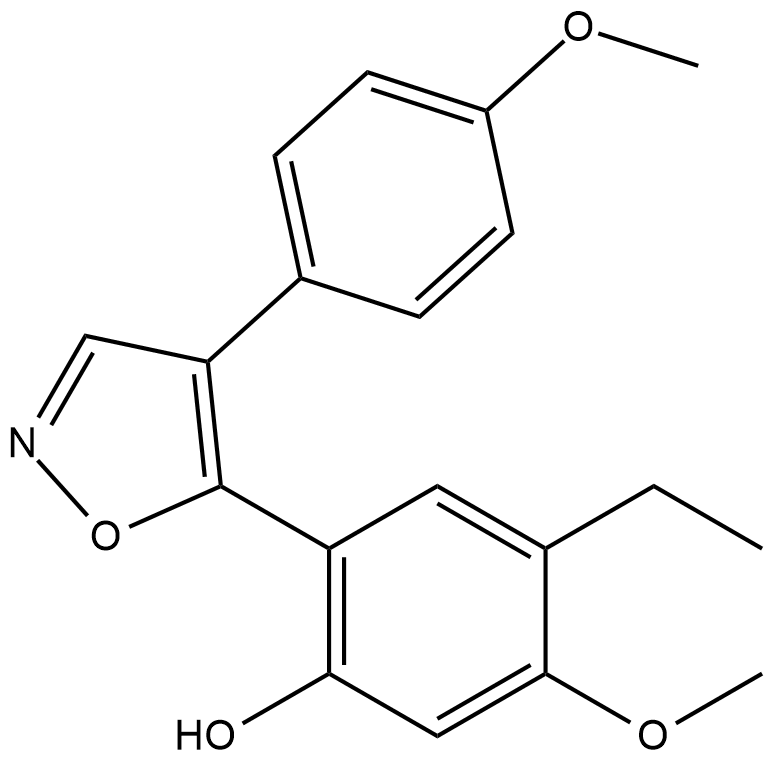
-
GC33377
KSI-3716
KSI-3716 es un potente inhibidor de c-Myc que bloquea la uniÓn de c-MYC/MAX a los promotores de genes diana. KSI-3716 es un agente de quimioterapia intravesical eficaz para el cÁncer de vejiga.
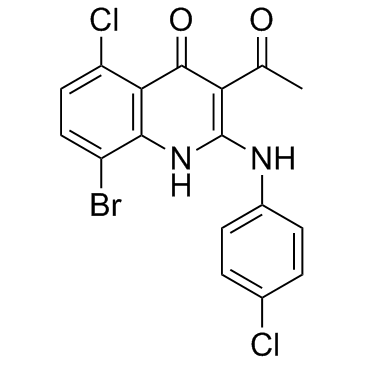
-
GC33404
KU 59403
KU 59403 es un potente inhibidor de ATM, con valores IC50 de 3 nM, 9,1 μM y 10 μM para ATM, DNA-PK y PI3K, respectivamente.
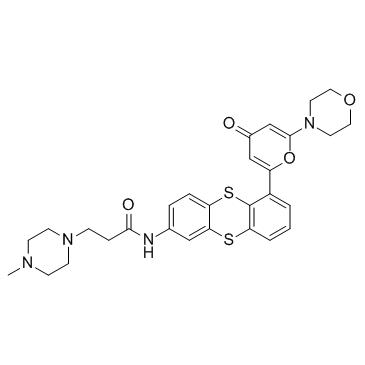
-
GC12054
KU-60019
KU-60019 es un inhibidor especÍfico de la quinasa ATM mejorado con IC50 de 6,3 nM.
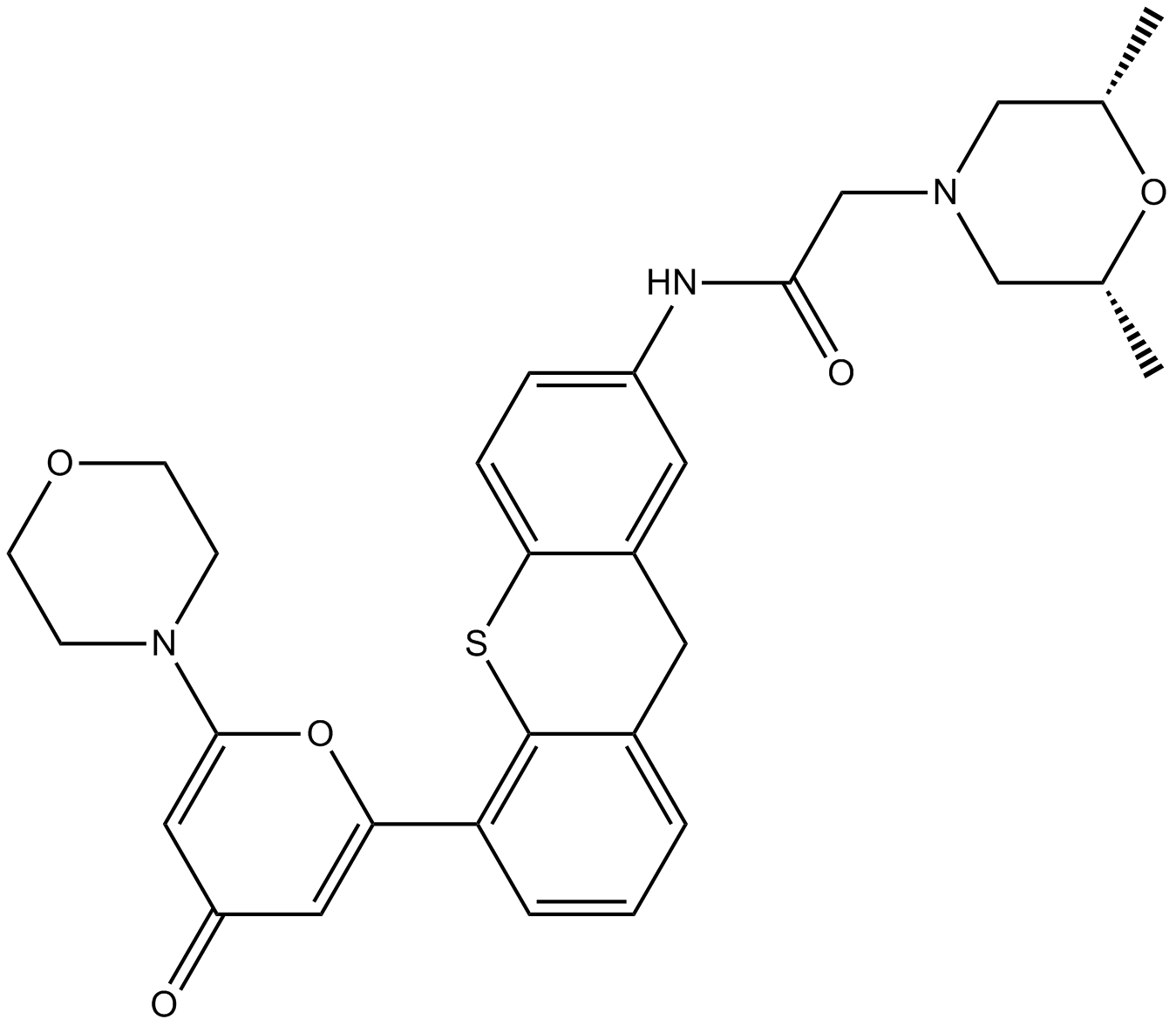
-
GC47532
KUS121
A VCP modulator
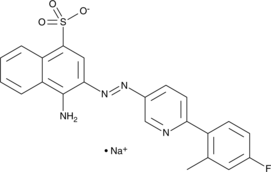
-
GC14288
KX2-391
KX2-391 (KX2-391) es un inhibidor de Src que se dirige al sitio del sustrato peptÍdico de Src, con GI50 de 9-60 nM en lÍneas celulares de cÁncer.
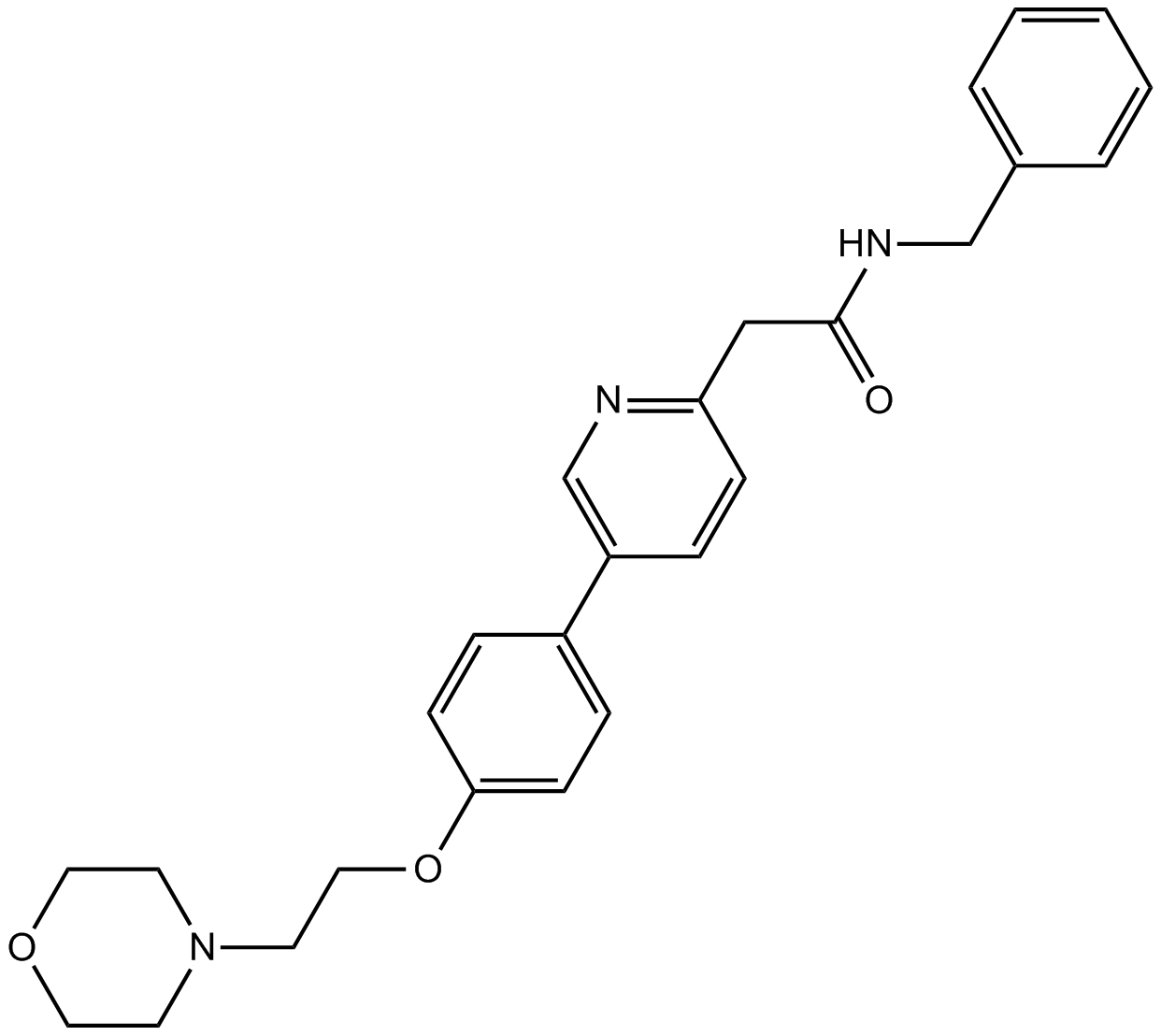
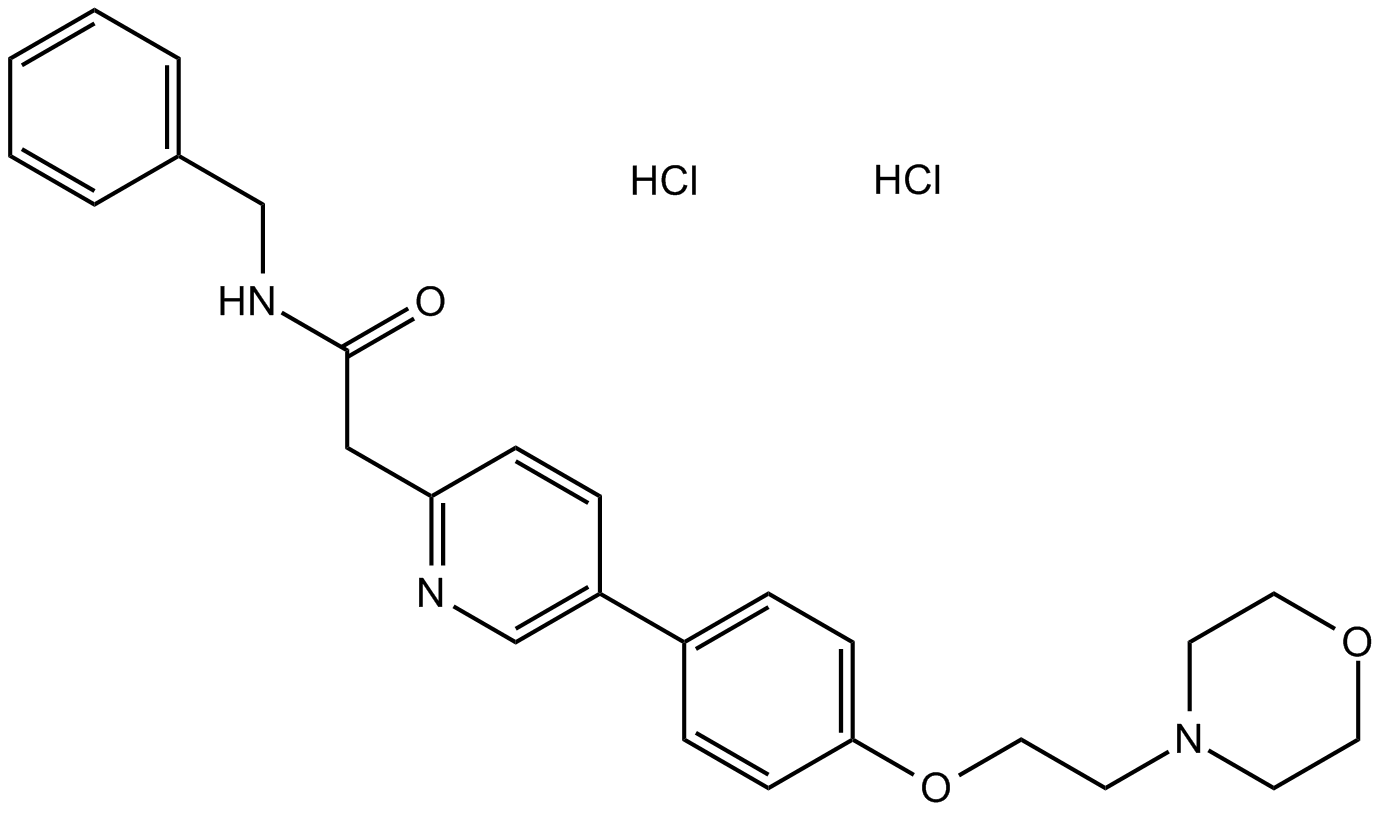
-
GC10222
KX2-391 dihydrochloride
A Src kinase inhibitor
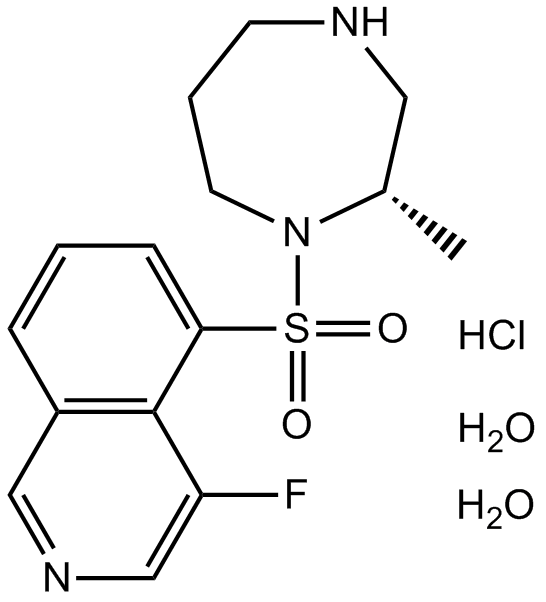
-
GC12817
K–115 hydrochloride dihydrate
El clorhidrato de K-115 dihidrato (K-115) es un inhibidor especÍfico de ROCK, con IC50 de 19 y 51 nM para ROCK2 y ROCK1, respectivamente.
-
GC44020
L-681,217
L-681,217 is an antibiotic originally isolated from S.
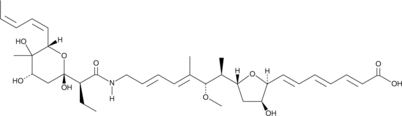
-
GC40793
L-erythro Sphinganine (d18:0)
L-erythro Sphinganine (d18:0) is a synthetic bioactive sphingolipid and stereoisomer of sphinganine (d18:0) and D-threo sphinganine.
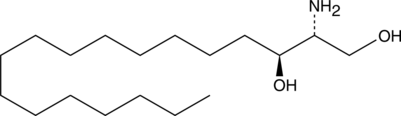
-
GC49547
L-Glutamic Acid γ-p-Nitroanilide (hydrate)
A colorimetric substrate for γ-glutamyl transpeptidase
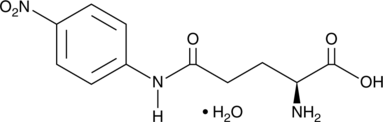
-
GC49205
L-Leu-AMC (hydrochloride)
El clorhidrato de L-leucina-7-amido-4-metilcumarina (Leu-AMC) es un sustrato peptÍdico fluorogénico azul brillante para LAP3 (leucina aminopeptidasa).
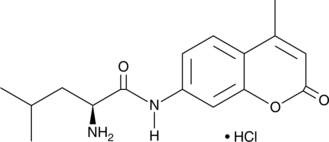
-
GC44081
L-myo-Inositol-1,4,5-triphosphate (sodium salt)
Ins(1,4,5)P3 is an isomer of the biologically important D-myo-inositol-1,4,5-triphosphate.
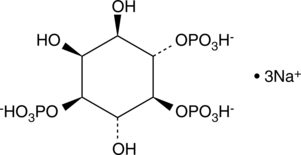
-
GC41520
Lachnone A
Lachnone A es un derivado de cromona aislado del hongo filamentoso Lachnum sp.
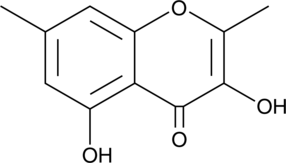
-
GC49471
LAMP1 Monoclonal Antibody (Clone Ly1C6)
For immunochemical analysis of LAMP1

-
GC41272
Lateropyrone
Lateropyrone is a fungal metabolite produced by Fusarium species.
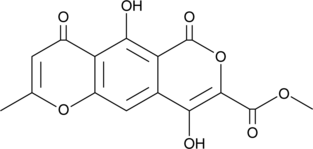
-
GC15671
Latrunculin A
Un inhibidor reversible del ensamblaje de actina.
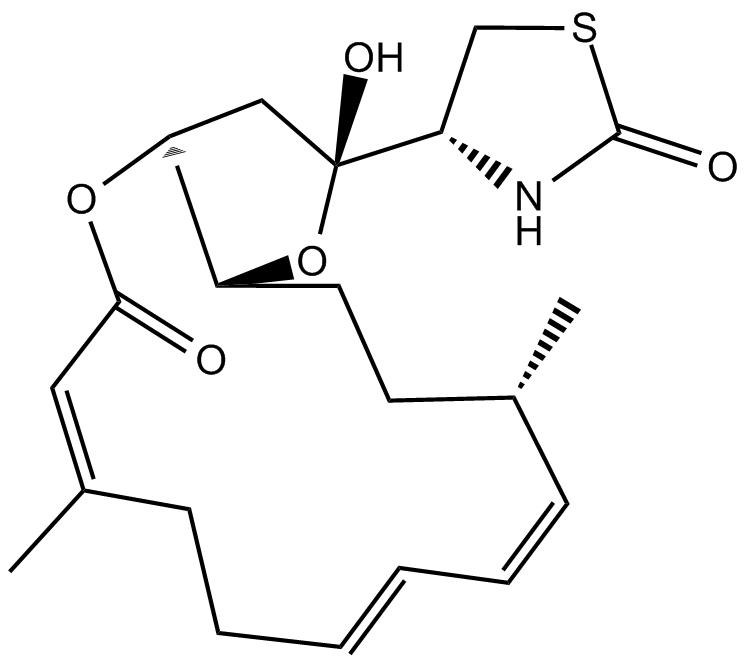
-
GC50052
Laulimalide
Microtubule stabilzing agent
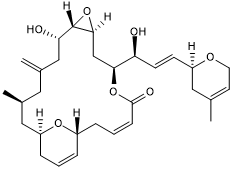
-
GC50214
LCB 03-0110 dihydrochloride
Potent c-SRC kinase inhibitor; also inhibits DDR2, BTK and Syk
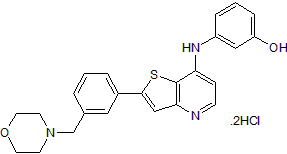
-
GC39209
LCH-7749944
LCH-7749944 (GNF-PF-2356) es un potente inhibidor de PAK4 con una IC50 de 14,93 μM. LCH-7749944 suprime eficazmente la proliferaciÓn de células de cÁncer gÁstrico humano a través de la regulaciÓn negativa de la vÍa PAK4/c-Src/EGFR/ciclina D1 e induce la apoptosis.
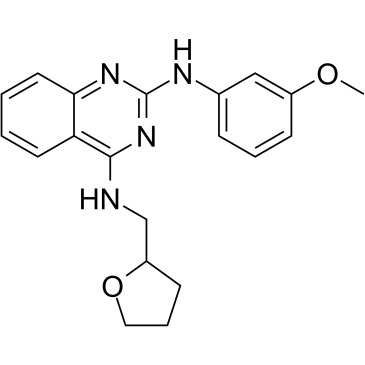
-
GC17067
LDC000067
LDC000067 es un inhibidor de CDK9 altamente especÍfico con un valor IC50 de 44±10 nM in vitro.
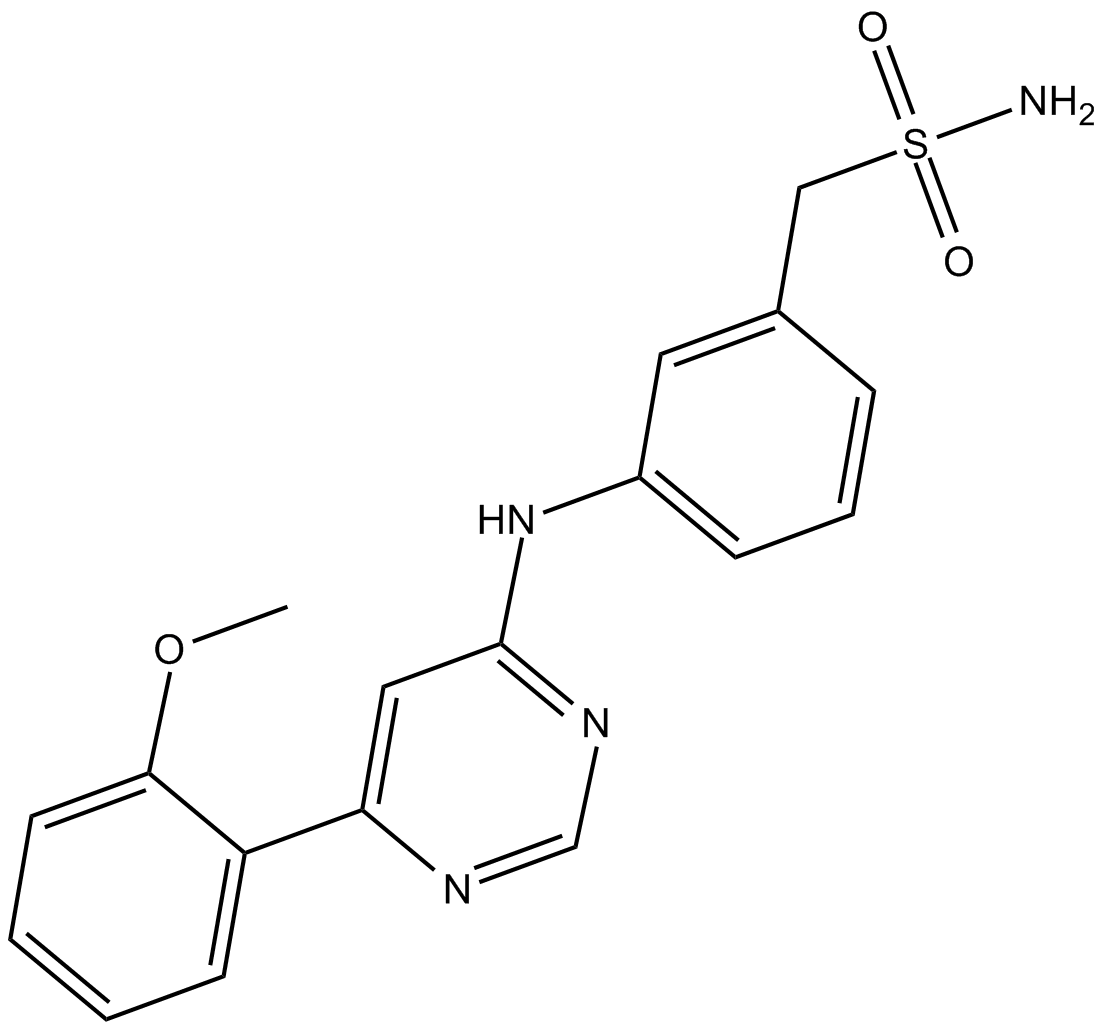
-
GC33086
LDN-192960
LDN-192960 es un inhibidor de la quinasa 2 regulada por tirosina de especificidad doble (DYRK2) y Haspin con IC50 de 10 nM y 48 nM, respectivamente.
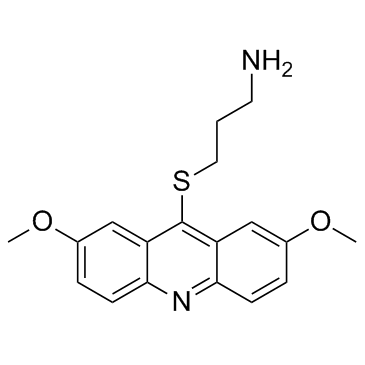
-
GC44047
LDN-192960 (hydrochloride)
LDN-192960 is an inhibitor of haspin protein kinase and dual-specificity tyrosine-(Y)-phosphorylation regulated kinase 2 (DYRK2) with IC50 values of 10 and 48 nM, respectively.
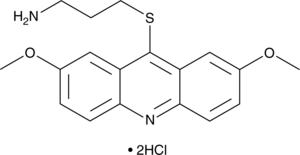
-
GC38403
LDN-192960 hydrochloride
El clorhidrato de LDN-192960 es un inhibidor de la quinasa 2 regulada por tirosina de especificidad dual (DYRK2) y Haspin con IC50 de 10 nM y 48 nM, respectivamente.
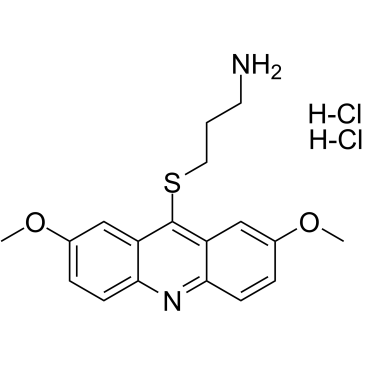
-
GC10842
LEE011
LEE011 (LEE01) es un inhibidor de CDK4/6 altamente especÍfico con valores IC50 de 10 nM y 39 nM, respectivamente, y es 1000 veces menos potente contra el complejo ciclina B/CDK1.
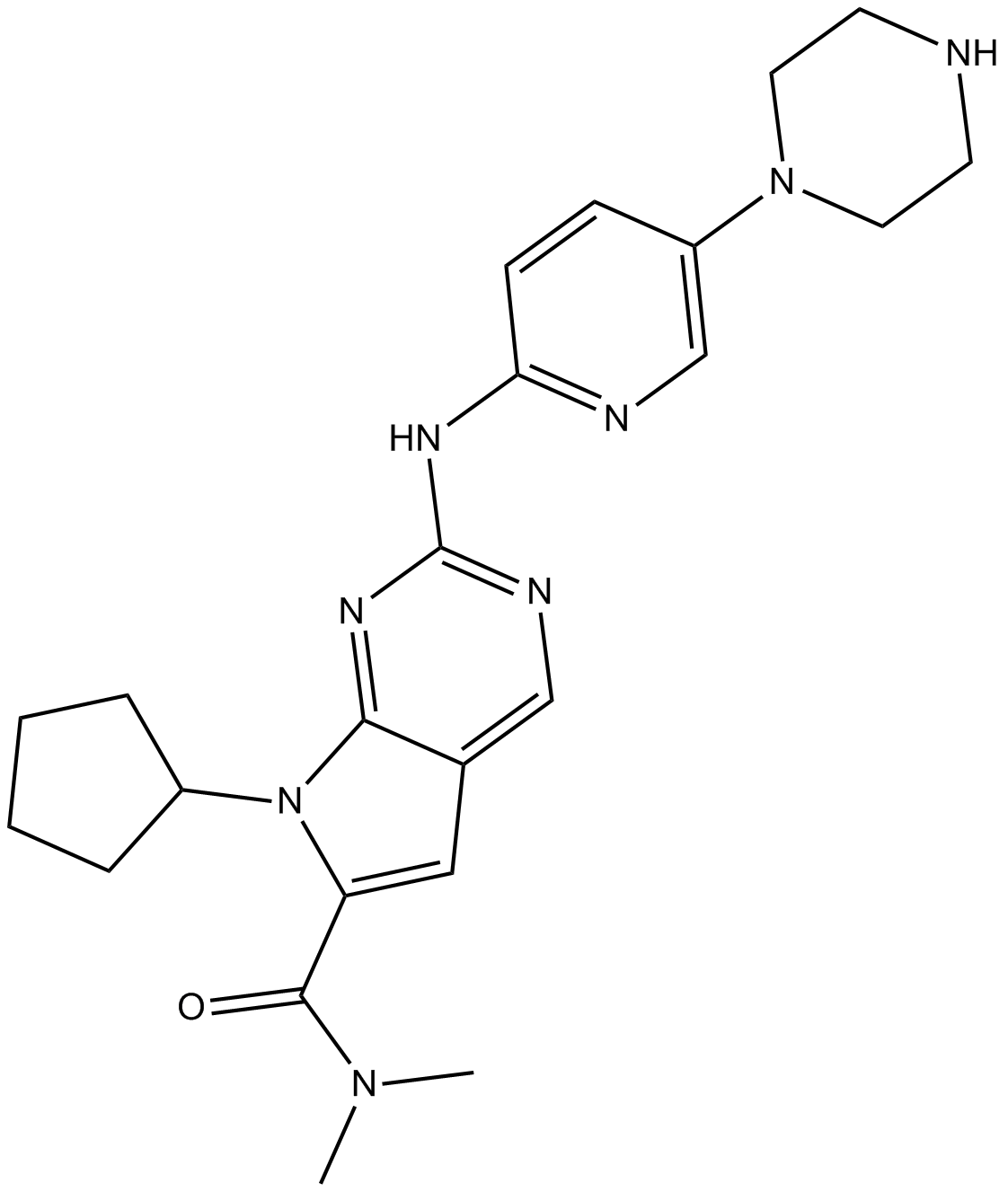
-
GC15922
LEE011 hydrochloride
El clorhidrato de LEE011 (clorhidrato de LEE011) es un inhibidor altamente especÍfico de CDK4/6 con valores de CI50 de 10 nM y 39 nM, respectivamente, y es mÁs de 1000 veces menos potente contra el complejo ciclina B/CDK1.
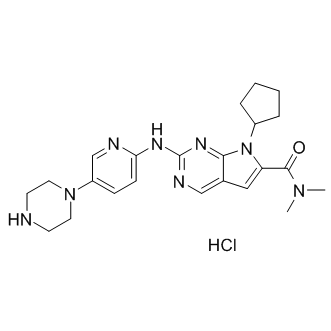
-
GC15377
LEE011 succinate
El succinato LEE011 (succinato LEE011) es un inhibidor altamente especÍfico de CDK4/6 con valores IC50 de 10 nM y 39 nM, respectivamente, y es 1000 veces menos potente contra el complejo ciclina B/CDK1.
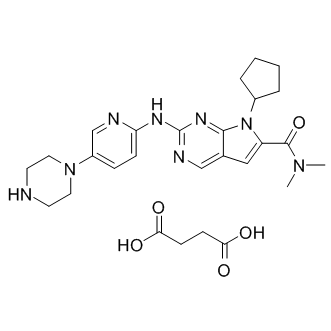
-
GC49751
Lefamulin
La lefamulina (BC-3781) es un antibiÓtico activo por vÍa oral.
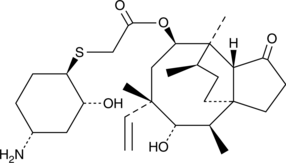
-
GC18345
Leptomycin A
La leptomicina A, un metabolito de Streptomyces, es un inhibidor de CRM1 (exportina 1) que bloquea la interacciÓn de CRM1 con las seÑales de exportaciÓn nuclear, impidiendo la exportaciÓn nuclear de una amplia gama de proteÍnas.
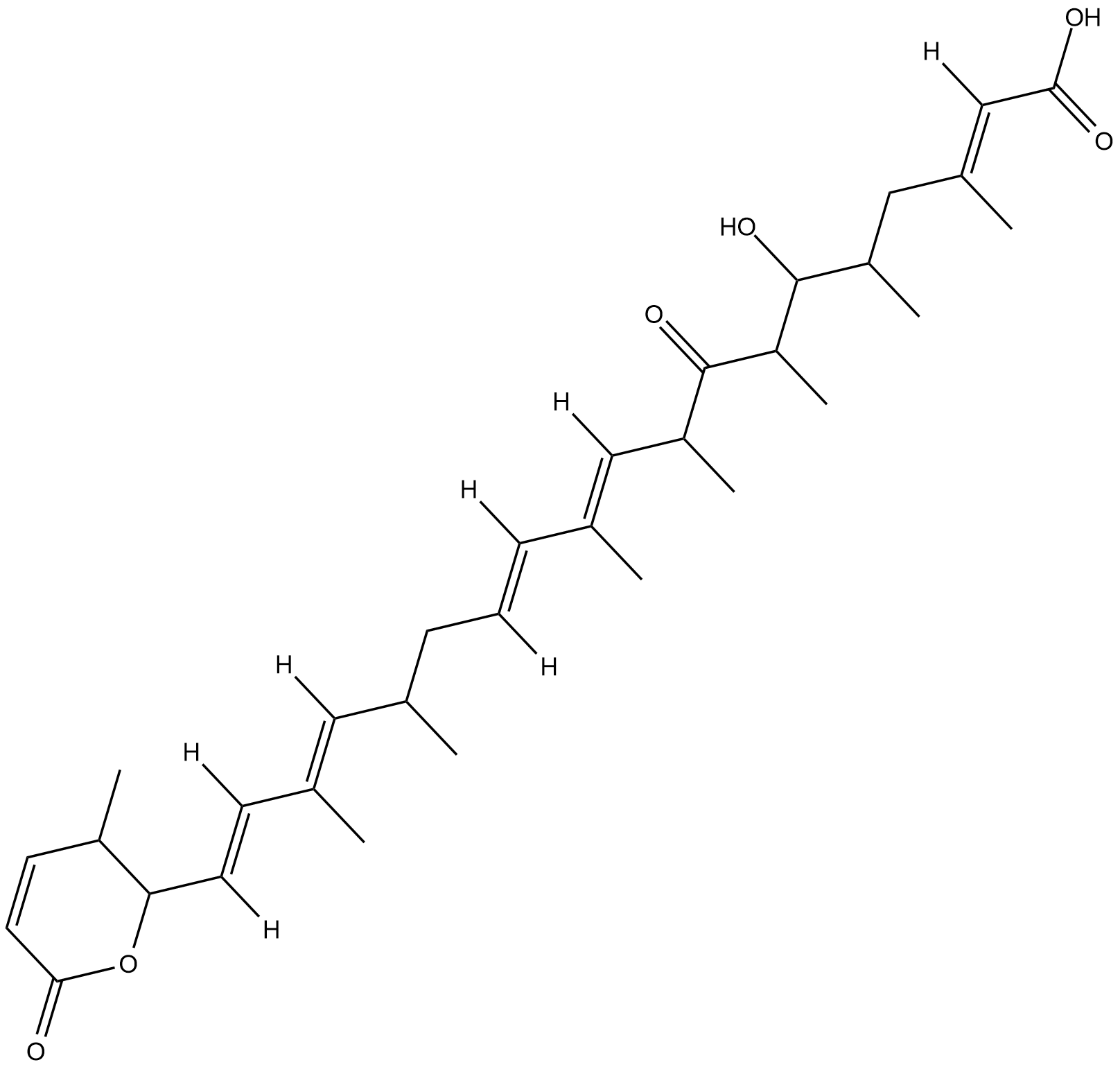
-
GC10954
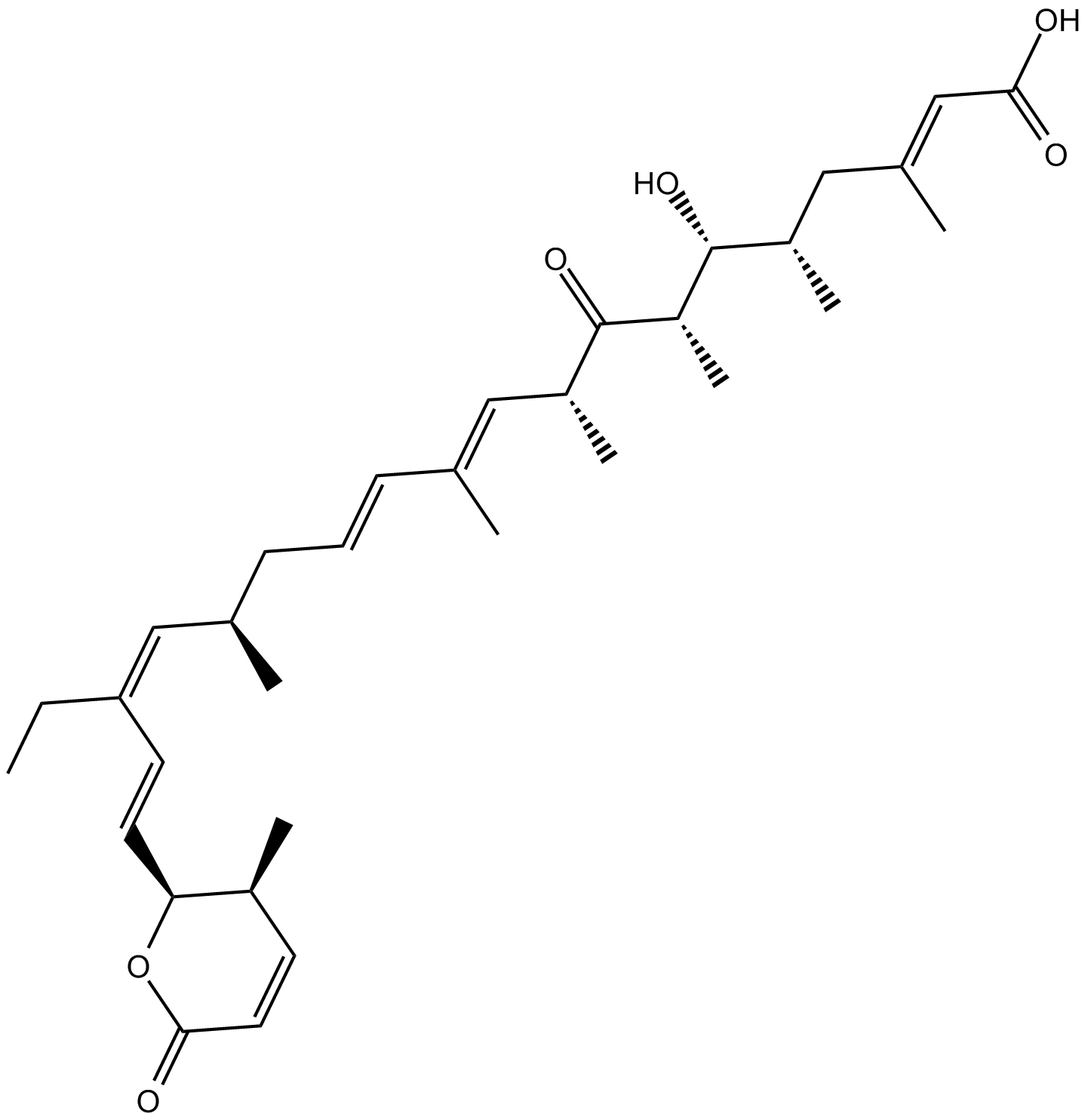
Leptomycin B
Leptomycin B (LMB) es un inhibidor de la proteína exportadora nuclear 1 de mantenimiento de la región cromosómica (CRM1).
-
GC33175
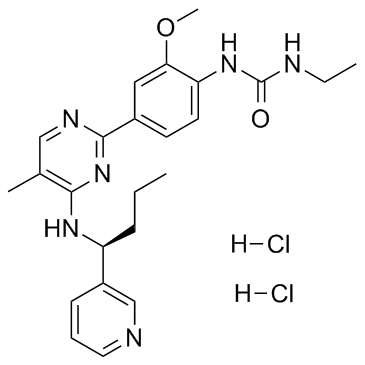
Lexibulin dihydrochloride (CYT-997 dihydrochloride)
El diclorhidrato de lexibulina (diclorhidrato de CYT-997) (diclorhidrato de CYT-997) es un inhibidor de la polimerizaciÓn de tubulina potente y activo por vÍa oral con IC50 de 10-100 nM en lÍneas de células cancerosas; con potente actividad citotÓxica y disruptiva vascular in vitro e in vivo. El diclorhidrato de lexibulina (diclorhidrato de CYT-997) induce la apoptosis celular e induce la generaciÓn de ROS mitocondrial en células GC.
-
GC14857
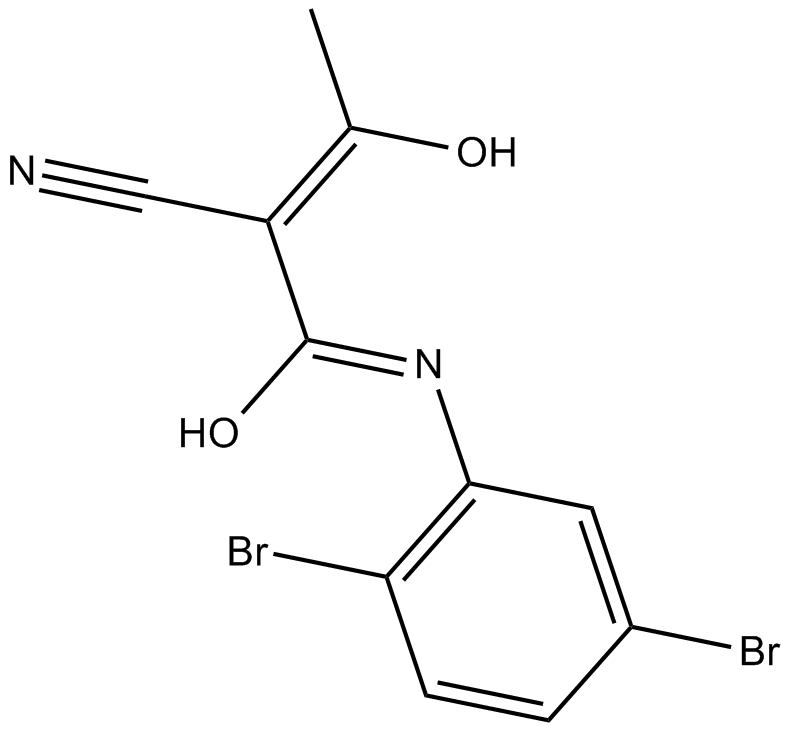
LFM-A13
LFM-A13 es un potente inhibidor de BTK, JAK2, PLK, inhibe BTK, Plx1 y PLK3 recombinantes con IC50 de 2,5 μM, 10 μM y 61 μM; LFM-A13 no muestra efectos sobre JAK1 y JAK3, la quinasa de la familia Src HCK, EGFR e IRK.
-
GC31803
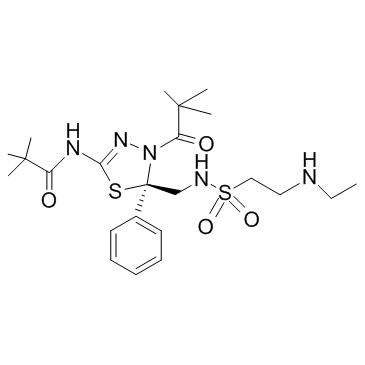
Litronesib (LY-2523355)
Litronesib (LY-2523355) (LY2523355) es un inhibidor selectivo de la cinesina Eg5 especÍfico de la mitosis, con actividad antitumoral.
-
GC30393
Litronesib Racemate (LY-2523355 Racemate)
El racemato de litronesib (LY-2523355 Racemate) (LY2523355 Racemate) es el racemato de litronesib.
-
GC61014
Lusianthridin
La lusiantridina, un compuesto puro de Dendrobium venustum, tiene un efecto antimigratorio. La lusiantridina mejora la degradaciÓn de c-Myc a través de la inhibiciÓn de la seÑalizaciÓn de Src-STAT3.
-
GC12402
LX7101 HCL
LX7101 HCL es un potente inhibidor de LIMK y ROCK2 con valores IC50 de 24, 1,6 y 10 nM para LIMK1, LIMK2 y ROCK2, respectivamente; también inhibe la PKA con una IC50 inferior a 1 nM.
-
GC15741
LY2603618
LY2603618 (LY2603618) es un inhibidor potente y selectivo de Chk1 con un IC50 de 7 nM.