TGF-β / Smad Signaling
Transforming growth factor-beta (TGF-beta) is a multifunctional cytokine that regulates proliferation, migration, differentiation, and survival of many different cell types. Deletion or mutation of different members of the TGF-β family have been shown to cause vascular remodeling defect and absence of mural cell formation, leading to embryonic lethality or severe vascular disorders. TGF-β induces smooth muscle differentiation via Notch or SMAD2 and SMAD3 signaling in ES cells or in a neural crest stem cell line. TGF-β binds to TGF-βRI and to induce phosphorylation of SMAD2/3, thereby inhibiting proliferation, tube formation, and migration of endothelial cells (ECs).
TGF-β is a pluripotent cytokine with dual tumour-suppressive and tumour-promoting effects. TGF-β induces the epithelial-to-mesenchymal transition (EMT) leading to increased cell plasticity at the onset of cancer cell invasion and metastasis.
Targets for TGF-β / Smad Signaling
Products for TGF-β / Smad Signaling
- Cat.No. Nombre del producto Información
-
GC15079
GNF 5
GNF 5, el análogo de N-hidroxietil carboxamida de GNF-2, es un inhibidor de Bcr-Abl activo por vía oral. GNF 5 tiene actividad de inhibición de Bcr-Abl con un valor IC50 de 0,22 µM. GNF 5 tiene buenas propiedades farmacocinéticas favorables. GNF 5 se puede utilizar para la investigación de tipos de cáncer, incluida la leucemia mielógena crónica (LMC) y el cáncer de mama.
-
GC10607
GNF-7
GNF-7 es un inhibidor multiquinasa. GNF-7 es un inhibidor de Bcr-Abl, con IC50 de 133 nM y 61 nM para Bcr-AblWT y Bcr-AblT315I, respectivamente. GNF-7 también posee actividad inhibidora frente a ACK1 (cinasa 1 de CDC42 activada) y GCK (cinasa del centro germinal) con IC50 de 25 nM y 8 nM, respectivamente. GNF-7 se puede utilizar para la investigaciÓn de neoplasias malignas hematolÓgicas.
-
GC15564
Go 6976
Go 6769 es un inhibidor de la Proteína Quinasa C (PKC), con una IC50 de 20 nM.
-
GC16907
Go 6983
Go 6983 (GÖ 6983) es uno del grupo bisindolilmaleimida de compuestos inhibidores de PKC, Go 6983 (GÖ 6983) pudo diferenciar entre PKC mu y otras isoenzimas de PKC.
-
GC38791
GSK-25
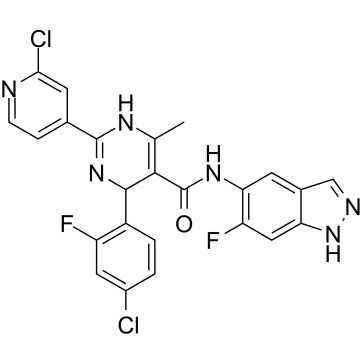
GSK-25 es un inhibidor de ROCK1 potente, selectivo y biodisponible por vÍa oral (IC50 = 7 nM).
-
GC19177
GSK180736A
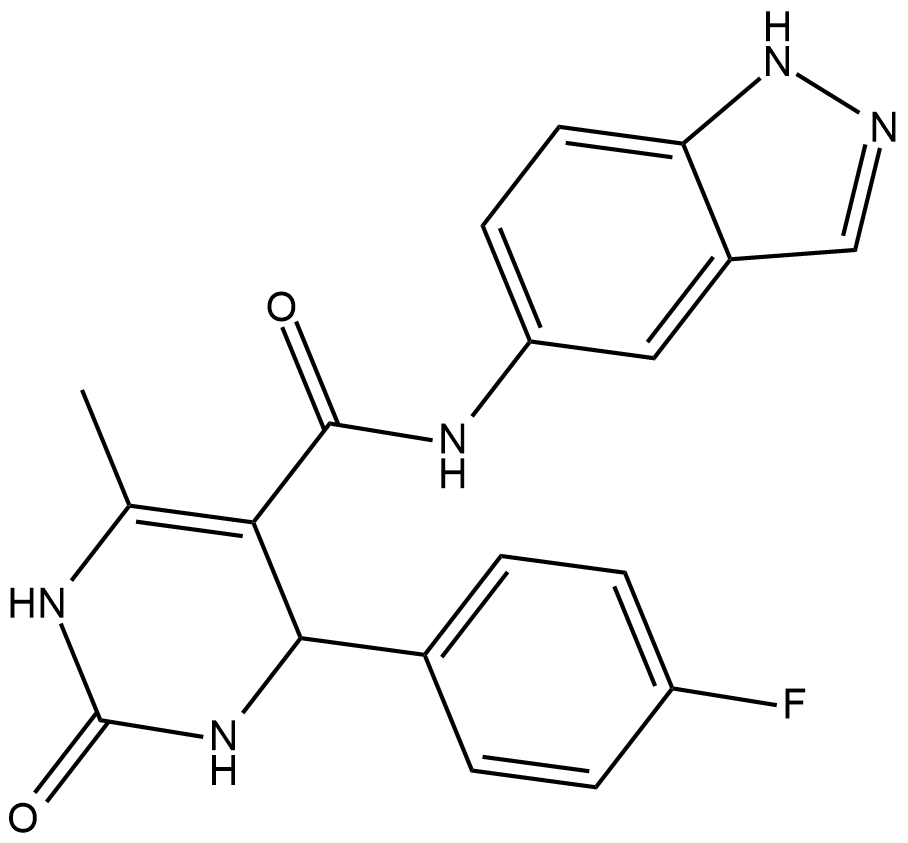
GSK180736A es un potente inhibidor de la quinasa 1 en espiral asociada a Rho (ROCK1) con una IC50 de 100 nM.
-
GC17936
GSK269962A
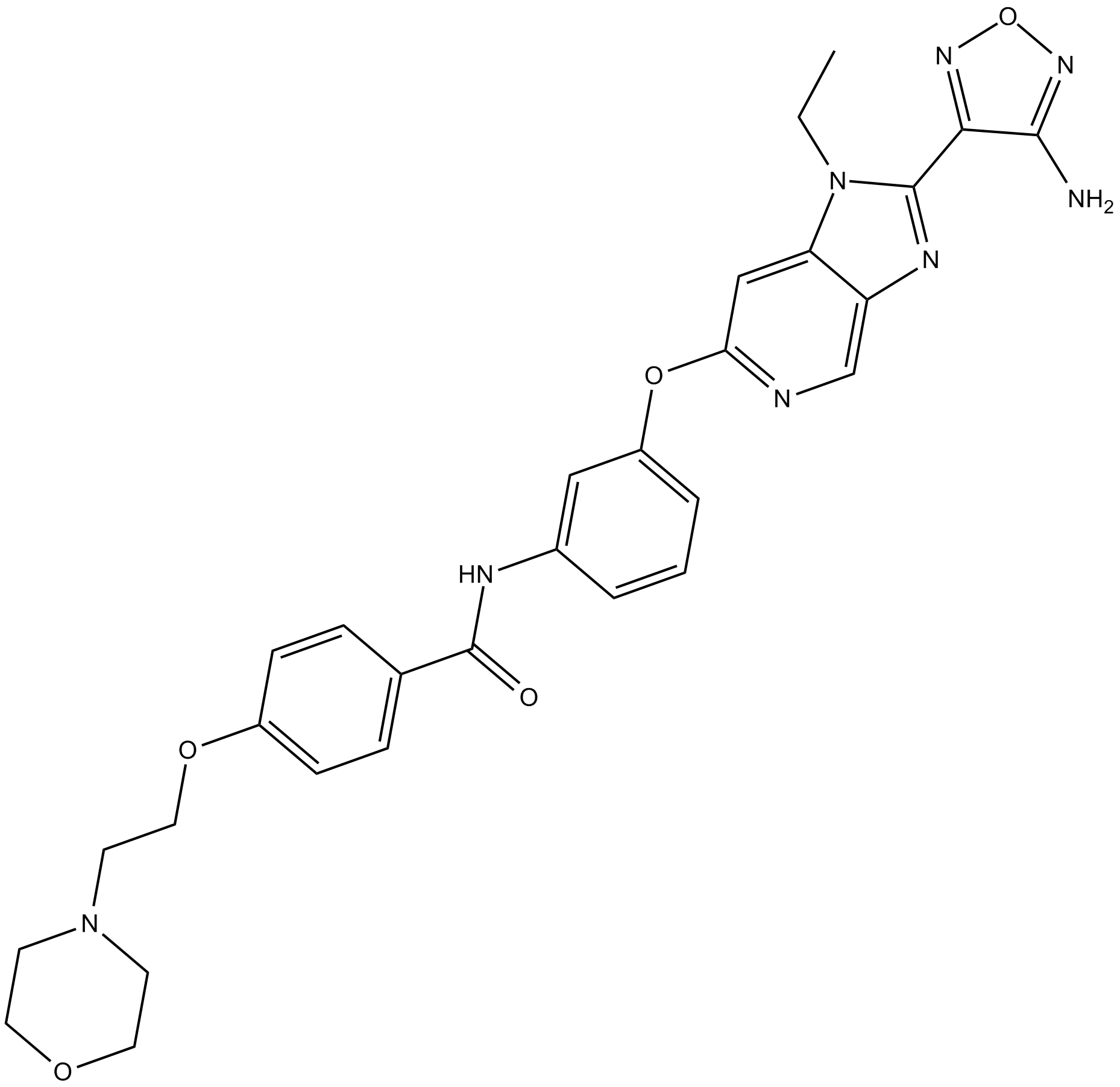
GSK269962A (GSK 269962) es un potente inhibidor de ROCK con IC50 de 1,6 y 4 nM para ROCK1 y ROCK2 humanos recombinantes, respectivamente.
-
GC25482
GSK269962A HCl
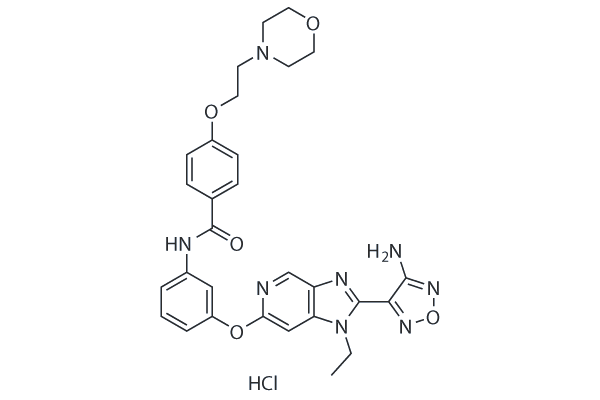
GSK269962A HCl (GSK269962B, GSK269962) is a selective ROCK(Rho-associated protein kinase) inhibitor with IC50 values of 1.6 and 4 nM for ROCK1 and ROCK2, respectively.
-
GC18119
GSK429286A
GSK429286A es un inhibidor selectivo de ROCK1 con un valor IC50 de 14 nM.
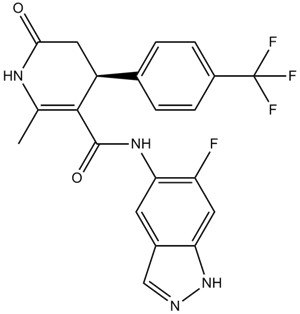
-
GC11878
GW788388
GW788388 es un inhibidor potente y selectivo de ALK5 con IC50 de 18 nM, y también inhibe las actividades del receptor de TGF-β tipo II y del receptor de activina tipo II, sin inhibir el receptor de BMP tipo II.
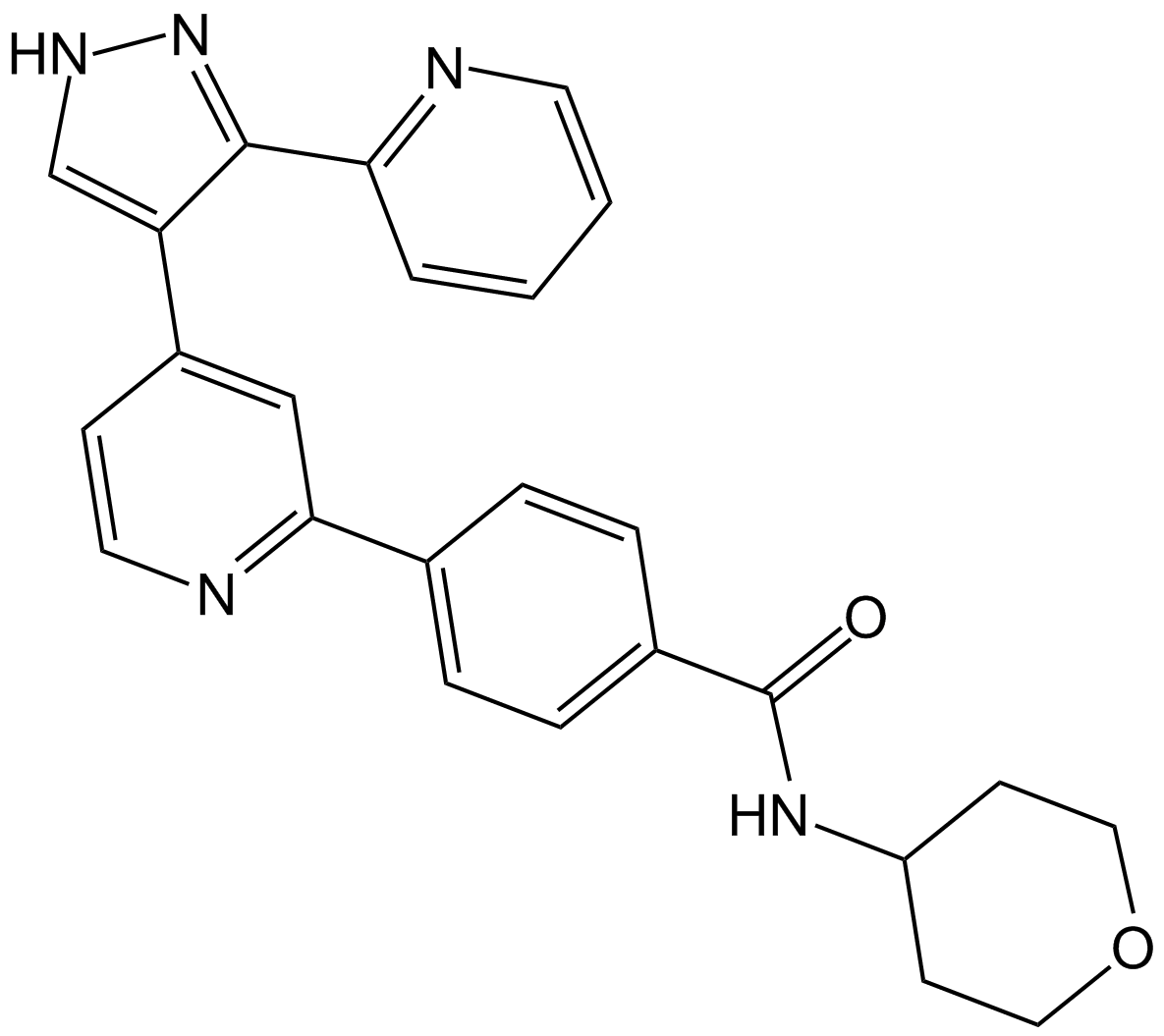
-
GC10915
GZD824
El dimesilato de olverembatinib (GZD824) es un inhibidor pan-Bcr-Abl potente y activo por vÍa oral. GZD824 inhibe potentemente un amplio espectro de mutantes Bcr-Abl. GZD824 inhibe fuertemente Bcr-Abl y Bcr-AblT315I nativos con IC50 de 0,34 nM y 0,68 nM, respectivamente. GZD824 tiene actividad antitumoral.
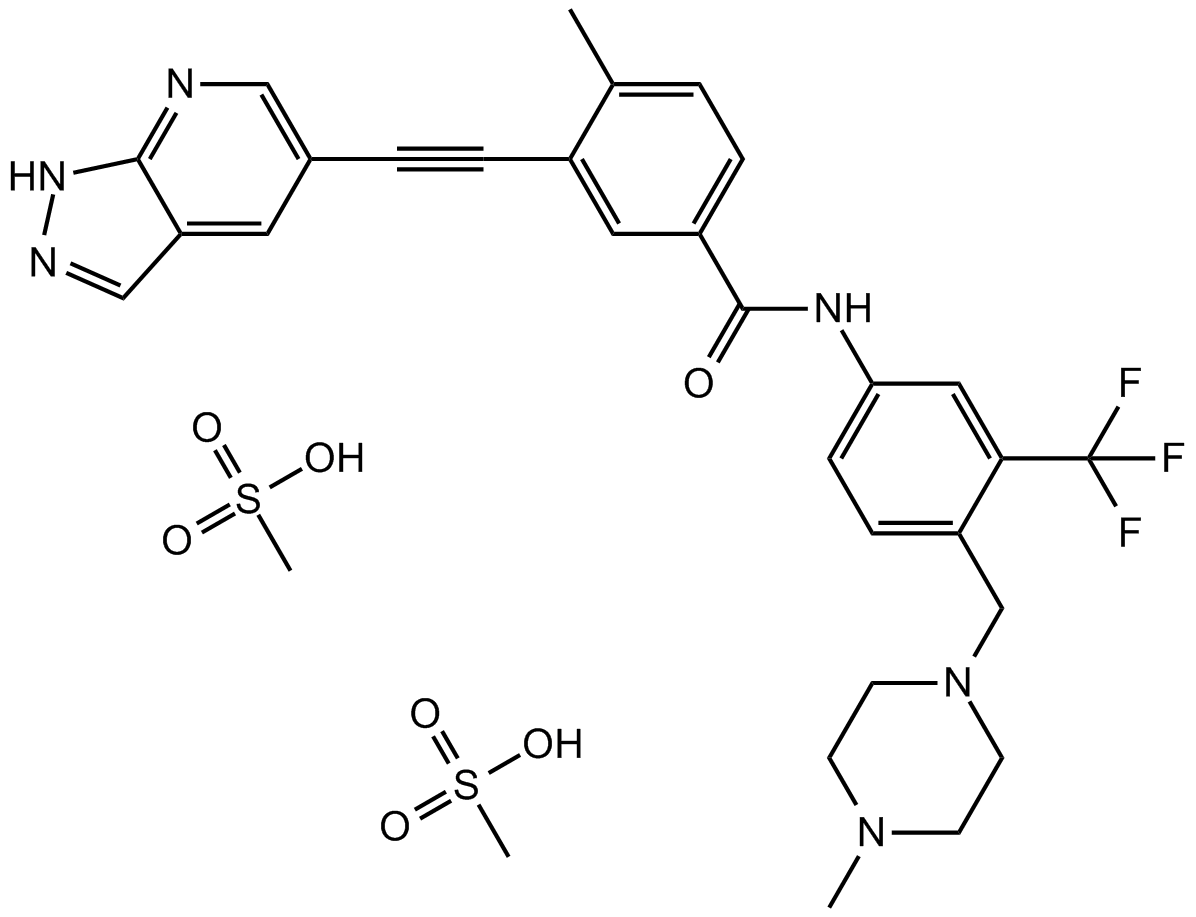
-
GC33201
GZD856
GZD856 fÓrmico es un inhibidor de PDGFRα/β potente y activo por vÍa oral, con IC50 de 68,6 y 136,6 nM, respectivamente. El fÓrmico GZD856 también es un inhibidor de Bcr-AblT315I, con IC50 de 19,9 y 15,4nM para Bcr-Abl nativo y el mutante T315I. El fÓrmico GZD856 tiene actividad antitumoral.
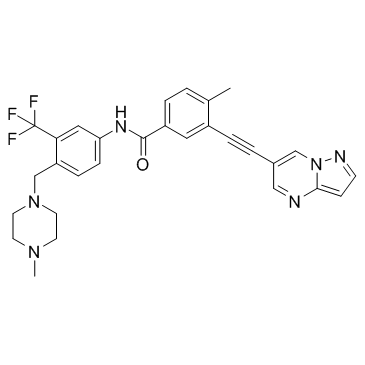
-
GC36207
H-1152
H-1152 es un inhibidor de ROCK selectivo y permeable a la membrana, con un valor Ki de 1,6 nM y un valor IC50 de 12 nM para ROCK2.
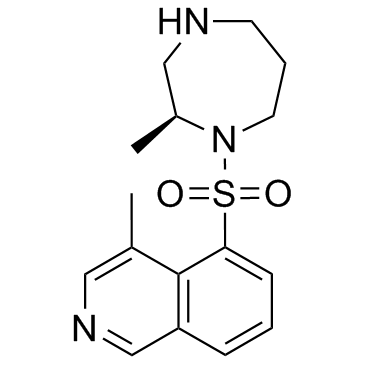
-
GC36208
H-1152 dihydrochloride
El diclorhidrato de H-1152 es un inhibidor de ROCK selectivo y permeable a la membrana, con un valor Ki de 1,6 nM y un valor IC50 de 12 nM para ROCK2.
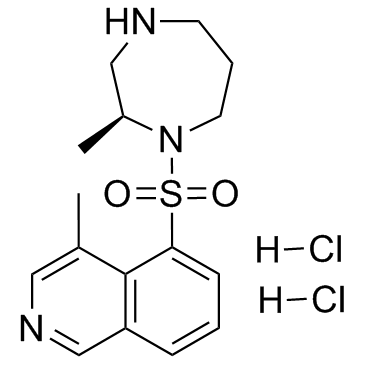
-
GC31650
Halofuginone (RU-19110)
La halofuginona (RU-19110) (RU-19110), un derivado de la febrifugina, es un inhibidor competitivo de la prolil-tRNA sintetasa con una Ki de 18,3 nM.
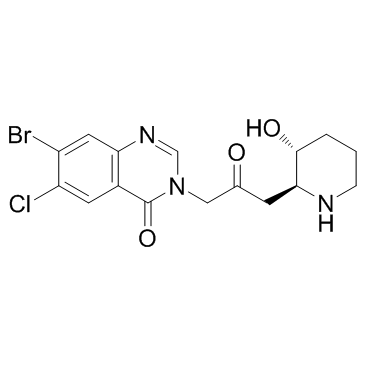
-
GC31950
Halofuginone hydrobromide (RU-19110 (hydrobromide))
El bromhidrato de halofuginona (RU-19110), un derivado de la febrifugina, es un inhibidor competitivo de la prolil-tRNA sintetasa con una Ki de 18,3 nM.
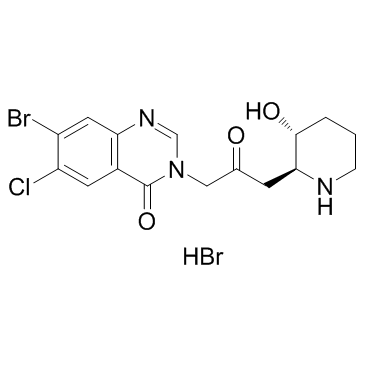
-
GC10299
Hexadecyl Methyl Glycerol
protein kinase C activity inhibitor
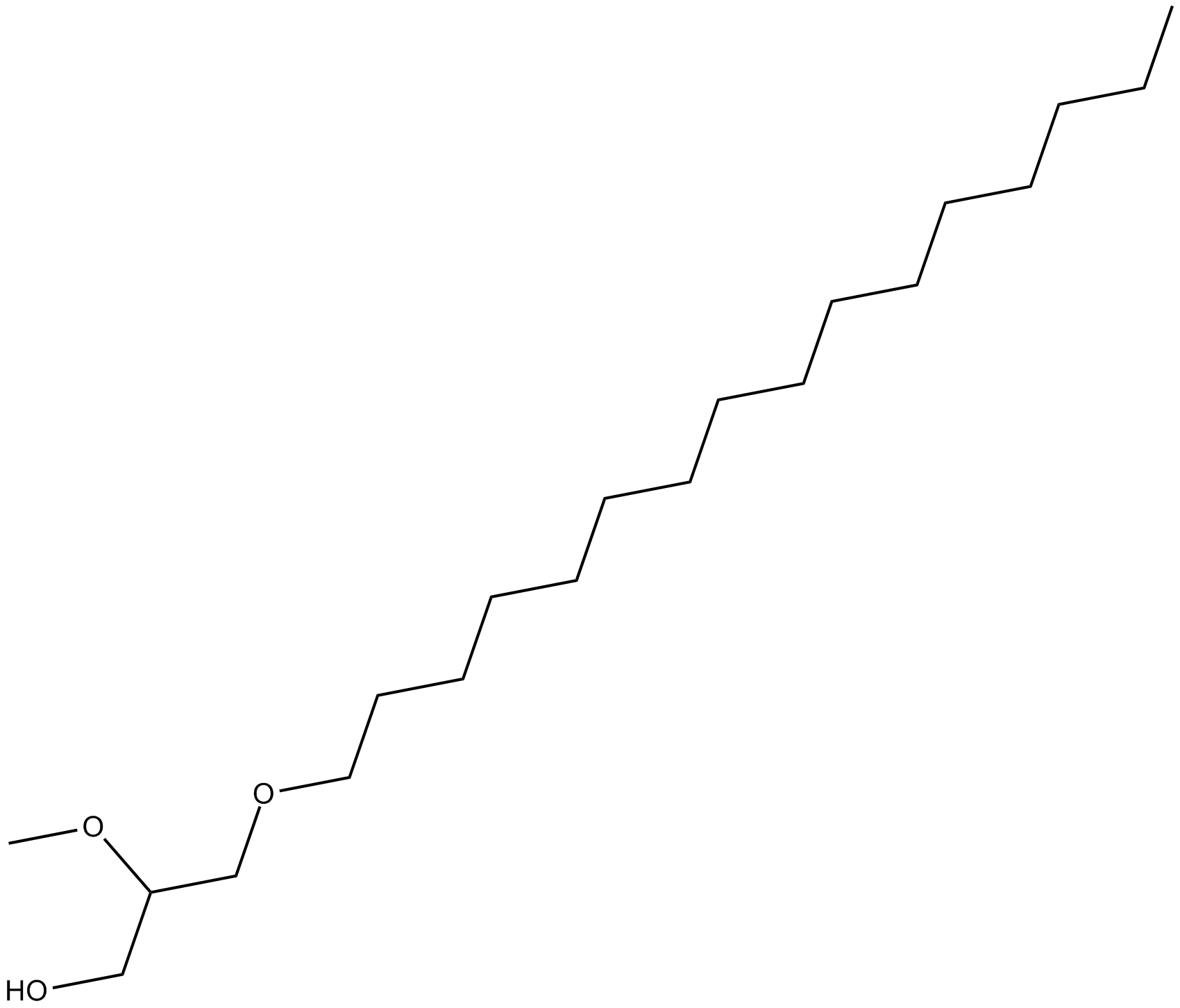
-
GC15018
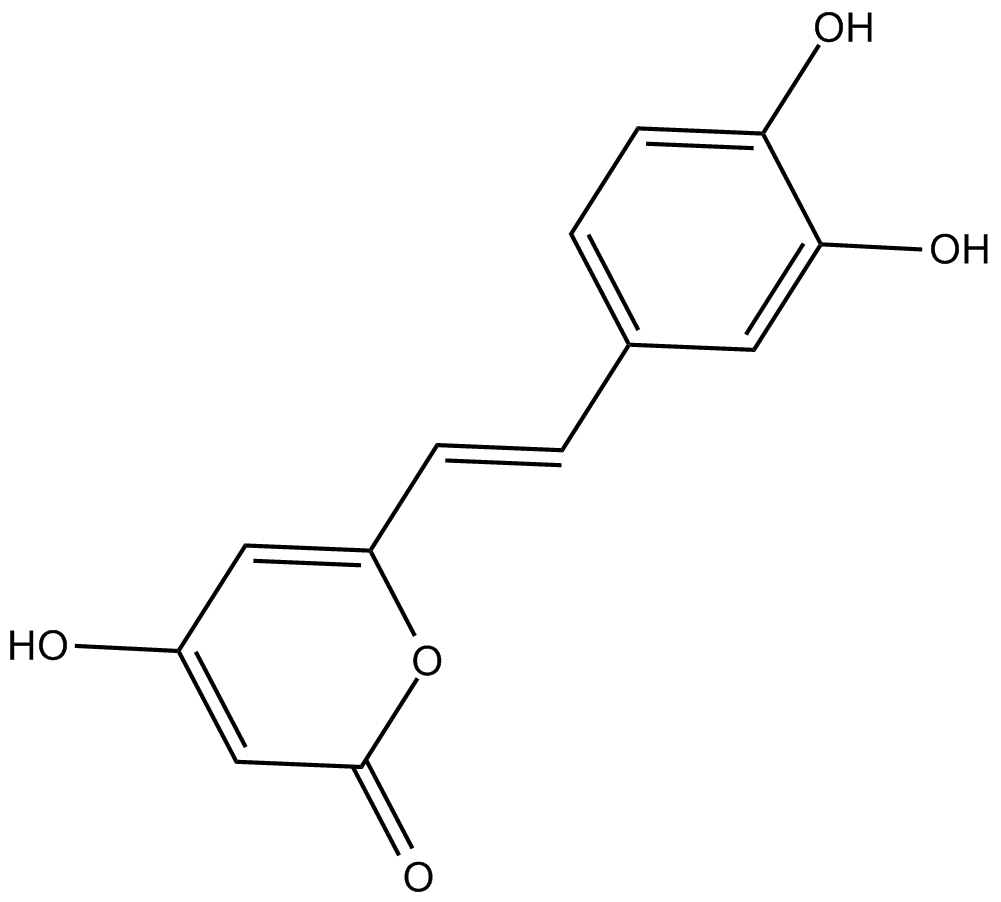
Hispidin
Se ha demostrado que la hispidina, un inhibidor de la PKC y un compuesto fenÓlico de Phellinus linteus, posee fuertes propiedades antioxidantes, anticancerÍgenas, antidiabéticas y antidemencia.
-
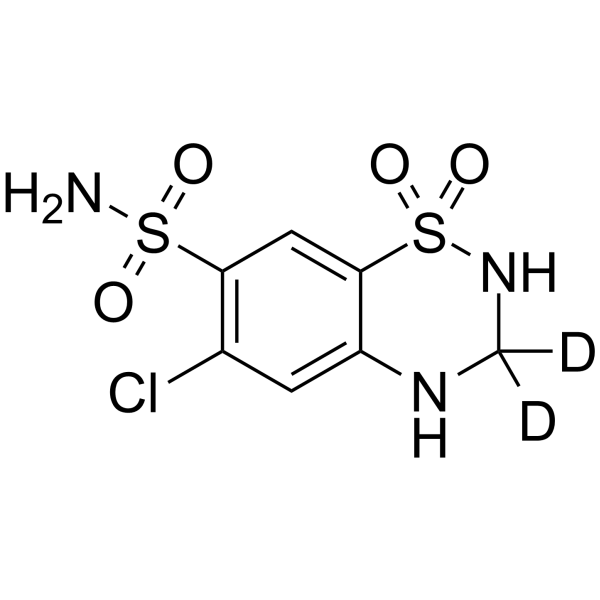
GC64326
Hydrochlorothiazid-d2
-
GC17523
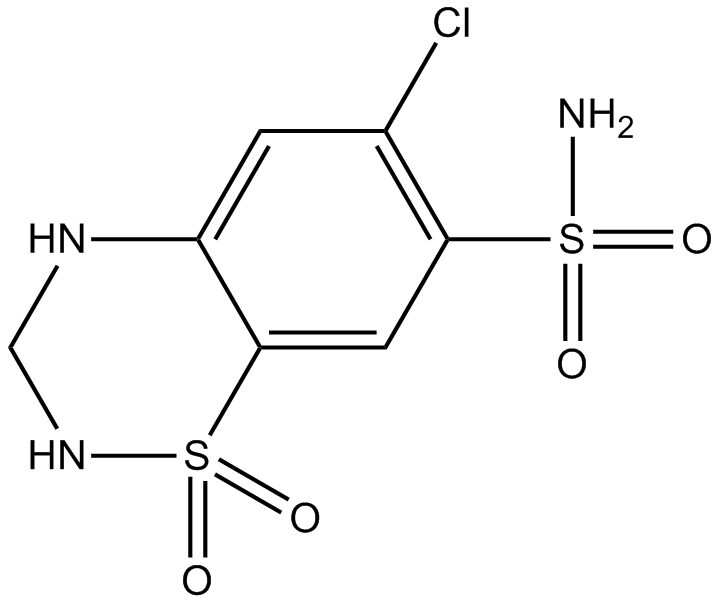
Hydrochlorothiazide
La hidroclorotiazida (HCTZ), un fÁrmaco diurético activo por vÍa oral de la clase de las tiazidas, inhibe la vÍa de seÑalizaciÓn transformante de TGF-β/Smad.
-
GC36282
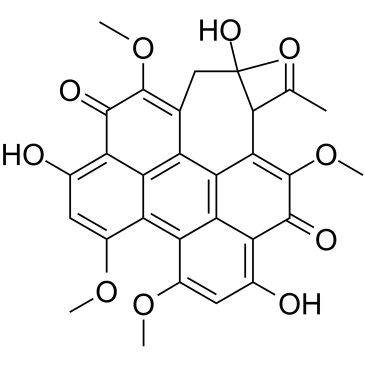
Hypocrellin A
La hipocrelina A, un inhibidor natural de la PKC, tiene muchas propiedades biolÓgicas y farmacolÓgicas, como actividades antitumorales, antivirales, antibacterianas y contra la leishmania.
-
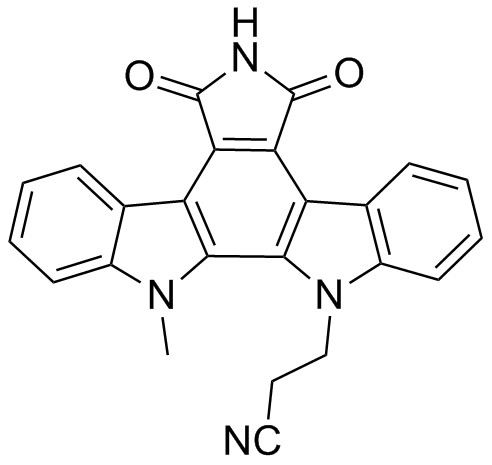
GC15420
ICP 103
Protein kinase inhibitor
-
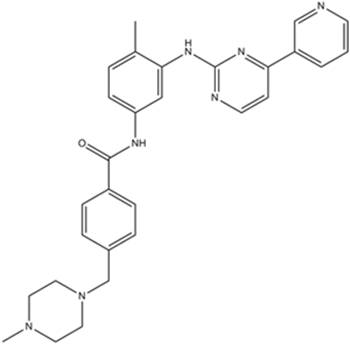
GC10314
Imatinib (STI571)
Imatinib (STI571) (STI571) es un inhibidor de tirosina quinasas biodisponible por vÍa oral que inhibe selectivamente la actividad de la quinasa BCR/ABL, v-Abl, PDGFR y c-kit. Imatinib (STI571) (STI571) funciona uniéndose cerca del sitio de uniÓn de ATP, bloqueÁndolo en una conformaciÓn cerrada o autoinhibida, lo que inhibe la actividad enzimÁtica de la proteÍna de forma semicompetitiva. Imatinib (STI571) también es un inhibidor de SARS-CoV y MERS-CoV.
-
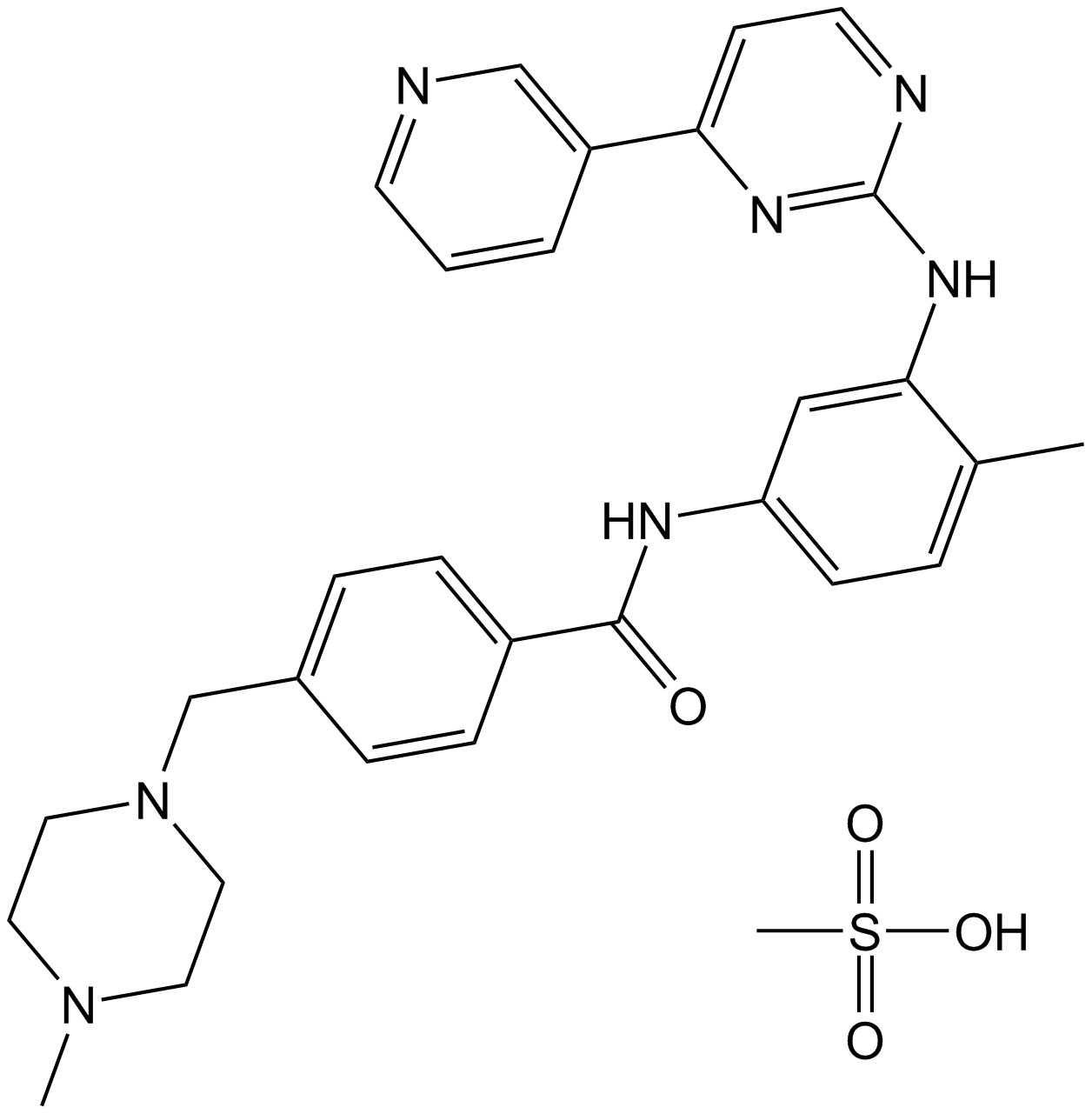
GC11759
Imatinib Mesylate (STI571)
El mesilato de imatinib (STI571) (STI571 Mesilato) es un inhibidor de las tirosina quinasas que inhibe las tirosina quinasas c-Kit, Bcr-Abl y PDGFR (IC50=100 nM).
-
GC50317
IN 1130
IN 1130 es un inhibidor de la cinasa del receptor tipo I del factor de crecimiento transformante β (ALK5) altamente selectivo con una IC50 de 5,3 nM para la fosforilación de Smad3 mediada por ALK5.
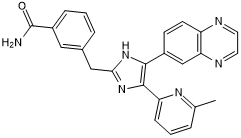
-
GC32870
Ingenol ((-)-Ingenol)
Ingenol ((-)-Ingenol) es un activador de PKC, con una Ki de 30 μM, con actividad antitumoral.
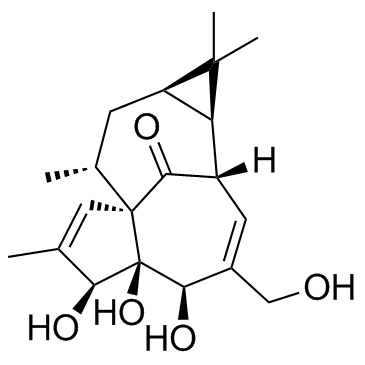
-
GC61776
Ingenol 3,20-dibenzoate
El 3,20-dibenzoato de ingenol es un potente agonista selectivo de la isoforma de la proteÍna quinasa C (PKC).

-
GC31656
Ingenol Mebutate (Ingenol 3-angelate)
El mebutato de ingenol (Ingenol 3-angelate) es un ingrediente activo en Euphorbia peplus, actÚa como un potente modulador de PKC, con Kis de 0.3, 0.105, 0.162, 0.376 y 0.171 nM para PKC-α, PKC-β, PKC-β γ, PKC-δ y PKC-ε, respectivamente, y tiene actividad antiinflamatoria y antitumoral.
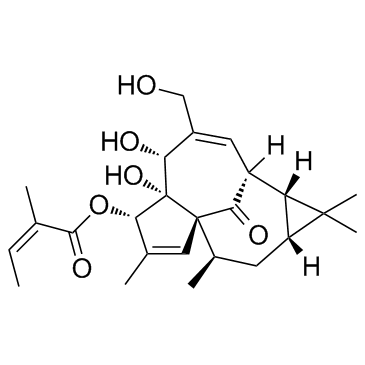
-
GC15148
Ionomycin calcium salt
Lonomycin es un ionóforo selectivo de calcio derivado de S. conglobatus que moviliza las reservas de calcio intracelular.
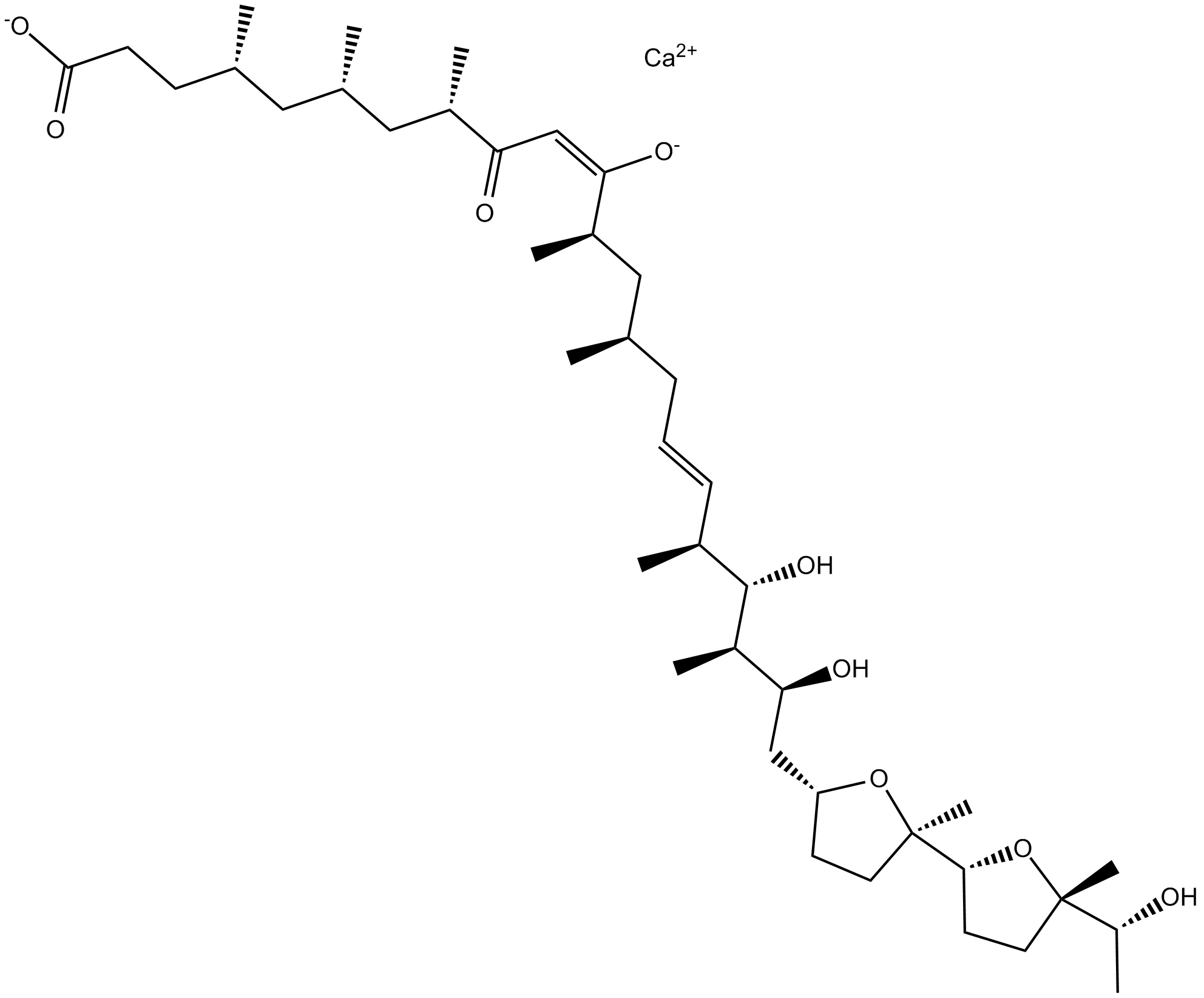
-
GC15446
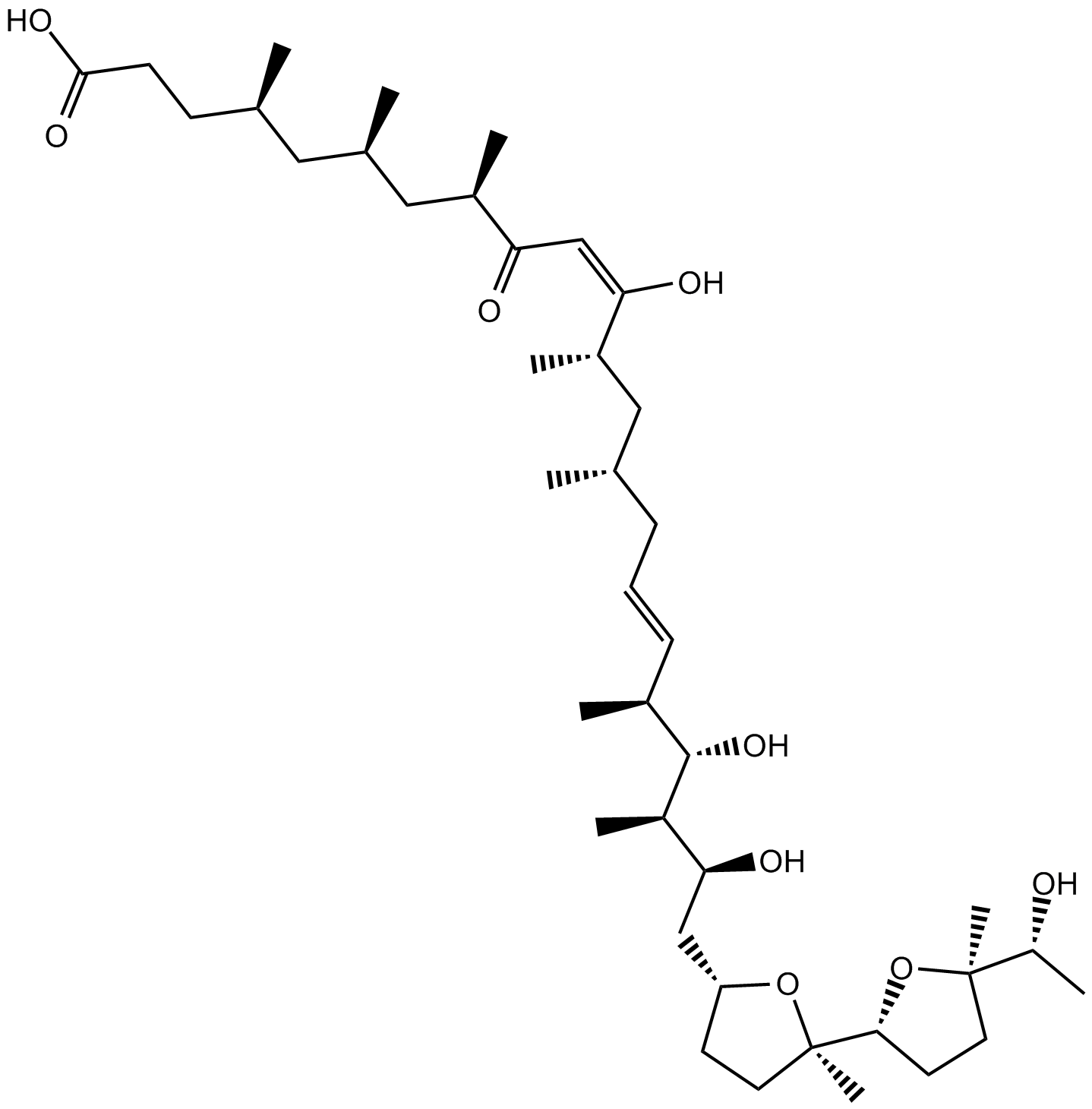
Ionomycin free acid
El ácido libre de ionomicina es un portador de iones de calcio potente y selectivo que actúa como portador activo de Ca2+.
-
GC60210
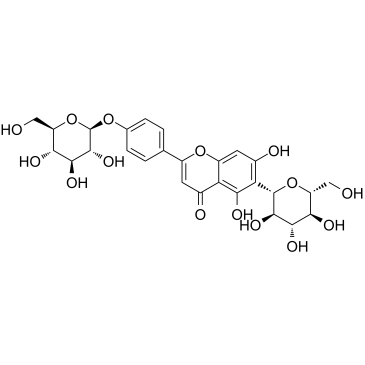
Isosaponarin
La isosaponarina es un glucÓsido de flavona aislado de las hojas de wasabi.
-
GC12108
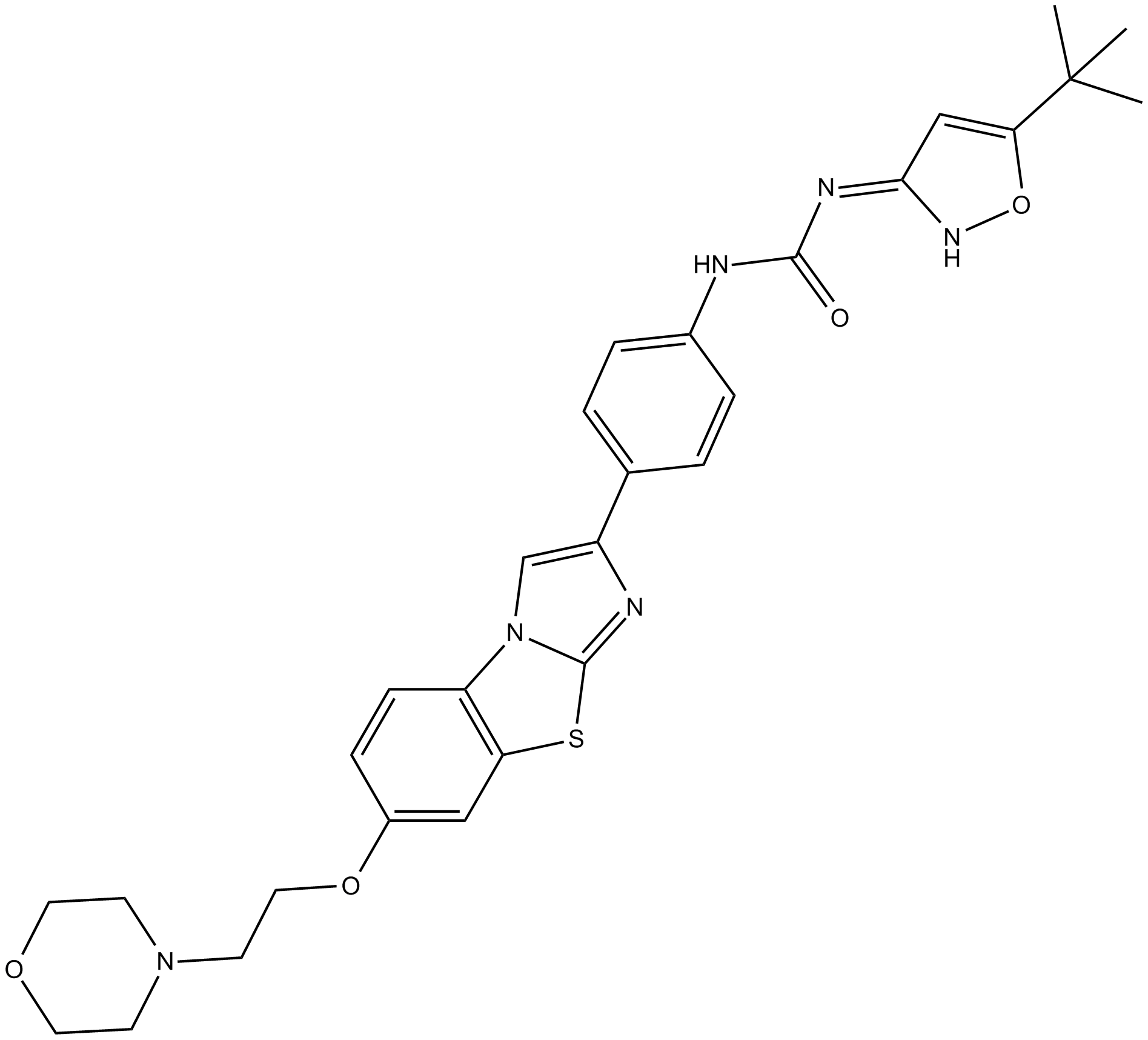
ITD 1
ITD 1 es el primer inhibidor selectivo del receptor de TGFβ con una IC50 de 460 nM.
-
GC11362
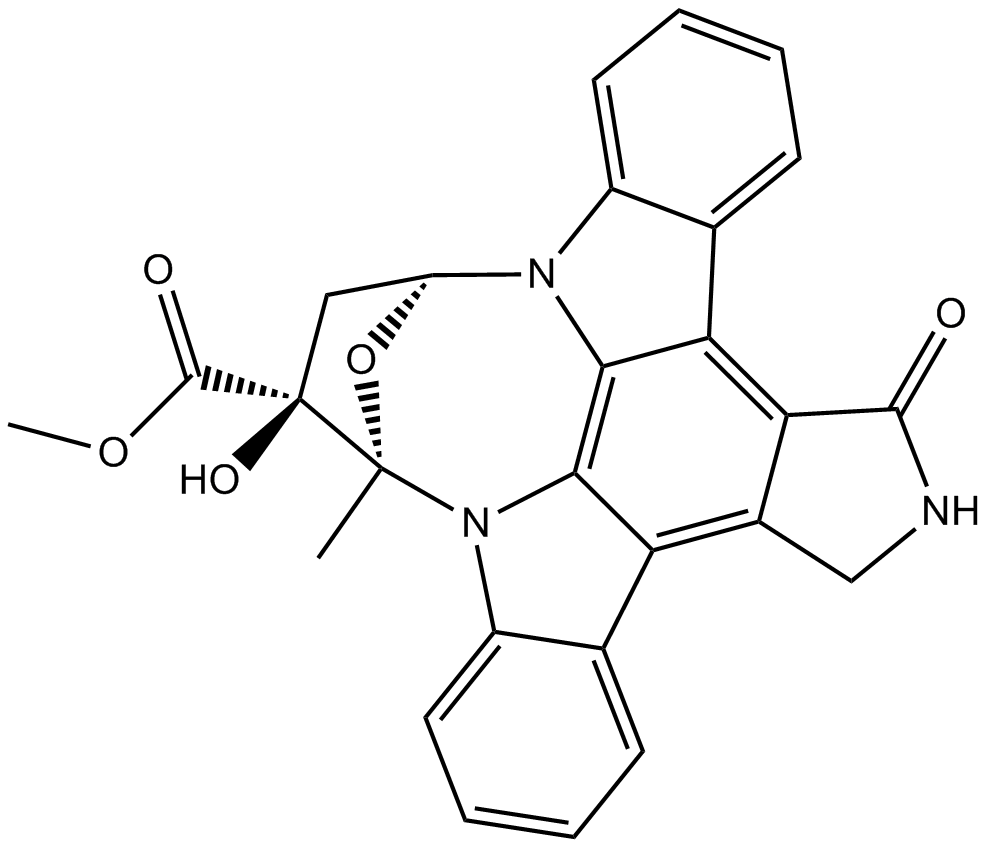
K 252a
Un inhibidor de la proteína quinasa.
-
GC15281
K-252c
K-252c, un anÁlogo de estaurosporina aislado de Nocardiopsis sp.
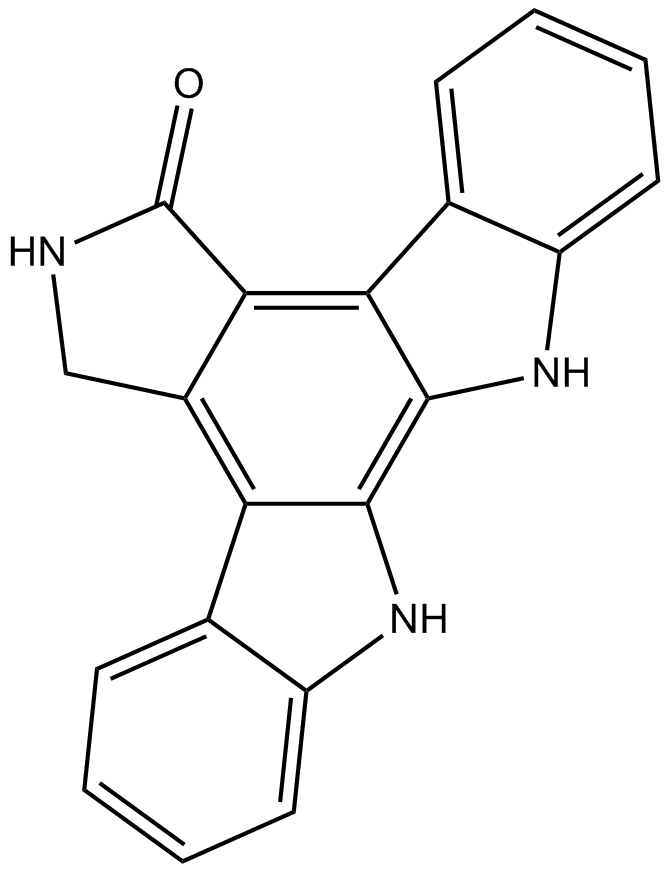
-
GC15567
K02288
K02288 es un potente inhibidor del receptor de proteÍna morfogenética Ósea (BMP) tipo I con IC50 de 1,8, 1,1, 6,4 nM para ALK1, ALK2 y ALK6, respectivamente.
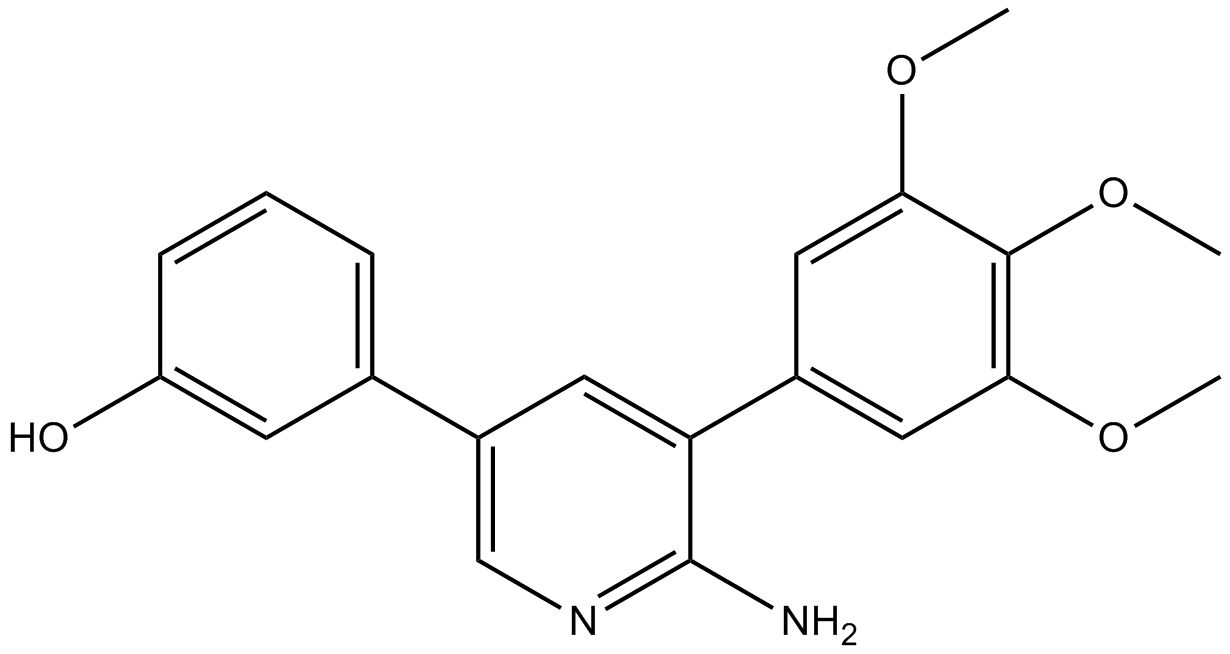
-
GC43993
K252b
K252b, un indolocarbazol aislado del actinomiceto Nocardiopsis, es un inhibidor de PKC.
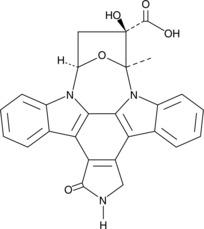
-
GC15057
Kartogenin
La kartogenina (KGN) es un inductor de la diferenciaciÓn de células madre mesenquimales humanas a condrocitos, con una EC50 de 100 nM.
-
GC64263
Kobophenol A
El kobofenol A, un estilbeno oligomérico, bloquea la interacciÓn entre el receptor ACE2 y S1-RBD con una IC50 de 1,81 μM e inhibe la infecciÓn viral por SARS-CoV-2 en células con una EC50 de 71,6 μM.
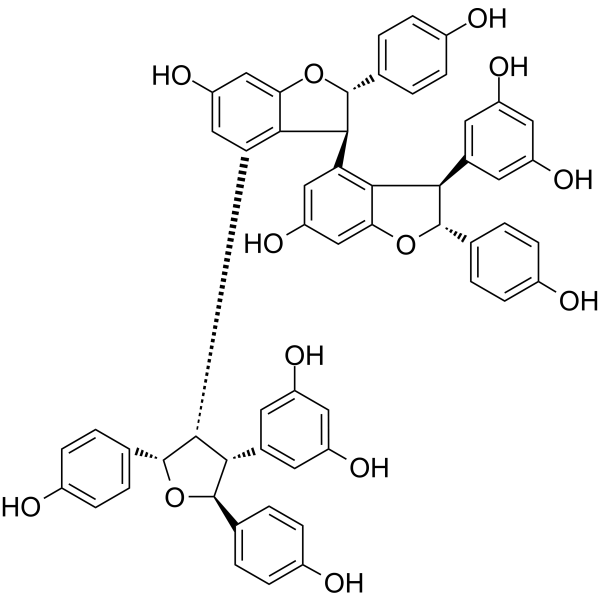
-
GC17346
KT 5823
KT 5823, un inhibidor selectivo de la proteína quinasa dependiente de cGMP (PKG) con un valor Ki de 0,23 μM, también inhibe PKA y PKC con valores Ki de 10 μM y 4 μM, respectivamente.
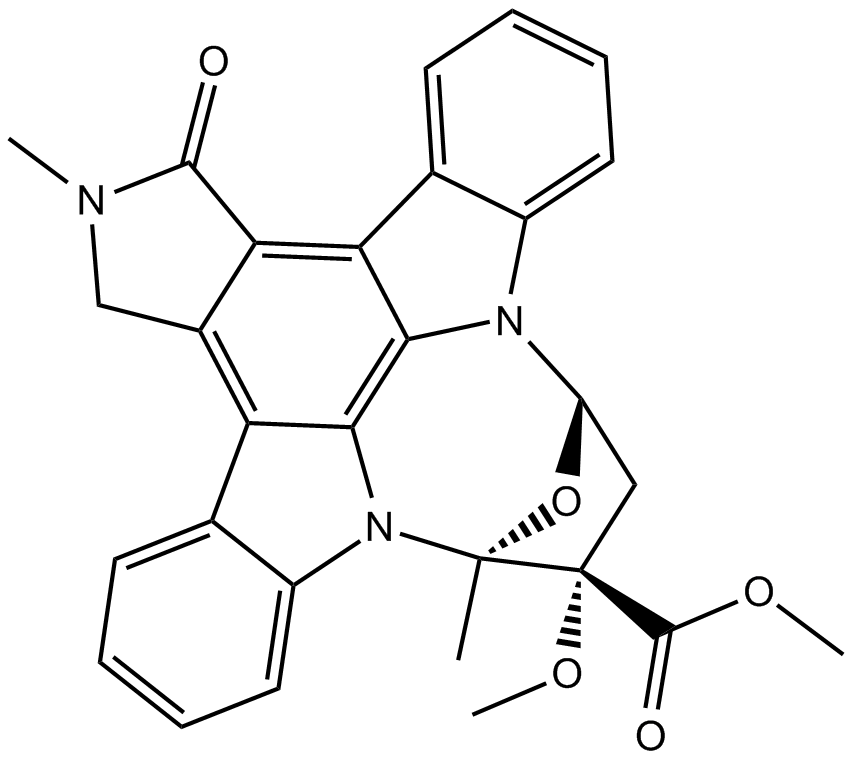
-
GC12817
K–115 hydrochloride dihydrate
El clorhidrato de K-115 dihidrato (K-115) es un inhibidor especÍfico de ROCK, con IC50 de 19 y 51 nM para ROCK2 y ROCK1, respectivamente.
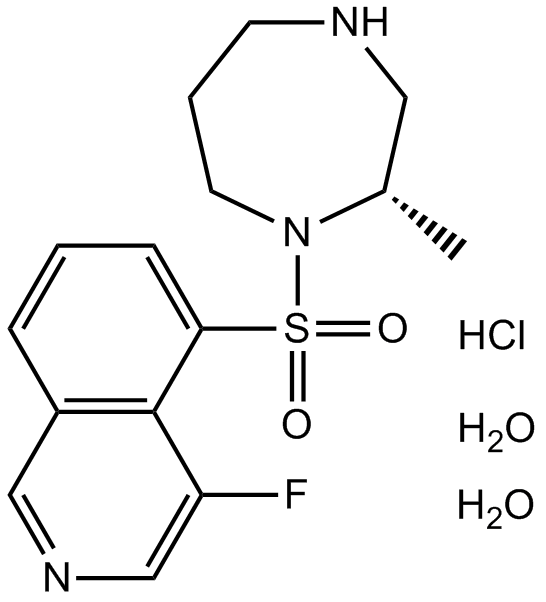
-
GC40770
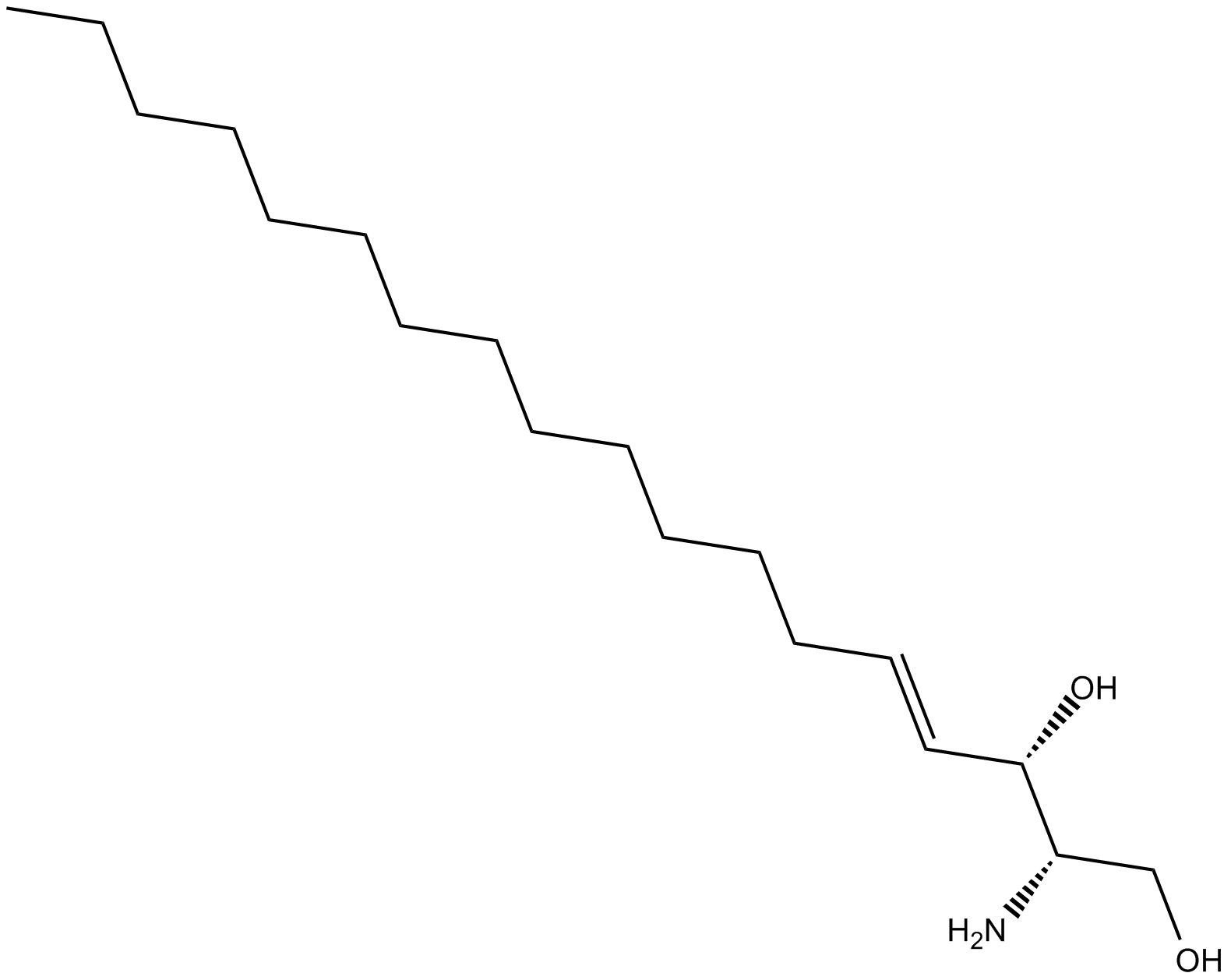
L-erythro Sphingosine (d18:1)
L-erythro Sphingosine is a synthetic stereoisomer of sphingosine (d18:1).
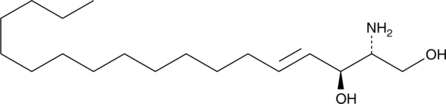
-
GC17815
L-threo-Sphingosine C-18
L-threo-Sphingosine C-18 es un potente inhibidor de MAPK. L-threo-Sphingosine C-18 induce la apoptosis y la fragmentaciÓn clara del ADN. L-threo-Sphingosine C-18 muestra un efecto anticancerÍgeno.
-
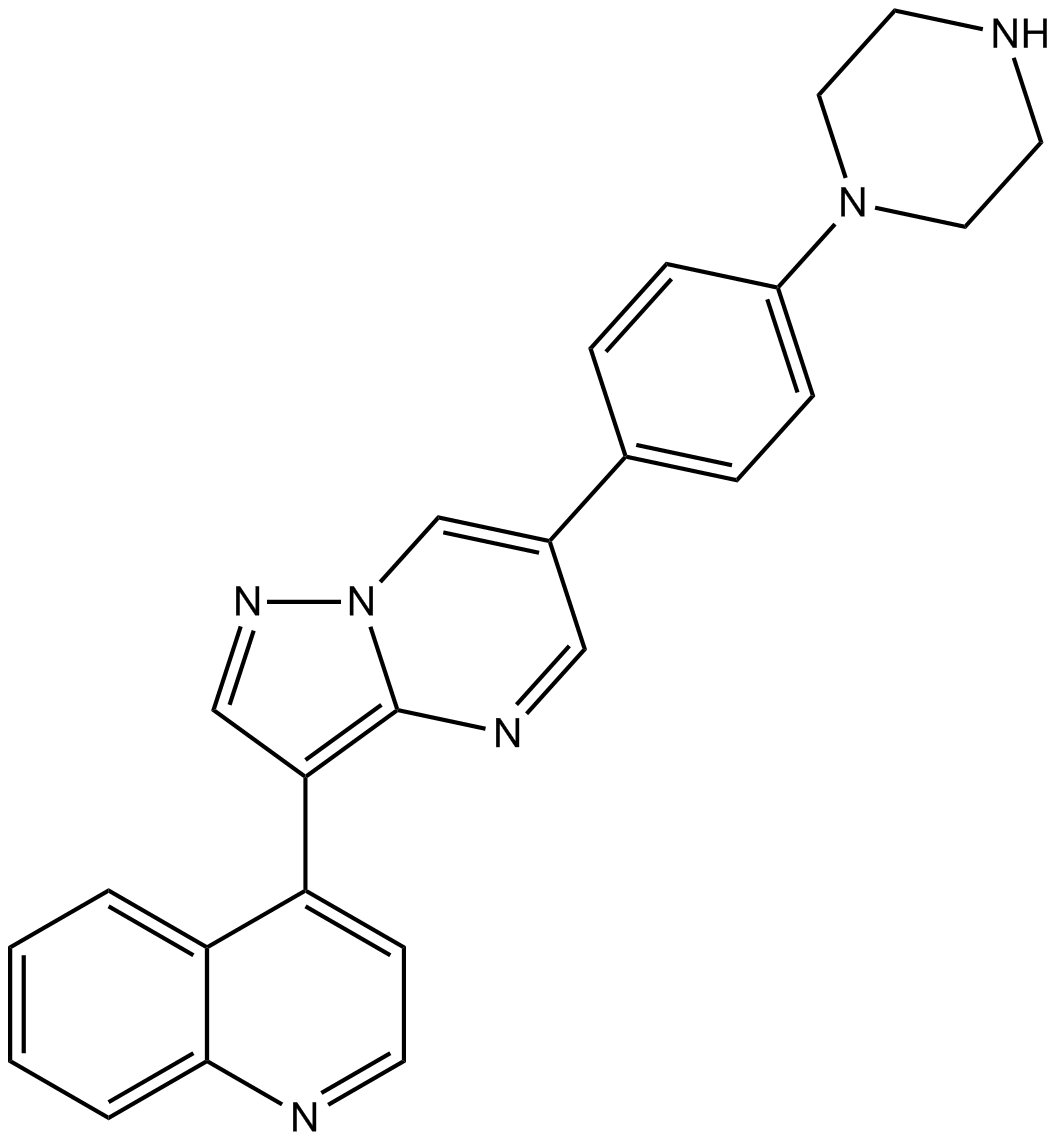
GC16580
LDN-193189
Inhibidor de ALK, potente y selectivo.
-
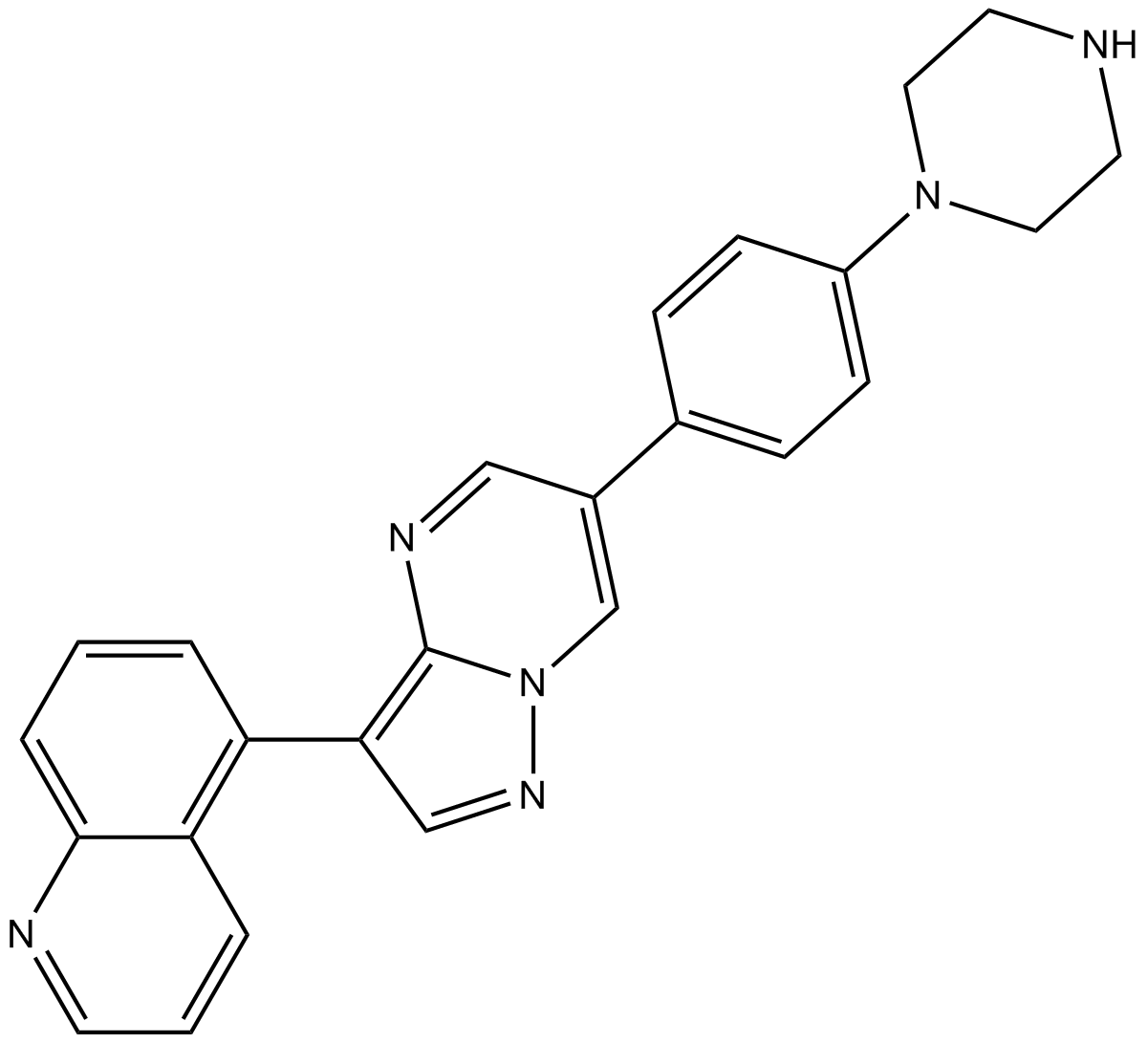
GC17035
LDN-212854
LDN-212854 es un inhibidor de la proteÍna morfogenética Ósea (BMP) que inhibe potentemente ALK2 (IC50: 1,3 nM). LDN-212854 también inhibe ALK1 (IC50: 2,40 nM). LDN-212854 se puede utilizar en la investigaciÓn de fibrodisplasia osificante progresiva y cÁnceres, como el carcinoma hepatocelular (HCC).
-
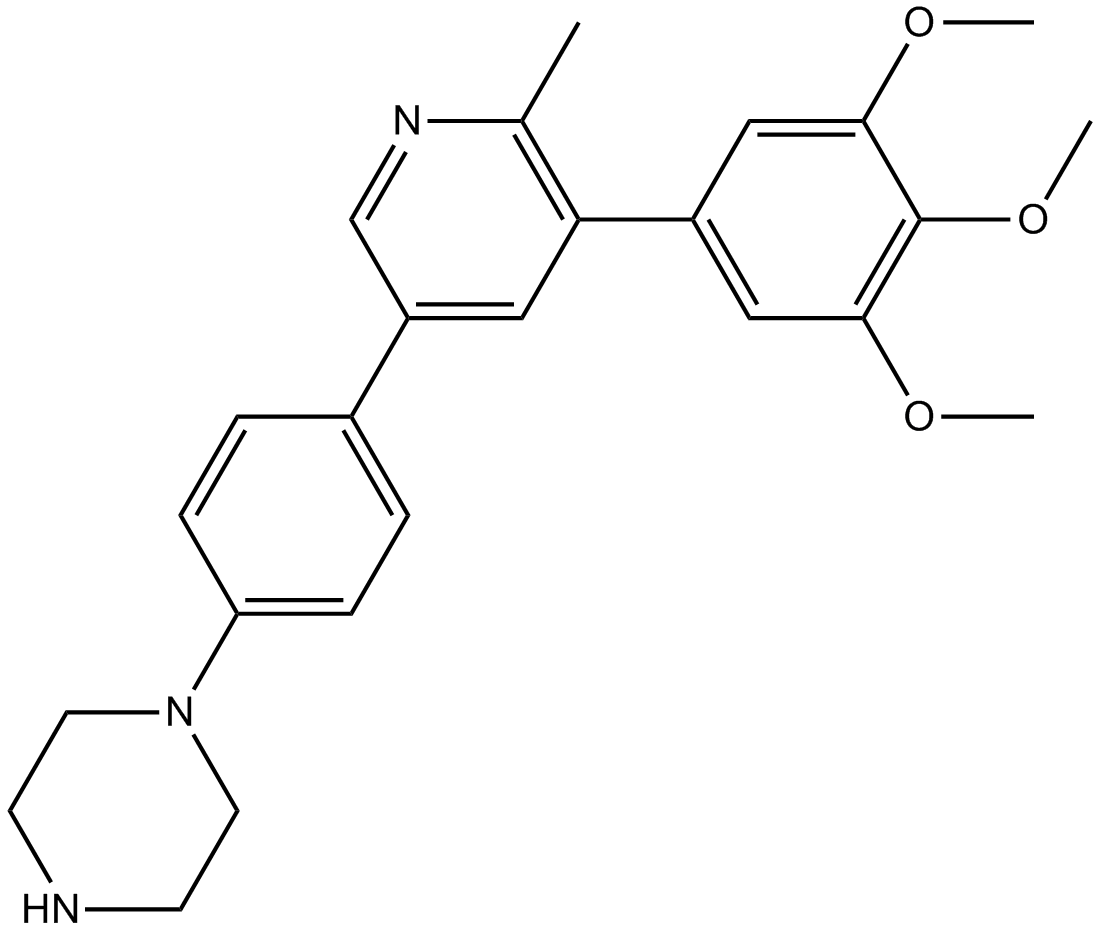
GC13225
LDN-214117
LDN-214117 es un inhibidor de ALK2 activo por vÍa oral con buena penetraciÓn cerebral y bien tolerado. LDN-214117 tiene una alta selectividad y baja citotoxicidad para ALK2 con un valor IC50 de 24 nM. LDN-214117 también es un inhibidor especÍfico de la seÑalizaciÓn de proteÍnas morfogenéticas Óseas (BMP) y tiene una inhibiciÓn relativamente selectiva para BMP6 con un valor IC50 de 100 nM. LDN-214117 se puede utilizar para la investigaciÓn de fibrodisplasia osificante progresiva (FOP), glioma pontino intrÍnseco difuso (DIPG) up.
-
GC14931
LDN193189 Hydrochloride
An inhibitor of BMP receptors ALK1, ALK2, ALK3, and ALK6

-
GC36433
LDN193189 Tetrahydrochloride
El tetraclorhidrato de LDN193189 es un inhibidor selectivo del receptor de BMP tipo I, que inhibe eficazmente ALK2 y ALK3 (IC50=5 nM y 30 nM, respectivamente), con efectos mÁs débiles sobre ALK4, ALK5 y ALK7 (IC50≥500 nM).
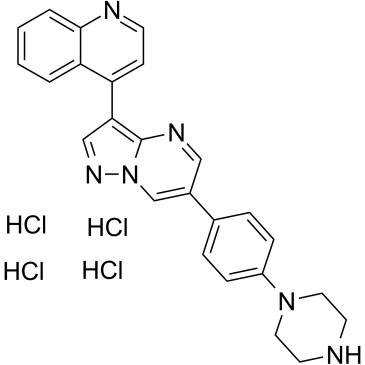
-
GC39398
LSKL, Inhibitor of Thrombospondin (TSP-1) (TFA)
LSKL, inhibidor de la trombospondina (TSP-1) TFA es un tetrapéptido derivado de la proteÍna asociada a la latencia (LAP)-TGFβ y un antagonista competitivo de TGF-β1. LSKL, inhibidor de trombospondina (TSP-1) TFA inhibe la uniÓn de TSP-1 a LAP y alivia la fibrosis intersticial renal y la fibrosis hepÁtica. LSKL, inhibidor de trombospondina (TSP-1) TFA suprime la fibrosis subaracnoidea a través de la inhibiciÓn de la actividad de TGF-β1 mediada por TSP-1, previene el desarrollo de hidrocefalia crÓnica y mejora los defectos neurocognitivos a largo plazo después de la hemorragia subaracnoidea (HSA). LSKL, inhibidor de trombospondina (TSP-1) TFA puede cruzar fÁcilmente la barrera hematoencefÁlica.

-
GC32728
LSKL, Inhibitor of Thrombospondin TSP-1
LSKL, inhibidor de la trombospondina TSP-1 es un tetrapéptido derivado de la proteína asociada a la latencia (LAP)-TGFβ y un antagonista competitivo de TGF-β1. LSKL, inhibidor de trombospondina TSP-1 inhibe la unión de TSP-1 a LAP y alivia la fibrosis intersticial renal y la fibrosis hepática. LSKL, inhibidor de la trombospondina TSP-1 suprime la fibrosis subaracnoidea a través de la inhibición de la actividad de TGF-β1 mediada por TSP-1, previene el desarrollo de hidrocefalia crónica y mejora los defectos neurocognitivos a largo plazo después de la hemorragia subaracnoidea (HSA). LSKL, inhibidor de la trombospondina TSP-1, puede atravesar fácilmente la barrera hematoencefálica.
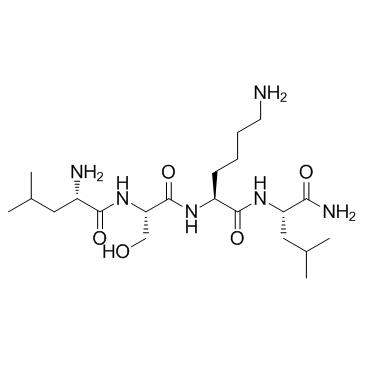
-
GC69412
Luspatercept
Luspatercept (ACE-536) es una proteína de fusión recombinante modificada que se une al ligando de la super familia del factor de crecimiento transformante beta β. Luspatercept aumenta el número de glóbulos rojos y promueve la maduración de los precursores de glóbulos rojos. Luspatercept se une a GDF11 e inhibe la vía de señalización Smad2/3. Luspatercept puede ser utilizado en investigaciones sobre anemia.

-
GC12402
LX7101 HCL
LX7101 HCL es un potente inhibidor de LIMK y ROCK2 con valores IC50 de 24, 1,6 y 10 nM para LIMK1, LIMK2 y ROCK2, respectivamente; también inhibe la PKA con una IC50 inferior a 1 nM.
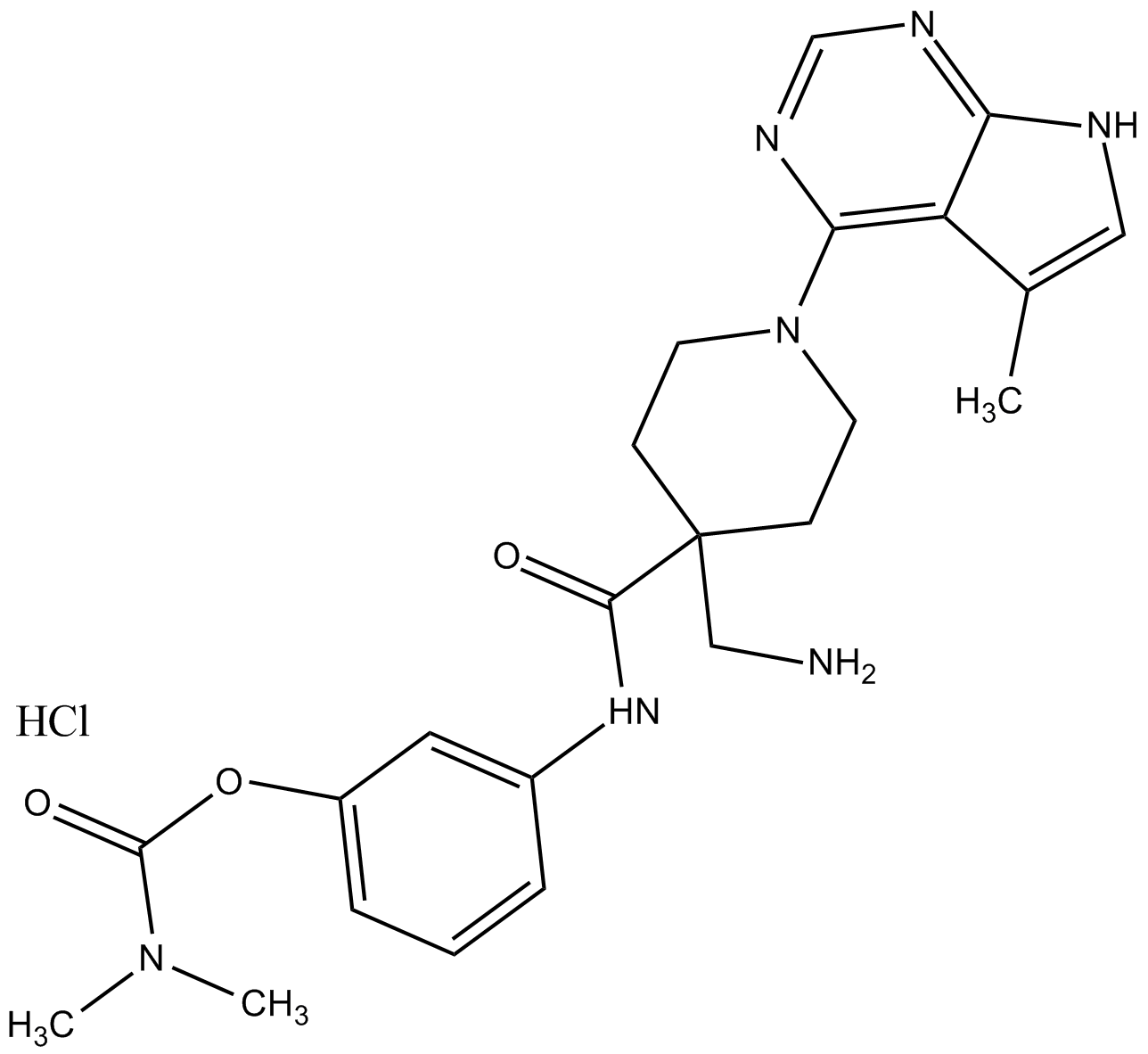
-
GC32811
LXS196
LXS196 (LXS196) es un inhibidor de la proteÍna quinasa C (PKC) potente, selectivo y activo por vÍa oral, con valores IC50 de 1,9 nM, 0,4 nM y 3,1 μM para PKCα, PKCθ y GSK3β, respectivamente. LXS196 tiene potencial para la investigaciÓn del melanoma uveal.
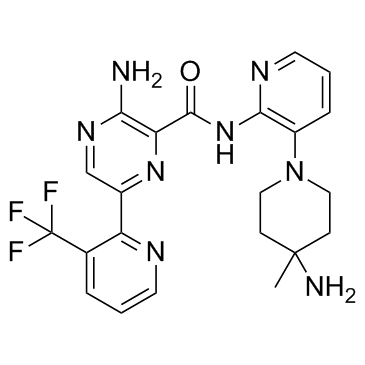
-
GC17563
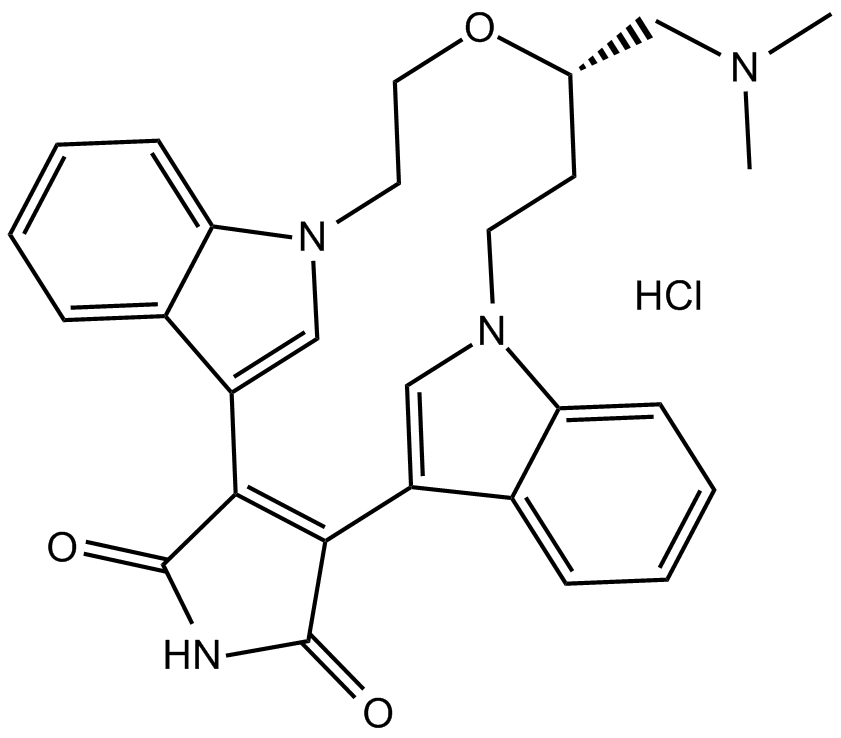
LY 333531 hydrochloride
El clorhidrato de ruboxistaurina (LY333531) es un inhibidor beta selectivo de la PKC activo por vÍa oral (Ki = 2 nM).
-
GC12363
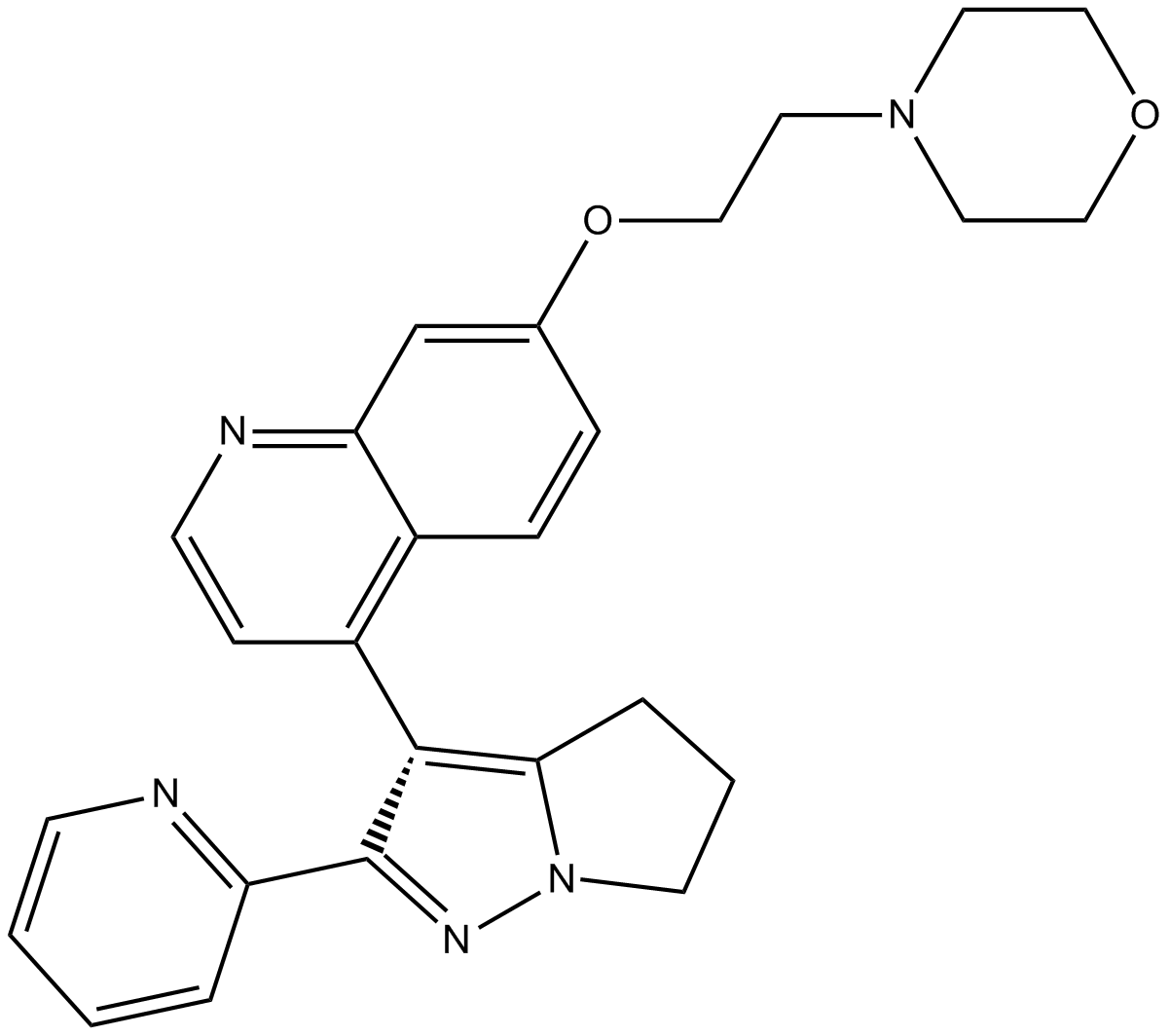
LY2109761
LY2109761 es un inhibidor selectivo del receptor de TGF-β tipo I/II activo por vÍa oral con Kis de 38 nM y 300 nM, respectivamente.
-
GC18015
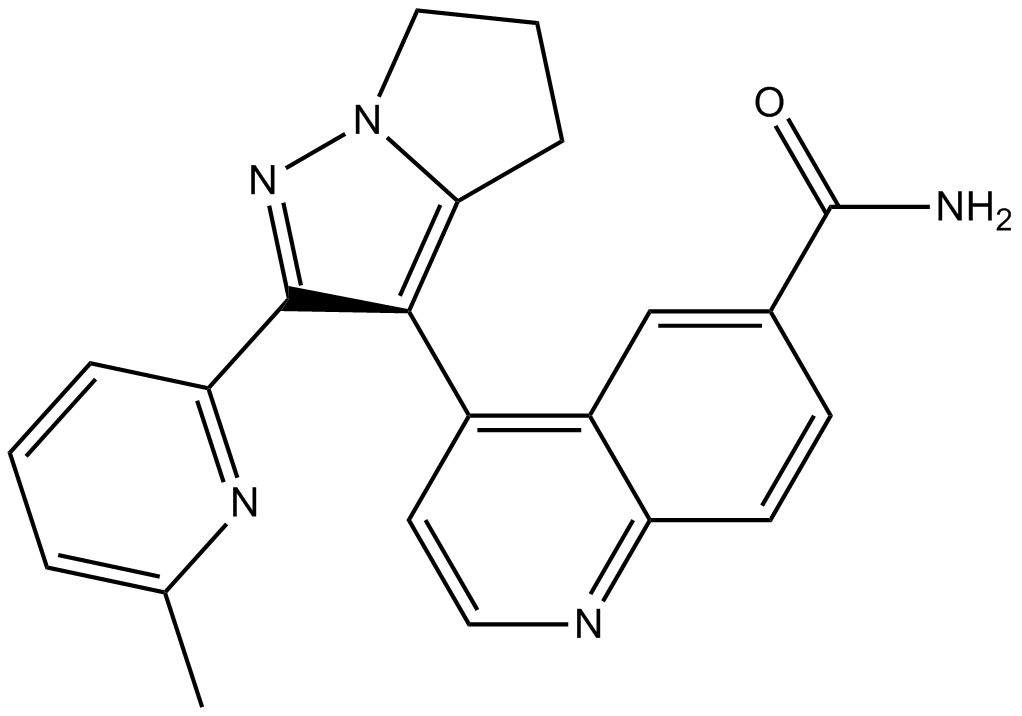
LY2157299
LY2157299 (LY2157299) es un TGF-β oral y selectivo; inhibidor de la cinasa del receptor tipo I (TGF-βRI) con una IC50 de 56 nM.
-
GC19234
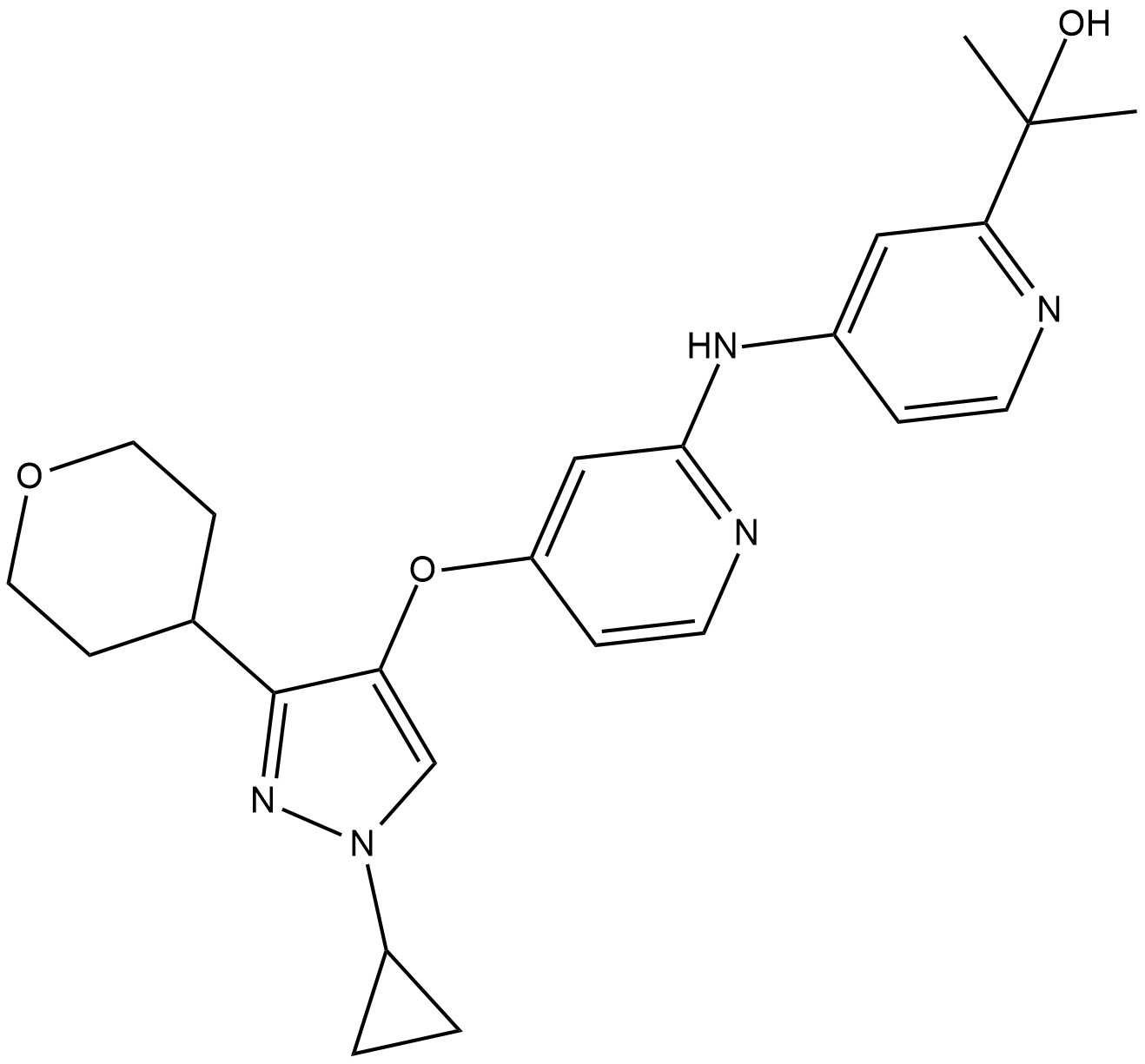
LY3200882
LY3200882 es un inhibidor del receptor de TGF-β tipo 1 (ALK5) potente, altamente selectivo, competitivo con ATP y activo por vÍa oral con una IC50 de 38,2 nM.
-
GC11604
LY364947
LY364947 (HTS466284) es un potente inhibidor competitivo de ATP de TGFβR-I con IC50 de 59 nM, y exhibe una selectividad de 7 veces sobre TGFβR-II.
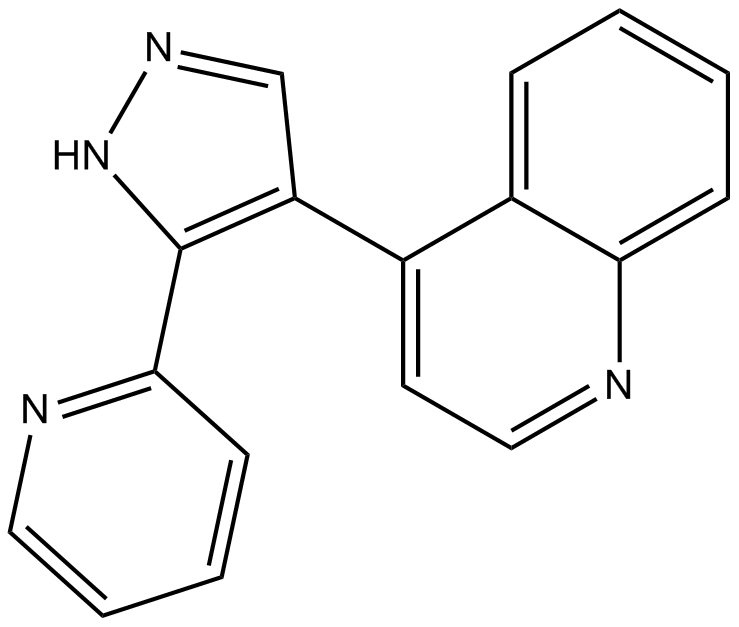
-
GC30545
Malantide
Malantide es un dodecapéptido sintético derivado del sitio fosforilado por la proteÍna quinasa dependiente de cAMP (PKA) en la subunidad β de la fosforilasa quinasa. Malantide es un sustrato altamente especÍfico para PKA con una Km de 15 μM y muestra una inhibiciÓn del inhibidor de proteÍnas (PKI) >90 % de la fosforilaciÓn del sustrato en varios extractos de tejido de rata. Malantide también es un sustrato eficiente para PKC con una Km de 16 μM.
-
GC10496
Midostaurin (PKC412)
La midostaurina (PKC412) (PKC412; CGP 41251) es un inhibidor reversible de la proteÍna quinasa multidiana activo por vÍa oral. Midostaurina (PKC412) inhibe PKCα/β/γ Syk, Flk-1, Akt, PKA, c-Kit, c-Fgr, c-Src, FLT3, PDFRβ y VEGFR1/2 con IC50 que oscilan entre 22 y 500 nM. La midostaurina (PKC412) también aumenta la expresiÓn del gen de la Óxido nÍtrico sintasa endotelial (eNOS). La midostaurina (PKC412) muestra poderosos efectos anticancerÍgenos.
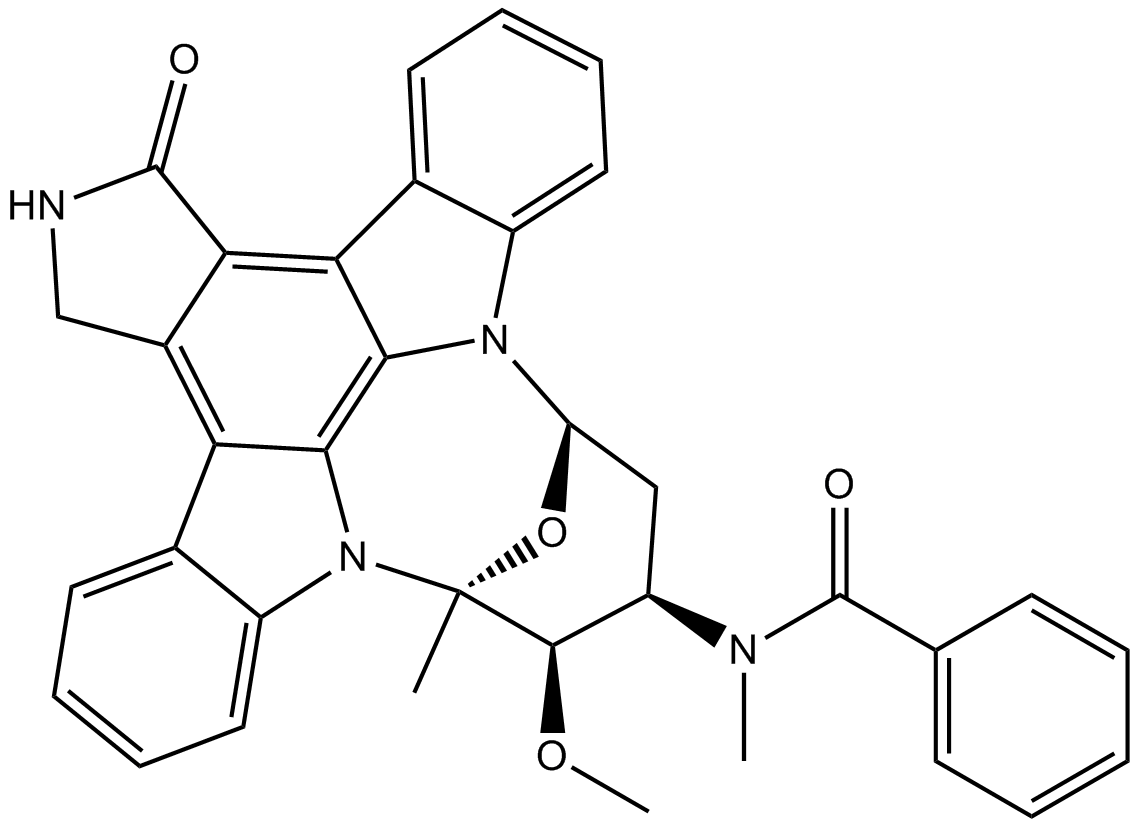
-
GC32714
Mitoxantrone (mitozantrone)
La mitoxantrona (mitozantrona) es un potente inhibidor de la topoisomerasa II.
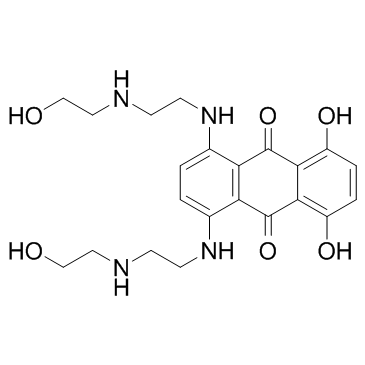
-
GC14363
Mitoxantrone HCl
Mitoxantrona HCl es un potente inhibidor de la topoisomerasa II.
-
GC17582
ML347
ML347 (LDN193719) es un inhibidor de ALK1/ALK2 altamente selectivo. ML347 tiene valores IC50 de 46 y 32 nM frente a ALK1 y ALK2, respectivamente, >300 veces mÁs selectivo que ALK3. ML347 bloquea la fosforilaciÓn de Smad1/5 por TGF-β1.
-
GC65585
Mongersen
Mongersen (GED-0301) es un oligonucleÓtido antisentido SMAD7 especÍfico y oralmente activo.

-
GC63778
Myelin Basic Protein TFA
ProteÍna bÁsica de mielina (MHP4-14) TFA, un péptido sintético que comprende los residuos 4-14 de la proteÍna bÁsica de mielina, es un sustrato de PKC muy selectivo (Km = 7 μM).
-
GC49269
Myr-ZIP
A PKMζ inhibitor
-
GC44303
N-Acetylpuromycin
N-Acetylpuromycin is a non-ribotoxic form of the antibiotic puromycin that is formed in puromycin-resistant S.
-
GC25662
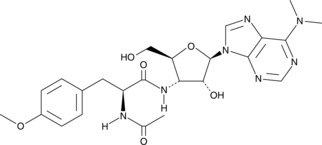
N-Desmethyltamoxifen
N-Desmethyltamoxifen, the major metabolite of Tamoxifen in humans and a ten-fold more potent protein kinase C (PKC) inhibitor than Tamoxifen, also is a potent regulator of ceramide metabolism in human AML cells, limiting ceramide glycosylation, hydrolysis, and sphingosine phosphorylation.
-
GC38931
N-Desmethyltamoxifen hydrochloride
El clorhidrato de N-desmetiltamoxifeno es el principal metabolito del tamoxifeno en humanos. El N-desmetiltamoxifeno, un antiestrÓgeno pobre, es un inhibidor de la proteÍna quinasa C (PKC) diez veces mÁs potente que el tamoxifeno. El clorhidrato de N-desmetiltamoxifeno también es un potente regulador del metabolismo de la ceramida en las células de LMA humana, lo que limita la glicosilaciÓn, la hidrÓlisis y la fosforilaciÓn de la esfingosina de la ceramida.

-
GC10716
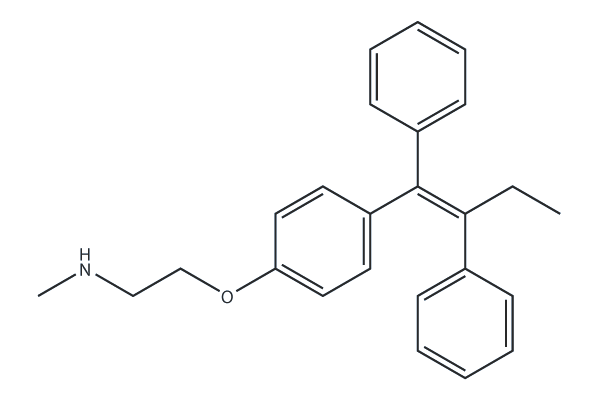
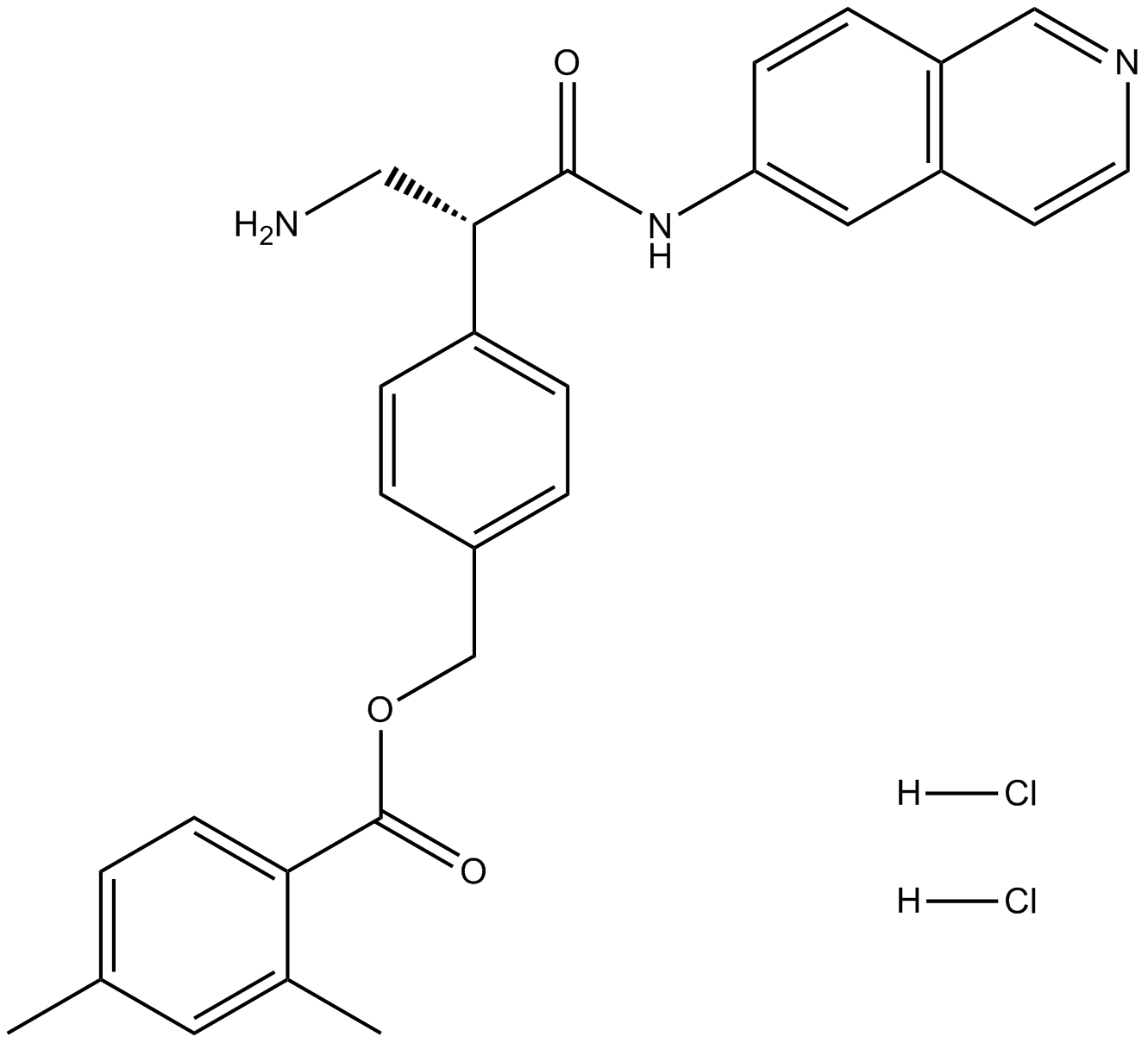
Netarsudil (AR-13324)
ROCK inhibitor
-
GC13514
NG25
An inhibitor of MAP4K2 and TAK1

-
GC25669
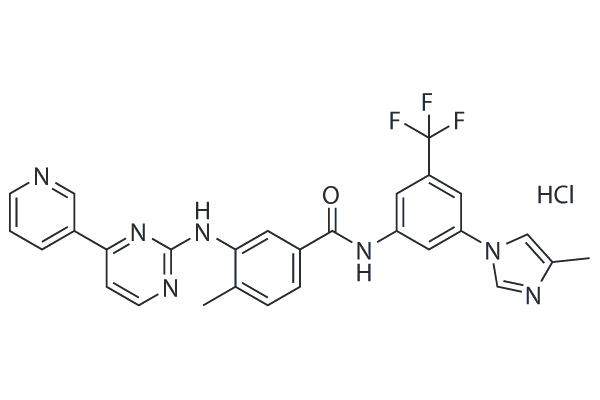
Nilotinib hydrochloride
Nilotinib hydrochloride (AMN-107) is the hydrochloride salt form of nilotinib, an orally bioavailable Bcr-Abl tyrosine kinase inhibitor with antineoplastic activity.
-
GC14075
Nocodazole
Un inhibidor de la producción de tubulina, agente antineoplásico.
-
GC60274
O-Desmethyl Midostaurin
La O-desmetil midostaurina (CGP62221; O-desmetil PKC412) es el metabolito activo de la midostaurina a través del metabolismo de las enzimas hepÁticas del citocromo P450. La O-desmetil midostaurina se puede utilizar como indicador del metabolismo de la midostaurina in vivo. La midostaurina es un inhibidor de la proteÍna quinasa multidirigidoconIC50que oscila entre 22 y 500 nM.

-
GC36807
ON 146040
ON 146040 es un potente inhibidor de PI3Kα y PI3Kδ (IC50≈14 y 20 nM, respectivamente). ON 146040 también inhibe Abl1 (IC50<150 nM).
-
GN10378
Oxymatrine
-
GC36833
p32 Inhibitor M36
El inhibidor de p32 M36 (M36) es un inhibidor de la proteÍna mitocondrial p32, que se une directamente a p32 e inhibe la asociaciÓn de p32 con LyP-1.
-
GC12637
PD 180970
PD 180970 es un inhibidor de la quinasa p210Bcr-Abl muy potente y competitivo con ATP, con una IC50 de 5 nM para inhibir la autofosforilación de p210Bcr-Abl.
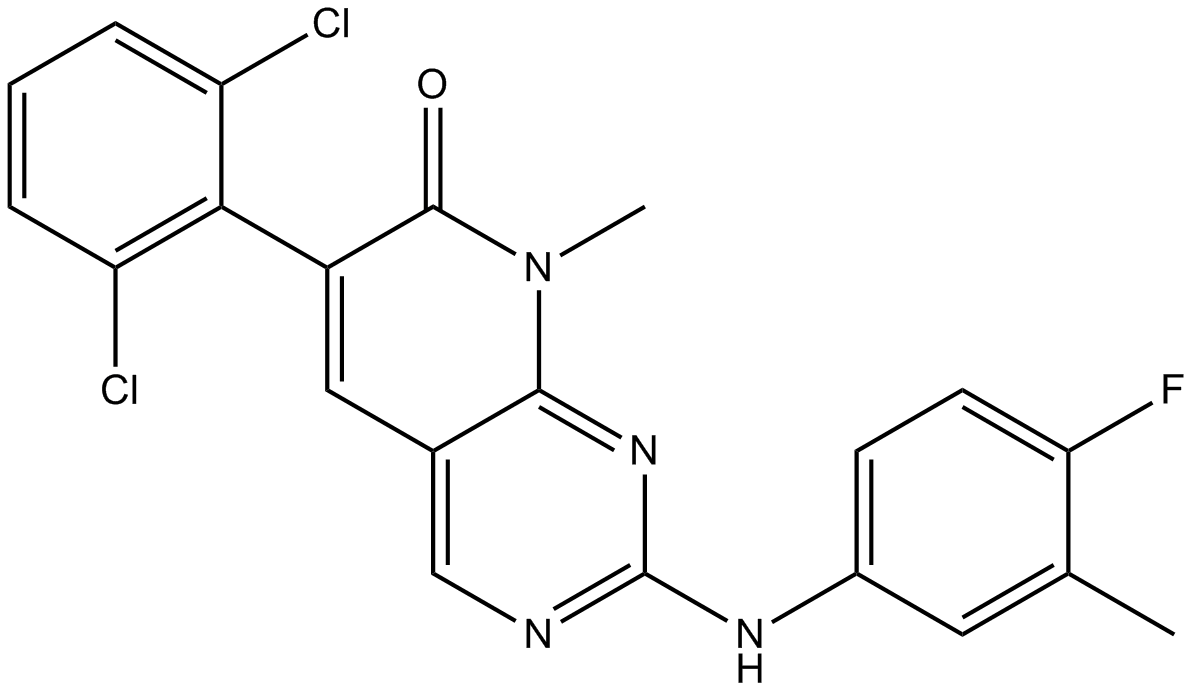
-
GC13592
PD173955
PD173955 es un inhibidor de la tirosina cinasa selectivo de la familia src con una IC50 de ~22 nM para las cinasas Src, Yes y Abl; menos potente para FGFRα y sin actividad sobre InsR y PKC.
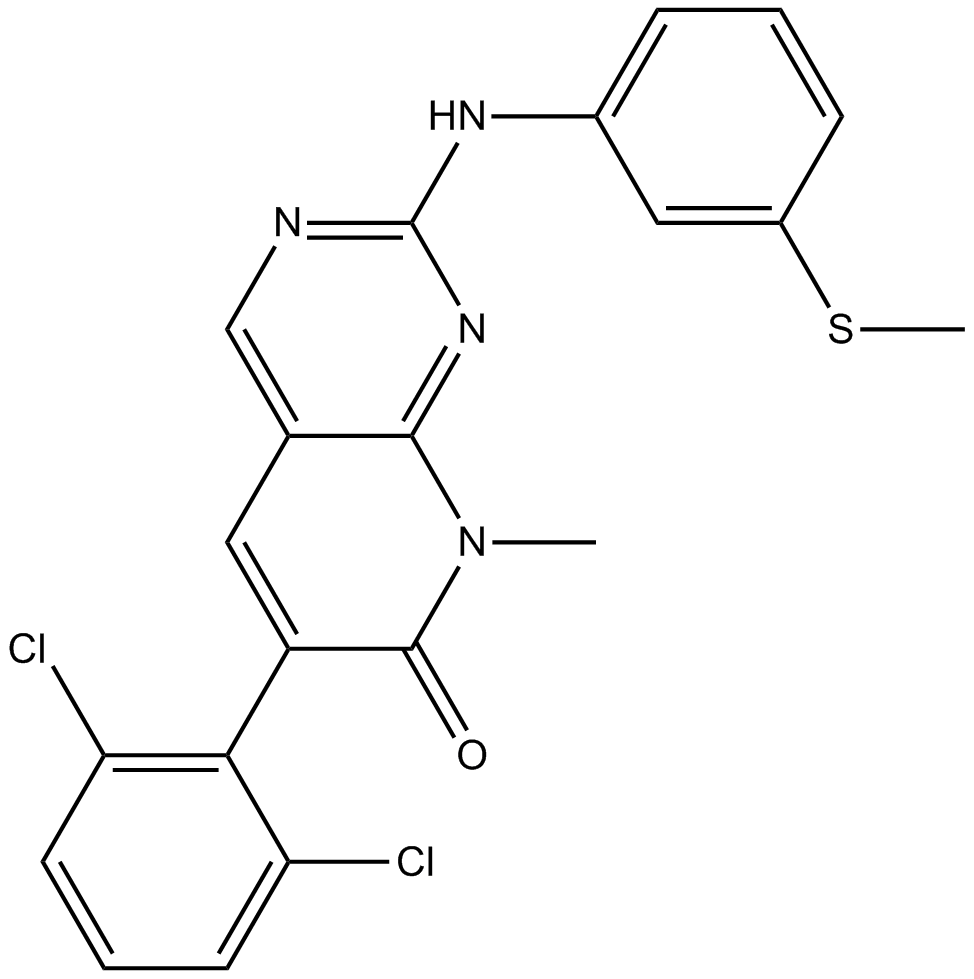
-
GC33901
Pentanoic acid
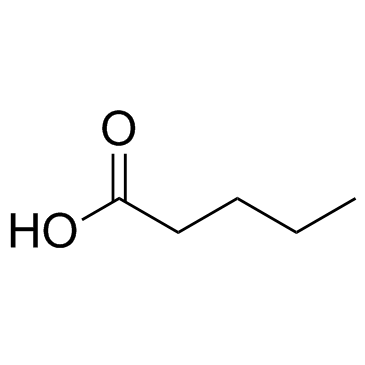
-
GC61585
Pep2m, myristoylated TFA
Pep2m, TFA miristoilado (Myr-Pep2m TFA) es un péptido permeable a las células.

-
GC14767
PF-00562271
El besilato PF-562271 (VS-6062) es un potente inhibidor reversible, competitivo con ATP, de las quinasas FAK y Pyk2, con una IC50 de 1,5 nM y 13 nM, respectivamente.
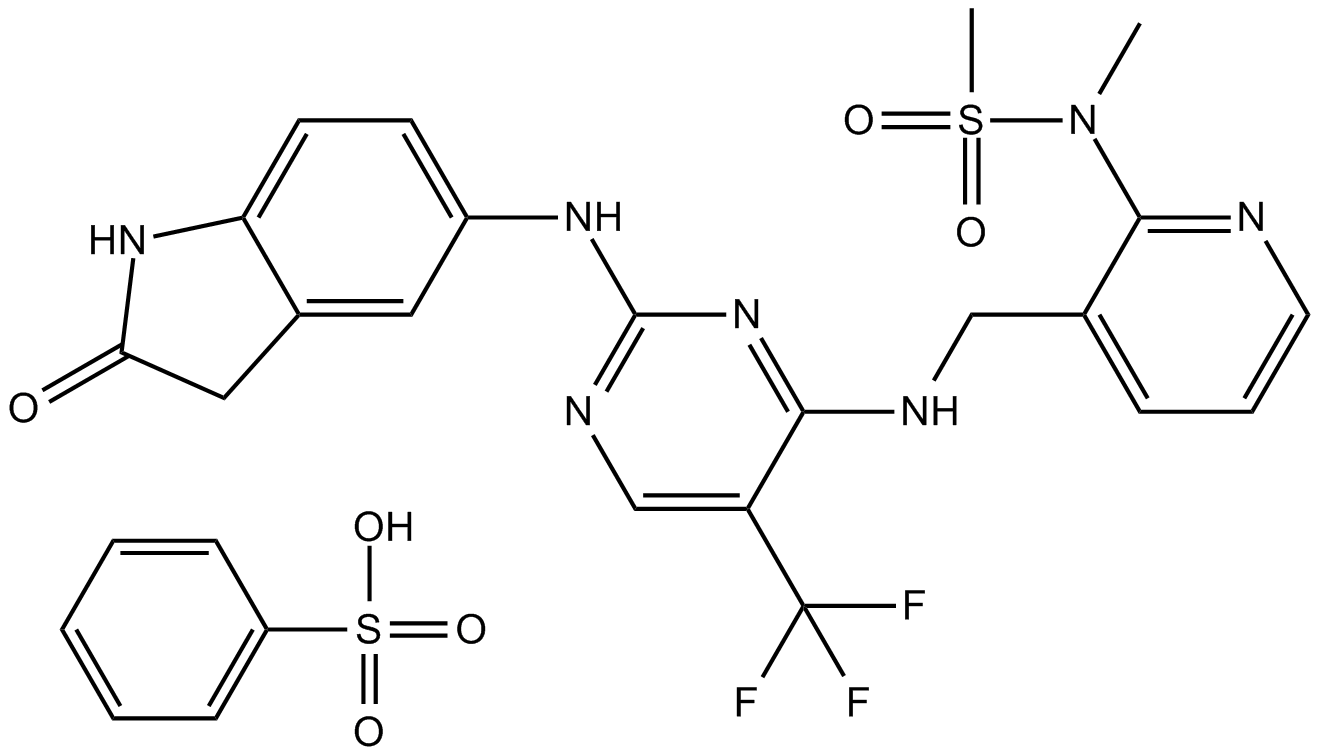
-
GC14407
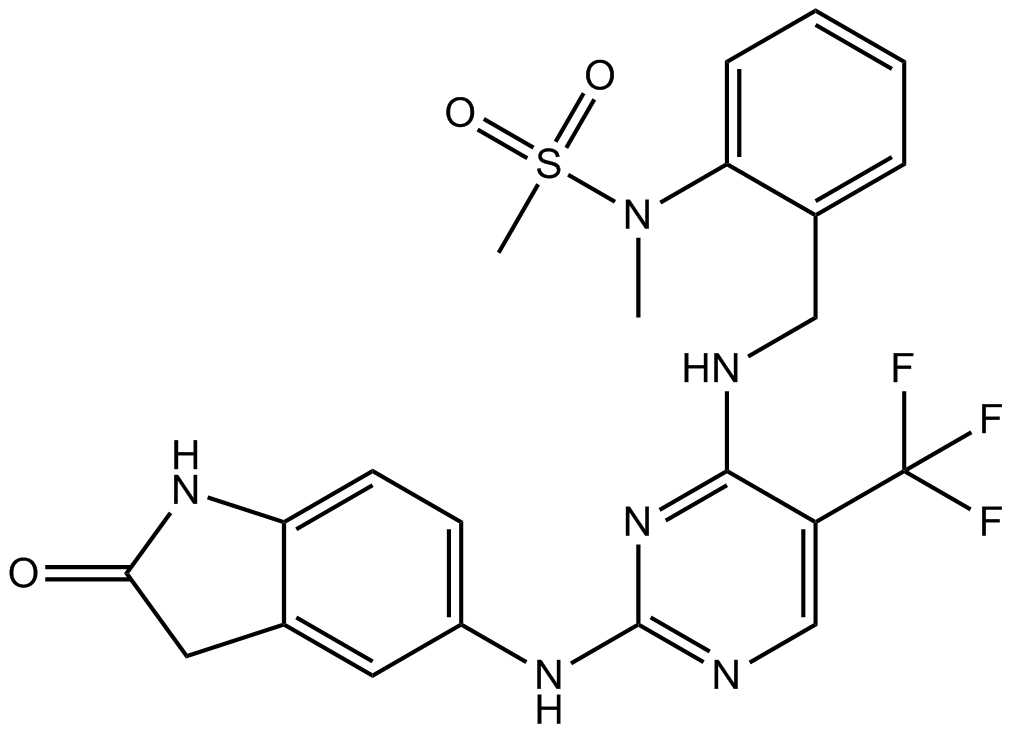
PF-431396
PF-431396 es un inhibidor activo por vÍa oral de la cinasa de adhesiÓn focal dual (FAK) y de la tirosina cinasa 2 rica en prolina (PYK2), con valores IC50 de 2 nM y 11 nM, respectivamente.
-
GC15380
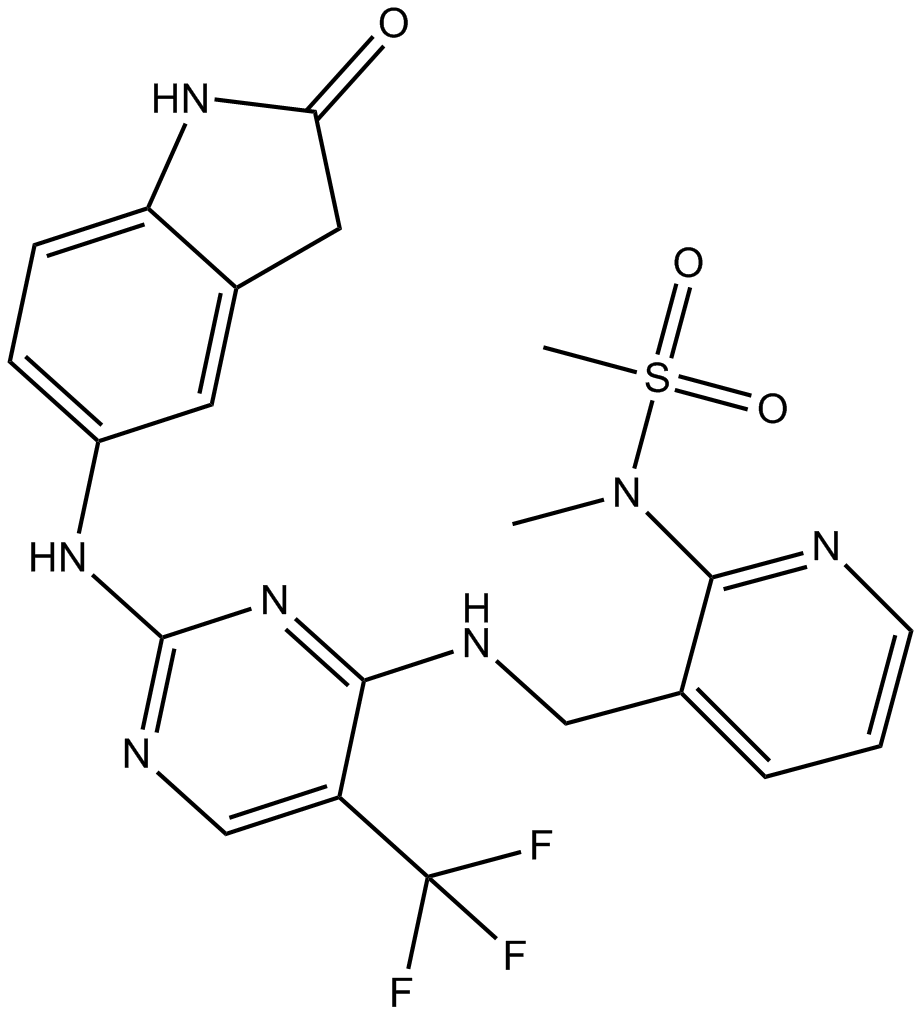
PF-562271
PF-562271 (VS-6062) es un inhibidor de la quinasa FAK y Pyk2 potente, competitivo con ATP y reversible con IC50 de 1,5 nM y 13 nM, respectivamente.
-
GC10810
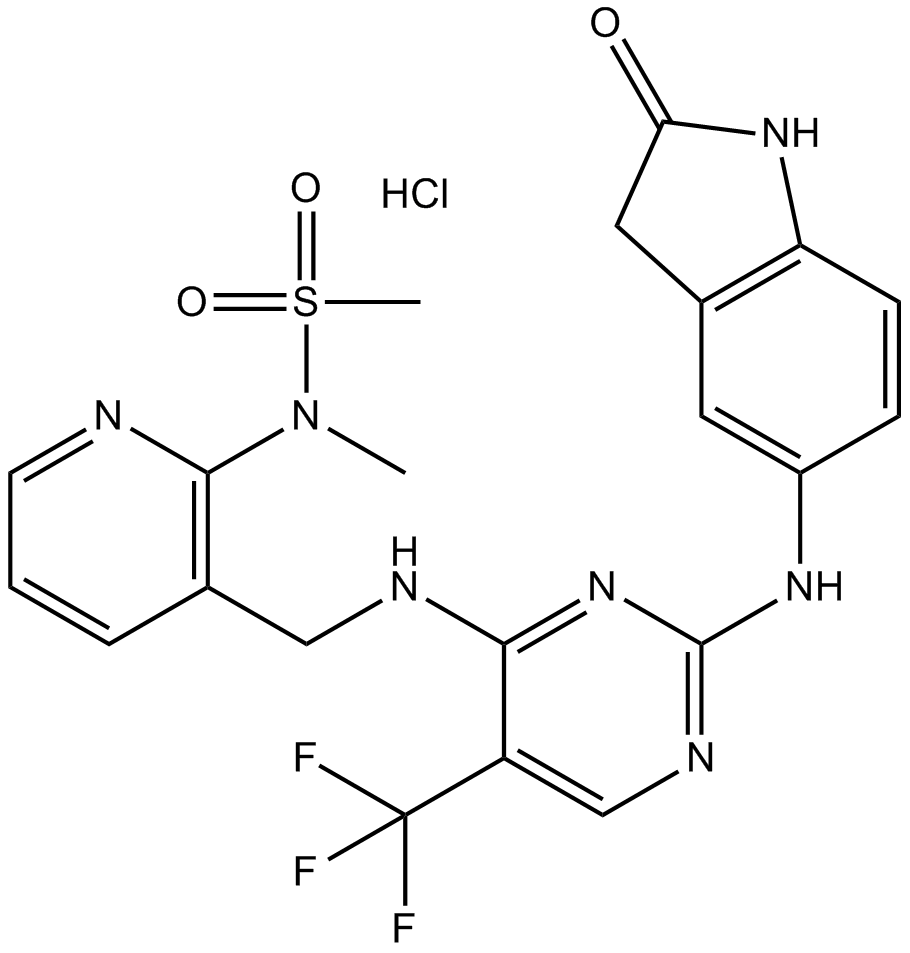
PF-562271 HCl
El clorhidrato de PF-562271 (VS-6062) es un inhibidor de la cinasa FAK y Pyk2 potente, competitivo con ATP y reversible con IC50 de 1,5 nM y 13 nM, respectivamente.
-
GC17352
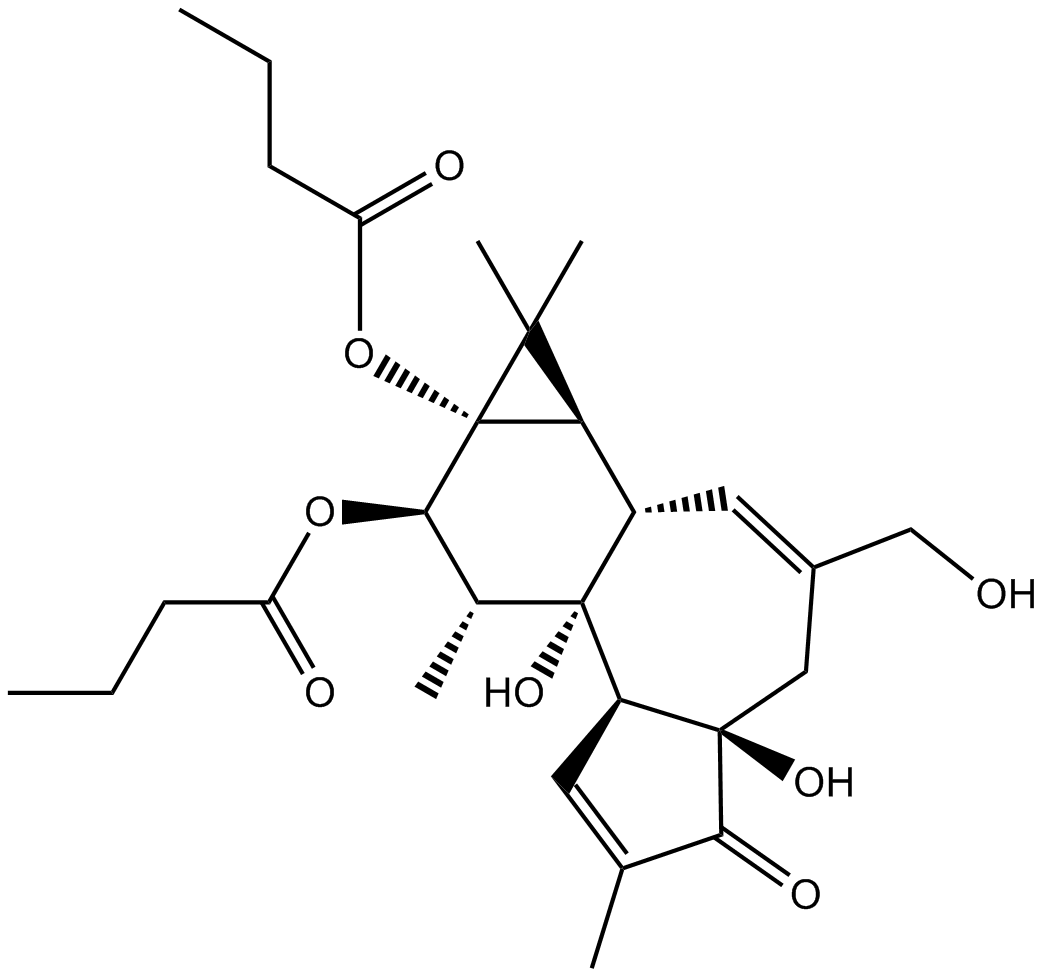
Phorbol 12,13-dibutyrate
El 12,13-dibutirato de forbol (dibutirato de forbol) es un activador de PKC y un potente promotor de tumores cutÁneos.
-
GC12790
Pirfenidone
La pirfenidona (AMR69) es un agente antifibrÓtico que atenÚa la producciÓn de CCL2 y CCL12 en las células de fibrocito.
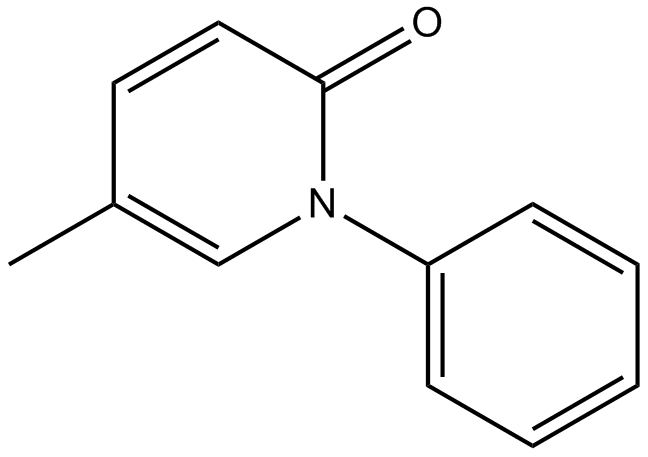
-
GC40197
Pirfenidone-d5
La pirfenidona d5 (AMR69 d5) es una pirfenidona marcada con deuterio.
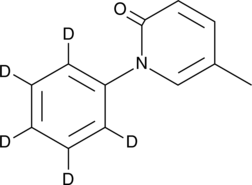
-
GC15471
PKC β pseudosubstrate
Selective cell-permeable peptide inhibitor of protein kinase C

-
GC10404
PKC ζ pseudosubstrate
PKCζ el pseudosustrato es un inhibidor selectivo de la PKC permeable a las células.

-
GC11671
PKC fragment (530-558)
Potent activator of protein kinase C

-
GC44655
PKCε Inhibitor Peptide
El péptido inhibidor PKCε, también llamado εV1-2, es un péptido derivado de la proteína quinasa C ε (PKCε), actúa como un inhibidor selectivo de PKCε e inhibe la translocación de PKCε.
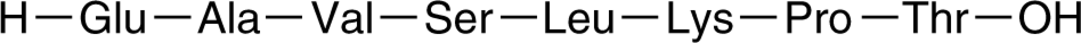
-
GC31711
PKC-IN-1
PKC-IN-1 es un inhibidor potente, competitivo con ATP y reversible de las enzimas PKC convencionales con Kis de 5,3 y 10,4 nM para PKCβ y PKCα humanas, y CI50 de 2,3, 8,1, 7,6, 25,6, 57,5, 314, 808 nM para PKCα, PKCβI, PKCβII, PKCθ, PKCγ, PKC mu y PKCε, respectivamente.
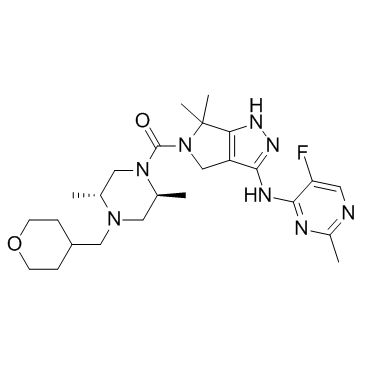
-
GC65126
PKC-iota inhibitor 1
El inhibidor 1 de PKC-iota (compuesto 19) es un inhibidor de la proteÍna quinasa C-iota (PKC-Ι ℩) con un valor IC50 de 0,34 μM.
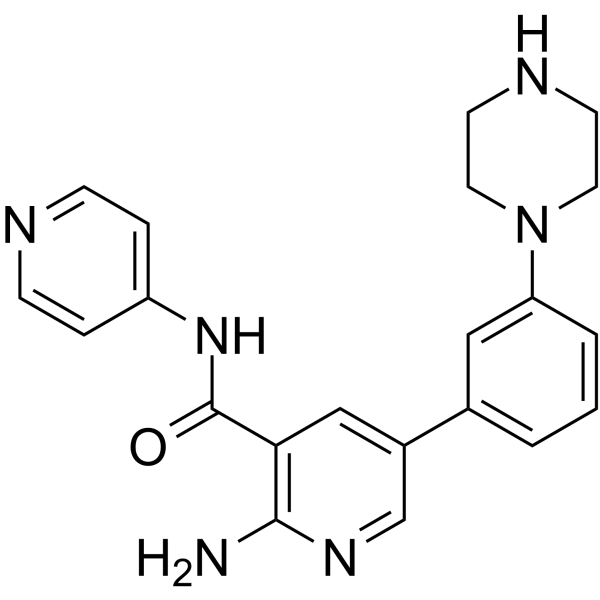
-
GC30200
PKC-theta inhibitor
El inhibidor de PKC-theta es un inhibidor selectivo de PKC-θ, con una IC50 de 12 nM.
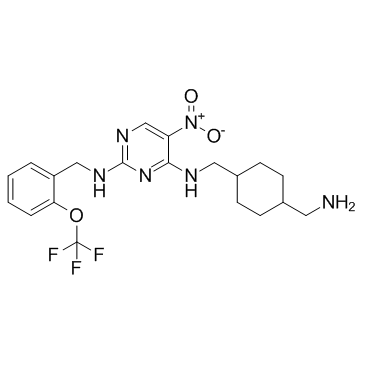
-
GC67686
PKCiota-IN-2 formic
-
GC14396
Ponatinib (AP24534)
Ponatinib (AP24534) (AP24534) es un inhibidor de cinasa multidiana activo por vÍa oral con IC50 de 0,37 nM, 1,1 nM, 1,5 nM, 2,2 nM y 5,4 nM para Abl, PDGFRα, VEGFR2, FGFR1 y Src, respectivamente.
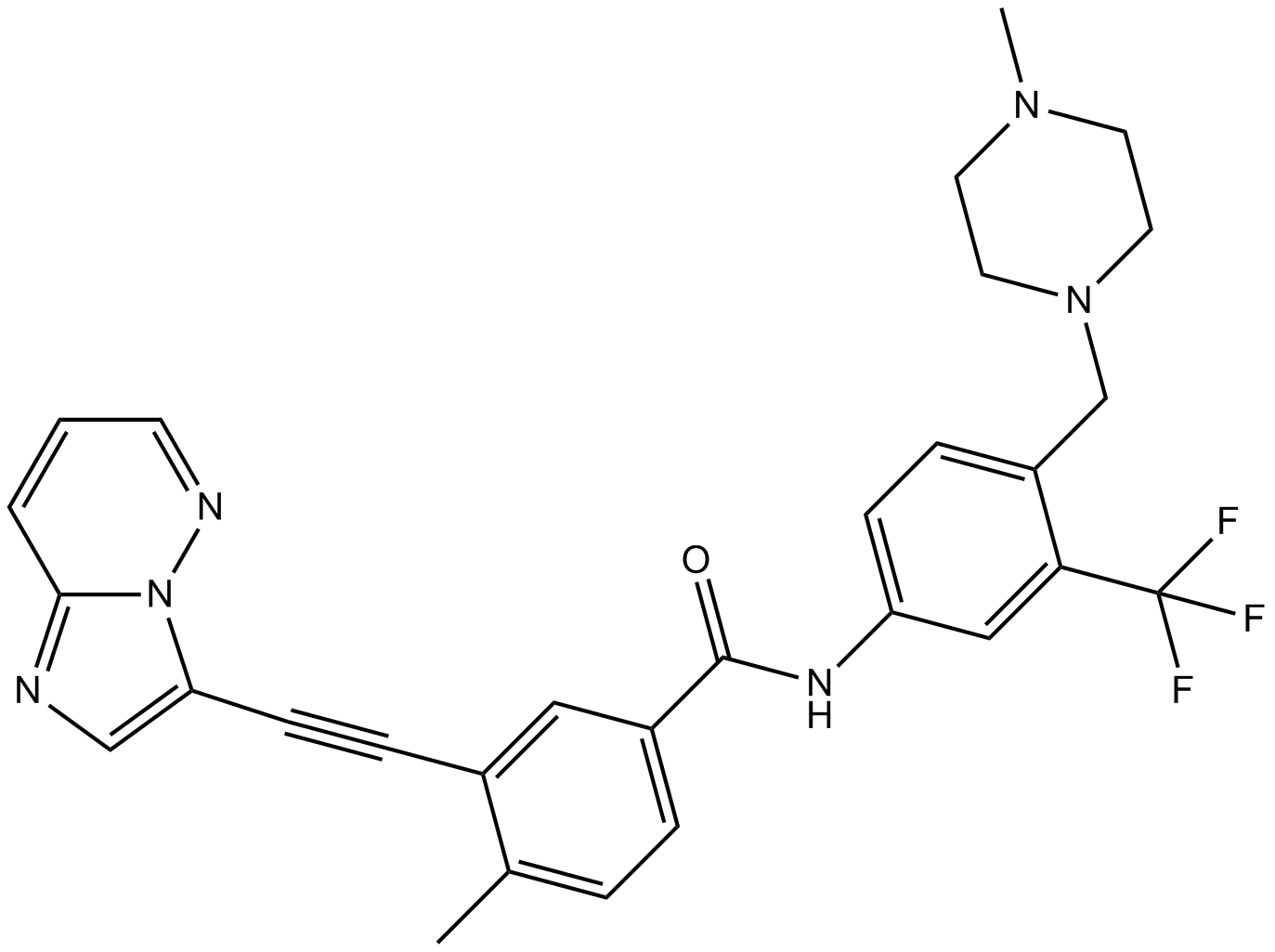
-
GC45828
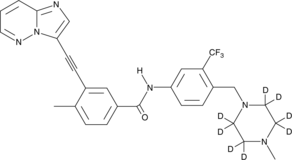
Ponatinib-d8
Ponatinib D8 (AP24534 D8) es un ponatinib marcado con deuterio. Ponatinib (AP24534) es un inhibidor de la cinasa multidiana activo por vÍa oral con IC50 de 0,37 nM, 1,1 nM, 1,5 nM, 2,2 nM y 5,4 nM para Abl, PDGFRα, VEGFR2, FGFR1 y Src, respectivamente.
-
GC68339
Ponsegromab

-
GC12779
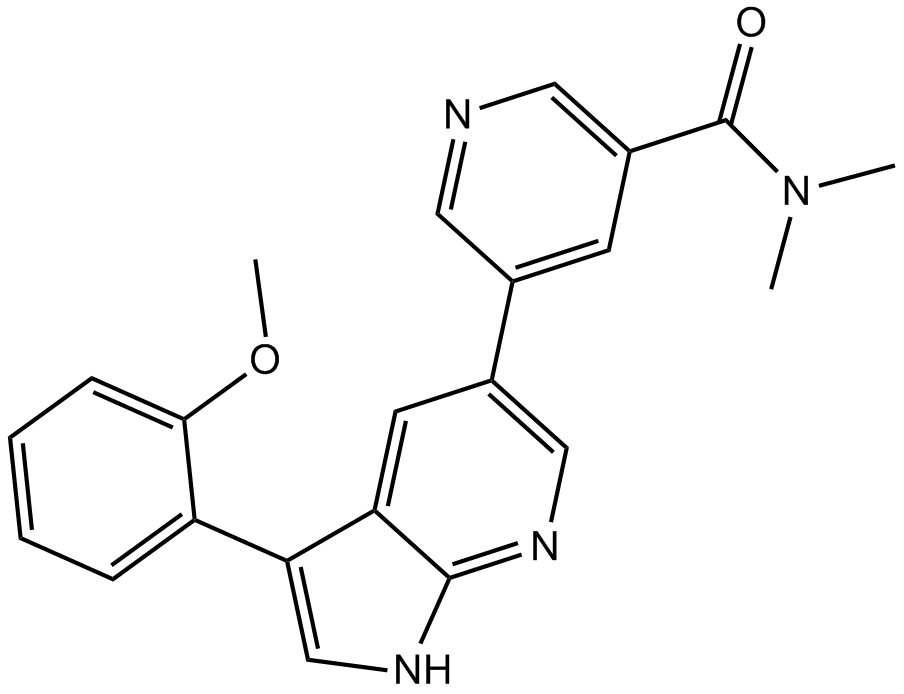
PPY A
PPY A es un potente inhibidor de quinasas Abl mutante T315I y de tipo salvaje con IC50 de 9 y 20 nM, respectivamente. PPY A inhibe las células Ba⁄F3 transformadas con Abl de tipo salvaje y Abl T315I mutante con IC50 de 390 y 180 nM, respectivamente. PPY A se puede utilizar para la investigación de la leucemia mieloide crónica (LMC).
-
GC36974
Procyanidin A1
La procianidina A1 (proantocianidina A1) es un dÍmero de procianidina que inhibe la desgranulaciÓn aguas abajo de la activaciÓn de la proteÍna quinasa C o la entrada de Ca2+ desde un almacén interno en las células RBL-213.



