Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
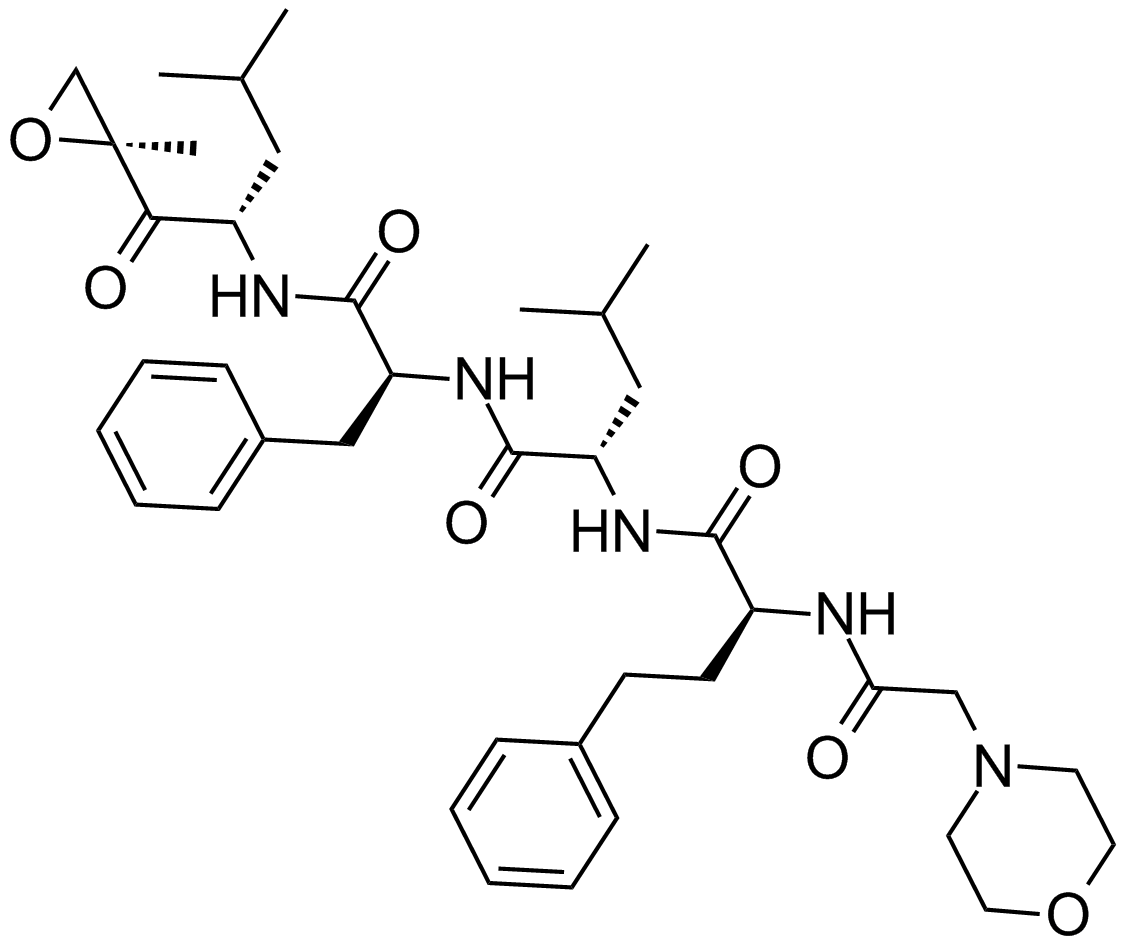
GC12426
Birinapant (TL32711)
Un antagoniste de cIAP1, cIAP2 et XIAP.
-
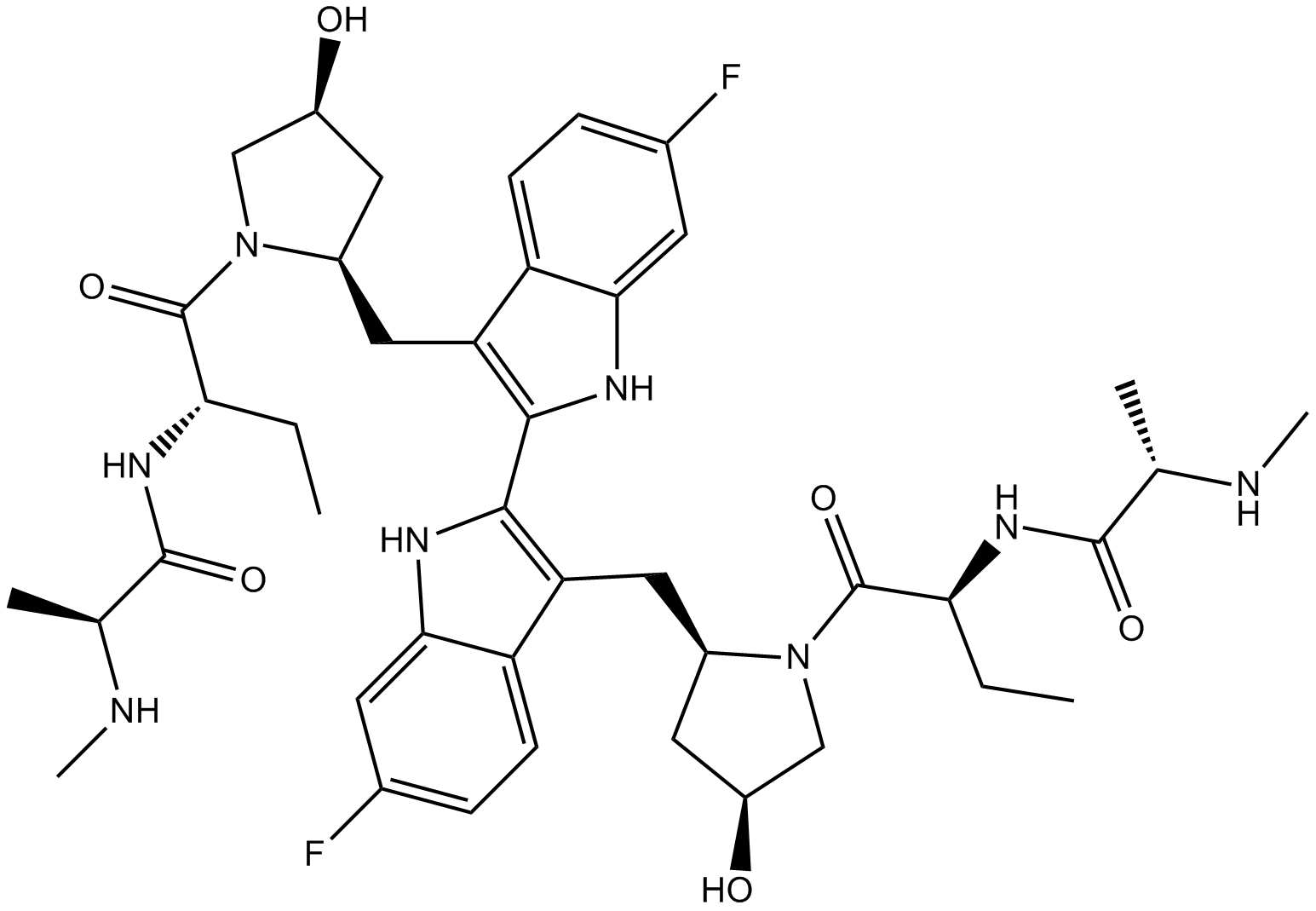
GN10037
Bisdemethoxycurcumin
-
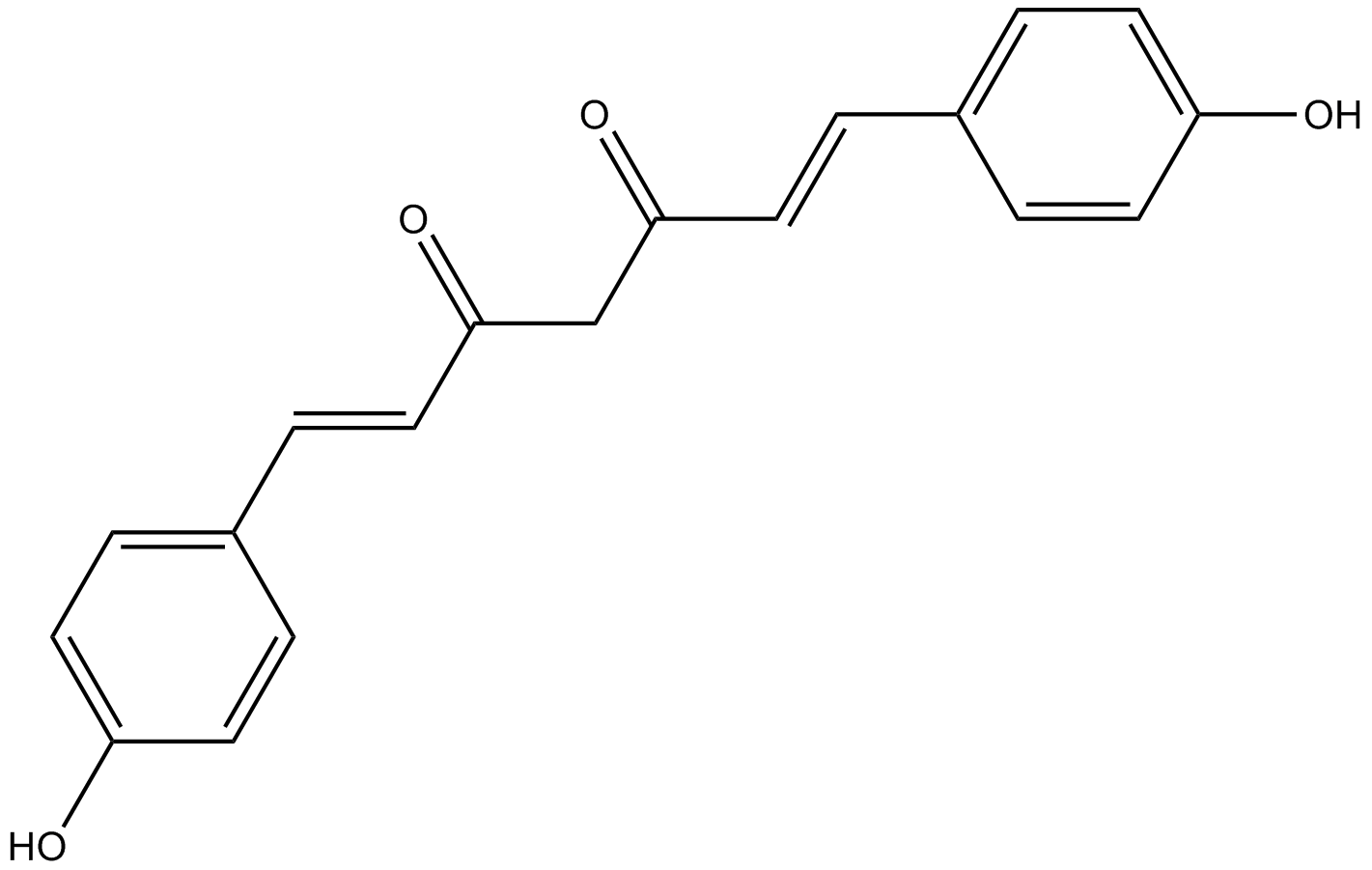
GC68308
Bisdemethoxycurcumin-d8
-
GC35530
BJE6-106
BJE6-106 (B106) est un puissant inhibiteur sélectif de PKCδ de 3e génération avec une IC50 de 0,05 μM et cible la sélectivité par rapport À l'isozyme PKC classique PKCα (IC50 = 50 μM).

-
GC11931
BKM120
An inhibitor of class I PI3K isoforms
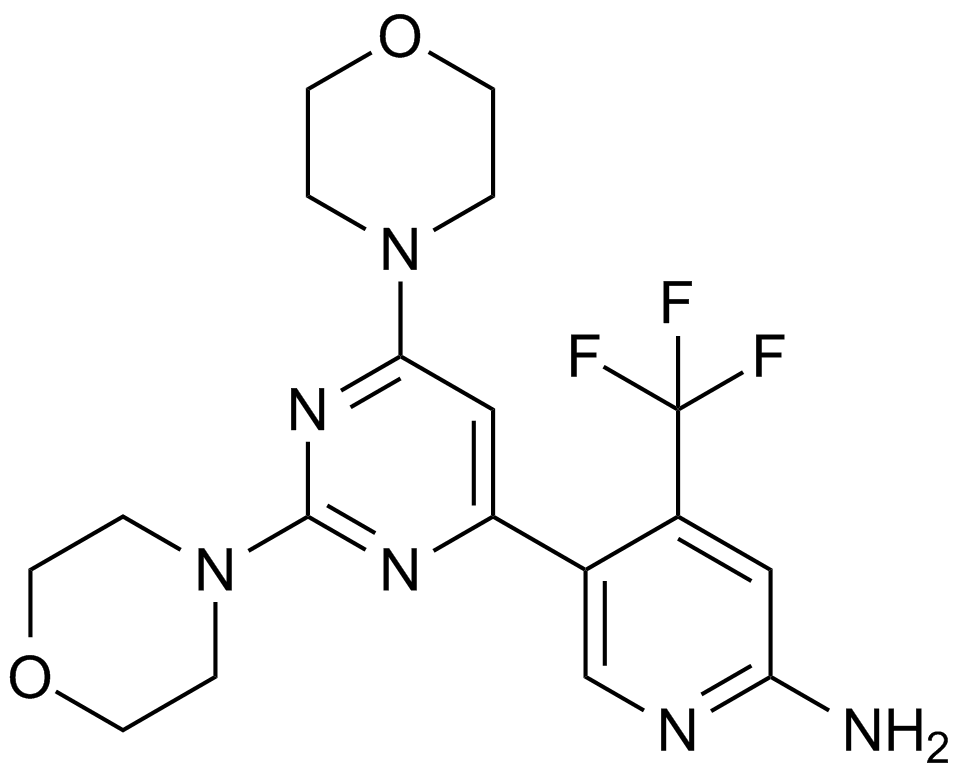
-
GC65428
BLM-IN-1
BLM-IN-1 (composé 29) est un inhibiteur efficace de la protéine du syndrome de Bloom (BLM), avec une forte liaison KD de 1,81 μM et une IC50 de 0,95 μM pour le BLM. Induit une réponse aux dommages À l'ADN, ainsi qu'un arrêt de l'apoptose et de la prolifération dans les cellules cancéreuses.
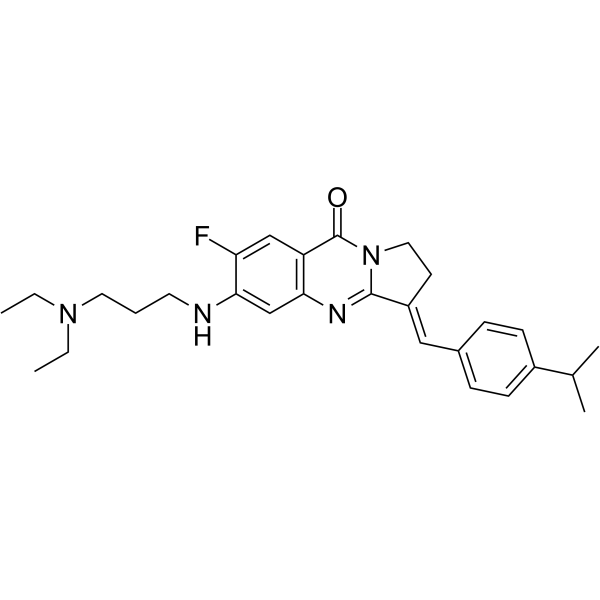
-
GC33407
BM 957
BM 957 est un puissant inhibiteur de Bcl-2 et Bcl-xL, avec Kis de 1,2, <1 nM et IC50 de 5,4, 6,0 nM respectivement.
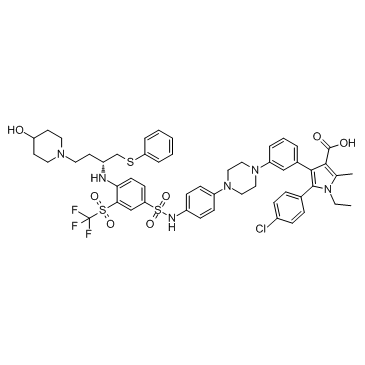
-
GC13498
BM-1074
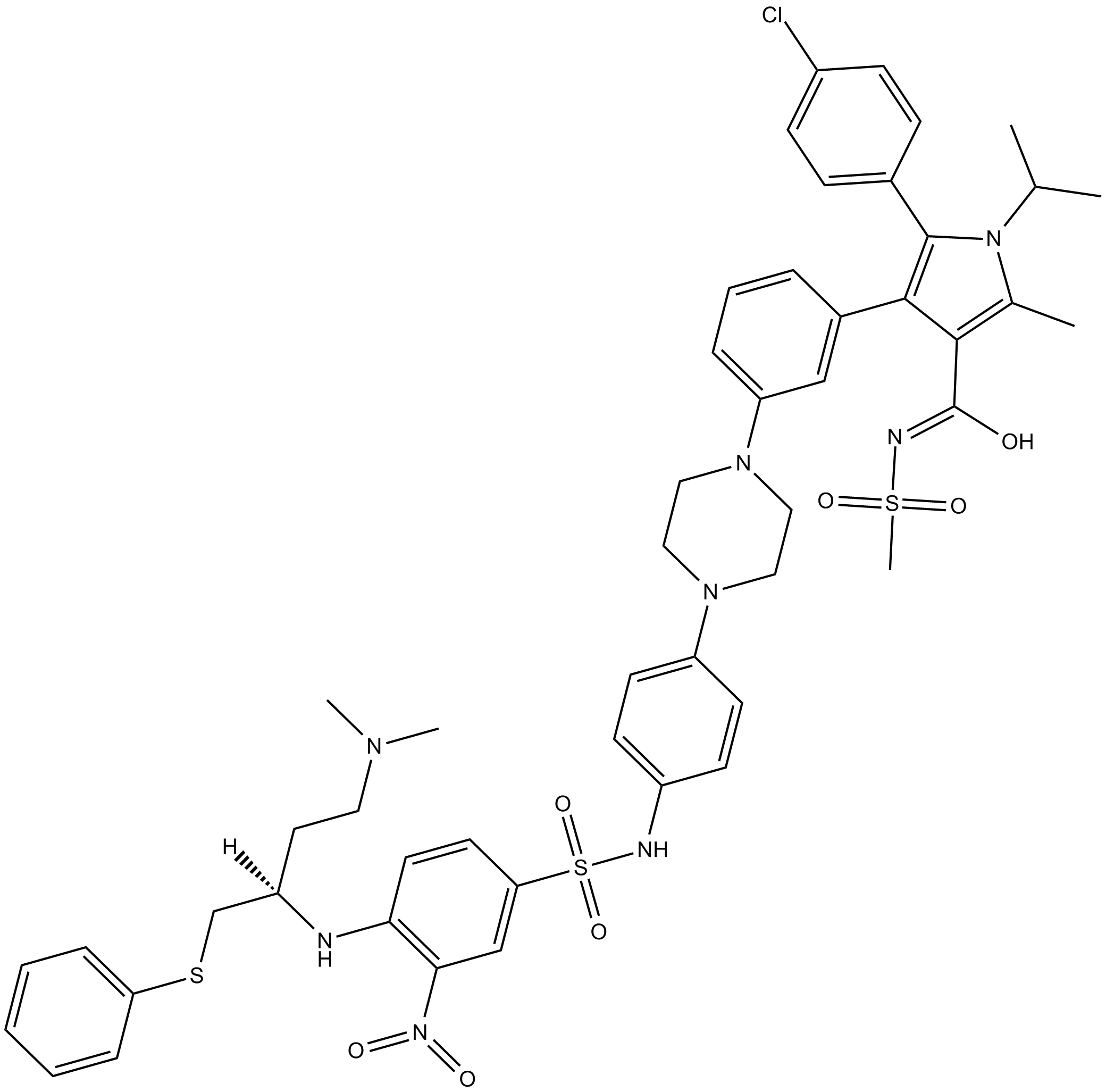
-
GC62871
BM-1244
BM-1244 (APG-1252-M1) est un puissant inhibiteur de Bcl-xL/Bcl-2 avec un Kis de 134 et 450 nM pour Bcl-xL et Bcl-2, respectivement. BM-1244 inhibe les fibroblastes sénescents (SnCs) avec une CE50 de 5 nM. (D'après le brevet WO2019033119A1).
-
GC12822
BML-210(CAY10433)
BML-210(CAY10433) est un nouvel inhibiteur d'HDAC, et son mécanisme d'action n'a pas été caractérisé.
-
GC11648
BML-277
BML-277 est un inhibiteur sélectif de la kinase de point de contrÔle 2 (Chk2) avec une IC50 de 15 nM.
-
GC42953
BMS 345541 (trifluoroacetate salt)
BMS 345541 is a cell permeable inhibitor of the IκB kinases IKKα and IKKβ (IC50s = 4 and 0.3 μM).
-
GC25160
BMS-1001
BMS-1001 is a potent inhibitor of PD-1/PD-L1 interaction with EC50 of 253 nM. BMS-1001 alleviates the inhibitory effect of the soluble PD-L1 on the T-cell receptor-mediated activation of T-lymphocytes.
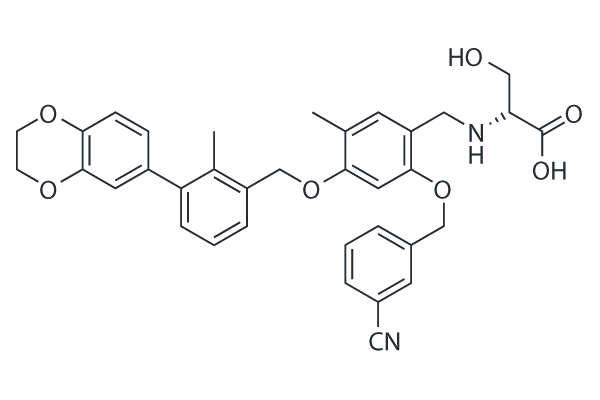
-
GC38740
BMS-1001 hydrochloride
Le chlorhydrate de BMS-1001 est un inhibiteur du point de contrÔle immunitaire humain PD-L1/PD-1 actif par voie orale.
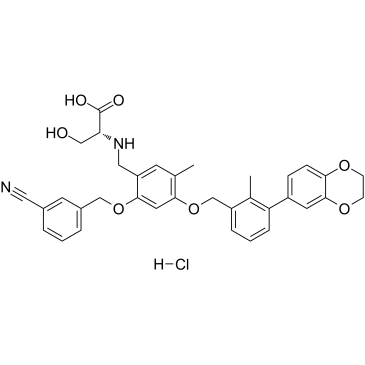
-
GC31753
BMS-1166 (PD-1/PD-L1-IN1)
BMS-1166 (PD-1/PD-L1-IN1) est un puissant inhibiteur du point de contrÔle immunitaire PD-1/PD-L1.
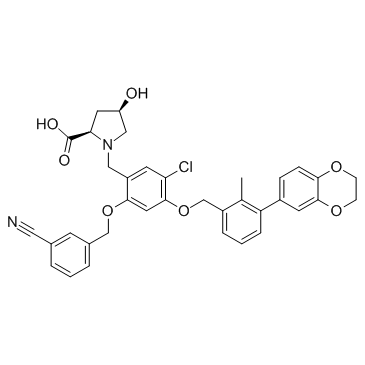
-
GC38131
BMS-1166 hydrochloride
Le chlorhydrate de BMS-1166 est un puissant inhibiteur du point de contrÔle immunitaire PD-1/PD-L1.

-
GC13628
BMS-833923
An orally bioavailable Smo inhibitor
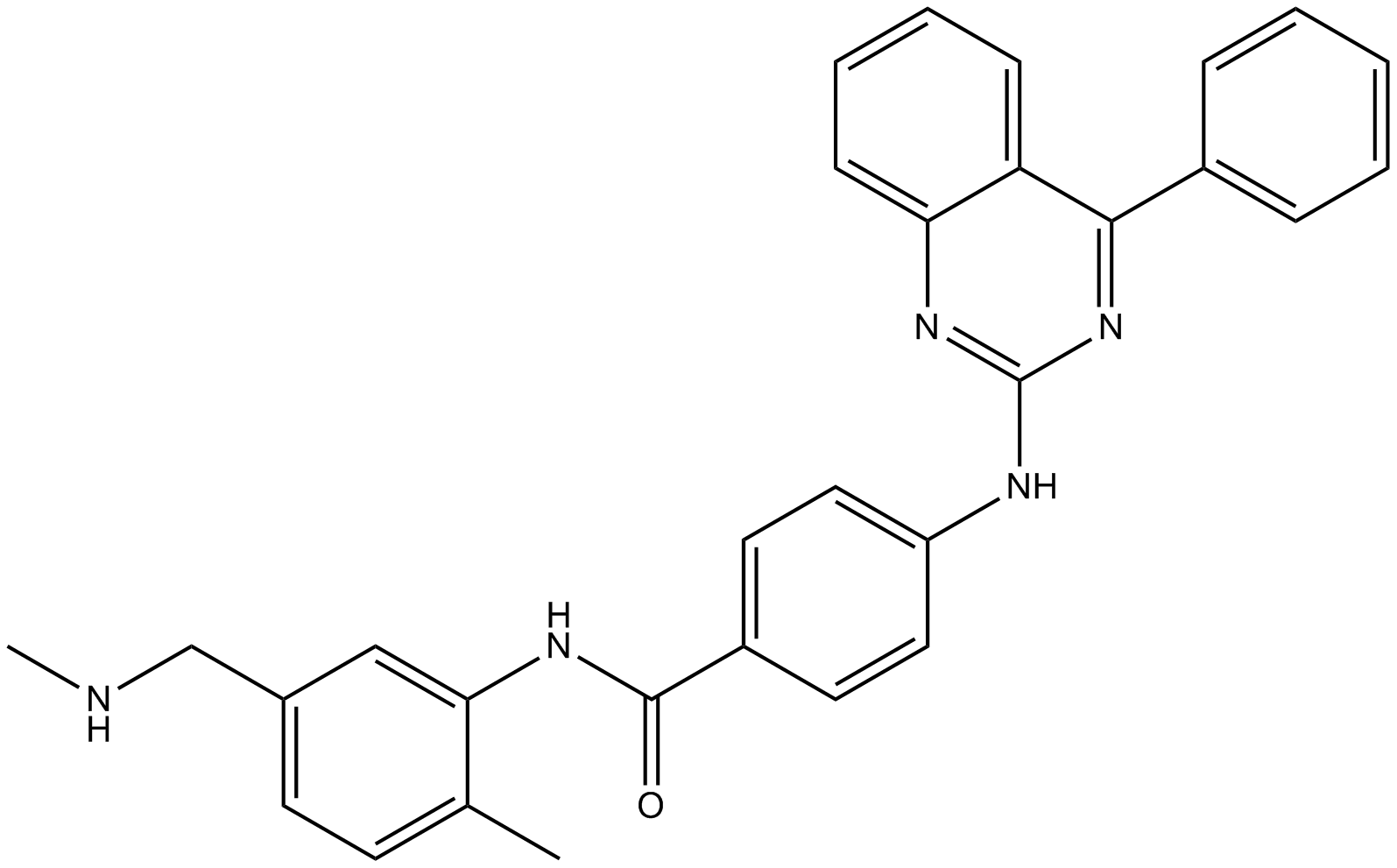
-
GC62682
BMSpep-57 hydrochloride
Le chlorhydrate de BMSpep-57 est un inhibiteur peptidique macrocyclique puissant et compétitif de l'interaction PD-1/PD-L1 avec une IC50 de 7,68nM. Le chlorhydrate de BMSpep-57 se lie À PD-L1 avec des Kd de 19 nM et 19,88 nM dans les tests MST et SPR, respectivement. Le chlorhydrate de BMSpep-57 facilite la fonction des lymphocytes T en augmentant la production d'IL-2 dans les PBMC.
-
GA20897
Boc-Arg(Boc)₂-OH
An amino acid building block

-
GC16774
Boc-D-FMK
An irreversible pan-caspase inhibitor
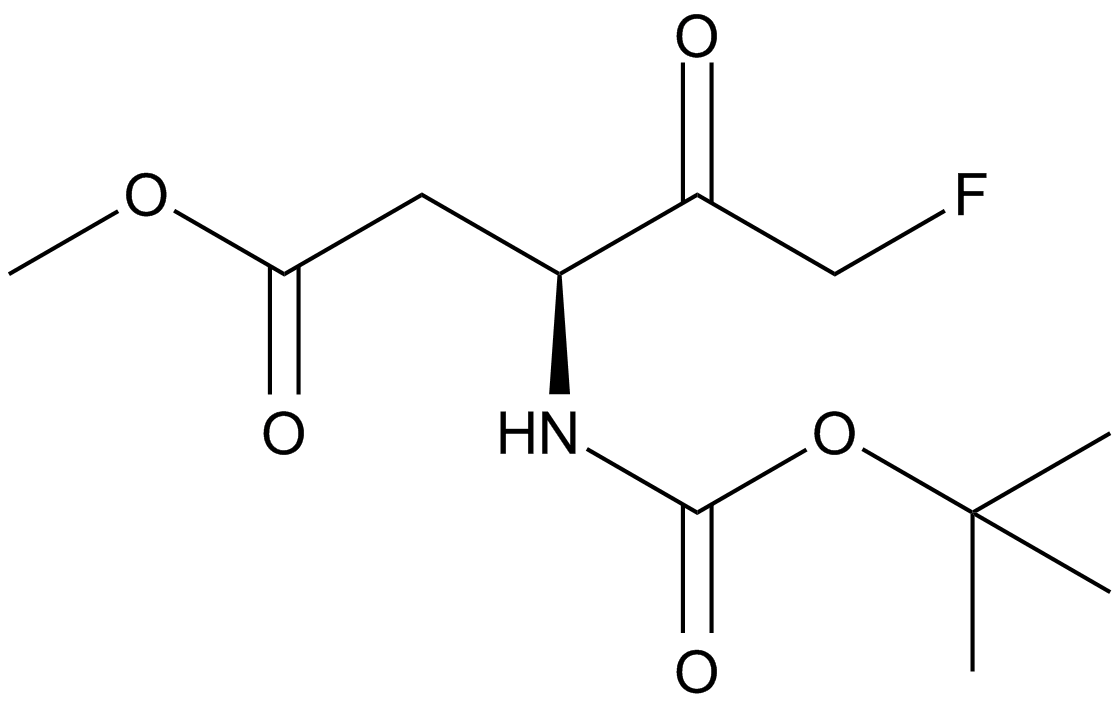
-
GC33501
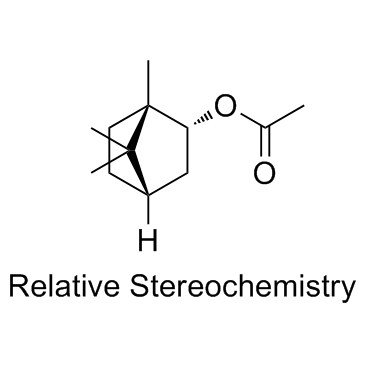
Bornyl acetate
L'acétate de bornyle est un odorant puissant, présentant l'un des facteurs de dilution de saveur les plus élevés (facteur FD). L'acétate de bornyle possède une activité anticancéreuse.
-
GC11040
Borrelidin
La borrélidine (tréponémycine) est un inhibiteur de la thréonyl-ARNt synthétase bactérienne et eucaryale qui est un antibiotique macrolide contenant des nitriles isolé de Streptomyces rochei .
-
GC17644
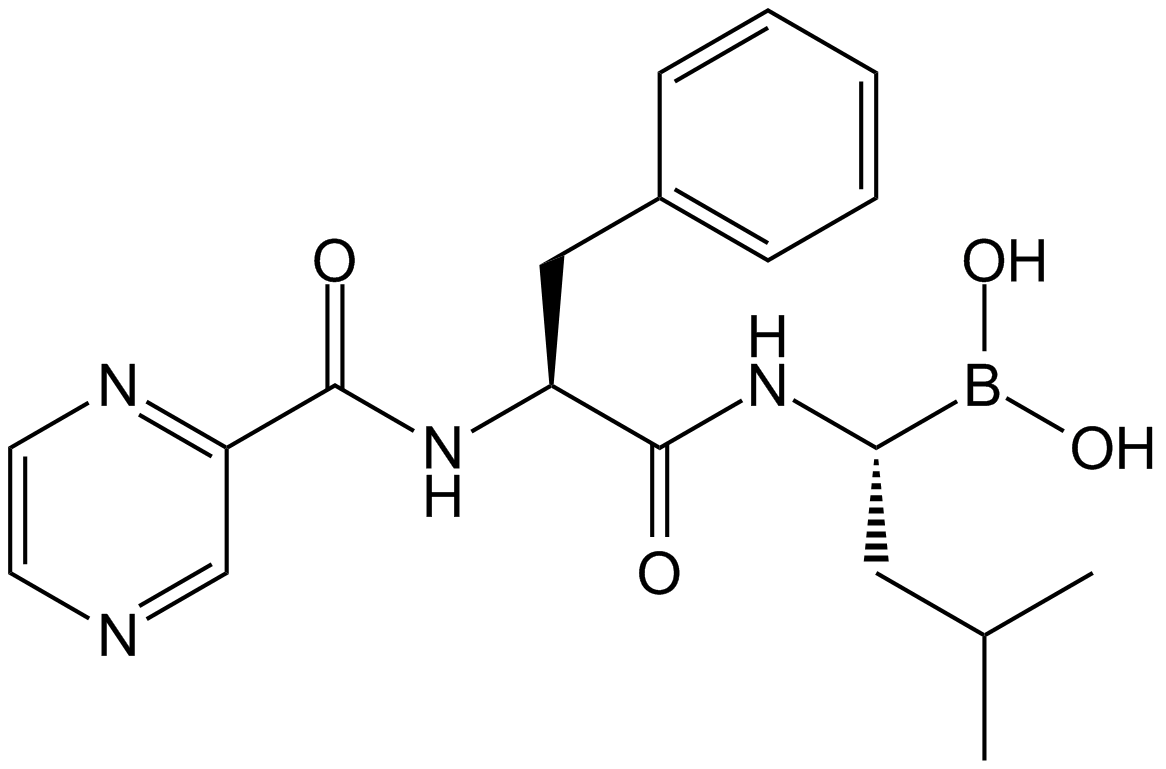
Bortezomib (PS-341)
Un inhibiteur puissant et réversible du protéasome 20S.
-
GC65010
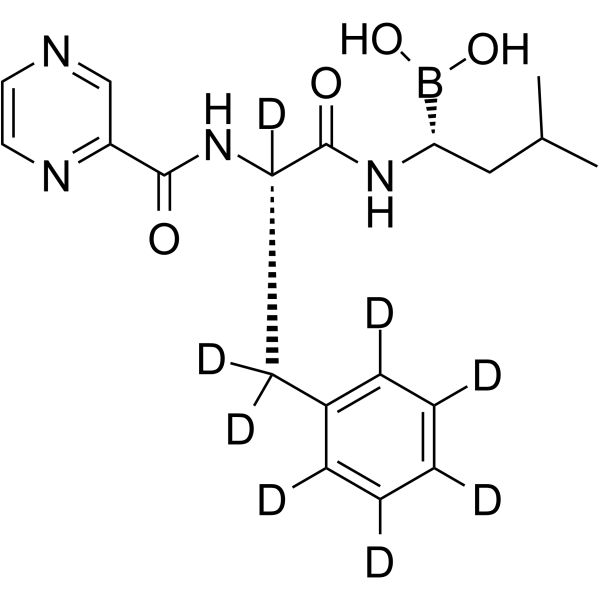
Bortezomib-d8
Bortezomib-d8 (PS-341-d8) est le bortézomib marqué au deutérium. Le bortézomib (PS-341) est un inhibiteur réversible et sélectif du protéasome et inhibe puissamment le protéasome 20S (Ki = 0,6 nM) en ciblant un résidu thréonine. Le bortézomib perturbe le cycle cellulaire, induit l'apoptose et inhibe le NF-κB. Le bortézomib est le premier agent anticancéreux inhibiteur du protéasome. Activité anticancéreuse.
-
GC40009
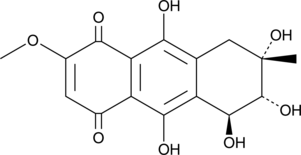
Bostrycin
Bostrycin is an anthraquinone originally isolated from B.
-
GC42969
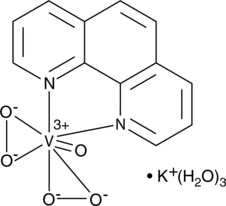
bpV(phen) (potassium hydrate)
bpV(phen) (hydrate de potassium), un agent insulino-mimétique, est un puissant inhibiteur de la protéine tyrosine phosphatase (PTP) et du PTEN avec des IC50 de 38 nM, 343 nM et 920 nM pour le PTEN, PTP-β ; et PTP-1B, respectivement.
-
GC42974
Brassinin
La brassinine est le métabolisme d'une phytoalexine de l'espèce Brassica.
-
GC11632
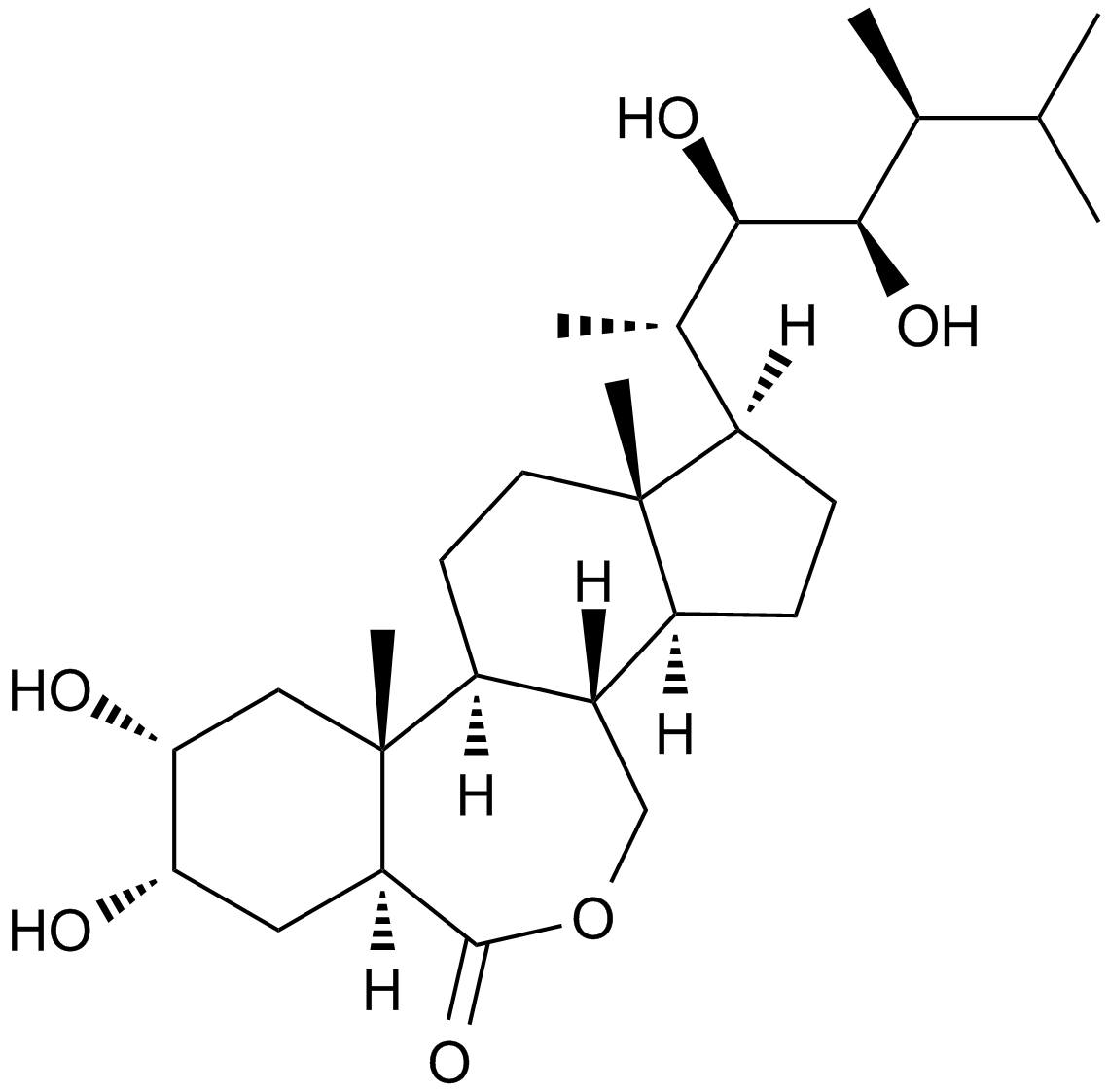
Brassinolide
A plant growth regulator
-
GC52101
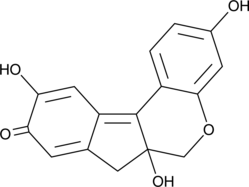
Brazilein
Brazilein est un composant immunosuppresseur important isolé de Caesalpinia sappan L.
-
GN10802
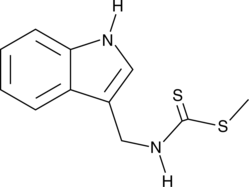
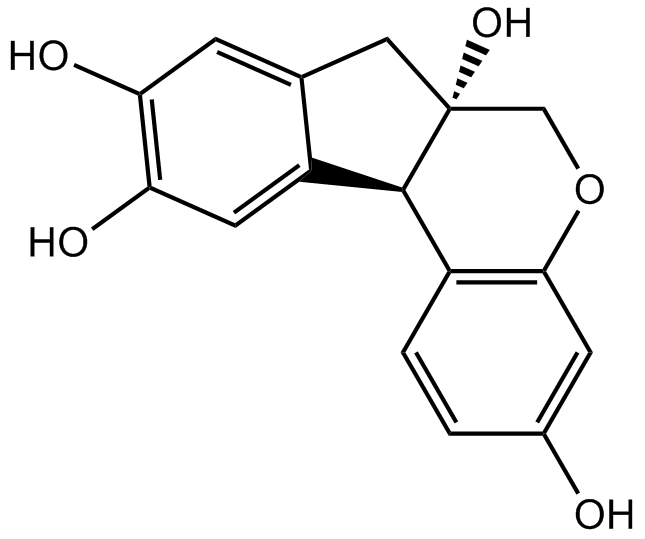
Brazilin
-
GC68288
Brentuximab

-
GC35554
Brevilin A
Brevilin A est un inhibiteur de STAT3/JAK actif par voie orale (STAT3 IC50=10,6 μM). La bréviline A présente une activité anti-tumorale, une activité anti-proliférative sur les cellules cancéreuses et peut induire l'apoptose et l'autophagie.

-
GC35555
Britannin
Britannin, isolé d'Inula aucheriana, est une lactone sesquiterpène.
-
GC62536
Bromelain
La bromélaÏne est un médicament anti-inflammatoire dérivé de la tige d'ananas qui agit par la régulation À la baisse du kininogène plasmatique, l'inhibition de l'expression de la prostaglandine E2, la dégradation des récepteurs avancés du produit final de glycation et la régulation des biomarqueurs angiogéniques ainsi que l'action antioxydante en amont dans la voie COX. .

-
GC35559
Bruceine D
Bruceine D est un inhibiteur de Notch avec une activité anticancéreuse et induit l'apoptose dans plusieurs cellules cancéreuses humaines.
-
GC34070
Brusatol (NSC 172924)
Le brusatol (NSC 172924) (NSC172924) est un inhibiteur unique de la voie Nrf2 qui sensibilise un large spectre de cellules cancéreuses au cisplatine et À d'autres agents chimiothérapeutiques. Le brusatol (NSC 172924) améliore l'efficacité de la chimiothérapie en inhibant le mécanisme de défense médié par Nrf2. Le brusatol (NSC 172924) peut être développé en agent chimiothérapeutique adjuvant. Le brusatol (NSC 172924) augmente l'apoptose cellulaire.
-
GC38014
BT2
BT2 est un inhibiteur de la BCKDC kinase (BDK) avec une IC50 de 3,19 μM.
-
GC38467
BTdCPU
BTdCPU est un puissant activateur de la kinase eIF2α régulée par l'hème (HRI). BTdCPU favorise la phosphorylation de eIF2α et induit l'apoptose dans les cellules résistantes.
-
GC34511
BTR-1
BTR-1 est un agent anticancéreux actif, provoque l'arrêt de la phase S et affecte la réplication de l'ADN dans les cellules leucémiques. BTR-1 active l'apoptose et induit la mort cellulaire.
-
GC11141
BTZO 1
BTZO 1 se lie au facteur inhibiteur de la migration des macrophages (MIF) avec une valeur de Kd de 68,6 nM, et sa liaison nécessite le Pro1 N-terminal.
-
GC48376
Burnettramic Acid A
A fungal metabolite with diverse biological activities
-
GC48409
Burnettramic Acid A aglycone
Acide burnettramique Un aglycone est un métabolite fongique présent dans Aspergillus burnettii.
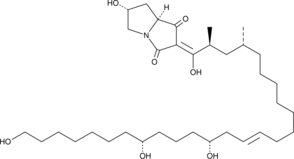
-
GC13671
Busulfan
Le busulfan est un puissant alkylant ayant un effet immunosuppresseur sélectif sur la moelle osseuse.
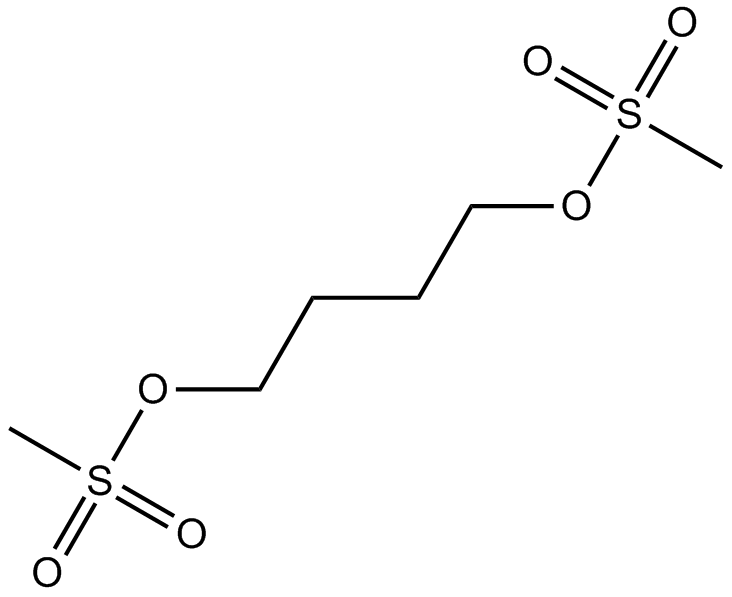
-
GC46962
Busulfan-d8
Le Busulfan-D8 est un Busulfan marqué au deutérium. Le busulfan est un sulfonate d'alkyle qui agit comme agent antinéoplasique alkylant. Le busulfan forme À la fois des réticulations intra et interbrin sur l'ADN. Chez les mammifères, le busulfan provoque une réduction profonde et prolongée de la génération de progéniteurs hématopoÏétiques sans affecter de manière significative les taux de lymphocytes ou les réponses humorales en anticorps.
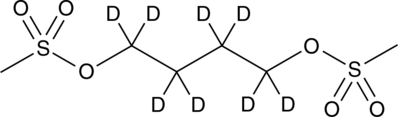
-
GC10944
Butein
La butéine est un inhibiteur de la PDE spécifique de l'AMPc avec une IC50 de 10,4 μM pour la PDE4. La butéine est un inhibiteur spécifique de la protéine tyrosine kinase avec des IC50 de 16 et 65 μM pour l'EGFR et le p60c-src dans les cellules HepG2. La butéine sensibilise les cellules HeLa au cisplatine par les voies AKT et ERK/p38 MAPK en ciblant FoxO3a. La butéine est un activateur SIRT1 (STAC).
-
GC46104
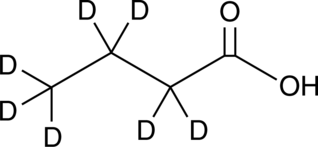
Butyric Acid-d7
An internal standard for the quantification of sodium butyrate
-
GC12333
BV6
BV6 est un antagoniste de cIAP1 et XIAP, membres de la famille des inhibiteurs de l'apoptose (IAP).
-
GC48433
BX-320
Le BX-320 est un inhibiteur sélectif, compétitif pour l'ATP, actif par voie orale et direct de PDK1 avec une CI50 de 30 nM dans un format de dosage direct de la kinase. Le BX-320 induit également l'apoptose. Effet anticancéreux.
-
GC16818
BX-912
Le BX-912 est un inhibiteur de PDK1 direct, sélectif et compétitif pour l'ATP (IC50 = 26 nM). Le BX-912 bloque la signalisation PDK1/Akt dans les cellules tumorales et inhibe la croissance dépendante de l'ancrage d'une variété de lignées cellulaires tumorales en culture ou induit l'apoptose.
-
GC31806
Bz 423 (BZ48)
Bz 423 (BZ48) est une 1,4-benzodiazépine pro-apoptotique aux propriétés thérapeutiques dans des modèles murins de lupus démontrant une sélectivité pour les lymphocytes autoréactifs, et active Bax et Bak.
-
GC33826
C 87
C 87 est un nouvel inhibiteur de TNFα À petite molécule; inhibe puissamment la cytotoxicité induite par le TNFα avec une IC50 de 8,73 μM.
-
GC19095
C-DIM12
C-DIM12 est un activateur synthétique de Nurr1 qui induit l'expression des gènes Nurr1 et DA dans les lignées cellulaires et les neurones primaires.
-
GC43028
C16 Ceramide (d18:1/16:0)
Les céramides sont générés à partir de la sphingomyéline par activation des sphingomyélinases ou par la voie de synthèse de novo, qui nécessite une action coordonnée de la sérine palmitoyl transférase et de la ceramide synthase.
-
GC46976
C16 Ceramide-d7 (d18:1-d7/16:0)
An internal standard for the quantification of C-16 ceramide
-
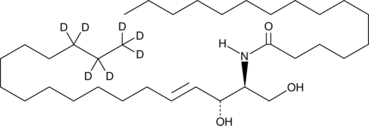
GC43052
C18 Phytoceramide (t18:0/18:0)
C18 Phytoceramide (t18:0/18:0) (Cer(t18:0/18:0)) is a bioactive sphingolipid found in S.
-
GC40141
C18 Phytoceramide-d3 (t18:0/18:0-d3)
C18 Phytoceramide-d3 (t18:0/18:0-d3) is intended for use as an internal standard for the quantification of C18 phytoceramide (t18:0/18:0) by GC- or LC-MS.

-
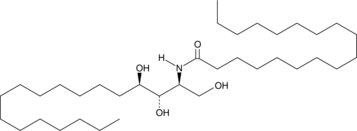
GC43065
C2 Phytoceramide (t18:0/2:0)
C2 Phytoceramide is a bioactive semisynthetic sphingolipid that inhibits formyl peptide-induced oxidant release (IC50 = 0.38 μM) in suspended polymorphonuclear cells.
-
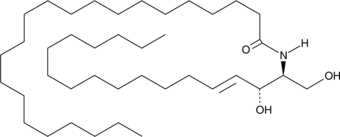
GC43069
C22 Ceramide (d18:1/22:0)
C-22 ceramide is an endogenous bioactive sphingolipid.
-
GC43075
C24 dihydro Ceramide (d18:0/24:0)
C24 dihydro Ceramide is a sphingolipid that has been found in the stratum corneum of human skin.
-
GC34513
C25-140
C25-140, un premier inhibiteur de TRAF6-Ubc13 actif par voie orale et assez sélectif, se lie directement À TRAF6 et bloque l'interaction de TRAF6 avec Ubc13.
-
GC43084
C4 Ceramide (d18:1/4:0)
C4 Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC40688
C6 D-threo Ceramide (d18:1/6:0)
C6 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides., C6 D-threo Ceramide is cytotoxic to U937 cells in vitro (IC50 = 18 μM).
-
GC40689
C6 L-erythro Ceramide (d18:1/6:0)
C6 L-erythro Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
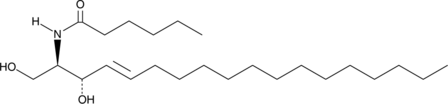
GC40690
C6 L-threo Ceramide (d18:1/6:0)
C6 L-threo Ceramide (d18:1/6:0) est un sphingolipide bioactif et un analogue perméable aux cellules des céramides naturels.
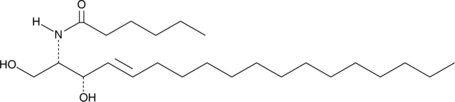
-
GC45616
C6 Urea Ceramide
An inhibitor of neutral ceramidase
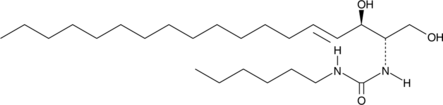
-
GC12733
C646
C646 est un inhibiteur sélectif et compétitif de l'histone acétyltransférase p300 avec Ki de 400 nM, et est moins puissant pour les autres acétyltransférases.
-
GC43105
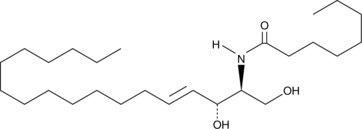
C8 Ceramide (d18:1.8:0)
C8 Céramide (d18:1.8:0) (N-octanoyl-D-érythro-sphingosine) est un analogue perméable aux cellules des céramides naturels.
-
GC43109
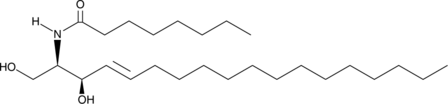
C8 D-threo Ceramide (d18:1/8:0)
C8 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC43110
C8 Galactosylceramide (d18:1/8:0)
C8 Galactosylceramide is a synthetic C8 short-chain derivative of known membrane microdomain-forming sphingolipids.

-
GC43111
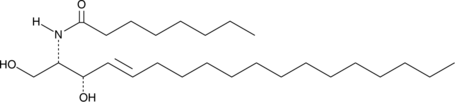
C8 L-threo Ceramide (d18:1/8:0)
C8 L-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC33218
CA-5f
CA-5f est un puissant inhibiteur de macroautophagie/autophagie À un stade avancé via l'inhibition de la fusion autophagosome-lysosome. CA-5f augmente LC3B-II (un marqueur pour surveiller l'autophagie) et la protéine SQSTM1, et augmente également la production de ROS. Activité anti-tumorale.
-
GC15779
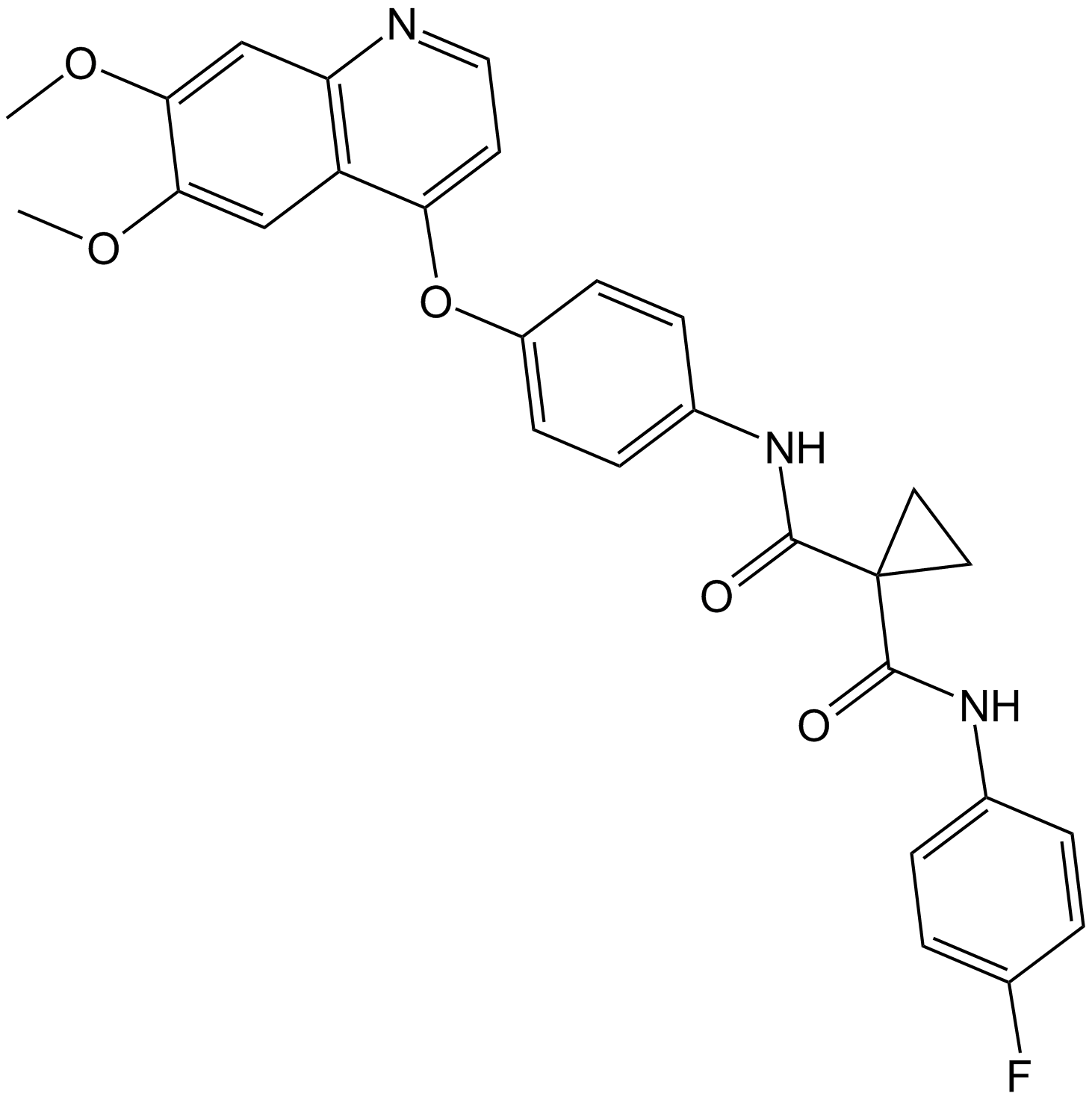
Cabozantinib (XL184, BMS-907351)
Le cabozantinib (XL184, BMS-907351) est un inhibiteur puissant et actif par voie orale du VEGFR2 et du MET, avec des valeurs IC50 de 0,035 et 1,3 nM, respectivement. Le cabozantinib (XL184, BMS-907351) affiche une forte inhibition de KIT, RET, AXL, TIE2 et FLT3 (IC50 = 4,6, 5,2, 7, 14,3 et 11,3 nM, respectivement). Le cabozantinib (XL184, BMS-907351) présente une activité anti-angiogénique. Le cabozantinib (XL184, BMS-907351) perturbe la vascularisation tumorale et favorise l'apoptose des cellules tumorales et endothéliales.
-
GC12531
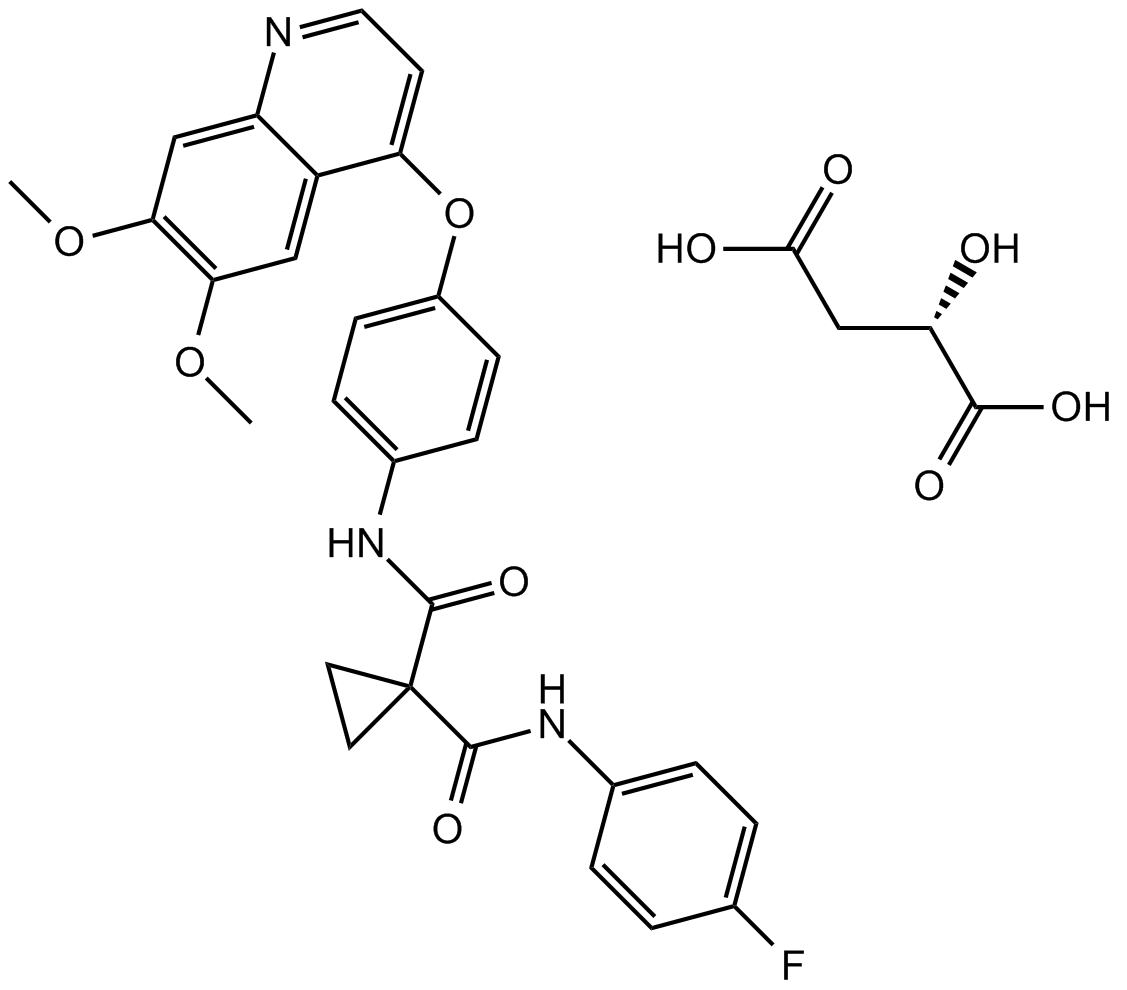
Cabozantinib malate (XL184)
Le cabozantinib malate (XL184) (XL184 S-malate) est un inhibiteur puissant de multiples récepteurs de tyrosine kinases qui inhibe VEGFR2, c-Met, Kit, Axl et Flt3 avec des IC50 de 0,035 ; 1,3 ; 4,6 ; 7 et 11,3 nM respectivement.
-
GC10692
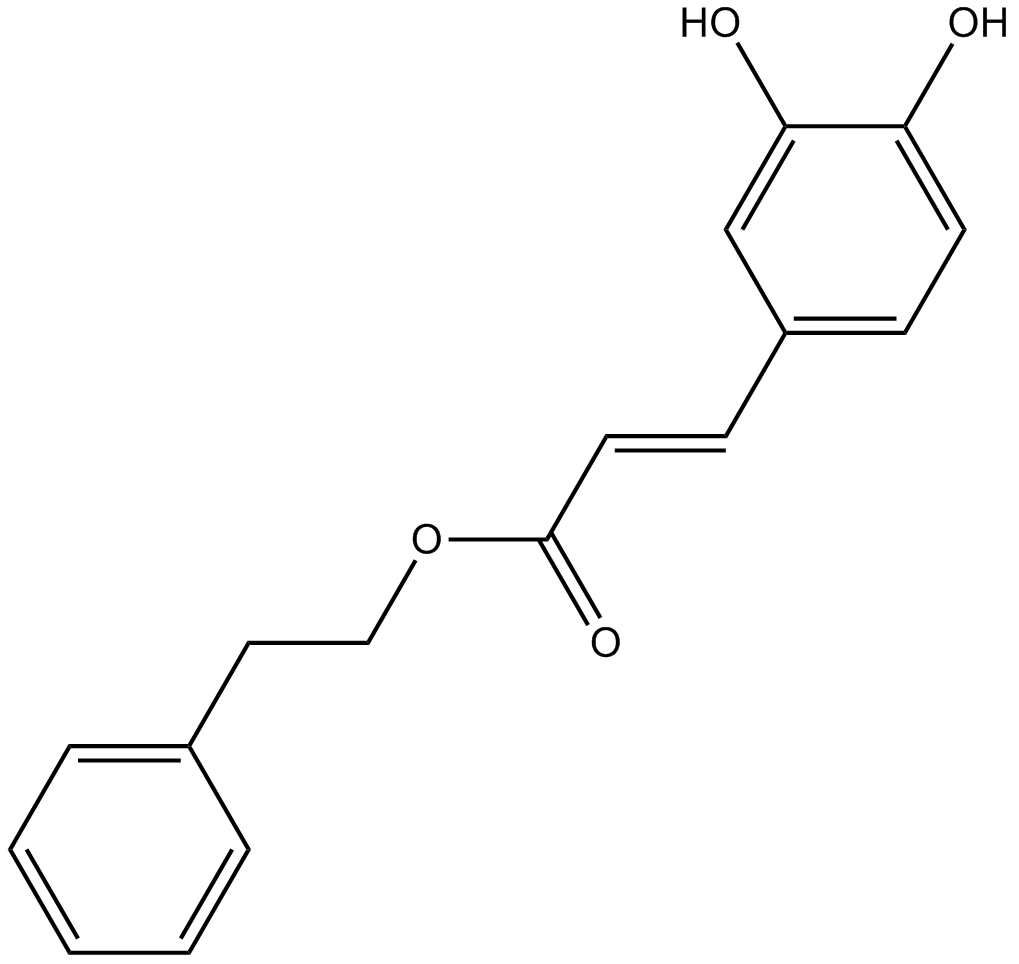
Caffeic Acid Phenethyl Ester
L'ester phénéthylique de l'acide caféique est un inhibiteur de NF-κB.
-
GC18604
Calcein Blue AM
Calcein Blue AM est un colorant cellulaire.
-
GC43121
Calcein Orange™ Diacetate
Calcein Orange Diacetate is a fluorogenic dye that is used to assess cell viability.

-
GC43123
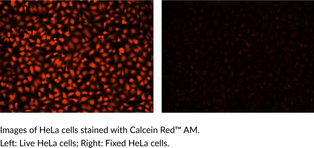
Calcein Red™ AM
Calcein Red? AM is a fluorogenic dye that is used to assess cell viability.
-
GC60668
Calcimycin hemimagnesium
La calcimycine (A-23187) hémimagnésium est un antibiotique et un ionophore cationique divalent unique (comme le calcium et le magnésium).
-
GC15161
Calcium D-Panthotenate
Le D-panthoténate de calcium (sel de calcium de la vitamine B5), une vitamine, peut réduire la teneur en patuline du jus de pomme.
-
GC30240
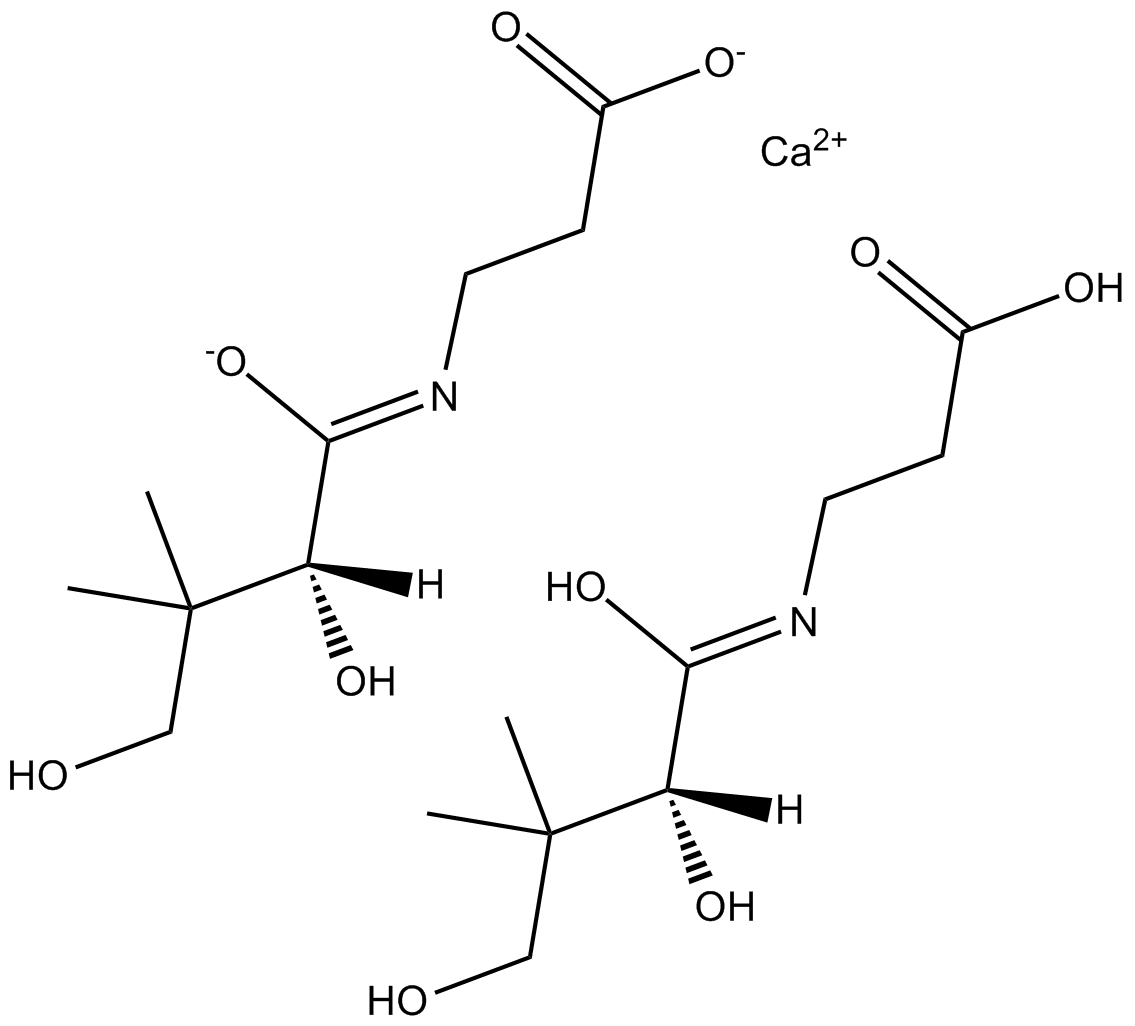
Calcium dobesilate
Le dobésilate de calcium, un vasoprotecteur, est largement utilisé dans les maladies veineuses chroniques, la rétinopathie diabétique et les symptÔmes de crise hémorroÏdaire dans de nombreux pays.
-
GC19086
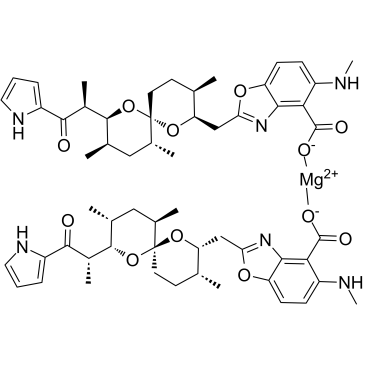
Calicheamicin
La calichéamicine, un antibiotique antitumoral, est un agent cytotoxique qui provoque des cassures double brin de l'ADN.
-
GC65081
CALP1 TFA
CALP1 TFA est un agoniste de la calmoduline (CaM) (Kd de 88 μM) avec liaison au site de liaison CaM EF-hand/Ca2+-.
-
GC18315
Calpain Inhibitor VI
Calpain inhibitor VI is an inhibitor of the calcium-dependent cysteine proteases u-calpain (calpain-1; IC50 = 7.5 nM) and m-calpain (calpain-2; IC50 = 78 nM).
-
GC10342
Calpeptin
A calpain inhibitor
-
GN10667
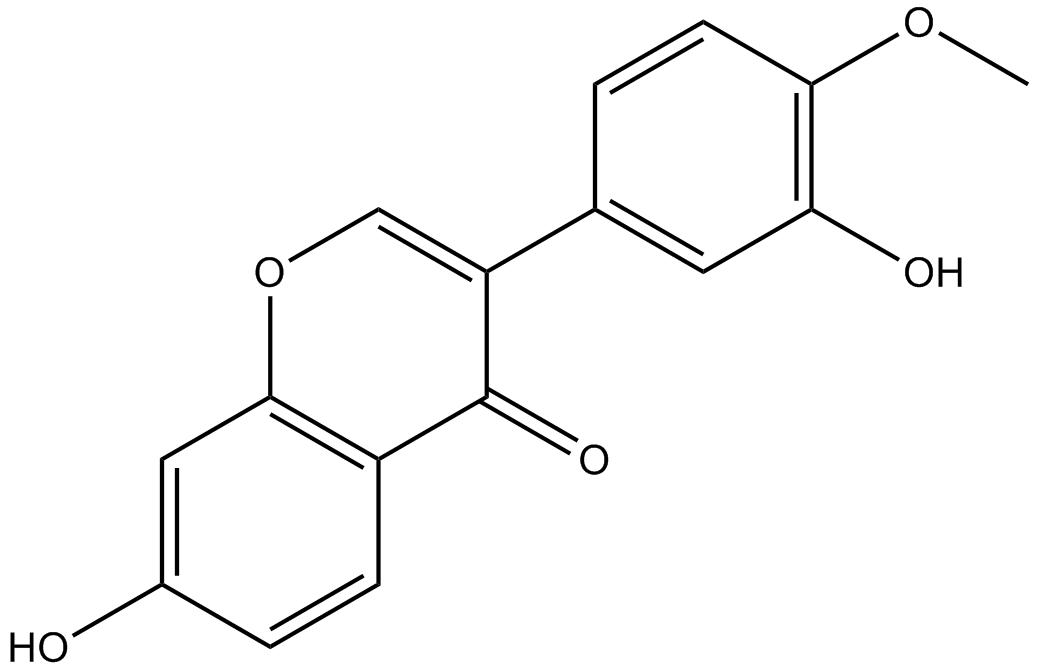
Calycosin
Calycosin (CA, 7, 3-dihydroxy-4-methoxy isoflavone, C16H12O5) is one of the flavonoids extracted from astragalus root, also known as the typical phytoestrogens.
-
GC35598
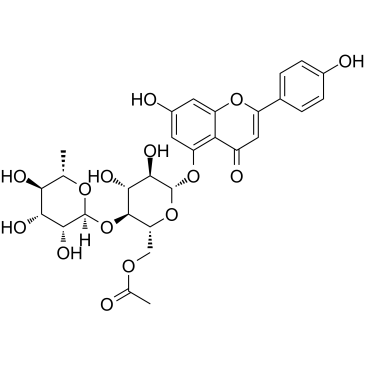
Camellianin A
La camellianine A, le principal flavonoÏde des feuilles d'A. nitida, présente une activité anticancéreuse et une activité inhibitrice de l'enzyme de conversion de l'angiotensine (ECA). La camellianine A inhibe la prolifération des lignées cellulaires humaines Hep G2 et MCF-7 et induit l'augmentation significative de la population de cellules G0/G1.
-
GC62253
Camrelizumab
Le camrelizumab (SHR-1210) est un puissant anticorps monoclonal (mAb) IgG4-κ humanisé de haute affinité contre PD-1. Le camrelizumab se lie À PD-1 À une affinité élevée de 3nM et inhibe l'interaction de liaison de PD-1 et PD-L1 avec une IC50 de 0,70nM. Le camrelizumab agit comme agent anti-PD-1/PD-L1 et peut être utilisé pour la recherche sur le cancer, y compris le NSCLC, l'ESCC, le lymphome hodgkinien et le CHC avancé et, al.

-
GC12318
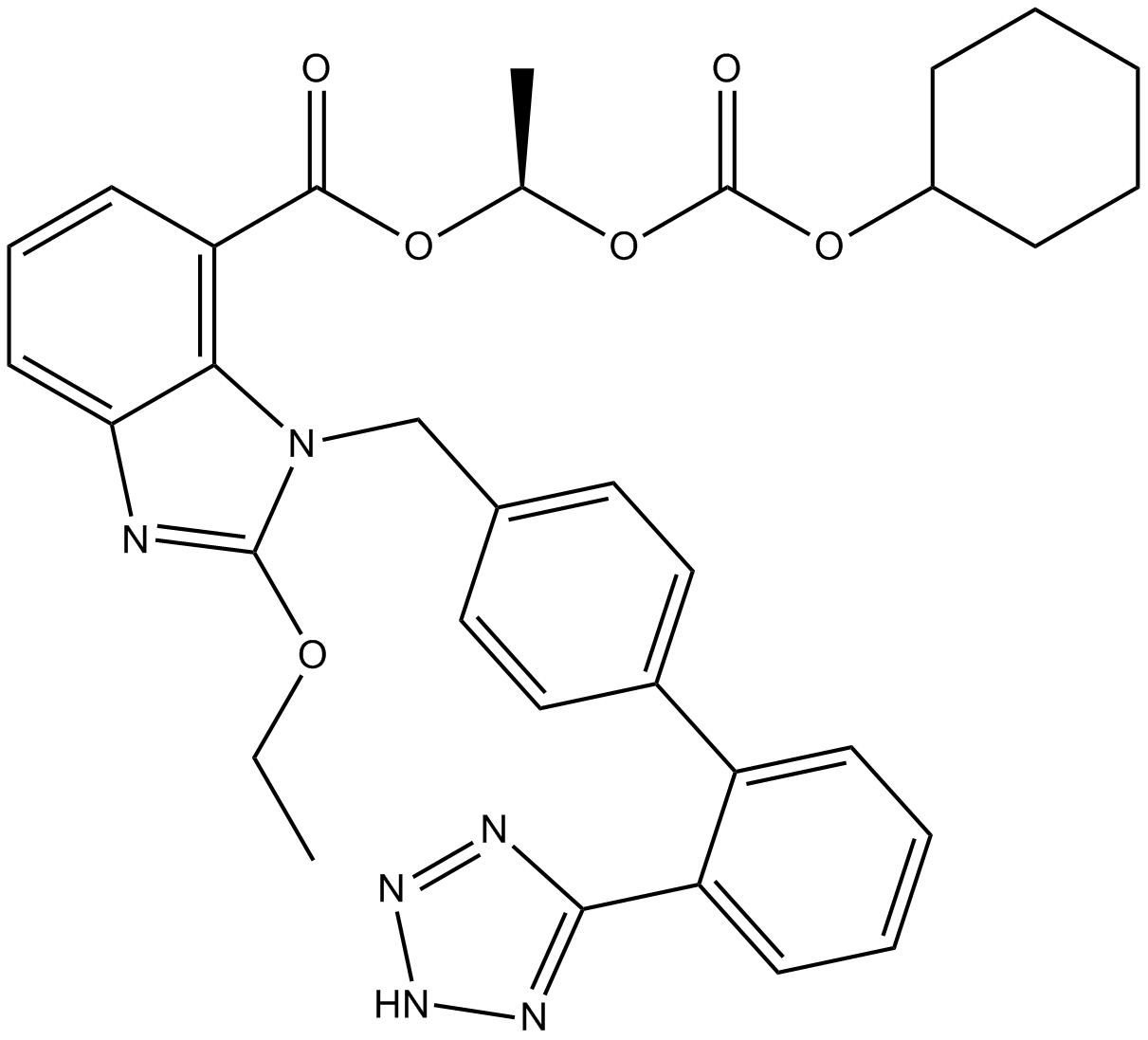
Candesartan Cilexetil
Le candésartan cilexétil (TCV-116) est un antagoniste des récepteurs de l'angiotensine II utilisé principalement pour le traitement de l'hypertension.
-
GC15866
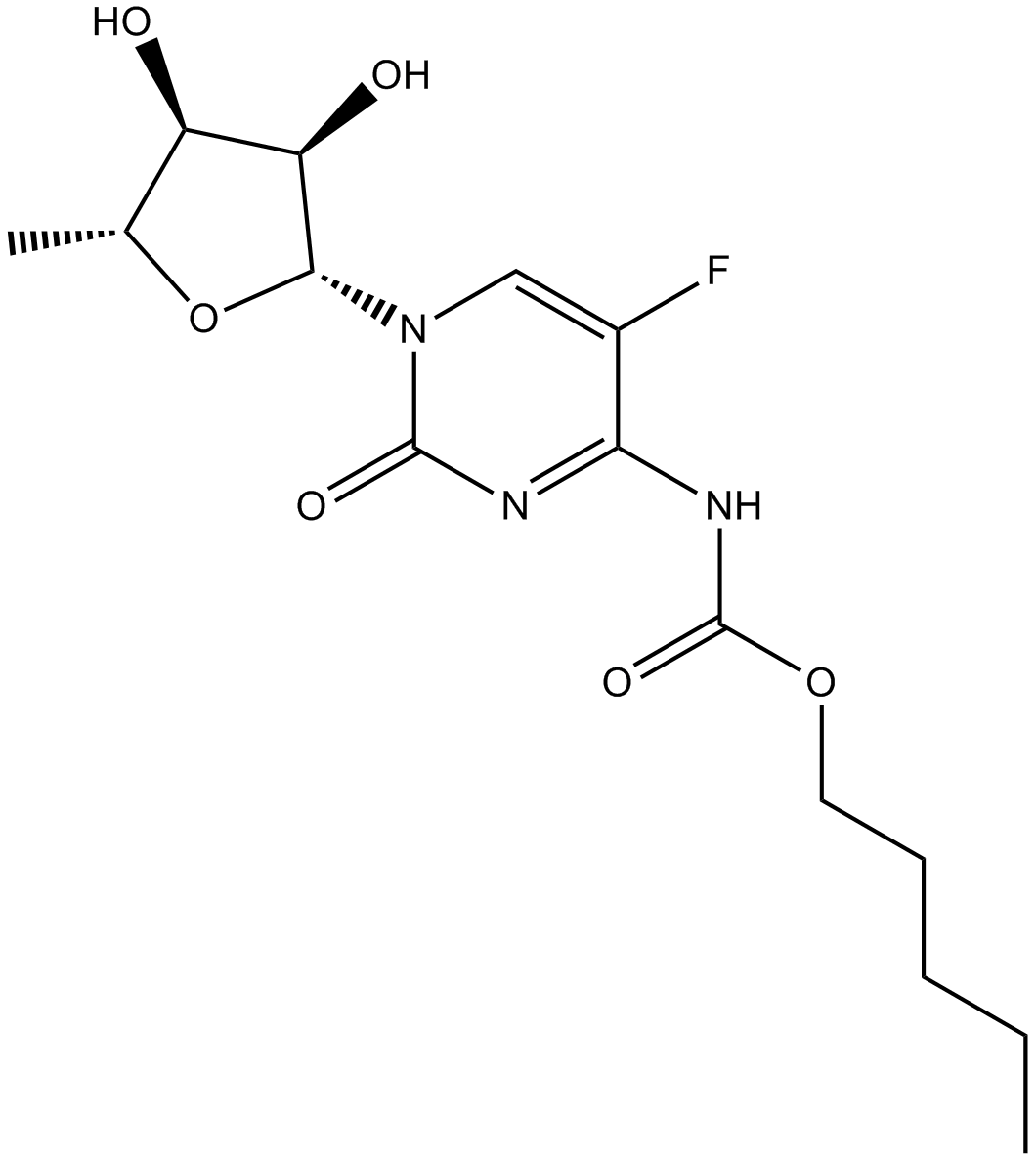
Capecitabine
La capécitabine est une prodrogue orale qui est convertie en son métabolite actif, le 5-FU, par la thymidine phosphorylase.
-
GC14065
Capsaicin
Un alcaloïde de terpène avec des activités biologiques diverses.
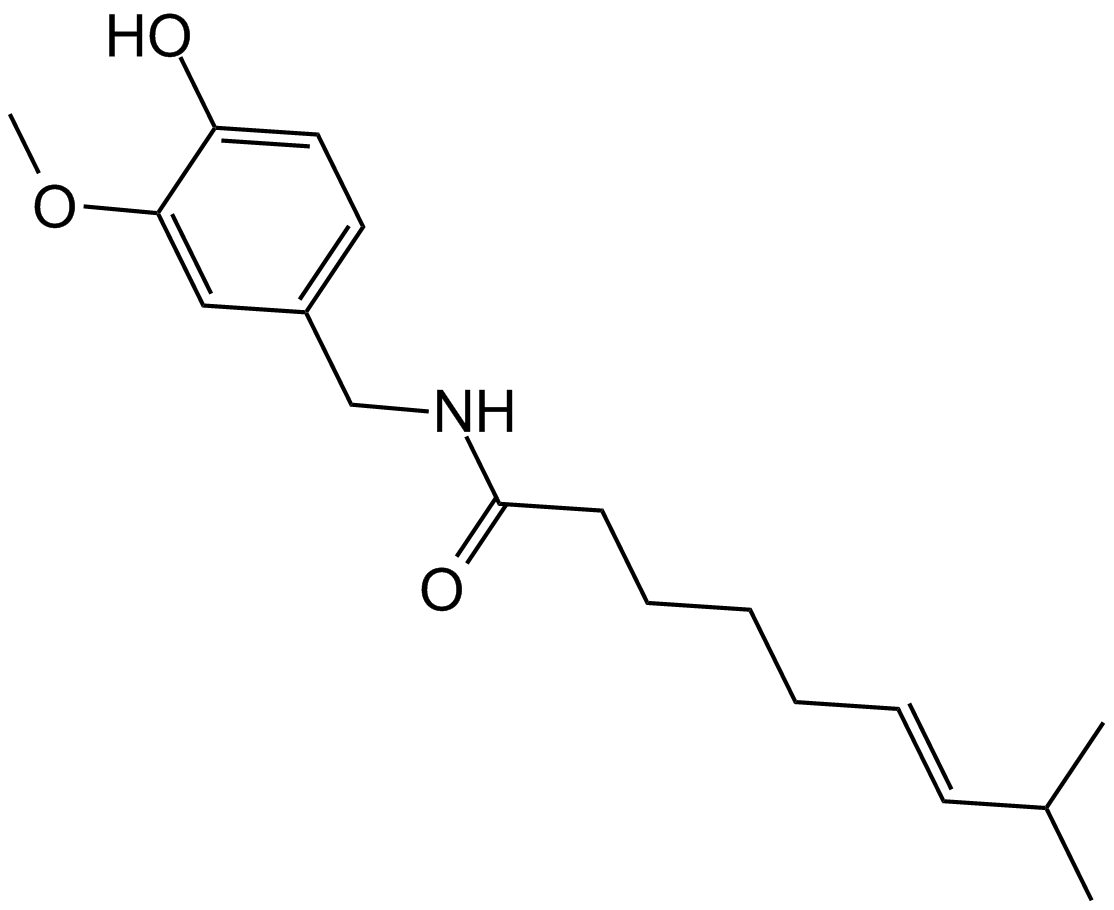
-
GC17918
Capsazepine
A TRPV1 antagonist
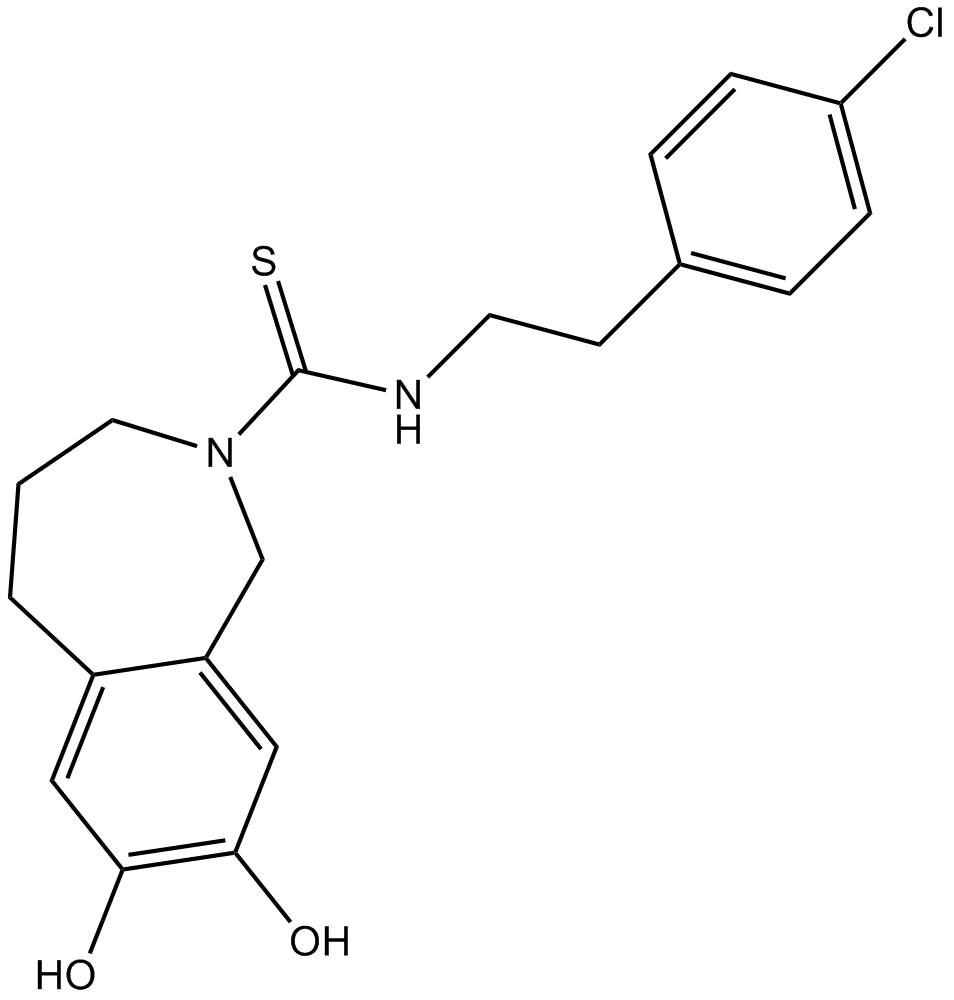
-
GC49415
Capsorubin
A carotenoid with diverse biological activities
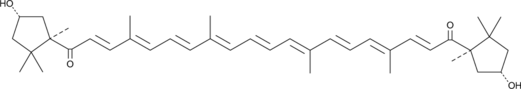
-
GC48878
Carbazomycin A
A bacterial metabolite with diverse biological activities
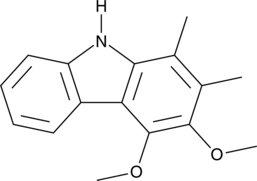
-
GC48893
Carbazomycin B
A bacterial metabolite with diverse biological activities
-
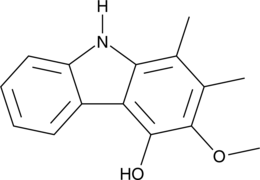
GC48850
Carbazomycin C
A bacterial metabolite with diverse biological activities
-
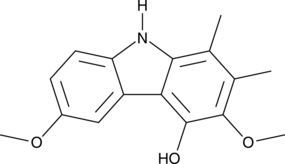
GC48826
Carbazomycin D
A bacterial metabolite with diverse biological activities
-
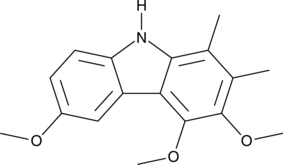
GC49147
Carboxyphosphamide
An inactive metabolite of cyclophosphamide
-
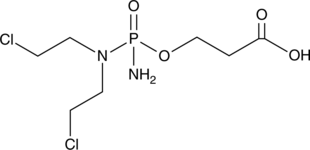
GC18069
Cardamonin
La cardamonine ((E)-cardamomine) est un nouvel antagoniste du canal cationique hTRPA1 avec une IC50 de 454 nM.
-
GC18449
Cardanol monoene
Le cardanol monoène (Cardanol C15:1) est un composé phénolique présent dans le liquide de coque de noix de cajou. Le monoène de cardanol peut induire l'apoptose associée aux mitochondries dans les cellules de mélanome humain.
-
GC15089
Carfilzomib (PR-171)
A proteasome inhibitor