Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Ziele für Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Produkte für Apoptosis
- Bestell-Nr. Artikelname Informationen
-
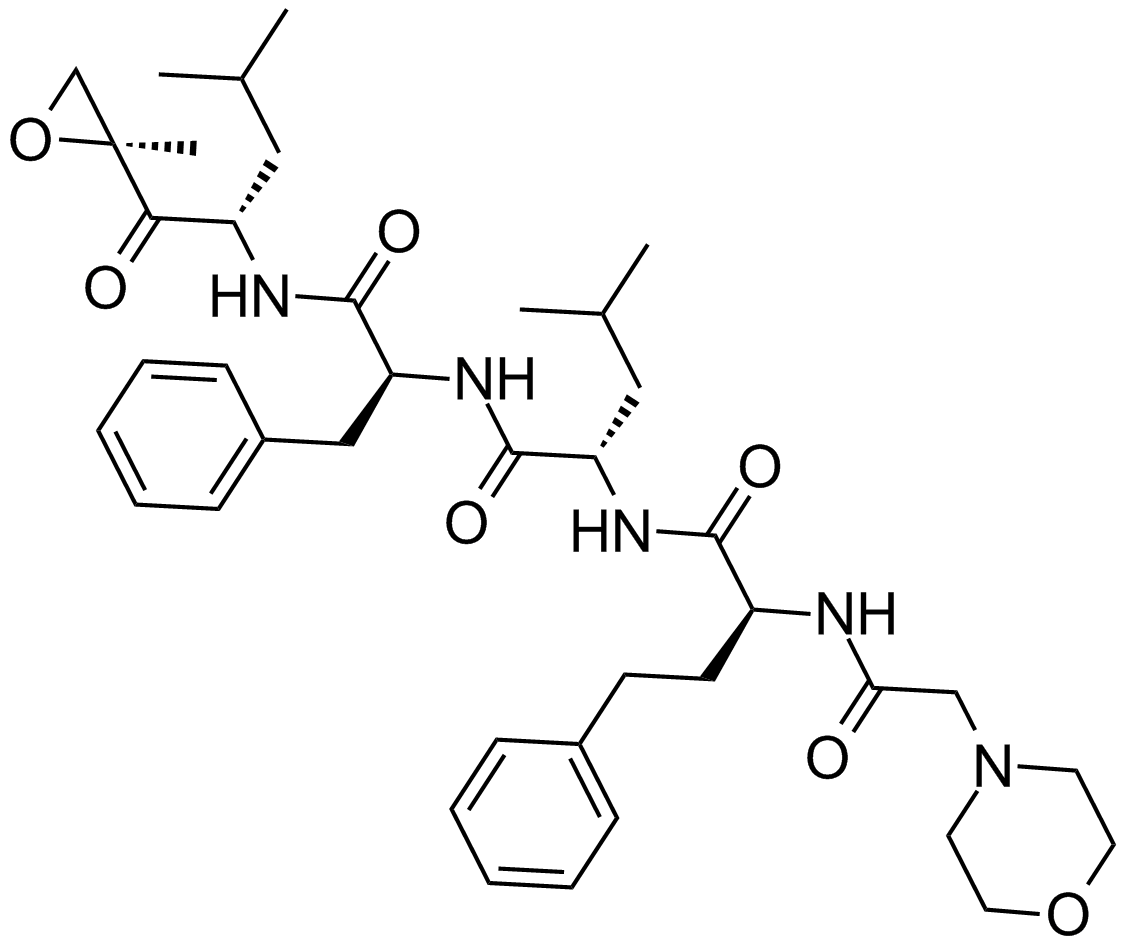
GC12426
Birinapant (TL32711)
Ein Antagonist von cIAP1, cIAP2 und XIAP.
-
GN10037
Bisdemethoxycurcumin
-
GC68308
Bisdemethoxycurcumin-d8
-
GC35530
BJE6-106
BJE6-106 (B106) ist ein potenter, selektiver PKCδ-Inhibitor der 3. Generation mit einem IC50 von 0,05 μM und zielt selektiv auf das klassische PKC-Isozym PKCα (IC50=50 μM).

-
GC11931
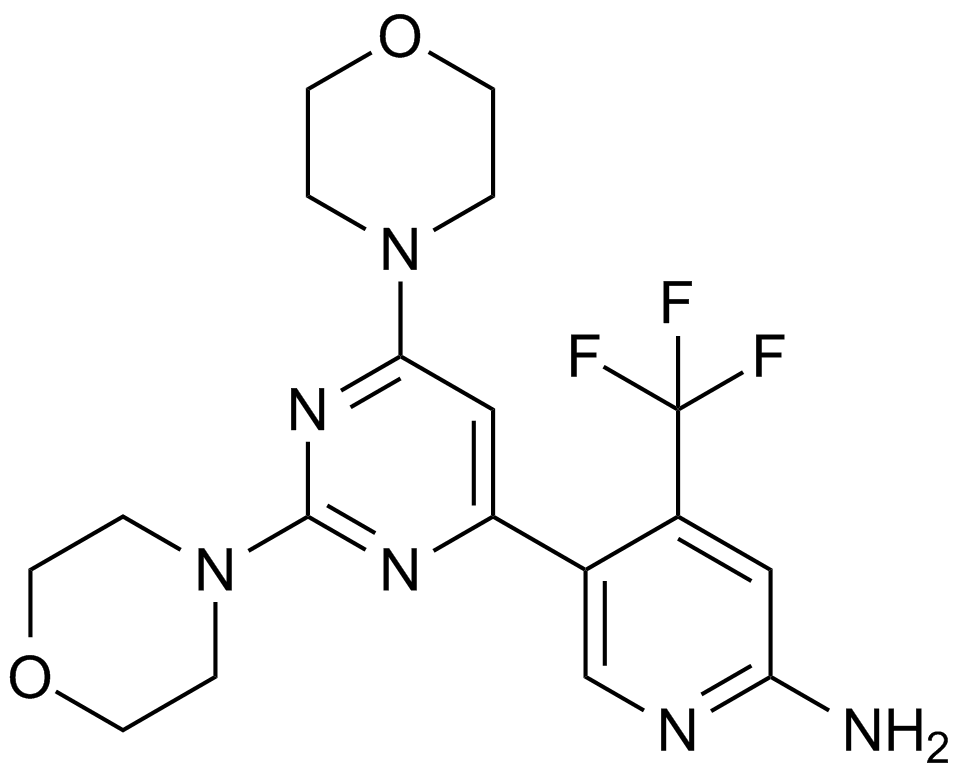
BKM120
An inhibitor of class I PI3K isoforms
-
GC65428
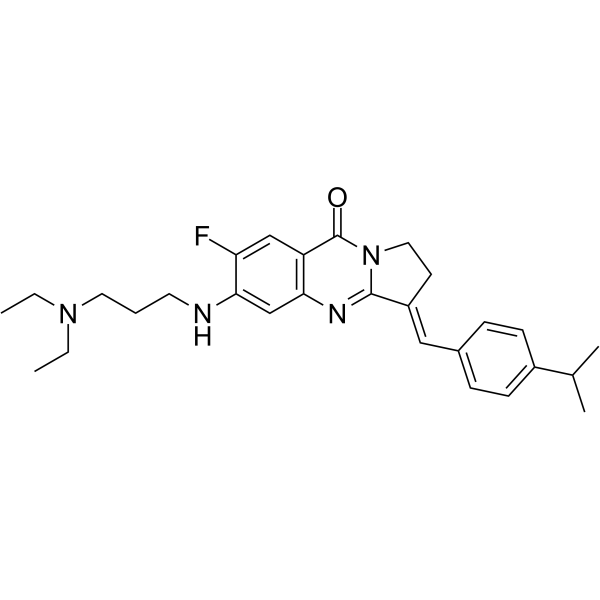
BLM-IN-1
BLM-IN-1 (Verbindung 29) ist ein wirksamer Inhibitor des Bloom-Syndrom-Proteins (BLM) mit einer starken BLM-Bindungs-KD von 1,81 μM und einem IC50 von 0,95 μM fÜr BLM. Induziert eine DNA-Schadensreaktion sowie Apoptose und Proliferationsstopp in Krebszellen.
-
GC33407
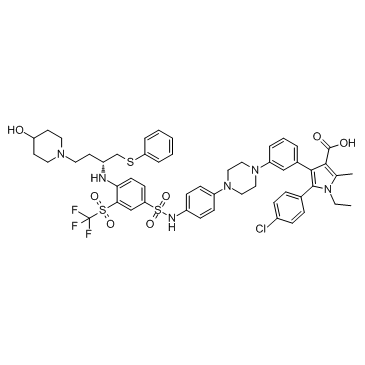
BM 957
BM 957 ist ein potenter Bcl-2- und Bcl-xL-Inhibitor mit Kis von 1,2, < 1 nM und IC50s von 5,4 bzw. 6,0 nM.
-
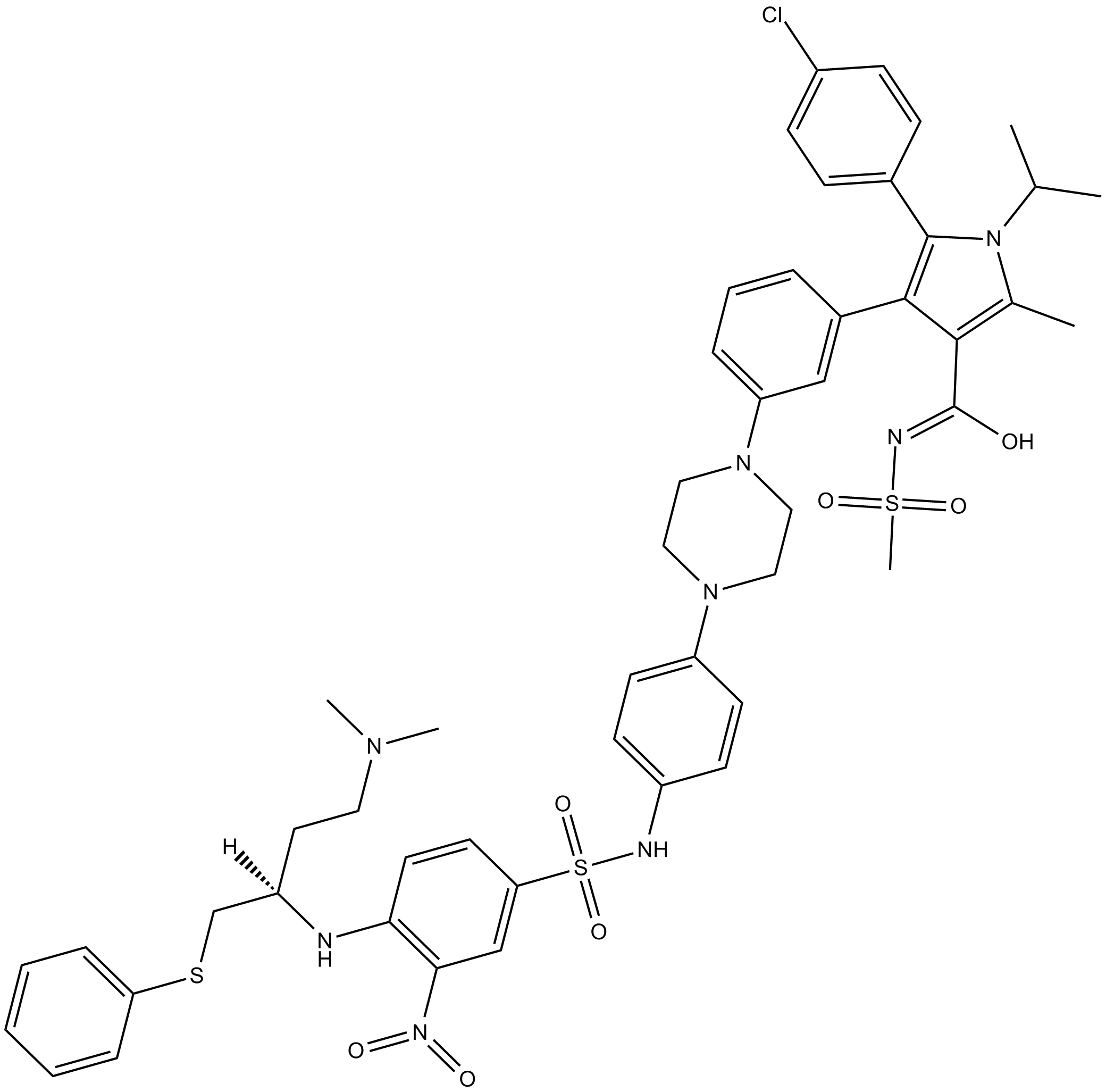
GC13498
BM-1074
-
GC62871
BM-1244
BM-1244 (APG-1252-M1) ist ein potenter Bcl-xL/Bcl-2-Inhibitor mit Kis von 134 und 450 nM fÜr Bcl-xL bzw. Bcl-2. BM-1244 hemmt seneszente Fibroblasten (SnCs) mit einem EC50 von 5 nM. (Aus dem Patent WO2019033119A1).
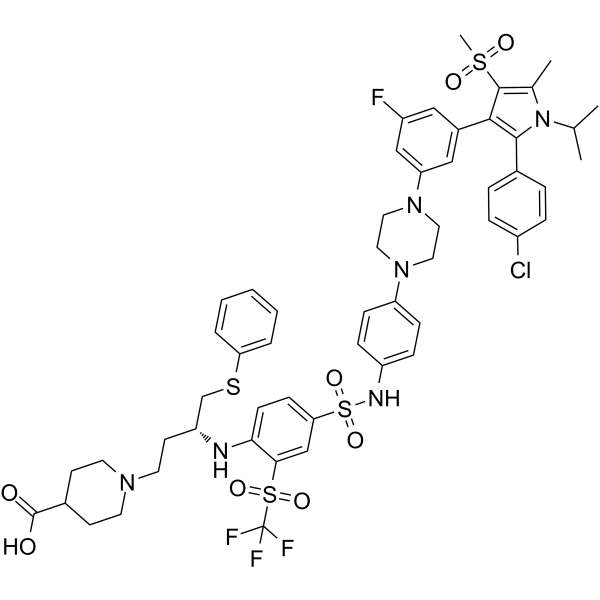
-
GC12822
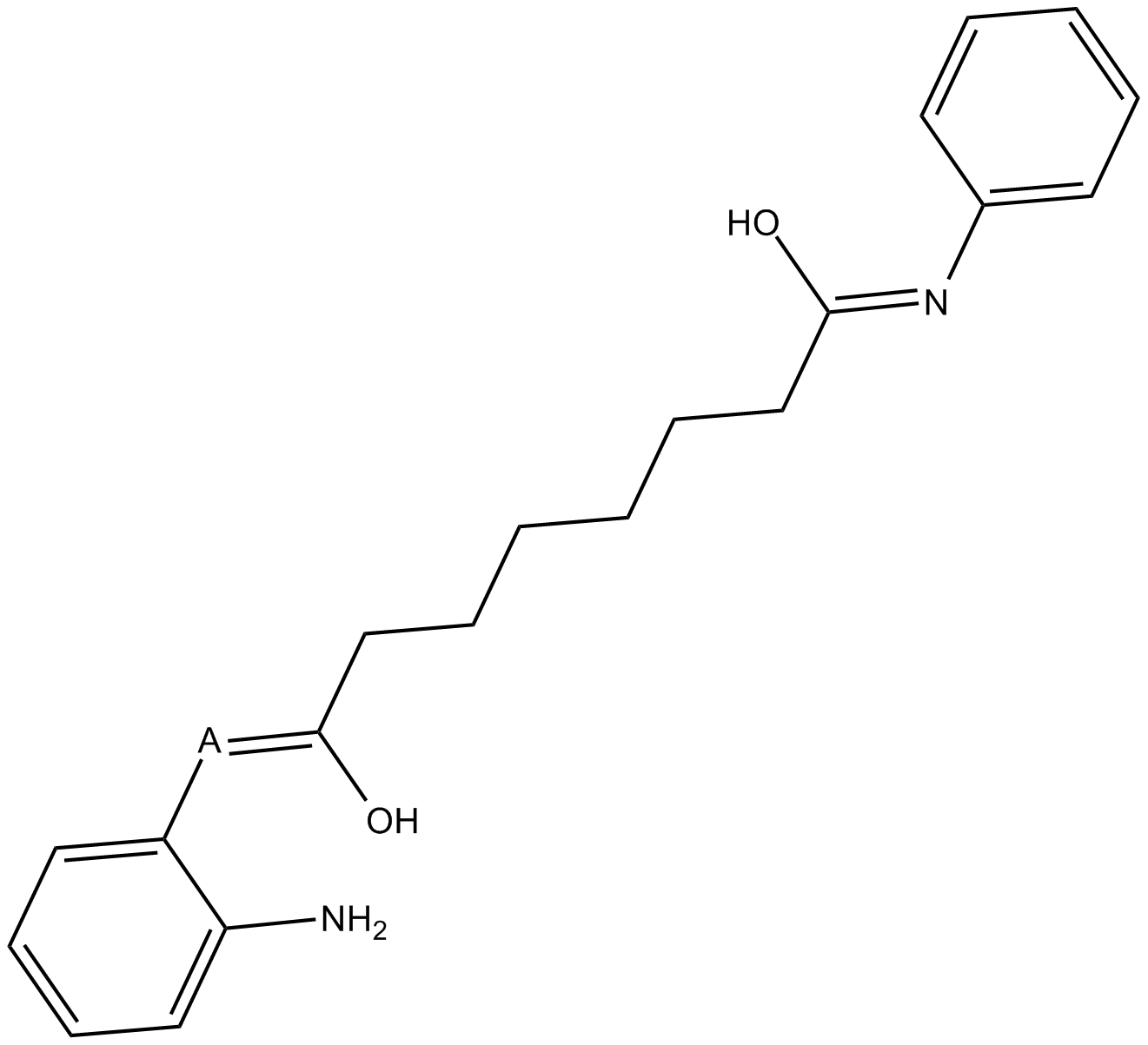
BML-210(CAY10433)
BML-210(CAY10433) ist ein neuer HDAC-Inhibitor, dessen Wirkungsmechanismus noch nicht charakterisiert wurde.
-
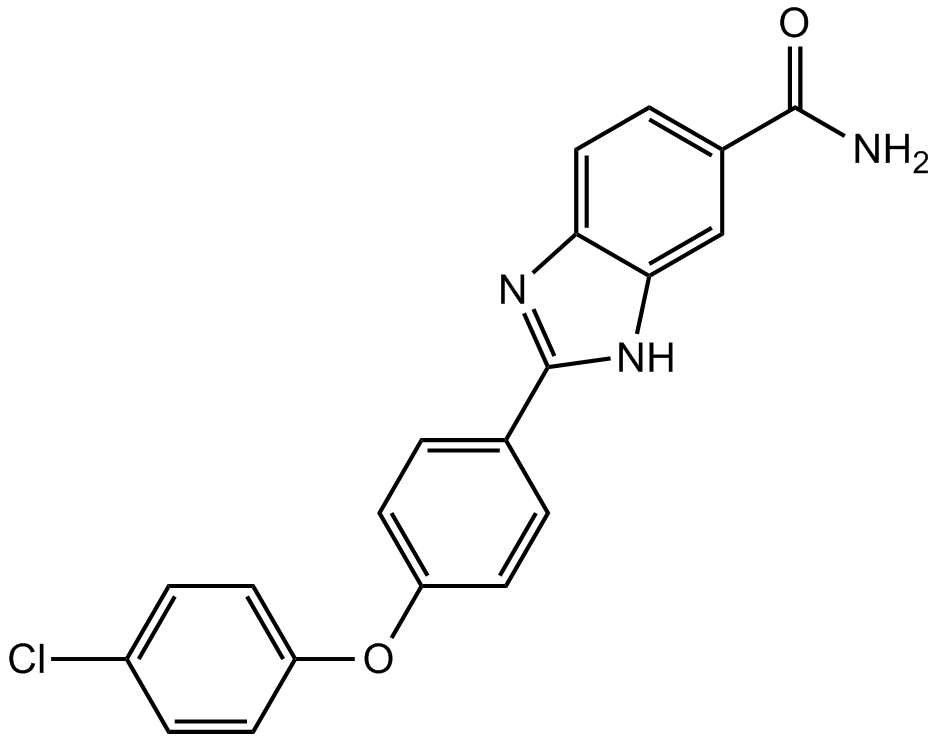
GC11648
BML-277
BML-277 ist ein selektiver Checkpoint-Kinase-2 (Chk2)-Inhibitor mit einem IC50 von 15 nM.
-
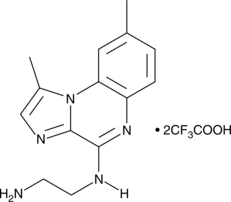
GC42953
BMS 345541 (trifluoroacetate salt)
BMS 345541 is a cell permeable inhibitor of the IκB kinases IKKα and IKKβ (IC50s = 4 and 0.3 μM).
-
GC25160
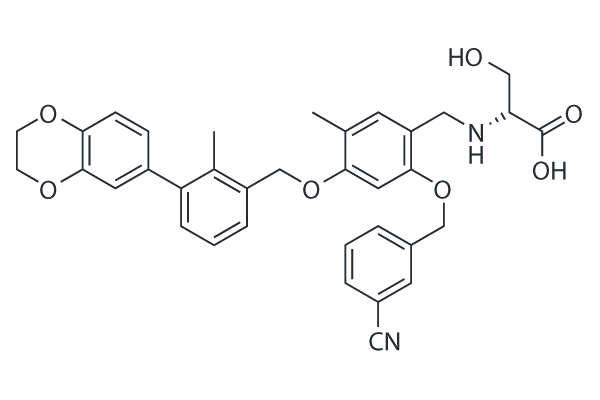
BMS-1001
BMS-1001 is a potent inhibitor of PD-1/PD-L1 interaction with EC50 of 253 nM. BMS-1001 alleviates the inhibitory effect of the soluble PD-L1 on the T-cell receptor-mediated activation of T-lymphocytes.
-
GC38740
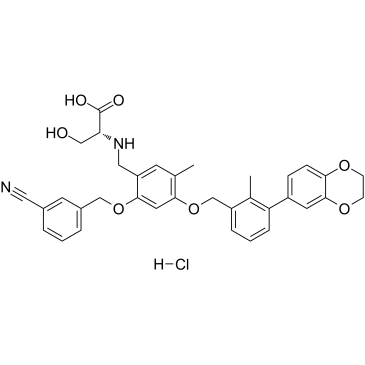
BMS-1001 hydrochloride
BMS-1001-Hydrochlorid ist ein oral aktiver humaner PD-L1/PD-1-Immun-Checkpoint-Inhibitor.
-
GC31753
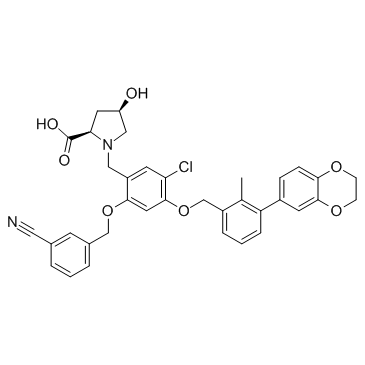
BMS-1166 (PD-1/PD-L1-IN1)
BMS-1166 (PD-1/PD-L1-IN1) ist ein potenter PD-1/PD-L1-Immun-Checkpoint-Inhibitor.
-
GC38131
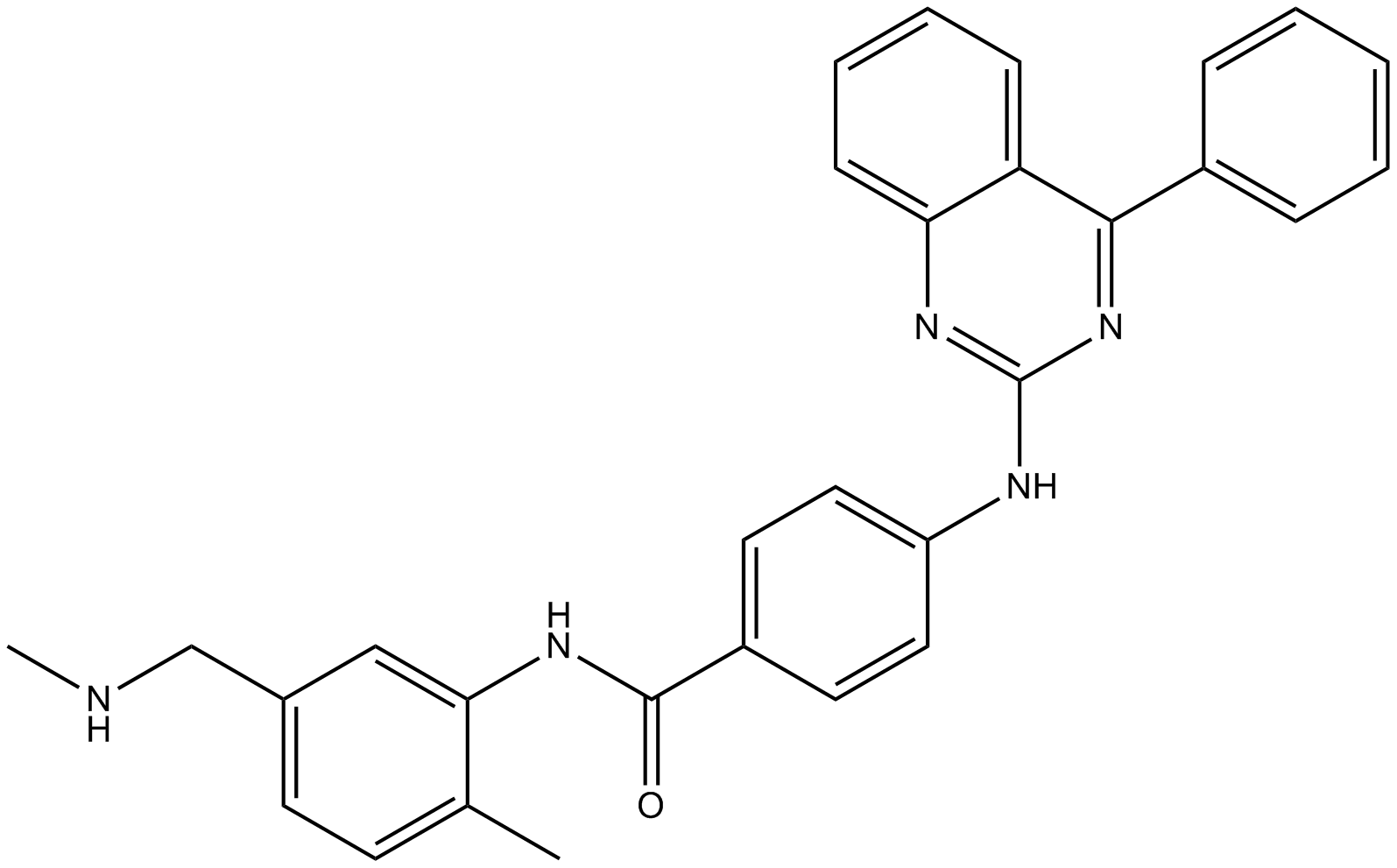
BMS-1166 hydrochloride
BMS-1166-Hydrochlorid ist ein potenter PD-1/PD-L1-Immun-Checkpoint-Inhibitor.

-
GC13628
BMS-833923
An orally bioavailable Smo inhibitor
-
GC62682
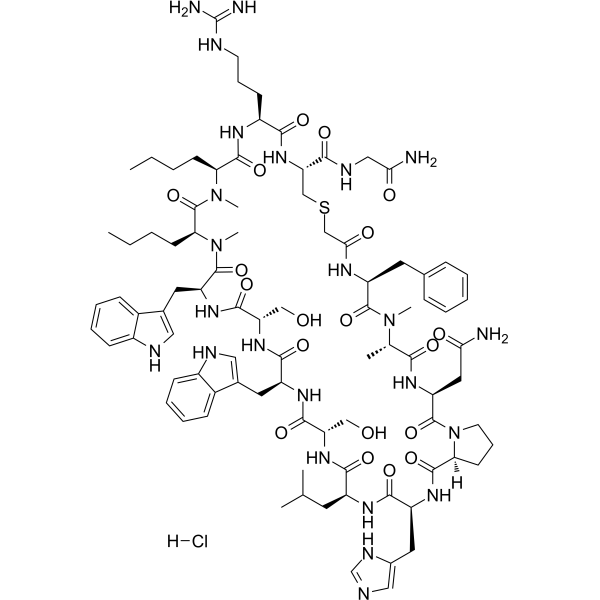
BMSpep-57 hydrochloride
BMSpep-57-Hydrochlorid ist ein potenter und kompetitiver makrozyklischer Peptid-Inhibitor der PD-1/PD-L1-Wechselwirkung mit einem IC50 von 7,68nM. BMSpep-57-Hydrochlorid bindet an PD-L1 mit Kds von 19 nM und 19,88 nM in MST- bzw. SPR-Assays. BMSpep-57-Hydrochlorid erleichtert die T-Zellfunktion, indem es die IL-2-Produktion in PBMCs erhÖht.
-
GA20897
Boc-Arg(Boc)₂-OH
An amino acid building block

-
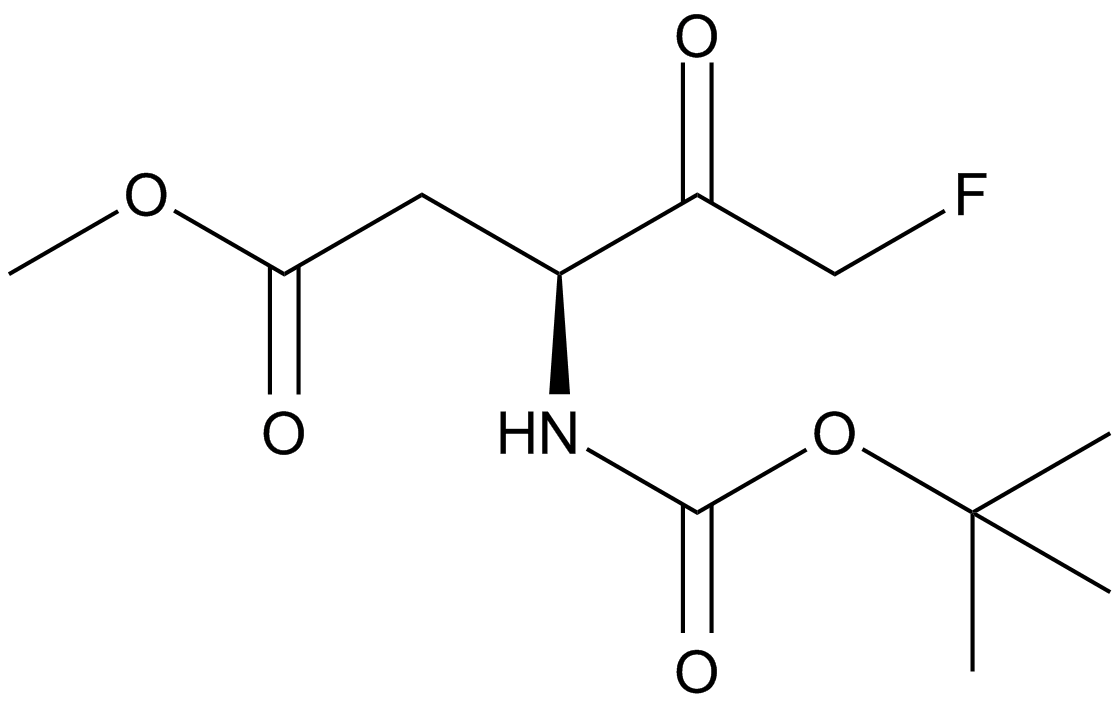
GC16774
Boc-D-FMK
An irreversible pan-caspase inhibitor
-
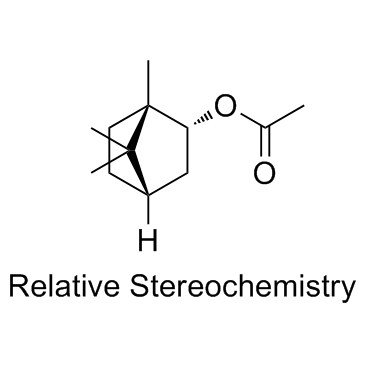
GC33501
Bornyl acetate
Bornylacetat ist ein starker Geruchsstoff, der einen der hÖchsten GeschmacksverdÜnnungsfaktoren (FD-Faktor) aufweist. Bornylacetat besitzt Anti-Krebs-AktivitÄt.
-
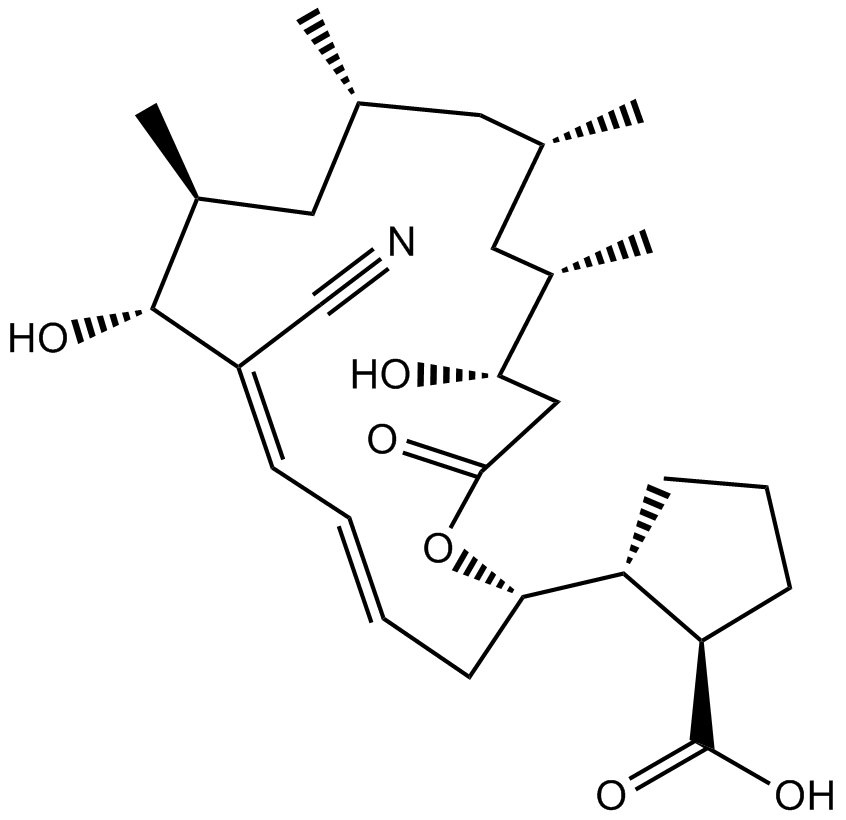
GC11040
Borrelidin
Borrelidin (Treponemycin) ist ein bakterieller und eukaryaler Threonyl-tRNA-Synthetase-Inhibitor, ein nitrilhaltiges Makrolid-Antibiotikum, das aus Streptomyces rochei isoliert wurde.
-
GC17644
Bortezomib (PS-341)
Ein potenter, reversibler 20S-Proteasom-Inhibitor.
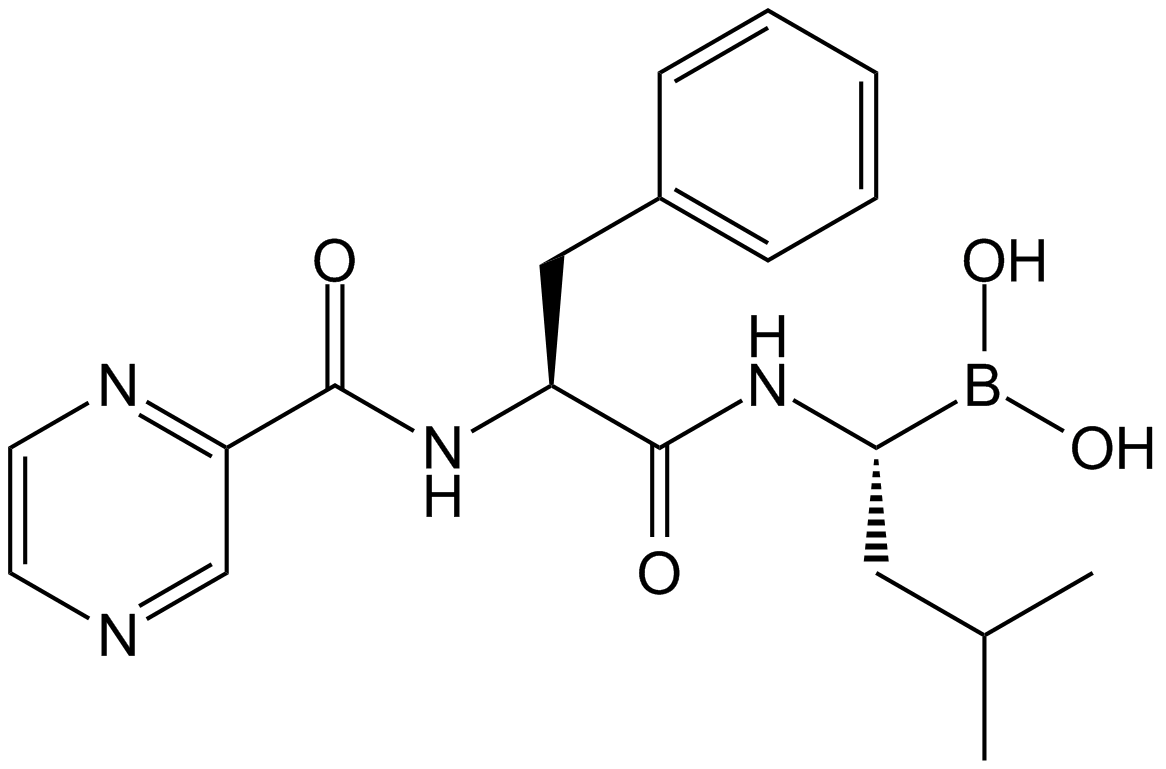
-
GC65010
Bortezomib-d8
Bortezomib-d8 (PS-341-d8) ist das mit Deuterium bezeichnete Bortezomib. Bortezomib (PS-341) ist ein reversibler und selektiver Proteasom-Inhibitor und hemmt wirksam das 20S-Proteasom (Ki=0,6 nM), indem es auf einen Threoninrest abzielt. Bortezomib unterbricht den Zellzyklus, induziert Apoptose und hemmt NF-κB. Bortezomib ist der erste Proteasom-Inhibitor gegen Krebs. Anti-Krebs-AktivitÄt.
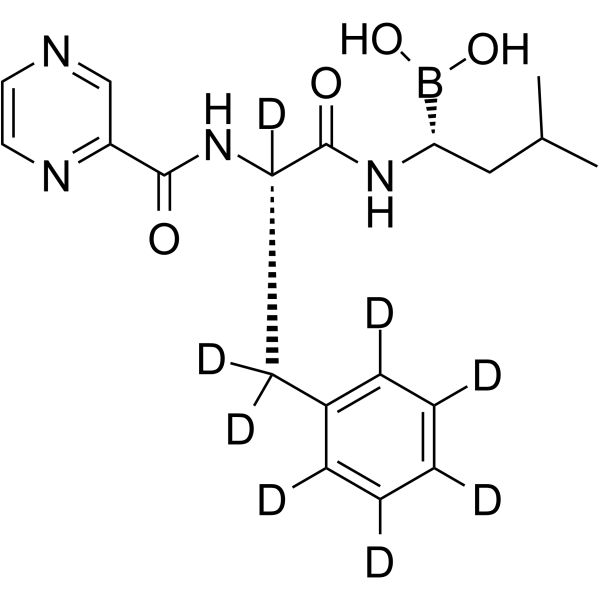
-
GC40009
Bostrycin
Bostrycin is an anthraquinone originally isolated from B.
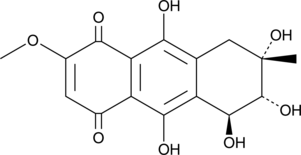
-
GC42969
bpV(phen) (potassium hydrate)
bpV(phen) (Kaliumhydrat), ein Insulin-Mimetikum, ist ein potenter Protein-Tyrosin-Phosphatase (PTP)- und PTEN-Inhibitor mit IC50-Werten von 38 nM, 343 nM und 920 nM fÜr PTEN, PTP-β bzw. PTP-1B.
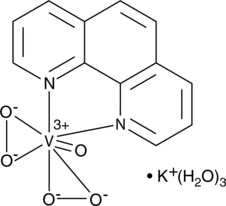
-
GC42974
Brassinin
Brassinin ist der Metabolismus eines Phytoalexins aus Brassica-Arten.
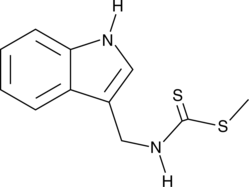
-
GC11632
Brassinolide
A plant growth regulator
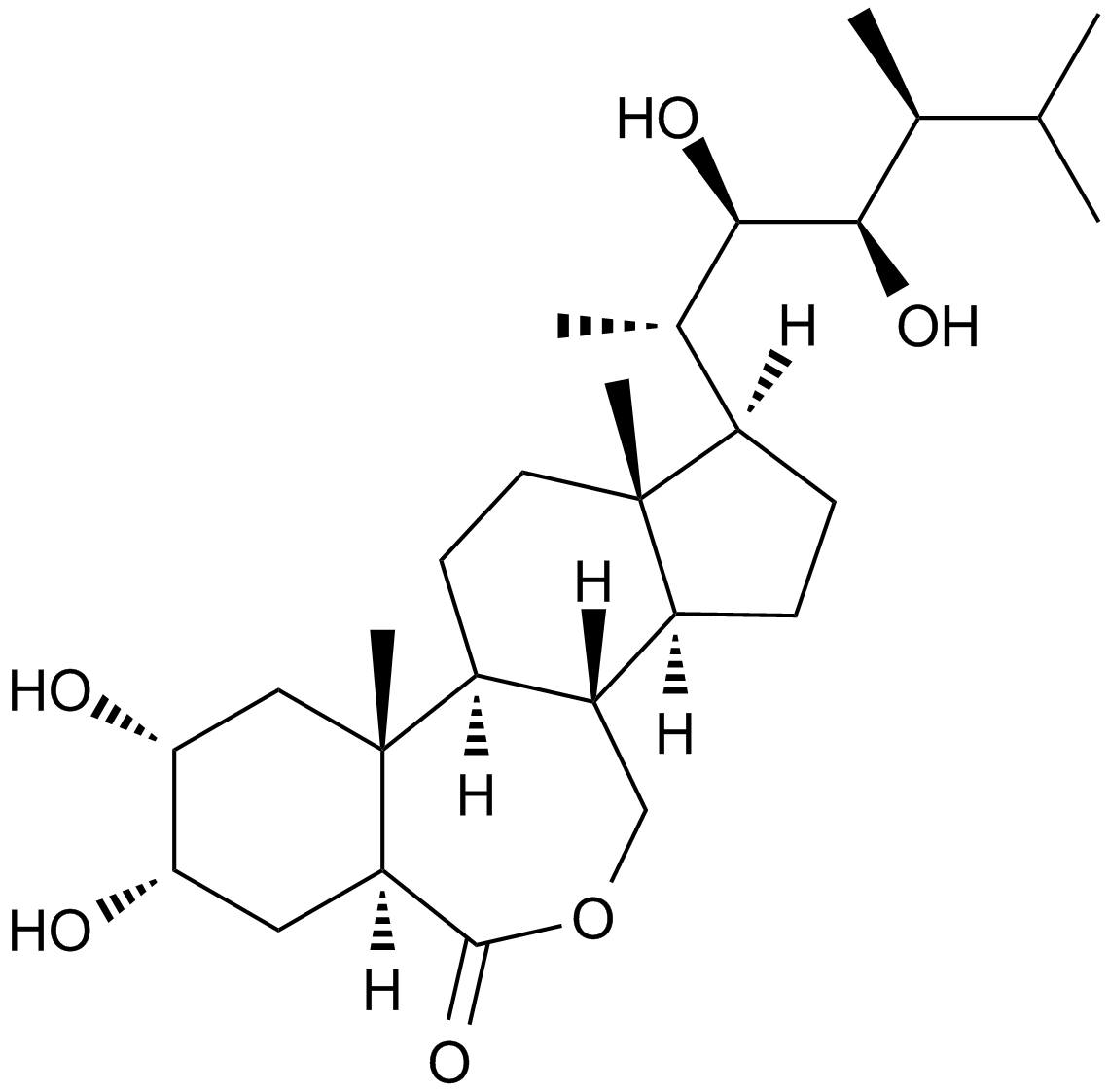
-
GC52101
Brazilein
Brazilein ist eine wichtige immunsuppressive Komponente, die aus Caesalpinia sappan L.
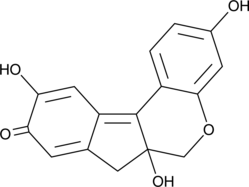
-
GN10802
Brazilin
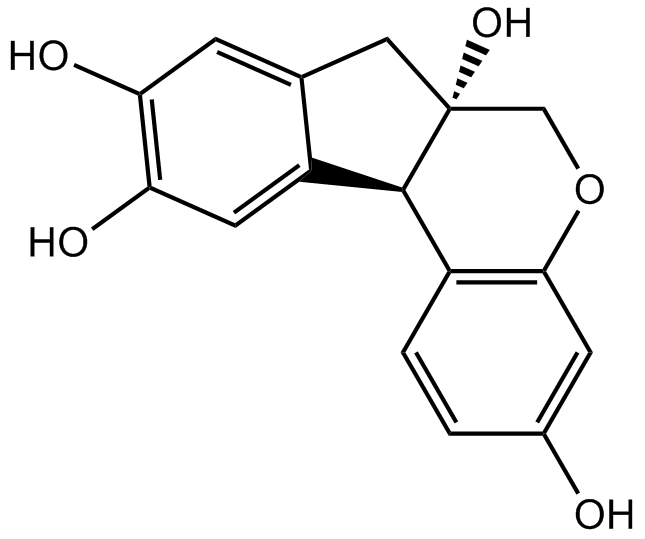
-
GC68288
Brentuximab

-
GC35554
Brevilin A
Brevilin A ist ein oral aktiver STAT3/JAK-Inhibitor (STAT3 IC50=10,6 μM). Brevilin A zeigt AntitumoraktivitÄt, antiproliferative AktivitÄt gegenÜber Krebszellen und kann Apoptose und Autophagie induzieren.

-
GC35555
Britannin
Britannin, isoliert aus Inula aucheriana, ist ein Sesquiterpenlacton.
-
GC62536
Bromelain
Bromelain ist ein entzÜndungshemmendes Medikament, das aus dem Ananasstamm gewonnen wird und durch Herunterregulierung von Plasmakininogen, Hemmung der Prostaglandin-E2-Expression, Abbau fortgeschrittener Glykationsendproduktrezeptoren und Regulierung angiogener Biomarker sowie durch antioxidative Wirkung stromaufwÄrts im COX-Weg wirkt .

-
GC35559
Bruceine D
Bruceine D ist ein Notch-Inhibitor mit Anti-Krebs-AktivitÄt und induziert Apoptose in mehreren menschlichen Krebszellen.
-
GC34070
Brusatol (NSC 172924)
Brusatol (NSC 172924) (NSC172924) ist ein einzigartiger Inhibitor des Nrf2-Signalwegs, der ein breites Spektrum von Krebszellen fÜr Cisplatin und andere Chemotherapeutika sensibilisiert. Brusatol (NSC 172924) verstÄrkt die Wirksamkeit der Chemotherapie, indem es den Nrf2-vermittelten Abwehrmechanismus hemmt. Brusatol (NSC 172924) kann zu einem adjuvanten Chemotherapeutikum entwickelt werden. Brusatol (NSC 172924) erhÖht die zellulÄre Apoptose.
-
GC38014
BT2
BT2 ist ein BCKDC-Kinase (BDK)-Inhibitor mit einem IC50 von 3,19 μM.
-
GC38467
BTdCPU
BTdCPU ist ein potenter HÄm-regulierter eIF2α-Kinase (HRI)-Aktivator. BTdCPU fÖrdert die eIF2α-Phosphorylierung und induzierte Apoptose in resistenten Zellen.
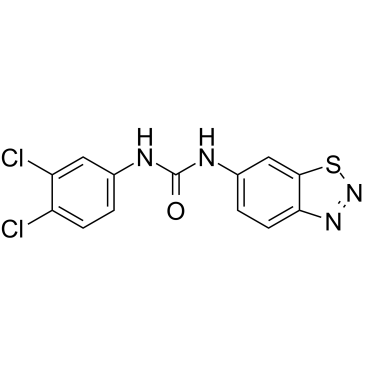
-
GC34511
BTR-1
BTR-1 ist ein aktives Antikrebsmittel, verursacht einen S-Phasen-Arrest und beeinflusst die DNA-Replikation in LeukÄmiezellen. BTR-1 aktiviert die Apoptose und induziert den Zelltod.
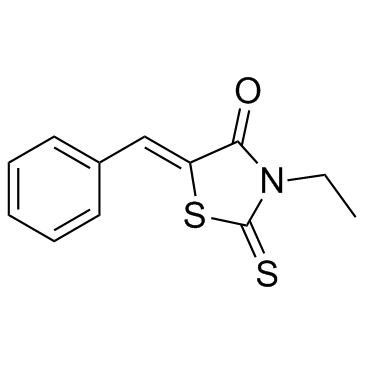
-
GC11141
BTZO 1
BTZO 1 bindet an den Hemmfaktor der Makrophagenmigration (MIF) mit einem Kd-Wert von 68,6 nM, und seine Bindung erfordert das N-terminale Pro1.
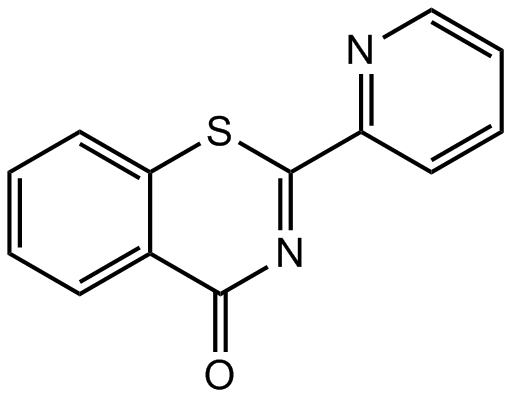
-
GC48376
Burnettramic Acid A
A fungal metabolite with diverse biological activities
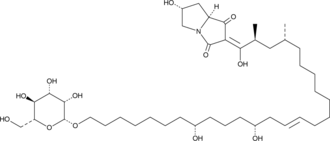
-
GC48409
Burnettramic Acid A aglycone
BurnetraminsÄure-A-Aglykon ist ein Pilzmetabolit, der in Aspergillus burnettii vorkommt.
-
GC13671
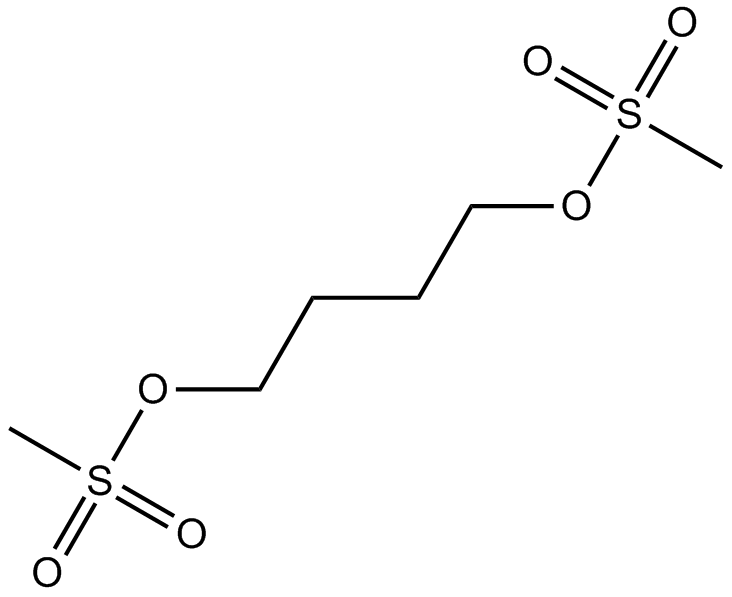
Busulfan
Busulfan ist ein starker Alkylator mit selektiver immunsuppressiver Wirkung auf das Knochenmark.
-
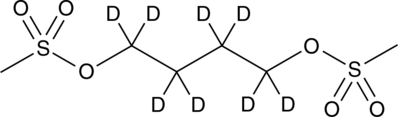
GC46962
Busulfan-d8
Busulfan-D8 ist ein Deuterium mit der Bezeichnung Busulfan. Busulfan ist ein Alkylsulfonat, das als alkylierendes antineoplastisches Mittel wirkt. Busulfan bildet sowohl Intra- als auch Interstrang-Crosslinks auf der DNA. Bei SÄugetieren bewirkt Busulfan eine tiefgreifende und anhaltende Verringerung der Bildung hÄmatopoetischer VorlÄufer, ohne die Lymphozytenspiegel oder die humoralen AntikÖrperreaktionen signifikant zu beeinflussen.
-
GC10944
Butein
Butein ist ein cAMP-spezifischer PDE-Inhibitor mit einer IC50 von 10,4 μM fÜr PDE4. Butein ist ein spezifischer Protein-Tyrosinkinase-Inhibitor mit IC50-Werten von 16 und 65 μM fÜr EGFR und p60c-src in HepG2-Zellen. Butein sensibilisiert HeLa-Zellen fÜr Cisplatin Über AKT- und ERK/p38-MAPK-Wege, indem es auf FoxO3a abzielt. Butein ist ein SIRT1-Aktivator (STAC).
-
GC46104
Butyric Acid-d7
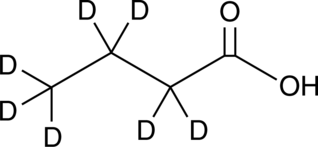
An internal standard for the quantification of sodium butyrate
-
GC12333
BV6
BV6 ist ein Antagonist von cIAP1 und XIAP, Mitgliedern der Familie der Inhibitoren der Apoptose (IAP).
-
GC48433
BX-320
BX-320 ist ein selektiver, ATP-kompetitiver, oral aktiver und direkter PDK1-Inhibitor mit einem IC50 von 30 nM in einem direkten Kinase-Assay-Format. BX-320 induziert auch Apoptose. Anti-Krebs-Wirkung.
-
GC16818
BX-912
BX-912 ist ein direkter, selektiver und ATP-kompetitiver PDK1-Inhibitor (IC50=26 nM). BX-912 blockiert die PDK1/Akt-Signalgebung in Tumorzellen und hemmt das verankerungsabhÄngige Wachstum einer Vielzahl von Tumorzelllinien in Kultur oder induziert Apoptose.
-
GC31806
Bz 423 (BZ48)
Bz 423 (BZ48) ist ein pro-apoptotisches 1,4-Benzodiazepin mit therapeutischen Eigenschaften in Mausmodellen von Lupus, das eine SelektivitÄt fÜr autoreaktive Lymphozyten zeigt und Bax und Bak aktiviert.
-
GC33826
C 87
C 87 ist ein neuartiger niedermolekularer TNFα-Inhibitor; hemmt wirksam die TNFα-induzierte ZytotoxizitÄt mit einem IC50 von 8,73 μM.
-
GC19095
C-DIM12
C-DIM12 ist ein synthetischer Nurr1-Aktivator, der die Nurr1- und DA-Genexpression in Zelllinien und primÄren Neuronen induziert.
-
GC43028
C16 Ceramide (d18:1/16:0)
Ceramide werden aus Sphingomyelin durch Aktivierung von Sphingomyelinase oder durch den de-novo-Syntheseweg generiert, der die koordinierte Wirkung von Serin-Palmitoyltransferase und Ceramidsynthase erfordert.
-
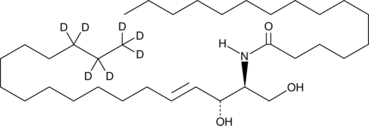
GC46976
C16 Ceramide-d7 (d18:1-d7/16:0)
An internal standard for the quantification of C-16 ceramide
-
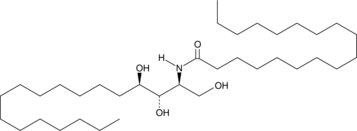
GC43052
C18 Phytoceramide (t18:0/18:0)
C18 Phytoceramide (t18:0/18:0) (Cer(t18:0/18:0)) is a bioactive sphingolipid found in S.
-
GC40141
C18 Phytoceramide-d3 (t18:0/18:0-d3)
C18 Phytoceramide-d3 (t18:0/18:0-d3) is intended for use as an internal standard for the quantification of C18 phytoceramide (t18:0/18:0) by GC- or LC-MS.

-
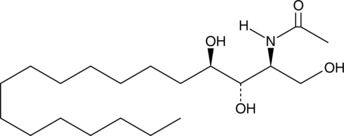
GC43065
C2 Phytoceramide (t18:0/2:0)
C2 Phytoceramide is a bioactive semisynthetic sphingolipid that inhibits formyl peptide-induced oxidant release (IC50 = 0.38 μM) in suspended polymorphonuclear cells.
-
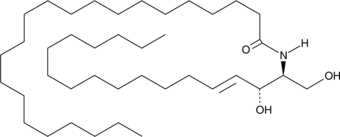
GC43069
C22 Ceramide (d18:1/22:0)
C-22 ceramide is an endogenous bioactive sphingolipid.
-
GC43075
C24 dihydro Ceramide (d18:0/24:0)
C24 dihydro Ceramide is a sphingolipid that has been found in the stratum corneum of human skin.
-
GC34513
C25-140
C25-140, ein erster oral aktiver und ziemlich selektiver TRAF6-Ubc13-Inhibitor seiner Klasse, bindet direkt an TRAF6 und blockiert die Wechselwirkung von TRAF6 mit Ubc13.
-
GC43084
C4 Ceramide (d18:1/4:0)
C4 Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC40688
C6 D-threo Ceramide (d18:1/6:0)
C6 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides., C6 D-threo Ceramide is cytotoxic to U937 cells in vitro (IC50 = 18 μM).
-
GC40689
C6 L-erythro Ceramide (d18:1/6:0)
C6 L-erythro Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
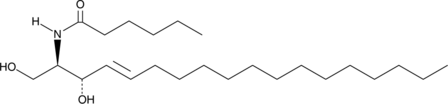
GC40690
C6 L-threo Ceramide (d18:1/6:0)
C6 L-threo Ceramide (d18:1/6:0) ist ein bioaktives Sphingolipid und zelldurchlÄssiges Analogon natÜrlich vorkommender Ceramide.
-
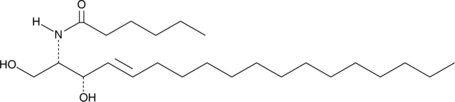
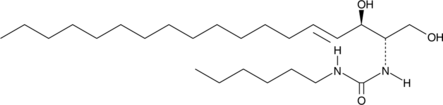
GC45616
C6 Urea Ceramide
An inhibitor of neutral ceramidase
-
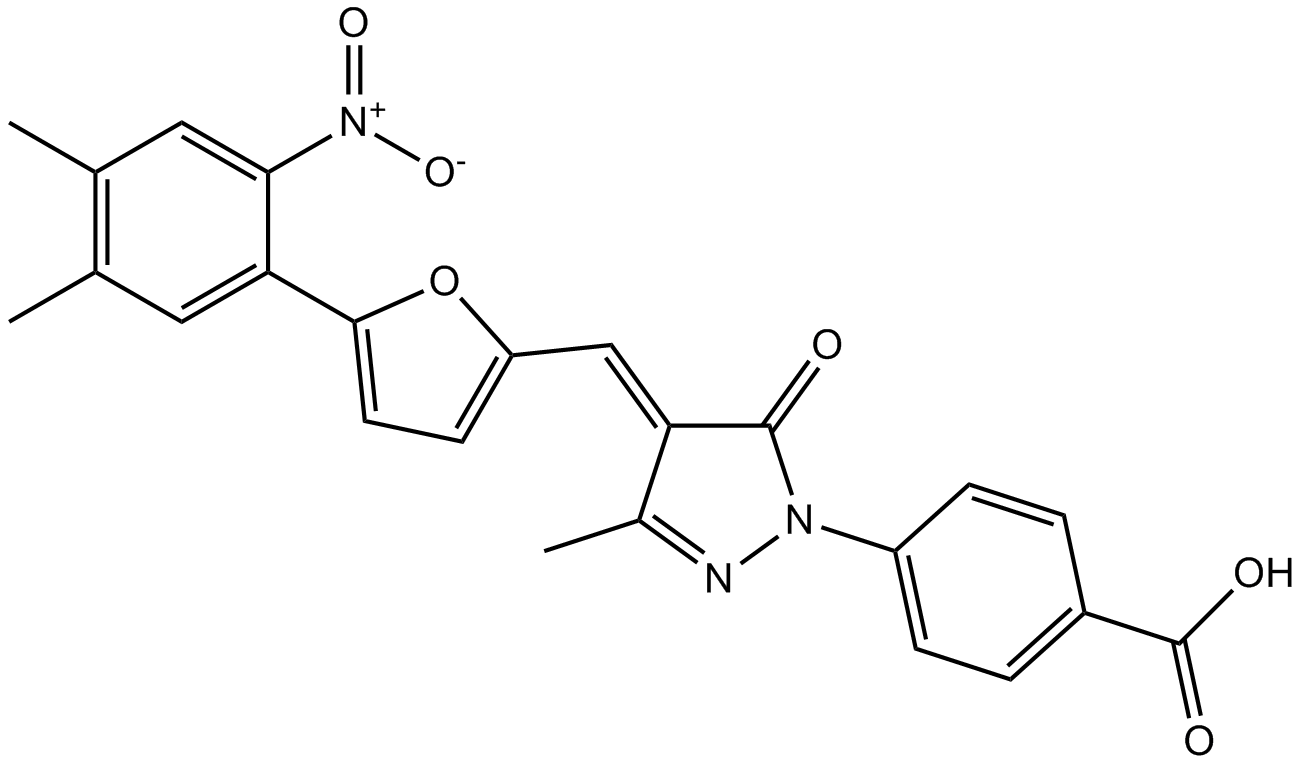
GC12733
C646
C646 ist ein selektiver und kompetitiver Histon-Acetyltransferase-p300-Inhibitor mit einem Ki von 400 nM und ist fÜr andere Acetyltransferasen weniger wirksam.
-
GC43105
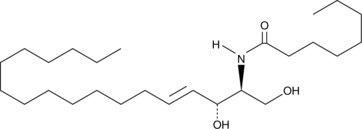
C8 Ceramide (d18:1.8:0)
C8 Ceramid (d18:1.8:0) (N-Octanoyl-D-erythro-sphingosin) ist ein zellpermeables Analogon von natürlich vorkommenden Ceramiden.
-
GC43109
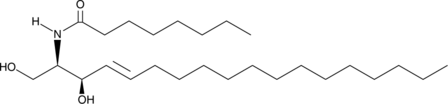
C8 D-threo Ceramide (d18:1/8:0)
C8 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC43110
C8 Galactosylceramide (d18:1/8:0)
C8 Galactosylceramide is a synthetic C8 short-chain derivative of known membrane microdomain-forming sphingolipids.

-
GC43111
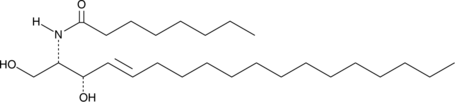
C8 L-threo Ceramide (d18:1/8:0)
C8 L-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC33218
CA-5f
CA-5f ist ein potenter Makroautophagie-/Autophagie-Hemmer im SpÄtstadium, indem es die Autophagosom-Lysosomen-Fusion hemmt. CA-5f erhÖht LC3B-II (ein Marker zur Überwachung der Autophagie) und das SQSTM1-Protein und erhÖht auch die ROS-Produktion. Anti-Tumor-AktivitÄt.
-
GC15779
Cabozantinib (XL184, BMS-907351)
Cabozantinib (XL184, BMS-907351) ist ein potenter und oral aktiver Inhibitor von VEGFR2 und MET mit IC50-Werten von 0,035 bzw. 1,3 nM. Cabozantinib (XL184, BMS-907351) zeigt eine starke Hemmung von KIT, RET, AXL, TIE2 und FLT3 (IC50 = 4,6, 5,2, 7, 14,3 bzw. 11,3 nM). Cabozantinib (XL184, BMS-907351) zeigt antiangiogene AktivitÄt. Cabozantinib (XL184, BMS-907351) stÖrt das GefÄßsystem des Tumors und fÖrdert die Apoptose von Tumoren und Endothelzellen.
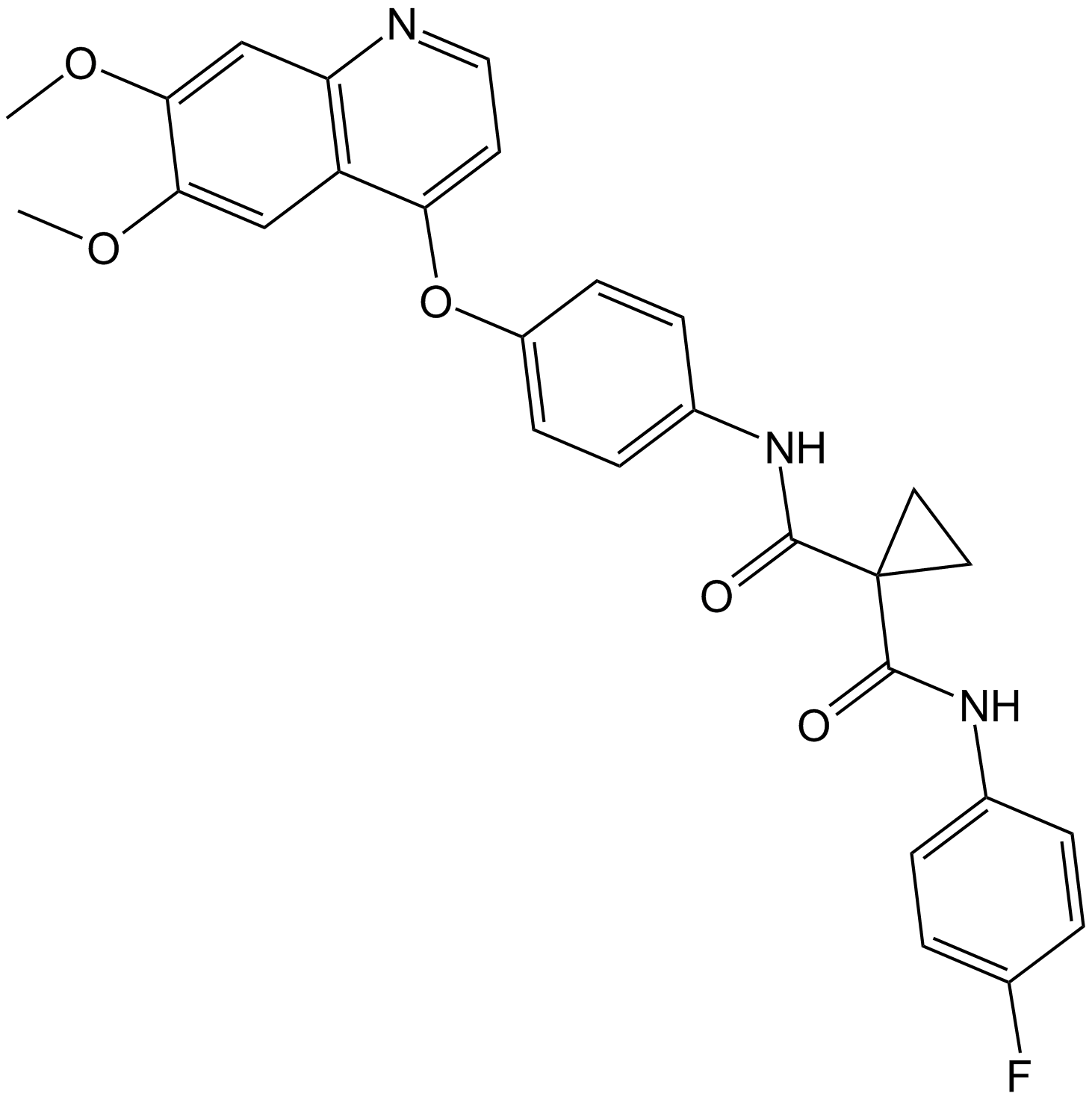
-
GC12531
Cabozantinib malate (XL184)
Cabozantinib Malat (XL184) (XL184 S-Malat) ist ein potenter Inhibitor von mehreren Rezeptor-Tyrosinkinasen, der VEGFR2, c-Met, Kit, Axl und Flt3 mit IC50-Werten von 0.035, 1.3, 4.6, 7 und 11.3 nM hemmt.
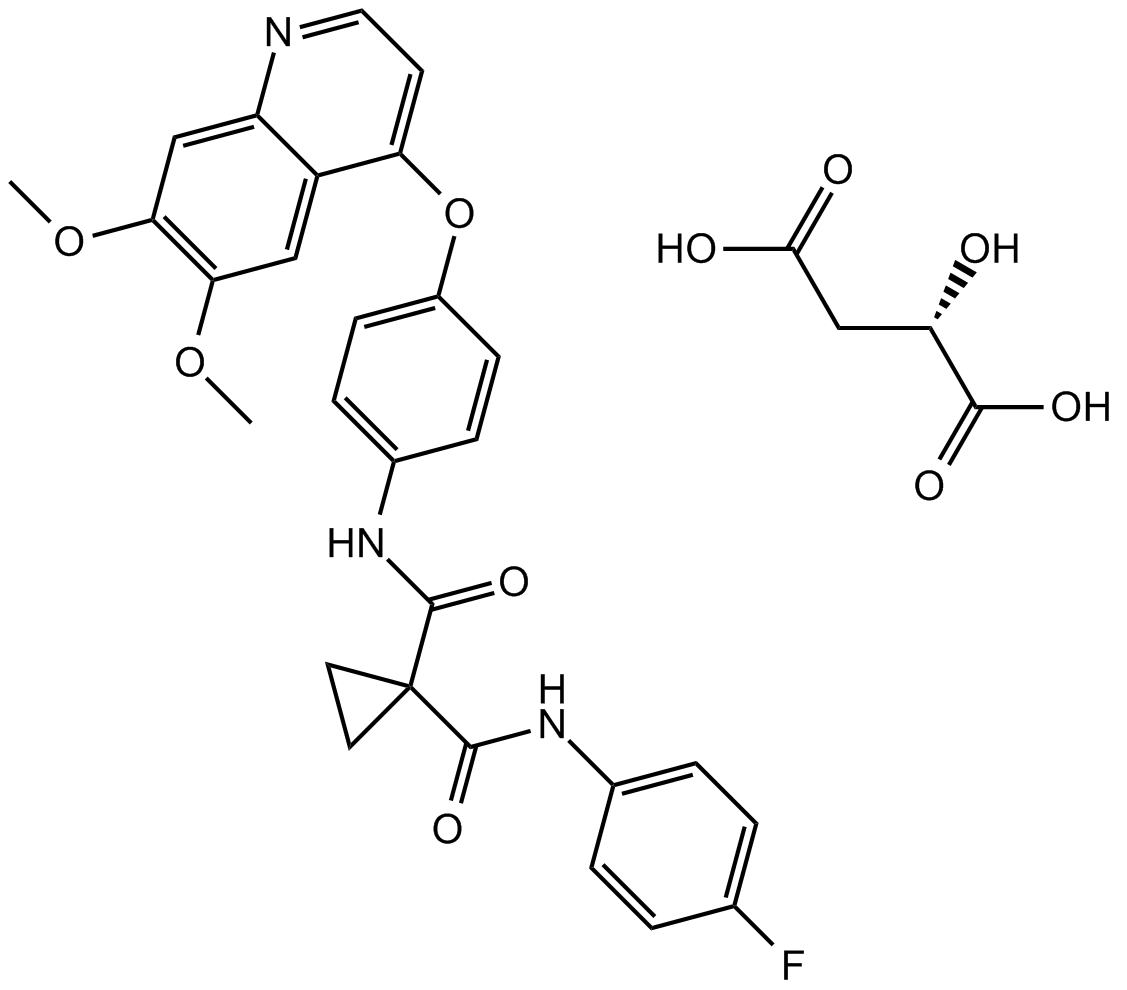
-
GC10692
Caffeic Acid Phenethyl Ester
KaffeesÄurephenethylester ist ein NF-κB-Hemmer.
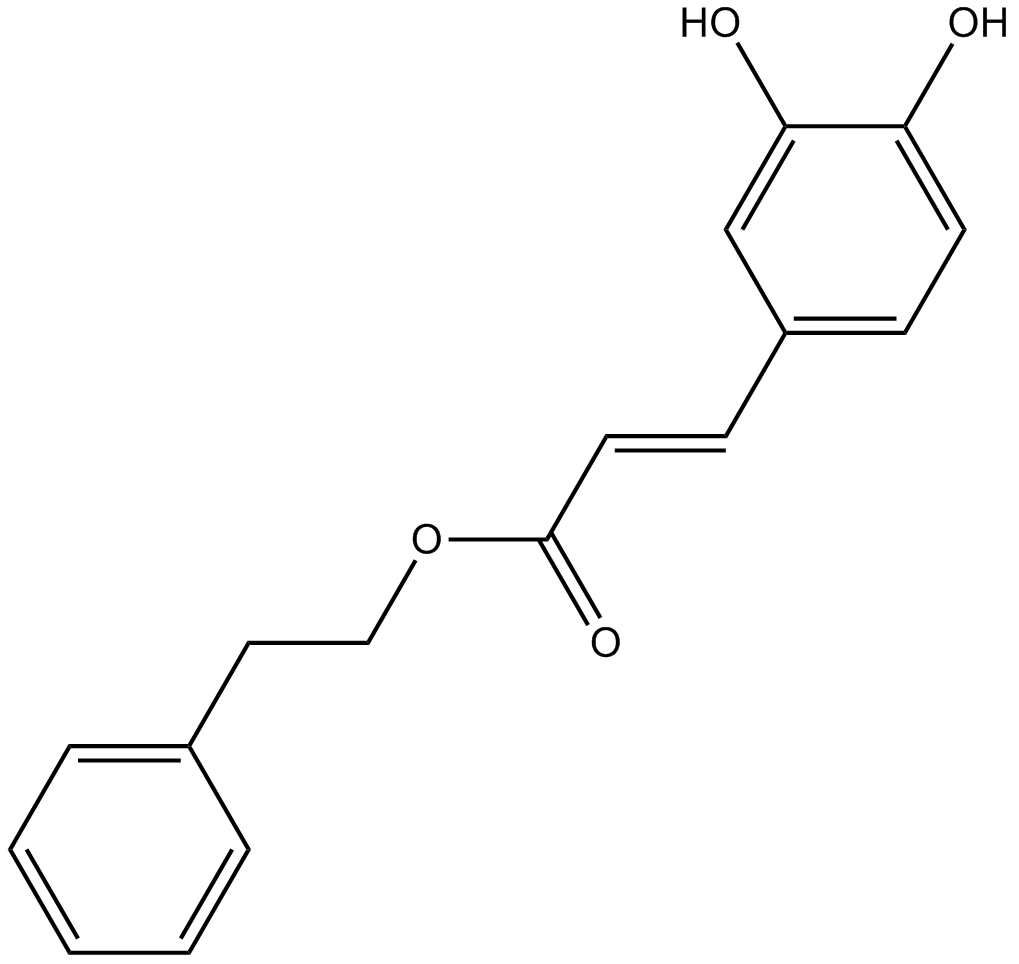
-
GC18604
Calcein Blue AM
Calcein Blue AM ist ein Zellfarbstoff.
-
GC43121
Calcein Orange™ Diacetate
Calcein Orange Diacetate is a fluorogenic dye that is used to assess cell viability.

-
GC43123
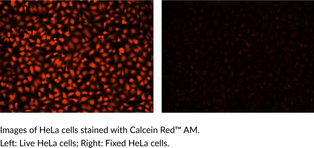
Calcein Red™ AM
Calcein Red? AM is a fluorogenic dye that is used to assess cell viability.
-
GC60668
Calcimycin hemimagnesium
Calcimycin (A-23187)-HÄmimagnesium ist ein Antibiotikum und ein einzigartiges Ionophor mit zweiwertigen Kationen (wie Calcium und Magnesium).
-
GC15161
Calcium D-Panthotenate
Calcium D-Panthotenat (Vitamin B5 Calciumsalz), ein Vitamin, kann den Patulingehalt des Apfelsaftes reduzieren.
-
GC30240
Calcium dobesilate
Calciumdobesilat, ein vasoprotektives Mittel, wird in vielen LÄndern hÄufig bei chronischen Venenerkrankungen, diabetischer Retinopathie und den Symptomen eines HÄmorrhoidenanfalls eingesetzt.
-
GC19086
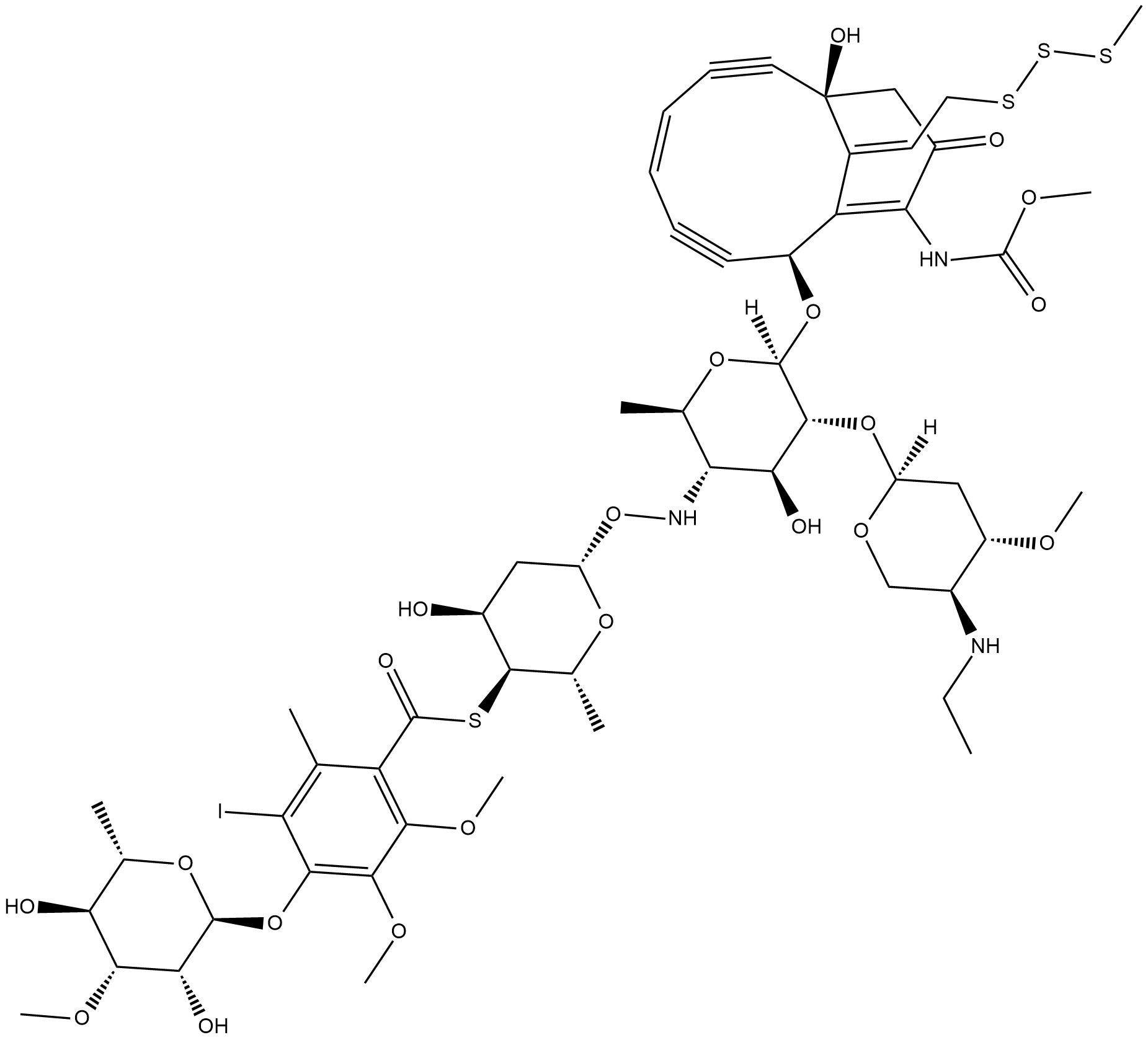
Calicheamicin
Calicheamicin, ein Antitumor-Antibiotikum, ist ein zytotoxisches Mittel, das DNA-DoppelstrangbrÜche verursacht.
-
GC65081
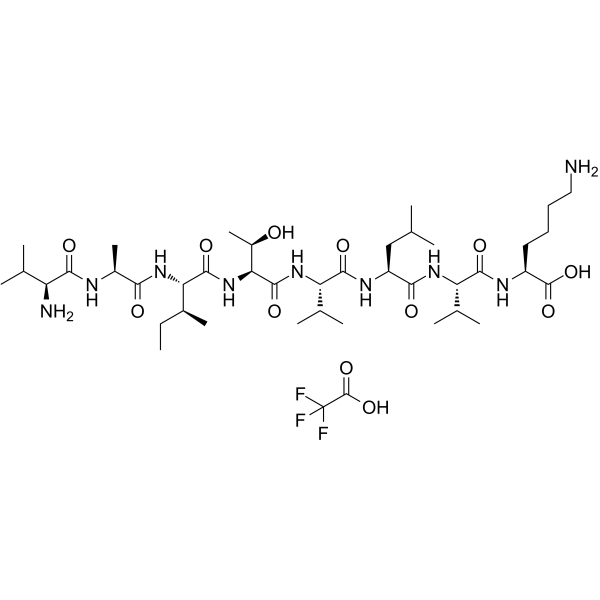
CALP1 TFA
CALP1 TFA ist ein Calmodulin (CaM)-Agonist (Kd von 88 μM) mit Bindung an die CaM EF-Hand/Ca2+--Bindungsstelle.
-
GC18315
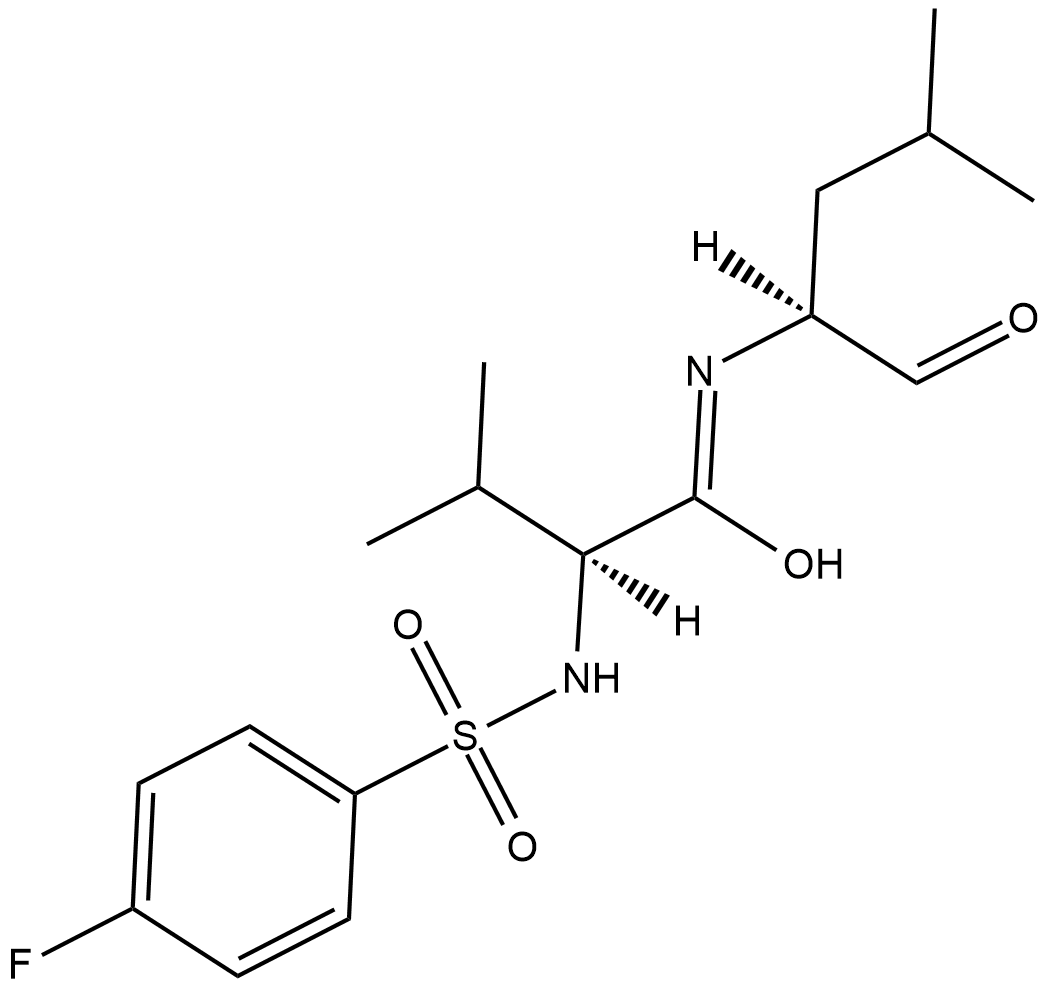
Calpain Inhibitor VI
Calpain inhibitor VI is an inhibitor of the calcium-dependent cysteine proteases u-calpain (calpain-1; IC50 = 7.5 nM) and m-calpain (calpain-2; IC50 = 78 nM).
-
GC10342
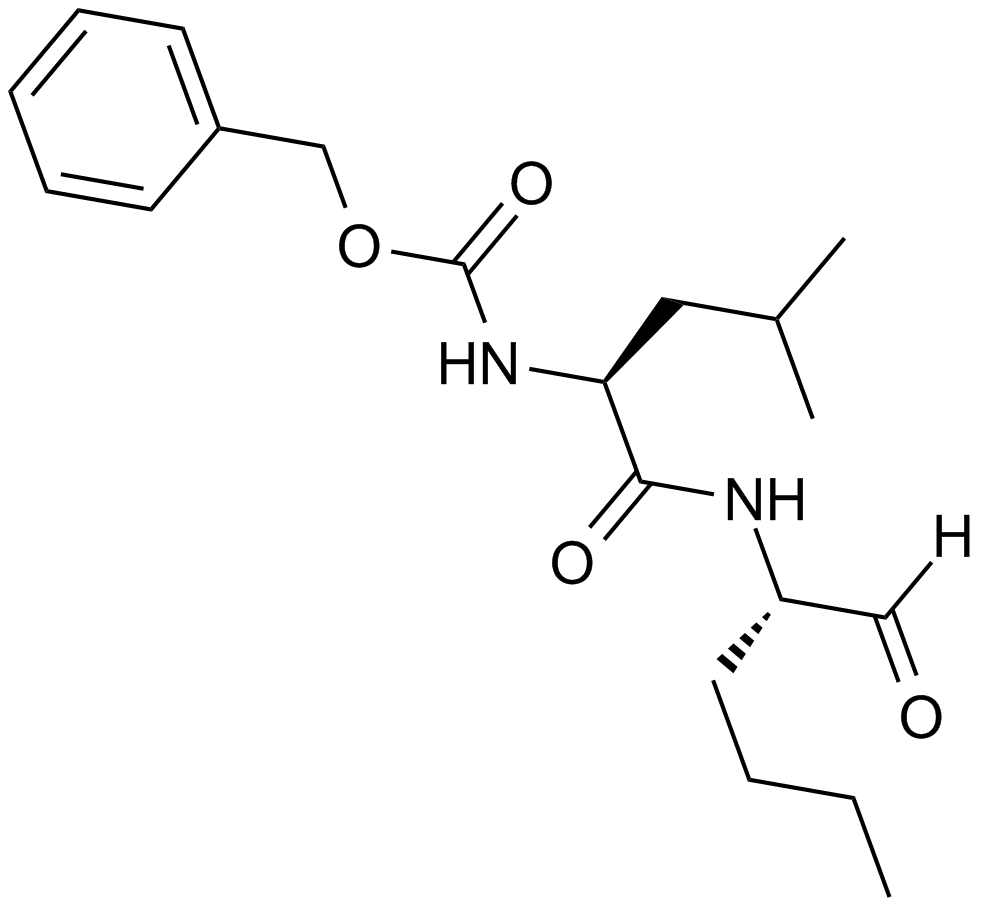
Calpeptin
A calpain inhibitor
-
GN10667
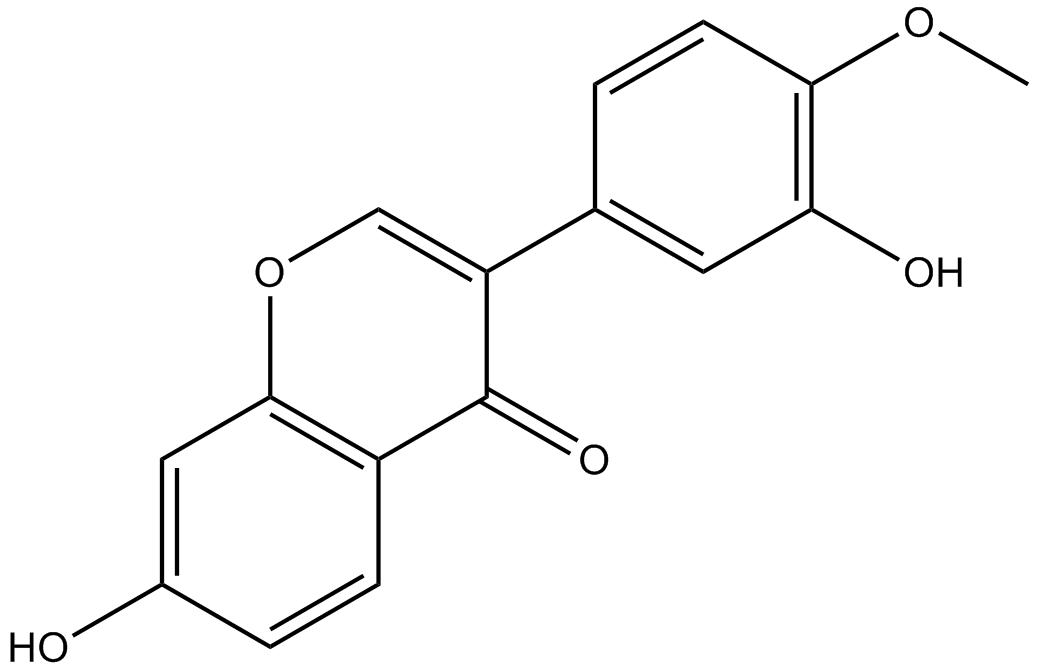
Calycosin
Calycosin (CA, 7, 3-dihydroxy-4-methoxy isoflavone, C16H12O5) is one of the flavonoids extracted from astragalus root, also known as the typical phytoestrogens.
-
GC35598
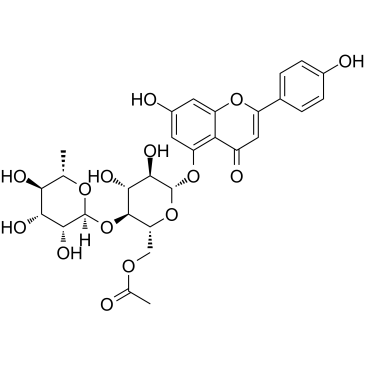
Camellianin A
Camellianin A, das wichtigste Flavonoid in A. nitida-BlÄttern, zeigt AntikrebsaktivitÄt und Angiotensin-Converting-Enzym (ACE)-hemmende AktivitÄt. Camellianin A hemmt die Proliferation der humanen Hep G2- und MCF-7-Zelllinien und induziert die signifikante Zunahme der G0/G1-Zellpopulation.
-
GC62253
Camrelizumab
Camrelizumab (SHR-1210) ist ein potenter humanisierter monoklonaler IgG4-κ-AntikÖrper (mAb) mit hoher AffinitÄt gegen PD-1. Camrelizumab bindet PD-1 mit einer hohen AffinitÄt von 3 nM und hemmt die Bindungswechselwirkung von PD-1 und PD-L1 mit einem IC50 von 0,70 nM. Camrelizumab wirkt als Anti-PD-1/PD-L1-Wirkstoff und kann fÜr die Krebsforschung verwendet werden, einschließlich NSCLC, ESCC, Hodgkin-Lymphom und fortgeschrittenem HCC et al.

-
GC12318
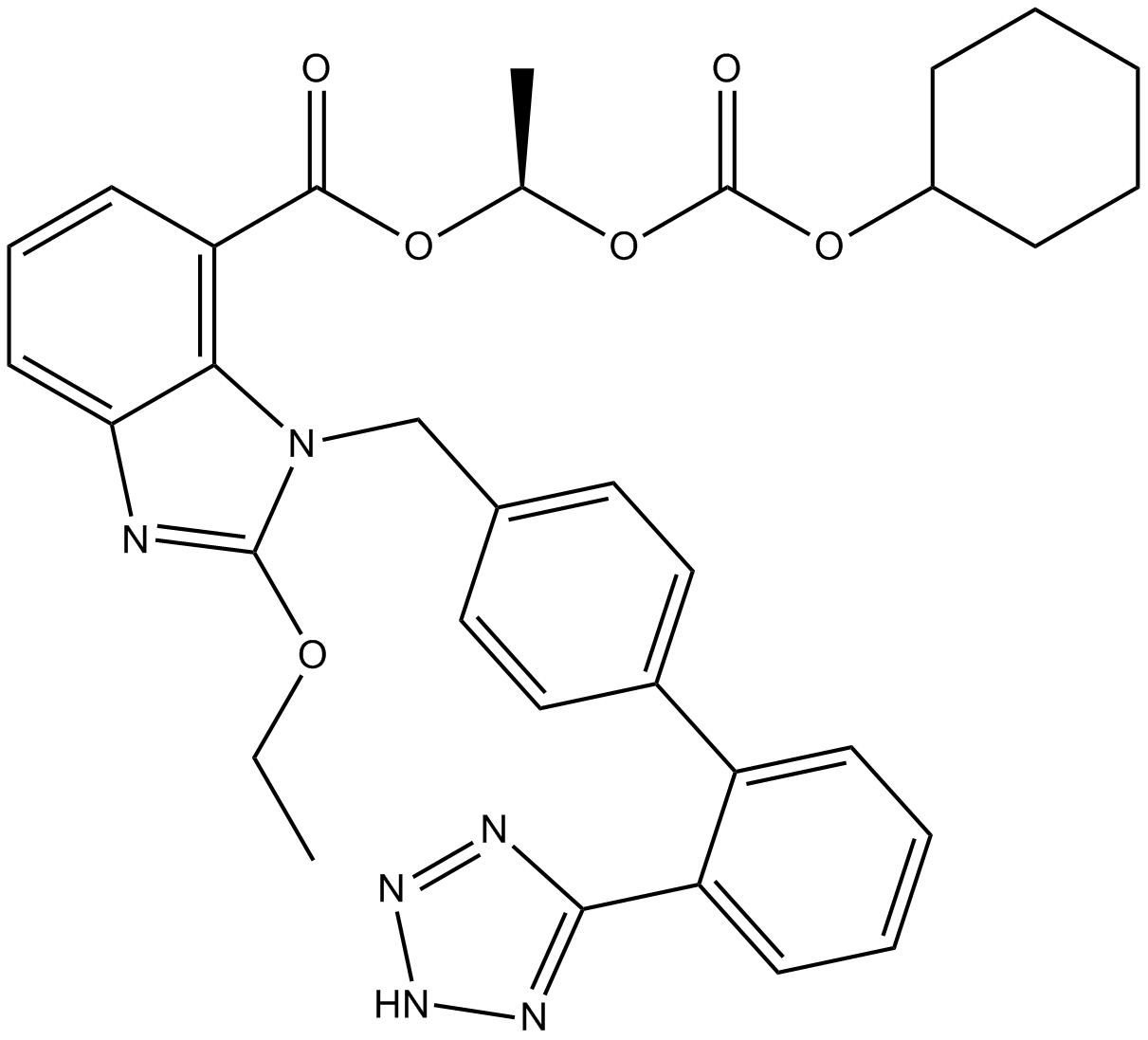
Candesartan Cilexetil
Candesartan Cilexetil (TCV-116) ist ein Angiotensin-II-Rezeptorantagonist, der hauptsÄchlich zur Behandlung von Bluthochdruck eingesetzt wird.
-
GC15866
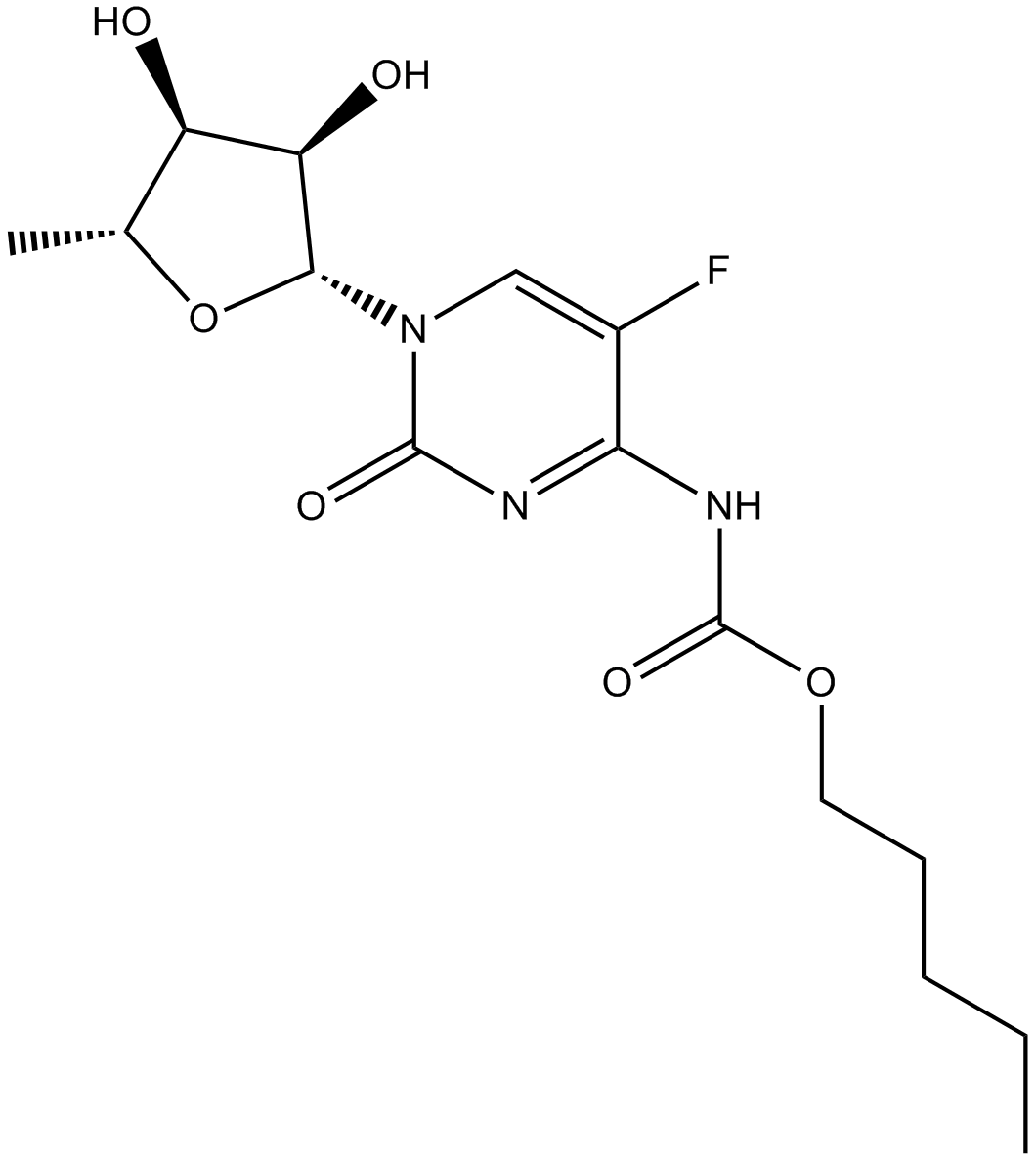
Capecitabine
Capecitabin ist ein orales Prodrug, das durch Thymidinphosphorylase in seinen aktiven Metaboliten 5-FU umgewandelt wird.
-
GC14065
Capsaicin
Ein Terpenalkaloid mit vielfältigen biologischen Aktivitäten.
-
GC17918
Capsazepine
A TRPV1 antagonist
-
GC49415
Capsorubin
A carotenoid with diverse biological activities
-
GC48878
Carbazomycin A
A bacterial metabolite with diverse biological activities
-
GC48893
Carbazomycin B
A bacterial metabolite with diverse biological activities
-
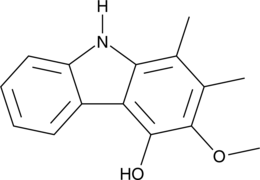
GC48850
Carbazomycin C
A bacterial metabolite with diverse biological activities
-
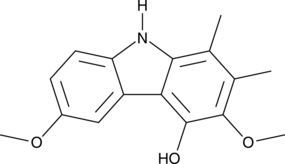
GC48826
Carbazomycin D
A bacterial metabolite with diverse biological activities
-
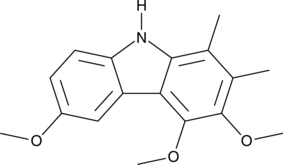
GC49147
Carboxyphosphamide
An inactive metabolite of cyclophosphamide
-
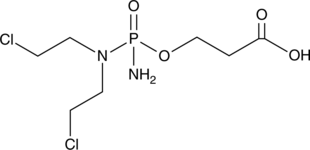
GC18069
Cardamonin
Cardamonin ((E)-Cardamomin) ist ein neuartiger Antagonist des hTRPA1-Kationenkanals mit einem IC50 von 454 nM.
-
GC18449
Cardanol monoene
Cardanolmonoen (Cardanol C15:1) ist eine phenolische Verbindung, die in CashewnussschalenflÜssigkeit zu finden ist. Cardanolmonoen kann eine mitochondrienassoziierte Apoptose in menschlichen Melanomzellen induzieren.
-
GC15089
Carfilzomib (PR-171)
A proteasome inhibitor