Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
GC17448
AT-406 (SM-406)
AT-406 (SM-406) (AT-406) est un mimétique Smac puissant et biodisponible par voie orale et un antagoniste des IAP, et il se lie aux protéines XIAP, cIAP1 et cIAP2 avec un Ki de 66,4, 1,9 et 5,1 nM, respectivement .
-
GC15870
AT7519
AT7519 (AT7519M) en tant qu'inhibiteur puissant des CDK, avec des IC50 de 210, 47, 100, 13, 170 et <10 nM pour CDK1, CDK2, CDK4 À CDK6 et CDK9, respectivement.
-
GC13998
AT7519 Hydrochloride
A Cdk inhibitor
-
GC10638
AT9283
A broad spectrum kinase inhibitor
-
GC18133
ATB-346
L'ATB-346 (ATB-346), un anti-inflammatoire non stéroÏdien (AINS) actif par voie orale, inhibe les cyclooxygénases-1 et 2 (COX-1 et 2).
-
GC32704
Atezolizumab (MPDL3280A)
Atezolizumab (MPDL3280A) est un anticorps monoclonal humanisé sélectif de type IgG1 contre le ligand de mort programmée 1 (PD-L1), utilisé pour la recherche sur le cancer.

-
GC62499
ATH686
ATH686 est un inhibiteur de FLT3 puissant, sélectif et compétitif pour l'ATP. ATH686 cible l'activité de la protéine kinase FLT3 mutante et inhibe la prolifération des cellules hébergeant des mutants FLT3 via l'induction de l'apoptose et l'inhibition du cycle cellulaire. ATH686 a des effets antileucémiques.
-
GC46892
ATRA-BA Hybrid
A prodrug form of all-trans retinoic acid and butyric acid
-
GN10394
Atractylenolide III

-
GC15878
Atractyloside Dipotassium Salt
Inhibitor of ADP/ATP translocases
-
GC39699
Aurintricarboxylic acid
L'acide aurintricarboxylique est un antagoniste allostérique de puissance nanomolaire avec une sélectivité envers les P2X1R et P2X3R sensibles À l'αβ-méthylène-ATP, avec des IC50 de 8,6 nM et 72,9 nM pour rP2X1R et rP2X3R, respectivement .
-
GC46895
Aurintricarboxylic Acid (ammonium salt)
A protein synthesis inhibitor with diverse biological activities
-
GC13332
Aurora A Inhibitor I
A potent and selective inhibitor of Aurora A kinase

-
GC15295
AUY922 (NVP-AUY922)
An Hsp90 inhibitor
-
GC31719
Avelumab (Anti-Human PD-L1, Human Antibody)
L'avelumab (Anti-Human PD-L1, Human Antibody) est un anticorps monoclonal IgG1 anti-PD-L1 entièrement humain avec une cytotoxicité potentielle À médiation cellulaire dépendante des anticorps.

-
GC42880
Avenanthramide-C methyl ester
Avenanthramide-C methyl ester is an inhibitor of NF-κB activation that acts by blocking the phosphorylation of IKK and IκB (IC50 ~ 40 μM).
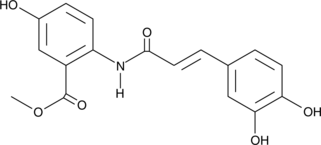
-
GC35440
AX-024
AX-024 est un inhibiteur de premier ordre disponible par voie orale de l'interaction TCR-Nck qui inhibe sélectivement l'activation des lymphocytes T déclenchée par le TCR avec une IC50 ~ 1 nM.
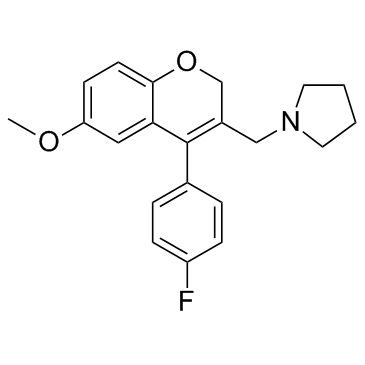
-
GC19046
AX-024 hydrochloride
Le chlorhydrate d'AX-024 est un inhibiteur de premier ordre disponible par voie orale de l'interaction TCR-Nck qui inhibe sélectivement l'activation des lymphocytes T déclenchée par le TCR avec une IC50 ~ 1 nM.
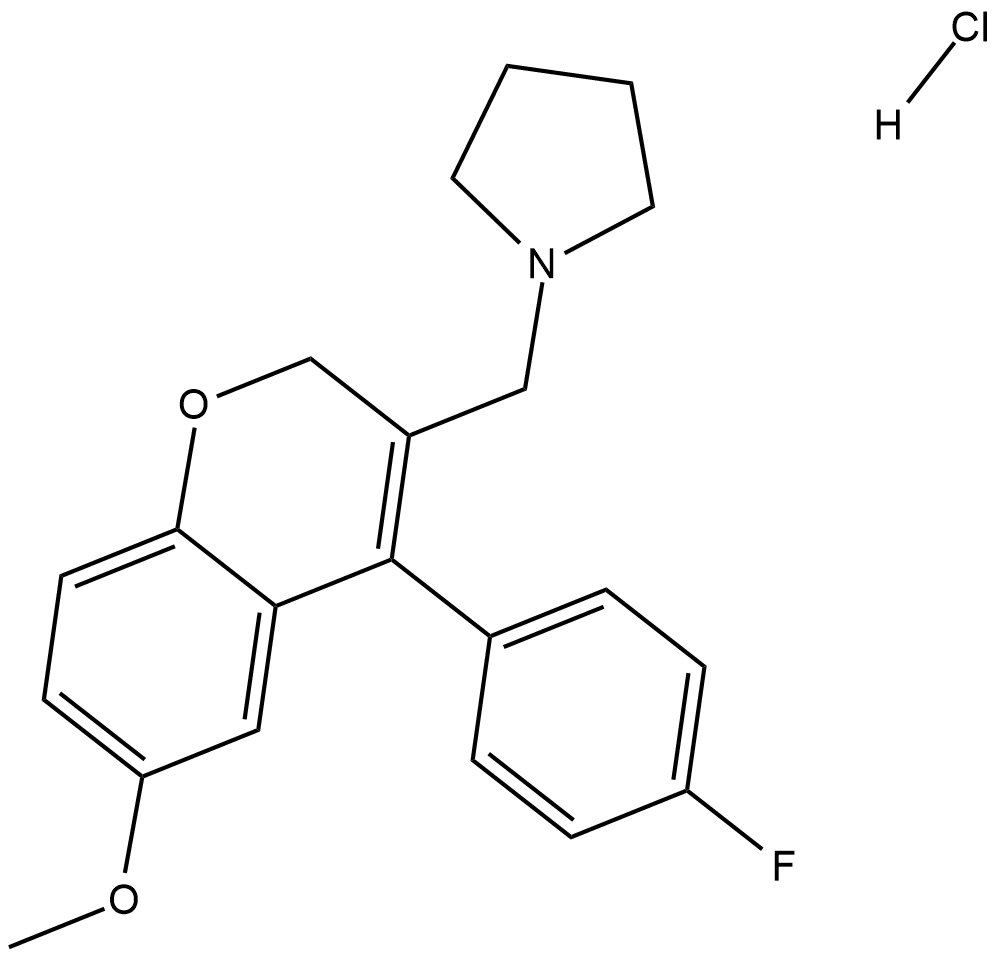
-
GC17045
AXL1717
A potent and selective inhibitor of IGF-1R
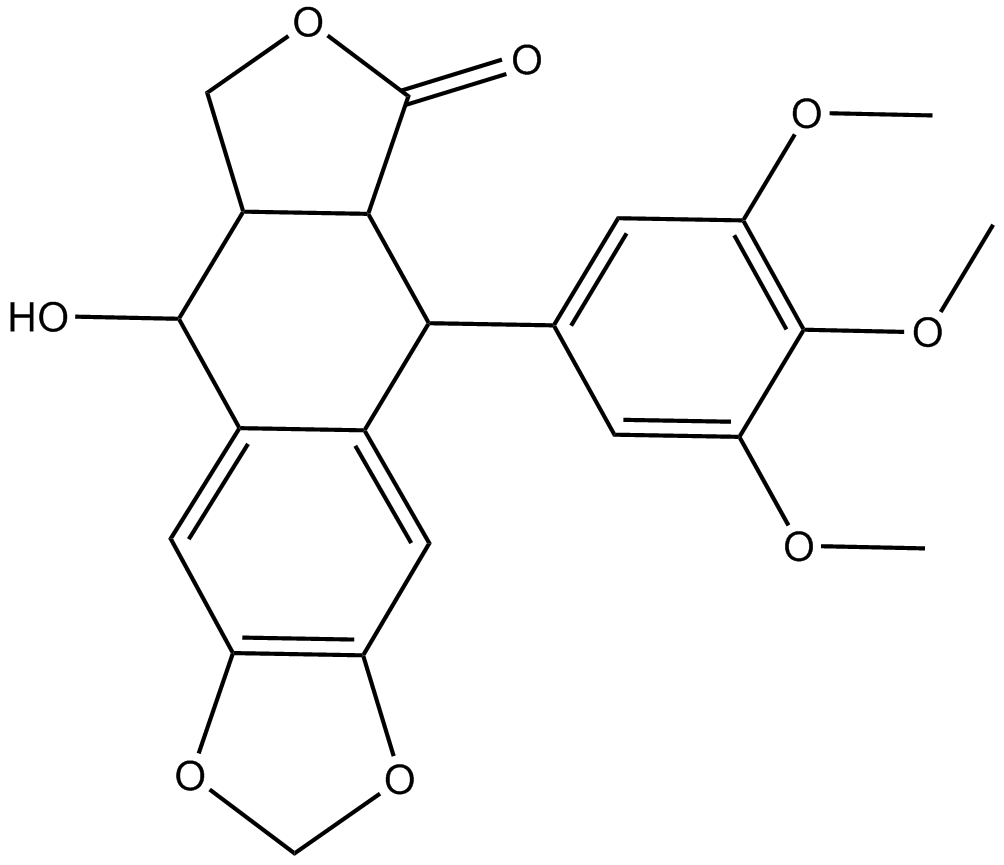
-
GC15055
AZ 628
AZ 628 est un inhibiteur de kinase pan-Raf avec des IC50 de 105, 34 et 29 nM pour B-Raf, B-RafV600E et c-Raf-1, respectivement.

-
GC13433
AZ 960
A JAK2 inhibitor
-
GC46901
Azadirachtin
L'azadirachtine, l'un des insecticides botaniques les plus prometteurs, est largement utilisé pour la lutte antiparasitaire.
-
GC15033
Azathioprine
L'azathioprine (BW 57-322) est un agent immunosuppresseur actif par voie orale.
-
GC48971
AZD 1152 (hydrochloride)
A prodrug for a potent Aurora B inhibitor
-
GC18566
AZD 3147
AZD 3147 est un puissant inhibiteur double sélectif, actif par voie orale, de mTORC1 et mTORC2 avec une valeur IC50 de 1,5 nM. AZD 3147 a également un effet sélectif sur PI3K.
-
GC50109
AZD 5582 dihydrochloride
Le dichlorhydrate d'AZD 5582 est un antagoniste de l'inhibiteur des protéines d'apoptose (IAP), qui se lie aux domaines BIR3 cIAP1, cIAP2 et XIAP avec des IC50 de 15, 21 et 15 nM, respectivement. AZD5582 induit l'apoptose.
-
GC33247
AZD-5991
AZD-5991 est un inhibiteur puissant et sélectif de Mcl-1 avec une IC50 de 0,7 nM dans le test FRET et un Kd de 0,17 nM dans le test de résonance plasmonique de surface (SPR).

-
GC33283
AZD-5991 Racemate
AZD-5991 Racemate est le racémate de AZD-5991. Le racémate AZD-5991 est un inhibiteur Mcl-1 avec une CI50 <3 nM dans le test FRET.
-
GC33239
AZD-5991 S-enantiomer
L'énantiomère S AZD-5991 est l'énantiomère le moins actif de l'AZD-5991. L'énantiomère S AZD-5991 est un inhibiteur de Mcl-1 avec une IC50 de 6,3 μM dans le test FRET et un Kd de 0,98 μM dans le test de résonance plasmonique de surface (SPR).
-
GC64938
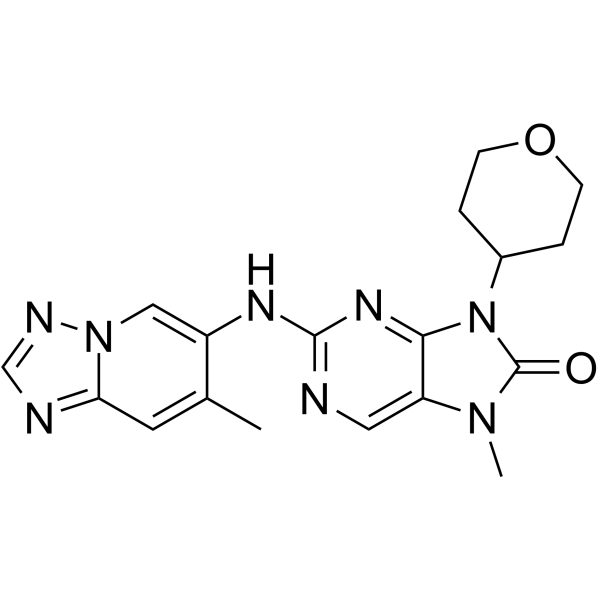
AZD-7648
L'AZD-7648 est un puissant inhibiteur sélectif de l'ADN-PK, actif par voie orale, avec une IC50 de 0,6 nM. L'AZD-7648 induit l'apoptose et présente une activité antitumorale.
-
GC12660
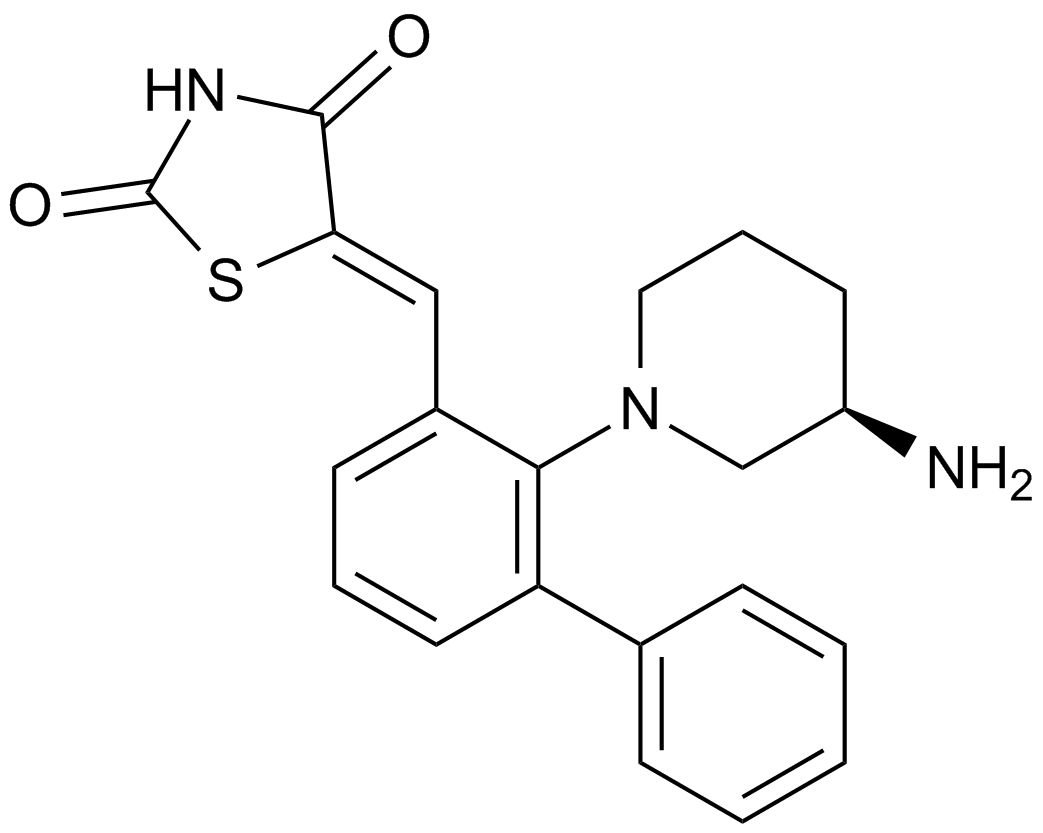
AZD1208
A pan-Pim kinase inhibitor
-
GC13029
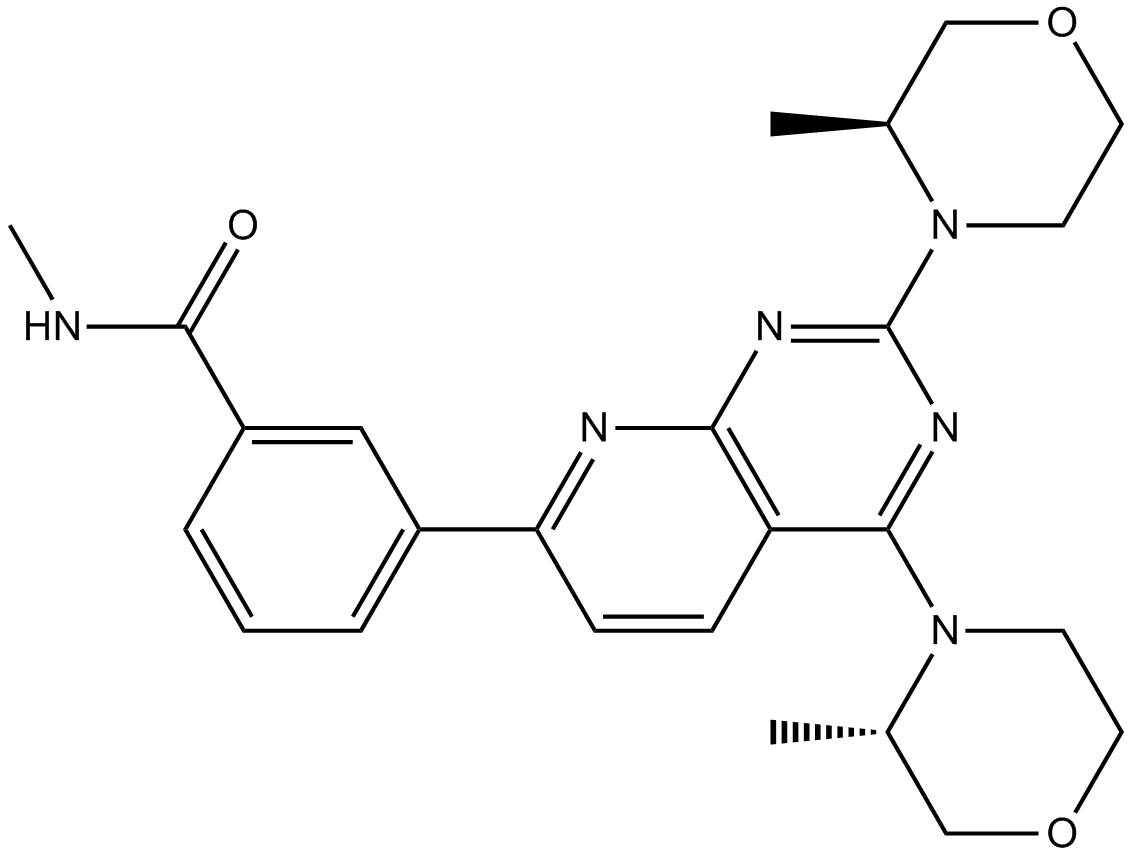
AZD2014
AZD2014 (AZD2014) est un inhibiteur mTOR compétitif de l'ATP avec une IC50 de 2,81 nM. AZD2014 inhibe À la fois les complexes mTORC1 et mTORC2.
-
GC33255
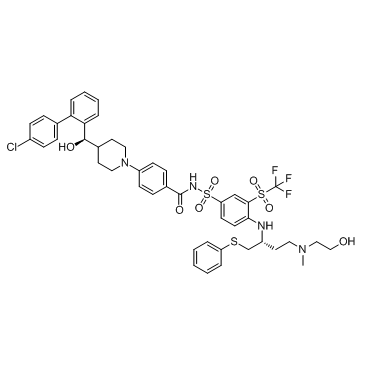
AZD4320
AZD4320 est un nouvel inhibiteur double BCL2/BCLxL imitant BH3 avec des IC50 de 26 nM, 17 nM et 170 nM pour les cellules KPUM-MS3, KPUM-UH1 et STR-428, respectivement.
-
GC19050
AZD5582
AZD5582 est un antagoniste de l'inhibiteur des protéines d'apoptose (IAP), qui se lie aux domaines BIR3 cIAP1, cIAP2 et XIAP avec des IC50 de 15, 21 et 15 nM, respectivement. AZD5582 induit l'apoptose.
-
GC16380
AZD8055
Inhibiteur de MTOR
-
GC19054
Azoramide
L'azoramide est un puissant modulateur À petite molécule actif par voie orale de la réponse protéique non repliée (UPR).
-
GC46904
Azoxystrobin
L'azoxystrobine est un fongicide β-méthoxyacrylate À large spectre.
-
GC60616
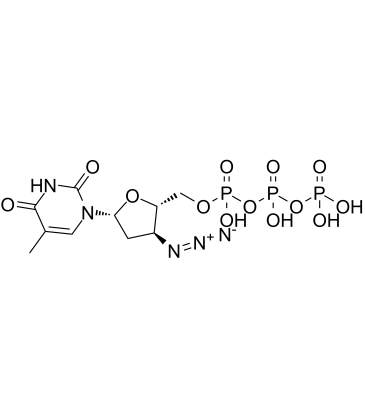
AZT triphosphate
L'AZT triphosphate (3'-azido-3'-désoxythymidine-5'-triphosphate) est un métabolite triphosphate actif de la zidovudine (AZT).
-
GC60617
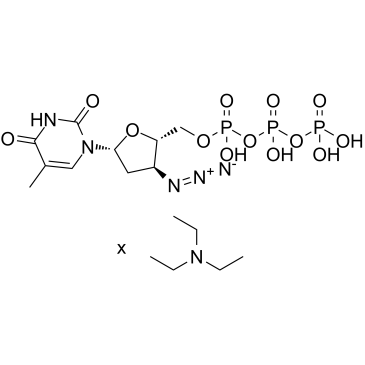
AZT triphosphate TEA
L'AZT triphosphate TEA (3'-azido-3'-désoxythymidine-5'-triphosphate TEA) est un métabolite triphosphate actif de la zidovudine (AZT).
-
GC35458
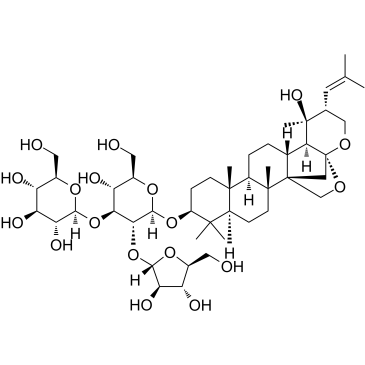
Bacopaside II
Le bacopaside II, un extrait de la plante médicinale Bacopa monnieri, bloque le canal de l'eau de l'Aquaporine-1 (AQP1) et altère la migration des cellules qui expriment l'AQP1. Le bacopaside II induit l'arrêt du cycle cellulaire et l'apoptose.
-
GC34263
Bak BH3
Bak BH3 est dérivé du domaine BH3 de Bak, peut antagoniser la fonction de Bcl-xL dans les cellules.

-
GC52344
Bak BH3 (72-87) (human) (trifluoroacetate salt)
A Bak-derived peptide
-
GC12053
BAM7
A direct activator of Bax
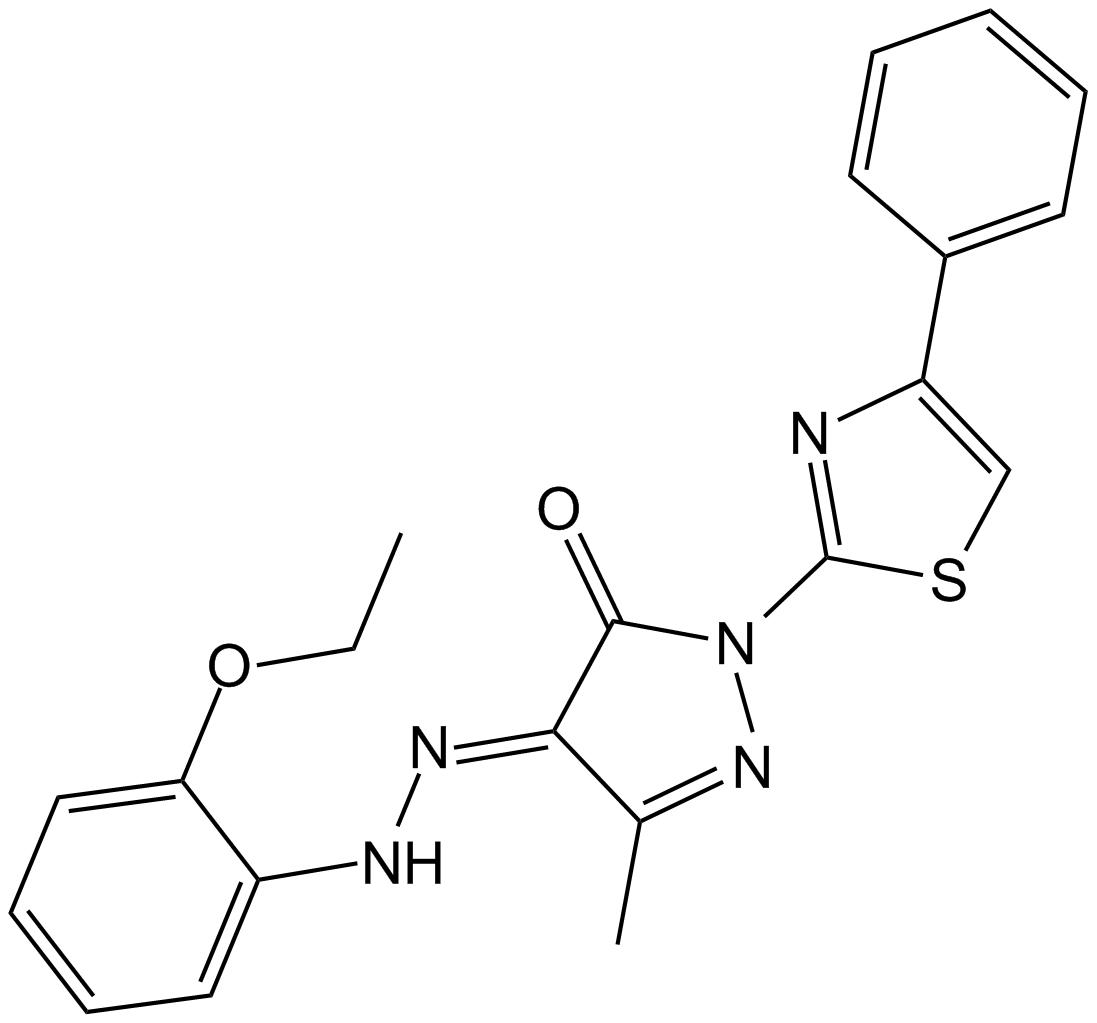
-
GN10507
Baohuoside I
Baohuoside I, a flavonoid isolated from Epimedium koreanum Nakai, acts as an inhibitor of CXCR4, downregulates CXCR4 expression, induces apoptosis and shows anti-tumor activity.
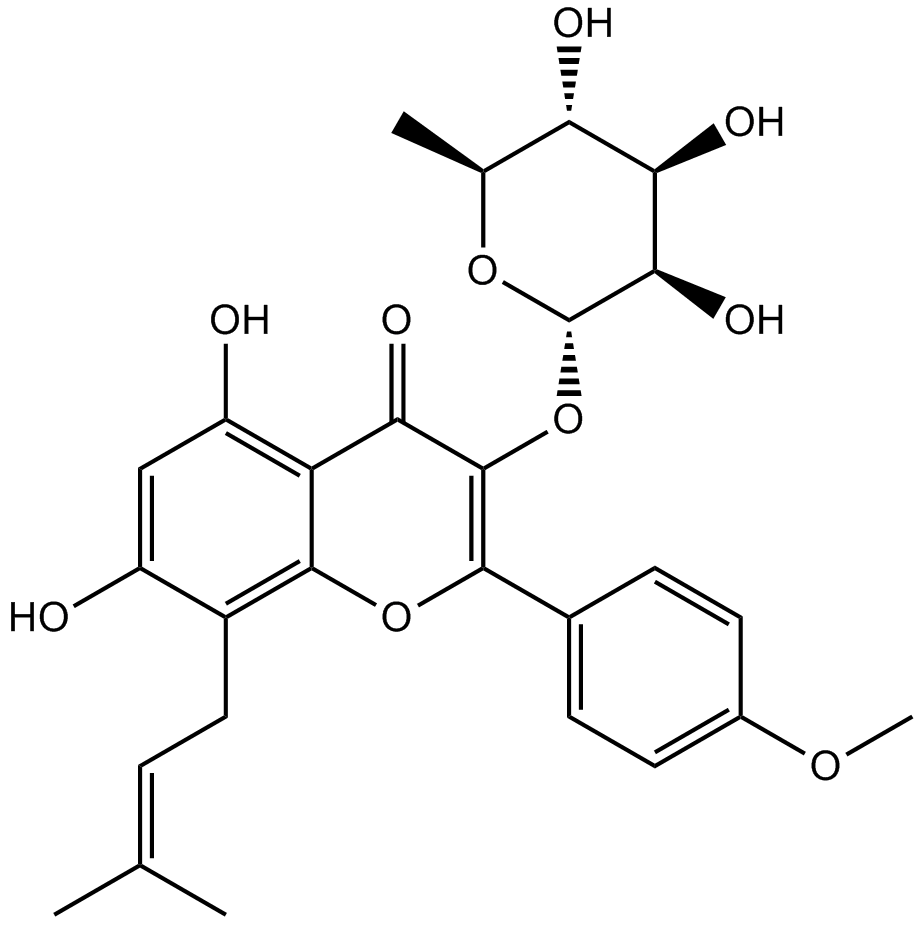
-
GC15371
Bardoxolone
An anti-inflammatory compound that activates Nrf2/ARE signaling
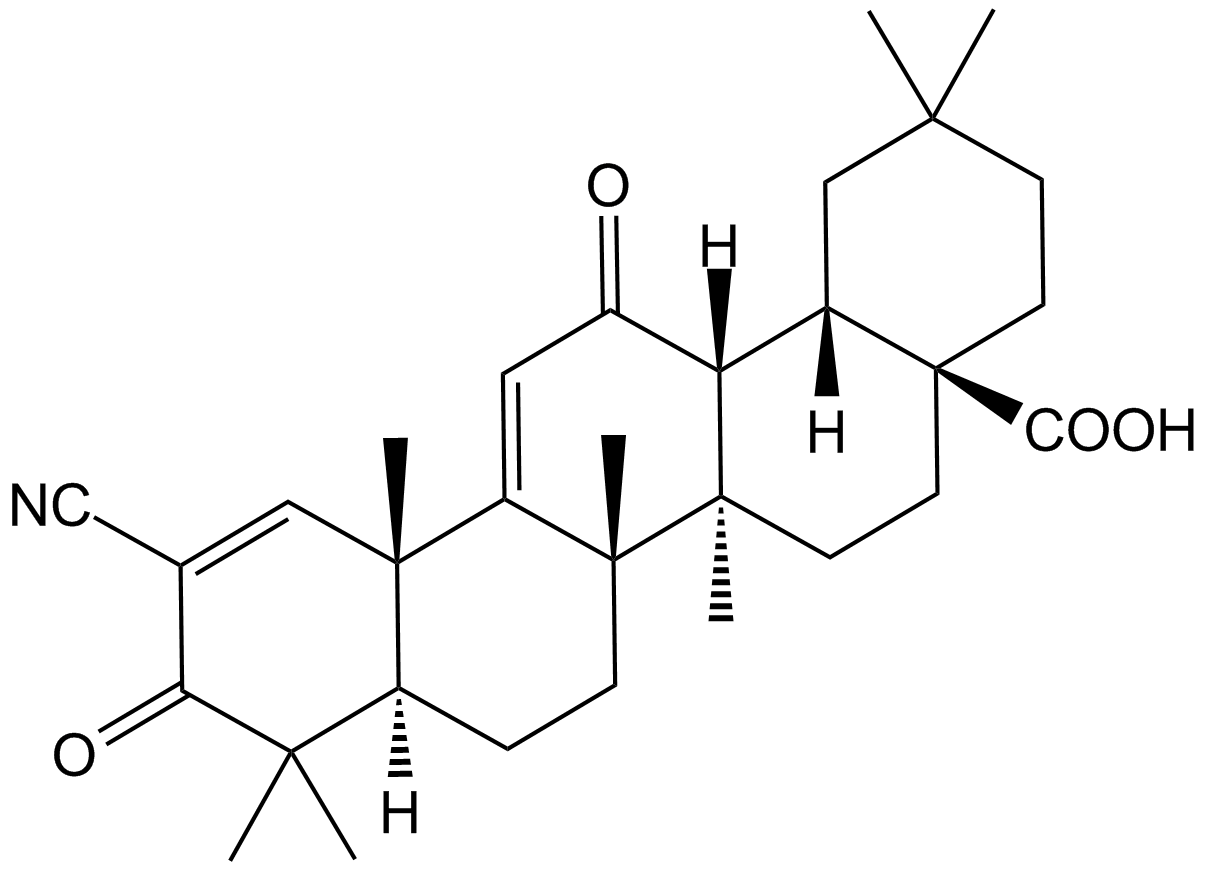
-
GC11572
Bardoxolone methyl
A synthetic triterpenoid with potent anticancer and antidiabetic activity
-
GC60620
Batabulin
La batabuline (T138067) est un agent antitumoral qui se lie de manière covalente et sélective À un sous-ensemble des isotypes de la β-tubuline, perturbant ainsi la polymérisation des microtubules. La batabuline affecte la morphologie cellulaire et conduit À l'arrêt du cycle cellulaire, induisant finalement la mort cellulaire apoptotique.
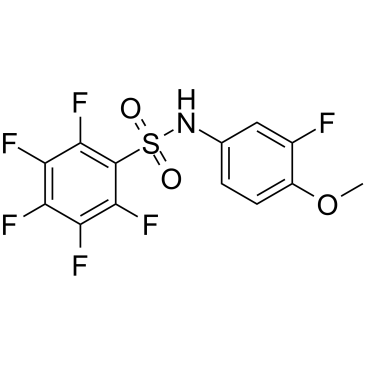
-
GC60621
Batabulin sodium
La batabuline sodique (T138067 sodique) est un agent antitumoral qui se lie de manière covalente et sélective À un sous-ensemble des isotypes de la β-tubuline, perturbant ainsi la polymérisation des microtubules. La batabuline sodique affecte la morphologie cellulaire et conduit À l'arrêt du cycle cellulaire, induisant finalement la mort cellulaire apoptotique.
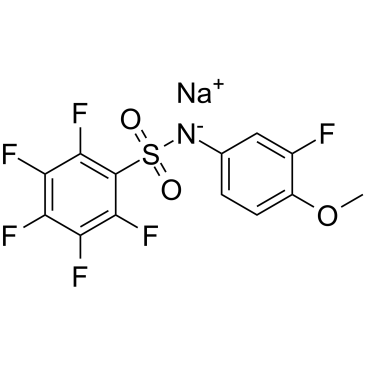
-
GC12763
Bax channel blocker
-
GC16023
Bax inhibitor peptide P5
Bax inhibitor
-
GC17195
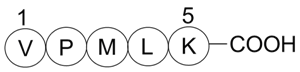
Bax inhibitor peptide V5
A Bax inhibitor
-
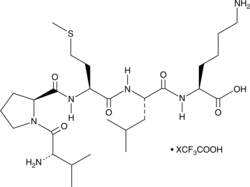
GC52476
Bax Inhibitor Peptide V5 (trifluoroacetate salt)
A Bax inhibitor
-
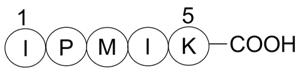
GC16695
Bax inhibitor peptide, negative control
Peptide inhibit Bax translocation to mitochondria
-
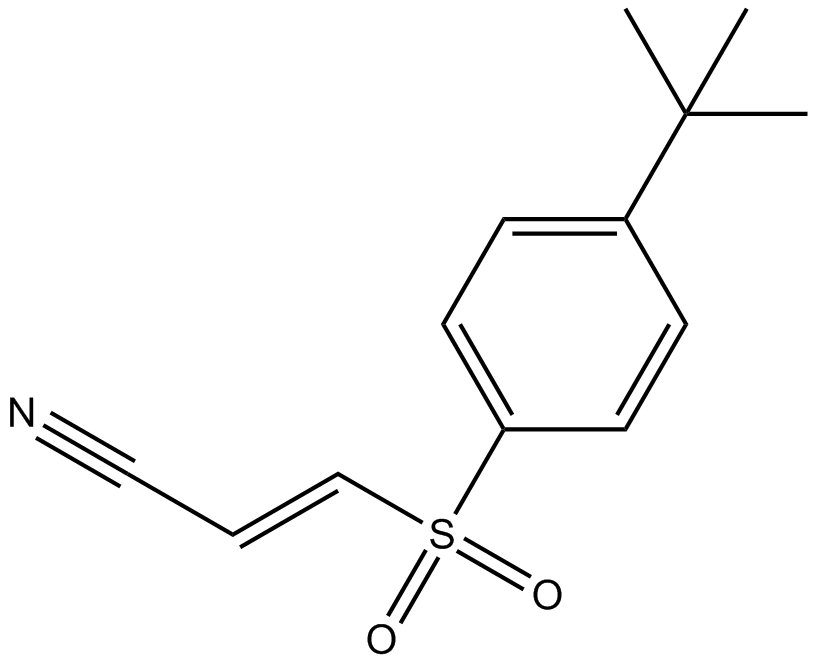
GC10345
Bay 11-7085
BAY 11-7085 (BAY 11-7083) est un inhibiteur de l'activation de NF-κB et de la phosphorylation de IκBα; il stabilise IκBα avec une IC50 de 10 μM.
-
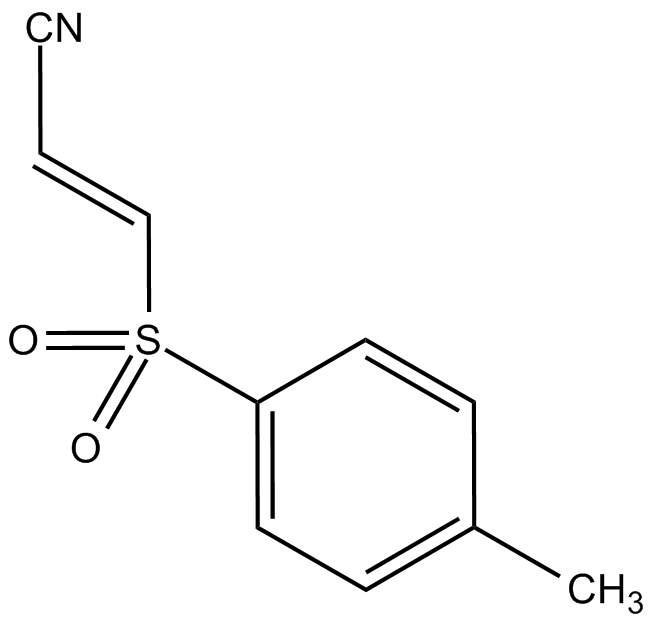
GC13035
Bay 11-7821
Un inhibiteur sélectif et irréversible de NF-κB
-
GC16389
BAY 61-3606
A Syk inhibitor

-
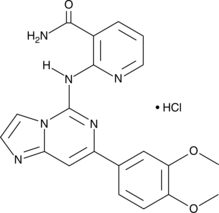
GC42897
BAY 61-3606 (hydrochloride)
BAY 61-3606 is a cell-permeable, reversible inhibitor of spleen tyrosine kinase (Syk; Ki = 7.5 nM; IC50 = 10 nM).
-
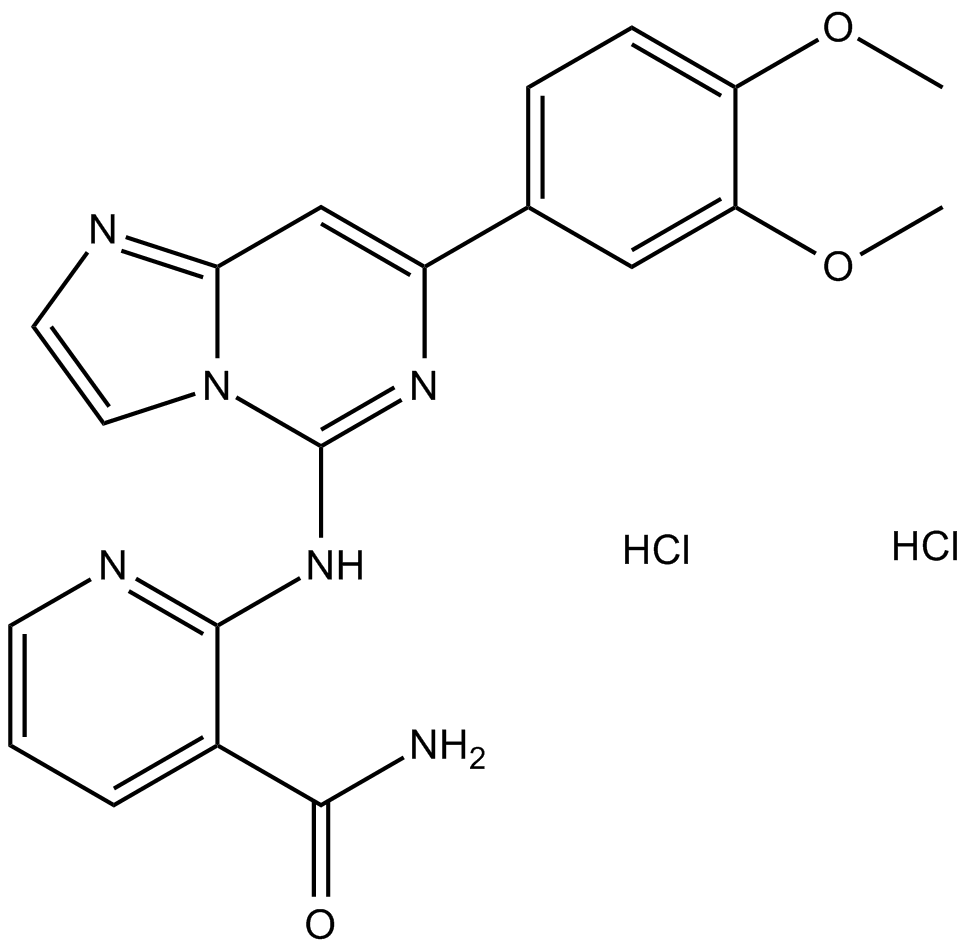
GC12136
BAY 61-3606 dihydrochloride
-
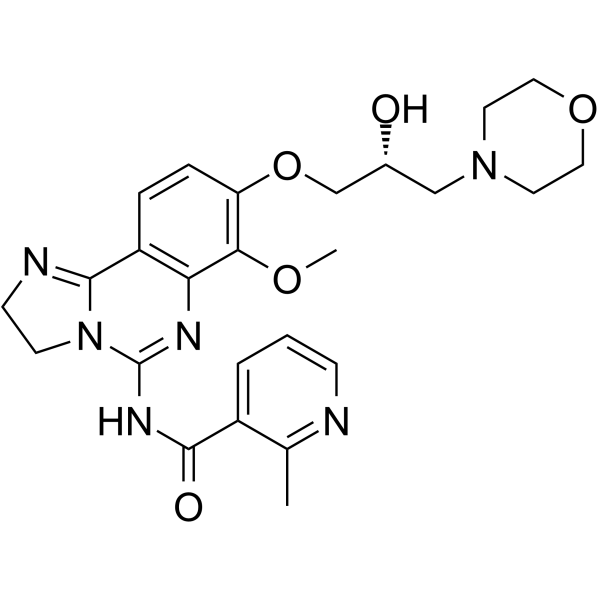
GC62164
BAY1082439
BAY1082439 est un inhibiteur sélectif de PI3Kα/β/δ biodisponible par voie orale. BAY1082439 inhibe également les formes mutées de PIK3CA. BAY1082439 est très efficace pour inhiber la croissance du cancer de la prostate Pten-null.
-
GC16516
BCH
Le BCH (BCH) est un inhibiteur sélectif et compétitif du grand transporteur d'acides aminés neutres 1 (LAT1) qui inhibe de manière significative l'absorption cellulaire des acides aminés et la phosphorylation de mTOR, ce qui induit la suppression de la croissance du cancer et de l'apoptose.
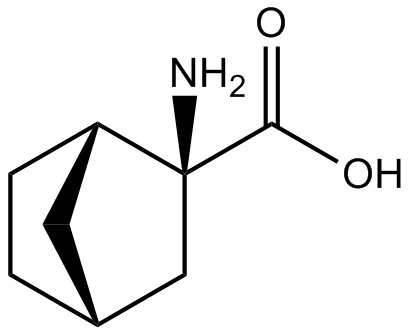
-
GC63325
Bcl-xL antagonist 2
L'antagoniste 2 de Bcl-xL est un antagoniste puissant, sélectif et actif par voie orale de BCL-XL avec une IC50 et un Ki de 0,091 μM et 65 nM, respectivement. L'antagoniste Bcl-xL 2 favorise l'apoptose des cellules cancéreuses. L'antagoniste Bcl-xL 2 a le potentiel pour la recherche sur la leucémie lymphoÏde chronique (LLC) et le lymphome non hodgkinien (LNH).
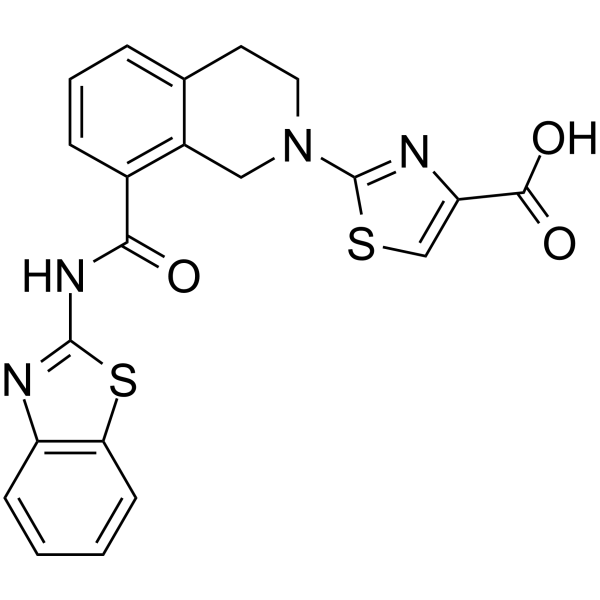
-
GC62599
BCL6-IN-4
BCL6-IN-4 est un puissant inhibiteur du lymphome 6 À cellules B (BCL6) avec une IC50 de 97 nM. BCL6-IN-4 a des activités antitumorales.
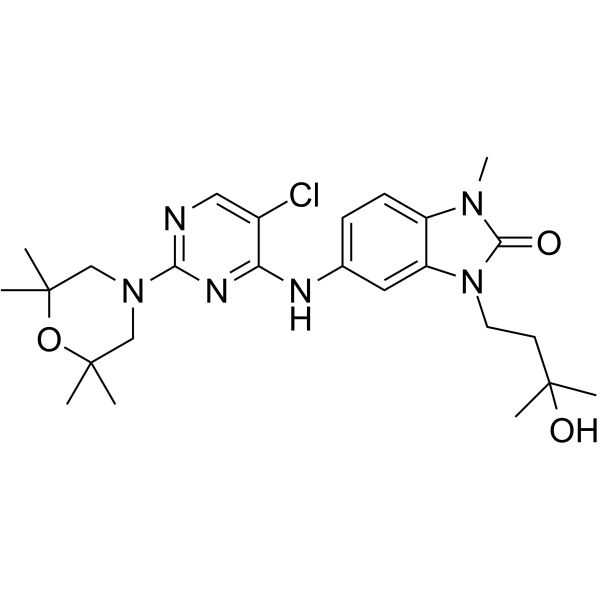
-
GC68012
BCL6-IN-7
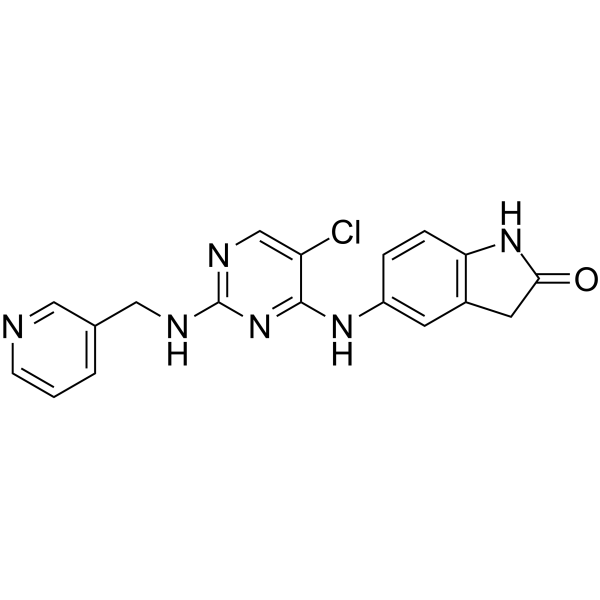
-
GC10721
BDA-366
Le BDA-366 est un puissant antagoniste de Bcl2 (Ki = 3,3 nM), liant le domaine Bcl2-BH4 avec une affinité et une sélectivité élevées. Le BDA-366 induit un changement conformationnel de Bcl2 qui annule sa fonction anti-apoptotique, le convertissant d'une molécule de survie en un inducteur de mort cellulaire. Le BDA-366 supprime la croissance des cellules cancéreuses du poumon.
-
GC42912
Becatecarin
La bécatécarine est un analogue de la rébeccamycine avec des effets antitumoraux. La bécatécarine s'intercale dans l'ADN et inhibe l'activité catalytique des topoisomérases I/II.
-
GC68369
Belantamab

-
GC65031
Belimumab
Le belimumab (LymphoStat B) est un anticorps monoclonal IgG1Λ humain qui inhibe le facteur d'activation des cellules B (BAFF).

-
GC49042
Benastatin A
A bacterial metabolite with diverse biological activities
-
GC64354
Bendamustine
La bendamustine (base libre SDX-105), un analogue purique, est un agent de réticulation de l'ADN. La bendamustine active la réponse au stress et l'apoptose des dommages À l'ADN. La bendamustine possède de puissantes propriétés alkylantes, anticancéreuses et antimétabolites.
-
GC10744
Bendamustine HCl
Bendamustine HCl (SDX-105), un analogue de la purine, est un agent de réticulation de l'ADN. Bendamustine HCl active la réponse au stress et l'apoptose des dommages À l'ADN. Le chlorhydrate de bendamustine possède de puissantes propriétés alkylantes, anticancéreuses et antimétabolites.
-
GC49781
Benomyl
A carbamate pesticide
-
GC62451
Benpyrine
La benpyrine est un inhibiteur du TNF-α hautement spécifique et actif par voie orale avec une valeur KD de 82,1 μM.
-
GC49403
Benzarone
Benzarone (Fragivix) est un puissant inhibiteur du transporteur 1 de l'acide urique humain (hURAT1), avec une IC50 de 2,8 μM dans l'ovocyte.
-
GC14930
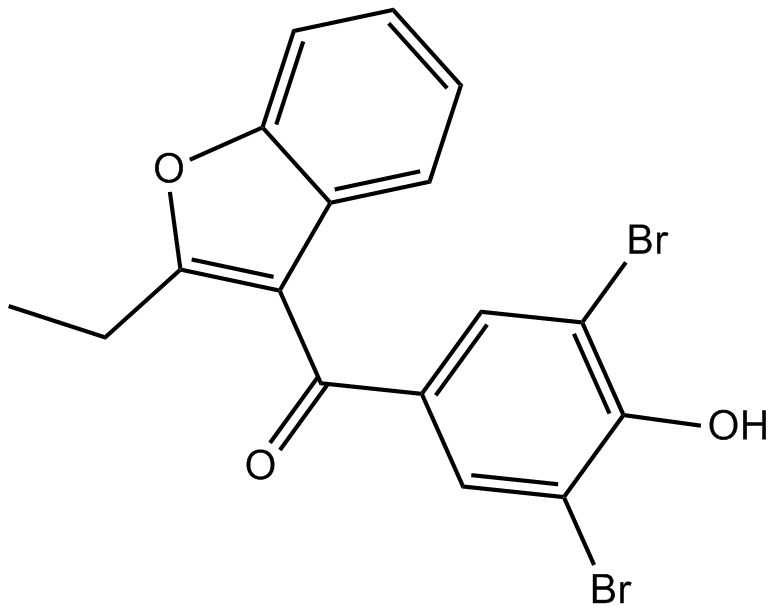
Benzbromarone
La benzbromarone est un inhibiteur non compétitif très efficace et bien toléré de la xanthine oxydase, utilisé comme agent uricosurique, utilisé dans le traitement de la goutte.
-
GN10520
Benzoylpaeoniflorin
-
GC38683
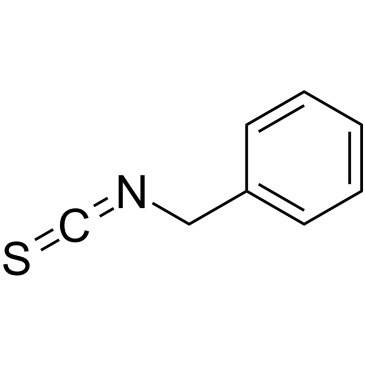
Benzyl isothiocyanate
L'isothiocyanate de benzyle fait partie des isothiocyanates naturels À activité antimicrobienne.
-
GN10358
Berbamine hydrochloride
-
GN10539
Bergenin

-
GC42925
Berteroin
La bertéroÏne, un analogue naturel du sulforaphane, est un agent antimétastatique.
-
GC10734
Beta-Lapachone
La bêta-lapachone (ARQ-501; NSC-26326) est une O-naphtoquinone naturelle, agit comme un inhibiteur de la topoisomérase I et induit l'apoptose en inhibant la progression du cycle cellulaire.
-
GC35504
Beta-Zearalanol
Le bêta-zéaralénol est une mycotoxine produite par Fusarium spp, qui provoque l'apoptose et le stress oxydatif dans les cellules reproductrices des mammifères.
-
GN10632
Betulin
-
GC10480
Betulinic acid
A plant triterpenoid similar to bile acids
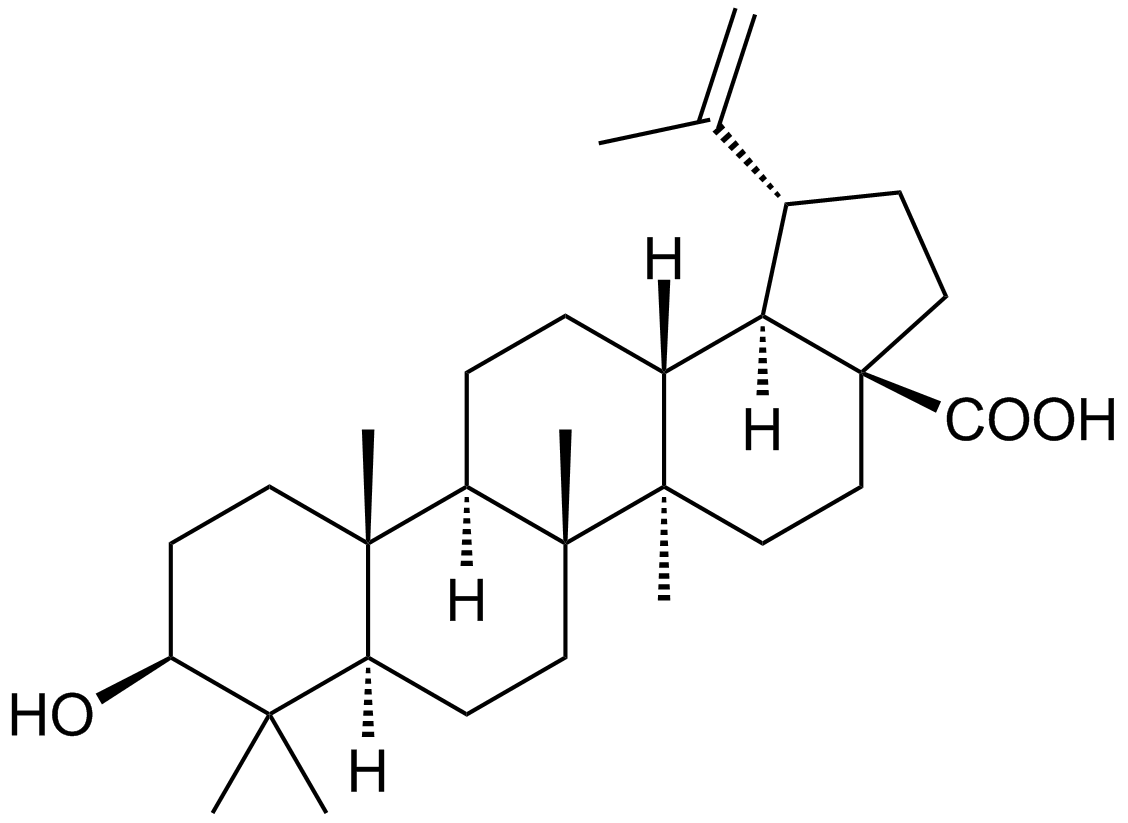
-
GC48477
Betulinic Acid propargyl ester
An alkyne derivative of betulinic acid
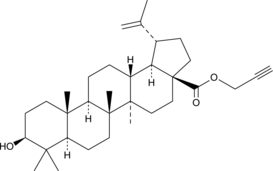
-
GC48504
Betulinic Aldehyde oxime
A derivative of betulin
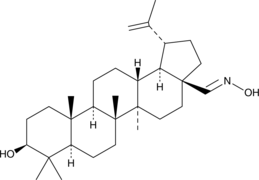
-
GC48520
Betulonaldehyde
A pentacyclic triterpenoid
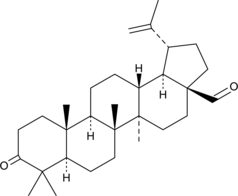
-
GC12074
BG45
BG45 est un inhibiteur de HDAC de classe I avec une sélectivité pour HDAC3 (IC50 = 289 nM).
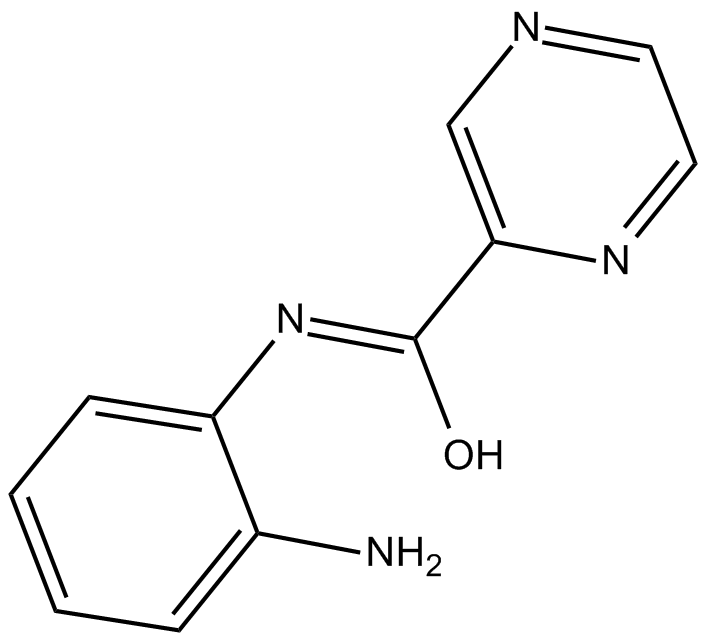
-
GC18136
BH3I-1
BH3I-1 est un antagoniste de la famille Bcl-2, qui inhibe la liaison du peptide Bak BH3 À Bcl-xL avec un Ki de 2,4 ± 0,2 μM dans le test FP. BH3I-1 a un Kd de 5,3 μM contre le couple p53/MDM2.
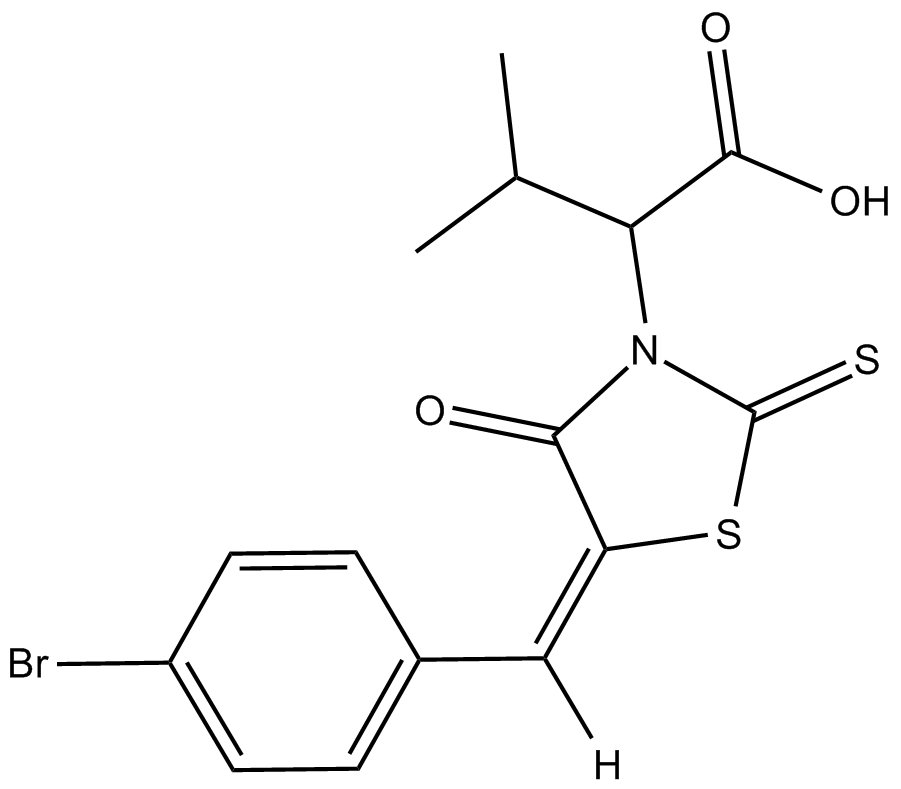
-
GC35511
BI-0252
Le BI-0252 est un inhibiteur sélectif de MDM2-p53 actif par voie orale avec une IC50 de 4 nM. Le BI-0252 peut induire des régressions tumorales chez tous les animaux d'une xénogreffe SJSA-1 de souris, avec induction concomitante des gènes cibles de la protéine tumorale p53 (TP53) et des marqueurs d'apoptose.
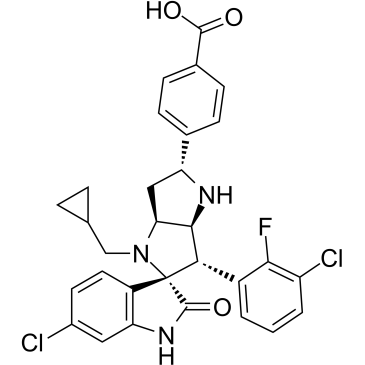
-
GC17828
BI-847325
Le BI-847325 est un double inhibiteur compétitif de l'ATP des kinases MEK et Aurora (AK) avec des valeurs IC50 de 4 et 15 nM pour la MEK2 humaine et l'AK-C, respectivement.

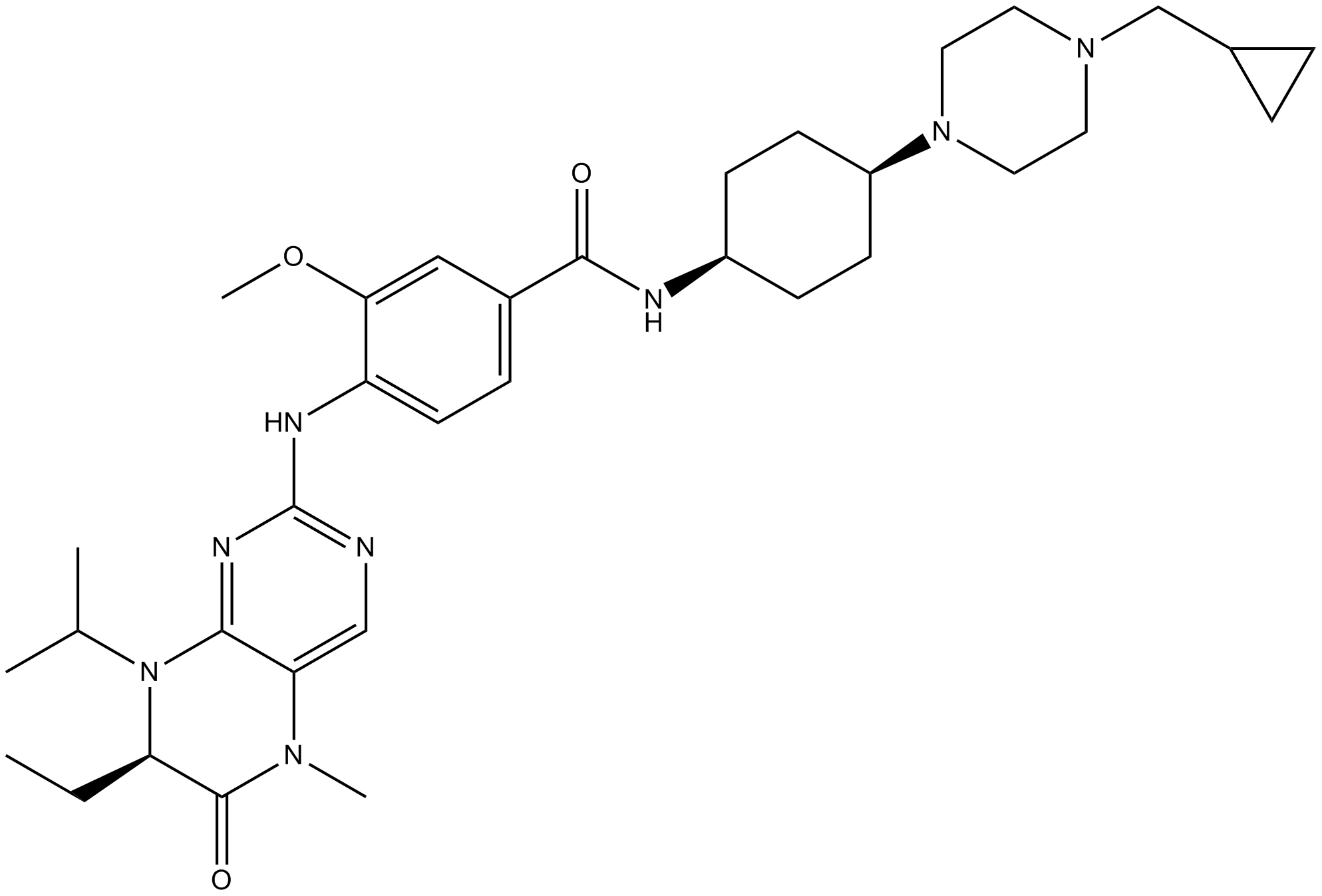
-
GC11224
BI6727(Volasertib)
Le BI6727 (Volasertib) (BI 6727) est un inhibiteur de la Polo-like kinase 1 (PLK1) actif par voie orale, très puissant et compétitif pour l'ATP avec une IC50 de 0,87 nM. BI6727(Volasertib) inhibe PLK2 et PLK3 avec des IC50 de 5 et 56 nM, respectivement. BI6727(Volasertib) induit l'arrêt de la mitose et l'apoptose. BI6727 (Volasertib), un dérivé de la dihydroptéridinone, montre une activité antitumorale marquée dans plusieurs modèles de cancer.
-
GC13636
BIBR 1532
Le BIBR 1532 est un inhibiteur de la télomérase puissant, sélectif et non compétitif avecIC50de 100nM dans un test acellulaire.
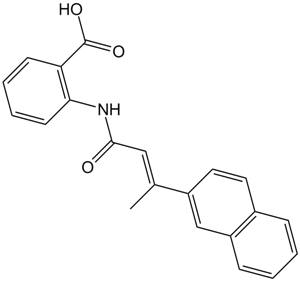
-
GC60076
Bigelovin
La bigélovine, une lactone sesquiterpénique isolée d'Inula helianthus-aquatica, est un agoniste sélectif des récepteurs α des rétinoÏdes X. Bigelovin supprime la croissance tumorale en induisant l'apoptose et l'autophagie via l'inhibition de la voie mTOR régulée par la génération de ROS.
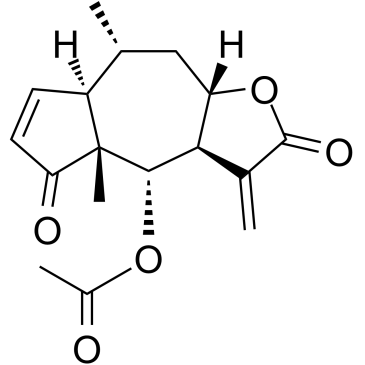
-
GC15987
BIM, Biotinylated
Bim peptide fragment with a biotin moiety attached
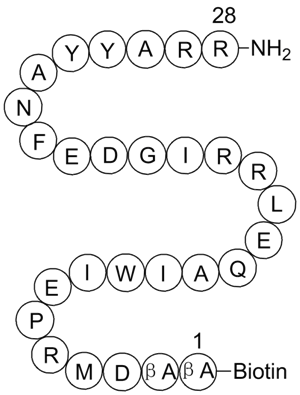
-
GC49513
Bim/BOD (IN) Polyclonal Antibody
For immunodetection of Bim-
related proteins 
-
GC52355
BimS BH3 (51-76) (human) (trifluoroacetate salt)
A Bim-derived peptide

-
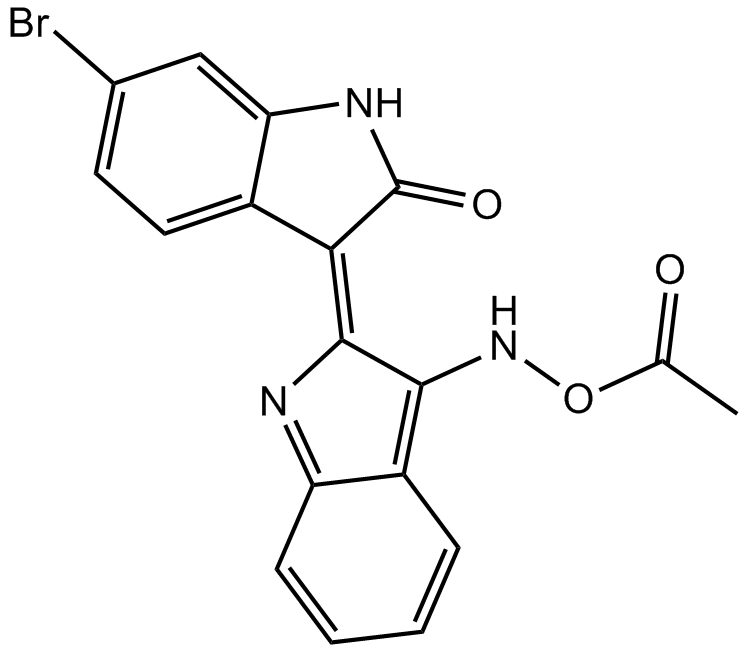
GC14233
BIO-acetoxime
Le BIO-acétoxime (BIA) est un inhibiteur puissant et sélectif de GSK-3, avec des IC50 de 10 nM pour GSK-3α/β.
-
GC67680
BIO8898
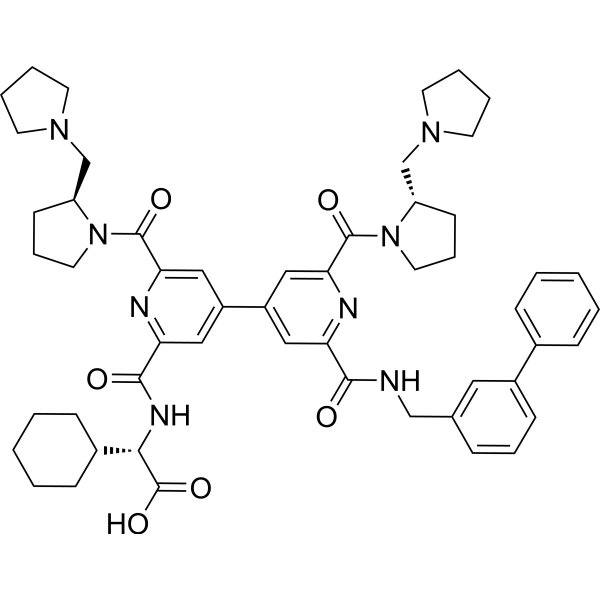
-
GC18476
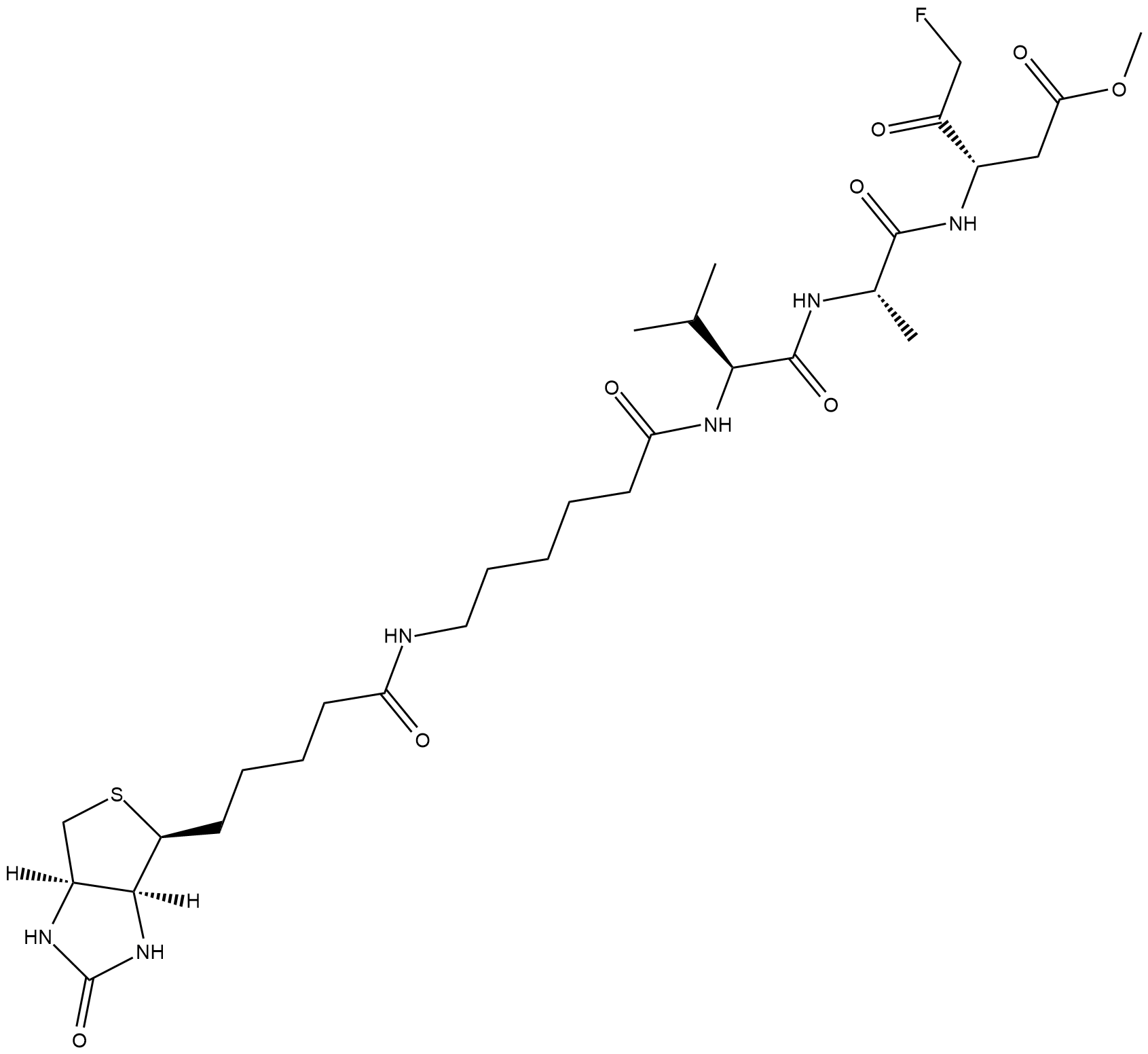
Biotin-VAD-FMK
Biotin-VAD-FMK est un inhibiteur de caspase marqué À la biotine, perméable aux cellules et irréversible, utilisé pour identifier les caspases actives dans les lysats cellulaires.
-
GC35523
Bioymifi
Bioymifi (DR5 Activator), un puissant activateur DR5 du récepteur TRAIL, se lie au domaine extracellulaire (ECD) de DR5 avec un Kd de 1,2 μM. Bioymifi peut agir comme un agent unique pour induire le regroupement et l'agrégation de DR5, conduisant À l'apoptose.