Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
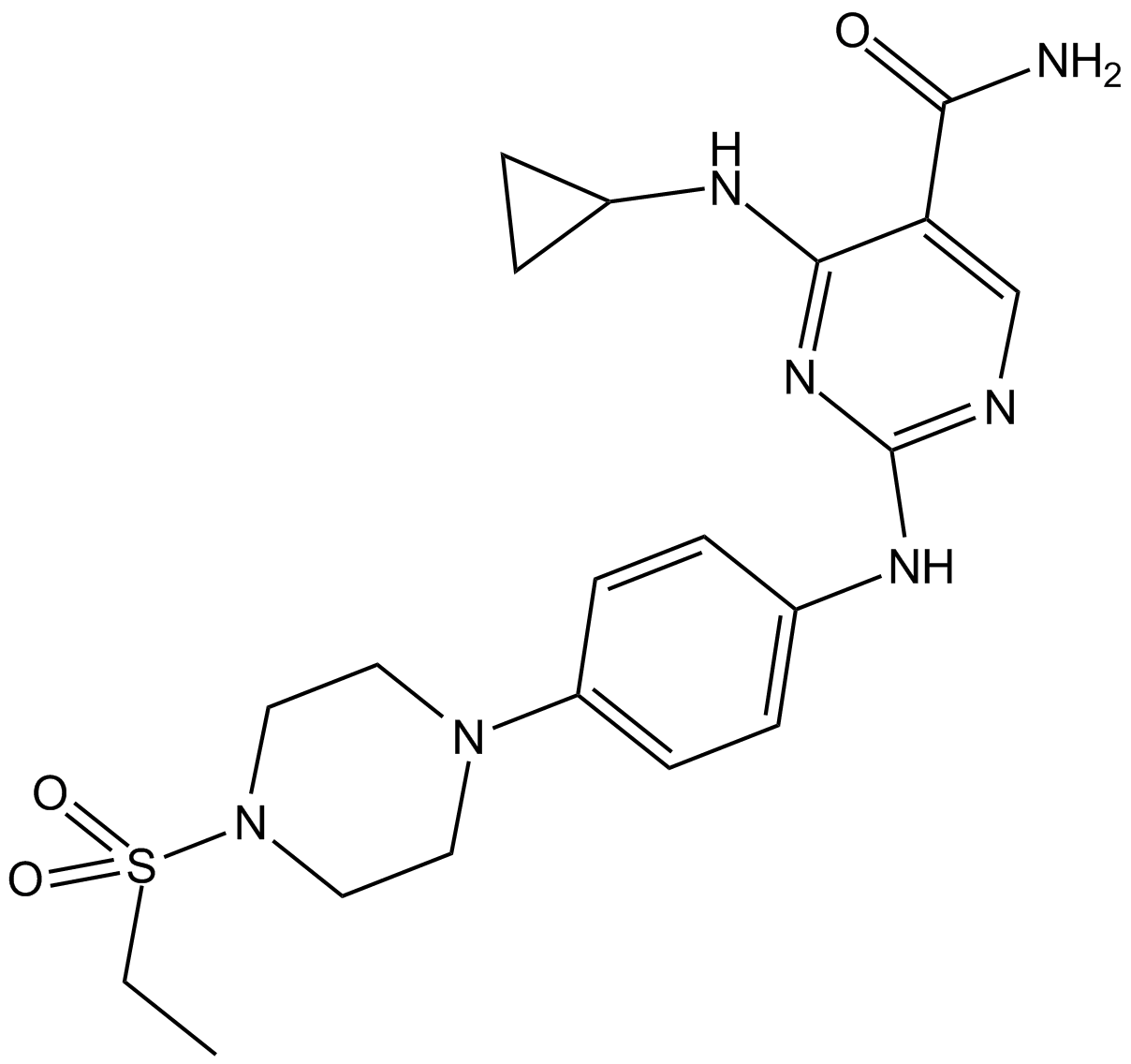
GC12171
BIX 01294
An inhibitor of G9a histone methyltransferase
-
GC50659
BIX NHE1 inhibitor
El inhibidor BIX NHE1 es un potente inhibidor de la isoforma 1 del intercambiador de sodio-hidrÓgeno (NHE1), con IC50 de 6 y 31 nM en ensayos de recuperaciÓn del pH intracelular (pHi) y de hinchazÓn de plaquetas humanas, respectivamente.
-
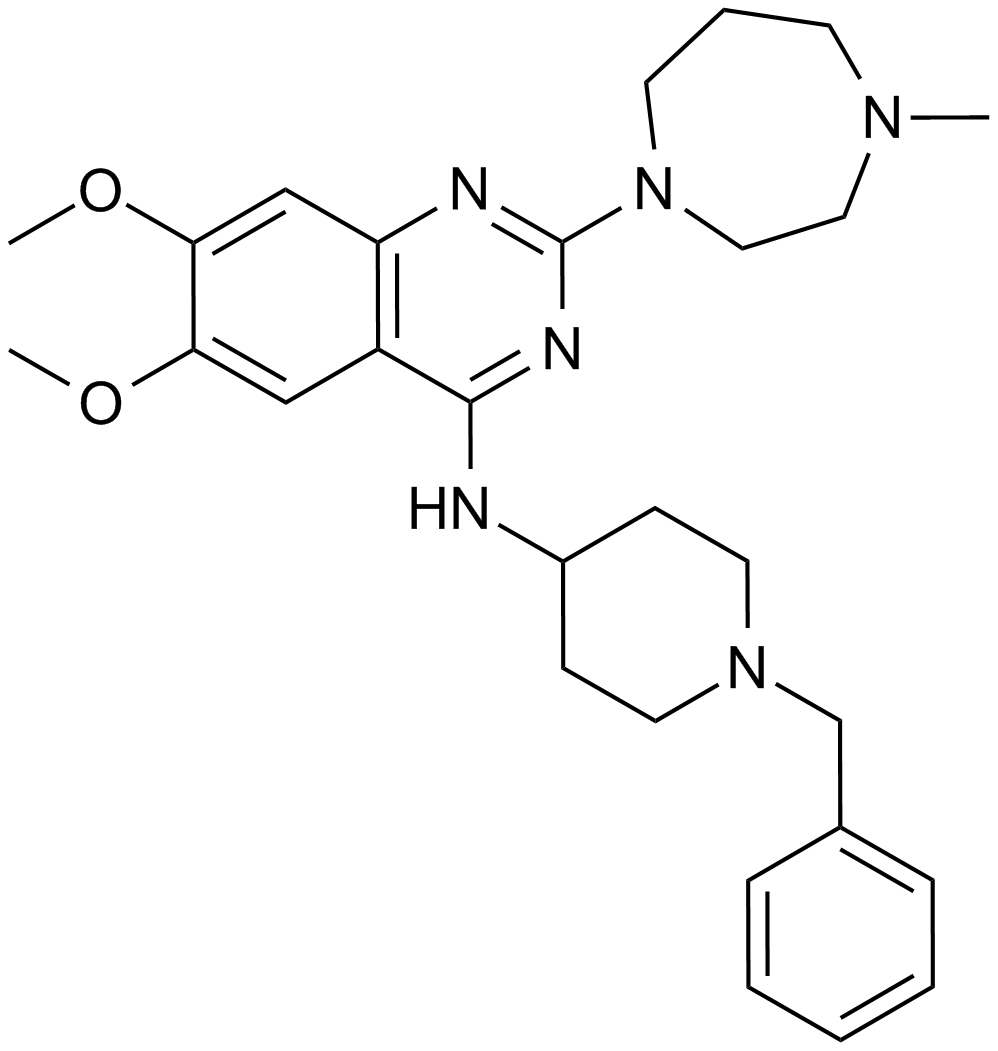
GC33301
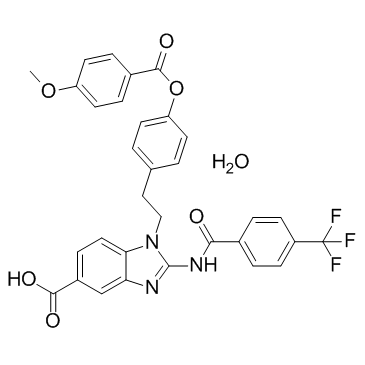
BIX-01338 hydrate (BIX01338 hydrate)
BIX-01338 hidrato (BIX01338 hidrato) es un inhibidor de la histona lisina metiltransferasa.
-
GC42944
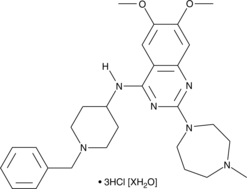
BIX01294 (hydrochloride hydrate)
The methylation of lysine residues on histones plays a central role in determining euchromatin structure and gene expression.
-
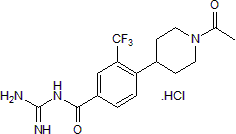
GC16114
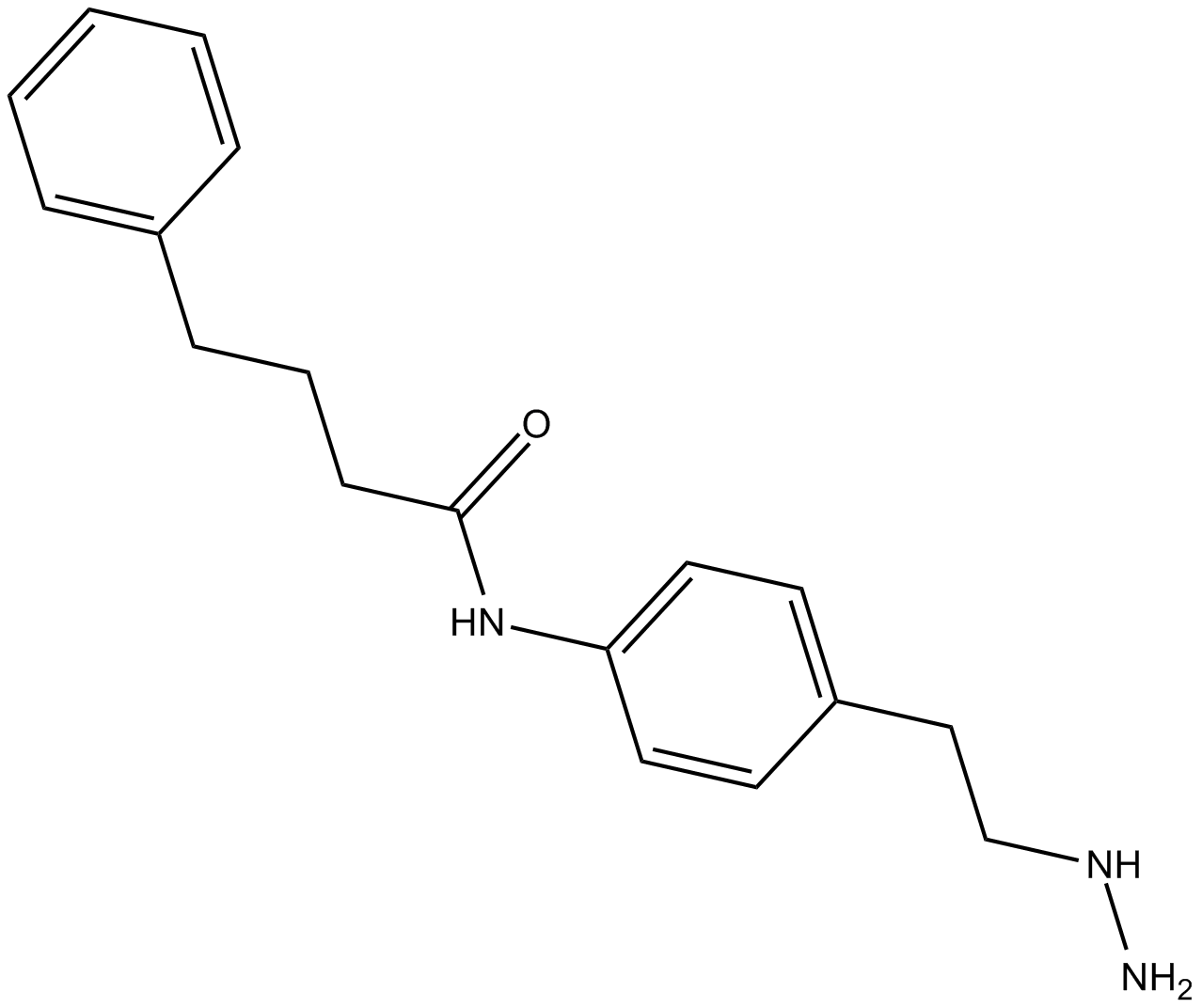
Bizine
Bizine, un anÁlogo de Phenelzine, es un inhibidor potente y selectivo de LSD1, con una b>Ki de 59 nM.
-
GC12822
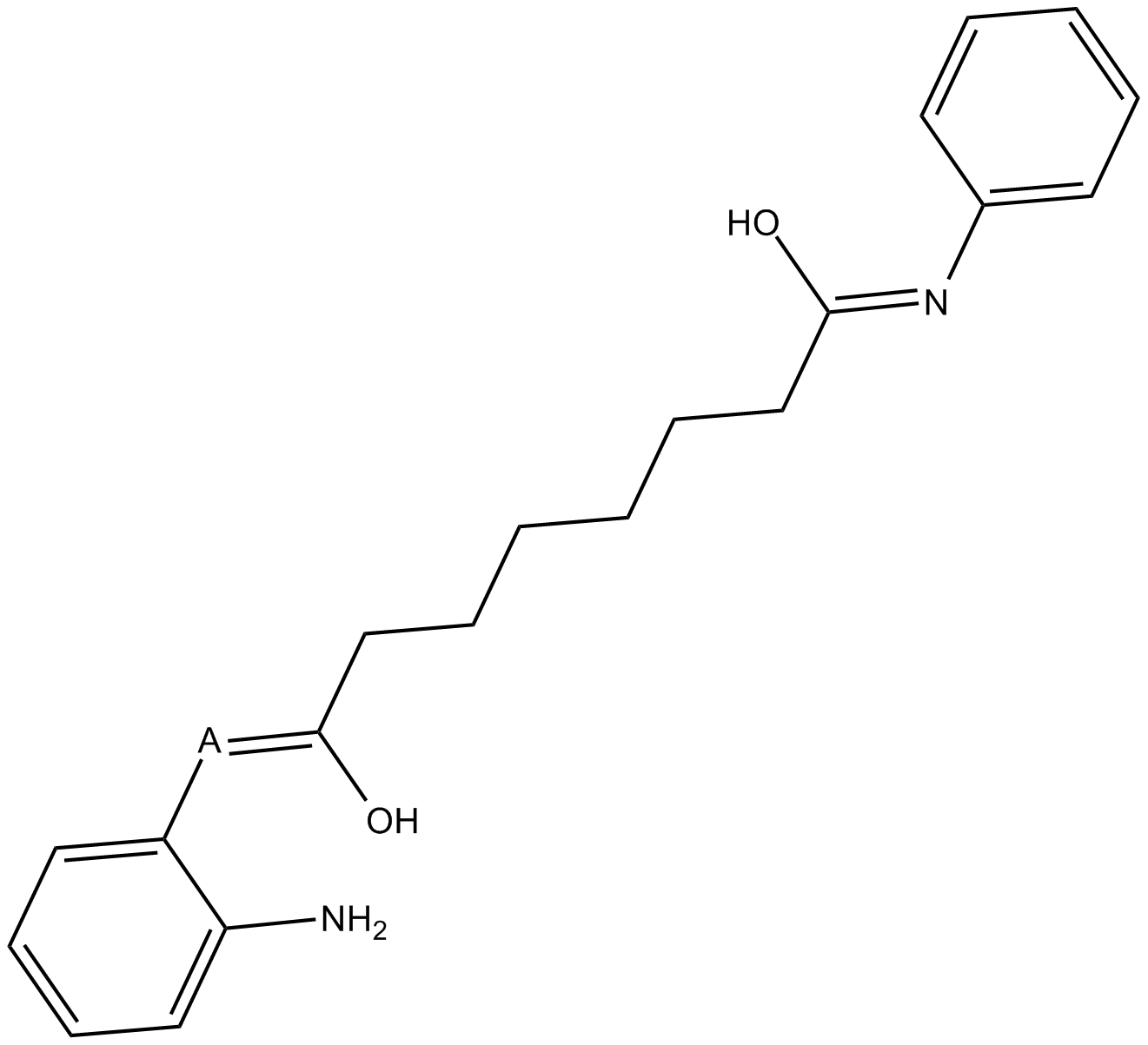
BML-210(CAY10433)
BML-210(CAY10433) es un nuevo inhibidor de HDAC y su mecanismo de acciÓn no ha sido caracterizado.
-
GC18408
BML-278
BML-278 is an activator of sirtuin 1 (SIRT1) that has an EC150 value (effective concentration able to increase the enzyme by 150%) of 1 uM.
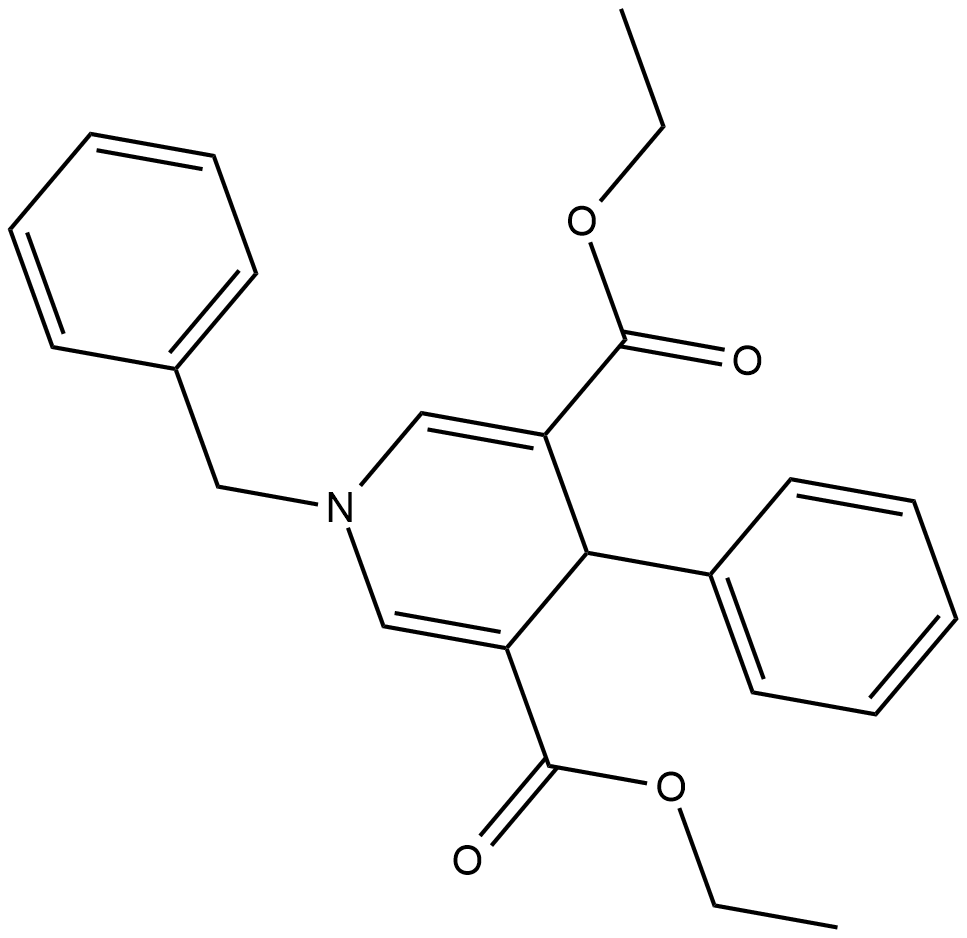
-
GC15932
BMN 673
A PARP inhibitor
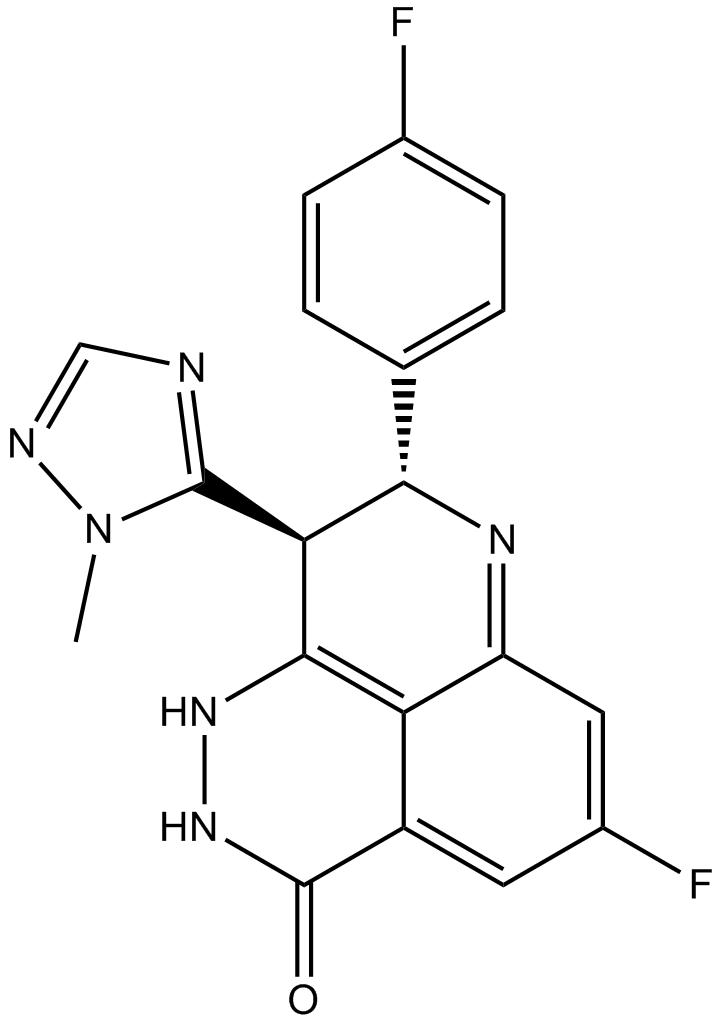
-
GC10920
BMN-673 8R,9S
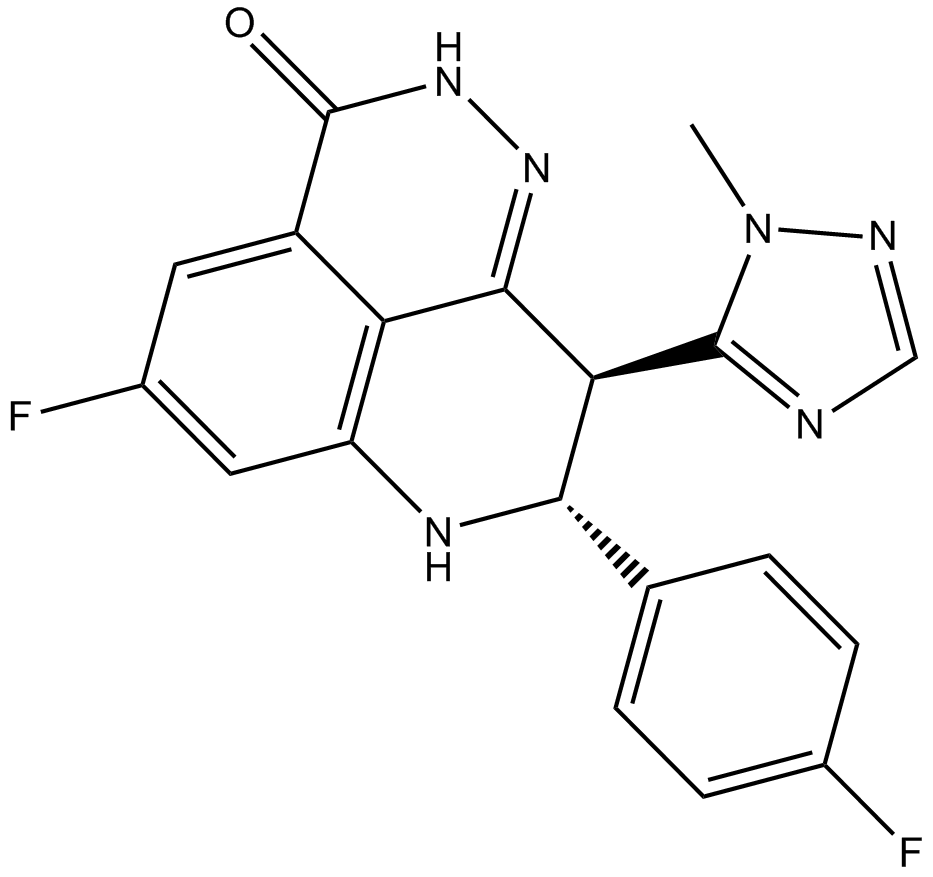
-
GC50633
BMS 986094
BMS 986094 (INX-08189) es un potente inhibidor de la replicación del virus de la hepatitis C (VHC), con una EC50 de 35 nM a las 24 h en células Huh-7.
-
GC32028
BMS-066
BMS-066 es un inhibidor de la pseudoquinasa IKKβ/Tyk2, con IC50 de 9 nM y 72 nM, respectivamente.
-
GC13926
BMS-345541(free base)
BMS-345541 (base libre) es un inhibidor selectivo de las subunidades catalÍticas de IKK (IKK-2 IC50=0,3 μM, IKK-1 IC50=4 μM). BMS-345541 (base libre) se une a un sitio alostérico de IKK.
-
GC12454
BMS-911543
A potent, selective JAK2 inhibitor

-
GC32812
BMS-986158
BMS-986158 es un potente inhibidor de BET con IC50 de 6,6 y 5nM en células de cÁncer de pulmÓn de células pequeÑas (SCLC) NCI-H211 y células de cÁncer de mama triple negativo (TNBC) MDA-MB231, respectivamente.
-
GC62491
BMS-986202
BMS-986202 es un inhibidor de Tyk2 potente, selectivo y activo por vÍa oral que se une a Tyk2 JH2 con una IC50 de 0,19 nM y una Ki de 0,02 nM.
-
GC48495
BMS-P5
BMS-P5 es un inhibidor de la peptidilarginina deiminasa 4 (PAD4) especÍfico y activo por vÍa oral. BMS-P5 bloquea la formaciÓn de NET inducida por MM y retrasa la progresiÓn de MM en un modelo de ratÓn singénico.
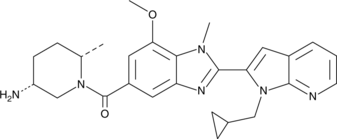
-
GC46935
Bobcat 339
Bobcat 339 es un inhibidor potente y selectivo basado en citosina de la enzima TET, con IC50 de 33 μM y 73 μM para TET1 y TET2, respectivamente. Bobcat 339 es útil en el campo de la epigenética y sirve como punto de partida para nuevas terapias dirigidas a la metilación del ADN y la transcripción de genes.
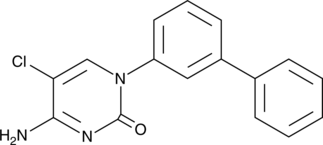
-
GC34498
Bobcat339 hydrochloride
El clorhidrato de Bobcat339 es un inhibidor potente y selectivo basado en citosina de la enzima TET, con IC50 de 33 μM y 73 μM para TET1 y TET2, respectivamente. El clorhidrato de Bobcat339 es Útil en el campo de la epigenética y sirve como punto de partida para nuevas terapias dirigidas a la metilaciÓn del ADN y la transcripciÓn de genes.
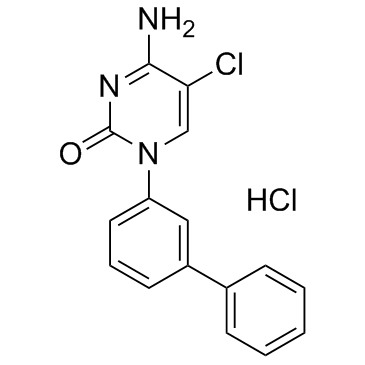
-
GC11641
Boc-Lys(Ac)-AMC
GPCR G2A/GPR132 agonist
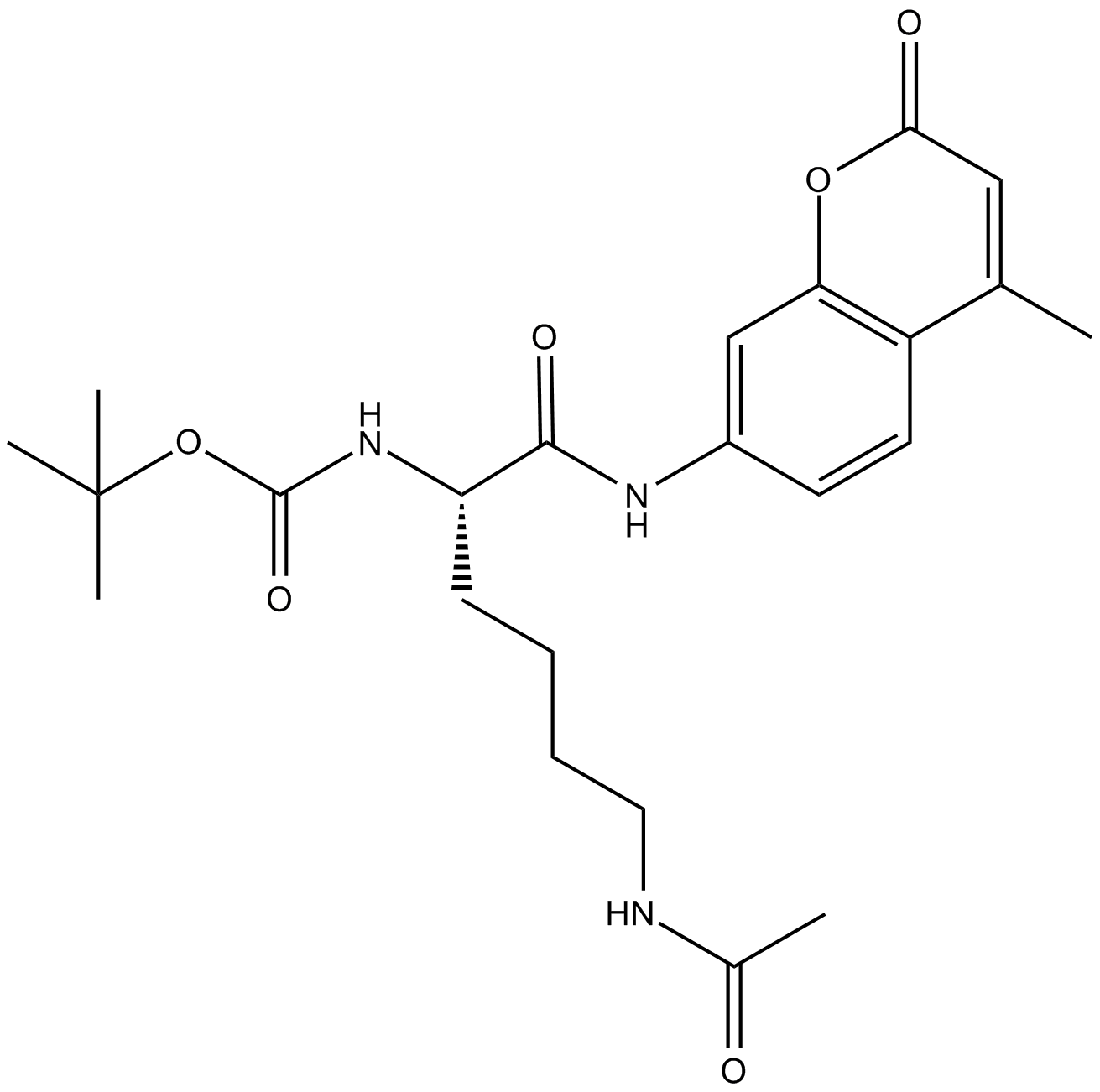
-
GC64865
Bomedemstat
Bomedemstat (IMG-7289) es un inhibidor de la desmetilasa 1 (LSD1) especÍfico de lisina activo e irreversible por vÍa oral. Bomedemstat puede aumentar la metilaciÓn de H3K4 y H3K9 y luego alterar la expresiÓn génica. Bomedemstat muestra actividades anticancerÍgenas, inhibe la proliferaciÓn de células cancerosas e induce la apoptosis.
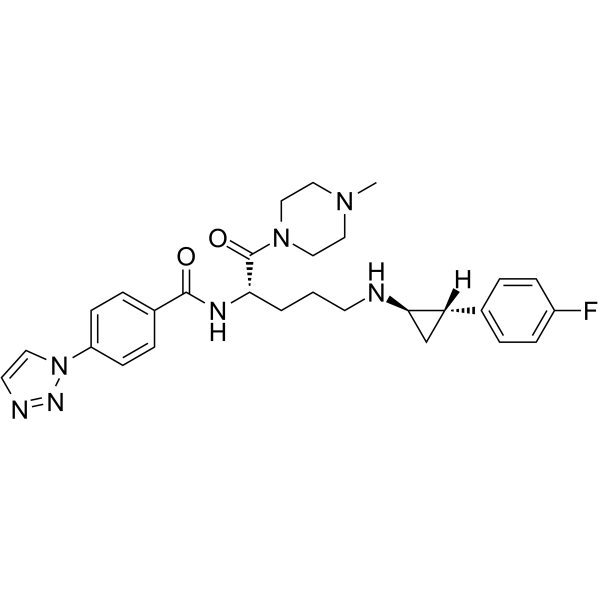
-
GC67921
Bomedemstat ditosylate

-
GC65879
Bomedemstat hydrochloride
El clorhidrato de bomedemstat (IMG-7289) es un inhibidor irreversible de la desmetilasa 1 (LSD1) especÍfico de la lisina activo por vÍa oral. El clorhidrato de bomedemstat puede aumentar la metilaciÓn de H3K4 y H3K9 y luego alterar la expresiÓn génica. El clorhidrato de bomedemstat muestra actividades anticancerÍgenas, inhibe la proliferaciÓn de células cancerosas e induce la apoptosis.
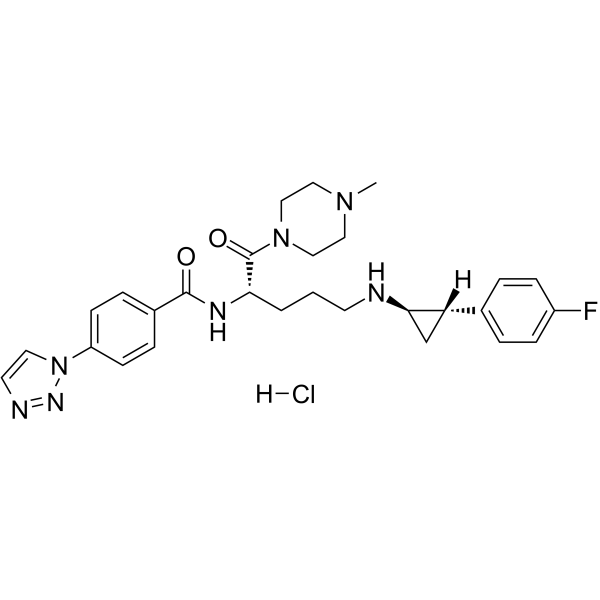
-
GC48620
BPKDi
BPKDi es un potente inhibidor de bipiridil PKD con IC50 de 1 nM, 9 nM y 1 nM para PKD1, PKD2 y PKD3, respectivamente.
-
GC64538
BPTF-IN-BZ1
BPTF-IN-BZ1, un inhibidor de BPTF, posee una alta potencia (Kd = 6,3 nM).
-
GC15974
bpV(HOpic) (potassium salt, technical grade)
bpV(HOpic) (sal de potasio, grado técnico) es un inhibidor potente y selectivo de PTEN con una IC50 de 14 nM.
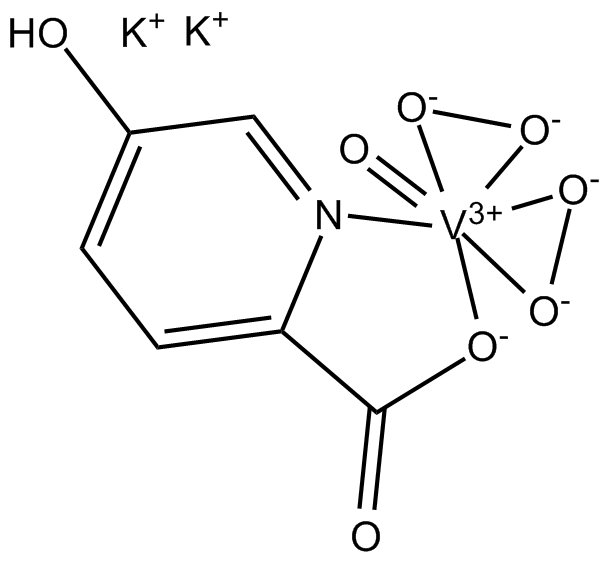
-
GC35547
BR102375
BR102375 es un agonista completo del receptor γ (PPAR γ) activado por el proliferador de peroxisomas sin TZD para el tratamiento de la diabetes tipo 2, revela un valor EC50 de 0,28 μM y una relaciÓn Amax del 98 %.
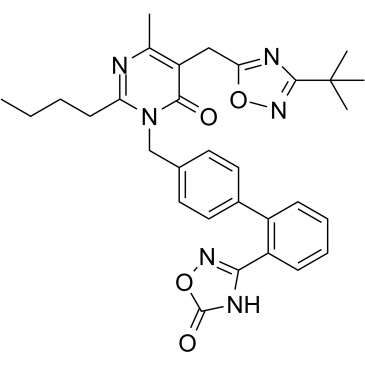
-
GC33223
BRCA1-IN-1
BRCA1-IN-1 es un nuevo inhibidor de BRCA1 similar a una molécula pequeÑa con IC50 y Ki de 0,53 μM y 0,71 μM, respectivamente.
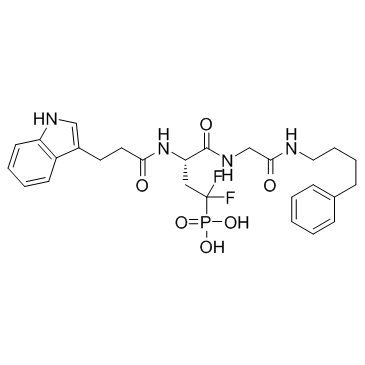
-
GC35550
BRCA1-IN-2
BRCA1-IN-2 (compuesto 15) es un inhibidor de la interacciÓn proteÍna-proteÍna (PPI) permeable a las células para BRCA1 con un IC50 de 0,31 μM y una Kd de 0,3 μM, que muestra actividades antitumorales a través de la interrupciÓn de BRCA1 (BRCT)2 /interacciones proteÍna.
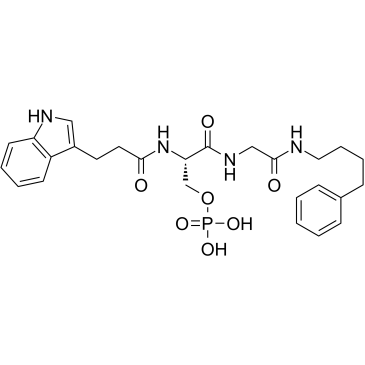
-
GC33331
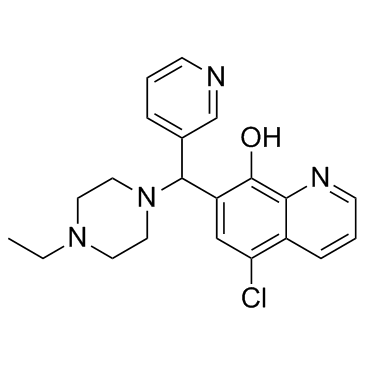
BRD 4354
BRD 4354 es un inhibidor moderadamente potente de HDAC5 y HDAC9, con IC50 de 0,85 y 1,88 μM, respectivamente.
-
GC35551
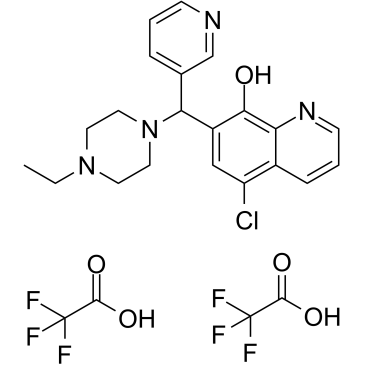
BRD 4354 ditrifluoroacetate
BRD 4354 (ditrifluoroacetato) es un inhibidor moderadamente potente de HDAC5 y HDAC9, con IC50 de 0,85 y 1,88 μM, respectivamente.
-
GC50322
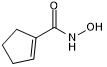
BRD 9757
Potent and selective HDAC6 inhibitor
-
GC38742
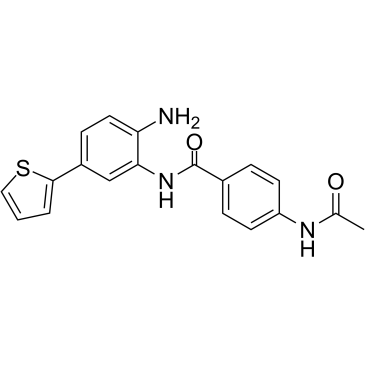
BRD-6929
BRD-6929 es un inhibidor potente y selectivo que penetra en el cerebro de la histona desacetilasa de clase I HDAC1 y el inhibidor de HDAC2 con IC50 de 1 nM y 8 nM, respectivamente.
-
GC35552
BRD-IN-3
BRD-IN-3 ((R,R)-36n) es un inhibidor de bromodominio PCAF (BRD) muy potente, con una IC50 de 7 nM. BRD-IN-3 también exhibe actividad contra GCN5 y FALZ.
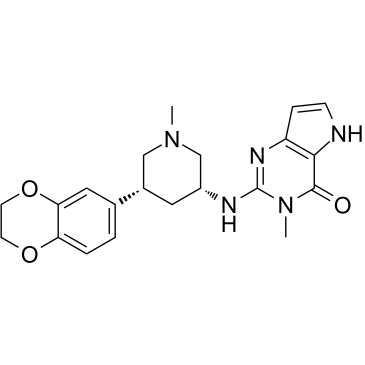
-
GC63731
BRD0639
BRD0639 es el primer inhibidor de su clase de la interacciÓn PRMT5-adaptador de sustrato. BRD0639 es un agente competitivo con motivo de uniÓn a PRMT5 (PBM) que puede respaldar los estudios de las actividades de PRMT5 dependientes de PBM.
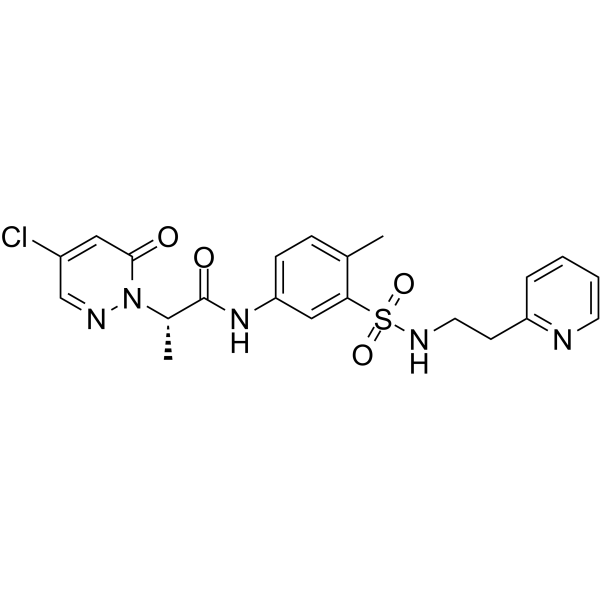
-
GC33017
BRD4 degrader AT1
El degradador de BRD4 AT1 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD4 como un degradador de Brd4 altamente selectivo, con una Kd de 44 nM para Brd4BD2 en las células.
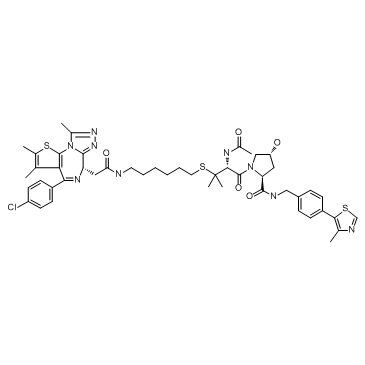
-
GC64960
BRD4 Inhibitor-10
BRD4 Inhibitor-10 es un potente inhibidor de BRD4-BD1 extraÍdo de la patente WO2015022332A1, Compound II-25, tiene un IC50 de 8 nM.
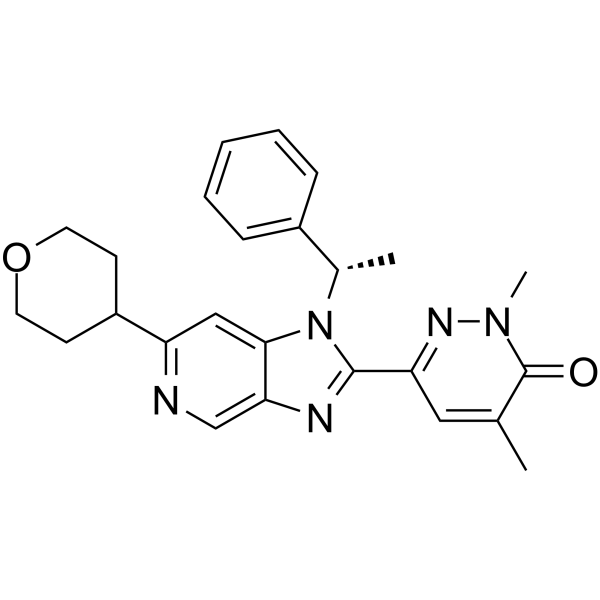
-
GC65569
BRD4 Inhibitor-24
BRD4 Inhibitor-24 (compuesto 3U) es un potente inhibidor de BRD4, BRD4 Inhibitor-24 muestra actividad antitumoral contra las células MCF7 y K652, con valores IC50 de 33,7 y 45,9 μM, respectivamente (extraÍdo de la patente CN107721975A).
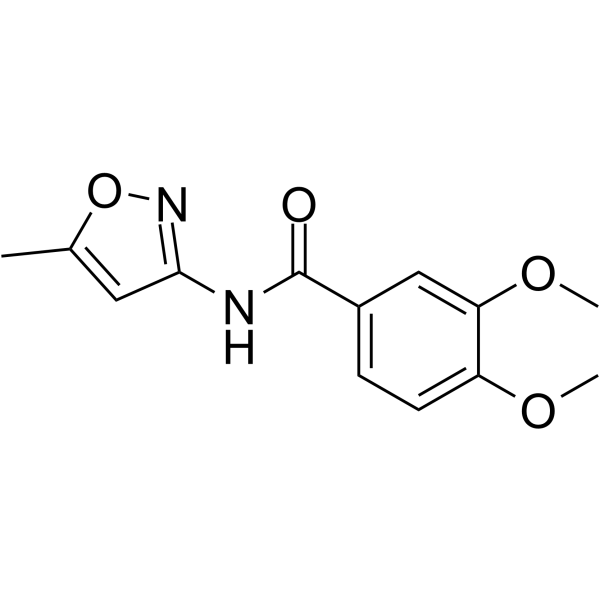
-
GC66441
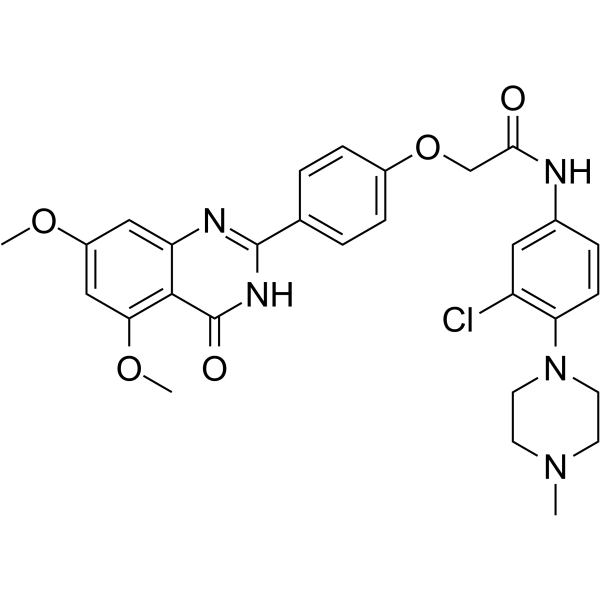
BRD4/CK2-IN-1
BRD4/CK2-IN-1 es el primer inhibidor de doble diana altamente eficaz y oral activo de BRD4/CK2 (proteÍna 4 que contiene bromodominio/caseÍna quinasa 2), con IC50 de 180 nM y 230 nM para BRD4 y CK2, respectivamente. BRD4/CK2-IN-1 tiene una fuerte actividad anticancerÍgena sin toxicidades obvias. BRD4/CK2-IN-1 induce la apoptosis y la muerte celular asociada a la autofagia en el cÁncer de mama triple negativo (TNBC)
-
GC10259
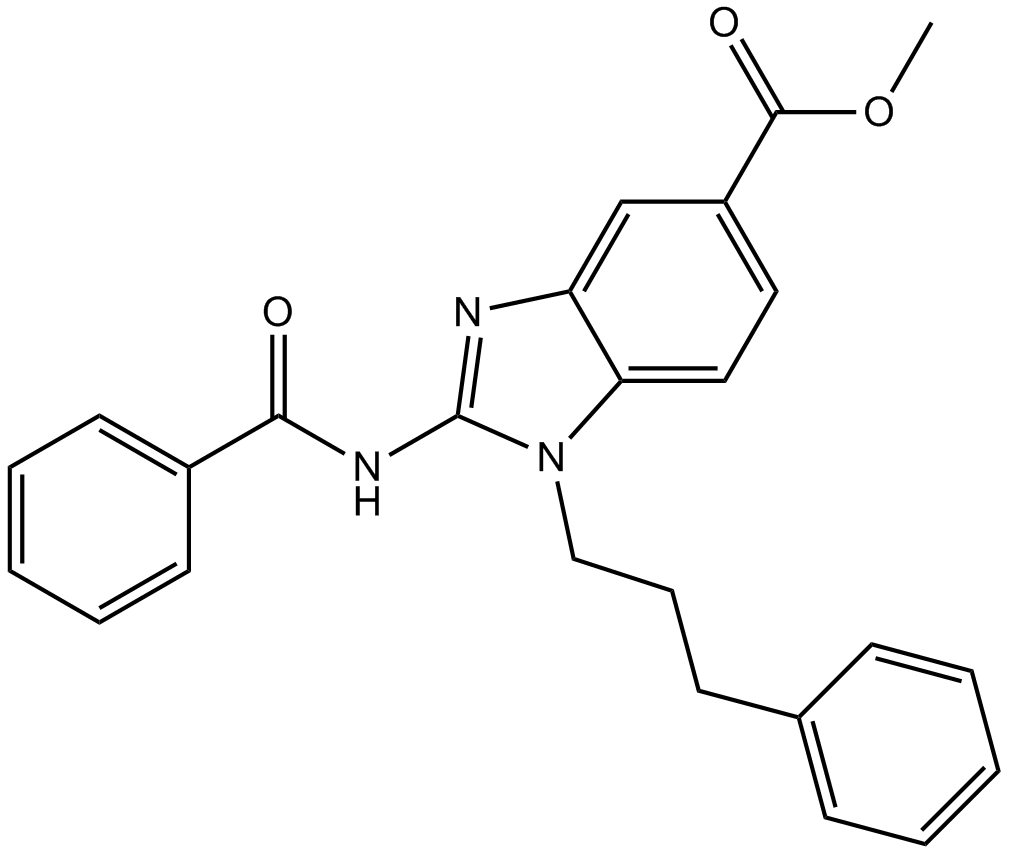
BRD4770
BRD4770 es un inhibidor de la histona metiltransferasa G9a. BRD4770 reduce la dimetilaciÓn y la trimetilaciÓn de la lisina 9 en la histona H3 (H3K9) con una CE50 de 5 μM y tiene un efecto menor o menor hacia H3K27me3, H3K36me3, H3K4me3 y H3K79me3. BRD4770 puede activar la vÍa mutada de ataxia telangiectasia (ATM) e inducir la senescencia celular.
-
GC40923
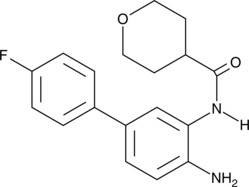
BRD4884
BRD4884 es un potente inhibidor de HDAC con valores IC50 de 29 nM, 62 nM y 1,09 μM para HDAC1, 2 y 3, respectivamente.
-
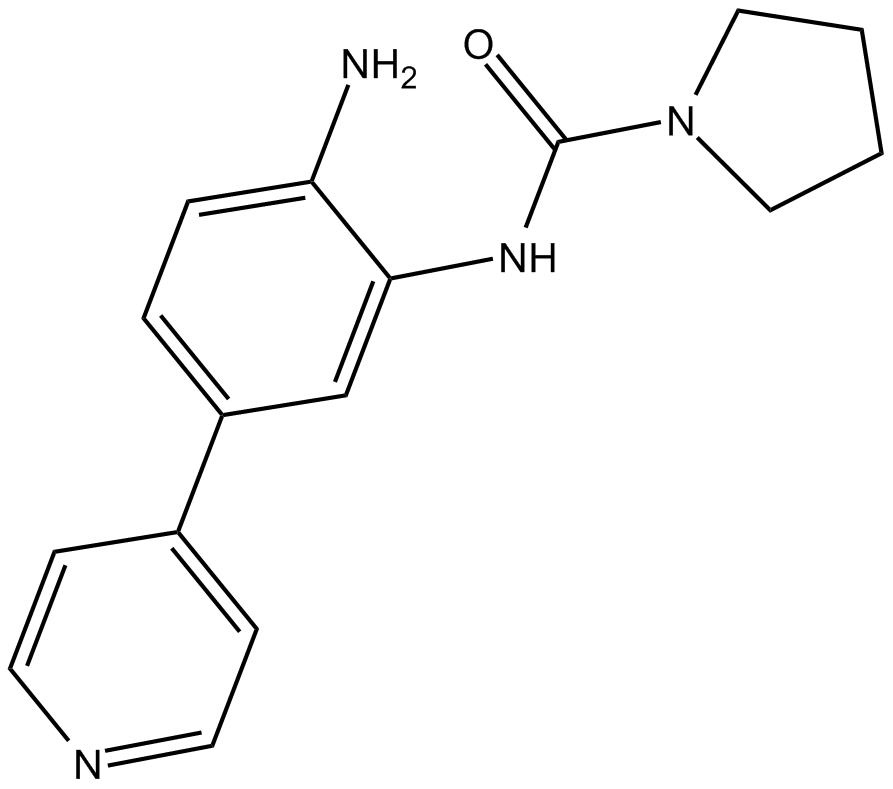
GC13088
BRD6688
BRD6688 es un inhibidor selectivo de HDAC2.
-
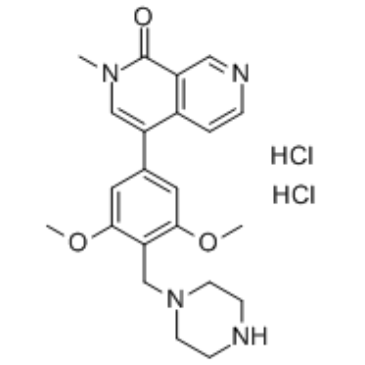
GC34505
BRD7-IN-1
BRD7-IN-1, un derivado modificado de BI7273 (inhibidor de BRD7/9), se une a un ligando VHL a través de un enlazador para formar un PROTAC VZ185 (VZ185 contra BRD7/9 con DC50 de 4,5 y 1,8 nM, respectivamente).
-
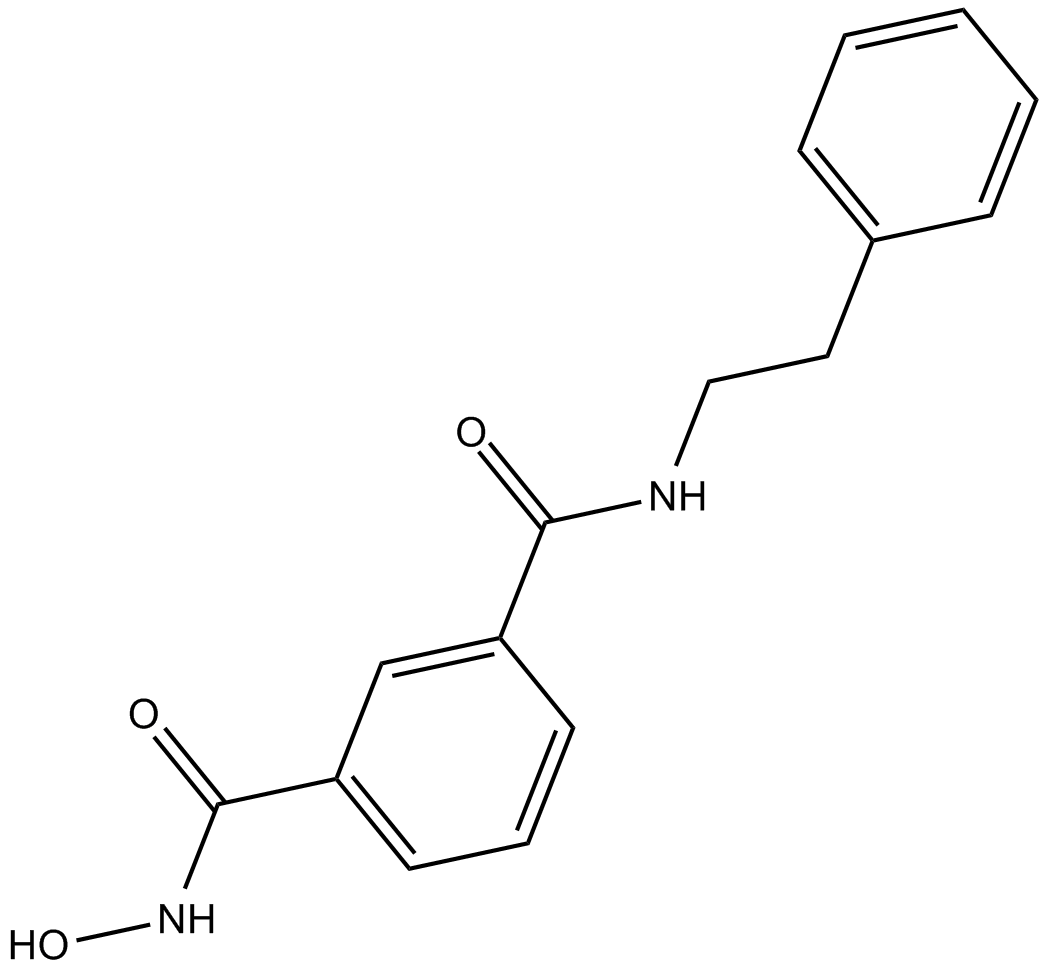
GC12484
BRD73954
BRD73954 es un potente inhibidor de HDAC e inhibe selectivamente HDAC6 y HDAC8 con valores IC50 de 0,0036, 0,12, 9, 12, 23 μM para HDAC6, HDAC8, HDAC2, HDAC1 y HDAC3, respectivamente. BRD73954 disminuye los niveles de HDAC6, asociado con la regulaciÓn positiva de Ac-Tubulin.
-
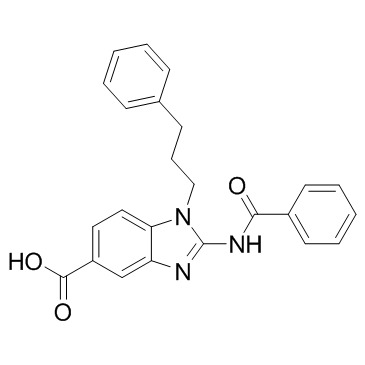
GC32988
BRD9539
BRD9539 es un inhibidor de la histona metiltransferasa G9a con una IC50 de 6,3 μM. BRD9539 también inhibe la actividad de PRC2 y es inactivo frente a SUV39H1, NSD2 y DNMT1.
-
GC25168
Brepocitinib (PF-06700841)
Brepocitinib (PF-06700841, PF-841) is a potent inhibitor of Tyk2 and Jak1 with IC50s of 23 nM, 17 nM, 77 nM for Tyk2, Jak1 and Jak2 respectively. It has appropriate in-family selectivity against JAK2 and JAK3.
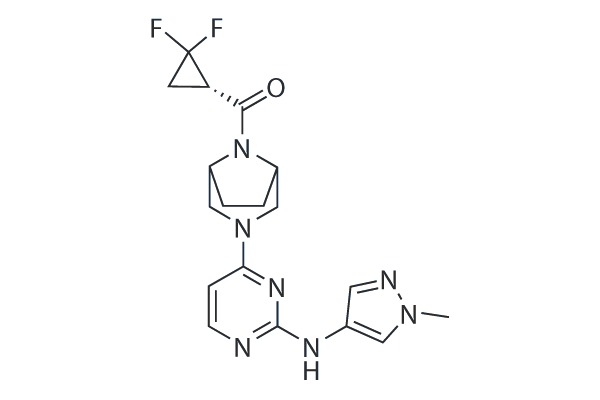
-
GC35554
Brevilin A
Brevilin A es un inhibidor de STAT3/JAK activo por vÍa oral (STAT3 IC50=10,6 μM). Brevilin A muestra actividad antitumoral, actividad antiproliferativa para las células cancerosas y puede inducir la apoptosis y la autofagia.

-
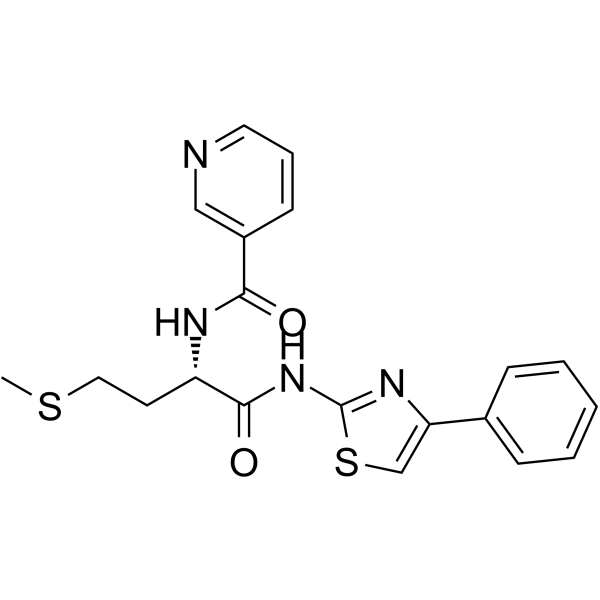
GC64541
BRM/BRG1 ATP Inhibitor-2
BRM/BRG1 ATP Inhibitor-2 es un inhibidor de ATPasa BRG1/BRM para el tratamiento de trastornos relacionados con BAF.
-
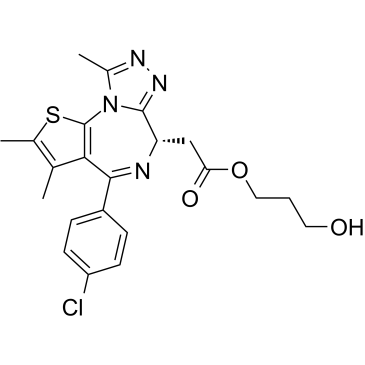
GC35557
Bromodomain IN-1
Bromodomain IN-1 es un inhibidor de bromodominio extraÍdo de la patente WO2016069578A1, compuesto 4.
-
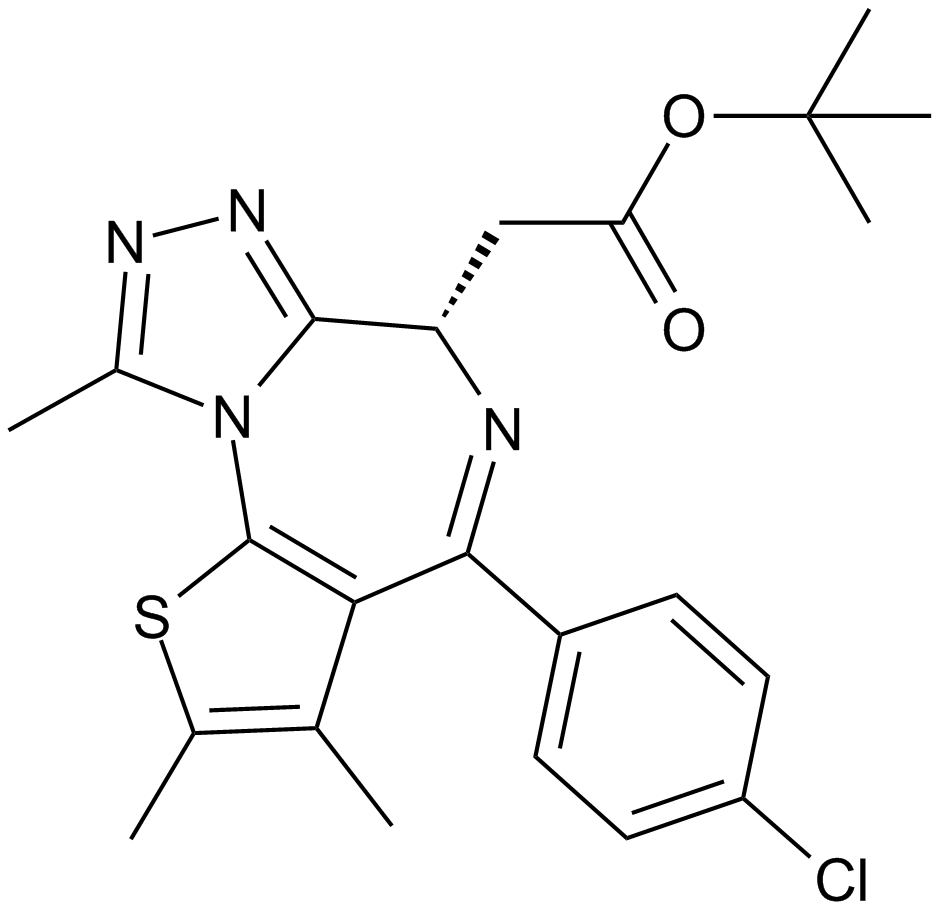
GC16531
Bromodomain Inhibitor, (+)-JQ1
A selective inhibitor of BET bromodomains
-
GC35558
Bromodomain inhibitor-8
El inhibidor de bromodominio-8 (intermedio 21) es un inhibidor de bromodominio BET para el tratamiento de enfermedades autoinmunes e inflamatorias.
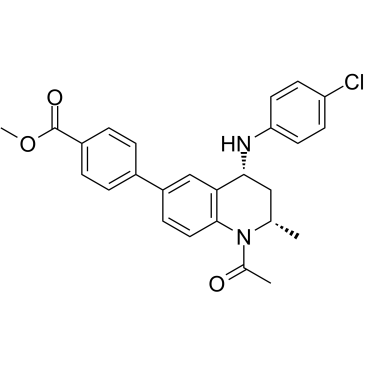
-
GC10402
Bromosporine
A non-specific bromodomain inhibitor
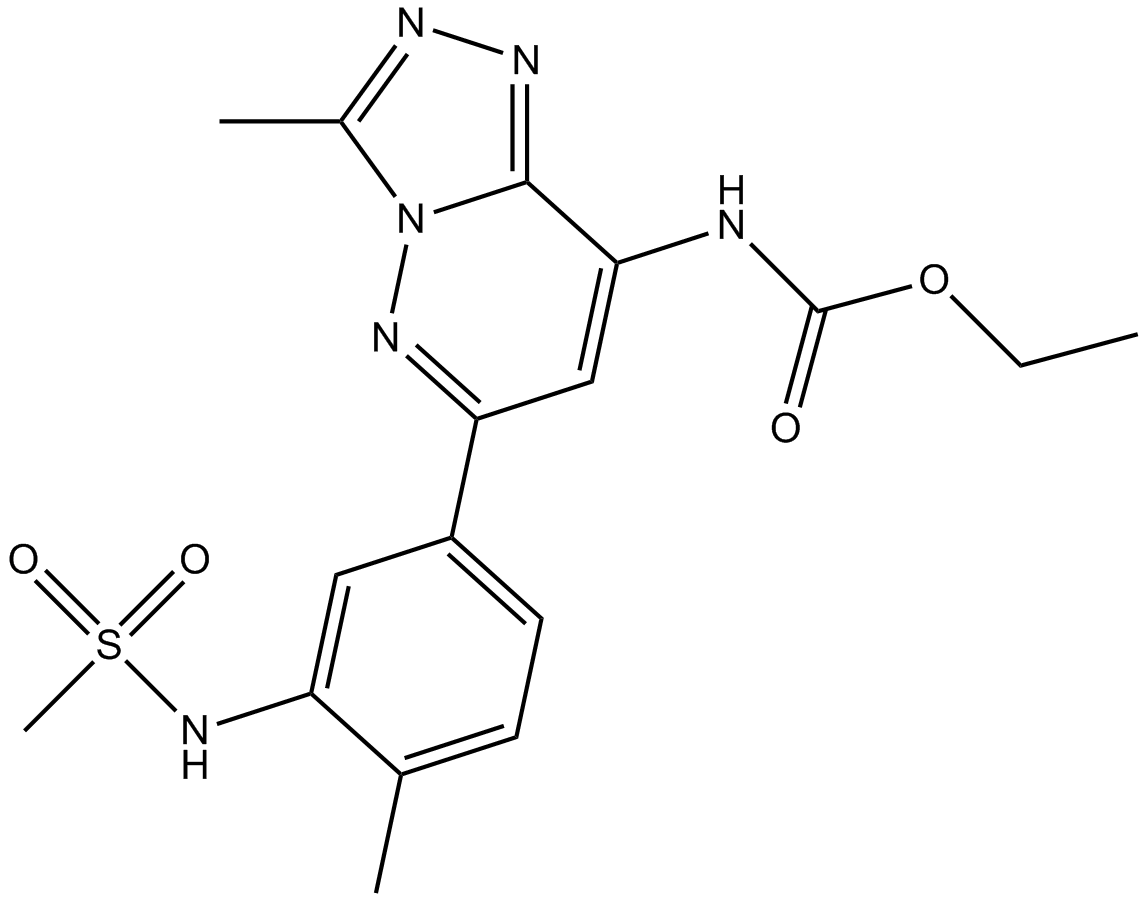
-
GC15410
Bufexamac
Bufexamac es un inhibidor de las histonas desacetilasas (HDAC6 y HDAC10) de clase IIB que se utiliza como agente antiinflamatorio.
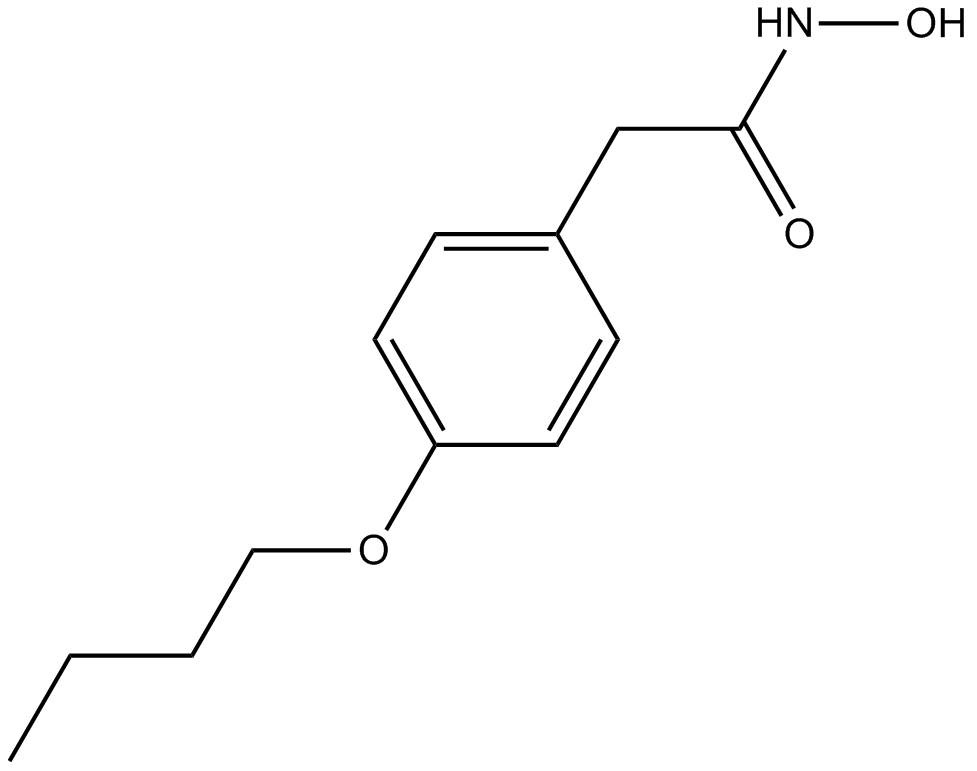
-
GC64761
Butyric acid-13C1
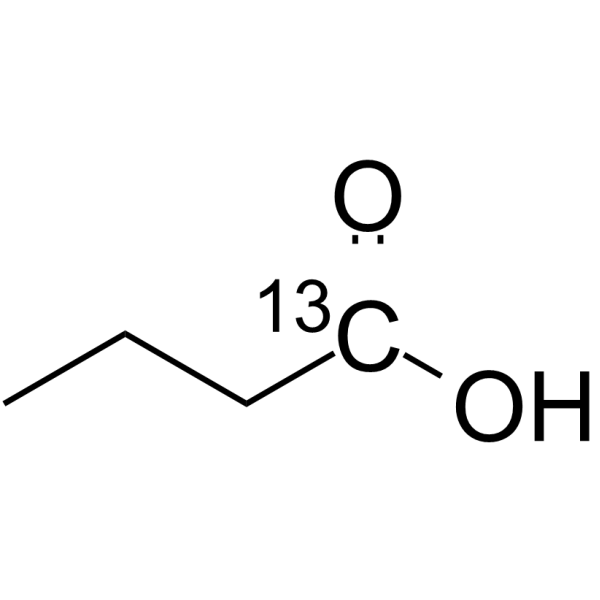
-
GC46104
Butyric Acid-d7
An internal standard for the quantification of sodium butyrate
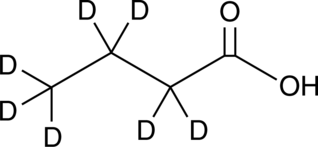
-
GC10226
Butyrolactone 3
La butirolactona 3 (MB-3) es un inhibidor especÍfico de molécula pequeÑa de la histona acetiltransferasa Gcn5 (IC50 = 100 μM), que tiene una alta afinidad por la enzima Gcn5 comparable a la de su sustrato natural, la histona H3.
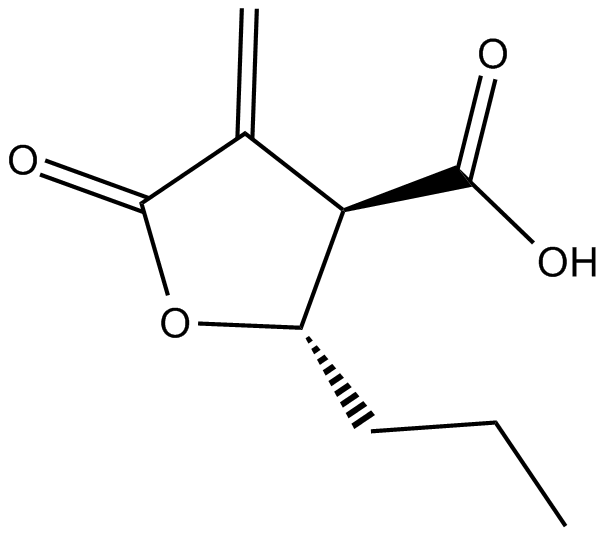
-
GC10690
BYK 204165
A selective inhibitor of PARP1
-
GC14434
BYK 49187
Potent PARP-1/PARP-2 inhibitor
-
GC16130
C 21
Protein arginine methyltransferase 1 (PRMT1) inhibitor

-
GC17011
C2 Ceramide
A cell-permeable analog of naturally occurring ceramides
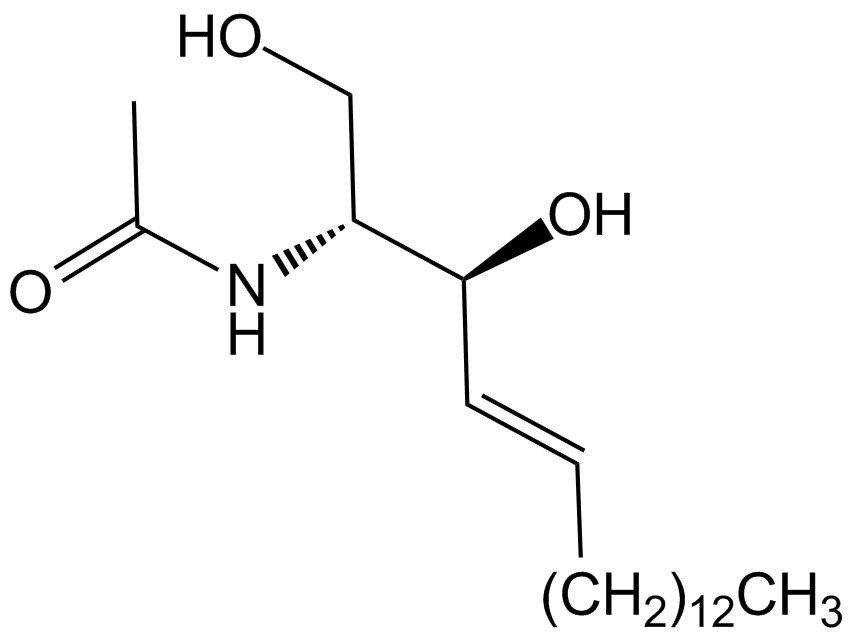
-
GC12733
C646
C646 es un inhibidor selectivo y competitivo de histona acetiltransferasa p300 con Ki de 400 nM, y es menos potente para otras acetiltransferasas.
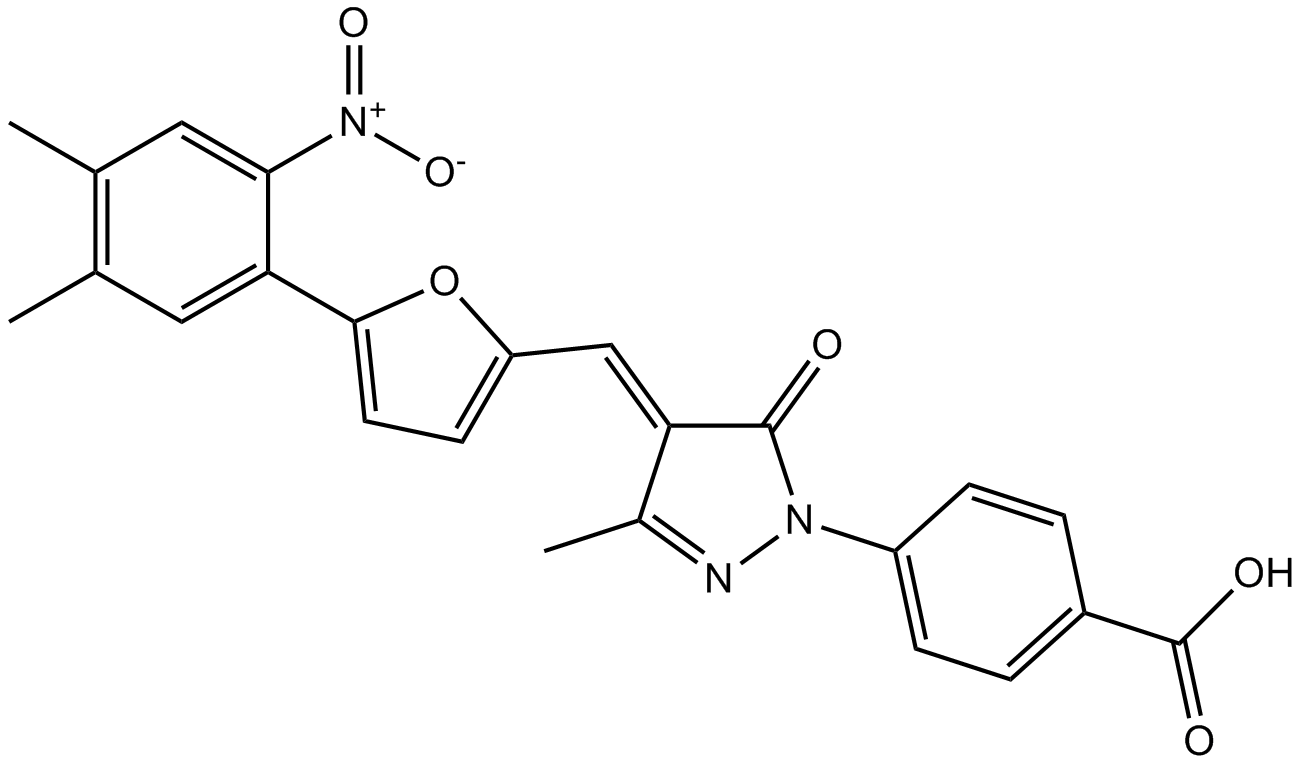
-
GC12005
C7280948
C7280948 es un inhibidor selectivo y potente de la proteína metiltransferasa 1 (PRMT1) con un valor IC50 de 12,75 μM.
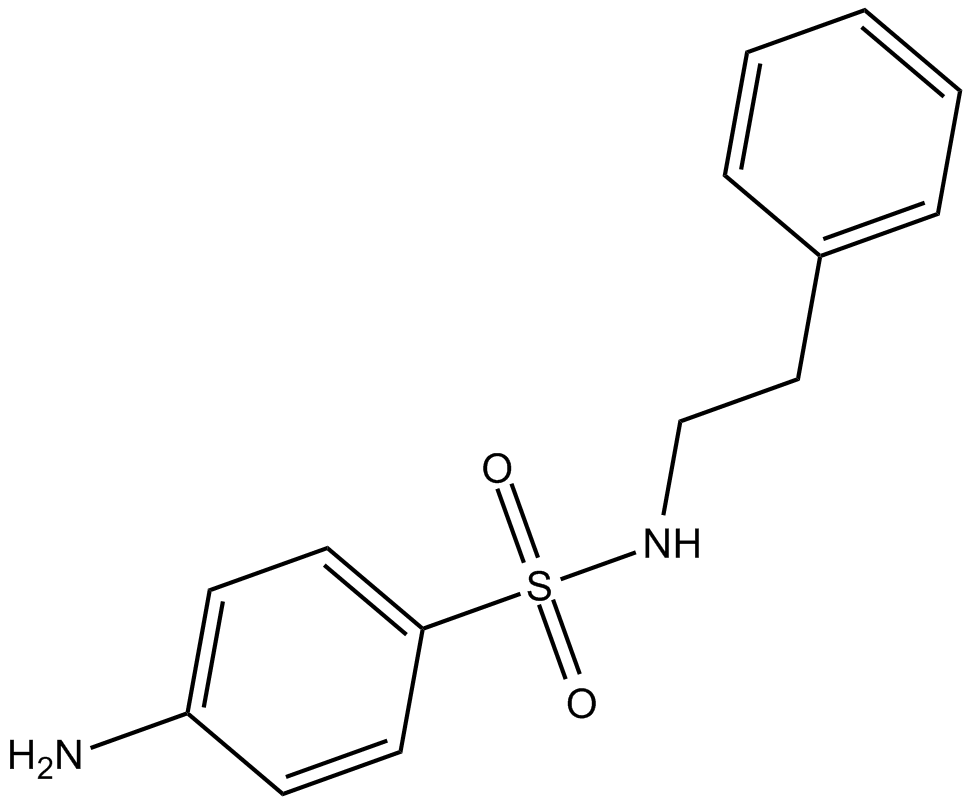
-
GC62886
Cannabisin F
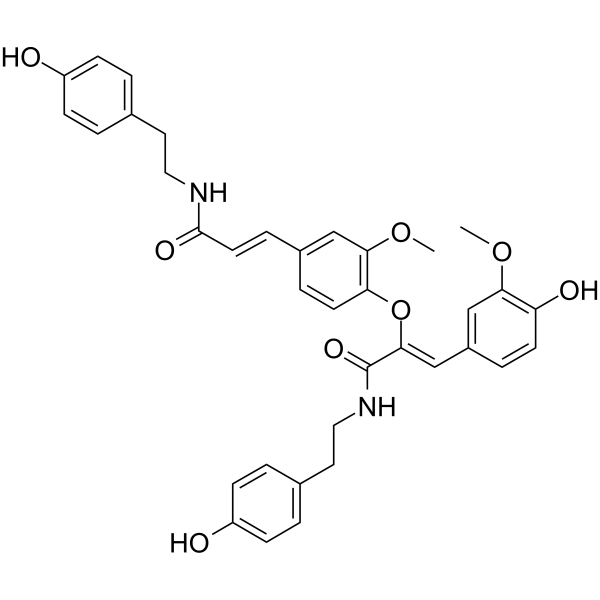
-
GC12082
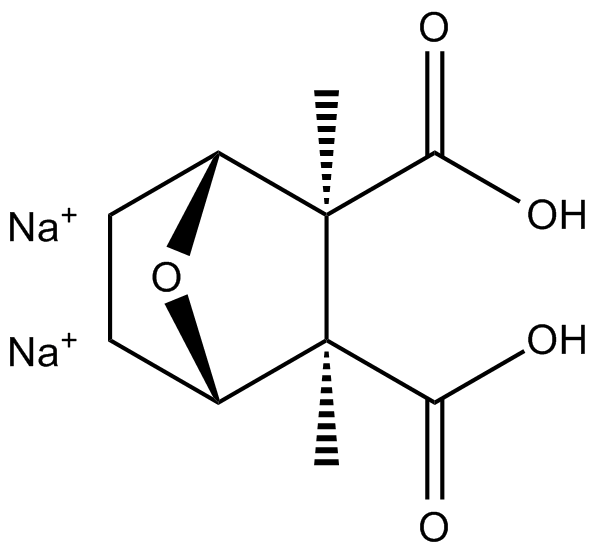
Cantharidic Acid (sodium salt)
PP1 and PP2A inhibitor
-
GC34108
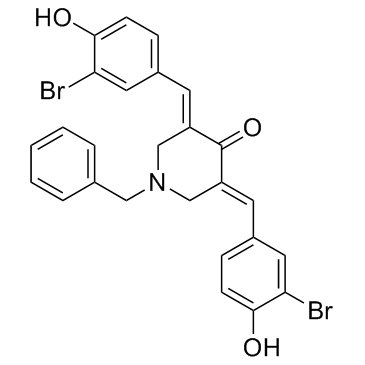
CARM1-IN-1
CARM1-IN-1 es un potente y especÍfico inhibidor de CARM1 (arginina metiltransferasa 1 asociada a coactivador) con IC50 de 8,6 uM; muestra una actividad muy baja frente a PRMT1 y SET7 (IC50 > 600 uM).
-
GC34424
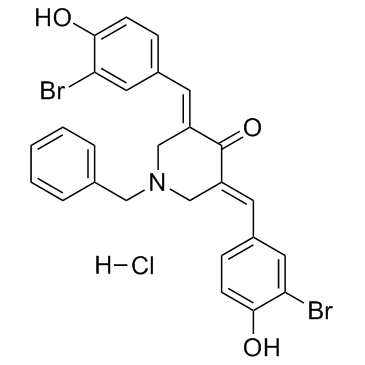
CARM1-IN-1 hydrochloride
El clorhidrato de CARM1-IN-1 es un inhibidor potente y especÍfico de CARM1 (arginina metiltransferasa 1 asociada a coactivador) con IC50 de 8,6 uM; muestra una actividad muy baja frente a PRMT1 y SET7 (IC50 > 600 uM).
-
GC43147
CAY10398
CAY10398 is an inhibitor of histone deacetylase (HDAC1) with an IC50 value of 10 μM.
-
GC41601
CAY10591
CAY10591 es un potente activador de Sirt1 y suprime TNF-α de manera dependiente de la dosis.
-
GC18228
CAY10602
CAY10602 es un activador SIRT1.
-
GC12971
CAY10603
CAY10603 (BML-281) es un inhibidor potente y selectivo de HDAC6, con una IC50 de 2 pM; CAY10603 (BML-281) también inhibe HDAC1, HDAC2, HDAC3, HDAC8, HDAC10, con IC50 de 271, 252, 0,42, 6851, 90,7 nM.
-
GC40767
CAY10669
CAY10669 is an inhibitor of the histone acetyltransferase PCAF (p300/CREB-binding protein-associated factor; IC50 = 662 μM), displaying a 2-fold improvement in inhibitory potency over anacardic acid.
-
GC18949
CAY10677
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.
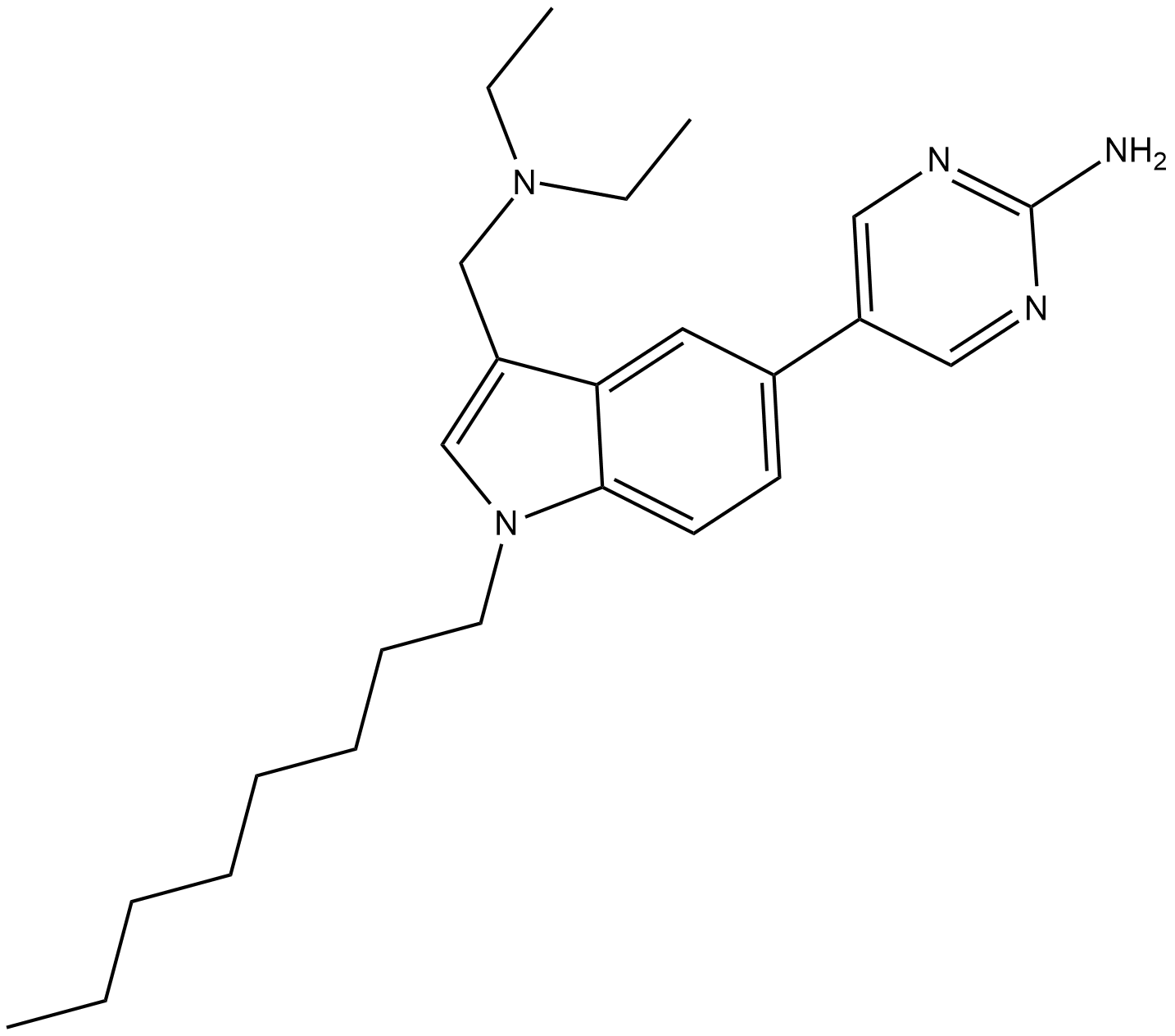
-
GC43192
CAY10685
CAY10685 is a cell-active analog of the lysine acetyltransferase inhibitor CPTH2 that contains an alkyne moiety for use in click chemistry reactions.
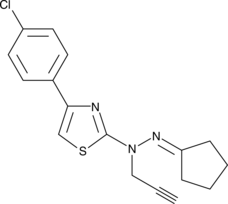
-
GC43200
CAY10721
CAY10721 is an inhibitor of sirtuin 3 (SIRT3), a class III HDAC (39% SIRT3 inhibition at 200 μM).
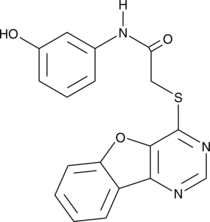
-
GC43201
CAY10722
CAY10722 is an inhibitor of sirtuin 3 (SIRT3), a class III HDAC (71% inhibition at 200 μM).
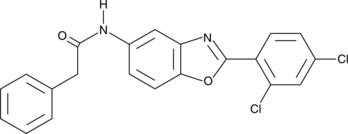
-
GC43202
CAY10723
CAY10723 es un potente inhibidor de la proteína arginina deiminasa 2 (PAD2) y tiene una excelente selectividad hacia PAD2.
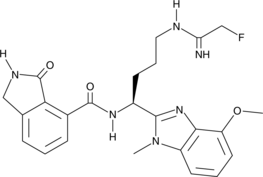
-
GC47050
CAY10727
A PAD3 inhibitor
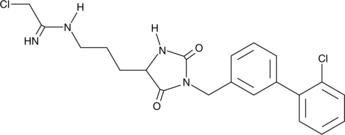
-
GC47055
CAY10749
CAY10749 (Compuesto 15) es un potente inhibidor PARP/PI3K con valores PIC50 de 8.22, 8.44, 8.25, 6.54, 8.13, 6.08 para PARP-1, PARP-2, PI3K⊵ ;, PI3K⋲ offlineefficient_models_2022q2.md.en_es_2021q4.mdCAY10749 is a highly effective anticancer compound targeted against a wide range of oncologic diseases.en_es_2021q4.md
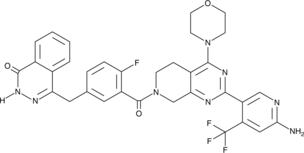
-
GC47056
CAY10753
A TNKS2 inhibitor
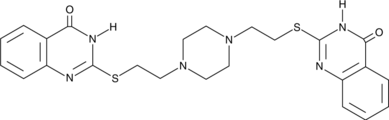
-
GC49029
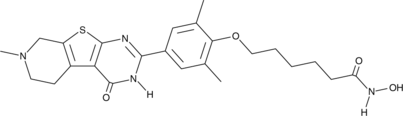
CAY17c
An inhibitor of BRD4 and class I and class IIb HDACs
-
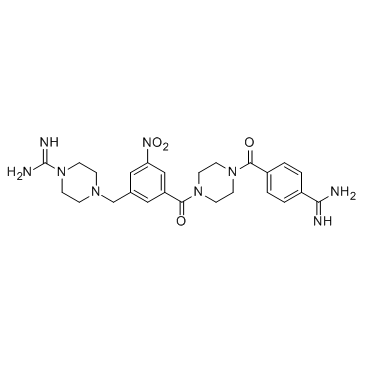
GC35621
CBB1003
CBB1003 es un nuevo inhibidor de histona desmetilasa LSD1 con IC50 de 10,54 uM.
-
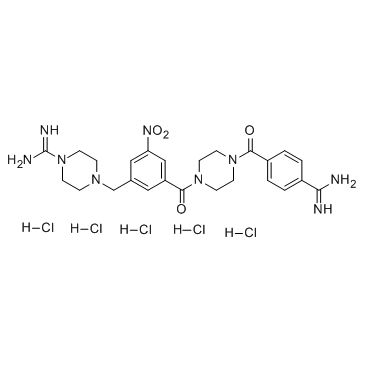
GC35622
CBB1003 hydrochloride
CBB1003 Hcl es un nuevo inhibidor de histona desmetilasa LSD1 con IC50 de 10,54 uM.
-
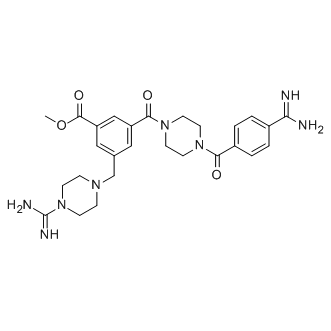
GC14151
CBB1007
CBB1007 es un compuesto de amidino-guanidinio permeable a las células que actÚa como un inhibidor selectivo de LSD1 potente, reversible y competitivo con el sustrato (IC50 = 5,27 μM para hLSD1).
-
GC35623
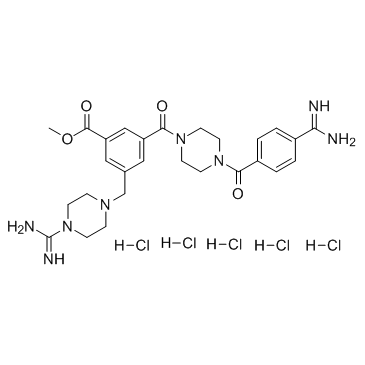
CBB1007 hydrochloride
CBB1007 Hcl es un compuesto de amidino-guanidinio permeable a las células que actÚa como un inhibidor selectivo de LSD1 potente, reversible y competitivo con el sustrato (IC50 = 5,27 μM para hLSD1).
-
GC35624
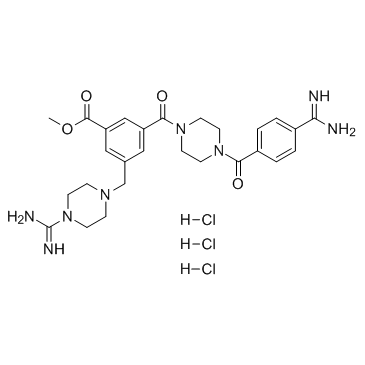
CBB1007 trihydrochloride
El trihidrocloruro de CBB1007 es un compuesto de amidino-guanidinio permeable a las células que actÚa como un inhibidor selectivo de LSD1 potente, reversible y competitivo con el sustrato (IC50 = 5,27 μM para hLSD1).
-
GC14058
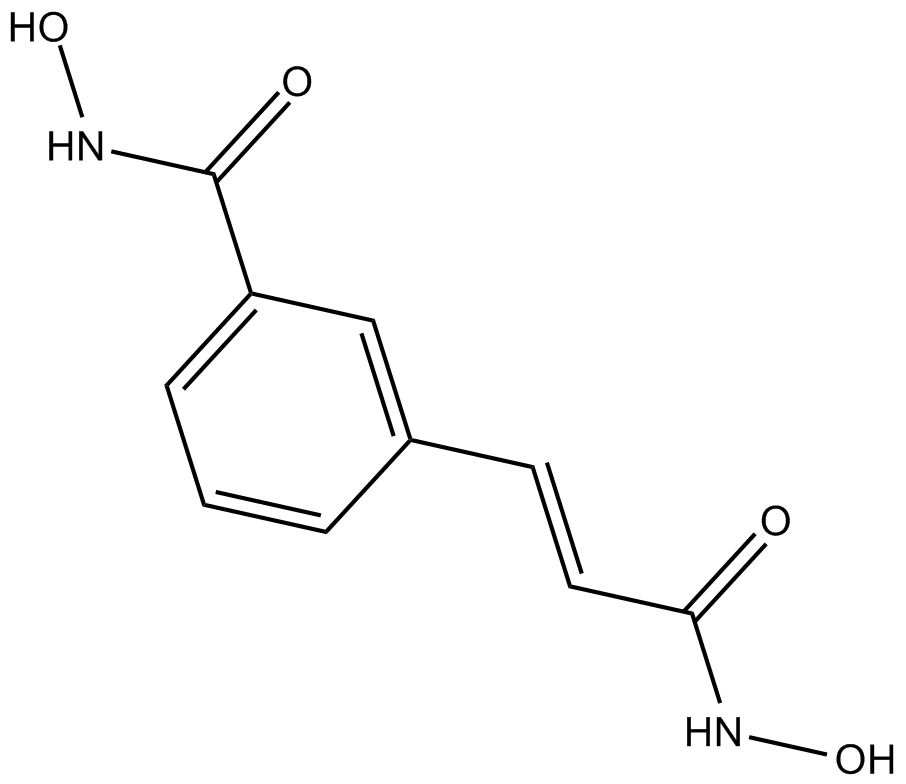
CBHA
CBHA es un potente inhibidor de HDAC, exhibiendo valores ID50 de 10 y 70 nM in vitro para HDAC1 y HDAC3, respectivamente. CBHA también induce la apoptosis y suprime el crecimiento tumoral.
-
GC34517
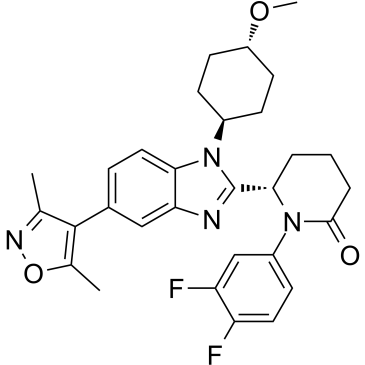
CBP-IN-1
CBP-IN-1 (CCS1477) es un inhibidor oralmente activo, potente y selectivo del bromodominio p300/CBP.
-
GC61577
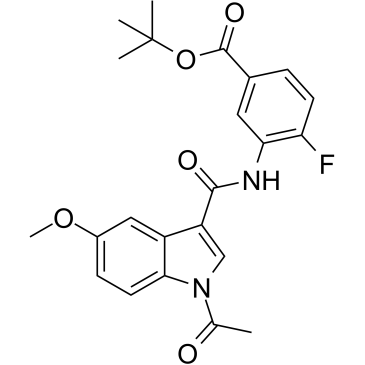
CBP/p300-IN-1
CBP/p300-IN-1 es un inhibidor del bromodominio CBP/EP300.
-
GC64696
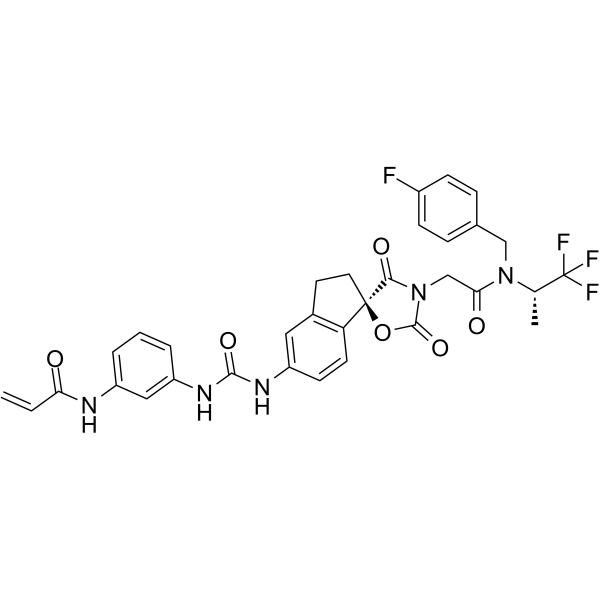
CBP/p300-IN-12
CBP/p300-IN-12 es un potente y selectivo covalente histona acetiltransferasas p300 (IC50 de 166 nM) e inhibidor de CBP. CBP/p300-IN-12 disminuye los niveles de H3K27Ac de las células PC-3 (EC50 de 37 nM). CBP/p300-IN-12 forma un aducto covalente con C1450.
-
GC38469
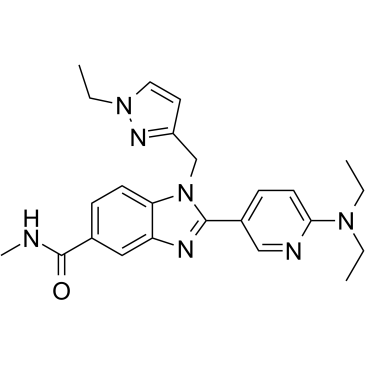
CBP/p300-IN-3
CBP/p300-IN-3, un inhibidor de histona acetiltransferasa p300/CBP, Compuesto 6, se obtiene de la patente WO 2019049061 A1.
-
GC62330
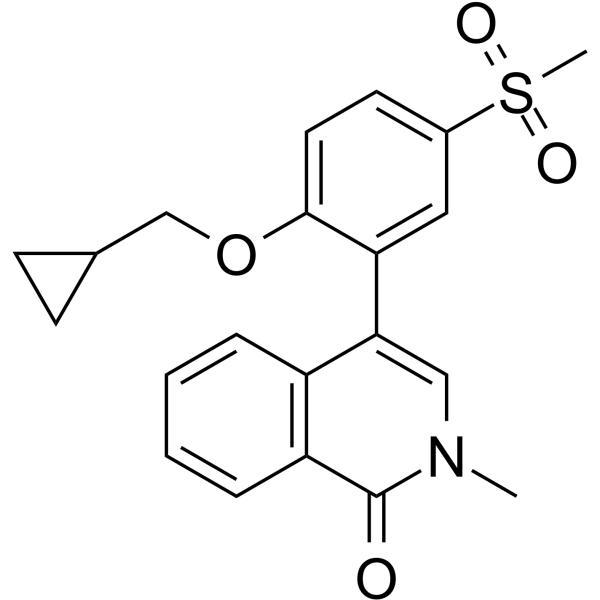
CC-90010
CC-90010 (compuesto 1) es un inhibidor de BET reversible y activo por vÍa oral. CC-90010 se aplica en el estudio de tumores sÓlidos avanzados.
-
GC12891
CCT007093
CCT007093 es un inhibidor eficaz de la proteÍna fosfatasa 1D (PPM1D Wip1). La inhibiciÓn de Wip1 puede activar la vÍa mTORC1 y mejorar la proliferaciÓn de hepatocitos después de la hepatectomÍa.
-
GC11310
CCT129202
An Aurora kinase inhibitor
-
GC14566
CCT137690
An inhibitor of Aurora kinases and FLT3
-
GC19092
CCT241736
CCT241736 es un inhibidor dual potente y biodisponible por vÍa oral de la quinasa Aurora y FLT3, que inhibe las quinasas Aurora (Aurora-A Kd, 7,5 nM, IC50, 38 nM; Aurora-B Kd, 48 nM), la quinasa FLT3 (Kd, 6,2 nM), y mutantes FLT3 que incluyen FLT3-ITD (Kd, 38 nM) y FLT3 (D835Y) (Kd, 14 nM).
-
GC48982
CD532
CD532 es un potente inhibidor de la cinasa Aurora A con una IC50 de 45 nM. CD532 tiene el doble efecto de bloquear la actividad de la quinasa Aurora A e impulsar la degradaciÓn de MYCN. CD532 también puede interactuar directamente con AURKA e induce un cambio conformacional global. CD532 se puede utilizar para la investigaciÓn del cÁncer.
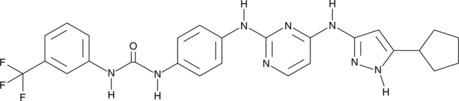
-
GC62189
CD532 hydrochloride
El clorhidrato de CD532 es un potente inhibidor de la cinasa Aurora A con una IC50 de 45 nM. El clorhidrato de CD532 tiene el doble efecto de bloquear la actividad de la quinasa Aurora A e impulsar la degradaciÓn de MYCN. El clorhidrato de CD532 también puede interactuar directamente con AURKA e induce un cambio conformacional global. El clorhidrato de CD532 se puede utilizar para la investigaciÓn del cÁncer.
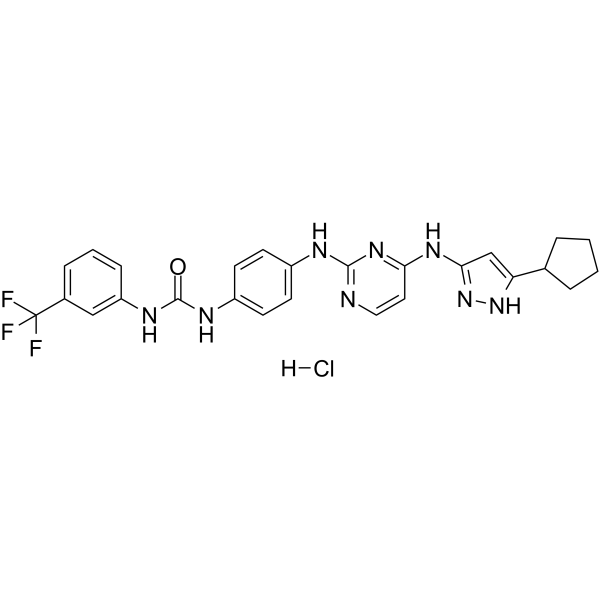
-
GC19098
CeMMEC1
CeMMEC1 es un inhibidor de BRD4 y también tiene una alta afinidad por TAF1, con una IC50 de 0,9 μM para TAF1 y una Kd de 1,8 μM para TAF1 (2).
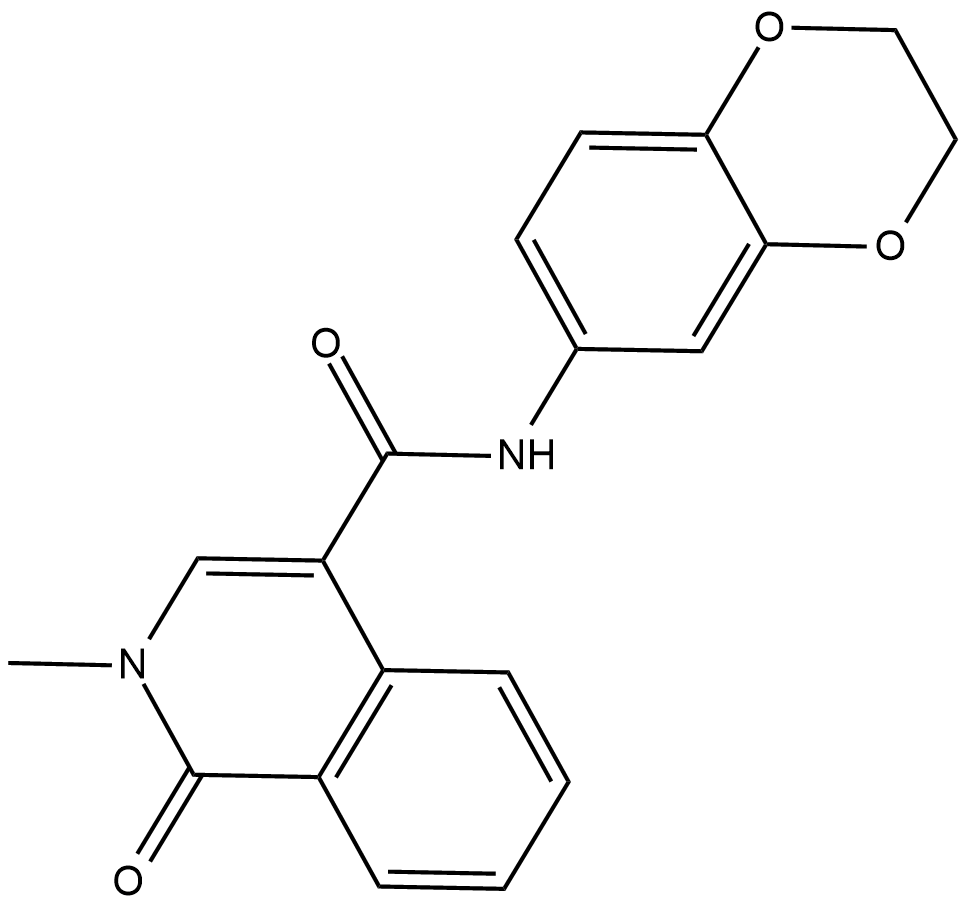
-
GC35651
Cenisertib
Cenisertib (AS-703569) es un inhibidor multicinasa competitivo con ATP que bloquea la actividad de Aurora-cinasa-A/B, ABL1, AKT, STAT5 y FLT3. Cenisertib induce importantes efectos inhibidores del crecimiento al bloquear la actividad de varios objetivos moleculares diferentes en los mastocitos neoplÁsicos (MC). Cenisertib inhibe el crecimiento tumoral en modelos de xenoinjerto de tumores de pÁncreas, mama, colon, ovario y pulmÓn y leucemia.
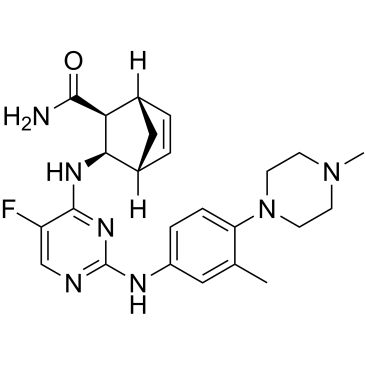
-
GC12083
CEP-33779
A potent, orally available inhibitor of JAK2
-
GC11209
Cerdulatinib (PRT062070)
Cerdulatinib (PRT062070) (PRT062070) es un inhibidor selectivo de Tyk2 con una IC50 de 0,5 nM. Cerdulatinib (PRT062070) (PRT062070) también es un inhibidor dual de JAK y SYK con IC50 de 12, 6, 8 y 32 para JAK1, 2, 3 y SYK, respectivamente.