MAPK Signaling

Targets for MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Products for MAPK Signaling
- Cat.No. Nombre del producto Información
-
GC13142
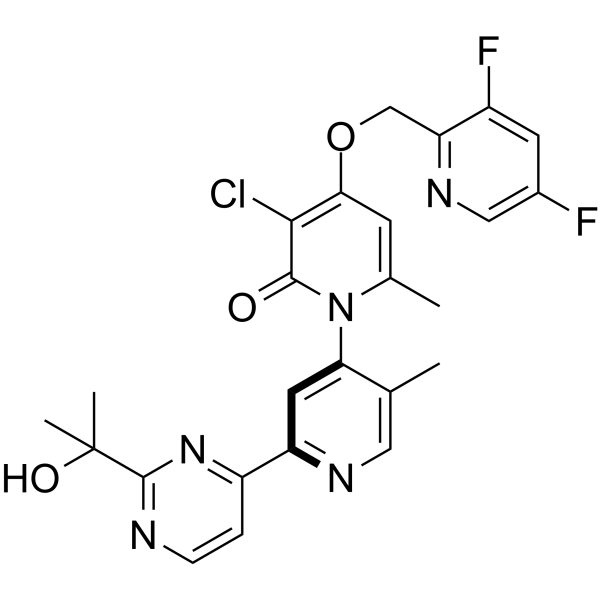
RWJ 67657
RWJ 67657 (JNJ 3026582) es un inhibidor de MAPK p38α y p38β selectivo y activo por vía oral con IC50 de 1 y 11 μM, respectivamente.
-
GC18273
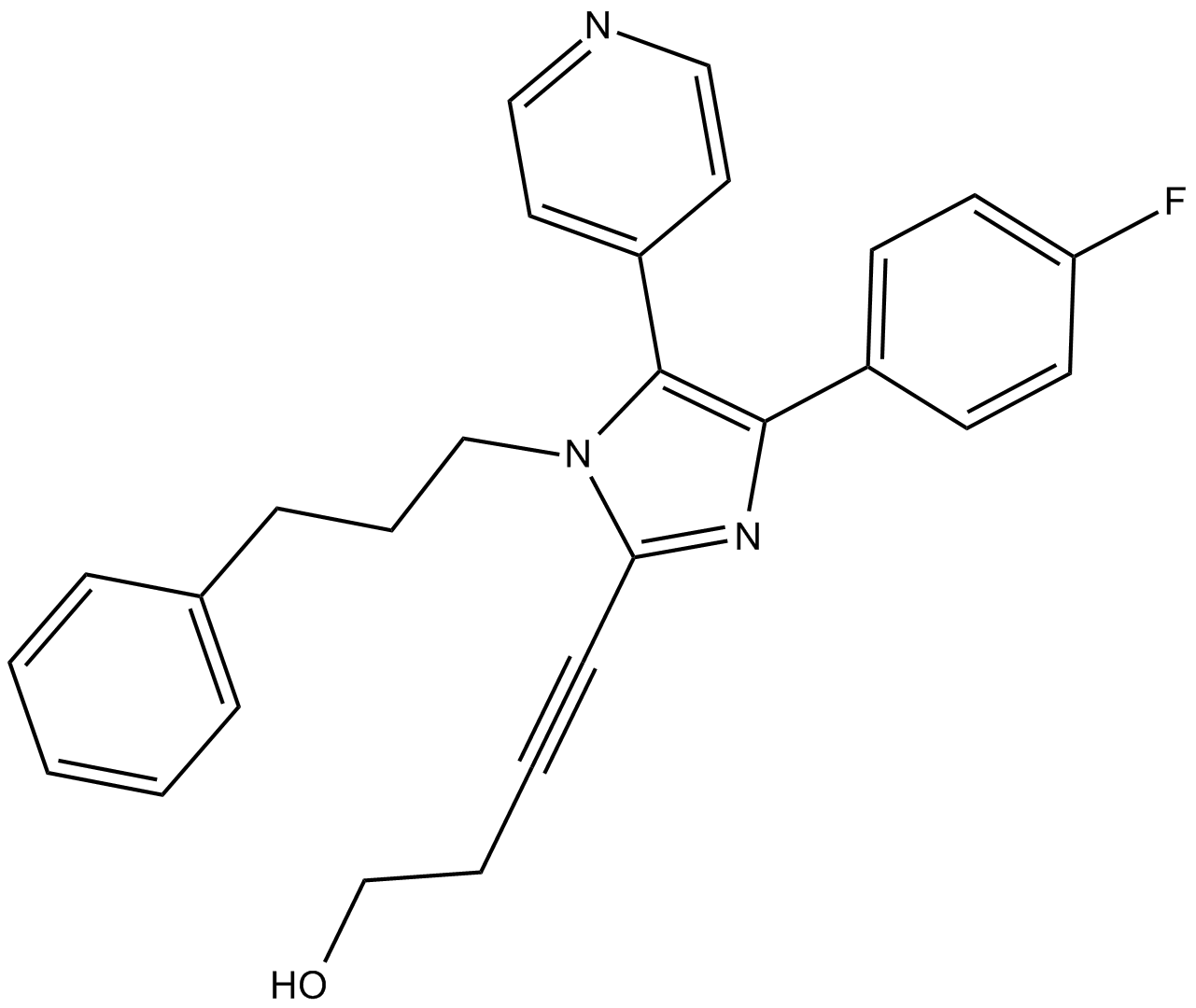
SB 202190 (hydrochloride)
SB 202190 (clorhidrato) es un inhibidor selectivo de la MAP quinasa p38 con IC50 de 50 nM y 100 nM para p38α y p38β2, respectivamente.
-
GC16019
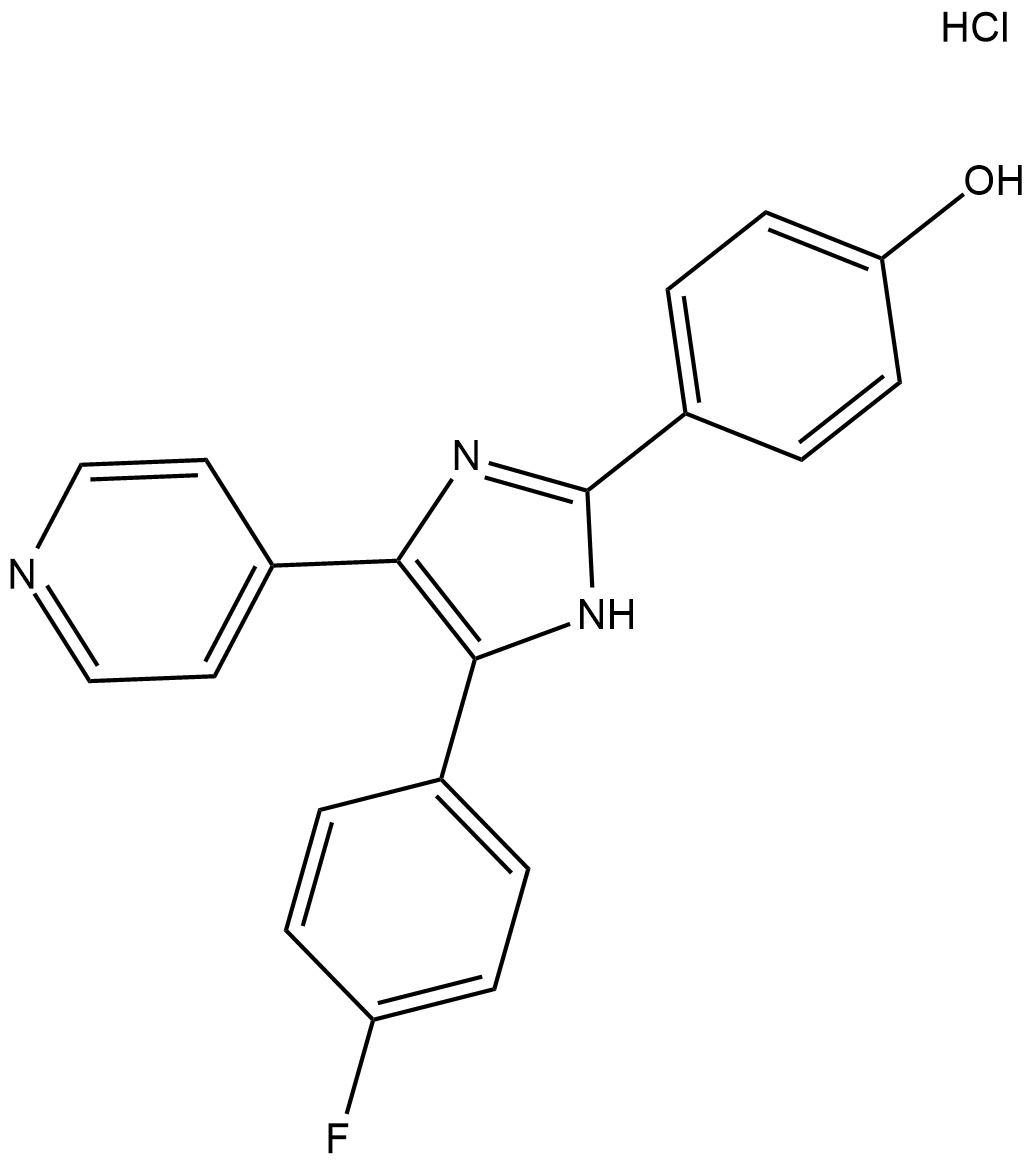
SB 202474
SB 202474, un anÁlogo negativo de SB203580. SB 202474, que no tiene capacidad para inhibir la actividad de p38 MAPK y se usa ampliamente como compuesto de control negativo en estudios de p38 MAPK, también suprimiÓ la inducciÓn de la sÍntesis de melanina.
-
GC13001
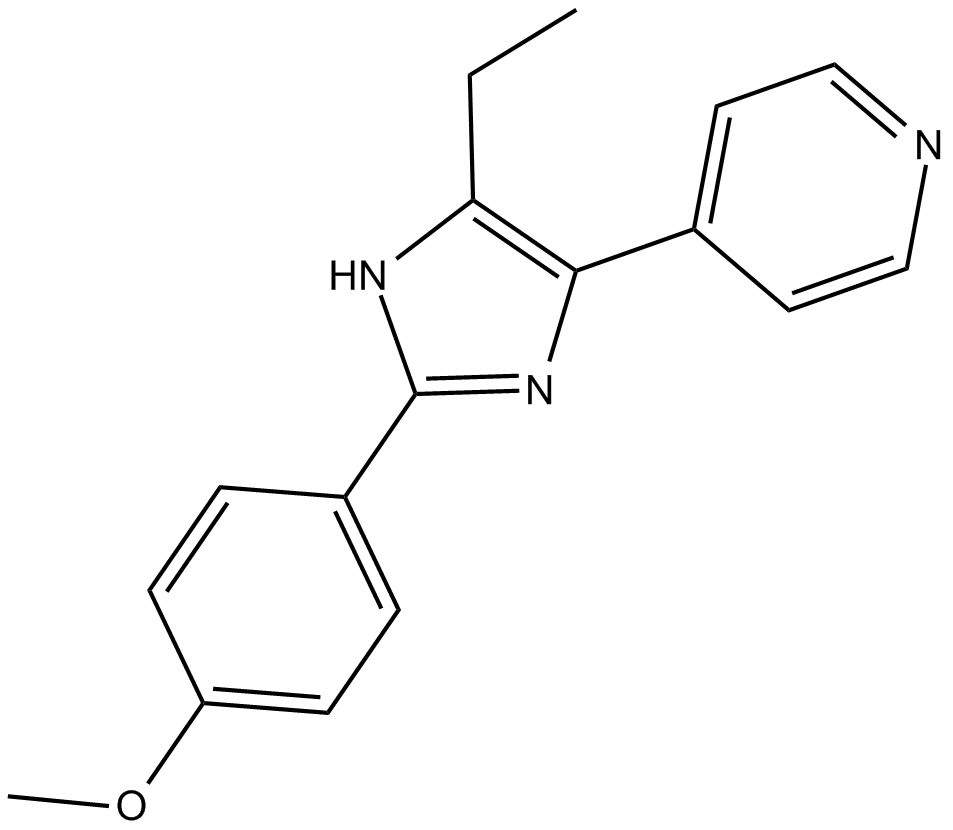
SB 203580 hydrochloride
El clorhidrato de adezmapimod (SB 203580) es un inhibidor de p38 MAPK selectivo y competitivo con ATP con IC50 de 50 nM y 500 nM para SAPK2a/p38 y SAPK2b/p38β2, respectivamente.
-
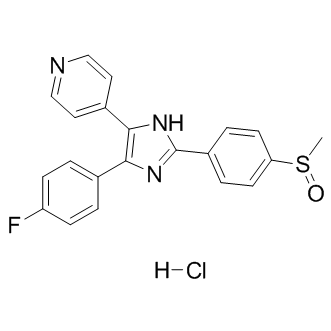
GC10054
SB 239063
A selective p38 MAPK inhibitor
-
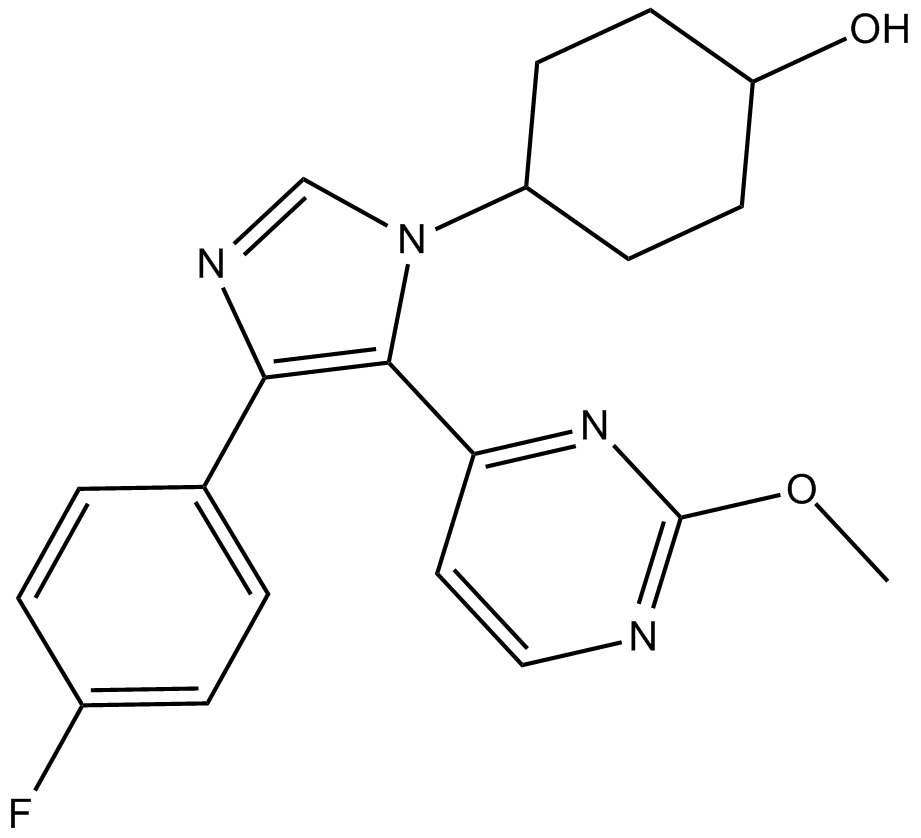
GC37595
SB 242235
SB-242235 es un inhibidor potente y selectivo de la MAP quinasa p38, con una IC50 de 1.0 μM en condrocitos humanos primarios.
-
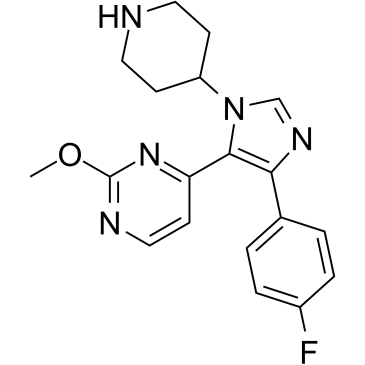
GC11922
SB 706504
p38 MAPK inhibitor
-
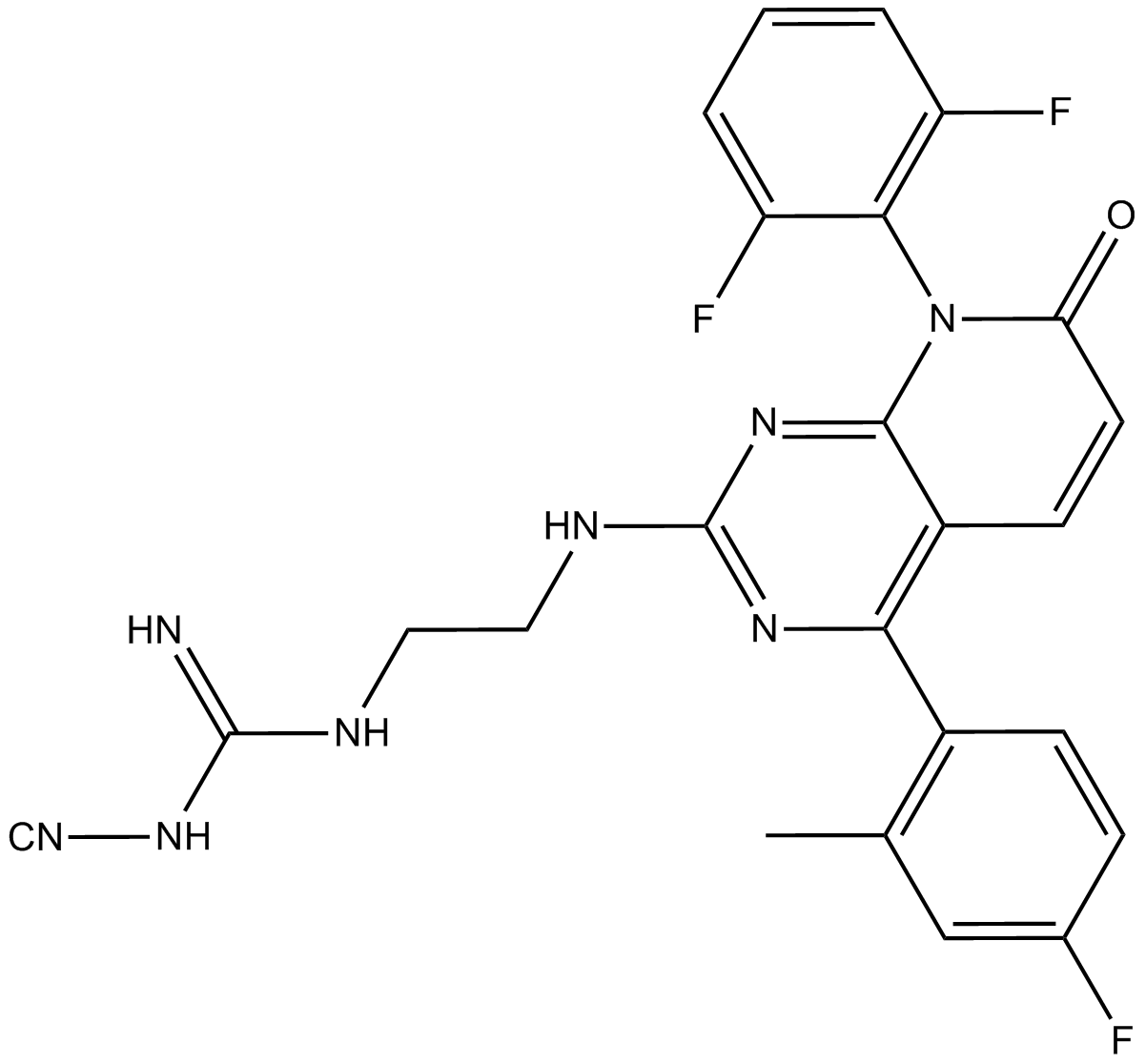
GC13968
SB202190 (FHPI)
SB202190 (FHPI) es un inhibidor selectivo de la quinasa p38 MAP con IC50s de 50 nM y 100 nM para p38α y p38β2, respectivamente.
-
GC11890
SB590885
SB590885 es un potente inhibidor de B-Raf con Ki de 0,16 nM y tiene una selectividad 11 veces mayor para B-Raf que para c-Raf, sin inhibición de otras quinasas humanas.
-
GC16001
SCH772984
Un inhibidor selectivo de ERK1/2.
-
GC14647
SCH772984 HCl
ERK1/2 inhibitor
-
GC15044
SCH772984 TFA
ERK1/2 inhibitor
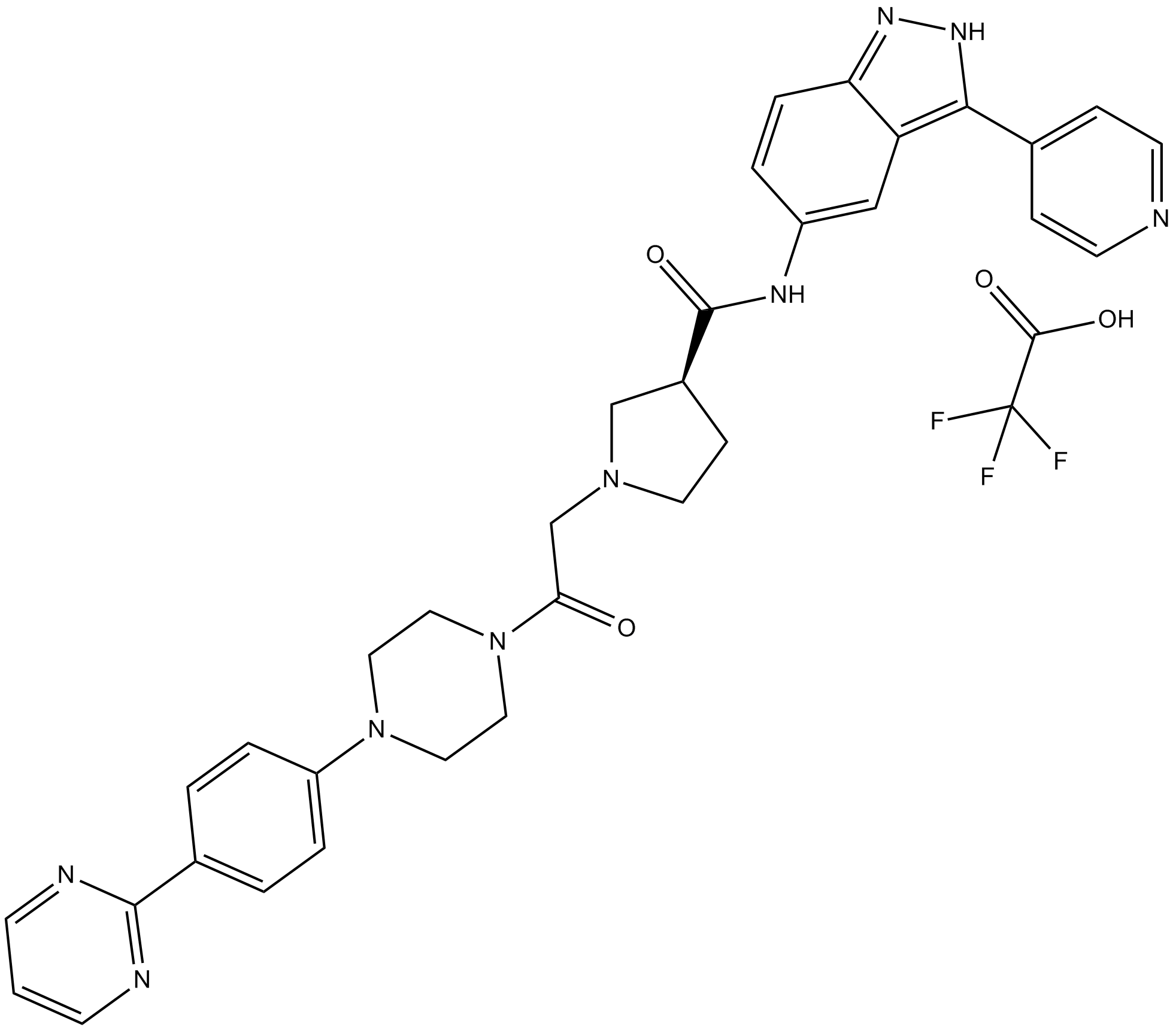
-
GC19325
SD 0006
SD 0006 (SD-06) es un inhibidor de diaril pirazol de p38α MAP quinasa, activo por vÍa oral, selectivo, ATP-competitivo y potente, con una IC50 de 110 nM para p38α.
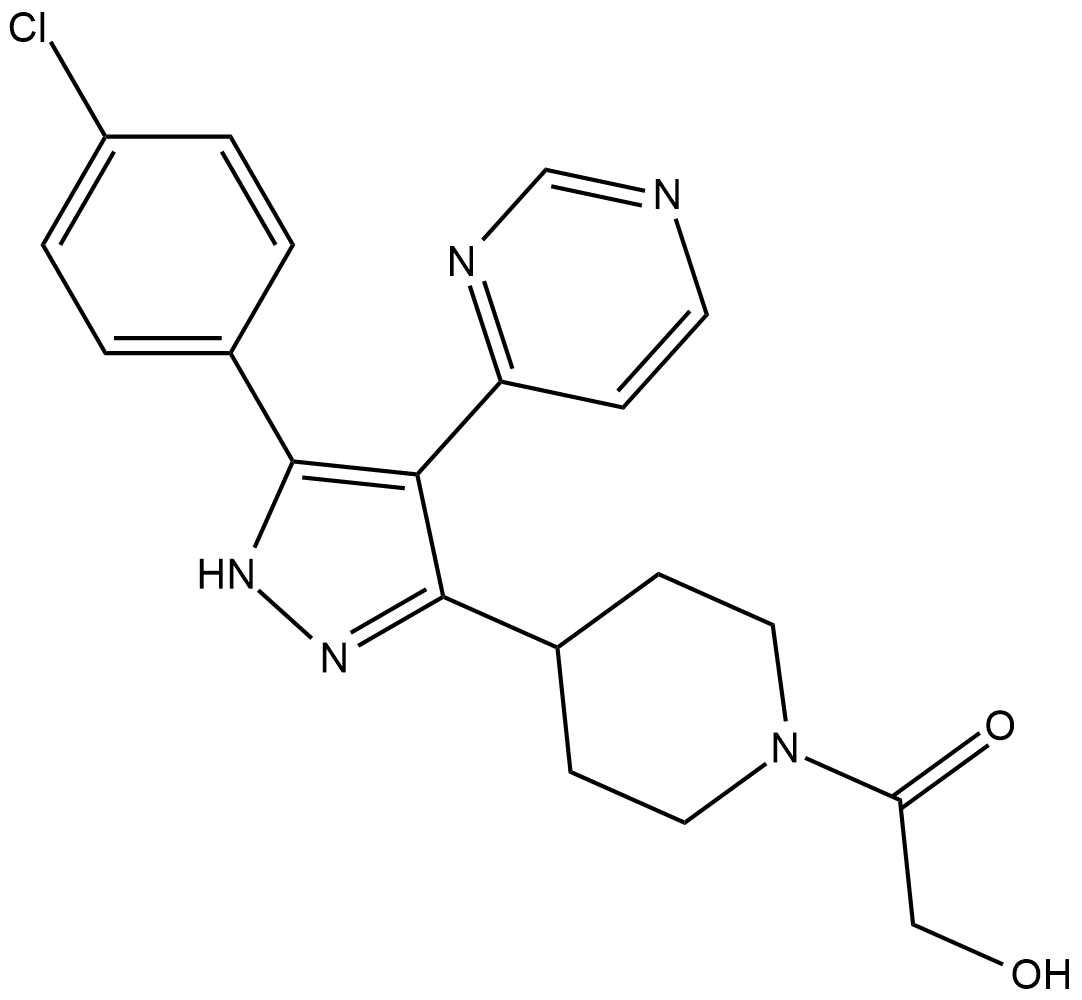
-
GC14982
SD 169
SD 169 es un inhibidor competitivo de ATP activo por vía oral de p38α MAPK, con una IC50 de 3,2 nM.
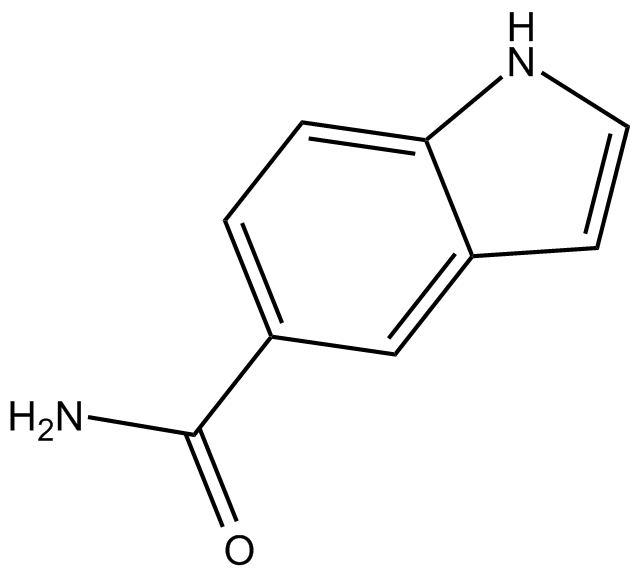
-
GC12124
Selonsertib (GS-4997)
Selonsertib (GS-4997) (GS-4997), un inhibidor selectivo de la cinasa 1 reguladora de la seÑal de apoptosis (ASK1) biodisponible por vÍa oral con una pIC50 de 8,3, se ha evaluado como tratamiento experimental para la nefropatÍa diabética y la fibrosis renal.
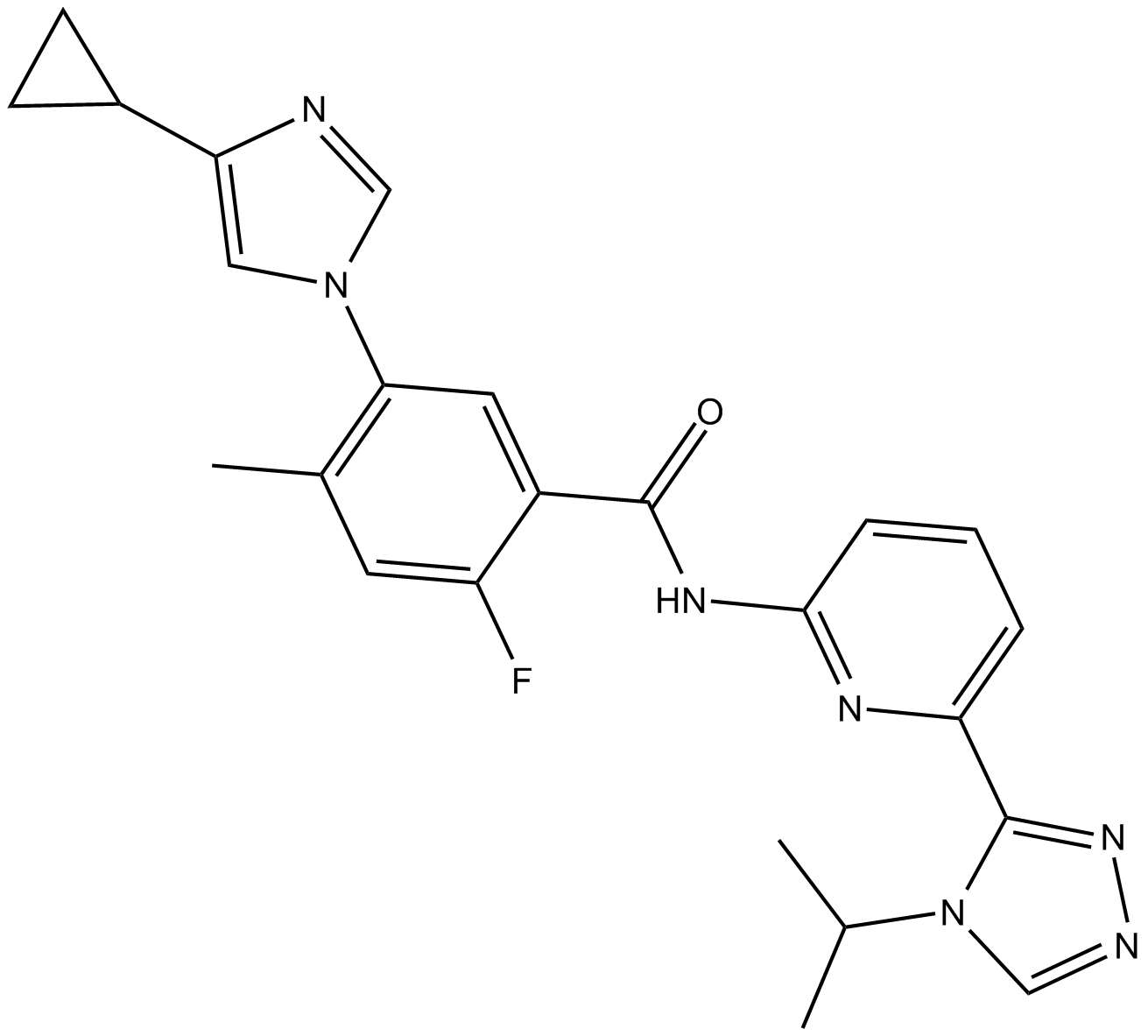
-
GC38653
Selumetinib sulfate
Selumetinib (AZD6244) es un inhibidor oral selectivo de MEK1/2 no competitivo con ATP, con una IC50 de 14 nM para MEK1. Selumetinib (AZD6244) inhibe la fosforilaciÓn de ERK1/2.
-
GC39815
Semapimod tetrahydrochloride
El tetraclorhidrato de semapimod (CNI-1493), un inhibidor de la producciÓn de citocinas proinflamatorias, puede inhibir el TNF-α, la IL-1β y la IL-6.
-
GC10005
SEP-0372814
A PDE10A inhibitor
-
GC33229
SJFα
SJF& alfa; es un conector PROTAC de 13 átomos basado en el ligando de von Hippel-Lindau. SJF& alfa; degrada p38α con una DC50 de 7,16 nM, pero es mucho menos eficaz para degradar p38δ (DC50\u003d299 nM) y no degrada las otras isoformas de p38 (β y γ) en concentraciones de hasta 2,5 µM.
-
GC33238
SJFδ
SJF& delta; es un conector PROTAC de 10 átomos basado en el ligando de von Hippel-Lindau. SJF& delta; degrada p38δ con una DC50 de 46,17 nM, pero no degrada p38α, p38β ni p38γ.
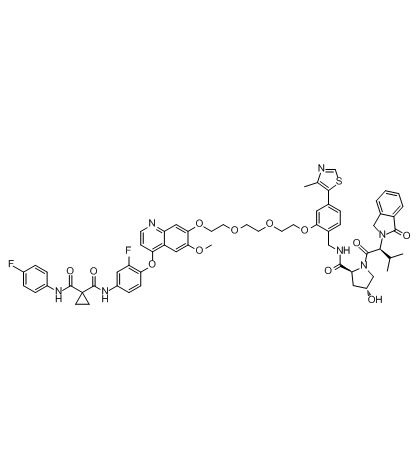
-
GC30646
Skatole(3-Methylindole)
El escatol (3-metilindol) es producido por las bacterias intestinales, regula las funciones celulares del epitelio intestinal mediante la activaciÓn de los receptores de hidrocarburo de arilo y p38.
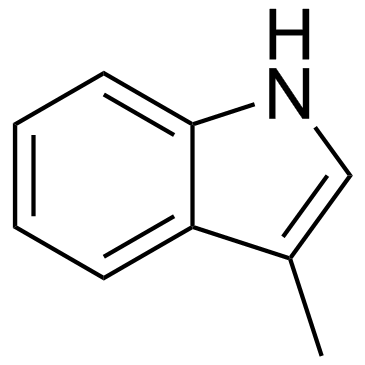
-
GC13578
Skepinone-L
An inhibitor of p38 MAPK
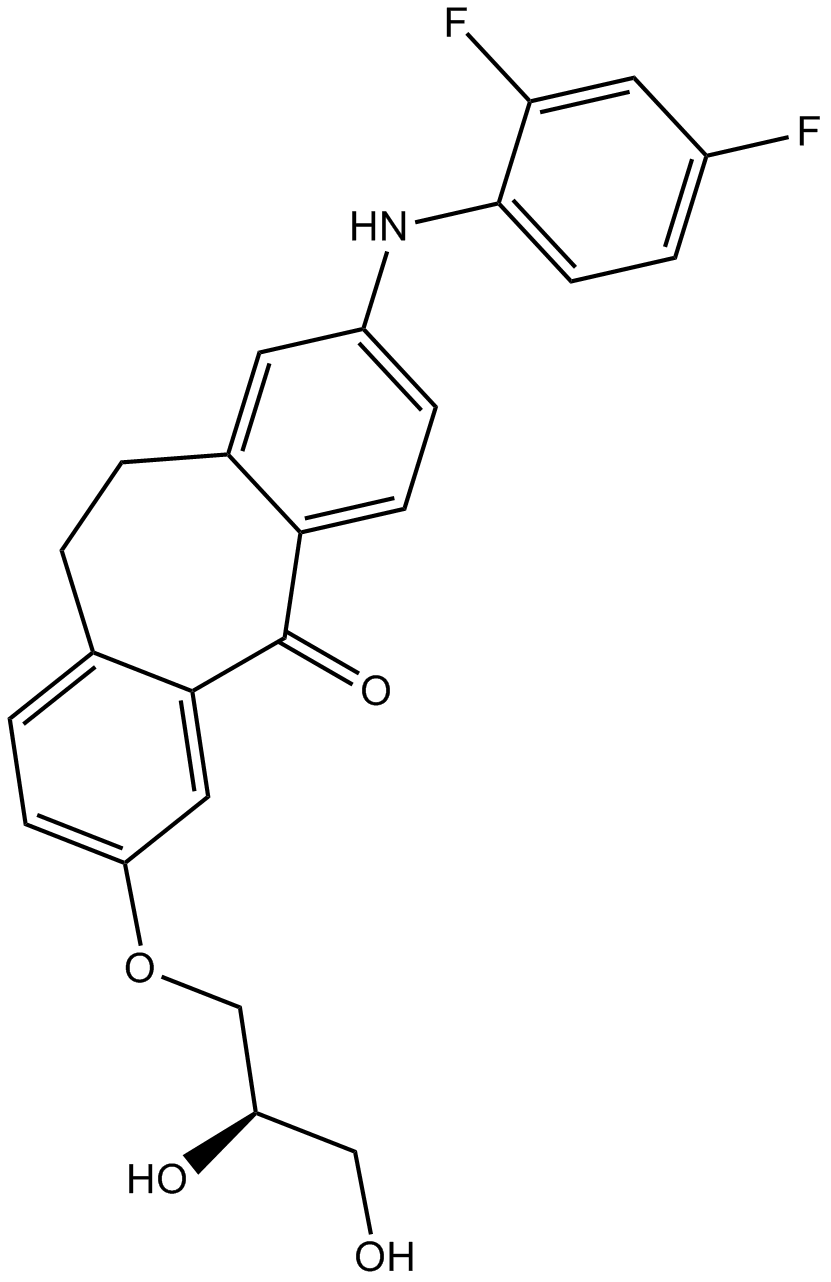
-
GC17725
SKF 86002 dihydrochloride
El diclorhidrato SKF 86002 es un inhibidor de p38 MAPK activo por vía oral, con actividades antiinflamatorias, antiartríticas y analgésicas.
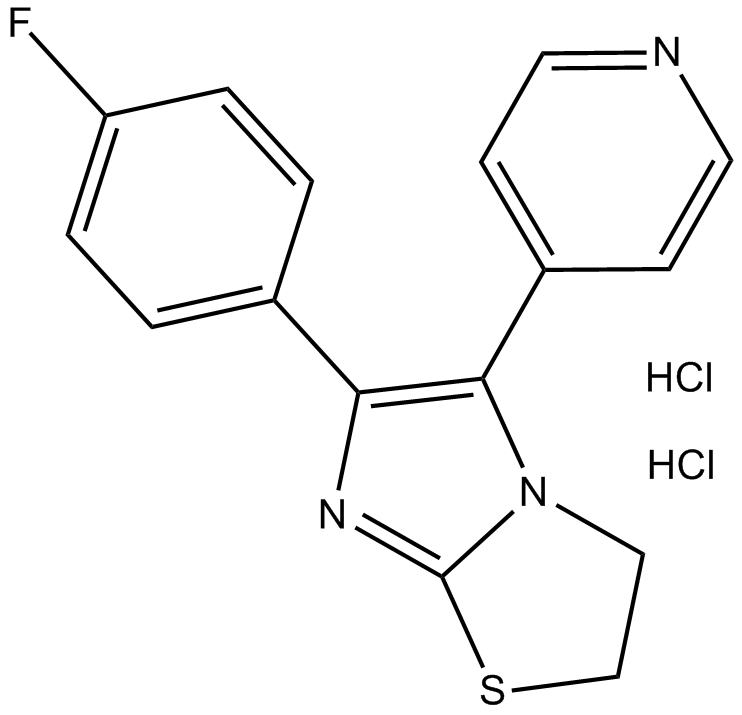
-
GC37646
SKF-86002
SKF-86002 es un inhibidor de p38 MAPK activo por vÍa oral, con actividades antiinflamatorias, antiartrÍticas y analgésicas.
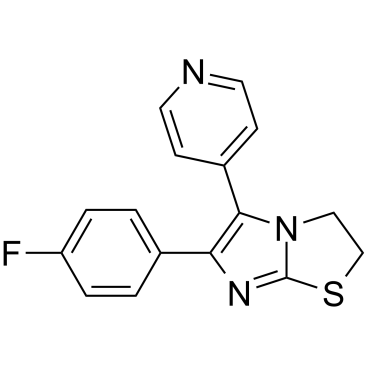
-
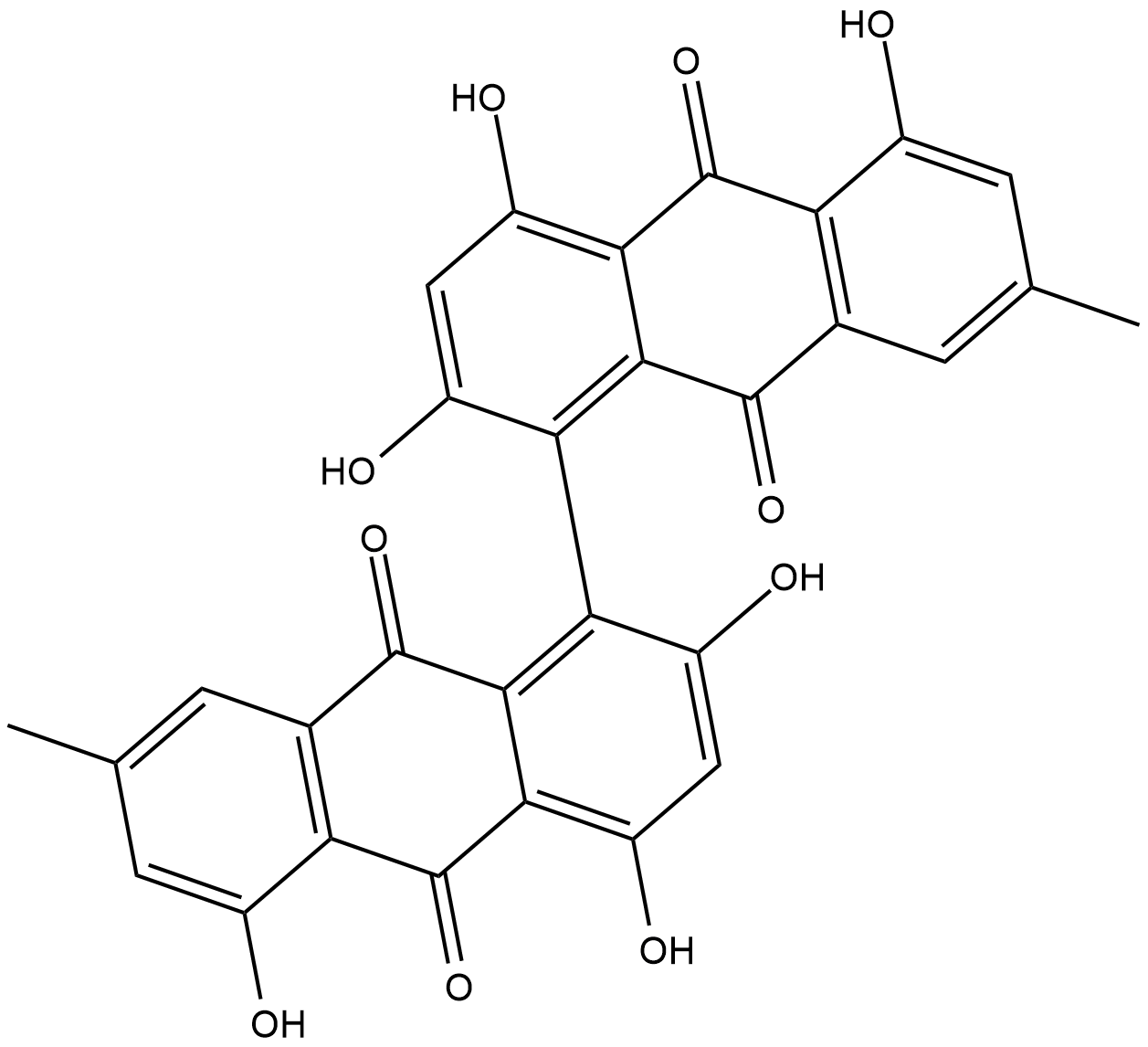
GC18532
Skyrin
Skyrin is a fungal metabolite characterized by a bisanthraquinone structure.
-
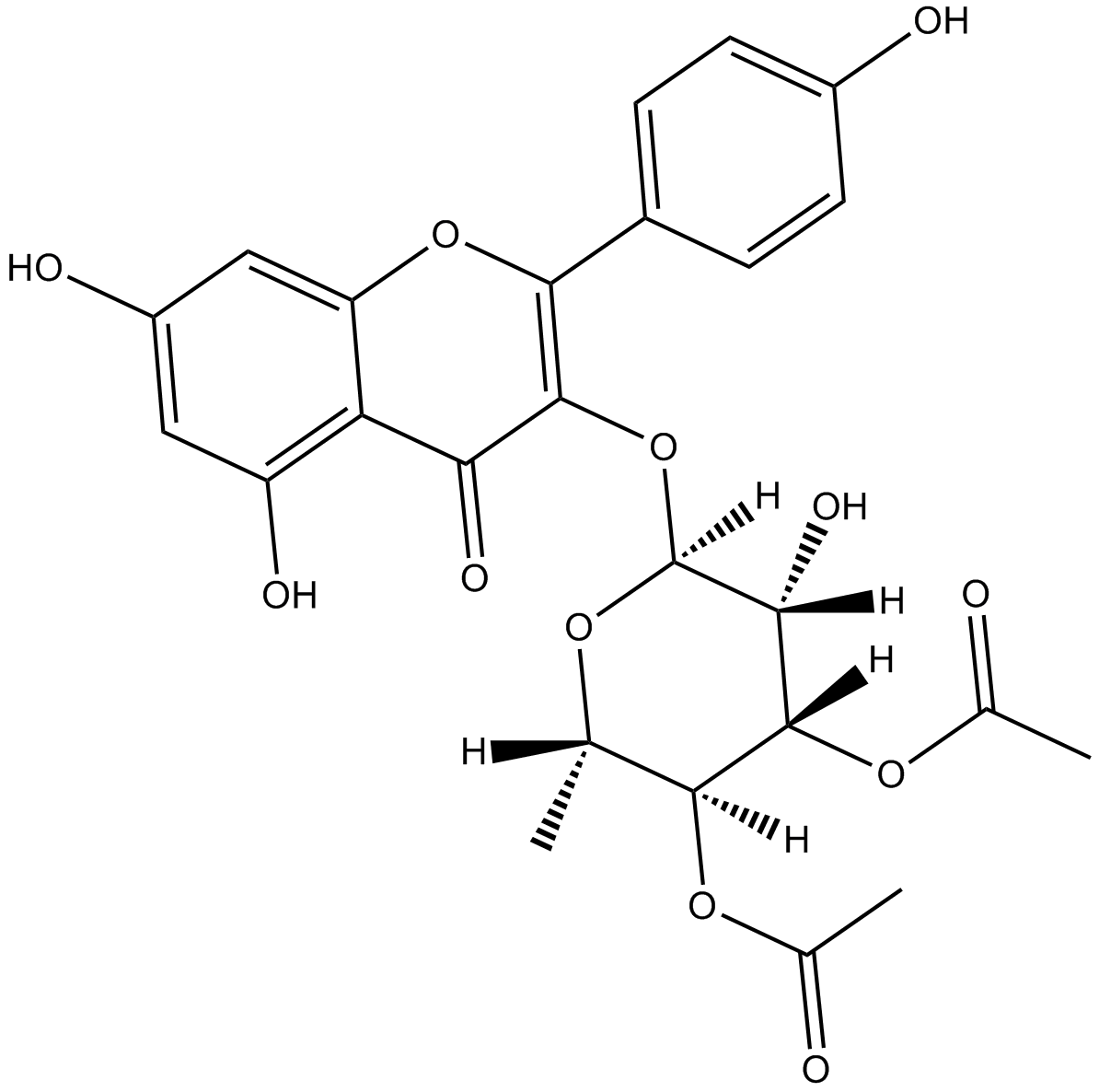
GC16313
SL 0101-1
SL 0101-1 (SL0101), un glucÓsido de kaempferol, aislado de la planta tropical F. refracta, es un inhibidor de la quinasa ribosomal S6 (RSK) p90 ribosomal S6 (RSK) permeable a las células, selectivo, reversible, con una IC50 de 89 nM. SL 0101-1 (SL0101) es un inhibidor selectivo de RSK1/2, con una Ki de 1 μM.
-
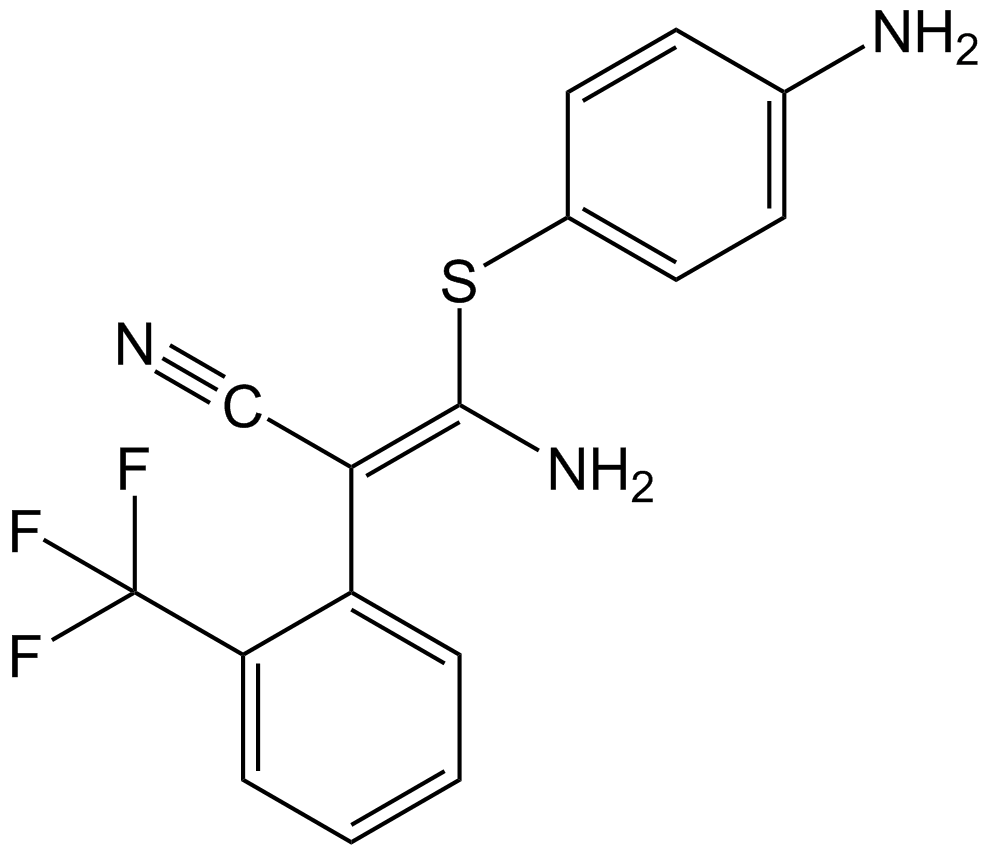
GC15359
SL-327
SL-327 inhibe MEK1 y MEK2, con valores IC50 de 180 nM y 220 nM, respectivamente.
-
GC32937
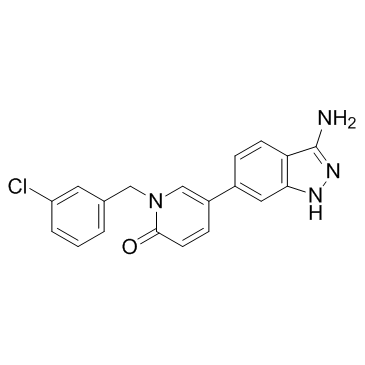
SLV-2436 (SEL201-88)
SLV-2436 (SEL201-88) es un inhibidor muy potente y competitivo con ATP de MNK1 y MNK2 con IC50 de 10,8 nM y 5,4 nM, respectivamente.
-
GC62675
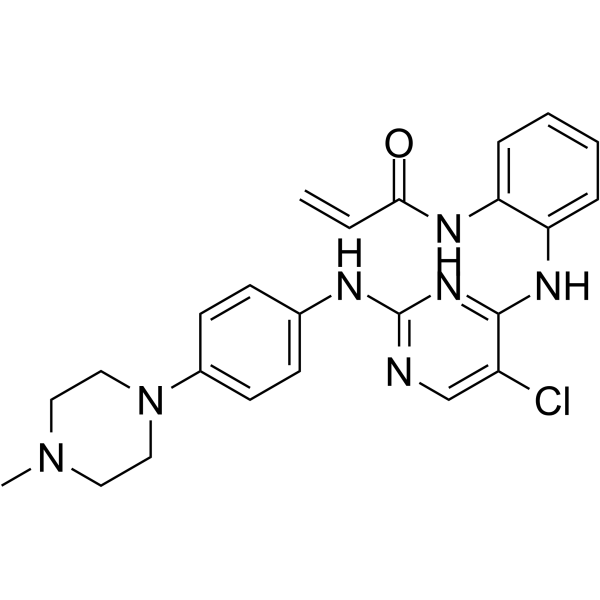
SM1-71
SM1-71 (compuesto 5) es un potente inhibidor de TAK1, con un Ki de 160 nM, también puede inhibir covalentemente MKNK2, MAP2K1/2/3/4/6/7, GAK, AAK1, BMP2K, MAP3K7, MAPKAPK5, GSK3A /B, MAPK1/3, SRC, YES1, FGFR1, ZAK (MLTK), MAP3K1, LIMK1 y RSK2. SM1-71 puede inhibir la proliferaciÓn de mÚltiples lÍneas celulares de cÁncer.
-
GC17425
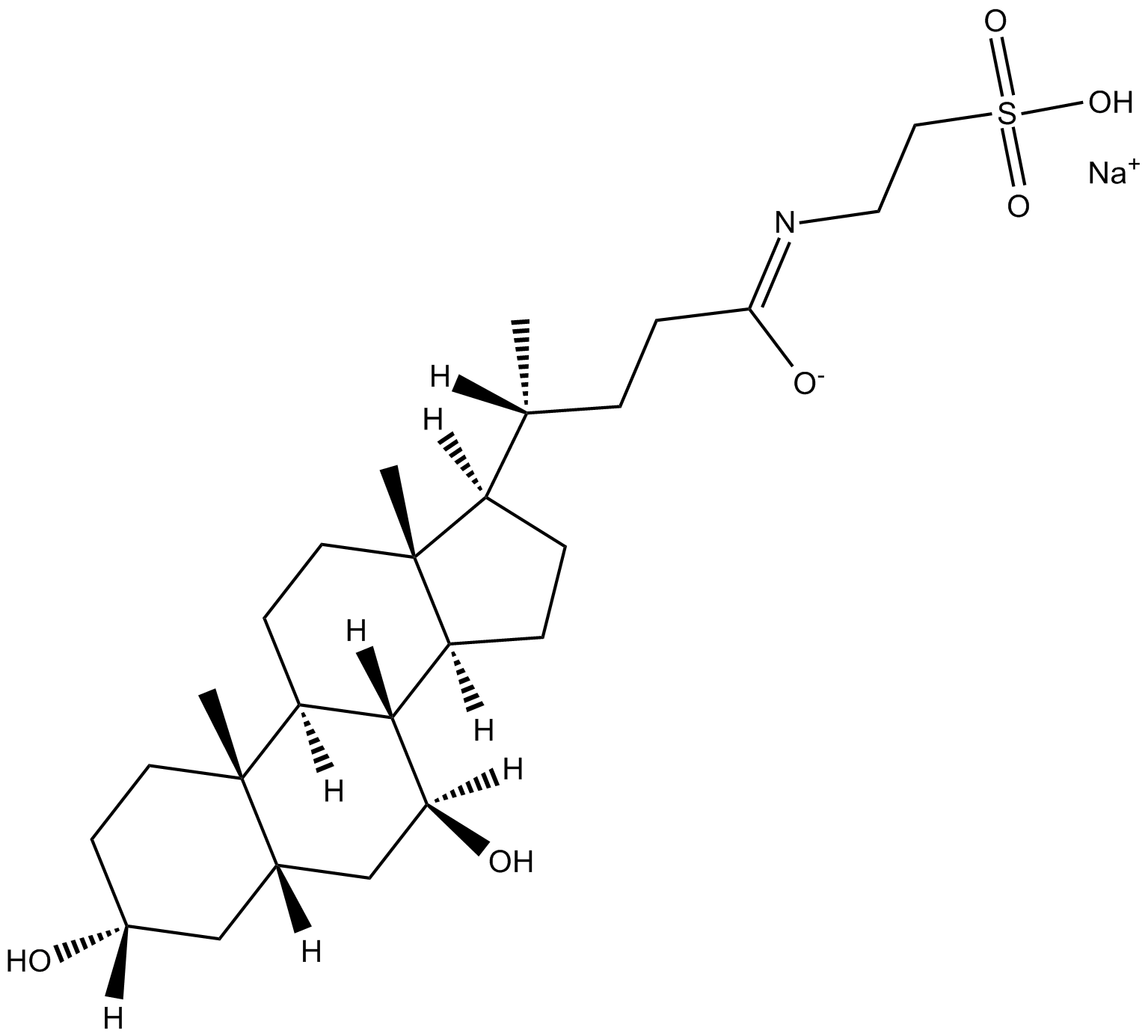
Sodium Tauroursodeoxycholate (TUDC)
El tauroursodesoxicolato (Ácido tauroursodesoxicÓlico; TUDCA) sÓdico es un inhibidor del estrés del retÍculo endoplÁsmico (RE). El tauroursodesoxicolato reduce significativamente la expresiÓn de moléculas de apoptosis, como la caspasa-3 y la caspasa-12. El tauroursodesoxicolato también inhibe la ERK.
-
GC17369
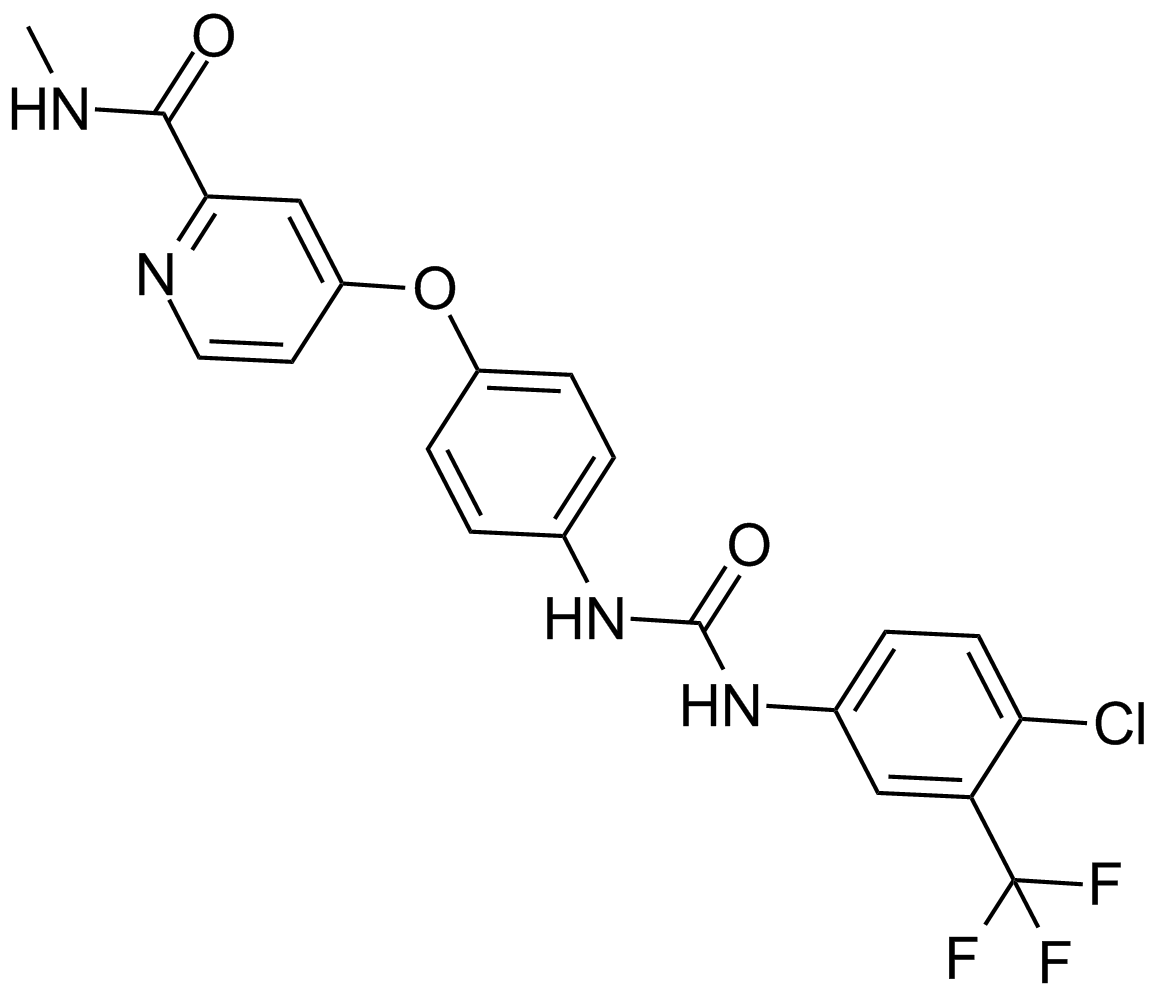
Sorafenib
Sorafenib actúa como un inhibidor de múltiples quinasas, dirigiéndose a Raf-1 y B-Raf con valores de IC50 de 6 nM y 22 nM, respectivamente.
-
GC37664
Sorafenib (D3)
Sorafenib (D3) (Bahía 43-9006-d3) es el deuterio etiquetado Sorafenib. Sorafenib es un inhibidor multiquinasa IC50 de 6 nM, 20 nM y 22 nM para Raf-1, B-Raf y VEGFR-3, respectivamente.
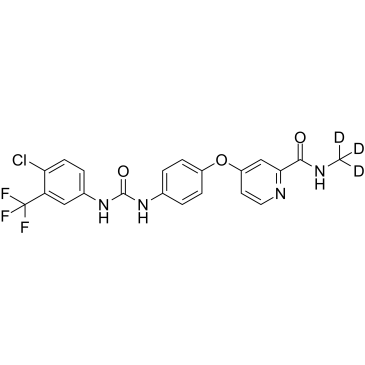
-
GC37665
Sorafenib (D4)
Sorafenib (D4) (Bahía 43-9006-d4) es el deuterio etiquetado Sorafenib. Sorafenib es un inhibidor multiquinasa IC50 de 6 nM, 20 nM y 22 nM para Raf-1, B-Raf y VEGFR-3, respectivamente.
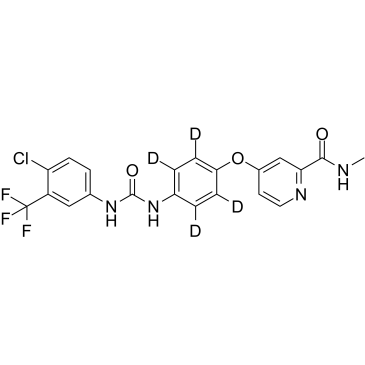
-
GC44917
Sorafenib N-oxide
Sorafenib N-oxide is an active metabolite of sorafenib, an inhibitor of Raf-1, B-RAF, and receptor tyrosine kinases.
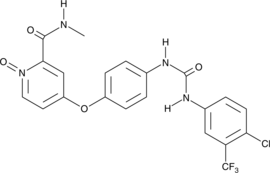
-
GC10428
Sp-5,6-dichloro-cBIMPS (sodium salt)
PKA activator
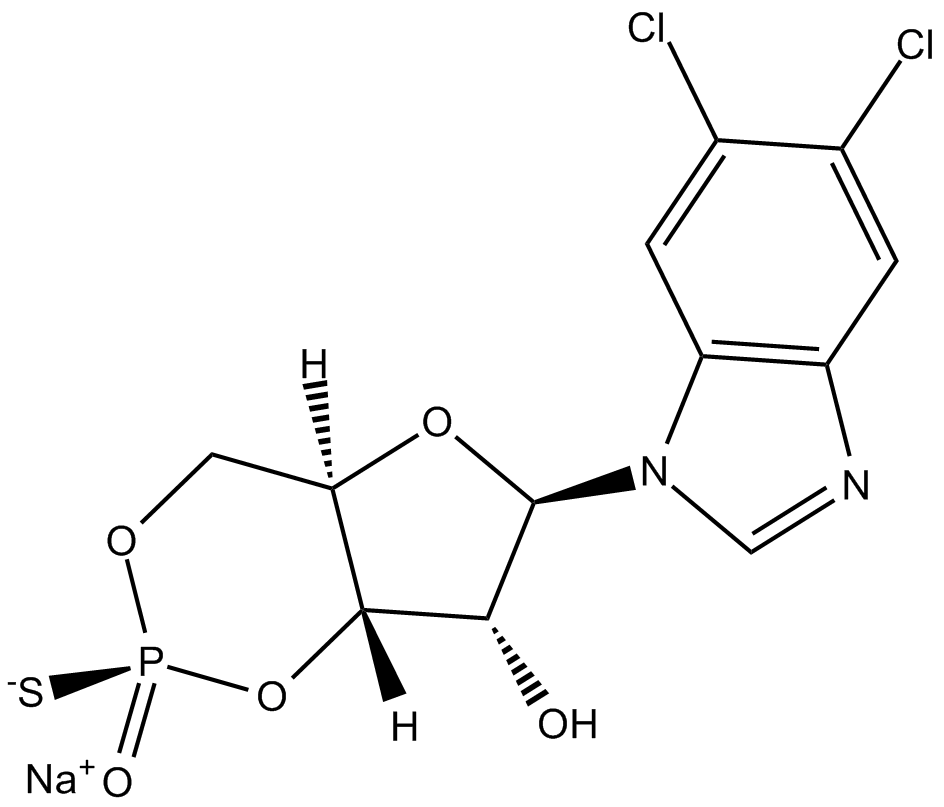
-
GC44920
Sp-8-bromo-Cyclic AMPS (sodium salt)
Sp-8-bromo-cyclic AMPS (Sp-8-bromo-cAMPS) is a cell-permeable, cAMP analog that combines an exocyclic sulfur substitution in the axial position of the cyclophosphate ring with a bromine substitution in the adenine base of cAMP.
-
GC61747
Sp-cAMPS
Sp-cAMPS, un anÁlogo de cAMP, es un potente activador de PKA I y PKA II dependientes de cAMP.
-
GC16652
SR 3576
SR 3576 es un inhibidor de JNK3 muy potente y selectivo con una IC50 de 7 nM.
-
GC30864
SR-3306
SR-3306 es un inhibidor de JNK selectivo, potente y altamente penetrante en el cerebro.
-
GC64287
SR15006
SR15006 es un inhibidor del factor 5 similar a KrÜppel (KLF5) con una IC50 de 41,6 nM en células DLD-1/pGL4.18hKLF5p).
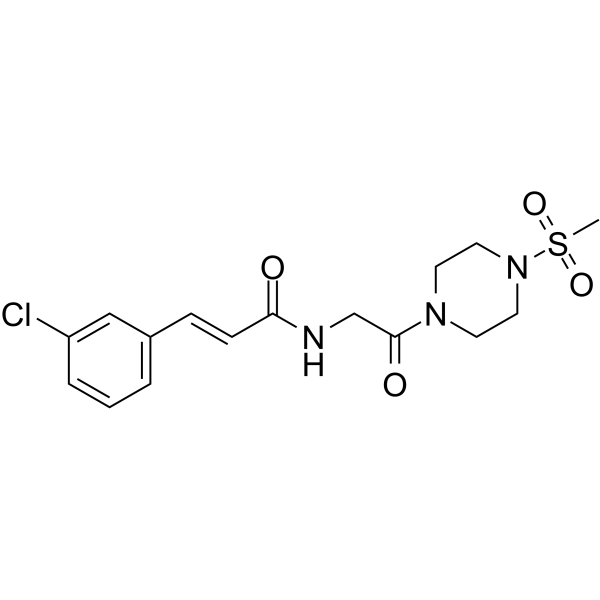
-
GC50406
st-Ht31
Inhibits PKA/AKAP interactions; cell permeable
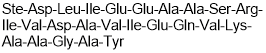
-
GC50407
st-Ht31 P
Negative control for st-Ht31
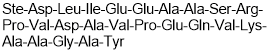
-
GC50501
STAD 2
STAD 2 es un disruptor potente y selectivo de PKA-RII, con una Kd de 6,2 nM.
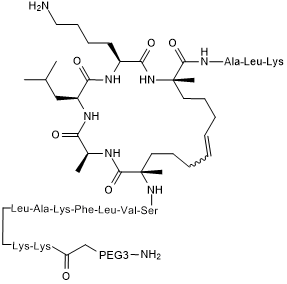
-
GC15299
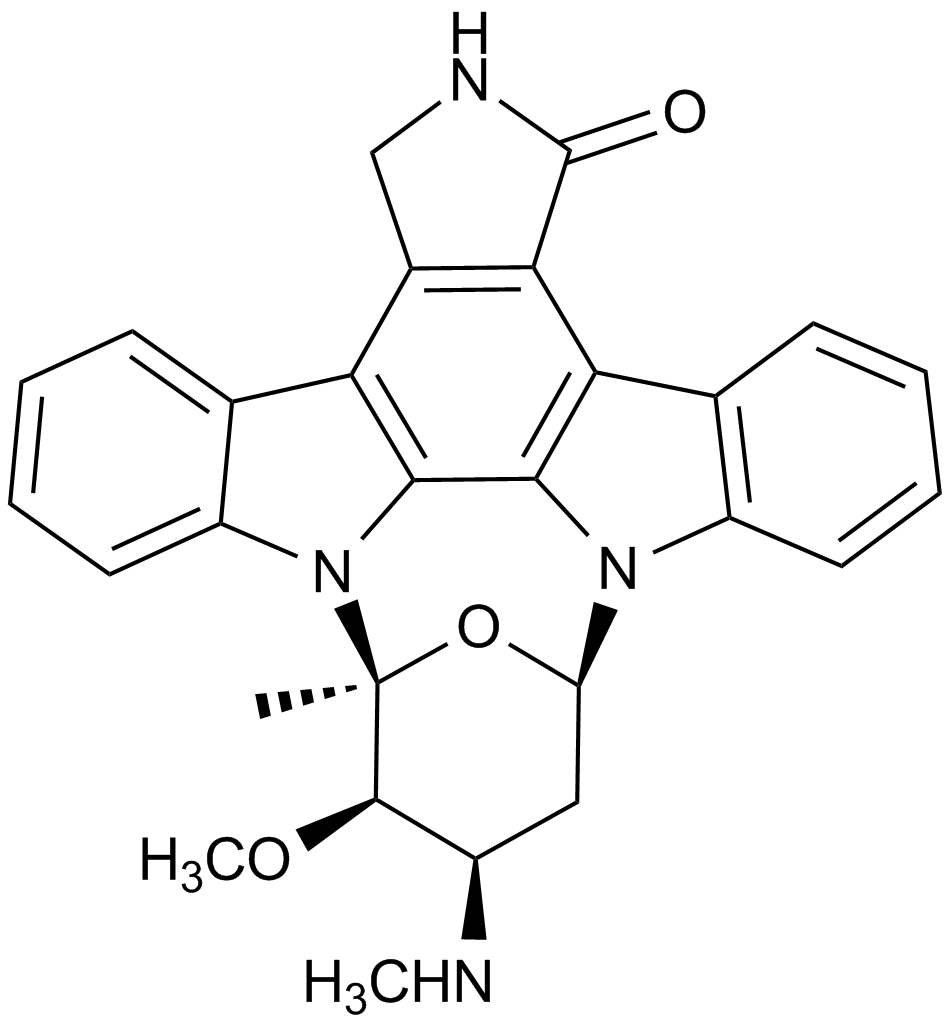
Staurosporine(CGP 41251)
A potent inhibitor of protein kinase C
-
GC11542
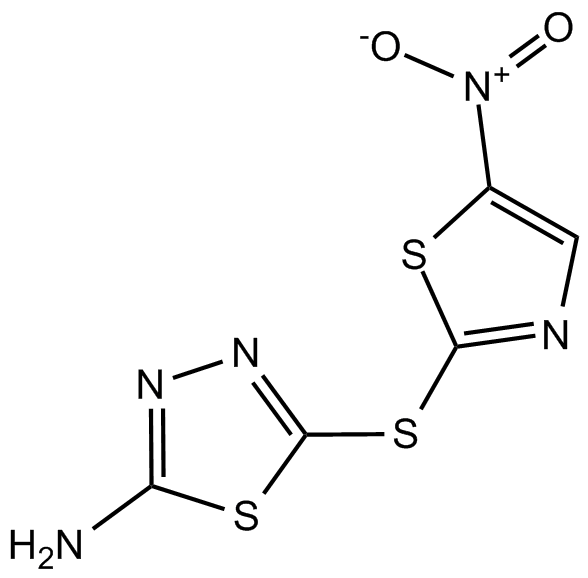
SU 3327
SU 3327 es un inhibidor de JNK potente, selectivo y competitivo con el sustrato con una IC50 de 0,7 μM.
-
GC13825
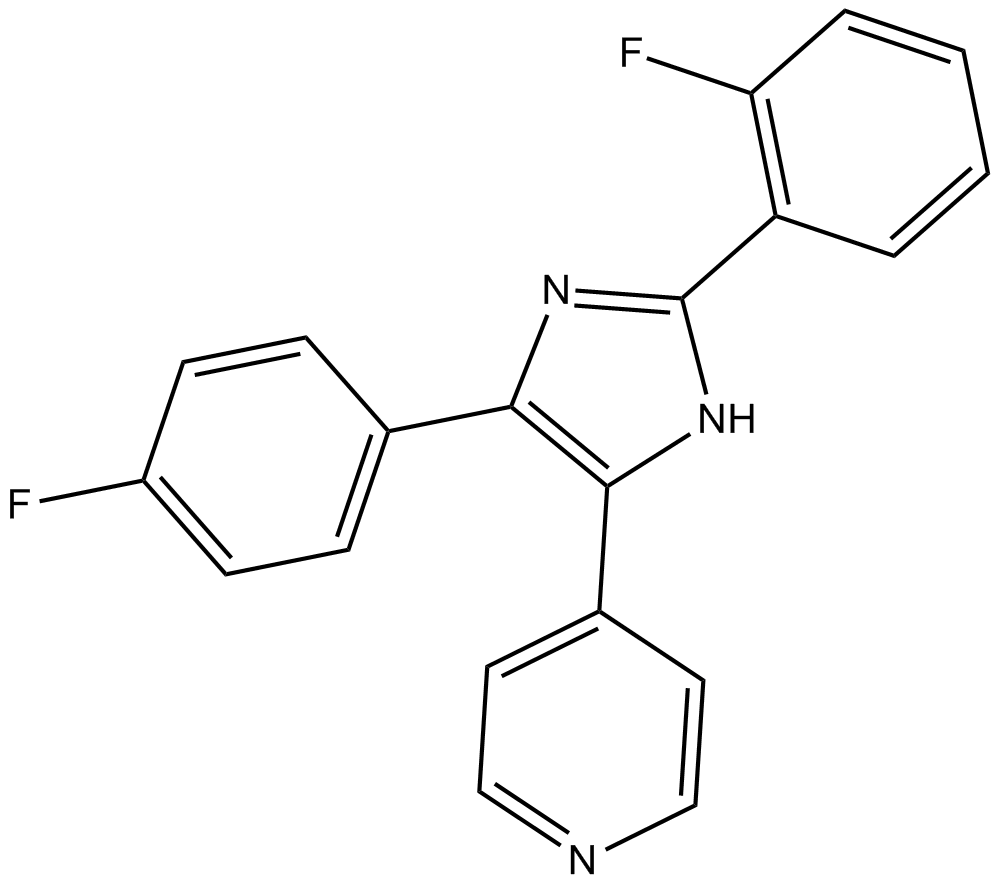
TA 01
TA 01 es un potente inhibidor de CK1 y p38 MAPK, con IC50 de 6,4 nM, 6,8 nM, 6,7 nM para CK1ε, CK1δ y p38 MAPK, respectivamente.
-
GC11635
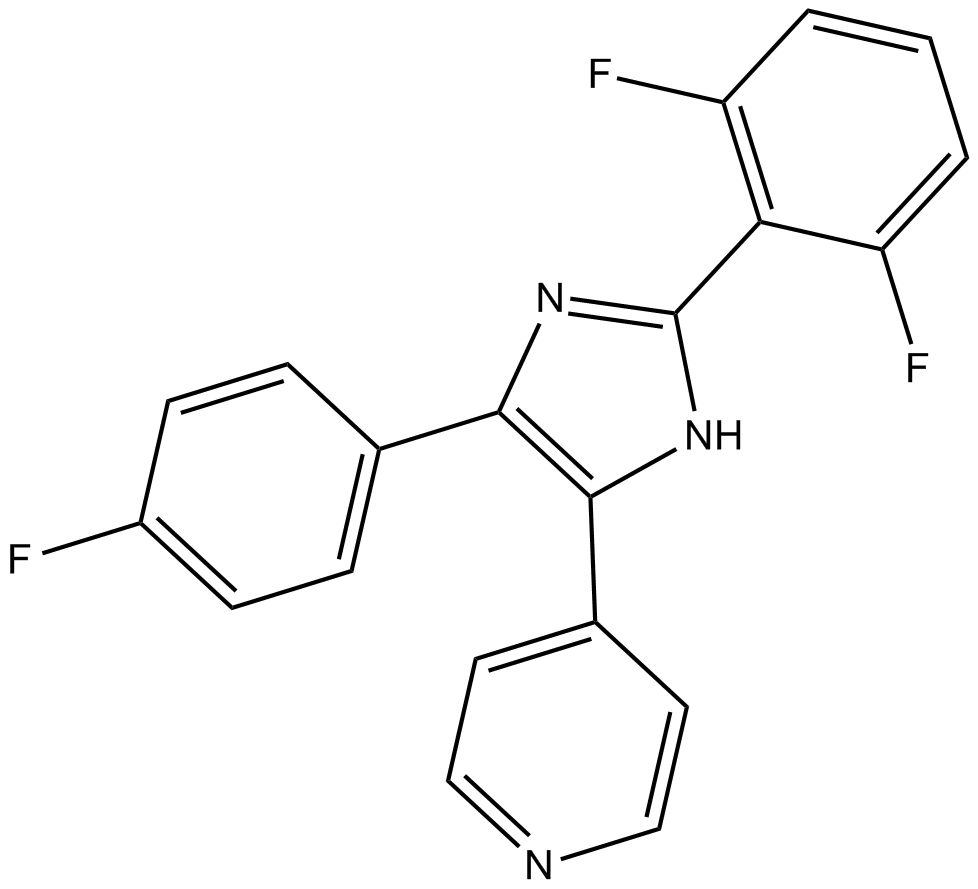
TA 02
TA 02, un análogo de SB 203580, es un inhibidor de p38 MAPK con un IC50 de 20 nM.
-
GC16543
TAK-715
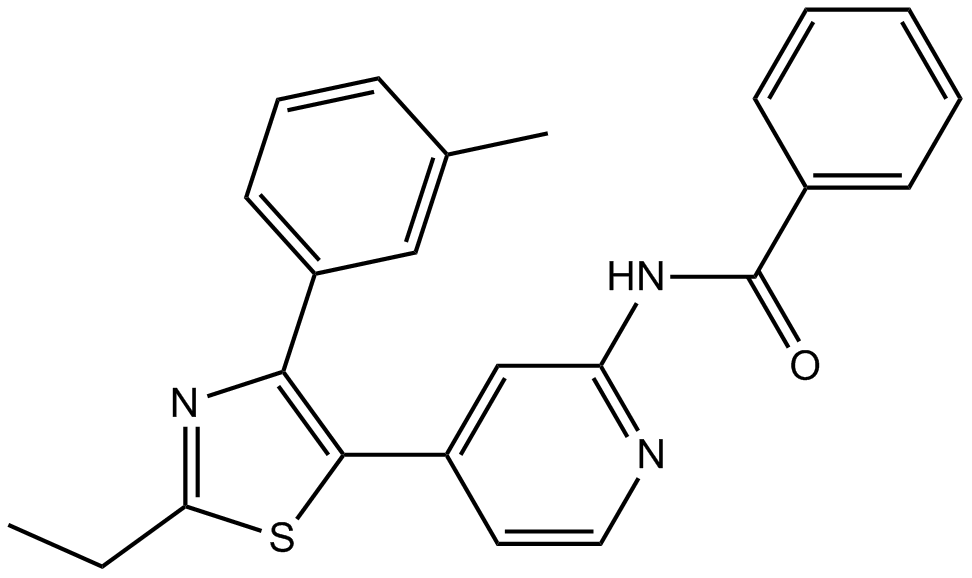
TAK-715 es un potente inhibidor de p38 MAPK activo por vÍa oral con IC50 de 7,1 nM, 200 nM para p38α y p38β, respectivamente.
-
GC10209
TAK-733
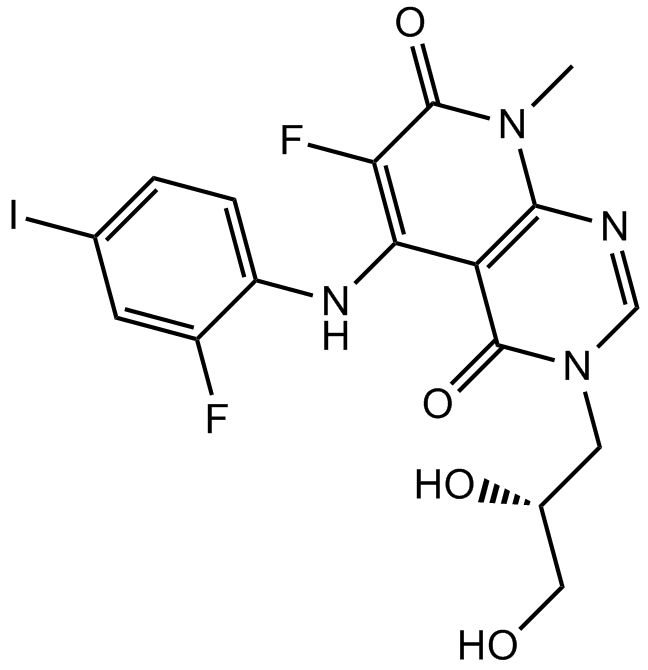
TAK-733 es un inhibidor del sitio alostérico MEK potente y selectivo con una IC50 de 3,2 nM.
-
GC62497
TAK1-IN-2
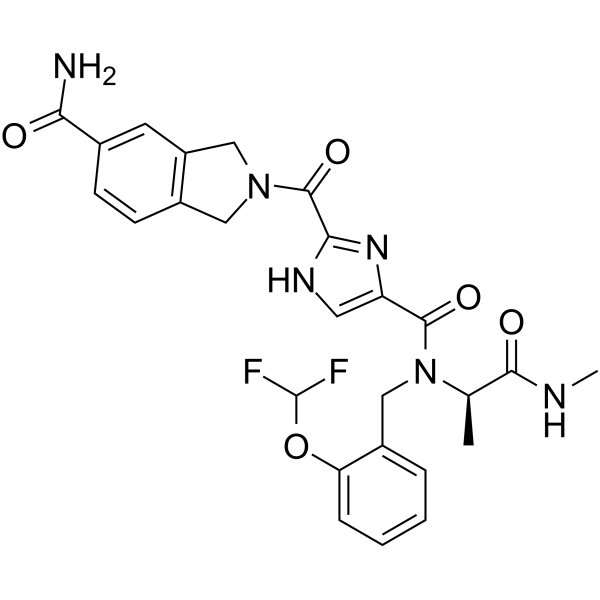
TAK1-IN-2 es un inhibidor potente y selectivo de TAK1, con una IC50> de 2 nM.
-
GC69984
TAK1-IN-4
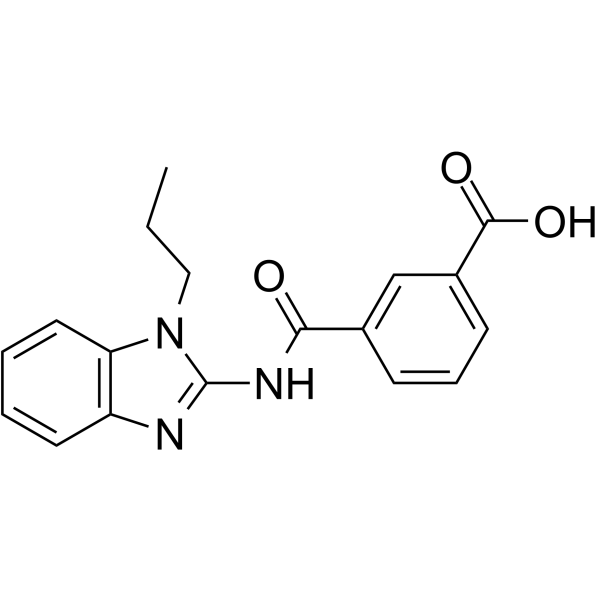
TAK1-IN-4 (Compuesto 14) es un inhibidor de TAK1.
-
GC49700
Takeda-6d
Takeda-6D (compuesto 6d) es un inhibidor de BRAF/VEGFR2 potente y activo por vÍa oral, con valores IC50 de 7,0 y 2,2 nM, respectivamente. Takeda-6D muestra antiangiogénesis al suprimir la vÍa VEGFR2 en 293/KDR y células HUVEC estimuladas por VEGF. Takeda-6D muestra una supresiÓn significativa de la fosforilaciÓn de ERK1/2. Takeda-6D muestra actividad antitumoral.
-
GC32687
Takinib
Takinib (EDHS-206) es un inhibidor de TAK1 selectivo y activo por vÍa oral (IC50 = 9,5 nM), mÁs de 1,5 log mÁs potente que los objetivos clasificados en segundo y tercer lugar, IRAK4 (120 nM) e IRAK1 (390 nM), respectivamente.
-
GC34072
Talmapimod (SCIO-469)
Talmapimod (SCIO-469) (SCIO-469) es un p38α oralmente activo, selectivo y competitivo con ATP; inhibidor con una IC50 de 9 nM. Talmapimod (SCIO-469) muestra una selectividad de aproximadamente 10 veces sobre p38β y una selectividad de al menos 2000 veces sobre un panel de otras 20 quinasas, incluidas otras MAPK.
-
GC25982
Tanzisertib(CC-930)
Tanzisertib (CC-930, JNK-930, JNKI-1) is kinetically competitive with ATP in the JNK-dependent phosphorylation of the protein substrate c-Jun and potent against all isoforms of JNK (Ki(JNK1) = 44 ± 3 nM, IC50(JNK1) = 61 nM, Ki(JNK2) = 6.2 ± 0.6 nM, IC50(JNK2) = 5 nM, IC50(JNK3) = 5 nM) and selective against MAP kinases ERK1 and p38a with IC50 of 0.48 and 3.4 μM respectively.

-
GC34181
Tauroursodeoxycholate (TUDCA)
El Tauroursodeoxicólico (TUDCA) es un inhibidor del estrés del retículo endoplásmico (ER). El Tauroursodeoxicólico (TUDCA) reduce significativamente la expresión de moléculas de apoptosis, como caspasa-3 y caspasa-12. Además, el Tauroursodeoxicólico (TUDCA) también inhibe ERK.
-
GC34831
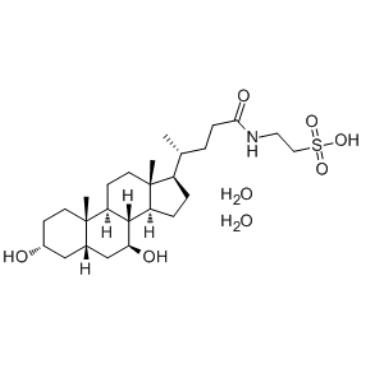
Tauroursodeoxycholate dihydrate
El dihidrato de tauroursodesoxicolato (Ácido tauroursodesoxicÓlico; TDUCA) es un inhibidor del estrés del retÍculo endoplÁsmico (RE). El tauroursodesoxicolato reduce significativamente la expresiÓn de moléculas de apoptosis, como la caspasa-3 y la caspasa-12. El tauroursodesoxicolato también inhibe la ERK.
-
GC34057
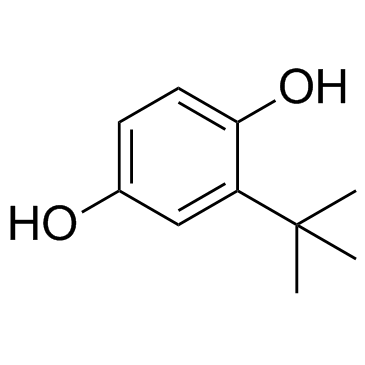
TBHQ (tert-Butylhydroquinone)
TBHQ (tert-Butylhydroquinone) (tert-Butylhydroquinone) es un activador de Nrf2 ampliamente utilizado, protege contra la cardiotoxicidad inducida por doxorrubicina (DOX) a través de la activaciÓn de Nrf2. TBHQ (tert-butilhidroquinona) (tert-butilhidroquinona) también es un activador de ERK; rescata la inhibiciÓn de la proliferaciÓn celular inducida por dehidrocoridalina (DHC) en el melanoma.
-
GC14853
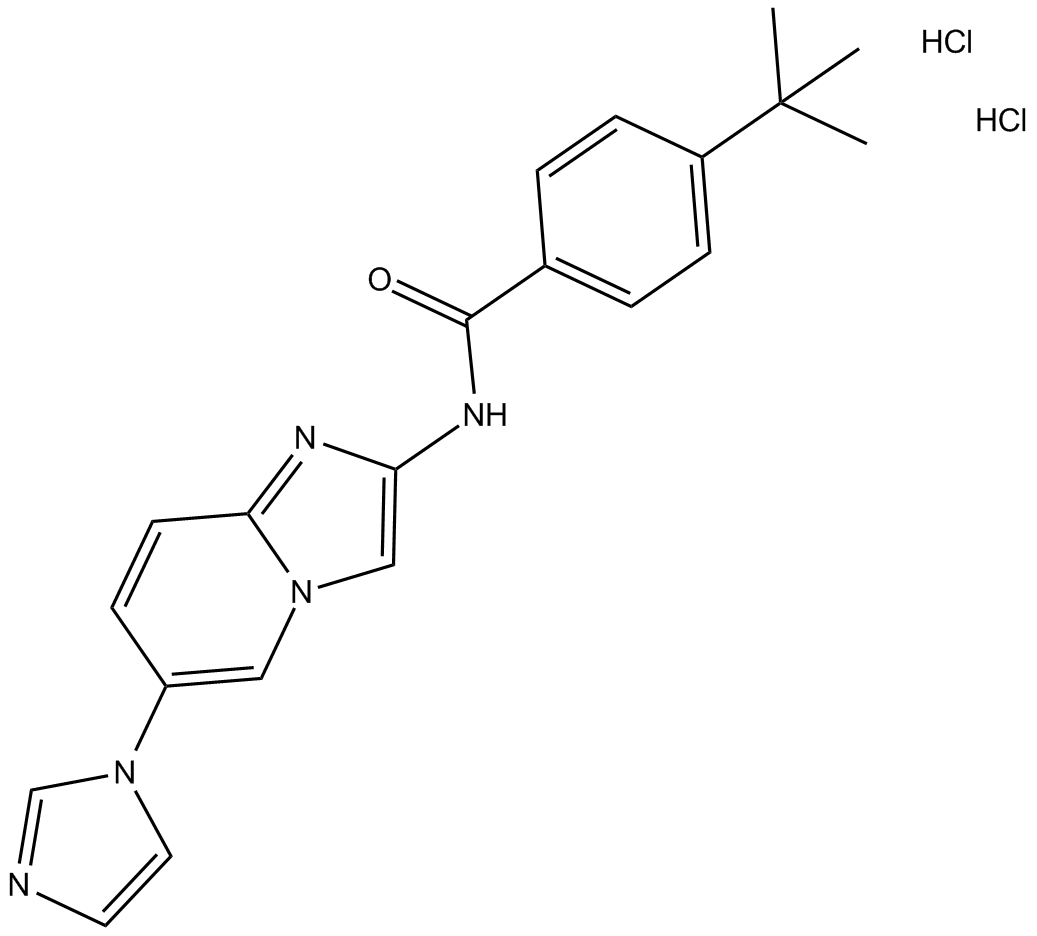
TC ASK 10
TC ASK 10 (Compuesto 10) es un inhibidor potente, selectivo y oralmente activo de la quinasa reguladora de la seÑal de apoptosis 1 (ASK1) con una IC50 de 14 nM. Las actividades inhibidoras de TC ASK 10 frente a otro panel representativo de quinasas son inferiores al 50 %, excepto para ASK2 (IC50 de 0,51 μM).
-
GC34140
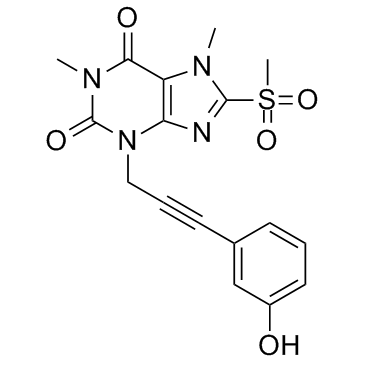
TC13172
TC13172 es un inhibidor de la proteÍna similar al dominio de la quinasa de linaje mixto (MLKL) con un valor EC50 de 2 nM para las células HT-29.
-
GC11488
TCS JNK 5a
TCS JNK 5a es un potente inhibidor de JNK3 con un pIC50 de 6,7. TCS JNK 5a también inhibe JNK2 con un pIC50 de 6,5.
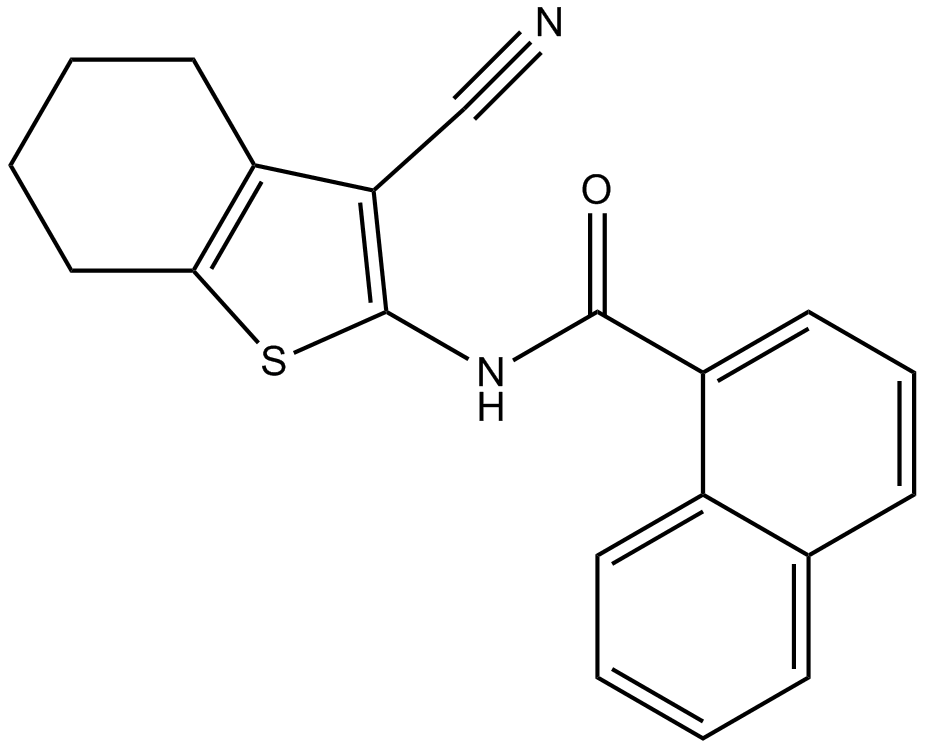
-
GC17282
TCS JNK 6o
TCS JNK 6o (TCS JNK 6o) es un inhibidor de quinasas c-Jun N-terminal (JNK-1, -2 y -3) con valores Ki de 2 nM, 4 nM, 52 nM, respectivamente, y tiene valores IC50 de 45 nM y 160 nM para JNK-1 y -2, respectivamente.
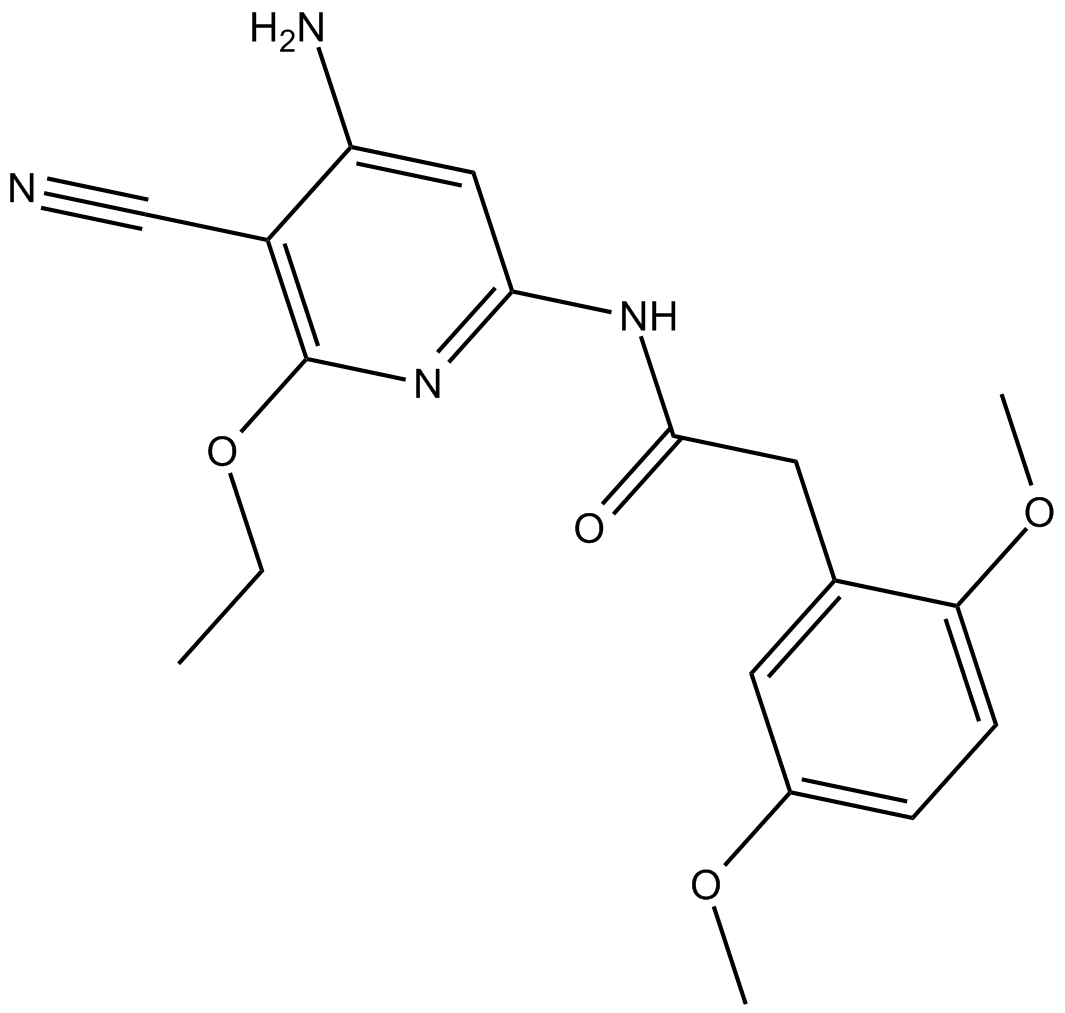
-
GC39074
Tenuifoliside A
Tenuifoliside A se aÍsla de Polygala tenuifolia, tiene efectos antiapoptÓticos y similares a los antidepresivos.
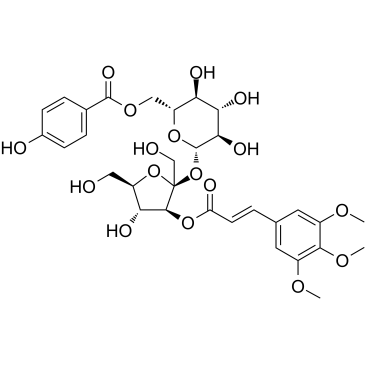
-
GC41573
Theaflavin 3,3'-digallate
El 3,3'-digalato de aflavina (TF-3) es un potente inhibidor de la proteasa del virus Zika (ZIKV) con una IC50 de 2,3 μM.
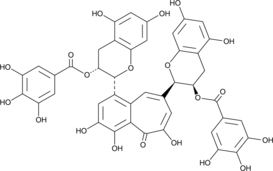
-
GC68350
Tinlorafenib
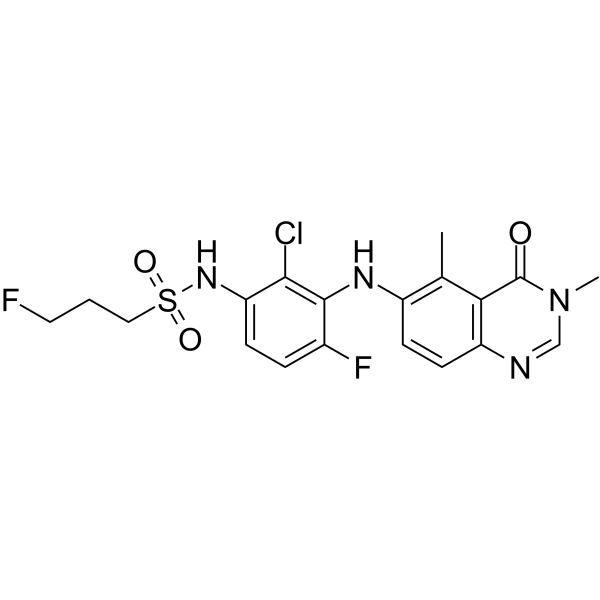
-
GC50295
TL4-12
TL4 12 es un inhibidor potente y selectivo de GCK (cinasa del centro germinal).
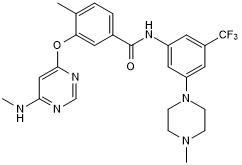
-
GC31647
Tomatidine
La tomatidina actÚa como un agente antiinflamatorio al bloquear la seÑalizaciÓn de NF-κB y JNK.
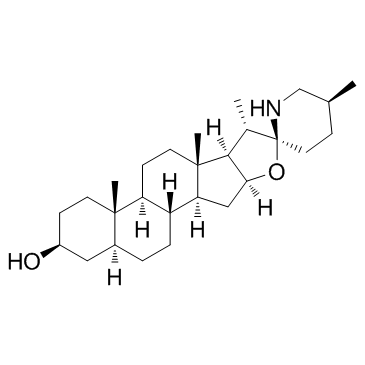
-
GC45064
Tomatidine (hydrochloride)
La tomatidina (clorhidrato) actúa como un agente antiinflamatorio al bloquear la señalización de NF-κB y JNK.
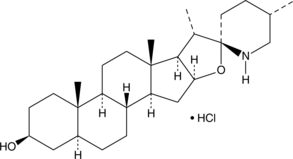
-
GC19131
Tomivosertib
Tomivosertib (eFT508) es un inhibidor de MNK1 y MNK2 potente, altamente selectivo y activo por vÍa oral, con IC50 de 1-2 nM contra ambas isoformas. El tratamiento con tomivosertib (eFT508) conduce a una reducciÓn dependiente de la dosis en la fosforilaciÓn de eIF4E en la serina 209 (IC50 = 2-16 nM) en lÍneas de células tumorales. Tomivosertib (eFT508) también reduce drÁsticamente la abundancia de proteÍna PD-L1.
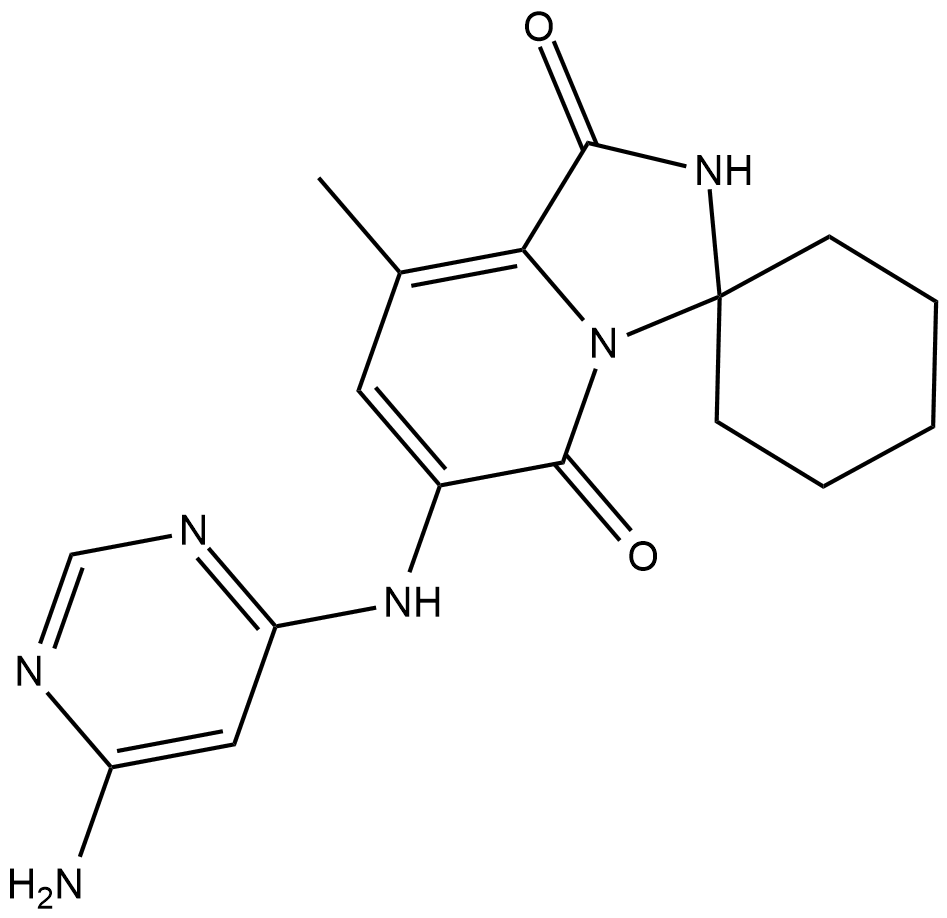
-
GC45067
Tpl2 Kinase Inhibitor
Tpl2 Kinase Inhibitor (Compuesto 1) es un potente y selectivo inhibidor de Tpl2 (COT quinasa, MAP3K8), juega un papel importante en la regulaciÓn de la respuesta inflamatoria y la progresiÓn de algunos tipos de cÁncer.
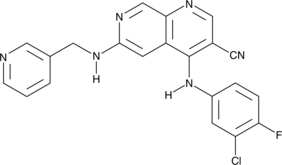
-
GC49150
Tpl2 Kinase Inhibitor (hydrochloride)
A Tpl2 inhibitor
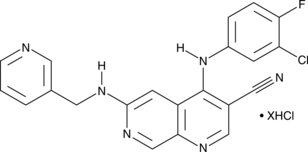
-
GC70053
Trametiglue
Trametiglue es un derivado de Trametinib que, a través de una interacción única en la interfaz, tiene una eficacia y selectividad sin precedentes para dirigirse específicamente a KSR-MEK y RAF-MEK.
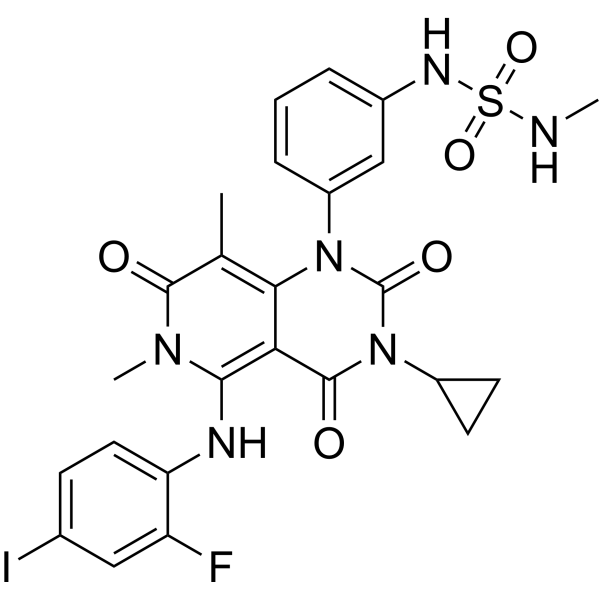
-
GC13508
Trametinib (GSK1120212)
Un inhibidor de MEK1 y MEK2.
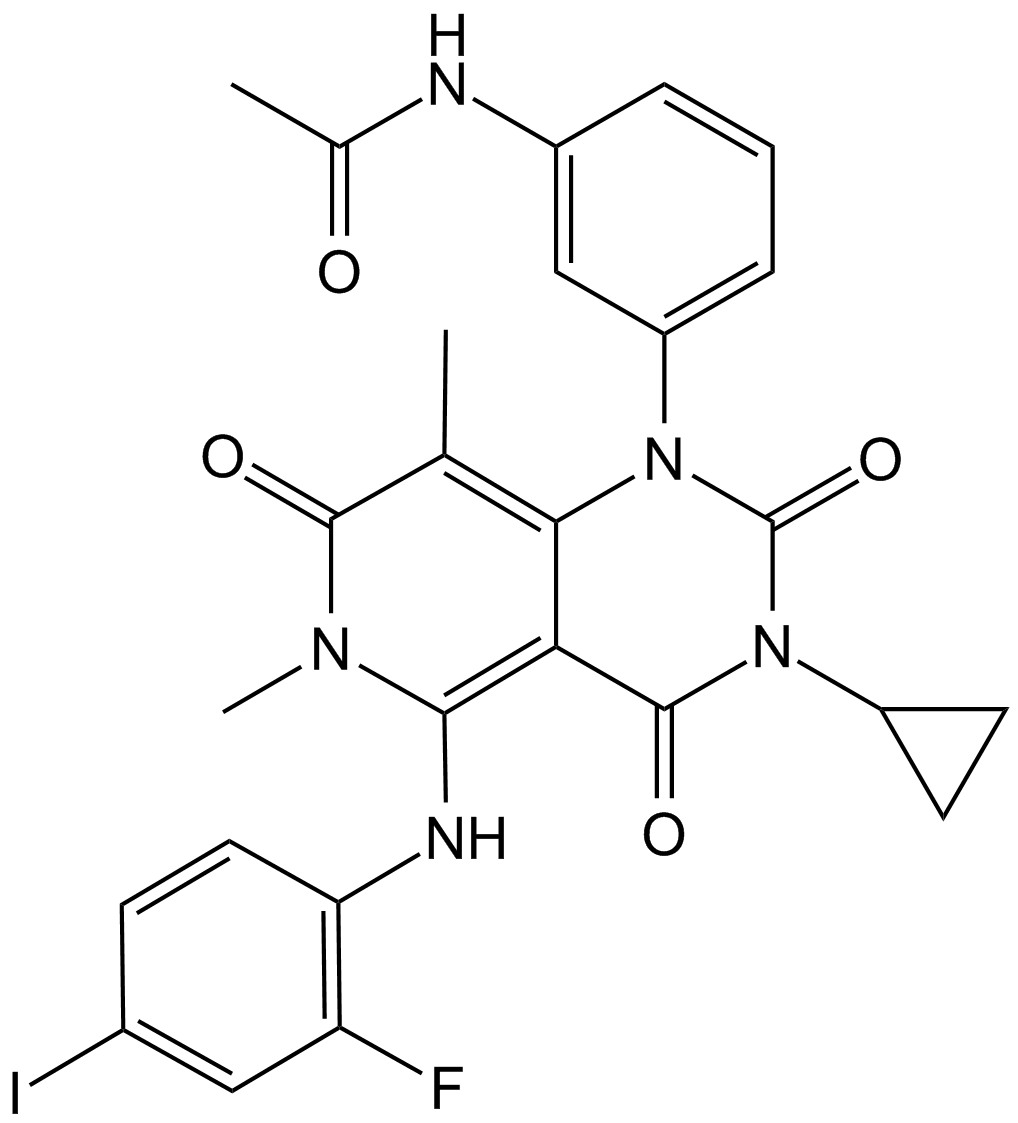
-
GC15260
Trametinib DMSO solvate
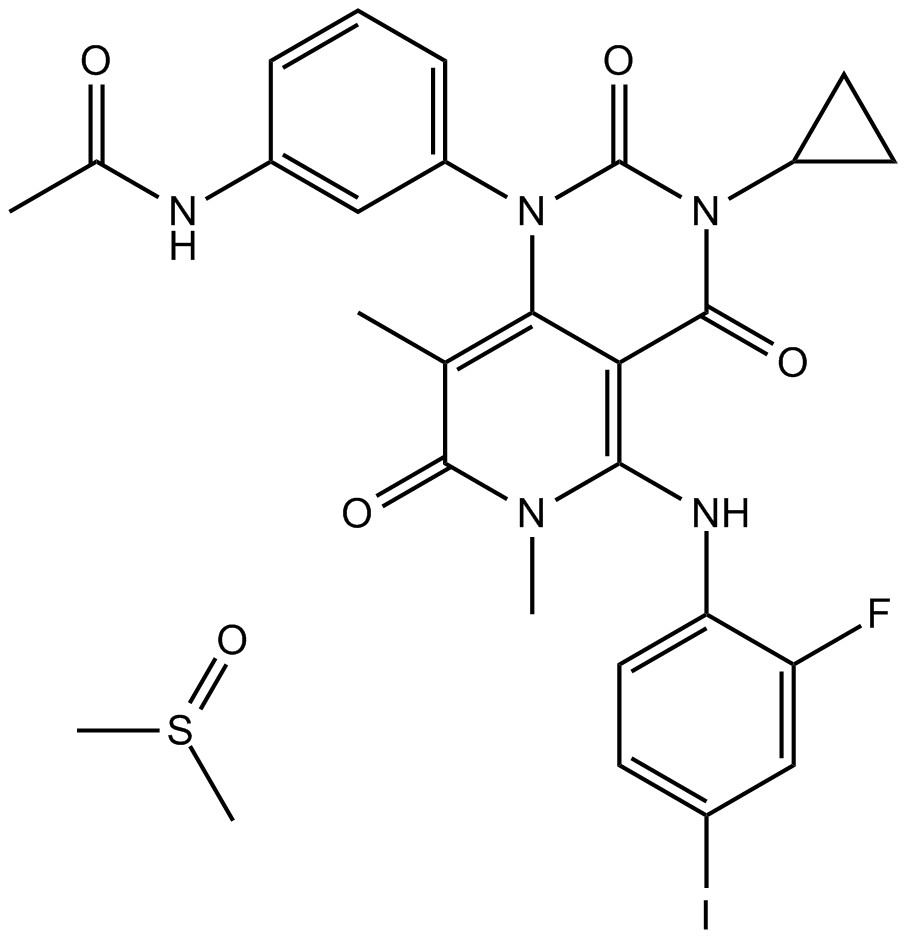
-
GC30162
trans-Zeatin
trans-Zeatin es una citoquinina vegetal, que juega un papel importante en el crecimiento, la diferenciaciÓn y la divisiÓn celular; trans-Zeatin también inhibe la activaciÓn de MEK/ERK inducida por UV.
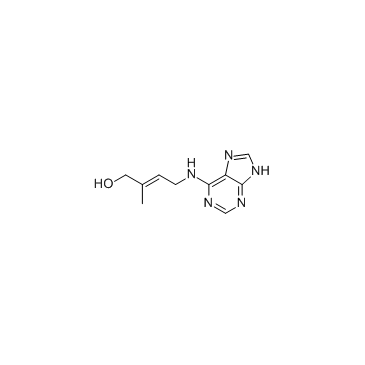
-
GC45099
U-0126
Un inhibidor de MEK
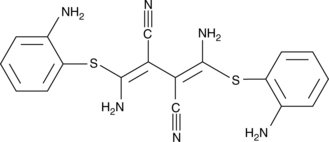
-
GC15061
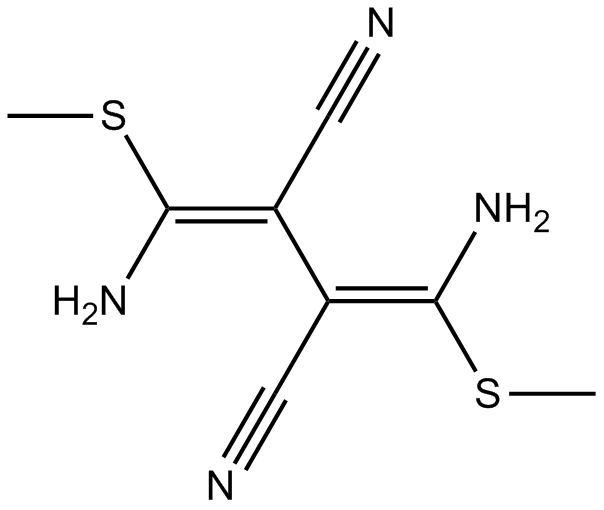
U0124
U0124, un anÁlogo inactivo de U0126, no tiene efecto sobre los niveles de proteÍna o ARNm de c-Fos y c-Jun. U0126 es un inhibidor de MEK. U0124 no inhibe MEK en concentraciones de hasta 100 μM.
-
GC12807
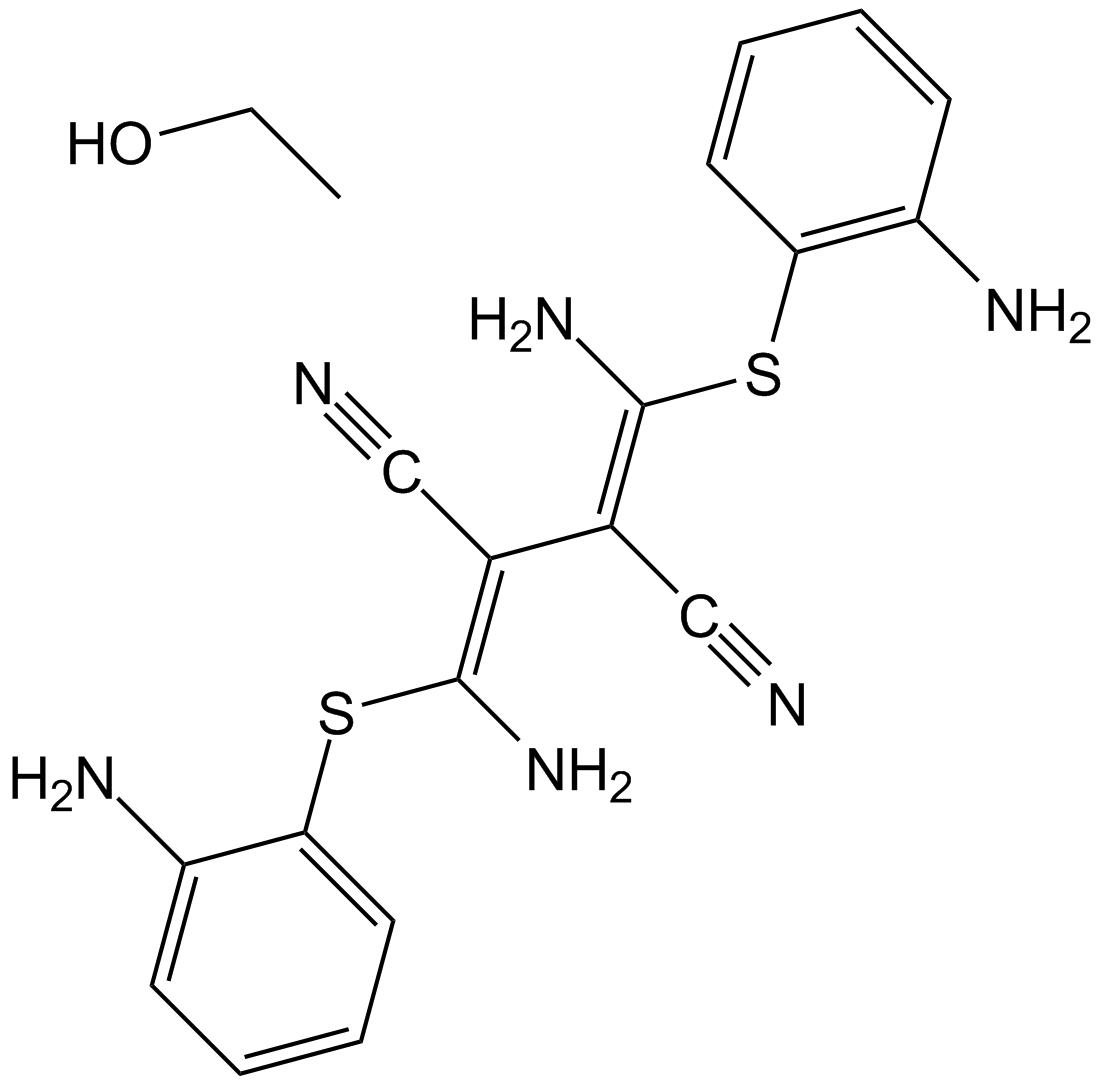
U0126-EtOH
Para MEK1y MEK2, U0126- etoh - etoh (U0126) tiene un fuerte efecto inhibidor, ic50 es repectivamente 72nM y 58nM.
-
GC16678
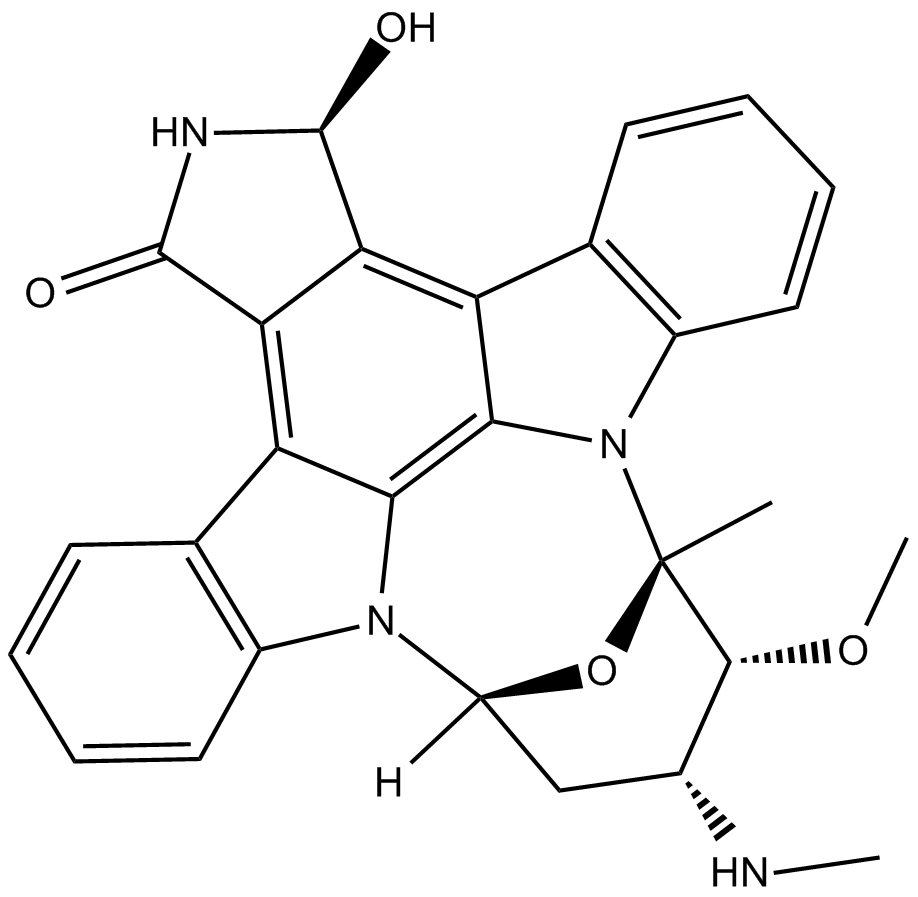
UCN-02
UCN-02 (7-epi-Hydroxystaurosporine) es un inhibidor selectivo de la proteÍna quinasa C (PKC) producido por la cepa N-12 de Streptomyces, con IC50 de 62 nM y 250 nM para PKC y proteÍna quinasa A (PKA), respectivamente. UCN-02 (7-epi-Hydroxystaurosporine) muestra un efecto citotÓxico en el crecimiento de las células HeLa S3.
-
GC11422
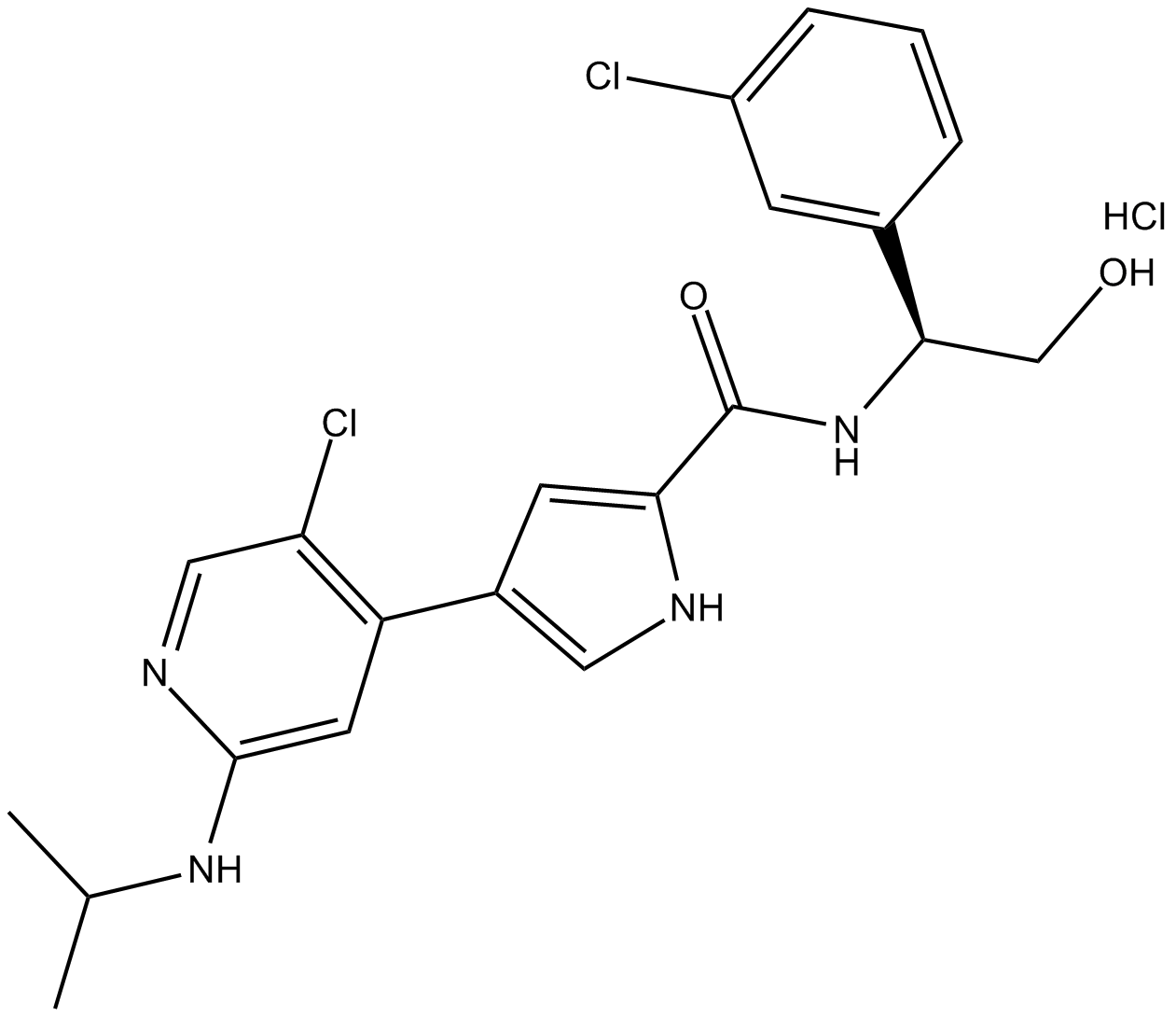
Ulixertinib (hydrochloride)
Ulixertinib (clorhidrato) (clorhidrato de BVD-523) es un inhibidor covalente potente, activo por vía oral, altamente selectivo, competitivo con ATP y reversible de las quinasas ERK1/2, con una IC50 de \u003c0,3 nM contra ERK2. Ulixertinib (clorhidrato) inhibe el ERK2 fosforilado (pERK) y la quinasa RSK corriente abajo (pRSK) en una línea celular de melanoma A375.
-
GC19366
UM-164
UM-164 (DAS-DFGO-II) es un inhibidor muy potente de c-Src con una Kd de 2,7 nM. UM-164 también inhibe potentemente p38α y p38β.
-
GC15004
URMC-099
URMC-099 es un potente inhibidor de la quinasa de linaje mixto tipo 3 (MLK3) (IC50 = 14 nM) biodisponible por vÍa oral con excelentes propiedades de penetraciÓn de la barrera hematoencefÁlica.
-
GC38679
Urolithin B
La urolitina B es uno de los metabolitos microbianos intestinales de los elagitaninos y tiene efectos antiinflamatorios y antioxidantes.
-
GC17818
Vacquinol-1
Vacquinol-1 (NSC13316) es un activador especÍfico de MKK4 que activa las vÍas MAPK. Vacquinol-1 induce especÍficamente la muerte de células de glioblastoma humano (GC), atenÚa la progresiÓn tumoral y prolonga la supervivencia en un modelo de ratÓn con glioblastoma multiforme (GBM). Vacquinol-1 también induce la apoptosis en células de carcinoma hepatocelular (HCC).
-
GC13412
Vemurafenib (PLX4032, RG7204)
Un inhibidor de la mutante V600E y del tipo salvaje B-Raf.
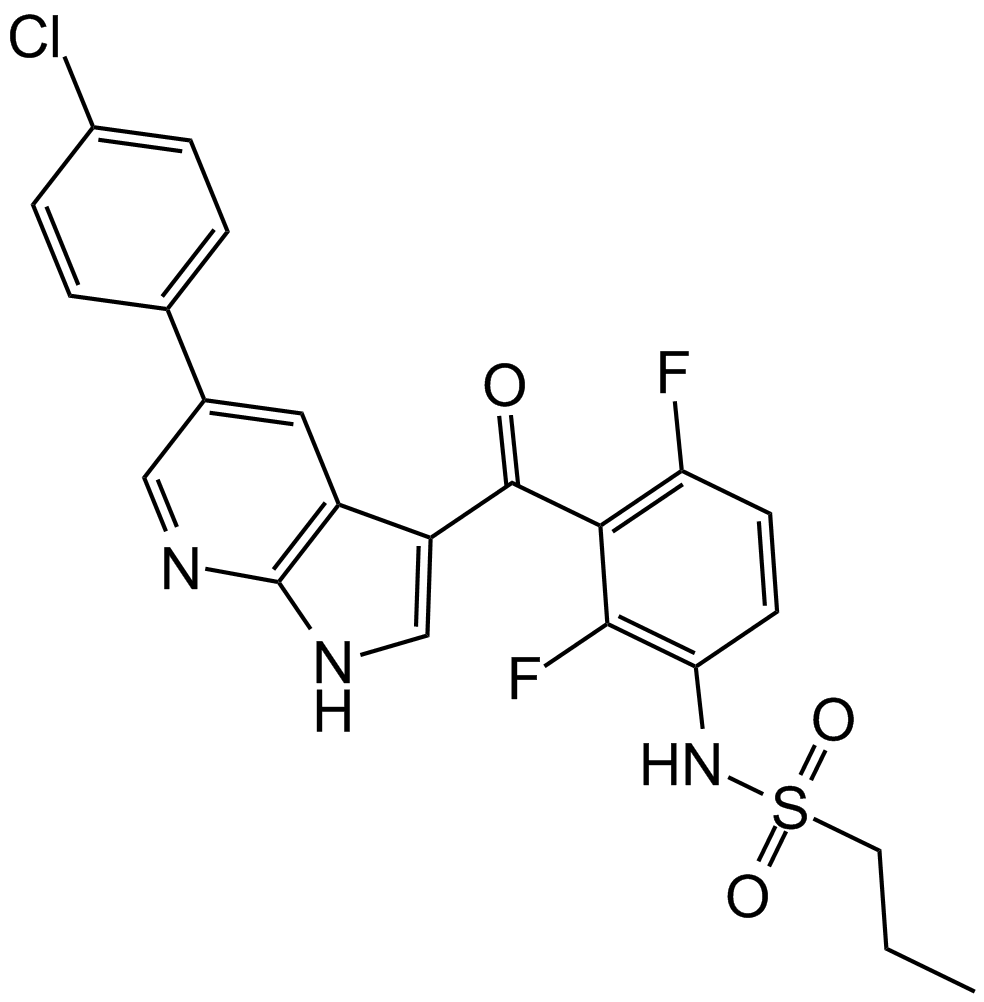
-
GC17031
VRT752271
VRT752271 (BVD-523; VRT752271) es un inhibidor covalente reversible, potente, activo por vÍa oral, altamente selectivo, competitivo con ATP de las quinasas ERK1/2, con una IC50 de <0,3 nM contra ERK2. VRT752271 (BVD-523; VRT752271) inhibe el ERK2 fosforilado (pERK) y la quinasa RSK corriente abajo (pRSK) en una lÍnea celular de melanoma A375.
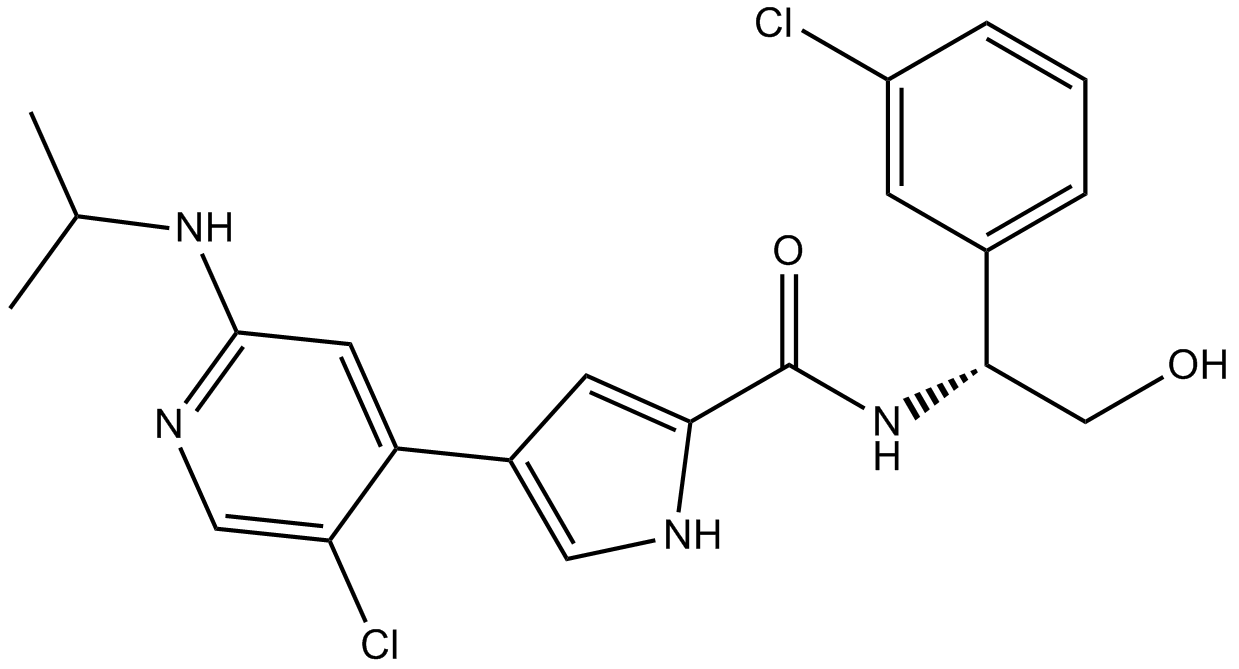
-
GC13115
VX-11e
A selective ERK inhibitor
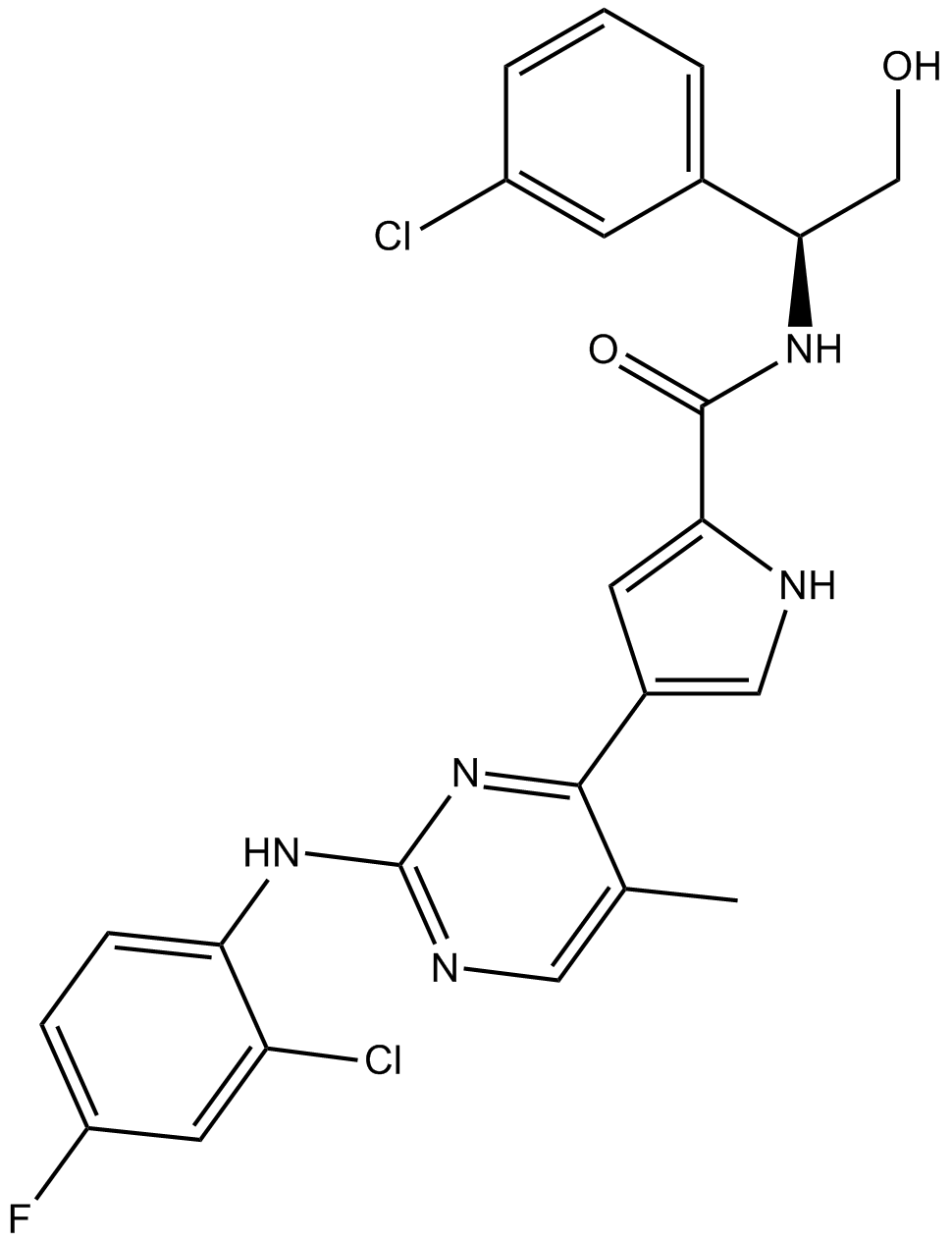
-
GC17508
VX-702
VX-702 es un inhibidor altamente selectivo de p38α MAPK, 14 veces mÁs potente contra p38α que contra p38β.
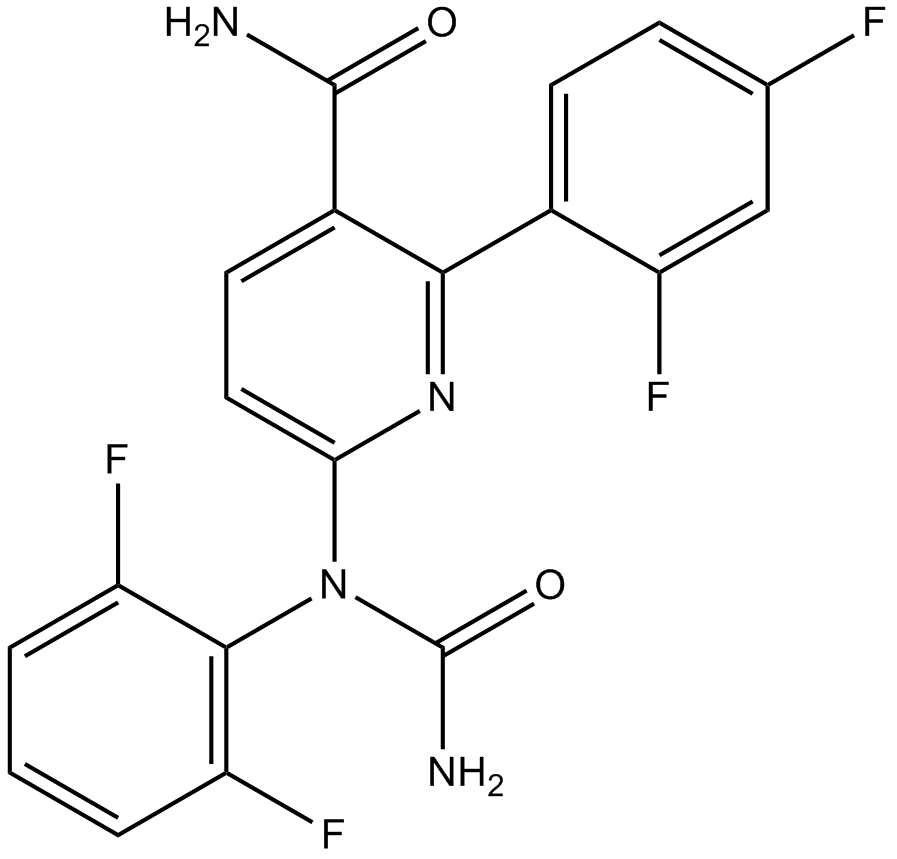
-
GC12926
VX-745
VX-745 (VX-745) es un potente inhibidor altamente selectivo que penetra la barrera hematoencefÁlica de p38α inhibidor con IC50 para p38α de 10 nM y para p38β de 220 nm.
-
GC30857
Warangalone (Scandenolone)
Warangalone (Scandenolone) es un compuesto contra la malaria que puede inhibir el crecimiento de ambas cepas del parÁsito 3D7 (sensible a la cloroquina) y K1 (resistente a la cloroquina) con IC50 de 4.8 μg/mL y 3.7 μg/mL, respectivamente.
-
GC38053
WHI-P258
WHI-P258, un compuesto de quinazolina, se une al sitio activo de JAK3 con una Ki estimada de 72 μM. WHI-P258 no inhibe JAK3 y no afecta la agregaciÓn de plaquetas inducida por trombina incluso a 100 μM.
-
GC12691
XMD17-109
A selective ERK5 inhibitor
-
GC11076
XMD8-92
A selective ERK5 inhibitor
-
GC16169
XRP44X
XRP44X inhibe la activaciÓn de la transcripciÓn inducida por Ras con el IC50 de 10 nM.
-
GC62146
XST-14
XST-14 es un inhibidor de ULK1 potente, competitivo y altamente selectivo con una IC50 de 26,6 nM. XST-14 induce la inhibiciÓn de la autofagia al reducir la fosforilaciÓn del sustrato aguas abajo de ULK1. XST-14 induce la apoptosis en células de carcinoma hepatocelular (CHC) y tiene efectos antitumorales.
-
GC63264
Zapnometinib
Zapnometinib (PD0184264), un metabolito activo de CI-1040, es un inhibidor de MEK, con una IC50 de 5,7 nM.
-
GC13286
ZM336372
ZM336372 es un potente inhibidor de la proteína quinasa c-Raf. El valor IC50 es 0,07 μM en el ensayo estándar, que contiene ATP 0,1 mM.
-
GC63425
Zunsemetinib
Zunsemetinib (CDD-450) es un inhibidor de la vÍa de la proteÍna quinasa 2 (MK2) activada por mitÓgeno p38α selectivo y activo por vÍa oral.