MAPK Signaling

Targets for MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Products for MAPK Signaling
- Cat.No. Nom du produit Informations
-
GC13142
RWJ 67657
RWJ 67657 (JNJ 3026582) est un inhibiteur de MAPK p38α et p38β actif et sélectif par voie orale avec des IC50 de 1 et 11 μM, respectivement.
-
GC18273
SB 202190 (hydrochloride)
SB 202190 (chlorhydrate) est un inhibiteur sélectif de la p38 MAP kinase avec des IC50 de 50 nM et 100 nM pour p38α et p38β2, respectivement.
-
GC16019
SB 202474
SB 202474, un analogue négatif de SB203580. Le SB 202474, qui n'a pas la capacité d'inhiber l'activité de p38 MAPK et est largement utilisé comme composé témoin négatif dans les études de p38 MAPK, a également supprimé l'induction de la synthèse de mélanine.
-
GC13001
SB 203580 hydrochloride
Le chlorhydrate d'adezmapimod (SB 203580) est un inhibiteur sélectif et compétitif de la MAPK p38 avec des CI50 de 50 nM et 500 nM pour SAPK2a/p38 et SAPK2b/p38β2, respectivement.
-
GC10054
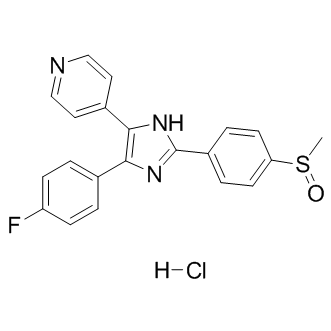
SB 239063
A selective p38 MAPK inhibitor
-
GC37595
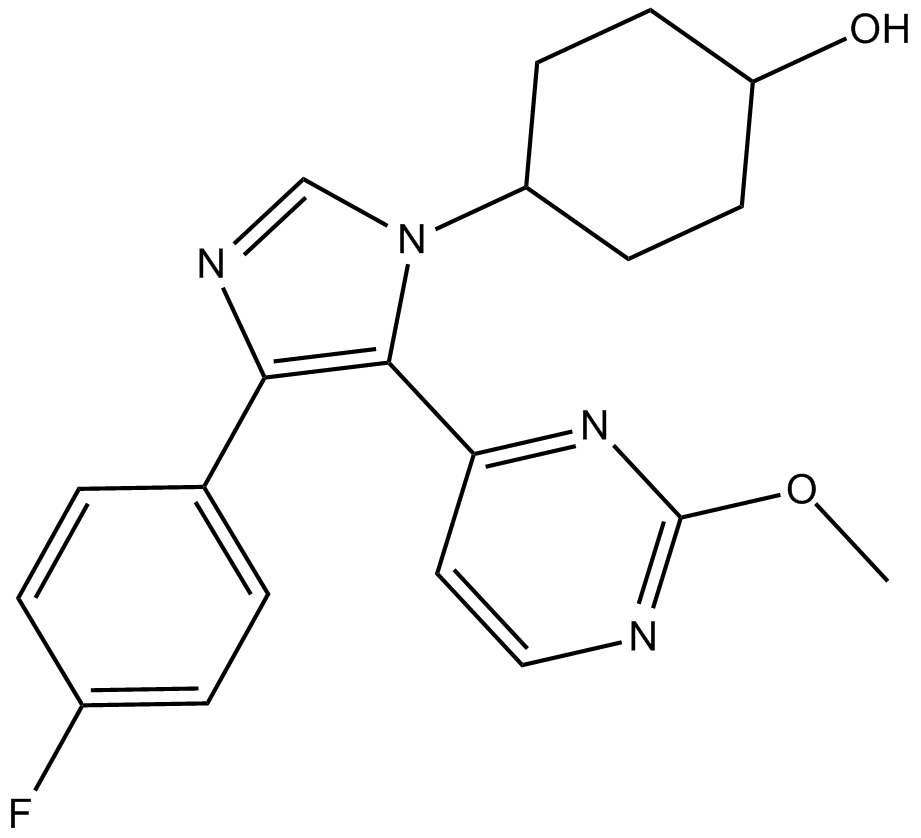
SB 242235
Le SB-242235 est un inhibiteur puissant et sélectif de la p38 MAP kinase, avec une IC50 de 1,0 μM dans les chondrocytes humains primaires.
-
GC11922
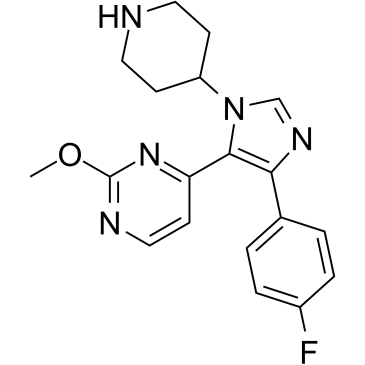
SB 706504
p38 MAPK inhibitor
-
GC13968
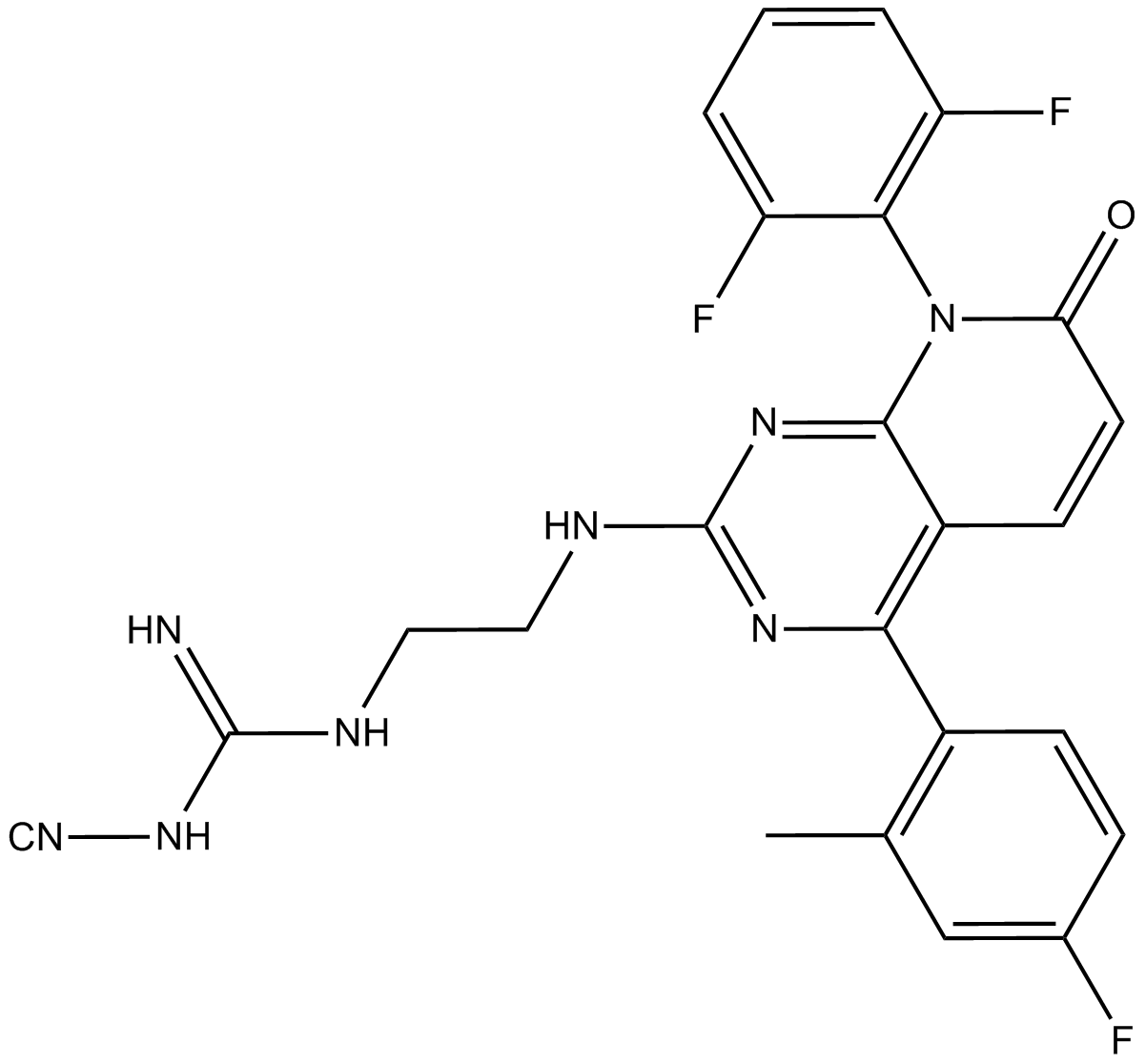
SB202190 (FHPI)
SB202190 (FHPI) est un inhibiteur sélectif de la kinase p38 MAP avec des IC50 de 50 nM et 100 nM pour p38α et p38β2, respectivement.
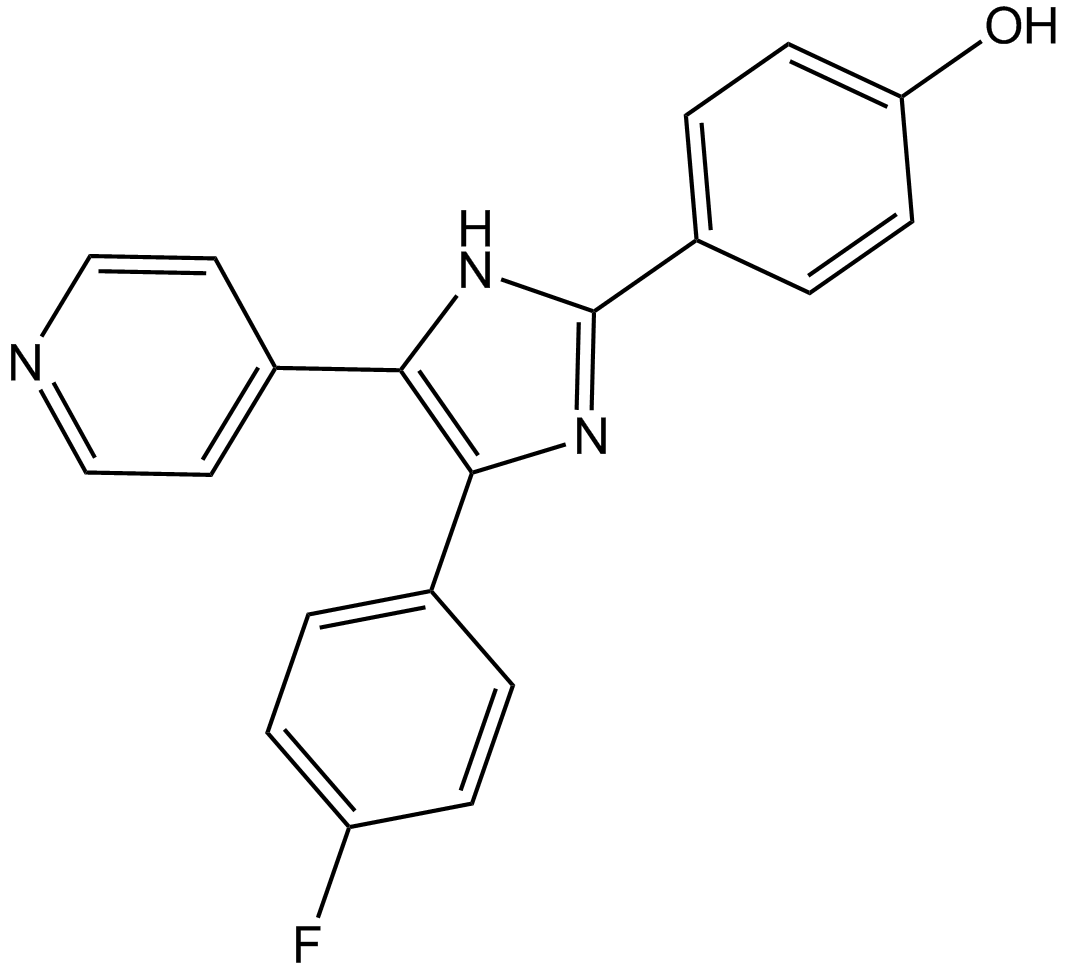
-
GC11890
SB590885
Le SB590885 est un puissant inhibiteur de B-Raf avec un Ki de 0,16 nM et a une sélectivité 11 fois plus élevée pour B-Raf que c-Raf, sans inhibition des autres kinases humaines.
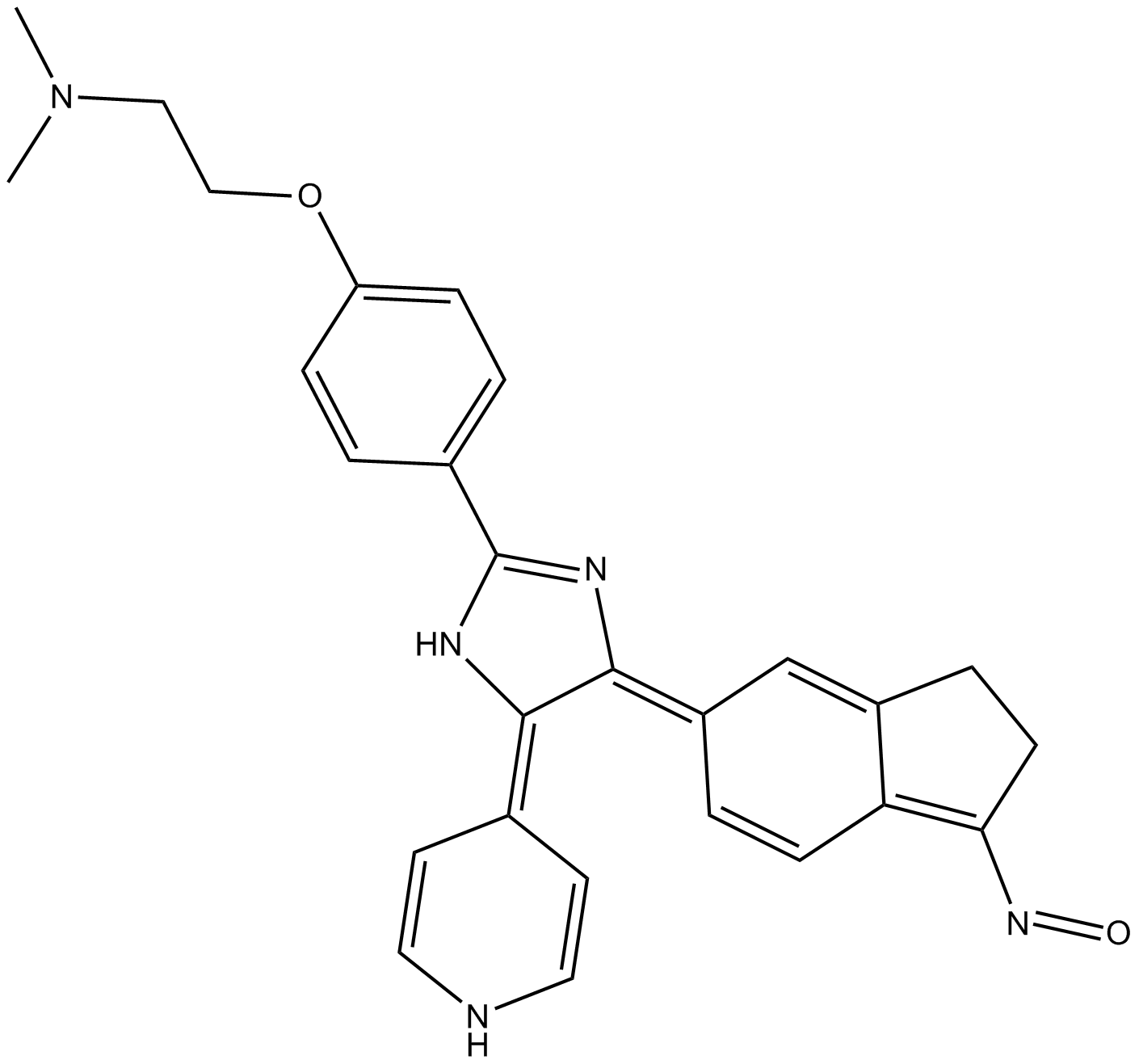
-
GC16001
SCH772984
Un inhibiteur sélectif d'ERK1/2
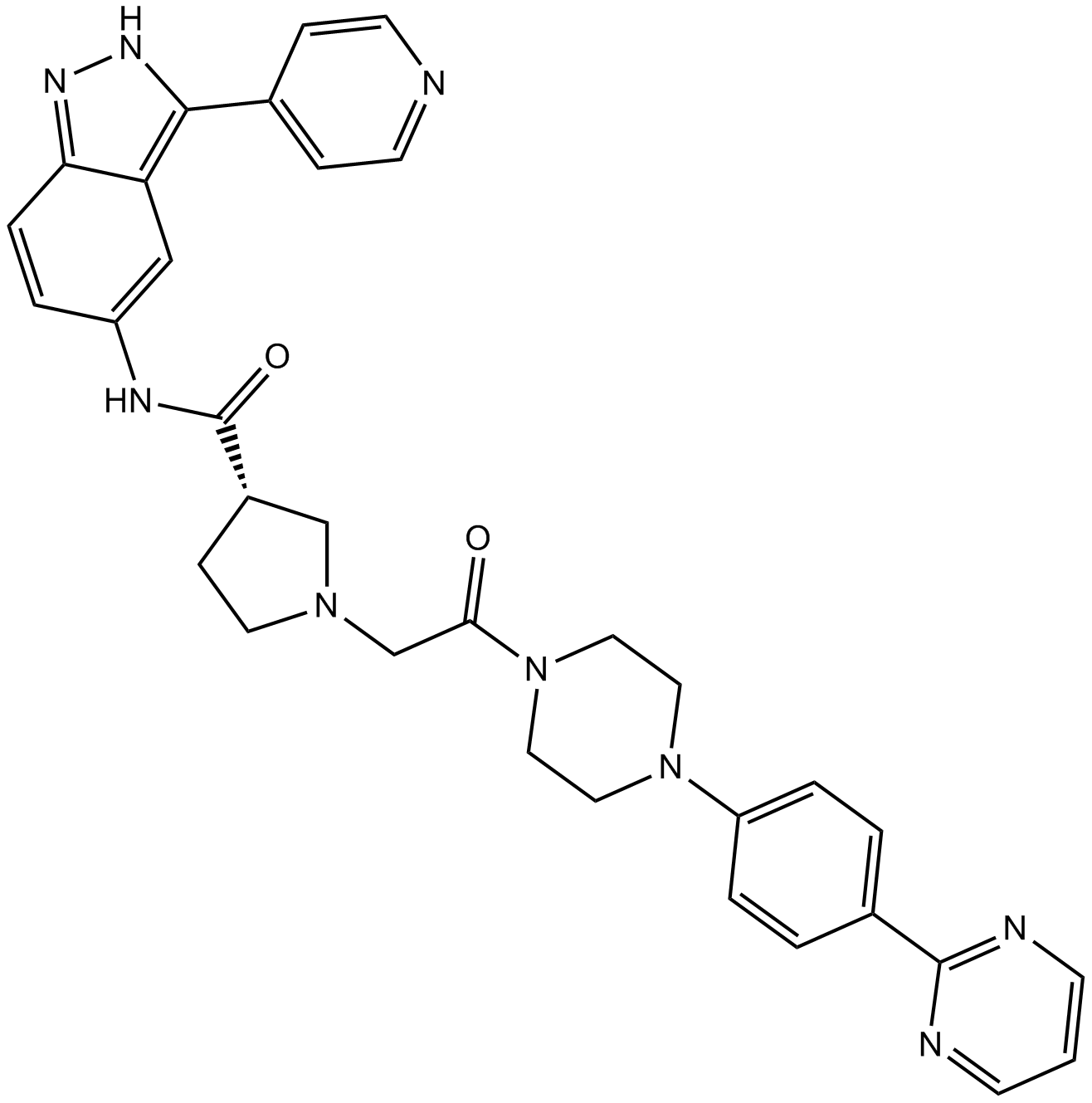
-
GC14647
SCH772984 HCl
ERK1/2 inhibitor
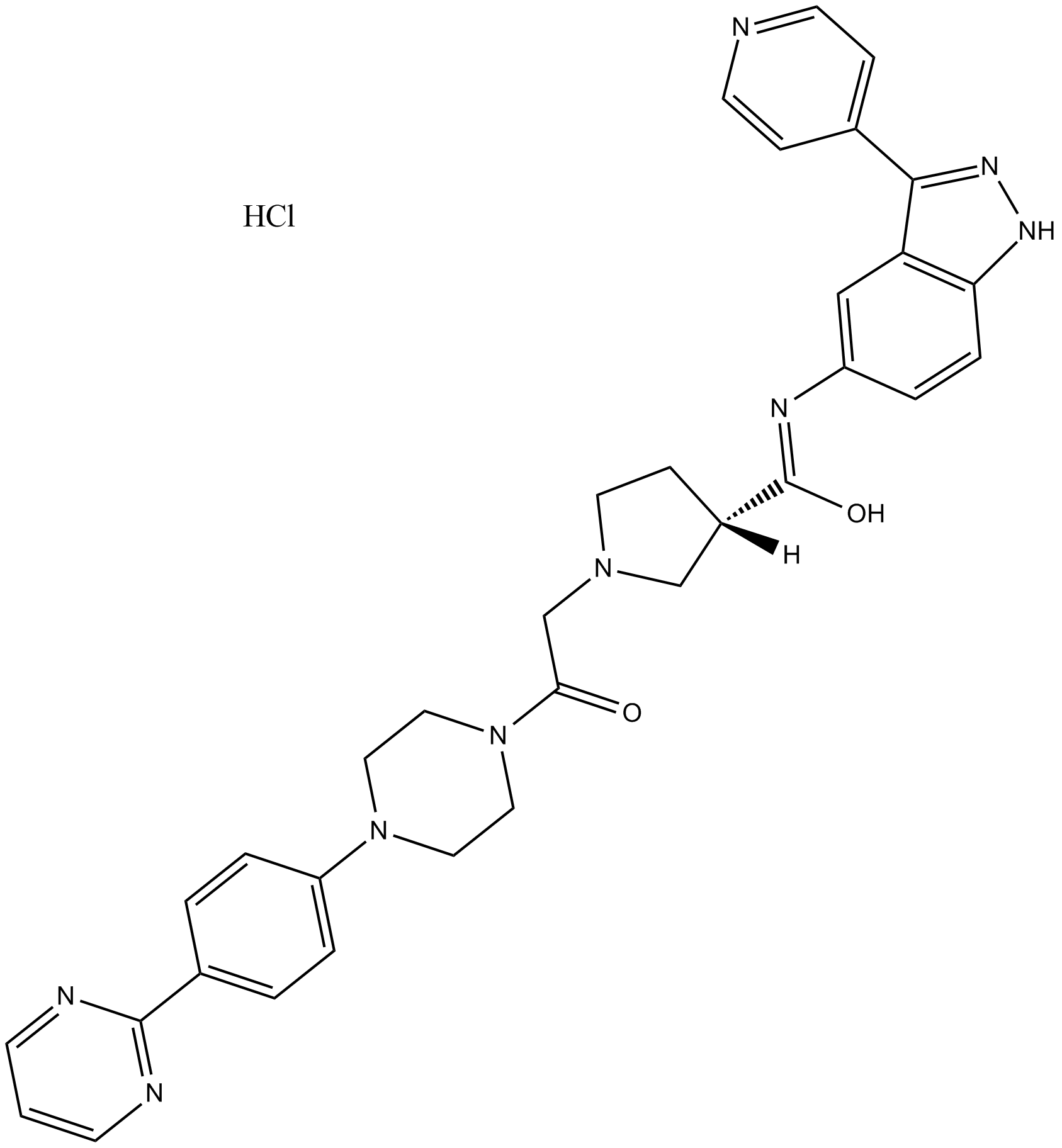
-
GC15044
SCH772984 TFA
ERK1/2 inhibitor
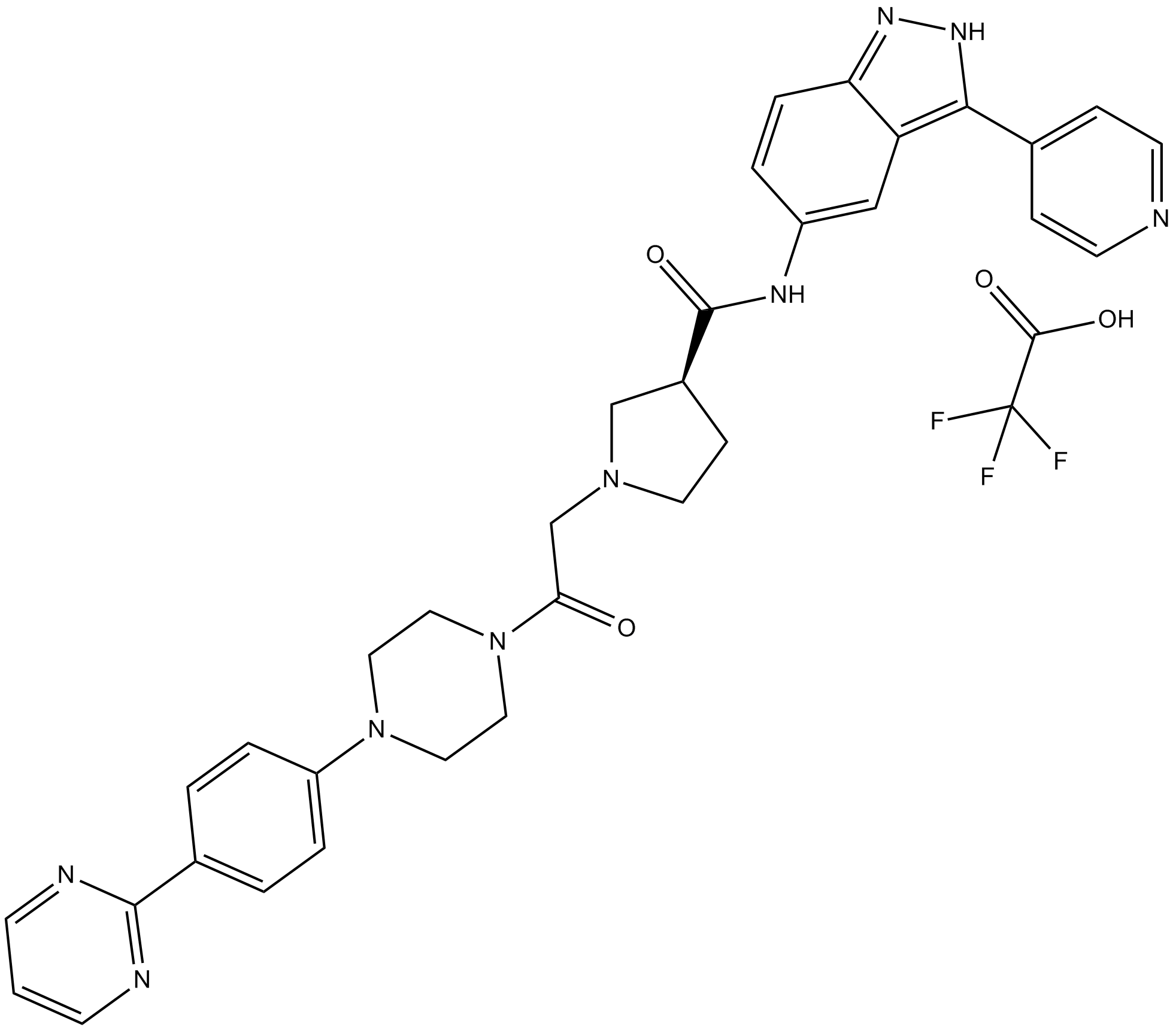
-
GC19325
SD 0006
SD 0006 (SD-06) est un inhibiteur diaryl pyrazole actif, sélectif, compétitif pour l'ATP et puissant de la p38α MAP kinase, avec une IC50 de 110 nM pour p38α .
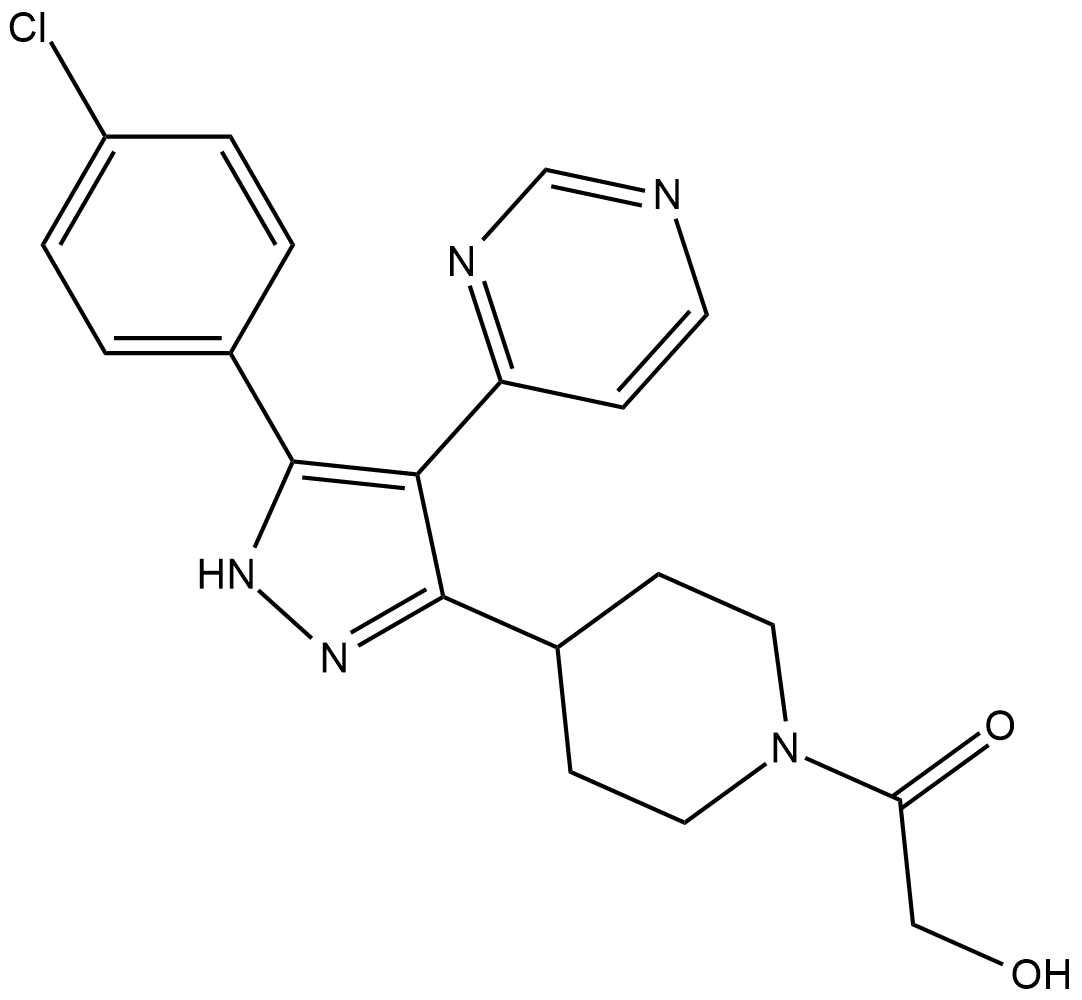
-
GC14982
SD 169
Le SD 169 est un inhibiteur compétitif de l'ATP actif par voie orale de p38α MAPK, avec une IC50 de 3,2 nM.
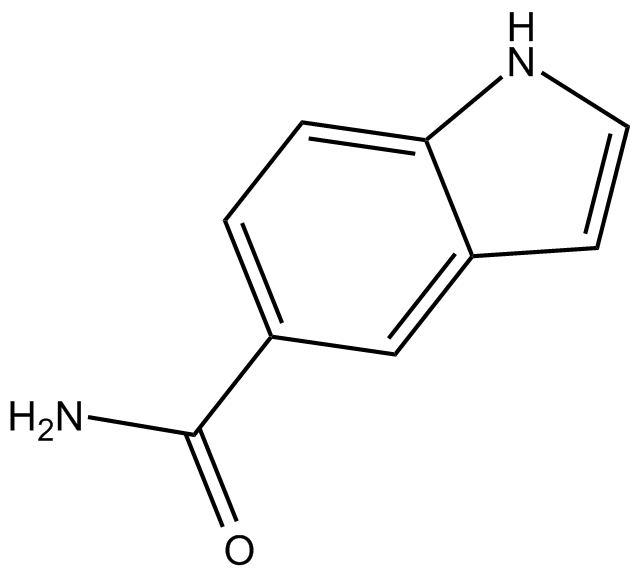
-
GC12124
Selonsertib (GS-4997)
Selonsertib (GS-4997) (GS-4997), un inhibiteur sélectif de la kinase 1 (ASK1) régulatrice du signal d'apoptose biodisponible par voie orale avec un pIC50 de 8,3, a été évalué comme traitement expérimental de la néphropathie diabétique et de la fibrose rénale.
-
GC38653
Selumetinib sulfate
Le sélumétinib (AZD6244) est un inhibiteur oral sélectif de MEK1/2 non compétitif pour l'ATP, avec une CI50 de 14nM pour MEK1. Le sélumétinib (AZD6244) inhibe la phosphorylation de ERK1/2.
-
GC39815
Semapimod tetrahydrochloride
Le tétrachlorhydrate de sémapimod (CNI-1493), un inhibiteur de la production de cytokines pro-inflammatoires, peut inhiber le TNF-α, l'IL-1β et l'IL-6.
-
GC10005
SEP-0372814
A PDE10A inhibitor
-
GC33229
SJFα
SJF&alpha ; est un lieur PROTAC à 13 atomes basé sur le ligand de von Hippel-Lindau. SJF&alpha ; dégrade p38α avec un DC50 de 7,16 nM, mais est beaucoup moins efficace pour dégrader p38δ (DC50\u003d299 nM) et ne dégrade pas les autres isoformes de p38 (β et γ) à des concentrations allant jusqu'à 2,5 µM.
-
GC33238
SJFδ
SJF&delta ; est un lieur PROTAC à 10 atomes basé sur le ligand de von Hippel-Lindau. SJF&delta ; dégrade p38δ avec un DC50 de 46,17 nM, mais ne dégrade pas p38α, p38β ou p38γ.
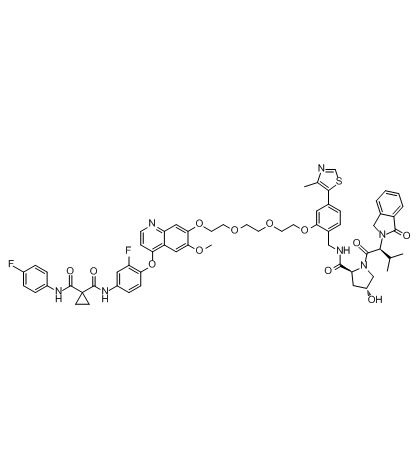
-
GC30646
Skatole(3-Methylindole)
Le scatole (3-méthylindole) est produit par des bactéries intestinales, régule les fonctions cellulaires épithéliales intestinales en activant les récepteurs d'hydrocarbures aryliques et p38.
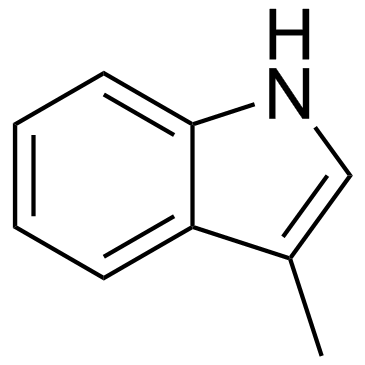
-
GC13578
Skepinone-L
An inhibitor of p38 MAPK
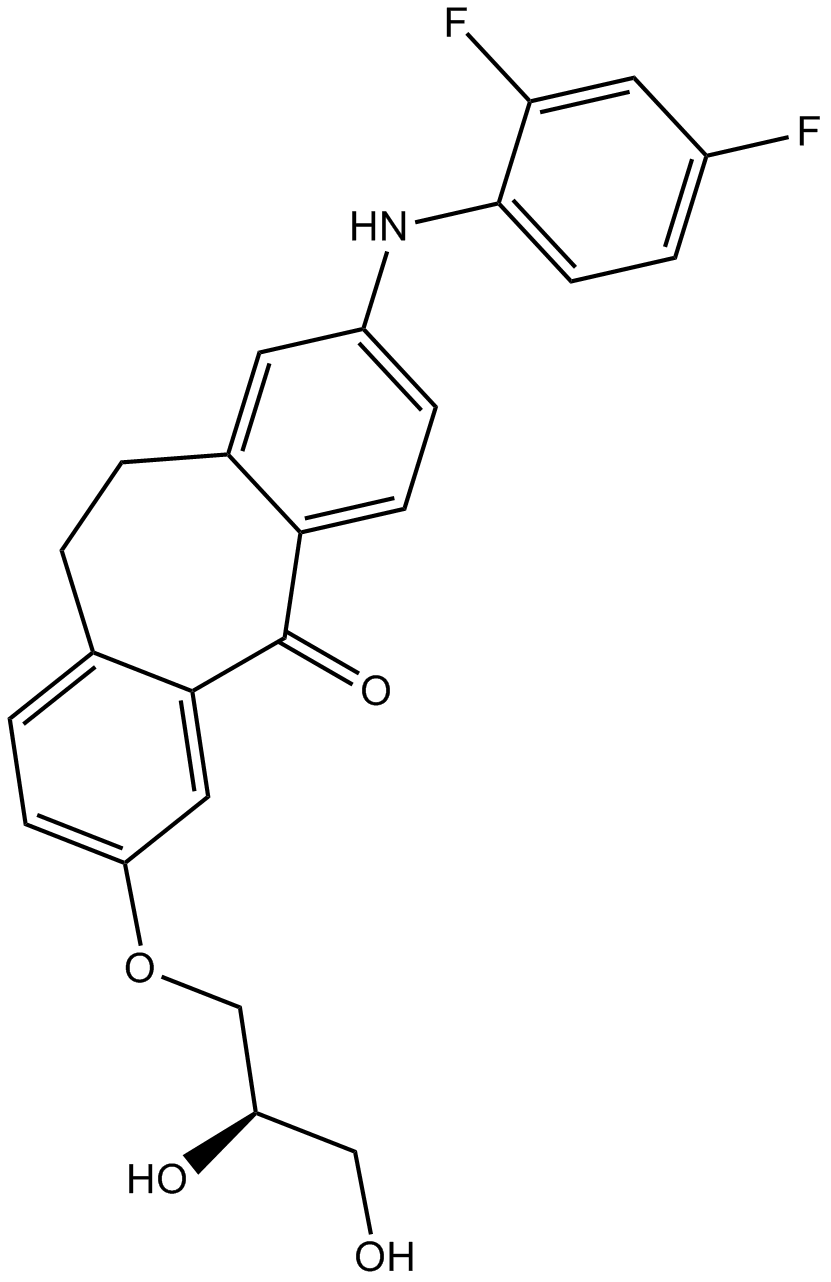
-
GC17725
SKF 86002 dihydrochloride
Le dichlorhydrate SKF 86002 est un inhibiteur de p38 MAPK actif par voie orale, avec des activités anti-inflammatoires, anti-arthritiques et analgésiques.
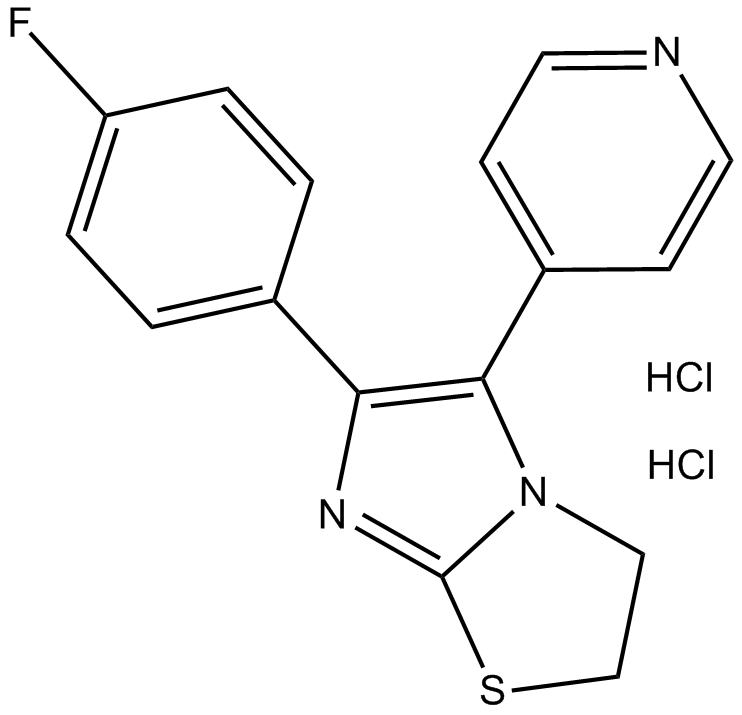
-
GC37646
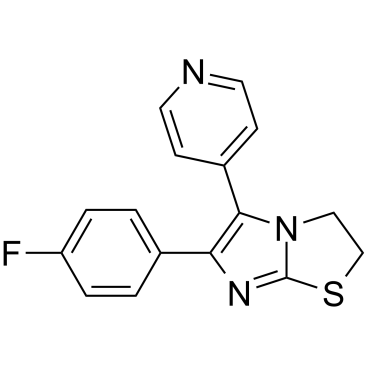
SKF-86002
SKF-86002 est un inhibiteur de p38 MAPK actif par voie orale, avec des activités anti-inflammatoires, anti-arthritiques et analgésiques.
-
GC18532
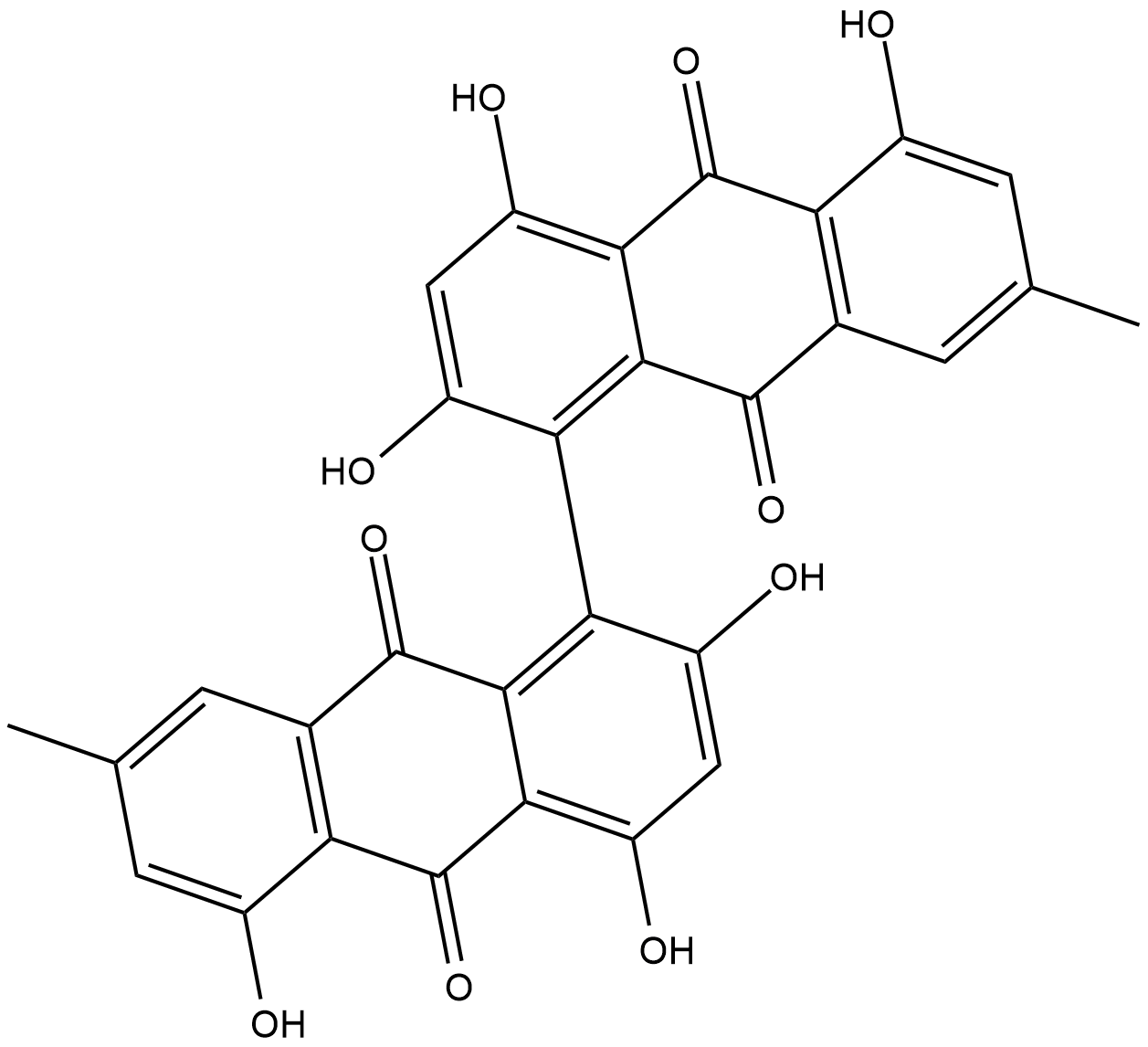
Skyrin
Skyrin is a fungal metabolite characterized by a bisanthraquinone structure.
-
GC16313
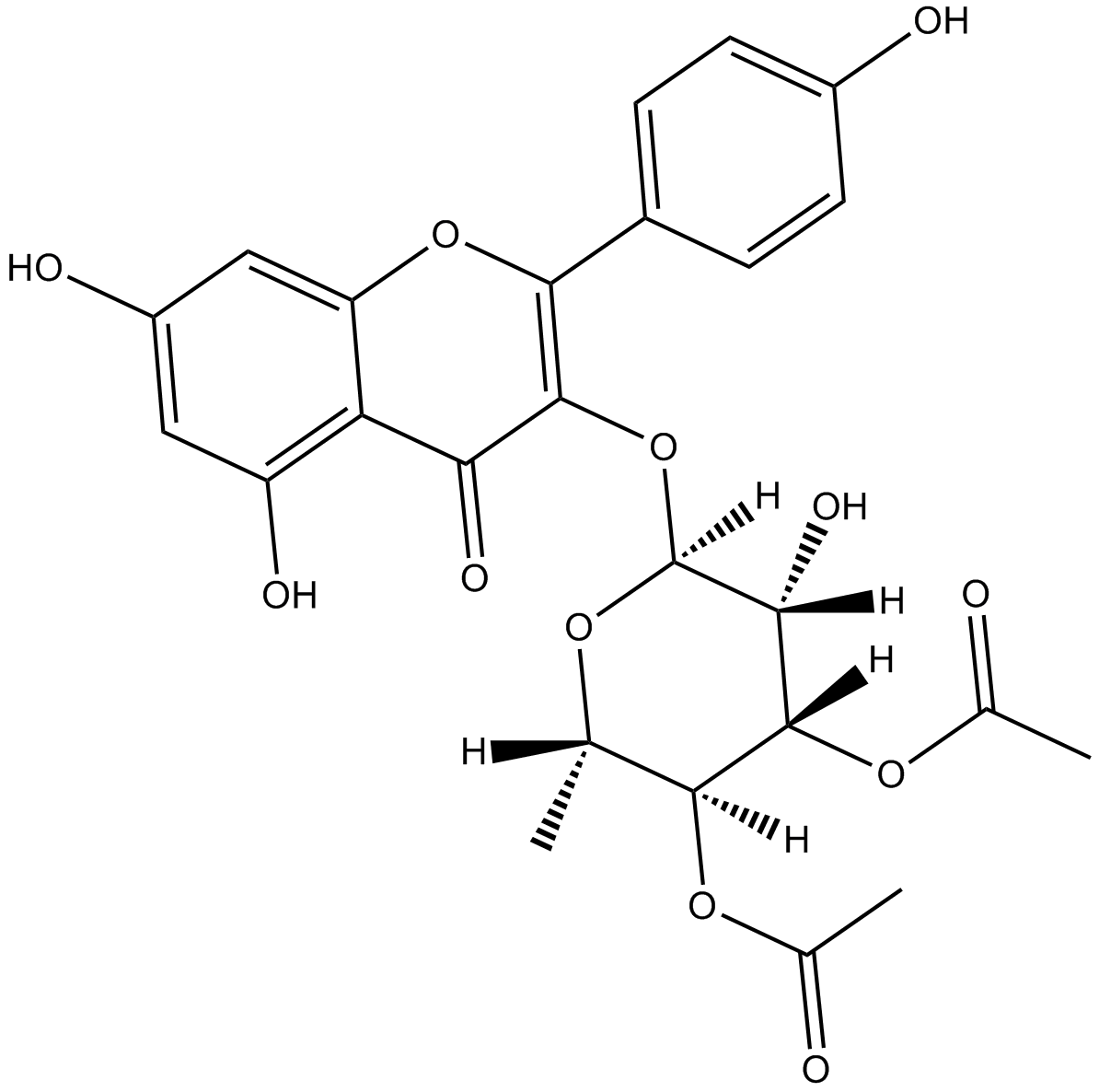
SL 0101-1
SL 0101-1 (SL0101), un glycoside de kaempférol, isolé de la plante tropicale F. réfractaire, est un inhibiteur sélectif, réversible et perméable aux cellules p90 Ribosomal S6 Kinase (RSK), avec une IC50 de 89 nM. SL 0101-1 (SL0101) est un inhibiteur sélectif de RSK1/2, avec un Ki de 1 μM.
-
GC15359
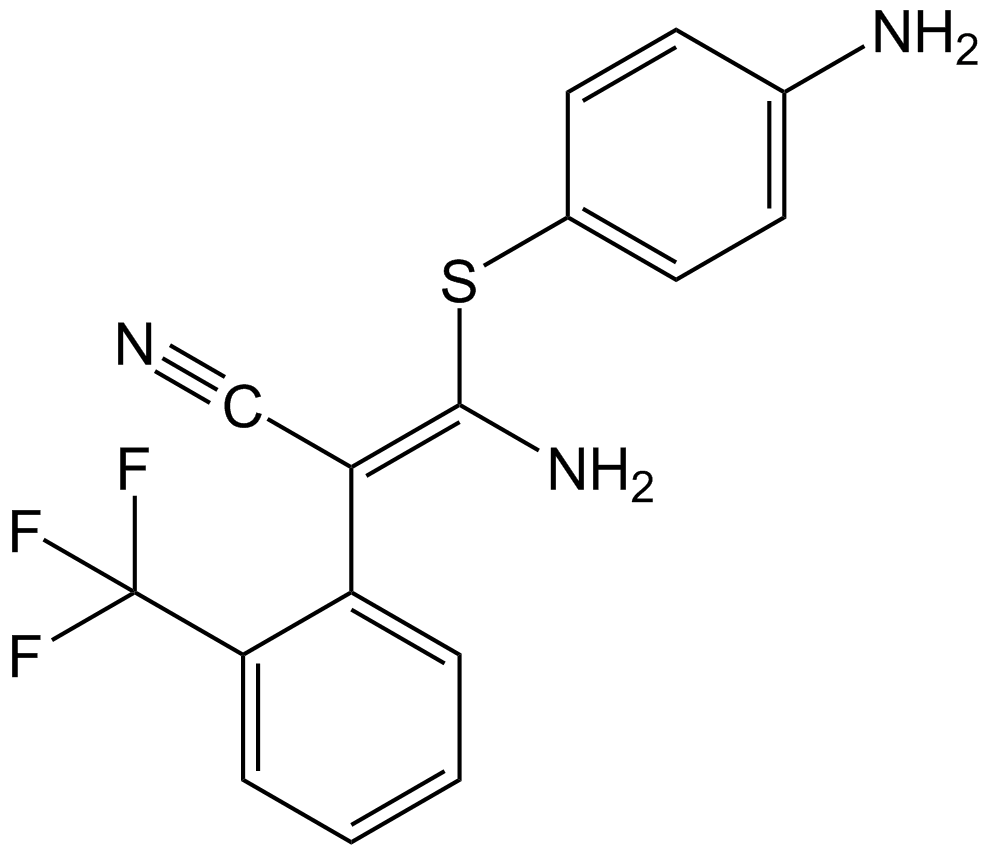
SL-327
SL-327 inhibe MEK1 et MEK2, avec des valeurs IC50 de 180 nM et 220 nM, respectivement.
-
GC32937
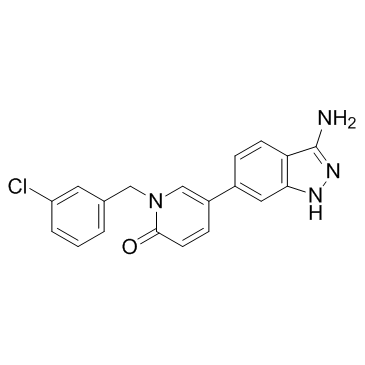
SLV-2436 (SEL201-88)
SLV-2436 (SEL201-88) est un inhibiteur très puissant et compétitif de l'ATP de MNK1 et MNK2 avec des IC50 de 10,8 nM et 5,4 nM, respectivement.
-
GC62675
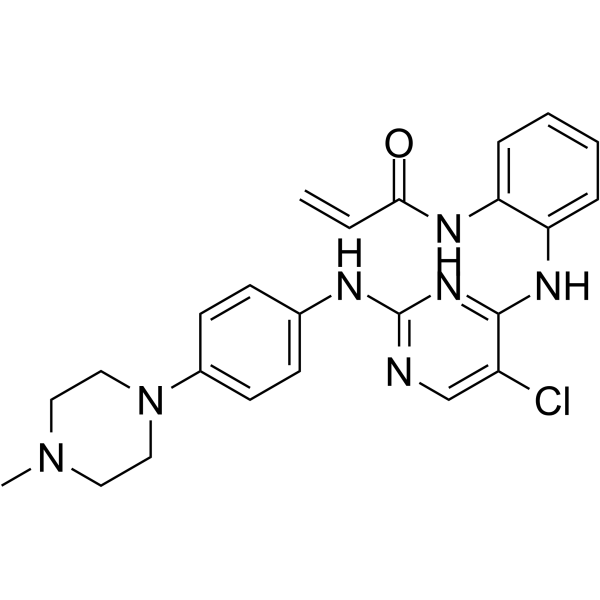
SM1-71
SM1-71 (composé 5) est un puissant inhibiteur de TAK1, avec un Ki de 160 nM, il peut également inhiber de manière covalente MKNK2, MAP2K1/2/3/4/6/7, GAK, AAK1, BMP2K, MAP3K7, MAPKAPK5, GSK3A /B, MAPK1/3, SRC, YES1, FGFR1, ZAK (MLTK), MAP3K1, LIMK1 et RSK2. SM1-71 peut inhiber la prolifération de plusieurs lignées cellulaires cancéreuses.
-
GC17425
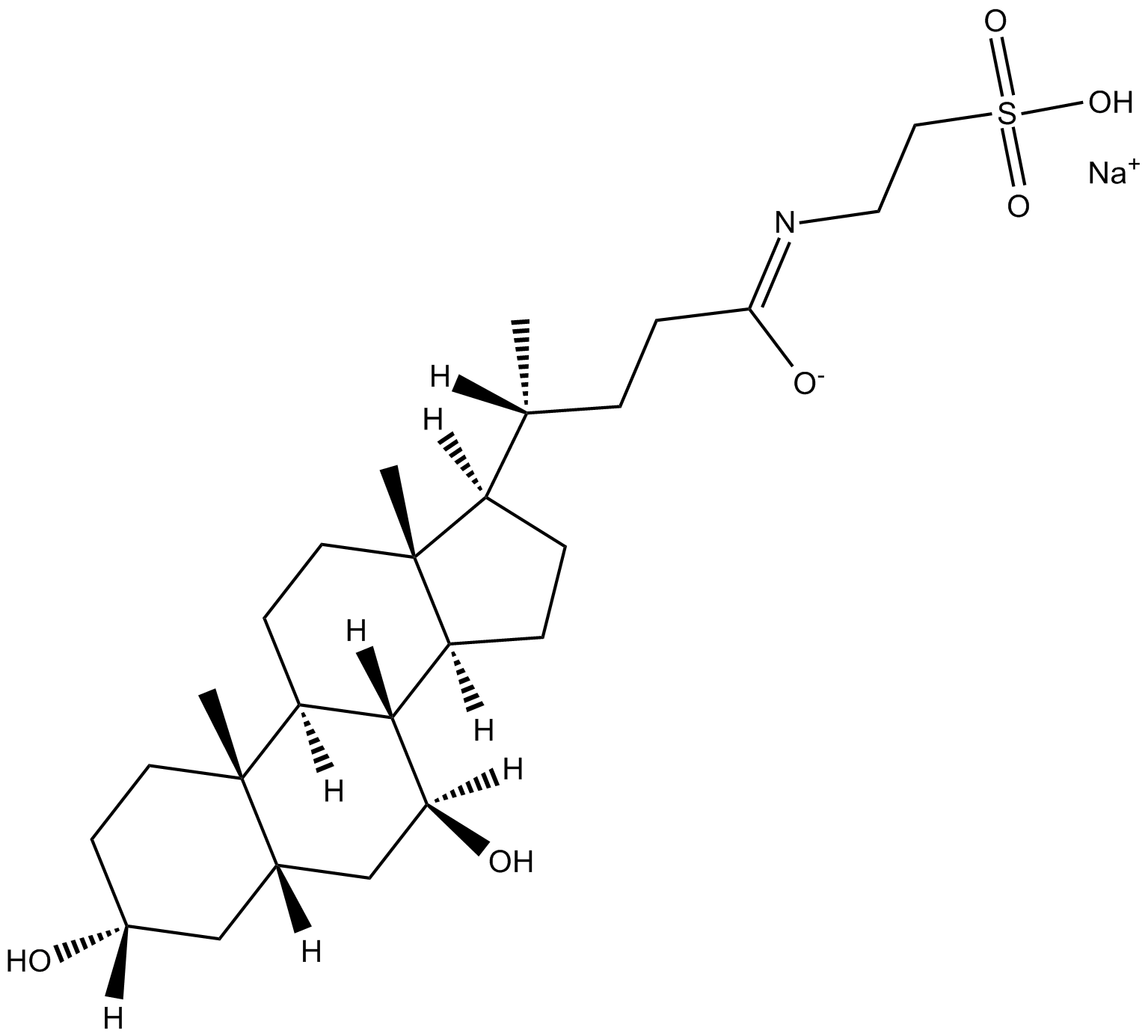
Sodium Tauroursodeoxycholate (TUDC)
Le tauroursodésoxycholate (acide tauroursodésoxycholique; TUDCA) de sodium est un inhibiteur du stress du réticulum endoplasmique (RE). Le tauroursodésoxycholate réduit significativement l'expression des molécules d'apoptose, telles que la caspase-3 et la caspase-12. Le tauroursodésoxycholate inhibe également l'ERK.
-
GC17369
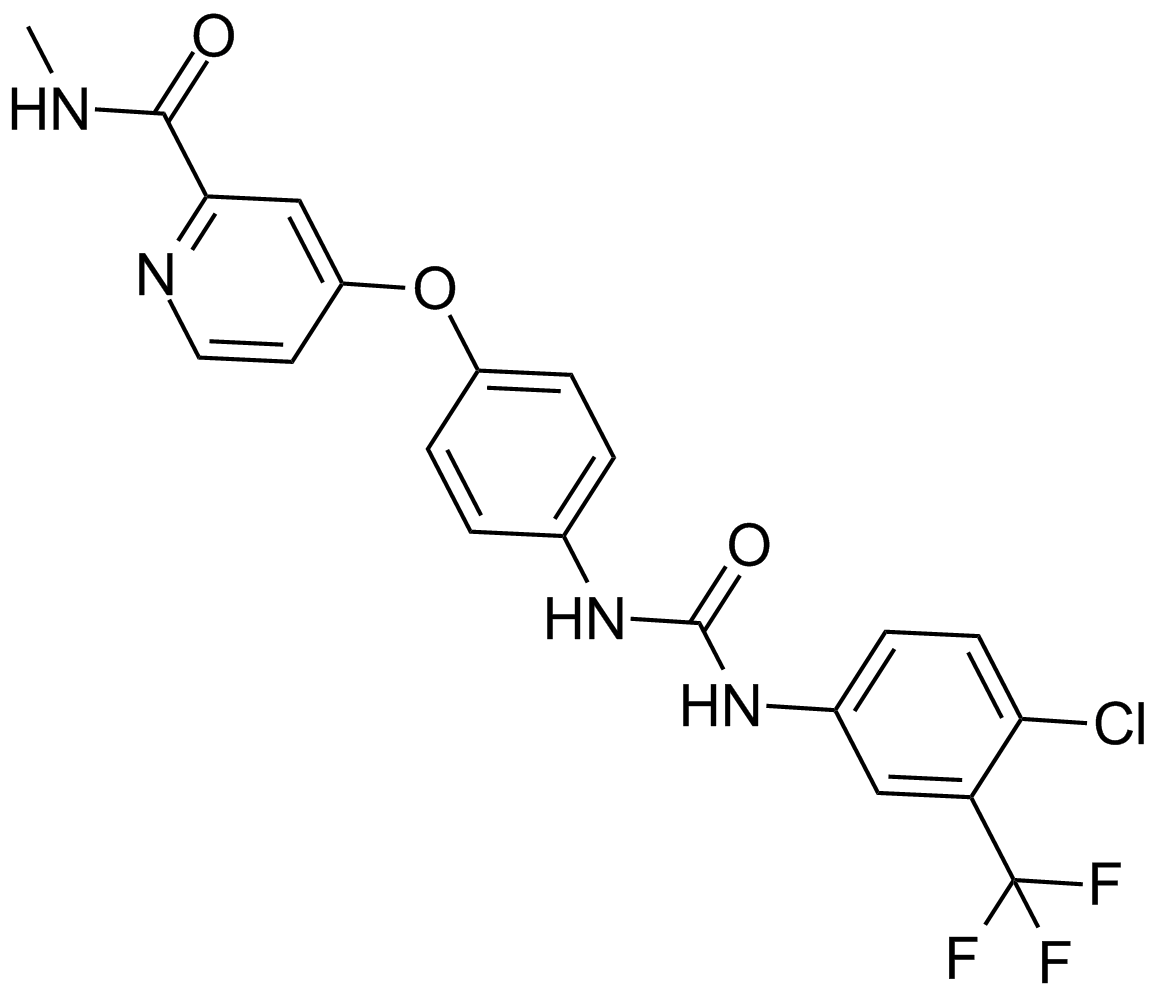
Sorafenib
Sorafenib agit en tant qu'inhibiteur multi-kinase, ciblant Raf-1 et B-Raf avec des valeurs d'IC50 de 6 nM et 22 nM, respectivement.
-
GC37664
Sorafenib (D3)
Sorafenib (D3) (Bay 43-9006-d3) est le Sorafenib marqué au deutérium. Le sorafénib est un inhibiteur multikinase IC50 de 6 nM, 20 nM et 22 nM pour Raf-1, B-Raf et VEGFR-3, respectivement.
-
GC37665
Sorafenib (D4)
Sorafenib (D4) (Bay 43-9006-d4) est le Sorafenib marqué au deutérium. Le sorafénib est un inhibiteur multikinase IC50 de 6 nM, 20 nM et 22 nM pour Raf-1, B-Raf et VEGFR-3, respectivement.
-
GC44917
Sorafenib N-oxide
Sorafenib N-oxide is an active metabolite of sorafenib, an inhibitor of Raf-1, B-RAF, and receptor tyrosine kinases.
-
GC10428
Sp-5,6-dichloro-cBIMPS (sodium salt)
PKA activator
-
GC44920
Sp-8-bromo-Cyclic AMPS (sodium salt)
Sp-8-bromo-cyclic AMPS (Sp-8-bromo-cAMPS) is a cell-permeable, cAMP analog that combines an exocyclic sulfur substitution in the axial position of the cyclophosphate ring with a bromine substitution in the adenine base of cAMP.
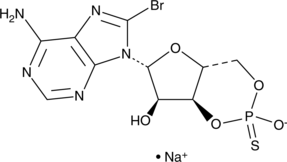
-
GC61747
Sp-cAMPS
Sp-cAMPS, un analogue de l'AMPc, est un puissant activateur des PKA I et PKA II dépendantes de l'AMPc.
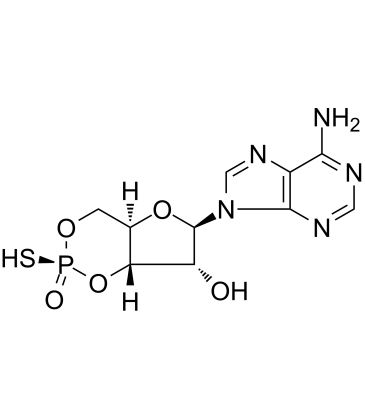
-
GC16652
SR 3576
SR 3576 est un inhibiteur JNK3 très puissant et sélectif avec une IC50 de 7 nM.
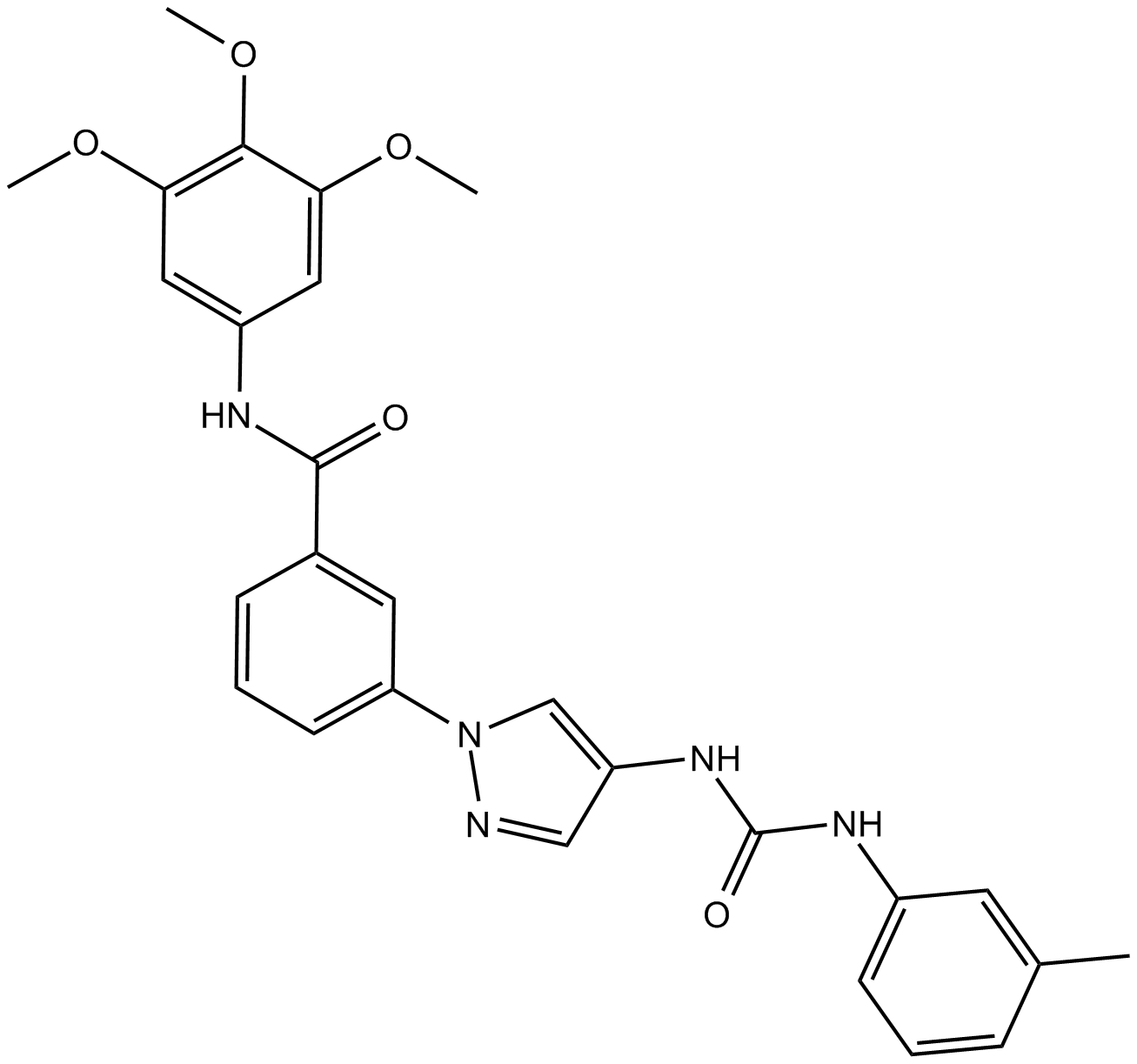
-
GC30864
SR-3306
Le SR-3306 est un inhibiteur JNK sélectif, puissant et hautement pénétrant dans le cerveau.
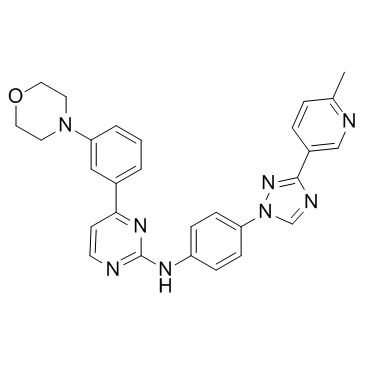
-
GC64287
SR15006
SR15006 est un inhibiteur du facteur 5 de type KrÜppel (KLF5) avec une IC50 de 41,6 nM dans les cellules DLD-1/pGL4.18hKLF5p).
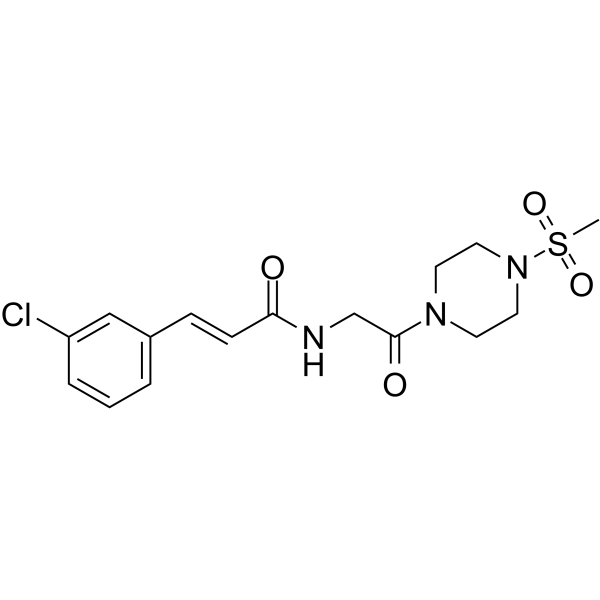
-
GC50406
st-Ht31
Inhibits PKA/AKAP interactions; cell permeable
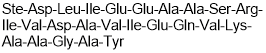
-
GC50407
st-Ht31 P
Negative control for st-Ht31
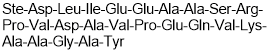
-
GC50501
STAD 2
STAD 2 est un perturbateur puissant et sélectif de PKA-RII, avec un Kd de 6,2 nM.
-
GC15299
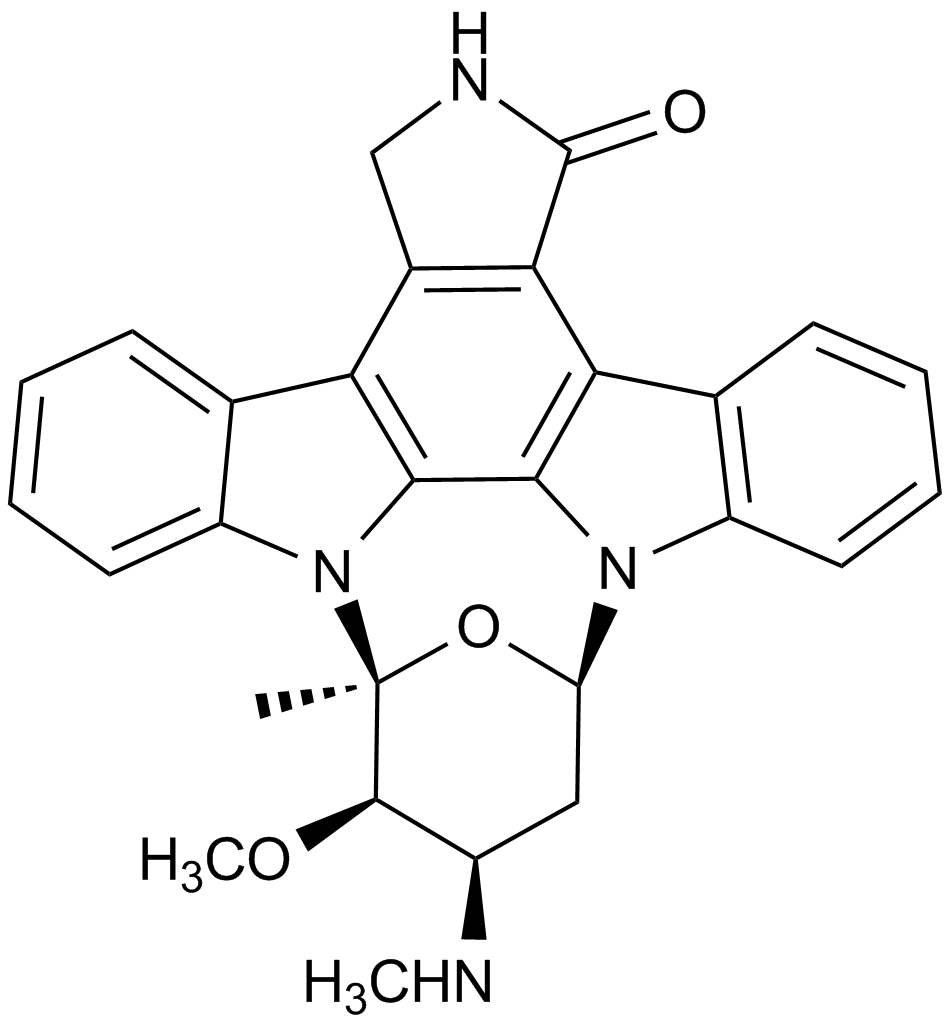
Staurosporine(CGP 41251)
A potent inhibitor of protein kinase C
-
GC11542
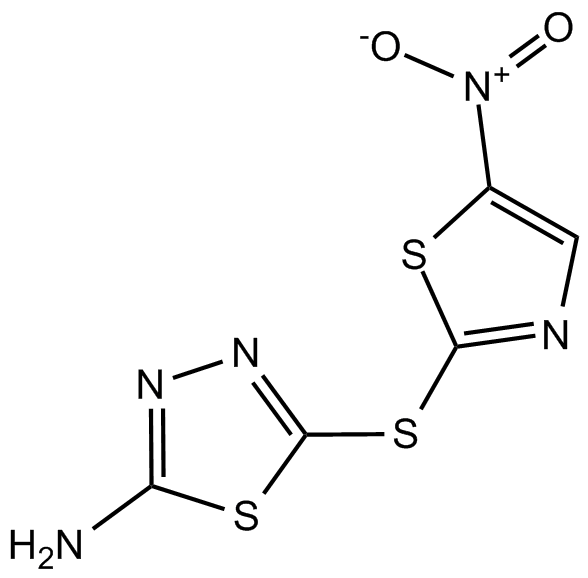
SU 3327
Le SU 3327 est un inhibiteur de JNK puissant, sélectif et compétitif avec un substrat avec une IC50 de 0,7 μM.
-
GC13825
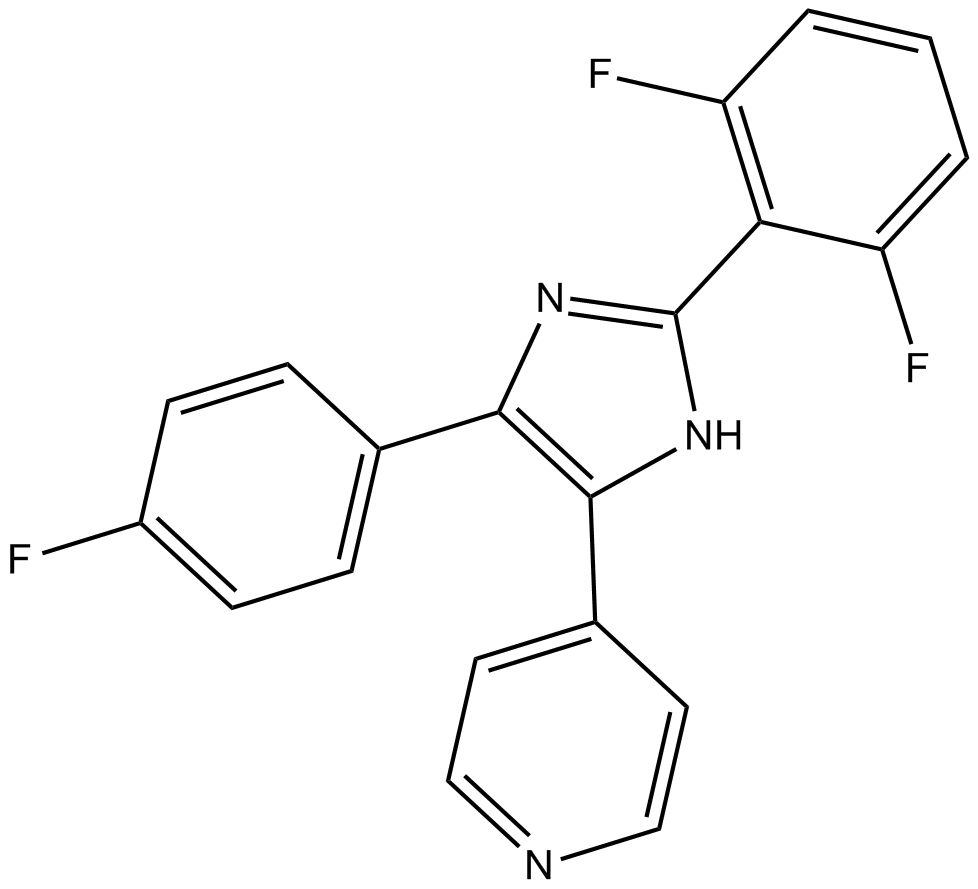
TA 01
TA 01 est un puissant inhibiteur de CK1 et p38 MAPK, avec des IC50 de 6,4 nM, 6,8 nM, 6,7 nM pour CK1ε, CK1δ et p38 MAPK, respectivement.
-
GC11635
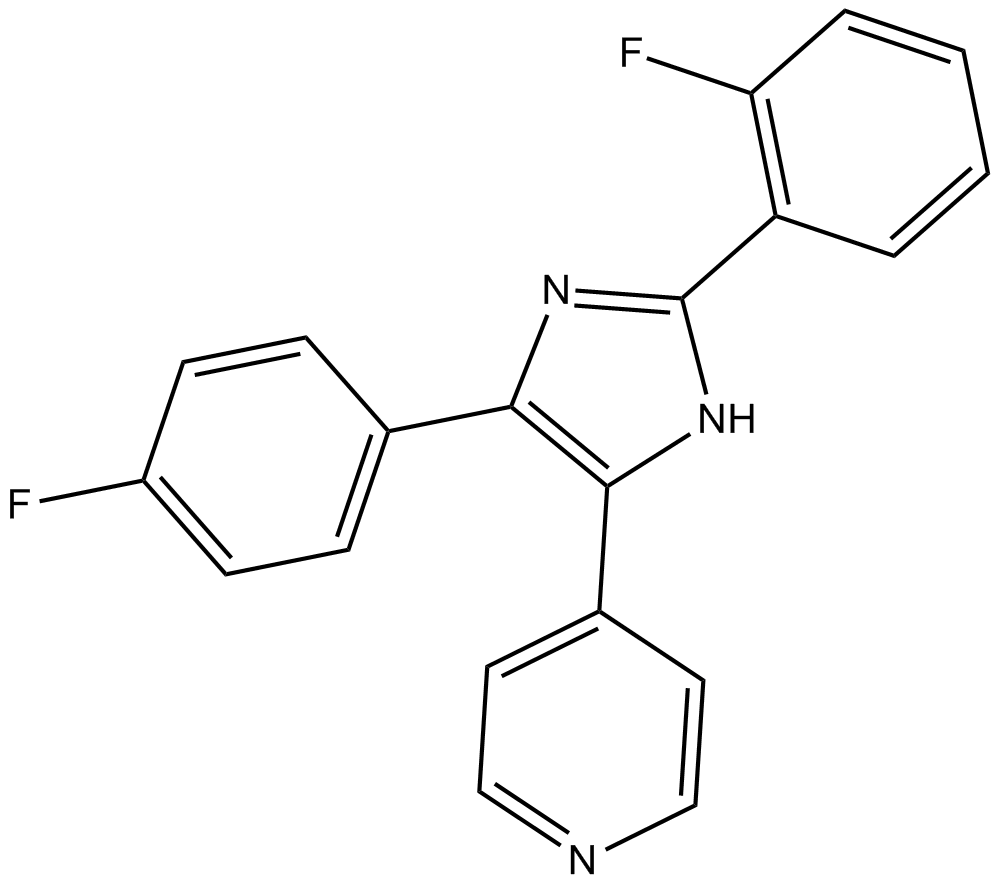
TA 02
Le TA 02, un analogue du SB 203580, est un inhibiteur de p38 MAPK avec une IC50 de 20 nM.
-
GC16543
TAK-715
TAK-715 est un inhibiteur de p38 MAPK actif et puissant par voie orale avec des IC50 de 7,1 nM, 200 nM pour p38α et p38β, respectivement.
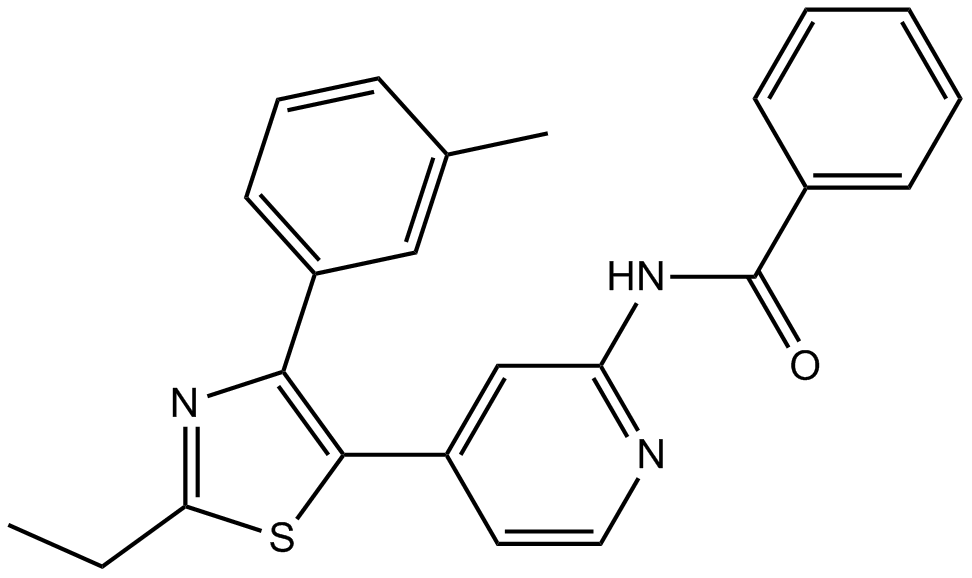
-
GC10209
TAK-733
TAK-733 est un inhibiteur puissant et sélectif du site allostérique MEK avec une IC50 de 3,2 nM.
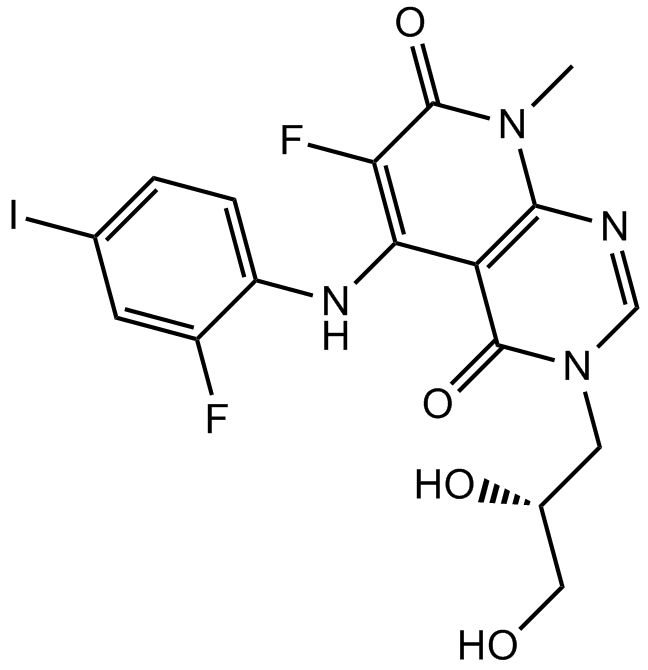
-
GC62497
TAK1-IN-2
TAK1-IN-2 est un inhibiteur puissant et sélectif de TAK1, avec une IC50> de 2 nM.
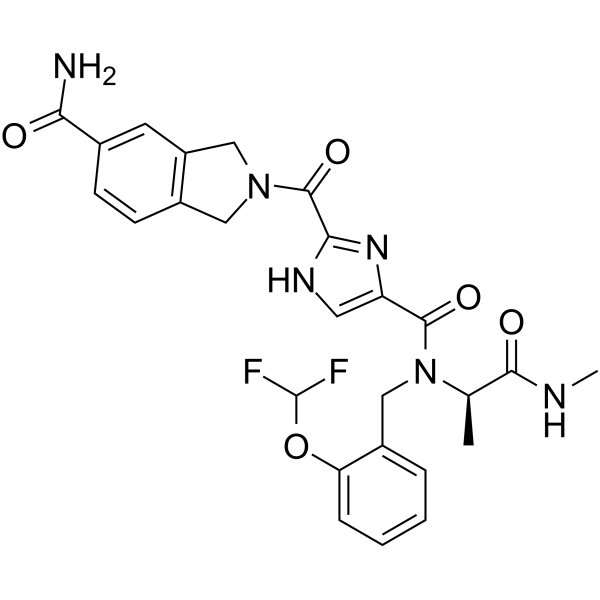
-
GC69984
TAK1-IN-4
TAK1-IN-4 (Composé 14) est un inhibiteur de TAK1.
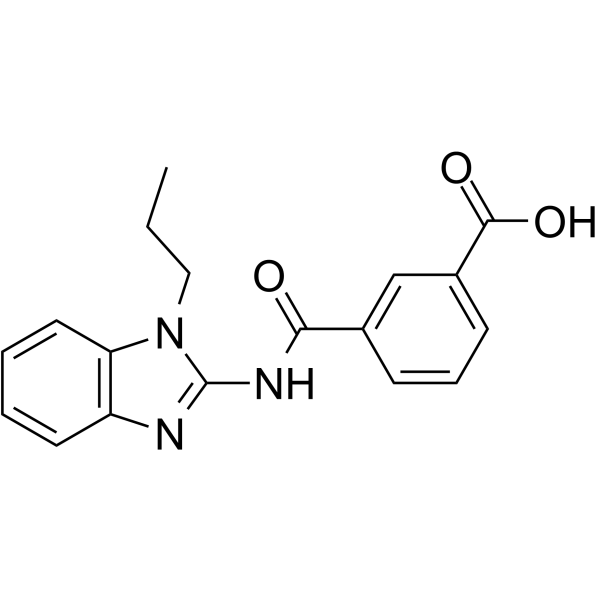
-
GC49700
Takeda-6d
Takeda-6D (composé 6d) est un inhibiteur puissant et actif par voie orale de BRAF/VEGFR2, avec des valeurs IC50 de 7,0 et 2,2 nM, respectivement. Takeda-6D montre une antiangiogenèse en supprimant la voie VEGFR2 dans les cellules HUVEC stimulées par 293/KDR et VEGF. Takeda-6D montre une suppression significative de la phosphorylation de ERK1/2. Takeda-6D montre une activité antitumorale.
-
GC32687
Takinib
Le takinib (EDHS-206) est un inhibiteur de TAK1 actif et sélectif par voie orale (IC50 = 9,5 nM), plus de 1,5 log plus puissant que les deuxième et troisième cibles classées, IRAK4 (120 nM) et IRAK1 (390 nM), respectivement.
-
GC34072
Talmapimod (SCIO-469)
Le talmapimod (SCIO-469) (SCIO-469) est un p38α actif par voie orale, sélectif et compétitif pour l'ATP ; inhibiteur avec une IC50 de 9 nM. Le talmapimod (SCIO-469) présente une sélectivité d'environ 10 fois par rapport À p38β et une sélectivité d'au moins 2000 fois sur un panel de 20 autres kinases, y compris d'autres MAPK.
-
GC25982
Tanzisertib(CC-930)
Tanzisertib (CC-930, JNK-930, JNKI-1) is kinetically competitive with ATP in the JNK-dependent phosphorylation of the protein substrate c-Jun and potent against all isoforms of JNK (Ki(JNK1) = 44 ± 3 nM, IC50(JNK1) = 61 nM, Ki(JNK2) = 6.2 ± 0.6 nM, IC50(JNK2) = 5 nM, IC50(JNK3) = 5 nM) and selective against MAP kinases ERK1 and p38a with IC50 of 0.48 and 3.4 μM respectively.

-
GC34181
Tauroursodeoxycholate (TUDCA)
Le tauroursodésoxycholate (TUDCA) (acide tauroursodésoxycholique) est un inhibiteur du stress endoplasmique réticulaire (ER). Le tauroursodésoxycholate (TUDCA) réduit significativement l'expression de molécules d'apoptose, telles que la caspase-3 et la caspase-12. Le tauroursodésoxycholate (TUDCA) inhibe également ERK.
-
GC34831
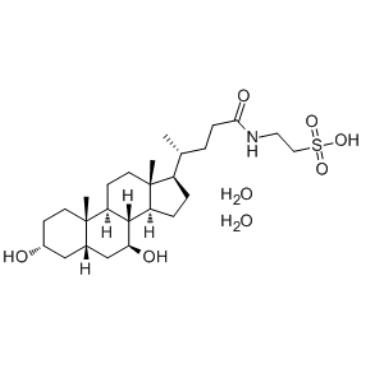
Tauroursodeoxycholate dihydrate
Le tauroursodésoxycholate (acide tauroursodésoxycholique; TDUCA) dihydraté est un inhibiteur du stress du réticulum endoplasmique (RE). Le tauroursodésoxycholate réduit significativement l'expression des molécules d'apoptose, telles que la caspase-3 et la caspase-12. Le tauroursodésoxycholate inhibe également l'ERK.
-
GC34057
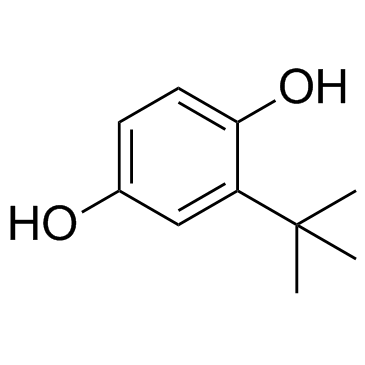
TBHQ (tert-Butylhydroquinone)
TBHQ (tert-butylhydroquinone) (tert-butylhydroquinone) est un activateur Nrf2 largement utilisé, protège contre la cardiotoxicité induite par la doxorubicine (DOX) par l'activation de Nrf2. TBHQ (tert-Butylhydroquinone) (tert-Butylhydroquinone) est également un activateur ERK; sauve l'inhibition de la prolifération cellulaire induite par la déhydrocorydaline (DHC) dans le mélanome.
-
GC14853
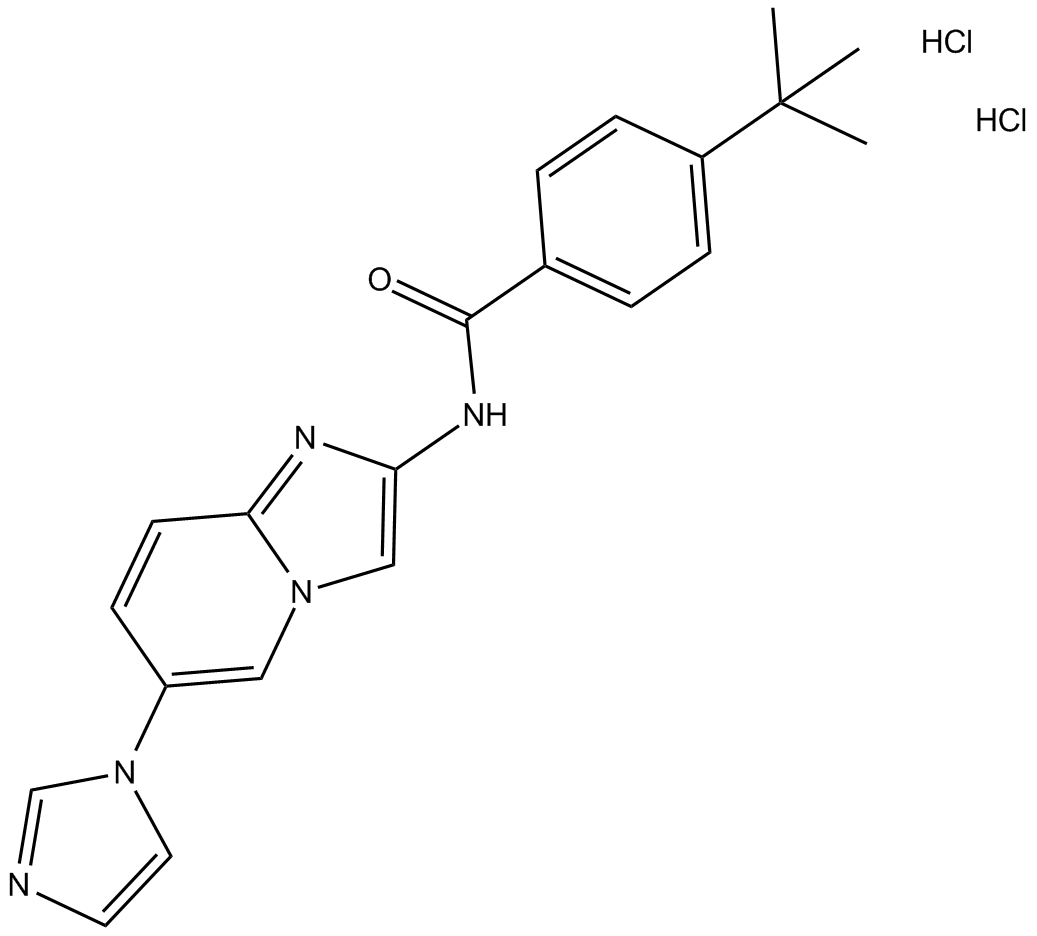
TC ASK 10
TC ASK 10 (composé 10) est un inhibiteur de la kinase 1 (ASK1) régulatrice du signal d'apoptose puissant, sélectif et actif par voie orale avec une IC50 de 14 nM. Les activités inhibitrices de TC ASK 10 vis-À-vis d'autres panel représentatifs de kinases sont inférieures À 50%, sauf pour ASK2 (IC50 de 0,51 μM).
-
GC34140
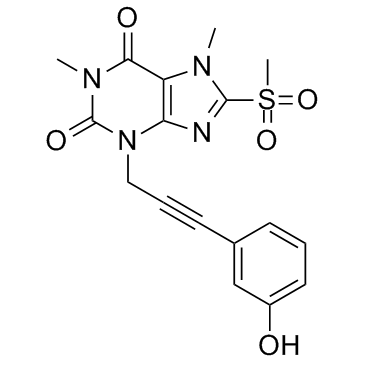
TC13172
TC13172 est un inhibiteur de protéine de type domaine de kinase de lignée mixte (MLKL) avec une valeur EC50 de 2 nM pour les cellules HT-29.
-
GC11488
TCS JNK 5a
Le TCS JNK 5a est un puissant inhibiteur de JNK3 avec un pIC50 de 6,7. TCS JNK 5a inhibe également JNK2 avec un pIC50 de 6,5.
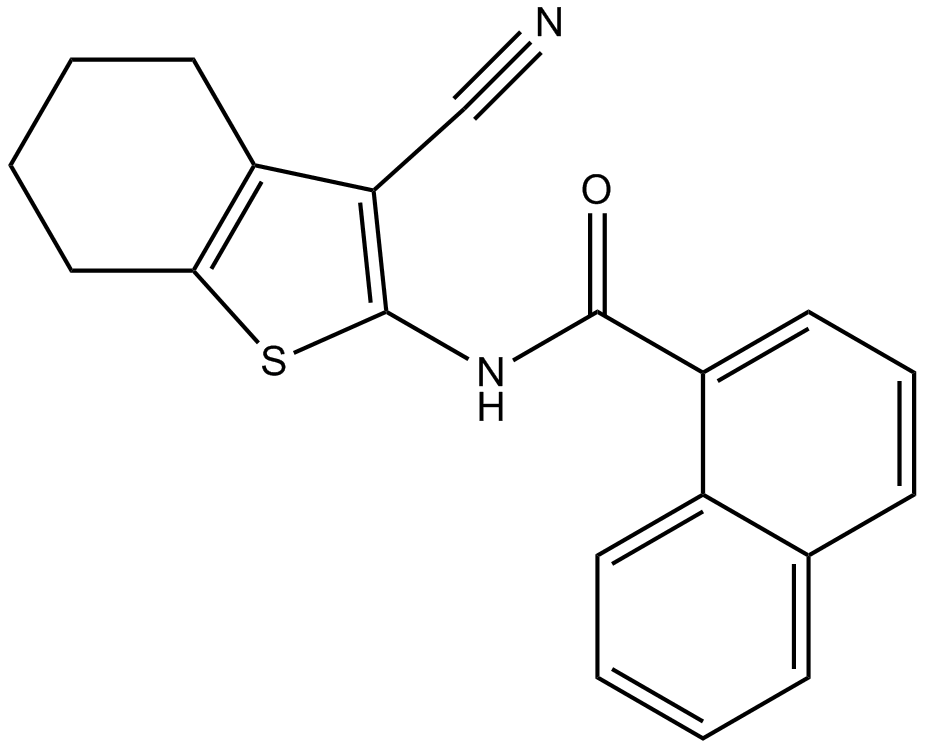
-
GC17282
TCS JNK 6o
TCS JNK 6o (TCS JNK 6o) est un inhibiteur des kinases N-terminales c-Jun (JNK-1, -2 et -3) avec des valeurs Ki de 2 nM, 4 nM, 52 nM, respectivement, et a des valeurs IC50 de 45 nM et 160 nM pour JNK-1 et -2, respectivement.
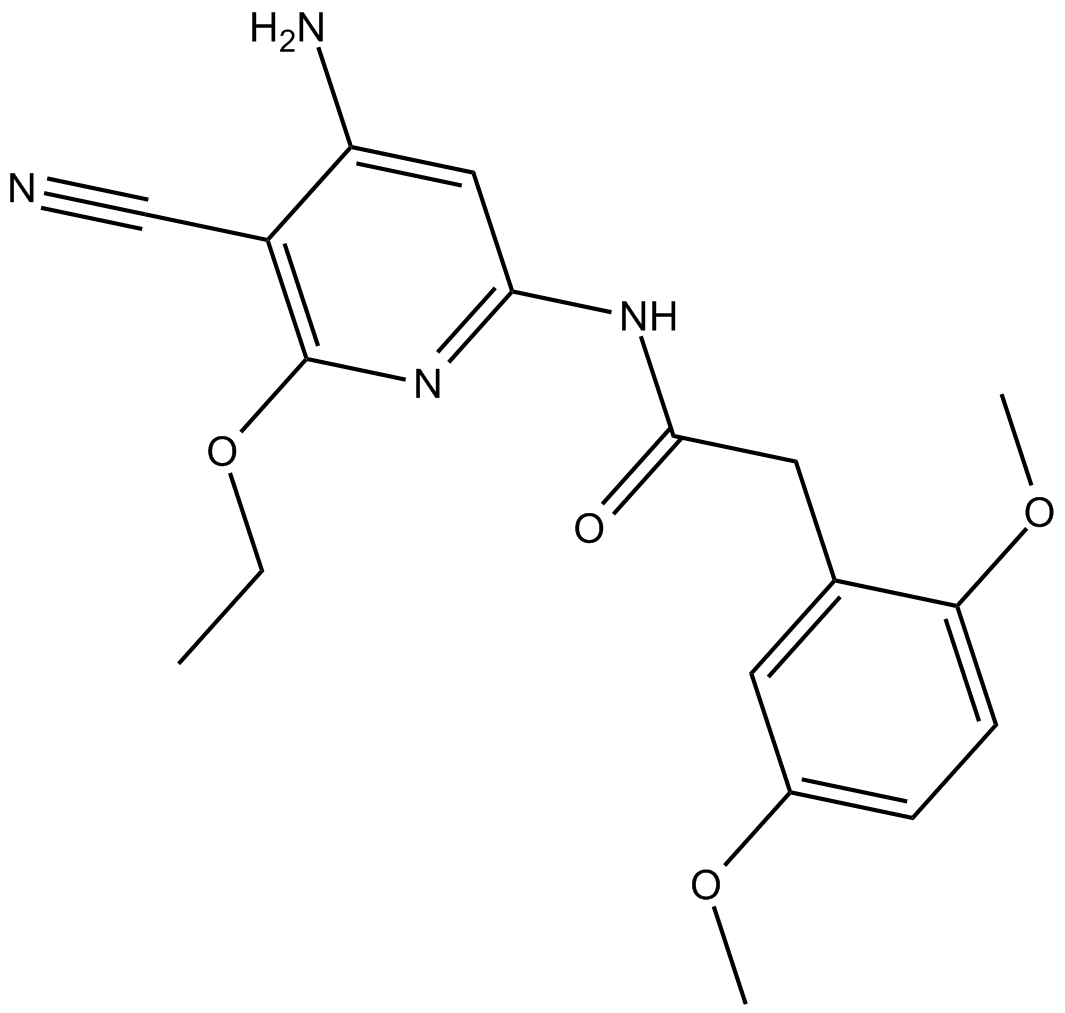
-
GC39074
Tenuifoliside A
Le ténuifoliside A est isolé de Polygala tenuifolia, a des effets anti-apoptotique et de type antidépresseur.
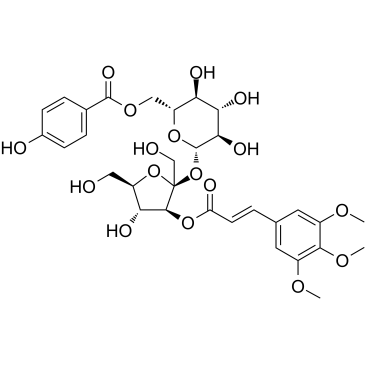
-
GC41573
Theaflavin 3,3'-digallate
Le 3,3'-digallate de théaflavine (TF-3) est un puissant inhibiteur de la protéase du virus Zika (ZIKV) avec une IC50 de 2,3 μM.
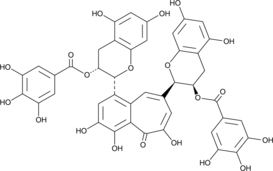
-
GC68350
Tinlorafenib
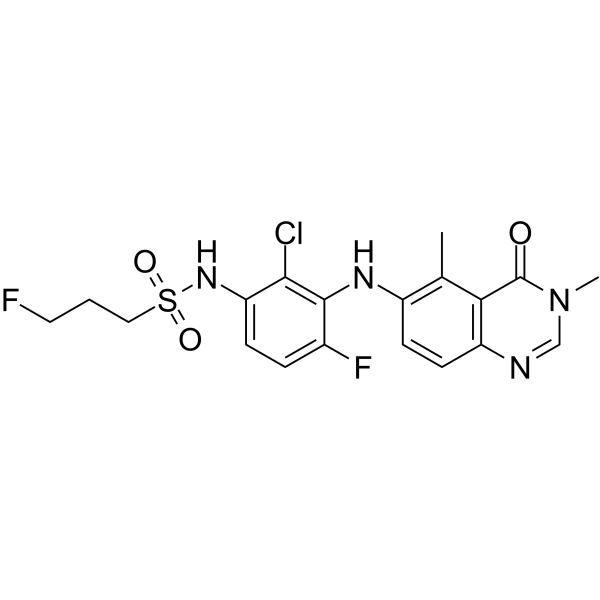
-
GC50295
TL4-12
TL4 12 est un inhibiteur puissant et sélectif de la GCK (germinal center kinase).
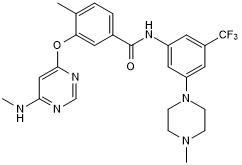
-
GC31647
Tomatidine
La tomatidine agit comme un agent anti-inflammatoire en bloquant la signalisation NF-κB et JNK.
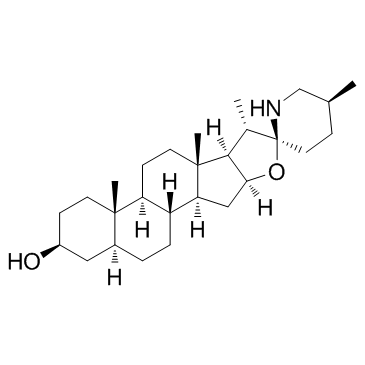
-
GC45064
Tomatidine (hydrochloride)
La tomatidine (chlorhydrate) agit comme un agent anti-inflammatoire en bloquant la signalisation NF-κB et JNK.
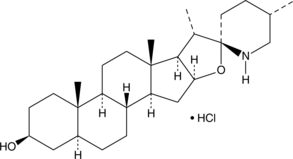
-
GC19131
Tomivosertib
Tomivosertib (eFT508) est un puissant inhibiteur de MNK1 et MNK2, hautement sélectif et actif par voie orale, avec des CI50 de 1 À 2 nM contre les deux isoformes. Le traitement par Tomivosertib (eFT508) entraÎne une réduction dose-dépendante de la phosphorylation de eIF4E au niveau de la sérine 209 (IC50 = 2-16 nM) dans les lignées cellulaires tumorales. Tomivosertib (eFT508) régule également considérablement À la baisse l'abondance de la protéine PD-L1.
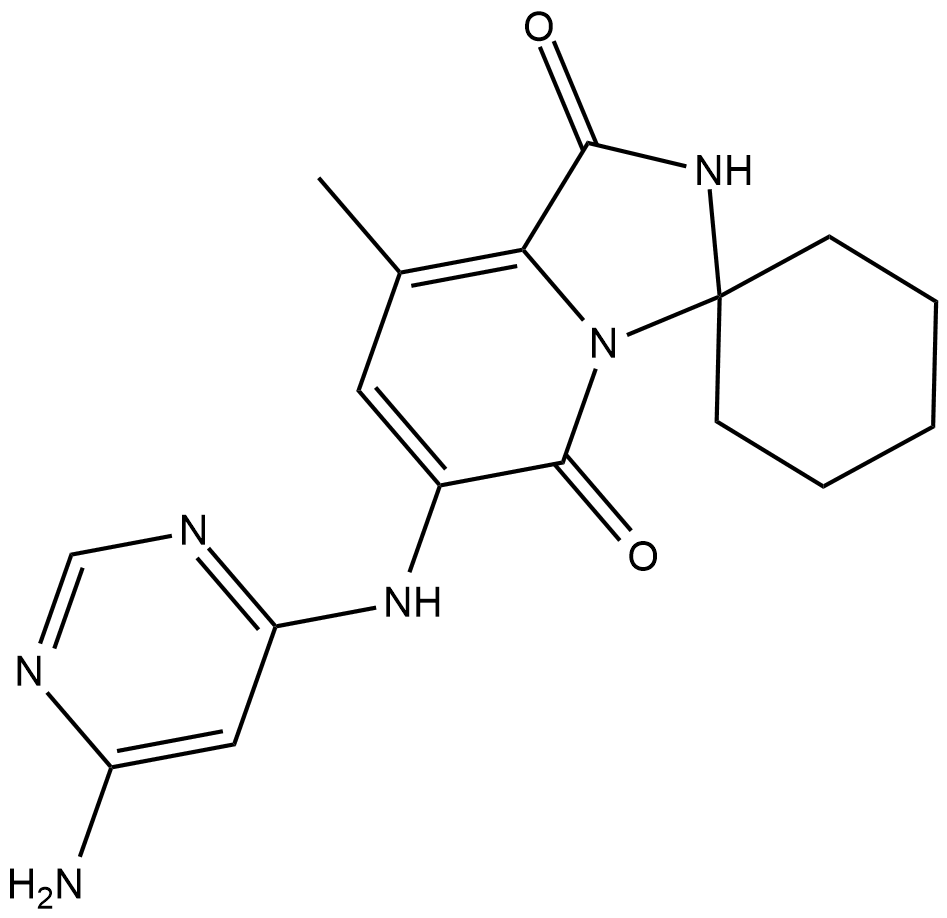
-
GC45067
Tpl2 Kinase Inhibitor
L'inhibiteur de la kinase Tpl2 (composé 1) est un inhibiteur puissant et sélectif de la Tpl2 (kinase COT, MAP3K8), qui joue un rÔle important dans la régulation de la réponse inflammatoire et la progression de certains cancers.
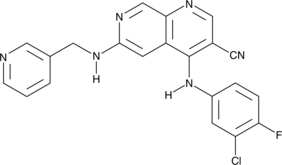
-
GC49150
Tpl2 Kinase Inhibitor (hydrochloride)
A Tpl2 inhibitor
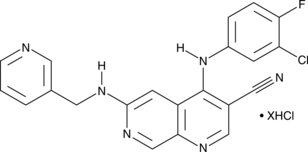
-
GC70053
Trametiglue
Trametiglue est un dérivé de Tramétinib qui se lie à une interface unique par interaction mutuelle, offrant une efficacité et une sélectivité sans précédent en ciblant KSR-MEK et RAF-MEK.
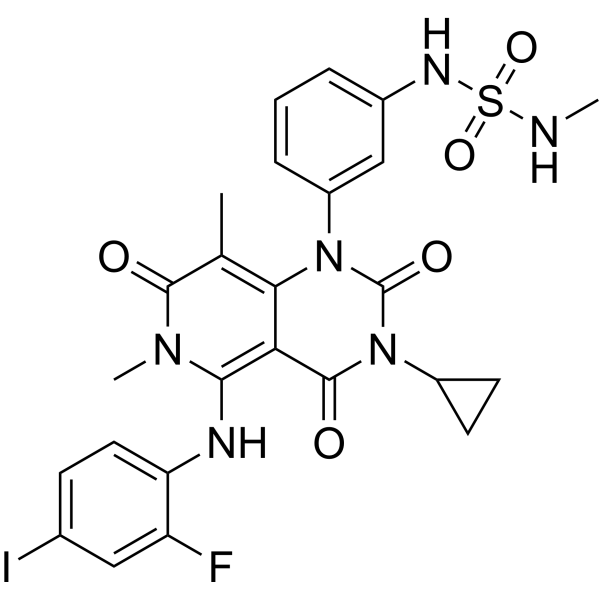
-
GC13508
Trametinib (GSK1120212)
Un inhibiteur de MEK1 et MEK2
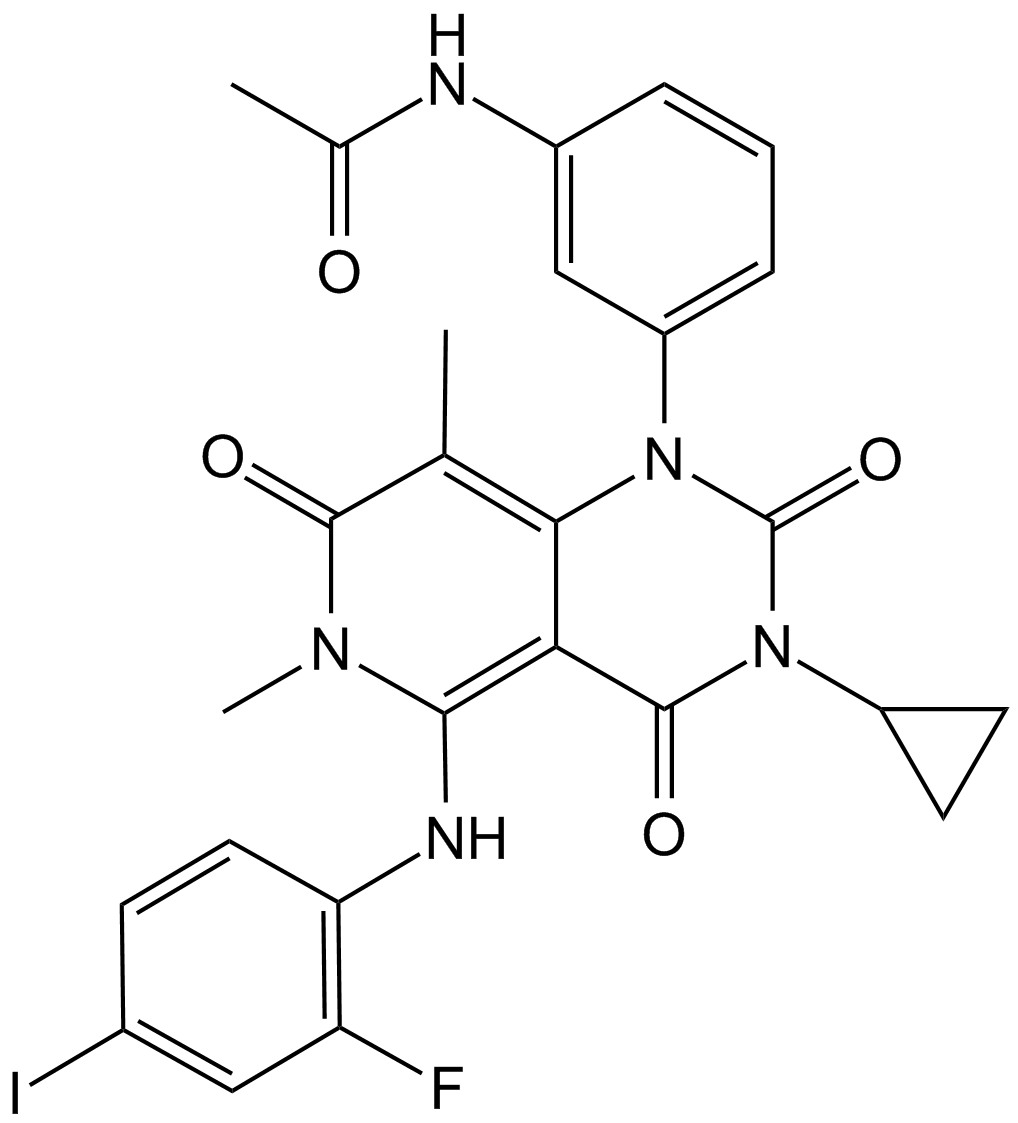
-
GC15260
Trametinib DMSO solvate
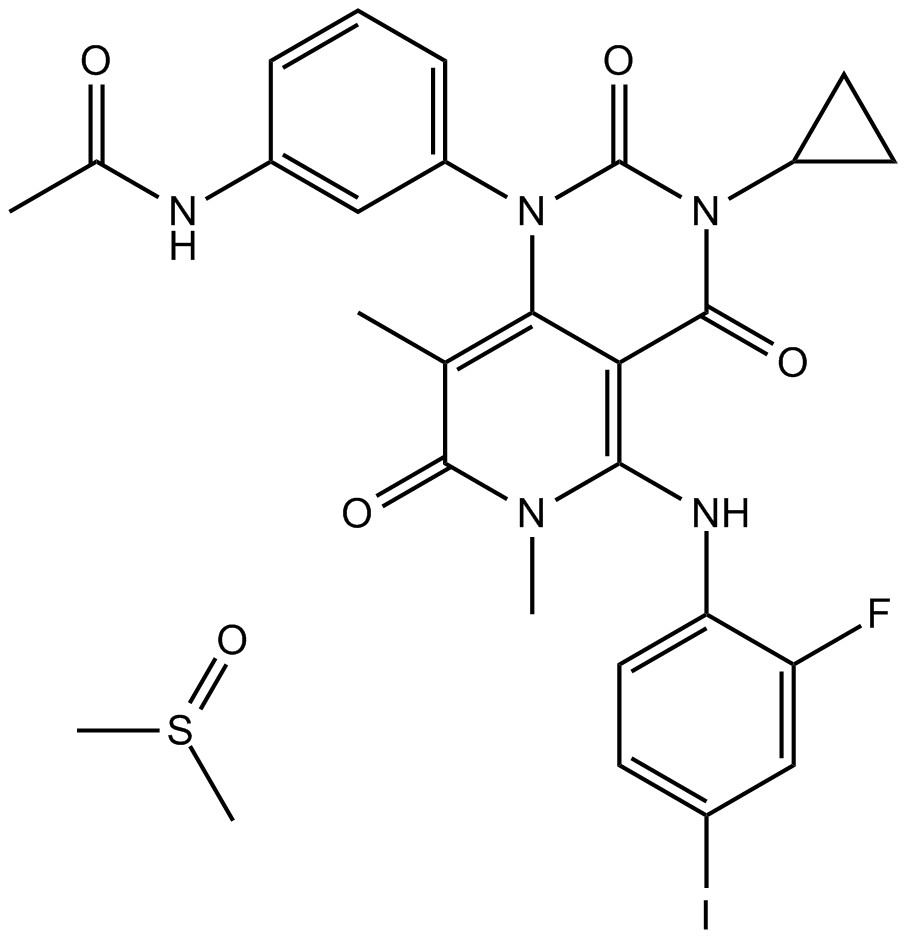
-
GC30162
trans-Zeatin
la trans-zéatine est une cytokinine végétale, qui joue un rÔle important dans la croissance, la différenciation et la division cellulaires; La trans-zéatine inhibe également l'activation de MEK/ERK induite par les UV.
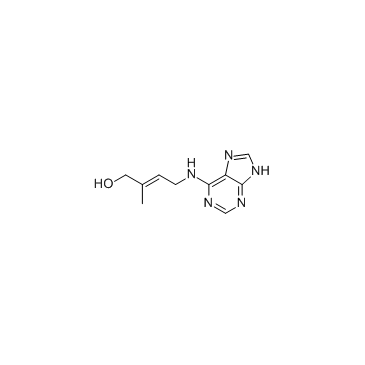
-
GC45099
U-0126
Un inhibiteur de MEK
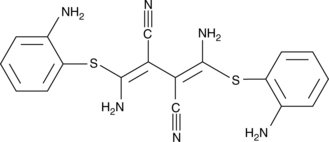
-
GC15061
U0124
U0124, un analogue inactif de U0126, n'a aucun effet sur les taux de protéines c-Fos et c-Jun ou d'ARNm. U0126 est un inhibiteur de MEK. U0124 n'inhibe pas la MEK À des concentrations allant jusqu'À 100 μM.
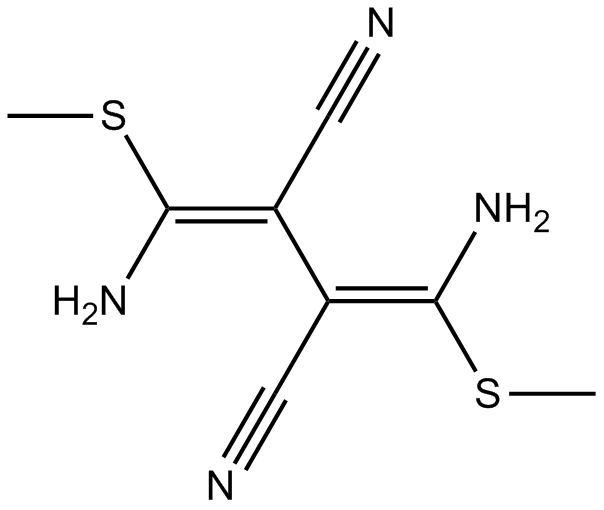
-
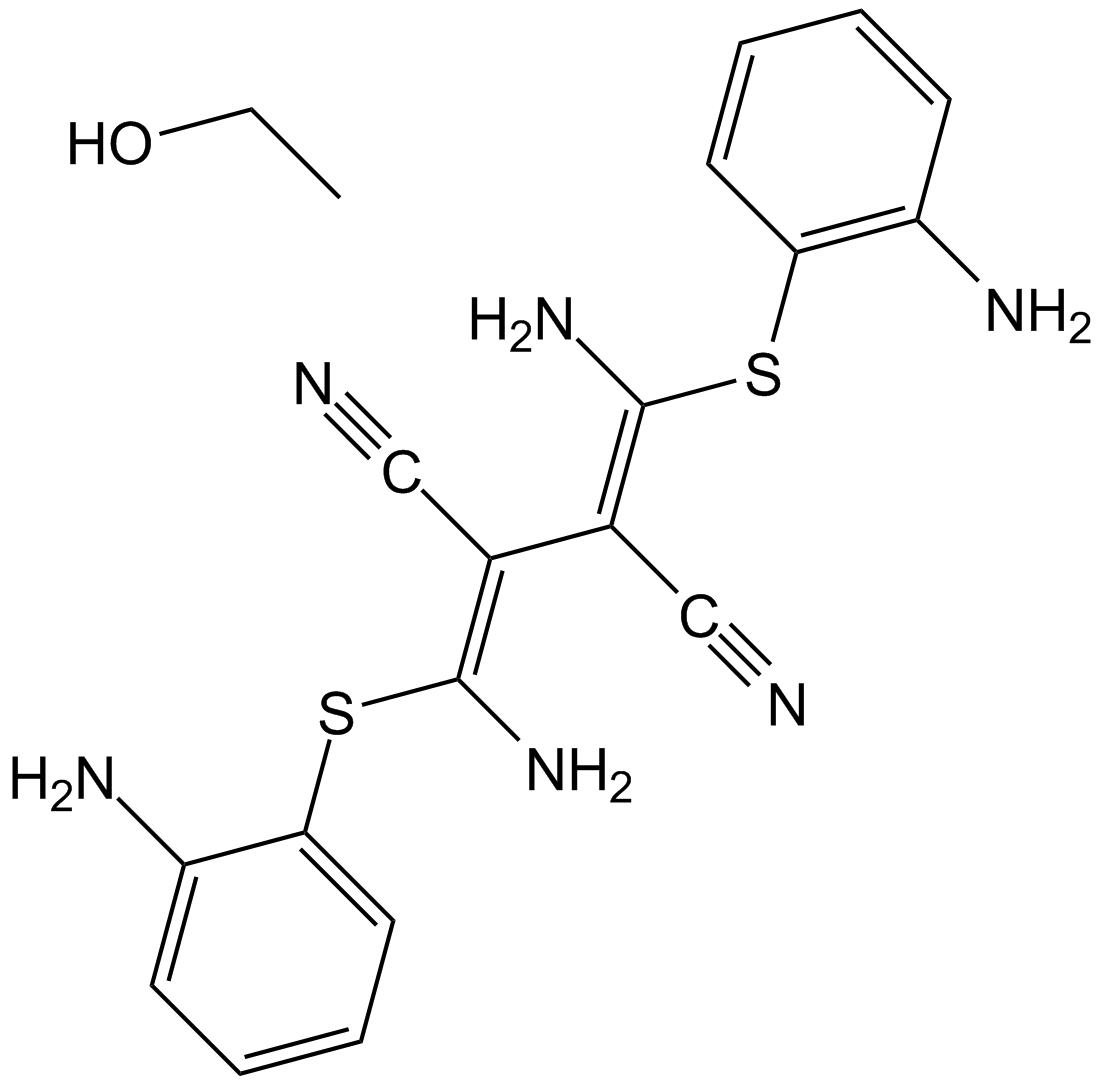
GC12807
U0126-EtOH
U0126 (U0126-EtOH) est un inhibiteur MEK1 et MEK2 puissant, non compétitif et sélectif de l'ATP, avec des IC50 de 72 nM et 58 nM, respectivement. U0126 est un inhibiteur de l'autophagie et de la mitophagie.
-
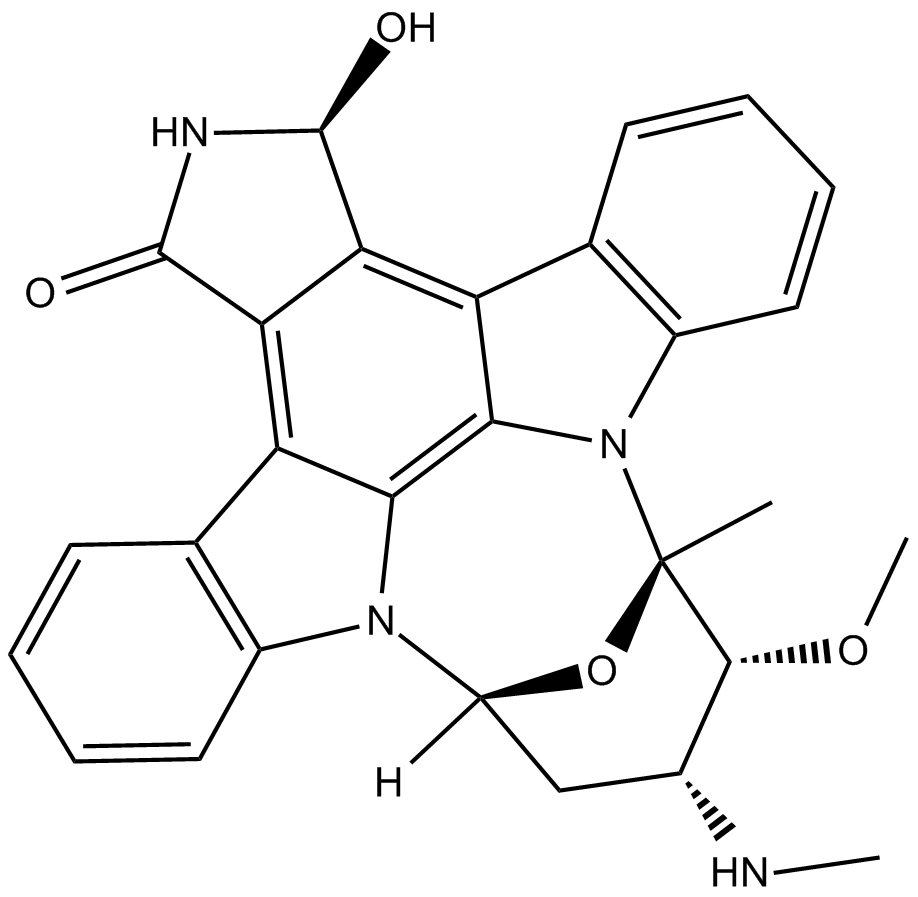
GC16678
UCN-02
UCN-02 (7-epi-Hydroxystaurosporine) est un inhibiteur sélectif de la protéine kinase C (PKC) produit par la souche Streptomyces N-12, avec des IC50 de 62 nM et 250 nM pour la PKC et la protéine kinase A (PKA), respectivement. UCN-02 (7-épi-Hydroxystaurosporine) présente un effet cytotoxique sur la croissance des cellules HeLa S3.
-
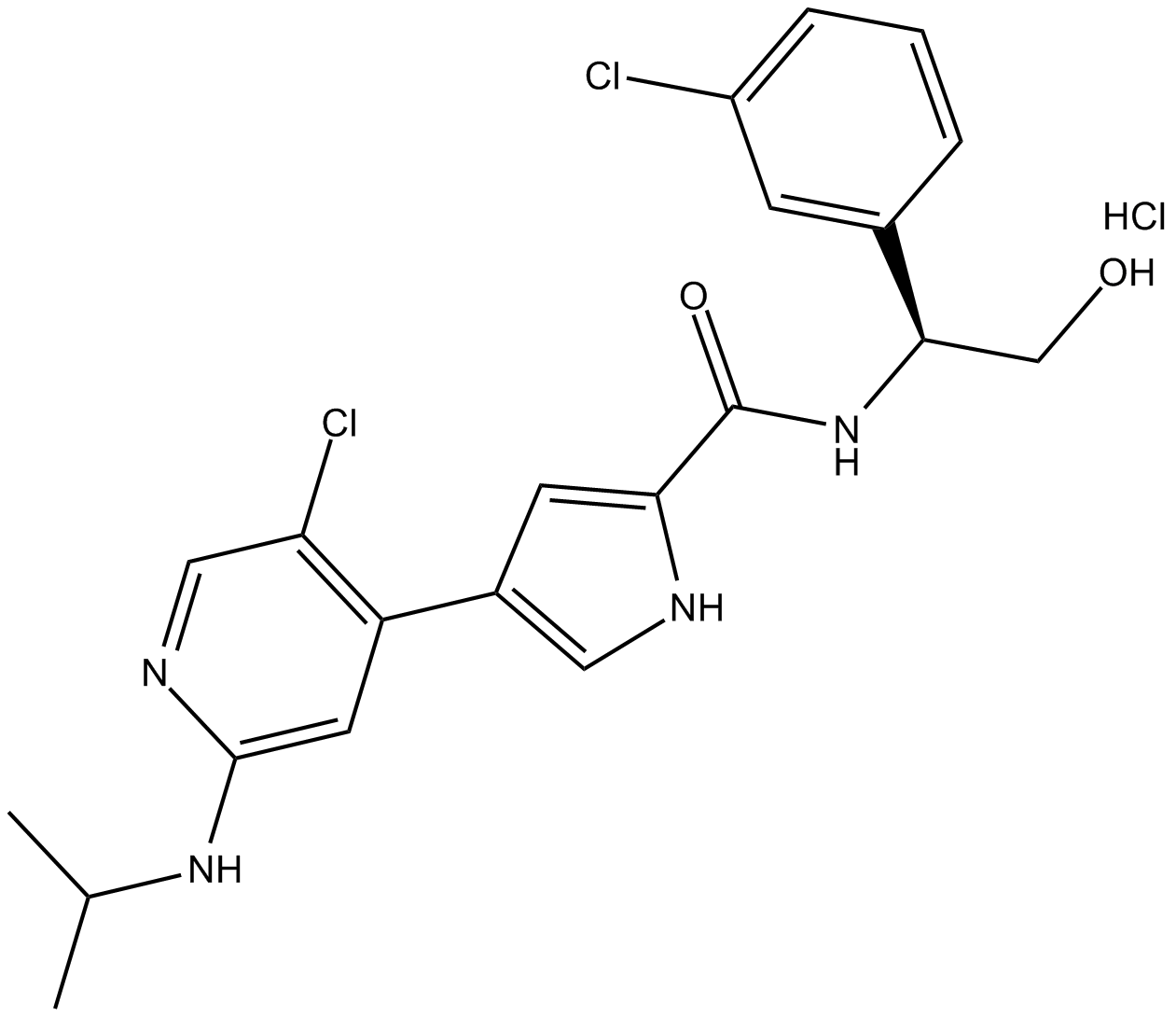
GC11422
Ulixertinib (hydrochloride)
L'ulixertinib (chlorhydrate) (chlorhydrate de BVD-523) est un inhibiteur covalent puissant, actif par voie orale, hautement sélectif, compétitif pour l'ATP et réversible des kinases ERK1/2, avec une IC50 \u003c0,3 nM contre ERK2. L'ulixertinib (chlorhydrate) inhibe l'ERK2 phosphorylée (pERK) et la kinase RSK en aval (pRSK) dans une lignée cellulaire de mélanome A375.
-
GC19366
UM-164
L'UM-164 (DAS-DFGO-II) est un inhibiteur très puissant de c-Src avec un Kd de 2,7 nM. L'UM-164 inhibe également puissamment p38α et p38β.
-
GC15004
URMC-099
L'URMC-099 est un inhibiteur de la lignée mixte kinase de type 3 (MLK3) (IC50 = 14 nM) biodisponible et puissant par voie orale, doté d'excellentes propriétés de pénétration de la barrière hémato-encéphalique.
-
GC38679
Urolithin B
L'urolithine B est l'un des métabolites microbiens intestinaux des ellagitanins et a des effets anti-inflammatoires et antioxydants.
-
GC17818
Vacquinol-1
Le Vacquinol-1 (NSC13316) est un activateur spécifique de MKK4 qui active les voies MAPK. Le Vacquinol-1 induit spécifiquement la mort des cellules de glioblastome humain (GC), atténue la progression tumorale et prolonge la survie dans un modèle murin de glioblastome multiforme (GBM). Le vacquinol-1 induit également l'apoptose dans les cellules de carcinome hépatocellulaire (HCC).
-
GC13412
Vemurafenib (PLX4032, RG7204)
Un inhibiteur de la mutation V600E et du type sauvage B-Raf.
-
GC17031
VRT752271
VRT752271 (BVD-523; VRT752271) est un inhibiteur covalent puissant, actif par voie orale, hautement sélectif, compétitif pour l'ATP et réversible des kinases ERK1/2, avec une IC50 <0,3 nM contre ERK2. VRT752271 (BVD-523; VRT752271) inhibe l'ERK2 phosphorylée (pERK) et la kinase RSK en aval (pRSK) dans une lignée cellulaire de mélanome A375.
-
GC13115
VX-11e
A selective ERK inhibitor
-
GC17508
VX-702
VX-702 est un inhibiteur hautement sélectif de p38α MAPK, 14 fois plus puissant contre p38α que p38β.
-
GC12926
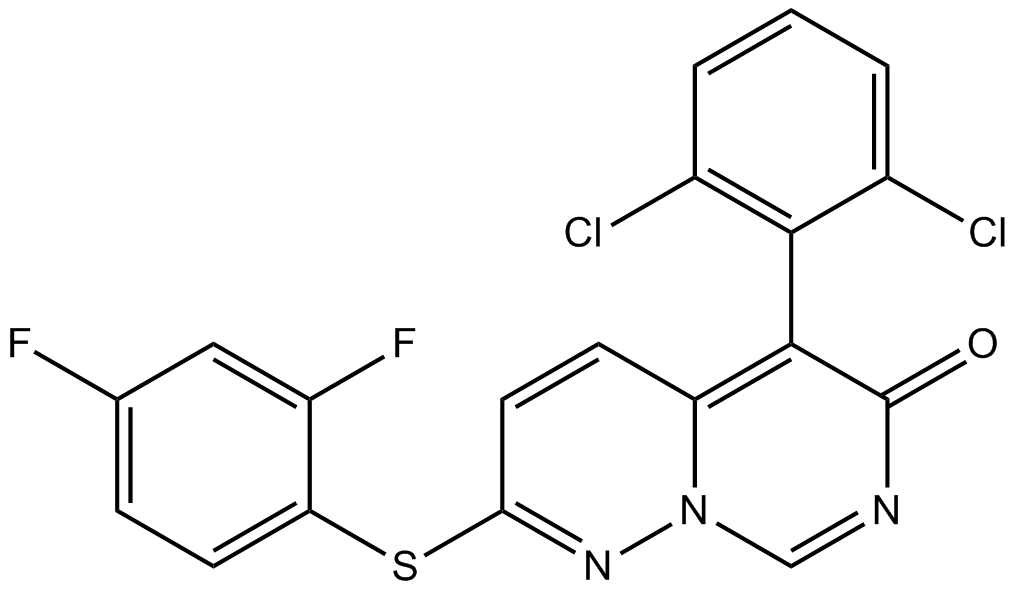
VX-745
VX-745 (VX-745) est un puissant pénétrant de la barrière hémato-encéphalique, inhibiteur hautement sélectif de p38α ; inhibiteur avec une IC50 pour p38α ; de 10 nM et pour p38β ; de 220nM.
-
GC30857
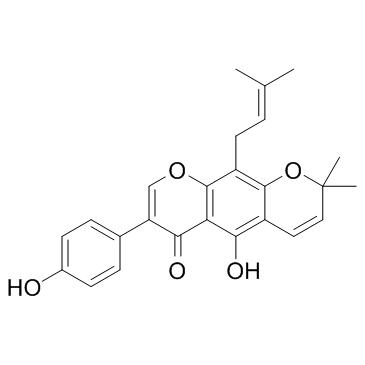
Warangalone (Scandenolone)
Warangalone (Scandénolone) est un composé antipaludéen qui peut inhiber la croissance des deux souches de parasites 3D7 (sensibles À la chloroquine) et K1 (résistantes À la chloroquine) avec des CI50 de 4,8 μ g/mL et 3,7 μ g/mL, respectivement.
-
GC38053
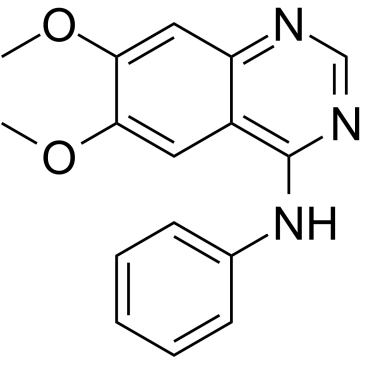
WHI-P258
WHI-P258, un composé quinazoline, se lie au site actif de JAK3 avec un Ki estimé de 72 μM. WHI-P258 n'inhibe pas JAK3 et n'affecte pas l'agrégation plaquettaire induite par la thrombine même À 100 μM.
-
GC12691
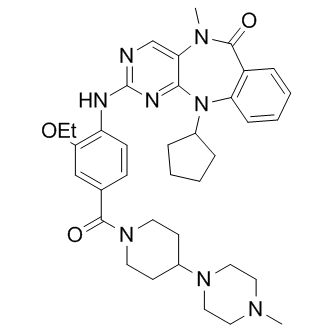
XMD17-109
A selective ERK5 inhibitor
-
GC11076
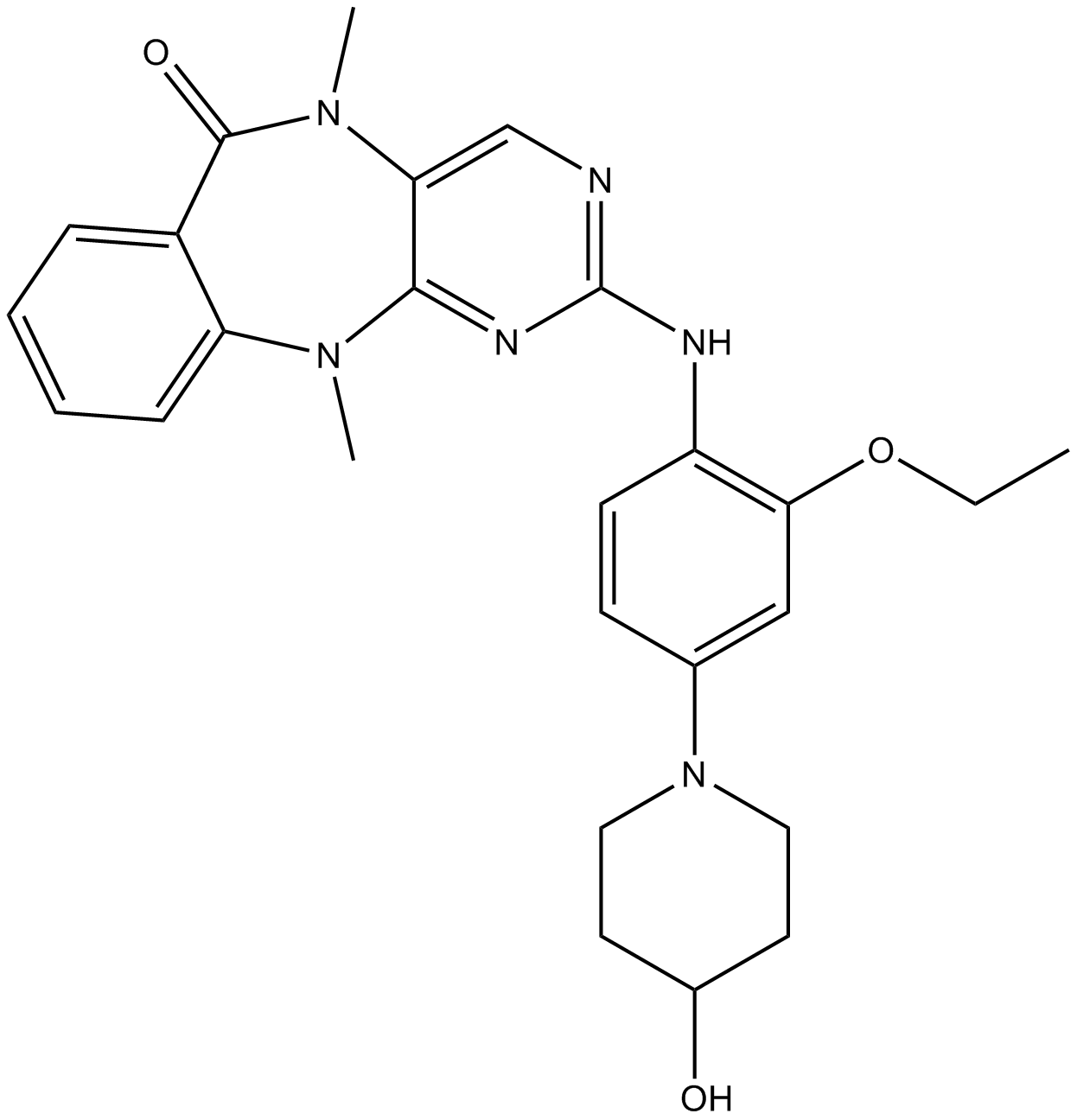
XMD8-92
A selective ERK5 inhibitor
-
GC16169
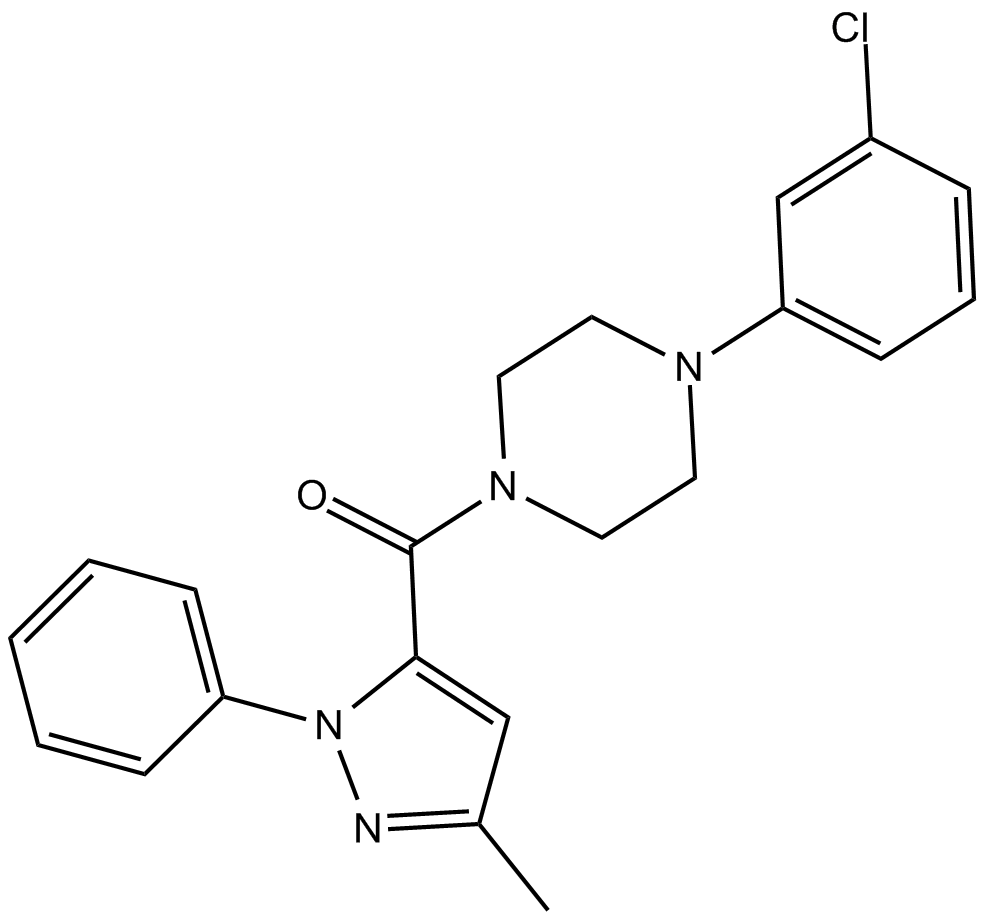
XRP44X
XRP44X inhibe l'activation de la transcription induite par Ras avec l'IC50 de 10 nM.
-
GC62146
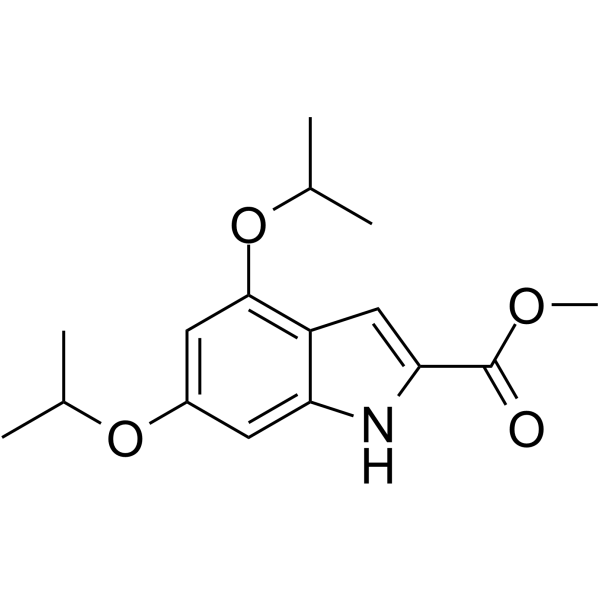
XST-14
XST-14 est un inhibiteur d'ULK1 puissant, compétitif et hautement sélectif avec une IC50 de 26,6 nM. XST-14 induit une inhibition de l'autophagie en réduisant la phosphorylation du substrat en aval ULK1. XST-14 induit l'apoptose dans les cellules de carcinome hépatocellulaire (HCC) et a des effets antitumoraux.
-
GC63264
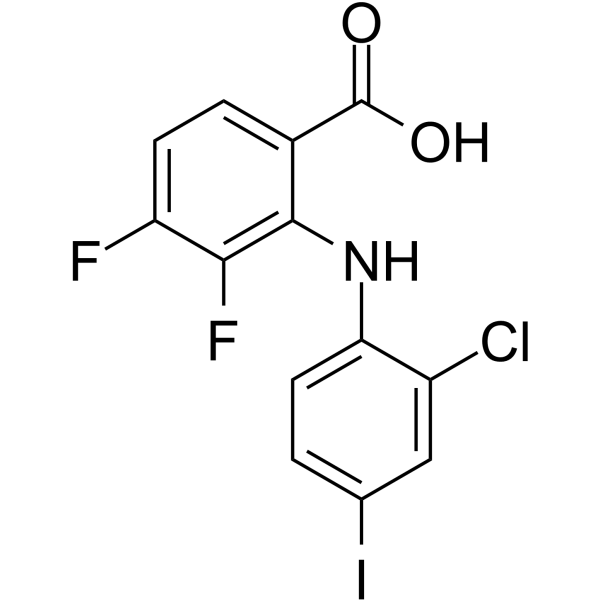
Zapnometinib
Le zapnometinib (PD0184264), un métabolite actif du CI-1040, est un inhibiteur de MEK, avec une IC50 de 5,7 nM.
-
GC13286
ZM336372
Le ZM336372 est un puissant inhibiteur de la protéine kinase c-Raf. La valeur IC50 est de 0,07 μM dans le test standard, qui contient 0,1 mM d'ATP.
-
GC63425
Zunsemetinib
Le zunsemetinib (CDD-450) est un inhibiteur de la voie de la protéine kinase 2 (MK2) activée par le mitogène p38α, actif et sélectif par voie orale.