Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nombre del producto Información
-
GC12426
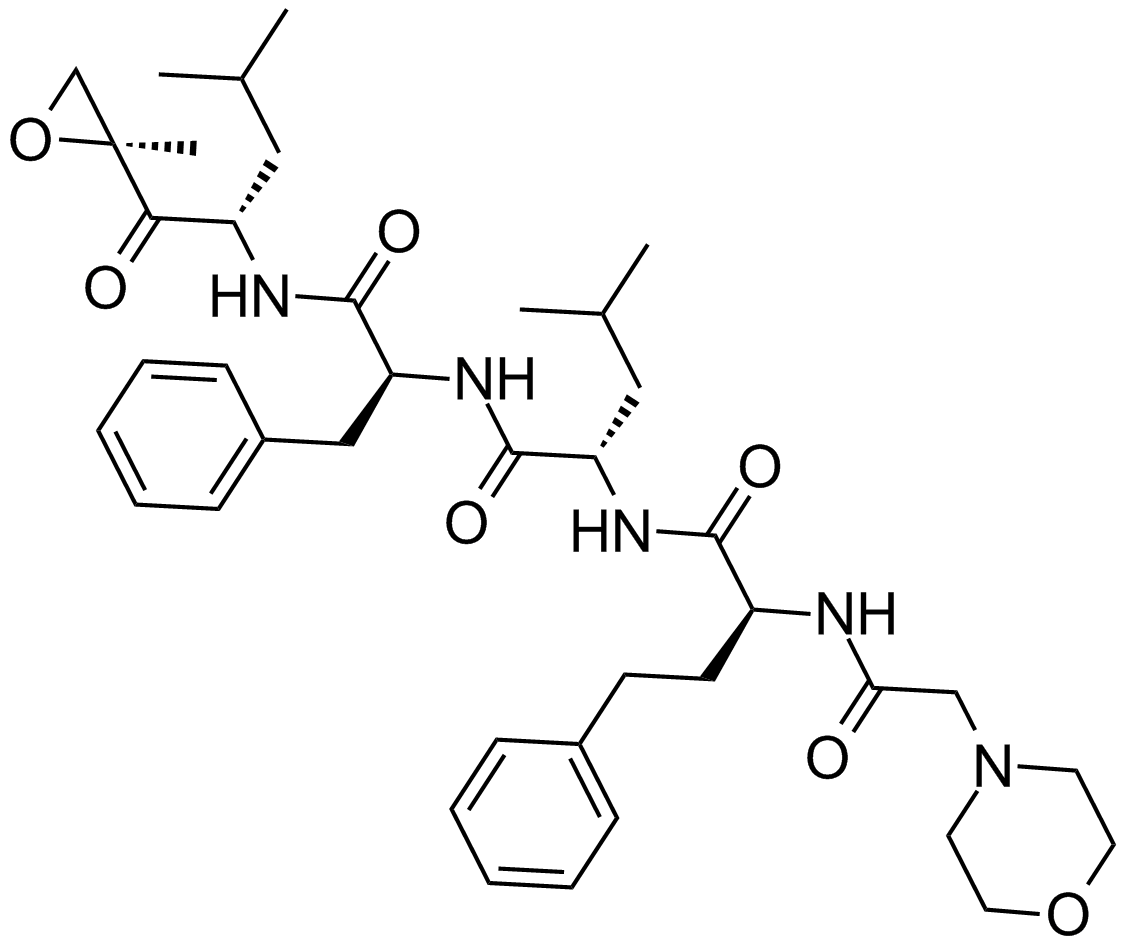
Birinapant (TL32711)
Un antagonista de cIAP1, cIAP2 y XIAP.
-
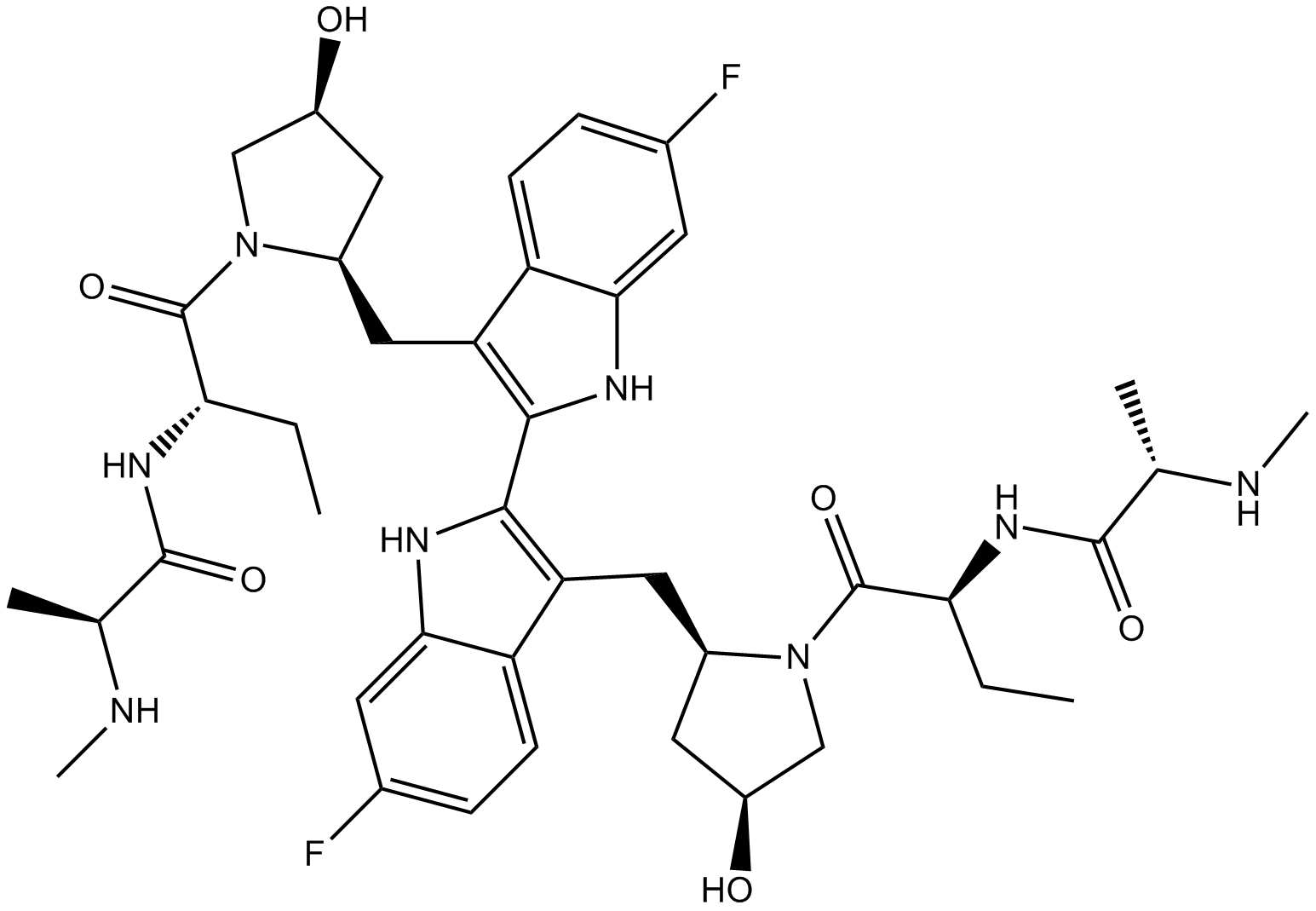
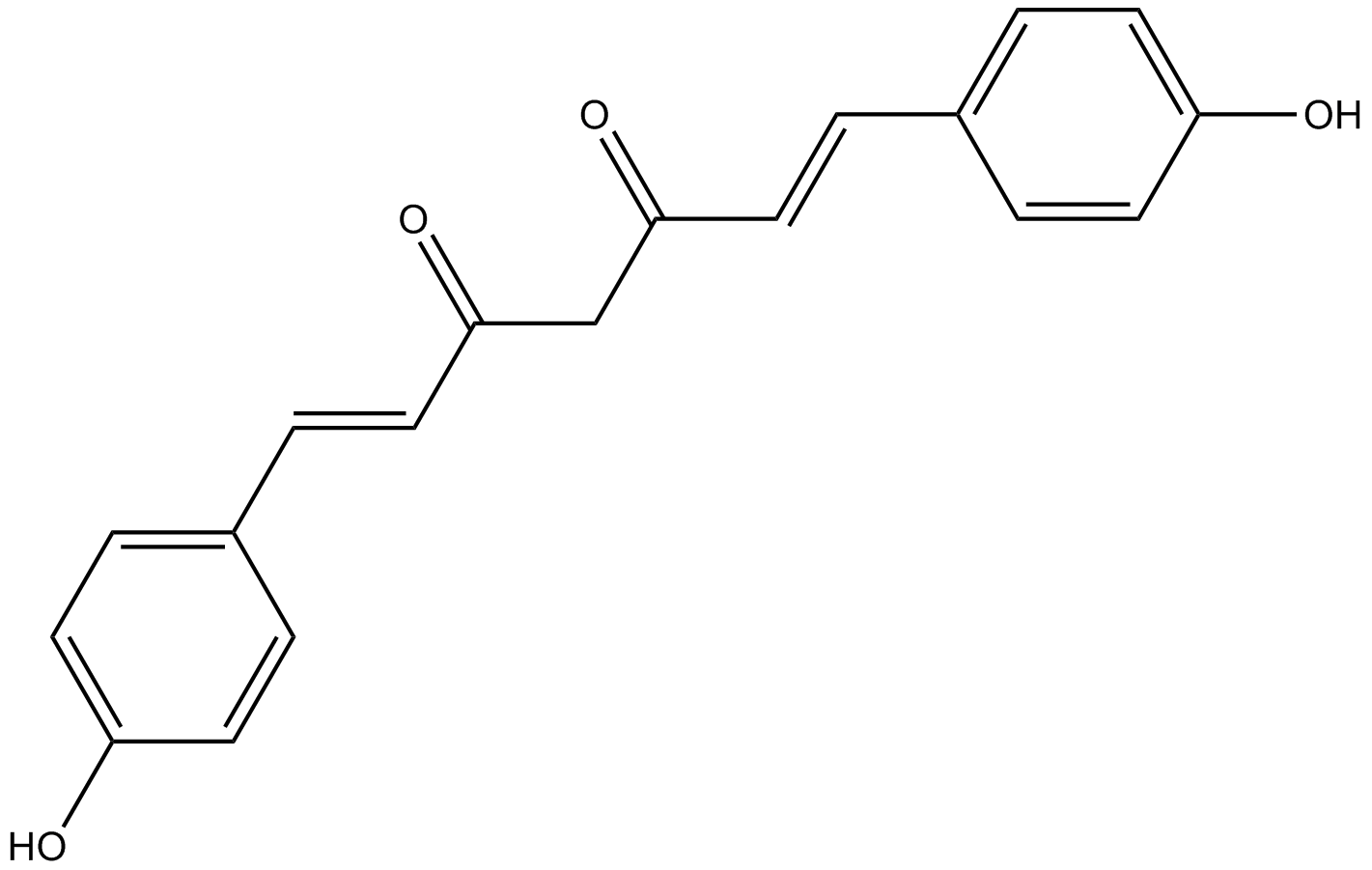
GN10037
Bisdemethoxycurcumin
-
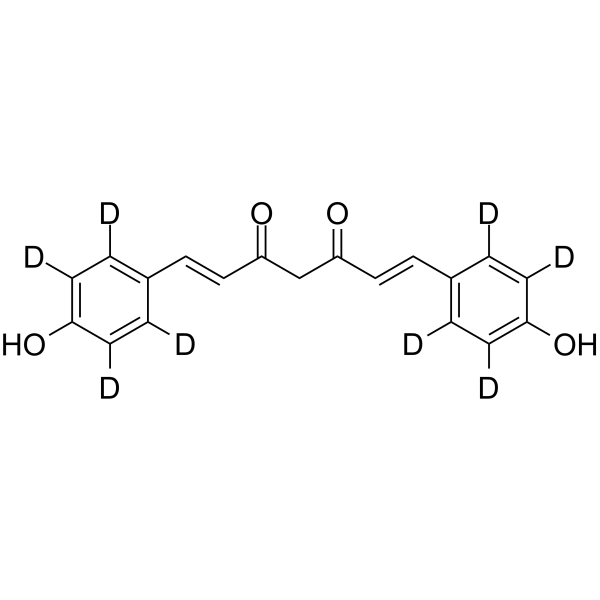
GC68308
Bisdemethoxycurcumin-d8
-
GC35530
BJE6-106
BJE6-106 (B106) es un potente inhibidor selectivo de PKCδ de tercera generaciÓn con una IC50 de 0,05 μM y una selectividad de objetivos sobre la isoenzima PKCα clÁsica de la PKC (IC50 = 50 μM).

-
GC11931
BKM120
An inhibitor of class I PI3K isoforms
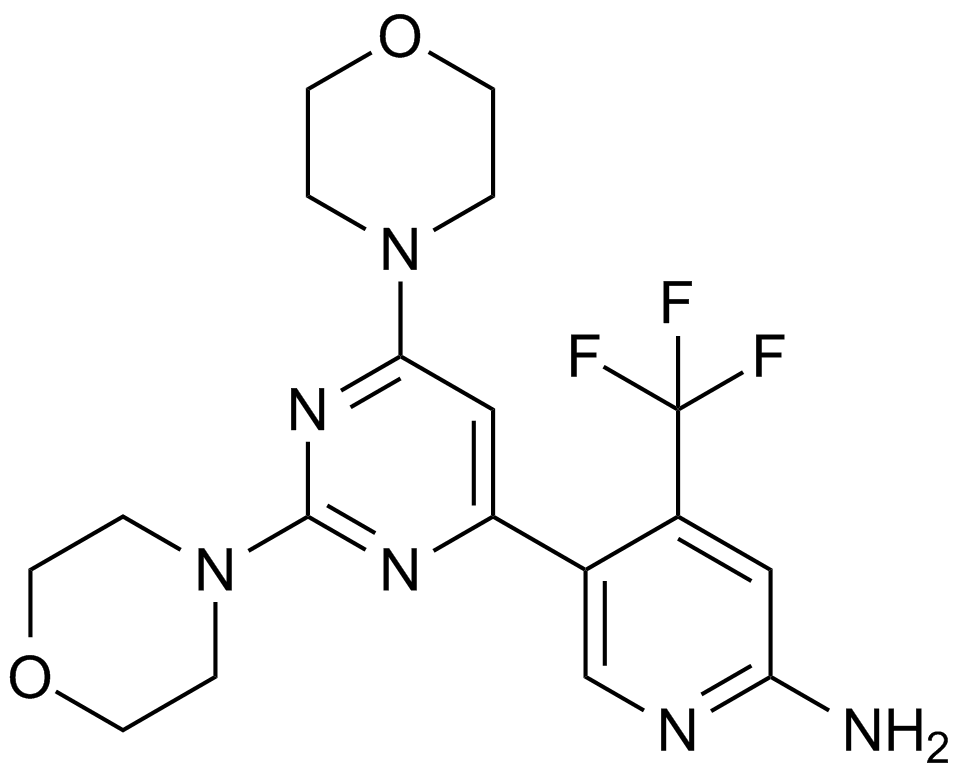
-
GC65428
BLM-IN-1
BLM-IN-1 (compuesto 29) es un inhibidor eficaz de la proteÍna del sÍndrome de Bloom (BLM), con un fuerte KD de uniÓn a BLM de 1,81 μM y una IC50 de 0,95 μM para BLM. Induce la respuesta al daÑo del ADN, asÍ como la apoptosis y la detenciÓn de la proliferaciÓn en las células cancerosas.
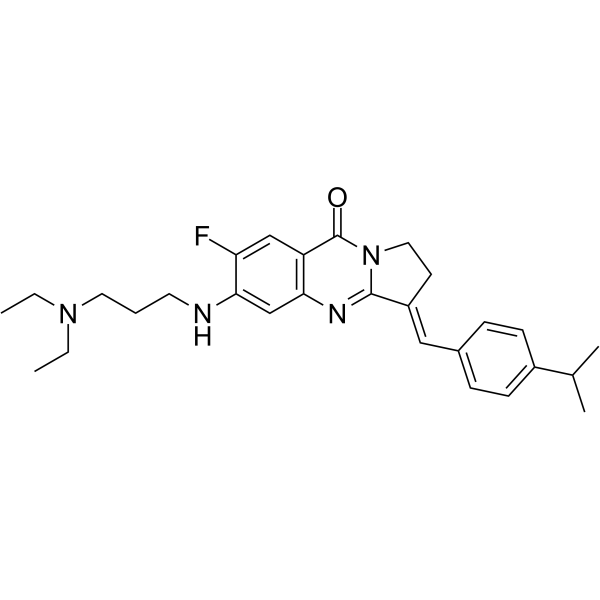
-
GC33407
BM 957
BM 957 es un potente inhibidor de Bcl-2 y Bcl-xL, con Kis de 1,2, <1 nM y IC50 de 5,4, 6,0 nM respectivamente.
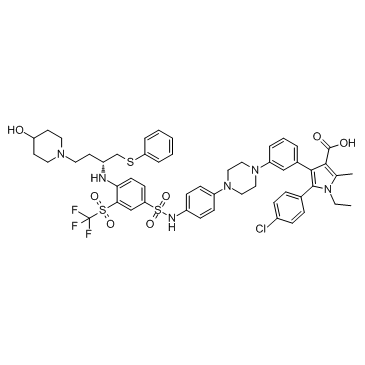
-
GC13498
BM-1074
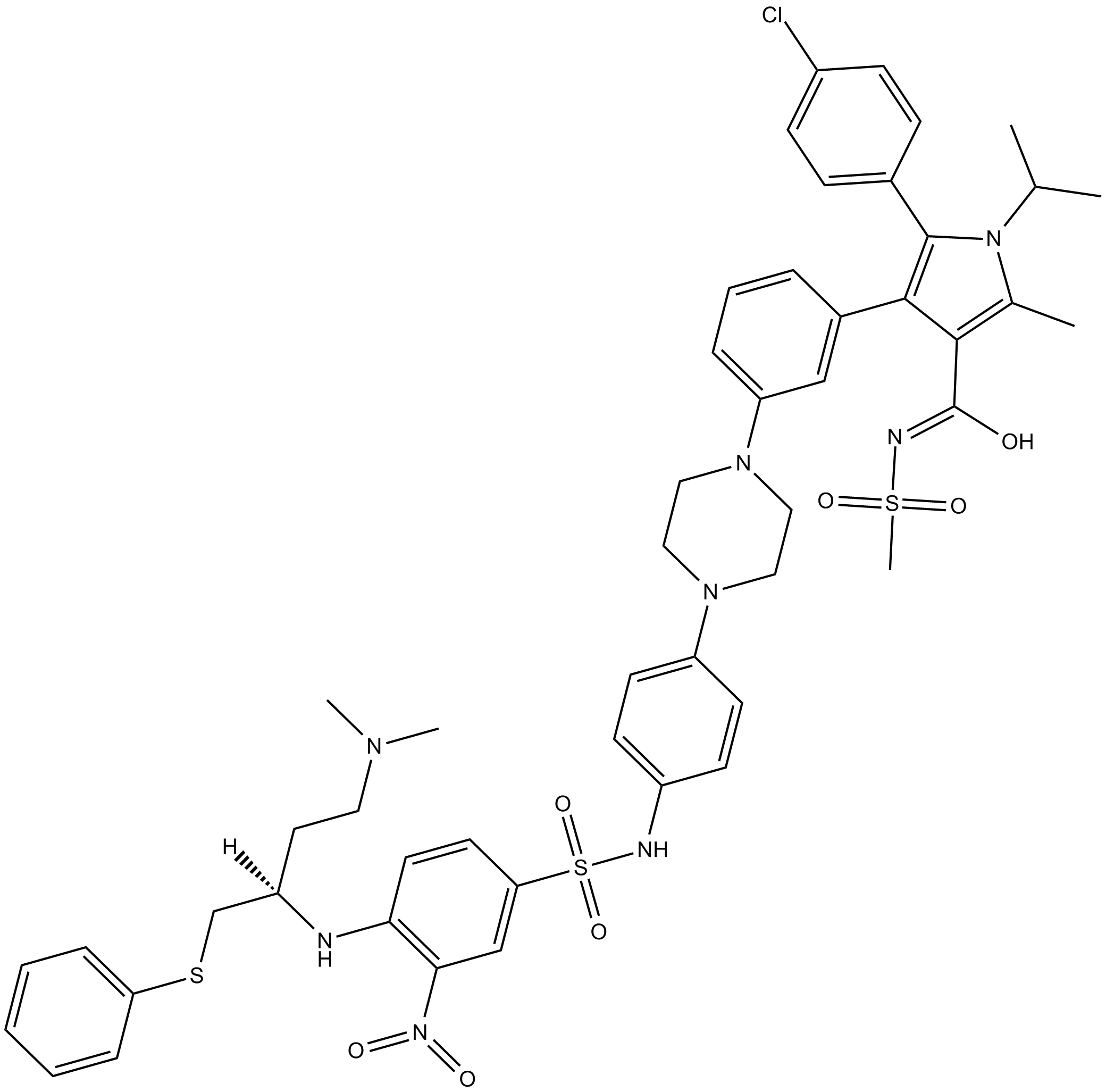
-
GC62871
BM-1244
BM-1244 (APG-1252-M1) es un potente inhibidor de Bcl-xL/Bcl-2 con Kis de 134 y 450 nM para Bcl-xL y Bcl-2, respectivamente. BM-1244 inhibe los fibroblastos senescentes (SnCs) con un EC50 de 5 nM. (De patente WO2019033119A1).
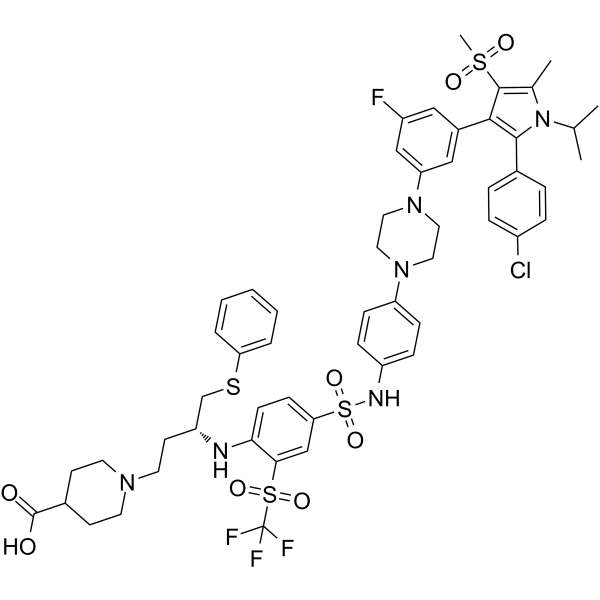
-
GC12822
BML-210(CAY10433)
BML-210(CAY10433) es un nuevo inhibidor de HDAC y su mecanismo de acciÓn no ha sido caracterizado.
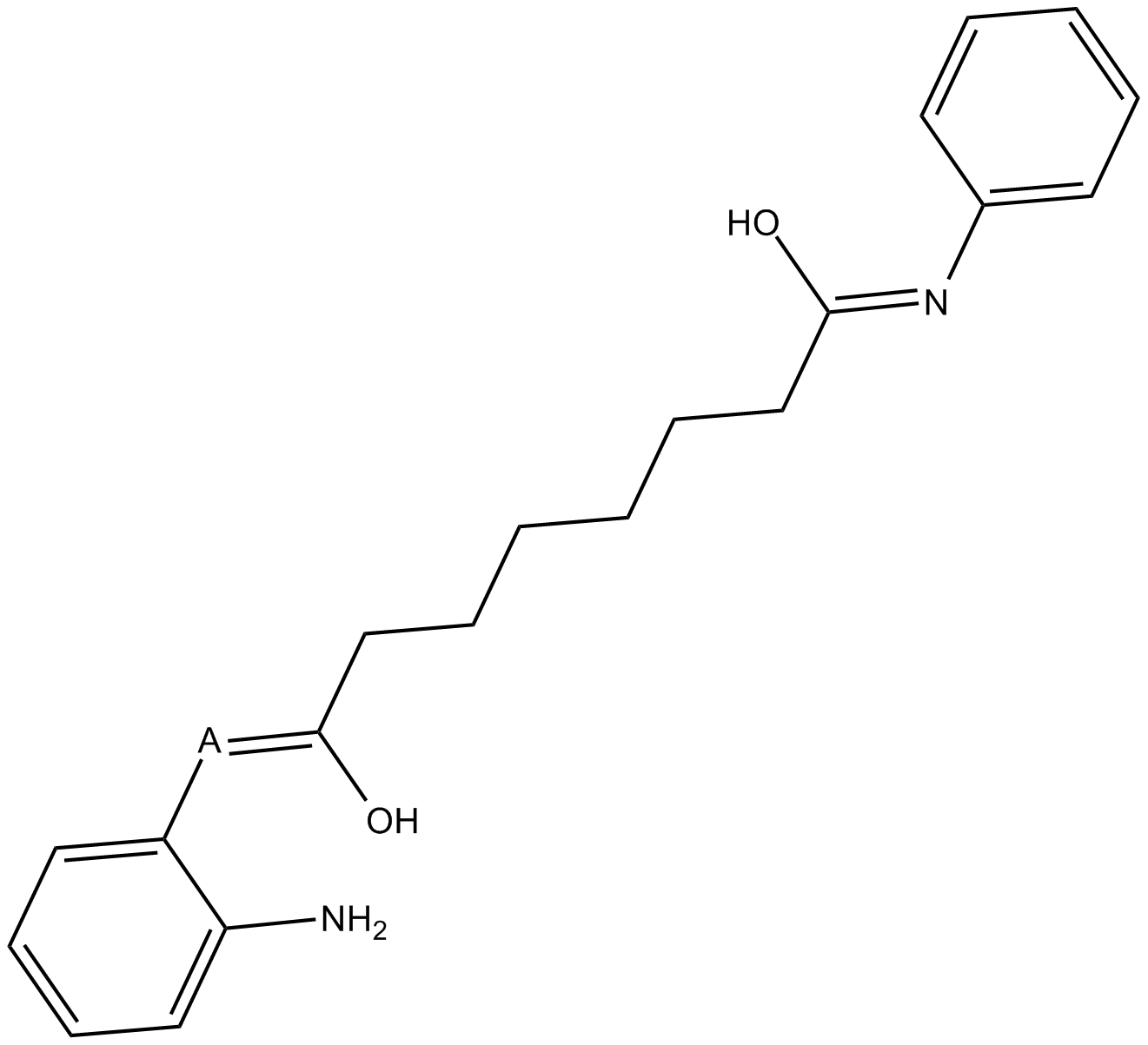
-
GC11648
BML-277
BML-277 es un inhibidor selectivo de la quinasa 2 del punto de control (Chk2) con una IC50 de 15 nM.
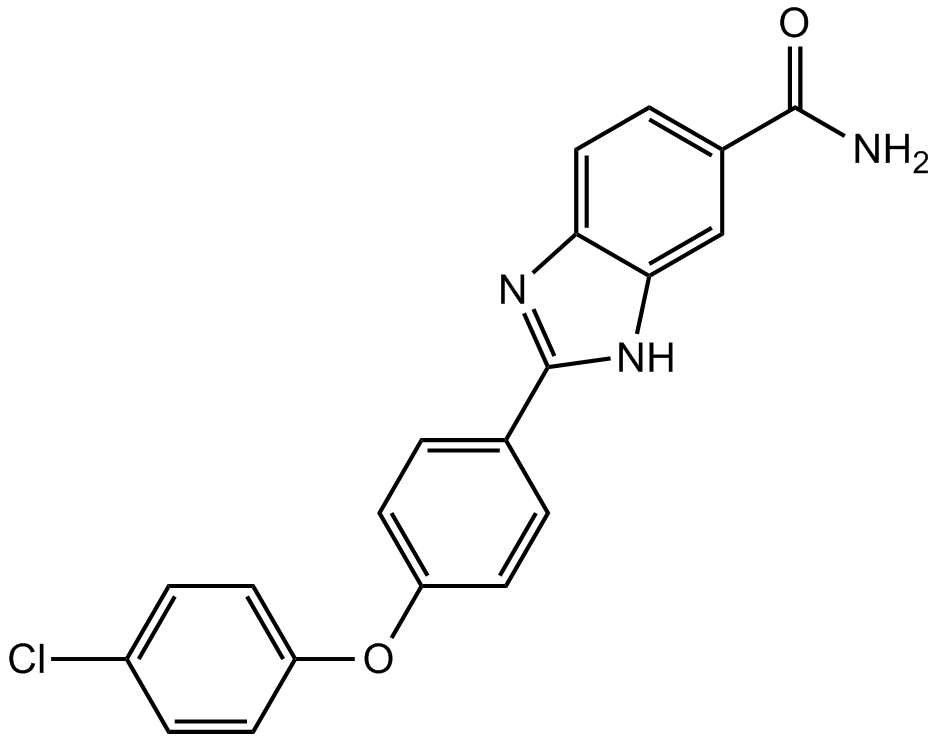
-
GC42953
BMS 345541 (trifluoroacetate salt)
BMS 345541 is a cell permeable inhibitor of the IκB kinases IKKα and IKKβ (IC50s = 4 and 0.3 μM).
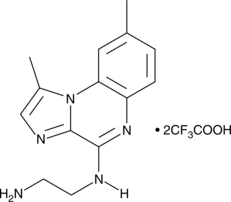
-
GC25160
BMS-1001
BMS-1001 is a potent inhibitor of PD-1/PD-L1 interaction with EC50 of 253 nM. BMS-1001 alleviates the inhibitory effect of the soluble PD-L1 on the T-cell receptor-mediated activation of T-lymphocytes.
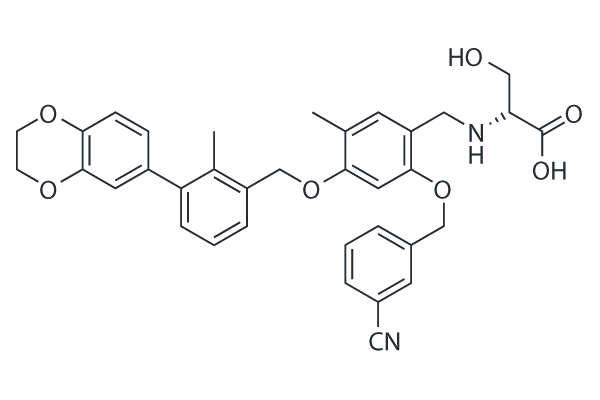
-
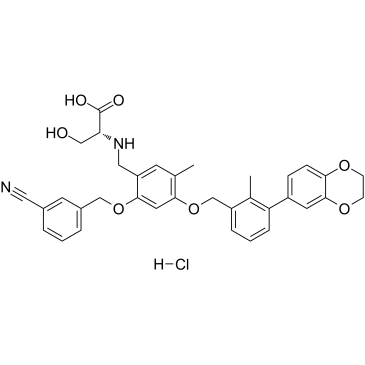
GC38740
BMS-1001 hydrochloride
El clorhidrato de BMS-1001 es un inhibidor del punto de control inmunitario PD-L1/PD-1 humano activo por vÍa oral.
-
GC31753
BMS-1166 (PD-1/PD-L1-IN1)
BMS-1166 (PD-1/PD-L1-IN1) es un potente inhibidor del punto de control inmunitario de PD-1/PD-L1.
-
GC38131
BMS-1166 hydrochloride
El clorhidrato de BMS-1166 es un potente inhibidor del punto de control inmunitario PD-1/PD-L1.

-
GC13628
BMS-833923
An orally bioavailable Smo inhibitor
-
GC62682
BMSpep-57 hydrochloride
El clorhidrato de BMSpep-57 es un inhibidor peptÍdico macrocÍclico potente y competitivo de la interacciÓn PD-1/PD-L1 con una IC50 de 7,68nM. El clorhidrato de BMSpep-57 se une a PD-L1 con Kds de 19 nM y 19,88 nM en ensayos MST y SPR, respectivamente. El clorhidrato de BMSpep-57 facilita la funciÓn de las células T al aumentar la producciÓn de IL-2 en las PBMC.
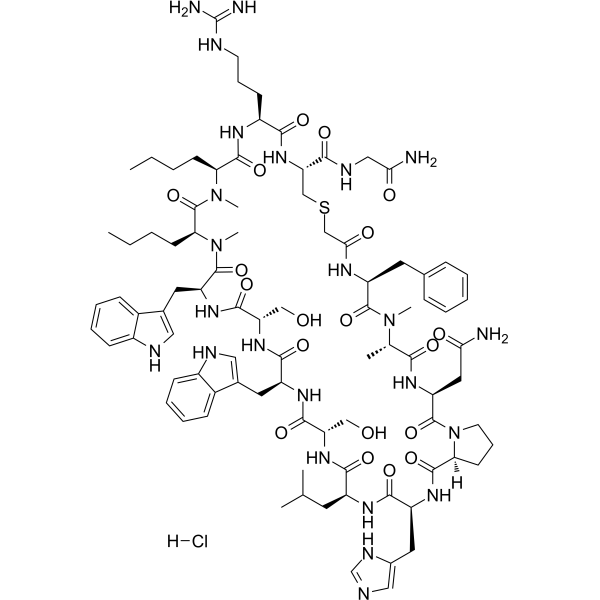
-
GA20897
Boc-Arg(Boc)₂-OH
An amino acid building block

-
GC16774
Boc-D-FMK
An irreversible pan-caspase inhibitor
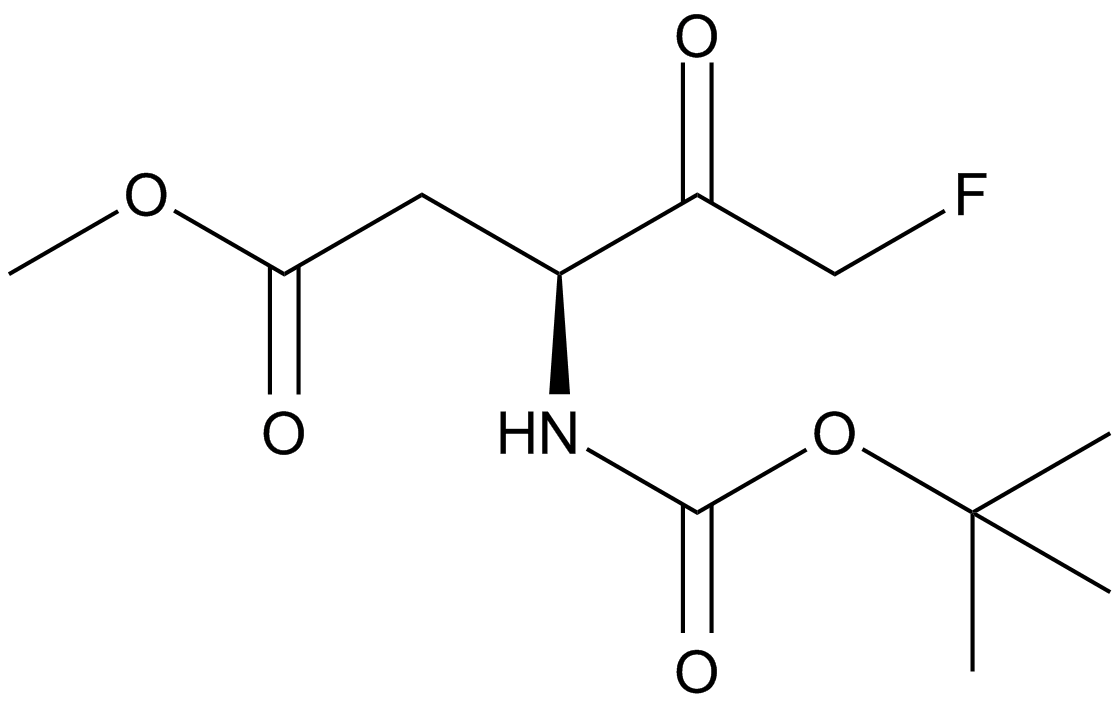
-
GC33501
Bornyl acetate
El acetato de bornilo es un odorante potente que exhibe uno de los factores de diluciÓn de sabor (factor FD) mÁs altos. El acetato de bornilo posee actividad anticancerÍgena.
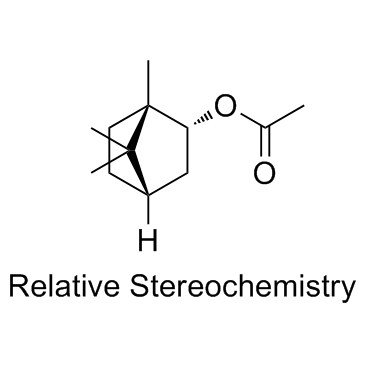
-
GC11040
Borrelidin
La borrelidina (treponemicina) es un inhibidor de la sintetasa de treonil-tRNA bacteriano y eucariota que es un antibiÓtico macrÓlido que contiene nitrilo aislado de Streptomyces rochei.
-
GC17644
Bortezomib (PS-341)
Un inhibidor potente y reversible del proteasoma 20S.
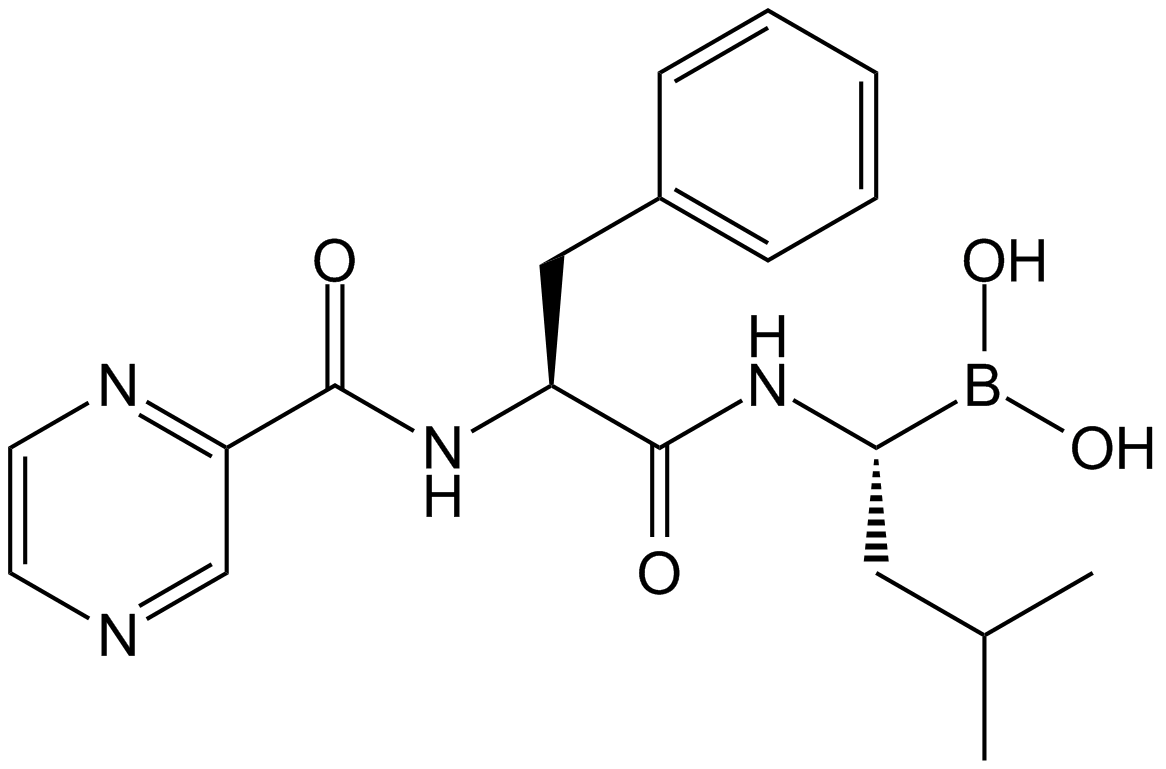
-
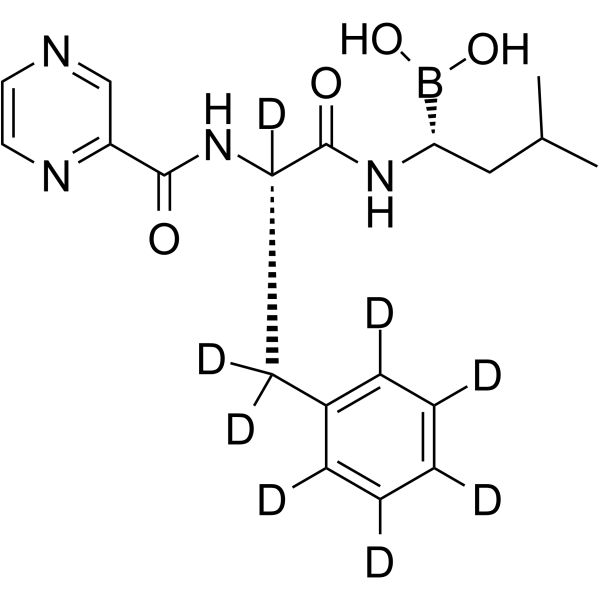
GC65010
Bortezomib-d8
Bortezomib-d8 (PS-341-d8) es el Bortezomib marcado con deuterio. Bortezomib (PS-341) es un inhibidor reversible y selectivo del proteasoma e inhibe potentemente el proteasoma 20S (Ki = 0,6 nM) al atacar un residuo de treonina. Bortezomib interrumpe el ciclo celular, induce la apoptosis e inhibe el NF-κB. Bortezomib es el primer agente anticancerÍgeno inhibidor del proteasoma. Actividad anticancerÍgena.
-
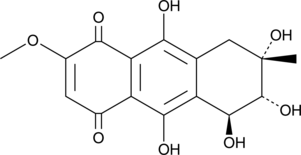
GC40009
Bostrycin
Bostrycin is an anthraquinone originally isolated from B.
-
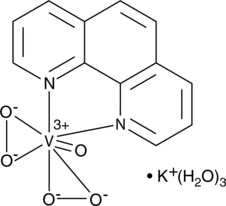
GC42969
bpV(phen) (potassium hydrate)
bpV(phen) (hidrato de potasio), un agente mimético de la insulina, es un potente inhibidor de la proteÍna tirosina fosfatasa (PTP) y PTEN con IC50 de 38 nM, 343 nM y 920 nM para PTEN, PTP-β y PTP-1B, respectivamente.
-
GC42974
Brassinin
La brassinina es el metabolismo de una fitoalexina de la especie Brassica.
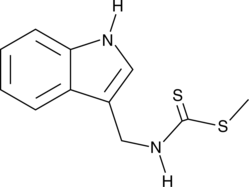
-
GC11632
Brassinolide
A plant growth regulator
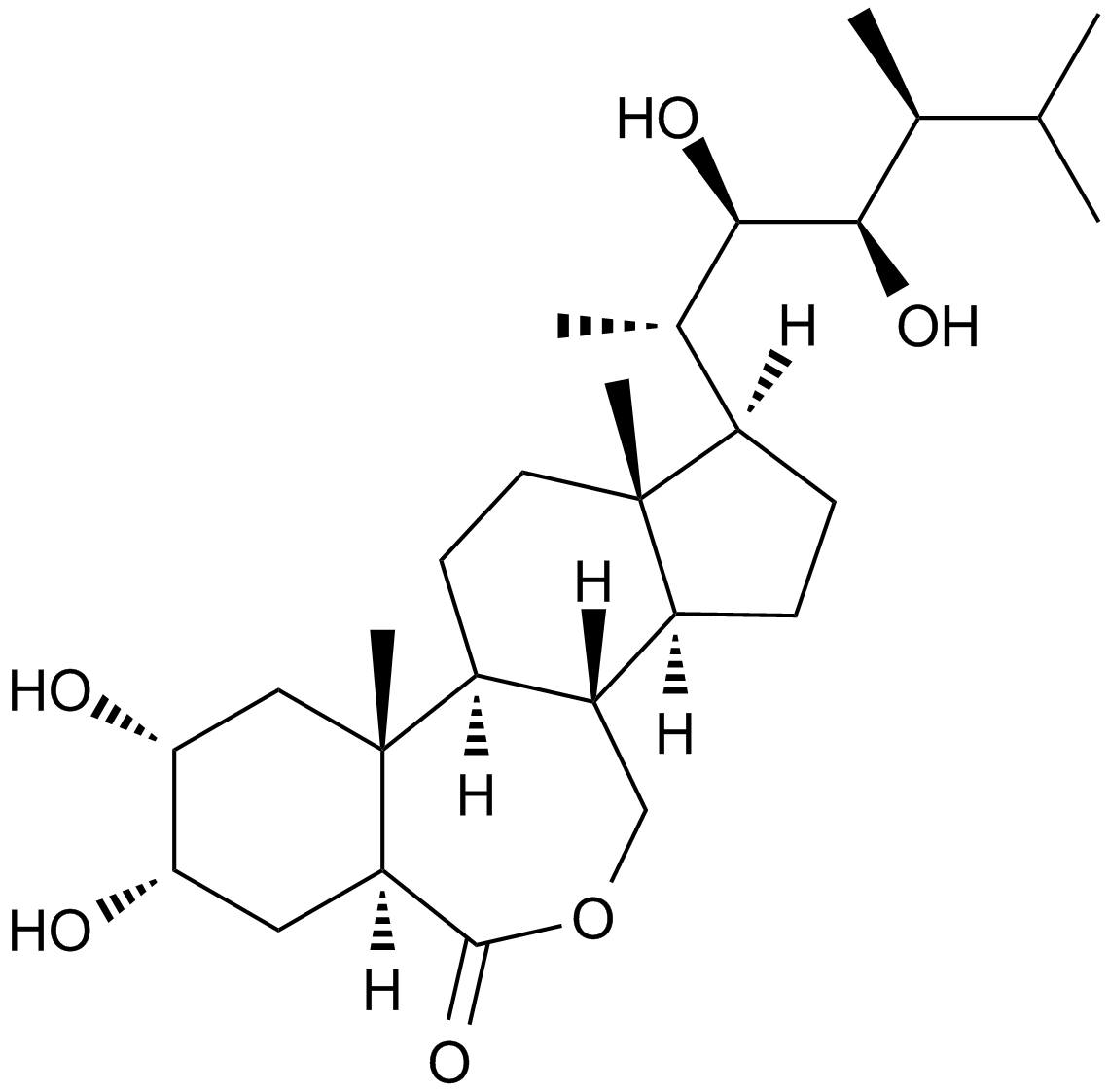
-
GC52101
Brazilein
La brazileÍna es un importante componente inmunosupresor aislado de Caesalpinia sappan L.
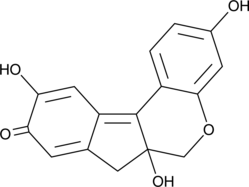
-
GN10802
Brazilin
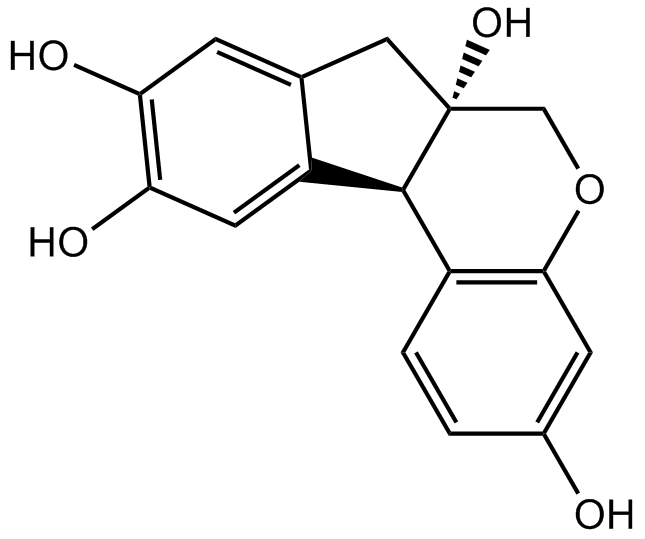
-
GC68288
Brentuximab

-
GC35554
Brevilin A
Brevilin A es un inhibidor de STAT3/JAK activo por vÍa oral (STAT3 IC50=10,6 μM). Brevilin A muestra actividad antitumoral, actividad antiproliferativa para las células cancerosas y puede inducir la apoptosis y la autofagia.

-
GC35555
Britannin
La britanina, aislada de Inula aucheriana, es una lactona sesquiterpénica.
-
GC62536
Bromelain
La bromelina es un fÁrmaco antiinflamatorio derivado del tallo de la piÑa que actÚa a través de la regulaciÓn a la baja del cininÓgeno plasmÁtico, la inhibiciÓn de la expresiÓn de prostaglandina E2, la degradaciÓn de los receptores de productos finales de glicaciÓn avanzada y la regulaciÓn de biomarcadores angiogénicos, asÍ como la acciÓn antioxidante aguas arriba en la vÍa COX. .

-
GC35559
Bruceine D
Bruceine D es un inhibidor de Notch con actividad anticancerÍgena e induce la apoptosis en varias células cancerosas humanas.
-
GC34070
Brusatol (NSC 172924)
Brusatol (NSC 172924) (NSC172924) es un inhibidor Único de la vÍa Nrf2 que sensibiliza un amplio espectro de células cancerosas al cisplatino y otros agentes quimioterapéuticos. Brusatol (NSC 172924) mejora la eficacia de la quimioterapia al inhibir el mecanismo de defensa mediado por Nrf2. Brusatol (NSC 172924) puede convertirse en un agente quimioterapéutico adyuvante. Brusatol (NSC 172924) aumenta la apoptosis celular.
-
GC38014
BT2
BT2 es un inhibidor de la quinasa BCKDC (BDK) con una IC50 de 3,19 μM.
-
GC38467
BTdCPU
BTdCPU es un potente activador de la quinasa eIF2α regulada por hemo (HRI). BTdCPU promueve la fosforilaciÓn de eIF2α y la apoptosis inducida en células resistentes.
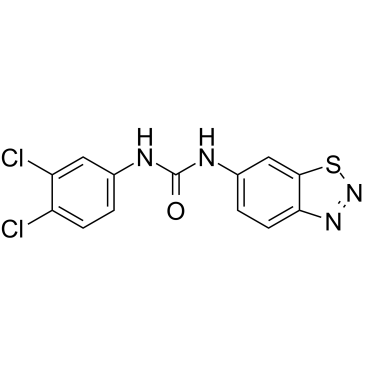
-
GC34511
BTR-1
BTR-1 es un agente anticancerÍgeno activo, provoca la detenciÓn de la fase S y afecta la replicaciÓn del ADN en las células leucémicas. BTR-1 activa la apoptosis e induce la muerte celular.
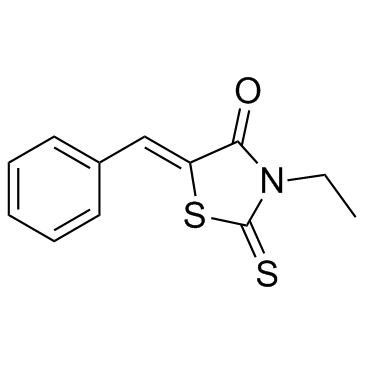
-
GC11141
BTZO 1
BTZO 1 se une al factor inhibidor de la migración de macrófagos (MIF) con un valor de Kd de 68,6 nM, y su unión requiere el N-terminal Pro1.
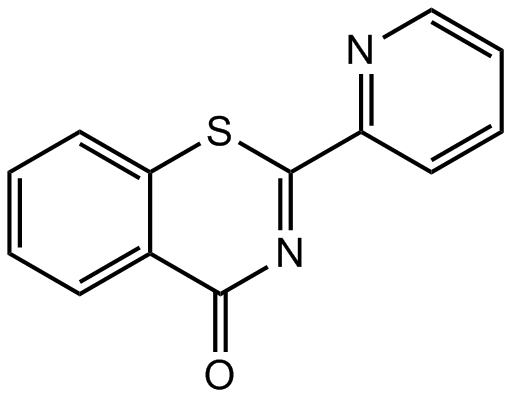
-
GC48376
Burnettramic Acid A
A fungal metabolite with diverse biological activities
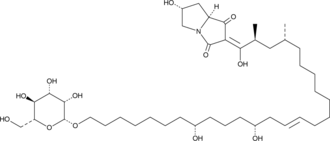
-
GC48409
Burnettramic Acid A aglycone
La aglicona del Ácido burnettrÁmico A es un metabolito fÚngico que se encuentra en Aspergillus burnettii.
-
GC13671
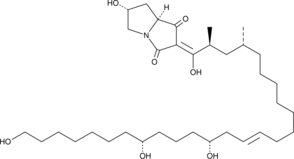
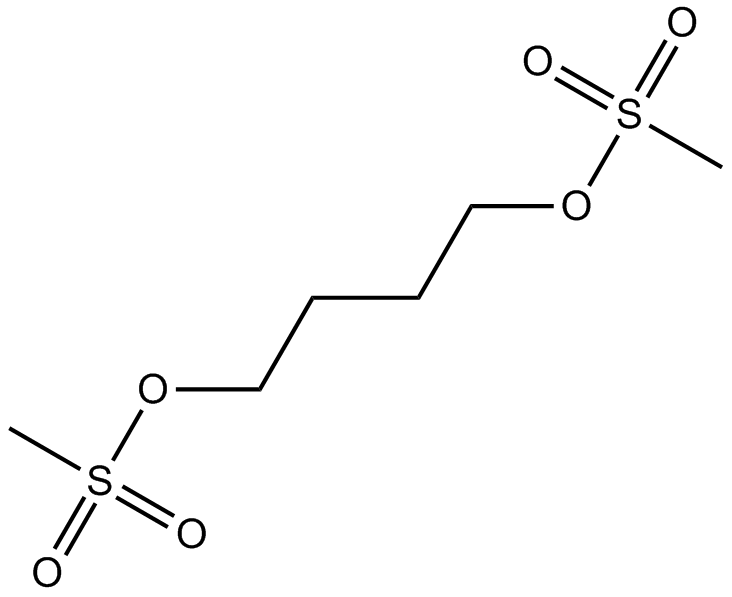
Busulfan
El busulfÁn es un potente alquilante con efecto inmunosupresor selectivo sobre la médula Ósea.
-
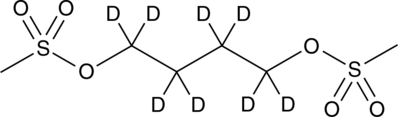
GC46962
Busulfan-d8
Busulfan-D8 es un Busulfan marcado con deuterio. El busulfÁn es un sulfonato de alquilo que actÚa como agente antineoplÁsico alquilante. El busulfÁn forma enlaces cruzados intra e intercatenarios en el ADN. En mamÍferos, Busulfan causa una reducciÓn profunda y prolongada en la generaciÓn de progenitores hematopoyéticos sin afectar significativamente los niveles de linfocitos o las respuestas de anticuerpos humorales.
-
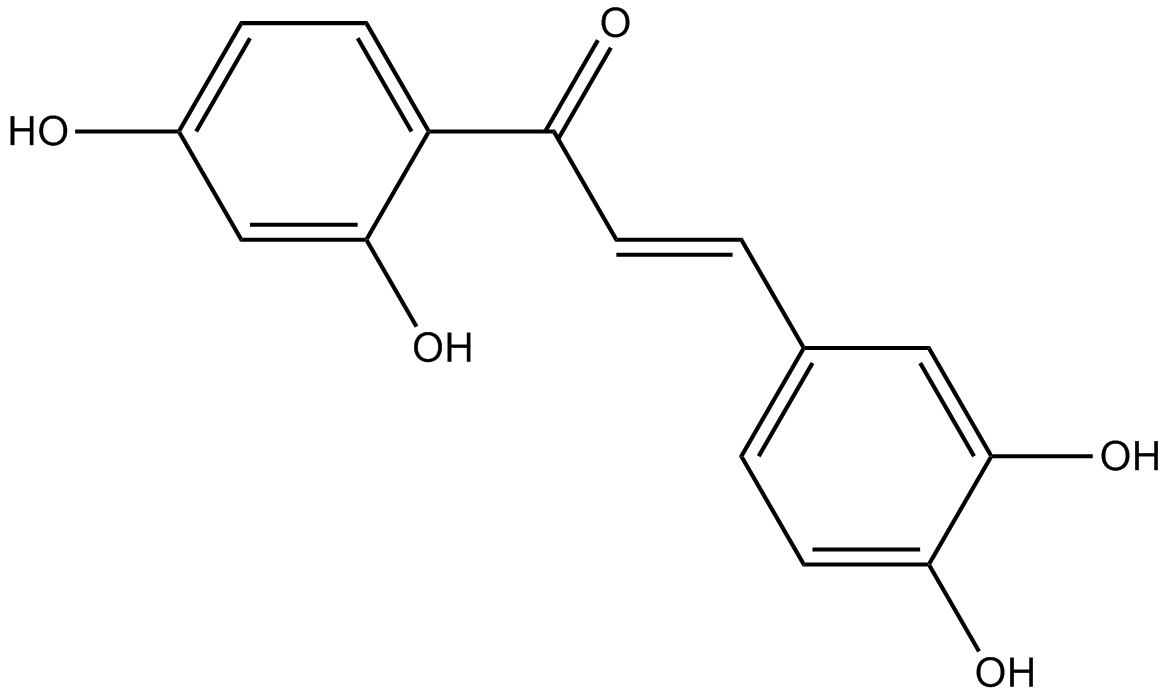
GC10944
Butein
La buteÍna es un inhibidor de la PDE especÍfico de AMPc con una IC50 de 10,4 μM para la PDE4. La buteÍna es un inhibidor especÍfico de la proteÍna tirosina quinasa con IC50 de 16 y 65 μM para EGFR y p60c-src en células HepG2. La buteÍna sensibiliza las células HeLa al cisplatino a través de las vÍas AKT y ERK/p38 MAPK al dirigirse a FoxO3a. Butein es un activador SIRT1 (STAC).
-
GC46104
Butyric Acid-d7
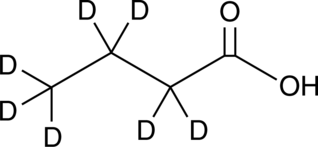
An internal standard for the quantification of sodium butyrate
-
GC12333
BV6
BV6 es un antagonista de cIAP1 y XIAP, miembros de la familia de inhibidores de la apoptosis (IAP).
-
GC48433
BX-320
BX-320 es un inhibidor directo de PDK1, activo por vÍa oral, competitivo con ATP y selectivo con una IC50 de 30 nM en un formato de ensayo de quinasa directo. BX-320 también induce apoptosis. Efecto anticancerÍgeno.
-
GC16818
BX-912
BX-912 es un inhibidor de PDK1 directo, selectivo y competitivo con ATP (IC50 = 26 nM). BX-912 bloquea la seÑalizaciÓn de PDK1/Akt en las células tumorales e inhibe el crecimiento dependiente del anclaje de una variedad de lÍneas de células tumorales en cultivo o induce la apoptosis.
-
GC31806
Bz 423 (BZ48)
Bz 423 (BZ48) es una 1,4-benzodiazepina proapoptÓtica con propiedades terapéuticas en modelos murinos de lupus que demuestra selectividad por los linfocitos autorreactivos y activa Bax y Bak.
-
GC33826
C 87
C 87 es un nuevo inhibidor de TNFα de molécula pequeÑa; inhibe potentemente la citotoxicidad inducida por TNFα con una IC50 de 8,73 μM.
-
GC19095
C-DIM12
C-DIM12 es un activador sintético de Nurr1 que induce la expresiÓn de los genes Nurr1 y DA en lÍneas celulares y neuronas primarias.
-
GC43028
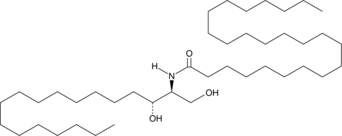
C16 Ceramide (d18:1/16:0)
Los ceramidas se generan a partir de la esfingomielina mediante la activación de las esfingomielinasas o a través de la vía de síntesis de novo, que requiere la acción coordinada de la serina palmitoil transferasa y la ceramida sintasa.
-
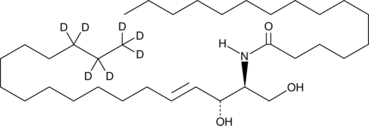
GC46976
C16 Ceramide-d7 (d18:1-d7/16:0)
An internal standard for the quantification of C-16 ceramide
-
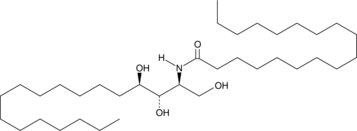
GC43052
C18 Phytoceramide (t18:0/18:0)
C18 Phytoceramide (t18:0/18:0) (Cer(t18:0/18:0)) is a bioactive sphingolipid found in S.
-
GC40141
C18 Phytoceramide-d3 (t18:0/18:0-d3)
C18 Phytoceramide-d3 (t18:0/18:0-d3) is intended for use as an internal standard for the quantification of C18 phytoceramide (t18:0/18:0) by GC- or LC-MS.

-
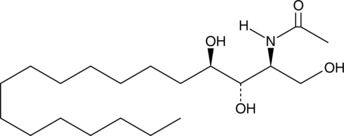
GC43065
C2 Phytoceramide (t18:0/2:0)
C2 Phytoceramide is a bioactive semisynthetic sphingolipid that inhibits formyl peptide-induced oxidant release (IC50 = 0.38 μM) in suspended polymorphonuclear cells.
-
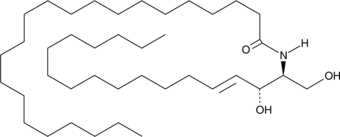
GC43069
C22 Ceramide (d18:1/22:0)
C-22 ceramide is an endogenous bioactive sphingolipid.
-
GC43075
C24 dihydro Ceramide (d18:0/24:0)
C24 dihydro Ceramide is a sphingolipid that has been found in the stratum corneum of human skin.
-
GC34513
C25-140
C25-140, un inhibidor de TRAF6-Ubc13 primero en su clase, activo por vÍa oral y bastante selectivo, se une directamente a TRAF6 y bloquea la interacciÓn de TRAF6 con Ubc13.
-
GC43084
C4 Ceramide (d18:1/4:0)
C4 Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC40688
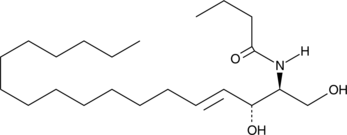
C6 D-threo Ceramide (d18:1/6:0)
C6 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides., C6 D-threo Ceramide is cytotoxic to U937 cells in vitro (IC50 = 18 μM).
-
GC40689
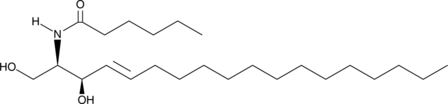
C6 L-erythro Ceramide (d18:1/6:0)
C6 L-erythro Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC40690
C6 L-threo Ceramide (d18:1/6:0)
C6 L-threo Ceramide (d18:1/6:0) es un esfingolÍpido bioactivo y un anÁlogo permeable a las células de las ceramidas naturales.
-
GC45616
C6 Urea Ceramide
An inhibitor of neutral ceramidase
-
GC12733
C646
C646 es un inhibidor selectivo y competitivo de histona acetiltransferasa p300 con Ki de 400 nM, y es menos potente para otras acetiltransferasas.
-
GC43105
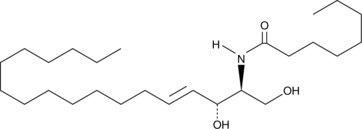
C8 Ceramide (d18:1.8:0)
C8 Ceramida (d18:1.8:0) (N-Octanoyl-D-eritro-esfingosina) es un análogo permeable a la célula de ceramidas que ocurren naturalmente.
-
GC43109
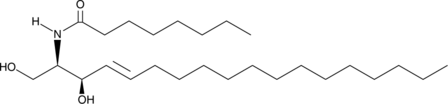
C8 D-threo Ceramide (d18:1/8:0)
C8 D-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC43110
C8 Galactosylceramide (d18:1/8:0)
C8 Galactosylceramide is a synthetic C8 short-chain derivative of known membrane microdomain-forming sphingolipids.

-
GC43111
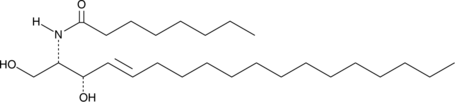
C8 L-threo Ceramide (d18:1/8:0)
C8 L-threo Ceramide is a bioactive sphingolipid and cell-permeable analog of naturally occurring ceramides.
-
GC33218
CA-5f
CA-5f es un potente inhibidor de macroautofagia/autofagia en etapa tardÍa a través de la inhibiciÓn de la fusiÓn autofagosoma-lisosoma. CA-5f aumenta LC3B-II (un marcador para monitorear la autofagia) y la proteÍna SQSTM1, y también aumenta la producciÓn de ROS. Actividad antitumoral.
-
GC15779
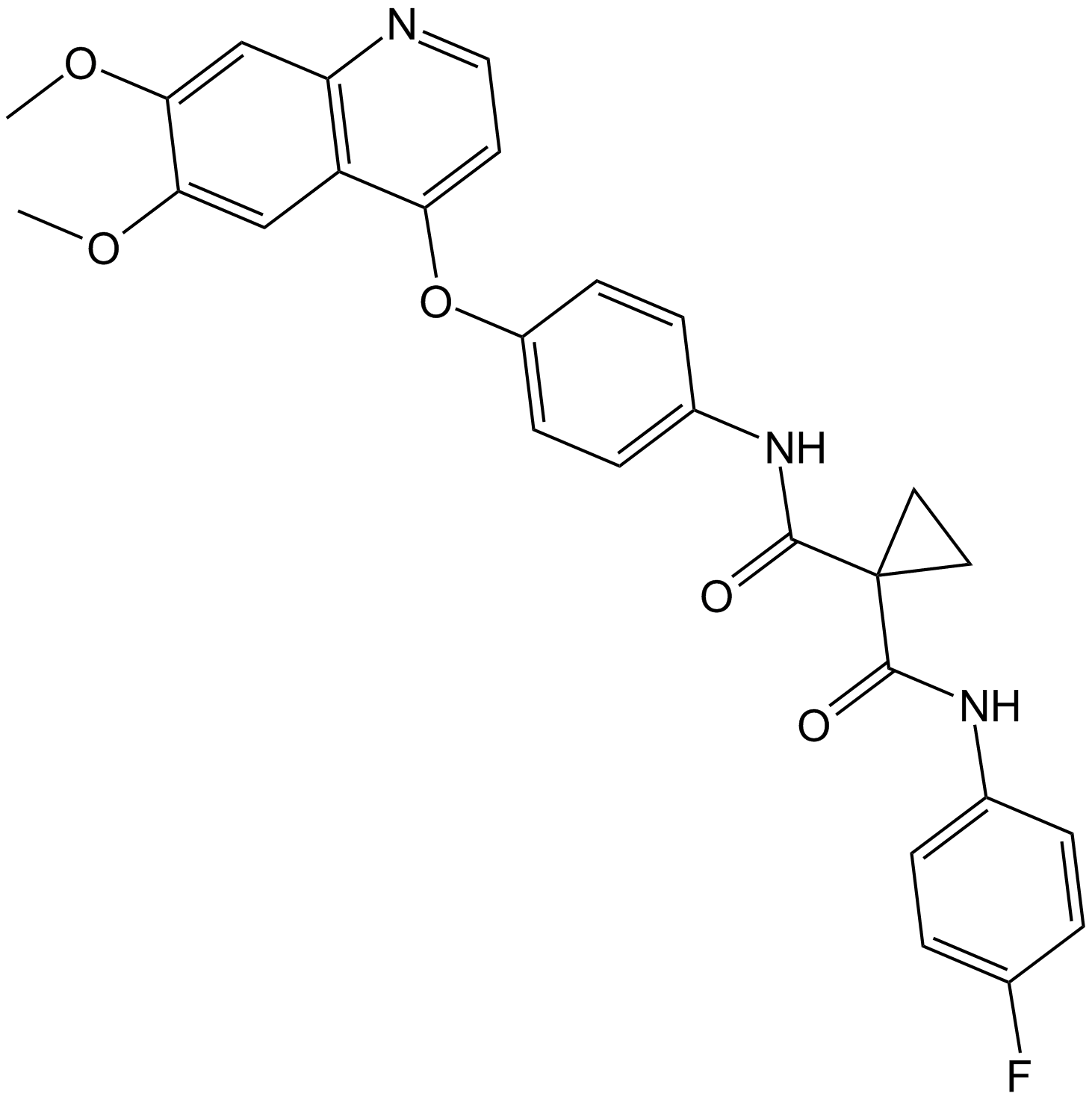
Cabozantinib (XL184, BMS-907351)
Cabozantinib (XL184,BMS-907351) es un nuevo inhibidor de MET y VEGFR2 que inhibe simultáneamente la metástasis, la angiogénesis y el crecimiento tumoral.
-
GC12531
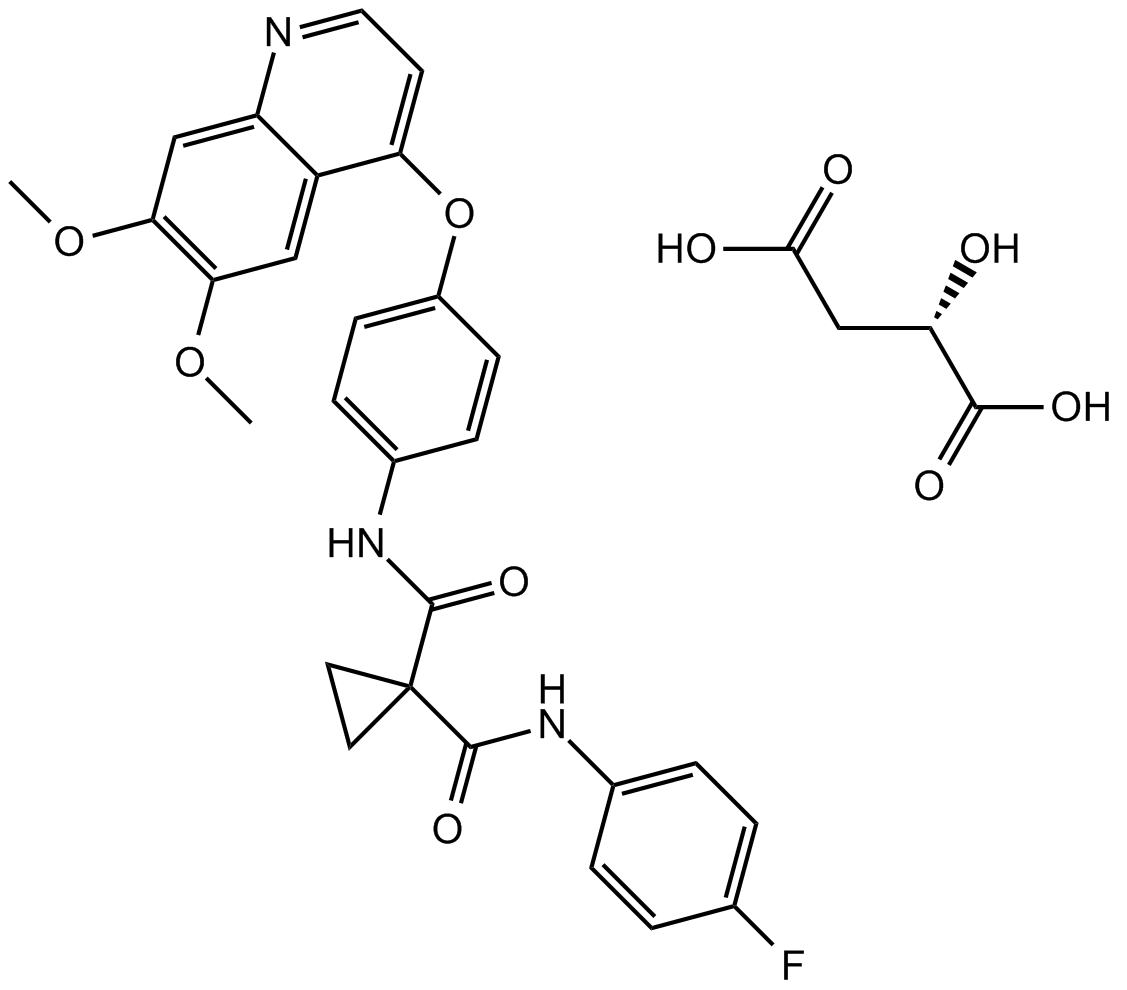
Cabozantinib malate (XL184)
El malato de cabozantinib (XL184) (malato de XL184 S) es un potente inhibidor de múltiples receptores tirosina quinasas que inhibe VEGFR2, c-Met, Kit, Axl y Flt3 con IC50s de 0.035, 1.3, 4.6, 7 y 11.3 nM respectivamente.
-
GC10692
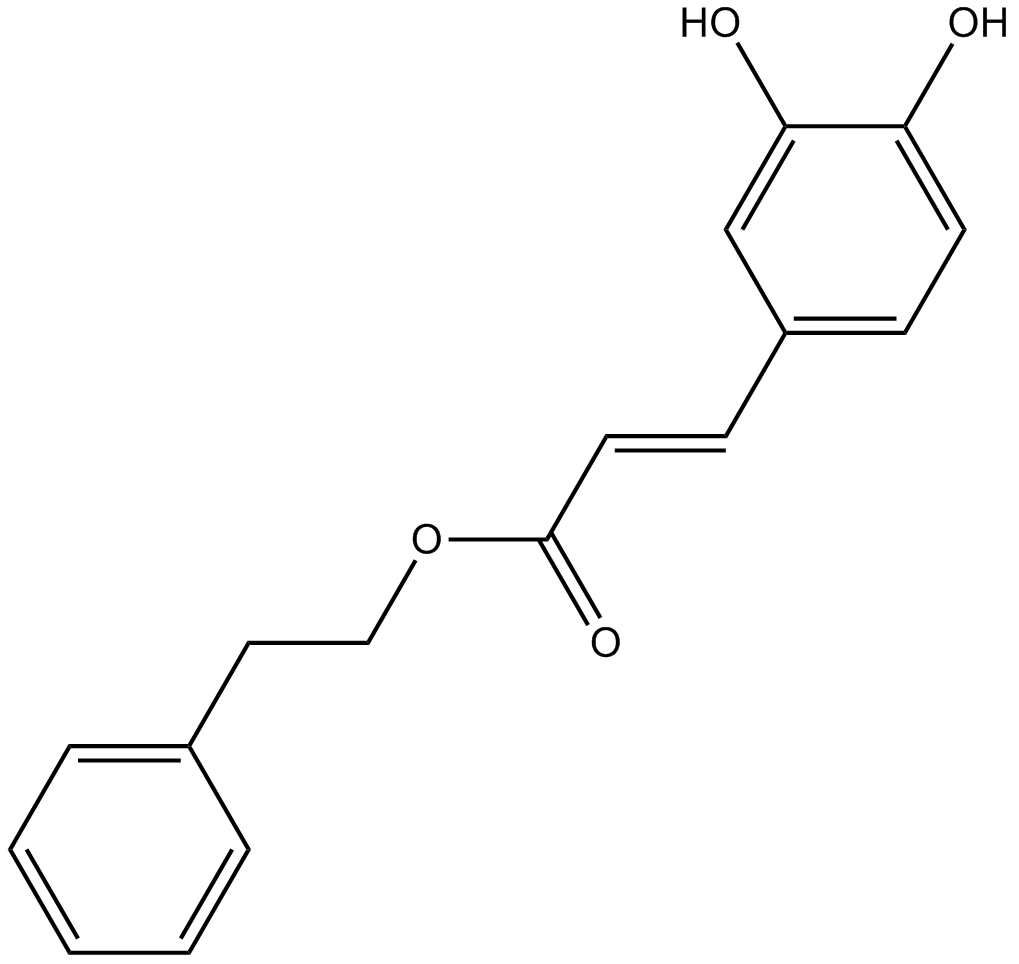
Caffeic Acid Phenethyl Ester
El éster fenetÍlico del Ácido cafeico es un inhibidor de NF-κB.
-
GC18604
Calcein Blue AM
Calcein Blue AM es un colorante celular.
-
GC43121
Calcein Orange™ Diacetate
Calcein Orange Diacetate is a fluorogenic dye that is used to assess cell viability.

-
GC43123
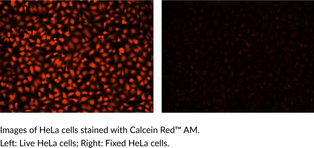
Calcein Red™ AM
Calcein Red? AM is a fluorogenic dye that is used to assess cell viability.
-
GC60668
Calcimycin hemimagnesium
Calcimicina (A-23187) hemimagnesio es un antibiÓtico y un ionÓforo de catiÓn divalente Único (como el calcio y el magnesio).
-
GC15161
Calcium D-Panthotenate
El D-pantotenato de calcio (sal de calcio de la vitamina B5), una vitamina, puede reducir el contenido de patulina del jugo de manzana.
-
GC30240
Calcium dobesilate
El dobesilato de calcio, un vasoprotector, se usa ampliamente en la enfermedad venosa crÓnica, la retinopatÍa diabética y los sÍntomas del ataque hemorroidal en muchos paÍses.
-
GC19086
Calicheamicin
La caliqueamicina, un antibiÓtico antitumoral, es un agente citotÓxico que provoca roturas del ADN de doble cadena.
-
GC65081
CALP1 TFA
CALP1 TFA es un agonista de calmodulina (CaM) (Kd de 88 μM) con uniÓn al sitio de uniÓn CaM EF-hand/Ca2+-.
-
GC18315
Calpain Inhibitor VI
Calpain inhibitor VI is an inhibitor of the calcium-dependent cysteine proteases u-calpain (calpain-1; IC50 = 7.5 nM) and m-calpain (calpain-2; IC50 = 78 nM).
-
GC10342
Calpeptin
A calpain inhibitor
-
GN10667
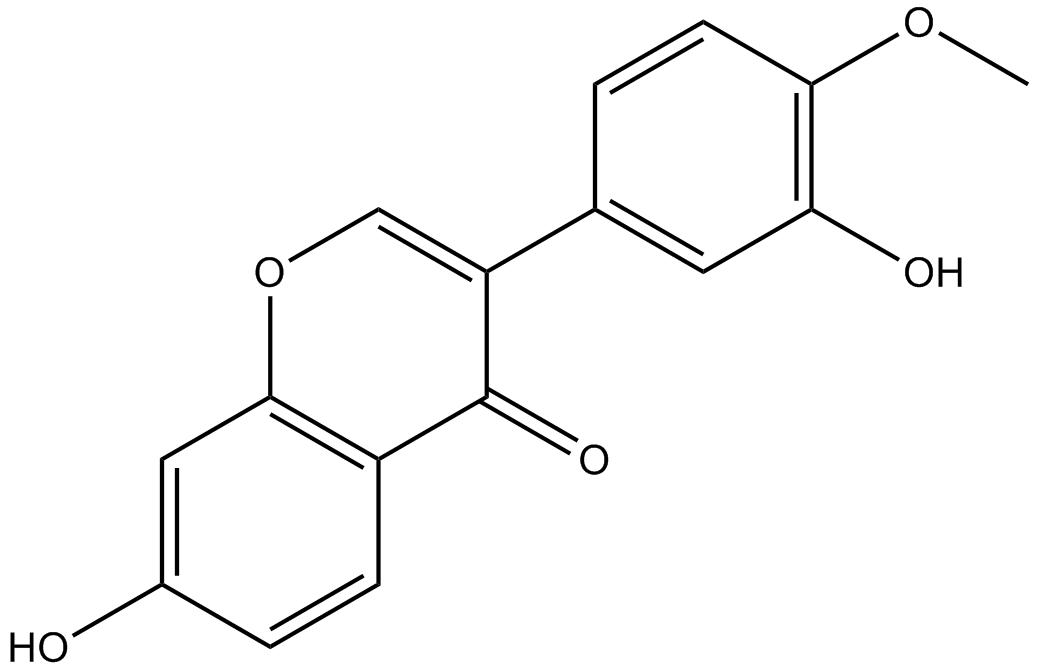
Calycosin
Calycosin (CA, 7, 3-dihydroxy-4-methoxy isoflavone, C16H12O5) is one of the flavonoids extracted from astragalus root, also known as the typical phytoestrogens.
-
GC35598
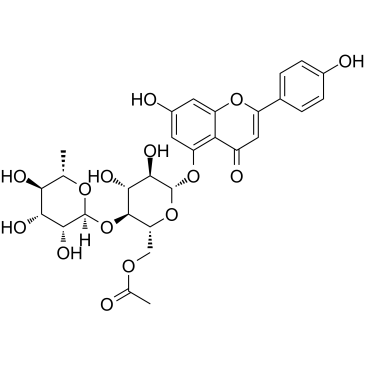
Camellianin A
La camelianina A, el principal flavonoide en las hojas de A. nitida, muestra actividad anticancerÍgena y actividad inhibidora de la enzima convertidora de angiotensina (ECA). La camelianina A inhibe la proliferaciÓn de las lÍneas celulares humanas Hep G2 y MCF-7 e induce el aumento significativo de la poblaciÓn de células G0/G1.
-
GC62253
Camrelizumab
Camrelizumab (SHR-1210) es un potente anticuerpo monoclonal (mAb) IgG4-κ humanizado de alta afinidad contra PD-1. Camrelizumabse une a PD-1 con una alta afinidad de 3 nM e inhibe la interacciÓn de uniÓn de PD-1 y PD-L1 con una IC50 de 0,70 nM. Camrelizumab actÚa como un agente anti-PD-1/PD-L1 y se puede utilizar para la investigaciÓn del cÁncer, incluidos NSCLC, ESCC, linfoma de Hodgkin y HCC avanzado et al.

-
GC12318
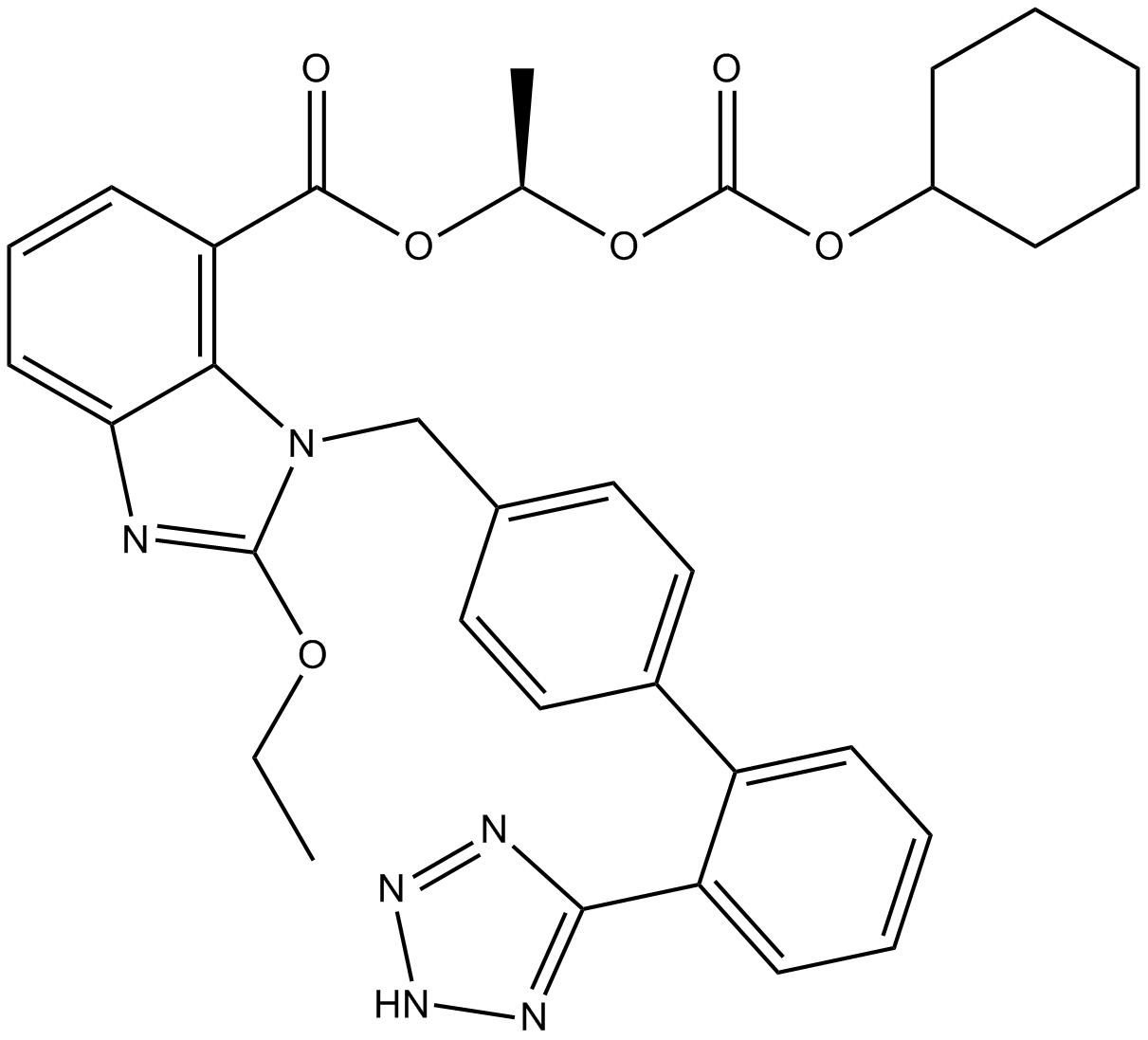
Candesartan Cilexetil
Candesartan Cilexetil (TCV-116) es un antagonista del receptor de la angiotensina II utilizado principalmente para el tratamiento de la hipertensiÓn.
-
GC15866
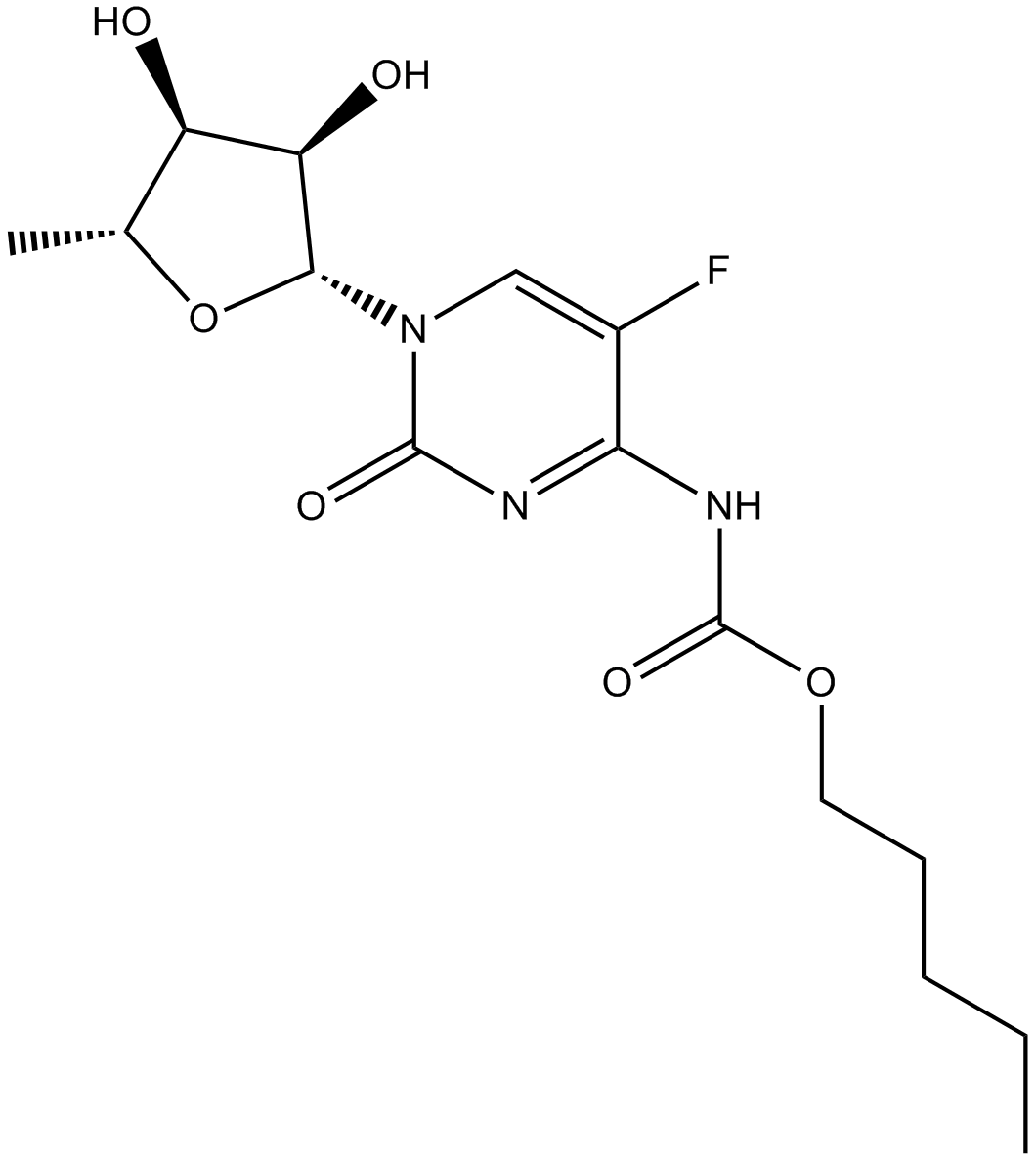
Capecitabine
La capecitabina es un profÁrmaco oral que se convierte en su metabolito activo, 5-FU, mediante la timidina fosforilasa.
-
GC14065
Capsaicin
Un alcaloide de terpeno con diversas actividades biológicas.
-
GC17918
Capsazepine
A TRPV1 antagonist
-
GC49415
Capsorubin
A carotenoid with diverse biological activities
-
GC48878
Carbazomycin A
A bacterial metabolite with diverse biological activities
-
GC48893
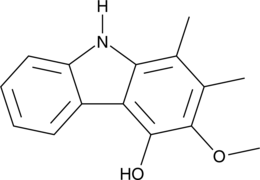
Carbazomycin B
A bacterial metabolite with diverse biological activities
-
GC48850
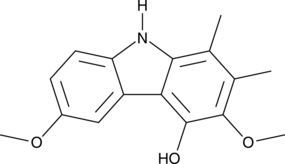
Carbazomycin C
A bacterial metabolite with diverse biological activities
-
GC48826
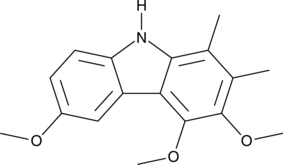
Carbazomycin D
A bacterial metabolite with diverse biological activities
-
GC49147
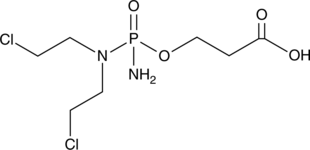
Carboxyphosphamide
An inactive metabolite of cyclophosphamide
-
GC18069
Cardamonin
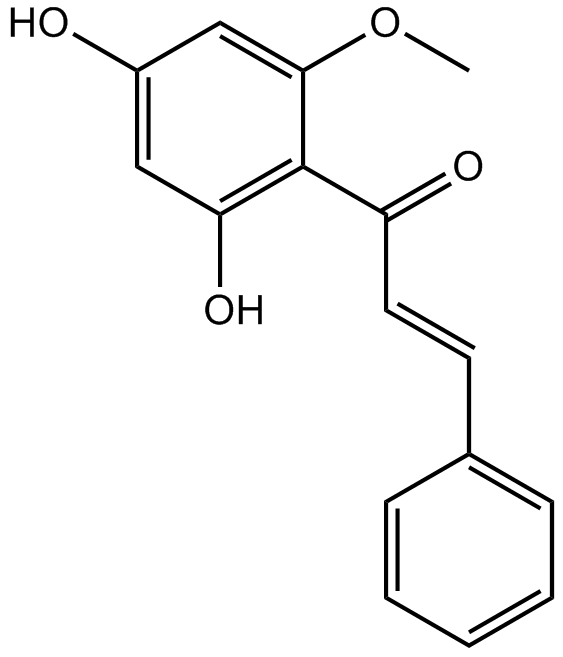
La cardamonina ((E)-Cardamomin) es un nuevo antagonista del canal catiÓnico hTRPA1 con una IC50 de 454 nM.
-
GC18449
Cardanol monoene
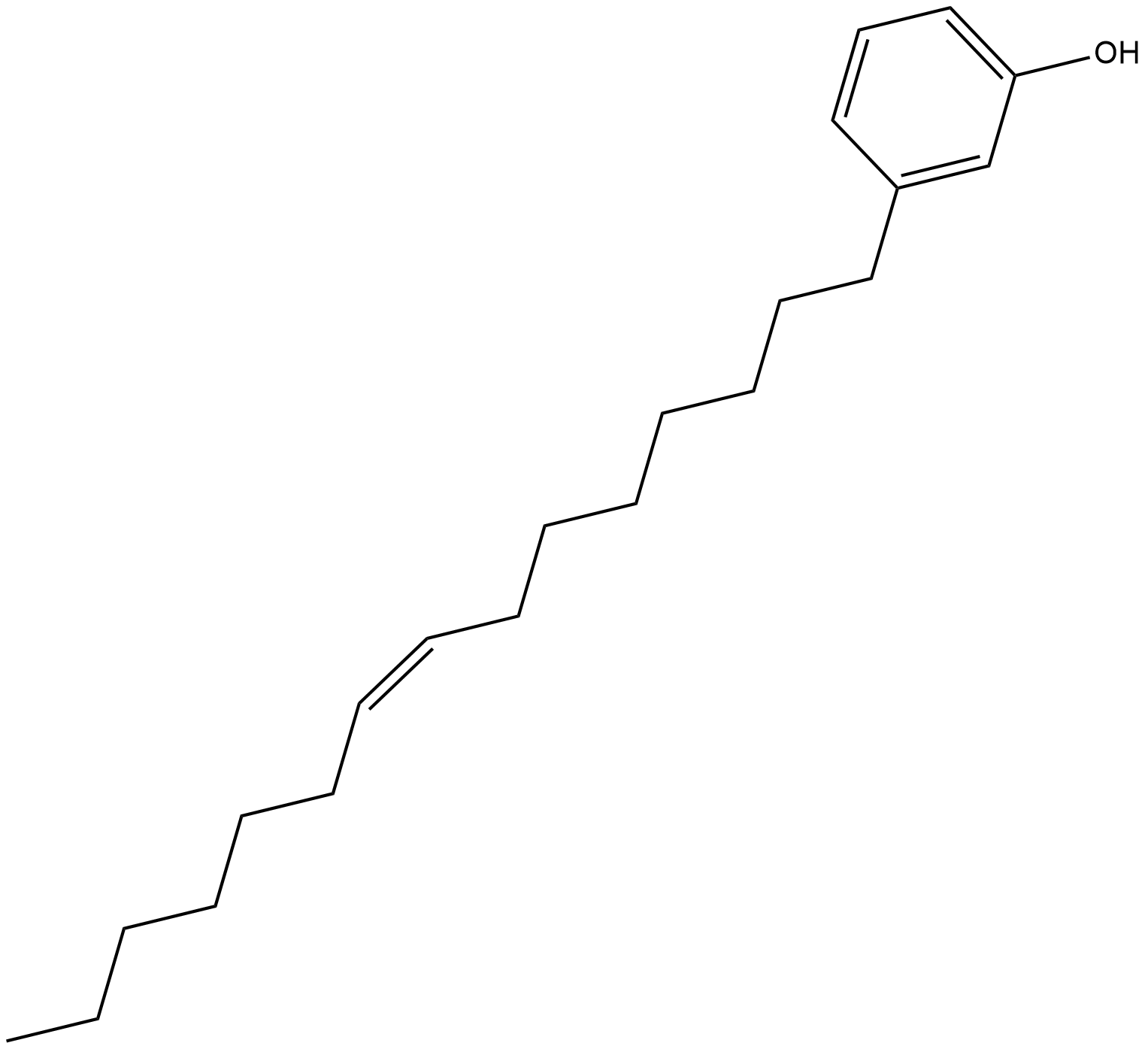
El cardanol monoeno (Cardanol C15:1) es un compuesto fenÓlico que se puede encontrar en el lÍquido de la cÁscara de la nuez de la India. El cardanol monoeno puede inducir la apoptosis asociada a las mitocondrias en células de melanoma humano.
-
GC15089
Carfilzomib (PR-171)
A proteasome inhibitor