Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nombre del producto Información
-
GC43274
Citromycetin
La citromicetina es un compuesto de policétido aromÁtico de Penicillium spp de origen marino y terrestre de Australia.

-
GC63393
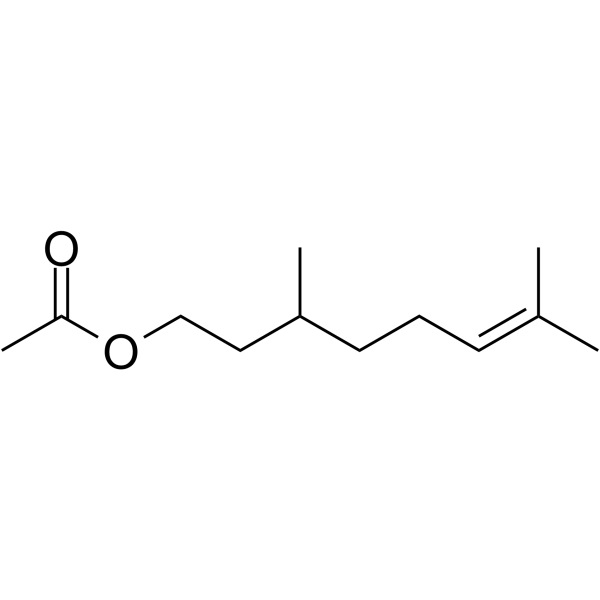
Citronellyl acetate
El acetato de citronelilo es un producto monoterpénico del metabolismo secundario de las plantas, con actividad antinociceptiva.
-
GC52367
Citrullinated Vimentin (G146R) (R144 + R146) (139-159)-biotin Peptide
A biotinylated and citrullinated mutant vimentin peptide

-
GC52370
Citrullinated Vimentin (R144) (139-159)-biotin Peptide
A biotinylated and citrullinated vimentin peptide

-
GN10219
Ciwujianoside-B
-
GC64649
Cjoc42
Cjoc42 es un compuesto capaz de unirse a gankyrin. Cjoc42 inhibe la actividad de la ganquirina de forma dependiente de la dosis. Cjoc42 previene la disminuciÓn en los niveles de proteÍna p53 normalmente asociados con altas cantidades de gankyrin. Cjoc42 restaura la transcripciÓn dependiente de p53 y la sensibilidad al daÑo del ADN.
-
GC39485
CK2/ERK8-IN-1
CK2/ERK8-IN-1 es un inhibidor dual de caseÍna quinasa 2 (CK2) (Ki de 0,25 μM) y ERK8 (MAPK15, ERK7) con IC50 de 0,50 μM. CK2/ERK8-IN-1 también se une a PIM1, HIPK2 (proteÍna quinasa 2 que interactÚa con el homeodominio) y DYRK1A con Kis de 8,65 μM, 15,25 μM y 11,9 μM, respectivamente. CK2/ERK8-IN-1 tiene eficacia proapoptÓtica.
-
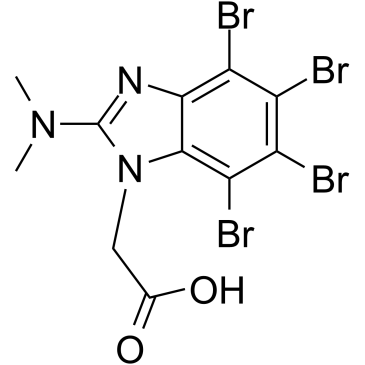
GC49556
Cl-Necrostatin-1
A RIPK1 inhibitor
-
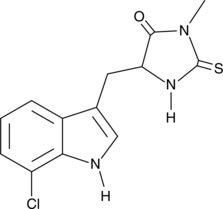
GC47098
CL2-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
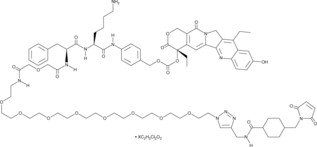
GC60714
CL2A-SN-38
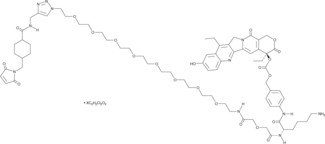
CL2A-SN-38 es un conjugado de fÁrmaco-enlazador compuesto por un potente inhibidor de la topoisomerasa I de ADN SN-38 y un enlazador CL2A para producir un conjugado de anticuerpo y fÁrmaco (ADC).
-
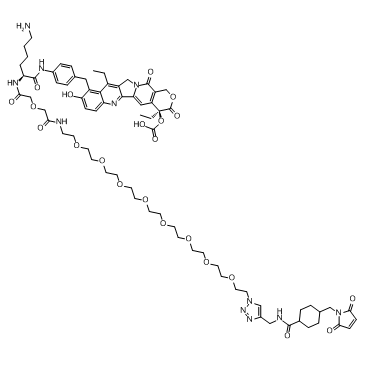
GC52469
CL2A-SN-38 (dichloroacetic acid salt)
An antibody-drug conjugate containing SN-38
-
GC10509
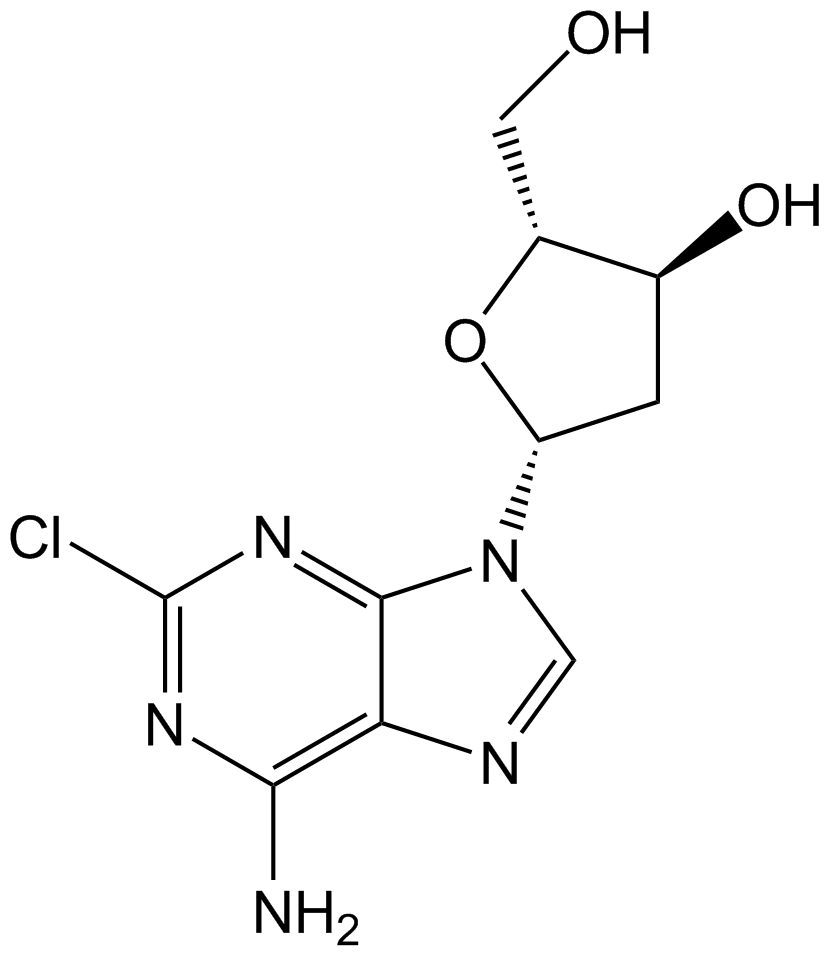
Cladribine
La cladribina (2-cloro-2′-desoxiadenosina), un anÁlogo de nucleÓsido de purina, es un inhibidor de la adenosina desaminasa activo por vÍa oral.
-
GC60111
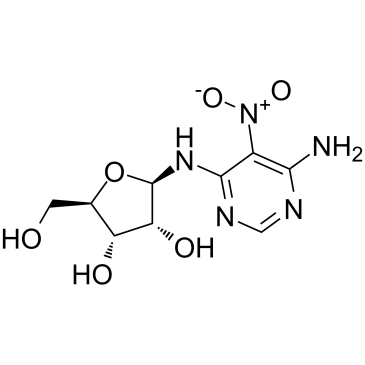
Clitocine
La clitocina, un anÁlogo de nucleÓsido de adenosina aislado de hongos, es un agente de lectura potente y eficaz. La clitocina actÚa como un supresor de mutaciones sin sentido y puede inducir la producciÓn de proteÍna p53 en células que albergan alelos mutados sin sentido de p53. La clitocina puede inducir la apoptosis en células cancerosas humanas resistentes a mÚltiples fÁrmacos al dirigirse a Mcl-1. Actividad anticancerÍgena.
-
GC15219
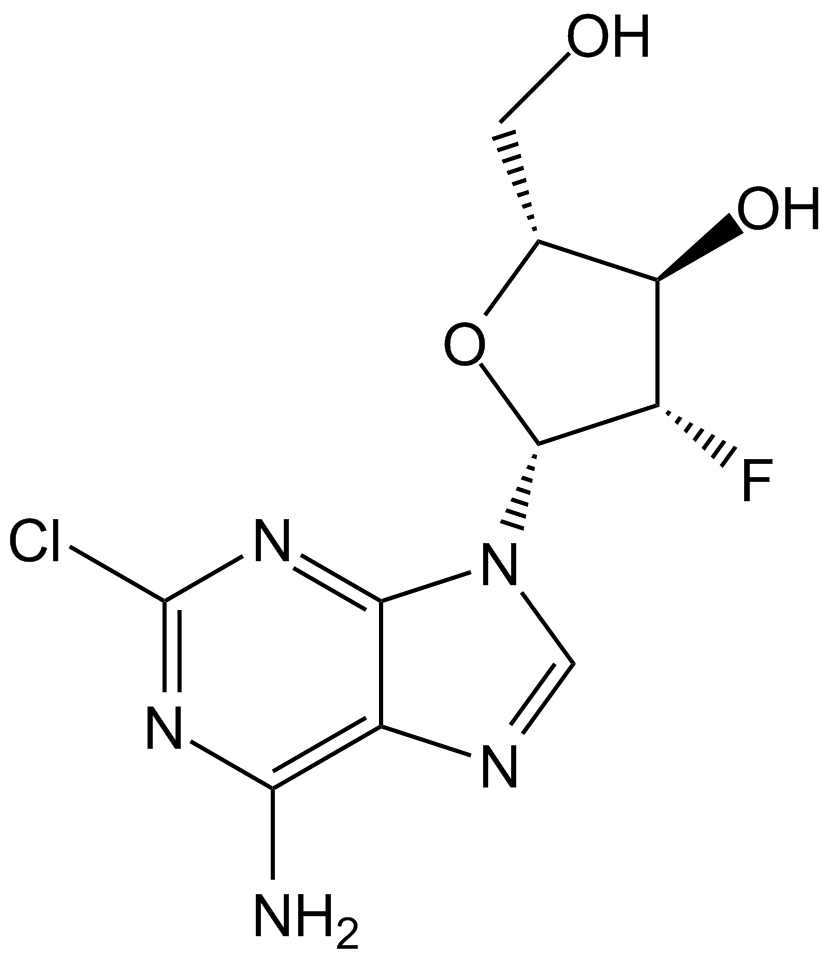
Clofarabine
La clofarabina, un anÁlogo de nucleÓsido para la investigaciÓn del cÁncer, es un potente inhibidor de la ribonucleÓtido reductasa (IC50=65 nM) al unirse al sitio alostérico de la subunidad reguladora.
-
GC10813
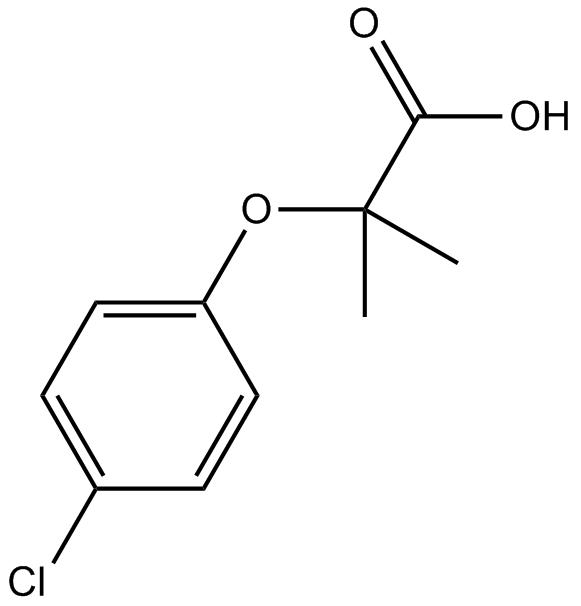
Clofibric Acid
El Ácido clofÍbrico (Ácido clorofibrÍnico), el metabolito farmacéuticamente activo de los reguladores de lÍpidos clofibrato, etofibrato y etofilinclofibrato, es un agonista de PPARα que exhibe efectos hipolipidémicos.
-
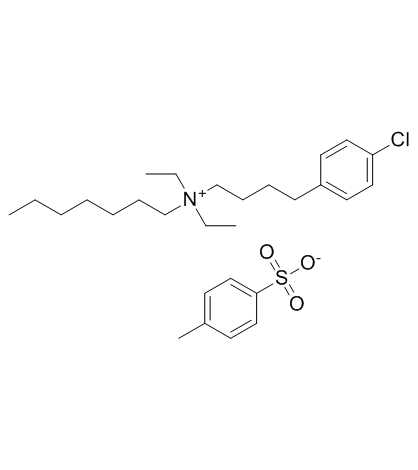
GC32587
Clofilium tosylate
El tosilato de clofilium, un bloqueador de los canales de potasio, induce la apoptosis de las células de leucemia promielocÍtica humana (HL-60) a través de la activaciÓn de caspasa-3 insensible a Bcl-2.
-
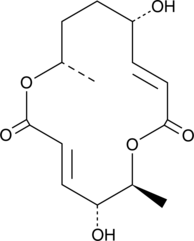
GC47105
Clonostachydiol
Clonostachydiol es un macrodiolido antihelmÍntico del hongo Clonostachys cylindrospora (cepa FH-A 6607).
-
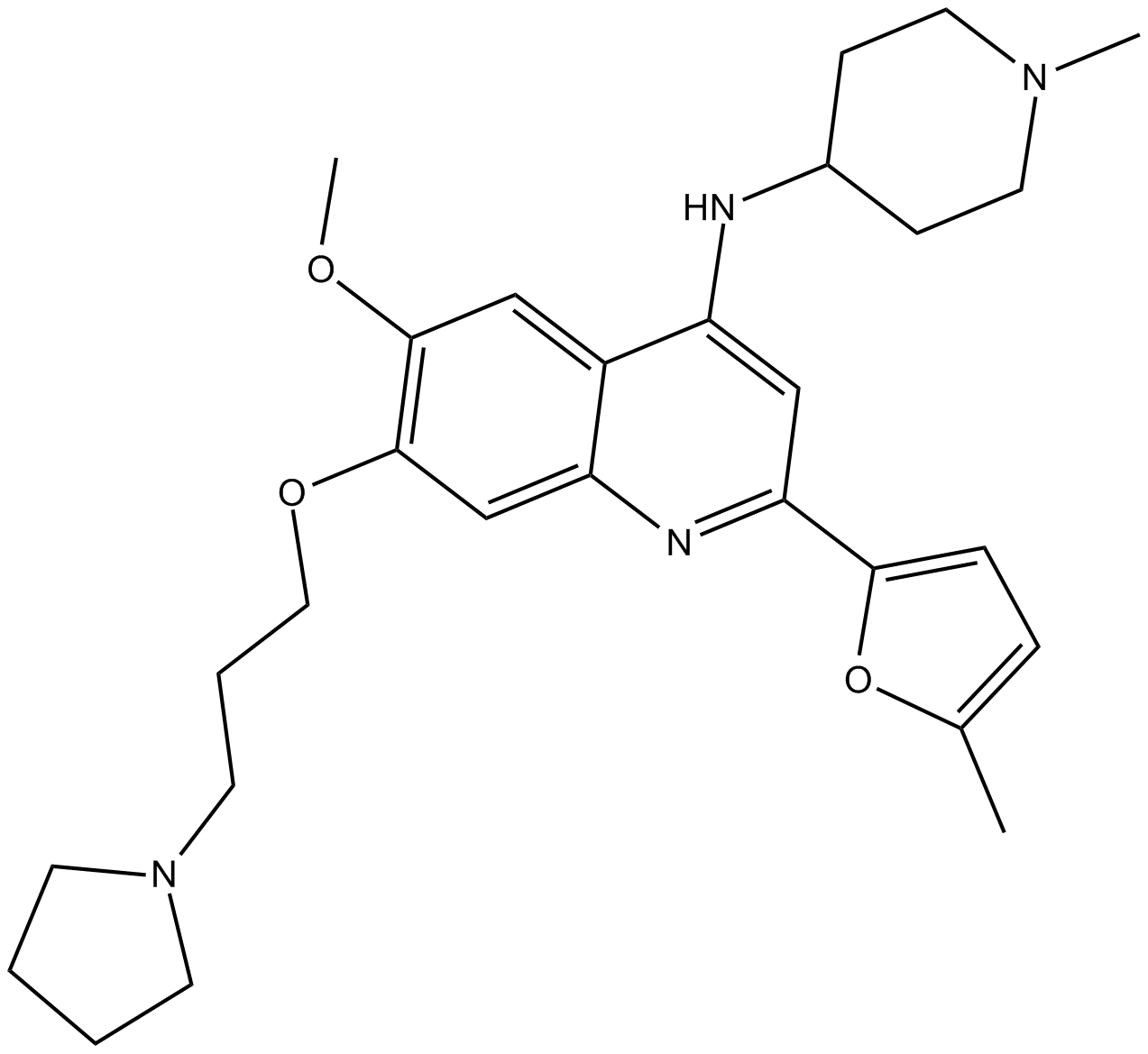
GC12367
CM-272
CM-272 es un inhibidor dual G9a/DNA metiltransferasas (DNMT) de primer nivel, potente, selectivo, competitivo con sustrato y reversible con actividades antitumorales. CM-272 inhibe G9a, DNMT1, DNMT3A, DNMT3B y GLP con IC50 de 8 nM, 382 nM, 85 nM, 1200 nM y 2 nM, respectivamente. CM-272 inhibe la proliferaciÓn celular y promueve la apoptosis, induciendo genes estimulados por IFN y muerte celular inmunogénica.
-
GC62347
CMC2.24
CMC2.24 (TRB-N0224), un agente de tricarbonilmetano activo por vÍa oral, es eficaz contra el tumor pancreÁtico en ratones al inhibir la activaciÓn de Ras y su vÍa efectora ERK1/2 corriente abajo.
-
GC61567
CMLD-2
CMLD-2, un inhibidor de la interacciÓn HuR-ARE, se une competitivamente a la proteÍna HuR interrumpiendo su interacciÓn con los ARNm que contienen elementos ricos en adenina-uridina (ARE) (Ki = 350 nM). CMLD-2 induce la apoptosis y exhibe actividad antitumoral en diferentes células cancerosas como lÍneas celulares de cÁncer de colon, pÁncreas, tiroides y pulmÓn. El antÍgeno Hu R (HuR) es una proteÍna de uniÓn a ARN que puede regular la estabilidad y la traducciÓn de los ARNm diana.
-
GC49096
Cobaltic Protoporphyrin IX (chloride)
An inducer of HO-1 activity
-
GC10033
Cobimetinib
A potent, orally available MEK1 inhibitor
-
GC43297
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
-
GC62192
COG1410
COG1410 es un péptido derivado de la apolipoproteÍna E.
-
GC40664
Colcemid
Colcemid es una inhibición de citoesqueleto, cuando las células de mamíferos y los ovocitos inducen la detención mitótica o la detención meiótica respectivamente durante la fase de ruptura de la vesícula.
-
GN10123
Columbianadin
-
GC49454
Complex 3
A fluorescent copper complex with anticancer activity
-
GC18572
Concanavalin A
La concanamicina A pertenece a las concanamicinas, una familia de antibióticos macrólidos aislados de Streptomyces diastatochromogenes que son inhibidores altamente activos y selectivos de la vacuolar proton-ATPasa (v-[H+] ATPasa).

-
GC18832
Conglobatin
Conglobatin is a dimeric macrolide dilactone originally isolated from S.
-
GC48483
Conglobatin B
A bacterial metabolite
-
GC48497
Conglobatin C1
A bacterial metabolite
-
GC38376
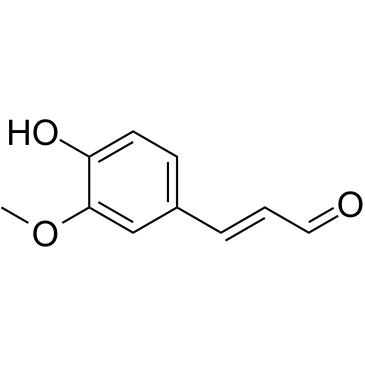
Coniferaldehyde
El coniferaldehÍdo (ferulaldehÍdo) es un inductor eficaz de la hemooxigenasa-1 (HO-1).
-
GC63379
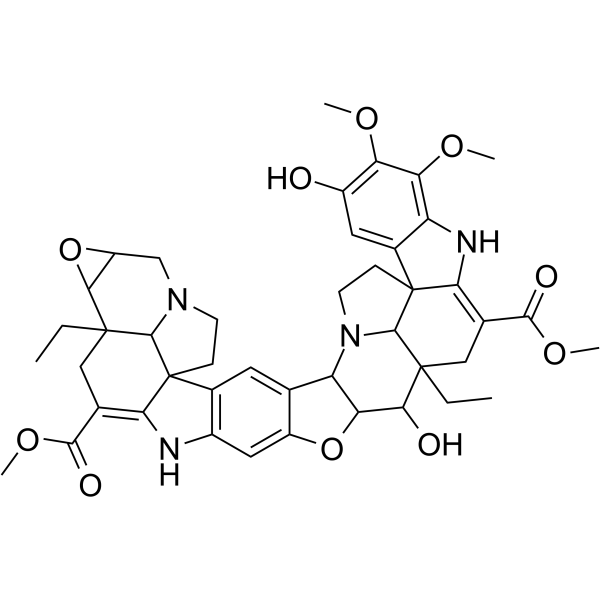
Conophylline
La conofilina es un alcaloide de la vinca extraÍdo de las hojas de una planta tropical Ervatamia microphylla.
-
GC16772
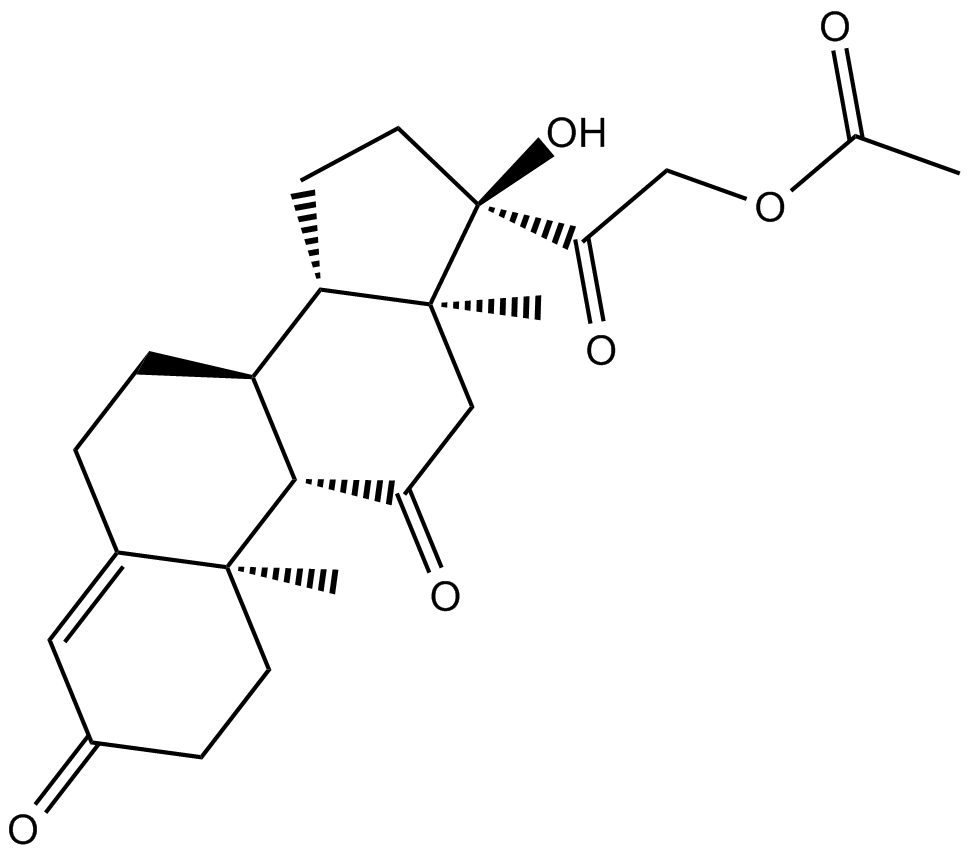
Cortisone acetate
Acetato de cortisona (21-acetato de cortisona), un metabolito oxidado del cortisol (un glucocorticoide).
-
GC16116
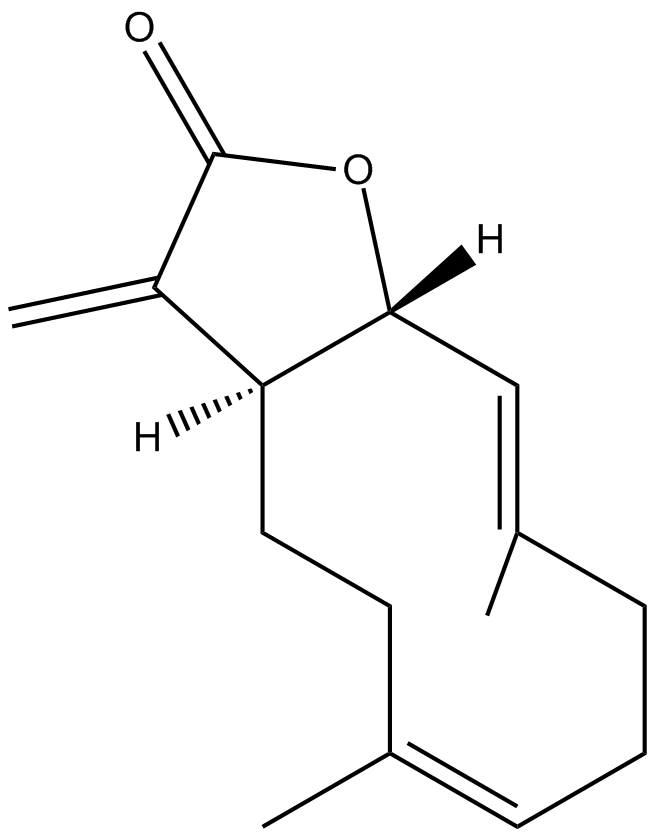
Costunolide
A natural sesquiterpene lactone
-
GC15225
COTI-2
COTI-2, un fÁrmaco contra el cÁncer con baja toxicidad, es un activador de tercera generaciÓn disponible por vÍa oral de formas mutantes de p53. COTI-2 actÚa reactivando p53 mutante e inhibiendo la vÍa PI3K/AKT/mTOR. COTI-2 induce la apoptosis en mÚltiples lÍneas de células tumorales humanas. COTI-2 exhibe actividad antitumoral en HNSCC a través de mecanismos dependientes e independientes de p53. COTI-2 convierte la p53 mutante en una conformaciÓn de tipo salvaje.
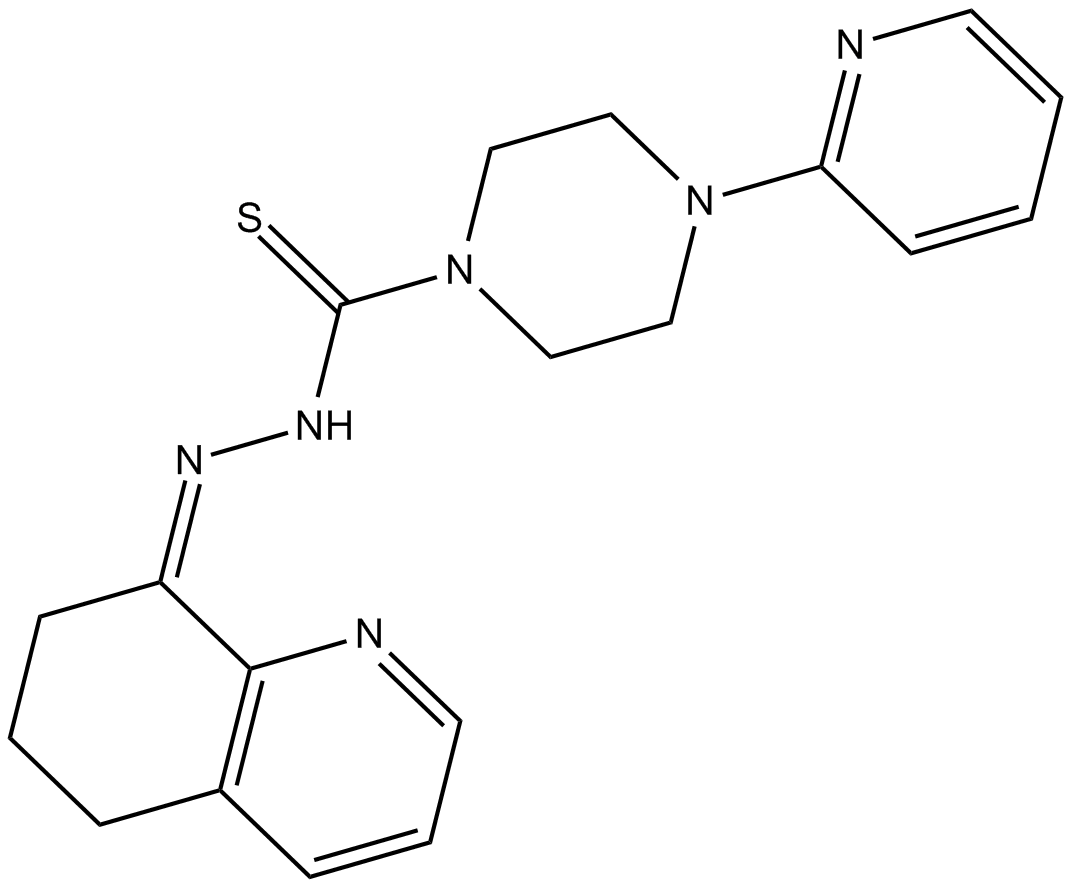
-
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
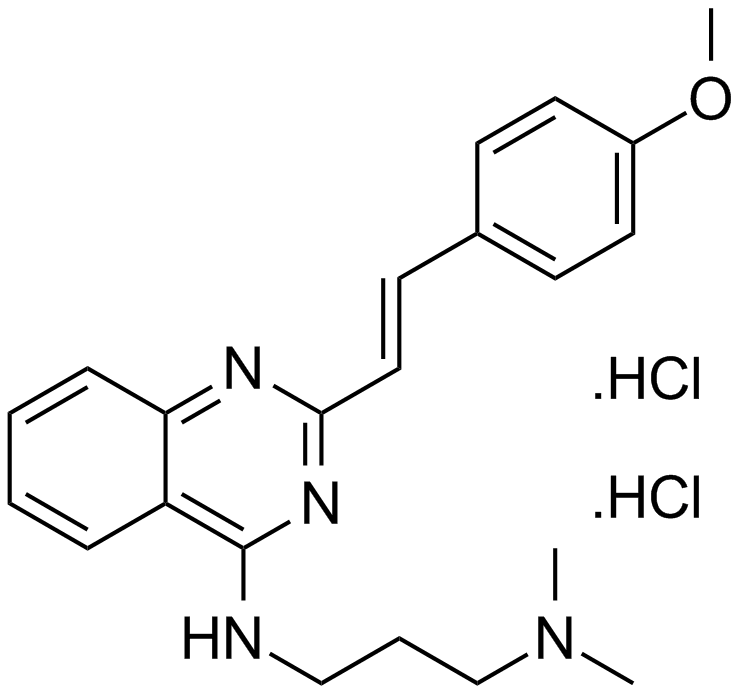
-
GC13091
CP-724714
CP-724714 es un inhibidor de la tirosina quinasa ErbB2 (HER2) potente, selectivo y activo por vÍa oral, con una IC50 de 10 nM. CP-724714 muestra una marcada selectividad frente a la quinasa EGFR (IC50=6400 nM). CP-724714 inhibe potentemente la autofosforilaciÓn del receptor ErbB2 en células intactas. Actividades antitumorales.
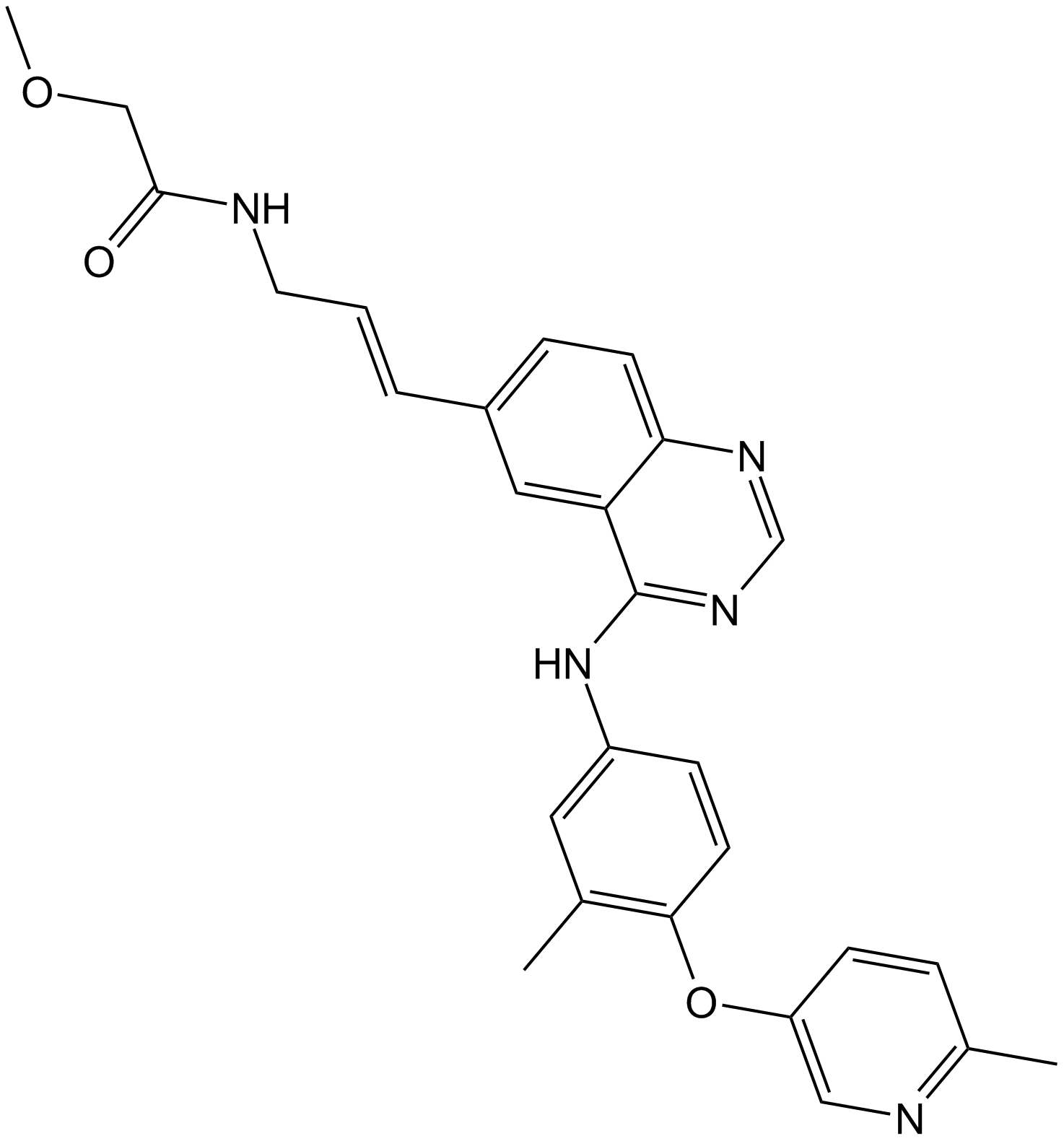
-
GC14500
CPI-1189
CPI-1189 es un inhibidor de la liberaciÓn de TNF-α con propiedades antioxidantes y neuroprotectoras.
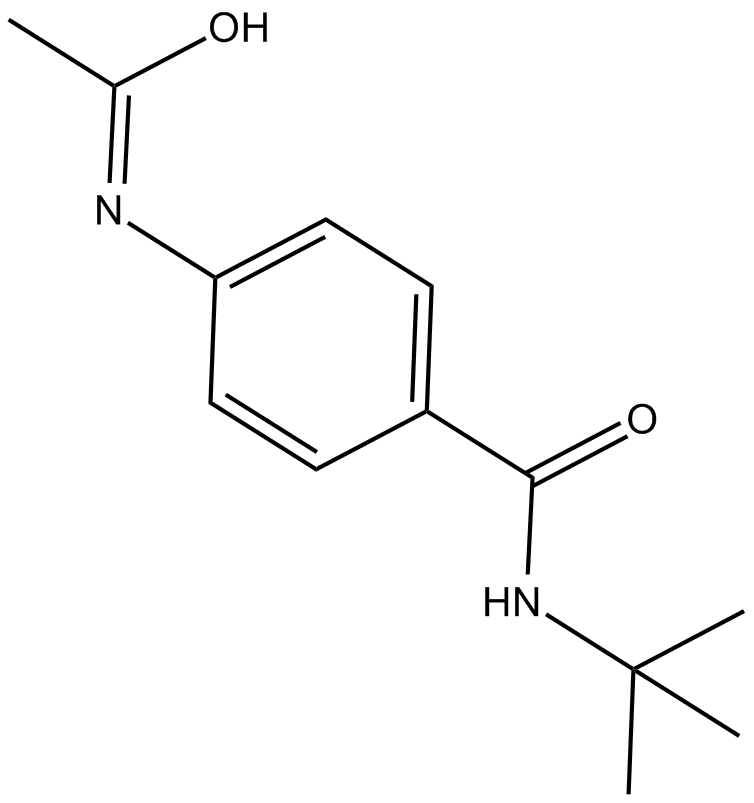
-
GC14699
CPI-203
CPI-203 es un nuevo inhibidor potente, selectivo y permeable a las células del bromodominio BET, con un valor IC50 de aproximadamente 37 nM (ensayo de pantalla α BRD4).
-
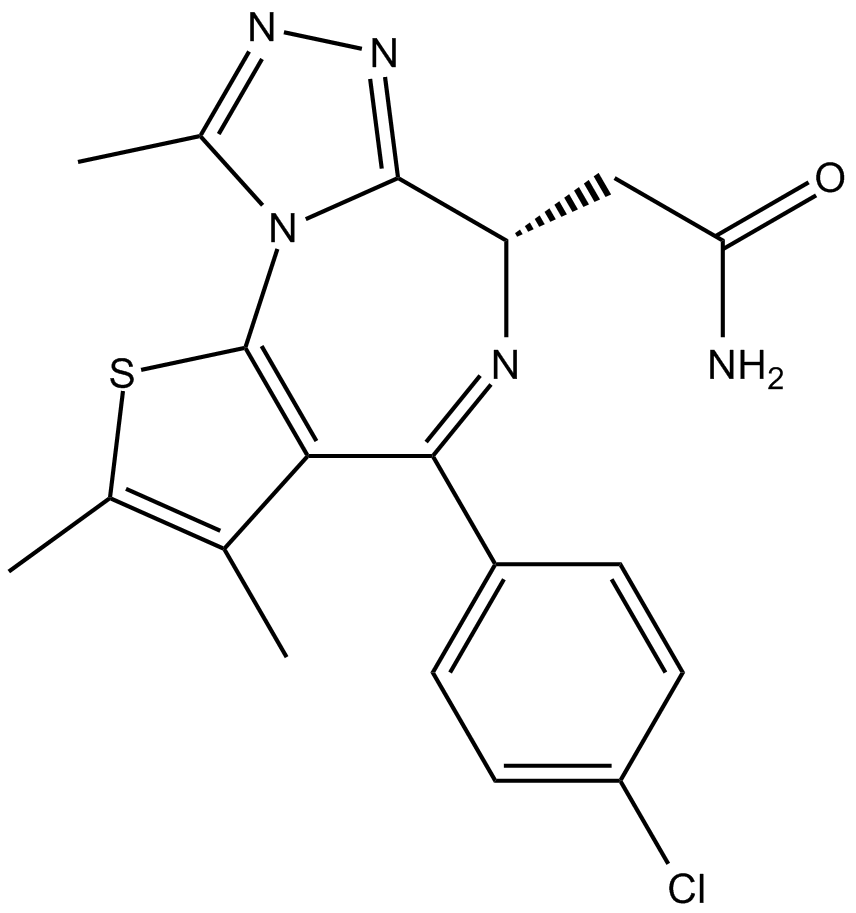
GC10021
CPI-360
EZH2 inhibitor
-
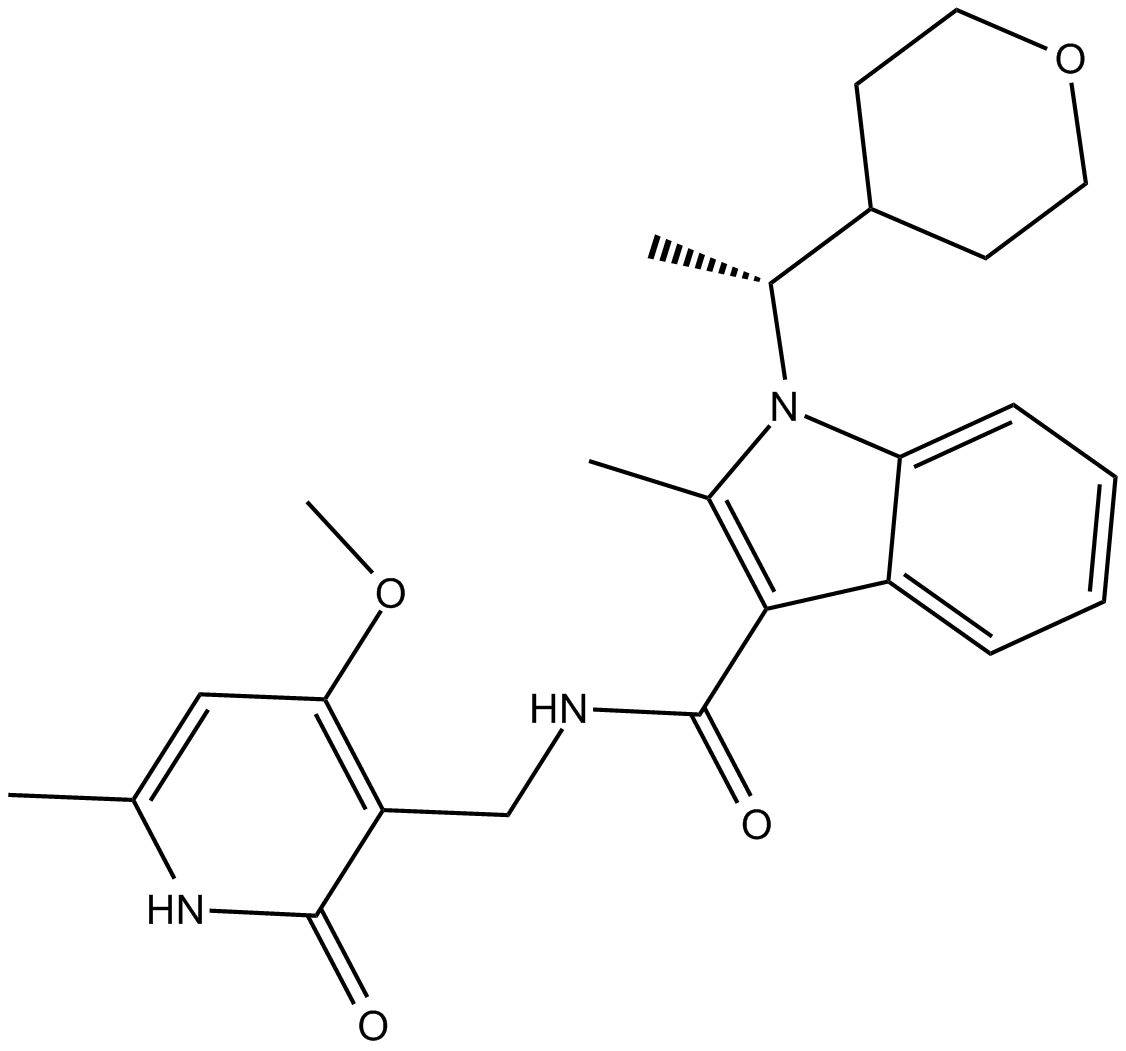
GC14921
CPI-613
An inhibitor of α-ketoglutarate dehydrogenase
-
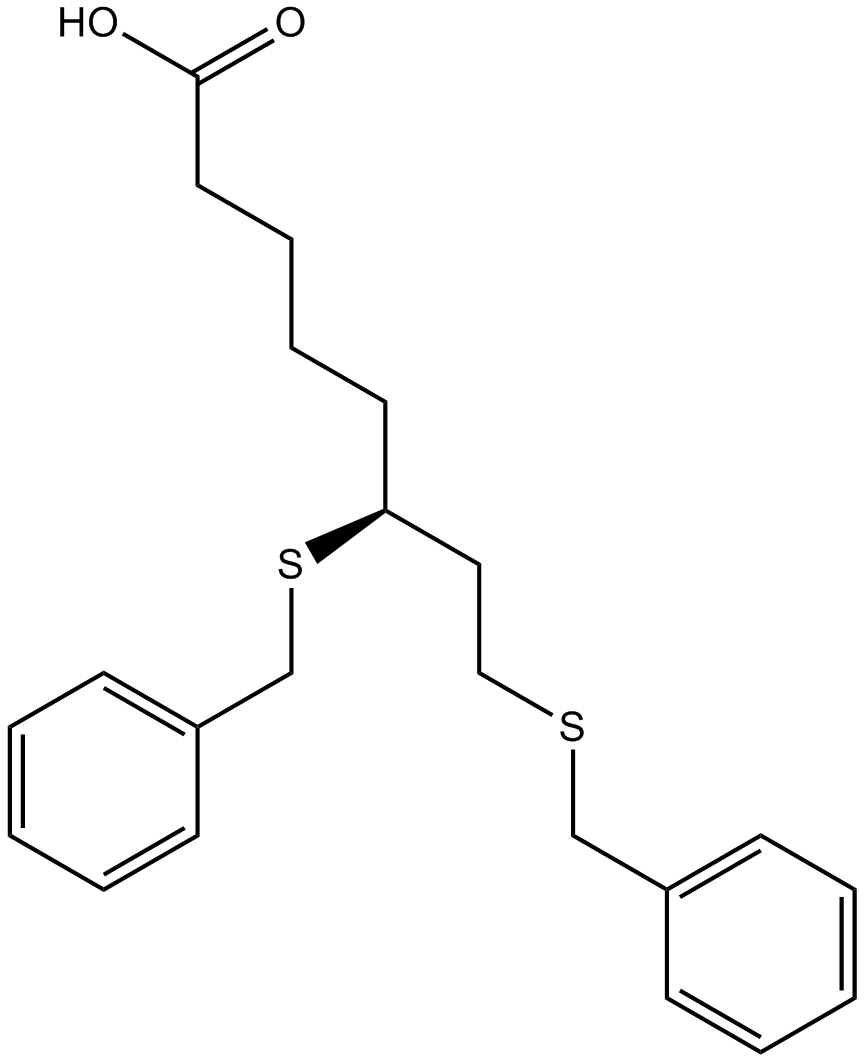
GC39365
CPTH2
CPTH2 es un potente inhibidor de histona acetiltransferasa (HAT). CPTH2 inhibe selectivamente la acetilaciÓn de la histona H3 por Gcn5. CPTH2 induce la apoptosis y disminuye la invasividad de una lÍnea celular de carcinoma renal de células claras (ccRCC) a través de la inhibiciÓn de la acetiltransferasa p300 (KAT3B).
-
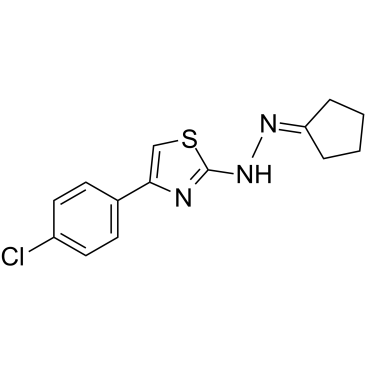
GC35747
Crebanine
La crebanina, un alcaloide de Stephania venosa, induce la detenciÓn de G1 y la apoptosis en células cancerosas humanas.
-
GC34543
cRIPGBM
cRIPGBM, un derivado proapoptÓtico de RIPGBM, un inductor selectivo de tipo celular de la apoptosis en células madre cancerosas (CSC) de GBM al unirse a la proteÍna quinasa 2 que interactÚa con el receptor (RIPK2), con un EC50 de 68 nM en células GBM-1.
-
GC13838
CRT 0066101
PKD inhibitor
-
GC35750
CRT0066101 dihydrochloride
El diclorhidrato de CRT0066101 es un inhibidor de PKD potente y especÍfico con valores IC50 de 1, 2,5 y 2 nM para PKD1, 2 y 3 respectivamente.
-
GC45414
CRT0066854
CRT0066854 es un potente y selectivo inhibidor de las isoenzimas PKC atÍpicas.
-
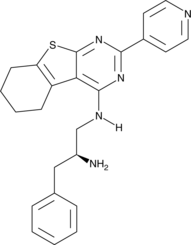
GC14355
CRT5
CRT5, una pirazina benzamida, es un inhibidor potente y selectivo de las tres isoformas de PKD en células endoteliales tratadas con VEGF (IC50 = 1, 2 y 1,5 nM para PKD1, PKD2 y PKD3, respectivamente).
-
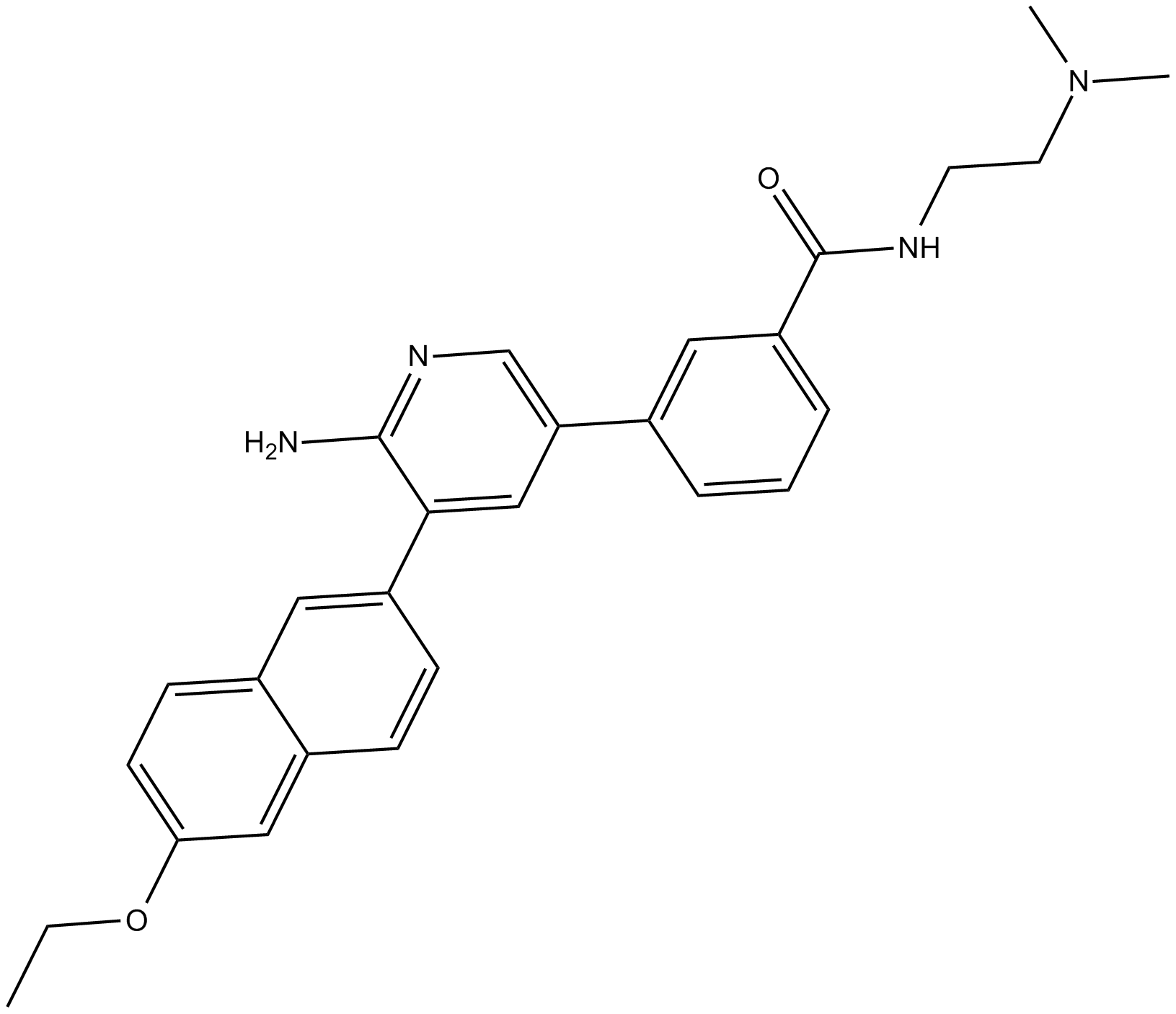
GC32911
CTX1
CTX1 es un activador de p53 que supera la represiÓn de p53 mediada por HdmX. CTX1 exhibe una potente actividad anticancerÍgena en un sistema modelo de leucemia mieloide aguda (LMA) de ratÓn.
-
GN10535
Cucurbitacin B
-
GC35758
Cucurbitacin IIa
La cucurbitacina IIa es un triterpeno aislado de Hemsleya amalils Diels, induce la apoptosis de las células cancerosas, reduce la expresiÓn de survivina, reduce la fosfohistona H3 y aumenta la PARP escindida en las células cancerosas.
-
GN10788
Cucurbitacin IIb
-
GC32781
CUDC-427 (GDC-0917)
CUDC-427 (GDC-0917) es un potente antagonista panselectivo de IAP de segunda generaciÓn, utilizado para el tratamiento de varios tipos de cÁncer.
-
GC12115
CUDC-907
A dual inhibitor of HDACs and PI3Ks
-
GC11217
CUR 61414
CUR 61414 es un nuevo inhibidor de la vía de señalización de Hedgehog, potente y permeable a las células (IC50 \u003d 100-200 nM).
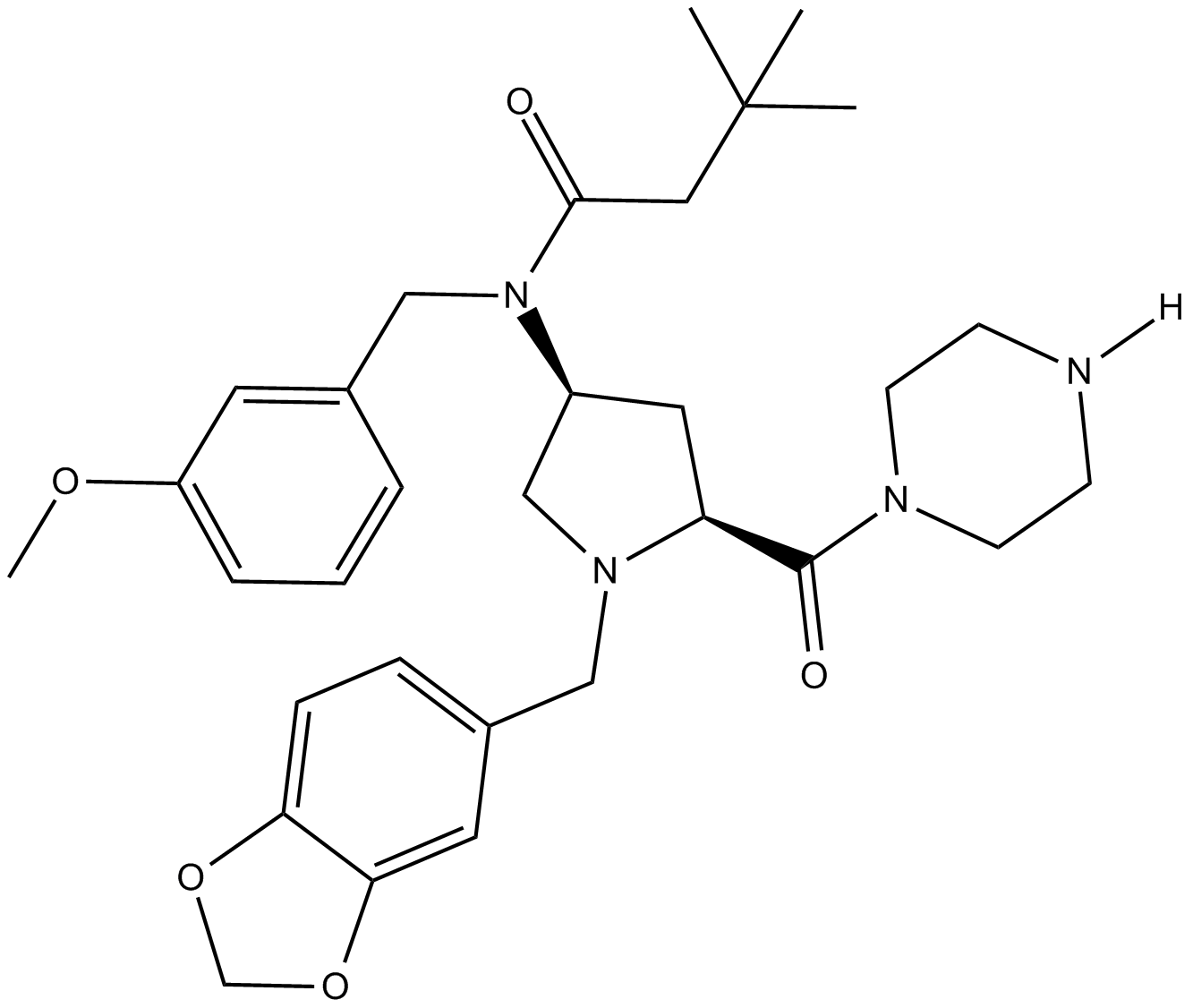
-
GC14787
Curcumin
Un pigmento amarillo con diversas actividades biológicas.
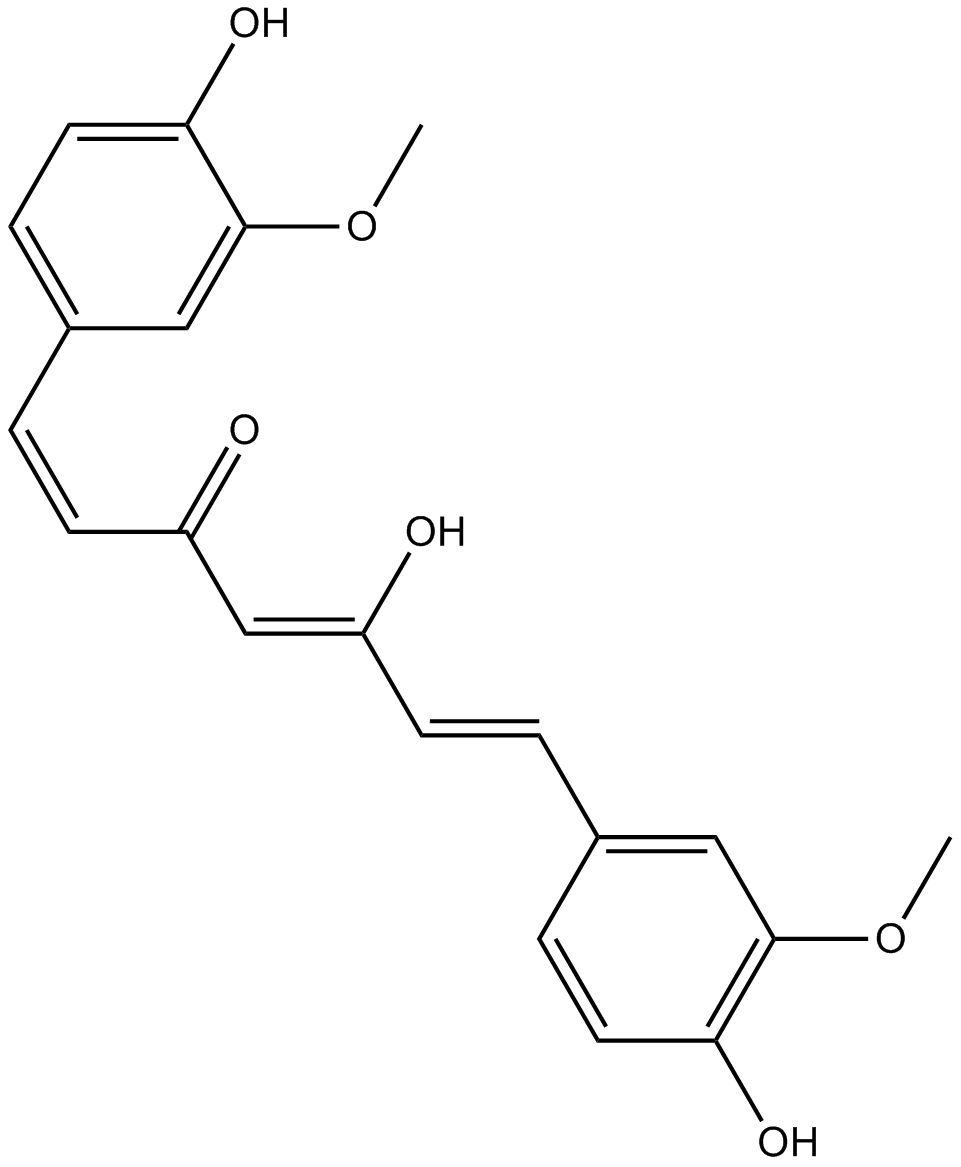
-
GC40226
Curcumin-d6
La curcumina D6 (diferuloilmetano D6) es una curcumina marcada con deuterio (cÚrcuma amarilla).
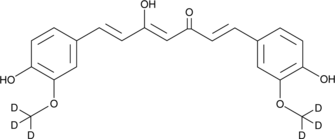
-
GN10521
Curcumol
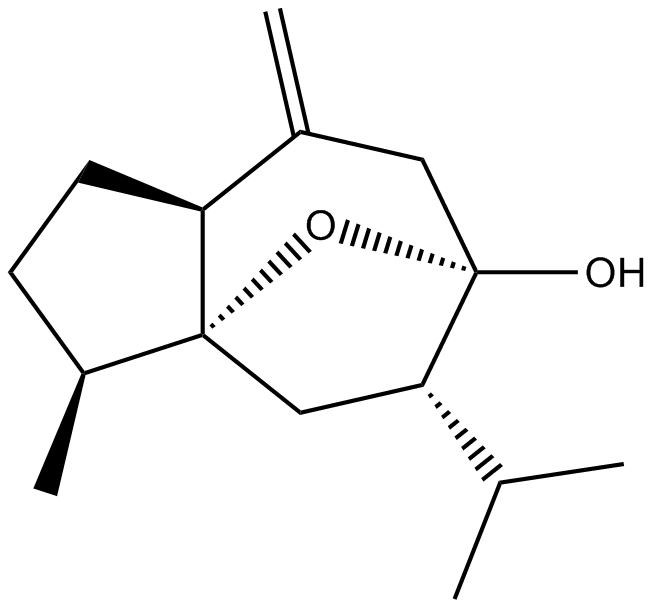
-
GC66356
Cusatuzumab
Cusatuzumab es un anticuerpo monoclonal humano αCD70. Cusatuzumab muestra actividad de citotoxicidad con células dependientes de anticuerpos mejoradas. Cusatuzumab reduce las células madre de leucemia (LSC) y activa firmas genéticas relacionadas con la diferenciaciÓn mieloide y la apoptosis. Cusatuzumab tiene potencial para la investigaciÓn de la leucemia mieloide aguda (LMA).

-
GC63967
Cycleanine
La ciclanina es un potente antagonista vascular selectivo del calcio.
-
GC49716
Cyclo(RGDyK) (trifluoroacetate salt)
A cyclic peptide ligand of αVβ3 integrin
-
GC17198
Cycloheximide
Cycloheximide is an antibiotic that inhibits protein synthesis at the translation level, acting exclusively on cytoplasmic (80s) ribosomes of eukaryotes.
-
GC43346
Cyclopamine-KAAD
La ciclopamina-KAAD, un inhibidor de la seÑalizaciÓn hedgehog, es un antagonista suavizado.

-
GC47148
Cyclophosphamide-d4
La ciclofosfamida-d4 es la ciclofosfamida marcada con deuterio. La ciclofosfamida es un agente alquilante sintético relacionado quÍmicamente con las mostazas nitrogenadas con actividad antineoplÁsica, inmunosupresora.
-
GC38419
Cyclovirobuxine D
La ciclovirobuxina D (CVB-D) es el principal componente activo de la medicina tradicional china Buxus microphylla.
-
GC33330
Cynaropicrin
La cinaropicrina es una lactona sesquiterpénica que puede inhibir la liberaciÓn del factor de necrosis tumoral (TNF-α) con IC50 de 8,24 y 3,18 μM para células macrÓfagas murinas y humanas, respectivamente. La cinaropicrina también inhibe el aumento del factor de degradaciÓn del cartÍlago (MMP13) y suprime la seÑalizaciÓn de NF-κB.
-
GC65565
Cyproheptadine
La ciproheptadina es un antagonista del receptor 5-HT2A potente y activo por vÍa oral, con efectos antidepresivos y antiserotonérgicos.
-
GC33779
Cysteamine (β-Mercaptoethylamine)
-
GC13502
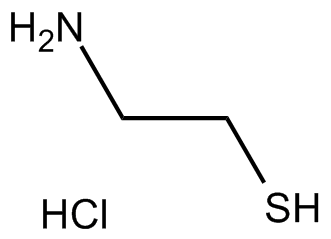
Cysteamine HCl
El clorhidrato de cisteamina (clorhidrato de 2-aminoetanotiol) es un agente activo por vÍa oral para el tratamiento de la cistinosis nefropÁtica y un antioxidante.
-
GC17050
CYT387
A potent inhibitor of JAK1 and JAK2
-
GC11383
CYT997 (Lexibulin)
CYT997 (Lexibulin) (CYT-997) es un inhibidor de polimerizaciÓn de tubulina potente y activo por vÍa oral con IC50 de 10-100 nM en lÍneas de células cancerosas; con potente actividad citotÓxica y disruptiva vascular in vitro e in vivo. CYT997 (Lexibulin) induce la apoptosis celular e induce la generaciÓn de ROS mitocondrial en células GC.
-
GC13070
Cytarabine
Agente citotóxico, bloquea la síntesis de ADN.
-
GC43356
CytoCalcein™ Violet 450
CytoCalcein? Violet 450 is a fluorogenic dye used to assess cell viability.
-
GC43357
CytoCalcein™ Violet 500
CytoCalcein? Violet 500 is a fluorogenic dye used to assess cell viability.
-
GC43361
Cytostatin (sodium salt)
Cytostatin is a natural antitumor inhibitor of cell adhesion to extracellular matrix, blocking adhesion of B16 melanoma cells to laminin and collagen type IV in vitro (IC50s = 1.3 and 1.4 μg/ml, respectively) and B16 cells metastatic activity in mice.
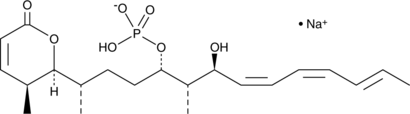
-
GC43368
D,L-1′-Acetoxychavicol Acetate
D,L-1′-Acetoxychavicol acetate is a natural compound first isolated from the rhizomes of ginger-like plants.
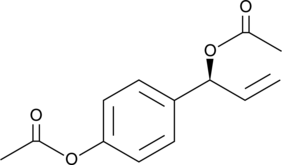
-
GC66824
D-α-Tocopherol Succinate
D-α-El succinato de tocoferol (succinato de vitamina E) es un tocoferol antioxidante y una forma de sal de la vitamina E. D-α-El succinato de tocoferol inhibe la citotoxicidad inducida por cisplatino. D-α-El succinato de tocoferol se puede utilizar para la investigación del cáncer.
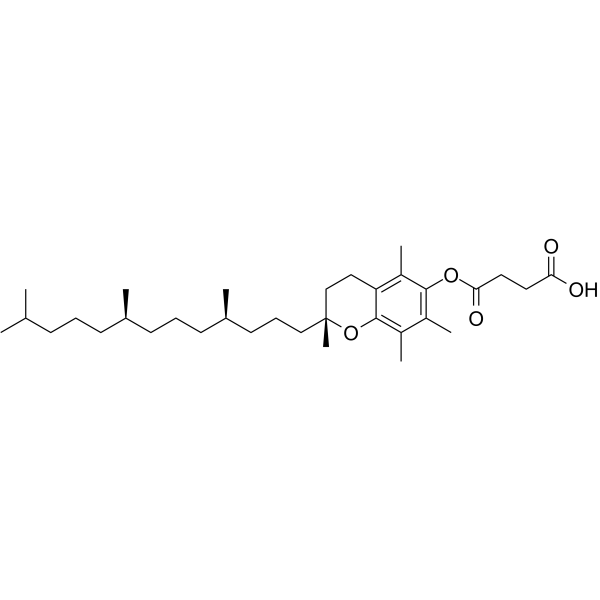
-
GC12256
D-Mannitol
El D-manitol es un agente diurético osmÓtico y un vasodilatador renal débil.
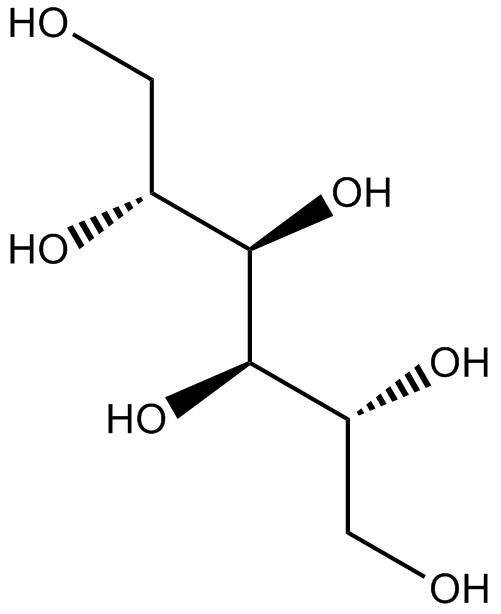
-
GC60796
D-Trimannuronic acid
El Ácido D-trimanurÓnico, un oligÓmero de alginato, se extrae de las algas.
-
GC13202
D4476
Inhibitor of CK1 and ALK5
-
GC17851
D609
A competitive inhibitor of PC-specific PLC
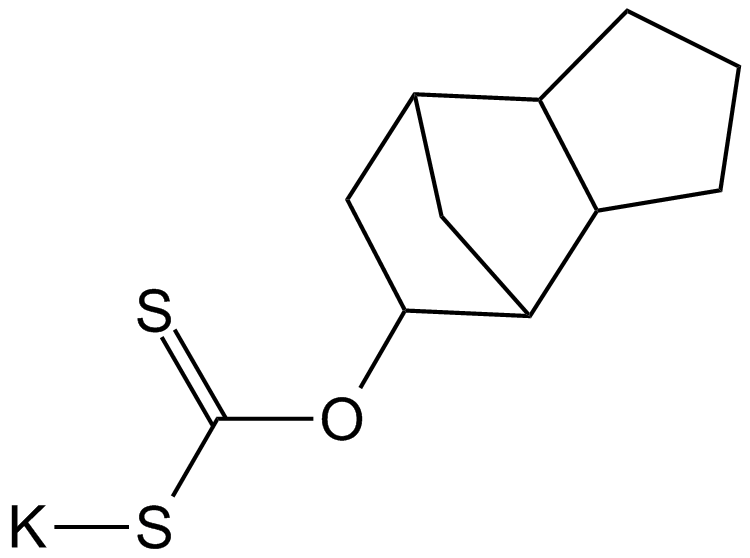
-
GC50296
D9
D9 es un inhibidor potente y selectivo de la tiorredoxina reductasa (TrxR), con una CE50 de 2,8 nM. D9 tiene la capacidad de inhibir la proliferaciÓn tumoral tanto in vitro como in vivo.
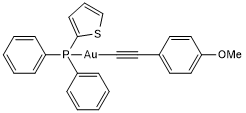
-
GC18421
Dabcyl-YVADAPV-EDANS
Dabcyl-YVADAPV-EDANS is a fluorogenic substrate for caspase-1.
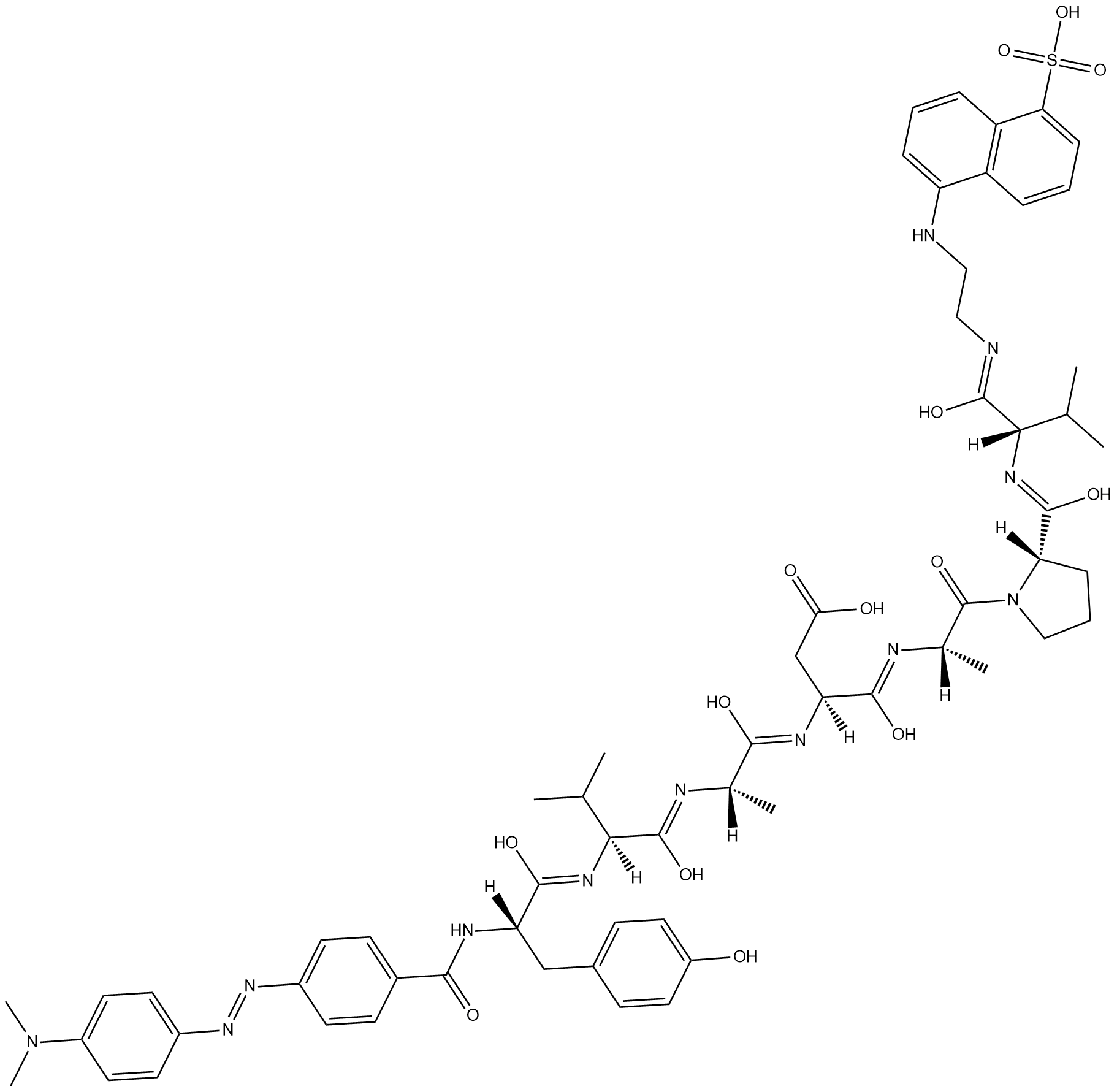
-
GC47166
Dabrafenib-d9
Dabrafenib-d9 (GSK2118436A-d9) es el deuterio etiquetado como Dabrafenib. Dabrafenib (GSK2118436A) es un inhibidor de Raf competitivo con ATP con IC50 de 5 nM y 0,6 nM para C-Raf y B-RafV600E, respectivamente.
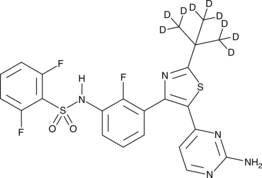
-
GC14485
Dacarbazine
La dacarbazina es un agente alquilante antineoplÁsico no especÍfico del ciclo celular. La dacarbazina inhibe la respuesta linfoblÁstica T y B, con valores de IC50 de 50 y 10 μg/ml, respectivamente. La dacarbazina se puede utilizar para la investigaciÓn del melanoma maligno metastÁsico.
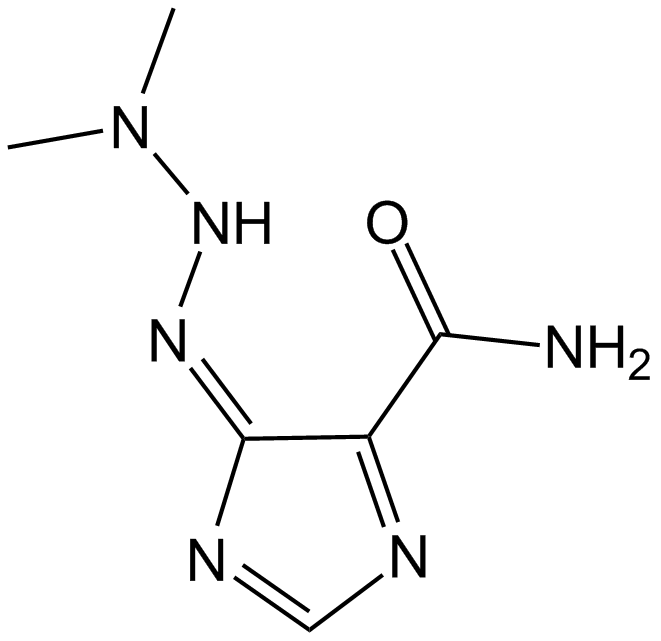
-
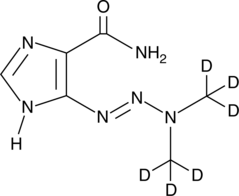
GC47167
Dacarbazine-d6
La dacarbazina-d6 (imidazol carboxamida-d6) es la dacarbazina marcada con deuterio. La dacarbazina (DTIC-Dome; DTIC) es un agente antineoplÁsico. Tiene una actividad significativa contra los melanomas.
-
GC68305
Dacetuzumab

-
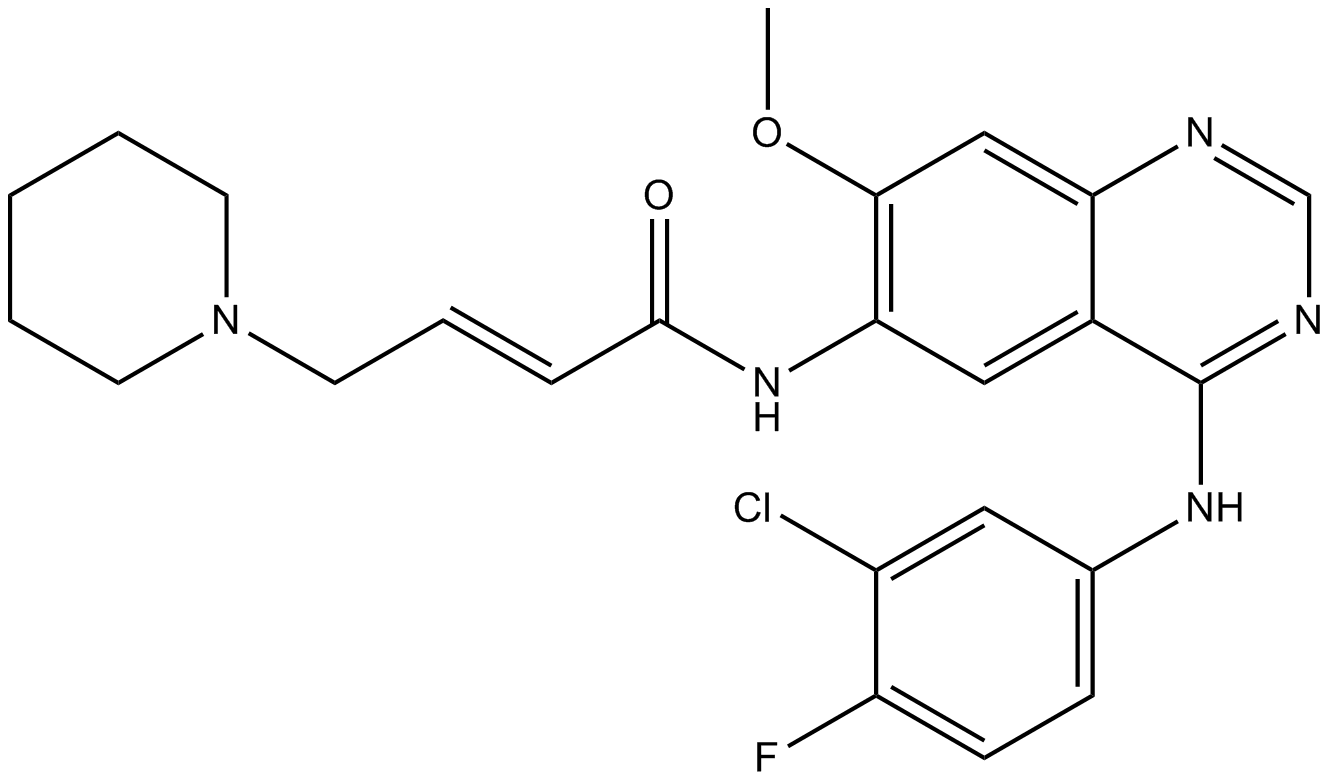
GC10225
Dacomitinib (PF299804, PF299)
Dacomitinib (PF299804, PF299) (PF-00299804) es un inhibidor especÍfico e irreversible de la familia de quinasas ERBB con IC50 de 6 nM, 45,7 nM y 73,7 nM para EGFR, ERBB2 y ERBB4, respectivamente.
-
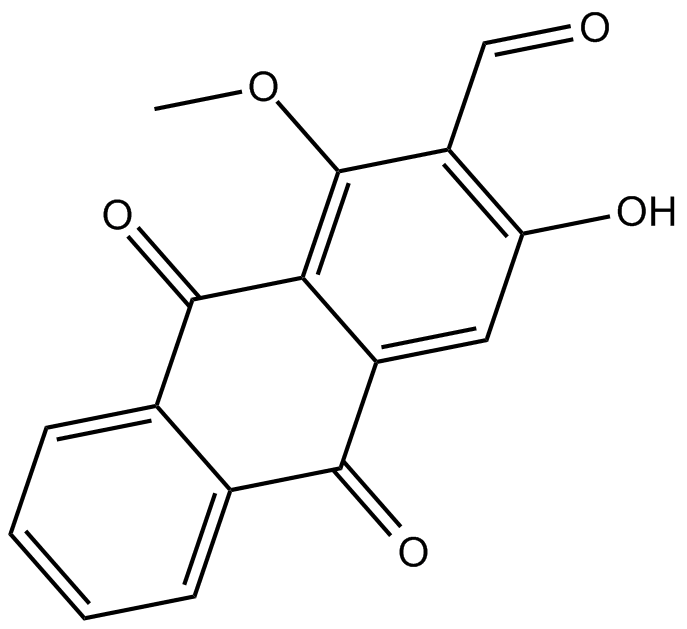
GC15211
Damnacanthal
Damnacanthal es una antraquinona aislada de la raÍz de Morinda citrifolia.
-
GN10318
Danshensu
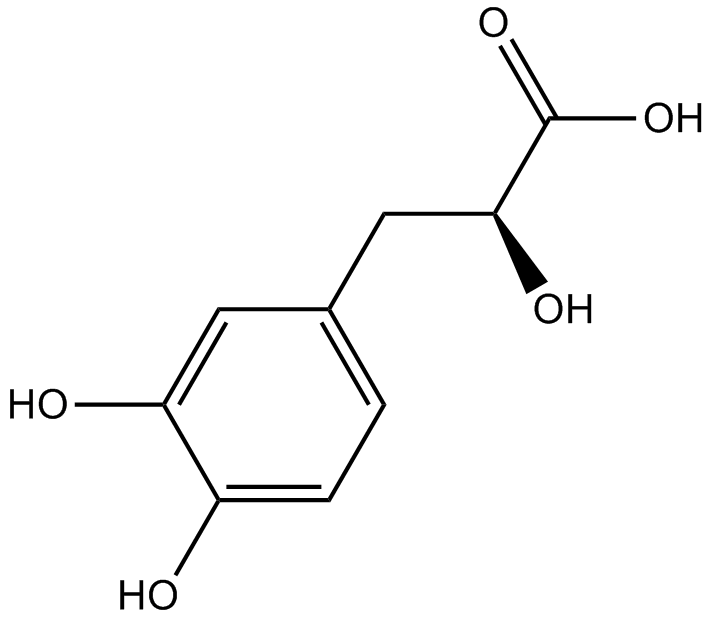
-
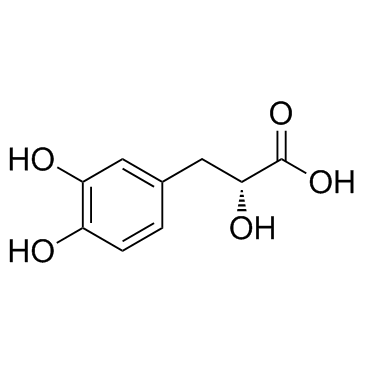
GC34010
Danshensu (Dan shen suan A)
Danshensu, an active ingredient of Salvia miltiorrhiza, shows wide cardiovascular benefit by activating Nrf2 signaling pathway.
-
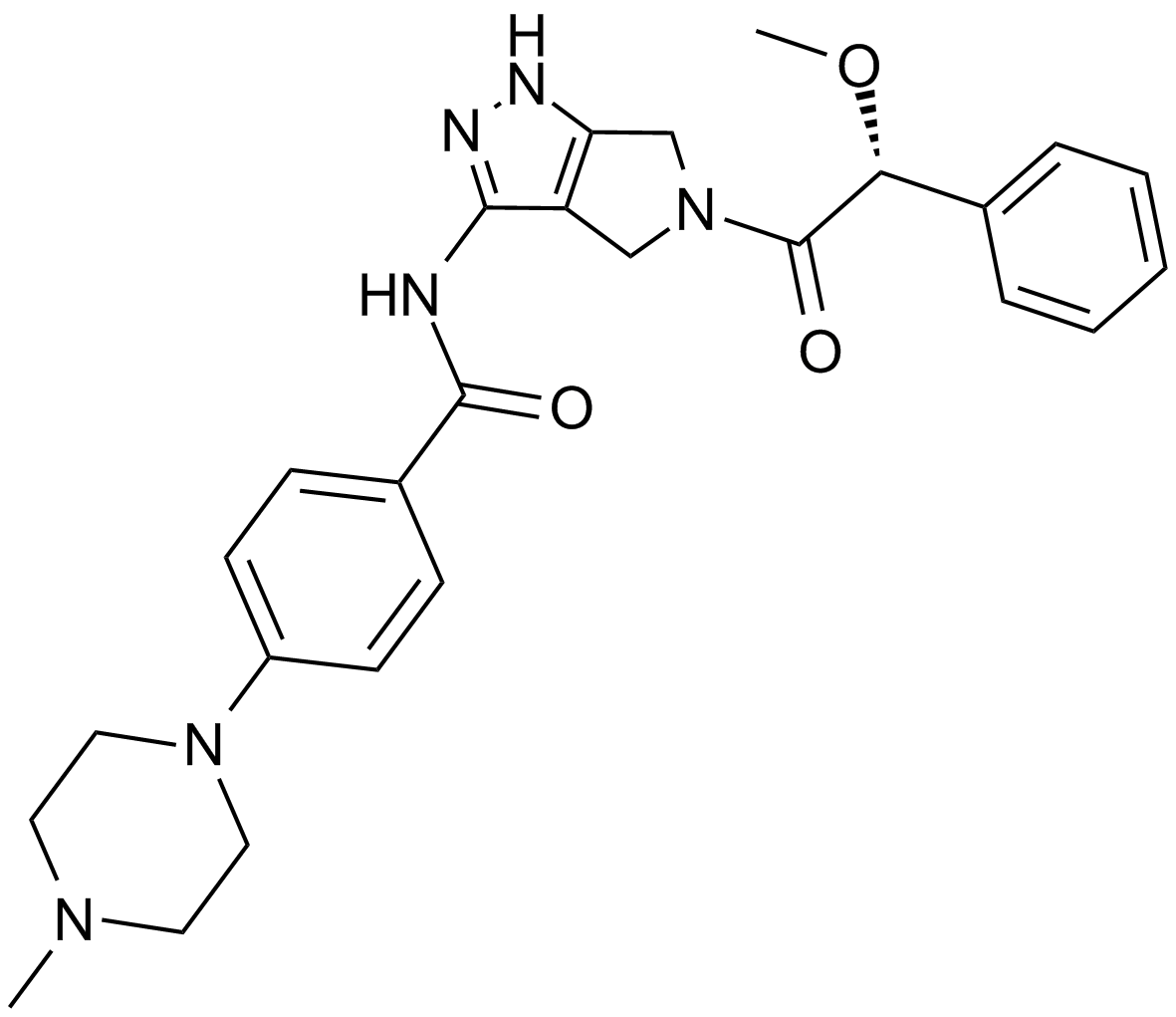
GC15217
Danusertib (PHA-739358)
A pan-Aurora kinase and Abl inhibitor
-
GC17650
DAPK Substrate Peptide
A DAPK1 peptide substrate
-
GC49883
DAPK Substrate Peptide (trifluoroacetate salt)
A DAPK1 peptide substrate

-
GC12942
DAPT (GSI-IX)
Inhibidor de la γ-secretasa
-
GC43379
Darinaparsin
Darinaparsin (ZIO-101), un arsénico orgÁnico, es un agente dirigido a las mitocondrias. La darinaparsina induce la apoptosis en las células cancerosas y tiene efectos anticancerÍgenos.
-
GC15568
Dasatinib (BMS-354825)
Un inhibidor de Abl y Src.
-
GC35812
Dasatinib hydrochloride
El clorhidrato de dasatinib (BMS-354825) es un inhibidor dual de Src/Bcr-Abl muy potente, competitivo con ATP, activo por vÍa oral y con potente actividad antitumoral. Los Kis son 16 pM y 30 pM para Src y Bcr-Abl, respectivamente. El clorhidrato de dasatinib inhibe Bcr-Abl y Src con IC50 de <1,0 nM y 0,5 nM, respectivamente. El clorhidrato de dasatinib también induce apoptosis y autofagia.
-
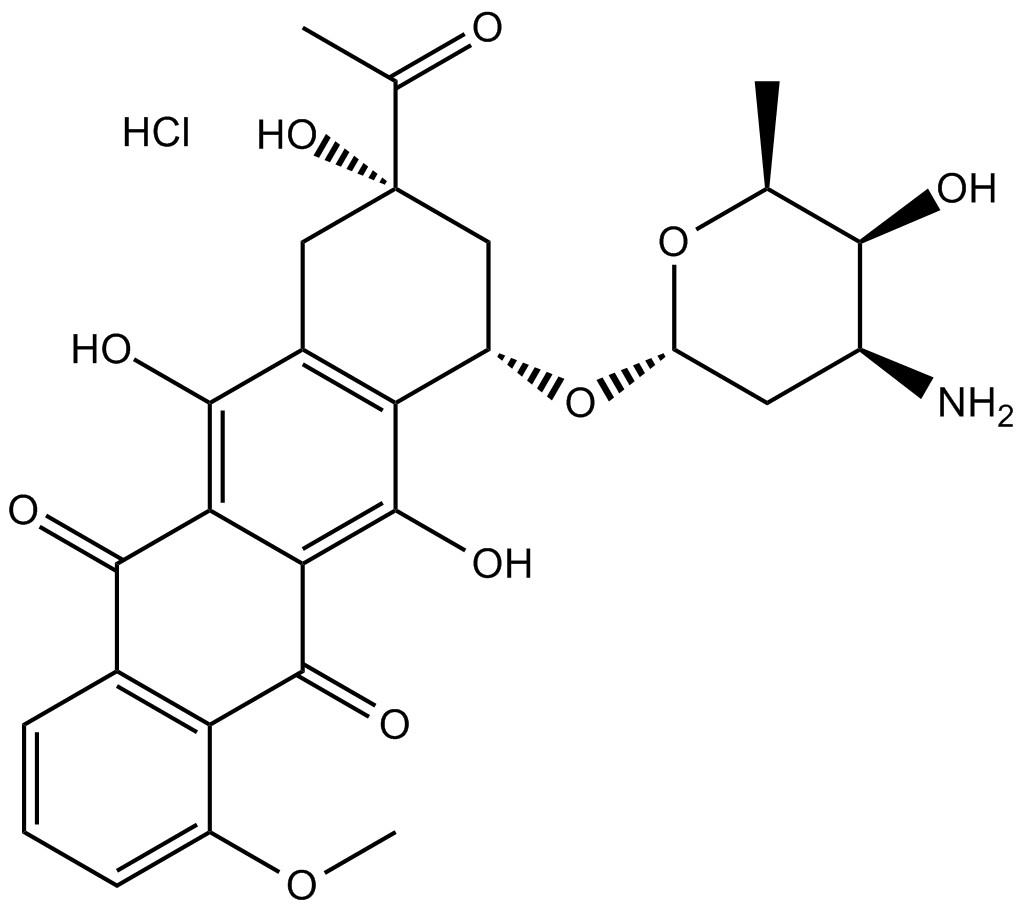
GC10354
Daunorubicin HCl
El clorhidrato de daunorrubicina (daunomicina) es un inhibidor de la topoisomerasa II con una potente actividad antitumoral.