Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
GC10610
Adapalene
L'adapalène (CD271), un rétinoÏde synthétique de troisième génération, est largement utilisé pour la recherche sur l'acné.

-
GC46798
Adapalene-d3
An internal standard for the quantification of adapalene
-
GC13959
Adarotene
An atypical retinoid
-
GC65880
ADH-6 TFA
ADH-6 TFA est un composé tripyridylamide. ADH-6 abroge l'auto-assemblage du sous-domaine d'agrégation-nucléation du mutant p53 DBD. L'ADH-6 TFA cible et dissocie les agrégats de p53 mutants dans les cellules cancéreuses humaines, ce qui restaure l'activité transcriptionnelle de p53', entraÎnant l'arrêt du cycle cellulaire et l'apoptose. L'ADH-6 TFA a le potentiel pour la recherche sur les maladies cancéreuses.
-
GC42735
Adipostatin A
L'adipostatine A (Adipostatine A) est un inhibiteur de la glycérol-3-phosphate déshydrogénase (GPDH) avec une IC50 de 4,1 μM.
-
GC11892
AEE788 (NVP-AEE788)
AEE788 (NVP-AEE788) est un inhibiteur de l'EGFR et ErbB2 avec des valeurs IC50 de 2 et 6 nM, respectivement.
-
GC42743
AEM1
AEM1 est un inhibiteur de Nrf2. AEM1 réduit les expressions des gènes dépendants de Nrf2 dans les cellules A549 et inhibe la croissance des cellules A549 in vitro et in vivo.
-
GC13168
AG 825
AG 825 (Tyrphostin AG 825) est un inhibiteur sélectif et compétitif d'ErbB2 qui supprime la phosphorylation de la tyrosine, avec une IC50 de 0,35 μM.
-
GC13697
AG-1024
AG-1024 (Tyrphostin AG 1024) est un inhibiteur réversible, compétitif et sélectif de l'IGF-1R avec une IC50 de 7 μM.
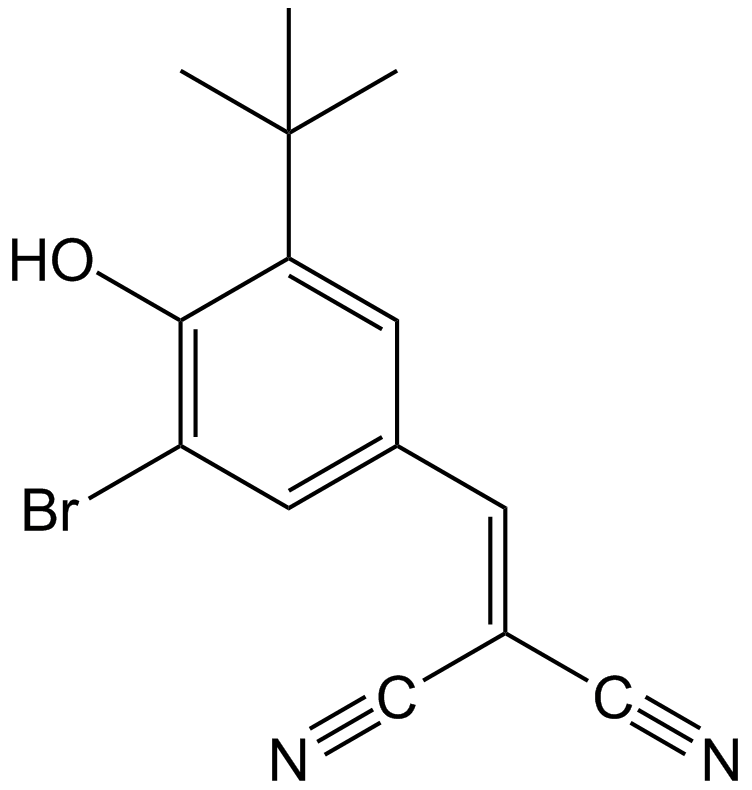
-
GC17881
AGK 2
AGK 2 est un inhibiteur sélectif de SIRT2 avec une IC50 de 3,5 μM. AGK 2 inhibe SIRT1 et SIRT3 avec des IC50 de 30 et 91 μM, respectivement.
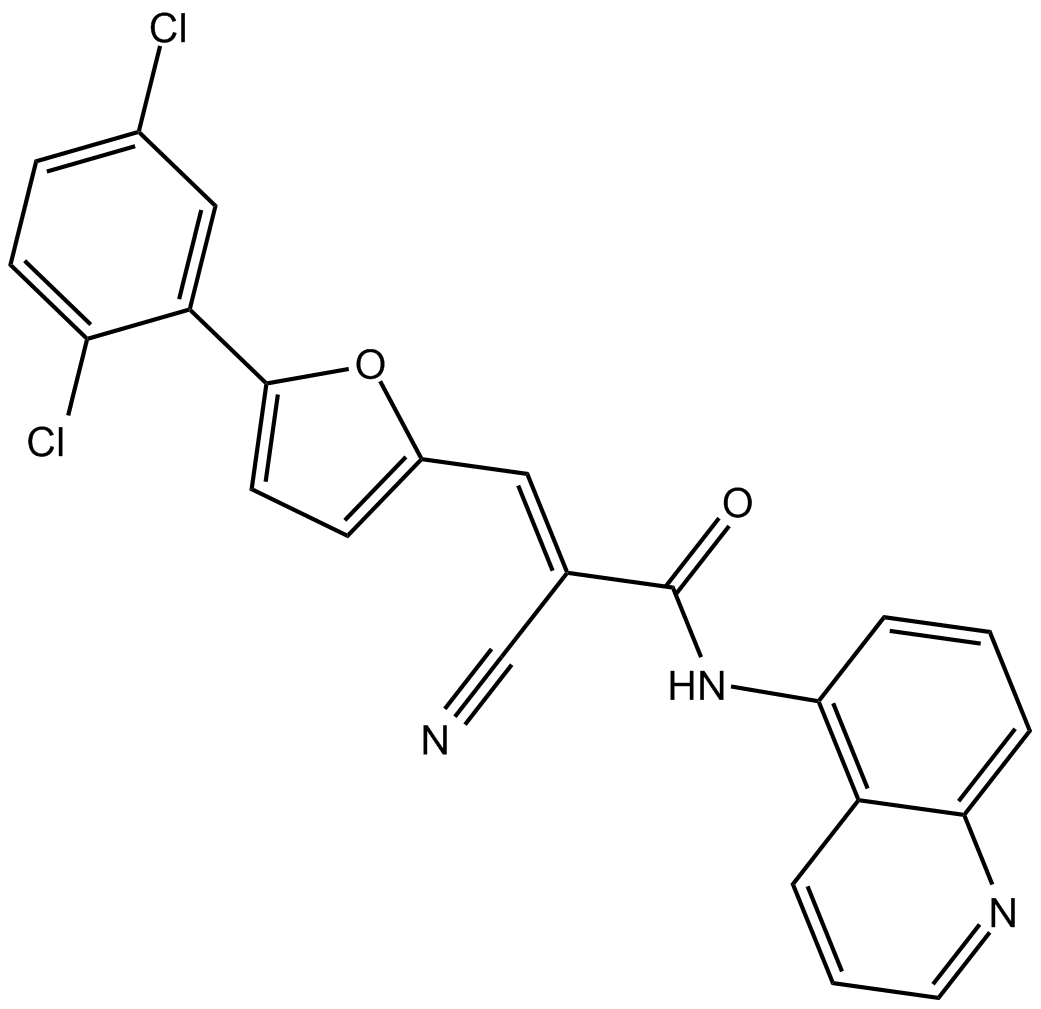
-
GC39584
AGN194204
AGN194204 (IRX4204) est un agoniste sélectif et actif par voie orale de RXR avec des valeurs Kd de 0,4 nM, 3,6 nM et 3,8 nM et des EC50s de 0,2 nM, 0,8 nM et 0.08 nM pour RXRα , RXRβ et RXRγ respectivement.
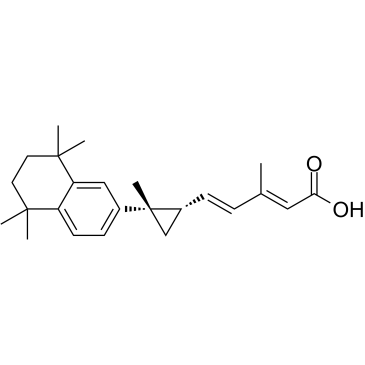
-
GC16120
AI-3
Nrf2/Keap1 and Keap1/Cul3 interaction inhibitor
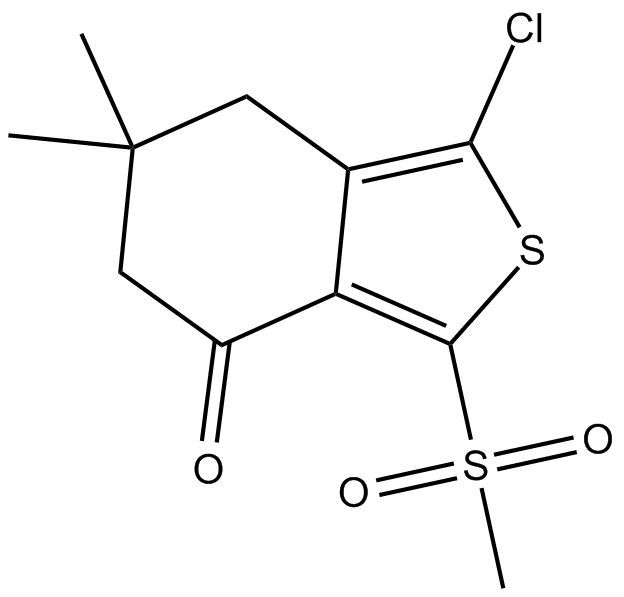
-
GC46821
Ajoene
L'ajoène, un composé dérivé de l'ail, est un agent antithrombotique et antifongique.
-
GC39620
AKOS-22
-
GC11589
AKT inhibitor VIII
A potent inhibitor of Akt1 and Akt2

-
GC35275
AKT-IN-3
AKT-IN-3 (composé E22) est un puissant inhibiteur d'Akt bloquant le hERG faiblement actif par voie orale, avec 1,4 nM, 1,2 nM et 1,7 nM pour Akt1, Akt2 et Akt3, respectivement. AKT-IN-3 (composé E22) présente également une bonne activité inhibitrice contre d'autres kinases de la famille AGC, telles que PKA, PKC, ROCK1, RSK1, P70S6K et SGK. AKT-IN-3 (composé E22) induit l'apoptose et inhibe la métastase des cellules cancéreuses.
-
GC49773
Albendazole sulfone-d3
An internal standard for the quantification of albendazole sulfone
-
GC48848
Albendazole-d7
L'albendazole-d7 (SKF-62979-d7) est l'albendazole marqué au deutérium.
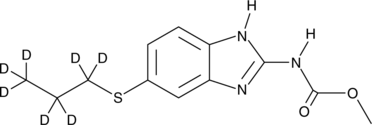
-
GC41080
Albofungin
Albofungin is a xanthone isolated from A.
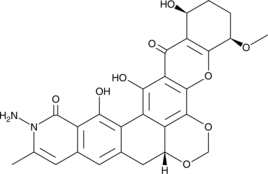
-
GC16597
Alda 1
Alda 1 est un agoniste puissant et sélectif de l'ALDH2, qui active l'ALDH2 de type sauvage et restaure l'activité quasi sauvage de l'ALDH2*2.
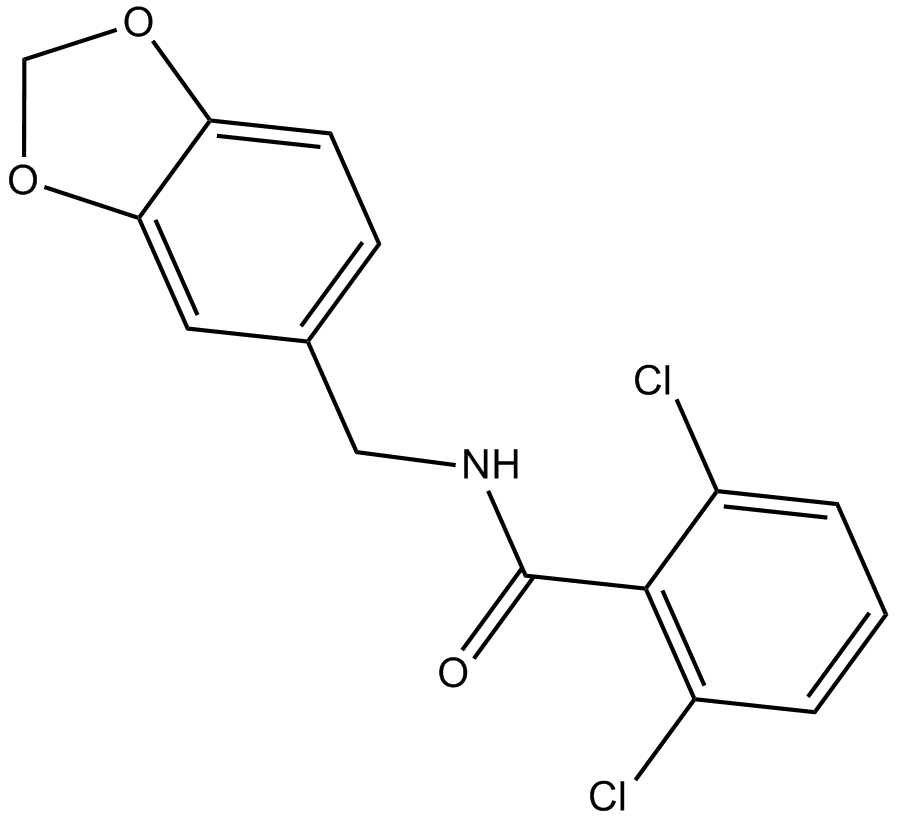
-
GC35288
Alkannin
L'alkannin, trouvé dans Alkanna tinctoria, est utilisé comme colorant alimentaire.
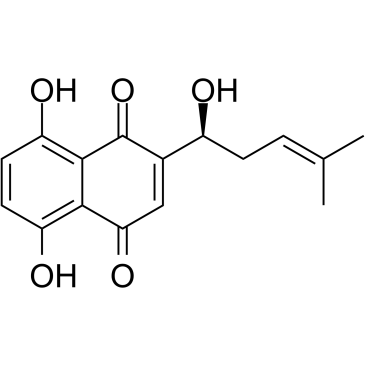
-
GC49393
all-trans-13,14-Dihydroretinol
A metabolite of all-trans retinoic acid

-
GC32127
Alofanib (RPT835)
L'alofanib (RPT835) (RPT835) est un inhibiteur allostérique puissant et sélectif du récepteur 2 du facteur de croissance des fibroblastes (FGFR2).

-
GC14314
Aloperine
L'alopérine est un alcaloÏde des plantes sophora telles que Sophora alopecuroides L, qui a montré des propriétés anticancéreuses, anti-inflammatoires et anti-virales.
-
GC35306
alpha-Mangostin
alpha-Mangostin (α-Mangostin) est une xanthone alimentaire avec de larges activités biologiques, telles que des effets antioxydants, anti-allergiques, antiviraux, antibactériens, anti-inflammatoires et anticancéreux. C'est un inhibiteur du mutant IDH1 (IDH1-R132H) avec un Ki de 2,85 μM.
-
GC18437
Alternariol monomethyl ether
L'éther monométhylique d'alternariol, isolé des racines d'Anthocleista djalonensis (Loganiaceae), est un important marqueur taxonomique de l'espèce végétale.
-
GC33356
AM-8735
L'AM-8735 est un inhibiteur puissant et sélectif de MDM2 avec une IC50 de 25 nM.
-
GC42776
Amarogentin
L'amarogentine est un glycoside secoiridoÏde principalement extrait des racines de Swertia et de Gentiana.
-
GN10484
Amentoflavone
-
GC42783
Ametantrone
L'amétantrone (NSC 196473) est un agent antitumoral qui s'intercale dans l'ADN et induit une rupture de l'ADN médiée par la topoisomérase II (TOP2).
-
GC19452
AMG-176
AMG-176 (Tapotoclax) est un inhibiteur de MCL-1 puissant, sélectif et actif par voie orale, avec un Ki de 0,13 nM.
-
GC15828
AMG232
AMG232 (AMG 232) est un inhibiteur puissant, sélectif et disponible par voie orale de l'interaction p53-MDM2, avec une IC50 de 0,6 nM. AMG232 se lie À MDM2 avec un Kd de 0,045 nM.
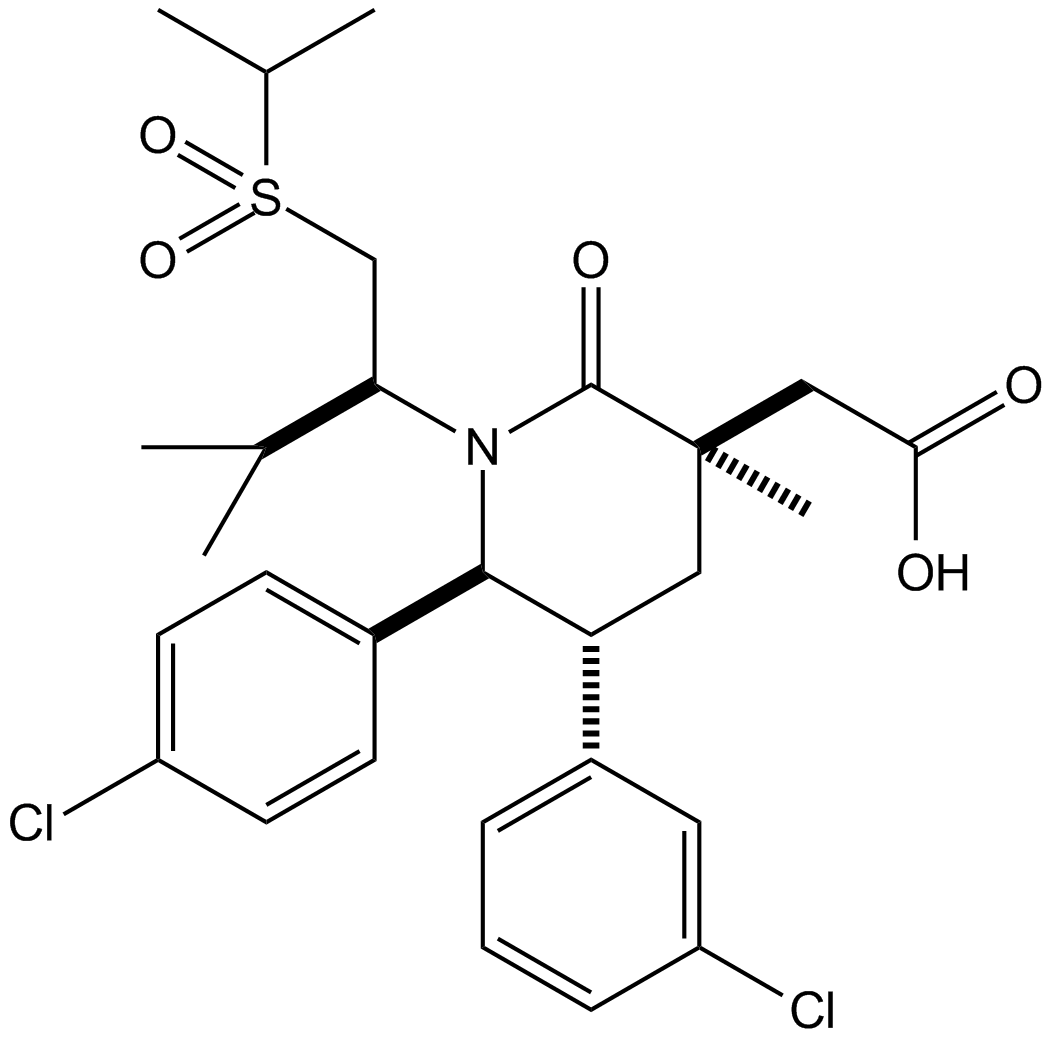
-
GC42785
Amifostine (hydrate)
L'amifostine (hydrate) (WR2721 trihydraté) est un agent cytoprotecteur À large spectre et un radioprotecteur. L'amifostine (hydrate) protège sélectivement les tissus normaux des dommages causés par la radiothérapie et la chimiothérapie. L'amifostine (hydrate) est un puissant facteur inductible par l'hypoxie-α1 (HIF-α1) et un inducteur de p53. L'amifostine (hydrate) protège les cellules contre les dommages en éliminant les radicaux libres dérivés de l'oxygène. L'amifostine (hydrate) réduit la toxicité rénale et a une action anti-angiogénique.
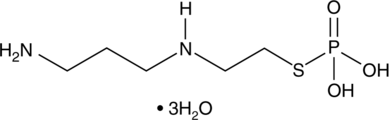
-
GC61804
Amifostine thiol
Amifostine thiol (WR-1065) est un métabolite actif du cytoprotecteur Amifostine. Le thiol d'amifostine est un agent cytoprotecteur doté de capacités radioprotectrices. Le thiol d'amifostine active p53 via une voie de signalisation dépendante de JNK.
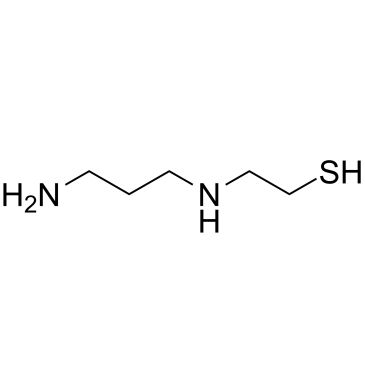
-
GC12051
Amiloride HCl dihydrate
L'amiloride HCl dihydraté (MK-870 chlorhydrate dihydraté) est un inhibiteur À la fois du canal sodique épithélial (ENaC[1]) et du récepteur de l'activateur du plasminogène de type urokinase (uTPA[2]).
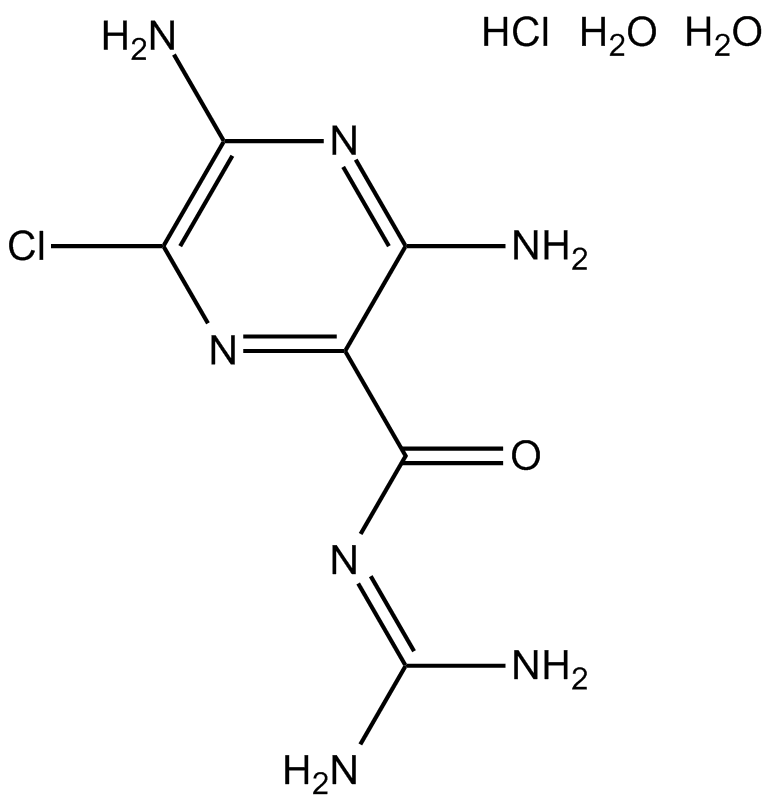
-
GC63932
Amsilarotene
L'amsilarotène (TAC-101 ; Am 555S), un rétinoÏde synthétique actif par voie orale, a une affinité sélective pour le récepteur de l'acide rétinoÏque α ; (RAR-α) liaison avec Ki de 2,4, 400 nM pour RAR-α et RAR-β ;. L'amsilarotène induit l'apoptose des cellules humaines du cancer gastrique, du carcinome hépatocellulaire et du carcinome ovarien. L'amsilarotène peut être utilisé pour la recherche sur le cancer.
-
GC16391
Amuvatinib (MP-470, HPK 56)
A multi-targeted RTK inhibitor
-
GC48339
Amycolatopsin A
A macrolide polyketide with antimycobacterial and anticancer activities
-
GC48341
Amycolatopsin B
A bacterial metabolite
-
GC48350
Amycolatopsin C
A polyketide macrolide with antimycobacterial and anticancer activities
-
GC42806
Andrastin A
Andrastin A is a meroterpenoid farnesyltransferase inhibitor.
-
GN10045
Angelicin
-
GC60584
Angiotensin II human acetate
L'acétate d'angiotensine II humaine (angiotensine II) est un vasoconstricteur et un peptide bioactif majeur du système rénine/angiotensine.
-
GC42813
Anguinomycin A
Anguinomycin A is an antibiotic first isolated from a Streptomyces sp.
-
GC40614
Anhydroepiophiobolin A
L'anhydroépiophiobolin A, un analogue de l'ophiobolin A, est un puissant inhibiteur de la photosynthèse (I50 de 6,1 et 1 mM pour la photosynthèse chez Chlorella et Spinach, respectivement).
-
GC40214
Anhydroophiobolin A
L'anhydroophiobolin A est un puissant inhibiteur de la photosynthèse avec des IC50 de 77 et 14 mM dans la photosynthèse de la chlorella et des épinards, respectivement.
-
GC11559
Anisomycin
Agoniste de JNK, puissant et spécifique.
-
GC49259
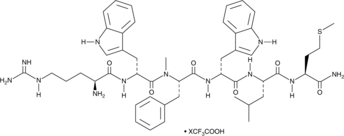
Antagonist G (trifluoroacetate salt)
A neuropeptide antagonist
-
GC66337
Anti-Mouse PD-L1 Antibody
Anti-L'anticorps PD-L1 de souris est un inhibiteur de l'anticorps IgG2b anti-PD-L1 de souris dérivé du rat hÔte.

-
GC35361
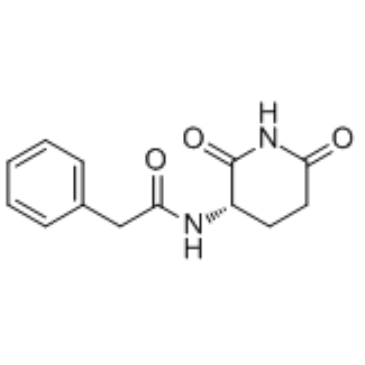
Antineoplaston A10
L'antinéoplaston A10, une substance naturellement présente dans le corps humain, est un inhibiteur de Ras potentiellement pour le traitement du gliome, du lymphome, de l'astrocytome et du cancer du sein.
-
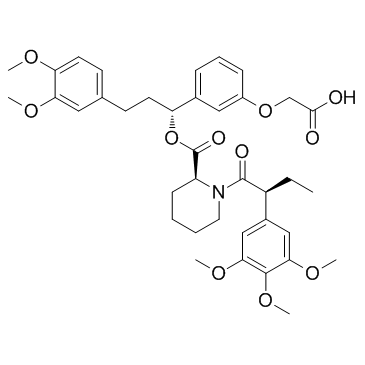
GC34172
AP1867
AP1867 est un ligand synthétique dirigé par FKBP12F36V.
-
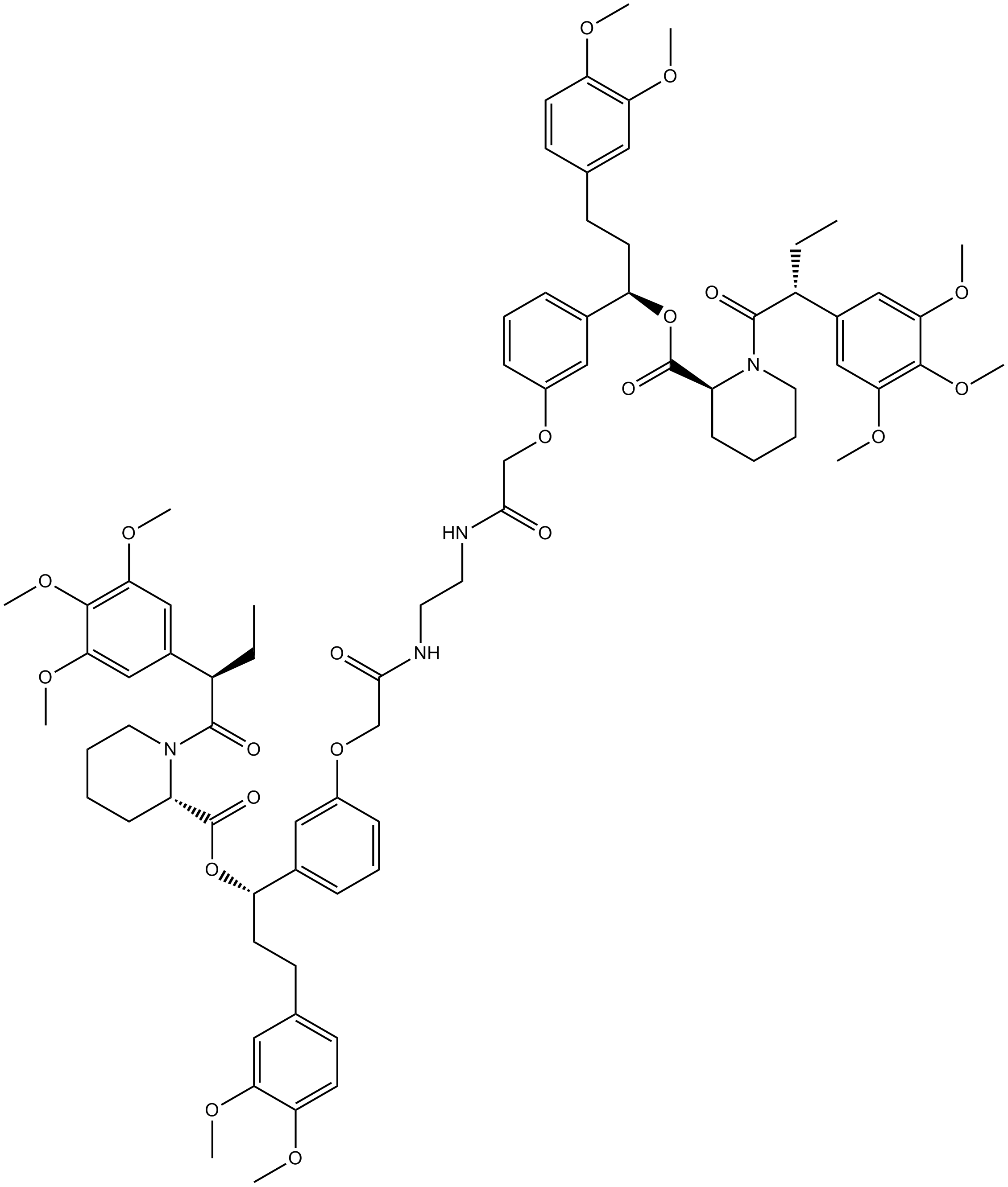
GC61745
AP1867-2-(carboxymethoxy)
AP1867-2-(carboxyméthoxy), le fragment basé sur AP1867 (un ligand synthétique dirigé par FKBP12F36V), se lie au ligand CRBN via un lieur pour former des molécules dTAG.

-
GC15586
AP1903
AP1903 (AP1903) est un agent dimérisant qui agit en réticulant les domaines FKBP. AP1903 (AP1903) dimérise l'interrupteur suicide Caspase 9 et induit rapidement l'apoptose.
-
GC14498
AP20187
AP20187 (B/B Homodimerizer) est un ligand perméable aux cellules utilisé pour dimériser les protéines de fusion de la protéine de liaison FK506 (FKBP) et initier des cascades de signalisation biologique et l'expression génique ou perturber les interactions protéine-protéine.
-
GC18518
Apcin
L'apcine, un ligand de Cdc20, est un inhibiteur puissant et compétitif de l'activité de la ligase E3 complexe/cyclosome (APC/C(Cdc20)) favorisant l'anaphase. Apcin inhibe de manière compétitive l'ubiquitylation dépendante d'APC/C en se liant À Cdc20 et en empêchant la reconnaissance du substrat. Apcin occupe la poche de liaison D-box sur la face latérale du domaine WD40 et peut prolonger la mitose.
-
GC62419
Apcin-A
L'apcine-A, un dérivé de l'apcine, est un inhibiteur du complexe promoteur de l'anaphase (APC). Apcin-A interagit fortement avec Cdc20 et inhibe l'ubiquitination des substrats Cdc20. Apcin-A peut être utilisé pour synthétiser le PROTAC CP5V.
-
GC35367
APG-115
APG-115 (APG-115) est un inhibiteur de la protéine MDM2 actif par voie orale se liant À la protéine MDM2 avec des valeurs IC50 et Ki de 3,8 nM et 1 nM, respectivement. L'APG-115 bloque l'interaction de MDM2 et de p53 et induit l'arrêt du cycle cellulaire et l'apoptose d'une manière dépendante de p53.
-
GC62640
APG-1387
APG-1387, un mimétique SMAC bivalent et un antagoniste IAP, bloque l'activité des protéines de la famille des IAP (XIAP, cIAP-1, cIAP-2 et ML-IAP). APG-1387 induit la dégradation des protéines cIAP-1 et XIAP, ainsi que l'activation de la caspase-3 et le clivage PARP, ce qui conduit À l'apoptose. L'APG-1387 peut être utilisé pour la recherche sur le carcinome hépatocellulaire, le cancer de l'ovaire et le carcinome du nasopharynx.
-
GC12961
Apicidin
Un inhibiteur d'HDAC perméable à la cellule.
-
GC46862
Apigenin-d5
An internal standard for the quantification of apigenin
-
GC16237
Apocynin
L'apocynine est un inhibiteur sélectif de la NADPH-oxydase avec une IC50 de 10 μM.
-
GC14080
Apogossypolone (ApoG2)
-
GC42827
Apoptolidin
L'apoptolidine est un polykétide isolé de la bactérie Nocardiopsis. L'apoptolidine est un inhibiteur sélectif de l'ATPase F1FO mitochondriale. L'apoptolidine est un inducteur de l'apoptose et induit la mort cellulaire apoptotique dans les cellules transformées avec les oncogènes de l'adénovirus de type 12, y compris ElA (IC50 = 10-17 ng/ml) mais pas dans les cellules normales.
-
GC14209
Apoptosis Activator 2
An activator of caspases
-
GC14411
Apoptozole
L'apoptazole (Apoptosis Activator VII) est un inhibiteur du domaine ATPase de Hsc70 et Hsp70, avec des Kd de 0,21 et 0,14μM, respectivement, et peut induire l'apoptose.
-
GC65004
Apostatin-1
L'apostatine-1 (Apt-1) est un puissant inhibiteur de TRADD.
-
GC35377
Apratastat
Apratastat (TMI-005) est un inhibiteur de TACE/MMPs actif par voie orale, non sélectif et réversible, qui peut inhiber la libération de TNF-α. Apratastat a le potentiel de surmonter la résistance À la radiothérapie dans le cancer du poumon non À petites cellules (NSCLC).
-
GC10420
Apremilast (CC-10004)
An orally available PDE4 inhibitor
-
GC32692
APTO-253 (LOR-253)
APTO-253 est une petite molécule innovante qui exerce une activité antitumorale puissante en induisant l'expression génique du facteur de transcription maître Kruppel-like factor 4 (KLF4), inhibant ainsi le cycle cellulaire et conduisant à la mort cellulaire programmée.
-
GC14590
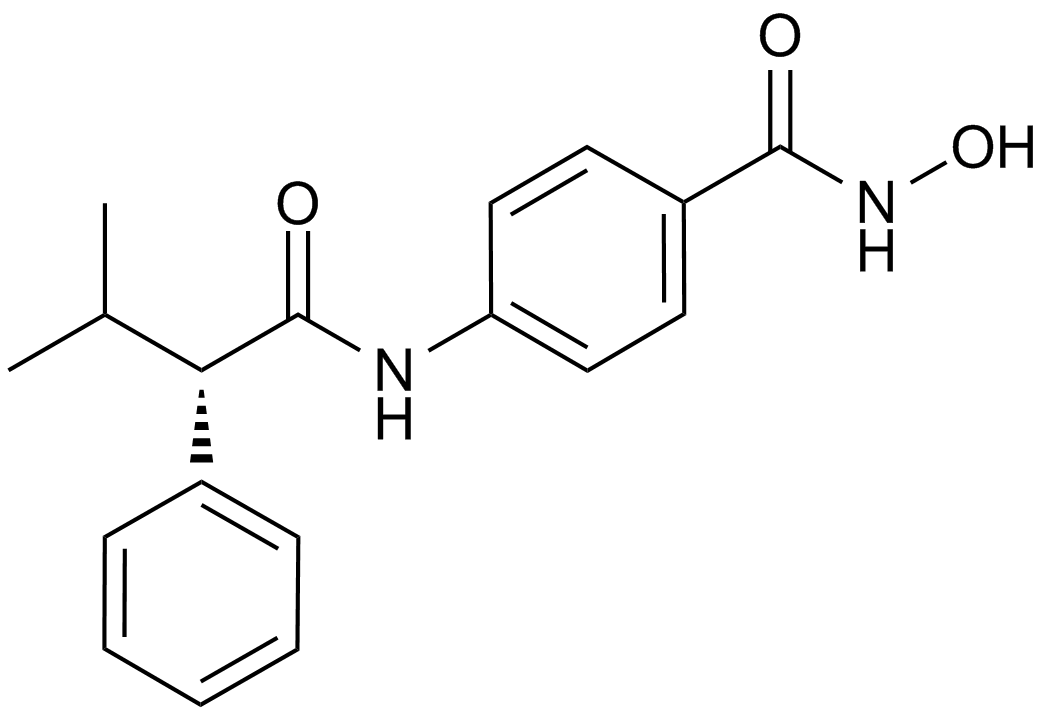
AR-42 (OSU-HDAC42)
HDAC inhibitor,novel and potent
-
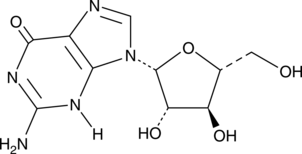
GC45385
Ara-G
-
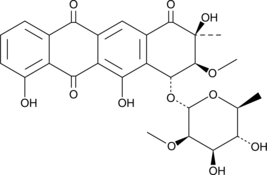
GC46878
Aranciamycin
A fungal metabolite with diverse biological activities
-
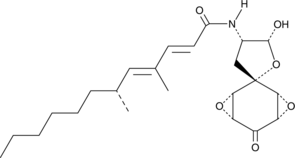
GC40116
Aranorosin
L'aranorosine, un antibiotique antifongique puissant, a été isolée du filtrat de culture et du mycélium d'une souche de Pseudoarachniotus roseus Kuehn.
-
GC65163
Ardisiacrispin B
L'ardisiacrispine B présente des effets cytotoxiques dans les cellules cancéreuses multifactorielles résistantes aux médicaments via la mort cellulaire ferroptotique et apoptotique.
-
GC49314
Arecaidine propargyl ester (hydrobromide)
A muscarinic M2 agonist
-
GC35388
Aristolactam I
L'aristololactame I (AL-I), est le principal métabolite de l'acide aristolochique I (AA-I), participe aux processus qui conduisent À des lésions rénales.
-
GC35395
Arnicolide D
L'arnicolide D est une lactone sesquiterpénique isolée de Centipeda minima. L'arnicolide D module le cycle cellulaire, active la voie de signalisation des caspases et inhibe les voies de signalisation PI3K/AKT/mTOR et STAT3. L'arnicolide D inhibe la viabilité des cellules du carcinome du nasopharynx (NPC) en fonction de la concentration et du temps.
-
GC19037
ARS-853
L'ARS-853 est un inhibiteur de KRAS G12C covalent, sélectif et actif sur les cellules avec une IC50 de 2,5 μM. L'ARS-853 inhibe la signalisation mutée par KRAS en se liant À l'oncoprotéine liée au GDP et en empêchant l'activation.
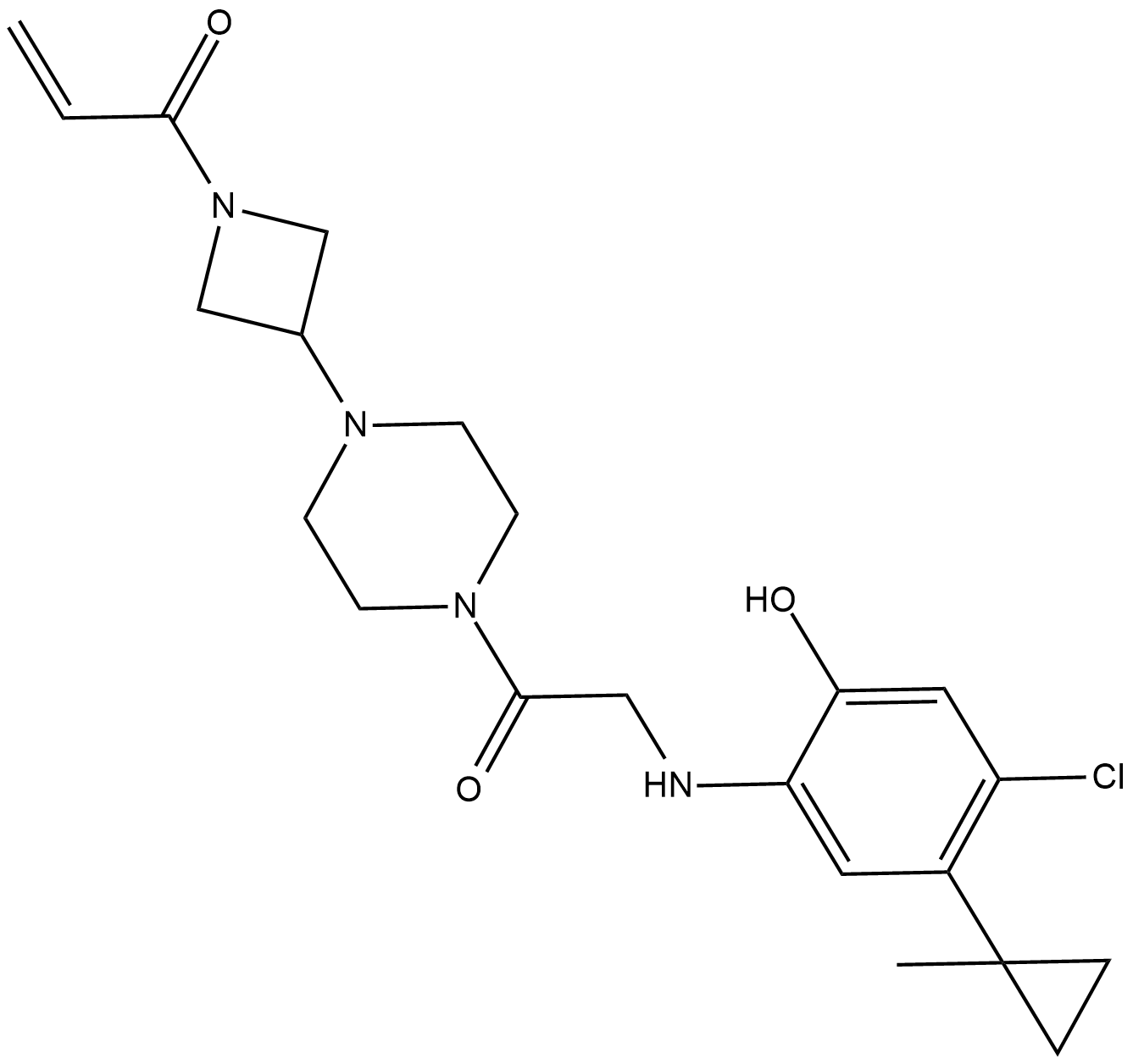
-
GC46882
Artemisinin-d3
An internal standard for the quantification of artemisinin
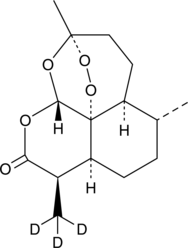
-
GC10040
Arylquin 1
Arylquin 1, un sécrétagogue de la réponse À l'apoptose de la prostate-4 (Par-4), cible la vimentine pour induire la sécrétion de Par-4. L'arylquine 1 induit la mort cellulaire non apoptotique dans les cellules cancéreuses par l'induction de la perméabilisation de la membrane lysosomale (LMP).
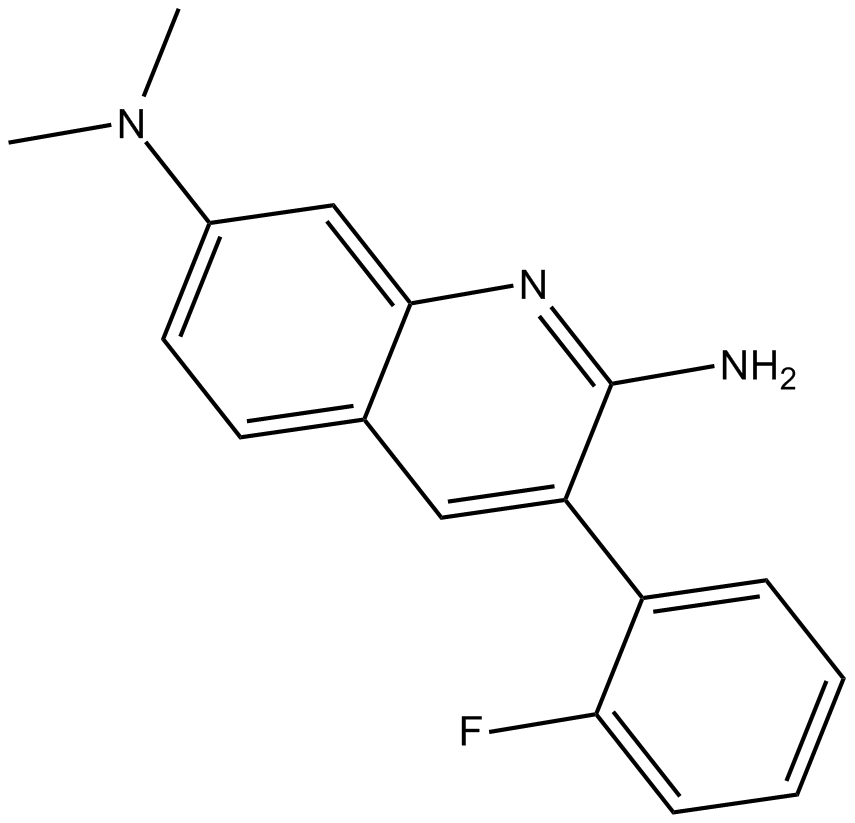
-
GC62615
AS-99
L'AS-99 est un inhibiteur puissant et sélectif de l'histone méthyltransférase ASH1L (IC50 = 0,79μ ; M, Kd = 0,89⋼ ; M) avec une activité anti-leucémique. L'AS-99 bloque la prolifération cellulaire, induit l'apoptose et la différenciation, régule À la baisse les gènes cibles de fusion MLL et réduit le fardeau de la leucémie in vivo.
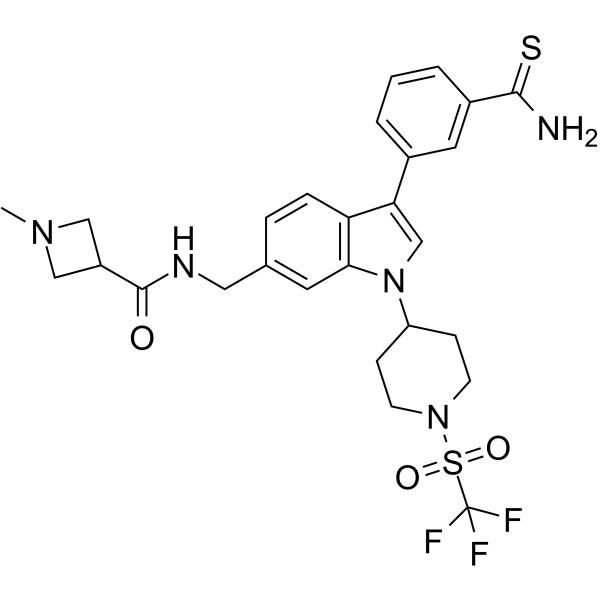
-
GC40715
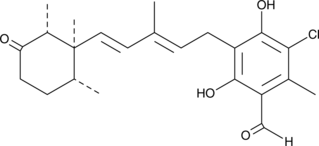
Ascochlorin
L'ascochlorine (ilicicoline D), un antibiotique isoprénoÏde, médie ses effets anti-tumoraux principalement par la suppression de la cascade de signalisation STAT3.
-
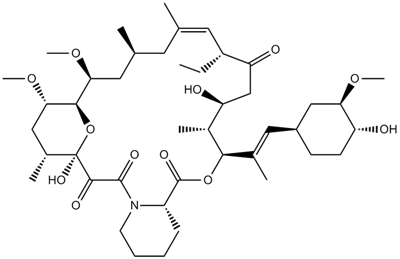
GC13215
Ascomycin(FK 520)
A potent macrolide immunosuppressant
-
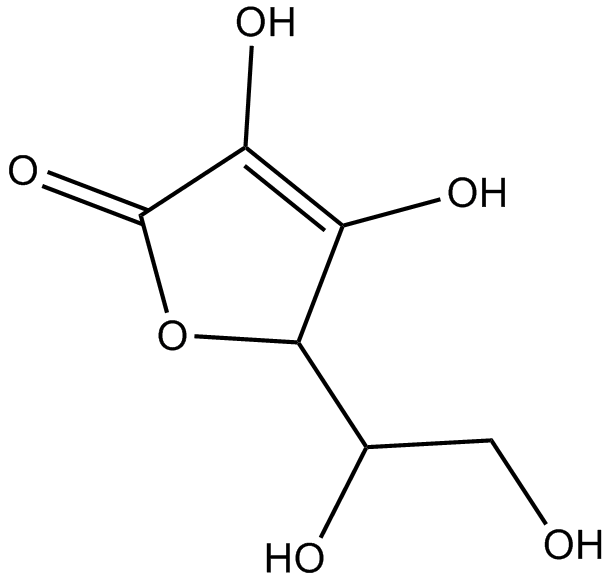
GC12070
Ascorbic acid
Un donneur d'électrons
-
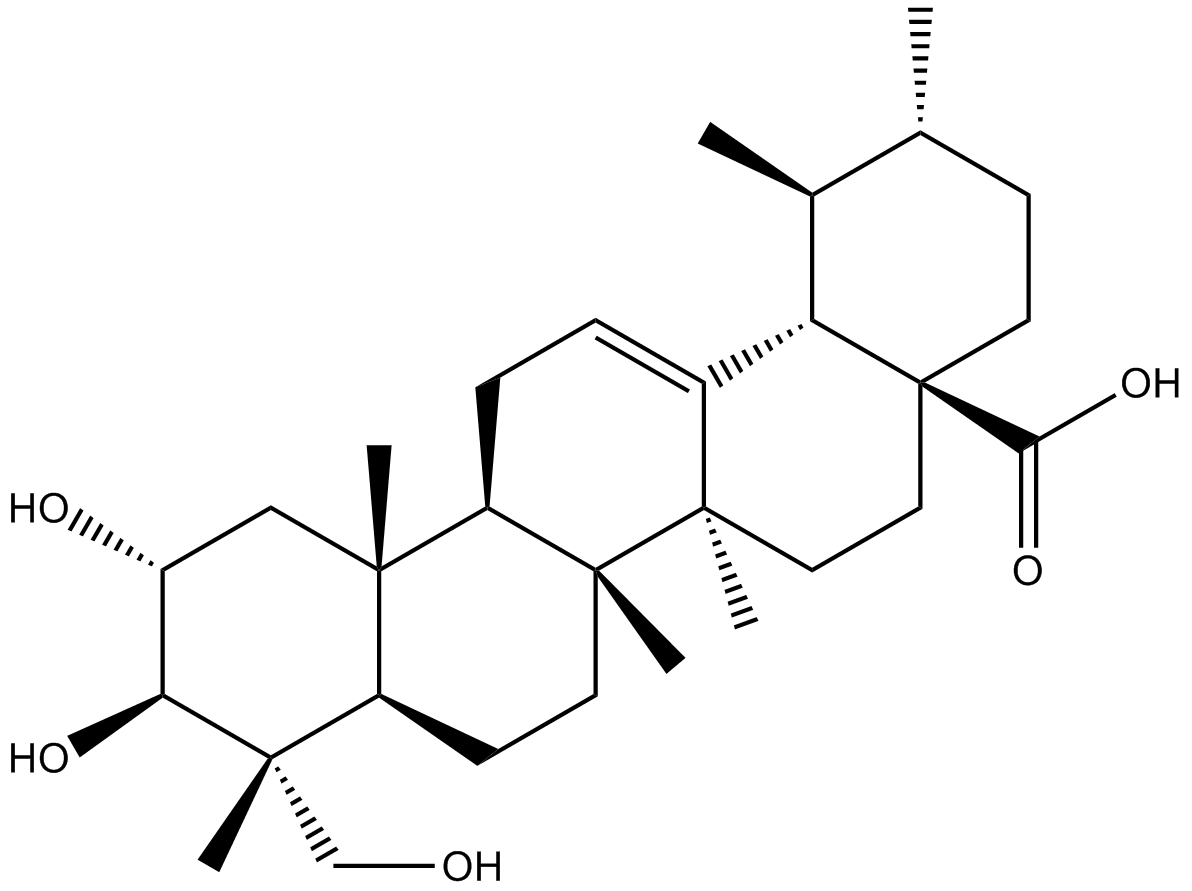
GN10702
Asiatic acid
-
GN10534
Asiaticoside
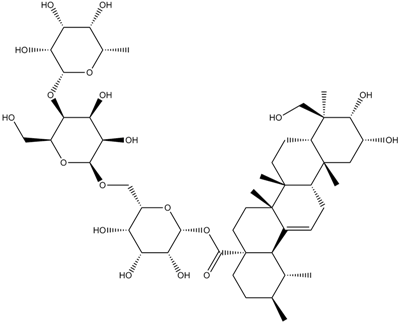
-
GC19041
ASK1-IN-1
ASK1-IN-1 est un puissant inhibiteur compétitif de l'ATP disponible par voie orale et sélectif de la kinase 1 régulatrice du signal d'apoptose (ASK1) avec une IC50 de 2,87 nM.
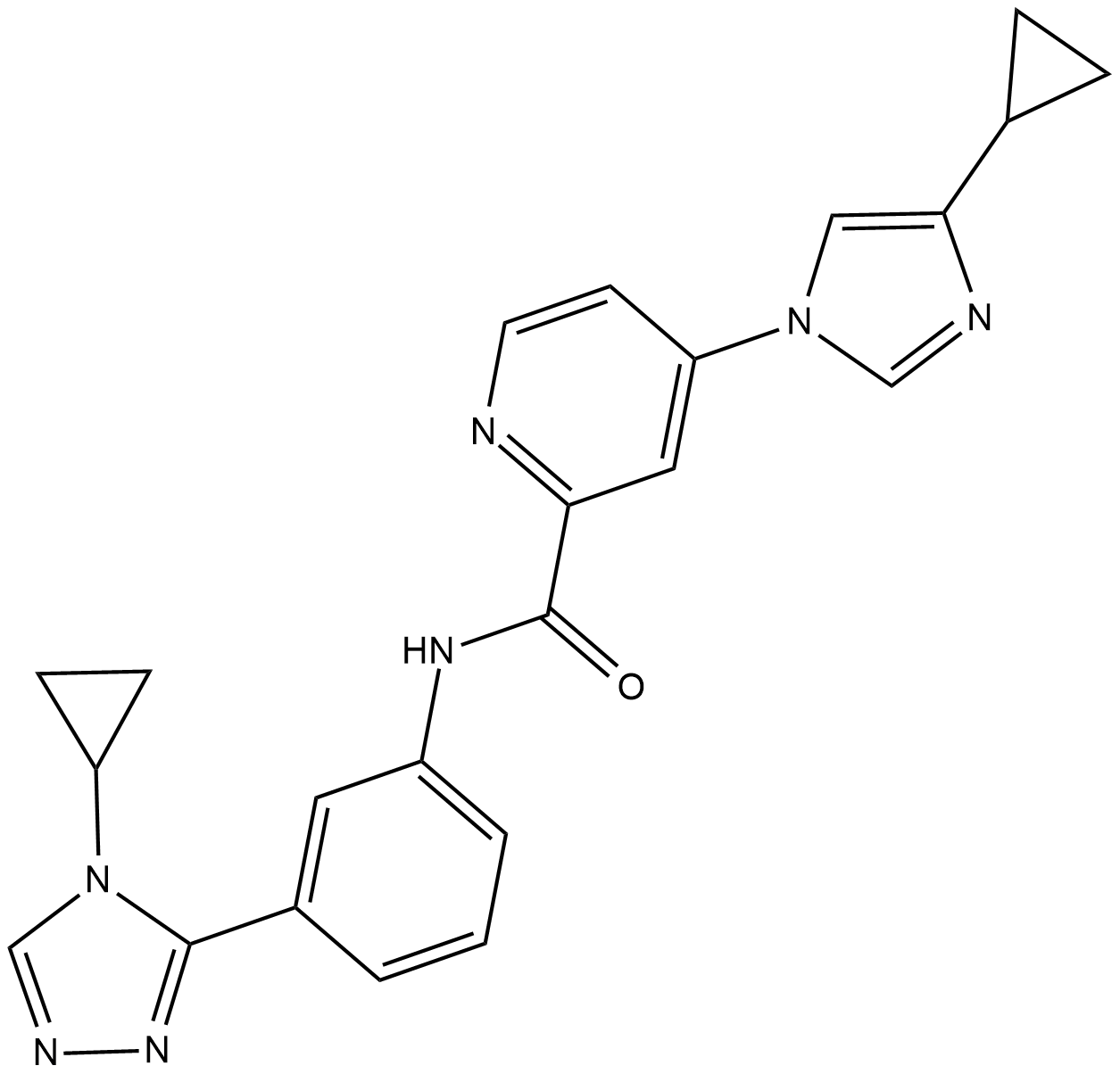
-
GC62426
ASK1-IN-2
ASK1-IN-2 est un inhibiteur puissant et actif par voie orale de la kinase 1 régulatrice du signal d'apoptose (ASK1), avec une IC50 de 32,8 nM.
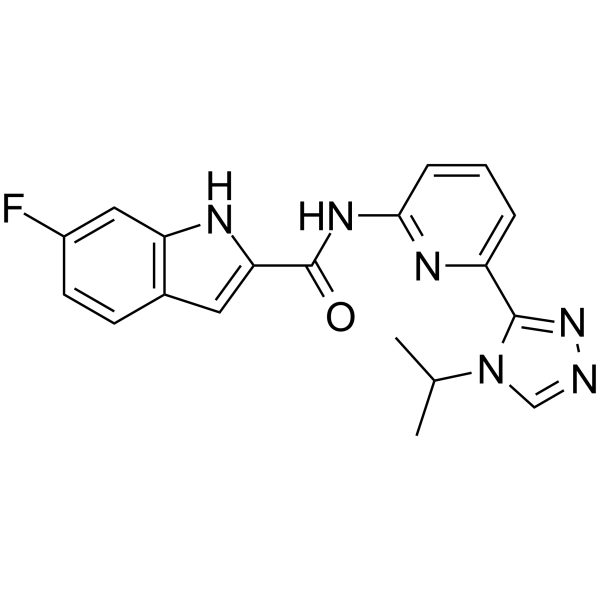
-
GC42858
Aspergillin PZ
L'aspergilline PZ est un nouvel isoindole-alcaloÏde d'Aspergillus awamori.
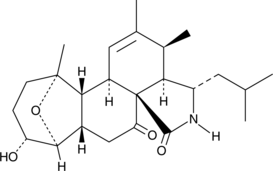
-
GN10064
Asperosaponin VI
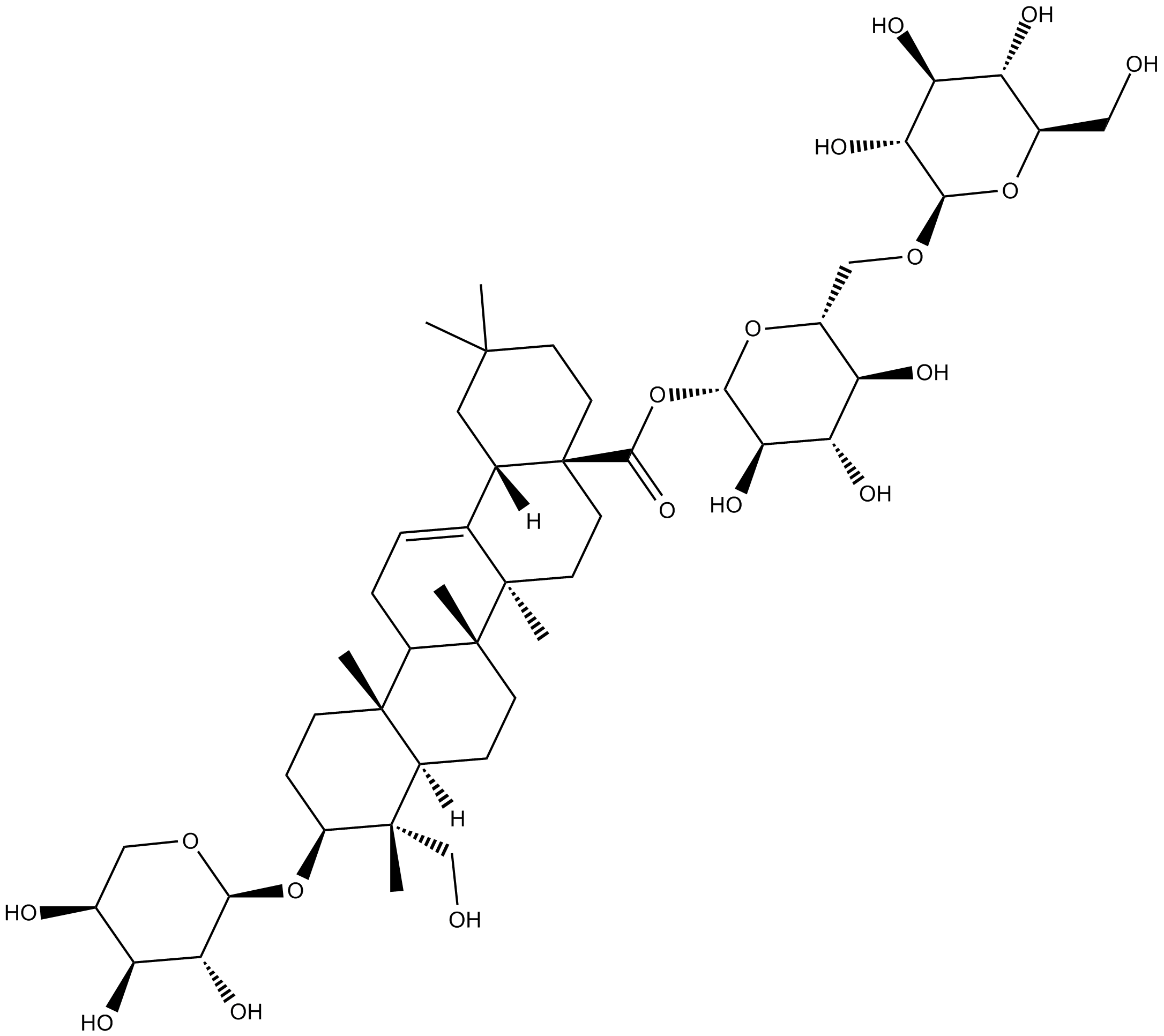
-
GC60603
Asperosaponin VI
A triterpenoid saponin with diverse biological activities
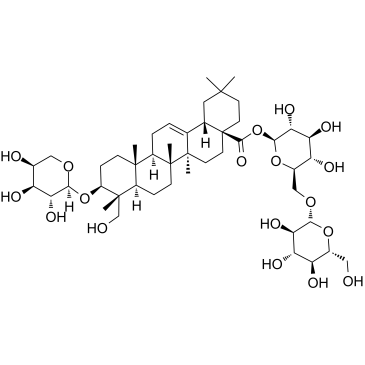
-
GC42860
Aspochalasin D
Aspochalasin D is a co-metabolite originally isolated from A.
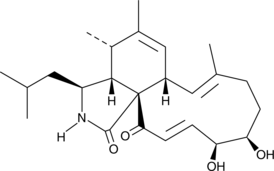
-
GC41640
Asterriquinol D dimethyl ether
L'éther diméthylique d'asterriquinol D est un métabolite fongique qui peut inhiber les lignées cellulaires de myélome de souris NS-1 avec une CI50 de 28 μg/mL.
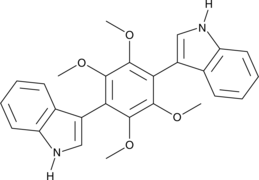
-
GN10415
Astilbin
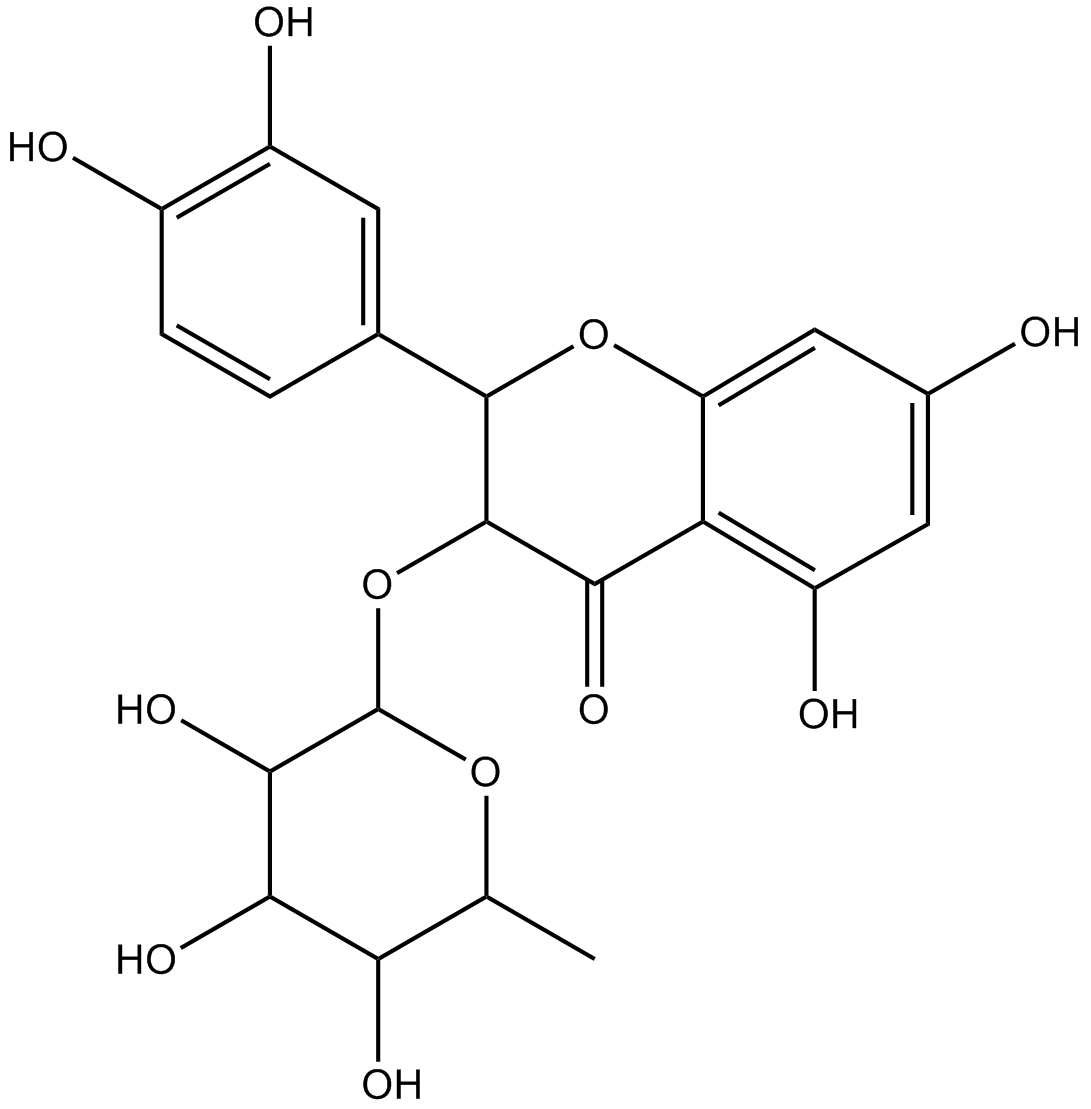
-
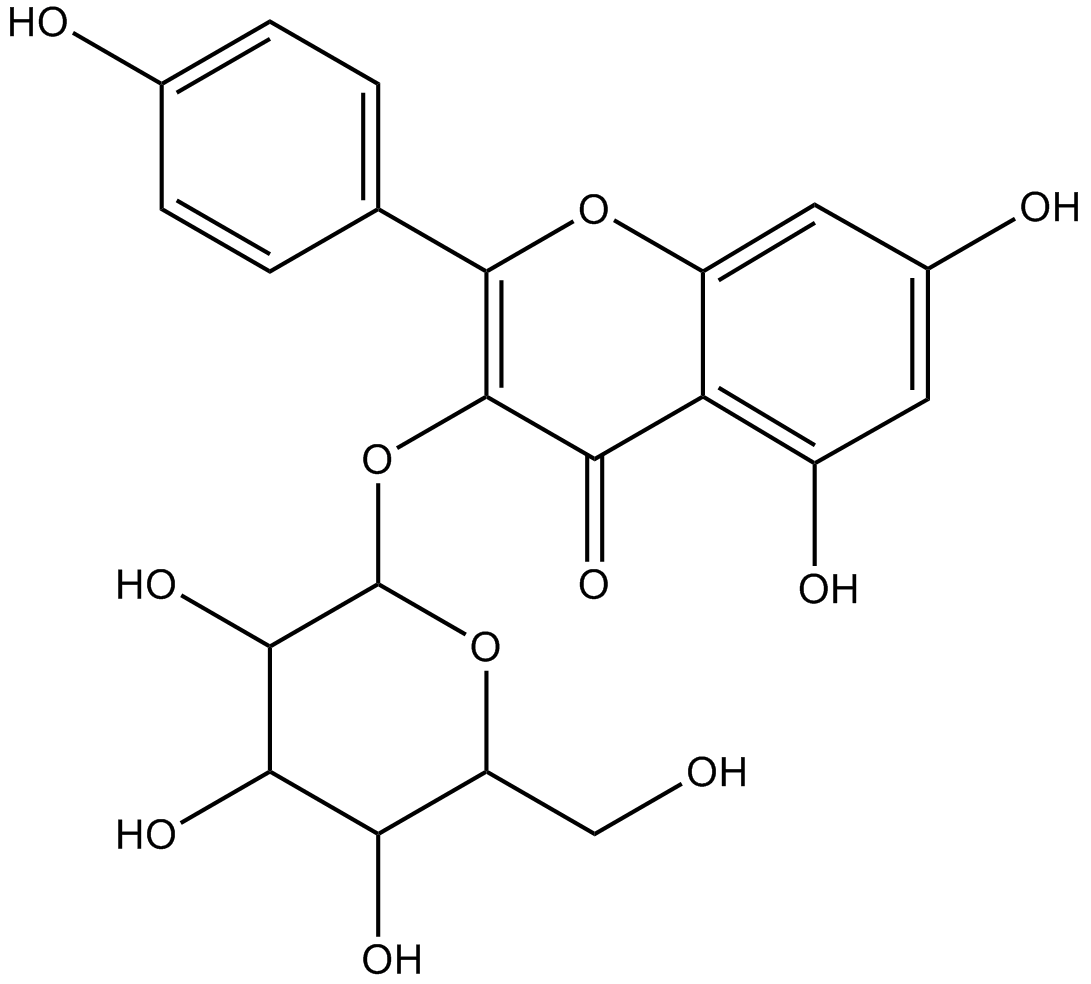
GN10561
astragalin
-
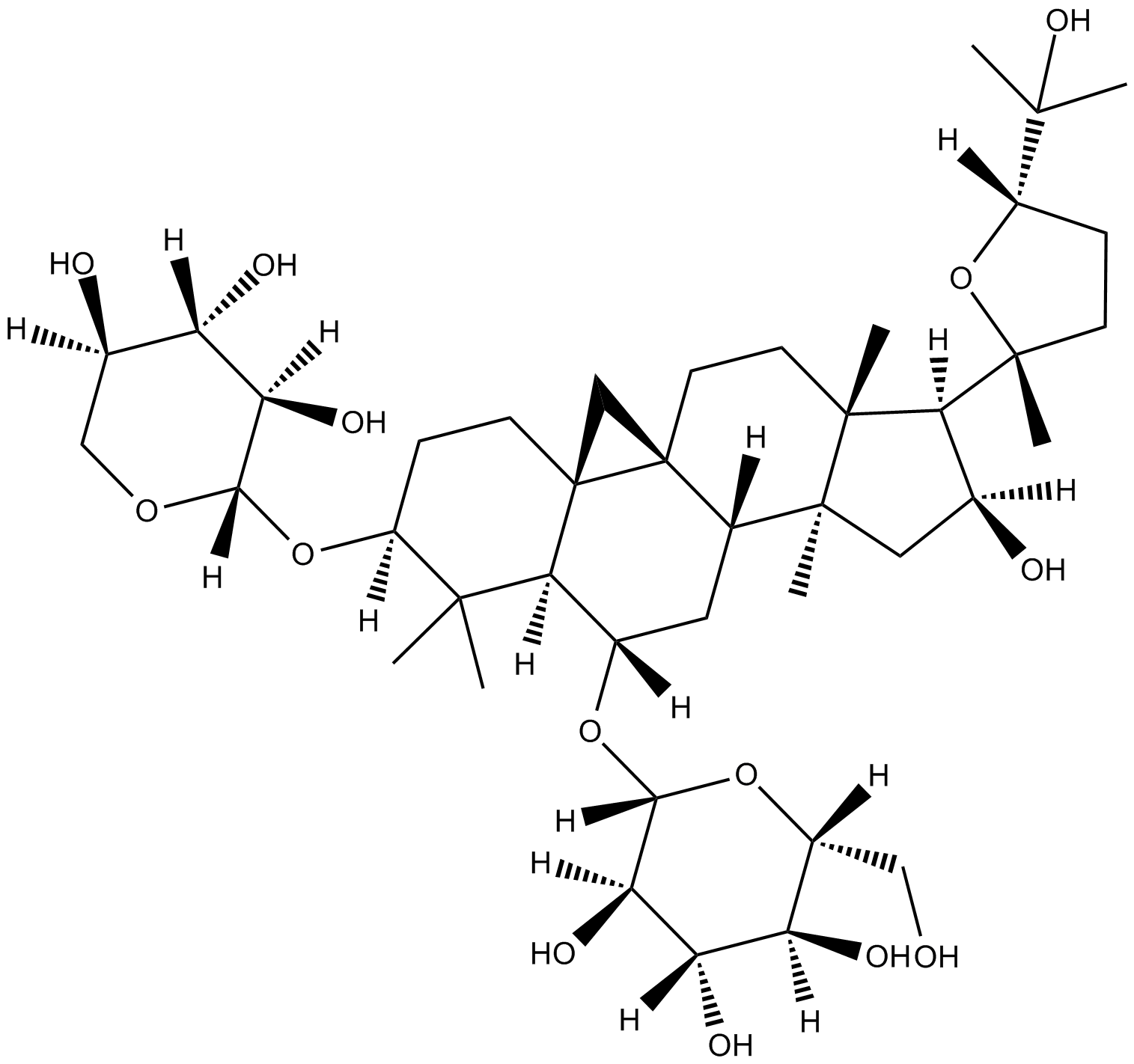
GC18109
Astragaloside A
anti-hypertension, positive inotropic action, anti-inflammation, and anti-myocardial injury
-
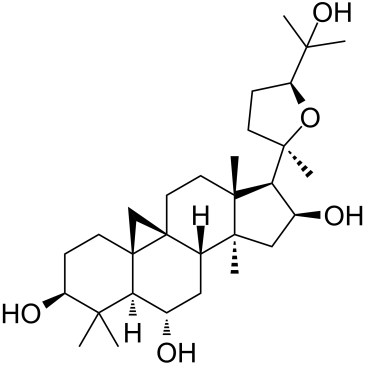
GC35415
Astramembrangenin
-
GC32803
ASTX660
ASTX660 est un double antagoniste biodisponible par voie orale de l'inhibiteur cellulaire de la protéine d'apoptose (cIAP) et de l'inhibiteur lié À l'X de la protéine d'apoptose (XIAP).
-
GC42863
Asukamycin
Asukamycin is polyketide isolated from the S.
-
GC11106
AT-101