DNA Damage/DNA Repair
- MTH1(4)
- PARP(58)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(94)
- Nucleoside Antimetabolite/Analogue(136)
- Telomerase(14)
- Topoisomerase(133)
- tankyrase(5)
- Antifolate(32)
- CDK(233)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(62)
- DNA Alkylator/Crosslinker(64)
- DNA/RNA Synthesis(388)
- Eukaryotic Initiation Factor (eIF)(22)
- IRE1(22)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(53)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nombre del producto Información
-
GC31950
Halofuginone hydrobromide (RU-19110 (hydrobromide))
El bromhidrato de halofuginona (RU-19110), un derivado de la febrifugina, es un inhibidor competitivo de la prolil-tRNA sintetasa con una Ki de 18,3 nM.
-
GC62328
HBV-IN-4
HBV-IN-4, un derivado de la ftalazinona, es un inhibidor de la replicaciÓn del ADN del VHB potente y activo por vÍa oral con una IC50 de 14 nM.
-
GC36211
HBX 19818
HBX 19818 es un inhibidor especÍfico de la proteasa 7 especÍfica de ubiquitina (USP7), con una IC50 de 28,1 μM.
-
GC19190
HDAC-IN-4
AZD 9468
HDAC-IN-4 es un inhibidor de HDAC de clase I potente, selectivo y activo por vÍa oral con IC50 de 63 nM, 570 nM y 550 nM para HDAC1, HDAC2 y HDAC3, respectivamente. HDAC-IN-4 no tiene actividad contra HDAC clase II. HDAC-IN-4 tiene actividad antitumoral.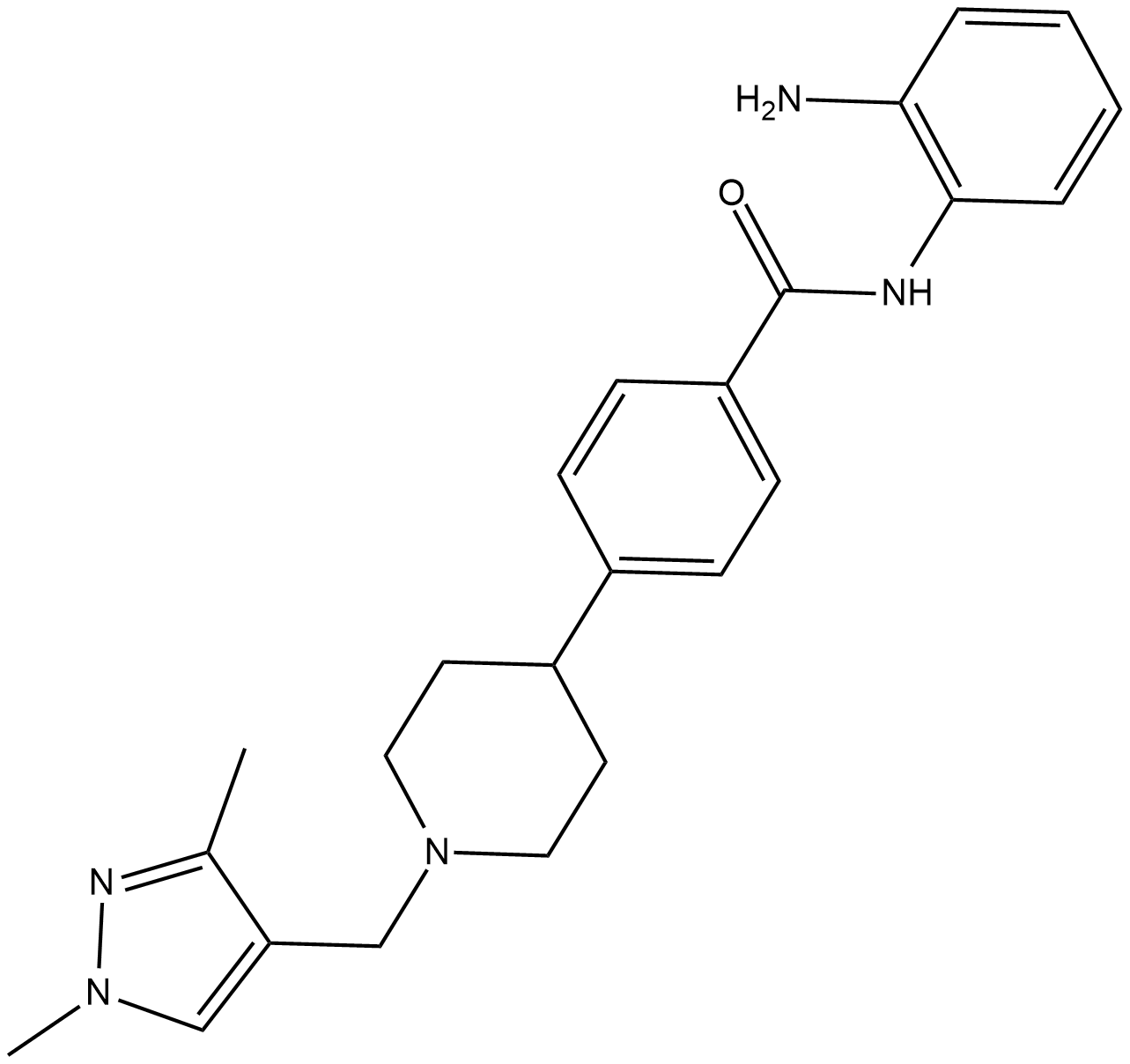
-
GC66052
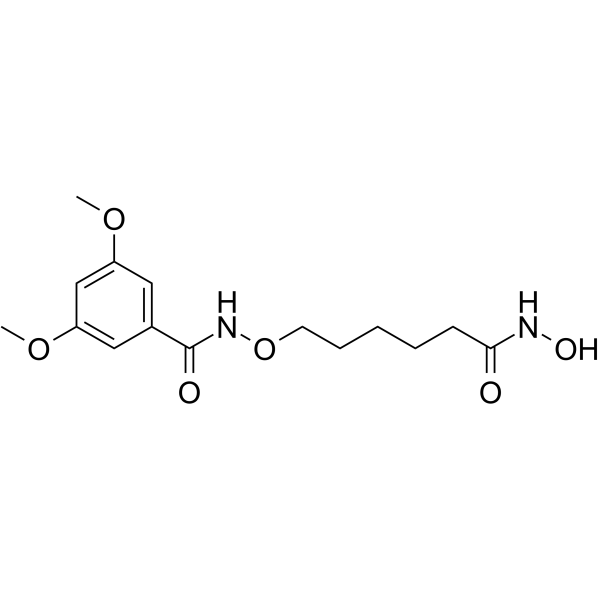
HDAC-IN-40
HDAC-IN-40 es un potente inhibidor de HDAC basado en alcoxiamida con valores Ki de 60 nM y 30 nM para HDAC2 y HDAC6, respectivamente. HDAC-IN-40 tuvo efectos antitumorales.
-
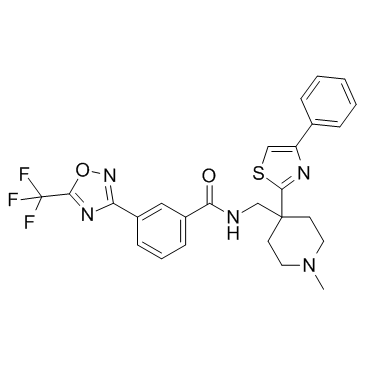
GC33395
HDAC-IN-5
HDAC-IN-5 es un inhibidor de la histona desacetilasa (HDAC).
-
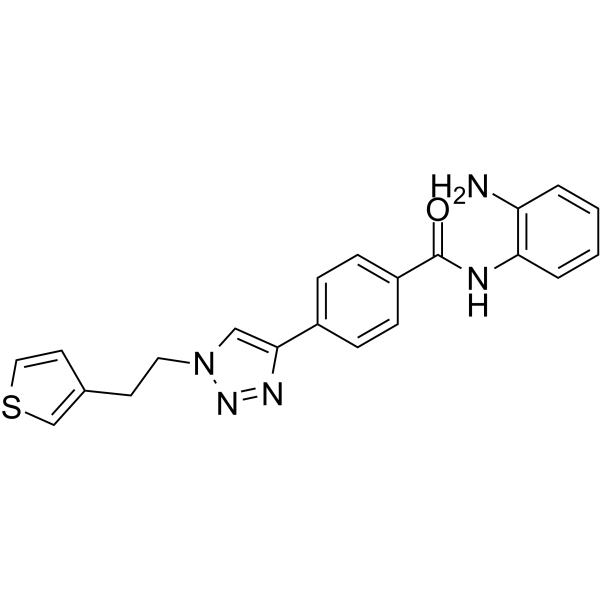
GC68010
HDAC3-IN-T247
-
GC41085
HDAC6 Inhibitor
Histone Deacetylase 6
HDAC6 Inhibitor es un potente y selectivo inhibidor de HDAC6 (IC50=36 nM). El inhibidor de HDAC6 inhibe débilmente otras isoformas de HDAC. El inhibidor de HDAC6 inhibe la acumulaciÓn de acil-tubulina en las células con un valor IC50 de 210 nM.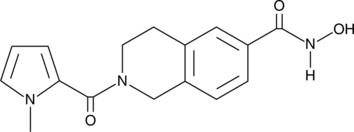
-
GC33317
HDAC6-IN-1
HDAC6-IN-1 es un inhibidor potente y selectivo de HDAC6 con una IC50 de 17 nM y muestra una selectividad de 25 y 200 veces en relaciÓn con HDAC1 (IC50=422 nM) y HDAC8 (IC50=3398 nM), respectivamente.
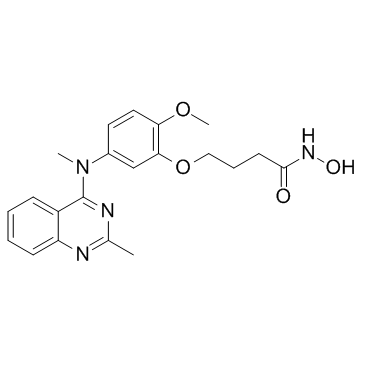
-
GC19189
HDAC8-IN-1
MDK-7933
HDAC8-IN-1 es un inhibidor de HDAC8 con un IC50 de 27,2 nM.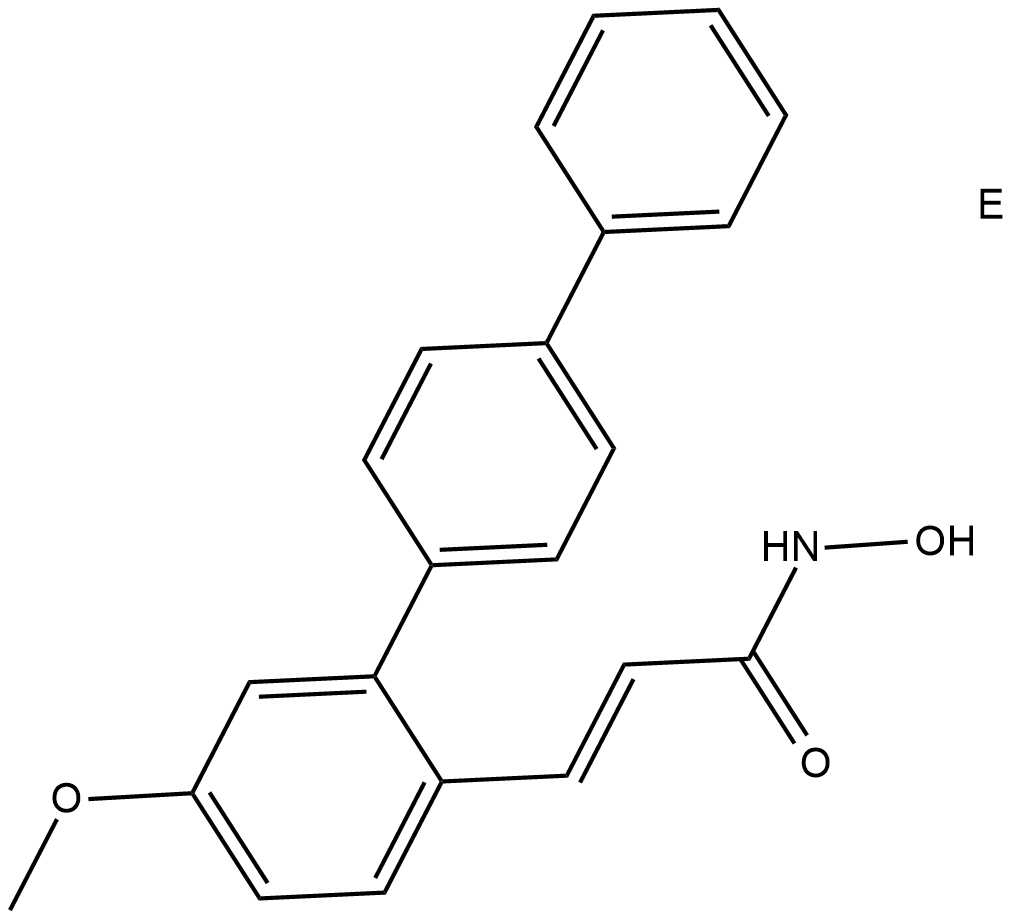
-
GC65460
HDACs/mTOR Inhibitor 1
HDACs/mTOR Inhibitor 1 es un inhibidor dual de histona desacetilasas (HDAC) y diana de rapamicina en mamÍferos (mTOR) para el tratamiento de neoplasias malignas hematolÓgicas, con IC50 de 0,19 nM, 1,8 nM, 1,2 nM y >500 nM para HDAC1, HDAC6, mTOR y PI3Kα, respectivamente. HDACs/mTOR Inhibitor 1 estimula la detenciÓn del ciclo celular en la fase G0/G1 e induce la apoptosis de las células tumorales con baja toxicidad in vivo.
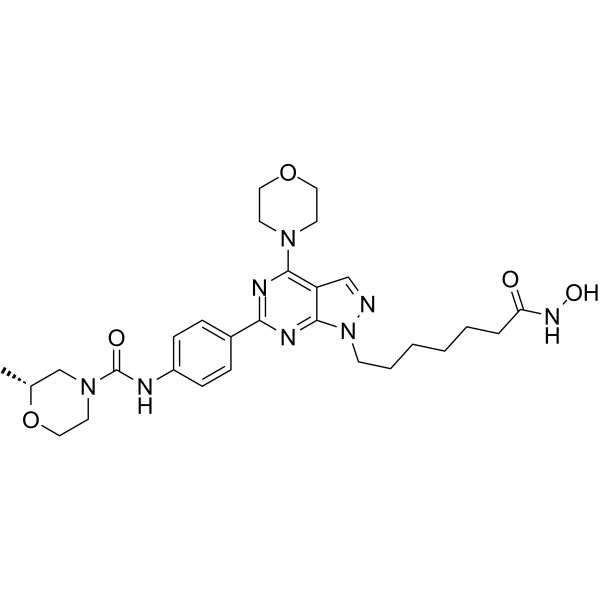
-
GC39279
hDHODH-IN-1
hDHODH-IN-1 es un inhibidor de la dihidroorotato deshidrogenasa humana (hDHODH).
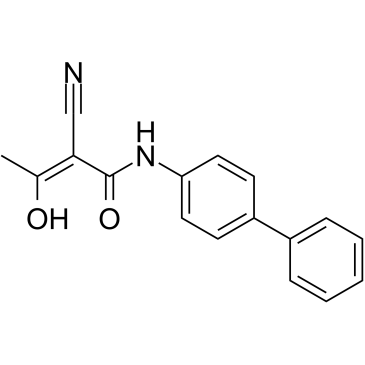
-
GC48388
Heliquinomycin
NSC 702208, Rubymycin
A bacterial metabolite with diverse biological activities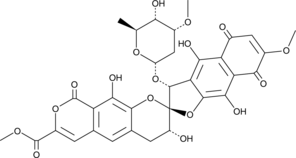
-
GC39266
Hematein
La hemateÍna es un producto de oxidaciÓn de la hematoxilina que actÚa como colorante. La hemateÍna es un inhibidor alostérico de la caseÍna cinasa II con una IC50 de 0,74 μM. La hemateÍna inhibe la fosforilaciÓn de Akt/PKB Ser129, la vÍa Wnt/TCF y aumenta la apoptosis en las células de cÁncer de pulmÓn.
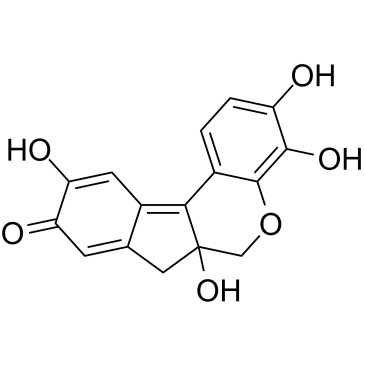
-
GC40103
Herboxidiene
GEX1A, Tan 1609
Herboxidieno, como un agente antitumoral potente, puede dirigirse a la subunidad SF3B del espliceosoma. Herboxidieno también induce arresto del ciclo celular en G1 y G2/M en una línea celular de fibroblastos humanos normales WI-38.
-
GC12201
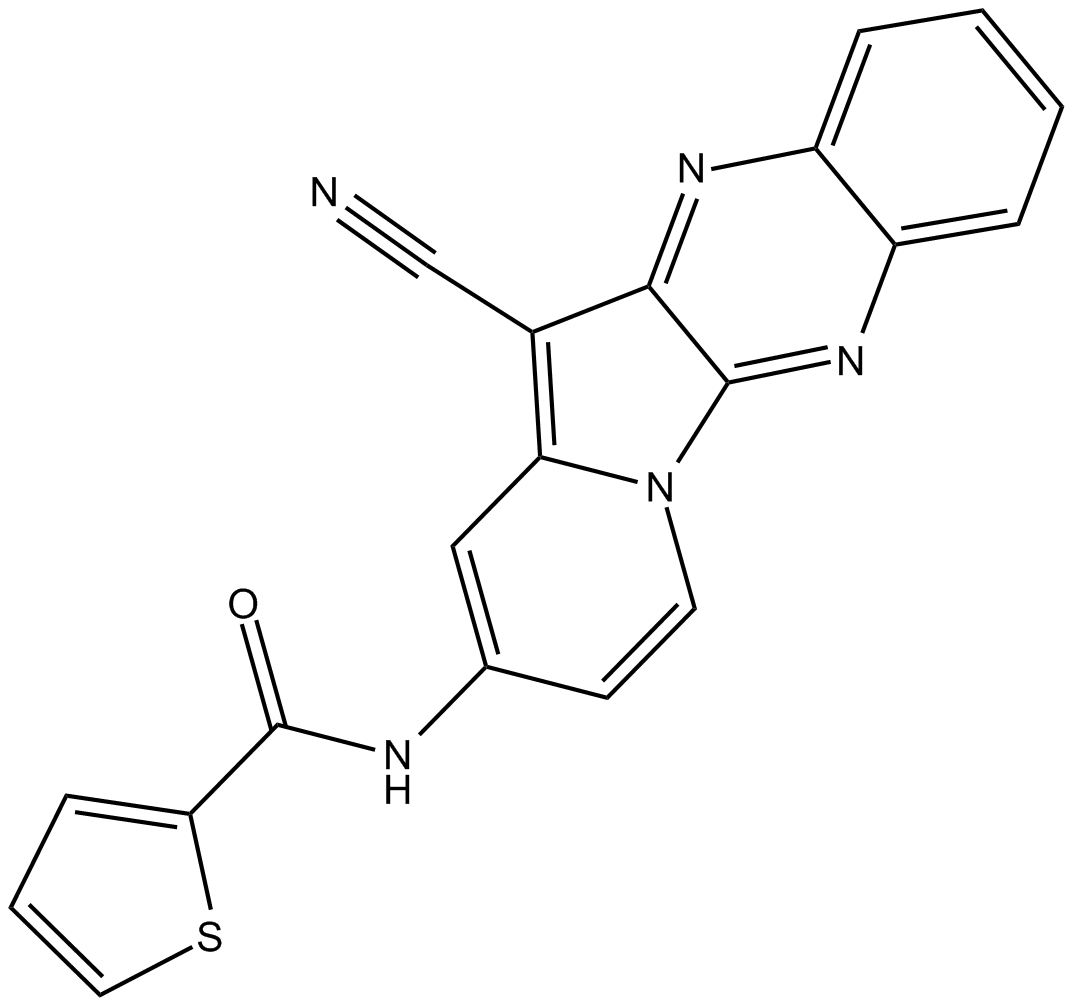
HI TOPK 032
HI TOPK 032 es un potente y específico inhibidor de TOPK.
-
GC11302
Hinokitiol
NSC 18804, β-Thujaplicin, 4-isopropyl Tropolone
El hinokitiol es un componente de los aceites esenciales aislado de Chymacyparis obtusa, reduce la expresiÓn de Nrf2 y disminuye la expresiÓn de proteÍnas y ARNm de DNMT1 y UHRF1, con actividades antiinfecciosas, antioxidantes y antitumorales.
-
GC12334
HNHA
Histone Deacetylase Inhibitor VI
HNHA es un potente inhibidor de la histona desacetilasa (HDAC). HNHA detiene el ciclo celular en la fase G1/S a través de la inducciÓn de p21. HNHA inhibe el crecimiento tumoral y la neovascularizaciÓn tumoral. HNHA puede ser un potente agente anticancerÍgeno contra el cÁncer de mama.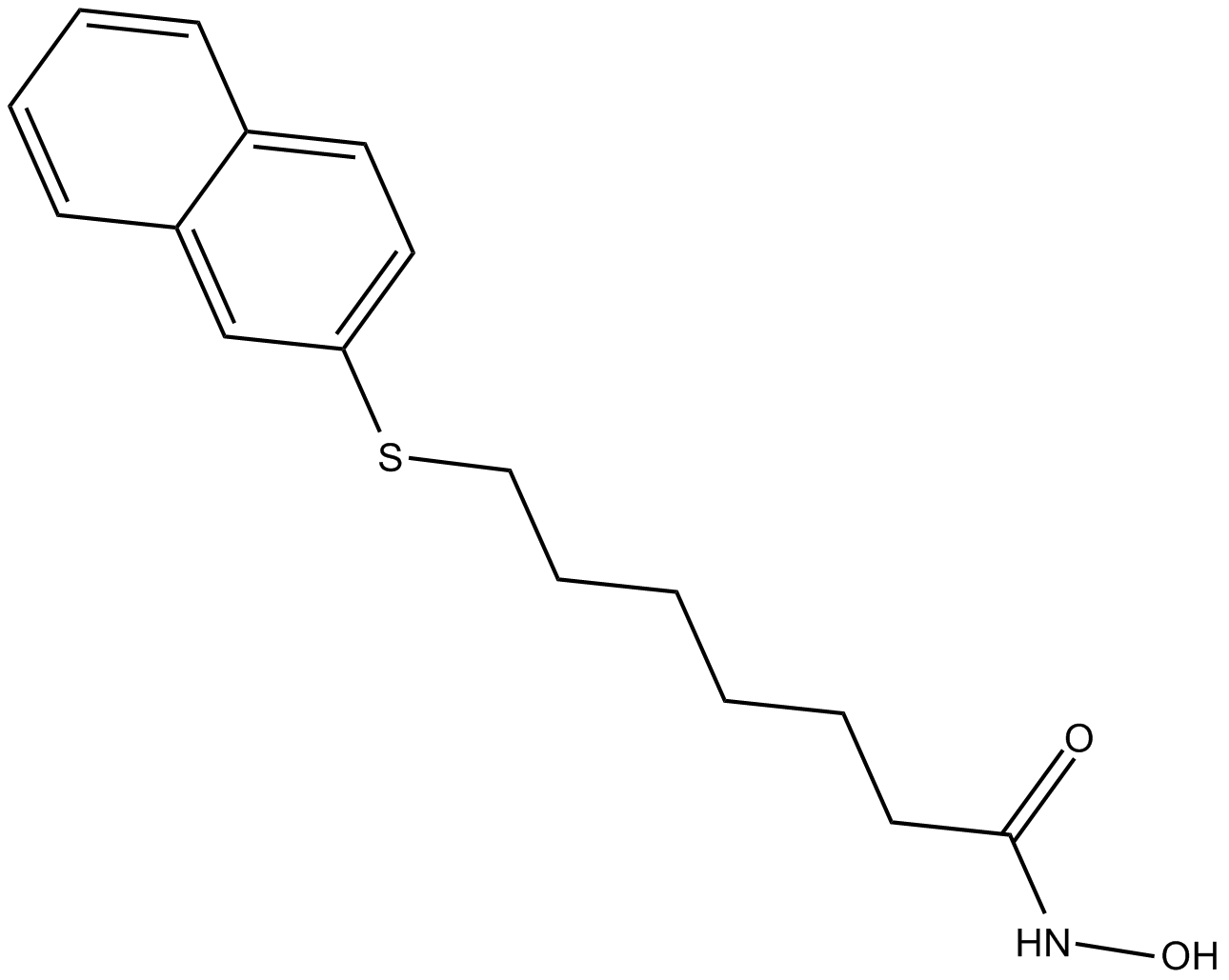
-
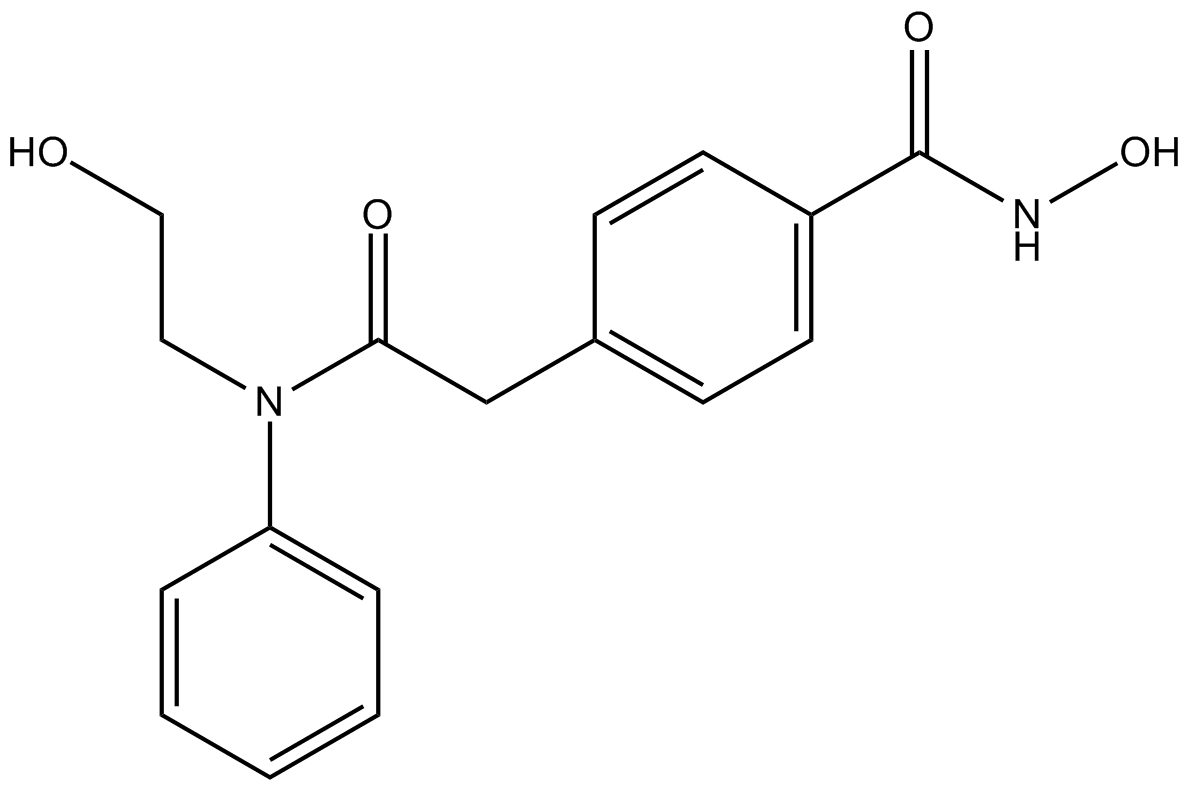
GC11574
HPOB
HPOB es un inhibidor altamente potente y selectivo de HDAC6 con un IC50 de 56 nM. HPOB muestra >30 veces menos potente que otros HDAC. HPOB mejora la eficacia de los agentes anticancerÍgenos que daÑan el ADN en las células transformadas, pero no en las células normales. HPOB no bloquea la actividad de uniÓn a ubiquitina de HDAC6.
-
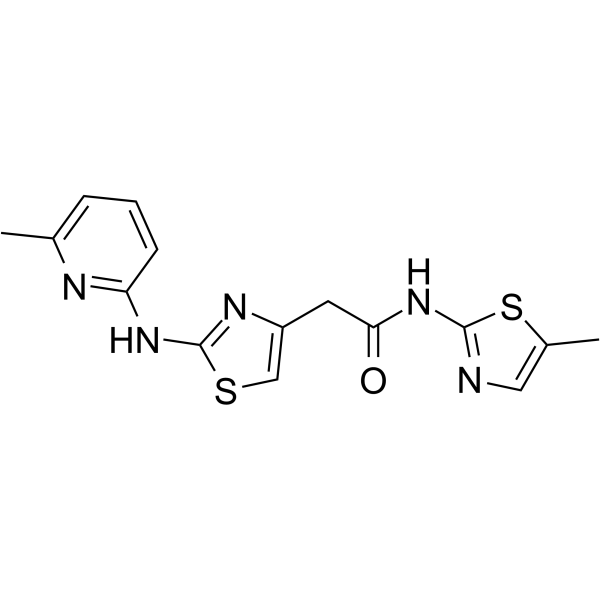
GC63854
HQ461
HQ461 es un pegamento molecular que promueve la interacciÓn CDK12-DDB1 para desencadenar la degradaciÓn de ciclina K. La degradaciÓn de la ciclina K mediada por HQ461 altera la funciÓn de CDK12, lo que resulta en una disminuciÓn de la fosforilaciÓn del sustrato de CDK12, regulaciÓn a la baja de los genes de respuesta al daÑo del ADN y muerte celular.
-
GC62572
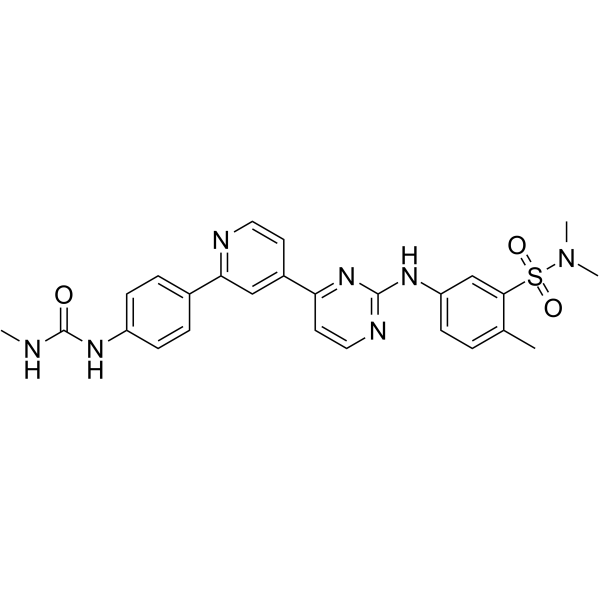
hSMG-1 inhibitor 11e
El inhibidor 11e de hSMG-1 es un inhibidor potente y selectivo de la quinasa hSMG-1 con una IC50 de 900 veces mÁs selectiva que mTOR (IC50 de 45 nM), PI3Kα/γ (IC50s de 61 nM y 92 nM) y CDK1/CDK2 (IC50s de 32 μM y 7,1 μM).
-
GC61925
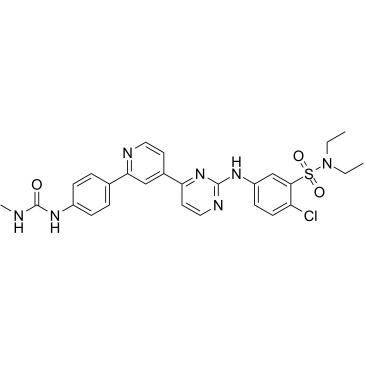
hSMG-1 inhibitor 11j
El inhibidor 11j de hSMG-1, un derivado de la pirimidina, es un inhibidor potente y selectivo de hSMG-1, con una IC50 de 0,11 nM. El inhibidor de hSMG-1 11j muestra una selectividad de >455 veces para hSMG-1 sobre mTOR (IC50 = 50 nM), PI3Kα/γ (IC50 = 92/60 nM) y CDK1/CDK2 (IC50 = 32/7,1 μM). El inhibidor de hSMG-1 11j se puede utilizar para la investigaciÓn del cÁncer.
-
GC16843
Hydroxyurea
NCI C04831, NSC 32065
La hidroxiurea es un inductor de la apoptosis celular que inhibe la sÍntesis de ADN mediante la inhibiciÓn de la ribonucleÓtido reductasa. La hidroxiurea muestra actividad anti-ortopoxvirus.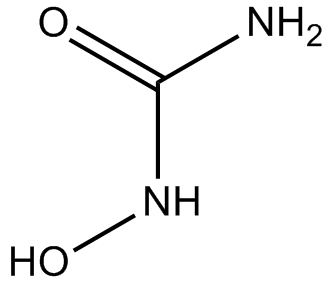
-
GC49694
Hygromycin A
(-)-Hygromycin A, Totomycin
An antibiotic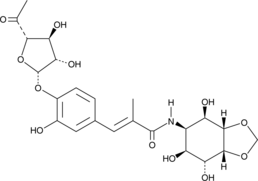
-
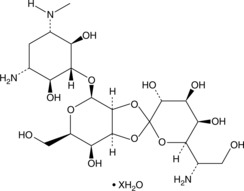
GC48445
Hygromycin B (hydrate)
An aminoglycoside antibiotic
-
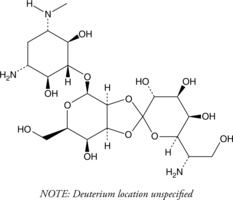
GC49267
Hygromycin B-d4
An internal standard for the quantification of hygromycin
-
GC14503
IC261
SU5607
IC261 es un inhibidor selectivo de CK1 competitivo con ATP, con IC50 de 1 μM, 1 μM, 16 μM para Ckiδ, Ckiε y Ckiα1, respectivamente.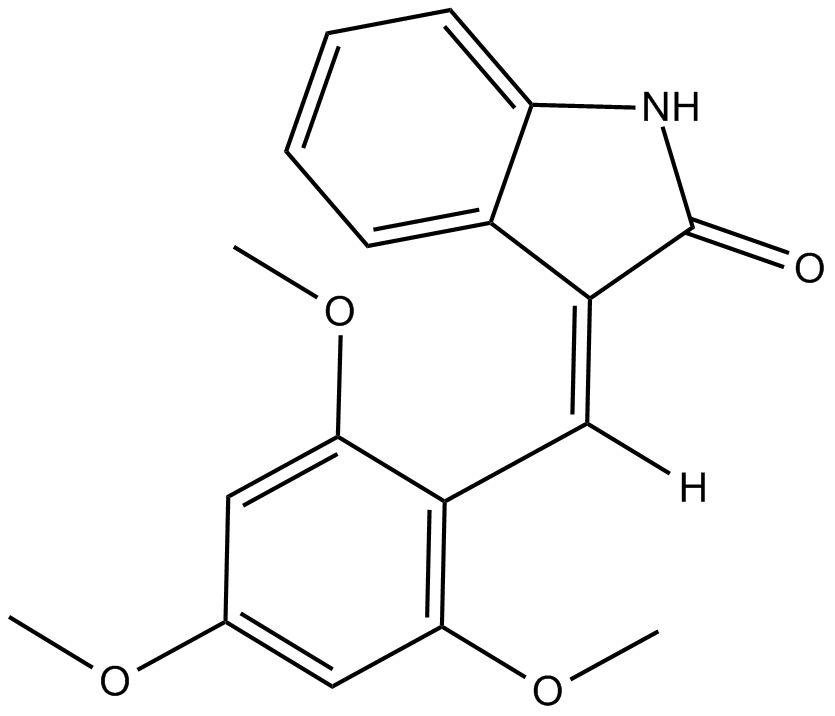
-
GC14969
Idarubicin HCl
4Demethoxydaunorubicin, 4DMD, NSC 256439
La idarubicina HCl es un fÁrmaco antileucémico de antraciclina.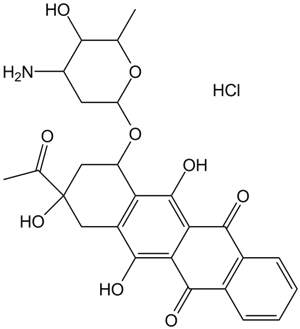
-
GC16003
Ifosfamide
Isophosphamide, NSC 109724
La ifosfamida es un agente quimioterapéutico alquilante con actividad contra una amplia gama de tumores.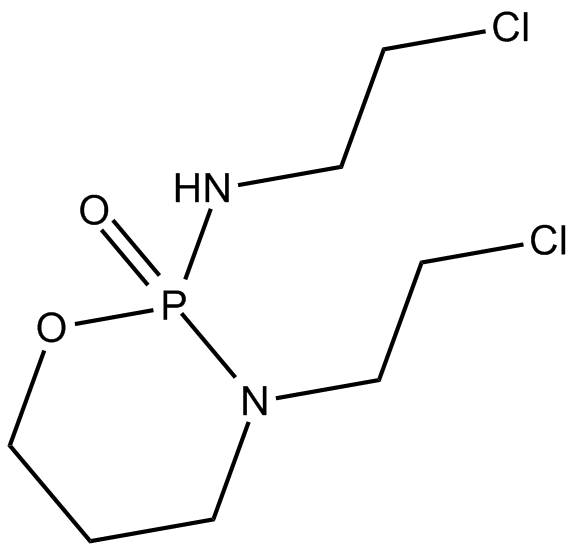
-
GC60929
Ilaprazole sodium
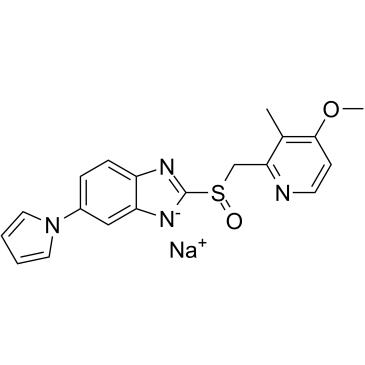
El ilaprazol (IY-81149) sÓdico es un inhibidor de la bomba de protones activo por vÍa oral.
-
GC41340
Illudin M
DR-15977, (-)-Illudin M, NSC 400978, NSC 626370
Illudin M es un sesquiterpeno fÚngico citotÓxico que se puede aislar del medio de cultivo de hongos Omphalotus olearius. Illudin M puede alquilar ADN. Illudin M tiene actividades antitumorales.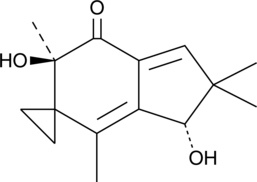
-
GC18100
Illudin S
Lampterol,NSC 400979,NSC 626369
Illudin S, un Illudin citotÓxico, es un sesquiterpeno natural con fuertes actividades antitumorales y antivirales.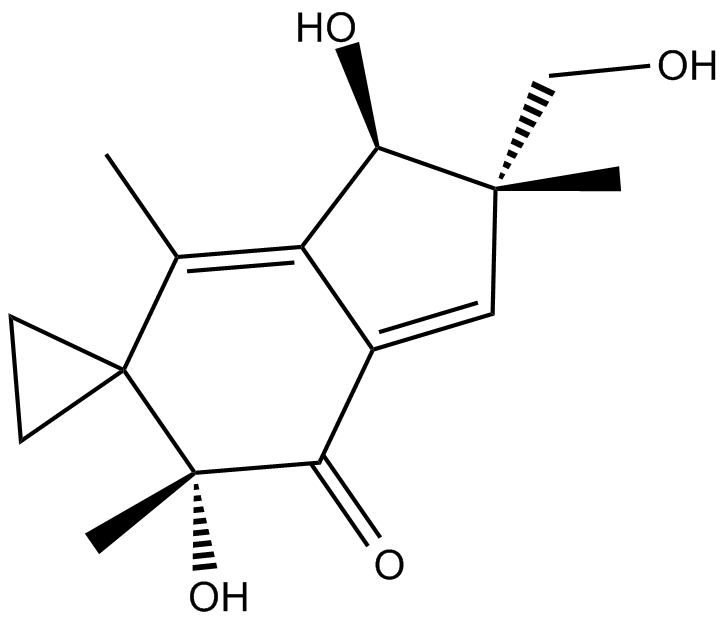
-
GC62137
IMT1
IMT1 es el primer inhibidor de la polimerasa de ARN mitocondrial humano (POLRMT) especÍfico y no competitivo de su clase.
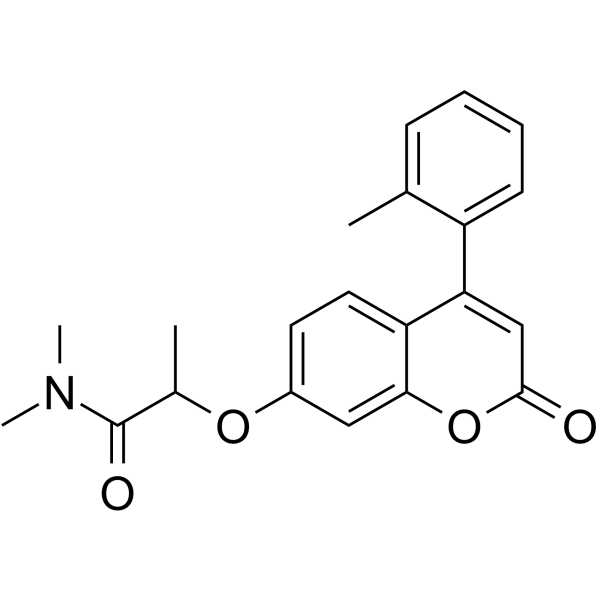
-
GC65939
Indimitecan
LMP776
El indimitecÁn (LMP776) es un inhibidor de la topoisomerasa I (Top1) con actividades anticancerÍgenas.
-
GC36312
Indirubin-3'-monoxime-5-sulphonic acid
El Ácido indirrubin-3'-monoxima-5-sulfÓnico es un inhibidor potente y selectivo de CDK1, CDK5 y GSK-3β con IC50 de 5 nM, 7 nM y 80 nM, respectivamente.
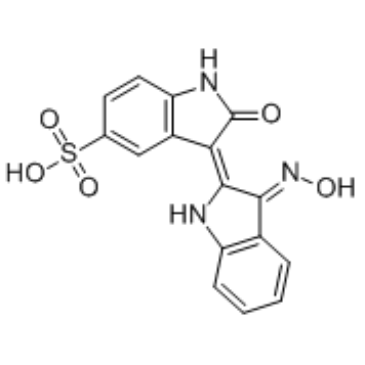
-
GC36313
Indirubin-5-sulfonate
El indirrubin-5-sulfonate es un inhibidor de la cinasa dependiente de ciclina (CDK), con valores IC50 de 55 nM, 35 nM, 150 nM, 300 nM y 65 nM para CDK1/ciclina B, CDK2/ciclina A, CDK2/ciclina E, CDK4/ciclina D1 y CDK5/p35, respectivamente. El indirrubin-5-sulfonate también muestra actividad inhibitoria contra GSK-3β.
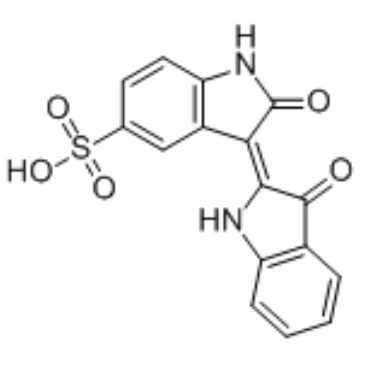
-
GC52513
Indolimine-214
Isoamylindolimine
A genotoxic gut microbiota metabolite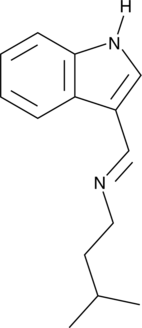
-
GC33205
Indotecan (LMP-400)
LMP-400; NSC-724998
Indotecan (LMP-400) (LMP-400) es un potente inhibidor de la topoisomerasa 1 (Top1) con valores IC50 de 300, 1200, 560 nM para lÍneas celulares P388, HCT116, MCF-7, respectivamente.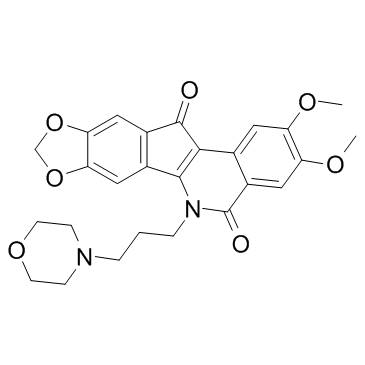
-
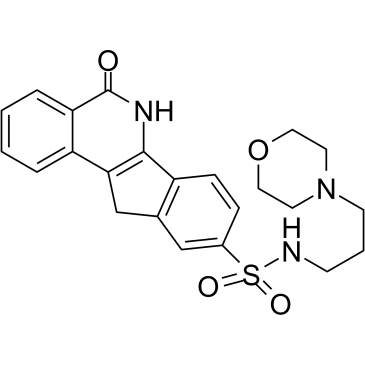
GC38385
INO-1001
-
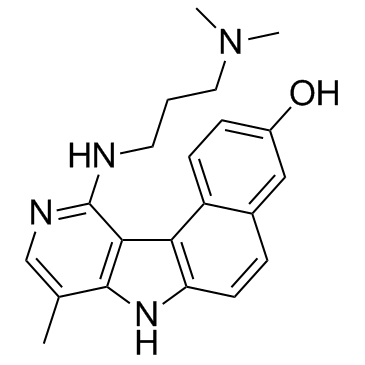
GC33165
Intoplicine
Intoplicine (RP 60475), un derivado antitumoral de la serie 7H-benzo[e]pirido[4,3-b]indol, es un inhibidor de la topoisomerasa I y II del ADN. Intoplicine se une fuertemente al ADN (KA = 2 x 105/M) y por lo tanto aumenta la longitud del ADN lineal.
-
GC63354
Intoplicine dimesylate
RP 60475 dimesylate
El dimesilato de intoplicina (RP 60475), un derivado antitumoral de la serie 7H-benzo[e]pirido[4,3-b]indol, es un inhibidor de la topoisomerasa I y II del ADN. El dimesilato de intoplicina se une fuertemente al ADN (KA = 2 x 105/M) y, por lo tanto, aumenta la longitud del ADN lineal.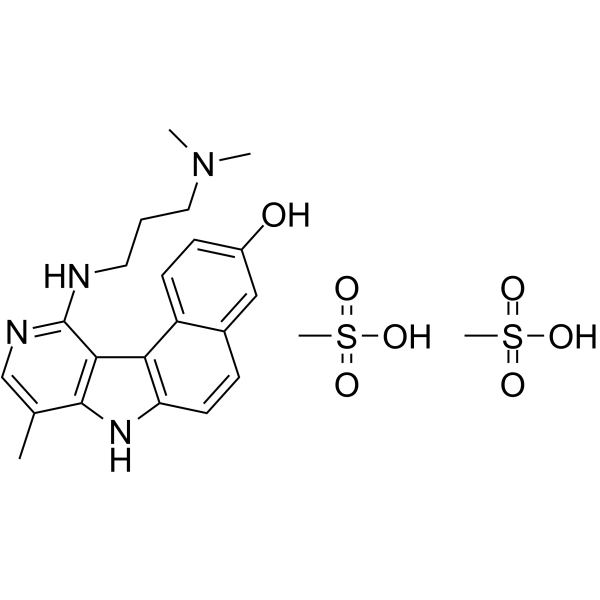
-
GC63023
Ipivivint
Ipivivint (compuesto 38) es un potente inhibidor de la quinasa similar a CDC (CLK) con EC50 de 1 nM, 7 nM para CLK2 y CLK3, respectivamente. Ipivivint inhibe la vÍa Wnt (EC50=13 nM).
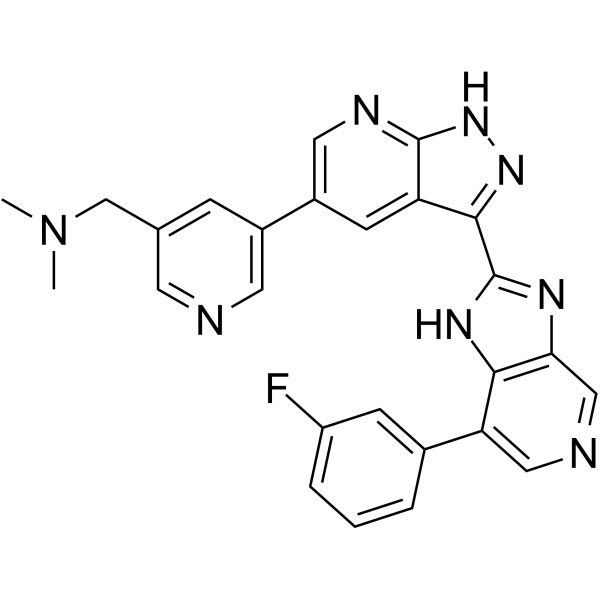
-
GC52175
IQA
CGP 029482
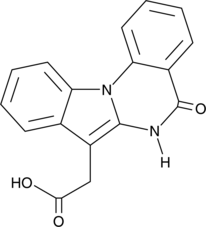
-
GC63513
IRE1α kinase-IN-1
IRE1α kinase-IN-1 es un IRE1α altamente selectivo; (ERN1), con un IC50 de 77 nM.
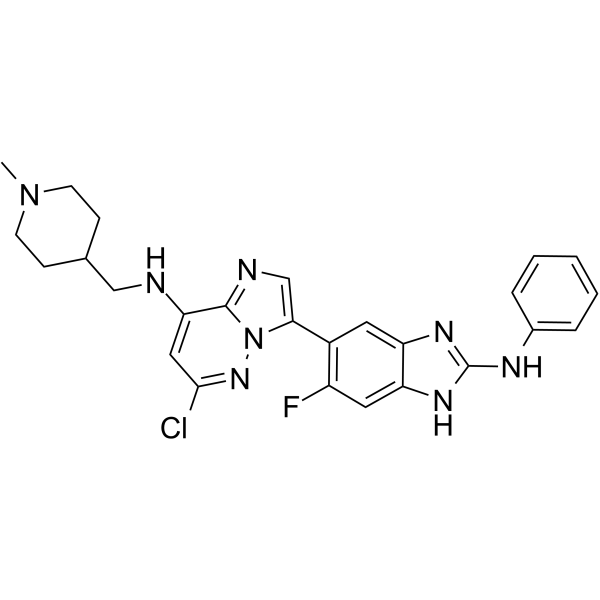
-
GC64232
IRE1α kinase-IN-2
IRE1α quinasa-IN-2 es un potente IRE1α inhibidor de quinasa, con un EC50 de 0,82 μM. IRE1α la quinasa-IN-2 inhibe IRE1α autofosforilaciÓn de cinasa (IC50 = 3,12 μ M). IRE1α quinasa-IN-2 inhibe el empalme del ARNm de XBP1 en las lÍneas celulares WT.
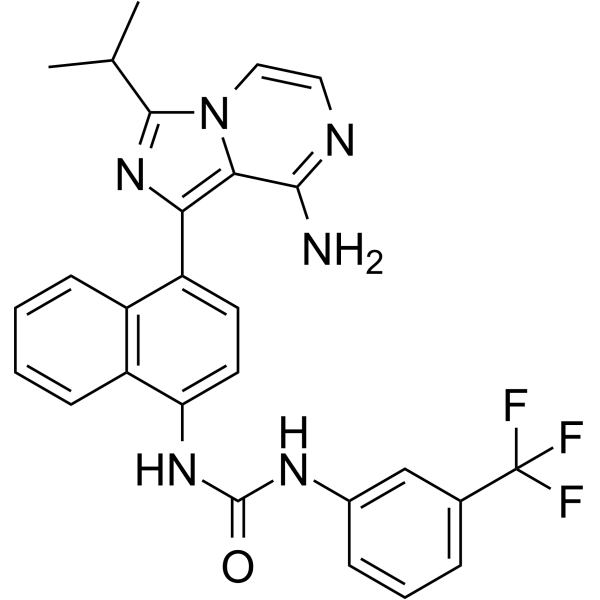
-
GC11473
Irinotecan
Inhibidor de la topoisomerasa I
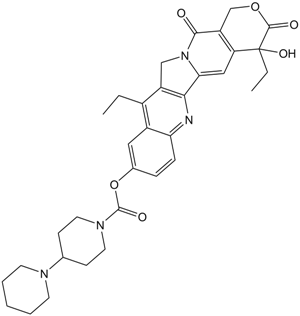
-
GC11048
Irinotecan HCl Trihydrate
El trihidrato de HCl de irinotecÁn ((+)-Trihidrato de HCl de irinotecÁn) es un inhibidor de la topoisomerasa I con actividad antitumoral.
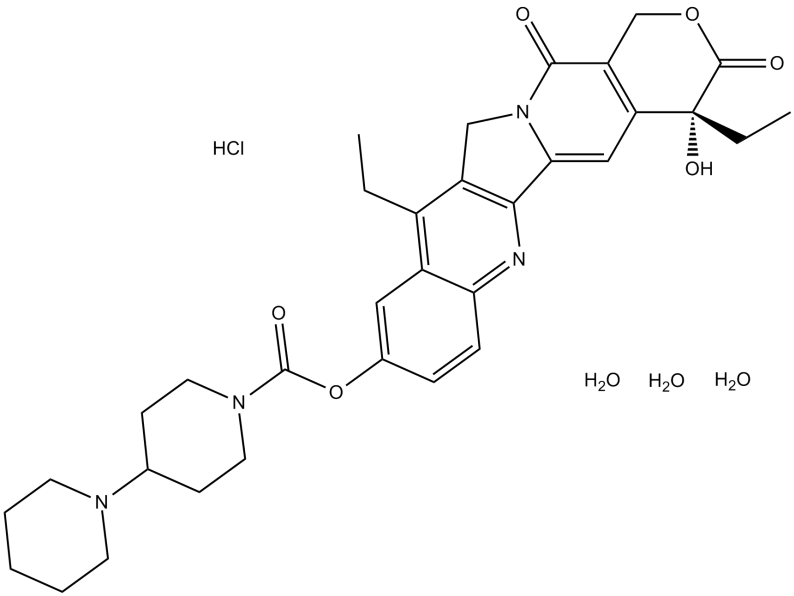
-
GC36329
Irinotecan hydrochloride
El clorhidrato de irinotecÁn ((+)-clorhidrato de irinotecÁn) es un inhibidor de la topoisomerasa I que se usa principalmente para tratar el cÁncer de colon y el cÁncer de recto.
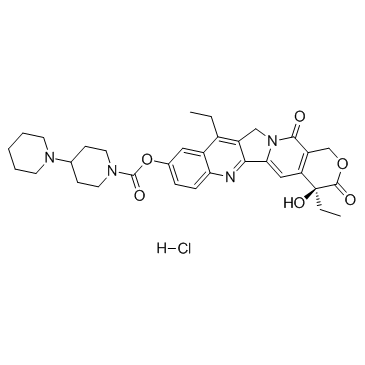
-
GC30554
Isocytosine
La isocitosina es una nucleobase no natural y un isÓmero de la citosina.
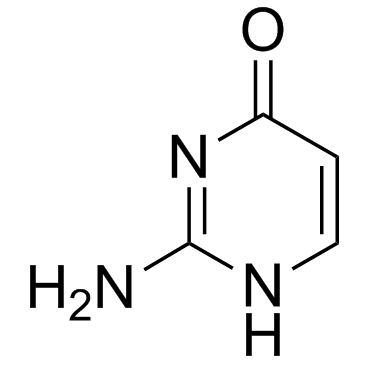
-
GC33662
Isoguanine
Crotonoside, 2-hydroxy-6-Aminopurine, 2-Hydroxyadenine, NSC 241501, 2-Oxoadenine
La isoguanina es una base de purina que es un isÓmero de la guanina.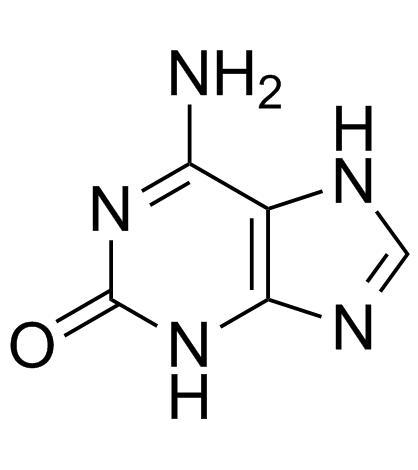
-
GC39140
Isopimpinellin
NSC 217988, NSC 401288
Isopimpinellin, un compuesto oralmente activo aislado de las raÍces de la saxifraga Pimpinella.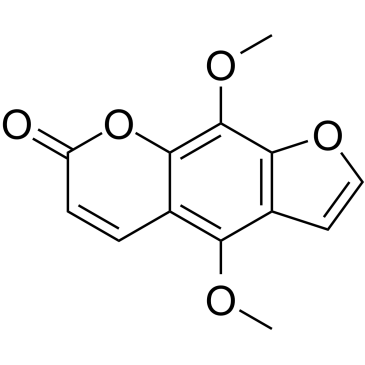
-
GC10834
Isoxanthopterin
NSC 118090,NSC 614991
interferes with RNA and DNA synthesis
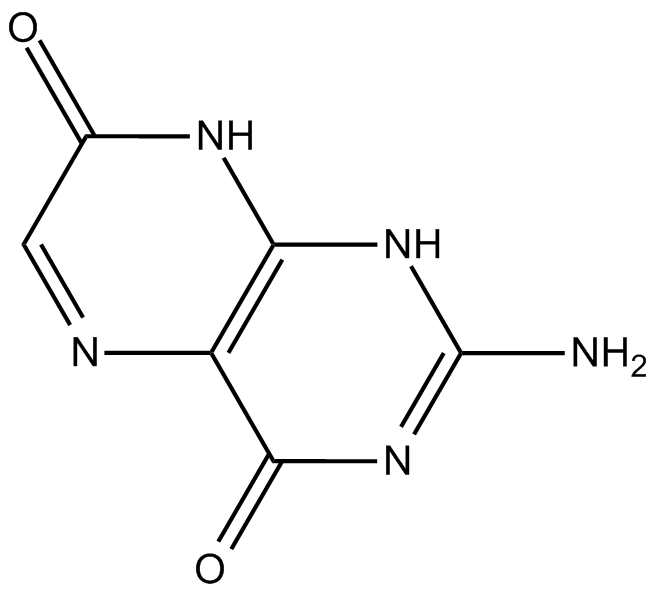
-
GC14597
ITSA-1 (ITSA1)
ITSA-1 (ITSA1) es un activador de la histona desacetilasa (HDAC) y contrarresta la detenciÓn del ciclo celular inducida por la tricostatina A (TSA), la acetilaciÓn de histonas y la activaciÓn transcripcional.
-
GC14797
IU1
Usp14 inhibitor
A deubiquitinating enzyme inhibitor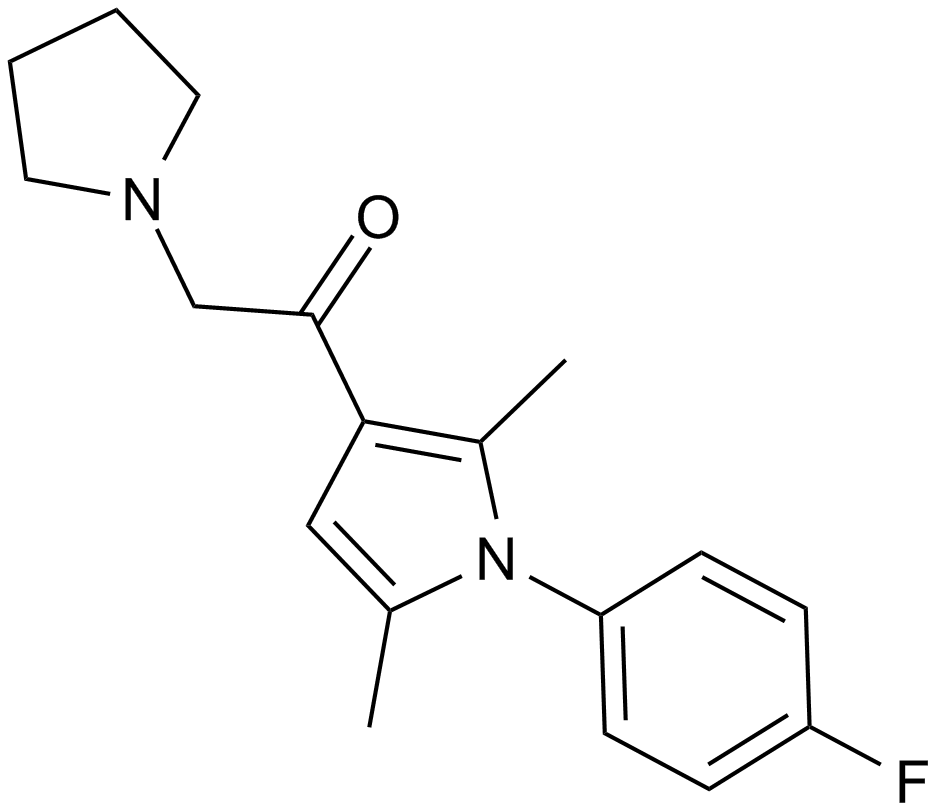
-
GC63027
IU1-248
IU1-248, un derivado de IU1, es un inhibidor potente y selectivo de USP14 con una IC50 de 0,83μM.
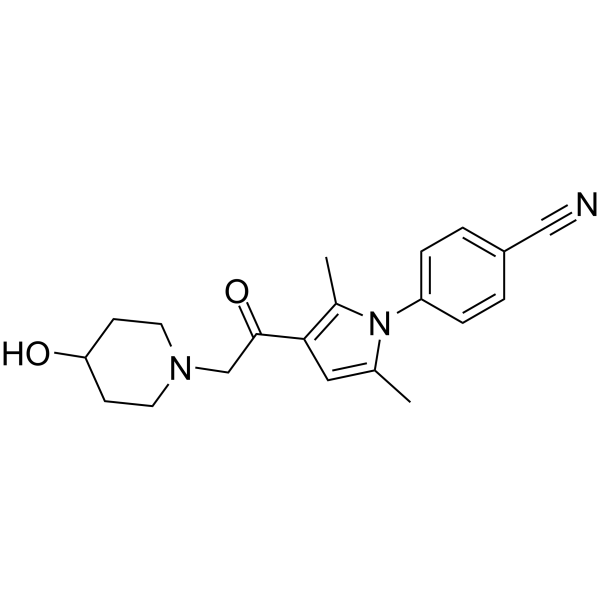
-
GC64956
IU1-47
IU1-47 es un inhibidor potente y especÍfico de USP14 con una IC50 de 0,6 μM.
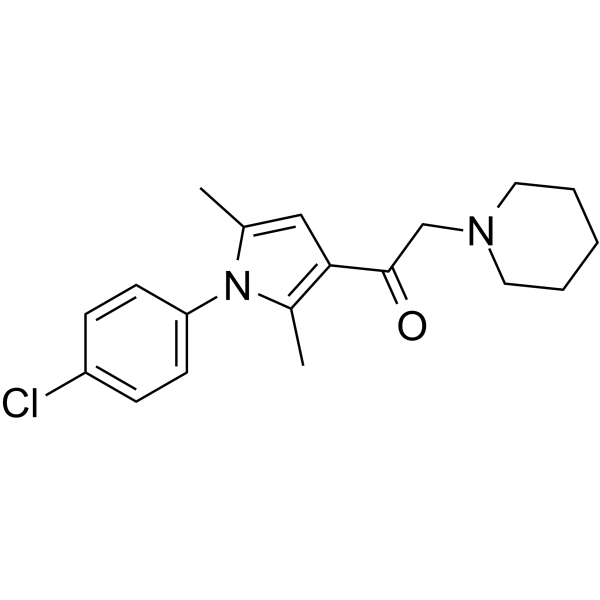
-
GC63460
IV-361
IV-361 es un inhibidor de CDK7 selectivo y activo por vÍa oral (Ki≤50 nM). IV-361 tiene actividad anticancerÍgena (US20190256531A1).
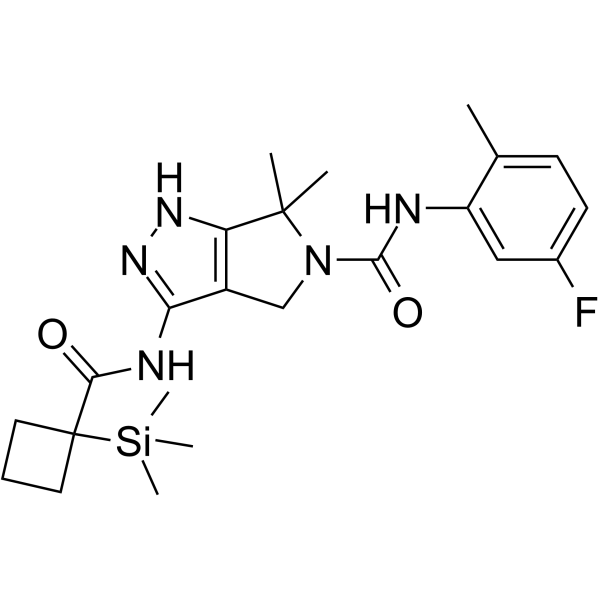
-
GC16454
IWP-2
IWP 2
IWP-2 is a inhibitor of the Wnt signaling pathway, with an IC50 value of 27 nM. IWP-2 is also an ATP-competitive inhibitor of CK1δ, with an IC50 value of 40 nM.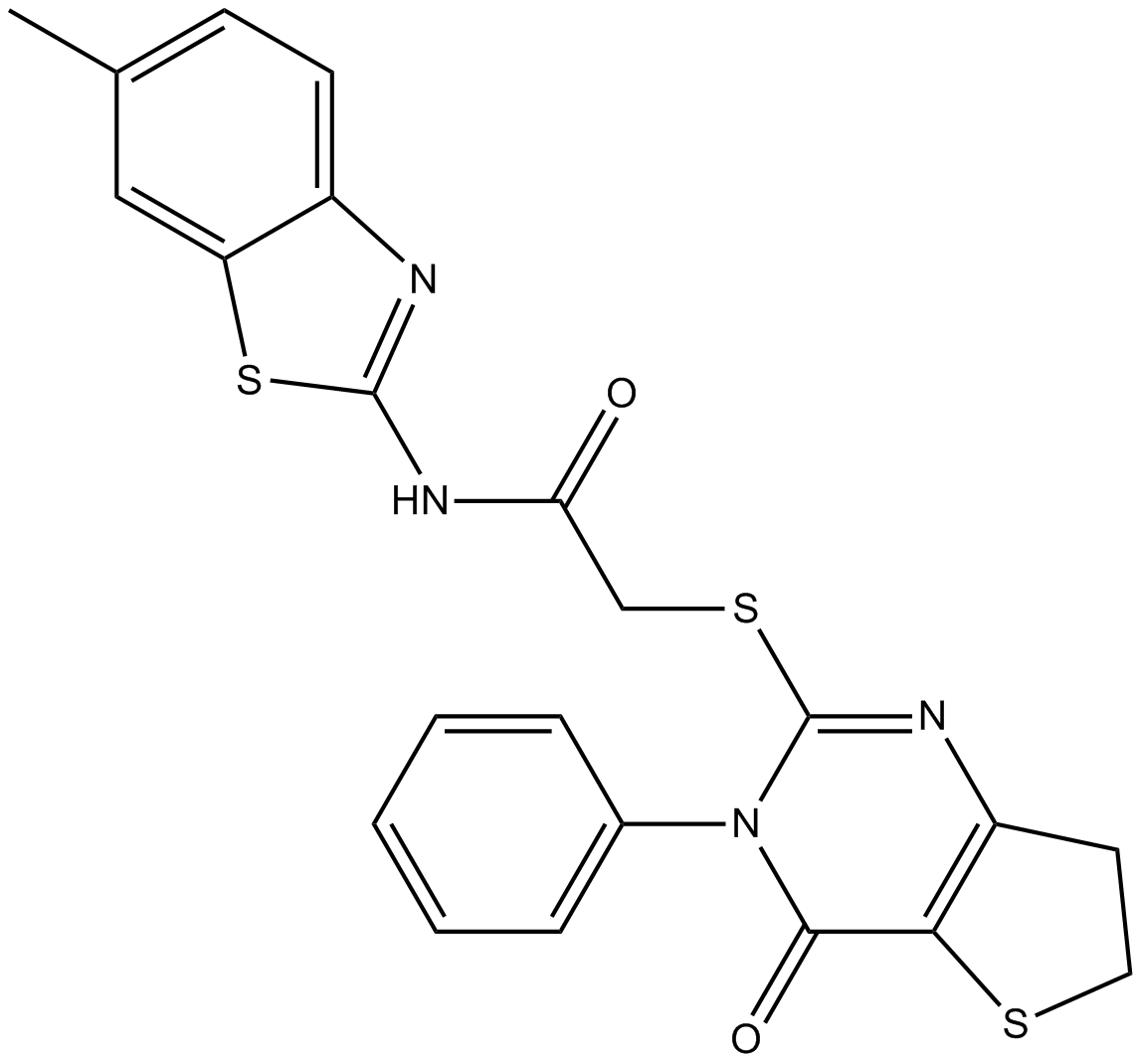
-
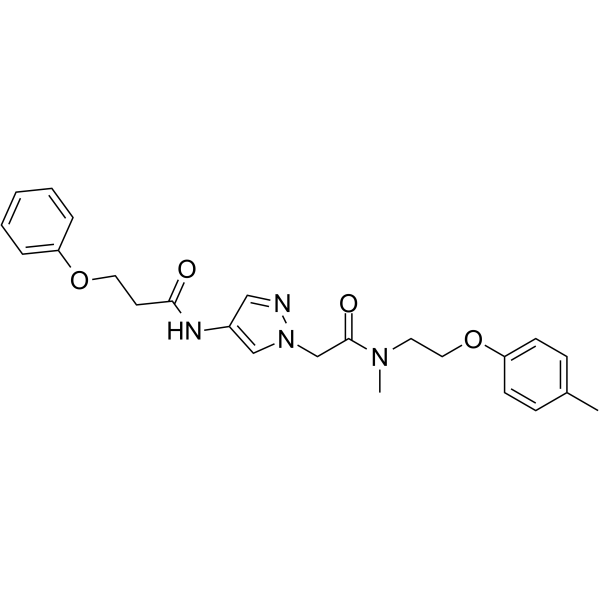
GC63785
IXA4
IXA4 es un activador de IRE1/XBP1s altamente selectivo y no tÓxico.
-
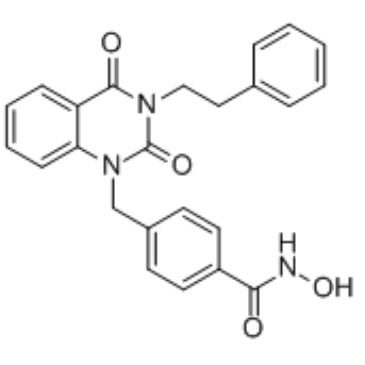
GC34629
J22352
J22352 es un inhibidor de HDAC6 altamente selectivo y similar a PROTAC (proteolysis-targetingchimeras) con un valor IC50 de 4,7 nM. J22352 promueve la degradaciÓn de HDAC6 e induce efectos anticancerÍgenos al inhibir la autofagia y provocar la respuesta inmunitaria antitumoral en los cÁnceres de glioblastoma, y conduce a la restauraciÓn de la actividad antitumoral del huésped al reducir la actividad inmunosupresora de PD-L1.
-
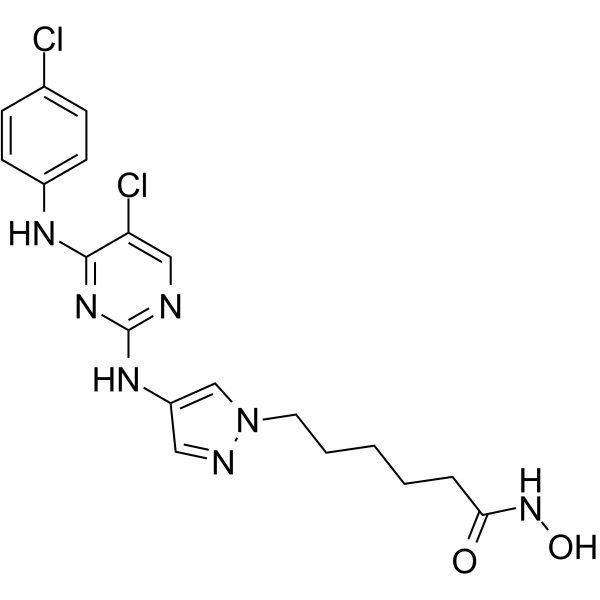
GC64959
JAK/HDAC-IN-1
JAK/HDAC-IN-1 es un potente inhibidor dual de JAK2/HDAC, exhibe actividades antiproliferativas y proapoptÓticas en varias lÍneas celulares hematolÓgicas. JAK/HDAC-IN-1 muestra IC50 de 4 y 2 nM para JAK2 y HDAC, respectivamente.
-
GC36367
JH-RE-06
JH-RE-06, un potente inhibidor de la interfaz REV1-REV7 (IC50 = 0,78 μM; Kd = 0,42 μM), apunta a REV1 que interactÚa con la subunidad REV7 de POLζ. JH-RE-06 interrumpe la sÍntesis de translesiÓn mutagénica (TLS) al evitar el reclutamiento de POLζ mutagénico. JH-RE-06 mejora la quimioterapia.
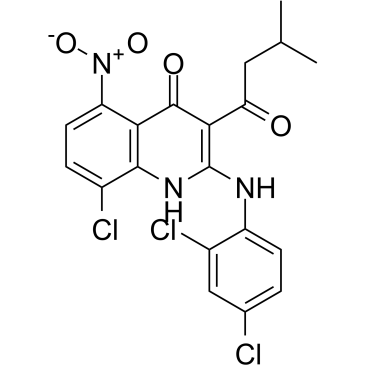
-
GC65471
JH-XI-10-02
JH-XI-10-02 es un PROTAC conectado por ligandos para Cereblon y CDK. JH-XI-10-02 es un degradador altamente potente y selectivo de CDK8, con una IC50 de 159 nM. JH-XI-10-02 causa la degradación proteasomal, no afecta los niveles de ARNm de CDK8. JH-XI-10-02 no muestra efecto sobre CDK19.
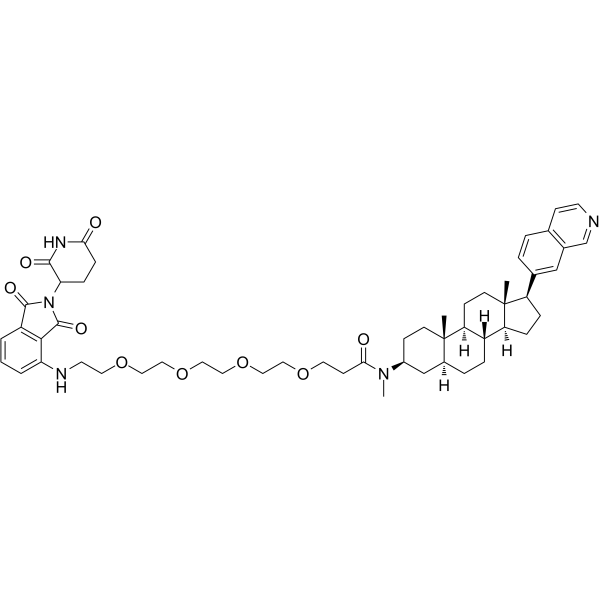
-
GC65577
JH-XVI-178
JH-XVI-178 es un inhibidor altamente potente y selectivo de CDK8/19 que muestra un aclaramiento bajo y propiedades farmacocinéticas orales moderadas.

-
GC12612
JNJ-7706621
JNJ7706621, JNJ 7706621
A dual inhibitor of CDKs and Aurora kinases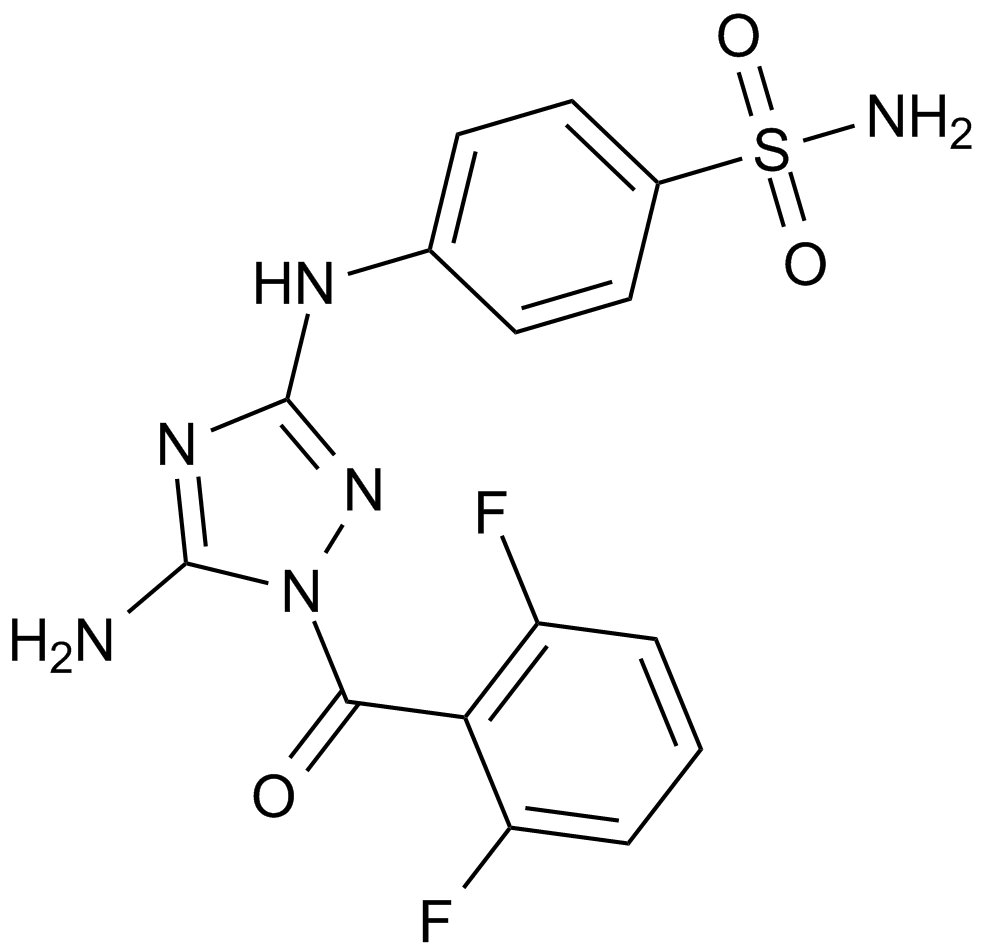
-
GC18734
JS-K
JS-K es un donante de NO que reacciona con el glutatiÓn para generar NO a pH fisiolÓgico. JS-K inhibe la proliferaciÓn, induce la apoptosis e interrumpe el ciclo celular de las células de leucemia linfoblÁstica aguda Jurkat T.
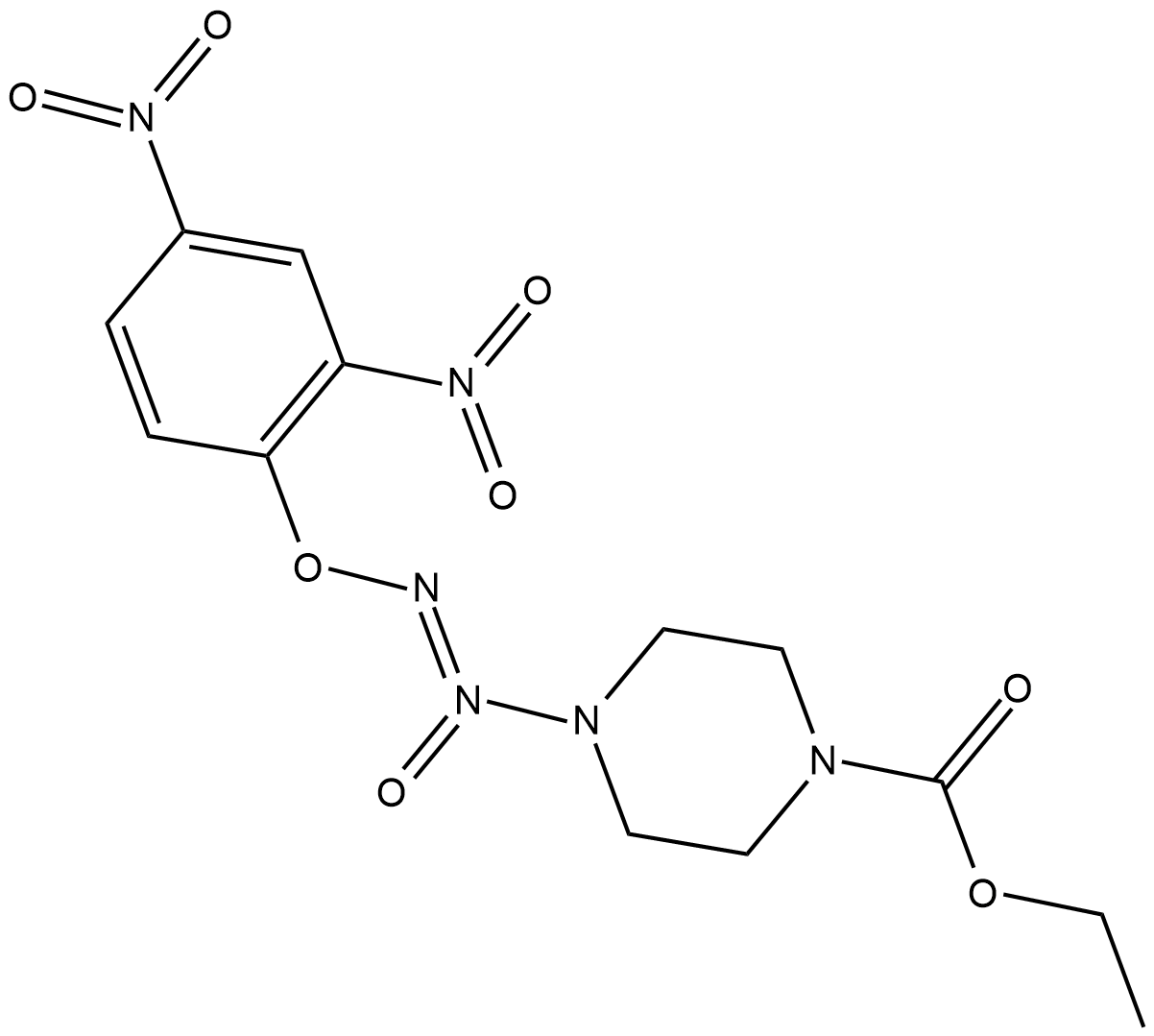
-
GC34204
JSH-150
JSH-150 es un inhibidor de CDK9 altamente selectivo y potente con un IC50 de 1 nM.
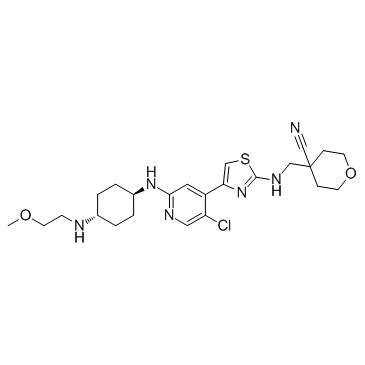
-
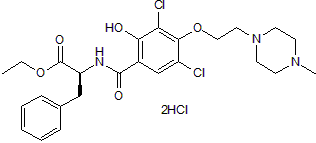
GC50117
JTE 607 dihydrochloride
El diclorhidrato de JTE 607, un inhibidor de la sÍntesis de citoquinas inflamatorias altamente selectivo, protege del shock de endotoxinas en ratones.
-
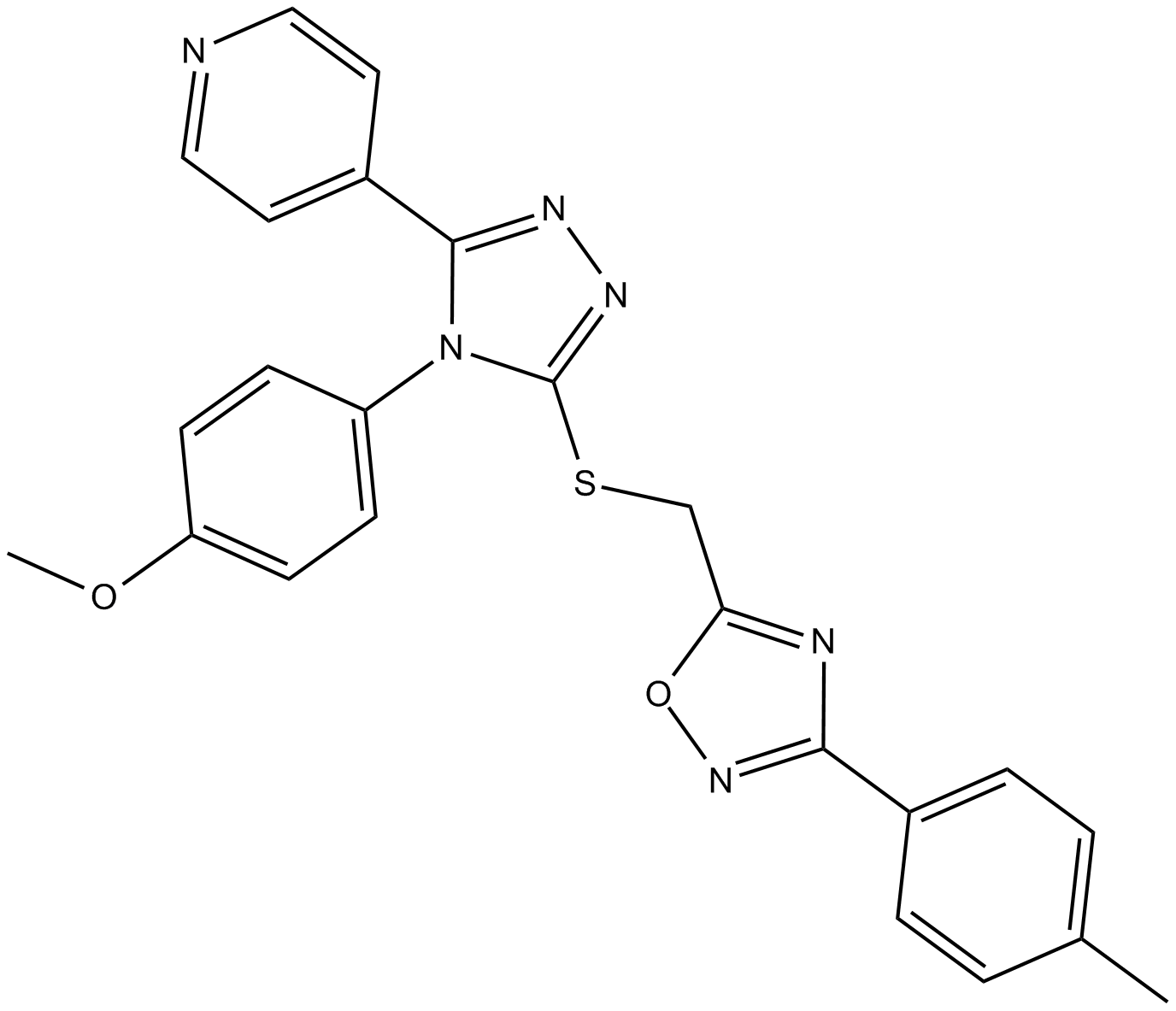
GC13978
JW 74
JW 74 antagoniza la activación inducida por LiCl de la señalización Wnt canónica con un IC50 de 420 nM.
-
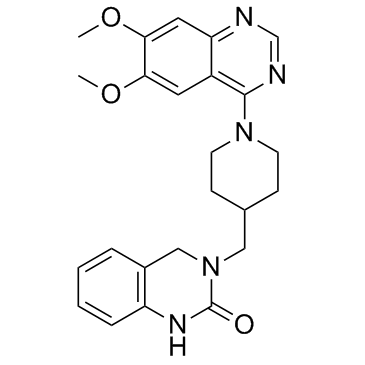
GC34195
K-756
K-756 es un inhibidor de tankirasa directo y selectivo (TNKS), que inhibe la actividad de ribosilaciÓn de ADP de TNKS1 y TNKS2 con IC50 de 31 y 36 nM, respectivamente.
-
GC41384
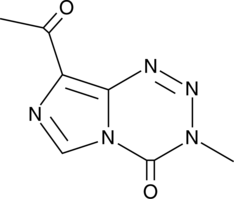
K-TMZ
K-TMZ is a DNA alkylating agent.
-
GC14230
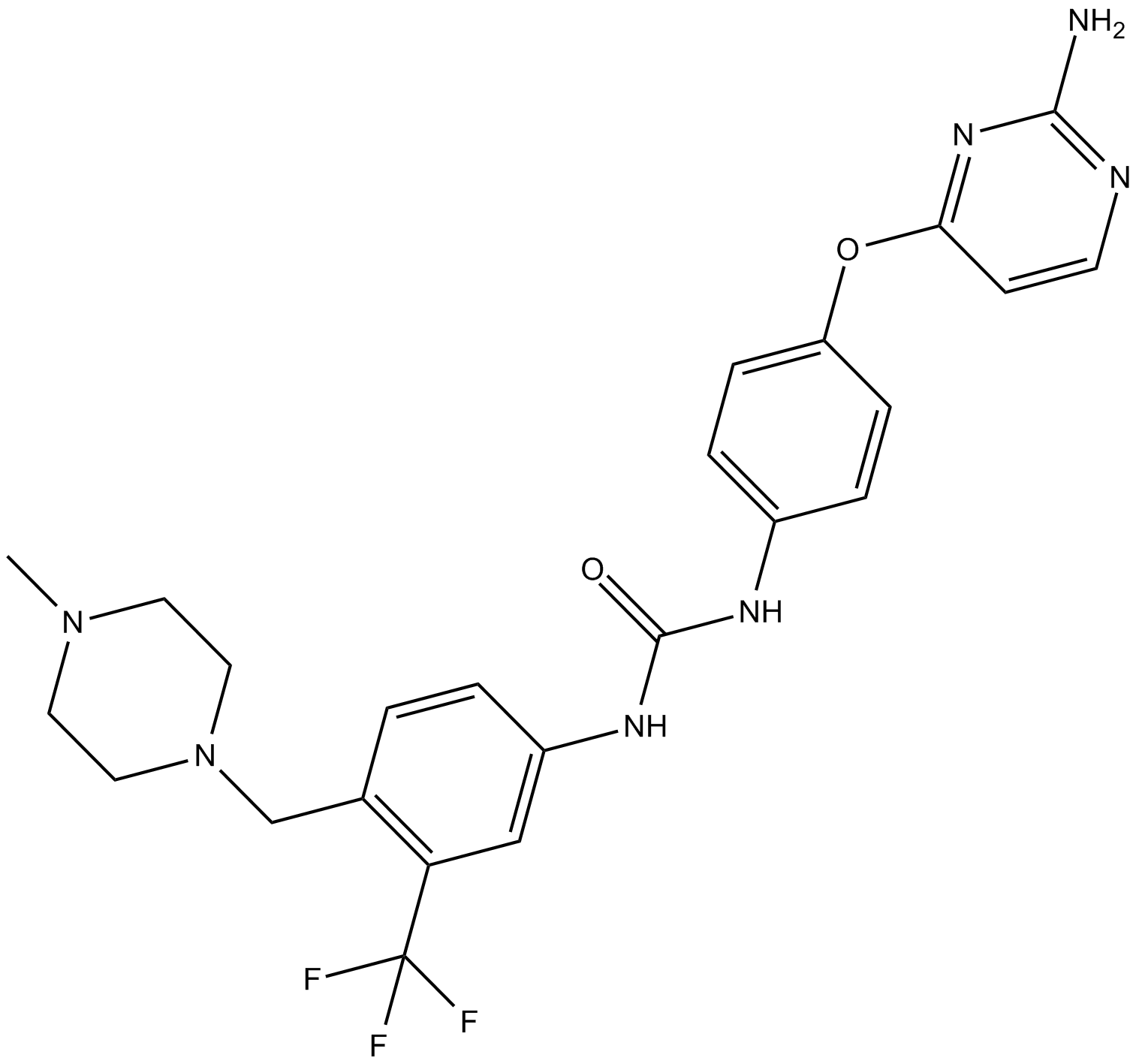
K03861
K03861 (K03861) es un inhibidor de CDK2 de tipo II con Kd de 8,2 nM. K03861 (K03861) inhibe la actividad de CDK2 al competir con la uniÓn de las ciclinas activadoras.
-
GC47521
K22
An antiviral agent
-
GC34144
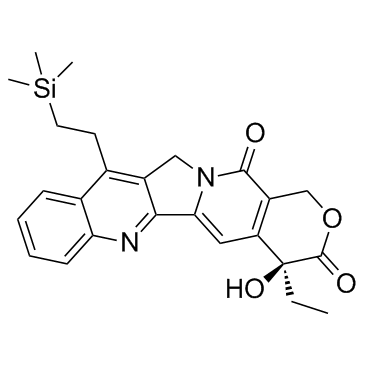
Karenitecin (Cositecan)
La karenitecina (Cositecan) (Cositecan) es un inhibidor de la topoisomerasa I, con una potente actividad anticancerÍgena.
-
GC62392
KB-0742 dihydrochloride
El diclorhidrato de KB-0742 es un inhibidor de CDK9 potente, selectivo y activo por vÍa oral con una IC50 de 6 nM para CDK9/ciclina T1. El diclorhidrato de KB-0742 es selectivo para CDK9/ciclina T1 con una selectividad >50 veces superior a otras CDK quinasas. El diclorhidrato de KB-0742 tiene una potente actividad antitumoral.
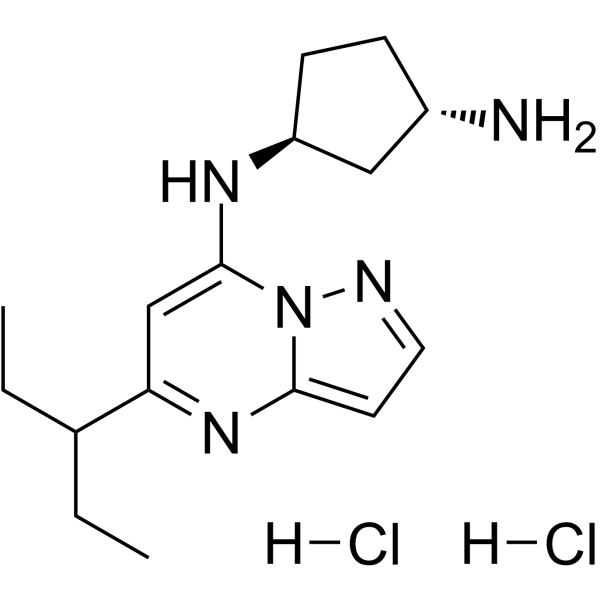
-
GC14182
Kenpaullone
9Bromopaullone, NSC 664704
La kenpaulona es un potente inhibidor de CDK1/ciclina B y GSK-3β, con IC50 de 0,4 μM y 23 nM, y también inhibe CDK2/ciclina A, CDK2/ciclina E y CDK5/p25 con IC50 de 0,68 μM, 7,5 μM, 0,85 μM, respectivamente. La kenpaulona, una molécula pequeÑa inhibidora de KLF4, reduce la autorrenovaciÓn de las células madre del cÁncer de mama y la motilidad celular in vitro.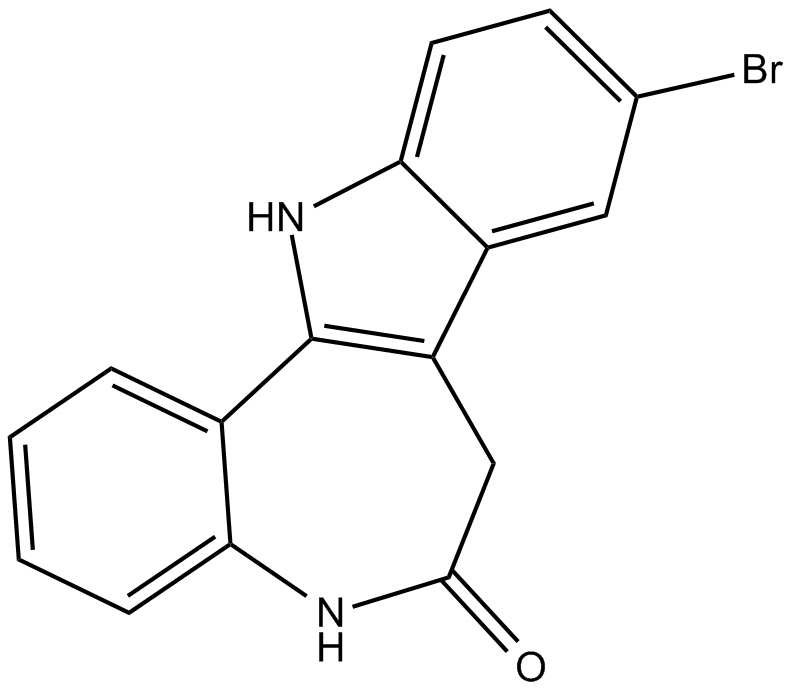
-
GC11774
KH CB19
KH CB19 es un potente inhibidor de CLK (cinasa similar a cdc2) (CLK1 IC50 \u003d 19,7 nM; CLK3 IC50 \u003d 530 nM).
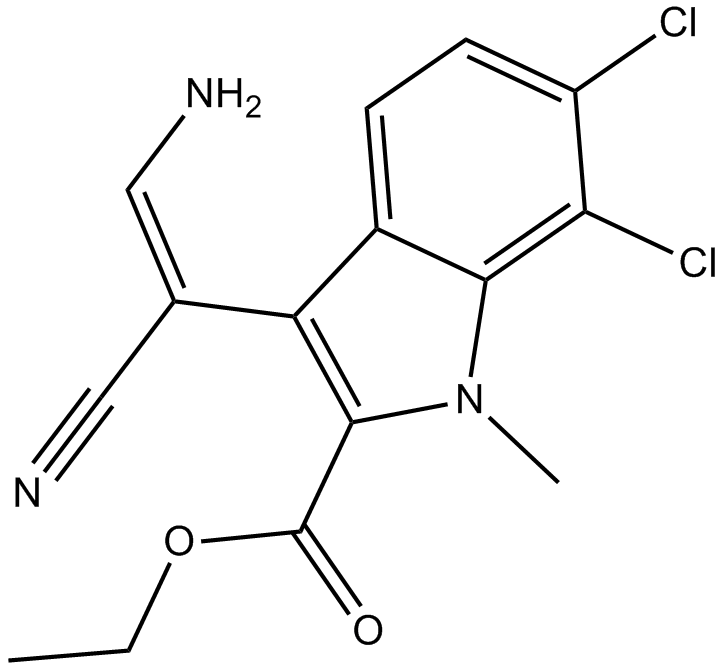
-
GC63943
KH-CB20
KH-CB20, una mezcla E/Z, es un inhibidor potente y selectivo de CLK1 y la isoforma CLK4 estrechamente relacionada, con una IC50 de 16,5 nM para CLK1.
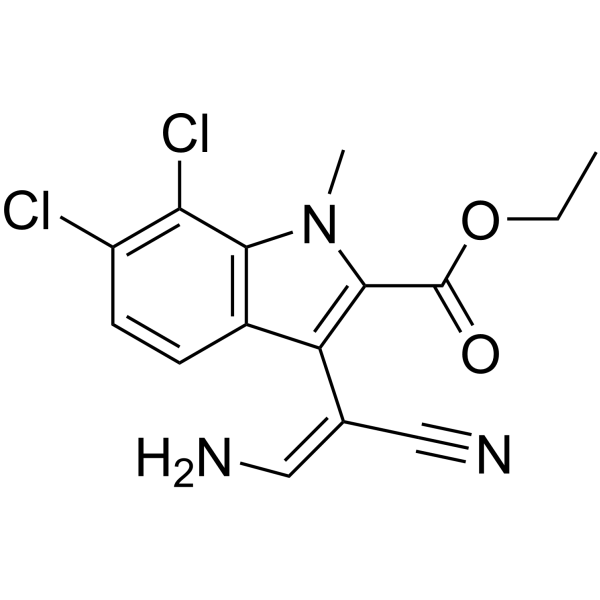
-
GC64238
KIRA-7
KIRA-7, un compuesto de imidazopirazina, se une a la quinasa IRE1α (IC50 de 110 nM) para inhibir alostéricamente su actividad de RNasa.
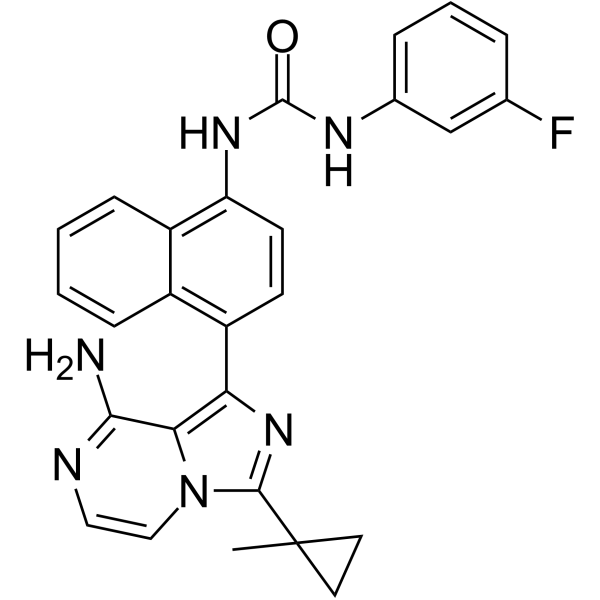
-
GC19212
KIRA6
IRE1 Inhibitor IV, IRE1α Kinase Inhibiting RNase Attenuator 6
KIRA6 es un inhibidor avanzado de la quinasa de la ARNasa IRE1α de molécula pequeÑa con una IC50 de 0,6 μM.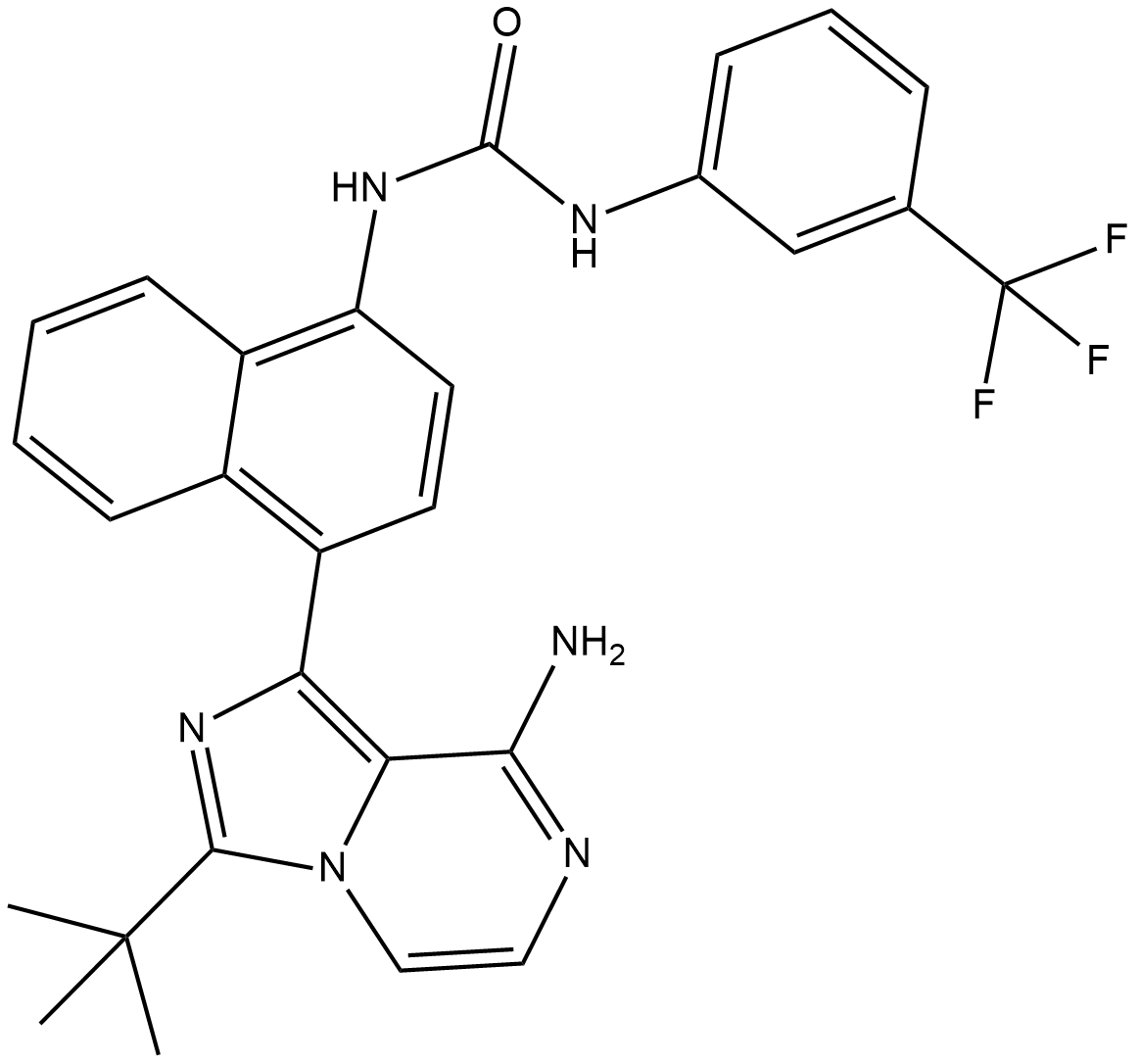
-
GC31879
Kira8
AMG-18
Kira8 (AMG-18) es un inhibidor monoselectivo de IRE1α que atenÚa alostéricamente la actividad de la RNasa de IRE1α con una IC50 de 5,9 nM.
-
GC65907
KSQ-4279
USP1-IN-1
KSQ-4279 (USP1-IN-1, FÓrmula I) es un inhibidor de USP1 y PARP (extraÍdo de la patente WO2021163530).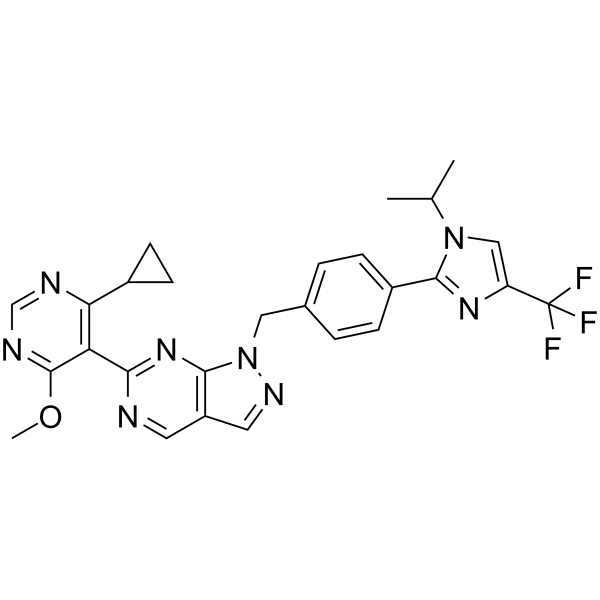
-
GC25552
KT-531
KT-531 (KT531) is a potent, selective HDAC6 inhibitor with IC50 of 8.5 nM, displays 39-fold selectivity over other HDAC isoforms.

-
GC33404
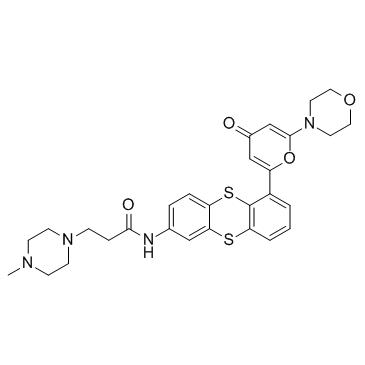
KU 59403
KU 59403 es un potente inhibidor de ATM, con valores IC50 de 3 nM, 9,1 μM y 10 μM para ATM, DNA-PK y PI3K, respectivamente.
-
GC14346
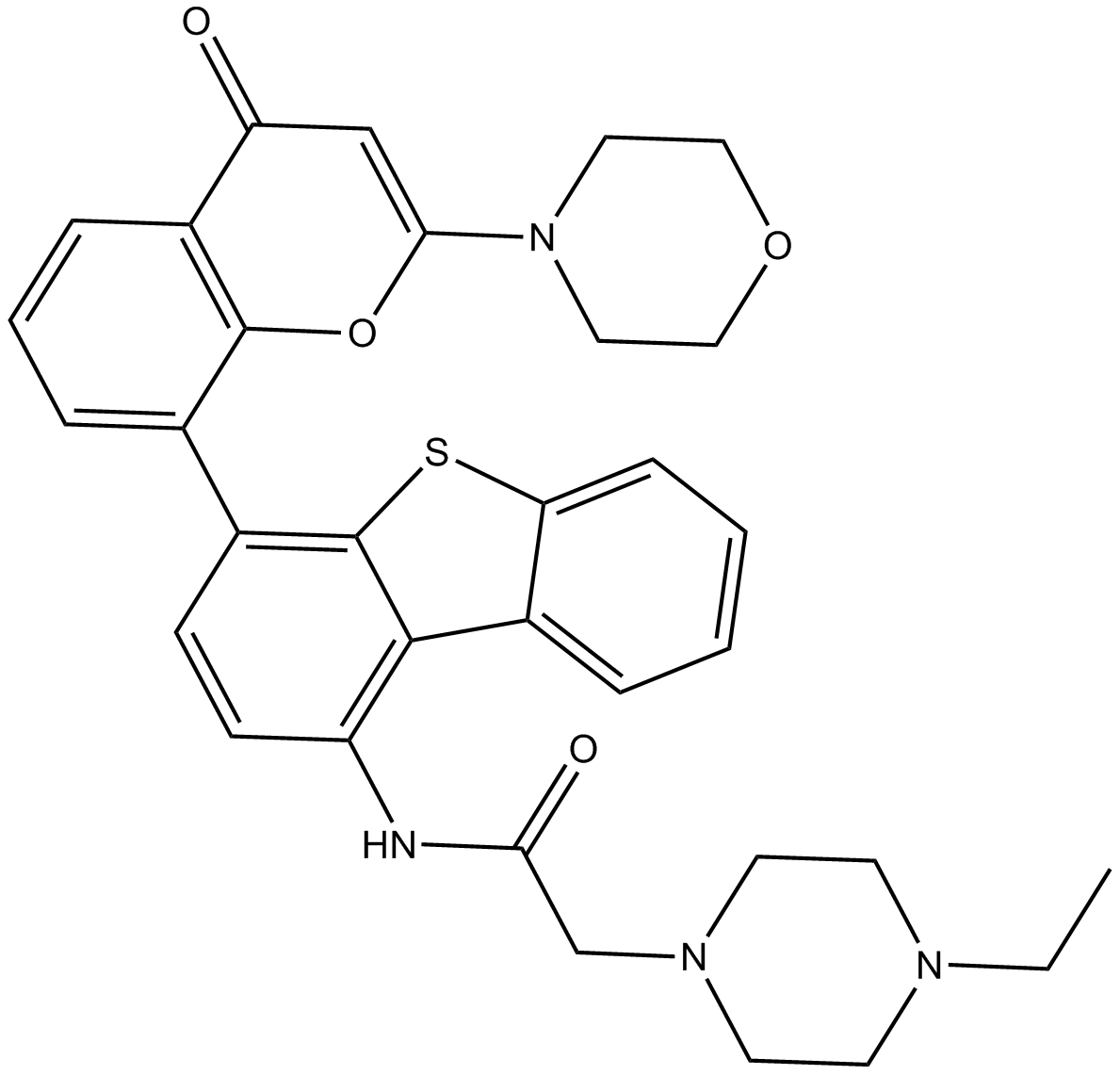
KU-0060648
KU-0060648 es un inhibidor dual de PI3K y DNA-PK con IC50 de 4 nM, 0,5 nM, 0,1 nM, 0,594 nM y 8,6 nM para PI3Kα, PI3Kβ, PI3Kγ, PI3Kδ y DNA-PK, respectivamente.
-
GC12054
KU-60019
KU60019;KU 60019
KU-60019 es un inhibidor especÍfico de la quinasa ATM mejorado con IC50 de 6,3 nM.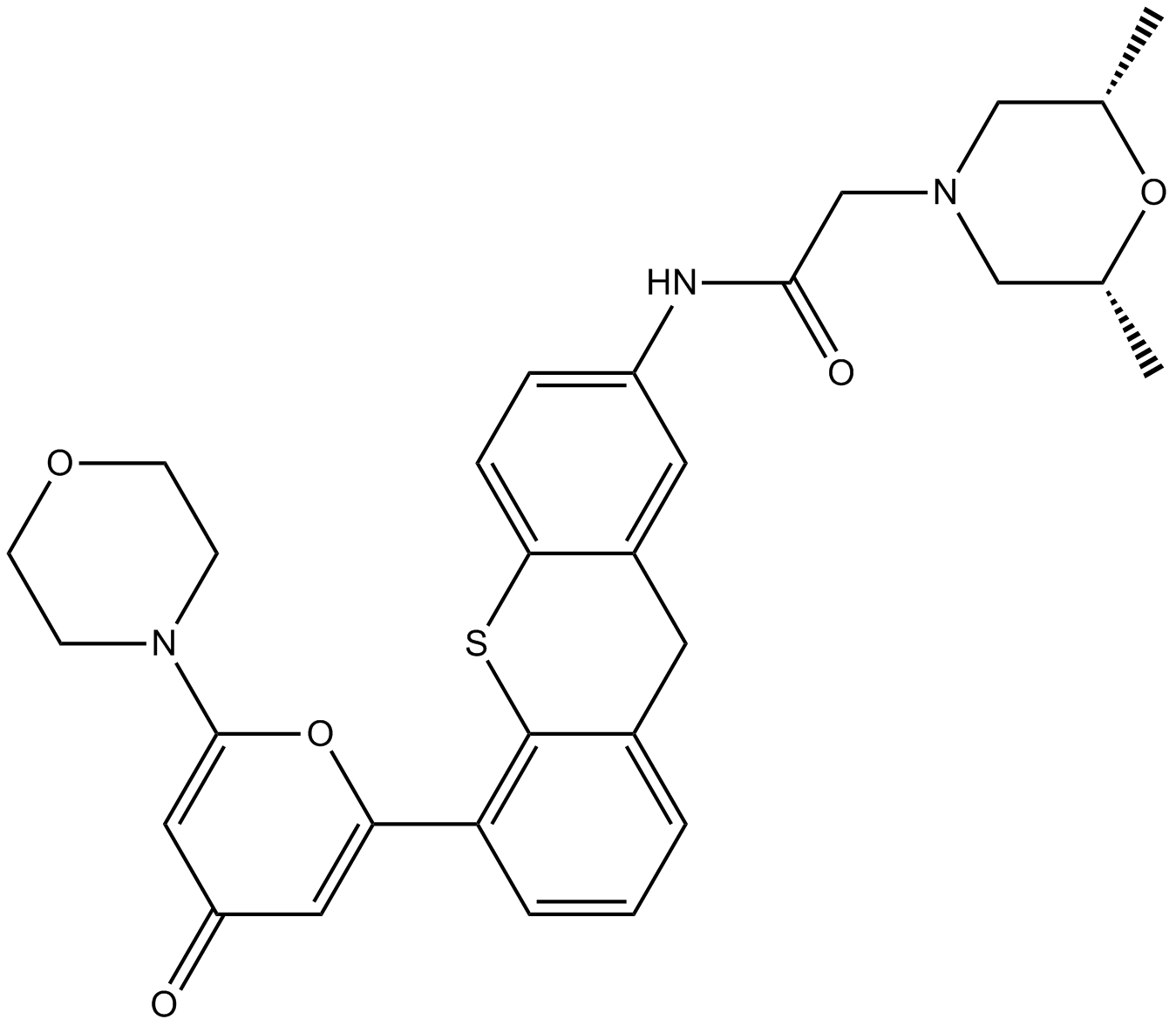
-
GC50532
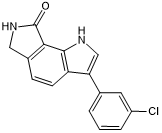
KuWal151
Potent and selective CLK inhibitor
-
GC15743
L-755,507
L-755,507 es un potente agonista selectivo de β3-AR con una CI50 de 35 nM.
-
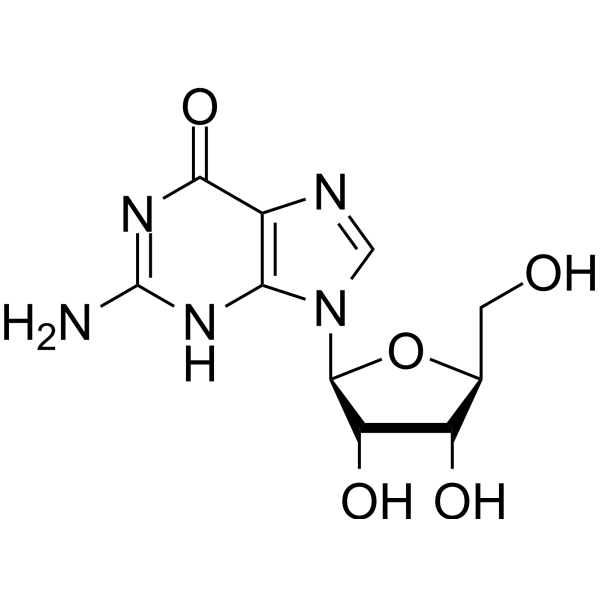
GC66622
L-Guanosine
L-guanosina es la configuración L de la guanosina. La guanosina es un nucleósido de purina con actividad anti-herpesvirus.
-
GC49381
L-Leucine-d10
Leu-d10, Pentanoic Acid-d10
An internal standard for the quantification of L-leucine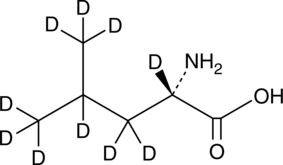
-
GC49368
L-Methionine-d3
(S)-Methionine-d3
L-Metionina-d3 es la L-Metionina marcada con deuterio.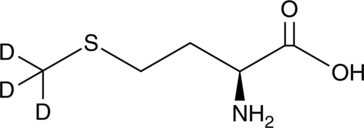
-
GC49384
L-Proline-d3
L-(–)-Proline-d3, (S)-(–)-Proline-d3
An internal standard for the quantification of L-proline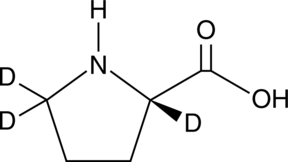
-
GC12131
L189
L189 es un inhibidor de la ADN ligasa. L189 tiene un efecto de inhibiciÓn de la ADN ligasa I, III y IV con valores IC50 de 5 μM, 9 μM y 5 μM, respectivamente. L189 no tiene citotoxicidad y aumenta individualmente la muerte celular. L189 se puede utilizar para la investigaciÓn del cÁncer.
-
GC18186
L67
DNA Ligase Inhibitor
L67 (inhibidor de ADN ligasa) es un inhibidor competitivo de ADN ligasa que inhibe eficazmente las ADN ligasas I/III (ambas IC50 son 10 μM). L67 (inhibidor de ADN ligasa) puede causar daÑos en el ADN nuclear al reducir los niveles de ADN mitocondrial y aumentar los niveles de ROS generados mitocondrialmente. L67 (inhibidor de ADN ligasa) también activa la vÍa de apoptosis dependiente de caspasa 1 en las células cancerosas, se puede utilizar en la investigaciÓn del cÁncer.
-
GC67785
L82

-
GC68436
L82-G17

-
GC39632
Laflunimus
Laflunimus (HR325) es un agente inmunosupresor y un anÁlogo del metabolito activo de leflunomida A77 1726.
-
GC62460
LCAHA
LCA hydroxyamide
LCAHA (LCA hidroxiamida) es un inhibidor de la deubiquitinasa USP2a con IC50 de 9,7 μM y 3,7 μM en el ensayo Ub-AMC y el ensayo Di-Ub, respectivamente. LCAHA desestabiliza la ciclina D1 e induce la detenciÓn de G0/G1 al inhibir la deubiquitinasa USP2a.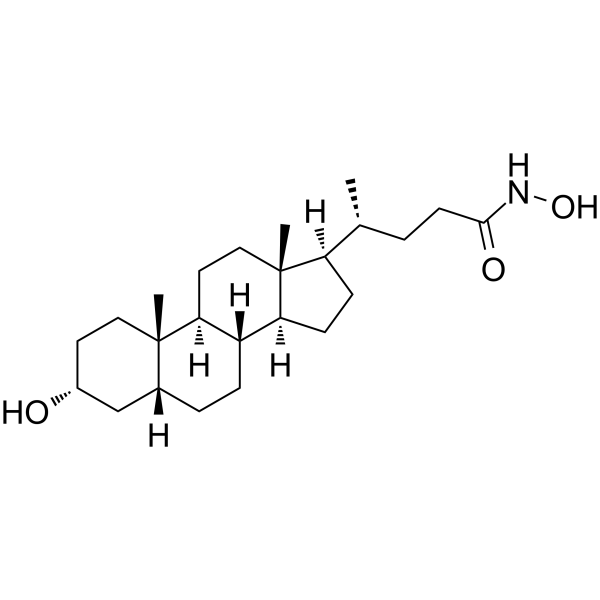
-
GC17067
LDC000067
LDC067
LDC000067 es un inhibidor de CDK9 altamente especÍfico con un valor IC50 de 44±10 nM in vitro.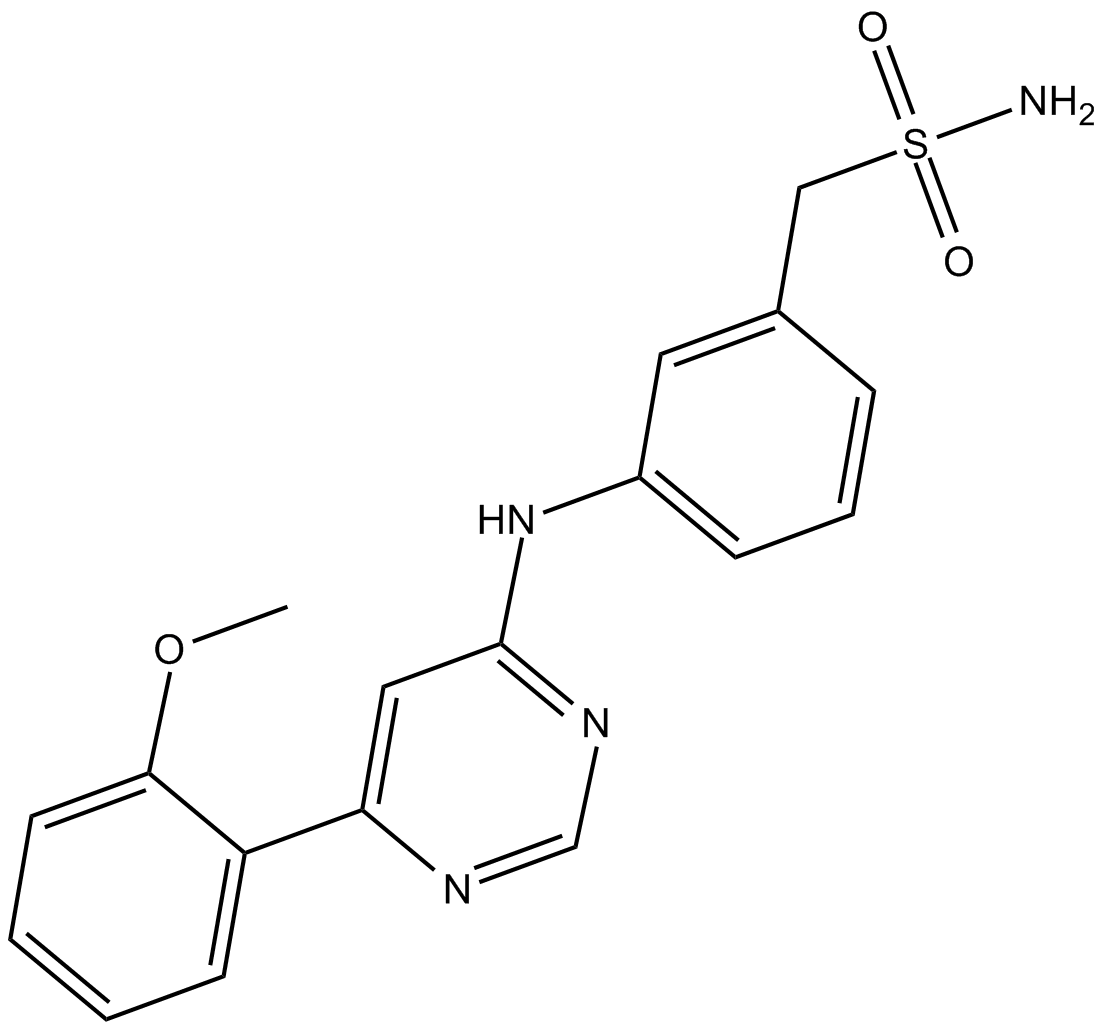
-
GC19219
LDC4297
LDC4297 es un inhibidor de CDK7 potente y selectivo con una IC50 de 0,13 nM.