Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
GC16509
Adox
Adox, un anÁlogo de nucleÓsido de purina, es un potente inhibidor de la S-adenosilhomocisteÍna hidrolasa (SAHH) (Ki = 3,3 nM). El dialdehÍdo de adenosina exhibe una potente actividad antitumoral in vivo y puede usarse para la investigaciÓn del cÁncer.
-
GC64527
ADTL-SA1215
ADTL-SA1215 es un activador de molécula pequeÑa especÍfico de primera clase de SIRT3 que modula la autofagia en el cÁncer de mama triple negativo.
-
GC65330
AES-135
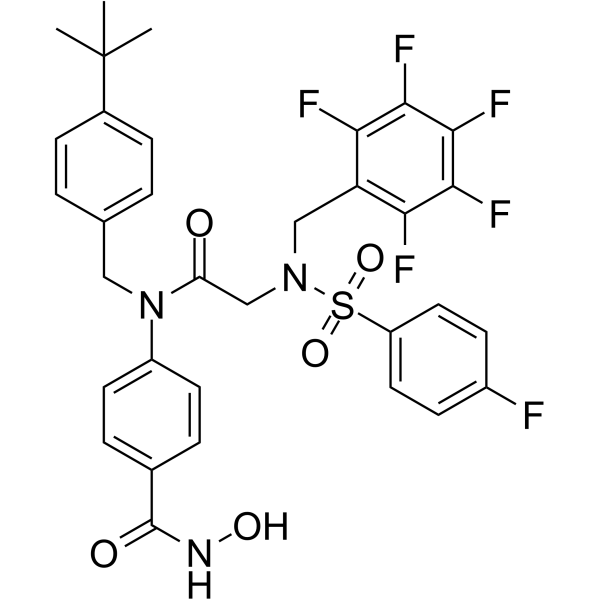
AES-135, un inhibidor pan-HDAC a base de Ácido hidroxÁmico, prolonga la supervivencia en un modelo de ratÓn ortotÓpico de cÁncer de pÁncreas. AES-135 inhibe HDAC3, HDAC6, HDAC8 y HDAC11 con IC50 que van desde 190-1100 nM.
-
GC48775
AES-350
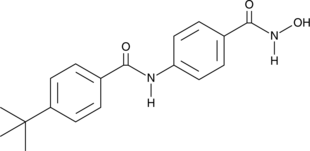
AES-350 es un inhibidor de HDAC6 potente y activo por vÍa oral con una IC50 y una Ki de 0,0244 μM y 0,035 μM, respectivamente. AES-350 también es contra HDAC3, HDAC8 en un ensayo de actividad enzimÁtica con valores IC50 de 0,187 μM y 0,245 μM, respectivamente. AES-350 desencadena la apoptosis en las células de AML a través de la inhibiciÓn de HDAC y se puede utilizar para la investigaciÓn de la leucemia mieloide aguda (AML).
-
GC66379
AFM-30a hydrochloride
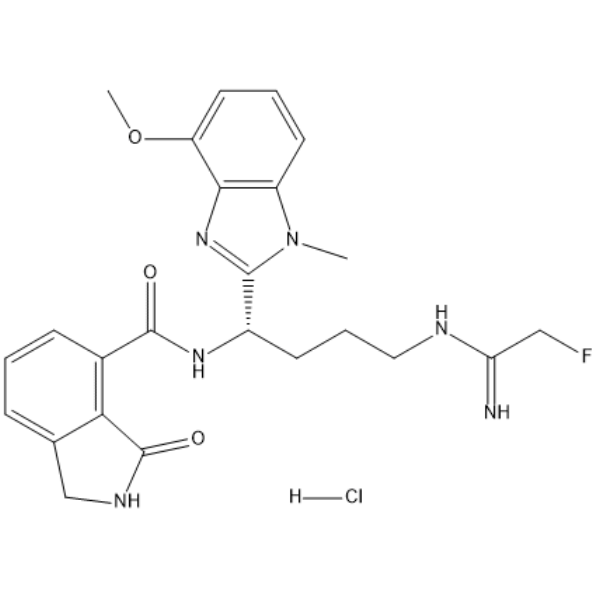
El clorhidrato de AFM-30a es un potente inhibidor de la proteÍna arginina deiminasa 2 (PAD2) y tiene una excelente selectividad para PAD2. El clorhidrato de AFM-30a se une a PAD2 con un valor EC50 de 9,5 μM. El clorhidrato de AFM-30a también inhibe la citrulinaciÓn H3 con un valor EC50 de 0,4 μM. El clorhidrato de AFM-30a se puede usar para la investigaciÓn de ciertos tipos de cÁncer y una variedad de enfermedades autoinmunes, incluida la artritis reumatoide (AR), la esclerosis mÚltiple, el lupus y la colitis ulcerosa.
-
GC33416
AFP464
AFP464 (NSC710464 base libre), es un HIF-1α activo; inhibidor con un IC50 de 0,25 μ M, también es un potente activador del receptor de hidrocarburos arÍlicos (AhR).

-
GC16318
AG-14361
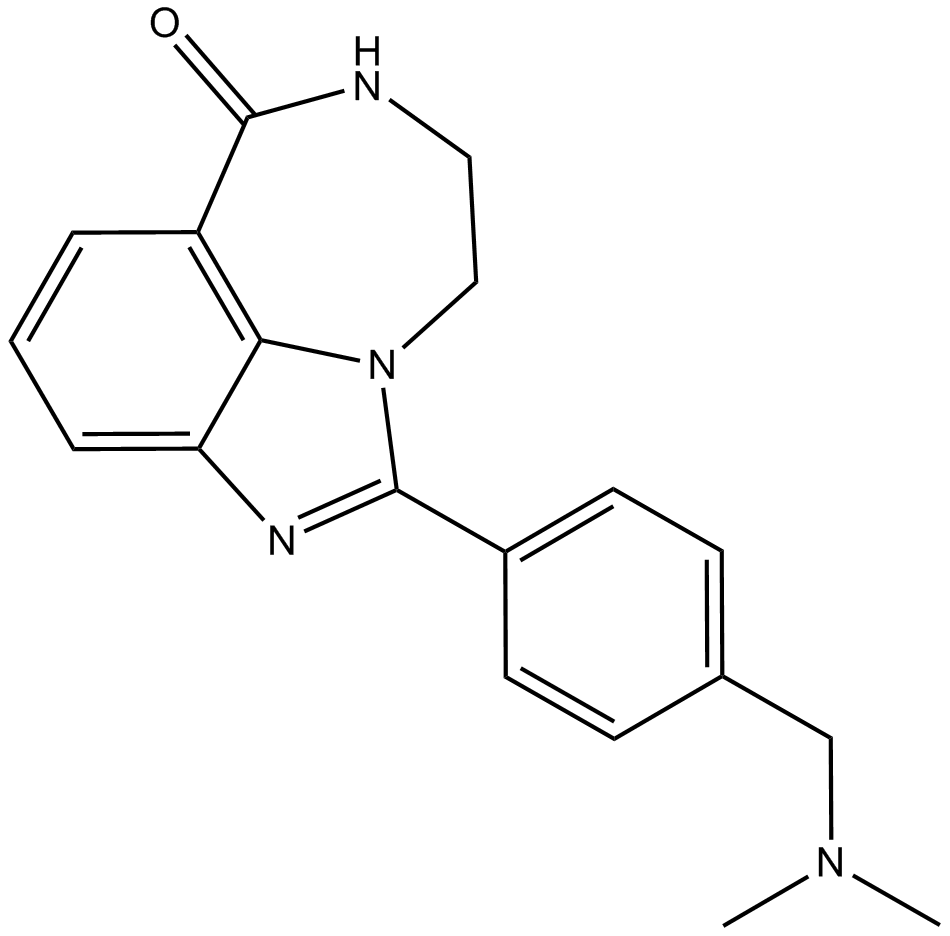
A PARP1 inhibitor
-
GC17881
AGK 2
AGK 2 es un inhibidor selectivo de SIRT2 con una IC50 de 3,5 μM. AGK 2 inhibe SIRT1 y SIRT3 con IC50 de 30 y 91 μM, respectivamente.
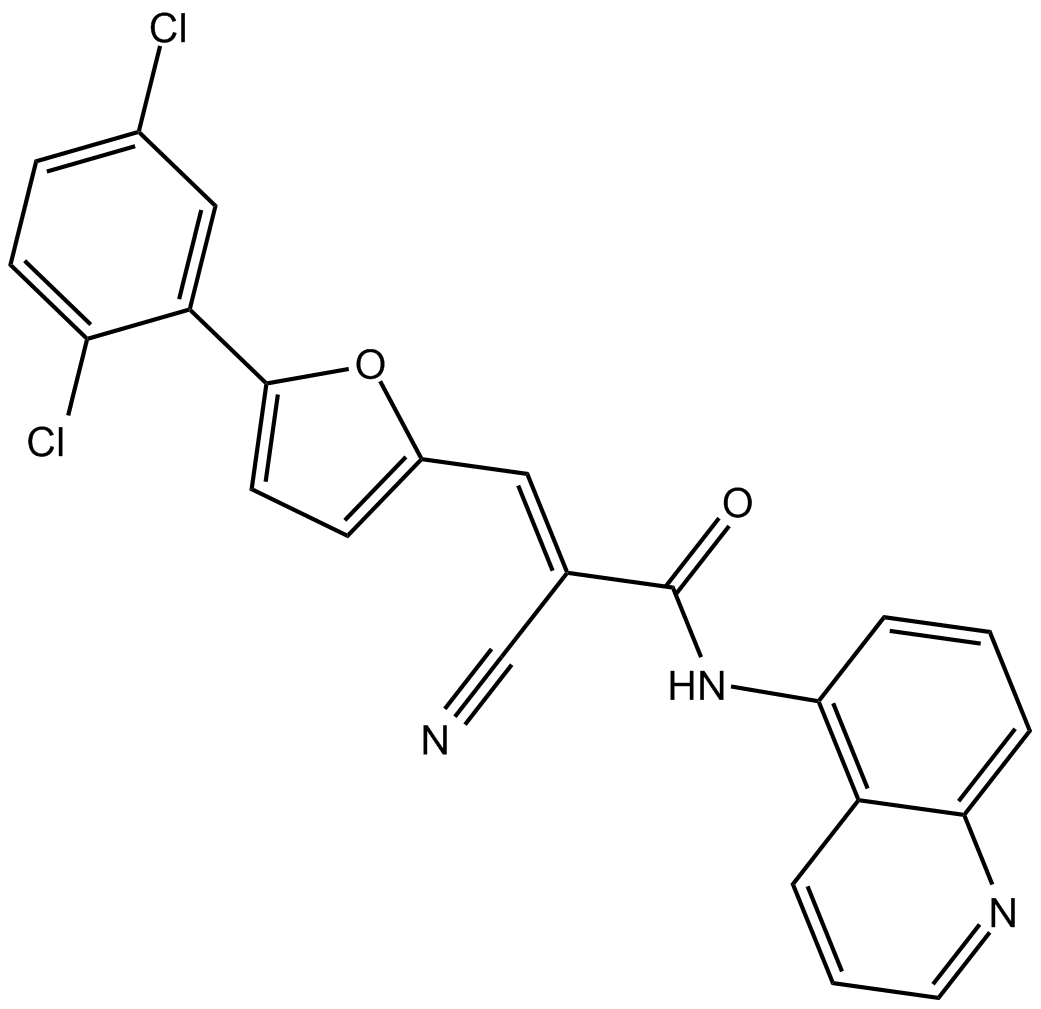
-
GC15931
AGK7
AGK7 es un potente inhibidor de la sirtuina 2 (SIRT2).
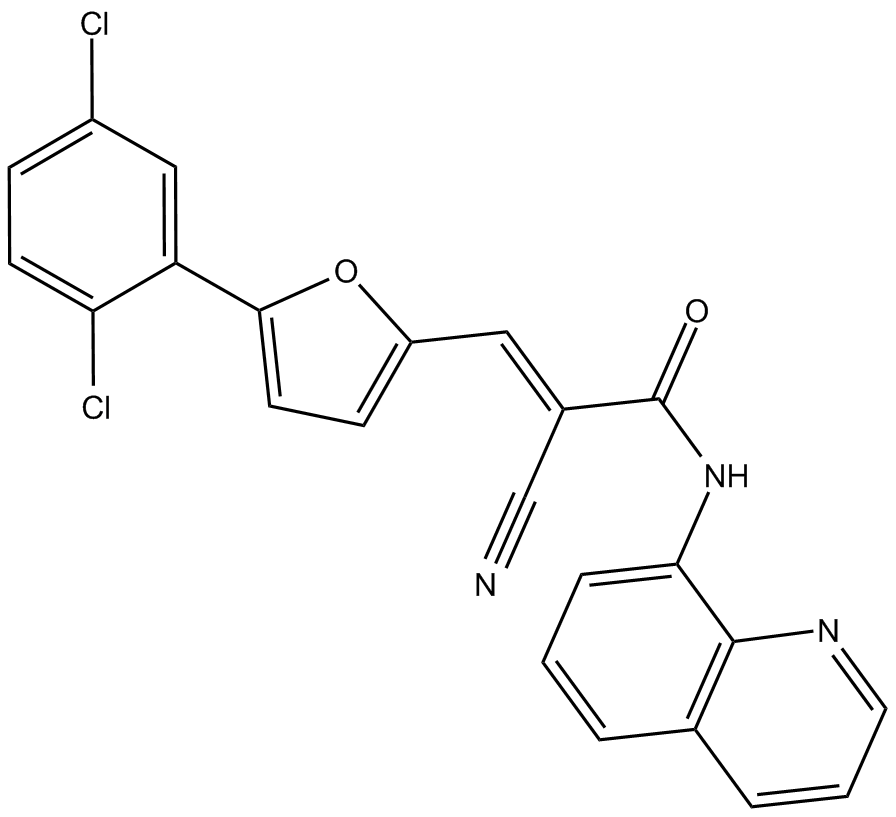
-
GN10413
Agrimol B
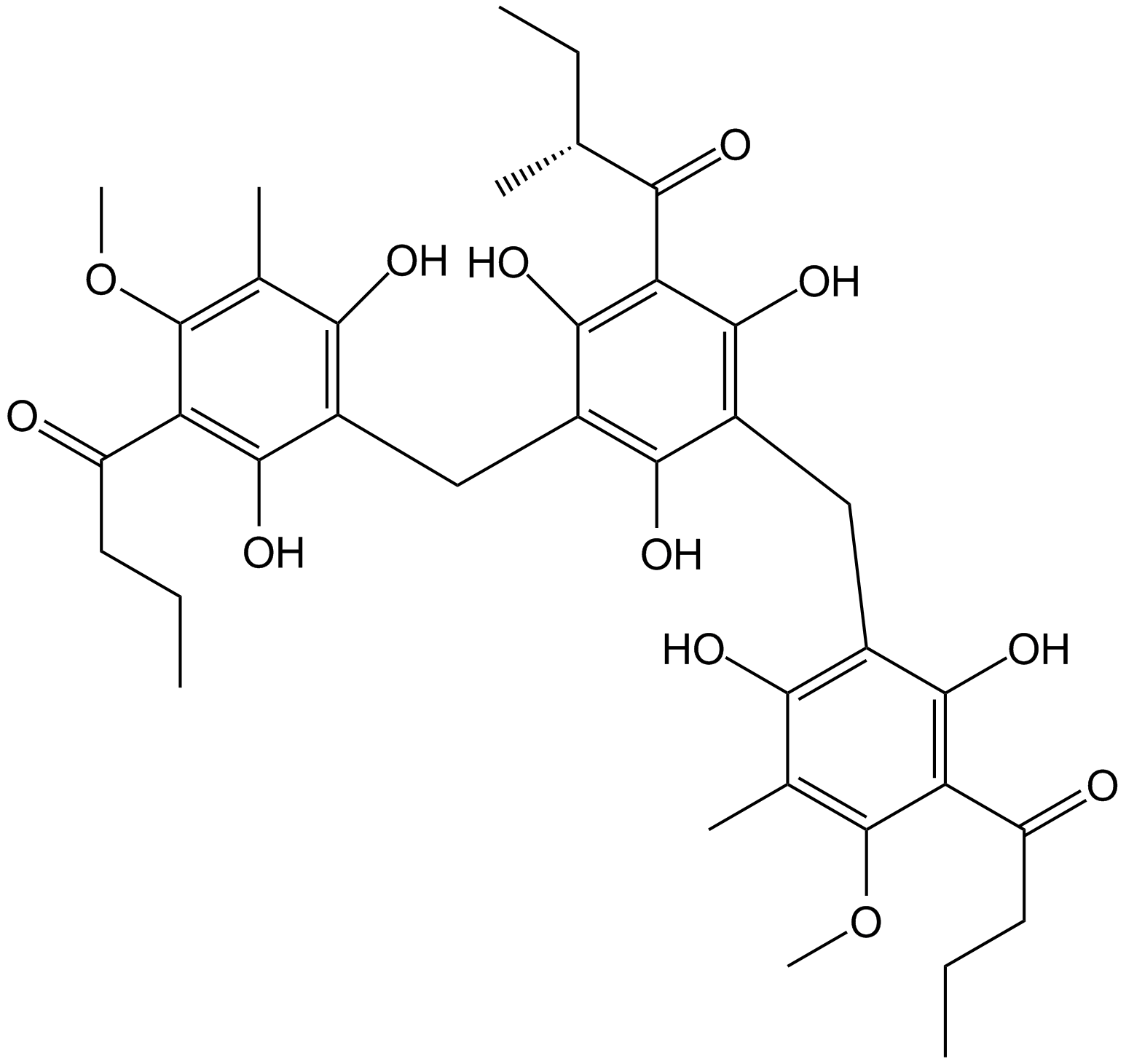
-
GC10676
AK-7
AK-7 es un inhibidor selectivo de SIRT2 permeable a las células y al cerebro, con una IC50 de 15,5 μM.
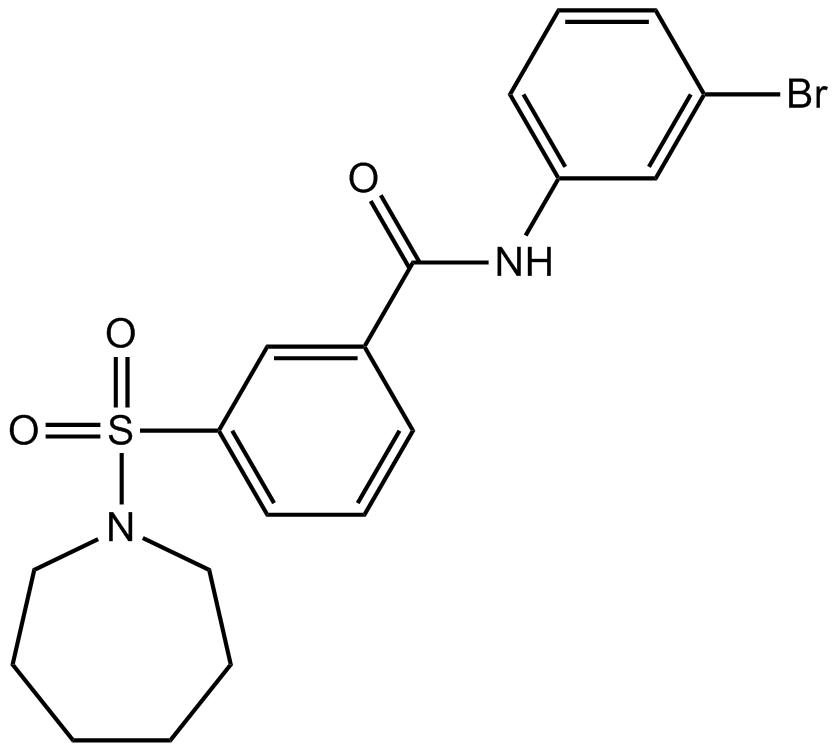
-
GC62277
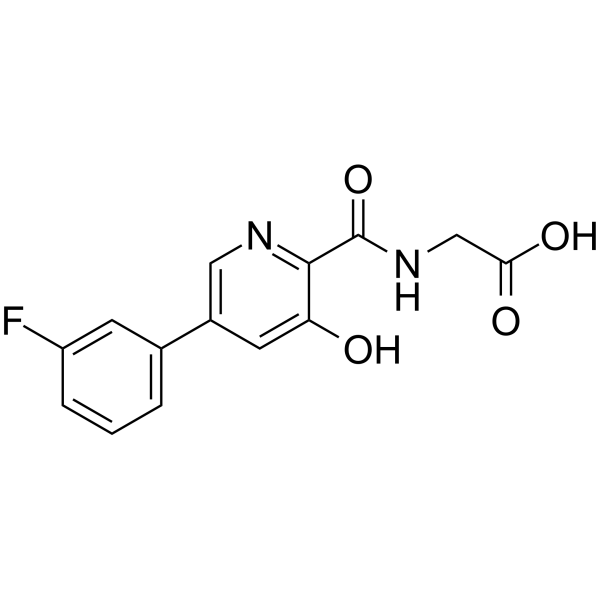
AKB-6899
Ro24-7429 es un antagonista Tat de la proteÍna transactivadora del VIH-1 potente y oralmente activo. Ro24-7429 también es un inhibidor del factor de transcripciÓn 1 relacionado con runt (RUNX1). Ro24-7429 tiene efectos anti-VIH, antifibrÓticos y antiinflamatorios.
-
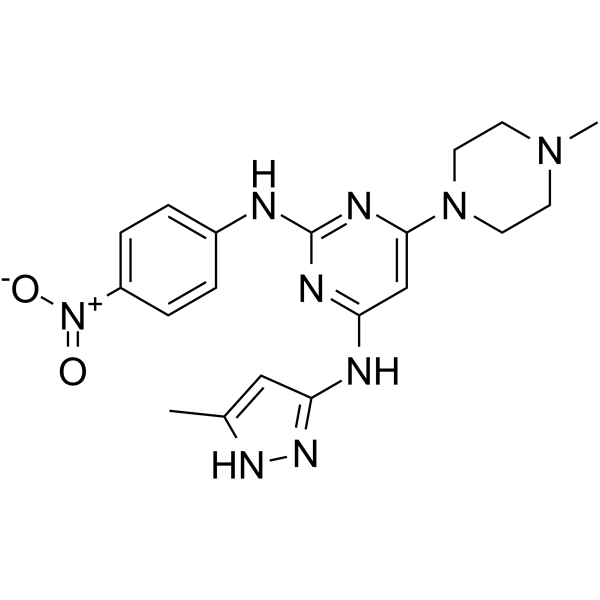
GC62433
AKI603
AKI603 es un inhibidor de Aurora quinasa A (AurA), con una IC50 de 12,3 nM. AKI603 estÁ desarrollado para superar la resistencia mediada por la mutaciÓn BCR-ABL-T315I. AKI603 exhibe una fuerte actividad antiproliferativa en células leucémicas.
-
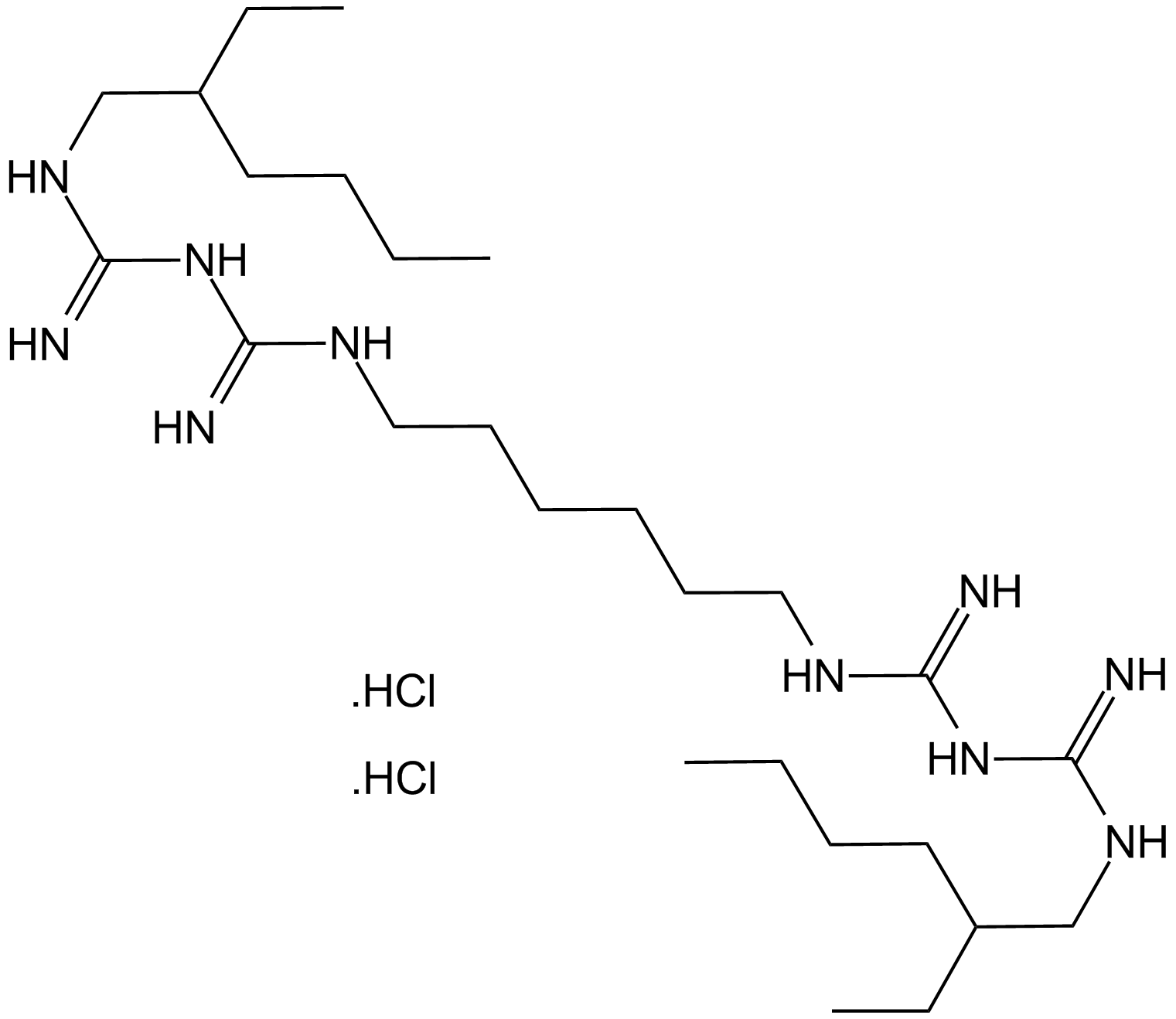
GC17344
Alexidine dihydrochloride
An alkyl bis(biguanide) antiseptic
-
GC49393
all-trans-13,14-Dihydroretinol
A metabolite of all-trans retinoic acid

-
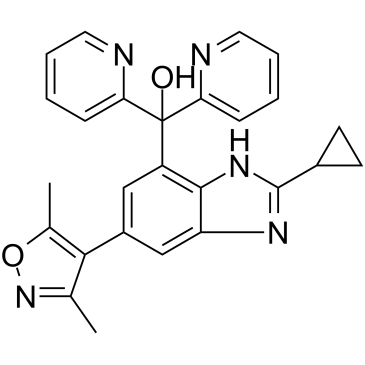
GC35297
Alobresib
Alobresib (GS-5829) es un inhibidor del bromodominio BET, que representa un agente terapéutico muy eficaz contra el carcinoma seroso uterino (USC) recurrente/resistente a la quimioterapia que sobreexpresa c-Myc.
-
GC67984
Alteminostat
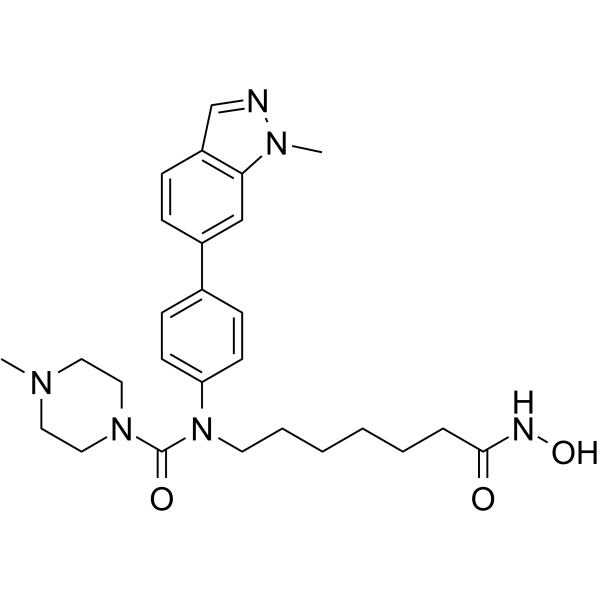
-
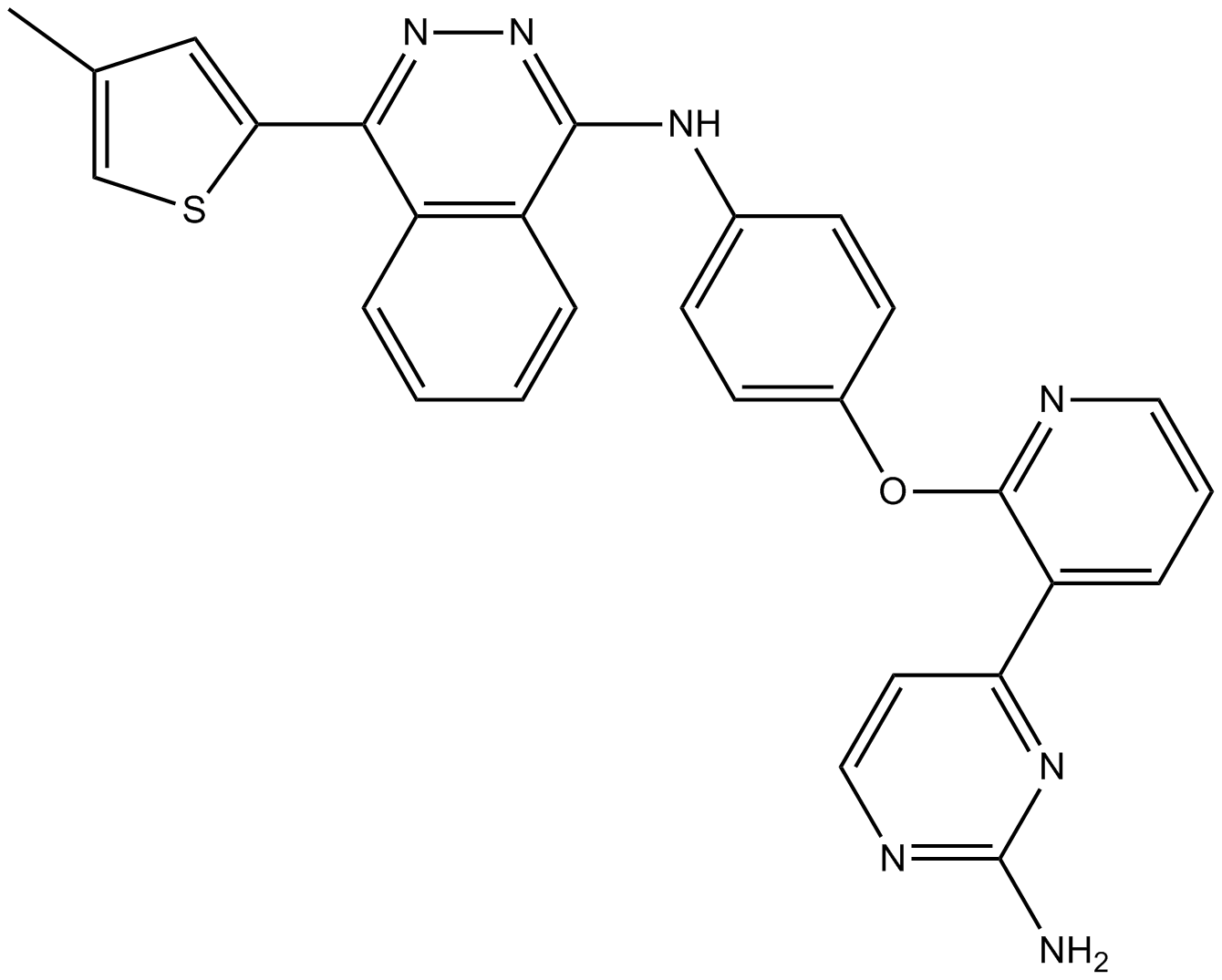
GC13198
AMG-900
A selective pan-Aurora kinase inhibitor
-
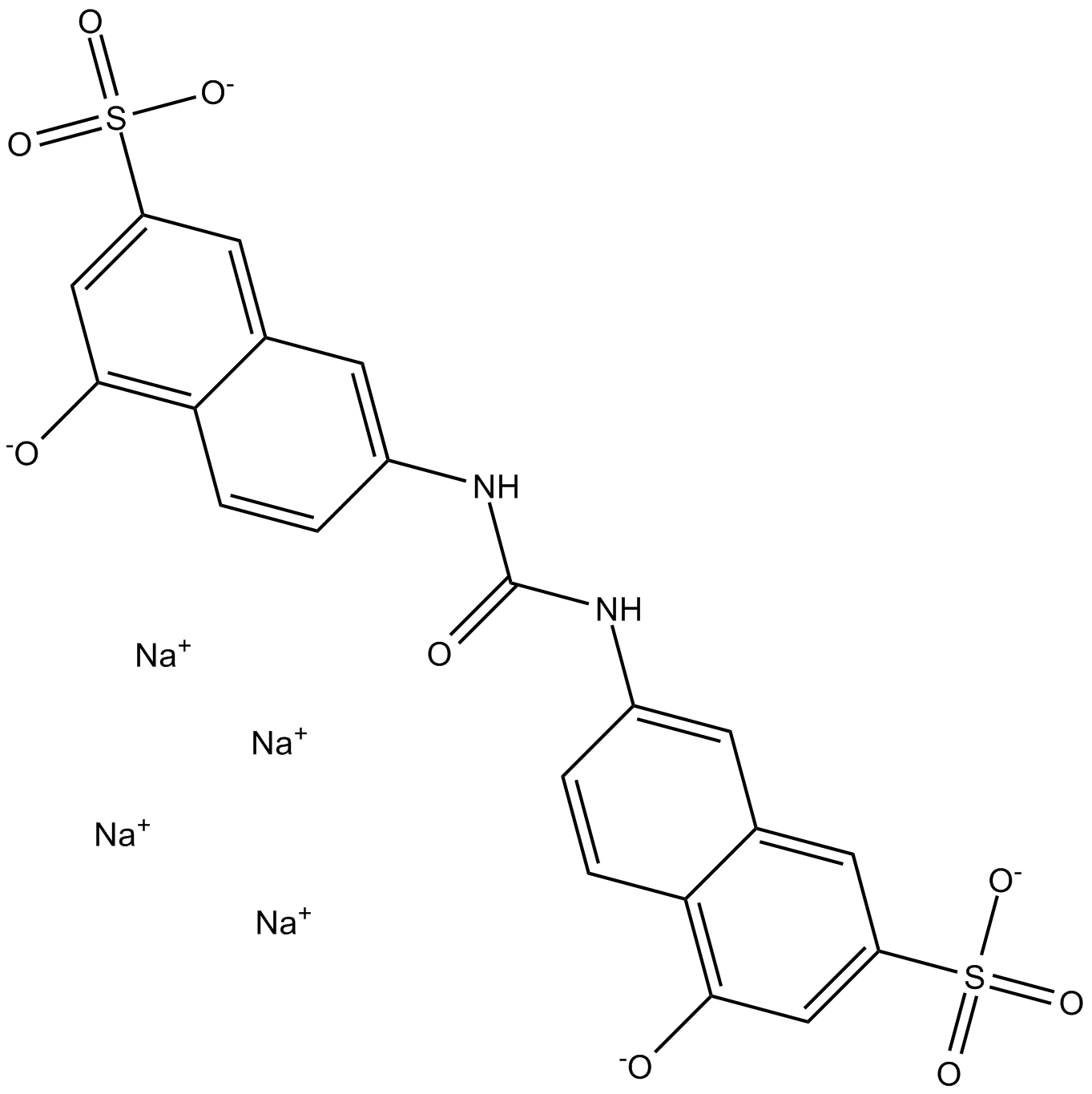
GC17275
AMI-1
A cell permeable inhibitor of PRMTs
-
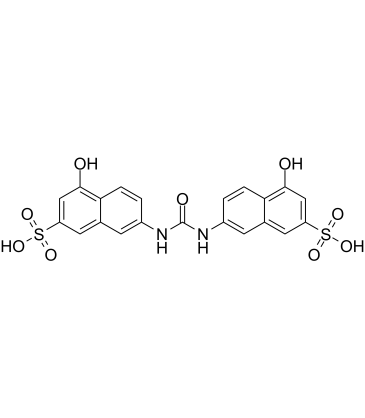
GC39840
AMI-1 free acid
El Ácido libre de AMI-1 es un inhibidor potente, permeable a las células y reversible de la proteÍna arginina N-metiltransferasas (PRMT), con IC50 de 8,8 μM y 3,0 μM para PRMT1 humano y levadura-Hmt1p, respectivamente. El Ácido libre de AMI-1 ejerce efectos inhibidores de PRMT al bloquear la uniÓn de péptido-sustrato.
-
GC17546
AMI5
La eosina Y (disÓdica) es una molécula de colorante rojo Ácido soluble.
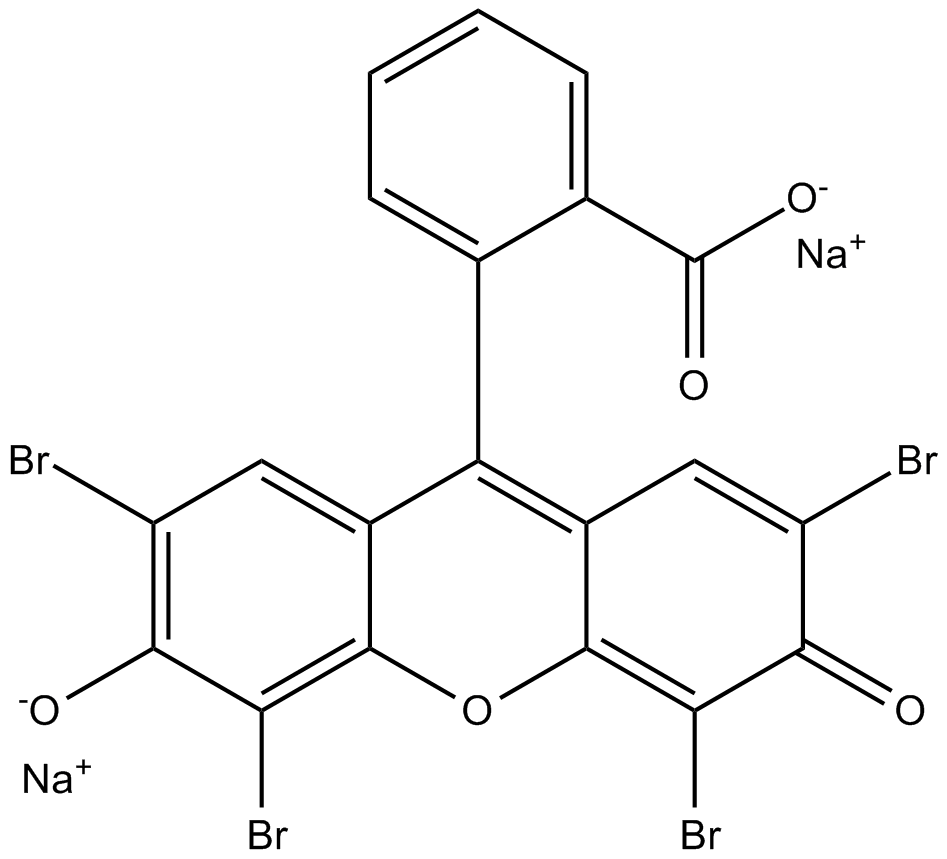
-
GC42785
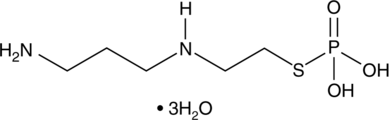
Amifostine (hydrate)
La amifostina (hidrato) (WR2721 trihidrato) es un agente citoprotector de amplio espectro y un radioprotector. La amifostina (hidrato) protege selectivamente los tejidos normales del daÑo causado por la radiaciÓn y la quimioterapia. La amifostina (hidrato) es un potente factor inducible por hipoxia-α1 (HIF-α1) e inductor de p53. La amifostina (hidrato) protege las células del daÑo al eliminar los radicales libres derivados del oxÍgeno. La amifostina (hidrato) reduce la toxicidad renal y tiene acciÓn antiangiogénica.
-
GC42790
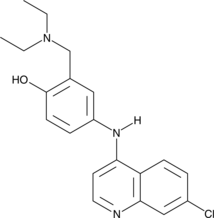
Amodiaquine
Amodiaquina es un compuesto antipalúdico aminoquinolínico.
-
GC60579
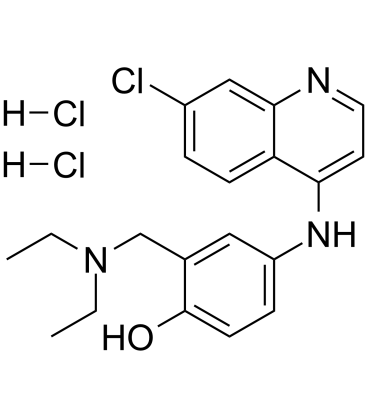
Amodiaquine dihydrochloride
El diclorhidrato de amodiaquina (diclorhidrato de amodiaquina), una clase de agente antipalÚdico de 4-aminoquinolina, es un inhibidor potente y activo por vÍa oral de la histamina N-metiltransferasa con una Ki de 18,6 nM.
-
GC10905
Amodiaquine dihydrochloride dihydrate
El dihidrocloruro de amodiaquina dihidrato (Dihidrocloruro de amodiaquina dihidrato), una clase de 4-aminoquinolina de agente antipalÚdico, es un inhibidor potente y activo por vÍa oral de la histamina N-metiltransferasa.
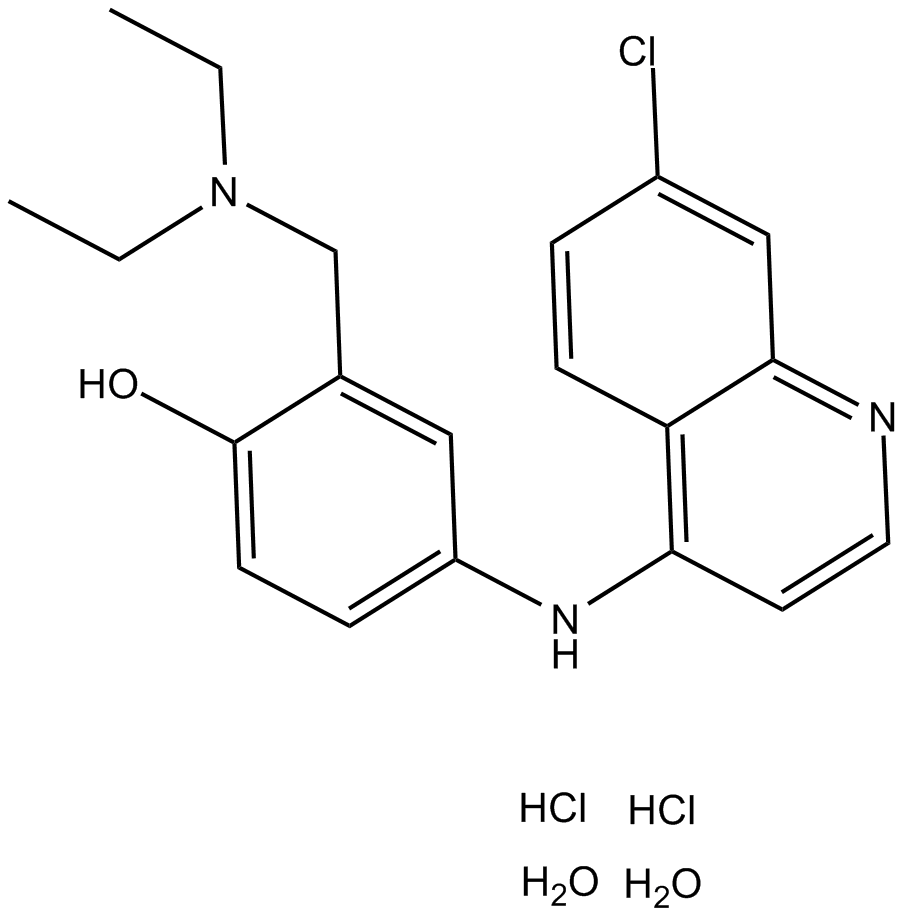
-
GC67912
Amredobresib

-
GC17284
Anacardic acid
A histone acetyltransferase inhibitor
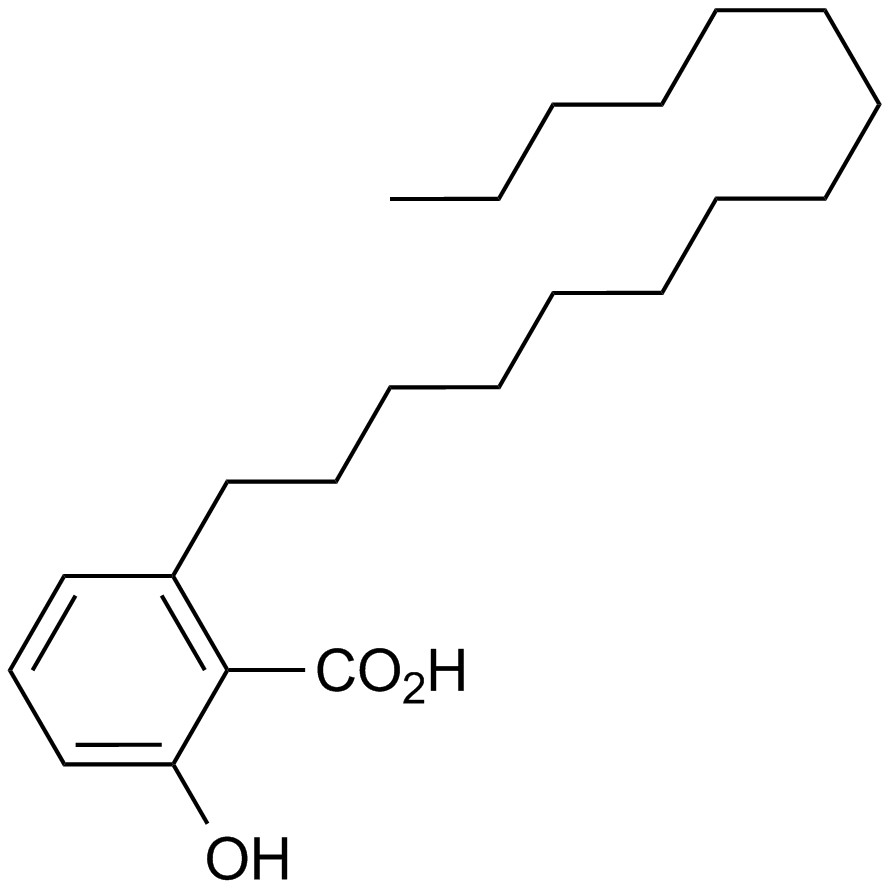
-
GC40674
APHA Compound 8
A class I and II HDAC inhibitor
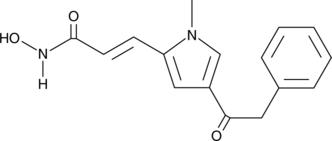
-
GC12961
Apicidin
Un inhibidor de HDAC permeable a través de la membrana celular.
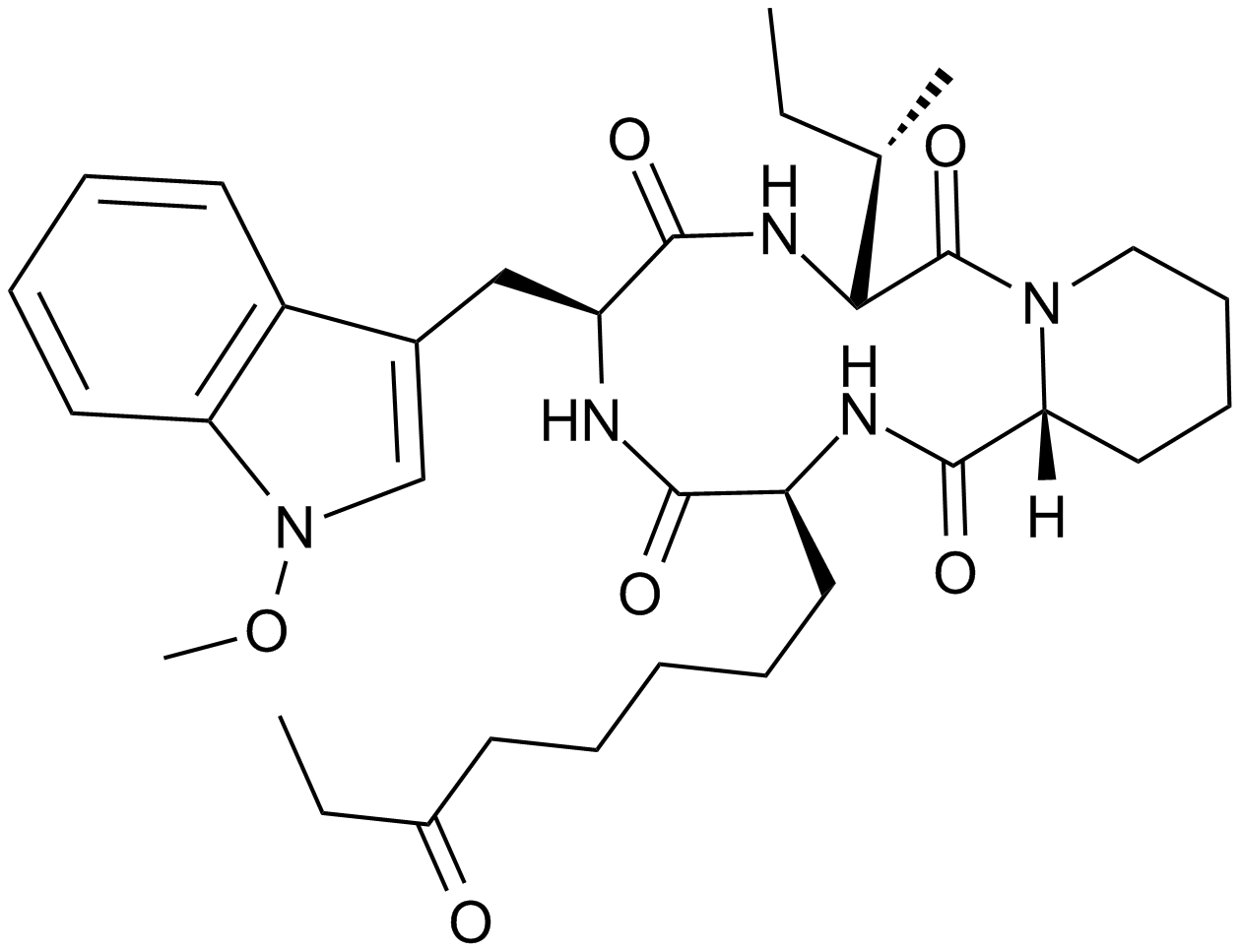
-
GC14590
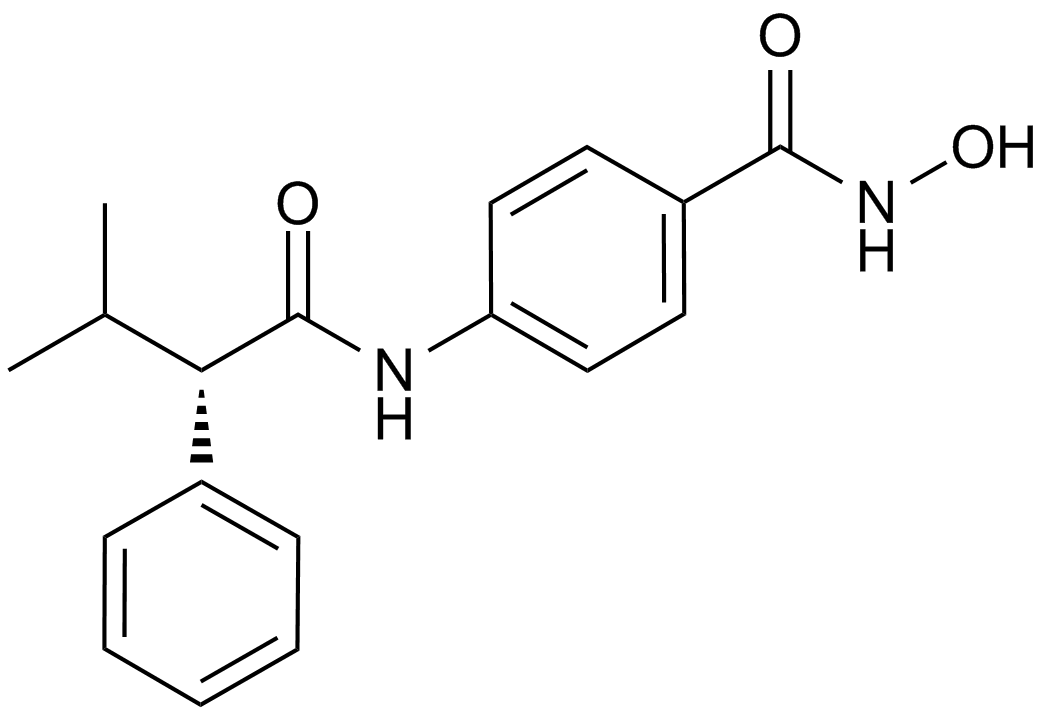
AR-42 (OSU-HDAC42)
HDAC inhibitor,novel and potent
-
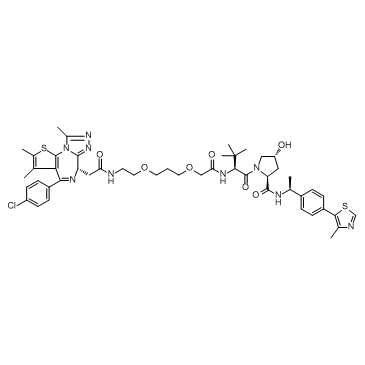
GC32685
ARV-771
ARV-771 es un potente BET PROTAC basado en E3 ligasa von Hippel-Lindau con Kds de 34 nM, 4,7 nM, 8,3 nM, 7,6 nM, 9,6 nM y 7,6 nM para BRD2(1), BRD2(2), BRD3( 1), BRD3(2), BRD4(1) y BRD4(2), respectivamente.
-
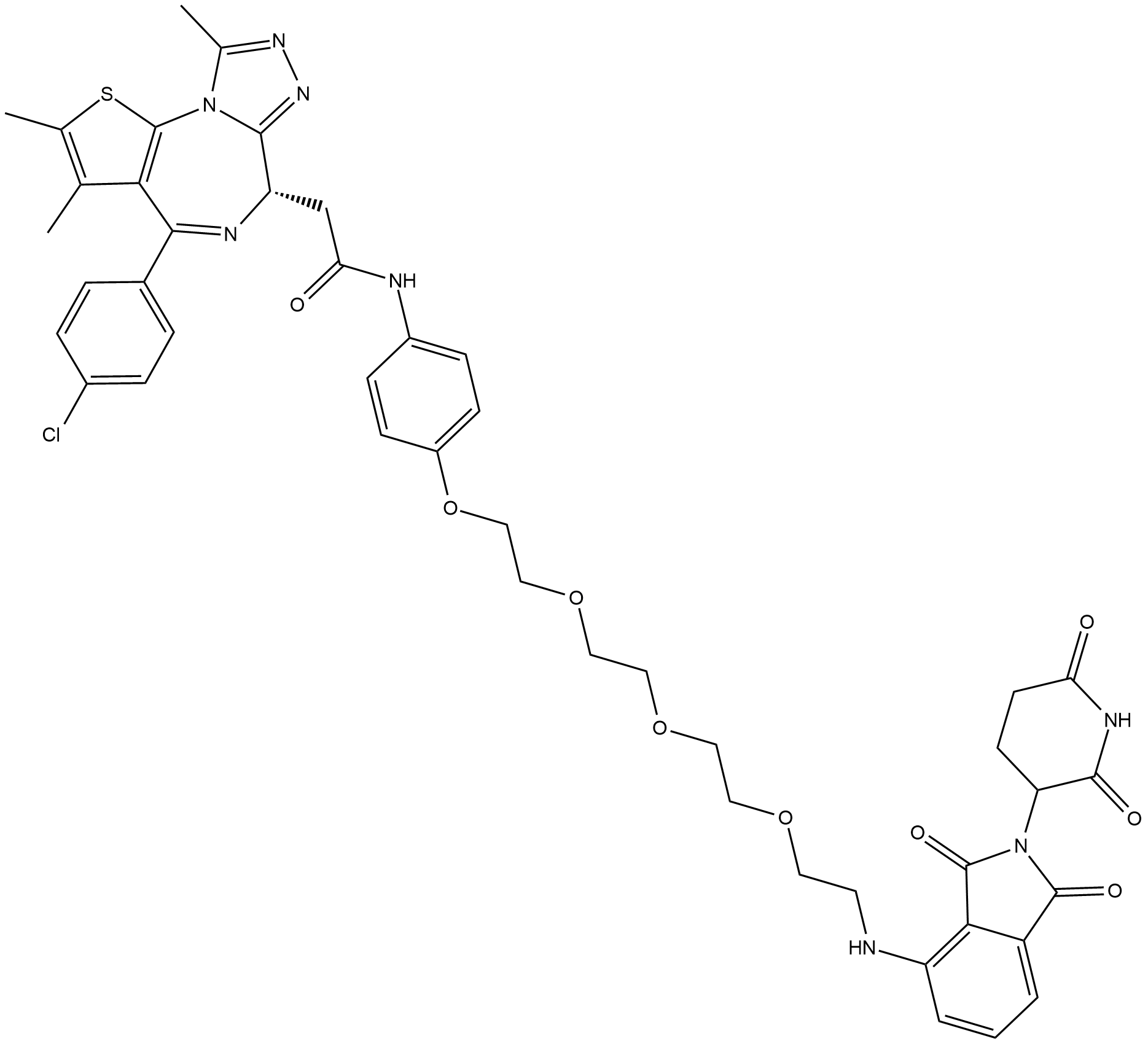
GC19038
ARV-825
ARV-825 es un PROTAC conectado por ligandos para Cereblon y BRD4. ARV-825 se une a BD1 y BD2 de BRD4 con Kds de 90 y 28 nM, respectivamente.
-
GC65918
AS-85
AS-85 es un potente inhibidor de la histona metiltransferasa ASH1L (IC50=0,6 μM) con actividad antileucémica. AS-85 se une fuertemente al dominio SET ASH1L, con el valor Kd de 0.78μM.
-
GC62615
AS-99
AS-99 es un inhibidor de histona metiltransferasa ASH1L selectivo, potente y de primera clase (IC50 = 0,79μ M, Kd = 0,89μ M) con actividad antileucémica. AS-99 bloquea la proliferaciÓn celular, induce la apoptosis y la diferenciaciÓn, regula a la baja los genes diana de la fusiÓn MLL y reduce la carga de leucemia in vivo.
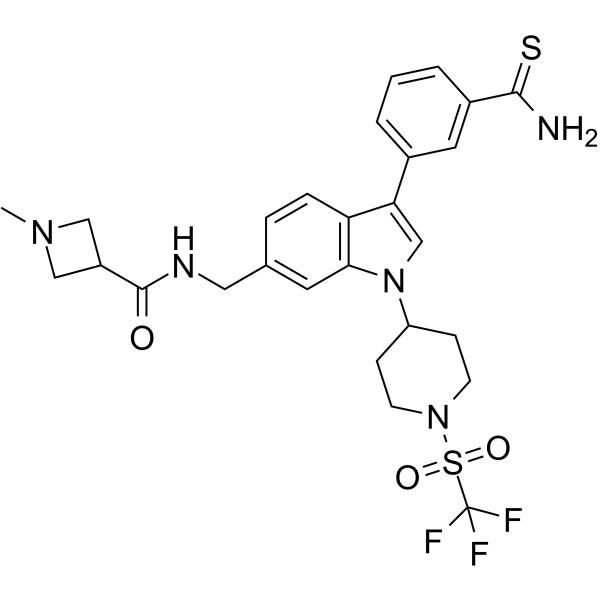
-
GC62849
AS-99 TFA
AS-99 TFA es un inhibidor de histona metiltransferasa ASH1L selectivo, potente y de primera clase (IC50=0,79μM, Kd=0,89μM) con actividad antileucémica. AS-99 TFA bloquea la proliferaciÓn celular, induce la apoptosis y la diferenciaciÓn, regula a la baja los genes diana de la fusiÓn MLL y reduce la carga de leucemia in vivo.
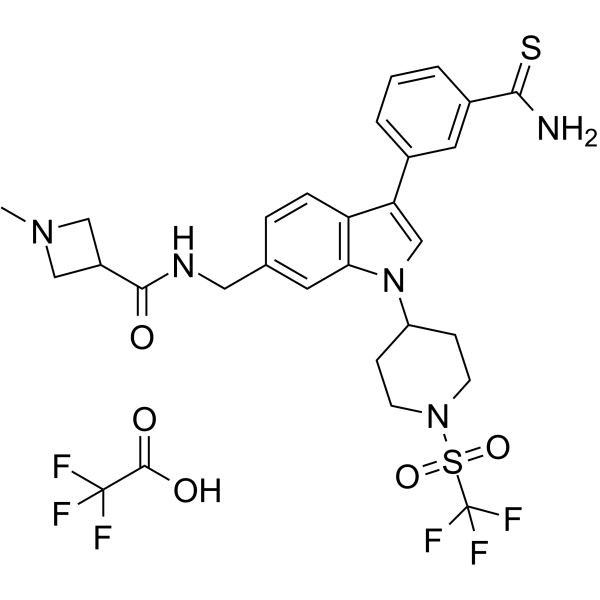
-
GC17820
AS8351
AS8351 (NSC51355) es un inhibidor de KDM5B, que puede inducir y mantener marcas activas de cromatina para facilitar la inducciÓn de células similares a cardiomiocitos.
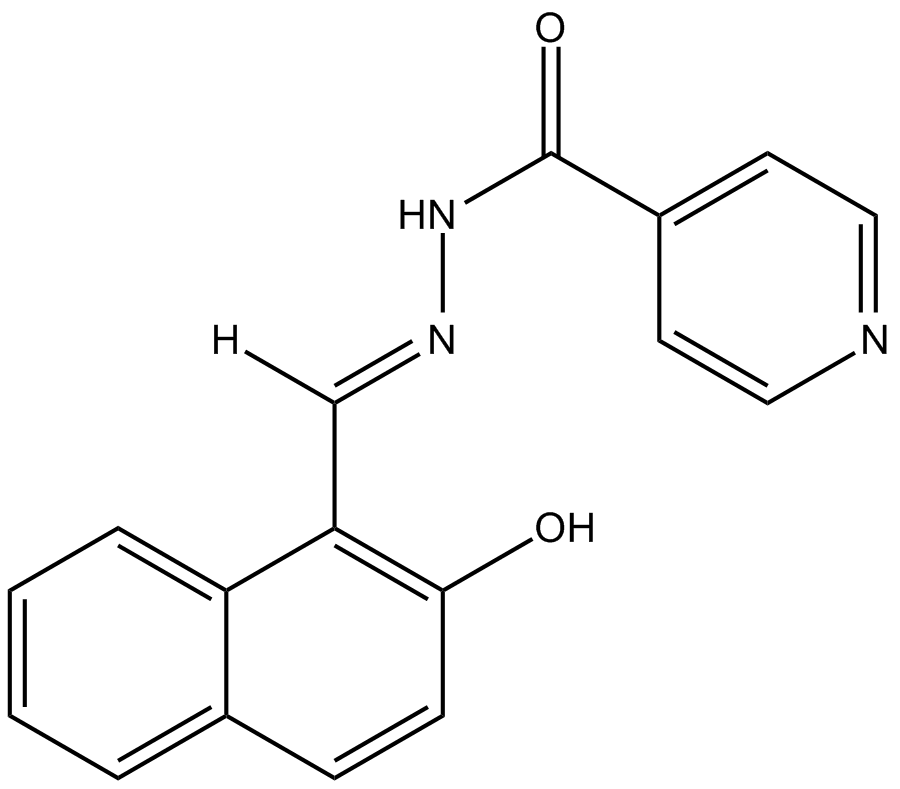
-
GC10638
AT9283
A broad spectrum kinase inhibitor
-
GC50066
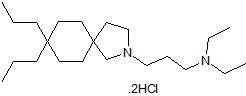
Atiprimod dihydrochloride
JAK2 inhibitor
-
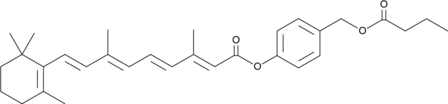
GC46892
ATRA-BA Hybrid
A prodrug form of all-trans retinoic acid and butyric acid
-
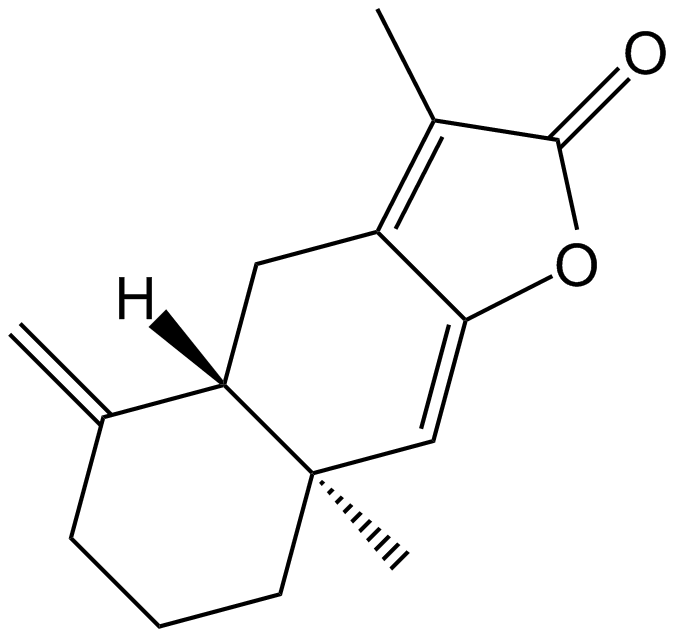
GN10627
Atractylenolide I
-
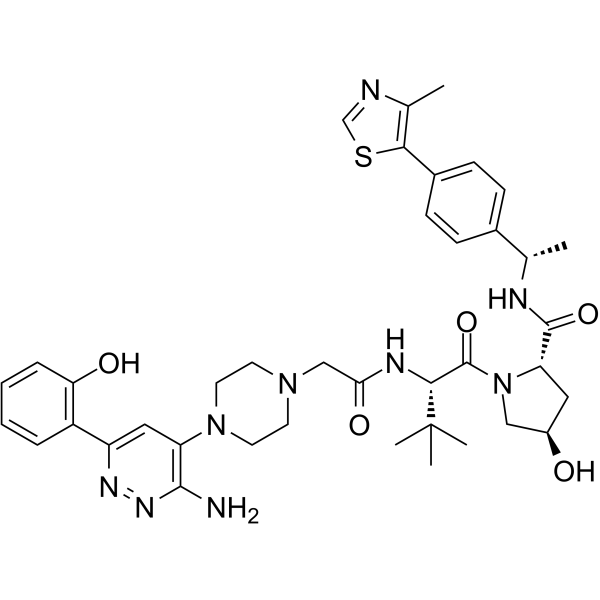
GC64271
AU-15330
AU-15330 es un degradador de quimera dirigida a proteÓlisis (PROTAC) de las subunidades SWI/SNF ATPasa, SMARCA2 y SMARCA4. AU-15330 induce una potente inhibiciÓn del crecimiento tumoral en modelos de xenoinjerto de cÁncer de prÓstata y actÚa sinérgicamente con el antagonista AR enzalutamida. AU-15330 induce la remisiÓn de la enfermedad en modelos de cÁncer de prÓstata resistente a la castraciÓn (CPRC) sin toxicidad.
-
GC39699
Aurintricarboxylic acid
El Ácido aurintricarboxÍlico es un antagonista alostérico de potencia nanomolar con selectividad hacia los P2X1R y P2X3R sensibles a αβ-metileno-ATP, con IC50 de 8,6 nM y 72,9 nM para rP2X1R y rP2X3R, respectivamente.
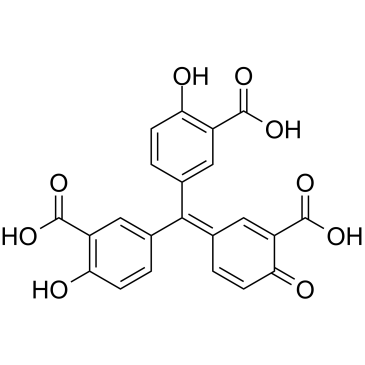
-
GC13332
Aurora A Inhibitor I
A potent and selective inhibitor of Aurora A kinase

-
GC33379
Aurora B inhibitor 1
El inhibidor 1 de Aurora B es un inhibidor de Aurora B (Aurora-1) extraÍdo de la patente WO2007059299A1, compuesto 1-3, tiene un valor Ki de <0,010 uM.
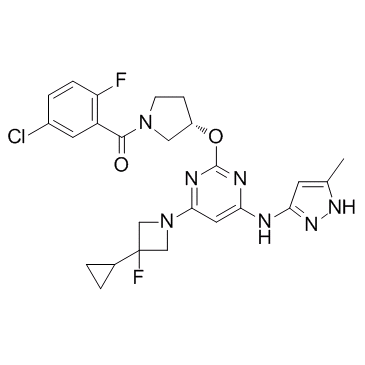
-
GC38441
Aurora Kinase Inhibitor 3
A potent inhibitor of Aurora A kinase
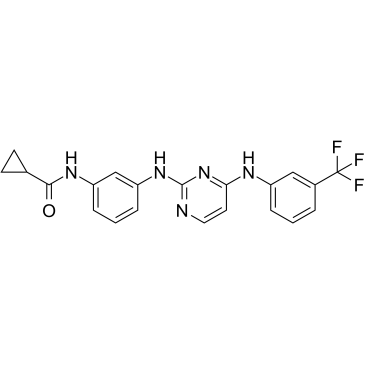
-
GC40667
Aurora Kinase Inhibitor II
Aurora Kinase Inhibitor II es un inhibidor de la cinasa Aurora selectivo y competitivo con ATP con IC50 de 310 nM y 240 nM para Aurora A y Aurora B, respectivamente.
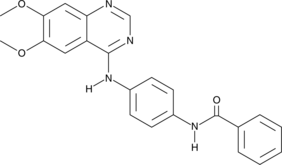
-
GC15711
Aurora Kinase Inhibitor III
Aurora Kinase Inhibitor III es un inhibidor fuerte y selectivo de la cinasa Aurora A con una IC50 de 42 nM e inhibe débilmente el EGFR con una IC50 de >10 μM.
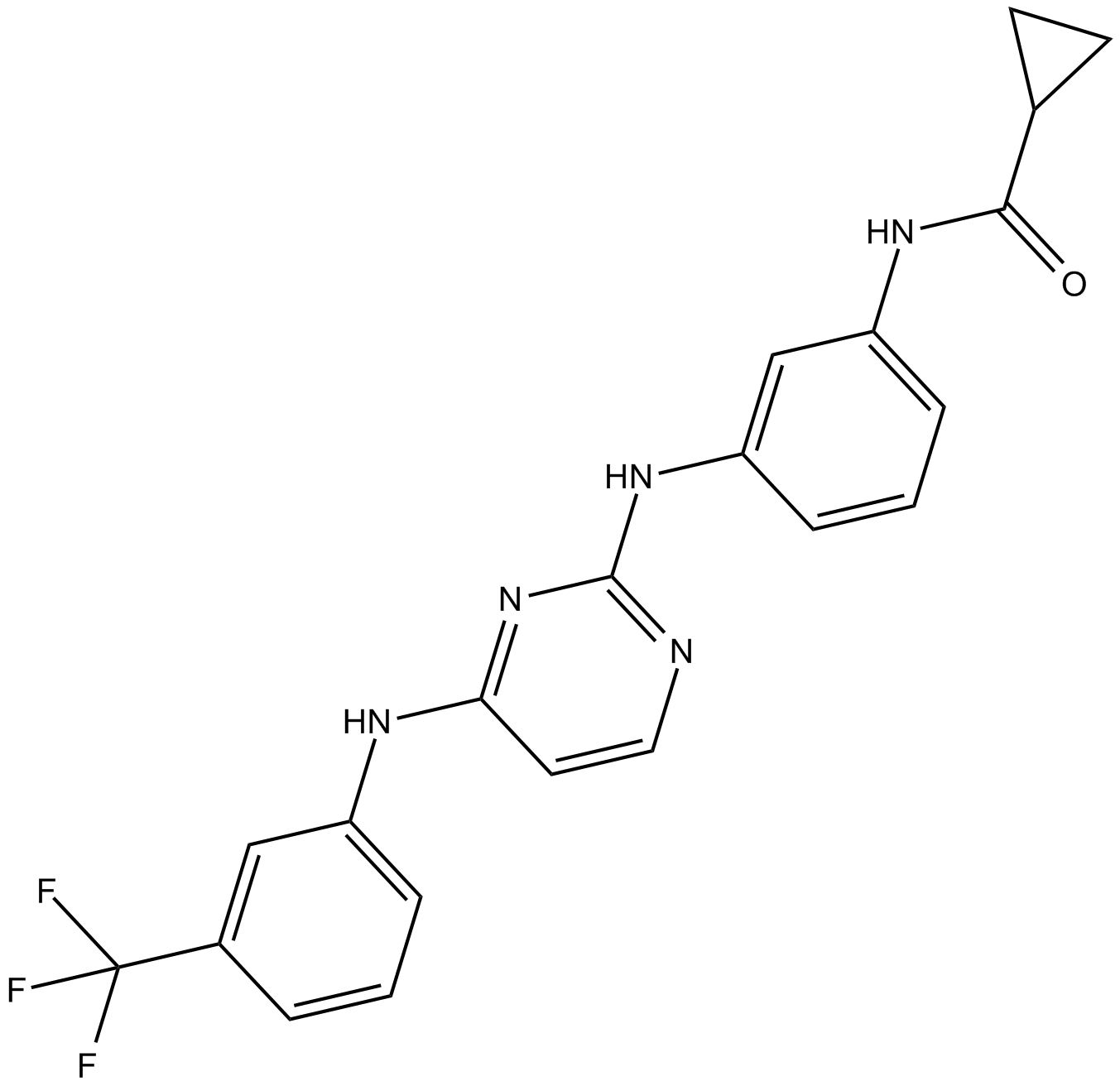
-
GC67899
Aurora kinase inhibitor-8

-
GC13433
AZ 960
A JAK2 inhibitor
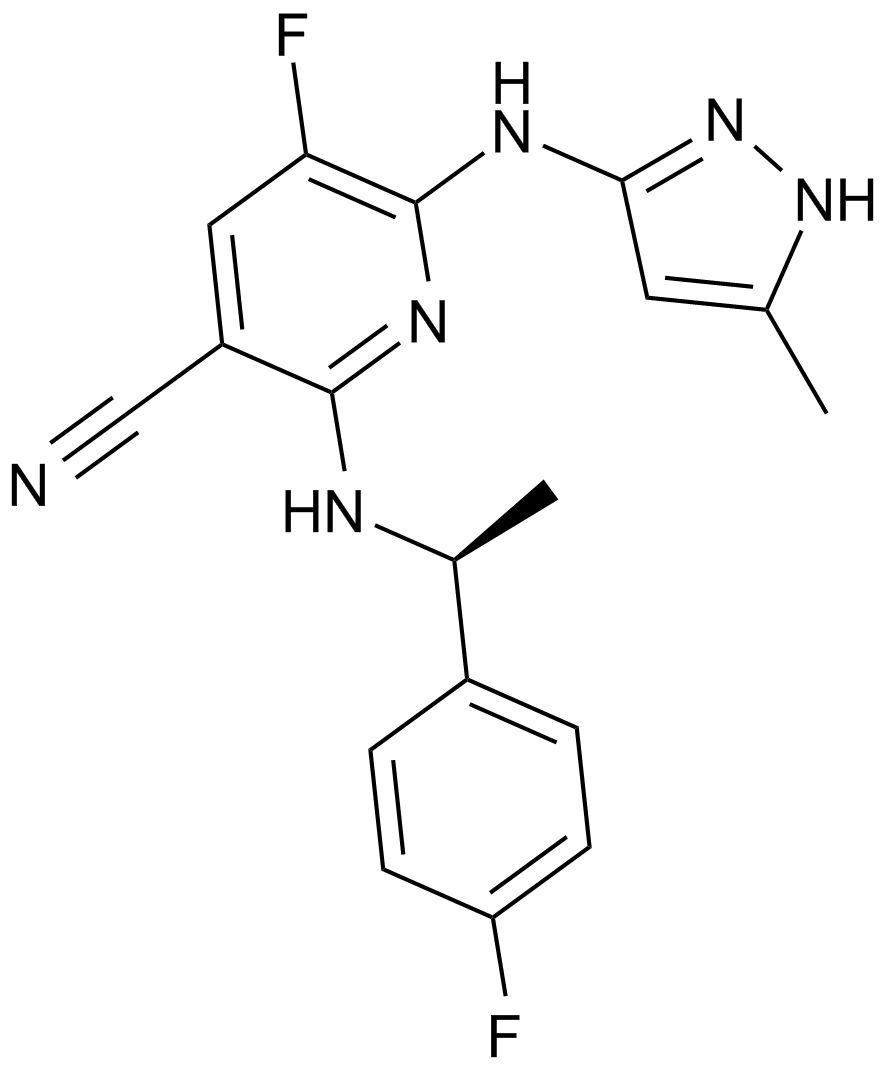
-
GC65899
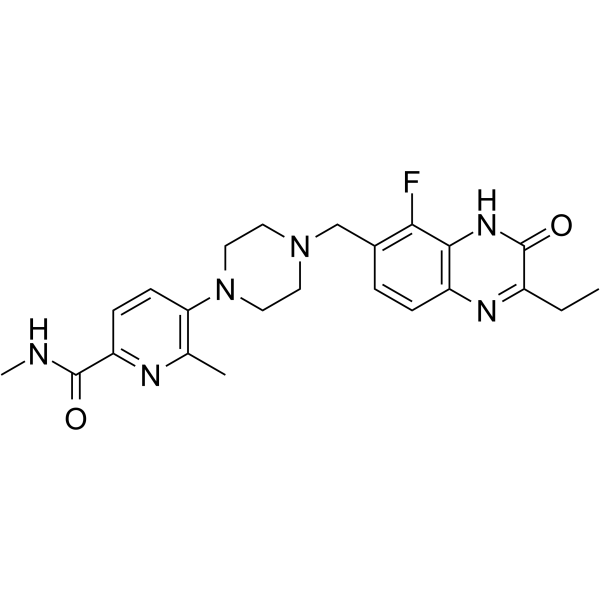
AZ3391
AZ3391 es un potente inhibidor de PARP. AZ3391 es un derivado de la quinoxalina. La familia de enzimas PARP desempeÑa un papel importante en una serie de procesos celulares, como la replicaciÓn, la recombinaciÓn, la remodelaciÓn de la cromatina y la reparaciÓn del daÑo del ADN. AZ3391 tiene el potencial para la investigaciÓn de enfermedades y condiciones que ocurren en los tejidos del sistema nervioso central, como el cerebro y la médula espinal (extraÍdo de la patente WO2021260092A1, compuesto 23).
-
GC13744
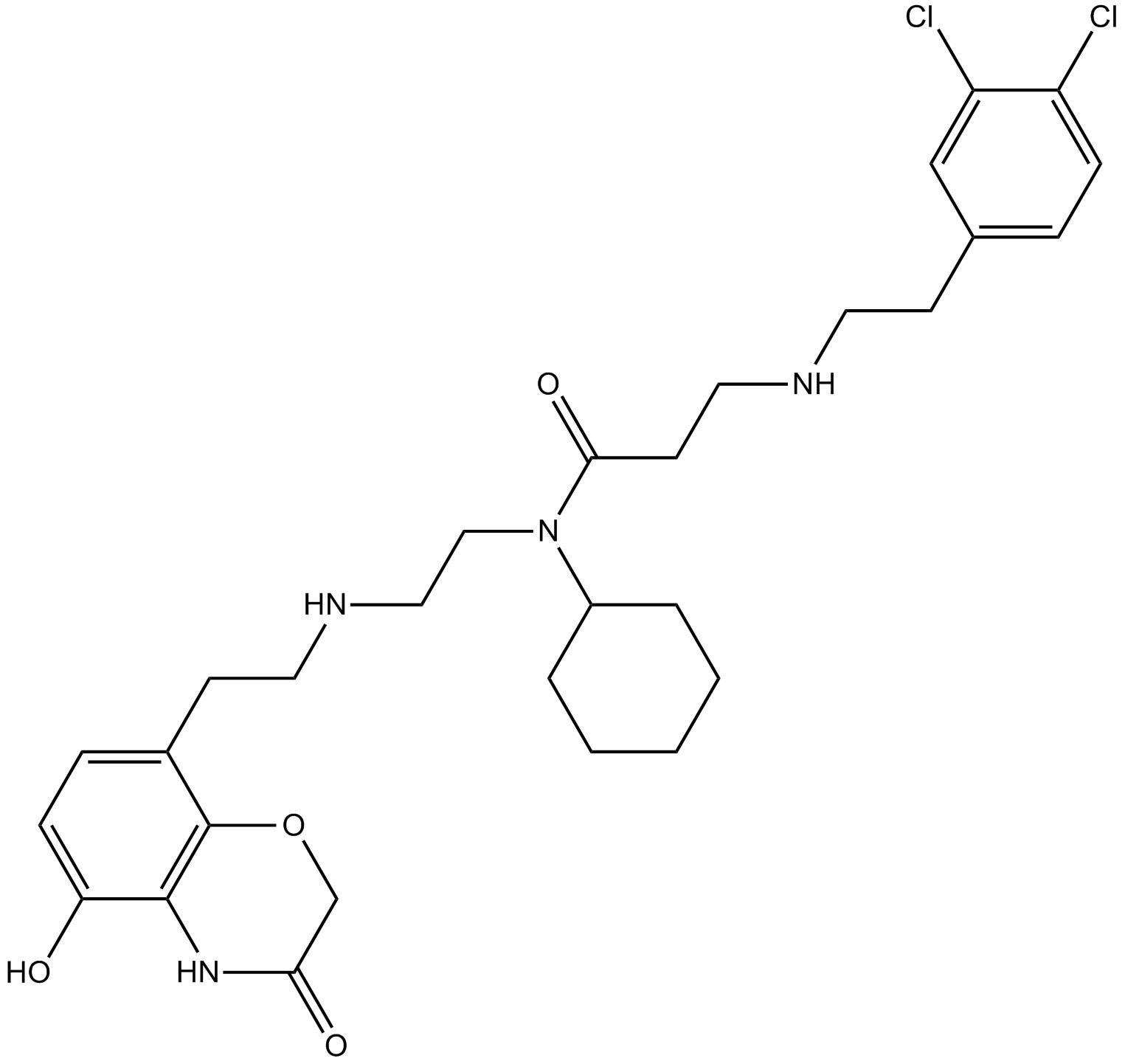
AZ505
AZ505 es un inhibidor potente y selectivo de SMYD2 con una IC50 de 0,12 μM.
-
GC13103
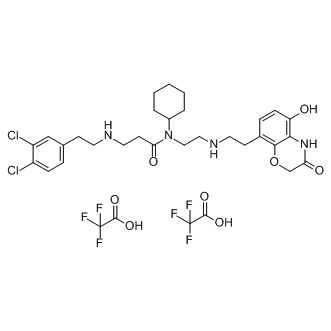
AZ505 ditrifluoroacetate
El ditrifluoroacetato AZ505 es un inhibidor potente y selectivo de SMYD2 con IC50 de 0,12 μM.
-
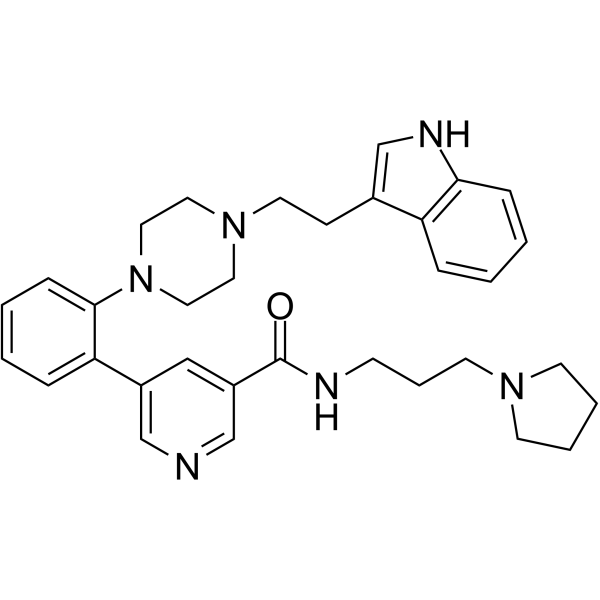
GC64124
AZ506
AZ506 es un potente inhibidor de SMYD2 con una IC50 de 17 nM. AZ506 inhibe la actividad de la metiltransferasa de SMYD2 en las células, lo que provoca una disminuciÓn de la seÑal de metilaciÓn mediada por SMYD2.
-
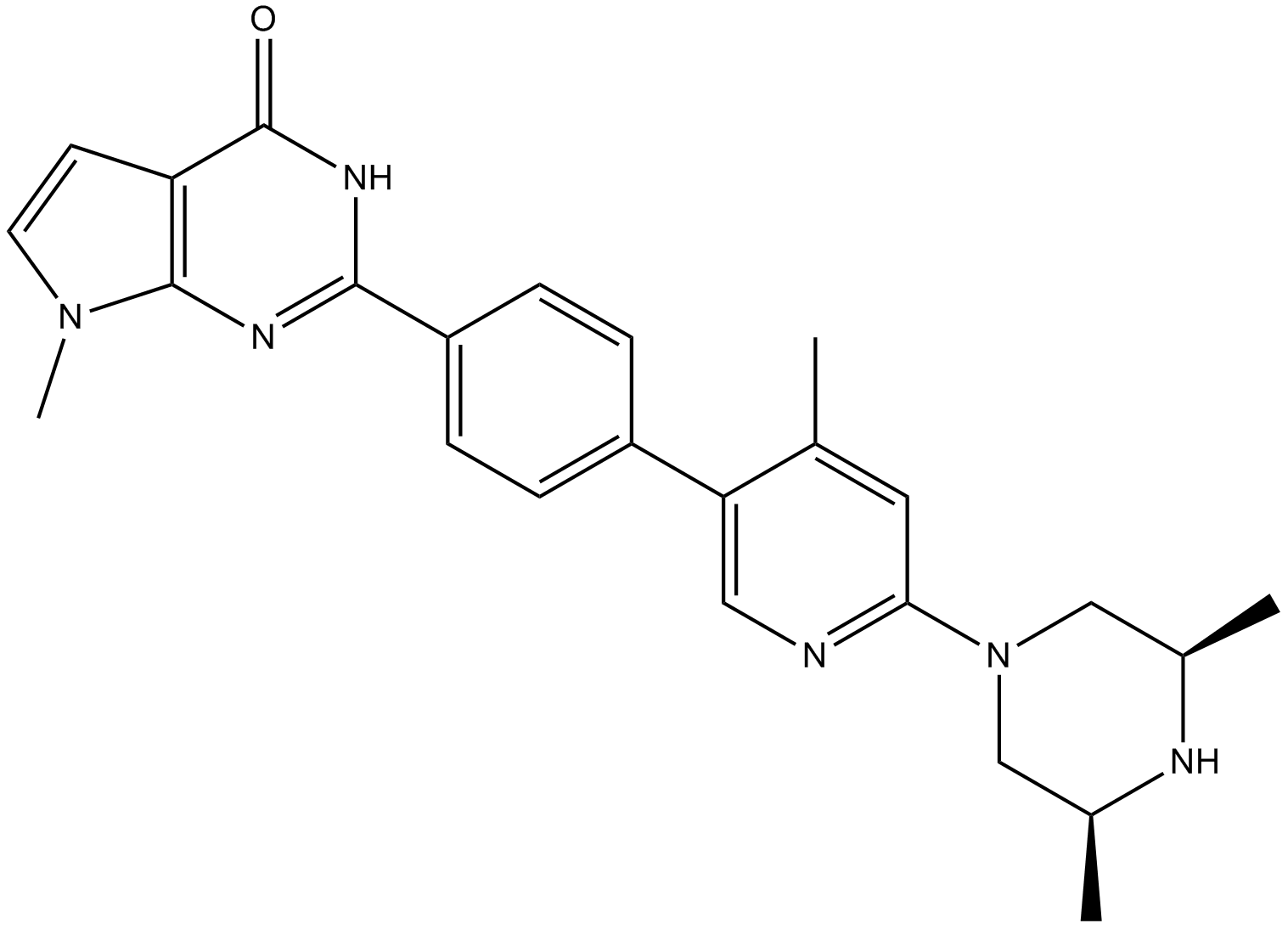
GC16725
AZ6102
AZ6102 es un potente inhibidor doble de TNKS1 y TNKS2, con IC50 de 3 nM y 1 nM, respectivamente, y tiene una selectividad 100 veces mayor frente a otras enzimas de la familia PARP, con IC50 de 2,0 μM, 0,5 μM y >3 μM, para PARP1 , PARP2 y PARP6, respectivamente.
-
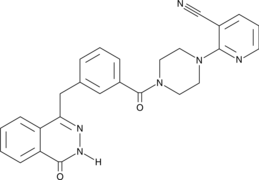
GC46900
AZ9482
AZ9482 es un triple inhibidor de PARP1/2/6, con valores IC50 de 1 nM, 1 nM y 640 nM para PARP1, PARP2 y PARP6, respectivamente.
-
GC48971
AZD 1152 (hydrochloride)
A prodrug for a potent Aurora B inhibitor
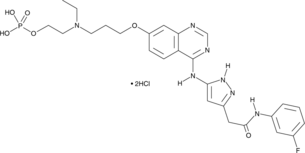
-
GC13014
AZD 5153
orally available, bivalent inhibitor of the bromodomain and extraterminal (BET) protein BRD4.
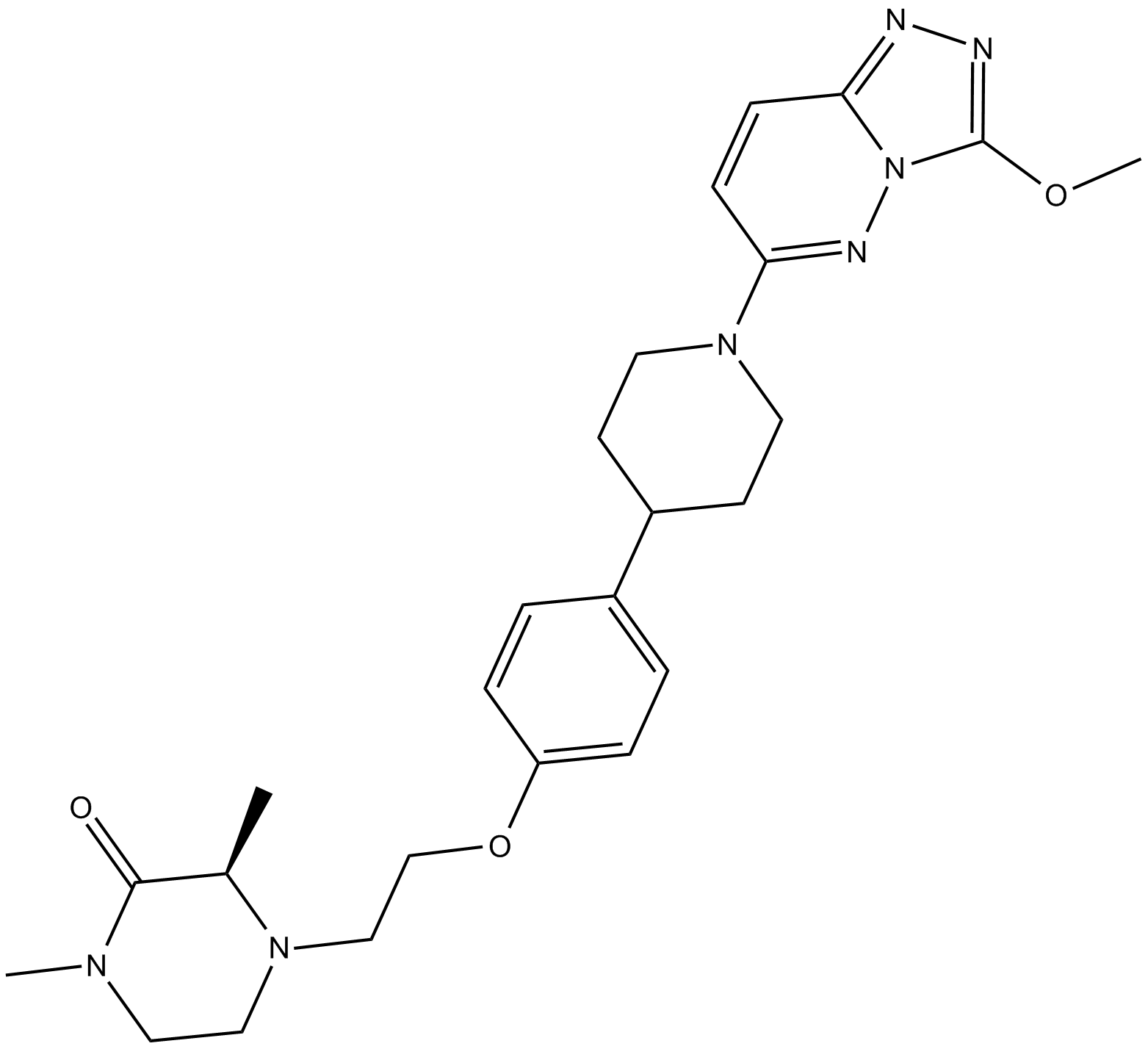
-
GC14709
AZD1152
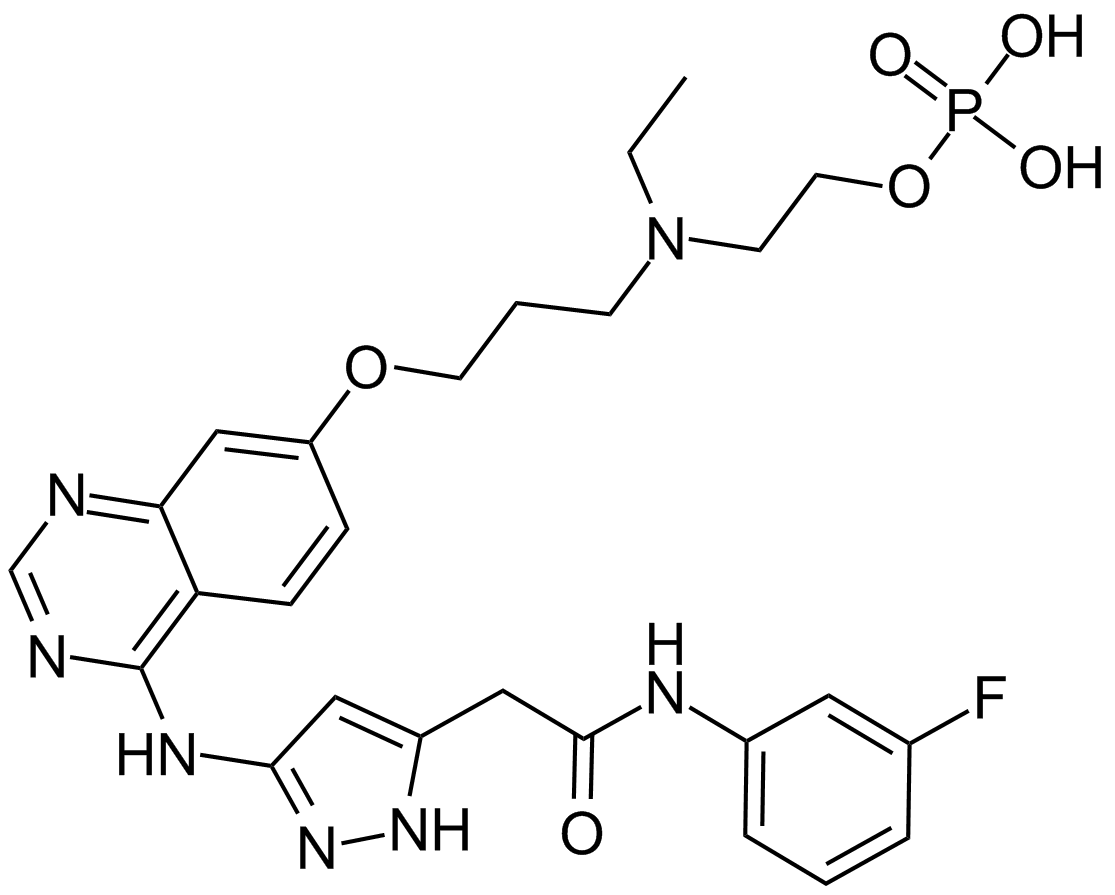
-
GC12660
AZD1208
A pan-Pim kinase inhibitor
-
GC35447
AZD1208 hydrochloride
El clorhidrato de AZD1208 es un inhibidor de cinasas PIM altamente selectivo biodisponible por vÍa oral.
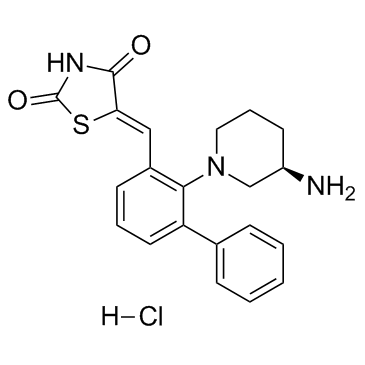
-
GC12504
AZD1480
A potent JAK2 inhibitor
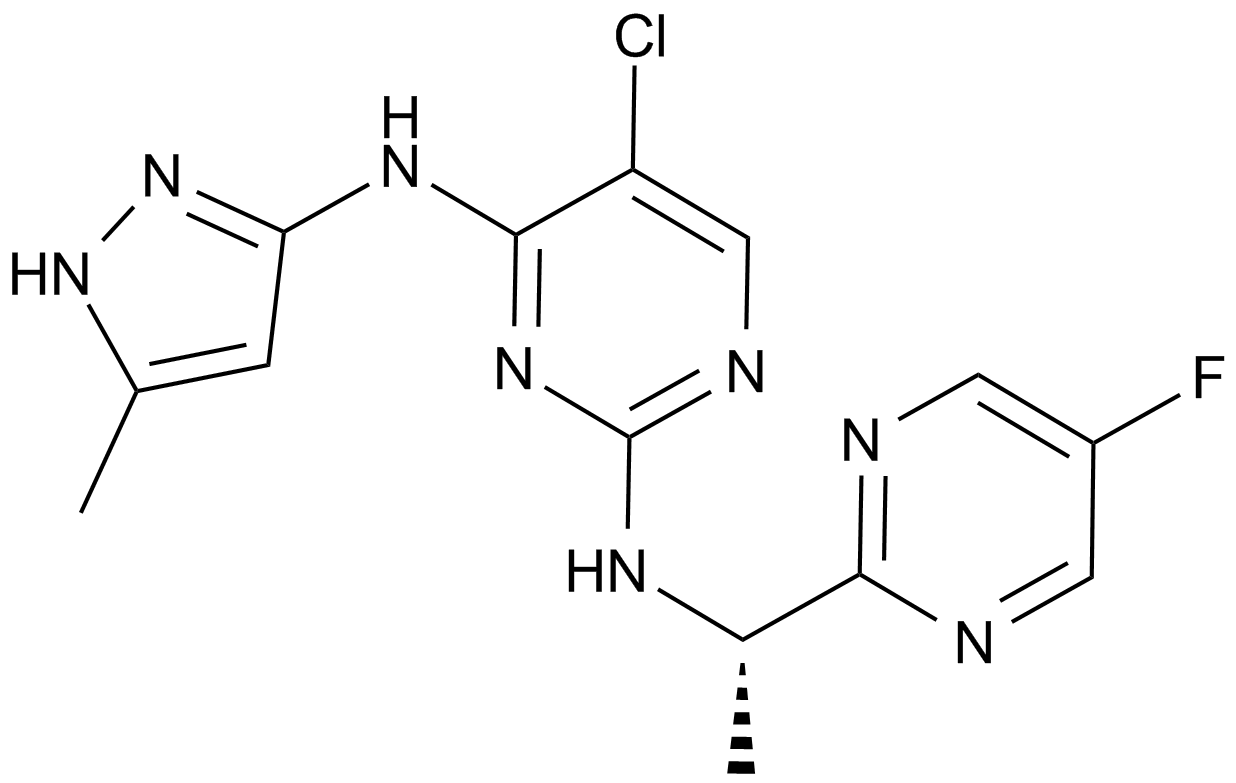
-
GC17965
AZD2461
A PARP inhibitor
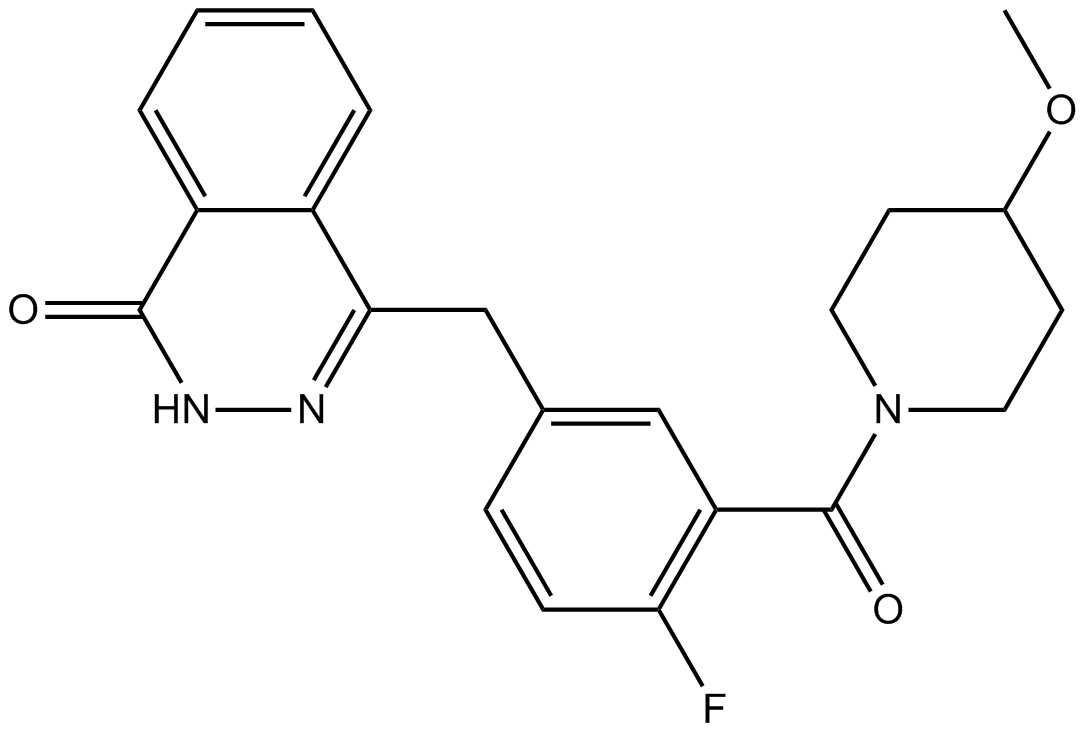
-
GC35448
AZD5153 6-Hydroxy-2-naphthoic acid
El Ácido 6-hidroxi-2-naftoico AZD5153 es el Ácido 6-hidroxi-2-naftoico de AZD5153. AZD5153 es un inhibidor de bromodominio BET/BRD4 potente, selectivo y disponible por vÍa oral; interrumpe BRD4 con un IC50 de 1,7 nM.
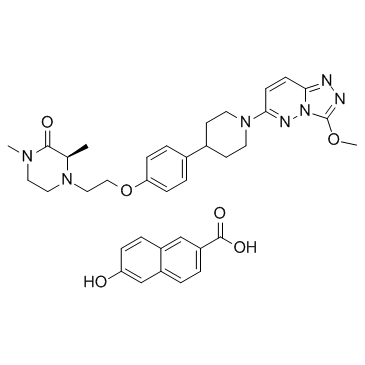
-
GC62310
AZD5305
AZD5305 es un inhibidor de PARP potente, selectivo y activo por vÍa oral. AZD5305 es potente y eficaz en xenoinjertos animales y modelos PDX.
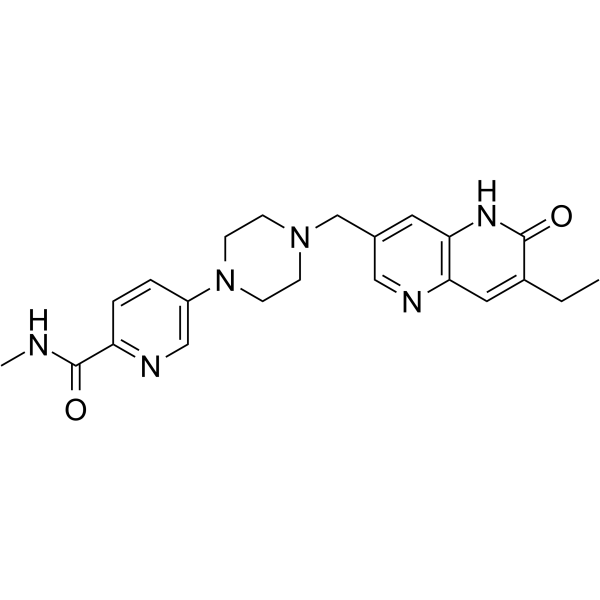
-
GC14955
Barasertib (AZD1152-HQPA)
A selective Aurora kinase B inhibitor
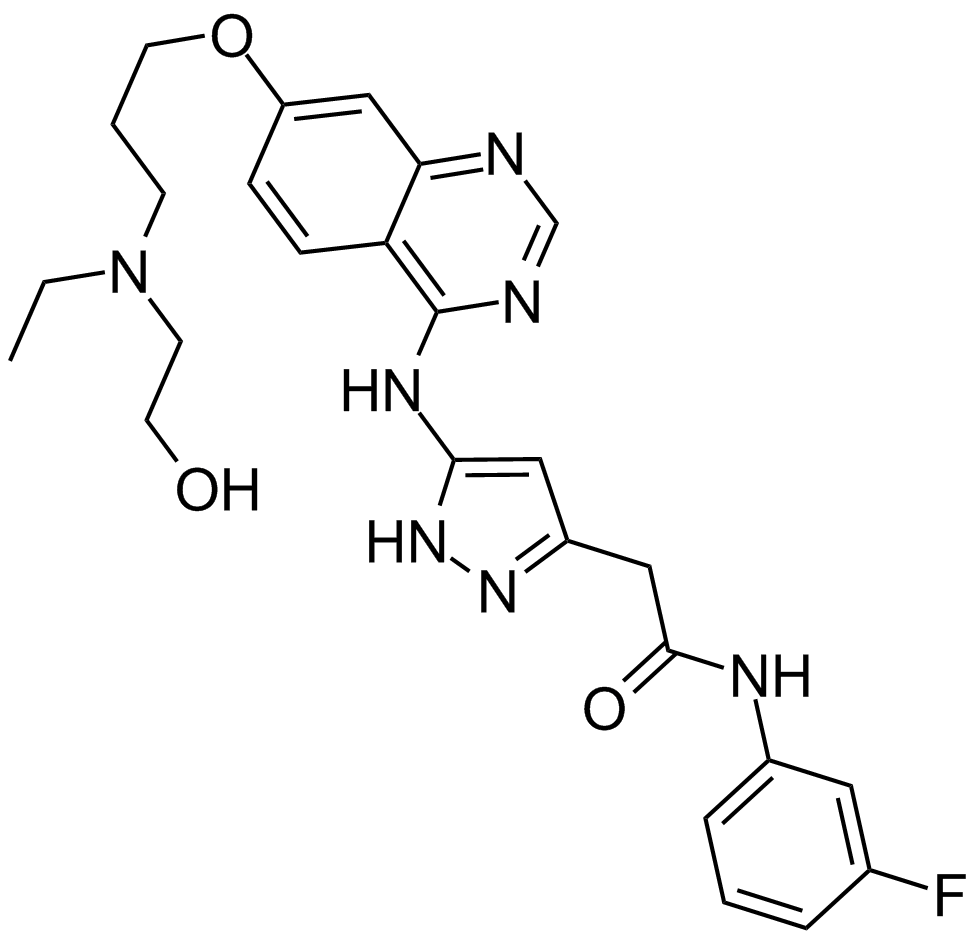
-
GC11572
Bardoxolone methyl
A synthetic triterpenoid with potent anticancer and antidiabetic activity
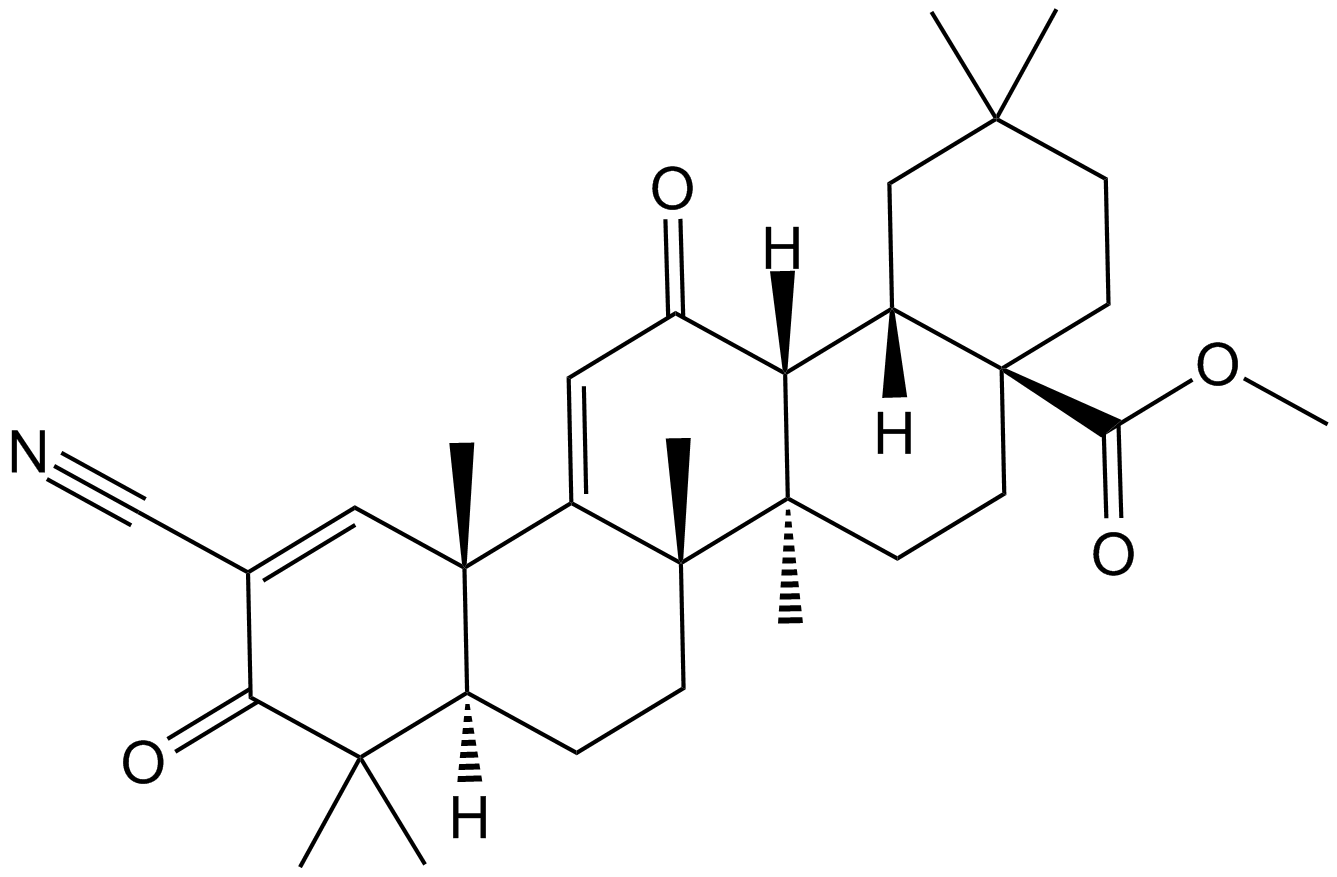
-
GC14844
Baricitinib (LY3009104, INCB028050)
A JAK1 and JAK2 inhibitor
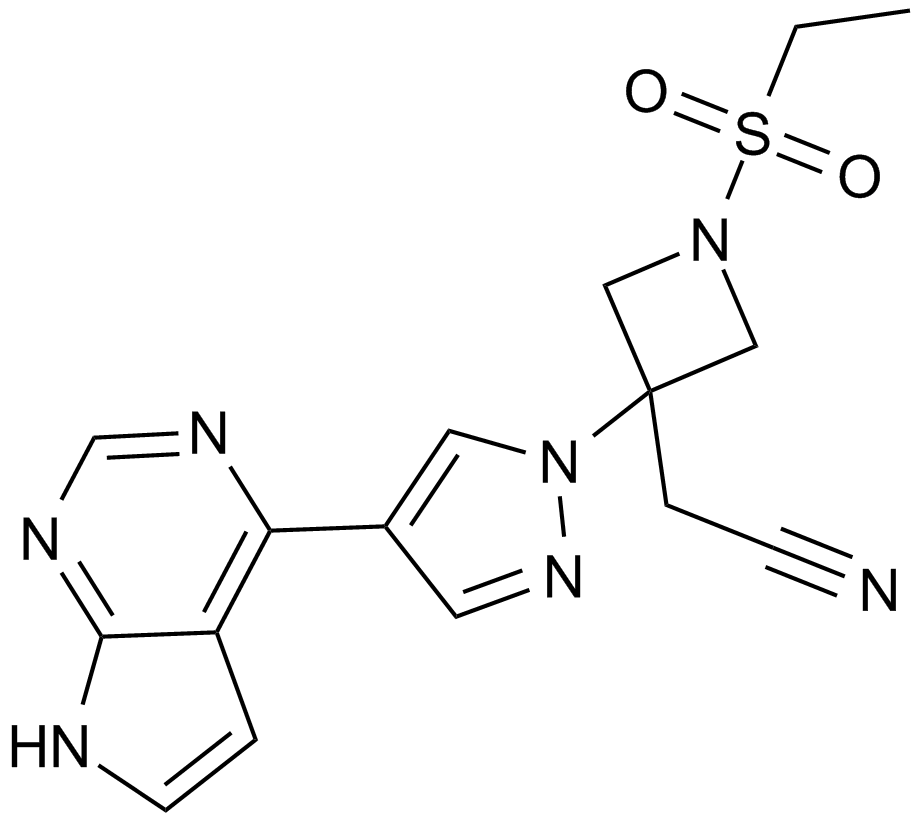
-
GC13830
Baricitinib phosphate
A JAK1 and JAK2 inhibitor

-
GC12698
BAY 87-2243
BAY 87-2243 es un inhibidor altamente potente y selectivo del factor 1 inducible por hipoxia (HIF-1).
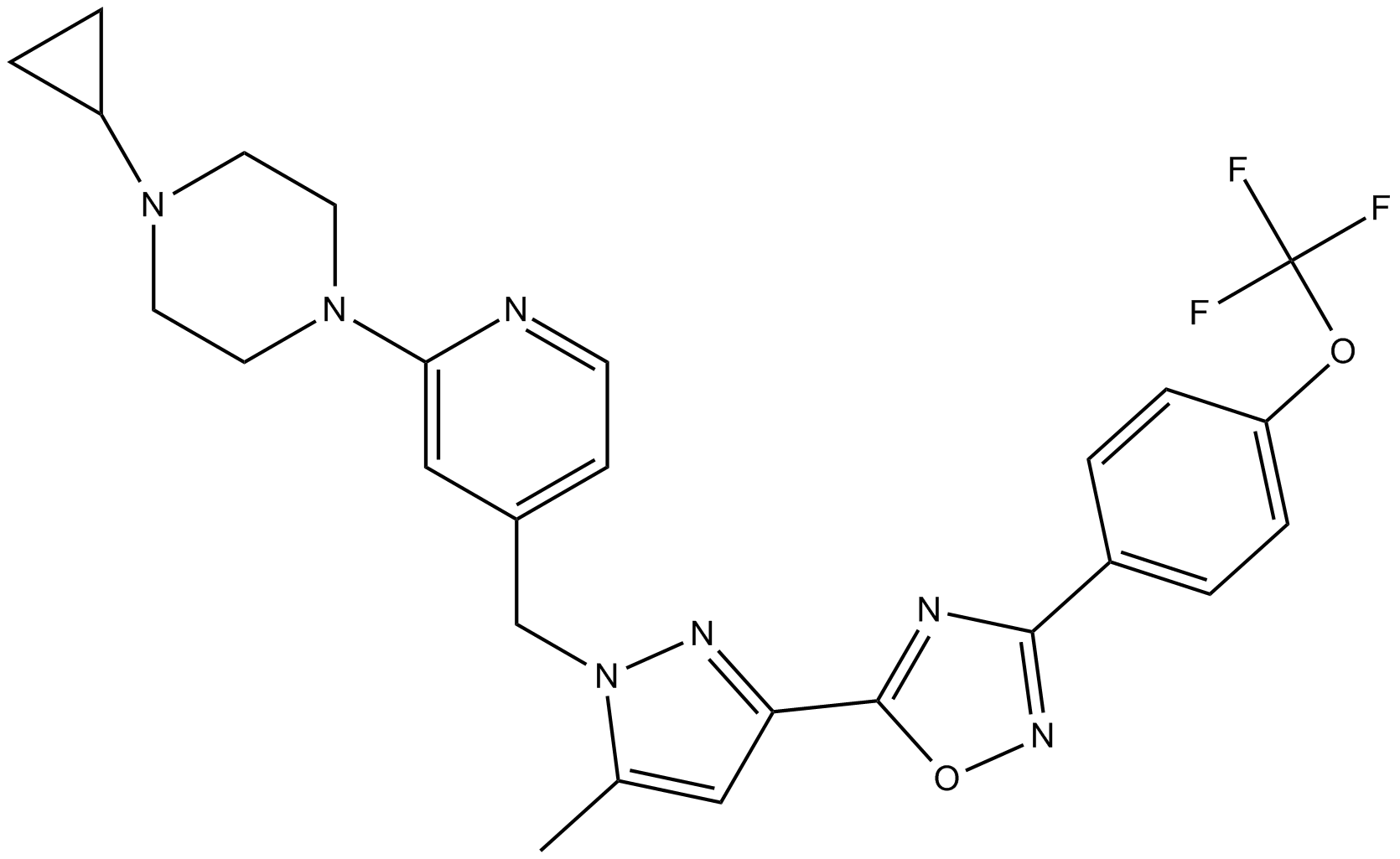
-
GC18508
BAY-299
BAY-299 es un inhibidor dual muy potente con IC50 de 67 nM para bromodominios BRPF2 (BD), 8 nM para TAF1 BD2 y 106 nM para TAF1L BD2.
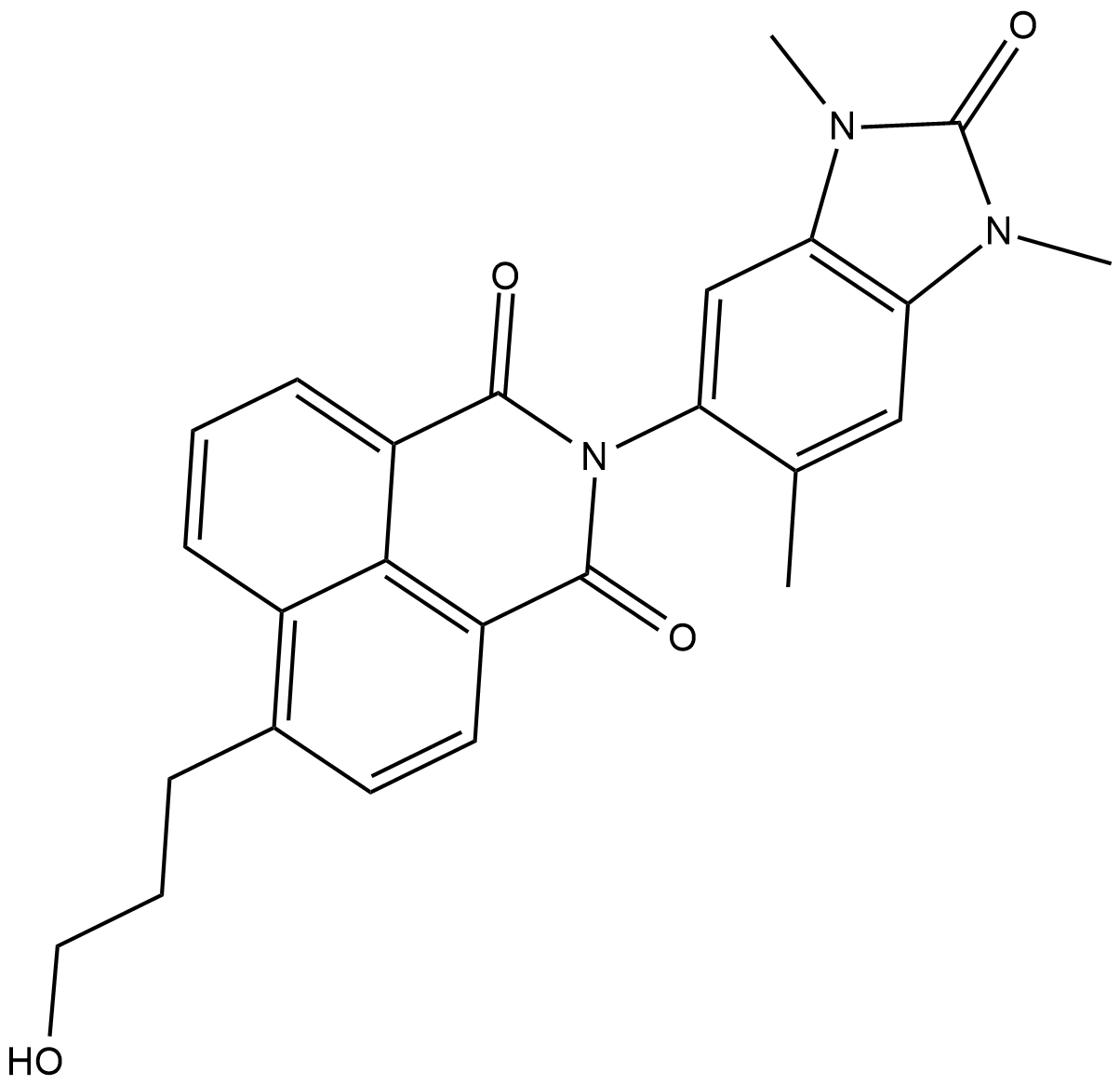
-
GC18159
BAY-598
BAY-598 es un inhibidor selectivo de molécula pequeÑa de SMYD2 con una IC50 de 27 nM.
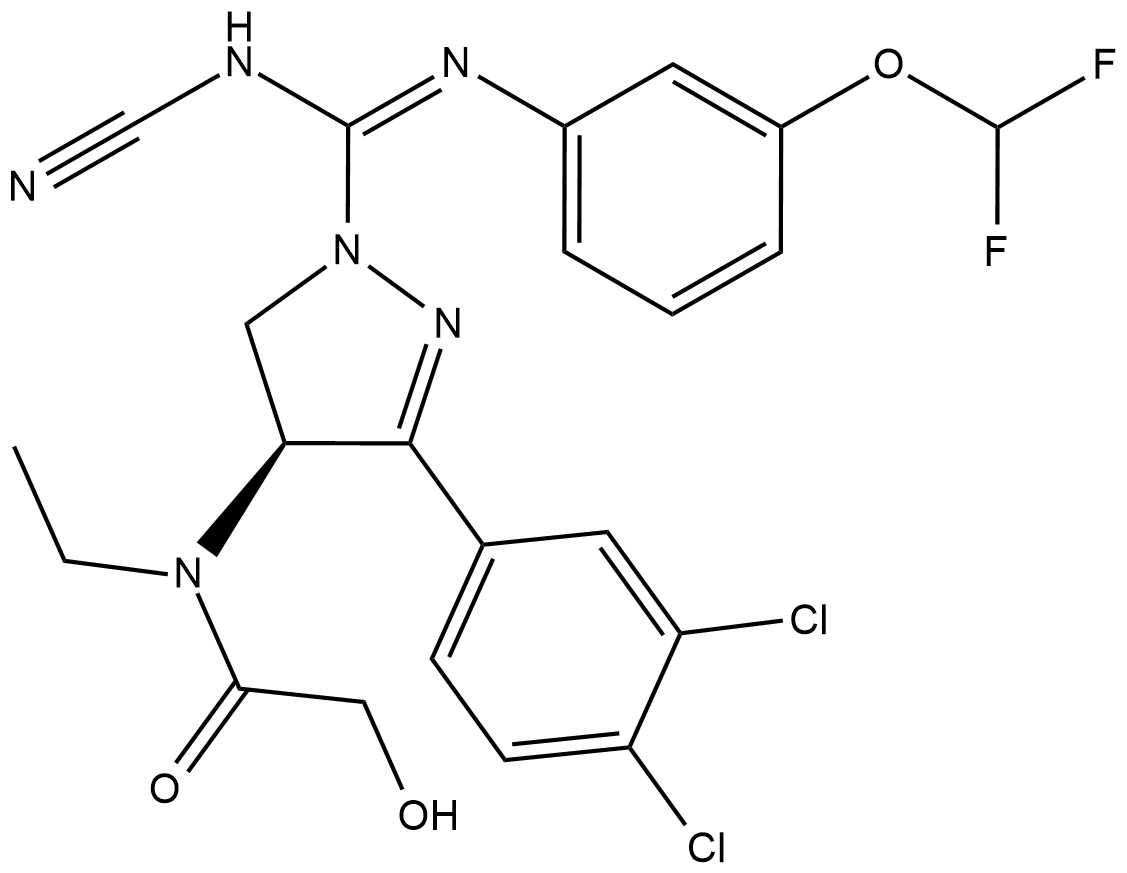
-
GC45389
BAY-6035
BAY-6035 es un inhibidor potente, selectivo y competitivo de sustrato de SMYD3. BAY-6035 inhibe la metilaciÓn del péptido MEKK2 con una IC50 de 88 nM.
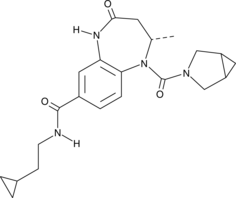
-
GC33104
BAY1238097
BAY1238097 es un inhibidor potente y selectivo de la uniÓn de BET a las histonas y tiene una fuerte actividad antiproliferativa en diferentes modelos de AML (leucemia mieloide aguda) y MM (mieloma mÚltiple) a través de la regulaciÓn negativa de los niveles de c-Myc y su transcriptoma aguas abajo (IC50 < 100 nM).
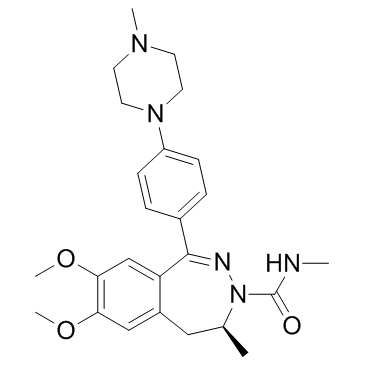
-
GC63694
BAZ1A-IN-1
BAZ1A-IN-1 es un potente inhibidor de BAZ1A (proteÍna que contiene bromodominio). BAZ1A-IN-1 muestra un valor KD de 0,52 μM frente al bromodominio BAZ1A. BAZ1A-IN-1 muestra una buena actividad antiviabilidad contra lÍneas de células cancerosas que expresan un alto nivel de BAZ1A, pero actividad débil o nula contra células cancerosas con un bajo nivel de expresiÓn de BAZ1A.
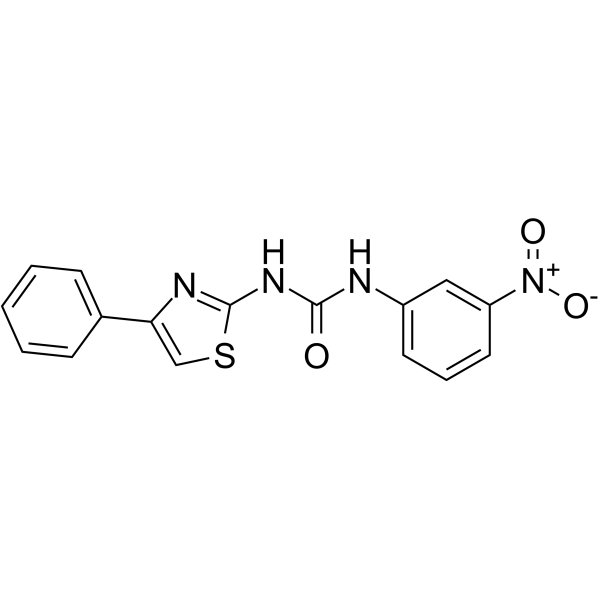
-
GC15175
BAZ2-ICR
BAZ2-ICR es un inhibidor de bromodominios BAZ2A/B potente, selectivo, activo celular y oralmente activo con IC50 de 130 nM y 180 nM, y Kds de 109 nM y 170 nM, respectivamente. BAZ2-ICR muestra una selectividad de 10-15 veces para unirse a BAZ2A/B sobre CECR2 y una selectividad de >100 veces sobre todos los demÁs bromodominios. BAZ2-ICR es una sonda quÍmica epigenética.
-
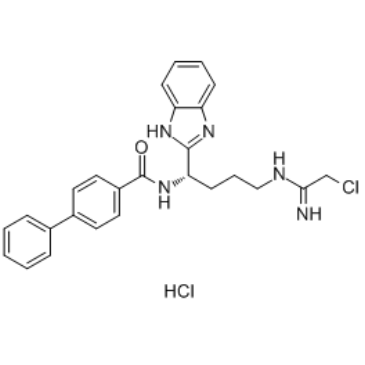
GC35480
BB-Cl-Amidine hydrochloride
El clorhidrato de BB-Cl-amidina es un inhibidor de la peptidilarginina deminasa (PAD).
-
GC42910
BB-F-Yne
BB-F-Yne is a cell-permeable derivative of the protein arginine diminase (PAD) inhibitor BB-Cl-amidine that contains an alkyne moiety for use in click chemistry reactions.
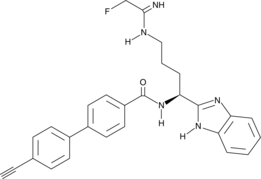
-
GC18161
BCI-121
BCI-121 es un inhibidor de SMYD3 que impide la proliferaciÓn de células cancerosas.
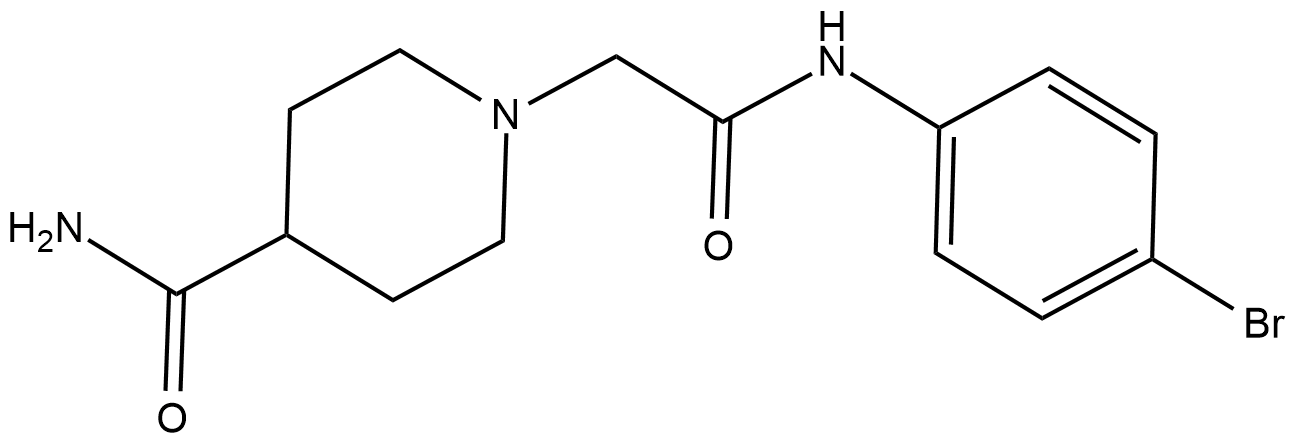
-
GC15962
Belinostat (PXD101)
Belinostat is an effective HDAC inhibitor with an IC50 of 27 nM in HeLa cell extracts.
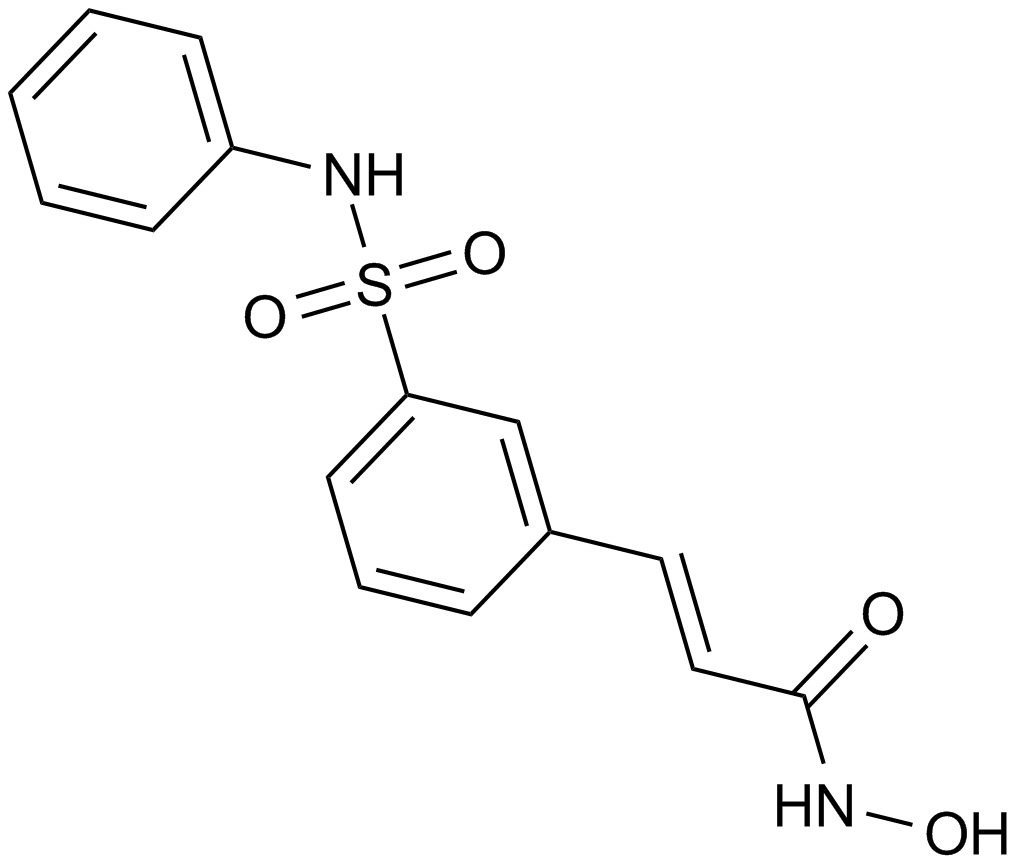
-
GC12844
Benzamide
La benzamida (bencenocarboxamida) es un potente inhibidor de la poli(ADP-ribosa) polimerasa (PARP).
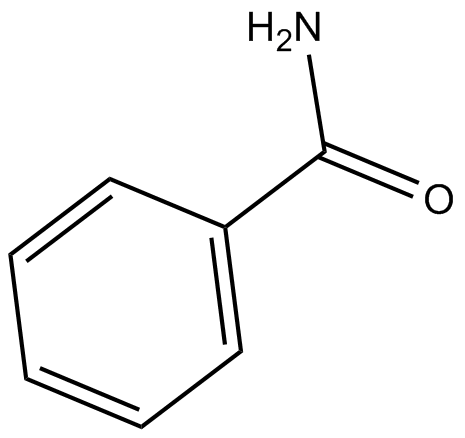
-
GC30763
Benzenebutyric acid (4-Phenylbutyric acid)
El ácido bencenobutírico (ácido 4-fenilbutírico) (4-PBA) es un inhibidor de HDAC y del estrés del retículo endoplásmico (ER), utilizado en la investigación sobre cáncer e infecciones.
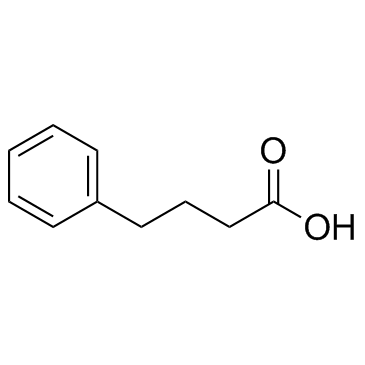
-
GC16299
BET bromodomain inhibitor
El inhibidor de bromodominio BET es un potente inhibidor de BET extraÍdo de la patente WO/2015/153871A2, compuesto del ejemplo 11.
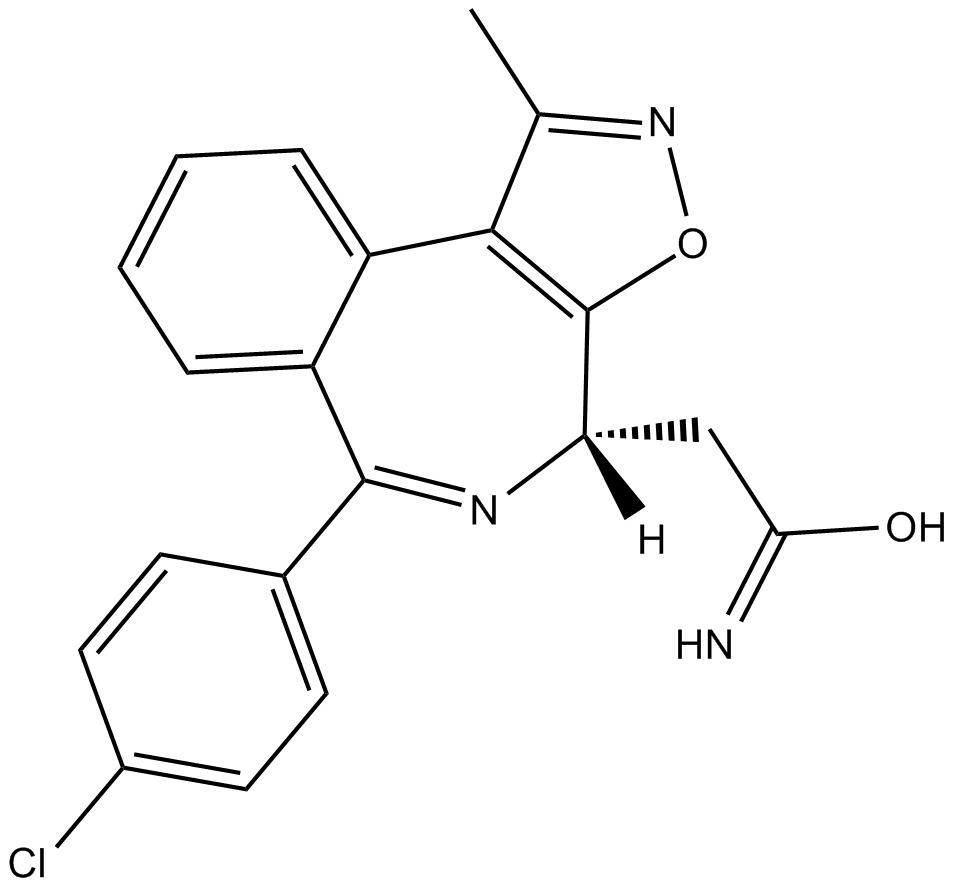
-
GC63528
BET bromodomain inhibitor 1
El inhibidor de bromodominio BET 1 es un inhibidor de bromodominio selectivo y extraterminal (BET) activo por vÍa oral con una IC50 de 2,6 nM para BRD4. El inhibidor de bromodominio BET 1 se une a BRD2(2), BRD3(2), BRD4(1), BRD4(2) y BRDT(2) con altas afinidades (valores de Kd de 1,3 nM, 1,0 nM, 3,0 nM, 1,6 nM, 2,1 nM, respectivamente). El inhibidor de bromodominio 1 tiene actividad anticancerÍgena.
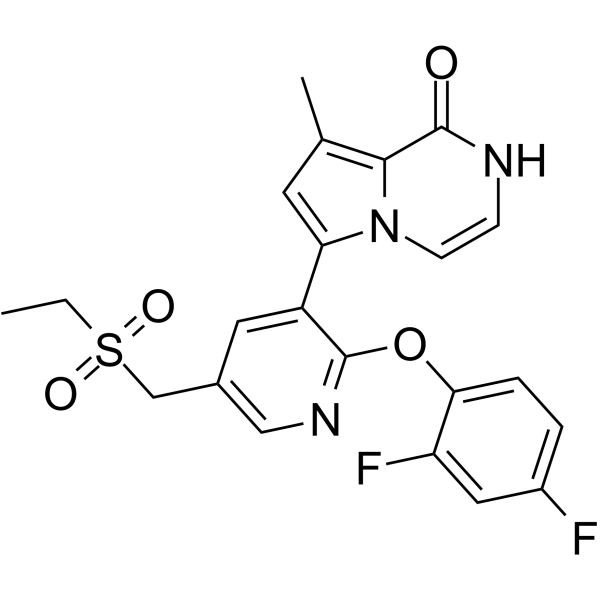
-
GC35505
BET-BAY 002
BET-BAY 002 es un potente inhibidor de BET; muestra eficacia en un modelo de mieloma mÚltiple.
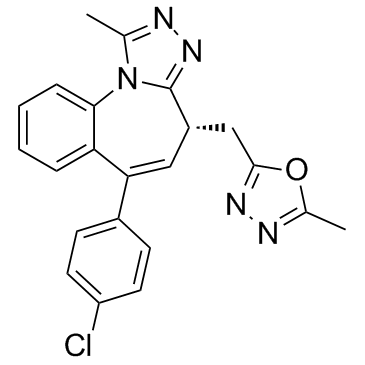
-
GC35506
BET-BAY 002 S enantiomer
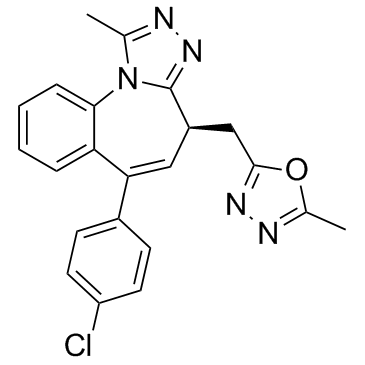
-
GC31831
BET-IN-1
BET-IN-1 es un pan-inhibidor de los ocho bromodominios BET y selectividad sobre otras proteÍnas representativas que contienen bromodominio.
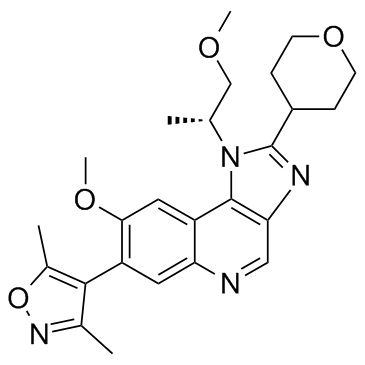
-
GC65506
BETd-246
BETd-246 es un inhibidor de bromodominio BET (BRD) de segunda generaciÓn y basado en PROTAC conectado por ligandos para Cereblon y BET, que muestra una selectividad, potencia y actividad antitumoral superior.
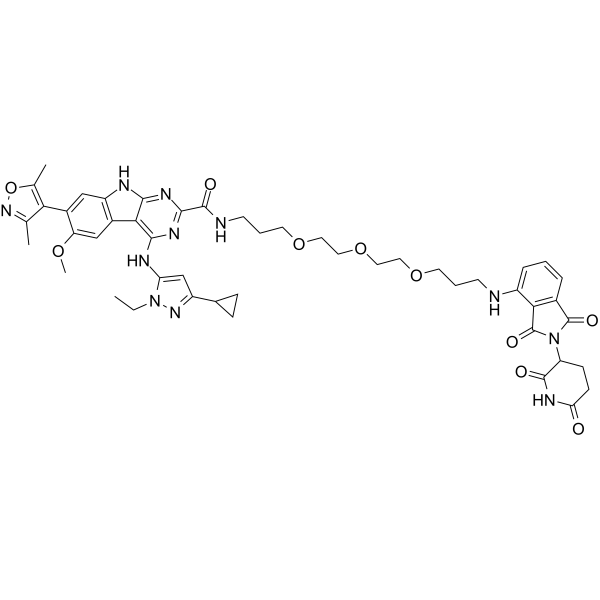
-
GC32791
BETd-260 (ZBC 260)
BETd-260 (ZBC 260) (ZBC 260) es un PROTAC conectado por ligandos para Cereblon y BET, con tan solo 30 pM contra la proteÍna BRD4 en la lÍnea celular de leucemia RS4;11. BETd-260 (ZBC 260) suprime de forma potente la viabilidad celular e induce de forma sÓlida la apoptosis en células de carcinoma hepatocelular (HCC).
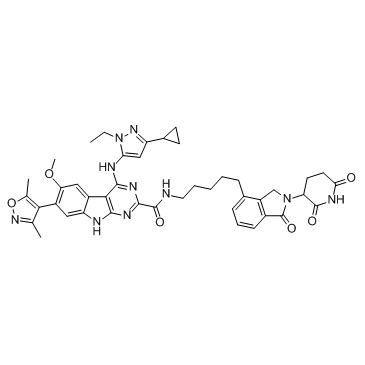
-
GC12074
BG45
BG45 es un inhibidor de HDAC clase I con selectividad por HDAC3 (IC50 = 289 nM).
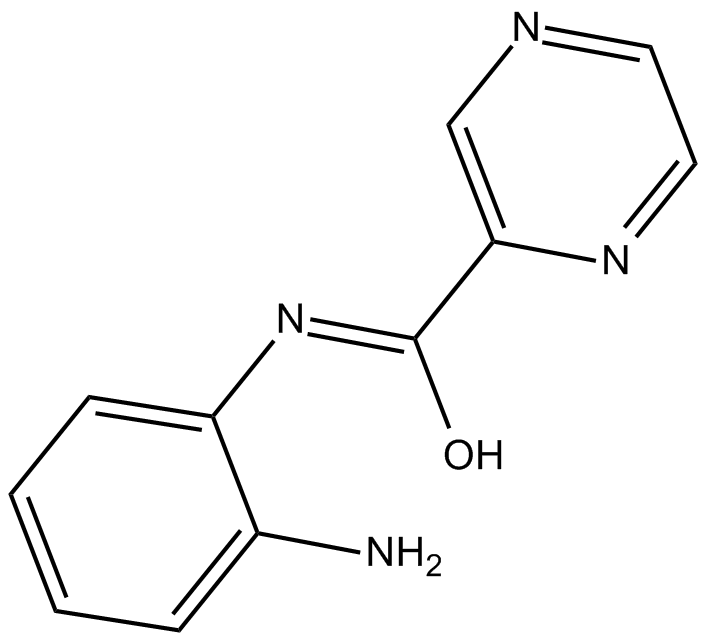
-
GC14380
BGP-15
BGP-15 es un inhibidor de PARP, con un IC50 y un Ki de 120 y 57 μM, respectivamente.
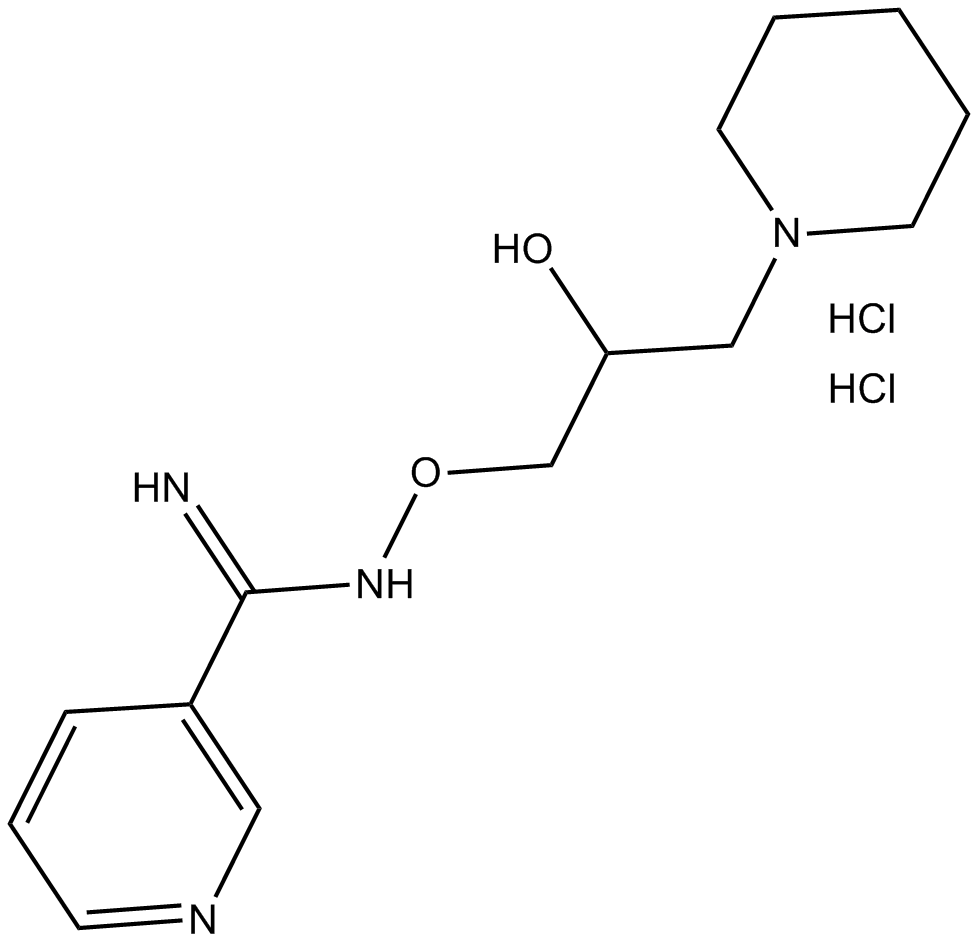
-
GC12450
BI 2536
Un potente inhibidor de Plk1.
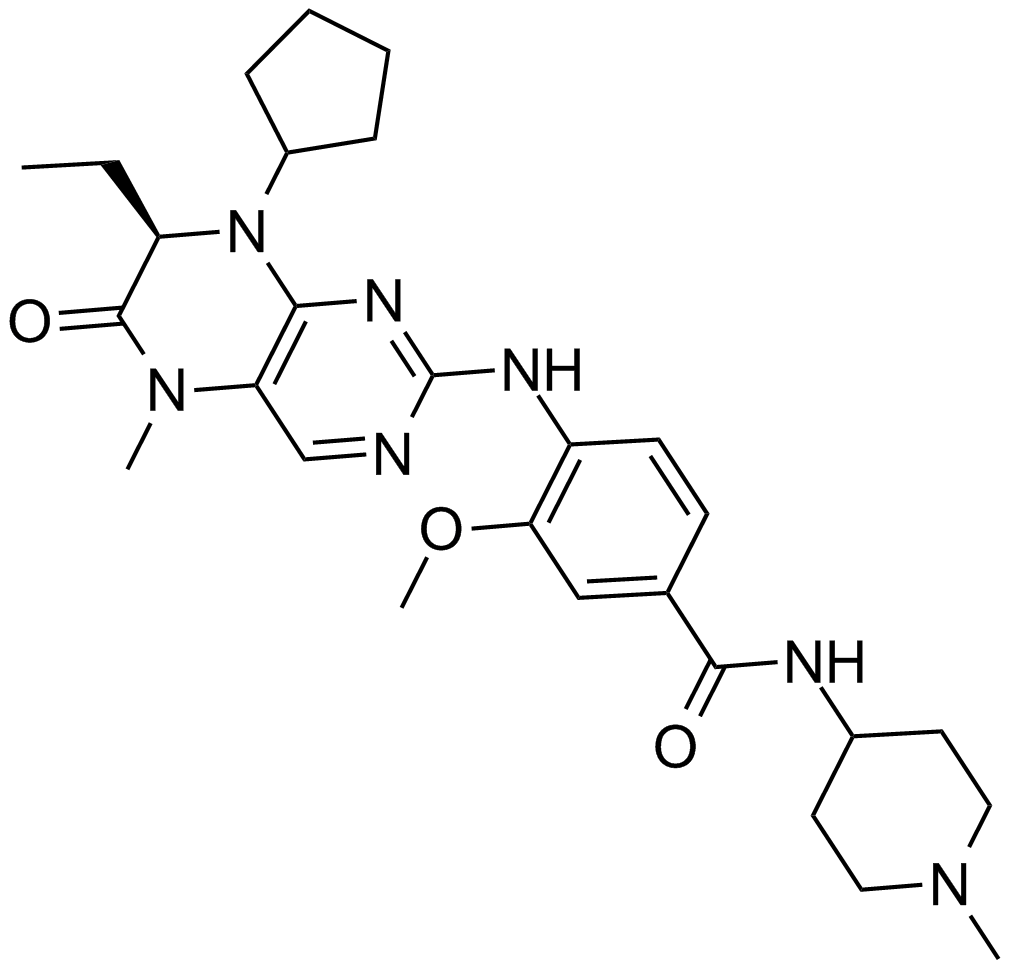
-
GC50540
BI 9321
Nuclear receptor-binding SET domain (NSD) 3 antagonist; selectively binds PWWP1 domain
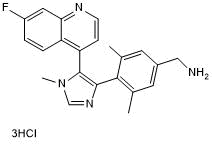
-
GC16726
BI-7273
BI-7273 es un inhibidor de BRD9 selectivo y permeable a las células, con una IC50 y una Kd de 19 y 0,75 nM; también muestra un alto efecto sobre BRD7, con un IC50 y un Kd de 117 nM y 0,3 nM.
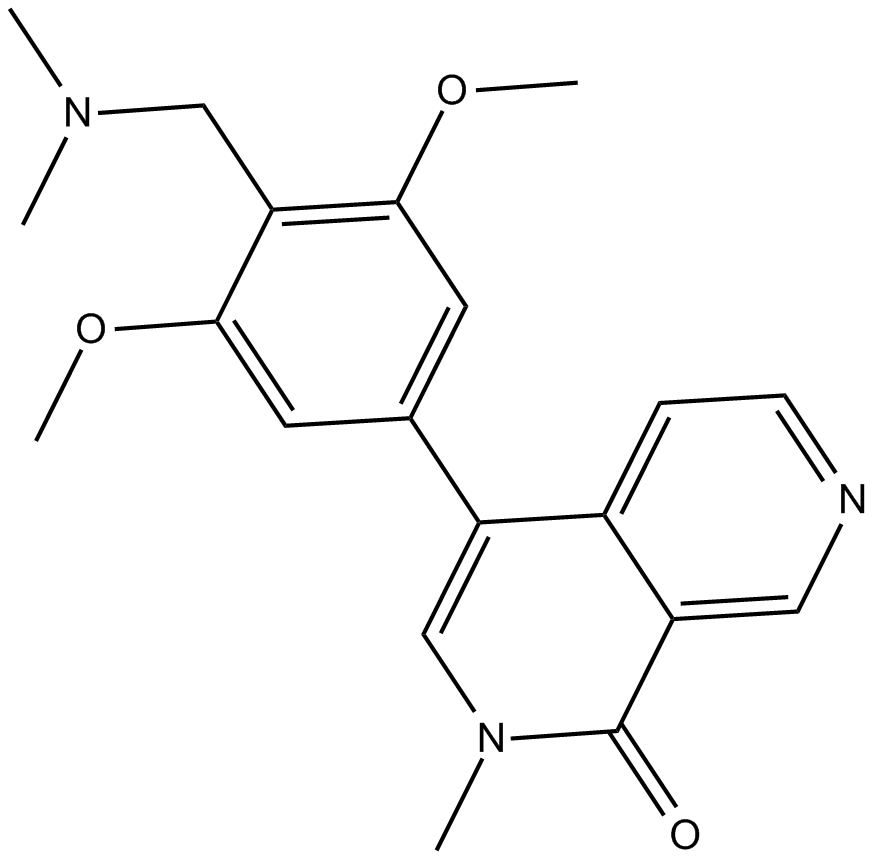
-
GC17828
BI-847325
BI-847325 es un inhibidor dual competitivo de ATP de MEK y aurora quinasas (AK) con valores IC50 de 4 y 15 nM para MEK2 y AK-C humanos, respectivamente.

-
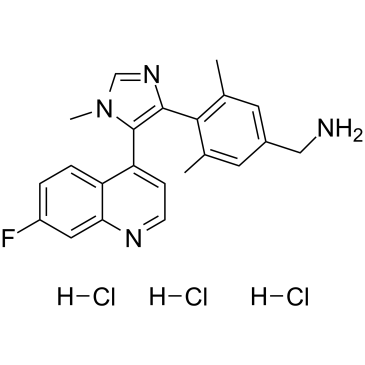
GC39663
BI-9321 trihydrochloride
El trihidrocloruro de BI-9321 es un antagonista del dominio SET 3 (NSD3)-PWWP1 potente, selectivo y celularmente activo con un valor de Kd de 166 nM. El trihidrocloruro de BI-9321 es inactivo frente a NSD2-PWWP1 y NSD3-PWWP2. El triclorhidrato de BI-9321 interrumpe especÍficamente las interacciones de histonas del dominio NSD3-PWWP1 con un IC50 de 1,2 μM en células U2OS.
-
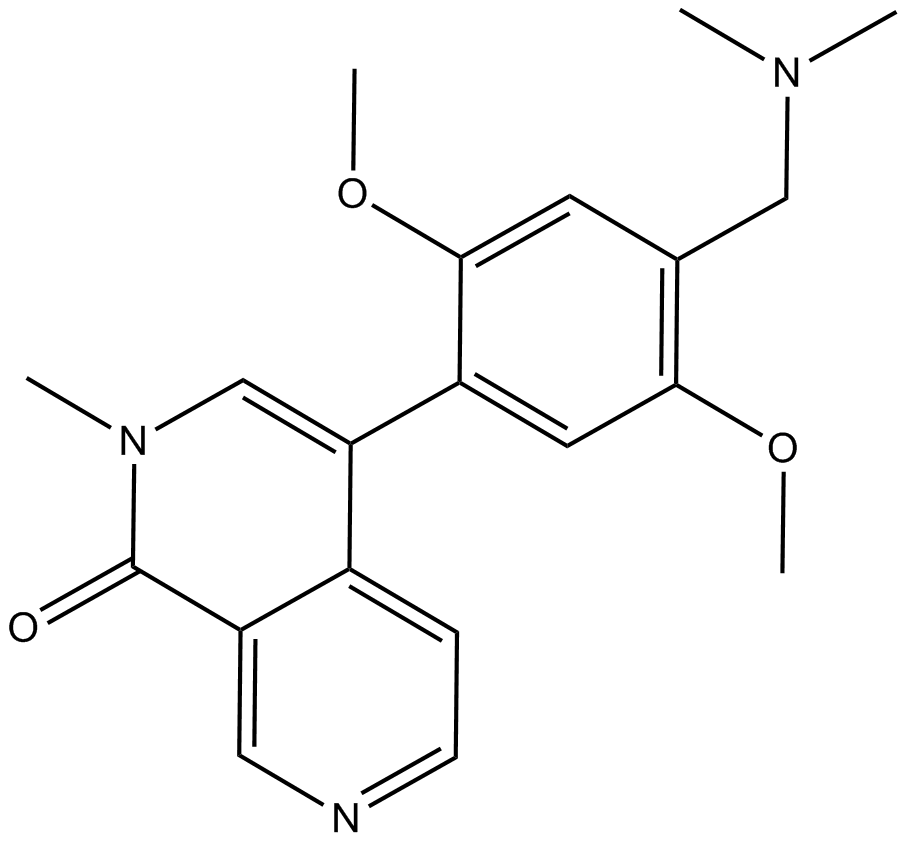
GC16817
BI-9564
BI-9564 es un inhibidor de bromodominios BRD9/BRD7 potente, selectivo y permeable a las células, con IC50 de 75 nM y 3,4 μM y Kds de 14 nM y 239 nM, respectivamente. BI-9564 tiene un IC50 de > 100 μM para la familia BET.
-
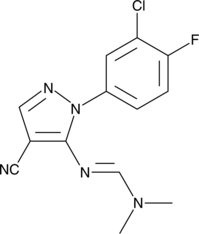
GC42933
Binucleine 2
Binucleine 2 is an isoform-specific and ATP-competitive inhibitor of Drosophila Aurora B kinase (Ki = 0.36 μM), a kinase involved in cell division.
-
GC64789
Biotinylated-JQ1
Biotinilated-JQ1 (Biotin-JQ1) es un derivado biotinilado de JQ1 con alta afinidad por el bromodominio de BRD4. El JQ1 biotinilado inhibe la proliferaciÓn de células de mieloma mÚltiple MM1.S con una EC50 de 0,4 μM.
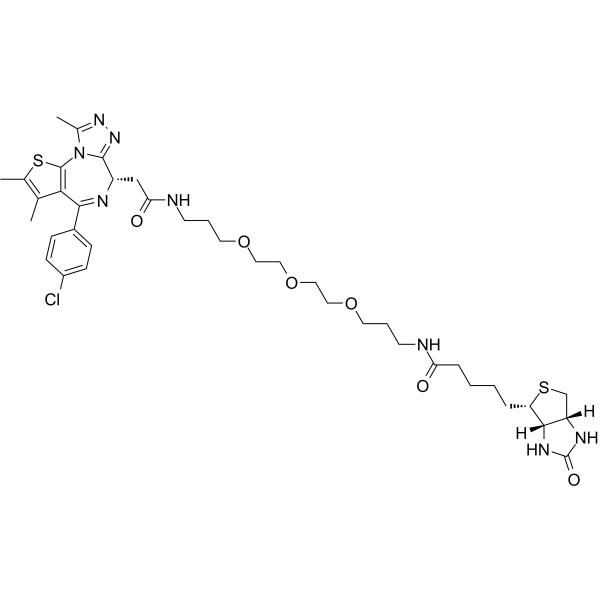
-
GC25139
Biphenyl-4-sulfonyl chloride
Biphenyl-4-sulfonyl chloride (p-Phenylbenzenesulfonyl, 4-Phenylbenzenesulfonyl, p-Biphenylsulfonyl) is a HDAC inhibitor with synthetic applications in palladium-catalyzed desulfitative C-arylation.
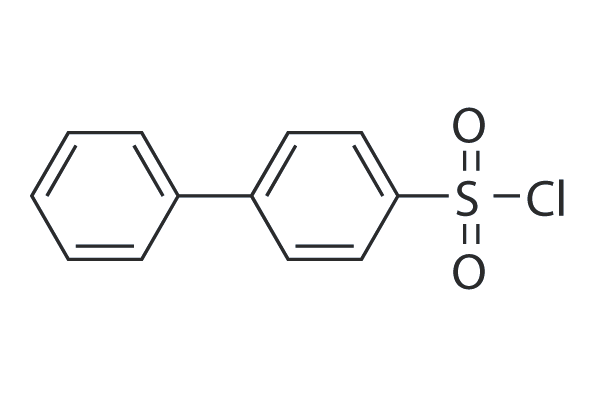
-
GC48463
Bisubstrate Inhibitor 78
An inhibitor of NNMT