Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nombre del producto Información
-
GC15355
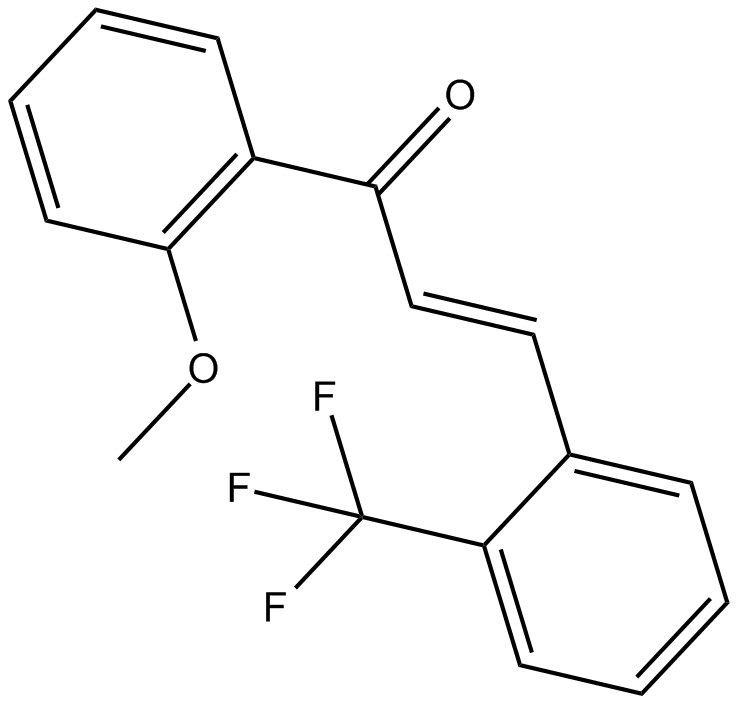
2-Trifluoromethyl-2'-methoxychalcone
Nrf2 activator
-
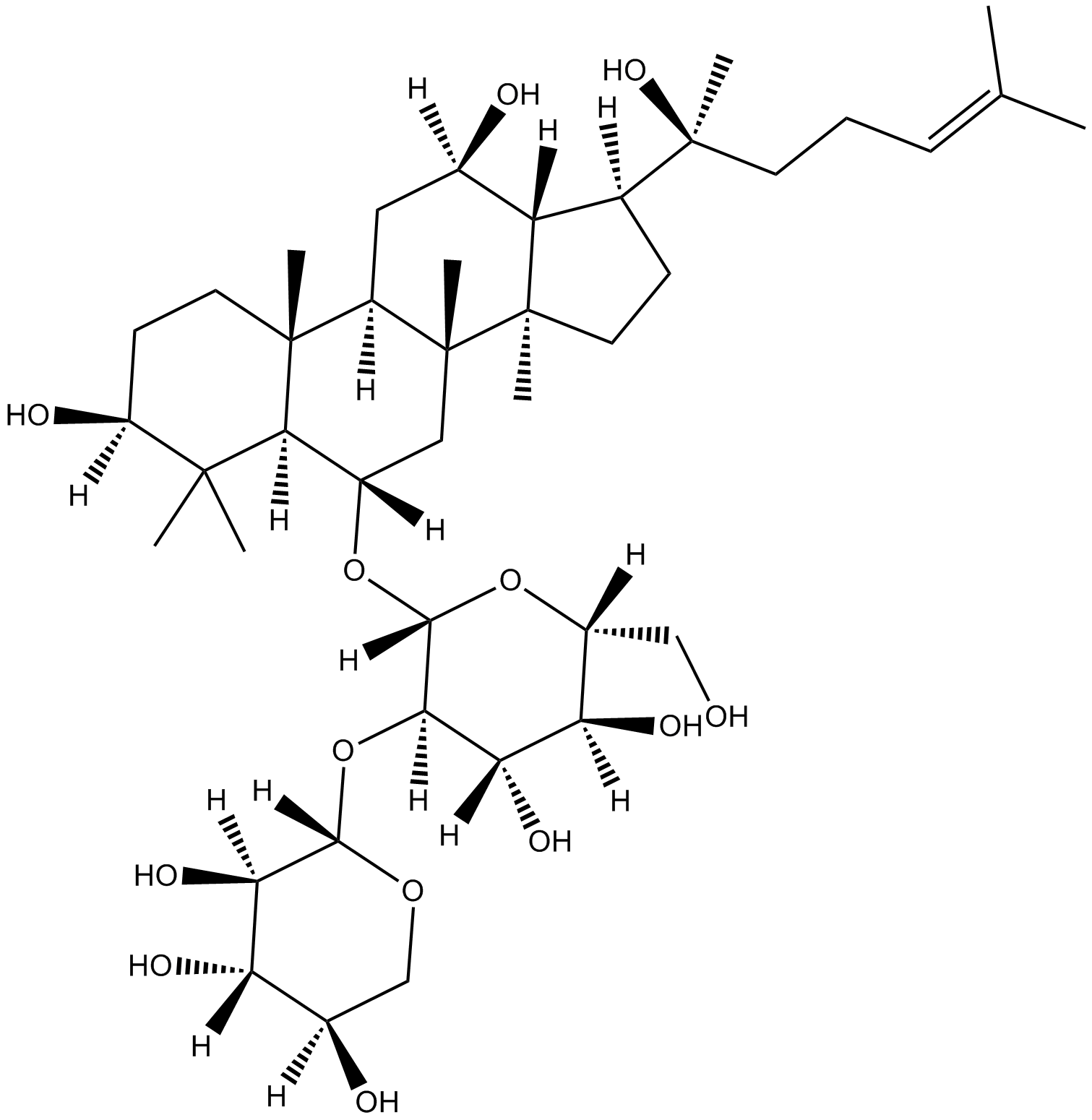
GN10800
20(S)-NotoginsenosideR2
-
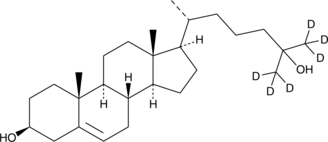
GC46528
25-hydroxy Cholesterol-d6
An internal standard for the quantification of 25hydroxy cholesterol
-
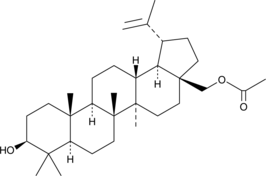
GC48482
28-Acetylbetulin
A lupane triterpenoid with anti-inflammatory and anticancer activities
-
GC35112
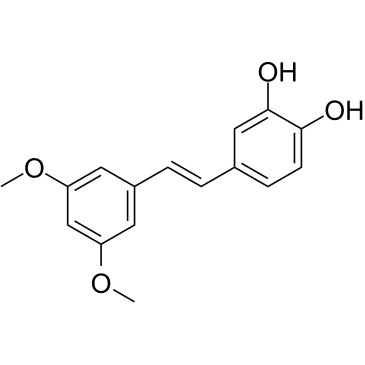
3'-Hydroxypterostilbene
El 3'-hidroxipterostilbeno es un anÁlogo de pterostilbeno. El 3'-hidroxipterostilbeno inhibe el crecimiento de células COLO 205, HCT-116 y HT-29 con IC50 de 9,0, 40,2 y 70,9 μM, respectivamente. El 3'-hidroxipterostilbeno regula significativamente a la baja las vÍas de seÑalizaciÓn de PI3K/Akt y MAPK e inhibe eficazmente el crecimiento de las células de cÁncer de colon humano al inducir la apoptosis y la autofagia. El 3'-hidroxipterostilbeno se puede utilizar para la investigaciÓn del cÁncer.
-
GC12791
3,3'-Diindolylmethane
A phytochemical with antiradiation and chemopreventative effects

-
GC42237
3,5-dimethyl PIT-1
PtdIns-(3,4,5)-P3 (PIP3) serves as an anchor for the binding of signal transduction proteins bearing pleckstrin homology (PH) domains such as phosphatidylinositol 3-kinase (PI3K) or PTEN.
-
GC64762
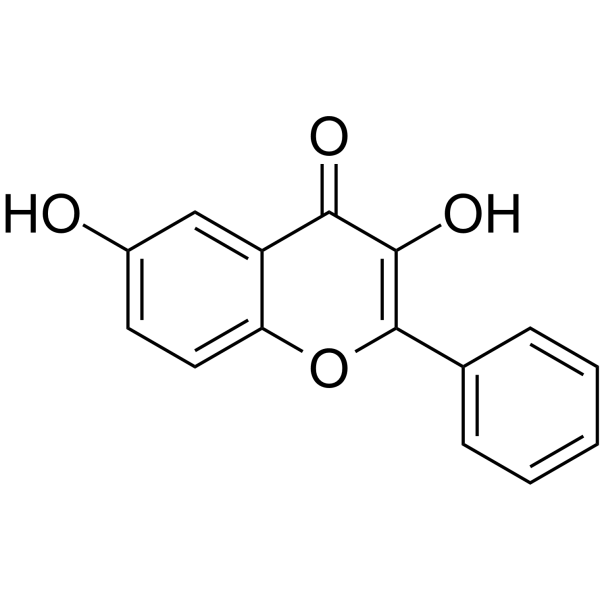
3,6-Dihydroxyflavone
La 3,6-dihidroxiflavona es un agente anticancerÍgeno. La 3,6-dihidroxiflavona, en funciÓn de la dosis y el tiempo, reduce la viabilidad celular e induce la apoptosis mediante la activaciÓn de la cascada de caspasas, escindiendo la poli(ADP-ribosa) polimerasa (PARP). La 3,6-dihidroxiflavona aumenta el estrés oxidativo intracelular y la peroxidaciÓn lipÍdica.
-
GC46583
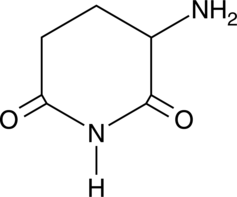
3-Amino-2,6-Piperidinedione
An active metabolite of (±)-thalidomide
-
GC49849
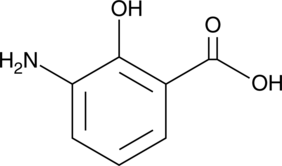
3-Aminosalicylic Acid
A salicylic acid derivative
-
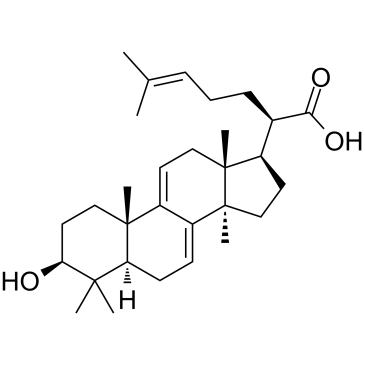
GC35106
3-Dehydrotrametenolic acid
El Ácido 3-dehidrotrametenÓlico, aislado del esclerocio de Poria cocos, es un inhibidor de la lactato deshidrogenasa (LDH). El Ácido 3-dehidrotrametenÓlico promueve la diferenciaciÓn de adipocitos in vitro y actÚa como sensibilizador de insulina in vivo. El Ácido 3-dehidrotrametenÓlico induce la apoptosis y tiene actividad anticancerÍgena.
-
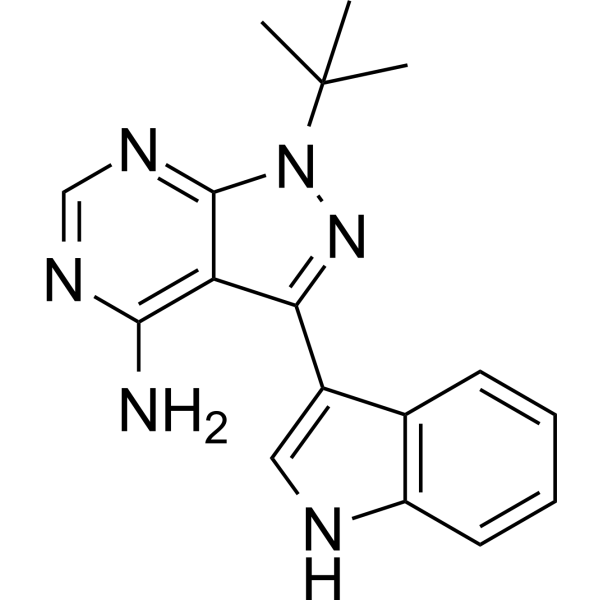
GC68537
3-IN-PP1
3-IN-PP1 es un inhibidor de la proteína quinasa D (PKD). 3-IN-PP1 tiene una amplia actividad inhibitoria sobre PKD1, PKD2 y PKD3, con valores de IC50 de 108, 94 y 108 nM respectivamente. También es un agente antitumoral de amplio espectro que inhibe el crecimiento de varias células tumorales. 3-IN-PP1 se puede utilizar en investigaciones sobre cáncer.
-
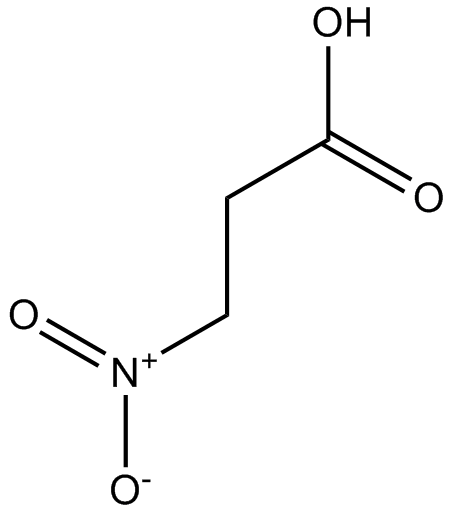
GC17394
3-Nitropropionic acid
El Ácido 3-nitropropiÓnico (β-Ácido nitropropiÓnico) es un inhibidor irreversible de la succinato deshidrogenasa.
-
GC35099
3-O-Acetyloleanolic acid
El ácido 3-O-acetiloleanólico (3AOA), un derivado del ácido oleanólico aislado de las semillas de Vigna sinensis K., induce el cáncer y también muestra actividad antiangiogénica.
-
GC60507
3-O-Methylgallic acid
El Ácido 3-O-metilgÁlico (Ácido 3,4-dihidroxi-5-metoxibenzoico) es un metabolito de antocianina y tiene una potente capacidad antioxidante. El Ácido 3-O-metilgÁlico inhibe la proliferaciÓn de células Caco-2 con un valor IC50 de 24,1 μM. El Ácido 3-O-metilgÁlico también induce la apoptosis celular y tiene efectos anticancerÍgenos.
-
GC32767
3BDO
3BDO es un nuevo activador de mTOR que también puede inhibir la autofagia.
-
GC45354
4β-Hydroxywithanolide E
4β-Hydroxywithanolide E, aislado de Physalis peruviana L.
-
GC48437
4'-Acetyl Chrysomycin A
A bacterial metabolite with antibacterial and anticancer activities
-
GC42346
4-bromo A23187
4-Bromo A23187 es un anÁlogo halogenado del ionÓforo de calcio altamente selectivo A-23187.
-
GC42401
4-hydroperoxy Cyclophosphamide
Un análogo activado de ciclofosfamida.
-
GC30896
4-Hydroxybenzyl alcohol
El alcohol 4-hidroxibencÍlico es un compuesto fenÓlico ampliamente distribuido en varios tipos de plantas.
-
GC33815
4-Hydroxyphenylacetic acid
El Ácido 4-hidroxifenilacético, un importante metabolito de los polifenoles derivado de la microbiota, participa en la acciÓn antioxidante.
-
GC35138
4-Methyldaphnetin
La 4-metildafnetina es un precursor en la sÍntesis de derivados de la 4-metil cumarina. La 4-metildafnetina tiene efectos potentes y selectivos antiproliferativos e inductores de apoptosis en varias lÍneas de células cancerosas. La 4-metildafnetina posee la propiedad de eliminar radicales e inhibe fuertemente la peroxidaciÓn lipÍdica de la membrana.
-
GC68231
4-Methylsalicylic acid

-
GC31648
4-Octyl Itaconate
El 4-Octil Itaconato (4-OI) es un derivado de itaconato permeable a la célula. El itaconato y el 4-Octil Itaconato tienen una reactividad similar con los tiolos, lo que hace que el 4-Octil Itaconato sea un sustituto adecuado del itaconato para estudiar su función biológica.
-
GC49127
4-oxo Cyclophosphamide
An inactive metabolite of cyclophosphamide
-
GC45352
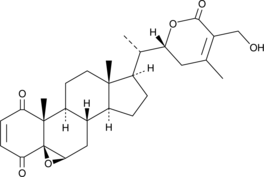
4-oxo Withaferin A
4-oxo Withaferin A es el anÁlogo de withaferin A. Withaferin A es una withanolida aislada de Withania somnifera. 4-oxo Withaferin A tiene potencial para la investigaciÓn del mieloma mÚltiple.
-
GC45353
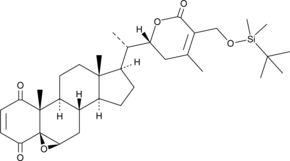
4-oxo-27-TBDMS Withaferin A
4-oxo-27-TBDMS Withaferin A, un derivado de withaferin A, muestra potentes efectos antiproliferativos en las células tumorales. 4-oxo-27-TBDMS Withaferin A induce la apoptosis de las células tumorales. 4-oxo-27-TBDMS Withaferin A es un agente anticancerÍgeno.
-
GC60525
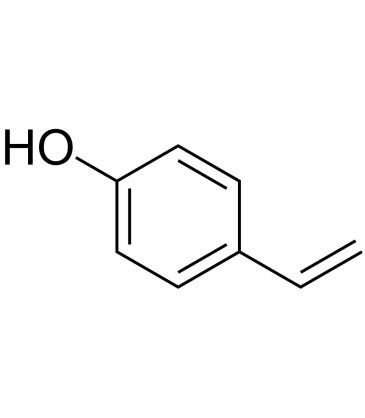
4-Vinylphenol (10%w/w in propylene glycol)
El 4-vinilfenol se encuentra en la hierba medicinal Hedyotis diffusa Willd, el arroz silvestre y también es el metabolito del Ácido p-cumÁrico y ferÚlico de las bacterias del Ácido lÁctico en el vino. El 4-vinilfenol induce la apoptosis e inhibe la formaciÓn de vasos sanguÍneos y suprime el crecimiento de tumores de mama invasivos in vivo.
-
GC10468
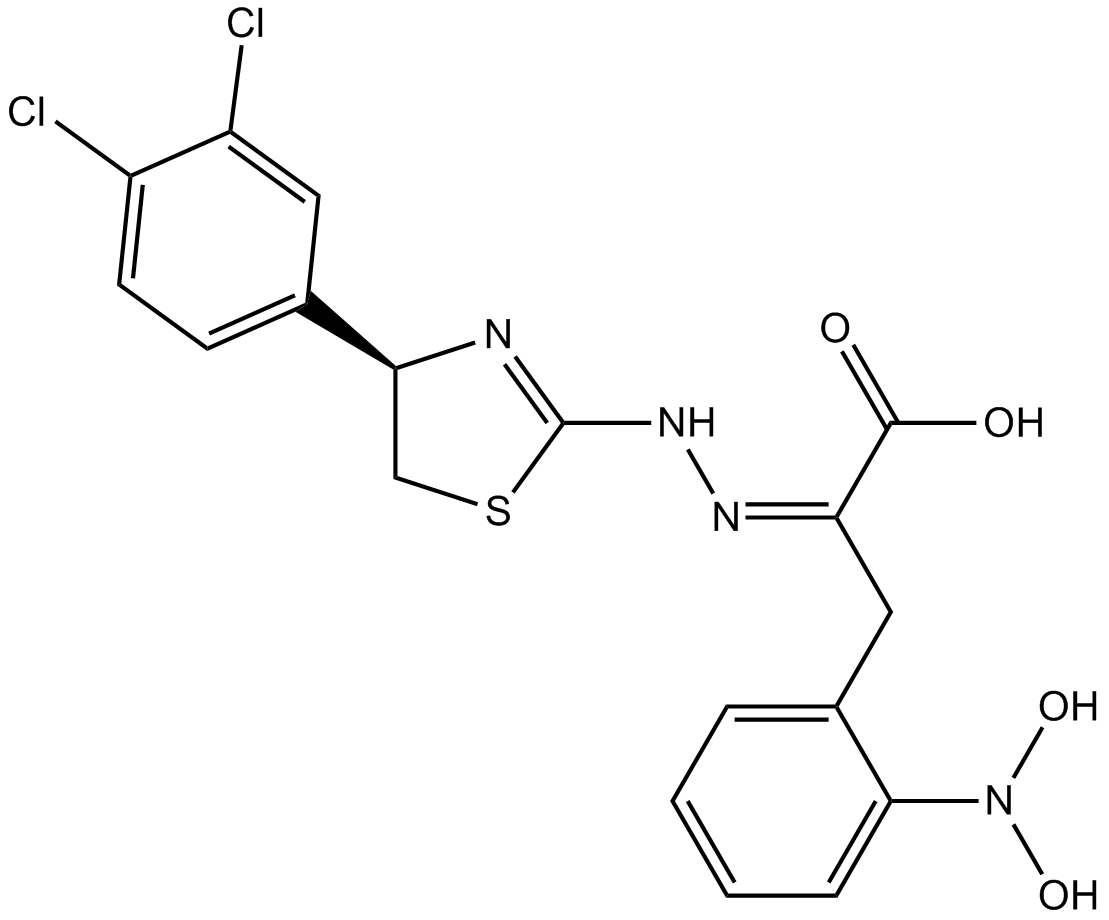
4EGI-1
4EGI-1 es un inhibidor de la interacciÓn eIF4E/eIF4G, con una Kd de 25 μM frente a la uniÓn de eIF4E.
-
GC35150
5,7,4'-Trimethoxyflavone
La 5,7,4'-trimetoxiflavona se aísla de Kaempferia parviflora (KP), una famosa planta medicinal de Tailandia. La 5,7,4'-trimetoxiflavona induce la apoptosis, como lo demuestran los incrementos de la fase sub-G1, la fragmentación del ADN, la tinción de anexina-V/PI, la relación Bax/Bcl-xL, la activación proteolítica de la caspasa-3 y la degradación de poli (ADP-ribosa) polimerasa (PARP) proteína. La 5,7,4'-trimetoxiflavona es significativamente eficaz para inhibir la proliferación de células de cáncer gástrico humano SNU-16 de una manera dependiente de la concentración.
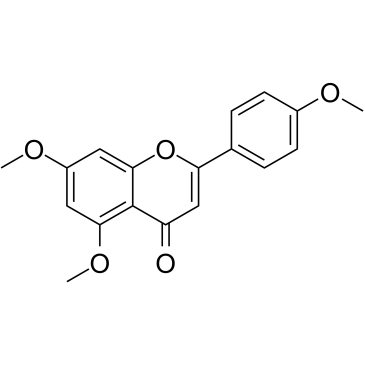
-
GN10629
5,7-dihydroxychromone
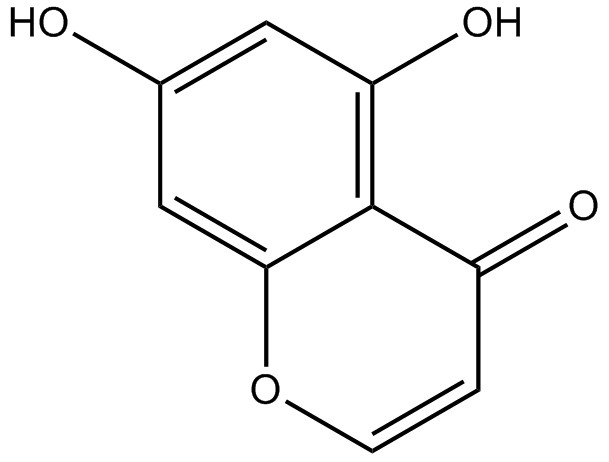
-
GC63972
5,7-Dimethoxyflavanone
La 5,7-dimetoxiflavanona muestra una potente actividad antimutagénica contra la mutagénesis de MeIQ en la prueba de Ames usando S.
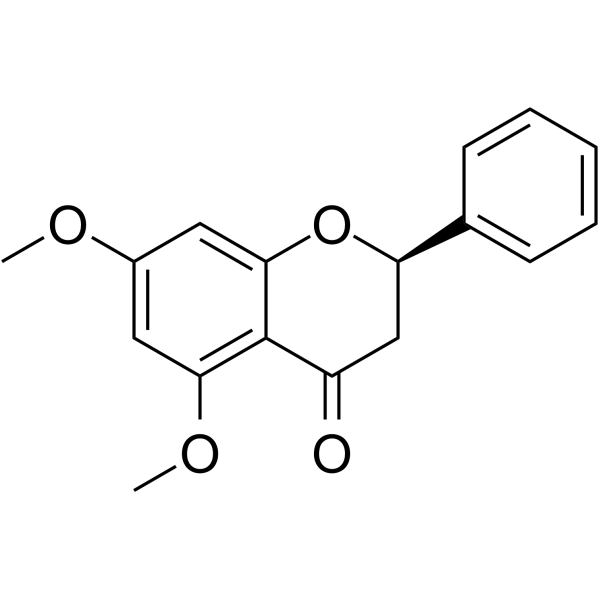
-
GC52227
5-(3',4'-Dihydroxyphenyl)-γ-Valerolactone
An active metabolite of various polyphenols
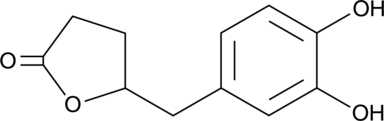
-
GC35147
5-(N,N-Hexamethylene)-amiloride
La 5-(N,N-hexametileno)-amilorida (hexametileno amilorida) deriva de una amilorida y es un potente inhibidor del intercambiador Na+/H+, que disminuye el pH intracelular (pHi) e induce la apoptosis en pacientes leucémicos. células.
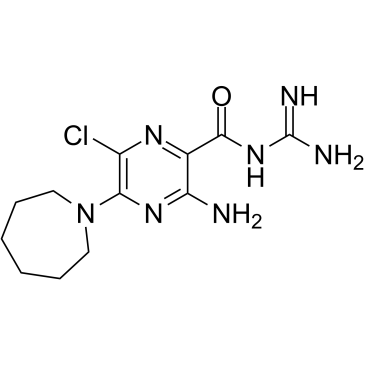
-
GC45356
5-Aminolevulinic Acid (hydrochloride)
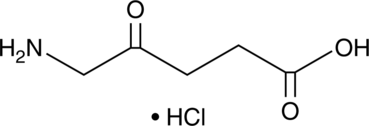
-
GC46681
5-Bromouridine
A brominated uridine analog
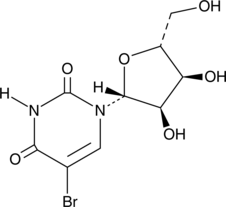
-
GC42545
5-Fluorouracil-13C,15N2
5-Fluorouracil-13C,15N2 is intended for use as an internal standard for the quantification of 5-flurouracil by GC- or LC-MS.
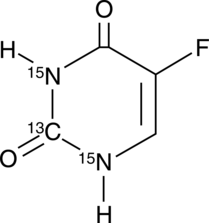
-
GC46705
5-Methoxycanthinone
La 5-metoxicantinona es un inhibidor activo por vÍa oral de las cepas de Leishmania.
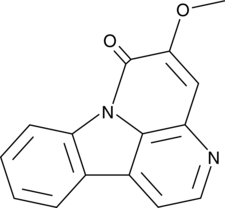
-
GC42586
6α-hydroxy Paclitaxel
6α-hidroxi Paclitaxel es un metabolito primario de Paclitaxel. 6α-hidroxi Paclitaxel conserva un efecto dependiente del tiempo sobre los polipéptidos transportadores de aniones orgÁnicos 1B1/SLCO1B1 (OATP1B1) con una potencia de inhibiciÓn similar a Paclitaxel, mientras que ya no mostrÓ inhibiciÓn dependiente del tiempo de OATP1B3. 6α-hidroxi Paclitaxel se puede utilizar para la investigaciÓn del cÁncer.
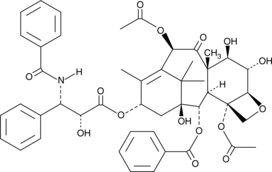
-
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
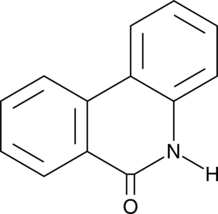
-
GN10093
6-gingerol
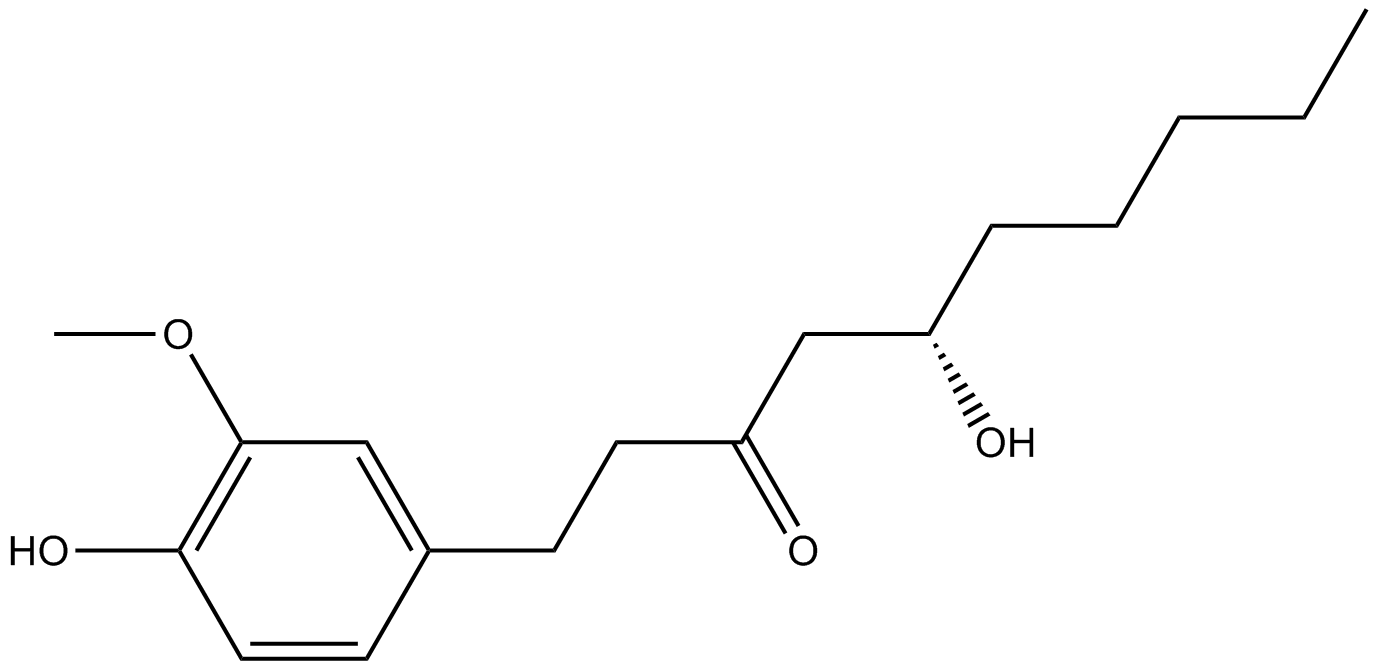
-
GC49429
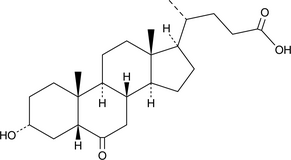
6-keto Lithocholic Acid
A metabolite of lithocholic acid
-
GC35184
7,3',4'-Tri-O-methylluteolin
La 7,3',4'-tri-O-metilluteolina (5-hidroxi-3',4',7-trimetoxiflavona), un compuesto flavonoide, posee potentes efectos antiinflamatorios en la lÍnea celular de macrÓfagos inducida por LPS mediada por la inhibiciÓn de liberaciÓn de mediadores inflamatorios, NO, PGE2 y citocinas proinflamatorias.
-
GC45673
7,8-Dihydroneopterin
La 7,8-dihidroneopterina, un marcador de inflamaciÓn, induce la apoptosis celular en astrocitos y neuronas a través de la mejora de la expresiÓn de Óxido nÍtrico sintasa (iNOS).
-
GC16853
7,8-Dihydroxyflavone
La 7,8-dihidroxiflavona es un agonista de TrkB potente y selectivo que imita las acciones fisiolÓgicas del factor neurotrÓfico derivado del cerebro (BDNF).
-
GC42616
7-oxo Staurosporine
7-oxo Staurosporine is an antibiotic originally isolated from S.
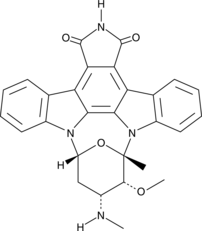
-
GC16037
7BIO
7BIO (7-Bromoindirrubin-3-Oxime) es el derivado de la indirrubina.
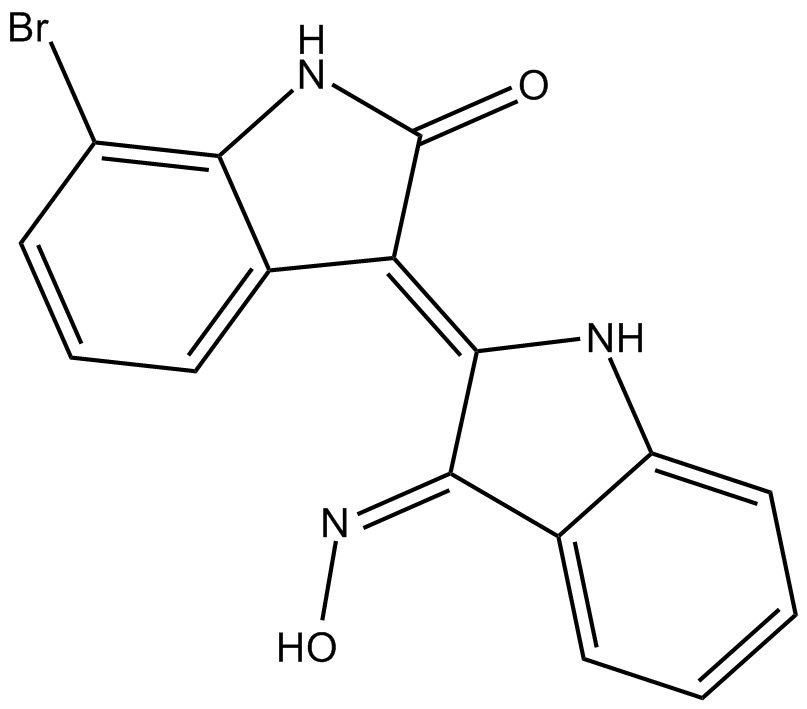
-
GC46741
8(E),10(E),12(Z)-Octadecatrienoic Acid
A conjugated PUFA

-
GC42622
8-bromo-Cyclic AMP
8-bromo-Cyclic AMP is a brominated derivative of cAMP that remains long-acting due to its resistance to degradation by cAMP phosphodiesterase.
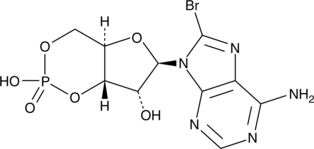
-
GC49275
8-Oxycoptisine
La 8-oxicoptisina es un alcaloide de protoberberina natural con actividad anticancerÍgena.
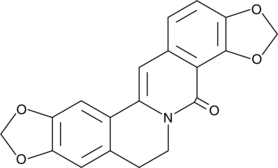
-
GC17119
8-Prenylnaringenin
La 8-prenilnaringenina es un prenilflavonoide aislado de conos de lúpulo (Humulus lupulus), con citotoxicidad. La 8-prenilnaringenina tiene actividad antiproliferativa contra las células de cáncer de colon HCT-116 a través de la inducción de la apoptosis mediada por la vía intrínseca y extrínseca. La 8-prenilnaringenina también promueve la recuperación de la atrofia muscular por desuso inducida por la inmovilización a través de la activación de la vía de fosforilación de Akt en ratones.
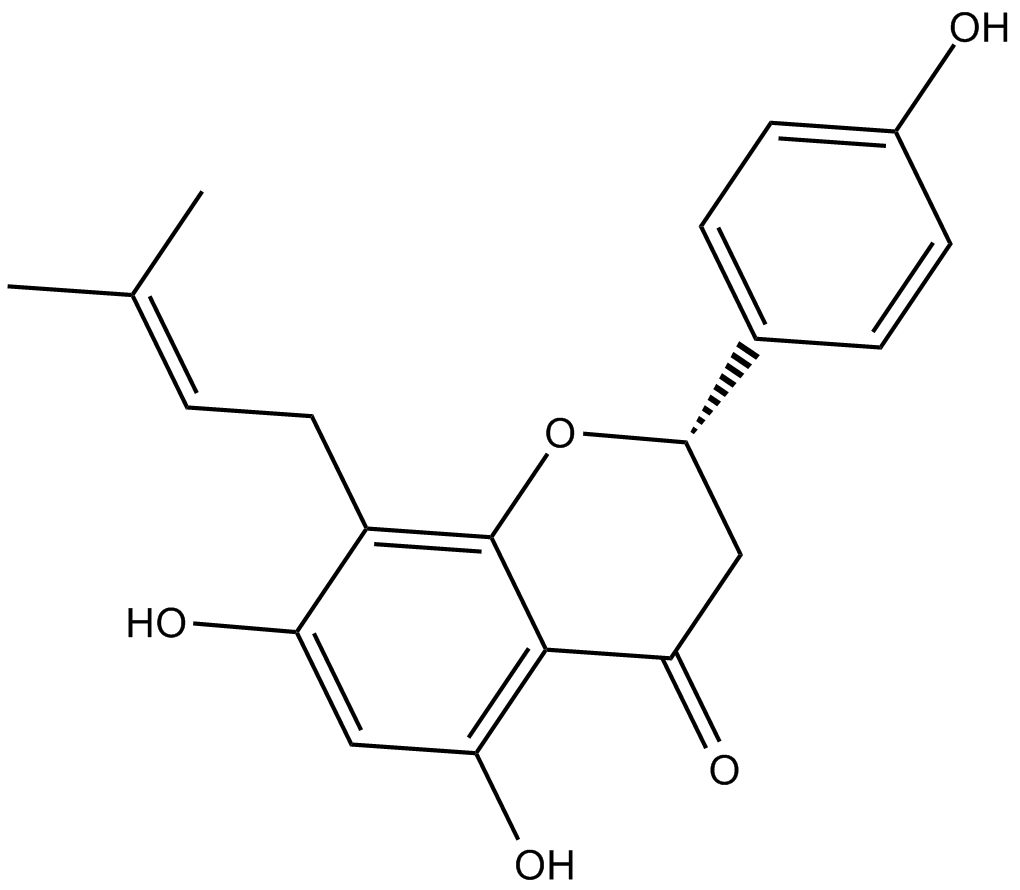
-
GC41642
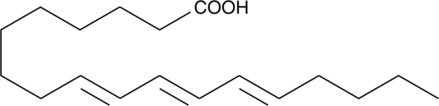
9(E),11(E),13(E)-Octadecatrienoic Acid
9(E),11(E),13(E)-Octadecatrienoic acid (β-ESA) is a conjugated polyunsaturated fatty acid that is found in plant seed oils and in mixtures of conjugated linolenic acids synthesized by the alkaline isomerization of linolenic acid.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
GC40785
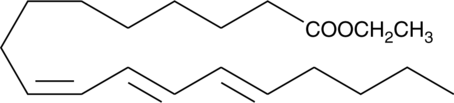
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC40710
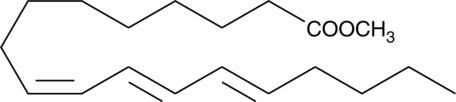
9(Z),11(E),13(E)-Octadecatrienoic Acid methyl ester
9Z,11E,13E-octadecatrienoic acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC39152
9-ING-41
9-ING-41 es un inhibidor de la glucÓgeno sintasa quinasa-3β (GSK-3β) selectivo y competitivo con ATP basado en maleimida con una IC50 de 0,71 μM. 9-ING-41 conduce significativamente a la detenciÓn del ciclo celular, la autofagia y la apoptosis en las células cancerosas. 9-ING-41 tiene actividad anticancerÍgena y tiene el potencial de potenciar los efectos antitumorales de los fÁrmacos quimioterapéuticos.
-
GN10035
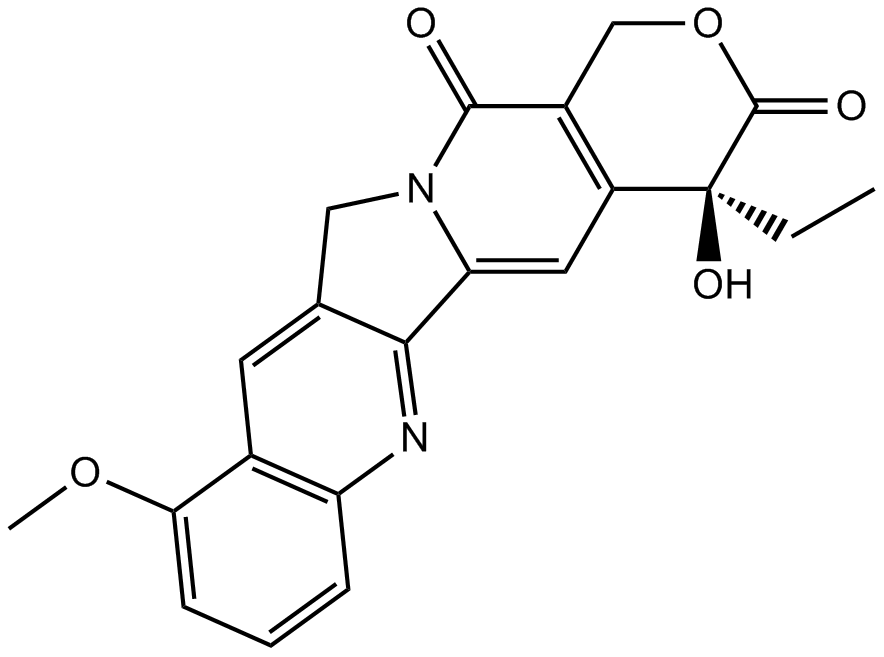
9-Methoxycamptothecin
-
GC45960
9c(i472)
9c(i472) es un potente inhibidor de 15-LOX-1 (15-lipoxigenasa-1) con un valor IC50 de 0,19 μM.
-
GC50465
A 410099.1
High affinity XIAP antagonist; active in vivo
-
GC17512
A-1155463
A-1155463 es un inhibidor de BCL-XL altamente potente y selectivo con un EC50 de 70 nM en células Molt-4.
-
GC16278
A-1210477
A-1210477 es un inhibidor potente y selectivo de MCL-1 con una Ki de 0,45 nM. A-1210477 se une especÍficamente a MCL-1 y promueve la apoptosis de las células cancerosas de manera dependiente de MCL-1.
-
GC17513
A-1331852
A-1331852 es un inhibidor selectivo de BCL-XL disponible por vÍa oral con una Ki de menos de 10 pM.
-
GC60544
A-192621
A-192621 es un potente antagonista del receptor de la endotelina B (ETB) selectivo, activo por vÍa oral, no peptÍdico y con una IC50 de 4,5 nM y una Ki de 8,8 nM.
-
GC32981
A-385358
A-385358 es un inhibidor selectivo de Bcl-XL con Kis de 0,80 y 67 nM para Bcl-XL y Bcl-2, respectivamente.
-
GC11200
A23187
A23187 (A-23187) es un antibiótico y un ionóforo de cationes divalentes único (como el calcio y el magnesio).
-
GC42659
A23187 (calcium magnesium salt)
A23187 is a divalent cation ionophore.
-
GC35216
AAPK-25
AAPK-25 es un inhibidor dual Aurora/PLK potente y selectivo con actividad antitumoral, que puede provocar un retraso mitÓtico y detener las células en una prometafase, reflejada por la fosforilaciÓn de la histona H3Ser10 del biomarcador y seguida de un aumento de la apoptosis. AAPK-25 se dirige a Aurora-A, -B y -C con valores de Kd que oscilan entre 23 y 289 nM, asÍ como a PLK-1, -2 y -3 con valores de Kd que oscilan entre 55 y 456 nM.
-
GC13805
Abacavir
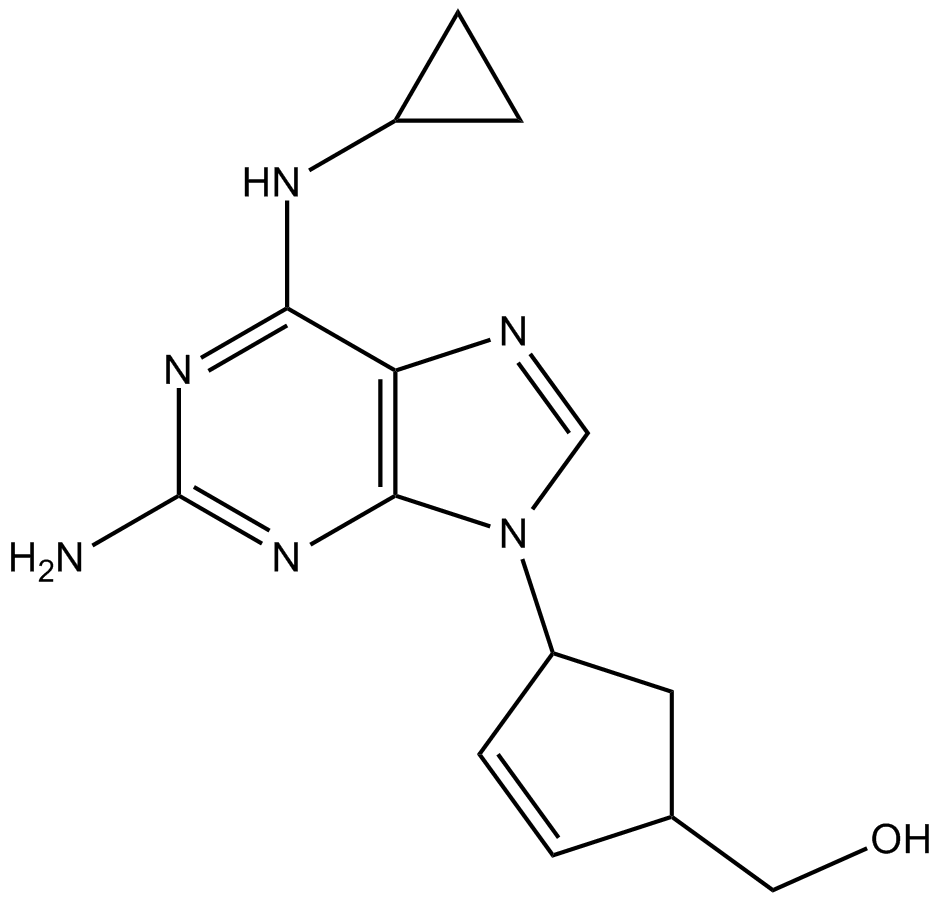
-
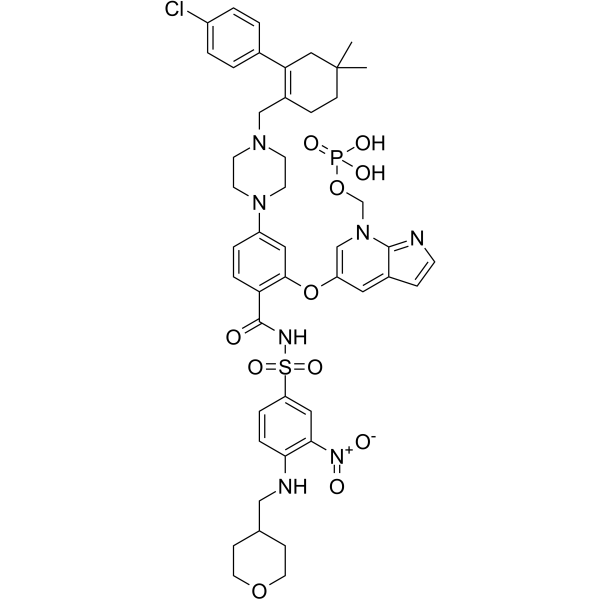
GC64674
ABBV-167
ABBV-167 es un profÁrmaco de fosfato del inhibidor de BCL-2 venetoclax.
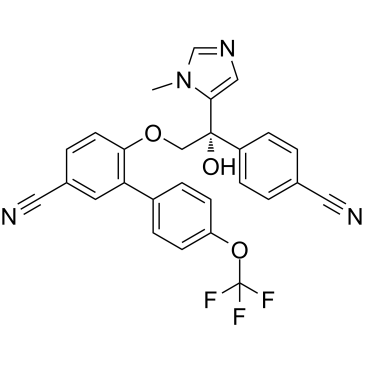
-
GC60548
ABT-100
ABT-100 es un inhibidor de farnesiltransferasa potente, altamente selectivo y activo por vÍa oral. ABT-100 inhibe la proliferaciÓn celular (IC50 de 2,2 nM, 3,8 nM, 5,9 nM, 6,9 nM, 9,2 nM, 70 nM y 818 nM para EJ-1, DLD-1, MDA-MB-231, HCT-116, MiaPaCa- 2, PC-3 y células DU-145, respectivamente), aumenta la apoptosis y disminuye la angiogénesis. ABT-100 posee actividad antitumoral de amplio espectro.
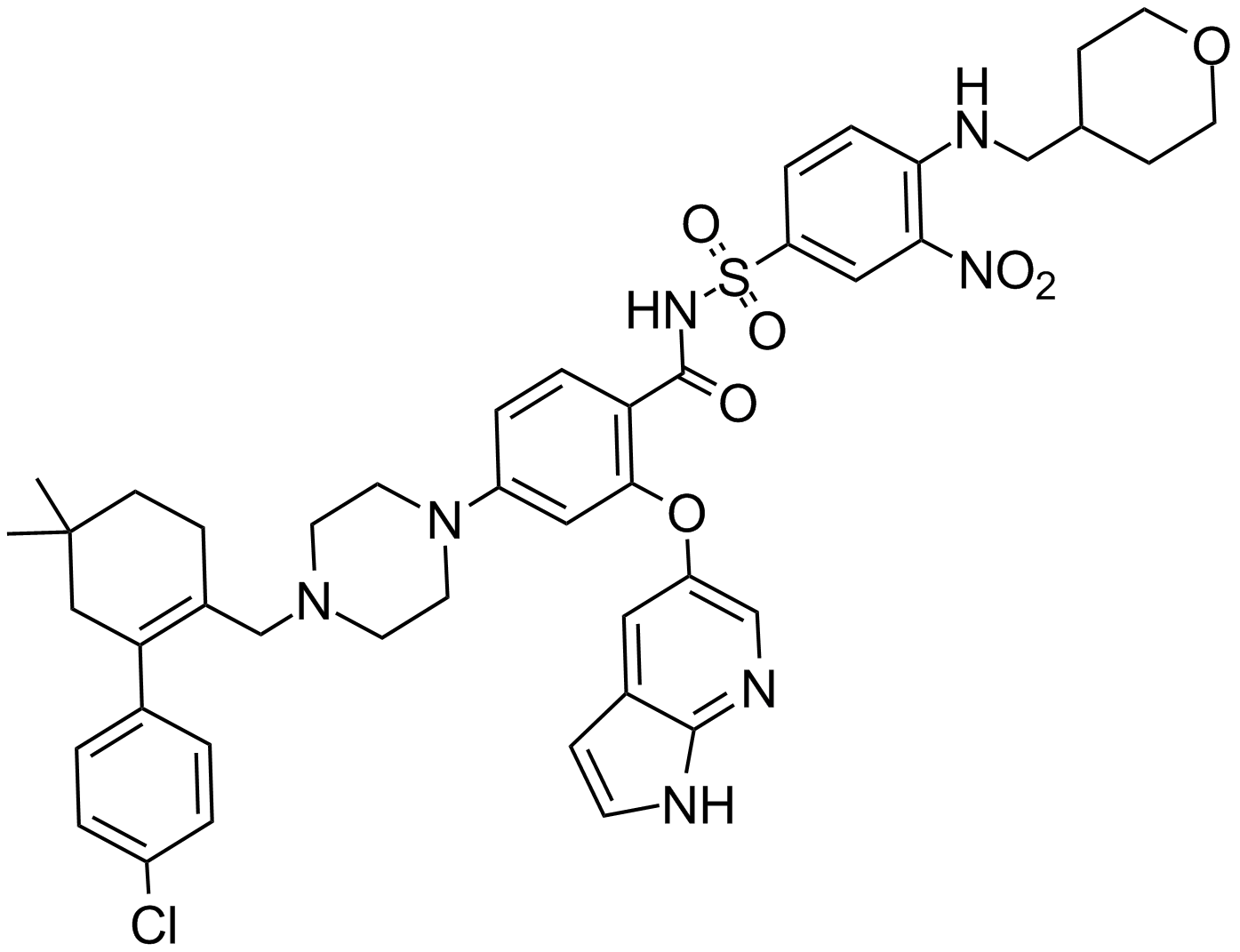
-
GC14069
ABT-199
Un inhibidor de Bcl-2
-
GC12405
ABT-263 (Navitoclax)
ABT-263 (Navitoclax) es un inhibidor de Bcl-xL, Bcl-2 y Bcl-w, con Ki ≤0,5 nM, ≤1 nM y ≤1 nM respectivamente.
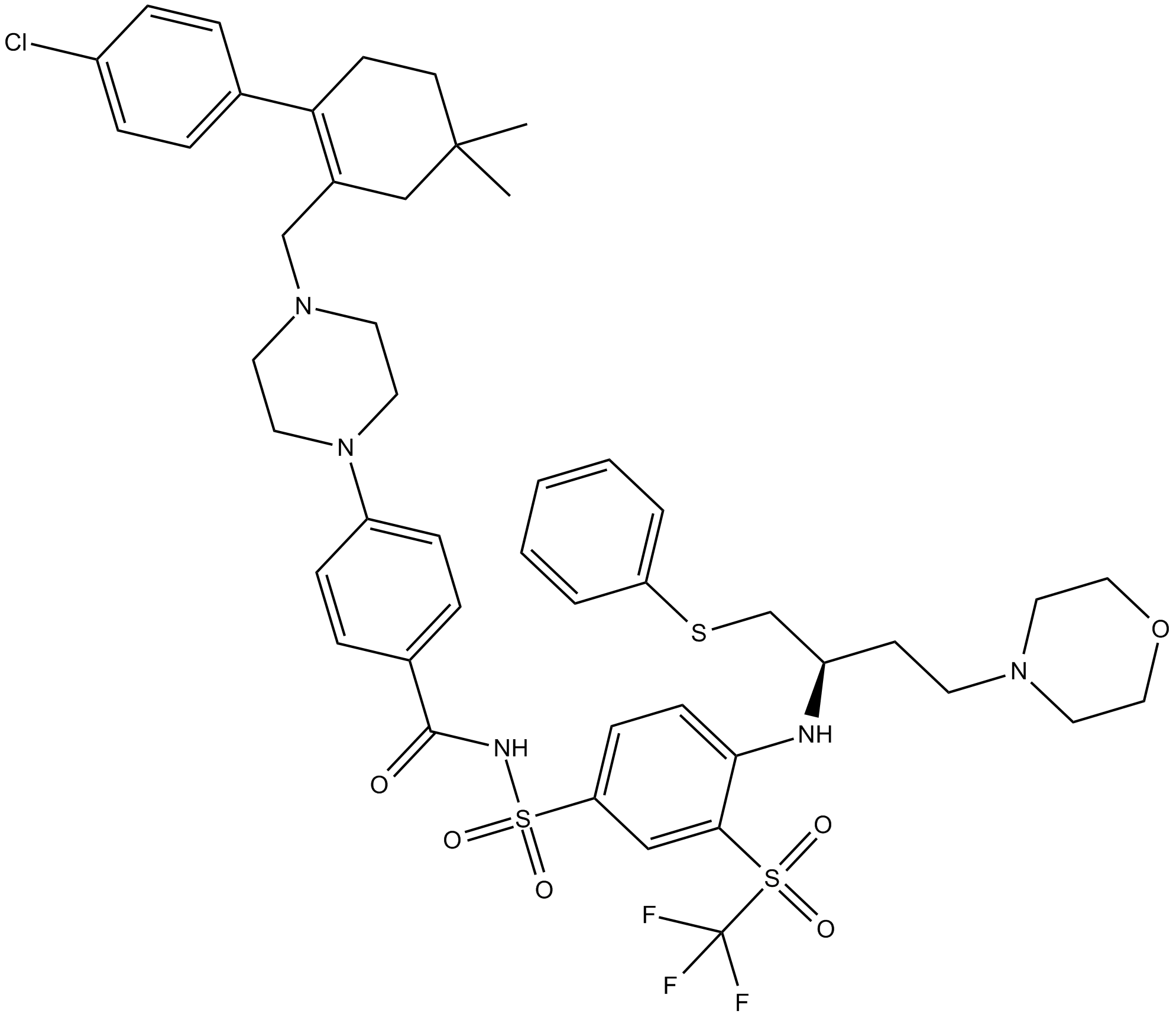
-
GC49745
ABT-263-d8
ABT-263-d8 es el deuterio etiquetado como Navitoclax. Navitoclax (ABT-263) es un inhibidor de proteÍnas de la familia Bcl-2 potente y activo por vÍa oral que se une a mÚltiples proteÍnas de la familia Bcl-2 antiapoptÓticas, como Bcl-xL, Bcl-2 y Bcl-w, con un Ki de menos de 1 nM.
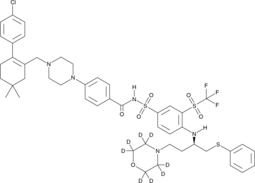
-
GC17234
ABT-737
An inhibitor of anti-apoptotic Bcl-2 proteins
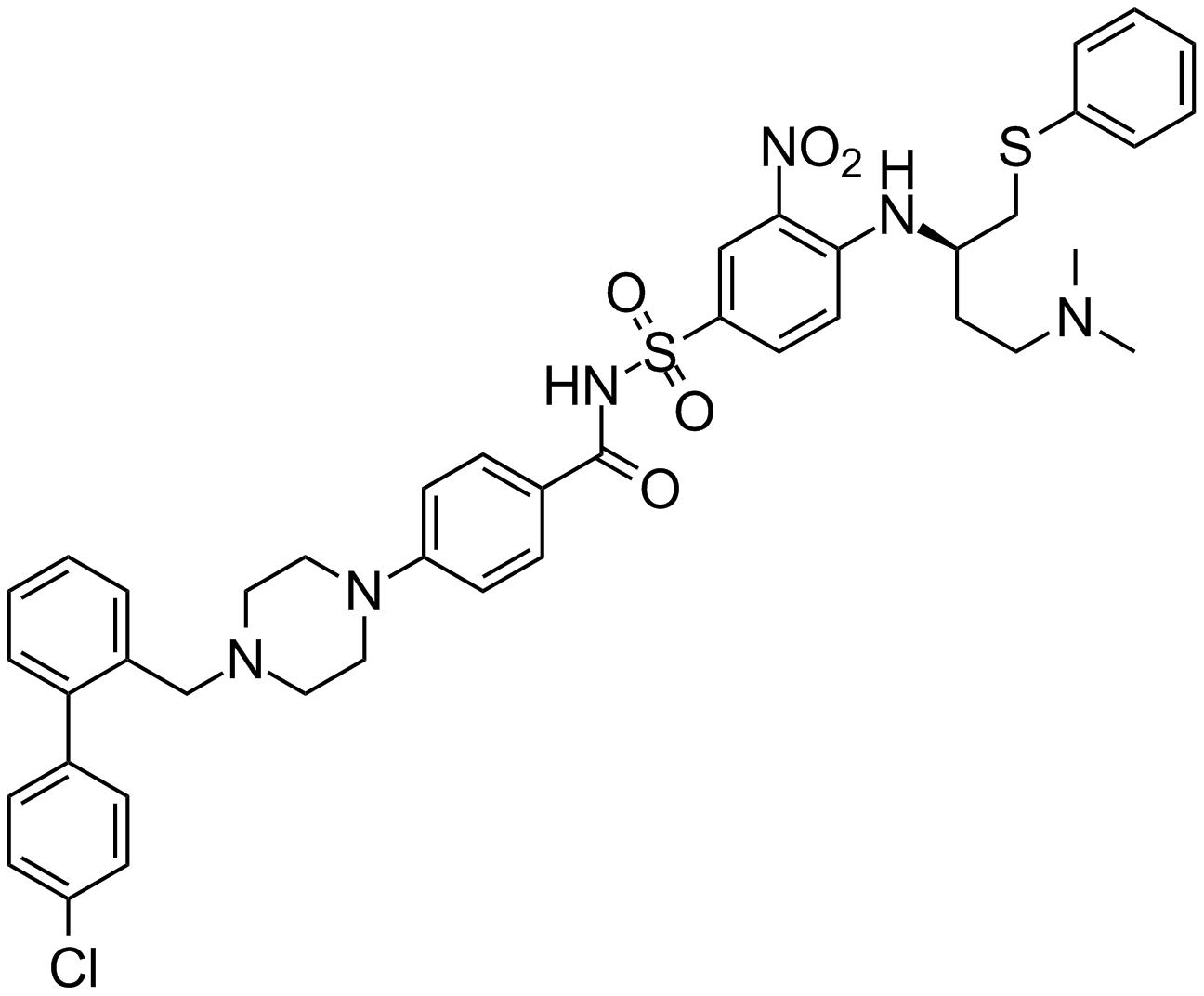
-
GA20494
Ac-Asp-Glu-Val-Asp-pNA
The cleavage of the chromogenic caspase-3 substrate Ac-DEVD-pNA can be monitored at 405 nm.

-
GC17602
Ac-DEVD-AFC
Ac-DEVD-AFC es un sustrato fluorogénico (Λex=400 nm, Λem=530 nm).
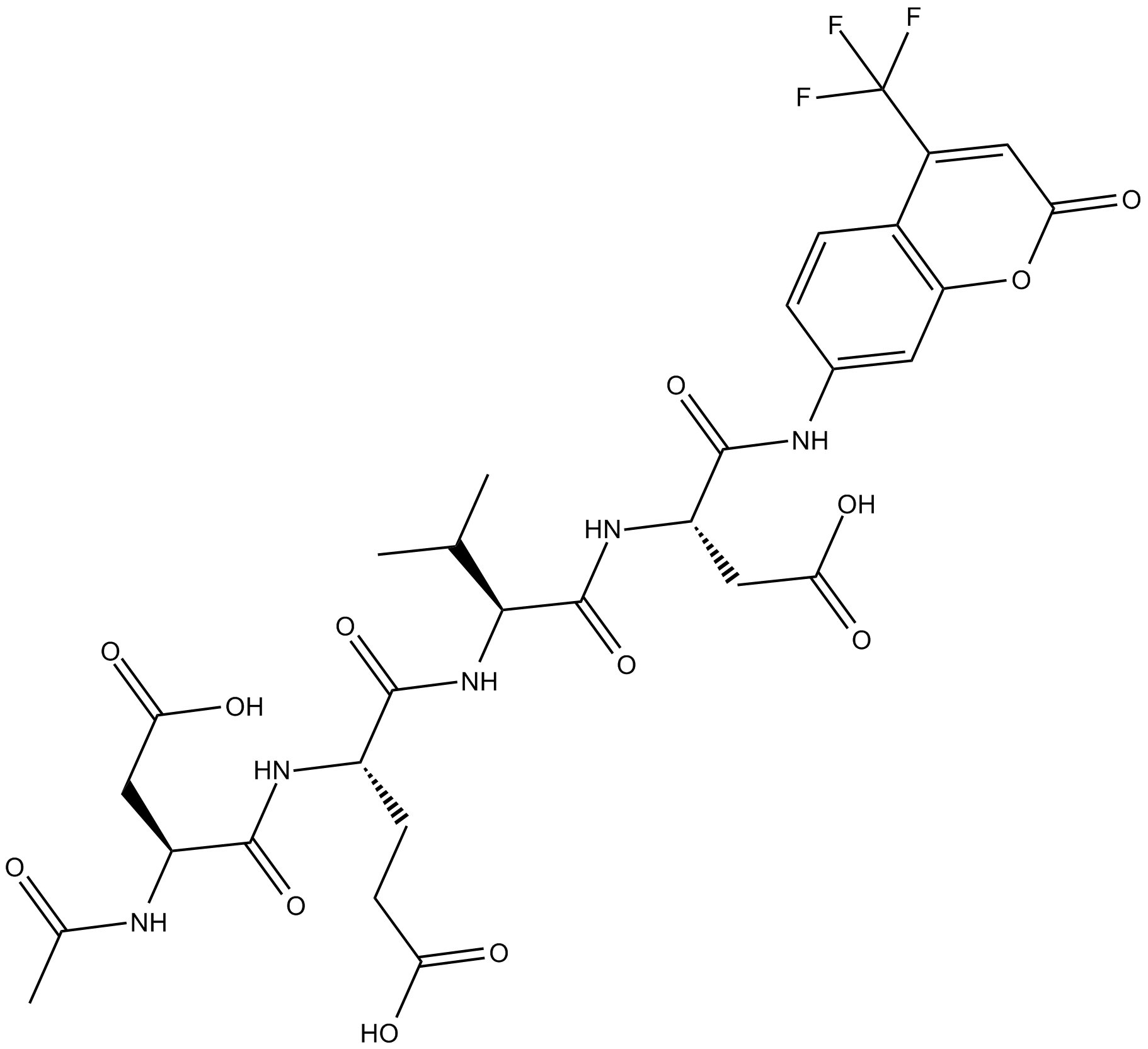
-
GC32695
Ac-DEVD-CHO
Ac-DEVD-CHO es un inhibidor especÍfico de Caspasa-3 con un valor de Ki de 230 pM.
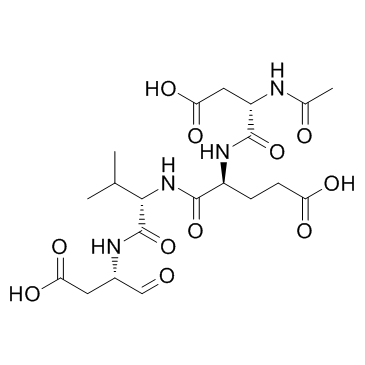
-
GC48470
Ac-DEVD-CHO (trifluoroacetate salt)
A dual caspase3/caspase7 inhibitor
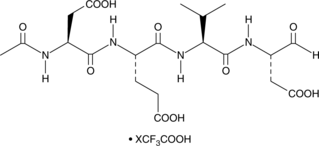
-
GC10951
Ac-DEVD-CMK
cell-permeable, and irreversible inhibitor of caspase
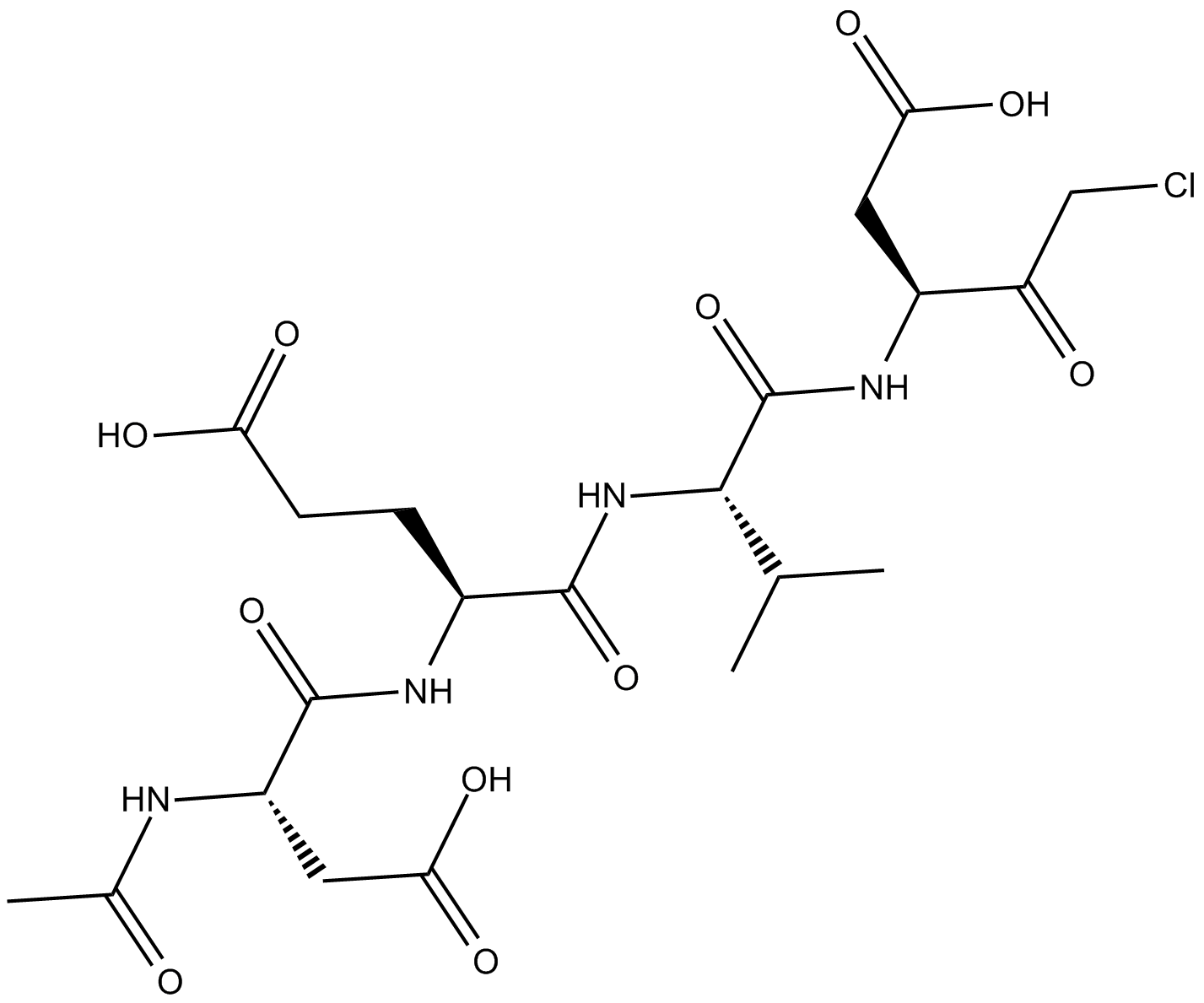
-
GC42689
Ac-DNLD-AMC
Ac-WLA-AMC es un sustrato fluorogénico de caspasa-3.
-
GC65107
Ac-FEID-CMK TFA
Ac-FEID-CMK TFA es un potente inhibidor peptÍdico derivado de GSDMEb especÍfico del pez cebra.
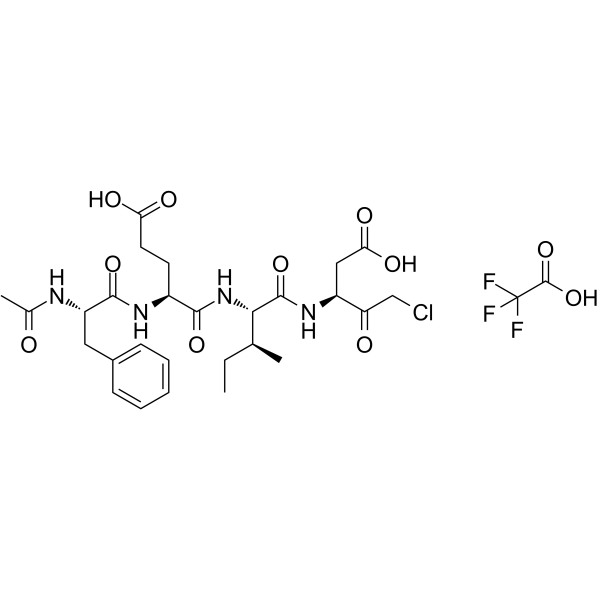
-
GC60558
Ac-FLTD-CMK
Ac-FLTD-CMK, un inhibidor derivado de la gasdermina D (GSDMD), es un inhibidor especÍfico de las caspasas inflamatorias.
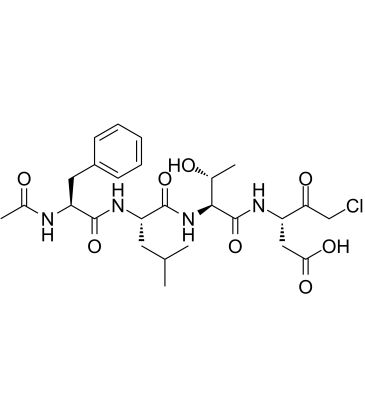
-
GC49704
Ac-FLTD-CMK (trifluoroacetate salt)
An inhibitor of caspase-1, -4, -5, and -11

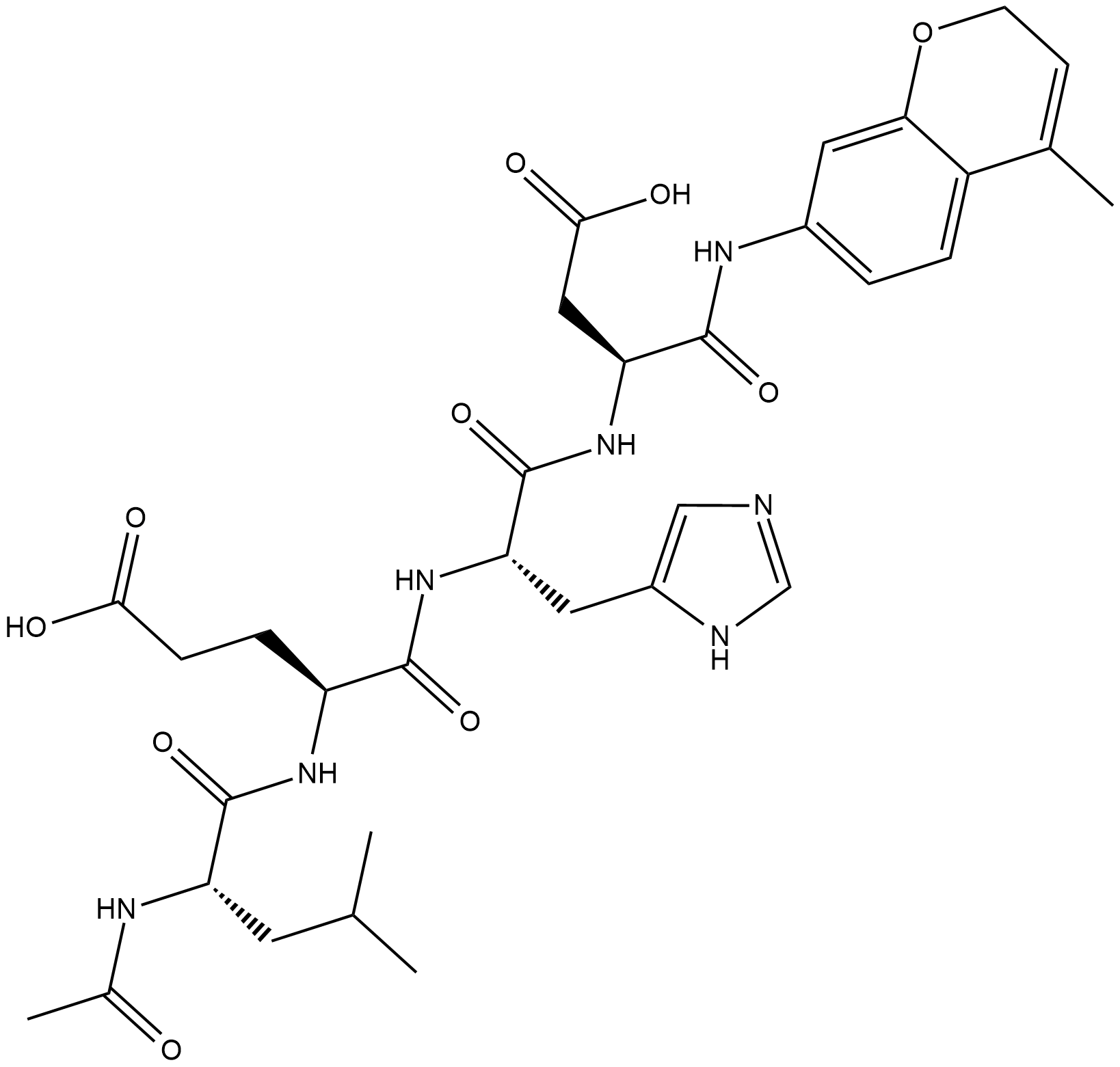
-
GC18226
Ac-LEHD-AMC (trifluoroacetate salt)
Ac-LEHD-AMC (sal de trifluoroacetato) es un sustrato fluorogénico para caspasa-9 (ExcitaciÓn: 341 nm; EmisiÓn: 441 nm).
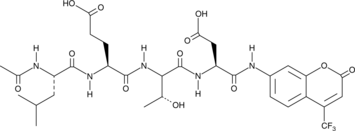
-
GC40556
Ac-LETD-AFC
Ac-LETD-AFC es un sustrato fluorogénico de caspasa-8.
-
GC13400
Ac-VDVAD-AFC
Ac-VDVAD-AFC es un sustrato fluorescente especÍfico de caspasa. Ac-VDVAD-AFC puede medir la actividad similar a la caspasa-3 y la actividad de la caspasa-2 y puede usarse para la investigaciÓn de tumores y cÁncer.
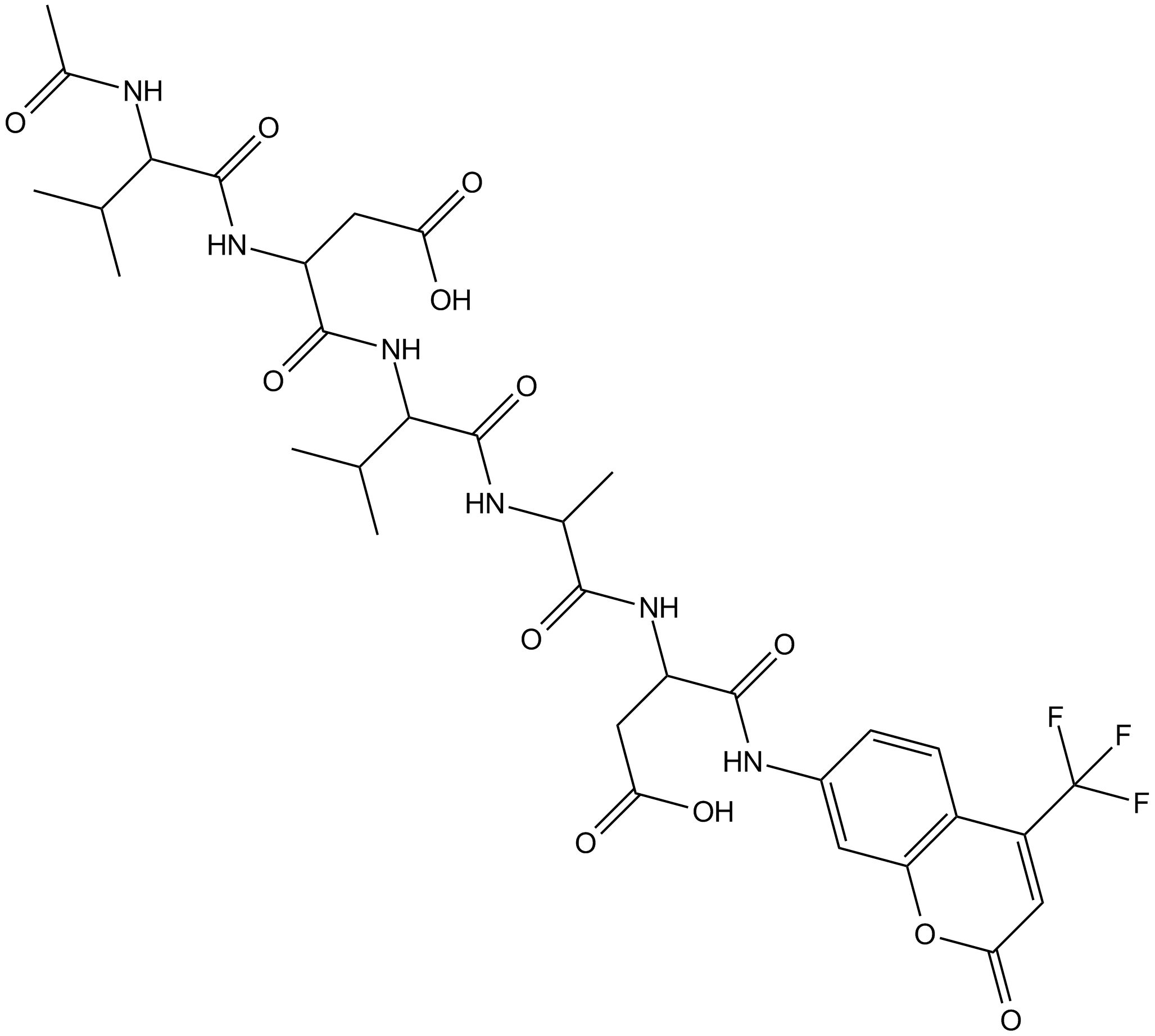
-
GC52372
Ac-VDVAD-AFC (trifluoroacetate salt)
A fluorogenic substrate for caspase-2
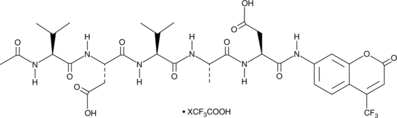
-
GC48974
Ac-VEID-AMC (ammonium acetate salt)
A caspase-6 fluorogenic substrate
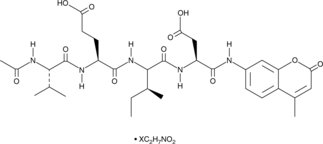
-
GC18021
Ac-YVAD-CHO
Ac-YVAD-CHO (L-709049) es un inhibidor de la enzima convertidora de interleucina-1β (ICE) tetrapeptÍdico, reversible, potente y especÍfico con valores Ki humanos y de ratÓn de 3,0 y 0,76 nM.
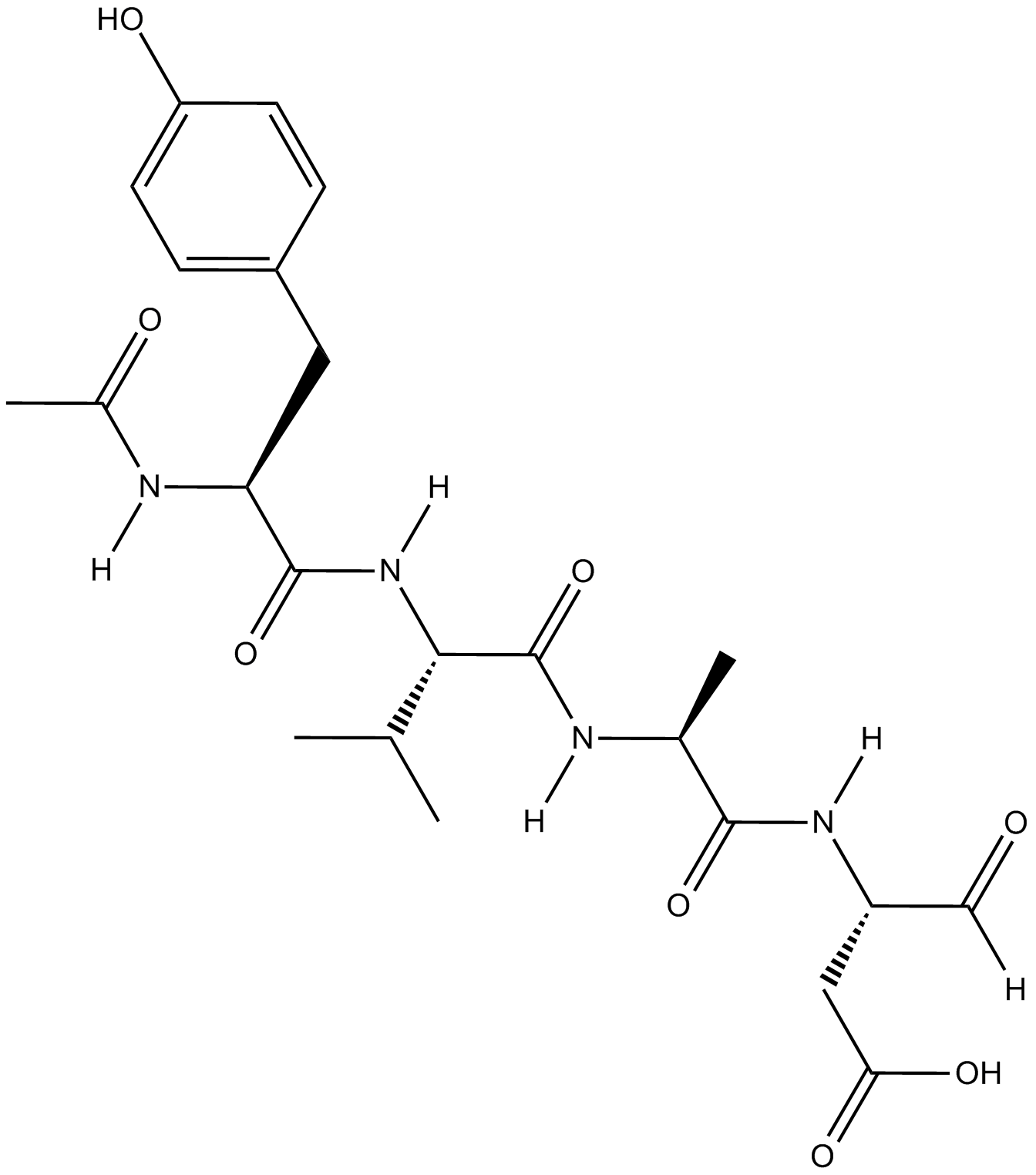
-
GC42721
Ac-YVAD-CMK
Ac-YVAD-CMK es una inhibición caspase-1 (Ki=0.8nM) que sin reversibilidad selectivacaspase-1 (Ki=0.8nM), se puede impedir la actividad de citoquinas proinflamatorias IL-1β. Ac-YVAD-CMK puede reducir la información e induce efectos neuroprotectores duraderos.
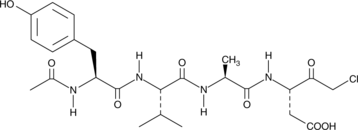
-
GC35227
ACBI1
ACBI1 es un degradador potente y cooperativo de SMARCA2, SMARCA4 y PBRM1 con DC50 de 6, 11 y 32 nM, respectivamente. ACBI1 es un degradador PROTAC. ACBI1 muestra actividad antiproliferativa. ACBI1 induce la apoptosis.
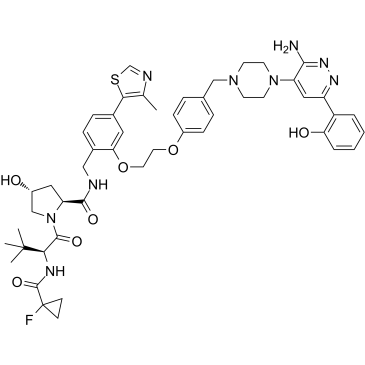
-
GN10341
Acetate gossypol
-
GC11786
Acetylcysteine
La acetilcisteína es el derivado N-acetil de la CISTEÍNA.
-
GC17094
Acitretin
La acitretina (Ro 10-1670) es un retinoide sistémico de segunda generaciÓn que se ha utilizado en el tratamiento de la psoriasis.
-
GC35242
Actein
La acteÍna es un glucÓsido triterpénico aislado de los rizomas de Cimicifuga foetida. La acteÍna suprime la proliferaciÓn celular, induce la autofagia y la apoptosis mediante la promociÓn de la activaciÓn de ROS/JNK y el bloqueo de la vÍa AKT en el cÁncer de vejiga humano. La acteÍna tiene poca toxicidad in vivo.
-
GC16866
Actinomycin D
Un bloqueador de transcripción que interactúa con el ADN y tiene actividad anticancerígena.

-
GC16350
Actinonin
La actinonina ((-)-actinonina) es un agente antibacteriano natural producido por Actinomyces.
-
GC16362
AD57 (hydrochloride)
polypharmacological cancer therapeutic that inhibits RET.
-
GC34214
Adalimumab (Anti-Human TNF-alpha, Human Antibody)
Adalimumab (anti-TNF-alfa humano, anticuerpo humano) es una de las terapias líderes para el tratamiento de la artritis reumatoide.



