Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nombre del producto Información
-
GC10610
Adapalene
El adapaleno (CD271), un retinoide sintético de tercera generaciÓn, se usa ampliamente para la investigaciÓn del acné.

-
GC46798
Adapalene-d3
An internal standard for the quantification of adapalene
-
GC13959
Adarotene
An atypical retinoid
-
GC65880
ADH-6 TFA
ADH-6 TFA es un compuesto de tripiridilamida. ADH-6 anula el autoensamblaje del subdominio de nucleaciÓn de agregaciÓn del mutante p53 DBD. ADH-6 TFA se dirige y disocia agregados de p53 mutantes en células cancerosas humanas, lo que restaura la actividad transcripcional de p53', lo que lleva a la detenciÓn del ciclo celular y la apoptosis. ADH-6 TFA tiene potencial para la investigaciÓn de enfermedades cancerosas.
-
GC42735
Adipostatin A
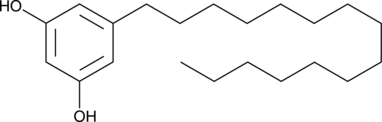
La adipostatina A (Adipostatina A) es un inhibidor de la glicerol-3-fosfato deshidrogenasa (GPDH) con una IC50 de 4,1 μM.
-
GC11892
AEE788 (NVP-AEE788)
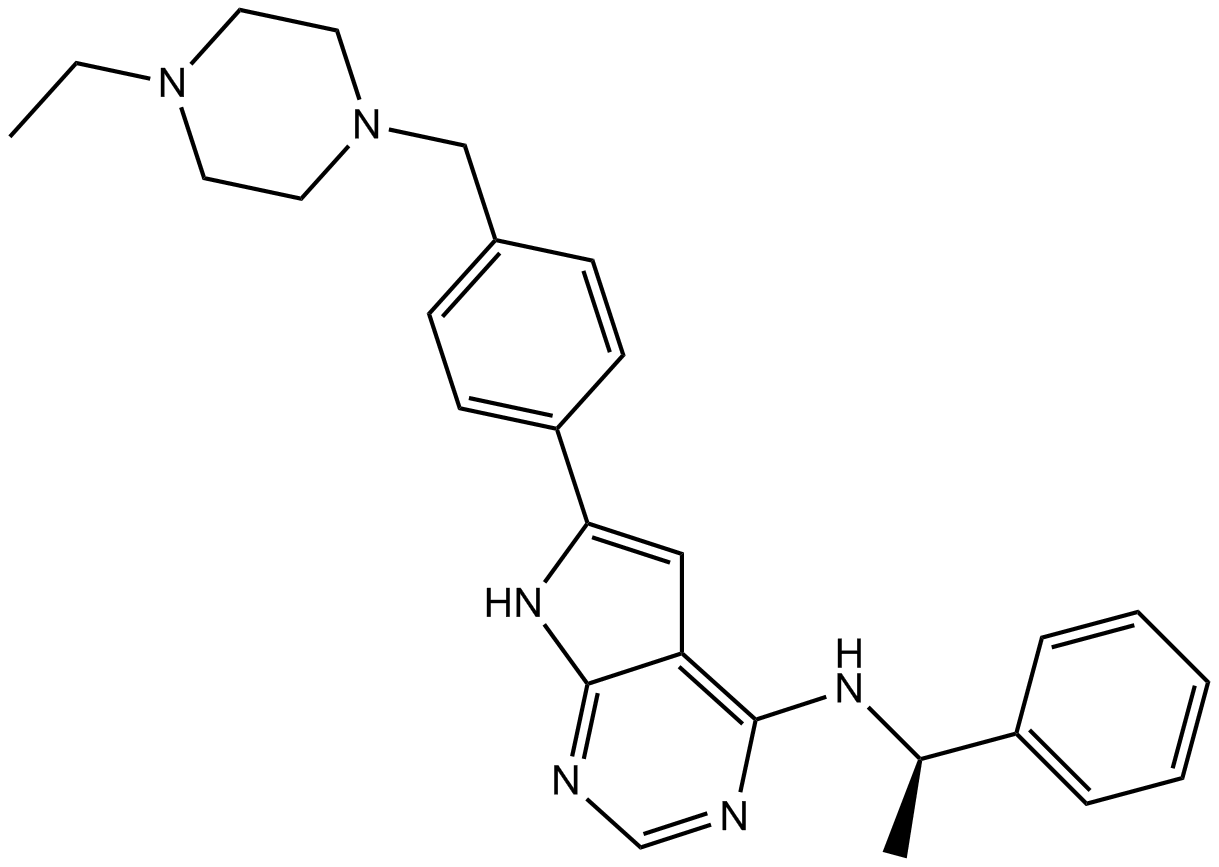
AEE788 (NVP-AEE788) es un inhibidor de EGFR y ErbB2 con valores IC50 de 2 y 6 nM, respectivamente.
-
GC42743
AEM1
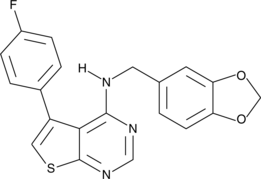
AEM1 es un inhibidor de Nrf2. AEM1 reduce las expresiones de genes dependientes de Nrf2 en células A549 e inhibe el crecimiento de células A549 in vitro e in vivo.
-
GC13168
AG 825
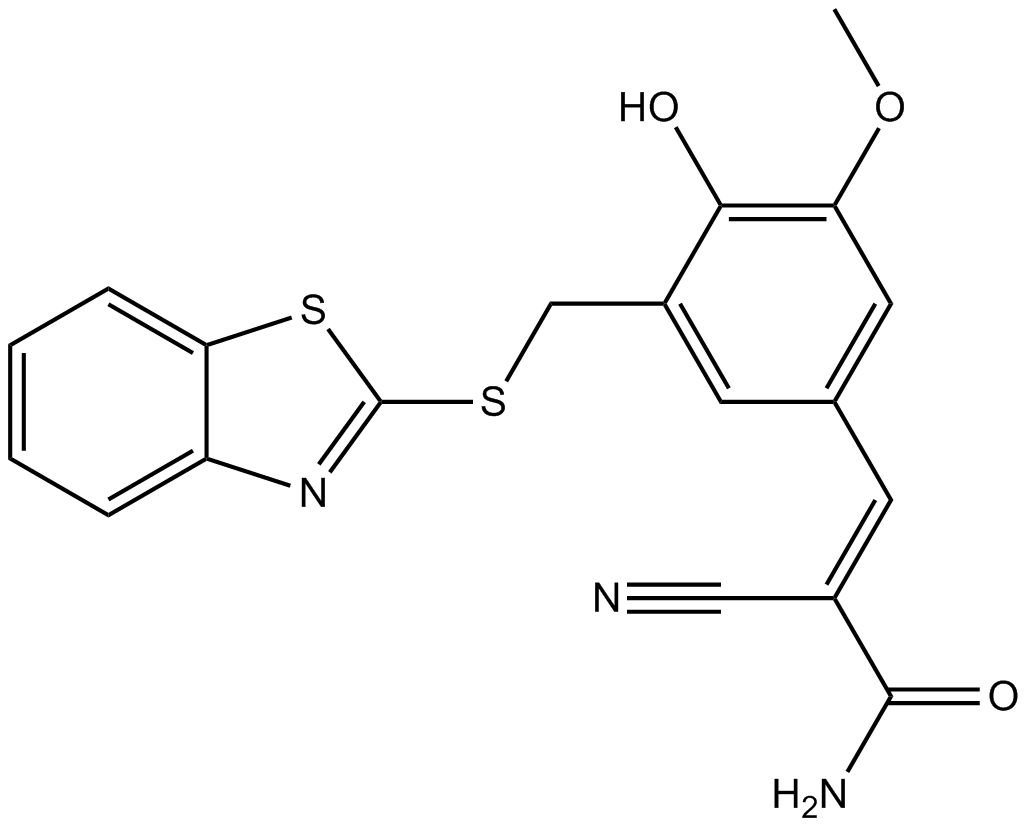
AG 825 (Tyrphostin AG 825) es un inhibidor de ErbB2 selectivo y competitivo con ATP que suprime la fosforilación de tirosina, con una IC50 de 0,35 μM.
-
GC13697
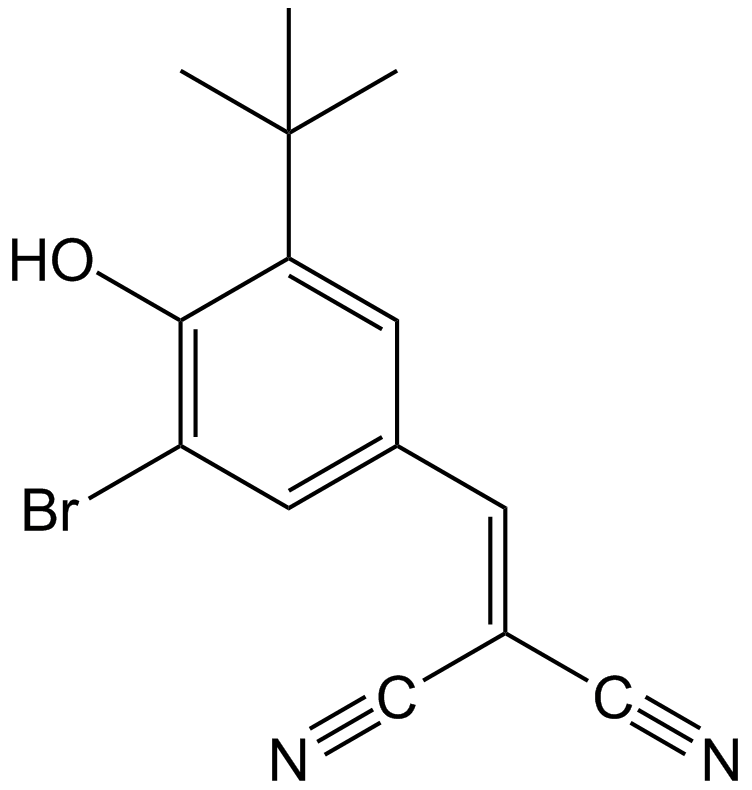
AG-1024
AG-1024 (Tyrphostin AG 1024) es un inhibidor de IGF-1R reversible, competitivo y selectivo con una IC50 de 7 μM.
-
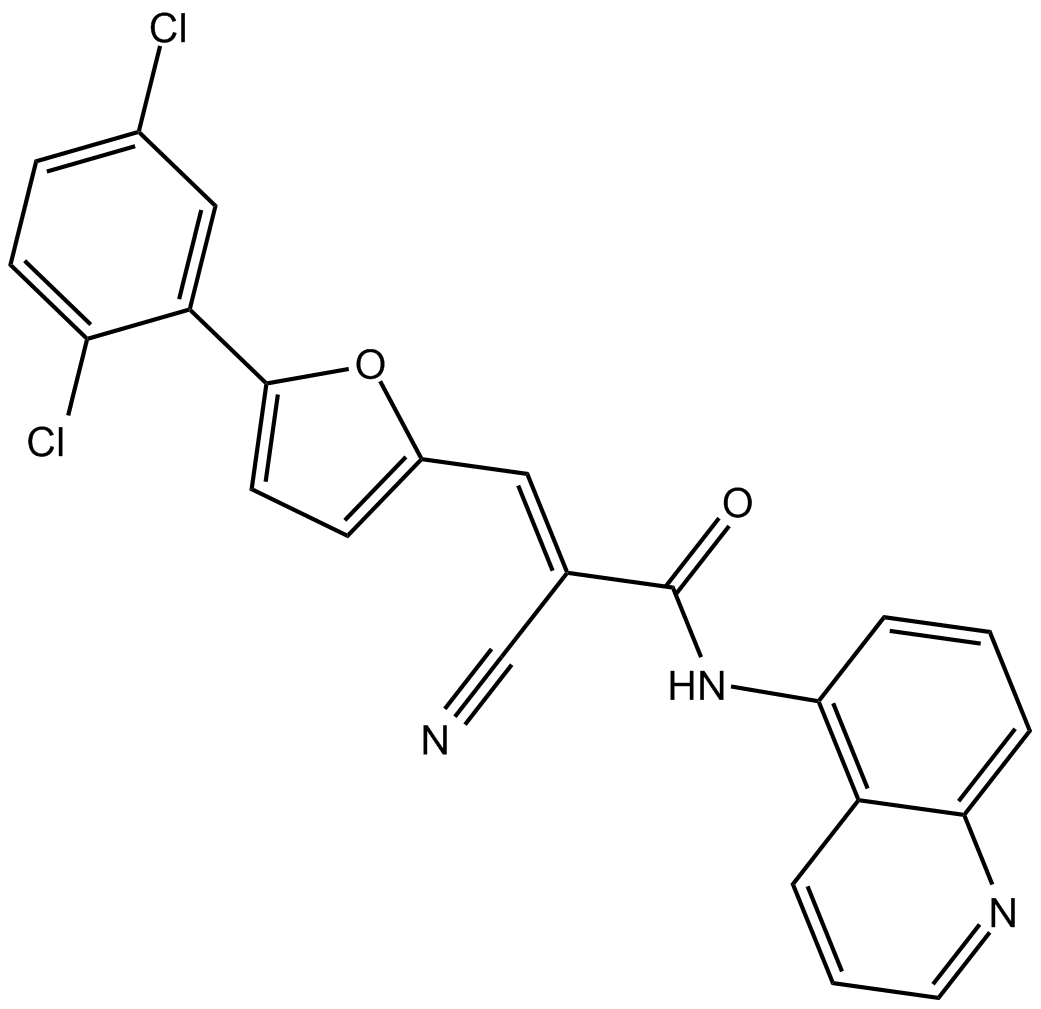
GC17881
AGK 2
AGK 2 es un inhibidor selectivo de SIRT2 con una IC50 de 3,5 μM. AGK 2 inhibe SIRT1 y SIRT3 con IC50 de 30 y 91 μM, respectivamente.
-
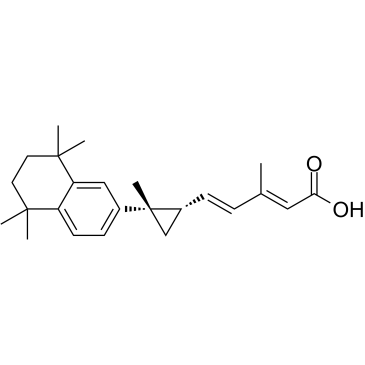
GC39584
AGN194204
AGN194204 (IRX4204) es un agonista selectivo y activo por vía oral de RXR con valores Kd de 0,4 nM, 3,6 nM y 3,8 nM y EC50s de 0,2 nM, 0,8 nM y 0.08 nM para RXRα, RXRβ y RXRγ respectivamente.
-
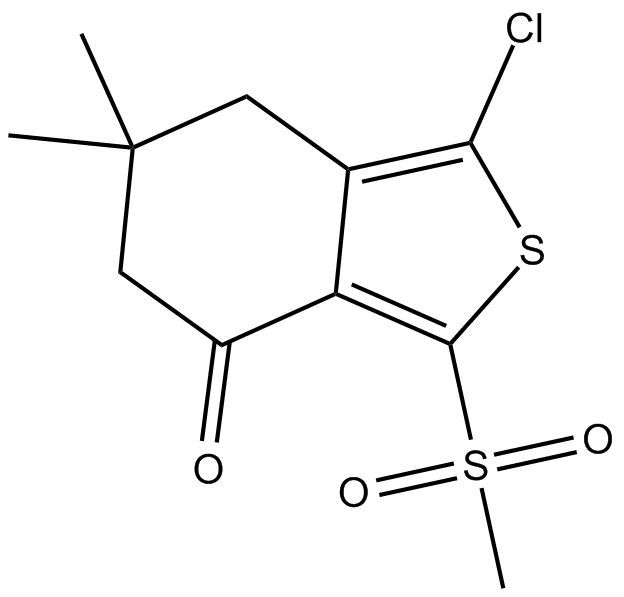
GC16120
AI-3
Nrf2/Keap1 and Keap1/Cul3 interaction inhibitor
-
GC46821
Ajoene
Ajoene, un compuesto derivado del ajo, es un agente antitrombÓtico y antifÚngico.
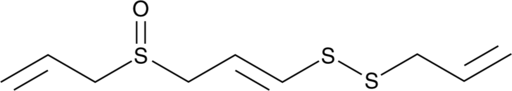
-
GC39620
AKOS-22
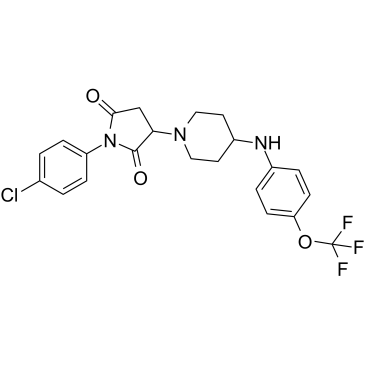
-
GC11589
AKT inhibitor VIII
A potent inhibitor of Akt1 and Akt2

-
GC35275
AKT-IN-3
AKT-IN-3 (compuesto E22) es un potente inhibidor de Akt bloqueador de hERG bajo activo por vÍa oral, con 1,4 nM, 1,2 nM y 1,7 nM para Akt1, Akt2 y Akt3, respectivamente. AKT-IN-3 (compuesto E22) también muestra una buena actividad inhibidora contra otras quinasas de la familia AGC, como PKA, PKC, ROCK1, RSK1, P70S6K y SGK. AKT-IN-3 (compuesto E22) induce la apoptosis e inhibe la metÁstasis de las células cancerosas.
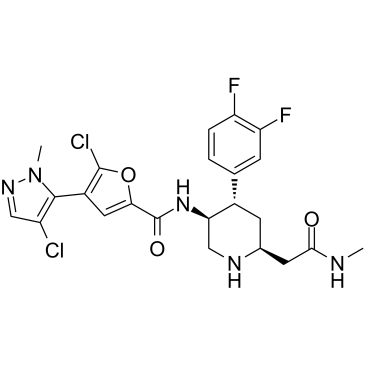
-
GC49773
Albendazole sulfone-d3
An internal standard for the quantification of albendazole sulfone
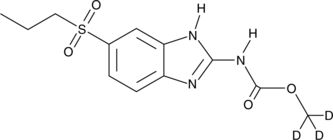
-
GC48848
Albendazole-d7
Albendazol-d7 (SKF-62979-d7) es el Albendazol marcado con deuterio.
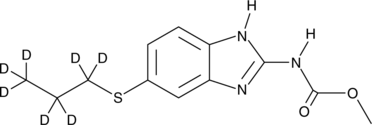
-
GC41080
Albofungin
Albofungin is a xanthone isolated from A.
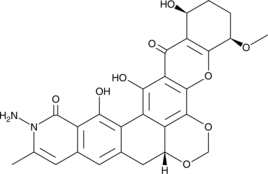
-
GC16597
Alda 1
Alda 1 es un agonista potente y selectivo de ALDH2, que activa ALDH2 de tipo salvaje y restaura la actividad casi natural de ALDH2*2.
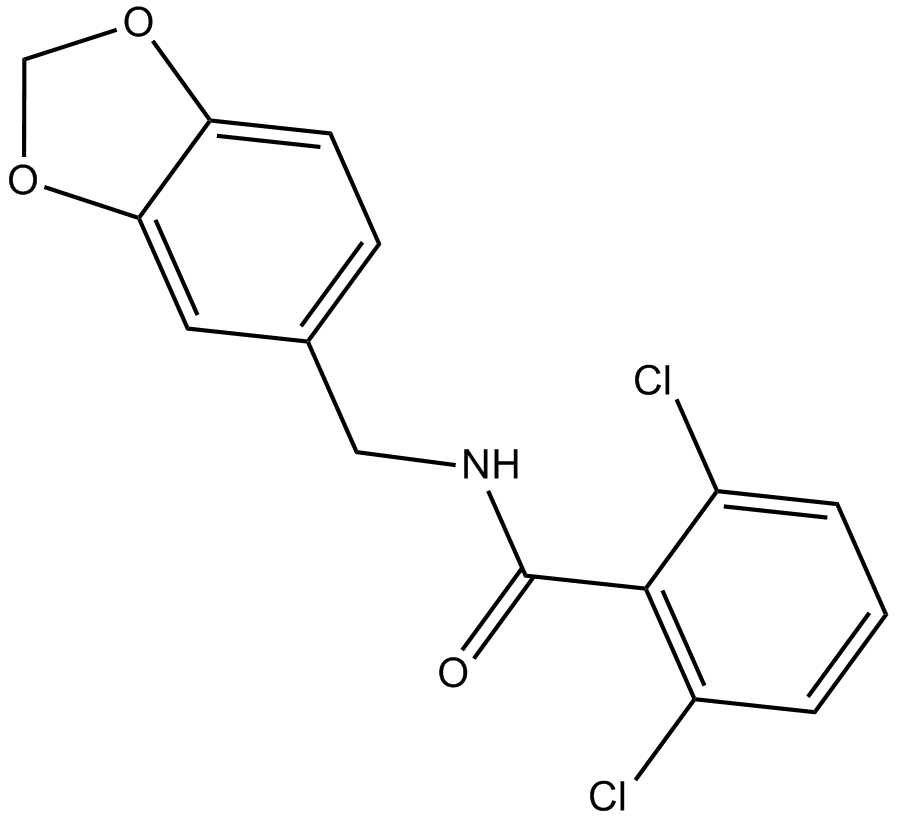
-
GC35288
Alkannin
Alkannin, que se encuentra en Alkanna tinctoria, se usa como colorante alimentario.
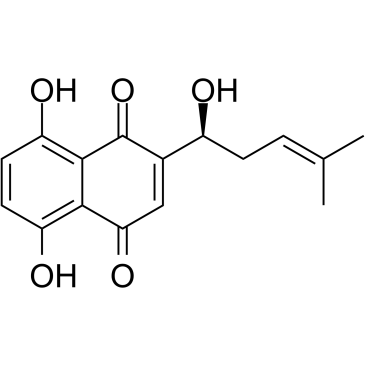
-
GC49393
all-trans-13,14-Dihydroretinol
A metabolite of all-trans retinoic acid

-
GC32127
Alofanib (RPT835)
Alofanib (RPT835) (RPT835) es un inhibidor alostérico potente y selectivo del receptor 2 del factor de crecimiento de fibroblastos (FGFR2).

-
GC14314
Aloperine
La aloperina es un alcaloide de plantas de sÓfora como Sophora alopecuroides L, que ha demostrado propiedades anticancerÍgenas, antiinflamatorias y antivirus.
-
GC35306
alpha-Mangostin
alpha-Mangostin (α-Mangostin) es una xantona dietética con amplias actividades biolÓgicas, como efectos antioxidantes, antialérgicos, antivirales, antibacterianos, antiinflamatorios y anticancerÍgenos. Es un inhibidor de la IDH1 mutante (IDH1-R132H) con una Ki de 2,85 μM.
-
GC18437
Alternariol monomethyl ether
El éter monometÍlico de alternariol, aislado de las raÍces de Anthocleista djalonensis (Loganiaceae), es un importante marcador taxonÓmico de la especie vegetal.
-
GC33356
AM-8735
AM-8735 es un inhibidor de MDM2 potente y selectivo con una IC50 de 25 nM.
-
GC42776
Amarogentin
La amarogentina es un glucÓsido secoiridoide que se extrae principalmente de las raÍces de Swertia y Gentiana.
-
GN10484
Amentoflavone
-
GC42783
Ametantrone
La ametantrona (NSC 196473) es un agente antitumoral que se intercala en el ADN e induce la ruptura del ADN mediada por la topoisomerasa II (TOP2).
-
GC19452
AMG-176
AMG-176 (Tapotoclax) es un inhibidor de MCL-1 potente, selectivo y oralmente activo, con un Ki de 0.13 nM.
-
GC15828
AMG232
AMG232 (AMG 232) es un inhibidor potente, selectivo y disponible por vÍa oral de la interacciÓn p53-MDM2, con una IC50 de 0,6 nM. AMG232 se une a MDM2 con una Kd de 0,045 nM.
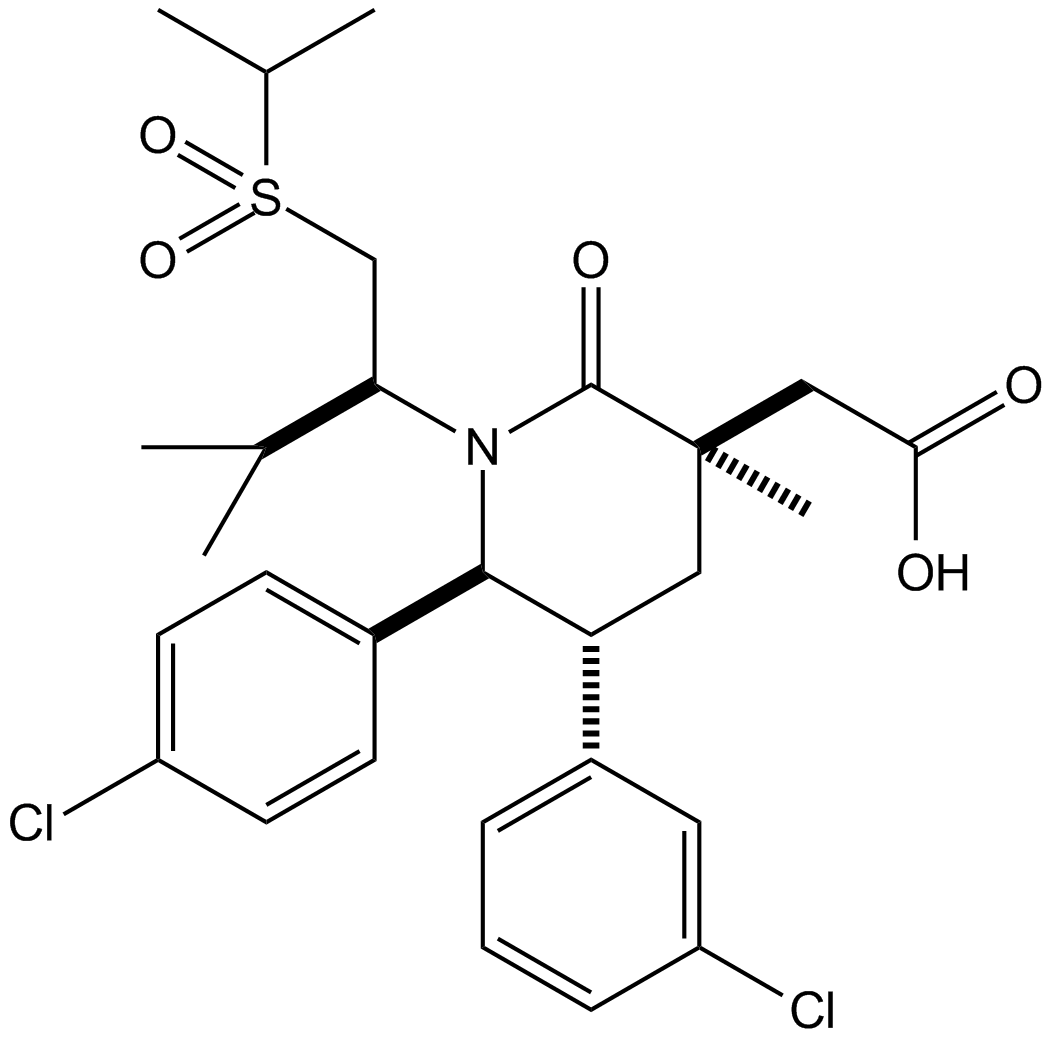
-
GC42785
Amifostine (hydrate)
La amifostina (hidrato) (WR2721 trihidrato) es un agente citoprotector de amplio espectro y un radioprotector. La amifostina (hidrato) protege selectivamente los tejidos normales del daÑo causado por la radiaciÓn y la quimioterapia. La amifostina (hidrato) es un potente factor inducible por hipoxia-α1 (HIF-α1) e inductor de p53. La amifostina (hidrato) protege las células del daÑo al eliminar los radicales libres derivados del oxÍgeno. La amifostina (hidrato) reduce la toxicidad renal y tiene acciÓn antiangiogénica.
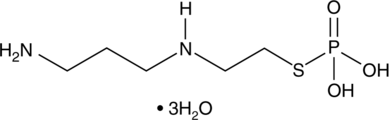
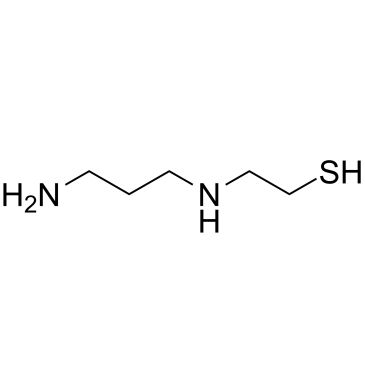
-
GC61804
Amifostine thiol
El tiol de amifostina (WR-1065) es un metabolito activo del citoprotector Amifostina. El tiol de amifostina es un agente citoprotector con capacidades radioprotectoras. El tiol de amifostina activa p53 a través de una vÍa de seÑalizaciÓn dependiente de JNK.
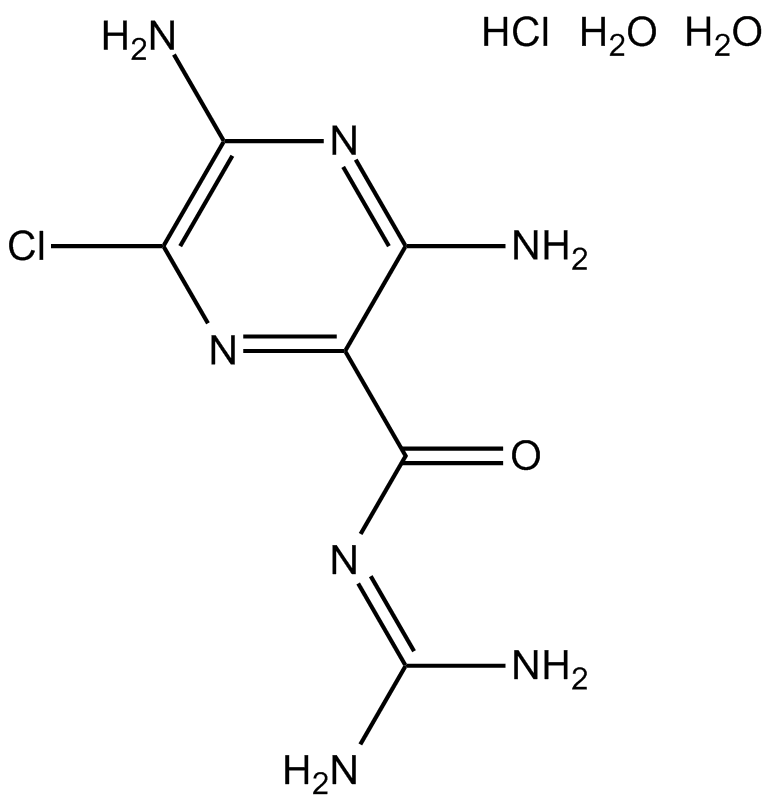
-
GC12051
Amiloride HCl dihydrate
El dihidrato de clorhidrato de amilorida (dihidrato de clorhidrato de MK-870) es un inhibidor tanto del canal de sodio epitelial (ENaC[1]) como del receptor del activador del plasminÓgeno de tipo uroquinasa (uTPA[2]).
-
GC63932
Amsilarotene
El amsilaroteno (TAC-101; Am 555S), un retinoide sintético activo por vÍa oral, tiene afinidad selectiva por el receptor de Ácido retinoico α (RAR-α) uniÓn con Ki de 2,4, 400 nM para RAR-α y RAR-β. El amsilaroteno induce la apoptosis de células de cÁncer gÁstrico humano, carcinoma hepatocelular y carcinoma de ovario. El amsilaroteno se puede utilizar para la investigaciÓn del cÁncer.
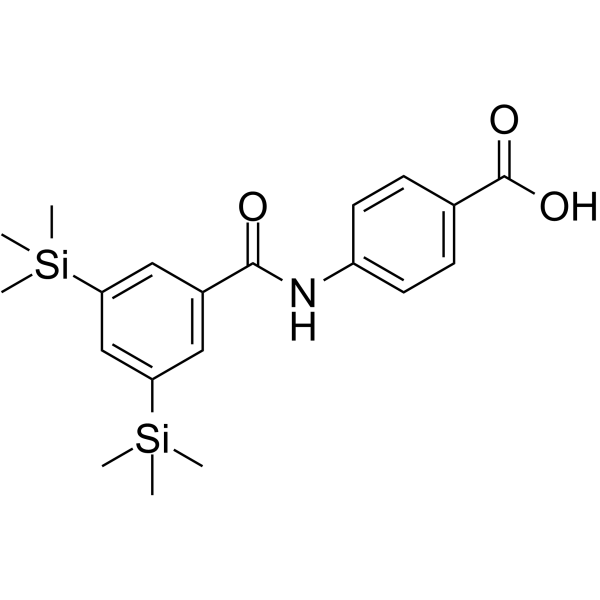
-
GC16391
Amuvatinib (MP-470, HPK 56)
A multi-targeted RTK inhibitor
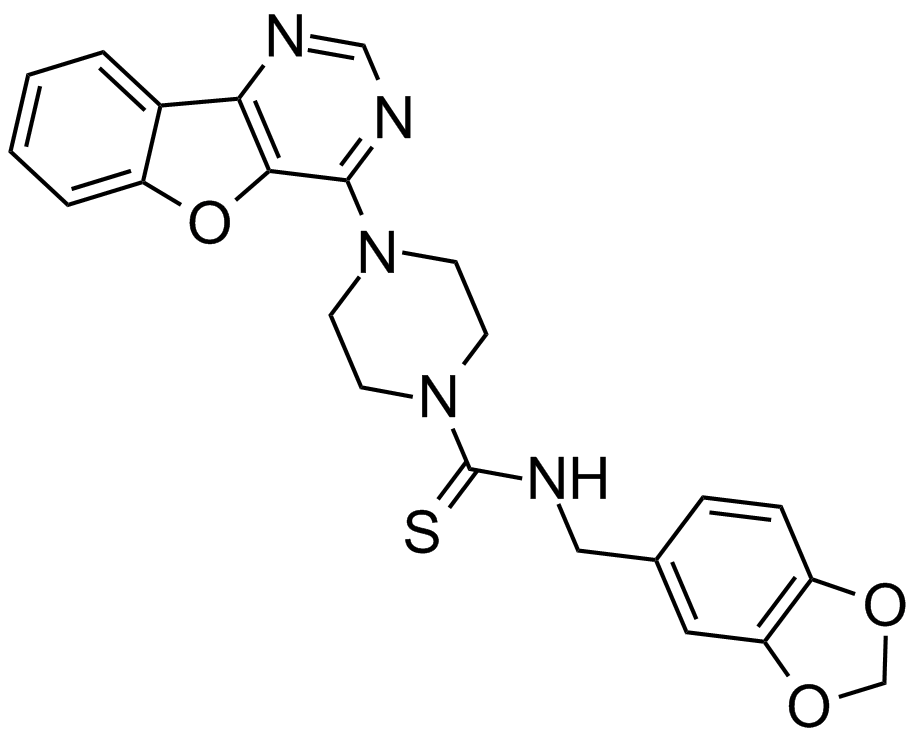
-
GC48339
Amycolatopsin A
A macrolide polyketide with antimycobacterial and anticancer activities
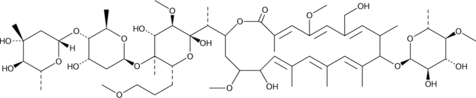
-
GC48341
Amycolatopsin B
A bacterial metabolite
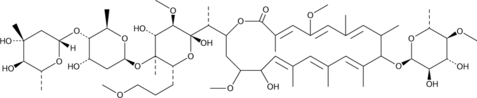
-
GC48350
Amycolatopsin C
A polyketide macrolide with antimycobacterial and anticancer activities
-
GC42806
Andrastin A
Andrastin A is a meroterpenoid farnesyltransferase inhibitor.
-
GN10045
Angelicin
-
GC60584
Angiotensin II human acetate
El acetato de angiotensina II humana (angiotensina II) es un vasoconstrictor y un péptido bioactivo principal del sistema renina/angiotensina.
-
GC42813
Anguinomycin A
Anguinomycin A is an antibiotic first isolated from a Streptomyces sp.
-
GC40614
Anhydroepiophiobolin A
Anhydroepiophiobolin A, un anÁlogo de Ophiobolin A, es un potente inhibidor de la fotosÍntesis (I50 de 6,1 y 1 mM para la fotosÍntesis en Chlorella y Spinach, respectivamente).
-
GC40214
Anhydroophiobolin A
Anhydroophiobolin A es un potente inhibidor de la fotosÍntesis con IC50 de 77 y 14 mM en la fotosÍntesis de chlorella y espinaca, respectivamente.
-
GC11559
Anisomycin
Agonista de JNK, potente y específico.
-
GC49259
Antagonist G (trifluoroacetate salt)
A neuropeptide antagonist
-
GC66337
Anti-Mouse PD-L1 Antibody
Anti-Mouse PD-L1 Antibody es un inhibidor de anticuerpos IgG2b anti-mouse PD-L1 derivado de la rata huésped.

-
GC35361
Antineoplaston A10
Antineoplaston A10, una sustancia natural en el cuerpo humano, es un inhibidor de Ras potencialmente para el tratamiento de glioma, linfoma, astrocitoma y cÁncer de mama.
-
GC34172
AP1867
AP1867 es un ligando dirigido por FKBP12F36V sintético.
-
GC61745
AP1867-2-(carboxymethoxy)
AP1867-2-(carboximetoxi), el resto basado en AP1867 (un ligando sintético dirigido por FKBP12F36V), se une al ligando CRBN a través de un conector para formar moléculas dTAG.

-
GC15586
AP1903
AP1903 (AP1903) es un agente dimerizador que actÚa entrecruzando los dominios FKBP. AP1903 (AP1903) dimeriza el interruptor suicida Caspase 9 e induce rÁpidamente la apoptosis.
-
GC14498
AP20187
AP20187 (B/B Homodimerizer) es un ligando permeable a las células que se utiliza para dimerizar las proteÍnas de fusiÓn de la proteÍna de uniÓn a FK506 (FKBP) e iniciar cascadas de seÑalizaciÓn biolÓgica y expresiÓn génica o interrumpir las interacciones proteÍna-proteÍna.
-
GC18518
Apcin
Apcin, un ligando de Cdc20, es un inhibidor potente y competitivo de la actividad de la ligasa E3 del complejo promotor de la anafase/ciclosoma (APC/C(Cdc20)). Apcin inhibe competitivamente la ubiquitilaciÓn dependiente de APC/C al unirse a Cdc20 y prevenir el reconocimiento del sustrato. Apcin ocupa el bolsillo de uniÓn de la caja D en la cara lateral del dominio WD40 y puede prolongar la mitosis.
-
GC62419
Apcin-A
Apcin-A, un derivado de Apcin, es un inhibidor del complejo promotor de la anafase (APC). Apcin-A interactÚa fuertemente con Cdc20 e inhibe la ubiquitinaciÓn de sustratos de Cdc20. Apcin-A se puede utilizar para sintetizar el PROTAC CP5V.
-
GC35367
APG-115
APG-115 (APG-115) es un inhibidor de la proteÍna MDM2 activo por vÍa oral que se une a la proteÍna MDM2 con valores IC50 y Ki de 3,8 nM y 1 nM, respectivamente. APG-115 bloquea la interacciÓn de MDM2 y p53 e induce la detenciÓn del ciclo celular y la apoptosis de manera dependiente de p53.
-
GC62640
APG-1387
APG-1387, un mimético bivalente de SMAC y un antagonista de IAP, bloquea la actividad de las proteÍnas de la familia de IAP (XIAP, cIAP-1, cIAP-2 y ML-IAP). APG-1387 induce la degradaciÓn de las proteÍnas cIAP-1 y XIAP, asÍ como la activaciÓn de caspasa-3 y la escisiÓn de PARP, lo que conduce a la apoptosis. APG-1387 se puede utilizar para la investigaciÓn de carcinoma hepatocelular, cÁncer de ovario y carcinoma nasofarÍngeo.
-
GC12961
Apicidin
Un inhibidor de HDAC permeable a través de la membrana celular.
-
GC46862
Apigenin-d5
An internal standard for the quantification of apigenin
-
GC16237
Apocynin
La apocinina es un inhibidor selectivo de la NADPH-oxidasa con una IC50 de 10 μM.
-
GC14080
Apogossypolone (ApoG2)
-
GC42827
Apoptolidin
La apoptolidina es un policétido aislado de la bacteria Nocardiopsis. La apoptolidina es un inhibidor selectivo de la F1FO ATPasa mitocondrial. La apoptolidina es un inductor de apoptosis e induce la muerte celular por apoptosis en células transformadas con oncogenes de adenovirus tipo 12, incluido ElA (CI50 = 10-17 ng/ml), pero no en células normales.
-
GC14209
Apoptosis Activator 2
An activator of caspases
-
GC14411
Apoptozole
El apoptozol (Apoptosis Activator VII) es un inhibidor del dominio ATPasa de Hsc70 y Hsp70, con Kds de 0,21 y 0,14μM, respectivamente, y puede inducir la apoptosis.
-
GC65004
Apostatin-1
La apostatina-1 (Apt-1) es un potente inhibidor de TRADD.
-
GC35377
Apratastat
Apratastat (TMI-005) es un inhibidor de TACE/MMP activo por vÍa oral, no selectivo y reversible, que puede inhibir la liberaciÓn de TNF-α. Apratastat tiene el potencial de superar la resistencia a la radioterapia en el cÁncer de pulmÓn de células no pequeÑas (NSCLC).
-
GC10420
Apremilast (CC-10004)
An orally available PDE4 inhibitor
-
GC32692
APTO-253 (LOR-253)
APTO-253 es una molécula pequeña novedosa que ejerce una potente actividad antitumoral induciendo la expresión del gen del factor de transcripción maestro Kruppel-like factor 4 (KLF4), inhibiendo así el ciclo celular y conduciendo a la muerte celular programada.
-
GC14590
AR-42 (OSU-HDAC42)
HDAC inhibitor,novel and potent
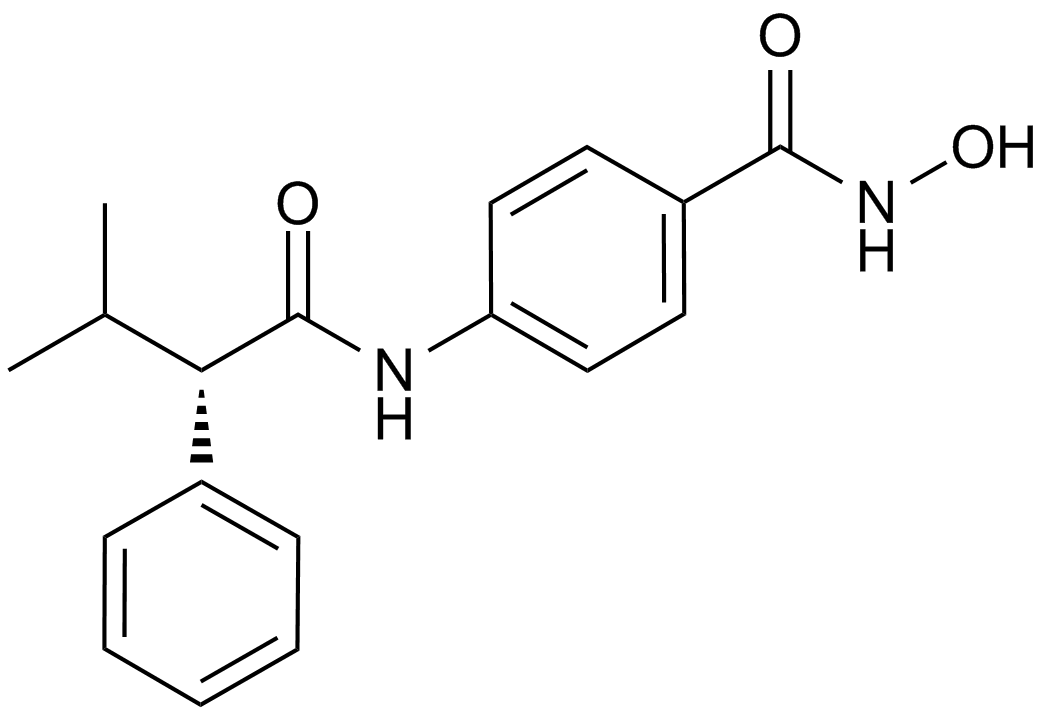
-
GC45385
Ara-G
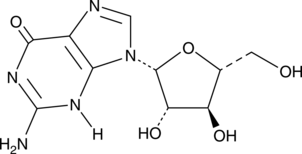
-
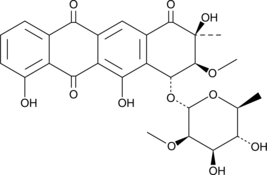
GC46878
Aranciamycin
A fungal metabolite with diverse biological activities
-
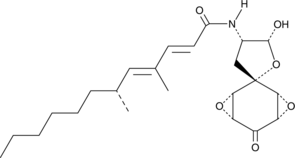
GC40116
Aranorosin
La aranorosina, un potente antibiÓtico antifÚngico, se ha aislado del filtrado de cultivo y del micelio de una cepa de Pseudoarachniotus roseus Kuehn.
-
GC65163
Ardisiacrispin B
Ardisiacrispin B muestra efectos citotÓxicos en células cancerosas resistentes a fÁrmacos multifactoriales a través de la muerte celular ferroptÓtica y apoptÓtica.
-
GC49314
Arecaidine propargyl ester (hydrobromide)
A muscarinic M2 agonist
-
GC35388
Aristolactam I
La aristololactama I (AL-I), es el principal metabolito del Ácido aristolÓquico I (AA-I), participa en los procesos que conducen al daÑo renal.
-
GC35395
Arnicolide D
Arnicolide D es una lactona sesquiterpénica aislada de Centipeda minima. Arnicolide D modula el ciclo celular, activa la vÍa de seÑalizaciÓn de caspasa e inhibe las vÍas de seÑalizaciÓn PI3K/AKT/mTOR y STAT3. Arnicolide D inhibe la viabilidad de las células del carcinoma nasofarÍngeo (NPC) de una manera dependiente de la concentraciÓn y el tiempo.
-
GC19037
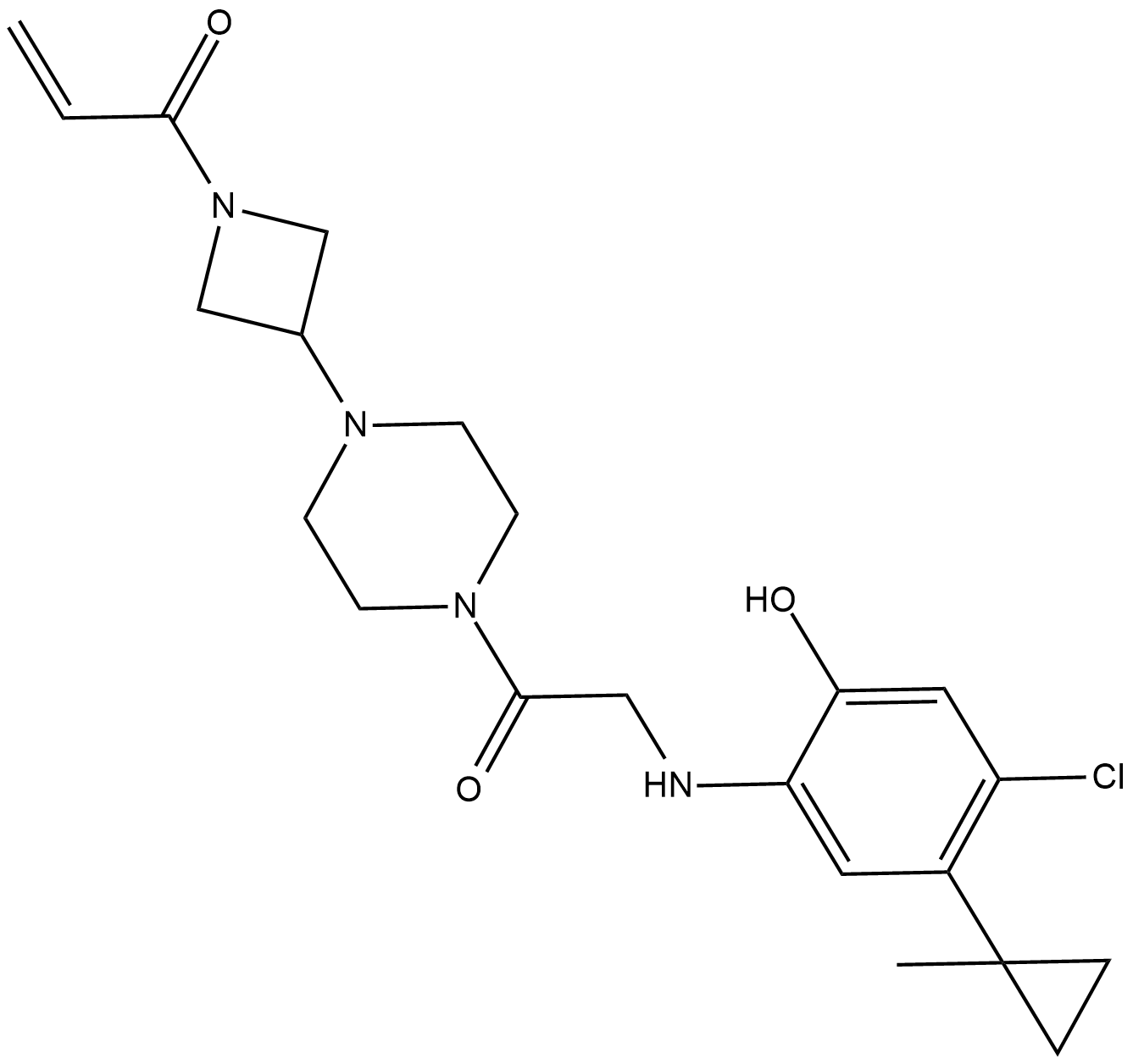
ARS-853
ARS-853 es un inhibidor de KRAS G12C covalente, selectivo y activo celular con una IC50 de 2,5 μM. ARS-853 inhibe la seÑalizaciÓn impulsada por KRAS mutante al unirse a la oncoproteÍna unida a GDP y prevenir la activaciÓn.
-
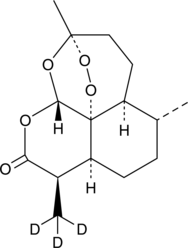
GC46882
Artemisinin-d3
An internal standard for the quantification of artemisinin
-
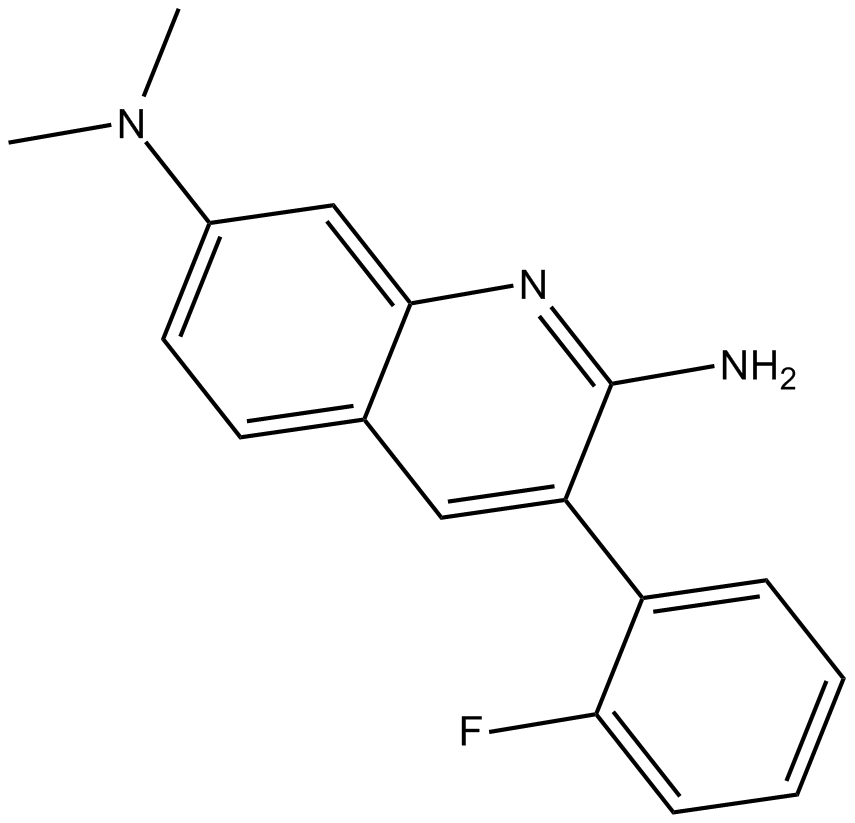
GC10040
Arylquin 1
Arylquin 1, un secretagogo de prÓstata-apoptosis-respuesta-4 (Par-4), se dirige a la vimentina para inducir la secreciÓn de Par-4. Arilquin 1 induce la muerte celular no apoptÓtica en las células cancerosas a través de la inducciÓn de la permeabilizaciÓn de la membrana lisosomal (LMP).
-
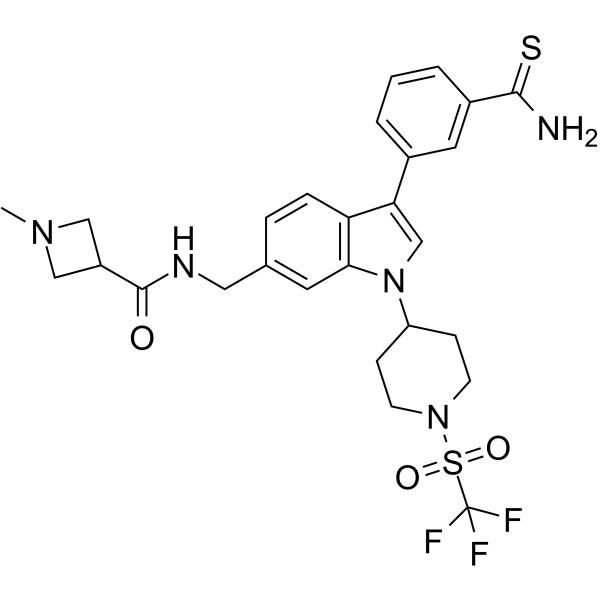
GC62615
AS-99
AS-99 es un inhibidor de histona metiltransferasa ASH1L selectivo, potente y de primera clase (IC50 = 0,79μ M, Kd = 0,89μ M) con actividad antileucémica. AS-99 bloquea la proliferaciÓn celular, induce la apoptosis y la diferenciaciÓn, regula a la baja los genes diana de la fusiÓn MLL y reduce la carga de leucemia in vivo.
-
GC40715
Ascochlorin
La ascoclorina (Ilicicolina D), un antibiÓtico isoprenoide, media sus efectos antitumorales predominantemente a través de la supresiÓn de la cascada de seÑalizaciÓn de STAT3.
-
GC13215
Ascomycin(FK 520)
A potent macrolide immunosuppressant
-
GC12070
Ascorbic acid
Un donante de electrones.
-
GN10702
Asiatic acid
-
GN10534
Asiaticoside
-
GC19041
ASK1-IN-1
ASK1-IN-1 es un potente inhibidor selectivo de ATP competitivo disponible por vÍa oral de la quinasa 1 reguladora de la seÑal de apoptosis (ASK1) con una IC50 de 2,87 nM.
-
GC62426
ASK1-IN-2
ASK1-IN-2 es un inhibidor potente y activo por vÍa oral de la quinasa 1 reguladora de la seÑal de apoptosis (ASK1), con una IC50 de 32,8 nM.
-
GC42858
Aspergillin PZ
Aspergillin PZ es un nuevo isoindol-alcaloide de Aspergillus awamori.
-
GN10064
Asperosaponin VI
-
GC60603
Asperosaponin VI
A triterpenoid saponin with diverse biological activities
-
GC42860
Aspochalasin D
Aspochalasin D is a co-metabolite originally isolated from A.
-
GC41640
Asterriquinol D dimethyl ether
El asterriquinol D dimetil éter es un metabolito fÚngico que puede inhibir las lÍneas celulares NS-1 del mieloma de ratÓn con una IC50 de 28 μg/mL.
-
GN10415
Astilbin
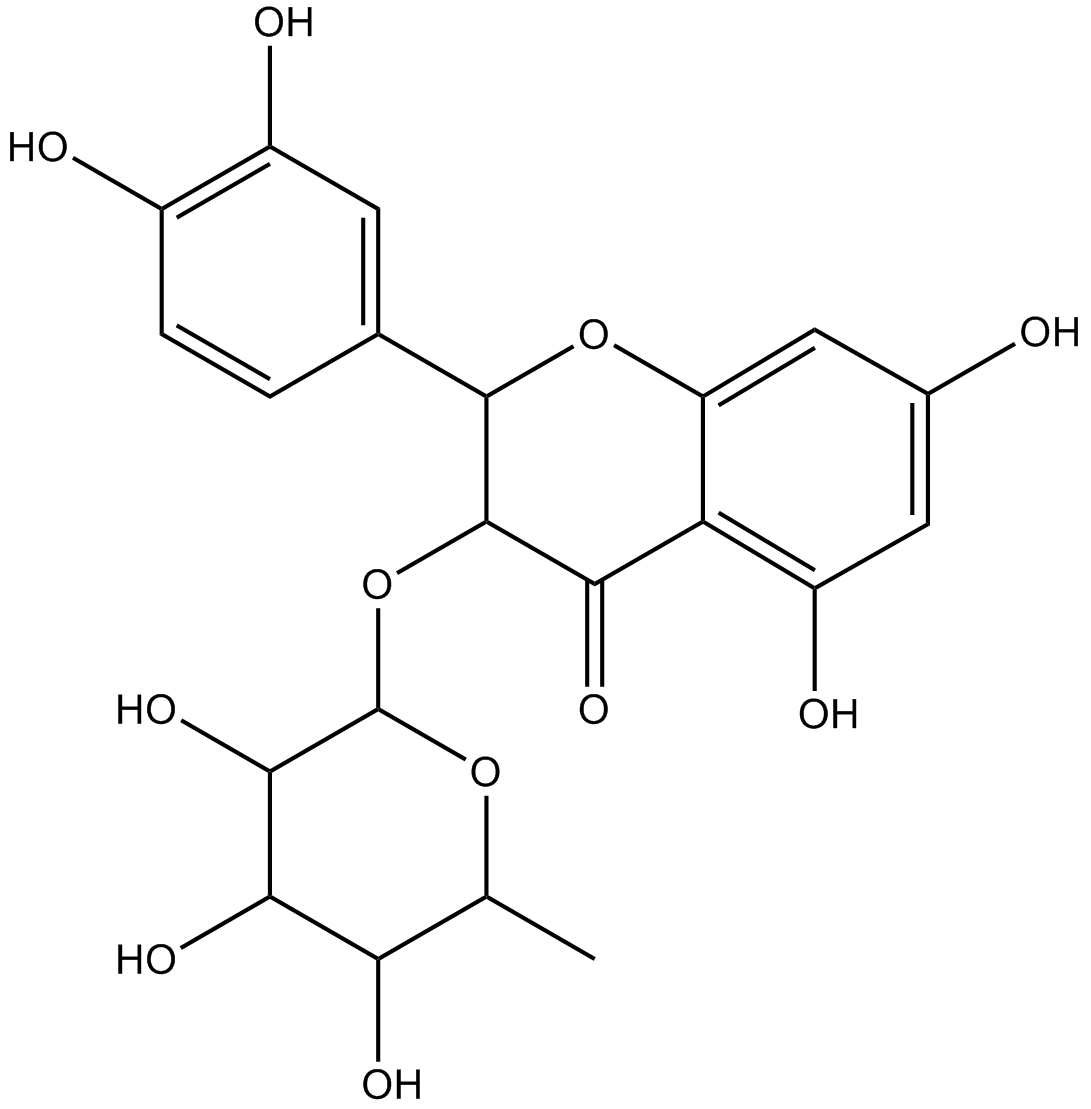
-
GN10561
astragalin
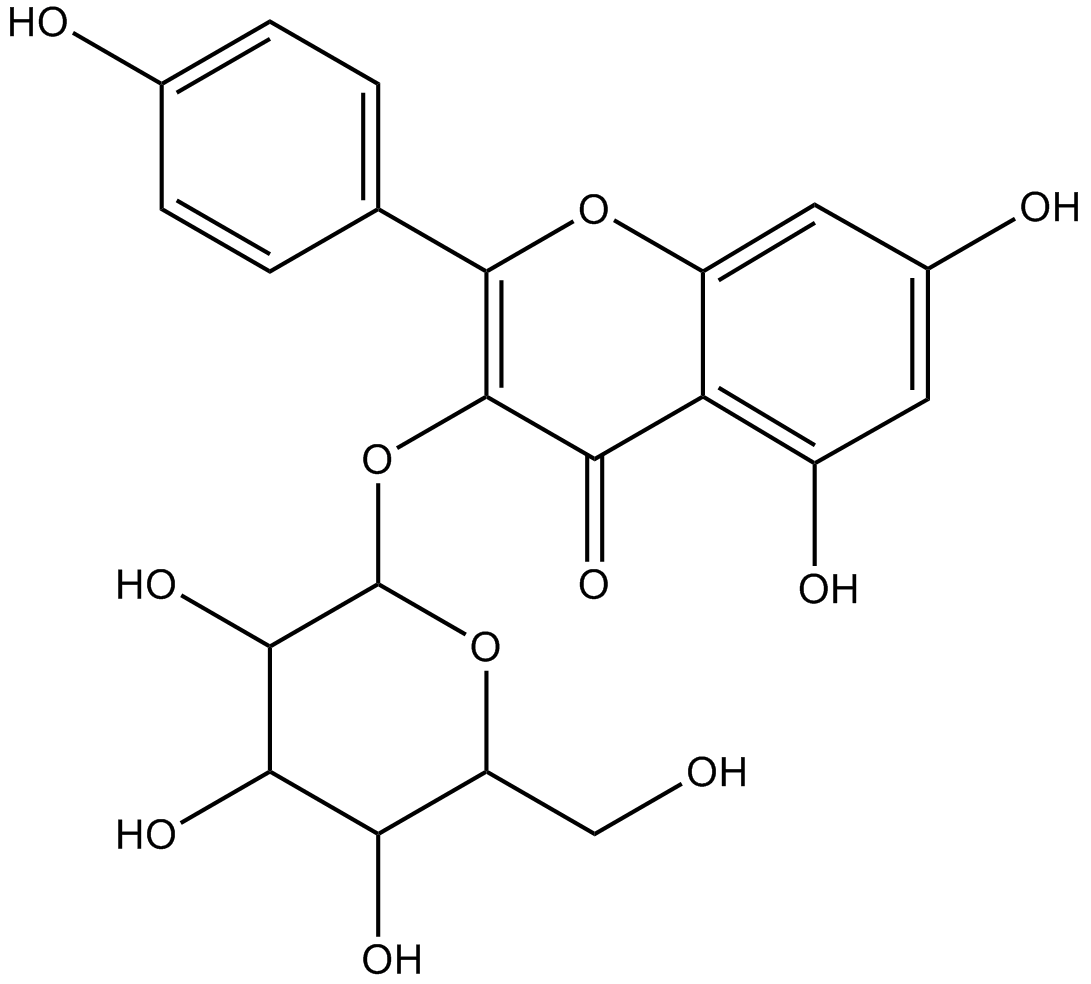
-
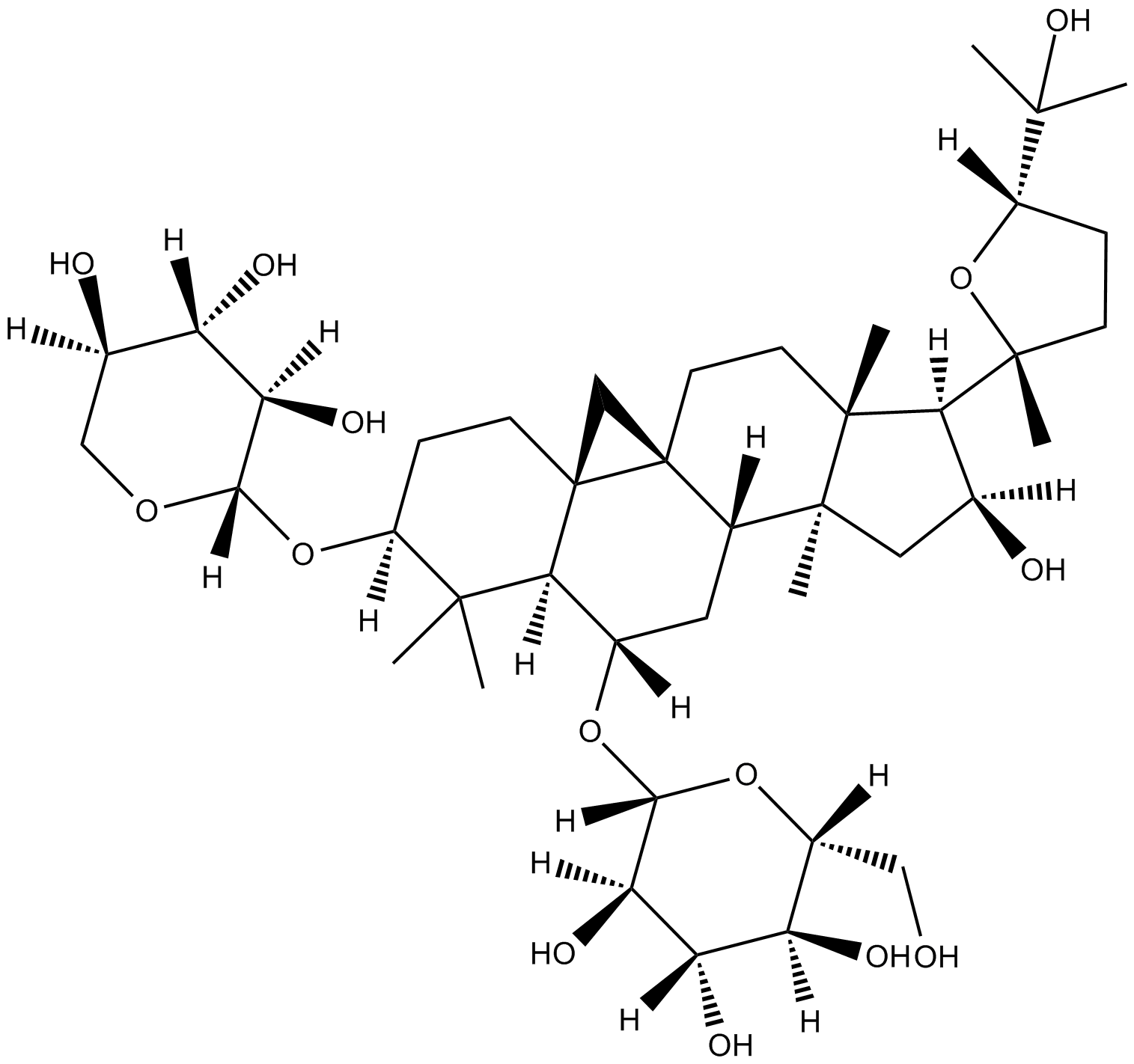
GC18109
Astragaloside A
anti-hypertension, positive inotropic action, anti-inflammation, and anti-myocardial injury
-
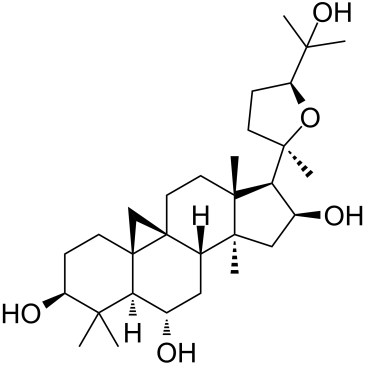
GC35415
Astramembrangenin
-
GC32803
ASTX660
ASTX660 es un antagonista dual biodisponible por vÍa oral del inhibidor celular de la proteÍna de apoptosis (cIAP) y el inhibidor de la proteÍna de apoptosis ligado al cromosoma X (XIAP).
-
GC42863
Asukamycin
Asukamycin is polyketide isolated from the S.
-
GC11106
AT-101