Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
GC30530
Dot1L-IN-2
Dot1L-IN-2 es un inhibidor potente, selectivo y biodisponible por vÍa oral de Dot1L (una histona metiltransferasa), con una IC50 y una Ki de 0,4 nM y 0,08 nM, respectivamente.
-
GC62613
Dot1L-IN-4
Dot1L-IN-4 es un potente disruptor del inhibidor de la proteÍna similar a 1 silenciador telomérico (DOT1L) con un IC50 SPA DOT1L de 0,11 nM.
-
GC65962
Dot1L-IN-5
Dot1L-IN-5 es un potente disruptor del inhibidor de la proteÍna similar al silenciador telomérico 1 (DOT1L) con un IC50 SPA DOT1L de 0,17 nM.
-
GC12680
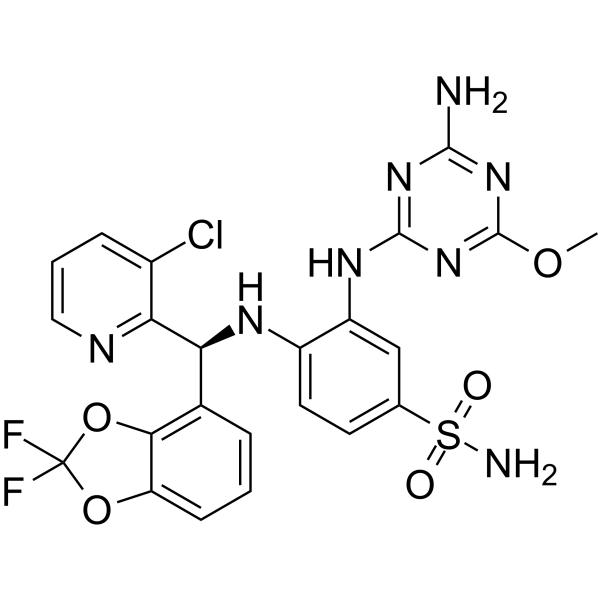
DR 2313
A PARP inhibitor
-
GC13706
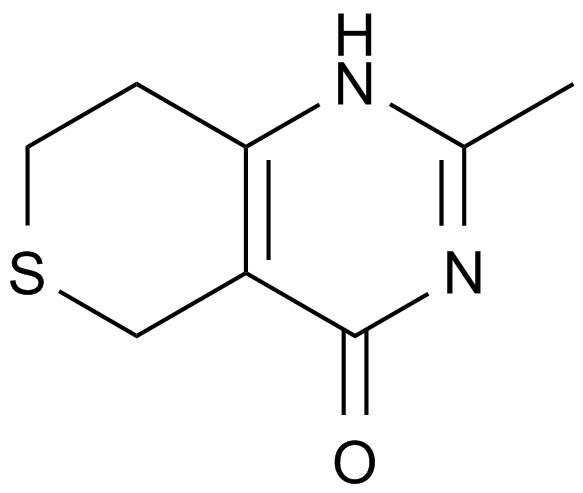
Droxinostat
An inhibitor of HDAC3, HDAC6, and HDAC8
-
GC45927
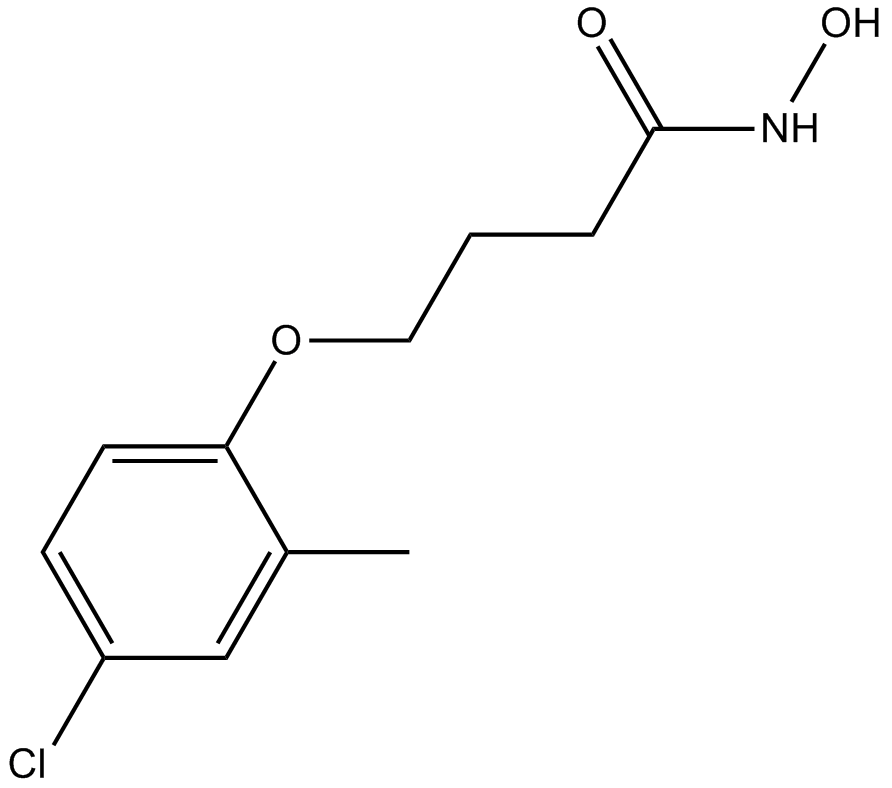
DS-437
DS-437 es un inhibidor dual de PRMT5/7 (CI50 de PRMT5/7 = 6 μM).
-
GC35904
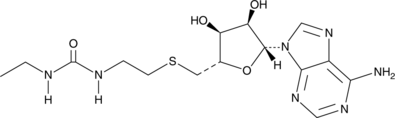
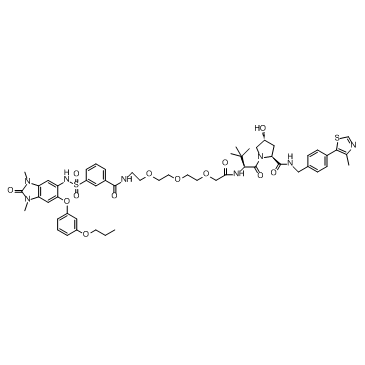
dTRIM24
dTRIM24 es un degradador bifuncional selectivo de TRIM24 basado en PROTAC, consta de ligandos para von Hippel-Lindau y TRIM24.
-
GC35914
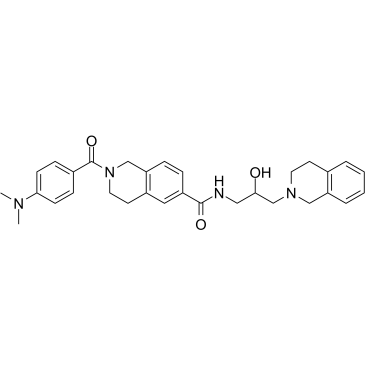
DW14800
DW14800 es un inhibidor de la proteÍna arginina metiltransferasa 5 (PRMT5), con una IC50 de 17 nM. DW14800 reduce los niveles de H4R3me2s y mejora la transcripciÓn de HNF4α, pero no altera la expresiÓn de PRMT5. Actividad anticancerÍgena.
-
GC33403
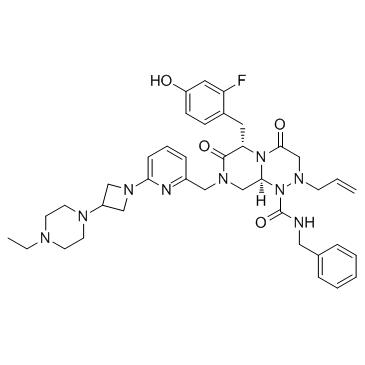
E-7386
E-7386 es un modulador de CBP/beta-catenina activo por vÍa oral.
-
GC64209
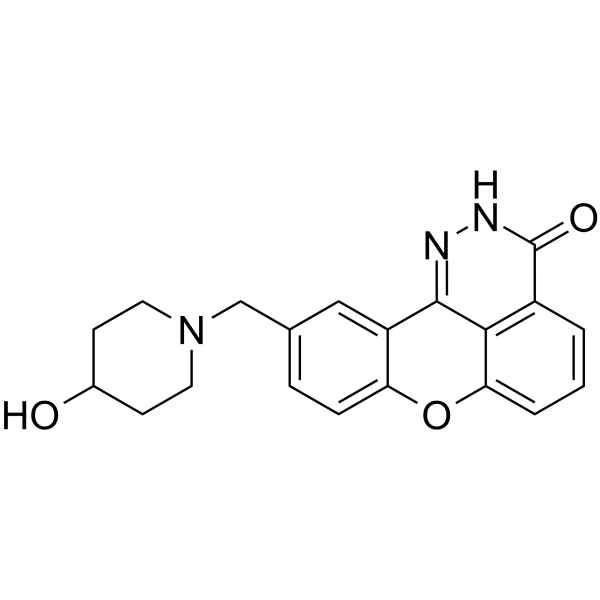
E7016
E7016 (GPI 21016) es un inhibidor de PARP disponible por vÍa oral. E7016 puede mejorar la radiosensibilidad de las células tumorales in vitro e in vivo a través de la inhibiciÓn de la reparaciÓn del ADN. E7016 actÚa como un potencial agente anticancerÍgeno.
-
GC18172
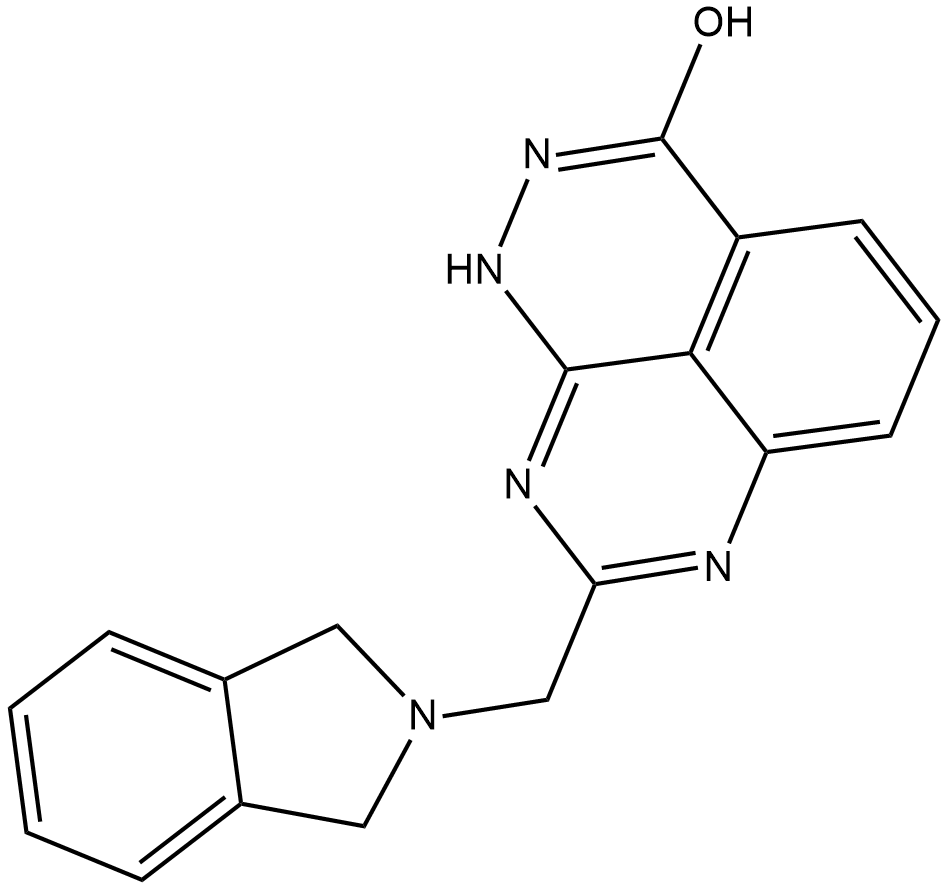
E7449
E7449 es un potente inhibidor de PARP1 y PARP2 y también inhibe TNKS1 y TNKS2, con IC50 de 2,0, 1,0, ~50 y ~50 nM para PARP1, PARP2, TNKS1 y TNKS2, respectivamente, usando 32P-NAD+ como sustrato.
-
GC12991
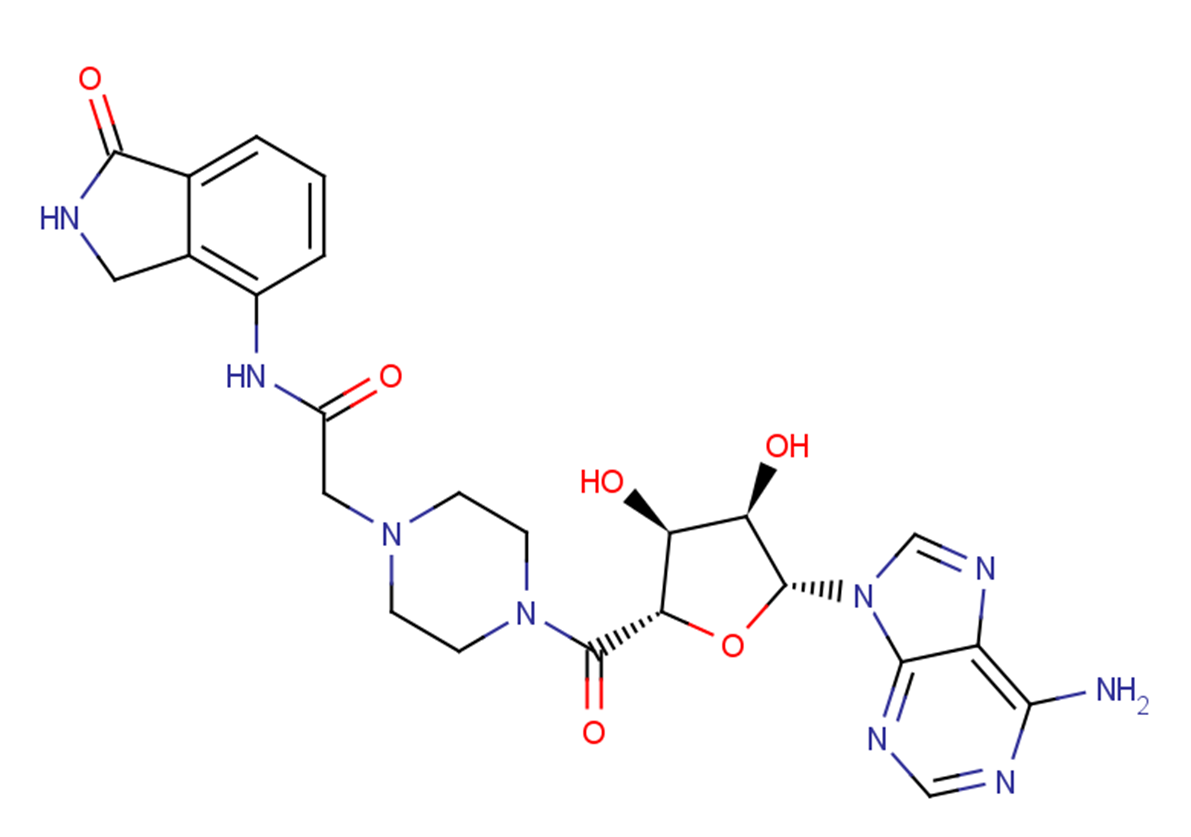
EB 47
A PARP1 and TNKS2 inhibitor
-
GC33220
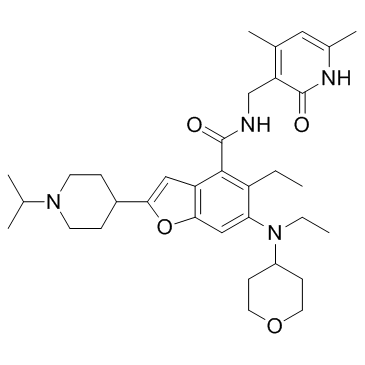
EBI-2511
EBI-2511 es un inhibidor de EZH2 muy potente y activo por vÍa oral, con una IC50 de 6 nM en lÍneas celulares Pfeffiera, respectivamente.
-
GC18236
Echinomycin
Un inhibidor de la transcripción génica mediada por HIF1.

-
GC19129
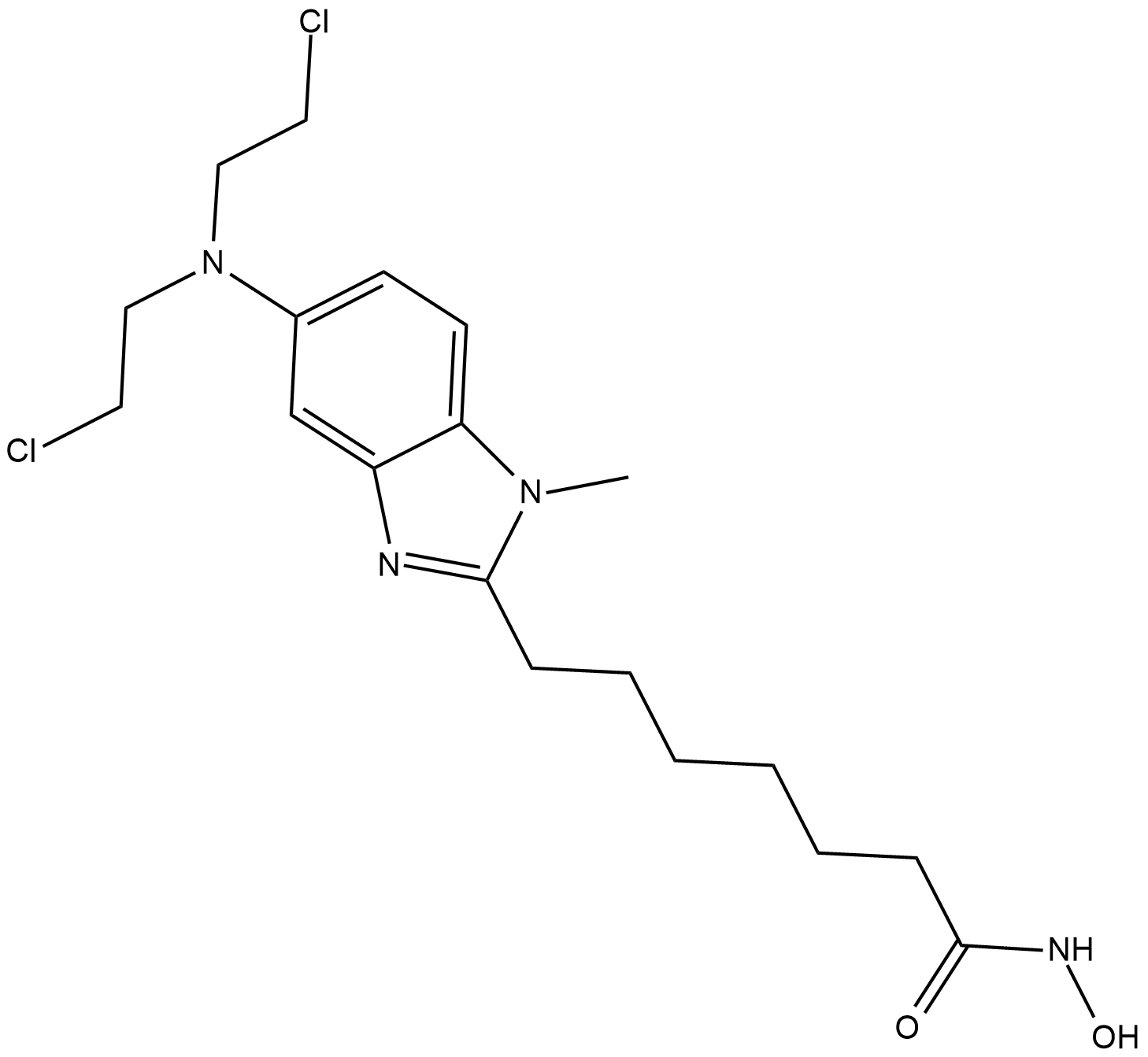
EDO-S101
EDO-S101 (Tinostamustina) es un inhibidor pan HDAC; inhibe HDAC6, HDAC1, HDAC2 y HDAC3 con valores IC50 de 6 nM, 9 nM, 9 nM y 25 nM, respectivamente.
-
GC19130
EED226
EED226 es un inhibidor del complejo represivo Polycomb 2 (PRC2), que se une al bolsillo K27me3 en el desarrollo del ectodermo embrionario (EED) y muestra una fuerte actividad antitumoral en el modelo de ratones con xenoinjerto. EED226 es un inhibidor de EED potente, selectivo y biodisponible por vÍa oral. EED226 inhibe PRC2 con una IC50 de 23,4 nM cuando se utiliza el péptido H3K27me0 como sustrato en los ensayos enzimÁticos in vitro.
-
GC67934
EEDi-5285
-
GC34567
EHMT2-IN-1
EHMT2-IN-1 es un potente inhibidor de EHMT, con IC50 de todos <100 nM para el péptido EHMT1, el péptido EHMT2 y el EHMT2 celular. Se utiliza en la investigaciÓn de trastornos sanguÍneos o cÁncer.
-
GC34568
EHMT2-IN-2
EHMT2-IN-2 es un potente inhibidor de EHMT, con IC50 de todos <100 nM para el péptido EHMT1, el péptido EHMT2 y el EHMT2 celular. Se utiliza en la investigaciÓn de enfermedades de la sangre o cÁncer.
-
GC14756
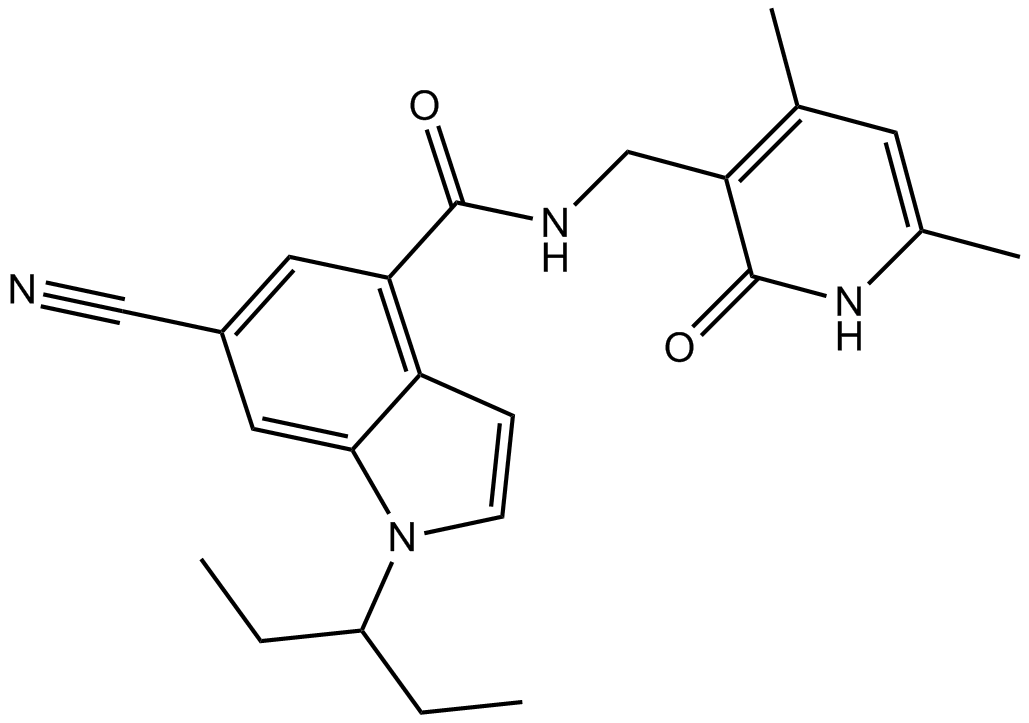
EI1
EI1 (KB-145943) es un inhibidor de EZH2 potente y selectivo con una IC50 de 15 nM y 13 nM para EZH2 (WT) y EZH2 (Y641F), respectivamente.
-
GC33043
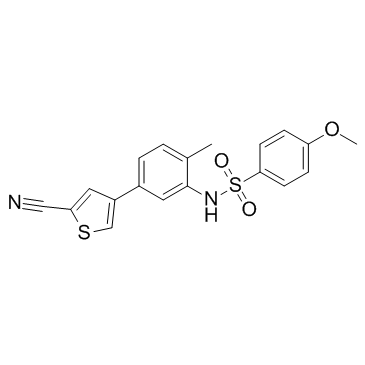
EL-102
EL-102 es un inhibidor del factor 1 inducido por hipoxia (Hif1α). EL-102 induce apoptosis, inhibe la polimerizaciÓn de tubulina y muestra actividad contra el cÁncer de prÓstata. EL-102 se puede utilizar para la investigaciÓn del cÁncer.
-
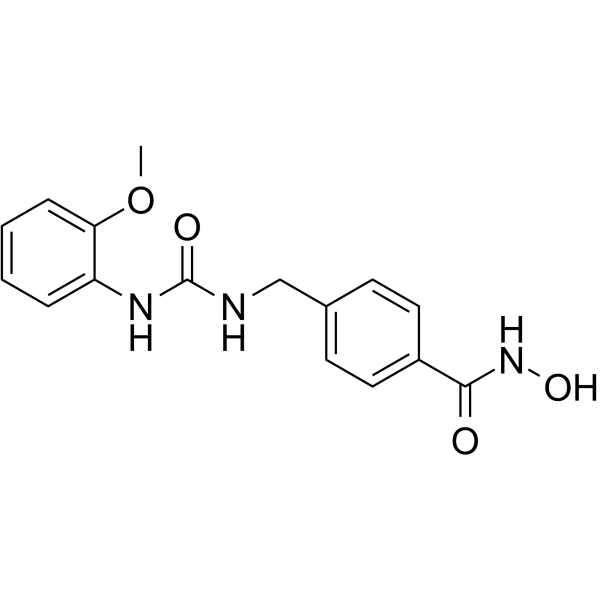
GC64191
Elevenostat
Elevenostat (JB3-22) es un inhibidor selectivo de HDAC11 (IC50=0.235μM). Actividad anti-mieloma mÚltiple (MM).
-
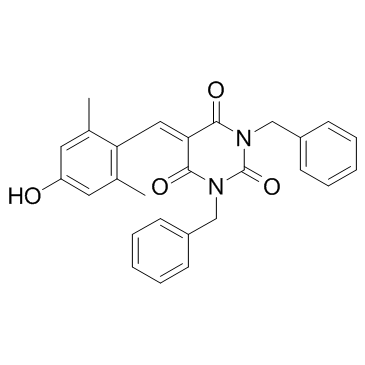
GC31245
EML 425
EML425 es un potente y selectivo inhibidor de la proteÍna de uniÓn a CREB (CBP)/p300 con IC50 de 2,9 y 1,1 μM, respectivamente.
-
GC49124
EN219
EN219 es un ligando covalente sintético moderadamente selectivo frente a una cisteÍna N-terminal (C8) de RNF114 con una IC50 de 470 nM. EN219 inhibe la autoubiquitinaciÓn mediada por RNF114 y la ubiquitinaciÓn de p21.
-
GC33634
Enarodustat (JTZ-951)
Enarodustat (JTZ-951) es un inhibidor de la prolil hidroxilasa del factor inducible por hipoxia potente y activo por vÍa oral, con un EC50 de 0,22 μM.
-
GC33170
ENMD-119 (ENMD 1198)
ENMD-119 (ENMD 1198) (IRC-110160), un agente desestabilizador de microtÚbulos activo por vÍa oral, es un anÁlogo de 2-metoxiestradiol con actividad antiproliferativa y antiangiogénica. ENMD-119 (ENMD 1198) es adecuado para inhibir HIF-1alfa y STAT3 en células HCC humanas y reduce el crecimiento tumoral y la vascularizaciÓn.
-
GC16519
ENMD-2076
A multi-kinase inhibitor
-
GC12145
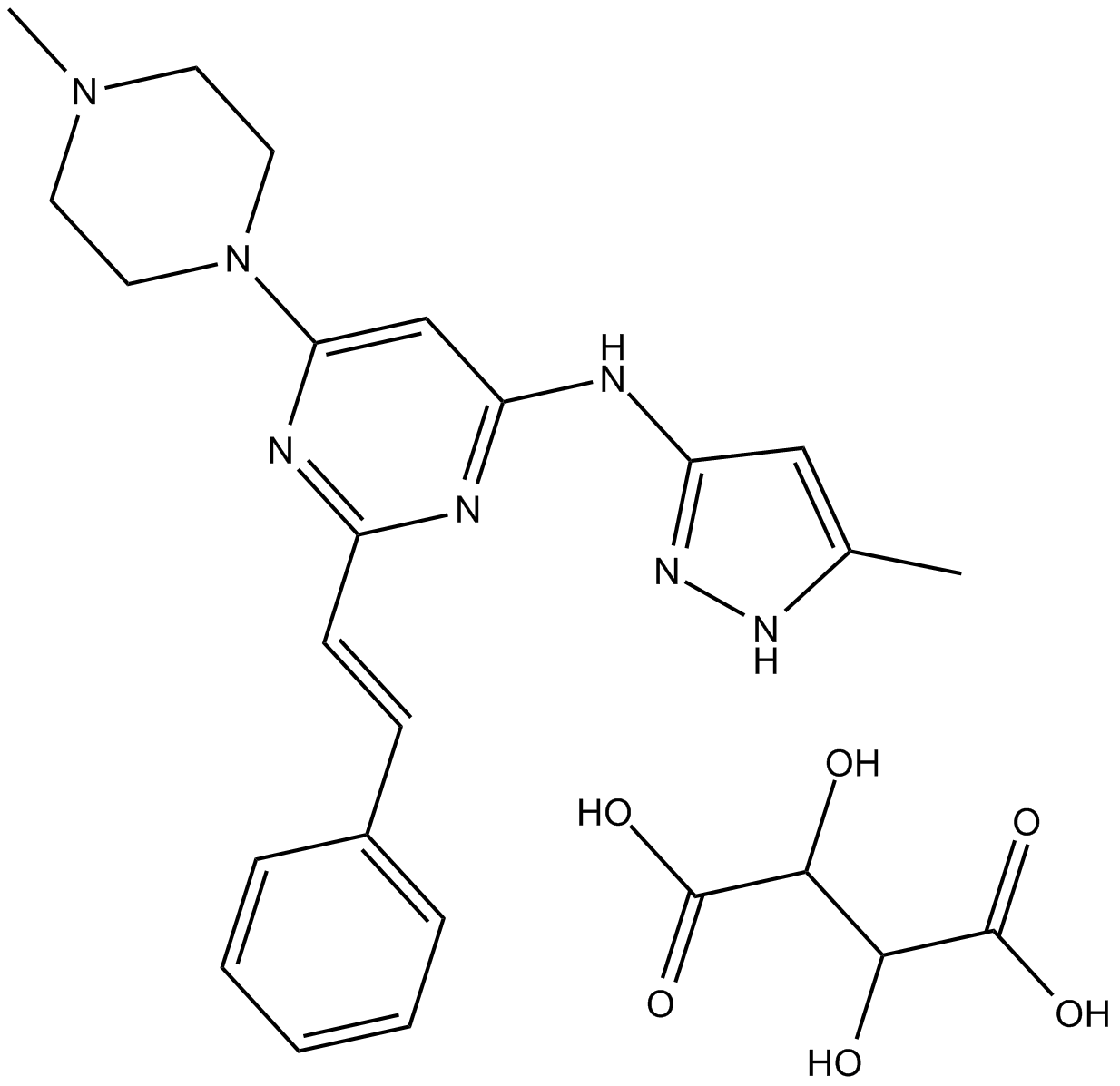
ENMD-2076 L-(+)-Tartaric acid
-
GC50182
ent-LP 99
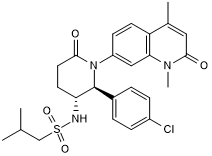
ent-LP 99 es un inhibidor potente y selectivo de BRD7 y BRD9 con una KD de 99 nM para BRD9.
-
GC17334
Entacapone
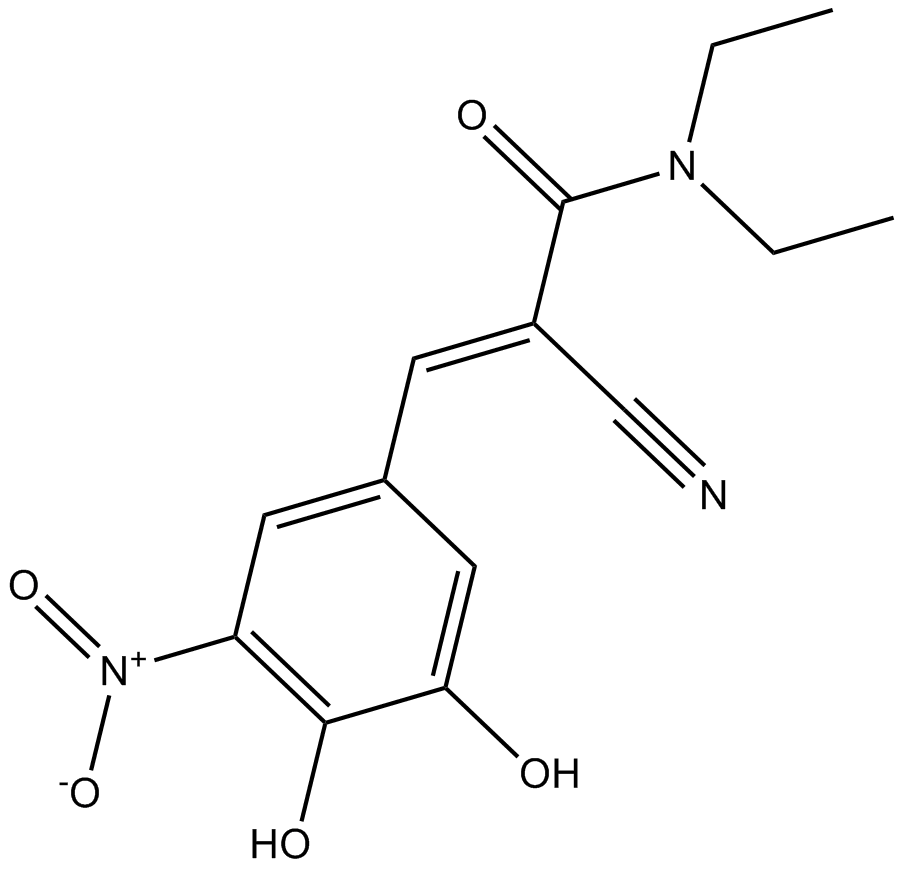
A reversible COMT inhibitor
-
GC47294
Entacapone-d10
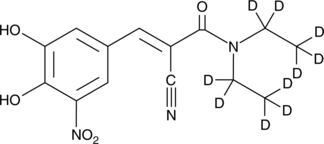
Entacapona-d10 es el deuterio etiquetado como entacapona.
-
GC11625
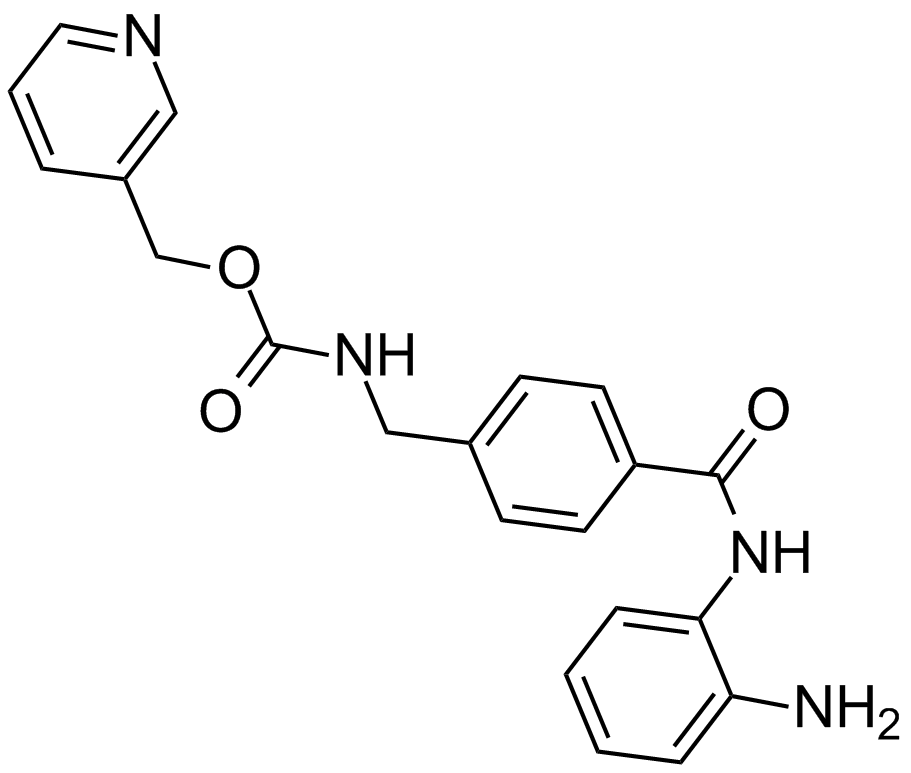
Entinostat (MS-275,SNDX-275)
A histone deacetylase inhibitor
-
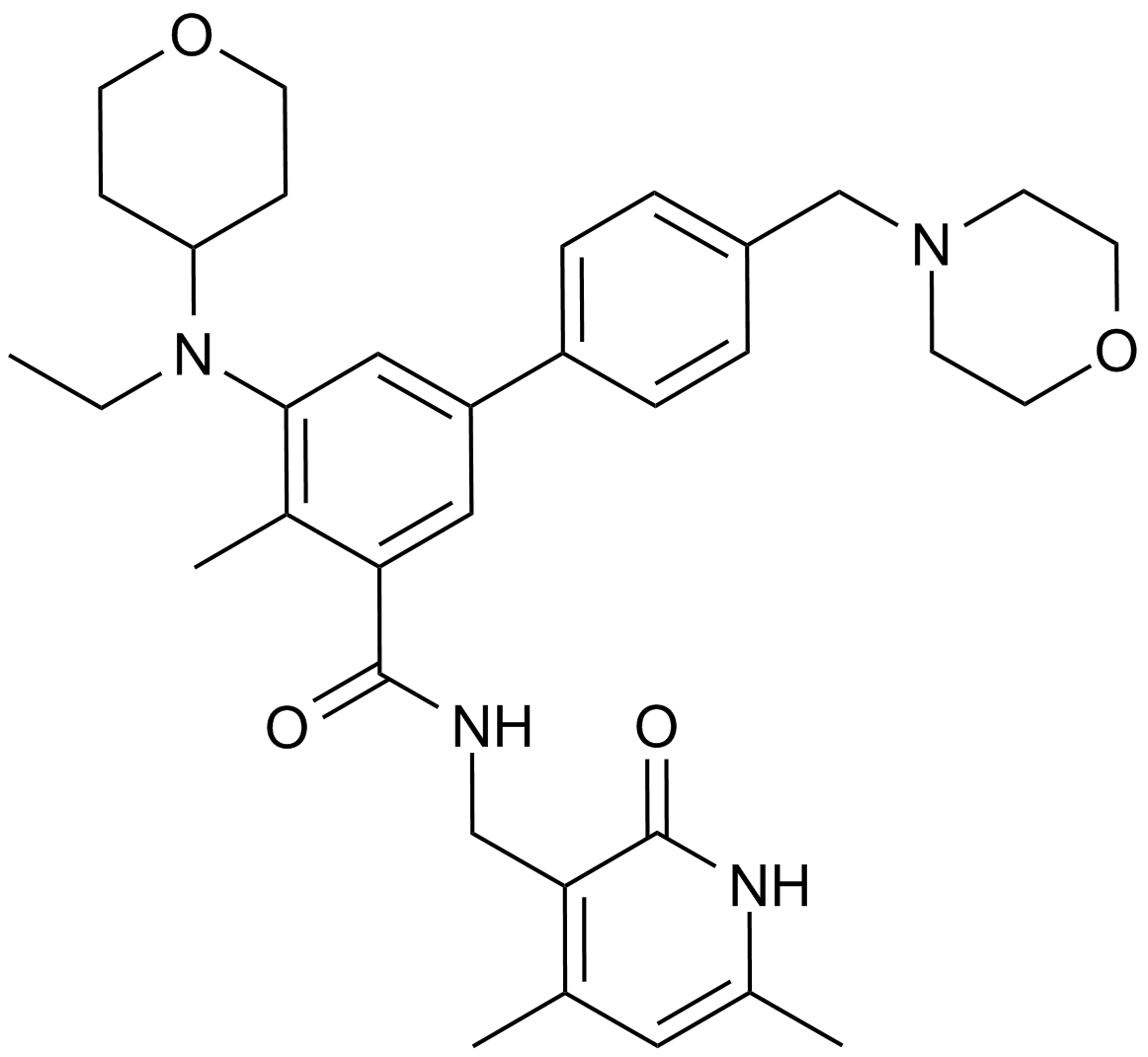
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
-
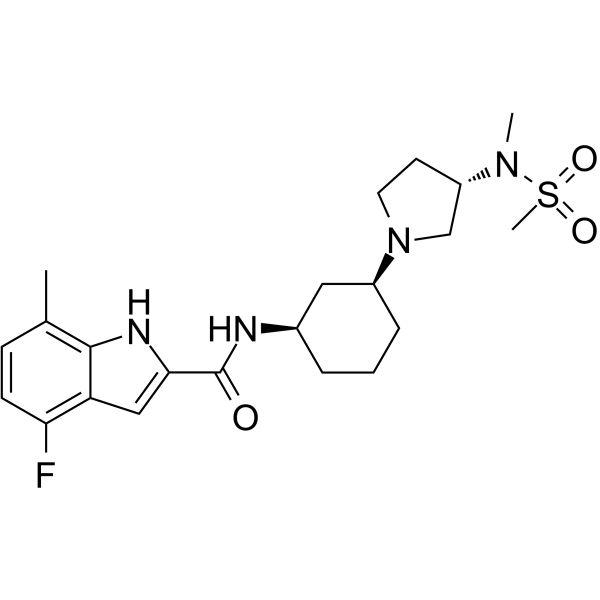
GC64343
EPZ-719
EPZ-719 es un nuevo y potente inhibidor de SETD2 (CI50 = 0,005 μM) con una alta selectividad sobre otras histonas metiltransferasas.
-
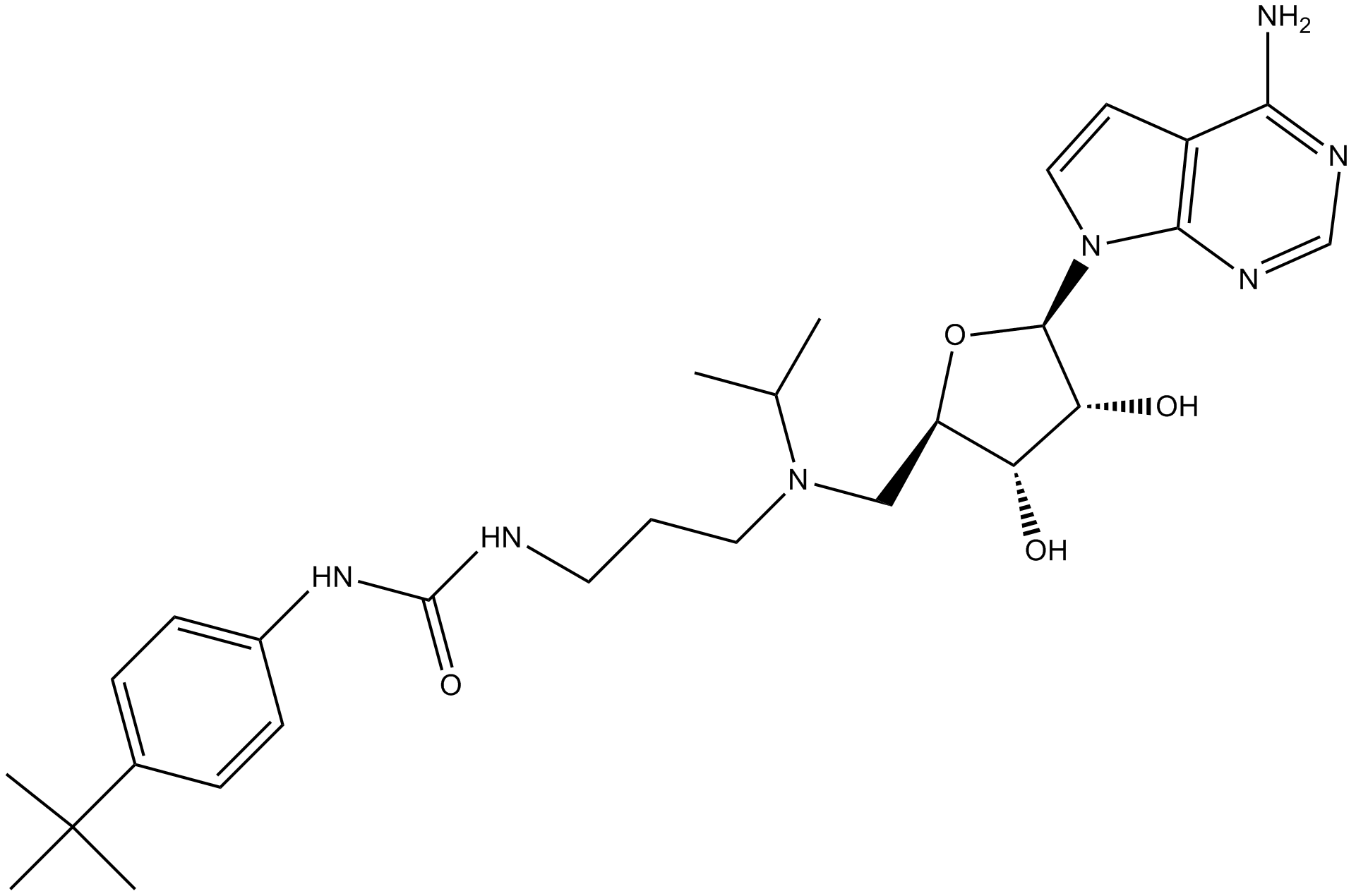
GC13383
EPZ004777
A potent inhibitor of DOT1L
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
GC15259
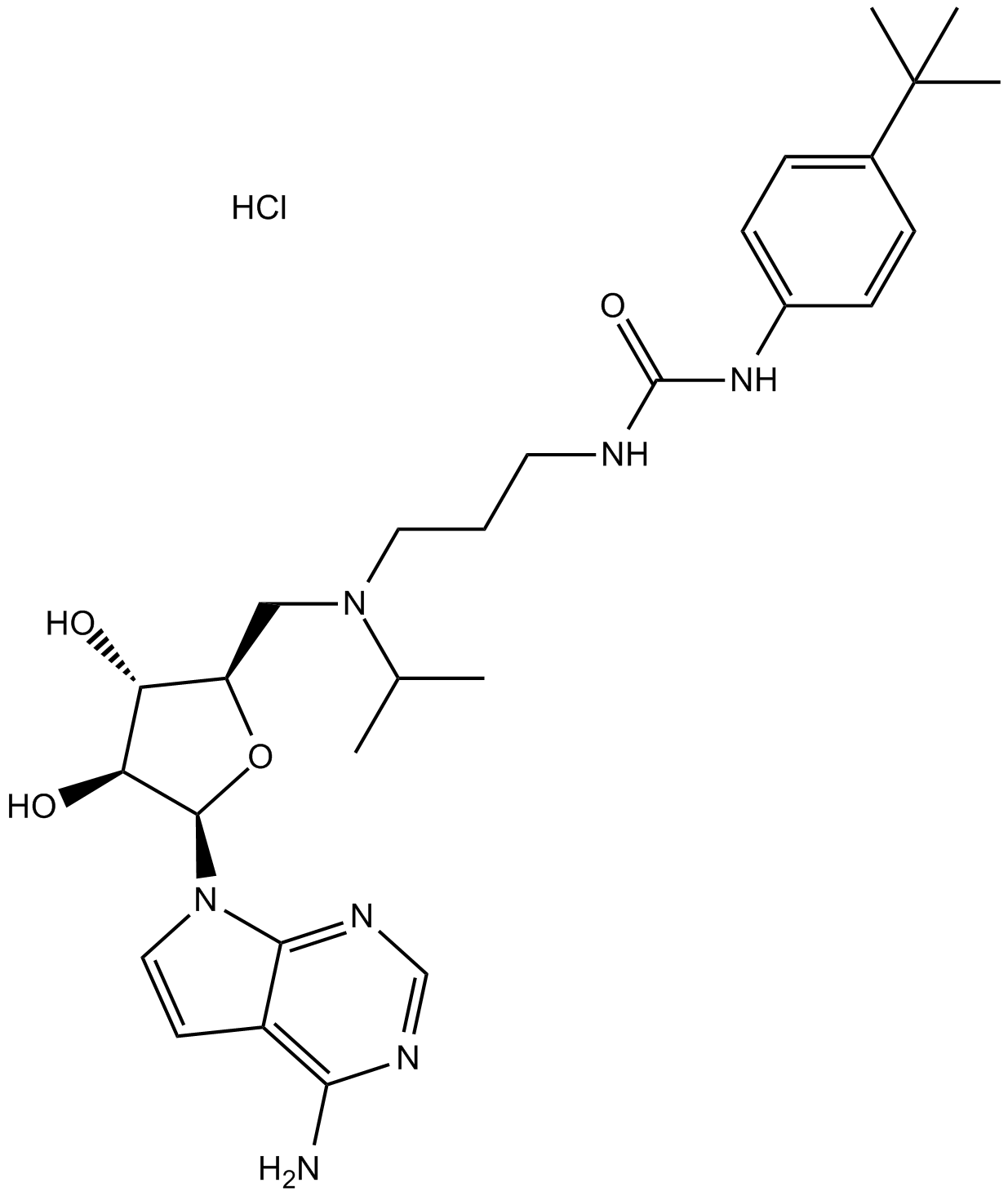
EPZ004777 HCl
EPZ004777 HCl es un potente inhibidor DOT1L selectivo con una IC50 de 0,4 nM.
-
GC13878
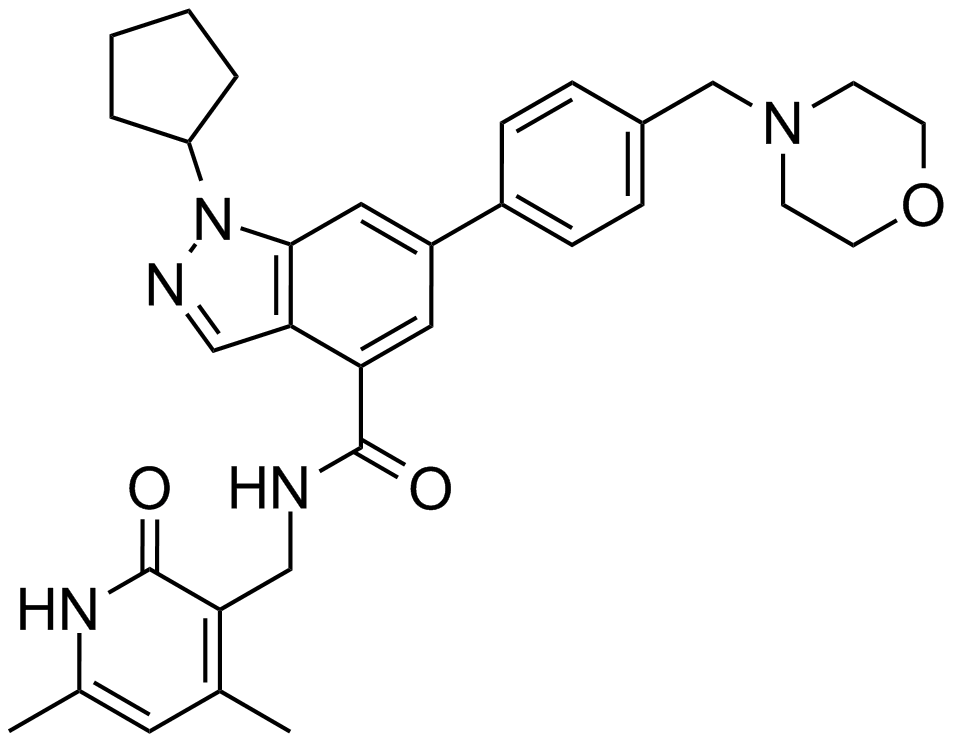
EPZ005687
A potent, selective inhibitor of EZH2
-
GC19141
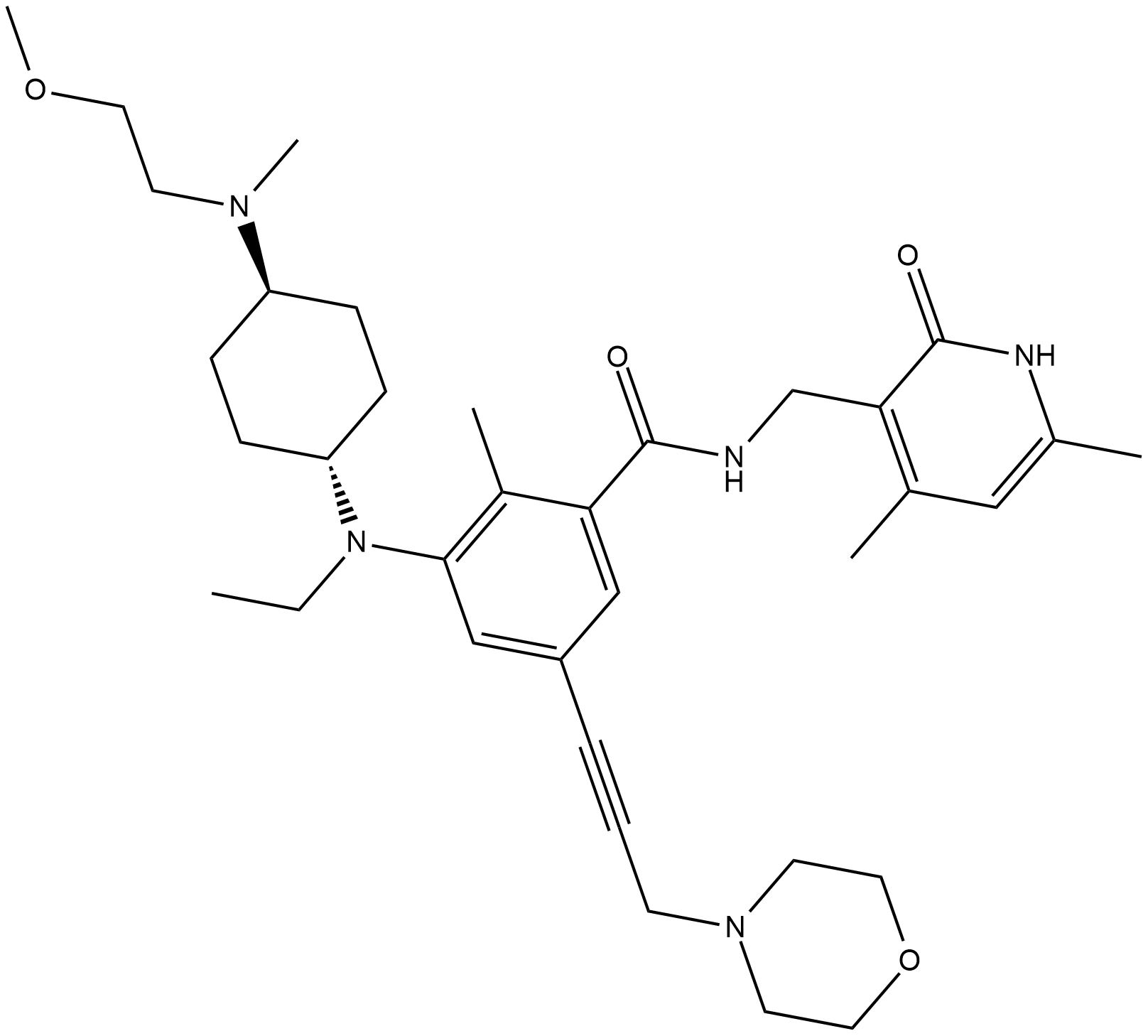
EPZ011989
EPZ011989 es un inhibidor potente y oralmente activo de Zeste Homolog 2 (EZH2) con estabilidad metabÓlica. EPZ011989 tiene una inhibiciÓn inhibitoria para EZH2 con un valor Ki de <3 nM. EPZ011989 muestra una fuerte inhibiciÓn de la marca de metilo y actividad antitumoral. EPZ011989 se puede utilizar para la investigaciÓn de varios tipos de cÁncer.
-
GC34136
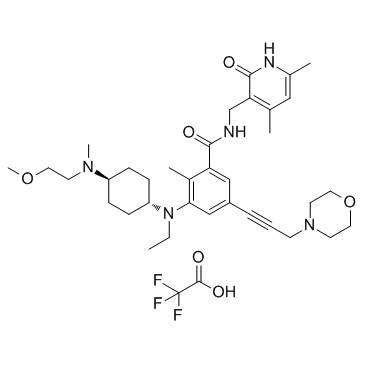
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
El trifluoroacetato EPZ-011989 es un inhibidor potente y oralmente activo de Zeste Homolog 2 (EZH2) con estabilidad metabÓlica. El trifluoroacetato EPZ-011989 tiene una inhibiciÓn inhibitoria para EZH2 con un valor Ki de <3 nM. El trifluoroacetato EPZ-011989 muestra una fuerte inhibiciÓn de la marca de metilo y actividad antitumoral. El trifluoroacetato EPZ-011989 se puede utilizar para la investigaciÓn de varios tipos de cÁncer.
-
GC15302
EPZ015666
EPZ015666 (GSK3235025) es un inhibidor de PRMT5 disponible por vÍa oral con una IC50 de 22 nM.
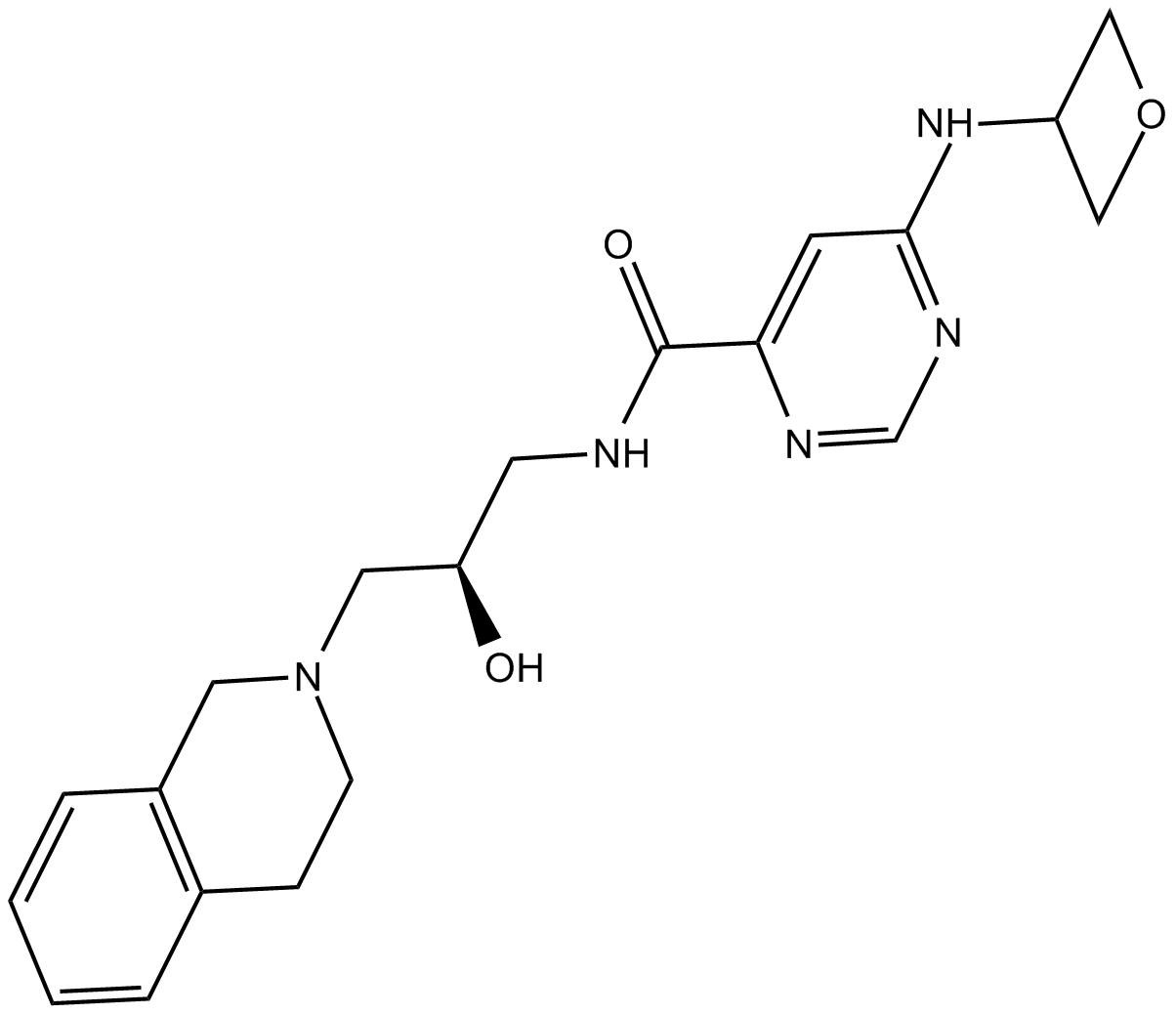
-
GC15102
EPZ020411
EPZ020411 es un inhibidor selectivo de PRMT6 con una IC50 de 10 nM, tiene una selectividad de >10 veces para PRMT6 sobre PRMT1 y PRMT8. EPZ020411 se puede utilizar para la investigaciÓn del cÁncer.
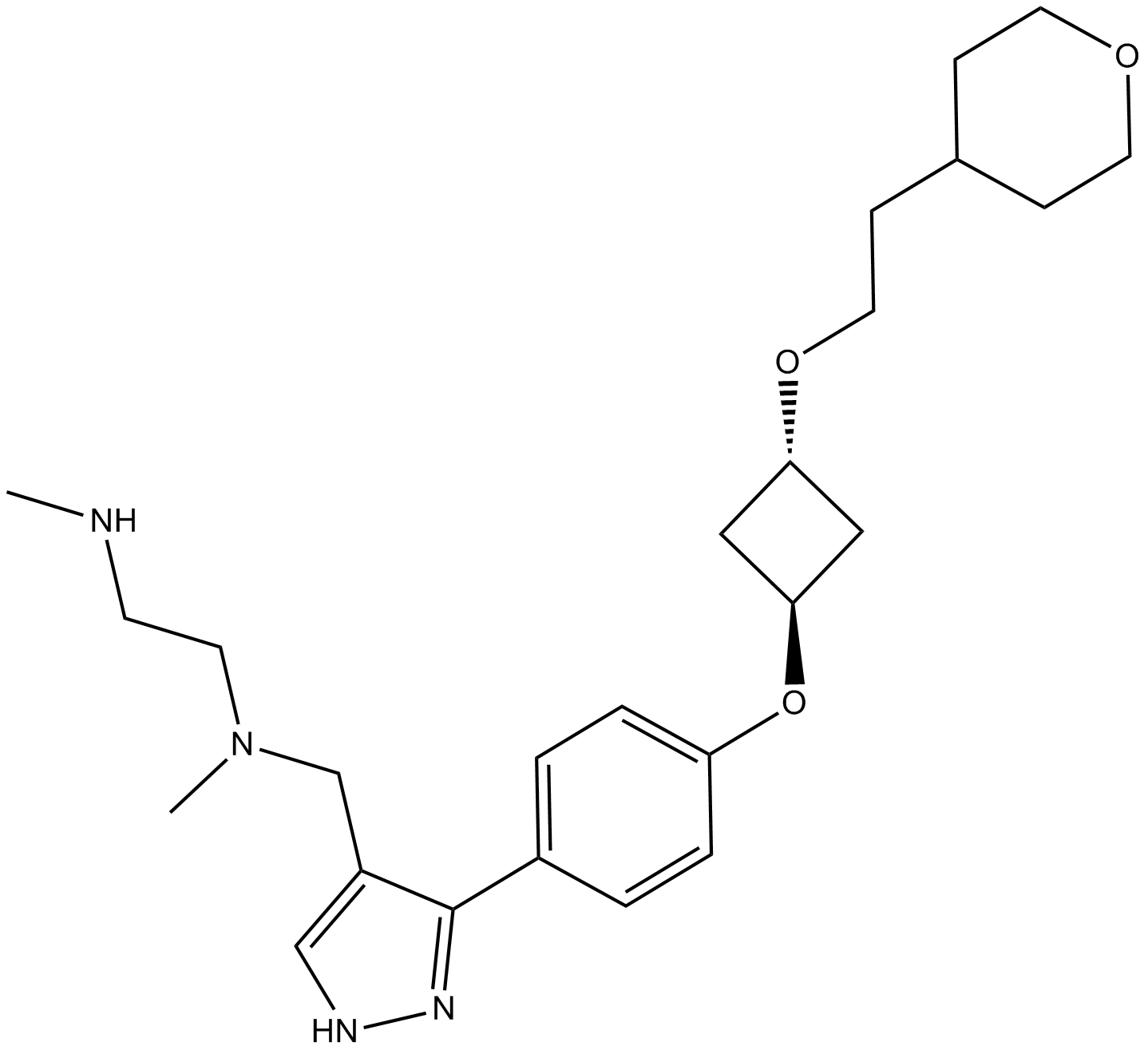
-
GC36000
EPZ020411 hydrochloride
El clorhidrato de EPZ020411 es un inhibidor selectivo de PRMT6 con una IC50 de 10 nM, tiene una selectividad de >10 veces para PRMT6 sobre PRMT1 y PRMT8. El clorhidrato de EPZ020411 se puede utilizar para la investigaciÓn del cÁncer.
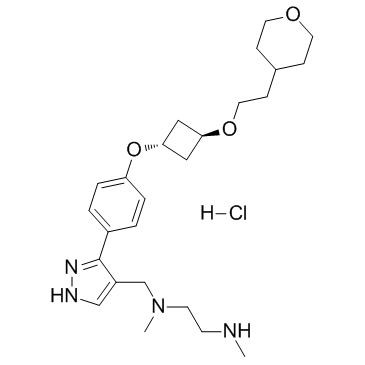
-
GC16224
EPZ031686
EPZ031686 es un inhibidor de SMYD3 potente y activo por vÍa oral y con un valor IC50 de 3 nM. EPZ031686 se puede utilizar para la investigaciÓn del cÁncer.
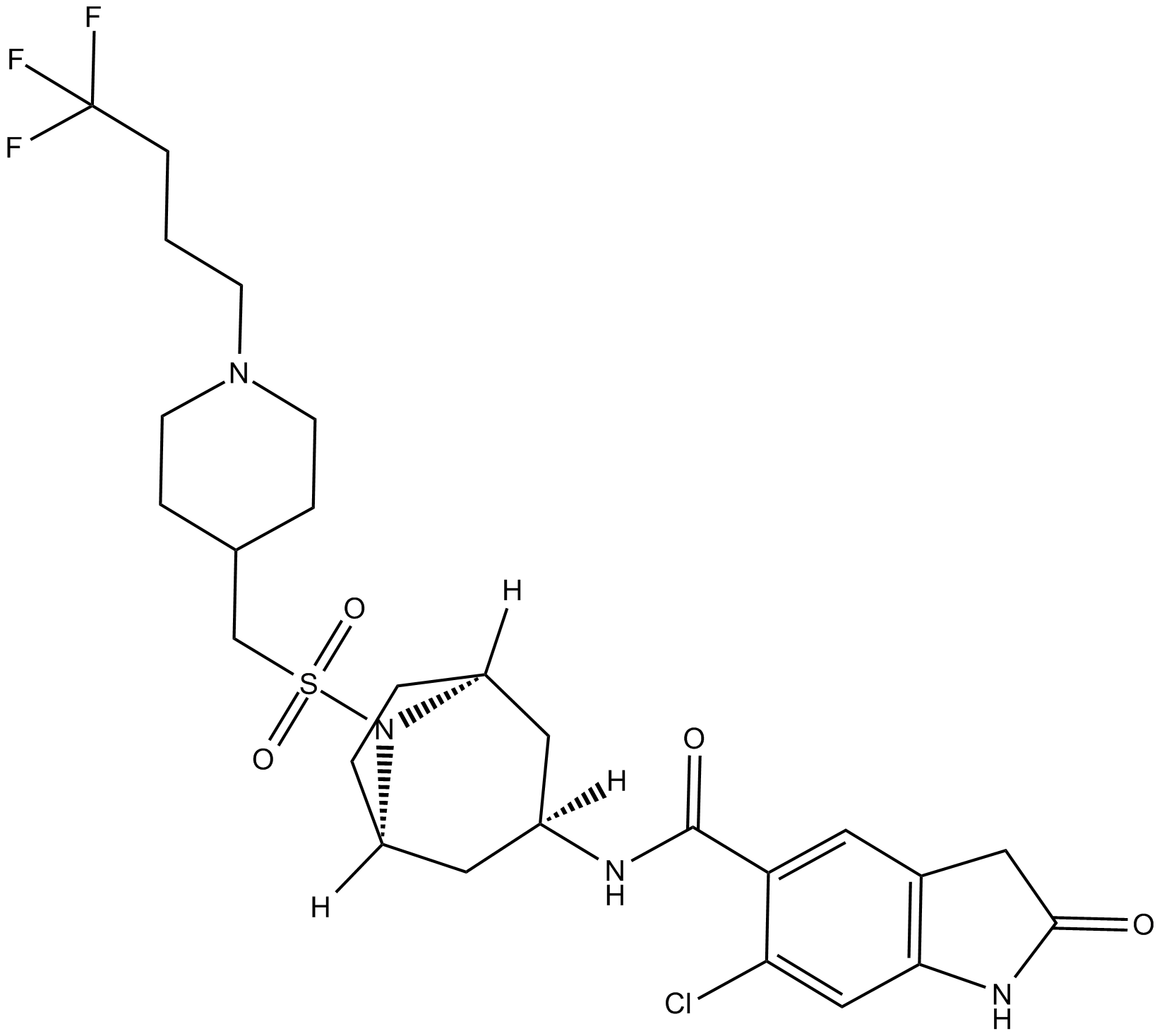
-
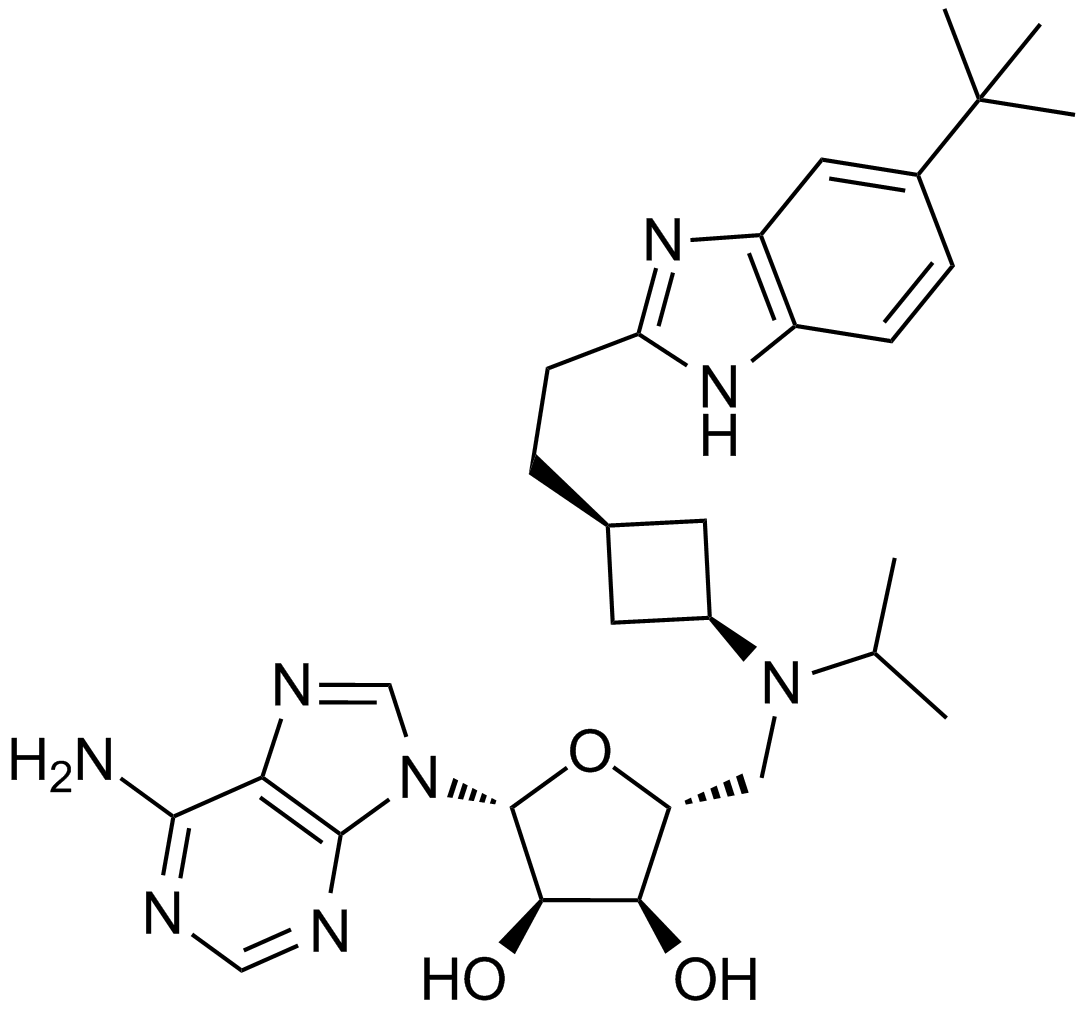
GC12932
EPZ5676
A highly potent DOT1L inhibitor
-
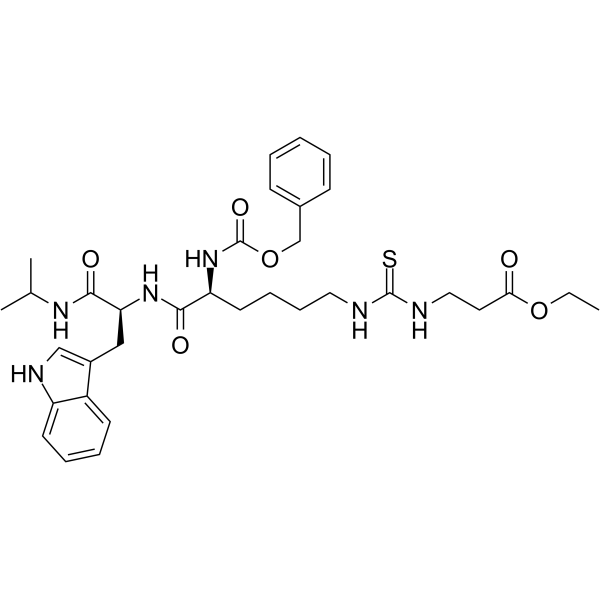
GC64575
Et-29
Et-29 es un inhibidor de SIRT5 potente y selectivo (Ki = 40 nM).
-
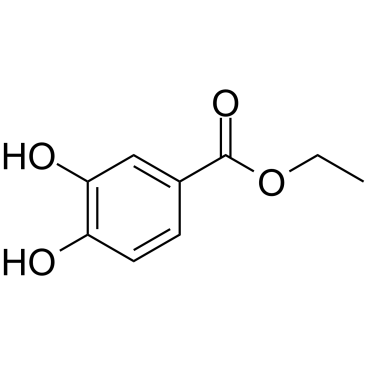
GC61669
Ethyl 3,4-dihydroxybenzoate
El 3,4-dihidroxibenzoato de etilo (protocatecuato de etilo), un antioxidante, es un inhibidor de la prolil-hidroxilasa que se encuentra en la testa de las semillas de manÍ.
-
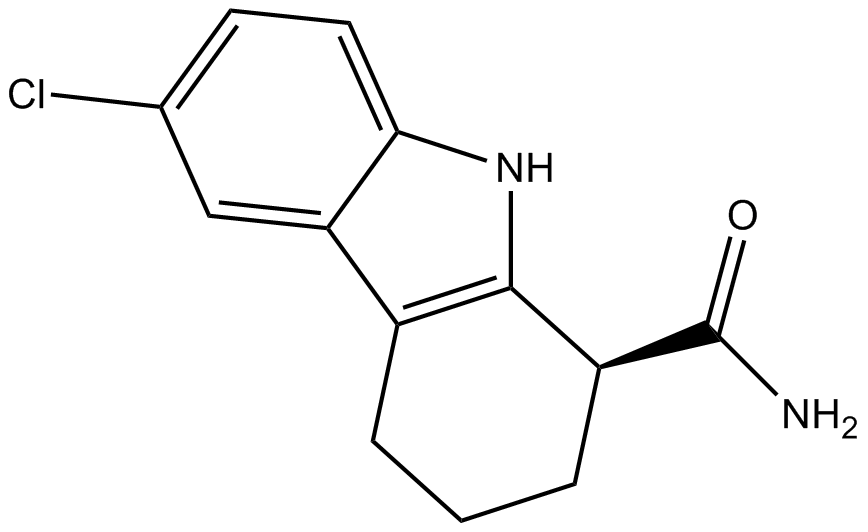
GC10635
EX 527 (SEN0014196)
A SIRT1 inhibitor
-
GC17126
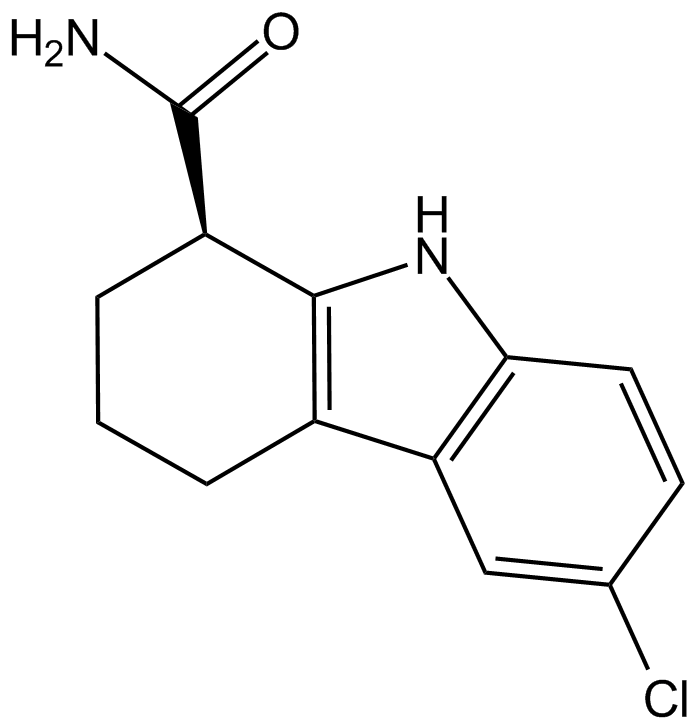
EX-527 R-enantiomer
El enantiÓmero EX-527 R ((R)-EX-527) es un enantiÓmero R de Selisistat. Selisistat (EX-527) es un inhibidor potente y selectivo de SIRT1 con IC50 de 98 nM.
-
GC13417
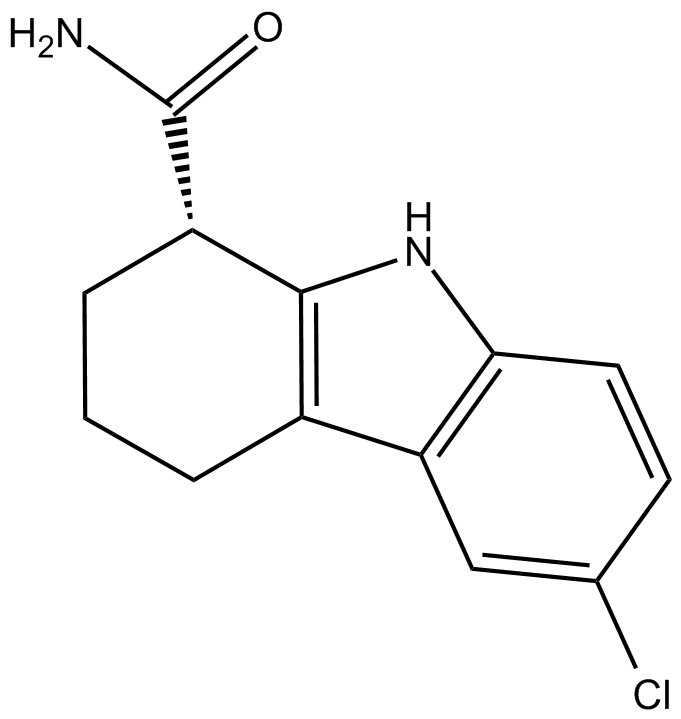
EX-527 S-enantiomer
El enantiÓmero EX-527 S ((S)-EX-527) es un inhibidor potente y selectivo de SIRT1, con una IC50 de 98 nM.
-
GC65980
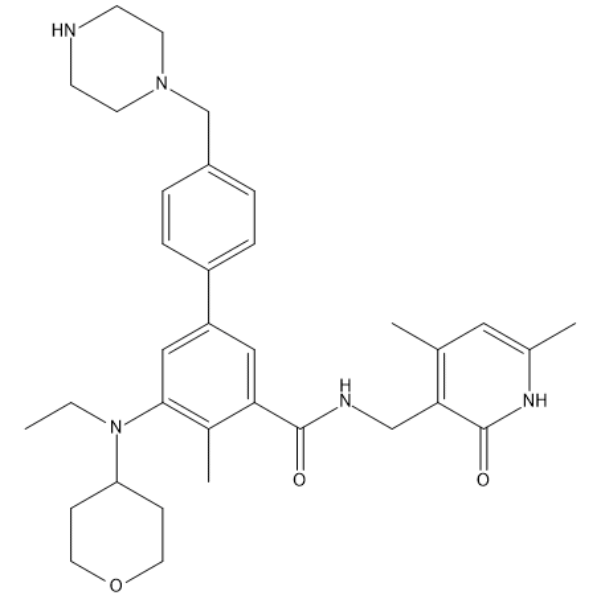
EZH2-IN-13
EZH2-IN-13 es un potente inhibidor de EZH2; para obtener mÁs informaciÓn, consulte el compuesto 73 en la patente WO2017139404. EZH2-IN-13 se puede utilizar para estudiar cÁnceres o lesiones precancerosas asociadas con la actividad de EZH2.
-
GC19149
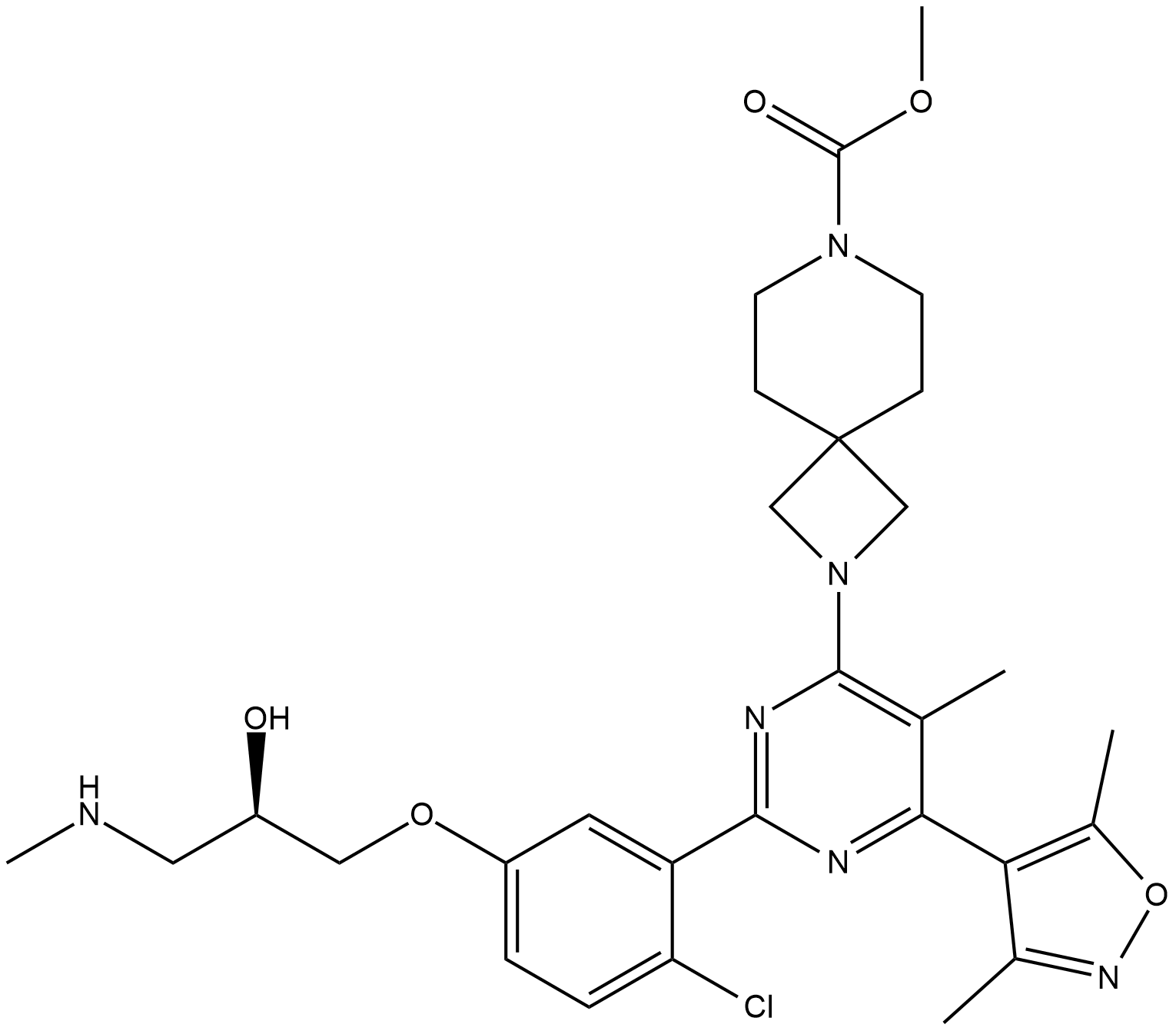
EZM 2302
EZM 2302 es un inhibidor de la arginina metiltransferasa 1 asociada al coactivador (CARM1) con una IC50 de 6nM.
-
GC64900
EZM0414
EZM0414 es un inhibidor potente, selectivo y biodisponible por vÍa oral de SETD2 (IC50 = 18 nM en el ensayo bioquÍmico de SETD2; IC50 = 34 nM en el ensayo celular). EZM0414 se puede utilizar para la investigaciÓn del mieloma mÚltiple en recaÍda o refractario y el linfoma difuso de células B grandes.
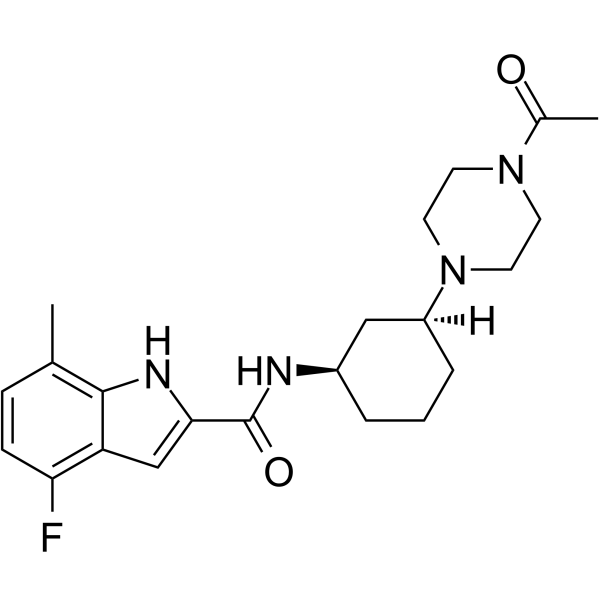
-
GC43651
F-Amidine (trifluoroacetate salt)
F-amidine is an inhibitor of protein arginine deiminases (PADs) that is selective for PAD1 and PAD4 (IC50s = 29.5, 350, and 21.6 μM for PAD1, PAD3, and PAD4 in vitro, respectively).
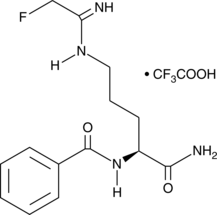
-
GC13139
FG-4592 (ASP1517)
Un inhibidor de las enzimas HIF-PH

-
GC16638
FG2216
FG2216 (IOX3) es un inhibidor potente y activo por vía oral de HIF prolil hidroxilasa-2 (PHD2), con una IC50 de 3,9 μM.
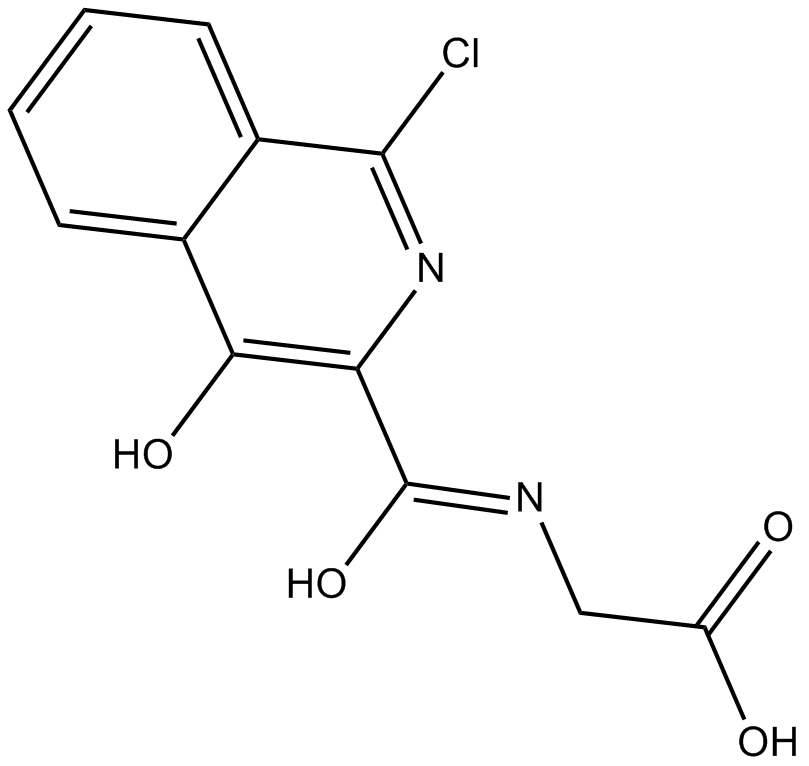
-
GC64821
FHD-286
FHD-286 es un inhibidor de ATPasa BRG1/BRM para el tratamiento de trastornos relacionados con BAF, como la leucemia mieloide aguda.
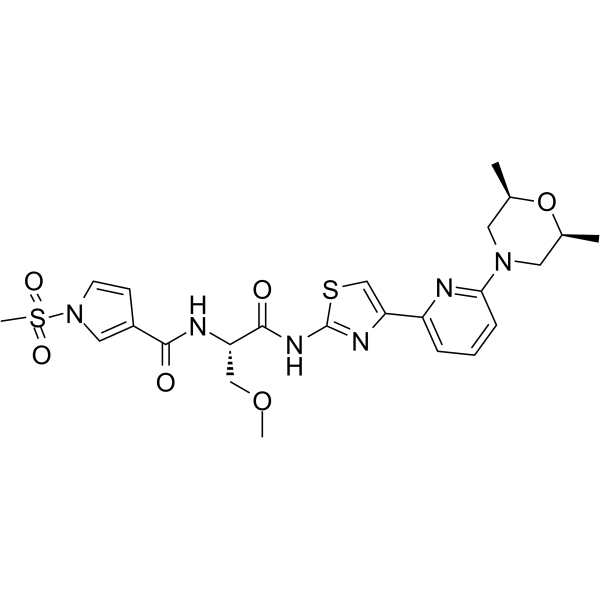
-
GC65160
FHT-1015
FHT-1205 es un potente inhibidor de ATPasa SMARCA4/SMARCA2 (BRG1 y BRM) con IC50 de ≤10 nM (WO2020160180A1; compuesto 67).
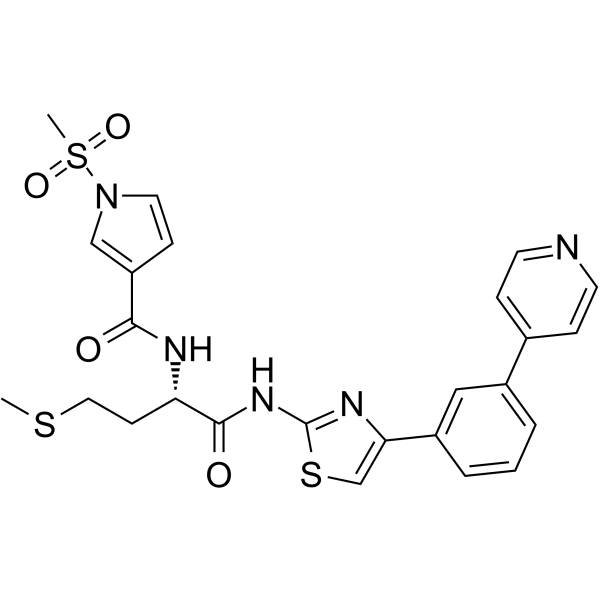
-
GC64777
FHT-1204
FHT-1204 es un potente inhibidor de ATPasa SMARCA4/SMARCA2 (BRG1 y BRM) con IC50 de ≤10 nM (WO2020160180A1; compuesto 70).
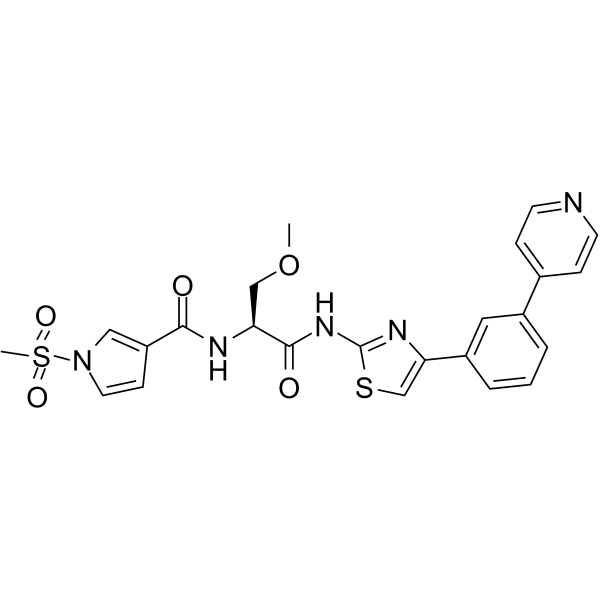
-
GC46148
Filgotinib-d4
Filgotinib-d4 (GLPG0634-d4) es el Filgotinib marcado con deuterio. Filgotinib (GLPG0634) es un inhibidor selectivo de JAK1 con IC50 de 10 nM, 28 nM, 810 nM y 116 nM para JAK1, JAK2, JAK3 y TYK2, respectivamente.
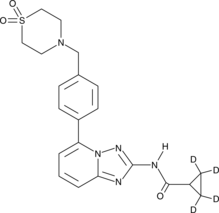
-
GN10030
Fisetin
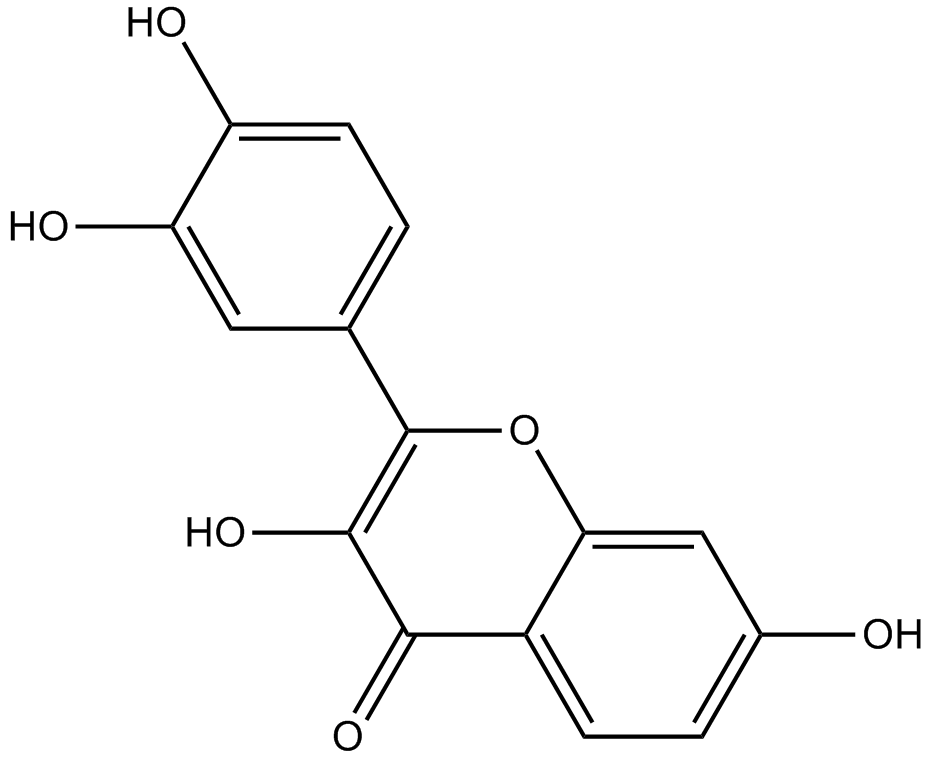
-
GC33015
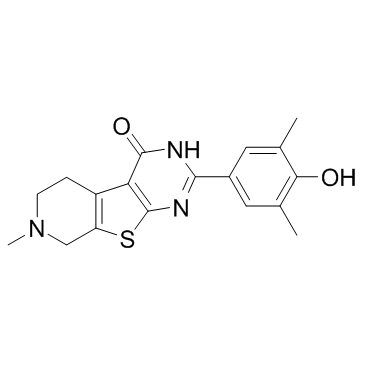
FL-411 (BRD4-IN-1)
FL-411 (BRD4-IN-1) es un inhibidor de BRD4 potente y selectivo con una IC50 de 0,43±0,09 μM para BRD4(1).
-
GC16875
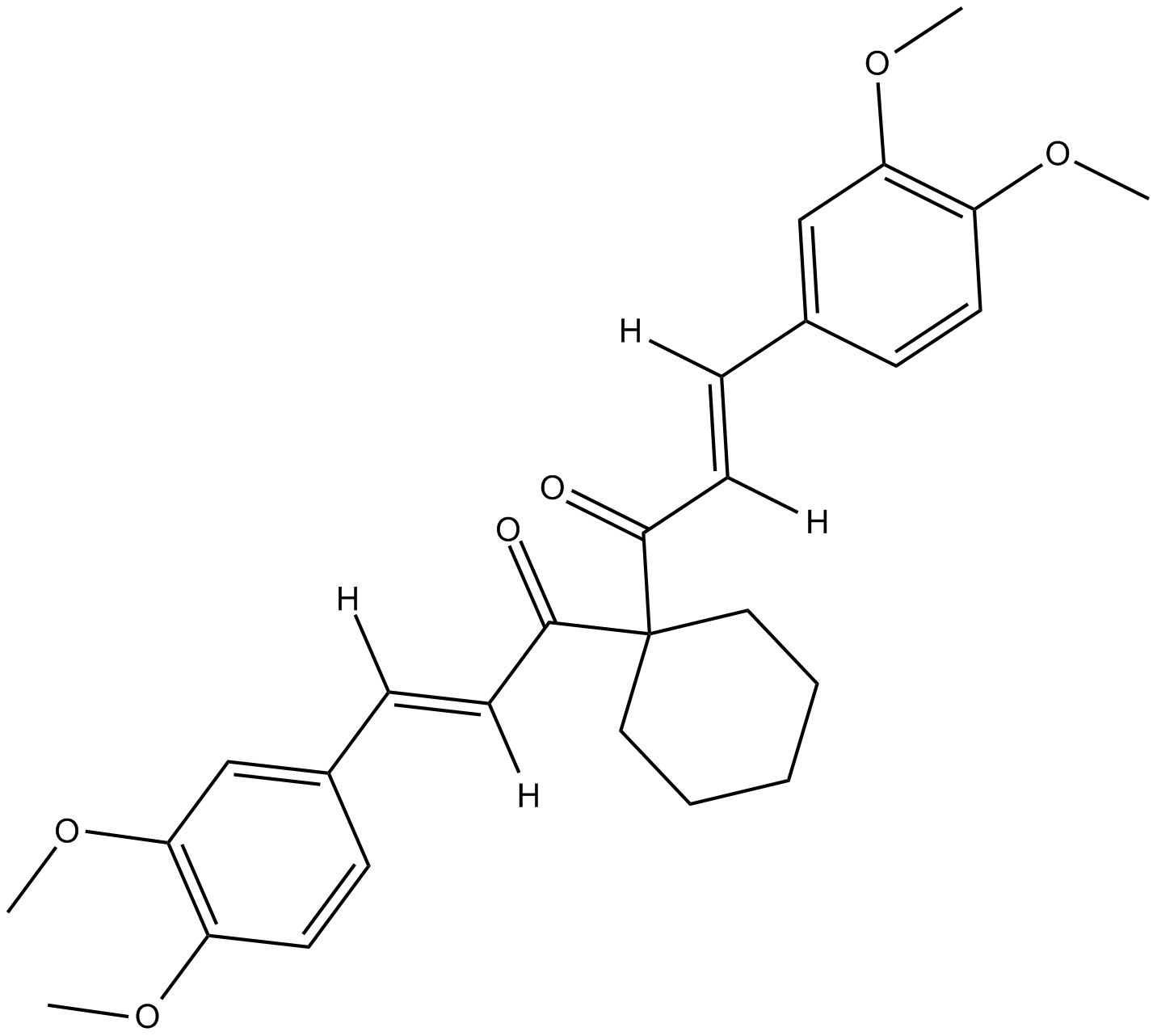
FLLL32
FLLL32, un anÁlogo sintético de la curcumina, es un inhibidor dual de JAK2/STAT3 con actividad antitumoral. FLLL32 puede inhibir la inducciÓn de la fosforilaciÓn de STAT3 por IFNα e IL-6 en células de cÁncer de mama.
-
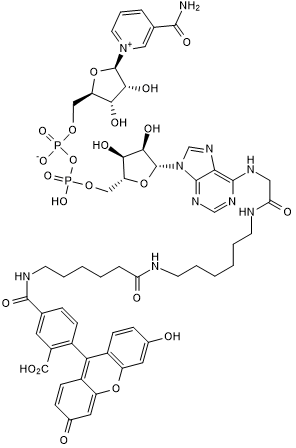
GC50506
Fluorescein-NAD+
La fluoresceÍna-NAD+ es una alternativa a la NAD radiomarcada y un sustrato para la ribosilaciÓn de ADP.
-
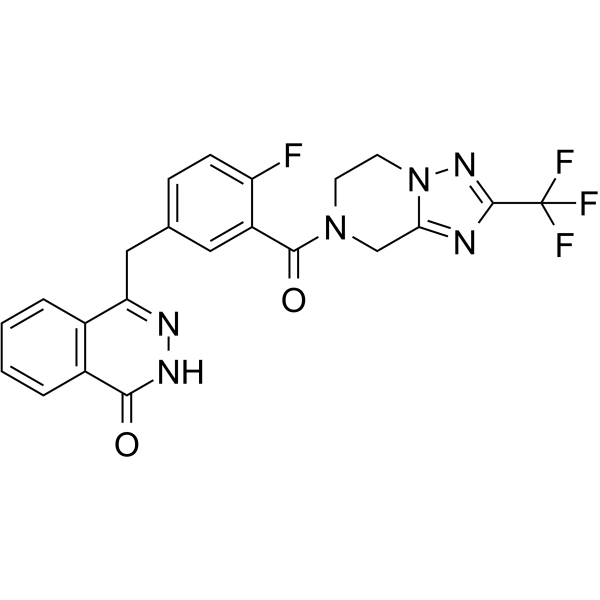
GC62121
Fluzoparib
Fluzoparib (SHR3162) es un inhibidor de PARP1 potente y activo por vÍa oral (IC50 = 1,46 ± 0,72 nM, un ensayo enzimÁtico libre de células) con una actividad antitumoral superior. Fluzoparib inhibe selectivamente la proliferaciÓn de células deficientes en reparaciÓn de recombinaciÓn homÓloga (HR) y sensibiliza tanto a las células con deficiencia de HR como a las células competentes con HR a los agentes citotÓxicos. Fluzoparib exhibe buenas propiedades farmacocinéticas in vivo y puede usarse para la investigaciÓn del cÁncer de ovario recidivante con mutaciÓn BRCA1/2.
-
GC50537
FM19G11
FM19G11 es un inhibidor del factor 1-alfa inducible por hipoxia (HIF-1α) e inhibe la actividad de luciferasa inducida por hipoxia con una IC50 de 80 nM en células HeLa.
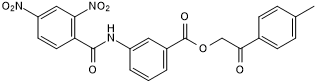
-
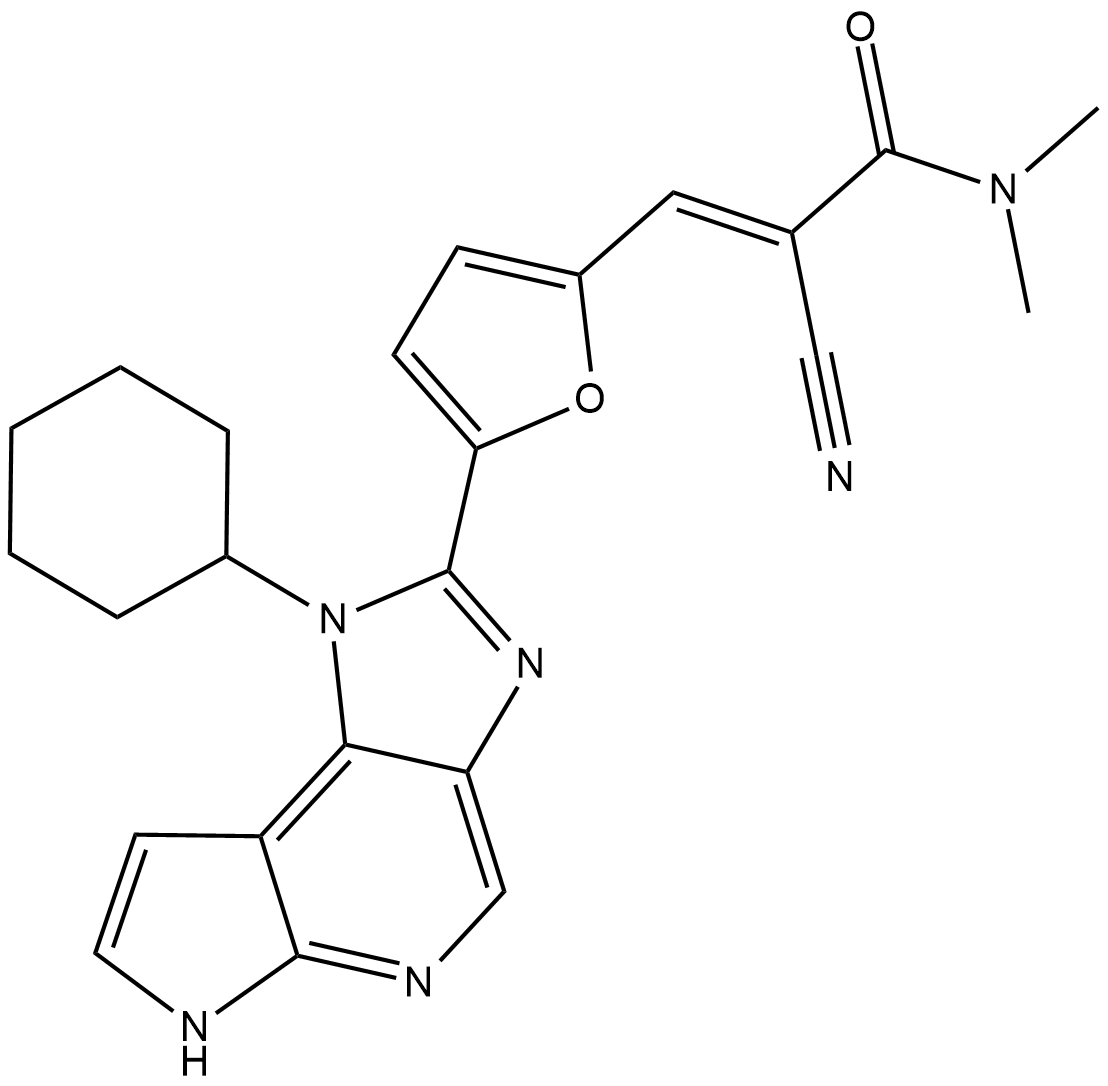
GC19408
FM381
FM-381 is a highly potent and JAK3-selective janus kinase (JAK) inhibitor
-
GC62461
FNDR-20123
FNDR-20123 es un inhibidor de HDAC antipalÚdico seguro, primero en su clase y activo por vÍa oral con IC50 de 31 nM y 3 nM para Plasmodium y HDAC humano, respectivamente.

-
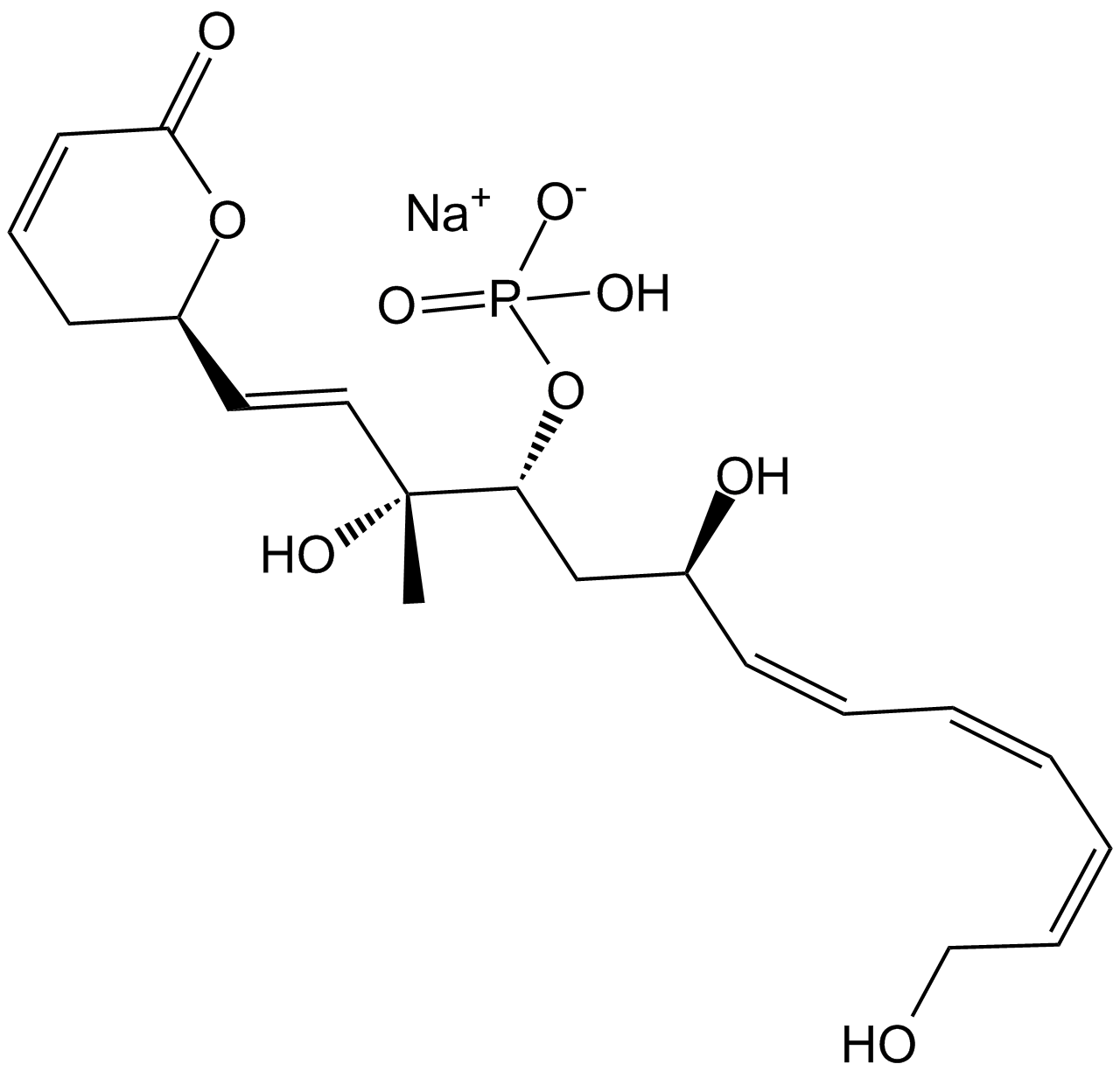
GC14395
Fostriecin sodium salt
A potent inhibitor of protein phosphatases 2A and 4
-
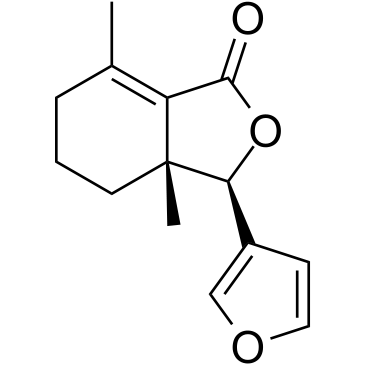
GC38044
Fraxinellone
-
GC65206
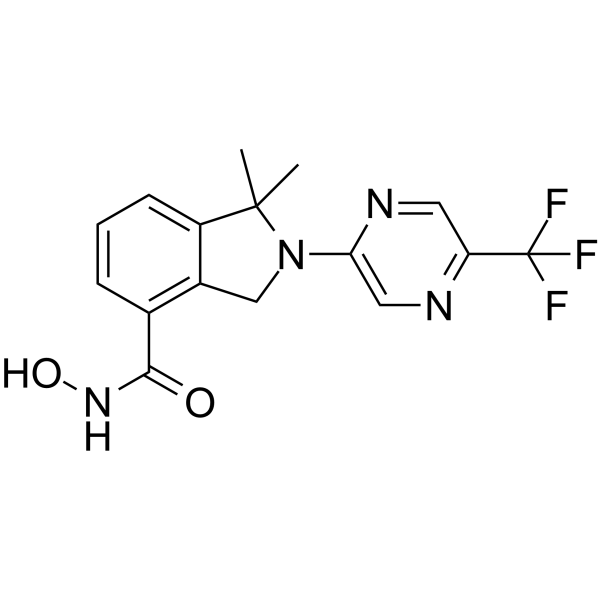
FT895
FT895 es un inhibidor de HDAC11 potente y selectivo con una IC50 de 3 nM.
-
GC63545
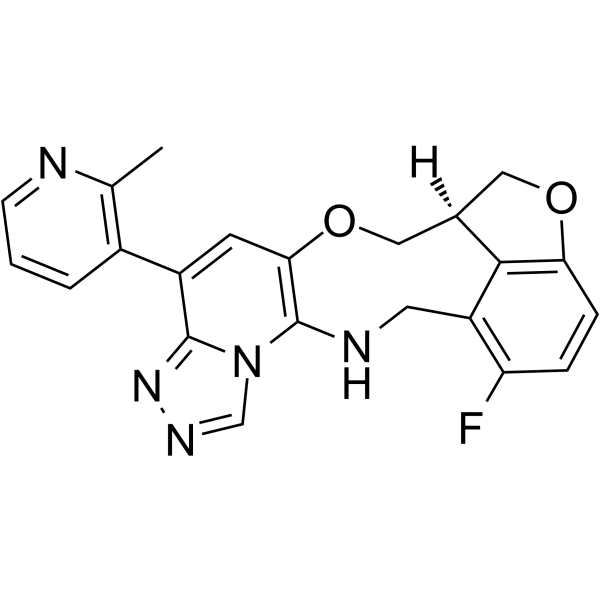
FTX-6058
FTX-6058 ((S)-FTX-6058) es un inhibidor potente y activo por vÍa oral del desarrollo del ectodermo embrionario (EED).
-
GC63546
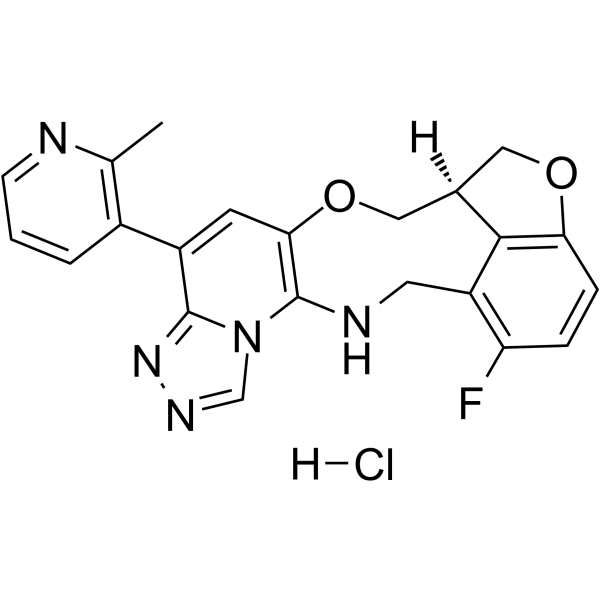
FTX-6058 hydrochloride
El clorhidrato de (S)-pociredir ((S)-FTX-6058) es un inhibidor potente y oralmente activo del desarrollo del ectodermo embrionario (EED).
-
GC10456
Fucosterol
El fucosterol es un esterol aislado de algas, algas o diatomeas.
-
GC43715
Furamidine (hydrochloride)
La furamidina (clorhidrato) (DB75 diclorhidrato) es un inhibidor selectivo de la proteÍna arginina metiltransferasa 1 (PRMT1) con una IC50 de 9,4 μM.
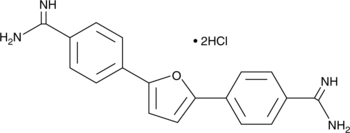
-
GC13541
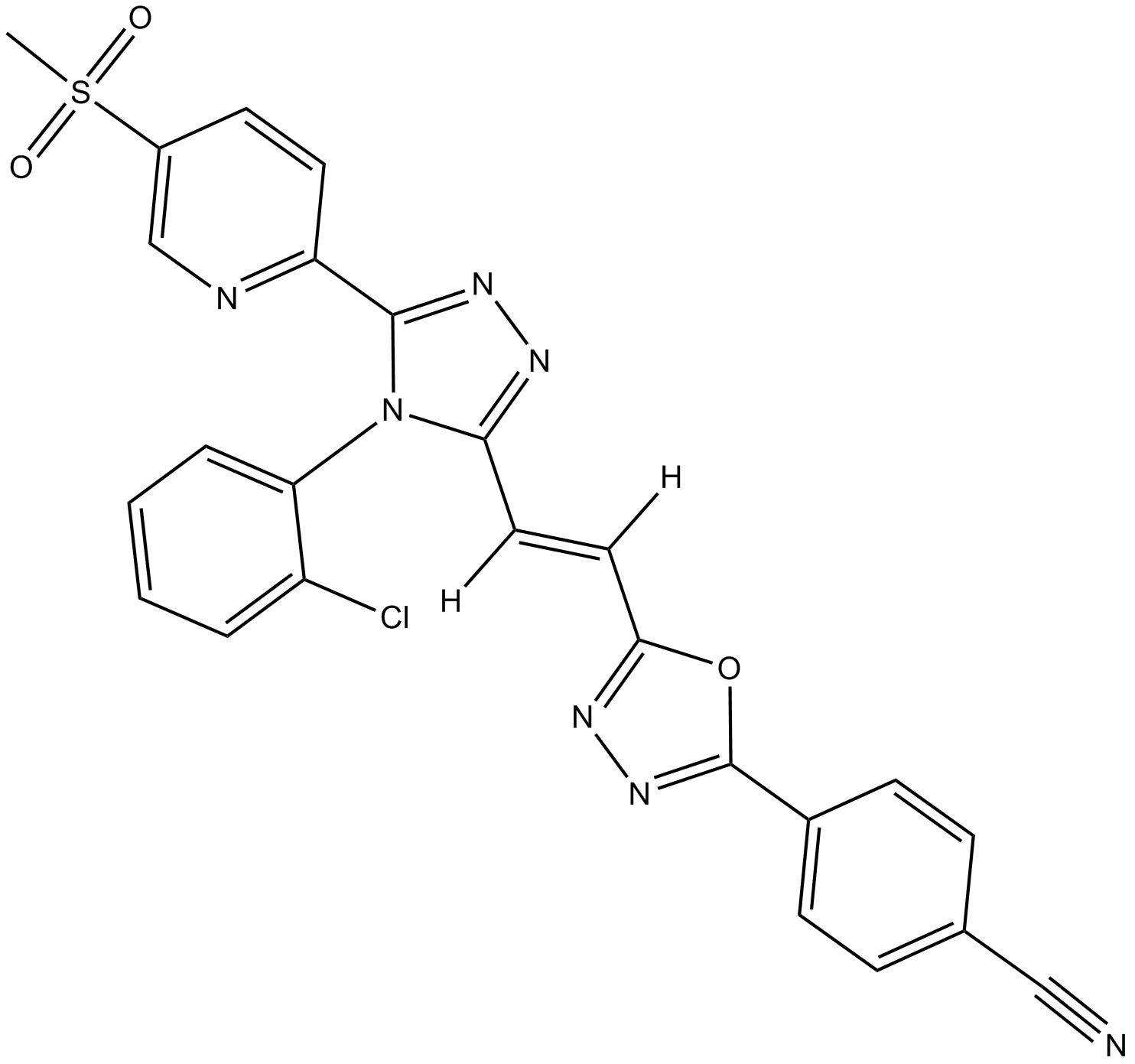
G007-LK
G007-LK es un inhibidor potente y selectivo de TNKS1 y TNKS2, con IC50 de 46 nM y 25 nM, respectivamente.
-
GC62246
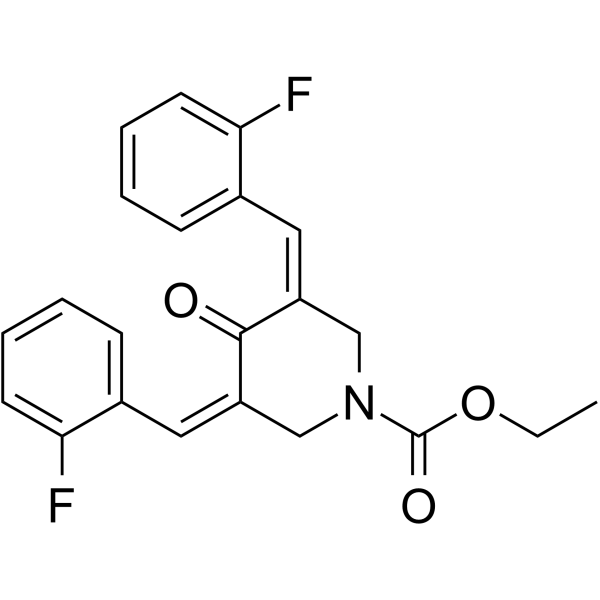
G5-7
G5-7, un inhibidor de JAK2 alostérico activo por vÍa oral, inhibe selectivamente la fosforilaciÓn mediada por JAK2 y la activaciÓn de EGFR (Tyr1068) y STAT3 al unirse a JAK2. G5-7 induce la detenciÓn del ciclo celular, la apoptosis y posee un efecto antiangiogénico. G5-7 tiene el potencial para el estudio de gliomas.
-
GC38062
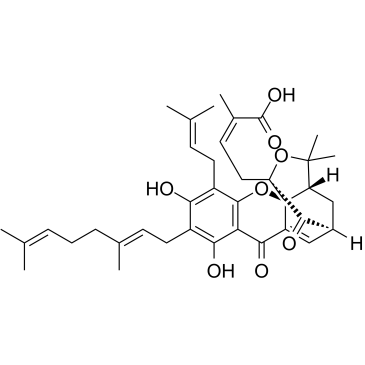
Gambogenic acid
El Ácido gambigénico es un ingrediente activo del gamboge, con actividad anticancerÍgena. El Ácido gambigénico actÚa como un inhibidor eficaz de EZH2, se une de manera especÍfica y covalente a Cys668 dentro del dominio EZH2-SET e induce la ubiquitinaciÓn de EZH2.
-
GC40900
Ganoderic Acid D
El Ácido ganodérico D, un triterpenoide tetracÍclico altamente oxigenado, es el principal componente activo de Ganoderma lucidum. El Ácido ganodérico D regula positivamente la expresiÓn proteica de SIRT3 e induce la ciclofilina D desacetilada (CypD) por SIRT3. El Ácido ganodérico D inhibe la reprogramaciÓn energética de las células de cÁncer de colon, incluida la absorciÓn de glucosa, la producciÓn de lactato, la producciÓn de piruvato y acetil-coenzima en las células de cÁncer de colon. El Ácido ganodérico D induce la apoptosis del carcinoma cervical humano HeLa.
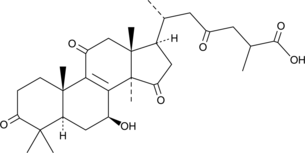
-
GC13474
Garcinol
El garcinol, una benzofenona poliisoprenilada extraÍda de Garcinia indica, ejerce propiedades anticolinesterasa frente a la acetilcolinesterasa (AChE) y la butirilcolinesterasa (BChE) con IC50 de 0,66 μM y 7,39 μM, respectivamente.
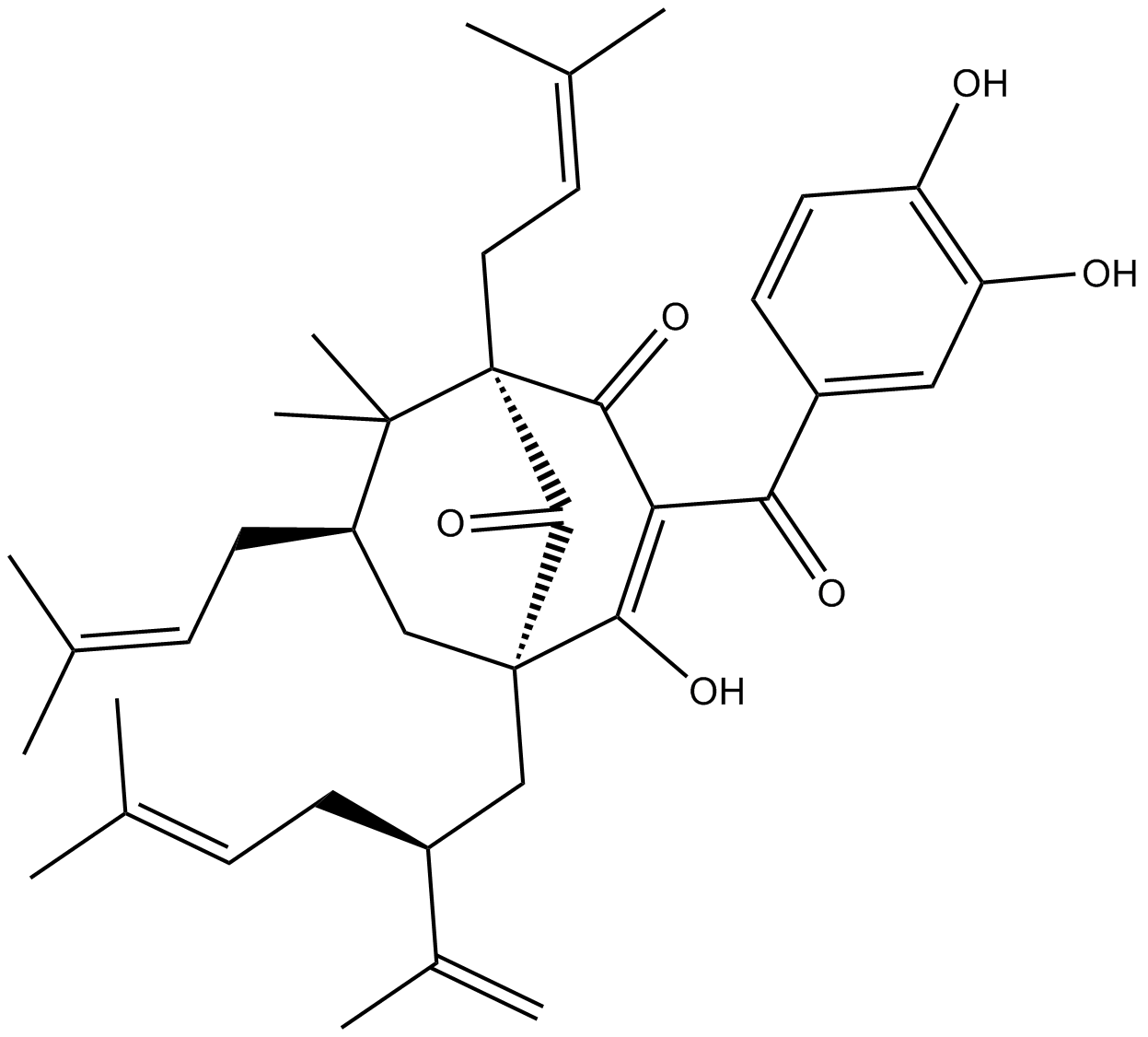
-
GC60172
Gardenia yellow
El amarillo de gardenia es un miembro activo de la crocina, aumenta la expresiÓn del ARNm de SIRT3 y actÚa como un agente antidepresivo activo por vÍa oral.

-
GC32958
GDC-0339
GDC-0339 es un potente inhibidor de la cinasa pan-Pim biodisponible por vÍa oral y bien tolerado, con Kis de 0,03 nM, 0,1 nM y 0,02 nM para Pim1, Pim2 y Pim3, respectivamente. GDC-0339 se descubre como un tratamiento potencial del mieloma mÚltiple.
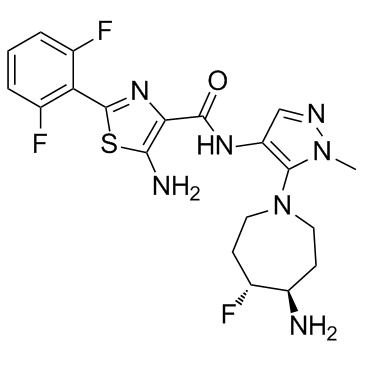
-
GC68378
GDC-4379

-
GC19542
GeA-69
GeA-69 es un inhibidor alostérico selectivo de la poliadenosina-difosfato-ribosa polimerasa 14 (PARP14) que se dirige al macrodominio 2 (MD2), con un valor de Kd de 2,1 µM. GeA-69 participa en los mecanismos de reparación de daños en el ADN y evita el reclutamiento de PARP14 MD2 en los sitios de daños en el ADN inducidos por láser.

-
GN10473
Ginkgolide C
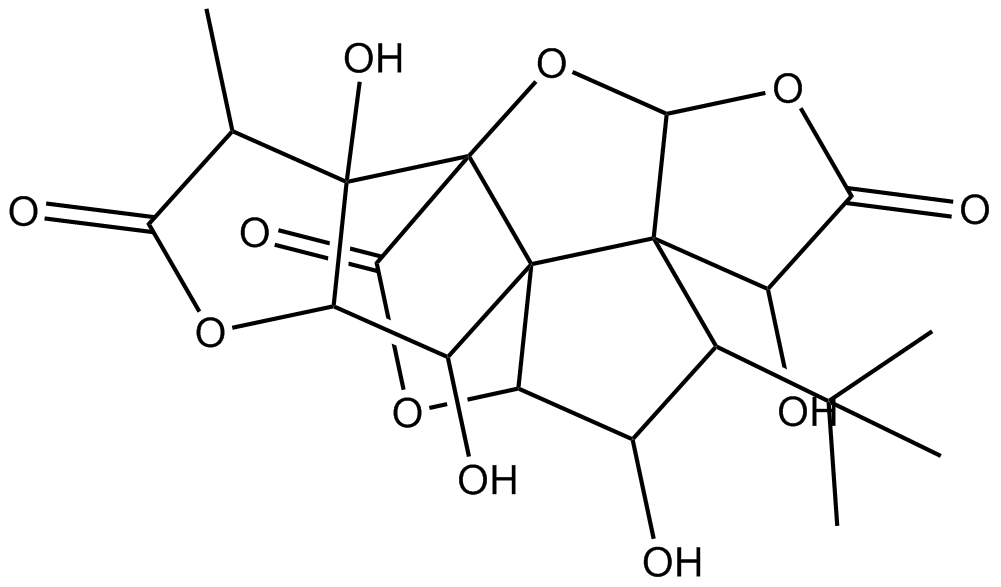
-
GN10584
Ginsenoside Rk1
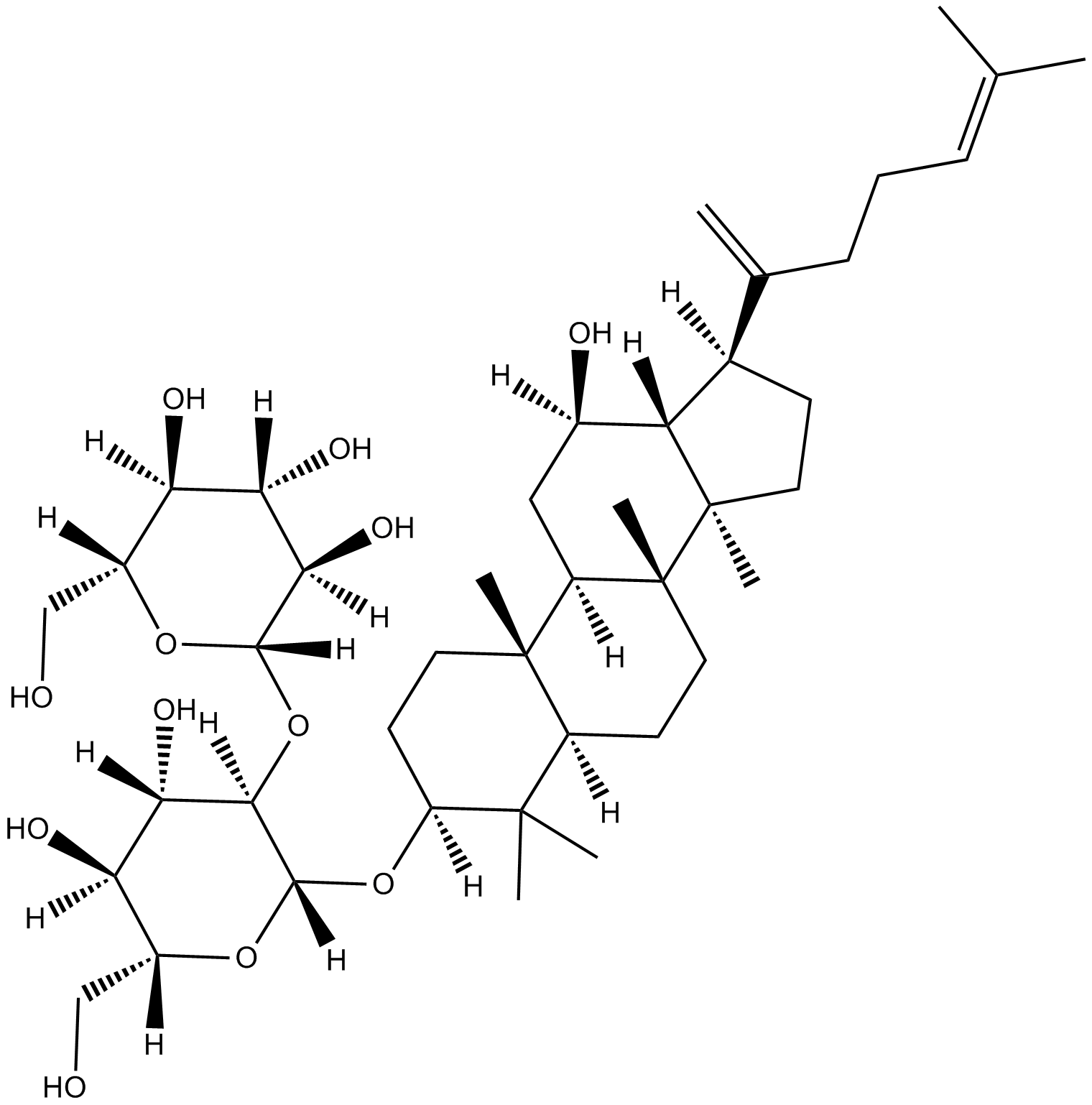
-
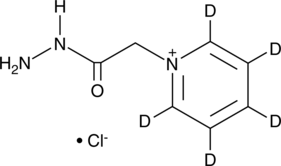
GC48994
Girard’s Reagent P-d5
An internal standard for the quantification of Girard’s reagent P
-
GC33050
Givinostat (ITF-2357)
Givinostat (ITF-2357) (ITF-2357) es un inhibidor de HDAC con una IC50 de 198 y 157 nM para HDAC1 y HDAC3, respectivamente.

-
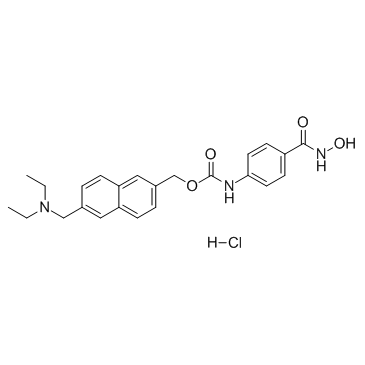
GC33159
Givinostat hydrochloride (ITF-2357 hydrochloride)
El clorhidrato de Givinostat (ITF-2357) es un inhibidor de HDAC con una IC50 de 198 y 157 nM para HDAC1 y HDAC3, respectivamente.
-
GC12579
GLPG0634
GLPG0634 (GLPG0634) es un inhibidor de JAK1 selectivo y activo por vÍa oral con IC50 de 10 nM, 28 nM, 810 nM y 116 nM para JAK1, JAK2, JAK3 y TYK2, respectivamente.
-
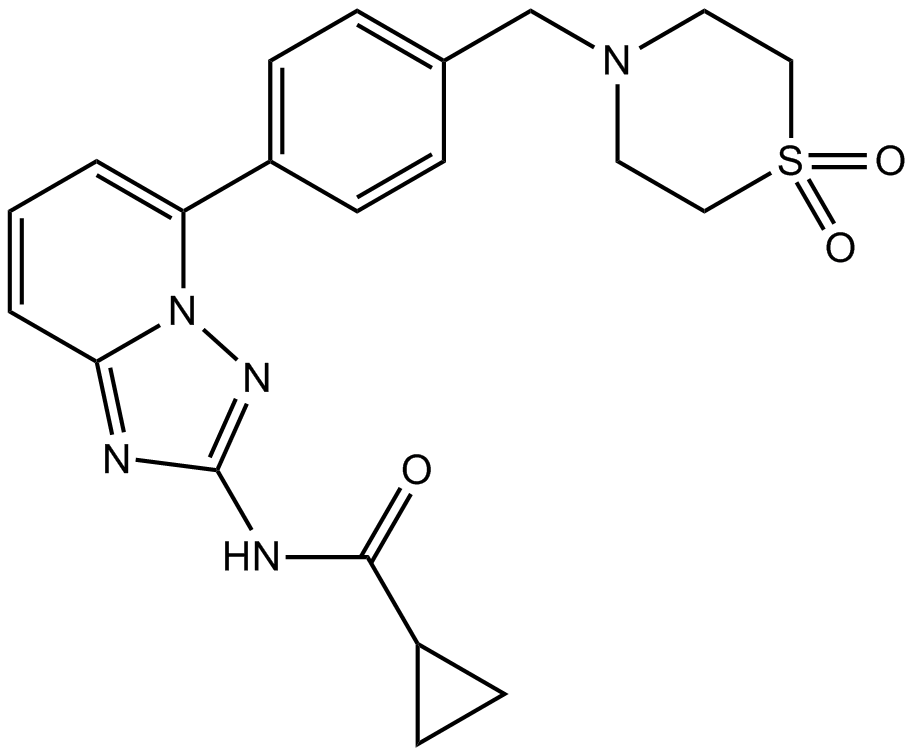
GC17222
GLPG0634 analogue
El anÁlogo GLPG0634 (Compuesto 176) es un inhibidor de JAK de amplio espectro con valores IC50 de <100 nM frente a JAK1, JAK2 y JAK3.
-
GC30263
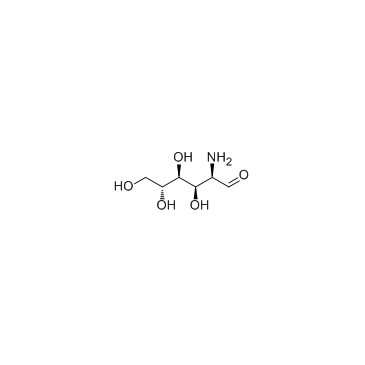
Glucosamine (D-Glucosamine)
La glucosamina (D-Glucosamina) (D-Glucosamina (D-Glucosamina)) es un aminoazÚcar y un precursor destacado en la sÍntesis bioquÍmica de proteÍnas y lÍpidos glicosilados, se utiliza como suplemento dietético.
-
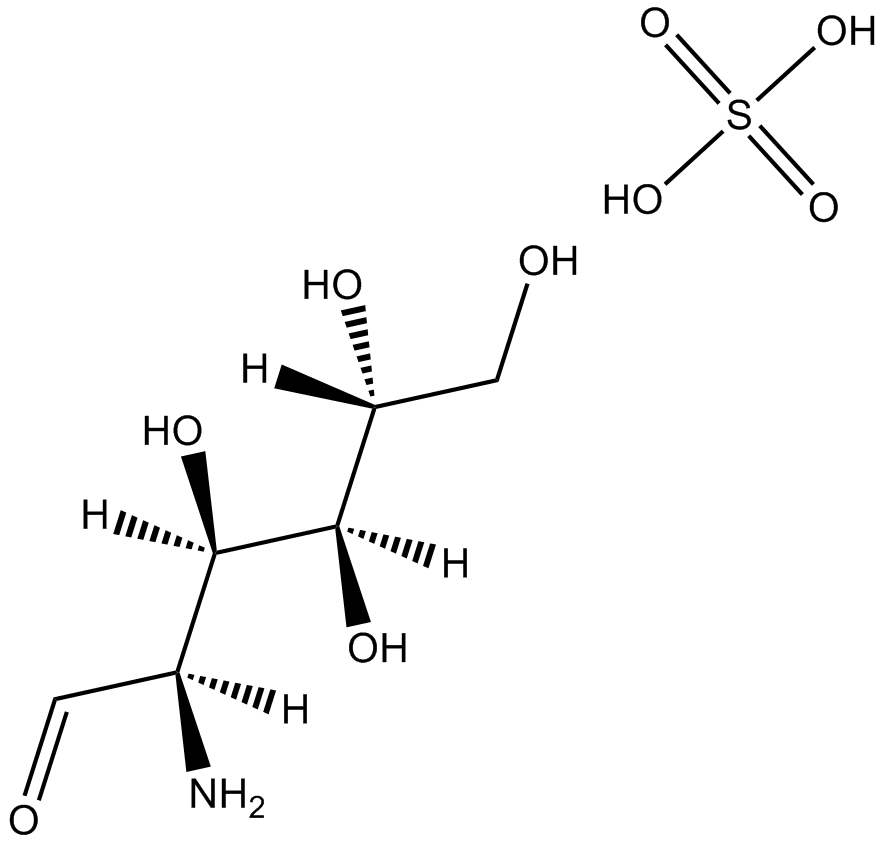
GN10669
Glucosamine sulfate
-
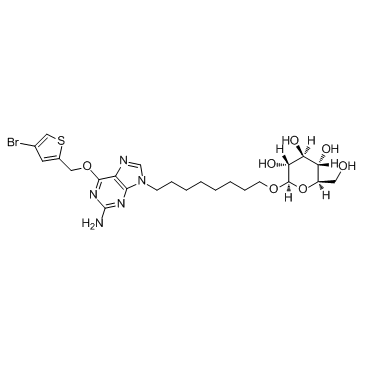
GC36153
Glucose-conjugated MGMT inhibitor
El inhibidor de MGMT conjugado con glucosa es un potente inhibidor de O6-metilguanina-DNAmetil-transferasa (MGMT), con IC50 de 32 nM in vitro (extractos celulares) y 10 nM en células HeLa S3.
-
GC34595
GN44028
GN44028 es un inhibidor del factor inducible por hipoxia (HIF)-1α potente y activo por vÍa oral, con una IC50 de 14 nM. GN44028 inhibe la actividad transcripcional de HIF-1α inducida por hipoxia sin suprimir la expresiÓn de ARNm de HIF-1α, la acumulaciÓn de proteÍna HIF-1α o la heterodimerizaciÓn de HIF-1α/HIF-1β. GN44028 se puede utilizar en la investigaciÓn de cÁnceres.
-
GC36168
GNA002
GNA002 es un inhibidor de EZH2 (potenciador del homÓlogo zeste 2) muy potente, especÍfico y covalente con una IC50 de 1,1 μM. GNA002 puede unirse de manera especÍfica y covalente a Cys668 dentro del dominio EZH2-SET, lo que desencadena la degradaciÓn de EZH2 a través del término COOH de la ubiquitinaciÓn mediada por la proteÍna que interactÚa con Hsp70 (CHIP). GNA002 reduce de manera eficiente la trimetilaciÓn de H3K27 mediada por EZH2, reactiva los genes supresores de tumores silenciados por el complejo represor polycomb 2 (PRC2).
-
GC32960
GNE-049
GNE-049 es un inhibidor de CBP muy potente y selectivo con una IC50 de 1,1 nM en el ensayo TR-FRET. GNE-049 también inhibe BRET y BRD4(1) con IC50 de 12 nM y 4200 nM, respectivamente.
-
GC68439
GNE-064

-
GC33212
GNE-207
GNE-207 es un inhibidor potente, selectivo y biodisponible por vÍa oral del bromodominio de CBP, con un IC50 de 1 nM, exhibe un Índice selectivo de> 2500 veces contra BRD4 (1). GNE-207 muestra una excelente potencia CBP, con un EC50 de 18 nM para la expresiÓn de MYC en células MV-4-11.
-
GC32747
GNE-272
GNE-272 es un inhibidor potente y selectivo de CBP/EP300 con valores IC50 de 0,02, 0,03 y 13 μM para CBP, EP300 y BRD4, respectivamente. GNE-272 también es una sonda in vivo selectiva para CBP/EP300.