DNA Damage/DNA Repair
- MTH1(4)
- PARP(58)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(94)
- Nucleoside Antimetabolite/Analogue(136)
- Telomerase(14)
- Topoisomerase(133)
- tankyrase(5)
- Antifolate(32)
- CDK(233)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(62)
- DNA Alkylator/Crosslinker(64)
- DNA/RNA Synthesis(388)
- Eukaryotic Initiation Factor (eIF)(22)
- IRE1(22)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(53)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
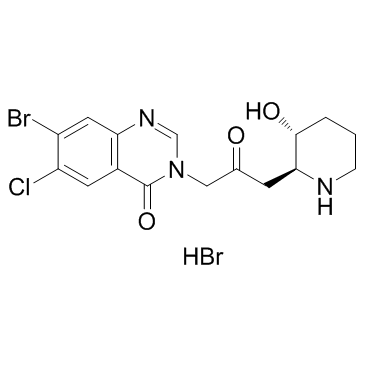
GC31950
Halofuginone hydrobromide (RU-19110 (hydrobromide))
Halofuginon (RU-19110) Hydrobromid, ein Febrifugin-Derivat, ist ein kompetitiver Prolyl-tRNA-Synthetase-Inhibitor mit einem Ki von 18,3 nM.
-
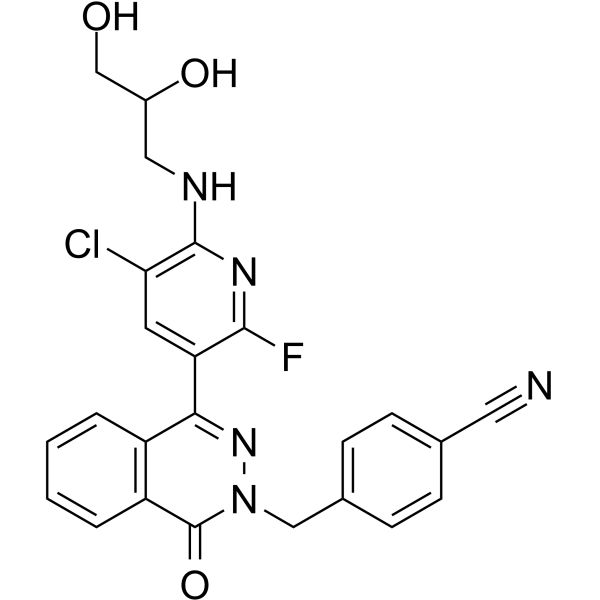
GC62328
HBV-IN-4
HBV-IN-4, ein Phthalazinon-Derivat, ist ein potenter und oral aktiver Hemmer der HBV-DNA-Replikation mit einem IC50-Wert von 14 nM.
-
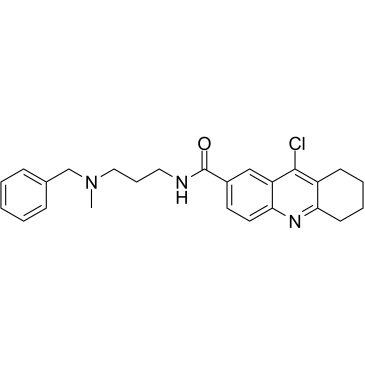
GC36211
HBX 19818
HBX 19818 ist ein spezifischer Inhibitor der Ubiquitin-spezifischen Protease 7 (USP7) mit einem IC50 von 28,1 μM.
-
GC19190
HDAC-IN-4
AZD 9468
HDAC-IN-4 ist ein potenter, selektiver und oral aktiver HDAC-Inhibitor der Klasse I mit IC50-Werten von 63 nM, 570 nM und 550 nM fÜr HDAC1, HDAC2 bzw. HDAC3. HDAC-IN-4 hat keine AktivitÄt gegen HDAC Klasse II. HDAC-IN-4 hat AntitumoraktivitÄt.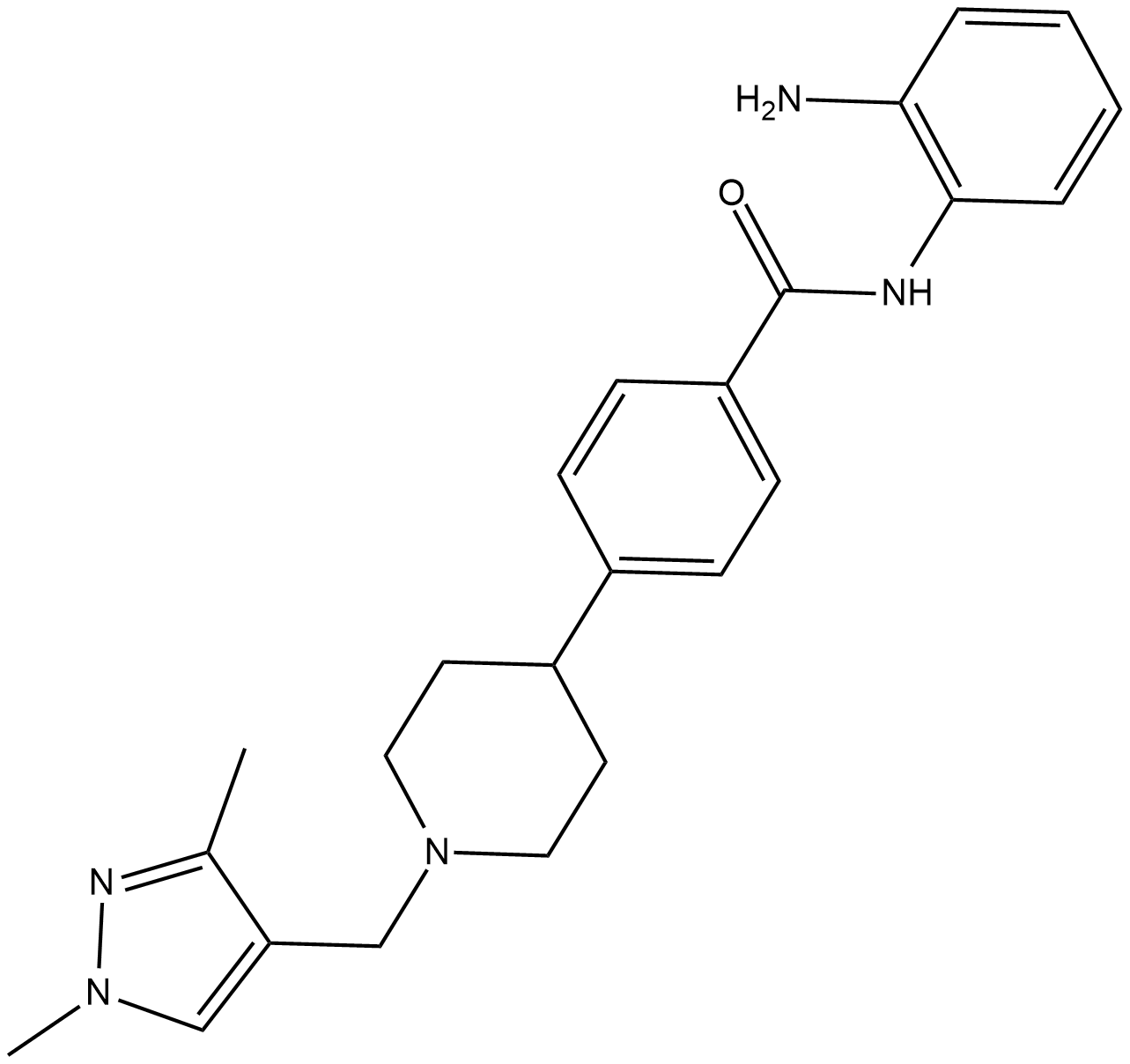
-
GC66052
HDAC-IN-40
HDAC-IN-40 ist ein potenter HDAC-Inhibitor auf Alkoxyamidbasis mit Ki-Werten von 60 nM und 30 nM fÜr HDAC2 bzw. HDAC6. HDAC-IN-40 hatte Antitumorwirkungen.
-
GC33395
HDAC-IN-5
HDAC-IN-5 ist ein Inhibitor der Histon-Deacetylase (HDAC).
-
GC68010
HDAC3-IN-T247
-
GC41085
HDAC6 Inhibitor
Histone Deacetylase 6
HDAC6-Inhibitor ist ein potenter und selektiver HDAC6-Inhibitor (IC50=36 nM). Der HDAC6-Inhibitor hemmt andere HDAC-Isoformen schwach. HDAC6-Inhibitor hemmt die Acyl-Tubulin-Akkumulation in Zellen mit einem IC50-Wert von 210 nM.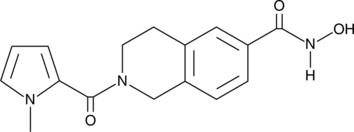
-
GC33317
HDAC6-IN-1
HDAC6-IN-1 ist ein potenter und selektiver Inhibitor fÜr HDAC6 mit einem IC50 von 17 nM und zeigt eine 25-fache bzw. 200-fache SelektivitÄt im Vergleich zu HDAC1 (IC50=422 nM) bzw. HDAC8 (IC50=3398 nM).
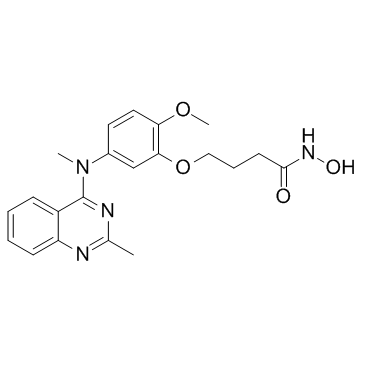
-
GC19189
HDAC8-IN-1
MDK-7933
HDAC8-IN-1 ist ein HDAC8-Inhibitor mit einem IC50 von 27,2 nM.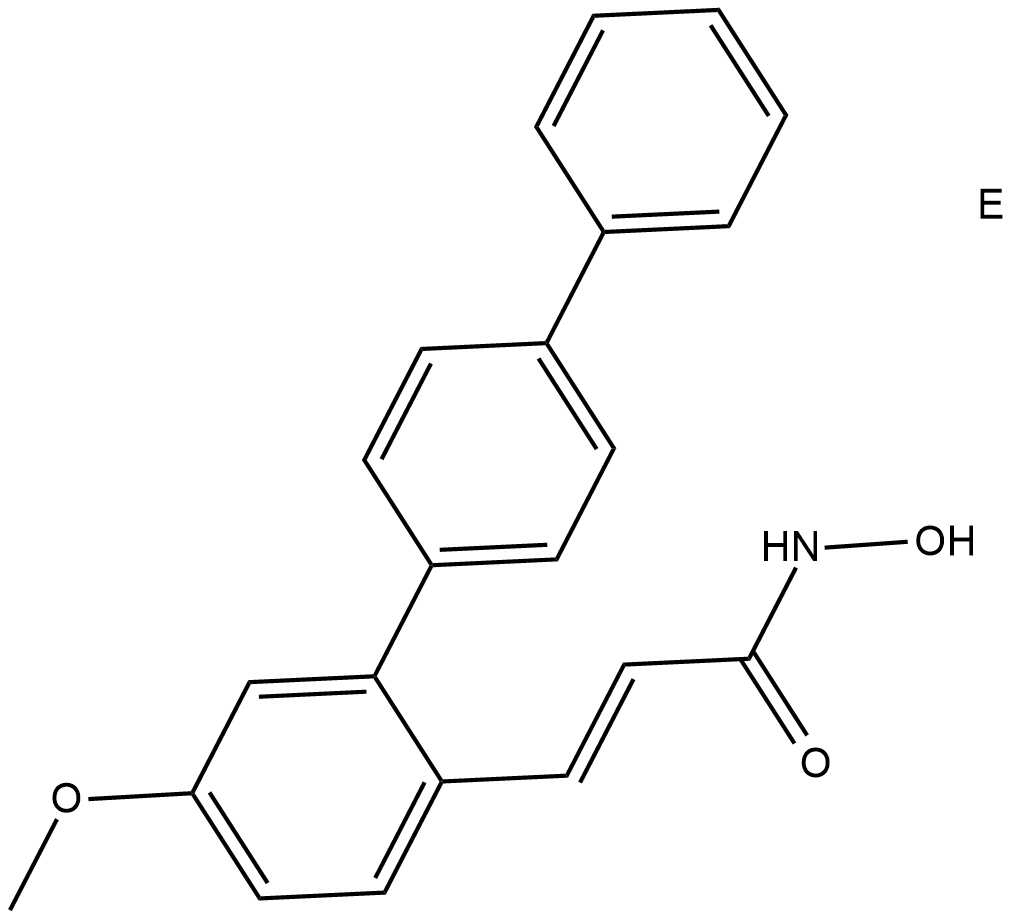
-
GC65460
HDACs/mTOR Inhibitor 1
HDACs/mTOR Inhibitor 1 ist ein dualer Histon-Deacetylasen (HDACs)- und SÄugetier-Target-Inhibitor von Rapamycin (mTOR) zur Behandlung hÄmatologischer Malignome mit IC50-Werten von 0,19 nM, 1,8 nM, 1,2 nM und >500 nM fÜr HDAC1, HDAC6, mTOR und PI3Kα. HDACs/mTOR Inhibitor 1 stimuliert den Zellzyklusarrest in der G0/G1-Phase und induziert die Apoptose von Tumorzellen mit geringer ToxizitÄt in vivo.
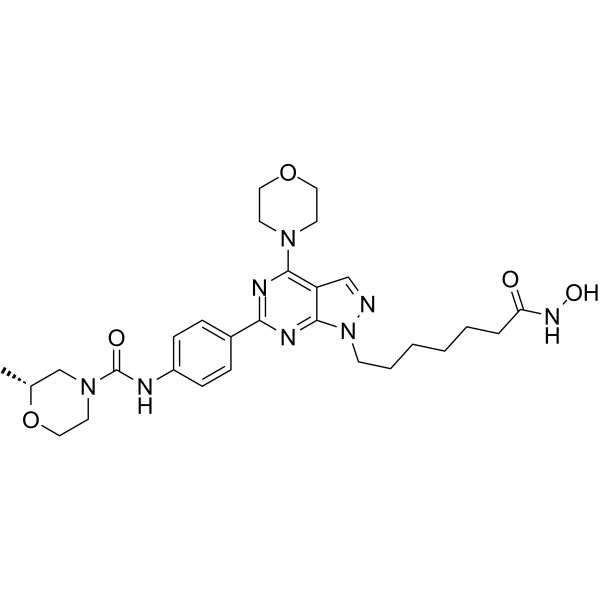
-
GC39279
hDHODH-IN-1
hDHODH-IN-1 ist ein Hemmer der humanen Dihydroorotat-Dehydrogenase (hDHODH).
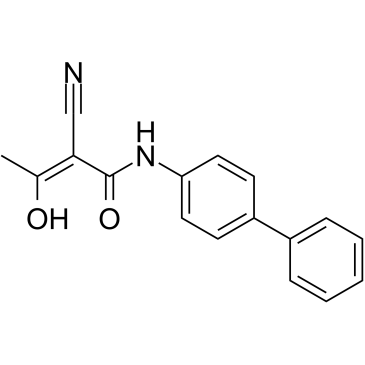
-
GC48388
Heliquinomycin
NSC 702208, Rubymycin
A bacterial metabolite with diverse biological activities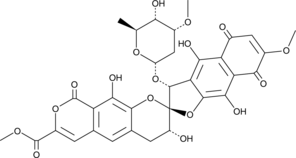
-
GC39266
Hematein
HÄmatein ist ein Oxidationsprodukt von HÄmatoxylin, das als Farbstoff wirkt. HÄmatein ist ein allosterischer Caseinkinase-II-Inhibitor mit einem IC50-Wert von 0,74 μM. HÄmatein hemmt die Akt/PKB-Ser129-Phosphorylierung, den Wnt/TCF-Weg und erhÖht die Apoptose in Lungenkrebszellen.
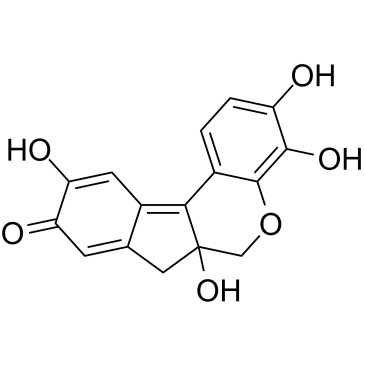
-
GC40103
Herboxidiene
GEX1A, Tan 1609
Herboxidiene, ein effektives Antitumormittel, zielt auf die SF3B-Untereinheit des Spliceosoms und bewirkt einen Zellzyklusarrest in G1 und G2/M in der Zelllinie WI-38.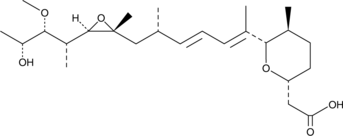
-
GC12201
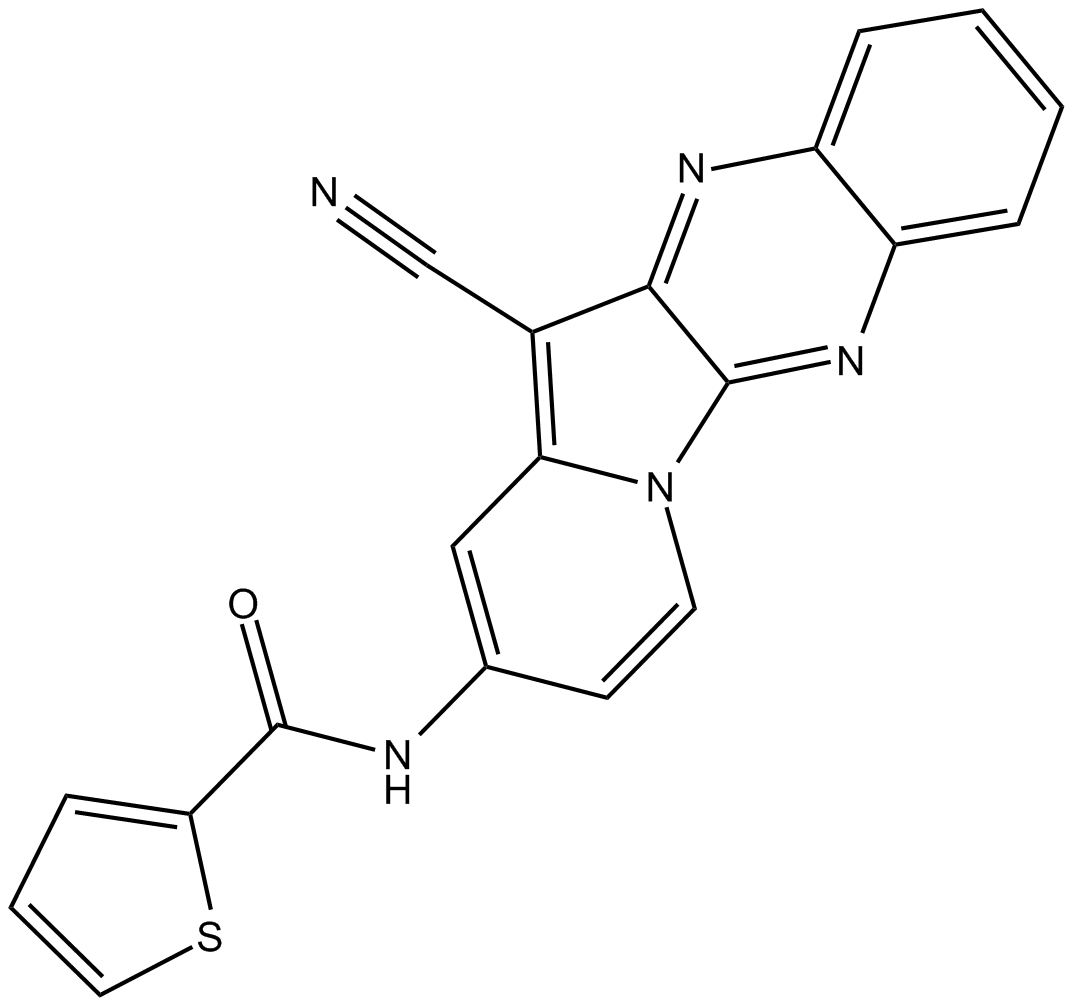
HI TOPK 032
HI TOPK 032 ist ein potenter und spezifischer TOPK-Hemmer.
-
GC11302
Hinokitiol
NSC 18804, β-Thujaplicin, 4-isopropyl Tropolone
Hinokitiol ist ein Bestandteil Ätherischer Öle, die aus Chymacyparis obtusa isoliert wurden, reduziert die Nrf2-Expression und verringert die mRNA- und Proteinexpression von DNMT1 und UHRF1 mit antiinfektiÖsen, antioxidativen und AntitumoraktivitÄten.
-
GC12334
HNHA
Histone Deacetylase Inhibitor VI
HNHA ist ein potenter Inhibitor der Histon-Deacetylase (HDAC). HNHA hÄlt den Zellzyklus in der G1/S-Phase Über p21-Induktion an. HNHA hemmt das Tumorwachstum und die Tumorneovaskularisation. HNHA kann ein wirksames Antikrebsmittel gegen Brustkrebs sein.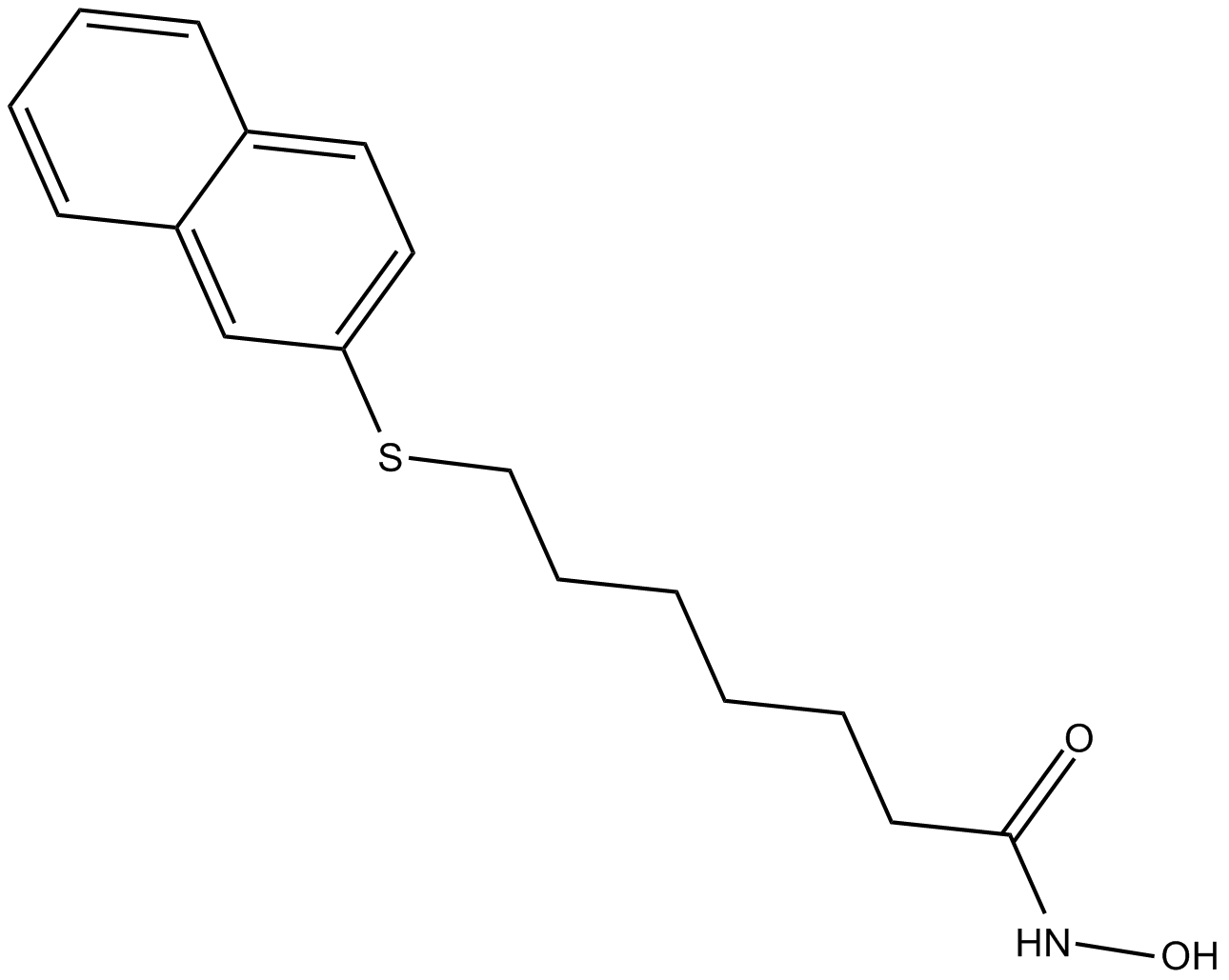
-
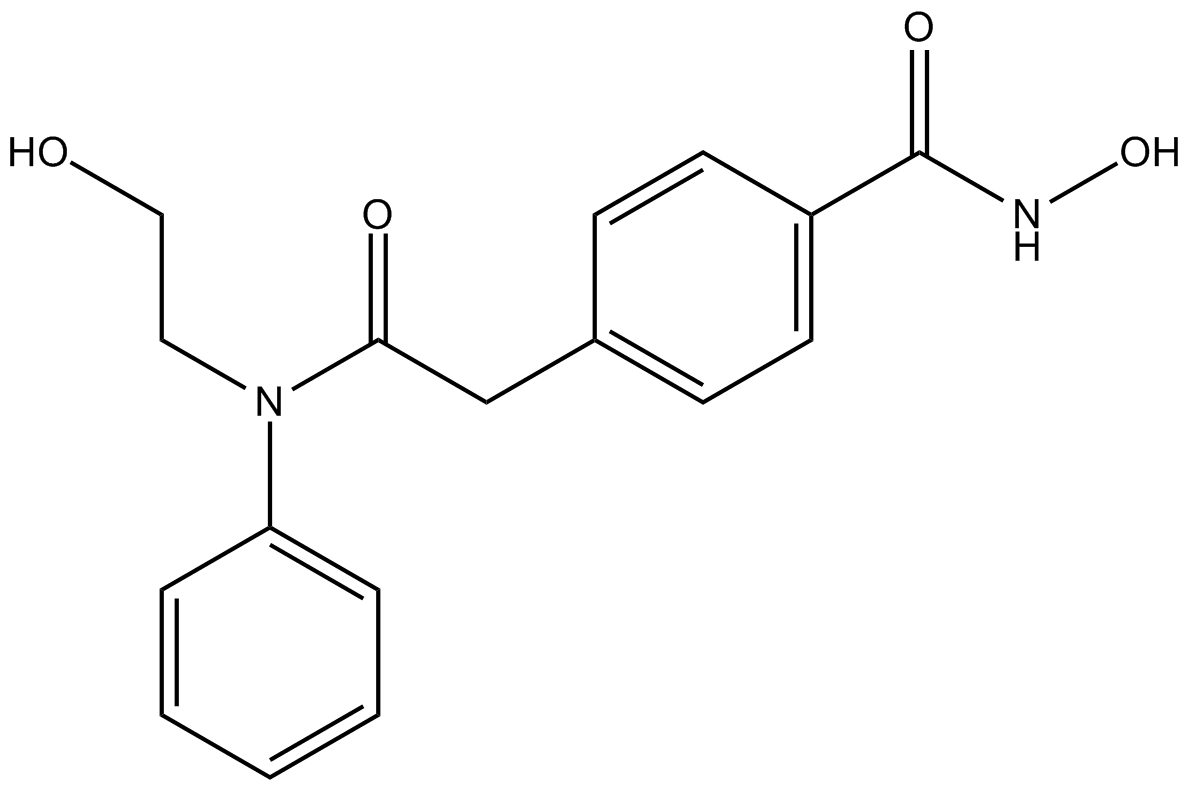
GC11574
HPOB
HPOB ist ein hochpotenter und selektiver Inhibitor von HDAC6 mit einem IC50 von 56 nM. HPOB zeigt eine >30-fach geringere Wirksamkeit gegenÜber anderen HDACs. HPOB verstÄrkt die Wirksamkeit von DNA-schÄdigenden Antikrebsmitteln in transformierten Zellen, aber nicht in normalen Zellen. HPOB blockiert nicht die Ubiquitin-bindende AktivitÄt von HDAC6.
-
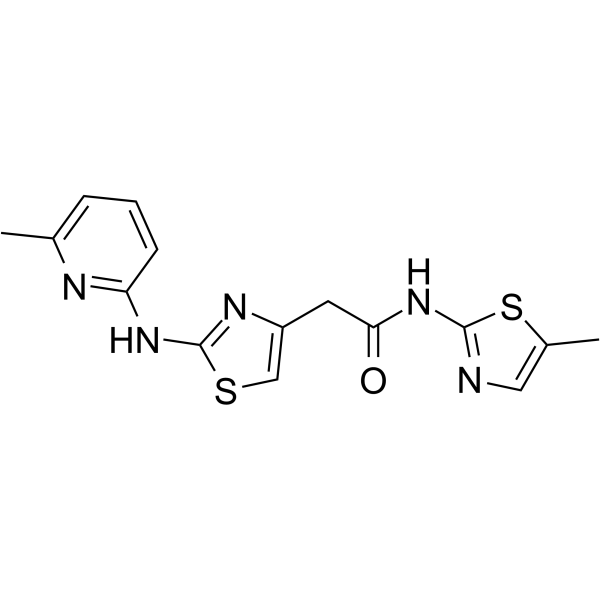
GC63854
HQ461
HQ461 ist ein molekularer Klebstoff, der die CDK12-DDB1-Interaktion fÖrdert, um den Abbau von Cyclin K auszulÖsen. Der HQ461-vermittelte Abbau von Cyclin K beeintrÄchtigt die Funktion von CDK12, was zu einer verminderten Phosphorylierung des CDK12-Substrats, einer Herunterregulierung von Genen fÜr die Reaktion auf DNA-SchÄden und zum Zelltod fÜhrt.
-
GC62572
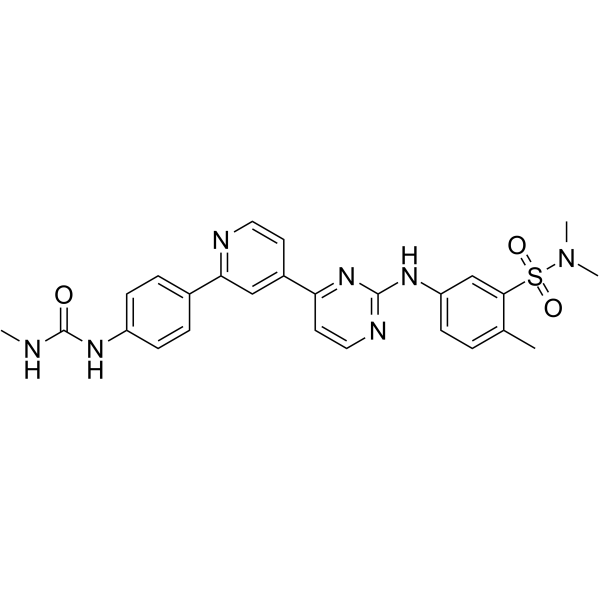
hSMG-1 inhibitor 11e
hSMG-1-Inhibitor 11e ist ein potenter und selektiver hSMG-1-Kinase-Inhibitor mit einer IC50 von 900-facher SelektivitÄt gegenÜber mTOR (IC50 von 45 nM), PI3Kα/γ (IC50 von 61 nM und 92 nM) und CDK1/CDK2 (IC50 von 32 μM und 7,1 μM).
-
GC61925
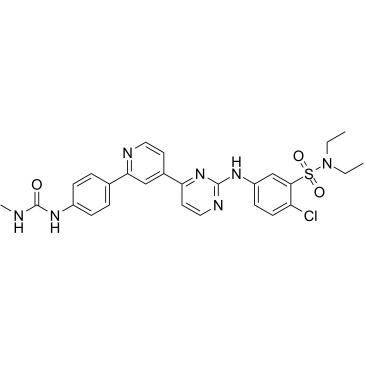
hSMG-1 inhibitor 11j
Der hSMG-1-Inhibitor 11j, ein Pyrimidinderivat, ist ein potenter und selektiver Inhibitor von hSMG-1 mit einem IC50-Wert von 0,11 nM. Der hSMG-1-Inhibitor 11j zeigt eine >455-fache SelektivitÄt fÜr hSMG-1 gegenÜber mTOR (IC50=50 nM), PI3Kα/γ (IC50=92/60 nM) und CDK1/CDK2 (IC50=32/7,1 μM). Der hSMG-1-Inhibitor 11j kann fÜr die Krebsforschung eingesetzt werden.
-
GC16843
Hydroxyurea
NCI C04831, NSC 32065
Hydroxyharnstoff ist ein Zellapoptoseinduktor, der die DNA-Synthese durch Hemmung der Ribonukleotidreduktase hemmt. Hydroxyharnstoff zeigt Anti-Orthopoxvirus-AktivitÄt.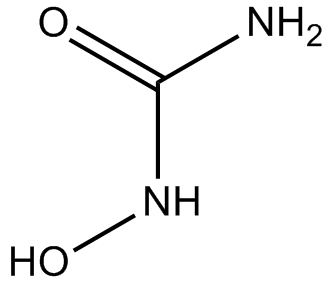
-
GC49694
Hygromycin A
(-)-Hygromycin A, Totomycin
An antibiotic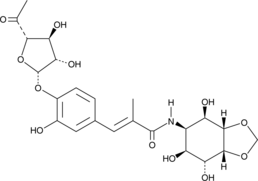
-
GC48445
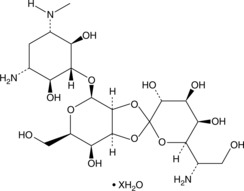
Hygromycin B (hydrate)
An aminoglycoside antibiotic
-
GC49267
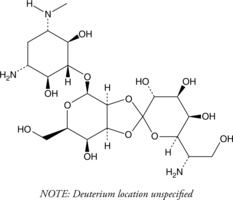
Hygromycin B-d4
An internal standard for the quantification of hygromycin
-
GC14503
IC261
SU5607
IC261 ist ein selektiver, ATP-kompetitiver CK1-Inhibitor mit IC50-Werten von 1 μM, 1 μM, 16 μM fÜr Ckiδ, Ckiε bzw. Ckiα1.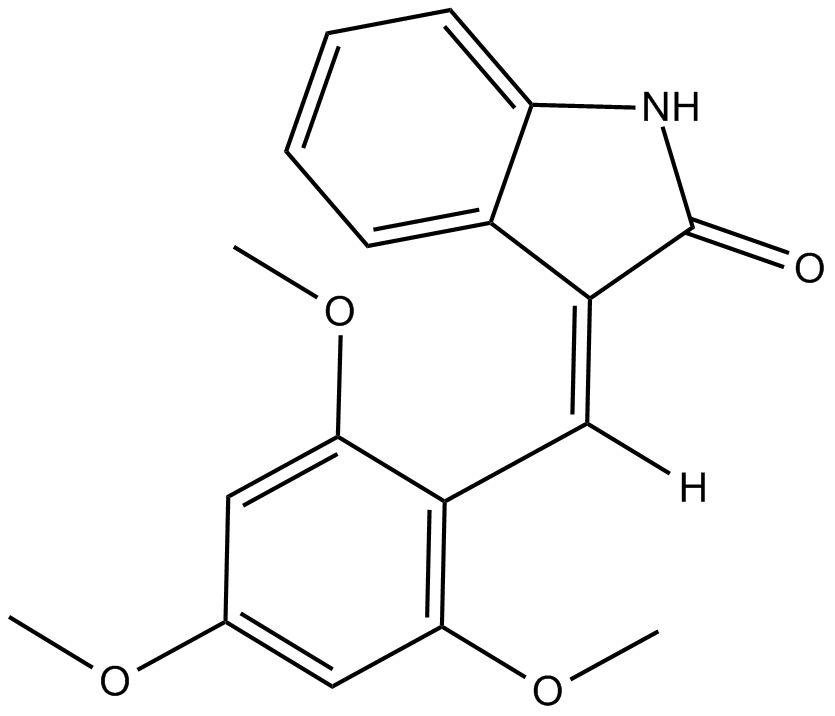
-
GC14969
Idarubicin HCl
4Demethoxydaunorubicin, 4DMD, NSC 256439
Idarubicin-HCl ist ein Anthracyclin-AntileukÄmie-Medikament.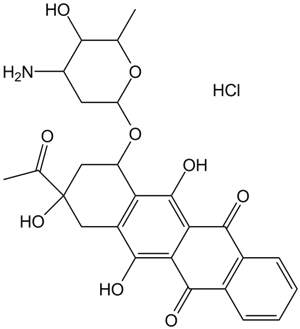
-
GC16003
Ifosfamide
Isophosphamide, NSC 109724
Ifosfamid ist ein alkylierendes Chemotherapeutikum mit AktivitÄt gegen ein breites Spektrum von Tumoren.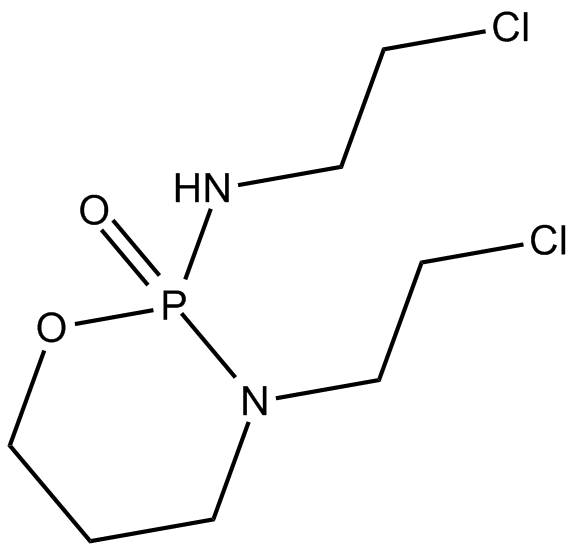
-
GC60929
Ilaprazole sodium
Ilaprazol (IY-81149) Natrium ist ein oral aktiver Protonenpumpenhemmer.
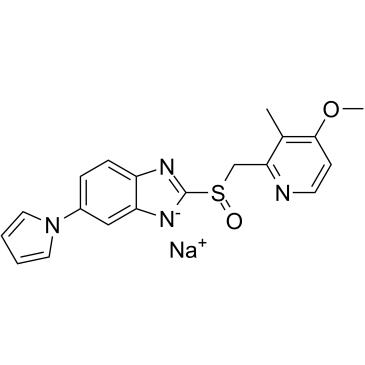
-
GC41340
Illudin M
DR-15977, (-)-Illudin M, NSC 400978, NSC 626370
Illudin M ist ein zytotoxisches Pilz-Sesquiterpen, das aus dem Kulturmedium von Omphalotus olearius-Pilzen isoliert werden kann. Illudin M kann DNA alkylieren. Illudin M hat Anti-Tumor-AktivitÄten.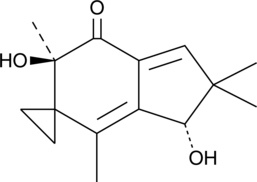
-
GC18100
Illudin S
Lampterol,NSC 400979,NSC 626369
Illudin S, ein zytotoxisches Illudin, ist ein natÜrliches Sesquiterpen mit starken Antitumor- und antiviralen AktivitÄten.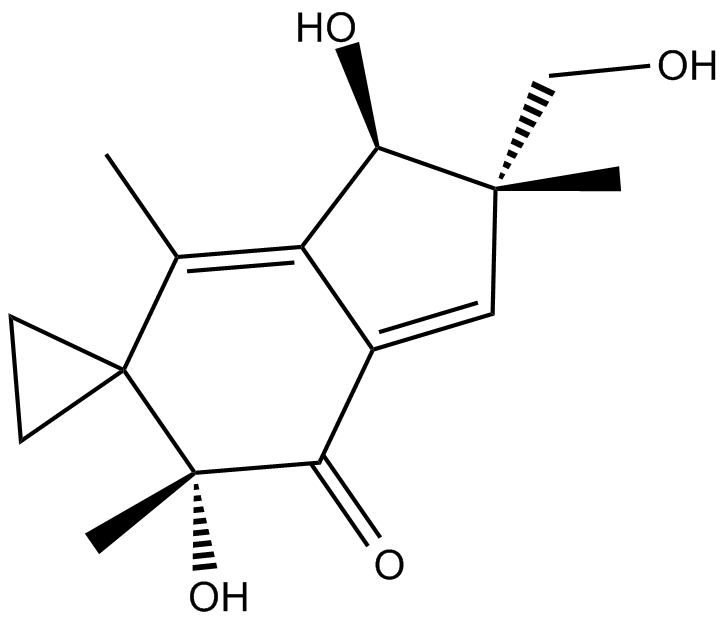
-
GC62137
IMT1
IMT1 ist ein erstklassiger spezifischer und nicht kompetitiver Hemmer der humanen mitochondrialen RNA-Polymerase (POLRMT).
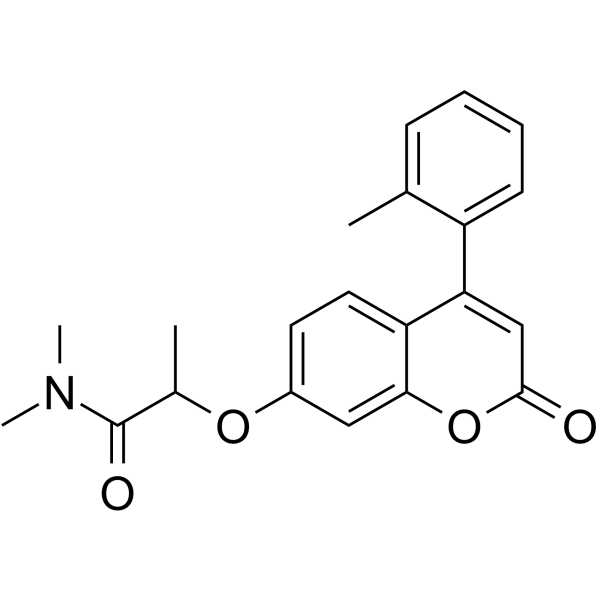
-
GC65939
Indimitecan
LMP776
Indimitecan (LMP776) ist ein Topoisomerase I (Top1)-Hemmer mit AntikrebsaktivitÄten.
-
GC36312
Indirubin-3'-monoxime-5-sulphonic acid
Indirubin-3'-Monoxim-5-sulfonsÄure ist ein potenter und selektiver Inhibitor von CDK1, CDK5 und GSK-3β mit IC50s von 5 nM, 7 nM bzw. 80 nM.
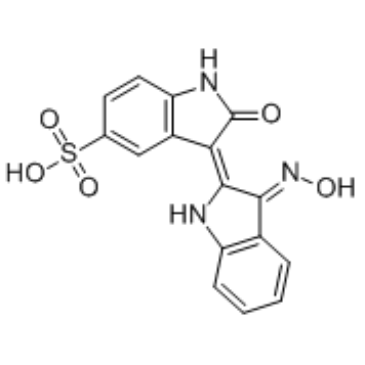
-
GC36313
Indirubin-5-sulfonate
Indirubin-5-sulfonat ist ein Cyclin-abhÄngiger Kinase (CDK)-Inhibitor mit IC50-Werten von 55 nM, 35 nM, 150 nM, 300 nM und 65 nM fÜr CDK1/Cyclin B, CDK2/Cyclin A, CDK2/Cyclin E, CDK4/Cyclin D1 bzw. CDK5/p35. Indirubin-5-sulfonat zeigt auch eine inhibitorische AktivitÄt gegen GSK-3β.
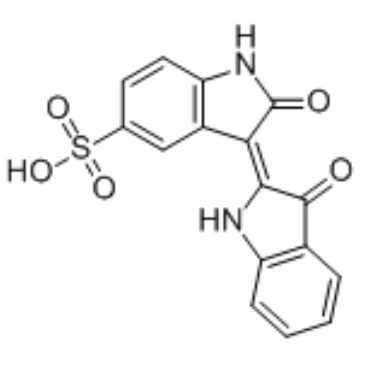
-
GC52513
Indolimine-214
Isoamylindolimine
A genotoxic gut microbiota metabolite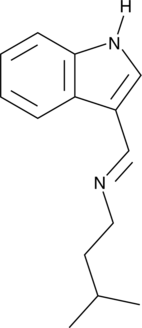
-
GC33205
Indotecan (LMP-400)
LMP-400; NSC-724998
Indotecan (LMP-400) (LMP-400) ist ein potenter Topoisomerase-1(Top1)-Inhibitor mit IC50-Werten von 300, 1200, 560 nM fÜr P388-, HCT116- bzw. MCF-7-Zelllinien.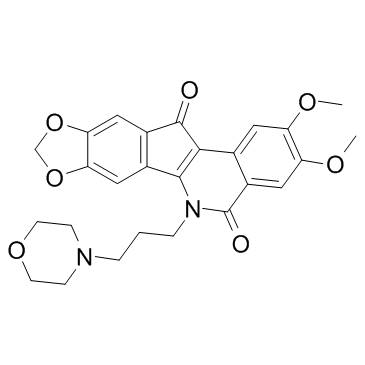
-
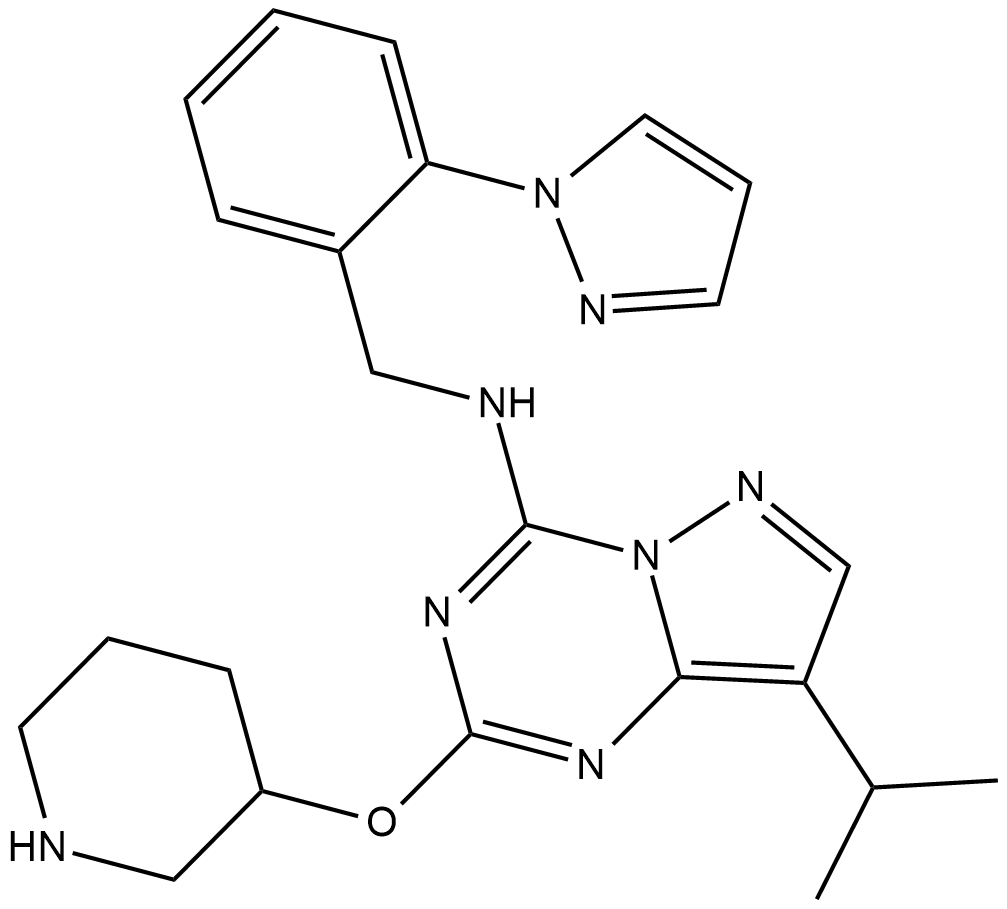
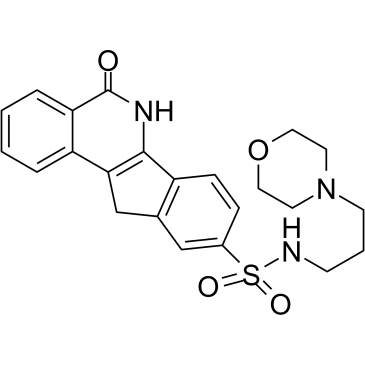
GC38385
INO-1001
-
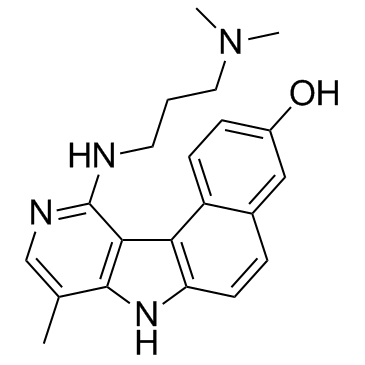
GC33165
Intoplicine
Intoplicine (RP 60475), ein Antitumorderivat aus der 7H-Benzo[e]pyrido[4,3-b]indol-Reihe, ist ein DNA-Topoisomerase-I- und -II-Inhibitor. Intoplicine bindet stark DNA (KA = 2 x 105 /M) und erhÖht dadurch die LÄnge der linearen DNA.
-
GC63354
Intoplicine dimesylate
RP 60475 dimesylate
Intoplicin (RP 60475) Dimesylat, ein Antitumorderivat aus der 7H-Benzo[e]pyrido[4,3-b]indol-Reihe, ist ein DNA-Topoisomerase-I- und -II-Inhibitor. Intoplicindimesylat bindet stark an DNA (KA = 2 x 105 /M) und erhÖht dadurch die LÄnge der linearen DNA.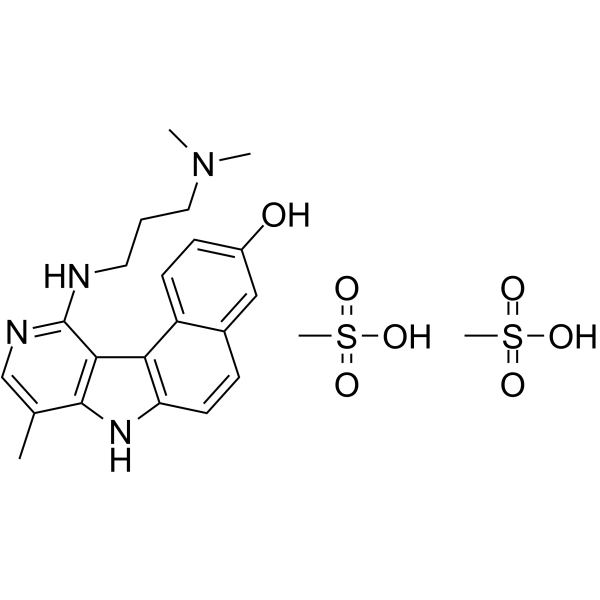
-
GC63023
Ipivivint
Ipivivint (Verbindung 38) ist ein potenter Inhibitor der CDC-Ähnlichen Kinase (CLK) mit EC50-Werten von 1 nM, 7 nM fÜr CLK2 bzw. CLK3. Ipivivint hemmt den Wnt-Signalweg (EC50=13 nM).
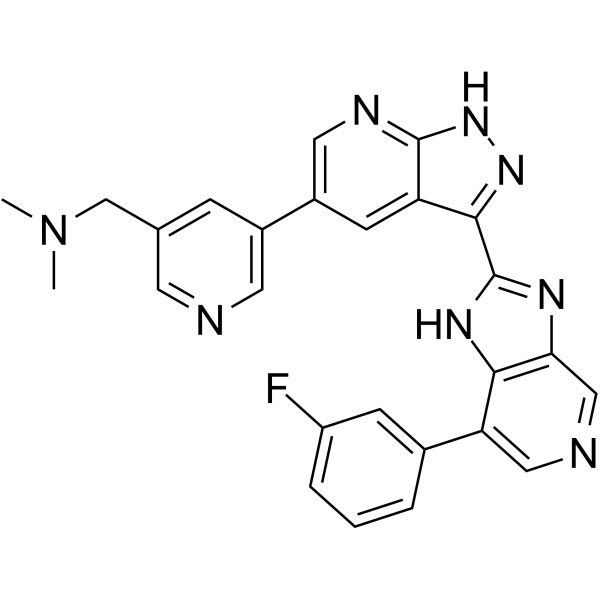
-
GC52175
IQA
CGP 029482
-
GC63513
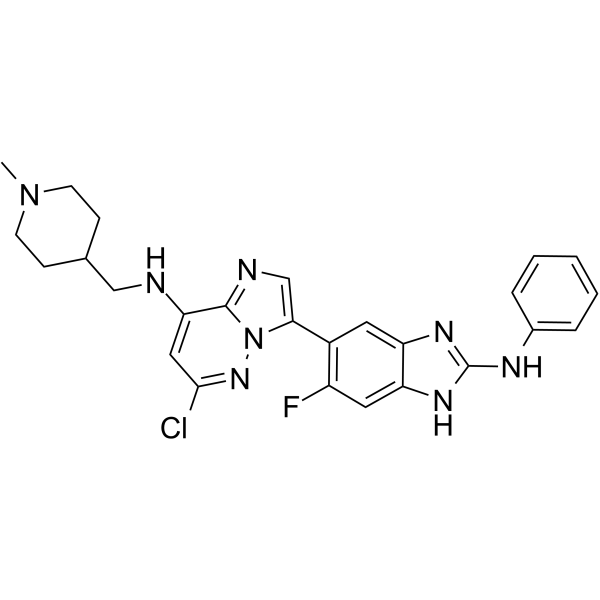
IRE1α kinase-IN-1
IRE1α Kinase-IN-1 ist ein hochselektives IRE1α (ERN1)-Inhibitor, mit einem IC50 von 77 nM.
-
GC64232
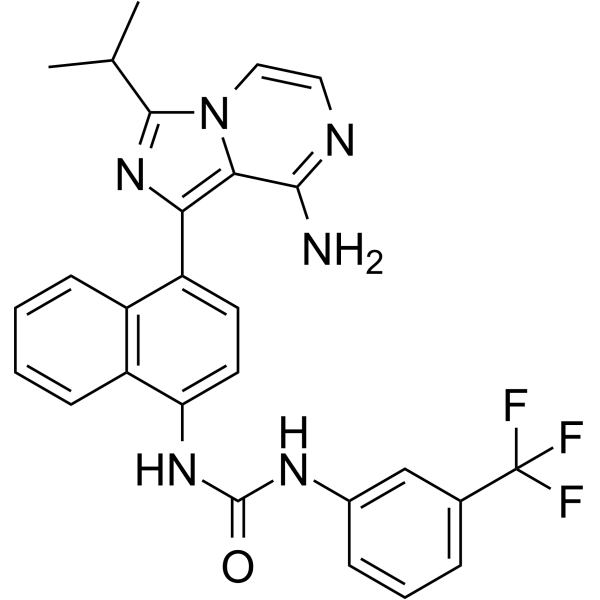
IRE1α kinase-IN-2
IRE1α Kinase-IN-2 ist ein starkes IRE1α Kinase-Inhibitor mit einem EC50-Wert von 0,82 μM. IRE1α Kinase-IN-2 hemmt IRE1α Kinase-Autophosphorylierung (IC50=3,12 μM). IRE1α Kinase-IN-2 hemmt das XBP1-mRNA-Spleißen in den WT-Zelllinien.
-
GC11473
Irinotecan
Topoisomerase-I-Inhibitor
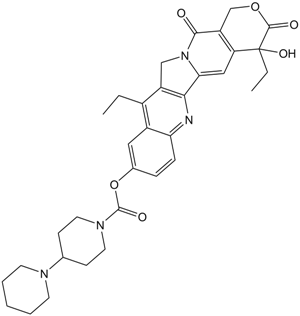
-
GC11048
Irinotecan HCl Trihydrate
Irinotecan-HCl-Trihydrat ((+)-Irinotecan-HCl-Trihydrat) ist ein Topoisomerase-I-Inhibitor mit AntitumoraktivitÄt.
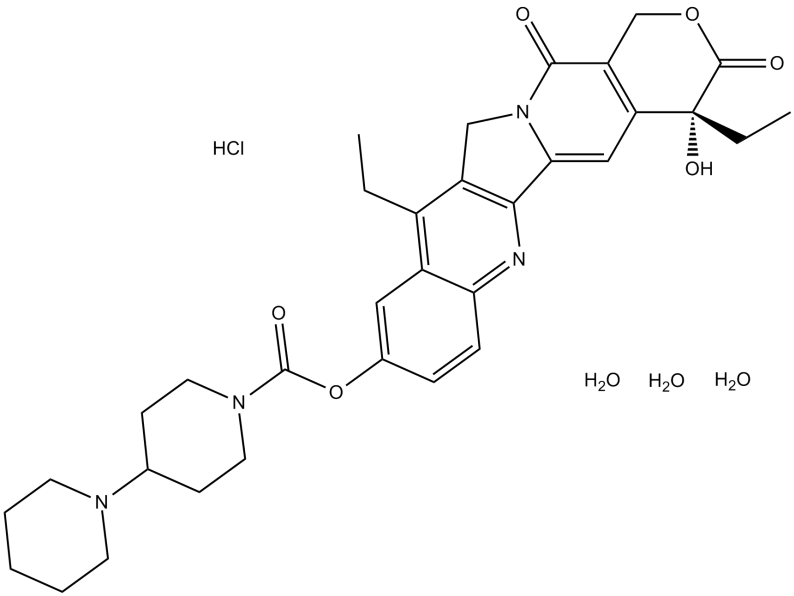
-
GC36329
Irinotecan hydrochloride
Irinotecan-Hydrochlorid ((+)-Irinotecan-Hydrochlorid) ist ein Topoisomerase-I-Inhibitor, der hauptsÄchlich zur Behandlung von Dickdarm- und Mastdarmkrebs eingesetzt wird.
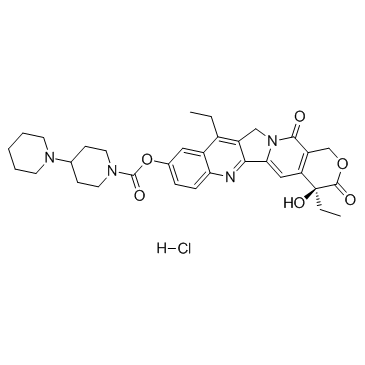
-
GC30554
Isocytosine
Isocytosin ist eine nicht natÜrliche Nukleobase und ein Isomer von Cytosin.
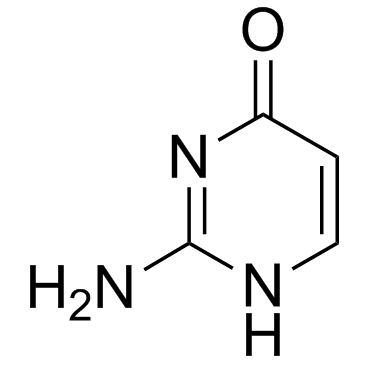
-
GC33662
Isoguanine
Crotonoside, 2-hydroxy-6-Aminopurine, 2-Hydroxyadenine, NSC 241501, 2-Oxoadenine
Isoguanin ist eine Purinbase, die ein Isomer von Guanin ist.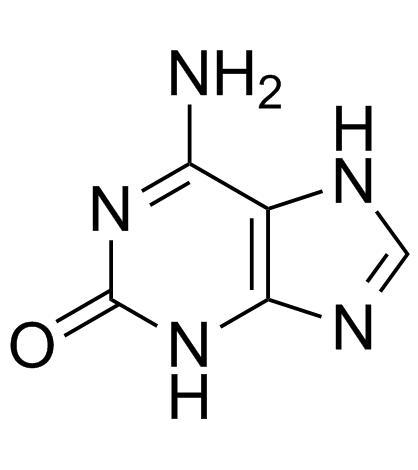
-
GC39140
Isopimpinellin
NSC 217988, NSC 401288
Isopimpinellin, eine oral wirksame Verbindung, die aus den Wurzeln des Pimpinella-Steinbrechs isoliert wurde.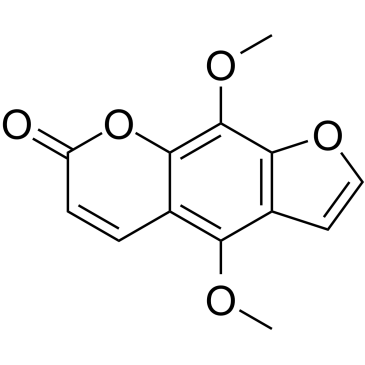
-
GC10834
Isoxanthopterin
NSC 118090,NSC 614991
interferes with RNA and DNA synthesis
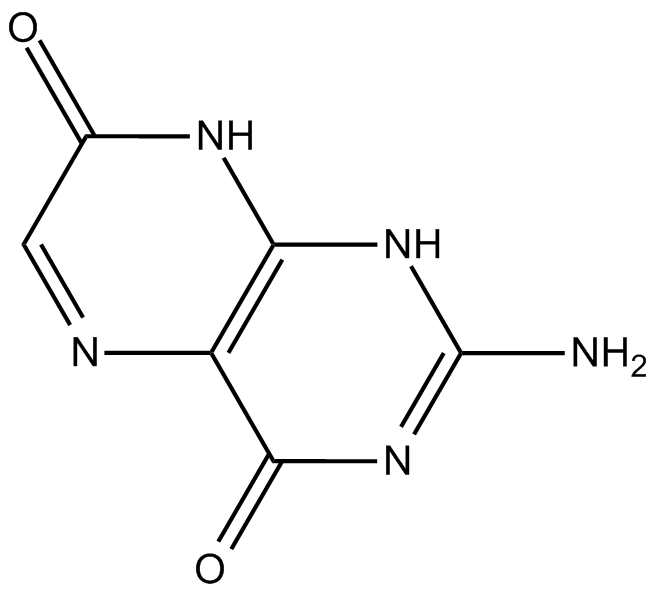
-
GC14597
ITSA-1 (ITSA1)
ITSA-1 (ITSA1) ist ein Aktivator der Histon-Deacetylase (HDAC) und wirkt dem durch Trichostatin A (TSA) induzierten Zellzyklusarrest, der Histonacetylierung und der Transkriptionsaktivierung entgegen.
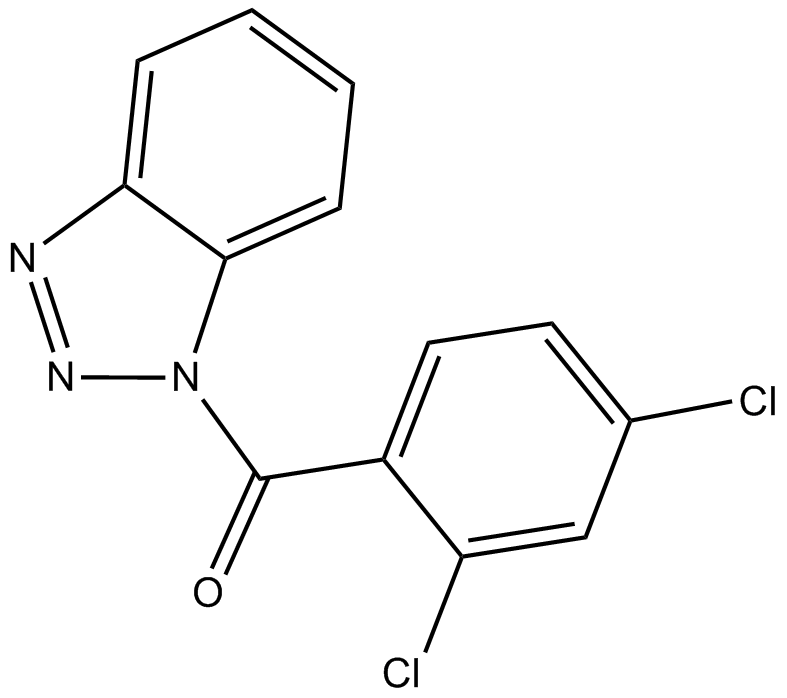
-
GC14797
IU1
Usp14 inhibitor
A deubiquitinating enzyme inhibitor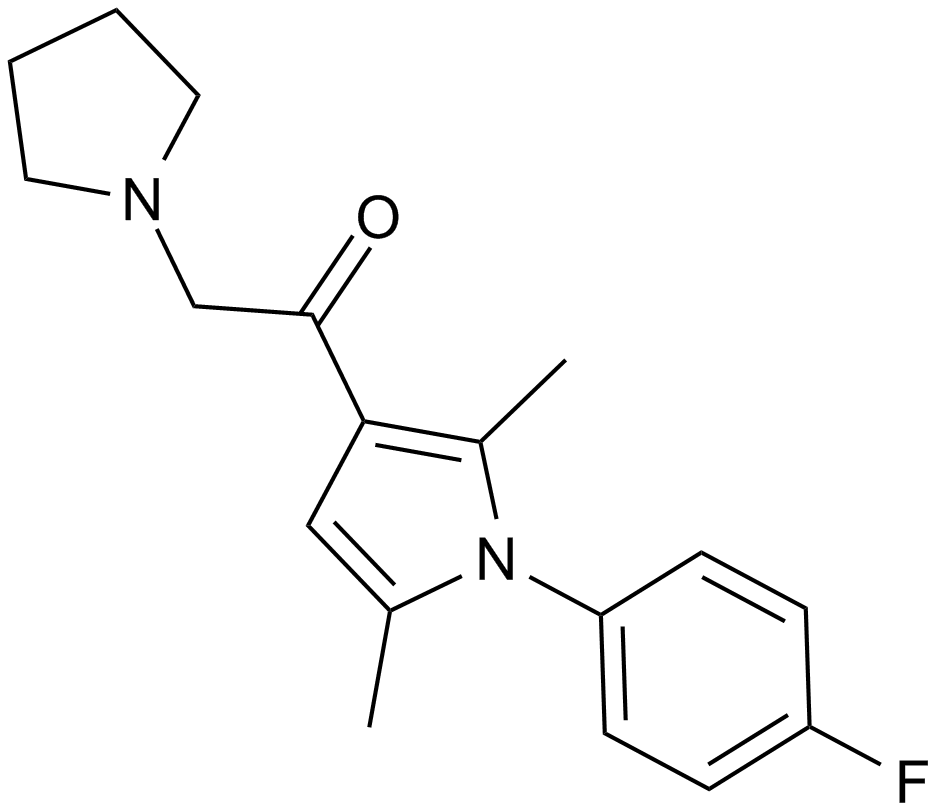
-
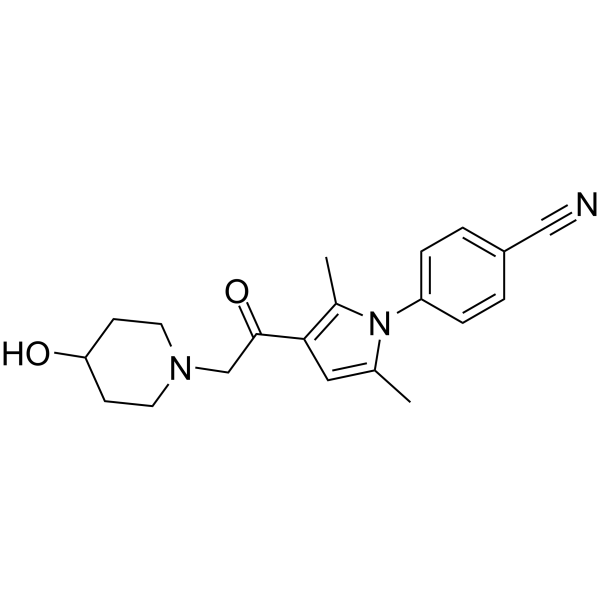
GC63027
IU1-248
IU1-248, ein Derivat von IU1, ist ein potenter und selektiver USP14-Inhibitor mit einem IC50-Wert von 0,83 μM.
-
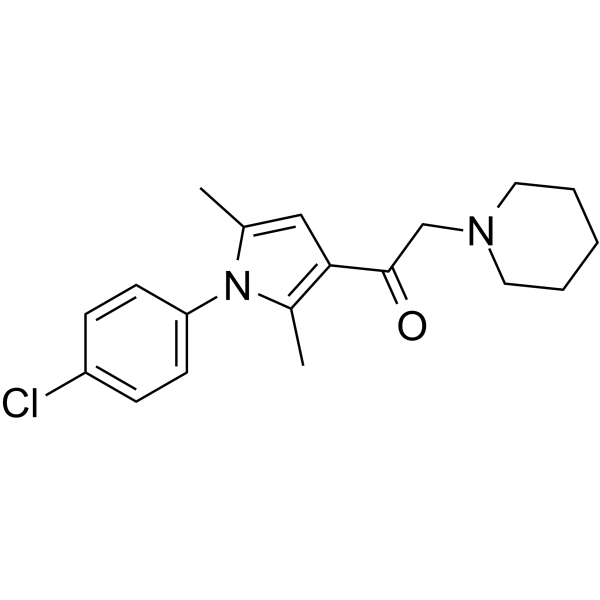
GC64956
IU1-47
IU1-47 ist ein potenter und spezifischer USP14-Inhibitor mit einem IC50 von 0,6 μM.
-
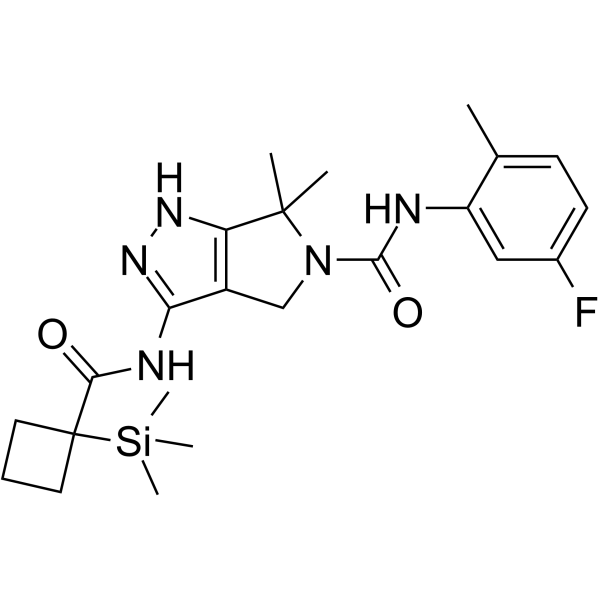
GC63460
IV-361
IV-361 ist ein oral aktiver und selektiver CDK7-Inhibitor (Ki≤50 nM). IV-361 hat Anti-Krebs-AktivitÄt (US20190256531A1).
-
GC16454
IWP-2
IWP 2
IWP-2 is a inhibitor of the Wnt signaling pathway, with an IC50 value of 27 nM. IWP-2 is also an ATP-competitive inhibitor of CK1δ, with an IC50 value of 40 nM.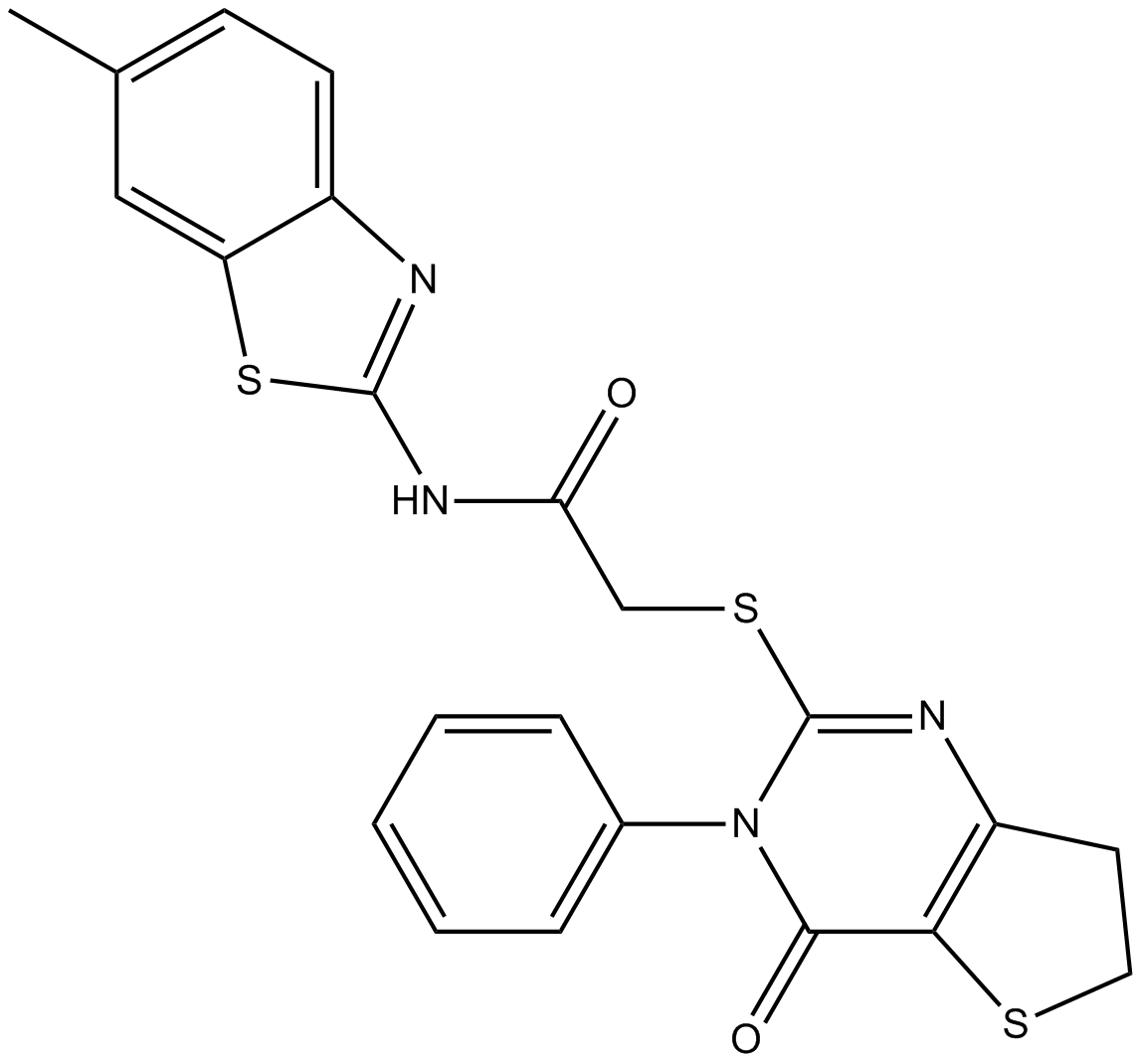
-
GC63785
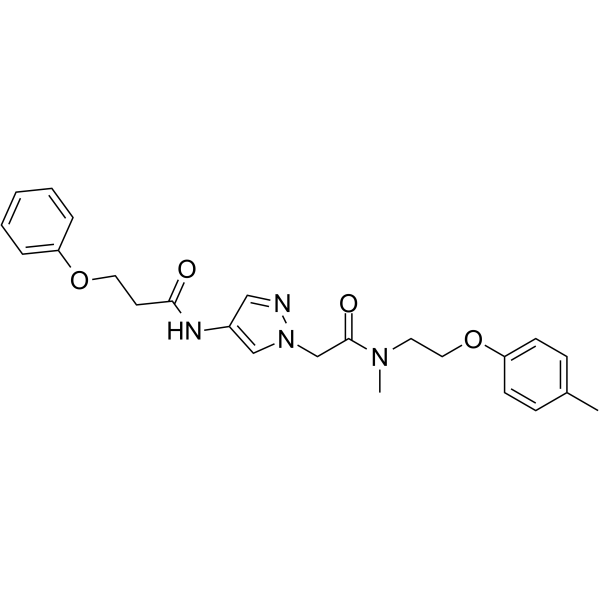
IXA4
IXA4 ist ein hochselektiver, ungiftiger IRE1/XBP1s-Aktivator.
-
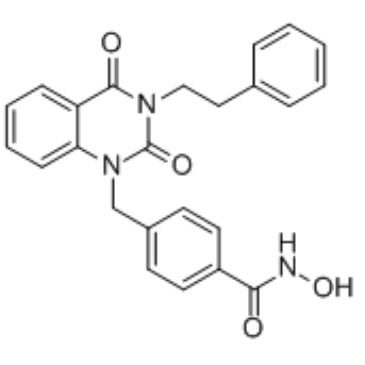
GC34629
J22352
J22352 ist ein PROTAC (proteolysis-targeting chimeras)-Ähnlicher und hochselektiver HDAC6-Inhibitor mit einem IC50-Wert von 4,7 nM. J22352 fÖrdert den Abbau von HDAC6 und induziert krebshemmende Wirkungen, indem es die Autophagie hemmt und die Antitumor-Immunantwort bei Glioblastom-Krebs hervorruft, und fÜhrt zur Wiederherstellung der AntitumoraktivitÄt des Wirts, indem es die immunsuppressive AktivitÄt von PD-L1 reduziert.
-
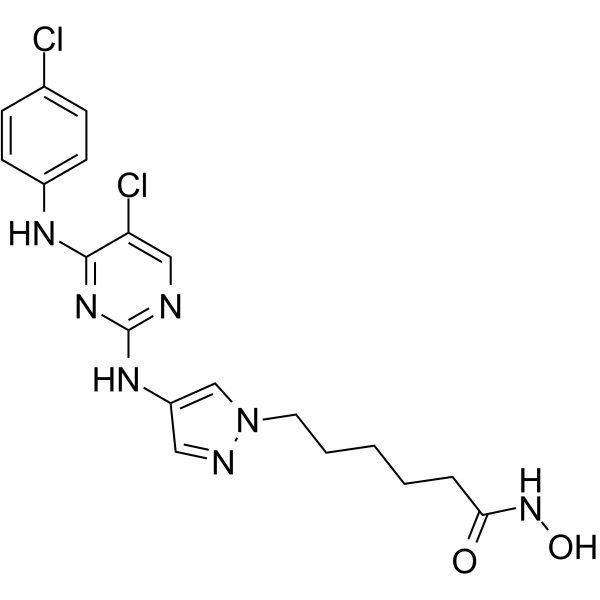
GC64959
JAK/HDAC-IN-1
JAK/HDAC-IN-1 ist ein potenter JAK2/HDAC-Doppelinhibitor, der in mehreren hÄmatologischen Zelllinien antiproliferative und proapoptotische AktivitÄten zeigt. JAK/HDAC-IN-1 zeigt IC50-Werte von 4 und 2 nM fÜr JAK2 bzw. HDAC.
-
GC36367
JH-RE-06
JH-RE-06, ein potenter REV1-REV7-Schnittstelleninhibitor (IC50=0,78 μM; Kd=0,42 μM), zielt auf REV1 ab, das mit der REV7-Untereinheit von POLζ interagiert. JH-RE-06 unterbricht die mutagene Translesionssynthese (TLS), indem es die Rekrutierung von mutagenem POLζ verhindert. JH-RE-06 verbessert die Chemotherapie.
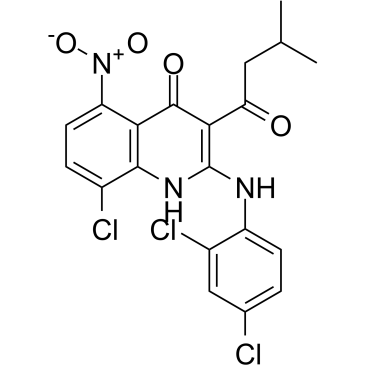
-
GC65471
JH-XI-10-02
JH-XI-10-02 ist ein PROTAC, der durch Liganden für Cereblon und CDK verbunden ist. JH-XI-10-02 ist ein hochpotenter und selektiver PROTAC CDK8-Degrader mit einer IC50 von 159 nM. JH-XI-10-02 verursacht proteasomale Degradation und beeinflusst nicht die mRNA-Level von CDK8. JH-XI-10-02 zeigt keine Wirkung auf CDK19.
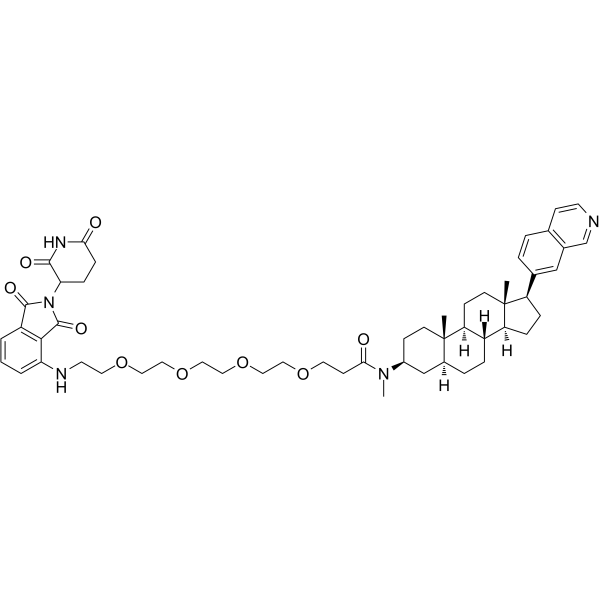
-
GC65577
JH-XVI-178
JH-XVI-178 ist ein hochwirksamer und selektiver Inhibitor von CDK8/19, der eine geringe Clearance und mÄßige orale pharmakokinetische Eigenschaften aufweist.

-
GC12612
JNJ-7706621
JNJ7706621, JNJ 7706621
A dual inhibitor of CDKs and Aurora kinases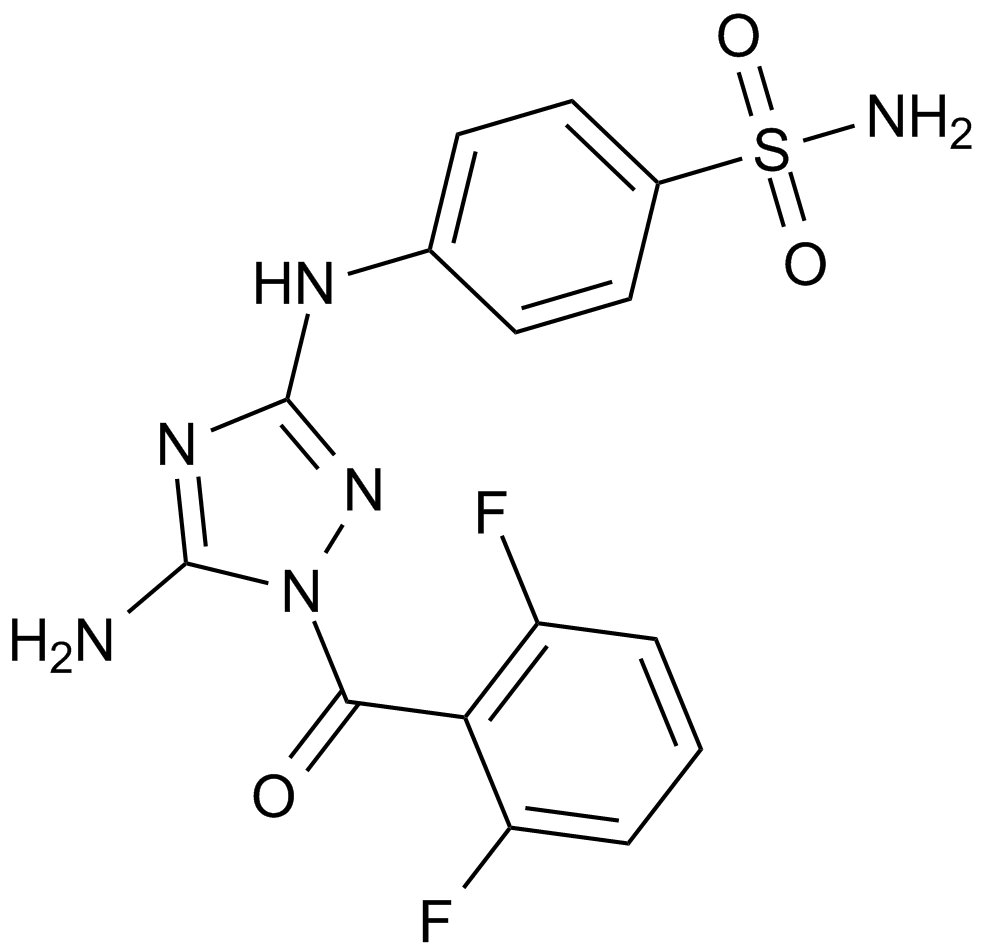
-
GC18734
JS-K
JS-K ist ein NO-Donor, der mit Glutathion reagiert, um NO bei physiologischem pH zu erzeugen. JS-K hemmt die Proliferation, induziert Apoptose und unterbricht den Zellzyklus von akuten lymphoblastischen Jurkat-T-LeukÄmiezellen.
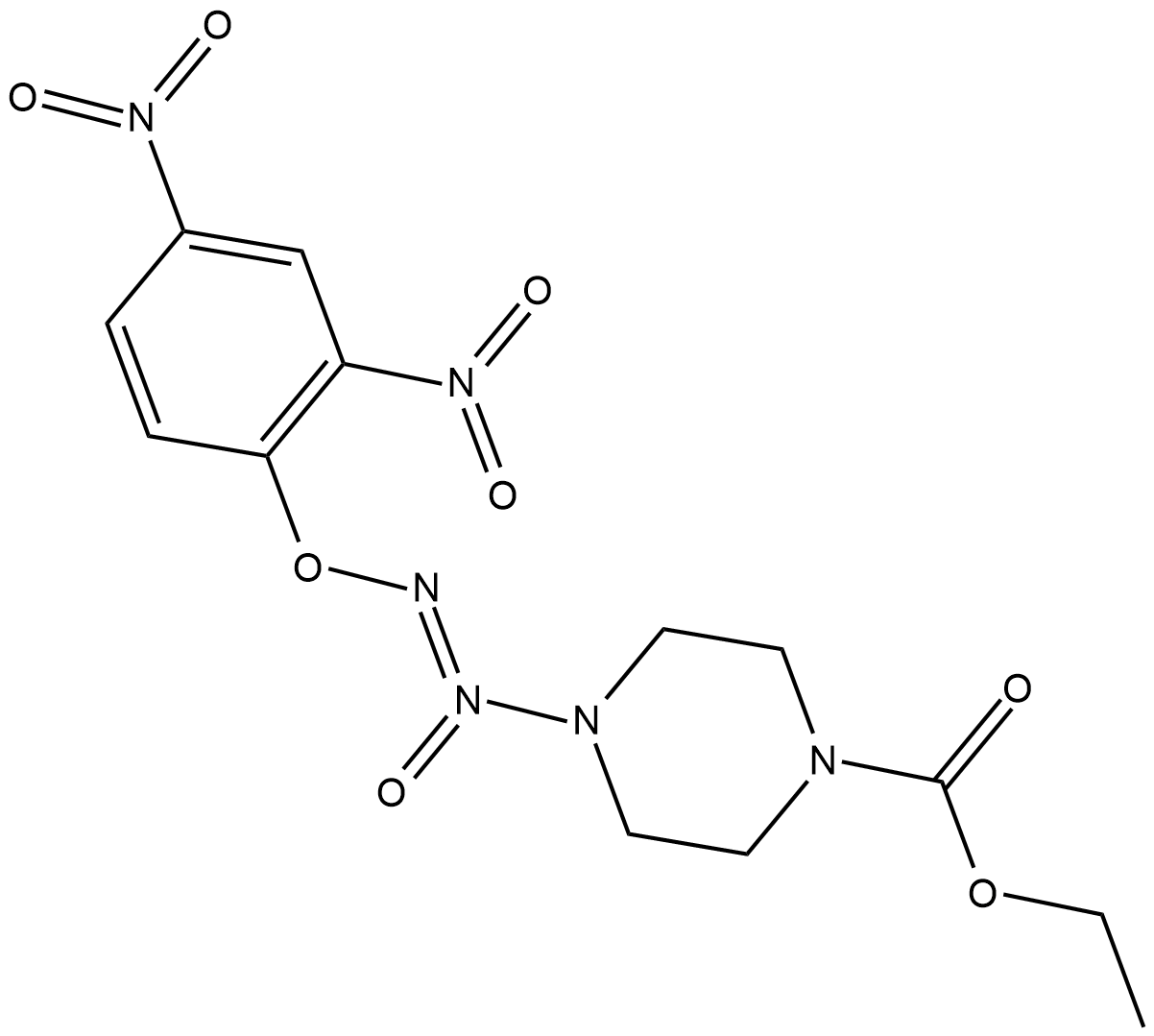
-
GC34204
JSH-150
JSH-150 ist ein hochselektiver und potenter CDK9-Inhibitor mit einem IC50 von 1 nM.
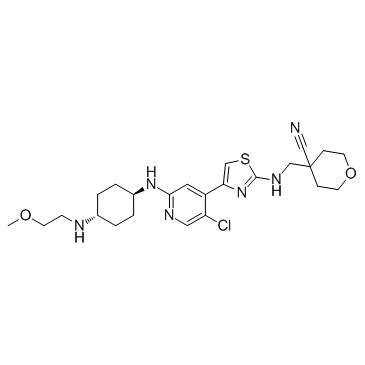
-
GC50117
JTE 607 dihydrochloride
JTE 607 Dihydrochlorid, ein hochselektiver Hemmer der entzÜndlichen Zytokinsynthese, schÜtzt MÄuse vor Endotoxinschock.
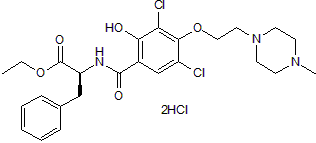
-
GC13978
JW 74
JW 74 antagonisiert die LiCl-induzierte Aktivierung des kanonischen Wnt-Signalwegs mit einem IC50 von 420 nM.
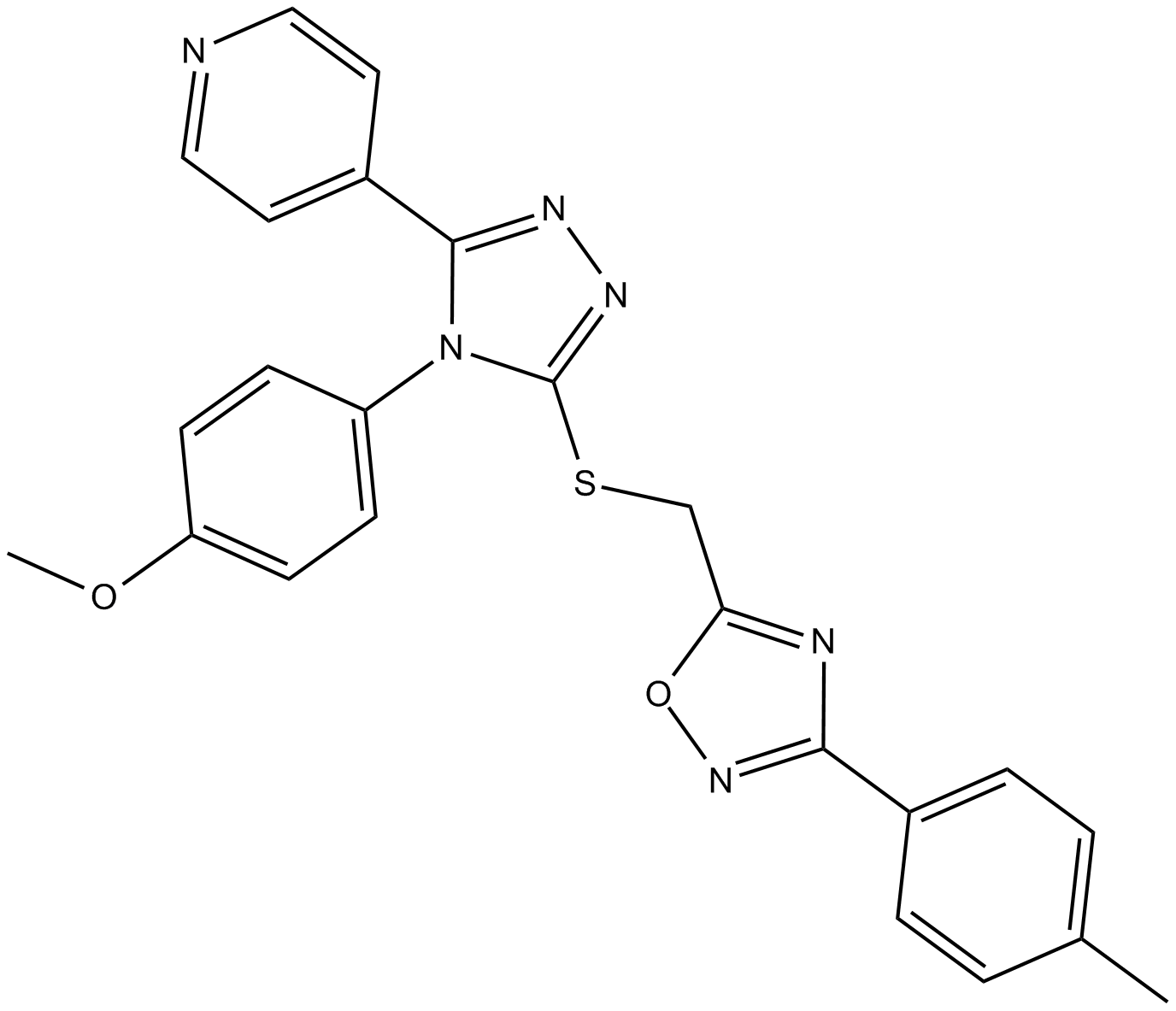
-
GC34195
K-756
K-756 ist ein direkter und selektiver Tankyrase (TNKS)-Inhibitor, der die ADP-RibosylierungsaktivitÄt von TNKS1 und TNKS2 mit IC50-Werten von 31 bzw. 36 nM hemmt.
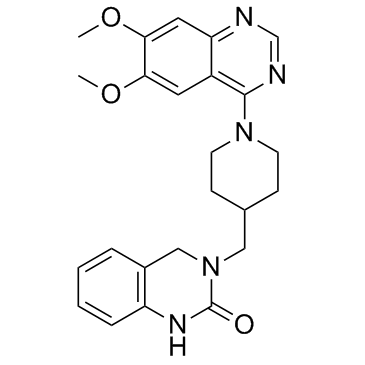
-
GC41384
K-TMZ
K-TMZ is a DNA alkylating agent.
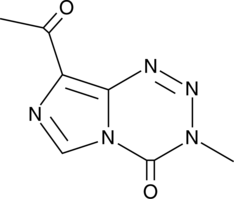
-
GC14230
K03861
K03861 (K03861) ist ein Typ-II-CDK2-Inhibitor mit einem Kd von 8,2 nM. K03861 (K03861) hemmt die CDK2-AktivitÄt, indem es mit der Bindung von aktivierenden Cyclinen konkurriert.
-
GC47521
K22
An antiviral agent
-
GC34144
Karenitecin (Cositecan)
Karenitecin (Cositecan) (Cositecan) ist ein Topoisomerase-I-Inhibitor mit starker Anti-Krebs-AktivitÄt.
-
GC62392
KB-0742 dihydrochloride
KB-0742 Dihydrochlorid ist ein potenter, selektiver und oral aktiver CDK9-Inhibitor mit einem IC50 von 6 nM fÜr CDK9/Cyclin T1. KB-0742-Dihydrochlorid ist selektiv fÜr CDK9/Cyclin T1 mit einer >50-fachen SelektivitÄt gegenÜber anderen CDK-Kinasen. KB-0742-Dihydrochlorid hat eine starke AntitumoraktivitÄt.
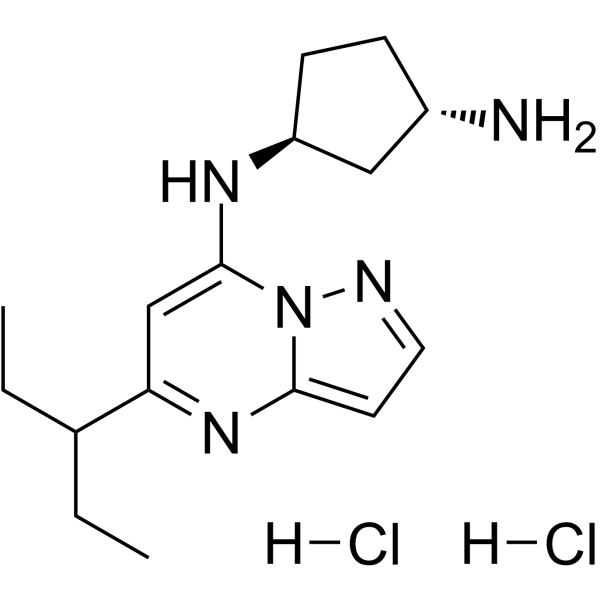
-
GC14182
Kenpaullone
9Bromopaullone, NSC 664704
Kenpaullon ist ein potenter Inhibitor von CDK1/Cyclin B und GSK-3β mit IC50-Werten von 0,4 μM und 23 nM und hemmt auch CDK2/Cyclin A, CDK2/Cyclin E und CDK5/p25 mit IC50-Werten von 0,68 μM, 7,5 μM, 0,85 μM. Kenpaullon, ein niedermolekularer Inhibitor von KLF4, reduziert die Selbsterneuerung von Brustkrebsstammzellen und die ZellmotilitÄt in vitro.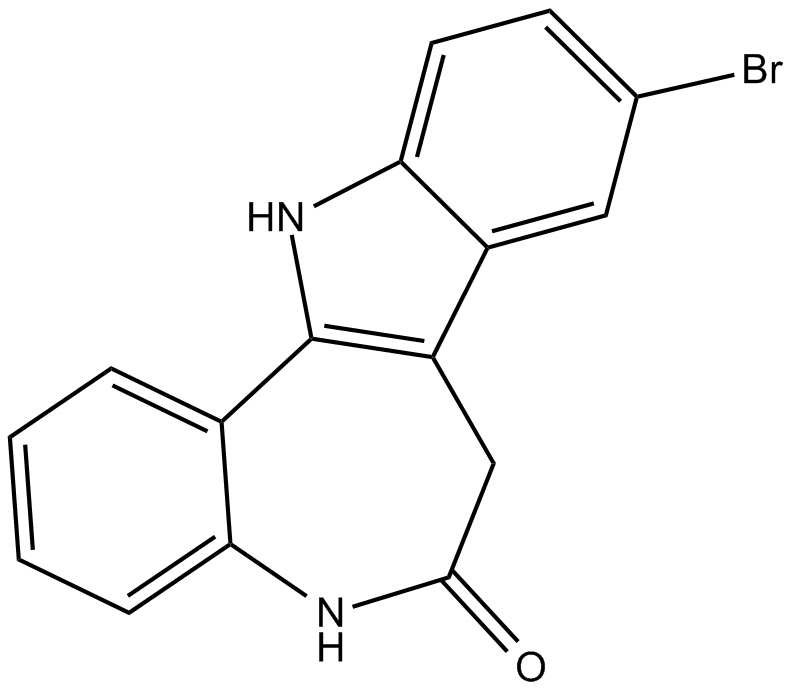
-
GC11774
KH CB19
KH CB19 ist ein potenter CLK (cdc2-like kinase)-Inhibitor (CLK1 IC50 \u003d 19,7 nM; CLK3 IC50 \u003d 530 nM).
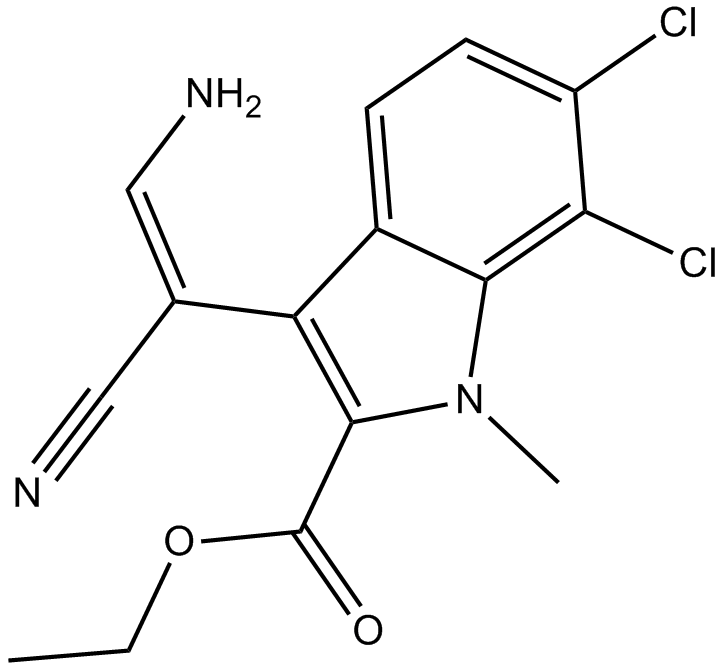
-
GC63943
KH-CB20
KH-CB20, eine E/Z-Mischung, ist ein potenter und selektiver Inhibitor von CLK1 und der eng verwandten Isoform CLK4 mit einem IC50-Wert von 16,5 nM fÜr CLK1.
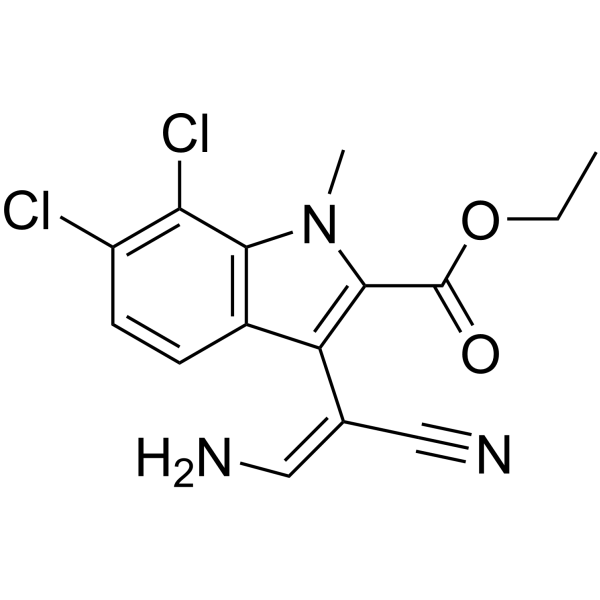
-
GC64238
KIRA-7
KIRA-7, eine Imidazopyrazin-Verbindung, bindet die IRE1α-Kinase (IC50 von 110 nM), um ihre RNase-AktivitÄt allosterisch zu hemmen.
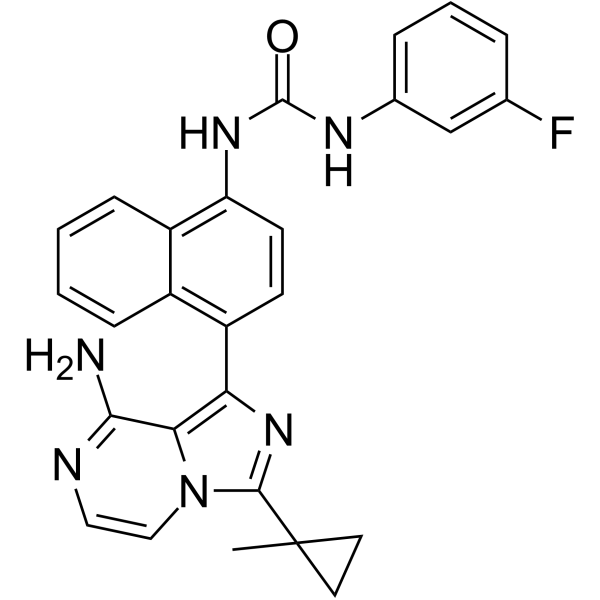
-
GC19212
KIRA6
IRE1 Inhibitor IV, IRE1α Kinase Inhibiting RNase Attenuator 6
KIRA6 ist ein fortschrittlicher niedermolekularer IRE1α-RNase-Kinase-Inhibitor mit einem IC50 von 0,6 μM.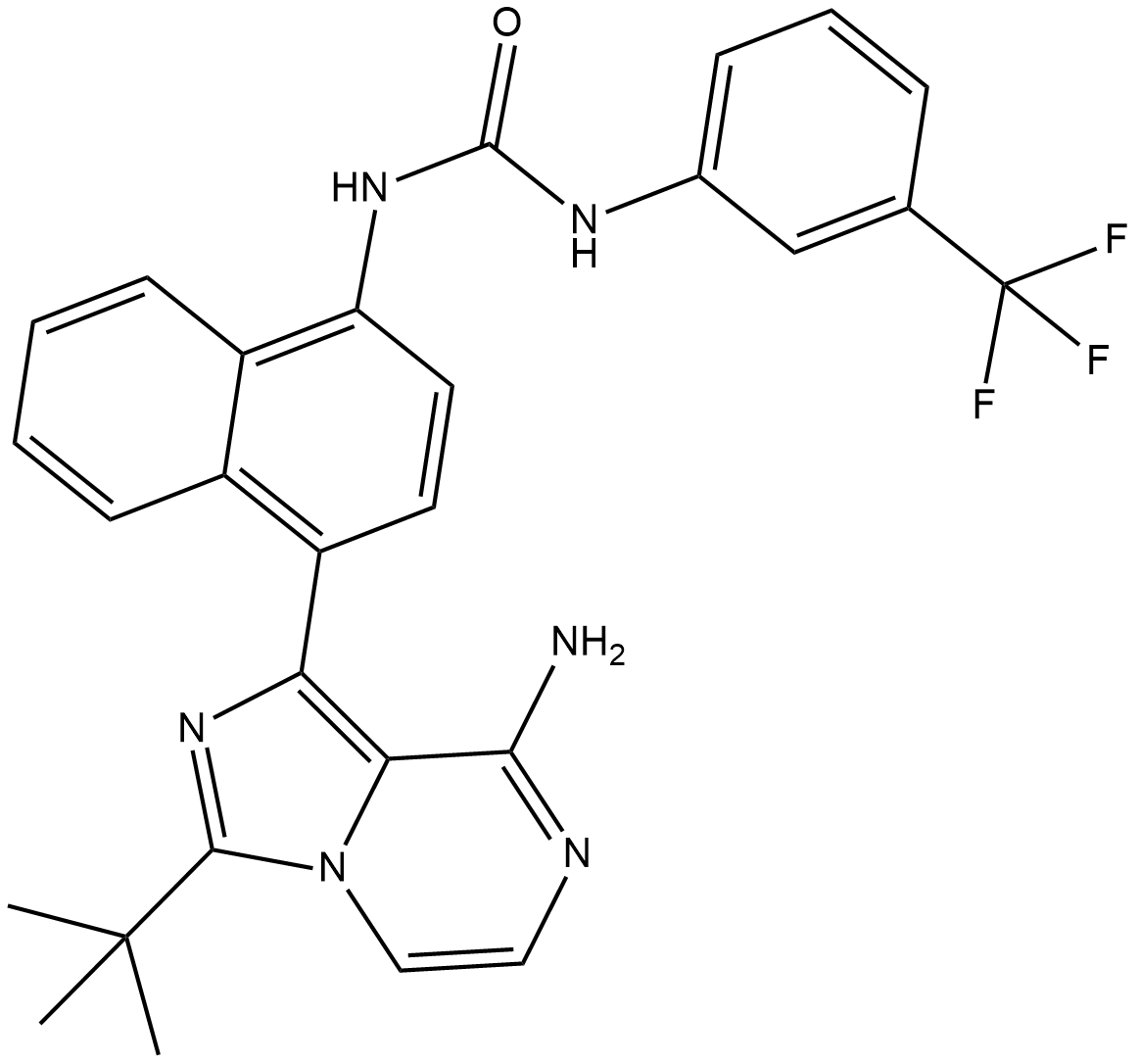
-
GC31879
Kira8
AMG-18
Kira8 (AMG-18) ist ein monoselektiver IRE1α-Inhibitor, der die IRE1α-RNase-AktivitÄt mit einem IC50 von 5,9 nM allosterisch abschwÄcht.
-
GC65907
KSQ-4279
USP1-IN-1
KSQ-4279 (USP1-IN-1, Formel I) ist ein USP1- und PARP-Inhibitor (aus dem Patent WO2021163530 entnommen).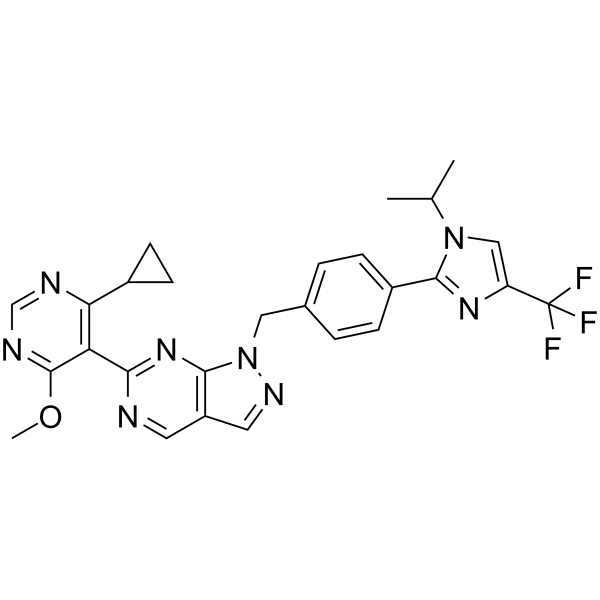
-
GC25552
KT-531
KT-531 (KT531) is a potent, selective HDAC6 inhibitor with IC50 of 8.5 nM, displays 39-fold selectivity over other HDAC isoforms.

-
GC33404
KU 59403
KU 59403 ist ein potenter ATM-Inhibitor mit IC50-Werten von 3 nM, 9,1 μM und 10 μM fÜr ATM, DNA-PK bzw. PI3K.
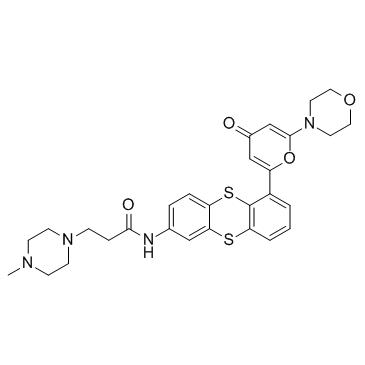
-
GC14346
KU-0060648
KU-0060648 ist ein dualer Inhibitor von PI3K und DNA-PK mit IC50-Werten von 4 nM, 0,5 nM, 0,1 nM, 0,594 nM und 8,6 nM fÜr PI3Kα, PI3Kβ, PI3Kγ, PI3Kδ bzw. DNA-PK.
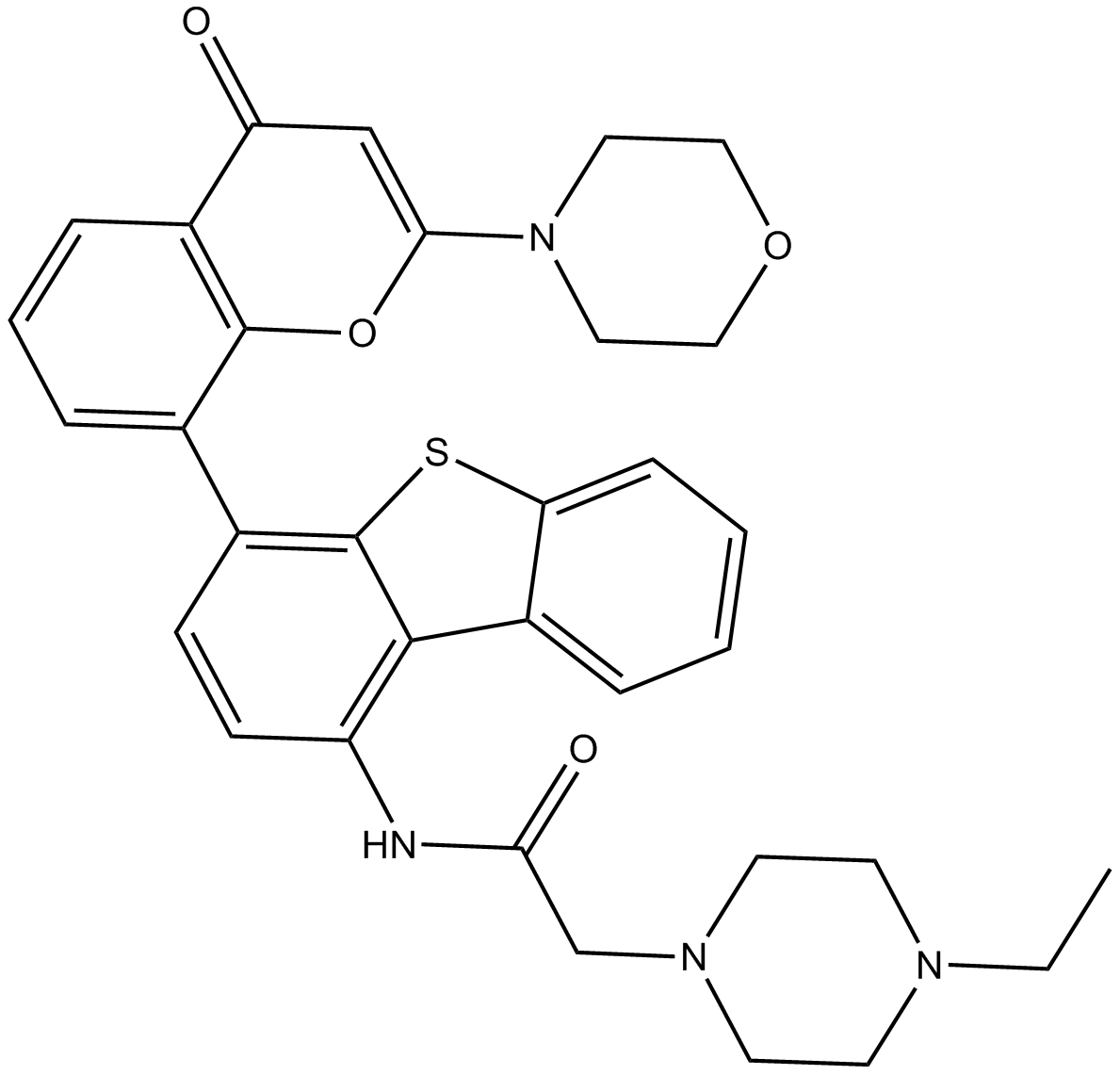
-
GC12054
KU-60019
KU60019;KU 60019
KU-60019 ist ein verbesserter ATM-Kinase-spezifischer Inhibitor mit IC50 von 6,3 nM.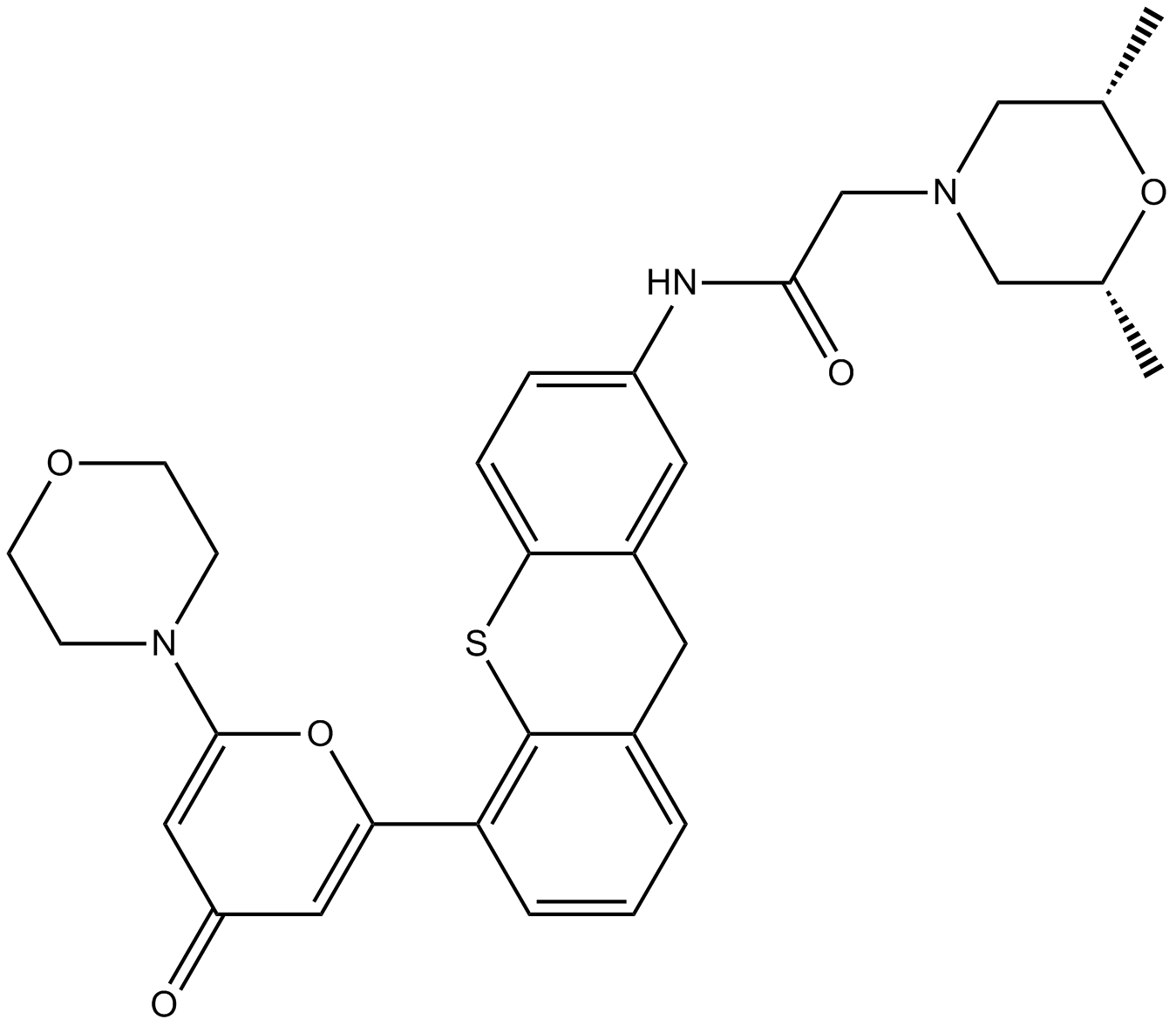
-
GC50532
KuWal151
Potent and selective CLK inhibitor
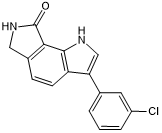
-
GC15743
L-755,507
L-755,507 ist ein potenter, selektiver Agonist von β3-AR mit einem IC50 von 35 nM.
-
GC66622
L-Guanosine
L-Guanosin ist die L-Konfiguration von Guanosin. Guanosin ist ein Purinnukleosid mit Anti-Herpesvirus-Aktivität.
-
GC49381
L-Leucine-d10
Leu-d10, Pentanoic Acid-d10
An internal standard for the quantification of L-leucine
-
GC49368
L-Methionine-d3
(S)-Methionine-d3
L-Methionin-d3 ist das mit Deuterium markierte L-Methionin.
-
GC49384
L-Proline-d3
L-(–)-Proline-d3, (S)-(–)-Proline-d3
An internal standard for the quantification of L-proline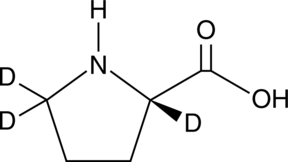
-
GC12131
L189
L189 ist ein DNA-Ligase-Inhibitor. L189 hat eine Hemmwirkung auf die DNA-Ligase I, III und IV mit IC50-Werten von 5 μM, 9 μM bzw. 5 μM. L189 hat keine ZytotoxizitÄt und erhÖht individuell den Zelltod. L189 kann fÜr die Krebsforschung verwendet werden.
-
GC18186
L67
DNA Ligase Inhibitor
L67 (DNA-Ligase-Inhibitor) ist ein kompetitiver DNA-Ligase-Inhibitor, der die DNA-Ligasen I/III wirksam hemmt (beide IC50 sind 10 μM). L67 (DNA-Ligase-Inhibitor) kann nukleÄre DNA-SchÄden verursachen, indem es die Menge an mitochondrialer DNA reduziert und die Menge an mitochondrial erzeugtem ROS erhÖht. L67 (DNA Ligase Inhibitor) aktiviert auch den Caspase 1-abhÄngigen Apoptoseweg in Krebszellen, kann in der Krebsforschung eingesetzt werden.
-
GC67785
L82

-
GC68436
L82-G17

-
GC39632
Laflunimus
Laflunimus (HR325) ist ein Immunsuppressivum und ein Analogon des Leflunomid-aktiven Metaboliten A77 1726.
-
GC62460
LCAHA
LCA hydroxyamide
LCAHA (LCA-Hydroxyamid) ist ein Deubiquitinase-USP2a-Inhibitor mit IC50-Werten von 9,7 μM und 3,7 μM im Ub-AMC-Assay bzw. Di-Ub-Assay. LCAHA destabilisiert Cyclin D1 und induziert den G0/G1-Arrest durch Hemmung der Deubiquitinase USP2a.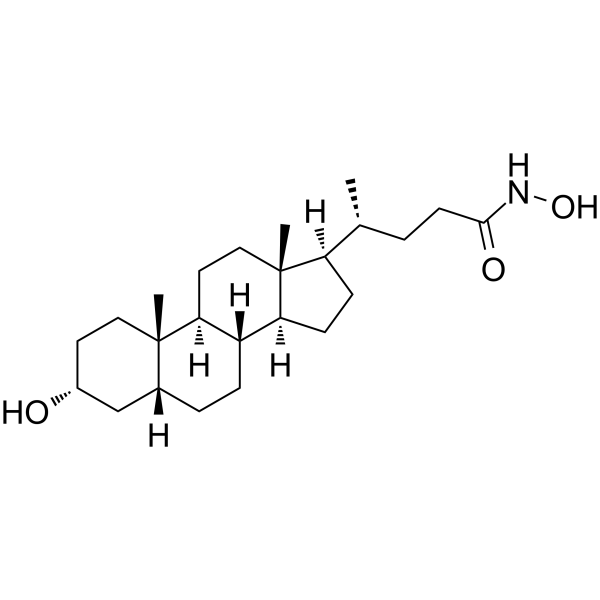
-
GC17067
LDC000067
LDC067
LDC000067 ist ein hochspezifischer CDK9-Inhibitor mit einem IC50-Wert von 44±10 nM in vitro.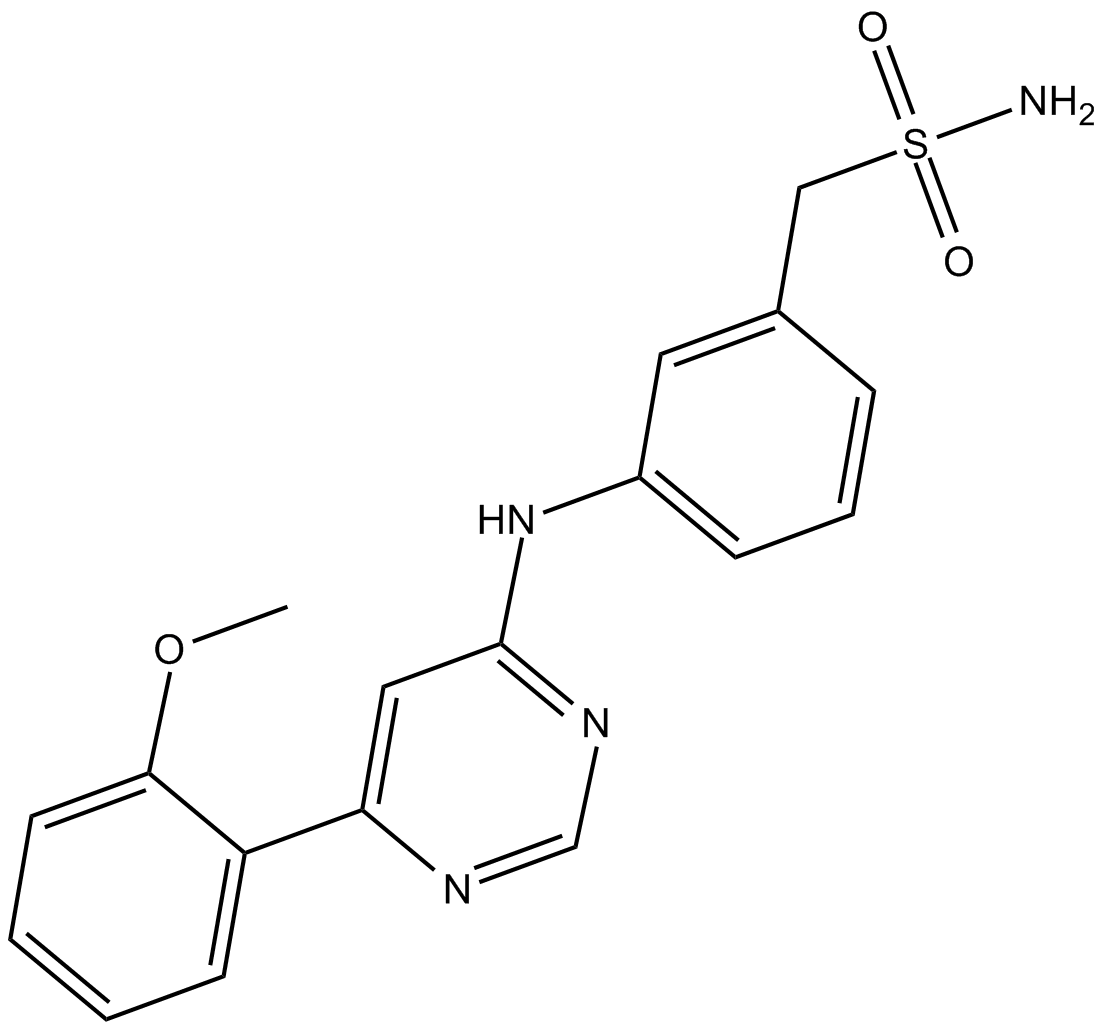
-
GC19219
LDC4297
LDC4297 ist ein potenter und selektiver CDK7-Inhibitor mit einem IC50 von 0,13 nM.