DNA Damage/DNA Repair
- MTH1(4)
- PARP(58)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(94)
- Nucleoside Antimetabolite/Analogue(136)
- Telomerase(14)
- Topoisomerase(133)
- tankyrase(5)
- Antifolate(32)
- CDK(233)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(62)
- DNA Alkylator/Crosslinker(64)
- DNA/RNA Synthesis(388)
- Eukaryotic Initiation Factor (eIF)(22)
- IRE1(22)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(53)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
GC44348
N-desmethyl Bendamustine
nor-Bendamustine
N-desmethyl Bendamustine is an active metabolite of the DNA alkylating agent bendamustine.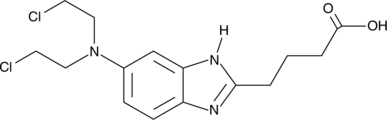
-
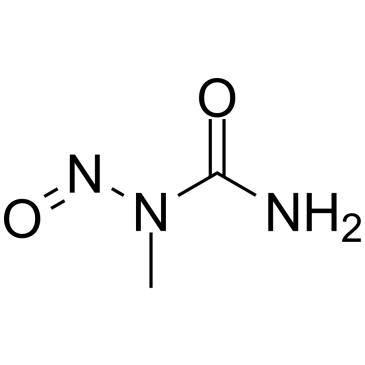
GC39498
N-Nitroso-N-methylurea
N-Nitroso-N-Methylharnstoff (NMU;MNU;NMH) ist ein starkes Karzinogen, Mutagen und Teratogen. N-Nitroso-N-Methylharnstoff ist ein direkt wirkendes Alkylierungsmittel, das mit DNA interagiert. N-Nitroso-N-Methylharnstoff zielt auf mehrere tierische Organe ab, um verschiedene Krebsarten und/oder degenerative Erkrankungen zu verursachen. N-Nitroso-N-methylharnstoff ist auch ein VorlÄufer bei der Synthese von Diazomethan.
-
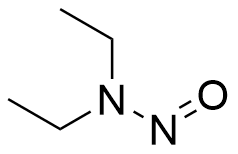
GC19566
N-Nitrosodiethylamine (950mg/mL)
N-Nitrosodiethylamin (Diethylnitrosamin) ist ein potentes hepatokarzinogenes Dialkylnitrosoamin. N-Nitrosodiethylamin ist hauptsächlich in Tabakrauch, Wasser, Cheddar-Käse, gepökelten, frittierten Gerichten und vielen alkoholischen Getränken enthalten. N-Nitrosodiethylamin ist verantwortlich für die Veränderungen in den Kernenzymen, die mit der DNA-Reparatur/Replikation verbunden sind. N-Nitrosodiethylamin führt bei allen Tierarten zu verschiedenen Tumoren. Die Hauptzielorgane sind Nasenhöhle, Luftröhre, Lunge, Speiseröhre und Leber.
-
GC69577
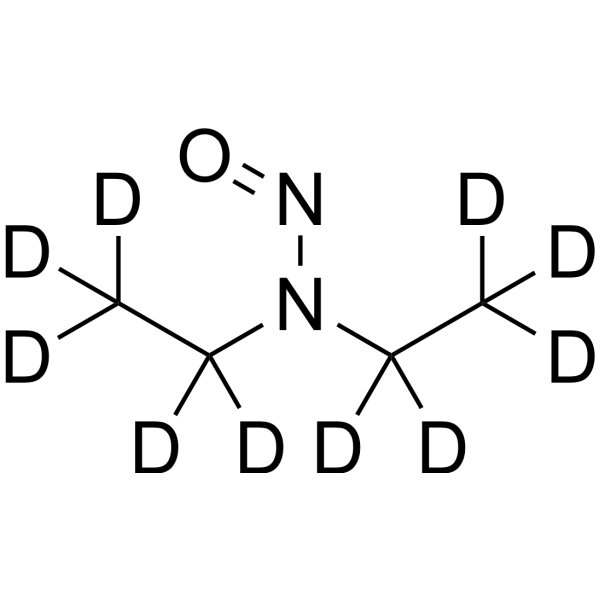
N-Nitrosodiethylamine-d10
Diethylnitrosamine-d10
N-Nitrosodiethylamine-d10 ist das Deuterium-Isotop von N-Nitrosodiethylamine. N-Nitrosodiethylamine (Diethylnitrosamin) ist eine wirksame krebserregende Substanz aus der Gruppe der Dialkylnitrosamine. N-Nitrosodiethylamin kommt hauptsächlich in Tabakrauch, Wasser, Cheddar-Käse, eingelegten Lebensmitteln, frittierten Lebensmitteln und vielen alkoholischen Getränken vor. N-Nitrosodiethylamin verursacht Veränderungen bei den nuklearen Enzymen, die mit DNA-Reparatur/-Replikation zusammenhängen. Es kann verschiedene Tumoren bei allen Tieren verursachen und hat als Hauptzielorgane die Nasenhöhle, Luftröhre, Lunge, Speiseröhre und Leber.
-
GC69589
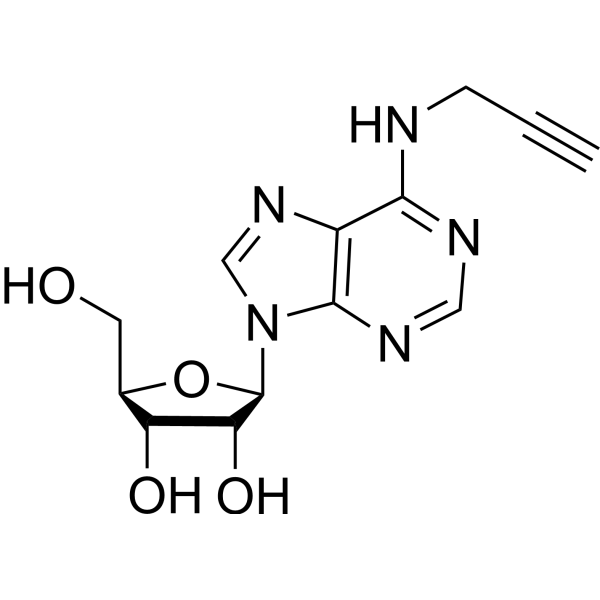
N-Propargyladenosine
N-Propargyladenosine ist ein Analogon von Adenosin-Nukleosiden. Adenosin-Analoga können in der Regel als Vasodilatatoren für glatte Muskeln verwendet werden und haben sich auch als wirksam bei der Hemmung des Fortschreitens von Krebs erwiesen. Zu dieser Gruppe beliebter Produkte gehören Adenosinphosphat, Acadesin, Clofarabin, Fludarabinphosphat und Vidarabin.
-
GC69525
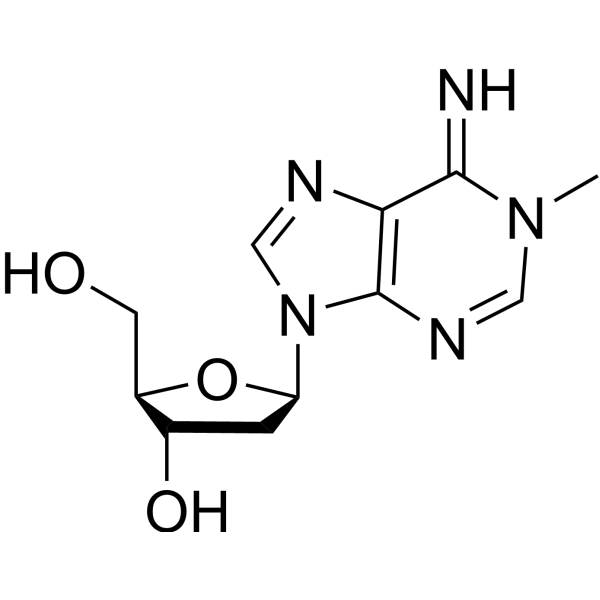
N1-Methyl-2'-deoxyadenosine
N1-Methyl-2'-deoxyadenosine, DNA adduct ist ein Purinnukleosid-Analogon. Purinnukleosid-Analoga haben eine breite antitumorale Aktivität und zielen auf inerte maligne Lymphsystemtumoren ab. Der Mechanismus der Antikrebswirkung in diesem Prozess beruht auf der Hemmung der DNA-Synthese, Induktion von Zellapoptose (Apoptose) usw.
-
GC66075
N1-Methylpseudouridine-5′-triphosphate trisodium
1-Methylpseudouridine-5′-triphosphate trisodium
N1-Methylpseudouridin-5′-triphosphat (1-Methylpseudouridin-5′-triphosphat) Trinatrium ist ein Nukleobasen-modifiziertes Nukleotid, das zur Synthese von mRNA mit reduzierter ImmunogenitÄt und verbesserter StabilitÄt verwendet wird.
-
GC66325
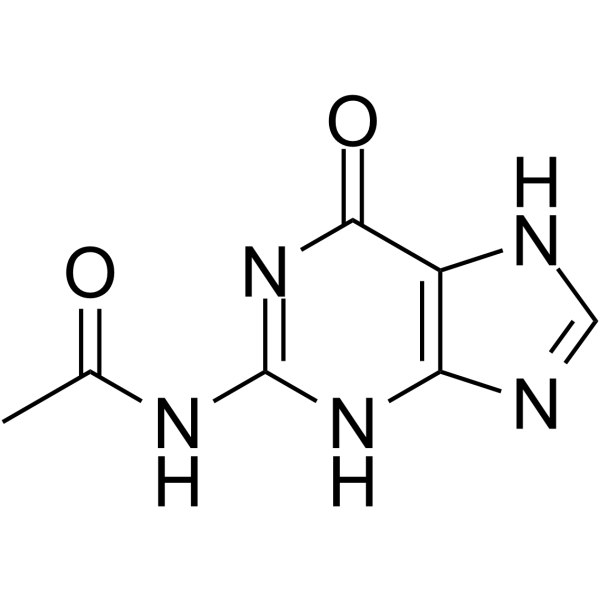
N2-Acetylguanine
N2-Acetylguanin ist ein C2-modifiziertes Guanin. N2-Acetylguanin bindet GR (Guanin-Guanin-Riboschalter) mit einem Kd-Wert von 300 nM. N2-Acetylguanin moduliert die Termination der Transkription. N2-Acetylguanin hat das Potenzial fÜr die Erforschung antimikrobieller Wirkstoffe.
-
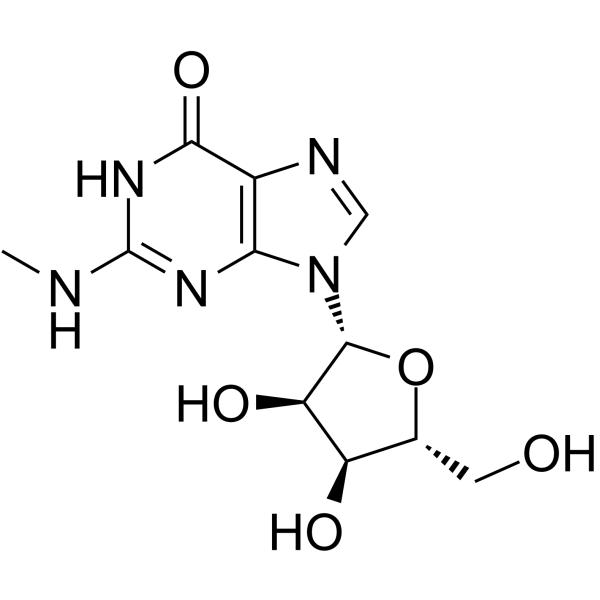
GC65127
N2-Methylguanosine
N2-Methylguanosin ist ein modifiziertes Nukleosid, das an mehreren spezifischen Stellen in vielen tRNAs vorkommt.
-
GC36681
N6,N6-Dimethyladenosine
6-N,N-Dimethyladenosine, 6-DMA, m62A
N6,N6-Dimethyladenosin ist ein modifiziertes Ribonukleosid, das zuvor in rRNA gefunden wurde und auch in Mycobacterium bovis Bacille Calmette-Guérin tRNA vorkommt.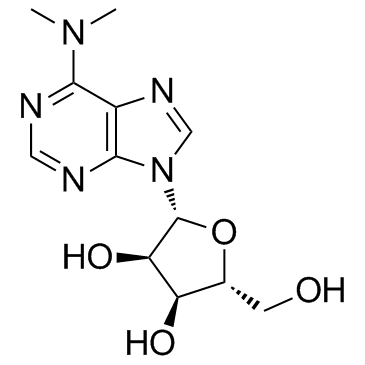
-
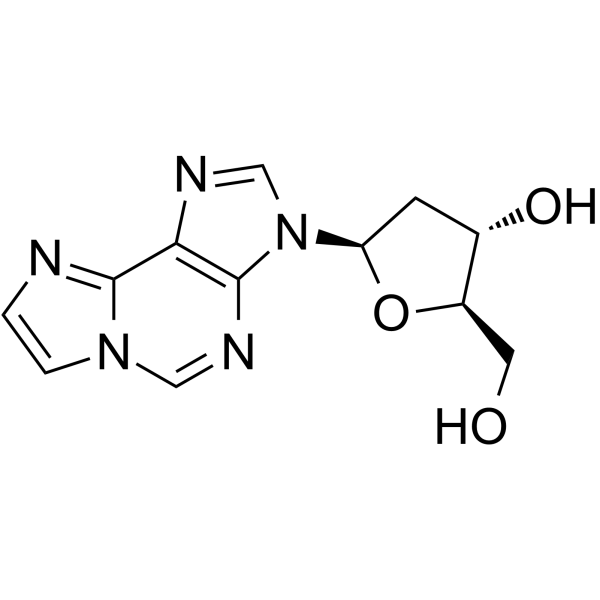
GC65241
N6-Etheno 2'-deoxyadenosine
N6-Etheno &2#39;-Desoxyadenosin ist ein durch reaktive Sauerstoffspezies (ROS)/reaktive Stickstoffspezies (RNS) induziertes DNA-Oxidationsprodukt, das als Biomarker zur Bewertung chronischer Entzündungen und Lipidperoxidation in tierischen oder menschlichen Geweben verwendet wird.
-
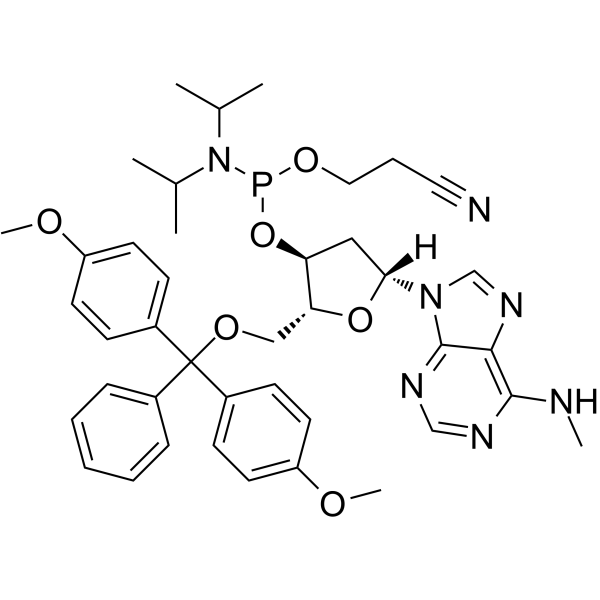
GC65561
N6-Methyl-dA phosphoramidite
N6-Methyl-dA-Phosphoramidit kann bei der Synthese von Oligodesoxyribonukleotiden verwendet werden.
-
GC34156
Namitecan (ST-1968)
ST-1968
Namitecan (ST-1968) ist ein potenter Topoisomerase-I-Hemmer mit Antitumor-Eigenschaften.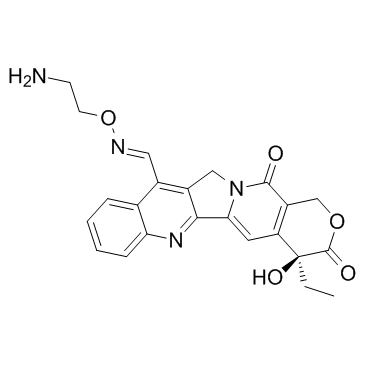
-
GC36690
Nampt-IN-3
Nampt-IN-3 (Verbindung 35) hemmt gleichzeitig Nicotinamid-Phosphoribosyltransferase (NAMPT) und HDAC mit IC50-Werten von 31 nM bzw. 55 nM. Nampt-IN-3 induziert effektiv Zellapoptose und Autophagie und fÜhrt letztendlich zum Zelltod.
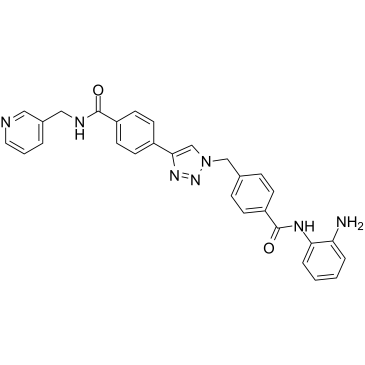
-
GC36691
Nanatinostat
CHR-3996
Nanatinostat (CHR-3996) ist ein potenter, Klasse-I-selektiver und oral aktiver Inhibitor der Histon-Deacetylase (HDAC) mit einem IC50-Wert von 8 nM.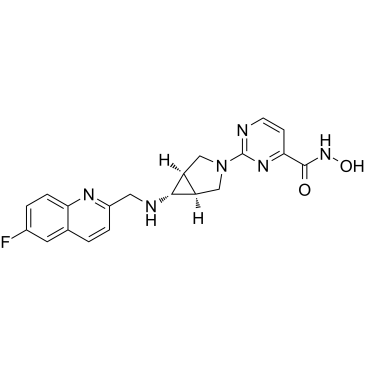
-
GC36708
NCC007
NCC007 ist ein dualer Caseinkinase-Iα (CKIα)- und δ (CKIδ)-Inhibitor mit IC50-Werten von 1,8 bzw. 3,6 μM.
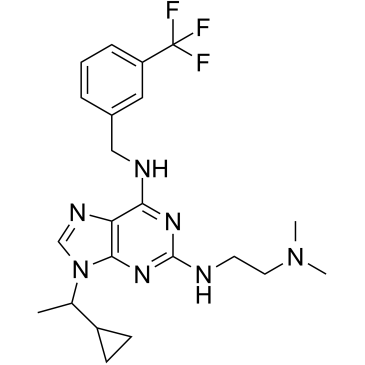
-
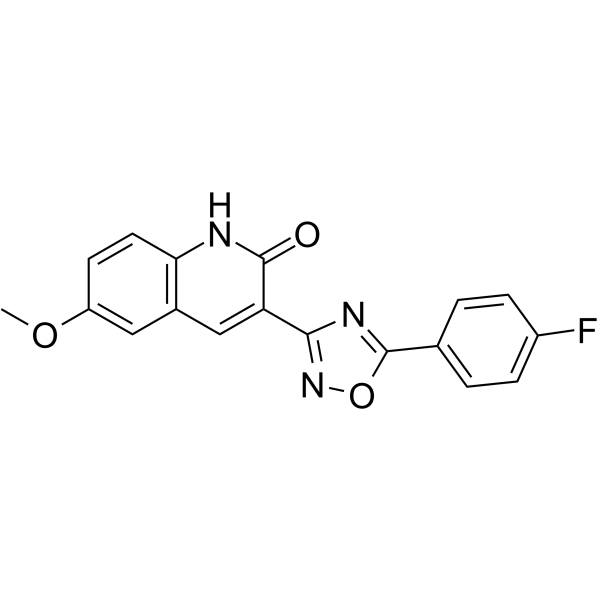
GC63653
NCGC00029283
NCGC00029283 ist ein Helicase-Nuclease (WRN)-Helicase-Inhibitor des Werner-Syndroms mit IC50-Werten von 2,3 μM, 12,5 μM und 3,4 μM fÜr WRN-, BLM- bzw. FANCJ-Helikase.
-
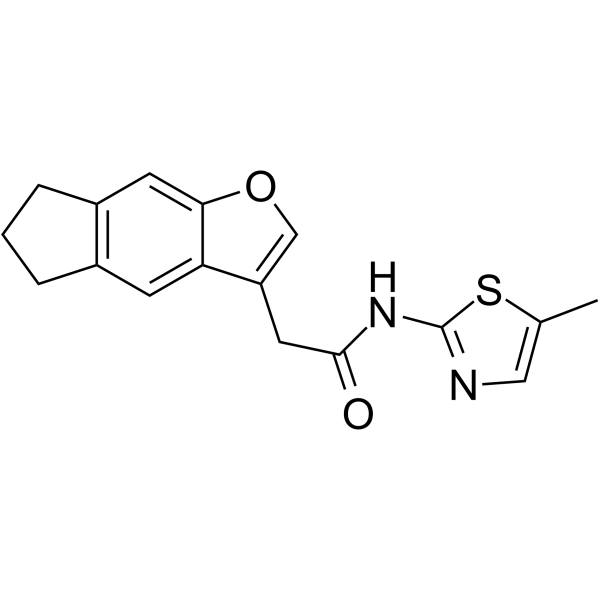
GC64393
NCT02
NCT02 ist ein Cyclin-K-Degrader. NCT02 induziert die Ubiquitinierung von Cyclin K (CCNK) und den proteasomalen Abbau von CCNK und seinem Komplexpartner CDK12. NCT02 hat das Potenzial für die Erforschung von metastasierendem Darmkrebs (CRC).
-
GC15786
Nedaplatin
NSC 375101
Nedaplatin (NSC 375101D) ist ein Derivat von Cisplatin und ein Mittel zur DNA-SchÄdigung.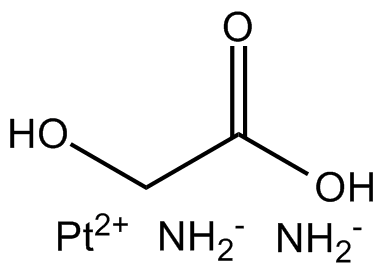
-
GC10591
Nelarabine
GW 506U78, Nelzarabine
Nelarabin (506U78) ist ein Nukleosid-Analogon und kann fÜr die Erforschung der akuten lymphoblastischen T-Zell-LeukÄmie (T-ALL) verwendet werden.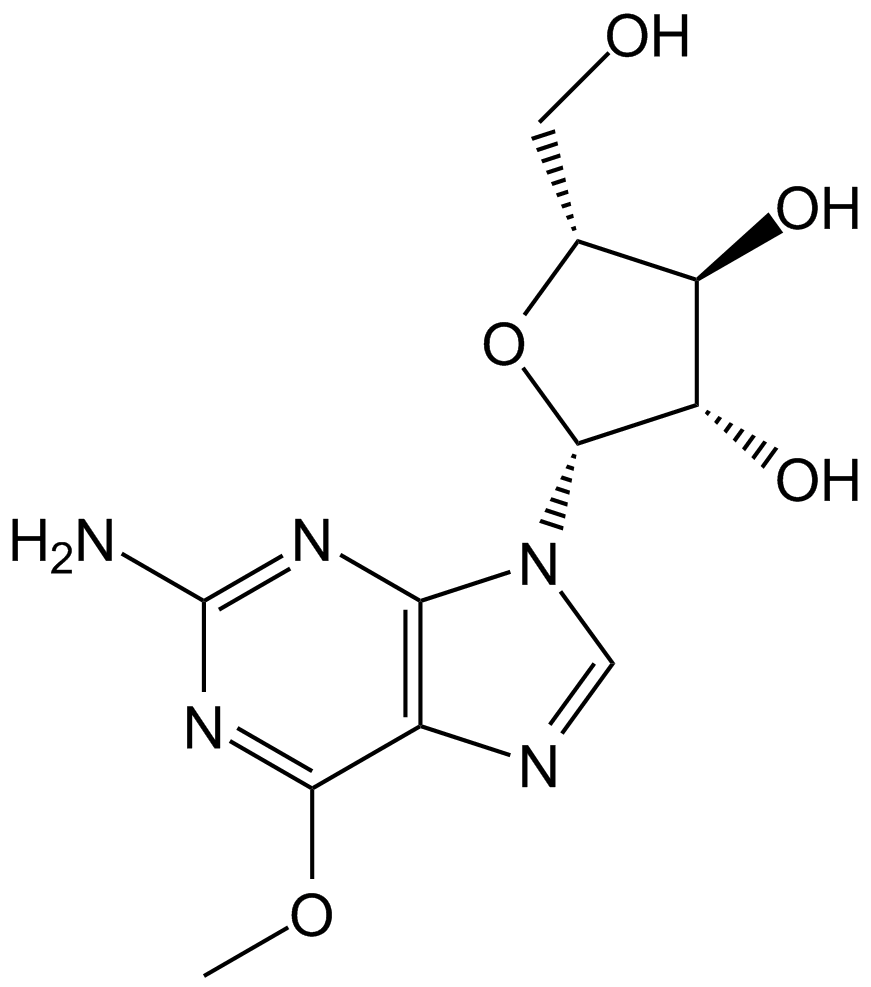
-
GC45523
Nemorosone
Nemoroson ist der Hauptbestandteil des BlÜtenharzes von Clusia rosea. Nemoroson hat eine antiproliferative Wirkung auf Krebszellen. Nemoroson induziert Apoptose in HT-29- und LoVo-Zellen.
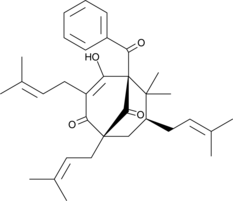
-
GC64493
Neocarzinostatin
Zinostatin; Vinostatin
Neocarzinostatin, ein starkes DNA-schÄdigendes Antitumor-Antibiotikum, erkennt doppelstrÄngige DNA-Ausbuchtungen und induziert DNA-DoppelstrangbrÜche (DSBs).
-
GC60268
Neoxanthin
Neoxanthin ist ein wichtiges Xanthophyll-Carotinoid und eine Vorstufe des Pflanzenhormons AbscisinsÄure in dunkelgrÜnem BlattgemÜse. Neoxanthin ist ein starkes Antioxidans und lichtsammelndes Pigment. Neoxanthin induziert Apoptose und hat Antikrebswirkungen.
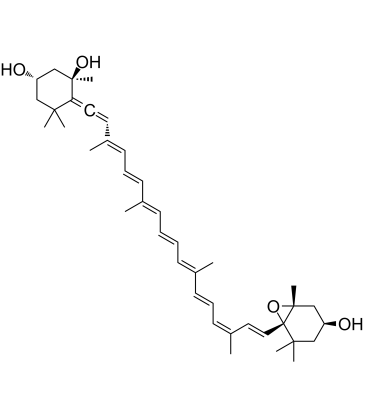
-
GC65202
Nesuparib
JPI-547/OCN-201
Nesuparib ist ein potenter Inhibitor von PARP.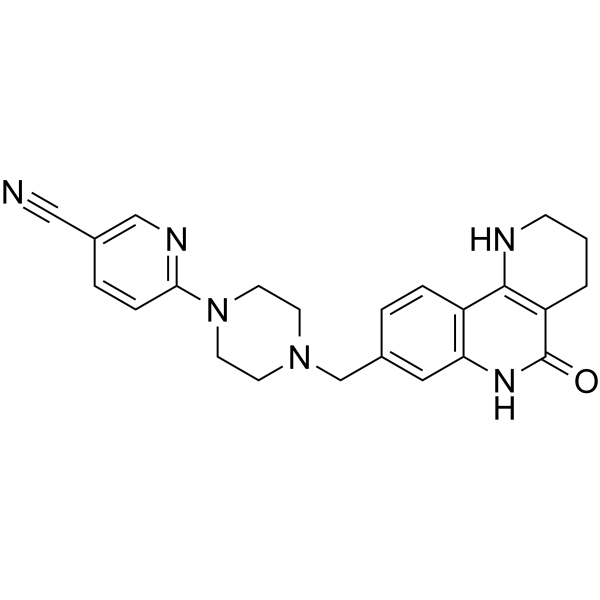
-
GC13764
Nexturastat A
Nexturastat A ist ein potenter HDAC6-Inhibitor mit einem IC50 von 5 nM. Nexturastat A hemmt auch HDAC10 und das die Metallo-β-Lactamase-DomÄne enthaltende Protein2 (MBLAC2).
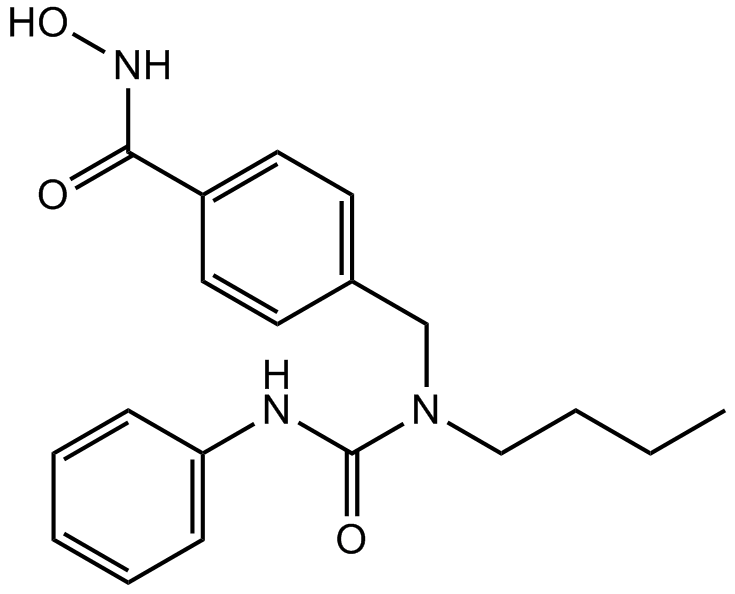
-
GC36732
NG 52
Compound 52
NG 52 ist ein potenter, zellgÄngiger, selektiver, ATP-kompatibler und oral aktiver Cdc28p- und Pho85p-Kinase-Inhibitor mit IC50-Werten von 7 μM bzw. 2 μM. NG 52 hemmt auch die AktivitÄt der Phosphoglyceratkinase 1 (PGK1) mit einer IC50 von 2,5 μM. NG 52 ist gegen die Hefekinasen Kin28p, Srb10 und Cak1p inaktiv.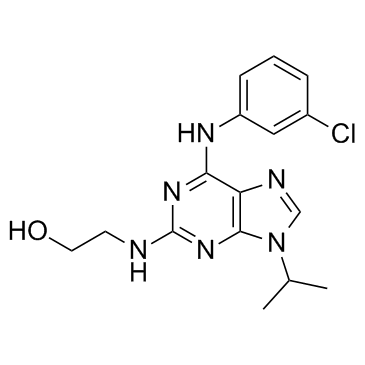
-
GC61956
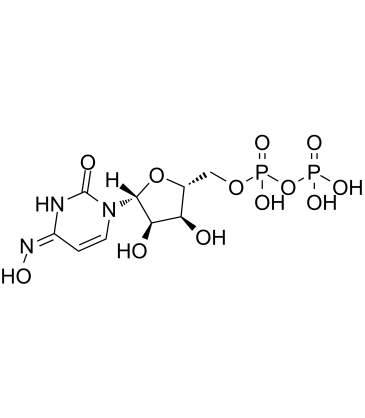
NHC-diphosphate
NHC-Diphosphat ist ein aktiver phosphorylierter intrazellulÄrer Metabolit von β-d-N4-Hydroxycytidin (NHC) als Diphosphatform.
-
GC61130
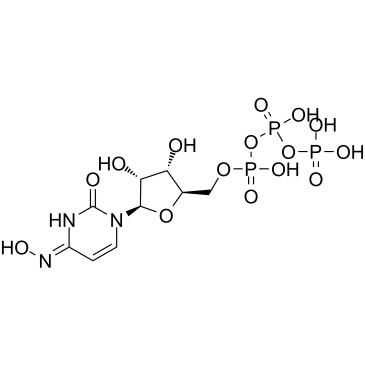
NHC-triphosphate
NHC-Triphosphat ist ein aktiver phosphorylierter intrazellulÄrer Metabolit von β-d-N4-Hydroxycytidin (NHC) als Triphosphatform.
-
GC61961
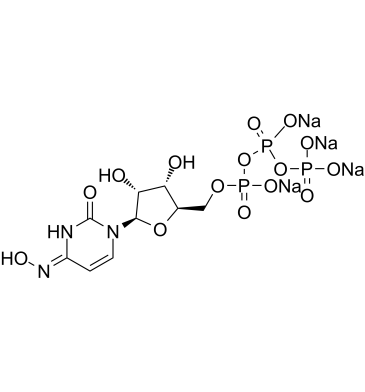
NHC-triphosphate tetrasodium
NHC-Triphosphat-Tetranatrium ist ein aktiver phosphorylierter intrazellulÄrer Metabolit von β-d-N4-Hydroxycytidin (NHC) als Triphosphatform.
-
GC36743
Nimustine hydrochloride
ACNU, NSC-D 245382
Nimustinhydrochlorid (ACNU) ist ein DNA-vernetzendes und DNA-alkylierendes Mittel, das LÄsionen und DNA-DoppelstrangbrÜche induziert, die die DNA-Replikation blockieren, und die DNA-Synthese hemmt, das Üblicherweise in der Chemotherapie von Glioblastomen verwendet wird.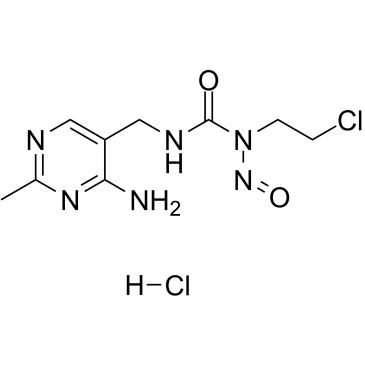
-
GC34120
Niraparib R-enantiomer (MK 4827 (R-enantiomer))
MK 4827 (R-enantiomer)
Niraparib R-Enantiomer (MK-4827 R-Enantiomer) ist ein ausgezeichneter PARP1-Inhibitor mit IC50 von 2,4 nM.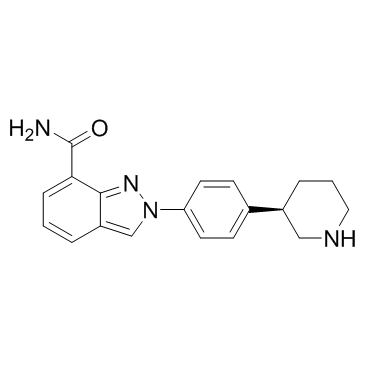
-
GC64559
NITD-2
NITD-2, ein Polymerase-Inhibitor des Dengue-Virus (DENV), hemmt die DENV-RdRp-vermittelte RNA-Elongation.
-
GC19506
NITD008
7-Deaza-2'-C-acetylene-adenosine
NITD008 ist ein potenter und selektiver Flavivirus-Inhibitor, der das Dengue-Virus Typ 2 (DENV-2) mit einem EC50-Wert von 0,64 μM hemmen kann.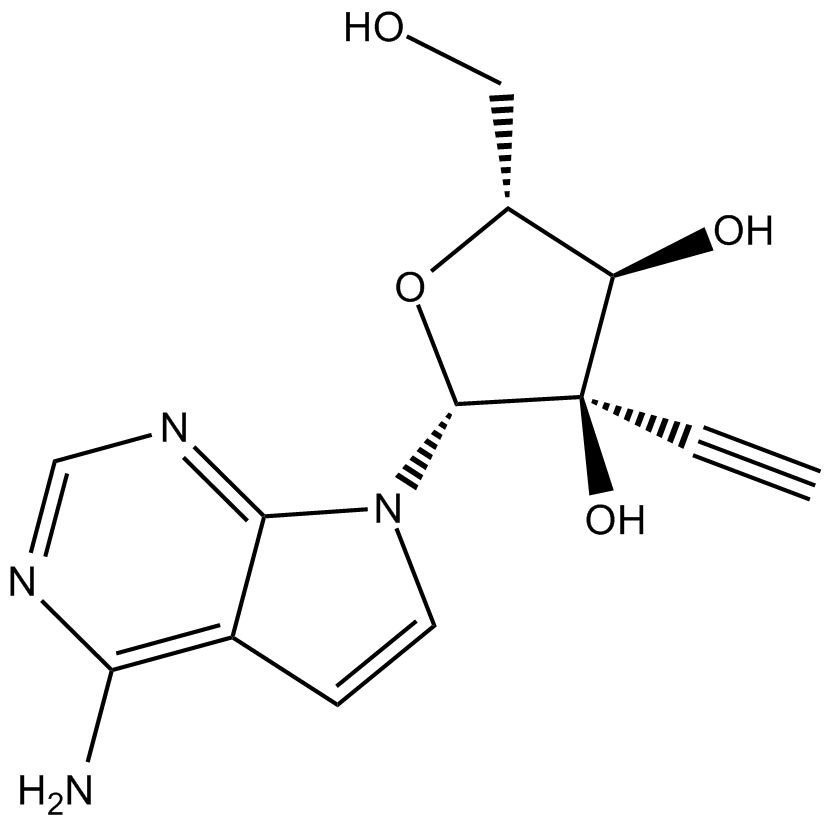
-
GC44408
Nitrosobenzene
NSC 66479
Nitrosobenzene is a spin trap that has been used in the study of oxidative DNA damage and nitroso-compound-induced respiratory burst in neutrophils.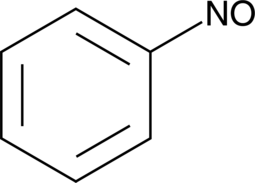
-
GC32964
NKL 22
Histone Deacetylase Inhibitor IV
NKL 22 (Verbindung 4b) ist ein potenter und selektiver Inhibitor von Histon-Deacetylasen (HDAC) mit einem IC50 von 199 und 69 nM fÜr HDAC1 bzw. HDAC3.
-
GC19263
NKP-1339
KP1339
NKP-1339 (IT-139; KP-1339) ist das erste Antikrebsmittel seiner Klasse auf Rutheniumbasis in der Entwicklung gegen soliden Krebs mit begrenzten Nebenwirkungen. NKP-1339 induziert den G2/M-Zellzyklusarrest, die Blockierung der DNA-Synthese und die Induktion der Apoptose Über den mitochondrialen Weg. NKP-1339 hat ein hohes Tumor-Targeting-Potenzial, bindet stark an Serumproteine wie Albumin und Transferrin und wird im reduktiven Tumormilieu aktiviert.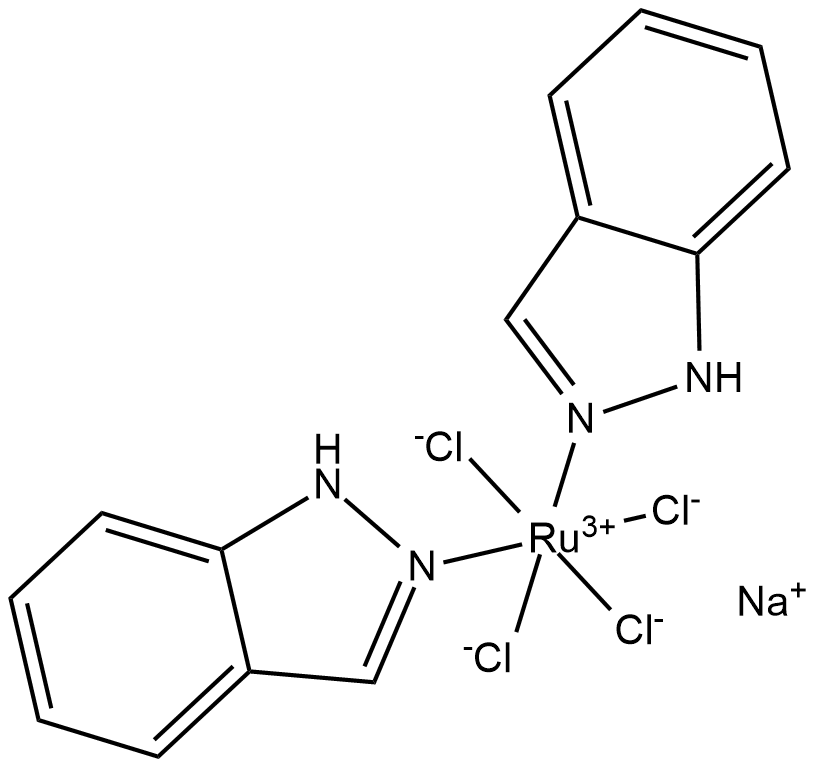
-
GC19264
NMS-P118
NMS-P118 ist ein potenter, oral verfÜgbarer und hochselektiver PARP-1-Inhibitor fÜr die Krebstherapie.
-
GC36751
NMS-P515
NMS-P515 ist ein potenter, oral aktiver und stereospezifischer PARP-1-Inhibitor mit einem Kd von 16 nM und einem IC50 von 27 nM (in Hela-Zellen). Anti-Tumor-AktivitÄt.
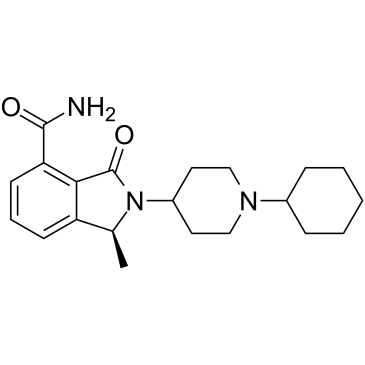
-
GC14075
Nocodazole
NSC 238159, Oncodazole, R 17934
Ein Tubulin-Produktionshemmer, ein antineoplastisches Mittel.
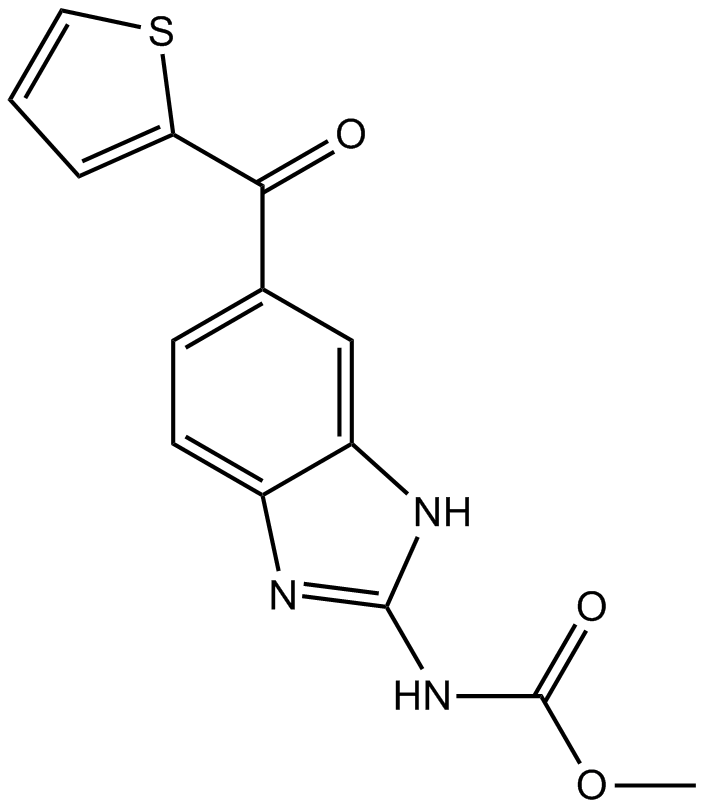
-
GC44438
Nogalamycin
NSC 70845, U 15167
CCG-273220 ist ein kovalenter Inhibitor der G-Protein-gekoppelten Rezeptor(GPCR)-Kinase 5 (GRK5) mit einem IC50-Wert von 220 nM. CCG-273220 ist hochgradig selektiv fÜr GRK5 gegenÜber GRK2 (IC50 = 350 μM), indem es Cys474, einen fÜr die Unterfamilie GRK5 spezifischen Rest, als kovalenten Griff bindet.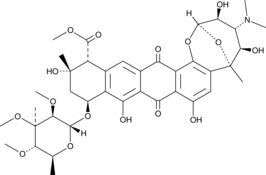
-
GC62679
NSAH
NSAH ist ein reversibler und kompetitiver Nichtnukleosid-Ribonukleotidreduktase (RR)-Inhibitor mit einer zellfreien IC50 von 32 μM bzw. einer zellbasierten IC50 von ~250 nM.
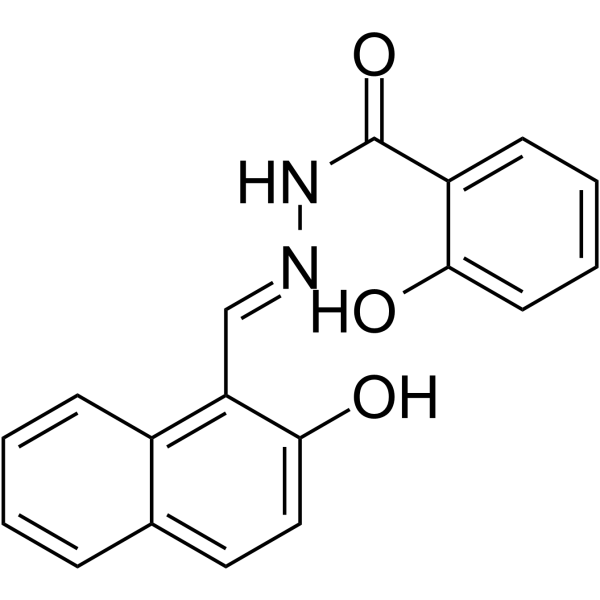
-
GC63874
NSC 107512
NSC 107512 ist ein potenter Inhibitor der Cyclin-abhÄngigen Kinase 9 (CDK9). NSC 107512 ist eine Klasse von Sangivamycin-Ähnlichen MolekÜlen (SLM). NSC 107512 hemmt das Wachstum und induziert die Apoptose von multiplen Myelomtumoren.
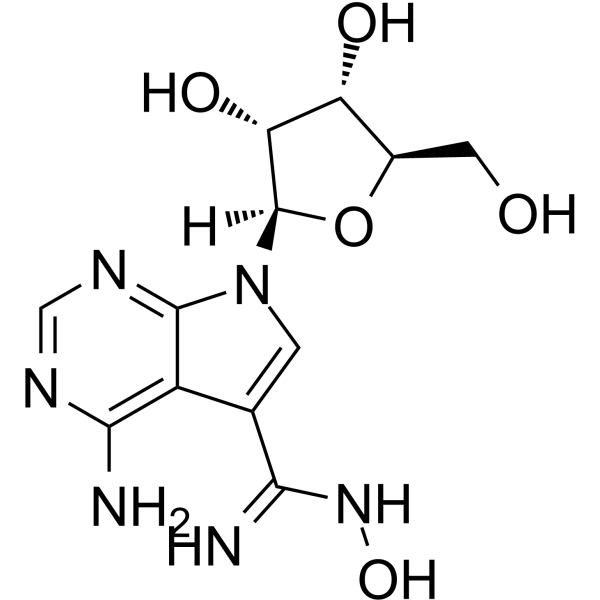
-
GC12475
NSC 632839 hydrochloride
F6, Ubiquitin Isopeptidase Inhibitor II
A deubiquitylase and deSUMOylase inhibitor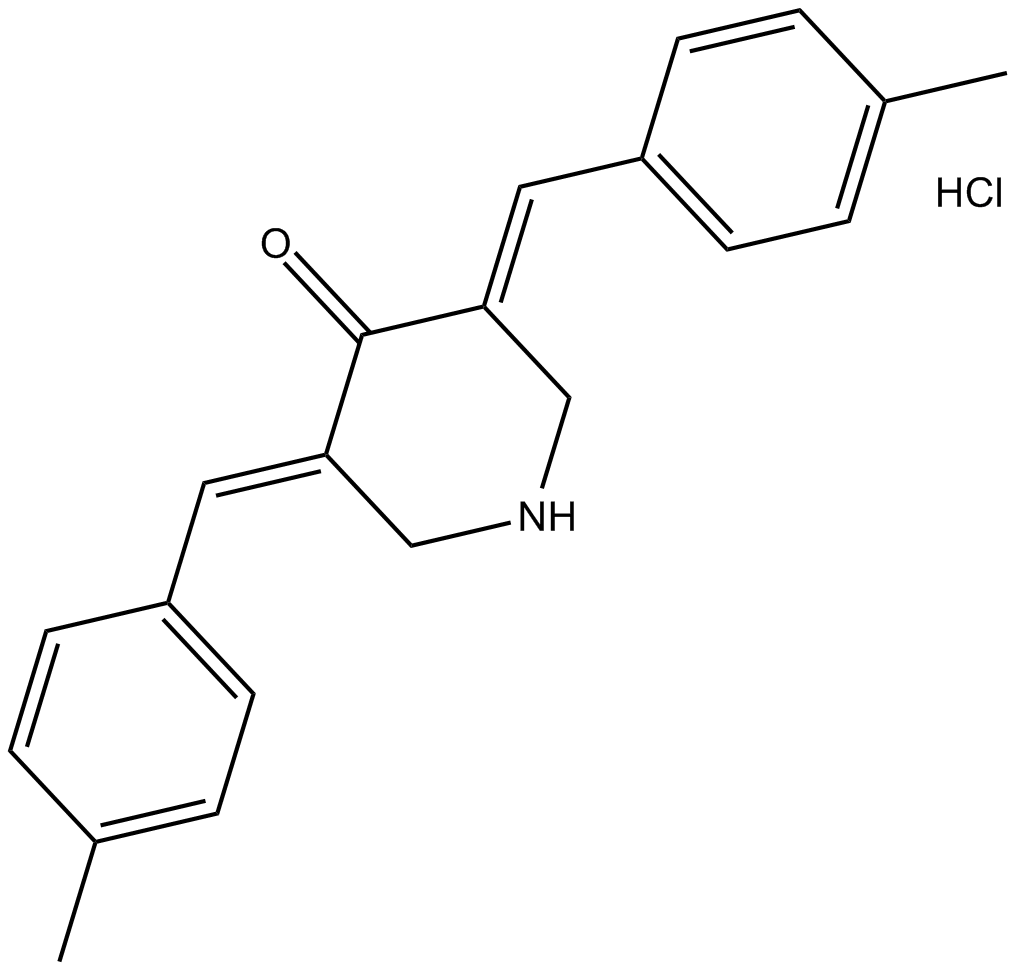
-
GC15503
NSC 687852 (b-AP15)
b-AP15
An inhibitor of the deubiquitinases USP14 and UCHL5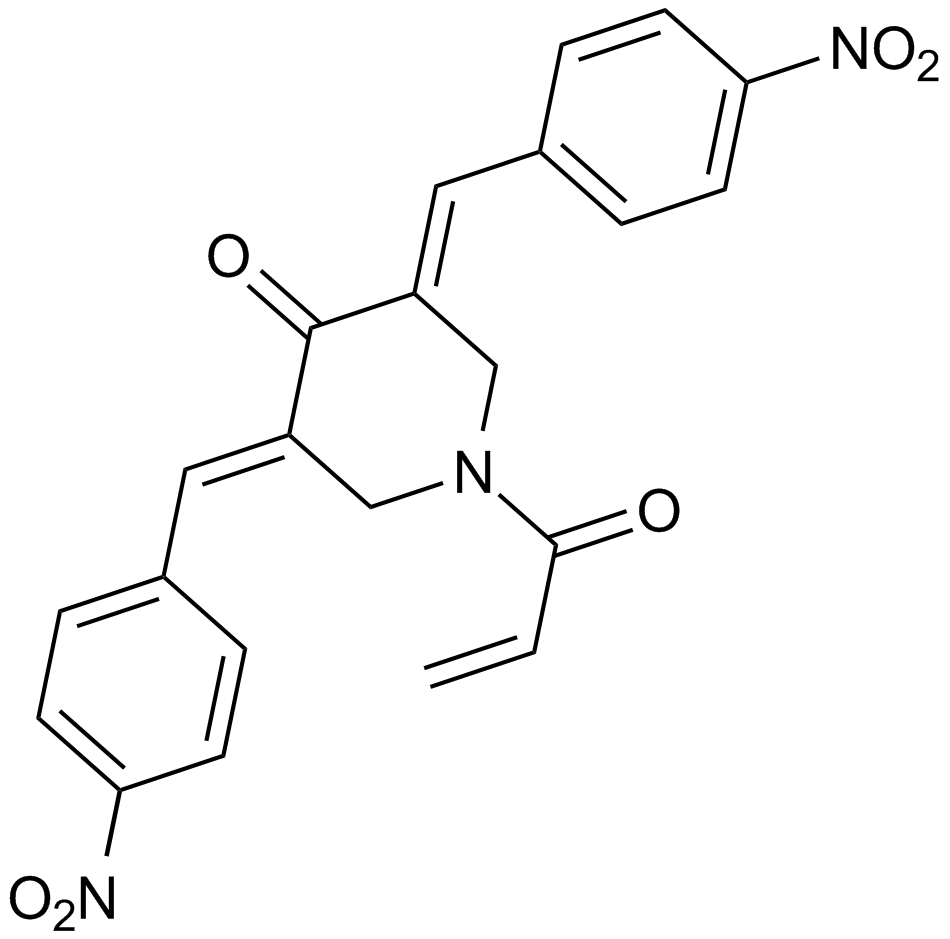
-
GC69598
NSC 80467
NSC 80467 ist ein DNA-Schädigungsmittel, das selektiv Survivin hemmt. NSC 80467 hemmt vorrangig die DNA-Synthese und induziert zwei Marker für DNA-Schäden, γH2AX und pKAP1.
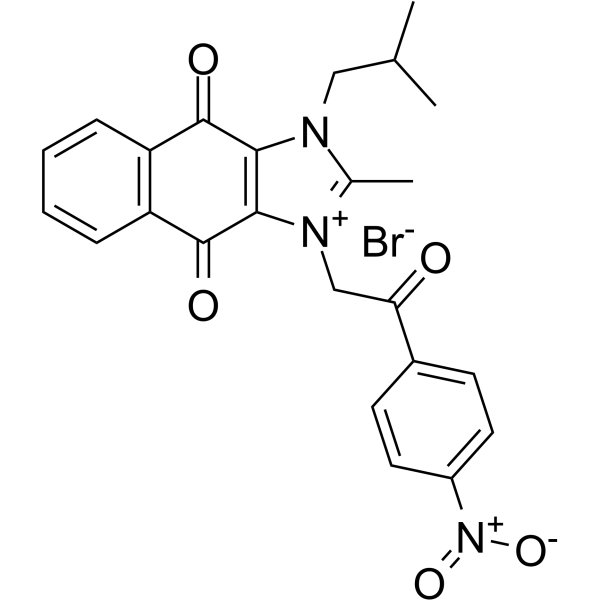
-
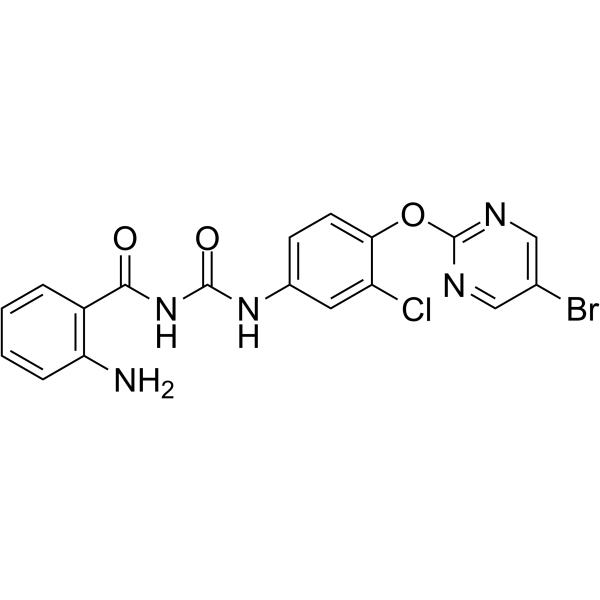
GC64103
NSC639828
NSC639828 ist ein potenter Inhibitor der DNA-Polymerase α mit einem IC50 von 70 μM. NSC639828 hat eine hohe AntitumoraktivitÄt. NSC639828 hat das Potenzial zur Erforschung von Krebserkrankungen.
-
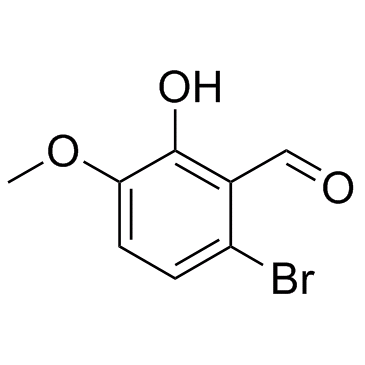
GC34111
NSC95682 (6-Bromo-2-hydroxy-3-methoxybenzaldehyde)
NSC95682 (6-Brom-2-hydroxy-3-methoxybenzaldehyd) (NSC95682) ist ein IRE-1&7#945; Inhibitor mit einem IC50 von 0,08 μM, entnommen aus Patent WO 2008154484 A1, IRE-1α Inhibitorverbindung 3-5.
-
GC12332
NU 7026
DNAPK Inhibitor II, LY293646
NU 7026 (LY293646) ist ein neuartiger spezifischer DNA-PK-Inhibitor mit einem IC50 von 0,23 μM, der auch PI3K mit einem IC50 von 13 μM hemmt.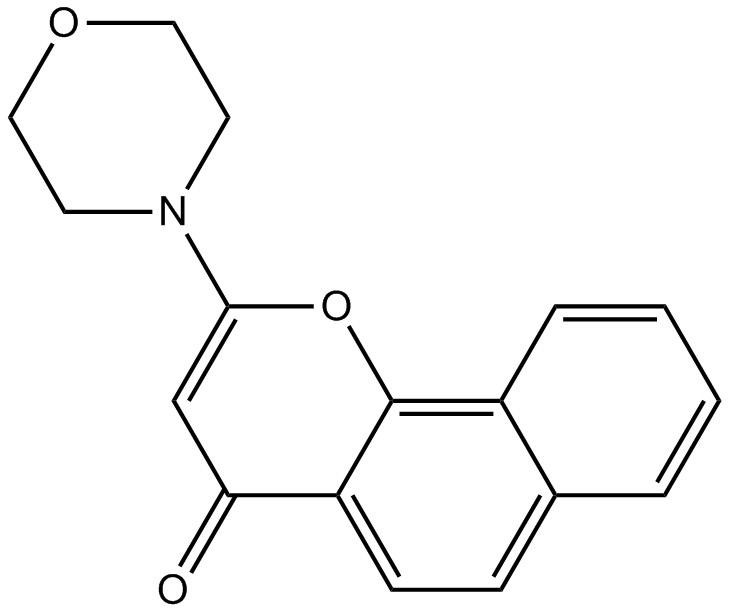
-
GC19267
NU2058
O6-(Cyclohexylmethyl)guanine
NU2058 (O6-(Cyclohexylmethyl)guanin) ist ein potenter, kompetitiver und auf Guanin basierender CDK-Inhibitor mit IC50-Werten von 17 μM und 26 μM fÜr CDK2 und CDK1. NU2058 hat Anti-Krebs-AktivitÄt.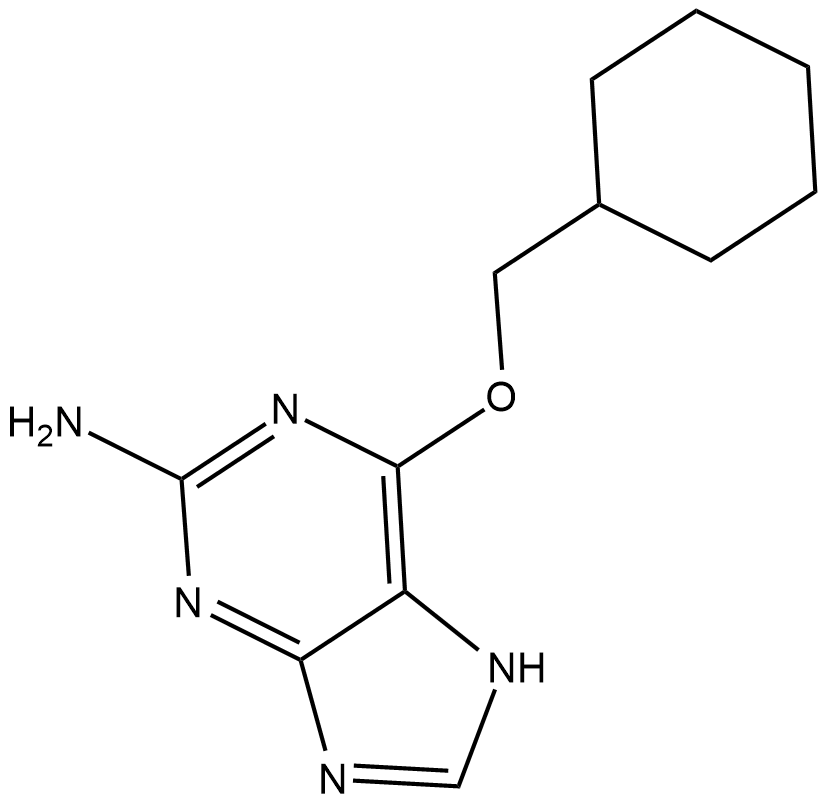
-
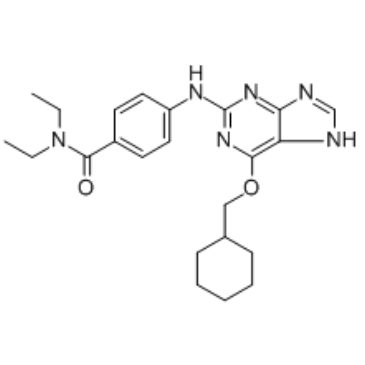
GC34692
NU6140
NU6140 ist ein selektiver CDK2-Cyclin-A-Inhibitor (IC50, 0,41 μM) und weist eine 10- bis 36-fache SelektivitÄt gegenÜber anderen CDKs auf. NU6140 hemmt auch stark Aurora A und Aurora B mit IC50-Werten von 67 bzw. 35 nM. VerstÄrkt die apoptotische Wirkung mit Anti-Krebs-AktivitÄt.
-
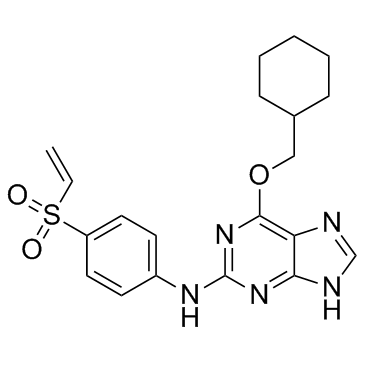
GC32829
NU6300
NU6300 ist ein kovalenter, irreversibler und ATP-kompetitiver CDK2-Inhibitor mit einem IC50-Wert von 0,16 μM. NU6300 kann für die Erforschung des eukaryotischen Zellzyklus und der Transkription verwendet werden.
-
GC11251
NU7441 (KU-57788)
KU 57788; NU-7441;KU57788;NU7441;NU 7441
NU7441 (KU-57788) (NU7441) ist ein hochpotenter und selektiver DNA-PK-Inhibitor mit einem IC50 von 14 nM. NU7441 (KU-57788) ist ein Inhibitor des NHEJ-Signalwegs. NU7441 (KU-57788) hemmt auch PI3K und mTOR mit IC50-Werten von 5,0 bzw. 1,7 μM.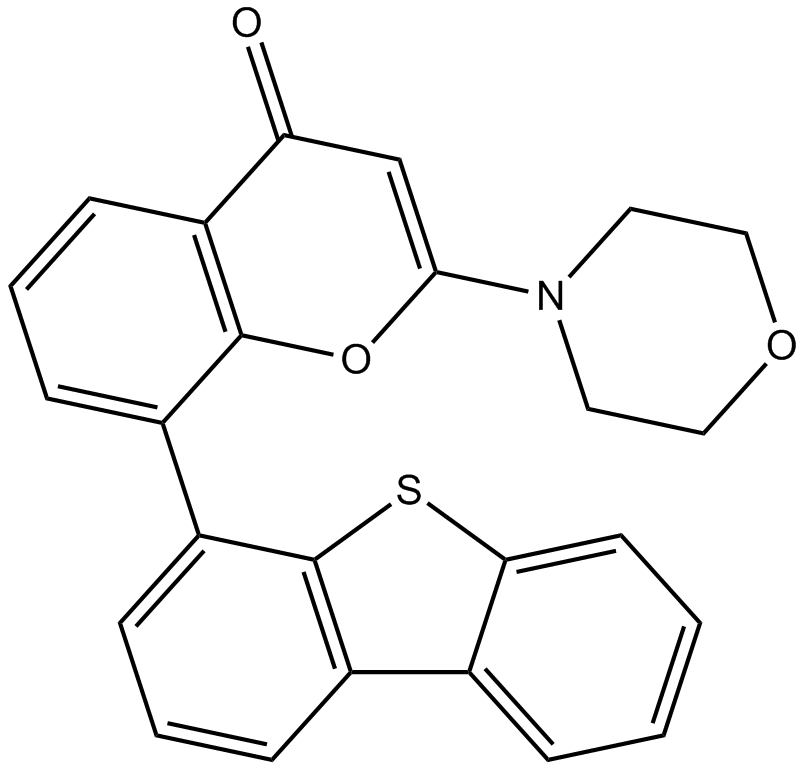
-
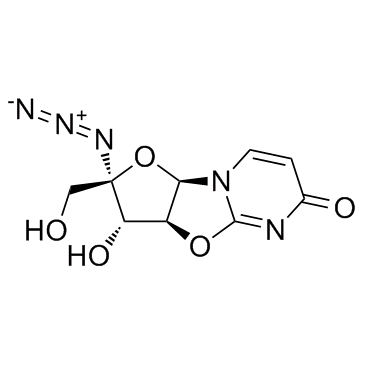
GC33956
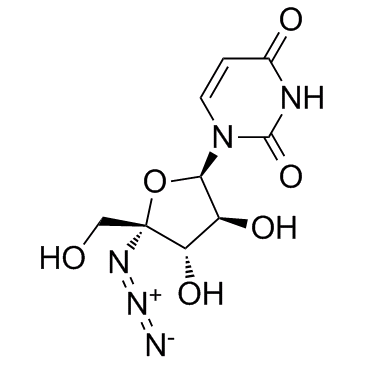
Nucleoside-Analog-1
Nucleosid-Analog-1 ist ein 4′-Azidocytidin-Analogon gegen die Replikation des Hepatitis-C-Virus.
-
GC33959
Nucleoside-Analog-2
Nucleosid-Analog-2 ist ein 4'-Azidocytidin-Analogon gegen die Replikation des Hepatitis-C-Virus (HCV).
-
GC13925
Nullscript
negative control of scriptaid, HDAC inhibitor
-
GC64293
Nusinersen
Nusinersen ist ein Antisense-Oligonukleotid-Medikament, das das Pre-Messenger-RNA-Spleißen des SMN2-Gens modifiziert und somit eine erhÖhte Produktion des SMN-Proteins in voller LÄnge fÖrdert.

-
GC34056
NVP-2
NVP-2 ist eine potente und selektive ATP-kompetitive Cyclin-abhÄngige Kinase 9 (CDK9)-Sonde, die die CDK9/CycT-AktivitÄt mit einem IC50 von 0,514 nM hemmt. NVP-2 zeigt inhibitorische Wirkungen auf CDK1/CycB-, CDK2/CycA- und CDK16/CycY-Kinasen mit IC50-Werten von 0,584 μM, 0,706 μM bzw. 0,605 μM. NVP-2 induziert Zellapoptose.
-
GC16959
NVP-LCQ195
-
GC17555
NVP-TNKS656
TNKS656
NVP-TNKS656 ist ein hochpotenter, selektiver und oral aktiver TNKS2-Inhibitor mit einem IC50 von 6 nM und einer > 300-fachen SelektivitÄt gegenÜber PARP1 und PARP2.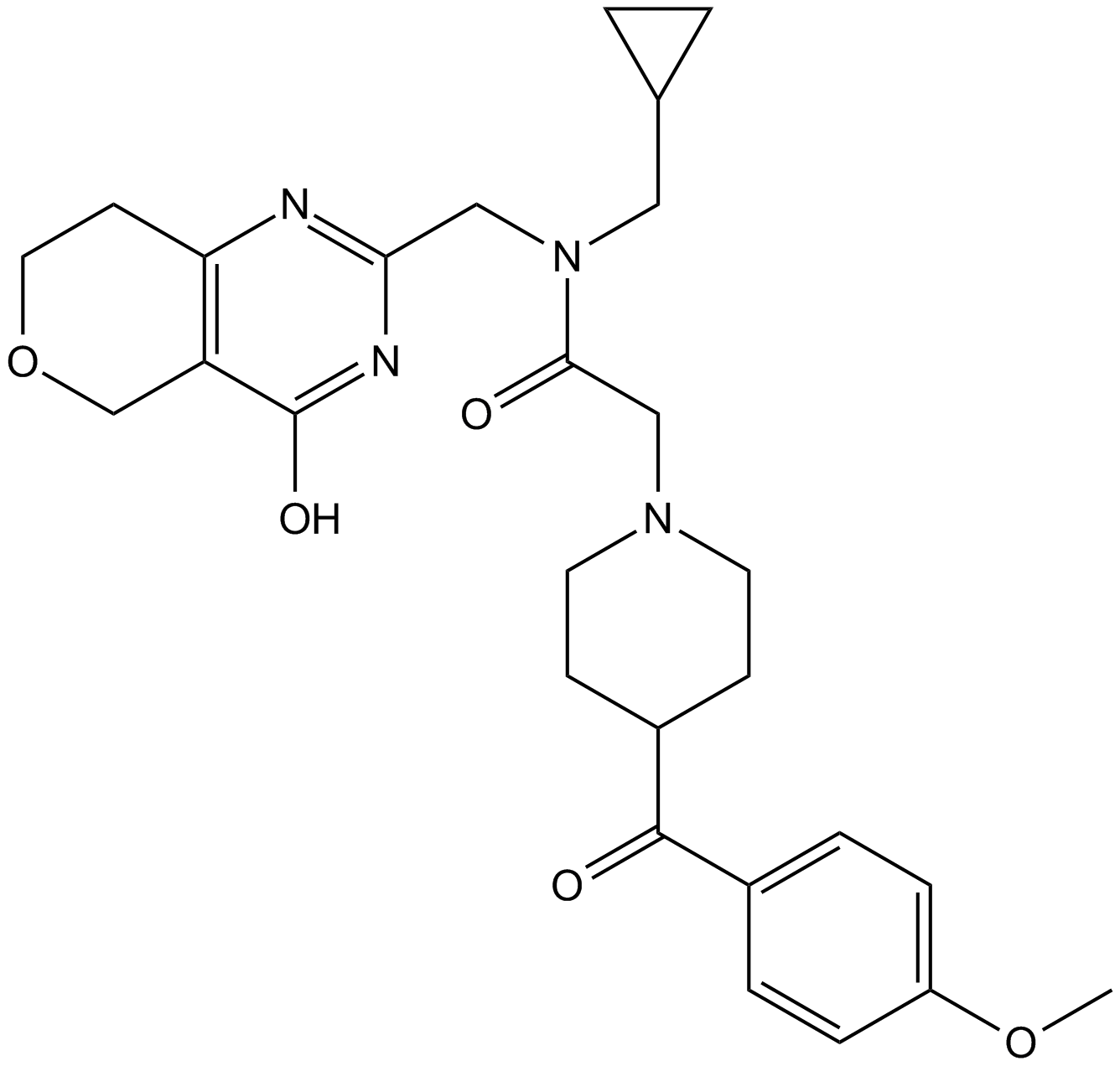
-
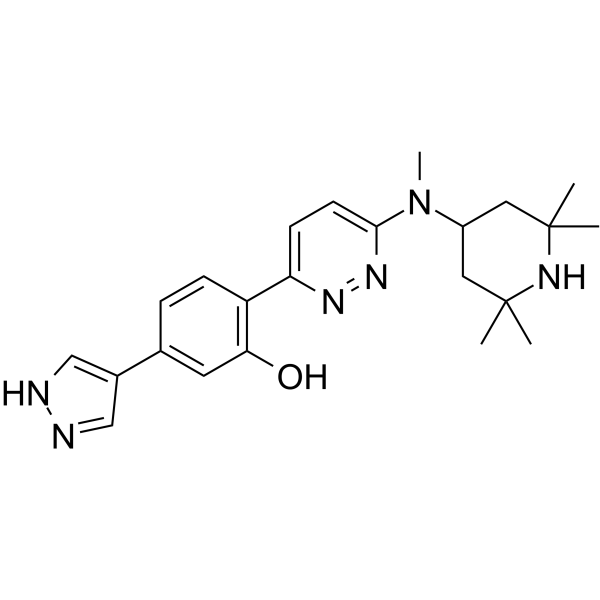
GC63123
NVS-SM2
NVS-SM2 ist ein potenter, oral aktiver und in das Gehirn eindringender SMN2-SpleißverstÄrker mit einem EC50 von 2 nM fÜr SMN.
-
GC62501
OBI-3424
TH-3424
OBI-3424 (TH-3424) ist ein Prodrug, das selektiv von AKR1C3 (Aldo-Keto-Reduktase 1C3) in ein wirksames DNA-alkylierendes Mittel umgewandelt wird. OBI-3424 kann in der Forschung zu hepatozellulÄrem Karzinom, kastrationsresistentem Prostatakrebs und akuter lymphoblastischer LeukÄmie (ALL) eingesetzt werden.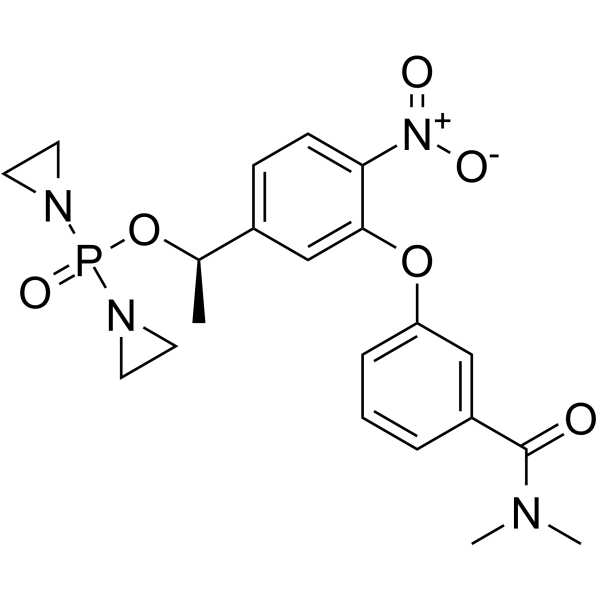
-
GC49743
Octapeptide-2
KLKKTETQ
A thymosin β4-derived peptide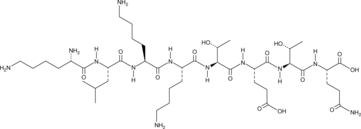
-
GC69618
Olaparib-d8
AZD2281-d8; KU0059436-d8
Olaparib-d8 ist das Deuterium-Isotop von Olaparib (AZD2281). Olaparib ist ein oral wirksamer PARP-Inhibitor, der die IC50-Werte von PARP-1 und PARP-2 bei 5 bzw. 1 nM hemmt. Olaparib ist ein Aktivator für Autophagie und Mitophagie.
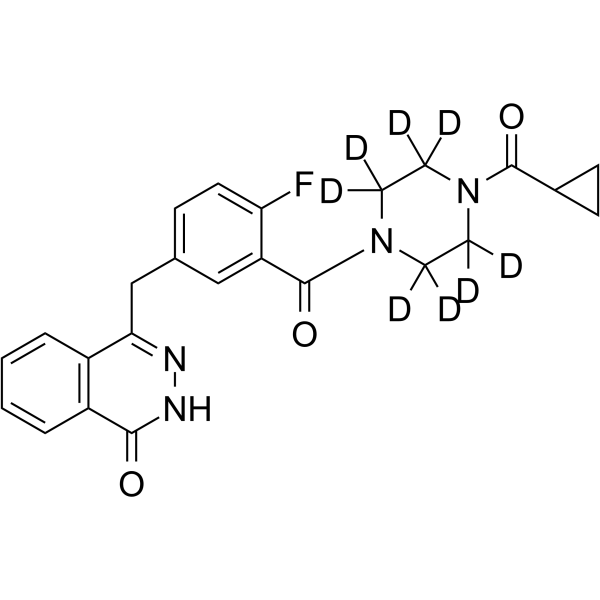
-
GC67906
OM-153

-
GC33353
ON-013100
ON-013100, ein antineoplastisches Medikament, wirkt als mitotischer Inhibitor, der die Cyclin D1-Expression hemmen kÖnnte.
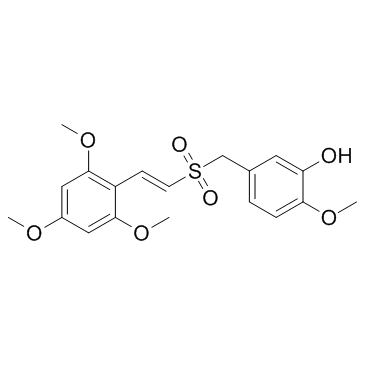
-
GC15607
ON123300
ON123300, ein starker und ins Gehirn eindringender Multikinase-Inhibitor, hemmt CDK4 (IC50=3,9 nM), Ark5 (IC50=5 nM), PDGFRβ (IC50=26 nM), FGFR1 (IC50=26 nM), RET (IC50= 9,2 nM) und FYN (IC50 = 11 nM). Der Einzelwirkstoff ON123300 bewirkt eine dosisabhÄngige UnterdrÜckung der Phosphorylierung von Akt sowie eine Aktivierung von Erk bei Hirntumoren. ON123300 hemmt CDK6 mit einem IC50 von 9,82 nM.
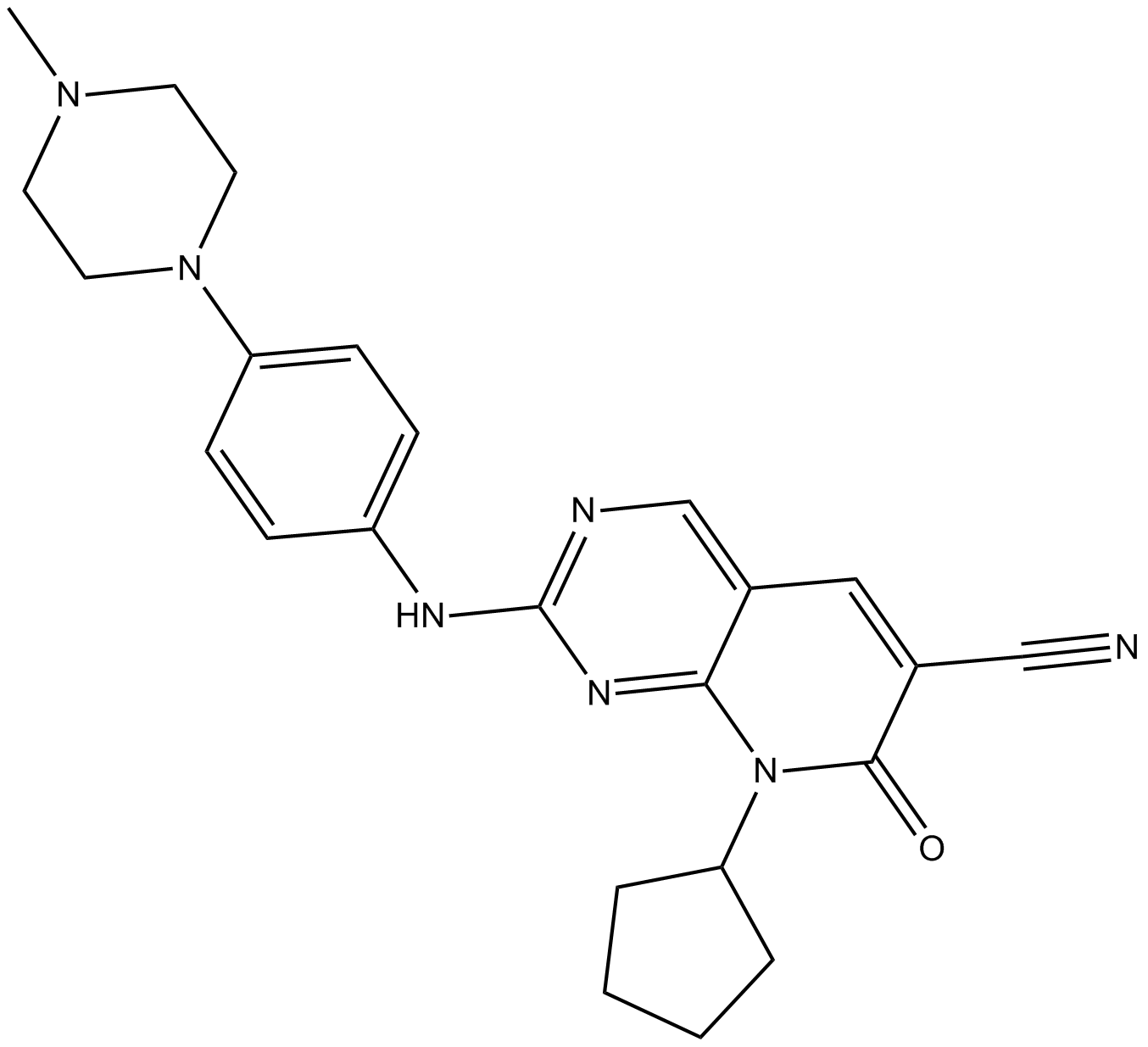
-
GC69633
Orobol
Orobol ist eine wichtige Isoflavon aus Sojabohnen mit verschiedenen pharmakologischen Aktivitäten, einschließlich Anti-Aging und Anti-Adipositas-Effekten. Orobol hemmt CK1ε, VEGFR2, MAP4K5, MNK1, MUSK, TOPK und TNIK (IC50=1.24-4.45 μM). Orobol hemmt auch PI3K-Subtypen (für PI3K α/β/γ/ K/δ, IC50=3.46-5.27 μM).
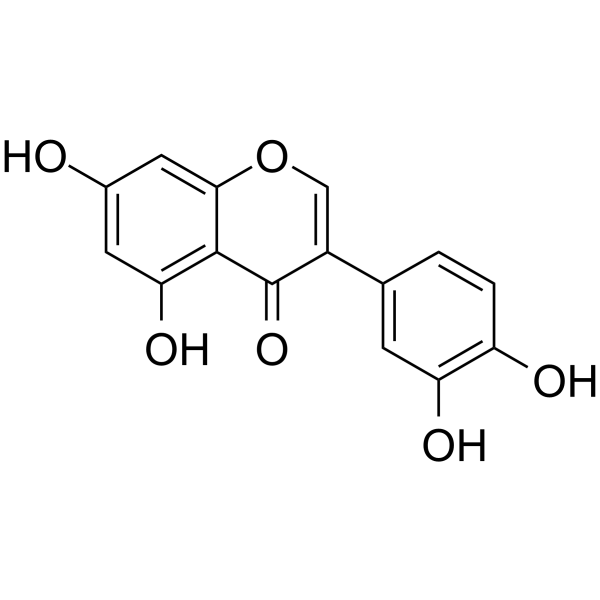
-
GC11528
Orotic acid
OrotsÄure (6-Carboxyuracil), ein VorlÄufer bei der Biosynthese von Pyrimidinnukleotiden und RNA, wird von der mitochondrialen Dihydroorotat-Dehydrogenase (DHODH) zur Umwandlung in UMP durch das zytoplasmatische UMP-Synthase-Enzym freigesetzt.
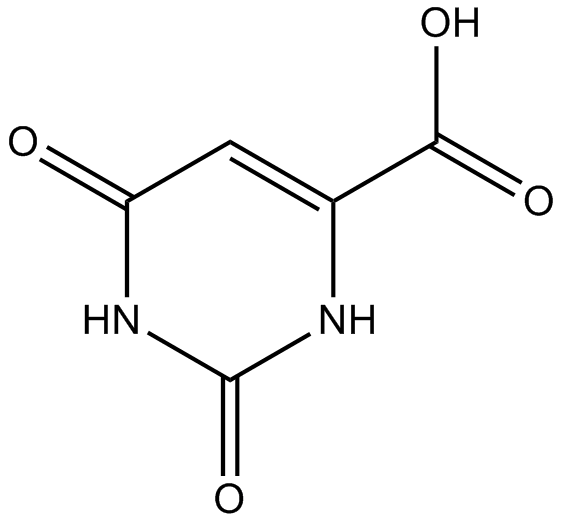
-
GC64296
Orotidine 5′-monophosphate trisodium
Orotidine monophosphate trisodium; Orotidylic acid trisodium
Orotidin-5'-Monophosphattrinatrium ist ein Pyrimidinnukleotid.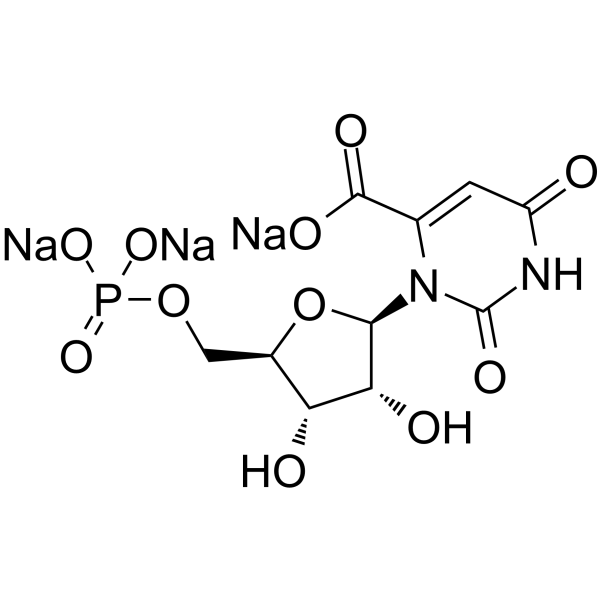
-
GC11360
ORY-1001
Iadademstat, RG-6016
Selective inhibitor of KDM1A.
-
GC12304
OTS514
OTS514 ist ein hochpotenter TOPK-Inhibitor mit einem IC50 von 2,6 nM. OTS514 unterdrÜckt stark das Wachstum von TOPK-positiven Krebszellen. OTS514 induziert Zellzyklusarrest und Apoptose.
-
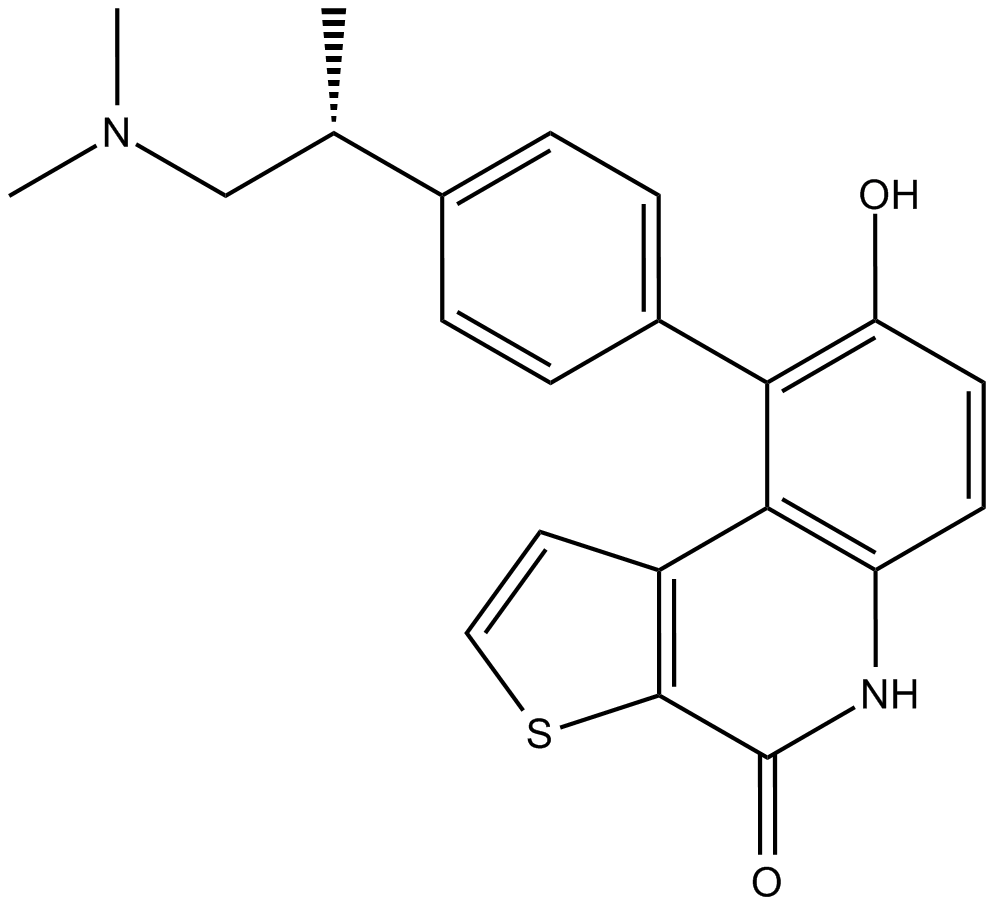
GC16511
OTS964
OTS964 ist ein oral aktiver, hochaffiner und selektiver TOPK-Inhibitor (T-lymphokin-activated killer cell-originated protein kinase) mit einem IC50 von 28 nM. OTS964 ist auch ein potenter Inhibitor der Cyclin-abhÄngigen Kinase CDK11, die mit einer Kd von 40 nM an CDK11B bindet.
-
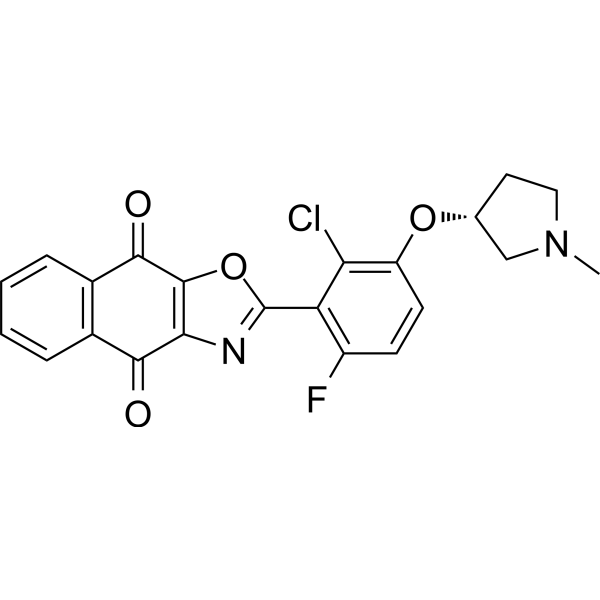
GC69637
OTUB1/USP8-IN-1
OTUB1/USP8-IN-1 ist ein wirksamer Dualhemmer von OTUB1/USP8, der die IC50-Werte von OTUB1 und USP8 bei 0,17 bzw. 0,28 nM hemmt. OTUB1/USP8-IN-1 kann für die Krebsforschung verwendet werden.
-
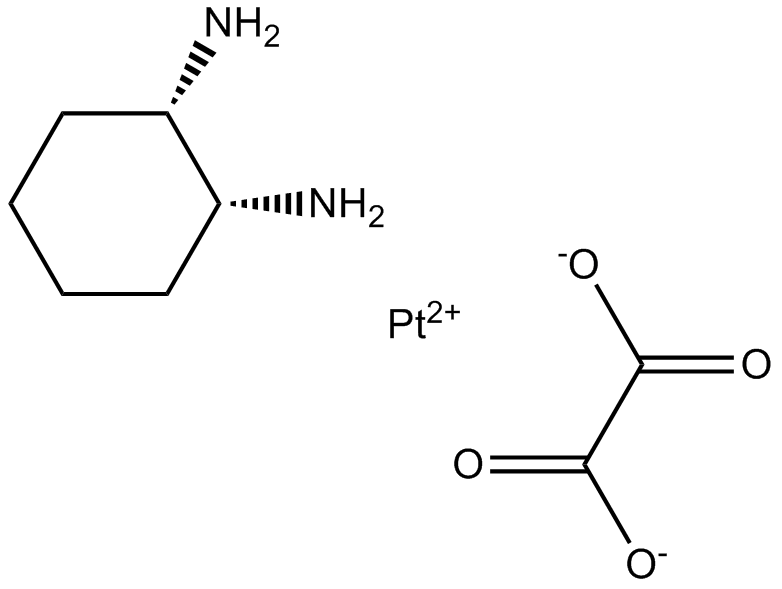
GC17716
Oxaliplatin
Lipoxal, NSC 266046, RP 54780
Oxaliplatin ist ein zytotoxisches Chemotherapeutikum, das zur Behandlung von Krebs eingesetzt wird.
-
GC49251
Oxaliplatin-d10
Lipoxal-d10
An internal standard for the quantification of oxaliplatin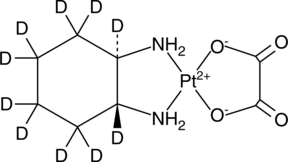
-
GC17472
Oxamflatin
Metacept 3
Oxamflatin (Metacept-3) ist ein potenter HDAC-Inhibitor mit einem IC50 von 15,7 nM.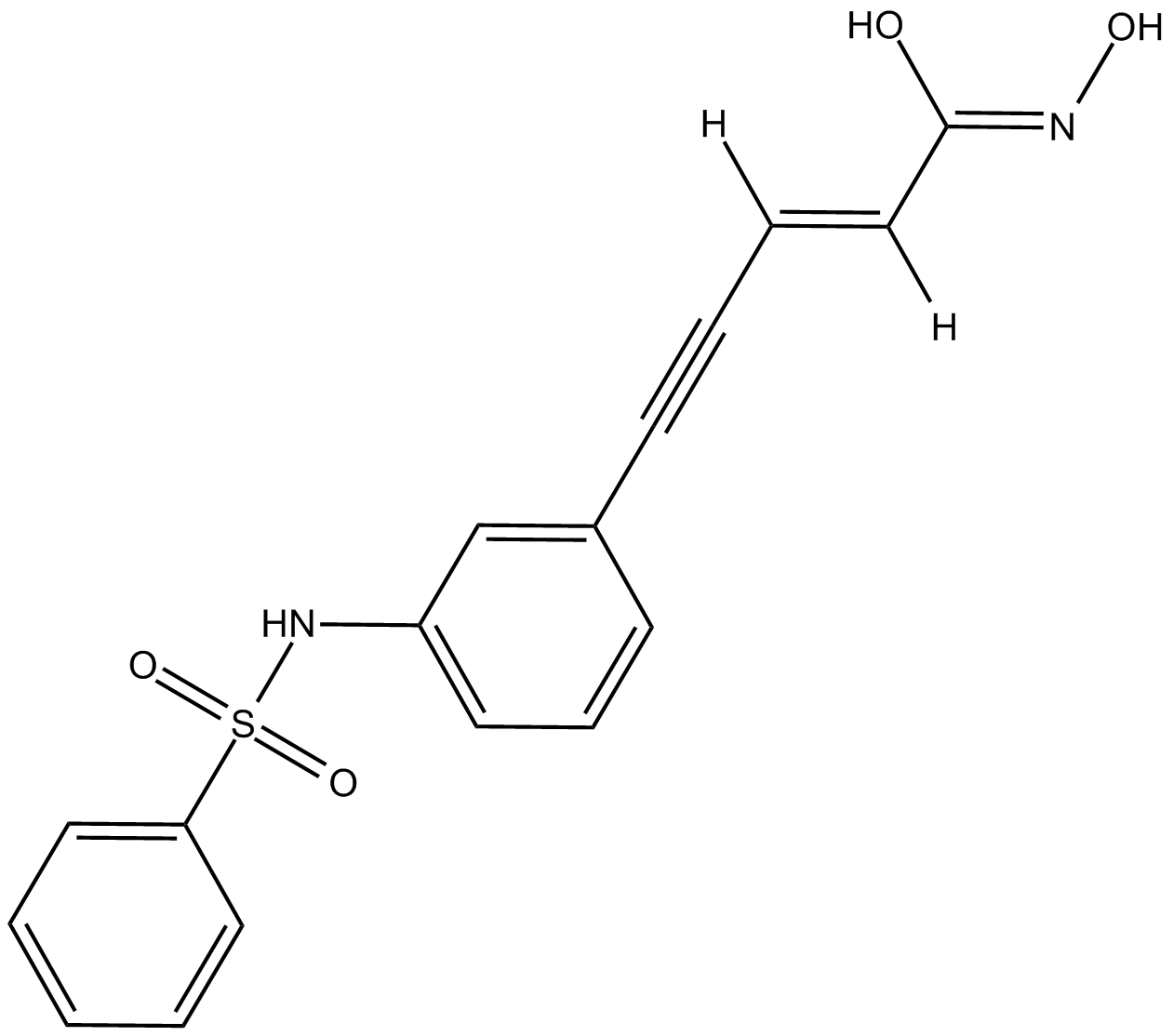
-
GC14107
Oxolinic acid
NSC 110364, Urinox
OxolinsÄure ist ein Antibiotikum gegen gramnegative und grampositive Bakterien.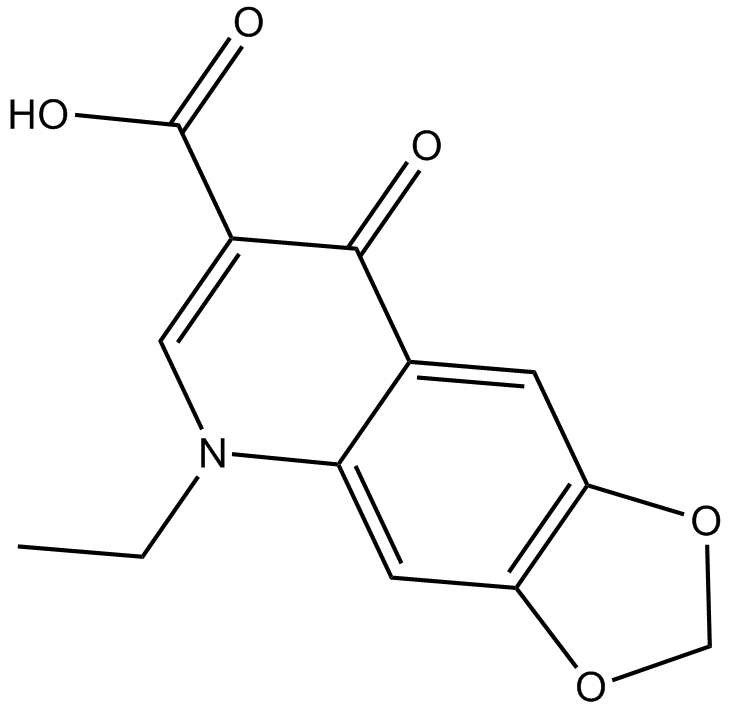
-
GC10379
P 22077
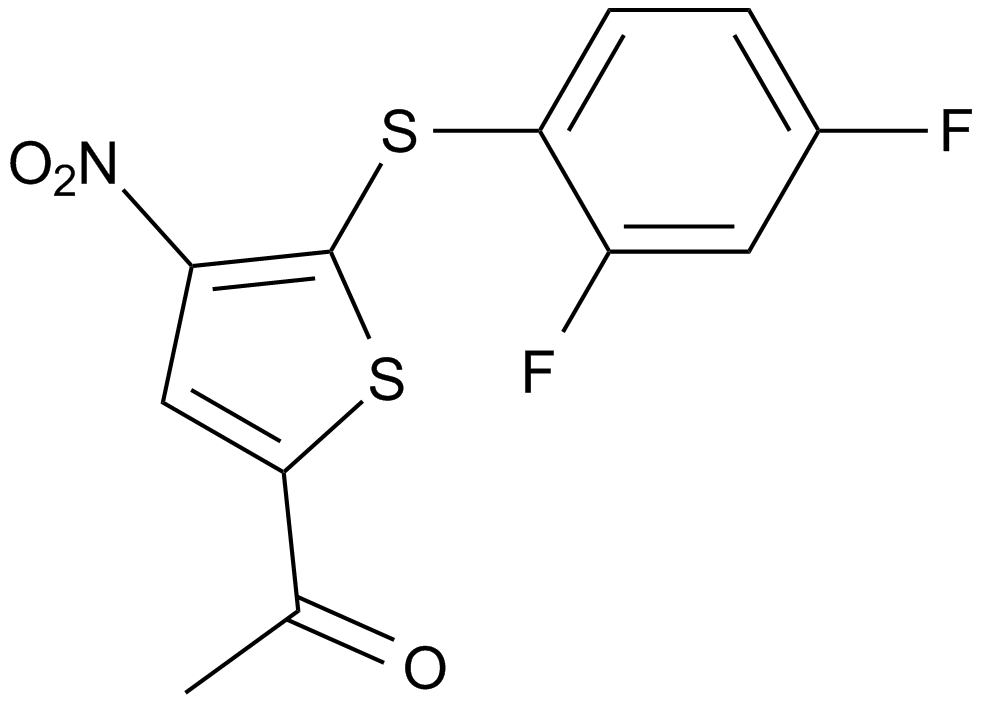
-
GC12067
P005091
P005091,P5091
P005091 is a potent and selective ubiquitin-specific proteinase 7 (USP7) inhibitor with an EC50 value of 4.2 μM.
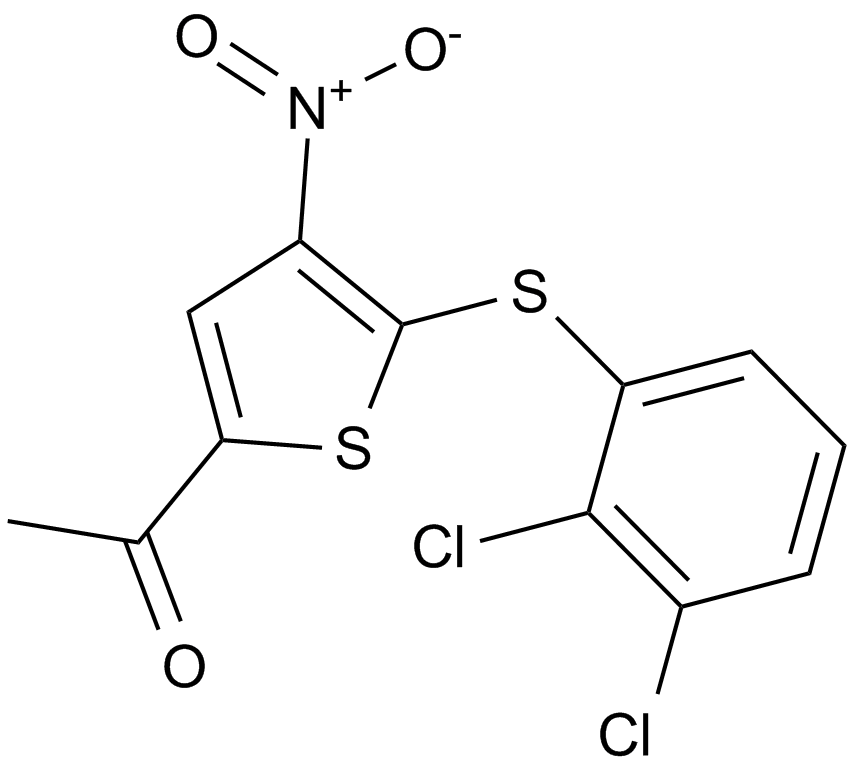
-
GC17232
P005672 hydrochloride
Sarecycline hydrochloride
P005672-Hydrochlorid ist ein Schmalspektrum-Antibiotikum der Tetracyclin-Klasse.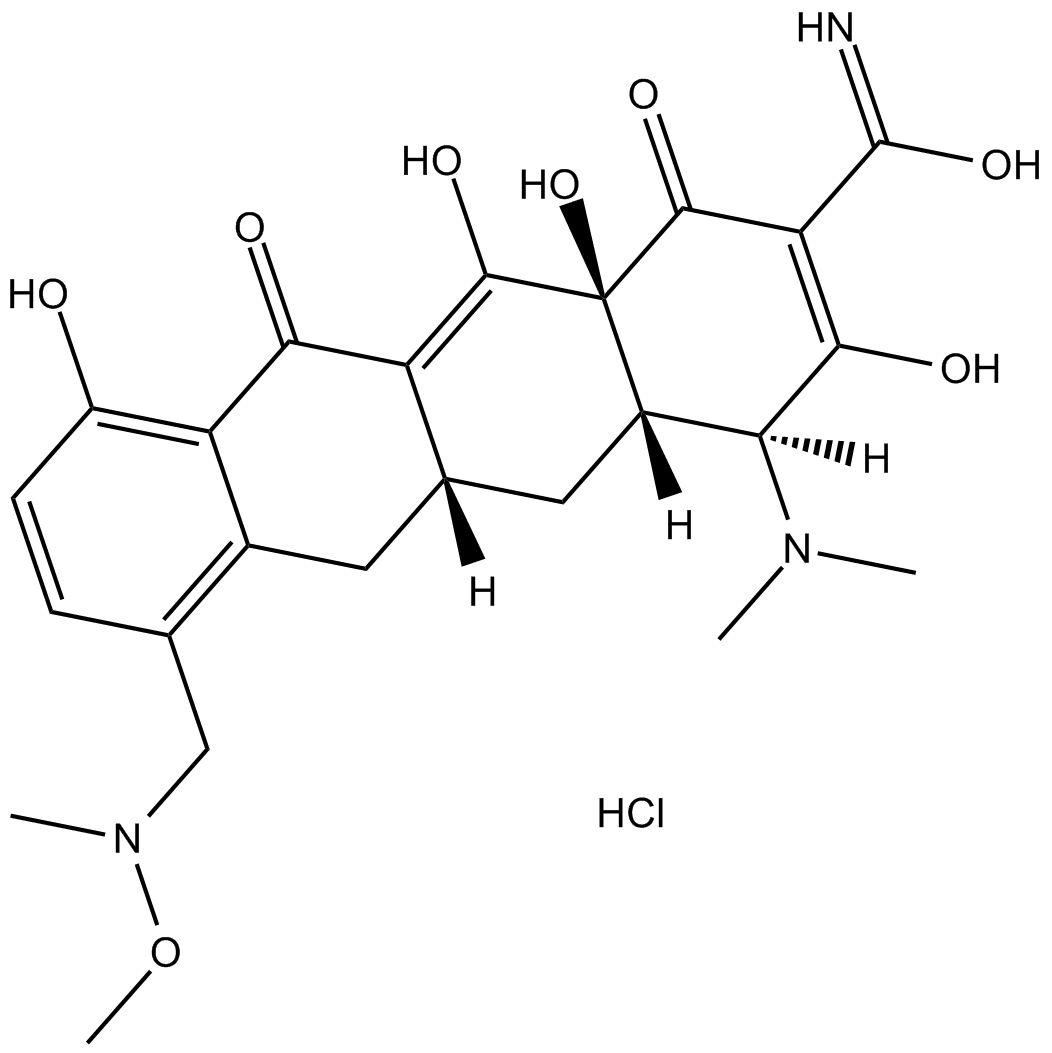
-
GC12011
P276-00
P276-00
P276-00 (P276-00) ist ein potenter Cyclin-abhÄngiger Kinase (CDK)-Inhibitor, der CDK9-CyclinT1, CDK4-Cyclin D1 und CDK1-CyclinB mit IC50-Werten von 20 nM, 63 nM bzw. 79 nM hemmt. P276-00 (P276-00) zeigt AntitumoraktivitÄt auf Cisplatin-resistente Zellen.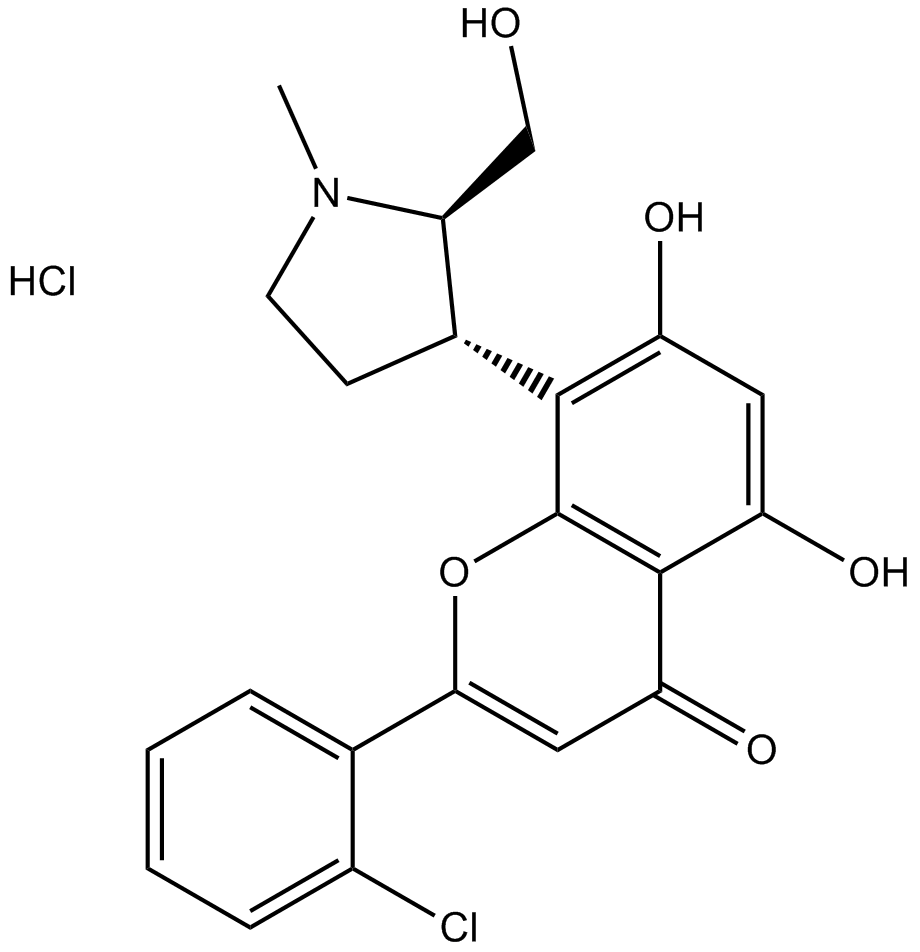
-
GC15421
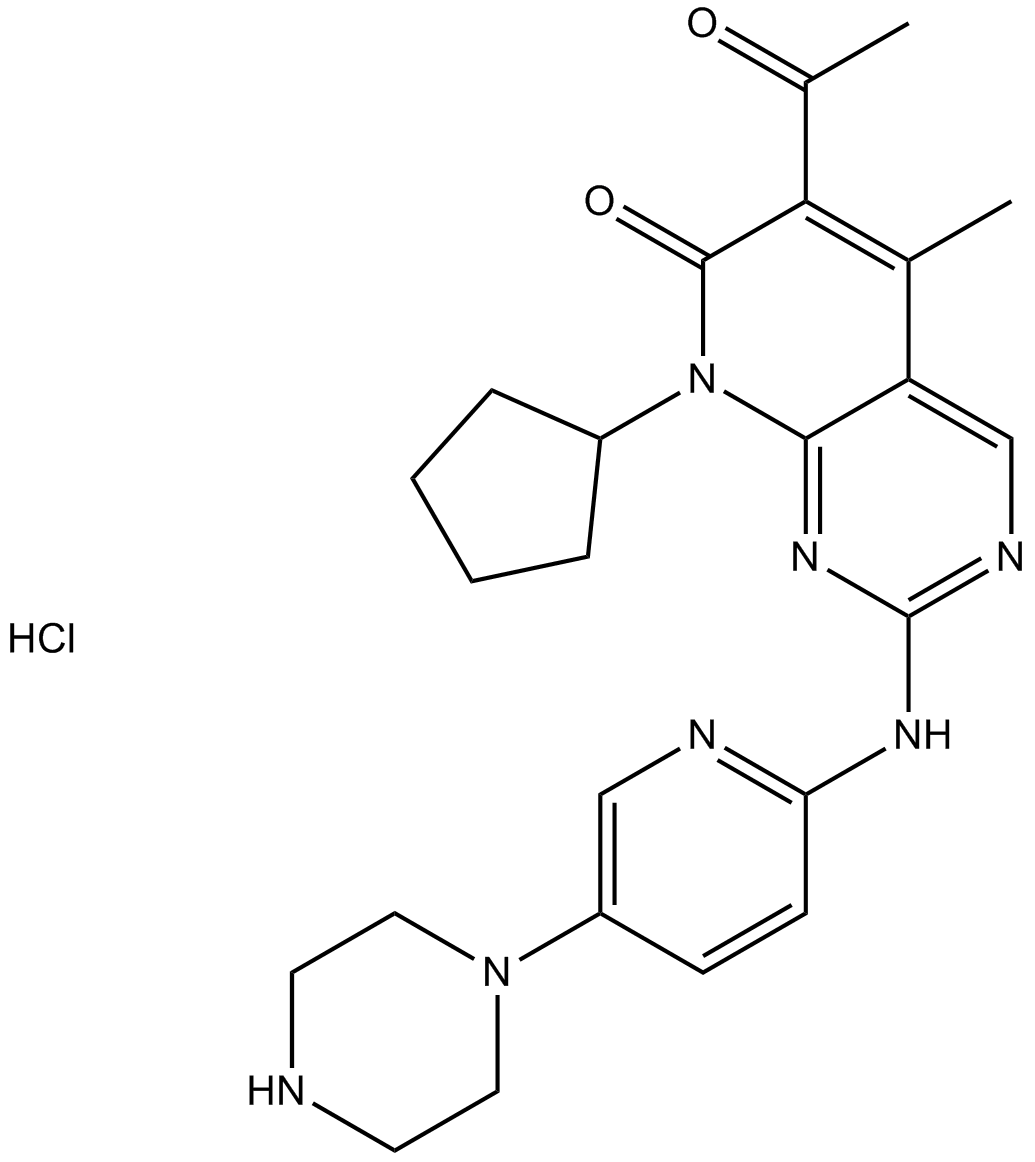
Palbociclib (PD0332991) Isethionate
Palbociclib Isethionate
Palbociclib (PD 0332991) Isethionat ist ein oral wirksamer selektiver CDK4- und CDK6-Inhibitor mit IC50-Werten von 11 bzw. 16 nM. Palbociclib (PD0332991) Isethionat hat eine starke antiproliferative AktivitÄt und induziert einen Zellzyklusarrest in Krebszellen, was bei der Erforschung von HR-positivem und HER2-negativem Brustkrebs und hepatozellulÄrem Karzinom verwendet werden kann.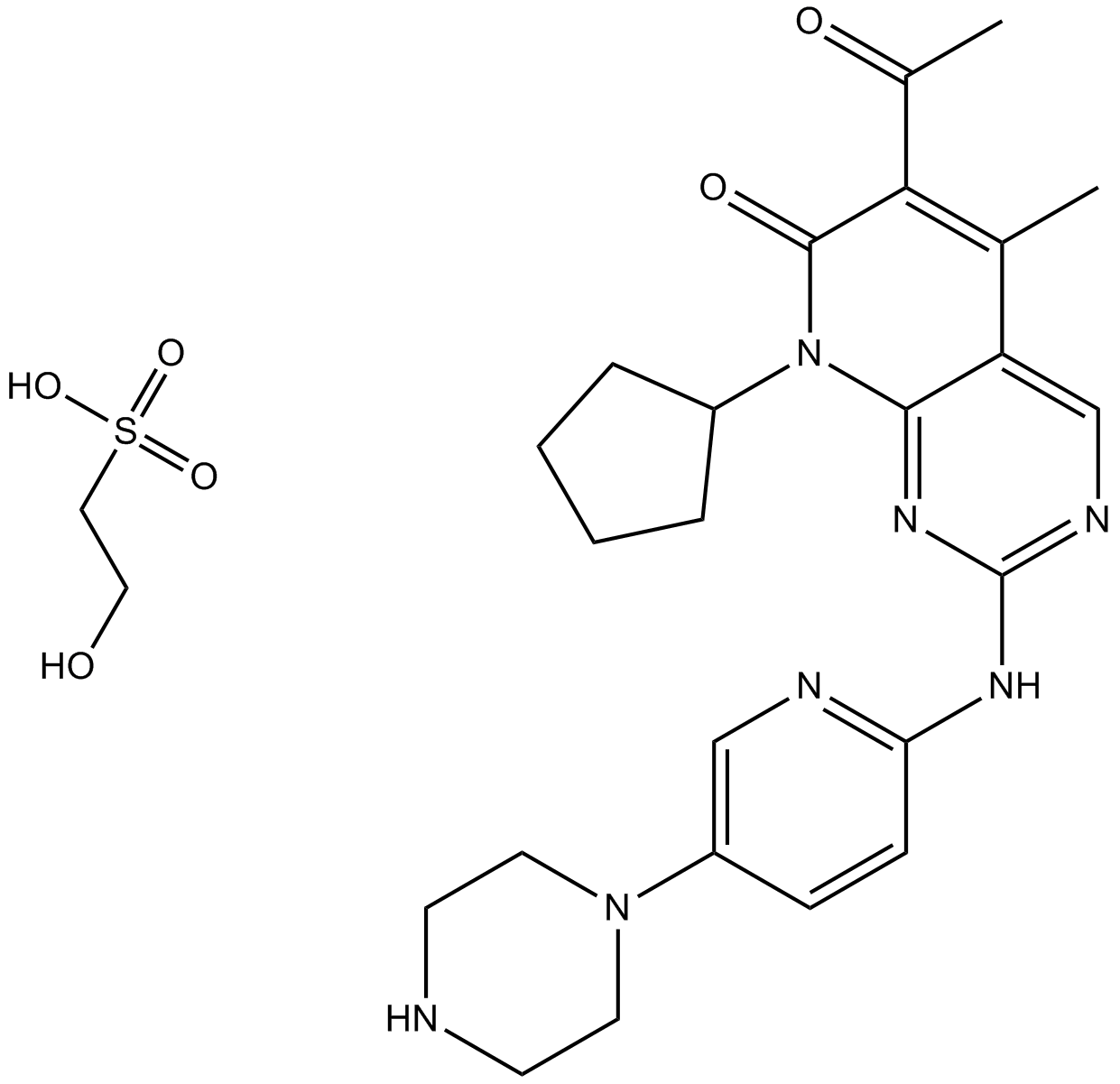
-
GC14762
Palifosfamide
Isophosphoramide Mustard, NSC 297900
An active metabolite of ifosfamide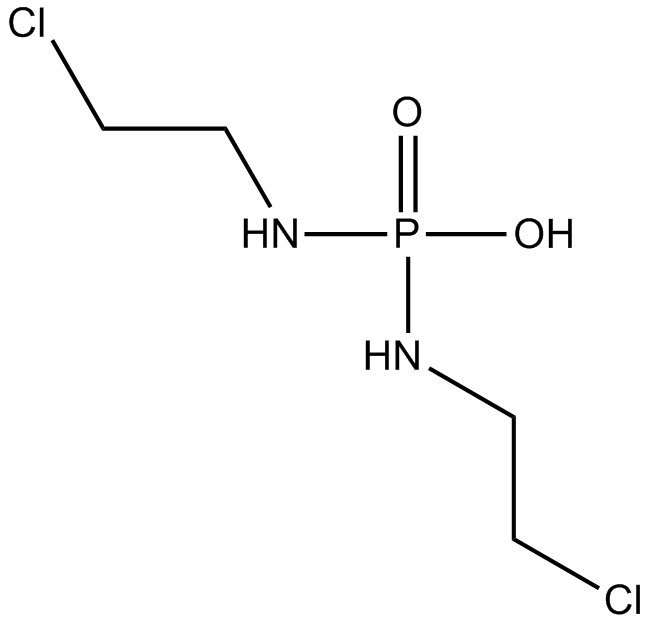
-
GC34071
Pamiparib (BGB-290)
BGB-290
Pamiparib (BGB-290) (BGB-290) ist ein oral aktiver, potenter, hochselektiver PARP-Inhibitor mit IC50-Werten von 0,9 nM und 0,5 nM fÜr PARP1 bzw. PARP2. Pamiparib (BGB-290) hat einen starken PARP-Einfang und die FÄhigkeit, in das Gehirn einzudringen, und kann fÜr die Erforschung verschiedener Krebsarten, einschließlich des soliden Tumors, verwendet werden.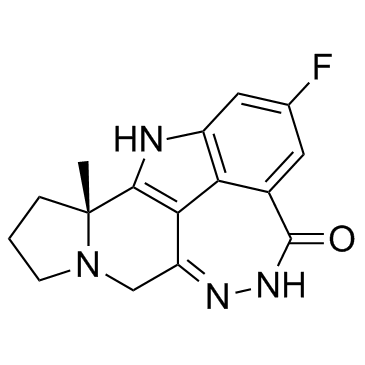
-
GC14849
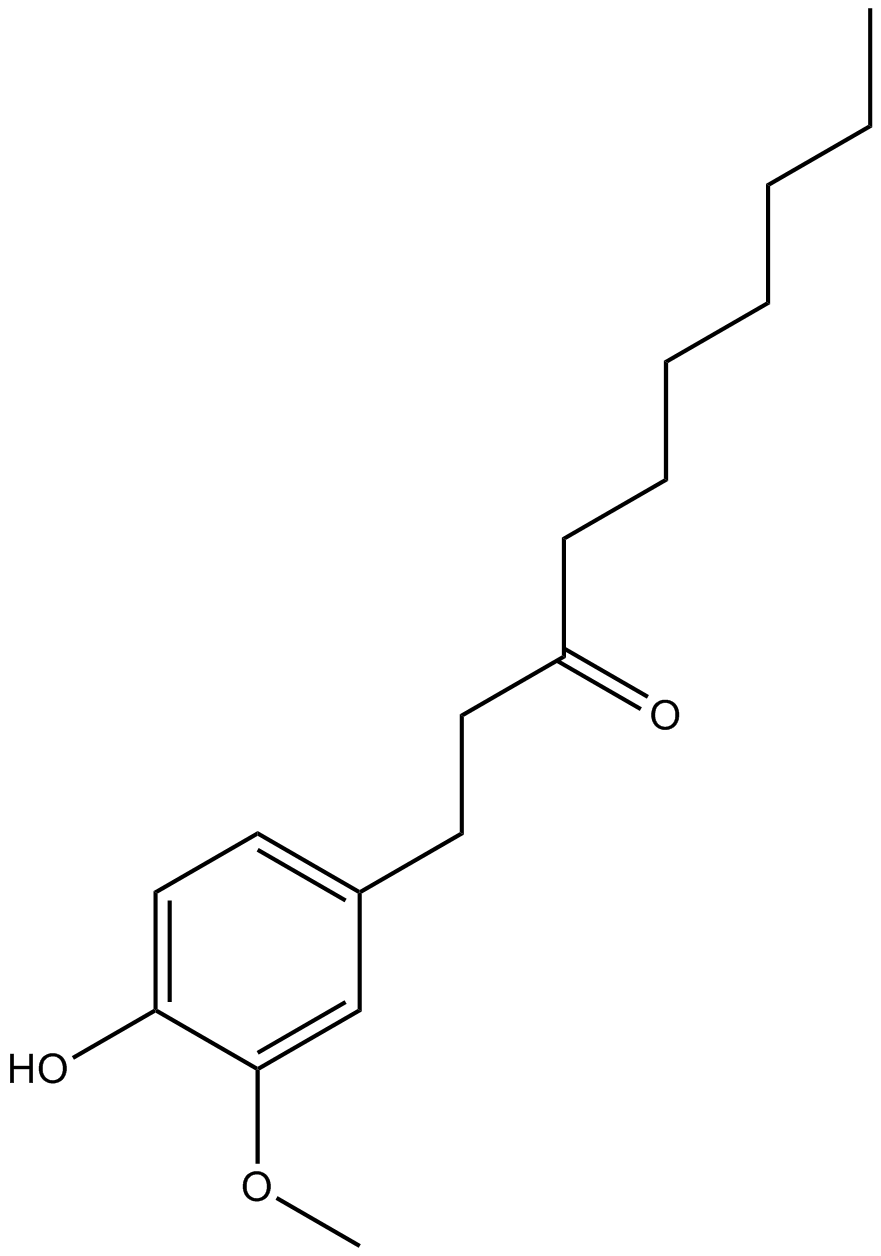
Paradol
A phenolic ketone with diverse biological activities
-
GC36855
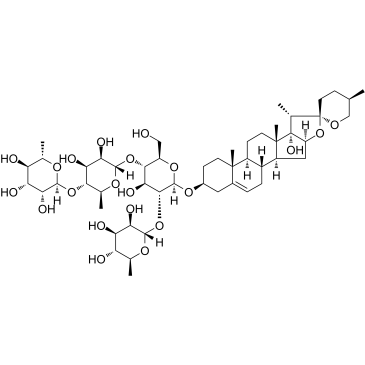
Paris saponin VII
Paris Saponin VII (Chonglou Saponin VII) ist ein steroidales Saponin, das aus den Wurzeln und Rhizomen von Trillium tschonoskii Maxim isoliert wird. Paris-Saponin-VII-induzierte Apoptose in K562/ADR-Zellen ist mit Akt/MAPK und der Hemmung von P-gp assoziiert. Paris-Saponin VII schwÄcht das mitochondriale Membranpotential ab, erhÖht die Expression von Apoptose-verwandten Proteinen wie Bax und Cytochrom c und verringert die Proteinexpressionsniveaus von Bcl-2, Caspase-9, Caspase-3, PARP-1 und p- Akt. Paris-Saponin VII induziert eine robuste Autophagie in K562/ADR-Zellen und liefert eine biochemische Grundlage fÜr die Behandlung von LeukÄmie.
-
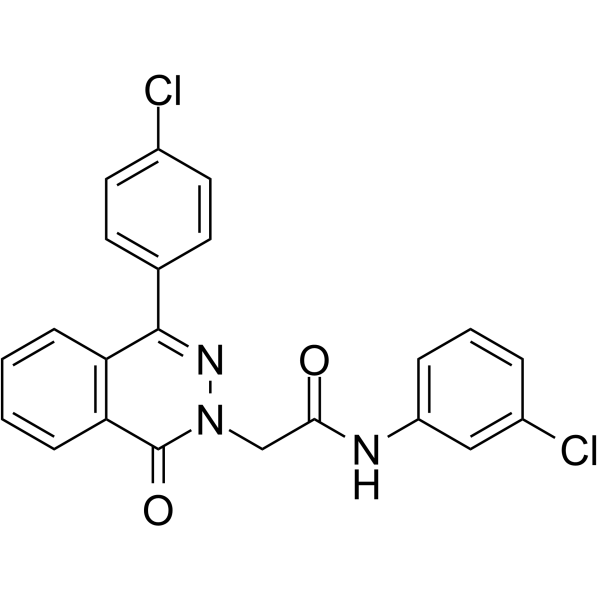
GC64579
PARP-1-IN-2
PARP-1-IN-2 (Verbindung 11g) ist ein potenter und BBB-penetrierter PARP1-Inhibitor mit einem IC50 von 149 nM. PARP1-IN-2 zeigt eine signifikant starke antiproliferative AktivitÄt gegen die humane Lungenadenokarzinom-Epithelzelllinie A549. PARP1-IN-2 kann die Apoptose von A549-Zellen induzieren.
-
GC65927
PARP-2-IN-1
PARP-2-IN-1 ist ein potenter und selektiver PARP-2-Inhibitor mit einem IC50 von 11,5 nM.
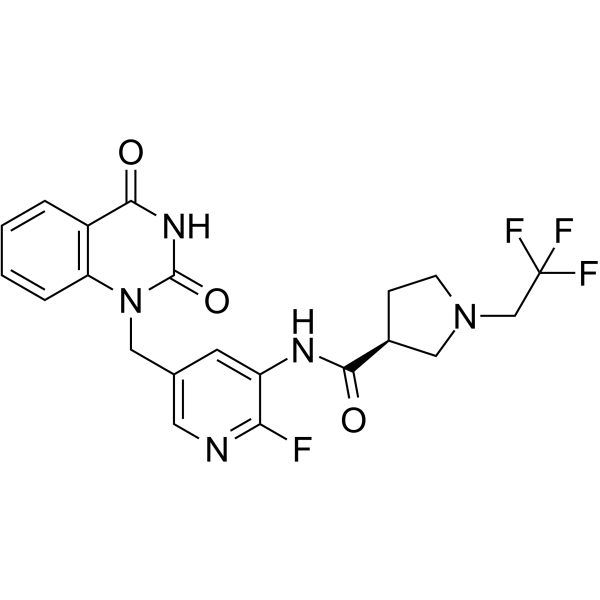
-
GC68006
PARP1-IN-11
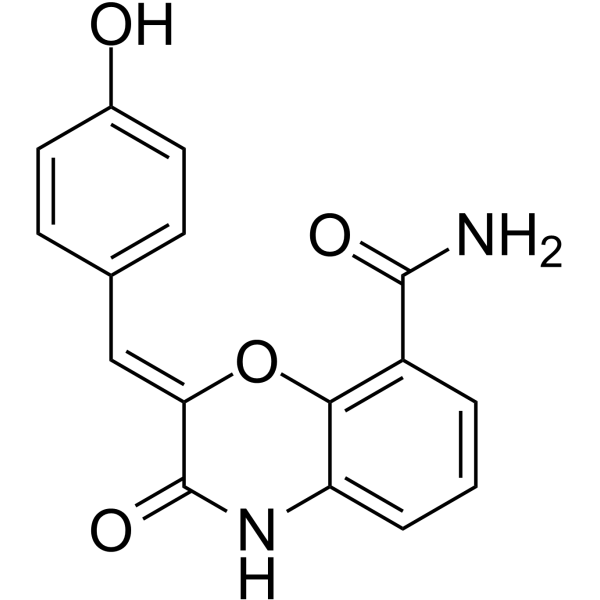
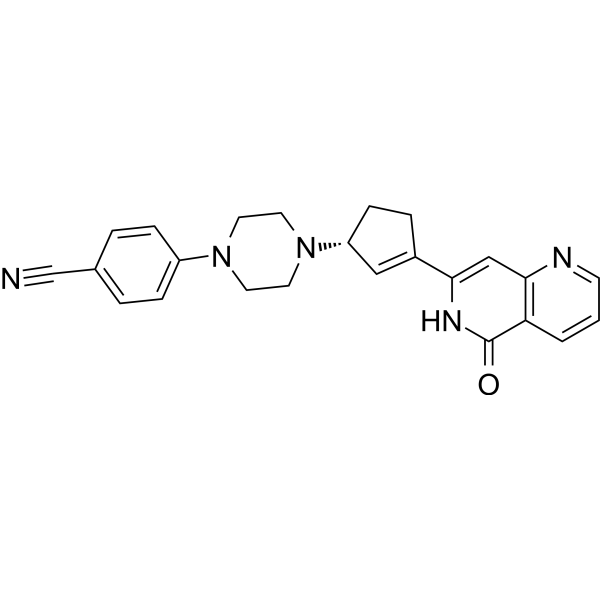
-
GC69660
PARP1-IN-7
PARP1-IN-7 is an inhibitor of poly ADP-ribose polymerase-1 (PARP1) and serves as an anticancer agent.
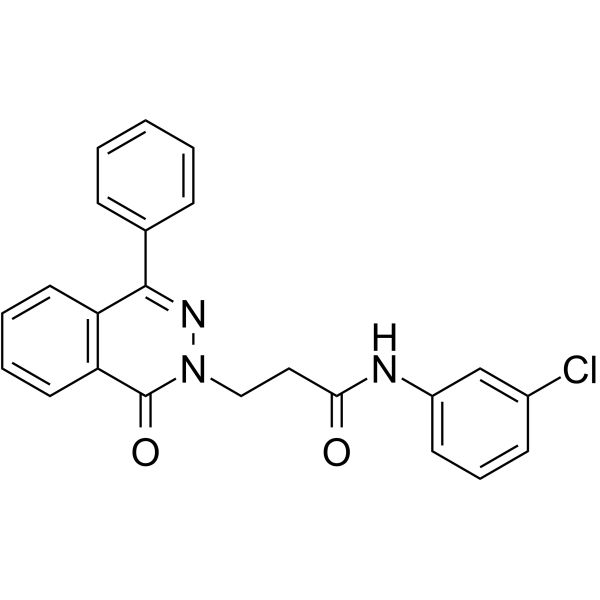
-
GC64578
PARP1-IN-8
PARP1-IN-8 (Verbindung 11c) ist ein potenter und BBB-penetrierter PARP1-Inhibitor mit einem IC50 von 97 nM. PARP1-IN-8 zeigt eine signifikant starke antiproliferative AktivitÄt gegen die humane Lungenadenokarzinom-Epithelzelllinie A549.
-
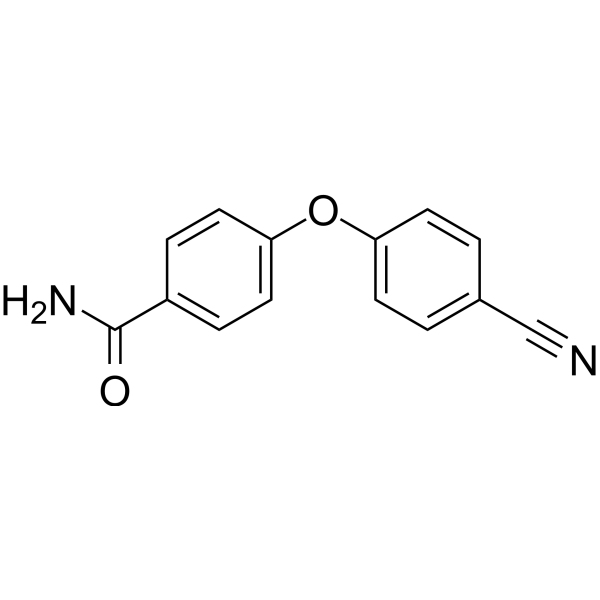
GC69657
PARP10-IN-2
PARP10-IN-2 ist ein wirksamer Inhibitor der Einzel-ADP-Ribosyltransferase PARP10 mit einer IC50 von 3,64 μM für menschliche PARP10. PARP10-IN-2 hemmt auch PARP2 und PARP15, mit einer IC50 von jeweils 27 μM für menschliche PARP2 und 11 μM für menschliche PARP15.
-
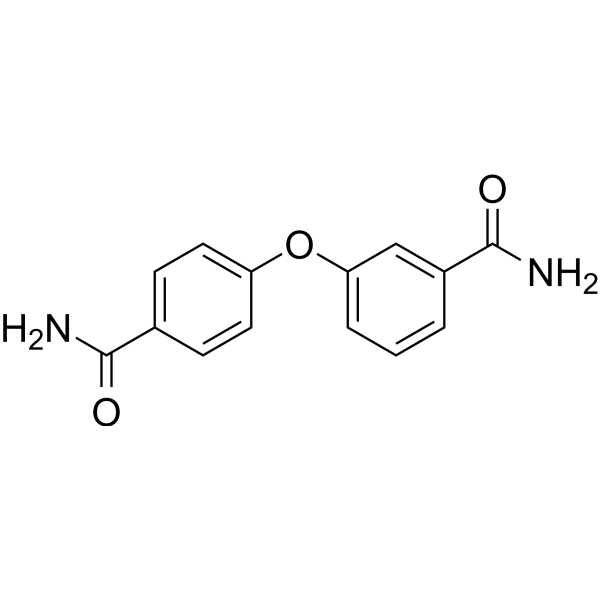
GC69658
PARP10-IN-3
PARP10-IN-3 ist ein selektiver PARP10-Inhibitor, der die Einzel-ADP-Ribosyltransferase hemmt. Die IC50 für menschliche PARP10 beträgt 480 nM. PARP10-IN-3 inhibiert auch PARP2 und PARP15 mit einer IC50 von jeweils 1,7 μM bei Menschen.
-
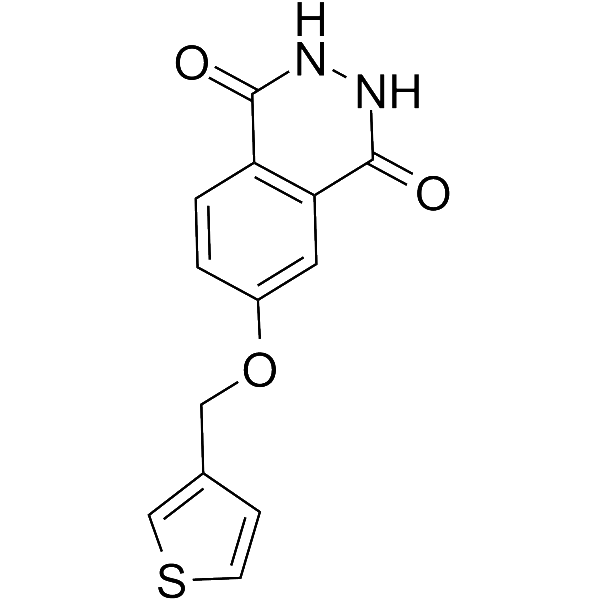
GC68035
PARP10/15-IN-1
-
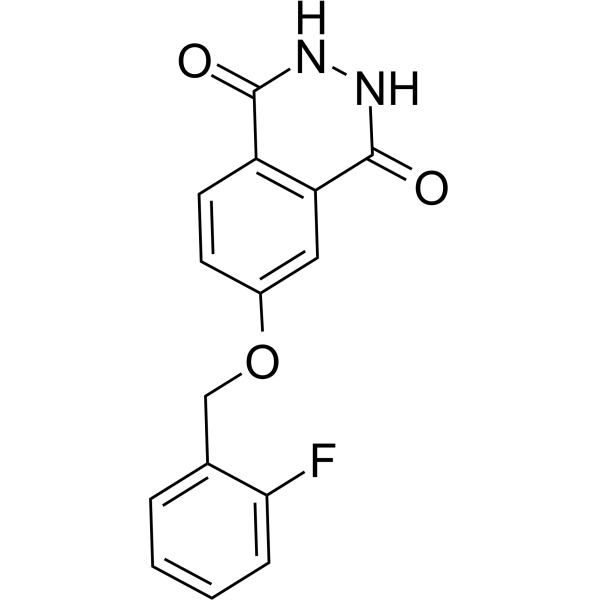
GC69655
PARP10/15-IN-2
PARP10/15-IN-2 (Verbindung 8h) ist ein wirksamer PARP10- und PARP15-Doppelinhibitor mit IC50-Werten von jeweils 0,15 µM und 0,37 µM. PARP10/15-IN-2 kann in Zellen eindringen und Apoptose verhindern.
-
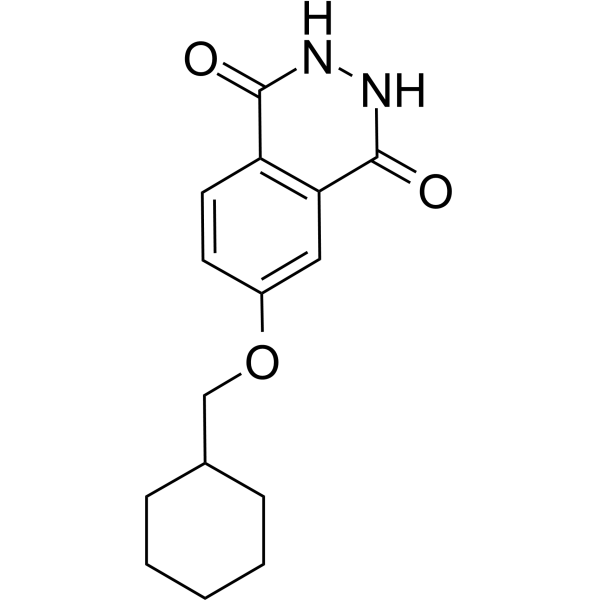
GC69656
PARP10/15-IN-3
PARP10/15-IN-3 (Verbindung 8a) ist ein wirksamer PARP10- und PARP15-Dualhemmer mit IC50-Werten von jeweils 0,14 µM und 0,40 µM. PARP10/15-IN-3 kann in Zellen eindringen und Apoptose verhindern.
-
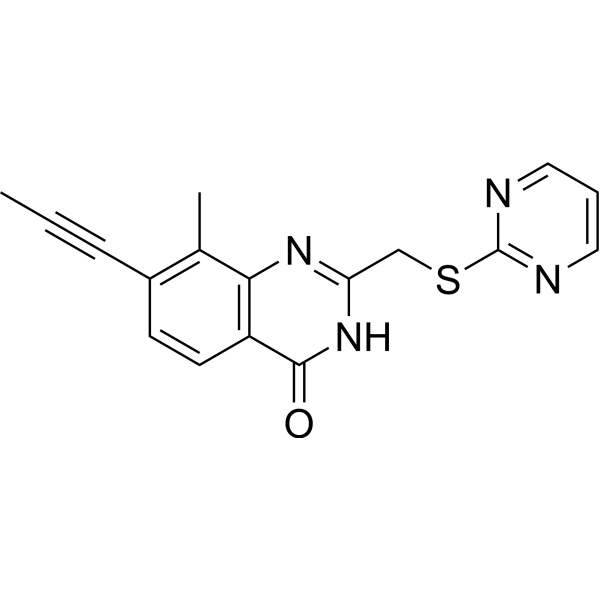
GC69659
PARP11 inhibitor ITK7
ITK7
Der PARP11-Inhibitor ITK7 (ITK7) ist ein wirksamer und selektiver Inhibitor von PARP11. Der IC50-Wert von PARP11-Inhibitor ITK7 zur effektiven Hemmung von PARP11 beträgt 14 nM. Der PARP11-Inhibitor ITK7 kann für die Erforschung der zellulären Lokalisierung verwendet werden.
-
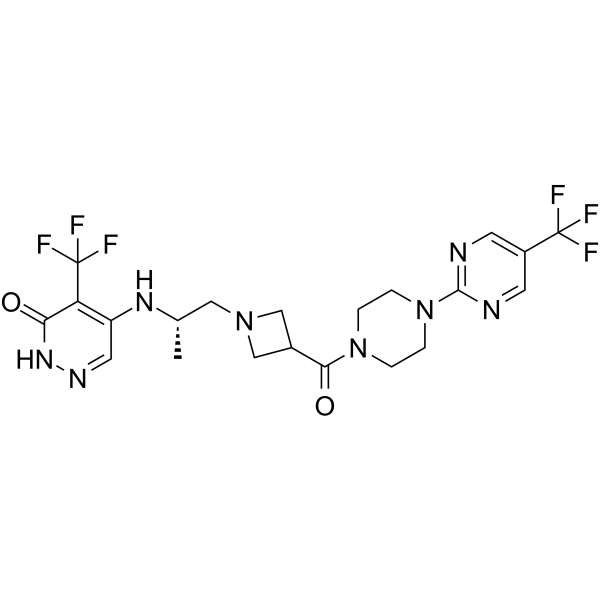
GC69661
PARP7-IN-14
PARP7-IN-14 (I-1) is an effective selective PARP7 inhibitor with an IC50 value of 7.6 nM. PARP7-IN-14 has anti-cancer activity.
-
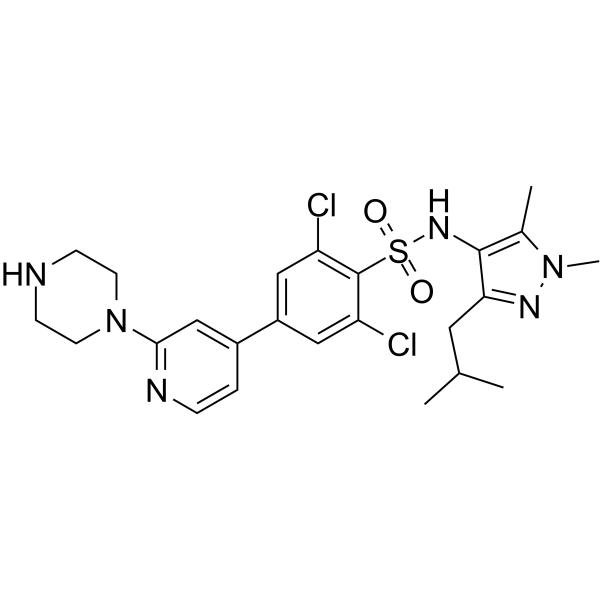
GC65891
PCLX-001
PCLX-001 ist ein oral aktiver, niedermolekularer, dualer N-Myristoyltransferase (NMT)-Hemmer mit IC50-Werten von 5 nM (NMT1) bzw. 8 nM (NMT2). PCLX-001 zeigt Antitumor-AktivitÄt und hemmt die frÜhe B-Zell-Rezeptor(BCR)-SignalÜbertragung und kann fÜr die Erforschung von bÖsartigen B-Zell-Erkrankungen verwendet werden.
-
GC17935
PD 0332991 (Palbociclib) HCl
PD0332991;PD-0332991;PD 0332991
Palbociclib (PD 0332991) Monohydrochlorid ist ein oral aktiver selektiver CDK4- und CDK6-Inhibitor mit IC50-Werten von 11 bzw. 16 nM. PD 0332991 (Palbociclib) HCl hat eine starke antiproliferative AktivitÄt und induziert einen Zellzyklusarrest in Krebszellen, was bei der Erforschung von HR-positivem und HER2-negativem Brustkrebs und hepatozellulÄrem Karzinom verwendet werden kann.