Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
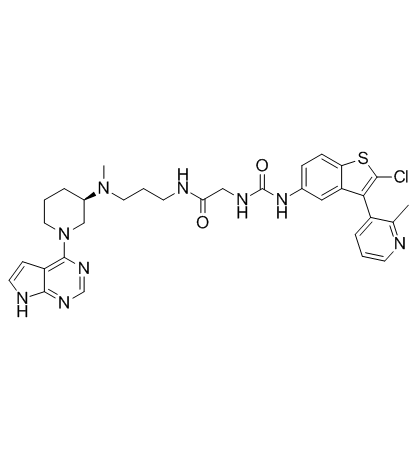
GC33028
CF53
CF53 es un inhibidor muy potente, selectivo y activo por vÍa oral de la proteÍna BET, con una Ki de <1 nM, una Kd de 2,2 nM y una IC50 de 2 nM para BRD4 BD1. CF53 se une a los dominios BD1 y BD2 de las proteÍnas BRD2, BRD3, BRD4 y BRDT BET con altas afinidades, muy selectivas sobre las proteÍnas que no contienen bromodominio BET. CF53 muestra una potente actividad antitumoral tanto in vitro como in vivo.
-
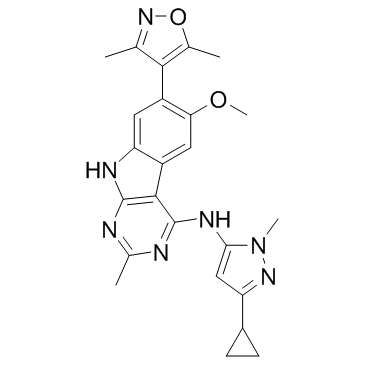
GC65602
CFT8634
CFT8634 es un degradador dirigido a BRD9 extraÍdo del compuesto 174 de la patente WO2021178920A1. CFT8634 se puede utilizar para la investigaciÓn de sarcoma sinovial y tumores sÓlidos con eliminaciÓn de SMARCB1.
-
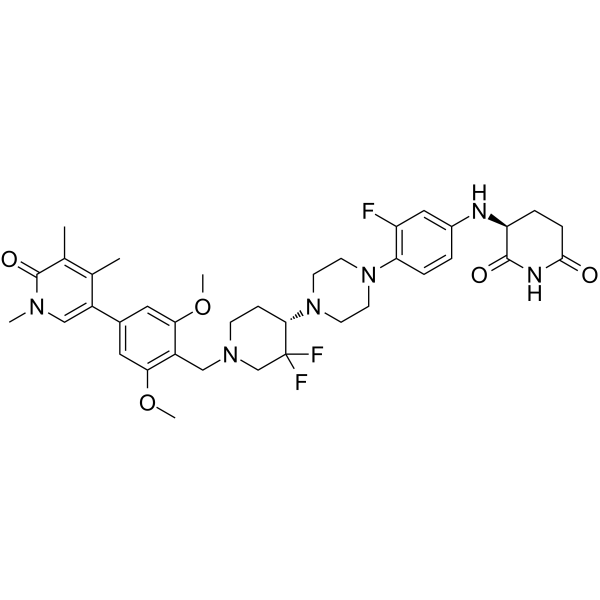
GC35668
CG-200745
CG-200745 (CG-200745) es un potente inhibidor pan-HDAC activo por vÍa oral que tiene la fracciÓn de Ácido hidroxÁmico para unirse al zinc en la parte inferior de la bolsa catalÍtica. CG-200745 inhibe la desacetilaciÓn de histona H3 y tubulina. CG-200745 induce la acumulaciÓn de p53, promueve la transactivaciÓn dependiente de p53 y mejora la expresiÓn de las proteÍnas MDM2 y p21 (Waf1/Cip1). CG-200745 mejora la sensibilidad de las células resistentes a la gemcitabina a la gemcitabina y al 5-fluorouracilo (5-FU; ). CG-200745 induce apoptosis y tiene efectos antitumorales.
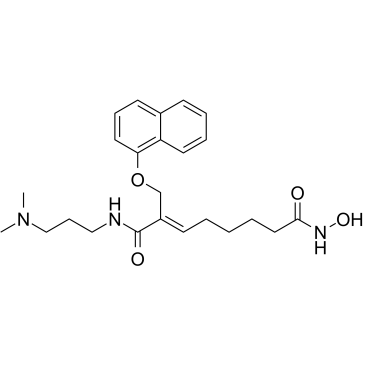
-
GC39679
CG347B
CG347B es un inhibidor selectivo de HDAC6, también participa en la sÍntesis de otros inhibidores de metaloenzimas.
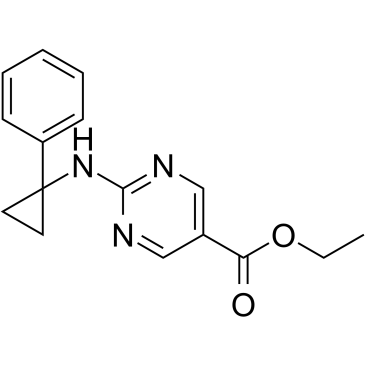
-
GC14936
Chaetocin
Inhibitor of lys9-specific HMTs
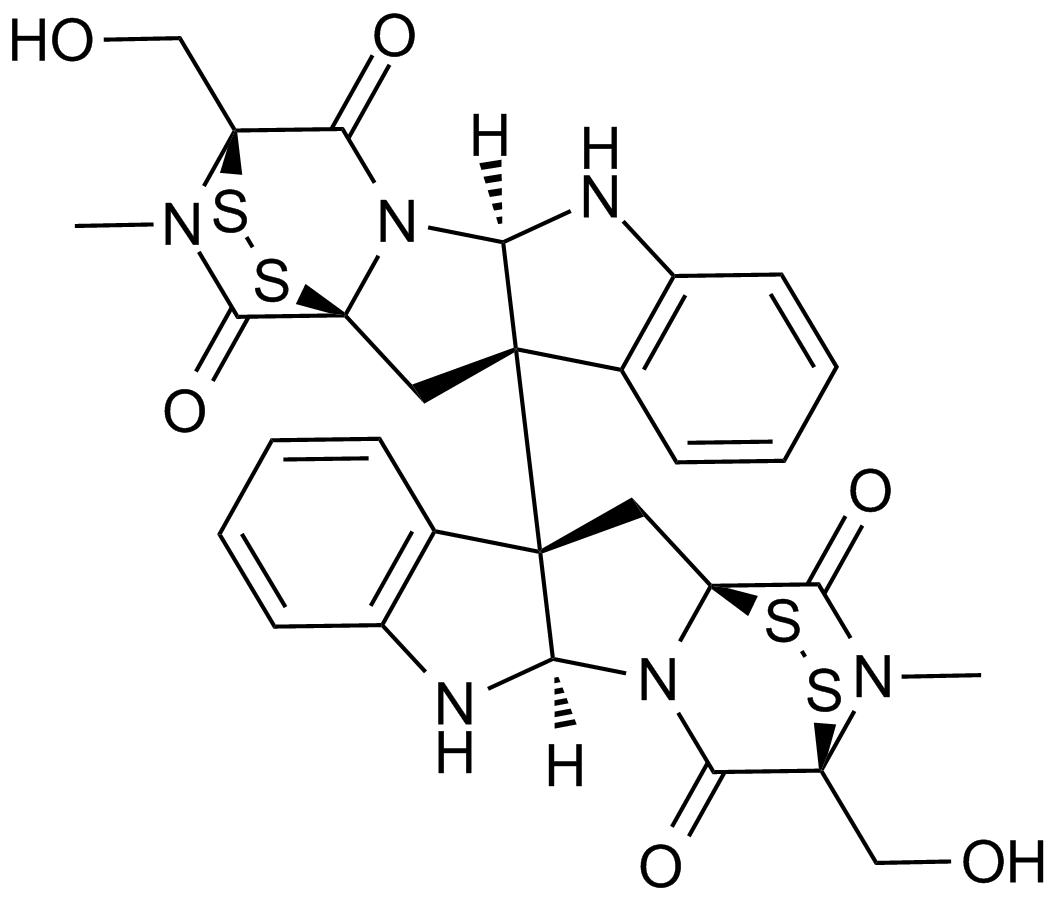
-
GC11975
CHAPS
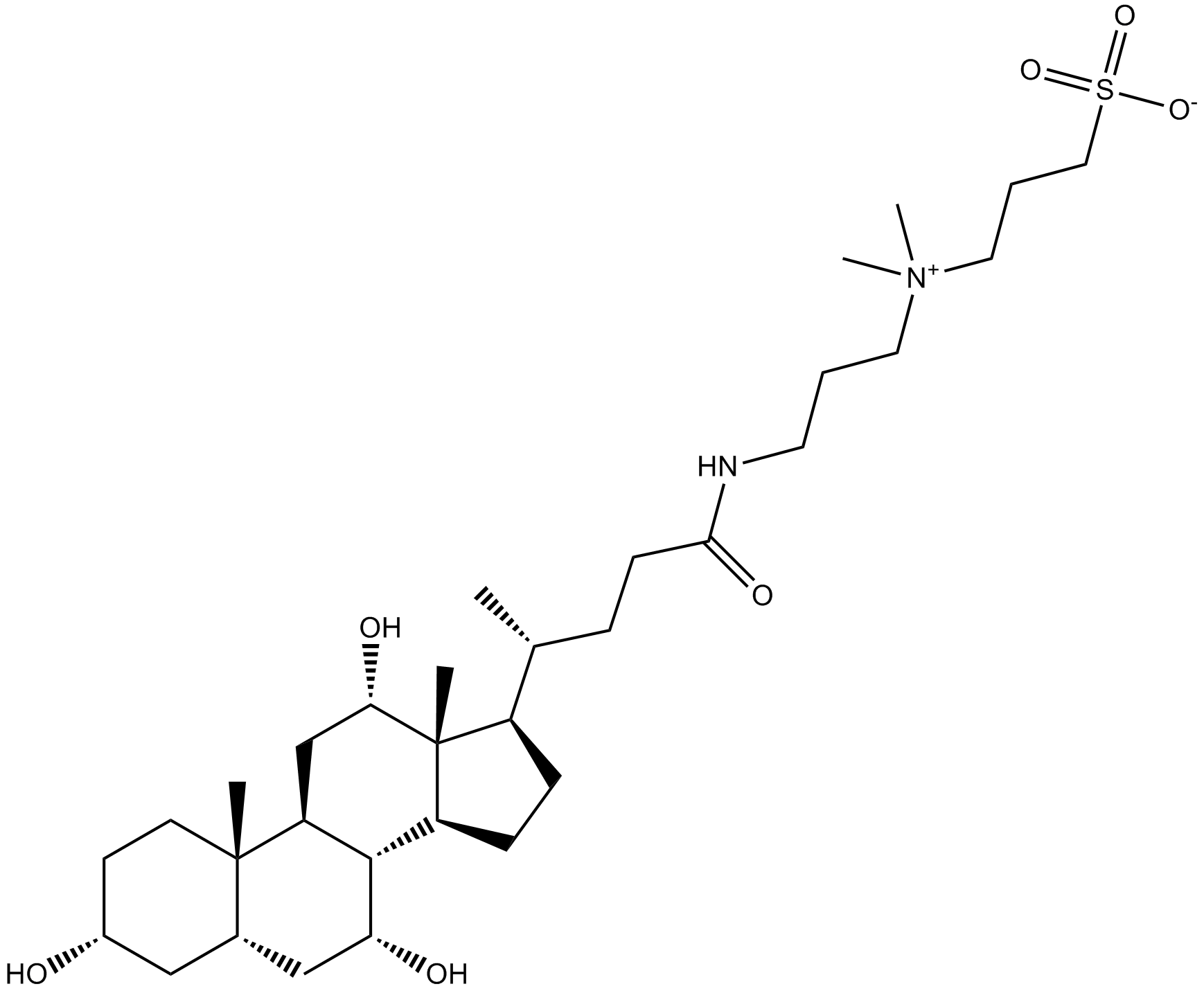
-
GC64942
CHDI-390576
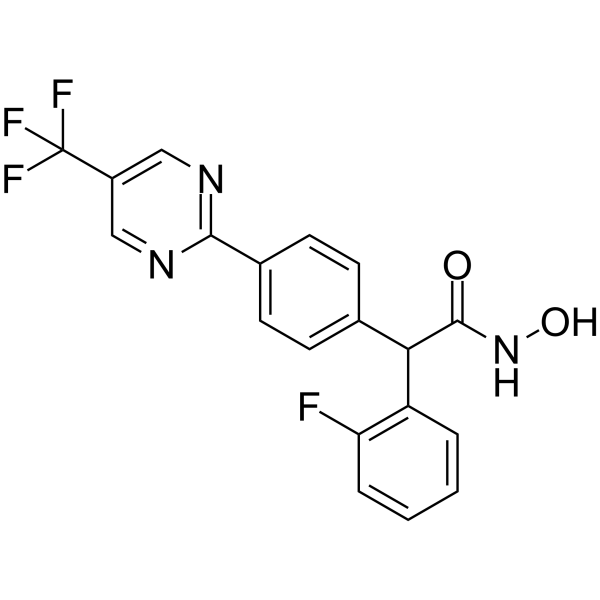
CHDI-390576, un potente inhibidor de histona desacetilasa de clase IIa (HDAC) permeable a las células y penetrante en el SNC con IC50 de 54 nM, 60 nM, 31 nM, 50 nM para clase IIa HDAC4, HDAC5, HDAC7, HDAC9, respectivamente, muestra >500 -selectividad de veces sobre HDAC de clase I (1, 2, 3) y selectividad de ~ 150 veces sobre HDAC8 y la isoforma clase IIb HDAC6.
-
GC17405
Chetomin
An inhibitor of HIF signaling
-
GC62145
Chiauranib
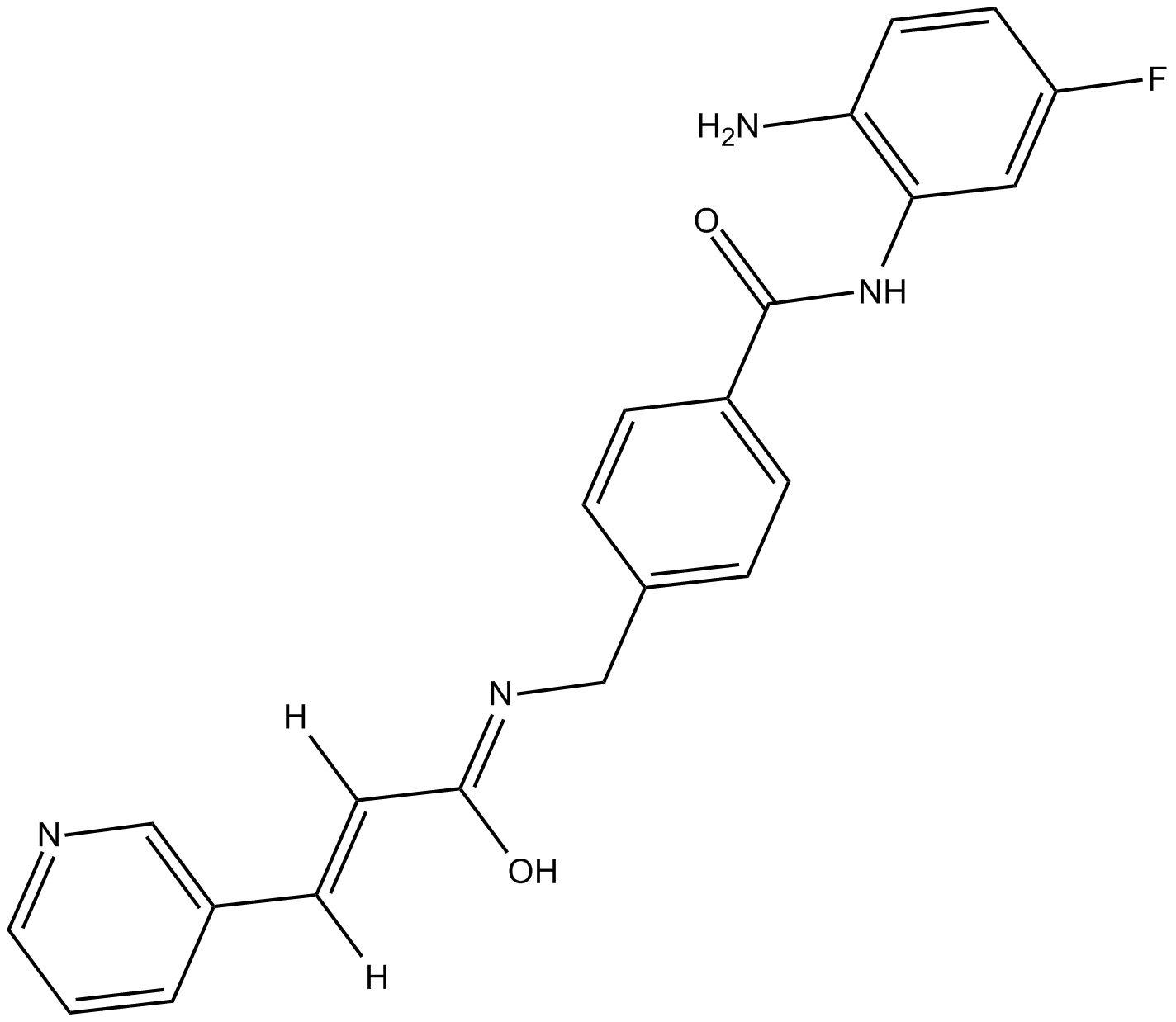
Chiauranib (CS2164) es un inhibidor multidiana activo por vÍa oral contra la angiogénesis tumoral. Chiauranib inhibe potentemente las quinasas relacionadas con la angiogénesis (VEGFR1, VEGFR2, VEGFR3, PDGFRα y c-Kit), la quinasa relacionada con la mitosis Aurora B y la quinasa relacionada con la inflamaciÓn crÓnica CSF-1R, con valores de IC50 que oscilan entre 1 y 9 nM. Chiauranib tiene fuertes efectos anticancerÍgenos.

-
GC64255
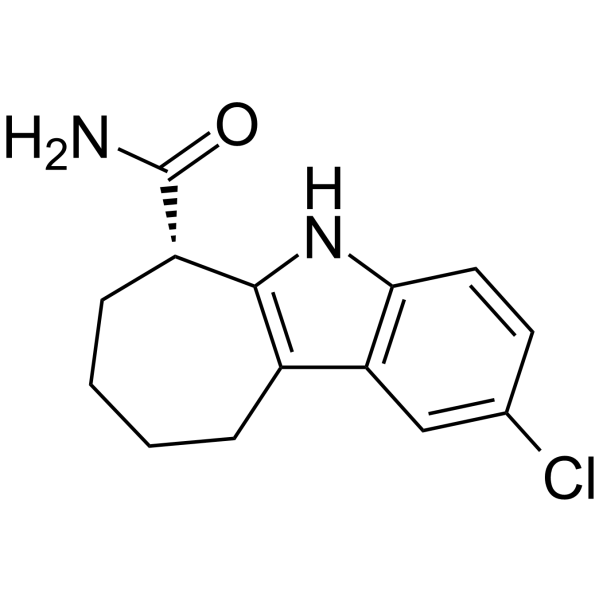
CHIC35
CHIC35, un anÁlogo de EX-527, es un inhibidor potente y selectivo de SIRT1 (IC50=0,124 μM).
-
GC16042
Chidamide
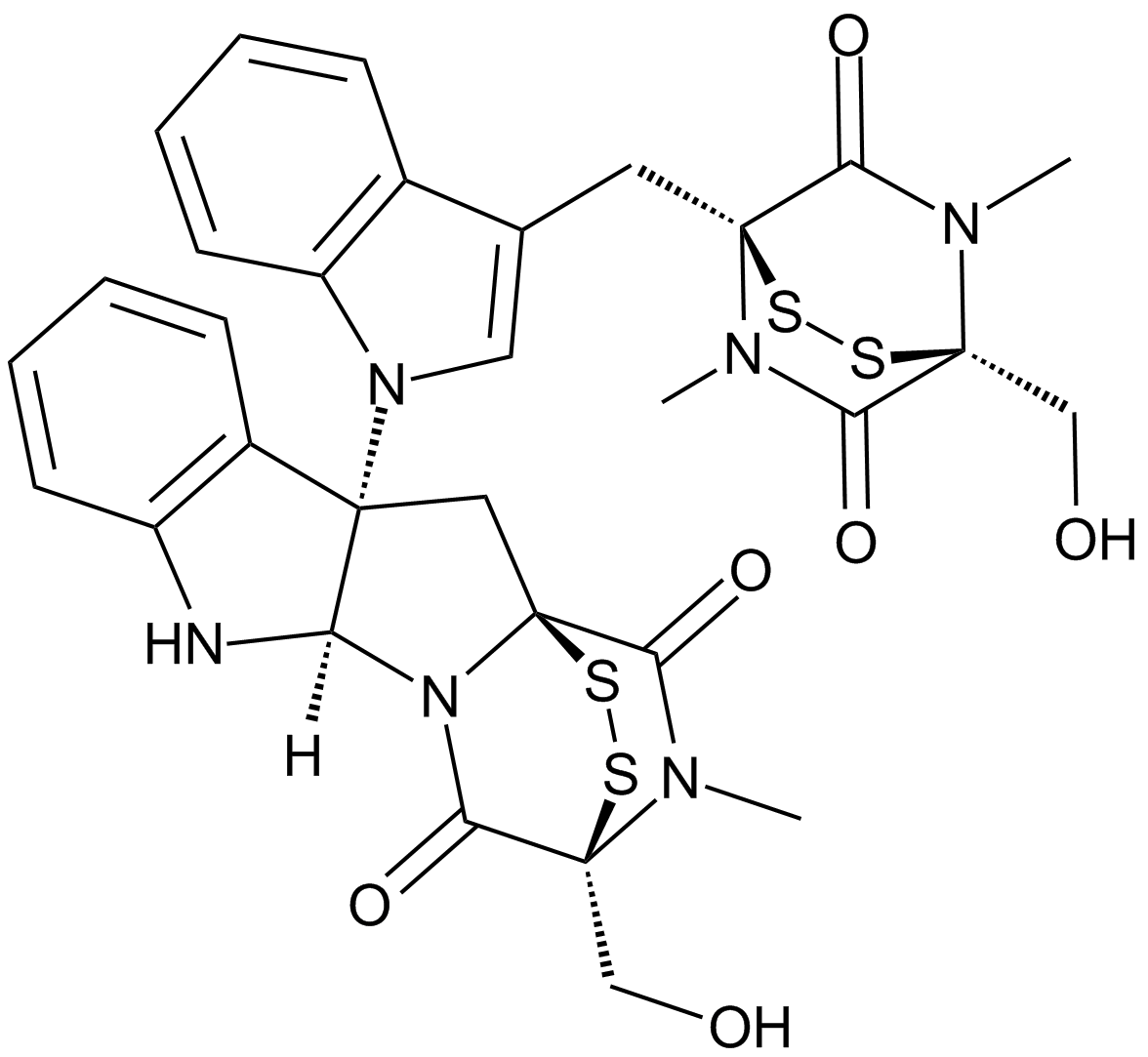
Chidamide (impureza de Chidamide) es una impureza de Chidamide. La chidamida es un inhibidor potente y biodisponible por vÍa oral de las enzimas HDAC clase I (HDAC1/2/3) y clase IIb (HDAC10).
-
GN10518
Chitosamine hydrochloride
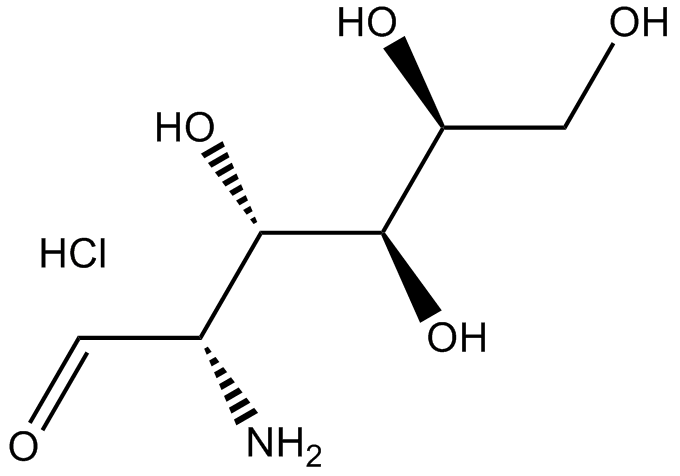
-
GC45717
Chlamydocin
La clamidocina, un metabolito fÚngico, es un inhibidor de HDAC muy potente, con una IC50 de 1,3 nM. La clamidocina exhibe potentes actividades antiproliferativas y anticancerÍgenas. La clamidocina induce la apoptosis al activar la caspasa-3.
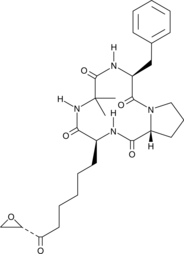
-
GN10308
Chlorogenic acid
El ácido clorogénico es un compuesto fenólico importante en el café y el té.
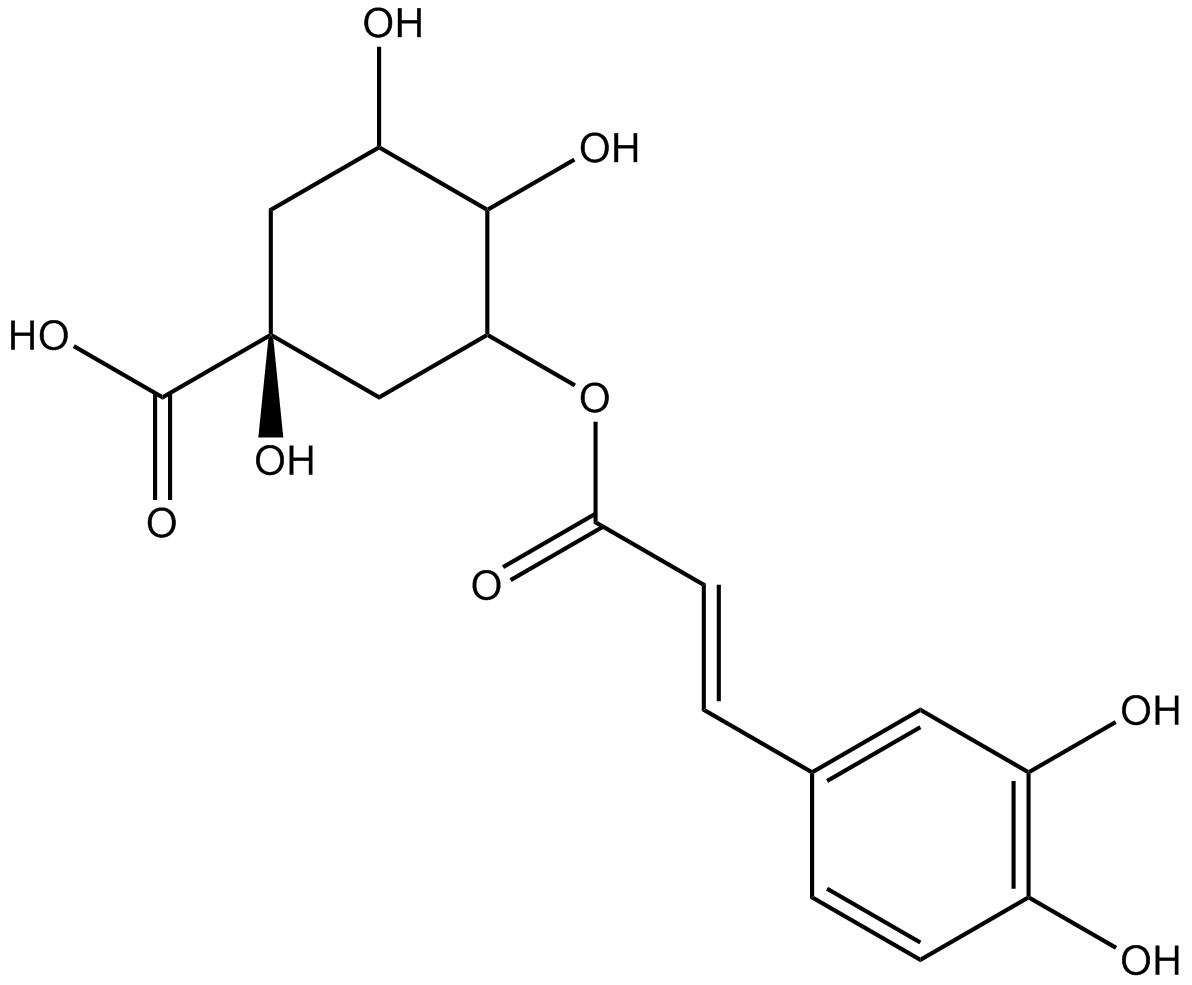
-
GC11652
CHZ868
CHZ868 es un inhibidor de JAK2 de tipo II con una IC50 de 0,17 μM en células EPOR JAK2 WT Ba/F3.
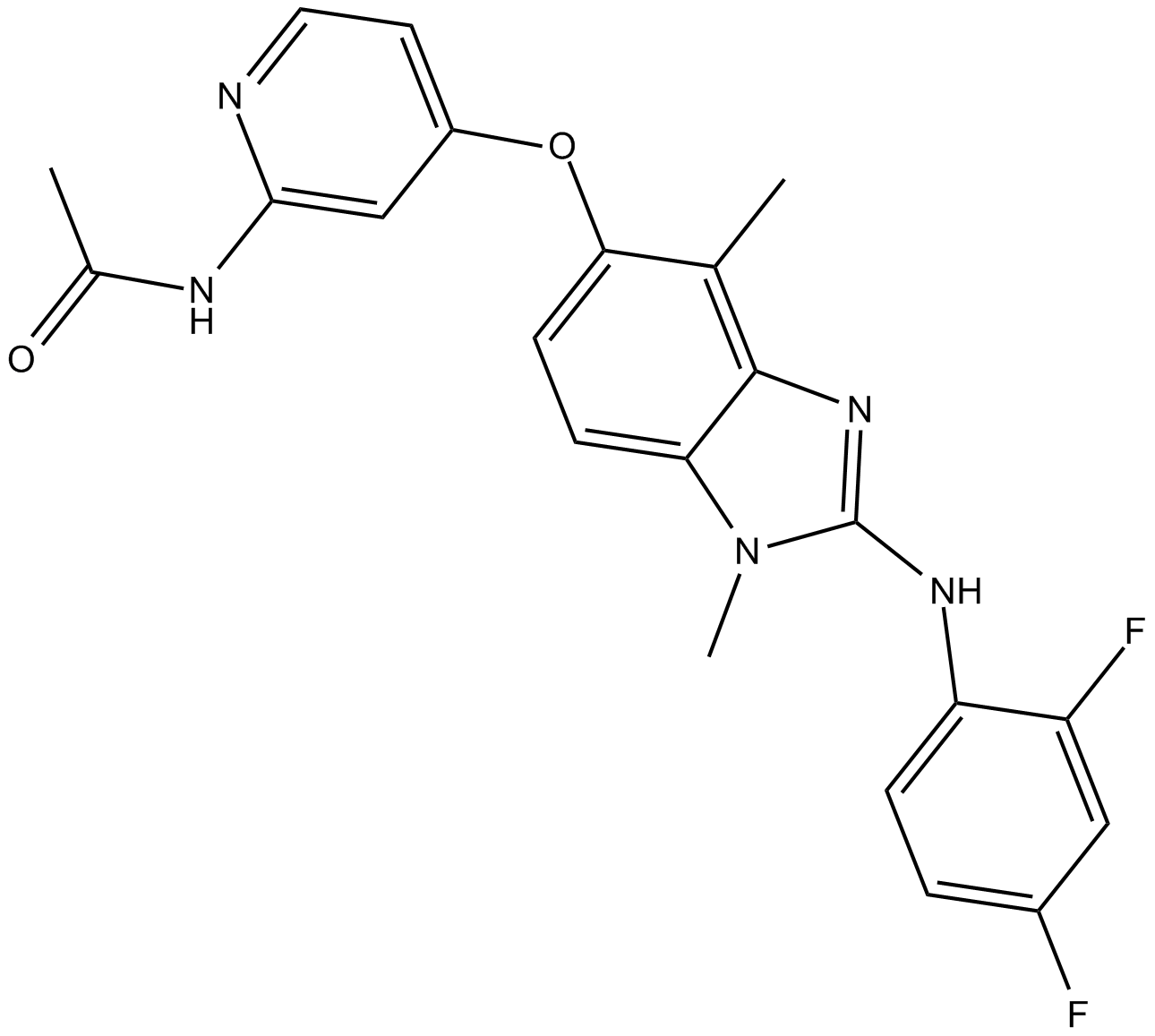
-
GC11539
CI 976
CI 769 (CI 769) es un inhibidor potente, oralmente activo y selectivo de ACAT (acil coenzima A: colesterol aciltransferasa) con una IC50 de 73 nM.
-
GC13408
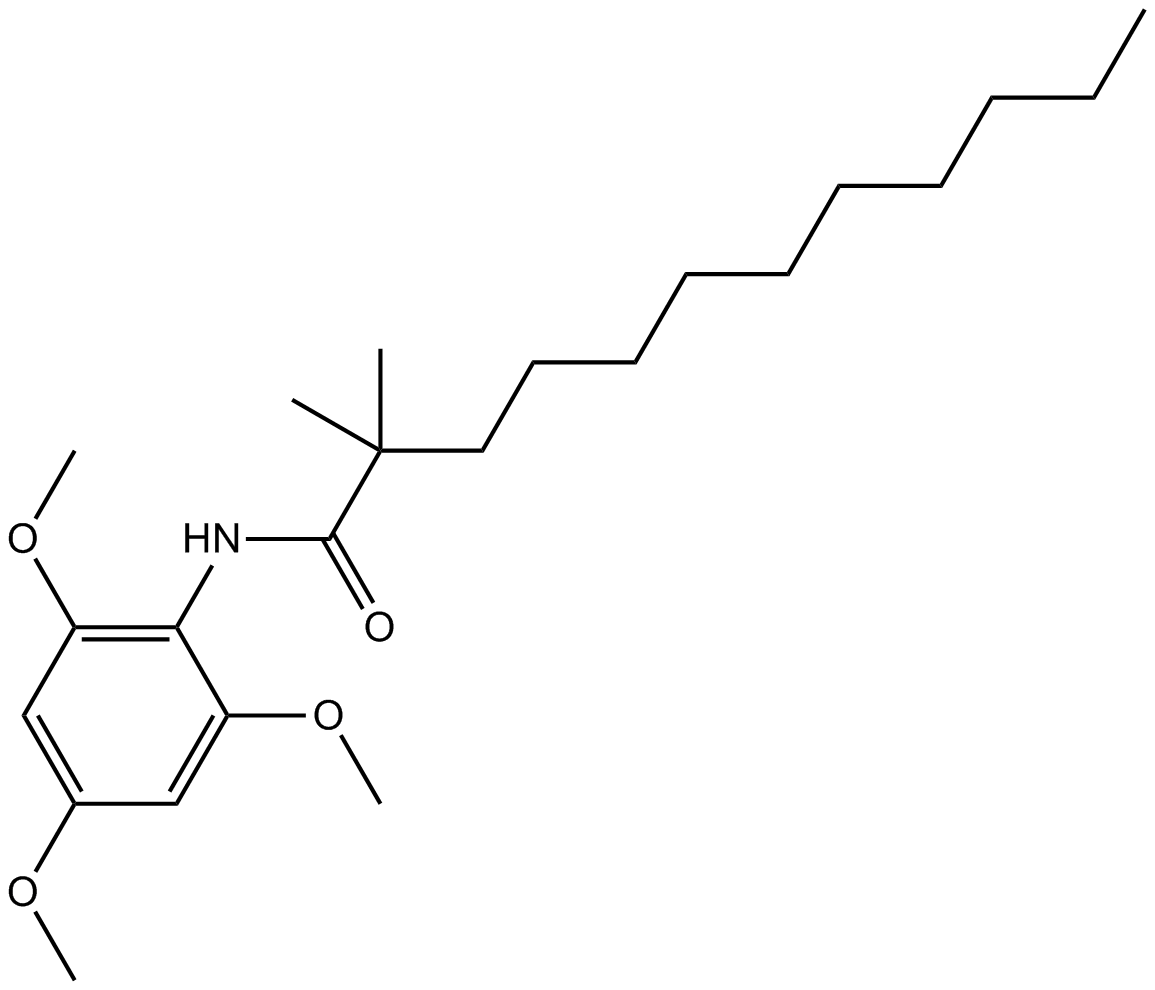
CI994 (Tacedinaline)
An inhibitor of HDAC1, -2, and -3
-
GC15718
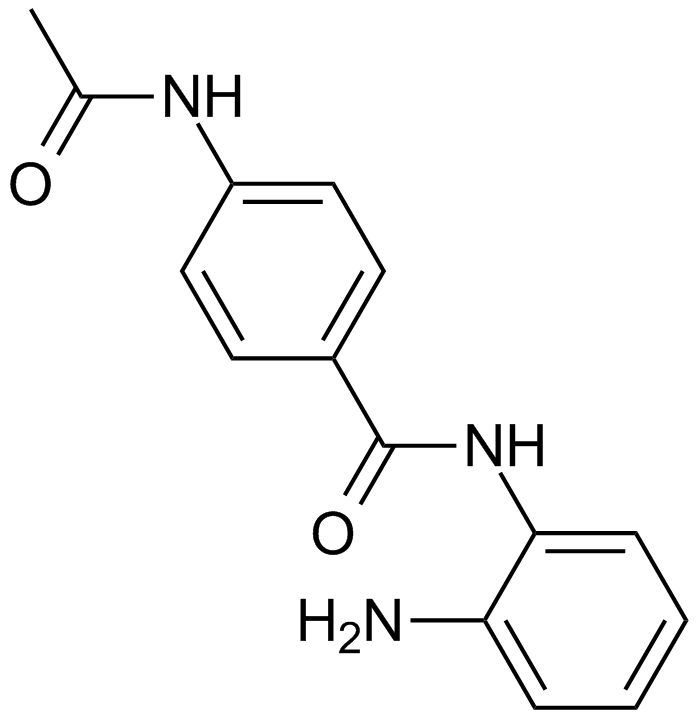
CID 2011756
An inhibitor of protein kinase D
-
GC18577
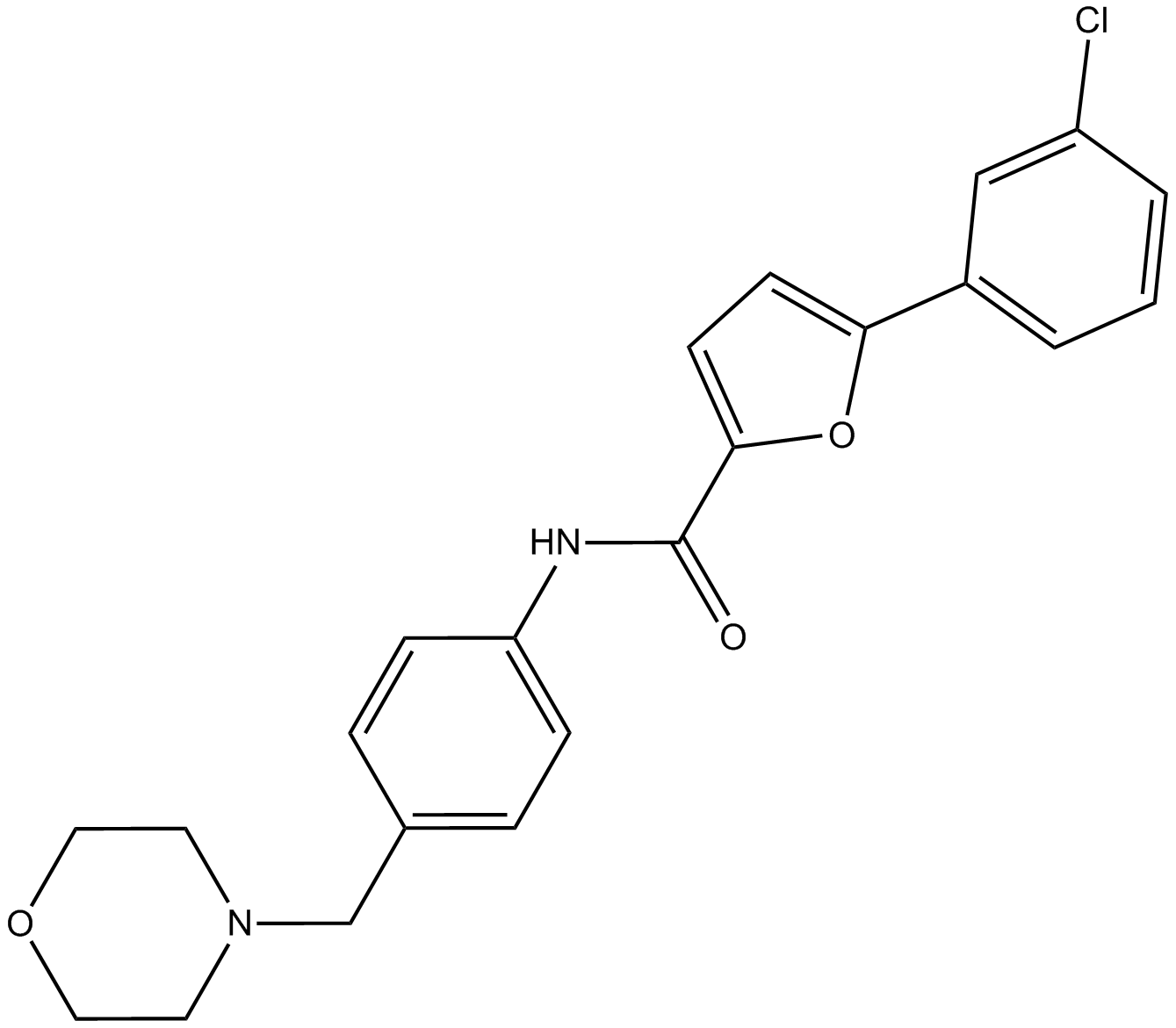
CID-2818500
An inhibitor of PRMT1
-
GC52351
Citrullinated α-Enolase (R8 + R14) (1-19)-biotin Peptide
A biotinylated and citrullinated α-enolase peptide

-
GC52363
Citrullinated Histone H3 (R2 + R8 + R17) (2-22)-biotin Peptide
A biotinylated and citrullinated histone H3 peptide

-
GC52367
Citrullinated Vimentin (G146R) (R144 + R146) (139-159)-biotin Peptide
A biotinylated and citrullinated mutant vimentin peptide

-
GC52370
Citrullinated Vimentin (R144) (139-159)-biotin Peptide
A biotinylated and citrullinated vimentin peptide

-
GC43275
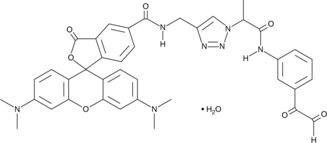
Citrulline-specific Probe-Rhodamine
Protein arginine deiminases (PADs) catalyze the posttranslational modification of arginine residues on proteins to form citrulline, which plays a large role in regulating gene expression.
-
GC39485
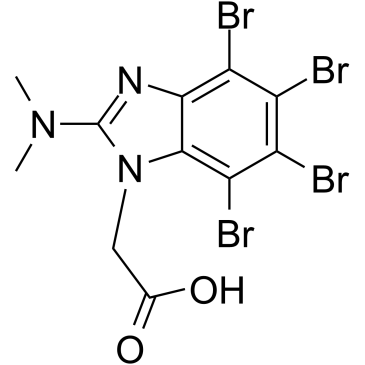
CK2/ERK8-IN-1
CK2/ERK8-IN-1 es un inhibidor dual de caseÍna quinasa 2 (CK2) (Ki de 0,25 μM) y ERK8 (MAPK15, ERK7) con IC50 de 0,50 μM. CK2/ERK8-IN-1 también se une a PIM1, HIPK2 (proteÍna quinasa 2 que interactÚa con el homeodominio) y DYRK1A con Kis de 8,65 μM, 15,25 μM y 11,9 μM, respectivamente. CK2/ERK8-IN-1 tiene eficacia proapoptÓtica.
-
GC35706
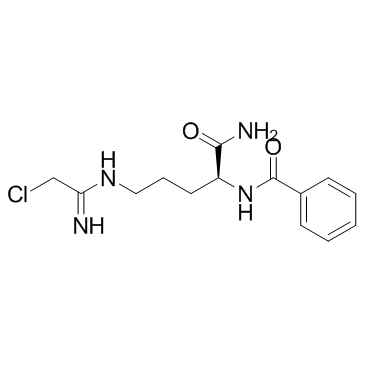
Cl-amidine
La Cl-amidina es un inhibidor de la peptidilarginina desminasa (PAD) activo por vÍa oral, con valores de CI50 de 0,8 μM, 6,2 μM y 5,9 μM para PAD1, PAD3 y PAD4, respectivamente.
-
GC18925
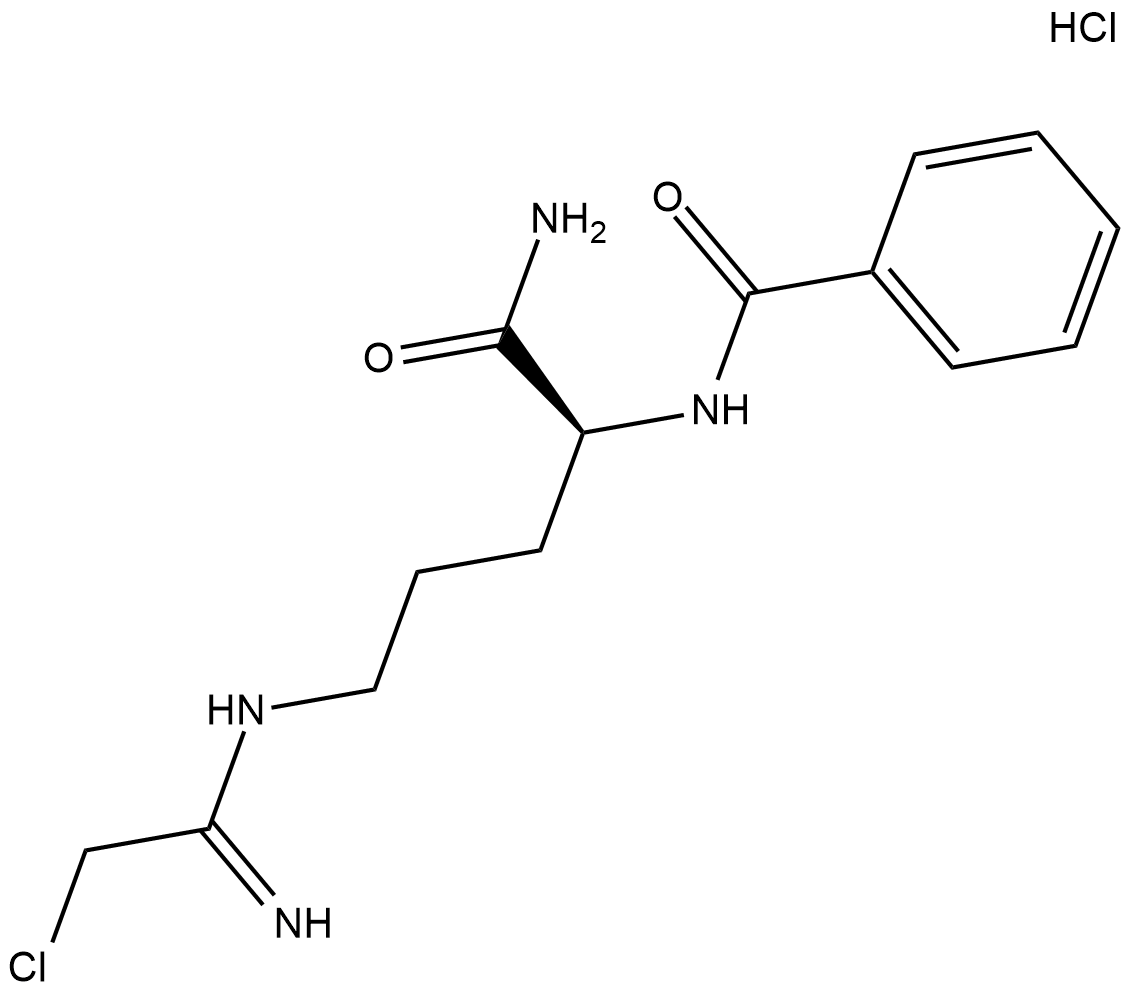
Cl-Amidine (hydrochloride)
La Cl-amidina (clorhidrato) es un inhibidor de la peptidilarginina desminasa (PAD) activo por vía oral, con valores de CI50 de 0,8 μM, 6,2 μM y 5,9 μM para PAD1, PAD3 y PAD4, respectivamente.
-
GC11032
Cl-Amidine (trifluoroacetate salt)
El inhibidor de la desoxigenación PAD4.
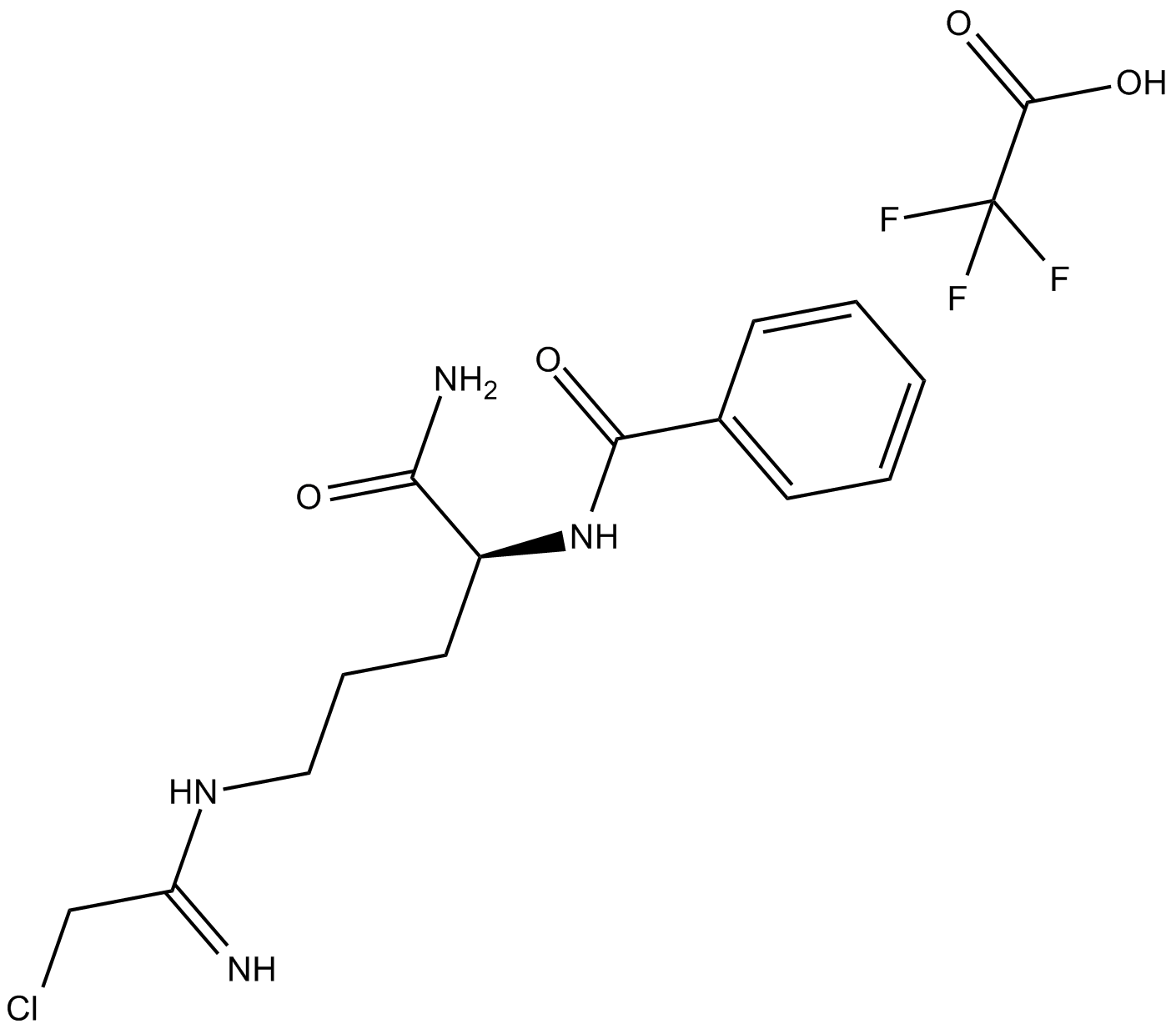
-
GC12367
CM-272
CM-272 es un inhibidor dual G9a/DNA metiltransferasas (DNMT) de primer nivel, potente, selectivo, competitivo con sustrato y reversible con actividades antitumorales. CM-272 inhibe G9a, DNMT1, DNMT3A, DNMT3B y GLP con IC50 de 8 nM, 382 nM, 85 nM, 1200 nM y 2 nM, respectivamente. CM-272 inhibe la proliferaciÓn celular y promueve la apoptosis, induciendo genes estimulados por IFN y muerte celular inmunogénica.
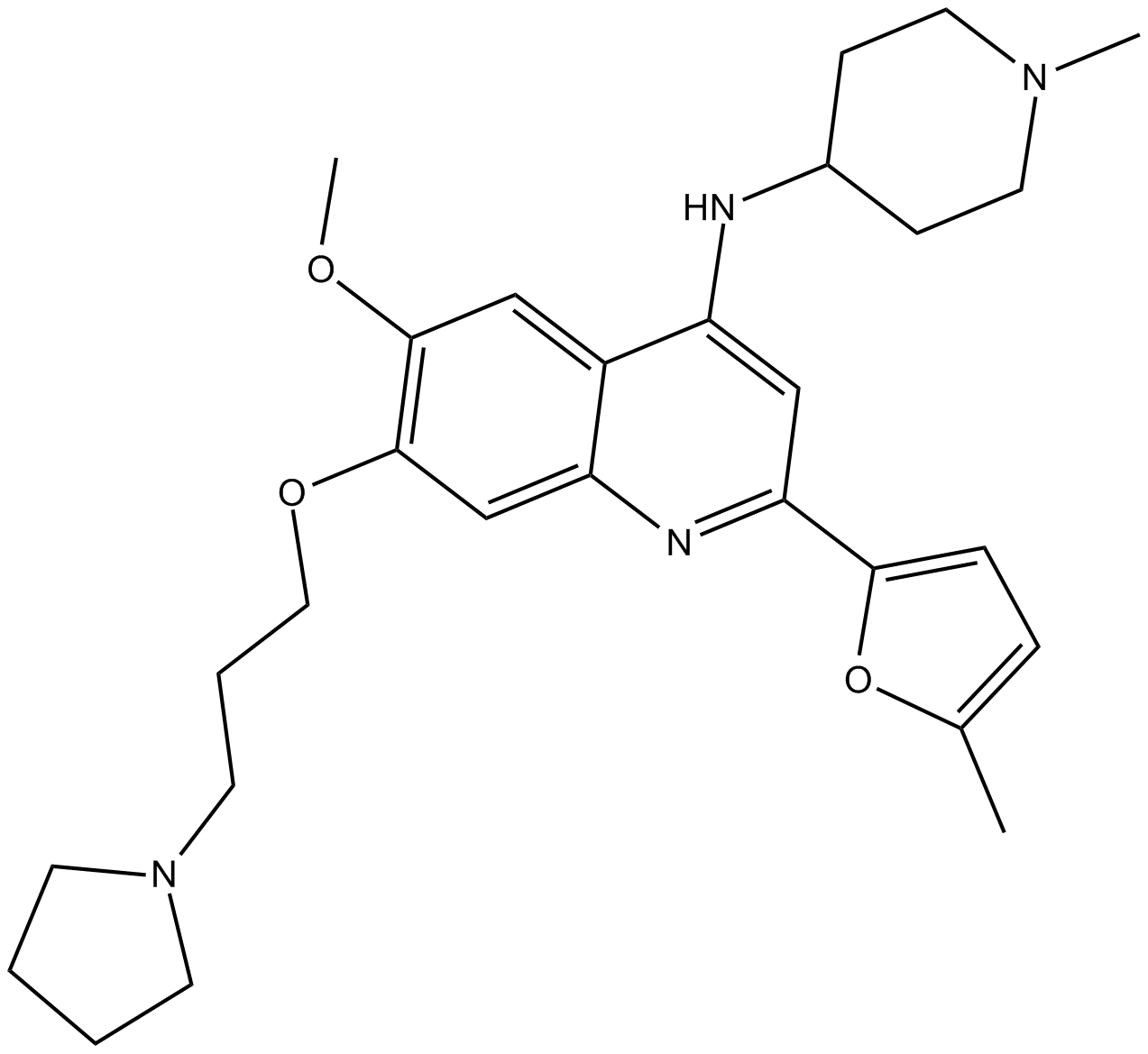
-
GC33320
CM-579
CM-579 es el primer inhibidor dual reversible de su clase de G9a y DNMT, con valores IC50 de 16 nM, 32 nM para G9a y DNMT, respectivamente. Tiene una potente actividad celular in vitro en una amplia gama de células cancerosas.
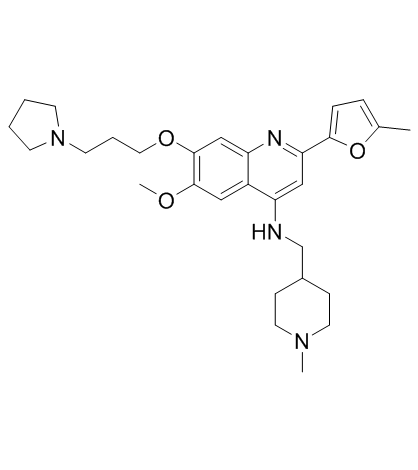
-
GC35714
CM-579 trihydrochloride
El trihidrocloruro de CM-579 es el primer inhibidor dual reversible de su clase de G9a y DNMT, con valores IC50 de 16 nM, 32 nM para G9a y DNMT, respectivamente. Tiene una potente actividad celular in vitro en una amplia gama de células cancerosas.
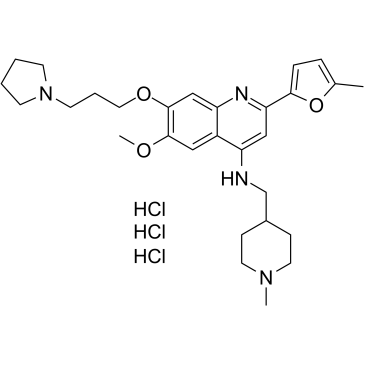
-
GC65426
CM-675
CM-675 es un inhibidor selectivo de la fosfodiesterasa 5 (PDE5) dual y de las histonas desacetilasas de clase I, con valores IC50 de 114 nM y 673 nM para PDE5 y HDAC1, respectivamente.
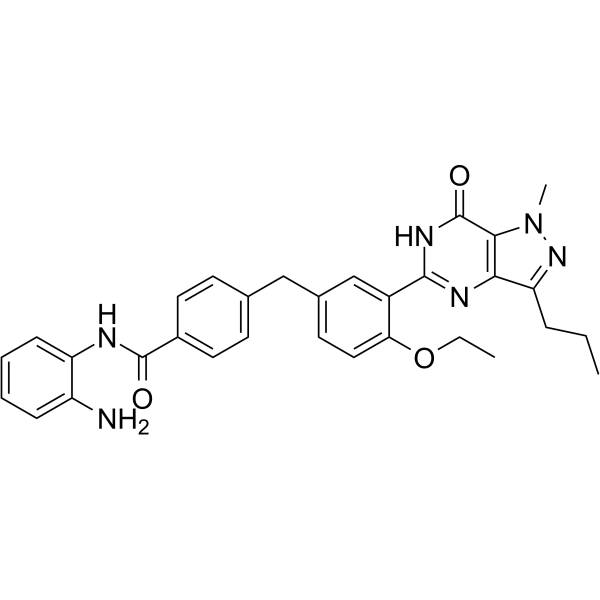
-
GC39665
CMP-5
CMP-5 es un inhibidor potente, especÍfico y selectivo de PRMT5, mientras que no muestra actividad contra las enzimas PRMT1, PRMT4 y PRMT7. CMP-5 bloquea selectivamente S2Me-H4R3 al inhibir la actividad de la metiltransferasa PRMT5 en las preparaciones de histonas. CMP-5 previene la transformaciÓn de linfocitos B impulsada por el virus de Epstein-Barr (EBV), pero no afecta a las células B normales.
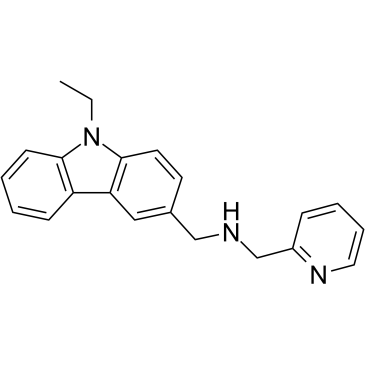
-
GC34165
Corin
Corin es un inhibidor dual de la desmetilasa especÍfica de histona lisina (LSD1) y la histona desacetilasa (HDAC), con una Ki (inactiva) de 110 nM para LSD1 y una IC50 de 147 nM para HDAC1.
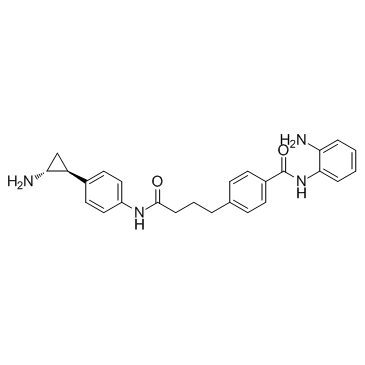
-
GC43318
coumarin-SAHA
Coumarin-SAHA es una sonda fluorescente para determinar las afinidades de uniÓn (kd) y las tasas de disociaciÓn (koff) de los complejos inhibidores de HDAC8.
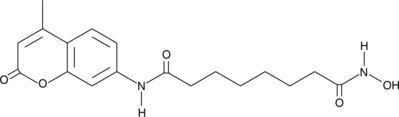
-
GC62908
Coumermycin A1
La cuumermicina A1 es un activador de la seÑal JAK2.
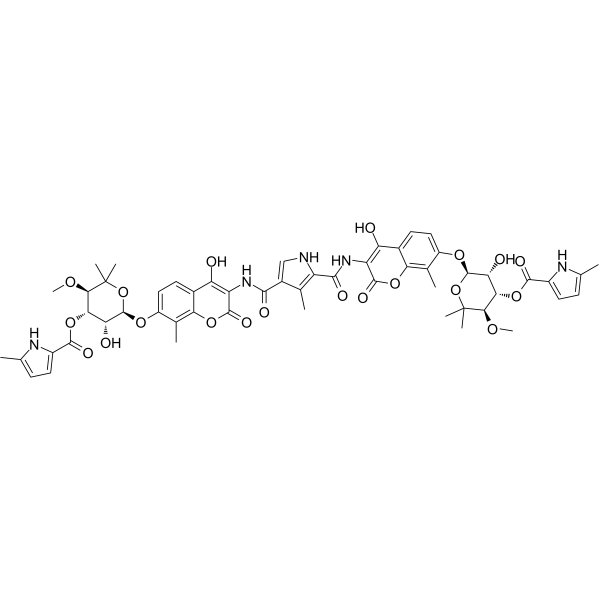
-
GC25304
CP2
CP2 is a cyclic peptide that inhibits the JmjC histone demethylases KDM4 with IC50 values of 42 nM and 29 nM for KDM4A and KDM4C, respectively.
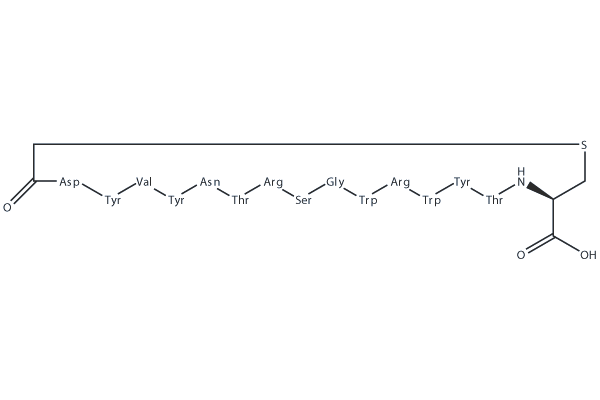
-
GC16298
CPI-1205
EZH2 inhibitor
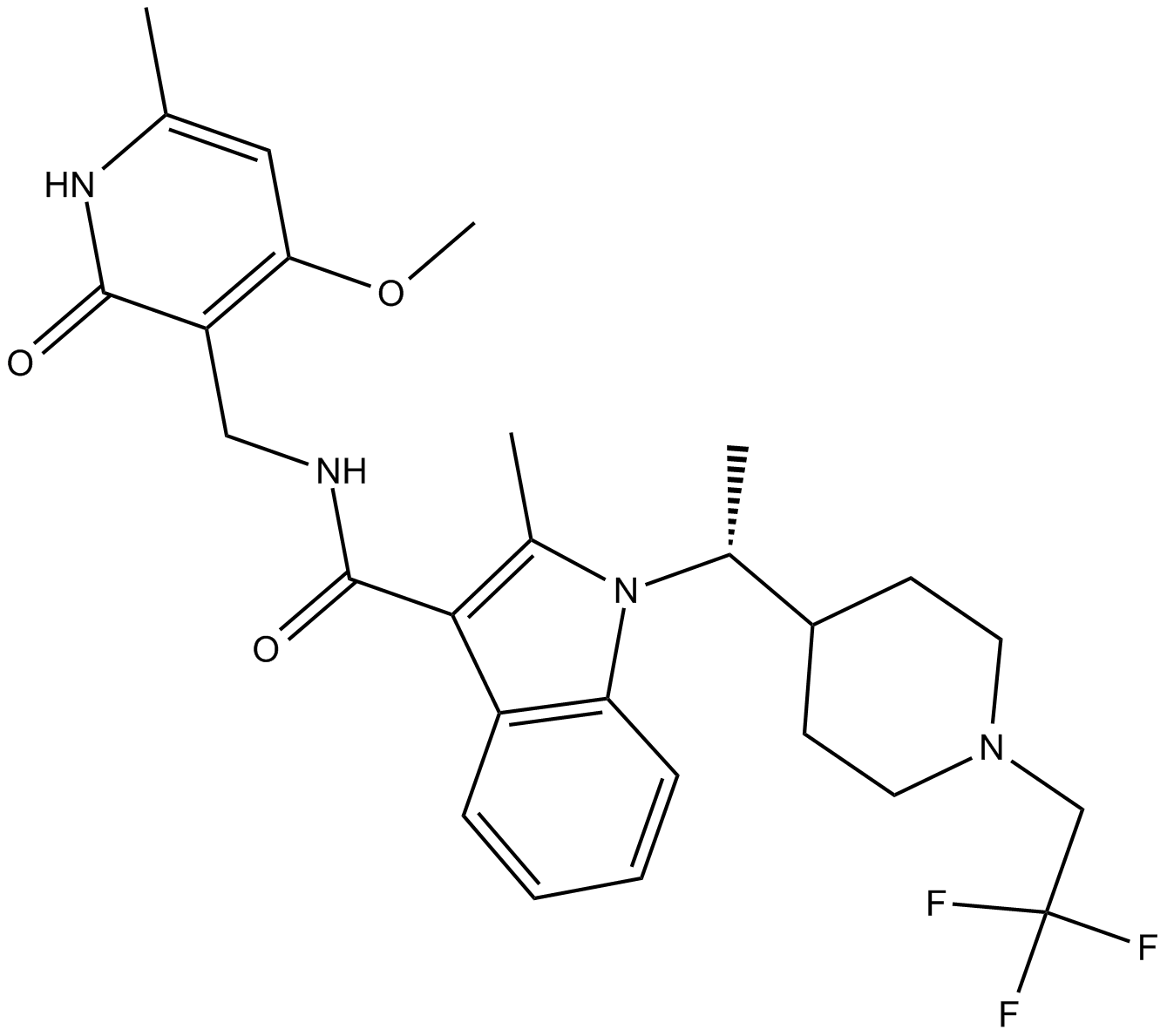
-
GC62669
CPI-1612
CPI-1612 es un inhibidor de la histona acetiltransferasa (HAT) EP300/CBP activo por vÍa oral muy potente con una IC50 de 8,1 nM para EP300 HAT. CPI-1612 tiene una actividad anticancerÍgena.
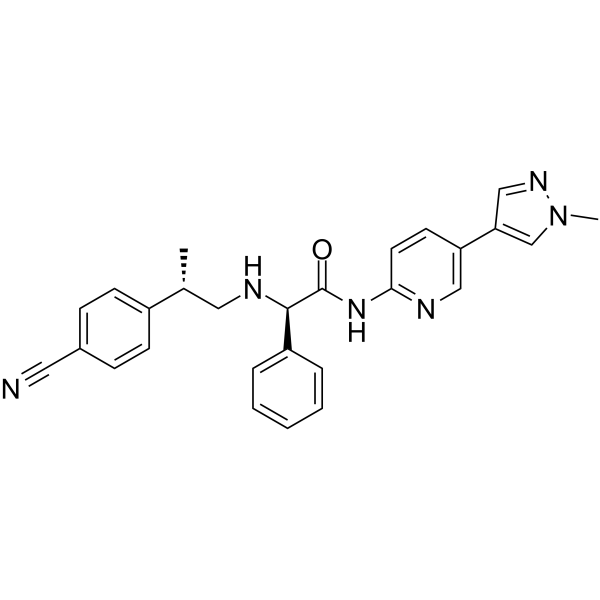
-
GC16599
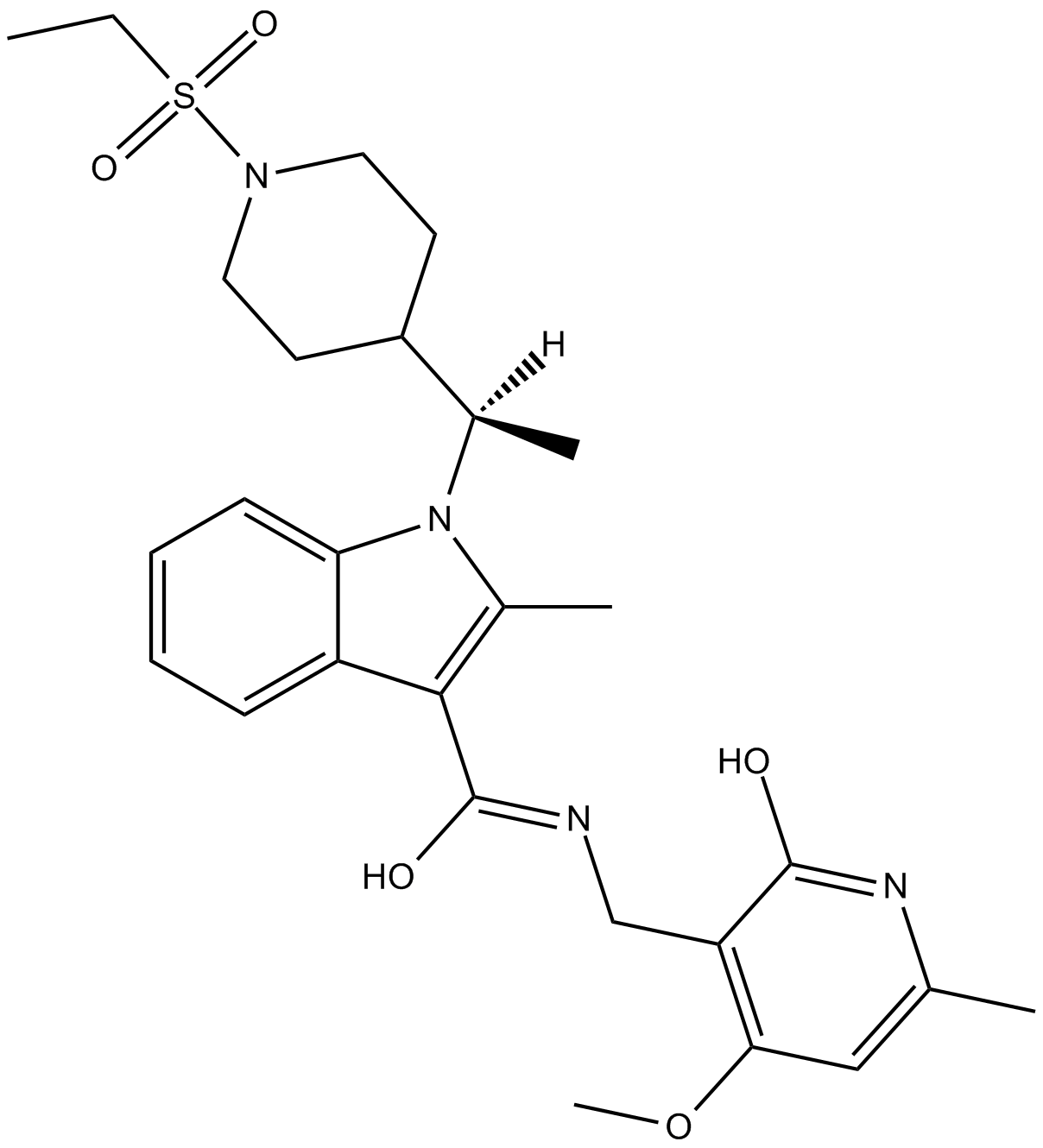
CPI-169
EZH2 inhibitor
-
GC14699
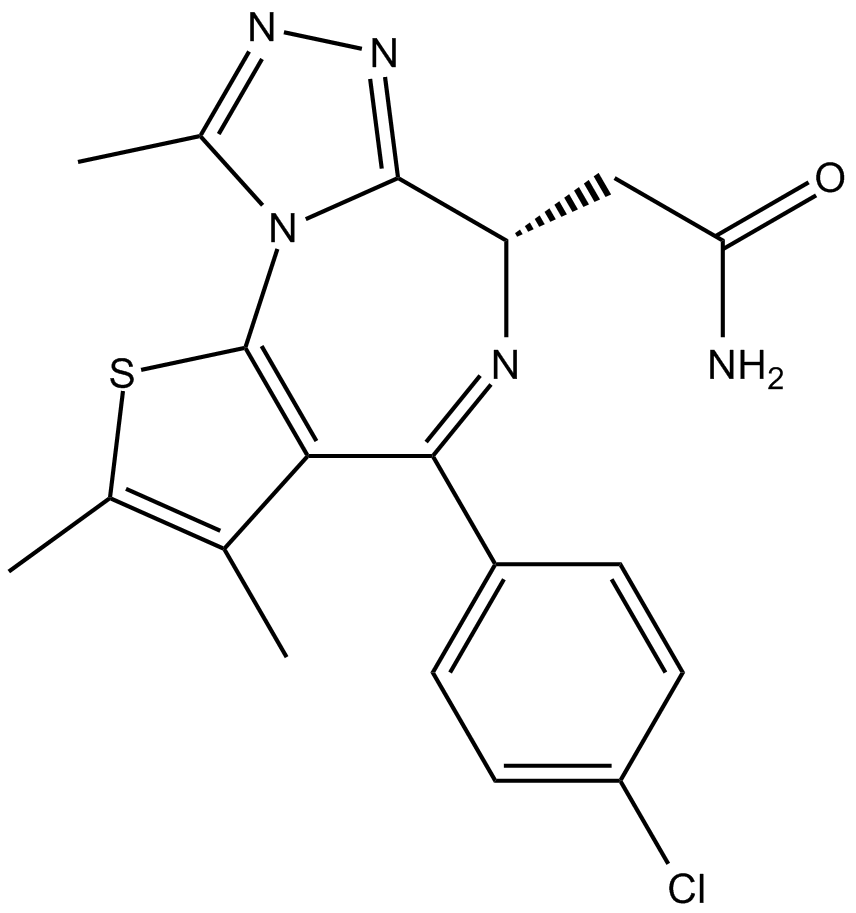
CPI-203
CPI-203 es un nuevo inhibidor potente, selectivo y permeable a las células del bromodominio BET, con un valor IC50 de aproximadamente 37 nM (ensayo de pantalla α BRD4).
-
GC43320
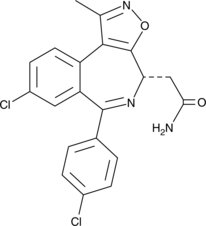
CPI-268456
CPI-268456 (compuesto 141) es un potente inhibidor de BRD4 con una IC50 de <0,5 μM.
-
GC10021
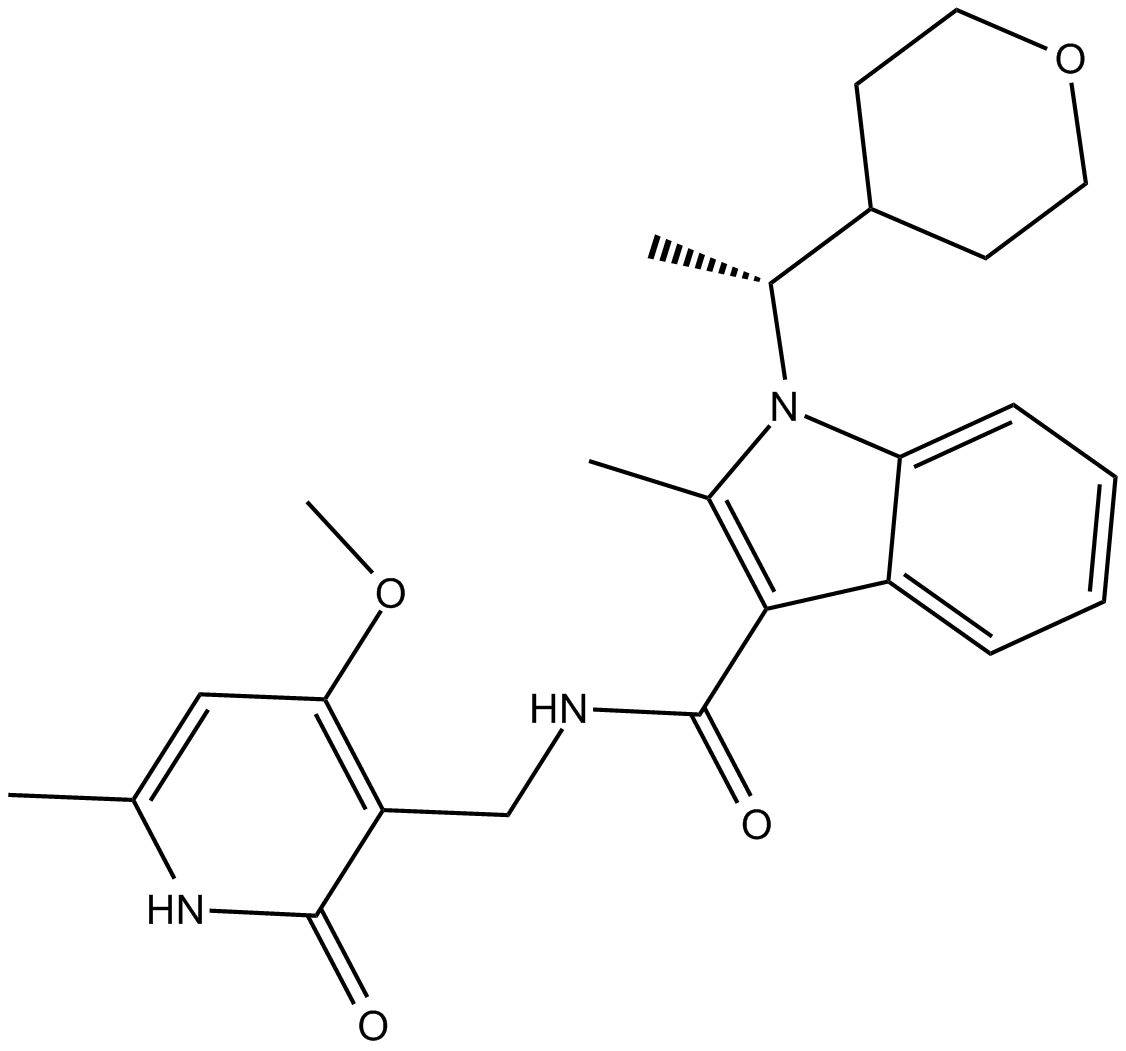
CPI-360
EZH2 inhibitor
-
GC10774
CPI-455
Inhibidor de KDM5
-
GC25305
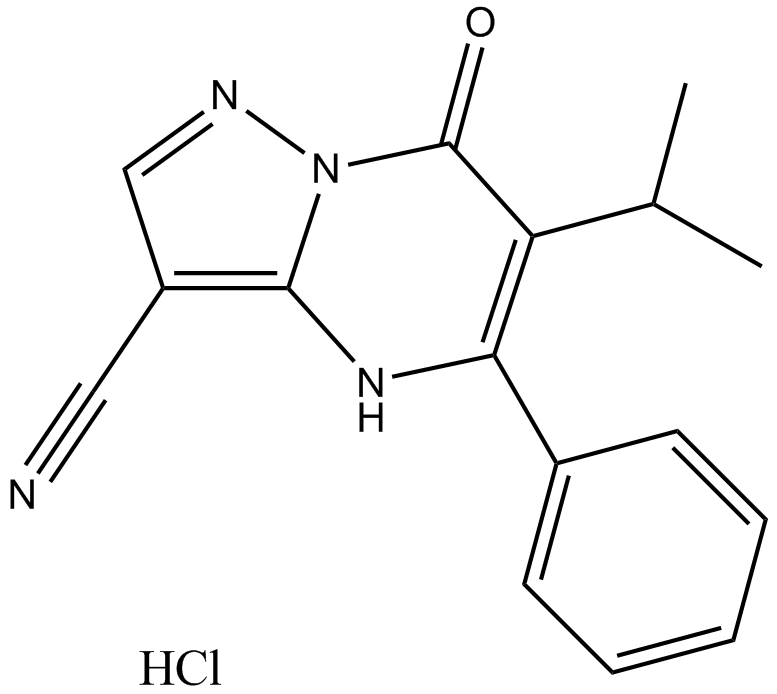
CPI-455 HCl
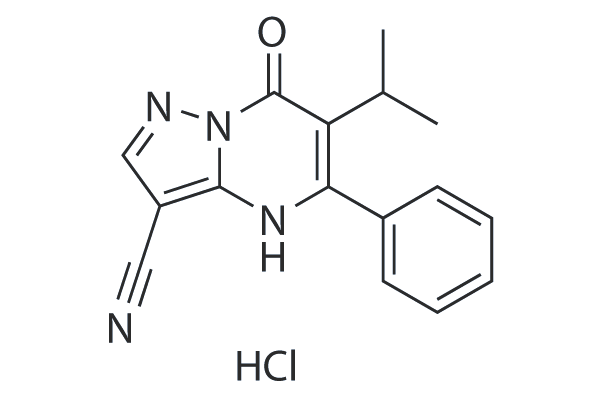
CPI-455 HCl is a specific KDM5 inhibitor with a half-maximal inhibitory concentration (IC50) of 10 ± 1 nM for full-length KDM5A in enzymatic assays, elevating global levels of H3K4 trimethylation (H3K4me3) and decreased the number of DTPs in multiple cancer cell line models treated with standard chemotherapy or targeted agents.This product has poor solubility, animal experiments are available, cell experiments please choose carefully!
-
GC10382
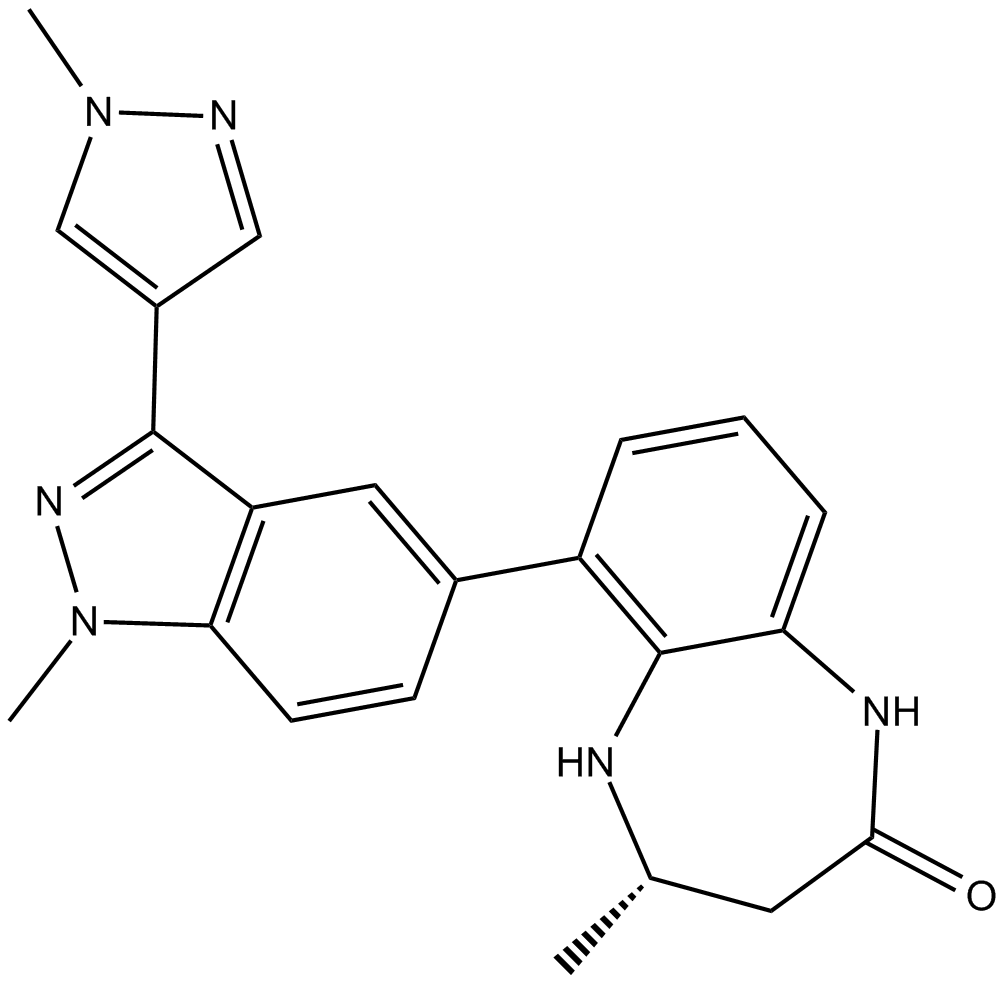
CPI-637
CPI-637 es un inhibidor selectivo y potente del bromodominio CBP/EP300 con valores IC50 de 0,03 μM, 0,051 μM y 11,0 μM para CBP, EP300 y BRD4 BD-1, respectivamente, y una EC50 de 0,3 μM para CBP.
-
GC39365
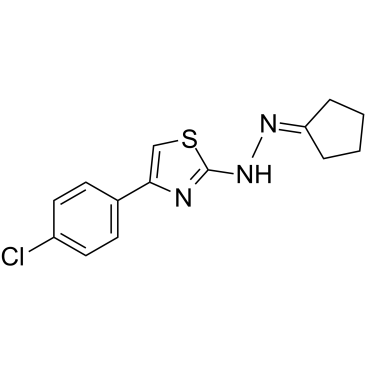
CPTH2
CPTH2 es un potente inhibidor de histona acetiltransferasa (HAT). CPTH2 inhibe selectivamente la acetilaciÓn de la histona H3 por Gcn5. CPTH2 induce la apoptosis y disminuye la invasividad de una lÍnea celular de carcinoma renal de células claras (ccRCC) a través de la inhibiciÓn de la acetiltransferasa p300 (KAT3B).
-
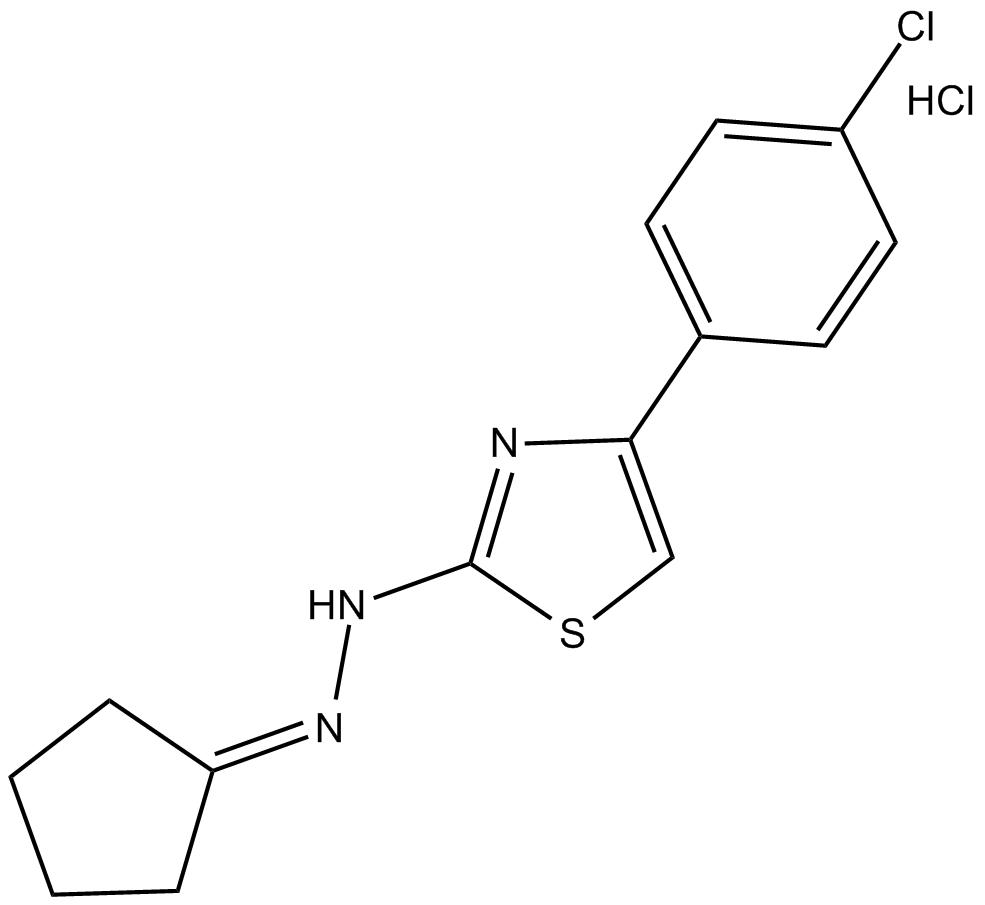
GC14428
CPTH2 (hydrochloride)
CPTH2 (clorhidrato) es un potente inhibidor de histona acetiltransferasa (HAT). CPTH2 (clorhidrato) inhibe selectivamente la acetilación de la histona H3 por Gcn5. CPTH2 (clorhidrato) induce la apoptosis y disminuye la invasividad de una línea celular de carcinoma renal de células claras (ccRCC) a través de la inhibición de la acetiltransferasa p300 (KAT3B).
-
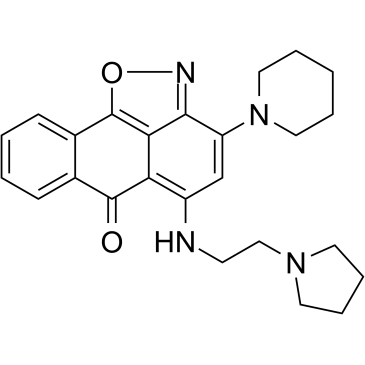
GC35742
CPUY074020
CPUY074020 es un inhibidor potente y biodisponible por vÍa oral de la histona metiltransferasa G9a, con una IC50 de 2,18 μM. CPUY074020 posee actividad antiproliferativa.
-
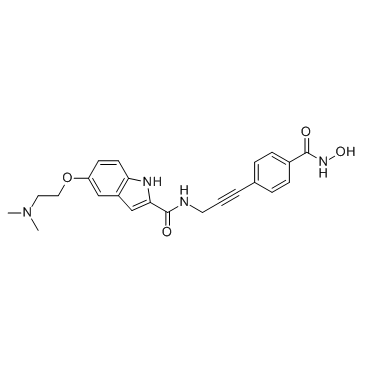
GC32565
CRA-026440
CRA-026440 es un potente inhibidor de HDAC de amplio espectro.
-
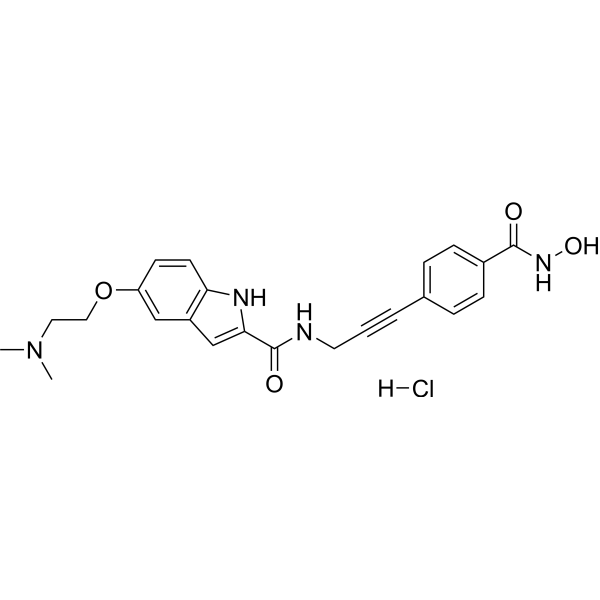
GC67674
CRA-026440 hydrochloride
-
GC38412
Crotonoside
A guanosine analog with diverse biological activities
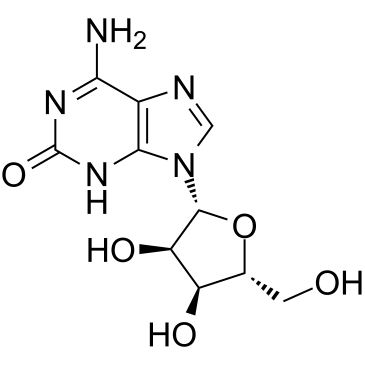
-
GC14524
CTPB
CTPB es un buen activador de la enzima p300 histona acetil transferasa (HAT).
-
GC16008
CUDC-101
A multi-target inhibitor of HDACs, EGFR, and HER2
-
GC12115
CUDC-907
A dual inhibitor of HDACs and PI3Ks
-
GN10442
Curculigoside
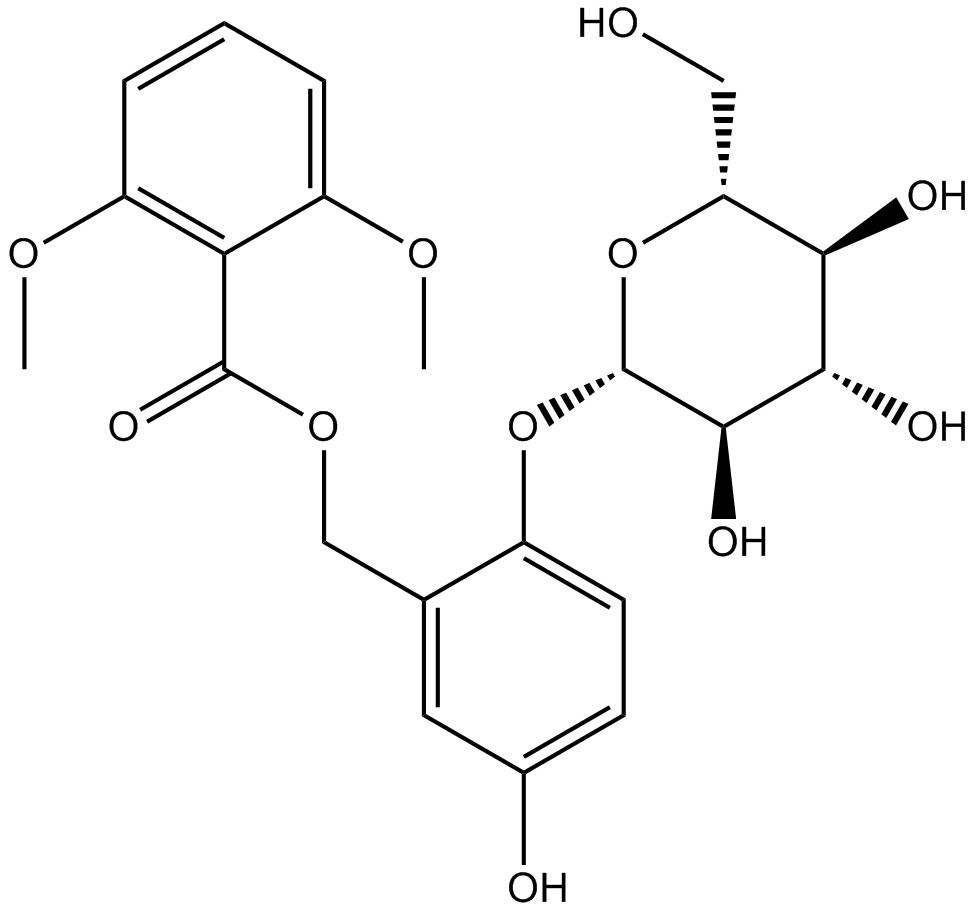
-
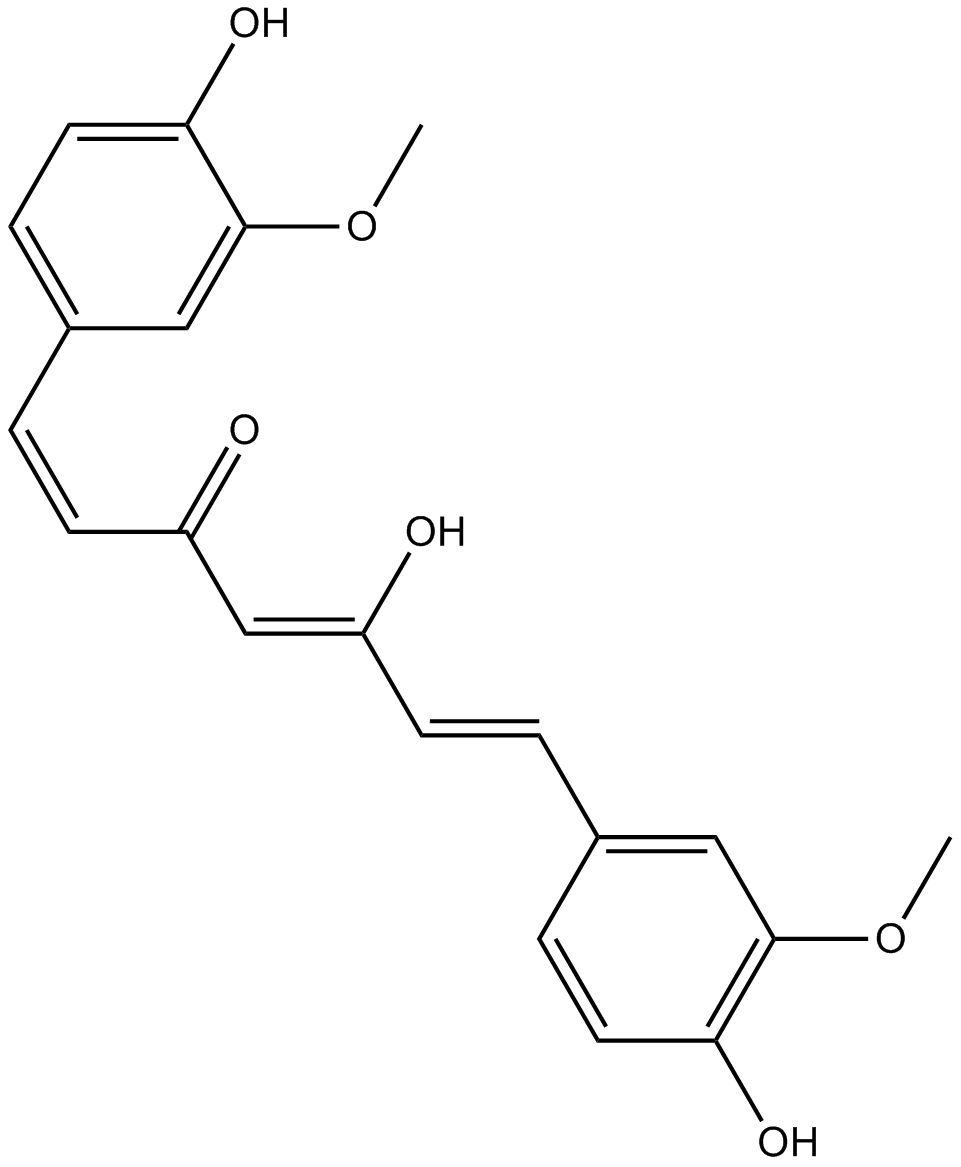
GC14787
Curcumin
Un pigmento amarillo con diversas actividades biológicas.
-
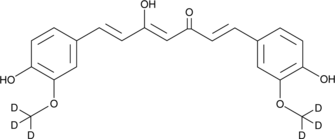
GC40226
Curcumin-d6
La curcumina D6 (diferuloilmetano D6) es una curcumina marcada con deuterio (cÚrcuma amarilla).
-
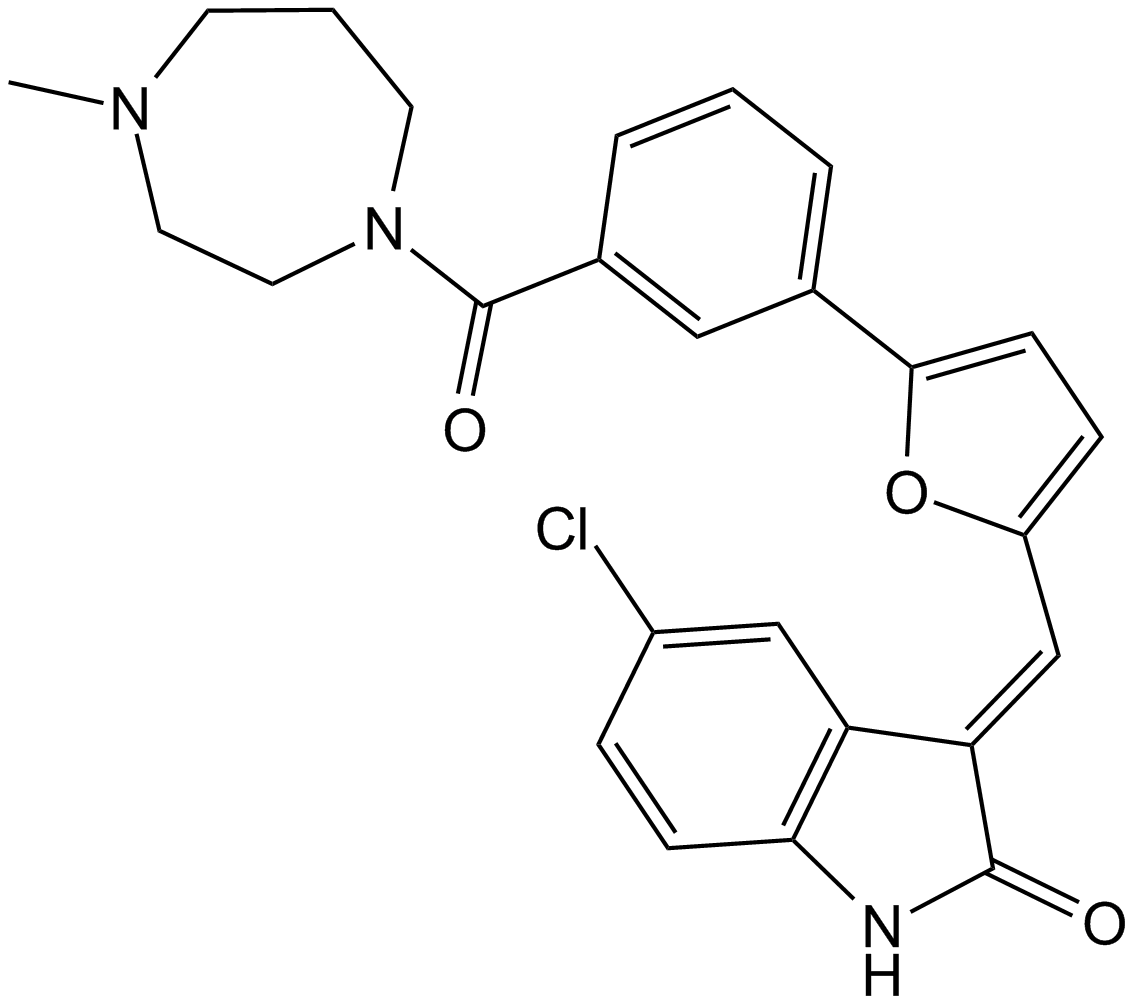
GC13056
CX-6258
A pan-Pim kinase inhibitor
-
GC19747
CX-6258 HCl
CX-6258 HCl es un inhibidor de cinasas pan-Pim potente y selectivo de cinasa, con IC50 de 5 nM, 25 nM y 16 nM para Pim-1, Pim-2 y Pim-3, respectivamente.
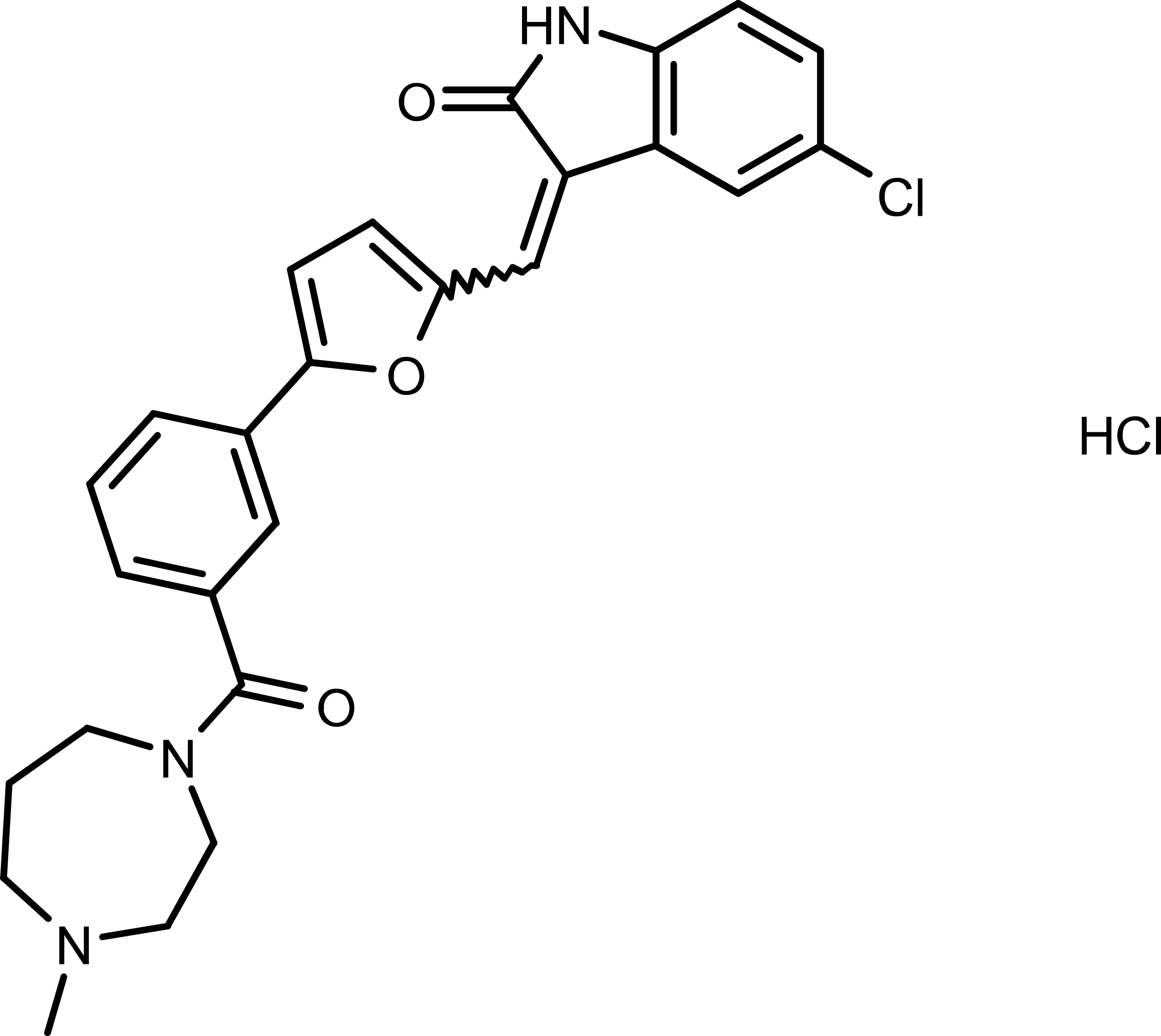
-
GC35762
CX-6258 hydrochloride hydrate
El hidrato de clorhidrato de CX-6258 es un inhibidor de cinasas pan-Pim potente y selectivo de cinasa, con IC50 de 5 nM, 25 nM y 16 nM para Pim-1, Pim-2 y Pim-3, respectivamente.
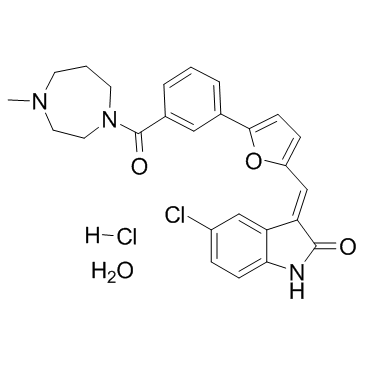
-
GC15106
CYC116
A potent Aurora kinase inhibitor
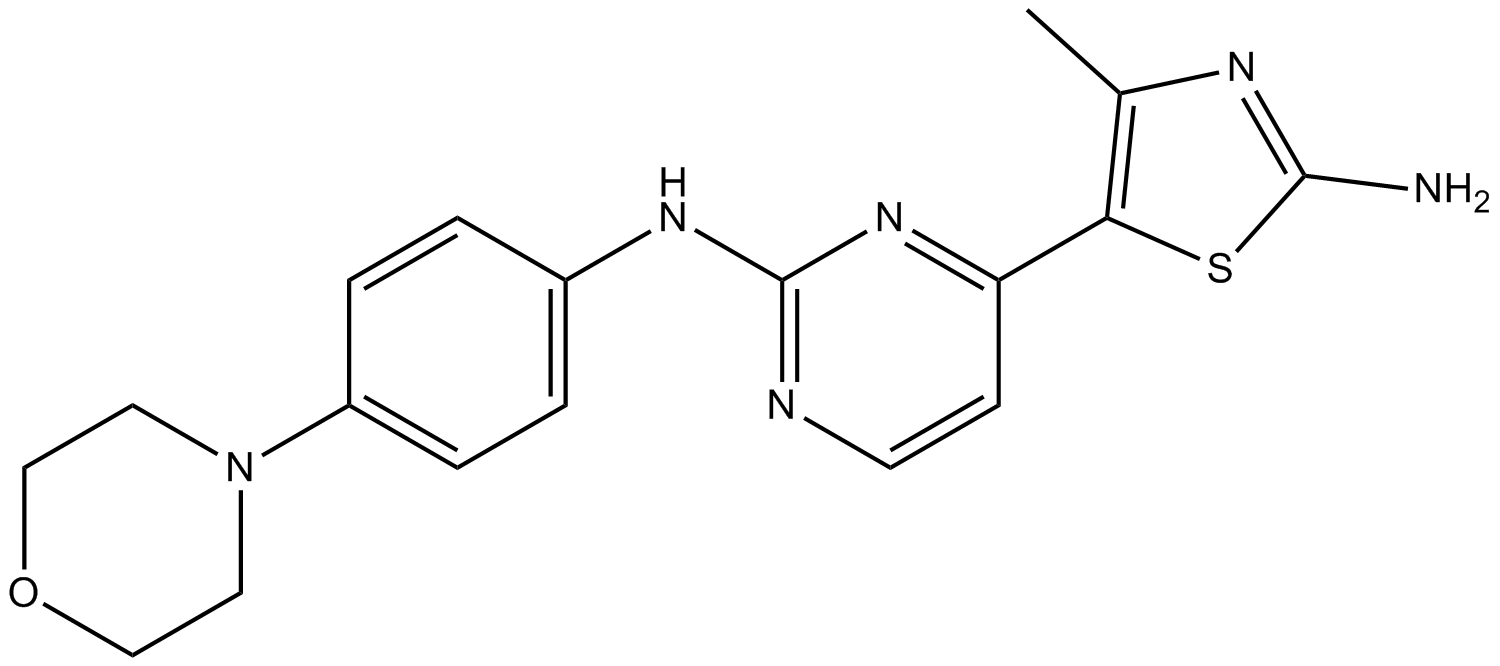
-
GC43354
Cysmethynil
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.

-
GC17050
CYT387
A potent inhibitor of JAK1 and JAK2
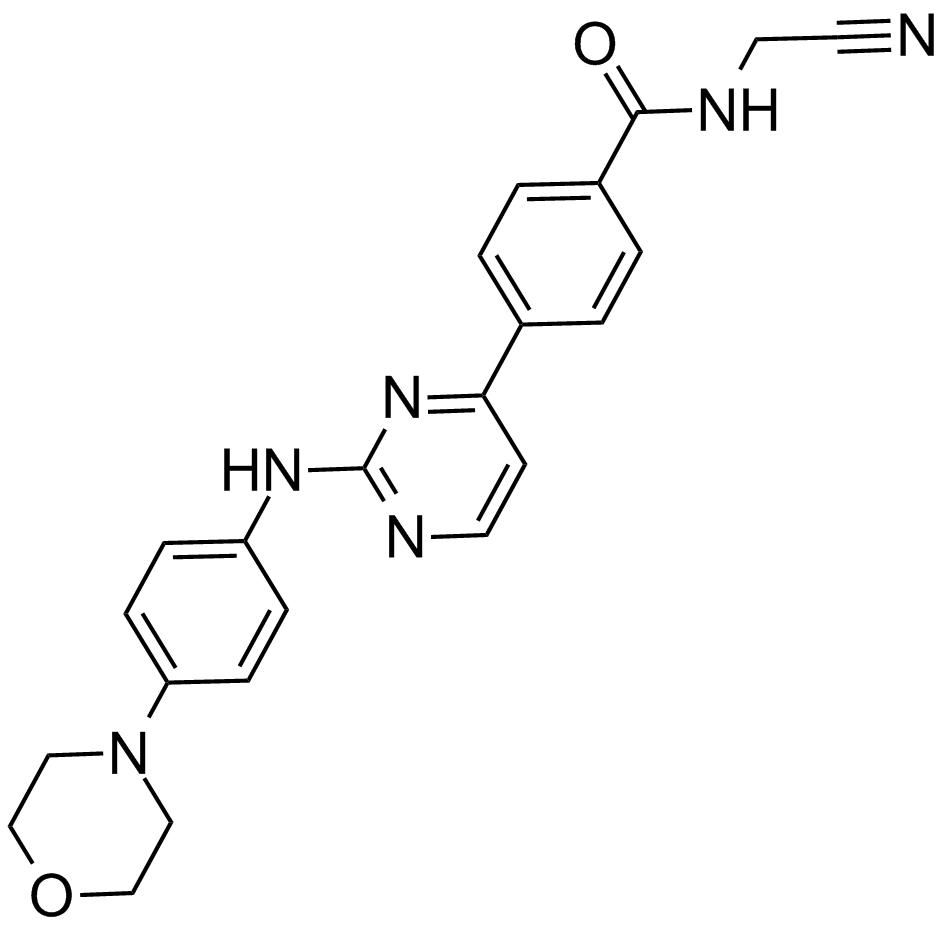
-
GC16468
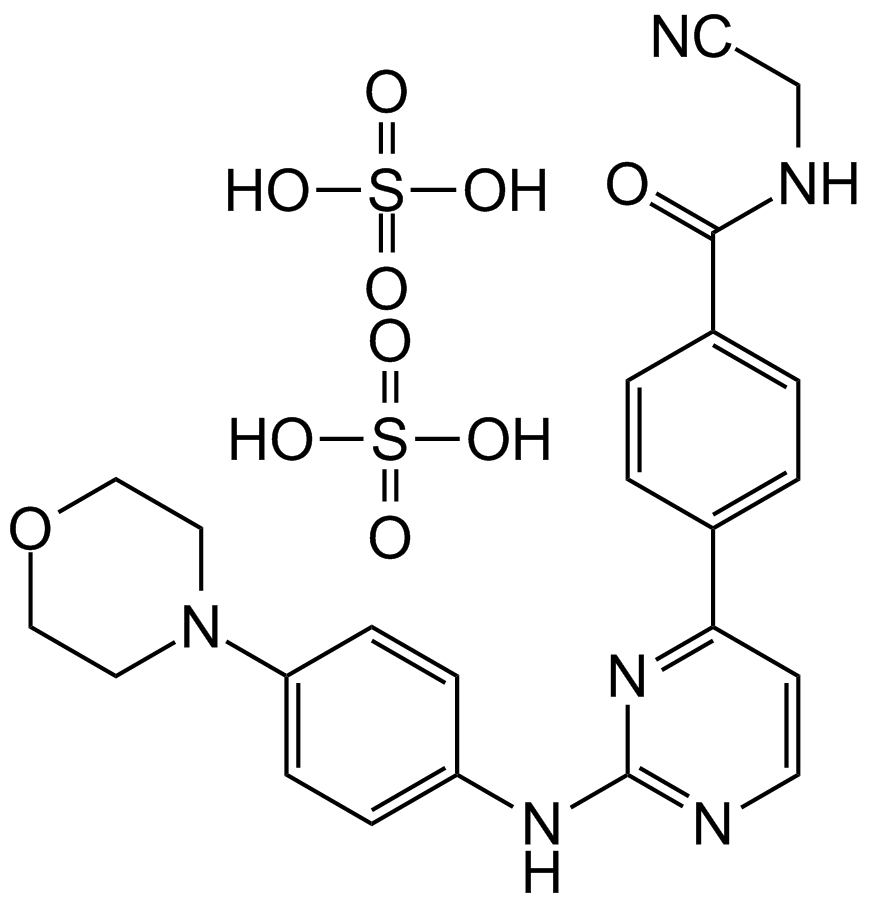
CYT387 sulfate salt
A potent inhibitor of JAK1 and JAK2
-
GC63780
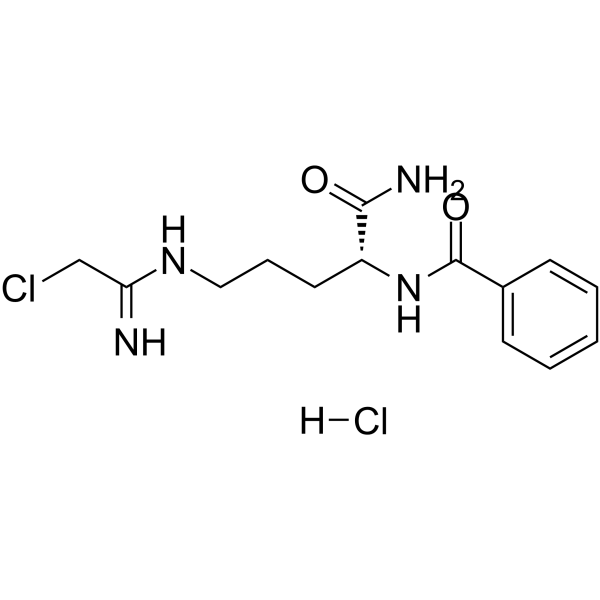
D-Cl-amidine hydrochloride
El clorhidrato de D-Cl-amidina es un inhibidor potente y altamente selectivo de PAD1. La D-Cl-amidina está bien tolerada sin toxicidad significativa.
-
GC48967
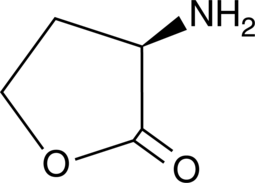
D-Homoserine lactone
An enantiomer of L-homoserine lactone
-
GC15830
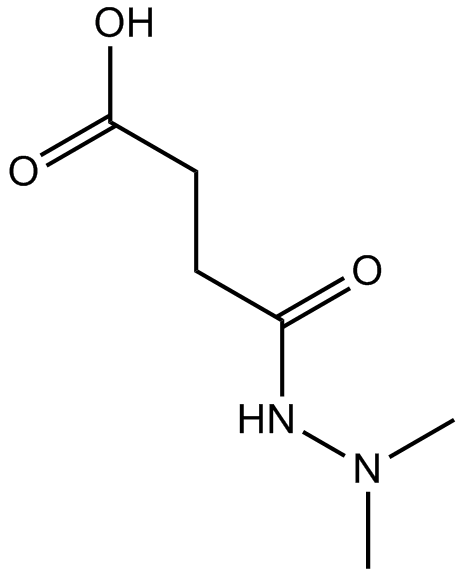
Daminozide
Selective inhibitor of KDM2/7 histone demethylases
-
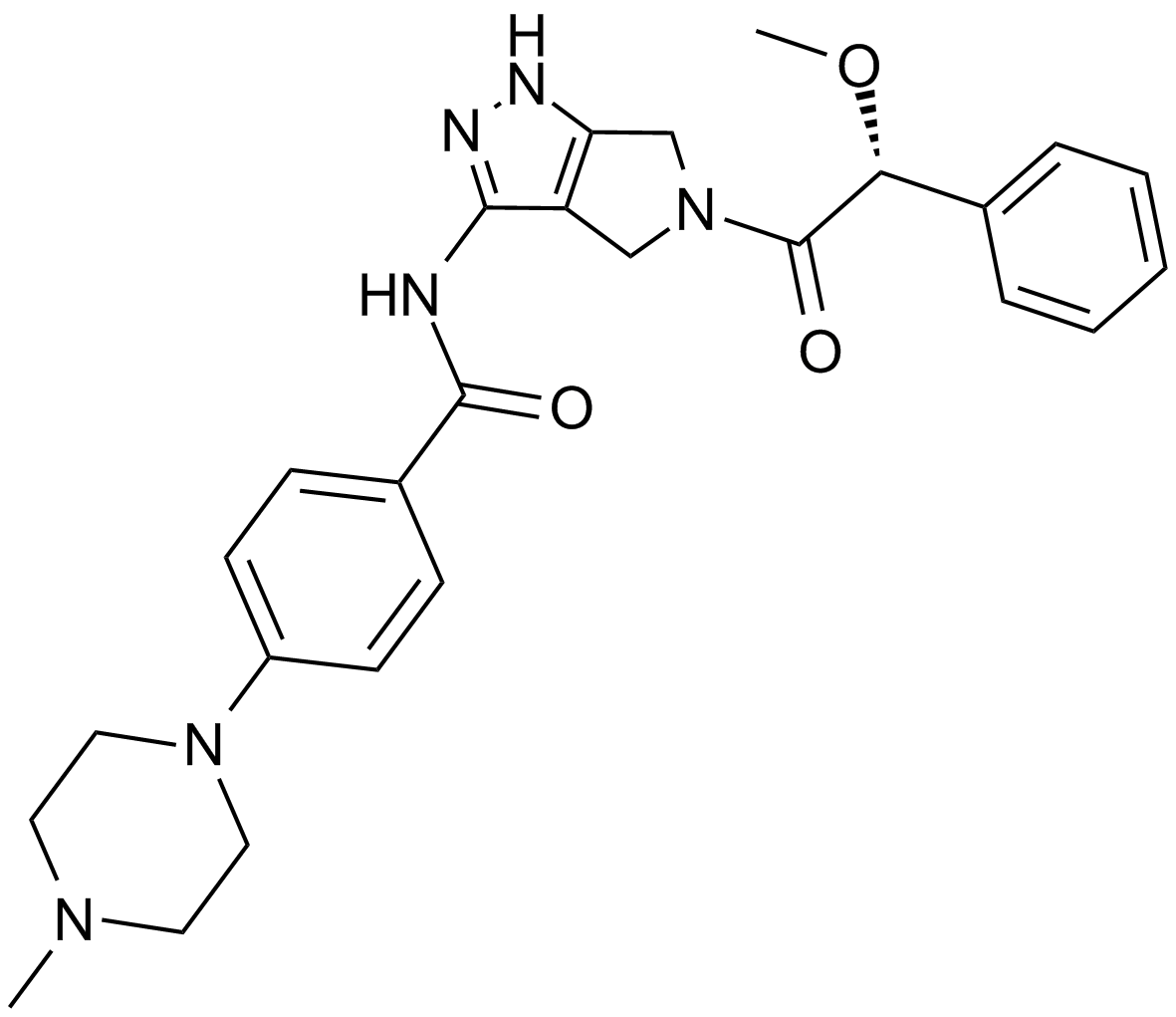
GC15217
Danusertib (PHA-739358)
A pan-Aurora kinase and Abl inhibitor
-
GC16647
Daprodustat(GSK1278863)
Daprodustat (GSK1278863) (GSK1278863) es un inhibidor de la prolil hidroxilasa (HIF-PH) del factor inducible por hipoxia activo por vÍa oral que se estÁ desarrollando para el tratamiento de la anemia asociada con la enfermedad renal crÓnica.

-
GC68147
dAURK-4 hydrochloride
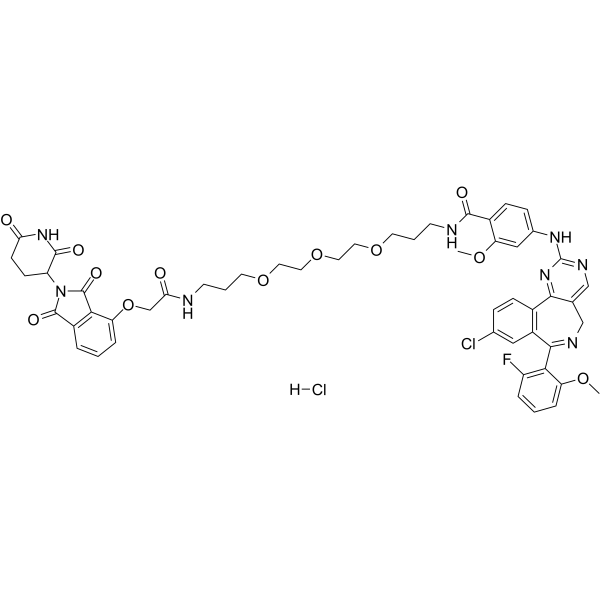
-
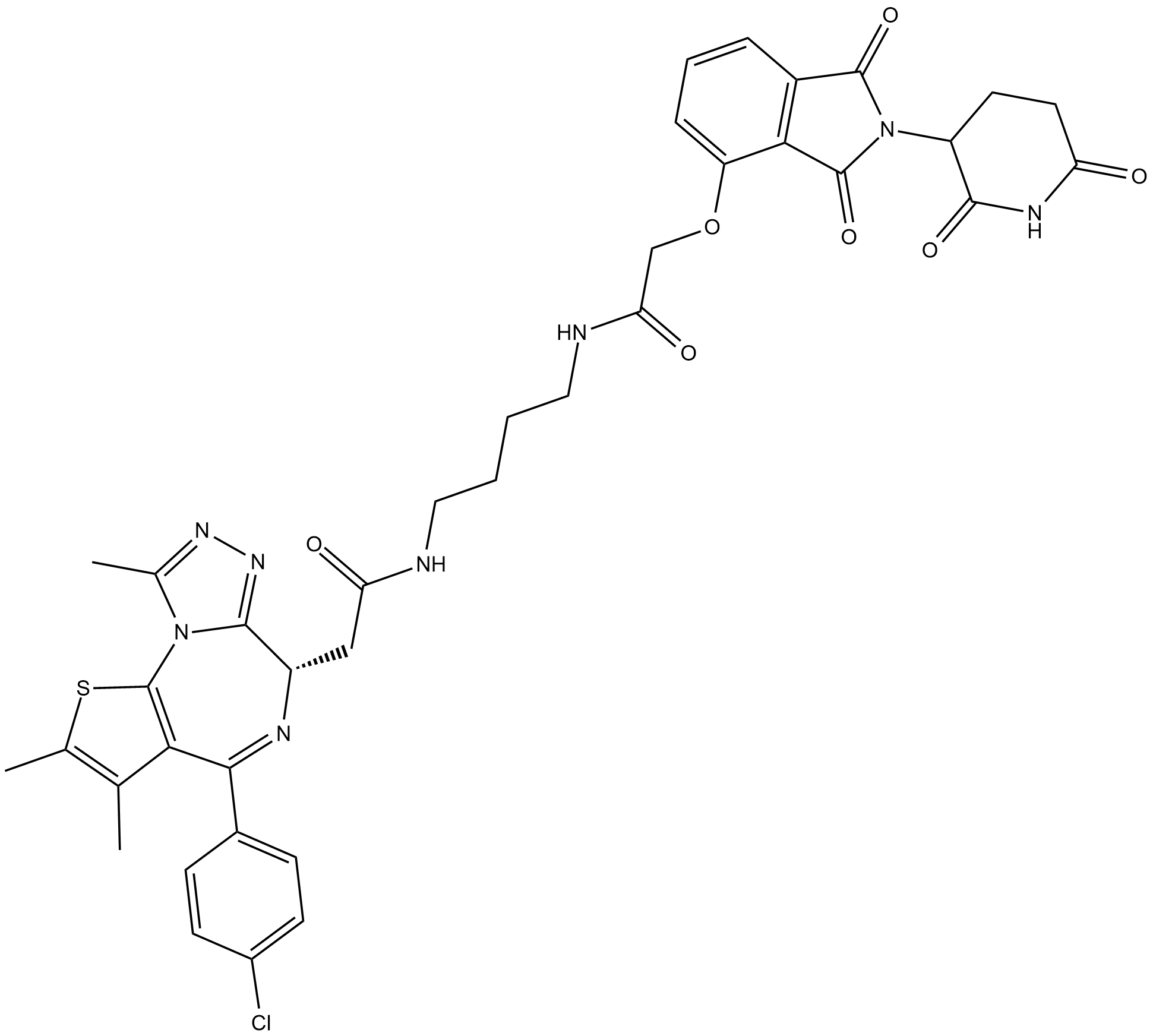
GC19119
dBET1
dBET1 es un PROTAC conectado por ligandos para Cereblon y BRD4 con un EC50 de 430 nM. dBET1 es un PROTAC que se compone de (+)-JQ1 vinculado a NSC 527179 con un enlazador.
-
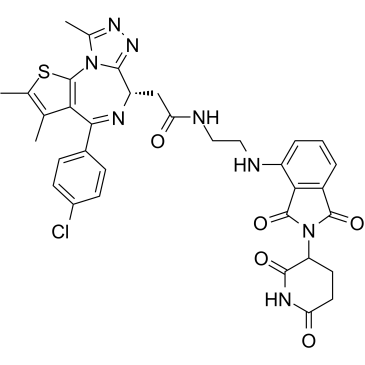
GC35815
dBET57
dBET57 es un degradador potente y selectivo de BRD4BD1 basado en la tecnologÍa PROTAC. dBET57 media el reclutamiento de la ligasa de ubiquitina CRL4Cereblon E3, con una DC50/5h de 500 nM para BRD4BD1, y es inactivo en BRD4BD2.
-
GC32719
dBET6
dBET6 es un PROTAC altamente potente, selectivo y permeable a las células conectado por ligandos para Cereblon y BET, con una IC50 de 14 nM y tiene actividad antitumoral.
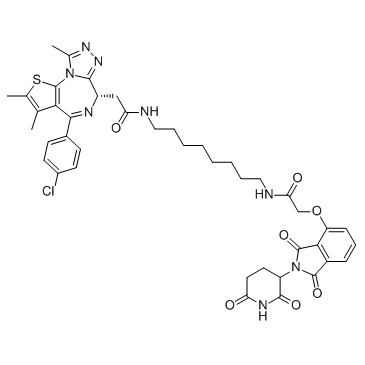
-
GC33148
DC-05
DC-05 es un inhibidor de la ADN metiltransferasa 1 (DNMT1), con una IC50 y una Kd de 10,3 μM y 1,09 μM, respectivamente.
-
GC65186
DC-S239
DC-S239 es un inhibidor selectivo de histona metiltransferasa SET7 con un valor IC50 de 4,59 μM. DC-S239 también muestra selectividad para DNMT1, DOT1L, EZH2, NSD1, SETD8 y G9a. DC-S239 tiene actividad anticancerÍgena.
-
GC62211
dCBP-1
dCBP-1 es un degradador heterobifuncional potente y selectivo de p300/CBP basado en el ligando Cereblon. dCBP-1 es excepcionalmente potente para matar células de mieloma mÚltiple y elimina la actividad potenciadora oncogénica que impulsa la expresiÓn de MYC.
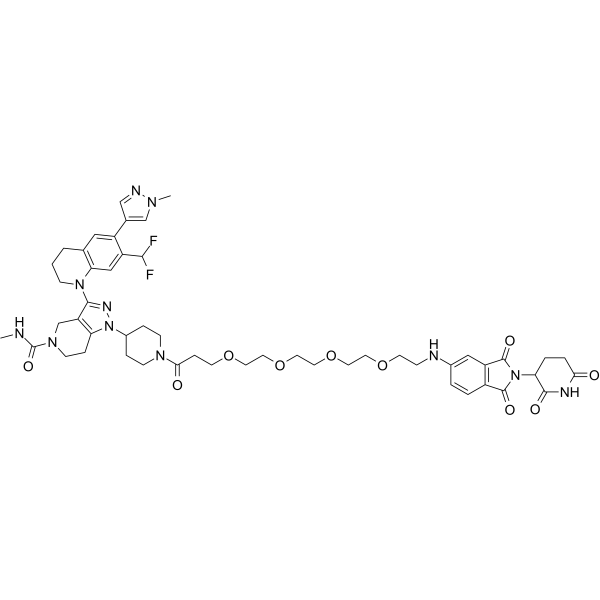
-
GC63627
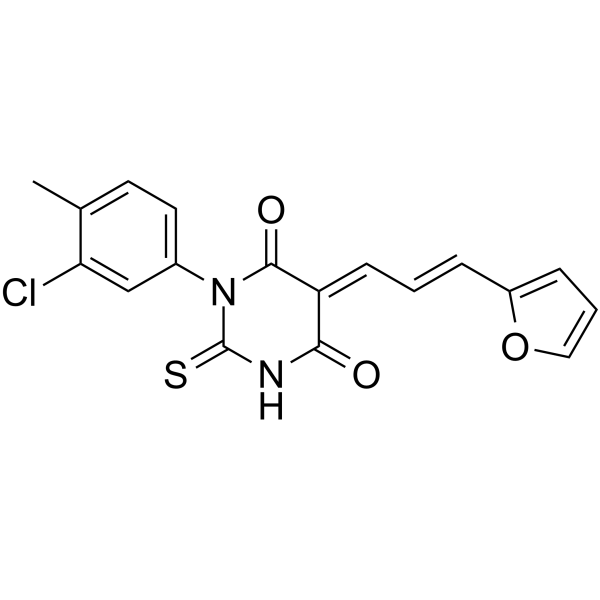
DCH36_06
DCH36_06 es un inhibidor potente y selectivo de p300/CBP con IC50 de 0,6 μM y 3,2 μM para p300 y CBP, respectivamente. InhibiciÓn de p300/CBP mediada por DCH36_06 que conduce a hipoacetilaciÓn en H3K18 en células leucémicas. Actividad antitumoral.
-
GC63662
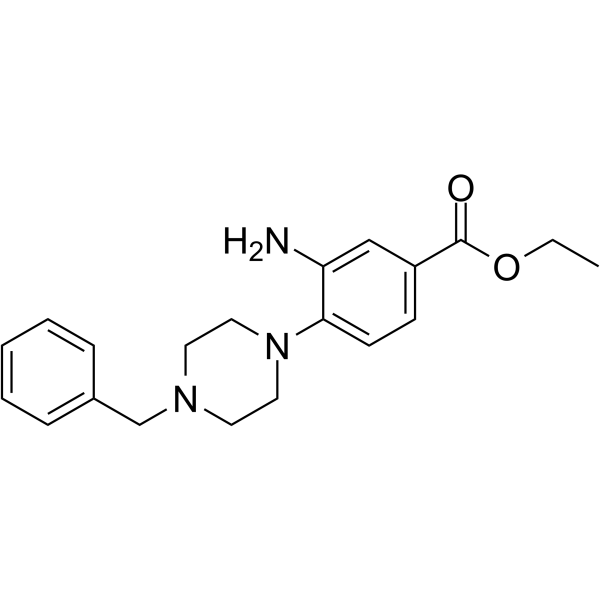
DCLX069
DCLX069 es un inhibidor selectivo de la proteÍna arginina metiltransferasa 1 (PRMT1) con un valor IC50 de 17,9 μM. DCLX069 se muestra menos activo frente a PRMT4 y PRMT6. DCLX069 tiene efectos anticancerÍgenos.
-
GC32872
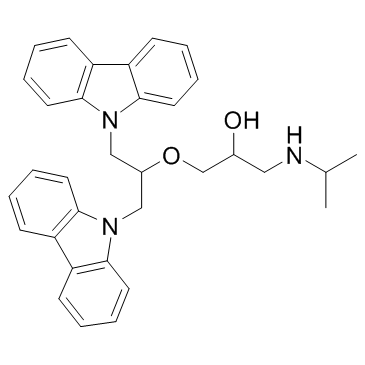
DC_517
DC_517 es un inhibidor de la ADN metiltransferasa 1 (DNMT1), con una IC50 y una Kd de 1,7 μM y 0,91 μM, respectivamente.
-
GC35816
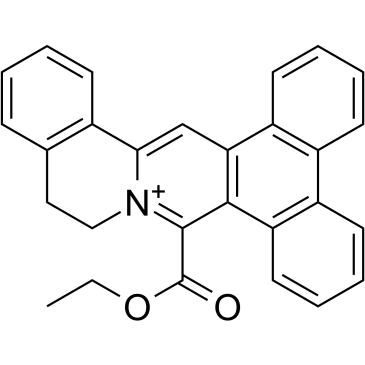
DC_C66
DC_C66 es un inhibidor selectivo de la arginina metiltransferasa 1 (CARM1) asociado a un coactivador permeable a las células con una IC50 de 1,8 μM. DC_C66 tiene una buena selectividad para CARM1 contra PRMT1 (IC50 = 21 μM), PRMT6 (IC50 = 47 μM) y PRMT5.
-
GC67863
DDO-2093 dihydrochloride

-
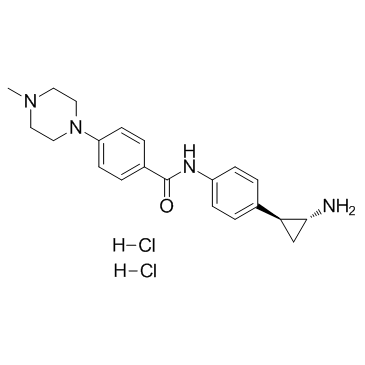
GC33013
DDP-38003 dihydrochloride
El diclorhidrato de DDP-38003 es un nuevo inhibidor disponible por vÍa oral de la desmetilasa 1A especÍfica de histona lisina (KDM1A/LSD1) con una IC50 de 84 nM.
-
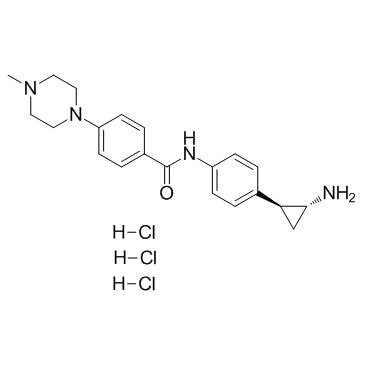
GC34296
DDP-38003 trihydrochloride
El trihidrocloruro de DDP-38003 es un nuevo inhibidor disponible por vÍa oral de la desmetilasa 1A especÍfica de histona lisina (KDM1A/LSD1) con una IC50 de 84 nM.
-
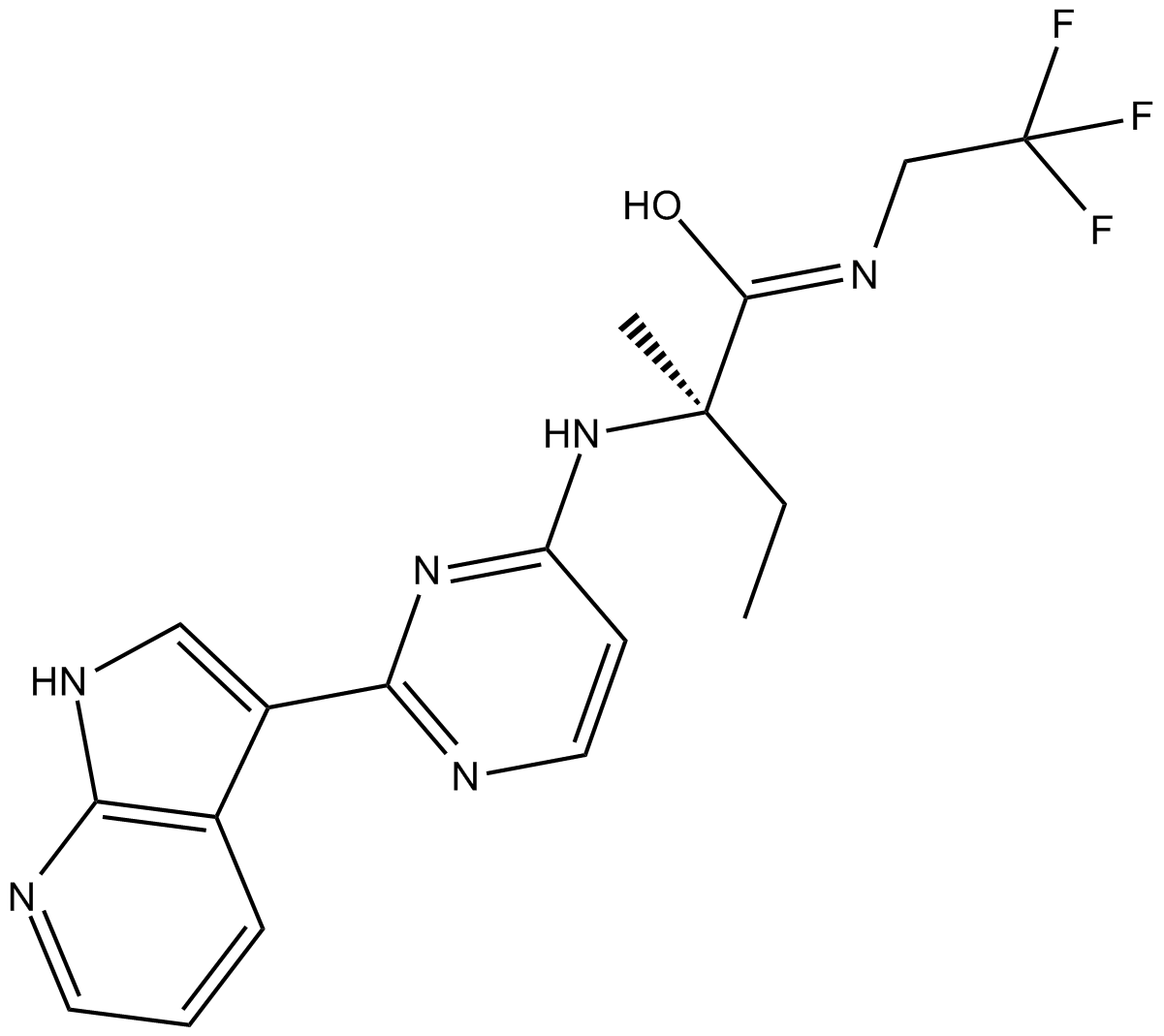
GC10770
Decernotinib(VX-509)
El decernotinib (VX-509) es un potente inhibidor de JAK3 activo por vÍa oral, con Kis de 2,5, 11, 13 y 11 nM para JAK3, JAK1, JAK2 y TYK2, respectivamente.
-
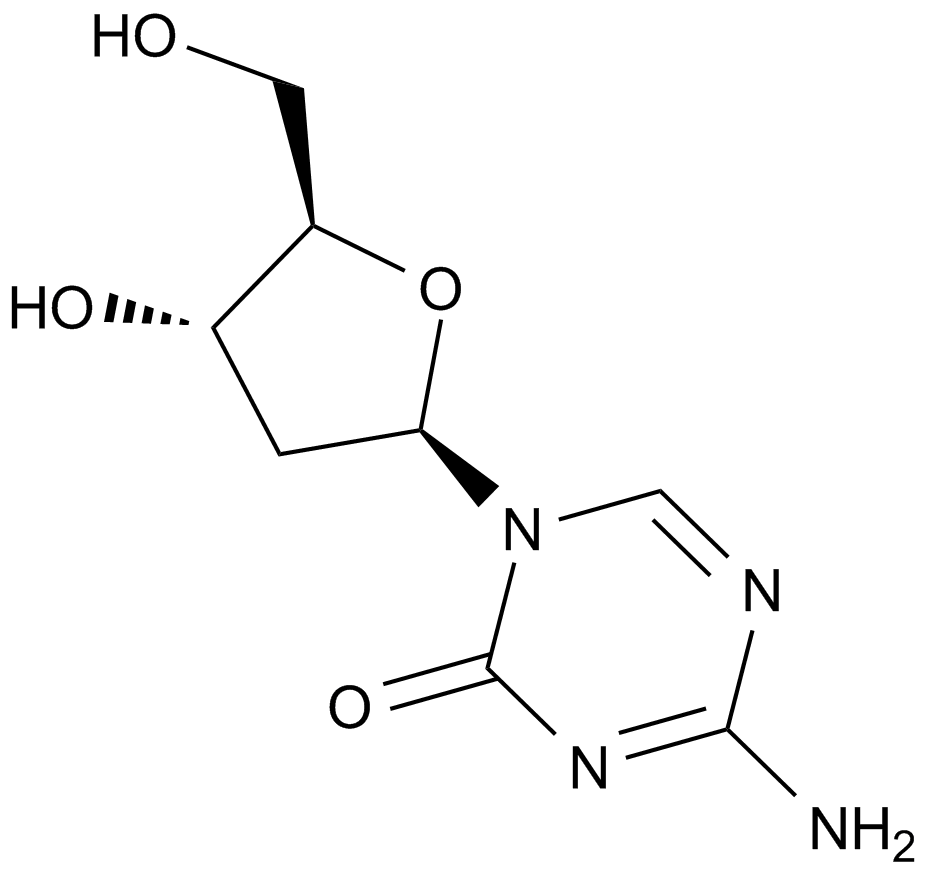
GC15255
Decitabine(NSC127716, 5AZA-CdR)
Un análogo de 2'-desoxi de 5-azacitidina.
-
GC68955
Deferoxamine
Deferoxamina (Deferoxamina B) is a chelating agent for iron (binding Fe(III) and many other metal cations), widely used to reduce the accumulation and deposition of iron in tissues. Deferoxamine has good antioxidant activity, can upregulate HIF-1α levels. Deferoxamine also has anti-proliferative activity, inducing cancer cell apoptosis and autophagy. Deferoxamine can be used in research on diabetes, neurodegenerative diseases, as well as anticancer and anti-COVID-19 studies.
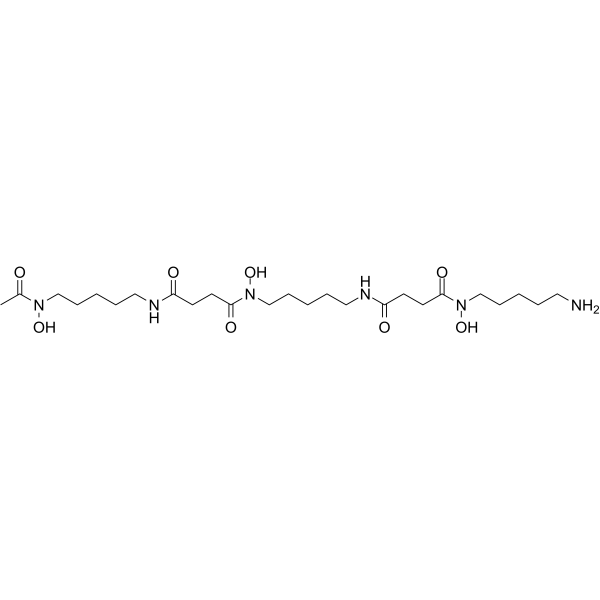
-
GN10040
Dehydrocorydaline
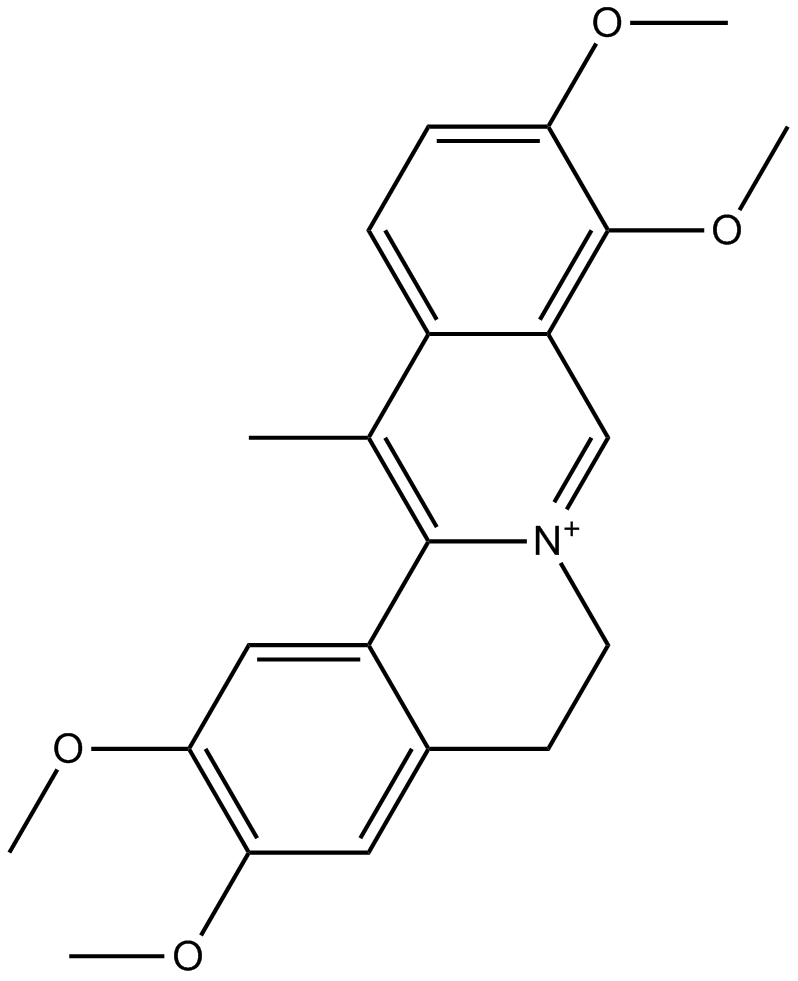
-
GC31660
Delgocitinib (JTE-052)
Delgocitinib (JTE-052) (JTE-052) es un inhibidor especÍfico de JAK con IC50 de 2,8, 2,6, 13 y 58 nM para JAK1, JAK2, JAK3 y Tyk2, respectivamente.
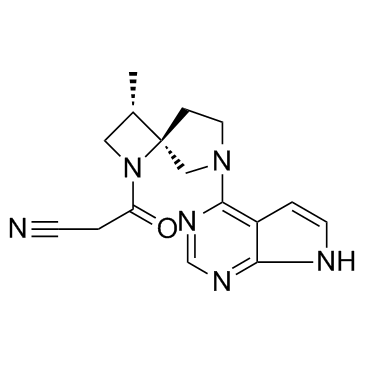
-
GC43406
Delphinidin (chloride)
La delfinidina (cloruro), una antocianidina, se aísla de las bayas y el vino tinto.
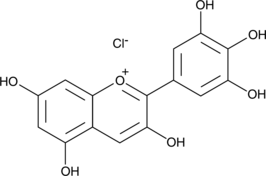
-
GC31230
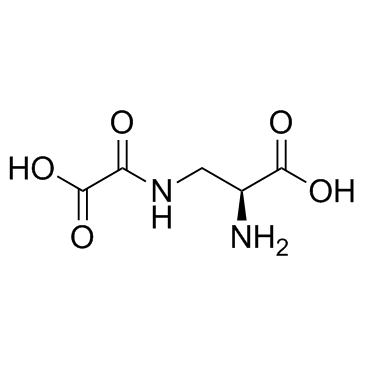
Dencichin (Dencichine)
Dencichin es un aminoÁcido no proteico extraÍdo originalmente de Panax notoginseng y puede inhibir la actividad de HIF-prolil hidroxilasa-2 (PHD-2).
-
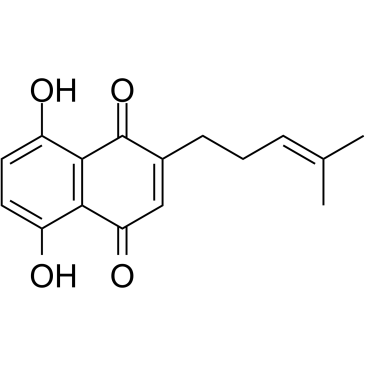
GC38767
Deoxyshikonin
La desoxishikonina se aÍsla de Lithospermum erythrorhizon Sieb con actividad antitumoral.
-
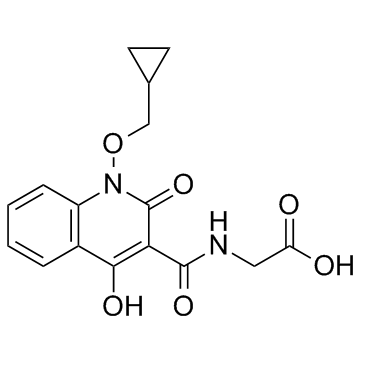
GC32449
Desidustat
Desidustat es un inhibidor de la hidroxilasa HIF activo por vÍa oral.
-
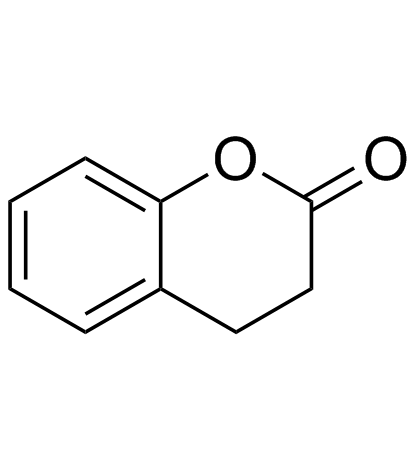
GC34187
Dihydrocoumarin (Hydrocoumarin)
La dihidrocumarina (Hydrocoumarin) es un compuesto que se encuentra en Melilotus officinalis. La dihidrocumarina (hidrocumarina) es un inhibidor de la levadura Sir2p. La dihidrocumarina (hidrocumarina) también inhibe la SIRT1 y la SIRT2 humanas con IC50 de 208 μM y 295 μM, respectivamente.
-
GC14024
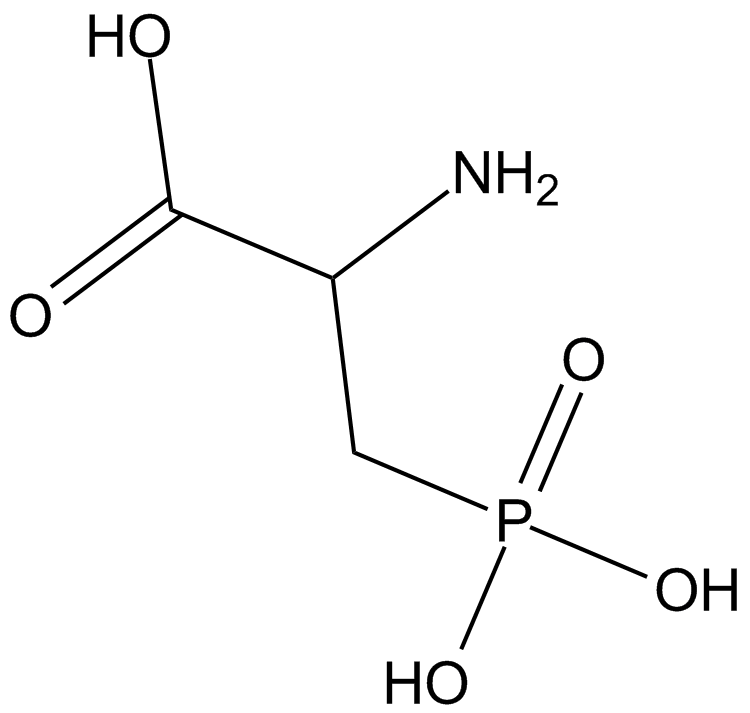
DL-AP3
A metabotropic glutamate receptor antagonist
-
GC52368
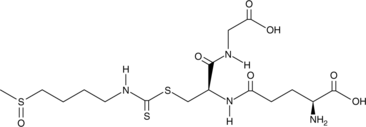
DL-Sulforaphane Glutathione
A metabolite of sulforaphane
-
GC65546
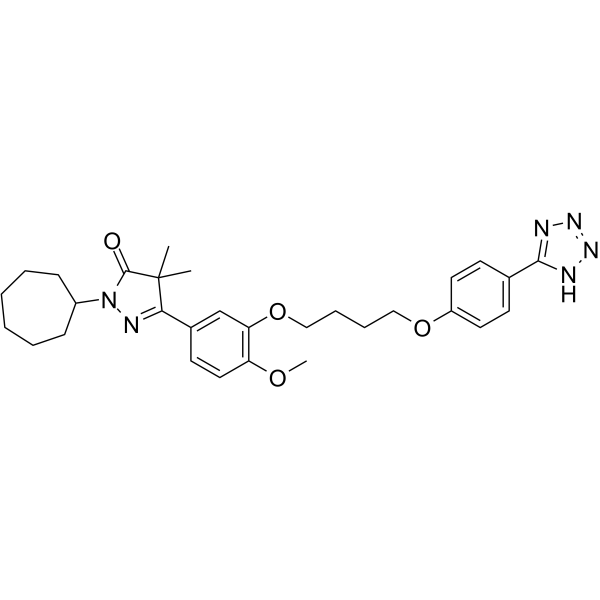
DNMT3A-IN-1
DNMT3A-IN-1 es un inhibidor potente y selectivo de DNMT3A.
-
GC35893
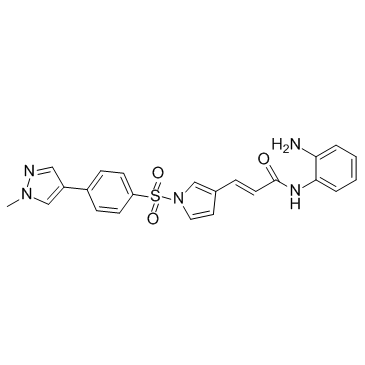
Domatinostat
Domatinostat (4SC-202 base libre) es un inhibidor selectivo de HDAC de clase I con IC50 de 1,20 μM, 1,12 μM y 0,57 μM para HDAC1, HDAC2 y HDAC3, respectivamente. También muestra actividad inhibitoria contra la desmetilasa 1 especÍfica de lisina (LSD1).
-
GC14490
Donepezil HCl
El clorhidrato de donepezilo (E2020) es un inhibidor selectivo y reversible de la AChE con una IC50 de 6,7 nM para la actividad de la AChE.
-
GC33208
Dot1L-IN-1
Dot1L-IN-1 es un inhibidor de Dot1L muy potente, selectivo y estructuralmente novedoso con una Ki de 2 pM.