Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Ziele für Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Produkte für Apoptosis
- Bestell-Nr. Artikelname Informationen
-
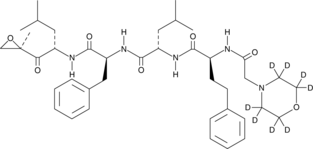
GC47042
Carfilzomib-d8
Ein interner Standard zur Quantifizierung von Carfilzomib.
-
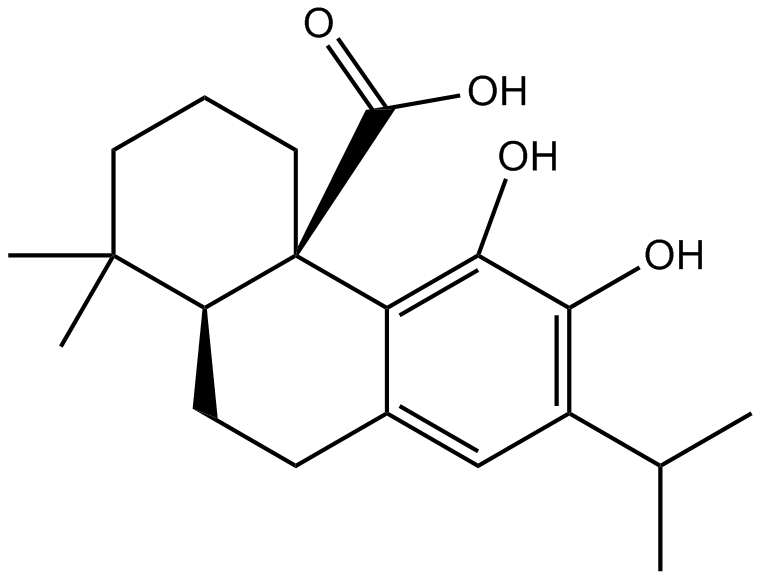
GN10733
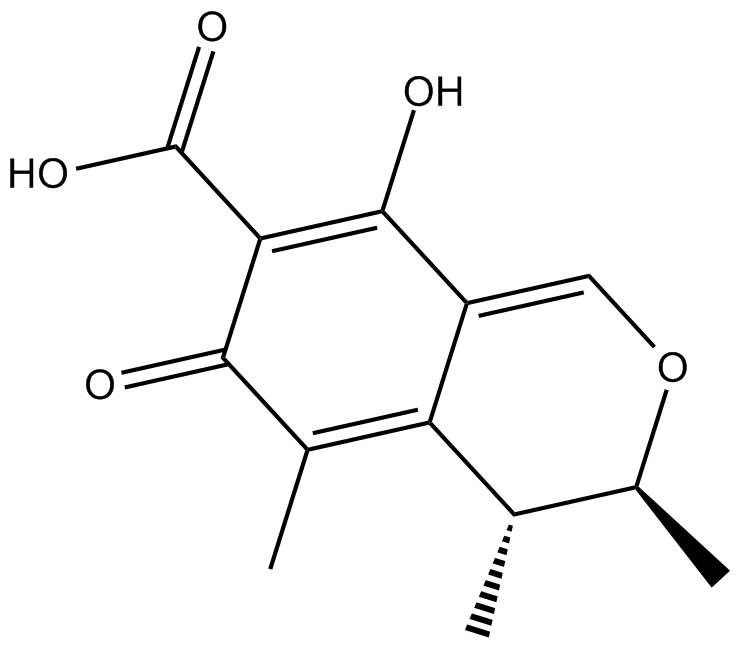
Carnosic acid
-
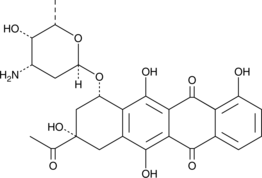
GC45679
Carubicin
Carubicin (Carminomycin) ist eine mikrobiell gewonnene Verbindung.
-
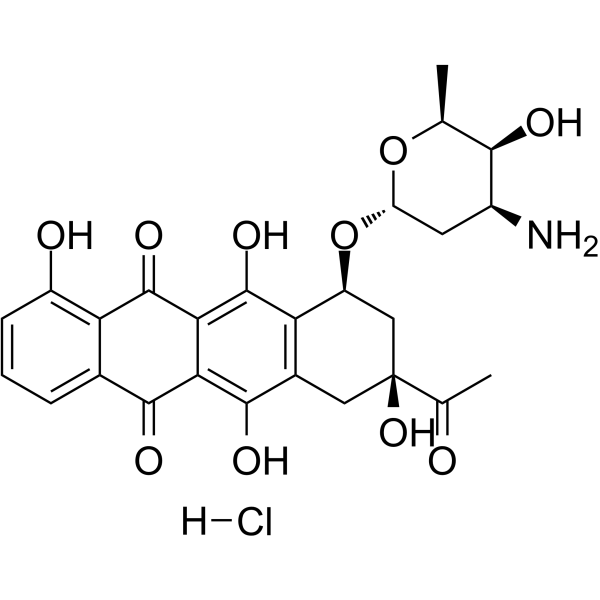
GC64110
Carubicin hydrochloride
Carubicinhydrochlorid ist eine mikrobiell gewonnene Verbindung.
-
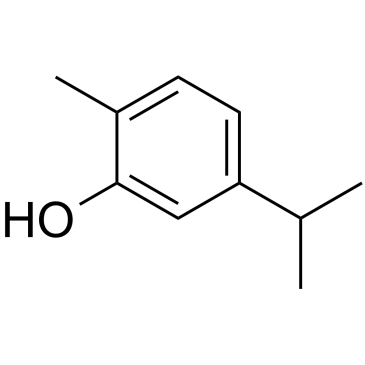
GC35612
Carvacrol
Carvacrol ist ein monoterpenoides Phenol, das aus Thymus mongolicus Ronn isoliert wird.
-
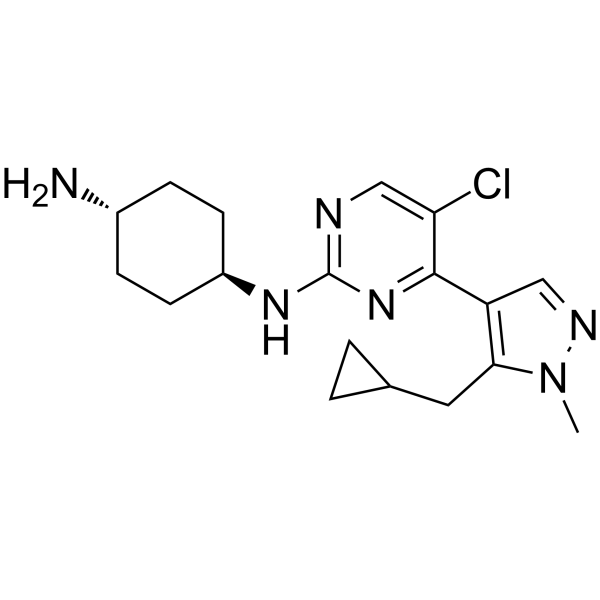
GC62442
Casein Kinase inhibitor A51
Caseinkinase-Inhibitor A51 ist ein potenter und oral aktiver Caseinkinase-1α (CK1α)-Inhibitor. Der Caseinkinase-Inhibitor A51 induziert die Apoptose von LeukÄmiezellen und hat starke antileukÄmische AktivitÄten.
-
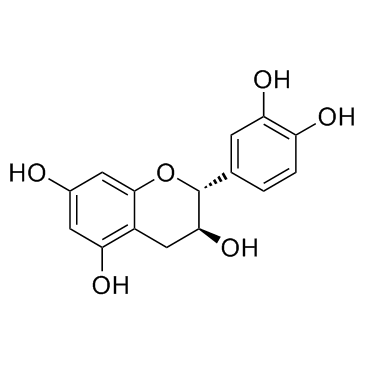
GC32841
Catechin ((+)-Catechin)
Catechin ((+)-Catechin) ((+)-Catechin ((+)-Catechin)) hemmt Cyclooxygenase-1 (COX-1) mit einem IC50 von 1,4 μM.
-
GN10543
caudatin
-
GC43149
CAY10404
CAY10404 ist ein potenter und selektiver Cyclooxygenase-2 (COX-2)-Inhibitor mit einem IC50 von 1 nM und einem SelektivitÄtsindex (SI; COX-1 IC50/COX-2 IC50) von >500000.
-
GC43150
CAY10406
CAY10406 is a trifluoromethyl analog of an isatin sulfonamide compound that selectively inhibits caspases 3 and 7.
-
GC43154
CAY10443
Mitochondrial release of cytochrome c triggers apoptosis via the assembly of a multimeric complex including caspase-9, Apaf-1, and other components, sometimes called the apoptosome.
-
GC43176
CAY10575
CAY10575 (Verbindung 8) ist ein IKK2-Inhibitor mit einem IC50 von 0,075 μM.
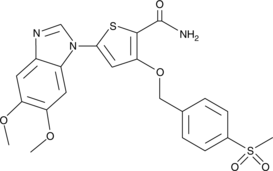
-
GC18530
CAY10616
Resveratrol is a natural polyphenolic antioxidant that has anti-cancer properties.
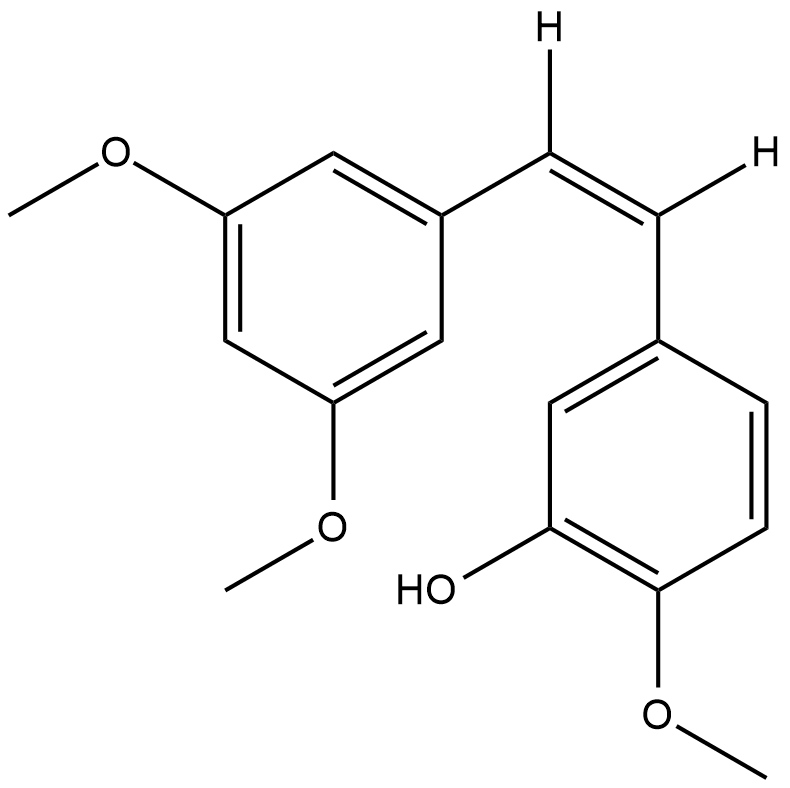
-
GC41317
CAY10625
Survivin is a cellular protein implicated in cell survival by interacting with and inhibiting the apoptotic function of several proteins including Smac/DIABLO, caspase-3, and caspase-7.
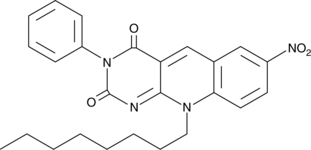
-
GC43189
CAY10681
Inactivation of the tumor suppressor p53 commonly coincides with increased signaling through NF-κB in cancer.
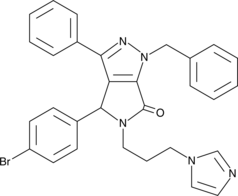
-
GC43190
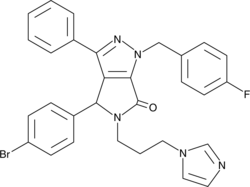
CAY10682
(±)-Nutlin-3 blocks the interaction of p53 with its negative regulator Mdm2 (IC50 = 90 nM), inducing the expression of p53-regulated genes and blocking the growth of tumor xenografts in vivo.
-
GC40650
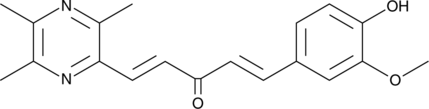
CAY10706
CAY10706 is a ligustrazine-curcumin hybrid that promotes intracellular reactive oxygen species accumulation preferentially in lung cancer cells.
-
GC43198
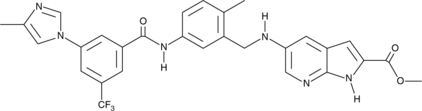
CAY10717
CAY10717 is a multi-targeted kinase inhibitor that exhibits greater than 40% inhibition of 34 of 104 kinases in an enzymatic assay at a concentration of 100 nM.
-
GC43203
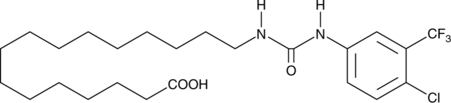
CAY10726
CAY10726 is an arylurea fatty acid.
-
GC46113
CAY10744
A topoisomerase II-α poison
-
GC47053
CAY10746
A ROCK1 and ROCK2 inhibitor
-
GC48392
CAY10747
An inhibitor of the Hsp90-Cdc37 protein-protein interaction
-
GC47055
CAY10749
Cay10749 (Verbindung 15) ist ein potenter PARP/PI3K-Inhibitor mit PIC50-Werten von 8,22, 8,44, 8,25, 6,54, 8,13, 6,08 fÜr PARP-1, PARP-2, PI3K&7777#945; offlineefficient_models_2022q2.md.en_de_2022q1.mdCAY10749 is a highly effective anticancer compound targeted against a wide range of oncologic diseases.en_de_2022q1.md
-
GC47057
CAY10755
A fungal metabolite with anticancer activity
-
GC47061
CAY10763
A dual inhibitor of IDO1 and STAT3 activation
-
GC47065
CAY10773
A derivative of sorafenib
-
GC49080
CAY10786
CAY10786 (Verbindung 43) ist ein GPR52-Antagonist mit einem IC50 von 0,63 μM.
-
GC52245
CAY10792
An anticancer agent
-
GC14634
CBL0137
curaxin that activates p53 and inhibits NF-κB
-
GC15394
CBL0137 (hydrochloride)
CBL0137 (Hydrochlorid) ist ein Inhibitor des Histon-Chaperons FACT. CBL0137 (Hydrochlorid) kann auch p53 aktivieren und hemmt NF-κB mit EC50-Werten von 0,37 bzw. 0,47 µM.
-
GC61636
CBR-470-2
CBR-470-2, ein Glycin-substituiertes Analogon, kann die NRF2-Signalgebung aktivieren.
-
GC13648
CC-223
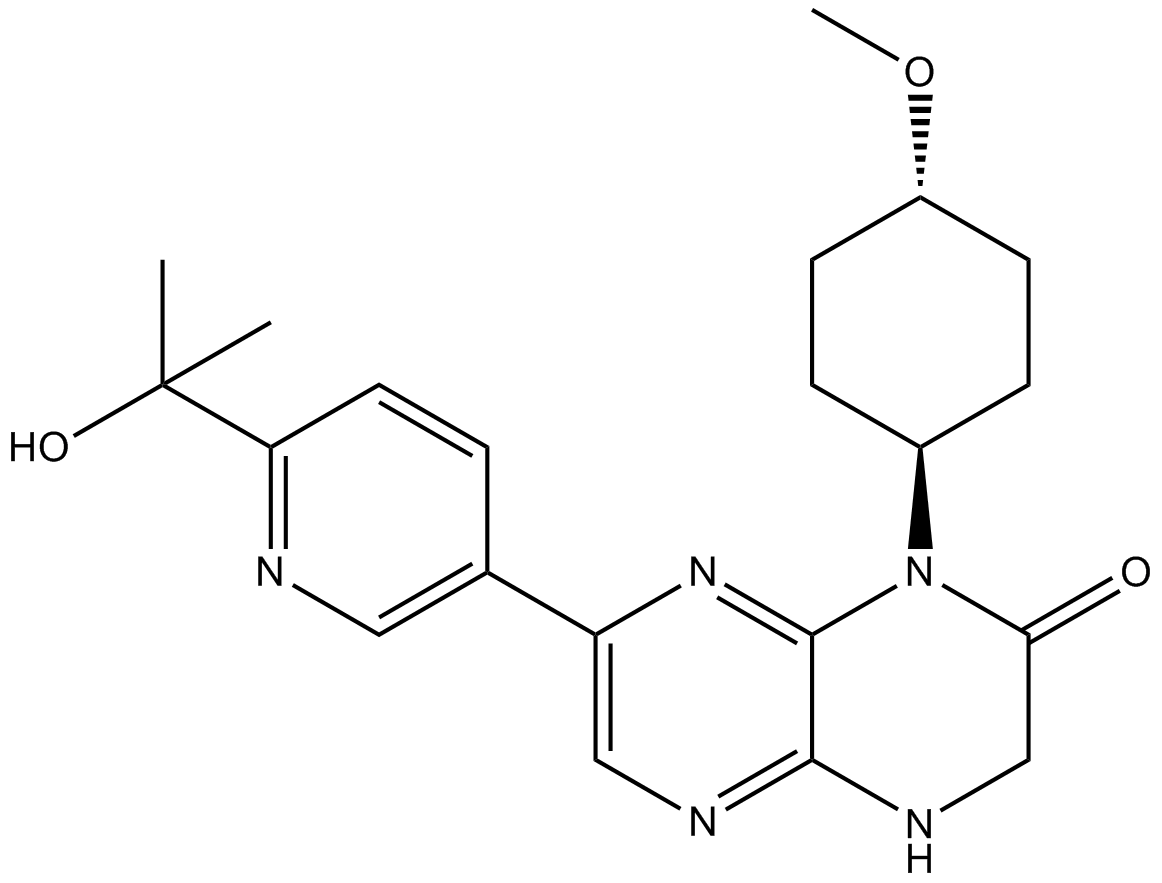
CC-223 (CC-223) ist ein potenter, selektiver und oral bioverfÜgbarer Inhibitor der mTOR-Kinase mit einem IC50-Wert fÜr mTOR-Kinase von 16 nM. CC-223 hemmt sowohl mTORC1 als auch mTORC2.
-
GC39169
CC-92480
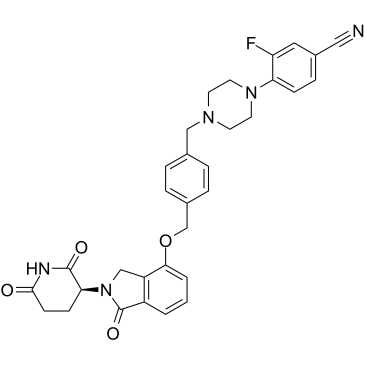
CC-92480 (CC-92480), ein Cereblon-E3-Ubiquitin-Ligase-modulierendes Medikament (CELMoD), wirkt als molekularer Klebstoff. CC-92480 zeigt eine hohe AffinitÄt zu Cereblon, was zu einer starken Antimyelom-AktivitÄt fÜhrt.
-
GC19088
CC122
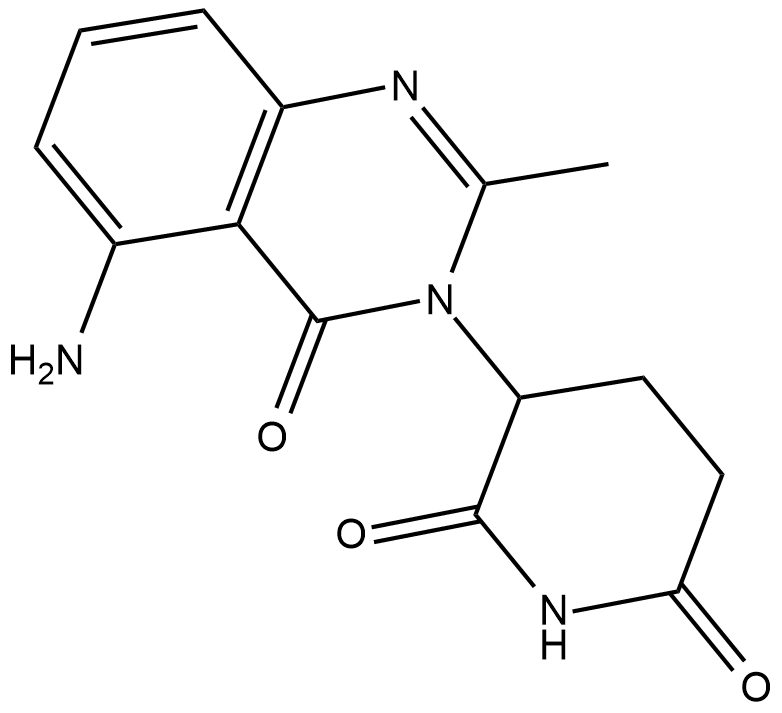
CC122 (CC 122) ist ein oral aktiver Cereblon-Modulator.
-
GC61532
CCI-007
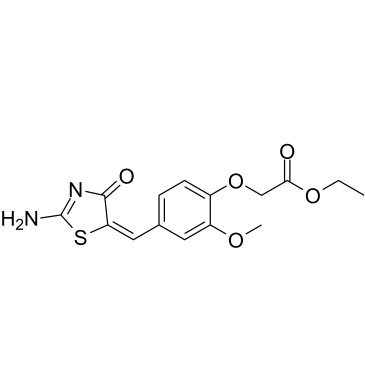
CCI-007 ist ein kleines MolekÜl mit zytotoxischer AktivitÄt gegen SÄuglingsleukÄmie mit MLL-Umlagerungen mit IC50-Werten von 2,5-6,2 μM in empfindlichen Zellen.
-
GC12891
CCT007093
CCT007093 ist ein wirksamer Inhibitor der Proteinphosphatase 1D (PPM1D Wip1). Die Wip1-Hemmung kann den mTORC1-Signalweg aktivieren und die Hepatozytenproliferation nach Hepatektomie verbessern.
-
GC14566
CCT137690
An inhibitor of Aurora kinases and FLT3
-
GC62561
CCT369260
CCT369260 (Verbindung 1) ist ein oral wirksamer Inhibitor des B-Zell-Lymphoms 6 (BCL6) mit AntitumoraktivitÄt. CCT369260 (Verbindung 1) weist einen IC50 von 520 nM auf.
-
GC33337
CDC801
CDC801 ist eine potente und oral aktive Phosphodiesterase 4 (PDE4) und ein Tumornekrosefaktor-α (TNF-α) Inhibitor mit IC50 von 1,1 μM bzw. 2,5 μM.
-
GC39555
CDDO-2P-Im
CDDO-2P-Im ist ein Analogon von CDDO-Imidazolid mit chemoprÄventiver Wirkung. CDDO-2P-Im kann die GrÖße und Schwere der Lungentumore im Maus-Lungenkrebsmodell reduzieren.
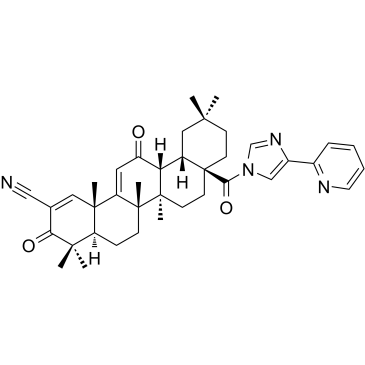
-
GC39556
CDDO-3P-Im
CDDO-3P-Im ist ein Analogon von CDDO-Imidazolid mit chemoprÄventiver Wirkung. CDDO-3P-Im kann die GrÖße und Schwere der Lungentumore im Maus-Lungenkrebsmodell reduzieren. CDDO-3P-Im ist ein oral aktiver Nekroptose-Inhibitor, der fÜr die Erforschung von IschÄmie/Reperfusion (I/R) verwendet werden kann.
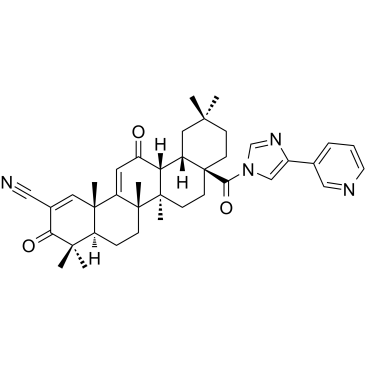
-
GC35629
CDDO-dhTFEA
CDDO-dhTFEA (RTA dh404) ist eine synthetische Oleanan-Triterpenoid-Verbindung, die Nrf2 wirksam aktiviert und den entzÜndungsfÖrdernden Transkriptionsfaktor NF-κB hemmt.
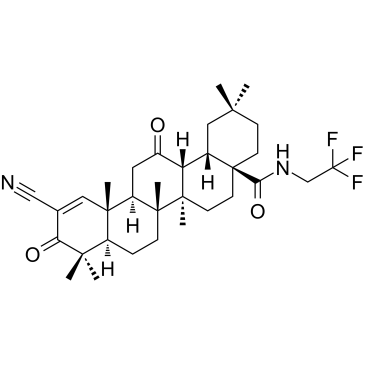
-
GC35630
CDDO-EA
CDDO-EA ist ein mit NF-E2 verwandter Faktor 2/antioxidatives Response-Element (Nrf2/ARE)-Aktivator.

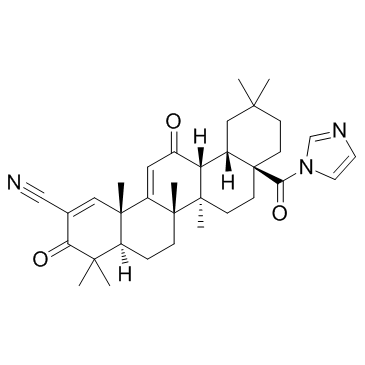
-
GC32723
CDDO-Im (RTA-403)
CDDO-Im (RTA-403) (RTA-403) ist ein Aktivator von Nrf2 und PPAR, mit Kis von 232 und 344 nM fÜr PPARα und PPARγ.
-
GC16625
CDDO-TFEA
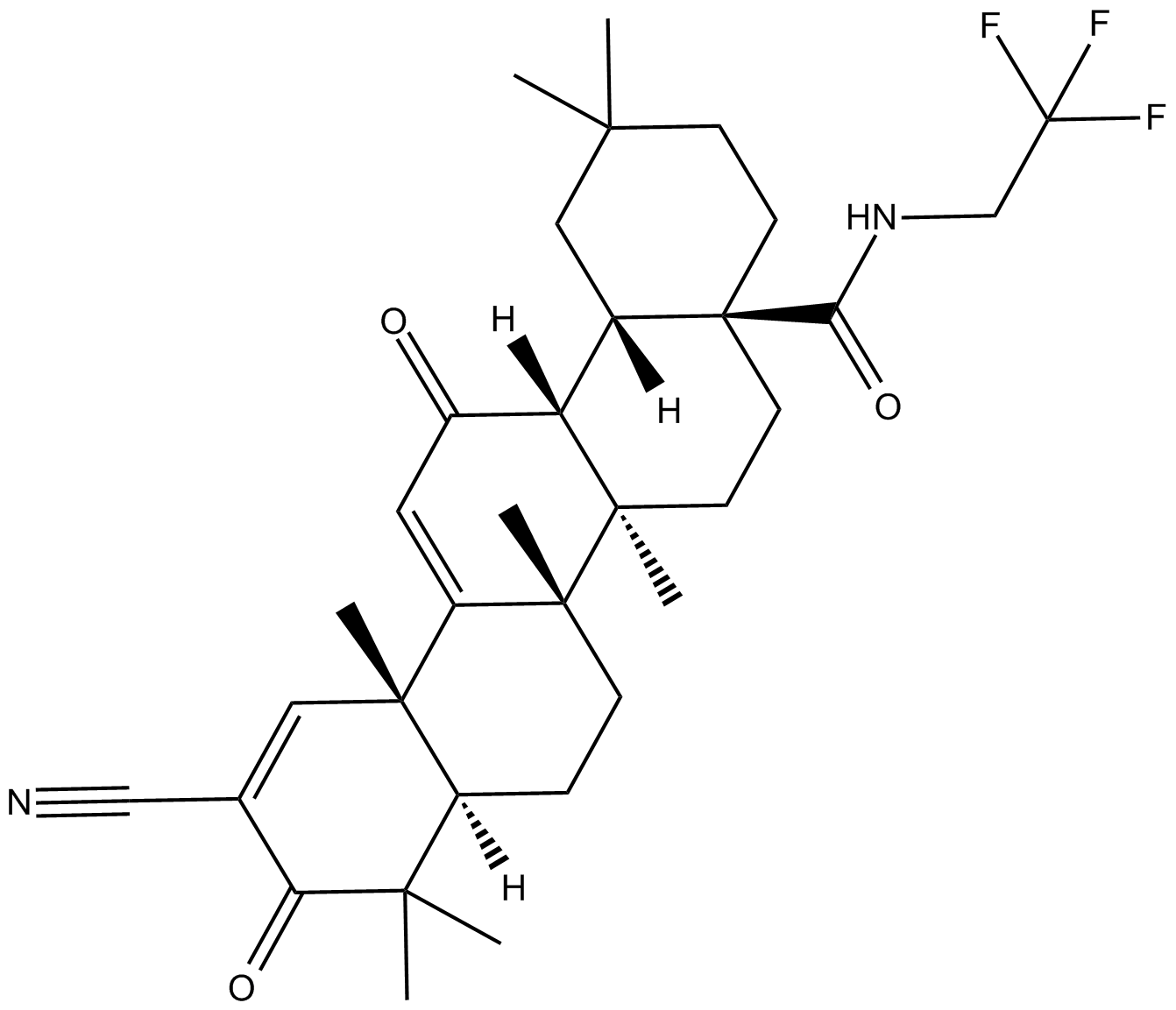
Nrf2 activator
-
GC43217
CDK/CRK Inhibitor
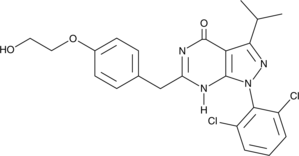
CDK/CRK inhibitor is an inhibitor of cyclin-dependent kinases (CDK) and CDK-related kinases (CRK) with IC50 values ranging from 9-839 nM in vitro.
-
GC62596
CDK7-IN-3
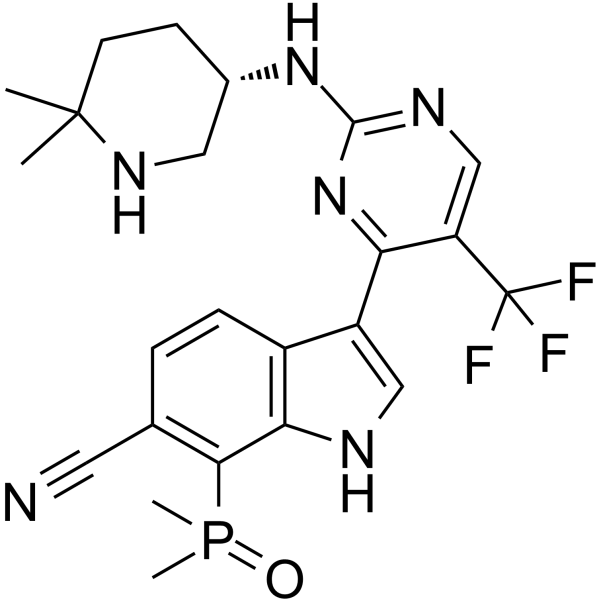
CDK7-IN-3 (CDK7-IN-3) ist ein oral aktiver, hochselektiver, nichtkovalenter CDK7-Inhibitor mit einer KD von 0,065 nM. CDK7-IN-3 zeigt eine schwache Hemmung von CDK2 (Ki = 2600 nM), CDK9 (Ki = 960 nM), CDK12 (Ki = 870 nM). CDK7-IN-3 induziert Apoptose in Tumorzellen und hat AntitumoraktivitÄt.
-
GC35636
CDK9-IN-7
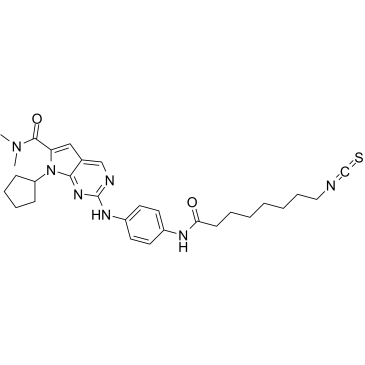
CDK9-IN-7 (Verbindung 21e) ist ein selektiver, hochpotenter und oral aktiver CDK9/Cyclin-T-Inhibitor (IC50=11 nM), der stÄrker als andere CDKs ist (CDK4/CyclinD=148 nM; CDK6/CyclinD= 145 nM). CDK9-IN-7 zeigt AntitumoraktivitÄt ohne offensichtliche ToxizitÄt. CDK9-IN-7 induziert NSCLC-Zellapoptose, hÄlt den Zellzyklus in der G2-Phase an und unterdrÜckt die Stammzelleneigenschaften von NSCLC.
-
GC19096
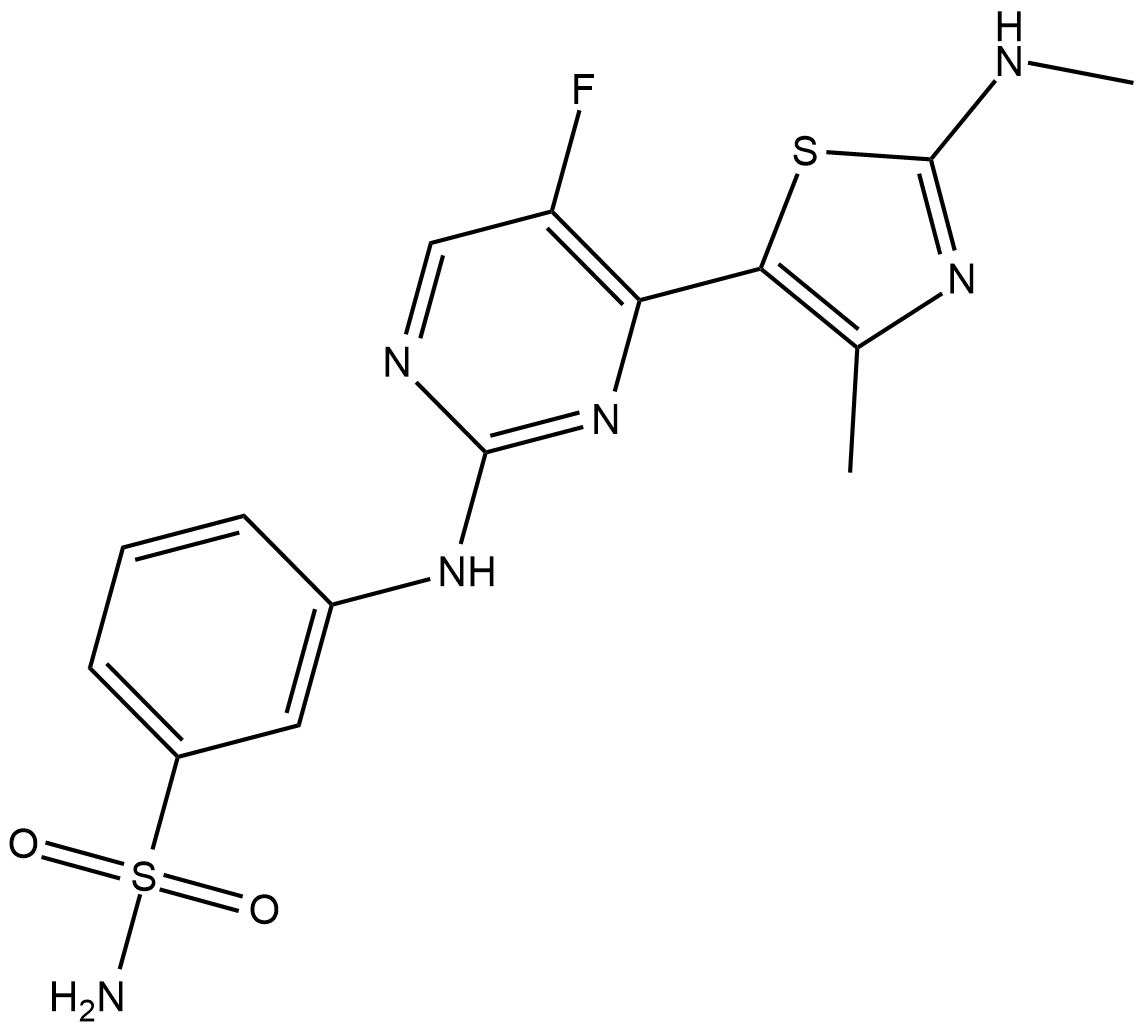
CDKI-73
CDKI-73 (LS-007) ist ein oral aktiver und hochwirksamer CDK9-Inhibitor mit Ki-Werten von 4 nM, 4 nM und 3 nM fÜr CDK9, CDK1 bzw. CDK2.
-
GC61865
Cearoin
Cearoin erhÖht die Autophagie und Apoptose durch die Produktion von ROS und die Aktivierung von ERK.
-
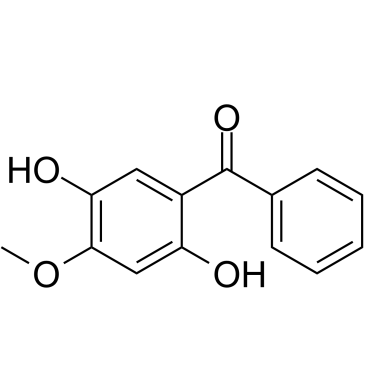
GC15083
Celastrol
A triterpenoid antioxidant
-
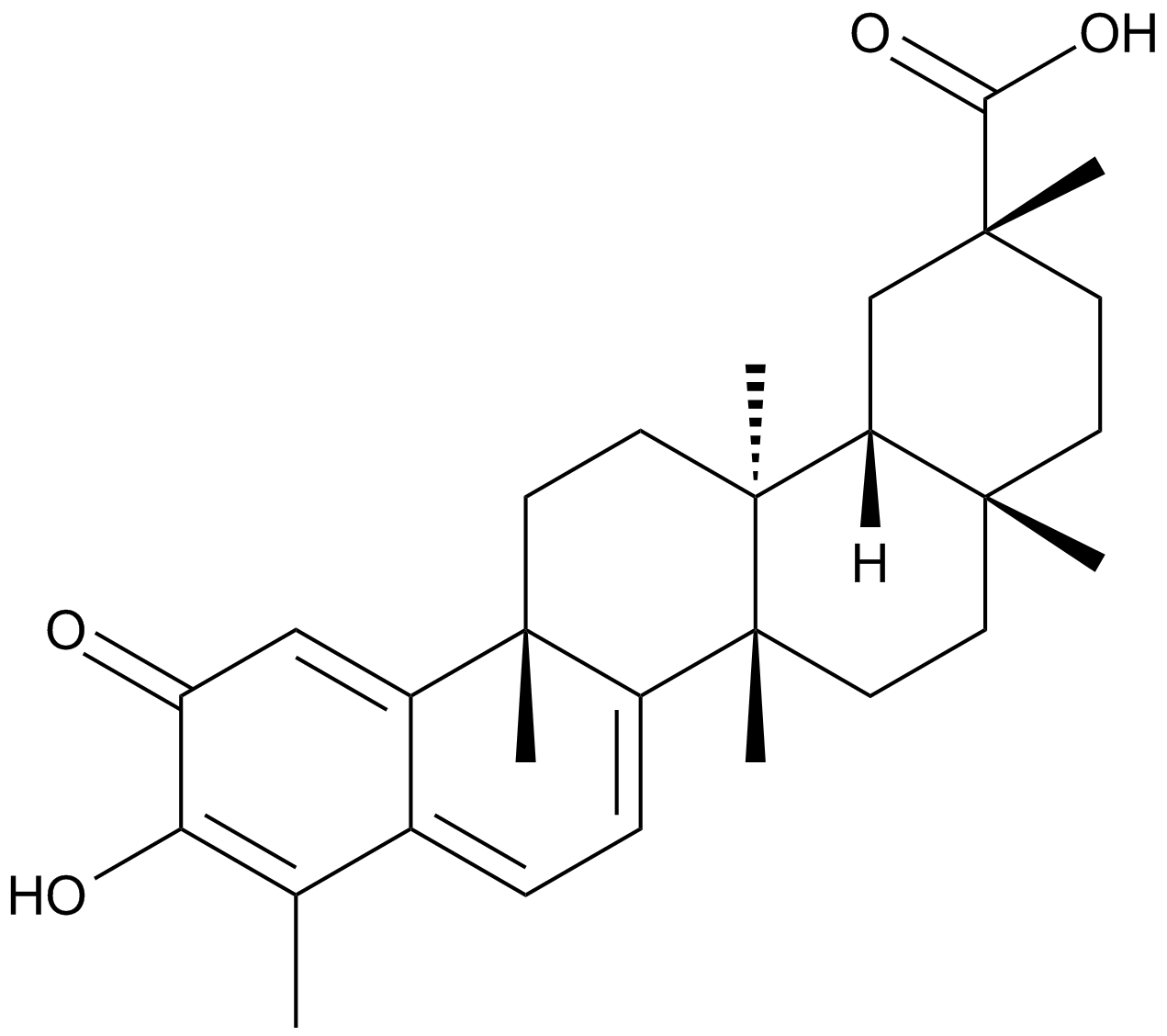
GC49152
Celecoxib Carboxylic Acid
An inactive metabolite of celecoxib
-
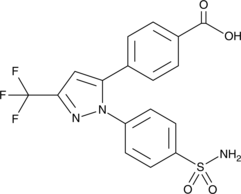
GC47070
Celecoxib-d7
An internal standard for the quantification of celecoxib
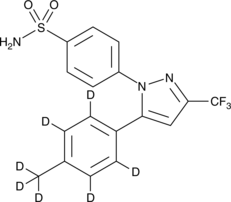
-
GC18392
Cellocidin
Cellocidin is an antibiotic originally isolated from S.
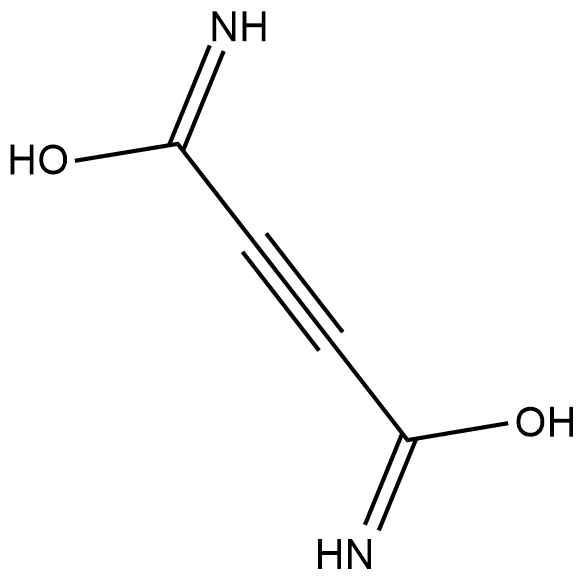
-
GN10113
Cepharanthine
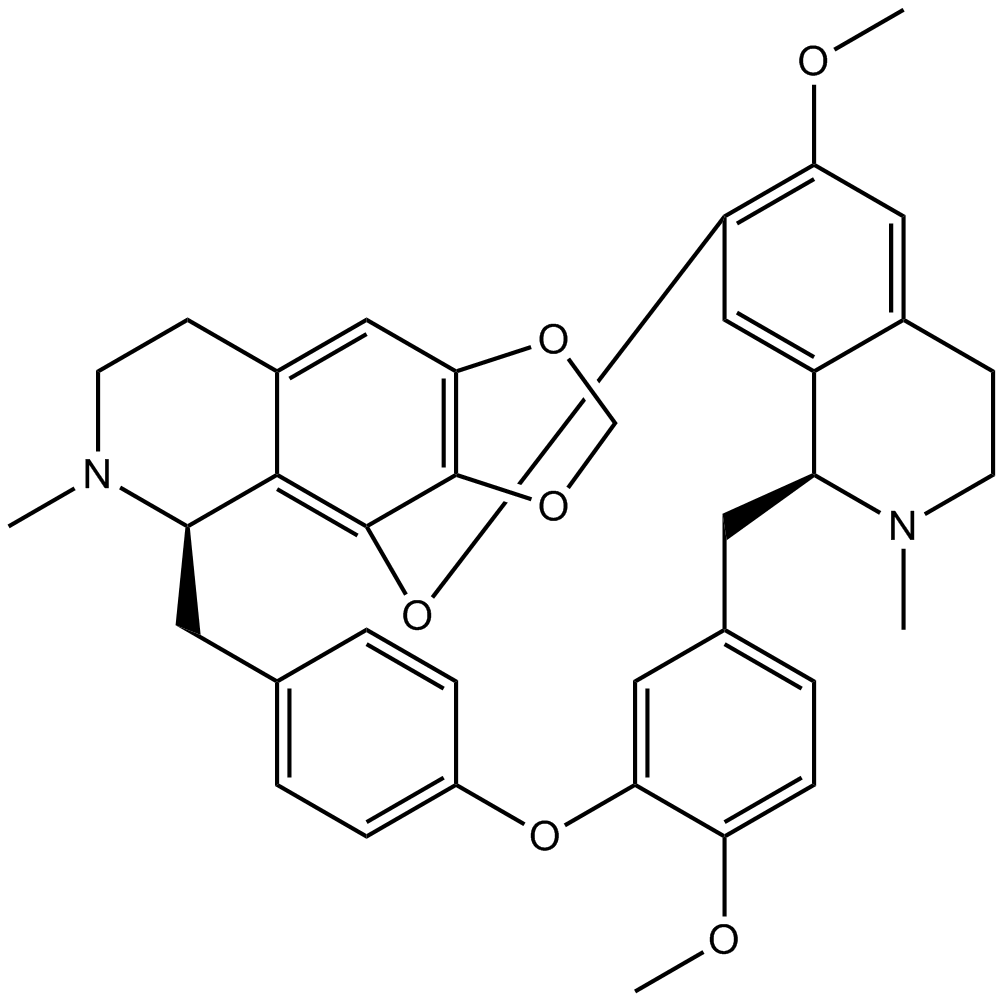
-
GC52489
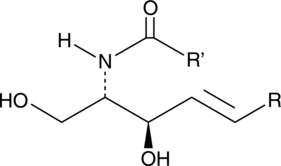
Ceramide (hydroxy) (bovine spinal cord)
A sphingolipid
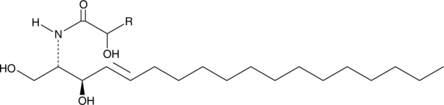
-
GC52485
Ceramide (non-hydroxy) (bovine spinal cord)
A sphingolipid
-
GC52486
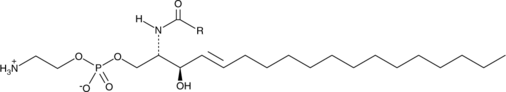
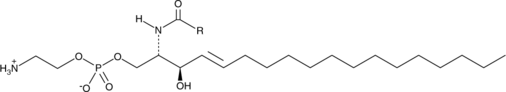
Ceramide Phosphoethanolamine (bovine)
A sphingolipid
-
GC43229
Ceramide Phosphoethanolamines (bovine)
Ceramide phosphoethanolamine (CPE) is an analog of sphingomyelin that contains ethanolamine rather than choline as the head group.
-
GC47073
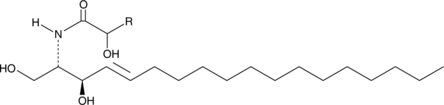
Ceramides (hydroxy)
A mixture of hydroxy fatty acid-containing ceramides
-
GC43230
Ceramides (non-hydroxy)
Ceramides are generated from sphingomyelin through activation of sphingomyelinases or through the de novo synthesis pathway, which requires the coordinated action of serine palmitoyl transferase and ceramide synthase.
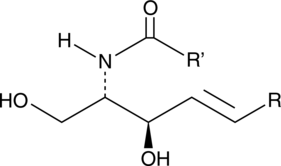
-
GC49706
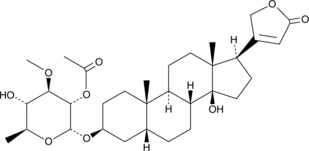
Cerberin
A cardiac glycoside with cytotoxic and cardiac modulatory activities
-
GC60688
Cereblon modulator 1
Cereblon Modulator 1 (CC-90009) ist ein erstklassiger GSPT1-selektiver Cereblon (CRBN) E3-Ligase-Modulator, der als molekularer Klebstoff fungiert.
-
GC65487
Certolizumab pegol
Certolizumab pegol (Certolizumab) ist ein rekombinanter, polyethylenglykolierter Antigen-bindender Fragment eines humanisierten monoklonalen Antikörpers, der selektiv Tumor-Nekrose-Faktor-α (TNF-α) zielt und neutralisiert.

-
GC11543
Cesium chloride
-
GC11710
CFM 4
CFM 4 ist ein potenter niedermolekularer Antagonist der CARP-1/APC-2-Bindung. CFM 4 verhindert die Bindung von CARP-1 an APC-2, verursacht einen Stillstand des G2M-Zellzyklus und induziert Apoptose mit einem IC50-Bereich von 10-15 μM. CFM 4 unterdrÜckt auch das Wachstum von arzneimittelresistenten menschlichen Brustkrebszellen.
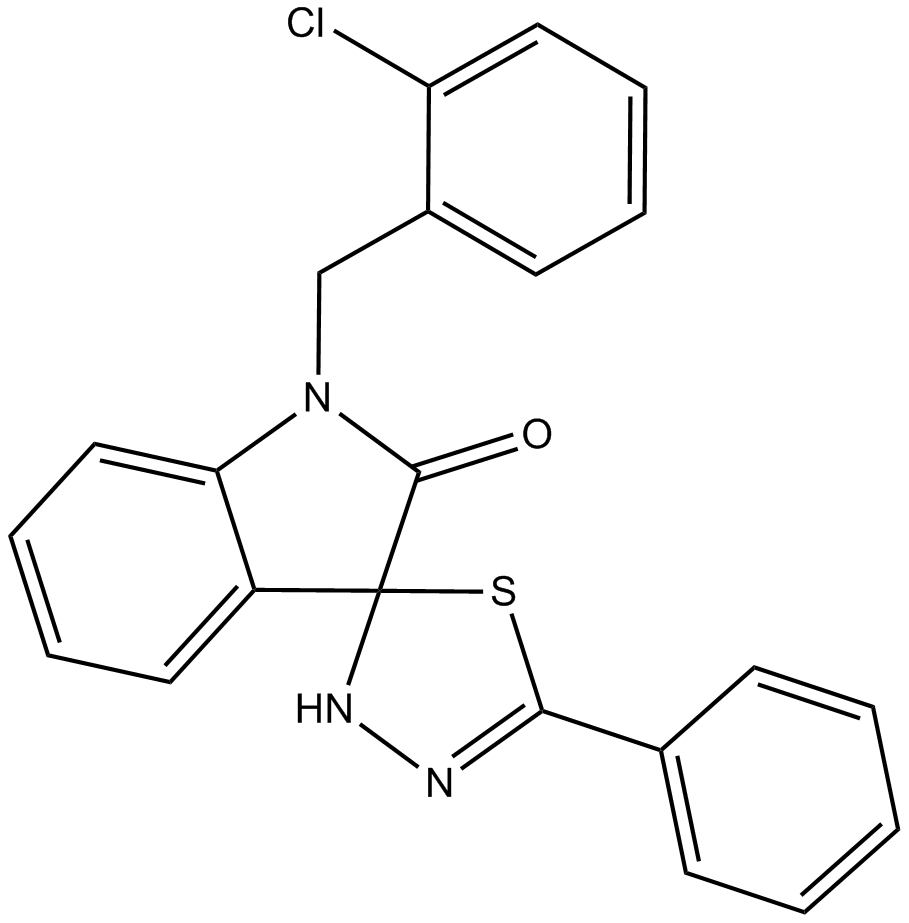
-
GC35668
CG-200745
CG-200745 (CG-200745) ist ein oral aktiver, potenter pan-HDAC-Inhibitor, der die HydroxamsÄureeinheit hat, um Zink am Boden der katalytischen Tasche zu binden. CG-200745 hemmt die Deacetylierung von Histon H3 und Tubulin. CG-200745 induziert die Akkumulation von p53, fÖrdert die p53-abhÄngige Transaktivierung und verstÄrkt die Expression der Proteine MDM2 und p21 (Waf1/Cip1). CG-200745 erhÖht die Empfindlichkeit von Gemcitabin-resistenten Zellen gegenÜber Gemcitabin und 5-Fluorouracil (5-FU; ). CG-200745 induziert Apoptose und hat Antitumorwirkungen.
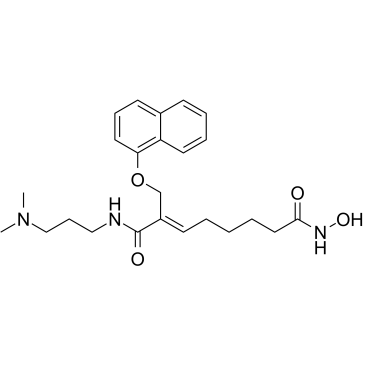
-
GC10666
CGP 57380
CGP 57380 ist eine zellgÄngige Pyrazolo-Pyrimidin-Verbindung, die mit einem IC50-Wert von 2,2 μM als selektiver Inhibitor von Mnk1 wirkt, aber keine inhibitorische AktivitÄt gegen p38, JNK1, ERK1/2, PKC oder Src-Ähnliche Kinasen aufweist.
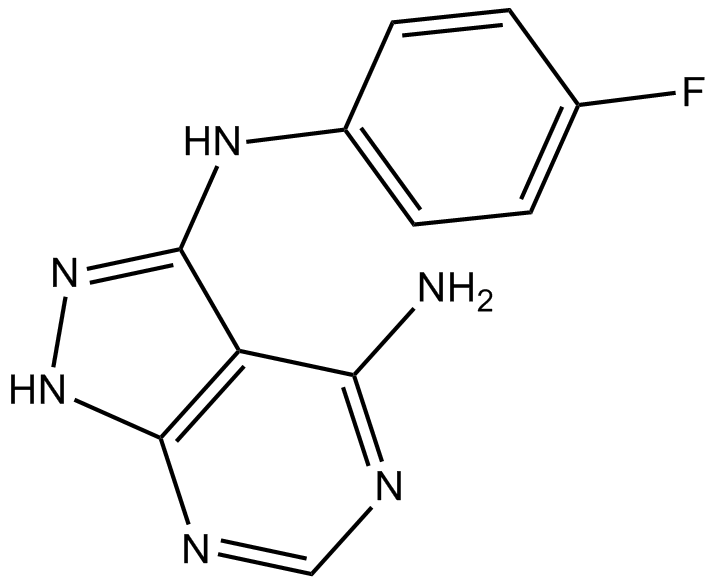
-
GC43234
Chaetoglobosin A
Chaetoglobosin A, der Wirkstoff im Extrakt von Penicillium aquamarinium, ist ein Mitglied der Familie der Cytochalasane.
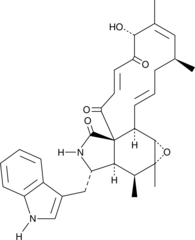
-
GC18536
Chartreusin
Chartreusin is an antibiotic originally isolated from S.
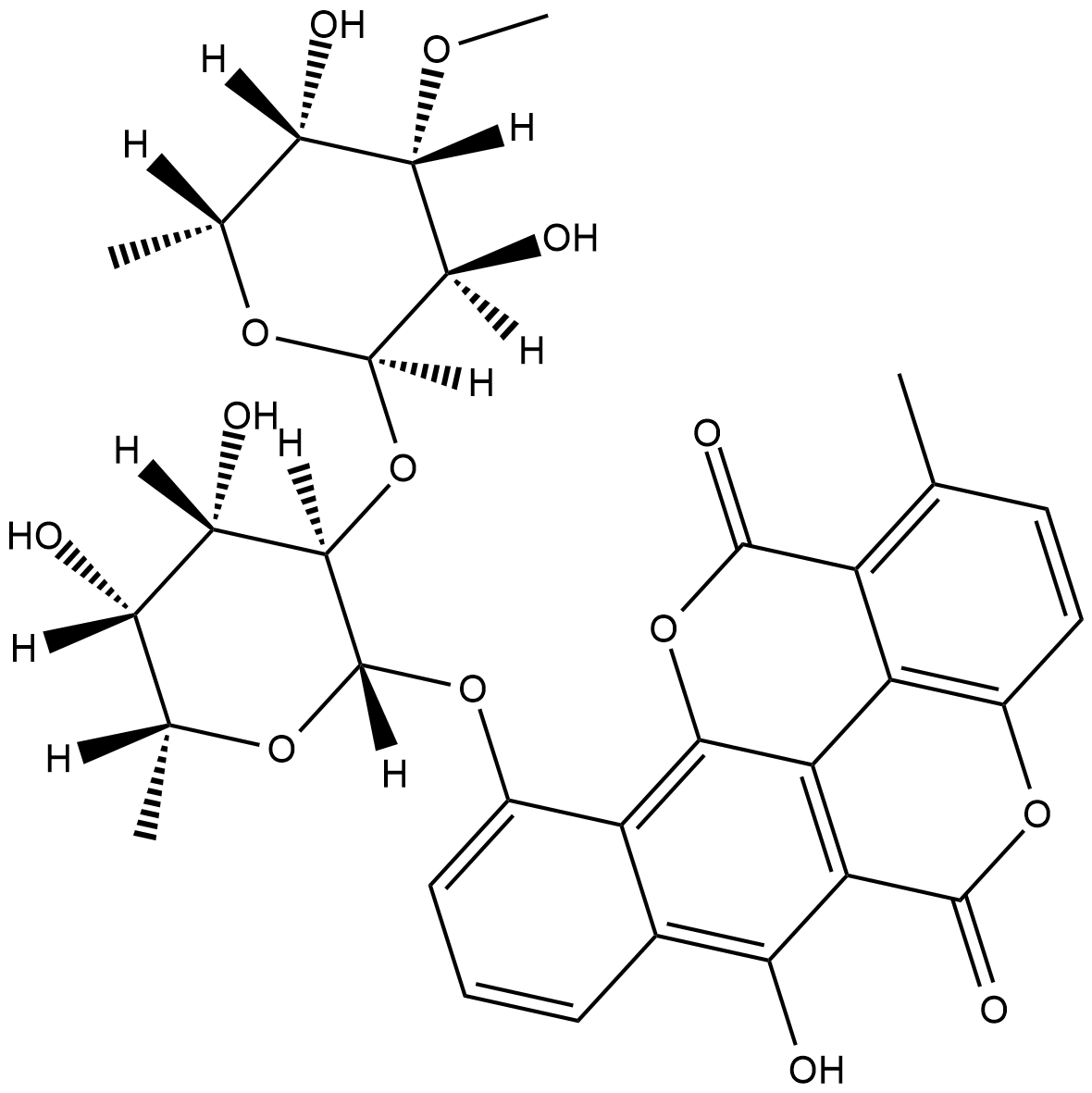
-
GN10463
Chelerythrine
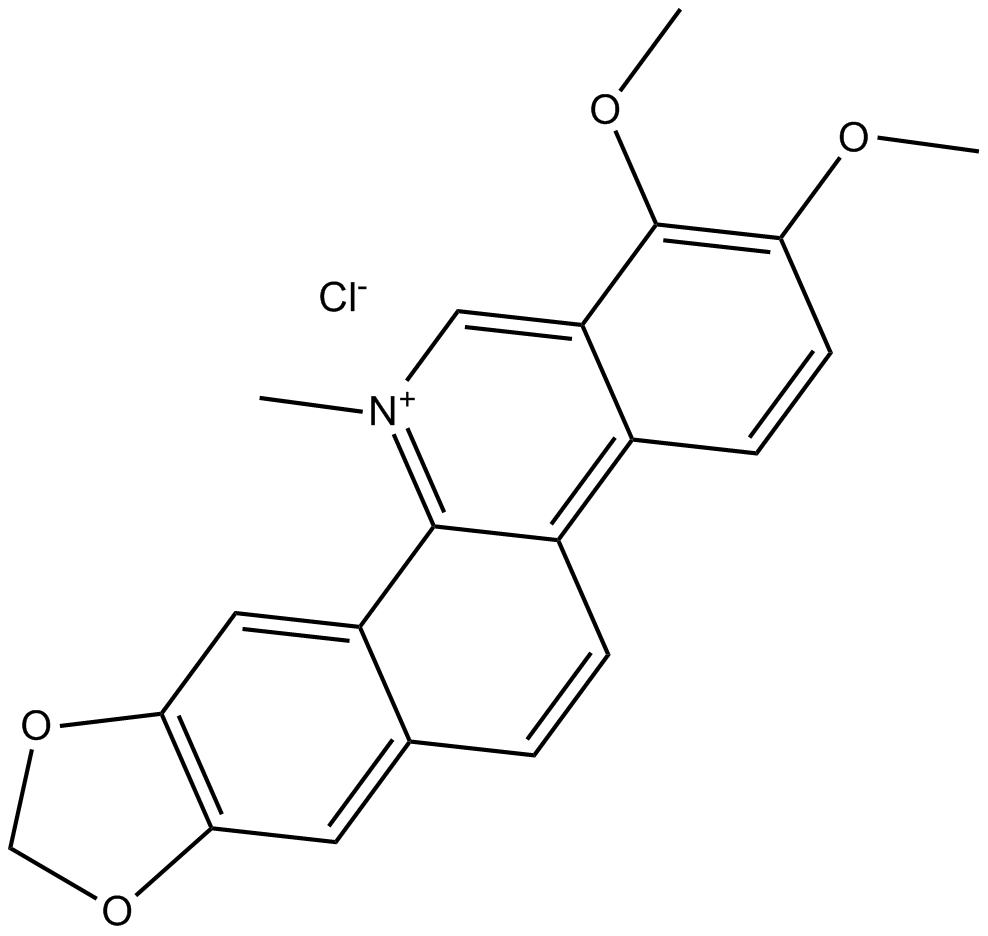
-
GC13065
Chelerythrine Chloride
Potent inhibitor of PKC and Bcl-xL
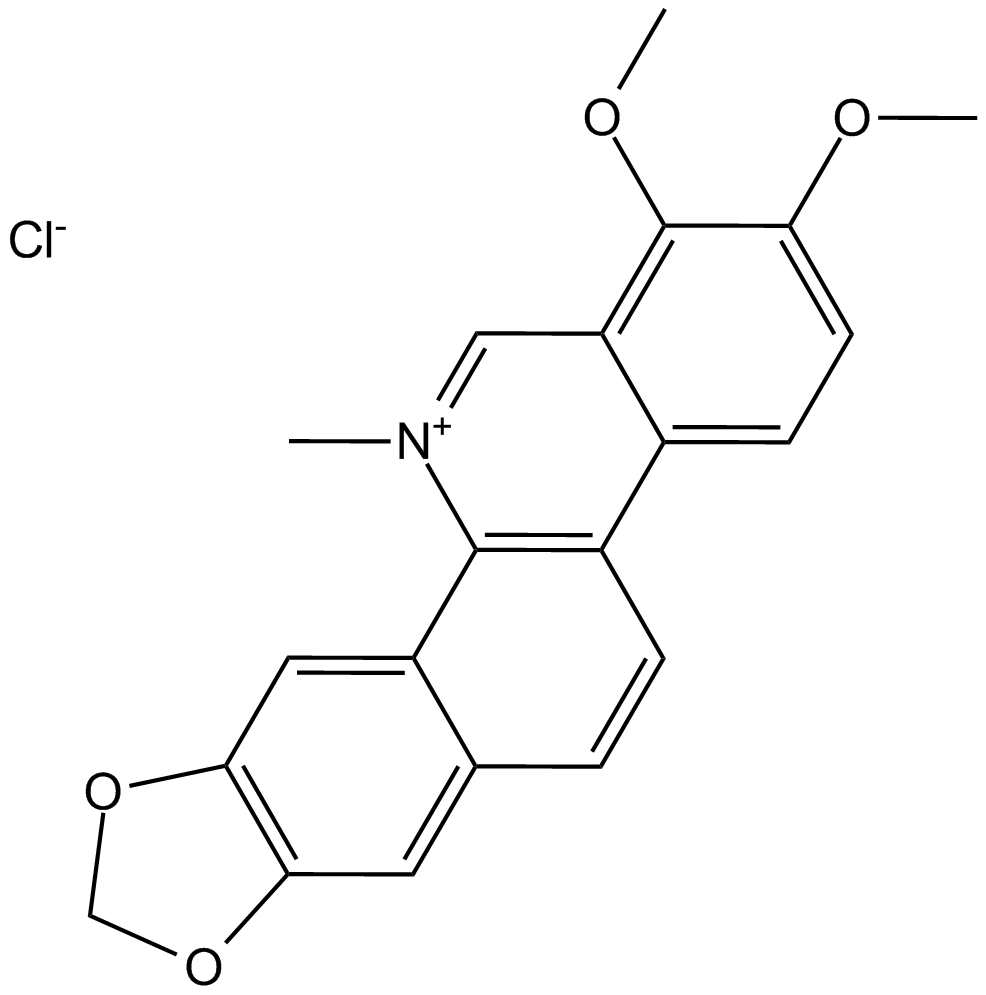
-
GC31886
Chelidonic acid
ChelidonsÄure ist ein Bestandteil von Chelidonium majus L.
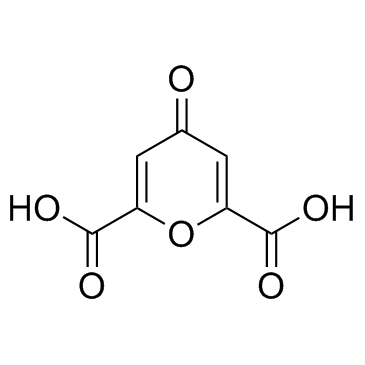
-
GC40878
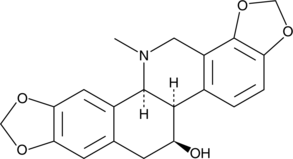
Chelidonine
Chelidonin ist ein Isochinolin-Alkaloid, das aus Chelidonium majus L isoliert werden kann.
-
GC43236
Chevalone B
Chevalone B is a meroterpenoid originally isolated from the fungus E.
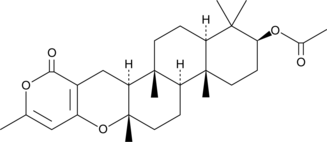
-
GC43237
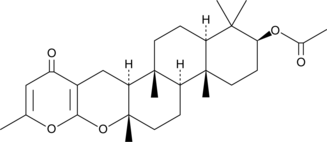
Chevalone C
Chevalone C, ein meroterpenoider Pilzmetabolit, zeigt mit einem IC50-Wert von 25,00 μg/ml eine Malaria-AktivitÄt.
-
GC64993
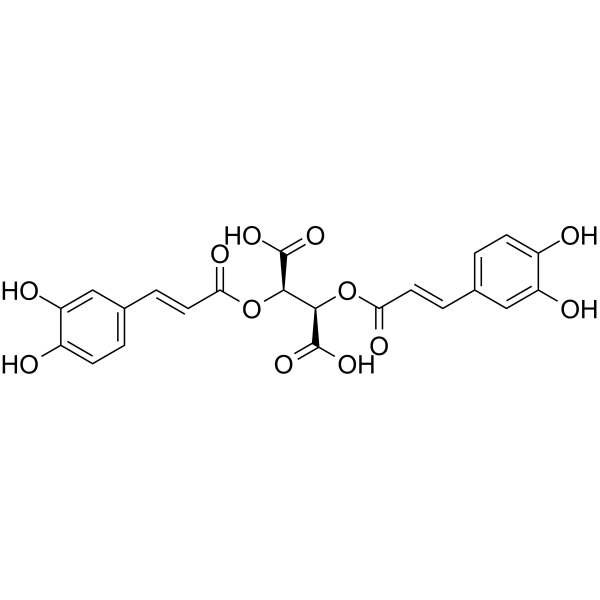
Chicoric acid
ChicorinsÄure (CichorinsÄure), eine oral aktive DicaffeylweinsÄure, induziert die Bildung reaktiver Sauerstoffspezies (ROS).
-
GC15739
CHIR-124
CHIR-124 ist ein potenter und selektiver Chk1-Inhibitor mit einem IC50-Wert von 0,3 nM und zielt mit IC50-Werten von 6,6 nM und 5,8 nM ebenfalls stark auf PDGFR und FLT3 ab.
-
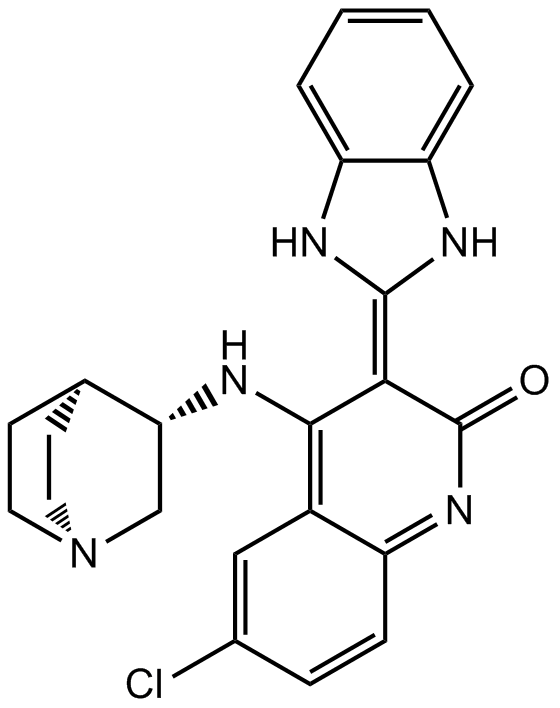
GC43239
Chk2 Inhibitor
Chk2-Inhibitor (Verbindung 1) ist ein potenter und selektiver Inhibitor der Checkpoint-Kinase 2 (Chk2) mit IC50-Werten von 13,5 nM und 220,4 nM fÜr Chk2 bzw. Chk1. Der Chk2-Inhibitor kann einen starken, von Ataxia telangiectasia mutated (ATM) abhÄngigen Chk2-vermittelten Strahlenschutzeffekt hervorrufen.
-
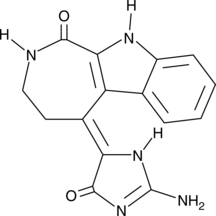
GC45717
Chlamydocin
Chlamydocin, ein Pilzmetabolit, ist ein hochpotenter HDAC-Inhibitor mit einem IC50 von 1,3 nM. Chlamydocin zeigt starke antiproliferative und AntikrebsaktivitÄten. Chlamydocin induziert Apoptose durch Aktivierung von Caspase-3.
-
GC17969
CHM 1
An inhibitor of tubulin polymerization
-
GC35682
CHMFL-ABL/KIT-155
CHMFL-ABL/KIT-155 (CHMFL-ABL-KIT-155; Verbindung 34) ist ein hochpotenter und oral aktiver ABL/c-KIT-Doppelkinase-Inhibitor vom Typ II (IC50s von 46 nM bzw. 75 nM) und es zeigt auch signifikante inhibitorische AktivitÄten gegenÜber BLK (IC50=81 nM), CSF1R (IC50=227 nM), DDR1 (IC50=116 nM), DDR2 (IC50=325 nM), LCK (IC50=12 nM) und PDGFRβ (IC50= 80 nM) Kinasen. CHMFL-ABL/KIT-155 (CHMFL-ABL-KIT-155) stoppt das Fortschreiten des Zellzyklus und induziert Apoptose.
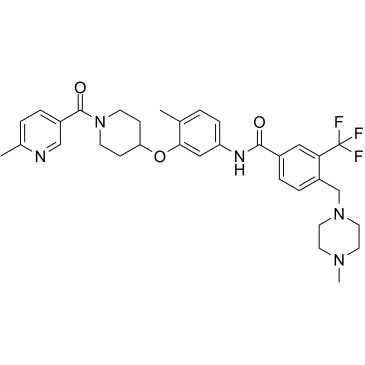
-
GC64028
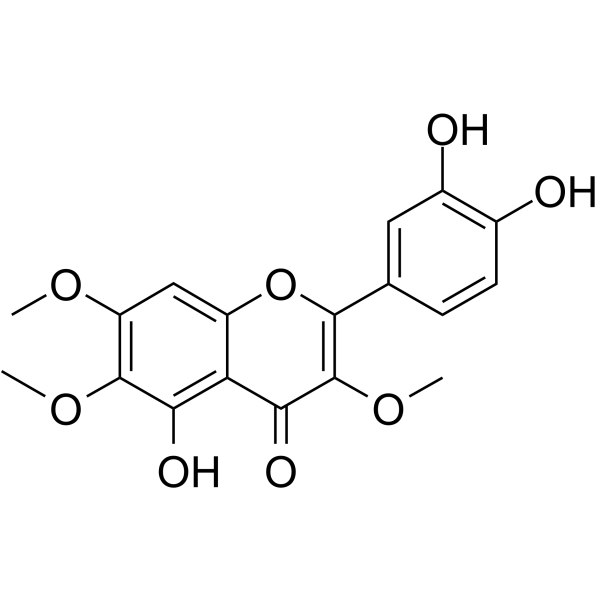
Chrysosplenol D
Chrysosplenol D ist ein Methoxyflavonoid, das ERK1/2-vermittelte Apoptose in dreifach negativen menschlichen Brustkrebszellen induziert.
-
GC13408
CI994 (Tacedinaline)
An inhibitor of HDAC1, -2, and -3
-
GC13589
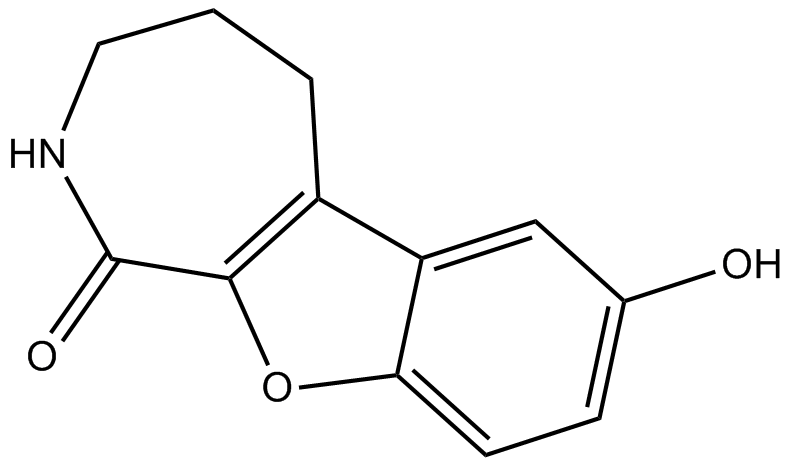
CID 755673
CID 755673 ist ein potenter PKD-Inhibitor mit IC50-Werten von 182 nM, 280 nM und 227 nM für PKD1, PKD2 bzw. PKD3.
-
GC19436
CID-5721353
CID-5721353 ist ein Inhibitor von BCL6 mit einem IC50-Wert von 212 μM, was einem Ki von 147 μM entspricht.
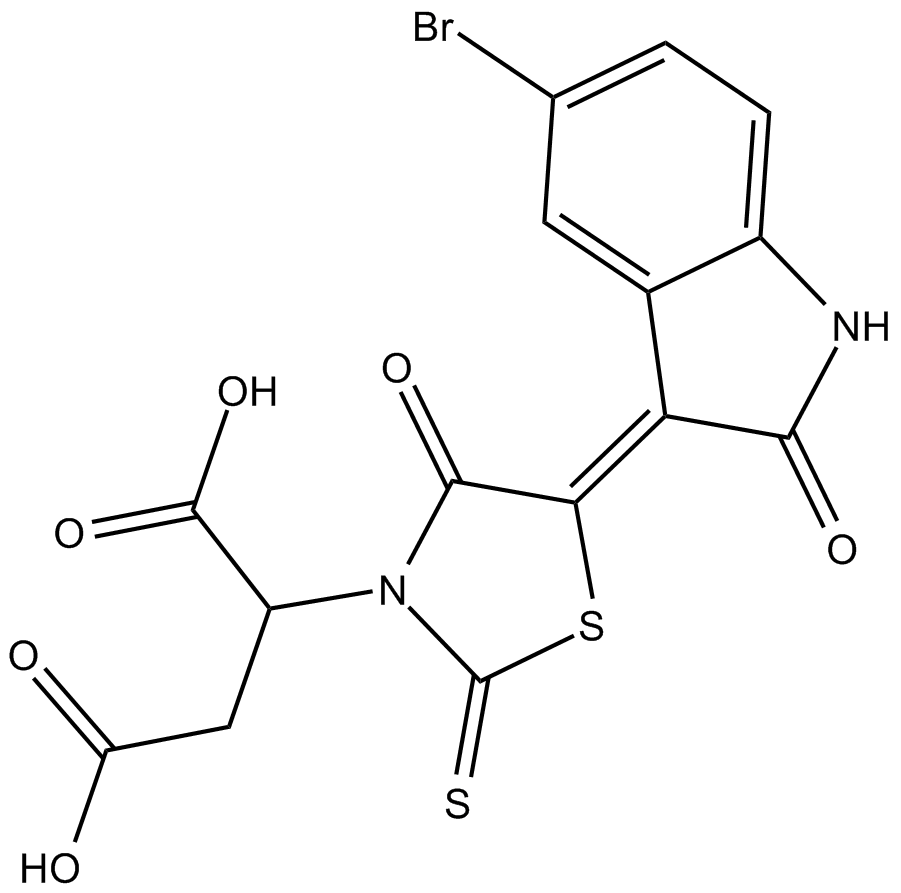
-
GC32997
Cinchonine ((8R,9S)-Cinchonine)
Cinchonin ((8R,9S)-Cinchonin) ist eine natÜrliche Verbindung, die in Chinarinde vorkommt. Cinchonin ((8R,9S)-Cinchonin) aktiviert die Stress-induzierte Apoptose des endoplasmatischen Retikulums in menschlichen Leberkrebszellen.
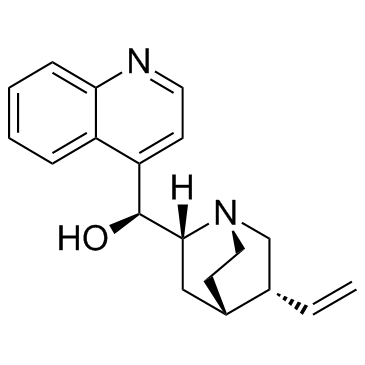
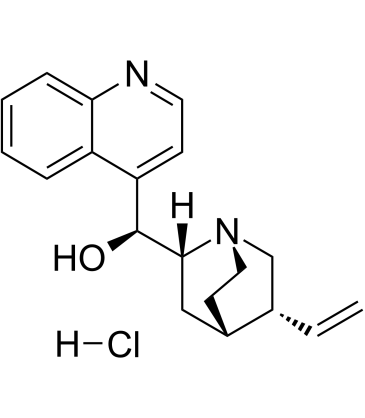
-
GC60708
Cinchonine hydrochloride
Cinchonin-Hydrochlorid ((8R,9S)-Cinchonin-Hydrochlorid) ist ein natÜrliches Alkaloid, das in Chinarinde vorkommt und Malaria-wirksam ist. Cinchoninhydrochlorid aktiviert die stressinduzierte Apoptose des endoplasmatischen Retikulums (ER) in menschlichen Leberkrebszellen.
-
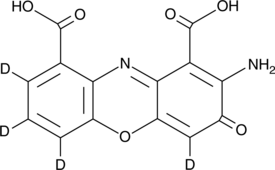
GC52269
Cinnabarinic Acid-d4
An internal standard for the quantification of cinnabarinic acid
-
GC40986
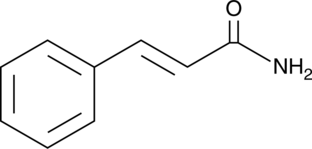
Cinnamamide
Cinnamamide is an amide form of of trans-cinnamic acid and a metabolite of Streptomyces.
-
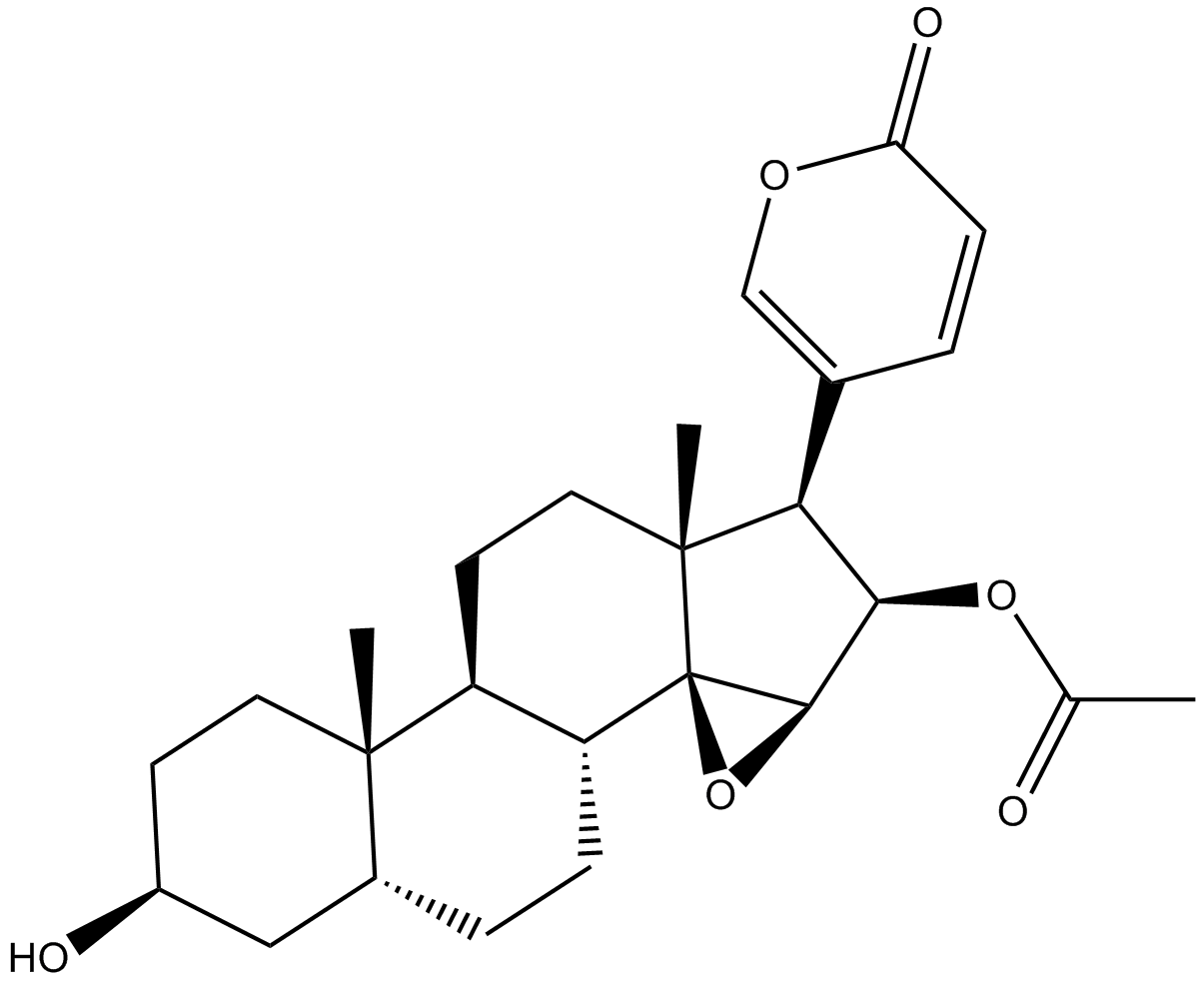
GN10189
Cinobufagin
-
GC11908
Cisplatin
Cisplatin ist eines der besten und ersten metallbasierten Chemotherapeutika, das für eine breite Palette von soliden Krebsarten wie Hoden-, Eierstock-, Blasen-, Lungen-, Gebärmutterhals-, Kopf- und Halskrebs, Magenkrebs und einigen anderen Krebsarten verwendet wird.

-
GC17491
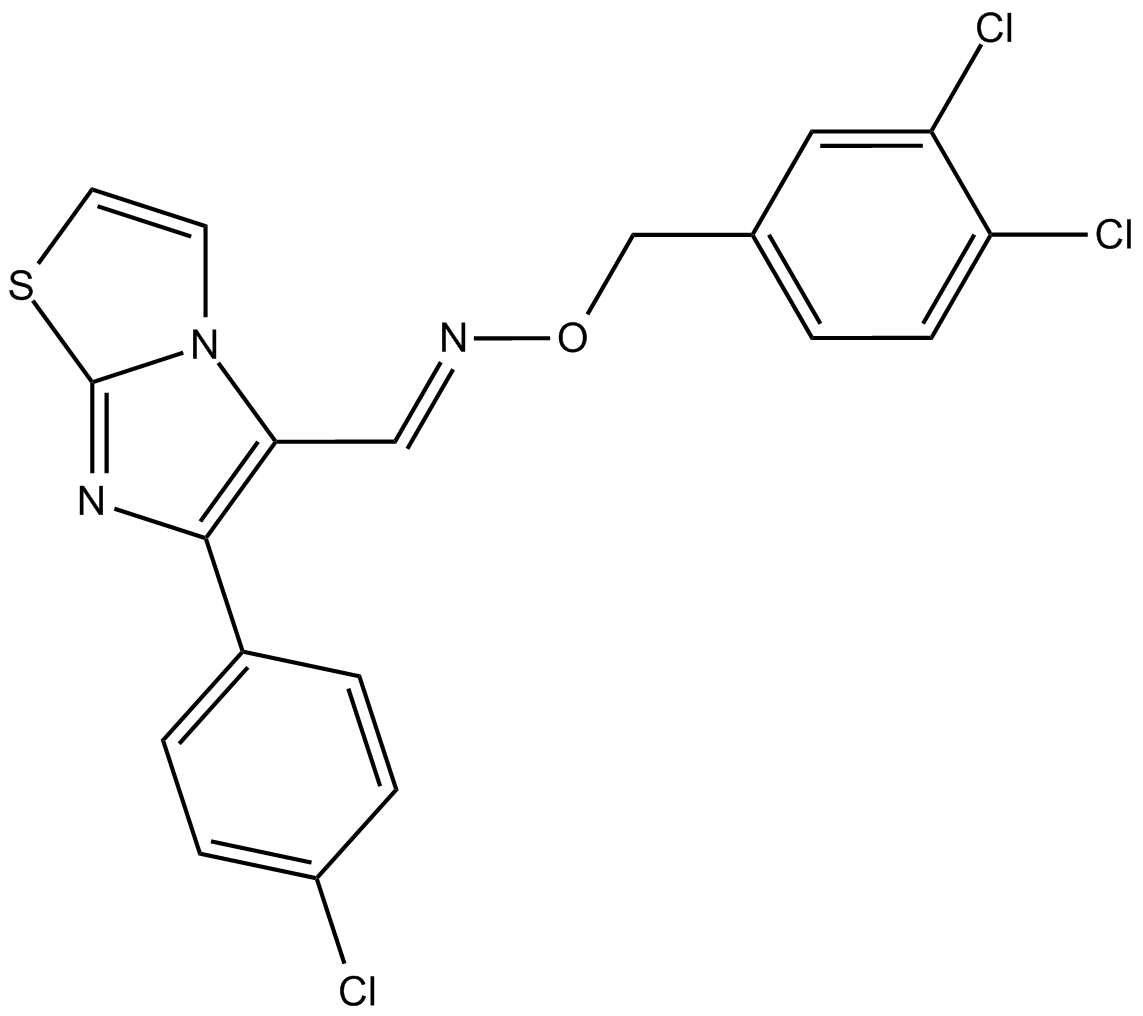
CITCO
CITCO, ein Imidazothiazol-Derivat, ist ein selektiver Agonist des konstitutiven Androstan-Rezeptors (CAR). CITCO hemmt das Wachstum und die Expansion von Hirntumorstammzellen (BTSCs) und hat einen EC50-Wert von 49nM Über dem Pregnane-X-Rezeptor (PXR) und keine AktivitÄt auf andere Kernrezeptoren.
-
GC35703
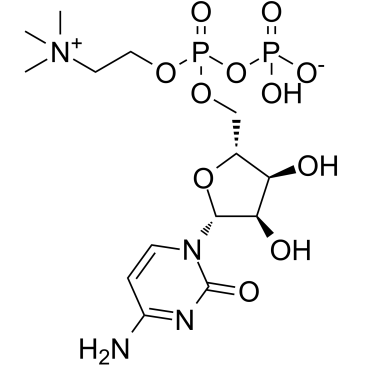
Citicoline
Citicolin (Cytidindiphosphat-Cholin) ist ein Zwischenprodukt bei der Synthese von Phosphatidylcholin, einem Bestandteil von Zellmembranen.
-
GC31186
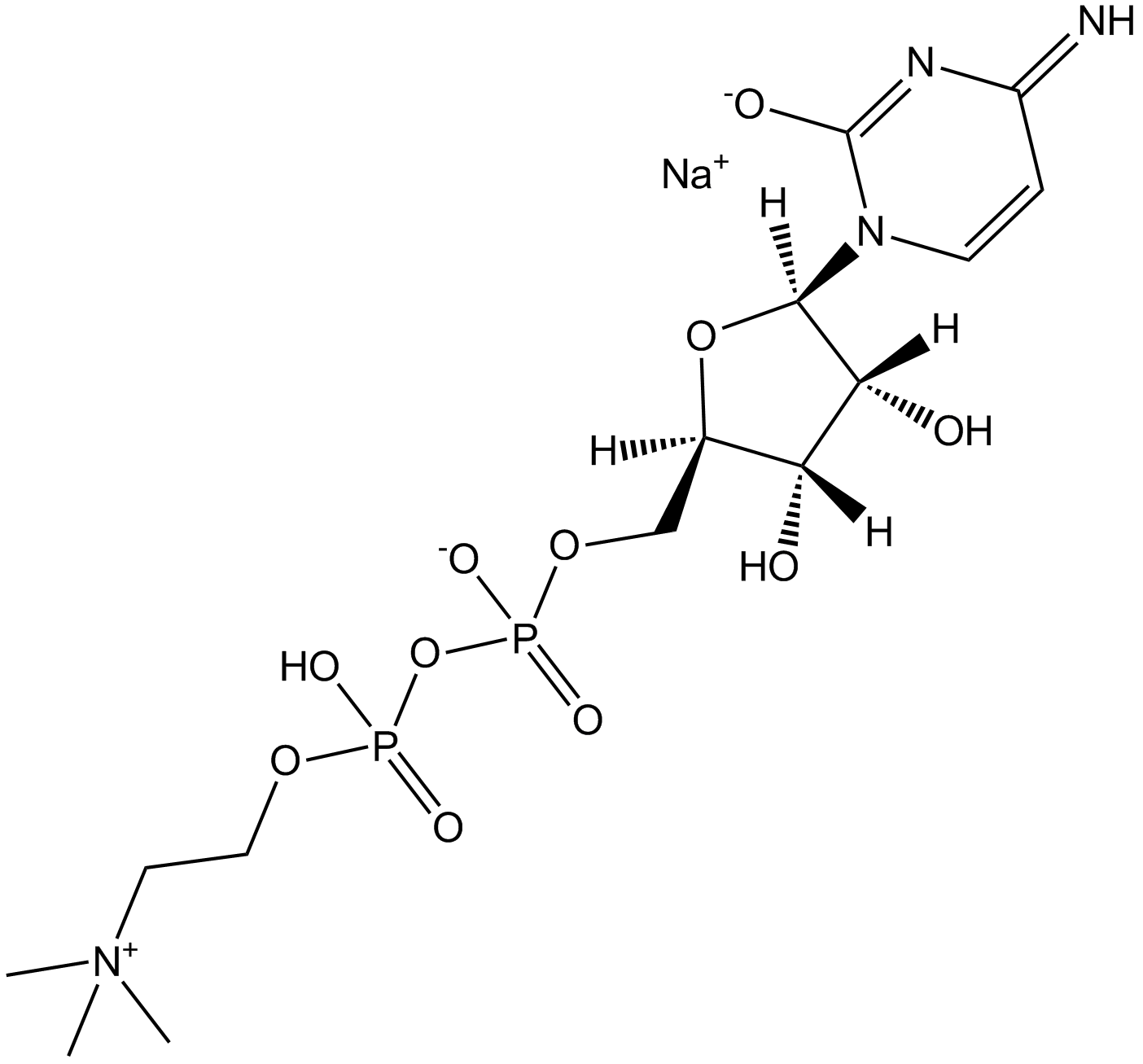
Citicoline sodium salt
Citicolin-Natriumsalz ist ein Zwischenprodukt bei der Synthese von Phosphatidylcholin, das ein Bestandteil von Zellmembranen ist und auch neuroprotektive Wirkungen hat.
-
GC43273
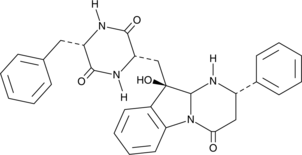
Citreoindole
Citreoindole is a diketopiperazine metabolite isolated from a hybrid cell fusion of two strains of P.
-
GC41514
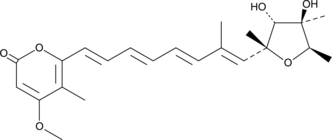
Citreoviridin
Citreoviridin, ein Toxin aus Penicillium citreoviride NRRL 2579, hemmt im Gehirn synaptosomale Na+/K+-ATPase, wÄhrend in Mikrosomen sowohl die Na+/K+-ATPase- als auch die Mg2+-ATPase-AktivitÄt dosisabhÄngig signifikant stimuliert werden.
-
GC14203
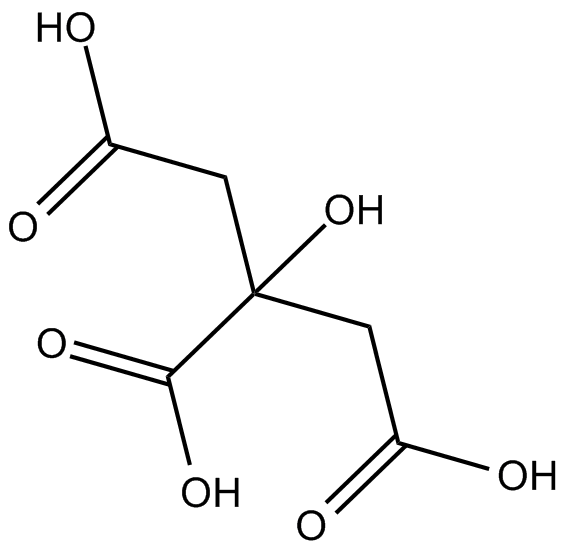
Citric acid
CitronensÄure ist ein natÜrliches Konservierungsmittel und ein SÄureverstÄrker fÜr Lebensmittel.
-
GC68051
Citric acid-d4

-
GC16661
Citrinin
Ein Mykotoxin, das Apoptose induziert.