Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Ziele für Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Produkte für Apoptosis
- Bestell-Nr. Artikelname Informationen
-
GC38182
Dauricine
-
GC49913
Davunetide (acetate)
A neuroprotective ADNP-derived peptide
-
GC63364
DB2115 tertahydrochloride
2-Desoxy-2-fluor-L-fucose, ein L-Fucose-Analogon, ist ein Fucosylierungsinhibitor. 2-Desoxy-2-fluor-L-fucose hemmt die de novo-Synthese von GDP-Fucose in SÄugerzellen. Die Fucosylierung ist ein relativ gut definierter Biomarker fÜr das Fortschreiten vieler Krebsarten beim Menschen; zum Beispiel Pankreas- und hepatozellulÄres Karzinom.
-
GC62222
DB2313
DB2313 ist ein potenter Inhibitor des Transkriptionsfaktors PU.1 mit einer Apoptose von 14 nM. DB2313 unterbricht die Wechselwirkung von PU.1 mit Zielgen-Promotoren. DB2313 induziert die Apoptose von Zellen der akuten myeloischen LeukÄmie (AML) und hat Antikrebswirkungen.
-
GC10824
DBeQ
DBeQ ist ein selektiver, potenter, reversibler und ATP-kompetitiver p97-Inhibitor mit einem IC50-Wert von 1,5 μM und 1,6 μM fÜr p97(wt) bzw. p97(C522A); DBeQ hemmt auch Vps4 mit einem IC50 von 11,5 μM.
-
GC32719
dBET6
dBET6 ist ein hochpotentes, selektives und zellgÄngiges PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit einem IC50 von 14 nM und AntitumoraktivitÄt hat.
-
GC19485
DC661
DC661 ist ein potenter Inhibitor der Palmitoyl-Protein-Thioesterase 1 (PPT1), hemmt die Autophagie und wirkt als anti-lysosomaler Wirkstoff. Anti-Krebs-Aktivität.

-
GC16795
DCA
DCA ist ein Stoffwechselregulator in den Mitochondrien von Krebszellen mit antikarzinogener Wirkung. DCA hemmt PDHK, was zu einer Verringerung der Milchsäure im Tumormikroenvironment führt. DCA erhöht die Produktion reaktiver Sauerstoffspezies (ROS) und fördert die Apoptose von Krebszellen. DCA wirkt auch als NKCC-Inhibitor.

-
GC14007
DCC-2036 (Rebastinib)
DCC-2036 (Rebastinib) (DCC-2036) ist ein oral aktiver, nicht ATP-kompetitiver Bcr-Abl-Inhibitor fÜr Abl1WT und Abl1T315I mit IC50-Werten von 0,8 nM bzw. 4 nM. DCC-2036 (Rebastinib) hemmt auch SRC, KDR, FLT3 und Tie-2 und hat eine geringe AktivitÄt gegenÜber c-Kit.
-
GC33934
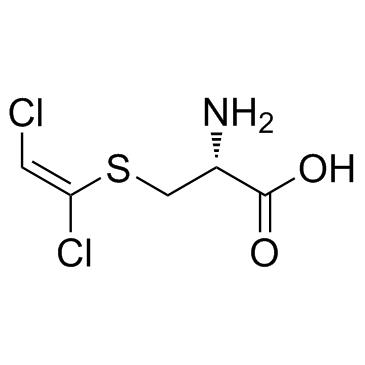
DCVC
DCVC (S-[(1E)-1,2-Dichlorethenyl]--L-Cystein) ist ein bioaktiver Metabolit von Trichlorethylen (TCE).
-
GC64971
DDO-7263
DDO-7263, ein 1,2,4-Oxadiazol-Derivat, ist ein potenter Nrf2-ARE-Aktivator.
-
GC52191
Deacetylanisomycin
Deacetylanisomycin ist ein potenter Wachstumsregulator in Pflanzen und ein inaktives Derivat von Anisomycin.
-
GC15255
Decitabine(NSC127716, 5AZA-CdR)
Ein 2'-Deoxy-Analogon von 5-Azacytidin.
-
GC31892
Decursin ((+)-Decursin)
Decursin ((+)-Decursin) ((+)-Decursin ((+)-Decursin)) ist ein starkes Antitumormittel.
-
GC18556
Degarelix (acetate)
Degarelix is a synthetic gonadotropin-releasing hormone receptor (GNRHR) antagonist (IC50 = 3 nM in HEK293 cells expressing the human receptor).
-
GC15484
Deguelin
A potent antiproliferative rotenoid compound
-
GC35829
Dehydroaltenusin
Dehydroaltenusin ist ein niedermolekularer selektiver Inhibitor der eukaryotischen DNA-Polymerase α, einer Art von Antibiotikum, das von einem Pilz mit einem IC50-Wert von 0,68 μM produziert wird. Die hemmende Wirkungsweise von Dehydroaltenusin gegen die Pol-α-AktivitÄt von SÄugetieren ist kompetitiv in Bezug auf den DNA-Template-Primer (Ki = 0,23 μM) und nicht kompetitiv in Bezug auf das 2'-Desoxyribonukleosid-5'-Triphosphat-Substrat (Ki = 0,18 μM). . Dehydroaltenusin hÄlt den Krebszellzyklus in der S-Phase an und lÖst Apoptose aus. Dehydroaltenusin besitzt in vivo eine Anti-Tumor-AktivitÄt gegen humanen Adenokarzinom-Tumor.
-
GN10040
Dehydrocorydaline
-
GC35834
Dehydroeffusol
Dehydroeffusol ist ein Phenanthren aus dem Heilkraut Juncus effuses. Dehydroeffusol hemmt das Wachstum und die TumorigenitÄt von Magenkrebszellen, indem es selektiv tumorunterdrÜckenden Stress des endoplasmatischen Retikulums und eine moderate Apoptose induziert. Es zeigt eine sehr geringe ToxizitÄt.
-
GC38452
Dehydrotrametenolic acid
DehydrotrametenolsÄure ist ein Sterin, das aus dem Sclerotium von Poria cocos isoliert wird.
-
GC16214
DEL-22379
DEL-22379 ist ein ERK-Dimerisierungsinhibitor. DEL-22379 bindet leicht an ERK2 mit einem im niedrigen mikromolaren Bereich geschÄtzten Kd, obwohl die Bindung selbst bei niedrigen nanomolaren Konzentrationen nachweisbar ist. Die Dimerisierung von ERK2 wird mit einer IC50 von ~0,5 μM zunehmend gehemmt.
-
GC43406
Delphinidin (chloride)
Delphinidin (Chlorid), ein Anthocyanidin, wird aus Beeren und Rotwein isoliert.
-
GN10529
Demethoxycurcumin
-
GC38160
Demethylzeylasteral
A nortriterpenoid with diverse biological activities
-
GC43408
Deoxycholic Acid (sodium salt hydrate)
DesoxycholsÄure (CholansÄure) Natriumhydrat, eine GallensÄure, ist ein Nebenprodukt des Darmstoffwechsels, das den G-Protein-gekoppelten GallensÄurerezeptor TGR5 aktiviert.
-
GC47187
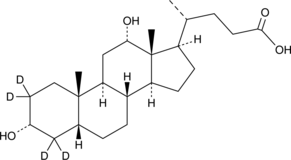
Deoxycholic Acid-d4
DesoxycholsÄure-d4 ist die mit Deuterium bezeichnete DesoxycholsÄure.
-
GC68306
Deoxynyboquinone
-
GC38564
Deoxypodophyllotoxin
Deoxypodophyllotoxin (DPT), ein Derivat von Podophyllotoxin, ist ein Lignan mit starken antimitotischen, entzÜndungshemmenden und antiviralen Eigenschaften, das aus Rhizomen von Sinopodophullumhexandrum (Berberidaceae) isoliert wird.
-
GC16077
Deracoxib
Deracoxib, ein selektiver Cyclooxygenase-2-Hemmer, ist ein nicht narkotisches, nichtsteroidales Antirheumatikum (NSAID).
-
GC34157
Desacetylcinobufotalin (Deacetylcinobufotalin)
Desacetylcinobufotalin (Deacetylcinobufotalin) ist eine natÜrliche Verbindung; Apoptoseinduktor und zeigt die deutliche Hemmwirkung auf HepG2-Zellen und der IC50-Wert betrÄgt 0,0279&7#956;mol/ml.
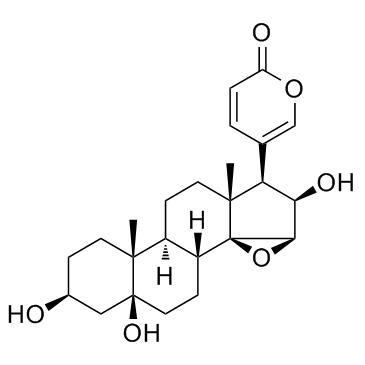
-
GC62927
Desmethylxanthohumol
Desmethylxanthohumol ist ein prenyliertes Hydroxychalcon, das aus Hopfenzapfen (Humulus lupulus L.) isoliert wird. Desmethylxanthohumol ist ein starker Apoptose-induzierender Wirkstoff. Desmethylxanthohumol hat antiplasmodiale, antiproliferative und antioxidative BioaktivitÄten.
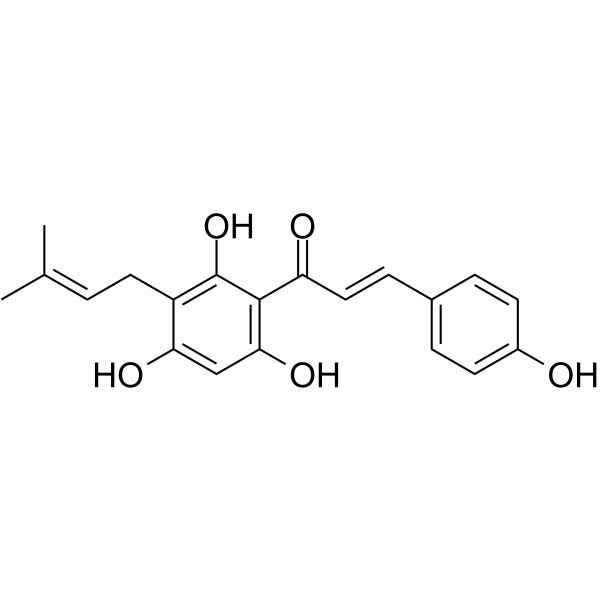
-
GC38221
Desoxyrhaponticin
Desoxyrhaponticin ist ein Stilbenglykosid aus dem tibetischen NahrungsergÄnzungsmittel Rheum tanguticum Maxim. Desoxyrhaponticin ist ein Inhibitor der FettsÄuresynthase (FASN) und hat eine apoptotische Wirkung auf menschliche Krebszellen.
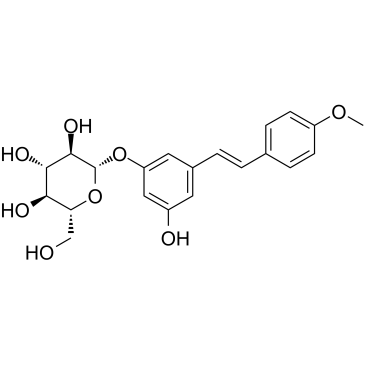
-
GC10661
Destruxin B
Destruxin B, isoliert aus dem entomopathogenen Pilz Metarhizium anisopliae, ist eines der Cyclodepsipeptide mit insektiziden und krebsbekÄmpfenden AktivitÄten.
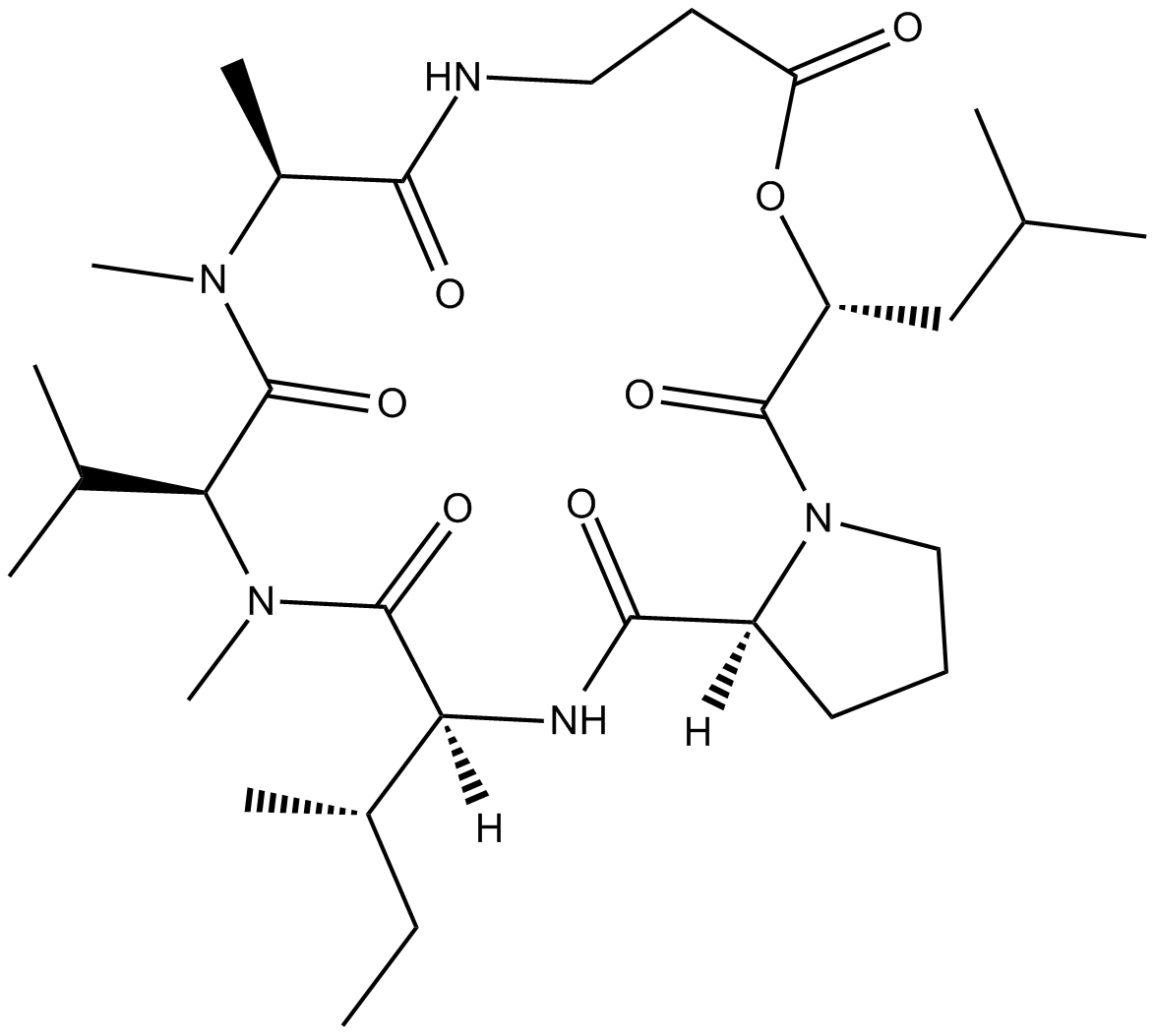
-
GC10520
Dextran sulfate sodium salt (M.W 200000)
Dextransulfat-Natriumsalz ist ein Polymer von Anhydroglucose mit einem Molekulargewichtsbereich von 35.000 bis 45.000.

-
GC38250
Dextran sulfate sodium salt (MW 16000-24000)
A sulfated polysaccharide

-
GC38252
Dextran sulfate sodium salt (MW 35000-45000)
A sulfated polysaccharide

-
GC38253
Dextran sulfate sodium salt (MW 4500-5500)
A sulfated polysaccharide

-
GC38251
Dextran sulfate sodium salt (MW 450000-550000)
A sulfated polysaccharide

-
GC43436
Diacetylcercosporin
Diacetylcercosporin is a perylenequinone produced by Cercospora and Septoria that has diverse biological activities.
-
GC45992
Diallyl Tetrasulfide
An organosulfur compound with diverse biological activities
-
GC43439
Diallyl Trisulfide
Diallyltrisulfid wird aus Knoblauch isoliert.
-
GC35859
Diazepinomicin
Diazepinomicin (TLN-4601) ist ein sekundÄrer Metabolit, der von Micromonospora sp. Diazepinomicin (TLN-4601) hemmt den EGF-induzierten Ras-ERK-MAPK-Signalweg und induziert Apoptose. Ein Antitumormittel fÜr K-Ras-Mutantenmodelle.
-
GC61419
Dibenzoylmethane
Dibenzoylmethan, ein Nebenbestandteil von SÜßholz, aktiviert Nrf2 und beugt verschiedenen Krebsarten und oxidativen SchÄden vor.
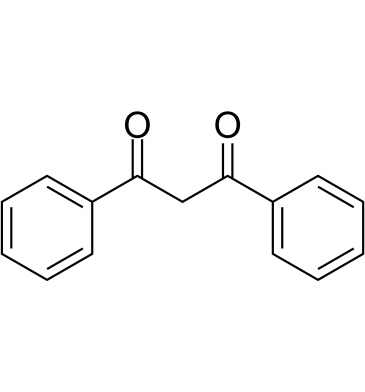
-
GC64139
Dibromoacetic acid
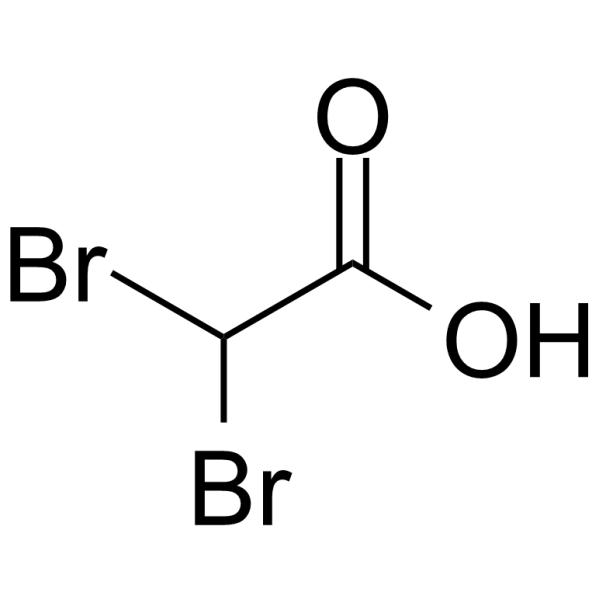
-
GC14622
Diclofenac Potassium
Diclofenac-Kalium ist ein starker und nicht selektiver EntzÜndungshemmer, wirkt als COX-Hemmer mit IC50-Werten von 4 und 1,3 nM fÜr menschliche COX-1 und COX-2 in CHO-Zellen und 5,1 und 0,84 μM fÜr Schaf-COX- 1 bzw. COX-2.
-
GN10751
Dictamnine
-
GC49153
Didemnin B
Didemnin B ist ein Depsipeptid, das aus dem marinen Manteltier Trididemnin cyanophorum extrahiert wird. Didemnin B kann fÜr die Krebsforschung verwendet werden.
-
GC39808
Didesmethylrocaglamide
Didesmethylrocaglamid, ein Derivat von Rocaglamid, ist ein potenter Hemmer des eukaryotischen Initiationsfaktors 4A (eIF4A). Didesmethylrocaglamid hat eine starke wachstumshemmende AktivitÄt mit einem IC50 von 5 nM. Didesmethylrocaglamid unterdrÜckt mehrere wachstumsfÖrdernde Signalwege und induziert Apoptose in Tumorzellen. Antitumor-AktivitÄt.
-
GC38179
Didymin
Didymin, ein diÄtetisches Flavonoidglykosid aus ZitrusfrÜchten, besitzt antioxidative Eigenschaften. Didymin induziert Apoptose durch Hemmung von N-Myc und Hochregulierung von RKIP im Neuroblastom.
-
GC15578
Dienogest
Dienogest (STS-557) ist ein oral aktiver und selektiver Progesteronrezeptor-Agonist, der die Genexpression von COX-2, mPGES-1 und Aromatase wirksam reduziert.
-
GC43452
Diffractaic Acid
DiffraktsÄure, ein Hauptbestandteil von U.
-
GC64375
Difopein TFA
Difopein (TFA), ein spezifischer und kompetitiver Inhibitor des 14-3-3-Proteins (ein hochkonserviertes eukaryontisches regulatorisches MolekÜl), blockiert die FÄhigkeit von 14-3-3, an Zielproteine zu binden, und hemmt 14-3-3/Ligand-Wechselwirkungen . Difopein (TFA) fÜhrt zur Induktion von Apoptose und verstÄrkt die FÄhigkeit von Cisplatin, Zellen abzutÖten.

-
GC49842
Digoxigenin Bisdigitoxoside
A Na+/K+-ATPase inhibitor
-
GN10056
Dihydroartemisinin
-
GC38617
Dihydrokaempferol
Dihydrokaempferol wird aus Bauhinia championii (Benth) isoliert.
-
GC43462
Dihydrolipoic Acid
DihydroliponsÄure (DHLA) ist ein hervorragendes Antioxidans, das nahezu jedes sauerstoffzentrierte Radikal abfangen kann.
-
GC38620
Dihydrorotenone
Dihydrorotenon, ein natürliches Pestizid, ist ein potenter Mitochondrienhemmer.
-
GC41112
Dihydroxy Melphalan
Dihydroxy melphalan is an inactive degradation product of melphalan.
-
GC43463
DiIC1(5)
DiIC1(5) is a signal-off fluorescent probe for the detection of mitochondrial membrane potential disruption.
-
GC38772
DIM-C-pPhOH
DIM-C-pPhOH ist ein Antagonist des Kernrezeptors 4A1 (NR4A1). DIM-C-pPhOH hemmt das Wachstum von Krebszellen und die mTOR-Signalgebung, induziert Apoptose und zellulÄren Stress. DIM-C-pPhOH reduziert die Zellproliferation mit IC50-Werten von 13,6 μM und 13,0 μM fÜr ACHN-Zellen bzw. 786-O-Zellen.
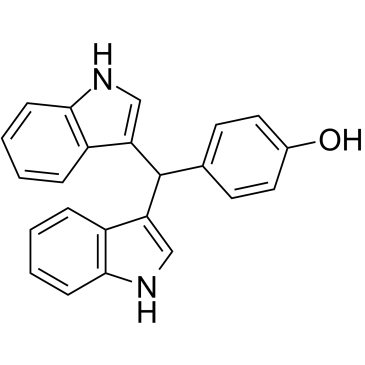
-
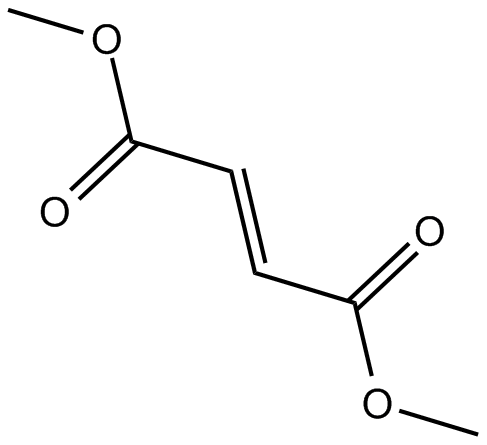
GC16590
Dimethyl Fumarate
Dimethylfumarat (DMF) ist ein oral aktiver und in das Gehirn eindringender Nrf2-Aktivator und induziert eine Hochregulierung der antioxidativen Genexpression.
-
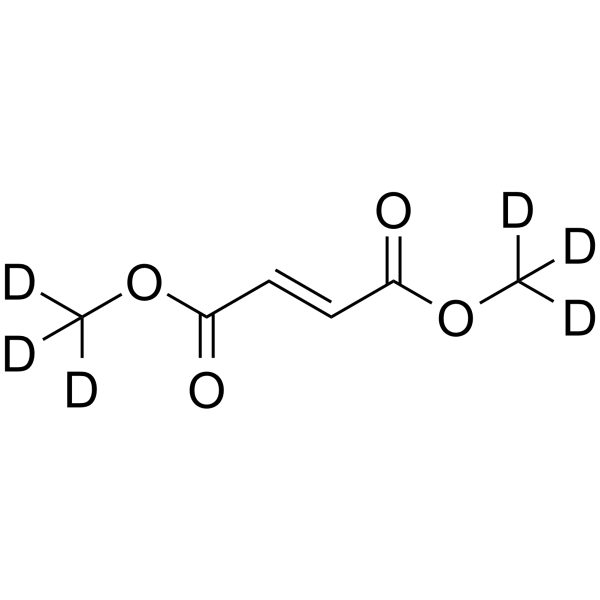
GC62936
Dimethyl fumarate D6
Dimethylfumarat D6 ist ein mit Deuterium gekennzeichnetes Dimethylfumarat.
-
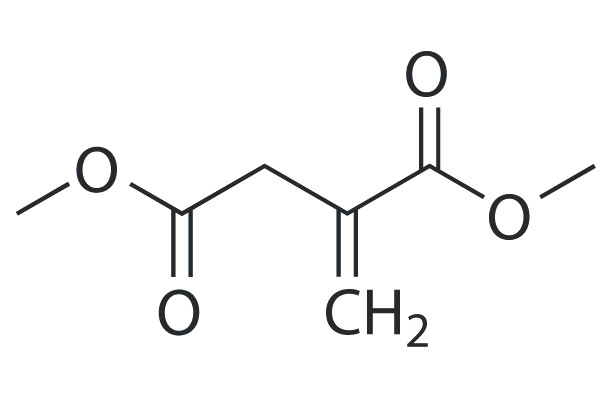
GC25351
Dimethyl itaconate
Dimethyl itaconate can reprogram neurotoxic to neuroprotective primary astrocytes through the regulation of LPS-induced Nod-like receptor protein 3 (NLRP3) inflammasome and nuclear factor 2/heme oxygenase-1 (NRF2/HO-1) pathways.
-
GN10531
Dimethylfraxetin
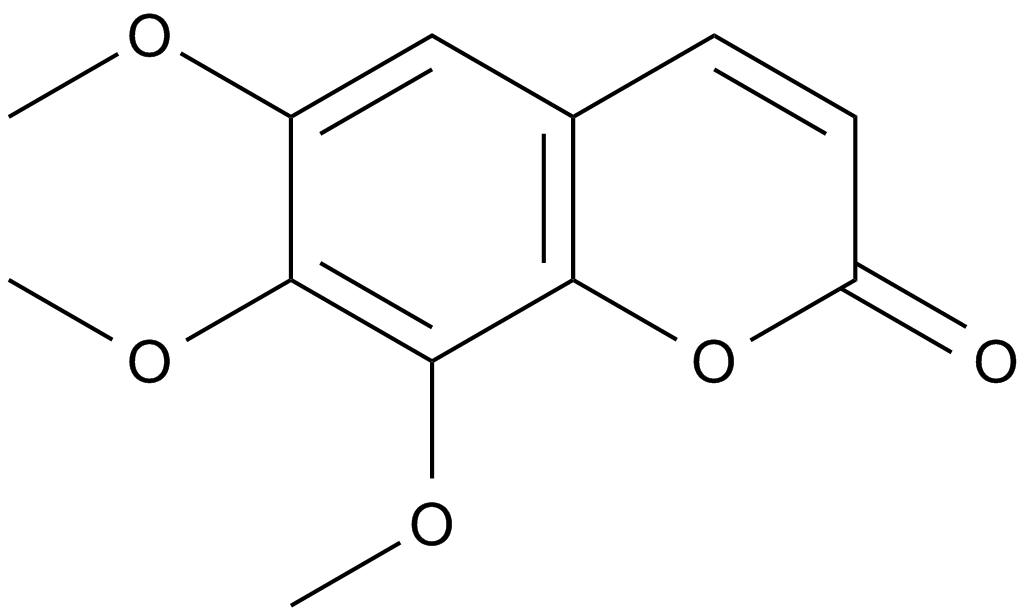
-
GC17648
Dinaciclib(SCH727965)
Dinaciclib(SCH727965) (SCH 727965) ist ein potenter Inhibitor von CDK mit IC50-Werten von 1 nM, 1 nM, 3 nM und 4 nM fÜr CDK2, CDK5, CDK1 bzw. CDK9.
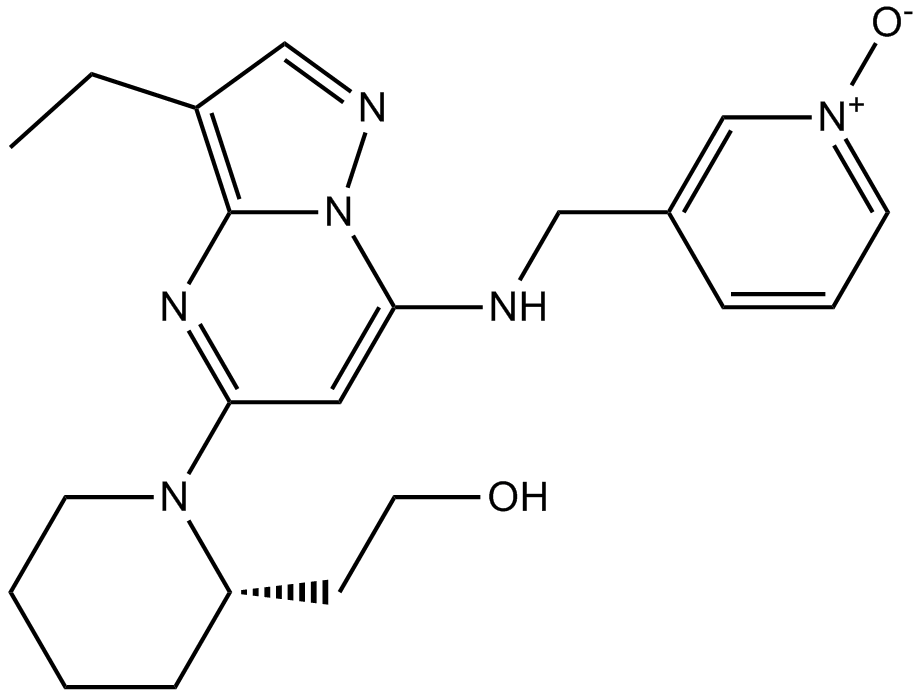
-
GN10284
Dioscin
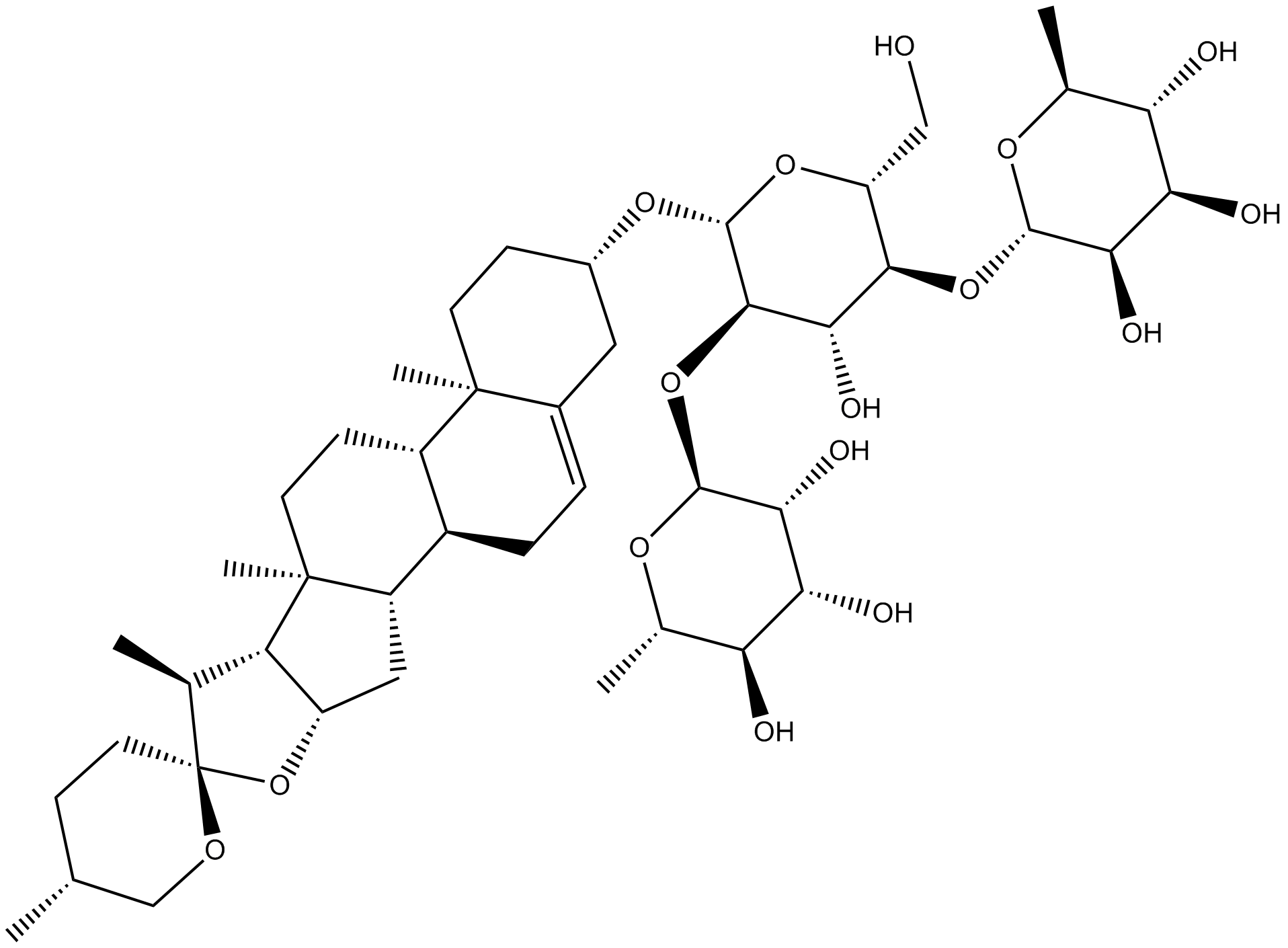
-
GC38408
Diosgenin glucoside
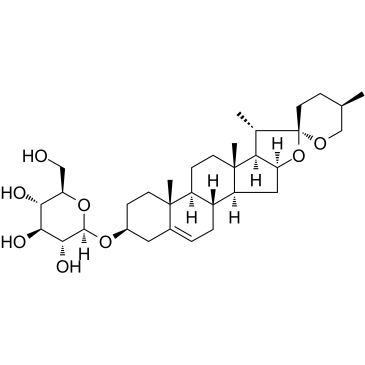
-
GC60782
Disitertide TFA
Disitertid (P144) TFA ist ein peptidischer Inhibitor des transformierenden Wachstumsfaktors Beta 1 (TGF-β1), der speziell entwickelt wurde, um die Wechselwirkung mit seinem Rezeptor zu blockieren. Disitertid (P144) TFA ist auch ein PI3K-Inhibitor und ein Apoptoseinduktor.

-
GC10801
Disulfiram
Ein irreversibler Inhibitor der Aldehyd-Dehydrogenase.
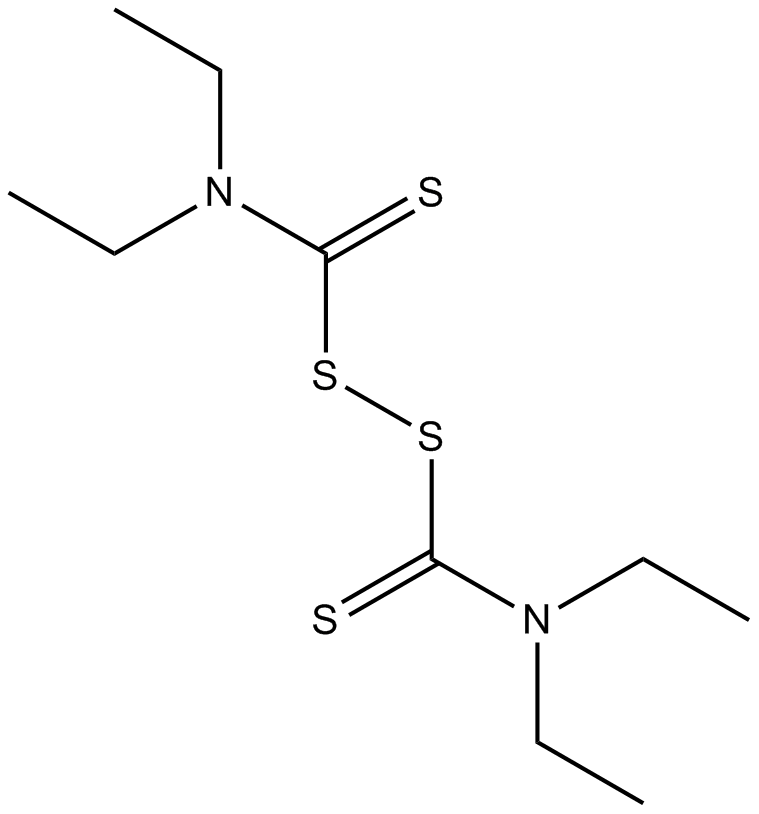
-
GC39295
DJ001
DJ001 ist ein hochspezifischer, selektiver und nicht-kompetitiver Protein-Tyrosin-Phosphatase-σ (PTPσ)-Inhibitor mit einem IC50 von 1,43 μM.
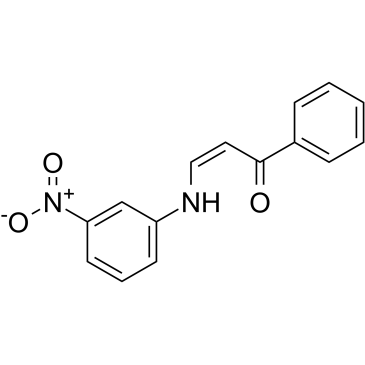
-
GC49164
DM4 (hydrate)
A derivative of maytansine
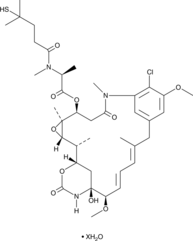
-
GC35882
dMCL1-2
dMCL1-2 ist ein potenter und selektiver PROTAC von myeloid cell leukemia 1 (MCL1) (Mitglied der Bcl-2-Familie) auf der Basis von Cereblon, das mit einer KD von 30 nM an MCL1 bindet. dMCL1-2 aktiviert die zellulÄre Apoptosemaschinerie durch Abbau von MCL1.
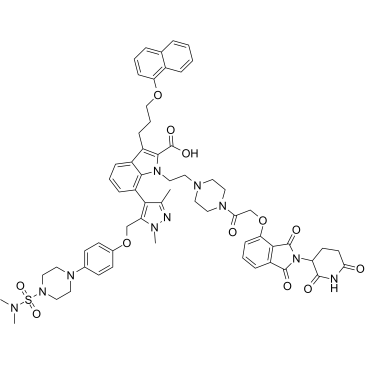
-
GC40192
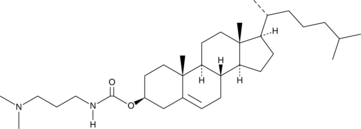
DMPAC-Chol
DMPAC-Chol is a cationic cholesterol derivative that has been used in liposome formation for gene transfection.
-
GC61466
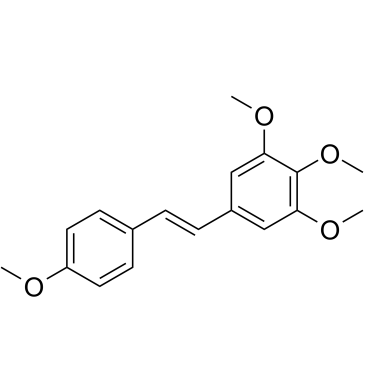
DMU-212
DMU-212 ist ein methyliertes Derivat von Resveratrol mit antimitotischen, antiproliferativen, antioxidativen und apoptosefÖrdernden AktivitÄten. DMU-212 induziert einen mitotischen Arrest durch Induktion von Apoptose und Aktivierung des ERK1/2-Proteins. DMU-212 ist oral aktiv.
-
GC16684
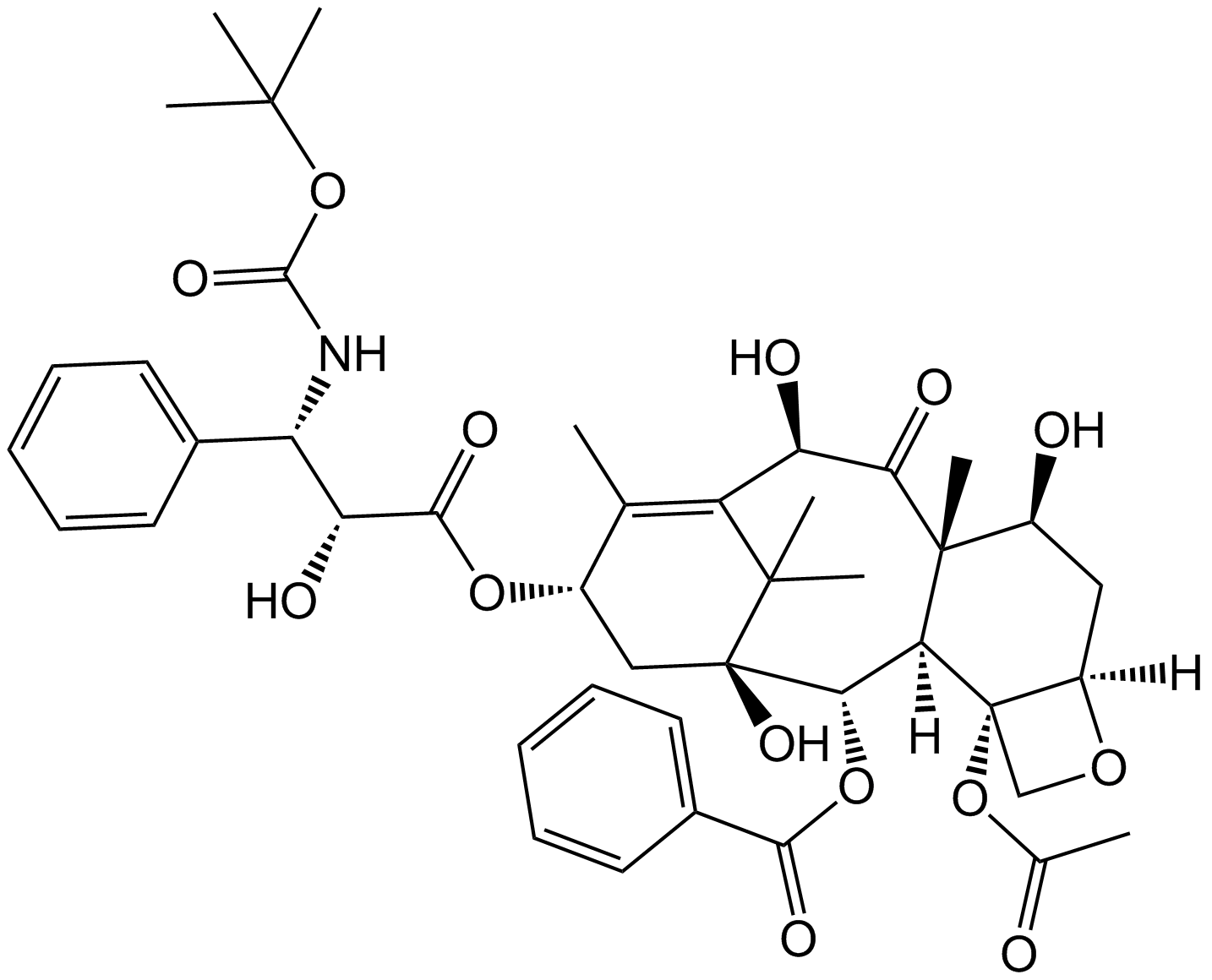
Docetaxel
Docetaxel ist ein antineoplastisches Medikament, das die Depolymerisation von Mikrotubuli hemmt und die Auswirkungen der Genexpression von bcl-2 und bcl-xL abschwächt.
-
GA21397
Dolastatin 15
Dolastatin 15 (DLS 15), ein von Dolabella auricularia abgeleitetes Depsipeptid, ist ein starkes Antimitosemittel, das strukturell mit dem Antitubulinmittel Dolastatin 10 verwandt ist. Dolastatin 15 induziert Zellzyklusstillstand und Apoptose in multiplen Myelomzellen. Dolastatin 15 kann als ADC-Zytotoxin verwendet werden.

-
GC11099
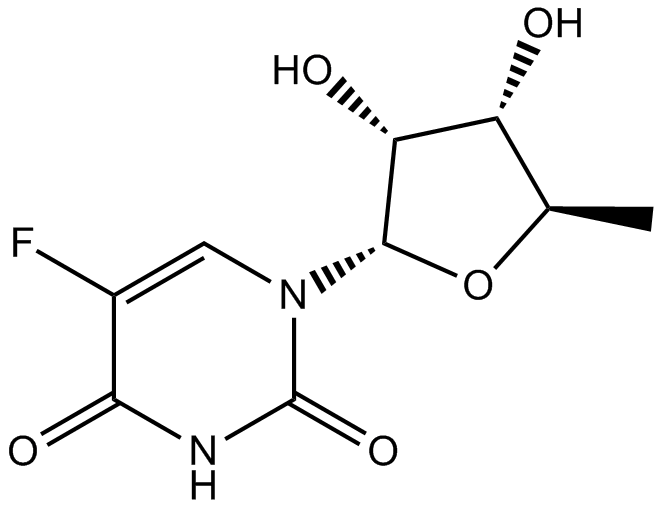
Doxifluridine
Doxifluridin (Ro 21-9738; 5'-DFUR) ist ein Thymidin-Phosphorylase-Aktivator fÜr PC9-DPE2-Zellen mit einem IC50-Wert von 0,62 μM.
-
GC18439
Doxorubicinone
Doxorubicinon ist ein Metabolit eines Chemotherapeutikums gegen Krebs, Doxorubicin. Doxorubicin ist ein potenter Human-DNA-Topoisomerase-I- und -Topoisomerase-II-Inhibitor mit IC50-Werten von 0,8μM bzw. 2,67μM.
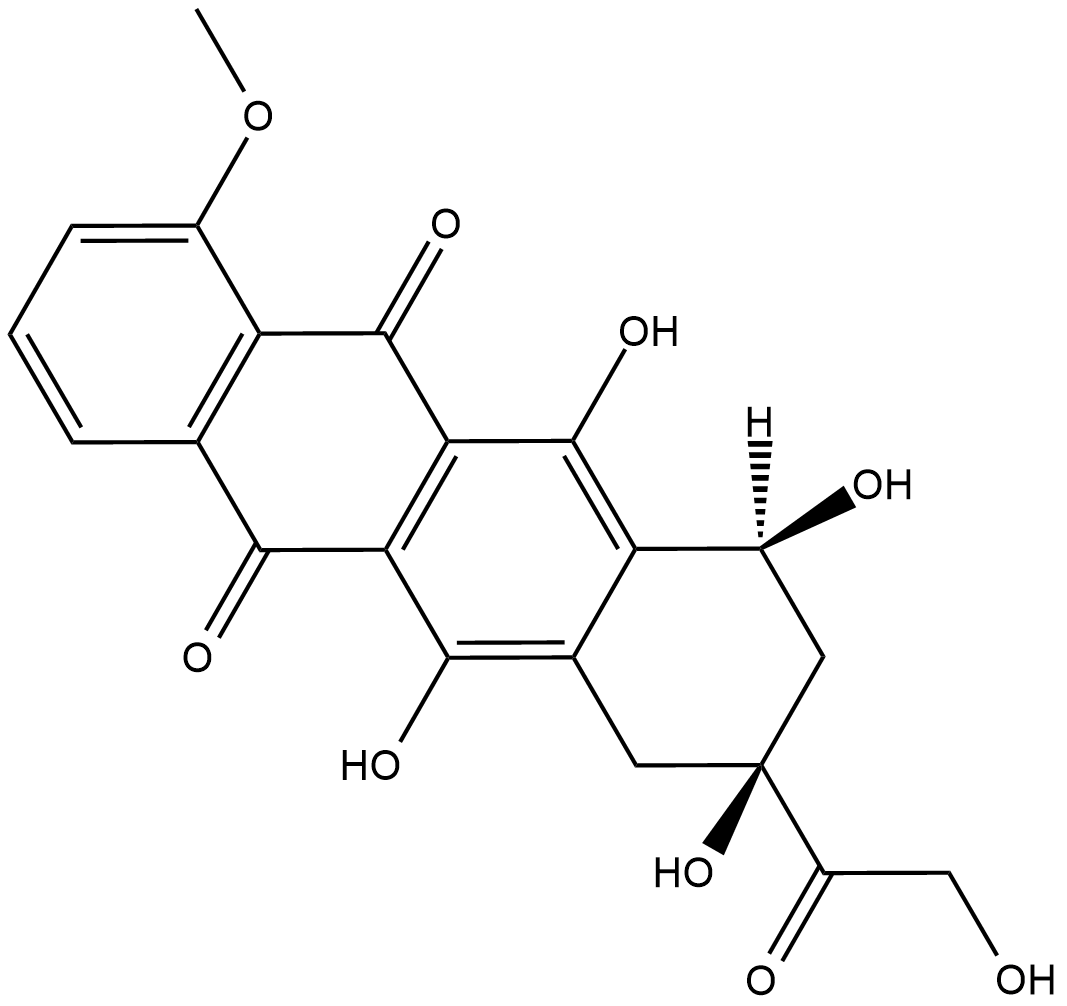
-
GC15399
Dp44mT
Dp44mT ist ein Eisenchelator mit selektiver AntikrebsaktivitÄt.
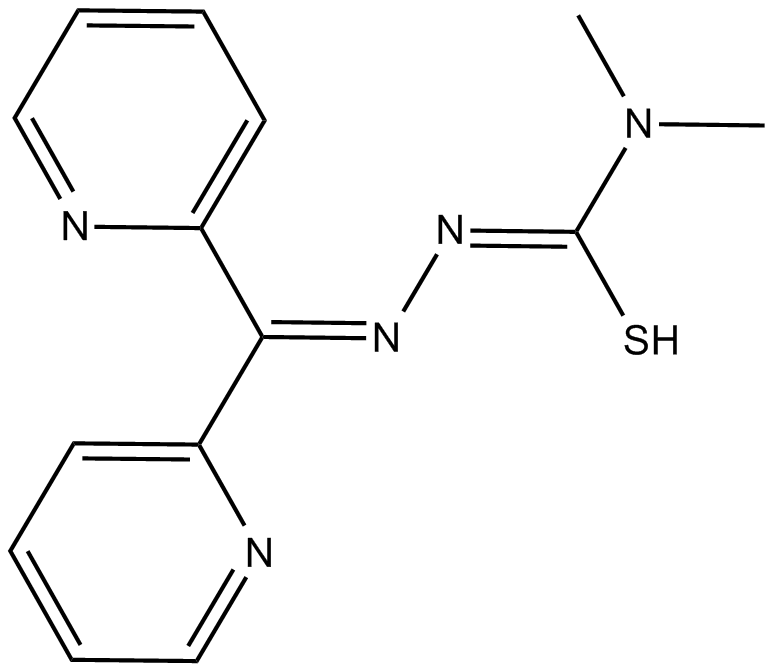
-
GC33384
DPBQ
DPBQ aktiviert p53 und lÖst polyploid-spezifisch Apoptose aus, hemmt aber weder Topoisomerase noch bindet es DNA. DPBQ lÖst die Expression und Phosphorylierung von p53 aus und diese Wirkung ist spezifisch fÜr tetraploide Zellen.
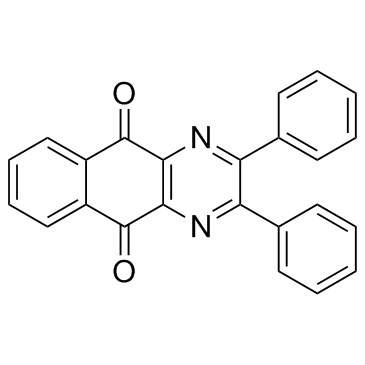
-
GC35898
Dracorhodin perchlorate
Dracorhodin-Perchlorat (Dracohodin-Perochlorat) ist ein Naturprodukt, das aus dem Drachenblut eines Naturheilmittels gewonnen wird. Dracorhodinperchlorat hemmt die Zellproliferation und induziert Zellzyklusstillstand und Apoptose.
-
GC39707
Droloxifene
Droloxifen, ein Tamoxifen-Derivat, ist ein oral aktiver und selektiver Östrogenrezeptor-Modulator. Droloxifen zeigt antiÖstrogene und antiimplantative Wirkungen. Droloxifen induziert die p53-Expression und Apoptose in MCF-7-Zellen. Droloxifen verhindert den Knochenschwund bei ovariektomierten Ratten.
-
GC13706
Droxinostat
An inhibitor of HDAC3, HDAC6, and HDAC8
-
GC35908
Duocarmycin A
Duocarmycin A, eines der bekannten Antitumor-Antibiotika, ist ein DNA-Alkylator und alkyliert effizient Adenin N3 am 3'-Ende von AT-reichen Sequenzen in der DNA. Duocarmycin A als Chemotherapeutikum fÜhrt bei HLC-2-Zellen typischerweise zu apoptotischen VerÄnderungen, einschließlich Chromatinkondensation, Sub-G1-Akkumulation im DNA-Histogrammmuster und Abnahme der Procaspase-3- und -9-Spiegel.
-
GC35913
Durvalumab
Durvalumab (MEDI 4736) ist ein humanisierter monoklonaler Anti-PD-L1-AntikÖrper. Durvalumab (MEDI4736) blockiert vollstÄndig die Bindung von PD-L1 an PD-1 und CD80 mit IC50-Werten von 0,1 bzw. 0,04 nM.

-
GC10893
Dutasteride
Dutasterid (GG745) ist ein potenter Inhibitor beider 5α-Reduktase-Isozyme. Dutasterid kann aufgrund seiner strukturellen Ähnlichkeit mit DHT Off-Target-Effekte auf den Androgenrezeptor (AR) besitzen.
-
GC62590
E64FC26
E64FC26 ist ein potenter Pan-Inhibitor der Protein-Disulfid-Isomerase (PDI)-Familie mit IC50-Werten von 1,9, 20,9, 25,9, 16,3 und 25,4 μM gegen PDIA1, PDIA3, PDIA4, TXNDC5 bzw. PDIA6. E64FC26 zeigt Anti-Myelom-AktivitÄt.
-
GC39377
EB-3D
EB-3D ist ein potenter und selektiver Hemmer der Cholinkinase α (ChoKα) mit einem IC50-Wert von 1 μM fÜr ChoKα1. EB-3D Übt Wirkungen auf die ChoKα-Expression, die AMPK-Aktivierung, die Apoptose, den Stress des endoplasmatischen Retikulums und den Lipidstoffwechsel aus. EB-3D zeigt eine starke antiproliferative AktivitÄt in einer Reihe von T-LeukÄmie-Zelllinien. Anti-Krebs-AktivitÄt.
-
GC50282
EC 19
Synthetic retinoid; induces differentiation of stem cells
-
GC39174
EC359
EC359 ist ein potenter, selektiver, hochaffiner und oral aktiver LeukÄmie-Hemmfaktor-Rezeptor (LIFR)-Inhibitor mit einer Kd von 10,2 nM, der direkt mit LIFR interagiert, um LIF/LIFR-Wechselwirkungen wirksam zu blockieren.
-
GC38149
Ecabet sodium
Ecabet-Natrium (TA-2711) wird derzeit bei einigen Magen-Darm-Erkrankungen eingesetzt, indem es die ROS-Produktion hemmt und die Eradikation von Helicobacter pylori verbessert.
-
GN10504
Echinocystic acid
-
GC47279
Echinosporin
A bacterial metabolite with antibacterial and anticancer activities
-
GC63871
Echitamine chloride
Echitaminchlorid ist das wichtigste in Alstonia vorkommende Monoterpen-Indol-Alkaloid mit starker Anti-Tumor-AktivitÄt. Echitaminchlorid induziert DNA-Fragmentierung und Zellapoptose. Echitaminchlorid hemmt die Pankreaslipase mit einem IC50 von 10,92 μM.
-
GC63470
Eclitasertib
Eclitasertib (DNL-758) ist ein potenter Hemmer der Rezeptor-interagierenden Proteinkinase 1 (RIPK1) mit einem IC50 von <1 μM (aus Patent WO2017136727A2, Beispiel 42).
-
GC33156
Ecteinascidin 770 (Ecteinascidine 770)
Ecteinascidin 770 (Ecteinascidin 770) (ET-770) ist ein 1,2,3,4-Tetrahydroisochinolin-Alkaloid mit starken Anti-Krebs-AktivitÄten; hemmt U373MG-Zellen mit einem IC50 von 4,83 nM.
-
GC12838
Edaravone
Edaravon ist ein starker neuartiger FÄnger freier Radikale und hemmt MMP-9-bedingte Gehirnblutungen bei Ratten, die mit Gewebe-Plasminogen-Aktivator behandelt wurden.
-
GC62562
EGFR-IN-11
EGFR-IN-11 ist ein EGFR-Tyrosinkinase-Inhibitor (EGFR-TKI) der vierten Generation mit einem IC50 von 18 nM fÜr die Dreifachmutante EGFRL858R/T790M/C797S. EGFR-IN-11 unterdrÜckt signifikant die EGFR-Phosphorylierung, induziert die Apoptose und hÄlt den Zellzyklus bei G0/G1 an.
-
GC14756
EI1
EI1 (KB-145943) ist ein potenter und selektiver EZH2-Inhibitor mit einem IC50-Wert von 15 nM bzw. 13 nM fÜr EZH2 (WT) und EZH2 (Y641F).
-
GC64864
EJMC-1
EJMC-1 ist ein mittelstarker TNF-α-Inhibitor mit einem IC50-Wert von 42 μM.