Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
GC34078
I-CBP112
I-CBP112 es un inhibidor de la interacciÓn proteÍna-proteÍna competitivo de acetil-lisina especÍfico y potente, que inhibe los bromodominios CBP/p300, mejora la acetilaciÓn por p300.
-
GC43889
I-CBP112 (hydrochloride)
I-CBP112 (clorhidrato) es un inhibidor selectivo de CBP/P300 que se une directamente a sus bromodominios (Kds \u003d 142 y 625 nM, respectivamente). I-CBP112 reduce significativamente el potencial iniciador de leucemia de las células de leucemia mieloide aguda MLL-AF9(+) de manera dependiente de la dosis in vitro e in vivo. I-CBP112 aumenta la actividad citotóxica del inhibidor del bromodominio BET JQ1, así como de la doxorrubicina.
-
GC33042
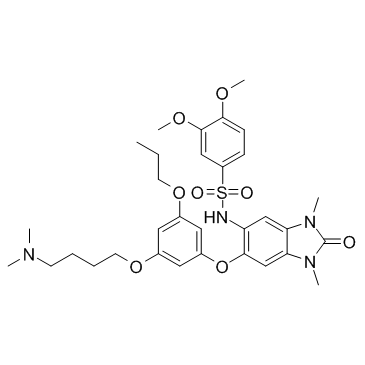
IACS-9571 (ASIS-P040)
IACS-9571 (ASIS-P040) es un inhibidor potente y selectivo de TRIM24 y BRPF1, con una IC50 de 8 nM para TRIM24, y Kds de 31 nM y 14 nM para TRIM24 y BRPF1, respectivamente.
-
GC60925
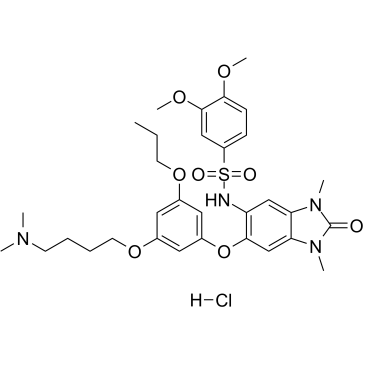
IACS-9571 hydrochloride
El clorhidrato de IACS-9571 (ASIS-P040) es un inhibidor potente y selectivo de TRIM24 y BRPF1, con una IC50 de 8 nM para TRIM24 y Kds de 31 nM y 14 nM para TRIM24 y BRPF1, respectivamente.
-
GC34437
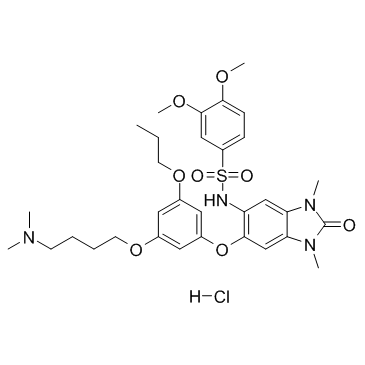
IACS-9571 Hydrochloride (ASIS-P040 Hydrochloride)
-
GC48548
iBET-BD2
A BD2 bromodomain inhibitor
-
GC32948
IDF-11774
IDF-11774 es un nuevo inhibidor del factor α inducible por hipoxia (HIFα)-1 con una IC50 de 3,65μM.
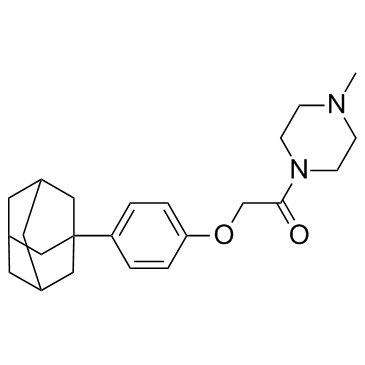
-
GC64108
Ifidancitinib
Ifidancitinib (ATI-50002) es un inhibidor potente y selectivo de las quinasas JAK 1/3.
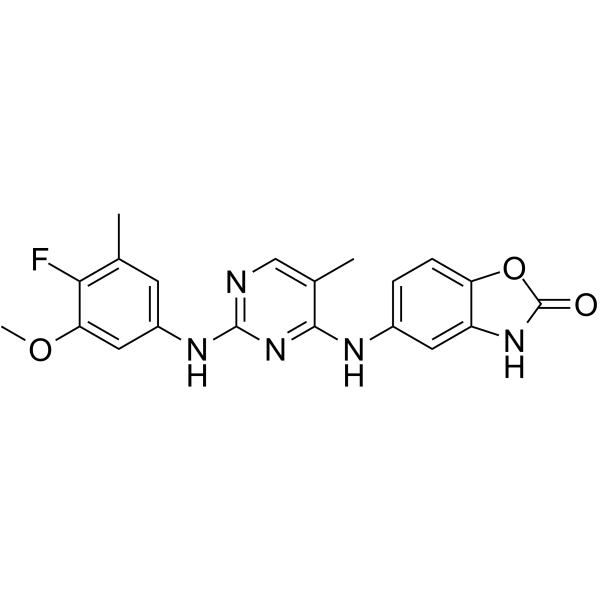
-
GC47450
IL-4 Inhibitor
El inhibidor de IL-4 (compuesto 52) es un inhibidor de IL-4, con un EC50 de 1,81 μM.
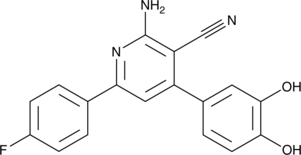
-
GC32809
Ilginatinib (NS-018)
Ilginatinib (NS-018) (NS-018) es un inhibidor de JAK2 altamente activo y biodisponible por vÍa oral, con una IC50 de 0,72 nM, una selectividad de 46, 54 y 31 veces para JAK2 sobre JAK1 (IC50, 33 nM), JAK3 (IC50, 39 nM) y Tyk2 (IC50, 22 nM).
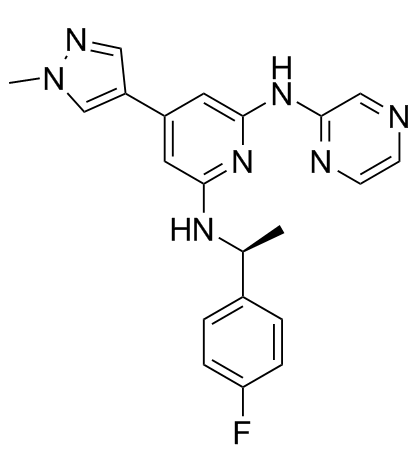
-
GC33037
Ilginatinib hydrochloride (NS-018 hydrochloride)
El clorhidrato de ilginatinib (clorhidrato de NS-018) (clorhidrato de NS-018) es un inhibidor de JAK2 altamente activo y biodisponible por vÍa oral, con una IC50 de 0,72 nM, selectividad de 46, 54 y 31 veces para JAK2 sobre JAK1 (IC50, 33 nM), JAK3 (IC50, 39 nM) y Tyk2 (IC50, 22 nM).
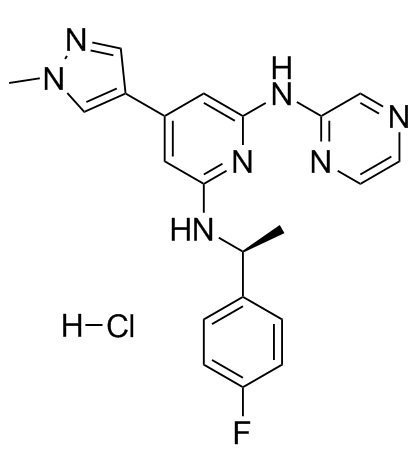
-
GC33018
Ilginatinib maleate (NS-018 (maleate))
El maleato de ilginatinib (NS-018 (maleato)) (maleato de NS-018) es un inhibidor de JAK2 altamente activo y biodisponible por vÍa oral, con una CI50 de 0,72 nM, una selectividad de 46, 54 y 31 veces para JAK2 sobre JAK1 (IC50 , 33 nM), JAK3 (IC50, 39 nM) y Tyk2 (IC50, 22 nM).
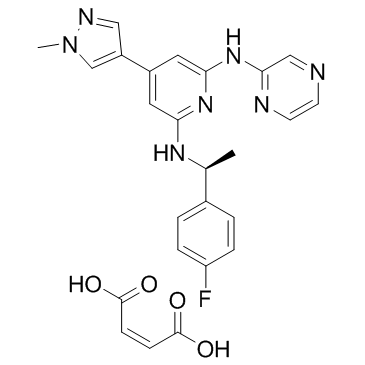
-
GC34159
Ilorasertib (ABT-348)
Ilorasertib (ABT-348) (ABT-348) es un inhibidor de aurora potente, activo por vÍa oral y competitivo con ATP con IC50 de 116, 5, 1 nM para aurora A, aurora B, aurora C, respectivamente. Ilorasertib (ABT-348) también es un potente inhibidor de VEGF, PDGF. Ilorasertib (ABT-348) tiene potencial para la investigaciÓn de la leucemia mieloide aguda (LMA) y el sÍndrome mielodisplÁsico (SMD).
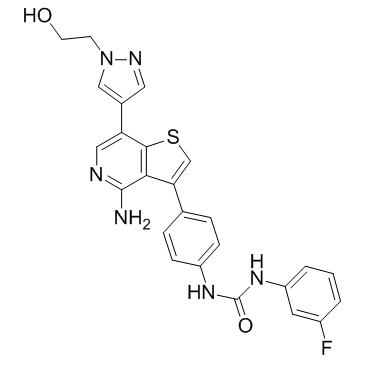
-
GC38519
Ilorasertib hydrochloride
El clorhidrato de ilorasertib (ABT-348) es un inhibidor de la aurora potente, oralmente activo y competitivo con ATP con IC50 de 116, 5, 1 nM para aurora A, aurora B, aurora C, respectivamente. El clorhidrato de ilorasertib también es un potente inhibidor de VEGF, PDGF. El clorhidrato de ilorasertib tiene potencial para la investigaciÓn de la leucemia mieloide aguda (LMA) y el sÍndrome mielodisplÁsico (SMD).
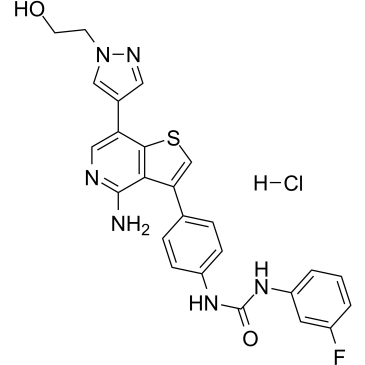
-
GC63309
Ilunocitinib
Ilunocitinib (compuesto 27) es un inhibidor de JAK (extraÍdo de la patente WO2009114512A1).
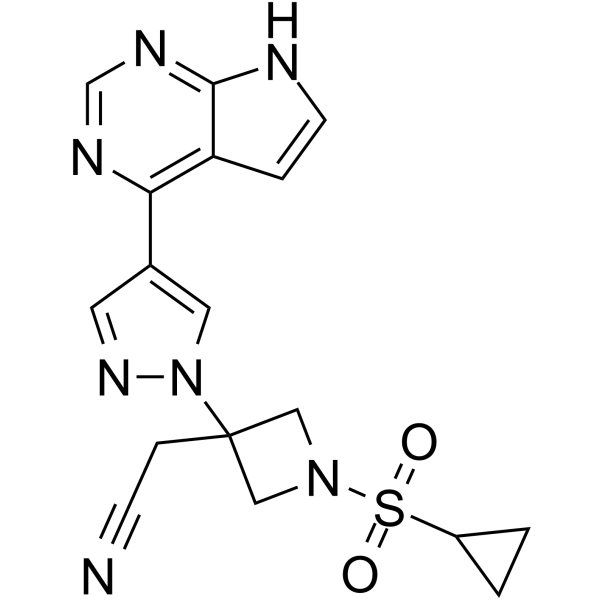
-
GC14755
Inauhzin
Inauhzin es un inhibidor dual SirT1/IMPDH2 y actÚa como un activador p53, utilizado en la investigaciÓn del cÁncer.
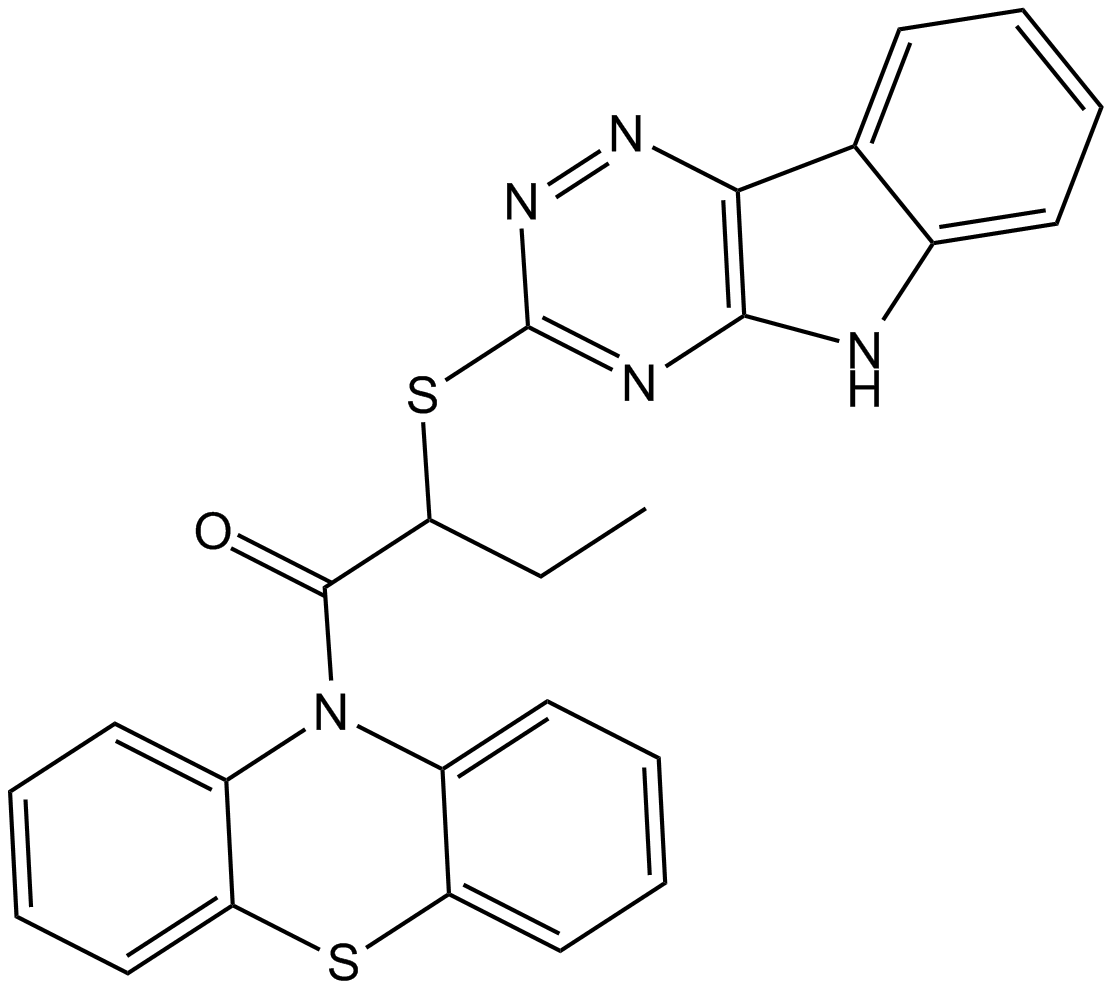
-
GC16117
INCA-6
A cell-permeant inhibitor of NFAT signaling
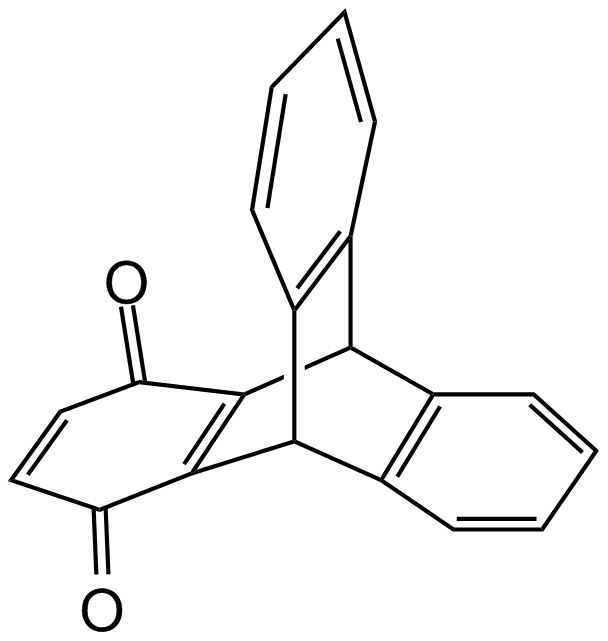
-
GC19200
INCB-057643
INCB-057643 es un nuevo inhibidor BET biodisponible por vÍa oral.
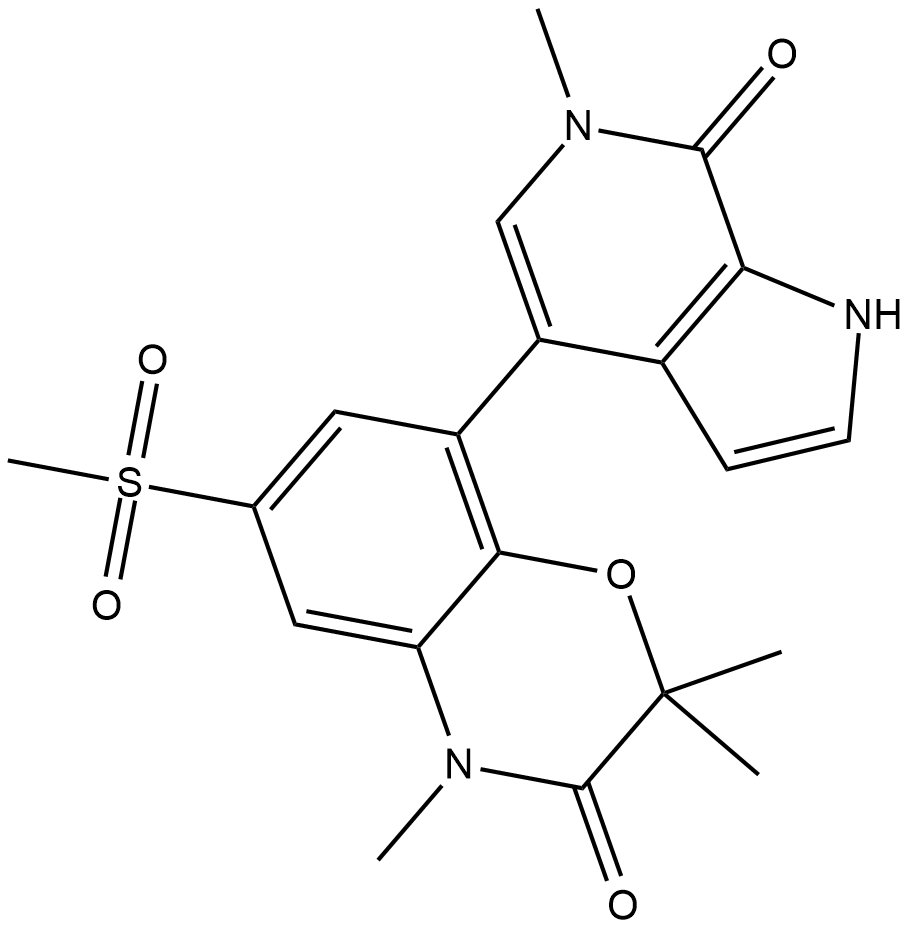
-
GC33026
INCB054329
INCB054329 es un potente inhibidor de BET.
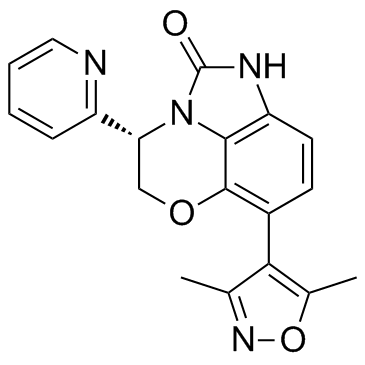
-
GC30644
INCB054329 Racemate
INCB054329 El racemato es un inhibidor de la proteÍna BET.
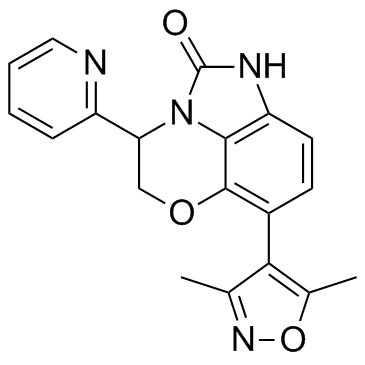
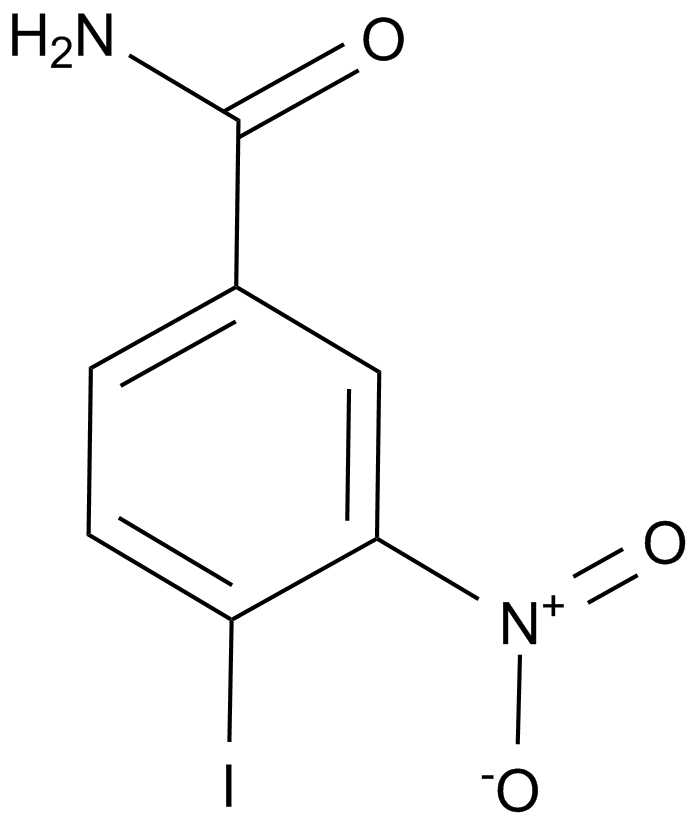
-
GC15353
Iniparib (BSI-201)
A PARP1 inhibitor
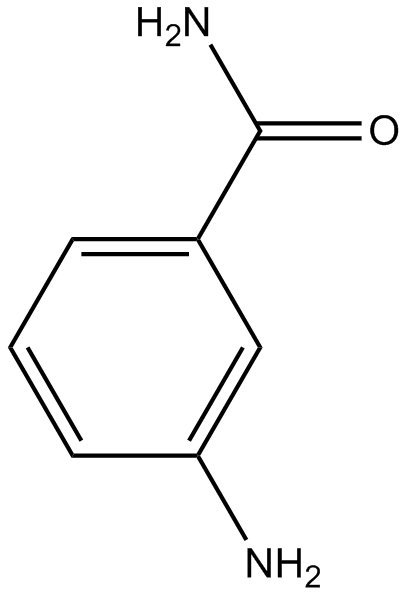
-
GC12496
INO-1001
A PARP inhibitor
-
GC38385
INO-1001
-
GC17754
IOX 1
IOX 1, 5-carboxi-8-hidroxiquinolina, es un potente inhibidor de amplio espectro de las oxigenasas 2OG, incluidas las desmetilasas JmjC. IOX 1 inhibe KDM4C, KDM4E, KDM2A, KDM3A y KDM6B con valores IC50 de 0,6 μM, 2,3 μM, 1,8 μM, 0,1 μM y 1,4 μM, respectivamente. IOX 1 también inhibe ALKBH5.
-
GC13018
IOX2(Glycine)
A selective PHD2 inhibitor
-
GC12255
IOX4
IOX4 es un inhibidor selectivo de HIF prolil-hidroxilasa 2 (PHD2) con un valor IC50 de 1,6 nM, induce HIFα en células y en ratones de tipo salvaje con una marcada inducciÓn en el tejido cerebral.
-
GC50721
iP300w
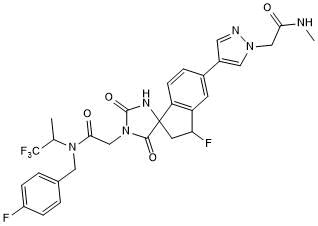
-
GC10727
Ischemin sodium salt
CBP bromodomain inhibitor
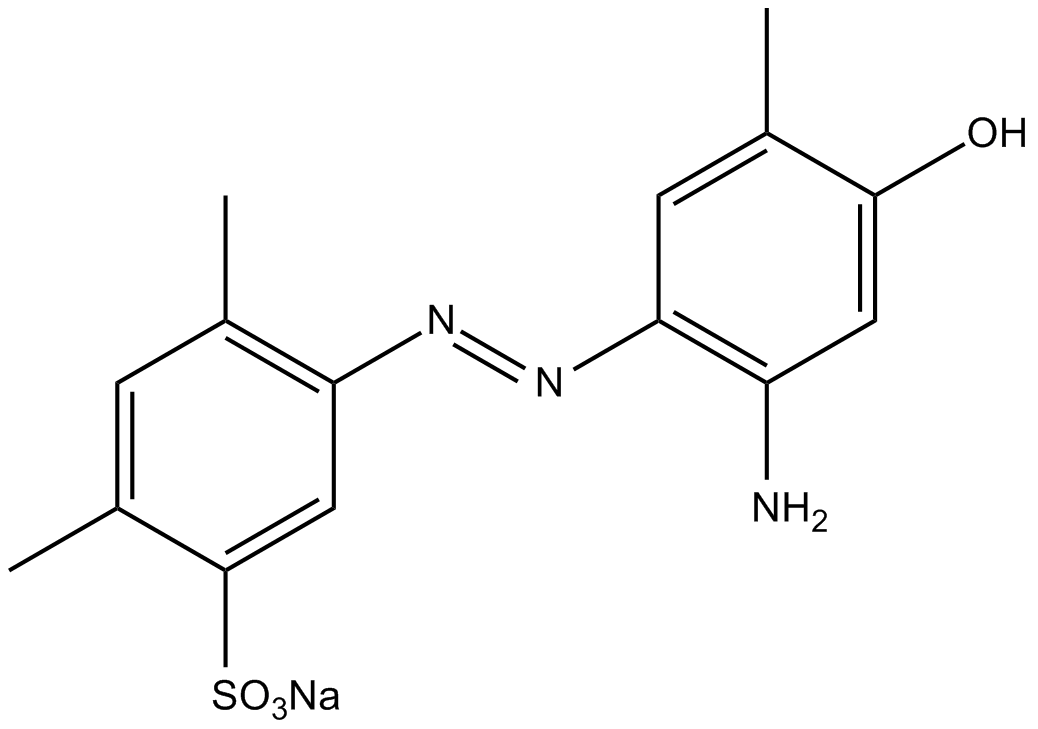
-
GC19204
Itacitinib
Itacitinib (INCB039110) es un inhibidor selectivo y activo por vÍa oral de JAK1 con una IC50 de 2 nM para JAK1 humano. Itacitinib muestra una selectividad >20 veces para JAK1 sobre JAK2 y >100 veces para JAK3 y TYK2; Itacitinib se utiliza en la investigaciÓn de la mielofibrosis.
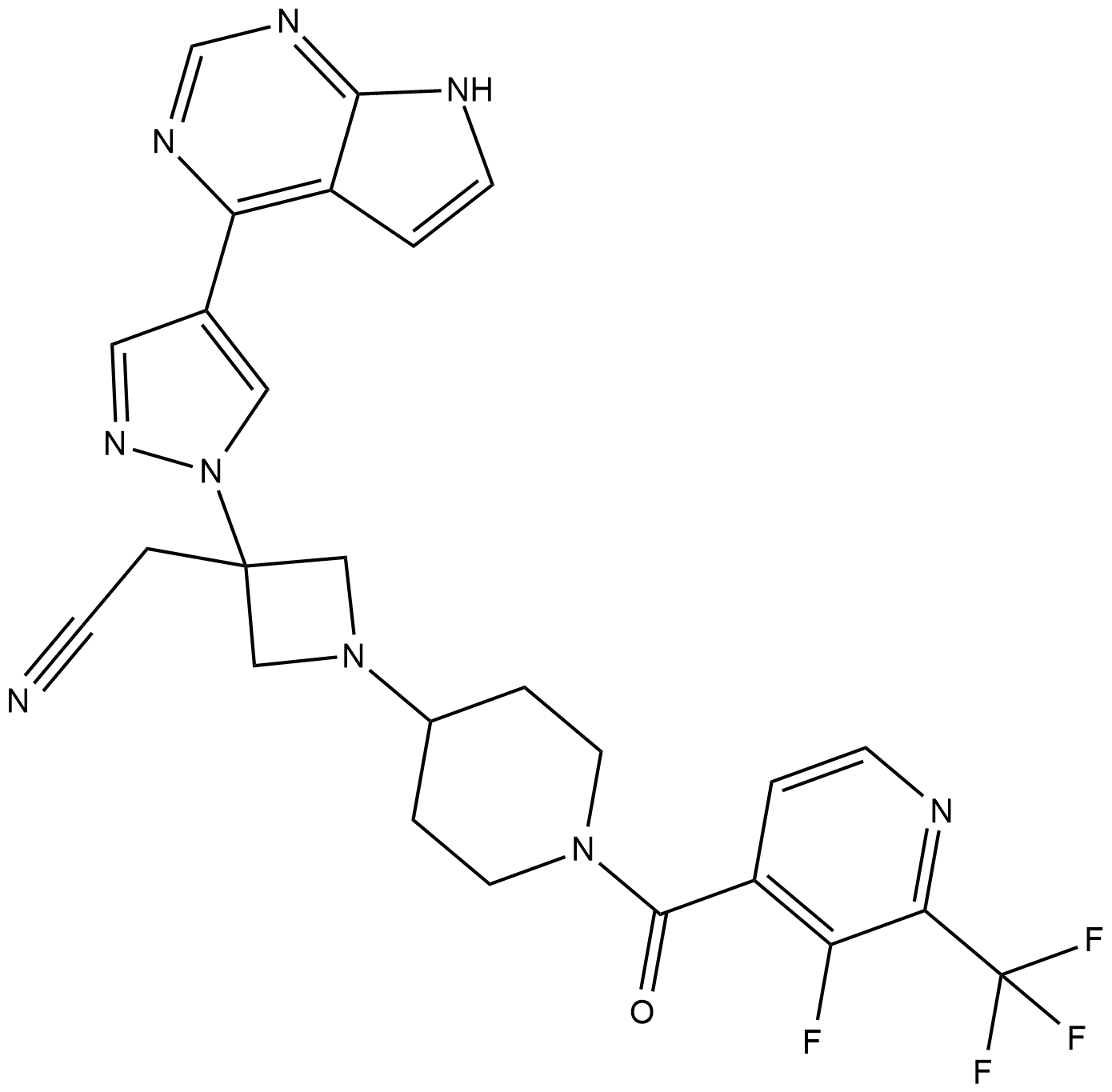
-
GC36351
Itacitinib adipate
El adipato de itacitinib es un inhibidor de JAK1 selectivo y biodisponible por vÍa oral cuya eficacia y seguridad se han probado en un ensayo de fase II en mielofibrosis.
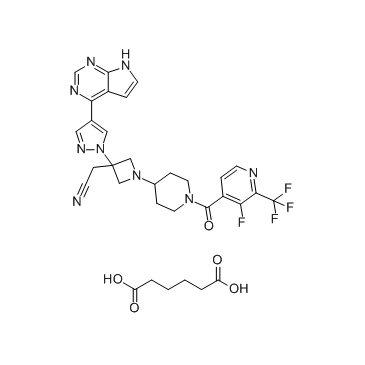
-
GC63416
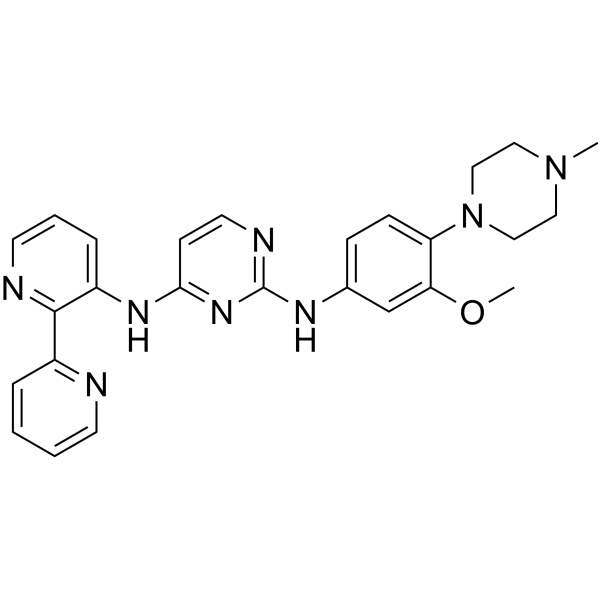
Itacnosertib
Itacnosertib (TP-0184) es un inhibidor de JAK2, ACVR1 (ALK2) y ALK5 como se describe en el documento WO2014151871.
-
GC17836
ITF2357 (Givinostat)
HDAC inhibitor with anti-inflammatory and antineoplastic activities
-
GC14597
ITSA-1 (ITSA1)
ITSA-1 (ITSA1) es un activador de la histona desacetilasa (HDAC) y contrarresta la detenciÓn del ciclo celular inducida por la tricostatina A (TSA), la acetilaciÓn de histonas y la activaciÓn transcripcional.
-
GC63703
Ivaltinostat formic
El valtinostat (CG-200745) fÓrmico es un inhibidor pan-HDAC potente y activo por vÍa oral que tiene la fracciÓn de Ácido hidroxÁmico para unirse al zinc en el fondo de la bolsa catalÍtica.

-
GC15836
IWP 12
IWP 12 es un potente inhibidor de puercoespín (PORCN) e inhibe la señalización Wnt autónoma celular con una IC50 de 15 nM.
-
GC62313
Izencitinib
Izencitinib (TD-1473) es un inhibidor de JAK activo por vÍa oral, no selectivo y restringido al intestino.
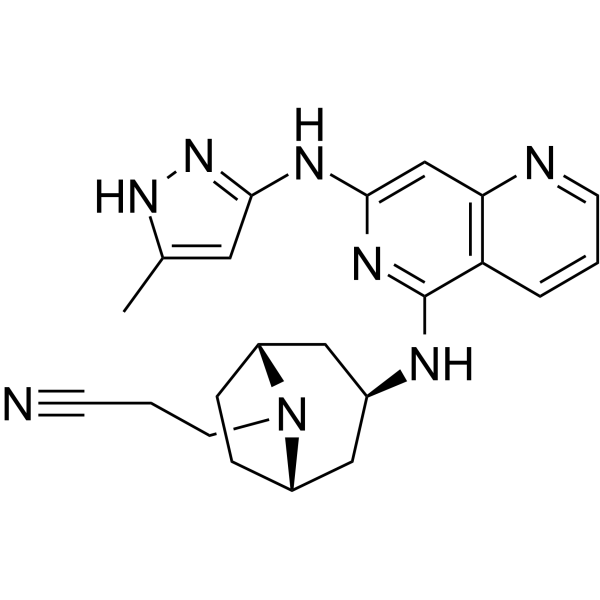
-
GC63828
Izilendustat
Izilendustat es un potente inhibidor de la prolil hidroxilasa que puede estabilizar el factor 1 alfa inducible por hipoxia (HIF-1α) y el factor 2 inducible por hipoxia (HIF-2).
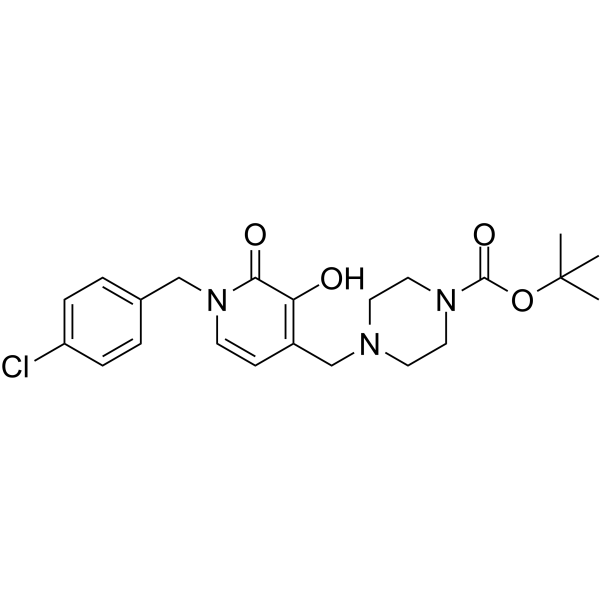
-
GC34629
J22352
J22352 es un inhibidor de HDAC6 altamente selectivo y similar a PROTAC (proteolysis-targetingchimeras) con un valor IC50 de 4,7 nM. J22352 promueve la degradaciÓn de HDAC6 e induce efectos anticancerÍgenos al inhibir la autofagia y provocar la respuesta inmunitaria antitumoral en los cÁnceres de glioblastoma, y conduce a la restauraciÓn de la actividad antitumoral del huésped al reducir la actividad inmunosupresora de PD-L1.
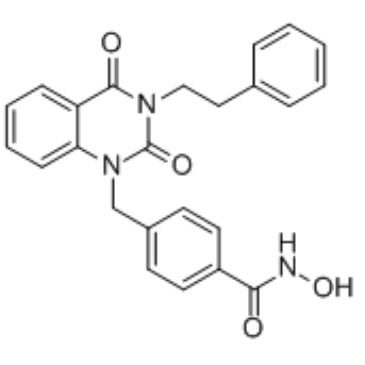
-
GC31866
JAK inhibitor 1
El inhibidor 1 de JAK es un inhibidor de JAK extraÍdo de la patente US20170121327A1, ejemplo de compuesto 283.
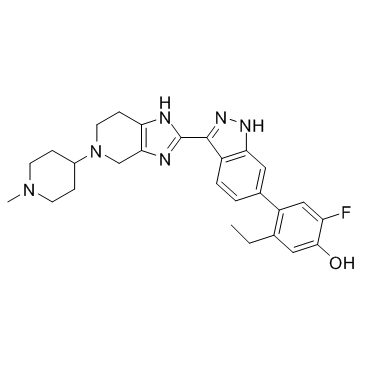
-
GC31999
JAK-IN-1
JAK-IN-1 es un inhibidor de JAK1/2/3 con IC50 de 0,26, 0,8 y 3,2 nM, respectivamente.
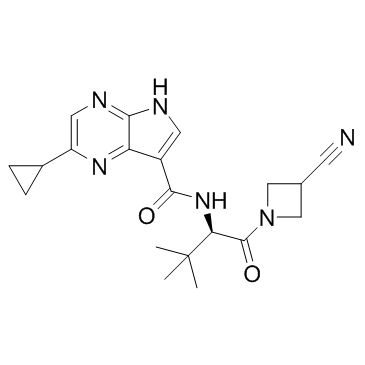
-
GC36366
JAK-IN-10
JAK-IN-10 es un inhibidor de JAK.
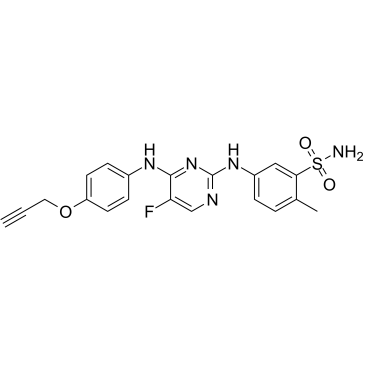
-
GC63448
JAK-IN-14
JAK-IN-14 es un inhibidor potente y selectivo de JAK1, con una IC50 de <5 μM.
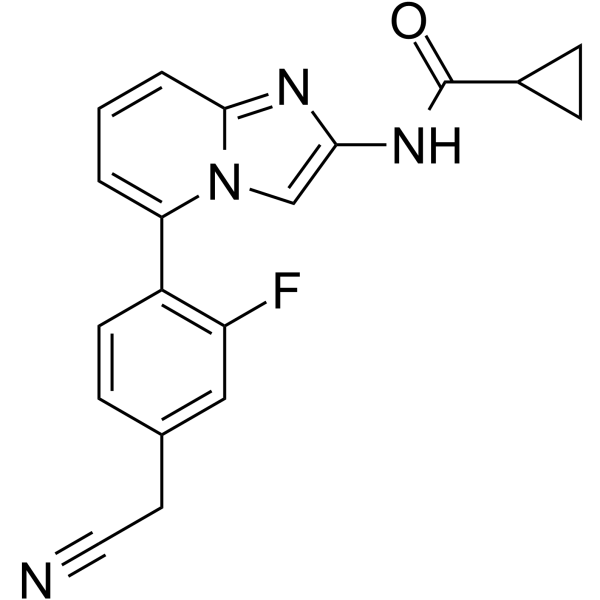
-
GC68000
JAK-IN-23
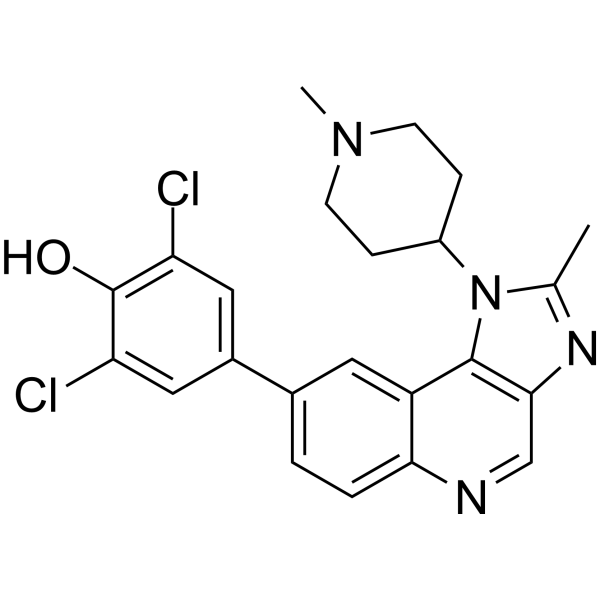
-
GC65453
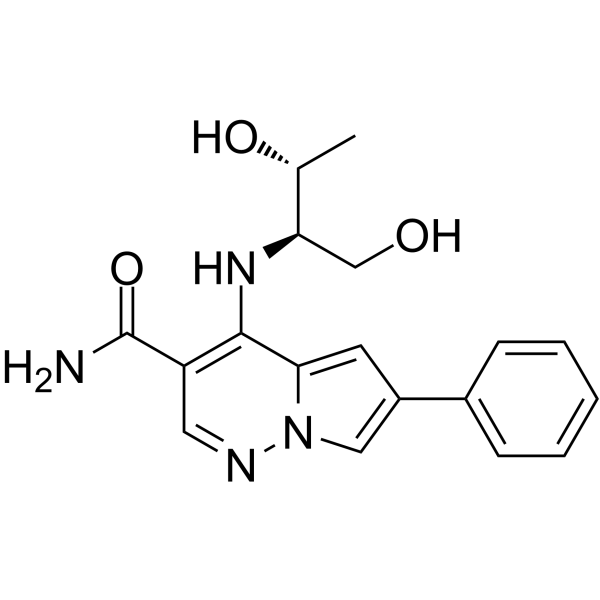
JAK-IN-3
JAK-IN-3 (compuesto 22) es un potente inhibidor de JAK, con valores IC50 de 3 nM, 5 nM, 34 nM y 70 nM para JAK3, JAK1, TYK2 y JAK2, respectivamente.
-
GC64959
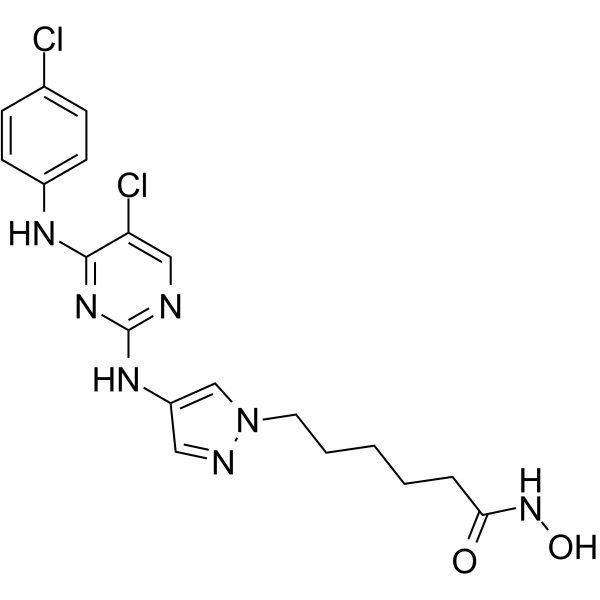
JAK/HDAC-IN-1
JAK/HDAC-IN-1 es un potente inhibidor dual de JAK2/HDAC, exhibe actividades antiproliferativas y proapoptÓticas en varias lÍneas celulares hematolÓgicas. JAK/HDAC-IN-1 muestra IC50 de 4 y 2 nM para JAK2 y HDAC, respectivamente.
-
GC33036
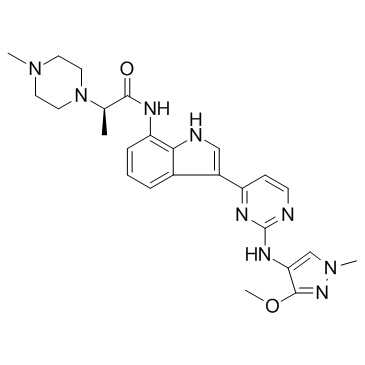
JAK1-IN-3
JAK1-IN-3 (AZD4205) es un inhibidor selectivo de JAK1, con una IC50 de 73 nM, inhibe débilmente JAK2 (IC50>14.7 μM) y muestra poca inhibiciÓn sobre JAK3 (IC50>30 μM).
-
GC33258
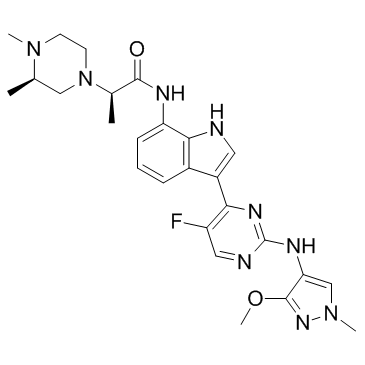
JAK1-IN-4
JAK1-IN-4 es un inhibidor potente y selectivo de JAK1, con IC50 de 85 nM, 12,8 μM y >30 μM para JAK1, JAK2 y JAK3, respectivamente. JAK1-IN-4 inhibe la fosforilaciÓn de STAT3 en células NCI-H 1975 (IC50, 227 nM).
-
GC36363
JAK1-IN-7
JAK1-IN-7 (JAK1-IN-7) es un inhibidor de la quinasa 1 asociada a Janus (JAK1) extraÍdo de la patente WO2018134213A1, ejemplo 63, tiene un efecto antiinflamatorio.
-
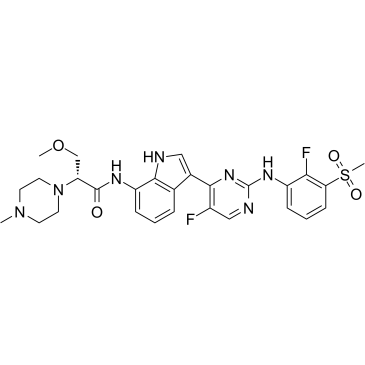
GC63029
JAK1-IN-8
JAK1-IN-8, un potente inhibidor de JAK1 (IC50<500 nM), compuesto 28, extraÍdo de la patente WO2016119700A1.
-
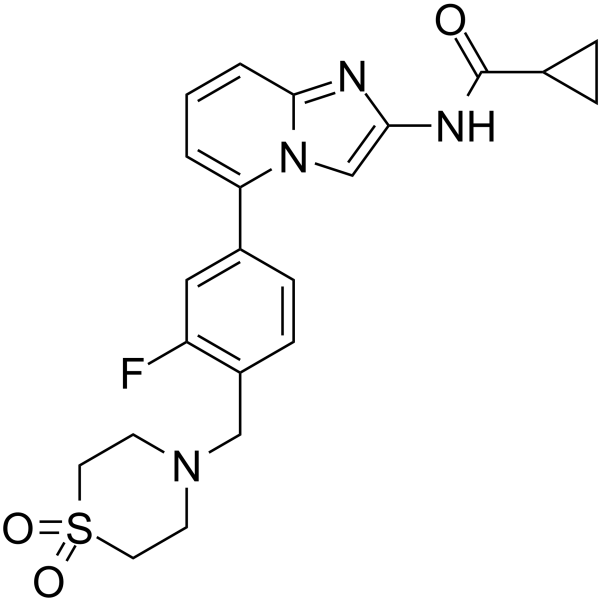
GC13826
JAK2 Inhibitor V, Z3
A selective inhibitor of the autophosphorylation of wild type and V617F mutant forms of JAK
-
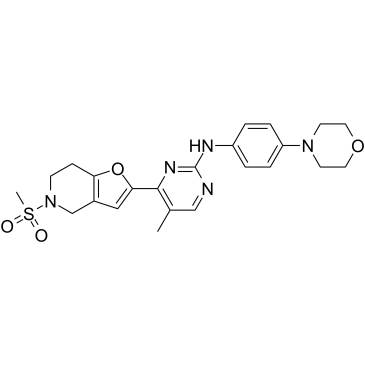
GC36364
JAK2-IN-4
JAK2-IN-4 (compuesto 16h) es un inhibidor selectivo de JAK2/JAK3, con valores de CI50 de 0,7 nM y 23,2 nM para JAK2 y JAK3, respectivamente.
-
GC65405
JAK2-IN-6
JAK2-IN-6, un derivado de aminotiazol con sustituciÓn mÚltiple, es un inhibidor potente y selectivo de JAK2 con una IC50 de 22,86 μg/mL. JAK2-IN-6 no muestra actividad contra JAK1 y JAK3. JAK2-IN-6 tiene un efecto antiproliferativo contra las células cancerosas.
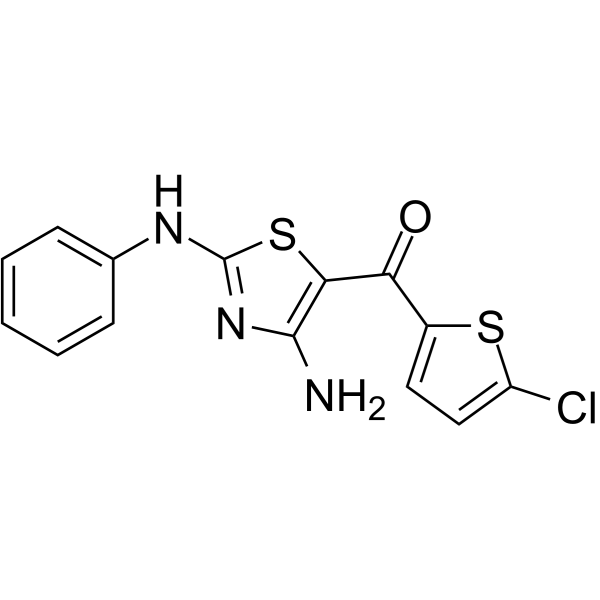
-
GC62500
JAK2-IN-7
JAK2-IN-7 es un inhibidor selectivo de JAK2 con IC50 de 3, 11,7 y 41 nM para células JAK2, SET-2 y Ba/F3V617F, respectivamente. JAK2-IN-7 posee una selectividad >14 veces superior a JAK1, JAK3, FLT3. JAK2-IN-7 estimula la detenciÓn del ciclo celular en la fase G0/G1 e induce la celapotosis tumoral. Actividades antitumorales.
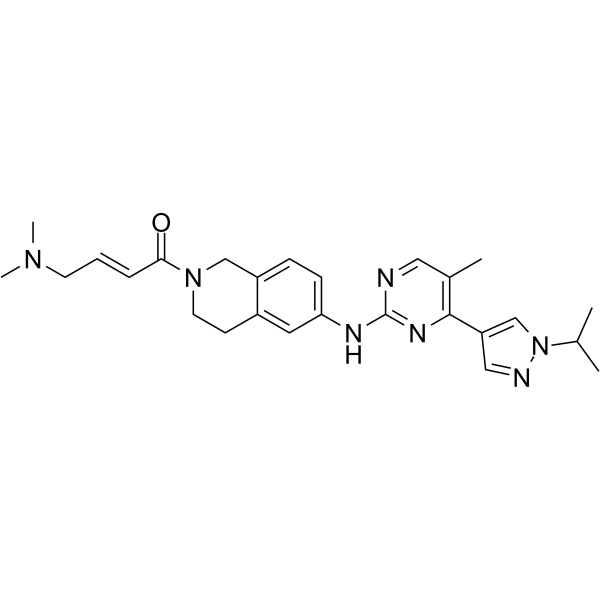
-
GC62665
JAK2/FLT3-IN-1 TFA
JAK2/FLT3-IN-1 (TFA) es un inhibidor dual potente y activo por vÍa oral de JAK2/FLT3 con valores IC50 de 0,7 nM, 4 nM, 26 nM y 39 nM para JAK2, FLT3, JAK1 y JAK3, respectivamente. JAK2/FLT3-IN-1 (TFA) tiene actividad anticancerÍgena.
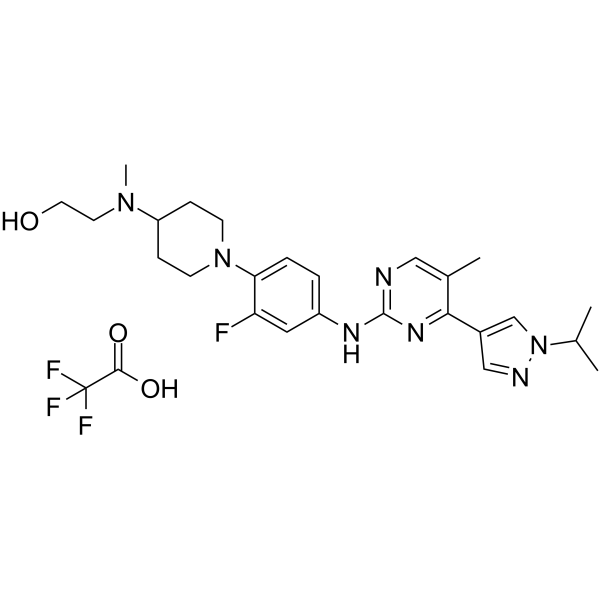
-
GC36365
JAK3 covalent inhibitor-1
El inhibidor covalente-1 de JAK3 es un inhibidor covalente potente y selectivo de la cinasa 3 de Janus (JAK3) con una IC50 de 11 nM y muestra una selectividad de 246 veces frente a otras JAK.
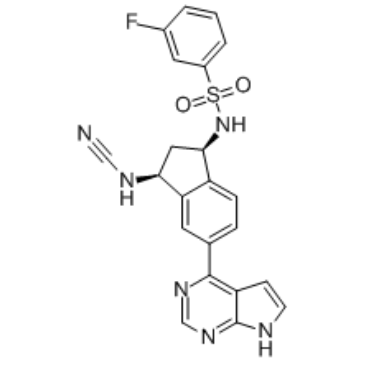
-
GC33046
JAK3-IN-1
JAK3-IN-1 es un inhibidor de JAK3 potente, selectivo y activo por vÍa oral con una IC50 de 4,8 nM. JAK3-IN-1 muestra mÁs de 180 veces mÁs selectividad para JAK3 que JAK1 (IC50 de 896 nM) y JAK2 (IC50 de 1050 nM).
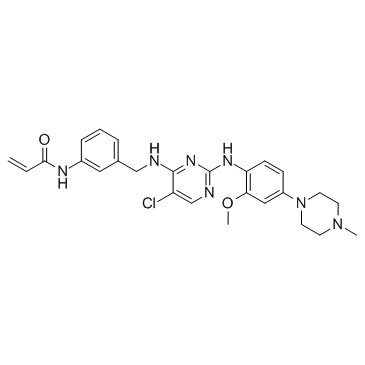
-
GC31902
JAK3-IN-6
JAK3-IN-6 es un inhibidor potente, selectivo e irreversible de la quinasa 3 asociada a Janus (JAK3), con una IC50 de 0,15 nM.
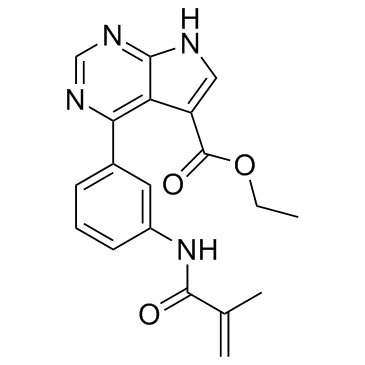
-
GC33382
JAK3-IN-7
JAK3-IN-7 es un potente y selectivo inhibidor de JAK3 extraÍdo de la patente WO2011013785A1, tiene un IC50 de <0.01 μM.
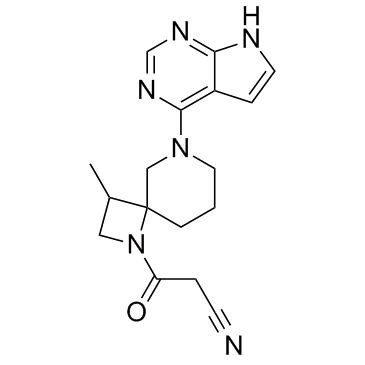
-
GC14671
JANEX-1
A selective JAK3 inhibitor
-
GC14686
JFD00244
JFD00244 es un inhibidor de la sirtuina 2 (SIRT2), con efecto antitumoral. JFD00244 también es un inhibidor de Nsp-16 contra el SARS-CoV-2.
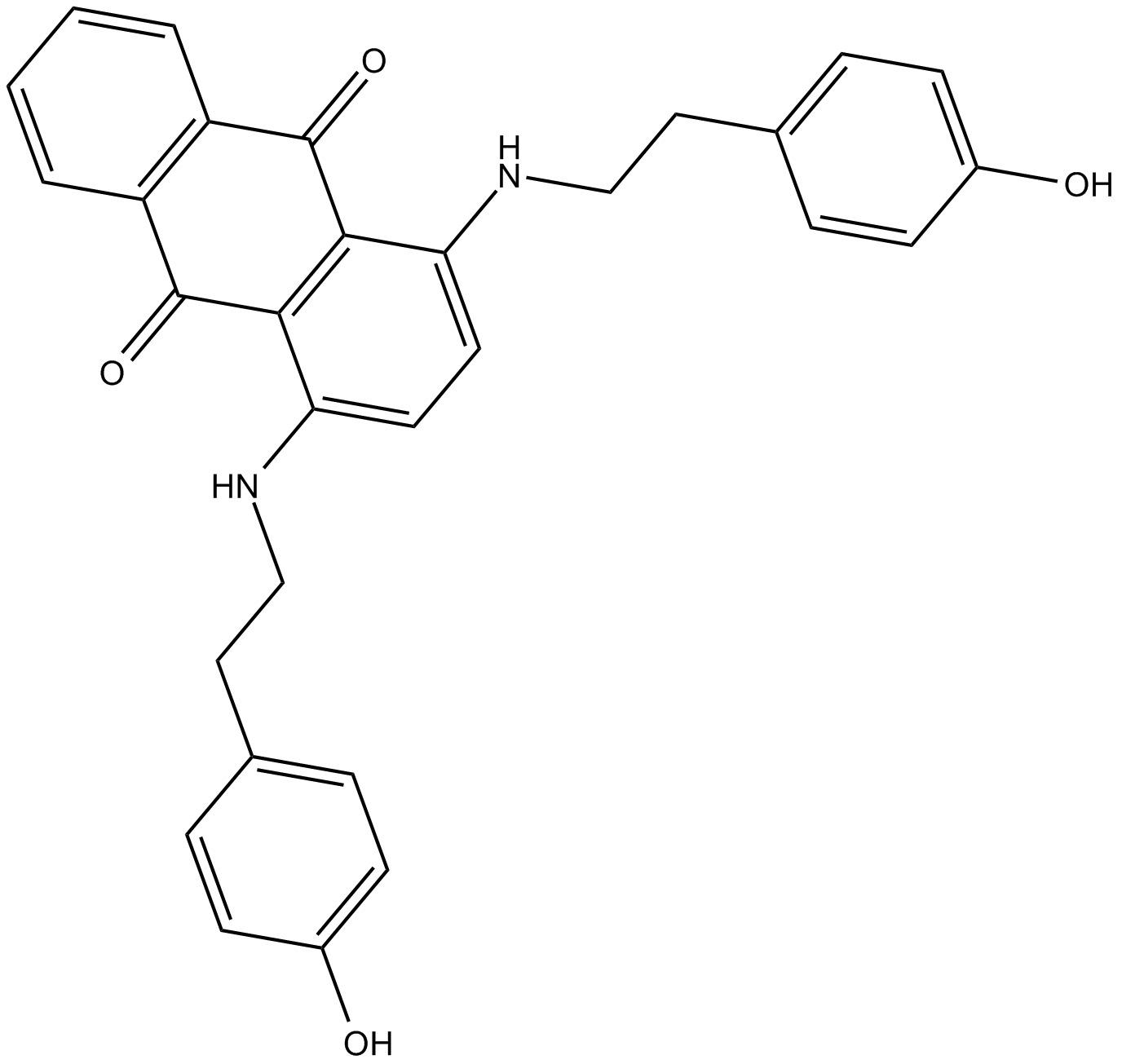
-
GC13062
JGB1741
JGB1741 (ILS-JGB-1741) es un inhibidor potente y especÍfico de la actividad de SIRT1 con una IC50 de ~15 μM. JGB1741 es un inhibidor débil de SIRT2 y SIRT3 con una IC50 >100 μM. JGB1741 aumenta los niveles de p53 acetilados que conducen a la apoptosis mediada por p53 con modulaciÓn de la relaciÓn Bax/Bcl2, liberaciÓn de citocromo c y escisiÓn de PARP. JGB1741 tiene potencial para la investigaciÓn del cÁncer de mama.
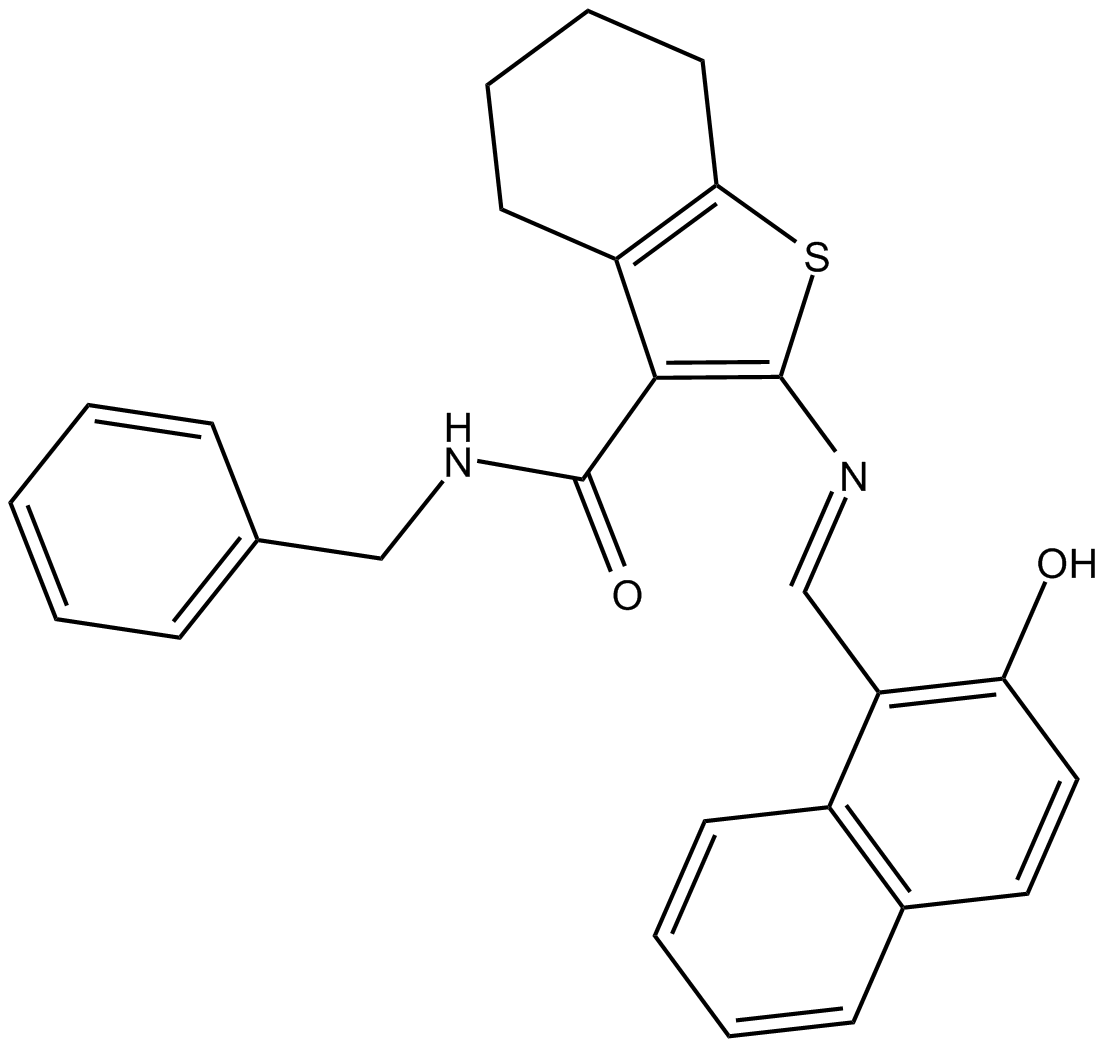
-
GC15603
JIB-04
JIB-04 es un inhibidor pan-selectivo de la histona desmetilasa Jumonji con IC50 de 230, 340, 855, 445, 435, 1100 y 290 nM para JARID1A, JMJD2E, JMJD3, JMJD2A, JMJD2B, JMJD2C y JMJD2D, respectivamente.
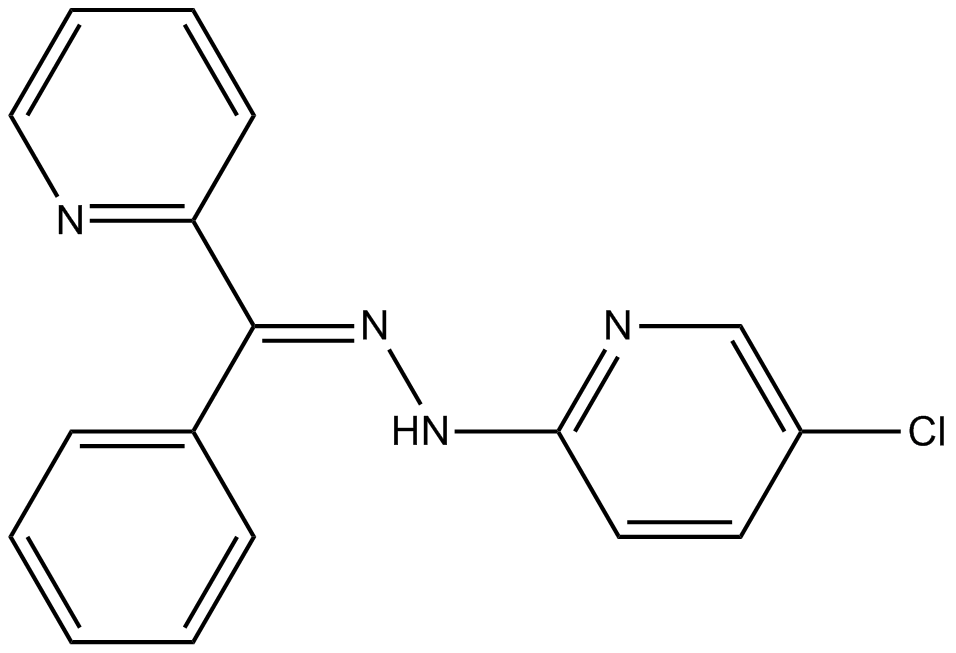
-
GC15476
JNJ-26481585
A pan-HDAC inhibitor
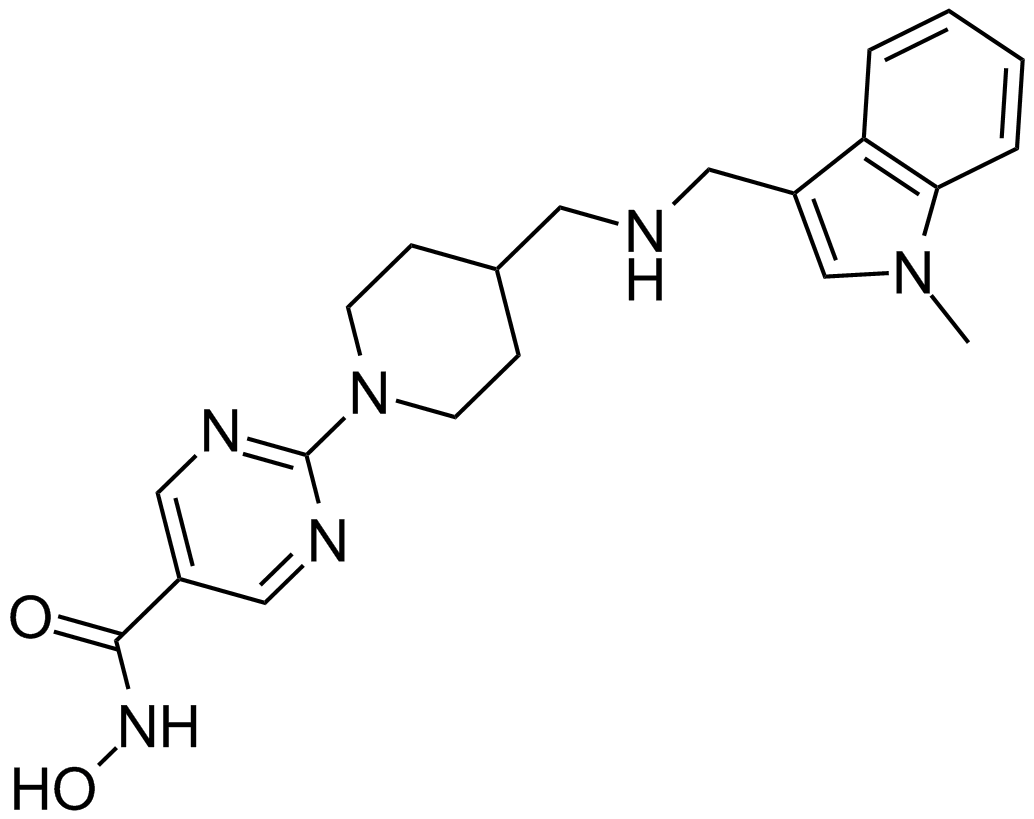
-
GC15379
JNJ-42041935
JNJ-42041935 es un inhibidor potente, competitivo y selectivo de la prolil hidroxilasa PHD; inhibe PHD1, PHD2 y PHD3 con valores de pKi de 7,91 ± 0,04, 7,29 ± 0,05 y 7,65 ± 0,09, respectivamente.
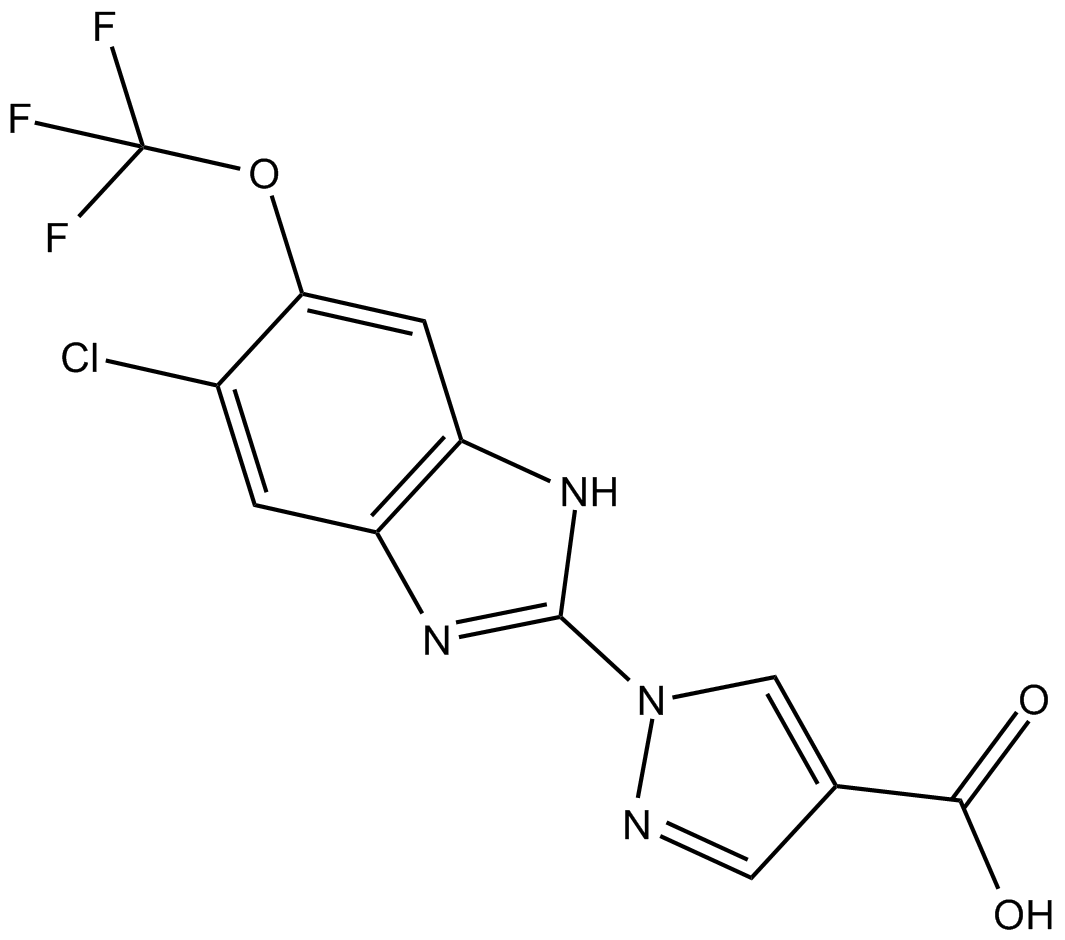
-
GC19465
JNJ-64619178
JNJ-64619178 (Onametostat) es un inhibidor de la proteína arginina metiltransferasa 5 (PRMT5) selectivo, activo por vía oral y pseudoirreversible con una IC50 de 0,14 nM. JNJ-64619178 tiene una potente actividad en el cáncer de pulmón.

-
GC12612
JNJ-7706621
A dual inhibitor of CDKs and Aurora kinases
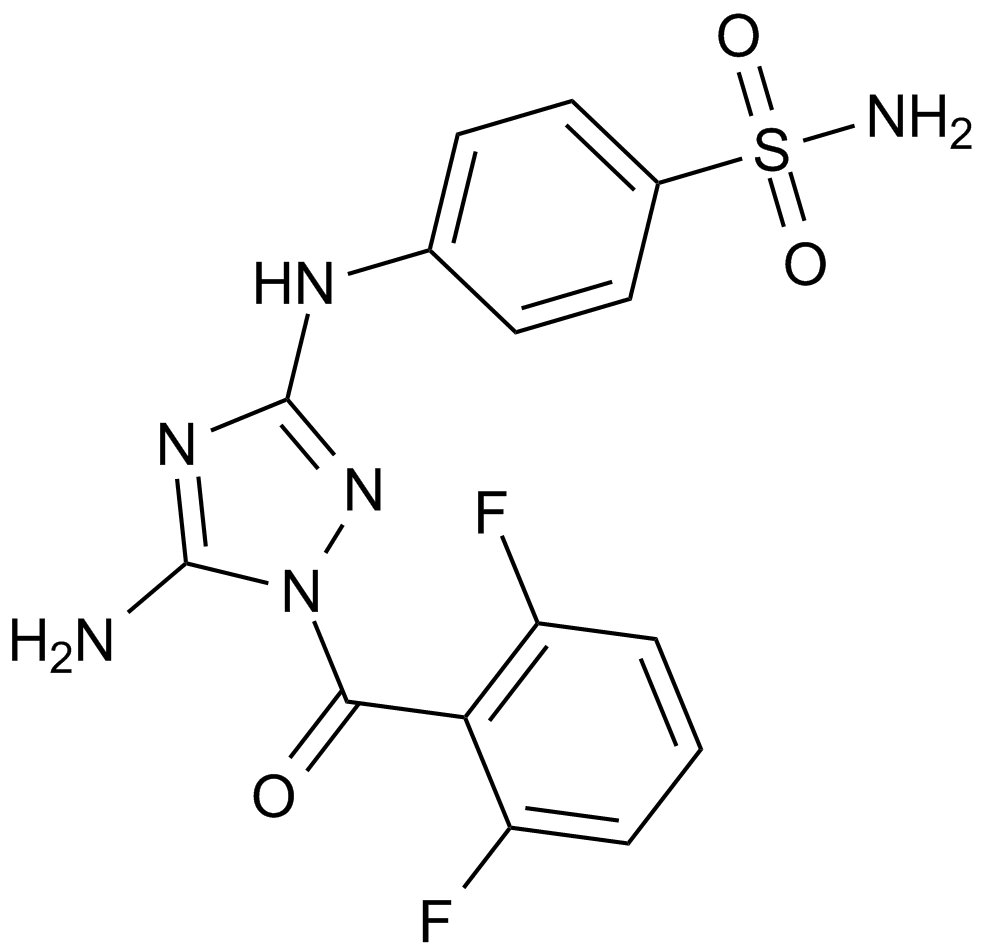
-
GC19210
JQ-1 carboxylic acid
El ácido carboxílico JQ-1, un tipo de derivado de (+)-JQ-1, es un potente inhibidor del bromodominio BET para regular a la baja la expresión de PD-L1 en la superficie de las células tumorales. El ácido carboxílico JQ-1 se puede utilizar como precursor para sintetizar PROTAC, que se dirige a los bromodominios BET.
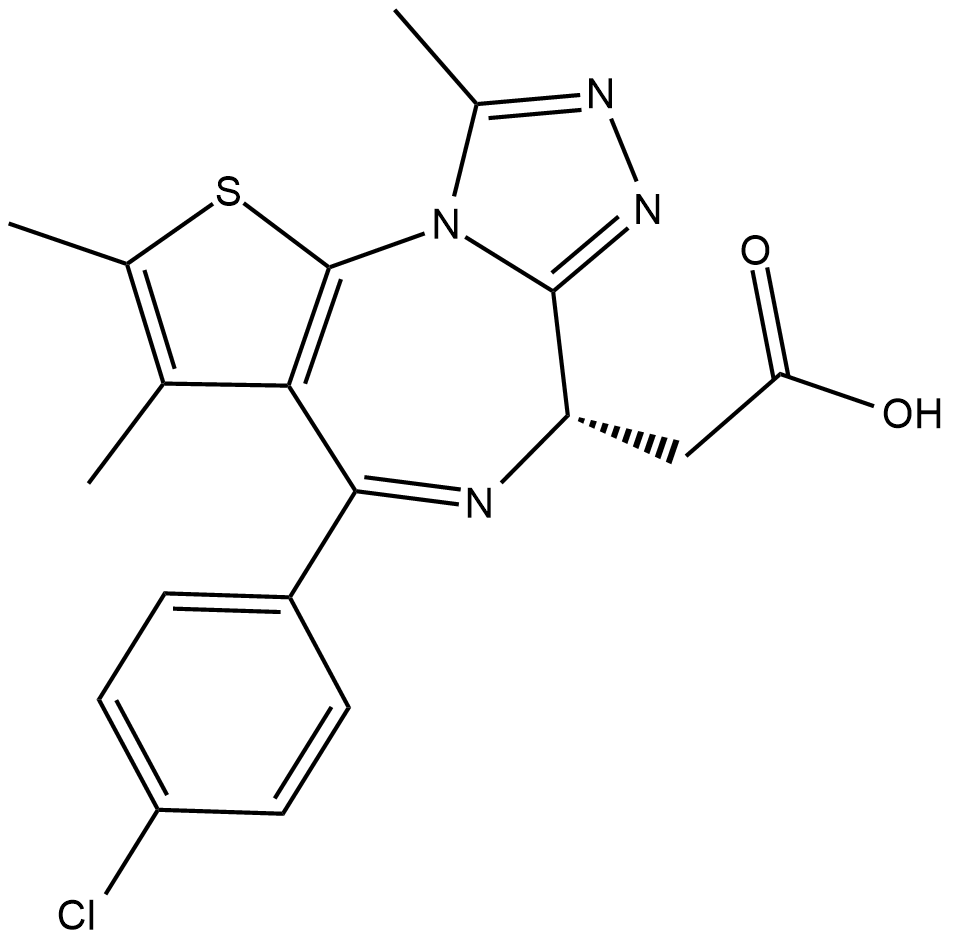
-
GC19211
JQEZ5
JQEZ5 es un inhibidor potente y selectivo de la lisina metiltransferasa EZH2. JQEZ5 InhibiciÓn competitiva de SAM del complejo represivo polycomb 2 (PRC2) con una IC50 de 80 nM. JQEZ5 tiene efectos antitumorales.
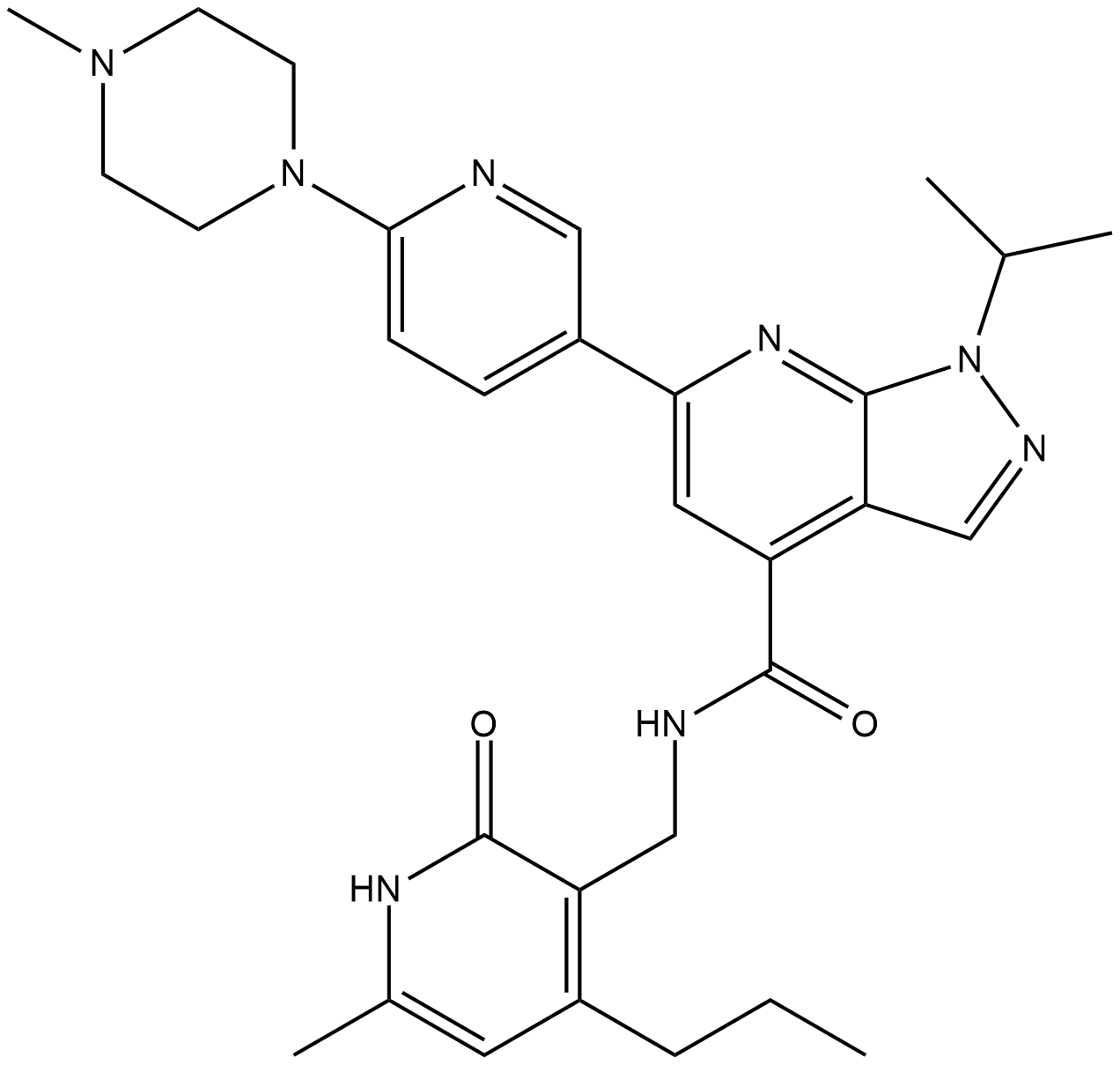
-
GC64841
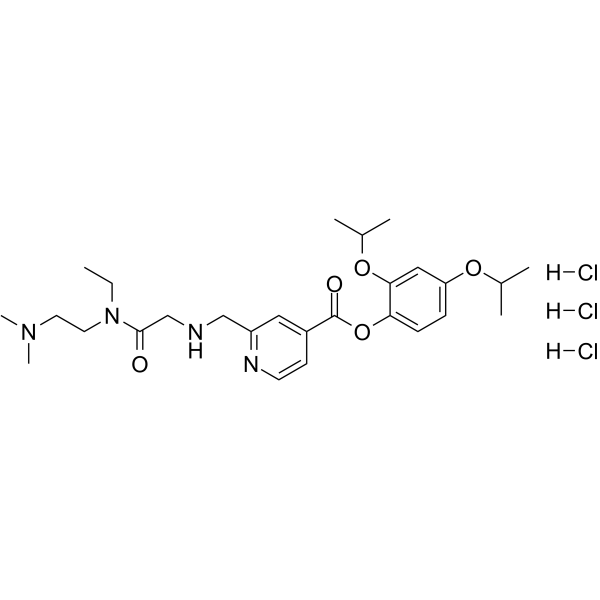
JQKD82 trihydrochloride
El trihidrocloruro de JQKD82 (JADA82) es un inhibidor de KDM5 selectivo y permeable a las células. El trihidrocloruro de JQKD82 aumenta H3K4me3 y se puede utilizar para la investigaciÓn del mieloma mÚltiple.
-
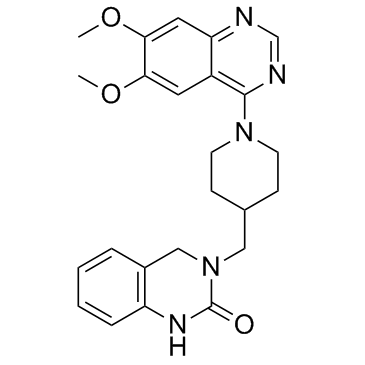
GC34195
K-756
K-756 es un inhibidor de tankirasa directo y selectivo (TNKS), que inhibe la actividad de ribosilaciÓn de ADP de TNKS1 y TNKS2 con IC50 de 31 y 36 nM, respectivamente.
-
GC45489
K-Biotin-W-Histone H2B (108-125) (trifluoroacetate salt)

-
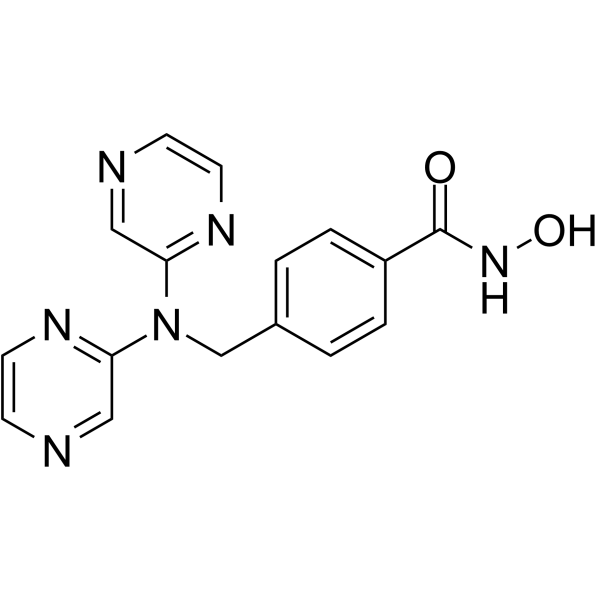
GC62430
KA2507
KA2507 es un inhibidor de HDAC6 potente, activo por vÍa oral y selectivo, con una IC50 de 2,5 nM. KA2507 muestra actividades antitumorales y efectos inmunomoduladores en modelos preclÍnicos.
-
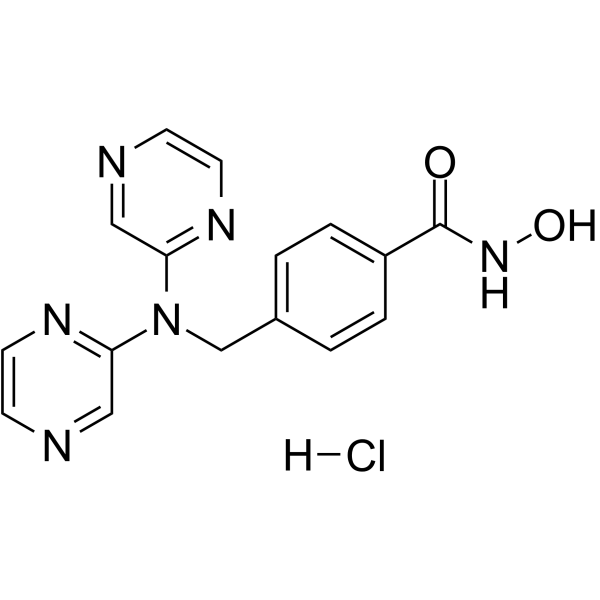
GC62374
KA2507 monohydrochloride
El clorhidrato de KA2507 es un inhibidor potente y altamente selectivo de HDAC6 (IC50 = 2,5 nM) sin toxicidades significativas.
-
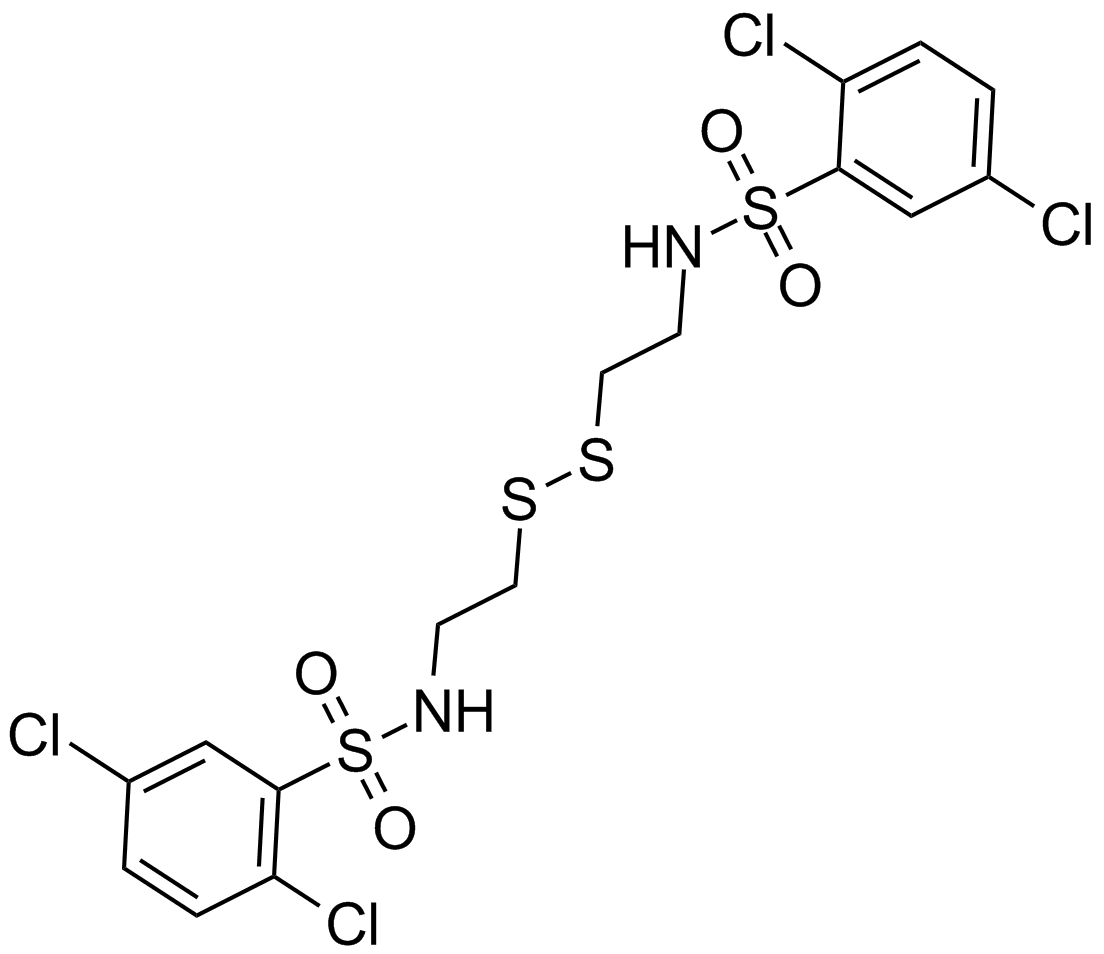
GC13141
KC7F2
An inhibitor of HIF-1a protein synthesis
-
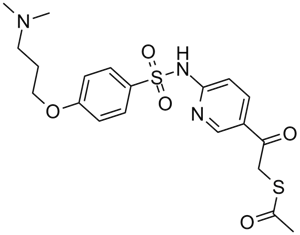
GC13435
KD 5170
An inhibitor of class I and II HDACs
-
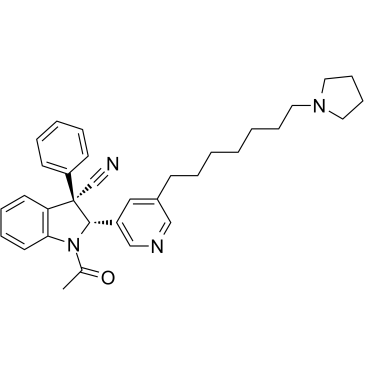
GC36388
KDM2A/7A-IN-1
KDM2A/7A-IN-1 es el primer inhibidor de su clase, selectivo y permeable a las células de las histonas lisina desmetilasas KDM2A/7A, con una CI50 de 0,16μM para KDM2A, exhibe una selectividad de 75 veces sobre otras lisina desmetilasas JmjC y es inactivo en metiltransferasas e histona acetiltransferasas.
-
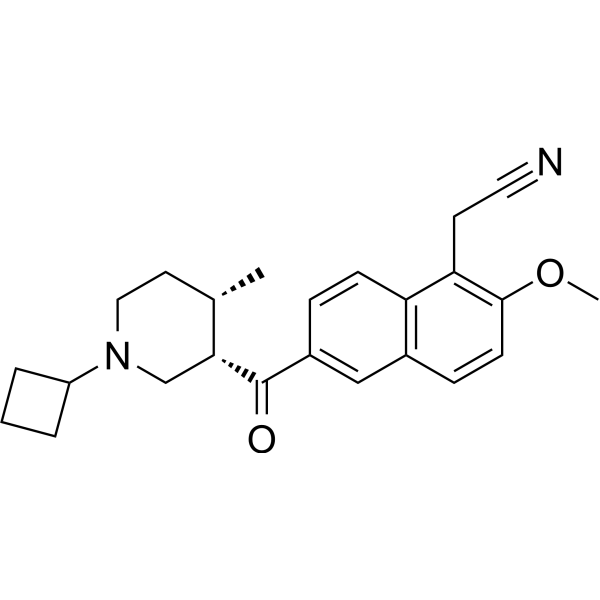
GC69327
KDM2B-IN-4
KDM2B-IN-4 es un inhibidor de la desmetilasa de histonas KDM2B. KDM2B-IN-4 se puede utilizar en investigaciones sobre el cáncer. Para obtener más información, consulte el compuesto 182b en el documento de patente WO2016112284A1.
-
GC36389
KDM4-IN-2
KDM4-IN-2 (Compuesto 19a) es un inhibidor dual potente y selectivo de KDM4/KDM5 con Kis de 4 y 7 nM para KDM4A y KDM5B, respectivamente.
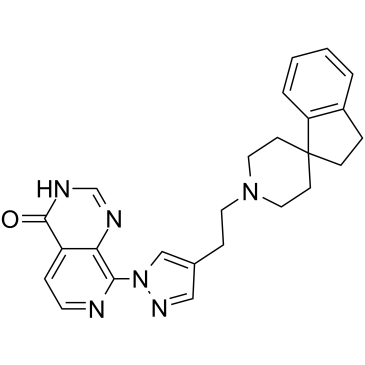
-
GC31887
KDM4D-IN-1
KDM4D-IN-1 es un nuevo inhibidor de la histona lisina desmetilasa 4D (KDM4D) con un valor IC50 de 0,41±0,03 μM.
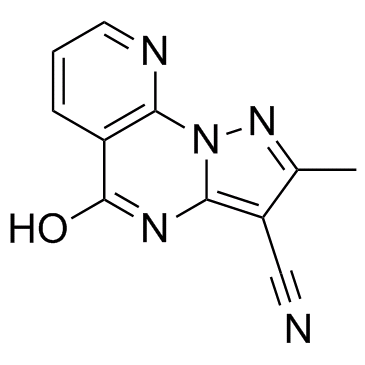
-
GC32790
KDM5-IN-1
KDM5-IN-1 es un inhibidor de KDM5 potente, selectivo y biodisponible por vÍa oral con una IC50 de 15,1 nM.
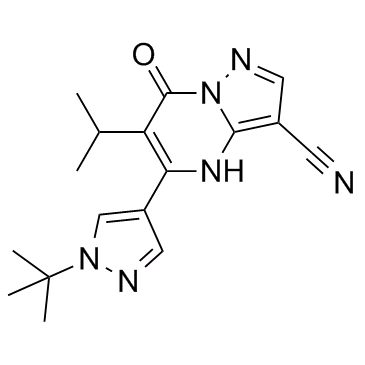
-
GC32842
KDM5A-IN-1
KDM5A-IN-1 es un potente inhibidor biodisponible por vÍa oral de panhistona lisina desmetilasas 5 (KDM5) con IC50 de 45 nM, 56 nM y 55 nM para KDM5A, KDM5B y KDM5C, respectivamente, y con un valor EC50 de 960 nM para PC9 H3K4Me3. KDM5A-IN-1 es significativamente menos potente frente a otras enzimas KDM5B (1A, 2B, 3B, 4C, 6A, 7B).
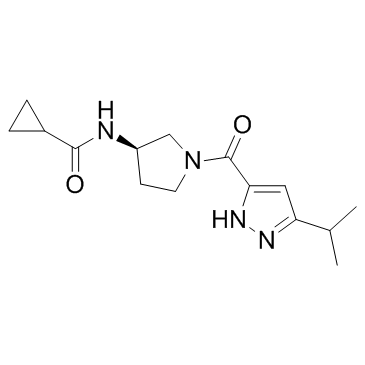
-
GC38709
KDOAM-25 citrate
El citrato KDOAM-25 es un inhibidor de histona lisina desmetilasas 5 (KDM5) potente y altamente selectivo con IC50 de 71 nM, 19 nM, 69 nM, 69 nM para KDM5A, KDM5B, KDM5C, KDM5D, respectivamente. El citrato de KDOAM-25 aumenta la metilaciÓn global de H3K4 en los sitios de inicio de la transcripciÓn y afecta la proliferaciÓn en células MM1S de mieloma mÚltiple.
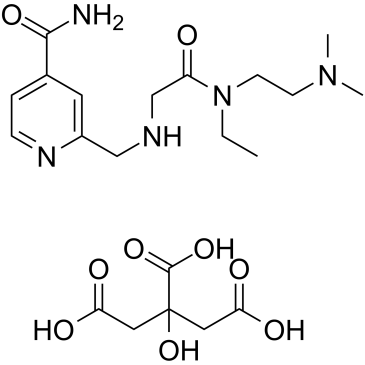
-
GC31654
KG-501 (Naphthol AS-E phosphate)
KG-501 (fosfato de naftol AS-E) es un inhibidor de CREB, con un IC50 de 6,89 μM.
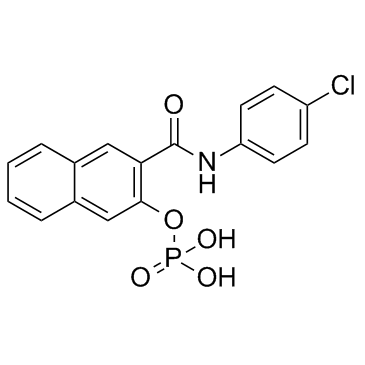
-
GC65907
KSQ-4279
KSQ-4279 (USP1-IN-1, FÓrmula I) es un inhibidor de USP1 y PARP (extraÍdo de la patente WO2021163530).
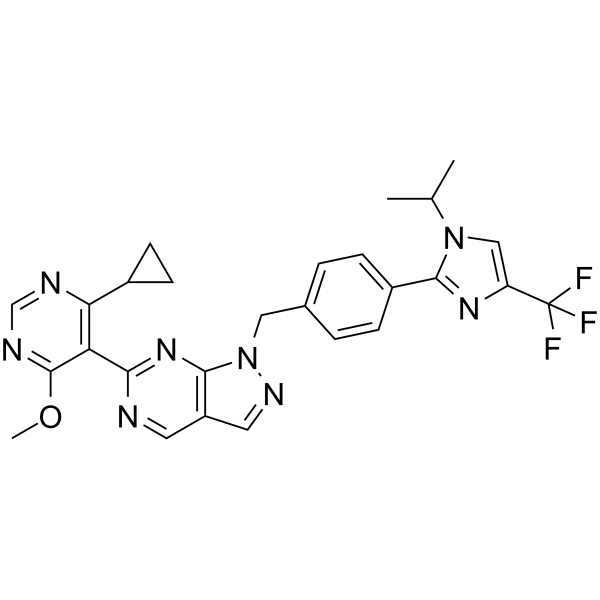
-
GC25552
KT-531
KT-531 (KT531) is a potent, selective HDAC6 inhibitor with IC50 of 8.5 nM, displays 39-fold selectivity over other HDAC isoforms.

-
GC14592
KW 2449
A multi-kinase inhibitor
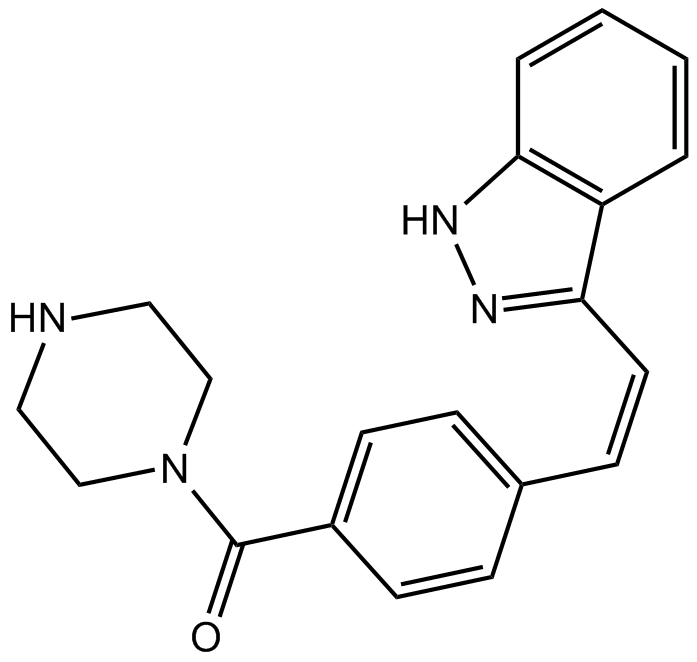
-
GC50395
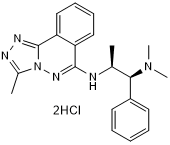
L Moses dihydrochloride
High affinity and selective PCAF bromodomain inhibitor
-
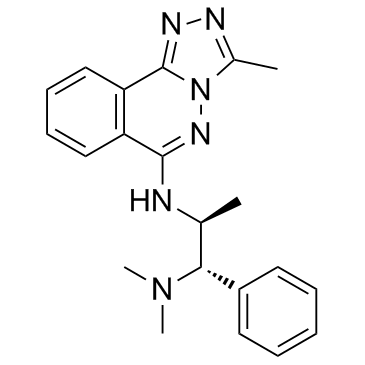
GC33183
L-45 (L-Moses)
L-45 (L-Moses) (L-45) es el primer inhibidor de bromodominio (Brd) del factor asociado a p300/CBP (PCAF) potente, selectivo y activo celular con una Kd de 126 nM.
-
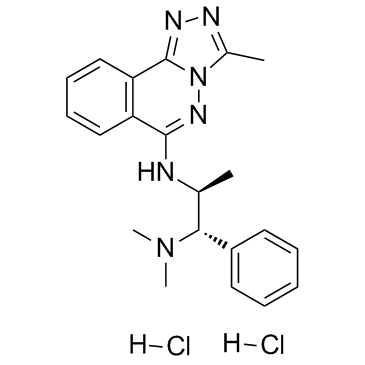
GC34377
L-45 dihydrochloride (L-Moses dihydrochloride)
-
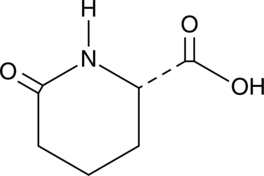
GC47579
L-Pyrohomoglutamic Acid
An amino acid building block
-
GC15731
L002
L002 es un inhibidor de la acetiltransferasa p300 (KAT3B) potente, permeable a las células, reversible y especÍfico con una IC50 de 1,98 μM.
-
GC65289
Lademirsen
Lademirsen (SAR339375; RG-012) es un oligonucleÓtido modificado quÍmicamente de una sola cadena que se une e inhibe la funciÓn de miR-21.

-
GC15114
LAQ824 (NVP-LAQ824,Dacinostat)
A hydroxamate-based HDAC inhibitor
-
GC12189
LB-100
protein phosphatase 2A(PP2A)inhibitor
-
GC14857
LFM-A13
LFM-A13 es un potente inhibidor de BTK, JAK2, PLK, inhibe BTK, Plx1 y PLK3 recombinantes con IC50 de 2,5 μM, 10 μM y 61 μM; LFM-A13 no muestra efectos sobre JAK1 y JAK3, la quinasa de la familia Src HCK, EGFR e IRK.
-
GC14362
Lin28 1632
Lin28 1632 (compuesto 1632) es un potente antagonista de la interacciÓn Lin28/pre-let-7.
-
GC36464
LIN28 inhibitor LI71
El inhibidor de LIN28 LI71 es un inhibidor de LIN28 potente y permeable a las células, que suprime la oligouridilaciÓn mediada por LIN28 con una IC50 de 7 uM. El inhibidor de LIN28 LI71 se une directamente al dominio de choque frÍo (CSD) para suprimir la actividad de LIN28 contra let-7 en células de leucemia y células madre embrionarias.
-
GC30501
Lin28-let-7a antagonist 1
El antagonista 1 de Lin28-let-7a muestra un claro efecto antagÓnico frente a la interacciÓn Lin28-let-7a con una IC50 de 4,03 μM para la interacciÓn Lin28A-let-7a-1.
-
GC13154
LKB1(AAK1 dual inhibitor)
-
GC33184
LLY-283
LLY-283 es un inhibidor potente, selectivo y oral de la proteÍna arginina metiltransferasa 5 (PRMT5), con una IC50 de 22 nM y una Kd de 6 nM para el complejo PRMT5:MEP50, y muestra actividad antitumoral.