Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
GC43889
I-CBP112 (hydrochloride)
I-CBP112 (chlorhydrate) est un inhibiteur sélectif de CBP/P300 qui lie directement leurs bromodomaines (Kds \u003d 142 et 625 nM, respectivement). I-CBP112 réduit significativement le potentiel d'initiation de la leucémie des cellules de leucémie myéloïde aiguë MLL-AF9 (+) de manière dose-dépendante in vitro et in vivo. I-CBP112 augmente l'activité cytotoxique de l'inhibiteur de bromodomaine BET JQ1 ainsi que de la doxorubicine.
-
GC33042
IACS-9571 (ASIS-P040)
IACS-9571 (ASIS-P040) est un inhibiteur puissant et sélectif de TRIM24 et BRPF1, avec une IC50 de 8 nM pour TRIM24, et des Kd de 31 nM et 14 nM pour TRIM24 et BRPF1, respectivement.
-
GC60925
IACS-9571 hydrochloride
Le chlorhydrate d'IACS-9571 (ASIS-P040) est un inhibiteur puissant et sélectif de TRIM24 et BRPF1, avec une IC50 de 8 nM pour TRIM24 et des Kd de 31 nM et 14 nM pour TRIM24 et BRPF1, respectivement.
-
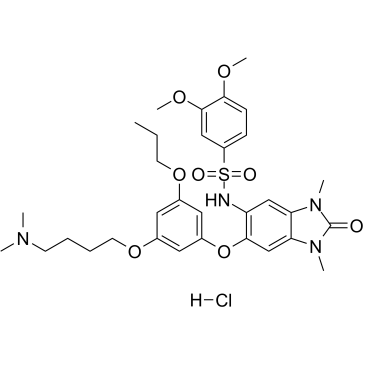
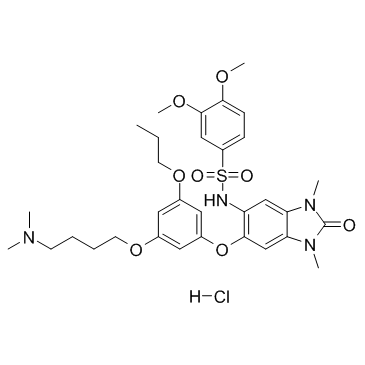
GC34437
IACS-9571 Hydrochloride (ASIS-P040 Hydrochloride)
-
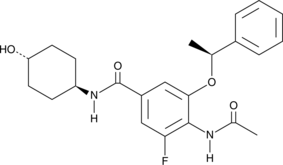
GC48548
iBET-BD2
A BD2 bromodomain inhibitor
-
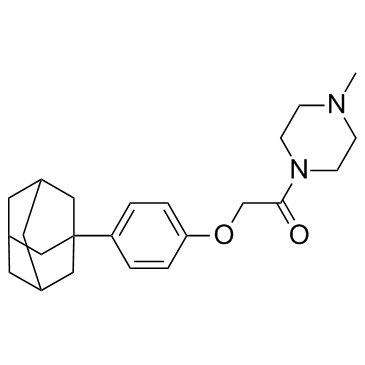
GC32948
IDF-11774
IDF-11774 est un nouvel inhibiteur du facteur α (HIFα)-1 inductible par l'hypoxie avec une IC50 de 3,65μM.
-
GC64108
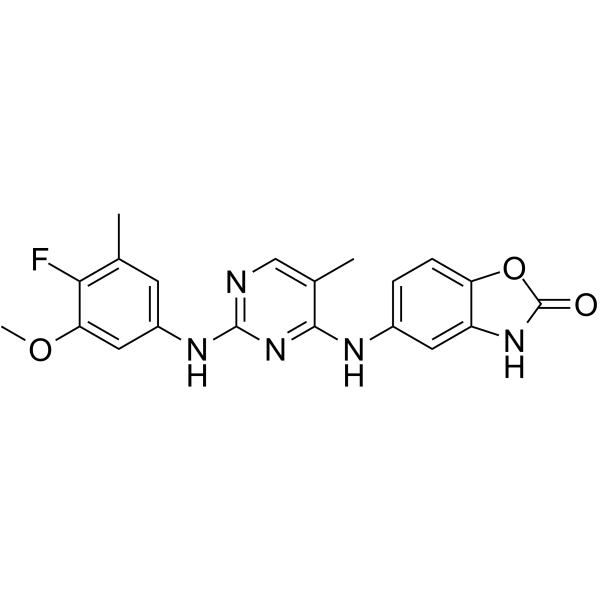
Ifidancitinib
L'ifidancitinib (ATI-50002) est un inhibiteur puissant et sélectif des kinases JAK 1/3.
-
GC47450
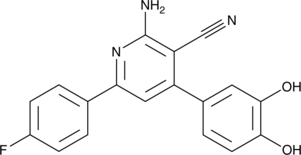
IL-4 Inhibitor
L'inhibiteur d'IL-4 (composé 52) est un inhibiteur d'IL-4, avec une EC50 de 1,81 μM.
-
GC32809
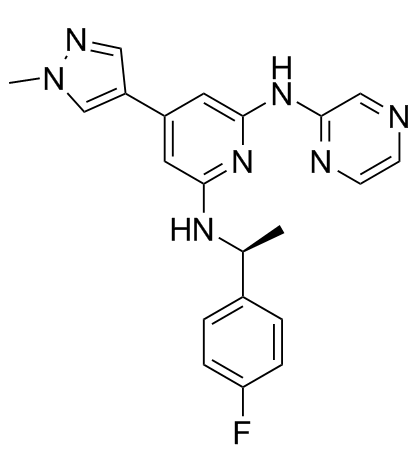
Ilginatinib (NS-018)
L'ilginatinib (NS-018) (NS-018) est un inhibiteur de JAK2 hautement actif et biodisponible par voie orale, avec une IC50 de 0,72 nM, une sélectivité de 46, 54 et 31 fois pour JAK2 par rapport À JAK1 (IC50, 33 nM), JAK3 (IC50, 39 nM) et Tyk2 (IC50, 22 nM).
-
GC33037
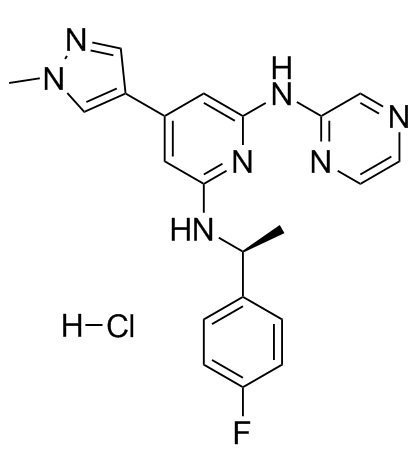
Ilginatinib hydrochloride (NS-018 hydrochloride)
Le chlorhydrate d'ilginatinib (chlorhydrate de NS-018) (chlorhydrate de NS-018) est un inhibiteur de JAK2 hautement actif et biodisponible par voie orale, avec une IC50 de 0,72 nM, une sélectivité de 46, 54 et 31 fois pour JAK2 par rapport À JAK1 (IC50, 33 nM), JAK3 (IC50, 39 nM) et Tyk2 (IC50, 22 nM).
-
GC33018
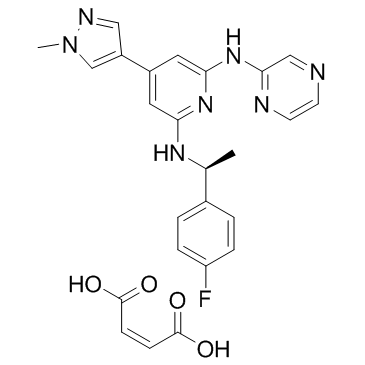
Ilginatinib maleate (NS-018 (maleate))
Le maléate d'ilginatinib (NS-018 (maléate)) (maléate NS-018) est un inhibiteur de JAK2 hautement actif et biodisponible par voie orale, avec une IC50 de 0,72 nM, une sélectivité de 46, 54 et 31 fois pour JAK2 par rapport À JAK1 (IC50 , 33 nM), JAK3 (IC50, 39 nM) et Tyk2 (IC50, 22 nM).
-
GC34159
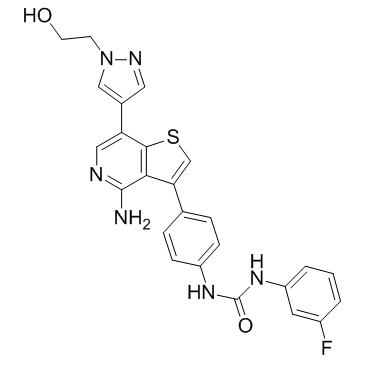
Ilorasertib (ABT-348)
L'ilorasertib (ABT-348) (ABT-348) est un inhibiteur d'aurore puissant, actif par voie orale et compétitif pour l'ATP avec des CI50 de 116, 5, 1 nM pour l'aurore A, l'aurore B, l'aurore C, respectivement. L'ilorasertib (ABT-348) est également un puissant inhibiteur du VEGF et du PDGF. L'ilorasertib (ABT-348) a le potentiel pour la recherche sur la leucémie myéloÏde aiguë (LMA) et le syndrome myélodysplasique (SMD).
-
GC38519
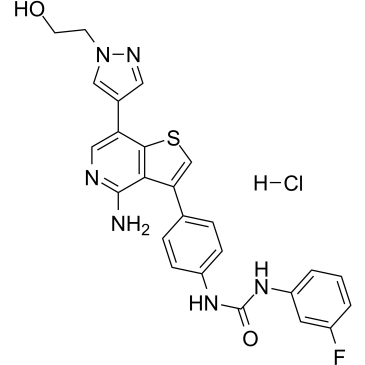
Ilorasertib hydrochloride
Le chlorhydrate d'ilorasertib (ABT-348) est un inhibiteur d'aurore puissant, actif par voie orale et compétitif pour l'ATP avec des CI50 de 116, 5, 1 nM pour l'aurore A, l'aurore B, l'aurore C, respectivement. Le chlorhydrate d'ilorasertib est également un puissant inhibiteur du VEGF et du PDGF. Le chlorhydrate d'ilorasertib a le potentiel pour la recherche sur la leucémie myéloÏde aiguë (LMA) et le syndrome myélodysplasique (SMD).
-
GC63309
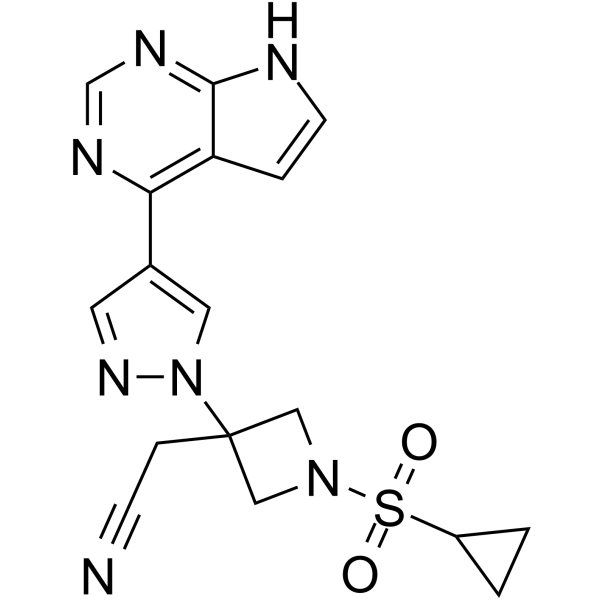
Ilunocitinib
L'ilunocitinib (composé 27) est un inhibiteur de JAK (extrait du brevet WO2009114512A1).
-
GC14755
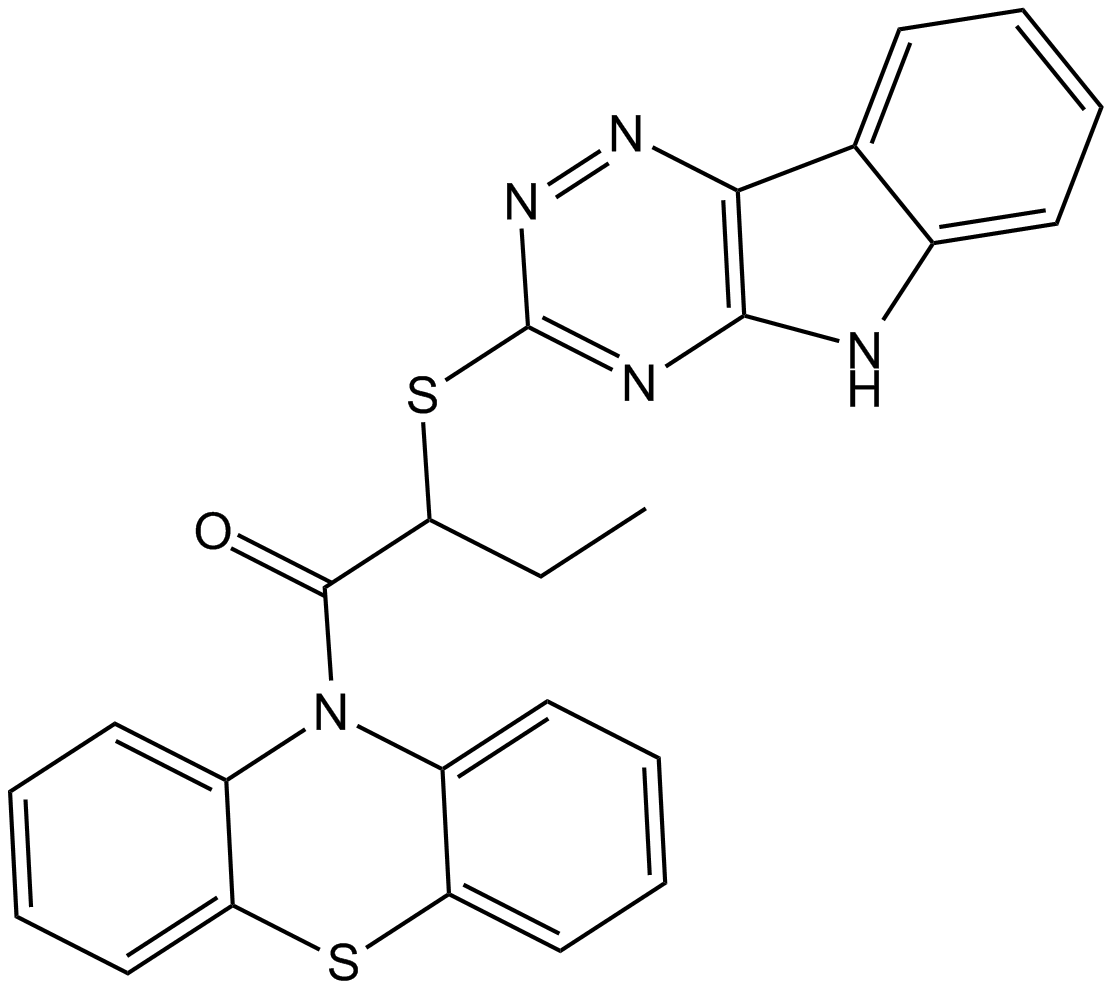
Inauhzin
L'inauhzine est un double inhibiteur SirT1/IMPDH2, et agit comme un activateur p53, utilisé dans la recherche sur le cancer.
-
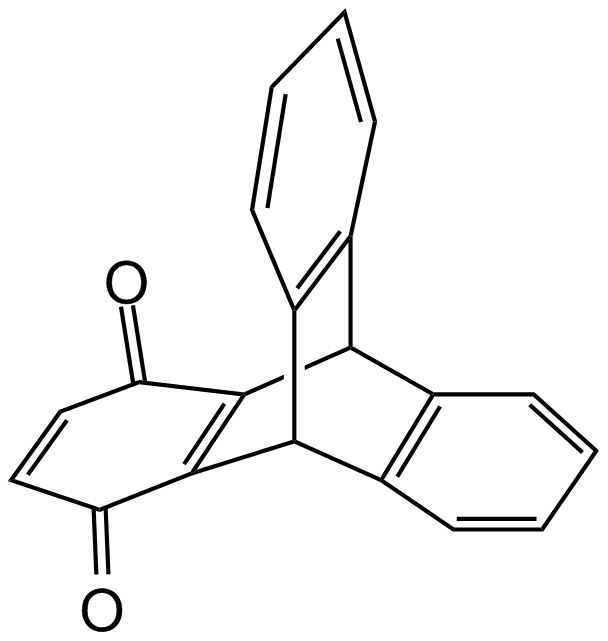
GC16117
INCA-6
A cell-permeant inhibitor of NFAT signaling
-
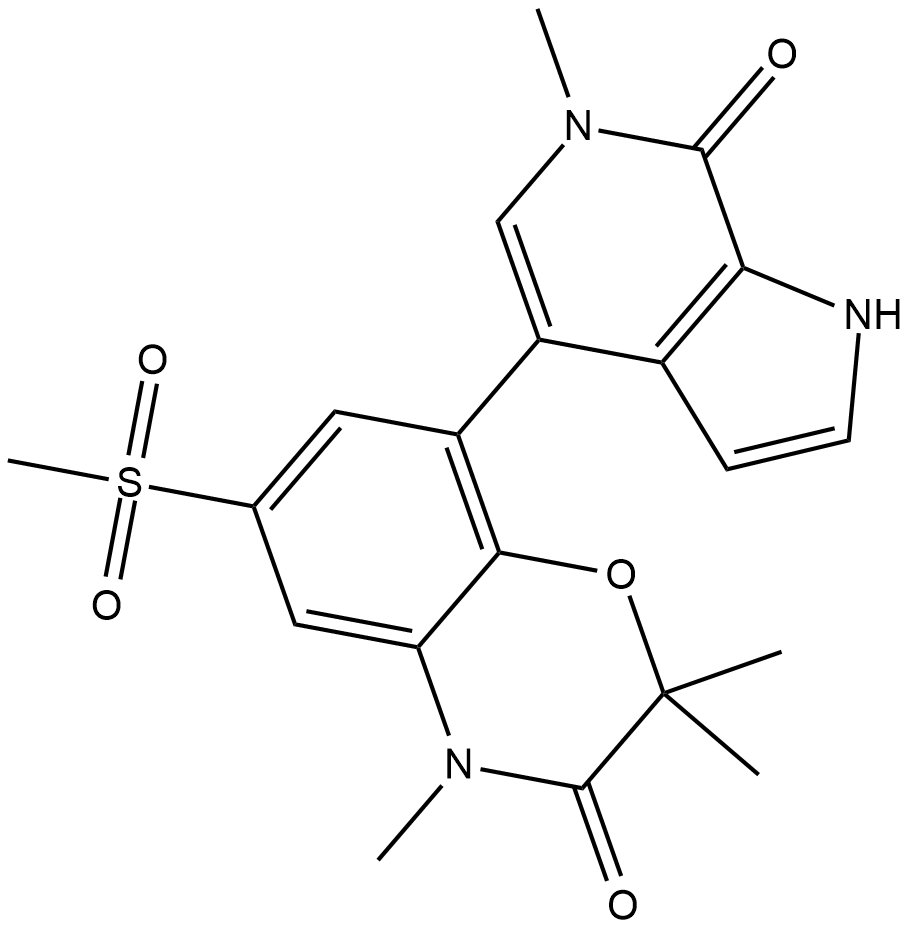
GC19200
INCB-057643
INCB-057643 est un nouvel inhibiteur de BET biodisponible par voie orale.
-
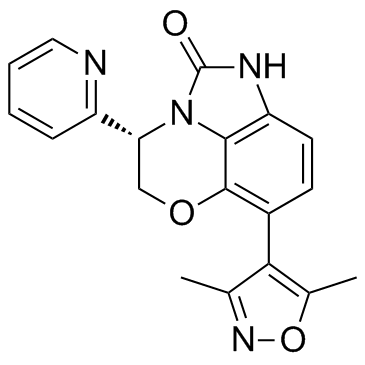
GC33026
INCB054329
INCB054329 est un puissant inhibiteur de BET.
-
GC30644
INCB054329 Racemate
INCB054329 Racemate est un inhibiteur de la protéine BET.
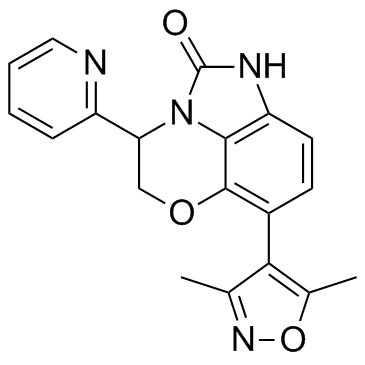
-
GC15353
Iniparib (BSI-201)
A PARP1 inhibitor
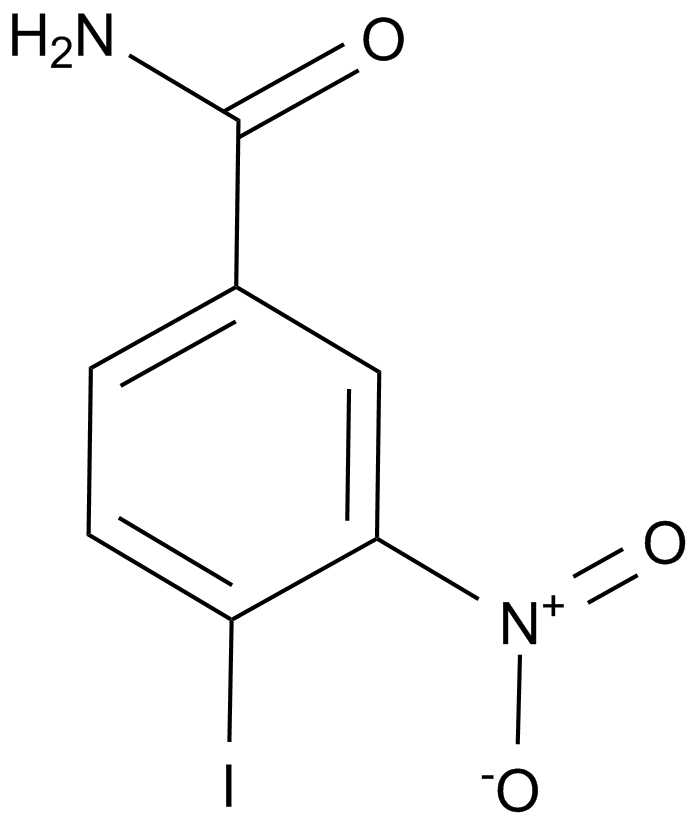
-
GC12496
INO-1001
A PARP inhibitor
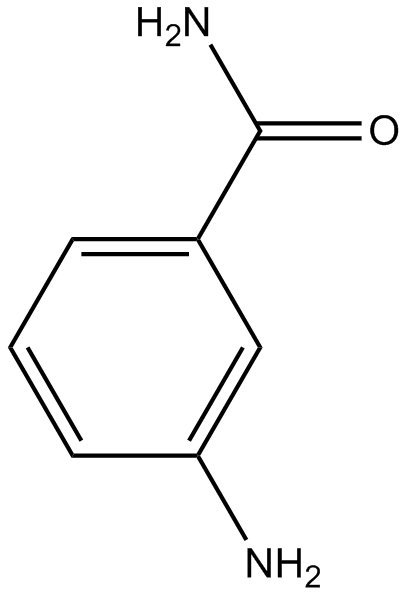
-
GC38385
INO-1001
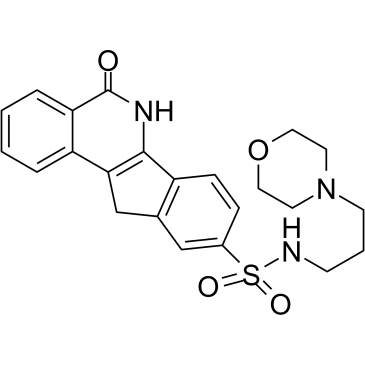
-
GC17754
IOX 1
IOX 1, 5-Carboxy-8-hydroxyquinoléine, est un puissant inhibiteur à large spectre des oxygénases 2OG, y compris les déméthylases JmjC. IOX 1 inhibe KDM4C, KDM4E, KDM2A, KDM3A et KDM6B avec des valeurs IC50 de 0,6 μM, 2,3 μM, 1,8 μM, 0,1 μM et 1,4 μM, respectivement. IOX 1 inhibe également ALKBH5.
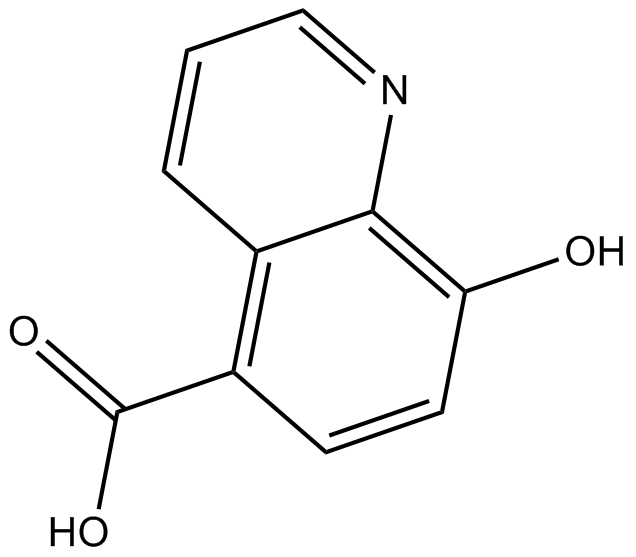
-
GC13018
IOX2(Glycine)
A selective PHD2 inhibitor
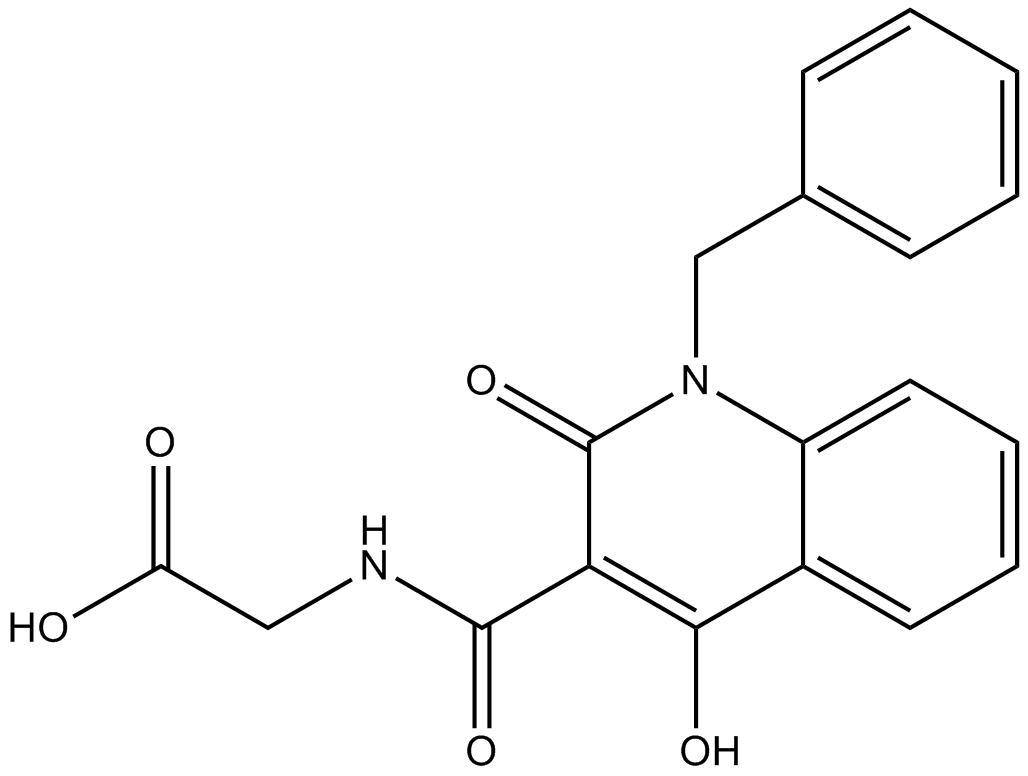
-
GC12255
IOX4
IOX4 est un inhibiteur sélectif de la prolyl-hydroxylase 2 (PHD2) HIF avec une valeur IC50 de 1,6 nM, induit HIFα dans les cellules et chez les souris de type sauvage avec une induction marquée dans le tissu cérébral.
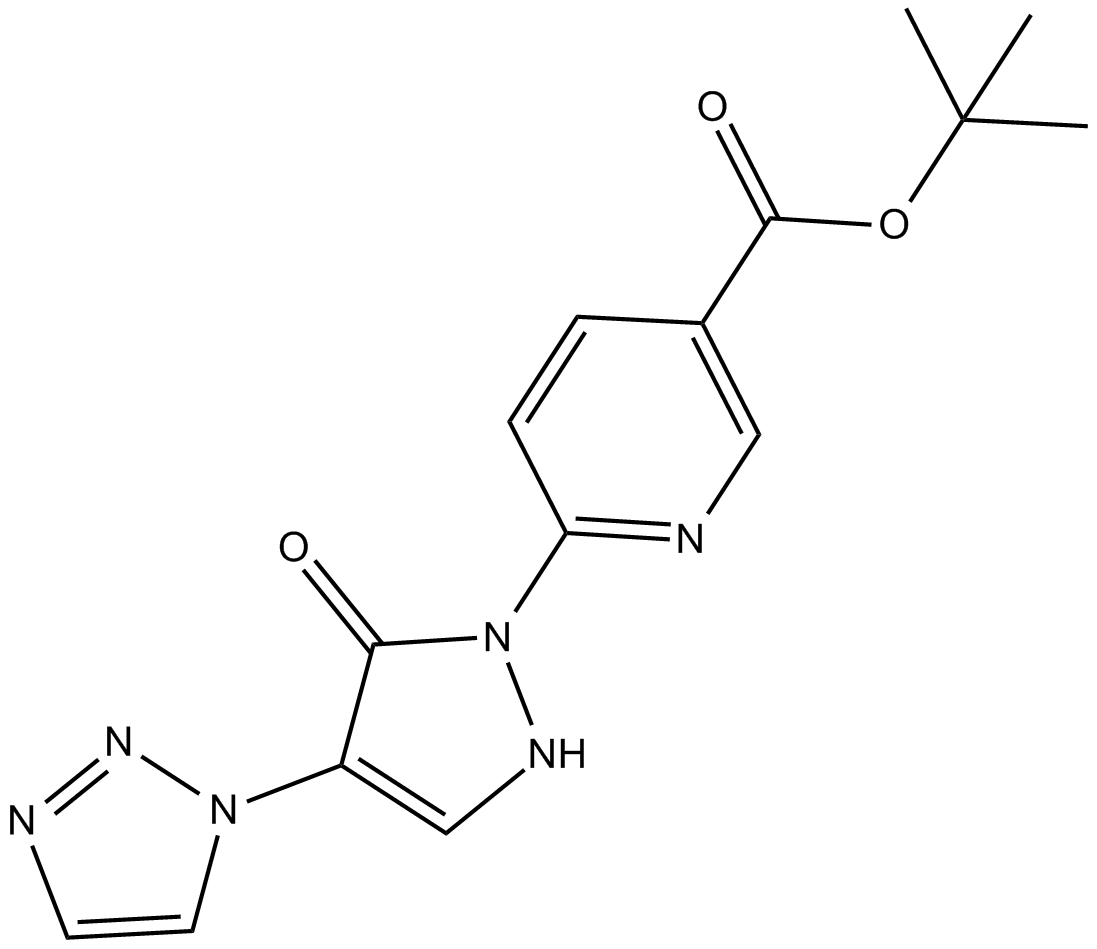
-
GC50721
iP300w
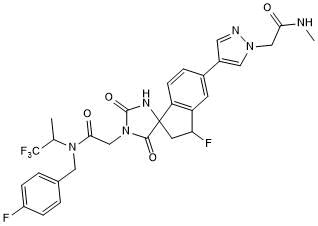
-
GC10727
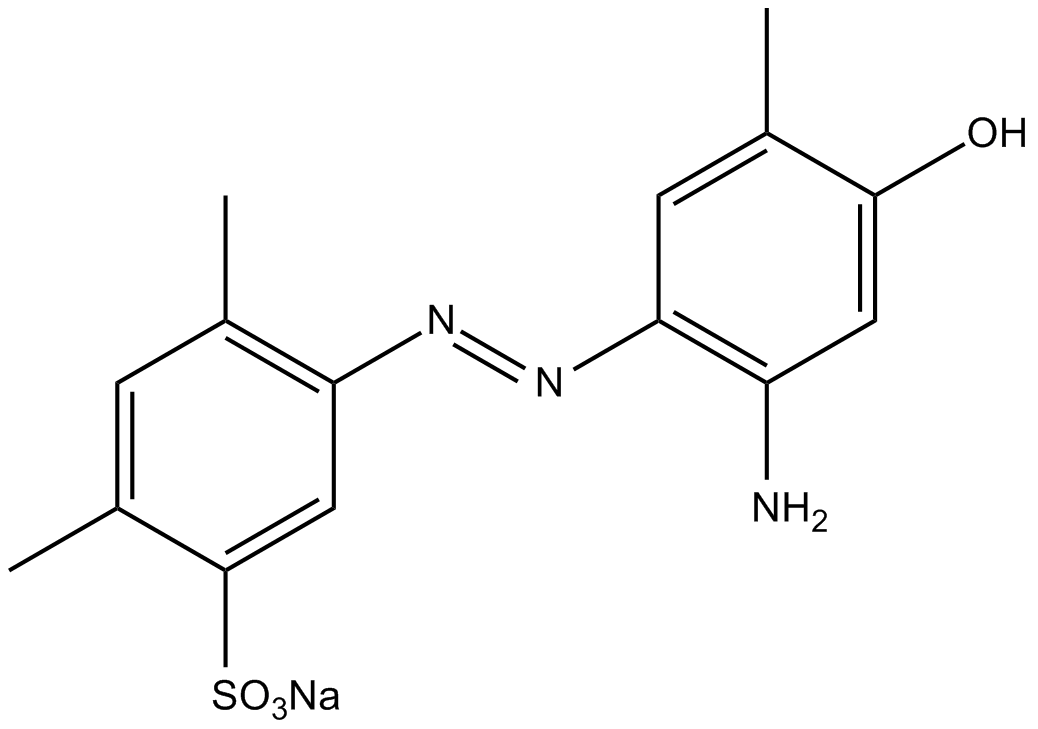
Ischemin sodium salt
CBP bromodomain inhibitor
-
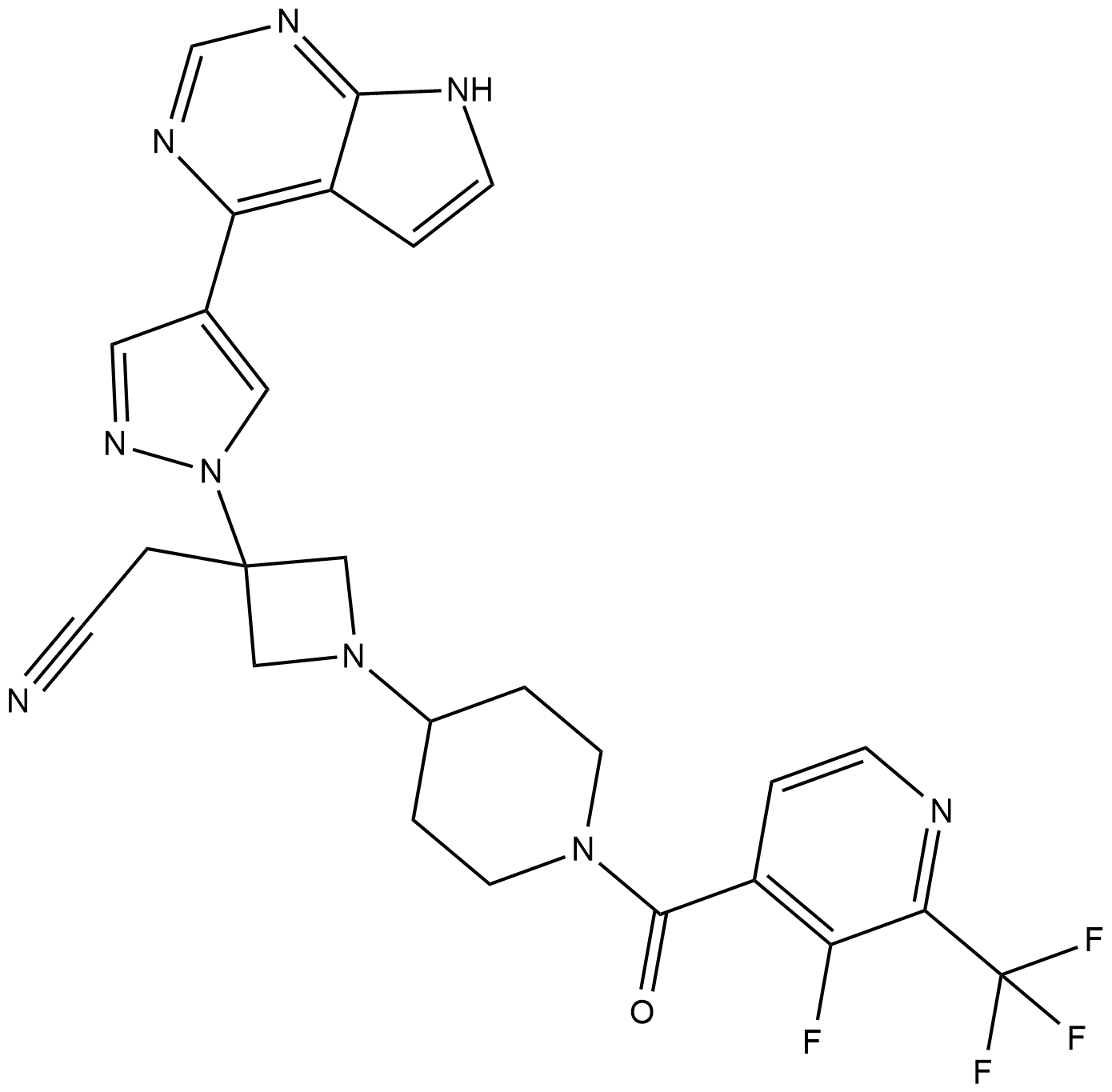
GC19204
Itacitinib
L'itacitinib (INCB039110) est un inhibiteur sélectif et actif par voie orale de JAK1 avec une IC50 de 2 nM pour JAK1 humain. L'itacitinib montre une sélectivité >20 fois pour JAK1 sur JAK2 et >100 fois sur JAK3 et TYK2; L'itacitinib est utilisé dans la recherche sur la myélofibrose.
-
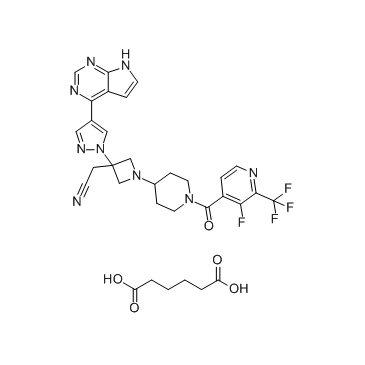
GC36351
Itacitinib adipate
L'adipate d'itacitinib est un inhibiteur de JAK1 biodisponible et sélectif par voie orale dont l'efficacité et l'innocuité ont été testées dans un essai de phase II sur la myélofibrose.
-
GC63416
Itacnosertib
L'itacnosertib (TP-0184) est À la fois un inhibiteur de JAK2, ACVR1 (ALK2) et ALK5 comme décrit dans WO2014151871.
-
GC17836
ITF2357 (Givinostat)
HDAC inhibitor with anti-inflammatory and antineoplastic activities
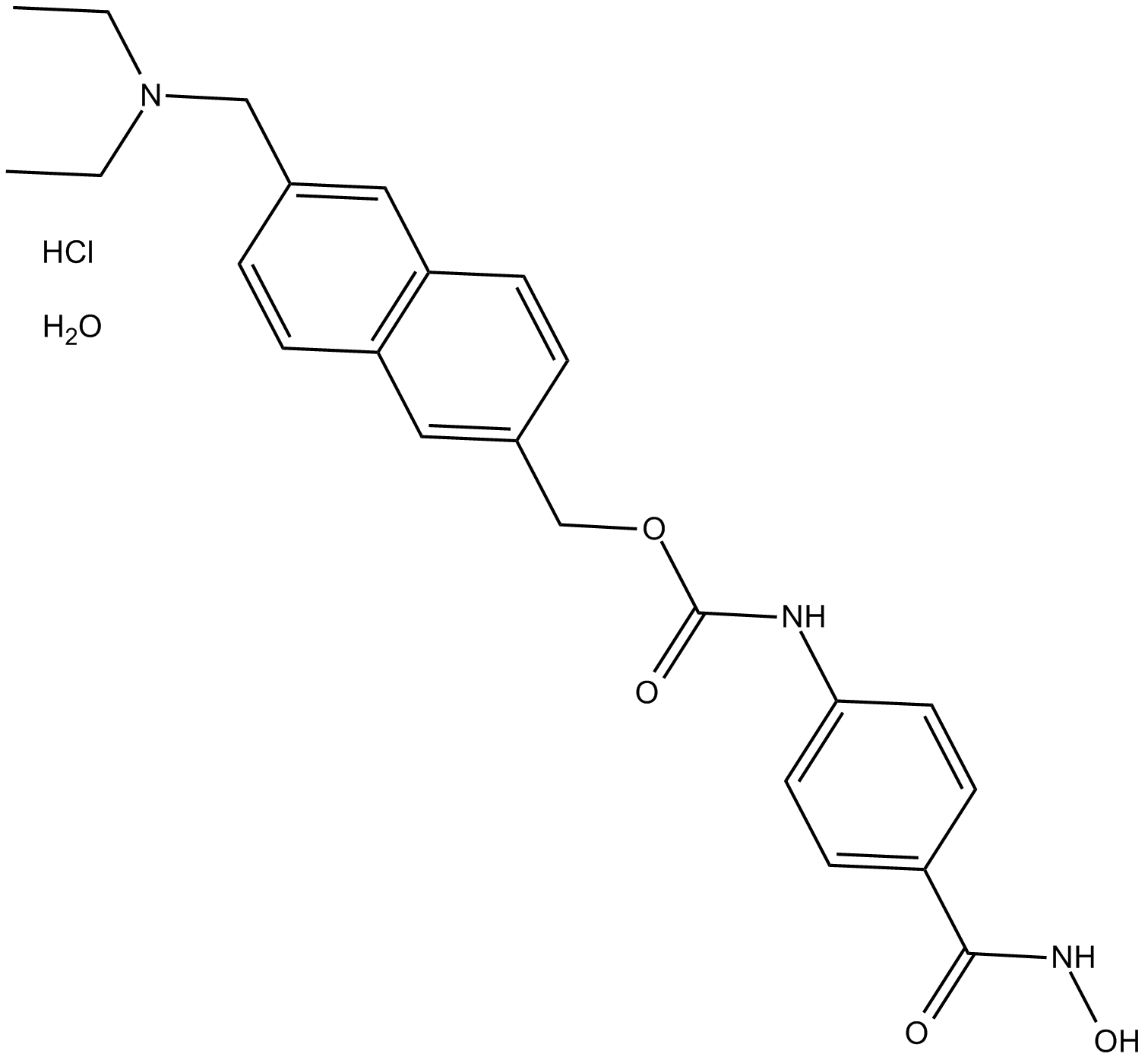
-
GC14597
ITSA-1 (ITSA1)
ITSA-1 (ITSA1) est un activateur de l'histone désacétylase (HDAC) et contrecarre l'arrêt du cycle cellulaire induit par la trichostatine A (TSA), l'acétylation des histones et l'activation de la transcription.
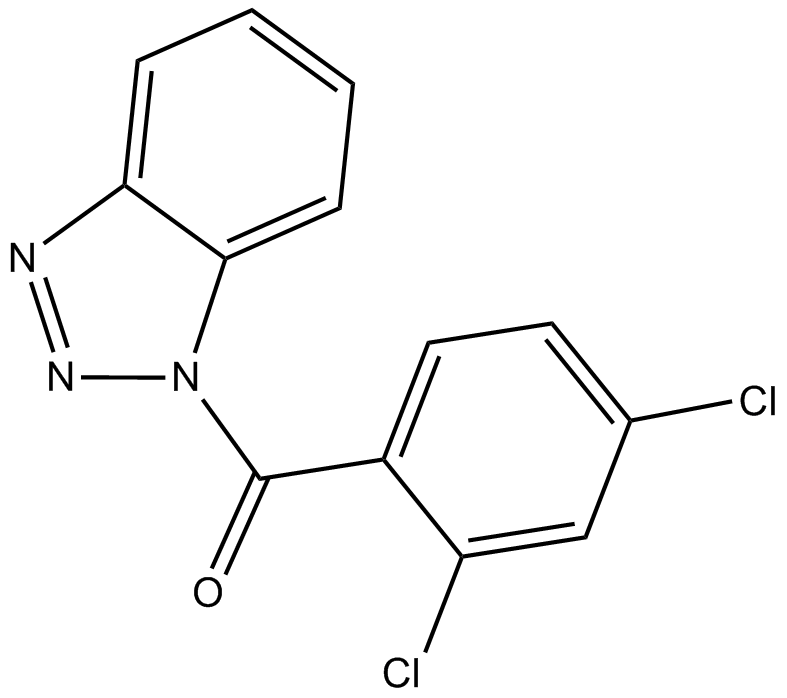
-
GC63703
Ivaltinostat formic
L'ivaltinostat (CG-200745) formique est un puissant inhibiteur pan-HDAC actif par voie orale qui possède la fraction acide hydroxamique pour lier le zinc au fond de la poche catalytique.

-
GC15836
IWP 12
IWP 12 est un puissant inhibiteur du porc-épic (PORCN) et inhibe la signalisation Wnt autonome des cellules avec une IC50 de 15 nM.
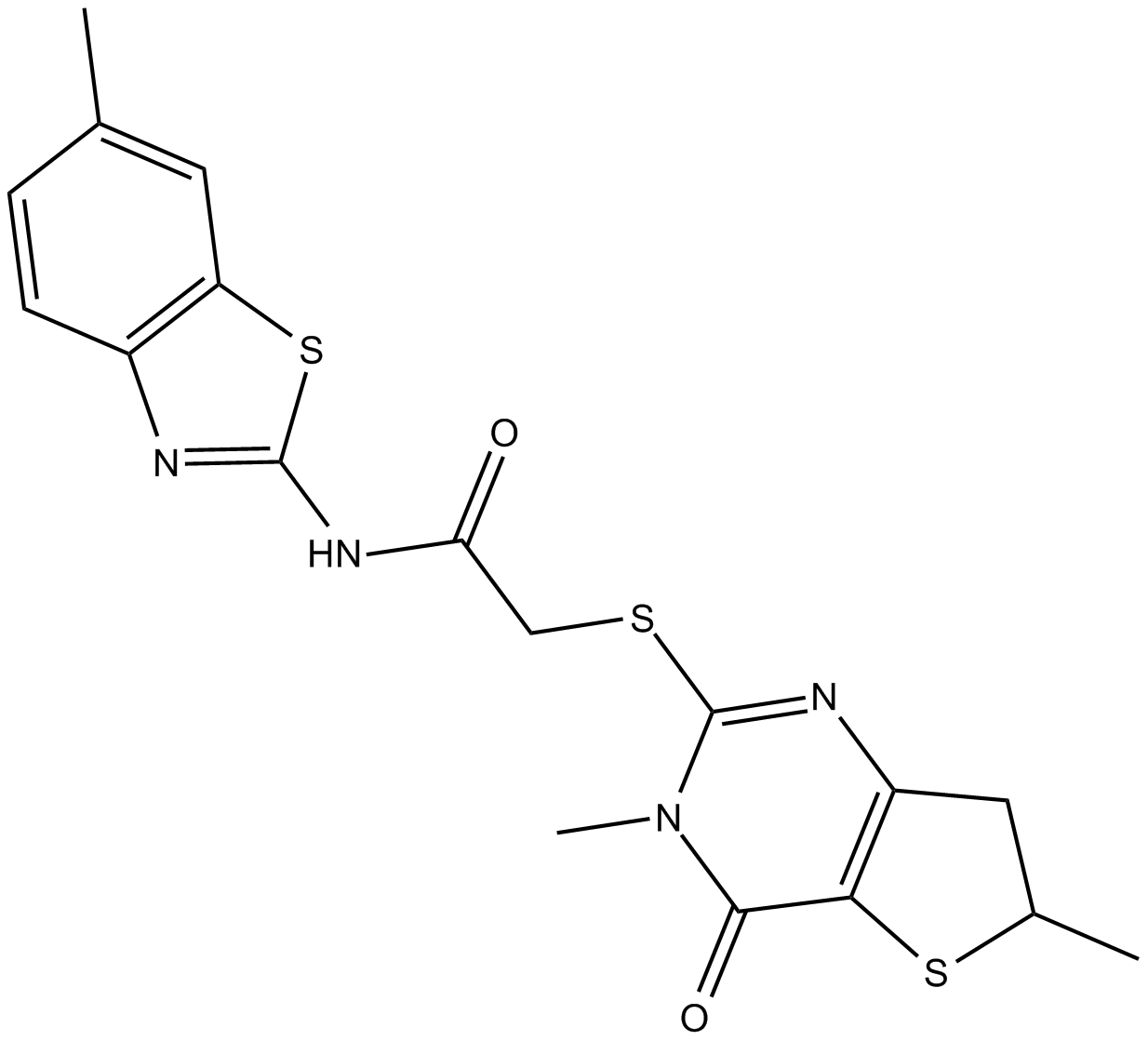
-
GC62313
Izencitinib
L'Izencitinib (TD-1473) est un inhibiteur de JAK actif par voie orale, non sélectif et restreint par l'intestin.
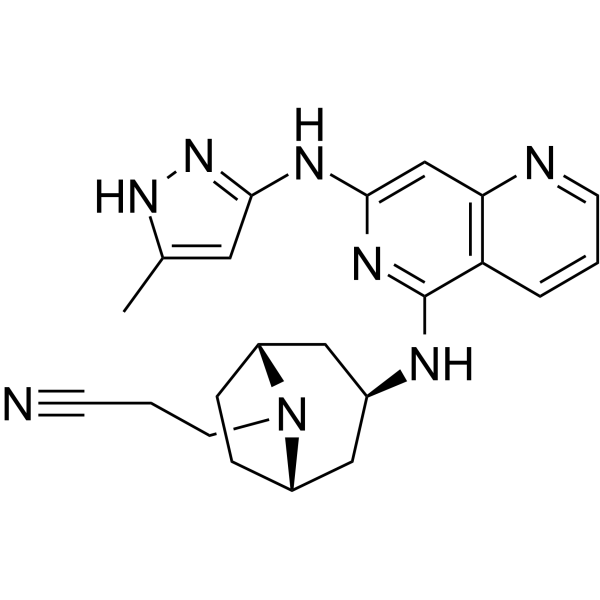
-
GC63828
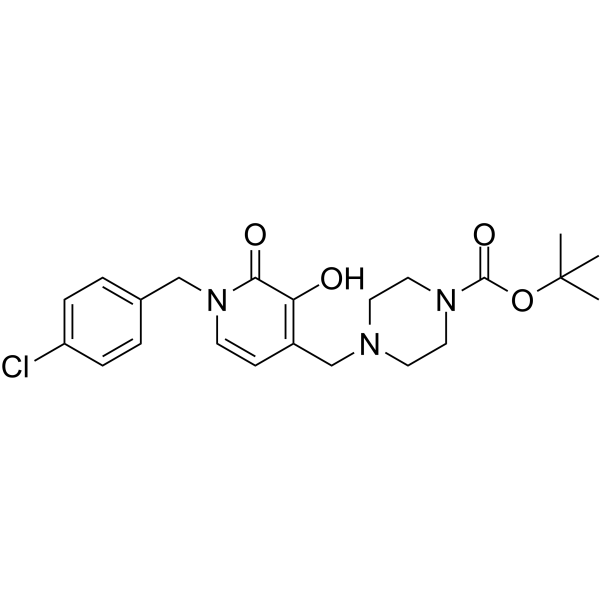
Izilendustat
L'izilendustat est un puissant inhibiteur de la prolyl hydroxylase qui peut stabiliser le facteur 1 alpha inductible par l'hypoxie (HIF-lα) et le facteur 2 inductible par l'hypoxie (HIF-2).
-
GC34629
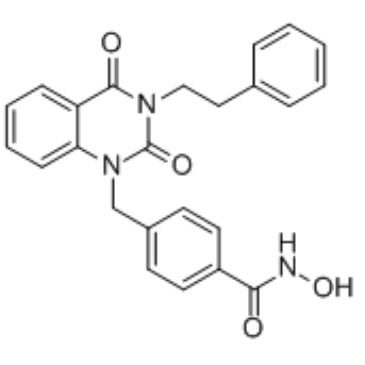
J22352
J22352 est un inhibiteur HDAC6 de type PROTAC (protéolyse-ciblage des chimères) et hautement sélectif avec une valeur IC50 de 4,7 nM. J22352 favorise la dégradation de HDAC6 et induit des effets anticancéreux en inhibant l'autophagie et en déclenchant la réponse immunitaire antitumorale dans les cancers du glioblastome, et en conduisant À la restauration de l'activité antitumorale de l'hÔte en réduisant l'activité immunosuppressive de PD-L1.
-
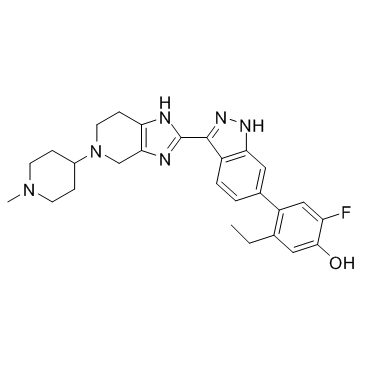
GC31866
JAK inhibitor 1
L'inhibiteur de JAK 1 est un inhibiteur de JAK extrait du brevet US20170121327A1, exemple composé 283.
-
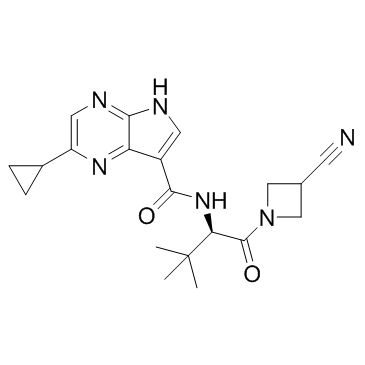
GC31999
JAK-IN-1
JAK-IN-1 est un inhibiteur de JAK1/2/3 avec des IC50 de 0,26, 0,8 et 3,2 nM, respectivement.
-
GC36366
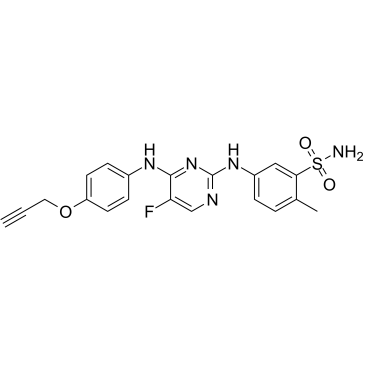
JAK-IN-10
JAK-IN-10 est un inhibiteur de JAK.
-
GC63448
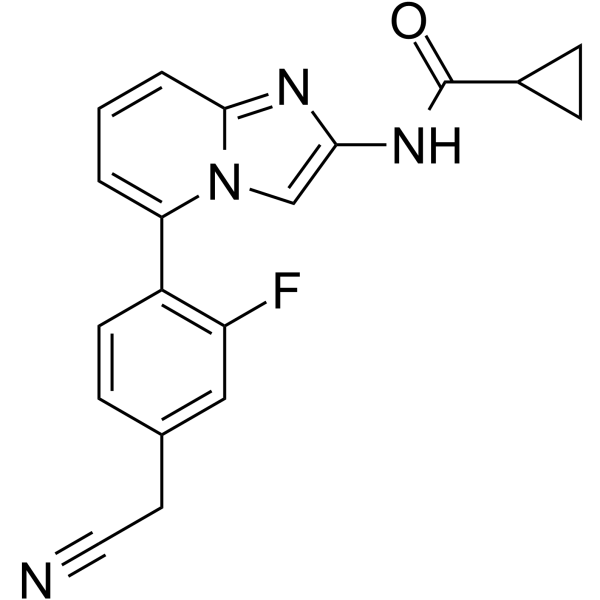
JAK-IN-14
JAK-IN-14 est un inhibiteur puissant et sélectif de JAK1, avec une IC50 <5 μM.
-
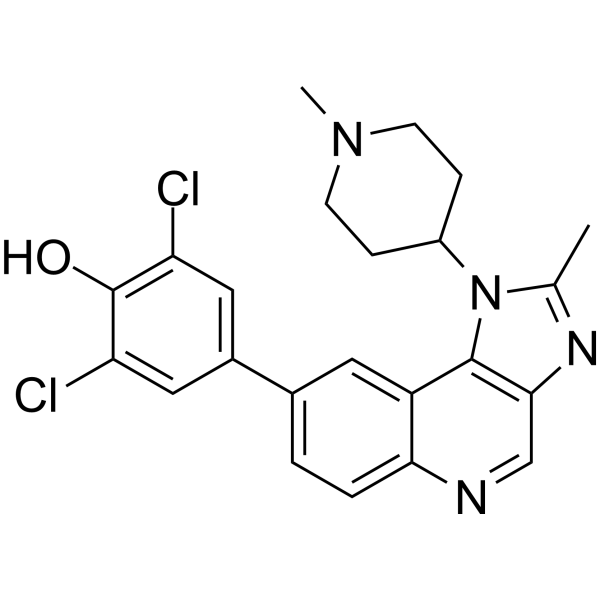
GC68000
JAK-IN-23
-
GC65453
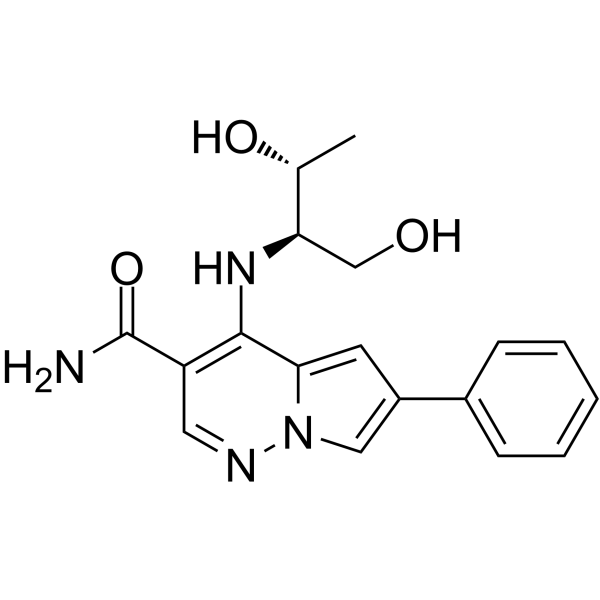
JAK-IN-3
JAK-IN-3 (composé 22) est un puissant inhibiteur de JAK, avec des valeurs IC50 de 3 nM, 5 nM, 34 nM et 70 nM pour JAK3, JAK1, TYK2 et JAK2, respectivement .
-
GC64959
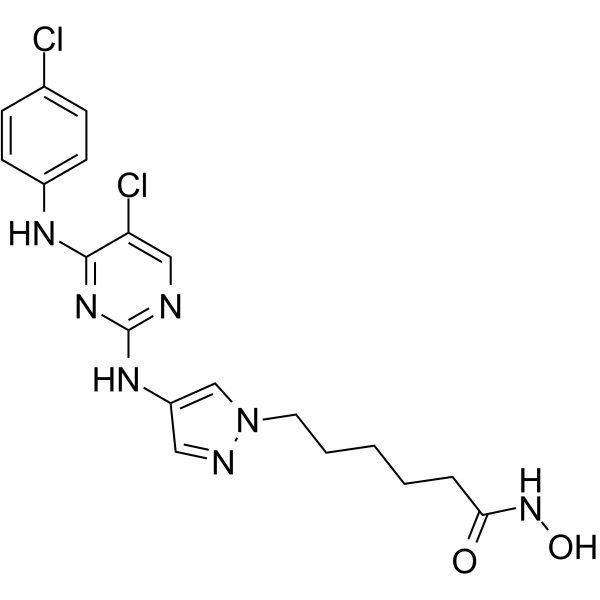
JAK/HDAC-IN-1
JAK/HDAC-IN-1 est un puissant double inhibiteur de JAK2/HDAC, présente des activités antiprolifératives et proapoptotiques dans plusieurs lignées cellulaires hématologiques. JAK/HDAC-IN-1 montre des IC50 de 4 et 2 nM pour JAK2 et HDAC, respectivement.
-
GC33036
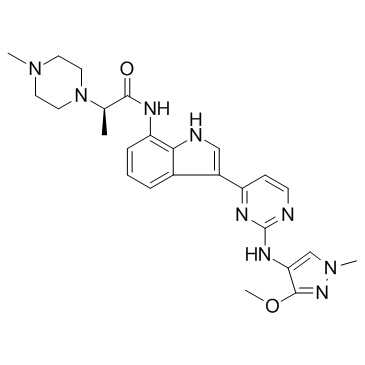
JAK1-IN-3
JAK1-IN-3 (AZD4205) est un inhibiteur sélectif de JAK1, avec une IC50 de 73 nM, inhibe faiblement JAK2 (IC50>14,7 μM) et montre peu d'inhibition sur JAK3 (IC50>30 μM).
-
GC33258
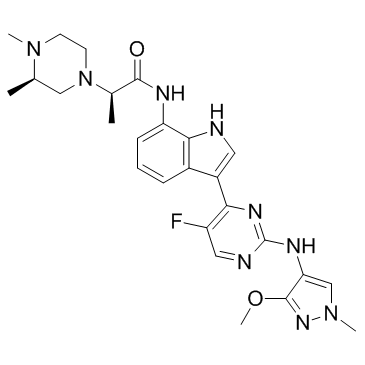
JAK1-IN-4
JAK1-IN-4 est un inhibiteur puissant et sélectif de JAK1, avec des IC50 de 85 nM, 12,8 μM et > 30 μM pour JAK1, JAK2 et JAK3, respectivement. JAK1-IN-4 inhibe la phosphorylation de STAT3 dans les cellules NCI-H 1975 (IC50, 227 nM).
-
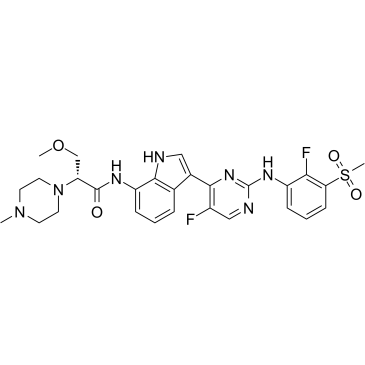
GC36363
JAK1-IN-7
JAK1-IN-7 (JAK1-IN-7) est un inhibiteur de Janus-associated kinase 1 (JAK1) extrait du brevet WO2018134213A1, exemple 63, a un effet anti-inflammatoire.
-
GC63029
JAK1-IN-8
JAK1-IN-8, un puissant inhibiteur de JAK1 (IC50<500 nM), composé 28, extrait du brevet WO2016119700A1.
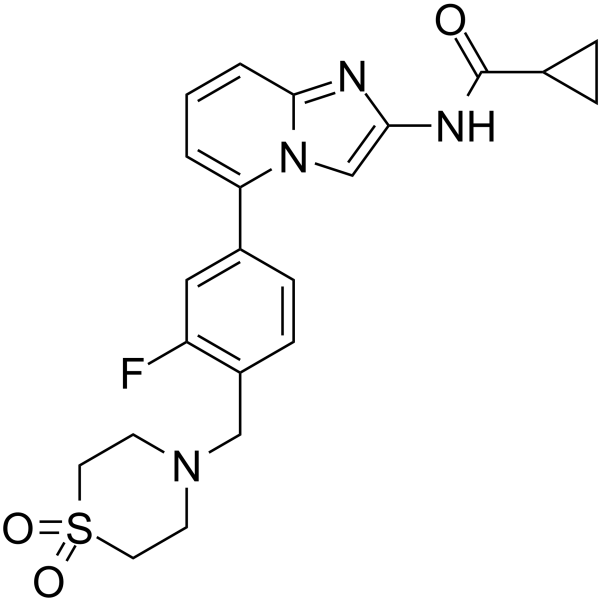
-
GC13826
JAK2 Inhibitor V, Z3
A selective inhibitor of the autophosphorylation of wild type and V617F mutant forms of JAK
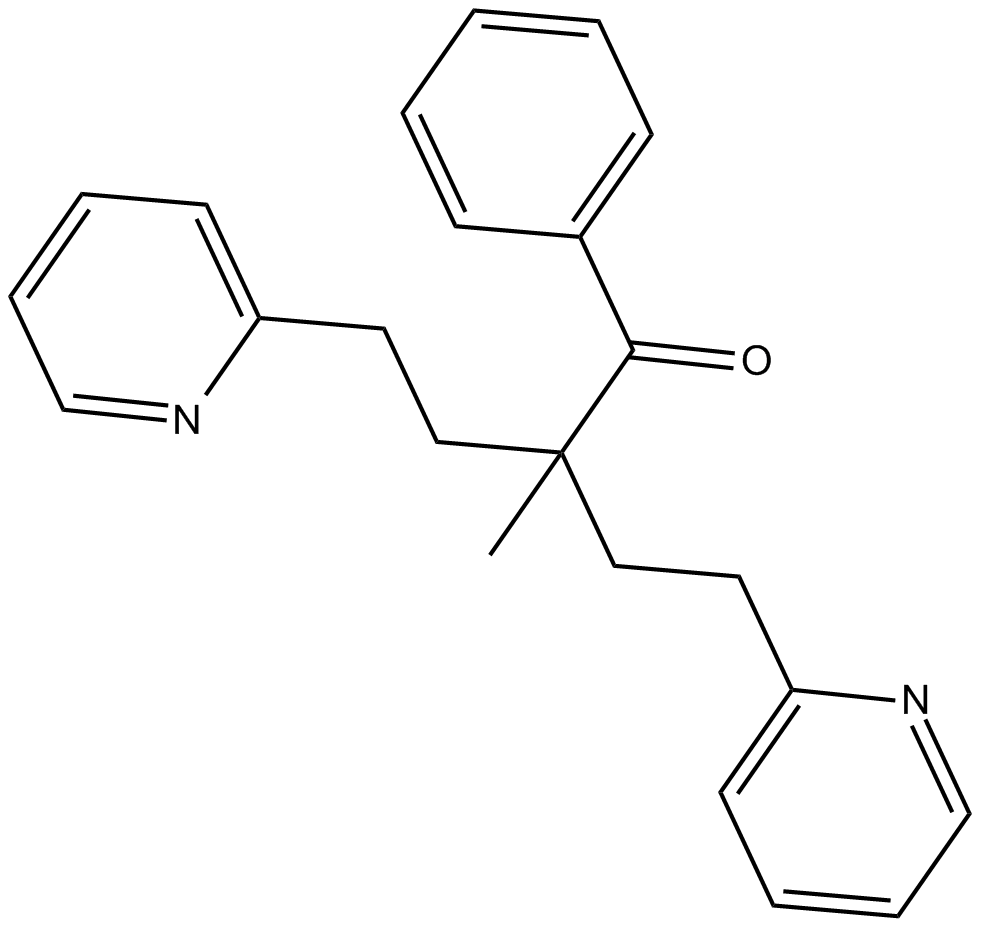
-
GC36364
JAK2-IN-4
JAK2-IN-4 (composé 16h) est un inhibiteur sélectif de JAK2/JAK3, avec des valeurs IC50 de 0,7 nM et 23,2 nM pour JAK2 et JAK3, respectivement.
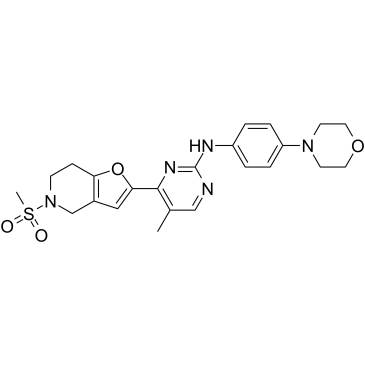
-
GC65405
JAK2-IN-6
JAK2-IN-6, un dérivé d'aminothiazole À plusieurs substitutions, est un inhibiteur puissant et sélectif de JAK2 avec une IC50 de 22,86 μg/mL. JAK2-IN-6 ne montre aucune activité contre JAK1 et JAK3. JAK2-IN-6 a un effet anti-prolifératif contre les cellules cancéreuses.
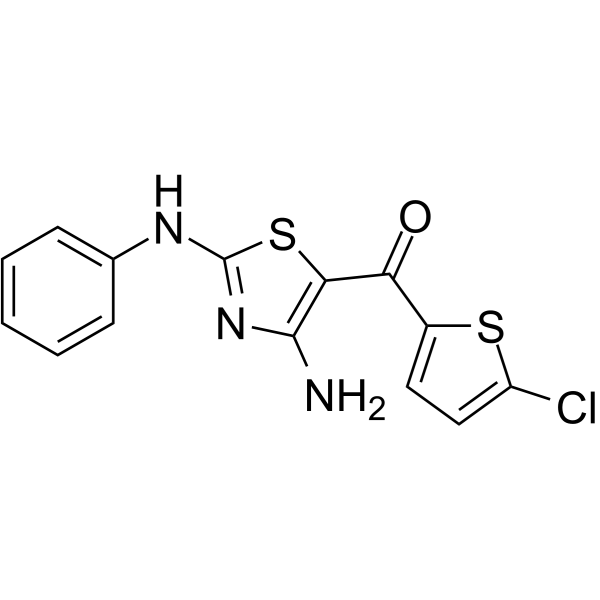
-
GC62500
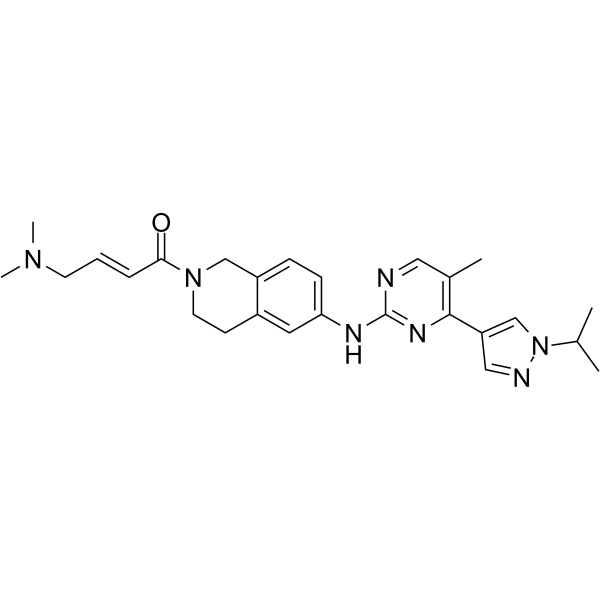
JAK2-IN-7
JAK2-IN-7 est un inhibiteur sélectif de JAK2 avec des IC50 de 3, 11,7 et 41 nM pour les cellules JAK2, SET-2 et Ba/F3V617F, respectivement. JAK2-IN-7 possède une sélectivité > 14 fois supérieure À JAK1, JAK3, FLT3. JAK2-IN-7 stimule l'arrêt du cycle cellulaire en phase G0/G1 et induit une cellapoptose tumorale. Activités antitumorales.
-
GC62665
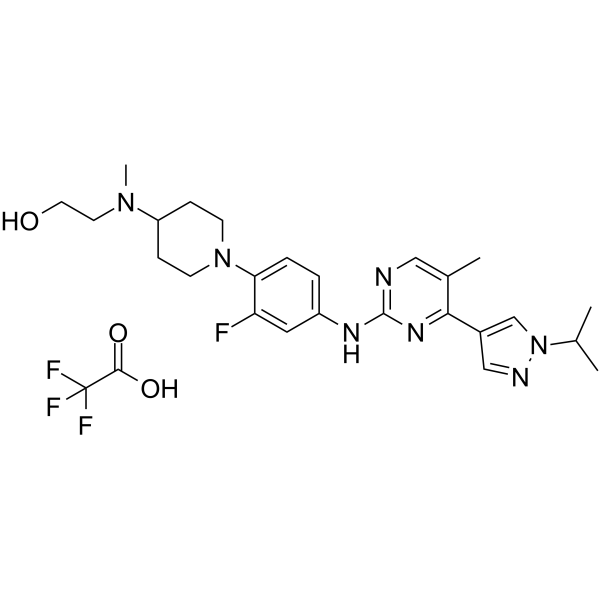
JAK2/FLT3-IN-1 TFA
JAK2/FLT3-IN-1 (TFA) est un double inhibiteur JAK2/FLT3 puissant et actif par voie orale avec des valeurs IC50 de 0,7 nM, 4 nM, 26 nM et 39 nM pour JAK2, FLT3, JAK1 et JAK3, respectivement. JAK2/FLT3-IN-1 (TFA) a une activité anticancéreuse.
-
GC36365
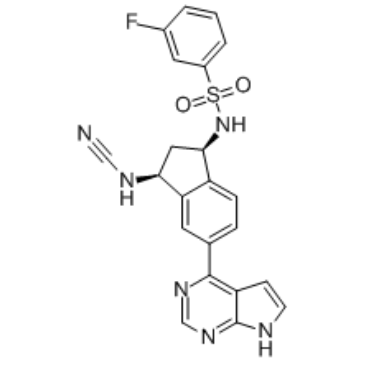
JAK3 covalent inhibitor-1
L'inhibiteur covalent JAK3-1 est un inhibiteur covalent puissant et sélectif de la janus kinase 3 (JAK3) avec une IC50 de 11 nM et présente une sélectivité de 246 fois par rapport aux autres JAK .
-
GC33046
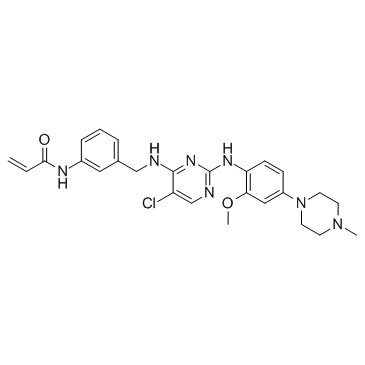
JAK3-IN-1
JAK3-IN-1 est un inhibiteur de JAK3 puissant, sélectif et actif par voie orale avec une IC50 de 4,8 nM. JAK3-IN-1 montre plus de 180 fois plus sélectif pour JAK3 que JAK1 (IC50 de 896 nM) et JAK2 (IC50 de 1050 nM).
-
GC31902
JAK3-IN-6
JAK3-IN-6 est un puissant inhibiteur sélectif et irréversible de la Janus Associated Kinase 3 (JAK3), avec une IC50 de 0,15 nM.
-
GC33382
JAK3-IN-7
JAK3-IN-7 est un inhibiteur puissant et sélectif de JAK3 extrait du brevet WO2011013785A1, a une IC50 <0,01 μM.
-
GC14671
JANEX-1
A selective JAK3 inhibitor
-
GC14686
JFD00244
JFD00244 est un inhibiteur de la sirtuine 2 (SIRT2), À effet anti-tumoral. JFD00244 est également un inhibiteur Nsp-16 contre le SRAS-CoV-2.
-
GC13062
JGB1741
JGB1741 (ILS-JGB-1741) est un inhibiteur puissant et spécifique de l'activité SIRT1 avec une IC50 d'environ 15 μM. JGB1741 est un inhibiteur faible de SIRT2 et SIRT3 avec un IC50>100 μM. JGB1741 augmente les niveaux de p53 acétylé conduisant À l'apoptose médiée par p53 avec modulation du rapport Bax/Bcl2, libération de cytochrome c et clivage PARP. JGB1741 a le potentiel pour la recherche sur le cancer du sein.
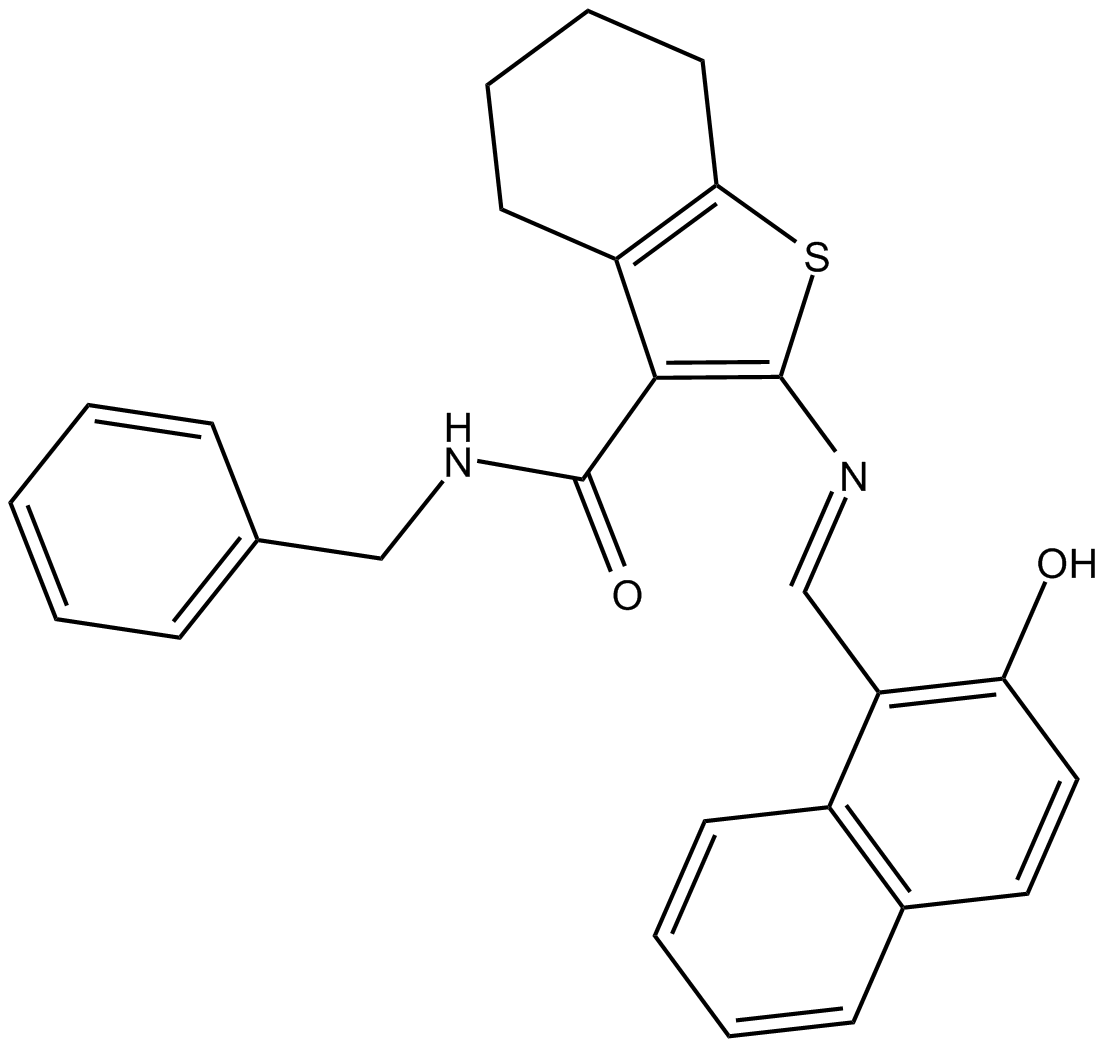
-
GC15603
JIB-04
JIB-04 est un inhibiteur pan-sélectif de l'histone déméthylase Jumonji avec des IC50 de 230, 340, 855, 445, 435, 1100 et 290 nM pour JARID1A, JMJD2E, JMJD3, JMJD2A, JMJD2B, JMJD2C et JMJD2D, respectivement.
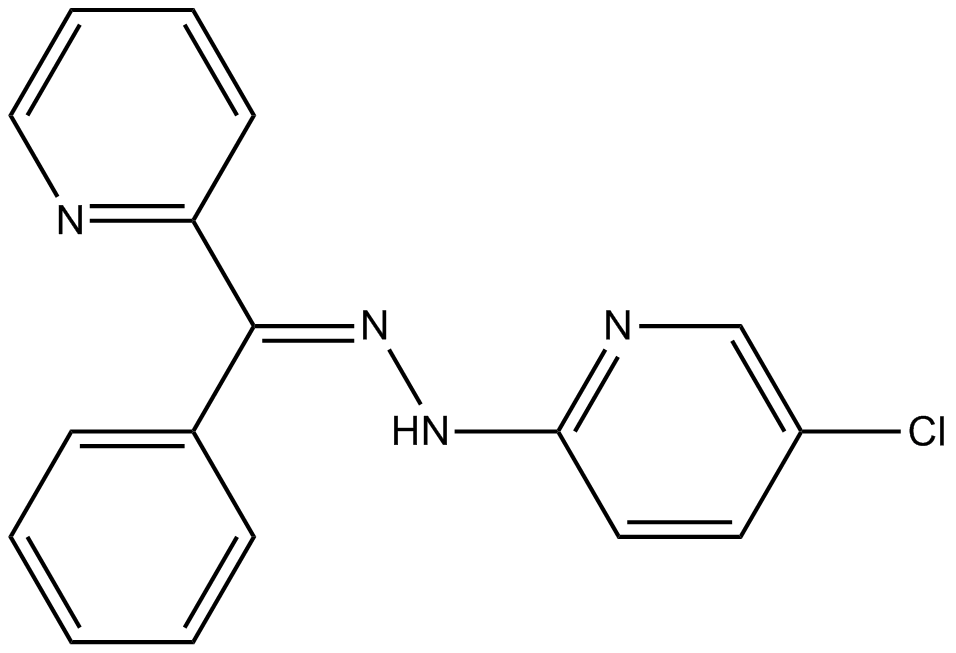
-
GC15476
JNJ-26481585
A pan-HDAC inhibitor
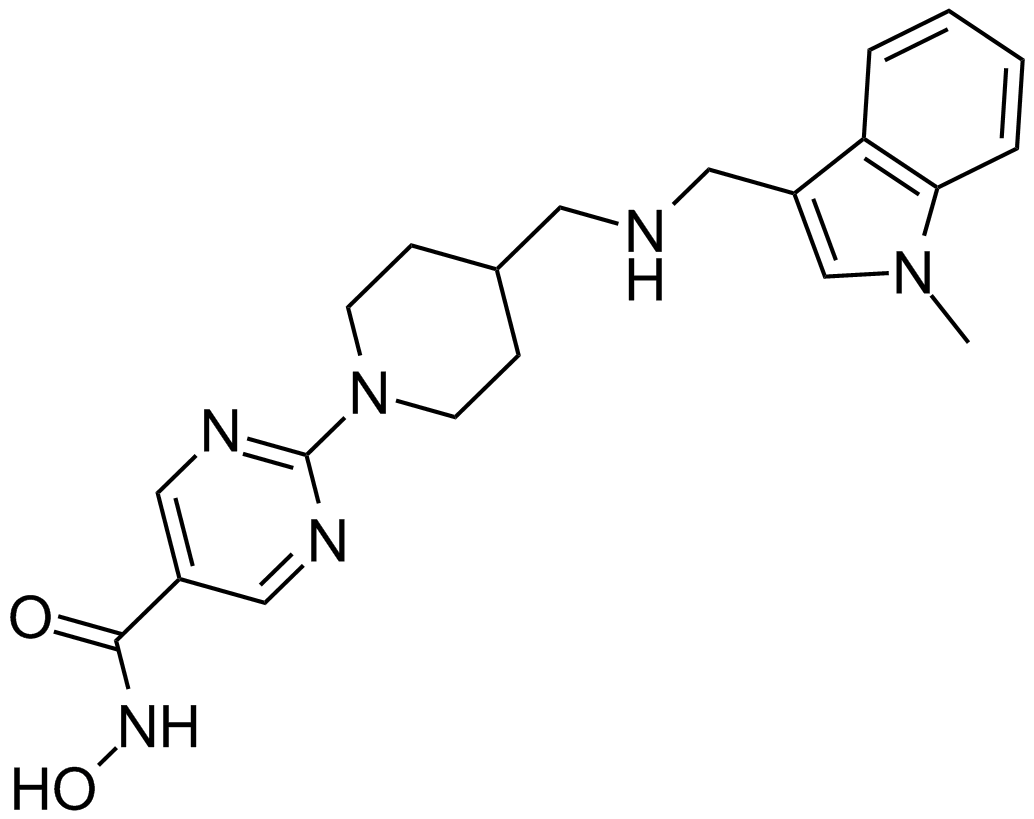
-
GC15379
JNJ-42041935
JNJ-42041935 est un inhibiteur puissant, compétitif et sélectif de la prolyl hydroxylase PHD; inhibe PHD1, PHD2 et PHD3 avec des valeurs de pKi de 7,91 ± 0,04, 7,29 ± 0,05 et 7,65 ± 0,09, respectivement.
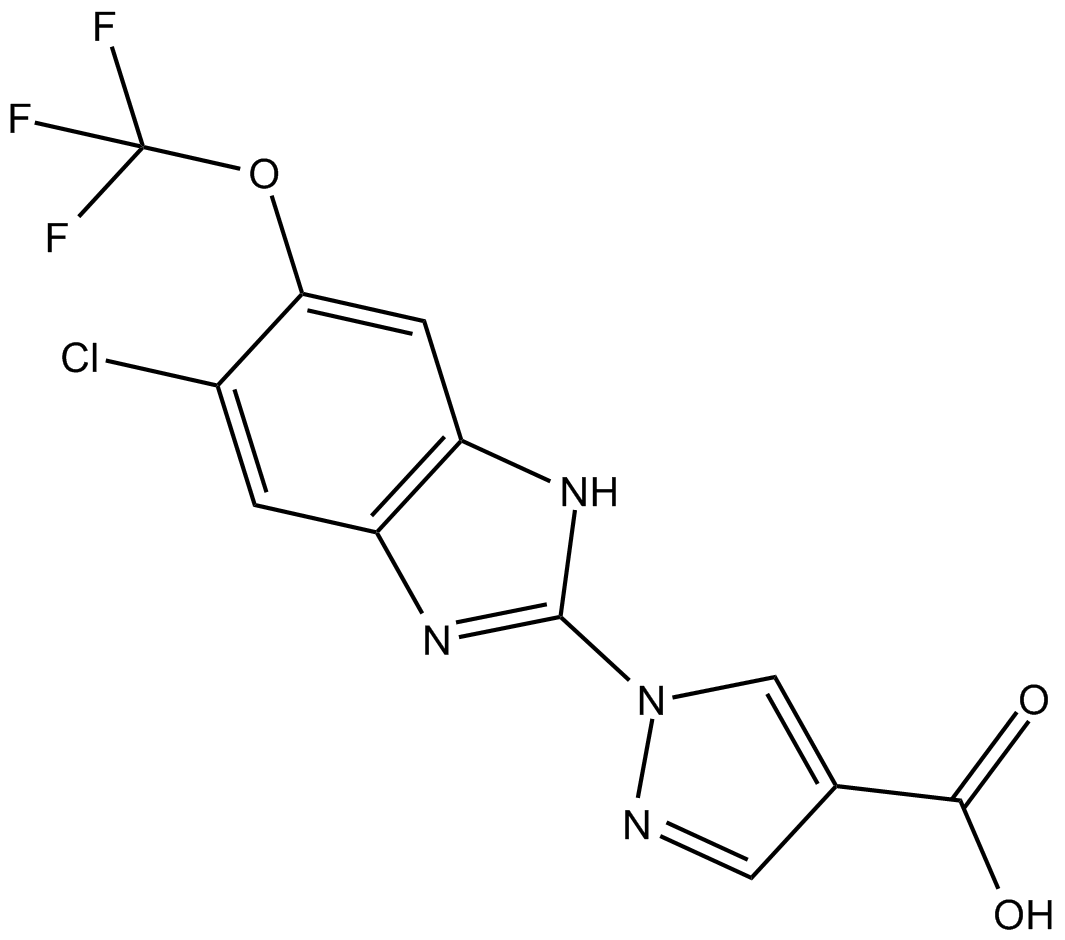
-
GC19465
JNJ-64619178
JNJ-64619178 (Onametostat) est un inhibiteur sélectif, actif par voie orale et pseudo-irréversible de la protéine arginine méthyltransférase 5 (PRMT5) avec une IC50 de 0,14 nM. JNJ-64619178 a une activité puissante dans le cancer du poumon.

-
GC12612
JNJ-7706621
A dual inhibitor of CDKs and Aurora kinases
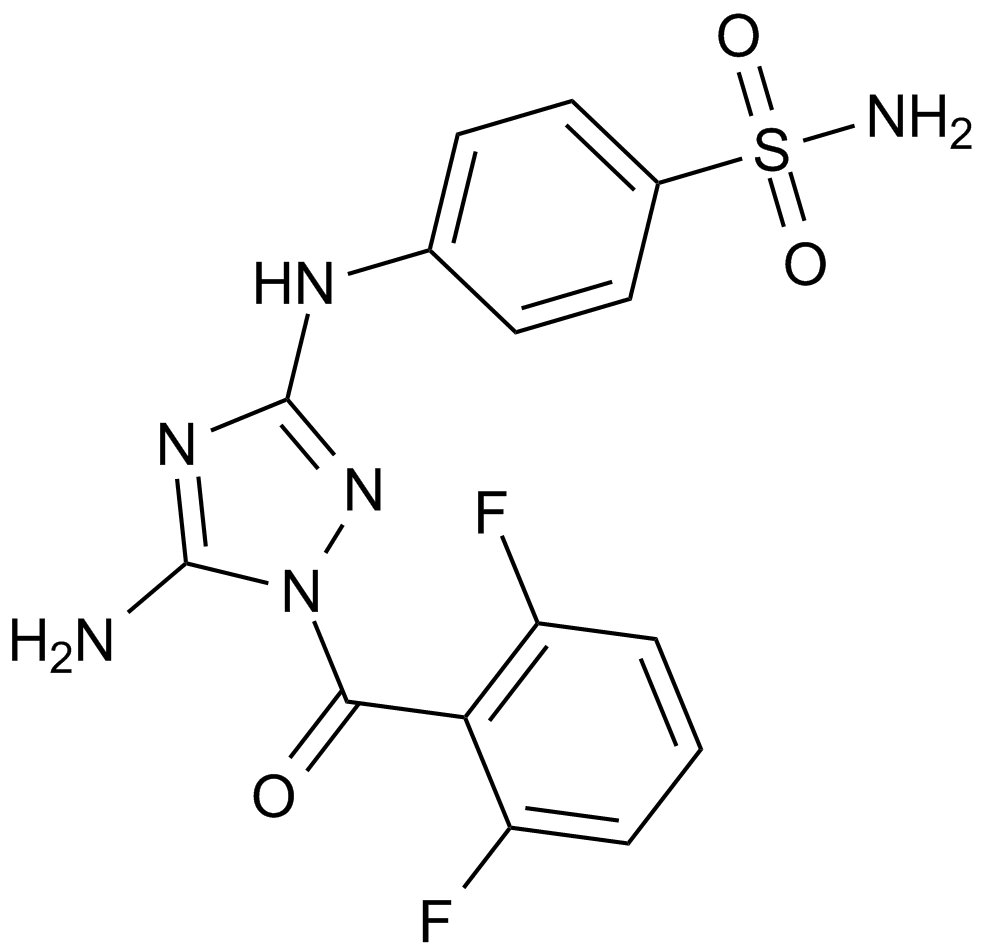
-
GC19210
JQ-1 carboxylic acid
L'acide carboxylique JQ-1, un type de dérivé (+)-JQ-1, est un puissant inhibiteur du bromodomaine BET pour la régulation négative de l'expression de PD-L1 à la surface des cellules tumorales. L'acide carboxylique JQ-1 peut être utilisé comme précurseur pour synthétiser les PROTAC, qui ciblent les bromodomaines BET.
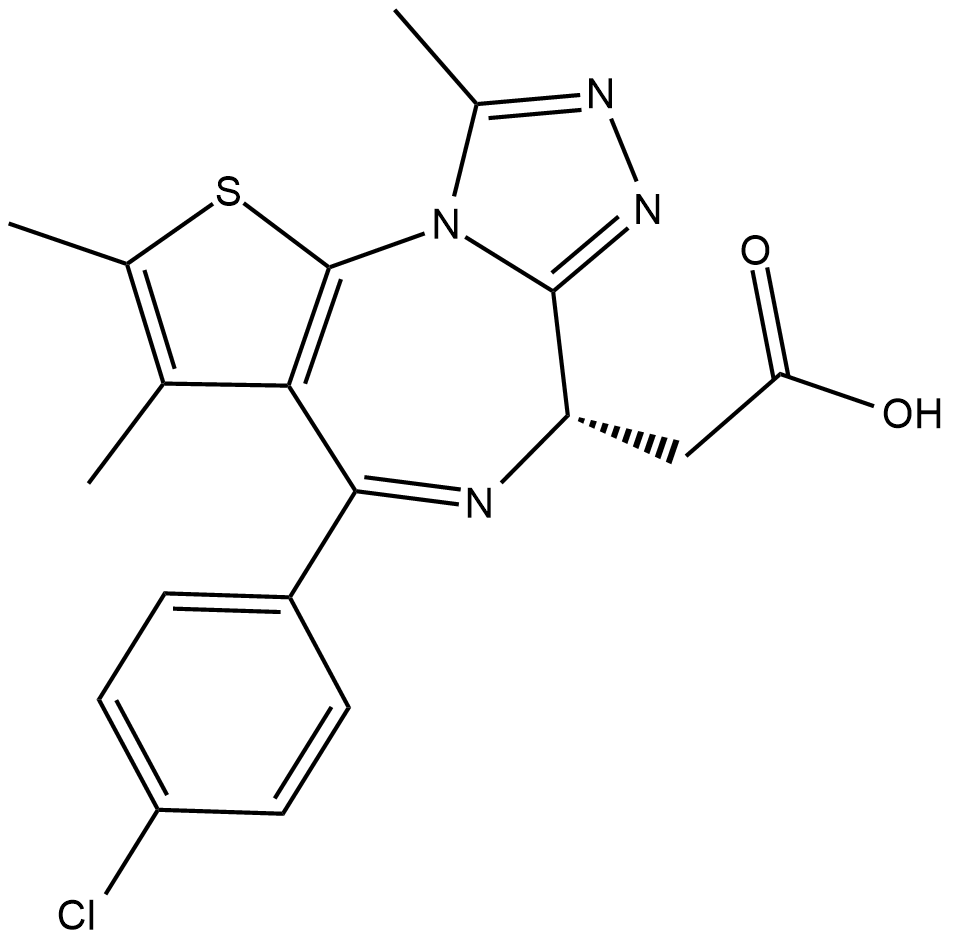
-
GC19211
JQEZ5
JQEZ5 est un inhibiteur puissant et sélectif de la lysine méthyltransférase EZH2. JQEZ5 SAM-inhibition compétitive du complexe répressif polycomb 2 (PRC2) avec une IC50 de 80 nM. JQEZ5 a des effets anti-tumoraux.
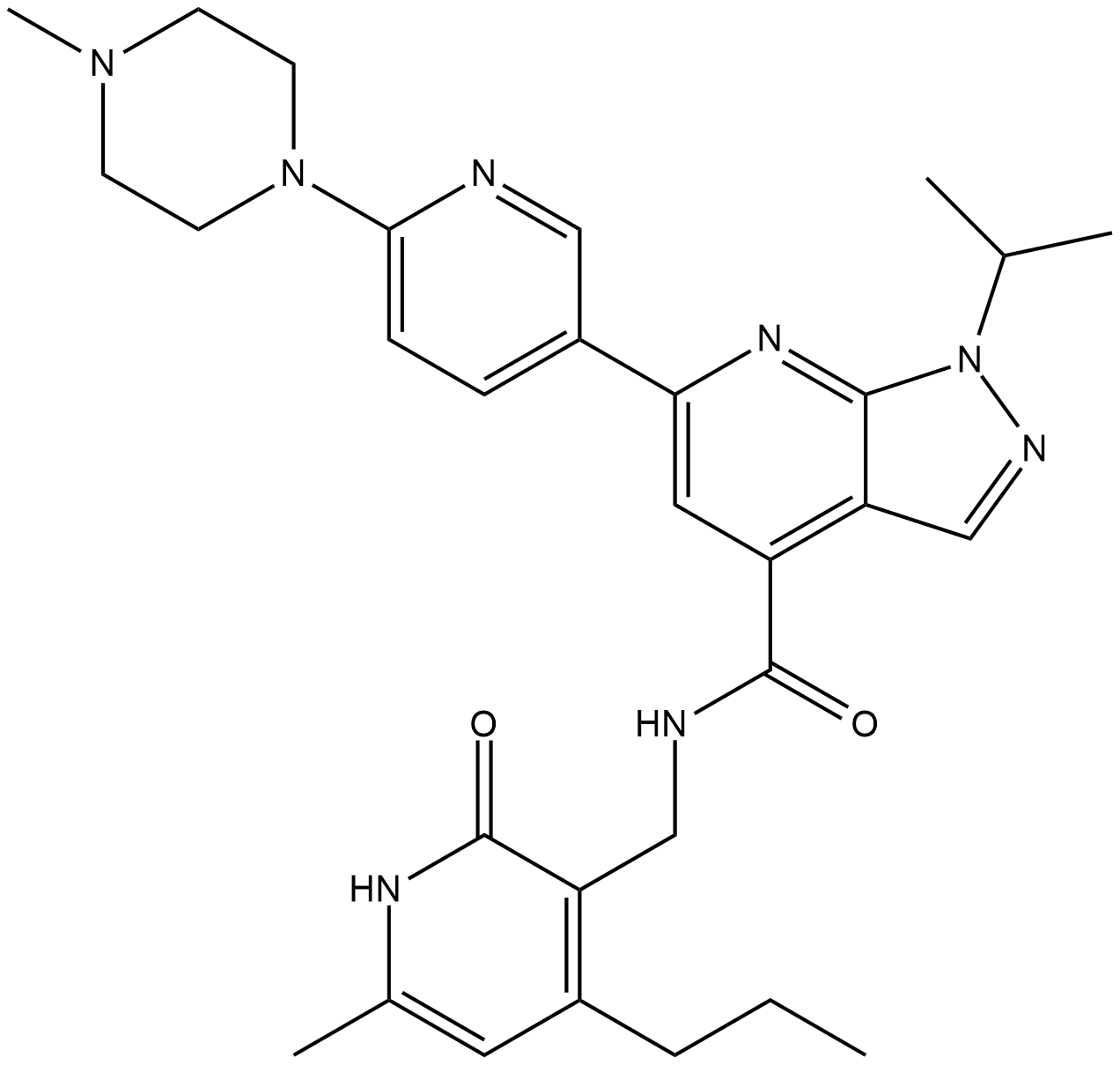
-
GC64841
JQKD82 trihydrochloride
Le trichlorhydrate de JQKD82 (JADA82) est un inhibiteur KDM5 sélectif et perméable aux cellules. Le trichlorhydrate de JQKD82 augmente le H3K4me3 et peut être utilisé pour la recherche du myélome multiple.
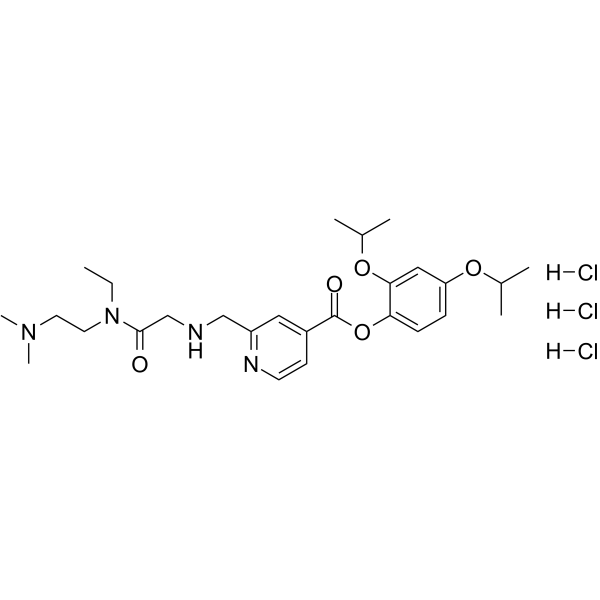
-
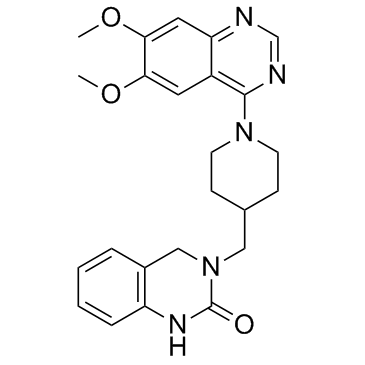
GC34195
K-756
Le K-756 est un inhibiteur direct et sélectif de la tankyrase (TNKS), qui inhibe l'activité d'ADP-ribosylation de TNKS1 et TNKS2 avec des IC50 de 31 et 36 nM, respectivement.
-
GC45489
K-Biotin-W-Histone H2B (108-125) (trifluoroacetate salt)

-
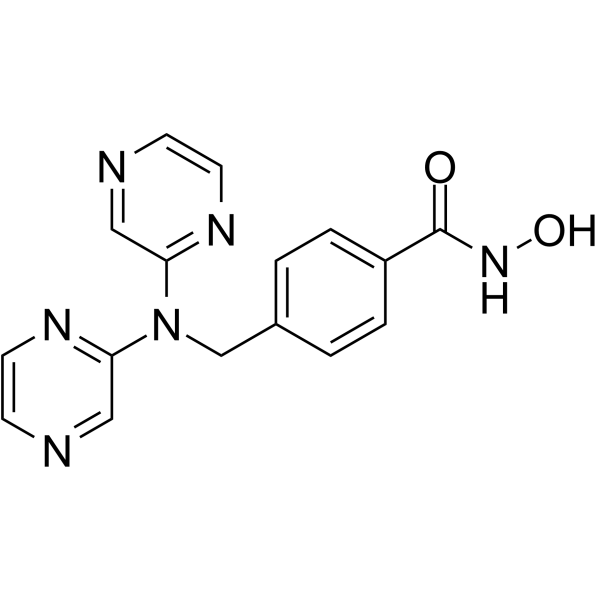
GC62430
KA2507
KA2507 est un inhibiteur HDAC6 puissant, actif par voie orale et sélectif, avec une IC50 de 2,5 nM. KA2507 montre des activités antitumorales et des effets immunomodulateurs dans des modèles précliniques.
-
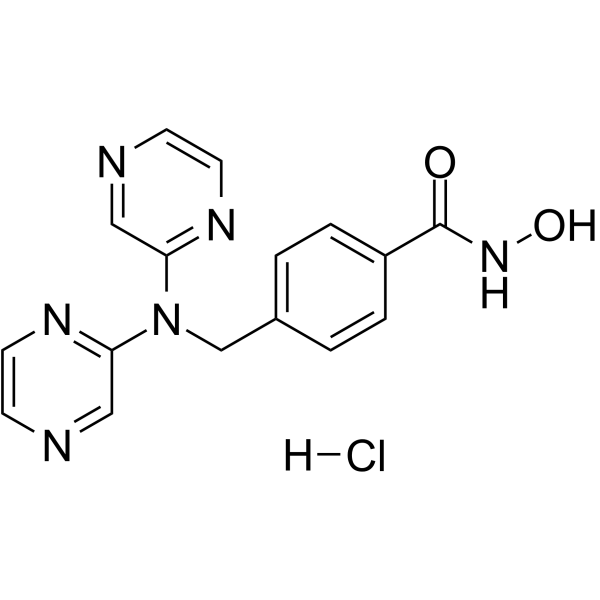
GC62374
KA2507 monohydrochloride
Le chlorhydrate de KA2507 est un inhibiteur puissant et hautement sélectif de HDAC6 (IC50 = 2,5 nM) sans toxicité significative.
-
GC13141
KC7F2
An inhibitor of HIF-1a protein synthesis
-
GC13435
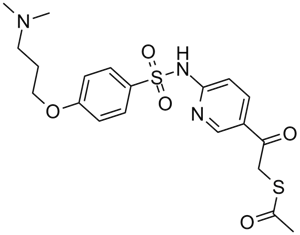
KD 5170
An inhibitor of class I and II HDACs
-
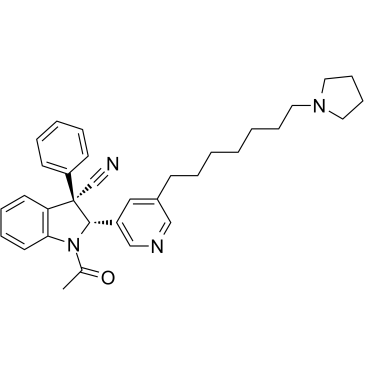
GC36388
KDM2A/7A-IN-1
KDM2A/7A-IN-1 est un inhibiteur sélectif et perméable aux cellules, premier de sa catégorie, des histones lysine déméthylases KDM2A/7A, avec une IC50 de 0,16μM pour KDM2A, présente une sélectivité 75 fois supérieure À celle des autres lysine déméthylases JmjC, et est inactif sur les méthyl transférases et les histone acétyl transférases.
-
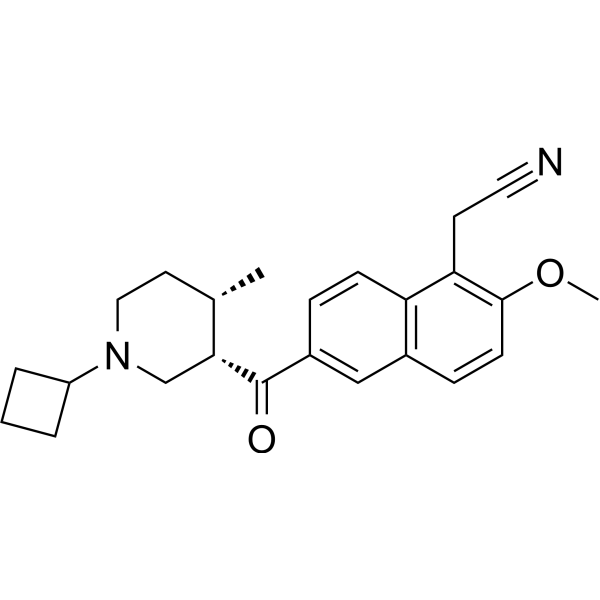
GC69327
KDM2B-IN-4
KDM2B-IN-4 est un inhibiteur de la déméthylation des histones KDM2B. Il peut être utilisé dans la recherche sur le cancer. Pour plus d'informations, veuillez vous référer au composé 182b dans le document de brevet WO2016112284A1.
-
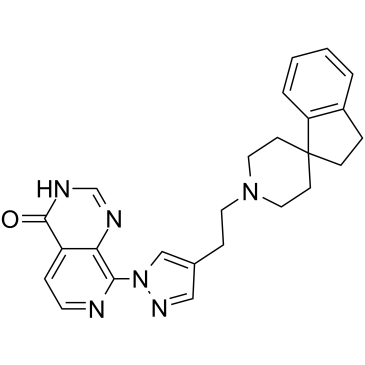
GC36389
KDM4-IN-2
KDM4-IN-2 (composé 19a) est un double inhibiteur puissant et sélectif de KDM4/KDM5 avec un Kis de 4 et 7 nM pour KDM4A et KDM5B, respectivement.
-
GC31887
KDM4D-IN-1
KDM4D-IN-1 est un nouvel inhibiteur de l'histone lysine déméthylase 4D (KDM4D) avec une valeur IC50 de 0,41 ± 0,03 μM.
-
GC32790
KDM5-IN-1
KDM5-IN-1 est un inhibiteur de KDM5 puissant, sélectif et biodisponible par voie orale avec une IC50 de 15,1 nM.
-
GC32842
KDM5A-IN-1
KDM5A-IN-1 est un inhibiteur puissant et biodisponible des pan-histones lysine déméthylases 5 (KDM5) avec des IC50 de 45 nM, 56 nM et 55 nM pour KDM5A, KDM5B et KDM5C, respectivement, et avec une valeur EC50 de 960 nM pour PC9H3K4Me3. KDM5A-IN-1 est significativement moins puissant contre les autres enzymes KDM5B (1A, 2B, 3B, 4C, 6A, 7B).
-
GC38709
KDOAM-25 citrate
Le citrate de KDOAM-25 est un inhibiteur puissant et hautement sélectif des histones lysine déméthylases 5 (KDM5) avec des IC50 de 71 nM, 19 nM, 69 nM, 69 nM pour KDM5A, KDM5B, KDM5C, KDM5D, respectivement. Le citrate de KDOAM-25 augmente la méthylation globale de H3K4 au niveau des sites d'initiation de la transcription et altère la prolifération dans les cellules MM1S du myélome multiple.
-
GC31654
KG-501 (Naphthol AS-E phosphate)
KG-501 (Naphtol AS-E phosphate) est un inhibiteur de CREB, avec une IC50 de 6,89 μM.
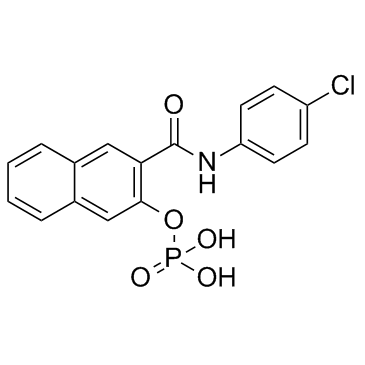
-
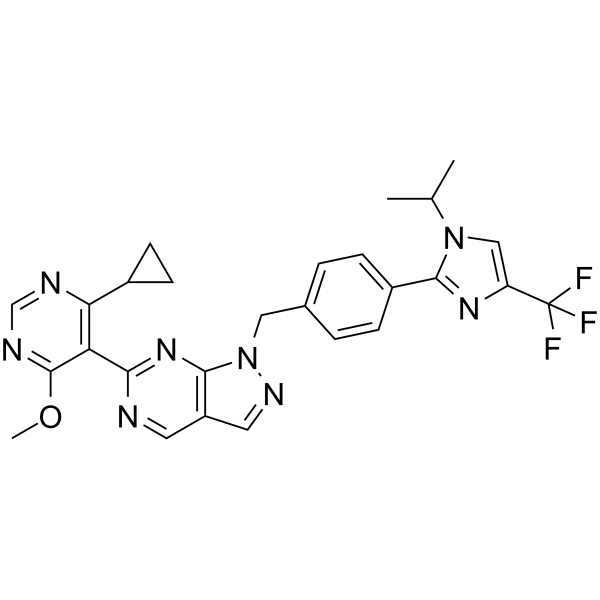
GC65907
KSQ-4279
KSQ-4279 (USP1-IN-1, Formule I) est un inhibiteur de USP1 et PARP (extrait du brevet WO2021163530).
-
GC25552
KT-531
KT-531 (KT531) is a potent, selective HDAC6 inhibitor with IC50 of 8.5 nM, displays 39-fold selectivity over other HDAC isoforms.

-
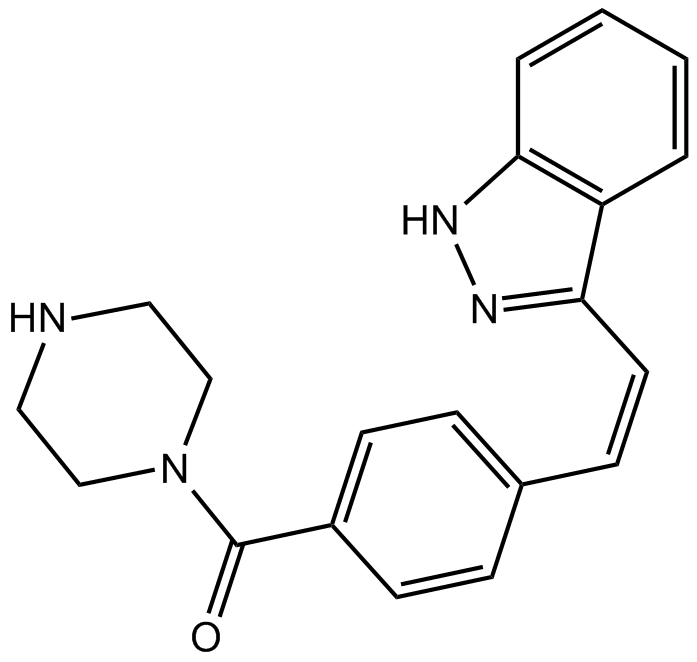
GC14592
KW 2449
A multi-kinase inhibitor
-
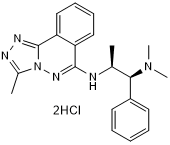
GC50395
L Moses dihydrochloride
High affinity and selective PCAF bromodomain inhibitor
-
GC33183
L-45 (L-Moses)
L-45 (L-Moses) (L-45) est le premier inhibiteur de bromodomaine (Brd) du facteur associé À p300/CBP (PCAF) puissant, sélectif et actif sur les cellules avec un Kd de 126 nM.
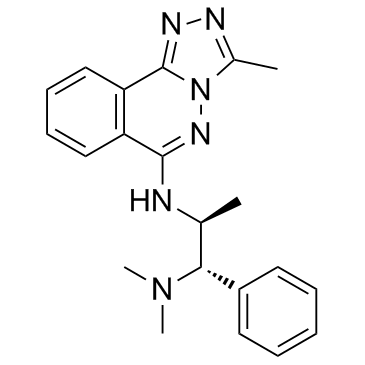
-
GC34377
L-45 dihydrochloride (L-Moses dihydrochloride)
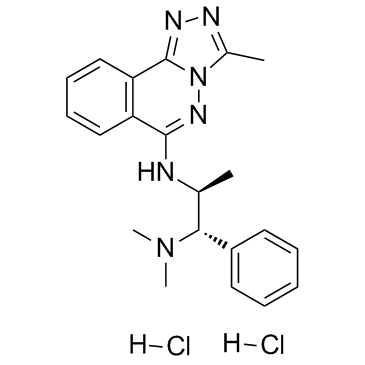
-
GC47579
L-Pyrohomoglutamic Acid
An amino acid building block
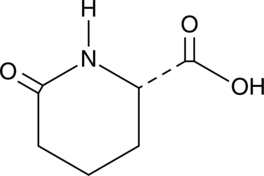
-
GC15731
L002
L002 est un inhibiteur puissant, perméable aux cellules, réversible et spécifique de l'acétyltransférase p300 (KAT3B) avec une IC50 de 1,98μM.
-
GC65289
Lademirsen
Lademirsen (SAR339375; RG-012) est un oligonucléotide simple brin modifié chimiquement qui se lie À et inhibe la fonction de miR-21.

-
GC15114
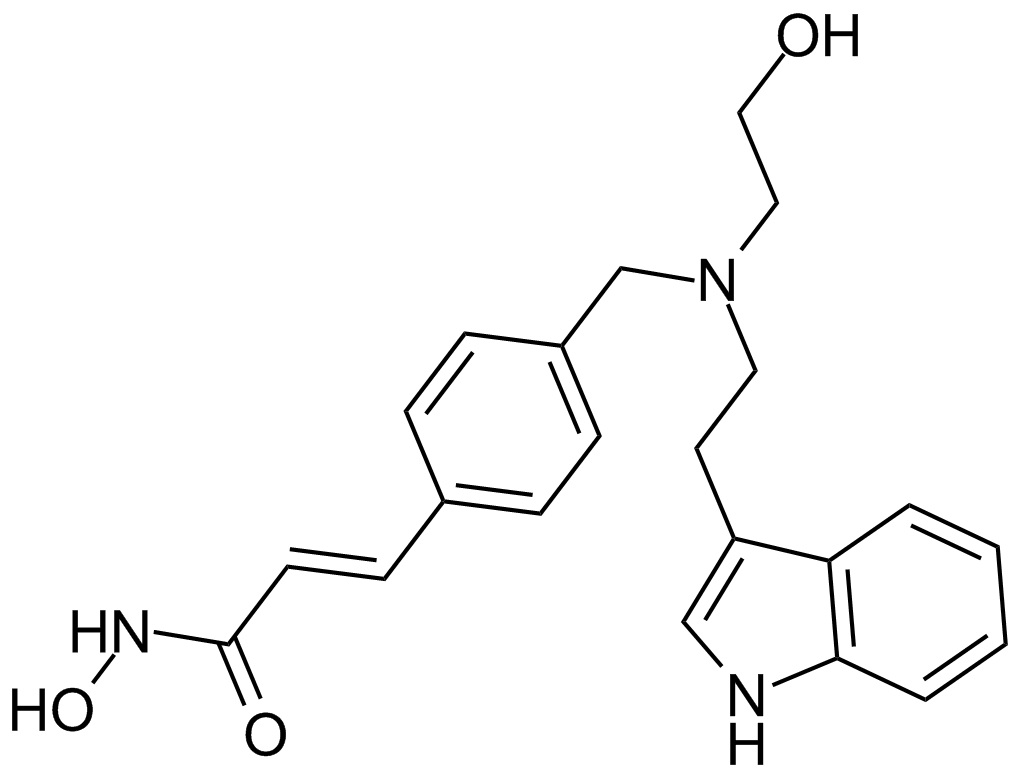
LAQ824 (NVP-LAQ824,Dacinostat)
A hydroxamate-based HDAC inhibitor
-
GC12189
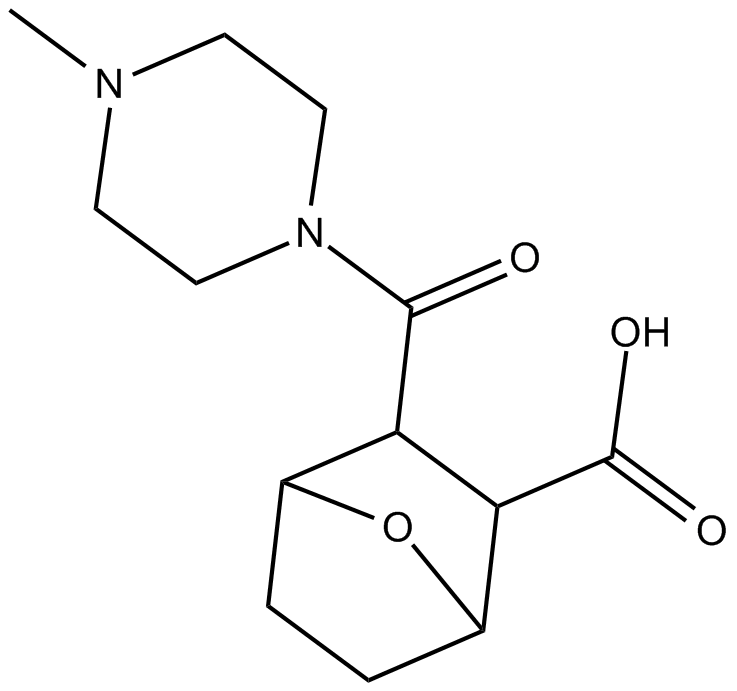
LB-100
protein phosphatase 2A(PP2A)inhibitor
-
GC14857
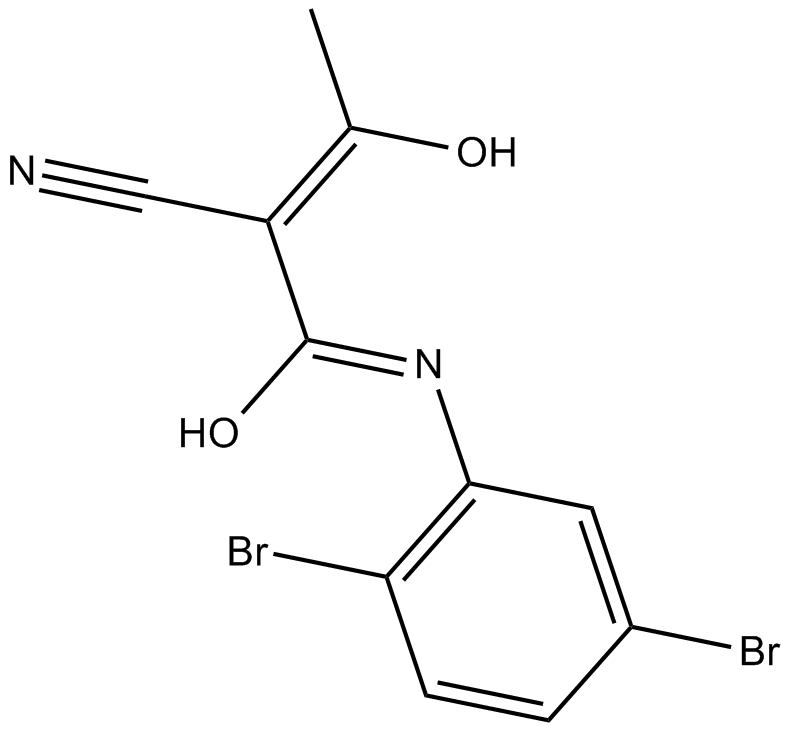
LFM-A13
LFM-A13 est un puissant inhibiteur de BTK, JAK2, PLK, inhibe la BTK recombinante, Plx1 et PLK3 avec des IC50 de 2,5 μM, 10 μM et 61 μM; LFM-A13 ne montre aucun effet sur JAK1 et JAK3, la kinase de la famille Src HCK, l'EGFR et l'IRK.
-
GC14362
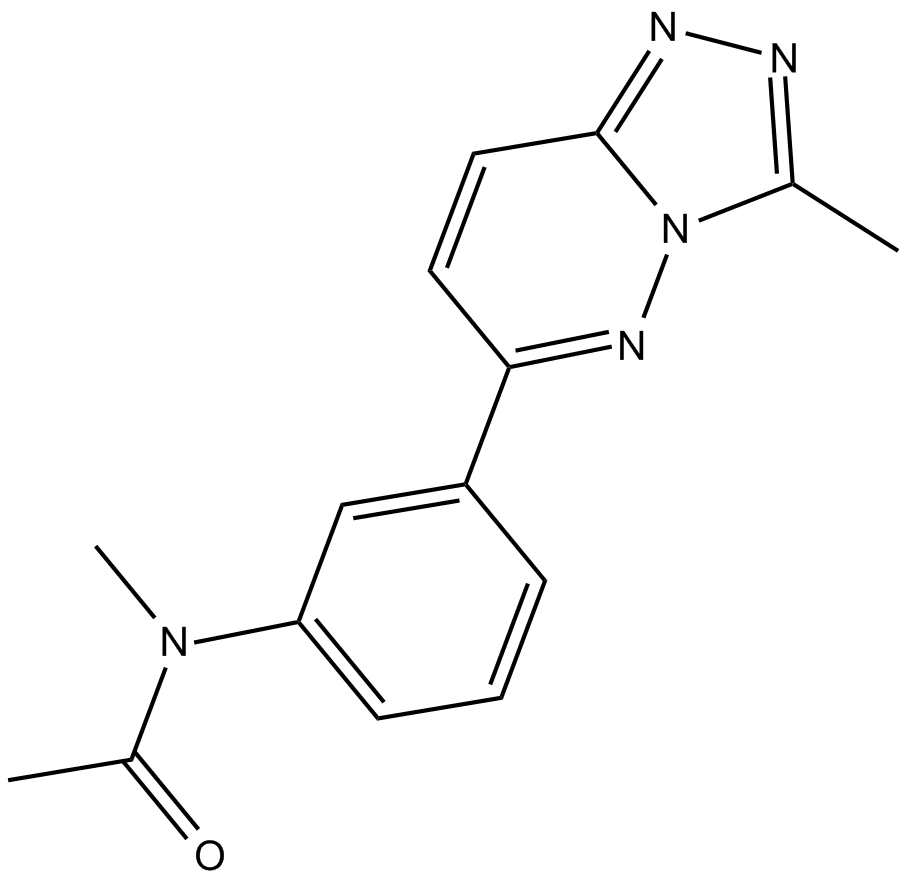
Lin28 1632
Lin28 1632 (composé 1632) est un puissant antagoniste de l'interaction Lin28/pre-let-7.
-
GC36464
LIN28 inhibitor LI71
L'inhibiteur de LIN28 LI71 est un inhibiteur de LIN28 puissant et perméable aux cellules, qui abolit l'oligouridylation médiée par LIN28 avec une IC50 de 7 uM. L'inhibiteur de LIN28 LI71 se lie directement au domaine de choc froid (CSD) pour supprimer l'activité de LIN28 contre let-7 dans les cellules leucémiques et les cellules souches embryonnaires.
-
GC30501
Lin28-let-7a antagonist 1
L'antagoniste Lin28-let-7a 1 montre un effet antagoniste clair contre l'interaction Lin28-let-7a avec une IC50 de 4,03 μM pour l'interaction Lin28A-let-7a-1.
-
GC13154
LKB1(AAK1 dual inhibitor)
-
GC33184
LLY-283
LLY-283 est un inhibiteur puissant, sélectif et oral de la protéine arginine méthyltransférase 5 (PRMT5), avec une IC50 de 22 nM et un Kd de 6 nM pour le complexe PRMT5:MEP50, et présente une activité antitumorale.
-
GC25580
LLY-284
LLY-284 is the diastereomer of LLY-283, which is a potent and selective SAM-competitive chemical probe for PRMT5. LLY-284 is much less active than LLY-283 and can be used as a negative control for LLY-283.