DNA Damage/DNA Repair
- MTH1(4)
- PARP(58)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(94)
- Nucleoside Antimetabolite/Analogue(136)
- Telomerase(14)
- Topoisomerase(133)
- tankyrase(5)
- Antifolate(32)
- CDK(233)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(62)
- DNA Alkylator/Crosslinker(64)
- DNA/RNA Synthesis(388)
- Eukaryotic Initiation Factor (eIF)(22)
- IRE1(22)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(53)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
GC40092
PD 0332991-d8
Palbociclib-d8
Palbociclib D8 (PD 0332991 D8) ist ein Deuterium mit der Bezeichnung Palbociclib. Palbociclib ist ein selektiver und oral aktiver CDK4- und CDK6-Inhibitor mit IC50-Werten von 11 bzw. 16 nM. Palbociclib hat das Potenzial fÜr die ER-positive und HER2-negative Brustkrebsforschung.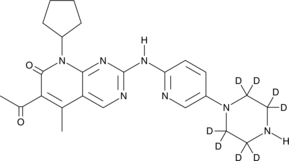
-
GC38216
PD 407824
-
GC18396
PD 407824
PD 407824 ist ein Checkpoint-Kinase-Chk1- und WEE1-Inhibitor mit IC50-Werten von 47 bzw. 97 nM.
-
GC61168
Peldesine
BCX 34
Peldesin (BCX 34) ist ein potenter, kompetitiver, reversibler und oral aktiver Purinnukleosid-Phosphorylase (PNP)-Inhibitor mit IC50-Werten von 36 nM, 5 nM und 32 nM fÜr rote BlutkÖrperchen (RBC) von Mensch, Ratte bzw. Maus (RBC). .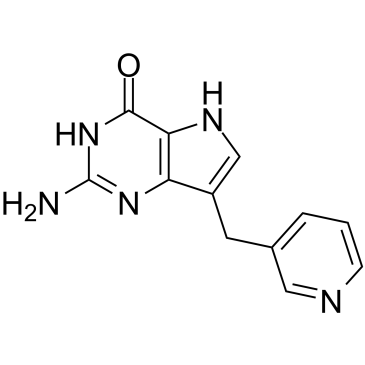
-
GC61633
Peldesine dihydrochloride
BCX 34 dihydrochloride
Peldesin (BCX 34) Dihydrochlorid ist ein potenter, kompetitiver, reversibler und oral aktiver Purinnukleosid-Phosphorylase (PNP)-Inhibitor mit IC50-Werten von 36 nM, 5 nM und 32 nM fÜr rote BlutkÖrperchen (RBC) von Mensch, Ratte und Maus PNP, beziehungsweise.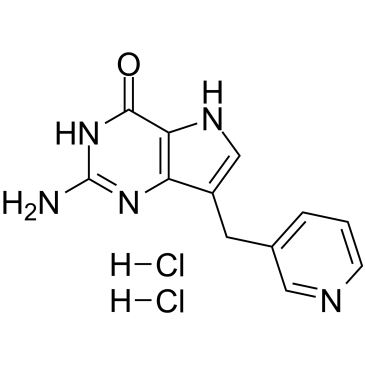
-
GC11610
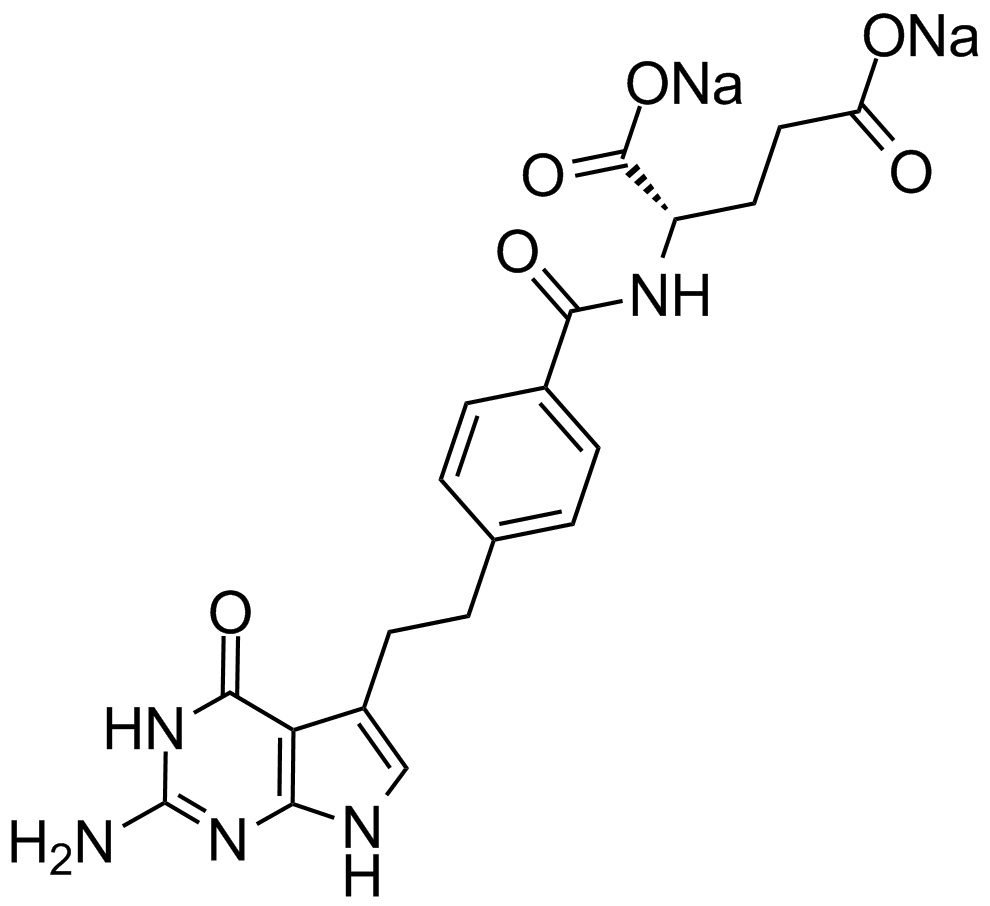
Pemetrexed
An antifolate with anticancer activity
-
GC44591
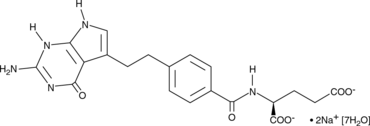
Pemetrexed (sodium salt hydrate)
Pemetrexed (Natriumsalzhydrat) ist ein neuartiges Antifolat, die Ki-Werte des Pentaglutamats von LY231514 betragen 1,3, 7,2 und 65 nM fÜr die Hemmung der Thymidylatsynthase (TS), der Dihydrofolatreduktase (DHFR) bzw. der Glycinamid-Ribonukleotid-Formyltransferase (GARFT). .
-
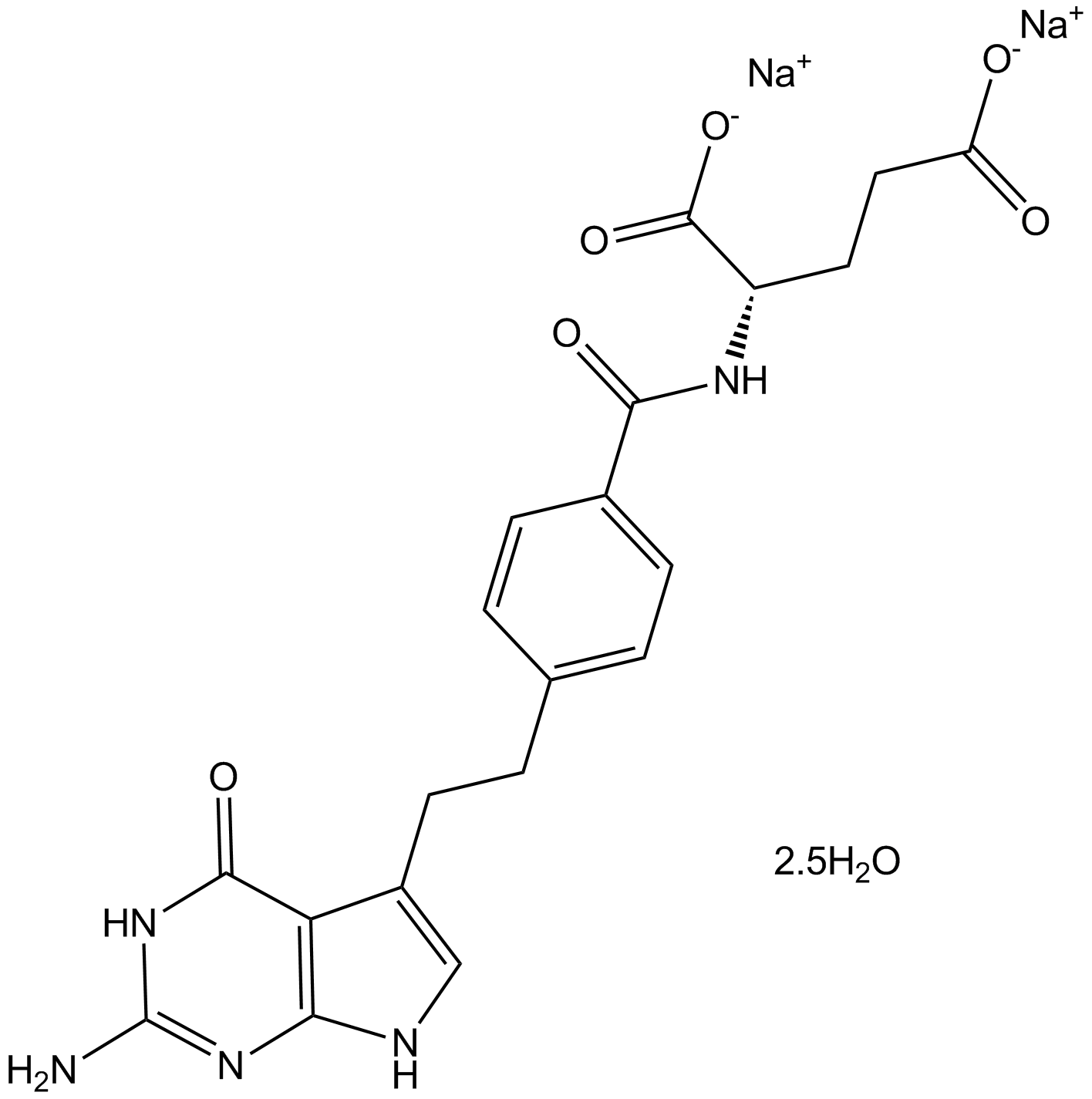
GC17694
Pemetrexed disodium hemipenta hydrate
-
GC17848
Penciclovir
BRL 39123
Penciclovir (VSA 671) ist ein potenter und selektiver Anti-Herpesvirus-Wirkstoff mit EC50-Werten von 0,5 bzw. 0,8 μg/ml fÜr HSV-1 (HFEM) bzw. HSV-2 (MS).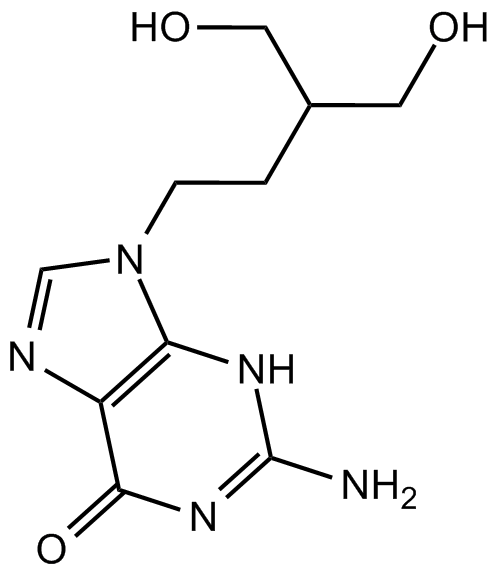
-
GC16514
Penciclovir Sodium
VSA 671 sodium; BRL 39123A; BRL 39123D
Penciclovir (VSA 671)-Natrium ist ein wirksames und selektives Anti-Herpesvirus-Mittel mit EC50-Werten von 0,5 bzw. 0,8 μg/ml fÜr HSV-1 (HFEM) bzw. HSV-2 (MS).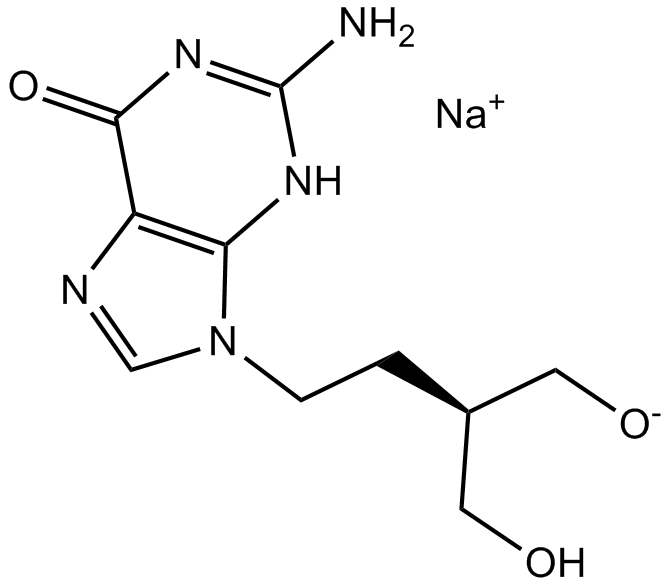
-
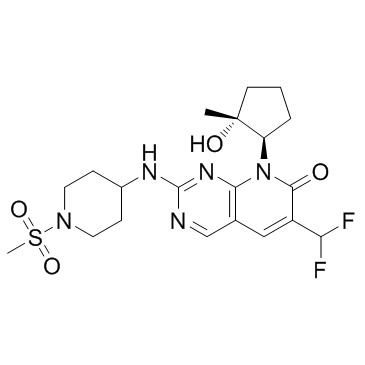
GC32832
PF-06873600
PF-3600
-
GC62660
PF-07104091
PF-07104091 ist ein CDK2/Cyclin E1-Inhibitor, der sich selektiv an die ATP-Stelle bindet und den aktivierten Zustand des Kinase-Komplexes im gebundenen Cyclin-Zustand hemmt [1].
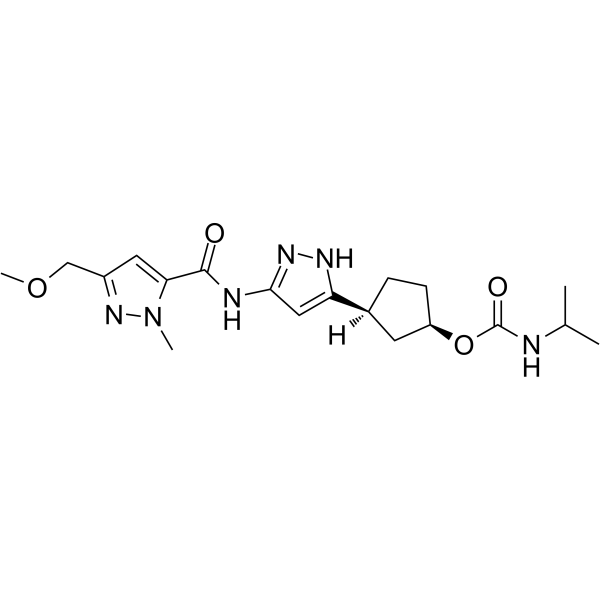
-
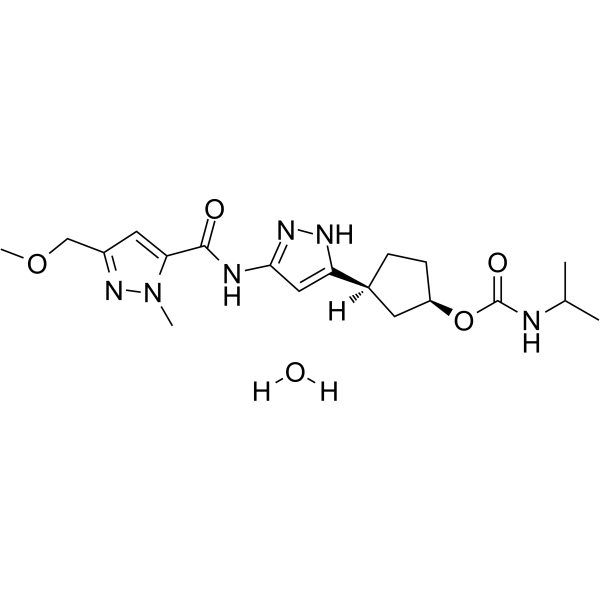
GC62661
PF-07104091 hydrate
PF-07104091 hydrate is a potent and selective CDK2/cyclin E1 and GSK3β inhibitor, with Kis of 1.16 and 537.81 nM, respectively.
-
GC14234
PF-477736
PF 00477736
PF-477736 (PF 00477736) ist ein potenter, selektiver und ATP-kompetitiver Inhibitor von Chk1 mit einem Ki von 0,49 nM, es ist auch ein Chk2-Inhibitor mit einem Ki von 47 nM. PF-477736 zeigt eine \u003c100-fache Selektivität für Chk1 gegenüber VEGFR2, Fms, Yes, Aurora-A, FGFR3, Flt3 und Ret (IC50\u003d8 (Ki), 10, 14, 23, 23, 25 und 39 nM, beziehungsweise). PF-477736 kann die Antitumoraktivität von Gemcitabin in vitro und in vivo verstärken.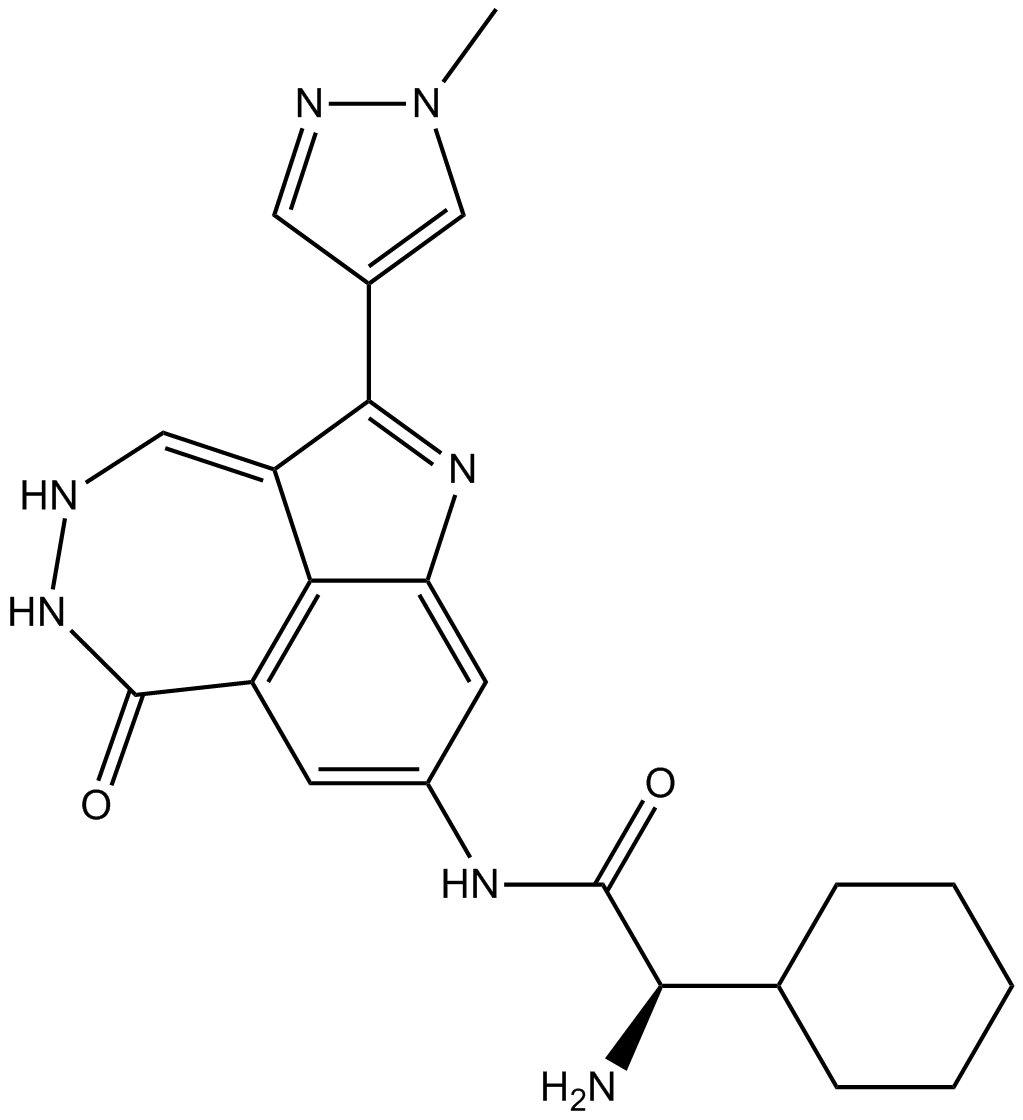
-
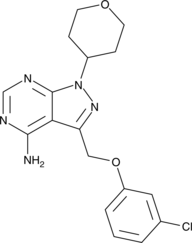
GC44612
PF-4800567
PF-4800567 ist ein potenter und selektiver Inhibitor der Caseinkinase 1ε (CK1ε) mit einem IC50 von 32 nM, was eine mehr als 20-fache SelektivitÄt gegenÜber CK1δ (IC50, 711 nM) bedeutet.
-
GC36885
PF-5006739
PF-5006739 ist ein potenter und selektiver Inhibitor von CK1δ/ε mit IC50-Werten von 3,9 nM bzw. 17,0 nM.
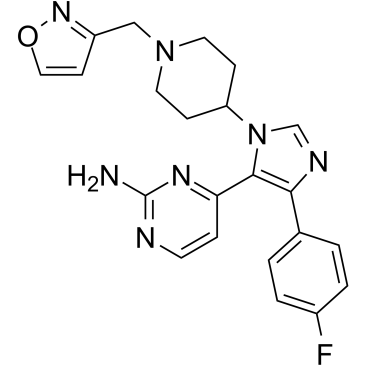
-
GC10556
PF-670462
An inhibitor of the CK1 isoforms CK1ε and CK1δ
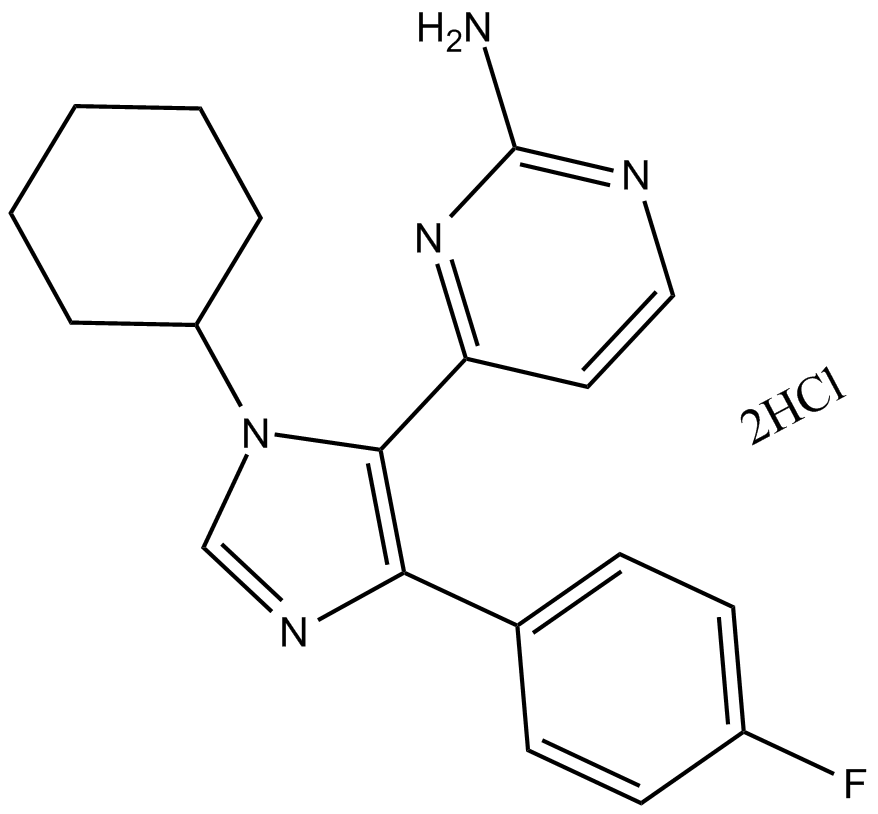
-
GC11324
PHA-767491
CAY10572;PHA767491;PHA 767491;CAY-10572;CAY 10572
Ein potenter Cdc7-Kinase-Inhibitor
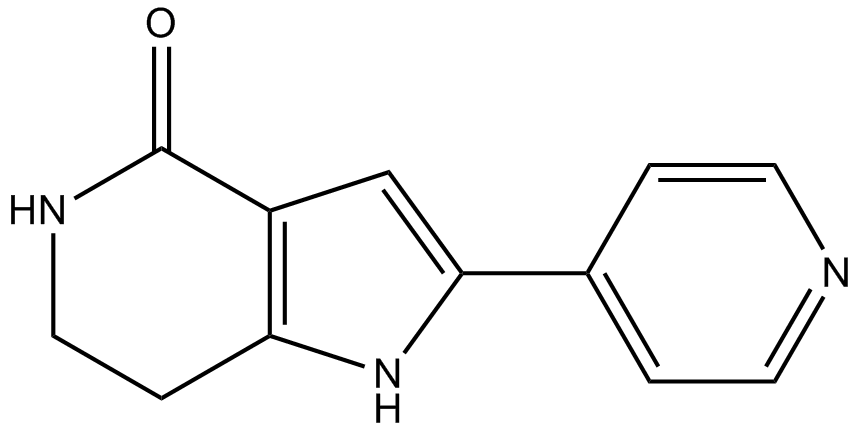
-
GC36892
PHA-767491 hydrochloride
CAY10572
PHA-767491-Hydrochlorid ist ein dualer Cdc7/Cdk9-Inhibitor mit IC50-Werten von 10 nM bzw. 34 nM.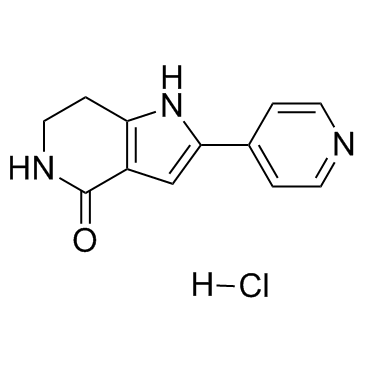
-
GC15588
PHA-848125
Milciclib
PHA-848125 (PHA-848125) ist ein potenter, ATP-kompetitiver und dualer Inhibitor von CDK und Tropomyosin-Rezeptorkinase (TRK) mit IC50-Werten von 45, 150, 160, 363, 398 nM und 53 nM fÜr Cyclin A/CDK2, Cyclin H/CDK7, Cyclin D1/CDK4, Cyclin E/CDK2, Cyclin B/CDK1 bzw. TRKA.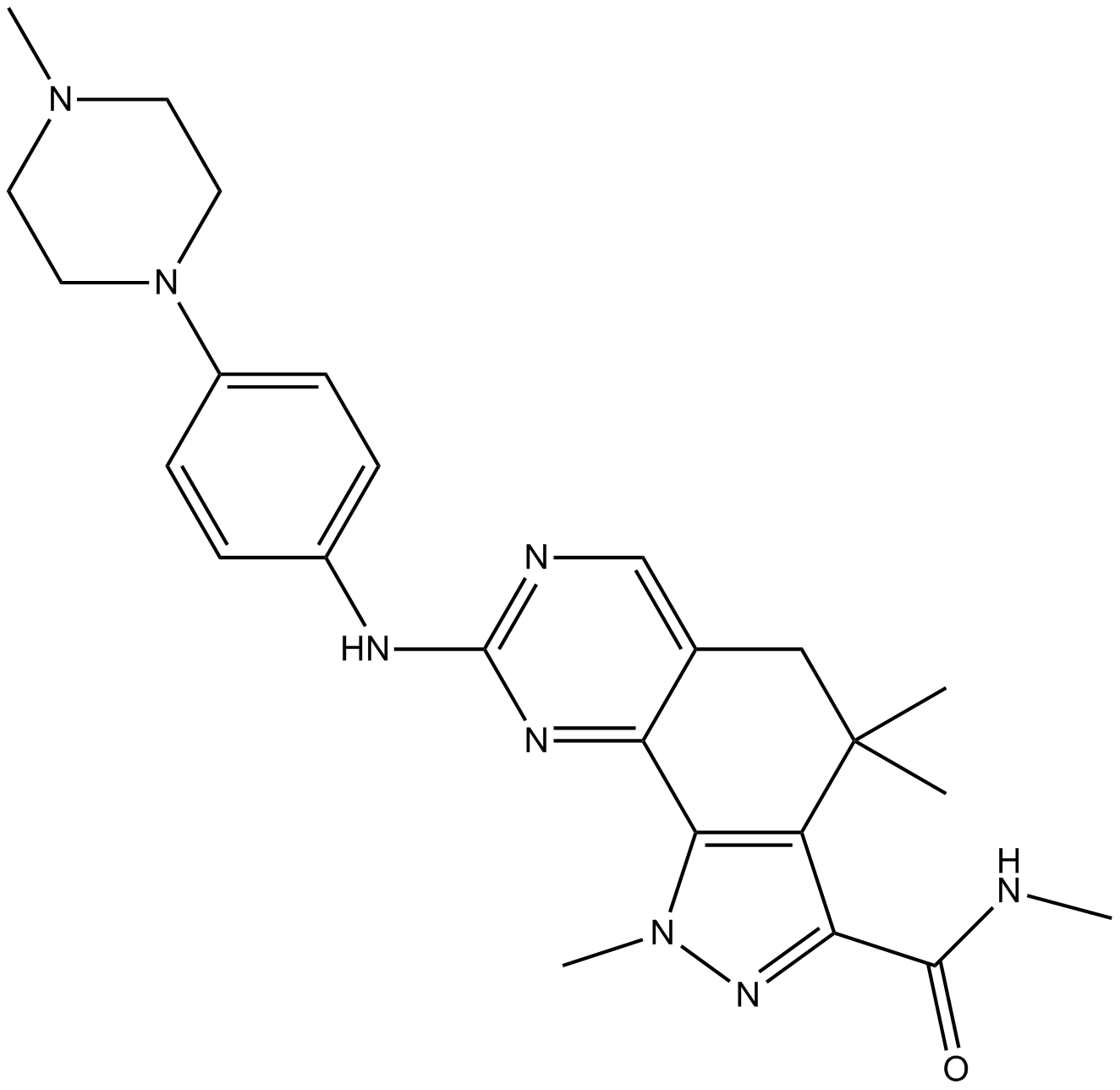
-
GC30695
Phen-DC3
Phen-DC3 ist ein G-Quadruplex (G4)-spezifischer Ligand, der FANCJ- und DinG-Helikasen mit IC50-Werten von 65±6 bzw. 50±10 nM hemmen kann.
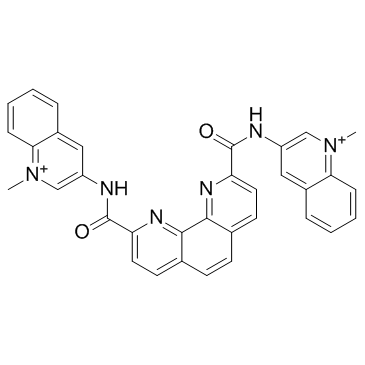
-
GC36895
Phenoxodiol
Haginin E, Phenoxodiol
Phenoxodiol (Idronoxil), ein synthetisches Analogon von Genestein, aktiviert das mitochondriale Caspase-System, hemmt XIAP (einen Apoptose-Hemmer) und sensibilisiert die Krebszellen fÜr Fas-vermittelte Apoptose. Phenoxodiol hemmt auch die DNA-Topoisomerase II, indem es den spaltbaren Komplex stabilisiert. Phenoxodiol induziert einen Zellzyklusarrest in der G1/S-Phase des Zellzyklus und reguliert p21WAF1 Über eine p53-unabhÄngige Weise hoch.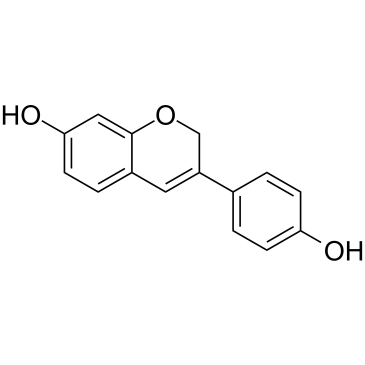
-
GC61182
Phosphoramide mustard (cyclohexanamine)
NSC 69945, PMC
Phosphoramid-Lost-Cyclohexanamin ist ein biologisch aktiver Metabolit von Cyclophosphamid mit krebsbekÄmpfender Wirkung. Phosphoramid-Senf-Cyclohexanamin induziert DNA-SchÄden.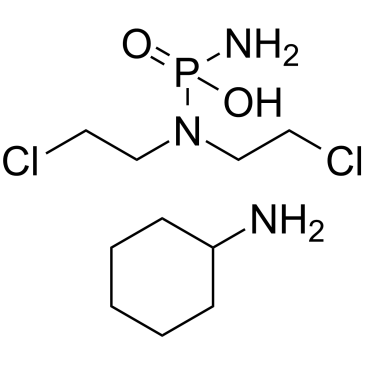
-
GC11165
PI-103
PI-103 ist ein potenter PI3K- und mTOR-Inhibitor mit IC50-Werten von 8 nM, 88 nM, 48 nM, 150 nM, 20 nM und 83 nM fÜr p110α, p110β, p110δ, p110γ, mTORC1 und mTORC2. PI-103 hemmt auch DNA-PK mit einem IC50 von 2 nM. PI-103 induziert Autophagie.
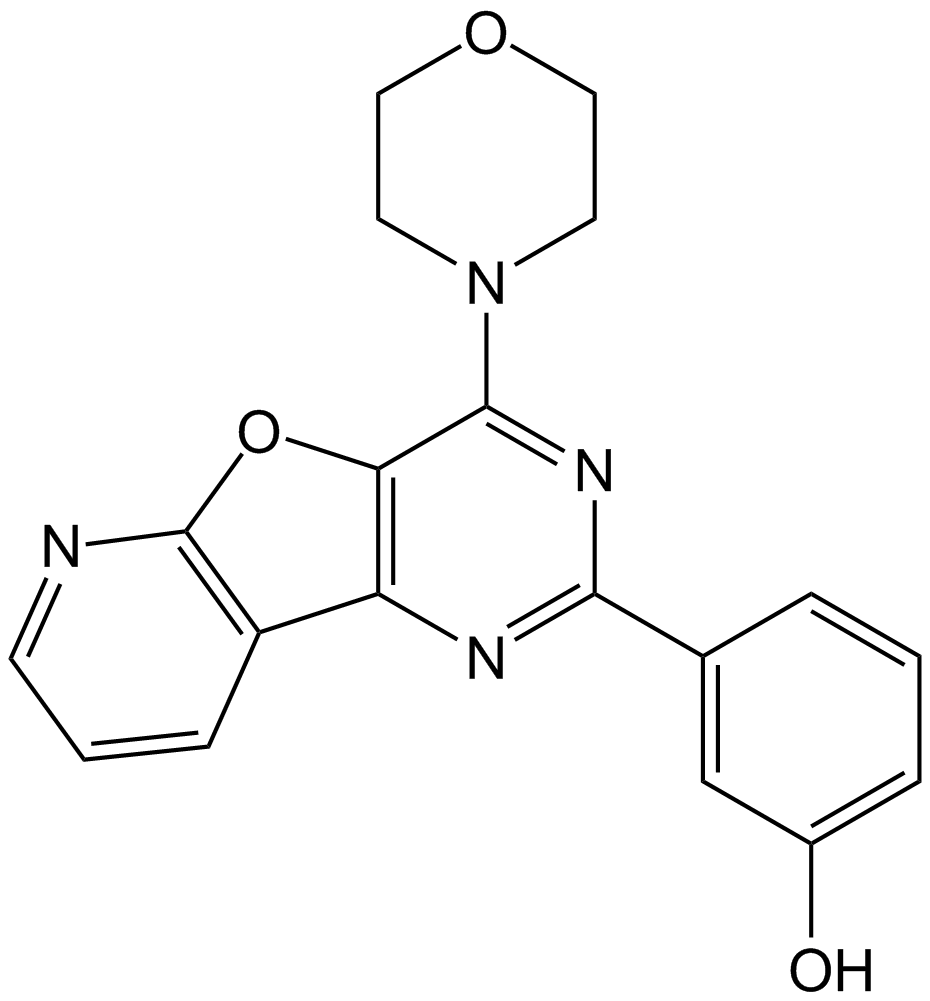
-
GC16379
PI-103 Hydrochloride
PI 103 hydrochloride;PI103 hydrochloride
A potent, cell-permeable PI3K inhibitor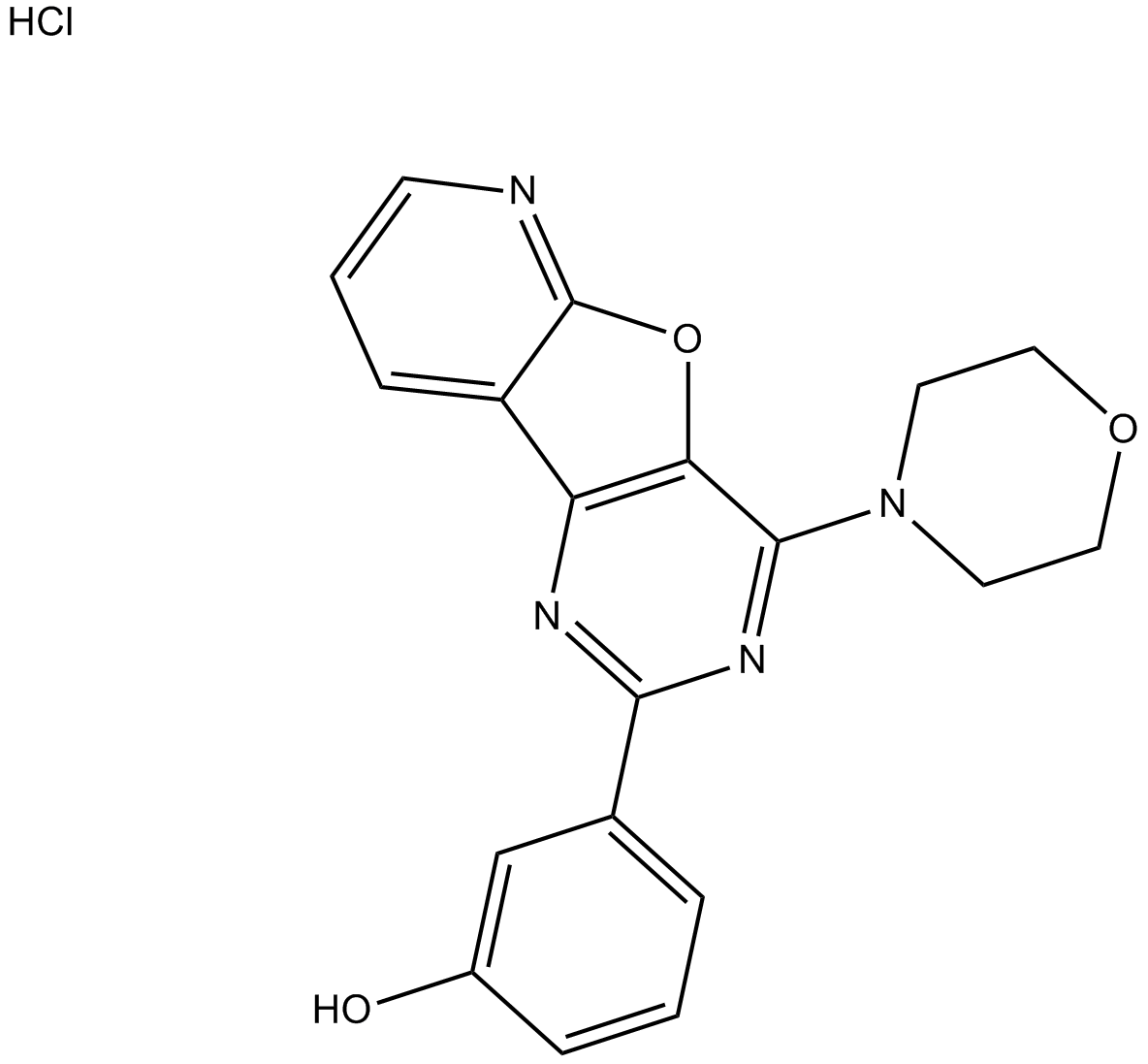
-
GC38578
PI-828
A PI3K inhibitor
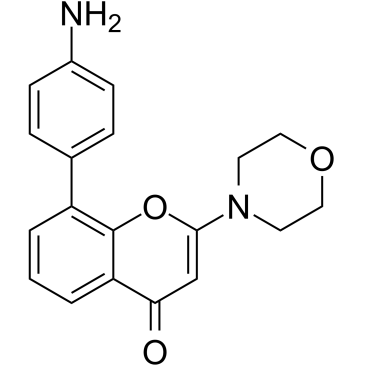
-
GC36905
PI3K/HDAC-IN-1
PI3K/HDAC-IN-1 ist ein potenter dualer Inhibitor von PI3K/HDAC, hemmt PI3Kδ und HDAC1 mit IC50-Werten von 8,1 nM bzw. 1,4 nM.
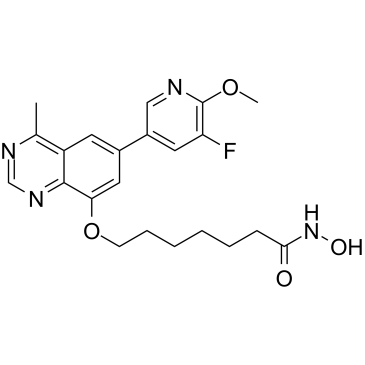
-
GC14251
Picolinamide
poly (ADP-ribose) synthetase inhibitor
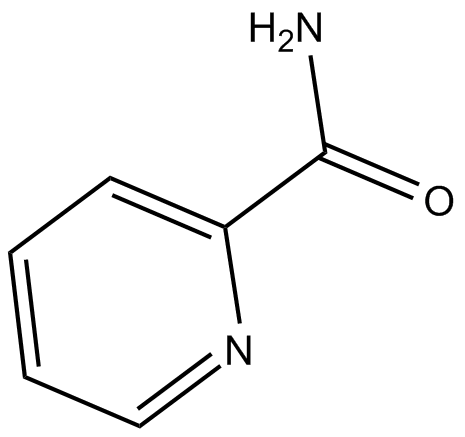
-
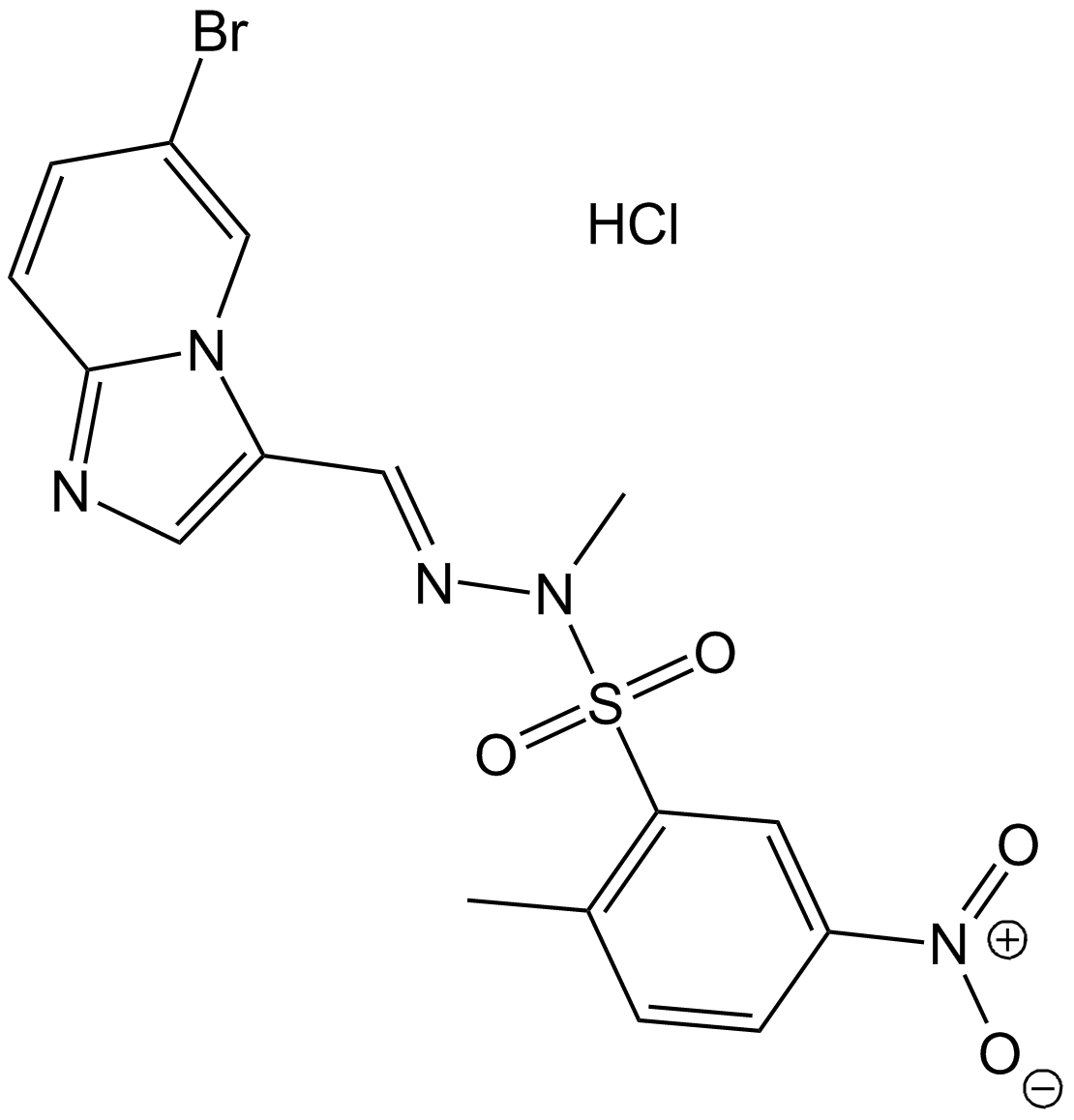
GC16904
PIK-75
PIK-75 ist ein reversibler DNA-PK- und p110α-selektiver Inhibitor, der DNA-PK hemmt, p110&7#945; und p110γ mit IC50s von 2, 5,8 bzw. 76 nM. PIK-75 hemmt p110α >200-mal stÄrker als p110&7#946; (IC50=1,3 μM). PIK-75 induziert Apoptose.
-
GC17199
PIK-90
PIK85
PIK-90 ist ein DNA-PK- und PI3K-Inhibitor, der p110α, p110γ und DNA-PK mit IC50-Werten von 11, 18 bzw. 13 nM hemmt.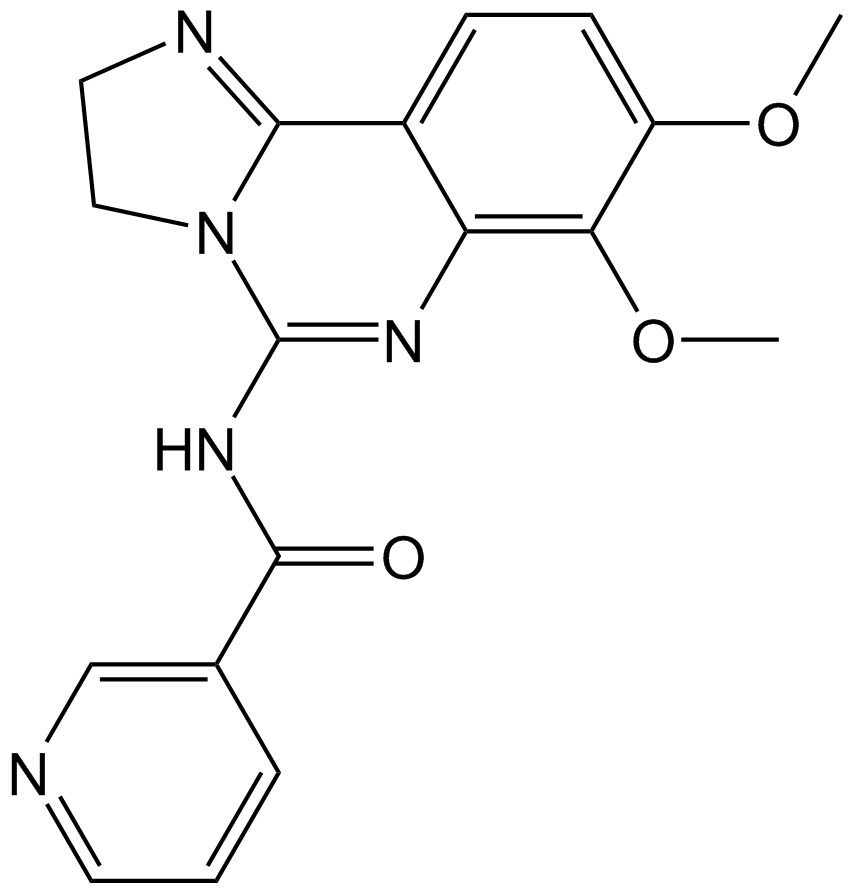
-
GC61188
PIP-199
PIP-199 ist ein selektiver Inhibitor der RMI (RecQ-mediated genome instability protein)-Kernkomplex/MM2-Interaktion mit einem IC50 von 36 μM. PIP-199 kann fÜr die Erforschung der Sensibilisierung resistenter Tumore gegenÜber DNA-vernetzenden Chemotherapeutika verwendet werden.
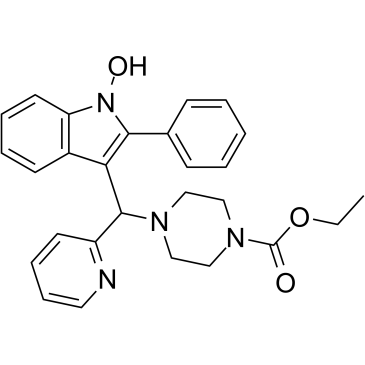
-
GC17082
Pirarubicin
Pirarubicin ist ein Anthracyclin-Antibiotikum, wirkt als Topoisomerase-II-Hemmer und wird weit verbreitet zur Behandlung verschiedener Krebsarten, insbesondere solider Tumore, verwendet.
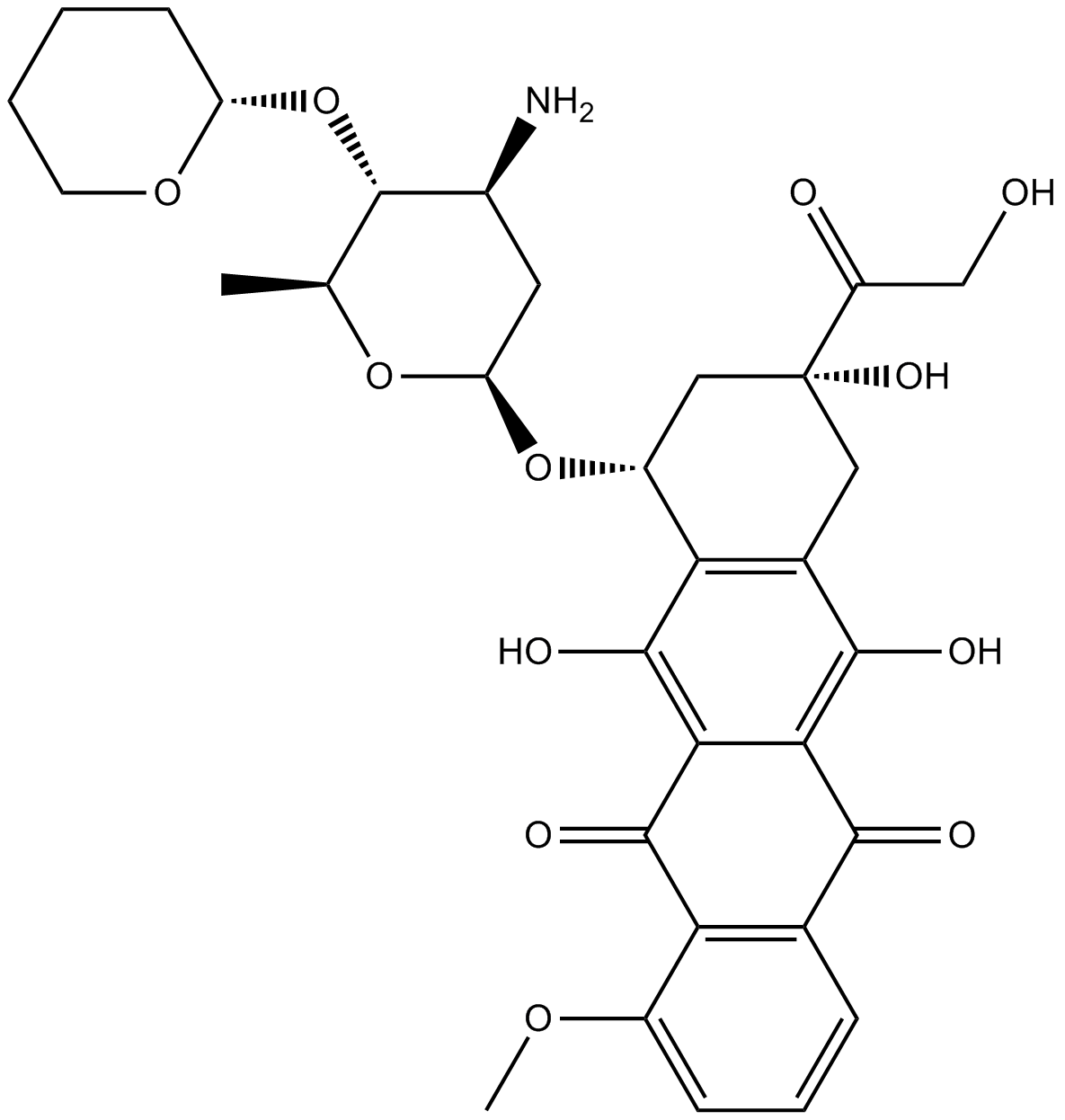
-
GC36928
Pirarubicin Hydrochloride
Pirarubicinhydrochlorid ist ein Anthracyclin-Antibiotikum, wirkt als Topoisomerase-II-Hemmer und wird hÄufig zur Behandlung verschiedener Krebsarten, insbesondere solider Tumore, eingesetzt.
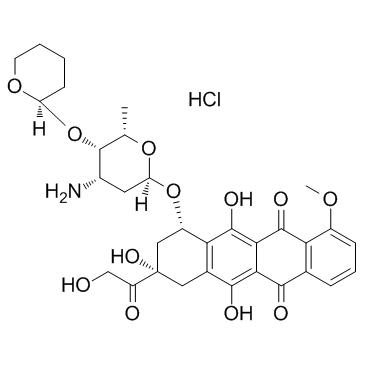
-
GC68474
Pivanex
AN-9; Pivalyloxymethyl butyrate
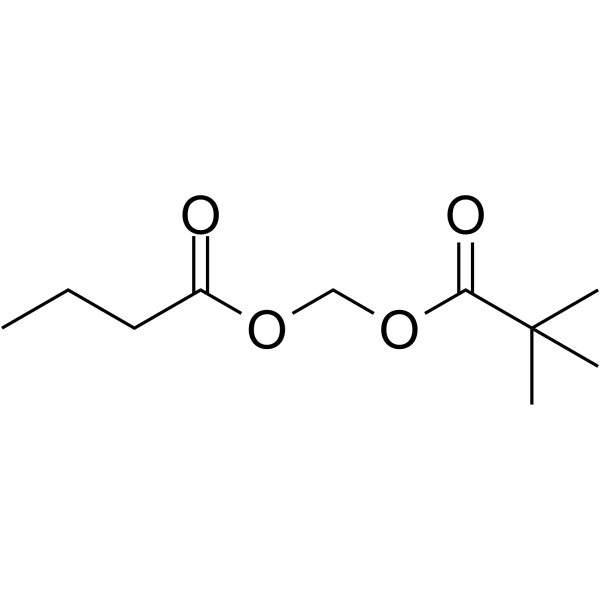
-
GC12503
Pixantrone
Pixantron (BBR 2778 (freie Base)), ein Mitoxantron-Analogon, ist ein Topoisomerase-II-Inhibitor und DNA-Interkalator mit AntitumoraktivitÄt.
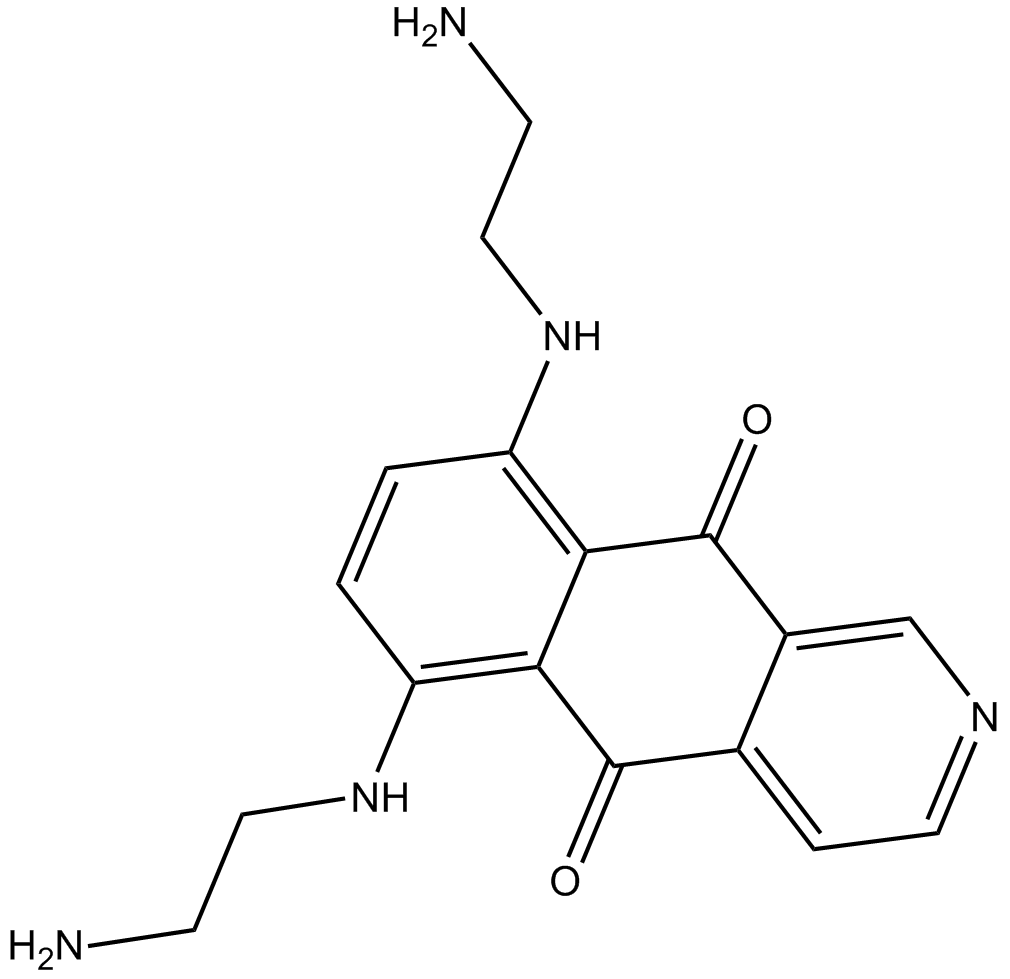
-
GC44651
Pixantrone (maleate)
BBR 2778
Pixantrone is an aza-anthracenedione analog that intercalates in DNA, directly alkylates DNA, and forms stable DNA adducts.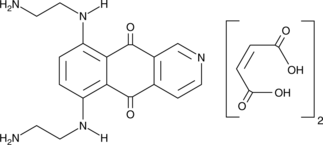
-
GC52455
Pixantrone-d8 (maleate)
An internal standard for the quantification of pixantrone
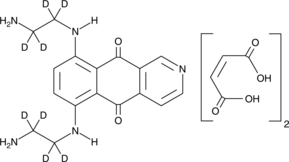
-
GC32946
PK11007
PK11007 ist ein milder Thiolalkylator mit AntikrebsaktivitÄt. PK11007 stabilisiert p53 durch selektive Alkylierung von zwei oberflÄchenexponierten Cysteinen, ohne seine DNA-BindungsaktivitÄt zu beeintrÄchtigen. PK11007 induziert den Tod von mutierten p53-Krebszellen, indem es die Werte der reaktiven Sauerstoffspezies (ROS) erhÖht.
-
GC61531
PluriSIn #2
PluriSIn #2 ist ein selektiver Transkriptionsinhibitor der Topoisomerase II α (TOP2A). PluriSIn #2 ist eine Verbindung, die selektiv undifferenzierte humane pluripotente Stammzellen (hPSCs) eliminiert.
-
GC39421
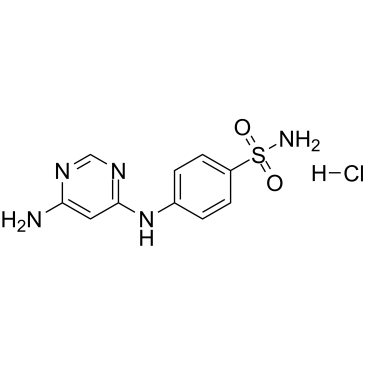
PNU112455A hydrochloride
PNU112455A-Hydrochlorid ist ein ATP-kompetitiver CDK2- und CDK5-Inhibitor. PNU112455A-Hydrochlorid bindet an die ATP-Stelle von CDK2 und CDK5 mit Kms von 3,6 bzw. 3,2 μM.
-
GC33143
Podocarpusflavone A
PCFA
Podocarpusflavone A ist ein DNA-Topoisomerase-I-Inhibitor. Podocarpusflavon A hat eine moderate antiproliferative AktivitÄt und induziert Zellapoptose in MCF-7. Podocarpusflavone A entwickelt Anti-Tumor-Medikamente.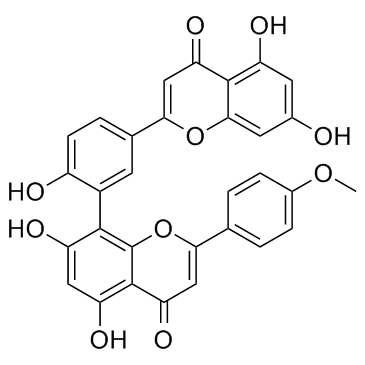
-
GN10416
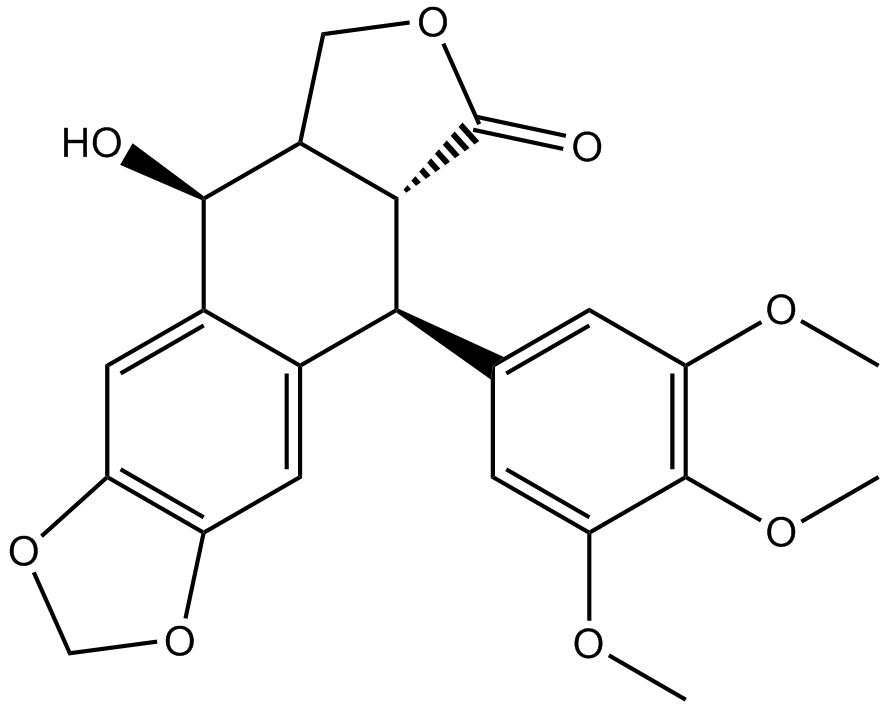
Podophyllotoxin
Condyline; Condylox; Podofilox
A lignan with diverse biological activities
-
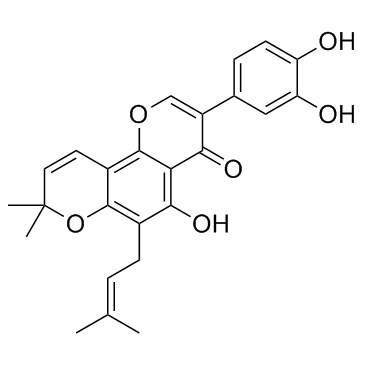
GC33115
Pomiferin (NSC 5113)
Pomiferin (NSC 5113) (NSC 5113) wirkt als potenzieller Inhibitor von HDAC mit einem IC50 von 1,05 μM und hemmt auch stark mTOR (IC50, 6,2 μM).
-
GC63154
PR-104
PR-104 ist ein selektives Hypoxie-aktiviertes DNA-Vernetzungsmittel und kann fÜr die Erforschung mehrerer Tumor-Xenograft-Modelle verwendet werden. PR-104 wird als Stickstoff-Lost-PrÄ-Prodrug effizient in den lipophileren Dinitrobenzamid-Lost-Alkohol PR-104A umgewandelt.
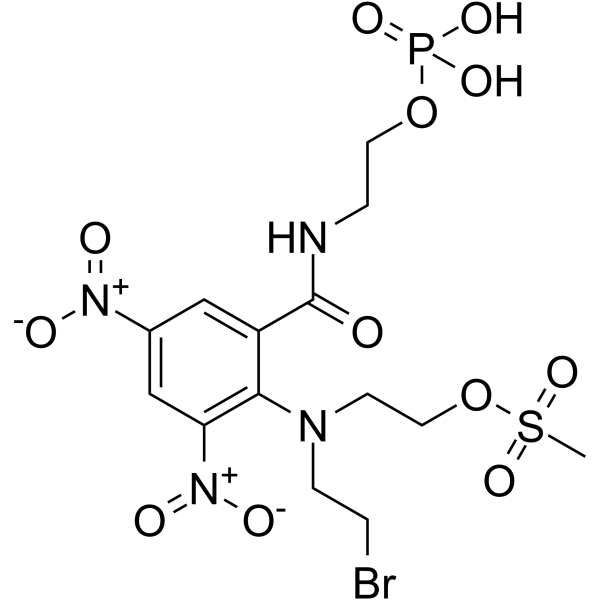
-
GC62490
PR-104A
SN 27858
PR-104A (SN 27858) ist der Alkoholmetabolit des Phosphat-Prodrugs PR-104. PR-104A ist ein Hypoxie-selektives DNA-Vernetzungsmittel/DNA-schÄdigendes Mittel und Zytotoxin. AntitumoraktivitÄt. PR-104A wird unter Hypoxie durch die 1-Elektronen-NADPH:Cytochrom-P450-Oxidoreduktase metabolisiert. PR-104A kann fÜr die Erforschung rezidivierter/refraktÄrer akuter lymphoblastischer LeukÄmie (T-ALL) der T-Linie verwendet werden.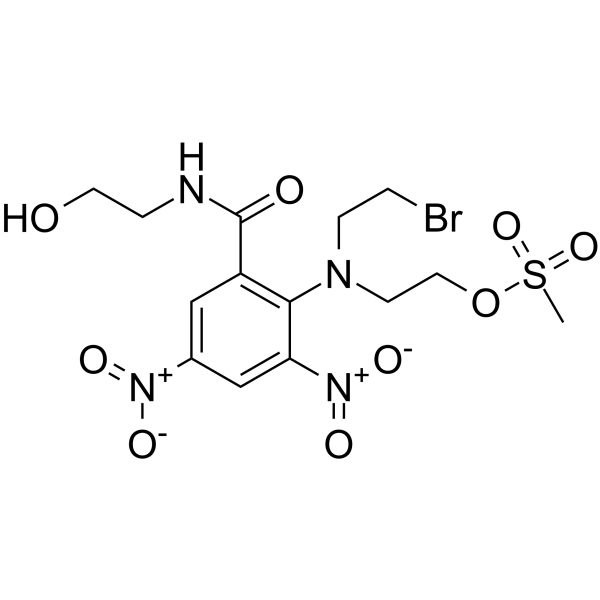
-
GC13208
PR-619
PR-619 is a broad-spectrum DUB (deubiquitinating enzyme) inhibitor. PR-619 has demonstrated robust DUB inhibitory activity (5-20 µM) and growth inhibitory activity with IC50 of 2 µM in HEK 293T cells.
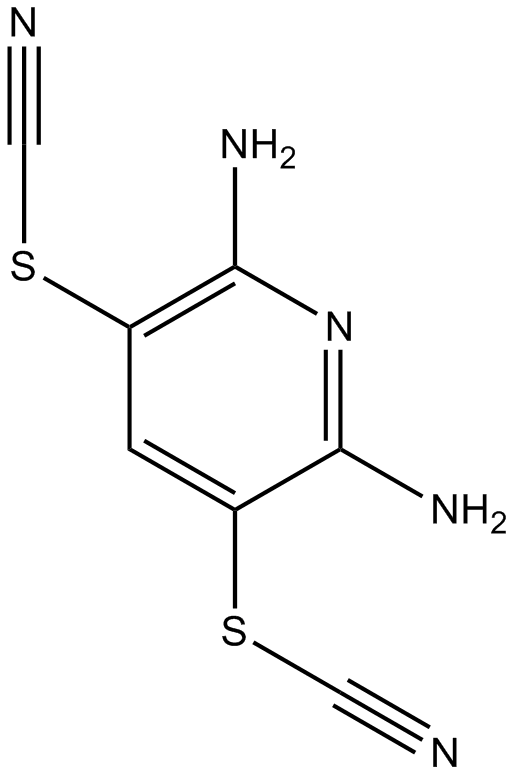
-
GC12919
Pralatrexate
NSC 754230, PDX, 10-Propargyl-10-deazaaminopterin
A dihydrofolate reductase inhibitor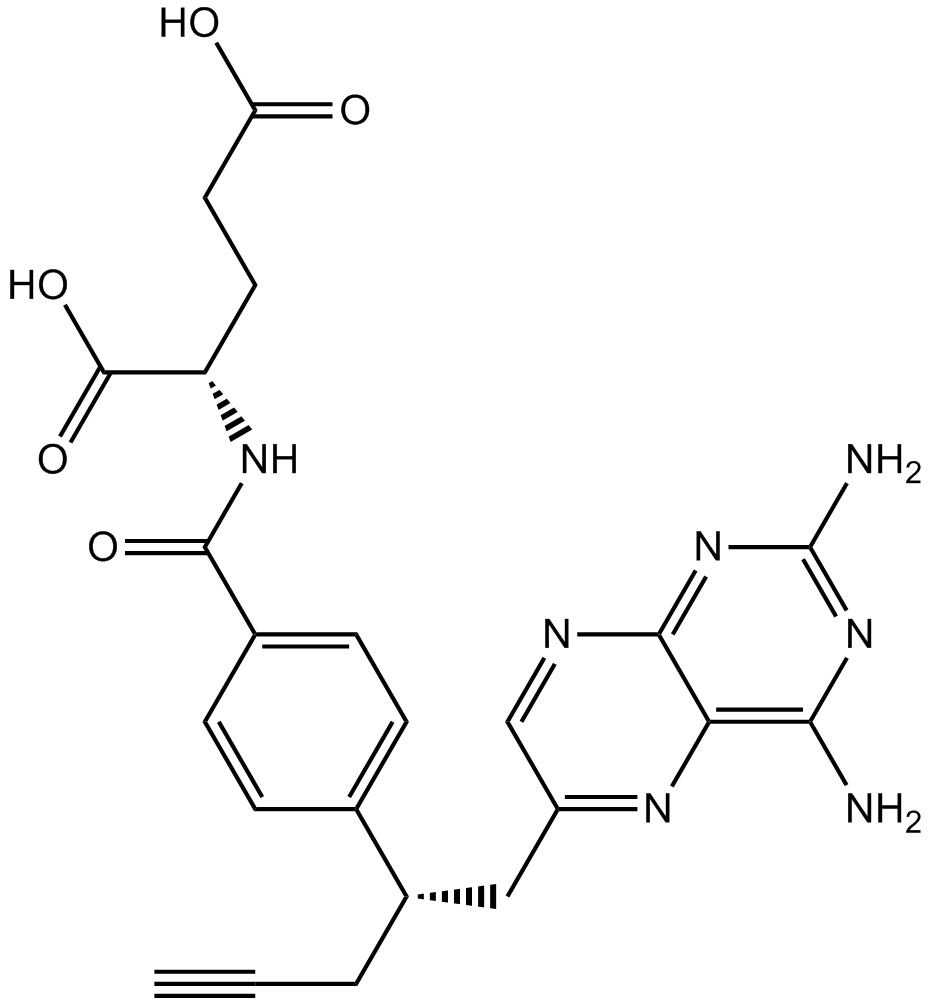
-
GC36966
Prexasertib dihydrochloride
Prexasertib-Dihydrochlorid (LY2606368-Dihydrochlorid) ist ein selektiver, ATP-kompetitiver Checkpoint-Kinase-1 (CHK1)-Inhibitor der zweiten Generation mit einem Ki von 0,9 nM und einem IC50 von < 1 nM. Prexasertib-Dihydrochlorid hemmt CHK2 (IC50 = 8 nM) und RSK1 (IC50 = 9 nM). Prexasertib-Dihydrochlorid verursacht einen Bruch der doppelstrÄngigen DNA und eine Replikationskatastrophe, die zu Apoptose fÜhrt. Prexasertib-Dihydrochlorid zeigt eine starke AntitumoraktivitÄt.
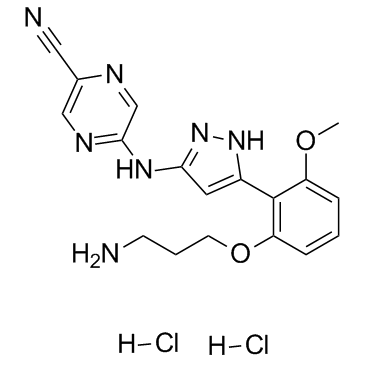
-
GC36967
Prexasertib Mesylate Hydrate
LY2606368 Mesylate Hydrate; LY2940930
Prexasertib Mesylathydrat (LY2606368 Mesylathydrat) ist ein selektiver, ATP-kompetitiver Checkpoint-Kinase-1 (CHK1)-Inhibitor der zweiten Generation mit einem Ki von 0,9 nM und einem IC50 von < 1 nM. Prexasertib Mesylathydrat hemmt CHK2 (IC50 = 8 nM) und RSK1 (IC50 = 9 nM). Prexasertib Mesylathydrat verursacht einen Bruch der doppelstrÄngigen DNA und eine Replikationskatastrophe, die zu Apoptose fÜhrt. Prexasertib Mesylathydrat zeigt eine starke AntitumoraktivitÄt.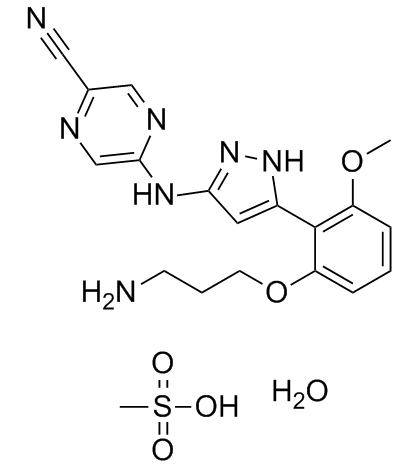
-
GC64733
Procainamide
Procainamid ist ein spezifischer und wirksamer Inhibitor der DNA-Methyltransferase 1 (DNMT1).
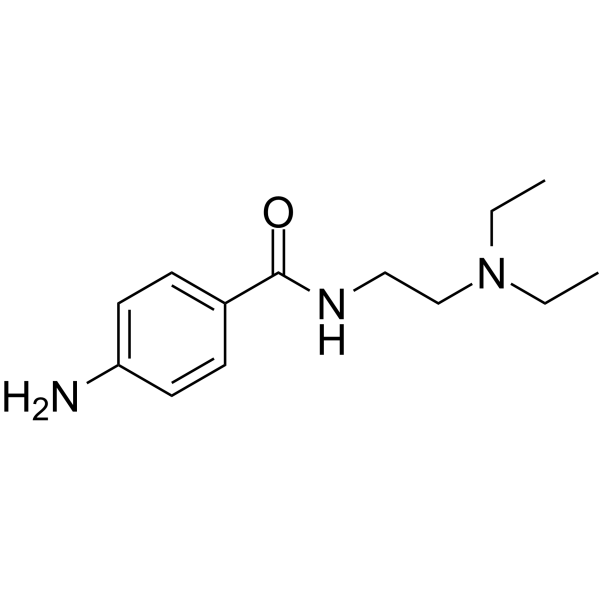
-
GC14066
Procaine
Procain ist ein DNA-demethylierendes Mittel.
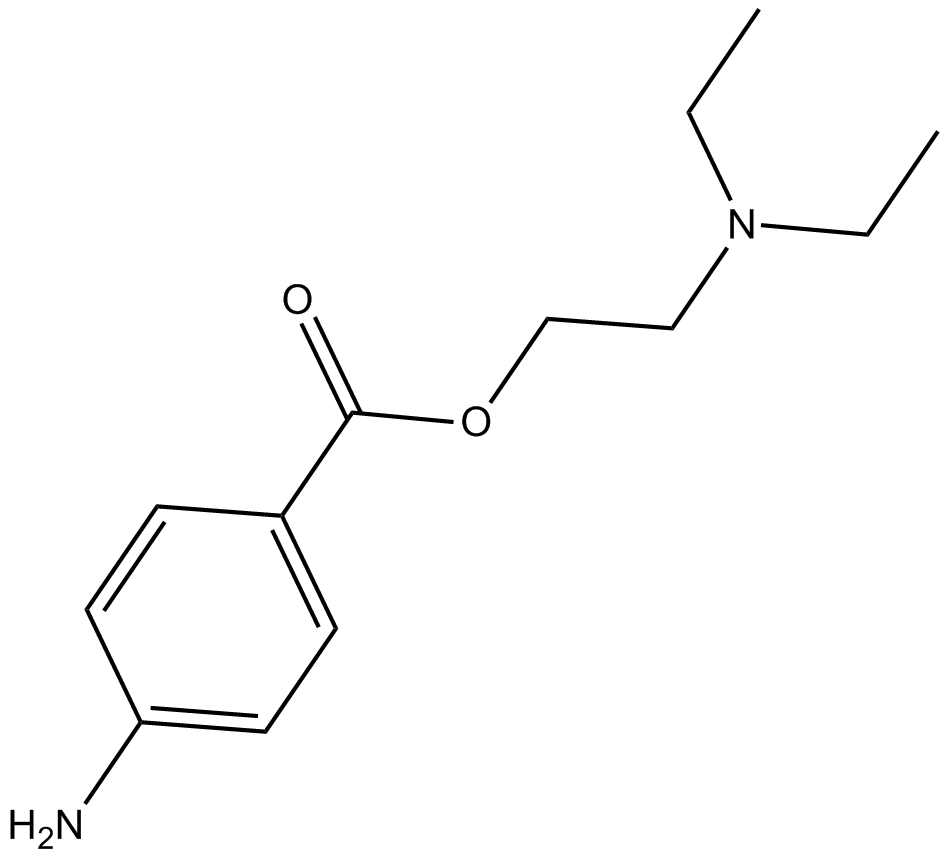
-
GC44685
Procaine (hydrochloride)
Novocaine
Procaine (hydrochloride) is an analytical reference standard categorized as a local anesthetic that is used as an adulterant.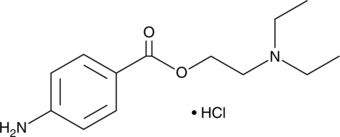
-
GC11793
Procarbazine HCl
Indicarb, Natulan, NSC 77213
Procarbazin HCl ist ein oral wirksames Alkylierungsmittel mit AntikrebsaktivitÄt. Procarbazin HCl kann in der Erforschung der Hodgkin-Krankheit verwendet werden.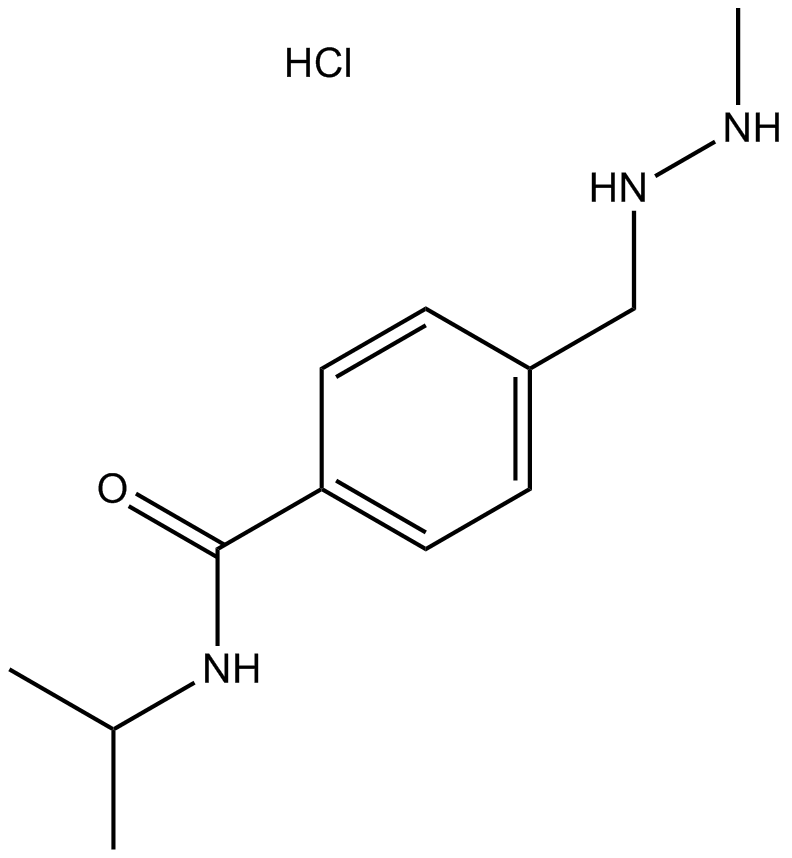
-
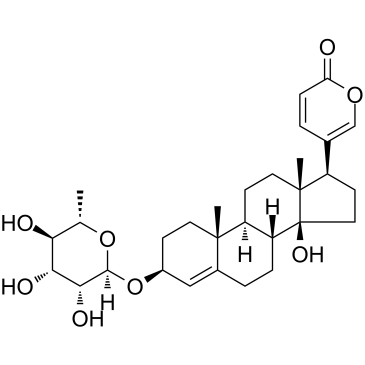
GC39425
Proscillaridin A
Proscillaridin A ist ein starkes Gift der Topoisomerase I/II-AktivitÄt mit IC50-Werten von 30 nM bzw. 100 nM.
-
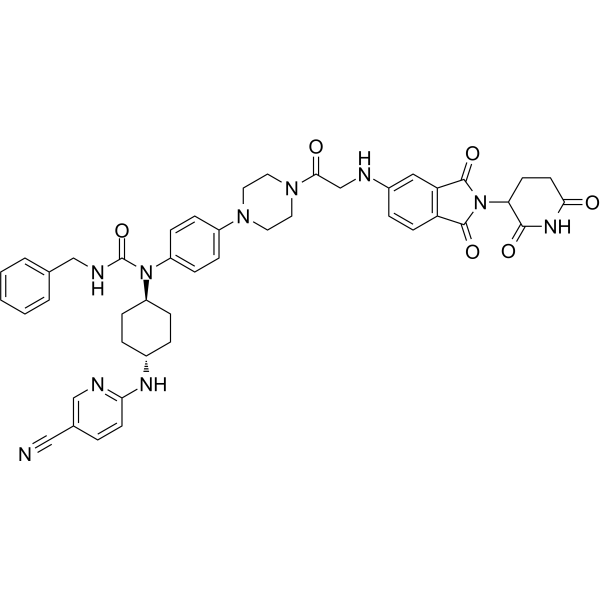
GC69747
PROTAC CDK12/13 Degrader-1
PROTAC CDK12/13 Degrader-1 (7f) is a highly efficient and selective dual degrader of cell cycle-dependent kinases CDK12/CDK13, with DC50 values of 2.2 nM and 2.1 nM, respectively. PROTAC CDK12/13 Degrader-1 has anti-proliferative activity and can be used for breast cancer research.
-
GC62666
PROTAC CDK2/9 Degrader-1
PROTAC CDK2/9 Abbauer-1 (Verbindung F3) ist ein potenter dualer Abbauer fÜr CDK2 (DC50=62 nM) und CDK9 (DC50=33 nM). PROTAC CDK2/9 Degrader-1 unterdrÜckt die Prostatakrebs-PC-3-Zellproliferation (IC50=0,12 μM), indem es den Zellzyklus in der S- und G2/M-Phase wirksam blockiert. PROTAC CDK2/9 Degrader-1 ist ein PROTAC, indem CDK-Inhibitor mit Cereblon-Ligand verknÜpft wird.
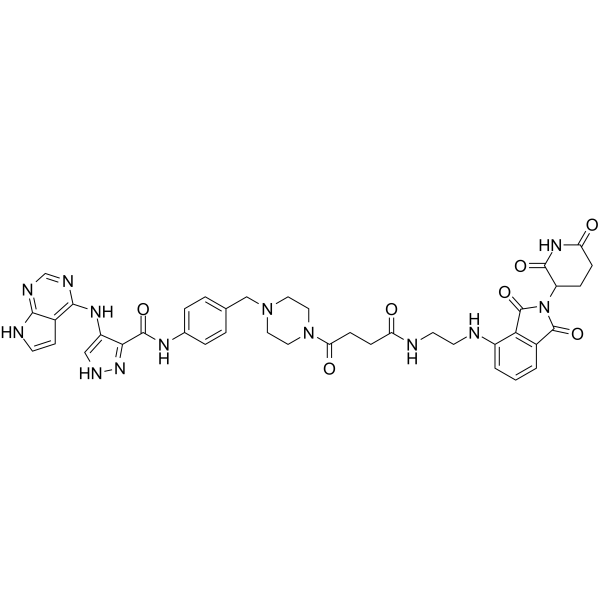
-
GC32845
PROTAC CDK9 Degrader-1
PROTAC CDK9 Degrader-1 ist ein PROTAC, das durch Liganden fÜr Cereblon und CDK als selektiver CDK9-Degrader verbunden ist.
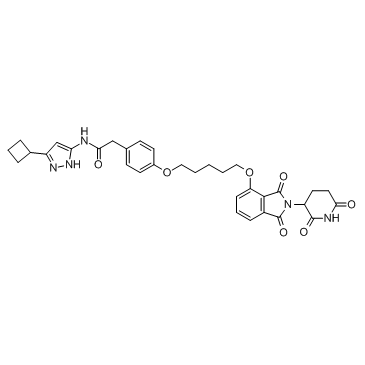
-
GC65147
PROTAC PARP1 degrader
PROTAC PARP1-Degrader ist ein PARP1-Degrader, der auf dem MDM2 E3-Liganden basiert. Es induziert eine signifikante PARP1-Spaltung und den programmierten Zelltod. PROTAC PARP1-Abbaumittel bei 10 μM bei 24 h hemmt die MDA-MB-231-Zelllinie mit einem IC50 von 6,12 μM.
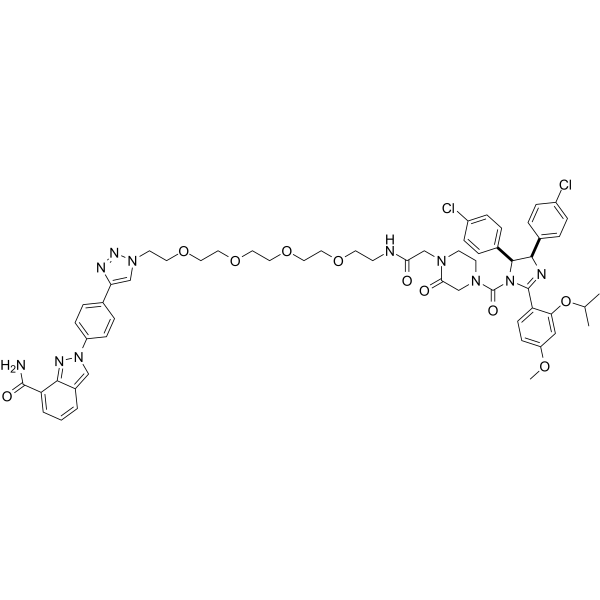
-
GC60310
Psammaplin A
Psammaplin A, ein mariner Metabolit, ist ein starker Inhibitor von HDAC und DNA-Methyltransferasen.
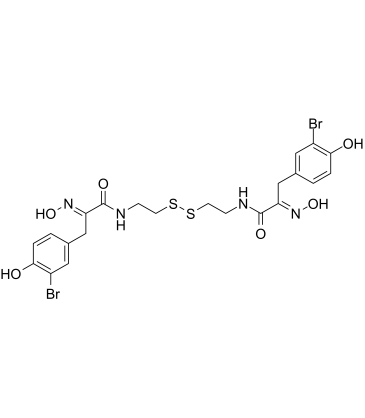
-
GC49053
Pseudomonic Acid (lithium salt)
An antibiotic
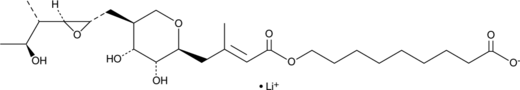
-
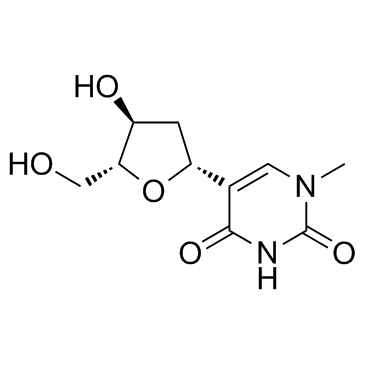
GC33944
Pseudothymidine (5-Methyl-2'-Deoxypseudouridin)
Pseudothymidin (5-Methyl-2'-Desoxypseudouridin) ist ein C-Nucleosid-Analogon von Thymidin.
-
GC38466
Pseudouridimycin
PUM
Pseudouridimycin (PUM), ein Antibiotikum, ist ein selektiver Inhibitor der bakteriellen RNA-Polymerase (RNAP).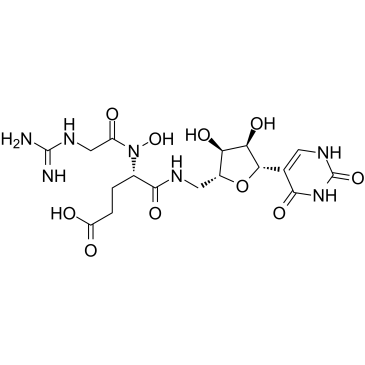
-
GC65372
Pseudouridine 5'-triphosphate trisodium
Pseudo-UTP trisodium
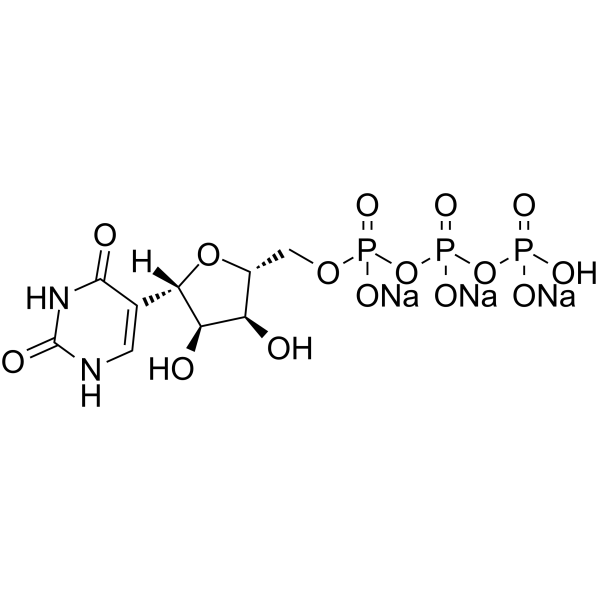
-
GC16367
PSI-7977
GI7977, Sofosbuvir
A prodrug form of PSI-7411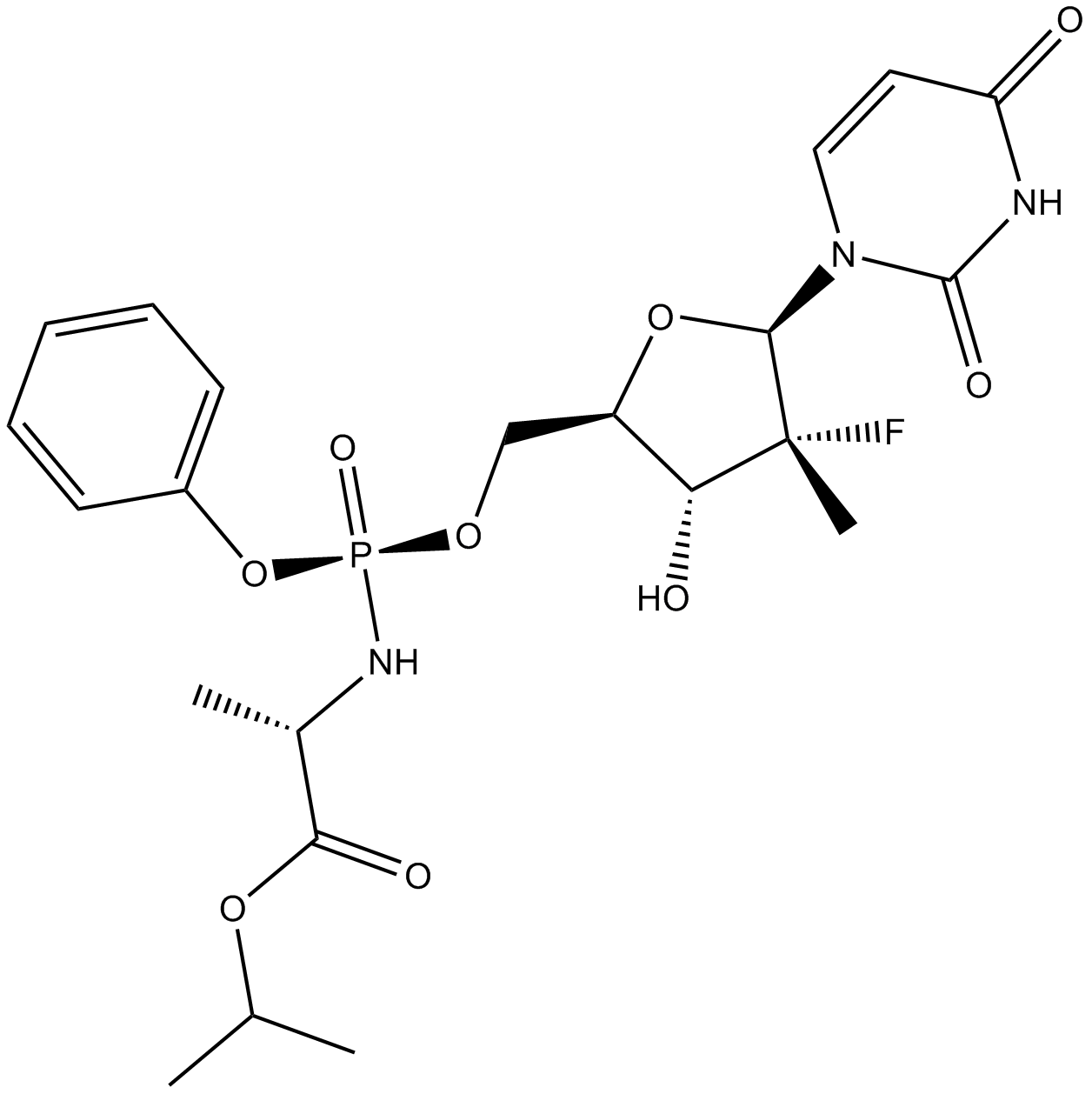
-
GC49914
Pt(II)-NHC Complex 2C
Platinum(II)-N-Heterocyclic Carbene Complex 2C
An inducer of immunogenic cancer cell death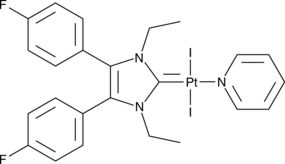
-
GC65335
PTC299
PTC-299
PTC299 ist ein oral aktiver Inhibitor der VEGFA-mRNA-Translation, der selektiv die VEGF-Proteinsynthese auf posttranskriptioneller Ebene hemmt. PTC299 ist auch ein starker Inhibitor der Dihydroorotat-Dehydrogenase (DHODH). PTC299 zeigt eine gute orale BioverfÜgbarkeit und das Fehlen einer Off-Target-Kinasehemmung und Myelosuppression. PTC299 kann fÜr die Erforschung hÄmatologischer Malignome nÜtzlich sein.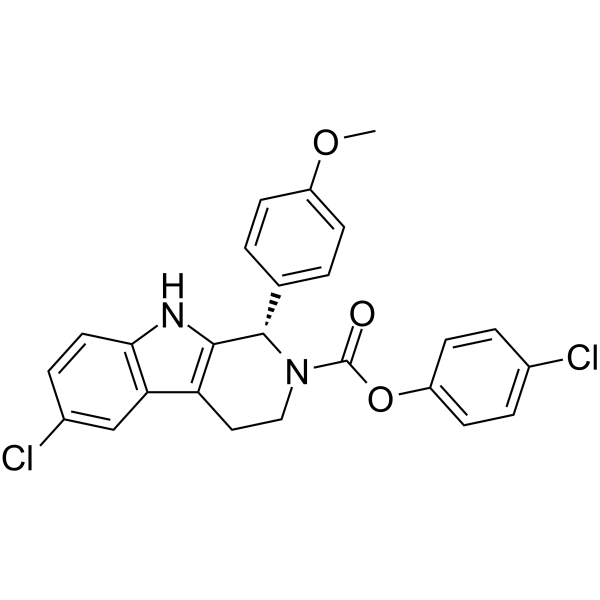
-
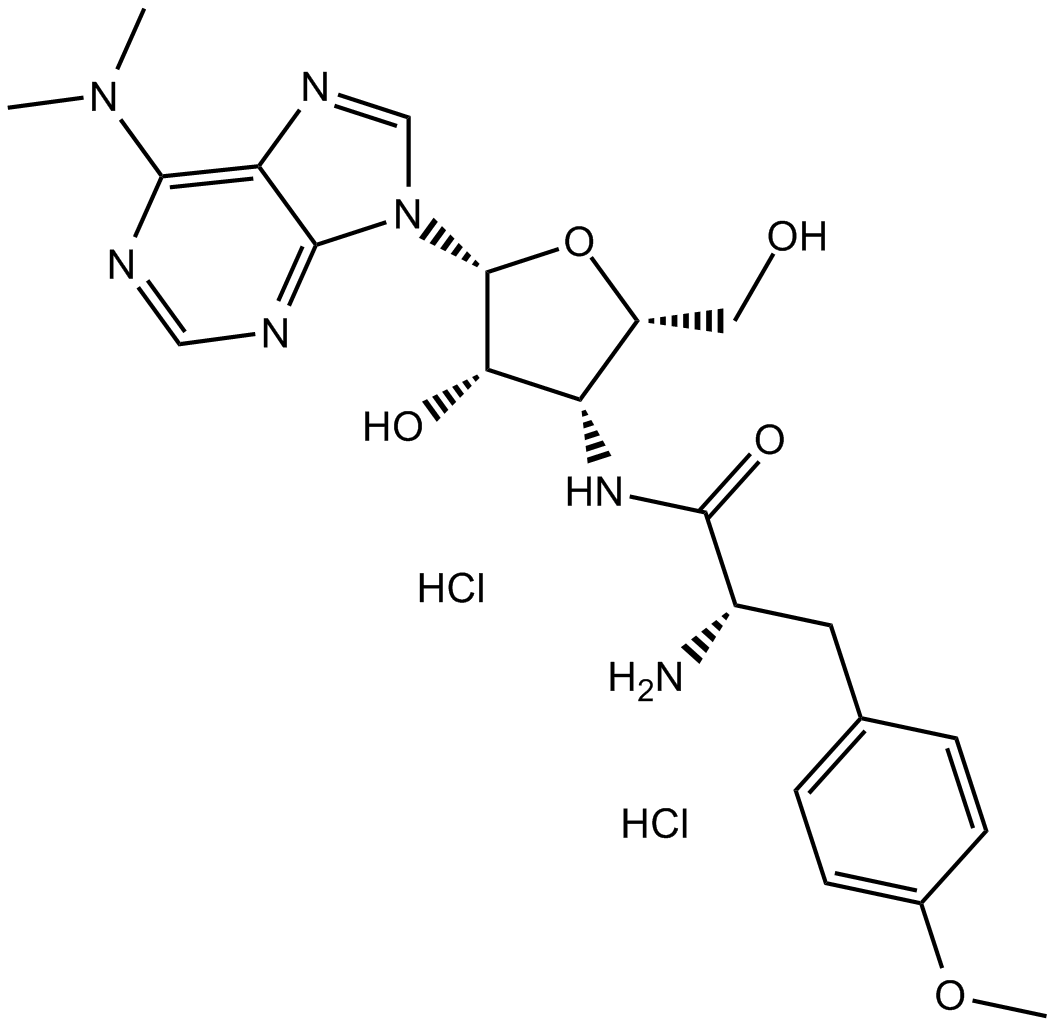
GC16384
Puromycin dihydrochloride
CL13900
Puromycin-Dihydrochlorid wird von Streptomyces alboniger, einem grampositiven Actinomyceten, durch eine Reihe von enzymatischen Reaktionen produziert.
-
GC14707
Purvalanol A
NG 60
Purvalanol A ist ein potenter CDK-Inhibitor, der cdc2-Cyclin B, cdk2-Cyclin A, cdk2-Cyclin E, cdk4-Cyclin D1 und cdk5-p35 mit IC50-Werten von 4, 70, 35, 850 bzw. 75 nM hemmt.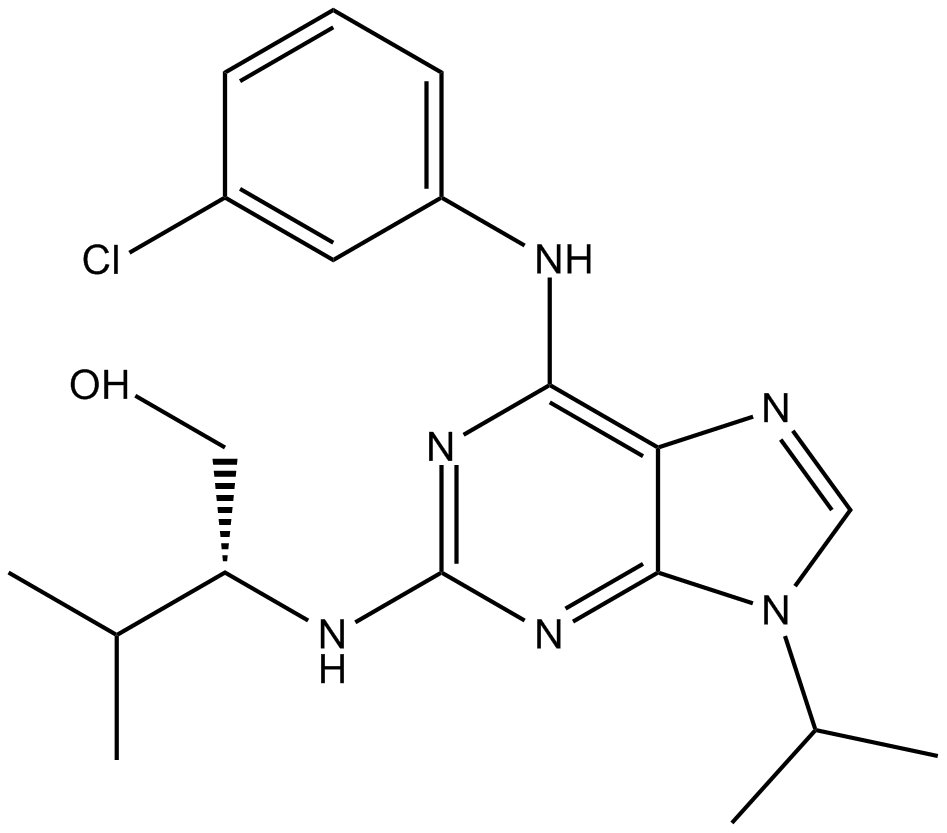
-
GC16268
Purvalanol B
NG 95; NG95; NG-95
Purvalanol B (NG 95) ist ein potenter, selektiver, reversibler und ATP-kompetitiver CDK-Inhibitor mit IC50-Werten von 6 nM, 6 nM, 9 nM, 6 nM fÜr cdc2-Cyclin B, CDK2-Cyclin A, CDK2-Cyclin E und CDK5-p35.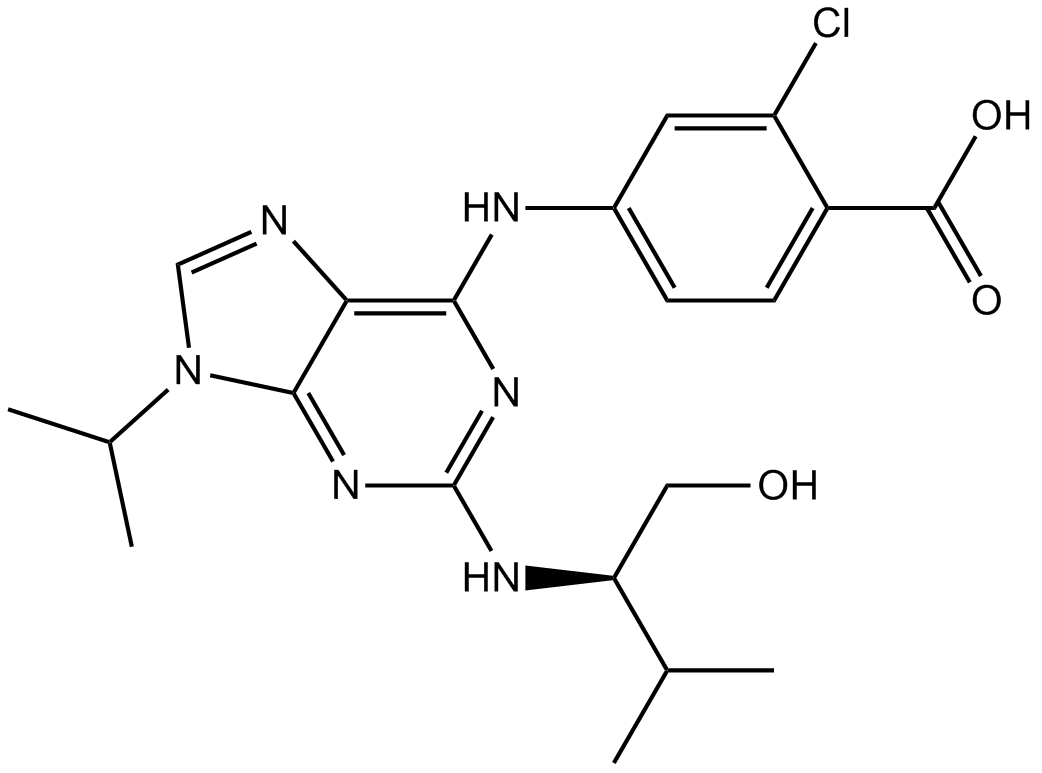
-
GC37042
Pyrazofurin
Pyrazofurin, ein Pyrimidin-Nucleosid-Analogon mit antineoplastischer AktivitÄt, hemmt die Zellproliferation und die DNA-Synthese in Zellen durch Hemmung der Uridin-5'-Phosphat (UMP)-Synthase. Pyrazofurin ist ein aktiver, sensitiver Orotat-Phosphoribosyltransferase-Inhibitor mit IC50-Werten zwischen 0,06 und 0,37 μM in den drei Plattenepithelkarzinom (SCC)-Zelllinien Hep-2, HNSCC-14B und HNSCC-14C.
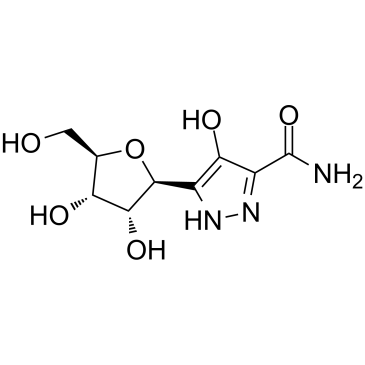
-
GC61224
Pyrazoloacridine
Pyrazoloacridin (NSC 366140), ein interkalierendes Mittel mit krebshemmender AktivitÄt, hemmt die AktivitÄt der Topoisomerasen 1 und 2. Pyrazoloacridin (NSC 366140) zeigt in myeloischen K562-LeukÄmiezellen einen IC50-Wert von 1,25 μM bei einer 24-stÜndigen Behandlung.
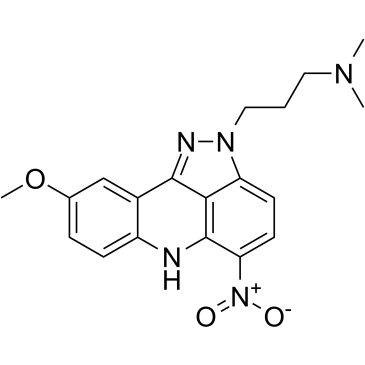
-
GC45552
Pyrenophorol
Pyrenophorol ist eine C9H12O3-Verbindung, die in geringer Ausbeute aus Kulturfiltraten des phytopathogenen Pilzes Stemphylium radicinum, C.
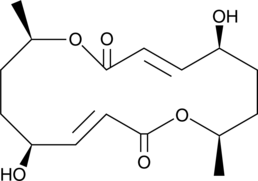
-
GC45553
Pyridoxine-d3 (hydrochloride)
Pyridoxol-d3 hydrochloride; Vitamin B6-d3 hydrochloride
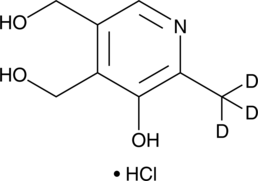
-
GC17867
Pyrimethamine
Darapram, Daraprim, Khloridin, NSC 3061
A dihydrofolate reductase inhibitor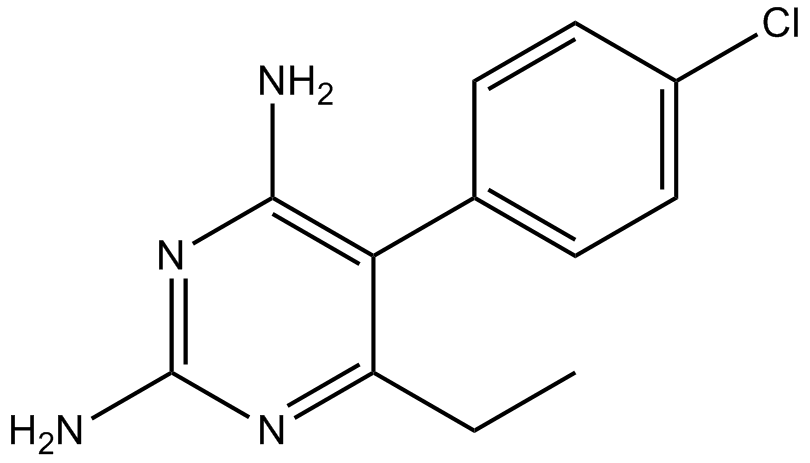
-
GC37044
Pyrindamycin A
Pyrindamycin A ist ein Antibiotikum, das die DNA-Synthese hemmt.
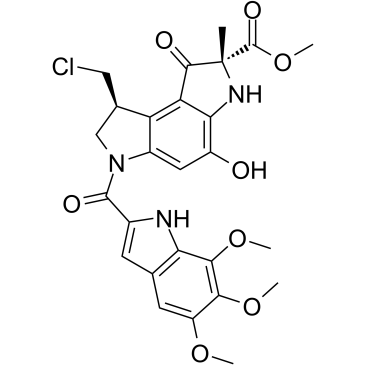
-
GC37051
Quarfloxin
CX-3543
Quarfloxin (CX-3543), ein Fluorchinolon-Derivat mit antineoplastischer AktivitÄt, zielt auf und hemmt die RNA-pol-I-AktivitÄt mit IC50-Werten im nanomolaren Bereich in Neuroblastomzellen. Quarfloxin unterbricht die Wechselwirkung zwischen dem Nucleolin-Protein und einer G-Quadruplex-DNA-Struktur in der ribosomalen DNA (rDNA)-Matrize.
-
GC61666
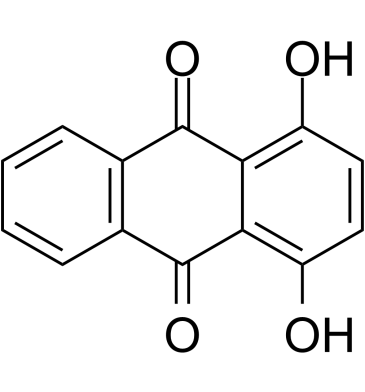
Quinizarin
Chinizarin (1,4-Dihydroxyanthrachinon), ein Bestandteil der Antikrebsmittel wie Doxorubicin, Daunorubicin und Adriamycin, interkaliert mit DNA durch Interkalationsmodus (Kd = 86,1 μM).
-
GC65207
R-10015
R-10015, eine antivirale Breitbandverbindung fÜr HIV-Infektionen, wirkt als potenter und selektiver Inhibitor der LIM-DomÄnenkinase (LIMK) und bindet an die ATP-Bindungstasche mit einem IC50 von 38 nM fÜr humanes LIMK1.
-
GC17400
R547
Ro 4584820
R547 ist ein potenter, selektiver und oral aktiver ATP-kompetitiver CDK-Inhibitor mit Kis von 2 nM, 3 nM und 1 nM fÜr CDK1/Cyclin B, CDK2/Cyclin E bzw. CDK4/Cyclin D1.
-
GC60317
RA-9
RA-9 ist ein potenter und selektiver Proteasom-assoziierter Deubiquitinating-Enzyme (DUBs)-Inhibitor mit gÜnstigem ToxizitÄtsprofil und AntikrebsaktivitÄt. RA-9 blockiert den Ubiquitin-abhÄngigen Proteinabbau, ohne die proteolytische AktivitÄt des 20S-Proteasoms zu beeintrÄchtigen. RA-9 induziert selektiv den Beginn der Apoptose in Eierstockkrebszelllinien und PrimÄrkulturen, die von Spendern stammen. RA-9 induziert Stressreaktionen des endoplasmatischen Retikulums (ER) in Eierstockkrebszellen.
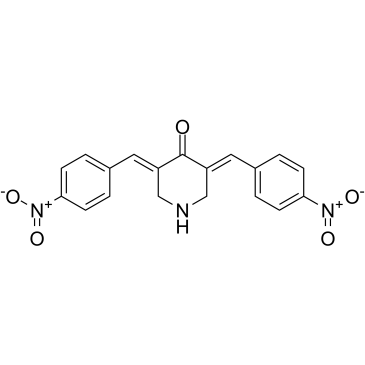
-
GC17750
Raltitrexed
ICI-D 1694, ZD 1694
Raltitrexed ist ein Antimetabolit-Medikament, das in der Chemotherapie verwendet wird und durch Hemmung der Thymidylat-Synthase wirkt.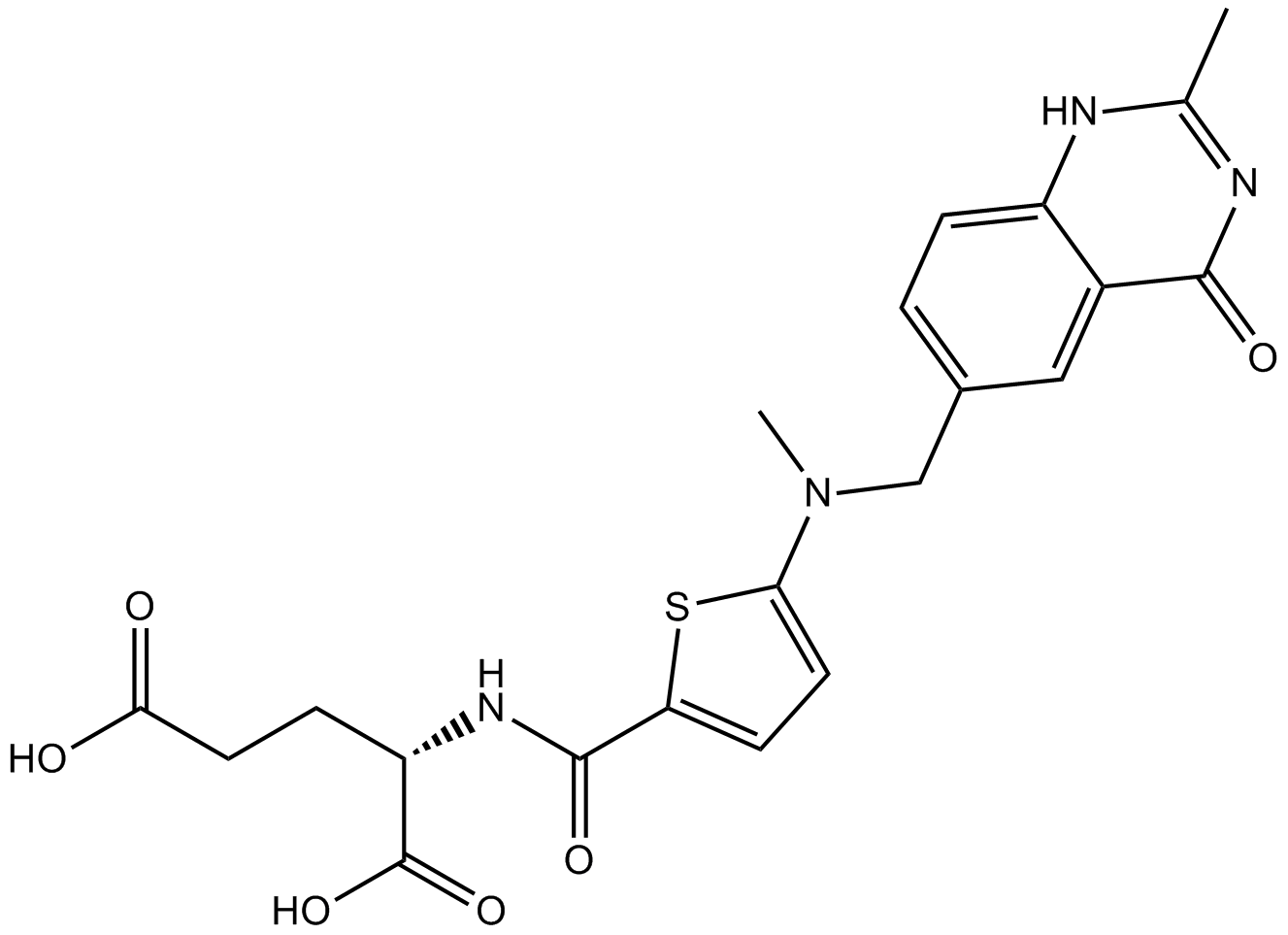
-
GC69804
RBN-3143
RBN-3143 ist ein wirksamer NAD+ kompetitiver katalytischer PARP14-Inhibitor mit einem IC50-Wert von 4 nM. RBN-3143 hemmt die ADP-Ribosylierung, die durch PARP14 vermittelt wird, und stabilisiert PARP14 in Zelllinien. RBN-3143 wird für die Erforschung von Lungenentzündungen eingesetzt.
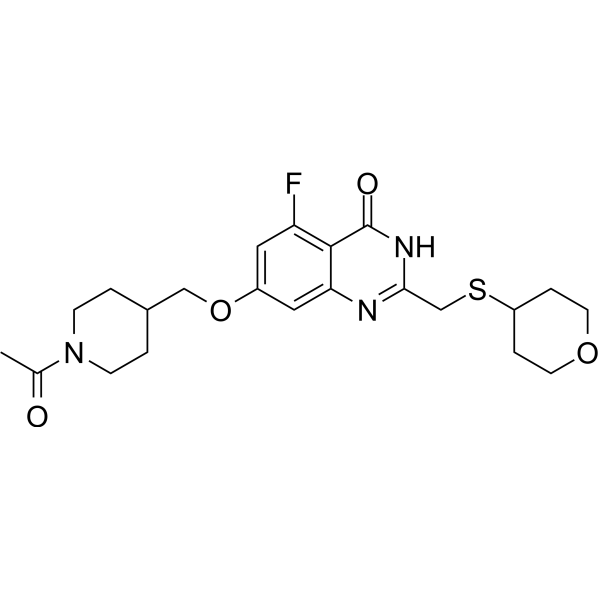
-
GC44809
Rebeccamycin
BRN 4732638, NSC 359079
Rebeccamycin, ein Antitumor-Antibiotikum, hemmt DNA-Topoisomerase I. Rebeccamycin scheint seine primÄre antineoplastische Wirkung durch Vergiftung von Topoisomerase I auszuÜben und hat eine vernachlÄssigbare Wirkung auf Proteinkinase C und Topoisomerase II.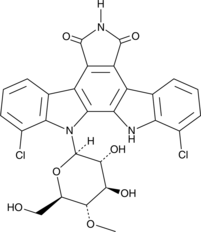
-
GC62639
rel-Zotatifin
rel-eFT226
rel-Zotatifin ist das racemische Isomer von Zotatifin und wirkt als eIF4A-Inhibitor mit einer geringeren AktivitÄt als Zotatifin.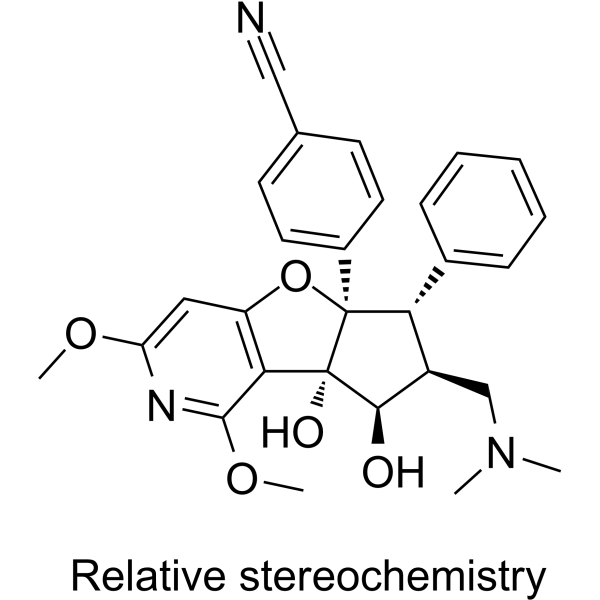
-
GC33290
Remetinostat (SHP-141)
SHP-141
Remetinostat (SHP-141) (SHP-141) ist ein auf HydroxamsÄure basierender Inhibitor der Histon-Deacetylase-Enzyme (HDAC), der zur Behandlung des kutanen T-Zell-Lymphoms entwickelt wird.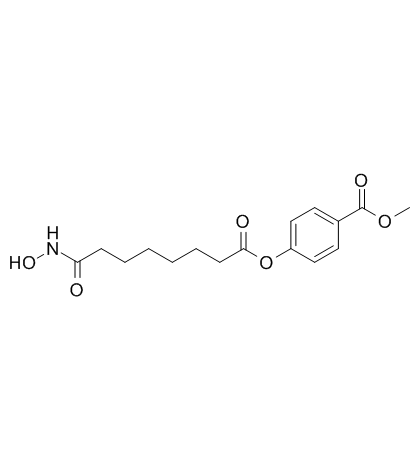
-
GC18751
Reticulol
6,8-dihydroxy-7-methoxy-3-methyl Isocoumarin, NSC 294978
Reticulol is an isocoumarin derivative produced by certain species of Streptomyces that inhibits cAMP phosphodiesterase (IC50 = 41 uM).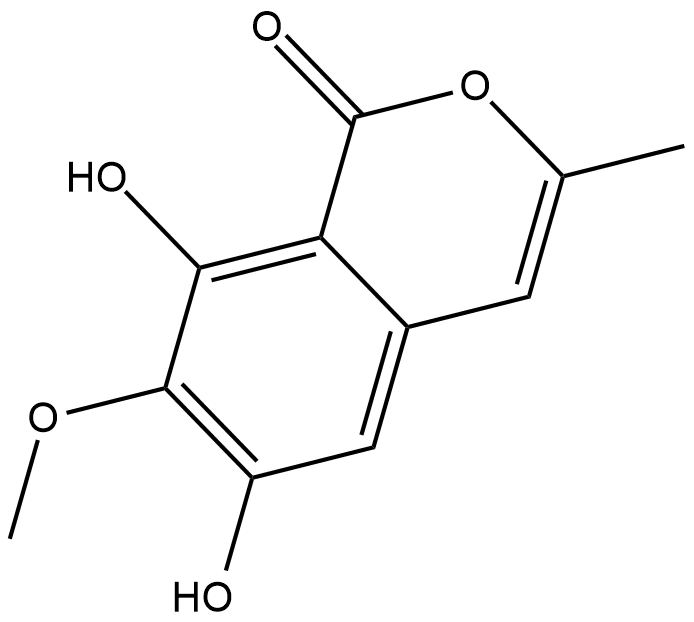
-
GC19309
RG7800
RO6885247
RG7800 ist ein SMN2-Spleißmodifikator.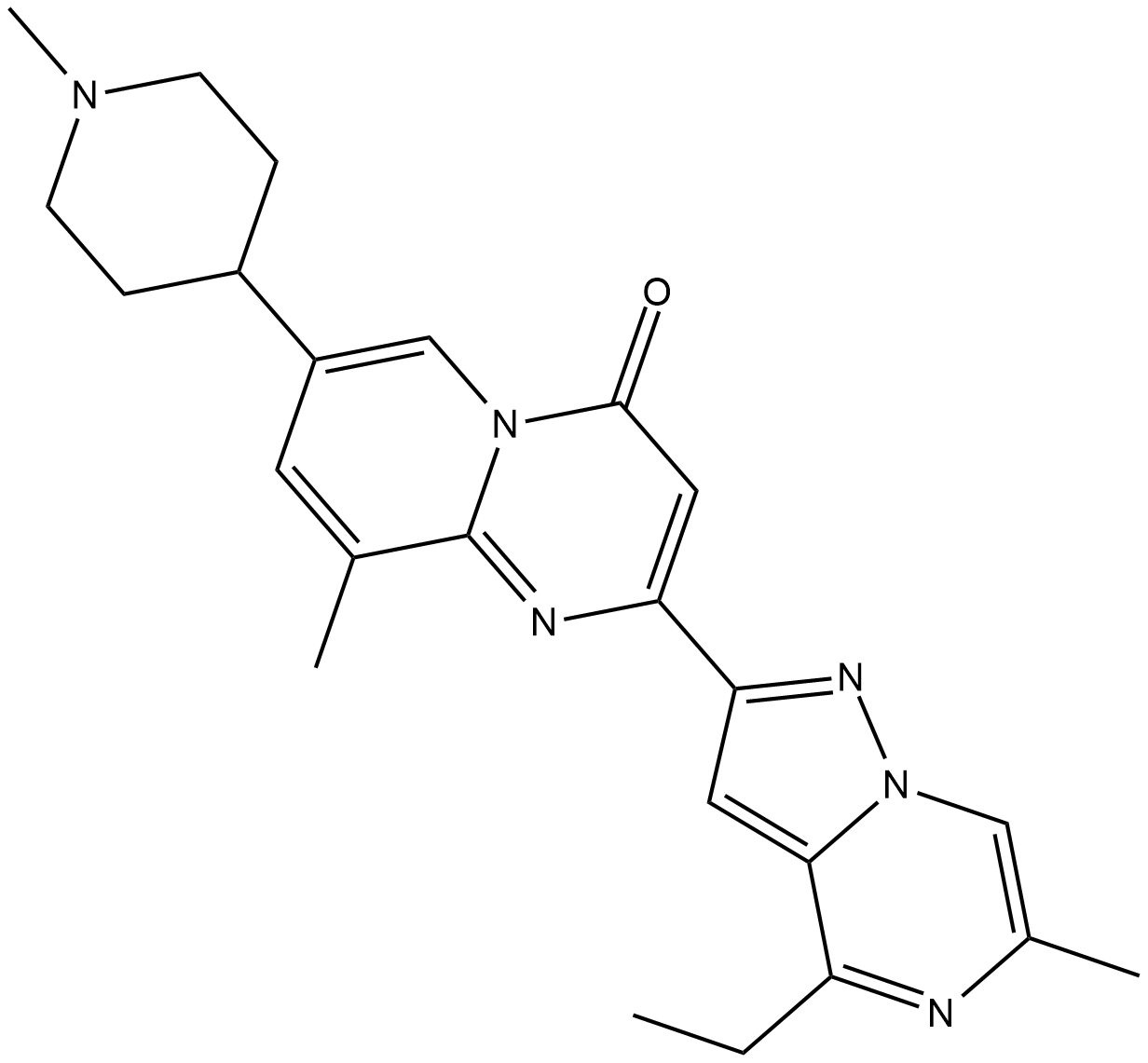
-
GC37521
RG7800 hydrochloride
RO6885247 hydrochloride
RG7800-Hydrochlorid ist ein oral aktiver SMN2-Splicing-Modulator mit EC1.5xs von 23 nM und 87 nM fÜr SMN2-Splicing und SMN-Protein; RG7800-Hydrochlorid hat das Potenzial, spinale Muskelatrophie zu behandeln.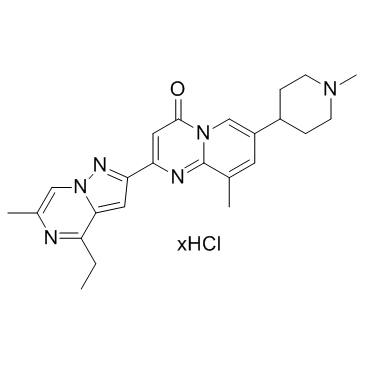
-
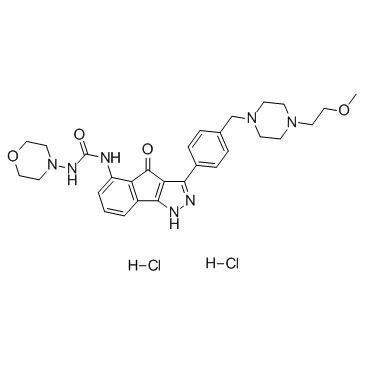
GC37522
RGB-286638
RGB-286638 ist ein CDK-Inhibitor, der die KinaseaktivitÄt von Cyclin T1-CDK9, Cyclin B1-CDK1, Cyclin E-CDK2, Cyclin D1-CDK4, Cyclin E-CDK3 und p35-CDK5 mit IC50-Werten von 1, 2, 3 hemmt , 4, 5 bzw. 5 nM; hemmt auch GSK-3β, TAK1, Jak2 und MEK1 mit IC50-Werten von 3, 5, 50 und 54 nM.
-
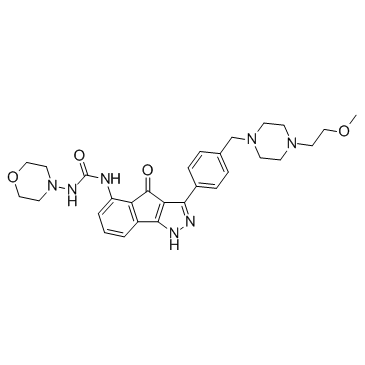
GC37523
RGB-286638 free base
RGB-286638 ist ein CDK-Inhibitor, der die KinaseaktivitÄt von Cyclin T1-CDK9, Cyclin B1-CDK1, Cyclin E-CDK2, Cyclin D1-CDK4, Cyclin E-CDK3 und p35-CDK5 mit IC50-Werten von 1, 2, 3 hemmt , 4, 5 bzw. 5 nM; hemmt auch GSK-3β, TAK1, Jak2 und MEK1 mit IC50-Werten von 3, 5, 50 und 54 nM.
-
GC14285
RGFP966
RGFP 966;RGFP-966
RGFP966 ist ein hochselektiver HDAC3-Inhibitor mit einem IC50 von 80 nM und zeigt keine Hemmung anderer HDACs bei Konzentrationen bis zu 15 μM. RGFP966 kann die Blut-Hirn-Schranke (BBB) durchdringen.
-
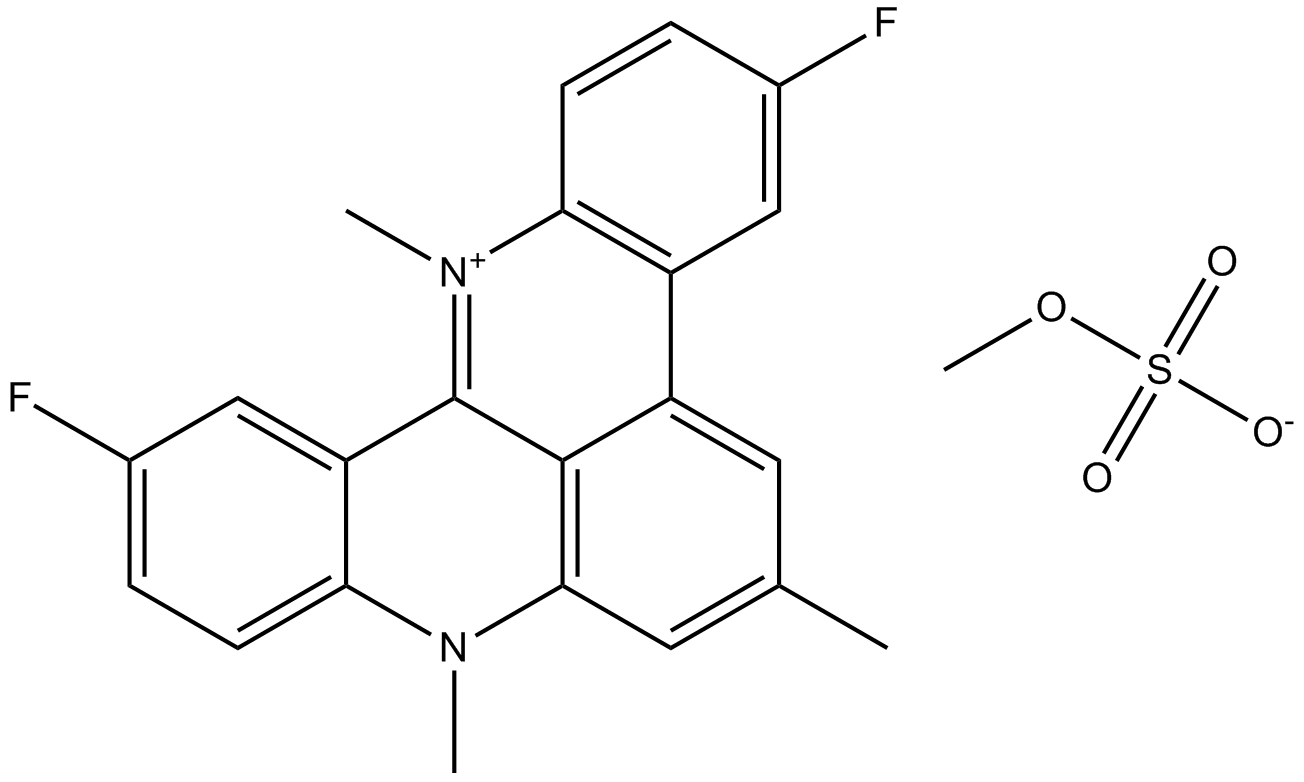
GC16485
RHPS4
RHPS4 ist ein potenter Telomerase-Inhibitor (IC50 = 0,33 μM). RHPS4 ist ein DNA-SchÄden-Induktor.
-
GC60324
Ribociclib D6
LEE011-d6
Ribociclib D6 (LEE011 D6) ist ein Deuterium mit der Bezeichnung Ribociclib. Ribociclib ist ein hochspezifischer CDK4/6-Inhibitor mit IC50-Werten von 10 nM bzw. 39 nM und über 1.000-mal weniger wirksam gegen den Cyclin B/CDK1-Komplex.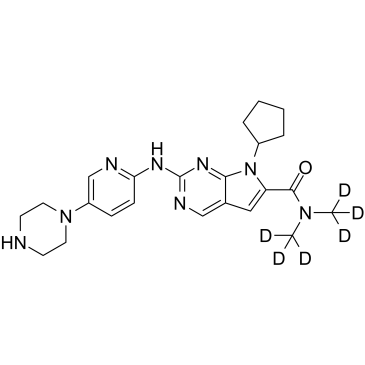
-
GC62638
Ribociclib D6 hydrochloride
LEE011-d6 hydrochloride
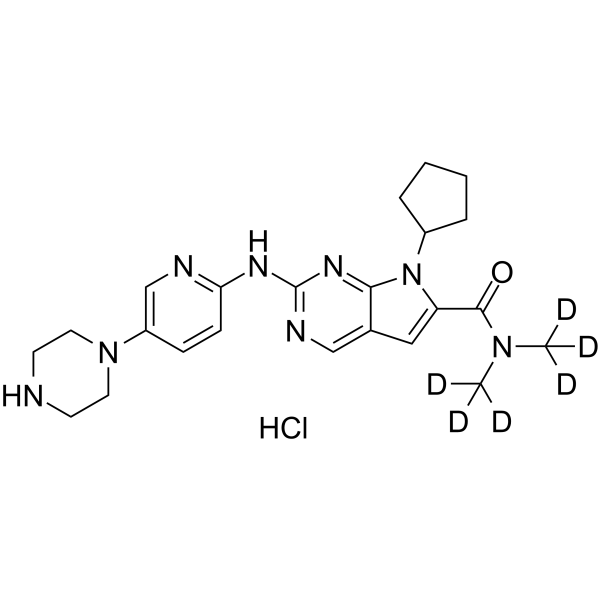
-
GC37529
Ribociclib succinate hydrate
Ribociclib-Succinathydrat (LEE011-Succinathydrat) ist ein hochspezifischer CDK4/6-Inhibitor mit IC50-Werten von 10 nM bzw. 39 nM und Über 1.000-mal weniger wirksam gegen den Cyclin B/CDK1-Komplex.
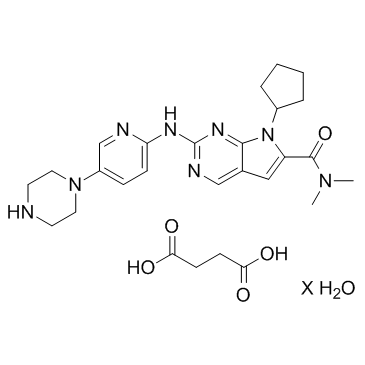
-
GC61246
Rifalazil
Rifalazil (KRM-1648; ABI-1648), ein Rifamycin-Derivat, hemmt die bakterielle DNA-abhÄngige RNA-Polymerase und tÖtet Bakterienzellen ab, indem es die β-Untereinheit in der RNA-Polymerase blockiert.
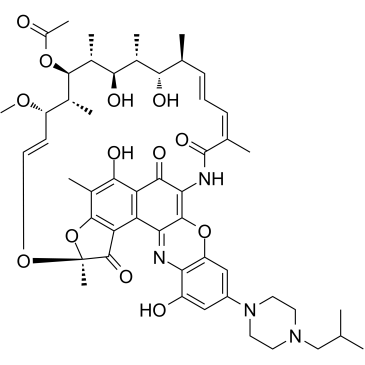
-
GC16970
Rifaximin (Xifaxan)
L 105SV, Rifamycin L 105
Rifaximin (Xifaxan), ein gastrointestinal-selektives Antibiotikum, bindet die ⋲-Untereinheit der bakteriellen DNA-abhÄngigen RNA-Polymerase, was zu einer Hemmung der bakteriellen RNA-Synthese fÜhrt.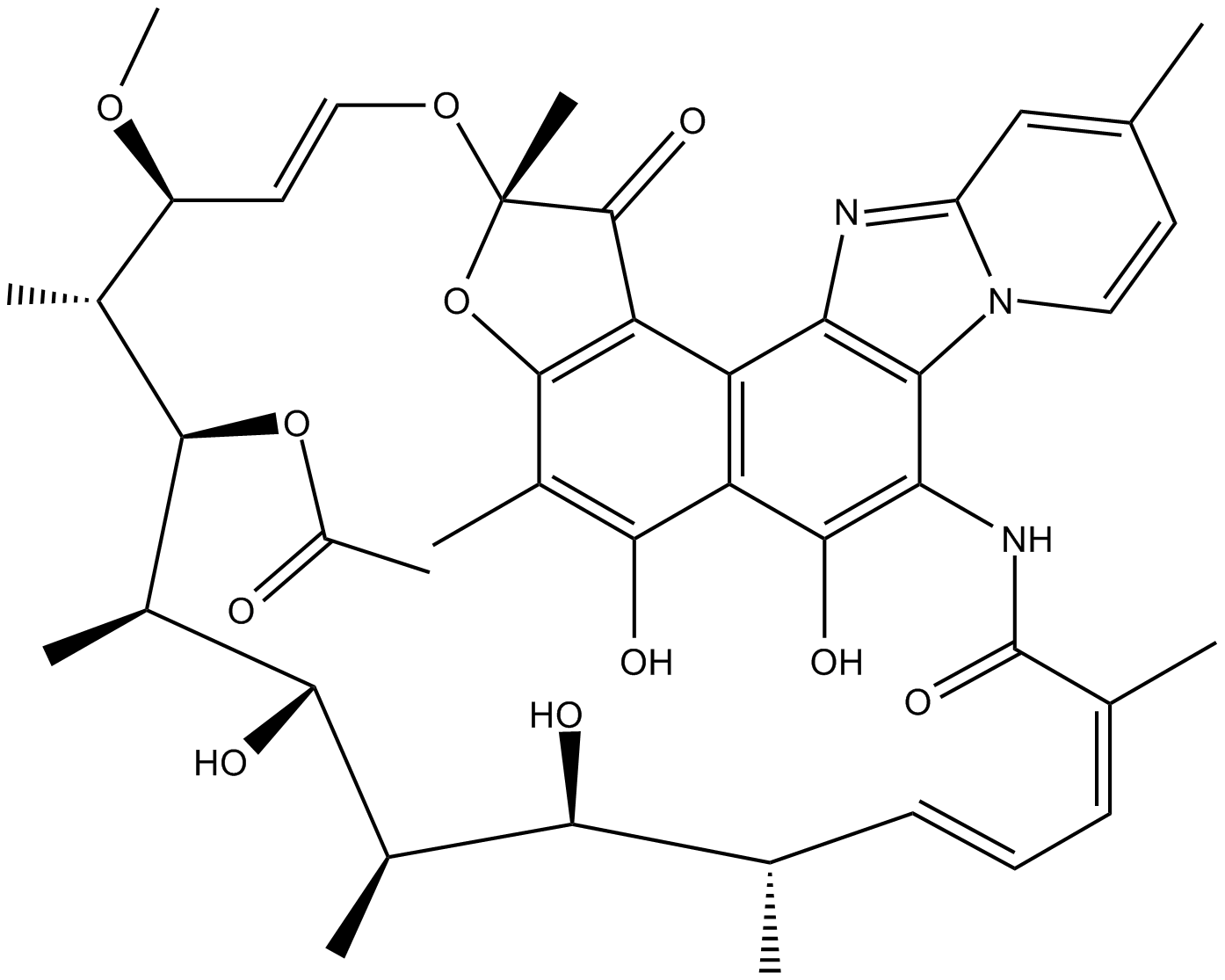
-
GC30845
Risdiplam (RG7916)
RG-7916, RO7034067
Risdiplam (RG7916) (RG7916) ist ein oral verabreichter, zentral und peripher verteilter SMN2-PrÄ-mRNA-Splicing-Modifikator, der die Überlebensproteinspiegel von Motoneuronen (SMN) erhÖht.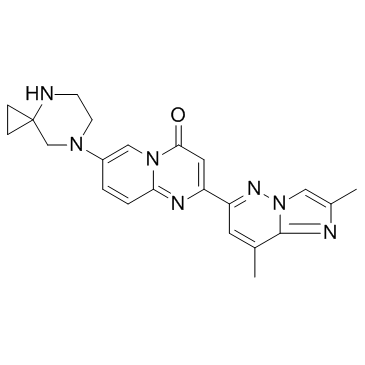
-
GC12793
RITA (NSC 652287)
2,5bis(5hydroxymethyl2thienyl) Furan, NSC 652287, Reactivation of p53 and Induction of Tumor Cell Apoptosis
An inhibitor of the p53-HDM-2 interaction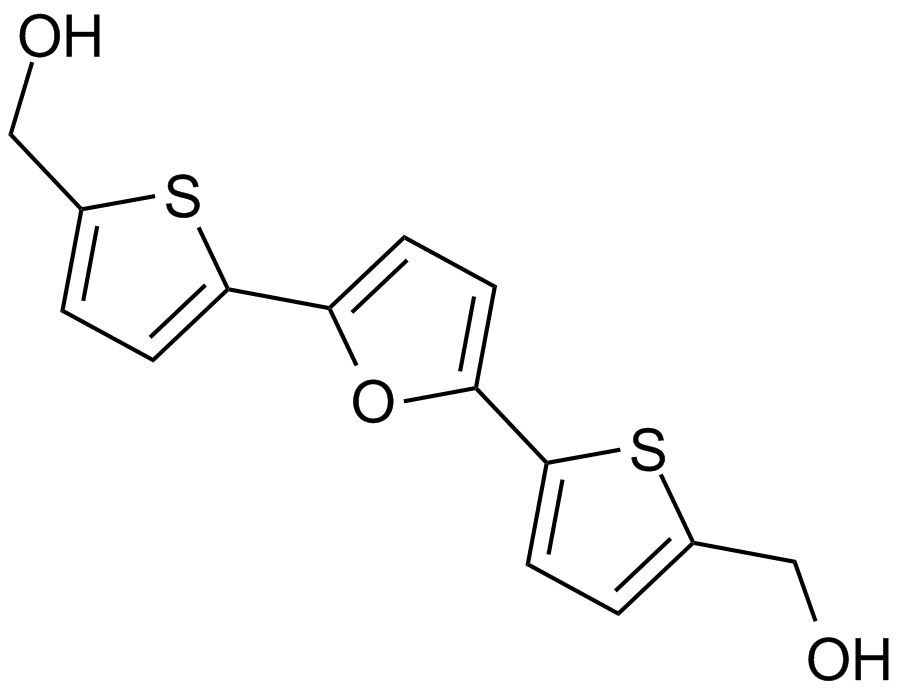
-
GC19505
RK-287107
RK-287107 ist ein potenter und spezifischer Tankyrase-Inhibitor mit IC50-Werten von 14,3 und 10,6 nM für Tankyrase-1 bzw. Tankyrase-2. RK-287107 blockiert das Wachstum von Darmkrebszellen.