Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Ziele für Cell Cycle/Checkpoint
- ATM/ATR(23)
- Aurora Kinase(17)
- Cdc42(4)
- Cdc7(3)
- Chk(14)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(77)
- E1 enzyme(1)
- G-quadruplex(11)
- Haspin(6)
- HMTase(1)
- Kinesin(23)
- Ksp(4)
- Microtubule/Tubulin(219)
- Mps1(15)
- Mitotic(7)
- RAD51(16)
- ROCK(60)
- Rho(13)
- PERK(10)
- PLK(33)
- PTEN(6)
- Wee1(7)
- PAK(20)
- Arp2/3 Complex(8)
- Dynamin(10)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Produkte für Cell Cycle/Checkpoint
- Bestell-Nr. Artikelname Informationen
-
GA21951
H-4-Nitro-Phe-OEt . HCl
A building block

-
GC49305
H-Arg-Gly-Asp-Cys-OH (trifluoroacetate salt)
H-RGDC-OH
An RGD-containing tetrapeptide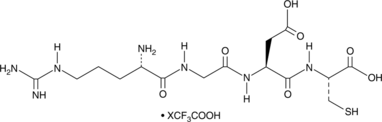
-
GA22303
H-D-Pro-Phe-Arg-pNA . 2 HCl
pFR-pNA, Plasma Kallikrein Chromogenic Substrate, D-Pro-Phe-Arg-p-nitroaniline
pFR-pNA, chromogenic substrate for the determination of plasma kallikrein-like activity. CAS Number (net): 64816-19-9.
-
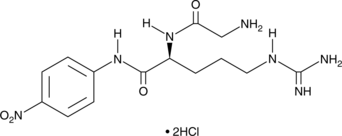
GC45471
H-Gly-Arg-pNA (hydrochloride)
Gly-Arg-4-NA, GR-pNA, GR p-nitroanilide
-
GC49605
H-Gly-Pro-Hyp-OH (acetate)
H-GP-Hyp-OH, Tripeptide-29
A peptide DPP-4 inhibitor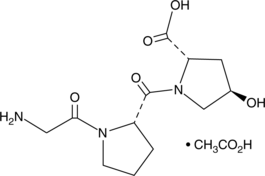
-
GC43803
HA-1077 (hydrochloride)
Fasudil
Fasudil (HA-1077; AT877) dihydrochloride is a nonspecific RhoA/ROCK inhibitor and also has inhibitory effect on protein kinases, with an Ki of 0.33 μM for ROCK1, IC50s of 0.158 μM and 4.58 μM, 12.30 μ ;M, 1.650 μM fÜr ROCK2 bzw. PKA, PKC, PKG. HA-1077 (Hydrochlorid) ist auch ein potenter Ca2+-Kanalantagonist und Vasodilatator.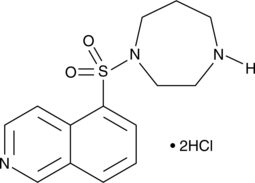
-
GC43805
HAPC-Chol
HAPC-Chol is a cationic cholesterol.
-
GC49855
Harmalol (hydrochloride hydrate)
11-hydroxy Harmalan, Harmidol
A β-carboline alkaloid and an active metabolite of harmaline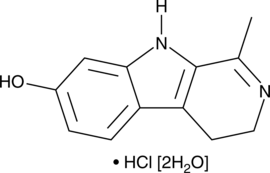
-
GC49915
Hexa-D-Arginine (trifluoroacetate salt)
Furin Inhibitor II, Hexa-D-Arg-NH2, Hexa-D-Arginine amide, D6R-NH2, (D)RRRRRR-NH2
A furin inhibitor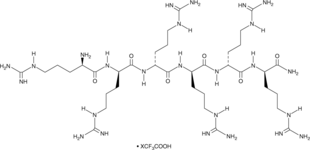
-
GC45639
Hexapeptide-11 (acetate)
FVAPFP, Phe-Val-Ala-Pro-Phe-Pro
A synthetic hexapeptide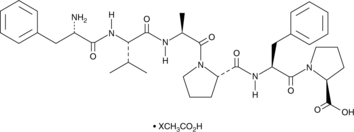
-
GC43832
Histone H3 (21-44)-GK-biotin amide (trifluoroacetate salt)
ATKAARKSAPSTGGVKKPHRYRPG-GK(Biotin)-NH2, Histone H3 (21-44)
Histone H3 (21-44)-GK-biotin is a peptide fragment of histone H3 that corresponds to amino acid residues 22-45 of the human histone H3.3 sequence and is biotinylated via a C-terminal GK linker.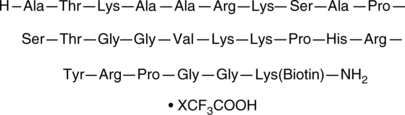
-
GC13928
HMN-214
IVX-214
HMN-214, ein oral bioverfÜgbares Prodrug von HMN-176, ist ein Inhibitor der Polo-Ähnlichen Kinase-1 (plk1) mit AntitumoraktivitÄt.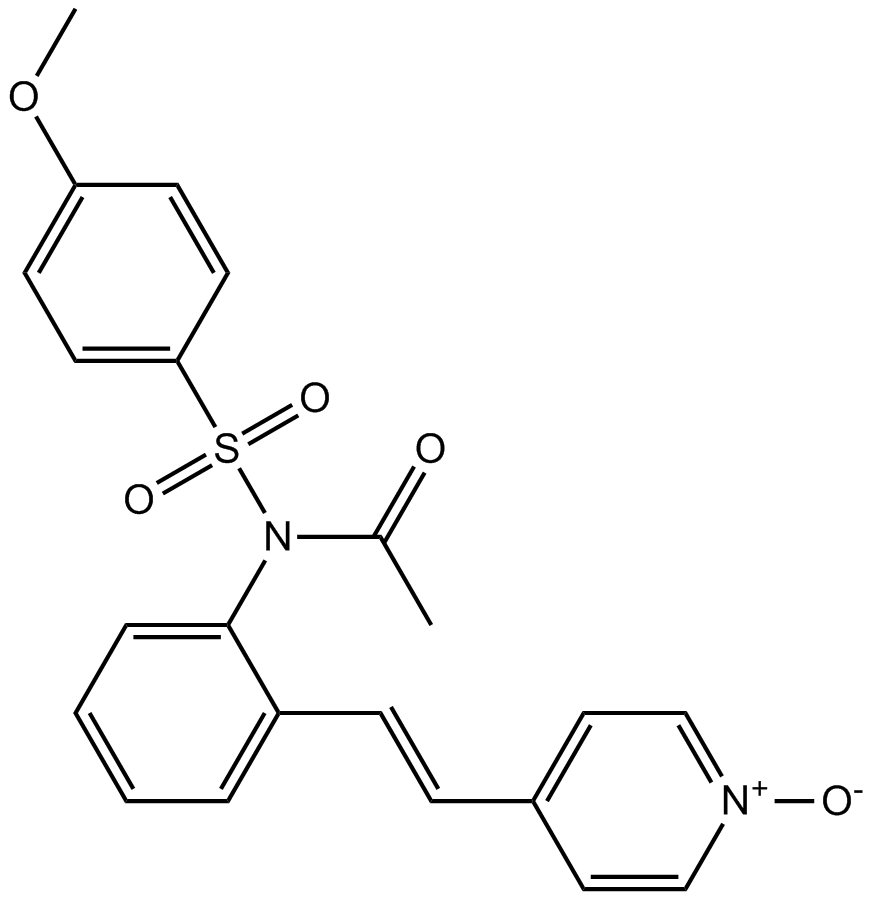
-
GC62594
hnRNPK-IN-1
hnRNPK-IN-1 ist ein heterogenes nukleÄres Ribonukleoprotein K (hnRNPK)-bindender Ligand mit Kd-Werten von 4,6 μM und 2,6 μM, gemessen mit SPR bzw. MST. hnRNPK-IN-1 hemmt die c-myc-Transkription durch Unterbrechung der Bindung von hnRNPK und c-myc-Promotor. hnRNPK-IN-1 induziert Hela-Zellen-Apoptose und hat starke Anti-Tumor-AktivitÄten.
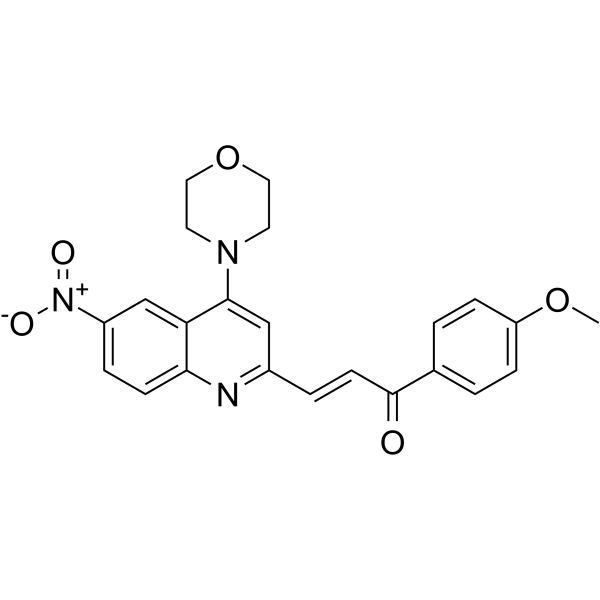
-
GC49742
Hsf1 Monoclonal Antibody (Clone 10H8)
For immunochemical analysis of Hsf1

-
GC47436
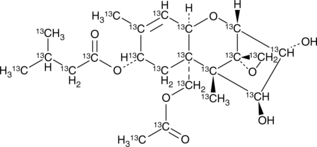
HT-2 Toxin-13C22
An internal standard for the quantification of HT-2 toxin
-
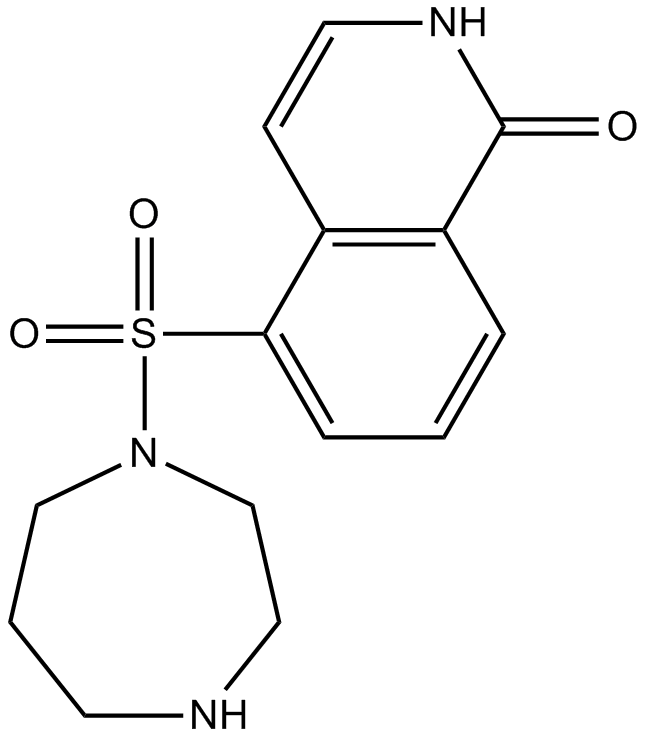
GC10901
Hydroxyfasudil
-
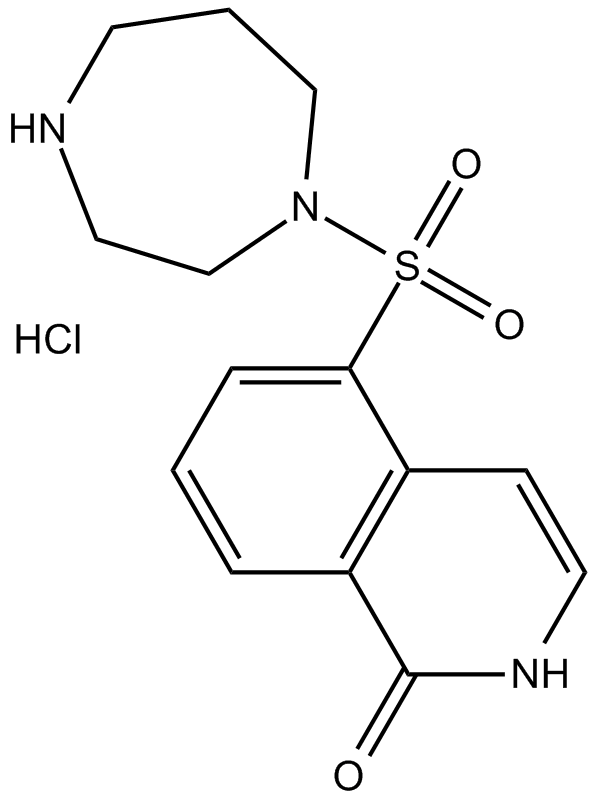
GC10338
Hydroxyfasudil hydrochloride
A ROCK inhibitor
-
GC32912
IBR2
IBR2 ist ein potenter und spezifischer RAD51-Inhibitor und hemmt die RAD51-vermittelte DNA-Doppelstrangbruchreparatur. IBR2 unterbricht die RAD51-Multimerisierung, beschleunigt den Proteasom-vermittelten RAD51-Proteinabbau, hemmt das Wachstum von Krebszellen und induziert Apoptose.
-
GC45743
ICAAc
3-amino-6-Isocyanoacridine
A solvatochromic fluorophore and pH probe
-
GC34159
Ilorasertib (ABT-348)
ABT-348
Ilorasertib (ABT-348) (ABT-348) ist ein potenter, oral aktiver und ATP-kompetitiver Aurora-Inhibitor mit IC50-Werten von 116, 5, 1 nM fÜr Aurora A, Aurora B bzw. Aurora C. Ilorasertib (ABT-348) ist auch ein starker VEGF- und PDGF-Hemmer. Ilorasertib (ABT-348) hat das Potenzial fÜr die Erforschung der akuten myeloischen LeukÄmie (AML) und des myelodysplastischen Syndroms (MDS).
-
GC38519
Ilorasertib hydrochloride
ABT-348 hydrochloride
Ilorasertib (ABT-348)-Hydrochlorid ist ein potenter, oral aktiver und ATP-kompetitiver Aurora-Inhibitor mit IC50-Werten von 116, 5, 1 nM fÜr Aurora A, Aurora B bzw. Aurora C. Ilorasertibhydrochlorid ist auch ein starker VEGF- und PDGF-Hemmer. Ilorasertib-Hydrochlorid hat das Potenzial fÜr die Erforschung der akuten myeloischen LeukÄmie (AML) und des myelodysplastischen Syndroms (MDS).
-
GC36308
Indibulin
D-24851, ZIO 301
Indibulin (ZIO 301), ein oral anwendbarer Inhibitor des Tubulinaufbaus, zeigt eine starke AntikrebsaktivitÄt mit minimaler NeurotoxizitÄt.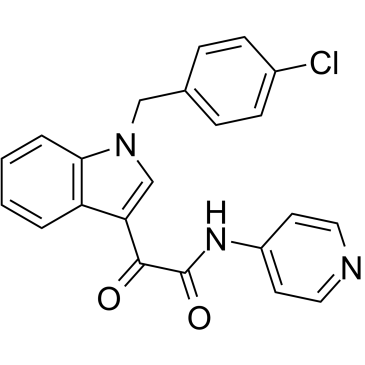
-
GC52513
Indolimine-214
Isoamylindolimine
A genotoxic gut microbiota metabolite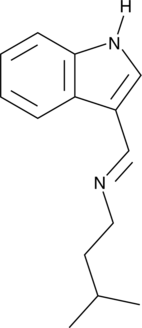
-
GC49460
ING-2 AM
ANG-2 Acetoxymethyl ester, ANG-2 AM, Asante Natrium Green-2 Acetoxymethyl ester, Asante Natrium Green-2 AM, ING-2 Acetoxymethyl ester, ION Natrium Green-2 Acetoxymethyl ester, ION Natrium Green-2 AM
A cell-permeable fluorescent sodium indicator
-
GC10334
INH6
INH6 ist ein potenter Nek2/Hec1-Inhibitor; hemmt das Wachstum von HeLa-Zellen mit einem IC50 von 2,4 μM.
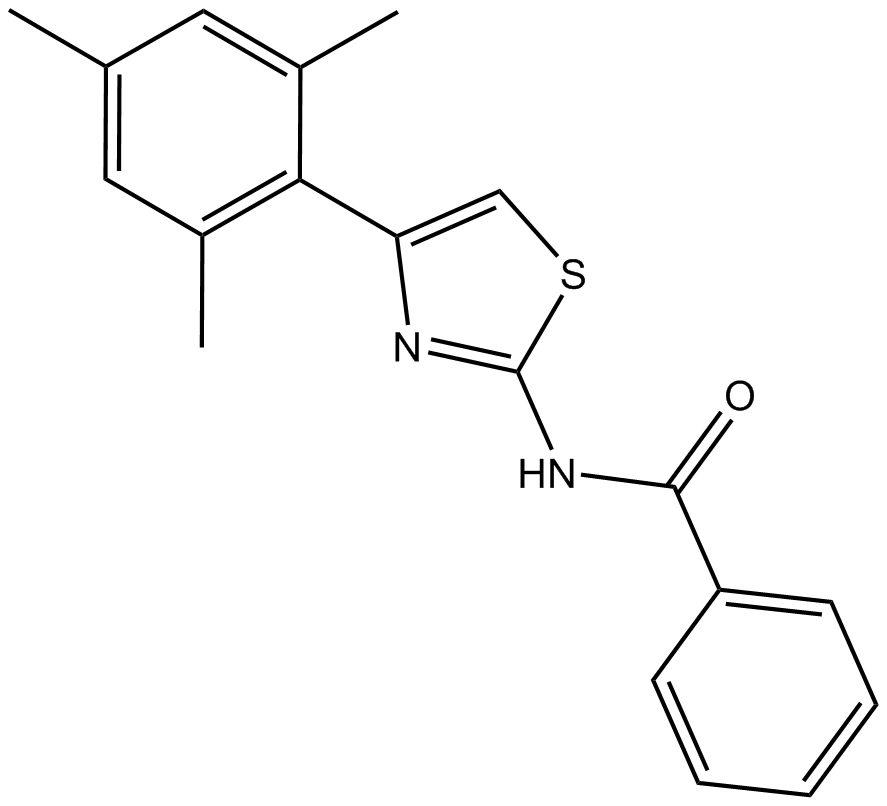
-
GC48387
Inostamycin A
A bacterial metabolite with anticancer activity
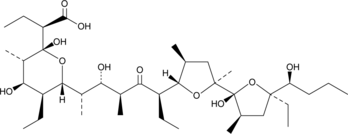
-
GC52472
Inostamycin A (sodium salt)
Inostamycin
A bacterial metabolite with anticancer activity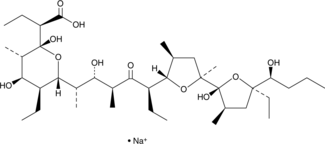
-
GC16225
IPA-3
IPA-3 ist ein selektiver nicht-ATP-kompetitiver PAK1-Inhibitor mit einem IC50-Wert von 2,5 μM und zeigt keine Hemmung von PAKs der Gruppe II (PAKs 4-6).
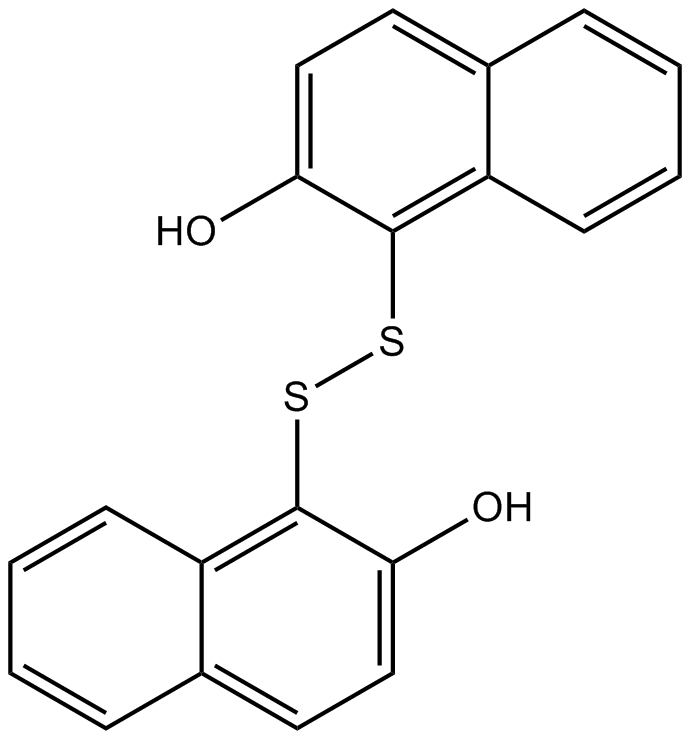
-
GC49468
IPG-1 AM
APG-1 AM, Asante Potassium Green–1 AM, ION Potassium Green–1 AM, IPG-1 Acetoxymethyl ester
A cell-permeable fluorescent potassium indicator
-
GC49469
IPG-1 TMA
APG-1, Asante Potassium Green-1, ION Potassium Green-1
A cell-impermeable fluorescent potassium indicator
-
GC49463
IPG-2 AM
APG-2 Acetoxymethyl ester, APG-2 AM, Asante Potassium Green-2 Acetoxymethyl ester, Asante Potassium Green-2 AM, ION Potassium Green-2 Acetoxymethyl ester, ION Potassium Green-2 AM, IPG-2 Acetoxymethyl ester
A cell-permeable fluorescent potassium indicator
-
GC49466
IPG-2 TMA
APG-2, Asante Potassium Green–2, ION Potassium Green–2, IPG-2
A cell-impermeable fluorescent potassium indicator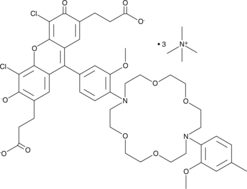
-
GC65188
IRES-C11
IRES-C11 ist ein spezifischer Translationsinhibitor der internen Ribosomeneintrittsstelle (IRES) von c-MYC. IRES-C11 blockiert die Wechselwirkung eines erforderlichen c-MYC-IRES-Transaktionsfaktors, des heterogenen nuklearen Ribonukleoproteins A1, mit seinem IRES. IRES-C11 hemmt BAG-1, XIAP und p53 IRESes nicht.
-
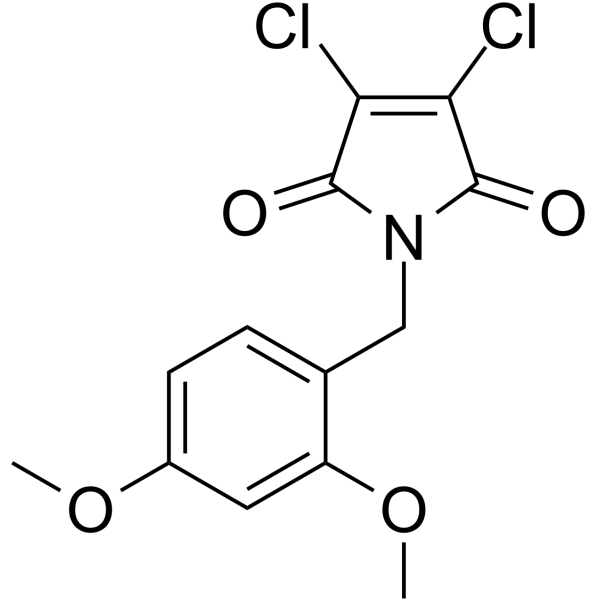
GC15215
Iso-Olomoucine
inactive stereoisomer of the Cdk5 inhibitor olomoucine
-
GC40901
Isogarcinol
Cambogin
Isogarcinol is a natural polyisoprenylated benzophenone first isolated from plant species in the genus Garcinia.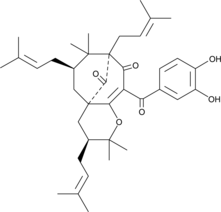
-
GC43917
Isopropyl Benzenesulfonate
Benzenesulfonic Acid isopropyl ester
Isopropyl benzenesulfonate is a sulfonate ester genotoxic impurity found in active pharmaceutical ingredients.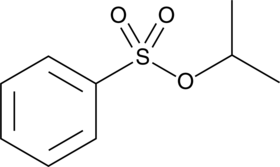
-
GC49142
Isorhoifolin
Apigenin-7-O-rutinoside
Isorhoifolin ist ein Flavonoidglykosid aus Periploca nigrescens-BlÄttern.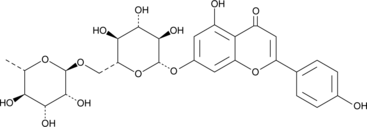
-
GC43922
Isovaleryl-L-carnitine (chloride)
CAR 5:0, C5:0 Carnitine, L-Carnitine isovaleryl ester, L-Isovalerylcarnitine
Isovaleryl-L-Carnitin (Chlorid), ein Produkt des Katabolismus von L-Leucin, ist ein starker Aktivator der Ca2+--abhÄngigen Proteinase (Calpain) menschlicher Neutrophiler.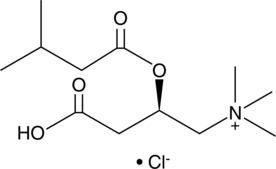
-
GC13394
Ispinesib (SB-715992)
SB-715992
Ispinesib (SB-715992) ist ein spezifischer Inhibitor des Kinesin-Spindelproteins (KSP) mit einem Ki-Wert von 1,7 nM.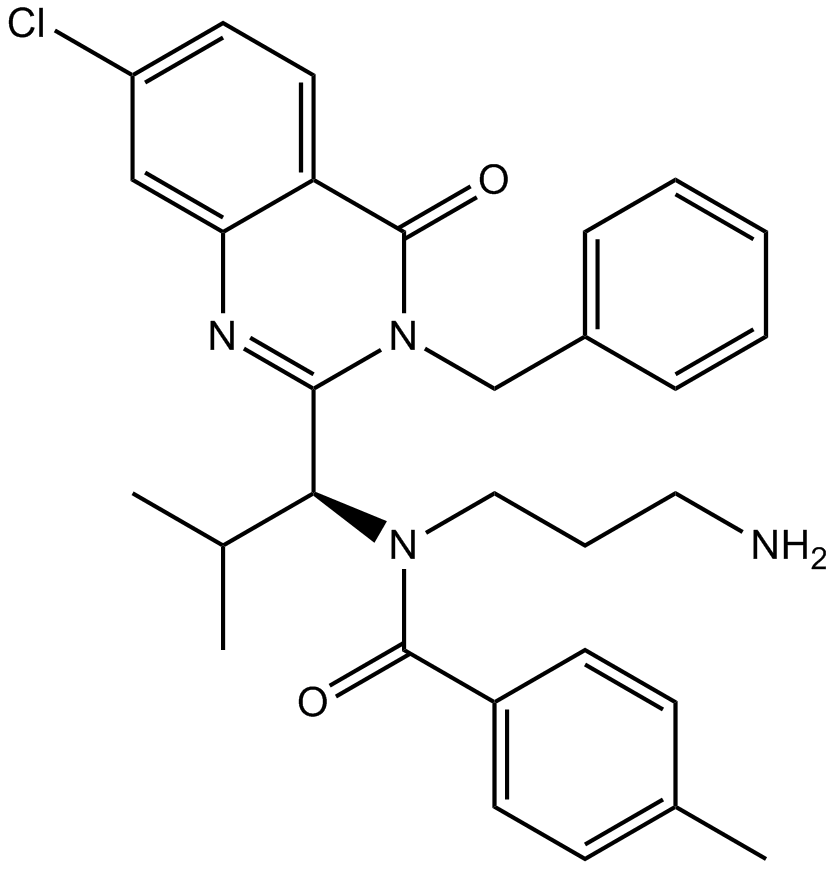
-
GC10581
ISRIB
PERK signaling inhibitor
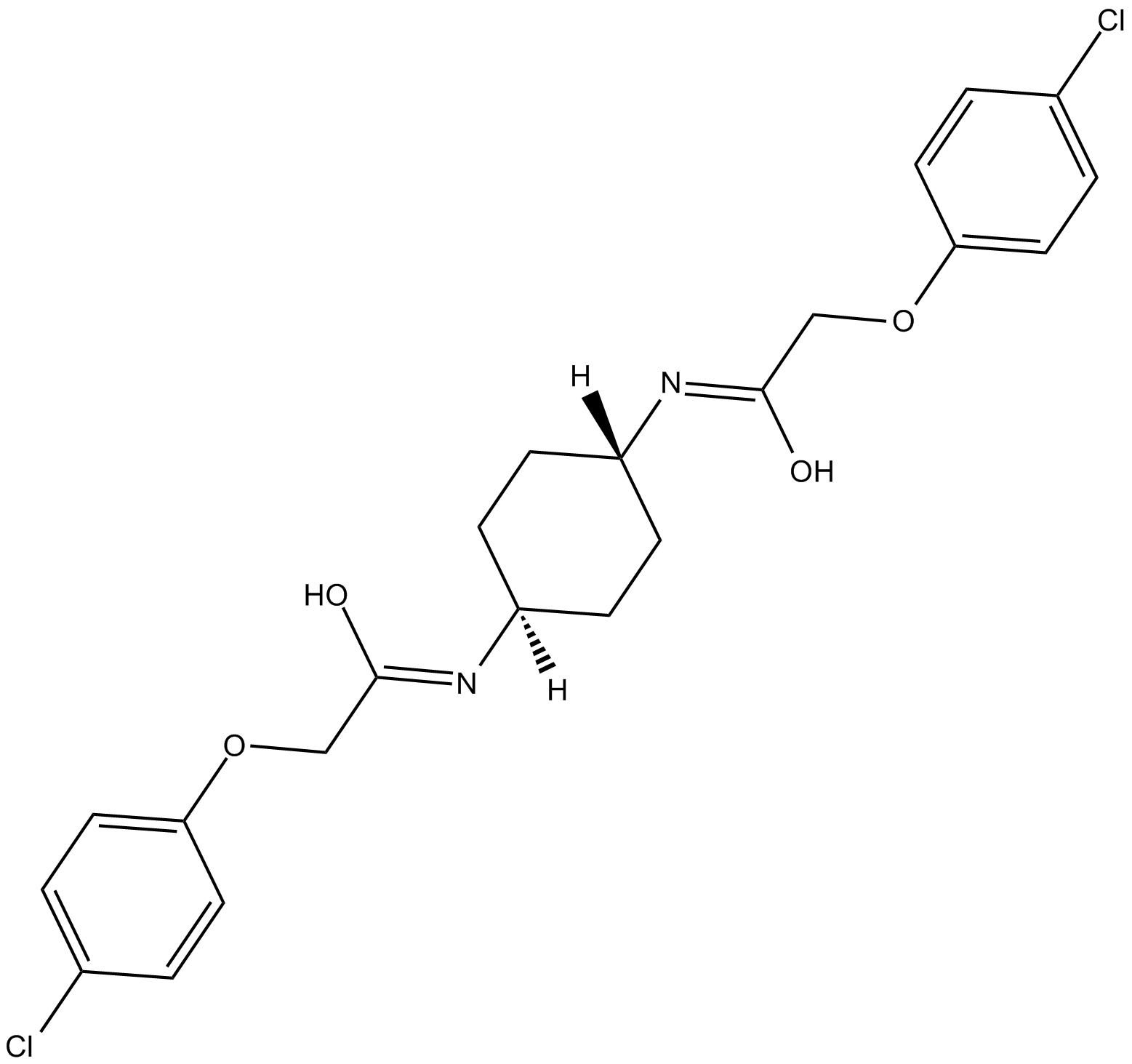
-
GC16869
Ixabepilone
Azaepothilone B, BMS-247550
A broad-spectrum anticancer agent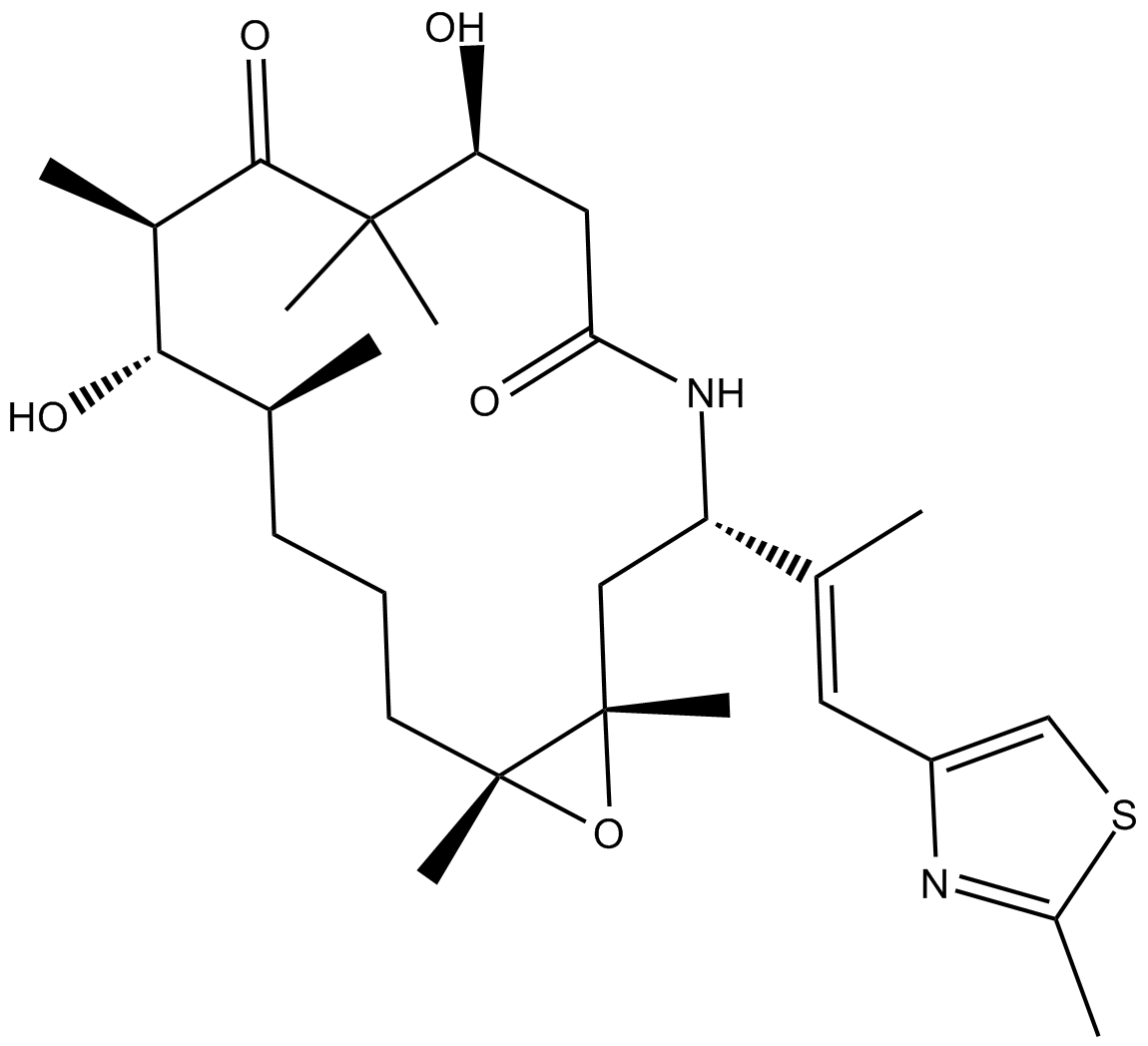
-
GC65331
IZCZ-3
IZCZ-3 ist ein potenter c-MYC-Transkriptionsinhibitor mit AntitumoraktivitÄt.
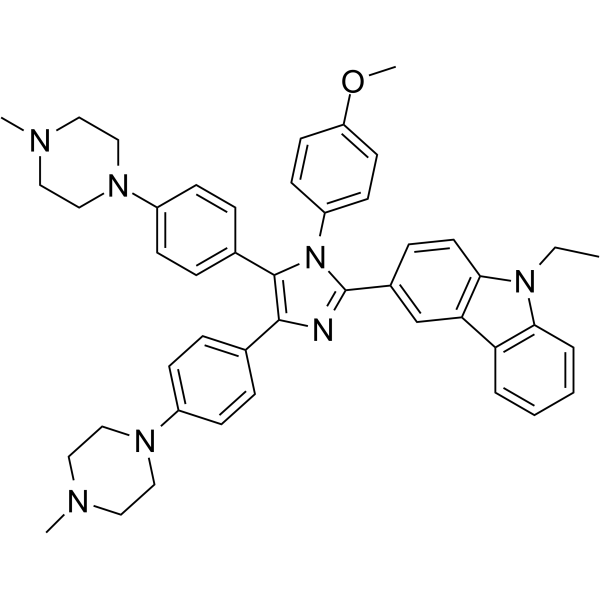
-
GC41645
Jacaric Acid
8(Z),10(E),12(Z)-Octadecatrienoic Acid
Jacaric acid is a conjugated polyunsaturated fatty acid first isolated from seeds of Jacaranda plants.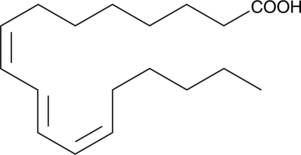
-
GC18699
JCP174
7-amino-4-chloro-3-Propoxyisocoumarin
JCP174 ist ein Inhibitor der Palmitoyl-Protein-Thioesterase-1 (TgPPT1), einer Depalmitoylase im Parasiten T.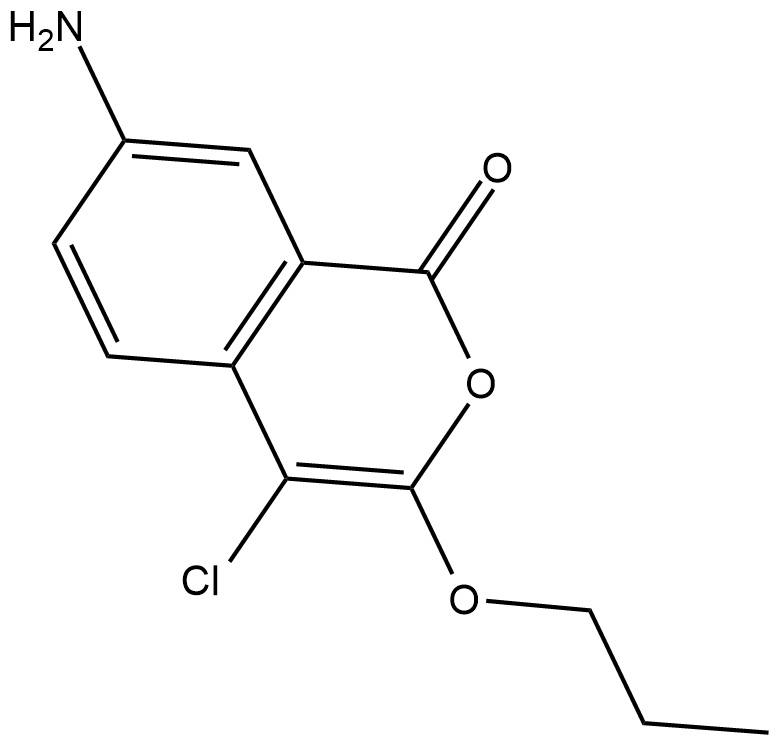
-
GC18734
JS-K
JS-K ist ein NO-Donor, der mit Glutathion reagiert, um NO bei physiologischem pH zu erzeugen. JS-K hemmt die Proliferation, induziert Apoptose und unterbricht den Zellzyklus von akuten lymphoblastischen Jurkat-T-LeukÄmiezellen.
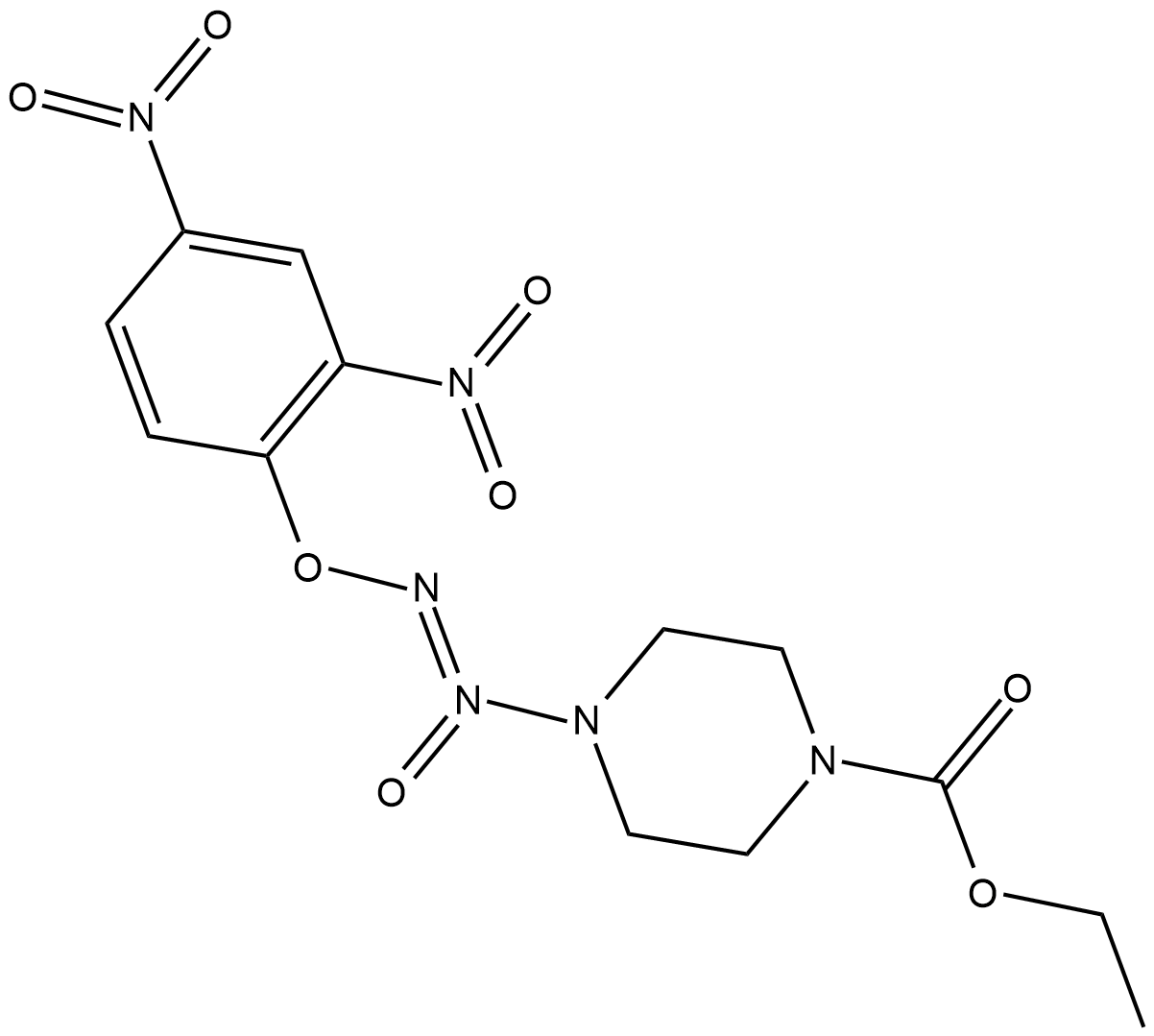
-
GC16167
K 858
K858 Racemic ist ein ATP-unkompetitiver Inhibitor von Kinesin Eg5 mit einem IC50 von 1,3 μM.
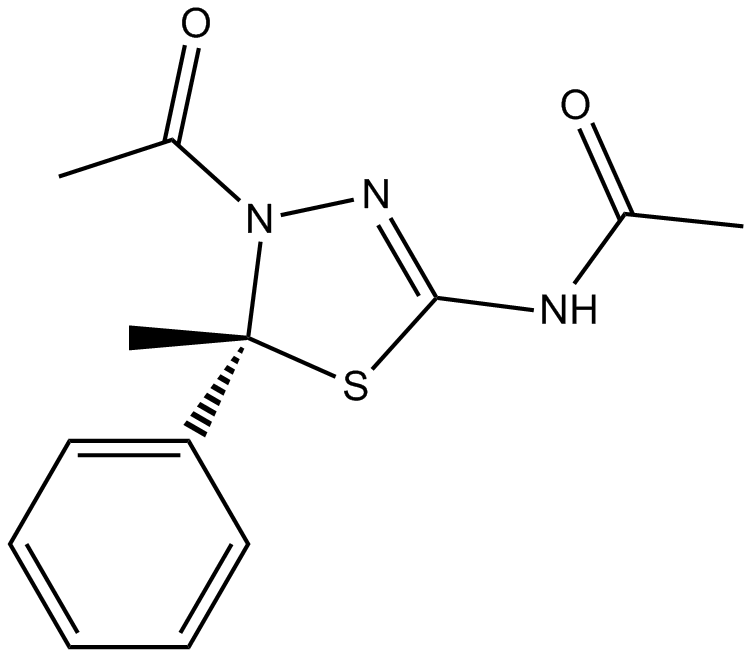
-
GC17388
K-Ras(G12C) inhibitor 6
K-Ras(G12C)-Inhibitor 6 ist ein irreversibler, allosterischer Inhibitor des K-Ras(G12C).
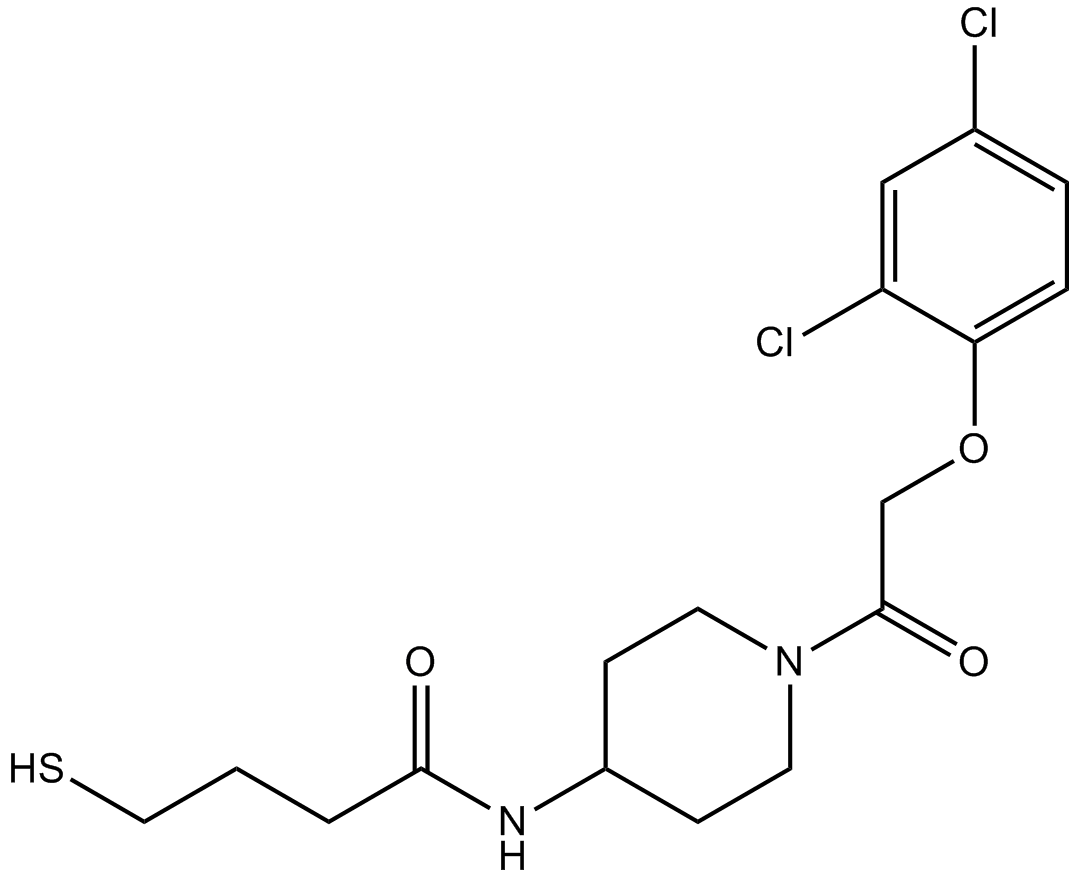
-
GC14230
K03861
K03861 (K03861) ist ein Typ-II-CDK2-Inhibitor mit einem Kd von 8,2 nM. K03861 (K03861) hemmt die CDK2-AktivitÄt, indem es mit der Bindung von aktivierenden Cyclinen konkurriert.
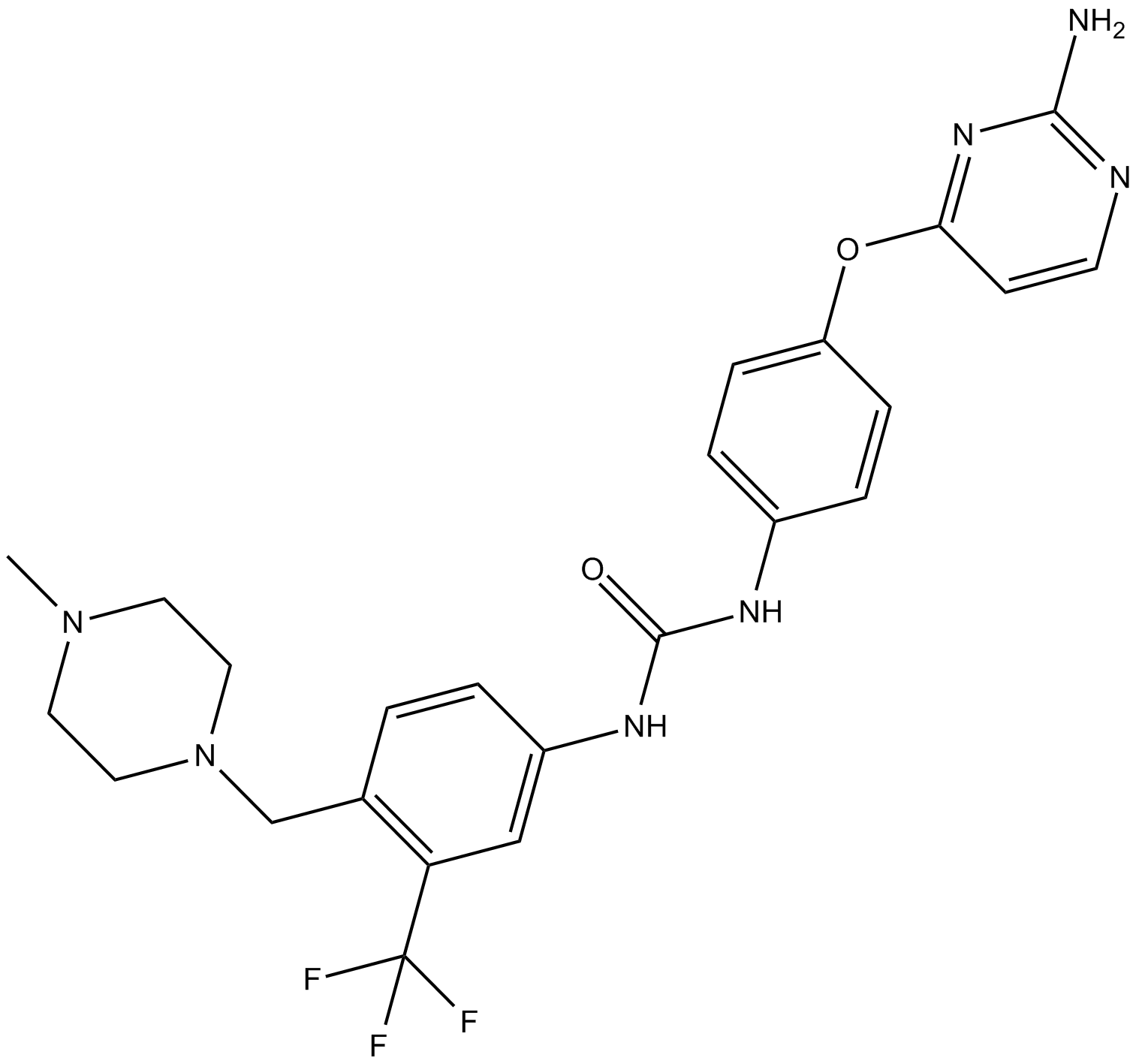
-
GC43994
KAPA (hydrochloride)
7-keto-8-Aminopelargomic Acid
Biotin is a growth factor that plays an important role in numerous reactions that are critical to microorganisms, plants, and animals.
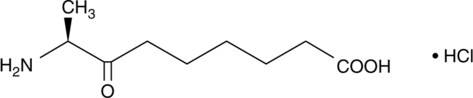
-
GC43995
Kazusamycin B
CL 1957E, Hydroxyleptomycin A, PD 124895
Kazusamycin B is a bacterial metabolite originally isolated from Streptomyces.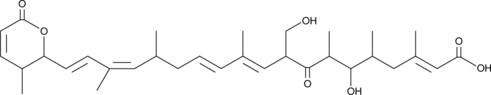
-
GC14182
Kenpaullone
9Bromopaullone, NSC 664704
Kenpaullon ist ein potenter Inhibitor von CDK1/Cyclin B und GSK-3β mit IC50-Werten von 0,4 μM und 23 nM und hemmt auch CDK2/Cyclin A, CDK2/Cyclin E und CDK5/p25 mit IC50-Werten von 0,68 μM, 7,5 μM, 0,85 μM. Kenpaullon, ein niedermolekularer Inhibitor von KLF4, reduziert die Selbsterneuerung von Brustkrebsstammzellen und die ZellmotilitÄt in vitro.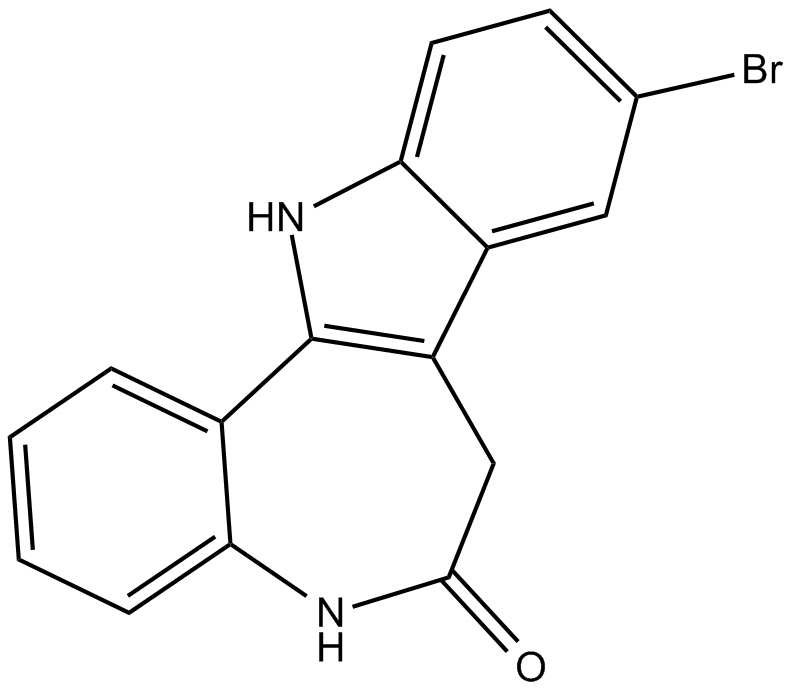
-
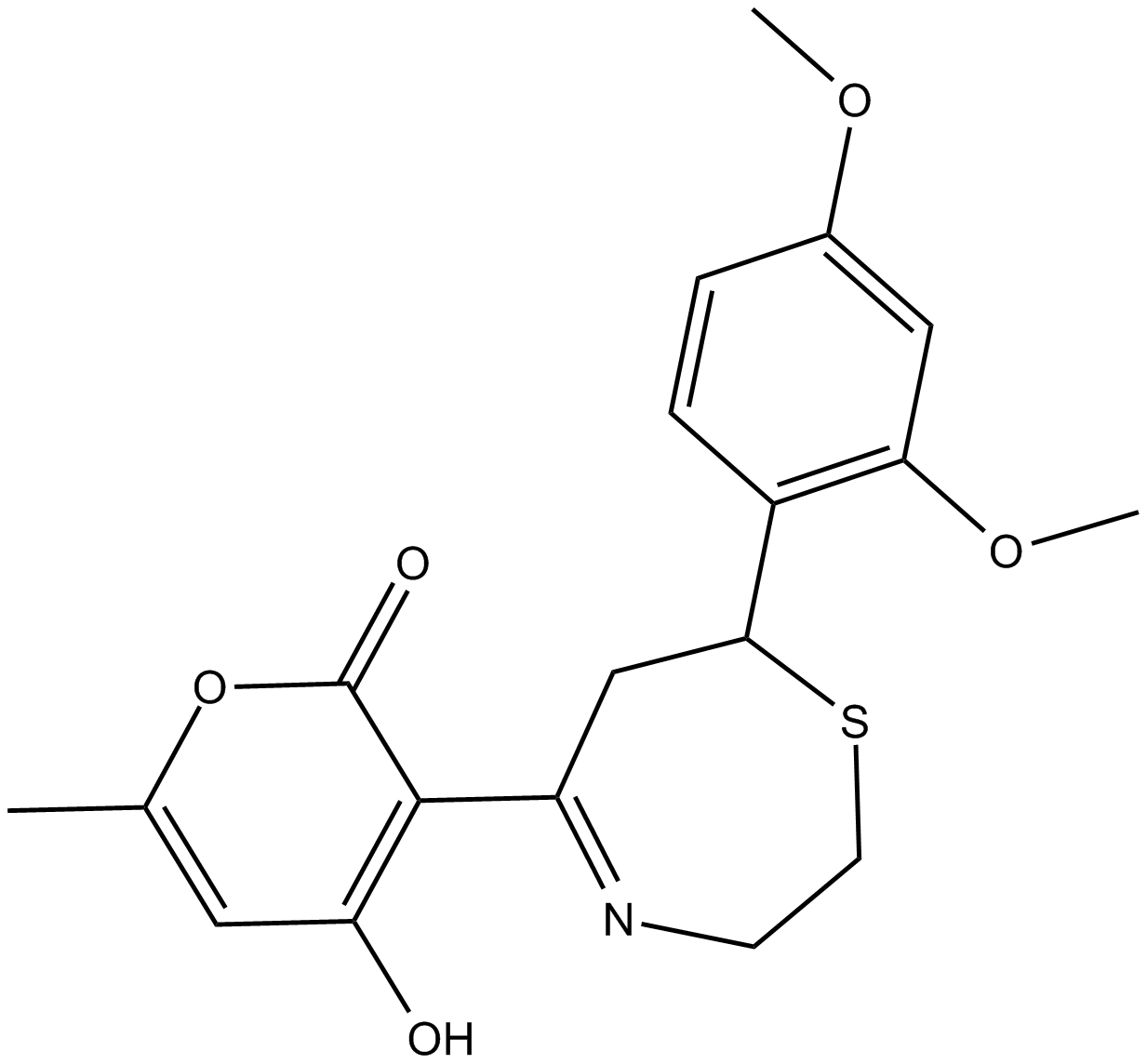
GC14589
KF 38789
KF 38789 ist ein selektiver Inhibitor der P-Selectin-PSGL-1-Bindung.
-
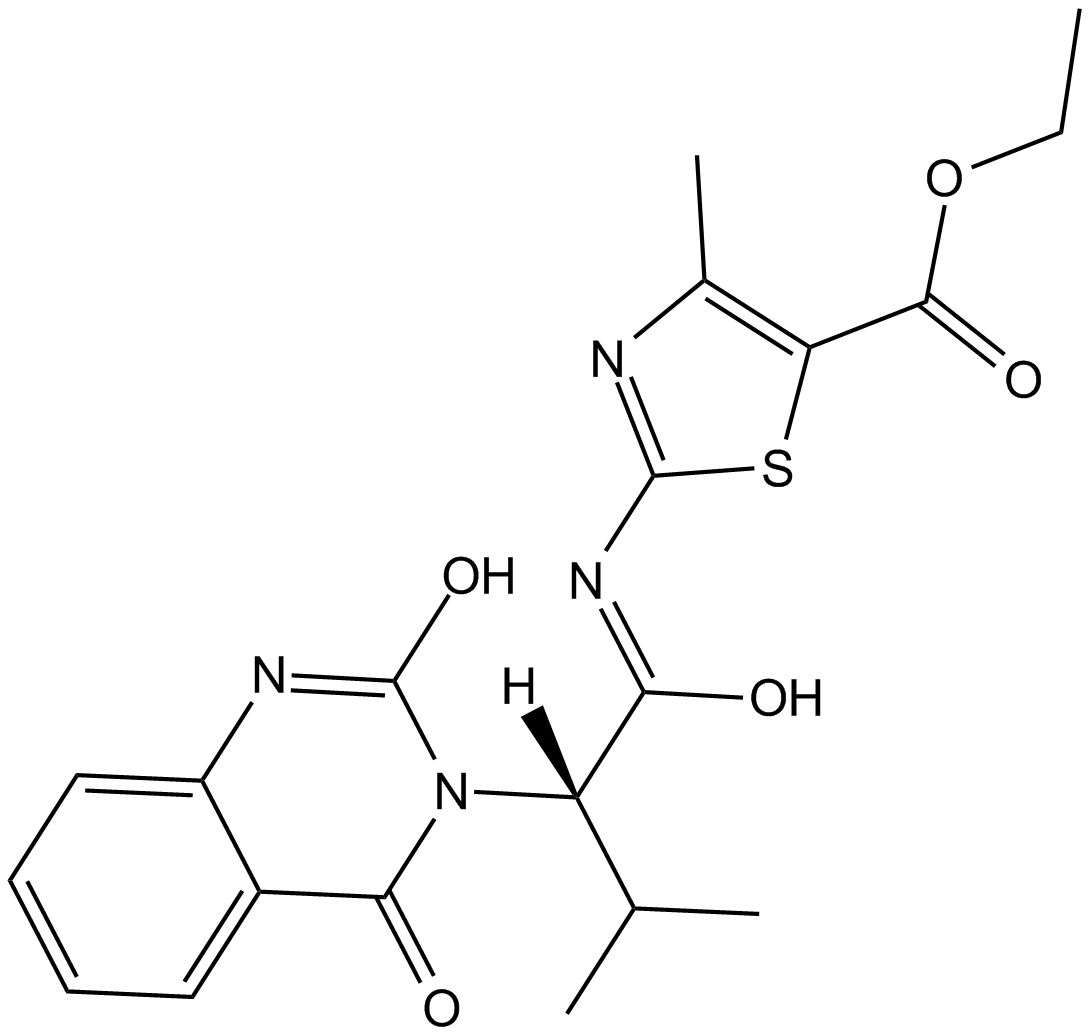
GC16603
Kif15-IN-1
Kif15-IN-1 ist ein Inhibitor des mitotischen Kinesin-Familienmitglieds 15 (Kif15) und wird zur Erforschung von zellulÄren proliferativen Erkrankungen eingesetzt.
-
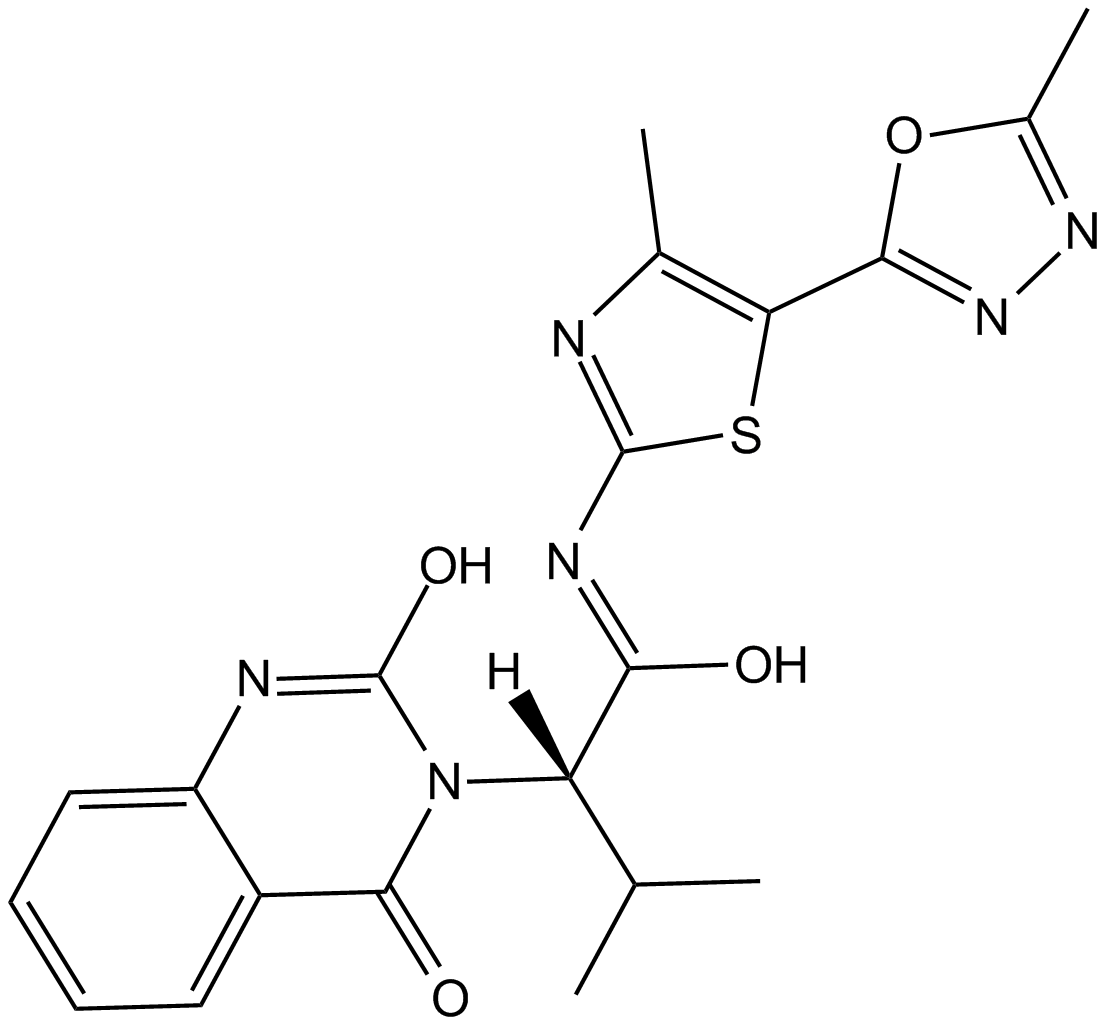
GC17994
Kif15-IN-2
Kif15-IN-2 ist ein Inhibitor des mitotischen Kinesins Kif15 und wird zur Erforschung von zellulÄren proliferativen Erkrankungen eingesetzt.
-
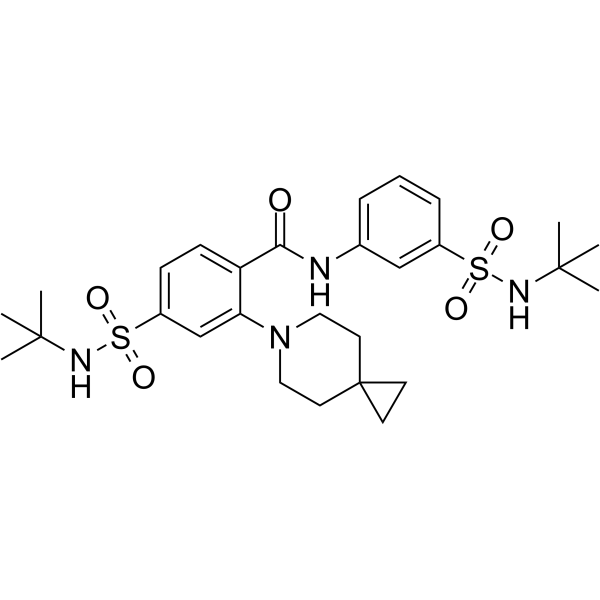
GC64085
KIF18A-IN-1
KIF18A-IN-1 ist ein mitotischer Kinesin-KIF18A-Inhibitor, extrahiert aus dem Patent WO2021026098A1, Beispiel 100-13.
-
GC64808
KIF18A-IN-2
KIF18A-IN-2 ist ein potenter KIF18A-Inhibitor (IC50=28 nM). KIF18A-IN-2 verursacht einen signifikanten mitotischen Arrest und erhÖht die Anzahl mitotischer Zellen in Tumorgeweben. KIF18A-IN-2 kann zur Krebsforschung eingesetzt werden.
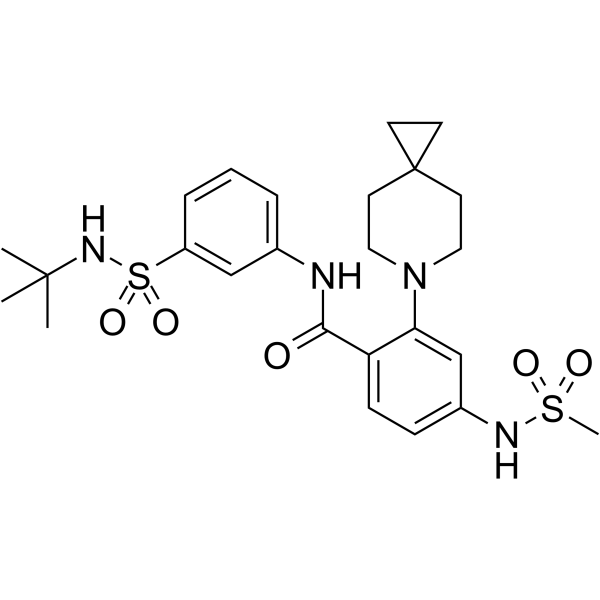
-
GC64857
KIF18A-IN-3
KIF18A-IN-3 ist ein potenter KIF18A-Inhibitor (IC50=61 nM). KIF18A-IN-3 verursacht einen signifikanten mitotischen Arrest und erhÖht die Anzahl mitotischer Zellen in Tumorgeweben. KIF18A-IN-3 kann zur Krebsforschung eingesetzt werden.
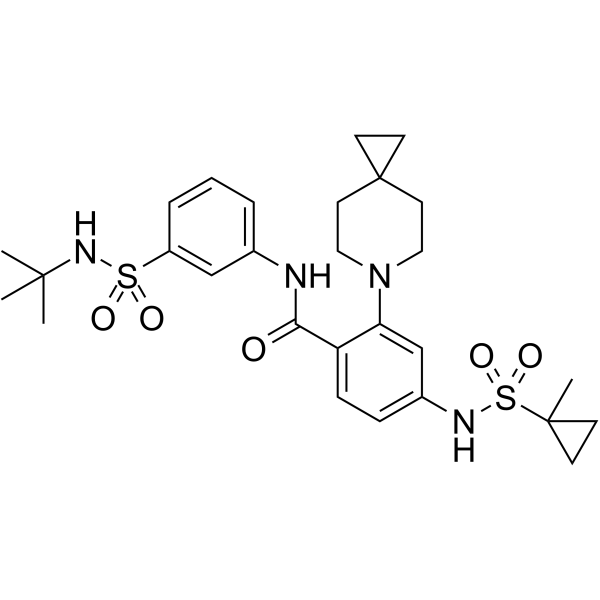
-
GC69333
KIF18A-IN-7
KIF18A-IN-7 (Verbindung 22) is a KIF18A inhibitor with oral activity, which inhibits the microtubule-dependent ATPase activity of KIF18A with an IC50 of 9.4 nM.
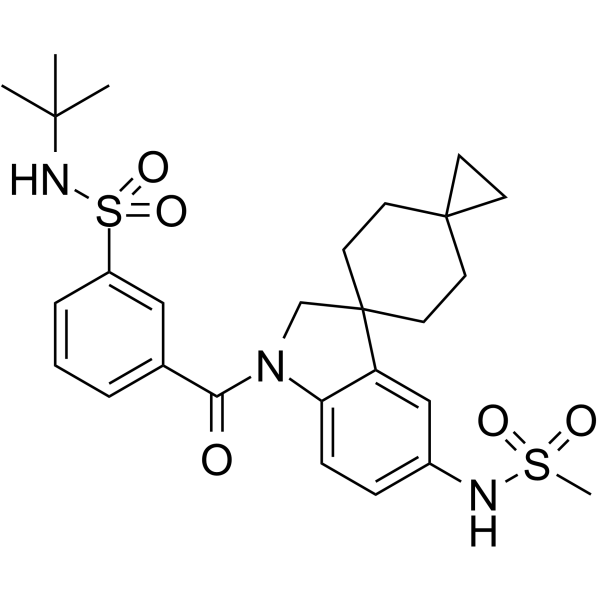
-
GC14413
KJ Pyr 9
KJ Pyr 9 ist ein Inhibitor von MYC mit einem Kd von 6,5 nM im In-vitro-Assay.
-
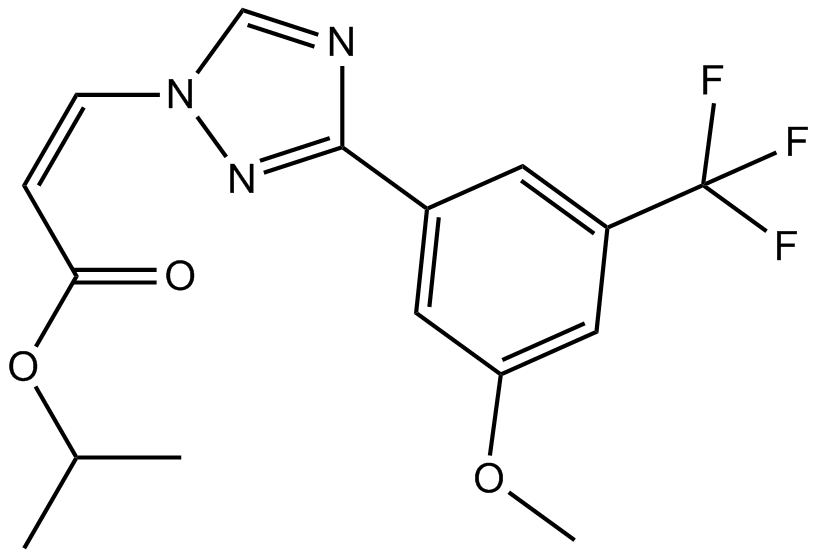
GC14517
KPT-185
CRM1 inhibitor,selective and irrversible
-
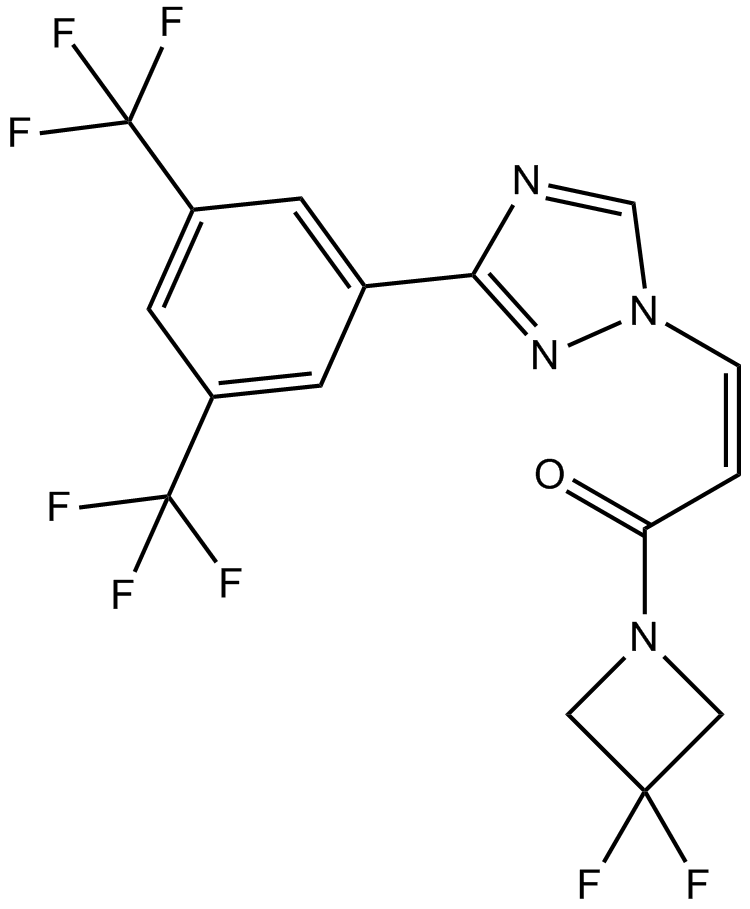
GC12142
KPT-276
inhibitor of nuclear export (SINE) and CRM1, orally bioavailable
-
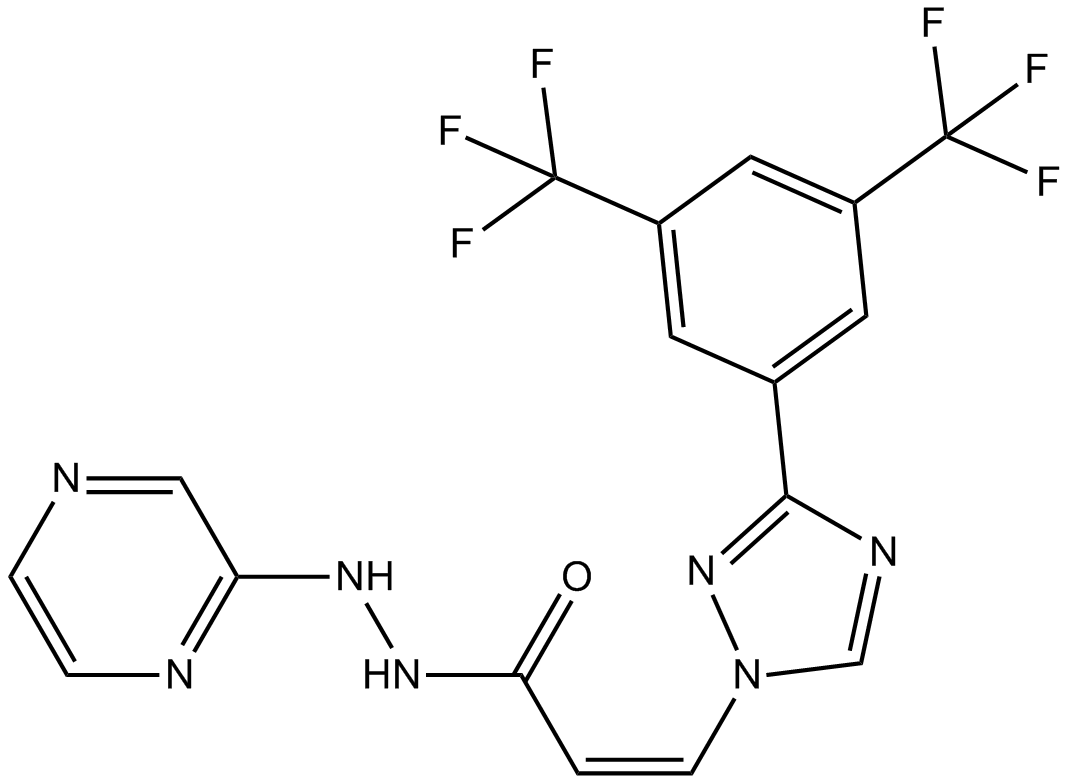
GC12467
KPT-330
CRM1-Inhibitor, oral bioverfügbar und selektiv
-
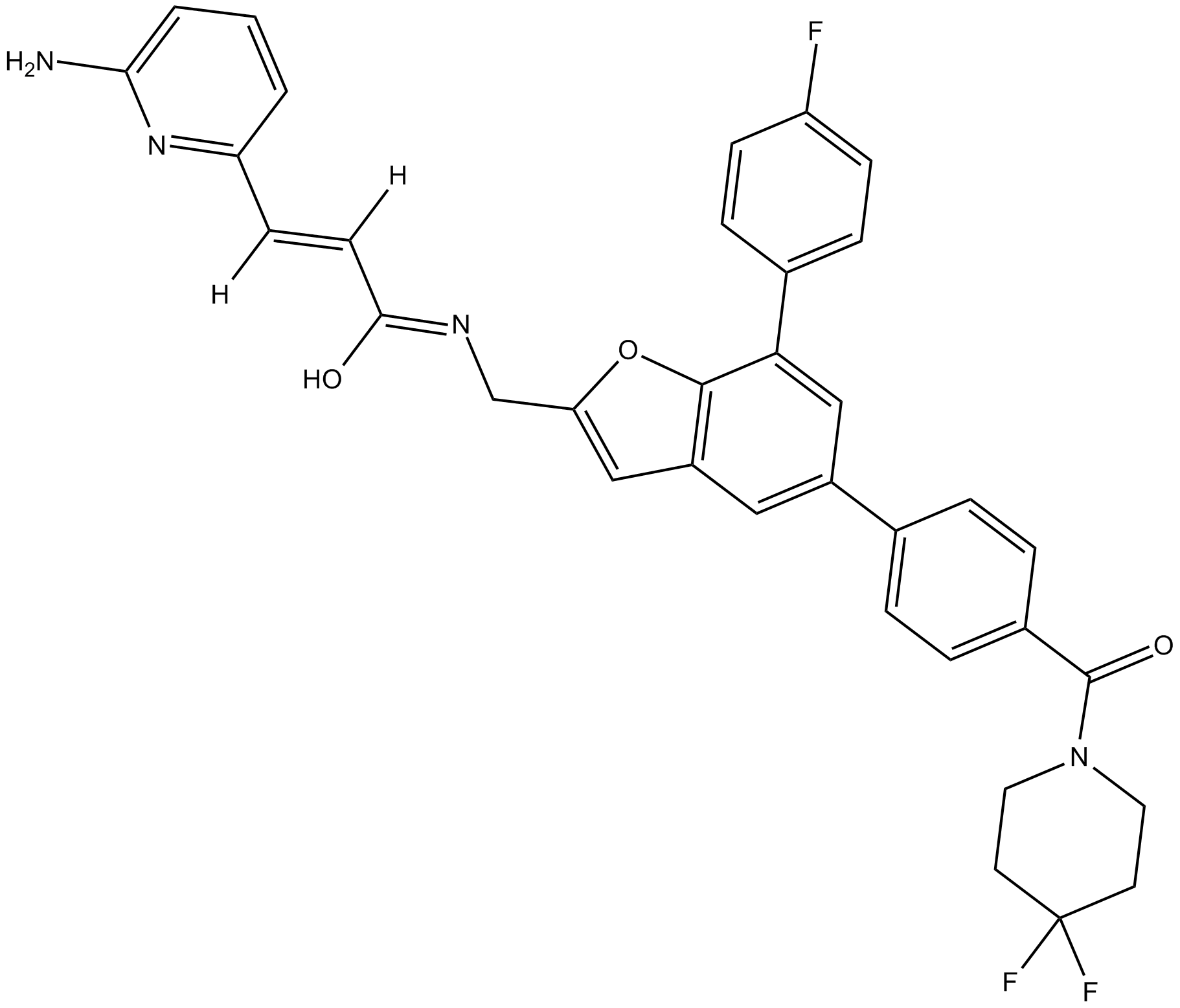
GC14615
KPT-9274
Orally acitve allosteric inhibitor of PAK4
-
GC18909
KRIBB3
KRIBB3 is an Hsp27 and microtubule inhibitor that inhibits migration and invasion of MDA-MB-231 cells in vitro in an Hsp27-dependent manner.
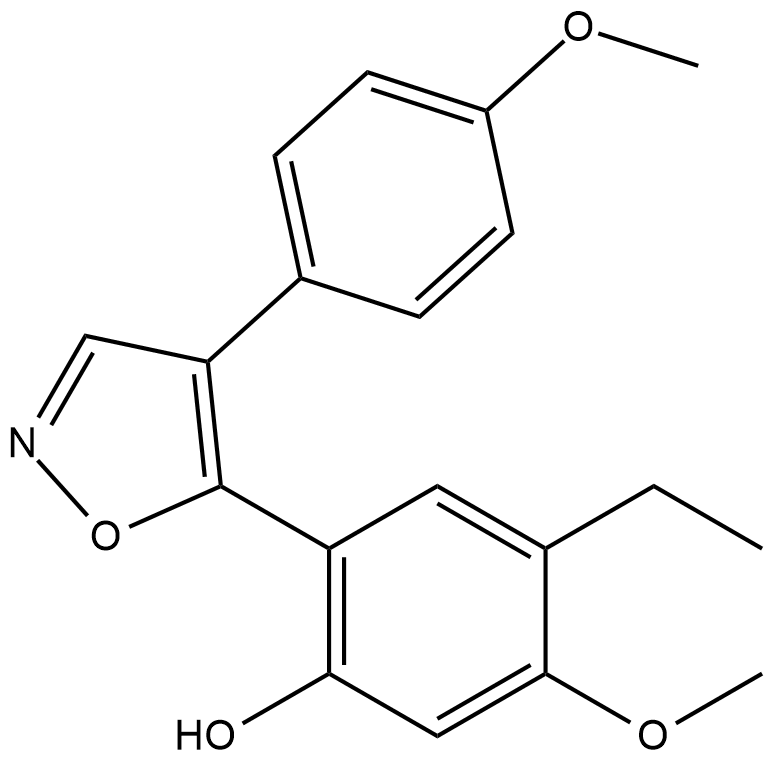
-
GC33377
KSI-3716
KSI-3716 ist ein potenter c-Myc-Inhibitor, der die c-MYC/MAX-Bindung an Zielgen-Promotoren blockiert. KSI-3716 ist ein wirksames intravesikales Chemotherapeutikum fÜr Blasenkrebs.
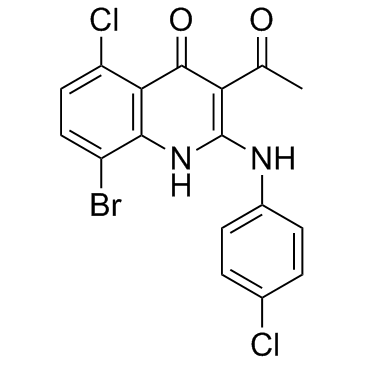
-
GC33404
KU 59403
KU 59403 ist ein potenter ATM-Inhibitor mit IC50-Werten von 3 nM, 9,1 μM und 10 μM fÜr ATM, DNA-PK bzw. PI3K.
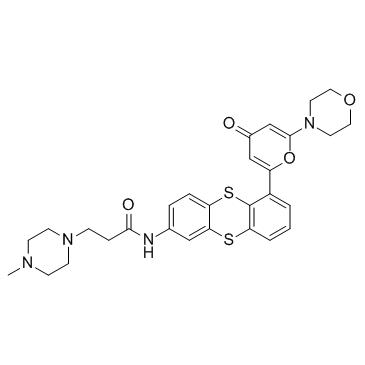
-
GC12054
KU-60019
KU60019;KU 60019
KU-60019 ist ein verbesserter ATM-Kinase-spezifischer Inhibitor mit IC50 von 6,3 nM.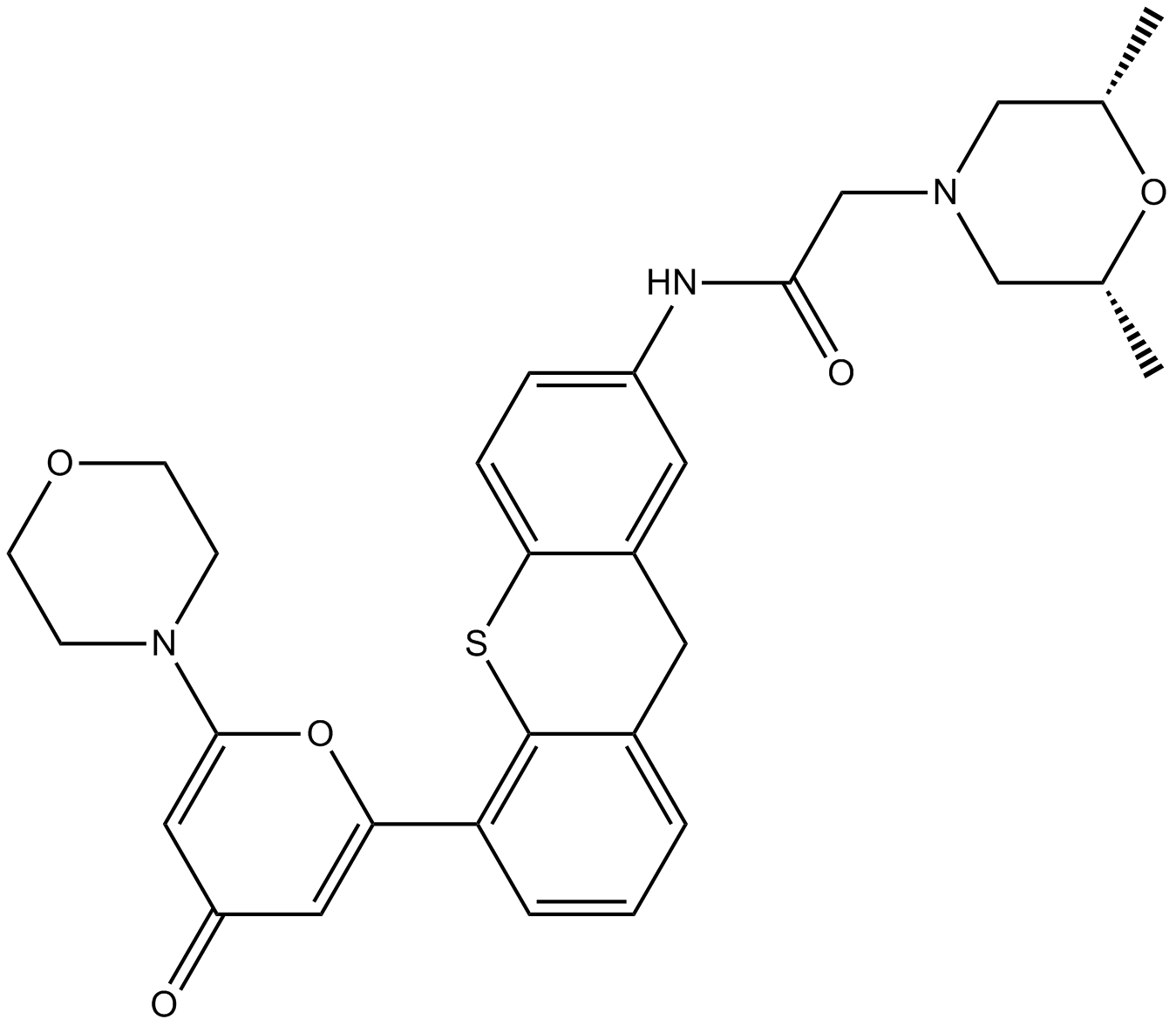
-
GC47532
KUS121
A VCP modulator
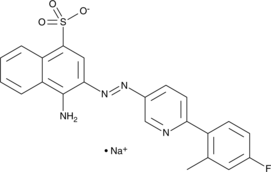
-
GC14288
KX2-391
KX 01, Tirbanibulin
KX2-391 (KX2-391) ist ein Inhibitor von Src, der auf die Peptidsubstratstelle von Src abzielt, mit einem GI50 von 9-60 nM in Krebszelllinien.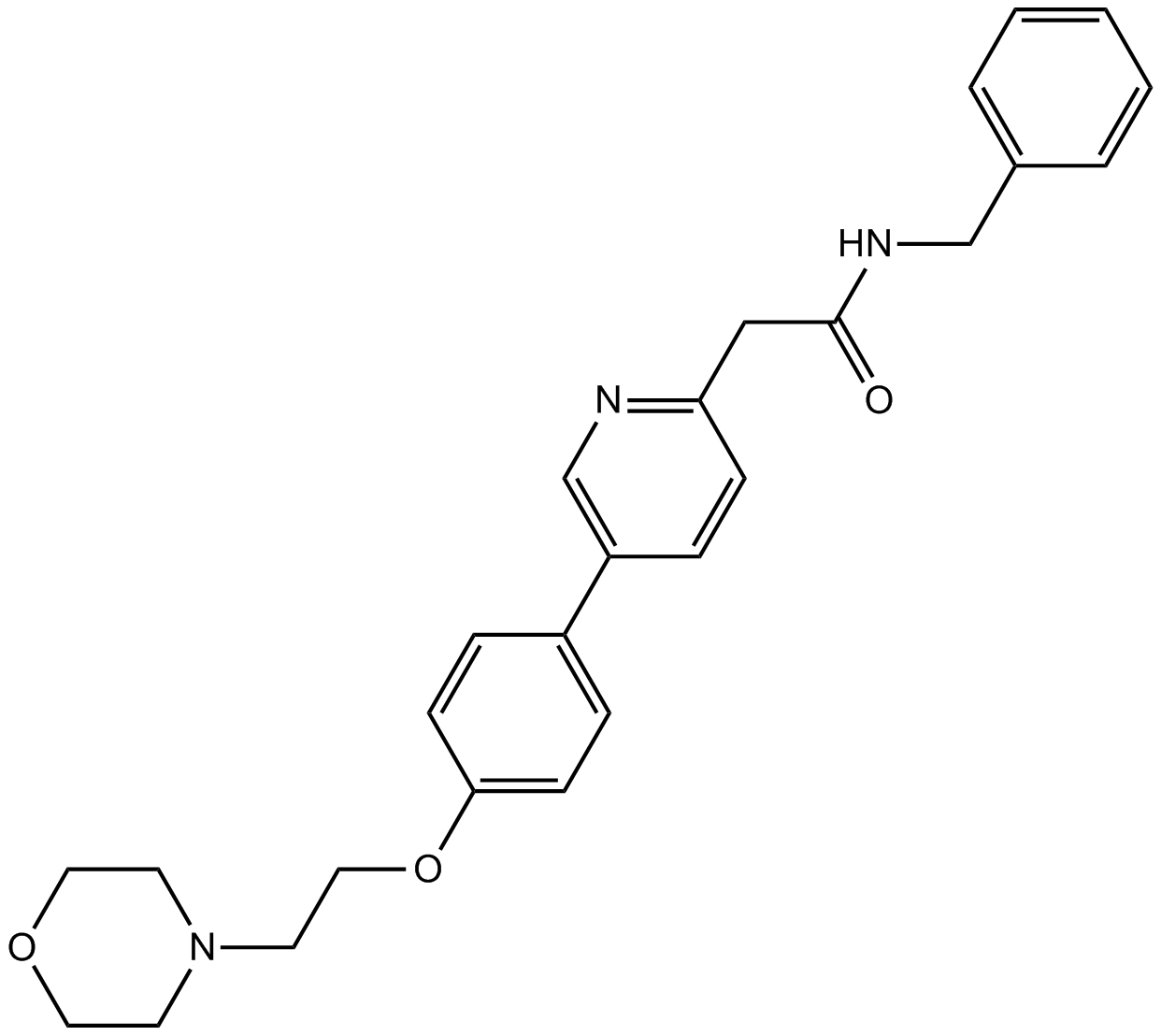
-
GC10222
KX2-391 dihydrochloride
A Src kinase inhibitor
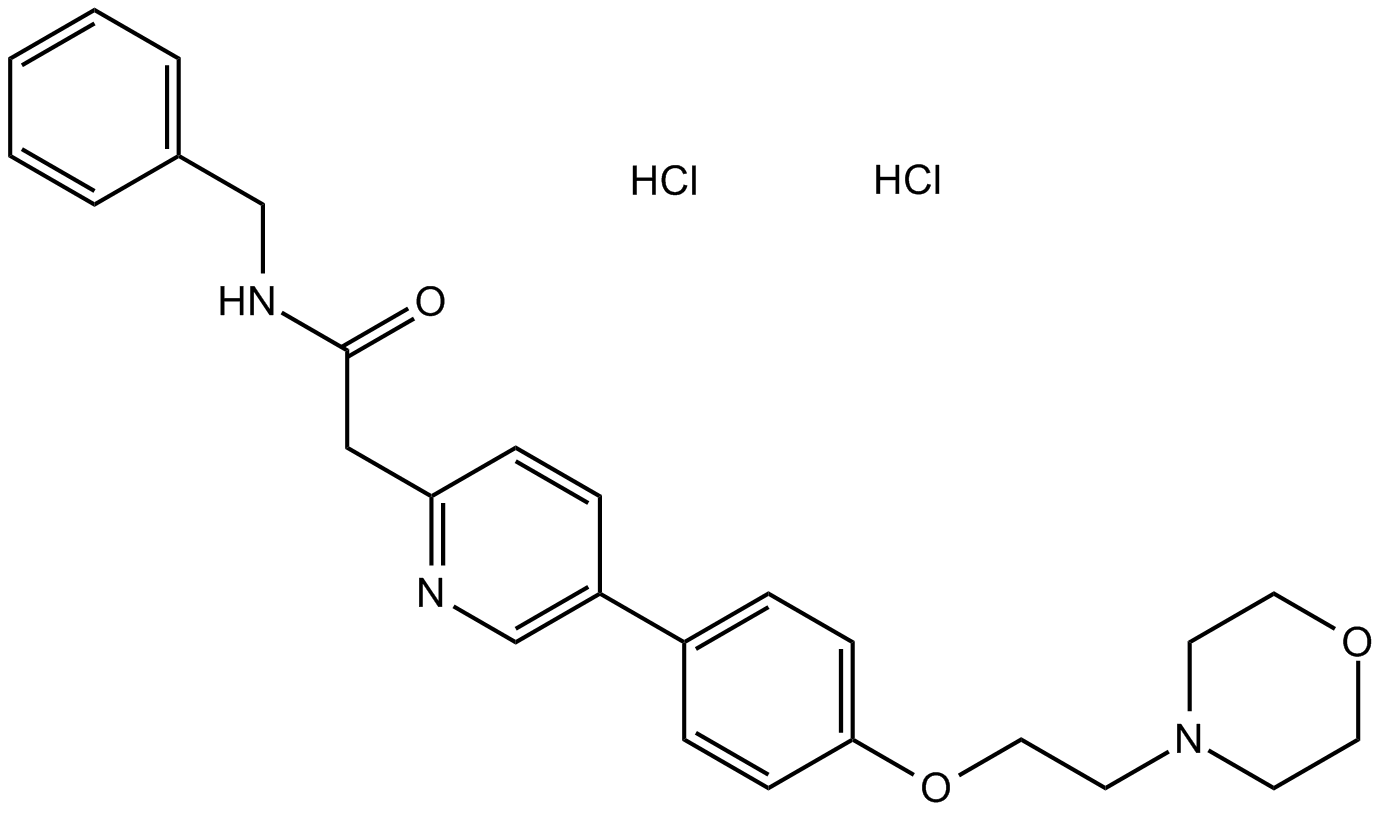
-
GC12817
K–115 hydrochloride dihydrate
K-115-Hydrochlorid-Dihydrat (K-115) ist ein spezifischer Inhibitor von ROCK mit IC50-Werten von 19 und 51 nM fÜr ROCK2 bzw. ROCK1.
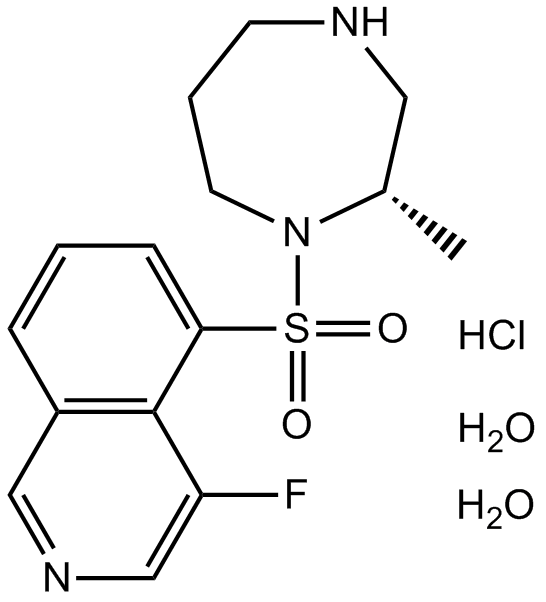
-
GC44020
L-681,217
L-681,217 is an antibiotic originally isolated from S.
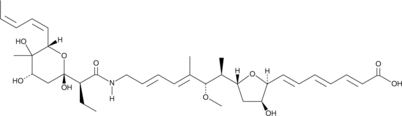
-
GC40793
L-erythro Sphinganine (d18:0)
L-erythro Dihydrosphingosine, C18 L-erythro Sphinganine (d18:0)
L-erythro Sphinganine (d18:0) is a synthetic bioactive sphingolipid and stereoisomer of sphinganine (d18:0) and D-threo sphinganine.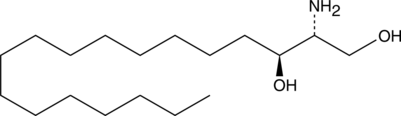
-
GC49547
L-Glutamic Acid γ-p-Nitroanilide (hydrate)
γ-Glu-pNA, Glutamate γ-(4-Nitroanilide), L-GPNA, E-pNA
A colorimetric substrate for γ-glutamyl transpeptidase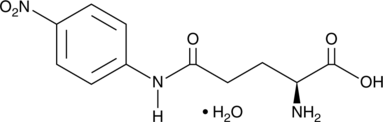
-
GC49205
L-Leu-AMC (hydrochloride)
Leu-MCA, L-Leucine-7-amido-4-methylcoumarin, 7-L-Leucyl-4-methylcoumarinylamide
L-Leucin-7-amido-4-methylcumarin (Leu-AMC)-Hydrochlorid ist ein hellblaues fluorogenes Peptidylsubstrat fÜr LAP3 (Leucin-Aminopeptidase).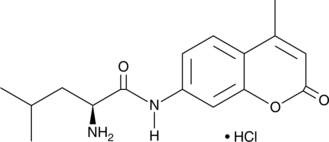
-
GC44081
L-myo-Inositol-1,4,5-triphosphate (sodium salt)
D-myo-Inositol-3,5,6-triphosphate, Ins(1,4,5)P3, 1,4,5IP3
Ins(1,4,5)P3 is an isomer of the biologically important D-myo-inositol-1,4,5-triphosphate.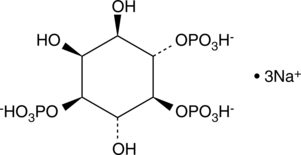
-
GC41520
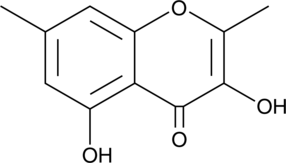
Lachnone A
Lachnone A ist ein Chromonderivat, das aus dem Fadenpilz Lachnum sp.
-
GC49471
LAMP1 Monoclonal Antibody (Clone Ly1C6)
For immunochemical analysis of LAMP1

-
GC41272
Lateropyrone
Avenacein Y
Lateropyrone is a fungal metabolite produced by Fusarium species.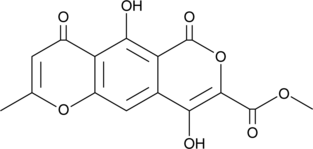
-
GC15671
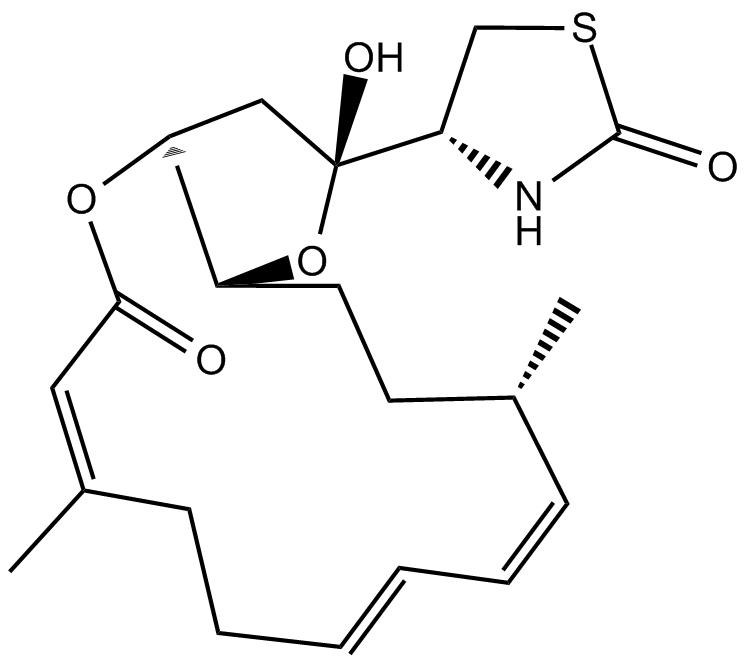
Latrunculin A
NSC 613011
Ein reversibler Hemmstoff der Aktin-Assemblierung.
-
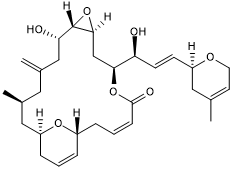
GC50052
Laulimalide
Microtubule stabilzing agent
-
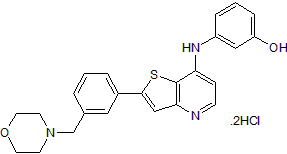
GC50214
LCB 03-0110 dihydrochloride
Potent c-SRC kinase inhibitor; also inhibits DDR2, BTK and Syk
-
GC39209
LCH-7749944
GNF-PF-2356
LCH-7749944 (GNF-PF-2356) ist ein potenter PAK4-Inhibitor mit einem IC50 von 14,93 μM. LCH-7749944 unterdrÜckt effektiv die Proliferation menschlicher Magenkrebszellen durch Herunterregulierung des PAK4/c-Src/EGFR/Cyclin-D1-Signalwegs und induziert Apoptose.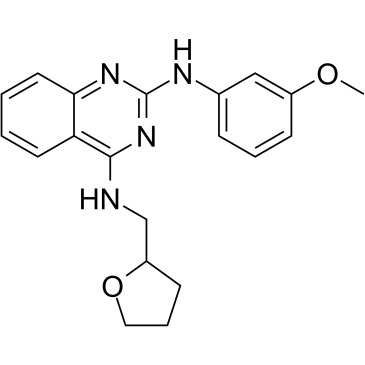
-
GC17067
LDC000067
LDC067
LDC000067 ist ein hochspezifischer CDK9-Inhibitor mit einem IC50-Wert von 44±10 nM in vitro.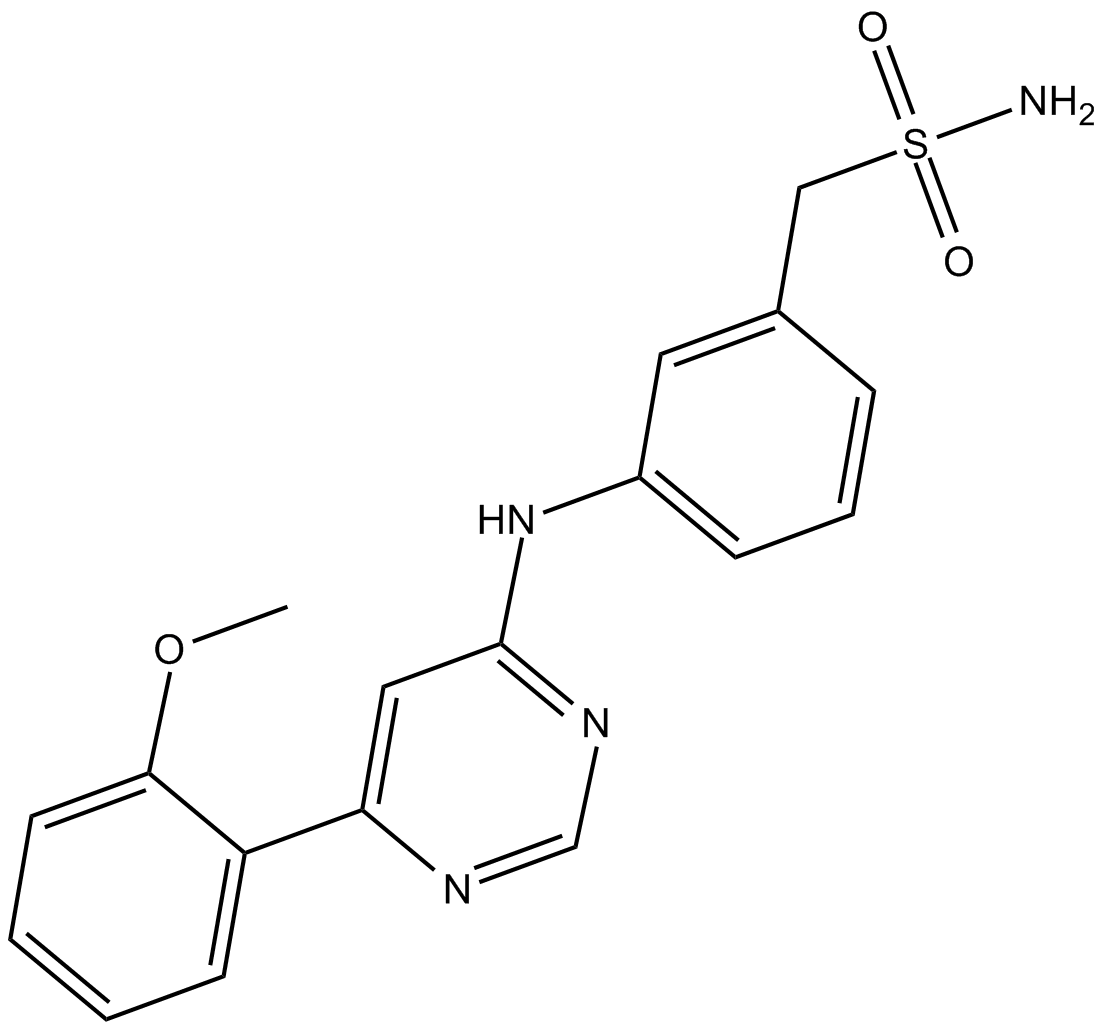
-
GC33086
LDN-192960
LDN-192960 ist ein Inhibitor von Haspin und Dual-SpezifitÄts-Tyrosin-regulierter Kinase 2 (DYRK2) mit IC50-Werten von 10 nM bzw. 48 nM.
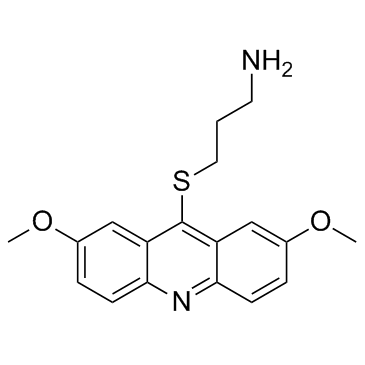
-
GC44047
LDN-192960 (hydrochloride)
LDN-192960 is an inhibitor of haspin protein kinase and dual-specificity tyrosine-(Y)-phosphorylation regulated kinase 2 (DYRK2) with IC50 values of 10 and 48 nM, respectively.
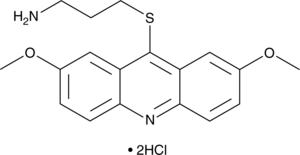
-
GC38403
LDN-192960 hydrochloride
LDN-192960-Hydrochlorid ist ein Inhibitor von Haspin und Dual-SpezifitÄts-Tyrosin-regulierter Kinase 2 (DYRK2) mit IC50-Werten von 10 nM bzw. 48 nM.
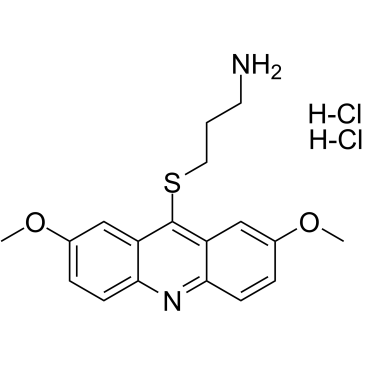
-
GC10842
LEE011
Ribociclib
LEE011 (LEE01) ist ein hochspezifischer CDK4/6-Inhibitor mit IC50-Werten von 10 nM bzw. 39 nM und Über 1.000-mal weniger wirksam gegen den Cyclin B/CDK1-Komplex.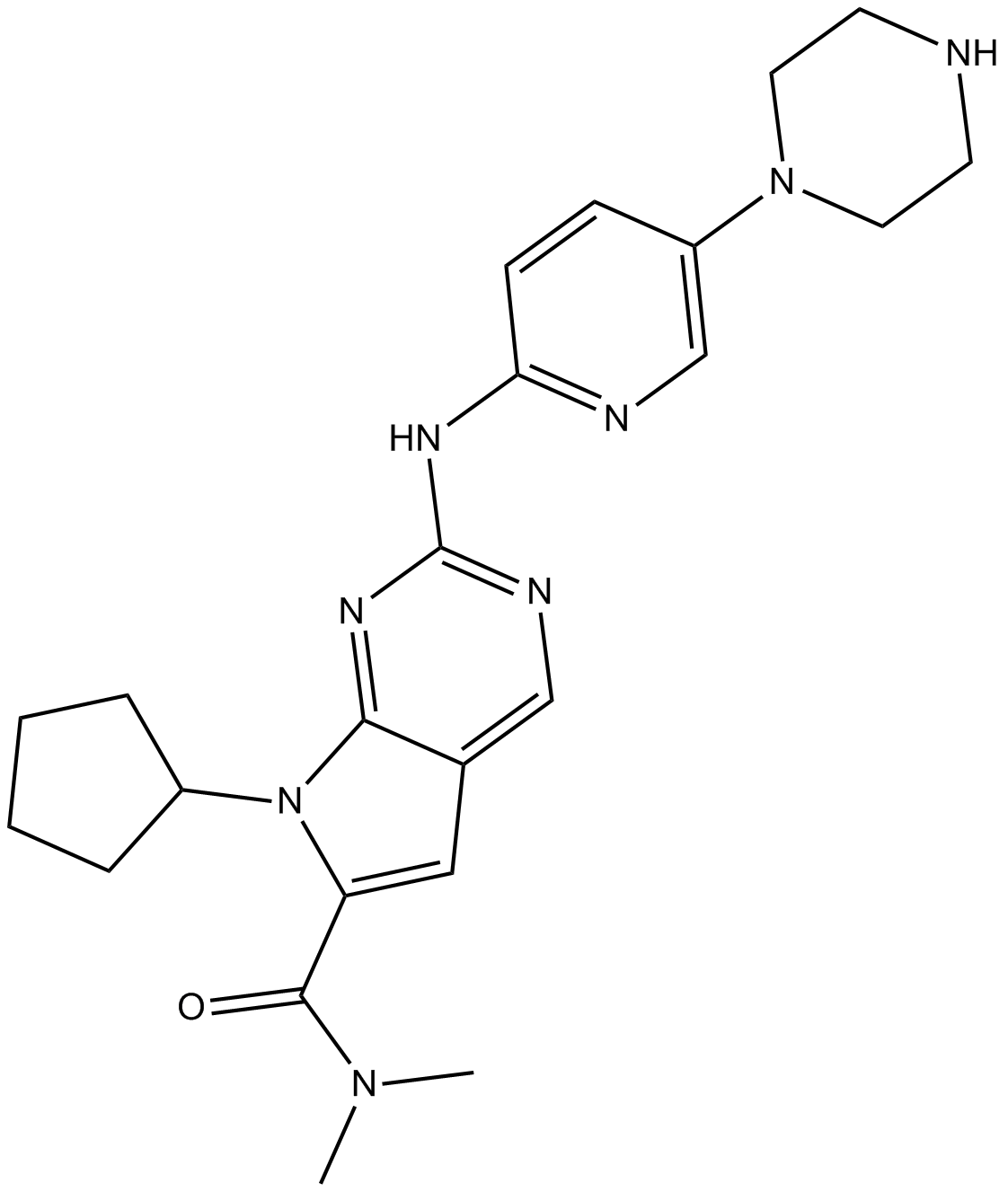
-
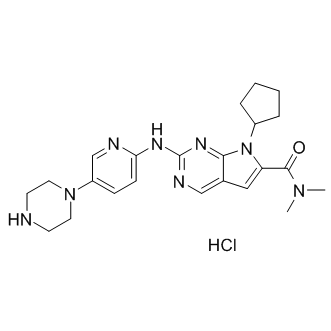
GC15922
LEE011 hydrochloride
LEE011-Hydrochlorid (LEE011-Hydrochlorid) ist ein hochspezifischer CDK4/6-Inhibitor mit IC50-Werten von 10 nM bzw. 39 nM und Über 1.000-mal weniger wirksam gegen den Cyclin B/CDK1-Komplex.
-
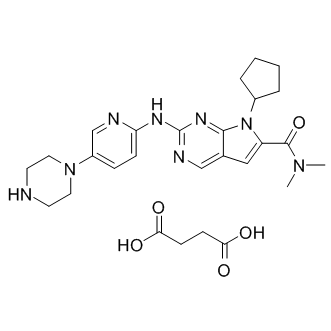
GC15377
LEE011 succinate
LEE011-Succinat (LEE011-Succinat) ist ein hochspezifischer CDK4/6-Inhibitor mit IC50-Werten von 10 nM bzw. 39 nM und Über 1.000-mal weniger wirksam gegen den Cyclin B/CDK1-Komplex.
-
GC49751
Lefamulin
BC-3781
Lefamulin (BC-3781) ist ein oral wirksames Antibiotikum.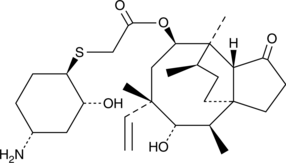
-
GC18345
Leptomycin A
NSC 369326
Leptomycin A, ein Streptomyces-Metabolit, ist ein Inhibitor von CRM1 (Exportin 1), der die CRM1-Interaktion mit nuklearen Exportsignalen blockiert und so den nukleÄren Export einer breiten Palette von Proteinen verhindert.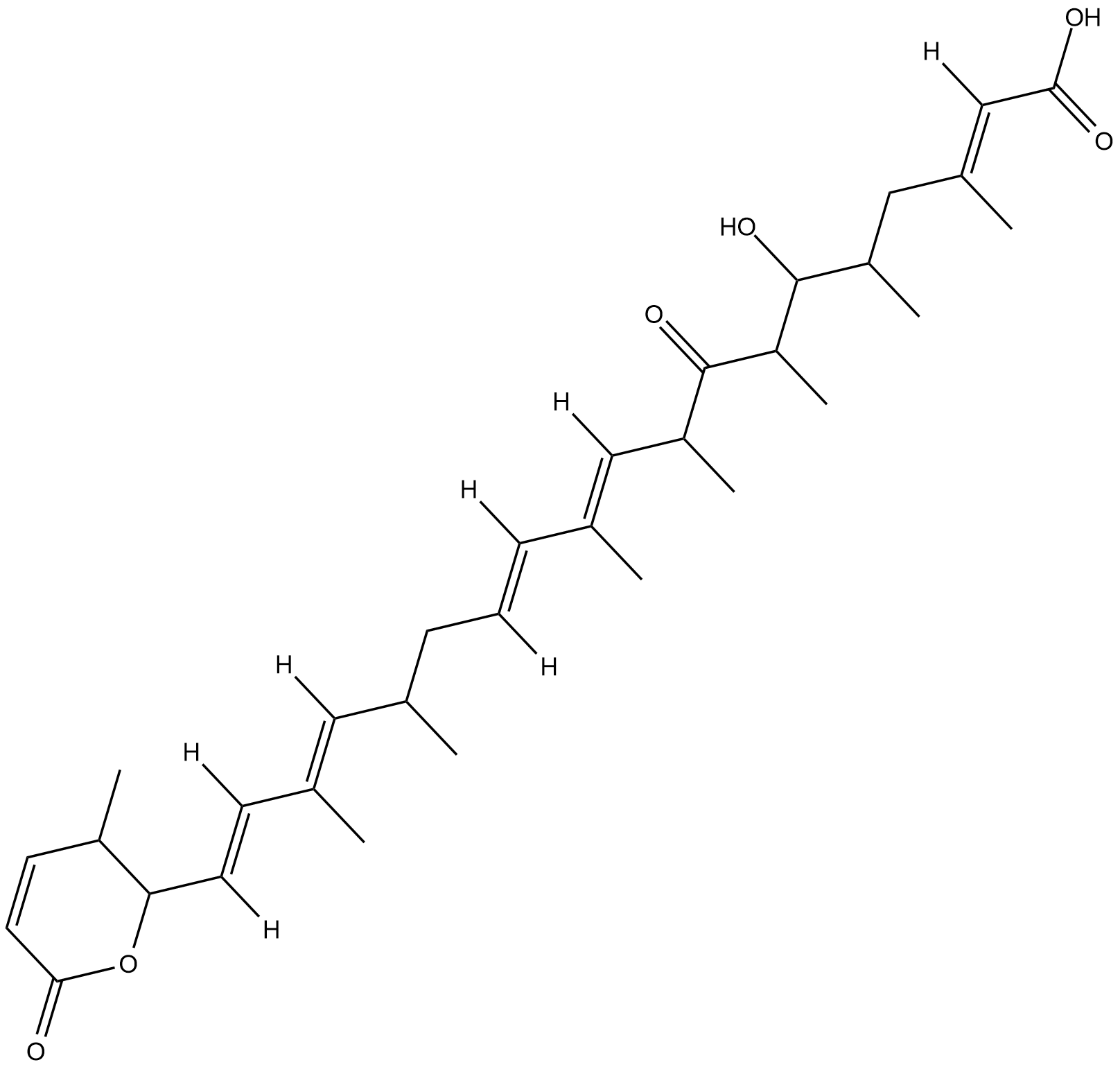
-
GC10954
Leptomycin B
Elactocin; LMB; Mantuamycin; NSC 364372
Leptomycin B (LMB) ist ein Inhibitor von Chromosomen-Regionserhaltung 1 (CRM1), der einer der Kernproteinexporteure ist.
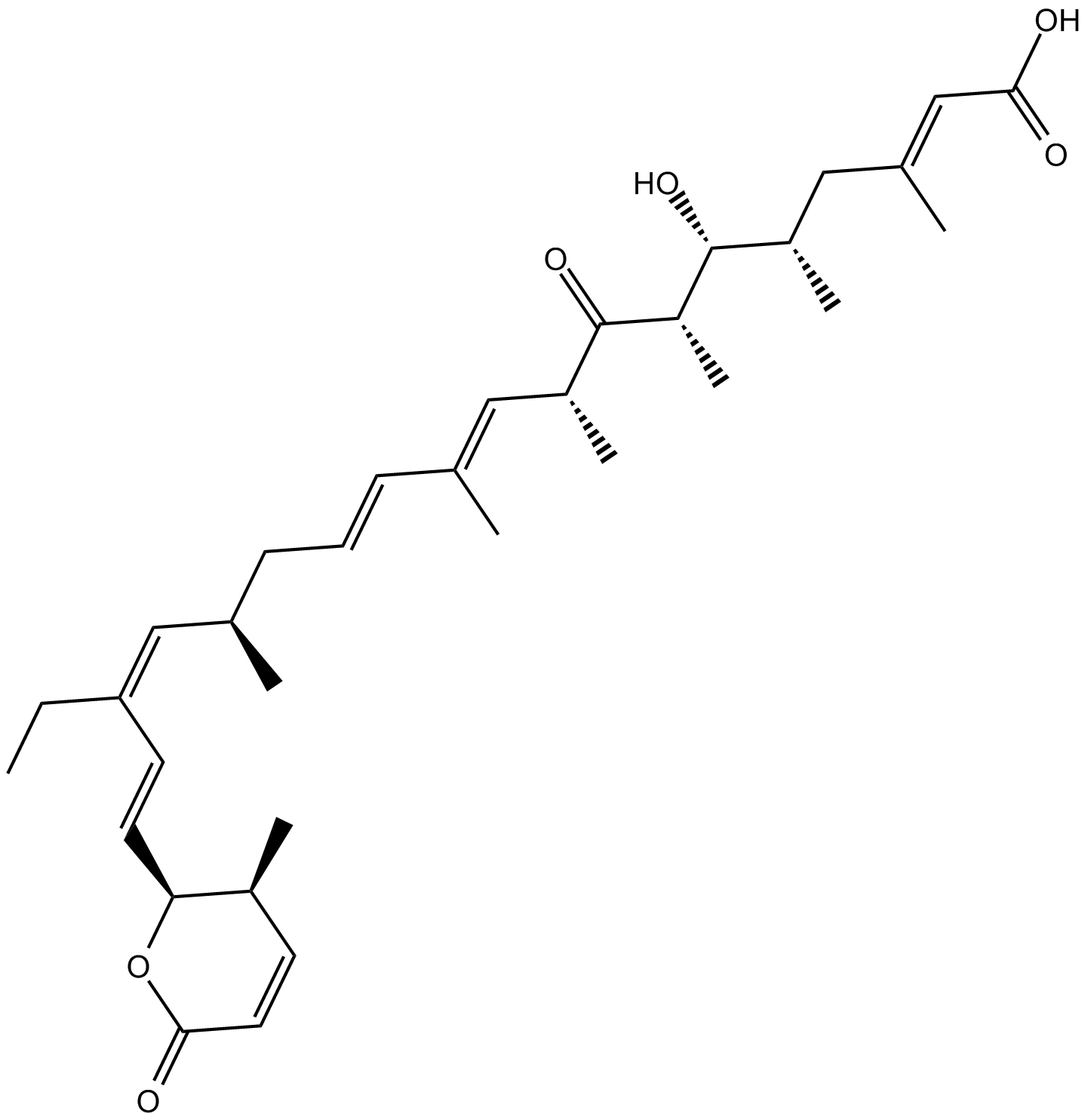
-
GC33175
Lexibulin dihydrochloride (CYT-997 dihydrochloride)
CYT-997 dihydrochloride
Lexibulindihydrochlorid (CYT-997-Dihydrochlorid) (CYT-997-Dihydrochlorid) ist ein potenter und oral aktiver Tubulin-Polymerisationshemmer mit IC50-Werten von 10-100 nM in Krebszelllinien; mit starker zytotoxischer und gefÄßzerstÖrender AktivitÄt in vitro und in vivo. Lexibulindihydrochlorid (CYT-997-Dihydrochlorid) induziert Zellapoptose und induziert die mitochondriale ROS-Erzeugung in GC-Zellen.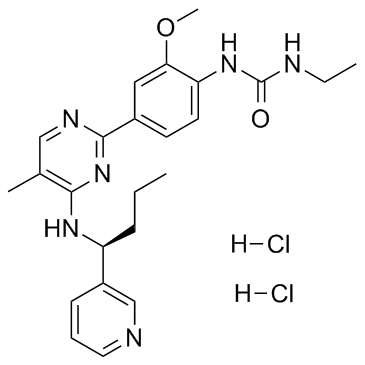
-
GC14857
LFM-A13
LFM-A13 ist ein potenter BTK-, JAK2-, PLK-Inhibitor, hemmt rekombinantes BTK, Plx1 und PLK3 mit IC50-Werten von 2,5 μM, 10 μM und 61 μM; LFM-A13 zeigt keine Wirkungen auf JAK1 und JAK3, die Kinase HCK der Src-Familie, EGFR und IRK.
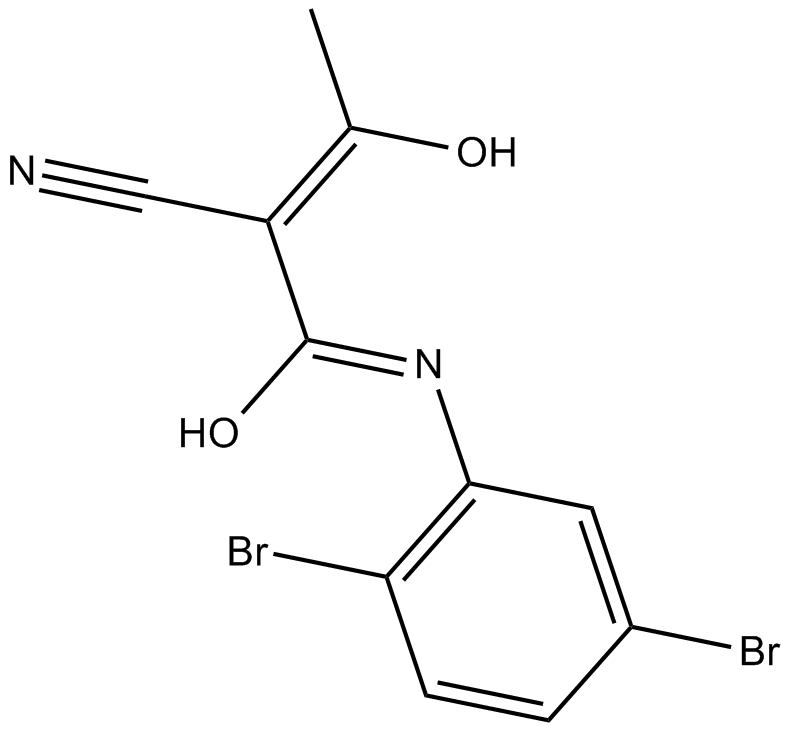
-
GC31803
Litronesib (LY-2523355)
Litronesib (LY-2523355) (LY2523355) ist ein selektiver Mitose-spezifischer Kinesin-Eg5-Inhibitor mit AntitumoraktivitÄt.
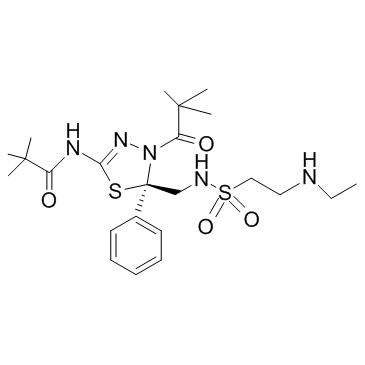
-
GC30393
Litronesib Racemate (LY-2523355 Racemate)
LY2523355 Racemate
Litronesib Racemate (LY-2523355 Racemate) (LY2523355 Racemate) ist das Racemat von Litronesib.
-
GC61014
Lusianthridin
Lusianthridin, eine reine Verbindung aus Dendrobium venustum, wirkt migrationshemmend. Lusianthridin verstÄrkt den Abbau von c-Myc durch die Hemmung der Src-STAT3-Signalgebung.
-
GC12402
LX7101 HCL
LX7101 HCL ist ein starker Inhibitor von LIMK und ROCK2 mit IC50-Werten von 24, 1,6 und 10 nM fÜr LIMK1, LIMK2 bzw. ROCK2; hemmt auch PKA mit einem IC50 von weniger als 1 nM.
-
GC15741
LY2603618
IC-83, Rabusertib
LY2603618 (LY2603618) ist ein potenter und selektiver Inhibitor von Chk1 mit einem IC50 von 7 nM.