DNA Damage/DNA Repair
- MTH1(4)
- PARP(64)
- ATM/ATR(28)
- DNA Alkylating(24)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(28)
- HDAC(99)
- Nucleoside Antimetabolite/Analogue(146)
- Telomerase(14)
- Topoisomerase(134)
- tankyrase(5)
- Antifolate(32)
- CDK(237)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(64)
- DNA Alkylator/Crosslinker(70)
- DNA/RNA Synthesis(445)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(22)
- LIM Kinase (LIMK)(11)
- TOPK(6)
- Casein Kinase(56)
- DNA Intercalating Agents(8)
- DNA/RNA Oxidative Damage(13)
- Poly(ADP-ribose) Glycohydrolase (PARG)(0)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
GC60714
CL2A-SN-38
CL2A-SN-38 ist ein Wirkstoff-Linker-Konjugat, das aus einem potenten DNA-Topoisomerase-I-Inhibitor SN-38 und einem Linker CL2A zur Herstellung eines AntikÖrper-Wirkstoff-Konjugats (ADC) besteht.
-
GC10509
Cladribine
2-Chlorodeoxyadenosine, Jk 6251, NSC 105014, RWJ 26251
Cladribin (2-Chlor-2′-desoxyadenosin), ein Purin-Nukleosid-Analogon, ist ein oral wirksamer Adenosin-Desaminase-Hemmer.
-
GC49852
Clindamycin (hydrochloride hydrate)
Clindamycin (Hydrochloridhydrat) ist ein oraler Hemmer der Proteinsynthese, der die FÄhigkeit besitzt, die Expression von Virulenzfaktoren in Staphylococcus aureus bei subinhibitorischen Konzentrationen (sub-MICs) zu unterdrÜcken.
-
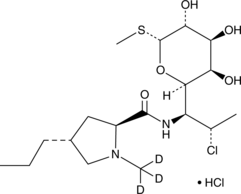
GC52171
Clindamycin-d3 (hydrochloride)
Clindamycin-d3 (Hydrochlorid) ist das mit Deuterium markierte Clindamycin.
-
GC30245
CLK1-IN-1
CLK1-IN-1 ist ein potenter und selektiver Inhibitor der Cdc2-Ähnlichen Kinase 1 (CLK1) mit einem IC50 von 2 nM.
-
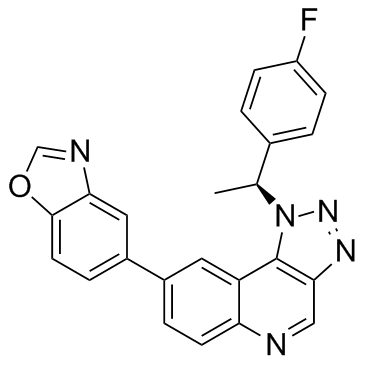
GC68885
CLK1-IN-2
CLK1-IN-2 ist ein metabolisch stabiler Inhibitor von Clk1. CLK1-IN-2 zeigt Selektivität gegenüber Clk1 mit einem IC50-Wert von 1,7 nM. CLK1-IN-2 kann für die Erforschung von Tumoren, Duchenne-Muskeldystrophie und Virusinfektionen wie HIV-1 und Grippe eingesetzt werden.

-
GC15219
Clofarabine
Clolar, Evoltra
Clofarabin, ein Nukleosid-Analogon fÜr die Krebsforschung, ist ein potenter Inhibitor der Ribonukleotid-Reduktase (IC50=65 nM), indem es an die allosterische Stelle der regulatorischen Untereinheit bindet.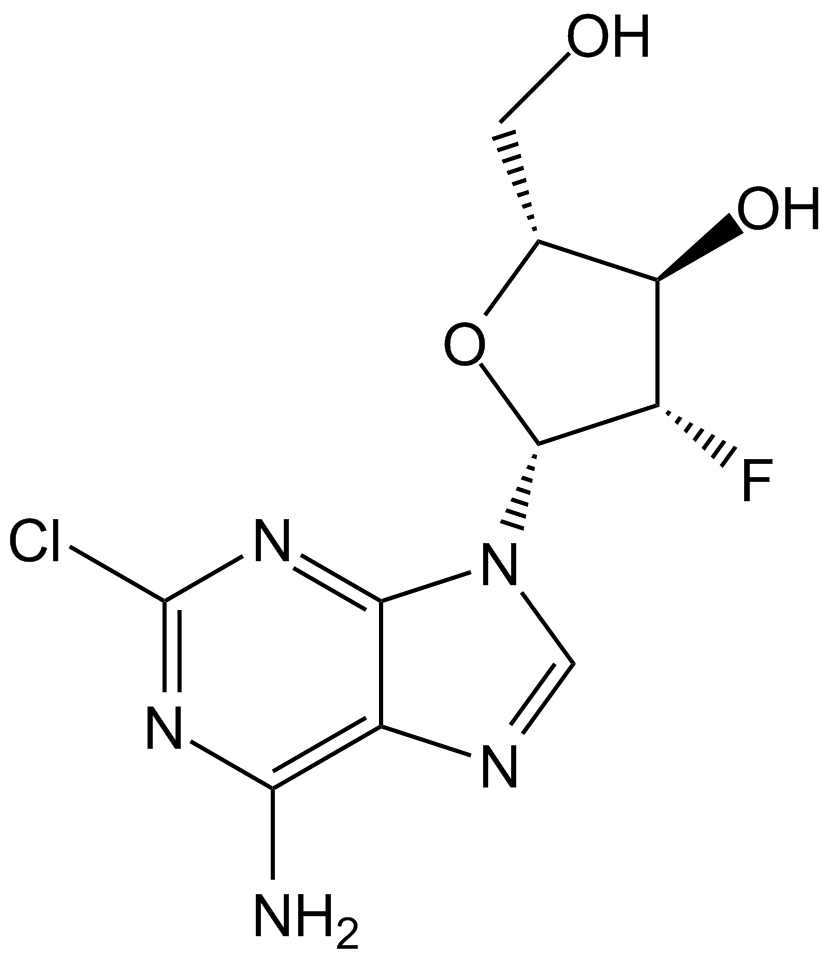
-
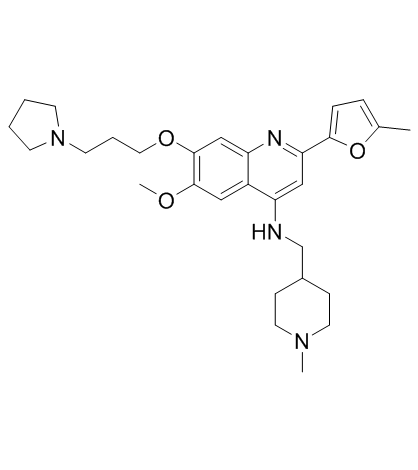
GC33320
CM-579
CM-579 ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM, 32 nM fÜr G9a bzw. DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
-
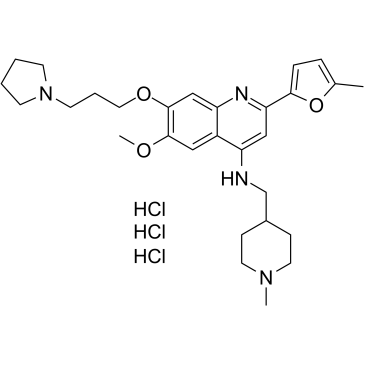
GC35714
CM-579 trihydrochloride
CM-579-Trihydrochlorid ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM bzw. 32 nM fÜr G9a und DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
-
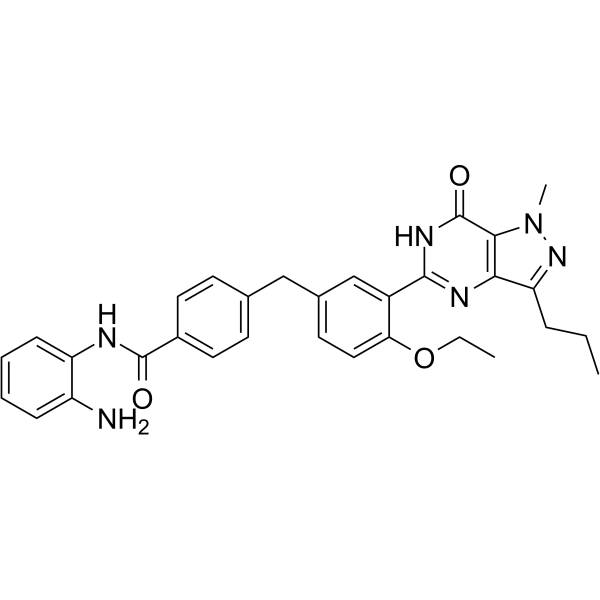
GC65426
CM-675
CM-675 ist ein dualer, fÜr Phosphodiesterase 5 (PDE5) und Histon-Deacetylasen der Klasse I selektiver Inhibitor mit IC50-Werten von 114 nM und 673 nM fÜr PDE5 bzw. HDAC1.
-
GC68887
CM03
CM03 ist ein wirksames Molekül, das sich an DNA G-Quadruplexe (G4s) bindet. CM03 kann G4s stabilisieren, mehr Gene mit G4 herunterregulieren und die Häufigkeit von Doppelstrangbrüchen (DSBs), die durch Verzerrungen der DNA- und Chromatinstruktur verursacht werden, erhöhen. CM03 hat eine selektive Wirkung auf Bauchspeicheldrüsenkrebszellen.

-
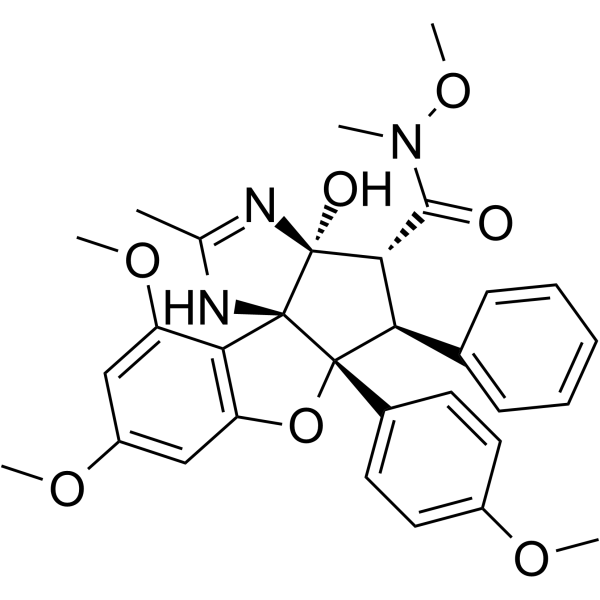
GC62698
CMLD012612
CMLD012612 ist ein Amidino-Rocaglat, das eine Hydroxamatgruppe enthÄlt und ein potenter Hemmer des eukaryotischen Initiationsfaktors 4A (eIF4A) ist. CMLD012612 hemmt die Zelltranslation und ist zytotoxisch fÜr NIH/3T3-Zellen mit einem IC50-Wert von 2 nM. CMLD012612 hemmt die eukaryotische Translationsinitiation durch Modifizierung des Verhaltens der RNA-Helikase (eIF4A) und besitzt eine starke antineoplastische AktivitÄt.
-
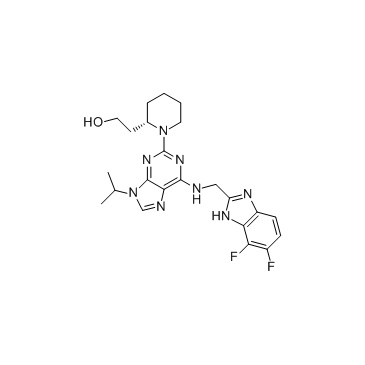
GC33327
CMPD 7
CMPD 7 ist ein potenter und selektiver CDK12-Inhibitor mit einem IC50-Wert von 491 nM im enzymatischen Assay.
-
GC33177
CNDAC
CNDAC ist ein Hauptmetabolit des oralen Medikaments Sapacitabin und ein Nukleosid-Analogon.
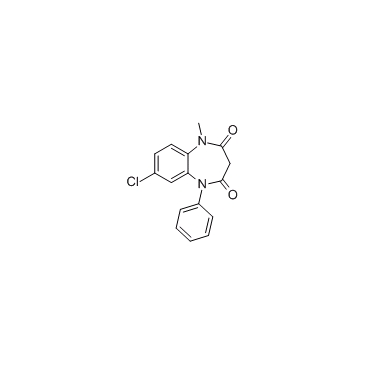
-
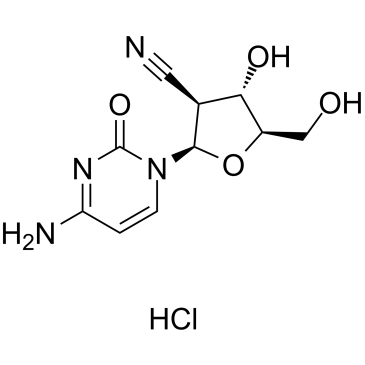
GC38382
CNDAC hydrochloride
CNDAC-Hydrochlorid ist ein Metabolit des oral wirksamen Wirkstoffs Sapacitabin und ein Nukleosid-Analogon.
-
GC19110
COH29
RNR Inhibitor COH29
COH29 (RNR-Inhibitor COH29) ist ein potenter Ribonukleotidreduktase (RNR)-Inhibitor mit AntikrebsaktivitÄt. COH29 hemmt die α- und β-Untereinheit von RNR mit IC50-Werten von 16 μM.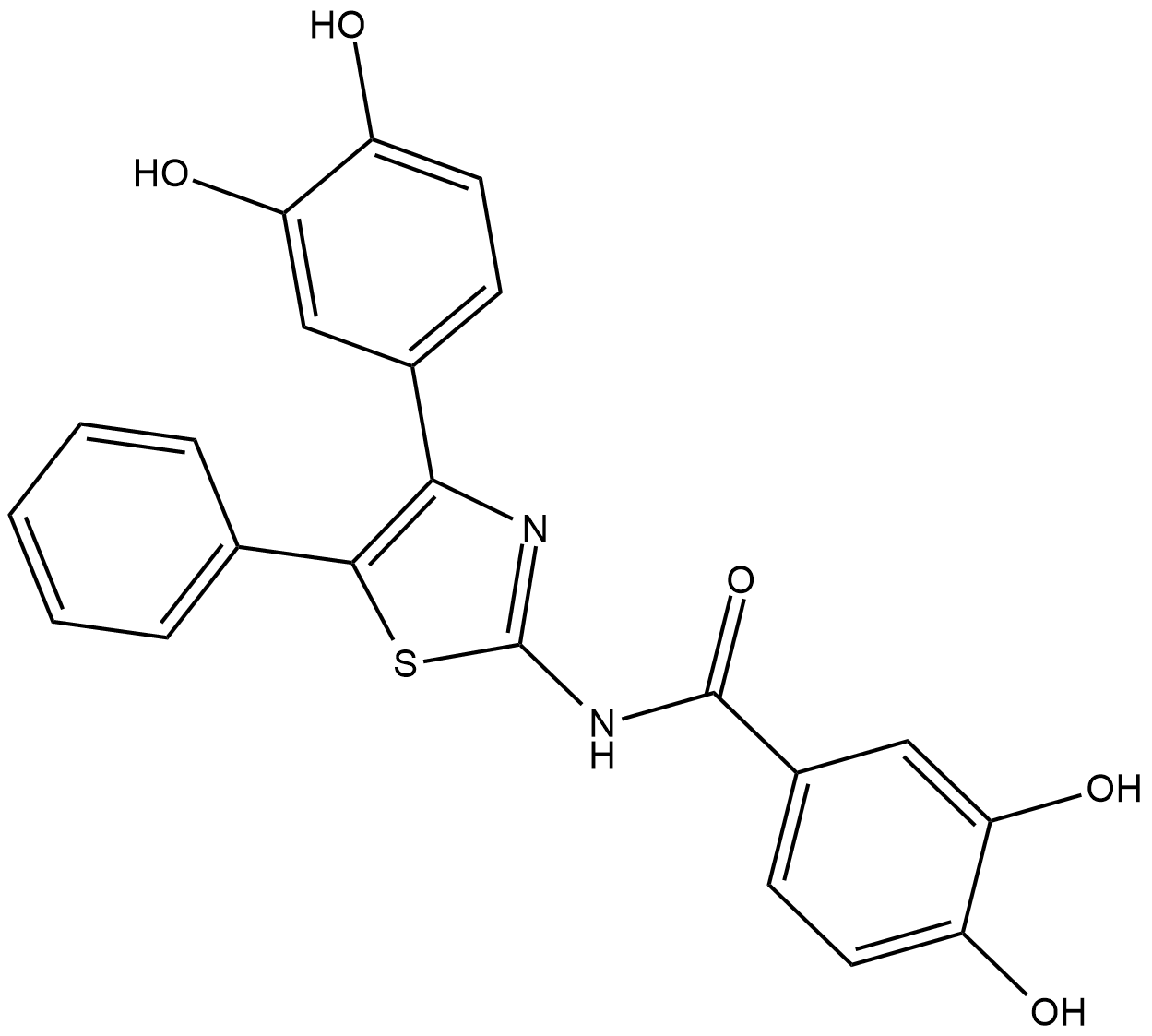
-
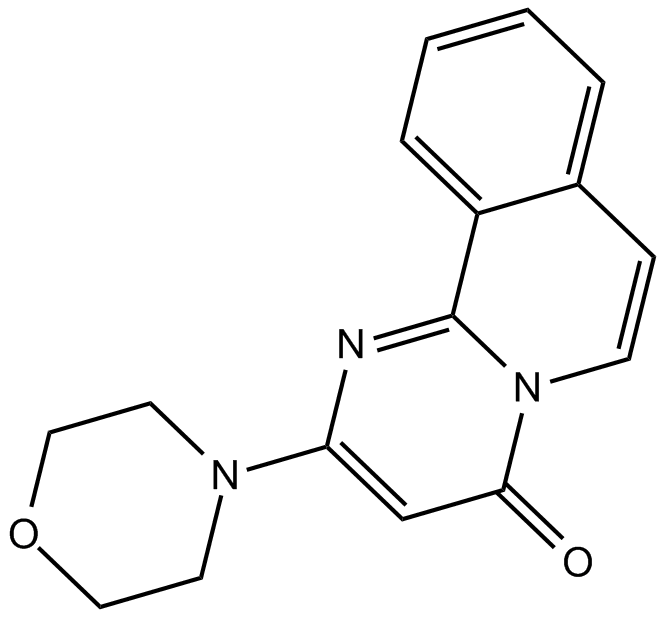
GC10812
Compound 401
Verbindung 401 ist ein synthetischer Inhibitor von DNA-PK (IC50 = 0,28 μM), der in vitro ebenfalls auf mTOR, aber nicht auf PI3K abzielt.
-
GC61965
Coralyne chloride
Coralyne Chlorid ist ein Protoberberin-Alkaloid mit starken Anti-Krebs-AktivitÄten. Coralynechlorid wirkt als potentes Topoisomerase-I-Gift und induziert Top-I-vermittelte DNA-Spaltung. Coralyne-Chlorid kann zur Herstellung von Coralyne-Derivaten als DNA-bindende fluoreszierende Sonden verwendet werden.
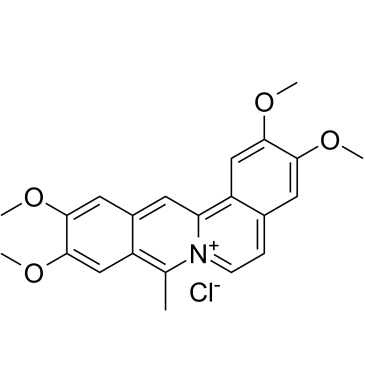
-
GC34165
Corin
Corin ist ein dualer Inhibitor der Histon-Lysin-spezifischen Demethylase (LSD1) und Histon-Deacetylase (HDAC) mit einem Ki(inact) von 110 nM fÜr LSD1 und einem IC50 von 147 nM fÜr HDAC1.
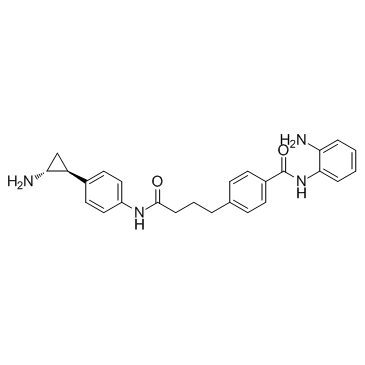
-
GC16116
Costunolide
Costus lactone, Melampolide, NSC 106404
A natural sesquiterpene lactone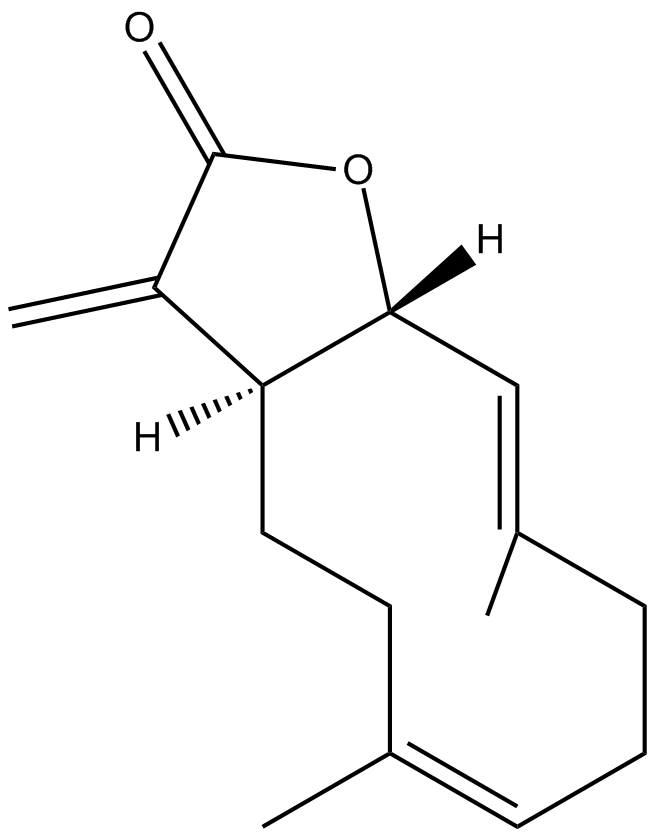
-
GC35739
CP-10
CP-10 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und CDK, mit hochselektivem, spezifischem und bemerkenswertem CDK6-Abbau (DC50=2,1 nM). Es hemmt die Proliferation mehrerer hÄmatopoetischer Krebszellen mit beeindruckender Potenz, einschließlich des multiplen Myeloms, und kann immer noch mutiertes und Überexprimiertes CDK6 abbauen.
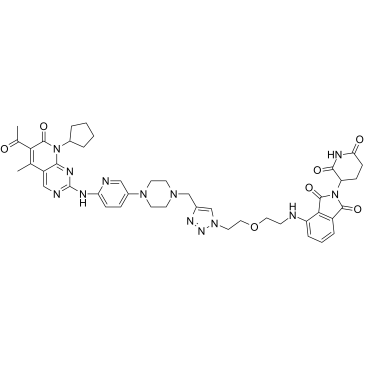
-
GC16489
CP-466722
CP-466722 ist ein schnell reversibler Inhibitor von ATM mit einem IC50 von 4,1 μM und hat keine Auswirkungen auf PI3K oder eng verwandte Familienmitglieder der PI3K-Ähnlichen Proteinkinase (PIKK).
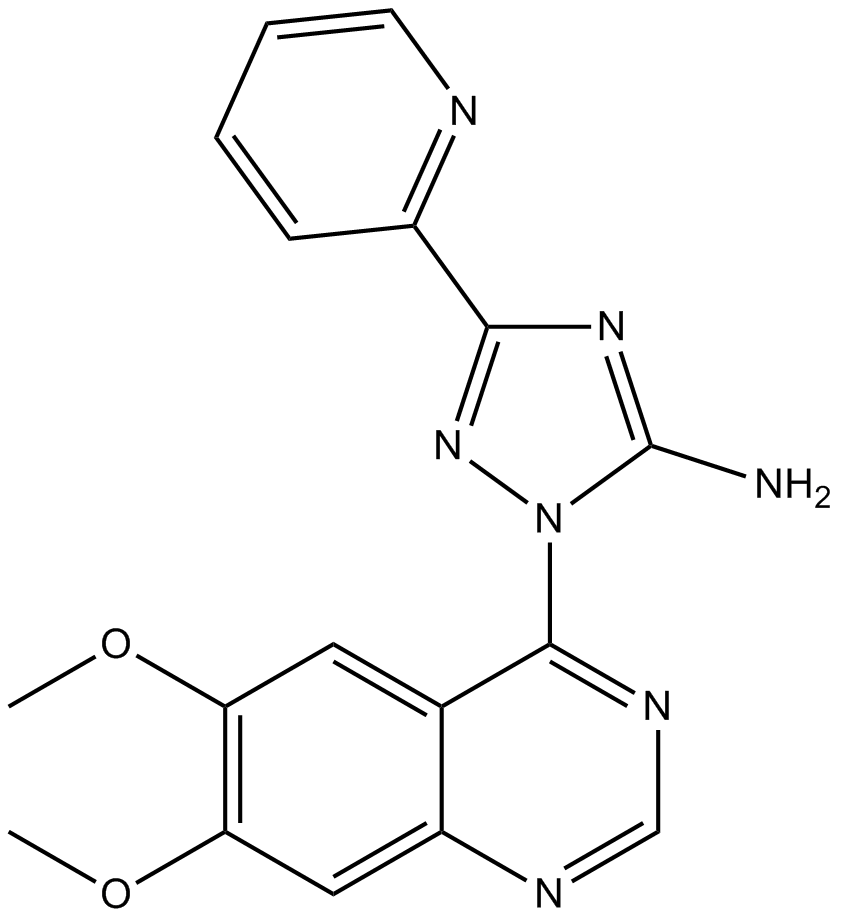
-
GC68455
CP681301

-
GC68898
CPS2
CPS2 ist ein effizientes, selektives und irreversibles PROTAC CDK2-Degradationsmittel (IC50 = 24 nM). CPS2 kann zur Erforschung akuter myeloischer Leukämie verwendet werden.

-
GC32565
CRA-026440
CRA-026440 ist ein potenter Breitspektrum-HDAC-Hemmer.
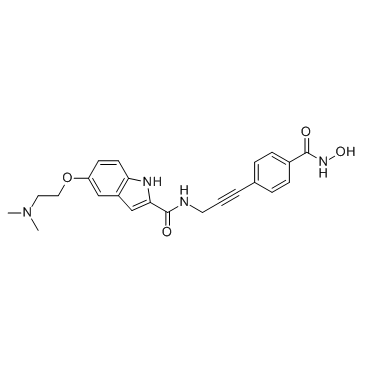
-
GC67674
CRA-026440 hydrochloride
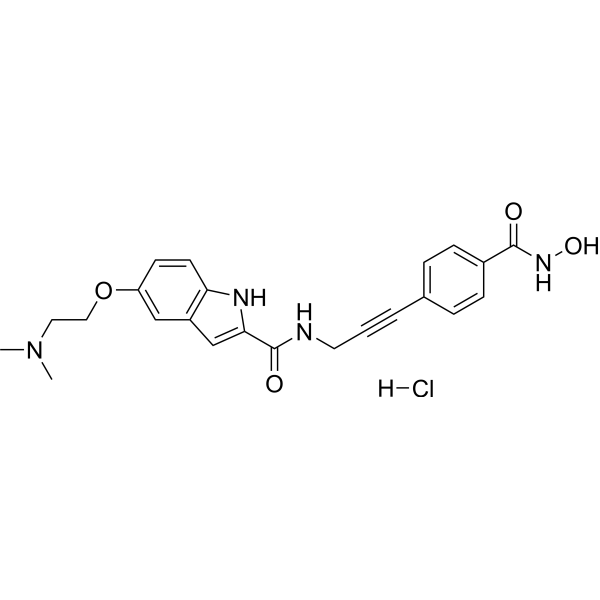
-
GC70432
Crebinostat
Crebinostat ist ein potenter Histondeacetylase (HDAC)-Inhibitor mit IC50-Werten von 0,7 nM, 1,0 nM, 2.0 nM und 9.3 nM für HDAC1, HDAC2, HDAC3 und HDAC6.

-
GC68904
Crisnatol
BWA770U
Crisnatol (BWA770U) is an orally effective anticancer agent and arylmethylamino-propanediol DNA intercalator. Crisnatol exhibits in vitro cytotoxicity against human breast cancer cells, but is ineffective against normal human skin fibroblasts.

-
GC38412
Crotonoside
2-Hydroxyadenosine, Isoguanine riboside, Isoguanosine, NSC 12161
A guanosine analog with diverse biological activities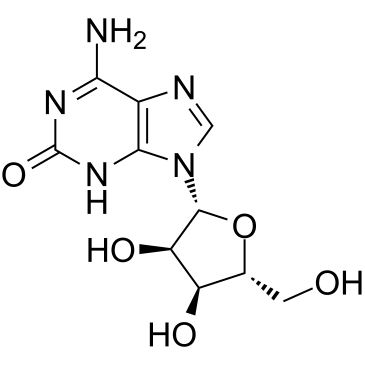
-
GC50404
CRT 0105950
CRT 0105950 ist ein potenter LIMK-Inhibitor mit IC50-Werten von 0,3 nM und 1 nM für LIMK1 bzw. LIMK2. CRT 0105950 kann für die Krebsforschung verwendet werden.
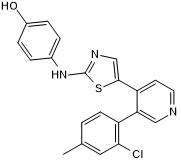
-
GC17231
CRT0044876
CRT0044876, 7-Nitroindole-2-Carboxylic Acid, NSC 69877
CRT0044876 ist ein potenter und selektiver Inhibitor der Apurin-/Apyrimidin-Endonuklease 1 (APE1) (IC50=~3 μM). CRT0044876 hemmt die AP-Endonuklease-, 3′-Phosphodiesterase- und 3′-Phosphatase-AktivitÄten von APE1 und ist ein spezifischer Inhibitor der Exonuklease-III-Enzymfamilie, zu der APE1 gehÖrt. CRT0044876 potenziert die ZytotoxizitÄt mehrerer auf DNA-Basen gerichteter Verbindungen.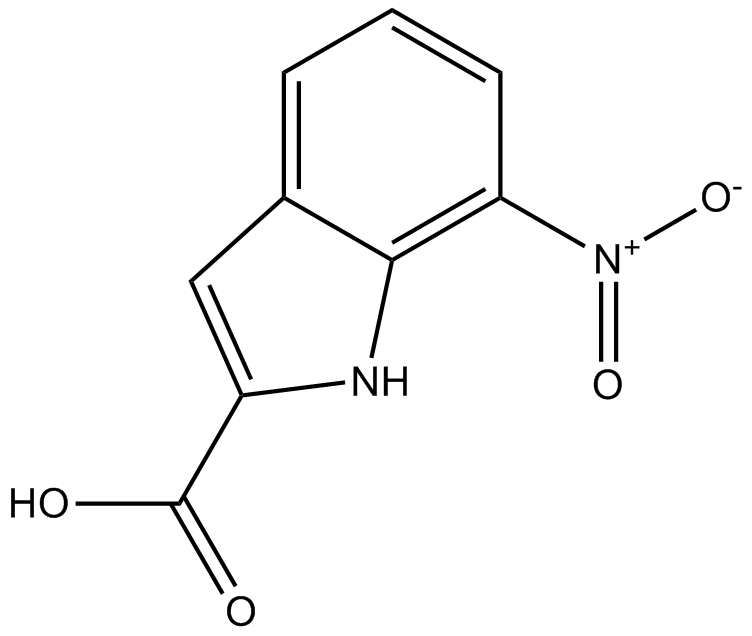
-
GC71196
CTP Synthetase-IN-1
CTP Synthetase-IN-1 ist ein potenter, oral aktiver Cytidin-5'-Triphosphat-Synthetase (CTPS)-Inhibitor mit IC50s von 32 nM und 18 nM für humane CTPS1 bzw. humane CTPS2.

-
GC65023
CTX-712
CTX-712 ist ein potenter Inhibitor der cdc2-Ähnlichen Kinase (CLK). CTX-712 hemmt die AktivitÄt der CLK-Kinase und hemmt somit das Überleben von Krebs und das Wachstum von Krebszellen. CTX-712 hat das Potenzial fÜr die Erforschung von Krebserkrankungen (aus Patent JPWO2017188374A1, Verbindung 286).
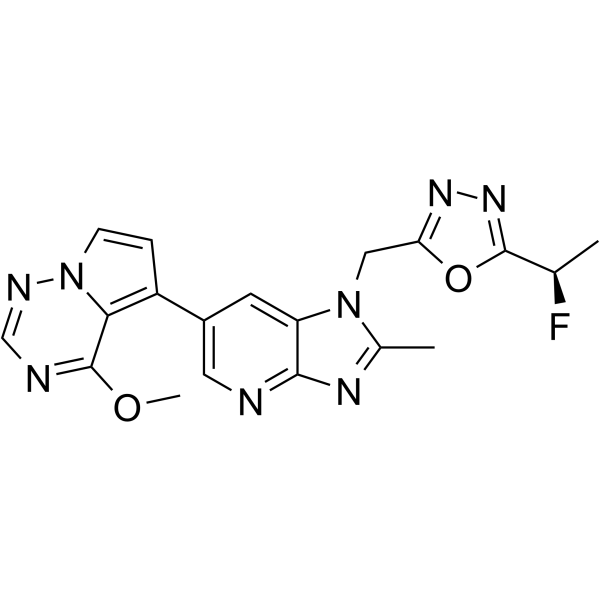
-
GN10526
Cucurbitacin E
NSC 106399, NSC 521775
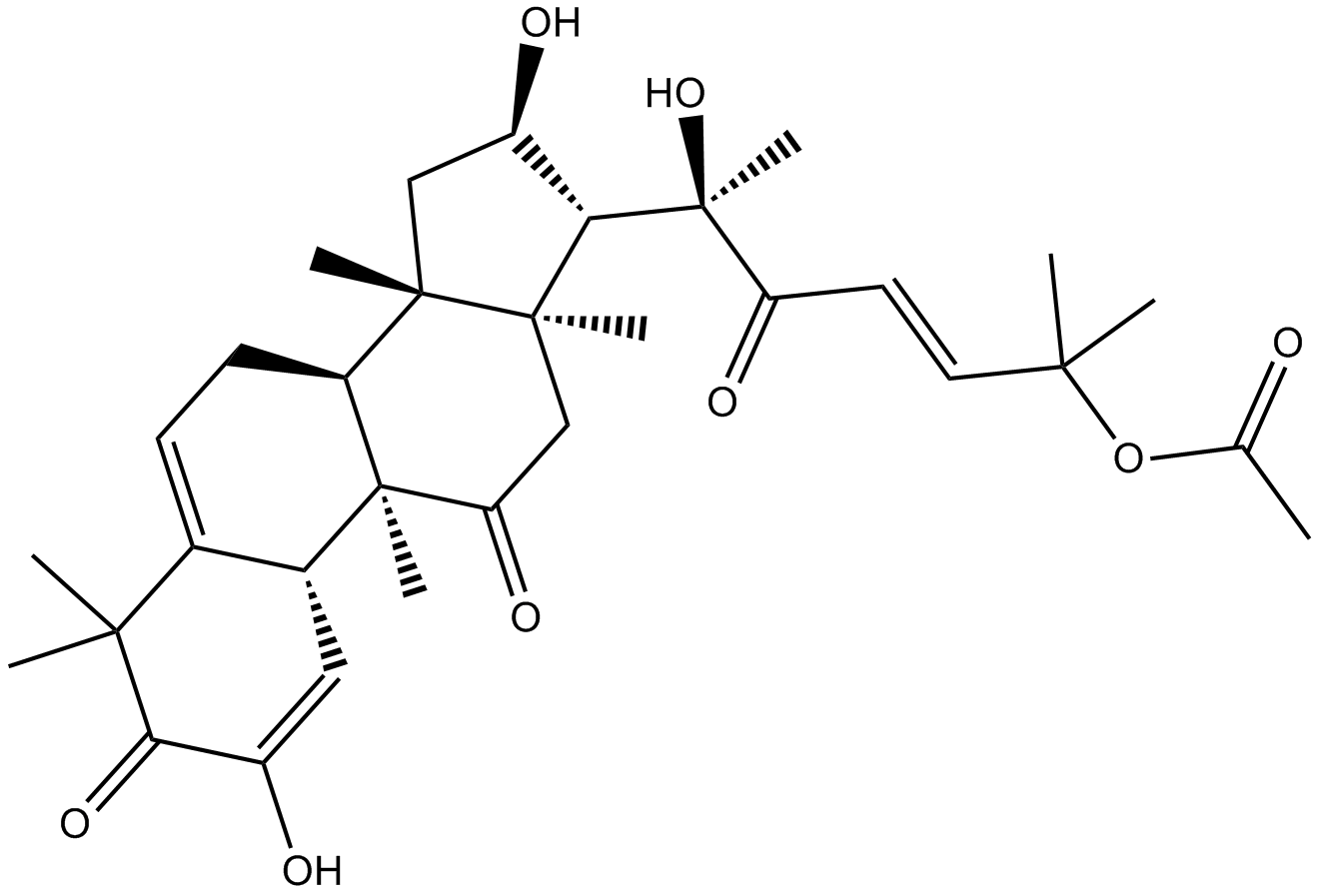
-
GC68913
Curcolonol
Curcolonol is a furanoid sesquiterpene. Curcolonol can be isolated from several herbs. Curcolonol has inhibitory activity against LIM kinase 1. Curcolonol can be used for research on breast cancer.

-
GC18028
CVT-313
CVT 313;NG 26;CVT313;NG26;NG-26
A Cdk2 inhibitor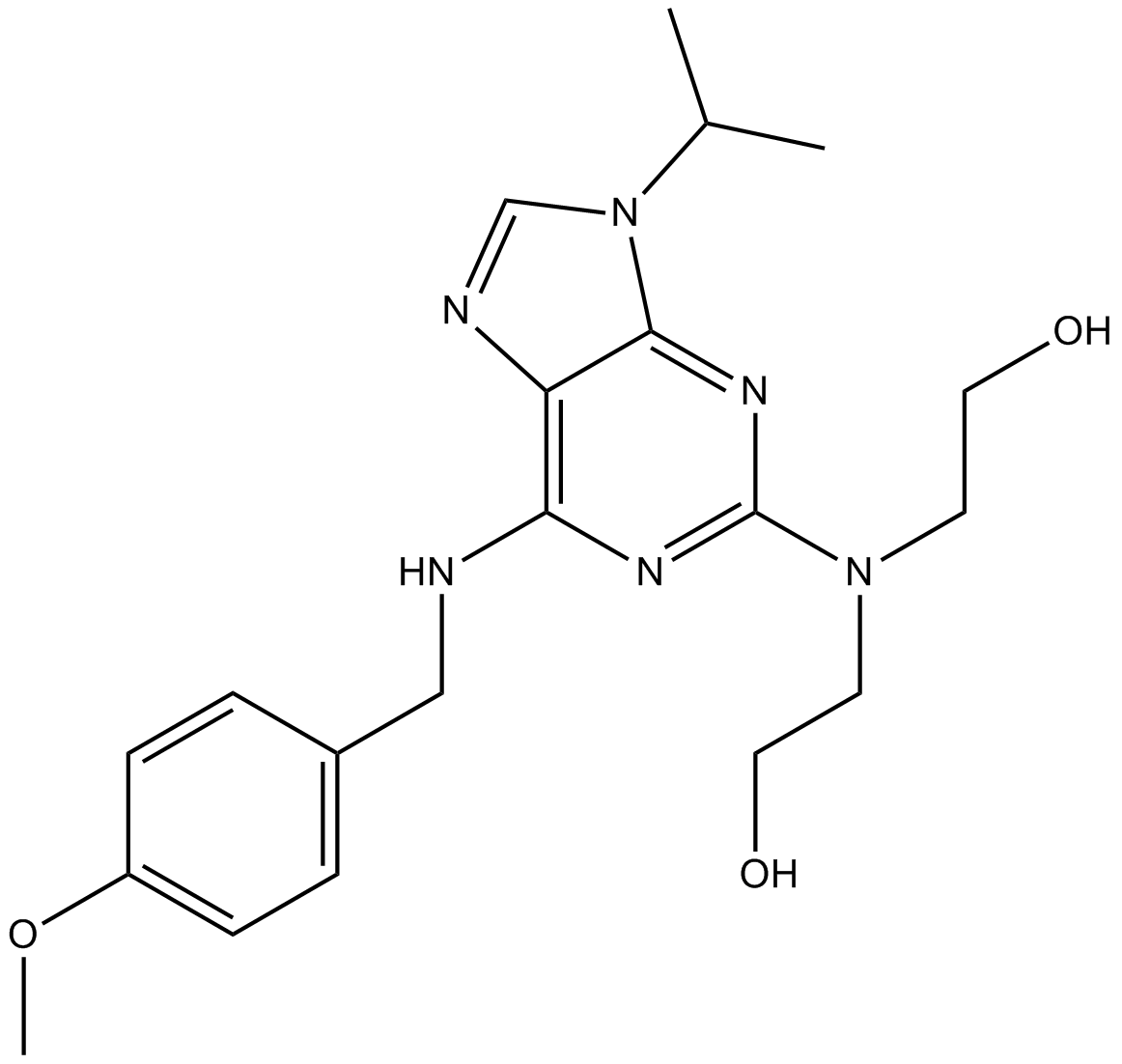
-
GC13037
CX-4945 (Silmitasertib)
CX 4945;CX4945
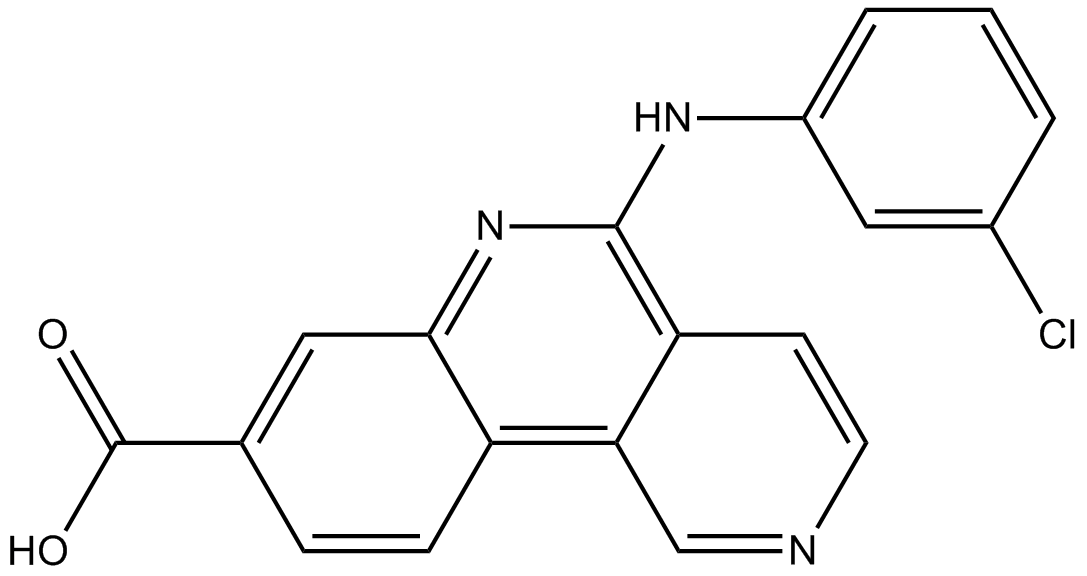
CX-4945 (Silmitasertib) ist ein oral verfügbarer, hochselektiver und potenter CK2-Inhibitor mit IC50-Werten von 1 nM gegenüber CK2α und CK2α'.
-
GC11325
CX-4945 sodium salt
CX-4945 sodium salt
CX-4945-Natriumsalz ist ein oral bioverfÜgbarer, hochselektiver und potenter CK2-Inhibitor mit IC50-Werten von 1 nM gegen CK2α und CK2α'.
-
GC14404
CX-5461
CX 5461;CX5461
CX-5461CX-5461 ist ein oral wirksamer Inhibitor der rRNA-Synthese, der die von der RNA-Polymerase (RNA Pol) I gesteuerte rRNA-Transkription mit IC50-Werten von 142, 113 bzw. 54 nM in HCT-116, A375 und MIA PaCa-2 Zellen hemmt.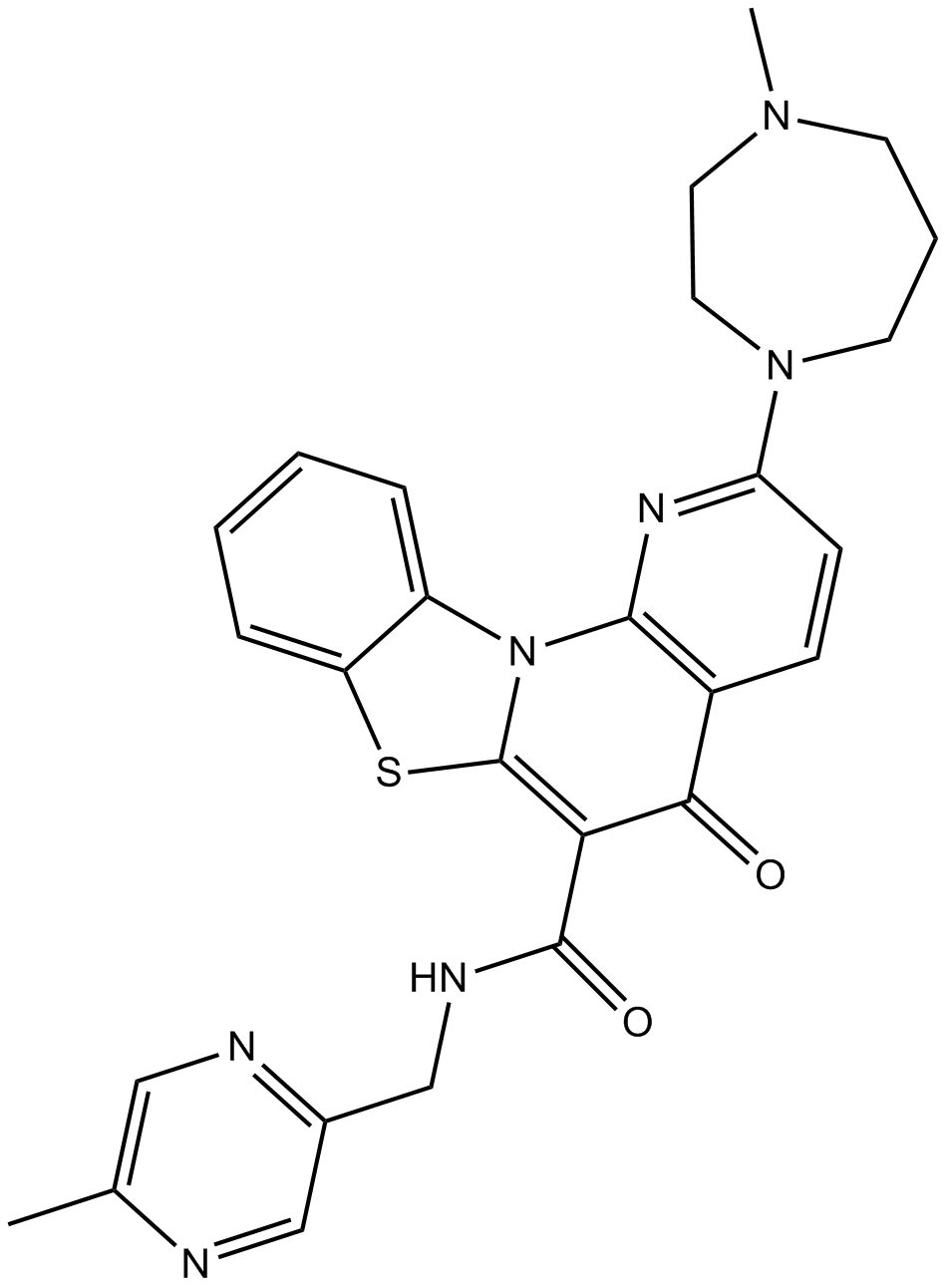
-
GC35761
CX-5461 dihydrochloride
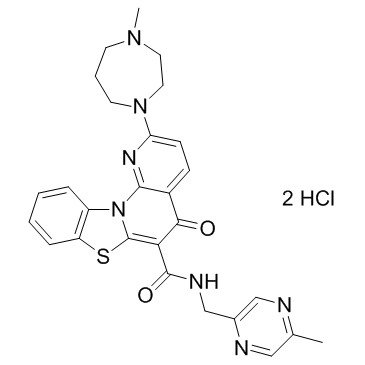
CX-5461-Dihydrochlorid ist ein potenter und oral bioverfÜgbarer Inhibitor der Pol I-vermittelten rRNA-Synthese mit IC50-Werten von 142 nM in HCT-116-, 113 nM in A375- und 54 nM in MIA-PaCa-2-Zellen und zeigt wenig oder keine Wirkung auf Pol II (IC50 ≥25 μM).
-
GC71778
Cy5-dATP
Cy5-dATP ist ein mit Cy5 gekennzeichneter dATP.

-
GC32700
CYC065
CYC065
CYC065 (CYC065) ist ein oral verfÜgbarer ATP-kompetitiver Inhibitor der zweiten Generation von CDK2/CDK9-Kinasen mit IC50-Werten von 5 bzw. 26 nM.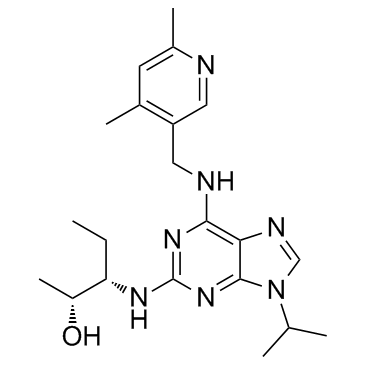
-
GC34309
Cycloguanil D6 (Chlorguanide triazine D6)
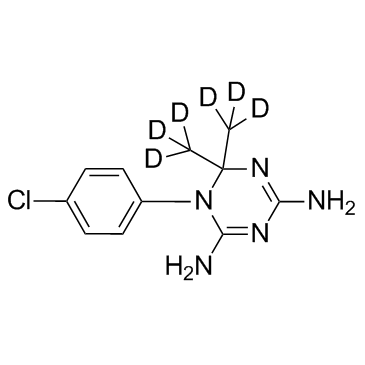
-
GC35781
Cycloguanil D6 Nitrate
Chlorguanide triazine D6 Nitrate
Cycloguanil D6 Nitrat ist das mit Deuterium markierte Cycloguanil, das ein Dihydrofolat-Reduktase-Hemmer ist.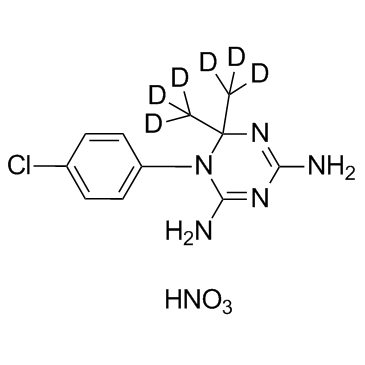
-
GC11145
Cyclophosphamide
Cyclophosphamide ist ein häufig verwendetes Chemotherapeutikum, das oft in Kombination mit anderen Chemotherapie-Arten zur Behandlung von Brustkrebs, malignen Lymphomen, multiplem Myelom und Neuroblastom eingesetzt wird.
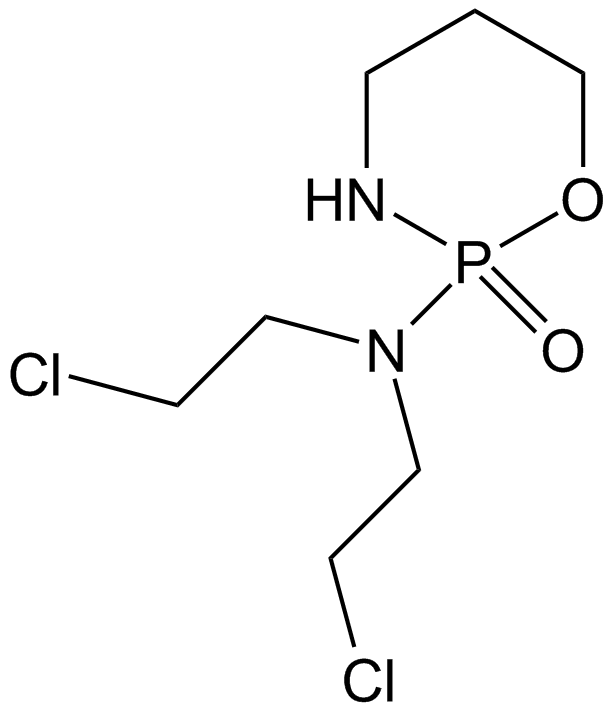
-
GC14077
Cyclophosphamide monohydrate
An alkylating agent
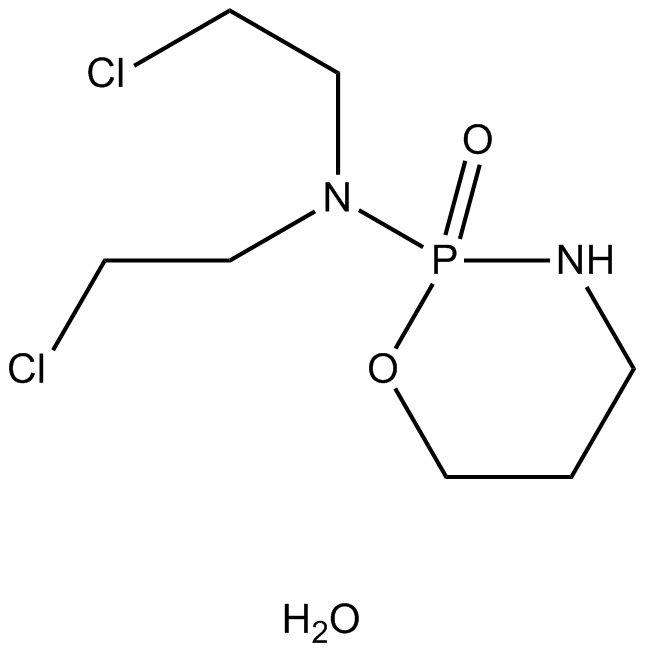
-
GC47148
Cyclophosphamide-d4
Cyclophosphamid-d4 ist das mit Deuterium markierte Cyclophosphamid. Cyclophosphamid ist ein synthetisches Alkylierungsmittel, das chemisch mit Stickstofflost mit antineoplastischer AktivitÄt, einem Immunsuppressivum, verwandt ist.
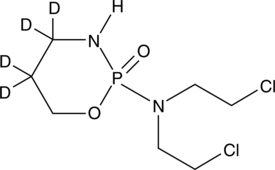
-
GC43351
Cylindrospermopsin
Cylindrospermopsin, a tricyclic uracil derivative, is a cyanobacterial toxin that was first discovered in an algal bloom contaminating a local drinking supply on Palm Island in Queensland, Australia after an outbreak of a mysterious disease.
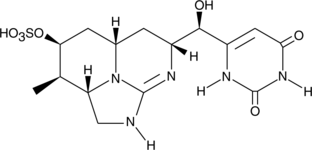
-
GC13070
Cytarabine
1βDArabinofuranosylcytosine, NSC 63878, NSC 287459, U19920A
Zytotoxisches Mittel, blockiert die DNA-Synthese.
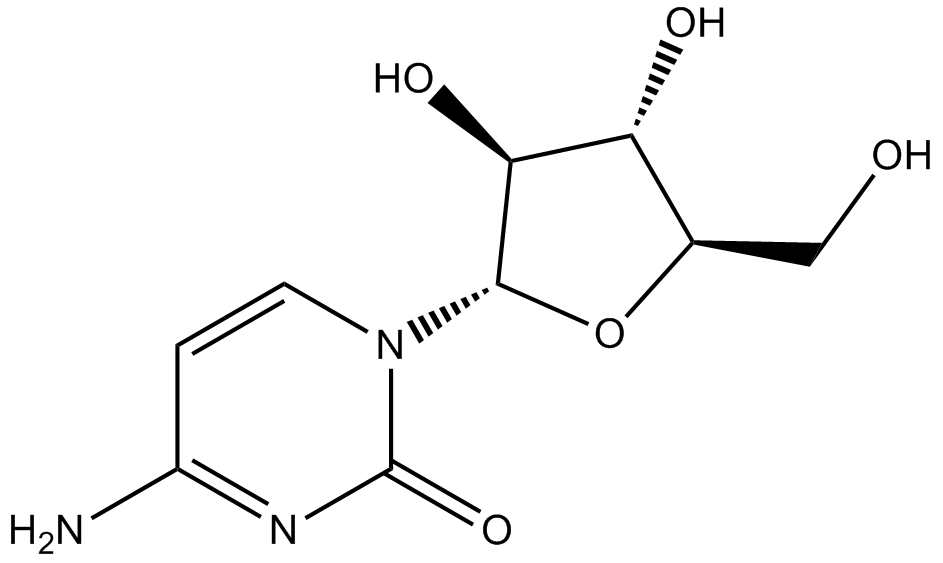
-
GC49863
Cytarabine 5′-monophosphate
ara-CMP, 1-β-D-Arabinofuranosylcytosine 5'-monophosphate, Cytosine Arabinoside monophosphate, NSC 99445
An active metabolite of cytarabine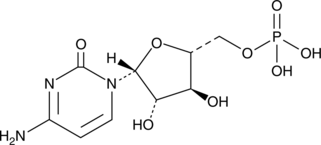
-
GC14961
Cytarabine hydrochloride
Cytarabin-Hydrochlorid, ein Nukleosid-Analogon, verursacht einen Stopp des Zellzyklus in der S-Phase und hemmt die DNA-Polymerase.
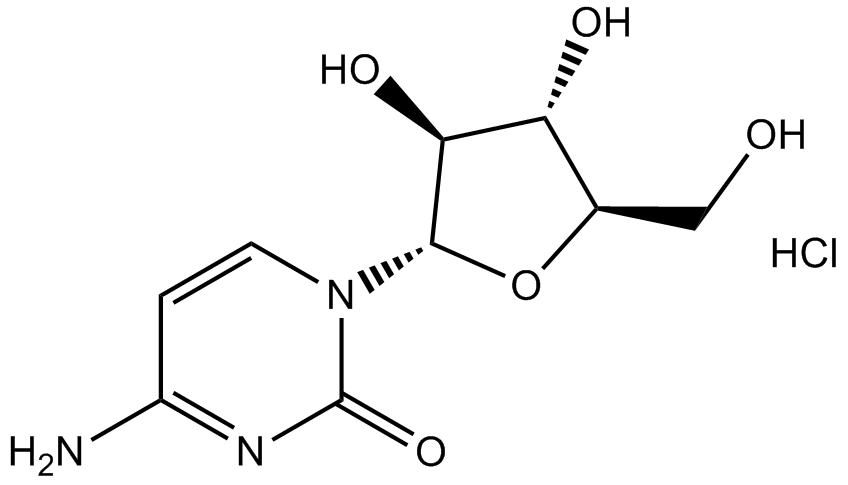
-
GC13729
Cytidine
β-D-Cytidine, NSC 20258
Cytidin ist ein Pyrimidinnukleosid und fungiert als Bestandteil der RNA.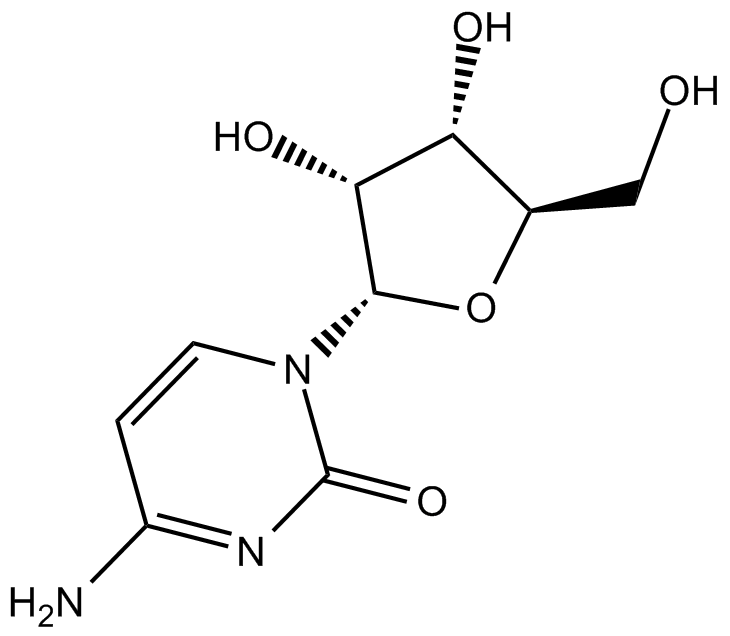
-
GC49104
Cytidine 3’-monophosphate
3'-CMP, Cytidine 3'-phosphate
A ribonucleotide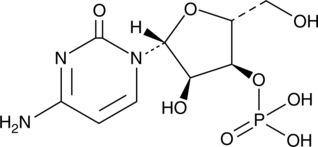
-
GC47162
Cytidine 5'-triphosphate (sodium salt hydrate)
CTP, NSC 20261
A pyrimidine nucleoside triphosphate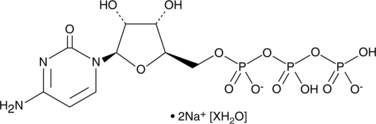
-
GC49526
Cytidine-d2
β-D-Cytidine-d2
An internal standard for the quantification of cytidine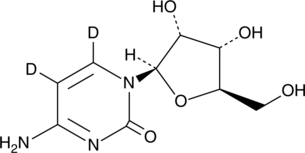
-
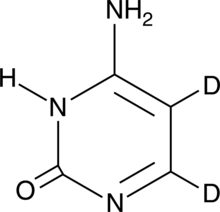
GC49464
Cytosine-d2
An internal standard for the quantification of cytosine
-
GC39149
D-I03
D-I03 ist ein selektiver RAD52-Inhibitor mit einem Kd von 25,8 μM. D-I03 hemmt spezifisch das RAD52-abhÄngige Single-Strand-Annealing (SSA) und die D-Loop-Bildung mit IC50-Werten von 5 μM bzw. 8 μM. D-I03 unterdrÜckt das Wachstum von BRCA1- und BRCA2-defizienten Zellen und hemmt die Bildung von schÄdigungsinduzierten RAD52-Foci, wirkt sich jedoch nicht auf durch Cisplatin induzierte RAD51-Foci aus.
-
GC47276
D-threo-PPMP (hydrochloride)
D-threo-1-phenyl-2-Palmitoylamino-3-morpholino-1-propanol
D-threo-PPMP (Hydrochlorid) ist ein potenter Inhibitor der Glucosylceramid (GlcCer)-Synthase.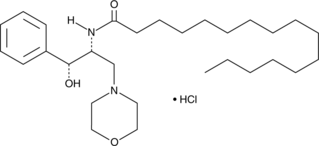
-
GC13202
D4476
Casein Kinase I Inhibitor
Inhibitor of CK1 and ALK5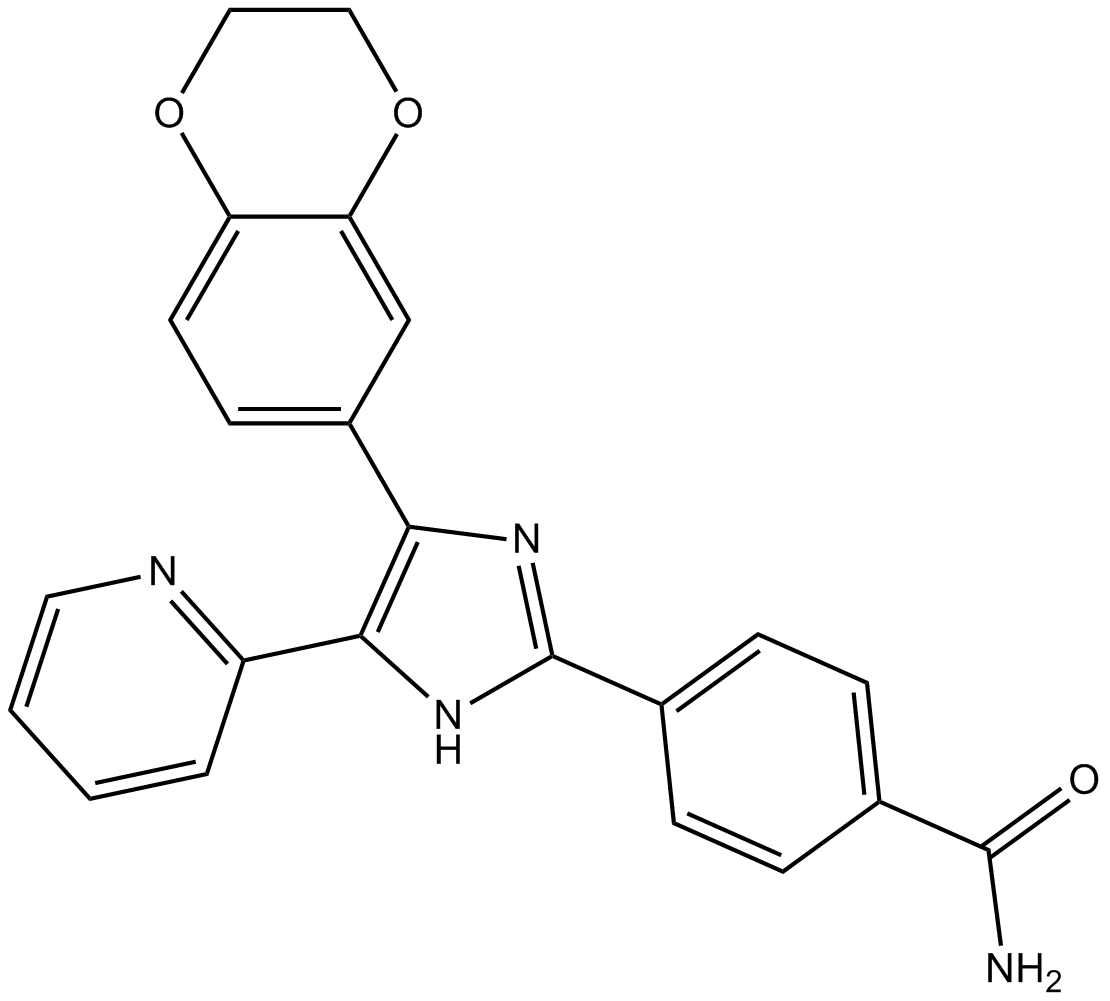
-
GC14485
Dacarbazine
NCI C04717, NSC 45388
Dacarbazin ist ein fÜr den Zellzyklus unspezifisches antineoplastisches Alkylierungsmittel. Dacarbazin hemmt die T- und B-lymphoblastische Reaktion mit IC50-Werten von 50 bzw. 10 μg/ml. Dacarbazin kann fÜr die Erforschung des metastasierten malignen Melanoms verwendet werden.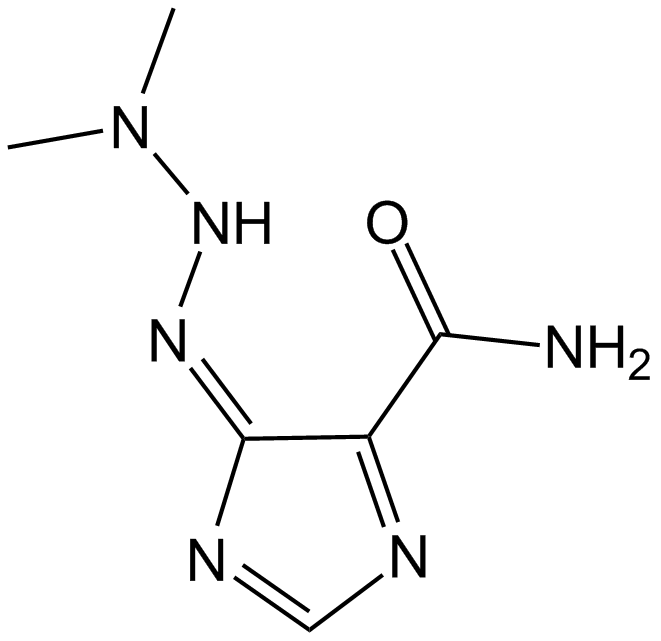
-
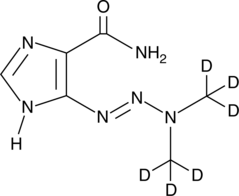
GC47167
Dacarbazine-d6
Dacarbazin-d6 (Imidazolcarboxamid-d6) ist das mit Deuterium bezeichnete Dacarbazin. Dacarbazin (DTIC-Dome; DTIC) ist ein antineoplastisches Mittel. Es hat eine signifikante AktivitÄt gegen Melanome.
-
GC63419
Dalpiciclib
SHR-6390
Dalpiciclib (SHR-6390) ist ein oral aktiver und hochselektiver Inhibitor von CDK4 und 6 mit IC50-Werten von 12,4nM bzw. 9,9nM. Dalpiciclib zeigt AntitumoraktivitÄt gegen Brustkrebs und Plattenepithelkarzinom des Ösophagus.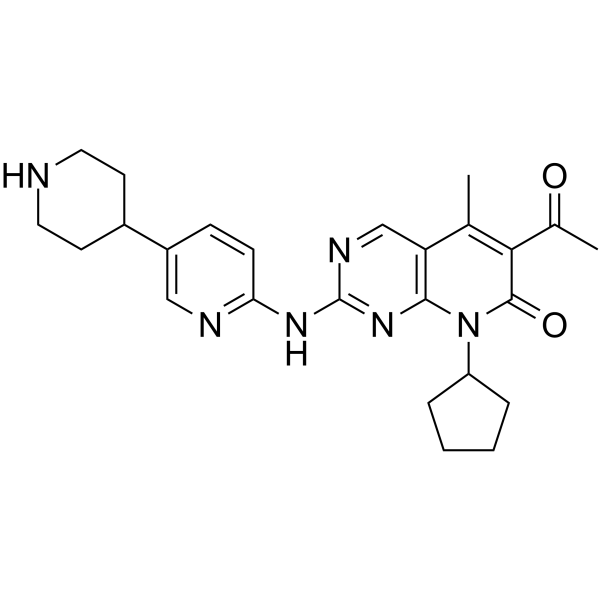
-
GC65369
Dalpiciclib hydrochloride
SHR-6390 hydrochloride
Dalpiciclib (SHR-6390) Hydrochlorid ist ein oral aktiver und hochselektiver Inhibitor von CDK4 und 6 mit IC50-Werten von 12,4nM bzw. 9,9nM. Dalpiciclib-Hydrochlorid zeigt AntitumoraktivitÄt gegen Brustkrebs und Plattenepithelkarzinom des Ösophagus.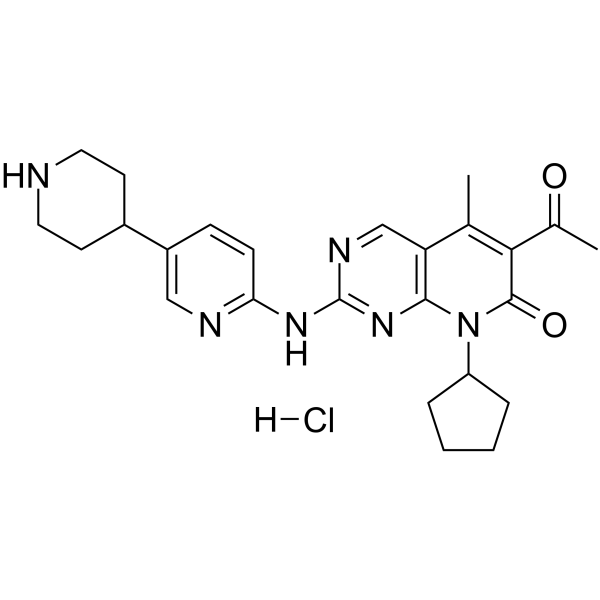
-
GC10214
Daptomycin
LY146032
Daptomycin ist ein Lipopeptid-Antibiotikum mit schneller bakterizider In-vitro-AktivitÄt gegen grampositive Organismen.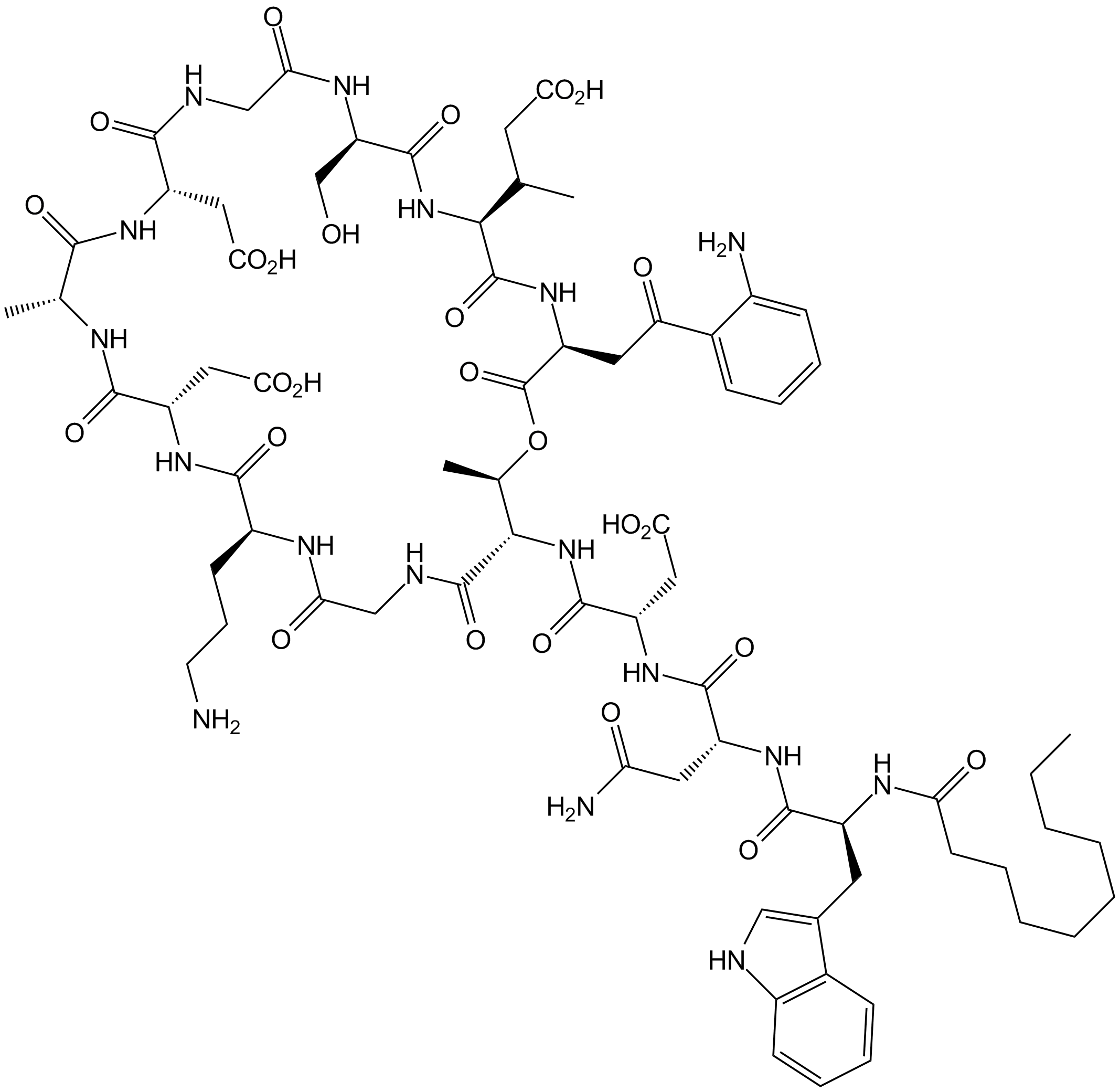
-
GC33124
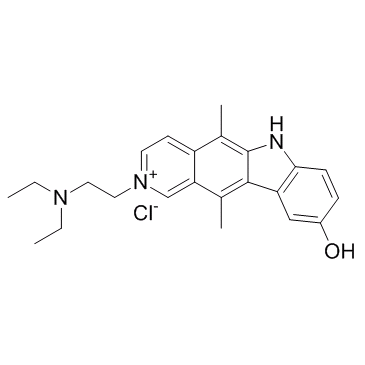
Datelliptium chloride
Datelliptiumchlorid ist ein von Ellipticin abgeleitetes DNA-interkalierendes Mittel mit Antitumorwirkung.
-
GC11175
Daun02
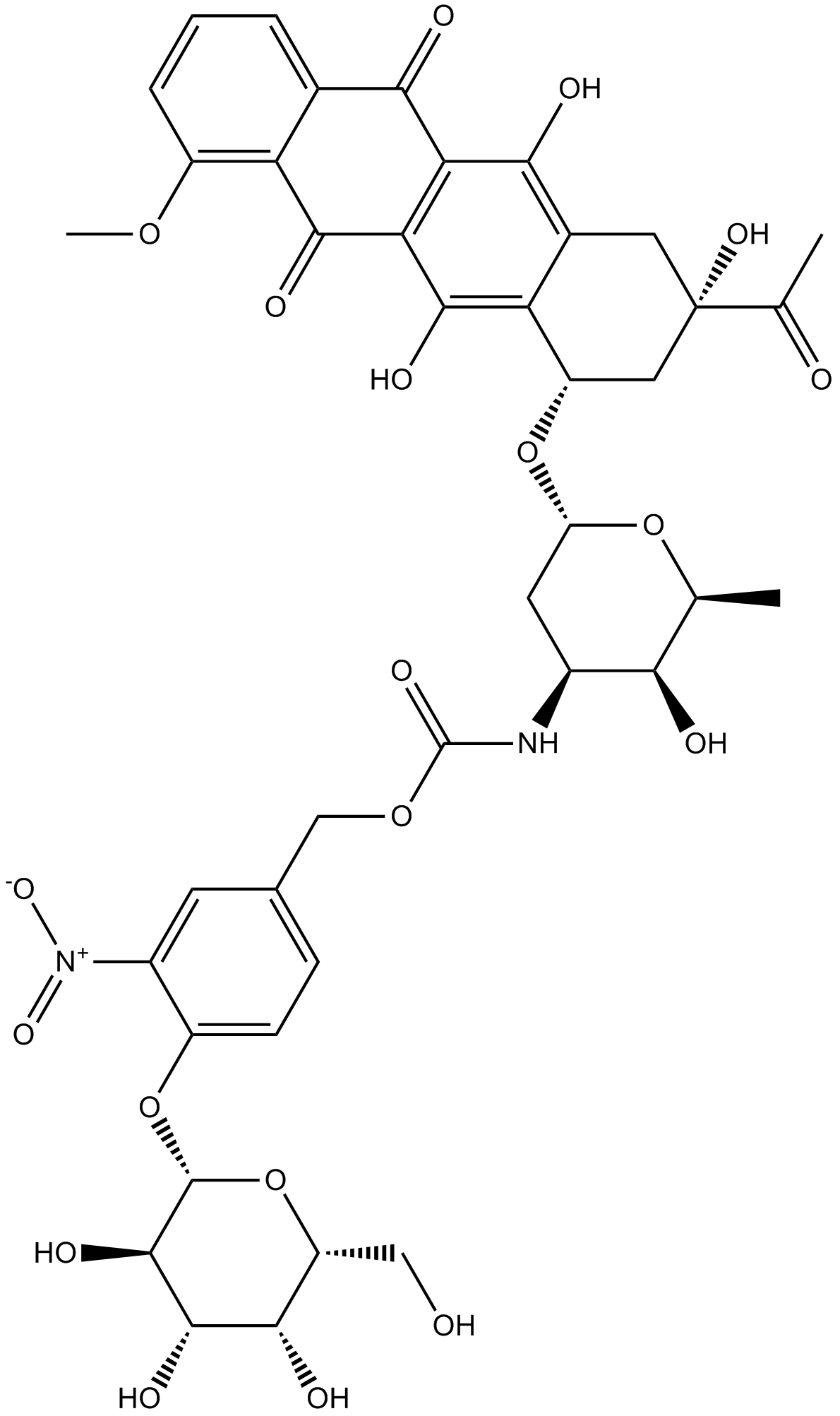
-
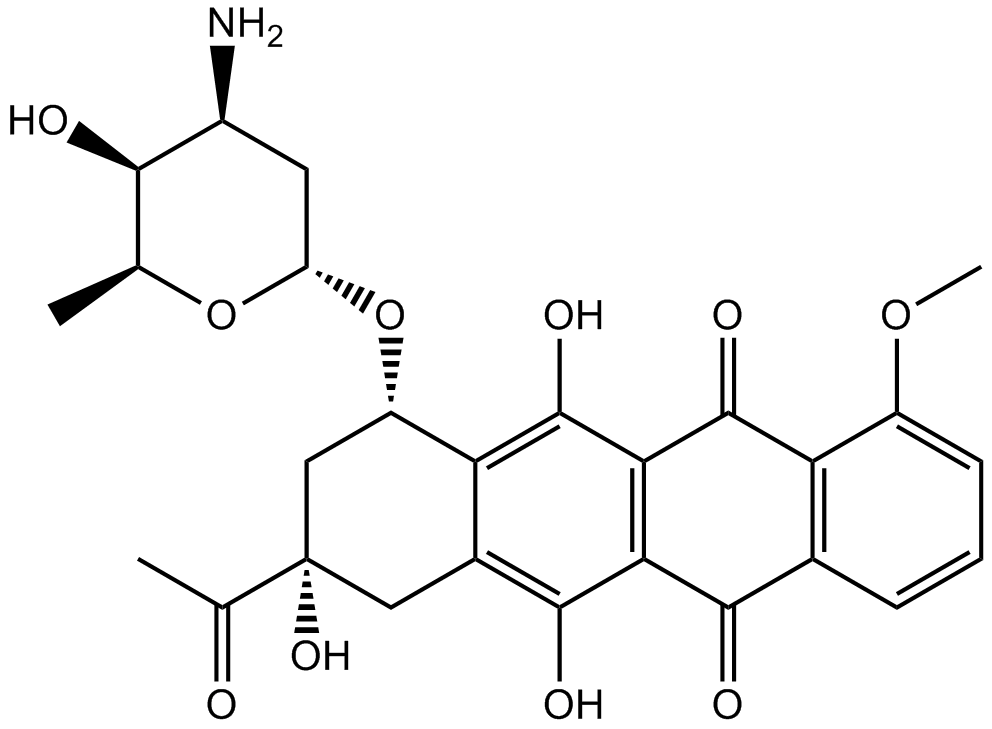
GC12828
Daunorubicin
DNA-Topoisomerase-II-Inhibitor
-
GC10354
Daunorubicin HCl
NDC 00824155, Ondena, RP 13057
Daunorubicin (Daunomycin)-Hydrochlorid ist ein Topoisomerase-II-Inhibitor mit starker AntitumoraktivitÄt.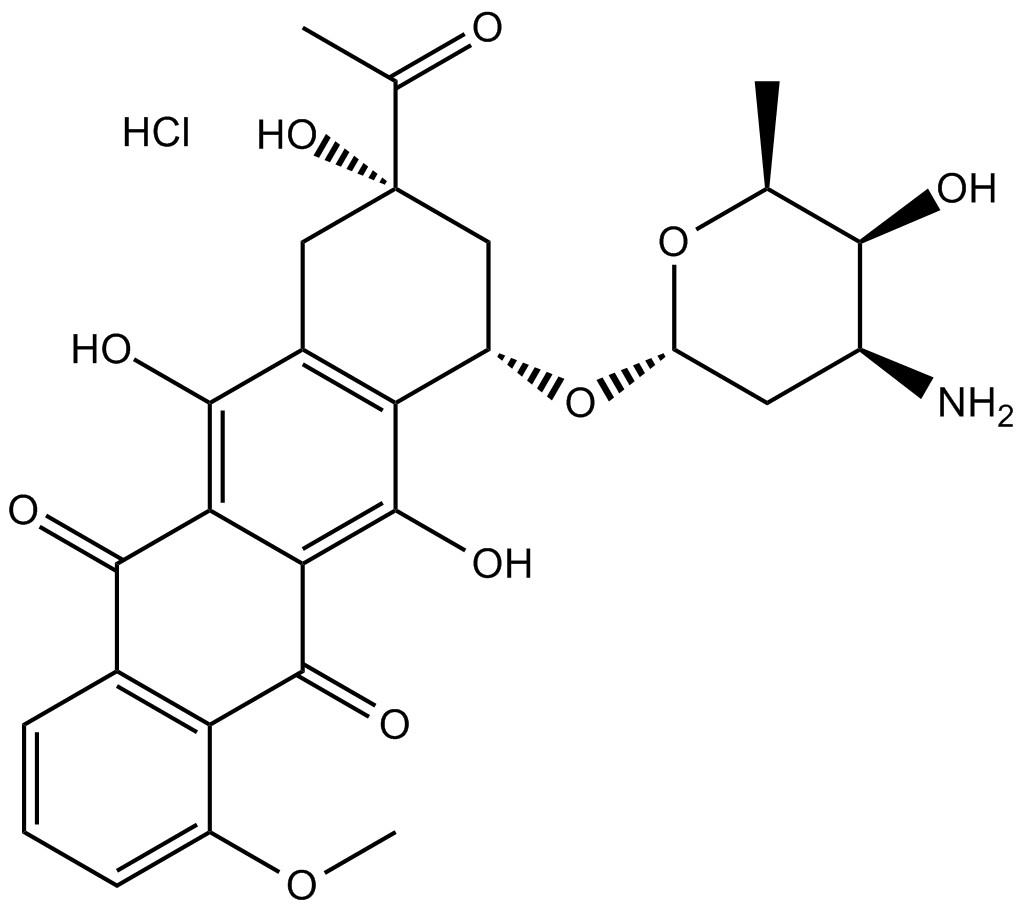
-
GC68940
DB008
DB008 ist ein wirksamer selektiver PARP16-Inhibitor mit einem IC50-Wert von 0,27 μM und enthält einen Acrylamid-Elektrophil. DB008 hat Membranpermeabilität und kann PARP16 selektiv markieren.

-
GC71011
DB0614
DB0614 (Beispiel 21) ist eine bifunktionelle Verbindung, die zielgerichteten Proteinabbau von Kinasen darstellt.

-
GC33148
DC-05
DC-05 ist ein DNA-Methyltransferase 1 (DNMT1)-Inhibitor mit einem IC50- und einem Kd-Wert von 10,3 μM bzw. 1,09 μM.
-
GC67886
DC-U4106

-
GC32872
DC_517
DC_517 ist ein Inhibitor der DNA-Methyltransferase 1 (DNMT1) mit einem IC50- und einem Kd-Wert von 1,7 μM bzw. 0,91 μM.
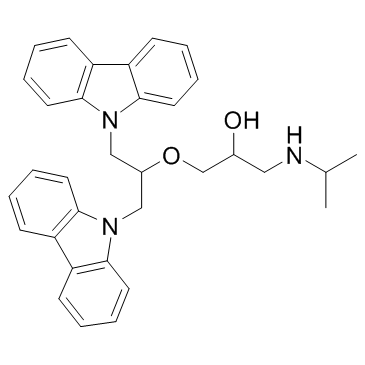
-
GC67970
DD-03-156
(S,R,S)-AHPC-Me-PEG2-dabrafenib
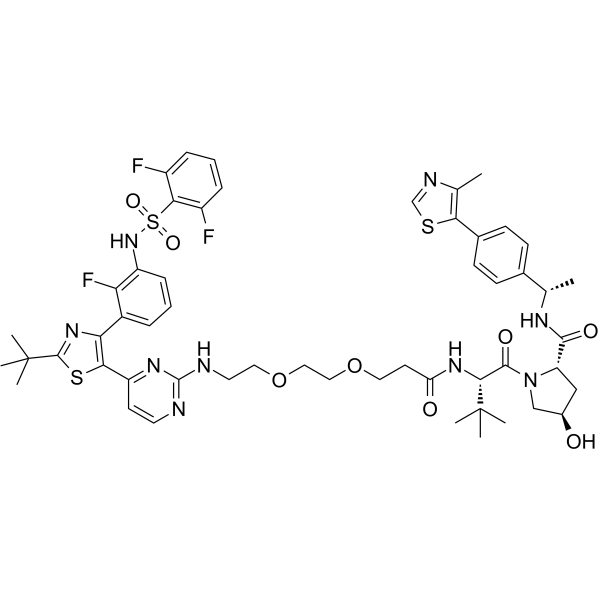
-
GC68952
ddATP trisodium
2',3'-Dideoxyadenosine 5'-triphosphate trisodium
ddATP (2',3'-Dideoxyadenosin-5'-triphosphat) Trinatrium ist ein aktives Stoffwechselprodukt von 2',3'-Didesoxyadenosin und ein Hemmstoff der Kettenverlängerung durch DNA-Polymerase. ddATP Trinatrium kann für die Sanger-Sequenzierung von DNA sowie für die Erforschung virusbedingter Infektionen verwendet werden.

-
GC65440
ddCTP trisodium
ddCTP-Trinatrium ist eines der 2',3'-Didesoxyribonukleosid-5'-Triphosphate (ddNTPs), das als kettenverlÄngernder Inhibitor der DNA-Polymerase fÜr die DNA-Sequenzierung wirkt.
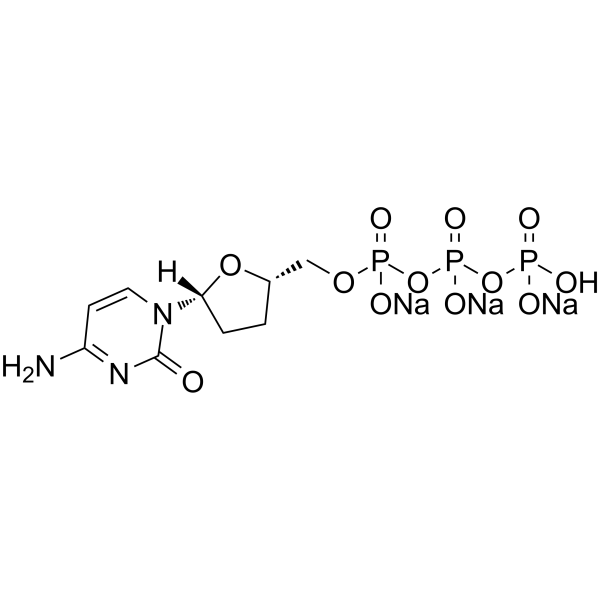
-
GC65295
ddGTP trisodium
2′,3′-Dideoxyguanosine 5′-triphosphate trisodium
ddGTP (2′,3′-Dideoxyguanosin-5′-triphosphat) Trinatrium ist eines der 2′,3′-Didesoxyribonukleosid-5′-triphosphate (ddNTPs), das als kettenverlÄngernder Inhibitor der DNA-Polymerase fÜr die DNA-Sequenzierung wirkt.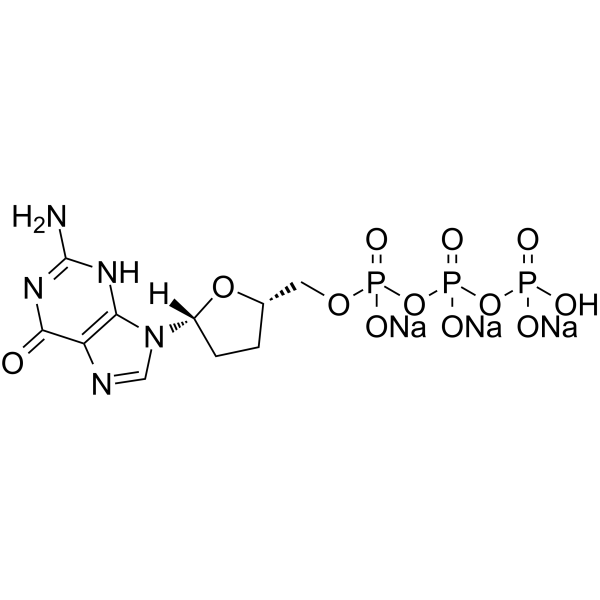
-
GC64020
ddTTP
ddTTP ist eines der 2',3'-Didesoxyribonukleosid-5'-Triphosphate (ddNTPs), das als kettenverlÄngernder Inhibitor der DNA-Polymerase fÜr die DNA-Sequenzierung fungiert.
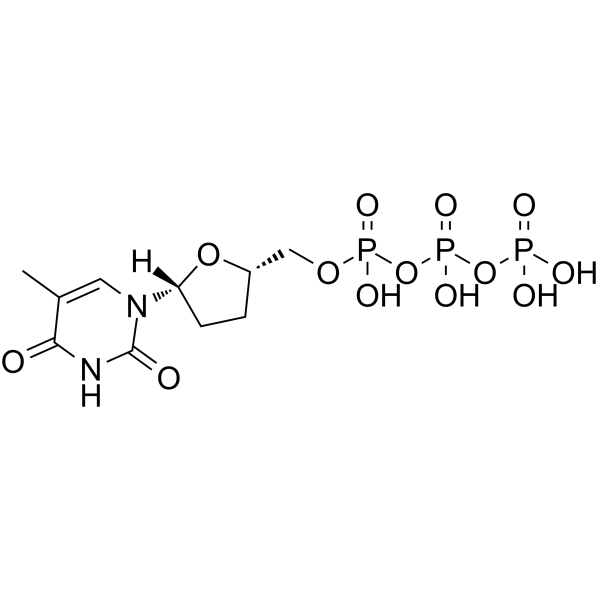
-
GC70789
ddTTP trisodium
ddTTP trisodium ist eines von 2',3'-Dideoxyribonucleosid-5'-Triphosphaten (ddNTPs), das als kettenverlängernder Inhibitor der DNA-Polymerase für die DNA-Sequenzierung wirkt.

-
GC18666
Debromohymenialdisine
DBH, SKF 108753
Damaged DNA in humans is detected by sensor proteins that transmit a signal through checkpoint kinases (Chks) Chk1 and Chk2.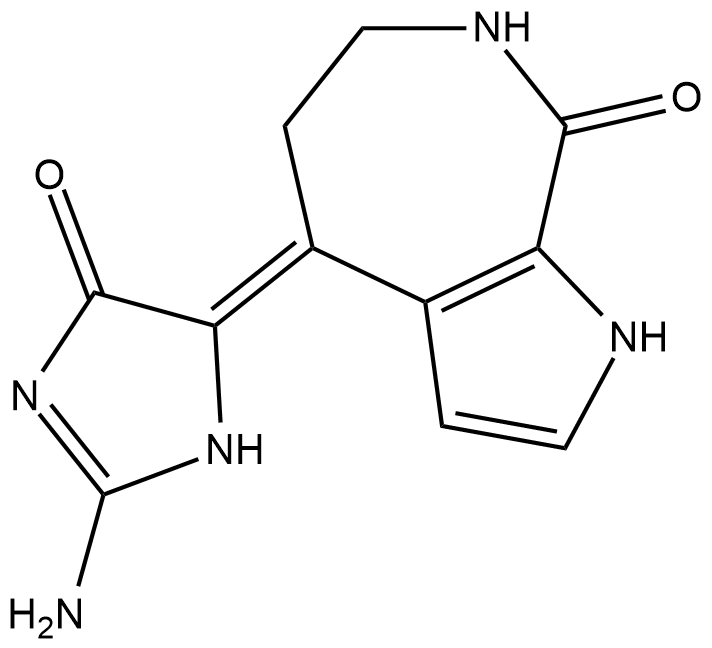
-
GC17412
Deferasirox Fe3+ chelate
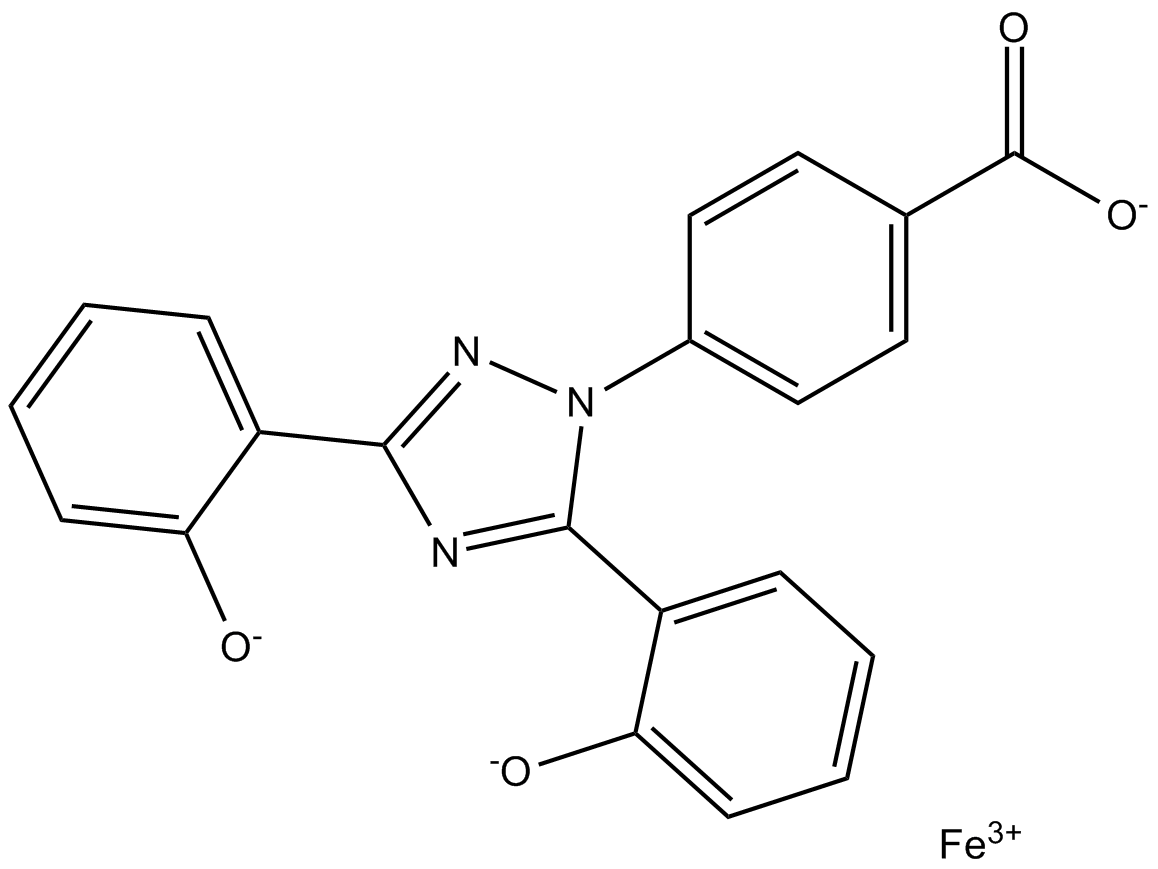
-
GC35829
Dehydroaltenusin
Dehydroaltenusin ist ein niedermolekularer selektiver Inhibitor der eukaryotischen DNA-Polymerase α, einer Art von Antibiotikum, das von einem Pilz mit einem IC50-Wert von 0,68 μM produziert wird. Die hemmende Wirkungsweise von Dehydroaltenusin gegen die Pol-α-AktivitÄt von SÄugetieren ist kompetitiv in Bezug auf den DNA-Template-Primer (Ki = 0,23 μM) und nicht kompetitiv in Bezug auf das 2'-Desoxyribonukleosid-5'-Triphosphat-Substrat (Ki = 0,18 μM). . Dehydroaltenusin hÄlt den Krebszellzyklus in der S-Phase an und lÖst Apoptose aus. Dehydroaltenusin besitzt in vivo eine Anti-Tumor-AktivitÄt gegen humanen Adenokarzinom-Tumor.
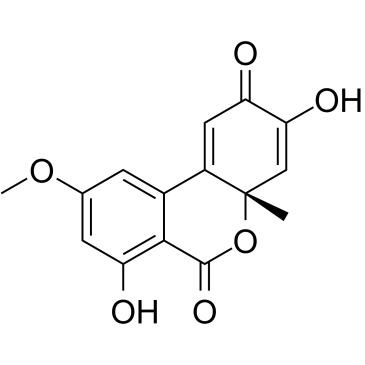
-
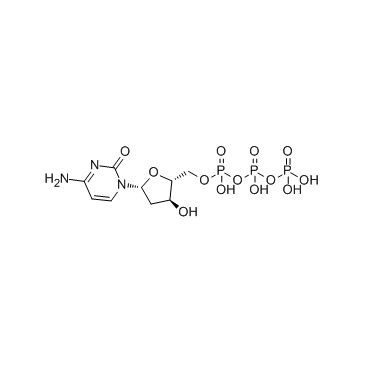
GC34123
Deoxycytidine triphosphate (dCTP)
Desoxycytidintriphosphat (dCTP) (dCTP) ist ein Nukleosidtriphosphat, das fÜr die DNA-Synthese verwendet werden kann.
-
GC12634
Deoxyguanosine 5-triphosphate
dGTP
Desoxyguanosintriphosphat (dGTP) Trinatriumsalz ist ein NukleotidvorlÄufer in Zellen fÜr die DNA-Synthese.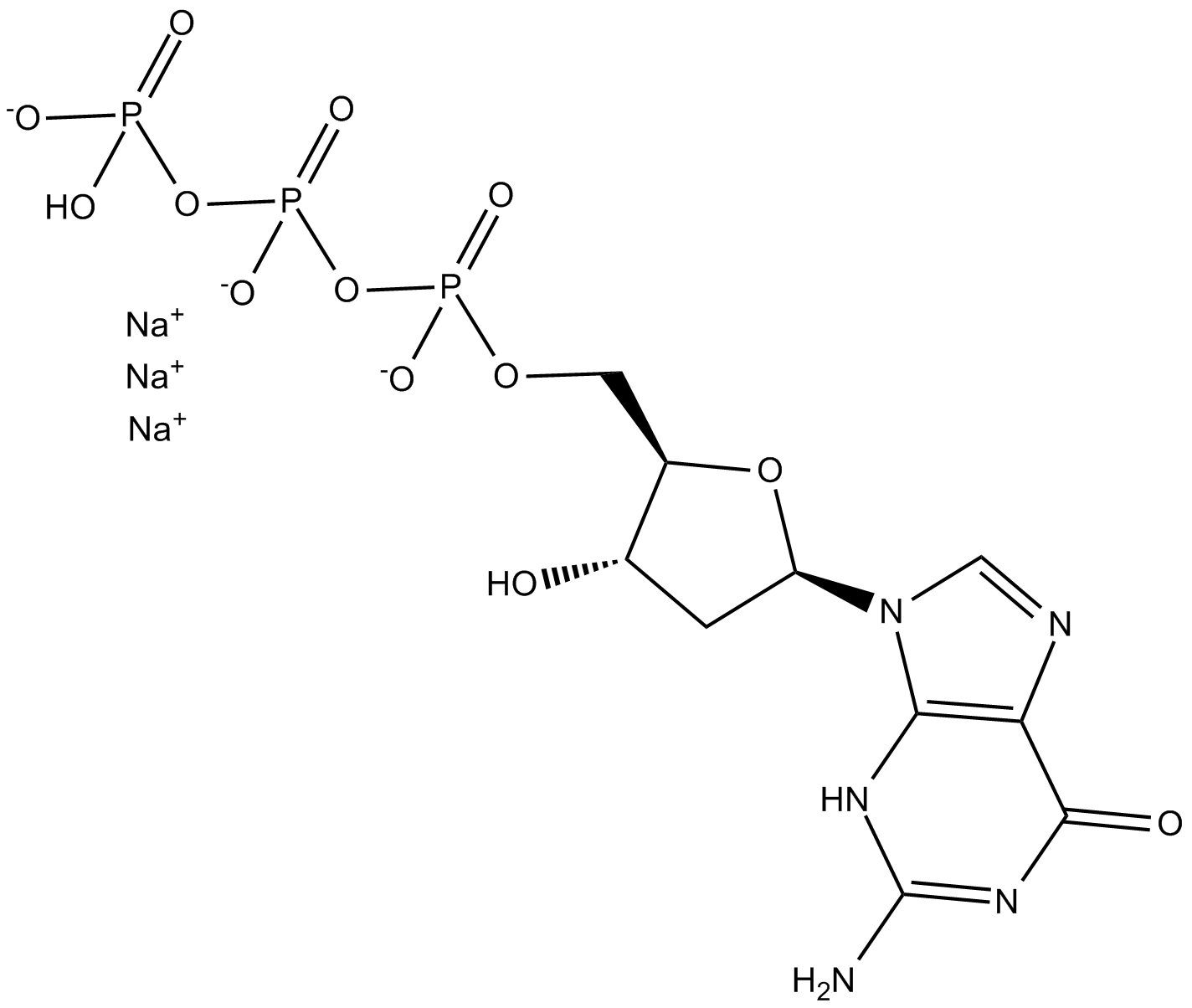
-
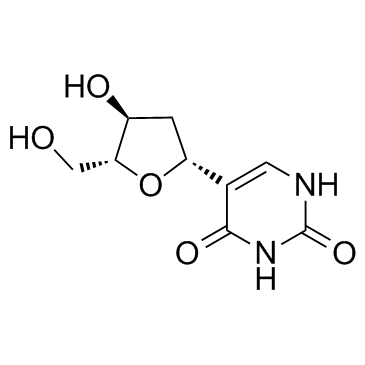
GC33494
Deoxypseudouridine
Desoxypseudouridin ist ein Nukleosid-Analogon.
-
GC68964
Deoxythymidine-5'-triphosphate sodium hydrate
dTTP sodium hydrate
Deoxythymidin-5'-triphosphat (dTTP) Natriumhydrat ist eine von vier Nukleosidtriphosphaten, die zur Synthese von DNA verwendet werden können.

-
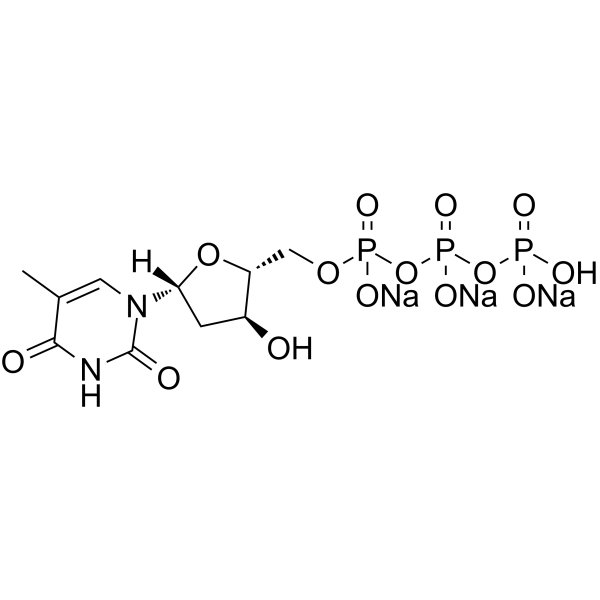
GC62925
Deoxythymidine-5’-triphosphate trisodium salt
Desoxythymidin-5'-Triphosphat (dTTP) Trinatrium ist eines der vier Nukleosidtriphosphate, die bei der Synthese von DNA verwendet werden.
-
GC49225
Deoxyuridine 5′-monophosphate (sodium salt)
dUMP
Desoxyuridin 5′-Monophosphat (Natriumsalz) wird durch das Bisubstrat-Enzym Thymidylat-Synthase (TS) reduktiv zu dTMP (2'-Desoxythymidin 5'-Monophosphat) methyliert.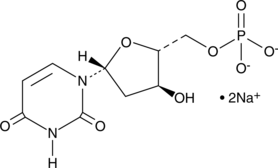
-
GC38482
Desmethylglycitein
6-hydroxy Daidzein, 6,7,4’-THIF
Desmethylglycitein (4',6,7-Trihydroxyisoflavone), ein Metabolit von Daidzein, der aus Glycine max gewonnen wird, mit antioxidativer und Anti-Krebs-AktivitÄt. Desmethylglycitein bindet in vivo direkt an CDK1 und CDK2, was zur UnterdrÜckung der CDK1- und CDK2-AktivitÄt fÜhrt. Desmethylglycitein ist ein direkter Inhibitor der Proteinkinase C (PKC)α gegen die durch solares UV (sUV) induzierte Matrix-Matrix-Metalloproteinase 1 (MMP1). Desmethylglycitein bindet an PI3K in ATP-kompetitiver Weise im Zytosol, wo es die AktivitÄt von PI3K und nachgeschalteten Signalkaskaden hemmt, was zur UnterdrÜckung der Adipogenese in 3T3-L1-PrÄadipozyten fÜhrt.
-
GC11619
Dexrazoxane HCl (ICRF-187, ADR-529)
Topoisomerase II inhibitor,intracellular ion chelator,cardioprotective agent
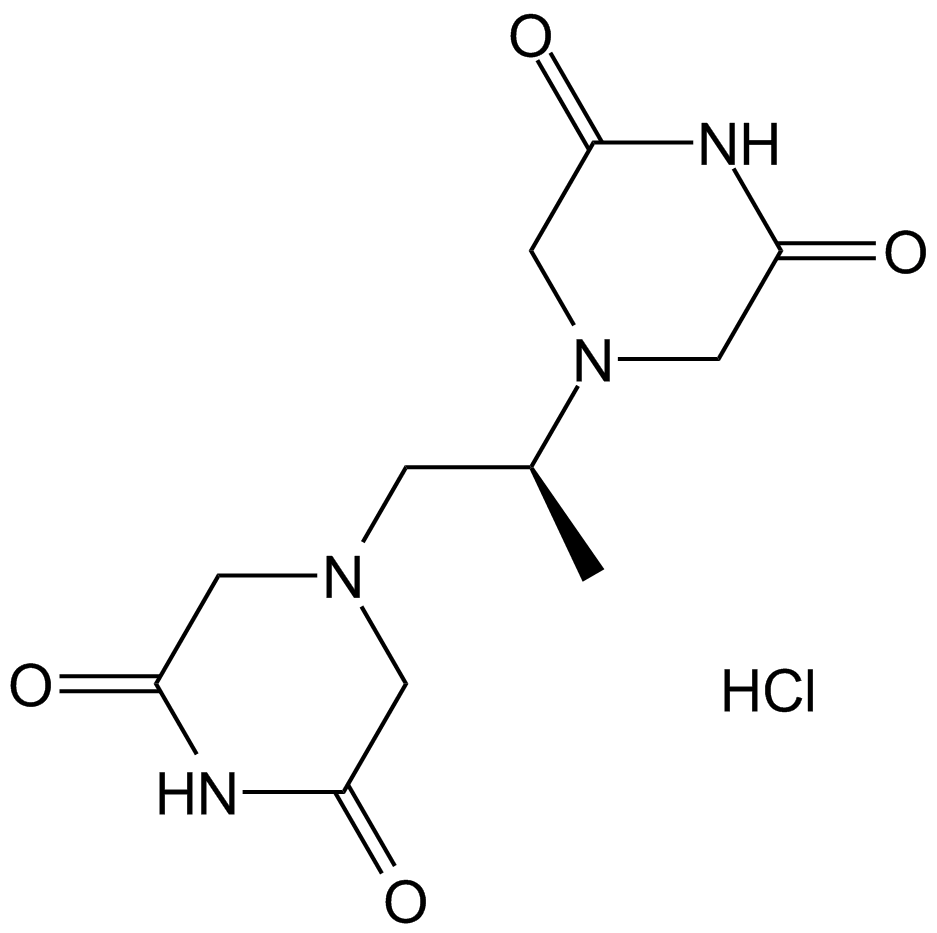
-
GC65617
DHFR-IN-3
DHFR-IN-3 ist ein Dihydrofolatreduktase (DHFR)-Inhibitor mit den IC50-Werten von 19 μM und 12 μM in Rattenleber bzw. P. carinii DHFR.

-
GC70726
DHODH-IN-1
DHODH-IN-1 (Verbindung 18d) ist ein potenter Dihydroorotatdehydrogenase (DHODH)-Inhibitor mit einem IC50 von 25 nM.

-
GC60763
DHODH-IN-11
Dihydroorotate Dehydrogenase Inhibitor 11
DHODH-IN-11 (Verbindung 14b) ist ein Leflunomid-Derivat und ein schwacher Dihydroorotat-Dehydrogenase (DHODH)-Inhibitor mit einem pKa-Wert von 5,03.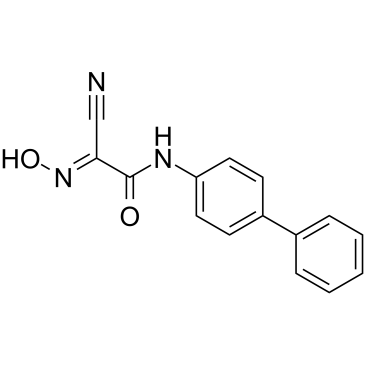
-
GC70741
DHODH-IN-12
DHODH-IN-12 (Verbindung 12b) ist ein Leflunomid-Derivat und ein schwacher Dihydroorotat-Dehydrogenase (DHODH)-Hemmer mit einem pKa von 5,07.

-
GC39234
DHODH-IN-2
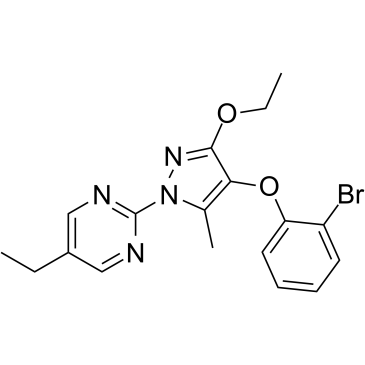
-
GC39374
DHODH-IN-5
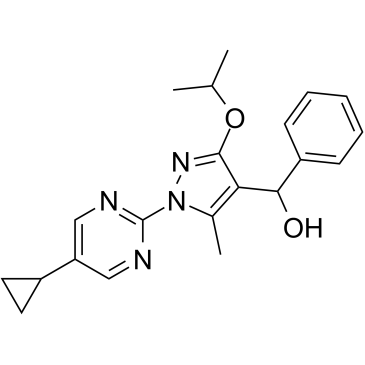
-
GC70740
DHODH-IN-9
DHODH-IN-9 (Verbindung 10k) ist ein azinhaltiges Analogon und ein humaner Dihydroorotat-Dehydrogenase-Inhibitor.

-
GC32343
Diaveridine (EGIS-5645)
NSC 408735
Diaveridin (EGIS-5645) (EGIS-5645) ist ein Dihydrofolatreduktase (DHFR)-Inhibitor mit einem Ki von 11,5 nM fÜr die Wildtyp-DHFR und auch ein antibakterielles Mittel.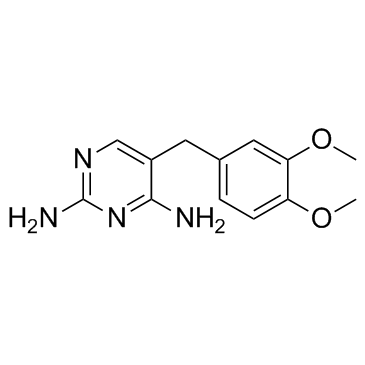
-
GC49022
Didanosine-d2
ddl-d2, 2′,3′-Dideoxyinosine-d2
An internal standard for the quantification of didanosine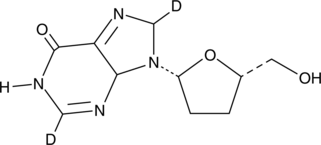
-
GC39808
Didesmethylrocaglamide
Didesmethylrocaglamid, ein Derivat von Rocaglamid, ist ein potenter Hemmer des eukaryotischen Initiationsfaktors 4A (eIF4A). Didesmethylrocaglamid hat eine starke wachstumshemmende AktivitÄt mit einem IC50 von 5 nM. Didesmethylrocaglamid unterdrÜckt mehrere wachstumsfÖrdernde Signalwege und induziert Apoptose in Tumorzellen. Antitumor-AktivitÄt.