Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
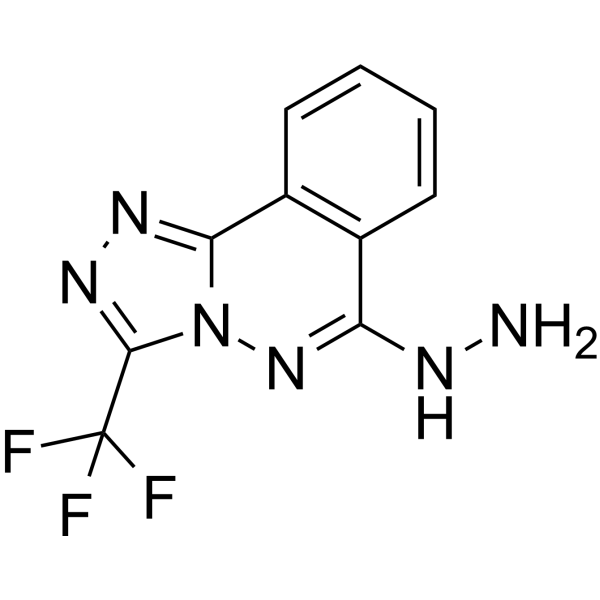
GC12458
N-Oxalylglycine
cell permeable inhibitor of α-ketoglutarate-dependent enzymes, including JMJD2A, JMJD2C, and JMJD2E.
-
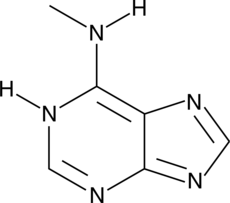
GC40496
N6-Methyladenine
N6-Methyladenine is a modified purine that is commonly found in genomes of prokaryotes, protists, and plants.
-
GC17676
Nafamostat
Inhibiteur de la sérine protéase à large spectre, inhibiteur de la kallikréine.
-
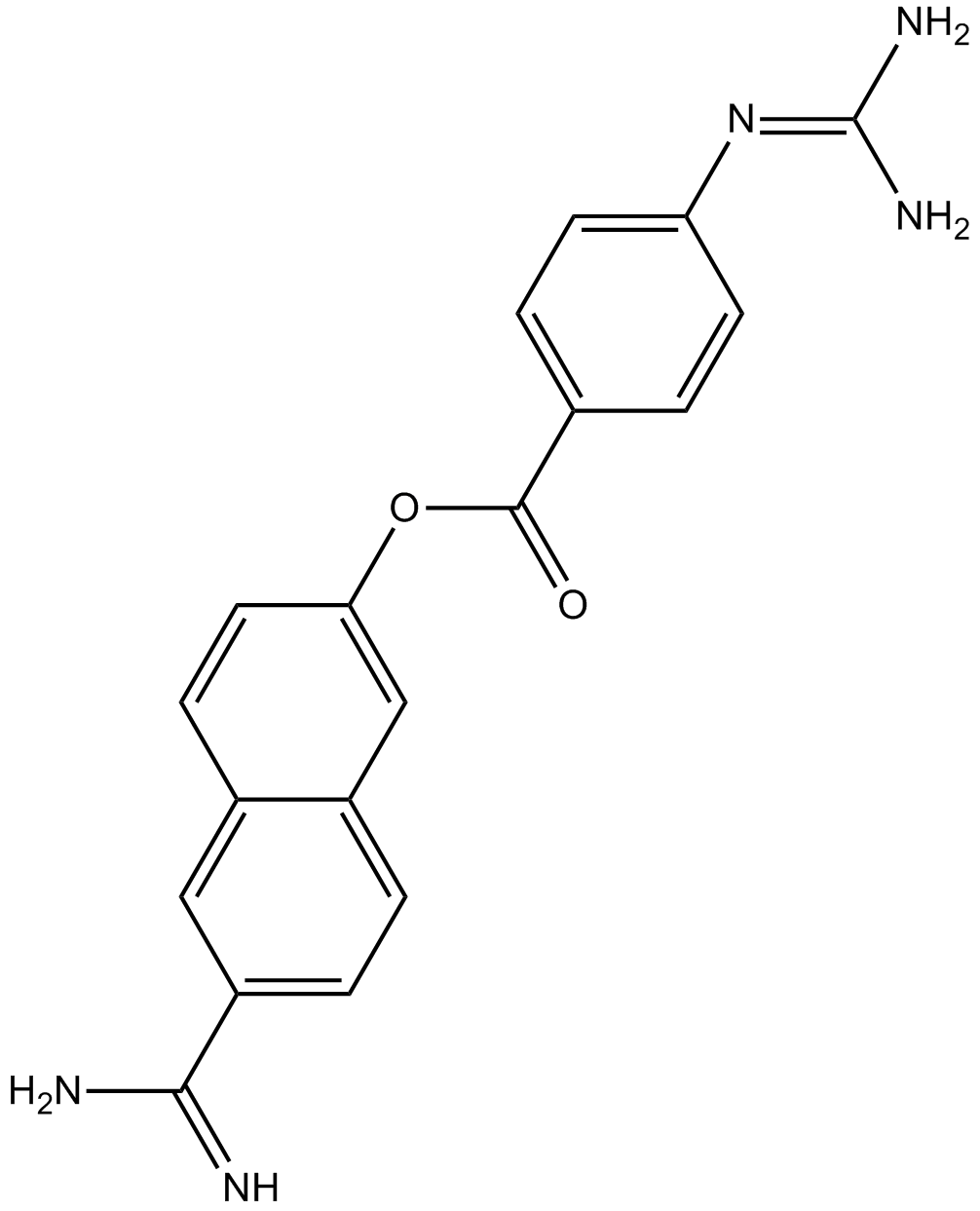
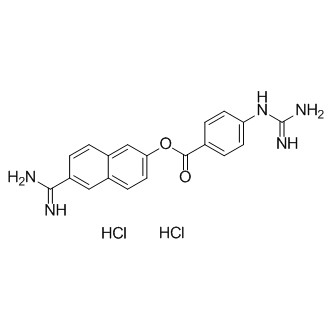
GC16290
Nafamostat hydrochloride
Le chlorhydrate de nafamostat, un inhibiteur synthétique de la sérine protéase, est un anticoagulant.
-
GC36690
Nampt-IN-3
Nampt-IN-3 (composé 35) inhibe simultanément la nicotinamide phosphoribosyltransférase (NAMPT) et l'HDAC avec des CI50 de 31 nM et 55 nM, respectivement. Nampt-IN-3 induit efficacement l'apoptose cellulaire et l'autophagie et conduit finalement À la mort cellulaire.
-
GC14562
Nanaomycin A
A bacterial metabolite
-
GC10243
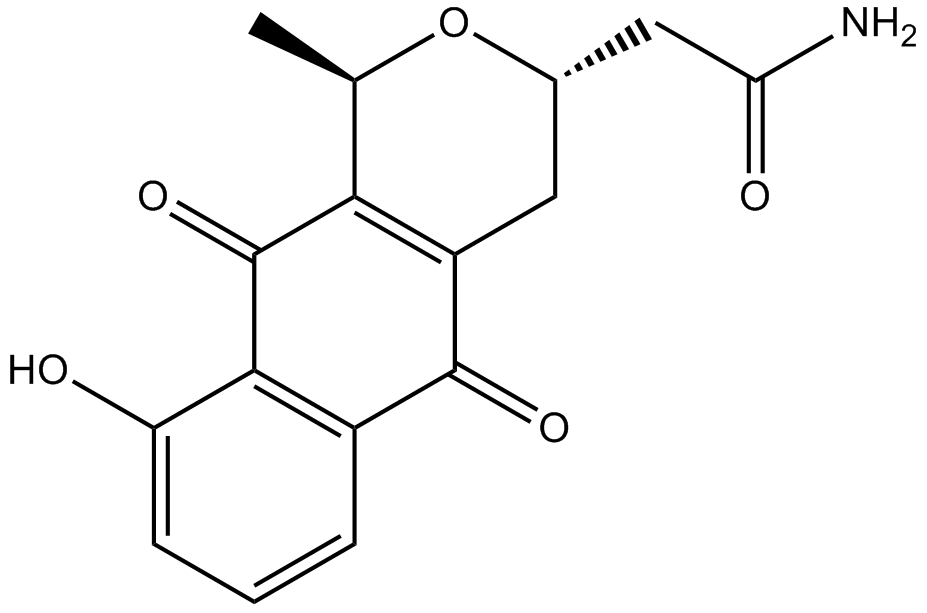
Nanaomycin C
Amide of nanaomycin A
-
GC36691
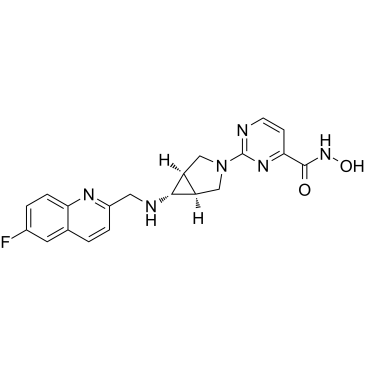
Nanatinostat
Le nanatinostat (CHR-3996) est un puissant inhibiteur de l'histone désacétylase (HDAC) sélectif de classe I et actif par voie orale avec une IC50 de 8 nM.
-
GC61841
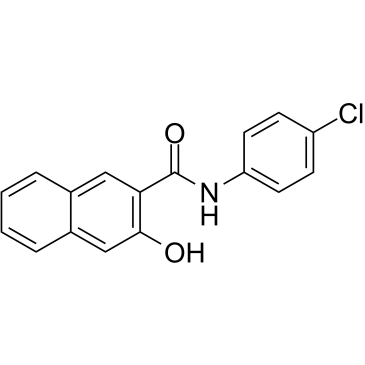
Naphthol AS-E
Le naphtol AS-E est un inhibiteur puissant et perméable aux cellules de l'interaction KIX-KID. Le naphtol AS-E se lie directement au domaine KIX de CBP (Kd : 8,6 μM), bloque l'interaction entre le domaine KIX et le domaine KID de CREB avec une IC50 de 2,26 μM. Le naphtol AS-E peut être utilisé pour la recherche sur le cancer.
-
GC65138
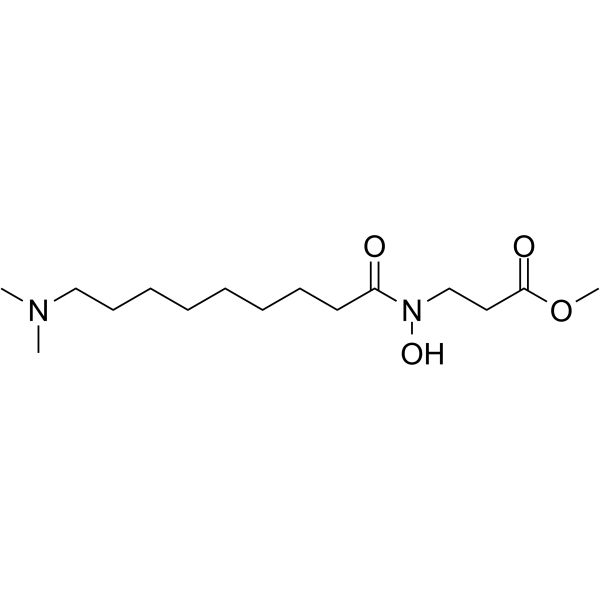
NCDM-32B
NCDM-32B est un inhibiteur puissant et sélectif de KDM4 qui altère la viabilité et transforme les phénotypes du cancer du sein.
-
GC19410
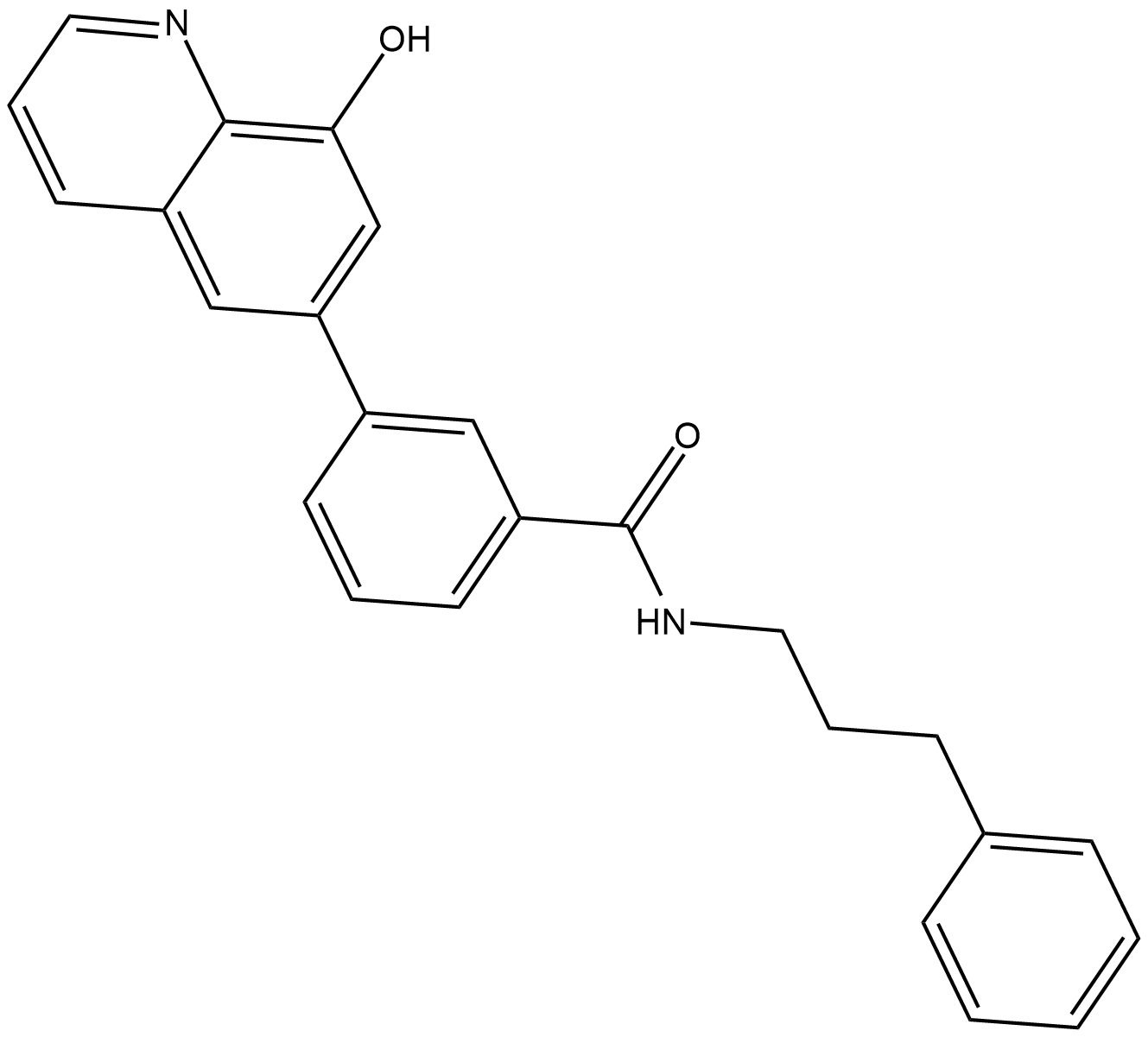
NCGC00244536
NCGC00244536 est un puissant inhibiteur de KDM4B avec une IC50 de 10 nM.
-
GC32896
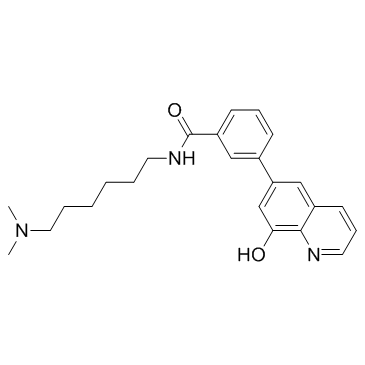
NCGC00247743
NCGC00247743 est un inhibiteur de l'histone lysine déméthylase KDM4.
-
GC15536
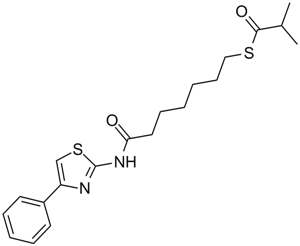
NCH 51
An HDAC inhibitor
-
GC66329
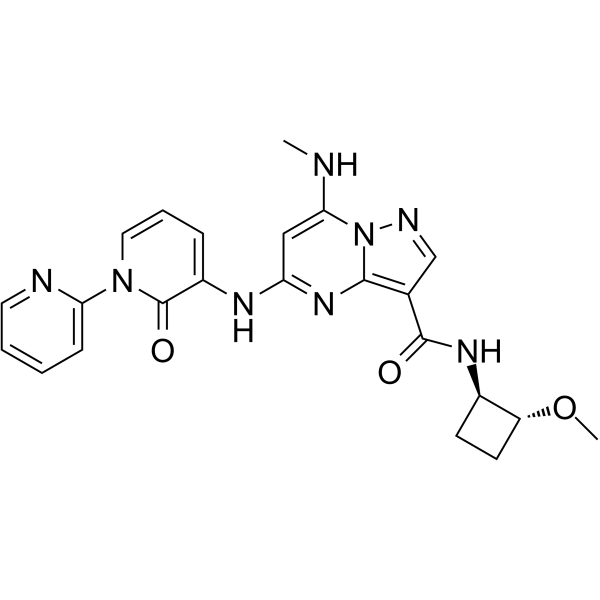
NDI-034858
NDI-034858 est un inhibiteur de TYK2, cible le domaine TYK2 JH2 avec une constante de liaison Kd <200 pM.
-
GC65202
Nesuparib
Le nésuparib est un puissant inhibiteur de la PARP.
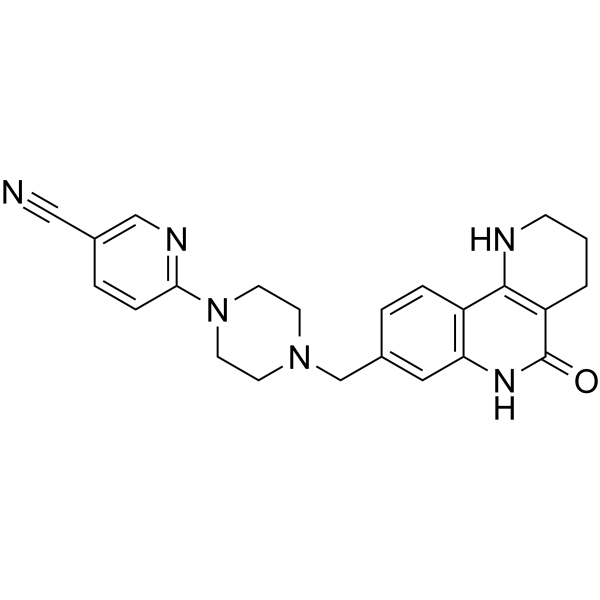
-
GC13764
Nexturastat A
Le Nexturastat A est un puissant inhibiteur de HDAC6 avec une IC50 de 5 nM. Nexturastat A inhibe également HDAC10 et la protéine contenant le domaine métallo-β-lactamase2 (MBLAC2).
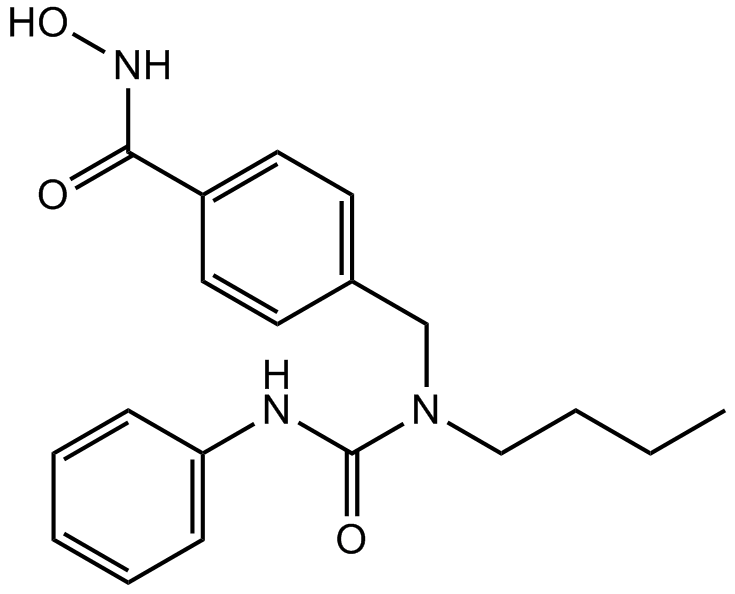
-
GC62620
NHWD-870
NHWD-870 est un inhibiteur de bromodomaine puissant, actif par voie orale et sélectif de la famille BET et ne lie que les bromodomaines de BRD2, BRD3, BRD4 (IC50 = 2,7 nM) et BRDT. Le NHWD-870 a une puissante efficacité de suppression des tumeurs et supprime l'interaction cellules cancéreuses-macrophages. Le NHWD-870 augmente l'apoptose tumorale et inhibe la prolifération tumorale.
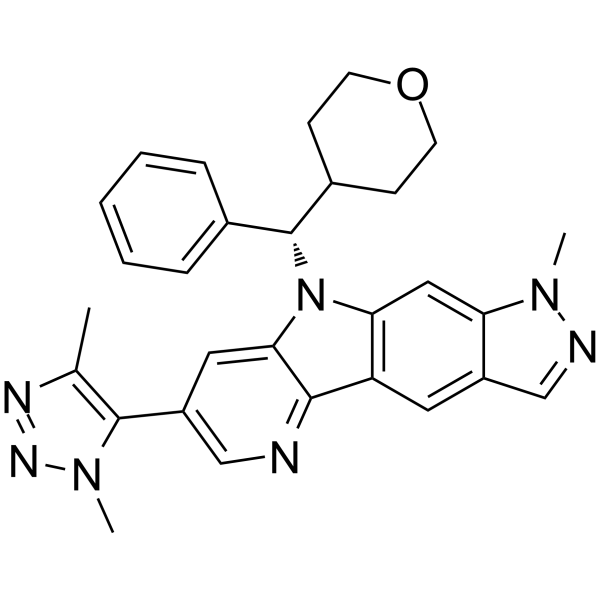
-
GC36734
NI-42
NI-42 (composé 13-d), une sonde chimique structurellement orthogonale pour les BRPF, est un puissant inhibiteur biaisé de la BRD des BRPF (IC50 de BRPF1/2/3 = 7,9/48/260 nM; Kds de BRPF1 /2/3=40/210/940 nM) avec une excellente sélectivité sur les protéines BRD non classe IV.
-
GC16763
NI-57
NI-57 est un inhibiteur de la famille de protéines bromodomaine et homéodomaine végétal contenant des doigts (BRPF), avec des IC50 de 3,1, 46 et 140 nM pour BRPF1, BRPF2 (BRD1) et BRPF3, respectivement.
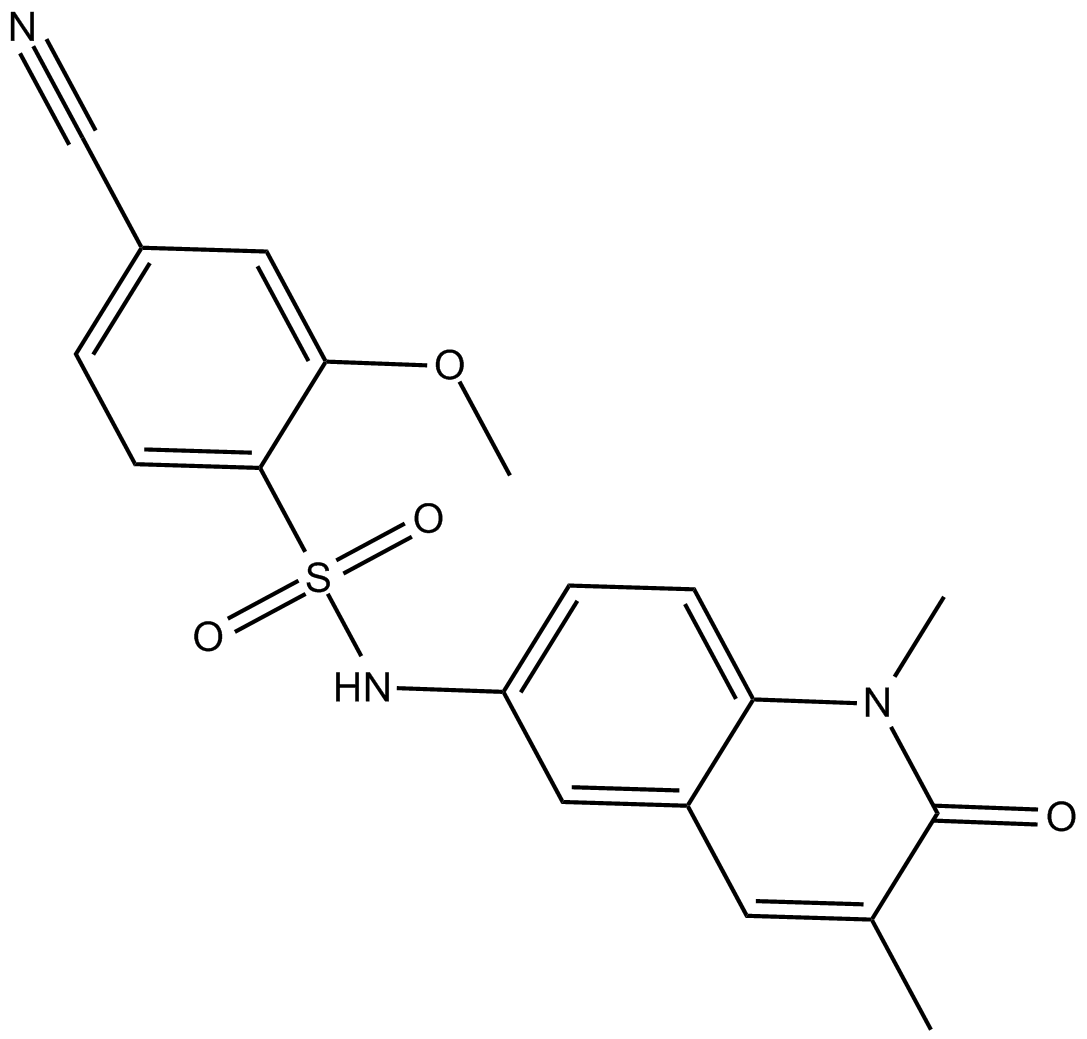
-
GN10347
Nicotinamide (Vitamin B3)
La nicotinamide est un inhibiteur des enzymes poly(ADP-ribose) polymérases (PARP-1).
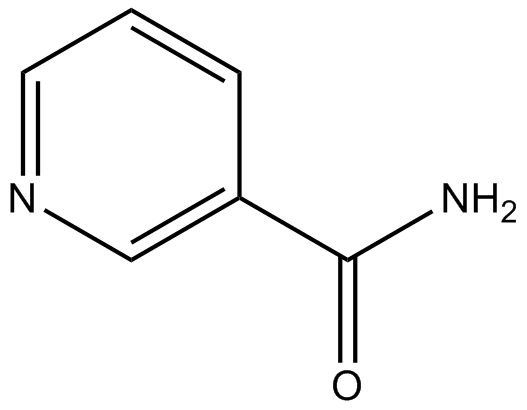
-
GC44401
Nicotinamide riboside
Le nicotinamide riboside, une forme de vitamine B3 et précurseur du NAD+, est converti en NAD+ bio-disponible, via la kinase du nicotinamide riboside (NRK) et NMNAT, ou par l'action de la nucléoside phosphorylase et du recyclage du NAM.

-
GC36738
Nicotinamide riboside chloride
Le chlorure de nicotinamide riboside, un précurseur de NAD+ actif par voie orale, augmente les niveaux de NAD+ et active SIRT1 et SIRT3.
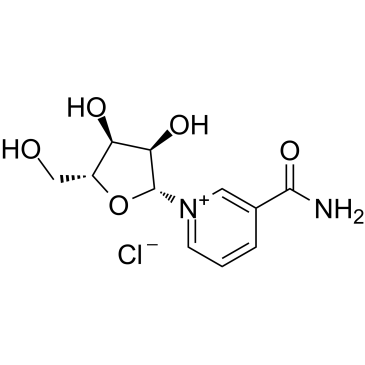
-
GC49270
Nicotinamide-d4
Le nicotinamide-d4 (niacinamide-d4) est le nicotinamide marqué au deutérium.
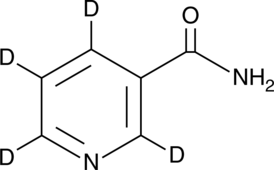
-
GC62248
NiCur
NiCur est un inhibiteur puissant et sélectif de l'histone acétyltransférase (HAT) de la CBP avec une valeur IC50 de 0,35 μμ. NiCur, qui bloque l'activité CBP HAT et régule À la baisse l'activation de p53 lors d'un stress génotoxique. NiCur peut être utilisé pour effectuer des études mécanistes sans affecter l'expression des protéines cibles.
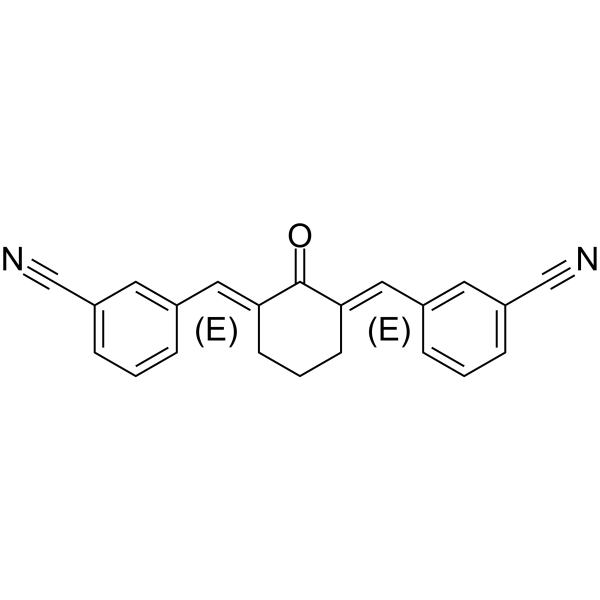
-
GC34120
Niraparib R-enantiomer (MK 4827 (R-enantiomer))
L'énantiomère R du niraparib (énantiomère MK-4827 R) est un excellent inhibiteur de PARP1 avec une IC50 de 2,4 nM.
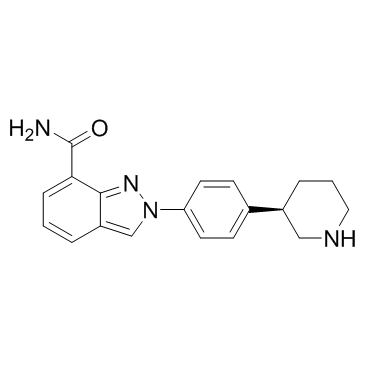
-
GC32964
NKL 22
NKL 22 (composé 4b) est un inhibiteur puissant et sélectif des histone désacétylases (HDAC), avec une IC50 de 199 et 69 nM pour HDAC1 et HDAC3, respectivement.
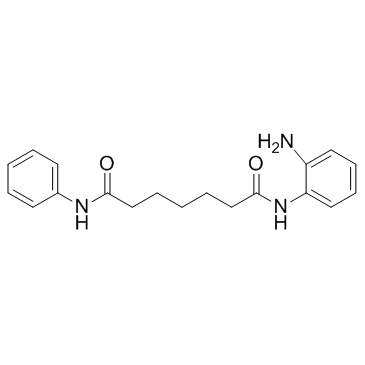
-
GC19264
NMS-P118
NMS-P118 est un puissant inhibiteur de PARP-1 disponible par voie orale et hautement sélectif pour le traitement du cancer.
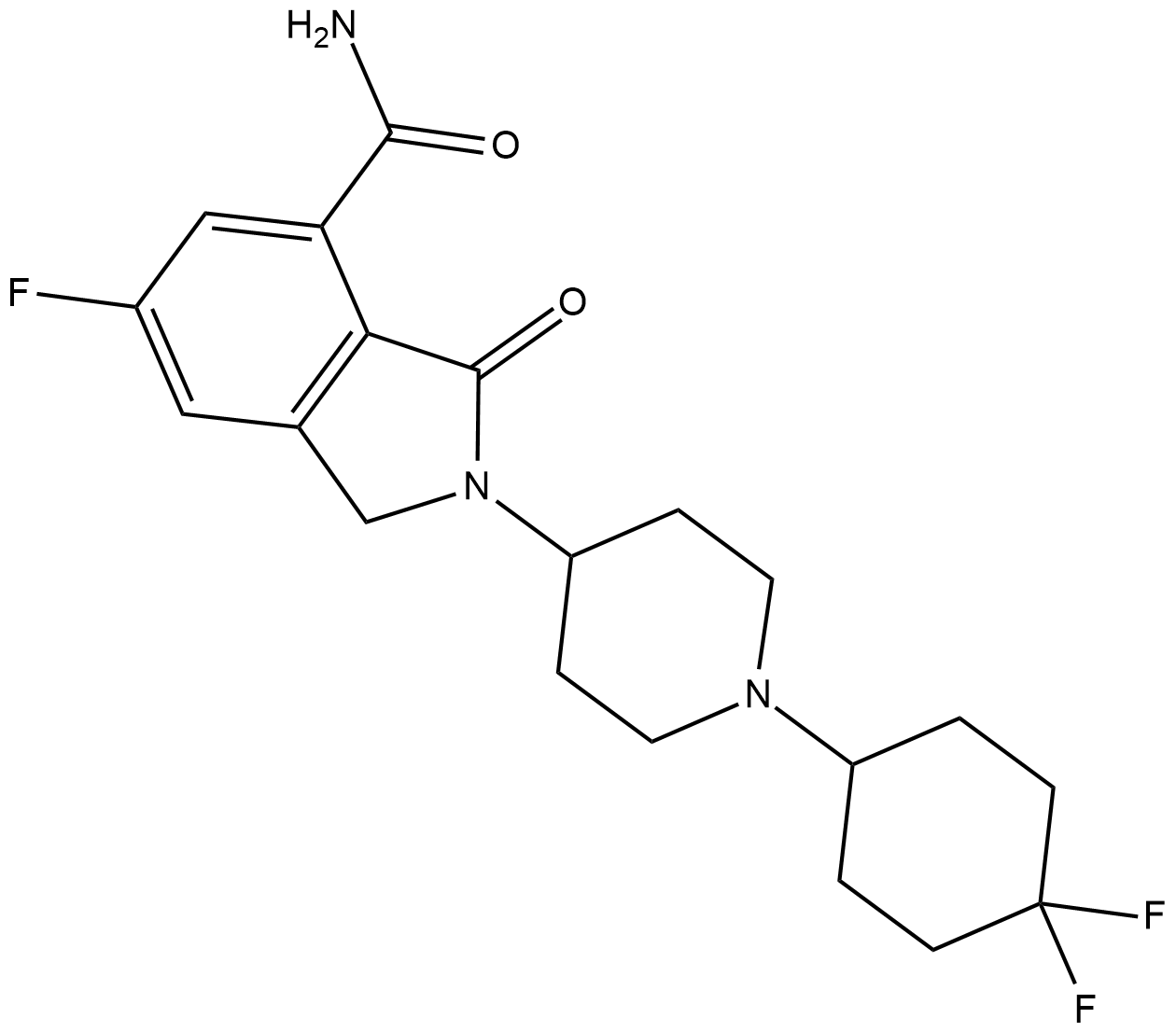
-
GC36751
NMS-P515
NMS-P515 est un inhibiteur de PARP-1 puissant, actif par voie orale et stéréospécifique, avec un Kd de 16 nM et une IC50 de 27 nM (dans les cellules Hela). Activité anti-tumorale.
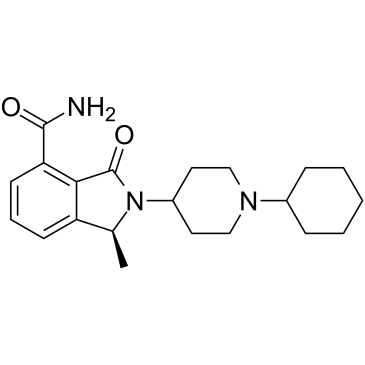
-
GC52166
NN-390
NN-390 est un inhibiteur HDAC6 puissant et sélectif, avec une IC50 de 9,8 nM. Le NN-390 traverse la barrière hémato-encéphalique (BBB). NN-390 montre un potentiel d'étude dans le groupe métastatique 3 MB (médulloblastome).

-
GC48460
NR-160
An inhibitor of HDAC6
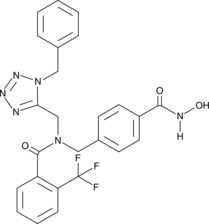
-
GC17762
NSC 33994
Selective inhibitor of JAK2 (IC50 = 60 nM). Displays no effect on Src and TYK2 tyrosine kinase activity at a concentration of 25 μM.
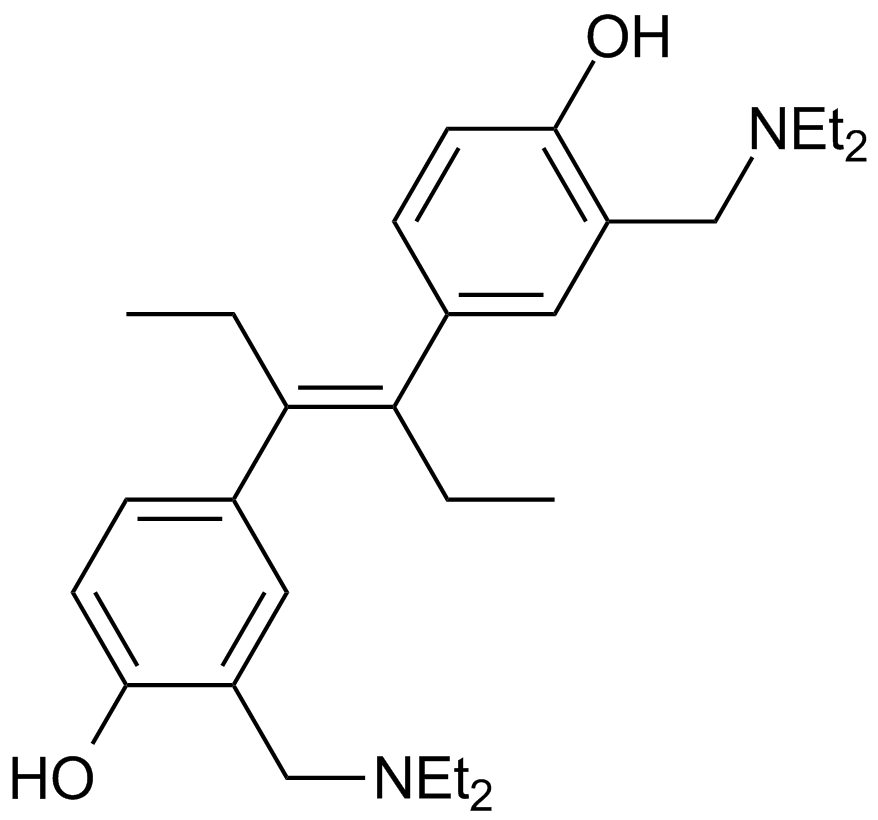
-
GC15040
NSC 3852
A tumor cell differentiating agent
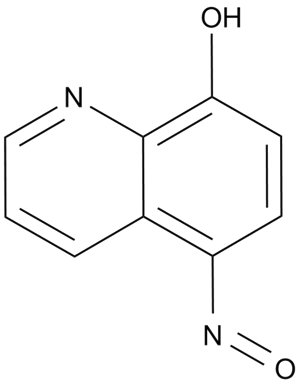
-
GC50136
NSC 636819
NSC 636819 est un inhibiteur compétitif et sélectif de KDM4A/KDM4B. KDM4A/KDM4B sont des facteurs potentiels de progression du cancer de la prostate. NSC 636819 a le potentiel pour la recherche sur les maladies cancéreuses, en particulier le cancer de la prostate.
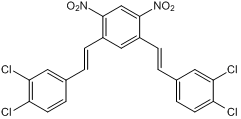
-
GC14875
NSC 663284
NSC 663284 (DA-3003-1) est un inhibiteur de la phosphatase À double spécificité Cdc25 puissant, perméable aux cellules et irréversible, a une IC50 pour Cdc25B2 de 0,21 μM. NSC 663284 présente une cinétique compétitive mixte contre Cdc25A, Cdc25B(2) et Cdc25C avec des valeurs Ki de 29, 95 et 89 nM, respectivement. NSC 663284 inhibe NSD2 (IC50 de 170 nM) par une interaction directe avec le domaine catalytique SET (Kd de 370 nM).
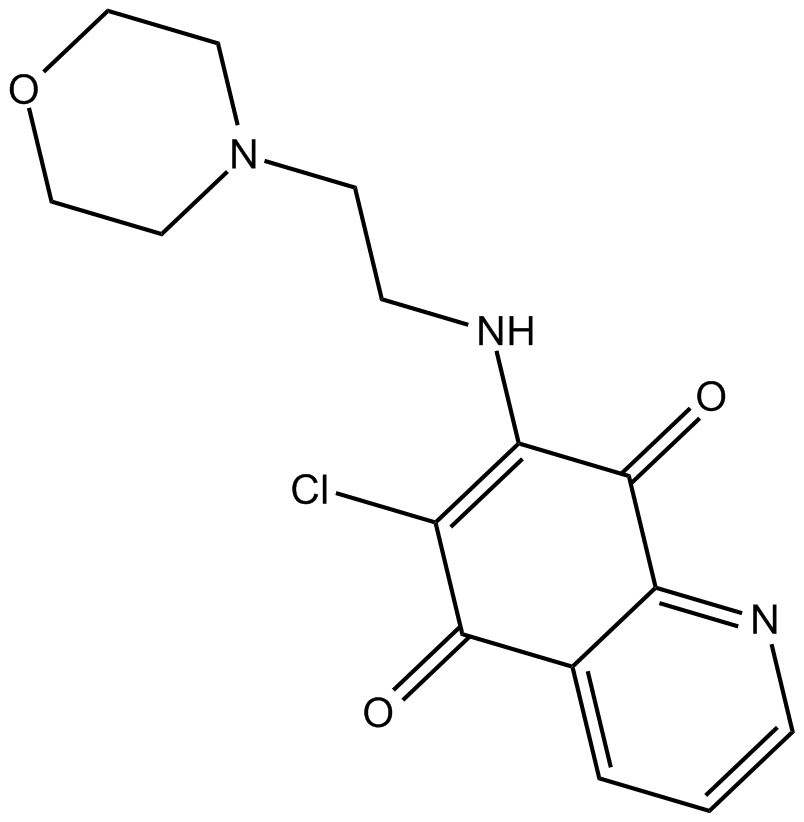
-
GC66049
NSC 694621
NSC 694621 est un puissant inhibiteur du PCAF, avec une IC50 de 5,71 μM (PCAF/H31-21). NSC 694621 présente une bonne activité d'inhibition de la prolifération des cellules cancéreuses.
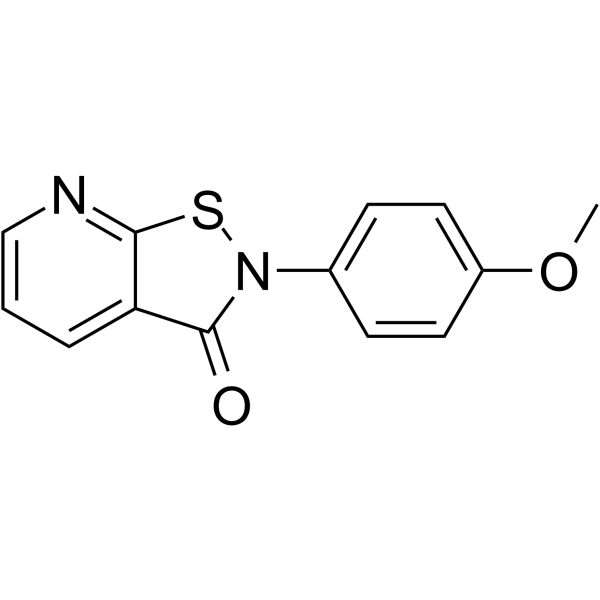
-
GC69597
NSC 694623
NSC 694623 est un inhibiteur efficace de l'histone acétyltransférase (HAT), avec une IC50 de 15,9 μM pour le facteur associé à la HAT recombinante p300/CBP (PCAF). NSC 694623 a une activité anti-proliférative sur certaines cellules cancéreuses. NSC 694623 peut être utilisé dans la recherche contre le cancer.
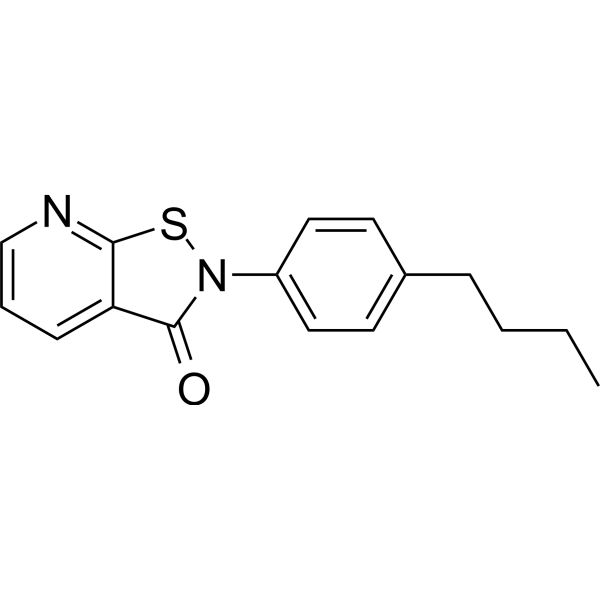
-
GC49412
NSC 756093
NSC 756093 est un puissant inhibiteur de l'interaction GBP1:PIM1. NSC 756093 peut potentiellement inverser la résistance au paclitaxel. NSC 756093 peut être utilisé pour la recherche sur le cancer de l'ovaire.
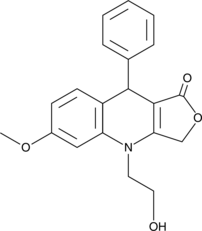
-
GC11561
NSC 95397
NSC 95397 est un puissant inhibiteur sélectif de la phosphatase À double spécificité Cdc25 (Ki = 32 nM (Cdc25A), 96 nM (Cdc25B), 40 nM (Cdc25C); IC50 = 22,3 nM (Cdc25A humain), 56,9 nM (Cdc25C humain), 125 nM (Cdc25B)). NSC 95397 inhibe la protéine kinase phosphatase-1 activée par les mitogènes (MKP-1), supprime la prolifération et induit l'apoptose dans les cellules cancéreuses du cÔlon par les voies MKP-1 et ERK1/2.
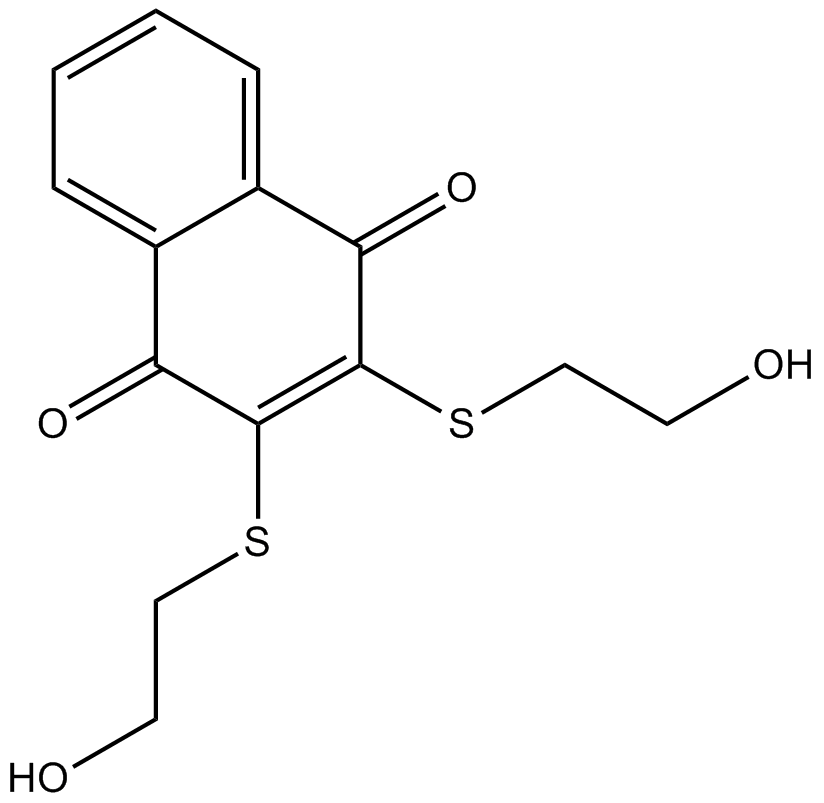
-
GC14103
NSC228155
NSC228155 est un activateur de l'EGFR, se lie à la région extracellulaire de l'EGFR et améliore la phosphorylation de la tyrosine de l'EGFR. NSC228155 est également un puissant inhibiteur de l'interaction KIX-KID, inhibe le domaine inductible par la kinase (KID) de CREB et le domaine interagissant avec le KID (KIX) de CBP, avec une IC50 de 0,36 μM.
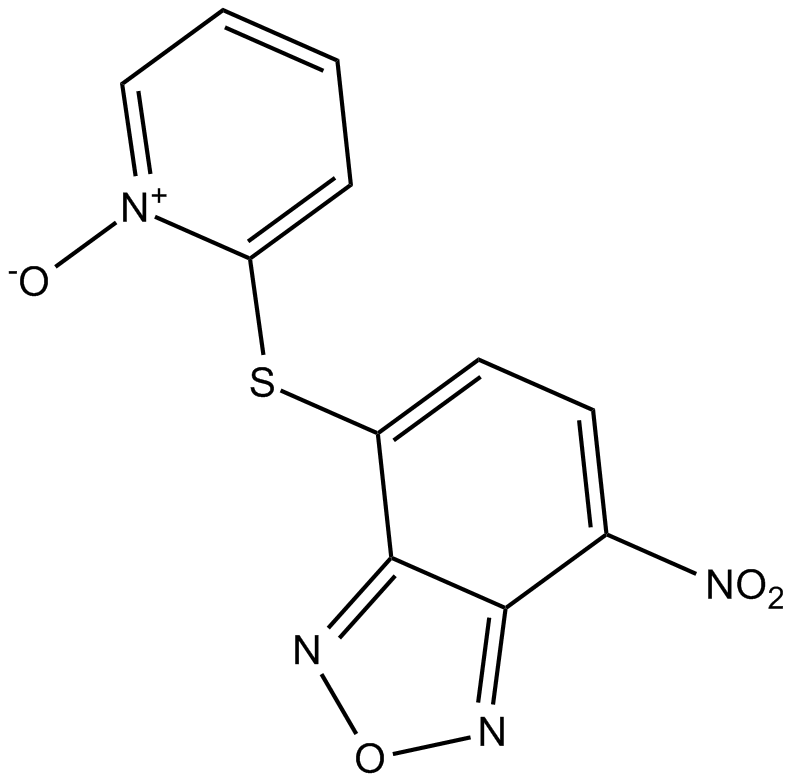
-
GC61142
NSC745885
NSC745885, un agent antitumoral efficace, présente une toxicité sélective contre plusieurs lignées de cellules cancéreuses, mais pas contre les cellules normales. NSC745885 est un régulateur À la baisse efficace d'EZH2 via la dégradation médiée par le protéasome. NSC745885 offre des possibilités pour l'étude des cancers avancés de la vessie et du carcinome épidermoÏde de la bouche (OSCC).
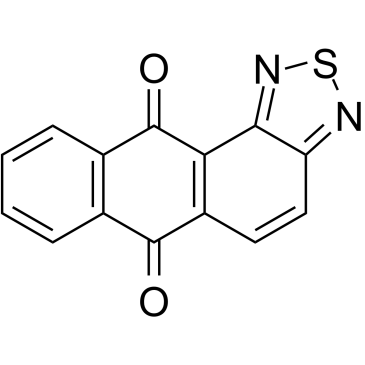
-
GC17775
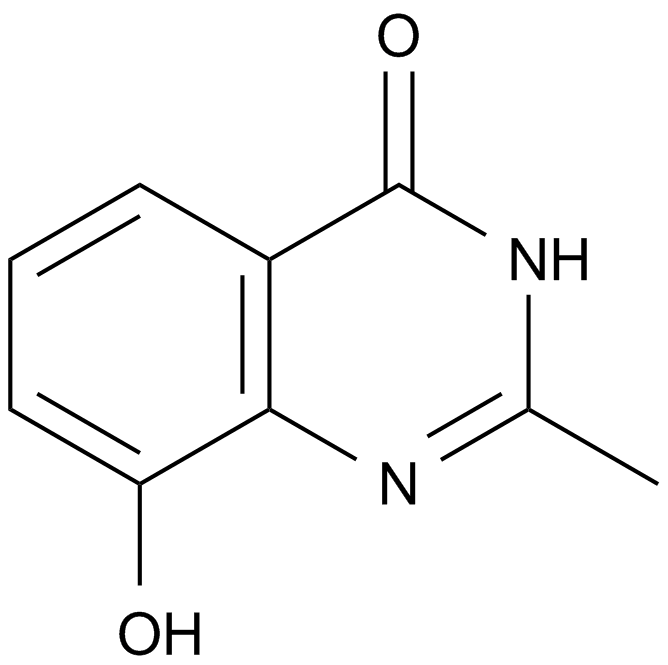
NU 1025
An inhibitor of PARP
-
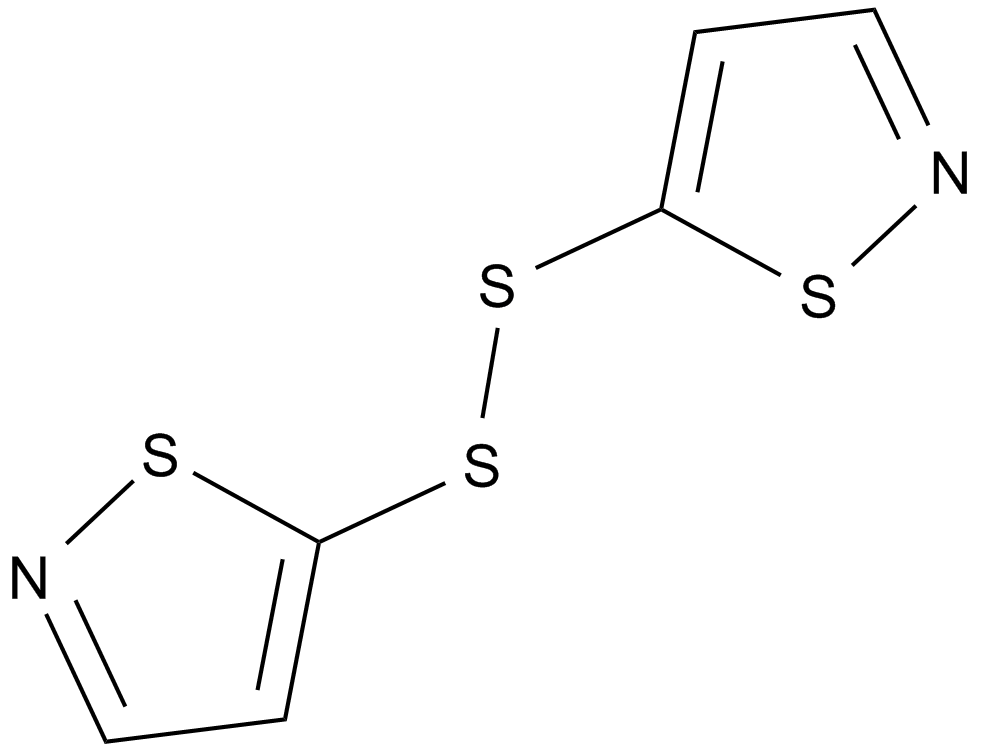
GC11972
NU 9056
Selective KAT5 (Tip60) histone acetyltransferase inhibitor (IC50 values are < 2, 60, 36, and >100 μM for KAT5, p300, pCAF and GCN5, respectively).
-
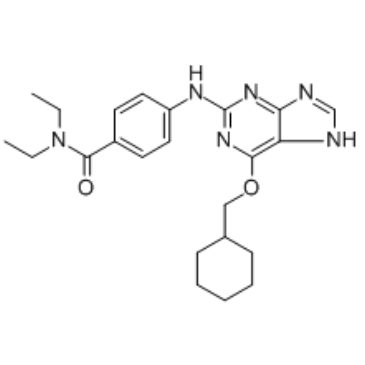
GC34692
NU6140
NU6140 est un inhibiteur sélectif de CDK2-cycline A (IC50, 0,41 μM), présente une sélectivité de 10 À 36 fois par rapport aux autres CDK. NU6140 inhibe également puissamment Aurora A et Aurora B, avec des IC50 de 67 et 35 nM, respectivement. Améliore l'effet apoptotique, avec une activité anticancéreuse.
-
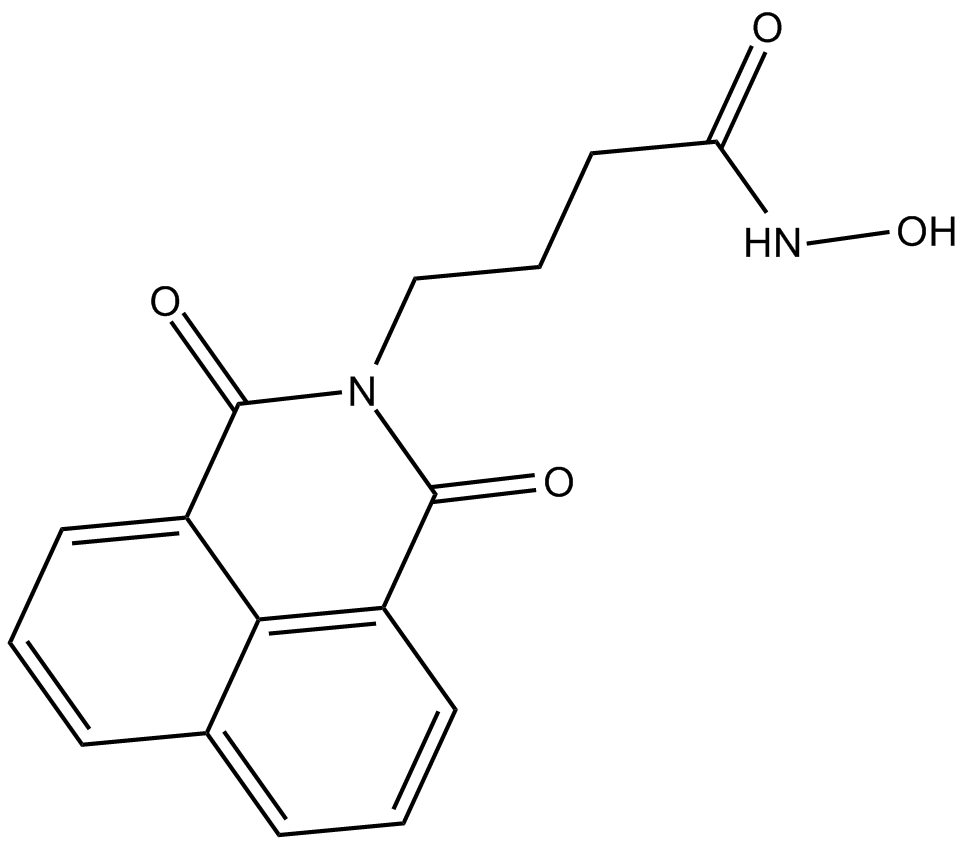
GC13925
Nullscript
negative control of scriptaid, HDAC inhibitor
-
GC64772
NV03
NV03 est un antagoniste puissant et sélectif de l'interaction UHRF1 (Ubiquitin-like with PHD and RING finger domains 1)-H3K9me3 en se liant au domaine tudor tandem UHRF1, avec un Kd de 2,4 μM. NV03 a une activité anticancéreuse.
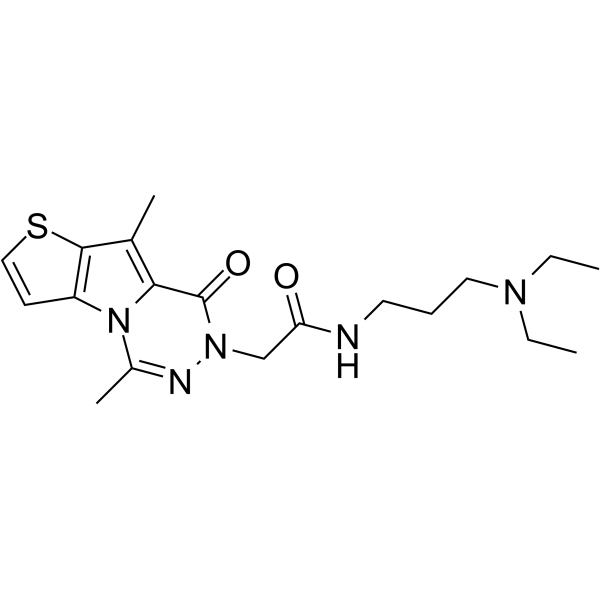
-
GC13229
NVP-BSK805
A potent, selective JAK2 inhibitor
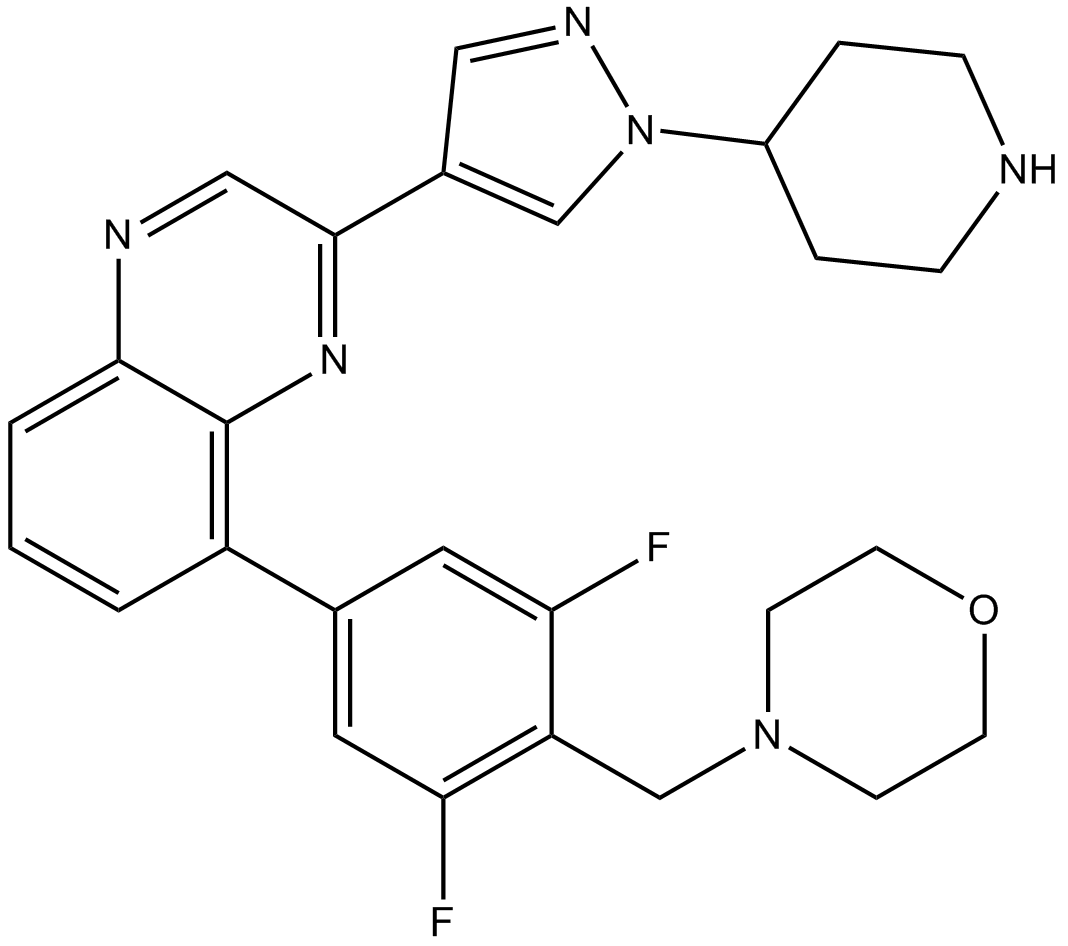
-
GC36783
NVP-BSK805 dihydrochloride
Le dichlorhydrate de NVP-BSK805 est un inhibiteur de JAK2 compétitif pour l'ATP, avec des IC50 de 0,48 nM, 31,63 nM, 18,68 nM et 10,76 nM pour JAK2 JH1 (homologie JAK 1), JAK1 JH1, JAK3 JH1 et TYK2 JH1, respectivement.
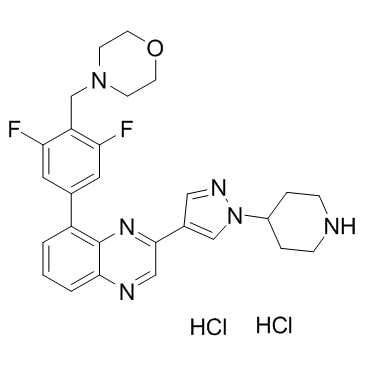
-
GC17555
NVP-TNKS656
NVP-TNKS656 est un inhibiteur de TNKS2 très puissant, sélectif et actif par voie orale avec une IC50 de 6 nM et une sélectivité > 300 fois contre PARP1 et PARP2.
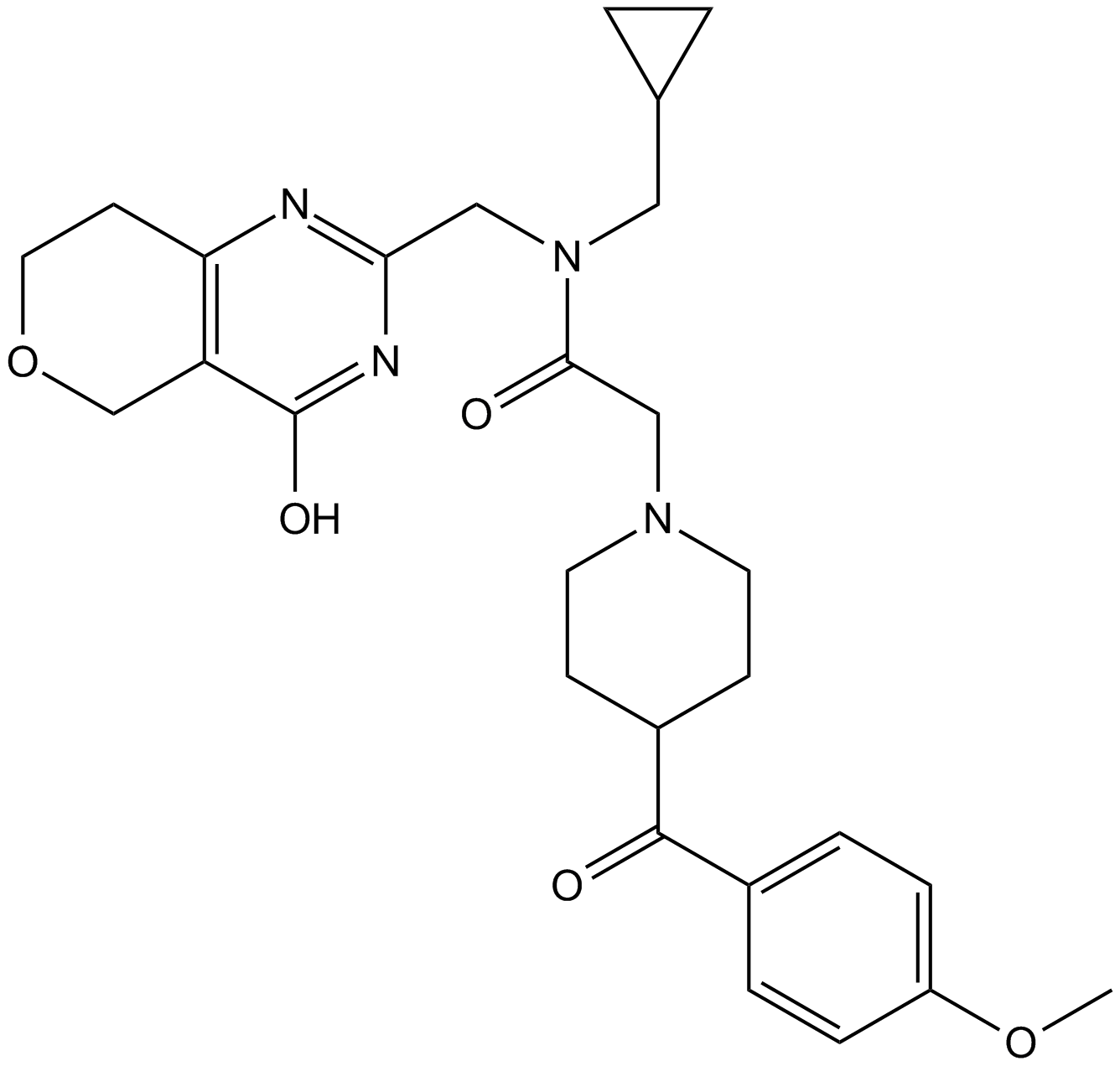
-
GC44477
NVS-CECR2-1
NVS-CECR2-1, un inhibiteur du bromodomaine (BRD) non de la famille BET, est un inhibiteur puissant et sélectif de la région chromosomique du syndrome de l'œil de chat, candidat 2 (CECR2). NVS-CECR2-1 se lie À CECR2 BRD avec une haute affinité (IC50 = 47 nM; KD = 80 nM). NVS-CECR2-1 présente une activité cytotoxique et induit l'apoptose contre diverses cellules cancéreuses en ciblant CECR2 ainsi que via un mécanisme indépendant de CECR2.
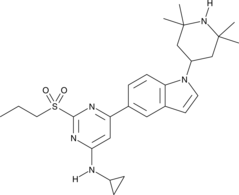
-
GC69601
OARV-771
OARV-771 est un dégradeur PROTAC basé sur VHL qui présente une meilleure perméabilité cellulaire. OARV-771 montre des DC50 de 6, 1 et 4 nM pour Brd4, Brd2 et Brd3 respectivement.
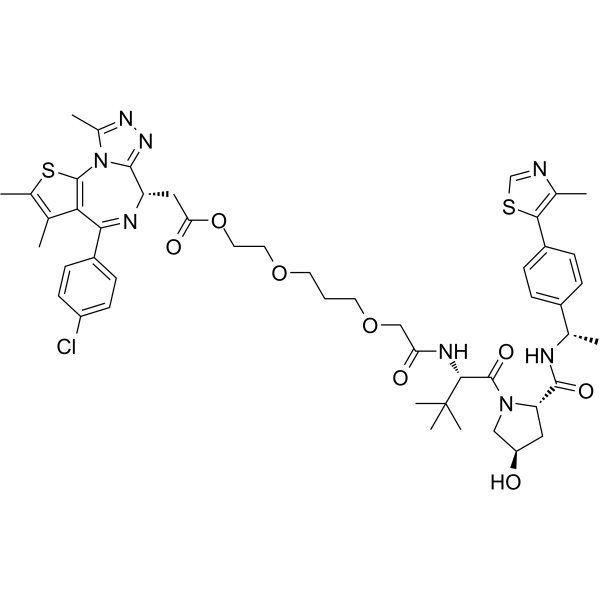
-
GC31677
Oclacitinib maleate (PF-03394197 maleate)
Le maléate d'oclacitinib (maléate PF-03394197) (maléate PF-03394197) est un nouvel inhibiteur de JAK.
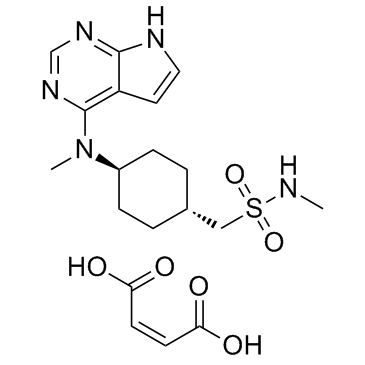
-
GC17358
Octyl-α-ketoglutarate
prolyl hydroxylases (PHD) activator
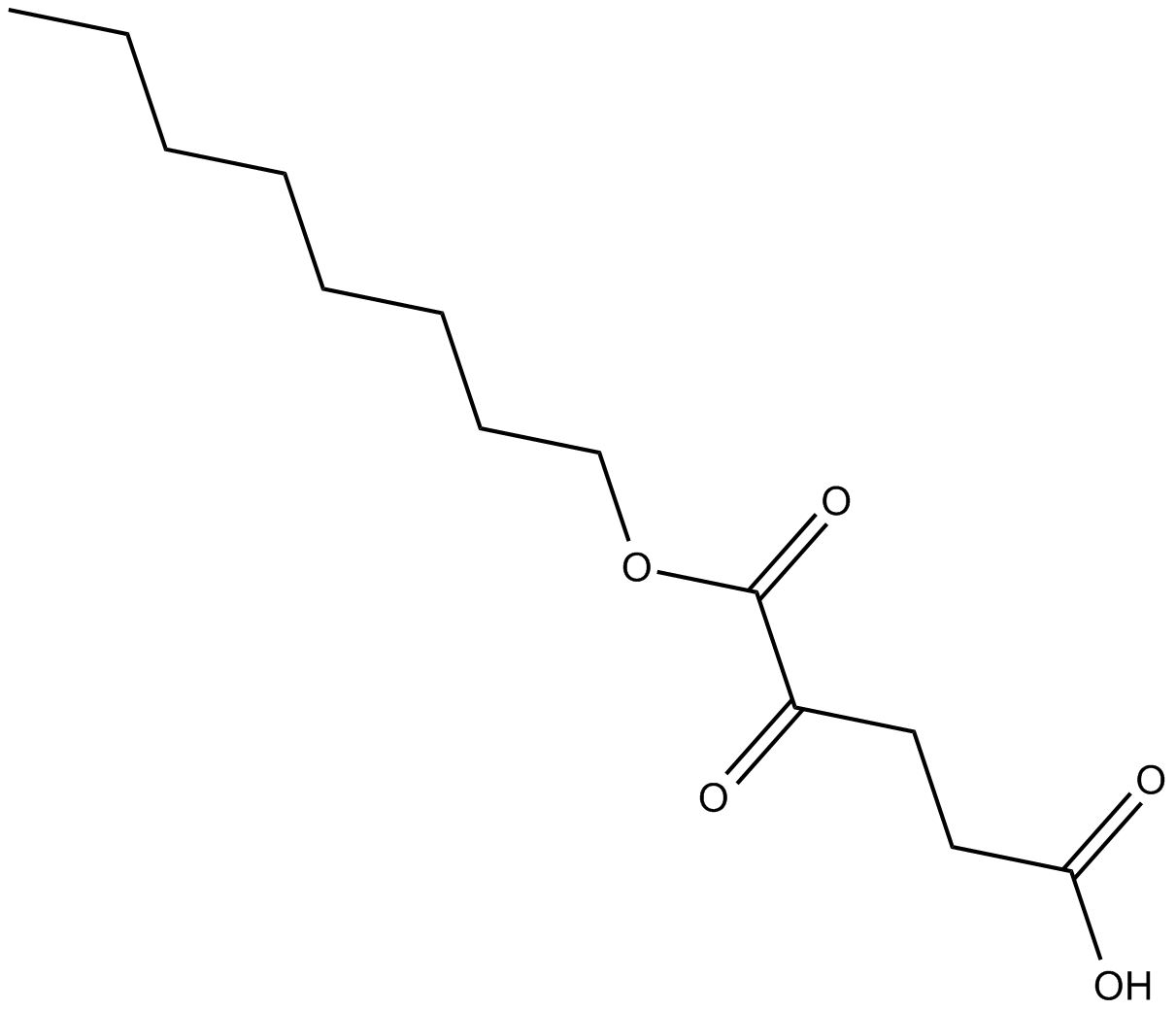
-
GC66067
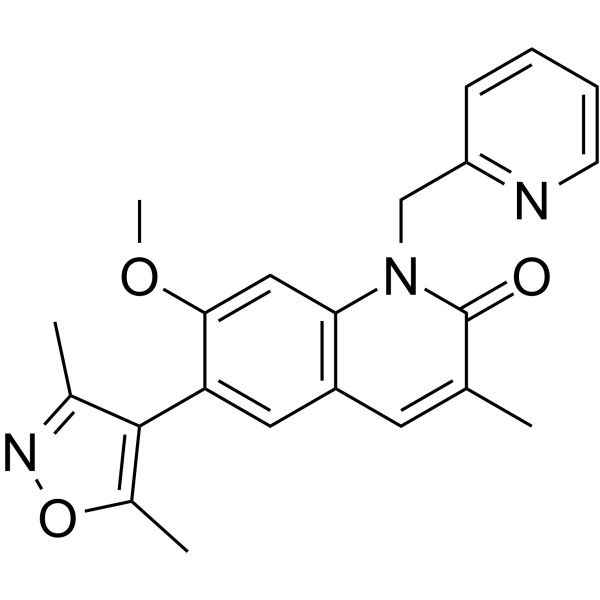
ODM-207
ODM-207 (BET-IN-4) est un puissant inhibiteur de la protéine bromodomaine BET (BRD4), avec une IC50 de ≤ ; 1 μM.
-
GC17482
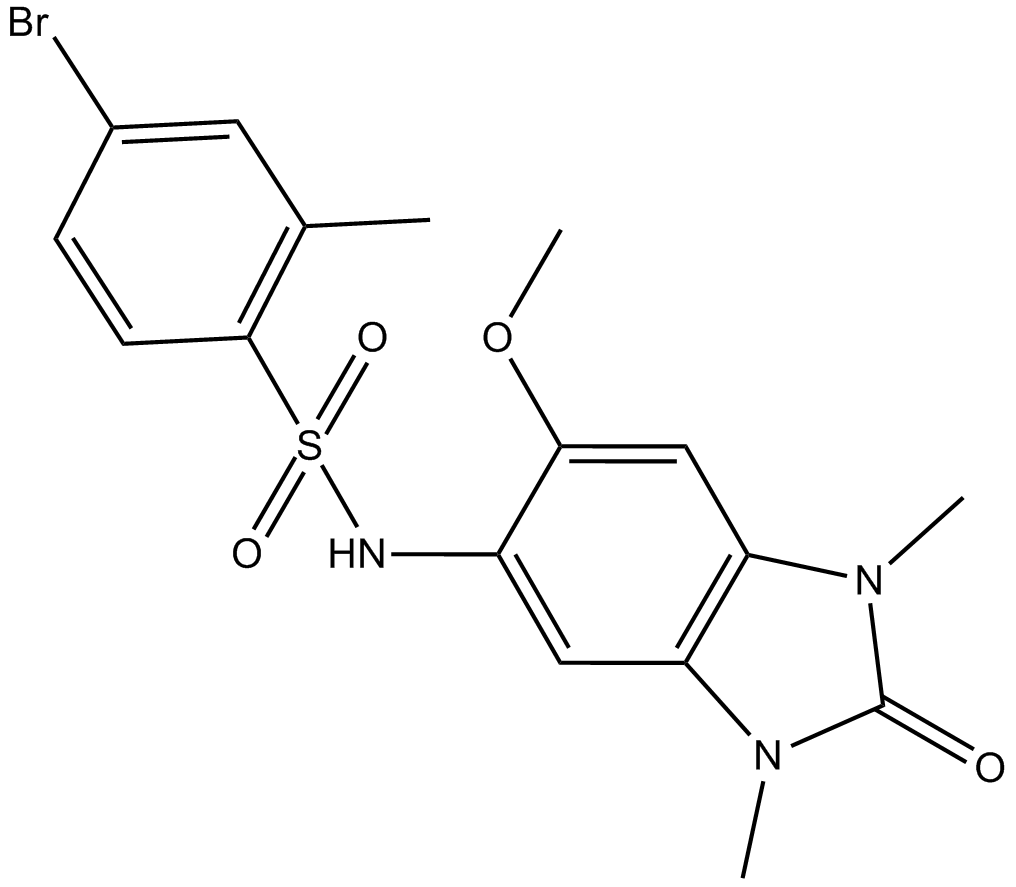
OF-1
OF-1 est un puissant inhibiteur du bromodomaine pan-BRPF (BRD), avec des valeurs IC50 de 270 nM, 1,2 μM pour TRIM24 et BRPF1B, respectivement.
-
GC17314
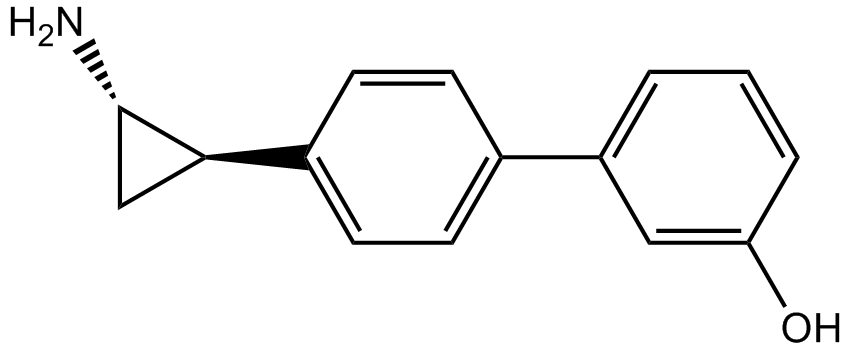
OG-L002
OG-L002 est un inhibiteur LSD1 puissant et hautement sélectif avec une IC50 de 0,02 μM.
-
GC11792
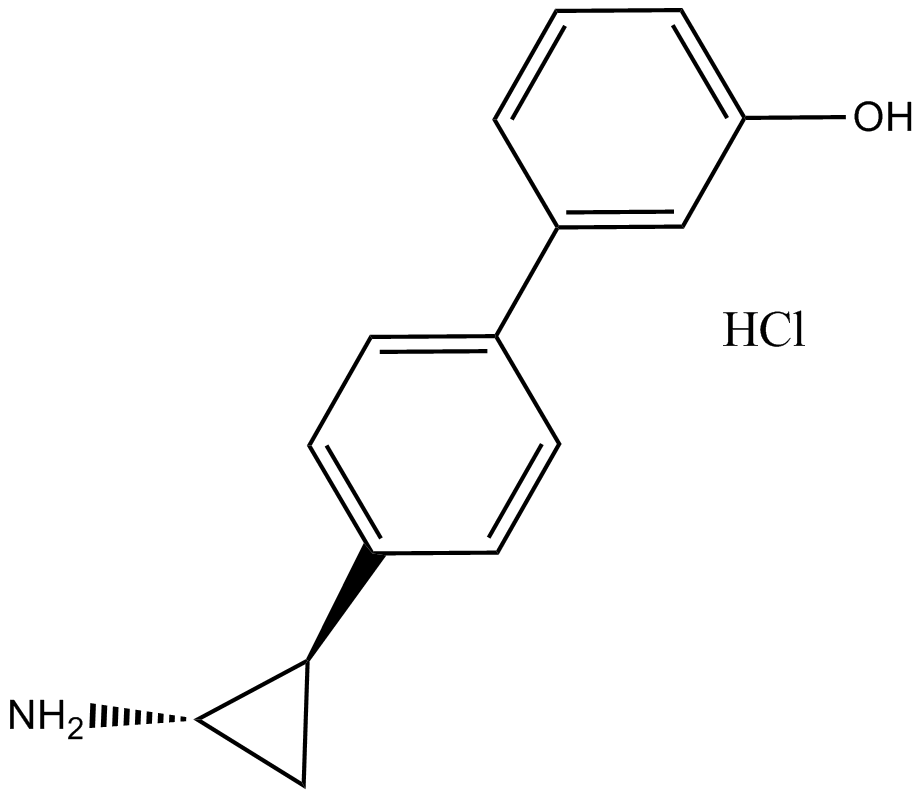
OG-L002 HCl
LSD1 inhibitor,potent and specific
-
GC16397
OICR-9429
OICR-9429 est un inhibiteur À haute affinité du domaine 5 de répétition WD (WDR5), qui bloque de manière compétitive l'interaction de WDR5 avec la protéine MLL via la liaison de la poche centrale de liaison peptidique de WDR5. L'IORC-9429 peut supprimer la triméthylation de l'histone H3K4 et peut être utilisé pour la recherche de divers cancers, notamment la leucémie non réarrangée par la MLL, le cancer du cÔlon, du pancréas, de la prostate et le cancer de la vessie (BCa).
-
GC16958
Okadaic acid
Un inhibiteur puissant de phosphatases protéiques
-
GC17580
Olaparib (AZD2281, Ku-0059436)
Un inhibiteur de PARP
-
GC69618
Olaparib-d8
Olaparib-d8 est le deutérium de Olaparib (AZD2281). Olaparib est un inhibiteur oral efficace de PARP qui inhibe PARP-1 et PARP-2 avec des IC50 respectifs de 5 et 1 nM. Olaparib est également un activateur d'autophagie et de mitophagie.
-
GC49815
Oleuropein aglycone
A polyphenol with diverse biological activities
-
GC12821
Oltipraz
Oltipraz a un effet inhibiteur sur l'activation de HIF-1α de manière dépendante du temps, annulant complètement l'induction de HIF-1α À des concentrations ≥ 10 μM, l'IC50 d'Oltipraz pour l'inhibition de HIF-1α est de 10 μM. Oltipraz est un puissant activateur Nrf2.
-
GC67906
OM-153

-
GN10420
Ophiopogonin D'
-
GN10114
Oroxin A

-
GC41625
Oroxylin A
L'oroxyline A est un flavonoÏde actif naturel avec de puissants effets anticancéreux.
-
GC11360
ORY-1001
Selective inhibitor of KDM1A.
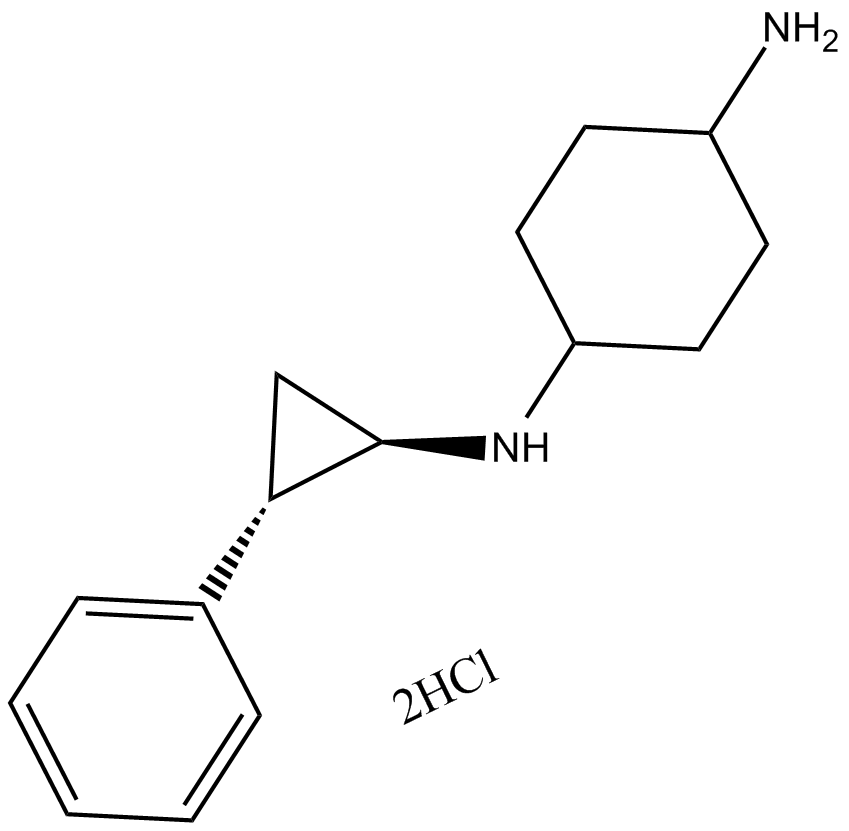
-
GC36818
ORY-1001(trans)
Le dichlorhydrate d'Iadademstat (ORY-1001) est un inhibiteur sélectif irréversible de la déméthylase spécifique de la lysine (K) 1A (KDM1A/LSD1).
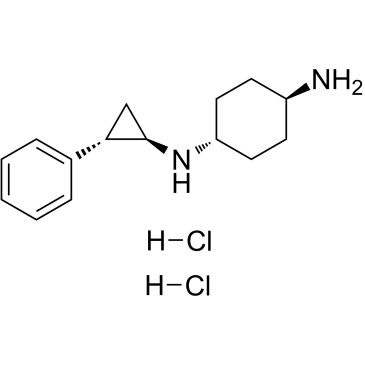
-
GC32745
OSS_128167
OSS_128167 est un puissant inhibiteur sélectif de la sirtuine 6 (SIRT6) avec des IC50 de 89 μM, 1578 μM et 751 μM pour SIRT6, SIRT1 et SIRT2, respectivement.
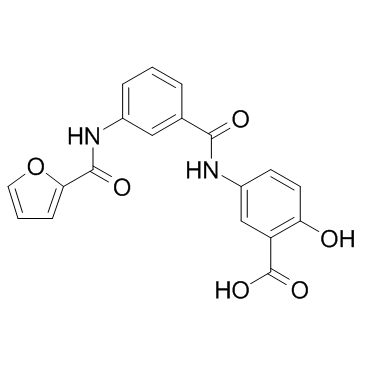
-
GC69636
OTS193320
OTS193320 est un composé d'imidazole et de [1,2-a] pyridine, un inhibiteur d'activité de la méthyltransférase SUV39H2. OTS193320 réduit le niveau de triméthylation globale de l'histone H3 lysine 9 dans les cellules cancéreuses du sein et induit la mort des cellules apoptotiques. Comparé à une seule dose d'OTS193320 ou DOX, la combinaison d'OTS193320 avec Doxorubicin (DOX; A) peut entraîner une diminution du niveau γ-H2AX ainsi que de la viabilité des cellules cancéreuses.
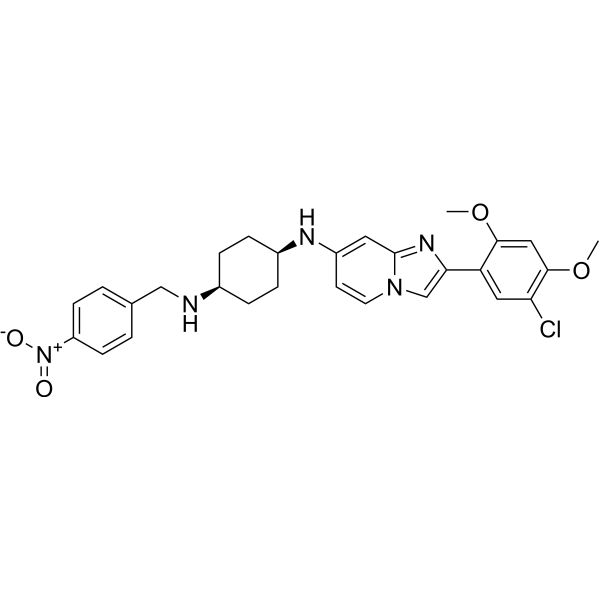
-
GC17973
OTX-015
A BRD2, BRD3, and BRD4 inhibitor
-
GC45808
OUL35
OUL35 (NSC39047) est un inhibiteur puissant et sélectif de ARTD10 (PARP-10), avec une IC50 de 329 nM.
-
GC17472
Oxamflatin
L'oxamflatine (Metacept-3) est un puissant inhibiteur d'HDAC avec une IC50 de 15,7 nM.
-
GC14655
OXF BD 02
OXF BD 02 est un inhibiteur sélectif de BRD4(1) (le premier bromodomaine de BRD4) avec une valeur IC50 de 382 nM.
-
GC39417
OXFBD04
OXFBD04 est un inhibiteur puissant et sélectif de BRD4 avec une IC50 de 166 nM. OXFBD04 est un puissant ligand du bromodomaine BET avec une affinité supplémentaire modeste pour le bromodomaine CREBBP. OXFBD04 a une activité anticancéreuse.
-
GC41329
Pacritinib
FMS-like tyrosine kinase 3 (FLT3) and Janus kinase 2 (JAK2) are tyrosine kinases that mediate cytokine signaling and are frequently mutated in cancers, particularly acute myeloid leukemia.
-
GC67873
PAD-IN-2
-
GC64367
PAD2-IN-1
PAD2-IN-1, un dérivé À base de benzimidazole, est un inhibiteur puissant et sélectif de la protéine arginine déiminase 2 (PAD2).
-
GC66335
PAD2-IN-1 hydrochloride
Le chlorhydrate de PAD2-IN-1, un dérivé À base de benzimidazole, est un inhibiteur puissant et sélectif de la protéine arginine déiminase 2 (PAD2). Le chlorhydrate de PAD2-IN-1 montre une sélectivité supérieure pour PAD2 par rapport À PAD4 (95 fois) et PAD3 (79 fois).
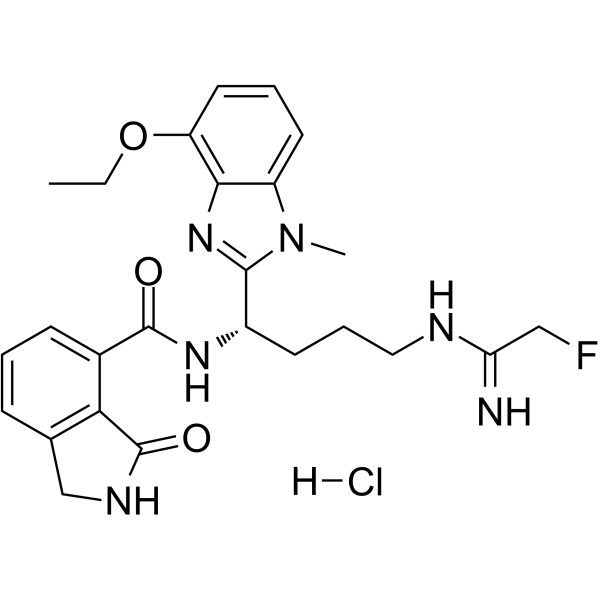
-
GN10452
Paeoniflorin
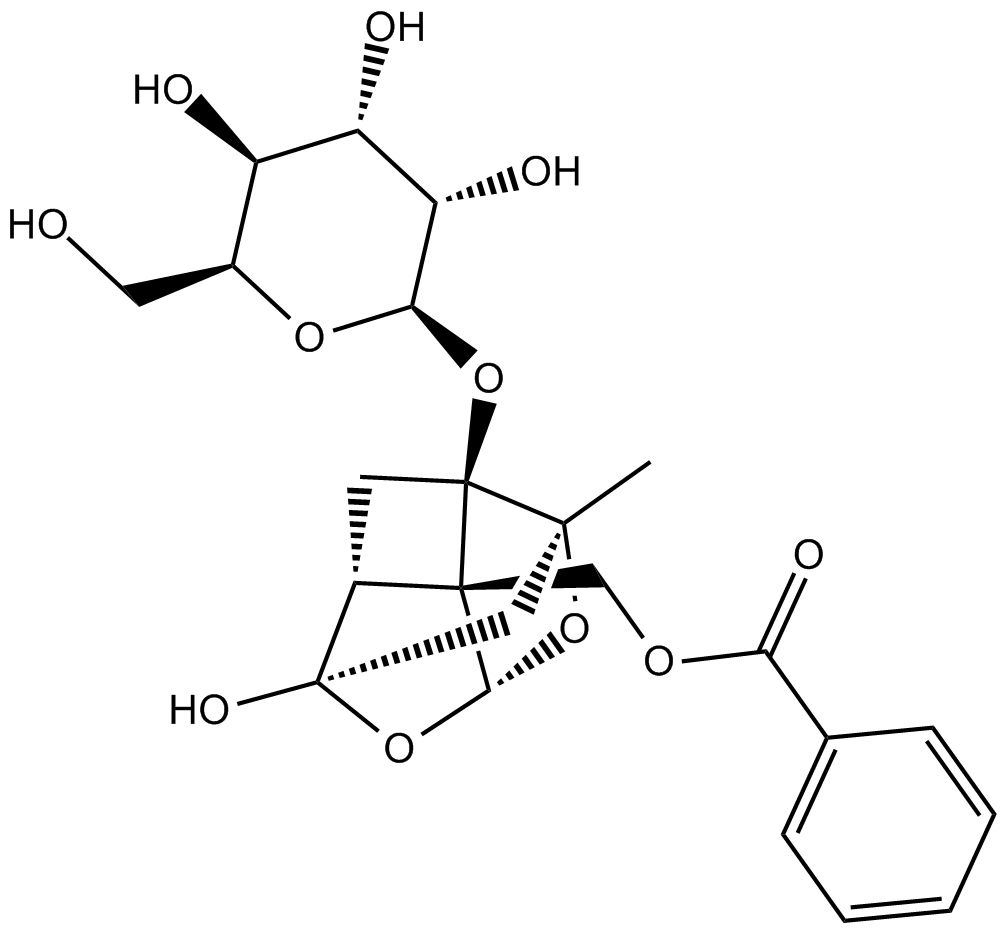
-
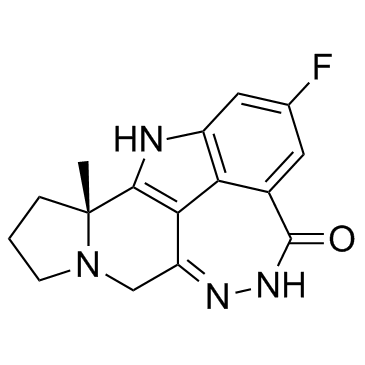
GC34071
Pamiparib (BGB-290)
Le pamiparib (BGB-290) (BGB-290) est un inhibiteur PARP actif par voie orale, puissant et hautement sélectif, avec des valeurs IC50 de 0,9 nM et 0,5 nM pour PARP1 et PARP2, respectivement. Le pamiparib (BGB-290) a un puissant piégeage PARP et une capacité À pénétrer dans le cerveau, et peut être utilisé pour la recherche de divers cancers, y compris la tumeur solide.
-
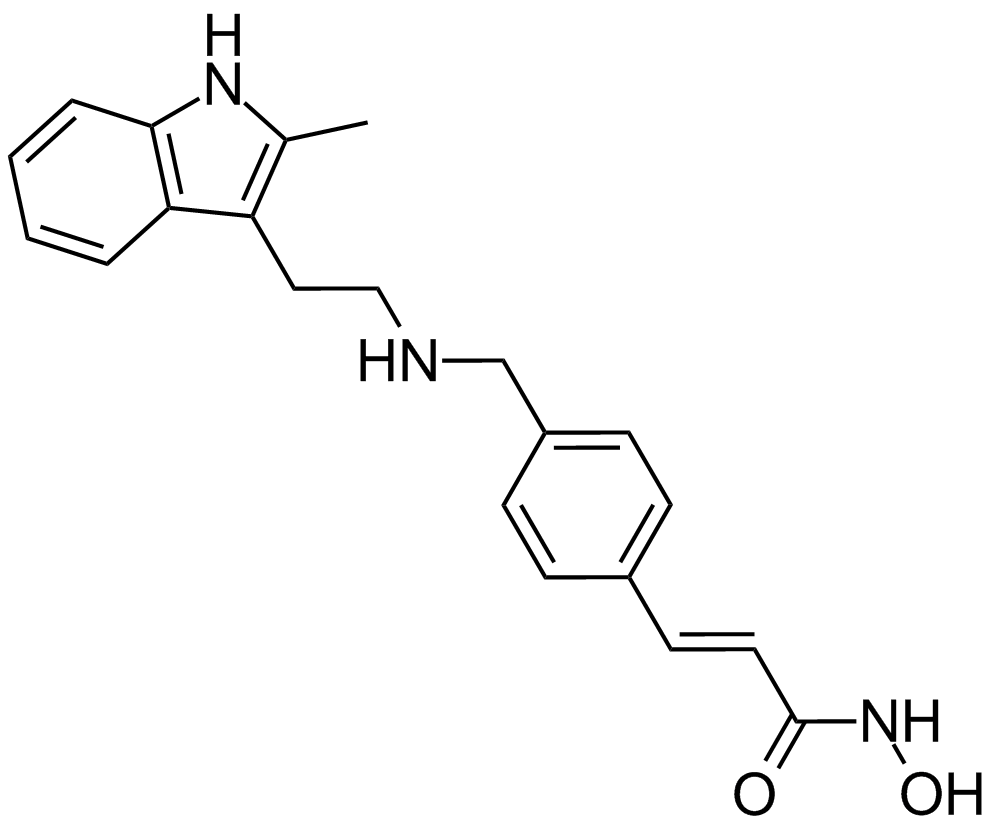
GC12257
Panobinostat (LBH589)
A pan-HDAC inhibitor
-
GC36855
Paris saponin VII
La saponine de Paris VII (Chonglou Saponin VII) est une saponine stéroÏdienne isolée des racines et des rhizomes de Trillium tschonoskii Maxim. L'apoptose induite par la saponine VII de Paris dans les cellules K562/ADR est associée À Akt/MAPK et À l'inhibition de la P-gp. La saponine de Paris VII atténue le potentiel de la membrane mitochondriale, augmente l'expression des protéines liées À l'apoptose, telles que Bax et le cytochrome c, et diminue les niveaux d'expression des protéines de Bcl-2, caspase-9, caspase-3, PARP-1 et p- Act. La saponine de Paris VII induit une autophagie robuste dans les cellules K562/ADR et fournit une base biochimique dans le traitement de la leucémie.
-
GC64579
PARP-1-IN-2
PARP-1-IN-2 (composé 11g) est un puissant inhibiteur de PARP1 pénétré dans la BBB, avec une IC50 de 149 nM. PARP1-IN-2 montre une activité anti-proliférative significativement puissante contre la lignée cellulaire épithéliale d'adénocarcinome pulmonaire humain A549. PARP1-IN-2 peut induire l'apoptose des cellules A549.
-
GC65927
PARP-2-IN-1
PARP-2-IN-1 est un inhibiteur puissant et sélectif de PARP-2 avec une IC50 de 11,5 nM.
-
GC68006
PARP1-IN-11
-
GC62275
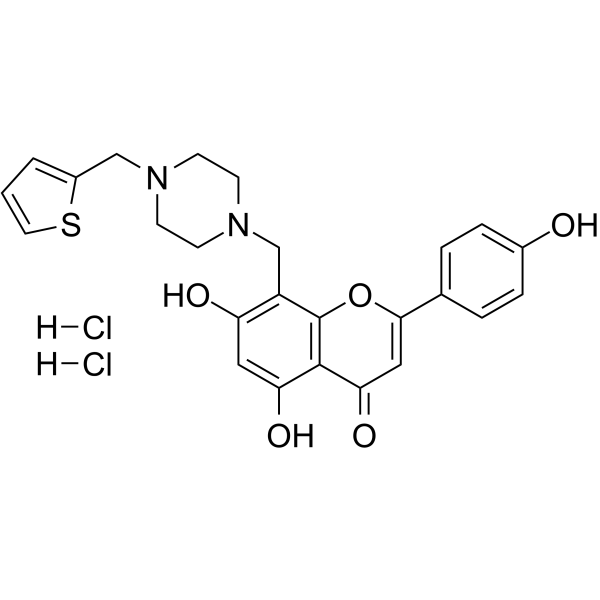
PARP1-IN-5 dihydrochloride
Le dichlorhydrate de PARP1-IN-5 est un inhibiteur de PARP-1 À faible toxicité, actif par voie orale, puissant et sélectif (IC50 = 14,7 nM). Le dichlorhydrate de PARP1-IN-5 peut être utilisé pour la recherche sur le cancer.
-
GC69660
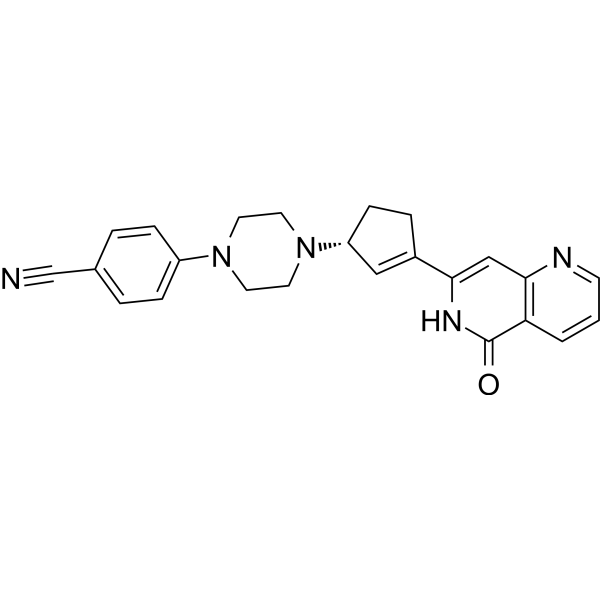
PARP1-IN-7
PARP1-IN-7 is an inhibitor of poly ADP-ribose polymerase-1 (PARP1), used as an anticancer agent.
-
GC64578
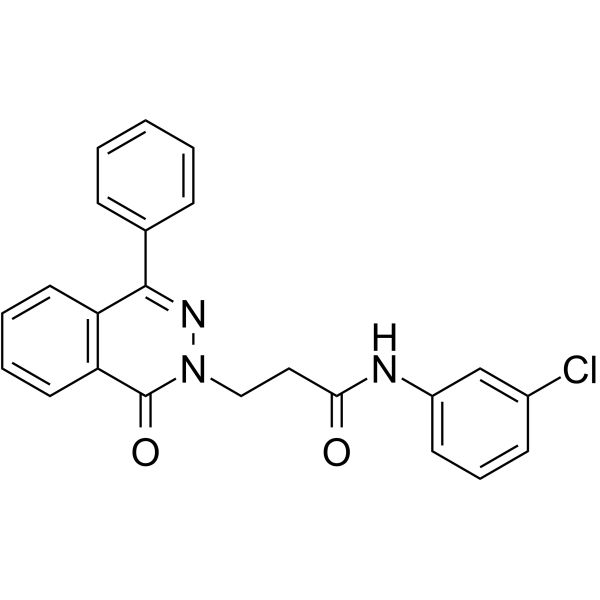
PARP1-IN-8
PARP1-IN-8 (composé 11c) est un puissant inhibiteur de PARP1 pénétré dans la BBB, avec une IC50 de 97 nM. PARP1-IN-8 montre une activité anti-proliférative significativement puissante contre la lignée cellulaire épithéliale d'adénocarcinome pulmonaire humain A549.
-
GC69657
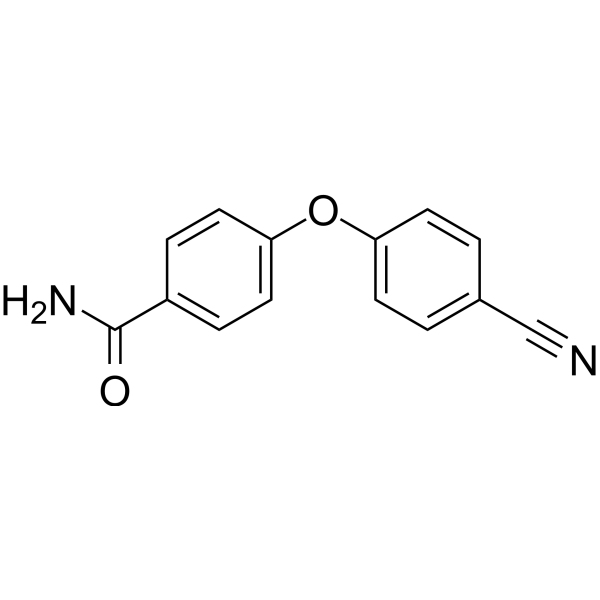
PARP10-IN-2
PARP10-IN-2 est un inhibiteur efficace de la PARP10, une mono-ADP-ribosyltransférase, avec une IC50 de 3,64 μM pour la PARP10 humaine. Il inhibe également la PARP2 et la PARP15, avec des IC50 respectifs de 27 μM et 11 μM pour les versions humaines de ces enzymes.
-
GC69658
PARP10-IN-3
PARP10-IN-3 est un inhibiteur sélectif de la poly(ADP-ribose) polymérase 10 (PARP10), une enzyme de transfert d'ADP-ribose monoétique. Son IC50 pour PARP10 humaine est de 480 nM. PARP10-IN-3 inhibe également les PARP2 et PARP15, avec des IC50 respectifs de 1,7 µM pour l'homme.
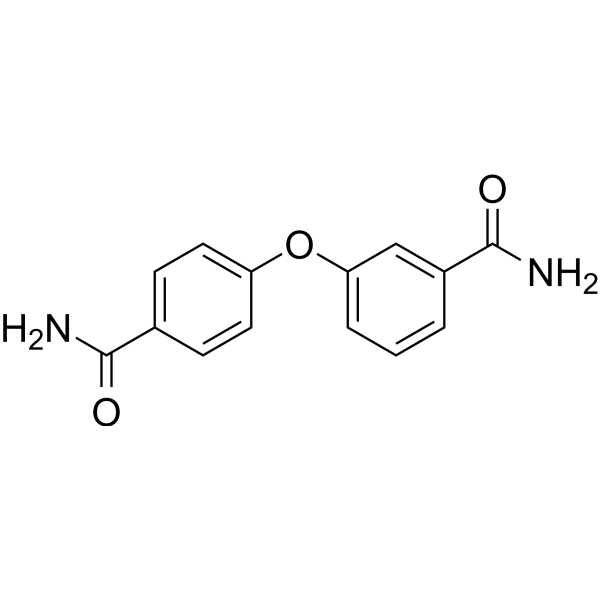
-
GC68035
PARP10/15-IN-1
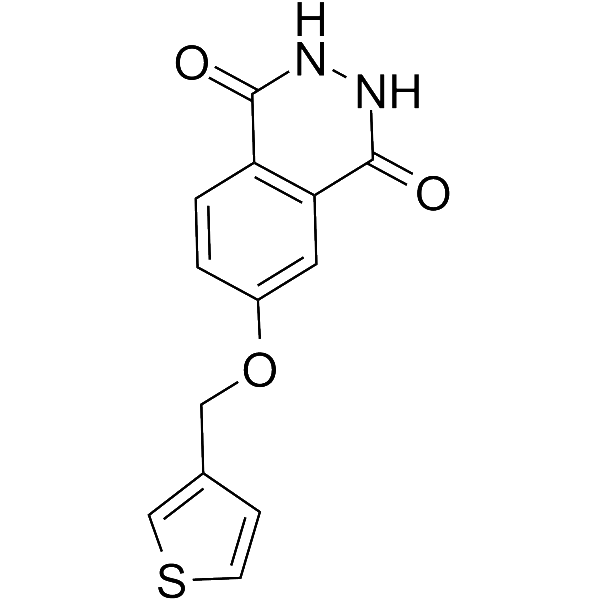
-
GC69655
PARP10/15-IN-2
PARP10/15-IN-2 (Composé 8h) est un inhibiteur doublement efficace de PARP10 et PARP15, avec des valeurs IC50 respectives de 0,15 µM et 0,37 µM. PARP10/15-IN-2 peut pénétrer dans les cellules et empêcher l'apoptose cellulaire.
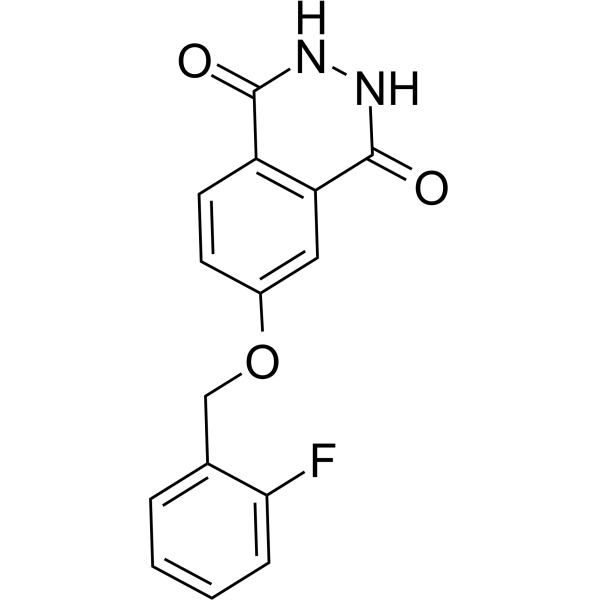
-
GC69656
PARP10/15-IN-3
PARP10/15-IN-3 (Composé 8a) est un inhibiteur doublement efficace de PARP10 et PARP15, avec des valeurs IC50 respectives de 0,14 µM et 0,40 µM. PARP10/15-IN-3 peut pénétrer dans les cellules et empêcher l'apoptose cellulaire.
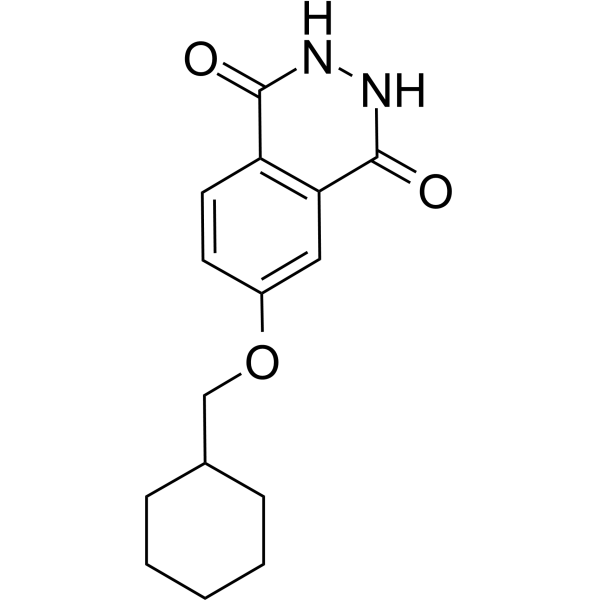
-
GC69659
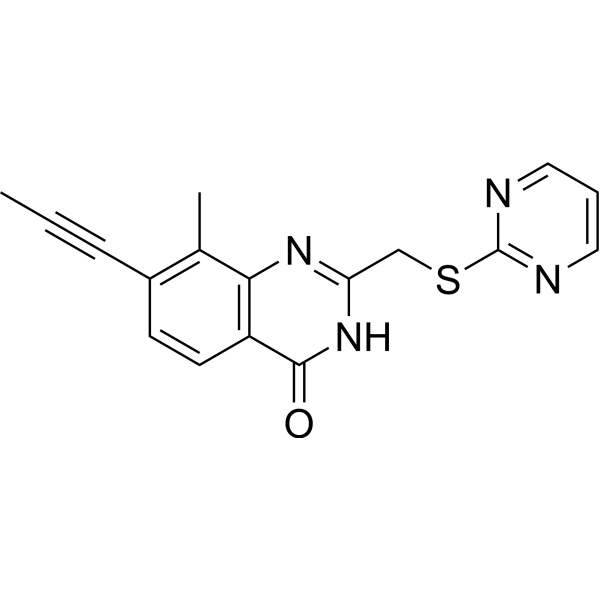
PARP11 inhibitor ITK7
L'inhibiteur de PARP11 ITK7 (ITK7) est un inhibiteur sélectif efficace de PARP11. L'ITK7 peut efficacement inhiber le PARP11 avec une valeur IC50 de 14 nM. L'ITK7 peut être utilisé pour des études de localisation cellulaire.
-
GC39302
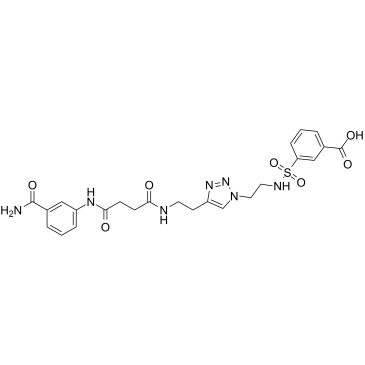
PARP14 inhibitor H10
L'inhibiteur de PARP14 H10, composé H 10, est un inhibiteur sélectif contre PARP14 (IC50 = 490 nM), par rapport aux autres PARP (≈ 24 fois par rapport À PARP1). L'inhibiteur de PARP14 H10 induit l'apoptose cellulaire médiée par la caspase-3/7.
-
GC69661
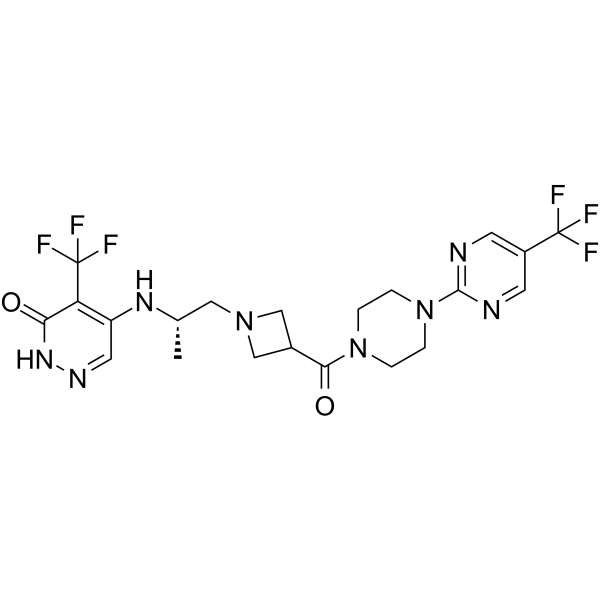
PARP7-IN-14
PARP7-IN-14 (I-1) est un inhibiteur sélectif efficace de PARP7 avec une valeur IC50 de 7,6 nM. PARP7-IN-14 possède une activité anticancéreuse.
-
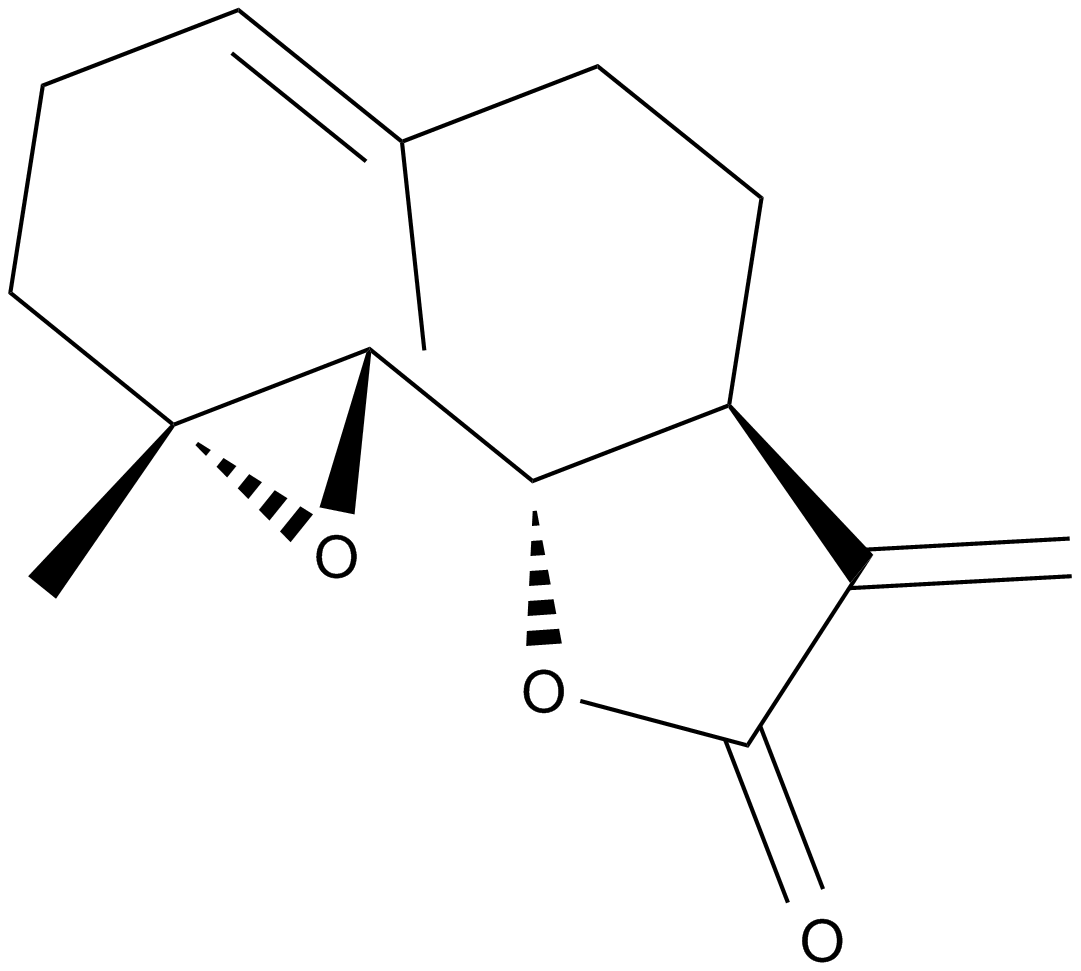
GN10357
Parthenolide
-
GC15301
PBIT
Le PBIT est un inhibiteur spécifique des enzymes Jumonji AT-rich Interactive Domain 1 (JARID1). Le PBIT inhibe l'histone déméthylase JARID1B (KDM5B ou PLU1) avec une IC50 d'environ 3 μM . PBIT inhibe également JARID1A et JARID1C avec des IC50 de 6 μM et 4,9 μM, respectivement.
-
GC69669
PCAF-IN-2
PCAF-IN-2 (composé 17) est un inhibiteur efficace de PCAF, avec une valeur IC50 de 5,31 µM. PCAF-IN-2 présente une activité anti-tumorale. CAF-IN-2 induit l'apoptose et bloque le cycle cellulaire en phase G2/M.