Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
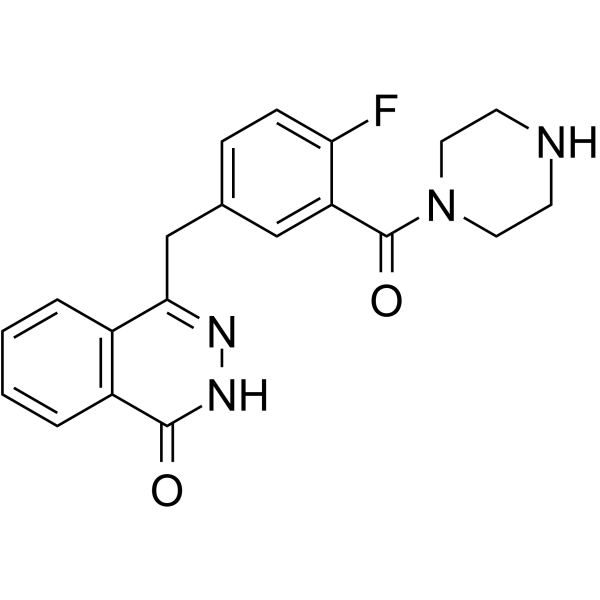
GC25580
LLY-284
LLY-284 is the diastereomer of LLY-283, which is a potent and selective SAM-competitive chemical probe for PRMT5. LLY-284 is much less active than LLY-283 and can be used as a negative control for LLY-283.
-
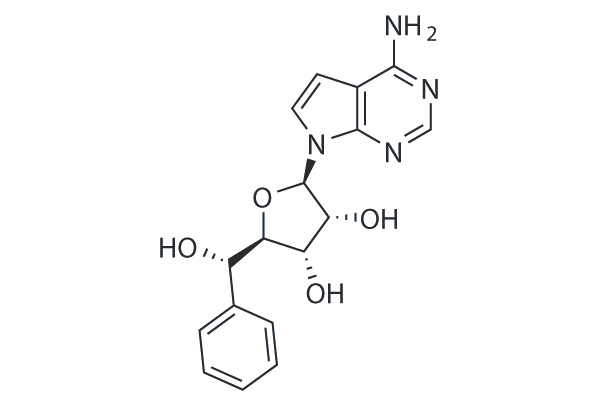
GC16261
LLY507
LLY507 est un inhibiteur puissant et sélectif de la protéine-lysine méthyltransférase SMYD2. LLY507 inhibe puissamment la capacité de SMYD2 à méthyler le peptide p53 avec une IC50 \u003c15 nM. LLY507 sert de sonde chimique précieuse pour aider à la dissection de la fonction SMYD2 dans le cancer et d'autres processus biologiques.
-
GC13237
LMK 235
A selective inhibitor of HDAC4 and HDAC5
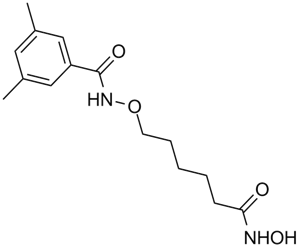
-
GC11778
Lomeguatrib
Inactivator of O6-methylguanine-DNA methyltransferase
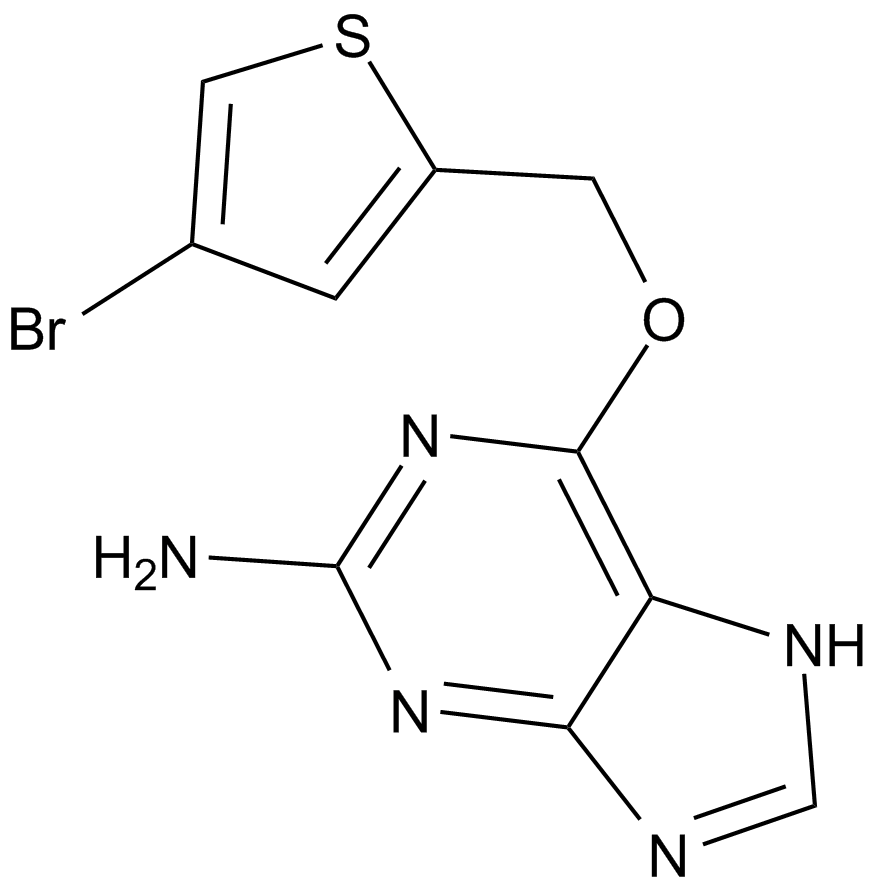
-
GC64372
Lorpucitinib
Le lorpucitinib est un inhibiteur de JAK À restriction intestinale pour la recherche sur les maladies inflammatoires de l'intestin.
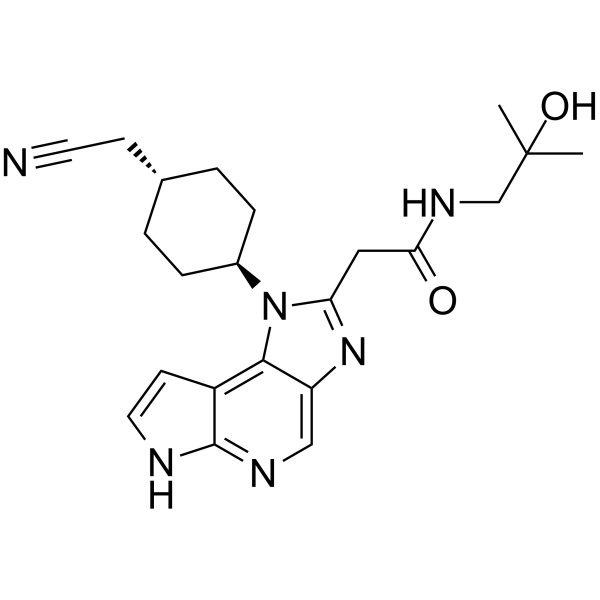
-
GC18731
LP99
LP99, une sonde épigénétique, est un inhibiteur puissant et sélectif des bromodomaines BRD7 et BRD9 avec un Kd de 99 nM contre BRD9.
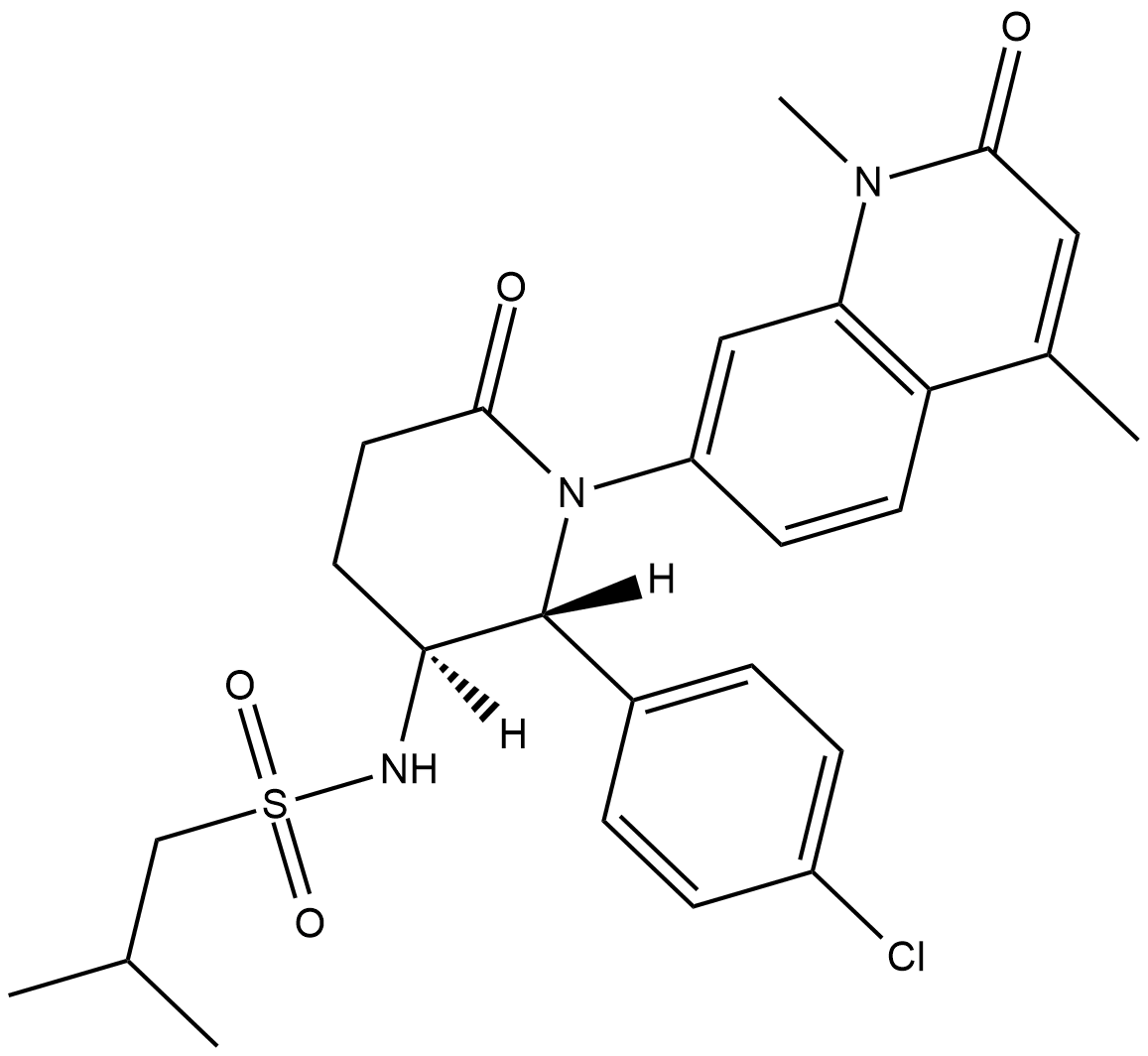
-
GC36484
LSD1-IN-5
LSD1-IN-5 (composé 4e) est un inhibiteur puissant et réversible de la déméthylase spécifique de la lysine 1 (LSD1), avec une IC50 de 121 nM. LSD1-IN-5 augmente la Lys4 diméthylée de l'histone H3, ne montre aucun effet sur l'expression de LSD1.
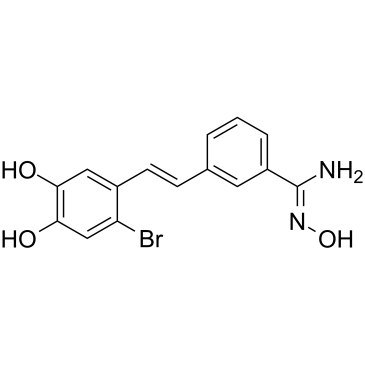
-
GC36485
LSD1-IN-6
LSD1-IN-6 (Composé 4m) est un inhibiteur puissant et réversible de la déméthylase spécifique de la lysine 1 (LSD1), avec une IC50 de 123 nM. LSD1-IN-6 augmente la Lys4 diméthylée de l'histone H3, ne montre aucun effet sur l'expression de LSD1.
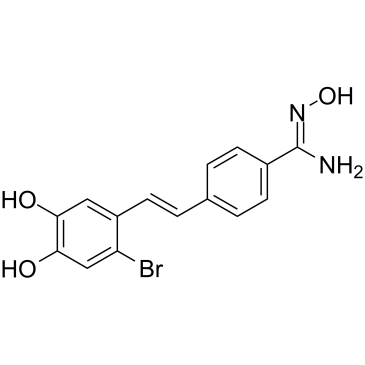
-
GC62434
LSD1/HDAC6-IN-1
LSD1/HDAC6-IN-1 est un double inhibiteur actif par voie orale de la déméthylase spécifique de la lysine 1 (LSD1)/Histone désacétylase 6 (HDAC6), avec une activité anti-tumorale. LSD1/HDAC6-IN-1 peut être utilisé pour la recherche sur le myélome multiple (MM).
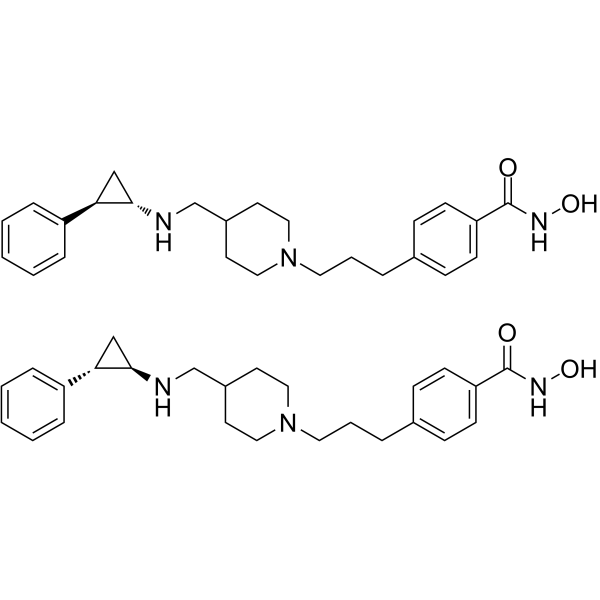
-
GC62591
LT052
LT052 est un inhibiteur hautement sélectif de BET BD1 avec une IC50 de 87,7 nM.
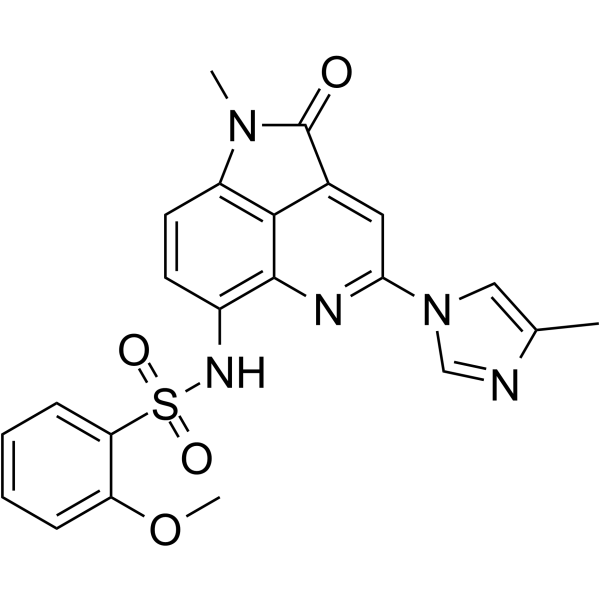
-
GC32724
LW6 (HIF-1α inhibitor)
LW6 (inhibiteur de HIF-1α) est un nouvel inhibiteur de HIF-1 avec une IC50 de 4,4 µM. LW6 (inhibiteur de HIF-1α) diminue l'expression des protéines HIF-1α sans affecter l'expression de HIF-1β.
-
GC13848
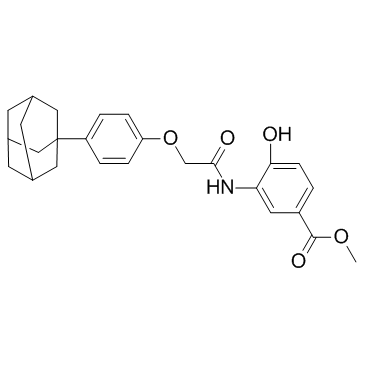
LY2784544
Potent inhibitor of JAK2
-
GC33057
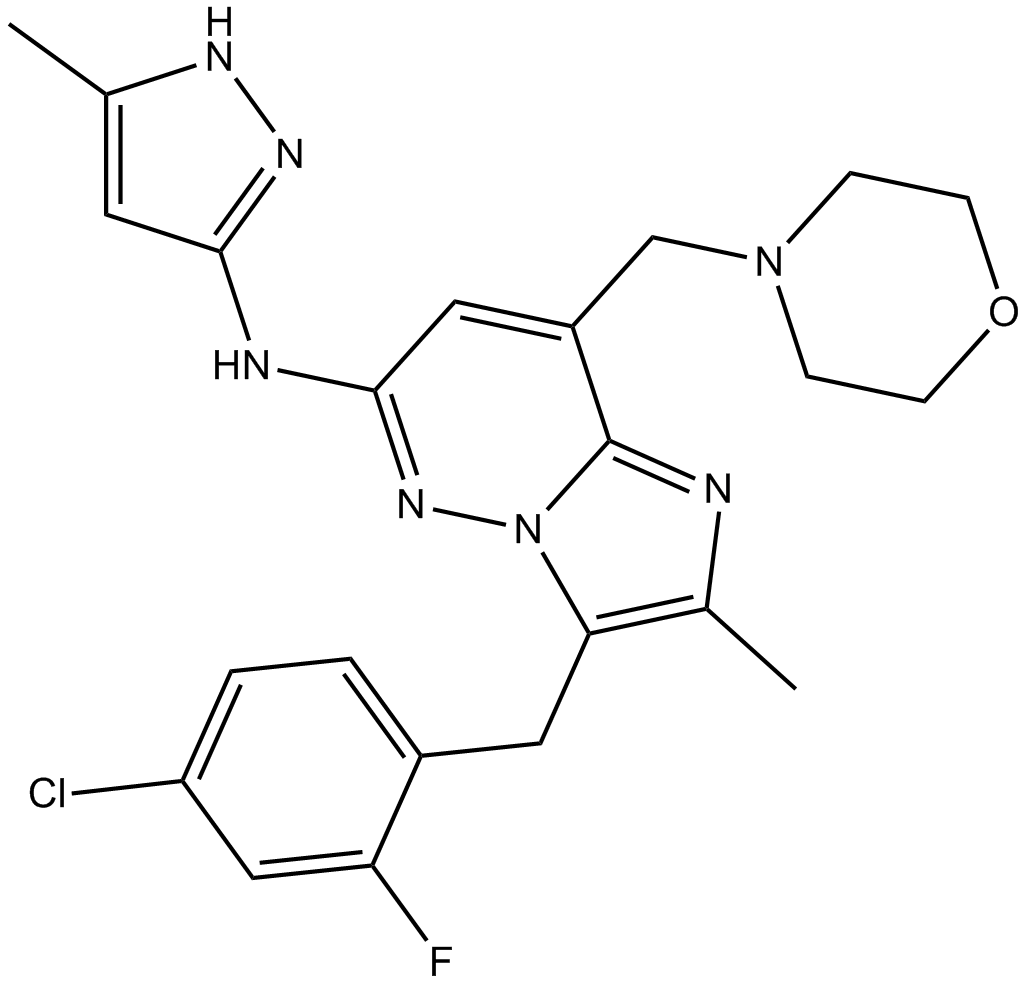
LY3295668 (AK-01)
LY3295668 (AK-01) (AK-01) est un inhibiteur de la kinase Aurora-A puissant, actif par voie orale et hautement spécifique, avec des valeurs Ki de 0,8 nM et 1038 nM pour AurA et AurB, respectivement.
-
GC68405
Lys-CoA TFA
-
GC13534
Lysine-specific Demethylase Inhibitor (1C)
lysine-specific demethylase Inhibitor
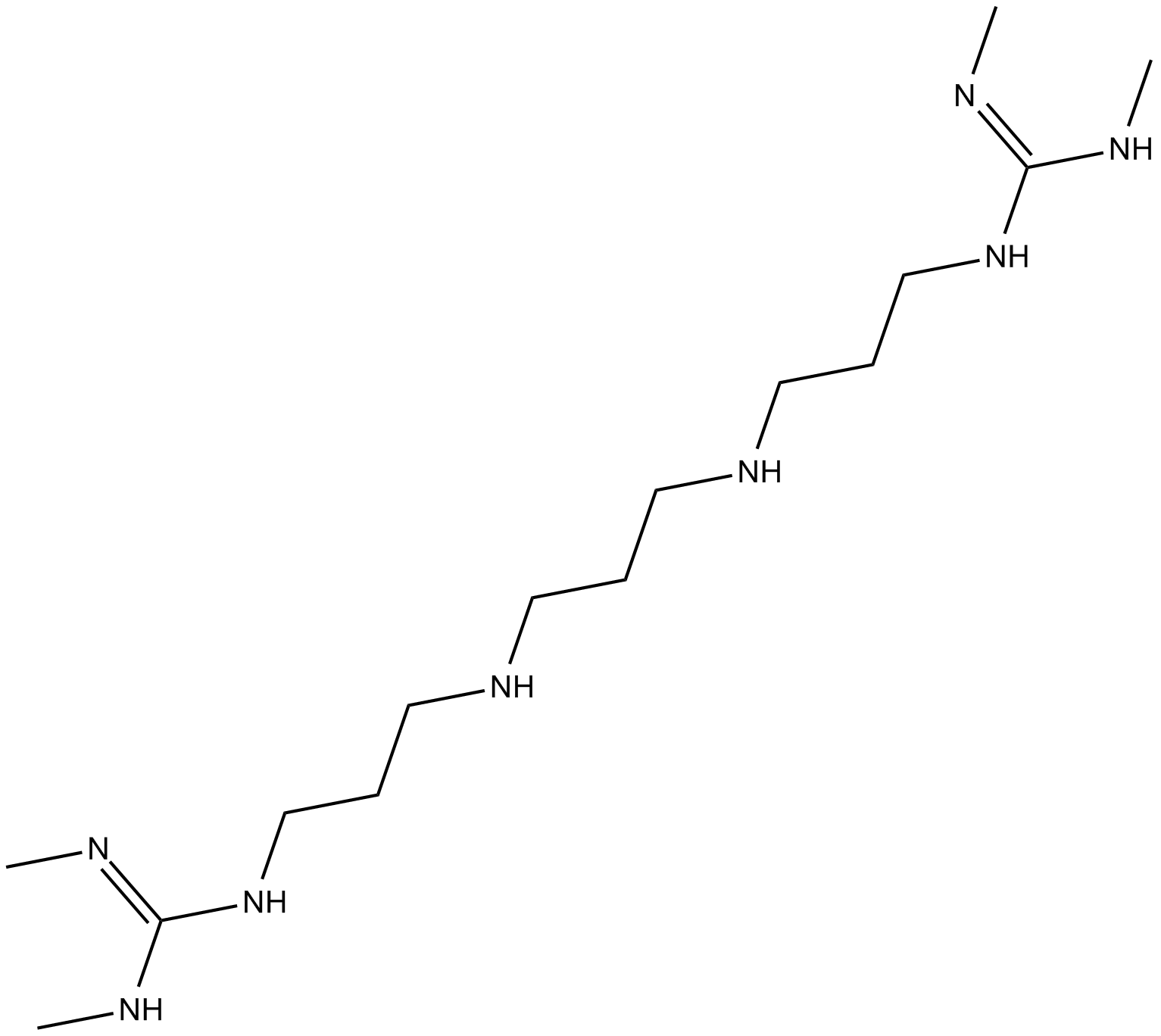
-
GC47586
Lysine-specific Demethylase Inhibitor (1C) (hydrochloride)
An LSD1 inhibitor
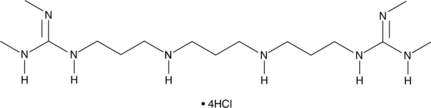
-
GC34138
M-110
M-110 est un inhibiteur hautement sélectif et compétitif de l'ATP des kinases PIM avec une préférence pour PIM-3 (IC50 = 47 nM). Le M-110 inhibe PIM-1 et PIM-2 avec des IC50 similaires de 2,5 μM. Le M-110 inhibe la prolifération des lignées cellulaires du cancer de la prostate avec des IC50 de 0,6 À 0,9 μM.
-
GC47693
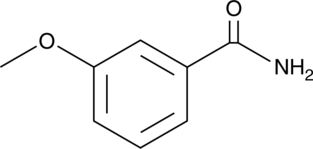
m-Methoxybenzamide
Le m-méthoxybenzamide (3-MBA), un inhibiteur de l'ADP-ribosyltransférase (ADPRT) et de la PARP, inhibe la division cellulaire chez Bacillus subtilis, entraÎnant la filamentation et éventuellement la lyse des cellules. Le m-méthoxybenzamide (3-MBA) améliore la croissance des plantes in vitro, la microtubérisation et l'efficacité de transformation de la pomme de terre bleue (Solanum tuberosum L. subsp. andigenum).
-
GC64945
M1001
M1001 est un agoniste faible du facteur 2α inductible par l'hypoxie (HIF-2α). M1001 peut se lier au domaine HIF-2α PAS-B, avec un Kd de 667 nM. Le M1001 peut être utilisé dans la recherche sur les maladies rénales chroniques.
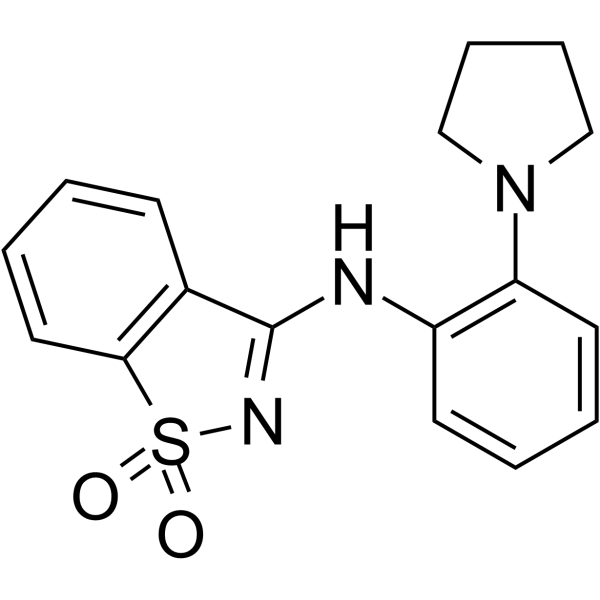
-
GC65046
M1002
M1002 est un agoniste du facteur 2 induit par l'hypoxie (HIF-2) et peut améliorer l'expression des gènes cibles de HIF-2. M1002 présente une synergie avec les inhibiteurs du domaine prolyl-hydroxylase (PHD).
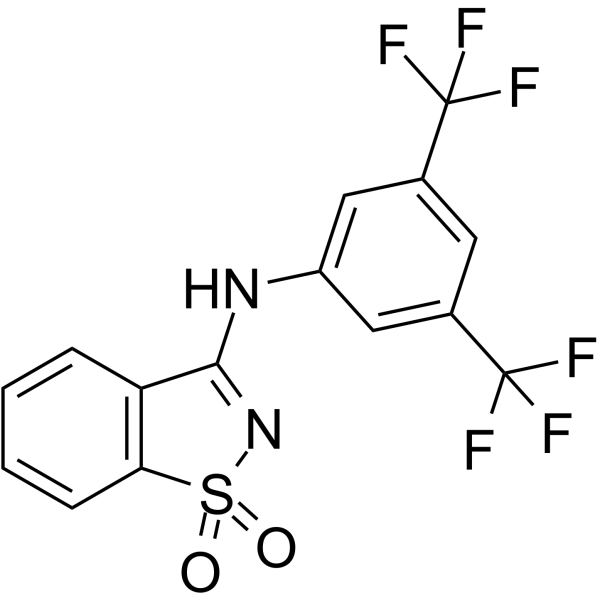
-
GC16456
M344
Inhibiteur d'HDAC
-
GC36521
M89
Le M-89 est un inhibiteur de la ménine très puissant et spécifique, avec un Kd de 1,4 nM pour la liaison à la ménine.
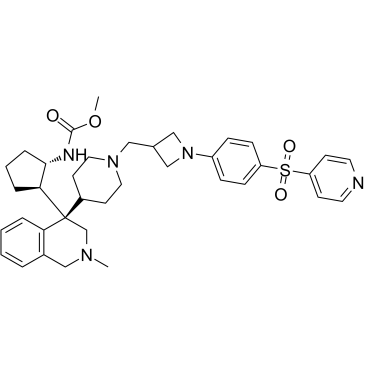
-
GC63056
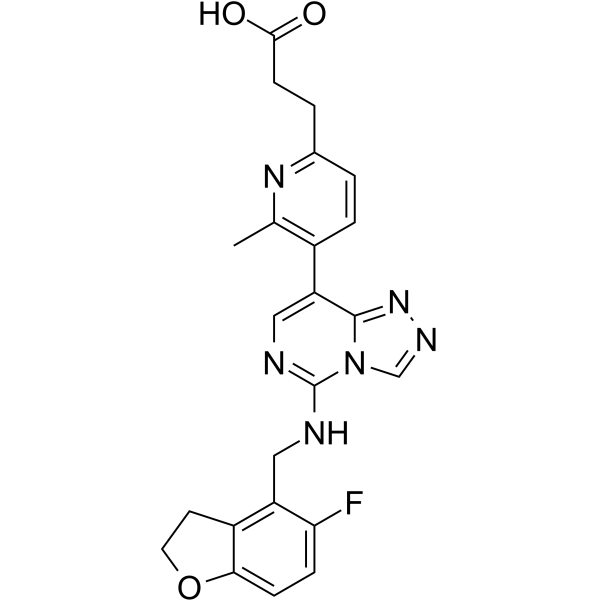
MAK683-CH2CH2COOH
MAK683-CH2CH2COOH se lie À l'EED (protéine de développement de l'ectoderme embryonnaire). MAK683-CH2CH2COOH et un ligand VHL pour l'ubiquitine ligase E3 ont été utilisés pour concevoir PROTAC EED degrader-1 et PROTAC EED degrader-2.
-
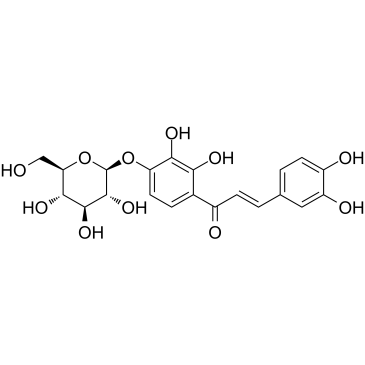
GC61029
Marein
Marein a l'effet neuroprotecteur dÛ À une réduction des dommages À la fonction des mitochondries et À l'activation de la voie du signal AMPK.
-
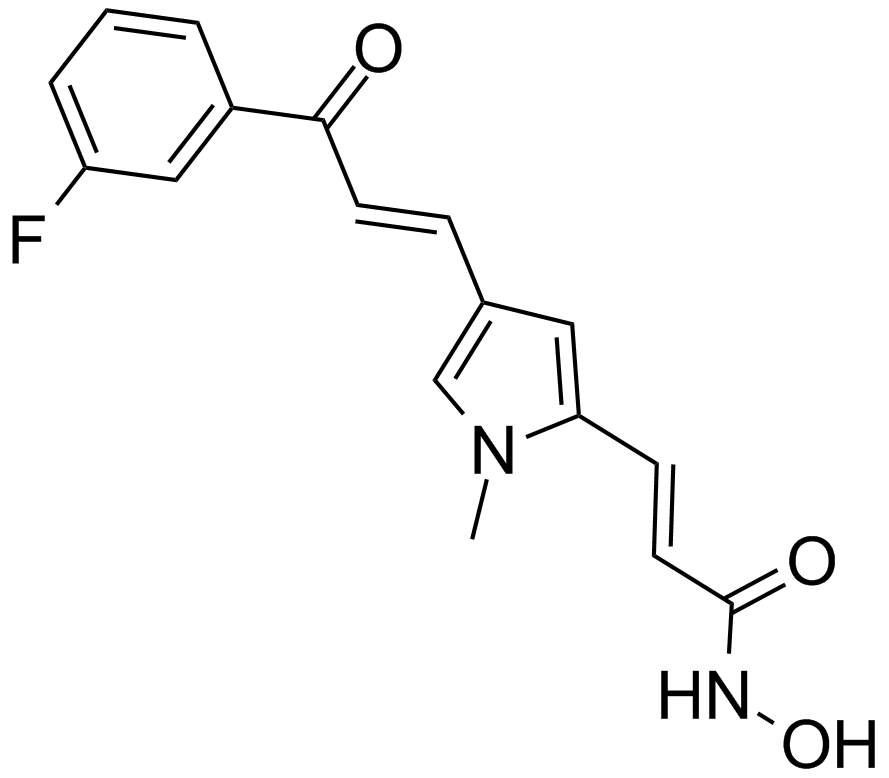
GC15965
MC1568
A selective class IIa HDAC inhibitor
-
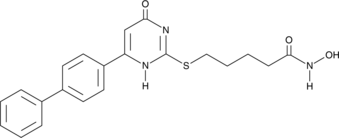
GC45906
MC1742
Le MC1742 est un puissant inhibiteur de HDAC, avec des IC50 de 0,1 μM, 0,11 μM, 0,02 μM, 0,007 μM, 0,61 μM, 0,04 μM et 0,1 μM pour HDAC1, HDAC2, HDAC3, HDAC6, HDAC8, HDAC10 et HDAC11, respectivement. Le MC1742 peut augmenter les niveaux d'acétyl-H3 et d'acétyl-tubuline et inhiber la croissance des cellules souches cancéreuses. Le MC1742 peut induire un arrêt de croissance, une apoptose et une différenciation dans le sarcome CSC.
-
GC34431
MC3482
Le MC3482 est un inhibiteur spécifique de la sirtuine5 (SIRT5).
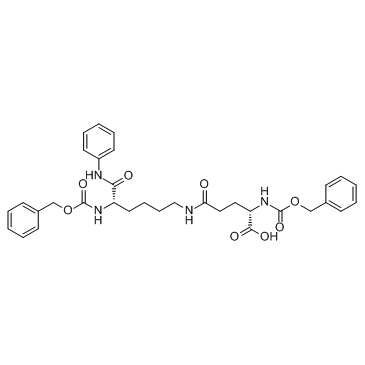
-
GC65254
MC4355
Le MC4355 est un double inhibiteur d'EZH2 et d'histone désacétylase (HDAC).
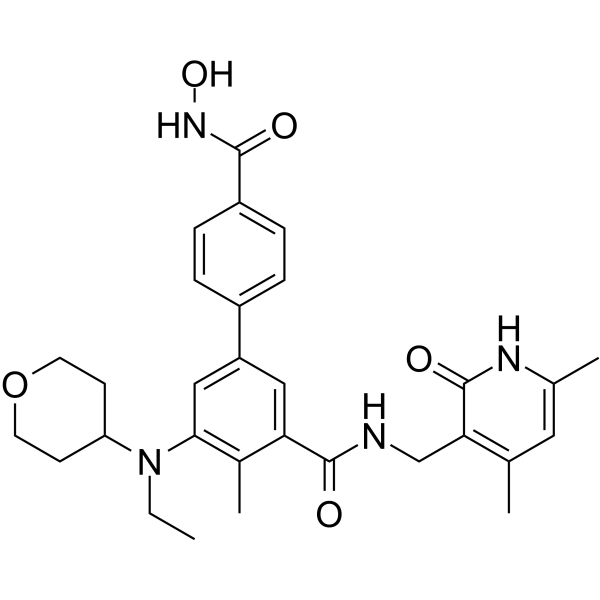
-
GC15049
MCB-613
Le MCB-613 est un puissant \MCB-613 is a potent Steroid receptor coactivator SRC small molecule 'stimulator' (SMS), super-stimulates SRCs' transcriptional activity.MCB-613 increases SRCs' interactions with other coactivators and markedly induces ER stress coupled to the generation of reactive oxygen speciesen_fr_2022q1.md(ROS).MCB-613 is a SMS that target oncogenes can be exploited as anti-cancer agents by over-stimulating the SRC oncogenic program.en_fr_2022q1.md
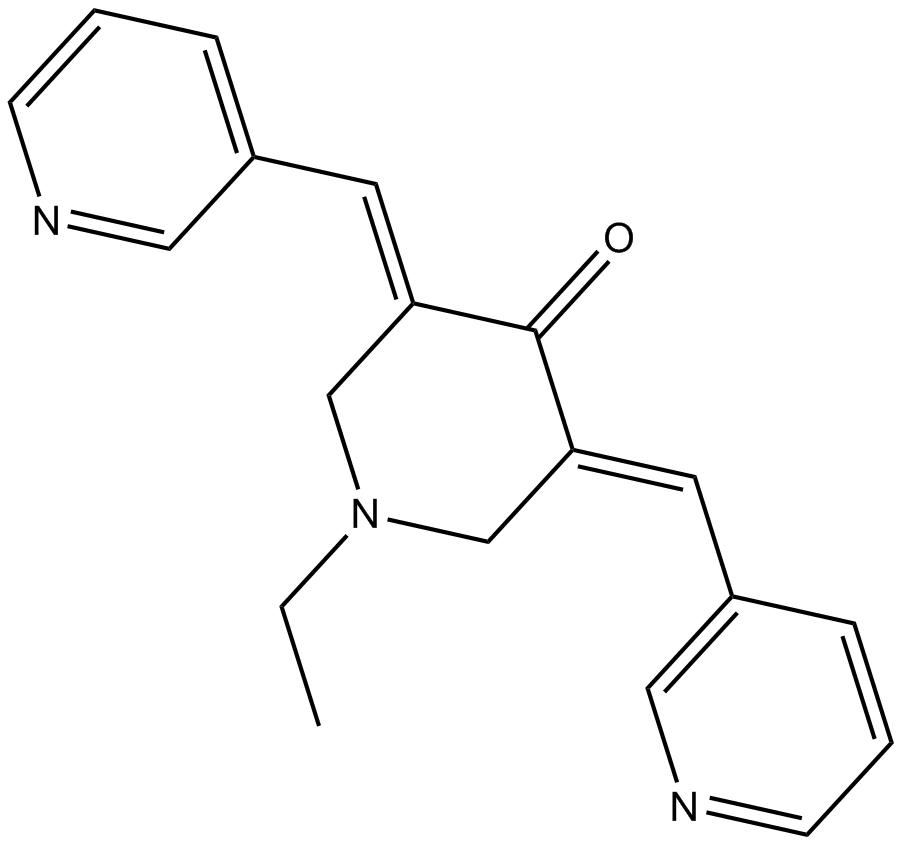
-
GC52188
MDL 800

-
GC13419
ME0328
ME0328 est un inhibiteur ARTD3/PARP3 puissant et sélectif avec une IC50 de 0,89 ± 0,28 μM.
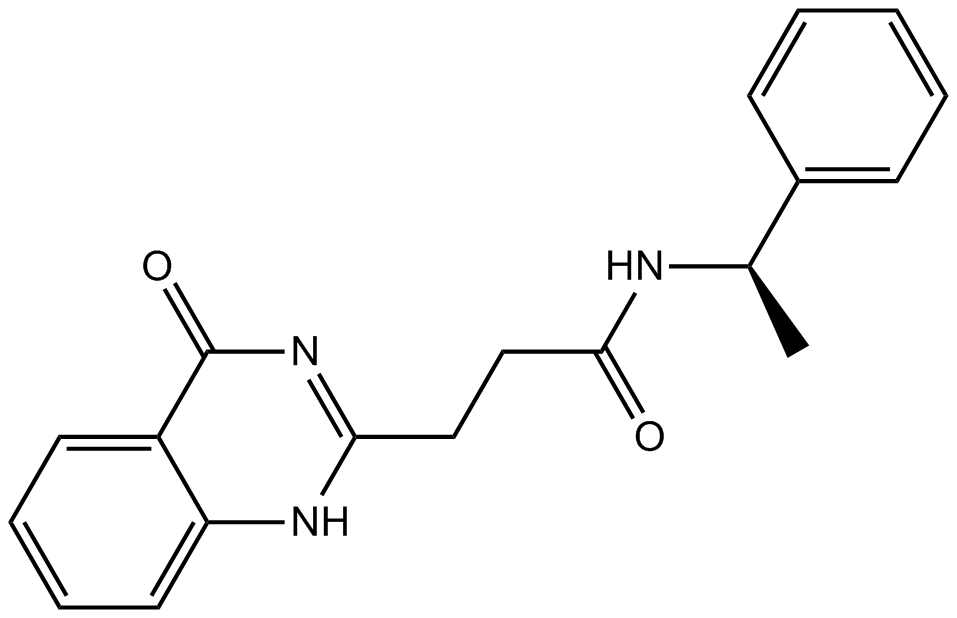
-
GC62252
Mefuparib hydrochloride
Le chlorhydrate de méfuparib (MPH) est un inhibiteur PARP1/2 actif par voie orale, compétitif avec le substrat et sélectif avec des CI50 de 3,2 nM et 1,9 nM, respectivement. Le chlorhydrate de méfuparib induit l'apoptose et possède une activité anticancéreuse importante in vitro et in vivo.
-
GC63064
Menin-MLL inhibitor 19
Inhibiteur de ménine-MLL 19, un puissant inhibiteur exo-aza spiro de l'interaction ménine-mll, exemple A17, extrait du brevet WO2019120209A1.
-
GC63538
Menin-MLL inhibitor 20
L'inhibiteur de ménine-MLL 20 est un inhibiteur irréversible de l'interaction ménine-MLL avec des activités antitumorales (WO2020142557A1, intermédiaire 6).

-
GC48805
Methapyrilene (hydrochloride)
Le chlorhydrate de méthapyrilène (Thenylpyramine) est un antihistaminique des récepteurs H1 actif par voie orale et un agent anticholinergique de la classe chimique des pyridines.
-
GC44184
Methylstat (hydrate)
Methylstat is a methyl ester prodrug of a Jumonji C domain-containing histone demethylase (JMJD) inhibitor that has favorable cell permeability.
-
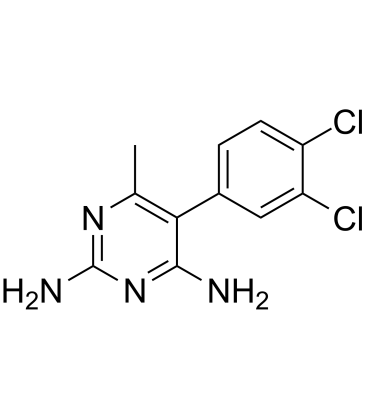
GC61058
Metoprine
La métoprine (BW 197U) est un puissant inhibiteur de l'histamine N-méthyltransférase (HMT).
-
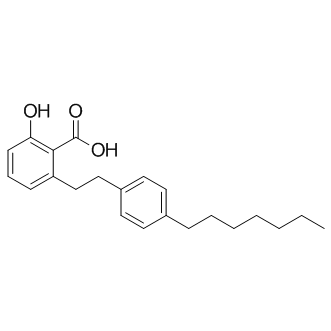
GC11439
MG 149
MG149 (inhibiteur de Tip60 HAT) est un inhibiteur sélectif et puissant de Tip60 avec IC50 de 74 uM, puissance similaire pour MOF (IC50 = 47 uM); peu puissant pour PCAF et p300 (IC50 > 200 uM).
-
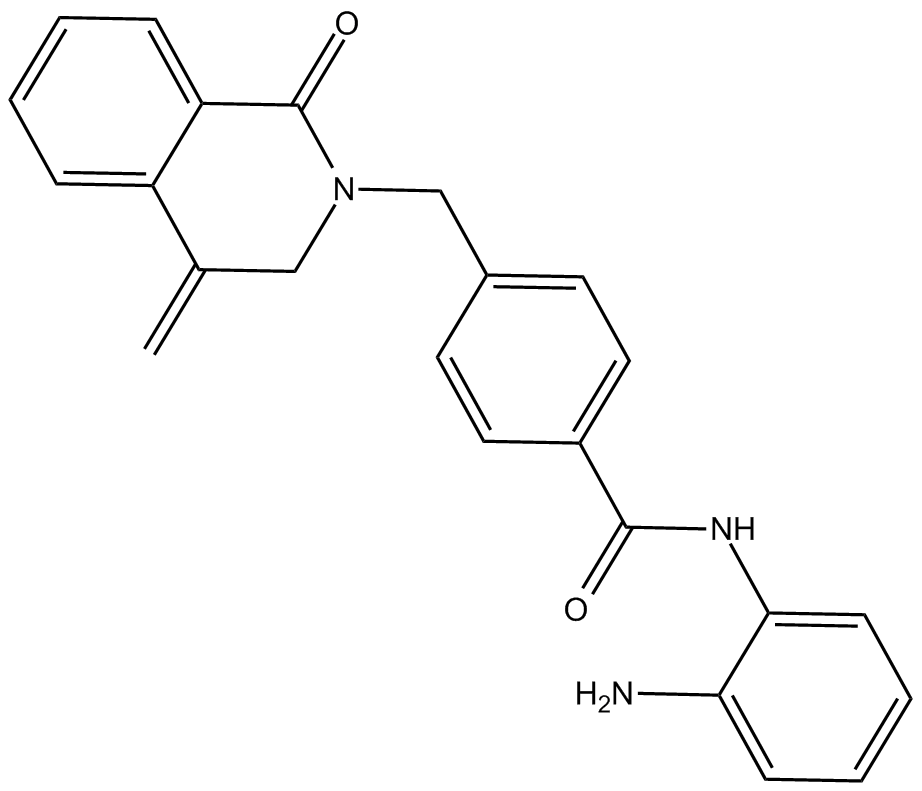
GC11949
MI-192
Le MI-192 est un inhibiteur sélectif de HDAC2 et HDAC3 avec des IC50 de 30 nM et 16 nM, respectivement.
-
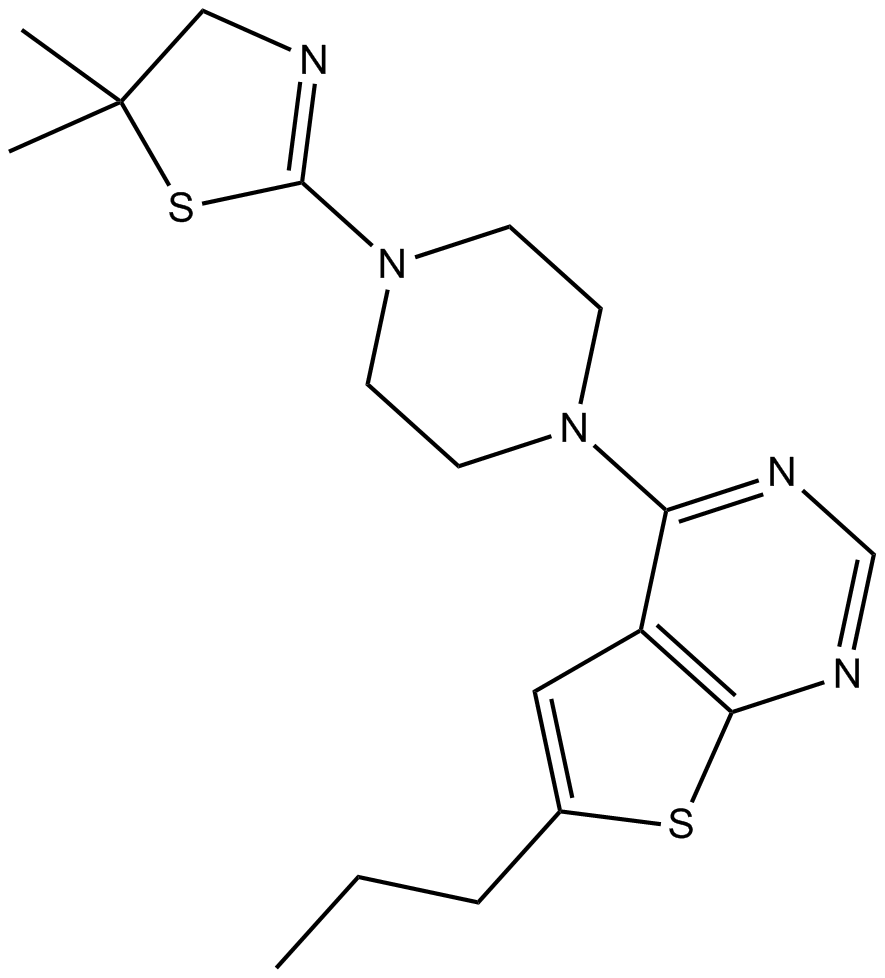
GC11418
MI-2
An inhibitor of menin-MLL fusion protein interactions
-
GC12199
MI-3
An inhibitor of menin-MLL fusion protein interactions
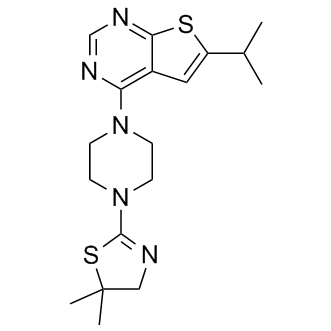
-
GC19246
MI-463
Le MI-463 est une petite molécule inhibitrice hautement puissante et biodisponible par voie orale de l'interaction ménine-mLL.
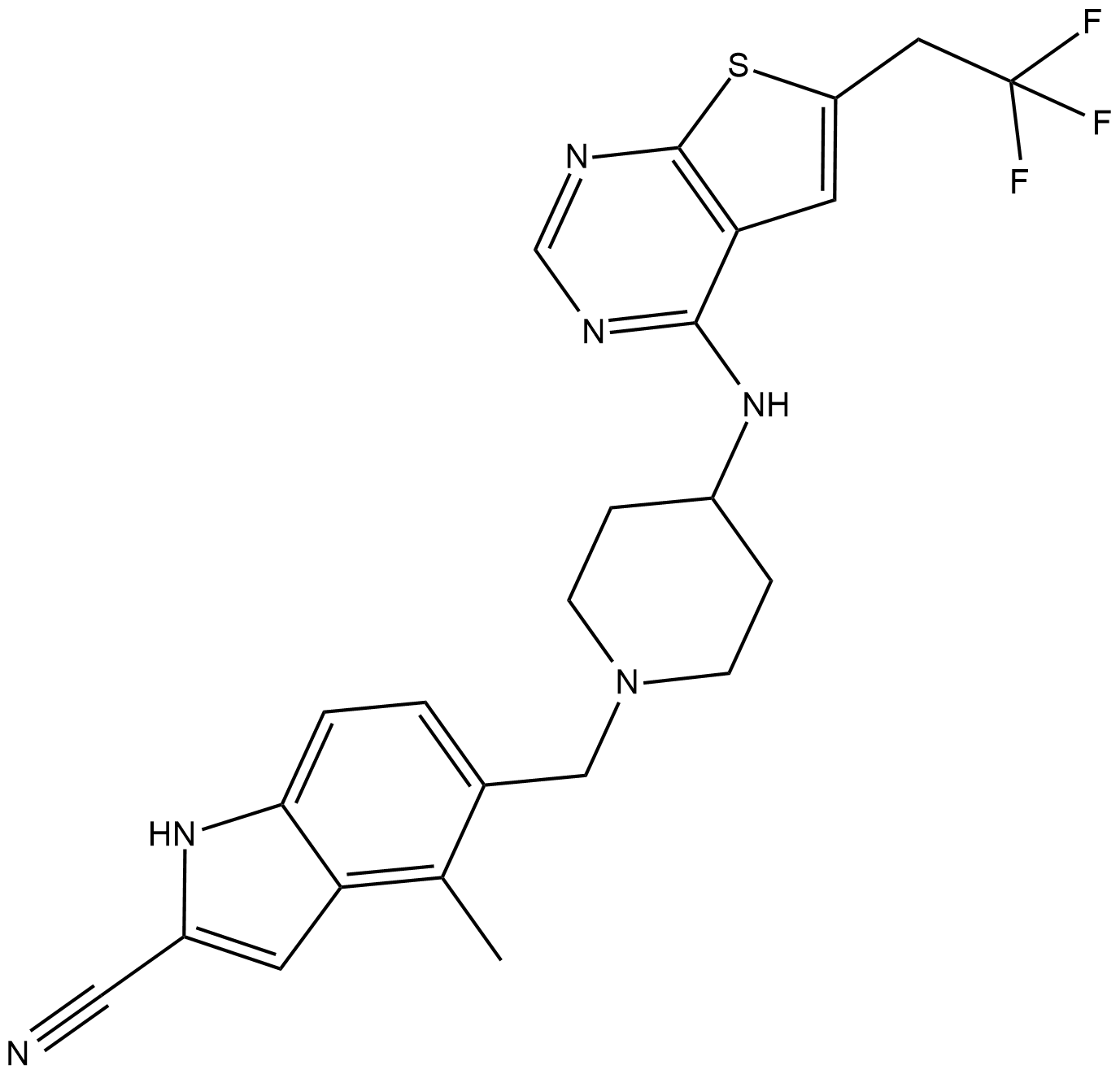
-
GC19247
MI-503
Le MI-503 est une petite molécule inhibitrice hautement puissante et biodisponible par voie orale de l'interaction ménine-mLL.
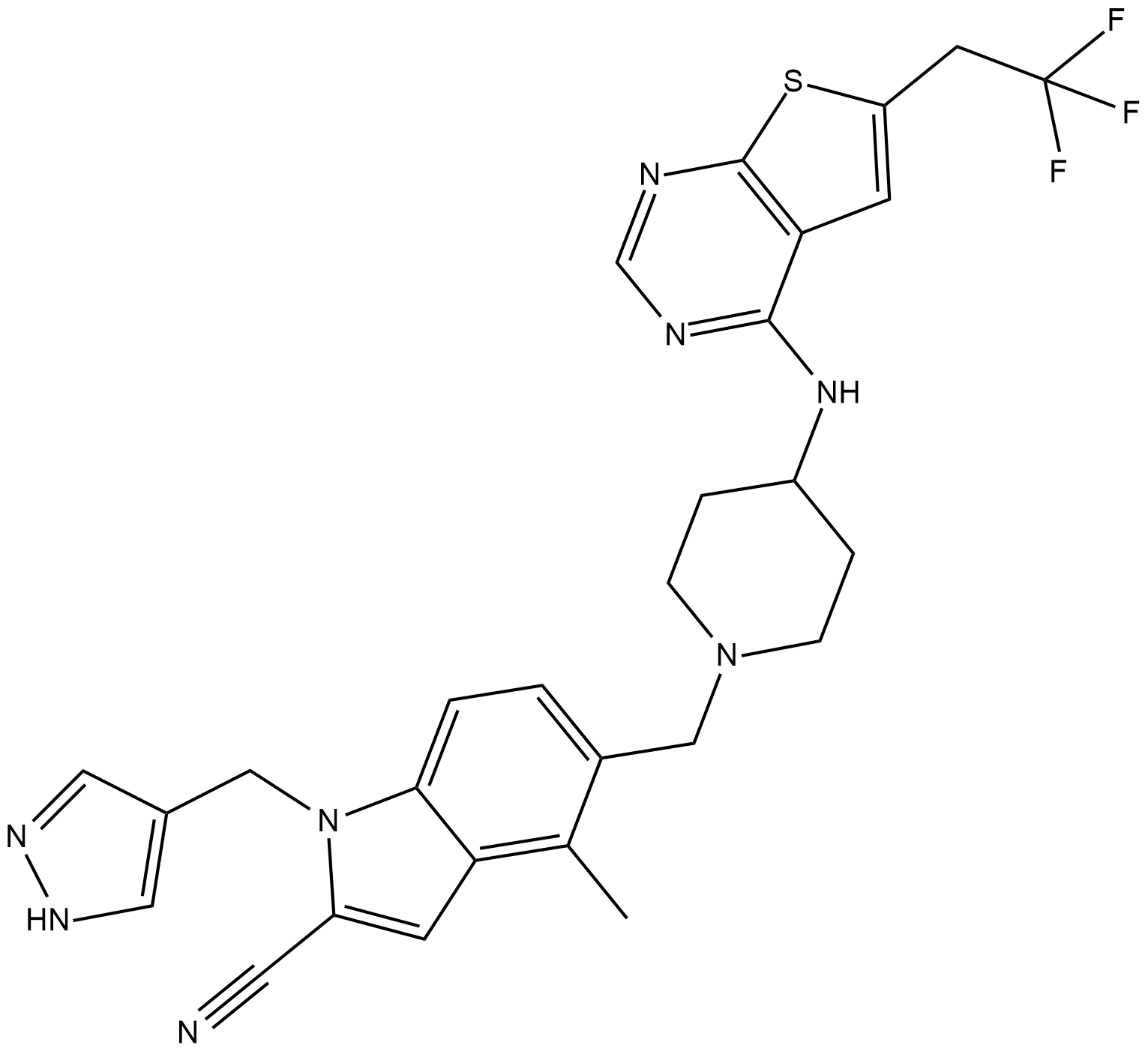
-
GC32798
MI-538
Le MI-538 est un inhibiteur de l'interaction entre la ménine et les protéines de fusion MLL avec une IC50 de 21 nM.
-
GC65581
MIND4-19
MIND4-19 est un puissant inhibiteur de SIRT2 avec une valeur IC50 de 7,0 μM.
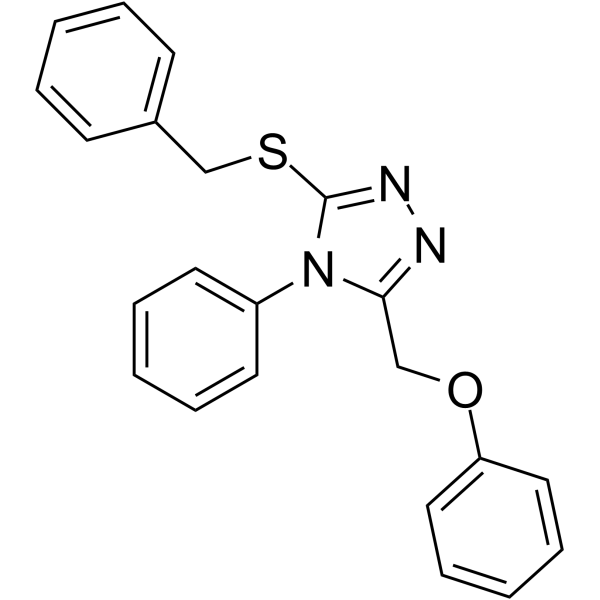
-
GC32929
MIR96-IN-1
MIR96-IN-1 cible le site Drosha dans le précurseur en épingle À cheveux miR-96 (miARN-96, microARN-96), inhibant sa biogenèse, déréprimant les cibles en aval et déclenchant l'apoptose dans les cellules cancéreuses du sein. MIR96-IN-1 se lie aux ARN avec des Kd de 1,3, 9,4, 3,4, 1,3 et 7,4 μM pour ARN1, ARN2, ARN3, ARN4 et ARN5, respectivement.
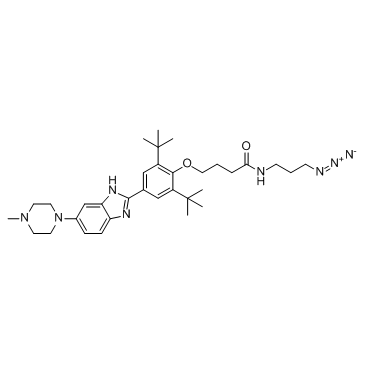
-
GC64487
Miravirsen
Miravirsen (SPC-3649), un oligonucléotide antisens phosphorothioate modifié par un acide nucléique bloqué par β-d-oxy, inhibe la biogenèse de miR-122.

-
GC19248
Mivebresib
Le mivébrésib (ABBV-075) est un inhibiteur de bromodomaine et de domaine extraterminal (BET) puissant et actif par voie orale. Le mivébrésib se lie À BRD4 avec un Ki de 1,5 nM.
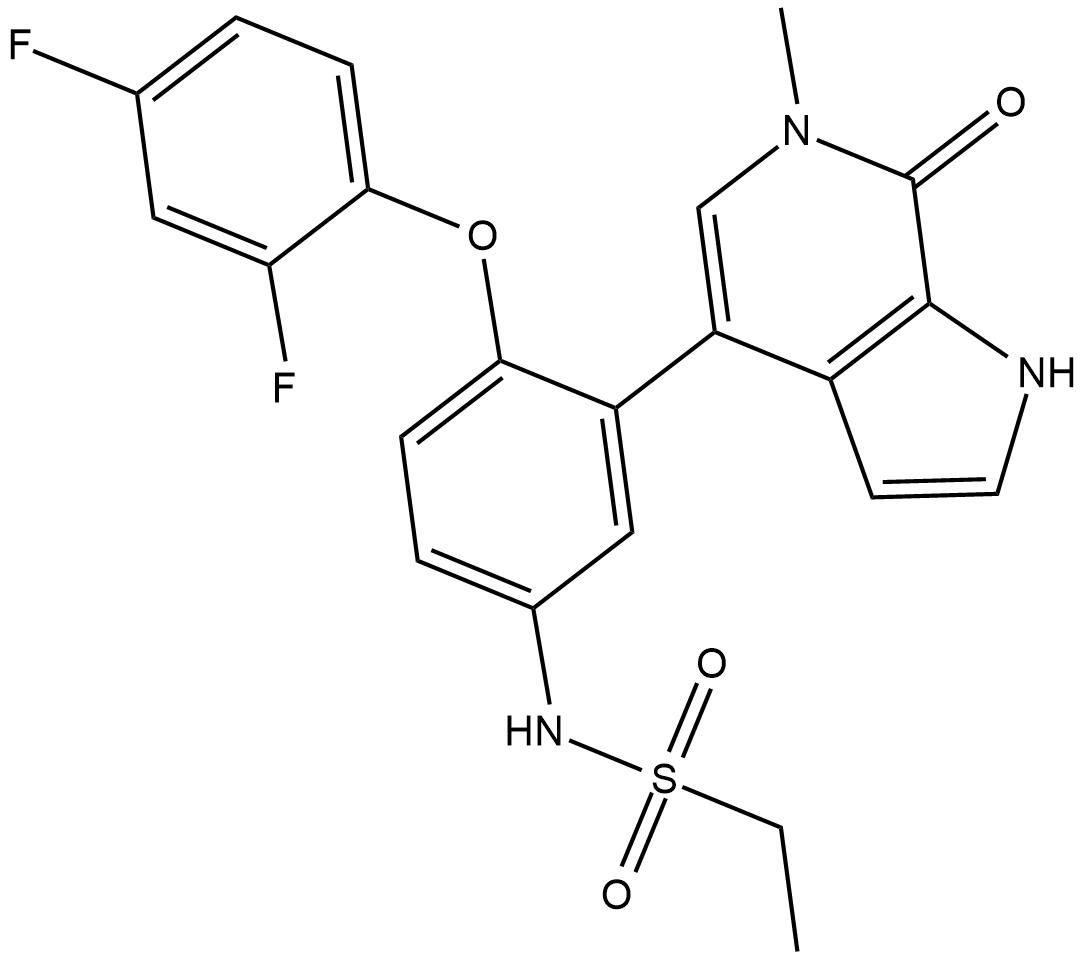
-
GC17802
MK-4827
An orally bioavailable PARP1/2 inhibitor
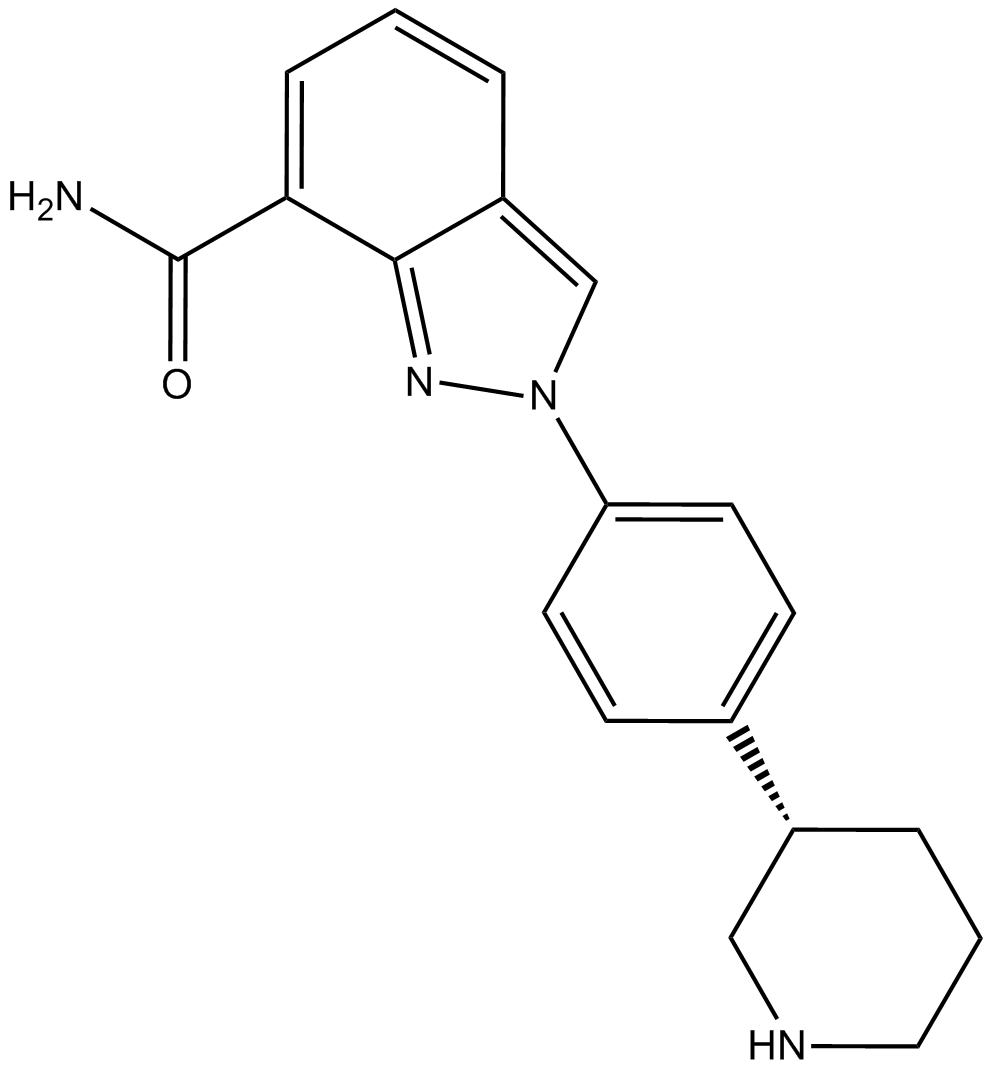
-
GC12756
MK-4827 hydrochloride
Le chlorhydrate de MK-4827 (chlorhydrate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec des CI50 de 3,8 et 2,1 nM, respectivement. Le chlorhydrate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.
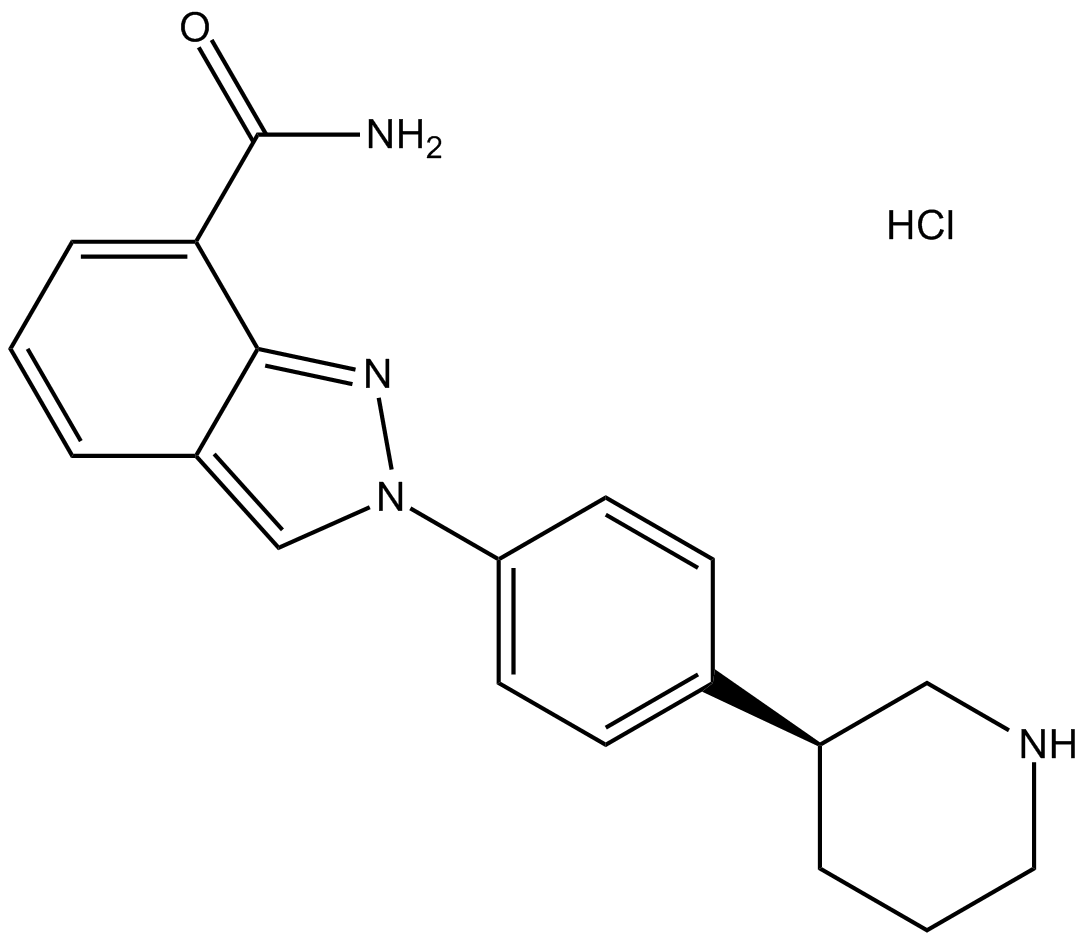
-
GC17052
MK-4827 Racemate
selective inhibitor of PARP1/PARP2
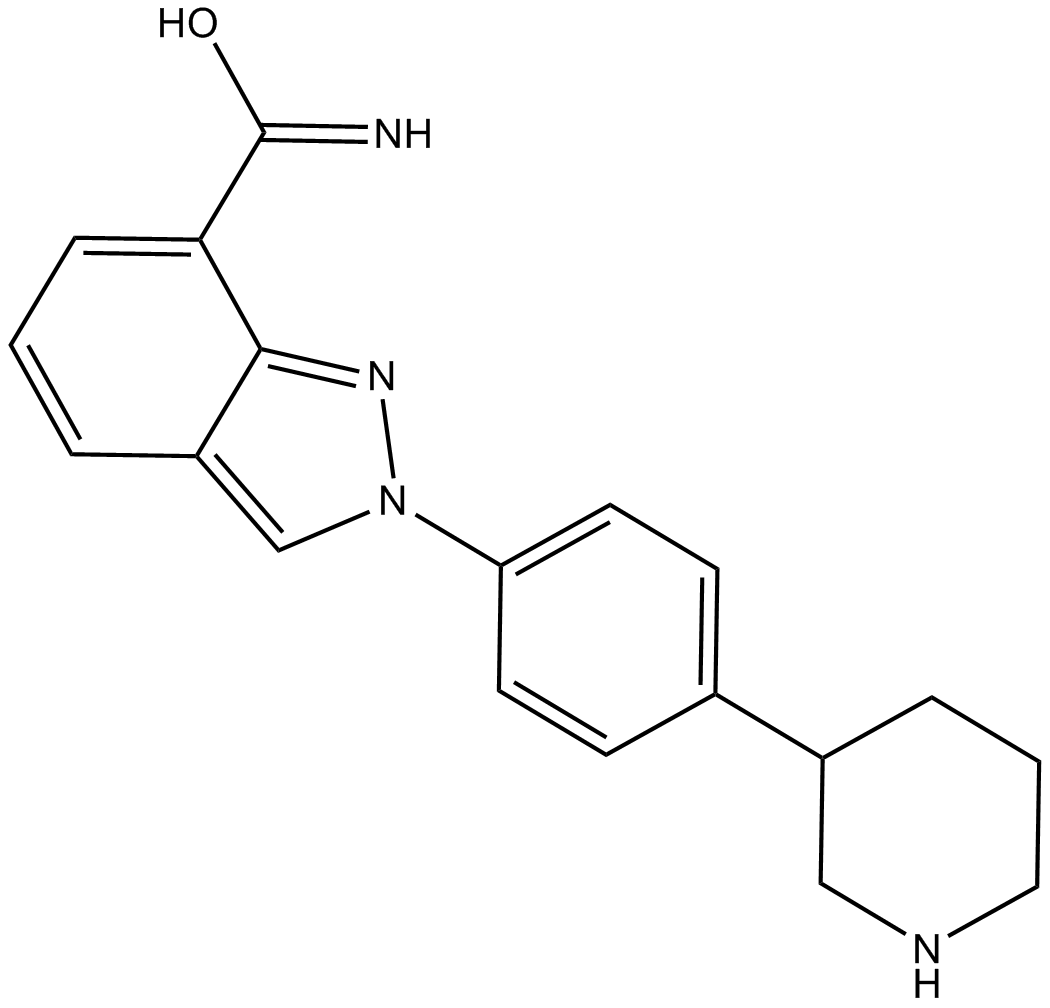
-
GC11537
MK-4827 tosylate
Le tosylate de MK-4827 (tosylate de MK-4827) est un inhibiteur PARP1 et PARP2 très puissant et biodisponible par voie orale avec une IC50 de 3,8 et 2,1 nM, respectivement. Le tosylate de MK-4827 conduit À l'inhibition de la réparation des dommages À l'ADN, active l'apoptose et présente une activité anti-tumorale.
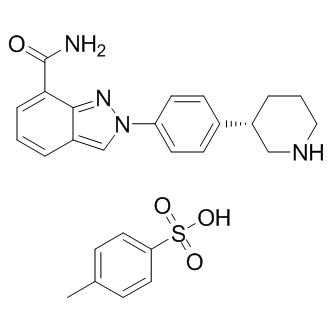
-
GC17162
MK-5108 (VX-689)
A potent inhibitor of Aurora kinases
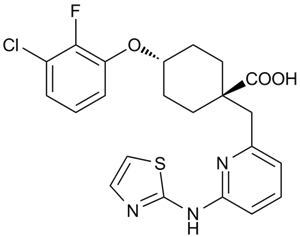
-
GC19251
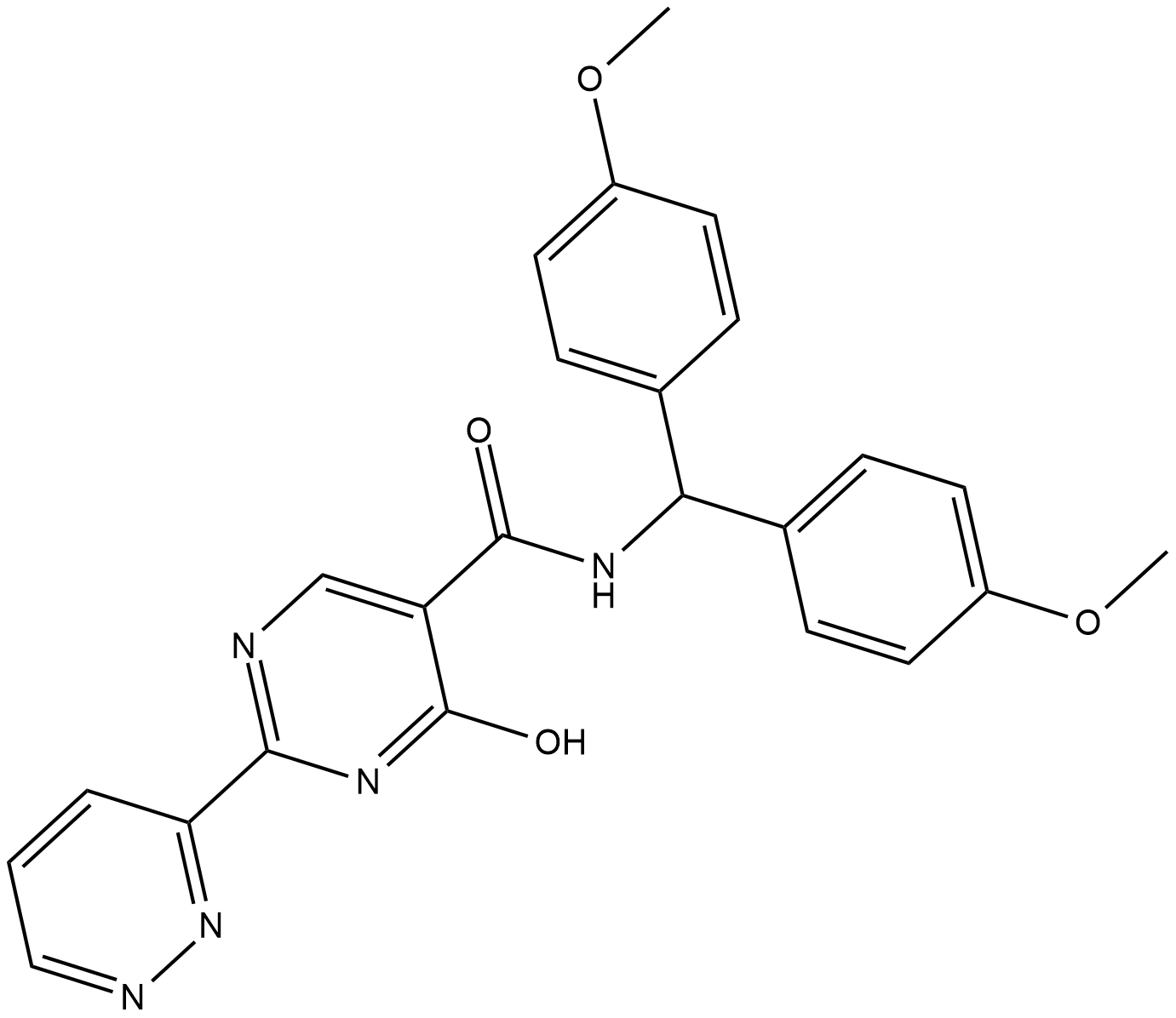
MK-8617
Le MK-8617 est un pan-inhibiteur actif par voie orale du facteur prolyl hydroxylase 1-3 induit par l'hypoxie (HIF PHD1-3) avec une IC50 de 1 nM pour PHD2.
-
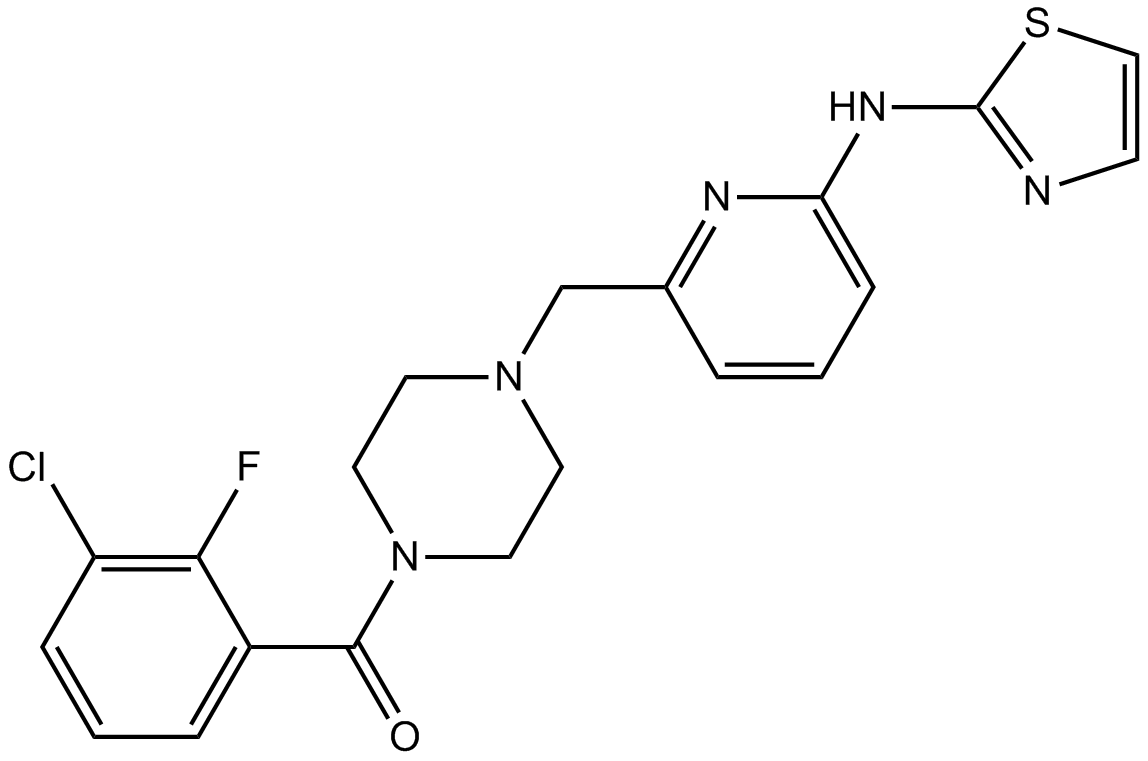
GC10442
MK-8745
Le MK-8745 est un inhibiteur de la kinase Aurora A avec une IC50 de 0,6 nM.
-
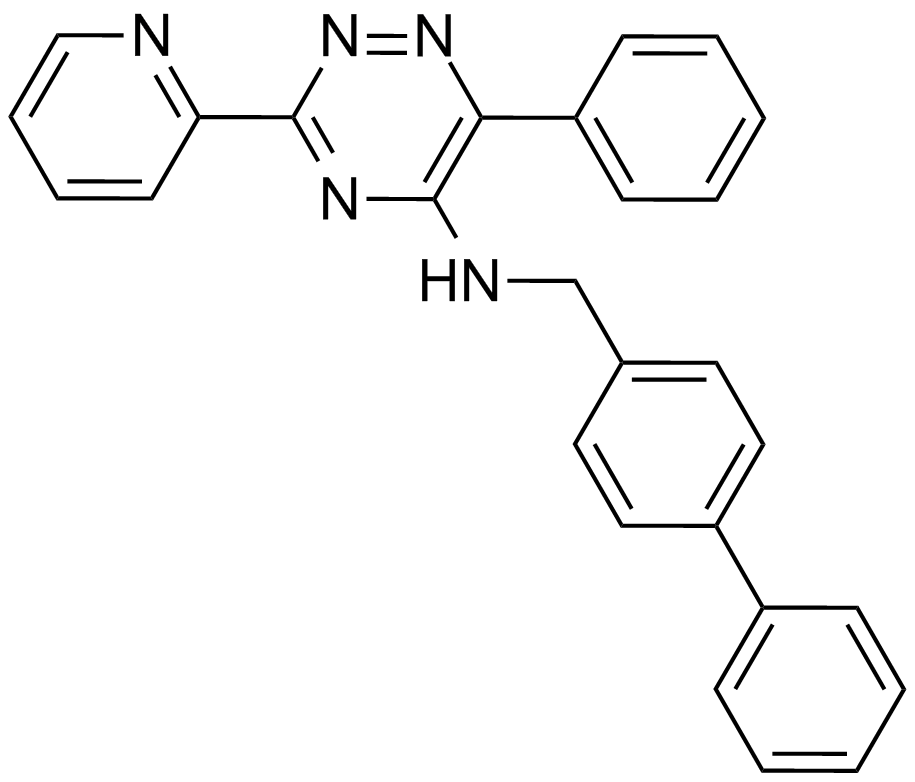
GC14319
ML 228
A HIF pathway activator
-
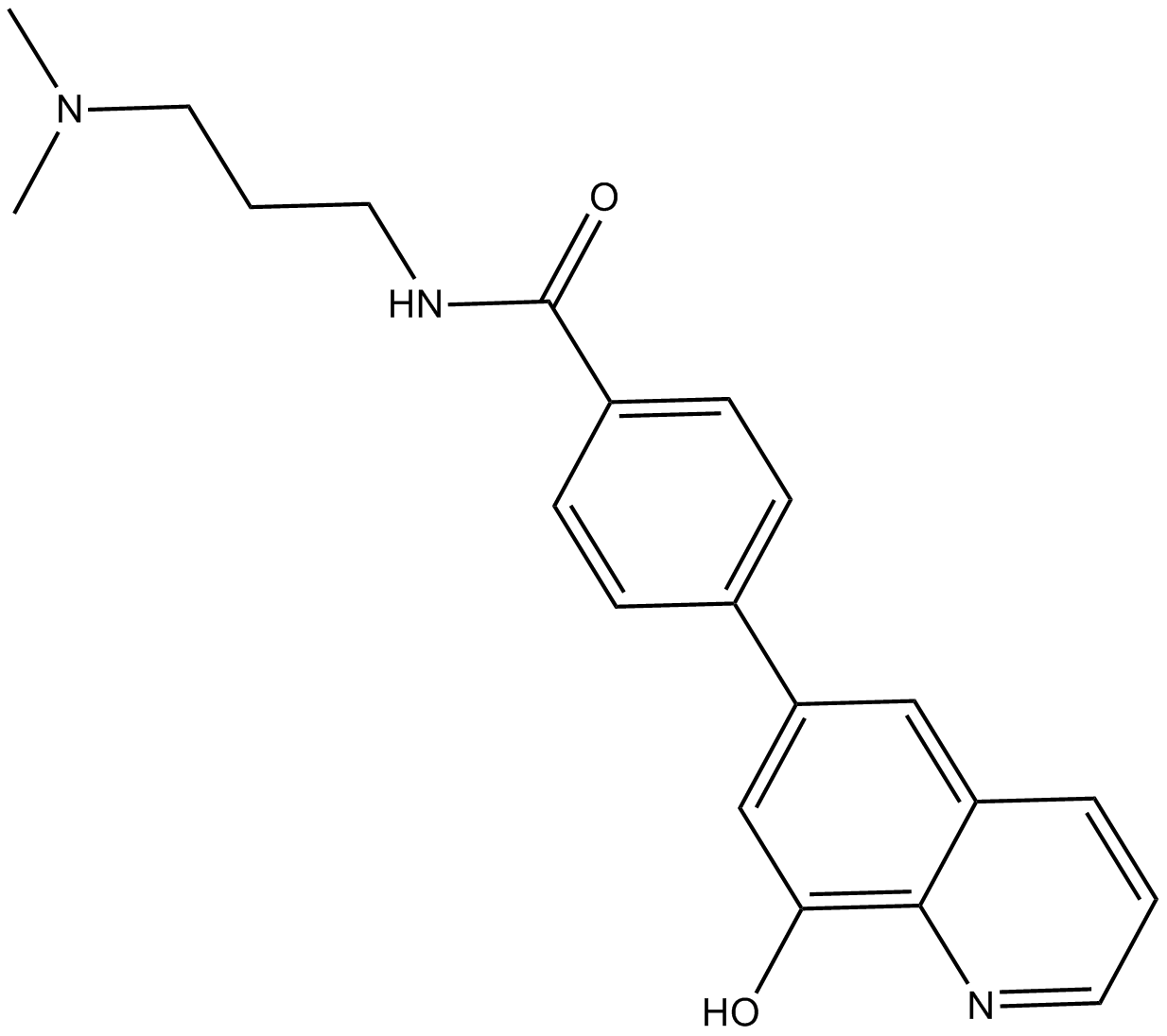
GC12557
ML324
ML324 est un puissant inhibiteur de la déméthylase JMJD2 avec une activité antivirale. ML324 présente également une inhibition de l'histone déméthylase KDM4B, avec une IC50 de 4,9 μM. ML324 a une activité antivirale puissante contre l'infection par le virus de l'herpès simplex (HSV) et le cytomégalovirus humain (hCMV) via l'inhibition de l'expression du gène viral IE.
-
GC12208
MLN8054
An inhibitor of Aurora A kinase
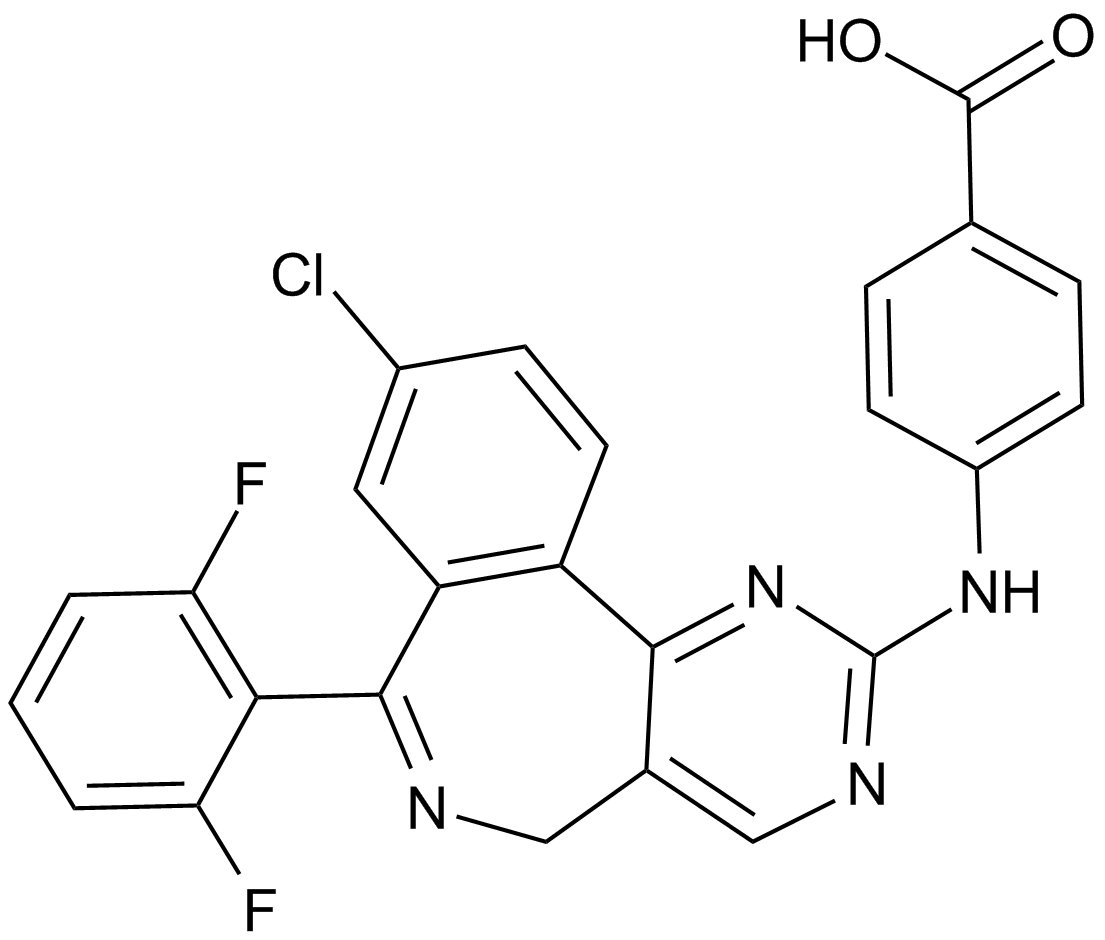
-
GC12690
MLN8237 (Alisertib)
Un inhibiteur de la kinase Aurora A
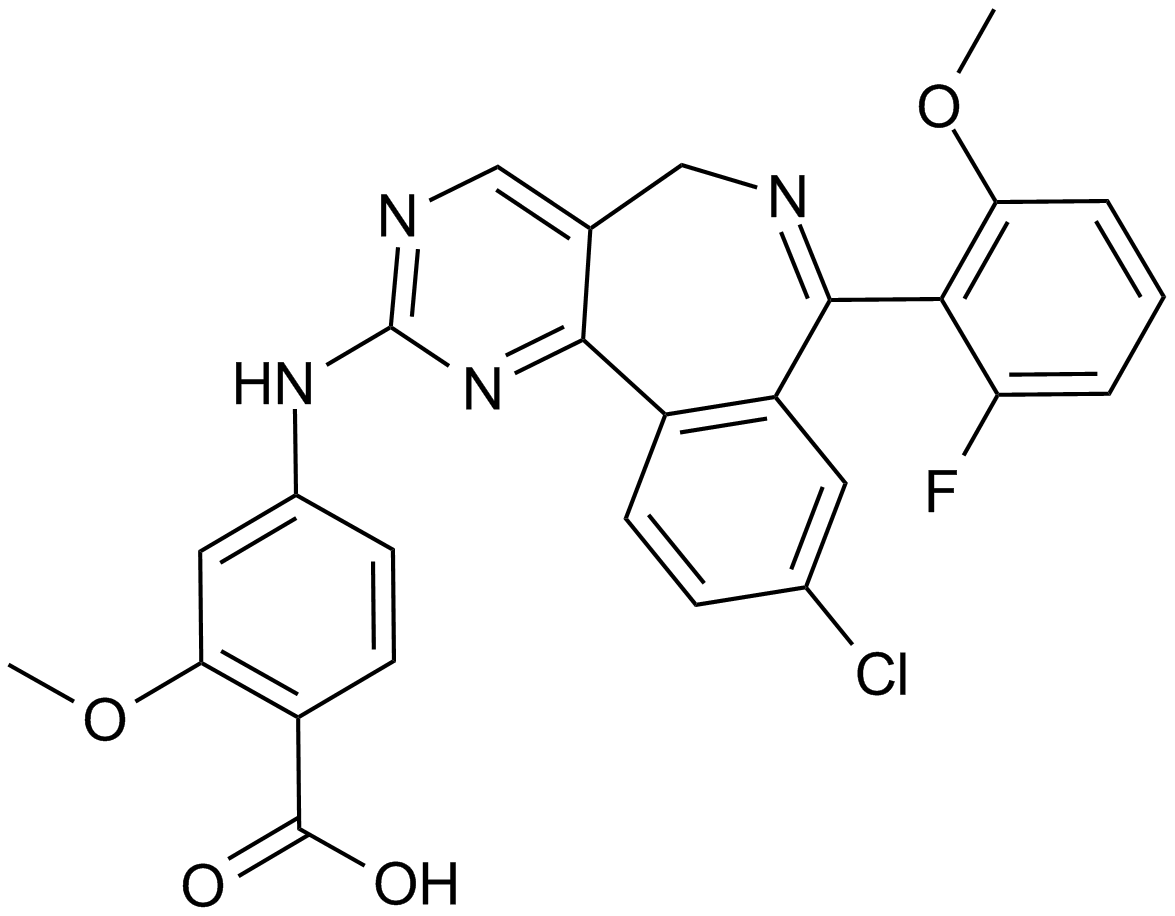
-
GC14652
MM-102
MM-102 (HMTase Inhibitor IX) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki200 fois plus puissant que le peptide ARA.
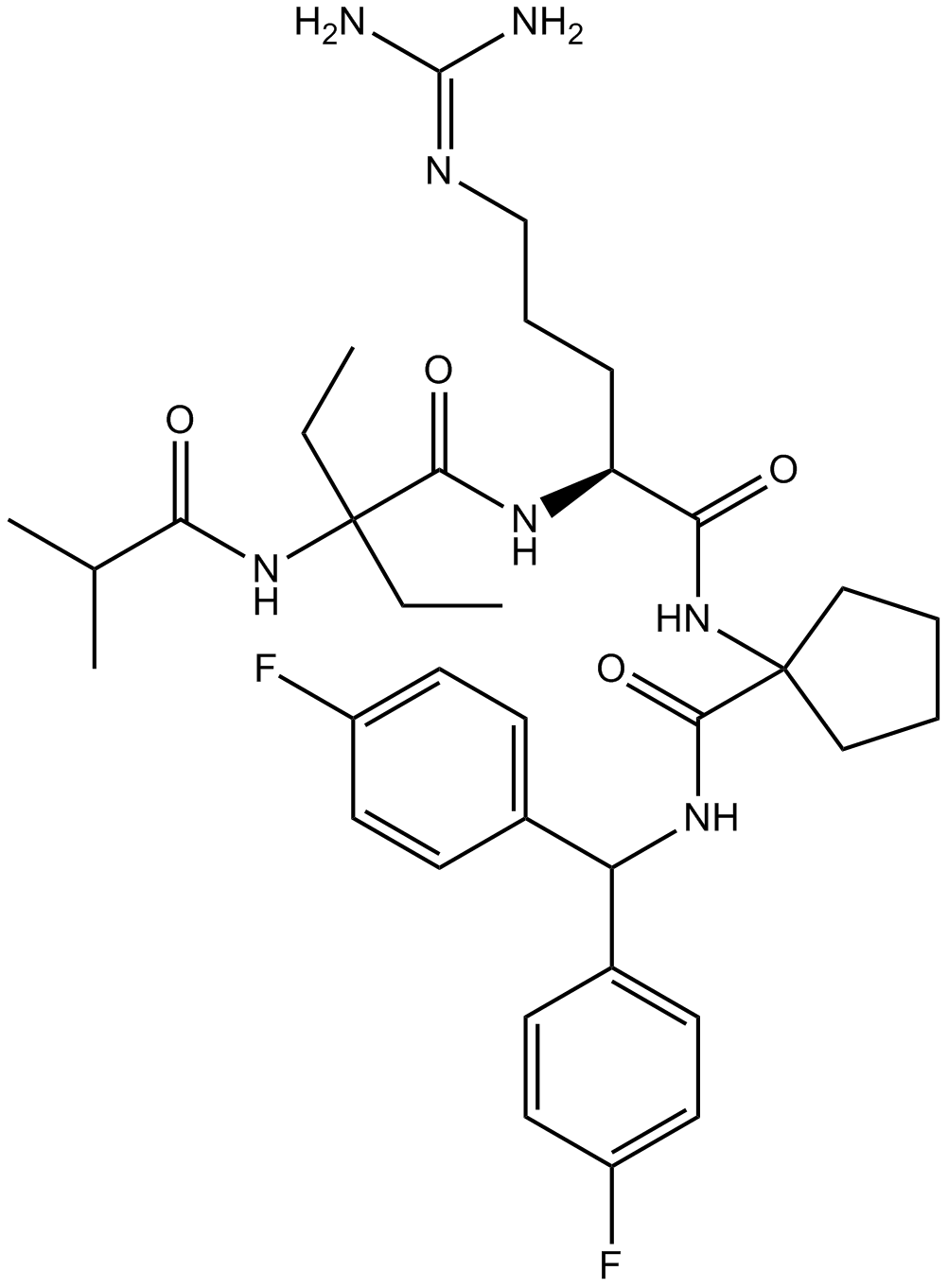
-
GC36632
MM-102 TFA
Le MM-102 TFA (HMTase Inhibitor IX TFA) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki estimé 200 fois plus puissant que le peptide ARA.
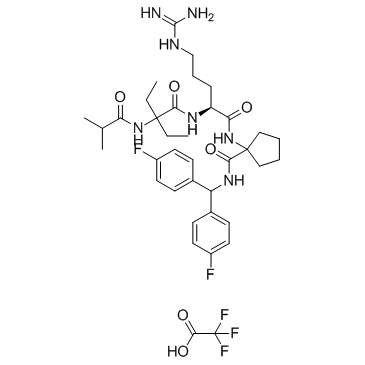
-
GC67758
MM-401 TFA
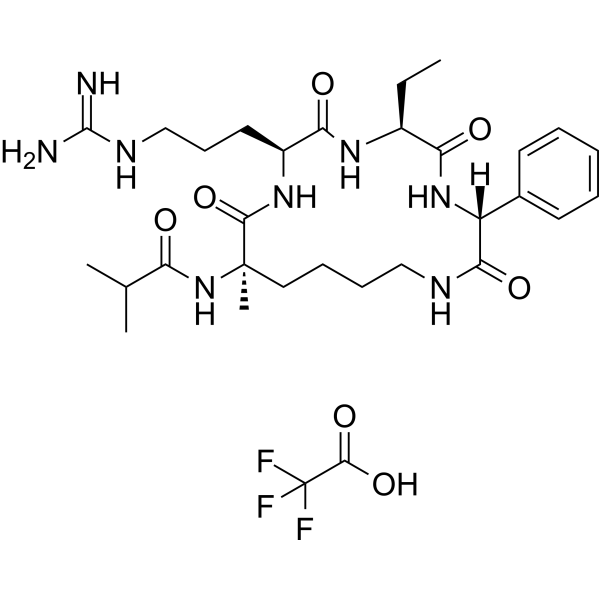
-
GC33278
MM-589
Le MM-589 est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.
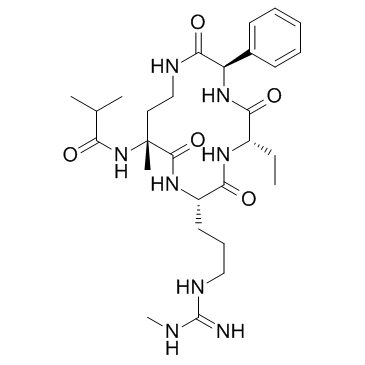
-
GC65037
MM-589 TFA
Le MM-589 TFA est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.
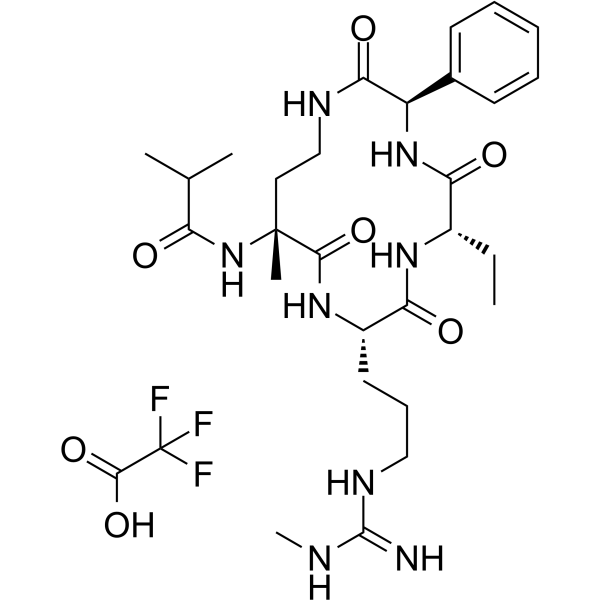
-
GC16914
MN 64
MN 64 est un puissant inhibiteur de la tankyrase 1, avec des CI50 de 6 nM, 72 nM, 19,1 μM et 39,4 μM pour TNKS1, TNKS2, ARTD1 et ARTD2, respectivement.
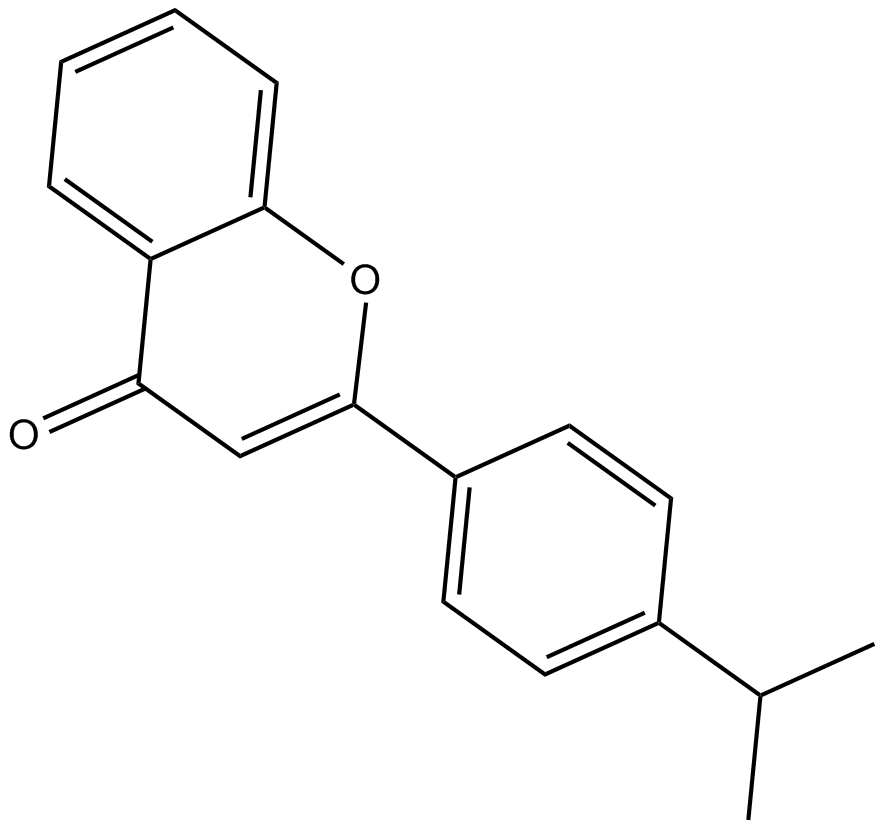
-
GC13055
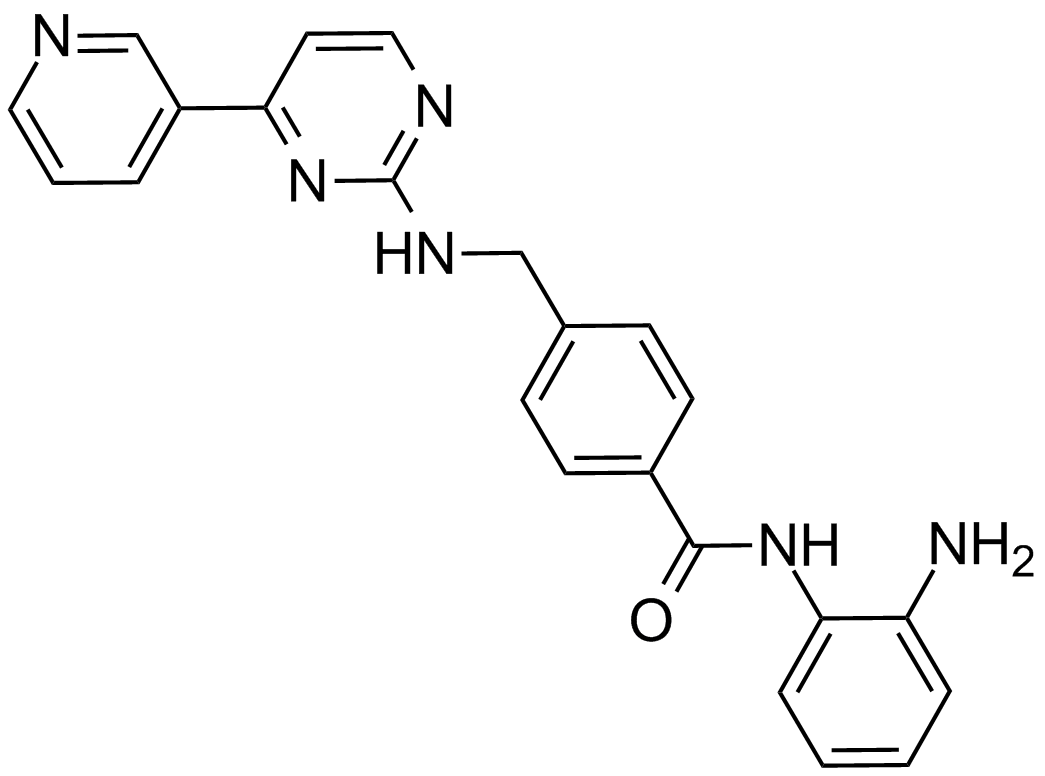
Mocetinostat (MGCD0103, MG0103)
An orally available HDAC inhibitor
-
GC34675
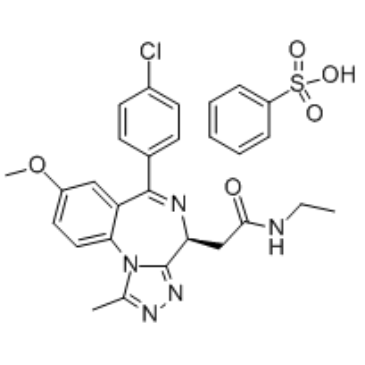
Molibresib besylate
Le bésylate de molibresib (GSK 525762C; bésylate I-BET 762) est un inhibiteur du bromodomaine BET avec une IC50 de 32,5 À 42,5nM.
-
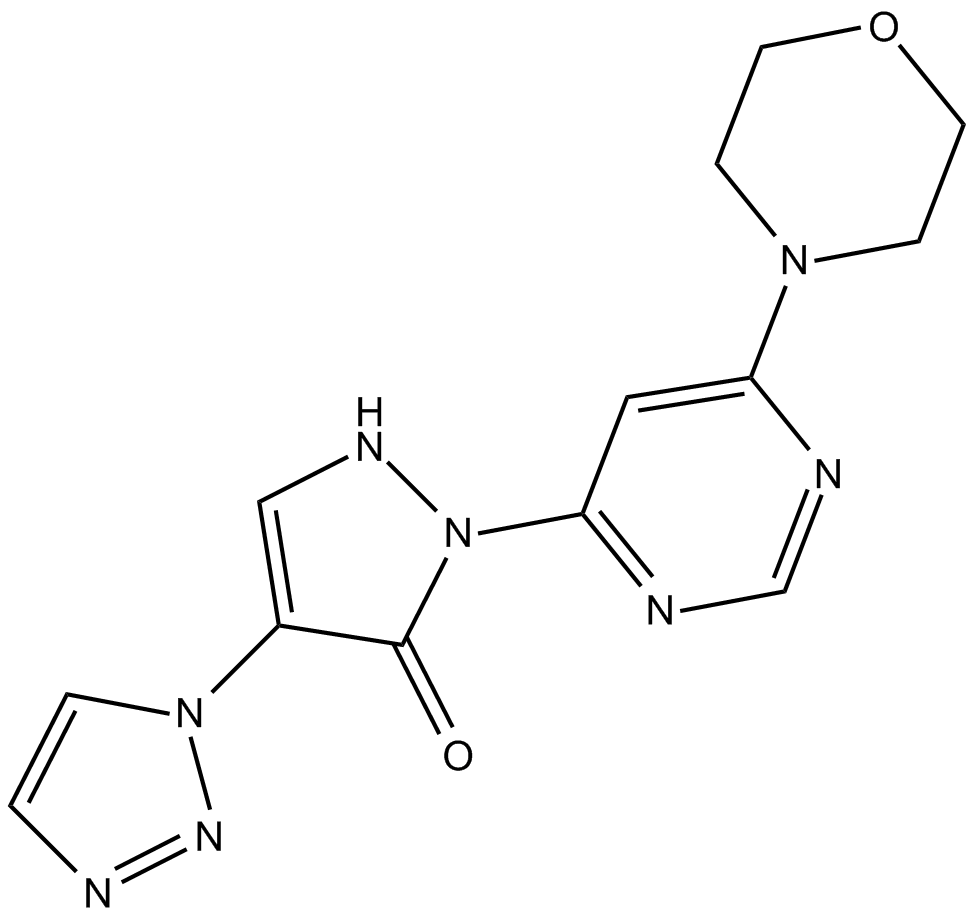
GC10046
Molidustat (BAY85-3934)
Molidustat (BAY85-3934) (BAY 85-3934) est un nouvel inhibiteur du facteur prolyl hydroxylase inductible par l'hypoxie (HIF-PH) avec des valeurs moyennes d'IC50 de 480 nM pour PHD1, 280 nM pour PHD2 et 450 nM pour PHD3.
-
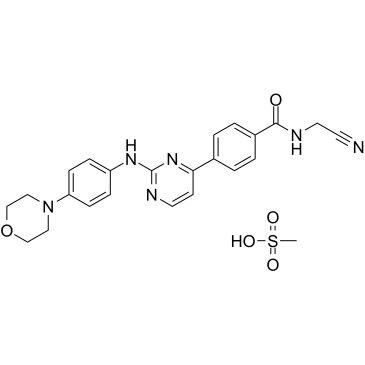
GC36646
Momelotinib Mesylate
Le mésylate de momelotinib (CYT387 Mesylate) est un inhibiteur compétitif de l'ATP de JAK1/JAK2 avec une IC50 de 11 nM/18 nM, une sélectivité d'environ 10 fois par rapport À JAK3.
-
GC62406
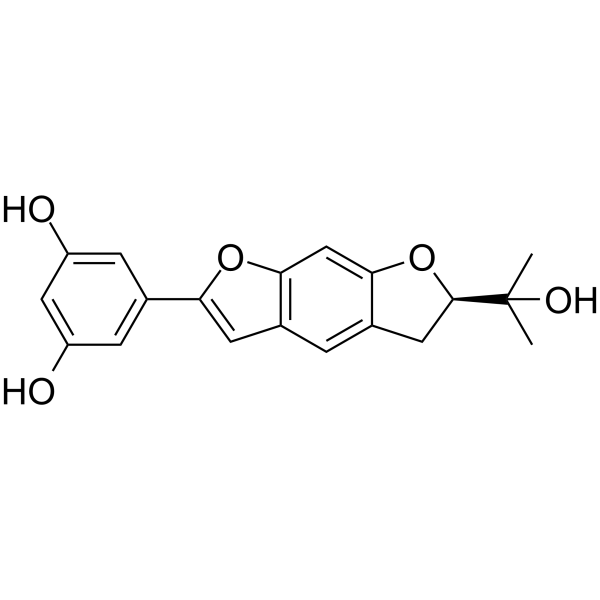
Moracin O
Moracin O est un 2-arylbenzofurane isolé du Mori Cortex Radicis.
-
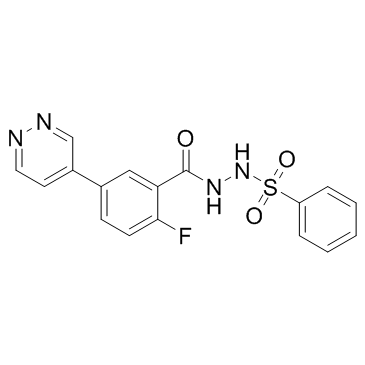
GC33371
MOZ-IN-2
MOZ-IN-2 est un inhibiteur de la protéine MOZ, membre des histones acétyltransférases, avec une IC50 de 125 μM.
-
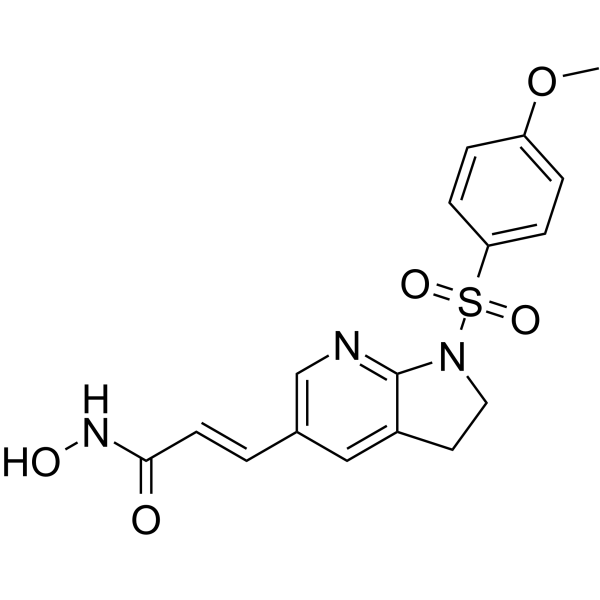
GC64729
MPT0B390
MPT0B390 est un dérivé À base d'arylsulfonamide doté d'une puissante capacité inhibitrice d'HDAC. MPT0B390, inducteur de TIMP3, inhibe la croissance tumorale, les métastases et l'angiogenèse. MPT0B390 montre une activité antiproliférative contre la lignée cellulaire de cancer du cÔlon humain HCT116 avec le GI50 de 0,03 μM.
-
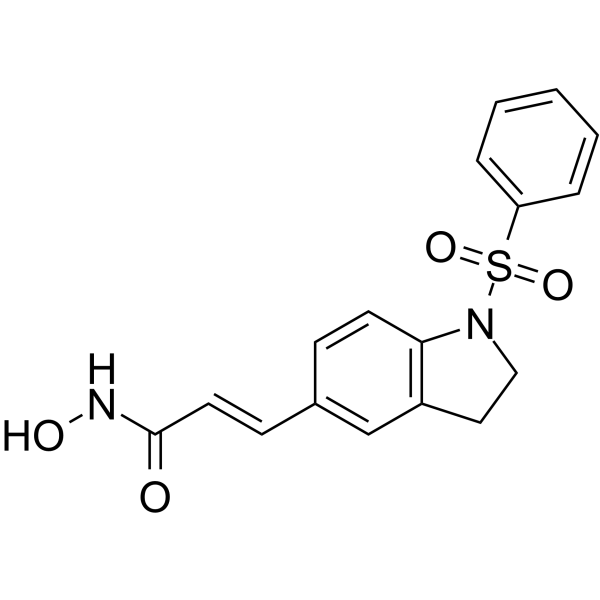
GC65965
MPT0E028
MPT0E028 est un inhibiteur HDAC actif et sélectif par voie orale avec des IC50 de 53,0 nM, 106,2 nM, 29,5 nM pour HDAC1, HDAC2 et HDAC6, respectivement. MPT0E028 réduit la viabilité des lymphomes À cellules B en induisant l'apoptose et possède une puissante capacité de ciblage direct Akt et réduit la phosphorylation d'Akt dans le lymphome À cellules B. MPT0E028 a une bonne activité anticancéreuse.
-
GC62308
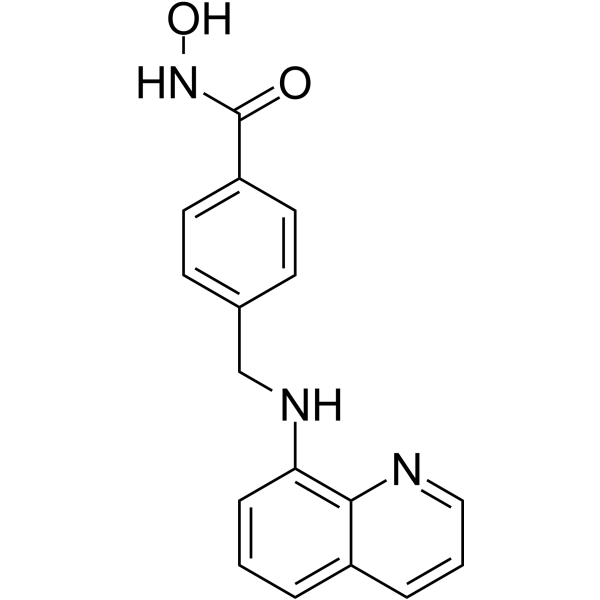
MPT0G211
MPT0G211 est un puissant inhibiteur HDAC6 actif par voie orale et sélectif (IC50 = 0,291nM).
-
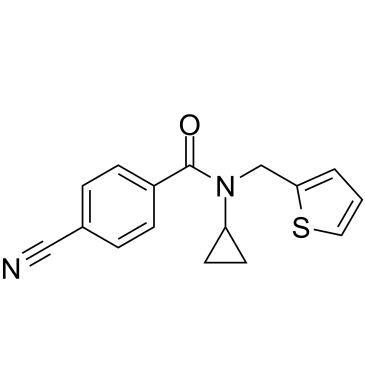
GC61468
MR837
MR837 est un inhibiteur de NSD2-PWWP1. MR837 peut se lier À la protéine 2 du domaine SET de liaison au récepteur nucléaire humain (domaine PWWP).
-
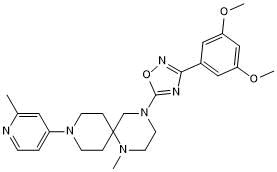
GC50576
MRK 740
Potent PRDM9 inhibitor
-
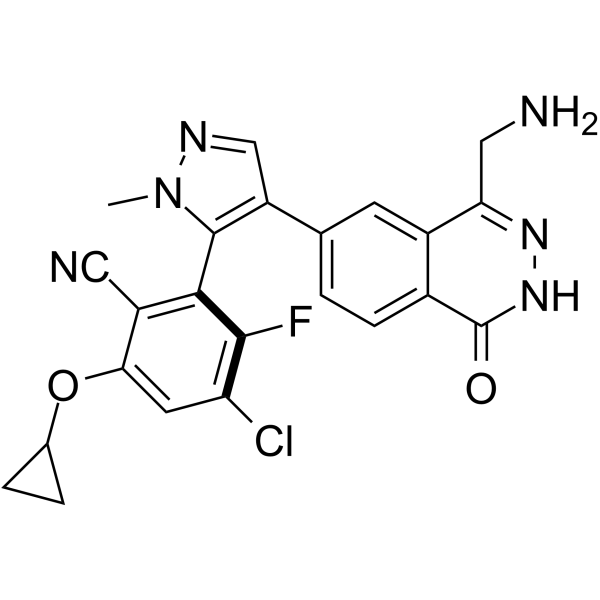
GC63558
MRTX-1719
MRTX-1719 est un inhibiteur sélectif de première classe puissant du complexe PRMT5/MTA, avec une IC50 de moins de 10 nM dans les cellules PRMT5/MTA MTAPDEL SDMA.
-
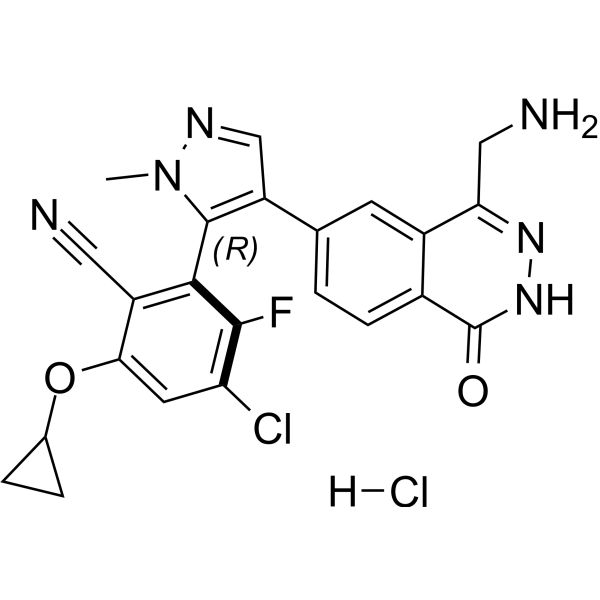
GC69499
MRTX-1719 hydrochloride
MRTX-1719 hydrochloride est un inhibiteur efficace, novateur et sélectif du complexe PRMT5/MTA. Son IC50 pour les cellules MTAPDEL SDMA de PRMT5/MTA est inférieur à 10 nM.
-
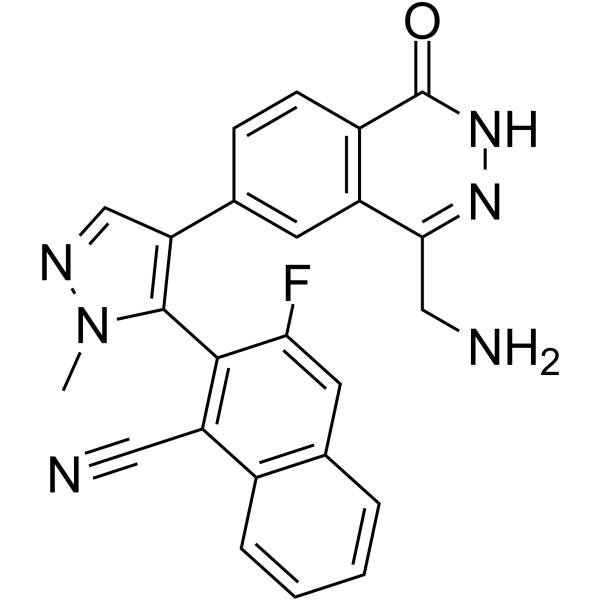
GC62715
MRTX9768
MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.
-
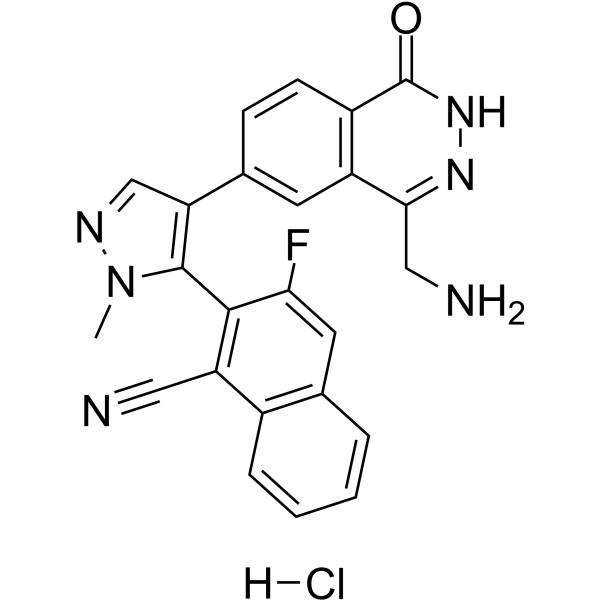
GC63080
MRTX9768 hydrochloride
Le chlorhydrate de MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.
-
GC40791
MS-1020
Le MS-1020 est un inhibiteur de JAK3 puissant et compétitif pour l'ATP. Le MS-1020 inhibe la signalisation JAK3/STAT et induit l'apoptose. MS-1020 favorise la mort cellulaire. Le MS-1020 diminue l'expression des niveaux de STAT3 phosphorylé par la tyrosine. MS-1020 a le potentiel pour la recherche de cancers hébergeant une signalisation JAK3 aberrante.
-
GC10099
MS023
MS023 est un inhibiteur puissant, sélectif et actif sur les cellules des protéines arginine méthyltransférases de type I (PRMTs).
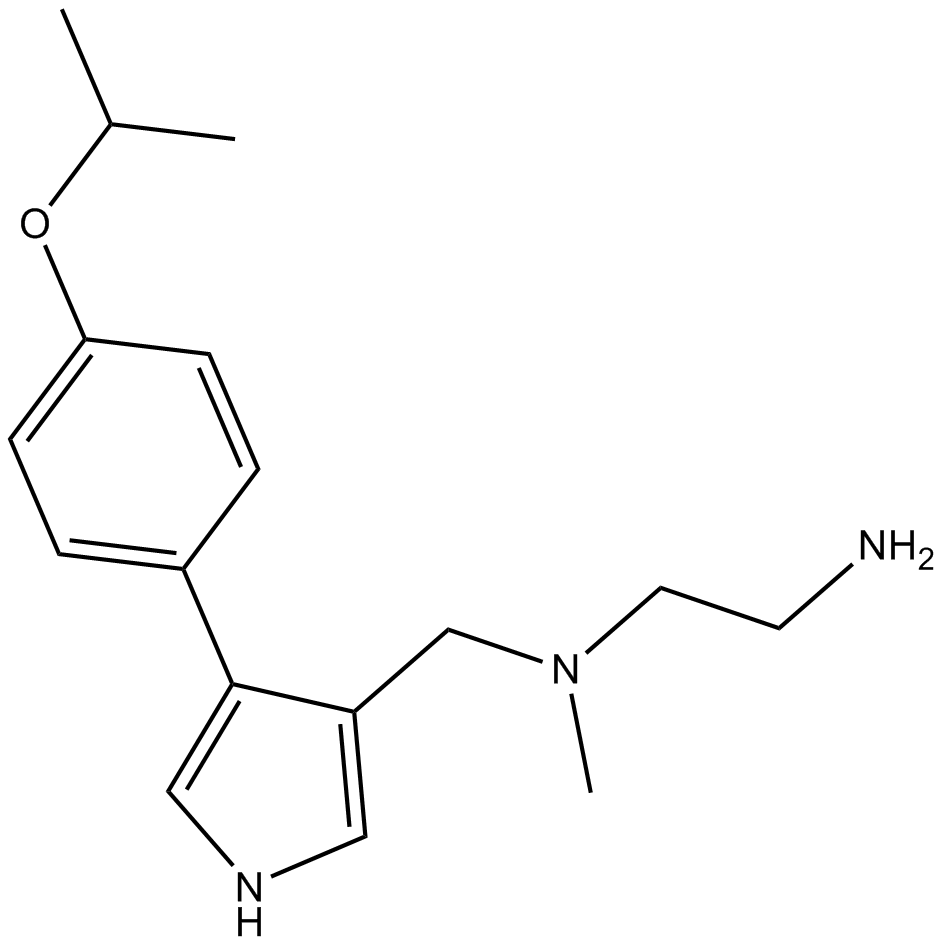
-
GC16432
MS023 (hydrochloride)
type I PRMTs inhibitor
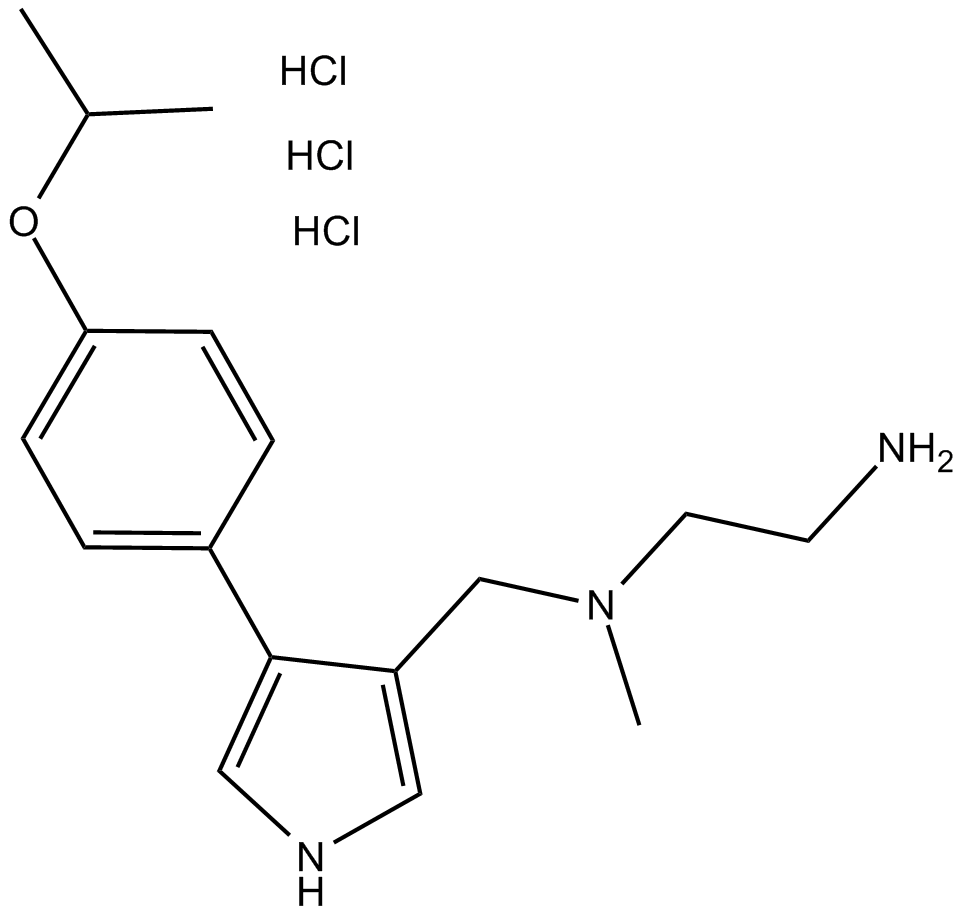
-
GC36655
MS023 dihydrochloride
Le dichlorhydrate de MS023 est un inhibiteur puissant, sélectif et actif sur les cellules de l'inhibiteur de la protéine arginine méthyltransférase (PRMT) humaine de type I, avec des IC50 de 30, 119, 83, 4 et 5 nM pour PRMT1, PRMT3, PRMT4, PRMT6 et PRMT8, respectivement.
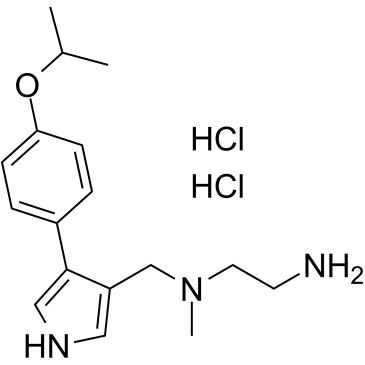
-
GC36656
MS049
MS049 est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 n'est pas toxique et n'affecte pas la croissance des cellules HEK293.
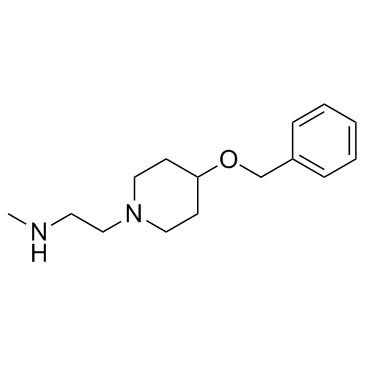
-
GC14240
MS049 (hydrochloride)
MS049 (chlorhydrate) est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 (chlorhydrate) réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 (chlorhydrate) n'est pas toxique et n'affecte pas la croissance des cellules HEK293.
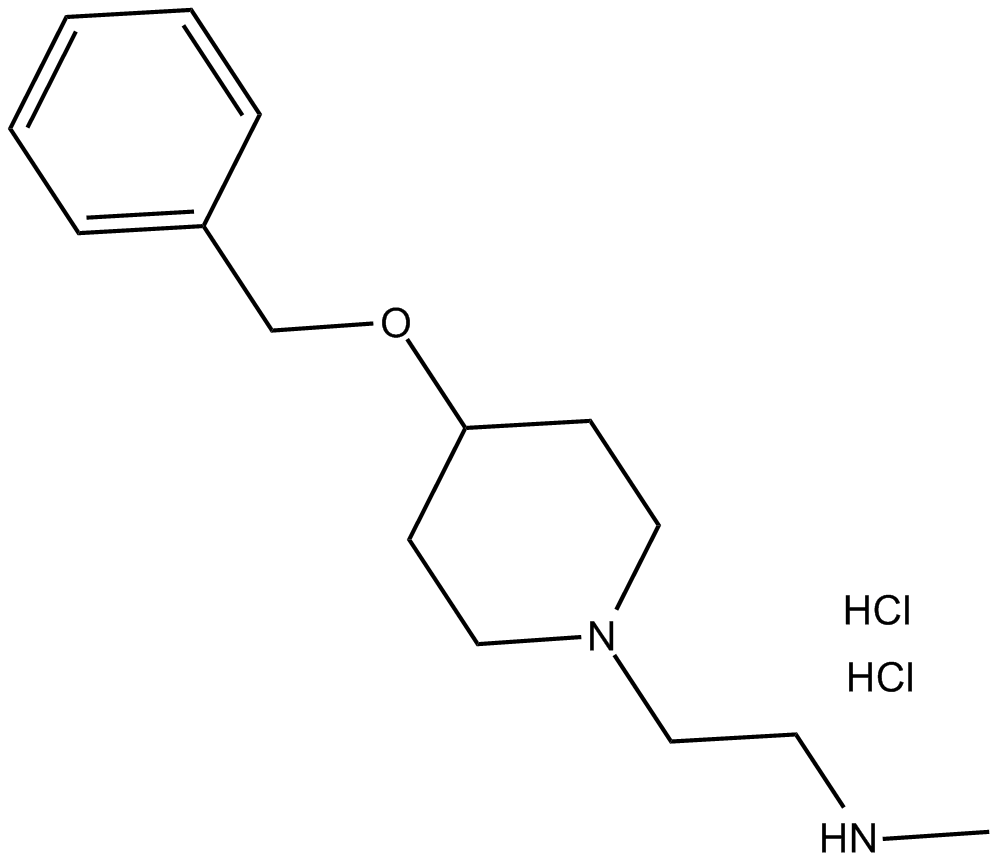
-
GC36657
MS31
MS31 est un puissant inhibiteur de spindline 1 (SPIN1) de la protéine lecteur de méthyllysine de type fragment, hautement affin et sélectif. MS31 inhibe puissamment les interactions entre SPIN1 et H3K4me3 (IC50 = 77 nM, AlphaLISA ; 243 nM, FP). MS31 se lie sélectivement au domaine Tudor II de SPIN1 (Kd = 91 nM). MS31 inhibe puissamment la liaison des peptides contenant de la triméthyllysine À SPIN1. MS31 n'est pas toxique pour les cellules non tumorigènes.
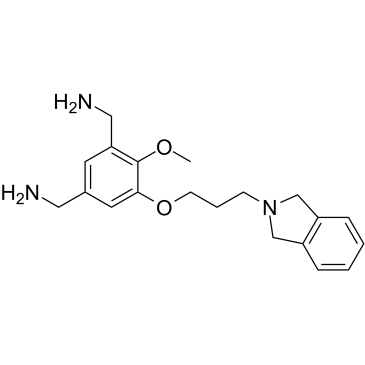
-
GC39252
MS31 trihydrochloride
Le trichlorhydrate de MS31 est un puissant inhibiteur de spindline 1 (SPIN1) de la protéine lecteur de méthyllysine, hautement affin et sélectif. Le trichlorhydrate de MS31 inhibe puissamment les interactions entre SPIN1 et H3K4me3 (IC50 = 77 nM, AlphaLISA ; 243 nM, FP). Le trichlorhydrate de MS31 se lie sélectivement au domaine Tudor II de SPIN1 (Kd = 91 nM). Le trichlorhydrate de MS31 inhibe puissamment la liaison des peptides contenant de la triméthyllysine À SPIN1 et n'est pas toxique pour les cellules non tumorigènes.
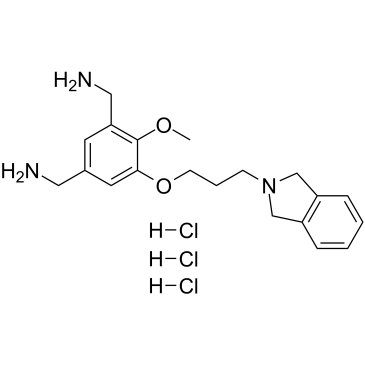
-
GC44250
MS351
MS351 is an antagonist of chromobox 7 (CBX7) that acts by binding the CBX7 chromodomain.
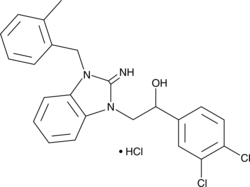
-
GC12630
MS37452
MS37452 est un puissant inhibiteur de la liaison du chromodomaine CBX7 À H3K27me3, avec un Kd de 27,7 μM. MS37452 peut déréprimer la transcription du gène cible du complexe répressif polycomb p16/CDKN2A en déplaÇant la liaison de CBX7 au locus INK4A/ARF dans les cellules cancéreuses de la prostate.
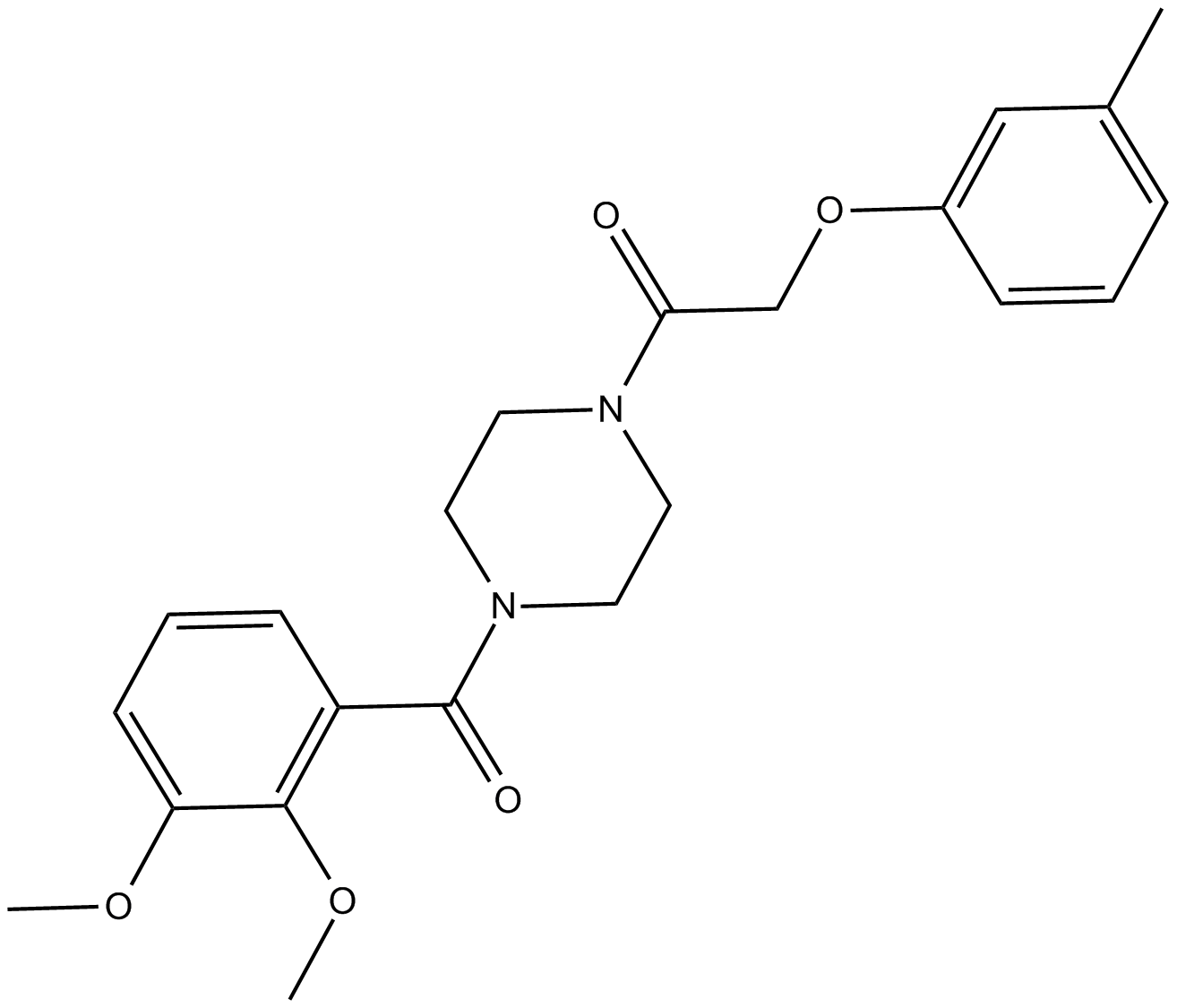
-
GC64999
MS402
MS402 est un inhibiteur BET BrD sélectif de BD1 avec Kis de 77 nM, 718 nM, 110 nM, 200 nM, 83 nM et 240 nM pour BRD4(BD1), BRD4(BD2), BRD3(BD1), BRD3(BD2) , BRD2(BD1) et BRD2(BD2), respectivement.

-
GC31881
MS417 (GTPL7512)
MS417 (GTPL7512) est un inhibiteur sélectif de BRD4 spécifique À BET, se lie À BRD4-BD1 et BRD4-BD2 avec des IC50 de 30, 46 nM et des Kd de 36,1, 25,4 nM, respectivement, avec une faible sélectivité À CBP BRD (IC50, 32,7 μ ;M).
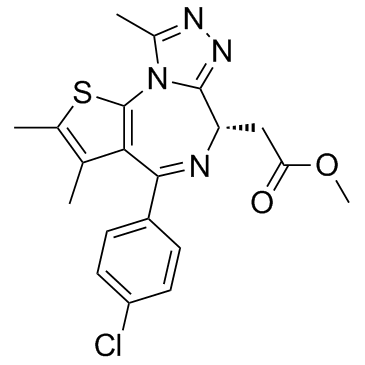
-
GC13148
MS436
MS436 est une nouvelle classe d'inhibiteurs de bromodomaine, présente une affinité puissante d'un Ki = 30-50 nM estimé pour le BRD4 BrD1 et une sélectivité de 10 fois par rapport au BrD2.
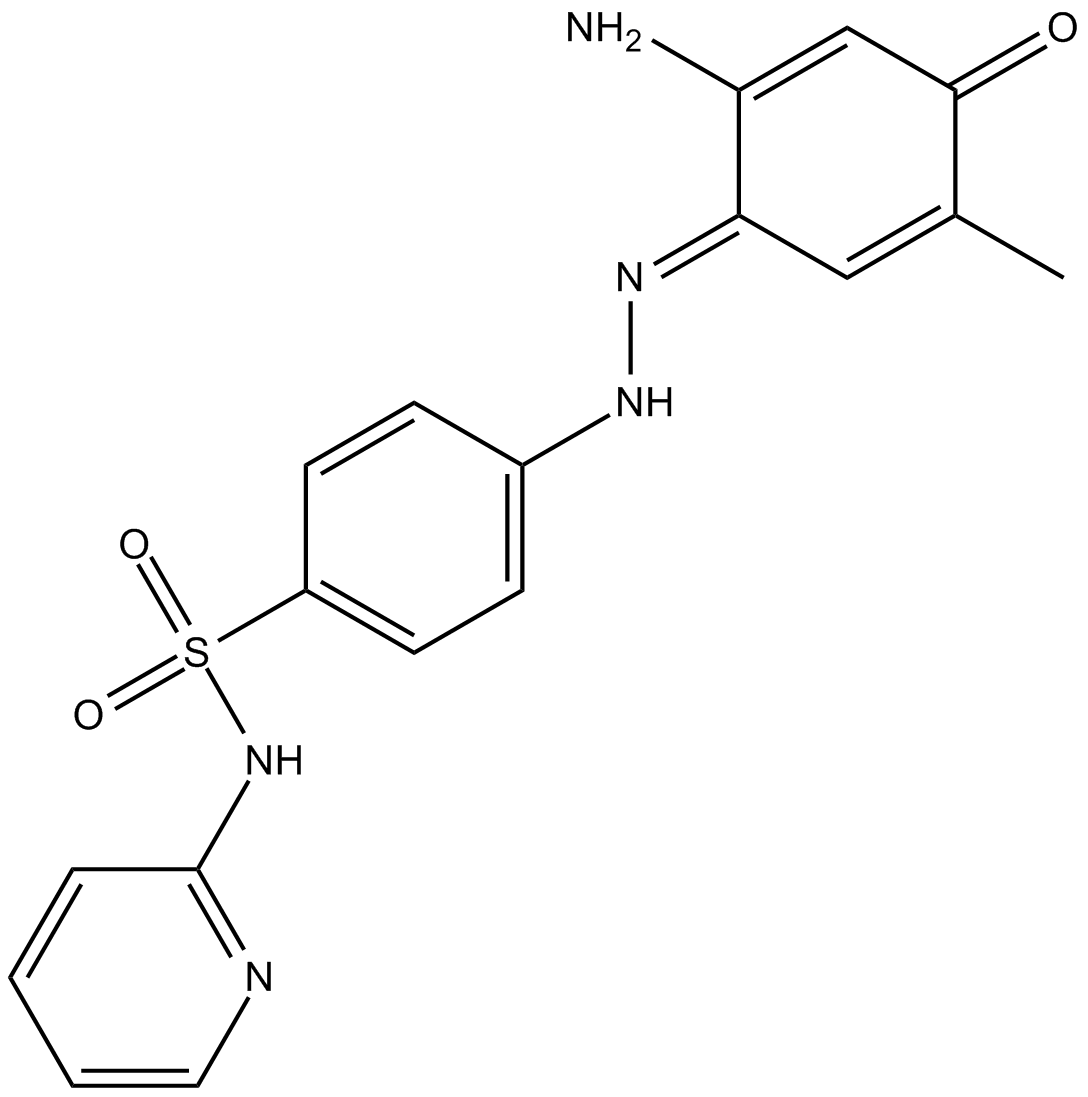
-
GC38474
MS645
MS645 est un inhibiteur bivalent des bromodomaines BET (BrD) avec un Ki de 18,4 nM pour BRD4-BD1/BD2. MS645 contraint spatialement l'inhibition bivalente des BrD de BRD4, ce qui entraÎne une répression soutenue de l'activité transcriptionnelle de BRD4 dans les cellules tumorales solides.
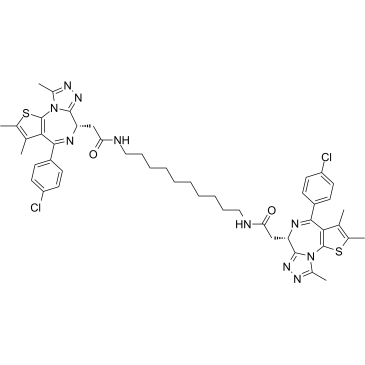
-
GC64295
MS67
MS67 est un dégradeur puissant et sélectif de la protéine 5 à répétition WD40 (WDR5) avec une Kd de 63 nM. MS67 est inactif contre d'autres méthyltransférases, kinases, RCPG, canaux ioniques et transporteurs. MS67 montre des effets anticancéreux puissants.

-
GC69504
MS8815
MS8815 est un dégradeur PROTAC sélectif de la protéine homologue 2 de zeste (EZH2). MS8815 présente une activité inhibitrice sur EZH2 avec une valeur IC50 de 8,6 nM. MS8815 peut être utilisé dans la recherche sur le cancer du sein triple négatif (TNBC).
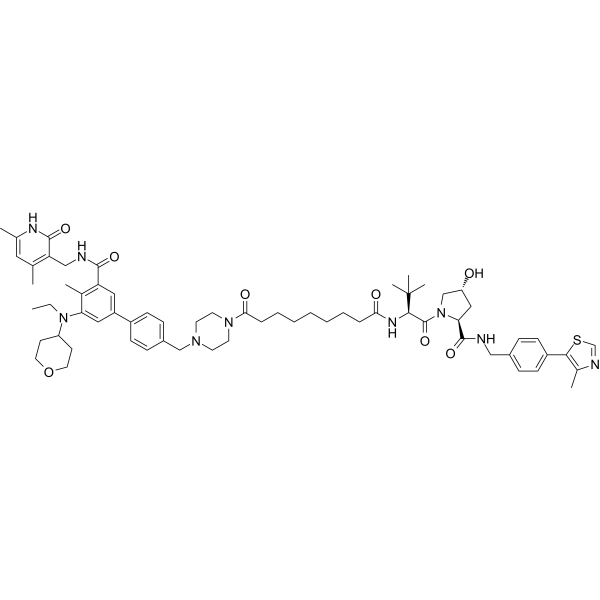
-
GC18729
MZ1
MZ1 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BRD4. MZ1 induit puissamment et rapidement une élimination réversible, durable et sélective de BRD4 sur BRD2 et BRD3. Kds de 382/120, 119/115 et 307/228 nM pour BRD4 BD1/2, BRD3 BD1/2 et BRD2 BD1/2, respectivement.
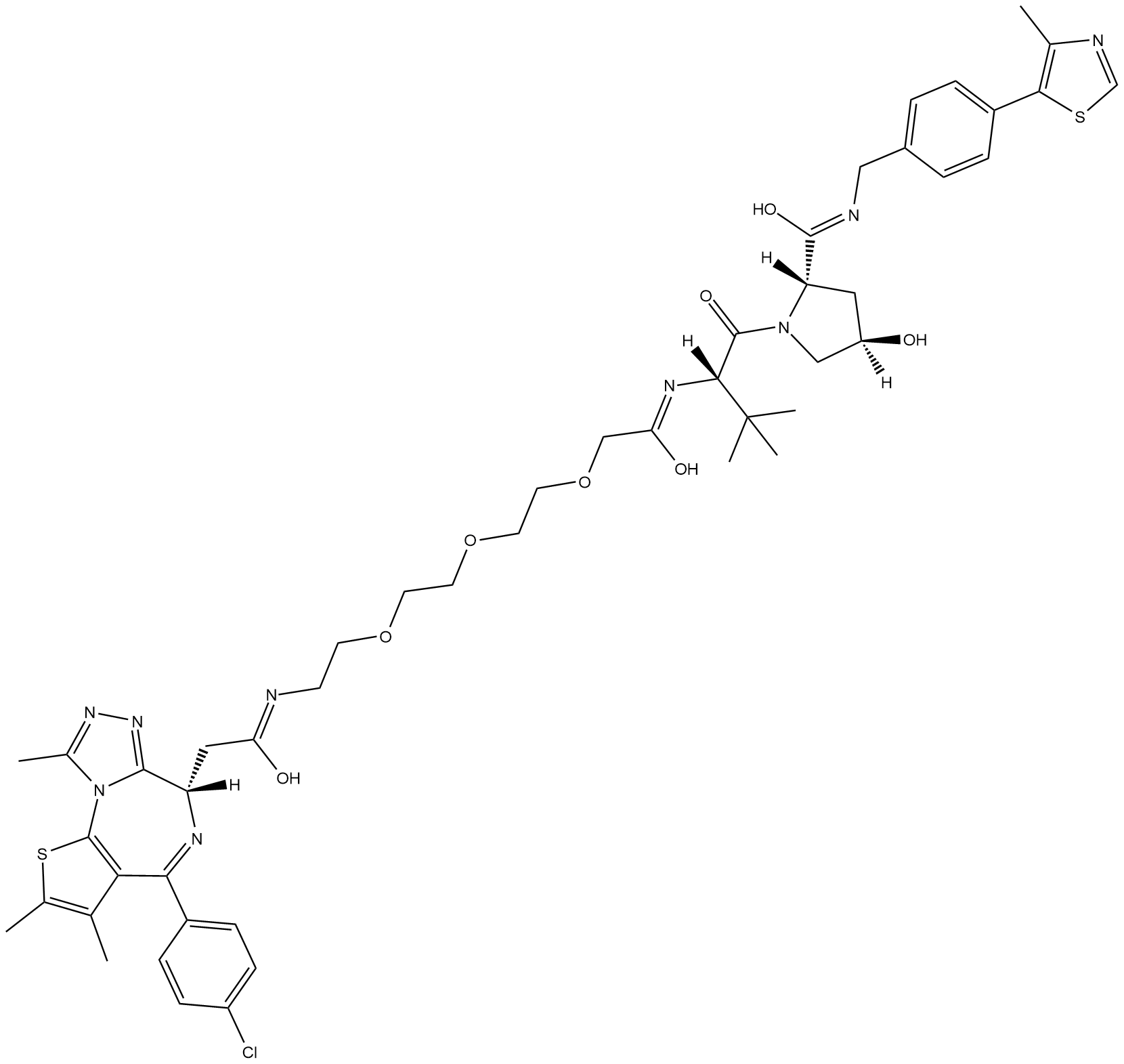
-
GC33102
MZP-54
MZP-54 est un PROTAC relié par des ligands pour von Hippel-Lindau et BRD3/4, avec un Kd de 4 nM pour Brd4BD2.
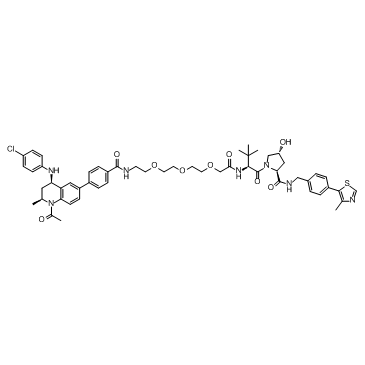
-
GC33363
MZP-55
MZP-55 est un PROTAC relié par des ligands pour von Hippel-Lindau et BRD3/4, avec un Kd de 8 nM pour Brd4BD2.
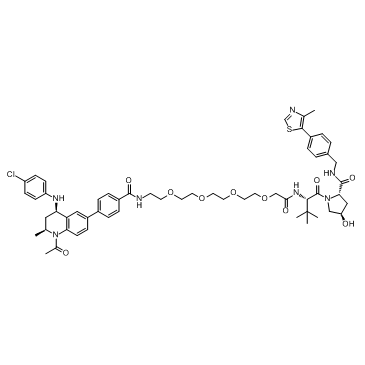
-
GC62154
N-Descyclopropanecarbaldehyde Olaparib
N-descyclopropanecarbaldéhyde L'olaparib est un analogue de l'olaparib contenant le fragment DOTA. Le N-descyclopropanecarbaldéhyde Olaparib est un ligand À base de CRBN pour la synthèse du nouveau double EGFR et PARP PROTAC, DP-C-4. N-Descyclopropanecarbaldéhyde L'olaparib peut être radiomarqué au F-18 ou au fluorophore pour la tomographie par émission de positrons (TEP) ou l'imagerie optique dans plusieurs types de tumeurs.