Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(43)
- Aurora Kinase(59)
- DNA Methyltransferase(32)
- HDAC(175)
- Histone Acetyltransferases(63)
- Histone Demethylases(94)
- Histone Methyltransferase(193)
- HIF(89)
- JAK(137)
- MBT Domains(0)
- PARP(102)
- Pim(26)
- Protein Ser/Thr Phosphatases(26)
- RNA Polymerase(5)
- Sirtuin(72)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(195)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
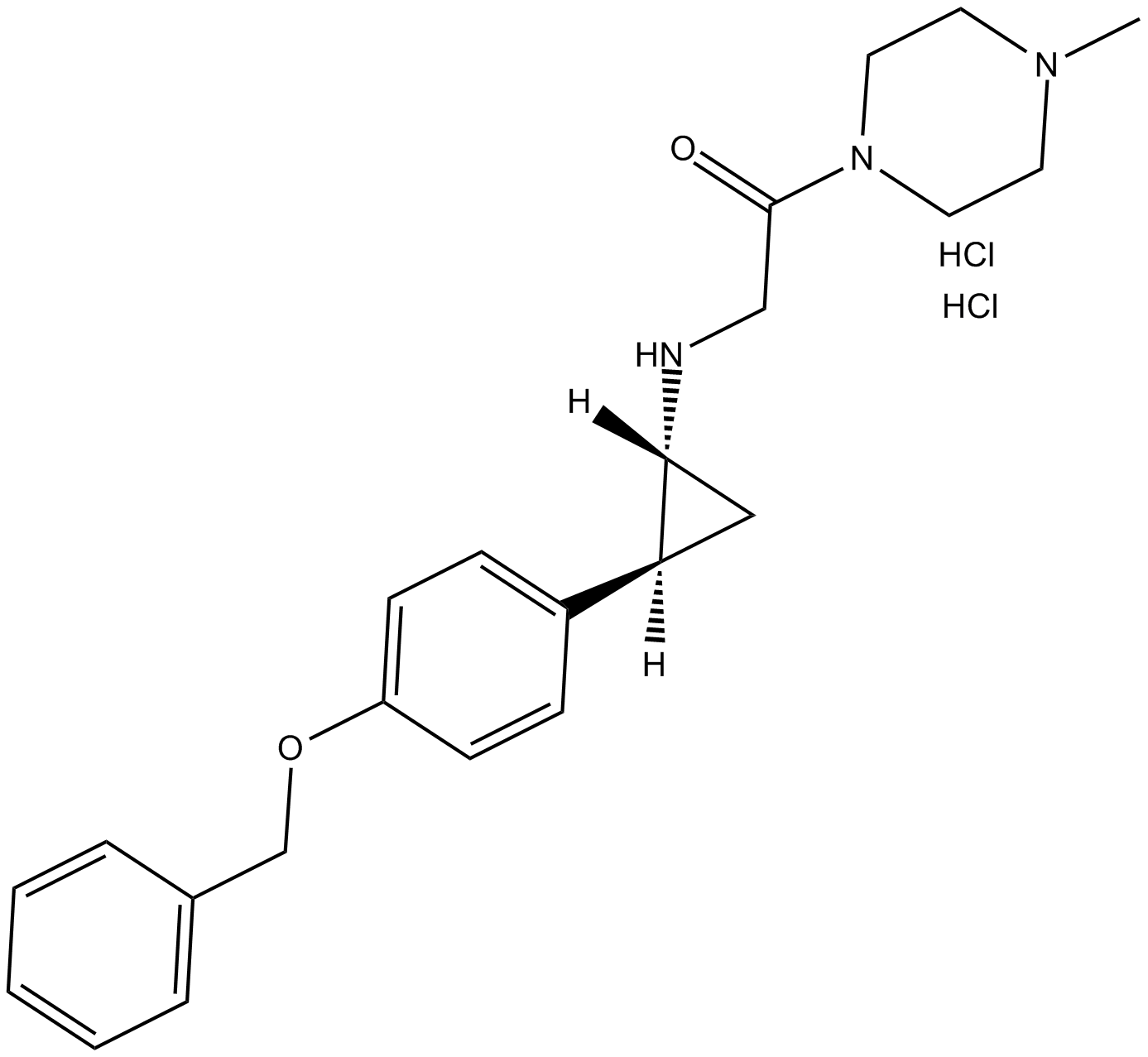
GC14682
PCI-24781 (CRA-024781)
CRA-024781, CRA-24781
An HDAC inhibitor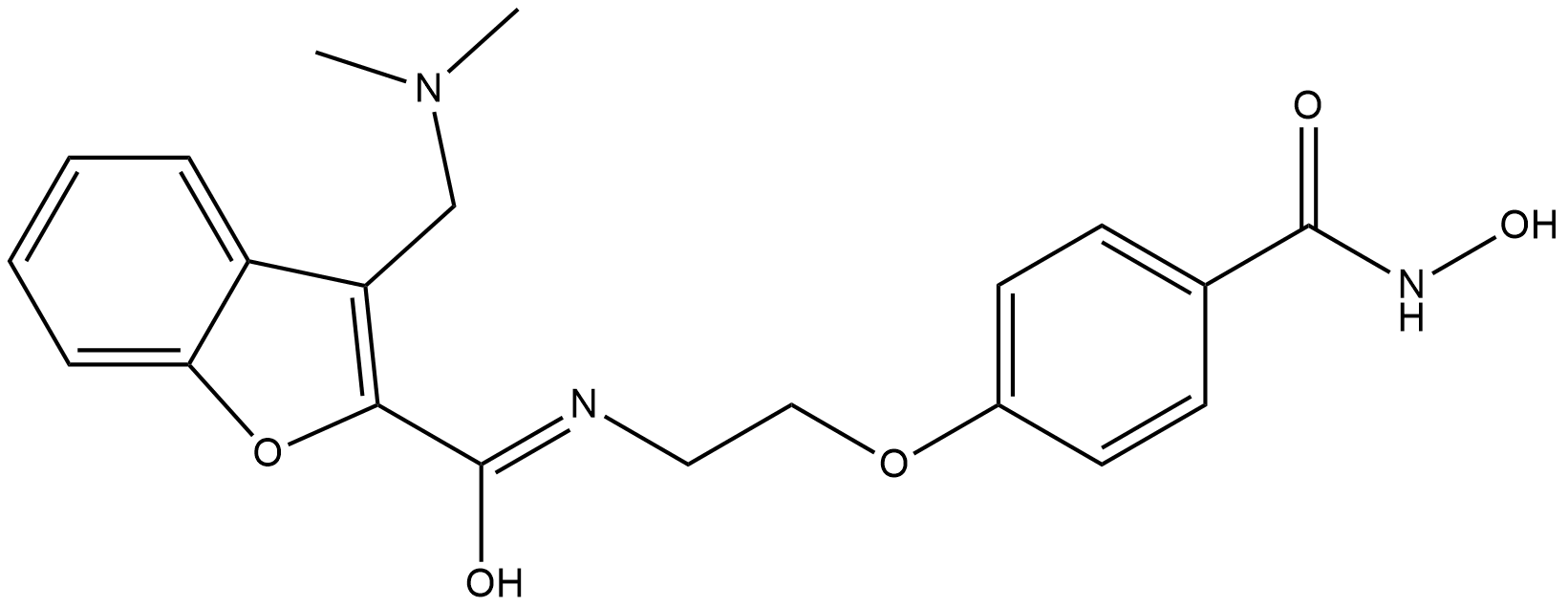
-
GC18008
PCI-34051
A potent, selective HDAC8 inhibitor
-
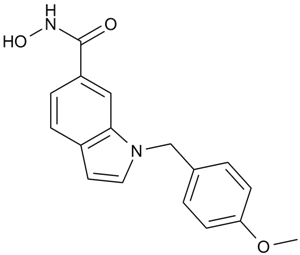
GC10341
Peficitinb (ASP015K, JNJ-54781532)
ASP015K, JNJ-54781532
Peficitinb (ASP015K, JNJ-54781532) (ASP015K) est un inhibiteur de JAK actif par voie orale, avec des CI50 de 3,9, 5,0, 0,7 et 4,8 nM pour JAK1, JAK2, JAK3 et Tyk2, respectivement.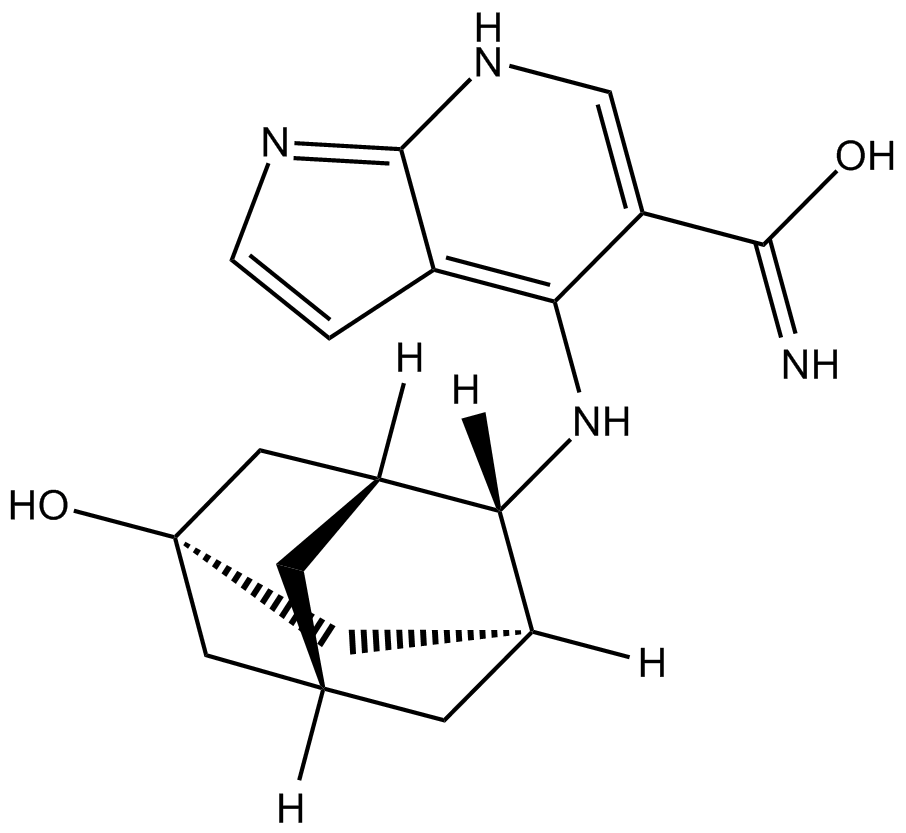
-
GC50480
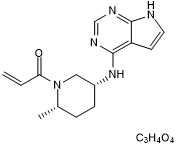
PF 06651600 malonate
Potent and selective JAK3 inhibitor
-
GC50367
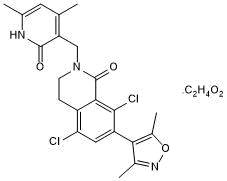
PF 06726304 acetate
Highly potent and SAM-competitive EZH2 inhibitor
-
GC14938
PF-03394197(Oclacitinib)
PF-03394197
Novel Janus kinase inhibitor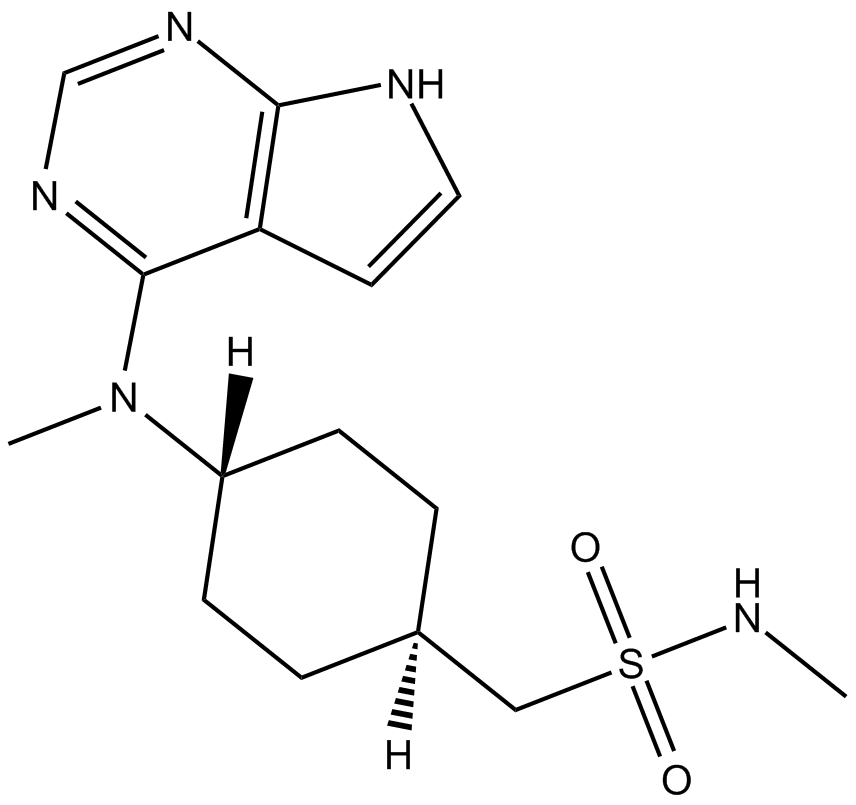
-
GC18074
PF-03814735
An inhibitor of Aurora A and B kinases
-
GC36882
PF-06263276
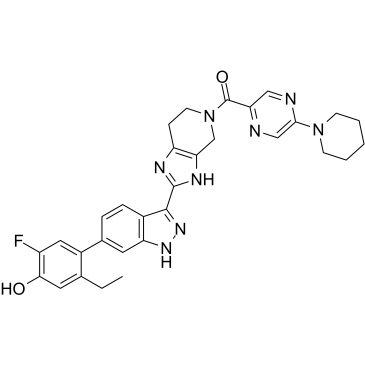
PF-06263276 (PF 6263276) est un inhibiteur pan-JAK puissant et sélectif, avec des IC50 de 2,2 nM, 23,1 nM, 59,9 nM et 29,7 nM pour JAK1, JAK2, JAK3 et TYK2, respectivement .
-
GC19288
PF-06651600
PF-06651600
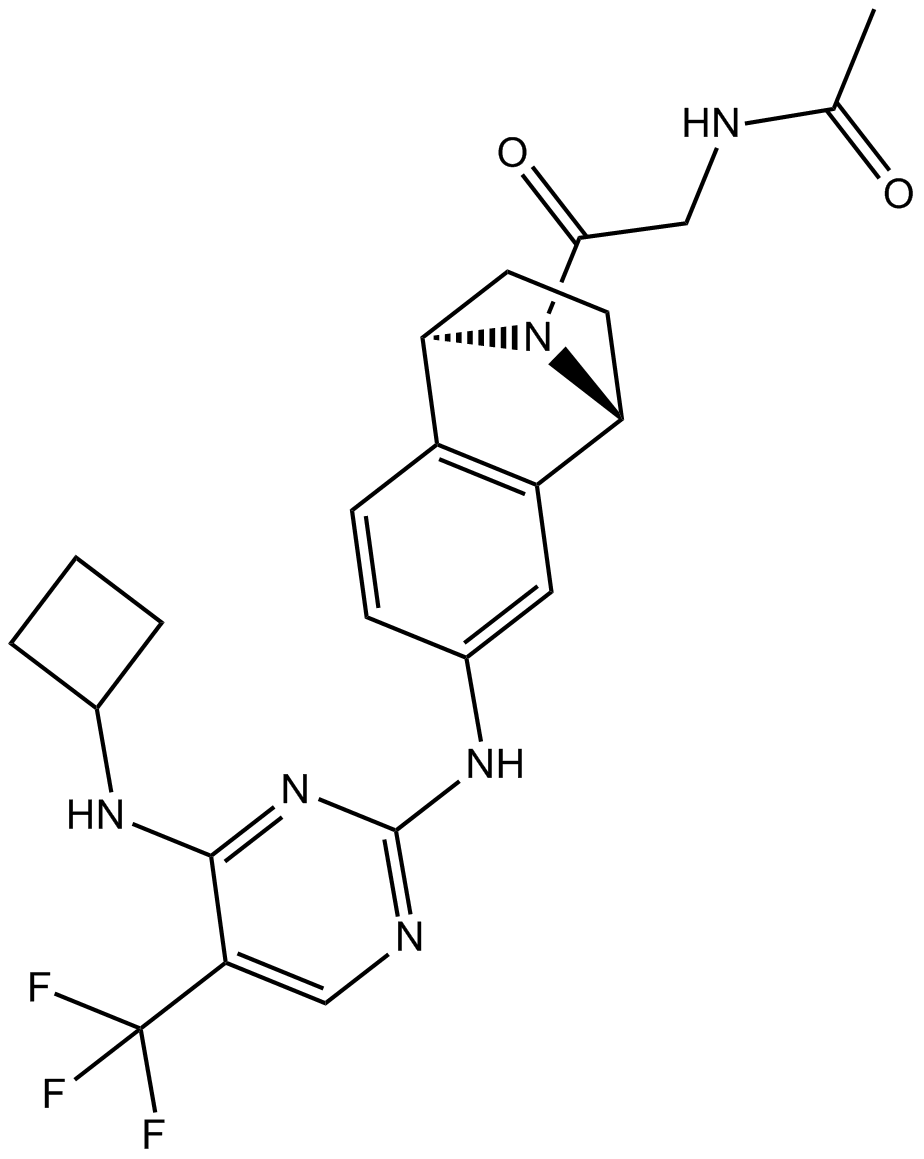
PF-06651600 (PF-06651600) est un inhibiteur de JAK3 actif et sélectif par voie orale avec une IC50 de 33,1 nM.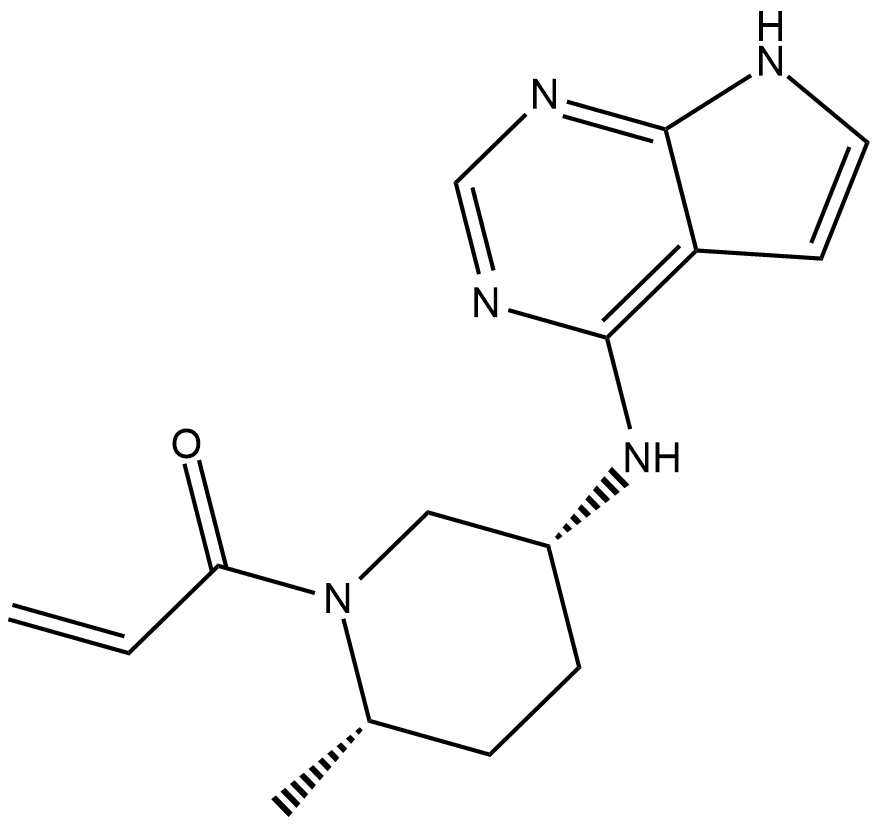
-
GC19405
PF-06700841 P-Tosylate
PF-06700841 P-Tosylate
Brepocitinib (PF-06700841) P-Tosylate est un puissant double inhibiteur de Janus kinase 1 (JAK1) et TYK2 avec des CI50 de 17 nM et 23 nM, respectivement.
-
GC32977
PF-06726304
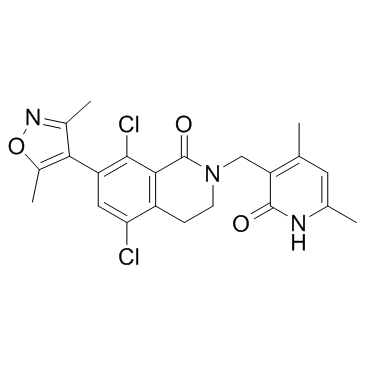
PF-06726304 est un inhibiteur EZH2 puissant et sélectif. PF-06726304 inhibe EZH2 de type sauvage et mutant Y641N avec Kis de 0,7 et 3,0 nM, respectivement. PF-06726304 affiche une activité de croissance antitumorale robuste.
-
GC62247
PF-9363
CTx-648
Le PF-9363 (CTx-648) est un inhibiteur puissant et hautement sélectif de KAT6A/KAT6B. Le PF-9363 peut être utilisé pour la recherche sur le cancer.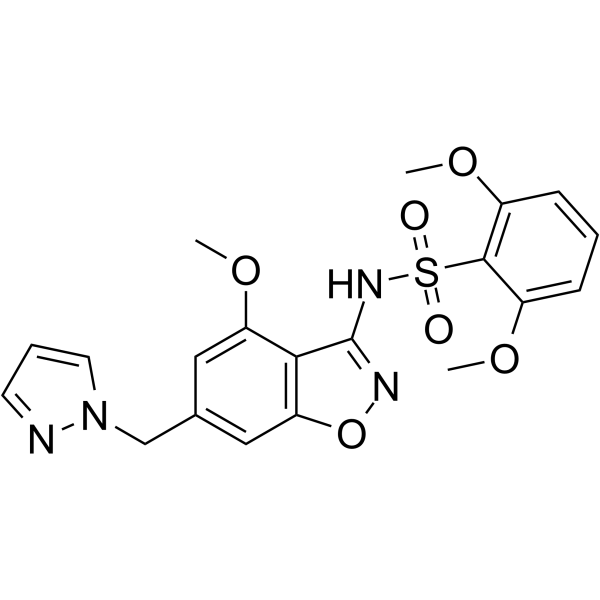
-
GC15152
PF-CBP1
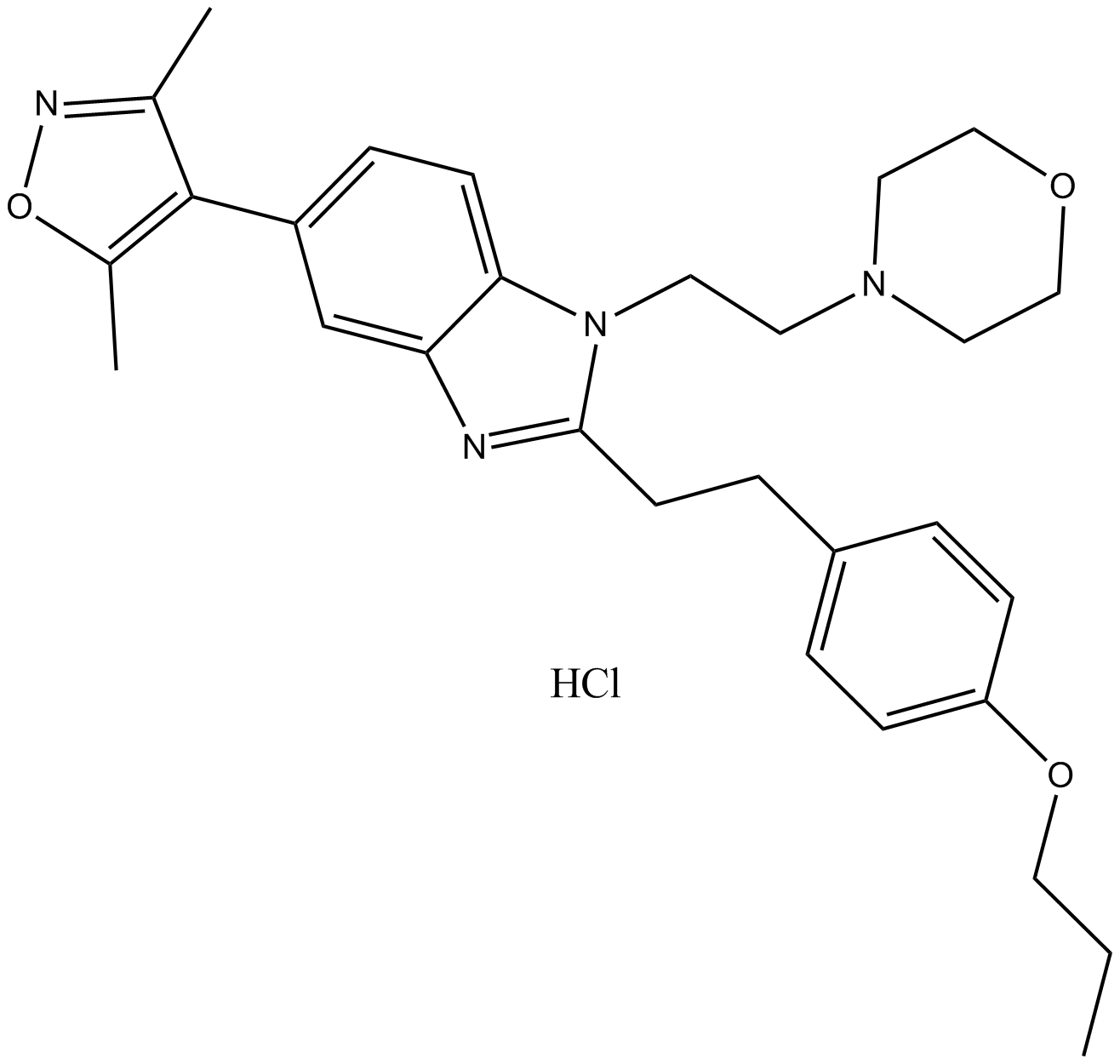
PF-CBP1 est un inhibiteur hautement sélectif du bromodomaine de la protéine de liaison CREB.
-
GC36887
PF-CBP1 hydrochloride
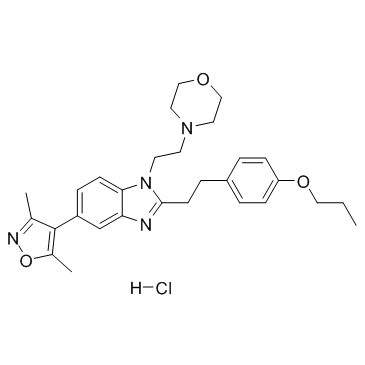
Le chlorhydrate de PF-CBP1 est un inhibiteur hautement sélectif du bromodomaine de la protéine de liaison CREB (CBP BRD).
-
GC14361
PFI 3
PFI 3 est un inhibiteur de bromodomaine SMARCA2/4 sélectif, puissant et perméable aux cellules avec un Kd de 89 nM.
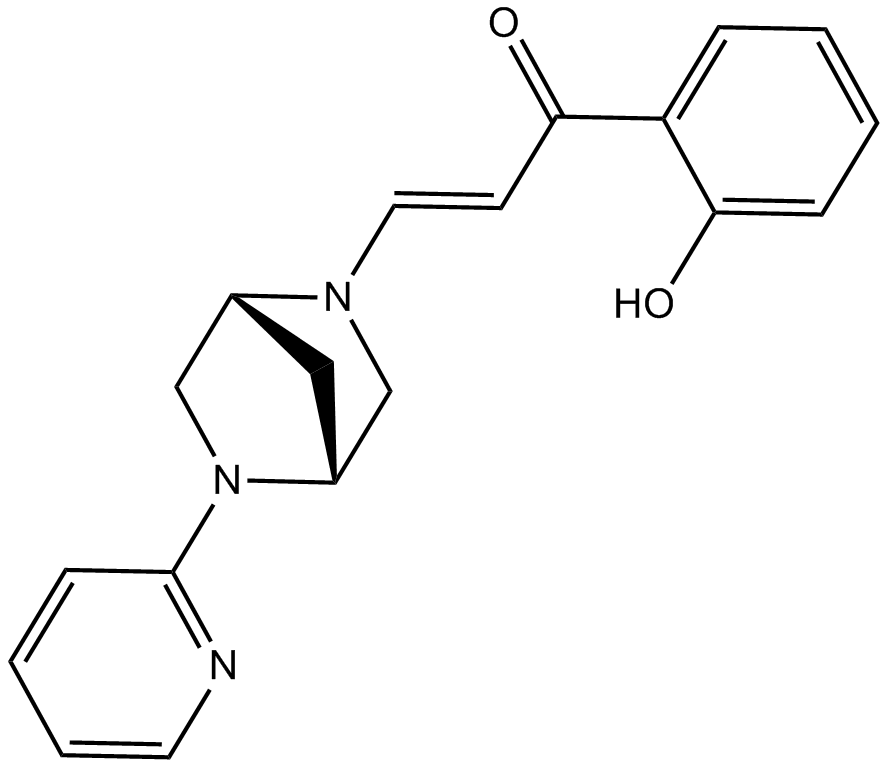
-
GC10979
PFI 4
PFI 4 (composé 11) est un inhibiteur puissant et hautement sélectif du bromodomaine BRPF1 (BRPF1B), avec une IC50 de 172 nM. PFI 4 permet d'explorer les mécanismes fonctionnels du complexe HBO1/BRPF1 et d'étudier la perte osseuse et les lésions osseuses malignes ostéolytiques.
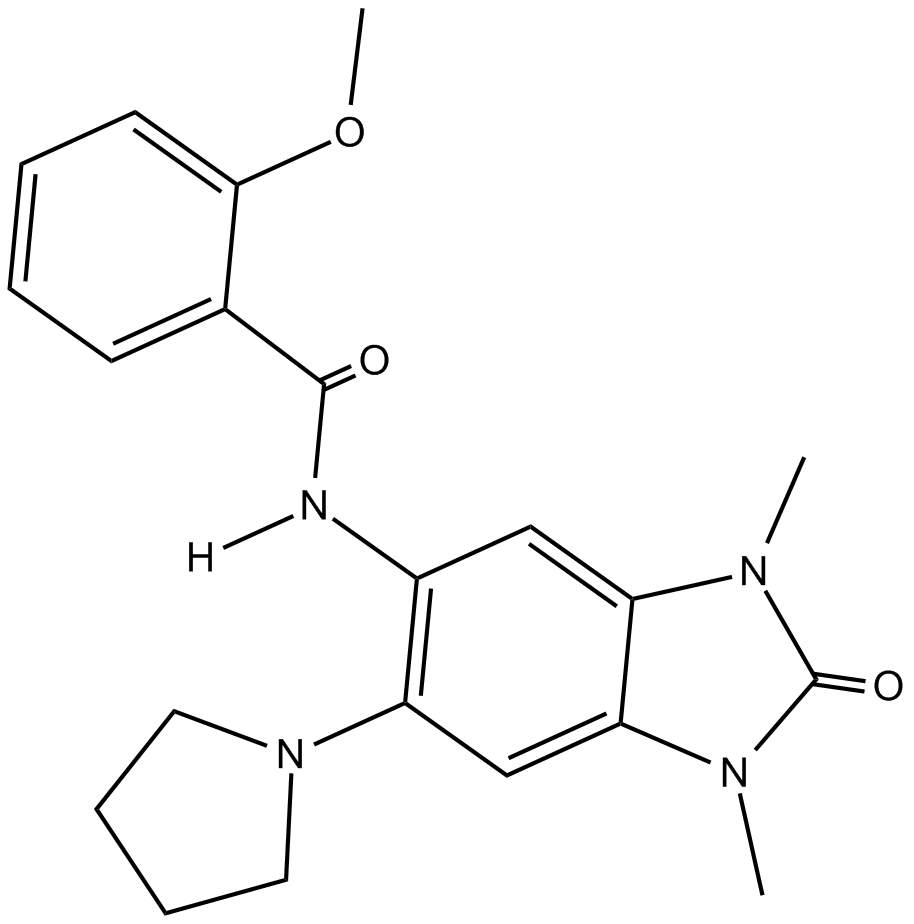
-
GC15375
PFI-1 (PF-6405761)
PF06405761
A BET bromodomain inhibitor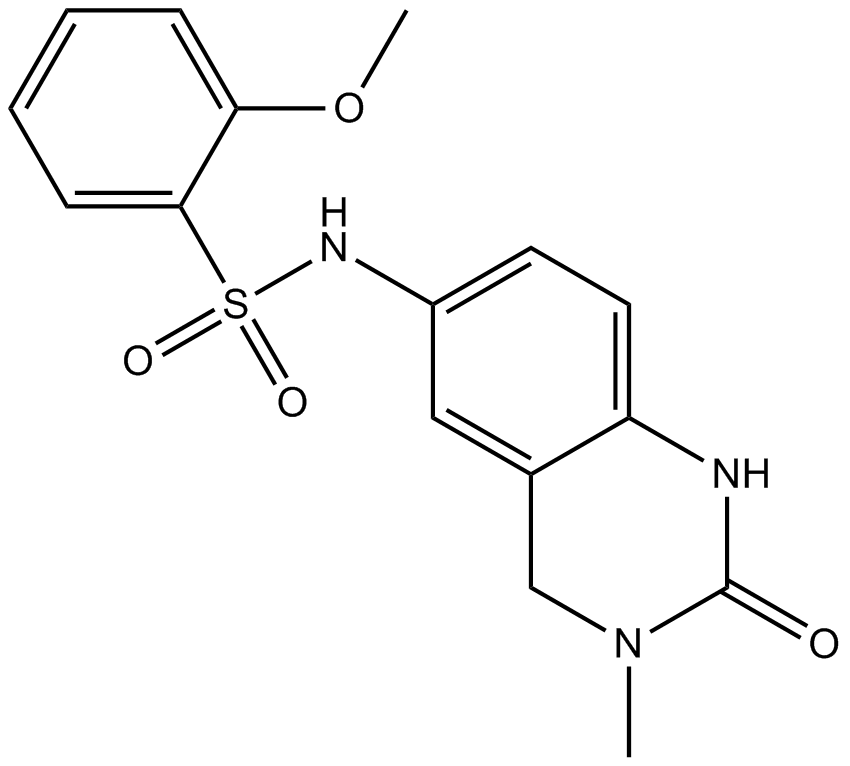
-
GC17956
PFI-2
(R)-PFI-2
Le chlorhydrate de PFI-2 ((R)-PFI-2 hydrochloride) est un domaine SET puissant et sélectif contenant un inhibiteur de la lysine méthyltransférase 7 (SETD7).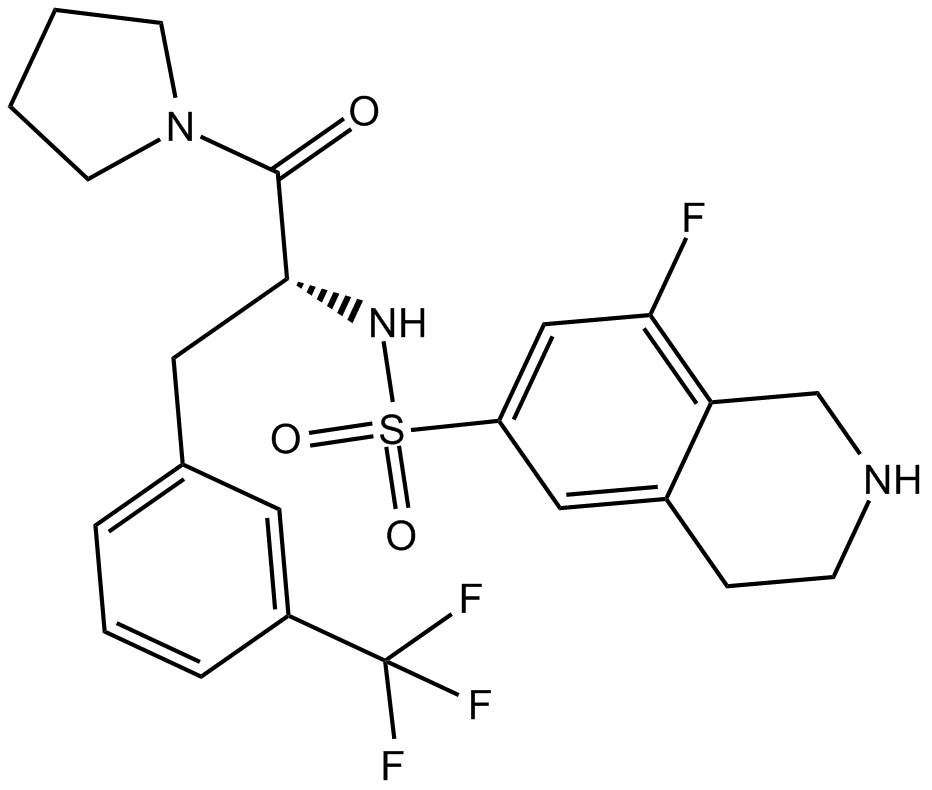
-
GC12530
PFI-2 (hydrochloride)
(R)-PFI-2 hydrochloride

-
GC62678
PFI-90
Le PFI-90 est un inhibiteur sélectif de l'histone déméthylase (KDM3B) qui inhibe l'action de PAX3-FOXO1. Le PFI-90 induit l'apoptose et la différenciation myogénique, entraÎnant une augmentation de la mort cellulaire. PFI-90 a le potentiel pour l'activité antitumorale. (brevet WO2021101929A1).
-
GC10479
PHA-680632
Pha 680632
An Aurora kinase inhibitor
-
GC64584
PHD-1-IN-1
PHD-1-IN-1 est un inhibiteur du domaine 1 de la prolylhydroxylase HIF (PHD-1) actif et puissant par voie orale avec une IC50 de 0,034 μM.
-
GC45548
Photoswitchable PAD Inhibitor (technical grade)
-
GC13668
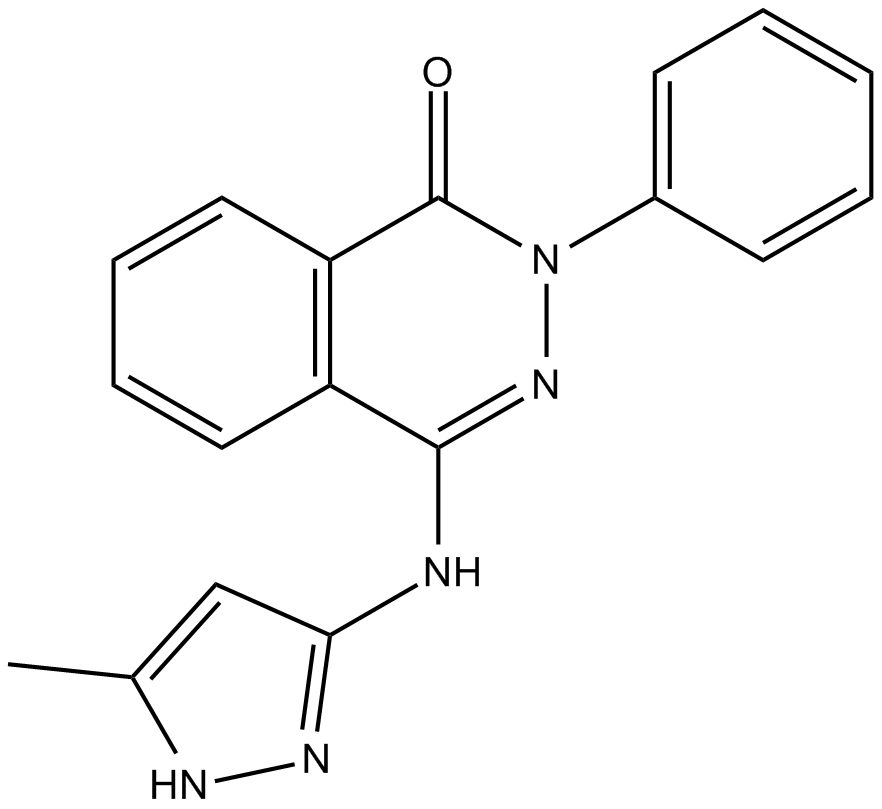
Phthalazinone pyrazole
Le phtalazinone pyrazole est un inhibiteur puissant, sélectif et actif par voie orale de la kinase Aurora-A avec une CI50 de 0,031 μM. Le phtalazinone pyrazole peut arrêter la mitose et ensuite inhiber la croissance tumorale via l'apoptose des cellules en prolifération. Le pyrazole de phtalazinone supprime la transition épithéliale-mésenchymateuse (EMT) lors de la différenciation des cellules de type hépatocyte (HLC) des cellules souches embryonnaires humaines.
-
GC36905
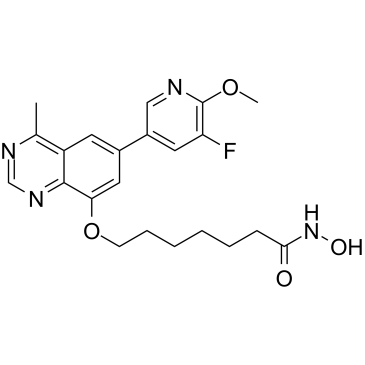
PI3K/HDAC-IN-1
PI3K/HDAC-IN-1 est un double inhibiteur puissant de PI3K/HDAC, inhibe puissamment PI3Kδ et HDAC1 avec des IC50 de 8,1 nM et 1,4 nM, respectivement.
-
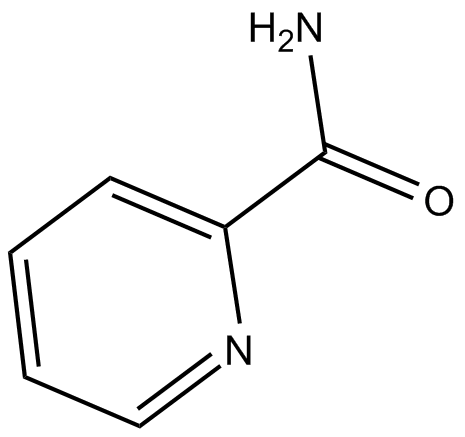
GC14251
Picolinamide
poly (ADP-ribose) synthetase inhibitor
-
GC19419
PIM inhibitor 1 phosphate
INCB053914 phosphate
Le phosphate d'Uzansertib (INCB053914) est un inhibiteur de pan-PIM kinase compétitif pour l'ATP, actif par voie orale, avec des IC50 de 0,24 nM, 30 nM, 0,12 nM pour PIM1, PIM2, PIM3, respectivement. L'inhibiteur de PIM 1 phosphate a une large activité anti-proliférative contre une variété de lignées cellulaires tumorales hématologiques.
-
GC13558
PIM-1 Inhibitor 2
PIMi II
A potent PIM-1 kinase inhibitor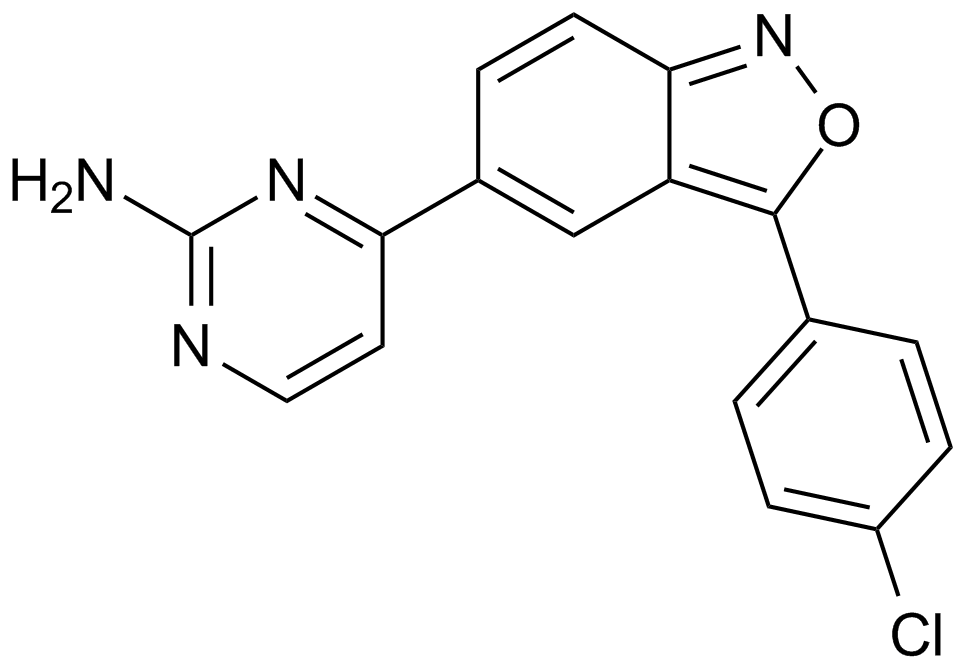
-
GC36918
PIM-447 dihydrochloride
LGH447 dihydrochloride
Le dichlorhydrate de PIM447 (dichlorhydrate de LGH447) est un inhibiteur de pan-PIM kinase puissant, disponible par voie orale et sélectif, avec des valeurs de Ki de 6, 18 et 9 pM pour PIM1, PIM2 et PIM3, respectivement. Le dichlorhydrate de PIM447 présente un double effet antimyélome et protecteur des os. Le dichlorhydrate de PIM447 induit l'apoptose.
-
GC65278
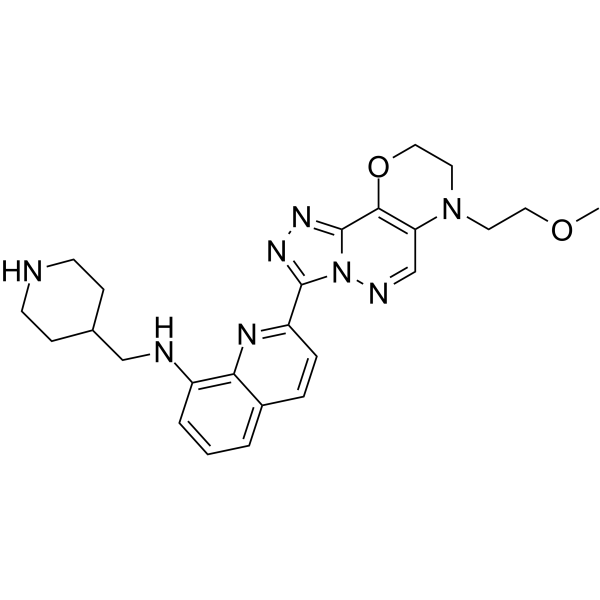
PIM1-IN-1
PIM1-IN-1 est un inhibiteur puissant et hautement sélectif de PIM1/3, avec des IC50 de 7, 5530 et 70 nM pour PIM1, PIM2 et PIM3, respectivement, inhibe la phosphorylation de BAD, une cible en aval de PIM, avec une CE50 de 262 nM. PIM1-IN-1 ne montre aucun effet évident sur la liaison de FLT3 ou de hERG. Activité antiproliférative et anticancéreuse.
-
GC19398
PIM447
LGH447
PIM447 (LGH447) est un inhibiteur de pan-PIM kinase puissant, disponible par voie orale et sélectif, avec des valeurs de Ki de 6, 18 et 9 pM pour PIM1, PIM2 et PIM3, respectivement. PIM447 affiche un double effet antimyélome et protecteur des os. PIM447 induit l'apoptose.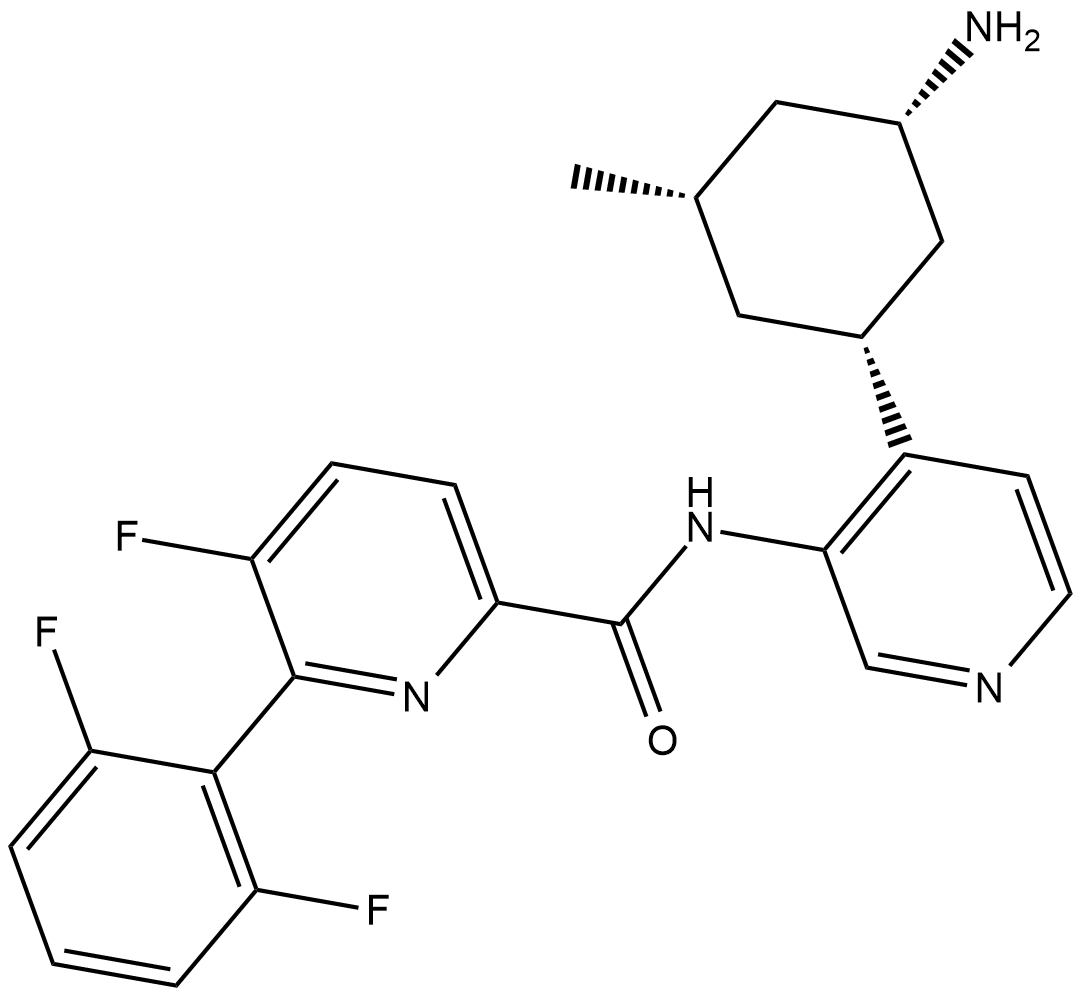
-
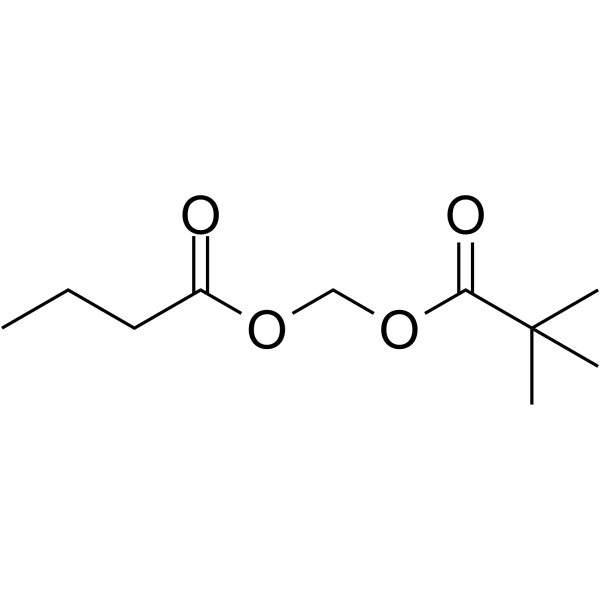
GC68474
Pivanex
AN-9; Pivalyloxymethyl butyrate
-
GC10995
PJ34
PJ-34;PJ 34
An inhibitor of poly (ADP-ribose) polymerases
-
GC10145
PJ34 hydrochloride
PJ 34 Hydrochloride
An inhibitor of poly (ADP-ribose) polymerases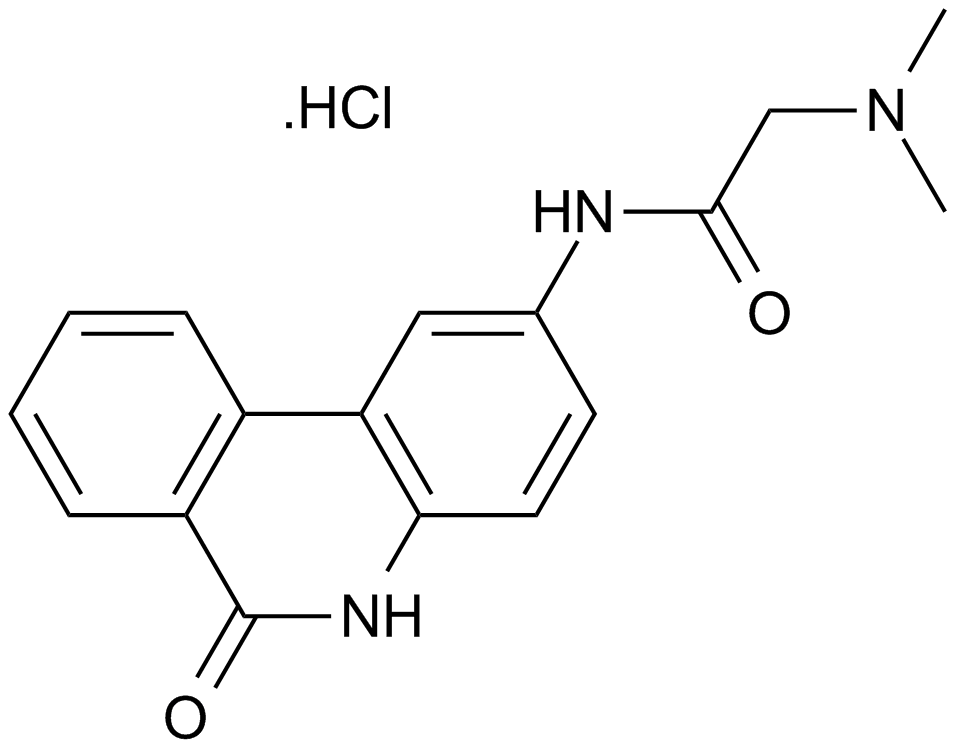
-
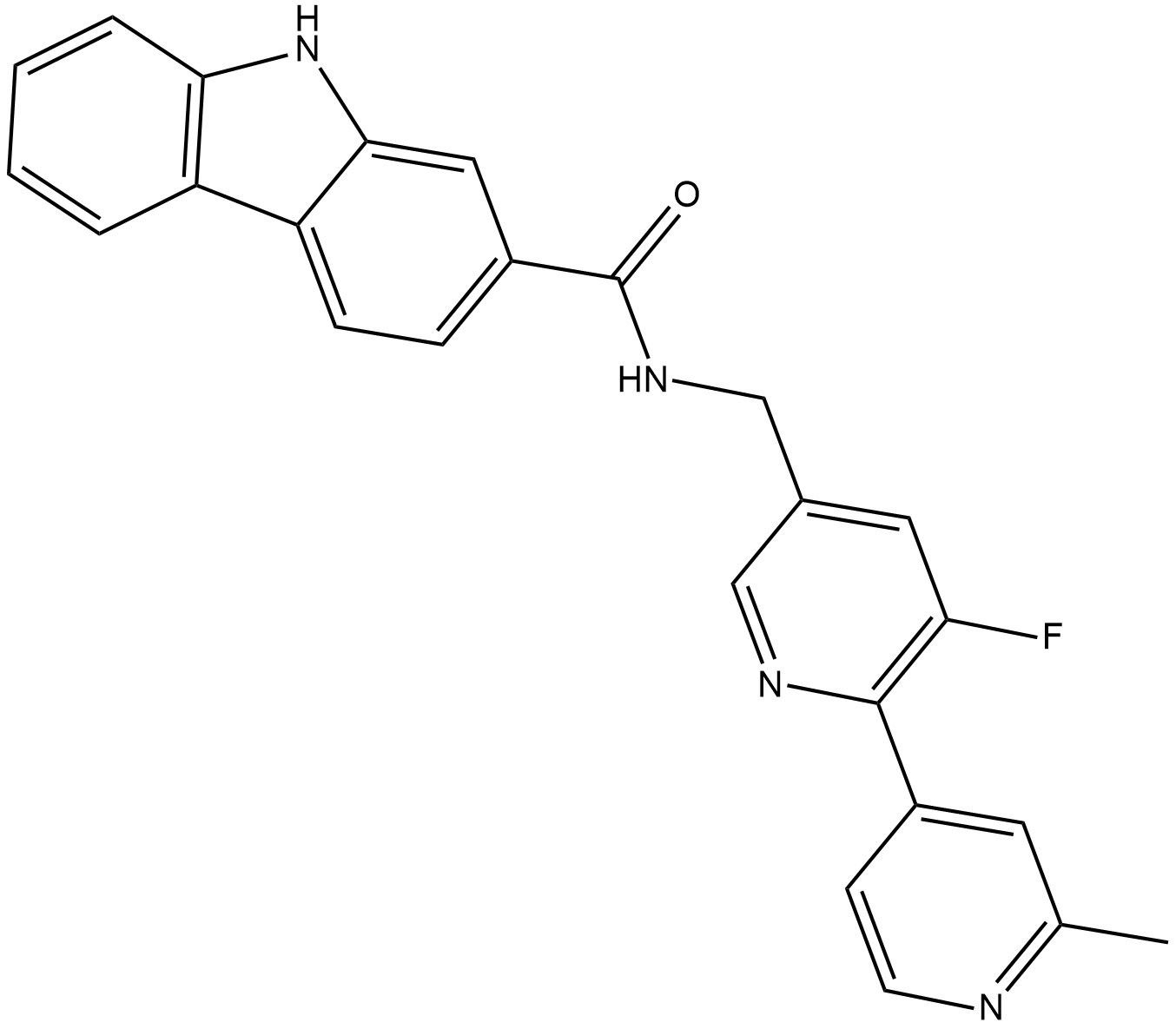
GC19298
PLX51107
PLX51107 est un inhibiteur puissant et sélectif de BET, avec des Kd de 1,6, 2,1, 1,7 et 5 nM pour BD1 et 5,9, 6,2, 6,1 et 120 nM pour BD2 de BRD2, BRD3, BRD4 et BRDT, respectivement; PLX51107 interagit également avec les bromodomaines de CBP et EP300 (Kd, dans la gamme 100 nM).
-
GC36943
PNZ5
PNZ5 est un inhibiteur pan-BET puissant À base d'isoxazole avec une sélectivité élevée et une puissance similaire au bien établi (+)-JQ1, avec un KD de 5,43 nM pour BRD4(1).

-
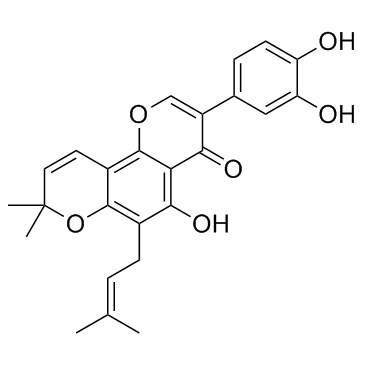
GC33115
Pomiferin (NSC 5113)
La pomiférine (NSC 5113) (NSC 5113) agit comme un inhibiteur potentiel de HDAC, avec une IC50 de 1,05 μM, et inhibe également puissamment mTOR (IC50, 6,2 μM).
-
GC64701
Povorcitinib
INCB54707
Le povorcitinib est un inhibiteur puissant et sélectif de JAK1.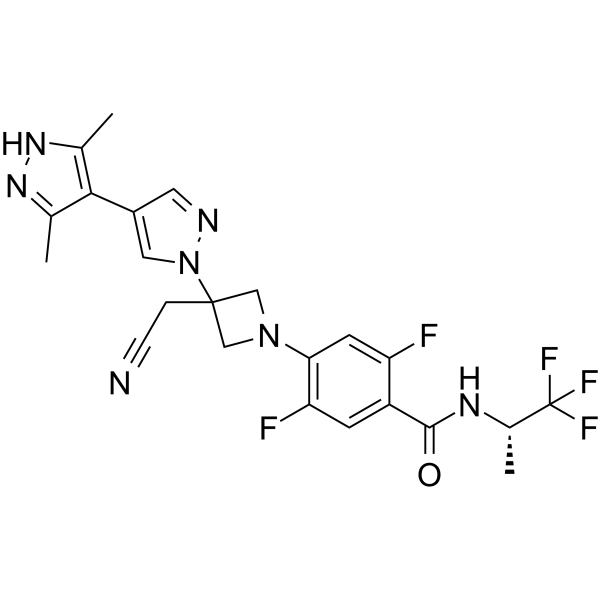
-
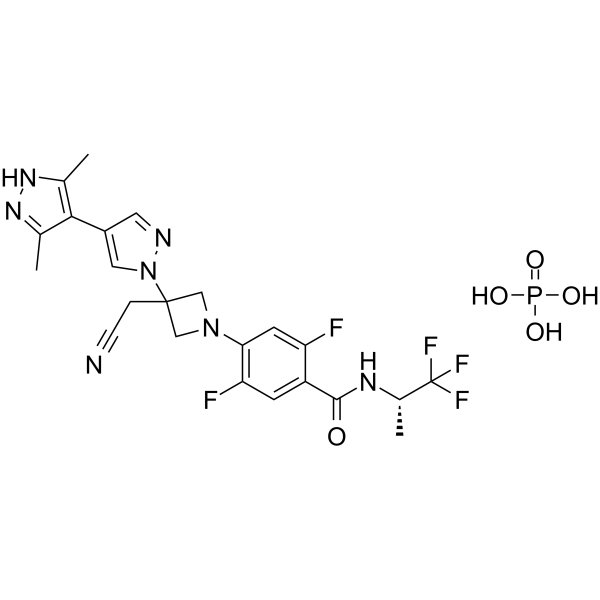
GC64700
Povorcitinib phosphate
-
GC64941
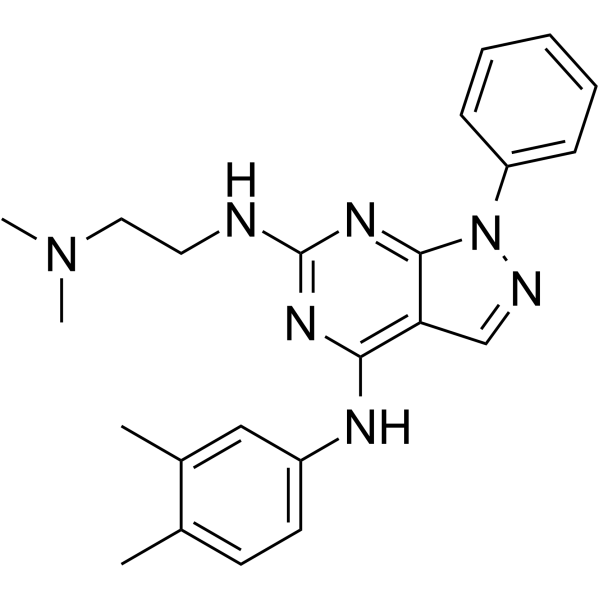
PR5-LL-CM01
PR5-LL-CM01 est un puissant inhibiteur de la protéine arginine méthyltransférase 5 (PRMT5) (IC50 = 7,5 μM). Activités anti-tumorales.
-
GC16734
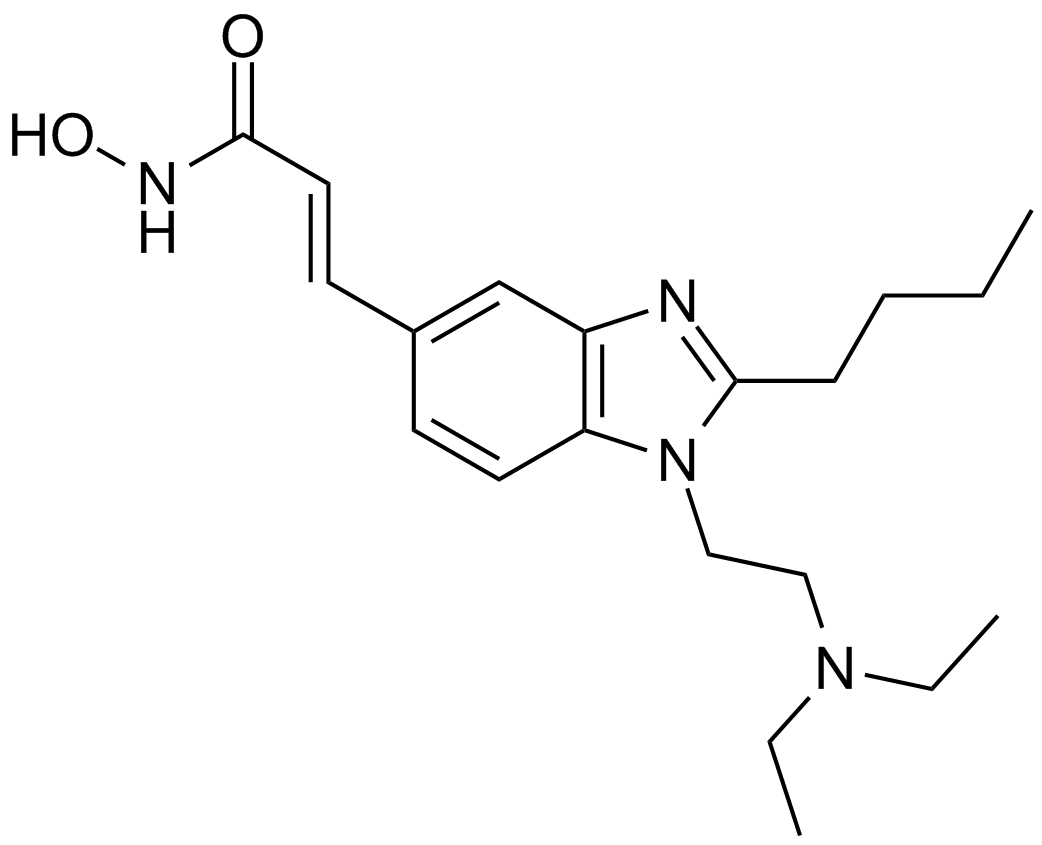
Pracinostat (SB939)
Pracinostat
A pan-HDAC inhibitor
-
GC16900
PRL-3 Inhibitor
BR-1,P0108,Phosphatase of Regenerating Liver 3 Inhibitor,PTP4A3 Inhibitor
L'inhibiteur de PRL-3 est un puissant inhibiteur de PRL-3 avec une CI50 de 0,9 μM. L'inhibiteur de PRL-3 montre une invasion réduite dans le dosage cellulaire.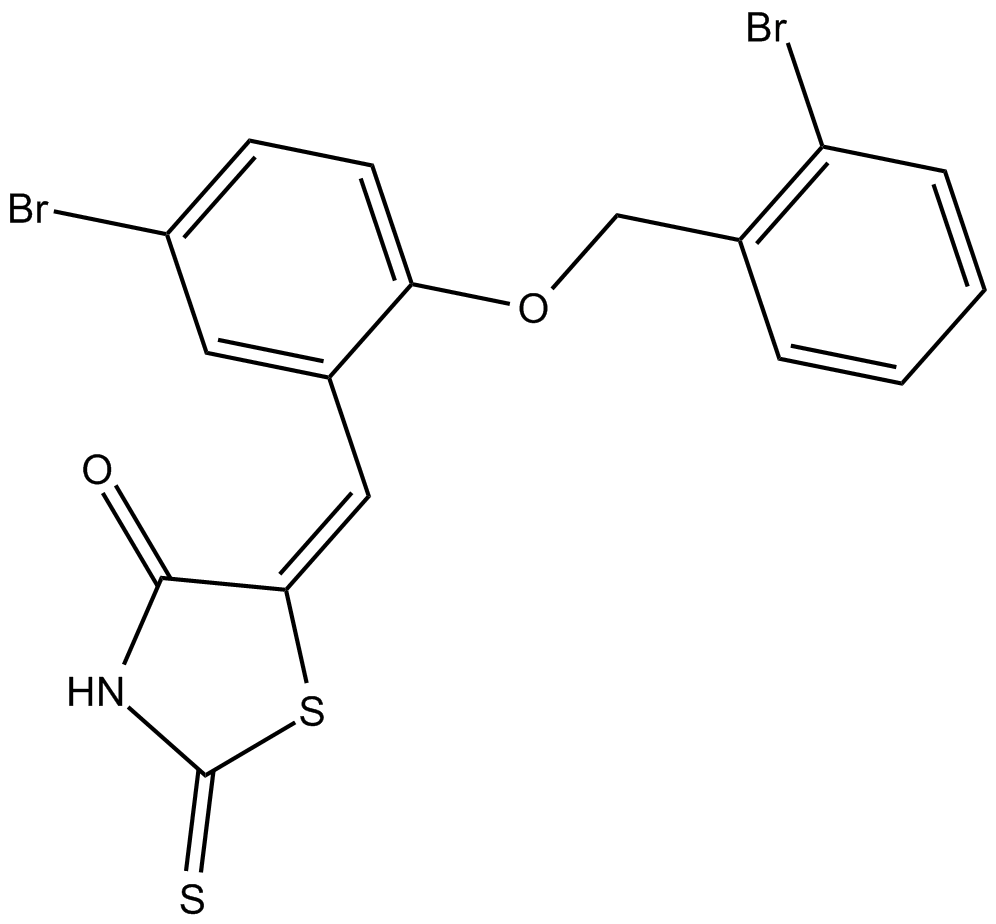
-
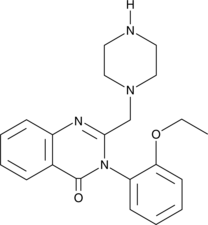
GC41572
PRLX-93936
PRLX-93936 is an analog of erastin that has antitumor activity.
-
GC36969
PRMT5-IN-1
PRMT5 IN-1, un hémiaminal, est un puissant inhibiteur sélectif de la protéine arginine méthyltransférase 5 (PRMT5) avec une IC50 de 11 nM pour PRMT5/MEP50. PRMT5 IN-1 peut être converti en aldéhydes et réagir avec C449 pour former des adduits covalents dans des conditions physiologiques.
-
GC65027
PRMT5-IN-20
PRMT5-IN-20 est un inhibiteur sélectif de la protéine arginine méthyltransférase 5 (PRMT5) avec une activité anti-tumorale.
-
GC64733
Procainamide
Le procaÏnamide est un inhibiteur spécifique et puissant de l'ADN méthyltransférase 1 (DNMT1).
-
GC14066
Procaine
La procaÏne est un agent de déméthylation de l'ADN.
-
GC44685
Procaine (hydrochloride)
Novocaine
Procaine (hydrochloride) is an analytical reference standard categorized as a local anesthetic that is used as an adulterant.
-
GC36975
Procyanidin B3
(+)-Catechin-(4α→8)-(+)-catechin, Proanthocyanidin B3, Procyanidol B3
La procyanidine B3 est un produit naturel, agit comme un inhibiteur spécifique de la HAT, se lie À l'autre site de p300 au lieu du site actif, inhibe sélectivement l'acétylation des récepteurs aux androgènes médiée par p300. La procyanidine B3 n'a aucun effet sur l'HDAC ou l'HMT (histone méthyltransférase).
-
GC49116
Prohexadione
BX-112
A plant growth regulator
-
GC30601
Propanamide
Le propanamide est un inhibiteur puissant et sélectif de JAK extrait du brevet WO2012122452A1, composé II, a le potentiel pour le traitement des troubles cutanés (tels que le lupus cutané).
-
GC33109
PROTAC BET Degrader-1
PROTAC BET Degrader-1 est un PROTAC connecté par des ligands pour Cereblon et BET, diminuant les niveaux de protéines BRD2, BRD3 et BRD4 À faible concentration.
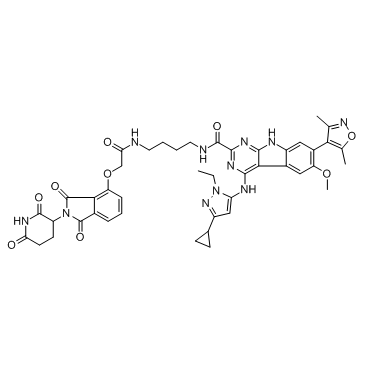
-
GC32980
PROTAC BET degrader-2
PROTAC BET degrader-2 est un PROTAC connecté par des ligands pour Cereblon et BET avec une valeur IC50 de 9,6 nM dans l'inhibition de la croissance cellulaire dans les cellules RS4;11 et capable d'obtenir une régression tumorale.
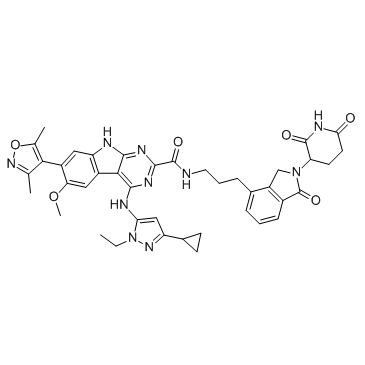
-
GC34325
PROTAC BET degrader-3
PROTAC BET Degrader-3 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BET.
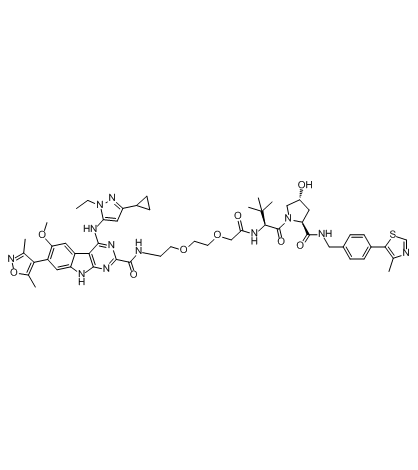
-
GC61212
PROTAC BRD4 Degrader-5
PROTAC BRD4 Degrader-5 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BRD4. PROTAC BRD4 Degrader-5 peut dégrader efficacement BRD4 dans les lignées cellulaires de cancer du sein HER2 positives et négatives.
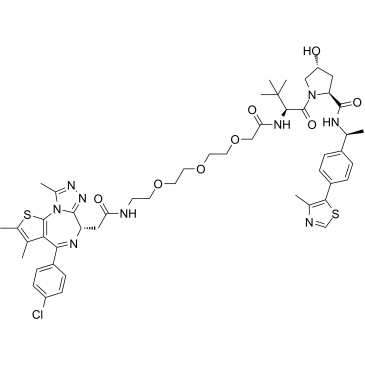
-
GC62606
PROTAC BRD4 Degrader-8
PROTAC BRD4 Degrader-8 est un PROTAC connecté par des ligands pour von Hippel-Lindau et BRD4, avec des IC50 de 1,1 nM et 1,4 nM pour BRD4 BD1 et BD2, respectivement. PROTAC BRD4 Degrader-8 est capable de dégrader puissamment la protéine BRD4 dans les cellules cancéreuses de la prostate PC3.
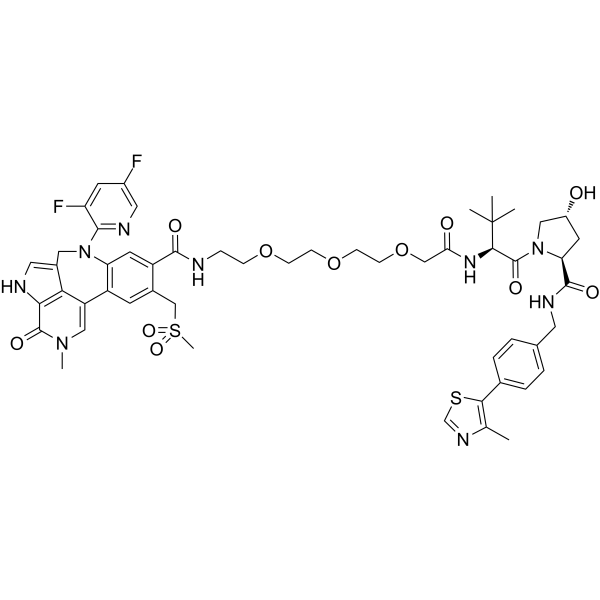
-
GC39180
PROTAC BRD4 ligand-1
PROTAC BRD4 ligand-1 est un puissant inhibiteur de BET et un ligand pour la protéine cible BRD4 pour PROTACT GNE-987.
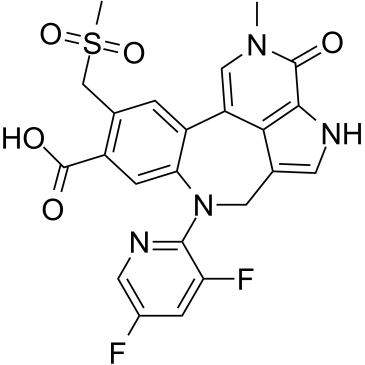
-
GC33372
PROTAC BRD9 Degrader-1
PROTAC BRD9 Degrader-1 est un PROTAC connecté par des ligands pour Cereblon et BRD9 (IC50 = 13,5 nM), qui peut être utilisé comme sonde sélective utile pour l'étude de la biologie du complexe BAF.
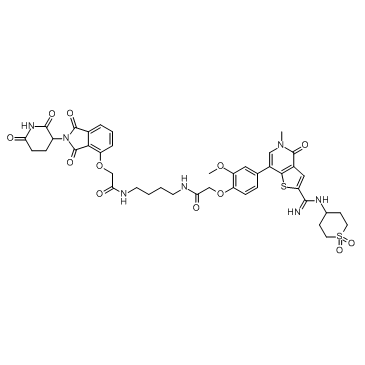
-
GC62187
PROTAC EED degrader-1
PROTAC EED degrader-1 est un PROTAC basé sur von Hippel-Lindau ciblant l'EED avec un pKD de 9,02. PROTAC EED degrader-1 est un inhibiteur du complexe répressif polycomb 2 (PRC2) (pIC50 = 8,17) ciblant la sous-unité EED.
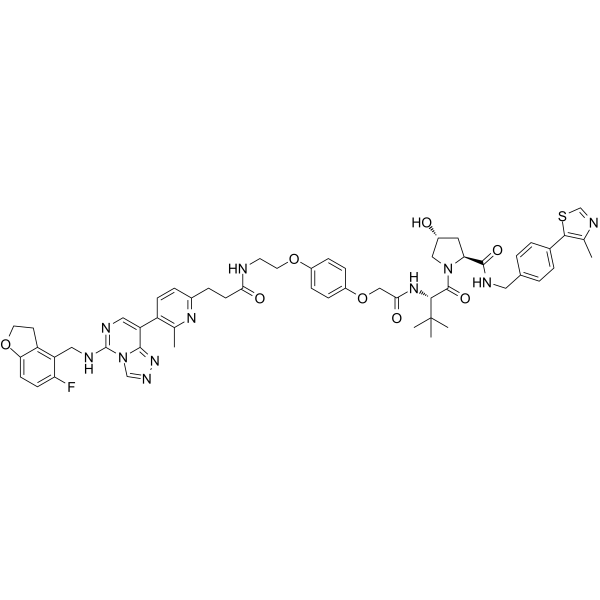
-
GC62717
PROTAC EED degrader-2
PROTAC EED degrader-2 est un PROTAC basé sur von Hippel-Lindau ciblant l'EED avec un pKD de 9,27. PROTAC EED degrader-2 est un inhibiteur du complexe répressif polycomb 2 (PRC2) (pIC50 = 8,11) ciblant la sous-unité EED.
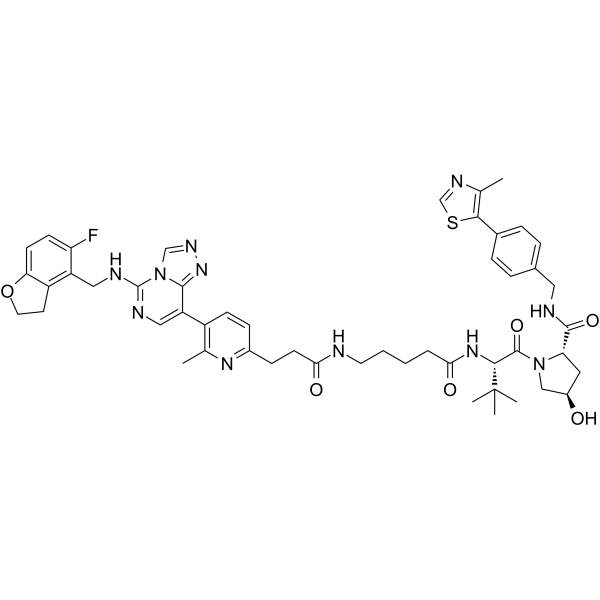
-
GC65509
PROTAC EZH2 Degrader-1
PROTAC EZH2 Degrader-1 (composé 150d), un puissant dégradeur PROTAC EZH2, exerce un effet inhibiteur sur l'activité de la méthyltransférase EZH2 avec une IC50 de 2,7 nM. EZH2 joue un rÔle important dans de nombreux processus de tumorigenèse et de développement.
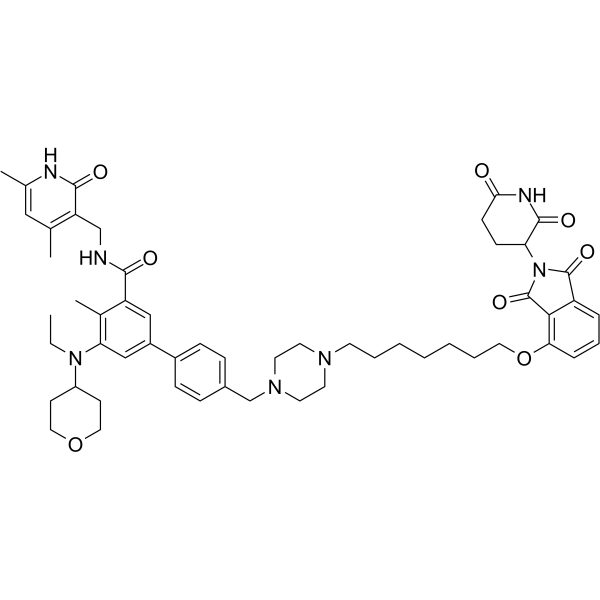
-
GC65147
PROTAC PARP1 degrader
Le dégradeur PROTAC PARP1 est un dégradeur PARP1 basé sur le ligand MDM2 E3. Il induit un clivage important de PARP1 et une mort cellulaire programmée. Le dégradeur PROTAC PARP1 À 10 μM À 24 h inhibe la lignée cellulaire MDA-MB-231 avec une IC50 de 6,12 μM.
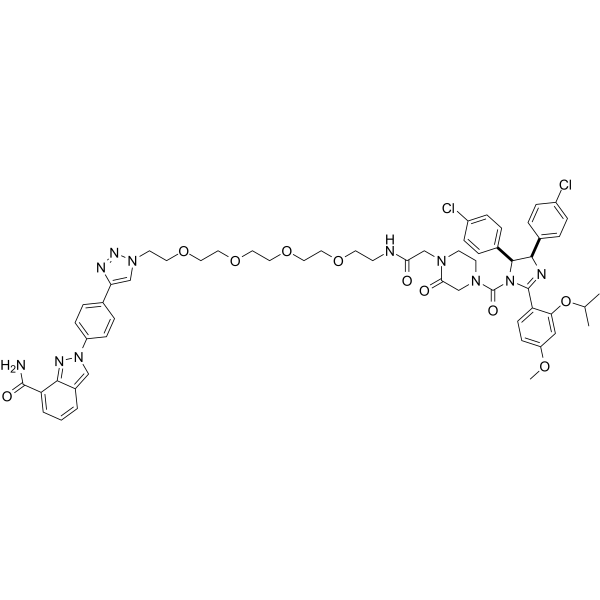
-
GC33418
PROTAC Sirt2 Degrader-1
PROTAC Sirt2 Degrader-1 est un PROTAC basé sur SirReal, agit comme un dégradeur de Sirt2, composé d'un inhibiteur de Sirt2 très puissant et isotype sélectif, d'un lieur et d'un véritable ligand Cereblon pour l'ubiquitine ligase E3. PROTAC Sirt2 Degrader-1 montre une IC50 de 0,25 μM pour Sirt2, sans effet sur Sirt1/Sirt3 (IC50s>100 μM).
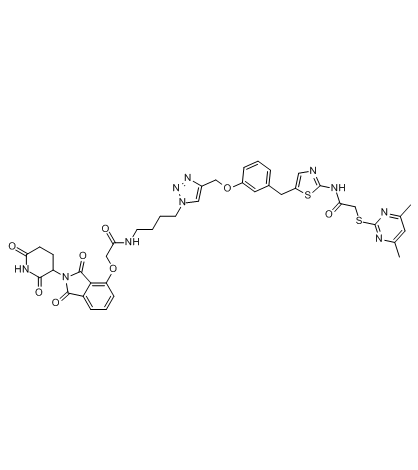
-
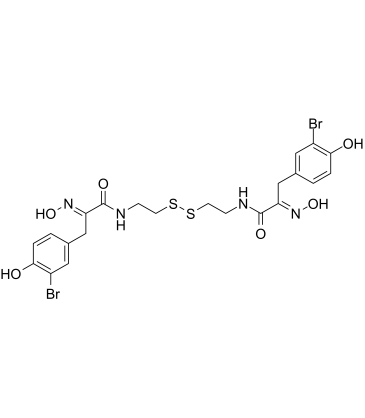
GC34742
Protosappanin A
La protosappanine A (PTA), un ingrédient immunosuppresseur et un composé biphényle majeur isolé de Caesalpinia sappan L, supprime la voie d'inflammation dépendante de JAK2/STAT3 en régulant À la baisse la phosphorylation de JAK2 et STAT3.
-
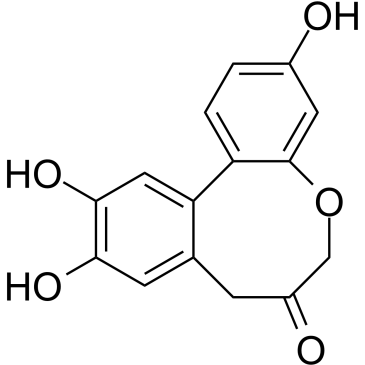
GC60310
Psammaplin A
La psammapline A, un métabolite marin, est un puissant inhibiteur des HDAC et des ADN méthyltransférases.
-
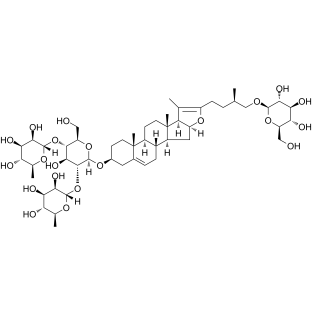
GC38839
Pseudoprotodioscin
La pseudoprotodioscine, un furostanoside, inhibe les niveaux de SREBP1/2 et de microARN 33a/b et réduit l'expression des gènes concernant la synthèse du cholestérol et des triglycérides.
-
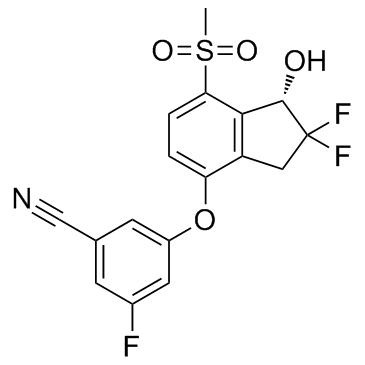
GC32680
PT-2385
PT-2385 est un inhibiteur sélectif de HIF-2α avec un Ki inférieur À 50 nM.
-
GC65333
PT2399
PT2399 est un antagoniste puissant et sélectif de HIF-2α, qui se lie directement au domaine HIF-2α PAS B avec une IC50 de 6 nM. PT2399 affiche une puissante activité antitumorale in vivo.

-
GC37034
PT2977
PT2977; MK-6482
PT2977 (PT2977) est un HIF-2α actif et sélectif par voie orale ; inhibiteur avec une IC50 de 9 nM. PT2977, en tant que HIF-2α de deuxième génération ; inhibiteur, augmente la puissance et améliore le profil pharmacocinétique. PT2977 est un traitement potentiel pour le carcinome À cellules claires du rein (ccRCC).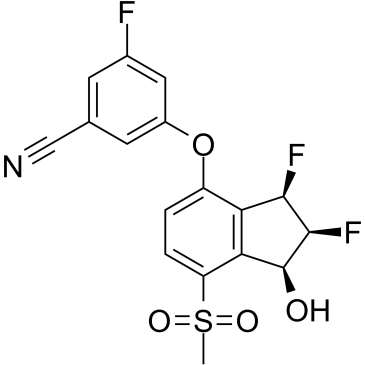
-
GC15120
PTP Inhibitor I
α-Bromo-4-hydroxyacetophenone|2-Bromo-4'-hydroxyacetophenone|4-Hydroxyphenacyl bromide|Protein Tyrosine Phosphatase Inhibitor I|SHP-1 Inhibitor II
PTP Inhibitor I un inhibiteur de PTP1B, avec un Ki de 42 μM.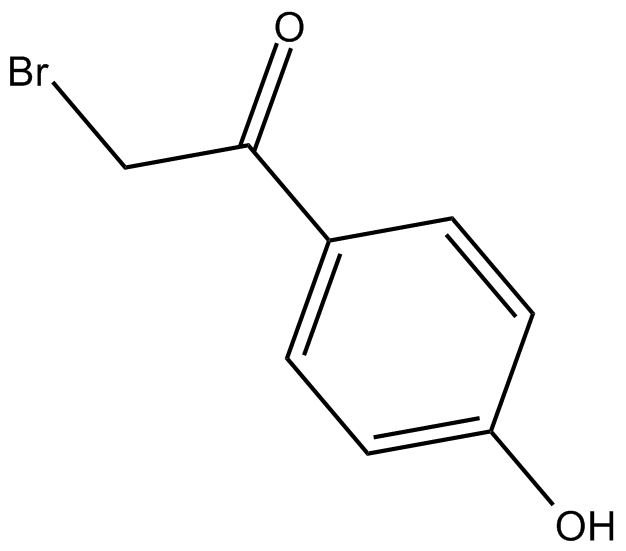
-
GC15804
PTP Inhibitor II
α-Bromo-4’-methoxyacetophenone|ω-Bromo-4’-methoxyacetophenone|4-(Bromoacetyl)anisole|4-Methoxyphenacyl bromide|NSC 129010|Protein Tyrosine Phosphatase Inhibitor II
PTP Inhibitor II est un inhibiteur de la protéine tyrosine phosphatase (PTP), avec un effet anti-angiogénique.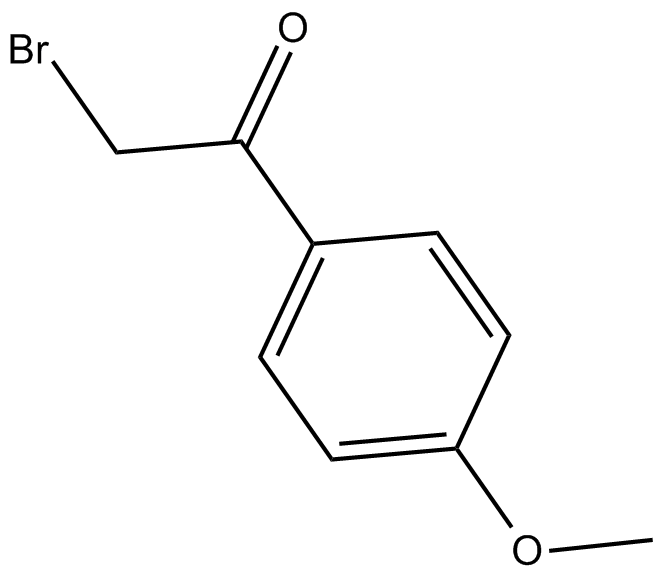
-
GC15969
PTP Inhibitor IV
Protein Tyrosine Phosphatase Inhibitor IV
PTP inhibitor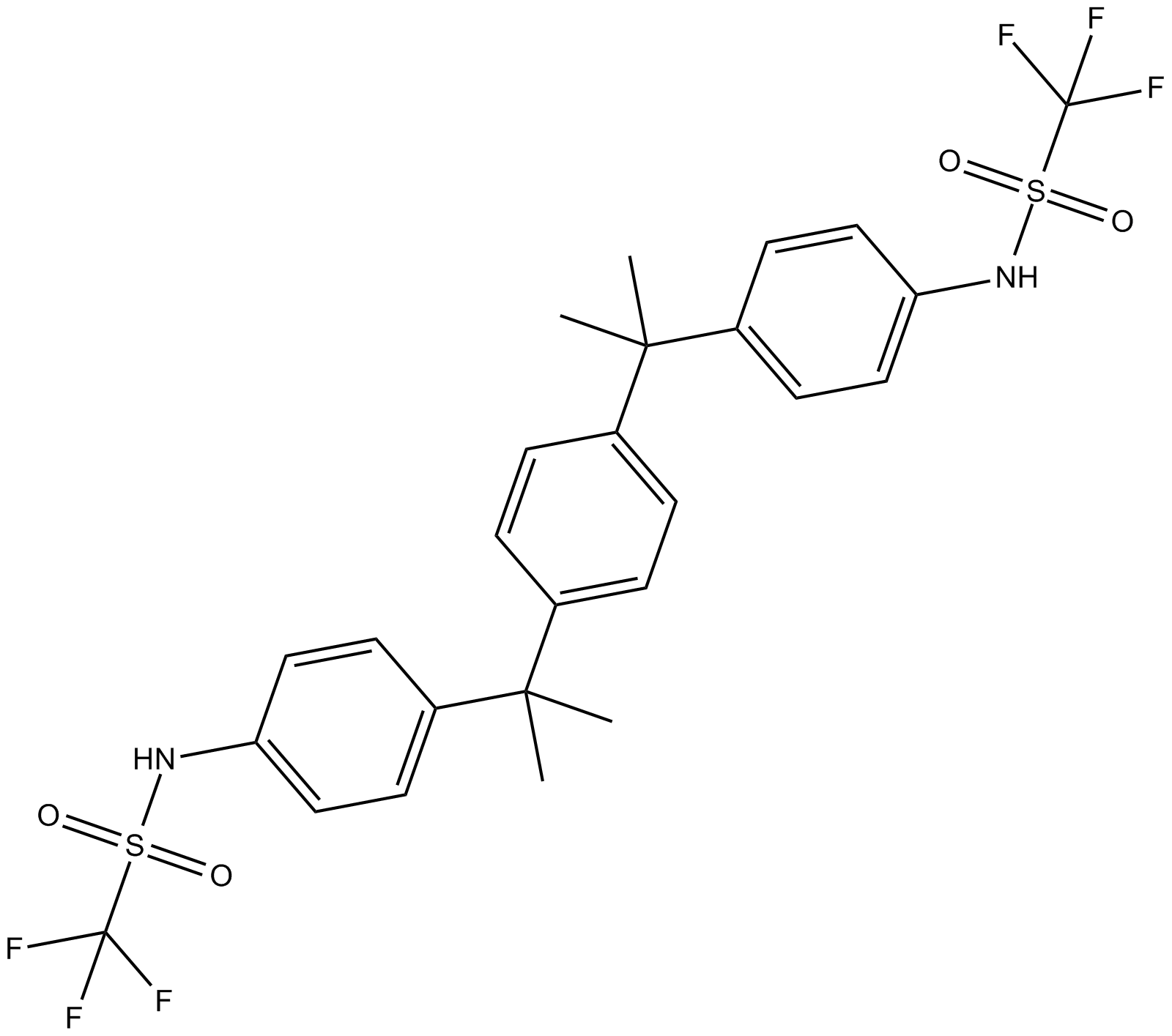
-
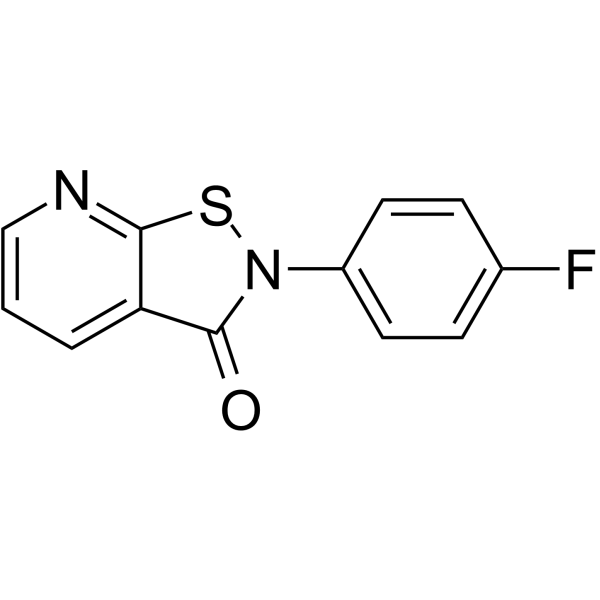
GC63405
PU139
Le PU139 est un puissant inhibiteur pan-histone de l'acétyltransférase (HAT). PU139 bloque les HAT Gcn5, p300/CBP-associated factor (PCAF), CREB (cAMP response element-binding) protein (CBP) et p300 avec des IC50 de 8,39, 9,74, 2,49 et 5,35 μM, respectivement.
-
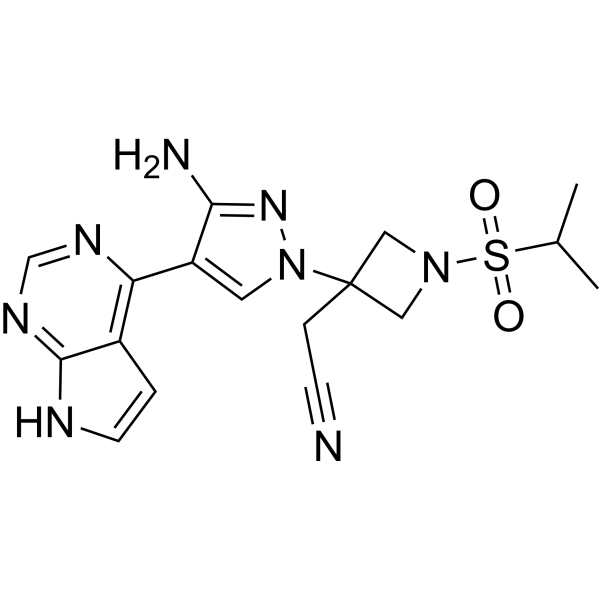
GC69780
Pumecitinib
Pumecitinib est un inhibiteur de JAK ayant une activité anti-inflammatoire.
-
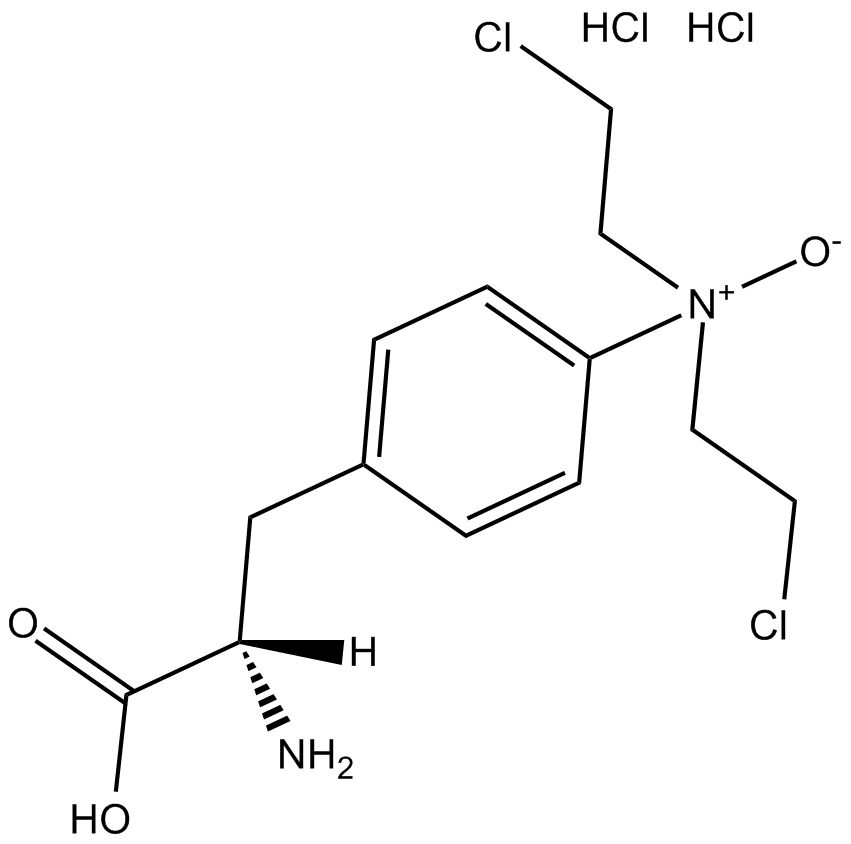
GC11031
PX-478 2HCl
PX-478 2HCl est un inhibiteur oral de HIF-1α avec des activités antitumorales puissantes. PX-478 2HCl peut traverser la barrière hémato-encéphalique.
-
GC11525
Pyridone 6
CMP 6, JanusAssociated Kinase Inhibitor I, Pyridone 6
A pan-JAK inhibitor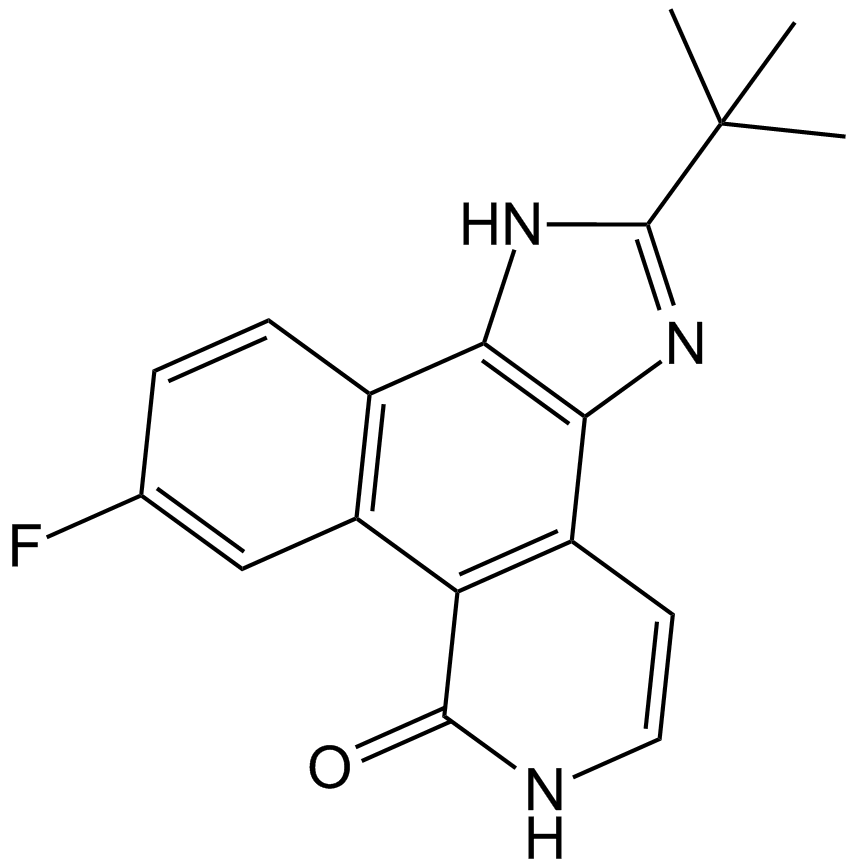
-
GC11095
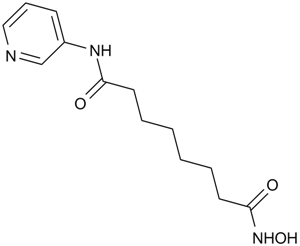
Pyroxamide
A HDAC inhibitor
-
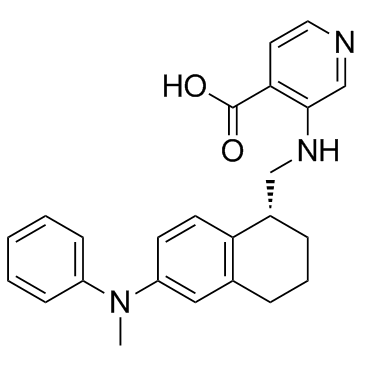
GC32699
QC6352
QC6352 est un inhibiteur de KDM4C sélectif et puissant disponible par voie orale avec une IC50 de 35 nM.
-
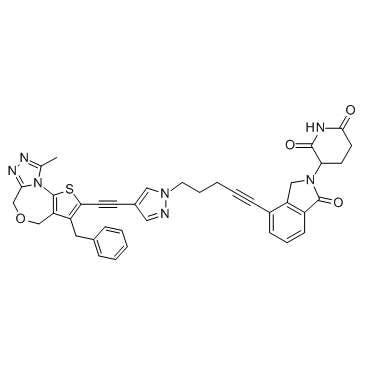
GC33217
QCA570
QCA570 est un PROTAC connecté par des ligands pour Cereblon et BET, avec une IC50 de 10 nM pour BRD4 BD1 Protein.
-
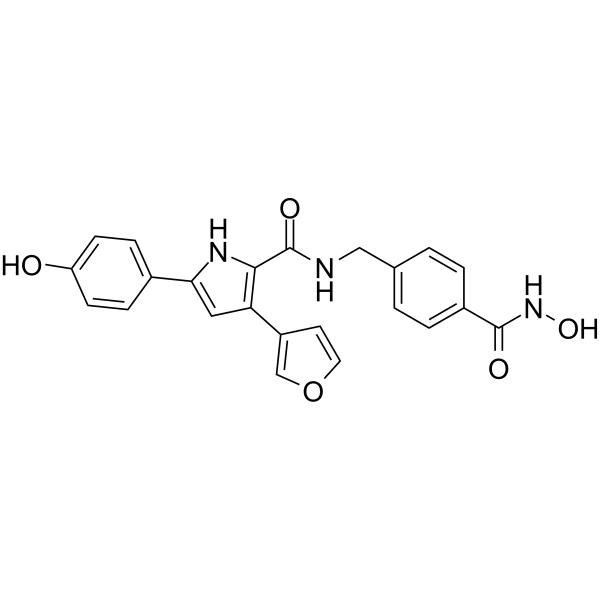
GC62411
QTX125
QTX125 est un inhibiteur de HDAC6 puissant et hautement sélectif. QTX125 présente une excellente sélectivité par rapport aux autres HDAC. QTX125 a des effets antitumoraux.
-
GC63730
QTX125 TFA
QTX125 TFA est un inhibiteur HDAC6 puissant et hautement sélectif. QTX125 TFA présente une excellente sélectivité par rapport aux autres HDAC. QTX125 a des effets antitumoraux.

-
GC65201
Quercetagetin
6-hydroxy Quercetin, NSC 115916
La quercétagetine (6-hydroxyquercétine) est un flavonoÏde.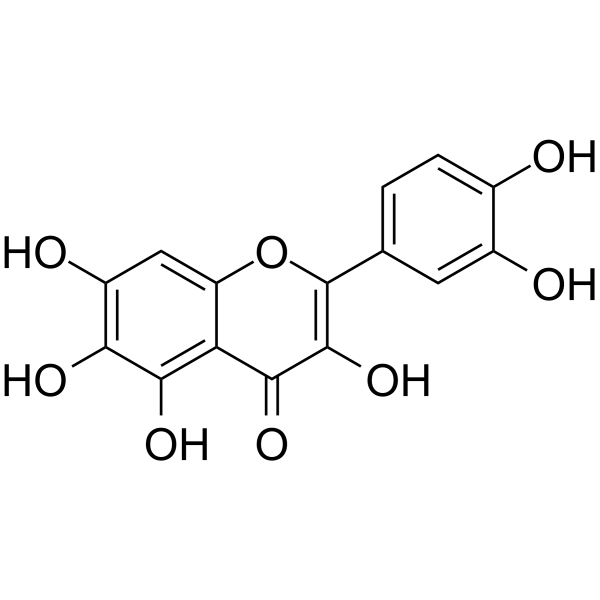
-
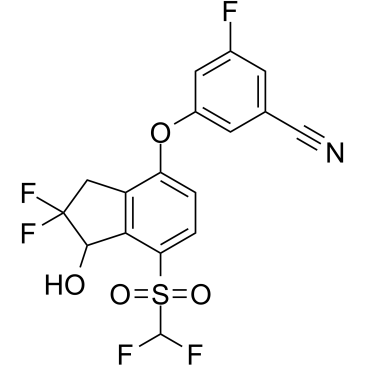
GC37066
Rac-PT2399
Rac-PT2399 (composé 10e), le racémate de PT2399, agit comme un inhibiteur puissant et spécifique du facteur 2a inductible par l'hypoxie (HIF-2α) avec une IC50 de 0,01 μM.
-
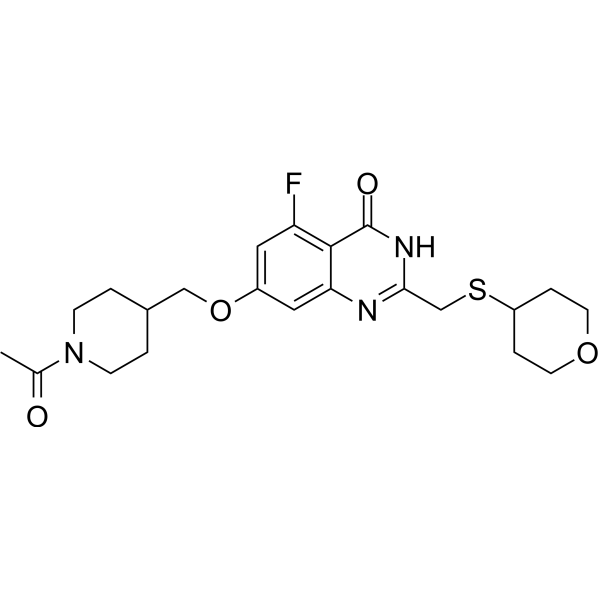
GC69804
RBN-3143
RBN-3143 est un inhibiteur compétitif efficace de PARP14 catalysant la NAD+ avec une valeur IC50 de 4 nM. RBN-3143 inhibe l'ADP-ribosylation médiée par PARP14 et stabilise PARP14 dans les lignées cellulaires. RBN-3143 est utilisé pour la recherche sur l'inflammation pulmonaire.
-
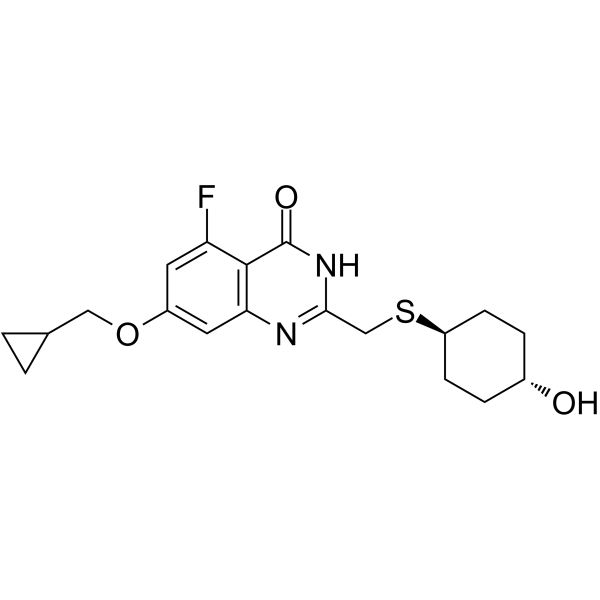
GC62473
RBN012759
RBN012759 est un inhibiteur puissant, sélectif et actif par voie orale de PARP14, avec une IC50 <3 nM.
-
GC33290
Remetinostat (SHP-141)
SHP-141
Remetinostat (SHP-141) (SHP-141) est un inhibiteur À base d'acide hydroxamique des enzymes histone désacétylase (HDAC) qui est en cours de développement pour le traitement du lymphome cutané À cellules T.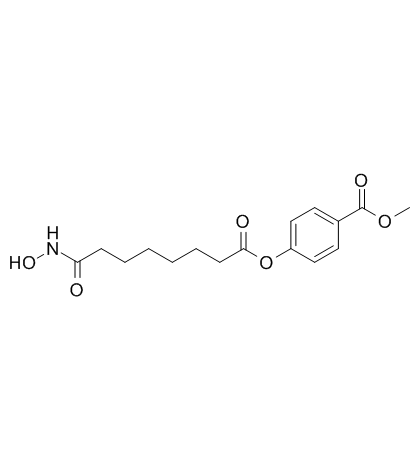
-
GC11430
Remodelin
La remodeline, un inhibiteur spécifique de la N-acétyltransférase NAT10, peut améliorer les phénotypes cellulaires du syndrome de Hutchinson-Gilford Progeria (HGPS). La remodeline agit dans une voie indépendante de la progérine et du FTI, en ciblant et en inhibant NAT10. NAT10 est une protéine ayant une activité d'acétylation des histones et principalement identifiée comme étant impliquée dans la régulation de l'activité de la télomérase.
-
GC15879
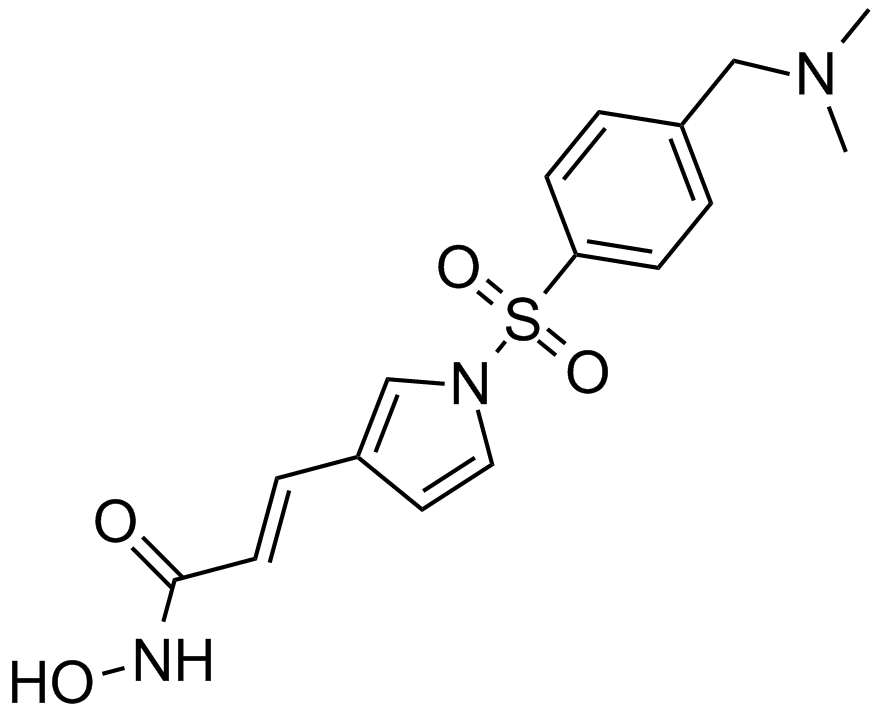
Resminostat (RAS2410)
RAS2410; 4SC-201
-
GC14243
Resminostat hydrochloride
4SC-201, RAS2410
An orally bioavailable HDAC inhibitor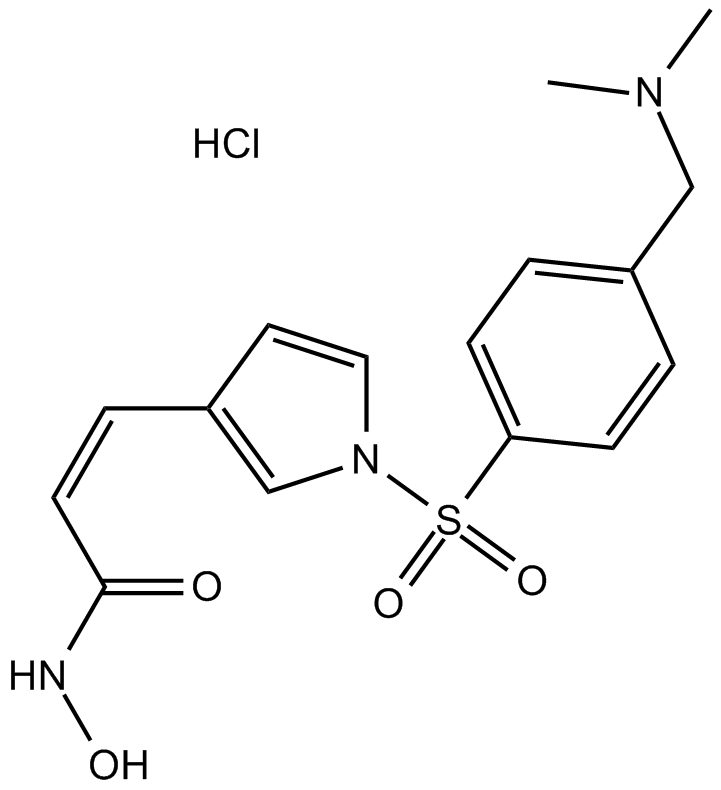
-
GC14553
Resveratrol
(E)Resveratrol
Un polyphénol aux activités biologiques diverses.
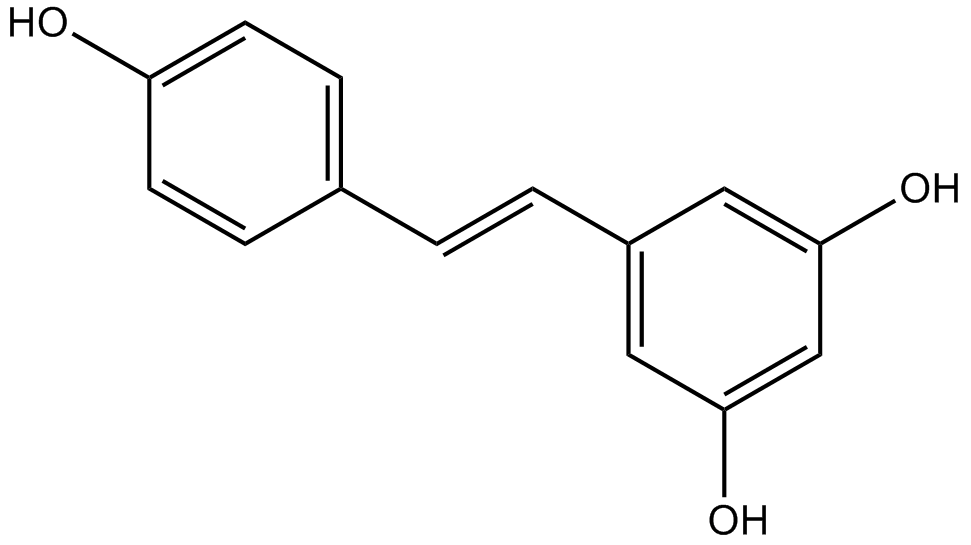
-
GC39081
Reticuline
La réticuline présente des effets anti-inflammatoires via les voies de signalisation JAK2/STAT3 et NF-κB.
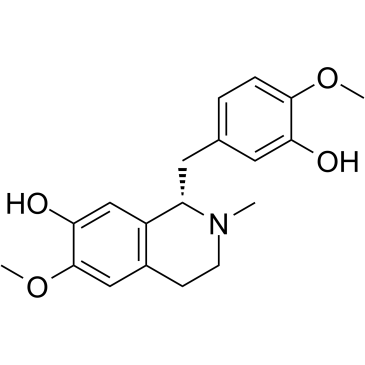
-
GC14651
Reversine
A purine derivative
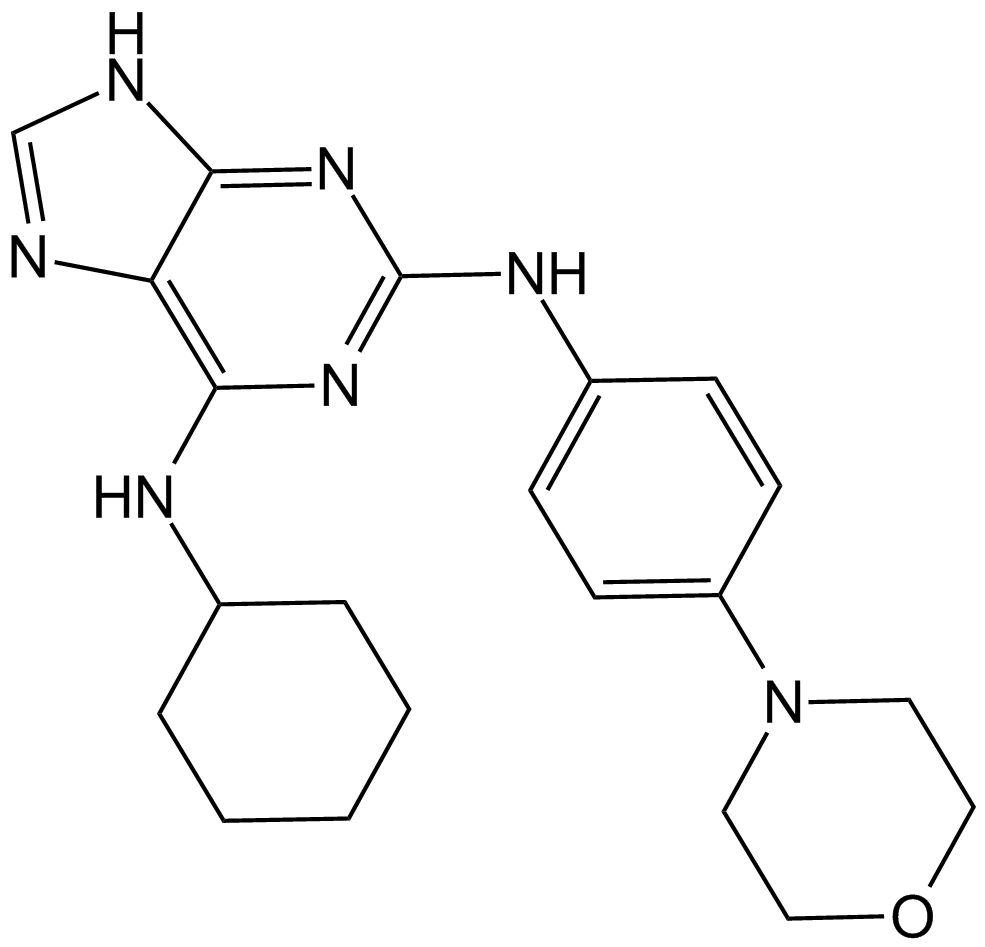
-
GC13398
RG 108
N-Phthalyl-L-tryptophan,RG108
DNA methyltransferase inhibitor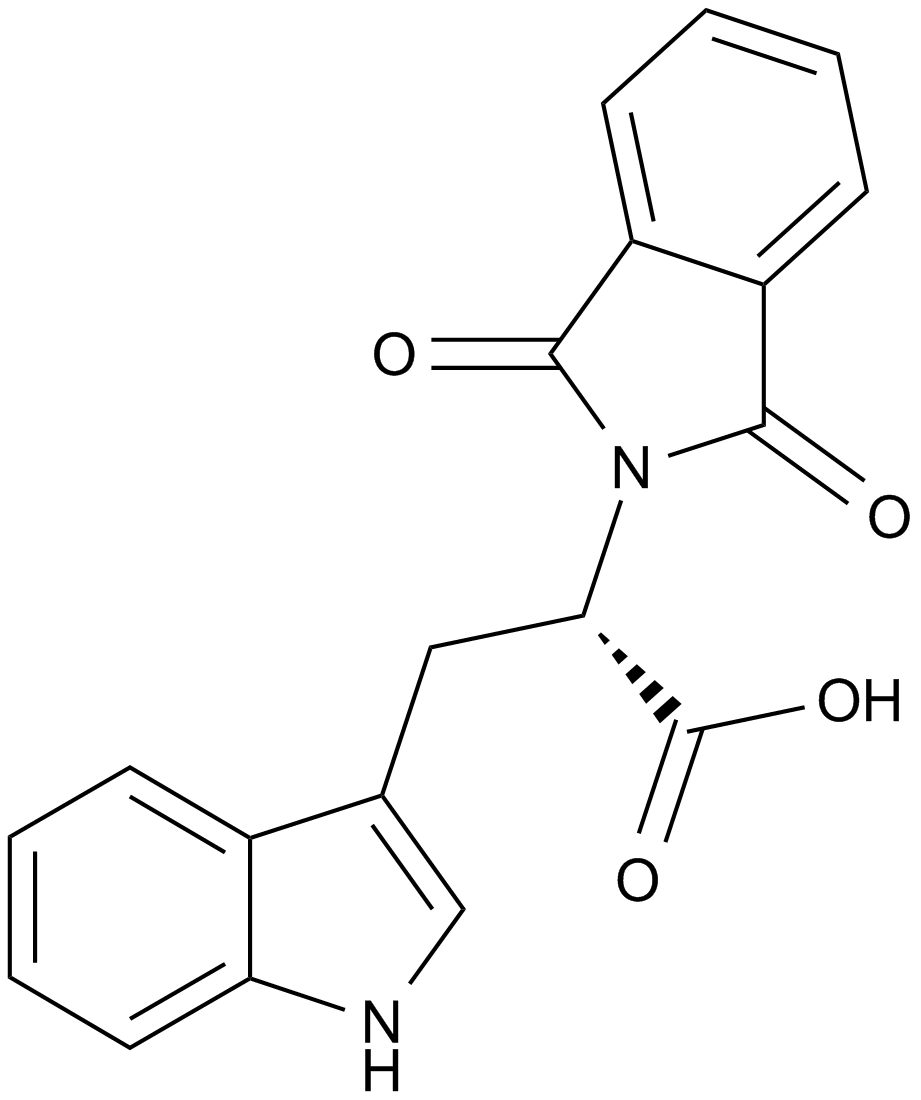
-
GC10423
RG2833
RG-2833
An HDAC inhibitor
-
GC37522
RGB-286638
RGB-286638 est un inhibiteur de CDK qui inhibe l'activité kinase de la cycline T1-CDK9, de la cycline B1-CDK1, de la cycline E-CDK2, de la cycline D1-CDK4, de la cycline E-CDK3 et de p35-CDK5 avec des IC50 de 1, 2, 3 , 4, 5 et 5 nM, respectivement; inhibe également GSK-3β, TAK1, Jak2 et MEK1, avec des IC50 de 3, 5, 50 et 54 nM.
-
GC37523
RGB-286638 free base
RGB-286638 est un inhibiteur de CDK qui inhibe l'activité kinase de la cycline T1-CDK9, de la cycline B1-CDK1, de la cycline E-CDK2, de la cycline D1-CDK4, de la cycline E-CDK3 et de p35-CDK5 avec des IC50 de 1, 2, 3 , 4, 5 et 5 nM, respectivement; inhibe également GSK-3β, TAK1, Jak2 et MEK1, avec des IC50 de 3, 5, 50 et 54 nM.
-
GC14285
RGFP966
RGFP 966;RGFP-966
RGFP966 est un inhibiteur HDAC3 hautement sélectif avec une IC50 de 80 nM et ne montre aucune inhibition des autres HDAC À des concentrations allant jusqu'À 15 μM. Le RGFP966 peut traverser la barrière hémato-encéphalique (BBB).
-
GC64499
RJW100
RJW100 est un puissant agoniste du récepteur hépatique homologue 1 (LRH-1, NR5A2) et du facteur stéroÏdogène 1 (SF-1, NR5A1) avec des pEC50 de 6,6 et 7,5, respectivement .
-
GC19505
RK-287107
Le RK-287107 est un inhibiteur de tankyrase puissant et spécifique avec des IC50 de 14,3 et 10,6 nM pour la tankyrase-1 et la tankyrase-2, respectivement. Le RK-287107 bloque la croissance des cellules cancéreuses colorectales.
-
GC10148
RN 1 dihydrochloride
LSD1 Inhibitor IV
Le dichlorhydrate de RN 1 est un inhibiteur puissant, pénétrant dans le cerveau, irréversible et sélectif de la déméthylase spécifique de la lysine 1 (LSD1) avec une IC50 de 70 nM.