DNA Damage/DNA Repair
- MTH1(4)
- PARP(84)
- ATM/ATR(30)
- DNA Alkylating(20)
- DNA Ligases(3)
- DNA Methyltransferase(25)
- DNA-PK(30)
- HDAC(136)
- Nucleoside Antimetabolite/Analogue(140)
- Telomerase(17)
- Topoisomerase(151)
- tankyrase(5)
- Antifolate(38)
- CDK(250)
- Checkpoint Kinase (Chk)(31)
- CRISPR/Cas9(9)
- Deubiquitinase(72)
- DNA Alkylator/Crosslinker(71)
- DNA/RNA Synthesis(400)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(23)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(61)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nombre del producto Información
-
GC30782
ACY-775
ACY-775 es un inhibidor potente y selectivo de la histona desacetilasa 6 (HDAC6) con una IC50 de 7,5nM.
-
GC30526
ACY-957
ACY-957 es un inhibidor selectivo y activo por vÍa oral de HDAC1 y HDAC2, con IC50 de 7 nM, 18 nM y 1300 nM contra HDAC1/2/3, respectivamente, y no muestra inhibiciÓn sobre HDAC4/5/6/7/8 /9.
-
GC17186
Acyclovir
El aciclovir (Aciclovir) es un potente agente antiviral activo por vÍa oral.
-
GC13432
Adenine
La adenina (6-aminopurina), una purina, es una de las cuatro bases nitrogenadas del Ácido nucleico del ADN. La adenina actÚa como un componente quÍmico del ADN y el ARN. La adenina también juega un papel importante en la bioquÍmica involucrada en la respiraciÓn celular, la forma tanto de ATP como de los cofactores (NAD y FAD) y la sÍntesis de proteÍnas.

-
GC11825
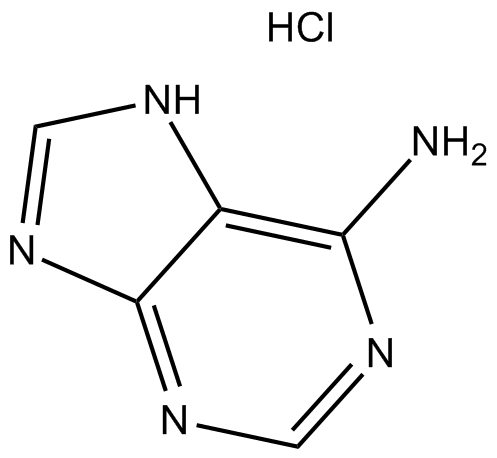
Adenine HCl
Adenine HCl (clorhidrato de 6-aminopurina), una purina, es una de las cuatro nucleobases en el Ácido nucleico del ADN. La adenina HCl actÚa como un componente quÍmico del ADN y el ARN. El clorhidrato de adenina también juega un papel importante en la bioquÍmica involucrada en la respiraciÓn celular, la forma tanto de ATP como de los cofactores (NAD y FAD) y la sÍntesis de proteÍnas.
-
GC17278
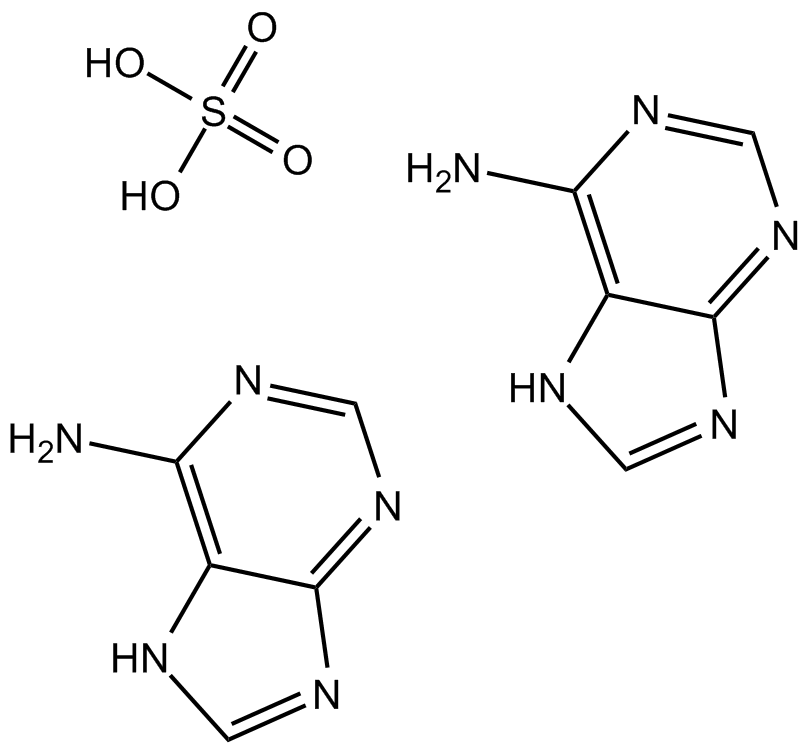
Adenine sulfate
El sulfato de adenina (hemisulfato de 6-aminopurina), una purina, es una de las cuatro bases nitrogenadas del Ácido nucleico del ADN. El sulfato de adenina actÚa como un componente quÍmico del ADN y el ARN. El sulfato de adenina también juega un papel importante en la bioquÍmica involucrada en la respiraciÓn celular, la forma tanto de ATP como de los cofactores (NAD y FAD) y la sÍntesis de proteÍnas.
-
GC67946
Adenine-d1
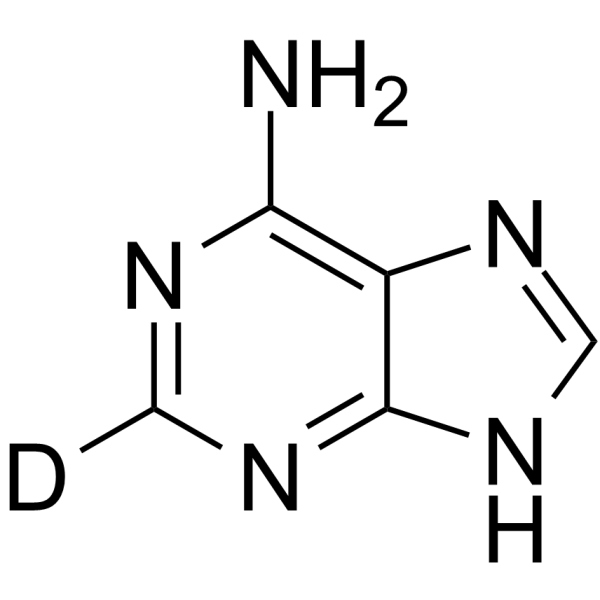
-
GC14106
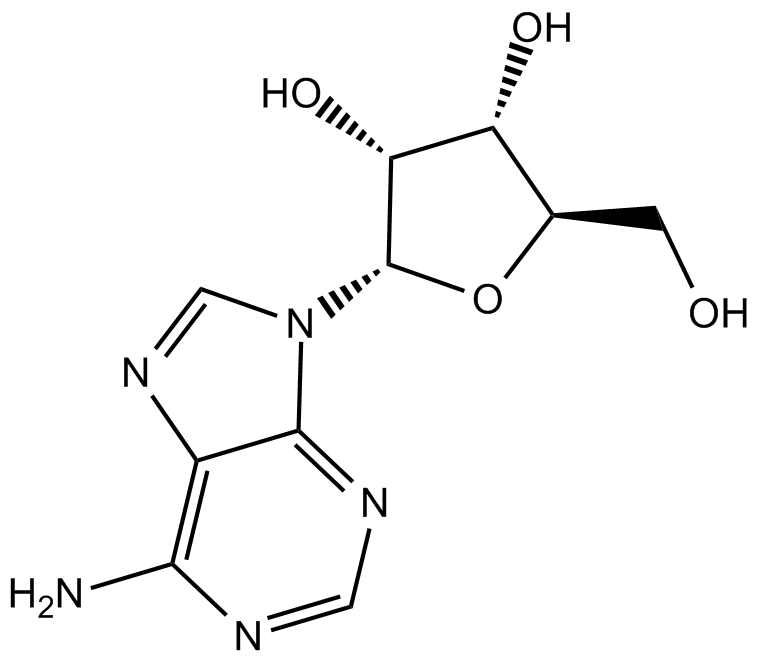
Adenosine
nucleósido
-
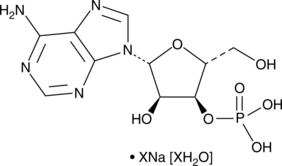
GC49004
Adenosine 3’-monophosphate (sodium salt hydrate)
A nucleotide
-
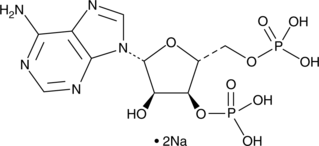
GC42732
Adenosine 3',5'-diphosphate (sodium salt)
La adenosina 3'5'-difosfato (sal de sodio) es un inhibidor de las sulfotransferasas de hidroxiesteroide.
-
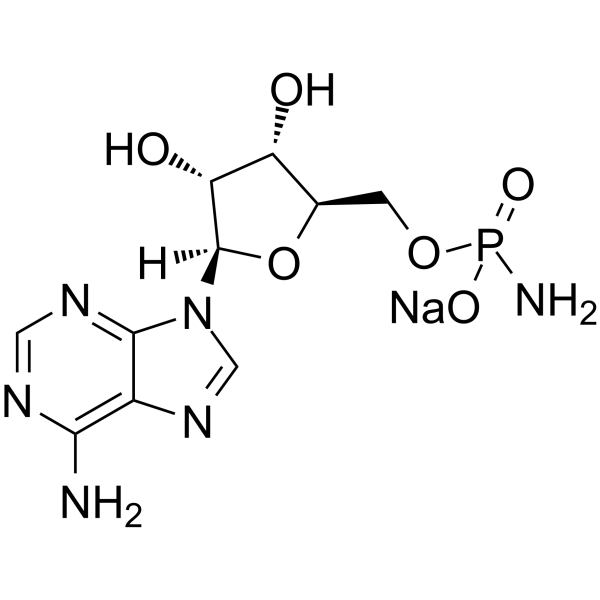
GC65462
Adenosine 5′-monophosphoramidate sodium
La adenosina 5′-monofosforamidato de sodio es un derivado de la adenosina y se puede utilizar como intermediario para la síntesis de nucleótidos.
-
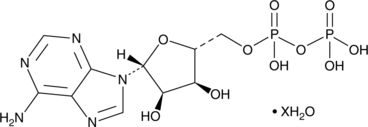
GC49285
Adenosine 5’-methylenediphosphate (hydrate)
An inhibitor of ecto-5’-nucleotidase
-
GC42733
Adenosine 5'-monophosphate (sodium salt hydrate)
Adenosine 5'-monophosphate (AMP) is a central nucleotide with functions in metabolism and cell signaling.
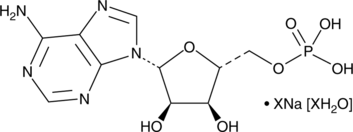
-
GC42734
Adenosine 5'-phosphosulfate (sodium salt)
Adenosine 5'-phosphosulfate is an ATP and sulfate competitive inhibitor of ATP sulfurylase in humans, S.
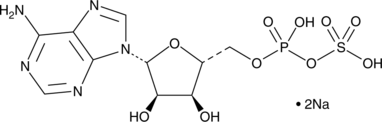
-
GC49231
Adenylosuccinic Acid (ammonium salt)
A purine nucleotide and an intermediate in the purine nucleotide cycle
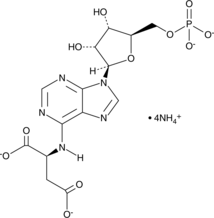
-
GC65330
AES-135
AES-135, un inhibidor pan-HDAC a base de Ácido hidroxÁmico, prolonga la supervivencia en un modelo de ratÓn ortotÓpico de cÁncer de pÁncreas. AES-135 inhibe HDAC3, HDAC6, HDAC8 y HDAC11 con IC50 que van desde 190-1100 nM.
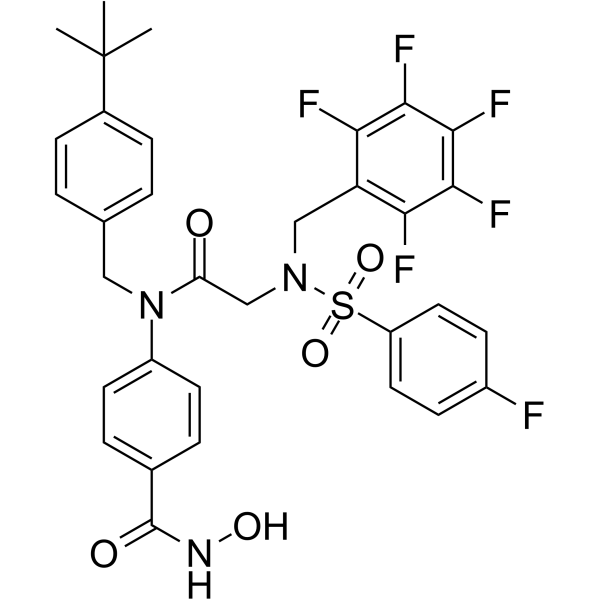
-
GC46810
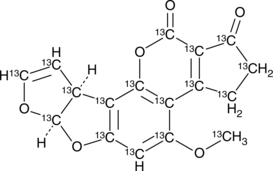
Aflatoxin B1-13C17
An internal standard for the quantification of aflatoxin B1
-
GC62470
AG-636
AG-636 es un inhibidor de la dihidroorotato deshidrogenasa (DHODH) potente, reversible, selectivo y activo por vÍa oral con una IC50 de 17 nM. AG-636 tiene fuertes efectos anticancerÍgenos.
-
GC42765
Aldehyde Reactive Probe (trifluoroacetate salt)
El ADN es continuamente dañado por agentes endógenos y ambientales, lo que lleva a la formación de sitios abásicos (apurínicos/apirimidínicos, AP) que son disruptivos para la síntesis del ADN.
-
GC14858
Aldoxorubicin
La aldoxorrubicina (INNO-206) es un profÁrmaco de la doxorrubicina (inhibidor de la ADN topoisomerasa II) que se une a la albÚmina, que se libera de la albÚmina en condiciones Ácidas. La aldoxorrubicina (INNO-206) tiene potentes actividades antitumorales en varias lÍneas de células cancerosas y en modelos de tumores murinos.
-
GC15841
Alsterpaullone
La alsterpaulona (9-nitropaulona) es un potente inhibidor de CDK, con IC50 de 35 nM, 15 nM, 200 nM y 40 nM para CDK1/ciclina B, CDK2/ciclina A, CDK2/ciclina E y CDK5/p35, respectivamente. La alsterpaulona también compite con ATP por unirse a GSK-3alpha/GSK-3beta con IC50 de ambos 4 nM. Alsterpaulone tiene actividad antitumoral y posee potencial para el estudio en trastornos neurodegenerativos y proliferativos. La alsterpaulona induce la apoptosis en la lÍnea celular de leucemia.
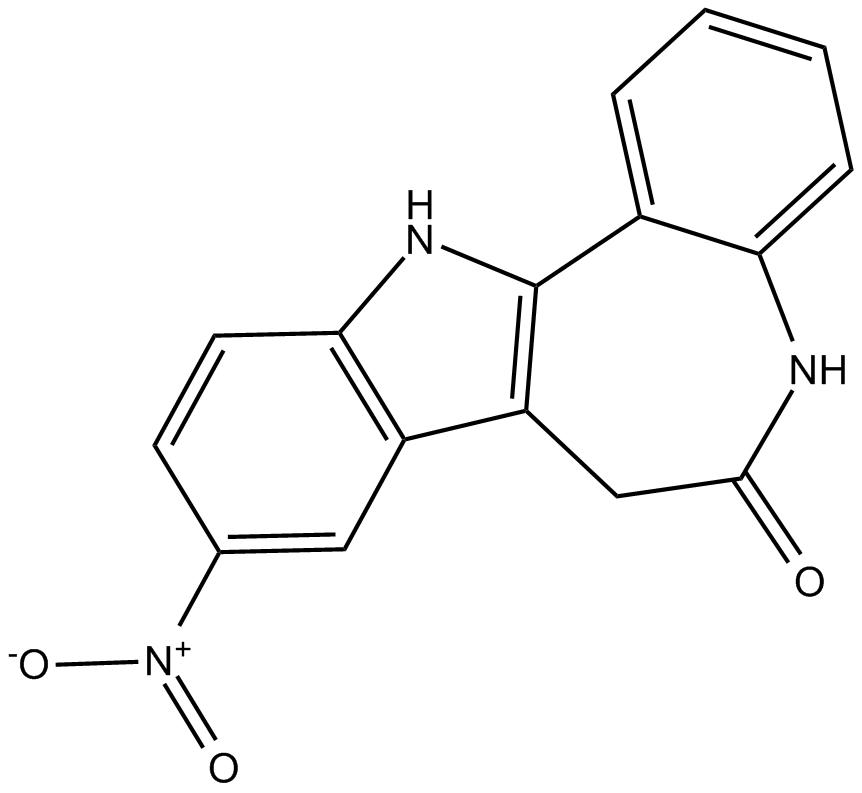
-
GC67984
Alteminostat
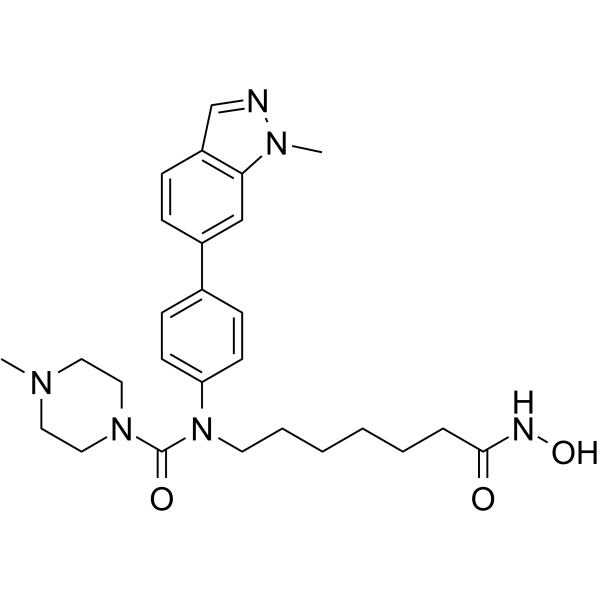
-
GC10779
Alternariol
El alternariol es una micotoxina producida por especies de Alternaria.
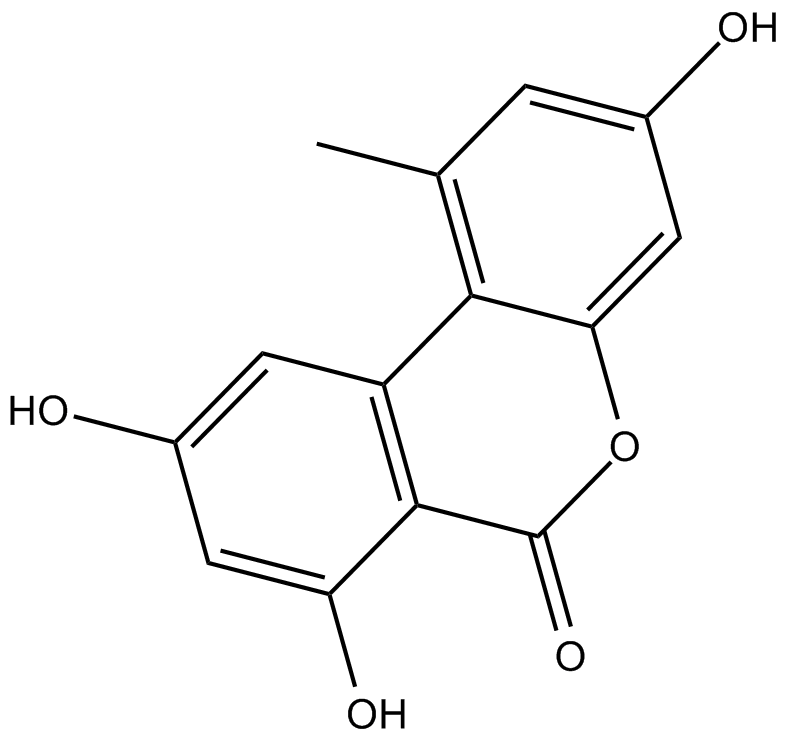
-
GC18437
Alternariol monomethyl ether
El éter monometÍlico de alternariol, aislado de las raÍces de Anthocleista djalonensis (Loganiaceae), es un importante marcador taxonÓmico de la especie vegetal.
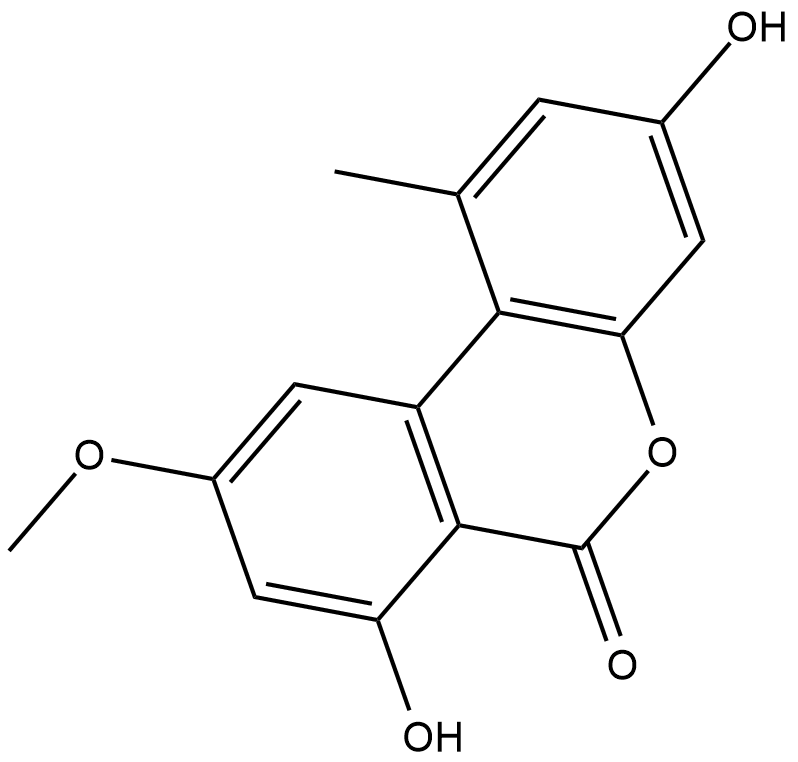
-
GC49039
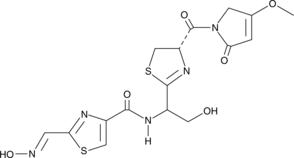
Althiomycin
A thiazole antibiotic
-
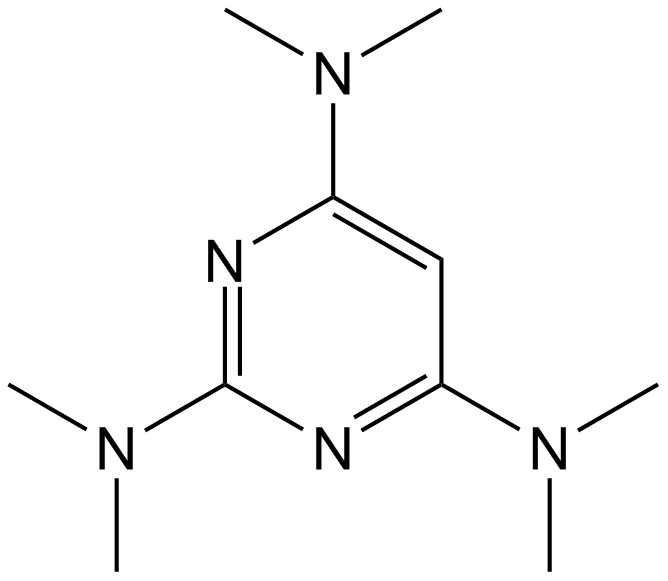
GC14564
Altretamine
La altretamina es un agente antineoplÁsico alquilante.
-
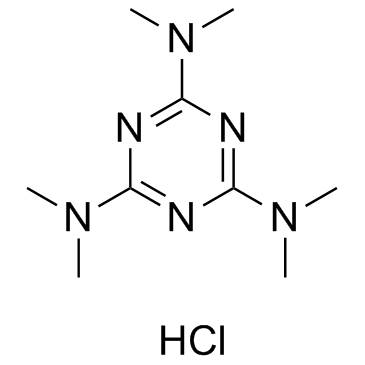
GC35310
Altretamine hydrochloride
El clorhidrato de altretamina es un agente antineoplÁsico alquilante.
-
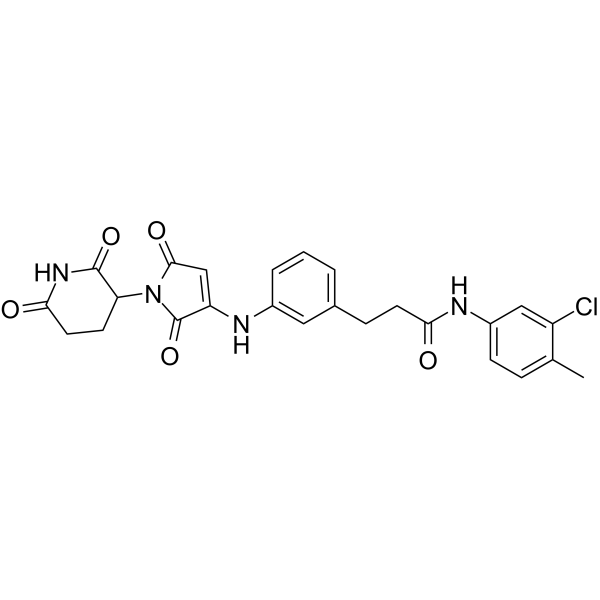
GC64638
ALV1
ALV1 es un potente degradador de Ikaros y Helios.
-
GC64818
AMA-37
AMA-37, un anÁlogo de arilmorfolina, es un inhibidor de ADN-PK competitivo con ATP, con valores de IC50 de 0,27 μM (DNA-PK), 32 μM (p110α), 3,7 μM (p110β) y 22 μM (p110γ), respectivamente.
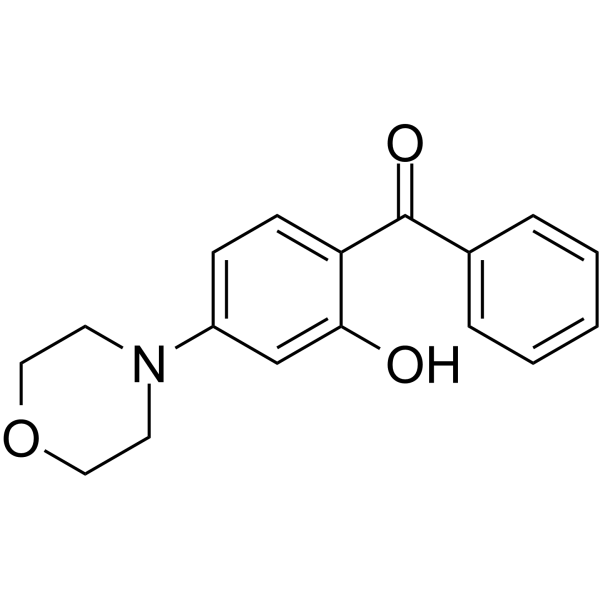
-
GC42783
Ametantrone
La ametantrona (NSC 196473) es un agente antitumoral que se intercala en el ADN e induce la ruptura del ADN mediada por la topoisomerasa II (TOP2).
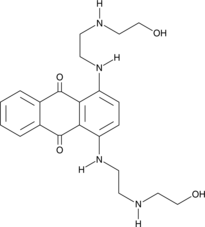
-
GC50366
AMG 18 hydrochloride
El clorhidrato de AMG 18 (clorhidrato de AMG-18) es un IRE1α monoselectivo; inhibidor que alostéricamente atenÚa IRE1α Actividad RNasa con una IC50 de 5,9 nM.
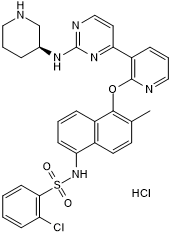
-
GC14899
AMG 548
AMG 548, un inhibidor de p38α selectivo y activo por vía oral (Ki \u003d 0,5 nM), se muestra ligeramente selectivo sobre p38β (Ki \u003d 36 nM) y \u003e1000 veces selectivo contra p38γ y p38δ.
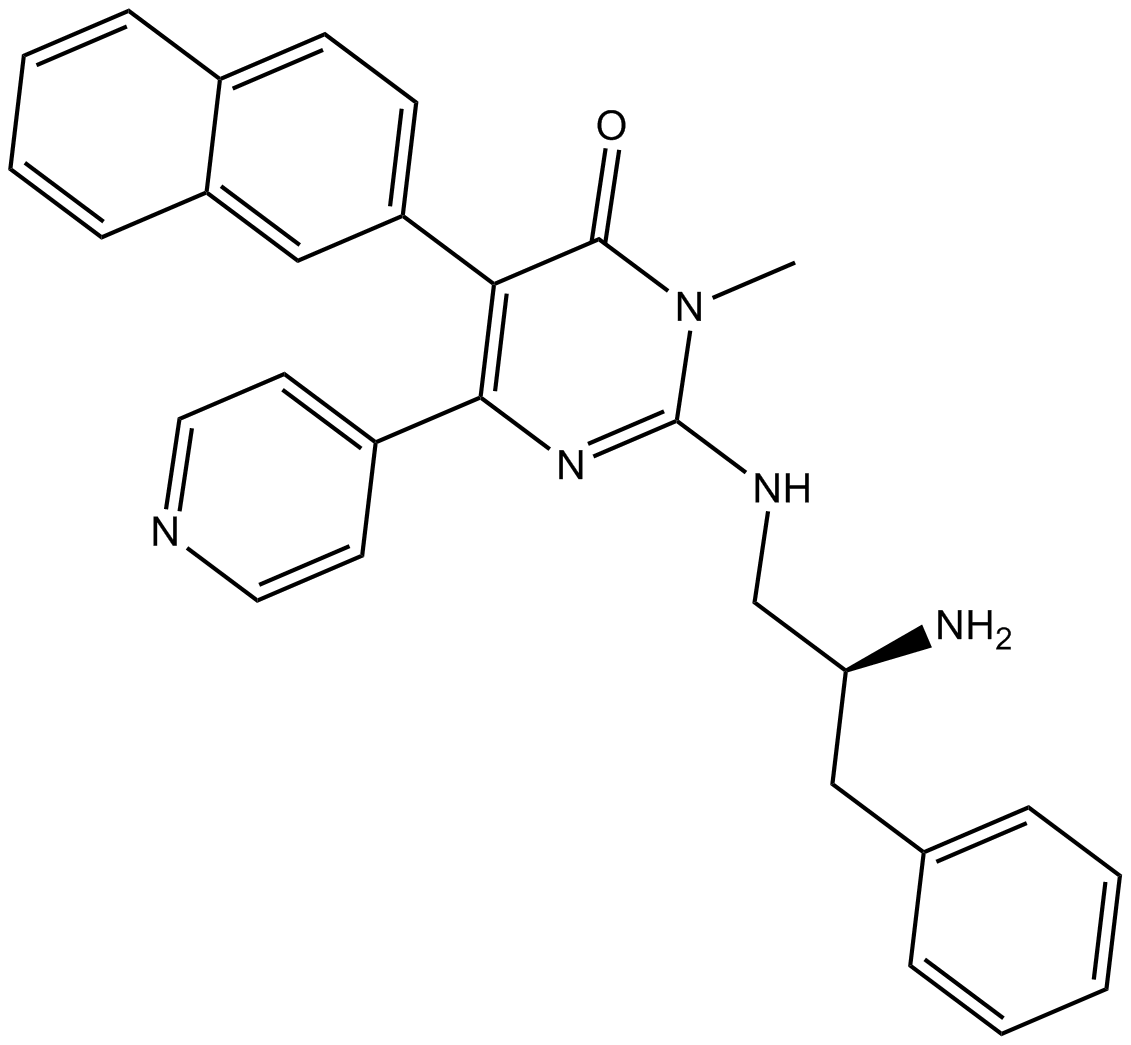
-
GC14974
AMG 925
AMG 925 es un inhibidor dual FLT3/CDK4 potente, selectivo y disponible por vÍa oral con IC50 de 2±1 nM y 3±1 nM, respectivamente.
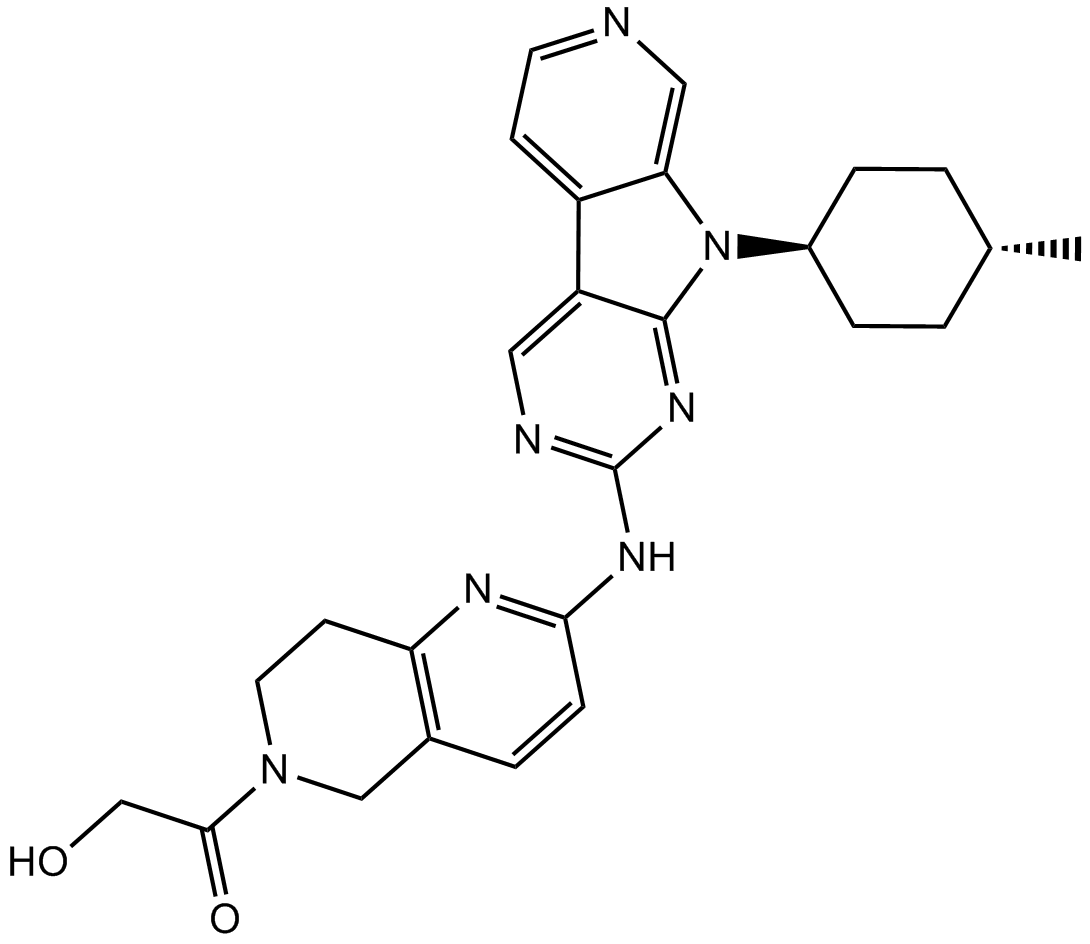
-
GC35315
AMG 925 HCl
AMG 925 HCl es un inhibidor dual de FLT3/CDK4 potente, selectivo y disponible por vÍa oral con IC50 de 2±1 nM y 3±1 nM, respectivamente.

-
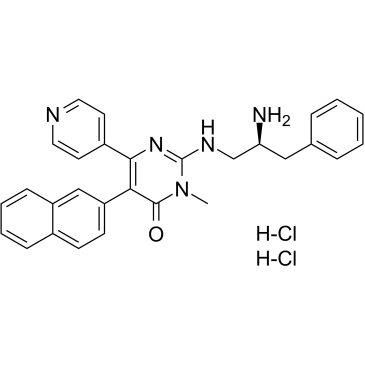
GC38518
AMG-548 dihydrochloride
El diclorhidrato de AMG-548, un inhibidor de p38α selectivo y activo por vÍa oral (Ki = 0,5 nM), se muestra ligeramente selectivo sobre p38β (Ki = 36 nM) y >1000 veces selectivo contra p38γ y p38δ.
-
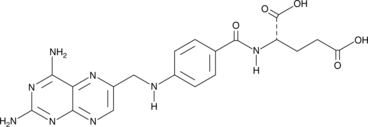
GC42789
Aminopterin
La aminopterina (Ácido 4-aminofÓlico), el derivado 4-amino del Ácido fÓlico, es un antagonista del Ácido fÓlico.
-
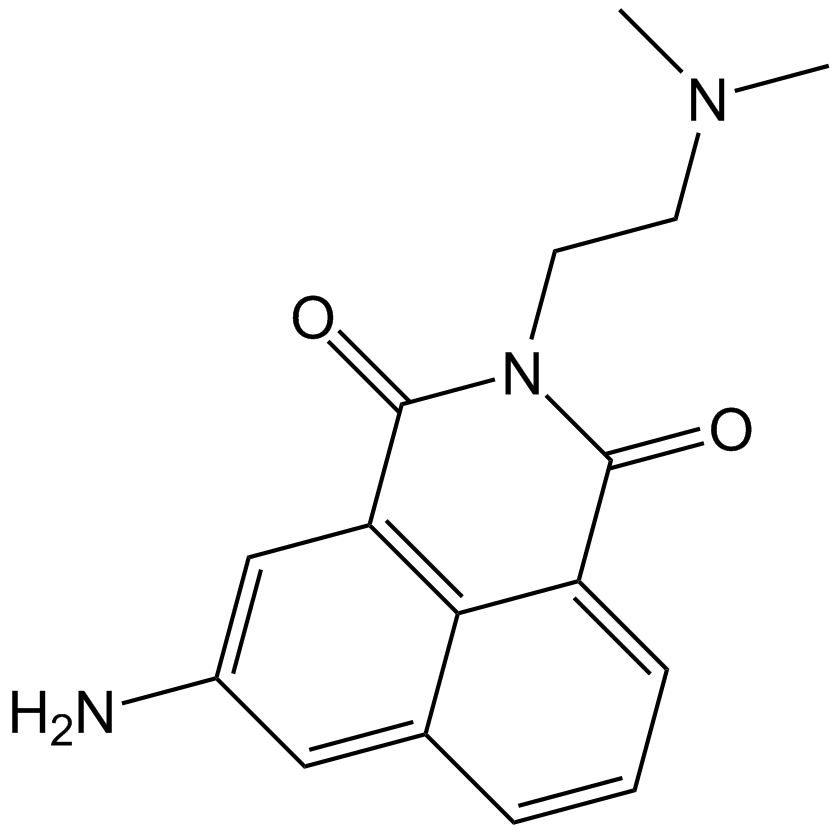
GC11019
Amonafide
Amonafide es un inhibidor de la topoisomerasa II y un intercalador de ADN que induce la seÑalizaciÓn apoptÓtica al bloquear la uniÓn de Topo II al ADN.
-
GC15644
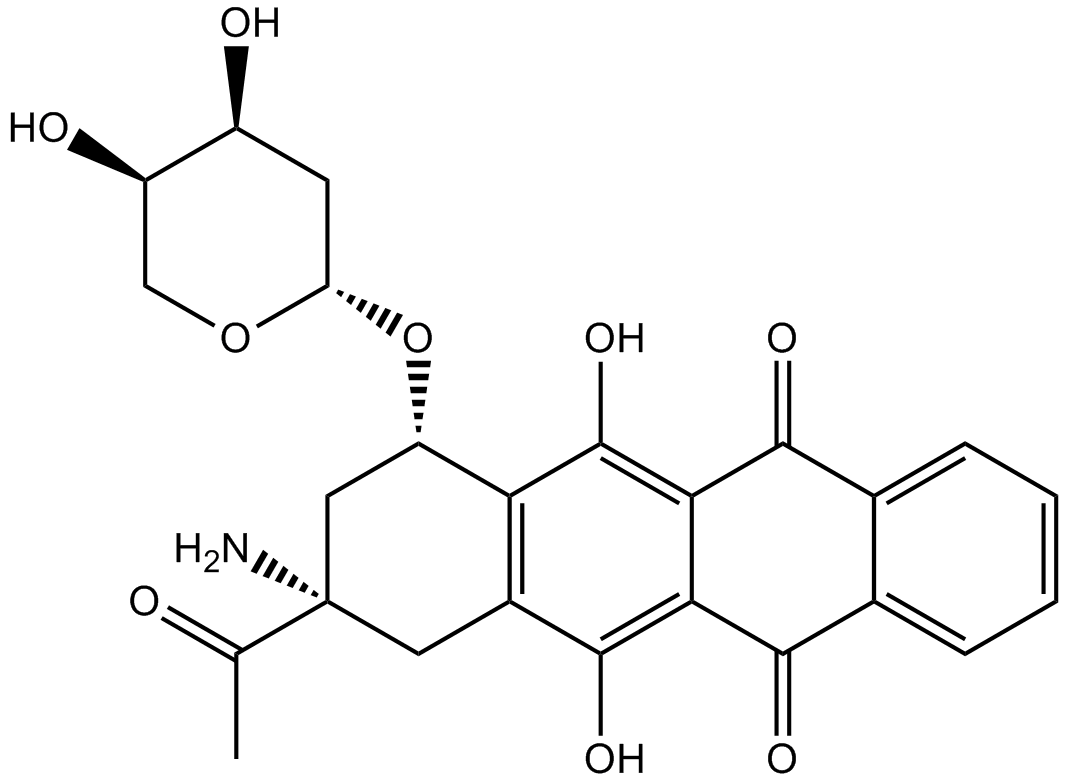
Amrubicin
A synthetic anthracycline antibiotic. It inhibits DNA topoisomerase II. Antineoplastic.
-
GC12326
Amsacrine
La amsacrina (m-AMSA; acridinil anisidida) es un inhibidor de la topoisomerasa II y actÚa como un agente antineoplÁsico que puede intercalarse en el ADN de las células tumorales.

-
GC11747
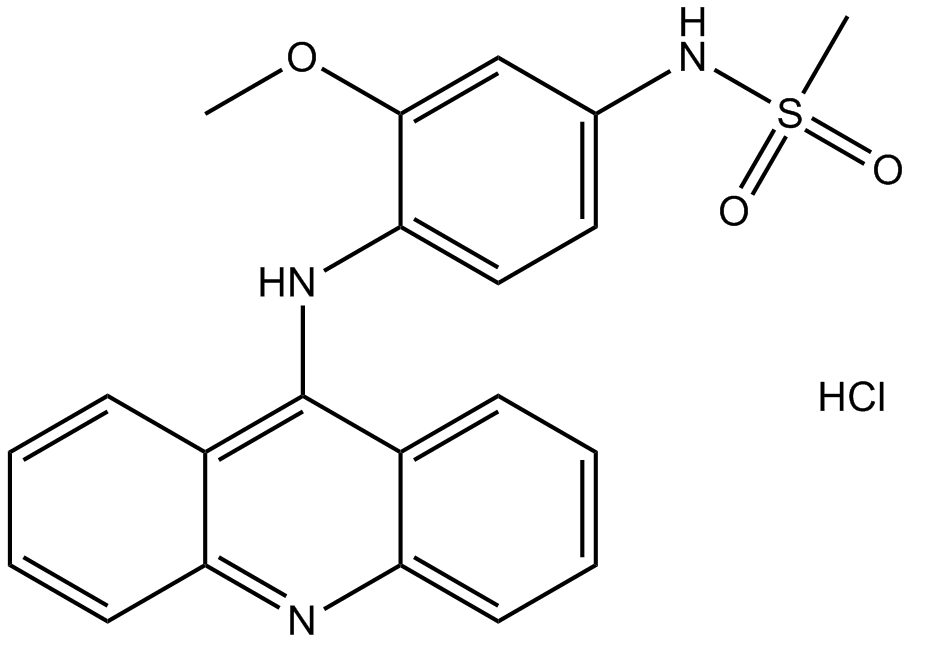
Amsacrine hydrochloride
El clorhidrato de amsacrina (clorhidrato de m-AMSA; clorhidrato de anisidida de acridinilo) es un inhibidor de la topoisomerasa II y actÚa como un agente antineoplÁsico que puede intercalarse en el ADN de las células tumorales.
-
GC39284
ANI-7
ANI-7 es un activador de la vÍa del receptor de hidrocarburos de arilo (AhR). ANI-7 inhibe el crecimiento de mÚltiples células cancerosas e inhibe potente y selectivamente el crecimiento de células de cÁncer de mama MCF-7 con un GI50 de 0,56 μM. ANI-7 induce monooxigenasas que metabolizan CYP1 mediante la activaciÓn de la vÍa AhR, y también induce daÑo en el ADN, activaciÓn del punto de control de la quinasa 2 (Chk2), detenciÓn del ciclo celular en fase S y muerte celular en lÍneas celulares de cÁncer de mama sensibles.
-
GC11559
Anisomycin
Agonista de JNK, potente y específico.
-
GC68667
Anticancer agent 73
El agente anticancerígeno 73 (compuesto CIB-3b) es un medicamento contra el cáncer que se dirige a la proteína de unión al ARN TAR 2 (TRBP) y destruye su interacción con Dicer. El agente anticancerígeno 73 puede reequilibrar el perfil de expresión de miARNs oncogénicos o supresores tumorales. El agente anticancerígeno 73 tiene efectos inhibidores sobre la proliferación y metástasis de células hepáticas cancerosas (HCC) tanto in vitro como in vivo.
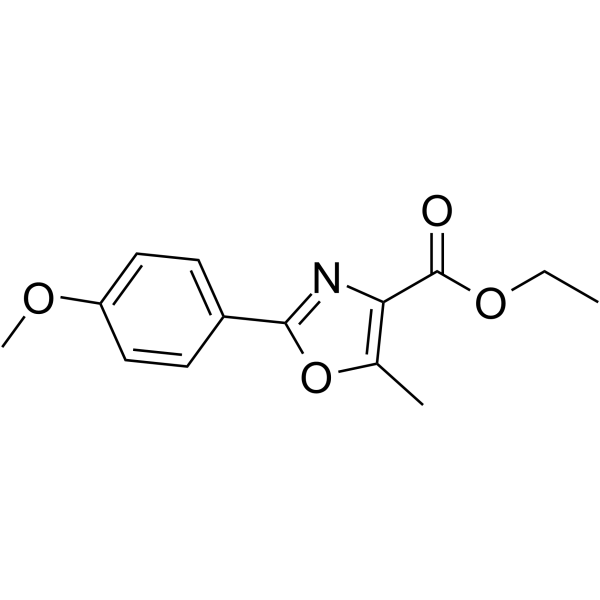
-
GC40032
Antipain (hydrochloride)
Antipain (clorhidrato) es un inhibidor de la proteasa aislado de Actinomycetes.
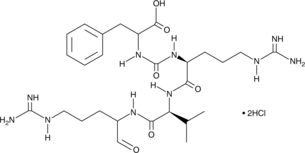
-
GC68448
AOH1160
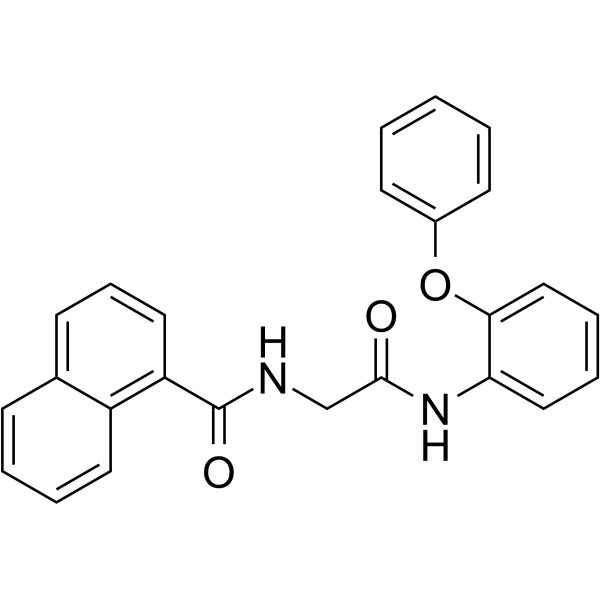
-
GC67876
APE1-IN-1

-
GC17139
APY29
APY29, un inhibidor competitivo de ATP, es un modulador alostérico de IRE1α que inhibe la autofosforilaciÓn de IRE1α al unirse al bolsillo de uniÓn de ATP con IC50 de 280 nM. APY29 actÚa como un ligando que activa alostéricamente el dominio de RNasa adyacente a IRE1α.
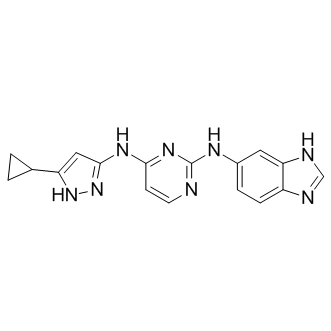
-
GC45385
Ara-G
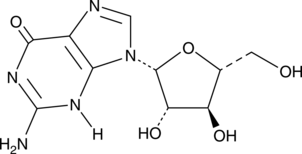
-
GC62425
ARN-21934
ARN-21934 es un potente inhibidor de penetraciÓn de la barrera hematoencefÁlica (BBB) altamente selectivo para la topoisomerasa II humana α sobre β. ARN-21934 inhibe la relajaciÓn del ADN con una IC50 de 2 μM en comparaciÓn con el agente anticancerÍgeno etopÓsido (IC50 = 120 μM). ARN-21934 exhibe un perfil farmacocinético in vivo favorable y es un compuesto lÍder prometedor para la investigaciÓn contra el cÁncer.
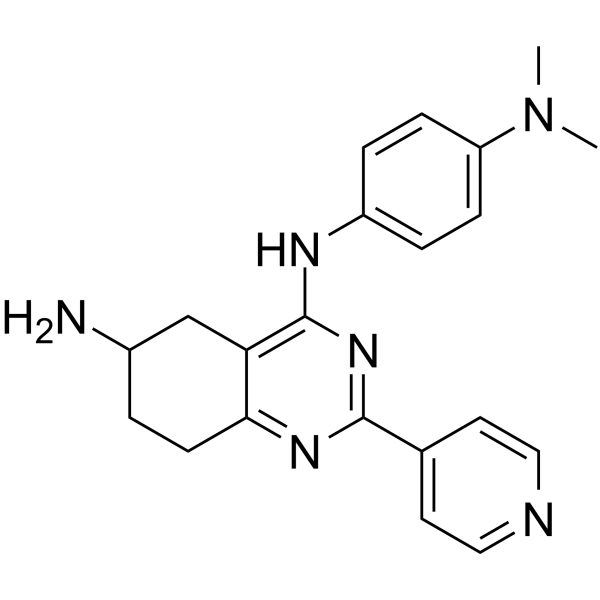
-
GC46880
ARN24139
A topoisomerase II poison
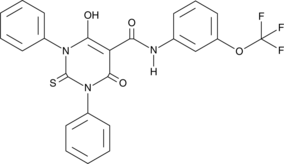
-
GC38736
AS2863619
AS2863619 permite la conversiÓn de células T efectoras/de memoria especÍficas de antÍgeno en células T reguladoras Foxp3+ (Treg) para el tratamiento de diversas enfermedades inmunolÓgicas.
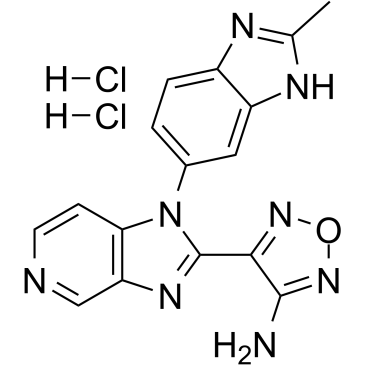
-
GC38737
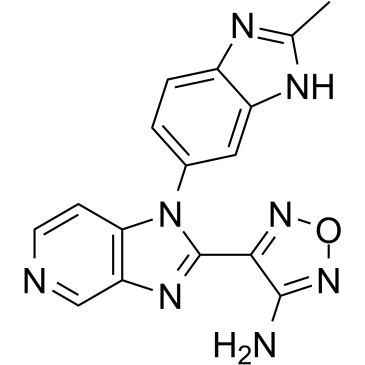
AS2863619 free base
La base libre AS2863619 permite la conversiÓn de células T efectoras/de memoria especÍficas de antÍgeno en células T reguladoras Foxp3+ (Treg) para el tratamiento de diversas enfermedades inmunolÓgicas.
-
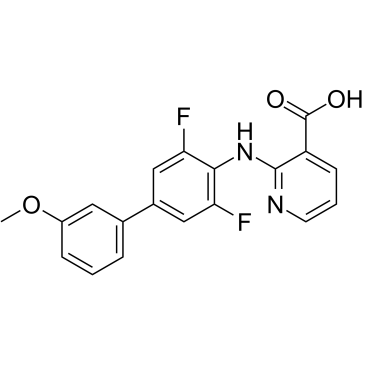
GC38463
ASLAN003
ASLAN003 (ASLAN003) es un potente inhibidor de la dihidroorotato deshidrogenasa (DHODH) activo por vÍa oral con una IC50 de 35 nM para la enzima DHODH humana. ASLAN003 inhibe la sÍntesis de proteÍnas mediante la activaciÓn de factores de transcripciÓn AP-1. ASLAN003 induce la apoptosis y prolonga sustancialmente la supervivencia en ratones con xenoinjerto de leucemia mieloide aguda (AML).
-
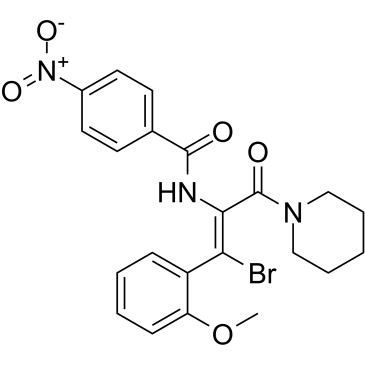
GC61821
AT-130
AT-130, un derivado de la fenilpropenamida, es un potente inhibidor no nucleÓsido de la replicaciÓn del virus de la hepatitis B (VHB).
-
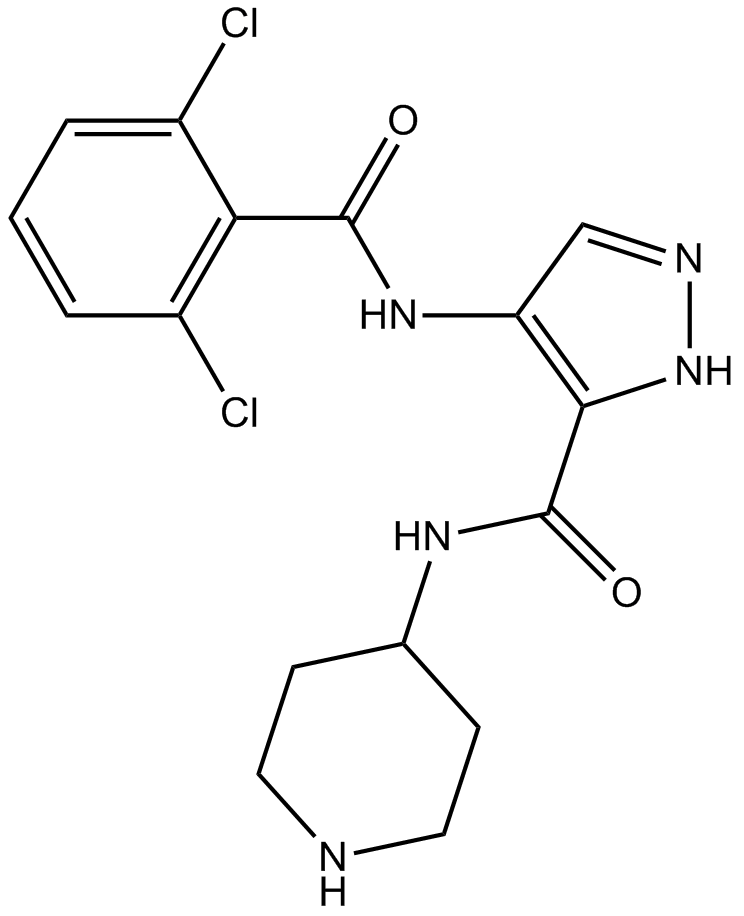
GC15870
AT7519
AT7519 (AT7519M) como un potente inhibidor de las CDK, con IC50 de 210, 47, 100, 13, 170 y <10 nM para CDK1, CDK2, CDK4 a CDK6 y CDK9, respectivamente.
-
GC13998
AT7519 Hydrochloride
A Cdk inhibitor
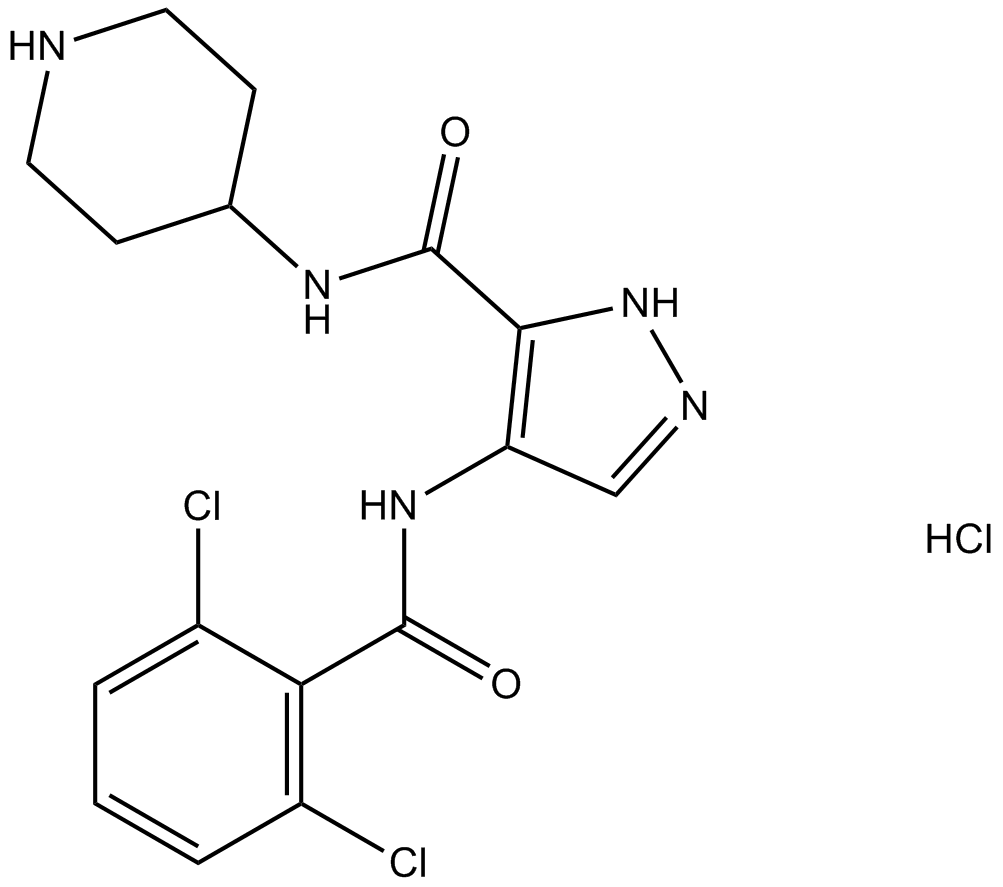
-
GC15597
AT7519 trifluoroacetate
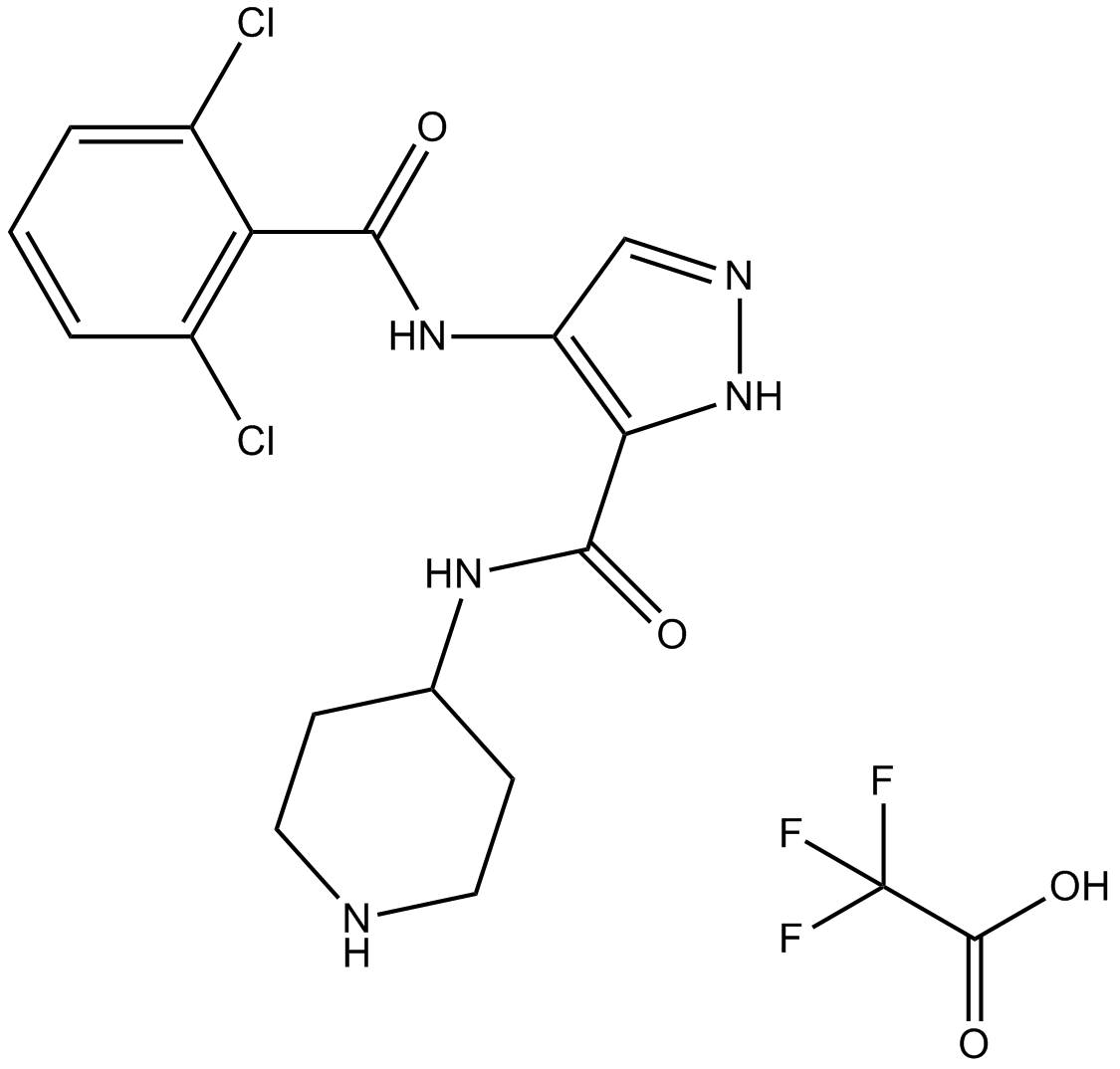
-
GC65327
ATM Inhibitor-5
ATM Inhibitor-5 [fÓrmula (1)] es un potente inhibidor de la serina/treonina proteÍna quinasa ATM (extraÍdo de la patente WO2022058351A1).
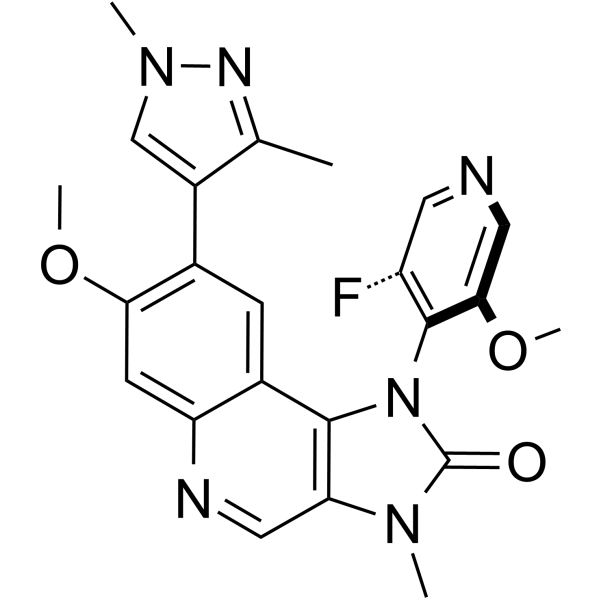
-
GC65514
ATR inhibitor 1
El inhibidor 1 de ATR es un inhibidor de ATR extraÍdo de la patente WO2015187451A1, compuesto I-1, tiene un valor de Ki por debajo de 1 μμ.
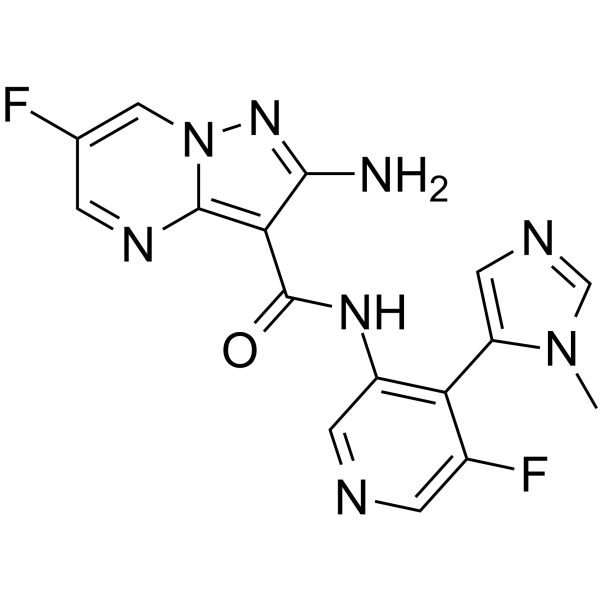
-
GC68707
ATR-IN-4
ATR-IN-4 es un inhibidor efectivo de ATR. ATR-IN-4 inhibe el crecimiento de células cancerosas de próstata humana DU145 y células cancerosas de pulmón humano NCI-H460, con IC50 respectivos de 130.9 nM y 41.33 nM (según se cita en la patente CN112142744A, compuesto 13).
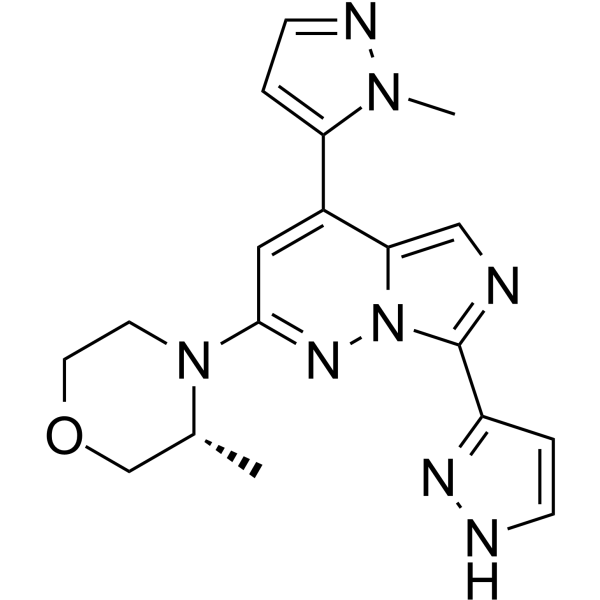
-
GC34422
Atuveciclib (BAY-1143572)
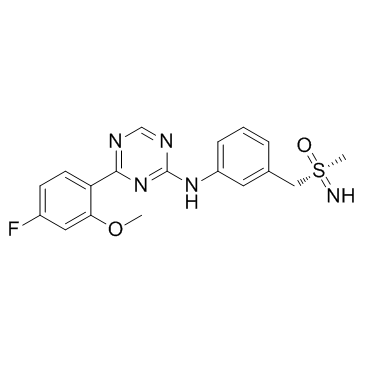
-
GC34059
Atuveciclib Racemate (BAY-1143572 Racemate)
Atuveciclib Racemate (BAY-1143572 Racemate) (BAY-1143572 Racemate) es la mezcla de racematos de Atuveciclib. Atuveciclib es un inhibidor oral potente y altamente selectivo de P-TEFb/CDK9 que suprime CDK9/CycT1 con una IC50 de 13 nM.
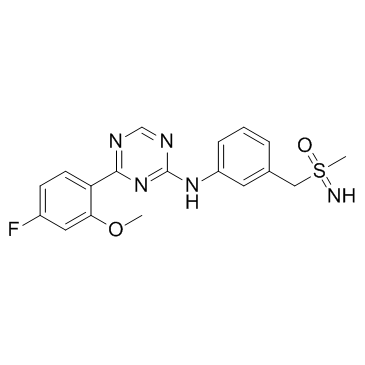
-
GC34421
Atuveciclib S-Enantiomer (BAY-1143572 S-Enantiomer)
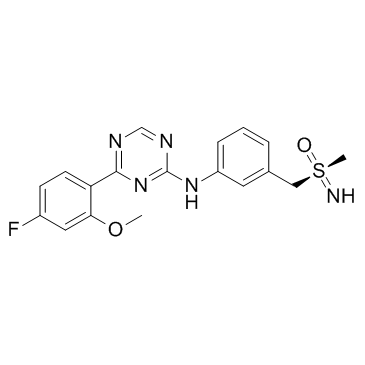
-
GC39699
Aurintricarboxylic acid
El Ácido aurintricarboxÍlico es un antagonista alostérico de potencia nanomolar con selectividad hacia los P2X1R y P2X3R sensibles a αβ-metileno-ATP, con IC50 de 8,6 nM y 72,9 nM para rP2X1R y rP2X3R, respectivamente.
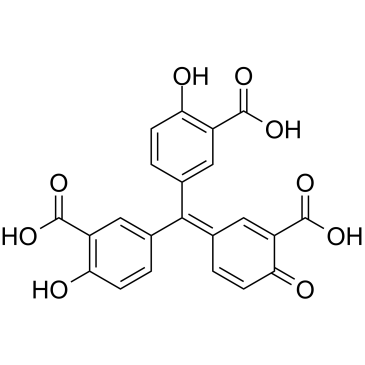
-
GC46895
Aurintricarboxylic Acid (ammonium salt)
A protein synthesis inhibitor with diverse biological activities
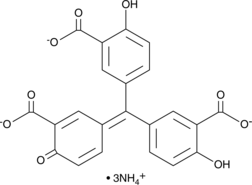
-
GC40005
Aurodox
Aurodox is a polyketide antibiotic originally isolated from S.
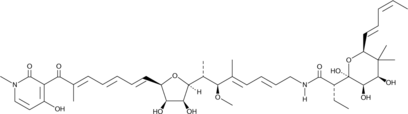
-
GC60062
AV-153 free base
La base libre AV-153, un derivado de la 1,4-dihidropiridina (1,4-DHP), es un antimutagénico. La base libre de AV-153 se intercala con el ADN en una rotura de una sola hebra y reduce el daÑo del ADN, estimula la reparaciÓn del ADN en células humanas in vitro. La base libre de AV-153 interactÚa con la timina y la citosina y tiene influencia en la poli(ADP)ribosilaciÓn. La base libre AV-153 tiene actividad anticancerÍgena.
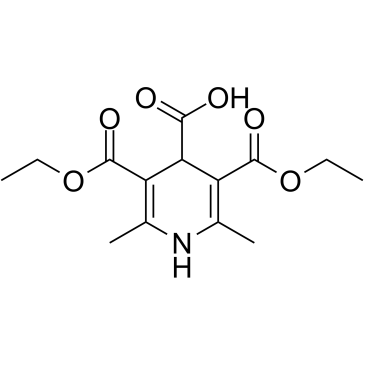
-
GC67960
AVG-233
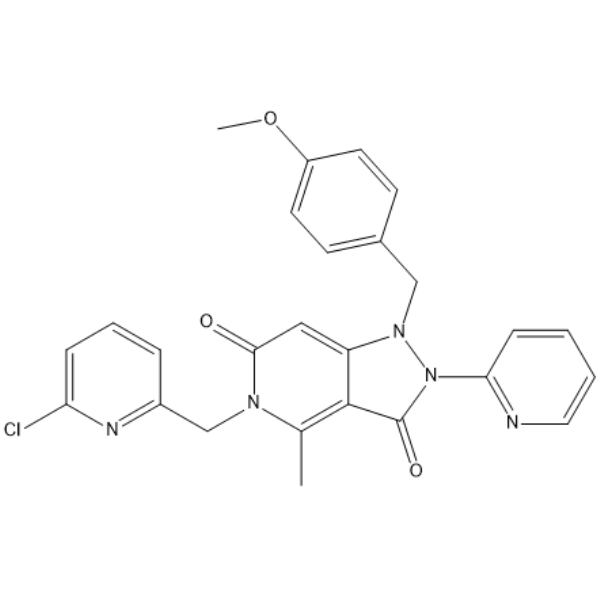
-
GC63449
Avotaciclib
Avotaciclib (BEY1107) es un inhibidor potente y activo por vÍa oral de la quinasa dependiente de ciclina 1 (CDK1). Avotaciclib se puede utilizar para la investigaciÓn del cÁncer de pÁncreas metastÁsico o localmente avanzado.
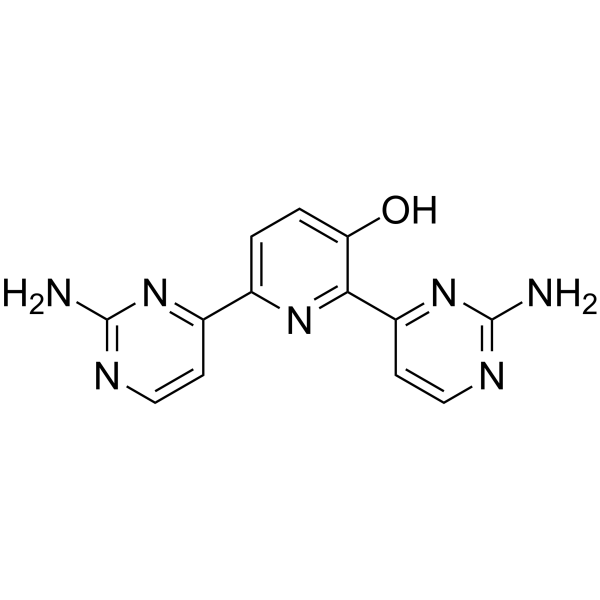
-
GC50424
AZ 5704
Potent and selective ATM kinase inhibitor; orally bioavailable
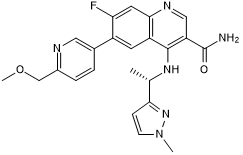
-
GC14949
AZ20
A potent, selective ATR inhibitor
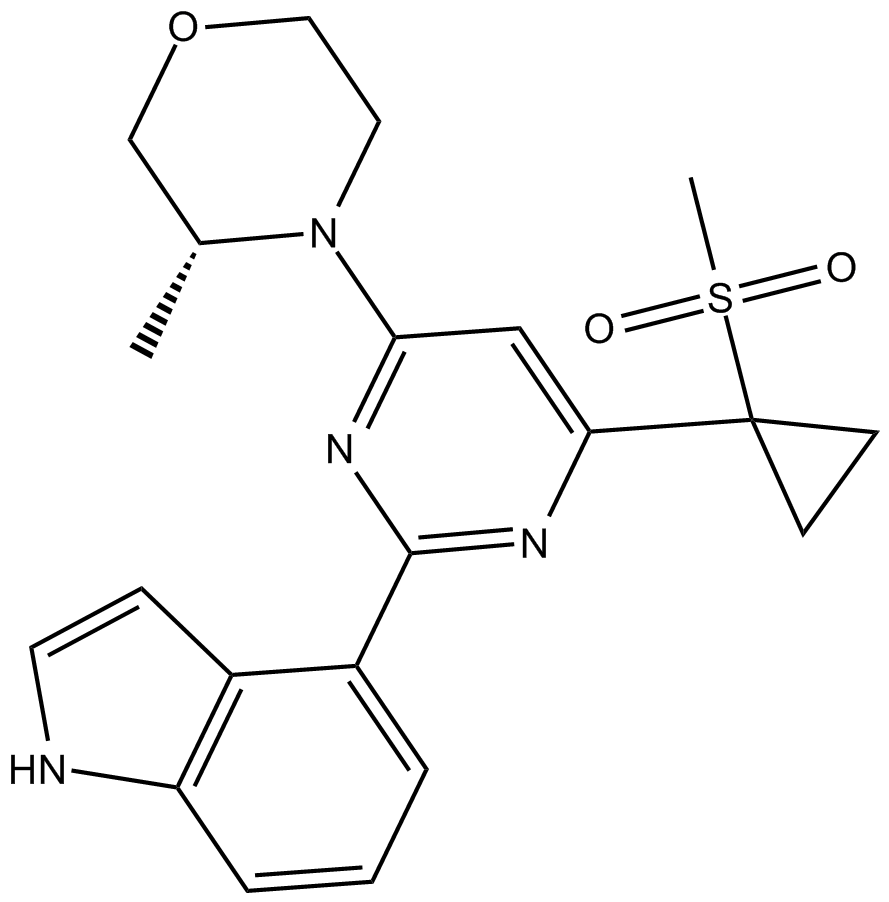
-
GC19047
AZ32
AZ32 es un inhibidor de ATM biodisponible por vÍa oral y que penetra la barrera hematoencefÁlica con una IC50 de <6,2 nM para la enzima ATM y una IC50 de 0,31 μM para ATM en la célula.
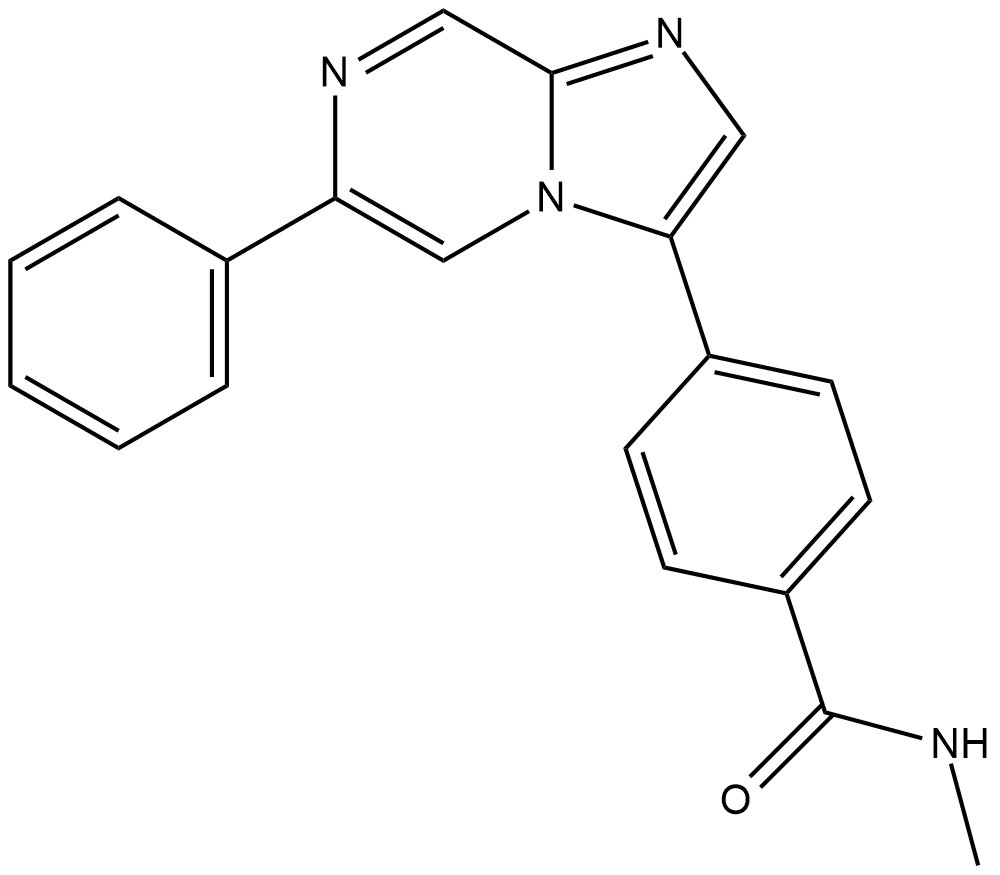
-
GC65899
AZ3391
AZ3391 es un potente inhibidor de PARP. AZ3391 es un derivado de la quinoxalina. La familia de enzimas PARP desempeÑa un papel importante en una serie de procesos celulares, como la replicaciÓn, la recombinaciÓn, la remodelaciÓn de la cromatina y la reparaciÓn del daÑo del ADN. AZ3391 tiene el potencial para la investigaciÓn de enfermedades y condiciones que ocurren en los tejidos del sistema nervioso central, como el cerebro y la médula espinal (extraÍdo de la patente WO2021260092A1, compuesto 23).
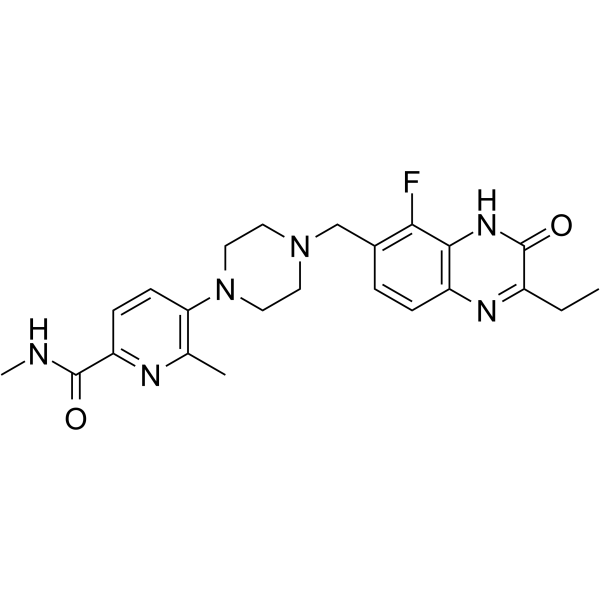
-
GC16725
AZ6102
AZ6102 es un potente inhibidor doble de TNKS1 y TNKS2, con IC50 de 3 nM y 1 nM, respectivamente, y tiene una selectividad 100 veces mayor frente a otras enzimas de la familia PARP, con IC50 de 2,0 μM, 0,5 μM y >3 μM, para PARP1 , PARP2 y PARP6, respectivamente.
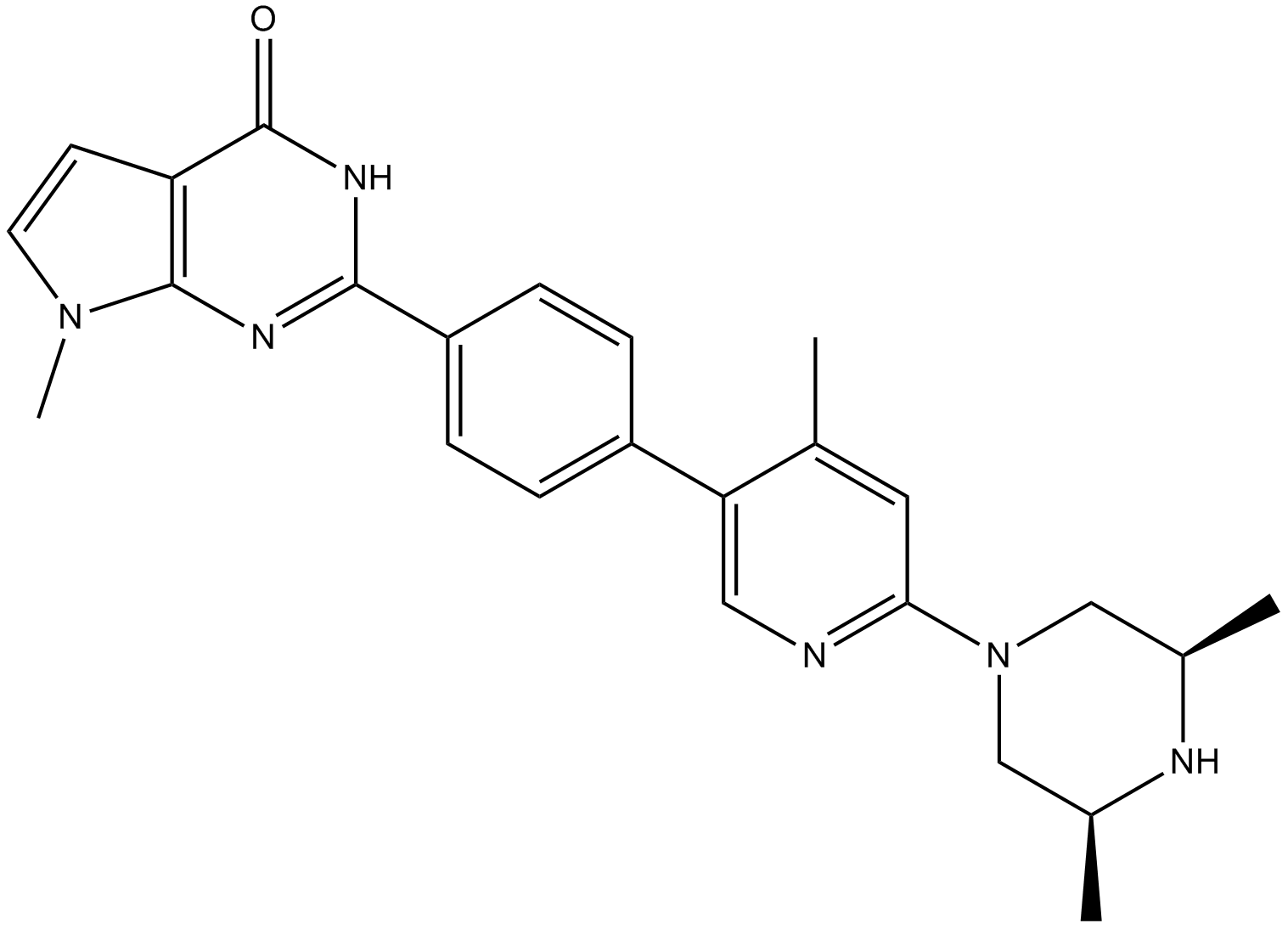
-
GC46900
AZ9482
AZ9482 es un triple inhibidor de PARP1/2/6, con valores IC50 de 1 nM, 1 nM y 640 nM para PARP1, PARP2 y PARP6, respectivamente.
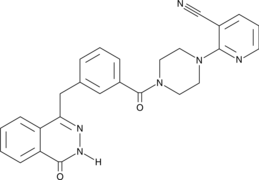
-
GC11843
Azaguanine-8
La azaguanina-8 es un anÁlogo de purina que muestra actividad antineoplÁsica. La azaguanina-8 funciona como un antimetabolito y se incorpora fÁcilmente a los Ácidos ribonucleicos, lo que interfiere con las vÍas biosintéticas normales y, por lo tanto, inhibe el crecimiento celular.
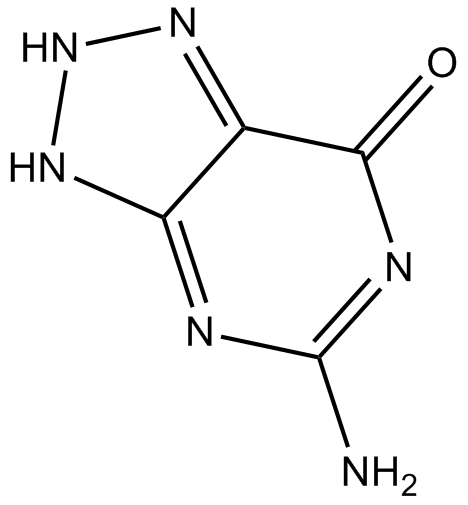
-
GC15033
Azathioprine
La azatioprina (BW 57-322) es un agente inmunosupresor activo por vÍa oral.
-
GC50119
AZD 7762 hydrochloride
Potent and selective ATP-competitive inhibitor of Chk1 and Chk2; also enhances CRISPR-Cpf1-mediated genome editing
-
GC12438
AZD-5438
AZD-5438 es un potente inhibidor de CDK1, CDK2 y CDK9, con IC50 de 16 nM, 6 nM y 20 nM en ensayos sin células, respectivamente. AZD-5438 muestra menos actividad de inhibiciÓn frente a GSK3β, CDK5 y CDK6.
-
GC64938
AZD-7648
AZD-7648 es un potente inhibidor selectivo de DNA-PK activo por vÍa oral con una IC50 de 0,6 nM. AZD-7648 induce apoptosis y muestra actividad antitumoral.
-
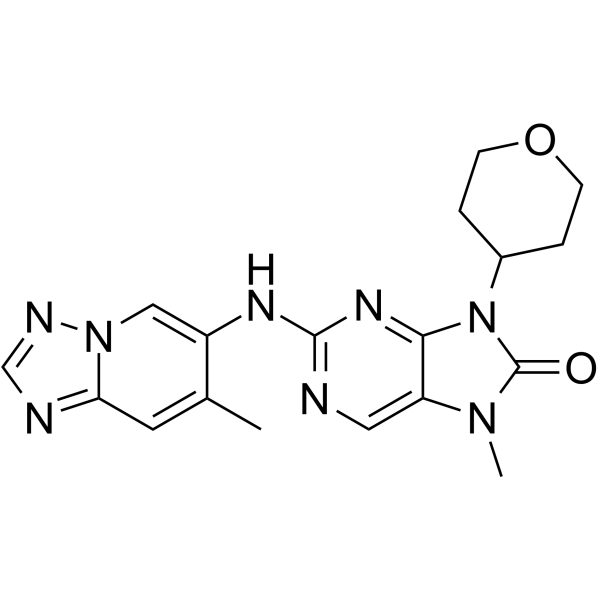
GC11593
AZD0156
AZD0156 es un inhibidor de ATM potente, selectivo y activo por vÍa oral con una IC50 de 0,58 nM. AZD0156 inhibe la seÑalizaciÓn mediada por ATM, previene la activaciÓn del punto de control de daÑos en el ADN, interrumpe la reparaciÓn de daÑos en el ADN e induce la apoptosis de las células tumorales.
-
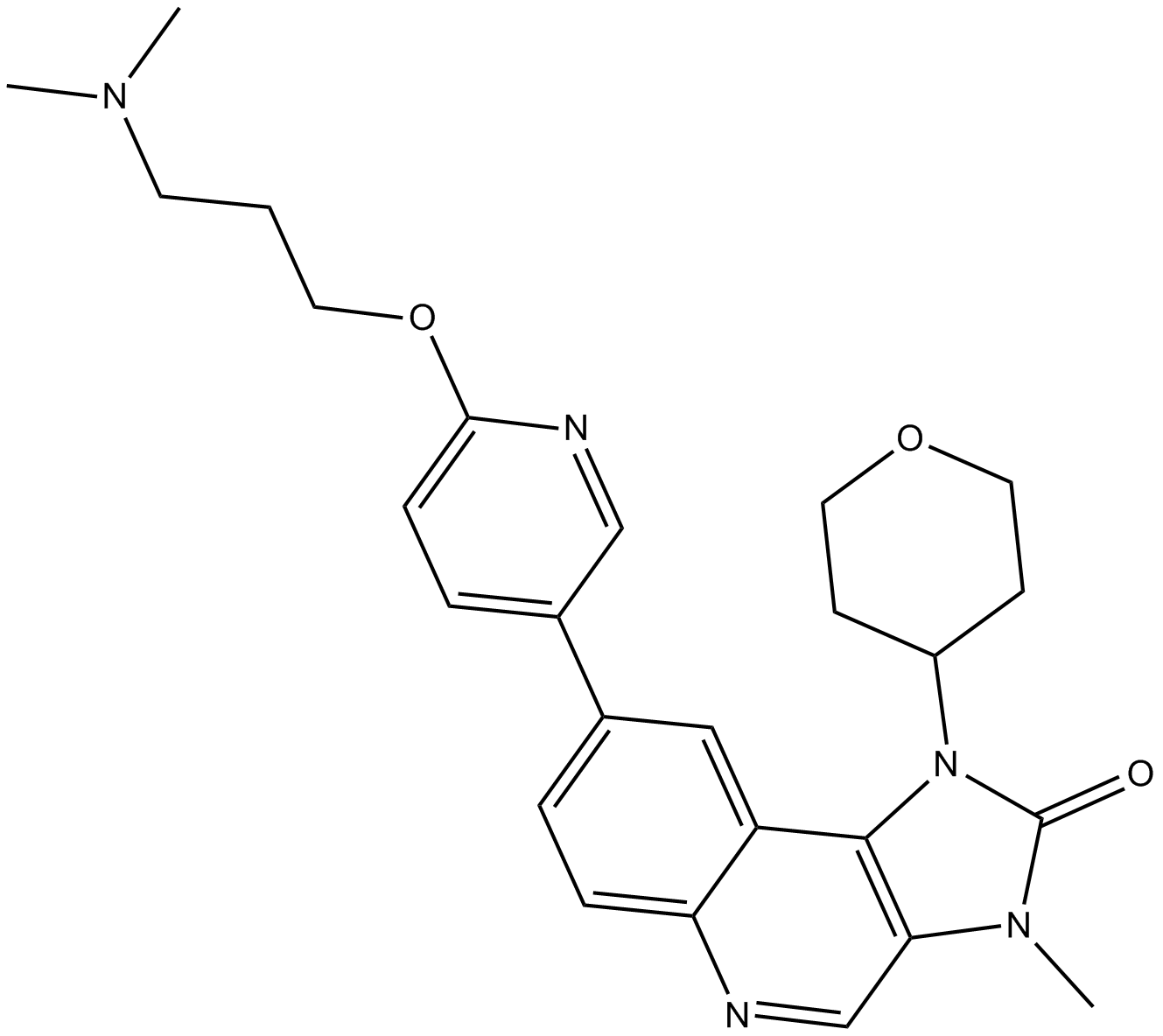
GC19468
AZD1390
AZD1390 es un inhibidor potente y selectivo de ATM.

-
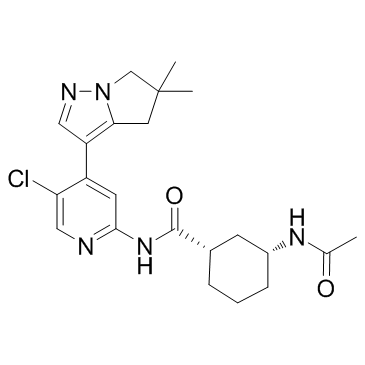
GC32717
AZD4573
AZD4573 es un inhibidor de CDK9 potente y altamente selectivo (IC50 de <4 nM) que permite el compromiso transitorio del objetivo para el tratamiento de neoplasias malignas hematolÓgicas.
-
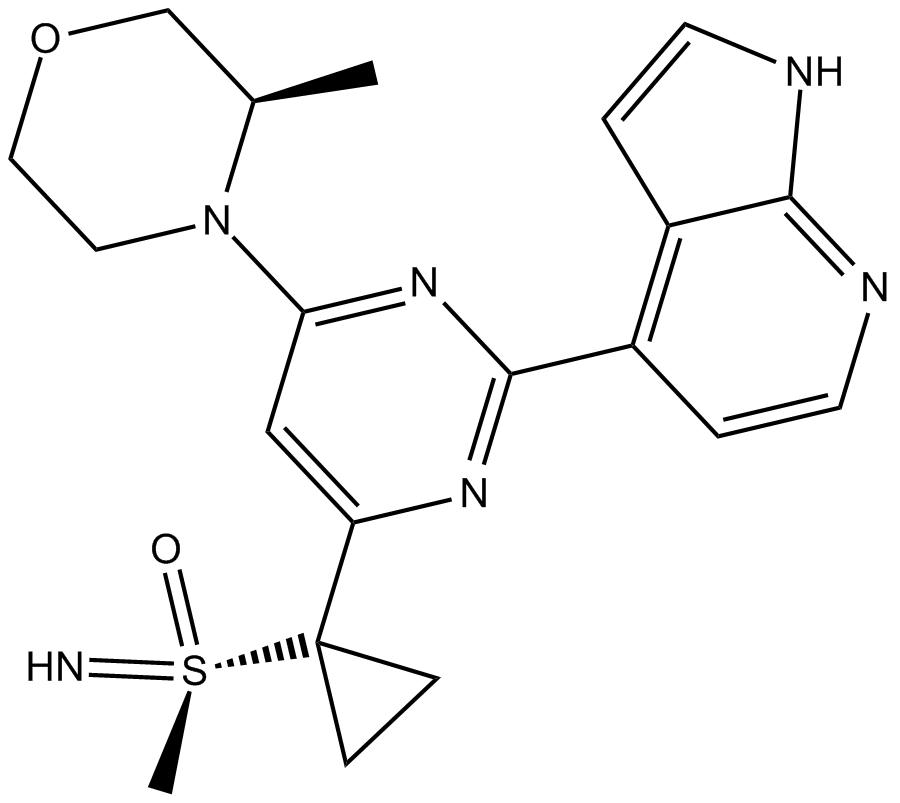
GC16941
AZD6738
AZD6738 (AZD6738) es un inhibidor biodisponible y activo por vÍa oral de la quinasa ATR con una IC50 de 1 nM.
-
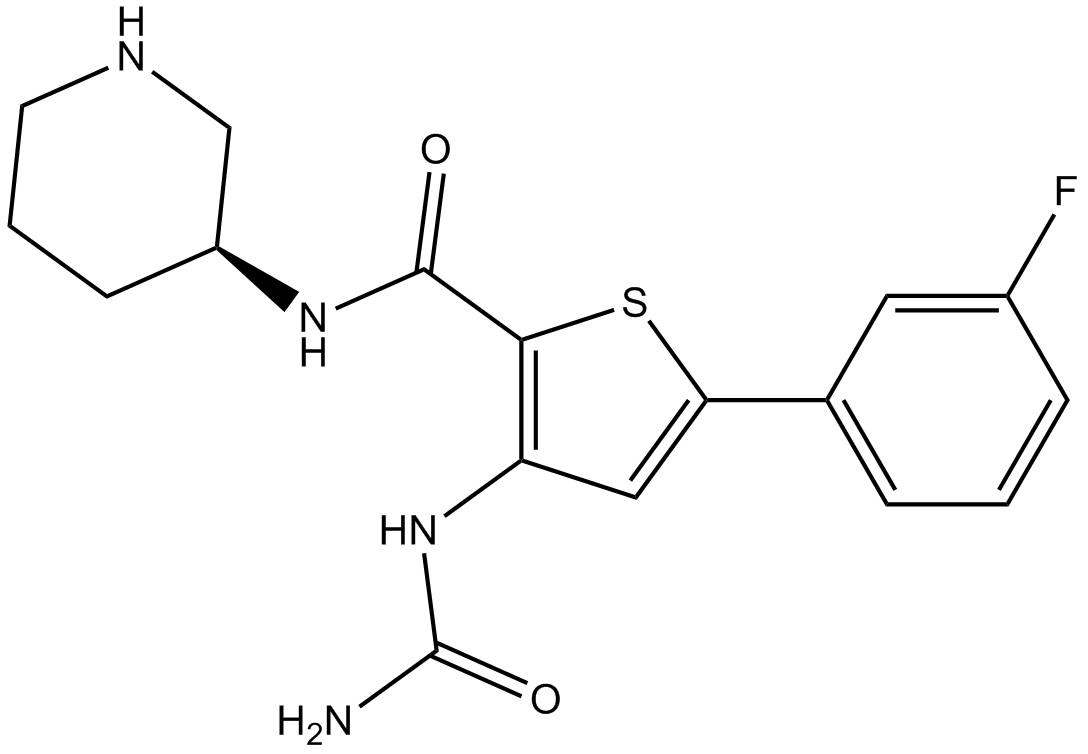
GC10546
AZD7762
AZD7762 es un potente inhibidor de la quinasa del punto de control (Chk) competitivo con ATP con una IC50 de 5 nM para Chk1.
-
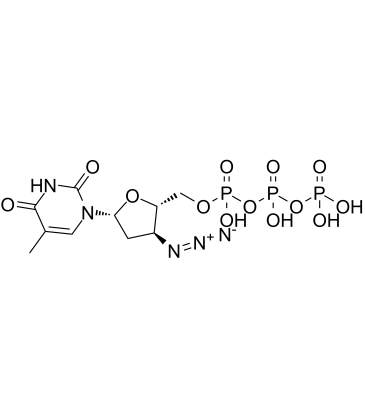
GC60616
AZT triphosphate
El trifosfato de AZT (3'-azido-3'-desoxitimidina-5'-trifosfato) es un metabolito trifosfato activo de la zidovudina (AZT).
-
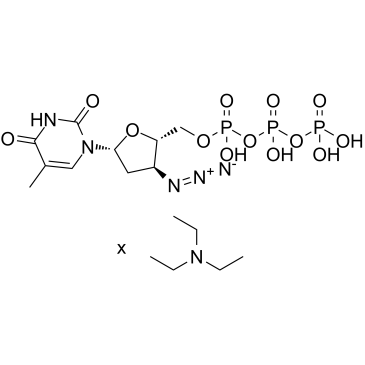
GC60617
AZT triphosphate TEA
AZT trifosfato TEA (3'-azido-3'-desoxitimidina-5'-trifosfato TEA) es un metabolito trifosfato activo de zidovudina (AZT).
-
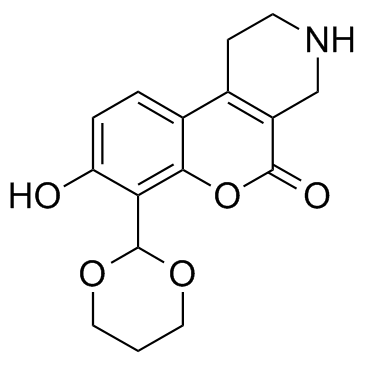
GC33406
B I09
B I09 es un inhibidor de la RNasa IRE-1, con una IC50 de 1230 nM.
-
GC33240
Banoxantrone D12 (AQ4N D12)
La banoxantrona D12 (AQ4N D12) es la banoxantrona marcada con deuterio. La banoxantrona es un agente biorreductor novedoso que se puede reducir a un compuesto AQ4 estable y afÍn al ADN, que es un potente inhibidor de la topoisomerasa II.
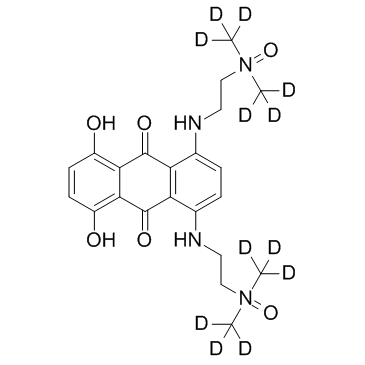
-
GC34169
Banoxantrone D12 dihydrochloride (AQ4N D12 dihydrochloride)
El diclorhidrato de banoxantrona-d12 (AQ4N-d12) es el diclorhidrato de banoxantrona marcado con deuterio. La banoxantrona es un agente biorreductor novedoso que se puede reducir a un compuesto AQ4 estable y afÍn al ADN, que es un potente inhibidor de la topoisomerasa II.
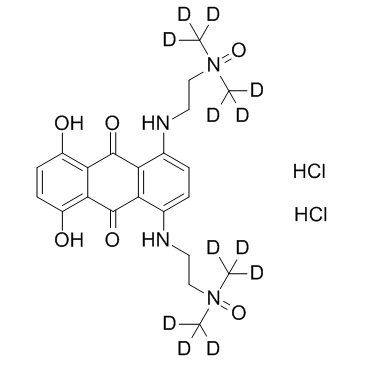
-
GC34085
Banoxantrone dihydrochloride (AQ4N dihydrochloride)
El dihidrocloruro de banoxantrona (dihidrocloruro de AQ4N) es un agente bioreductor novedoso que puede reducirse a un compuesto estable y afín al ADN llamado AQ4, el cual es un potente inhibidor de la topoisomerasa II.
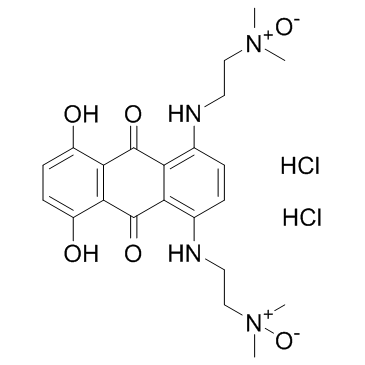
-
GC13035
Bay 11-7821
Un inhibidor selectivo e irreversible de NF-κB.
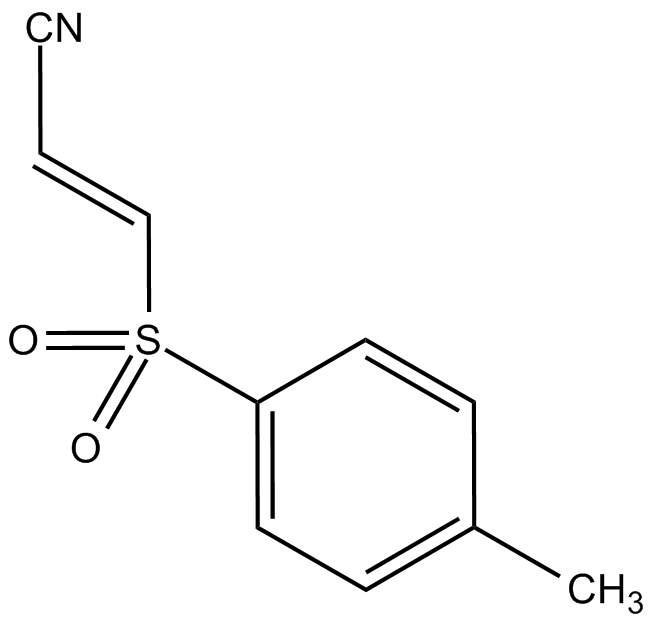
-
GC50656
BAY 707
BAY 707 es un inhibidor de MTH1 (NUDT1) altamente potente y selectivo competitivo con el sustrato con una IC50 de 2,3 nM. BAY 707 tiene un buen perfil farmacocinético (FC) frente a otros compuestos MTH1 y es bien tolerado en ratones, pero muestra una clara falta de eficacia anticancerígena in vitro o in vivo.
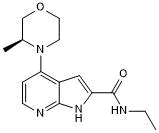
-
GC33420
BAY-1895344
BAY-1895344 (BAY-1895344) es un inhibidor de ATR potente, activo por vÍa oral y selectivo con una IC50 de 7 nM. BAY-1895344 tiene actividad antitumoral. BAY-1895344 se puede utilizar para la investigaciÓn de tumores sÓlidos y linfomas.
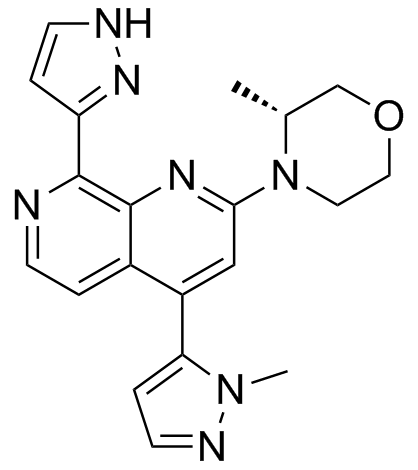
-
GC34374
BAY-1895344 hydrochloride
An ATR inhibitor
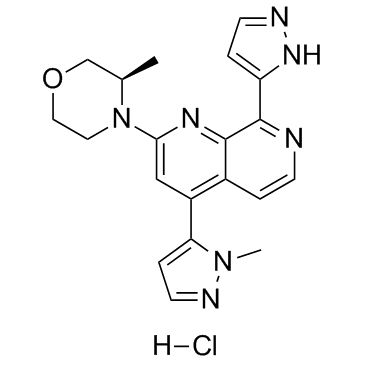
-
GC32838
BAY-2402234
BAY-2402234 es un inhibidor selectivo de la dihidroorotato deshidrogenasa (DHODH) para el tratamiento de tumores malignos mieloides.
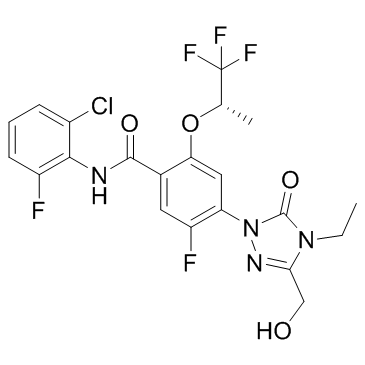
-
GC63763
BAY-8400
BAY-8400 es un inhibidor de la proteÍna quinasa dependiente de ADN (ADN-PK) activo por vÍa oral, potente y selectivo (IC50 = 81 nM). BAY-8400 se puede utilizar para la investigaciÓn del cÁncer.
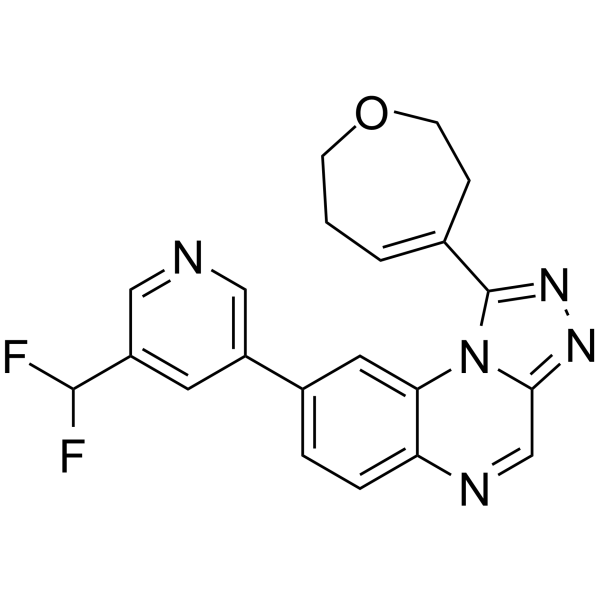
-
GC65259
BC-1471
BC-1471 es un inhibidor de la deubiquitinasa de la proteÍna de uniÓn a STAM (STAMBP).
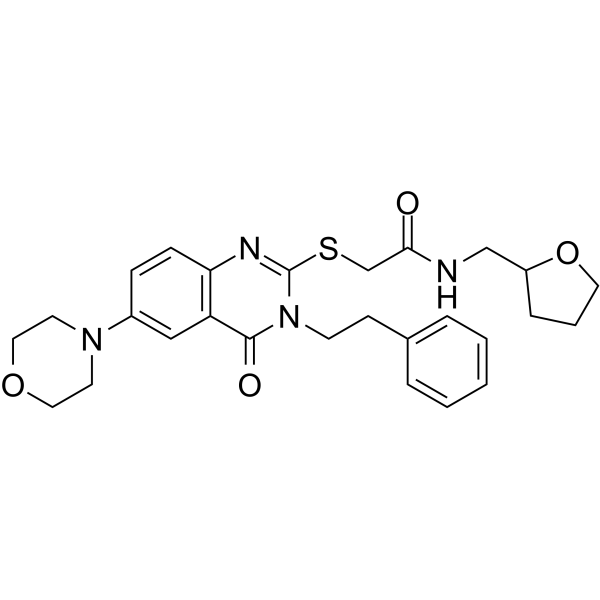
-
GC50572
BC-LI-0186
BC-LI-0186 es un inhibidor potente y selectivo de la leucil-tRNA sintetasa (LRS; LeuRS) y la interacciÓn de la proteÍna D de uniÓn a GTP relacionada con Ras (RagD) (IC50 = 46,11 nM). BC-LI-0186 se une de forma competitiva al sitio de interacciÓn RagD de LRS (Kd=42,1 nM) y tiene efectos sobre LRS-Vps34, LRS-EPRS, asociaciÓn RagB-RagD, formaciÓn del complejo mTORC1 o las actividades de 12 quinasas. BC-LI-0186 puede suprimir eficazmente la actividad de los mutantes MTORasociados al cÁncer y el crecimiento de células cancerosas resistentes a la rapamicina. BC-LI-0186 es un agente prometedor para la investigaciÓn del cÁncer de pulmÓn.
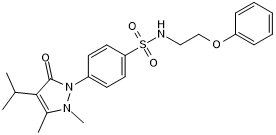
-
GC62863
BCH001
BCH001, un derivado de la quinolina, es un inhibidor especÍfico de PAPD5.