Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Ziele für Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Produkte für Chromatin/Epigenetics
- Bestell-Nr. Artikelname Informationen
-
GC33208
Dot1L-IN-1
Dot1L-IN-1 ist ein hochwirksamer, selektiver und strukturell neuer Dot1L-Inhibitor mit einem Ki von 2 pM.
-
GC30530
Dot1L-IN-2
Dot1L-IN-2 ist ein potenter, selektiver und oral bioverfÜgbarer Inhibitor von Dot1L (einer Histon-Methyltransferase) mit einem IC50 und Ki von 0,4 nM bzw. 0,08 nM.
-
GC62613
Dot1L-IN-4
Dot1L-IN-4 ist ein potenter Disruptor des telomeric silencing 1-like protein (DOT1L)-Inhibitors mit einem IC50 SPA DOT1L von 0,11 nM.
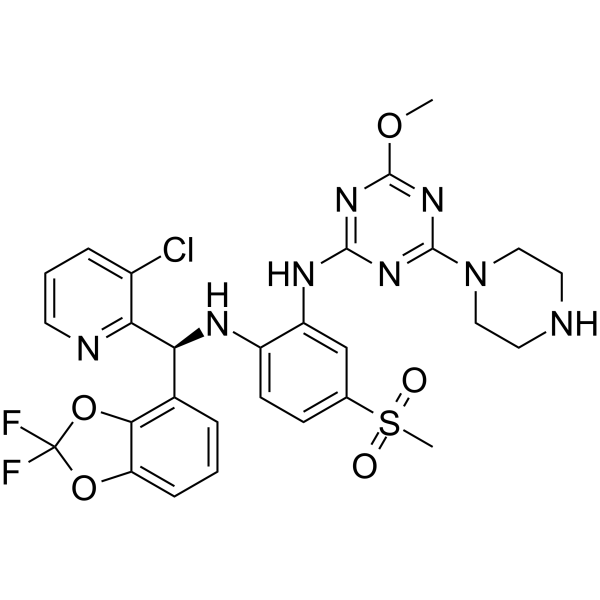
-
GC65962
Dot1L-IN-5
Dot1L-IN-5 ist ein potenter Disruptor des telomeric silencing 1-like protein (DOT1L)-Inhibitors mit einem IC50 SPA DOT1L von 0,17 nM.
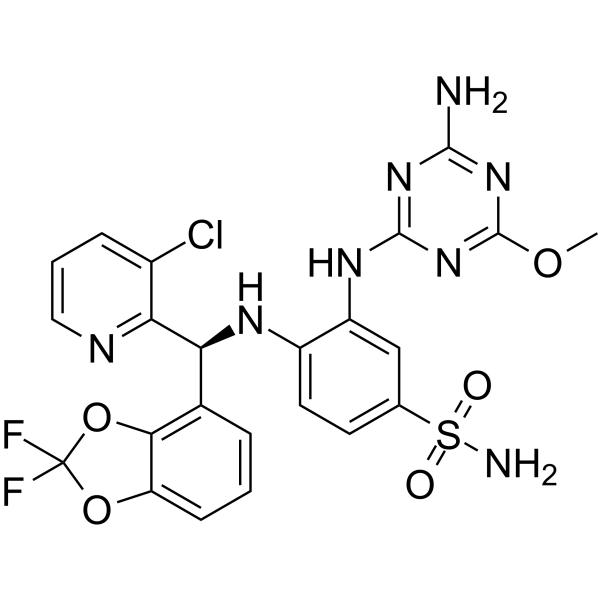
-
GC12680
DR 2313
A PARP inhibitor
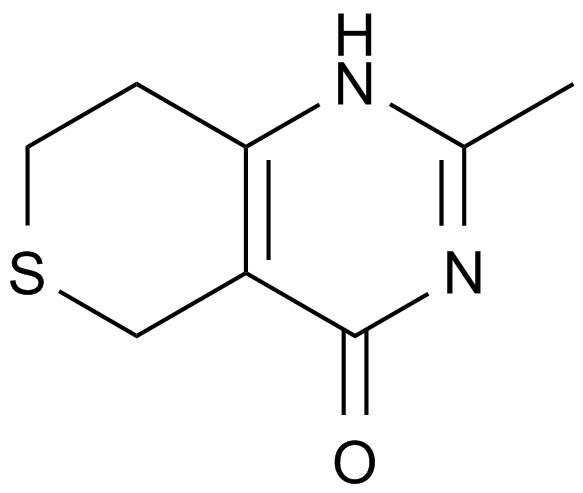
-
GC13706
Droxinostat
An inhibitor of HDAC3, HDAC6, and HDAC8
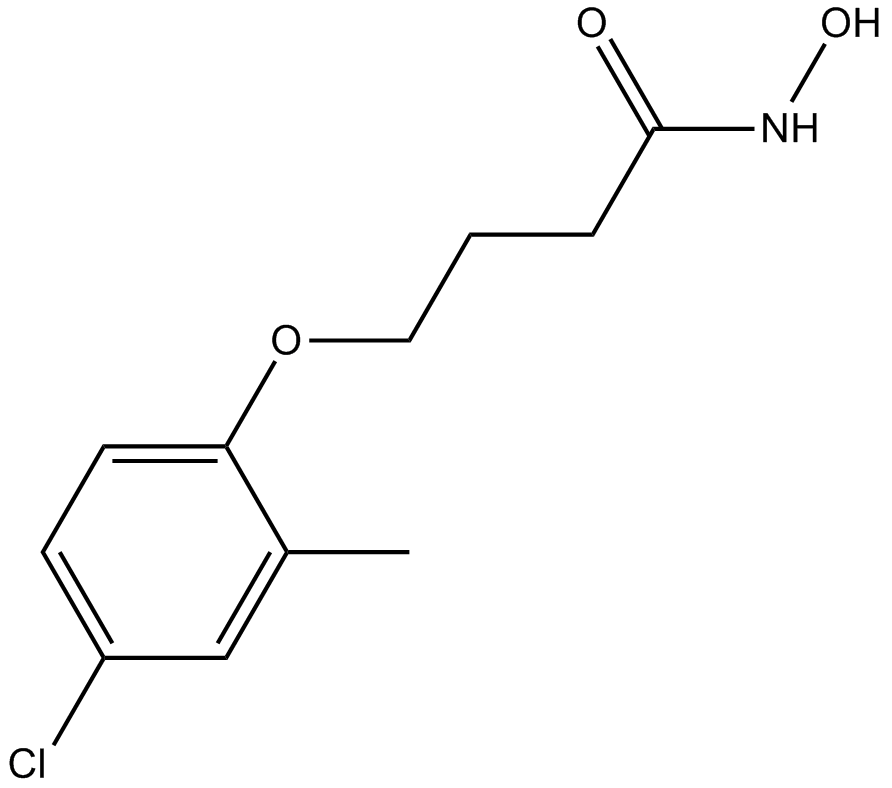
-
GC45927
DS-437
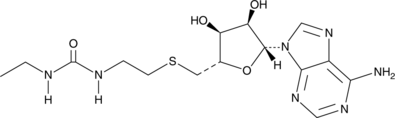
DS-437 ist ein dualer PRMT5/7-Inhibitor (IC50s von PRMT5/7 = 6 μM).
-
GC35904
dTRIM24
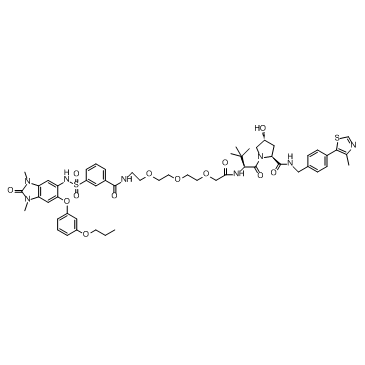
dTRIM24 ist ein selektiver bifunktionaler Abbauer von TRIM24 auf Basis von PROTAC, bestehend aus Liganden fÜr von Hippel-Lindau und TRIM24.
-
GC35914
DW14800
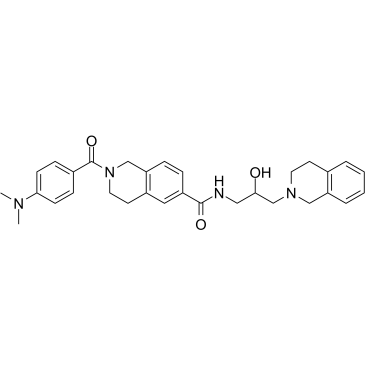
DW14800 ist ein Inhibitor der Protein-Arginin-Methyltransferase 5 (PRMT5) mit einem IC50-Wert von 17 nM. DW14800 reduziert die H4R3me2s-Spiegel und verstÄrkt die Transkription von HNF4α, verÄndert jedoch nicht die PRMT5-Expression. Anti-Krebs-AktivitÄt.
-
GC33403
E-7386
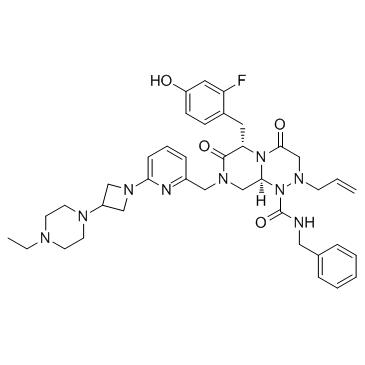
E-7386 ist ein oral aktiver CBP/Beta-Catenin-Modulator.
-
GC64209
E7016
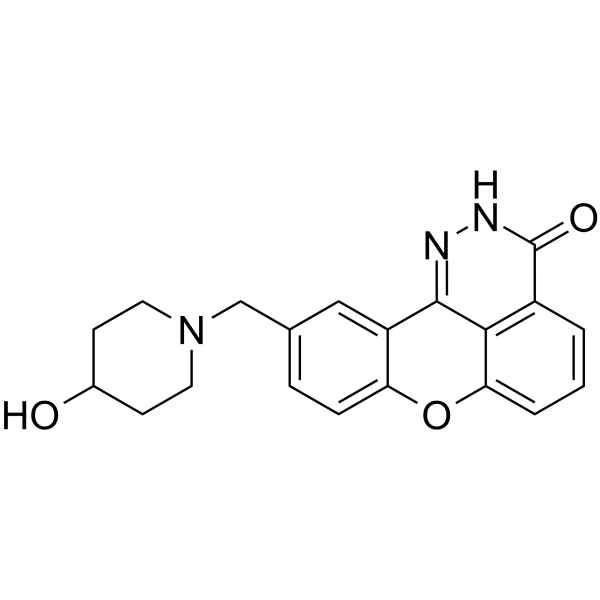
E7016 (GPI 21016) ist ein oral verfÜgbarer PARP-Hemmer. E7016 kann die Strahlenempfindlichkeit von Tumorzellen in vitro und in vivo durch die Hemmung der DNA-Reparatur erhÖhen. E7016 wirkt als potenzielles Antikrebsmittel.
-
GC18172
E7449
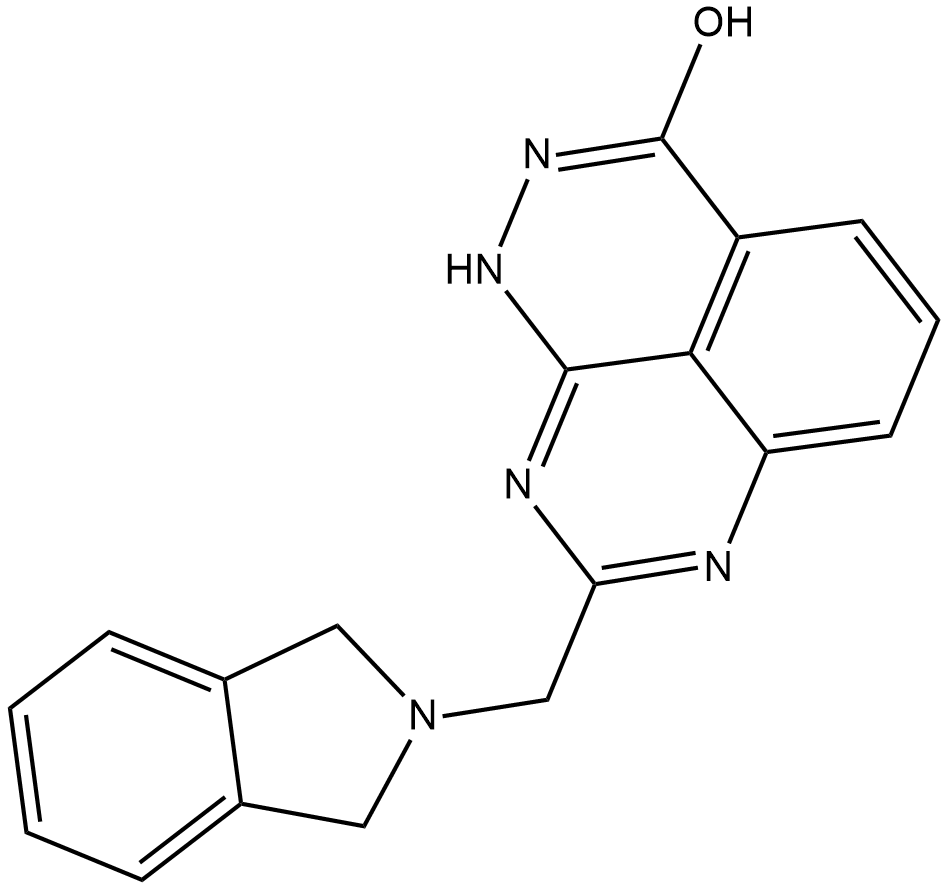
E7449 ist ein potenter PARP1- und PARP2-Inhibitor und hemmt auch TNKS1 und TNKS2 mit IC50-Werten von 2,0, 1,0, ~50 und ~50 nM fÜr PARP1, PARP2, TNKS1 bzw. TNKS2 unter Verwendung von 32P-NAD+ als Substrat.
-
GC12991
EB 47
A PARP1 and TNKS2 inhibitor
-
GC33220
EBI-2511
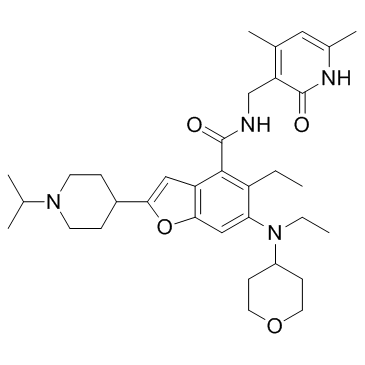
EBI-2511 ist ein hochpotenter und oral aktiver EZH2-Inhibitor mit einem IC50-Wert von 6 nM in Pfeffiera-Zelllinien.
-
GC18236
Echinomycin
Ein Hemmstoff der HIF1-vermittelten Gen-Transkription.

-
GC19129
EDO-S101
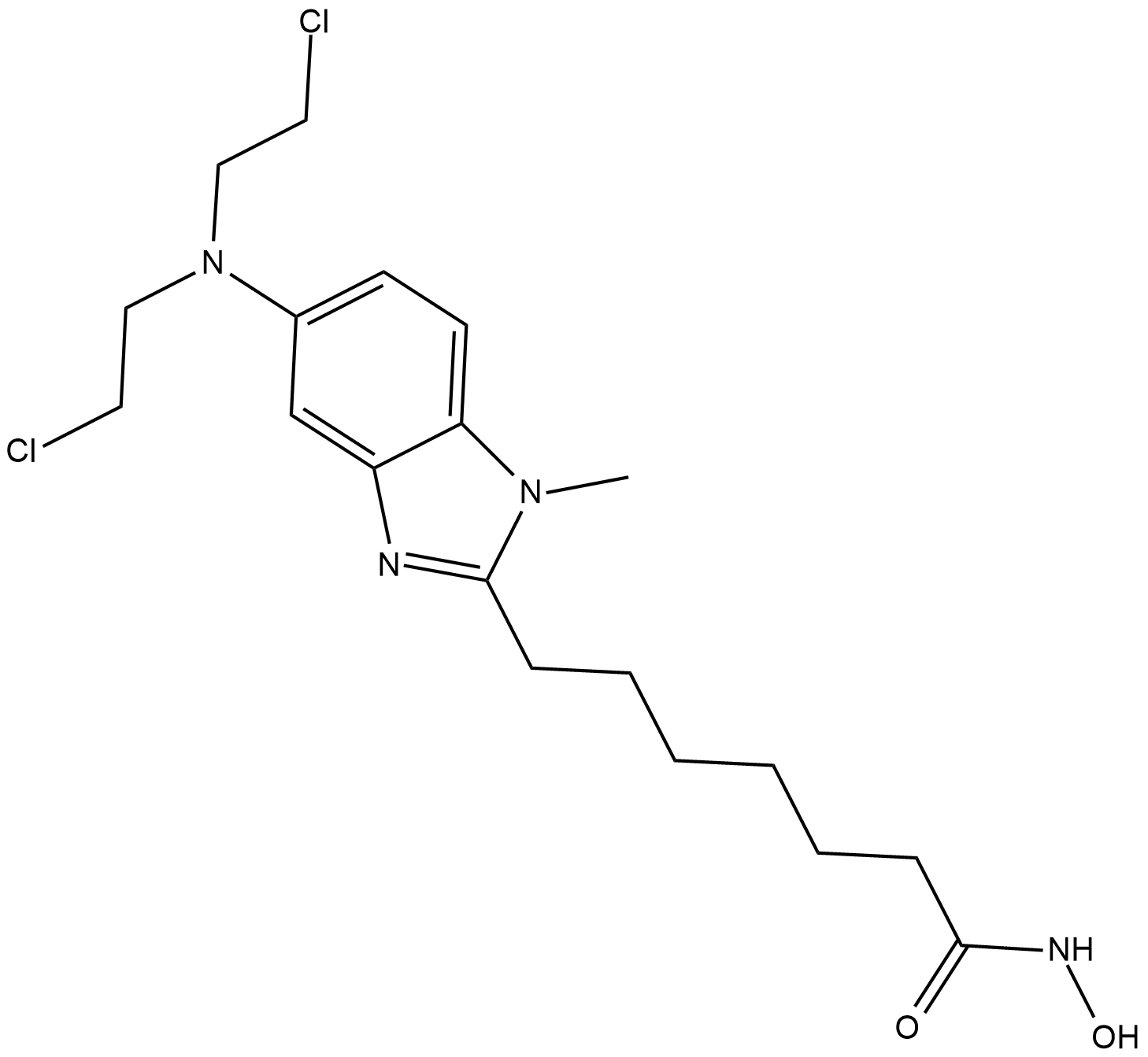
EDO-S101 (Tinostamustin) ist ein Pan-HDAC-Hemmer; hemmt HDAC6, HDAC1, HDAC2 und HDAC3 mit IC50-Werten von 6 nM, 9 nM, 9 nM bzw. 25 nM.
-
GC19130
EED226
EED226 ist ein Polycomb Repressive Complex 2 (PRC2)-Inhibitor, der bei der embryonalen Ektodermentwicklung (EED) an die K27me3-Tasche bindet und im Xenograft-Mausmodell eine starke AntitumoraktivitÄt zeigt. EED226 ist ein potenter, selektiver und oral bioverfÜgbarer EED-Inhibitor. EED226 hemmt PRC2 mit einem IC50 von 23,4 nM, wenn das H3K27me0-Peptid als Substrat in den enzymatischen In-vitro-Assays verwendet wird.
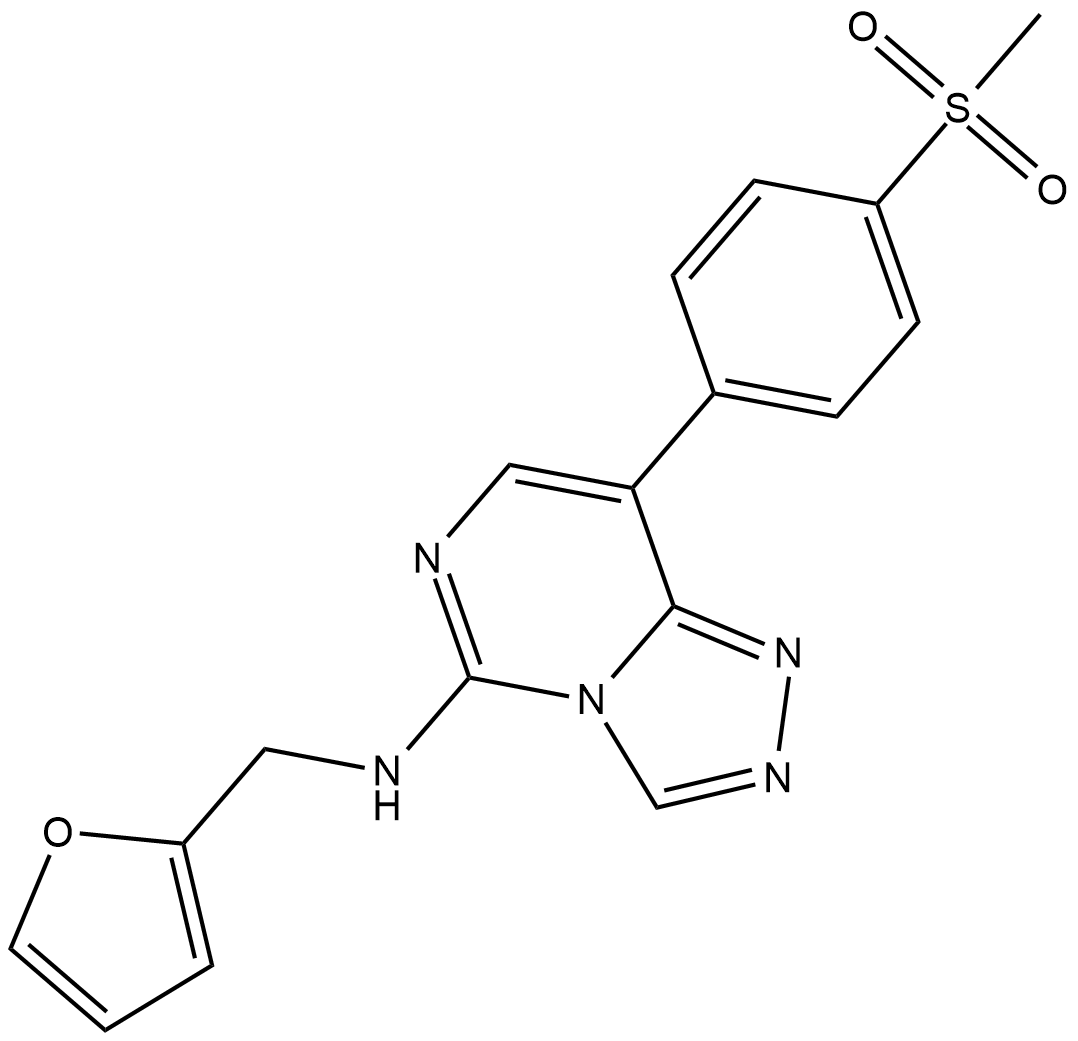
-
GC67934
EEDi-5285
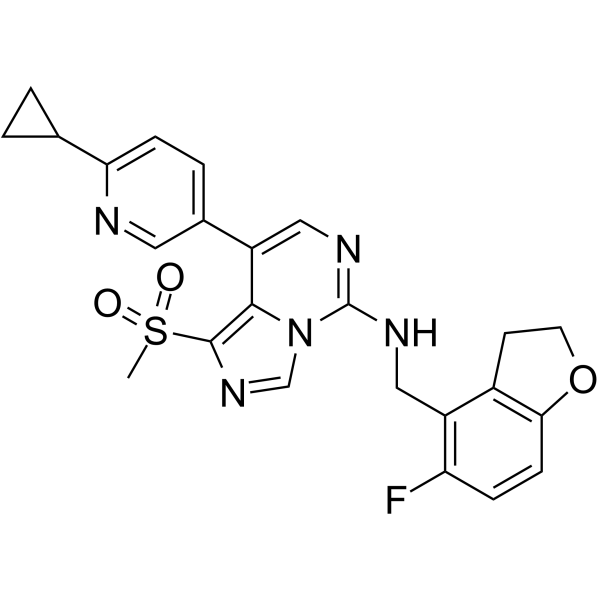
-
GC34567
EHMT2-IN-1
EHMT2-IN-1 ist ein potenter EHMT-Inhibitor mit IC50-Werten von allen < 100 nM fÜr EHMT1-Peptid, EHMT2-Peptid und zellulÄres EHMT2. Wird in der Erforschung von Blutkrankheiten oder Krebs verwendet.
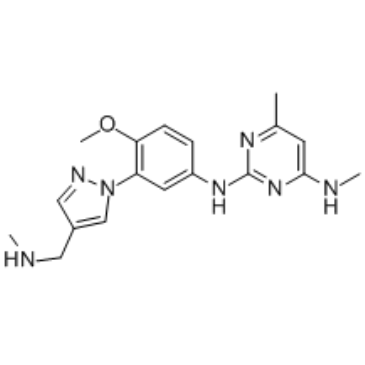
-
GC34568
EHMT2-IN-2
EHMT2-IN-2 ist ein potenter EHMT-Inhibitor mit IC50-Werten von allen < 100 nM fÜr EHMT1-Peptid, EHMT2-Peptid und zellulÄres EHMT2. Wird in der Erforschung von Blutkrankheiten oder Krebs verwendet.
-
GC14756
EI1
EI1 (KB-145943) ist ein potenter und selektiver EZH2-Inhibitor mit einem IC50-Wert von 15 nM bzw. 13 nM fÜr EZH2 (WT) und EZH2 (Y641F).
-
GC33043
EL-102
EL-102 ist ein Hypoxie-induzierter Faktor 1 (Hif1α)-Inhibitor. EL-102 induziert Apoptose, hemmt die Tubulin-Polymerisation und zeigt AktivitÄten gegen Prostatakrebs. EL-102 kann fÜr die Krebsforschung eingesetzt werden.
-
GC64191
Elevenostat
Elevenostat (JB3-22) ist ein selektiver HDAC11-Inhibitor (IC50=0,235μM). AktivitÄt gegen multiples Myelom (MM).
-
GC31245
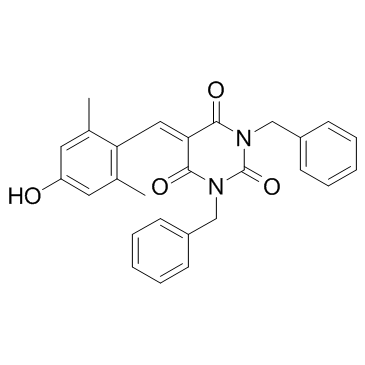
EML 425
EML425 ist ein potenter und selektiver CREB-Bindungsprotein (CBP)/p300-Inhibitor mit IC50-Werten von 2,9 bzw. 1,1 μM.
-
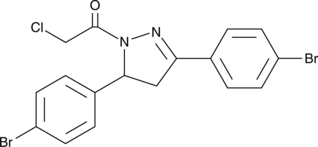
GC49124
EN219
EN219 ist ein mÄßig selektiver synthetischer kovalenter Ligand gegen ein N-terminales Cystein (C8) von RNF114 mit einem IC50 von 470 nM. EN219 hemmt die RNF114-vermittelte Autoubiquitinierung und p21-Ubiquitinierung.
-
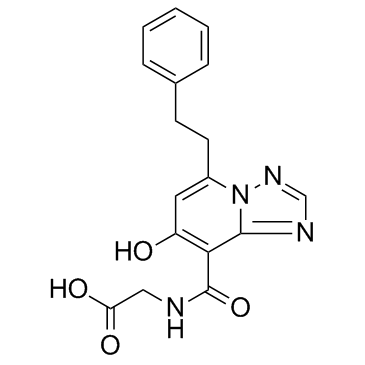
GC33634
Enarodustat (JTZ-951)
Enarodustat (JTZ-951) ist ein potenter und oral aktiver Hypoxie-induzierbarer Prolylhydroxylase-Inhibitor mit einem EC50-Wert von 0,22 μM.
-
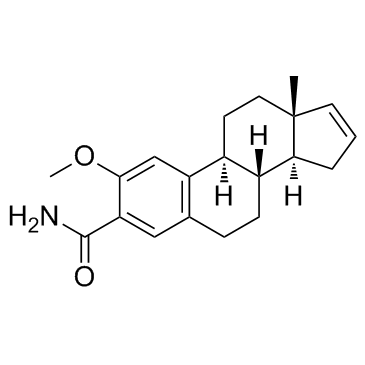
GC33170
ENMD-119 (ENMD 1198)
ENMD-119 (ENMD 1198) (IRC-110160), ein oral aktiver Wirkstoff zur Destabilisierung von Mikrotubuli, ist ein 2-Methoxyestradiol-Analogon mit antiproliferativer und antiangiogener AktivitÄt. ENMD-119 (ENMD 1198) eignet sich zur Hemmung von HIF-1alpha und STAT3 in humanen HCC-Zellen und fÜhrt zu einer Verringerung des Tumorwachstums und der Vaskularisierung.
-
GC16519
ENMD-2076
A multi-kinase inhibitor
-
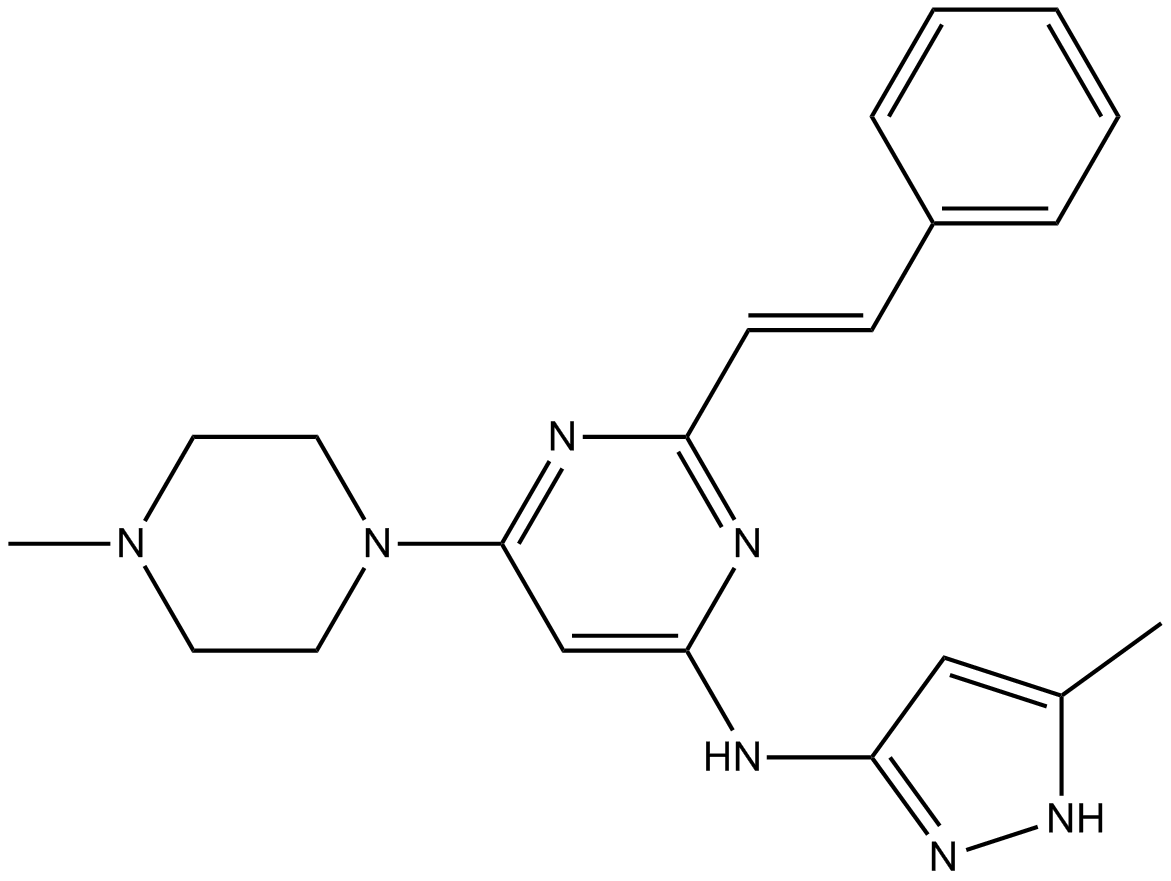
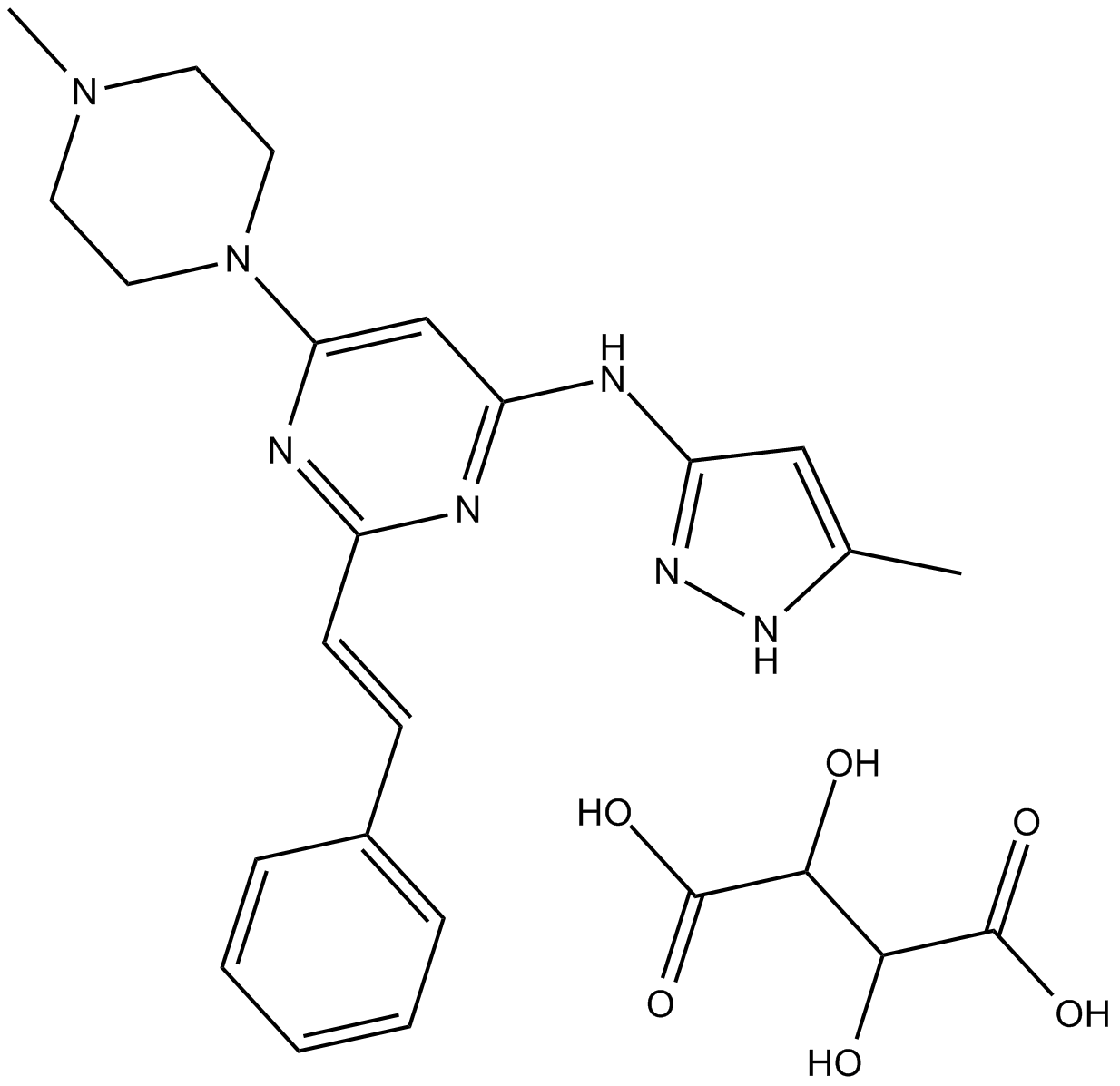
GC12145
ENMD-2076 L-(+)-Tartaric acid
-
GC50182
ent-LP 99
ent-LP 99 ist ein potenter und selektiver BRD7- und BRD9-Inhibitor mit einer KD von 99 nM fÜr BRD9.
-
GC17334
Entacapone
A reversible COMT inhibitor
-
GC47294
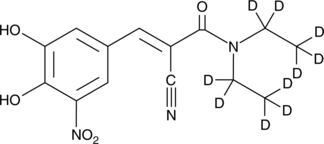
Entacapone-d10
Entacapone-d10 ist das Deuterium mit der Bezeichnung Entacapone.
-
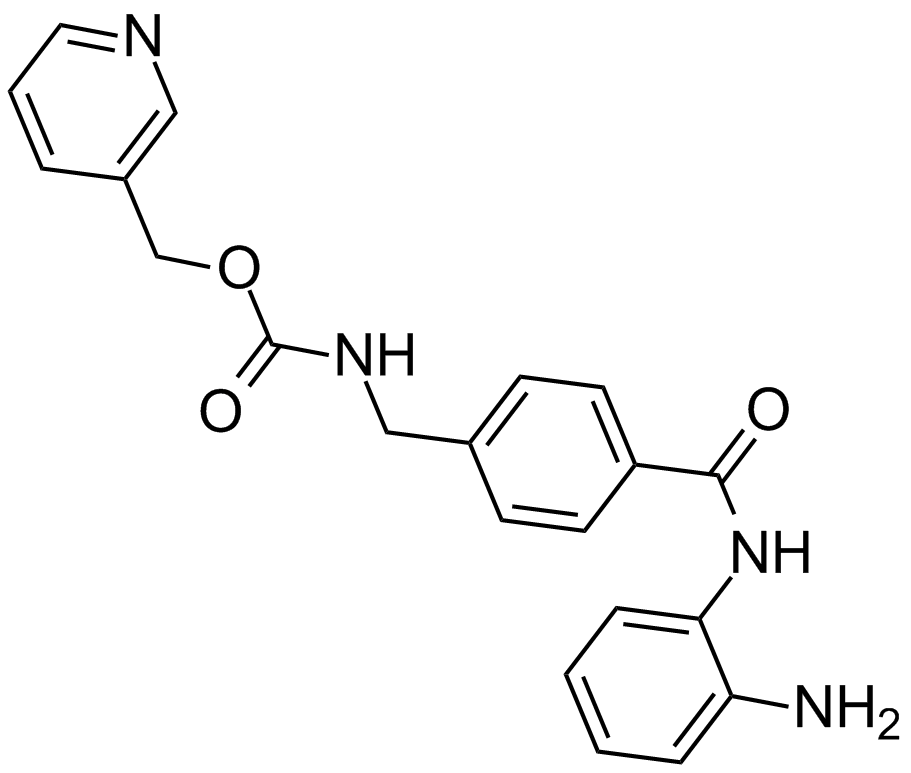
GC11625
Entinostat (MS-275,SNDX-275)
A histone deacetylase inhibitor
-
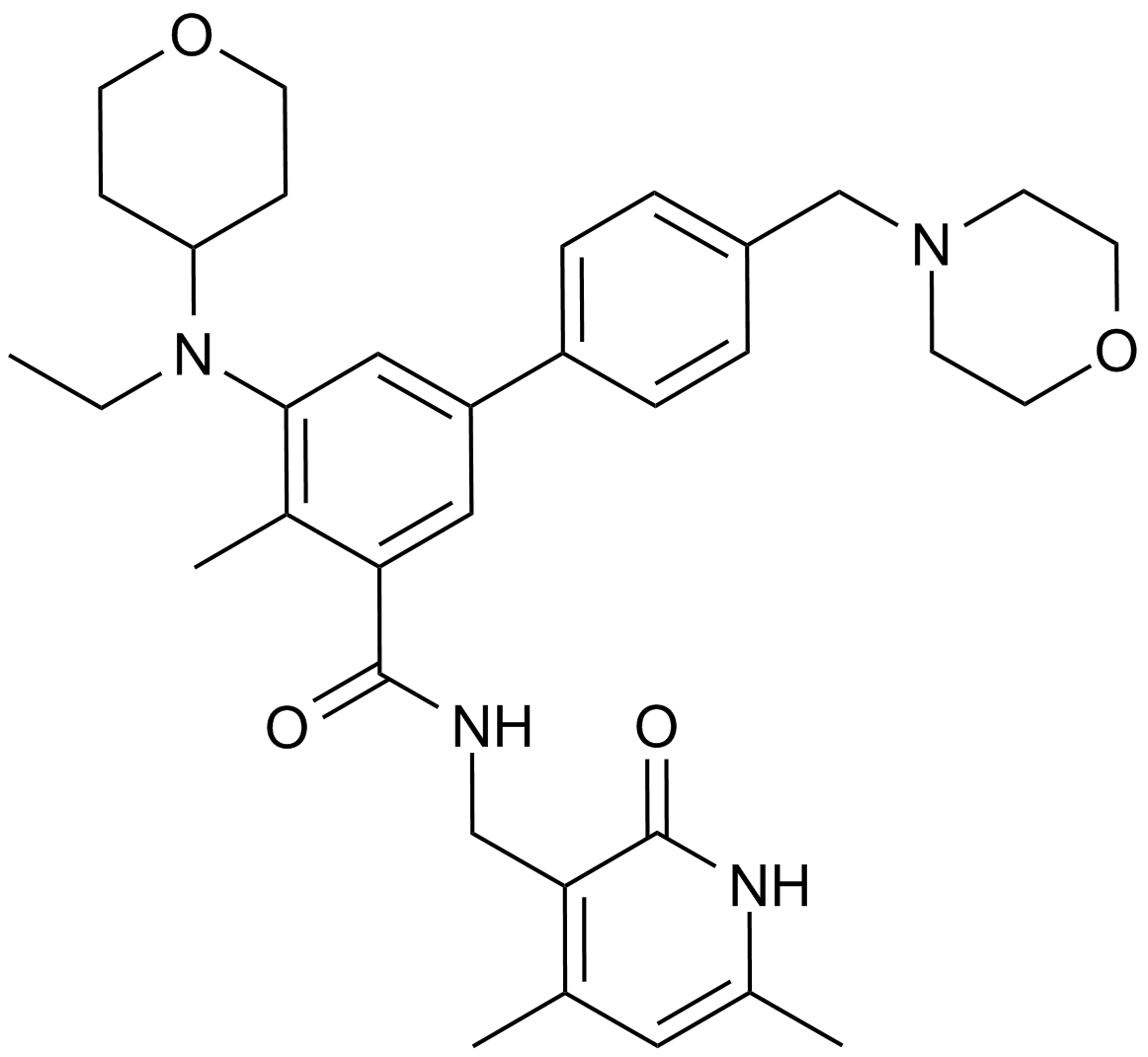
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
-
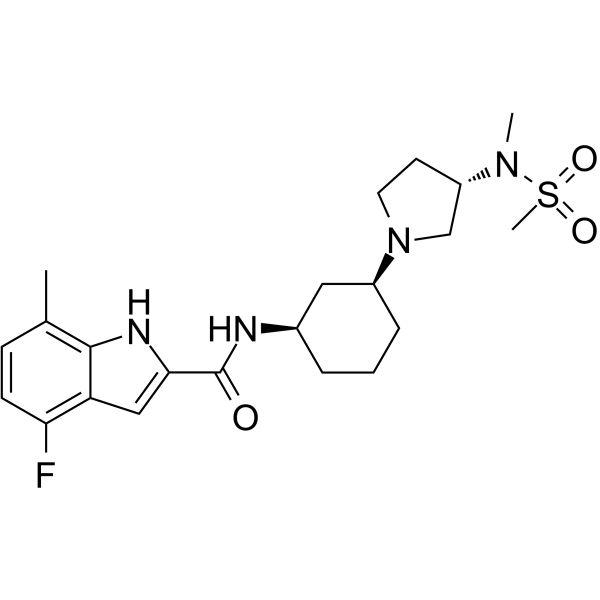
GC64343
EPZ-719
EPZ-719 ist ein neuartiger und potenter SETD2-Inhibitor (IC50 = 0,005 μM) mit einer hohen SelektivitÄt gegenÜber anderen Histon-Methyltransferasen.
-
GC13383
EPZ004777
A potent inhibitor of DOT1L
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
GC15259
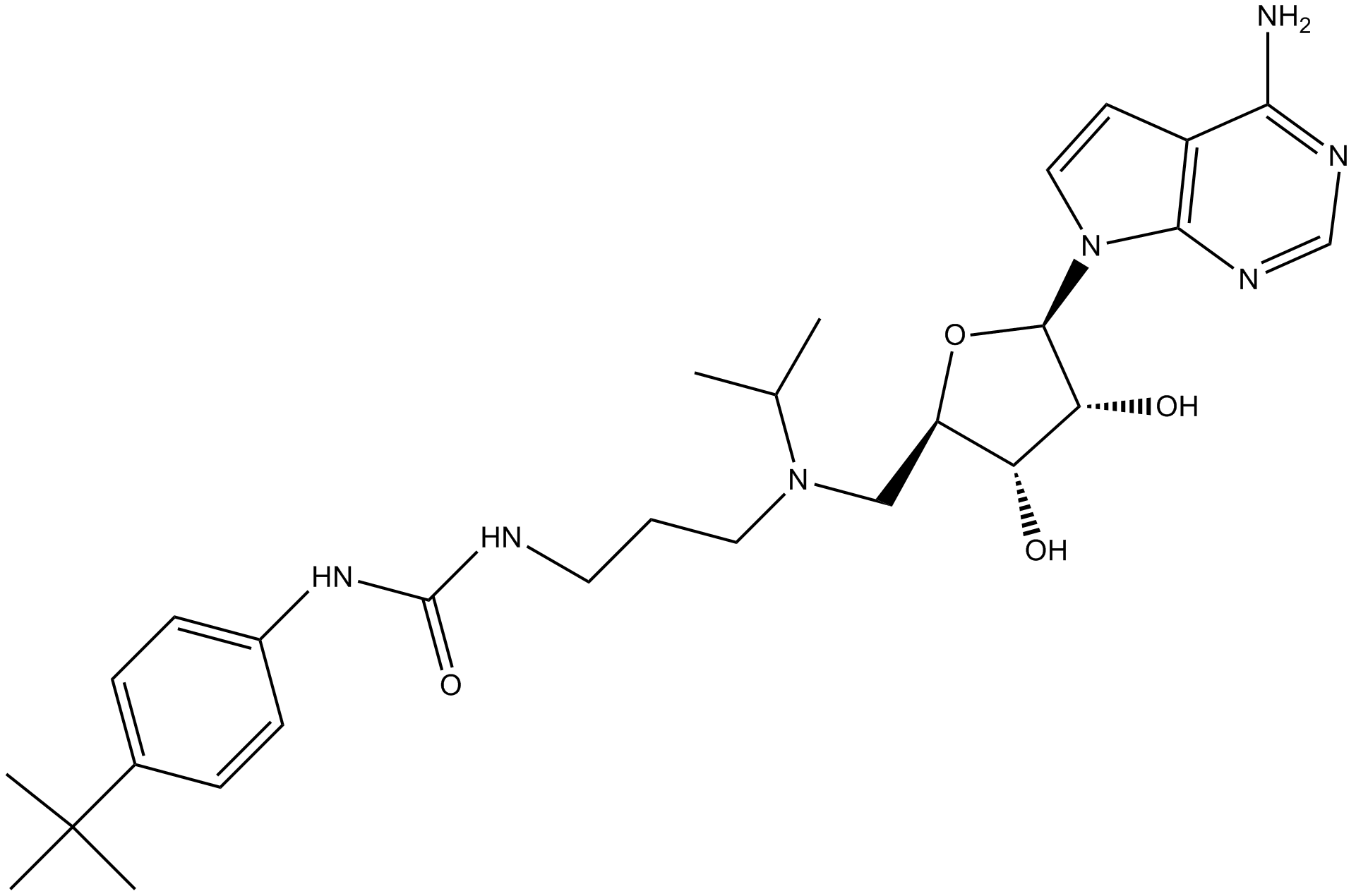
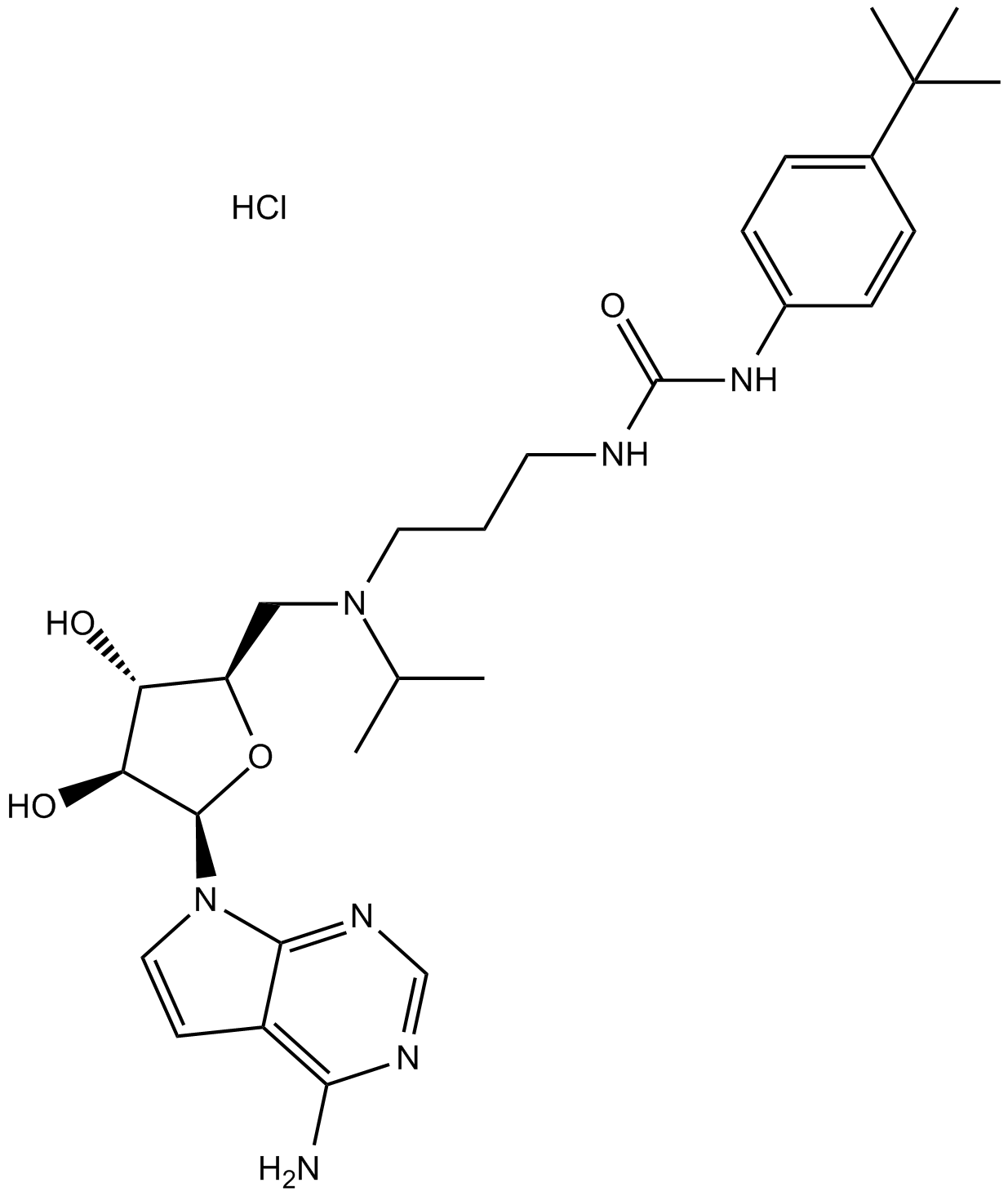
EPZ004777 HCl
EPZ004777 HCl ist ein potenter, selektiver DOT1L-Inhibitor mit einem IC50 von 0,4 nM.
-
GC13878
EPZ005687
A potent, selective inhibitor of EZH2
-
GC19141
EPZ011989
EPZ011989 ist ein potenter und oral aktiver Hemmer von Zeste Homolog 2 (EZH2) mit metabolischer StabilitÄt. EPZ011989 hat eine hemmende Hemmung fÜr EZH2 mit einem Ki-Wert von <3 nM. EPZ011989 zeigt eine robuste Methylmarkierungshemmung und AntitumoraktivitÄt. EPZ011989 kann fÜr die Erforschung verschiedener Krebsarten verwendet werden.
-
GC34136
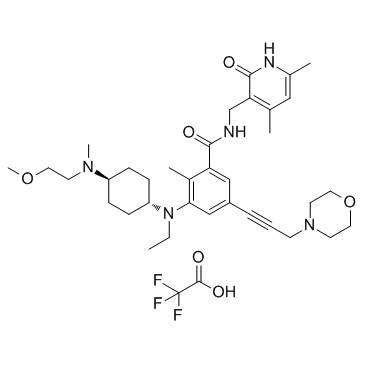
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
EPZ-011989 Trifluoracetat ist ein potenter und oral aktiver Hemmer von Zeste Homolog 2 (EZH2) mit metabolischer StabilitÄt. EPZ-011989 Trifluoracetat hat eine hemmende Hemmung fÜr EZH2 mit einem Ki-Wert von <3 nM. EPZ-011989 Trifluoracetat zeigt eine robuste Methylmarkierungshemmung und AntitumoraktivitÄt. EPZ-011989 Trifluoracetat kann fÜr die Erforschung verschiedener Krebsarten verwendet werden.
-
GC15302
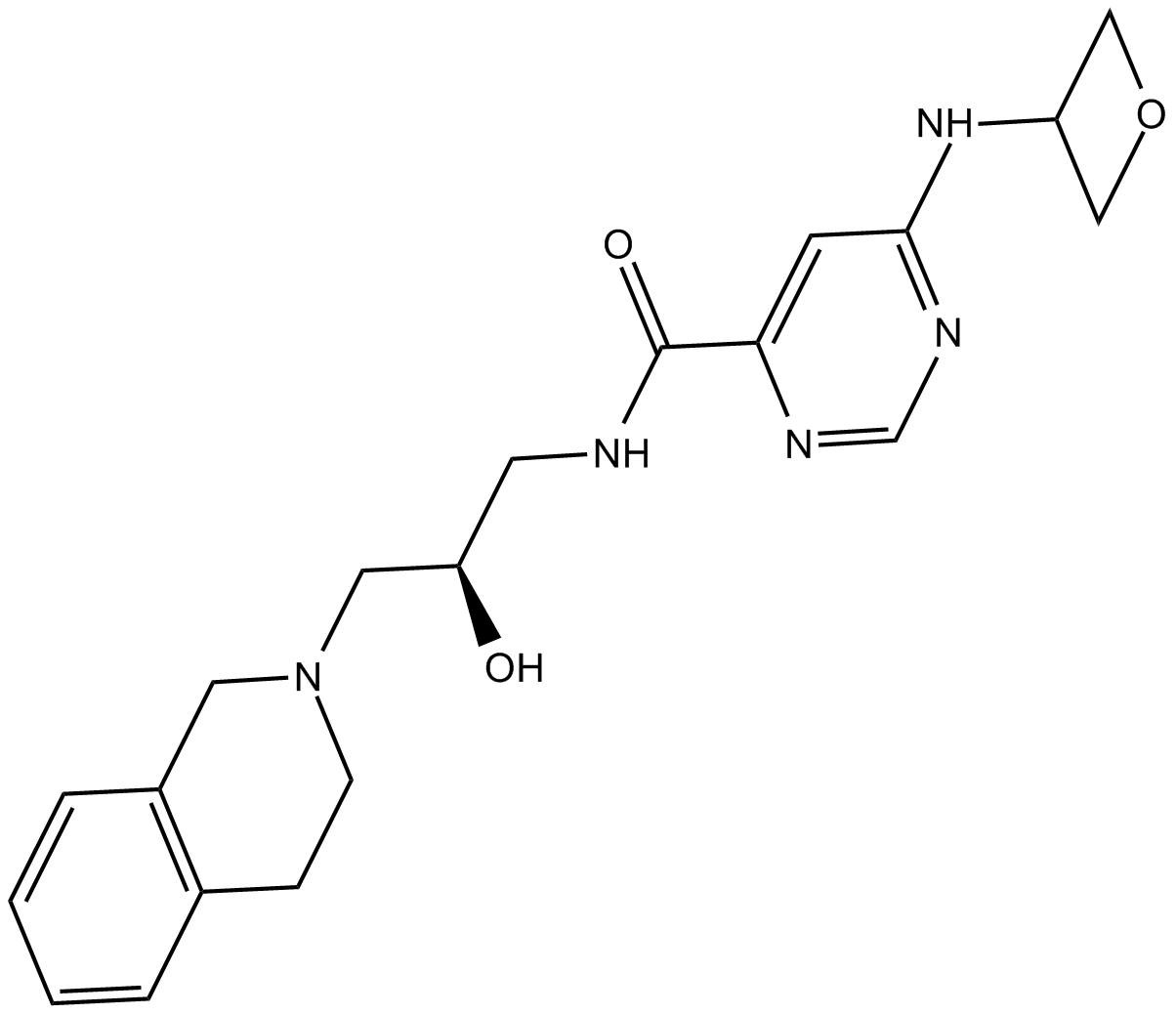
EPZ015666
EPZ015666 (GSK3235025) ist ein oral verfÜgbarer Inhibitor von PRMT5 mit einem IC50 von 22 nM.
-
GC15102
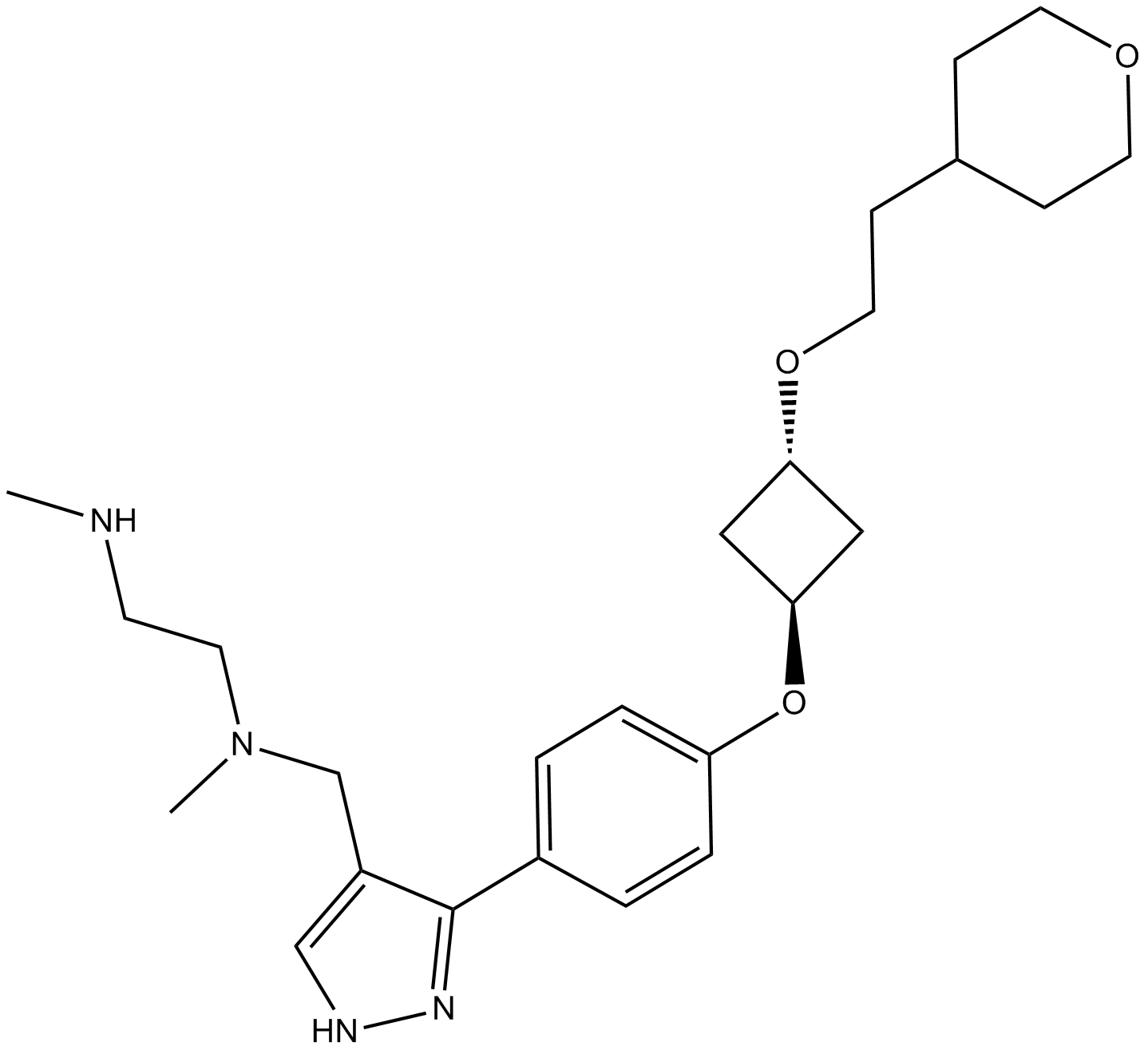
EPZ020411
EPZ020411 ist ein selektiver Inhibitor von PRMT6 mit einem IC50 von 10 nM und hat eine >10-fache SelektivitÄt fÜr PRMT6 gegenÜber PRMT1 und PRMT8. EPZ020411 kann fÜr die Krebsforschung verwendet werden.
-
GC36000
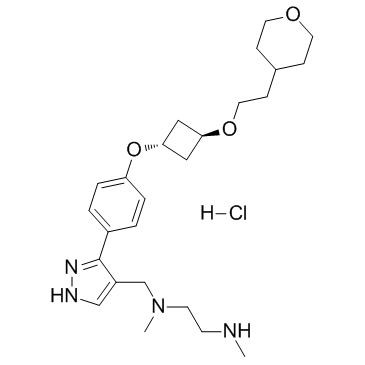
EPZ020411 hydrochloride
EPZ020411 Hydrochlorid ist ein selektiver Inhibitor von PRMT6 mit einem IC50 von 10 nM, es hat eine >10-fache SelektivitÄt fÜr PRMT6 gegenÜber PRMT1 und PRMT8. EPZ020411 Hydrochlorid kann fÜr die Krebsforschung verwendet werden.
-
GC16224
EPZ031686
EPZ031686 ist ein potenter und oral aktiver SMYD3-Inhibitor mit einem IC50-Wert von 3 nM. EPZ031686 kann fÜr die Krebsforschung verwendet werden.
-
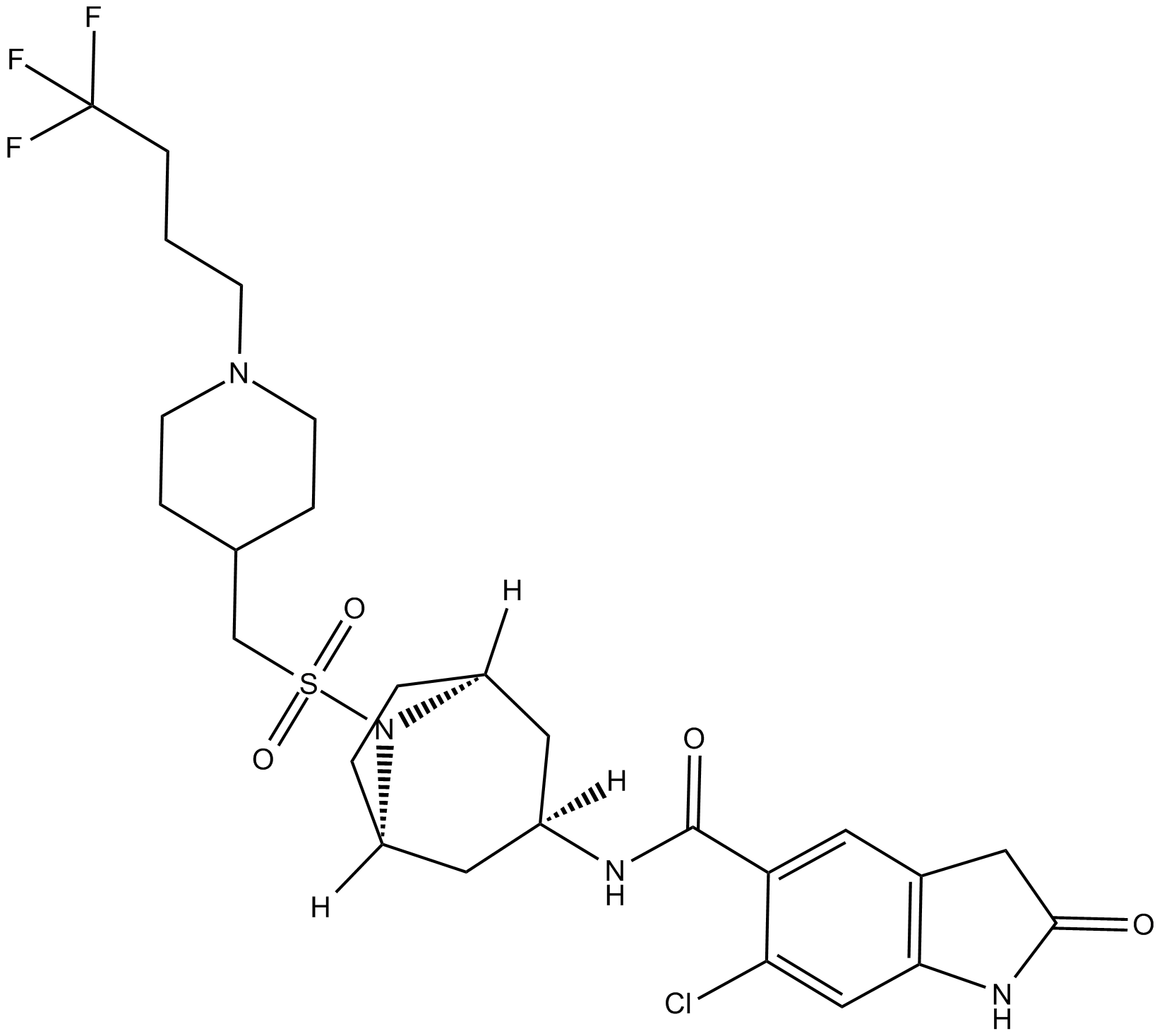
GC12932
EPZ5676
A highly potent DOT1L inhibitor
-
GC64575
Et-29
Et-29 ist ein potenter und selektiver SIRT5-Inhibitor (Ki=40 nM).
-
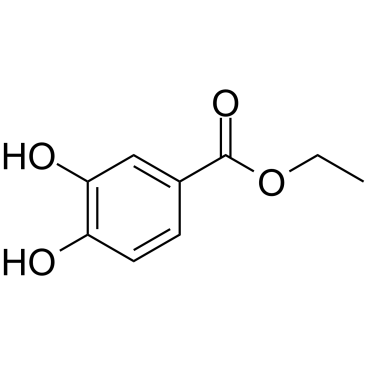
GC61669
Ethyl 3,4-dihydroxybenzoate
Ethyl 3,4-Dihydroxybenzoat (Ethyl Protocatechuate), ein Antioxidans, ist ein Prolyl-Hydroxylase-Hemmer, der in der Testa von Erdnusssamen vorkommt.
-
GC10635
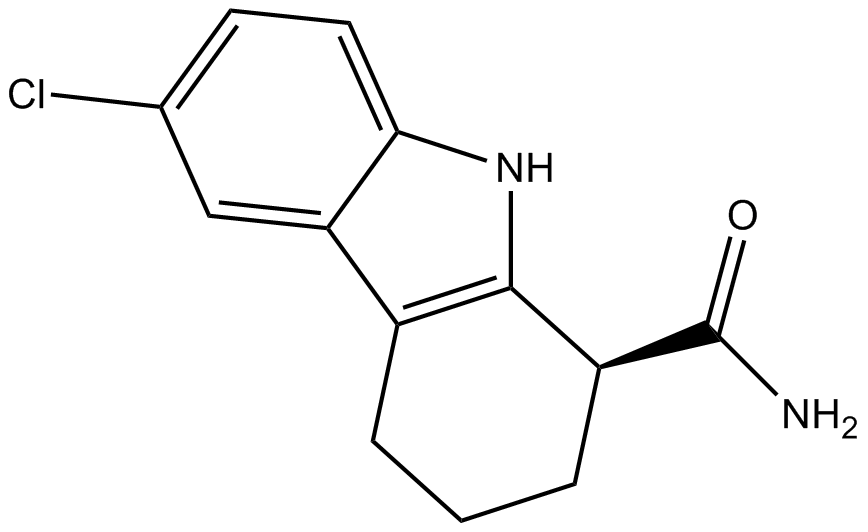
EX 527 (SEN0014196)
A SIRT1 inhibitor
-
GC17126
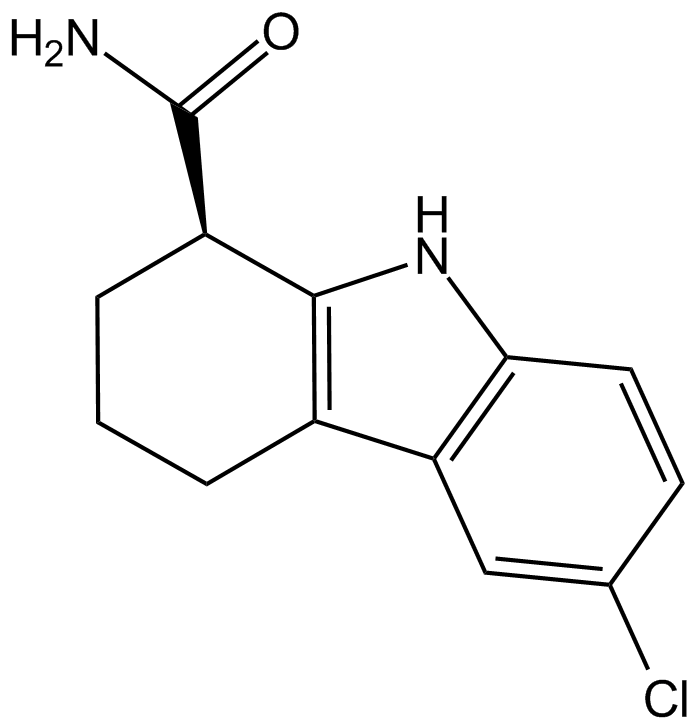
EX-527 R-enantiomer
EX-527 R-Enantiomer ((R)-EX-527) ist ein R-Enantiomer von Selisistat. Selisistat (EX-527) ist ein potenter und selektiver SIRT1-Inhibitor mit einem IC50-Wert von 98 nM.
-
GC13417
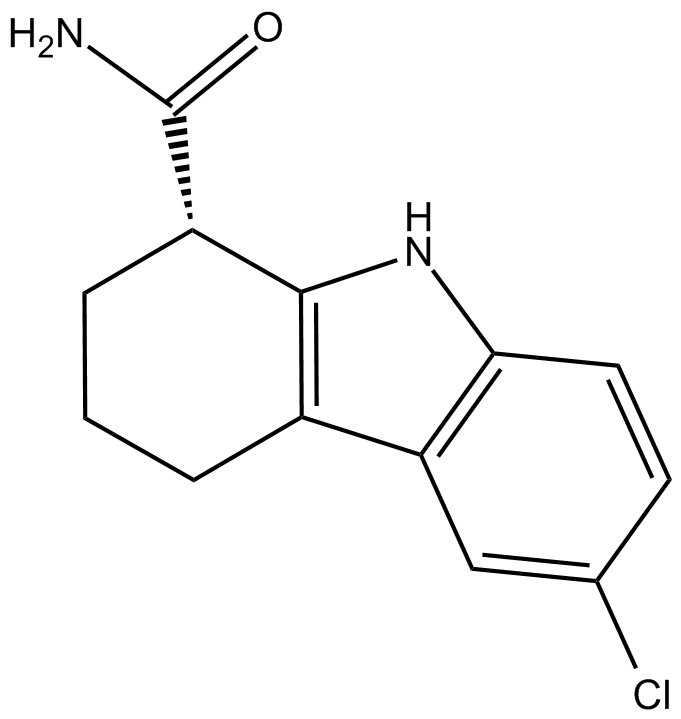
EX-527 S-enantiomer
EX-527 S-Enantiomer ((S)-EX-527) ist ein potenter und selektiver SIRT1-Inhibitor mit einem IC50 von 98 nM.
-
GC65980
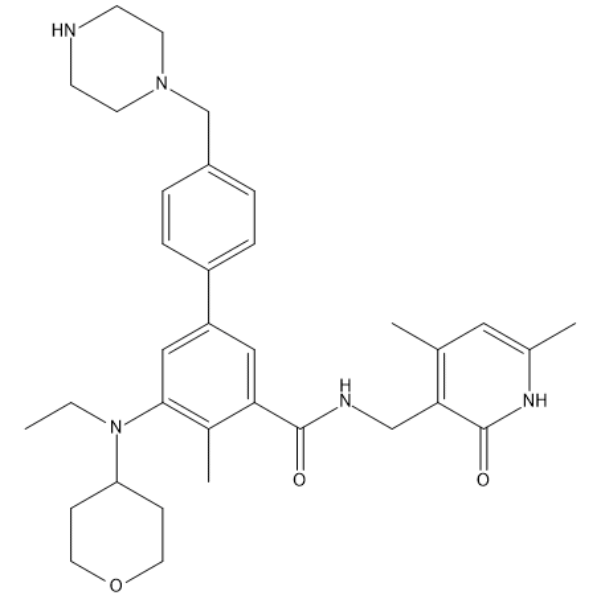
EZH2-IN-13
EZH2-IN-13 ist ein potenter EZH2-Inhibitor, fÜr Details siehe Verbindung 73 im Patent WO2017139404. EZH2-IN-13 kann verwendet werden, um Krebserkrankungen oder prÄkanzerÖse LÄsionen im Zusammenhang mit EZH2-AktivitÄt zu untersuchen.
-
GC19149
EZM 2302
EZM 2302 ist ein Inhibitor der Coaktivator-assoziierten Arginin-Methyltransferase 1 (CARM1) mit einem IC50 von 6nM.
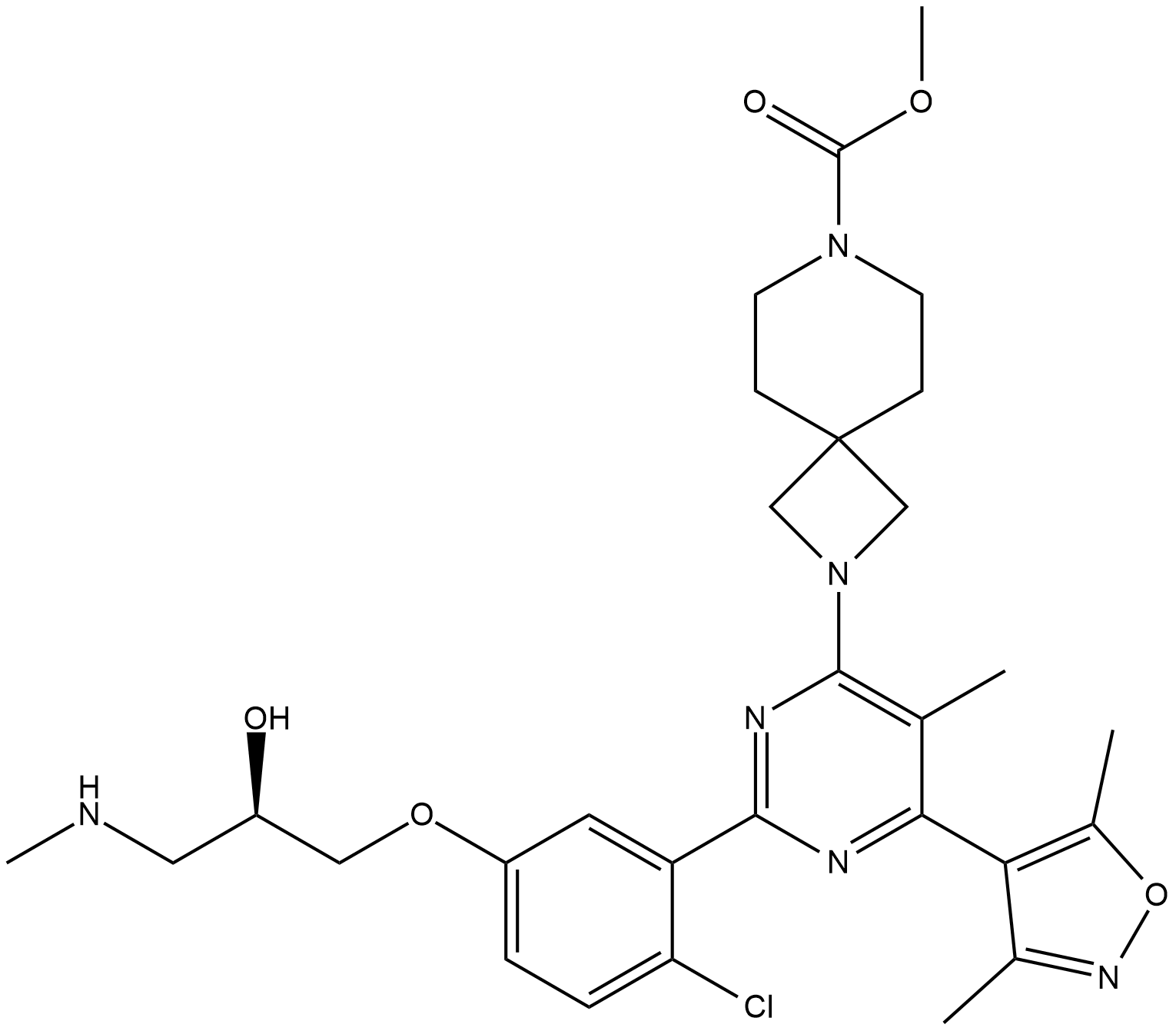
-
GC64900
EZM0414
EZM0414 ist ein potenter, selektiver, oral bioverfÜgbarer Inhibitor von SETD2 (IC50 = 18 nM im biochemischen SETD2-Assay; IC50 = 34 nM im zellulÄren Assay). EZM0414 kann fÜr die Erforschung des rezidivierten oder refraktÄren multiplen Myeloms und des diffusen großzelligen B-Zell-Lymphoms verwendet werden.
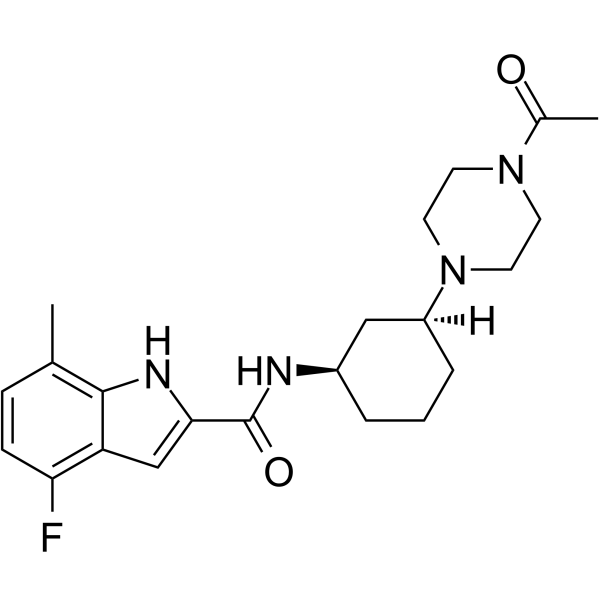
-
GC43651
F-Amidine (trifluoroacetate salt)
F-amidine is an inhibitor of protein arginine deiminases (PADs) that is selective for PAD1 and PAD4 (IC50s = 29.5, 350, and 21.6 μM for PAD1, PAD3, and PAD4 in vitro, respectively).
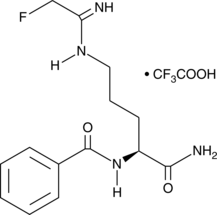
-
GC13139
FG-4592 (ASP1517)
Ein Hemmstoff von HIF-PH-Enzymen.

-
GC16638
FG2216
FG2216 (IOX3) ist ein potenter und oral aktiver Inhibitor der HIF-Prolylhydroxylase-2 (PHD2) mit einem IC50 von 3,9 μM.
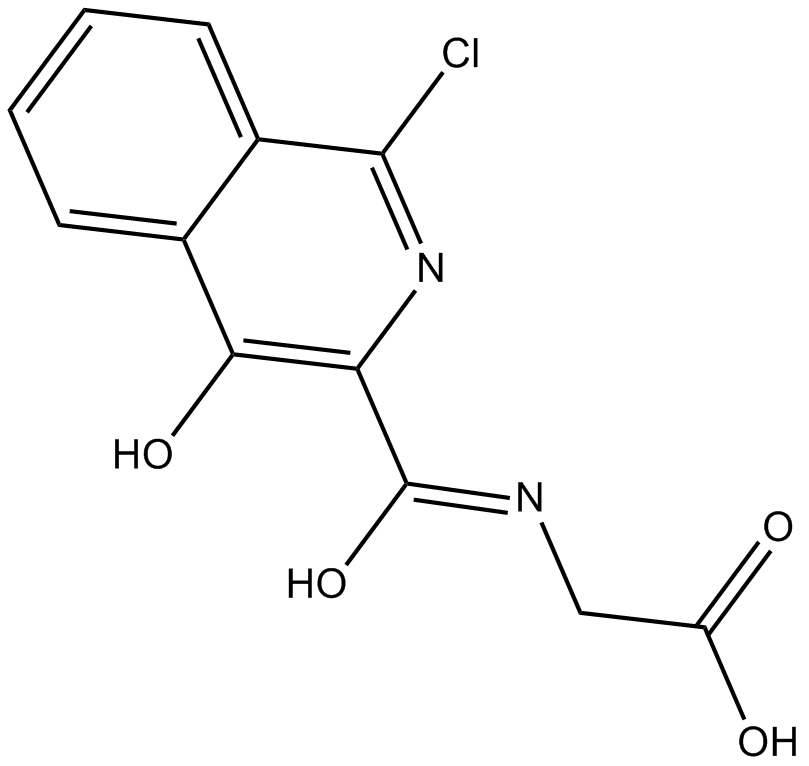
-
GC64821
FHD-286
FHD-286 ist ein BRG1/BRM-ATPase-Inhibitor zur Behandlung von BAF-bedingten Erkrankungen wie akuter myeloischer LeukÄmie.
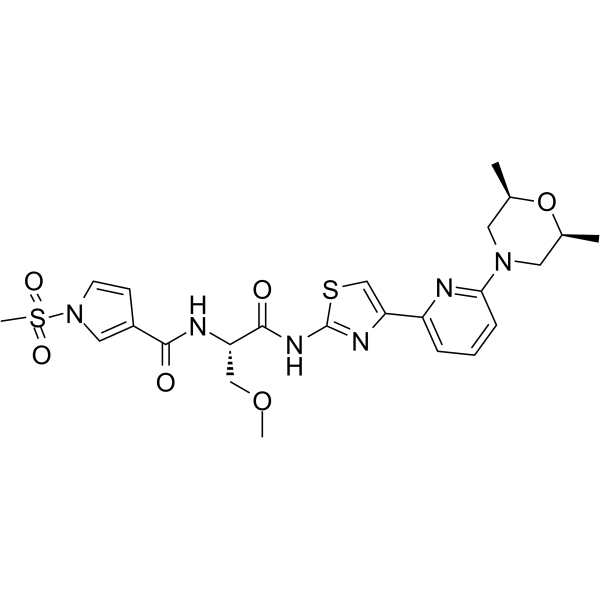
-
GC65160
FHT-1015
FHT-1205 ist ein potenter SMARCA4/SMARCA2 ATPase (BRG1 und BRM) Inhibitor mit IC50s von ≤10 nM (WO2020160180A1; Verbindung 67).
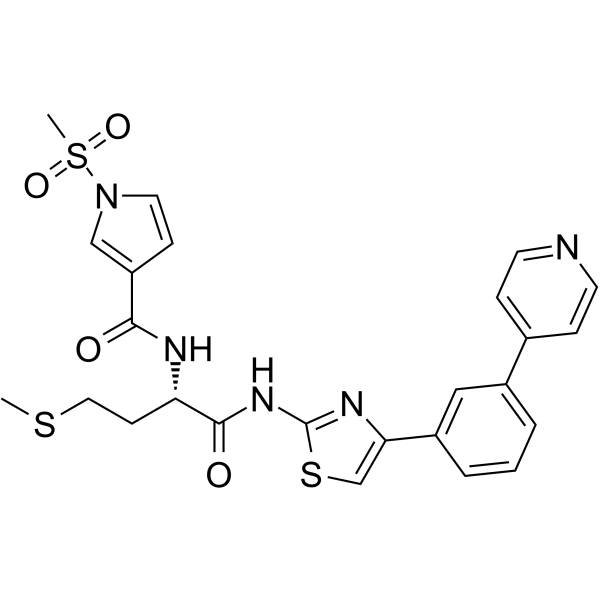
-
GC64777
FHT-1204
FHT-1204 ist ein potenter SMARCA4/SMARCA2 ATPase (BRG1 und BRM) Inhibitor mit IC50s von ≤10 nM (WO2020160180A1; Verbindung 70).
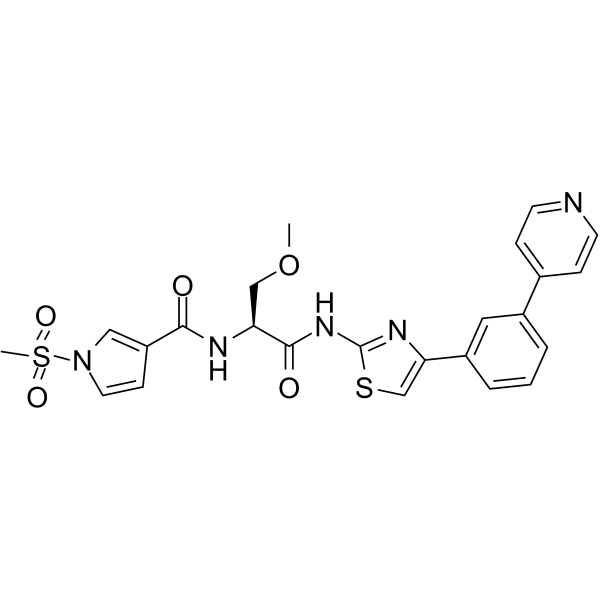
-
GC46148
Filgotinib-d4
Filgotinib-d4 (GLPG0634-d4) ist das mit Deuterium gekennzeichnete Filgotinib. Filgotinib (GLPG0634) ist ein selektiver JAK1-Inhibitor mit IC50-Werten von 10 nM, 28 nM, 810 nM und 116 nM fÜr JAK1, JAK2, JAK3 bzw. TYK2.
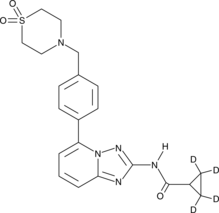
-
GN10030
Fisetin
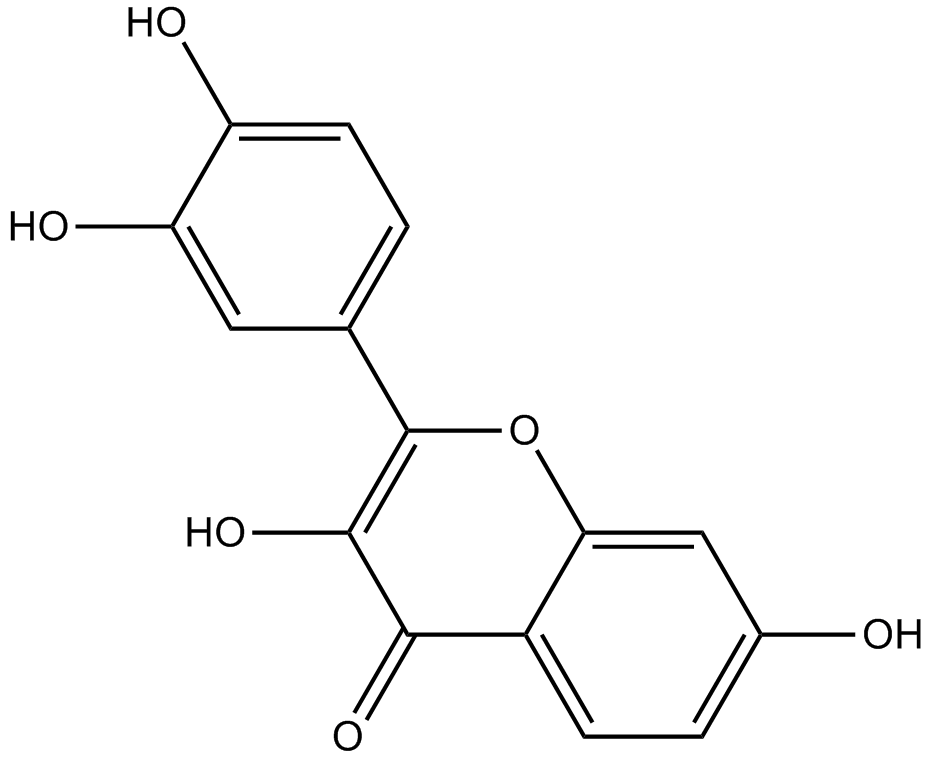
-
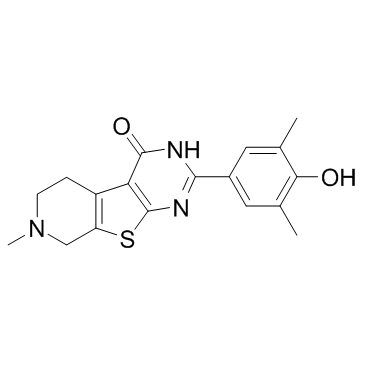
GC33015
FL-411 (BRD4-IN-1)
FL-411 (BRD4-IN-1) ist ein potenter und selektiver BRD4-Inhibitor mit einem IC50 von 0,43&7#177;0,09 μM fÜr BRD4(1).
-
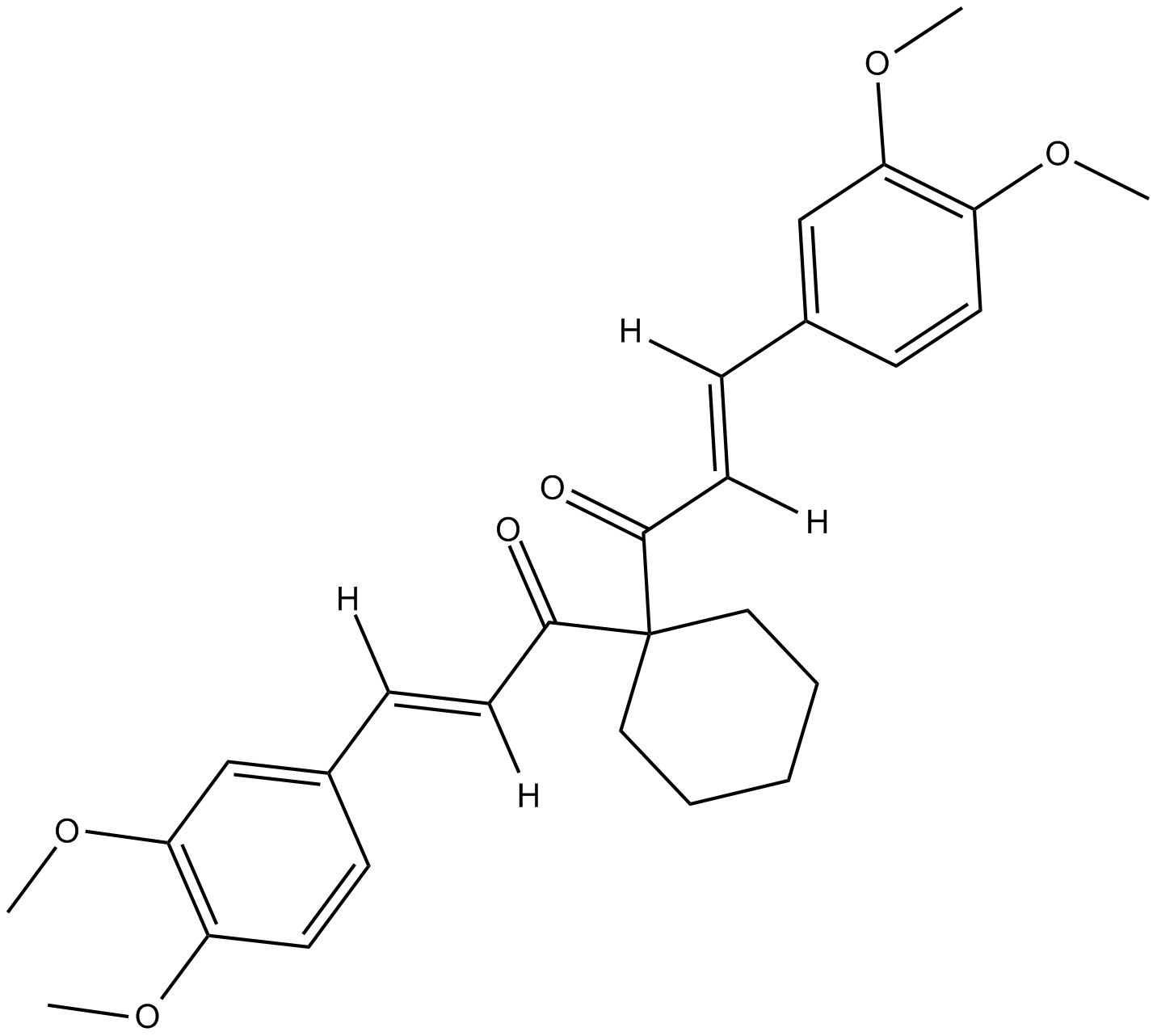
GC16875
FLLL32
FLLL32, ein synthetisches Analogon von Curcumina, ist ein dualer JAK2/STAT3-Inhibitor mit AntitumoraktivitÄt. FLLL32 kann die Induktion der STAT3-Phosphorylierung durch IFNα und IL-6 in Brustkrebszellen hemmen.
-
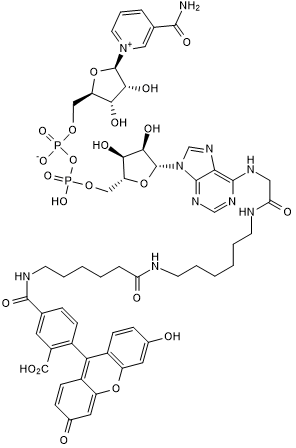
GC50506
Fluorescein-NAD+
Fluorescein-NAD+ ist eine Alternative zu radioaktiv markiertem NAD und ein Substrat fÜr die ADP-Ribosylierung.
-
GC62121
Fluzoparib
Fluzoparib (SHR3162) ist ein potenter und oral aktiver PARP1-Inhibitor (IC50=1,46±0,72 nM, ein zellfreier enzymatischer Assay) mit Überlegener AntitumoraktivitÄt. Fluzoparib hemmt selektiv die Proliferation von Zellen mit einem Mangel an homologer Rekombinationsreparatur (HR) und sensibilisiert sowohl HR-defiziente als auch HR-fÄhige Zellen gegenÜber zytotoxischen Wirkstoffen. Fluzoparib weist in vivo gute pharmakokinetische Eigenschaften auf und kann fÜr die Erforschung von rezidiviertem Eierstockkrebs mit BRCA1/2-Mutation verwendet werden.
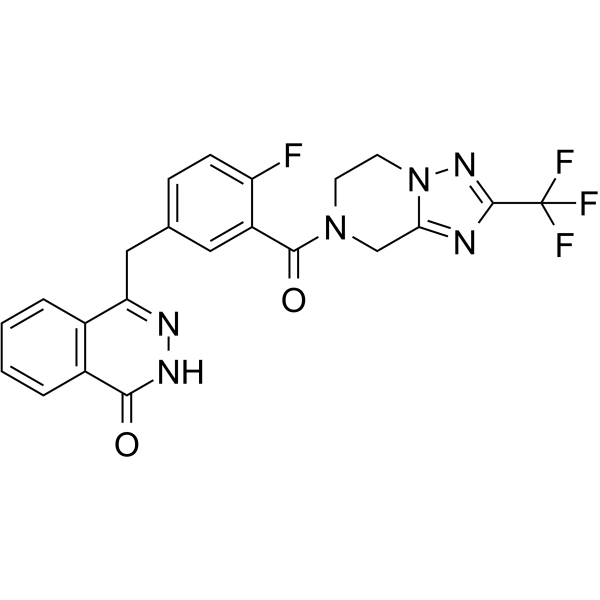
-
GC50537
FM19G11
FM19G11 ist ein Hypoxie-induzierbarer Faktor-1-alpha (HIF-1α)-Inhibitor und hemmt die Hypoxie-induzierte Luciferase-AktivitÄt mit einem IC50 von 80 nM in HeLa-Zellen.
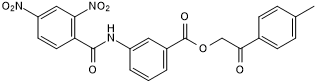
-
GC19408
FM381
FM-381 is a highly potent and JAK3-selective janus kinase (JAK) inhibitor
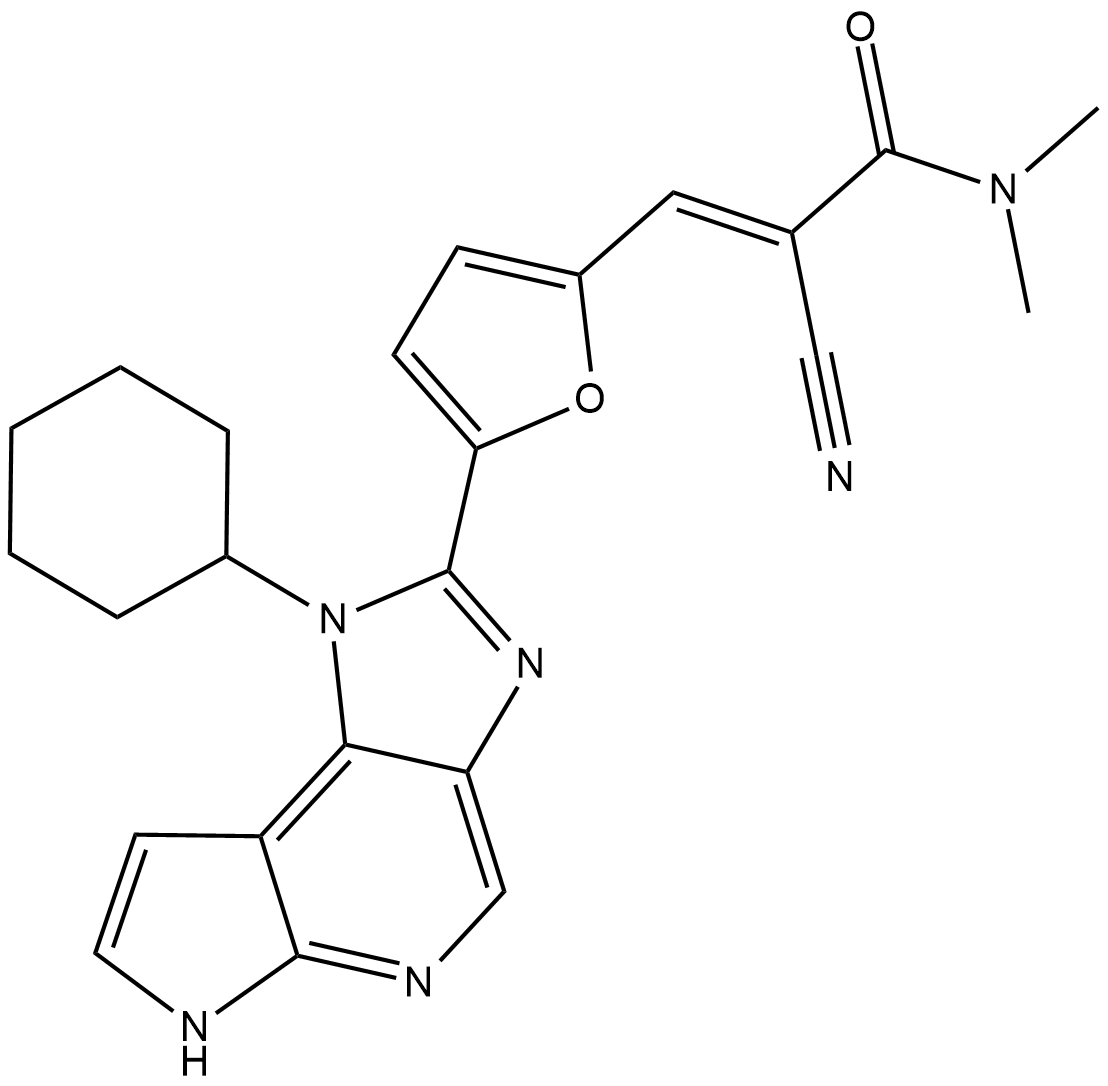
-
GC62461
FNDR-20123
FNDR-20123 ist ein sicherer, erstklassiger und oral aktiver Anti-Malaria-HDAC-Inhibitor mit IC50-Werten von 31 nM bzw. 3 nM fÜr Plasmodium bzw. humanes HDAC.

-
GC14395
Fostriecin sodium salt
A potent inhibitor of protein phosphatases 2A and 4
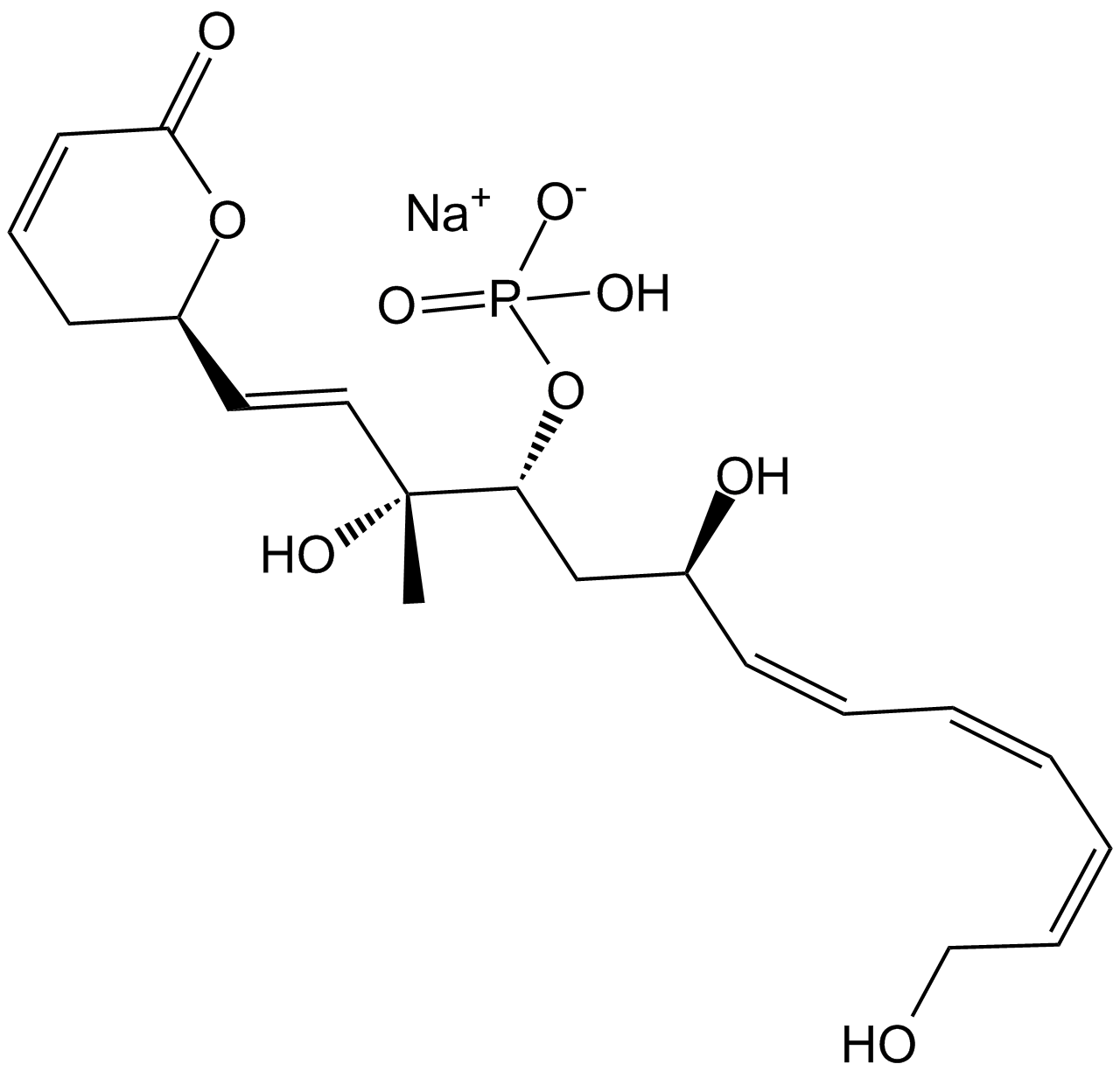
-
GC38044
Fraxinellone
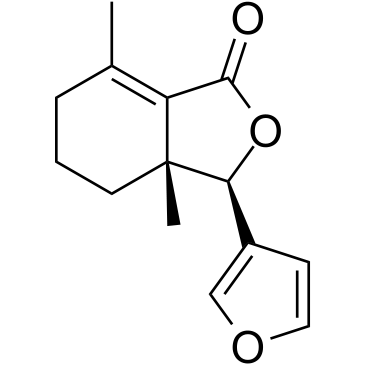
-
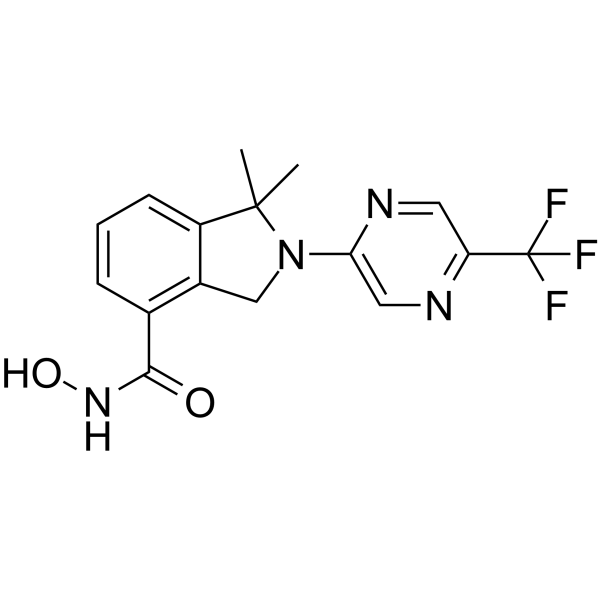
GC65206
FT895
FT895 ist ein potenter und selektiver HDAC11-Inhibitor mit einem IC50 von 3 nM.
-
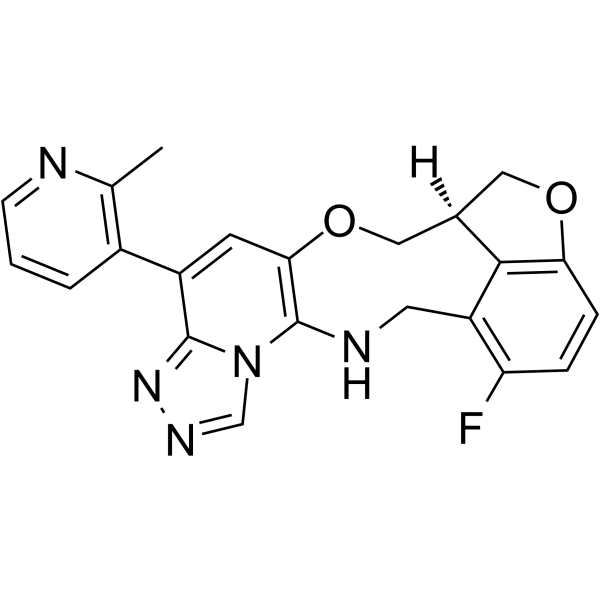
GC63545
FTX-6058
FTX-6058 ((S)-FTX-6058) ist ein potenter und oral aktiver Inhibitor der embryonalen Ektodermentwicklung (EED).
-
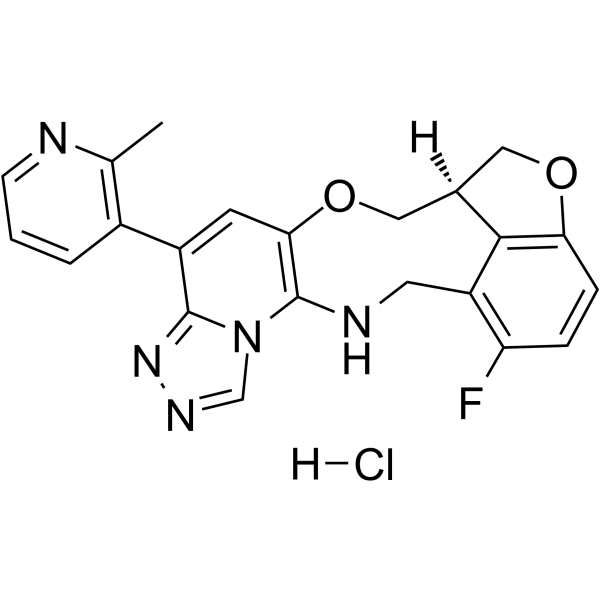
GC63546
FTX-6058 hydrochloride
(S)-Pociredir ((S)-FTX-6058) Hydrochlorid ist ein potenter und oral aktiver Inhibitor der embryonalen Ektodermentwicklung (EED).
-
GC10456
Fucosterol
Fucosterol ist ein Sterin, das aus Algen, Algen oder Diatomeen isoliert wird.
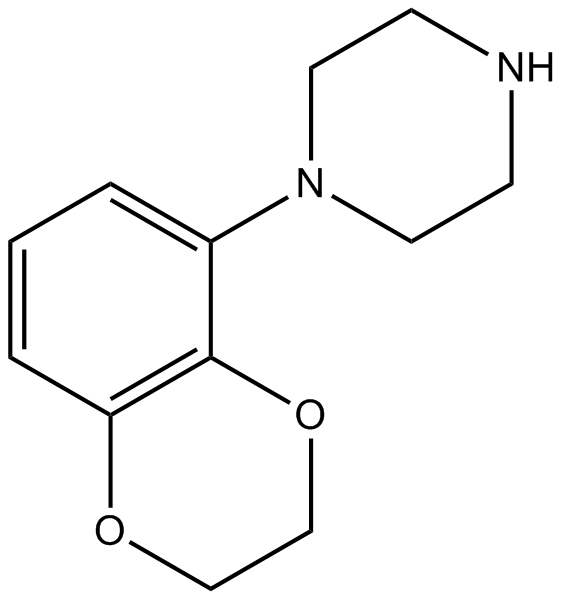
-
GC43715
Furamidine (hydrochloride)
Furamidin (Hydrochlorid) (DB75-Dihydrochlorid) ist ein selektiver Inhibitor der Protein-Arginin-Methyltransferase 1 (PRMT1) mit einem IC50-Wert von 9,4 μM.
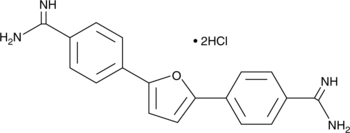
-
GC13541
G007-LK
G007-LK ist ein potenter und selektiver Inhibitor von TNKS1 und TNKS2 mit IC50-Werten von 46 nM bzw. 25 nM.
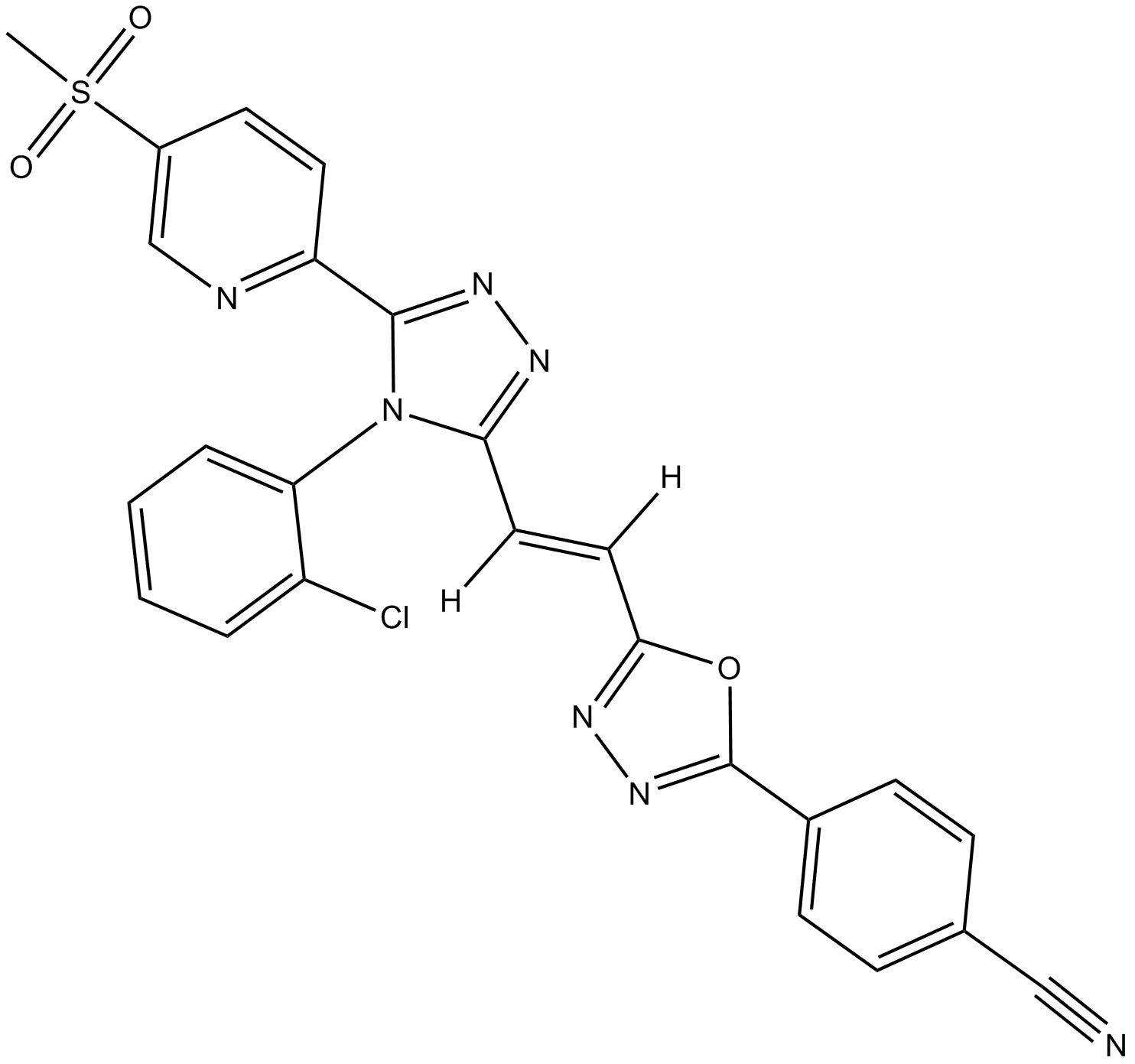
-
GC62246
G5-7
G5-7, ein oral aktiver und allosterischer JAK2-Inhibitor, hemmt selektiv die JAK2-vermittelte Phosphorylierung und Aktivierung von EGFR (Tyr1068) und STAT3 durch Bindung an JAK2. G5-7 induziert Zellzyklusarrest, Apoptose und besitzt antiangiogene Wirkung. G5-7 hat das Potenzial fÜr Gliomstudien.
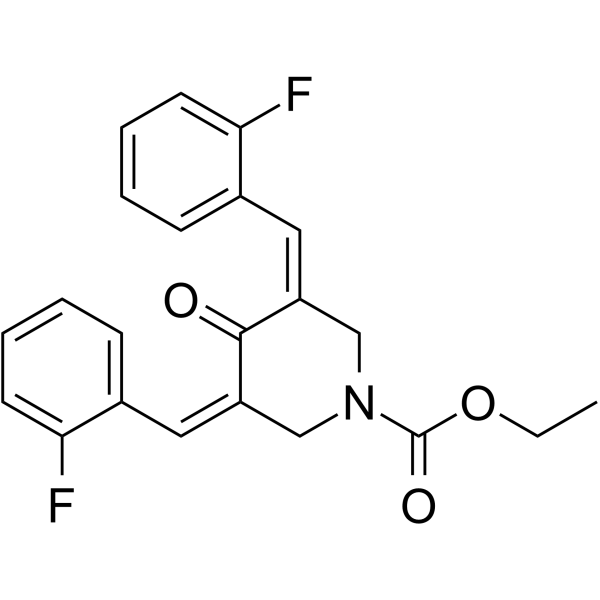
-
GC38062
Gambogenic acid
GambogensÄure ist ein Wirkstoff in Gamboge mit krebsbekÄmpfender Wirkung. GambogensÄure wirkt als wirksamer Inhibitor von EZH2, bindet spezifisch und kovalent an Cys668 innerhalb der EZH2-SET-DomÄne und induziert die EZH2-Ubiquitinierung.
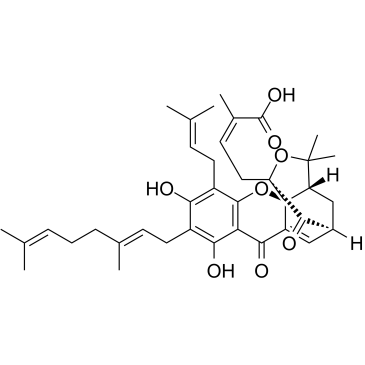
-
GC40900
Ganoderic Acid D
GanoderinsÄure D, ein stark sauerstoffreiches tetrazyklisches Triterpenoid, ist der Hauptwirkstoff von Ganoderma lucidum. GanoderinsÄure D reguliert die Proteinexpression von SIRT3 hoch und induziert das deacetylierte Cyclophilin D (CypD) durch SIRT3. GanoderinsÄure D hemmt die Energieumprogrammierung von Dickdarmkrebszellen, einschließlich Glukoseaufnahme, Laktatproduktion, Pyruvat- und Acetyl-Coenzym-Produktion in Dickdarmkrebszellen. GanoderinsÄure D induziert die Apoptose von menschlichem HeLa-Zervixkarzinom.
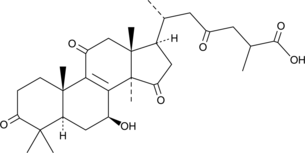
-
GC13474
Garcinol
Garcinol, ein aus Garcinia indica gewonnenes polyisoprenyliertes Benzophenon, Übt Anti-Cholinesterase-Eigenschaften gegenÜber Acetylcholinesterase (AChE) und Butyrylcholinesterase (BChE) mit IC50-Werten von 0,66 μM bzw. 7,39 μM aus.
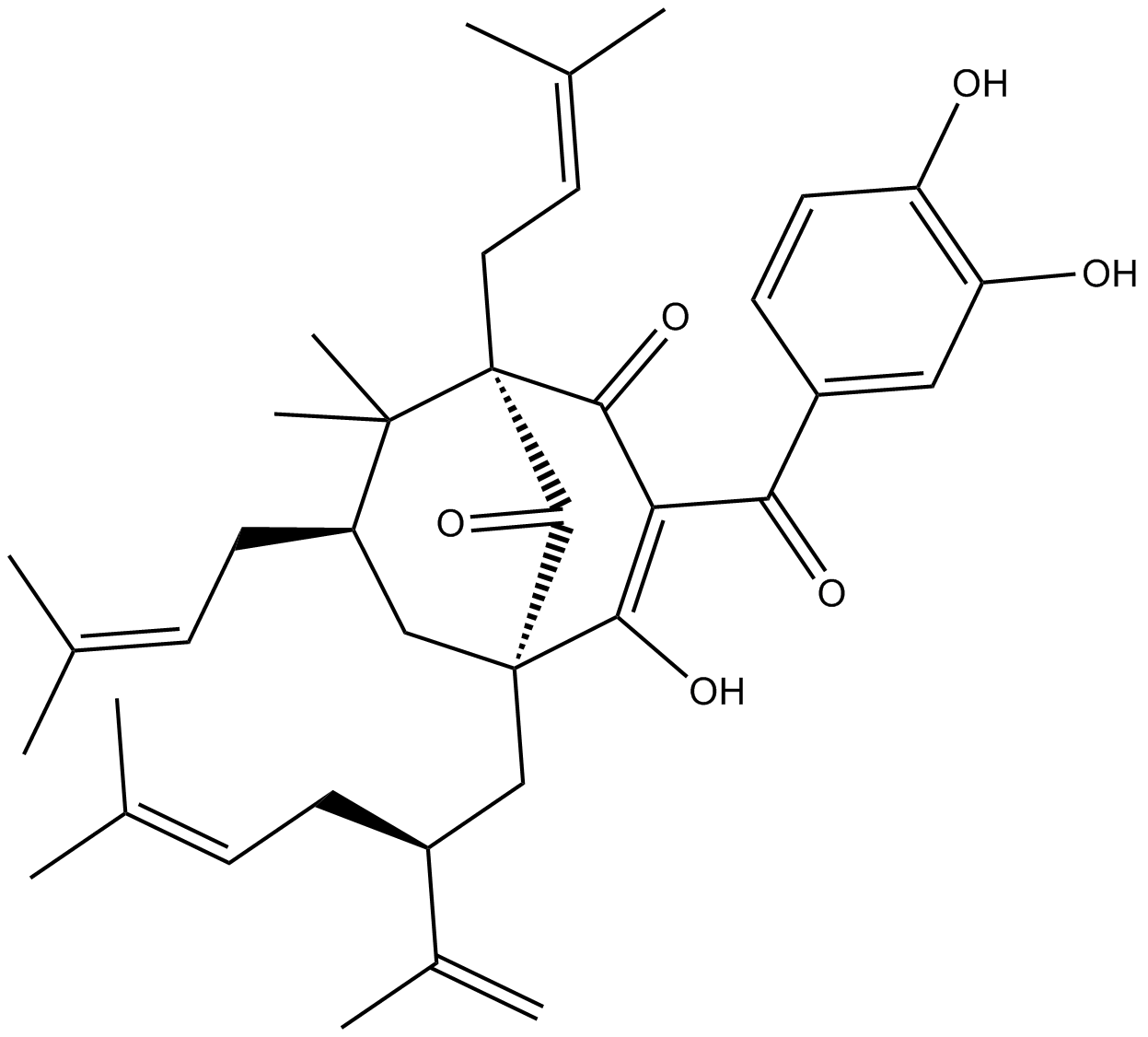
-
GC60172
Gardenia yellow
Gardeniengelb ist ein aktives Mitglied von Crocin, erhÖht die mRNA-Expression von SIRT3 und wirkt als oral aktives Antidepressivum.

-
GC32958
GDC-0339
GDC-0339 ist ein potenter, oral bioverfÜgbarer und gut vertrÄglicher Pan-Pim-Kinase-Inhibitor mit Kis von 0,03 nM, 0,1 nM und 0,02 nM fÜr Pim1, Pim2 bzw. Pim3. GDC-0339 wird als mÖgliche Behandlung des multiplen Myeloms entdeckt.
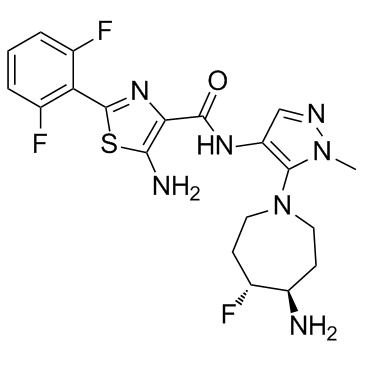
-
GC68378
GDC-4379

-
GC19542
GeA-69
GeA-69 ist ein selektiver, allosterischer Inhibitor der Poly-Adenosin-Diphosphat-Ribose-Polymerase 14 (PARP14), der auf die Makrodomäne 2 (MD2) abzielt, mit einem Kd-Wert von 2,1 µM. GeA-69 ist an Mechanismen zur Reparatur von DNA-Schäden beteiligt und verhindert die Rekrutierung von PARP14 MD2 an Stellen laserinduzierter DNA-Schäden.

-
GN10473
Ginkgolide C
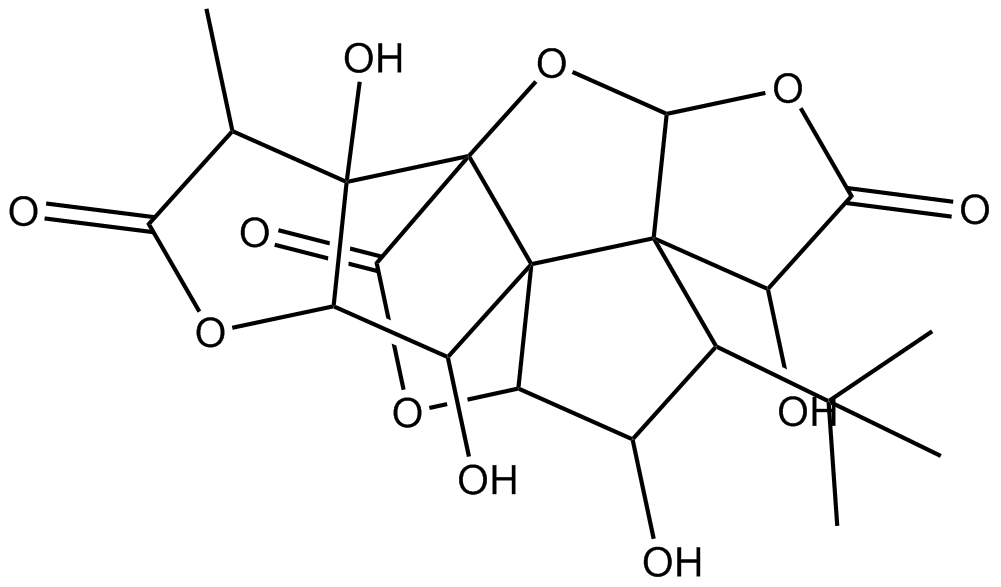
-
GN10584
Ginsenoside Rk1
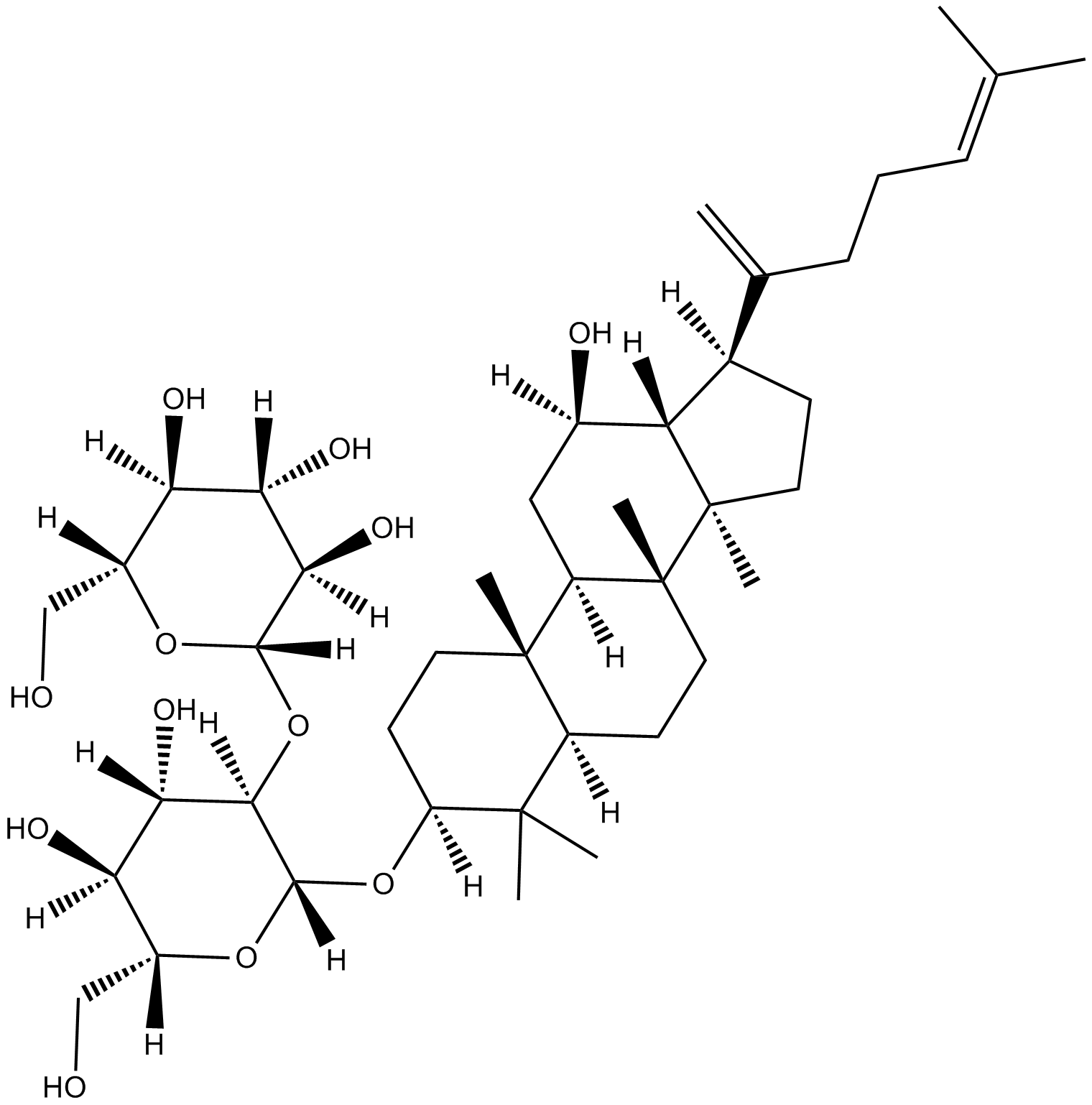
-
GC48994
Girard’s Reagent P-d5
An internal standard for the quantification of Girard’s reagent P
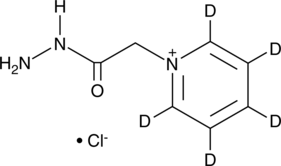
-
GC33050
Givinostat (ITF-2357)
Givinostat (ITF-2357) (ITF-2357) ist ein HDAC-Inhibitor mit einem IC50 von 198 und 157 nM fÜr HDAC1 bzw. HDAC3.

-
GC33159
Givinostat hydrochloride (ITF-2357 hydrochloride)
Givinostat (ITF-2357) Hydrochlorid ist ein HDAC-Inhibitor mit einem IC50-Wert von 198 und 157 nM fÜr HDAC1 bzw. HDAC3.
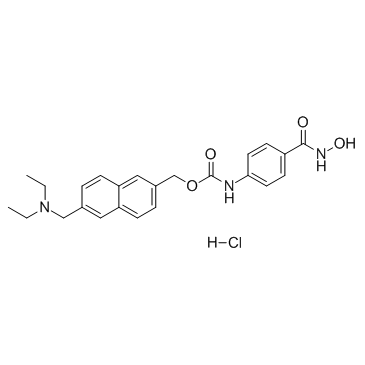
-
GC12579
GLPG0634
GLPG0634 (GLPG0634) ist ein selektiver und oral aktiver JAK1-Inhibitor mit IC50 von 10 nM, 28 nM, 810 nM und 116 nM fÜr JAK1, JAK2, JAK3 bzw. TYK2.
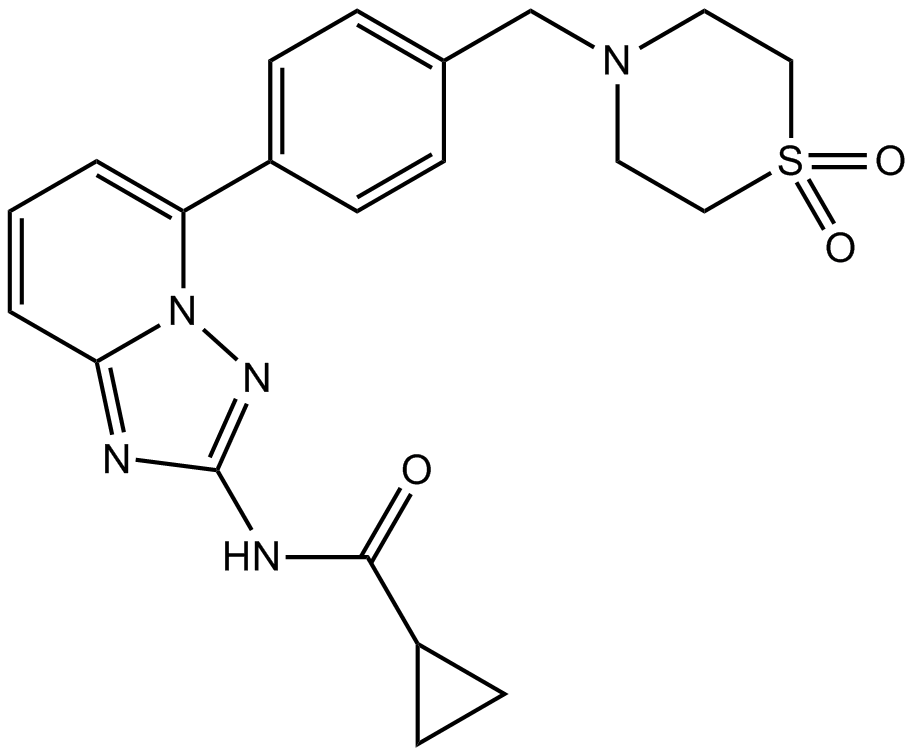
-
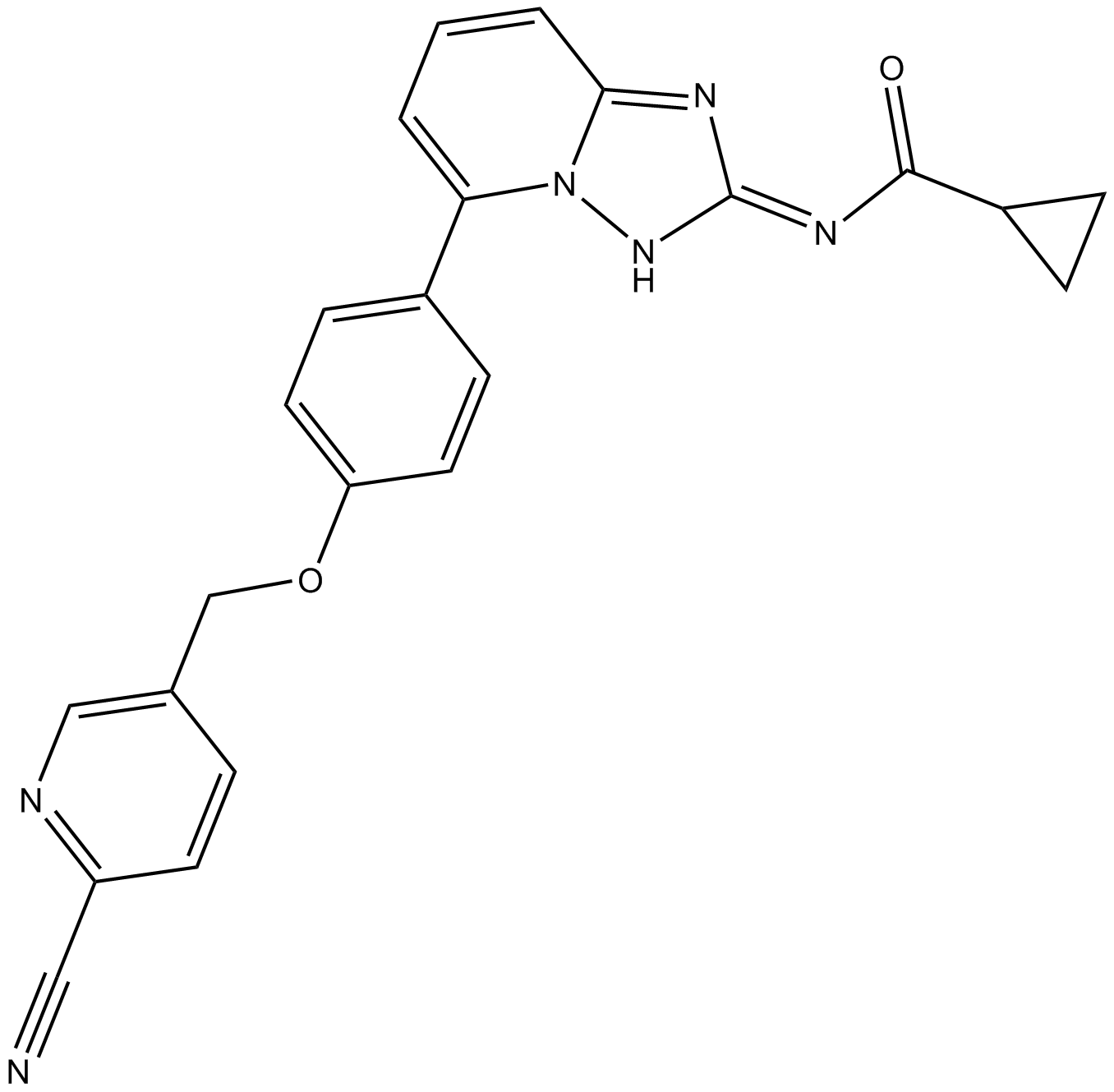
GC17222
GLPG0634 analogue
Das GLPG0634-Analogon (Compoun 176) ist ein Breitband-JAK-Inhibitor mit IC50-Werten von < 100 nM gegen JAK1, JAK2 und JAK3.
-
GC30263
Glucosamine (D-Glucosamine)
Glucosamin (D-Glucosamin) (D-Glucosamin (D-Glucosamin)) ist ein Aminozucker und ein wichtiger VorlÄufer in der biochemischen Synthese von glykosylierten Proteinen und Lipiden und wird als NahrungsergÄnzungsmittel verwendet.
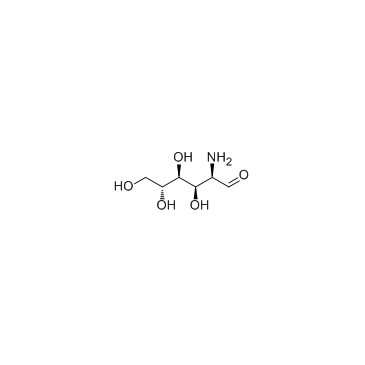
-
GN10669
Glucosamine sulfate
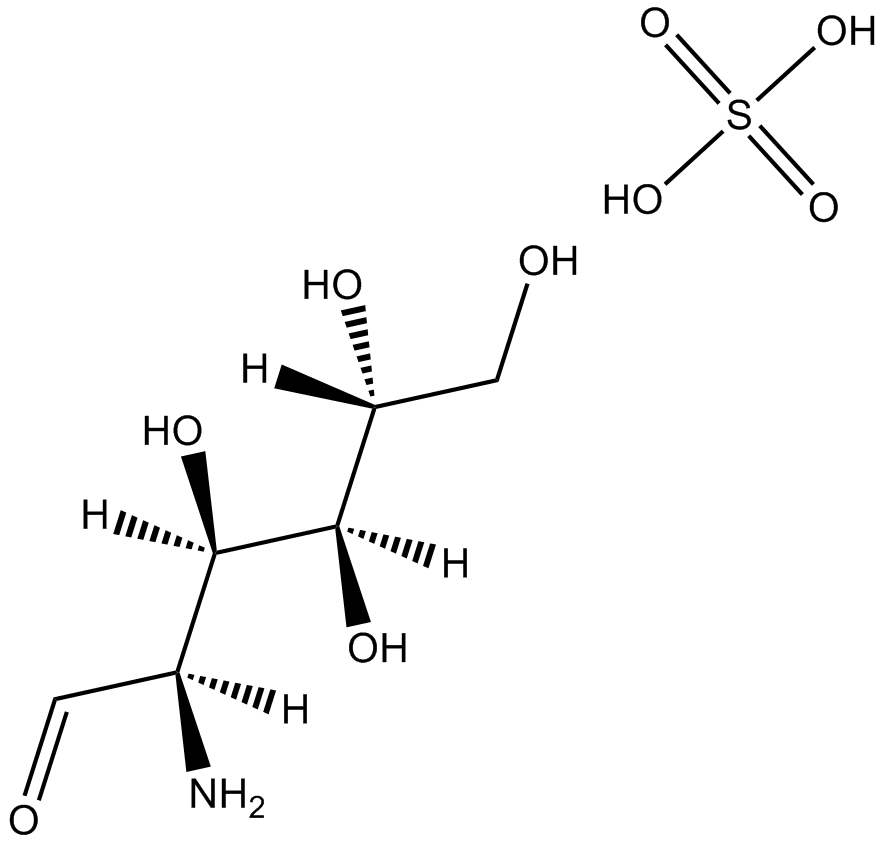
-
GC36153
Glucose-conjugated MGMT inhibitor
Glucose-konjugierter MGMT-Inhibitor ist ein potenter O6-Methylguanin-DNAmethyltransferase (MGMT)-Inhibitor mit IC50-Werten von 32 nM in vitro (Zellextrakte) und 10 nM in HeLa S3-Zellen.
-
GC34595
GN44028
GN44028 ist ein potenter und oral aktiver Hypoxie-induzierbarer Faktor (HIF)-1α-Inhibitor mit einem IC50 von 14 nM. GN44028 hemmt die Hypoxie-induzierte HIF-1α-TranskriptionsaktivitÄt, ohne die HIF-1α-mRNA-Expression, die HIF-1α-Proteinakkumulation oder die HIF-1α/HIF-1β-Heterodimerisierung zu unterdrÜcken. GN44028 kann in der Krebsforschung eingesetzt werden.
-
GC36168
GNA002
GNA002 ist ein hochpotenter, spezifischer und kovalenter EZH2 (Enhancer of zeste homolog 2)-Inhibitor mit einem IC50 von 1,1 μM. GNA002 kann spezifisch und kovalent an Cys668 innerhalb der EZH2-SET-DomÄne binden und den EZH2-Abbau durch den COOH-Terminus der Hsp70-interagierenden Protein (CHIP)-vermittelten Ubiquitinierung auslÖsen. GNA002 reduziert effizient die EZH2-vermittelte H3K27-Trimethylierung und reaktiviert durch den Polycomb-Repressor-Komplex 2 (PRC2) stummgeschaltete Tumorsuppressorgene.
-
GC32960
GNE-049
GNE-049 ist ein hochpotenter und selektiver CBP-Inhibitor mit einem IC50-Wert von 1,1 nM im TR-FRET-Assay. GNE-049 hemmt auch BRET und BRD4(1) mit IC50-Werten von 12 nM bzw. 4200 nM.
-
GC68439
GNE-064

-
GC33212
GNE-207
GNE-207 ist ein potenter, selektiver und oral bioverfÜgbarer Inhibitor der BromodomÄne von CBP mit einem IC50 von 1 nM, der einen >2500-fachen selektiven Index gegenÜber BRD4 aufweist (1). GNE-207 zeigt eine hervorragende CBP-Potenz mit einem EC50-Wert von 18 nM fÜr die MYC-Expression in MV-4-11-Zellen.