Proteases
Proteases is a general term for a class of enzymes that hydrolyze protein peptide chains. According to the way they degrade polypeptides, they are divided into two categories: endopeptidases and telopeptidases. The former can cut the large molecular weight polypeptide chain from the middle to form prions and peptones with smaller molecular weights; the latter can be divided into carboxypeptidase and aminopeptidase, which respectively remove the peptide from the free carboxyl terminus or free amino terminus of the polypeptide one by one. Chain hydrolysis produces amino acids.
A general term for a class of enzymes that hydrolyze peptide bonds in proteins. According to the way they hydrolyze polypeptides, they can be divided into endopeptidases and exopeptidases. Endopeptidase cleaves the interior of the protein molecule to form smaller molecular weight peptones and peptones. Exopeptidase hydrolyzes peptide bonds one by one from the end of the free amino group or carboxyl group of protein molecules, and frees amino acids, the former is aminopeptidase and the latter is carboxypeptidase. Proteases can be classified into serine proteases, sulfhydryl proteases, metalloproteases and aspartic proteases according to their active centers and optimum pH. According to the optimum pH value of its reaction, it is divided into acidic protease, neutral protease and alkaline protease. The proteases used in industrial production are mainly endopeptidases.
Proteases are widely found in animal offal, plant stems and leaves, fruits and microorganisms. Microbial proteases are mainly produced by molds and bacteria, followed by yeast and actinomycetes.
Enzymes that catalyze the hydrolysis of proteins. There are many kinds, the important ones are pepsin, trypsin, cathepsin, papain and subtilisin. Proteases have strict selectivity for the reaction substrates they act on. A protease can only act on certain peptide bonds in protein molecules, such as the peptide bonds formed by the hydrolysis of basic amino acids catalyzed by trypsin. Proteases are widely distributed, mainly in the digestive tract of humans and animals, and are abundant in plants and microorganisms. Due to limited animal and plant resources, the industrial production of protease preparations is mainly prepared by fermentation of microorganisms such as Bacillus subtilis and Aspergillus terrestris.
Targets for Proteases
- Caspase(114)
- Aminopeptidase(24)
- ACE(74)
- Calpains(20)
- Carboxypeptidase(10)
- Cathepsin(81)
- DPP-4(31)
- Elastase(26)
- Gamma Secretase(67)
- HCV Protease(59)
- HSP(113)
- HIV Integrase(37)
- HIV Protease(47)
- MMP(228)
- NS3/4a protease(8)
- Serine Protease(18)
- Thrombin(58)
- Urokinase(4)
- Cysteine Protease(0)
- Other Proteases(18)
- Tyrosinases(47)
- 15-PGDH(1)
- Acetyl-CoA Carboxylase(13)
- Acyltransferase(59)
- Aldehyde Dehydrogenase (ALDH)(28)
- Aminoacyl-tRNA Synthetase(9)
- ATGL(1)
- Dipeptidyl Peptidase(56)
- Drug Metabolite(457)
- E1/E2/E3 Enzyme(90)
- Endogenous Metabolite(1636)
- FABP(30)
- Farnesyl Transferase(23)
- Glutaminase(14)
- Glutathione Peroxidase(14)
- Isocitrate Dehydrogenase (IDH)(28)
- Lactate Dehydrogenase(17)
- Lipoxygenase(234)
- Mitochondrial Metabolism(207)
- NEDD8-activating Enzyme(7)
- Neprilysin(12)
- PAI-1(13)
- Ser/Thr Protease(41)
- Tryptophan Hydroxylase(13)
- Xanthine Oxidase(18)
- MALT1(10)
- PCSK9(1)
Products for Proteases
- Cat.No. Nom du produit Informations
-
GC39474
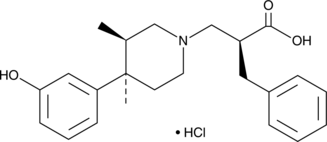
7-Ketolithocholic acid
L'acide 7-cétolithocholique (acide 3α-hydroxy-7-oxo-5β-cholanique), un acide biliaire, peut être absorbé et supprime la production endogène d'acide biliaire et la sécrétion de cholestérol biliaire.
-
GC33616
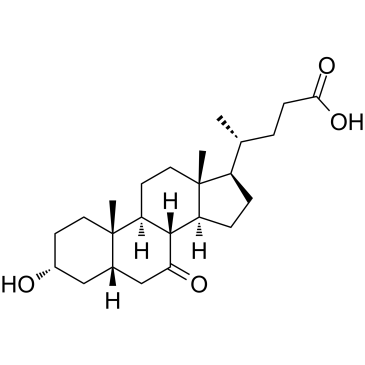
7-Methylguanine
La 7-méthylguanine est un métabolite de la méthylation de l'ADN.
-
GC62817
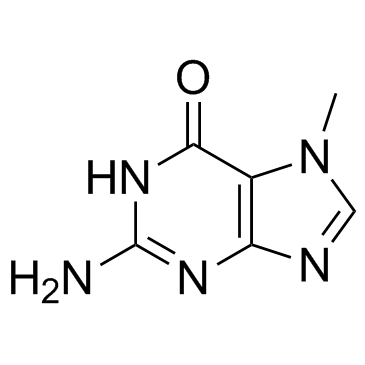
7-Methylguanosine 5’-diphosphate sodium
Le 7-méthylguanosine 5'-diphosphate (7-méthyl-GDP) sodium, un analogue de coiffe, peut être utilisé dans la synthèse d'analogues de coiffe d'ARNm.
-
GC31617
7-Methylxanthine
La 7-méthylxanthine, un dérivé méthyle de la xanthine, est l'un des composants puriques des calculs urinaires.
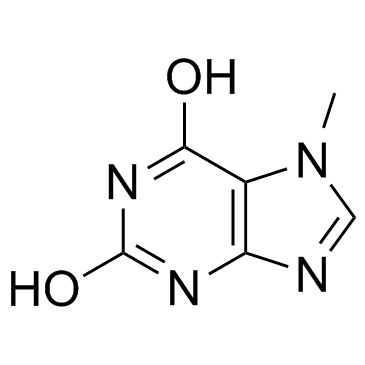
-
GC14433
7α,25-dihydroxy Cholesterol
Le 7α, 25-dihydroxycholestérol (7α,25-OHC) est un agoniste puissant et sélectif et un ligand endogène du récepteur GPCR orphelin EBI2 (GPR183).
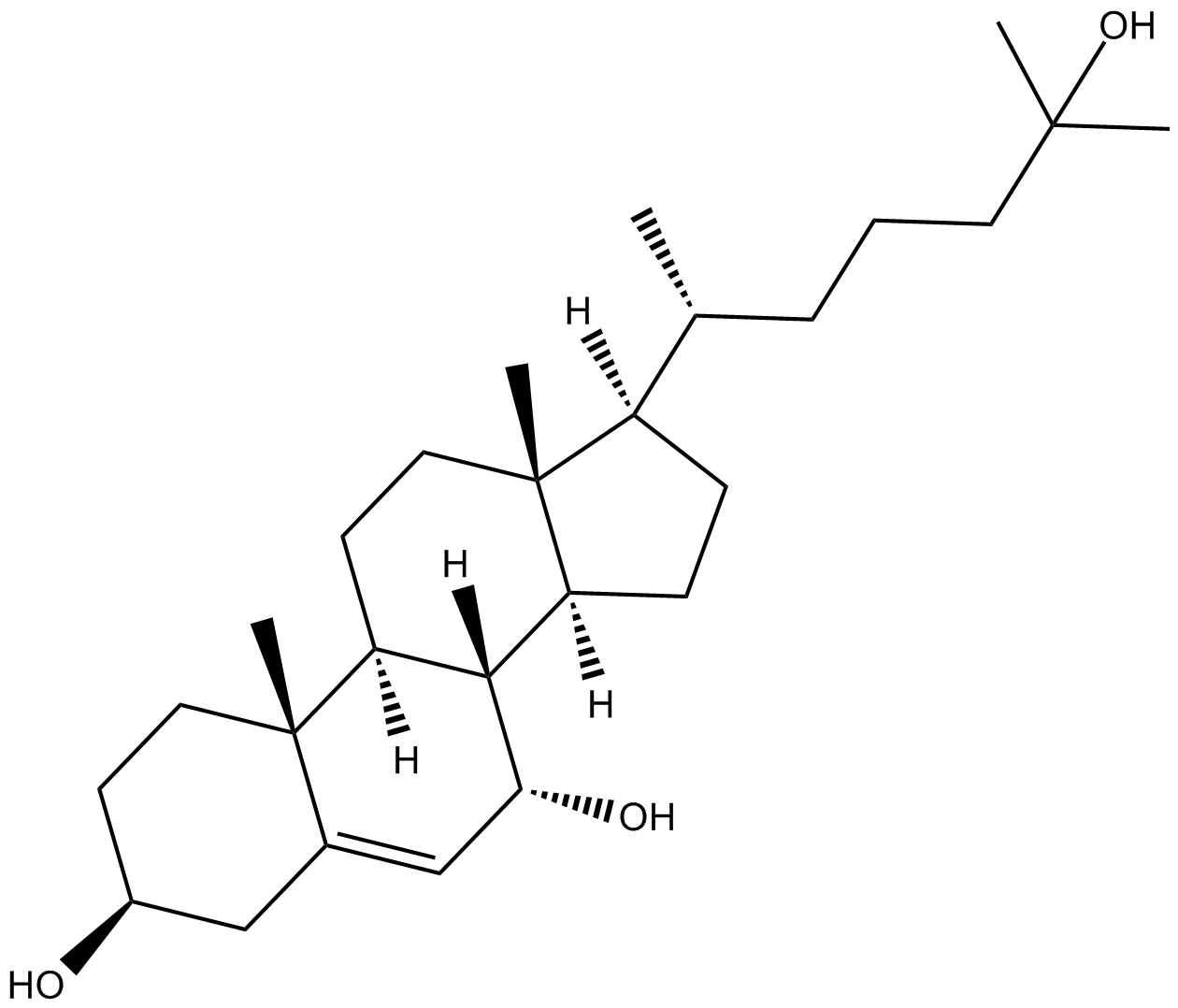
-
GC40462
8(R)-HETE
8(R)-HETE is biosynthesized by lipoxygenation of arachidonic acid in marine invertebrates such as gorgonian corals and starfish.
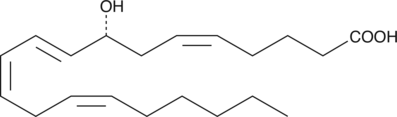
-
GC41136
8(S),15(S)-DiHETE
8(S),15(S)-DiHETE is formed when 15(S)-HETE is subjected to further oxidation by 15-LO.
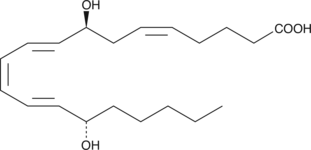
-
GC40382
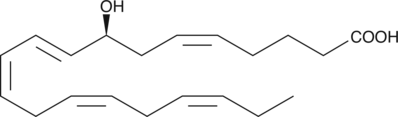
8(S)-HEPE
8(S)-HEPE is a monohydroxy fatty acid produced by lipoxygenase oxidation of EPA.
-
GC40463
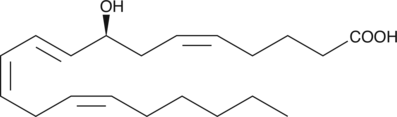
8(S)-HETE
8(S)-HETE is a major lipoxygenase product in PMA-treated murine epidermis.
-
GC42619
8(S)-HETrE
8(S)-HETrE is a monohydroxy polyunsaturated fatty acid produced by rabbit neutrophil lipoxygenase when dihomo-γ-linolenic acid is used as a substrate.

-
GC18544
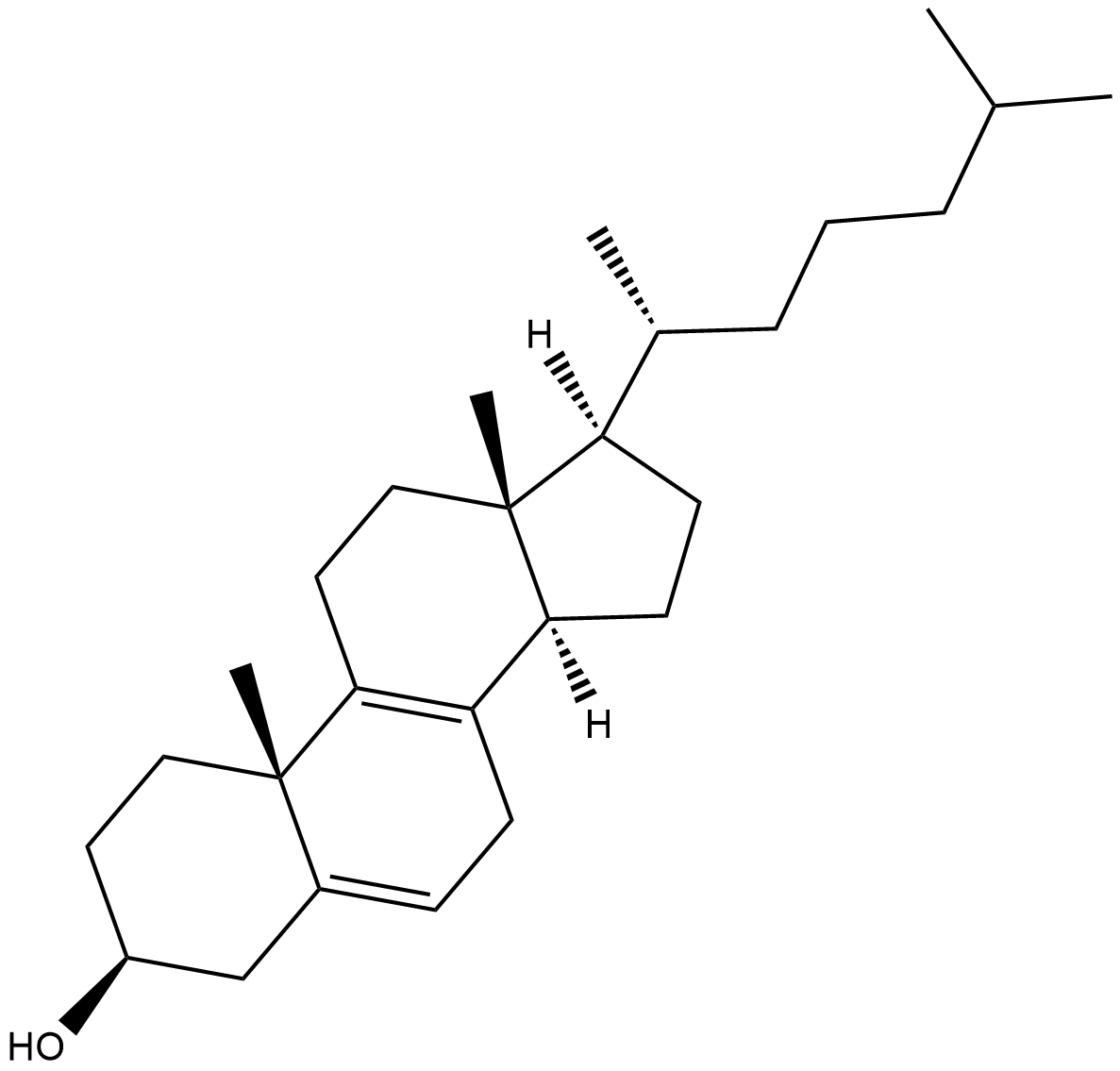
8-dehydro Cholesterol
La concentration élevée de 8-déhydro-cholestérol est l'une des caractéristiques biochimiques diagnostiques du syndrome de Smith-Lemli-Opitz classique (SLOS).
-
GC60541
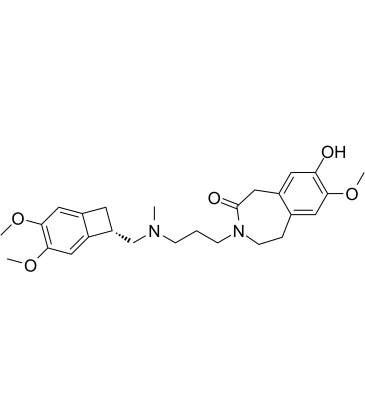
8-Demethyl Ivabradine
La 8-déméthyl ivabradine est un métabolite de l'ivabradine.
-
GC49017
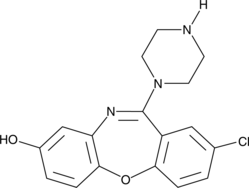
8-hydroxy Amoxapine
A metabolite of amoxapine
-
GC42626
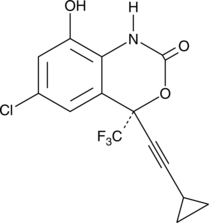
8-hydroxy Efavirenz
8-hydroxy Efavirenz is a major oxidative metabolite of the non-nucleoside reverse transcriptase inhibitor efavirenz.
-
GC18654
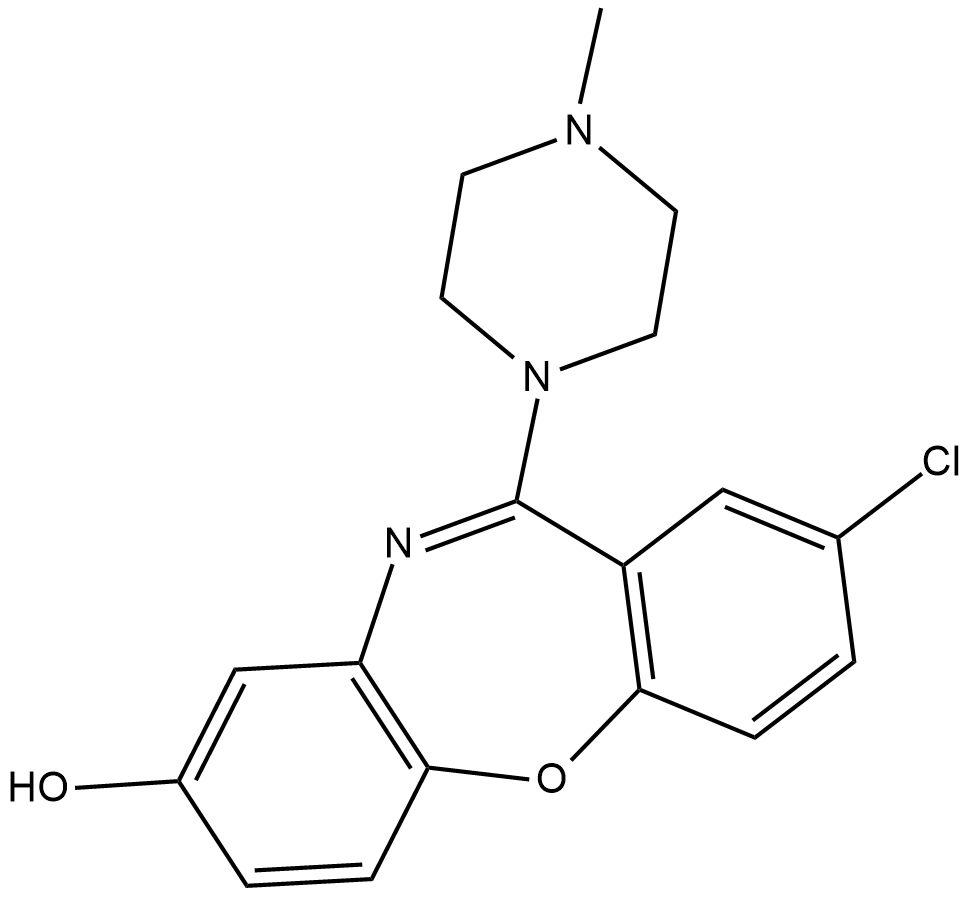
8-hydroxy Loxapine
8-hydroxy Loxapine (8-OH loxapine) is a metabolite formed when loxapine , an atypical antipsychotic, is metabolized by the cytochrome P450 isoform CYP1A2.
-
GC49544
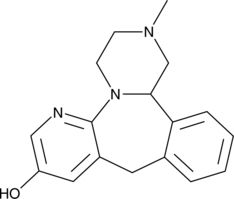
8-hydroxy Mirtazapine
A metabolite of mirtazapine
-
GC40926
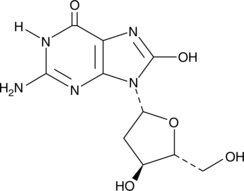
8-Hydroxy-2'-deoxyguanosine
La 8-hydroxy-2'-désoxyguanosine est un biomarqueur essentiel du stress oxydatif et de la carcinogenèse.
-
GC35204
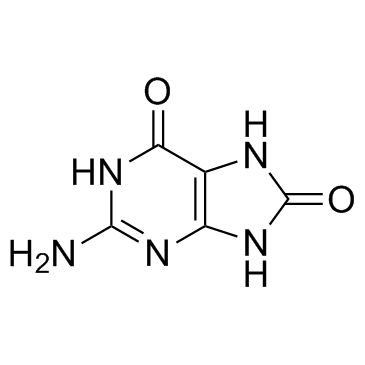
8-Hydroxyguanine
La 8-hydroxyguanine est une lésion pré-mutagène majeure générée À partir d'espèces réactives de l'oxygène.
-
GC31219
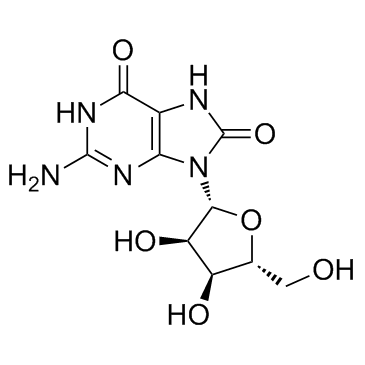
8-Hydroxyguanosine
La 8-hydroxyguanosine est un marqueur systématique du stress oxydatif et un marqueur des dommages causés par les radicaux hydroxyles À l'ARN.
-
GC18515
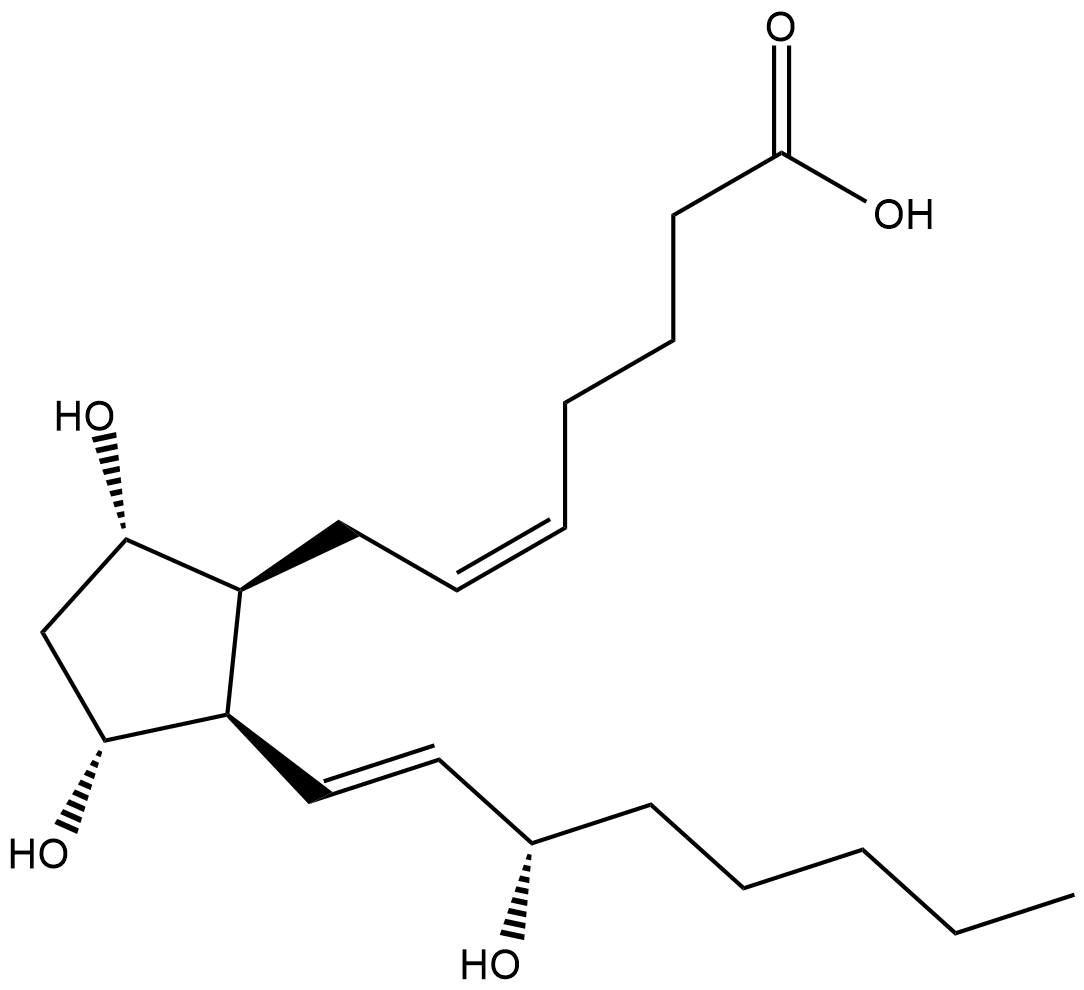
8-iso Prostaglandin F2α
La 8-iso prostaglandine F2α est un isoprostane produit par la peroxydation non enzymatique de l'acide arachidonique dans les phospholipides membranaires.
-
GC18994
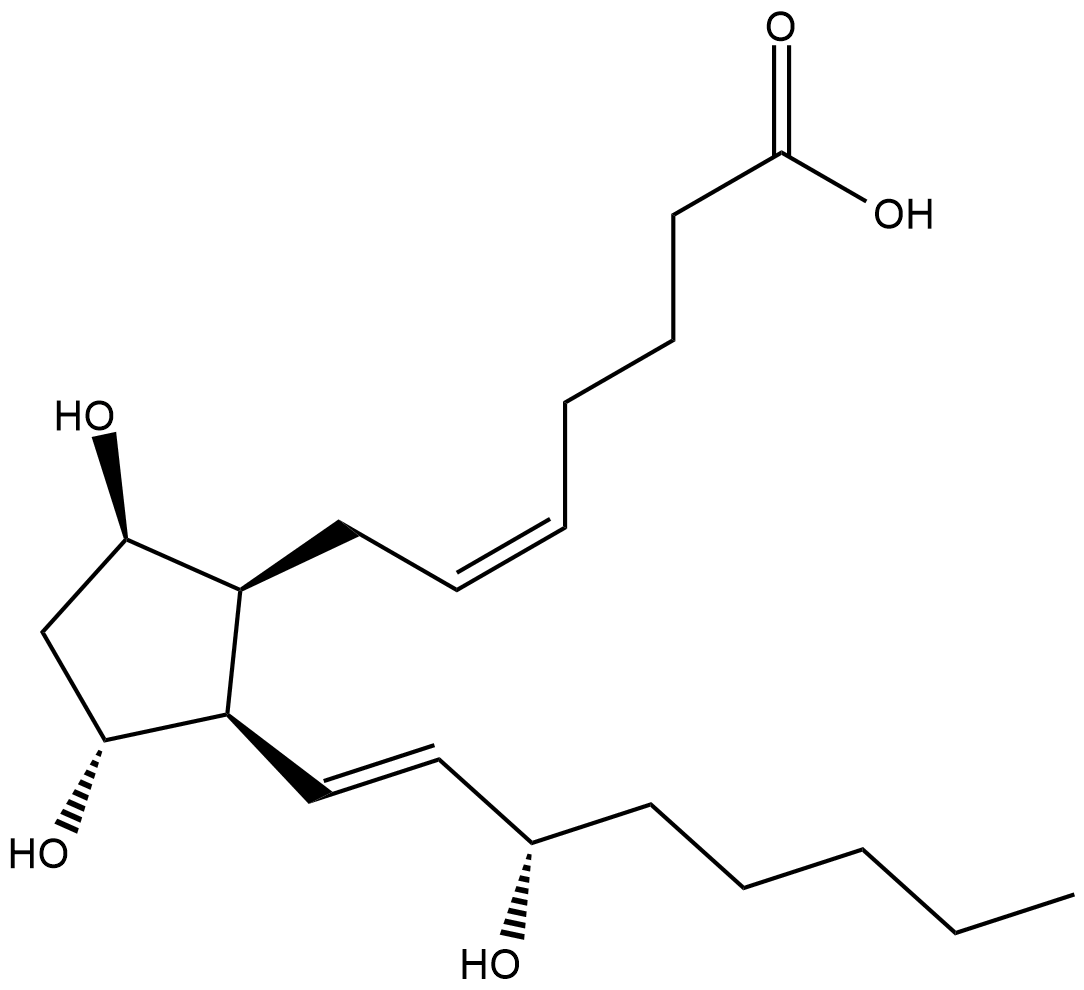
8-iso Prostaglandin F2β
8-iso Prostaglandin F2β (8-iso PGF2β) is an isomer of PGF2α with a non-enzymatic, non-cyclooxygenase origin.
-
GC41642
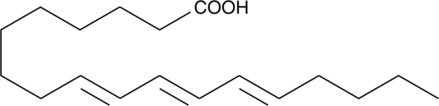
9(E),11(E),13(E)-Octadecatrienoic Acid
9(E),11(E),13(E)-Octadecatrienoic acid (β-ESA) is a conjugated polyunsaturated fatty acid that is found in plant seed oils and in mixtures of conjugated linolenic acids synthesized by the alkaline isomerization of linolenic acid.
-
GC42633
9(E)-Erythromycin A oxime
(9E)-Erythromycin A oxime is a metabolite of the semisynthetic antibiotic roxithromycin .
-
GC40542
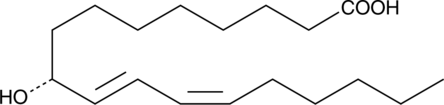
9(R)-HODE
9(R)-HODE is one of several monohydroxylated products of linoleic acid.
-
GC40029
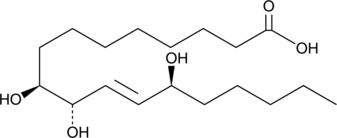
9(S),10(S),13(S)-TriHOME
9(S),10(S),13(S)-TriHOME is an oxylipin derived from linoleic acid.
-
GC46753
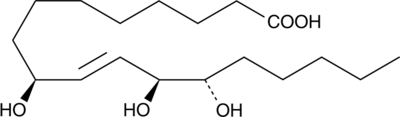
9(S),12(S),13(S)-TriHOME
An oxylipin
-
GC40383
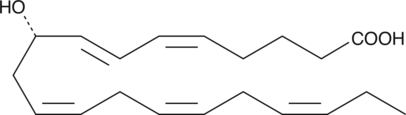
9(S)-HEPE
9(S)-HEPE is a monohydroxy fatty acid derived from EPA.
-
GC19460
9(S)-HODE
9(S)-HODE is produced by the lipoxygenation of linoleic acid in both plants and animals.

-
GC40250
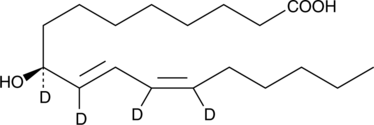
9(S)-HODE-d4 MaxSpec® Standard
9(S)-HODE-d4 is intended for use as an internal standard for the quantification of 9(S)-HODE by GC- or LC-mass spectrometry.
-
GC42636
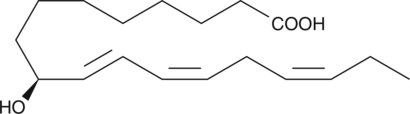
9(S)-HOTrE
9(S)-HOTrE is a monohydroxy polyunsaturated fatty acid produced by the action of 5-lipoxygenase on α-linolenic acid.
-
GC40357
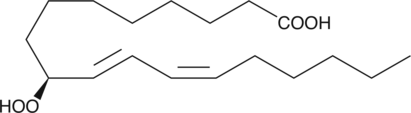
9(S)-HpODE
9(S)-HpODE is produced by the action of arachidonate 5-LO on linoleic acid.
-
GC42637
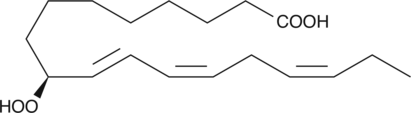
9(S)-HpOTrE
9(S)-HpOTrE is a monohydroperoxy polyunsaturated fatty acid produced by the action of 5-lipoxygenase (5-LO) on α-linolenic acid.
-
GC18389
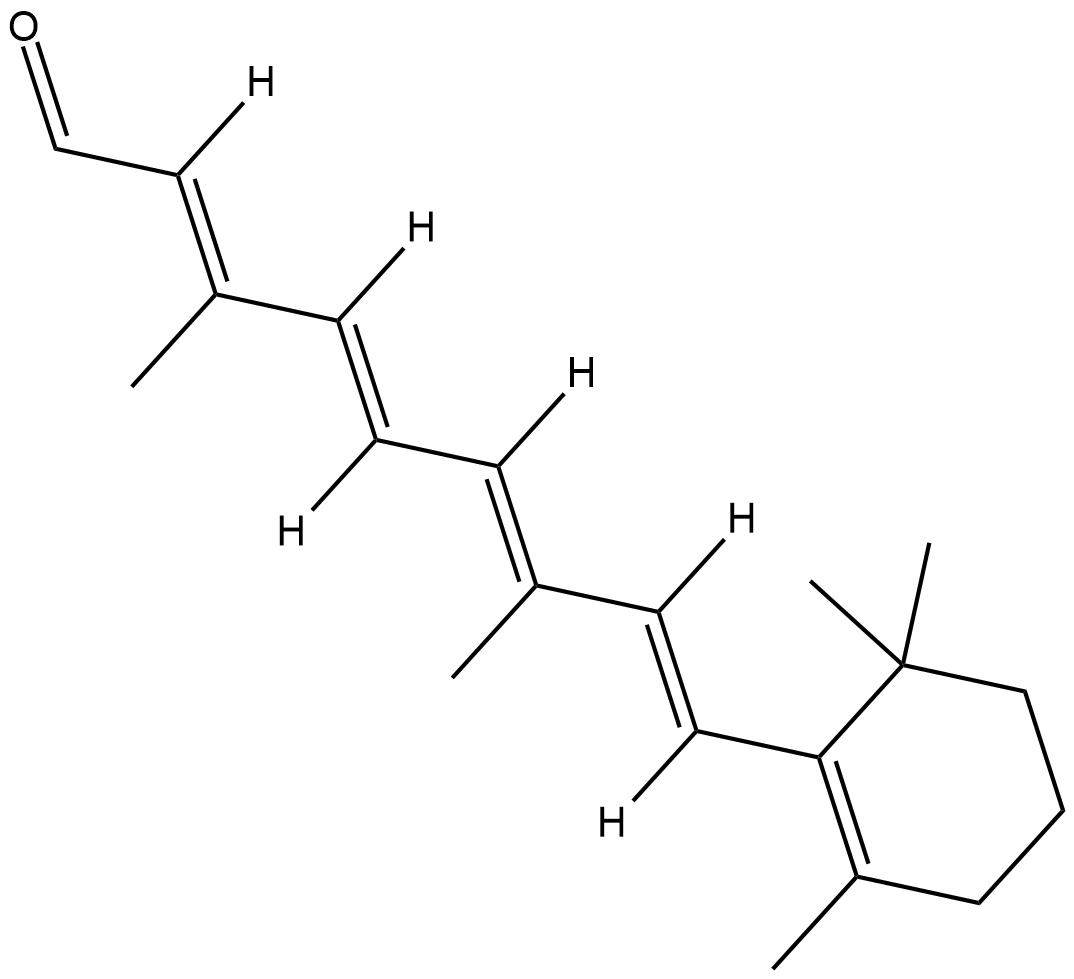
9-cis Retinal
Le 9-cis Retinal est un rétinoïde naturel.
-
GC38883
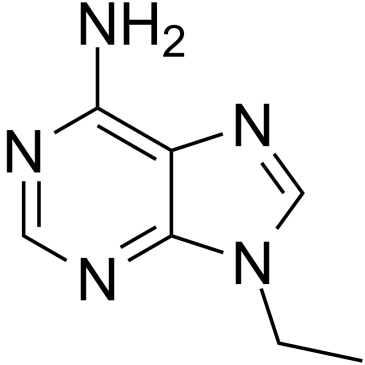
9-Ethyladenine
La 9-éthyladénine est un inhibiteur partiellement efficace de l'APRT (adénine phosphoribosyltransférase).
-
GC42651
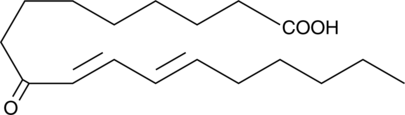
9-oxo-10(E),12(E)-Octadecadienoic Acid
9-oxo-10(E),12(E)-Octadecadienoic acid (9-oxoODA) is a natural agonist, abundant in tomatoes, that activates PPARα at 10-20 μM.
-
GC42653
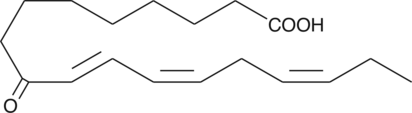
9-OxoOTrE
9-OxoOTrE is produced by the oxidation of 9-HpOTrE.
-
GC45960
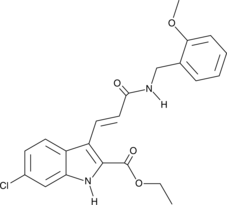
9c(i472)
9c(i472) est un puissant inhibiteur de 15-LOX-1 (15-lipoxygénase-1) avec une valeur IC50 de 0,19 μM.
-
GC62859
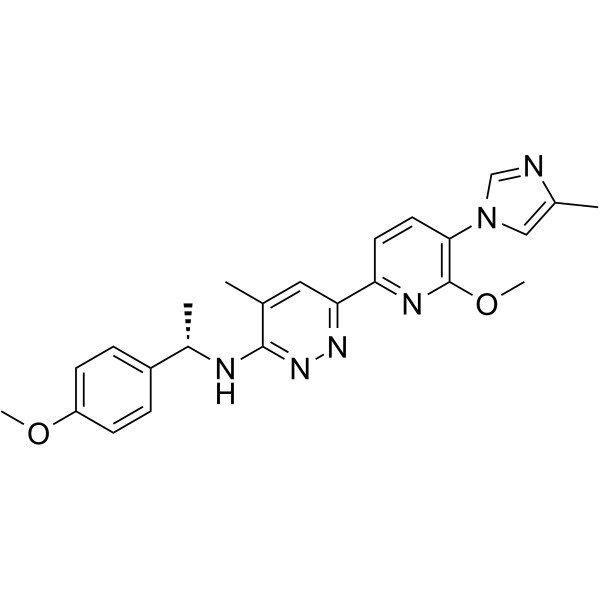
Aβ42-IN-2
Aβ42-IN-2 est un γ-modulateur de sécrétase extrait du brevet WO2016070107, exemple composé 36.
-
GC31938
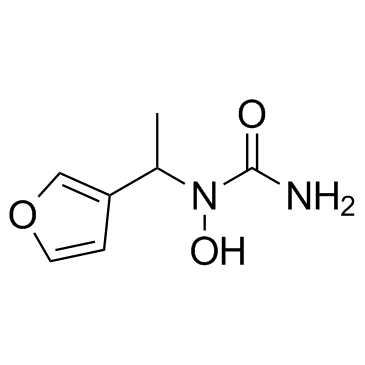
A-69412
A-69412 est un inhibiteur spécifique réversible de la 5-lipoxygénase (5-LO).
-
GC68587
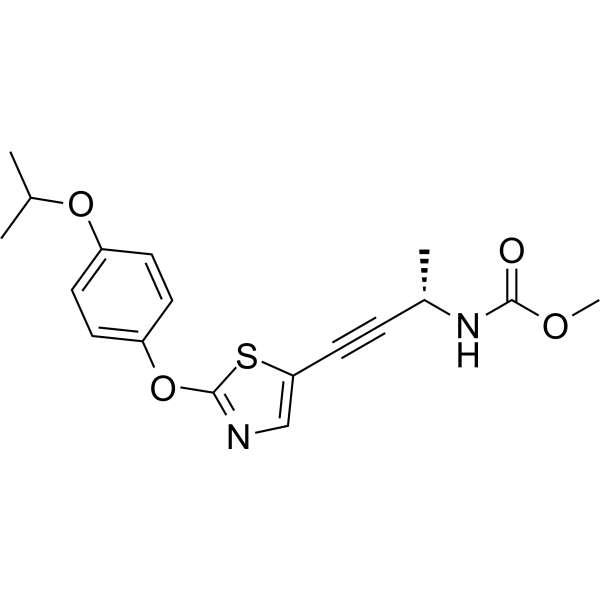
A-908292
A-908292 est un inhibiteur efficace et sélectif de l'acétyl-CoA carboxylase 2 (ACC2), avec une valeur IC50 de 23 nM pour l'inhibition de hACC2. A-908292 peut être utilisé dans la recherche sur le métabolisme des acides gras.
-
GC15614
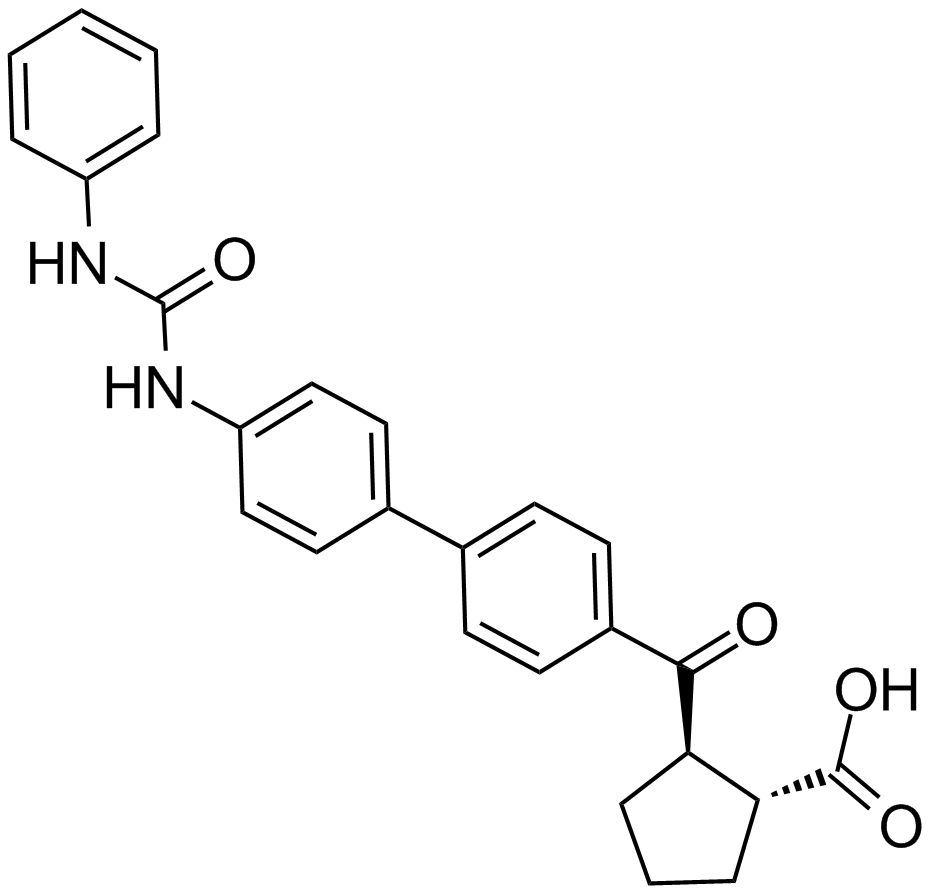
A922500
A diacylglycerol acyltransferase 1 inhibitor
-
GC42665
AAF-CMK (trifluoroacetate salt)
Tripeptidyl peptidase II (TPPII) is a serine peptidase of the subtilisin-type which removes tripeptides from the free NH2 terminus of oligopeptides.

-
GC42667
Abacavir Carboxylate
Abacavir carboxylate is an inactive metabolite of the HIV-1 reverse transcriptase inhibitor abacavir.
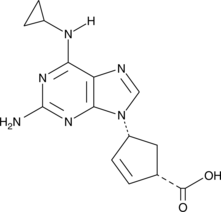
-
GC46769
Abametapir
L'abamétapir est un inhibiteur des métalloprotéinases (MMP) capable de cibler les métalloprotéinases essentielles À l'éclosion des œufs et au développement des poux.
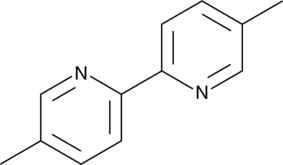
-
GC35219
Abiraterone metabolite 1
Le métabolite 1 de l'abiratérone est un métabolite réduit en 5β de l'abiratérone. L'abiratérone, un médicament stéroÏdien, inhibe le CYP17A1, bloque la synthèse des androgènes et prolonge la survie dans le cancer de la prostate.
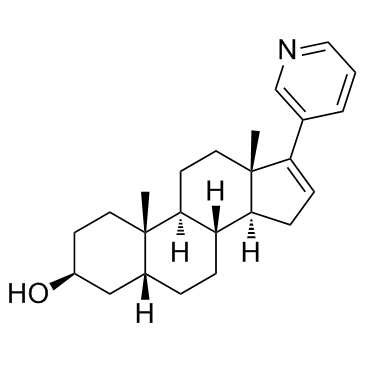
-
GC67766
Abiraterone sulfate

-
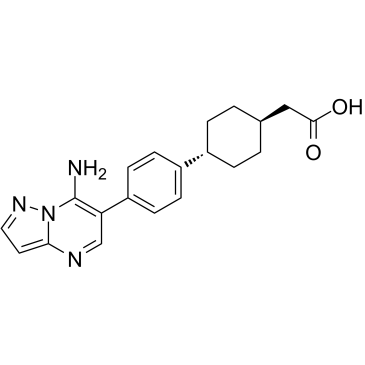
GC35221
ABT-046
ABT-046 est un inhibiteur puissant, sélectif et actif par voie orale de l'acyl CoA:diacylglycérol acyltransférase 1 (DGAT-1) avec des IC50 de 8 nM contre la DGAT-1 humaine et la souris .
-
GC60548
ABT-100
ABT-100 est un inhibiteur de farnésyltransférase puissant, hautement sélectif et actif par voie orale. ABT-100 inhibe la prolifération cellulaire (IC50 de 2,2 nM, 3,8 nM, 5,9 nM, 6,9 nM, 9,2 nM, 70 nM et 818 nM pour EJ-1, DLD-1, MDA-MB-231, HCT-116, MiaPaCa- 2, PC-3 et cellules DU-145, respectivement), augmente l'apoptose et diminue l'angiogenèse. ABT-100 possède une activité antitumorale À large spectre.
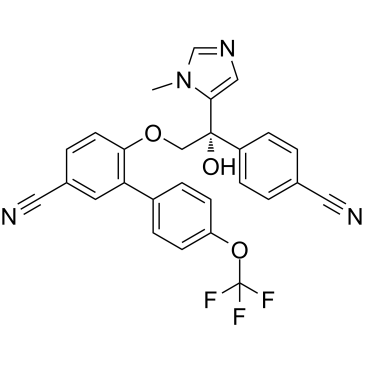
-
GC38264
Ac-Ala-OH
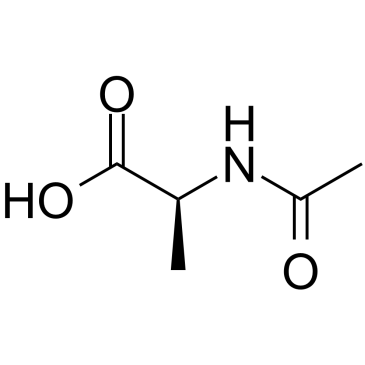
-
GA11174
Ac-Arg-OH.2H2O
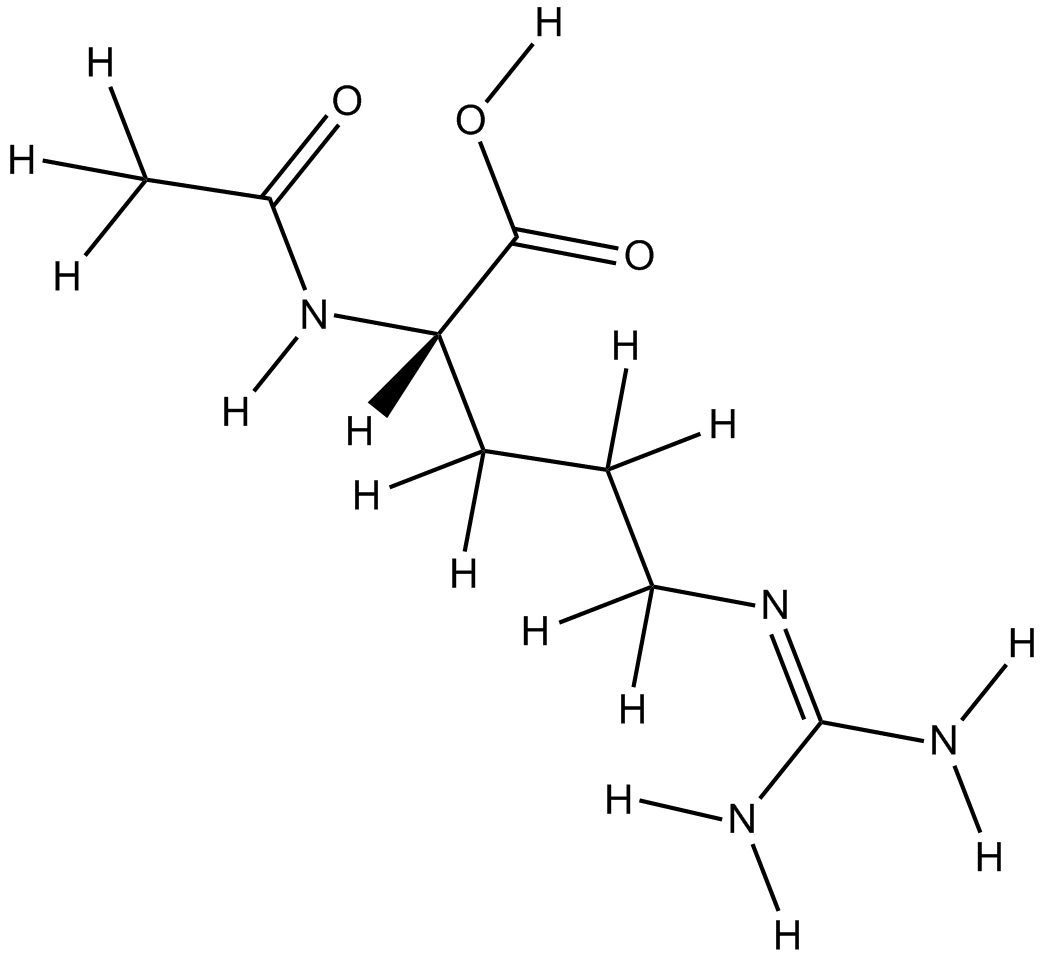
-
GA20489
Ac-Asn-OH
Ac-Asn-OH est un métabolite endogène.

-
GC17602
Ac-DEVD-AFC
Ac-DEVD-AFC est un substrat fluorogène (Λex = 400 nm, Λem = 530 nm).
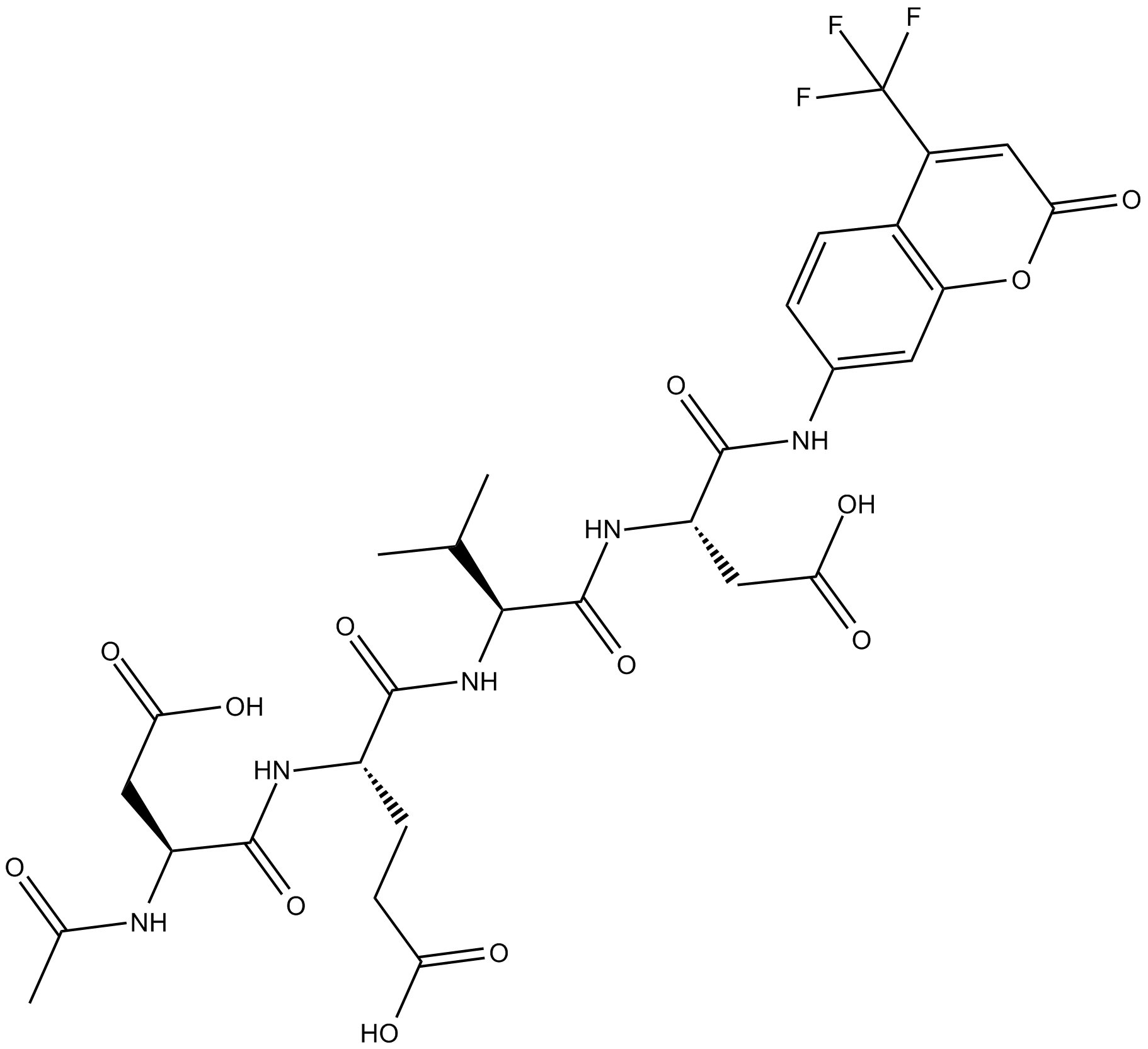
-
GC32695
Ac-DEVD-CHO
Ac-DEVD-CHO est un inhibiteur spécifique de la caspase-3 avec une valeur Ki de 230 pM.
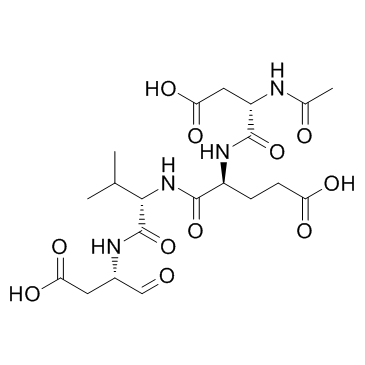
-
GC10951
Ac-DEVD-CMK
cell-permeable, and irreversible inhibitor of caspase
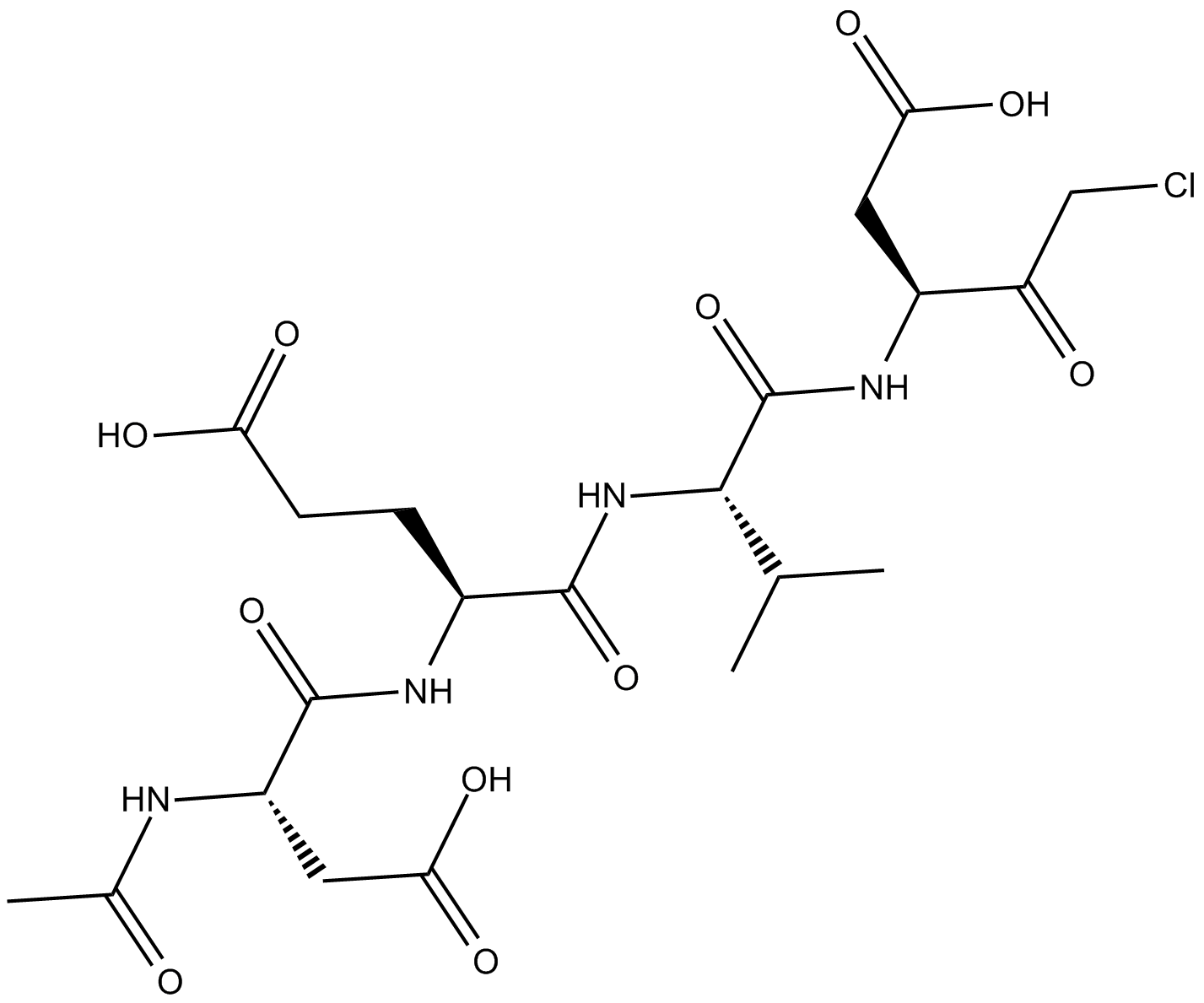
-
GA10446
Ac-DL-Trp-OH
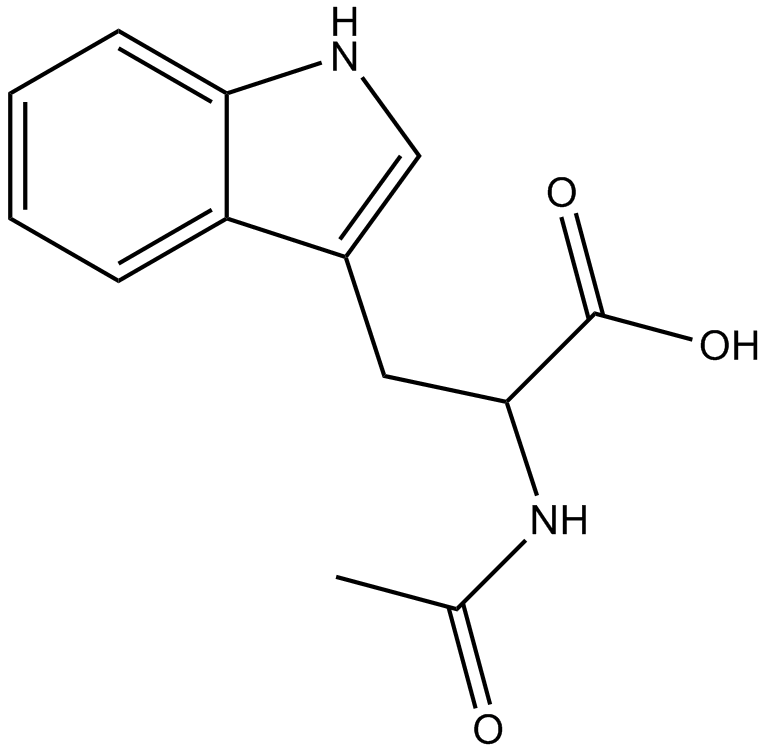
-
GC60558
Ac-FLTD-CMK
Ac-FLTD-CMK, un inhibiteur dérivé de la gasdermine D (GSDMD), est un inhibiteur spécifique des caspases inflammatoires.
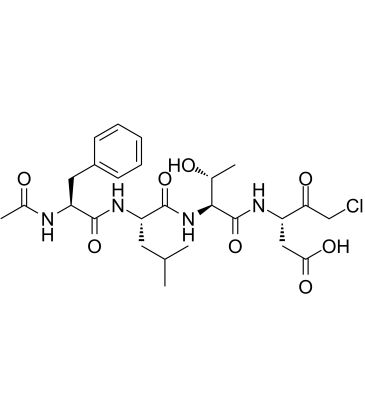
-
GC49704
Ac-FLTD-CMK (trifluoroacetate salt)
An inhibitor of caspase-1, -4, -5, and -11

-
GA10356
Ac-Gly-OH
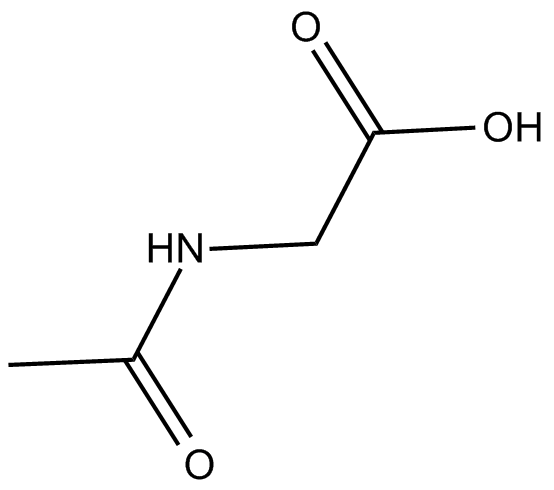
-
GA10825
Ac-His-OH
Ac-His-OH

-
GC10258
Ac-IEPD-AFC
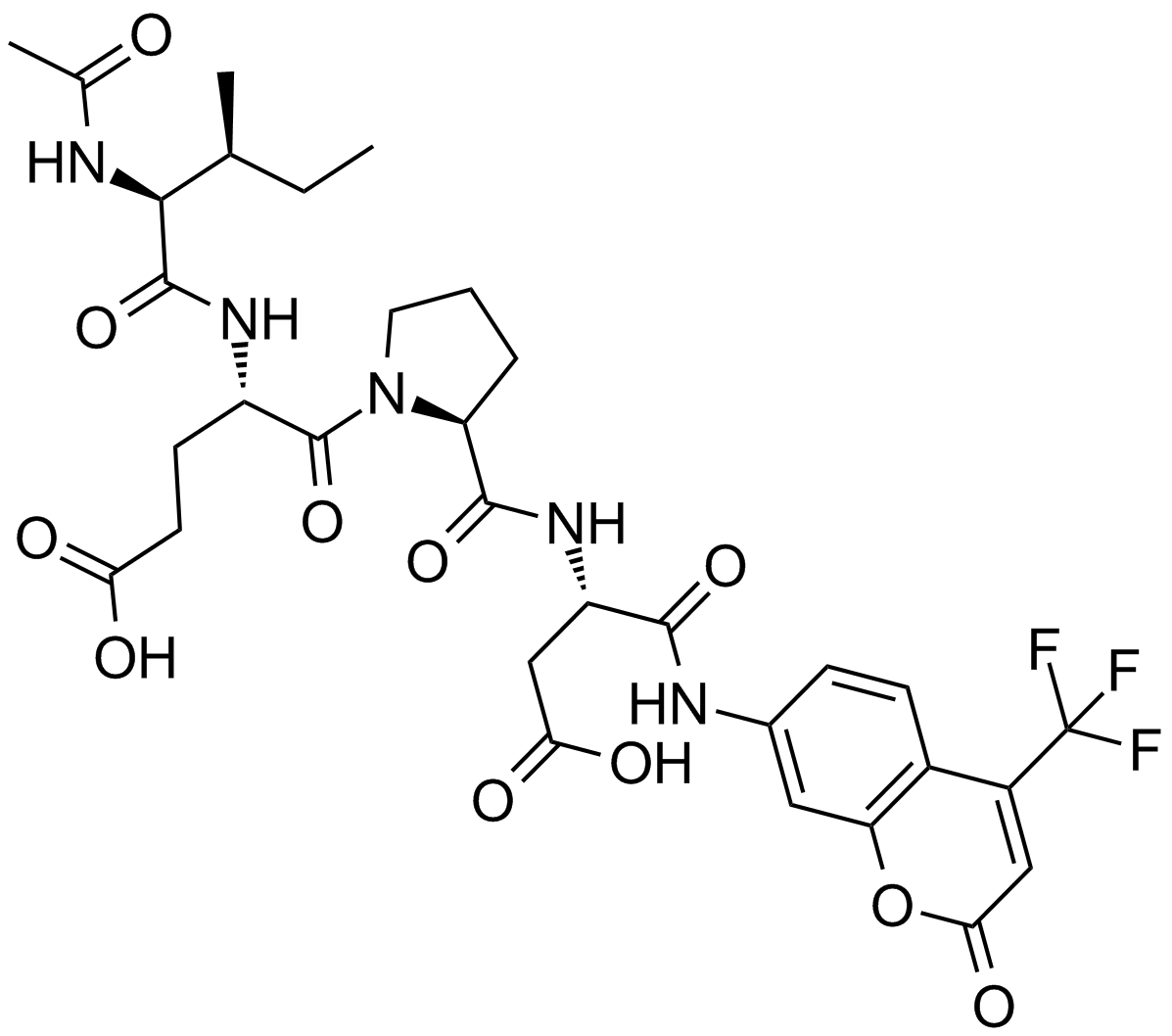
-
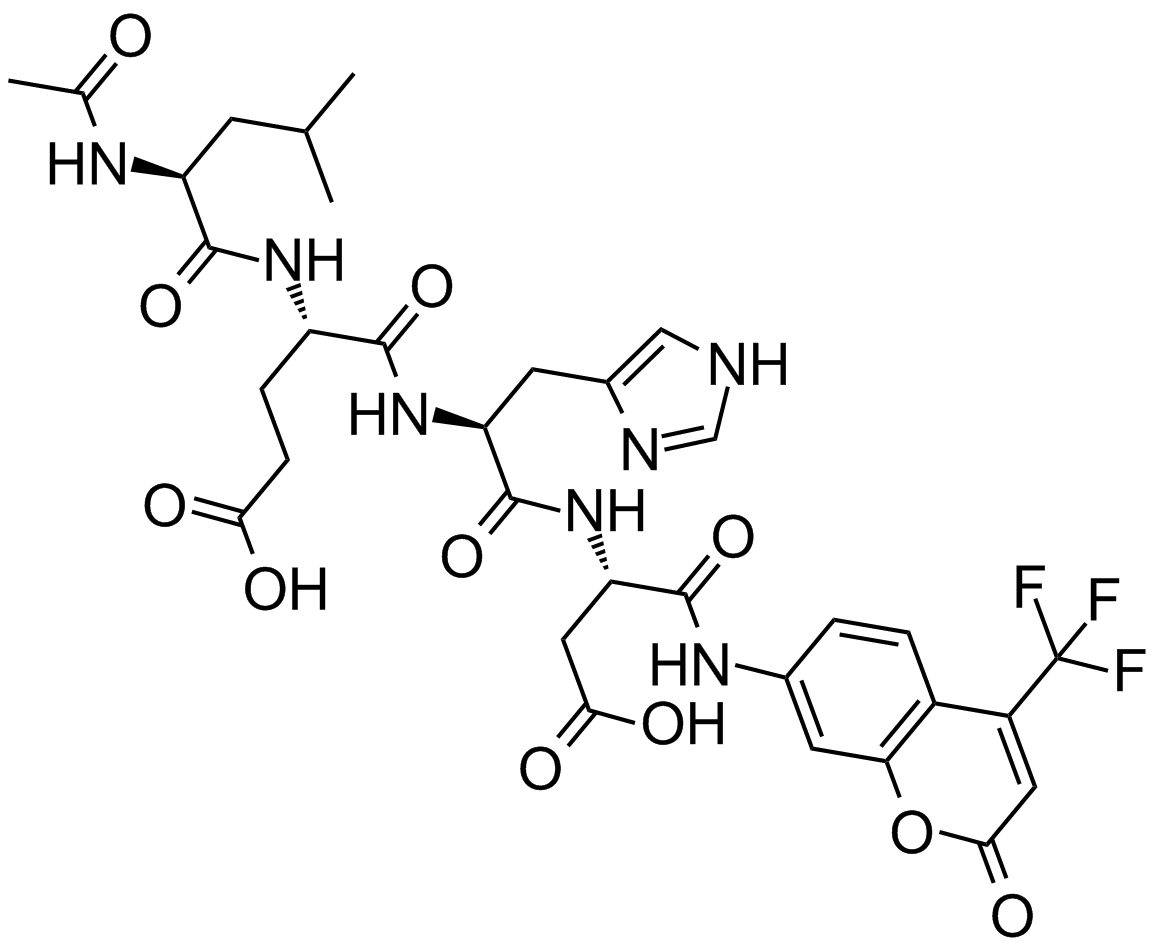
GC10968
Ac-LEHD-AFC
A caspase-4, -5, and -9 fluorogenic substrate
-
GA20612
Ac-Lys-OH
Ac-Lys-OH est un métabolite endogène.

-
GA20680
Ac-Val-OH

-
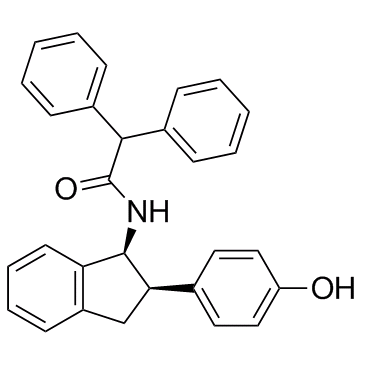
GC31548
ACAT-IN-1 cis isomer
L'isomère cis de l'ACAT-IN-1 est un puissant inhibiteur de l'ACAT avec une IC50 de 100 nM.
-
GC42690
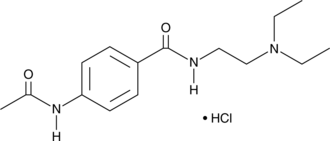
Acecainide (hydrochloride)
Acecainide is an active metabolite of the class III antiarrhythmic procainamide.
-
GC35228
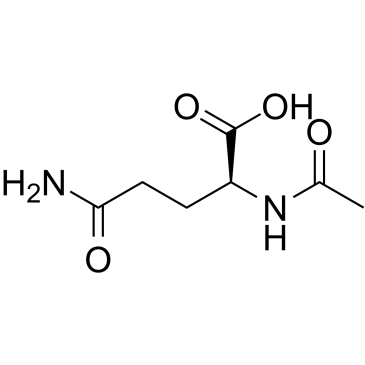
Aceglutamide
L'acéglutamide (α-N-acétyl-L-glutamine) est un psychostimulant et nootropique, utilisé pour améliorer la mémoire et la concentration.
-
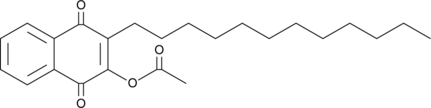
GC46779
Acequinocyl
A naphthoquinone acaricide
-
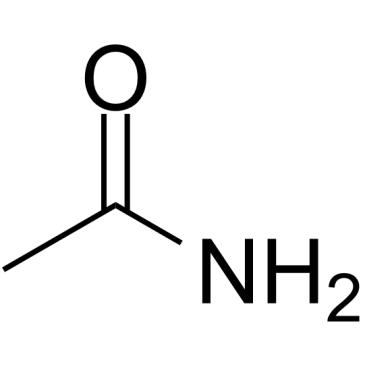
GC38317
Acetamide
L'acétamide est utilisé comme intermédiaire dans la synthèse de la méthylamine, du thioacétamide et des insecticides, et comme plastifiant dans le cuir, les tissus et les revêtements. L'acétamide est cancérigène.
-
GC12917
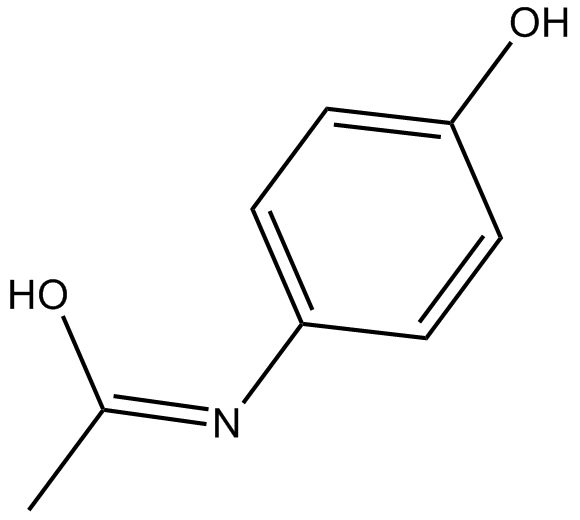
Acetaminophen
Un inhibiteur de COX
-
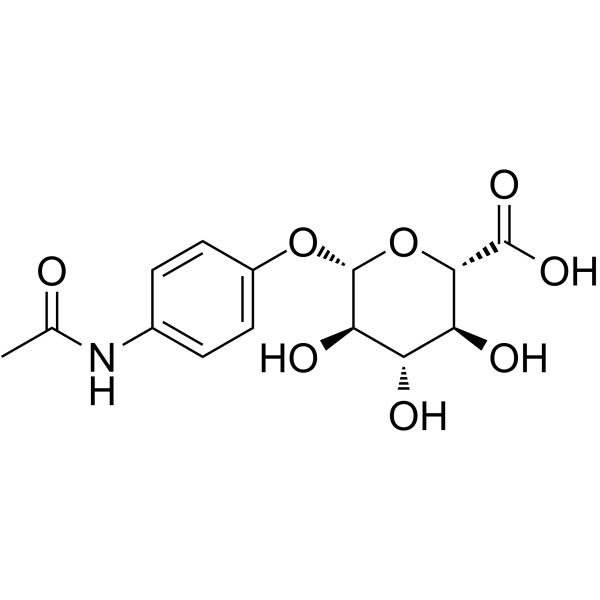
GC62824
Acetaminophen glucuronide
Le glucuronide d'acétaminophène (APAP-glu) est un métabolite glucuronide inactif de l'acétaminophène.
-
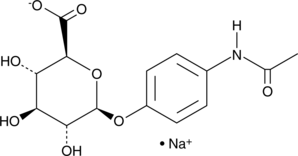
GC42693
Acetaminophen Glucuronide (sodium salt)
Acetaminophen glucuronide is an inactive metabolite of the analgesic and antipyretic agent acetaminophen.
-
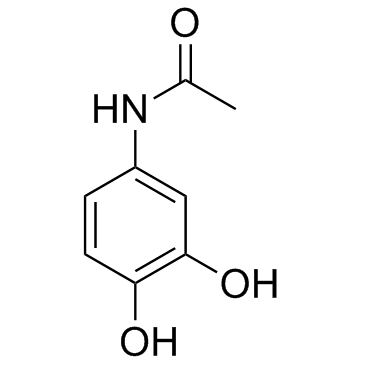
GC33558
Acetaminophen metabolite 3-hydroxy-acetaminophen (3-Hydroxyacetaminophen)
Le 3-hydroxy-acétaminophène est un métabolite de l'acétaminophène, qui est un analgésique.
-
GC42694
Acetaminophen sulfate (potassium salt)
Acetaminophen sulfate is a metabolite of acetaminophen.
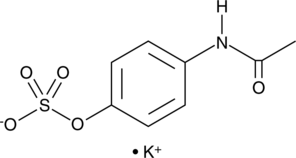
-
GC64137
Acetaminophen-d3
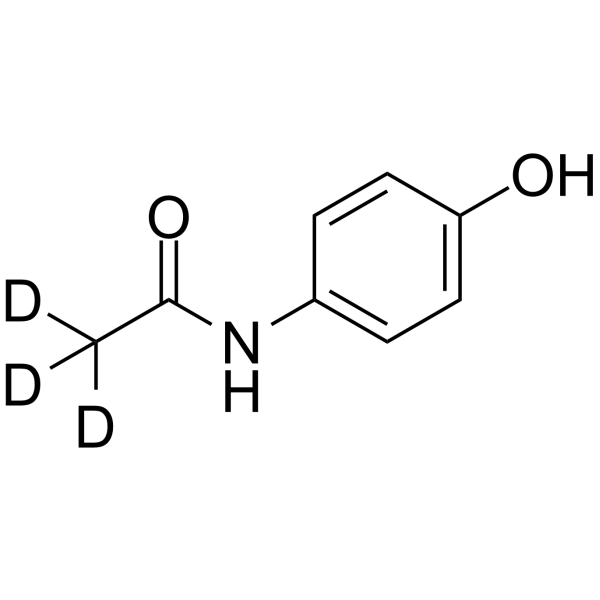
-
GC42695
Acetoacetyl Coenzyme A (sodium salt hydrate)
Acetoacetyl coenzyme A (acetoacetyl-CoA) is a precursor to HMG-CoA in the isoprenoid pathway.
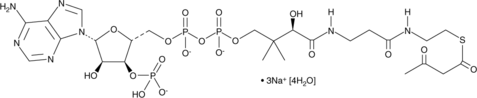
-
GC42697
Acetyl Coenzyme A (sodium salt)
L'acétyl-coenzyme A (acétyl-CoA) trisodique est un intermédiaire métabolique central imperméable À la membrane, participe au cycle du TCA et au métabolisme de la phosphorylation oxydative.
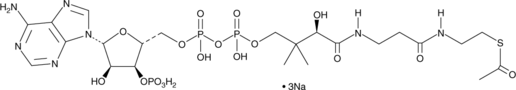
-
GC60555
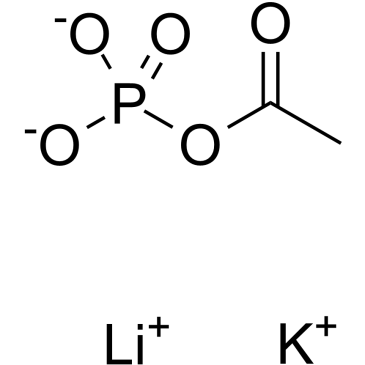
Acetyl phosphate(lithium potassium)
Le phosphate d'acétyle (lithium potassium) est un métabolite endogène.
-
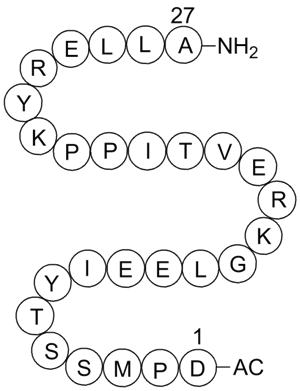
GC12152
Acetyl-Calpastatin (184-210) (human)
-
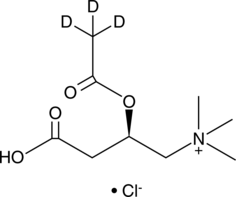
GC46789
Acetyl-L-carnitine-d3 (chloride)
An internal standard for the quantification of L-acetylcarnitine
-
GC68097
Acetyl-L-carnitine-d3 hydrochloride

-
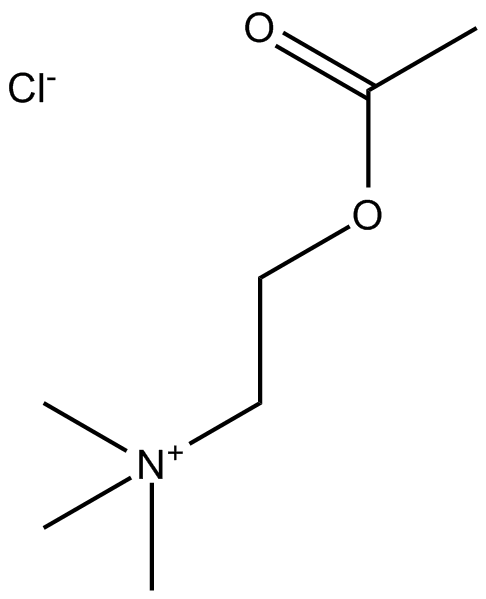
GC14142
Acetylcholine Chloride
Le chlorure d'acétylcholine (chlorure d'ACh), un neurotransmetteur, est un puissant agoniste cholinergique.
-
GC11786
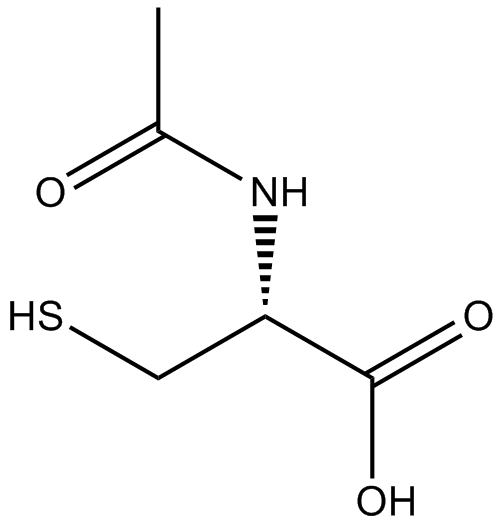
Acetylcysteine
L'acétylcystéine est le dérivé N-acétyl de la CYSTEINE.
-
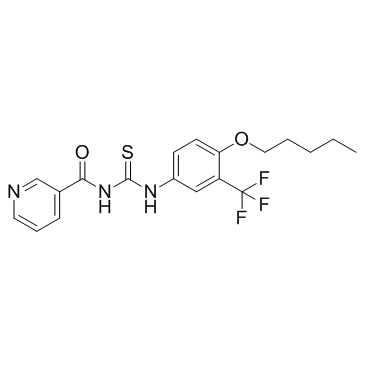
GC32305
ACH-806 (GS9132)
ACH-806 (GS9132) est un antagoniste de la NS4A qui peut inhiber la réplication du virus de l'hépatite C (VHC) avec une EC50 de 14 nM.
-
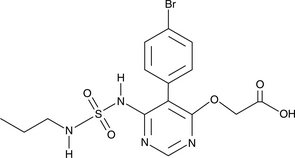
GC49406
ACT-373898
ACT-373898 est un métabolite acide carboxylique inactif du macitentan.
-
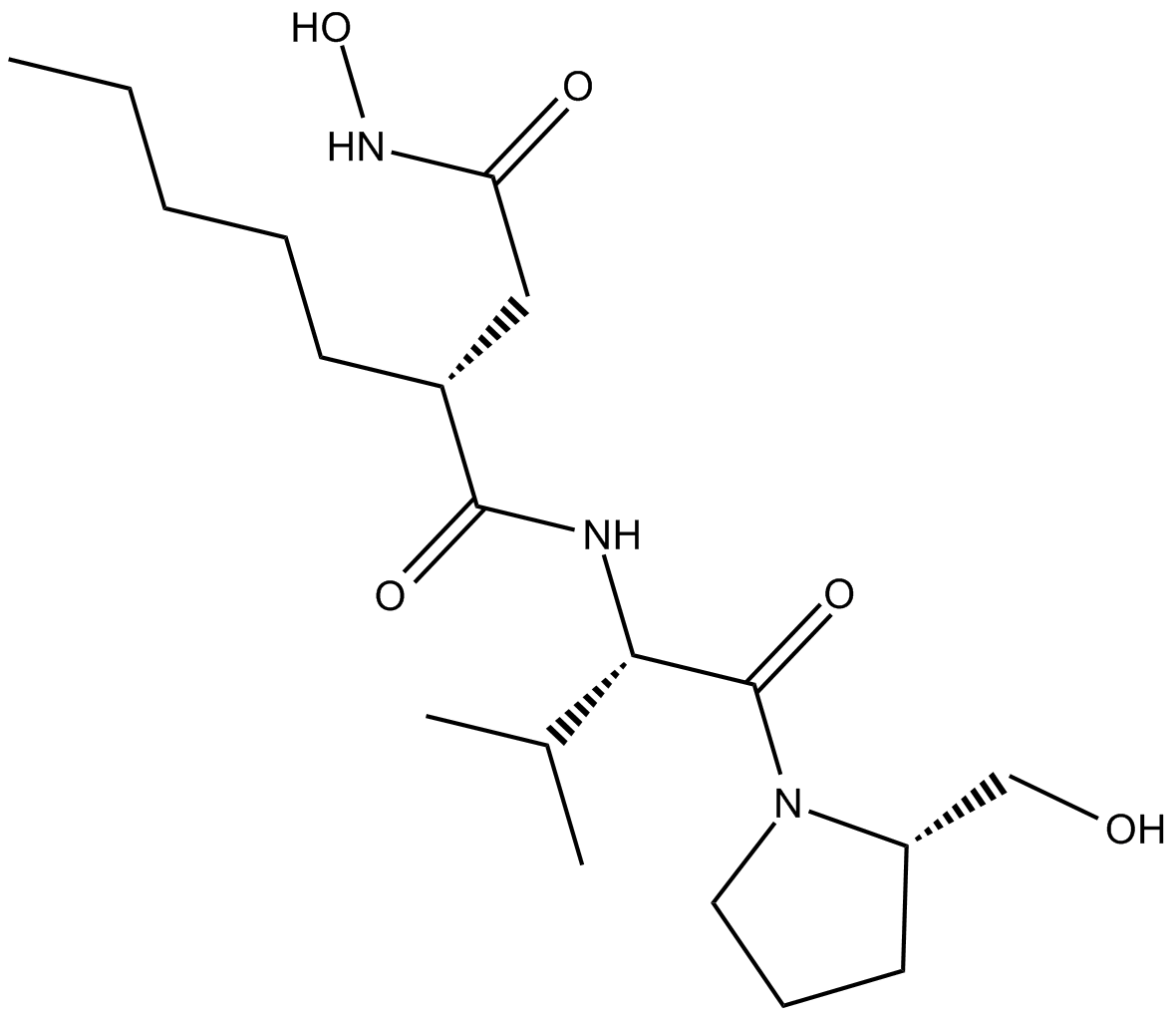
GC16350
Actinonin
L'actinonine ((-)-Actinonine) est un agent antibactérien naturel produit par Actinomyces.
-
GC46798
Adapalene-d3
An internal standard for the quantification of adapalene
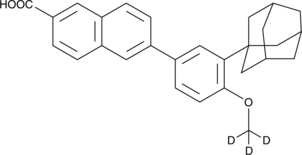
-
GC13432
Adenine
L'adénine (6-aminopurine), une purine, est l'une des quatre nucléobases de l'acide nucléique de l'ADN. L'adénine agit comme un composant chimique de l'ADN et de l'ARN. L'adénine joue également un rÔle important dans la biochimie impliquée dans la respiration cellulaire, la forme À la fois de l'ATP et des cofacteurs (NAD et FAD), et la synthèse des protéines.

-
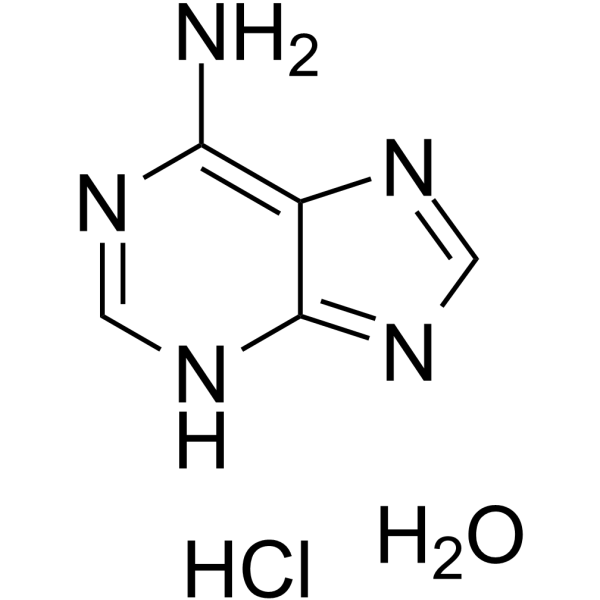
GC62828
Adenine monohydrochloride hemihydrate
L'hémihydrate de monochlorhydrate d'adénine est un métabolite endogène.
-
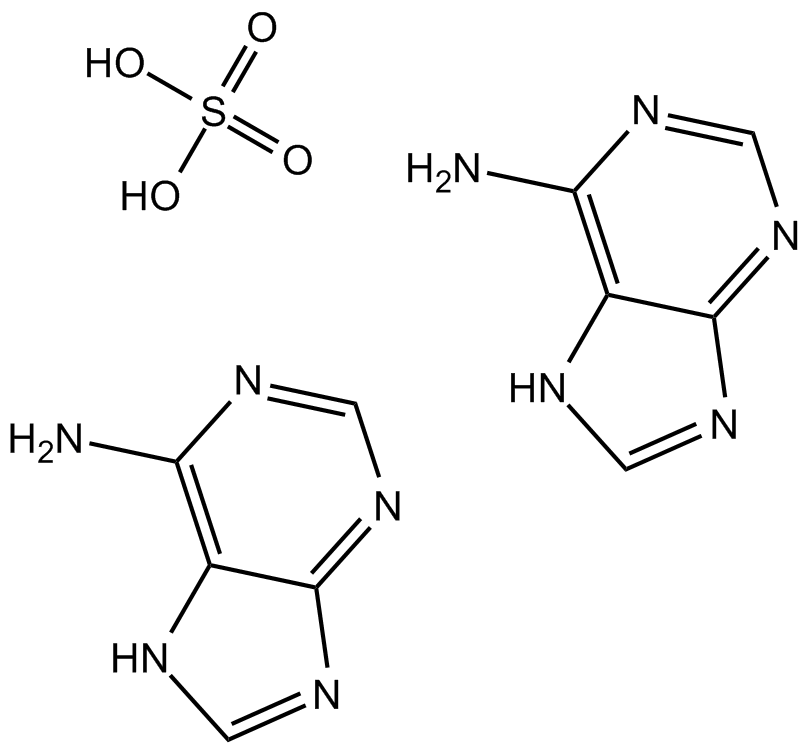
GC17278
Adenine sulfate
Le sulfate d'adénine (hémisulfate de 6-aminopurine), une purine, est l'une des quatre nucléobases de l'acide nucléique de l'ADN. Le sulfate d'adénine agit comme un composant chimique de l'ADN et de l'ARN. Le sulfate d'adénine joue également un rÔle important dans la biochimie impliquée dans la respiration cellulaire, la forme À la fois de l'ATP et des cofacteurs (NAD et FAD), et la synthèse des protéines.
-
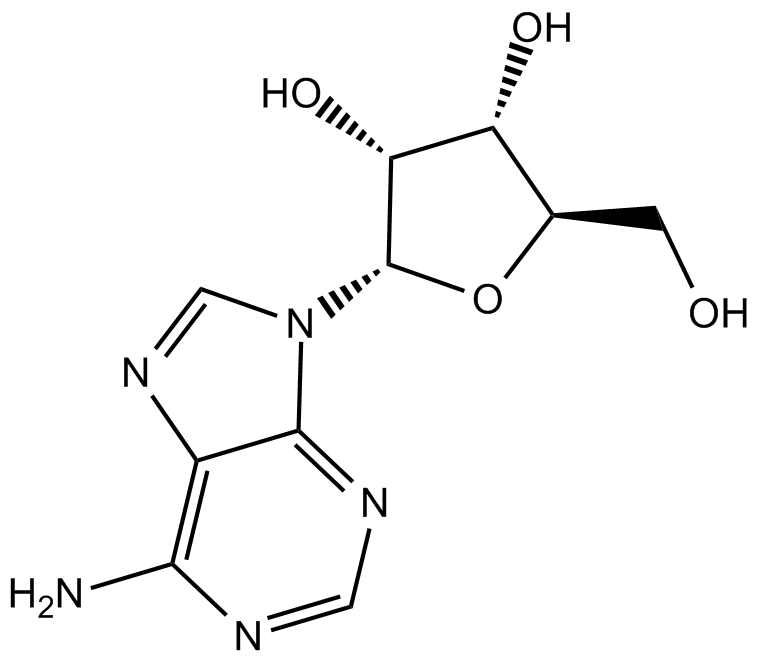
GC14106
Adenosine
nucléoside
-
GC19630
Adenosine 2′-monophosphate
L'adénosine &2#8242;-monophosphate (2'-AMP) est convertie par le 2',3'-CAMP extracellulaire.
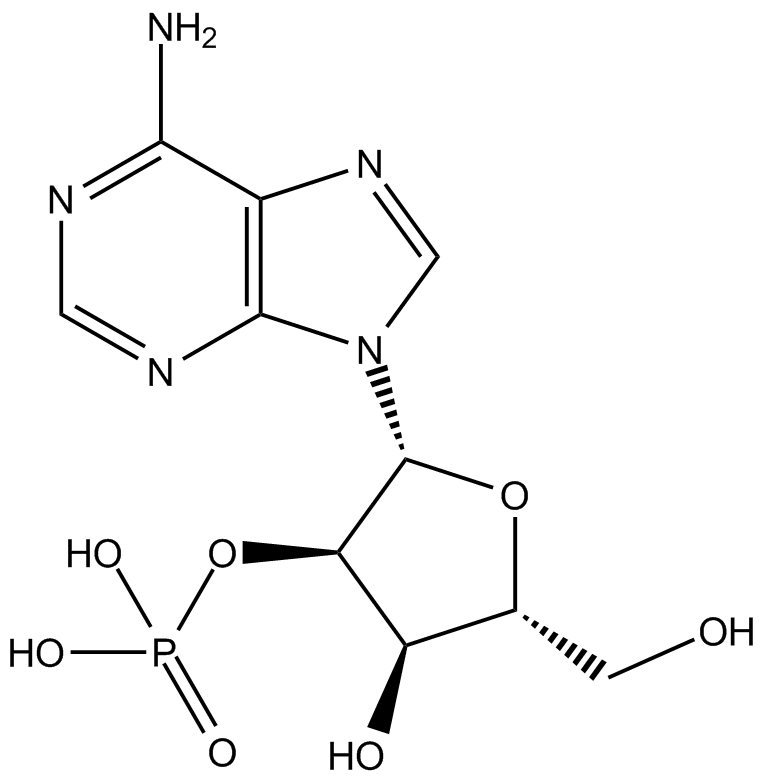
-
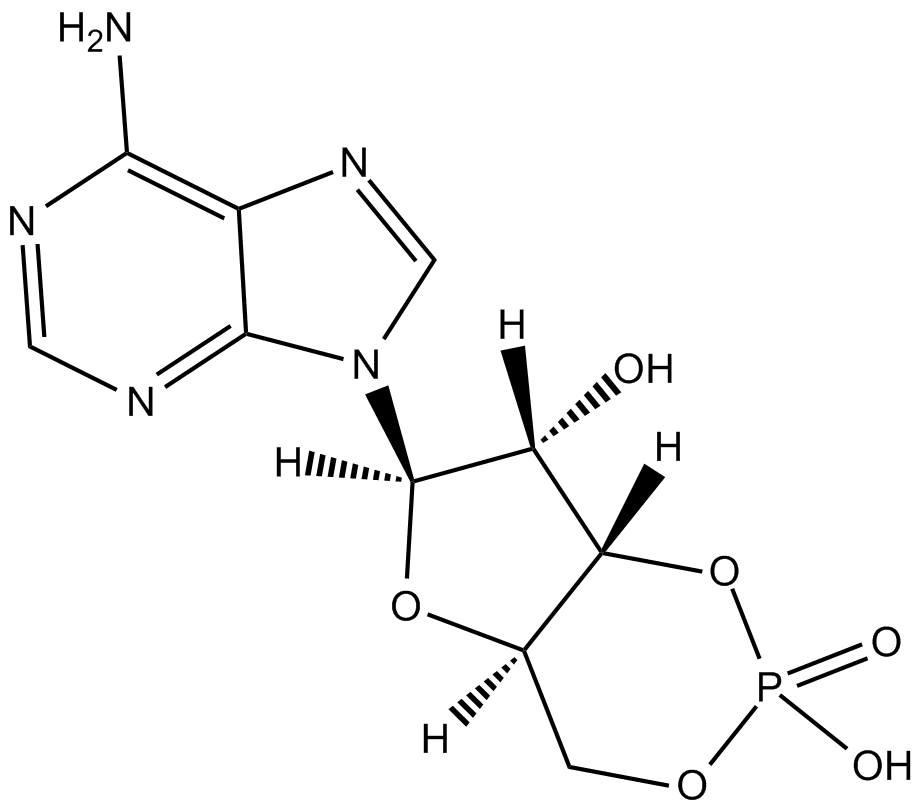
GC10880
Adenosine 3-5-cyclic monophosphate
A second messenger
-
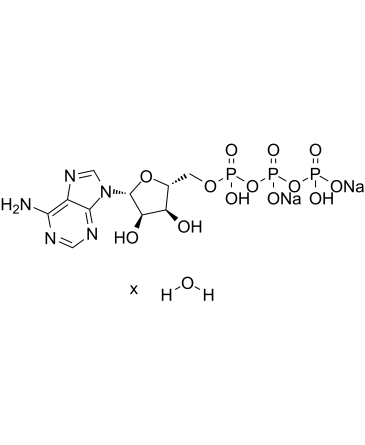
GC17558
Adenosine 5'-triphosphate disodium salt hydrate
L'adénosine 5'-triphosphate disodium salt hydrate (Adenosine 5'-triphosphatedisodium salt hydrate) est un composant central du stockage de l'énergie et du métabolisme in vivo, fournit l'énergie métabolique pour entraÎner les pompes métaboliques et sert de coenzyme dans les cellules.
-
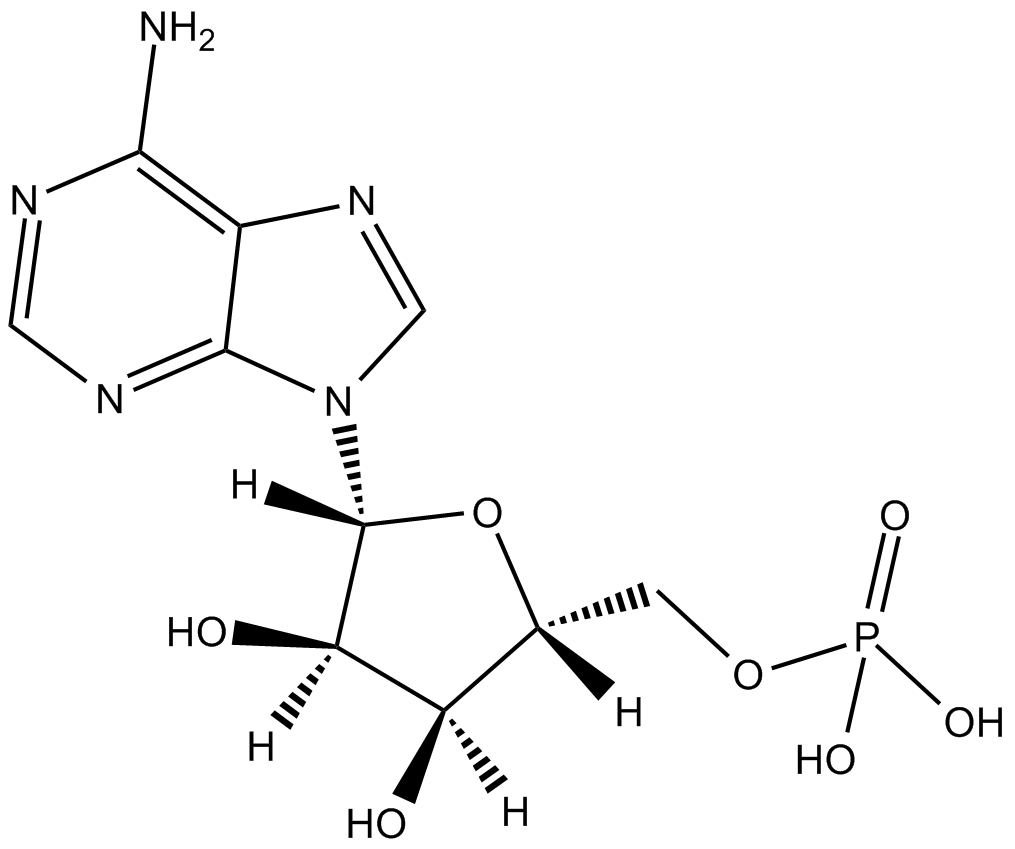
GC13172
Adenosine 5-monophosphate
L'adénosine 5-monophosphate est un métabolite cellulaire clé régulant l'homéostasie énergétique et la transduction du signal.
-
GC11969
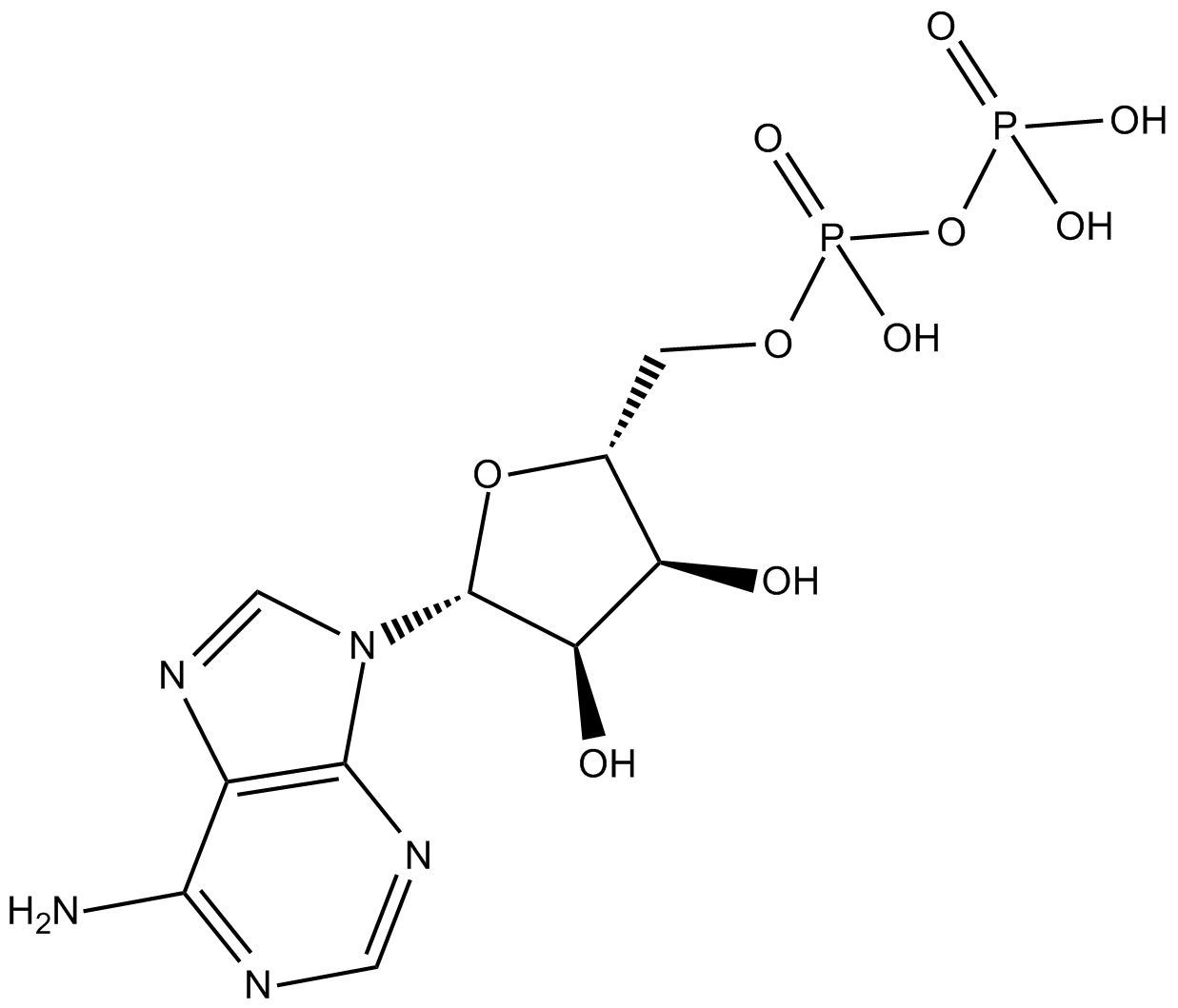
Adenosine-5'-diphosphate
Agoniste des récepteurs purinergiques
-
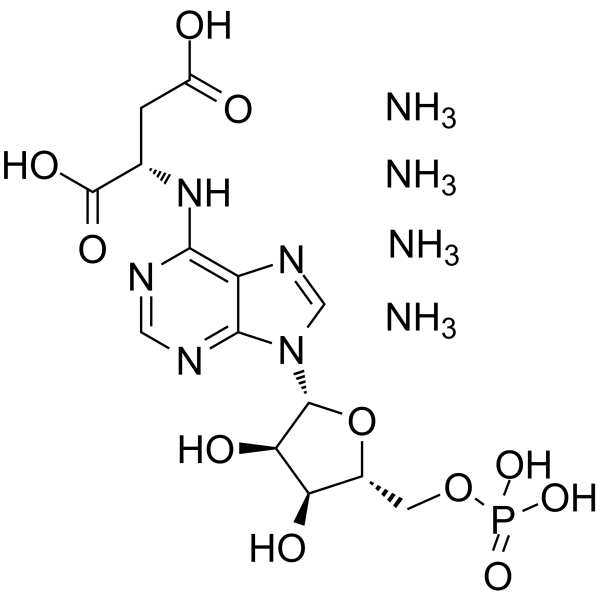
GC64916
Adenylosuccinic acid tetraammonium
L'acide adénylosuccinique tétraammonium (adénylosuccinate; adénylate d'aspartyle) est un ribonucléoside monophosphate de purine et joue un rÔle dans le métabolite du cycle nucléotidique.
-
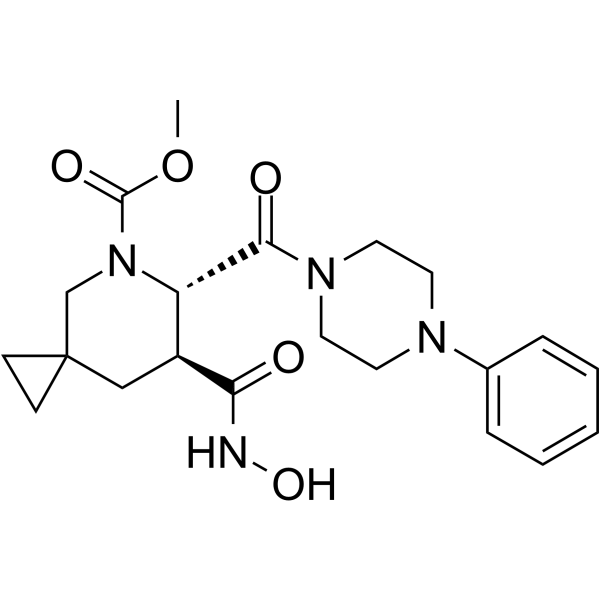
GC63661
Aderbasib
L'aderbasib (INCB007839) est un inhibiteur À base d'hydroxamate nanomolaire puissant, actif par voie orale et spécifique À la cible d'ADAM10 et ADAM17. L'aderbasib présente une activité antinéoplasique robuste et peut être utilisé pour la recherche sur le cancer, y compris le lymphome non hodgkinien diffus À grandes cellules B, le cancer du sein HER2+, les gliomes, et al.
-
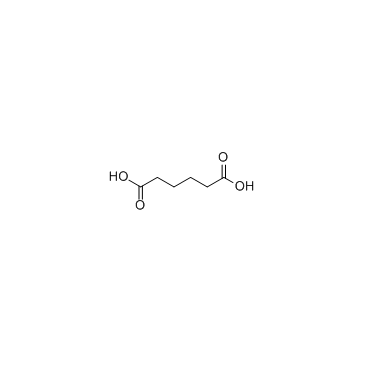
GC33805
Adipic acid
L'acide adipique est associé À un déficit en HMG-CoA lyase, un déficit en carnitine-acylcarnitine translocase, un déficit en malonyl-Coa décarboxylase et un déficit en acyl-CoA déshydrogénase À chaÎne moyenne, qui sont des erreurs innées du métabolisme.
-
GC19021
Adjudin
Adjudin est un contraceptif masculin largement étudié avec un effet supérieur d'inhibition des mitochondries. Adjudin est également un puissant bloqueur du canal Cl.
-
GC49216
ADL 08-0011 (hydrochloride)
An active metabolite of alvimopan